Микробиология, 2023, T. 92, № 4, стр. 347-357
PALUDISPHAERA MUCOSA SP. NOV., новый планктомицет семейства ISOSPHAERACEAE из бореального низинного болота
А. А. Иванова a, *, Д. Г. Наумов a, И. С. Куличевская a, А. А. Мещерякова a, b, С. Н. Дедыш a
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
b Московский государственный университет им. М.В. Ломоносова, почвенный факультет
119991 Москва, Россия
* E-mail: ivanovastasja@gmail.com
Поступила в редакцию 16.03.2023
После доработки 28.03.2023
Принята к публикации 29.03.2023
- EDN: RKYILM
- DOI: 10.31857/S0026365623600104
Аннотация
Планктомицеты являются типичными обитателями северных болотных экосистем. В настоящей работе новый планктомицет рода Paludisphaera, штамм Pla2T, был выделен из низинного болота бореальной зоны России. Изолят был представлен розово-пигментированными, одиночными или собранными в небольшие группы неподвижными сферическими клетками, размножающимися почкованием. Штамм Pla2T являлся хемоорганотрофным психротолерантным мезофилом с оптимумом роста при pH 5.5‒6 и температуре 15‒20°C. Предпочтительными ростовыми субстратами нового планктомицета являлись полисахариды ксилан, ксантановая камедь и фитагель, а также некоторые сахара. Наиболее высокое сходство последовательности гена 16S рРНК штамма Pla2T (97.9%) наблюдалось с последовательностью планктомицета “Paludisphaera soli” JC670Т, выделенного из почвы высокогорья Гималаев. С другими представителями рода Paludisphaera, “P. rhizosphae-rae” JC665Т и P. borealis PX4Т, это сходство составляло 97.0 и 93.8% соответственно. Геном штамма Pla2T, размером 8.21 млн п.о., содержал около 6.5 тыс. белок-кодирующих генов и 3 копии оперона рРНК. Содержание пар Г + Ц в ДНК составляло 67 мол. %. Сходство нуклеотидных последовательностей генома штамма Pla2Т и ранее описанных представителей рода Paludisphaera составило от 79.4 до 82.6%. Эти генотипические отличия, а также ряд фенотипических особенностей позволили отнести планктомицет из низинного болота к новому виду рода Paludisphaera, Paludisphaera mucosa sp. nov. с типовым штаммом Pla2T (=KCTC92668T = VKM B-3698T).
Семейство Isosphaeraceae было сформировано в 2016 г. на основании данных сравнительного анализа последовательностей генов 16S рРНК культивируемых представителей филума Planctomycetota (Kulichevskaya et al., 2016). В настоящее время это единственное семейство порядка Isosphaerales (класс Planctomycetia). В состав семейства входят планктомицеты c клетками сферической формы, которые могут быть собраны в короткие цепочки, длинные нити или образовывать бесформенные агрегаты. Клетки этих планктомицетов покрыты кратериформными структурами, расположенными по всей поверхности. Деление клеток происходит путем почкования; дочерние клетки неподвижны. Представители семейства Isosphaeraceae являются хемоорганотрофными аэробами, хотя некоторые из них способны расти в микроаэробных условиях. Типовым родом данного семейства является род Isosphaera с единственным представителем Isosphaera pallida IS1BT (Giovannoni et al., 1987), выделенным из горячего источника Северной Америки. Помимо Iso-sphaera, в состав семейства Isosphaeraceae входят роды Singulisphaera (Kulichevskaya et al., 2008), Aquisphaera (Bondoso et al., 2011), Paludisphaera (Kulichevskaya et al., 2016), Tundrisphaera (Kulichevskaya et al., 2017) и Tautonia (Kovaleva et al., 2019). Еще один род, Candidatus Nostocoida, представлен нитчатыми планктомицетами, которые пока не получены в чистых культурах (Liu et al., 2001; Kulichevskaya et al., 2012).
Размеры геномов представителей Isosphaera-ceae варьируют от 5.53 млн п.о. у Isosphaera pallida IS1BT до 9.74 млн п.о. у Singulisphaera acidiphila DSM 18658T (Göker et al., 2011; Ivanova et al., 2017). Примечательно, что во всех полных геномах этих бактерий были выявлены плазмиды в количестве от 1 до 4 (Ivanova et al., 2017).
Одной из характерных особенностей планктомицетов семейства Isosphaeraceae является наличие гидролитического потенциала. Таксономически охарактеризованные представители этого семейства способны утилизировать широкий спектр полисахаридов растительного и микробного происхождения (Dedysh, Ivanova, 2019). Геномы этих бактерий содержат большое количество генов гликозил-гидролаз, многие из которых не принадлежат ни к одному из известных семейств классификации CAZy (Ivanova et al., 2017).
Планктомицеты семейства Isosphaeraceae населяют широкий спектр наземных и водных экосистем, богатых органическим веществом. По данным молекулярных исследований, Isosphaeraceae является одной из численно доминирующих групп планктомицетов в бореальных и субарктических торфяных болотах (Serkebaeva et al., 2013; Moore et al., 2015; Dedysh, Ivanova, 2019). Работы по выделению новых микроорганизмов из этих экосистем имели своим результатом описание трех новых родов семейства (Kulichevskaya et al., 2008, 2016, 2017).
Настоящая работа была посвящена изучению планктомицета, штамма Pla2T, выделенного из низинного болота бореальной зоны России и идентифицированного по данным анализа гена 16S рРНК как представитель рода Paludisphaera. В настоящее время этот род включает 3 таксономически описанных представителя – P. borealis PX4T (Kulichevskaya et al., 2016), выделенный из торфа верхового болота, “P. soli” JC670T из почвы высокогорья Гималаев (Kaushik et al., 2020) и “P. rhizosphae-rae” JC665T из почвы ризосферы (Lhingjakim et al., 2022).
Целью настоящего исследования явилось изучение совокупности морфологических, экофизиологических и геномных характеристик нового штамма Pla2T, отличающих его от ранее описанных представителей рода Paludisphaera, и установление его таксономического статуса.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Источник выделения и условия культивирования. Штамм Pla2T был выделен из образца торфяной почвы (рН 7.1), отобранной в августе 2019 г. с глубины 5‒10 см профиля низинного болота Чарозерское (Вологодская обл., 60°30′42″ N, 38°38′59″ E). Растительное сообщество места отбора образца было представлено ассоциацией Carex lasiocarpa и Campylium stellatum. Электропроводность воды в этом низинном болоте варьировала в диапазоне 236‒279 мкСм/см. Содержание общего органического углерода в отобранном образце торфа составляло 66.2%, общего азота ‒ 2.4%. Более подробная характеристика места отбора образца приведена в ранее опубликованном исследовании (Dedysh et al., 2020).
Отобранный образец торфа был использован для получения накопительной культуры микроорганизмов, участвующих в гидролизе одного из ключевых биополимеров болотных экосистем – ксилана. Для этого в стеклянные флаконы объемом 160 мл, содержащие 2 г измельченного стерильными ножницами торфа и 0.1% березового ксилана (“Sigma-Aldrich”), было добавлено 30 мл нативной болотной воды. Инкубацию накопительной культуры проводили при комнатной температуре (20‒22°С) в течение 8 нед. Изолят, штамм Pla2T, был получен путем высева аликвоты (0.2 мл) накопительной культуры на среду M31 следующего состава (г/л дистиллированной воды): KH2PO4 – 0.1, N-ацетилглюкозамин – 0.5, пептон – 0.1, дрожжевой экстракт ‒ 0.1, раствор солей Хатнера – 20 мл; раствор витаминов № 6 (Staley et al., 1992) – 1 мл; рН 7.0. В качестве желирующего агента использовали 1% раствор полисахарида микробного происхождения фитагеля (PhytaGel, “Fluka”). Очистку изолята проводили путем его рассева на аналогичную среду с добавлением ампициллина (200 мг/л). Посевы инкубировали при 22°С в течение трех недель.
Идентификация изолята. Выделение тотальной ДНК из клеток штамма Pla2T производили с использованием набора FastDNA SPIN kit for soil (“Biol 101”, США) в соответствии с рекоменда-цией производителя. Полученную ДНК использовали в качестве матрицы в ПЦР-амплификации генов 16S рРНК со стандартными бактериальными праймерами 9F/1492R (Lane, 1991). Очистку ПЦР-продукта осуществляли с использованием набора Wizard® SV Gel and PCR Clean-Up System (“Promega”, США). Секвенирование проводили на базе ЦКП “Биоинженерия” ФИЦ Биотехнологии РАН. Редактирование полученных нуклеотидных последовательностей проводили с помощью программы BioEdit. Построение филогенетических дендрограмм производили с использованием программного пакета MEGAX (Kumar et al., 2018) методом максимального правдоподобия. Статистическую достоверность дендрограмм рассчитывали с помощью “bootstrap”-анализа путем построения 100 альтернативных деревьев. Определенная в работе последовательность гена 16S рРНК штамма Pla2T депонирована в GenBank под номером OQ533426.
Геномное секвенирование и аннотирование. Выделение ДНК для геномного секвенирования осуществляли по стандартному протоколу CTAB – фенол/хлороформ. Часть полученной геномной ДНК секвенировали на ячейке R9.4 прибора Mi-nION (“Oxford Nanopore”, Великобритания), используя Ligation Sequencing kit 1D согласно рекомендациям производителя. Другая часть геномной ДНК была секвенирована на платформе Illumina MiSeq. Подготовка библиотеки и процедура секвенирования осуществлялись коммерчески, компанией “ReaGen” (Москва, Россия). Гибридная сборка прочтений Illumina и Nanopore была выполнена с использованием программы Unicycler (Wick et al., 2017). Последовательность генома штамма Pla2T депонирована в GenBank под номером PRJNA940391.
Аннотацию генома проводили с помощью программного пакета PROKKA (Seemann, 2014) и BLASTKoala (Kanehisa et al., 2016). Поиск генов синтеза вторичных метаболитов осуществляли в программе AntiSmash (Medema et al., 2011). Построение геномного дерева производили в программе GTDB-Tk (Chaumeil et al., 2020), путем множественного выравнивания 120 маркерных генов. В анализ были также взяты геномы других охарактеризованных представителей семейства Isosphaeraceae.
Поиск генов гликозил-гидролаз и их гомологов. В работе использовали основанную на гомологии аминокислотных последовательностей каталитических доменов классификацию гликозил-гидролаз CAZy (Drula et al., 2022). Поиск потенциальных гликозил-гидролаз среди белков, закодированных в геноме штамма Pla2T, проводили с помощью сервера dbCAN3 (https://bcb.unl.edu/dbCAN2/index.php), используя три алгоритма: HMMER:dbCAN, DIAMOND:CAZy и HMMER:dbCAN-sub. Все найденные хотя бы одним из алгоритмов белки, распознанные как гликозил-гидролазы семейств GH1–GH173 или как неклассифицированные гликозил-гидролазы (условное семейство GH0 или GH_NC), проверяли вручную на предмет принадлежности семействам GH1–GH180. Список обсуждаемых в работе гликозил-гидролаз штамма Pla2T приведен во вспомогательной таблице S1. По алгоритму blastp на сайте NCBI (http://www.ncbi.nlm.nih.gov/) проводили скрининг пула белков, закодированных в штамме Pla2T, с помощью 37 планктомицетных белков из 24 семейств: GH1, GH3, GH9, GH17, GH23, GH25, GH26, GH35, GH55, GH62, GH65, GH73, GH76, GH79, GH88, GH92, GH104, GH108, GH125, GH133, GH135, GH136, GH137 и GH144.
Оценка фенотипических характеристик. Определение ростовых характеристик изолята Pla2T проводили при культивировании в жидкой среде, указанной выше, во флаконах на шейкере (120 об./мин) в диапазоне температур от 4 до 35°С (с шагом в 5°С), значений рН от 4 до 8 (шаг 0.5) и концентрации NaCl в среде 0–3.0% (шаг 1%). Окислительное и ферментативное использование углеводов определяли с использованием набора API 20NE kit (“bioMérieux”). Энзиматическую активность проверяли с помощью набора API ZYM (“bioMérieux”). Спектр используемых источников углерода определяли на минеральной среде MM следующего состава (г/л дистиллированной воды): KH2PO4 ‒ 0.1; (NH4)2SO4 ‒ 0.1; MgSO4 · 7H2O ‒ 0.1; CaCl2 · 2H2O – 0.02; 1 мл раствора “44” (Staley et al., 1992) и 1 мл витаминов № 6 (Staley et al., 1992). Культивирование проводили в 100 мл флаконах, содержащих 10 мл среды в 3-х повторностях. В качестве источников углерода проверяли сахара (глюкоза, сахароза, ксилоза, лактоза, манноза, раффиноза, мелибиоза) и полимерные соединения (фитагель, крахмал, пектин, ксилан, ксантановая камедь, хитин и микрокристаллическая целлюлоза). Тестируемые субстраты вносили в концентрации 0.5 г/л.
Эксперимент по проверке используемых источников азота проводили на жидкой среде ММ, в которой (NH4)2SO4 был заменен одним из следующих соединений в концентрации 0.01%: KNO3, NaNO2, мочевиной, дрожжевым экстрактом, N-ацетилглюкозамином, пептоном, хлоридом аммония или одной из аминокислот ‒ изолейцином, аланином, треонином, триптофаном, пролином, валином, фенилаланином, глутамином, лизином, аспарагином, аргинином, глицином.
Рост штамма Pla2T оценивали путем регулярных измерений оптической плотности культуры на спектрофотометре Eppendorf Biophotometer AG 22331 (“Eppendorf”, Германия) при длине волны 600 нм. Перед измерением культуру гомогенизировали путем активного встряхивания. Чувствительность штамма Pla2T к антибиотикам проверяли на твердой среде путем наложения на газоны культуры тест-дисков с различными антибиотиками и последующим измерением зон подавления роста.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
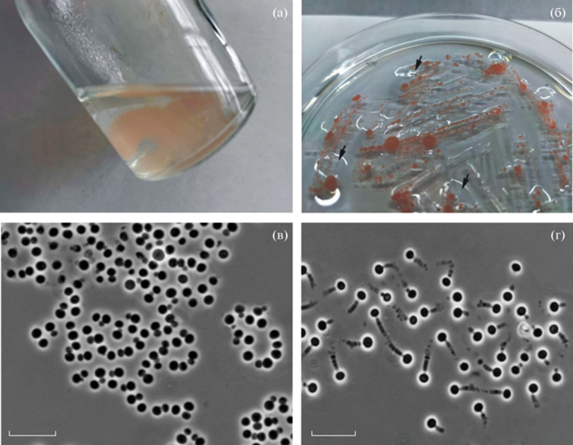
Выделение, анализ морфологии и идентификация нового изолята. Полученный из торфа низинного болота изолят Pla2T рос в жидкой среде M31 в виде слизевого агрегата розового цвета (рис. 1а). На твердой среде с фитагелем этот штамм формировал бледно-розовые колонии плотной консистенции, несколько погруженные в толщу среды (рис. 1б). Аналогичный эффект деградации фитагеля вокруг формирующихся колоний был ранее описан для Paludisphaera borealis PX4T (Kulichevskaya et al., 2016). Микроскопический анализ показал, что колонии образованы шаровидными неподвижными клетками размером 1.5‒2.8 мкм, которые размножались почкованием (рис. 1в). В культуре редко встречались одиночные клетки, чаще они формировали плотные скопления в виде бесформенных агрегатов. В некоторых старых культурах (более 3-х нед. инкубации) можно было наблюдать образование внеклеточных аморфных структур типа “выростов”, которые, по всей видимости, служат для прикрепления клеток к различным поверхностям, однако условия их формирования определить не удалось (рис. 1г).
Рис. 1.
Особенности роста и морфология клеток штамма Pla2T: (а) – рост культуры в жидкой среде с формированием слизевого агрегата; (б) – рост планктомицета на твердой среде с фитагелем (колонии, погруженные в толщу среды, указаны стрелками); (в) − морфология клеток 10-суточной культуры штамма Pla2T на твердой среде; (г) – клетки со структурами – “выростами” – в старой жидкой культуре планктомицета. Маcштабные метки – 10 мкм.
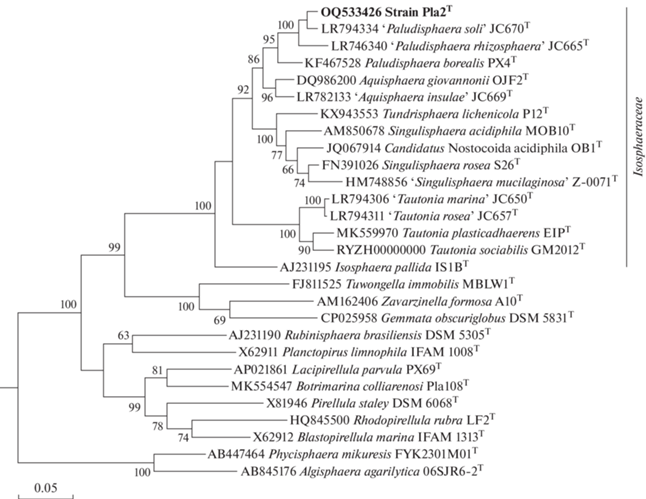
Определение и сравнительный анализ последовательности гена 16S рРНК штамма Pla2T позволили идентифицировать его в качестве представителя рода Paludisphaera (рис. 2). Ближайшим филогенетическим родственником штамма Pla2T со сходством последовательностей гена 16S рРНК в 97.9% оказался планктомицет “Paludisphaera soli” JC670Т, выделенный из почвы высокогорного участка западной цепи Гималаев (Kaushik et al., 2020). С другими представителями рода Paludisphaera сходство составляло 97.0% в случае “Paludisphaera rhizosphaera” JC665Т из ризосферы растения Erianthus ravennae (Lhingjakim et al., 2022) и 93.8% с Paludisphaera borealis PX4Т, выделенной из торфа верхового болота (Kulichevskaya et al., 2016).
Рис. 2.
Филогенетическая дендрограмма, построенная методом максимального подобия на основе сравнительного анализа нуклеотидных последовательностей гена 16S рРНК штамма Pla2T и других представителей филума Planctomycetota. В качестве внешней группы использованы нуклеотидные последовательности генов 16S рРНК пяти anammox-планктомицетов (AF375994, AF375995, AY254883, AY254882, AY257181). Показаны значения бутстрэп-анализа >60.
Физиологические характеристики. Штамм Pla2T рос в интервале pH среды от 6 до 8 (оптимум рН 5.5‒6.5) и солености среды от 0 до 2%. Температурный диапазон роста этого планктомицета составил 4‒28°С, с оптимумом при 15‒20°С. Тесты на отношение к кислороду показали, что штамм Pla2T является строго аэробным микроорганизмом. Попытки вырастить его в анаэробных условиях не увенчались успехом.
Анализ ферментативных свойств штамма Pla2T, проведенный с использованием стандартного набора API ZYM (“bioMérieux”), показал наличие активности ряда ферментов, включая, эстеразу (С4), липазу (С8), лейцин-, валин-цистин-ариламидазы, трипсин, α-химотрипсин, кислую фосфатазу и нафтол-AS-BI-фосфогидролазу. Активности щелочной фосфатазы, α- и β-глюкозидазы, α- и β-галактозидазы, β-глюкуронидазы, N-ацетил-β-глюкозаминидазы, α-маннозидазы и α-фукозидазы выявлены не были. По результатам тестов системы API NE новый планктомицет не способен к брожению и к восстановлению нитратов до нитритов и молекулярного азота.
Проверка спектра используемых субстратов показала, что предпочтительными источниками углерода и энергии штамма Pla2T являются полисахариды ксилан, фитагель, ксантановая камедь. Из простых сахаров хороший рост нового изолята был зафиксирован на глюкозе, маннозе, ксилозе и мелибиозе. В качестве источников азота планктомицет мог использовать нитрат, пептон, дрожжевой экстракт, N-ацетилглюкозамин, лизин, глицин, аспарагин, аспартат, валин, пролин, триптофан, треонин, изолейцин, аргинин.
Проверка устойчивости штамма Pla2T к ряду антибиотиков выявила устойчивость к хлорамфениколину, гентамицинину, ампициллину, канамицину, имипениму, в то же время он оказался чувствительным к тетрациклину.
Геномные характеристики. По итогам секвенирования ДНК штамма Pla2T на платформе Nanopore было получено 140 874 прочтения с общей длиной 1.4 ГБ. Дополнительный раунд секвенирования на платформе Illumina MiSeq сгенерировал в общей сложности 10 517 034 парных прочтений со средней длиной считывания 150 п.о. По итогам гибридной сборки генома удалось получить 9 контигов длиной от 15 502 п.о. до 7 626 770 п.о. Содержание пар Г + Ц в геноме штамма Pla2T составило 67 мол. % (табл. 1). Геномным аннотатором Prokka было предсказано около 6.5 тыс. потенциальных белок-кодирующих последовательностей, 3 копии оперона рРНК и 93 гена тРНК. Полученная геномная сборка не позволила точно определить количество плазмид. Однако нам удалось выявить сходство части контигов генома штамма Pla2Т с последовательностями плазмид других представителей семейства Isosphaeraceae. Наибольшее покрытие и сходство наблюдались с плазмидой PALBO1 из Paludisphaera borealis PX4Т (78.6%) и двумя плазмидами неохарактеризованного штамма SH-PL62 (84.5 и 86.9%).
Таблица 1.
Отличительные характеристики штамма Pla2Т от ранее описанных видов рода Paludisphaera
| Характеристика | Штамм Pla2Т | “P. rhizosphaerae” JC665Т | “P. soli” JC670Т | P. borealis PX4T |
|---|---|---|---|---|
| Размер клеток, мкм | 1.5−2.8 | 1.8–1.7 | 1.6–1.7 | 1.5−2.5 |
| Диапазон pH, (оптимум) | 6−8 (5.5−6) | 6–9 (7.0) | 7.0–8.0 (7.0) | 3.8–8.0 (5.0–5.5) |
| Диапазон T, °C, (оптимум) | 4−28 (15−20) | 4–34 (26–28) | 4–30 (22–25) | 4–37 (15–25) |
| Соленость, % | 0–2 (0) | 0–2 (0) | 0–2 (0) | 0–3 (0) |
| Источники углерода: | ||||
| сахароза | − | + | − | + |
| мелибиоза | + | ND | ND | − |
| лактоза | − | + | − | + |
| Источники азота: | ||||
| фенилаланин | − | + | + | − |
| лизин | + | + | + | − |
| треонин | + | + | + | − |
| глицин | + | + | + | − |
| изолейцин | + | + | + | − |
| глутамин | − | − | + | − |
| пролин | + | + | + | − |
| аспартат | + | − | − | − |
| валин | + | − | − | − |
| Ферментативная активность: | ||||
| щелочная фосфатаза | − | − | + | − |
| β-галактозидаза | − | − | − | + |
| N-ацетил-β-глюкозаминидаза | − | − | − | + |
| Размер генома, млн п.о. | 8.21 | 8.05 | 7.97 | 7.65 |
| Г + Ц, мол. % | 67 | 66.4 | 70.4 | 66.3 |
| Количество белок-кодирующих генов | 6468 | 6364 | 6389 | 5785 |
| Кластеры генов синтеза вторичных метаболитов, шт. | 6 | 6 | 6 | 5 |
| Источник выделения | Торф низинного болота | Почва ризосферы | Почва высокогорья | Торф верхового болота |
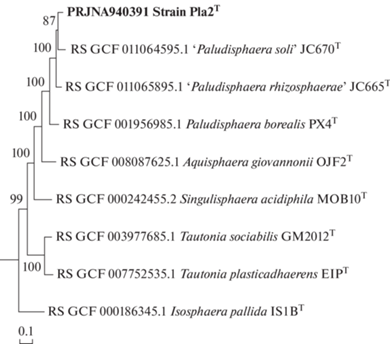
ДНК‒ДНК гибридизация in silico выявила следующие величины сходства штамма Pla2Т с тремя представителями рода Paludisphaera: 24.4 ± 2.3% c “P. soli” JC670Т, 22.0 ± 2.3% c “P. rhizosphaerae” JC665Т и 21.0 ± 2.3% c P. borealis PX4Т. Согласно калькулятору ANI (average nucleotide identity) сходство последовательности генома Pla2Т с таковыми у представителей рода Paludisphaera составило 82.6% (“P. soli” JC670Т), 80.8% (“P. rhizosphaera” JC665Т) и 79.4% (P. borealis PX4T). Согласно ныне принятым стандартам, такие величины ANI свидетельствуют о принадлежности изолята к новому виду (Chun et al., 2018). Аналогичное заключение позволил сделать и филогеномный анализ, согласно которому штамм Pla2Т образует общий кластер с другими представителями рода Paludisphaera в семействе Isosphaeraceae (рис. 3).
Рис. 3.
Филогеномная дендрограмма, построенная на основании результатов сравнительного анализа 120 конкатенированных последовательностей консервативных маркерных белков штамма Pla2T и других представителей семейства Isosphaeraceae. В качестве внешней группы использованы геномы anammox-планктомицетов, доступные в базе данных GTDB (Parks et al., 2022).
Гены, кодирующие основные метаболические пути хемоорганотрофных бактерий, такие как гликолиз, ЦТК, пентозо-фосфатный путь и окислительное фосфорилирование, присутствуют в геноме штамма Pla2Т. У этого планктомицета имеется геномный потенциал для синтеза всех аминокислот. Большинство генов, ответственных за хемотаксис, включая cheB, cheR и cheW, присутствуют в геноме Pla2Т.
Анализ генов, кодирующих синтез вторичных метаболитов. Геномы планктомицетов кодируют большое количество генных кластеров, отвечающих за синтез вторичных метаболитов. Данное свойство делает эту группу микроорганизмов потенциально интересным объектом для биотехнологии (Wiegand et al., 2018). С помощью программы Antismash в геноме штамма Pla2Т был произведен поиск таких генных кластеров. По итогам анализа в геноме было выявлено 6 кластеров, содержащих гены, потенциально кодирующие синтез вторичных метаболитов. Из них три кластера отвечали за синтез поликетидсинтаз (PKS), один кластер отвечал за синтез индола и два за синтез терпенов. Поликетидсинтазы ‒ ключевые ферменты синтеза биологически-активных веществ поликетидов (Donadio et al., 2007). Ближайшими гомологами PKS штамма Pla2Т были таковые у планктомицетов P. borealis PX4Т (100% сходства) и I. pallida ATCC43644Т (16% сходства), а также цианобактерии Scytonema hofmannii (8% сходства).
Индол повсеместно распространен в природе и представляет собой азотсодержащее гетероциклическое ароматическое соединение. У бактерий индол влияет на образование спор, вирулентность и формирование биопленок. В промышленности используется как усилитель вкуса и отдушка (Ferrer et al., 2022). Ближайшим гомологом индольного генного кластера у Pla2Т был обнаруженный у планктомицета Aquisphaera giovannonii OF2Т. Терпены представляют собой структурно наиболее разнообразный класс природных соединений. Их структурное многообразие привело к широкому диапазону функциональных ролей, к которым относятся витамины, каротиноиды, гормоны, ароматические вещества и защитные метаболиты (Helfrich et al., 2019). Для двух терпеновых кластеров Pla2T гомологичным был генный кластер неохарактеризованного планктомицета семейства Isosphaeraceae штамма SH-PL62 со сходством 50 и 100%.
Репертуар гликозил-гидролаз, закодированных в геноме штамма Pla2T. Поиск потенциальных гликозил-гидролаз с помощью сервера dbCAN3 и дальнейшая их верификация позволили обнаружить 108 белков, принадлежащих 40 семействам: GH2 (2 белка), GH5 (11), GH10 (3), GH13 (11), GH15 (2), GH16 (3), GH18 (2), GH20 (1), GH27 (2), GH29 (3), GH32 (3), GH33 (4), GH36 (2), GH38 (2), GH39 (4), GH43 (5), GH50 (1), GH51 (2), GH57 (4), GH77 (1), GH78 (3), GH94 (2), GH95 (1), GH97 (1), GH99 (1), GH106 (1), GH116 (1), GH127 (5), GH130 (2), GH140 (2), GH141 (2), GH146 (1), GH148 (3), GH151 (1), GH163 (2), GH165 (1), GH171 (1), GH172 (3), GH176 (1) и GH177 (6). Сравнение этого списка семейств с размещенными на сайте CAZy списками семейств, представленных у шести планктомицетов семейства Isosphaeraceae (A. giovannonii OJF2T, I. pallida ATCC 43644T, P. borealis PX4T, S. acidiphila DSM 18658T, T. plasticadhaerens ElPT и штамма SH-PL62), выявило 24 семейства гликозил-гидролаз, представители которых не были обнаружены алгоритмами dbCAN3 у штамма Pla2T. 37 соответствующих планктомицетных белков были использованы нами в качестве запросов для скрининга пула белков штамма Pla2T, его результаты не позволили обнаружить близких гомологов ни в одном из случаев.
Семейства GH27 и GH36 гликозил-гидролаз находятся в близком родстве (клан GH-D) и объединяют почти исключительно ферменты, обладающие α-D-галактозидазной (КФ 3.2.1.22) активностью (Наумов, 2004, 2011). Гены белков этих двух семейств крайне редко обнаруживаются в геномах планктомицетов. Среди 101 генома, представленных в базе данных CAZy, лишь в 11 закодированы белки семейства GH27 и в 16 – GH36. В семействе Isosphaeraceae только у штамма A. giovannonii OJF2T закодирован белок семейства GH27 (GenPept, QEH34490.1). Он на 48% и 28% идентичен с белками Pla2_02372 и Pla2_03258 штамма Pla2T (между собой они имеют 30% идентичности). Белки Pla2_03686 и Pla2_05159 семейства GH36 из штамма Pla2T имеют 41% и 37% идентичности на N- и C-концевом фрагментах, которые у второго разделены длинной инсерцией. Оба белка принадлежат одному подсемейству GH36A. Низкий уровень сходства аминокислотных последовательностей позволяет сделать вывод, что все пять генов α-галактозидаз попали в планктомицеты семейства Isosphaeraceae в результате независимых горизонтальных переносов. Накопление четырех α-галактозидазных генов у штамма Pla2T явно имеет адаптивное значение, природа которого пока остается неясной. Проведенные нами тесты позволили подтвердить способность Pla2T к росту на мелибиозе, аналогичные данные имеются в литературе для штамма A. giovannonii OJF2T (Bondoso et al., 2011).
Семейство GH10 гликозил-гидролаз представлено у планктомицетов двумя четко обособленными подсемействами (Наумов, 2016; Наумов и др., 2022). Гены представителей одного из них закодированы в каждом геноме в одной копии, на филогенетическом древе они образуют компактный кластер, а в аминокислотных последовательностях отсутствуют два каталитически важных остатка Glu, что указывает на отсутствие у них какой-либо ферментативной активности. К этому подсемейству относится белок Pla2_04228 из штамма Pla2T. Гены второго подсемейства закодированы лишь в редких штаммах планктомицетов, но обычно представлены в нескольких копиях. Сюда относятся белки Pla2_03346 и Pla2_05951 из штамма Pla2T, имеющие 71% идентичности аминокислотных последовательностей и сохранные каталитически значимые остатки. Они, вероятно, обладают эндо-β-ксиланазной активностью и частично ответственны за способность штамма Pla2T к росту на ксилане.
Семейство GH32 гликозил-гидролаз содержит разнообразные β-фруктозидазы. Гены его белков закодированы в 43 из 101 генома планктомицетов, представленного в базе данных CAZy. Ранее нами было предложено разбиение этого семейства на несколько подсемейств (Naumoff, 2001). Белки Pla2_05839 и Pla2_05968 имеют 38% идентичности, содержат оба каталитически важных остатка и относятся к подсемейству GH32b, для бактериальных представителей которого характерно наличие эндо-β-фруктозидазных активностей (расщепление полимеров фруктозы). Белок Pla2_05993 не относится ни к одному из ранее выделенных подсемейств, но находится в близком родстве (71% идентичности) с белком из P. borealis PX4T (GenPept, APW62066.1), для которого нами (Ivanova et al., 2017) на основании необычного строения предполагаемого активного центра было сделано предположение о вероятном отсутствии каталитической активности. Низкий уровень сходства трех рассмотренных белков штамма Pla2T указывает на независимую эволюционную историю, исключающую недавнюю дупликацию их генов.
Проведенный нами сравнительный анализ морфологических, физиолого-биохимических и генетических характеристик нового изолята Pla2T с описанными ранее у представителей рода Paludisphaera позволил выявить ряд различий, приведенных в табл. 1. Новый штамм филоге-нетически наиболее близок к выделенному из почвы планктомицету “Paludisphaera soli” JC670T, но, в отличие от последнего, имеет оптимум роста при 15‒20°С и предпочитает условия с более низкими значениями pH. Также имеется ряд различий в способности к росту на разных источниках углерода и азота. Отличия в температурном и рН-диапазонах роста, а также ростовых субстратах наблюдались и с “P. rhizosphaerae” JC665Т. Значения ДНК‒ДНК гибридизации и калькулятора ANI соответствуют стандартам таксономической классификации (Chun et al., 2018) для отнесения штамма Pla2T к новому виду рода Paludisphaera.
Диагноз нового вида – Paludisphaera mucosa sp. nov.
Paludisphaera mucosa sp. nov. (mu.co′sa. L. fem. adj. mucosa слизистый). Шаровидные неподвижные клетки диаметром 1.5–2.8 мкм, одиночные или объединенные в бесформенные скопления. Колонии розово-пигментированные. В жидкой культуре растут в виде слизевого агрегата. Облигатные аэробы. Психротолерантные мезофилы и нейтрофилы с оптимумом роста при 15‒20°С и рН 5.5‒6. Предпочтительные ростовые субстраты ‒ полисахариды, в том числе ксилан, фитагель и ксантановая камедь. Хороший рост наблюдается на глюкозе, ксилозе, маннозе и мелибиозе. Обнаружены активности ряда ферментов: эстеразы (С4), липазы (С8), лейцин-, валин-цистин-арил-амидазы, трипсина, α-химотрипсина, кислой фосфатазы и нафтол-AS-BI-фосфогидролазы. Активности щелочной фосфатазы, α- и β-глюкозидазы, α- и β-галактозидазы, β-глюкуронидазы, N-ацетил-β-глюкозаминидазы, α-маннозидазы и α-фукозидазы не выявлены. Типовым штаммом вида является штамм Pla2T (=KCTC92668T = VKM B-3698T). Местообитания представителей вида – низинные болота.
Список литературы
Наумов Д.Г. Филогенетический анализ α-галактозидаз семейства GH27 // Мол. биология. 2004. Т. 38. С. 463–476.
Naumoff D.G. Phylogenetic analysis of α-galactosidases of the GH27 family // Mol. Biol. (Moscow). 2004. V. 38. P. 388–399. https://doi.org/10.1023/B:MBIL.0000032210.97006.de
Наумов Д.Г. Иерархическая классификация гликозил-гидролаз // Биохимия. 2011. Т. 76. С. 764–780.
Naumoff D.G. Hierarchical classification of glycoside hydrolases // Biochemistry (Moscow). 2011. V. 76. P. 622–635. https://doi.org/10.1134/S0006297911060022
Наумов Д.Г. Семейство GH10 гликозилгидролаз: структура и эволюционные связи // Мол. биология. 2016. Т. 50. С. 151–160.
Naumoff D.G. GH10 family of glycoside hydrolases: structure and evolutionary connections // Mol. Biol. (Moscow). 2016. V. 50. P. 132–140. https://doi.org/10.1134/S0026893315060205
Наумов Д.Г., Куличевская И.С., Дедыш С.Н. Генетические детерминанты утилизации ксилана у планктомицета класса Phycisphaerae, Humisphaera borealis M1803T // Микробиология. 2022. Т. 91. С. 300–311.
Naumoff D.G., Kulichevskaya I.S., Dedysh S.N. Genetic determinants of xylane utilization in Humisphaera borealis M1803T, a planctomycete of the class Phycisphaerae // Microbiology (Moscow). 2022. V. 91. P. 249–258. https://doi.org/10.1134/S002626172230004X
Bondoso J., Albuquerque L., Nobre M.F., Lobo-da-Cunha A., da Costa M.S., Lage O.M. Aquisphaera giovannonii gen. nov., sp. nov., a planctomycete isolated from a freshwater aquarium // Int. J. Syst. Evol. Microbiol. 2011. V. 61. P. 2844–2850. https://doi.org/10.1099/ijs.0.027474-0
Chaumeil P.-A., Mussig A.J., Hugenholtz P., Parks D.H. GTDB-Tk: a toolkit to classify genomes with the Genome Taxonomy Database // Bioinformatics. 2020. V. 36. P. 1925–1927. https://doi.org/10.1093/bioinformatics/btz848
Chun J., Oren A., Ventosa A., Christensen H., Arahal D.R., da Costa M.S. et al. Proposed minimal standards for the use of genome data for the taxonomy of prokaryotes // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 461–466. https://doi.org/10.1099/ijsem.0.002516
Dedysh S.N., Beletsky A.V., Ivanova A.A., Kulichevskaya I.S., Suzina N.E., Philippov D.A. et al. Wide distribution of Phycisphaera-like planctomycetes from WD2101 soil group in peatlands and genome analysis of the first cultivated representative // Environ. Microbiol. 2020. V. 23. P. 1510–1526. https://doi.org/10.1111/1462-2920.15360
Dedysh S.N., Ivanova A.A. Planctomycetes in boreal and subarctic wetlands: diversity patterns and potential ecological functions // FEMS Microbiol. Ecol. 2019. V. 95. Art. fiy227. https://doi.org/10.1093/femsec/fiy227
Donadio S., Monciardini P., Sosio M. Polyketide synthases and nonribosomal peptide synthetases: the emerging view from bacterial genomics // Nat. Prod. Rep. 2007. V. 24. P. 1073–1109. https://doi.org/10.1039/b514050c
Drula E., Garron M.-L., Dogan S., Lombard V., Henrissat B., Terrapon N. The carbohydrate-active enzyme database: functions and literature // Nucl. Acids Res. 2022. № Database issue (50). P. D571–D577. https://doi.org/10.1093/nar/gkab1045
Ferrer L., Mindt M., Suarez-Diez M., Jilg T., Zagorščak M., Lee J.H. et al. Fermentative indole production via bacterial tryptophan synthase alpha subunit and plant indole-3-glycerol phosphate lyase enzymes // J. Agric. Food Chem. 2022. V. 70. P. 5634–5645. https://doi.org/10.1021/acs.jafc.2c01042
Giovannoni S.J., Schabtach E., Castenholz R.W. Isosphaera pallida, gen. and comb. nov., a gliding, budding eubacterium from hot springs // Arch. Microbiol. 1987. V. 147. P. 276–284. https://doi.org/10.1007/BF00463488
Göker M., Cleland D., Saunders E., Lapidus A., Nolan M., Lucas S. et al. Complete genome sequence of Isosphaera pallida type strain (IS1B) // Stand. Genom. Sci. 2011. V. 4. P. 63–71. https://doi.org/10.4056/sigs.1533840
Helfrich E.J.N., Lin G.M., Voigt C.A., Clardy J. Bacterial terpene biosynthesis: challenges and opportunities for pathway engineering // Beilstein J. Org. Chem. 2019. V. 15. P. 2889–2906. https://doi.org/10.3762/bjoc.15.283
Ivanova A.A., Naumoff D.G., Miroshnikov K.K., Liesack W., Dedysh S.N. Comparative genomics of four Isosphaeraceae planctomycetes: a common pool of plasmids and glycoside hydrolase genes shared by Paludisphaera borealis PX4T, Isosphaera pallida IS1BT, Singulisphaera acidiphila DSM 18658T, and strain SH-PL62 // Front. Microbiol. 2017. V. 8. P. 412. https://doi.org/10.3389/fmicb.2017.00412
Kanehisa M., Sato Y., Morishima K. BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences // J. Mol. Biol. 2016. V. 428. P. 726–731. https://doi.org/10.1016/j.jmb.2015.11.006
Kaushik R., Sharma M., Gaurav K., Jagadeeshwari U., Shabbir A., Sasikala C. et al. Paludisphaera soli sp. nov., a new member of the family Isosphaeraceae isolated from high altitude soil in the Western Himalaya // Antonie van Leeuwenhoek. 2020. V. 113. P. 1663–1674. https://doi.org/10.1007/s10482-020-01471-w
Kovaleva O.L., Elcheninov A.G., Toshchakov S.V., Novikov A.A., Bonch-Osmolovskaya E.A., Kublanov I.V. Tautonia sociabilis gen. nov., sp. nov., a novel thermotolerant planctomycete, isolated from a 4000 m deep subterranean habitat // Int. J. Syst. Evol. Microbiol. 2019. V. 69. P. 2299–2304. https://doi.org/10.1099/ijsem.0.003467
Kulichevskaya I.S., Ivanova A.A., Detkova E.N., Rijpstra W.I.C., Sinninghe Damsté J.S., Dedysh S.N. Tundrisphaera lichenicola gen. nov., sp. nov., a psychrotolerant representative of the family Isosphaeraceae from lichen-dominated tundra soils // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 3583–3589. https://doi.org/10.1099/ijsem.0.002172
Kulichevskaya I.S., Ivanova A.A., Suzina N.E., Rijpstra W.I.C., Damsté J.S.S., Dedysh S.N. Paludisphaera borealis gen. nov., sp. nov., a hydrolytic planctomycete from northern wetlands, and the proposal of Isosphaeraceae fam. nov. // Int. J. Syst. Evol. Microbiol. 2016. V. 66. P. 837–844. https://doi.org/10.1099/ijsem.0.000799
Kulichevskaya I.S., Ivanova A.O., Baulina O.I., Bodelier P.L.E., Damsté J.S.S., Dedysh S.N. Singulisphaera acidiphila gen. nov., sp. nov., a non-filamentous, Isosphaera-like planctomycete from acidic northen wetlands // Int. J. Syst. Evol. Microbiol. 2008. V. 58. P. 1186–1193. https://doi.org/10.1099/ijs.0.65593-0
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X : molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. P. 1547–1549. https://doi.org/10.1093/molbev/msy096
Lane D.J. 16S/23S rRNA sequencing // Nucleic Acid Techniques in Bacterial Systematic / Eds. Stackebrandt E., Goodfellow M., New York: John Wiley & Sons, 1991. P. 115–175.
Lhingjakim K.L., Smita N., Kumar G., Jagadeeshwari U., Ahamad S., Sasikala C. et al. Paludisphaera rhizosphaereae sp. nov., a new member of the family Isosphaeraceae, isolated from the rhizosphere soil of Erianthus ravennae // Antonie van Leeuwenhoek. 2022. V. 115. P. 1073–1084. https://doi.org/10.1007/s10482-022-01758-0
Medema M.H., Blin K., Cimermancic P., De Jager V., Zakrzewski P., Fischbach M.A. et al. AntiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences // Nucl. Acids Res. 2011. V. 39 (Web Server issue) P. W339–W346. https://doi.org/10.1093/nar/gkr466
Moore E.K., Villanueva L., Hopmans E.C., Rijpstra W.I.C., Mets A., Dedysh S.N. et al. Abundant trimethylornithine lipids and specific gene sequences are indicative of planctomycete importance at the oxic/anoxic interface in Sphagnum-dominated northern wetlands // Appl. Environ. Microbiol. 2015. V. 81. P. 6333–6344. https://doi.org/10.1128/AEM.00324-15
Naumoff D.G. β-Fructosidase superfamily: homology with some α-L-arabinases and β-D-xylosidases // Proteins. 2001. V. 42. P. 66–67. https://doi.org/10.1002/1097-0134(20010101)42:1<66::AID- PROT70>3.0.CO;2-4
Seemann T. Prokka: rapid prokaryotic genome annotation // Bioinformatics. 2014. V. 30. P. 2068–2069. https://doi.org/10.1093/bioinformatics/btu153
Serkebaeva Y.M., Kim Y., Liesack W., Dedysh S.N. Pyrosequencing-based assessment of the bacteria diversity in surface and subsurface peat layers of a northern wetland, with focus on poorly studied phyla and candidate divisions // PloS One. 2013. V. 8. P. e63994. https://doi.org/10.1371/journal.pone.0063994
Staley J.T., Fuerst J.A., Giovannoni S., Schlesner H. The order Planctomycetales and the genera Planctomyces, Pirellula, Gemmata, and Isosphaera // The Prokaryotes / Eds. Balows A. et al. New York, NY: Springer New York, 1992. P. 3710–3731.
Wick R.R., Judd L.M., Gorrie C.L., Holt K.E. Unicycler: resolving bacterial genome assemblies from short and long sequencing reads // PLoS Comput. Biol. 2017. V. 13. P. e1005595. https://doi.org/10.1371/journal.pcbi.1005595
Wiegand S., Jogler M., Jogler C. On the maverick Planctomycetes // FEMS Microbiol. Rev. 2018. V. 42. P. 739–760. https://doi.org/10.1002/adsc.201
Дополнительные материалы
- скачать ESM.zip
- Приложение 1.
Таблица S1. Гликозил-гидролазы штамма Pla2T, проанализированные в работе
Инструменты
Микробиология


