БИОФИЗИКА, 2020, том 65, № 1, с. 29-35
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.323
ДИНАМИЧЕСКИЕ И СТАТИСТИЧЕСКИЕ СВОЙСТВА КИНКОВ ДНК
© 2020 г. Л.А. Краснобаева*, **, Л.В. Якушевич***
*Сибирский государственный медицинский университет, 634050, Томск, Московский тракт, 2
**Томский государственный университет, 634050, Томск, пр. Ленина, 36
E-mail: kla1983@mail.ru
***Институт биофизики клетки Российской академии наук - обособленное подразделение Федерального
исследовательского центра «Пущинский научный центр биологических исследований Российской академии наук»,
142290, Пущино Московской области, ул. Институтская, 3
E-mail: yakushev@icb.psn.ru
Поступила в редакцию 04.05.2019 г.
После доработки 28.10.2019 г.
Принята к публикации 11.11.2019 г.
Конформационная подвижность - одно из важнейших свойств молекулы ДНК. Ярким примером
этой подвижности является образование локально расплетенных участков двойной спирали,
называемых открытыми состояниями ДНК. Такие состояния играют важную роль в процессах
транскрипции, репликации, денатурации. В
«нерелятивистском» приближении открытые
состояния ДНК часто моделируют как квазичастицы - кинки, обладающие определенной массой
(mk), скоростью (uk) и энергией покоя (E0k). Если в молекуле ДНК образовалось не одно, а N
открытых состояний, то правомерно поставить вопрос о статистике ансамбля из N кинков ДНК.
Статистические свойства такого ансамбля до сих пор остаются малоизученными. В настоящей
работе мы исследуем эти свойства, опираясь на полученные недавно данные о динамических
характеристиках кинков ДНК. Предположив, что число кинков (N) фиксировано, взаимодействие
между кинками мало и все кинки одинаковые, мы рассчитали статистическую сумму (Zk),
свободную энергию (Fk), функцию распределения скоростей (ρ1(υk), среднюю энергию (εk),
теплоемкость (Cv,k) и энтропию (Sk) ансамбля из N кинков ДНК, а также построили и сравнили
между собой графики температурной зависимости этих характеристик для четырех однородных
последовательностей poly(A), poly(T), poly(G), poly(С) и для последовательности плазмиды pBR322.
Ключевые слова: кинки ДНК, статистика ансамбля кинков, статистическая сумма, свободная энер-
гия, энтропия, плазмида pBR322.
DOI: 10.31857/S0006302920010044
Изучая внутреннюю конформационную по-
зи между комплементарными основаниями разо-
движность молекулы ДНК, исследователи часто
рваны [7, 8].
используют инструменты теоретической физики
Если молекула ДНК достаточно длинная, в
и нелинейной математики, включая представле-
ней могут образоваться не одно, а несколько не-
ние о кинках, антикинках и бризерах, которые
линейных конформационных возмущений и пра-
вомерно поставить вопрос о статистических
моделируют нелинейные конформационные воз-
свойствах такого ансамбля. Статистическая зада-
мущения, возникающие в двойной спирали ДНК
ча особенно важна и интересна, поскольку слу-
в результате воздействия температуры, столкно-
жит ключом к интерпретации эксперименталь-
вений с молекулами раствора, действия радиа-
ных данных по рассеянию нейтронов или света
ции, взаимодействия с белками [1-4]. В частно-
молекулой ДНК [9], а также данных по денатура-
сти, бризеры оказались достаточно эффективным
ции ДНК [10]. В случае бризеров такая задача уже
математическим инструментом в исследованиях
ставилась и решалась [2, 11]. В случае кинков
процессов денатурации ДНК [5, 6], а кинки - в
вопрос о статистике все еще остается мало изу-
исследованиях динамики открытых состояний
ченным.
ДНК, называемых также пузырьками (bubbles) и
Так, в монографии [12] ставился вопрос о ста-
представляющих собой локально расплетенные
тистике кинков ДНК, а в работе [9] рассматрива-
участки двойной спирали длиной около десяти
лась возможность применения статистических
пар оснований, внутри которых водородные свя-
свойств кинков для расчета динамического
29
30
КРАСНОБАЕВА, ЯКУШЕВИЧ
Таблица 1. Параметры модели (3)
Вид цепочки
I
×10-44, кг · м2
K′
×10-18, Дж
V
×10-20, Н/м
a ×10-10, м
poly(A)
7.61
2.35
2.09
3.4
poly(T)
4.86
1.61
1.43
3.4
poly(G)
8.22
2.27
3.12
3.4
poly(C)
4.11
1.54
2.12
3.4
pBR322
6.14
1.93
2.21
3.4
форм-фактора рассеяния нейтронов на ДНК.
МОДЕЛЬ И ПАРАМЕТРЫ
Однако отсутствие в то время полного набора
В КВАЗИОДНОРОДНОМ ПРИБЛИЖЕНИИ
данных о динамических параметрах ДНК не дало
Для моделирования внутренней динамики мо-
авторам возможности в полной мере применить
лекулы ДНК воспользуемся уравнением синус-
полученные результаты к реальным последова-
Гордона:
тельностям ДНК. Появление в литературе полно-
го набора данных [13, 14] позволяет сейчас по-но-
Iφtt = K′a2φzz - Vsinφ,
(1)
вому подойти к решению задачи о статистике ан-
где φ - угловое отклонение азотистого основа-
самбля кинков ДНК. Кроме того, за это время
ния; I - момент инерции; K′ - константа, харак-
заметно изменилась и сама динамическая модель
теризующая крутильную жесткость сахаро-фос-
ДНК, и методы исследования динамики кинков
фатной цепочки; V - константа, характеризую-
[15-19]. Они позволяют в «нерелятивистском»
щая взаимодействие между основаниями внутри
приближении моделировать кинки как квазича-
пар; a - расстояние между ближайшими парами
стицы, обладающие определенной массой, ско-
оснований.
ростью и энергией покоя, движущиеся в потен-
В квазиоднородном приближении коэффици-
циальном поле ДНК с профилем, полностью
енты уравнения (1) усредняют по всей длине по-
определяемом последовательностью оснований
следовательностей ДНК [13, 20]:
[20, 21].
I
=
I c
+
I c
+
I c
+
I c ,
A A
T T
G G
C C
В настоящей работе исследуется вопрос о ста-
K′=K′
c
+
K′c
+
K′
c
+
K′c
,
(2)
A A
T T
G G
C C
тистике ансамбля кинков ДНК с учетом новых
данных о динамических характеристиках ДНК.
V =V
A A
c
+
V
cT T
+
V
cG G
+
V
cC C
,
Для определенности все расчеты выполняются
где cj = nj/n - концентрация оснований j-го типа
для кинков, активированных в плазмиде pBR322,
(j = А, Т, G, С); nj - количество азотистых основа-
которая представляет собой небольшую кольце-
ний j-го типа; n - общее количество оснований.
вую ДНК, широко используемую в исследовани-
С учетом соотношений (2) уравнение (1) пре-
ях генов, а ее компоненты применяются для со-
образуется к следующему виду:
здания новых плазмид [22]. Последовательность
плазмиды неоднородна. Она содержит 4361 азо-
2
Iϕ
=K′
ϕ
−
V
sinϕ
(3)
tt
zz
тистое основание, включая
983 аденинов,
1034 тиминов, 1134 гуанинов и 1210 цитозинов.
Мы рассчитали значения коэффициентов
Предположив (в первом приближении), что чис-
уравнения (3) для последовательности плазмиды
ло кинков, активированныех в этой последова-
pBR322. Результаты этих расчетов, а также полу-
тельности, фиксировано, взаимодействие между
ченные ранее данные о значениях коэффициен-
тов уравнения (3) для однородных последователь-
кинками пренебрежимо мало и все активирован-
ностей poly(A), poly(T), poly(G) и poly(С) [13]
ные кинки одинаковые, мы рассчитываем стати-
представлены в табл. 1.
стическую сумму (Zk), свободную энергию (Fk),
Решение уравнения (3) в виде кинка определя-
функцию распределения скоростей (ρ1(υk), сред-
ется формулой:
нюю энергию (εk), теплоемкость (Cv,k) и энтро-
φk(z,t) = 4arctg{exp[(γk/dk)⋅(z - υk⋅t - z0)]},
(4)
пию (Sk). Полученные результаты сравниваются с
данными о статистических свойствах ансамблей
2
1/2
где υk - скорость кинка,
d
= (K ′
/V)
- раз-
k
кинков, активированных в четырех однородных
2
2
−1/2
2
1/2
последовательностях: poly(A), poly(T), poly(G) и
мер кинка,
γ
=
(1−υ
/
C
)
,C = (K 'a
/I)
-
k
k
poly(С).
скорость звука в ДНК.
БИОФИЗИКА том 65
№ 1
2020
ДИНАМИЧЕСКИЕ И СТАТИСТИЧЕСКИЕ СВОЙСТВА КИНКОВ ДНК
31
Таблица 2. Динамические параметры кинка
Вид цепочки
mk ×10-24, кг
E0k ×10-17, Дж
dk ×10-8, м
poly(A)
0.226
0.177
0.361
poly(T)
0.211
0.121
0.361
poly(G)
0.253
0.213
0.290
poly(C)
0.186
0.145
0.290
pBR322
0.224
0.165
0.318
Гамильтониан, отвечающий уравнению (3),
родных последовательностях poly(A), poly(T),
имеет следующий вид:
poly(G) и poly(С), представлены в табл. 2.
∞
2
Из табл. 2 видно, что массы кинков, активиро-
⎡
K′a
⎤
I
2
2
dz
ванных в рассматриваемых пяти последователь-
H
=
⎢
φ
t
+
φ
z
+V(1
− φ
⎥
(5)
∫
2
2
a
ностях, соотносятся следующим образом:
−∞
⎣
⎦
Подставив решение (4) в формулу (5), вычис-
poly(G)
poly(A)
poly(pBR322)
poly(T)
poly(C)
m
k
>m
k
>m
k
>m
k
>m
k
лим полную энергию кинка ДНК:
Видно, что самой большой массой обладают
E
0k
E
=
,
(6)
кинки, активированные в poly(G), а самые «лег-
k
2
υ
кие» кинки активируются в poly(C). Из табл. 2
k
1
-
2
также следует, что энергии покоя кинков соотно-
C
сятся как:
где
E
=
8
′
- энергия покоя кинка.
0k
poly(G)
poly(A)
poly(pBR322)
poly(C)
poly(T)
E
0k
>E
0k
>E
0k
>E
0k
>E
0k
,
В «нерелятивистском» пределе, когда скорость
кинка υk мала по сравнению со скоростью звука
т.е. кинки, имеющие наибольшую энергию по-
C, формула (6) примет следующий вид:
коя, активируются в poly(G), а наименьшую - в
poly(T). В то же время размеры кинков , активи-
2
8
K′
υ
k
рованных в poly(G) и poly(C) равны, так же как и
E
k
=
E
0k
+
(7)
2
C
2
равны между собой размеры кинков, активиро-
ванных в poly(A) и poly(T), причем последние
Из формулы (7) получим массу покоя (mk), ки-
больше чем первые. Размер кинков, активиро-
нетическую (Tk) и потенциальную (Uk) энергии
ванных в плазмиде pBR322, занимает промежу-
кинка:
точное значение.
8
K′
m
=
,
(8)
k
2
C
СТАТИСТИЧЕСКИЕ ХРАРАКТЕРИСТИКИ
АНСАМБЛЯ КИНКОВ ДНК
2
8
K′
υ
k
T
=
,
(9)
k
2
Статистическая сумма и функция распределения
C
2
Гиббса. В соответствии с законами классической
U
= 8( K
′
(10)
статистической физики [23] состояние ансамбля
k
из N кинков ДНК определим набором координат
Импульс кинка запишем в виде:
и импульсов кинков:
pk = mk υk.
(11)
Q = {qk,1,qk,2,...qk,N},
(12)
Эти динамические характеристики позволяют
P = {pk,1,pk,2,...pk,N},
(13)
предложить простую модель ансамбля кинков
ДНК в виде обычной классической системы, со-
а статистическую сумму - формулой
стоящей из N взаимодействующих частиц, обла-
дающих массой mk, импульсом pk и энергией по-
E P,Q)
−
1
k
B
T
dPdQ
коя E0k.
Z
=
e
,
(14)
k
∫
N
!
(2πℏ
)N
Мы рассчитали значения основных динамиче-
ских параметров кинков плазмиды pBR322. Ре-
где kB
- постоянная Больцмана, а E(P,Q) -
зультаты этих расчетов, а также значения этих па-
раметров для кинков, активированных в одно-
полная энергия ансамбля.
БИОФИЗИКА том 65
№ 1
2020
32
КРАСНОБАЕВА, ЯКУШЕВИЧ
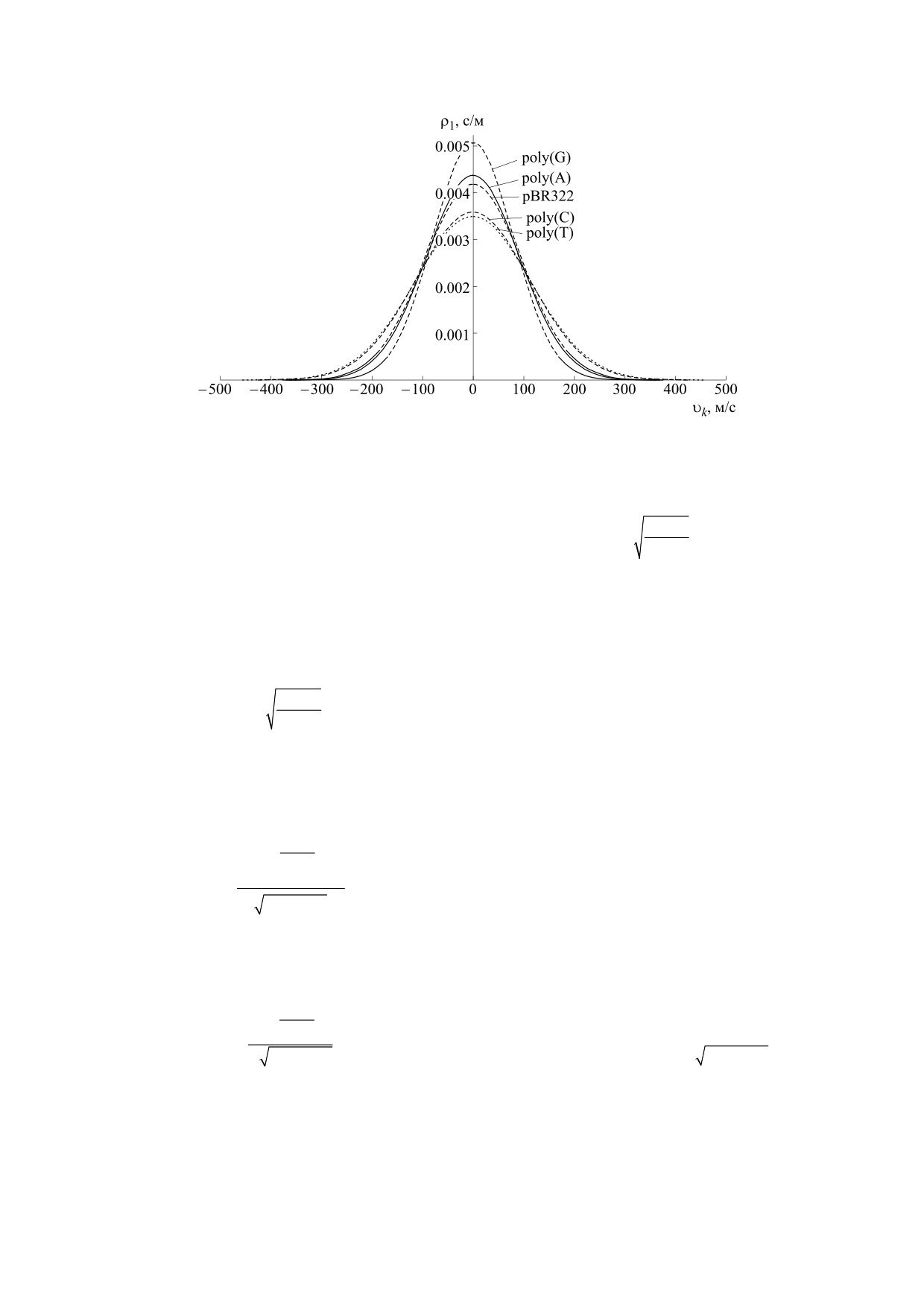
Рис. 1. Функция распределения кинков ДНК по скоростям. Расчеты выполнены при фиксированной температуре
T = 300 К для пяти видов последовательностей ДНК.
Если число кинков фиксировано, взаимодей-
⎛
2
⎞
m
υk k
−⎜
⎟
ствие между ними пренебрежимо мало и все кин-
1
m
2k
T
k
⎝
B
⎠
ρ
(υ
)
=
e
(19)
1
k
ки одинаковые, полная энергия ансамбля кинков
L
2πk
B
T
ДНК равна
С помощью рассчитанных выше динамиче-
N
2
p
ских характеристик кинков ДНК (см. табл. 2) мы
i
E P,Q)
=
NE
+
,
(15)
0k
∑ k,
построили распределения кинков по скоростям
i
2m
k
для случая плазмиды pBR322 и для четырех одно-
и формула (14) для статистической суммы ансам-
родных последовательностей: poly(A), poly(T),
бля кинков ДНК приобретает следующий вид:
poly(G) и poly(С) (рис. 1).
N
E
0k
Из рис. 1 видно, что функция распределения
N
⎛
−
⎞
L
k
B
T m
k B
kT
имеет форму колокола, самое большое значение
Z
=
⎜e
⎟
,
(16)
k
2
N
!⎜
2πℏ
⎟
высоты колокола наблюдается для кинков, акти-
⎝
⎠
вированных в последовательности poly(G), а ми-
где L - длина последовательности ДНК.
нимальное - в последовательности poly(T). Вид-
Другая важная характеристика ансамбля кин-
но также, что кривая, отвечающая распределе-
ков ДНК - функция распределения Гиббса [24]
нию скоростей кинков в плазмиде pBR322,
имеет следующий вид:
расположилась между кривыми, относящимися к
N
кинкам, активированным в poly(A) и в poly(C).
⎛
2
⎞
⎜
∑
p
k,i
⎟
i
Такое расположение кривых функций распреде-
−⎜
⎟
⎜2m
k B
k T
⎟
ления согласуется с соотношениями между зна-
⎜
⎟
⎝
⎠
e
чениями энергий покоя кинков, активированных
ρ
(P,Q)
=
(17)
k
N
в пяти рассматриваемых последовательностях
(
L 2πm kT
k B
)
(см. табл. 2).
Так как в рассматриваемом приближении кин-
Свободная энергия. Согласно законам класси-
ки ДНК являются статистически независимыми,
ческой статистической физики [25] свободная
то можно записать функцию распределении
энергия ансамбля кинков в рассматриваемом
Гиббса отдельного кинка ДНК:
приближении будет иметь следующий вид:
⎛
p
2
⎞
k
−⎜
⎟
F
=-k
T
ln
Z
=
2m
k T
k
B
k
⎝
k B
⎠
e
ρ
(
p q)
=
(18)
⎡
⎛
⎞⎤
(20)
1
k
L e
L
2π
m
kT
=N⎢E
−
k
T
ln
2π
m
kT
k B
0k
B
⎜
k B
⎟⎥
2π
ℏ
N
⎣
⎝
⎠⎦
После замены переменных
p
k
=m
k k
υ
в форму-
ле
(18), получаем функцию распределения
кинков ДНК по скоростям (распределение Макс-
Графики температурной зависимости свобод-
велла):
ной энергии ансамбля кинков, активированных в
БИОФИЗИКА том 65
№ 1
2020
ДИНАМИЧЕСКИЕ И СТАТИСТИЧЕСКИЕ СВОЙСТВА КИНКОВ ДНК
33
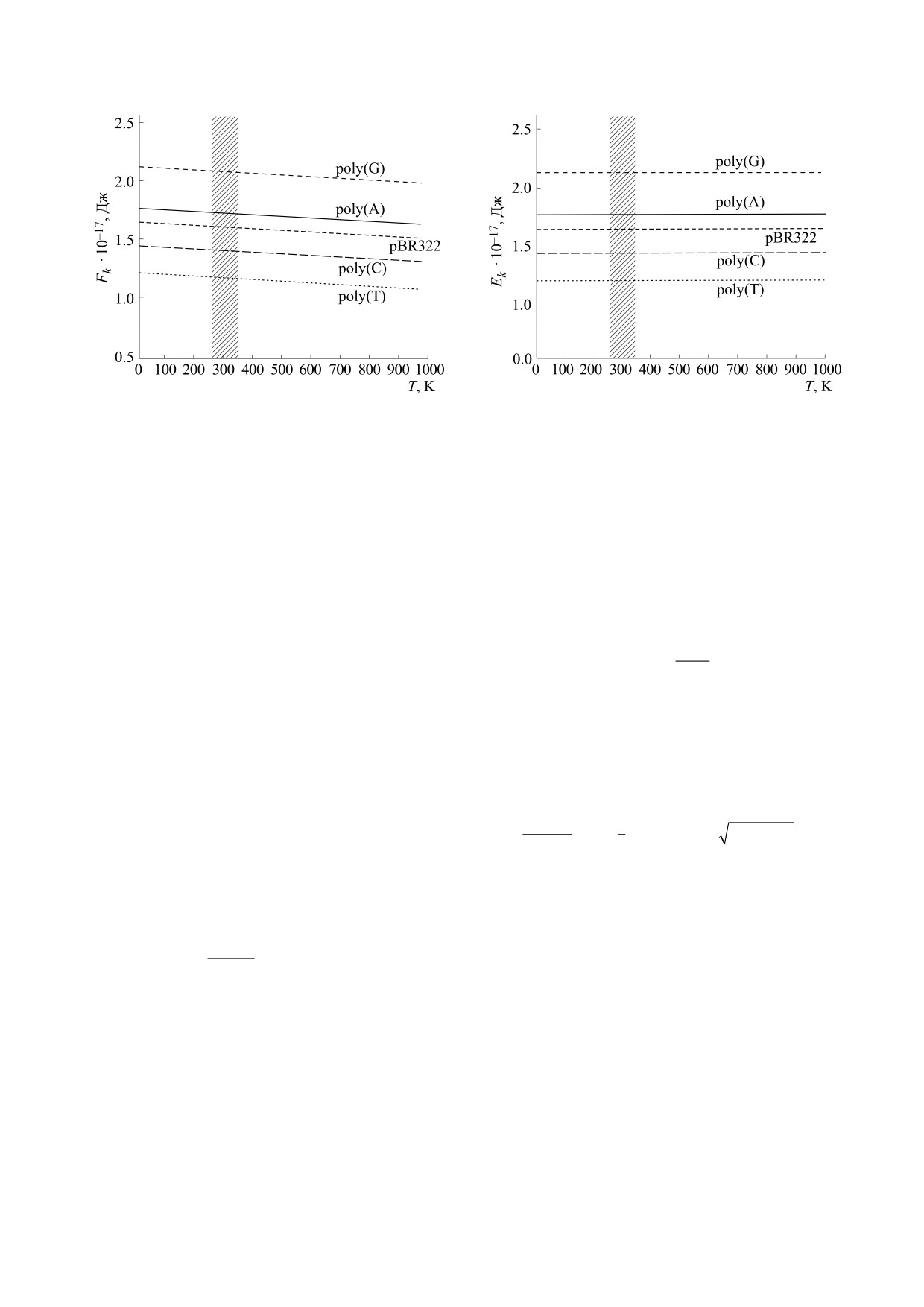
Рис. 2. Свободная энергия ансамбля из N кинков
Рис. 3. Средняя энергия ансамбля из N кинков ДНК.
ДНК. Заштрихованной полосой показан температур-
Заштрихованной полосой показан температурный
ный интервал (280…320 K), характерный для боль-
интервал (280…320 K), характерный для большинства
шинства живых систем. Расчеты сделаны для пяти
живых систем. Расчеты сделаны для пяти видов
видов последовательностей ДНК.
последовательностей ДНК.
различных последовательностях ДНК, а также в
ходит очень медленно и практически не заметно.
последовательности плазмиды pBR322, представ-
Что касается взаимного расположения кривых, то
лены на рис. 2. При проведении расчетов мы ис-
оно такое же, как и предыдущем случае.
пользовали модельные значения: L = 1000 пар ос-
Теплоемкость и энтропия. В рассматриваемом
нований и N = 10.
приближении теплоемкость ансамбля из N кин-
Из рис. 2 видно, что для всех рассматриваемых
ков ДНК равна константе:
последовательностей при повышении температу-
∂ε
Nk
ры T свободная энергия ансамбля кинков ДНК
k
В
C
ν,k
=
=
(22)
уменьшается, причем при любом фиксирован-
∂T
2
ном значении температуры самые высокие значе-
Для случая с N = 10 теплоемкость ансамбля
ния свободной энергии наблюдаются в случае po-
кинков равна Cv,k = 0.69×10-22 Дж/К. Очевидно,
ly(G), а самые низкие - в случае poly(T). Также
что это значение не зависит также и от вида по-
видно, что кривая, отвечающая свободной энер-
следовательности.
гии ансамбля кинков в плазмиде pBR322, распо-
лагается между кривыми, относящимися к ан-
Энтропия ансамбля кинков будет определять-
самблям кинков, активированных в poly(A) и в
ся формулой [27]:
poly(C), а общее расположение кривых согласует-
ε
−
F
⎡
⎛
⎞⎤
ся с соотношениями между значениями энергий
k
k
1
L e
S
=
=
N
+
ln
2πm
kT
(23)
k
⎢
⎜
k B
⎟⎥
покоя кинков в этих пяти последовательностях
k
T
2
2πℏN
B
⎣
⎝
⎠⎦
(см. табл. 2).
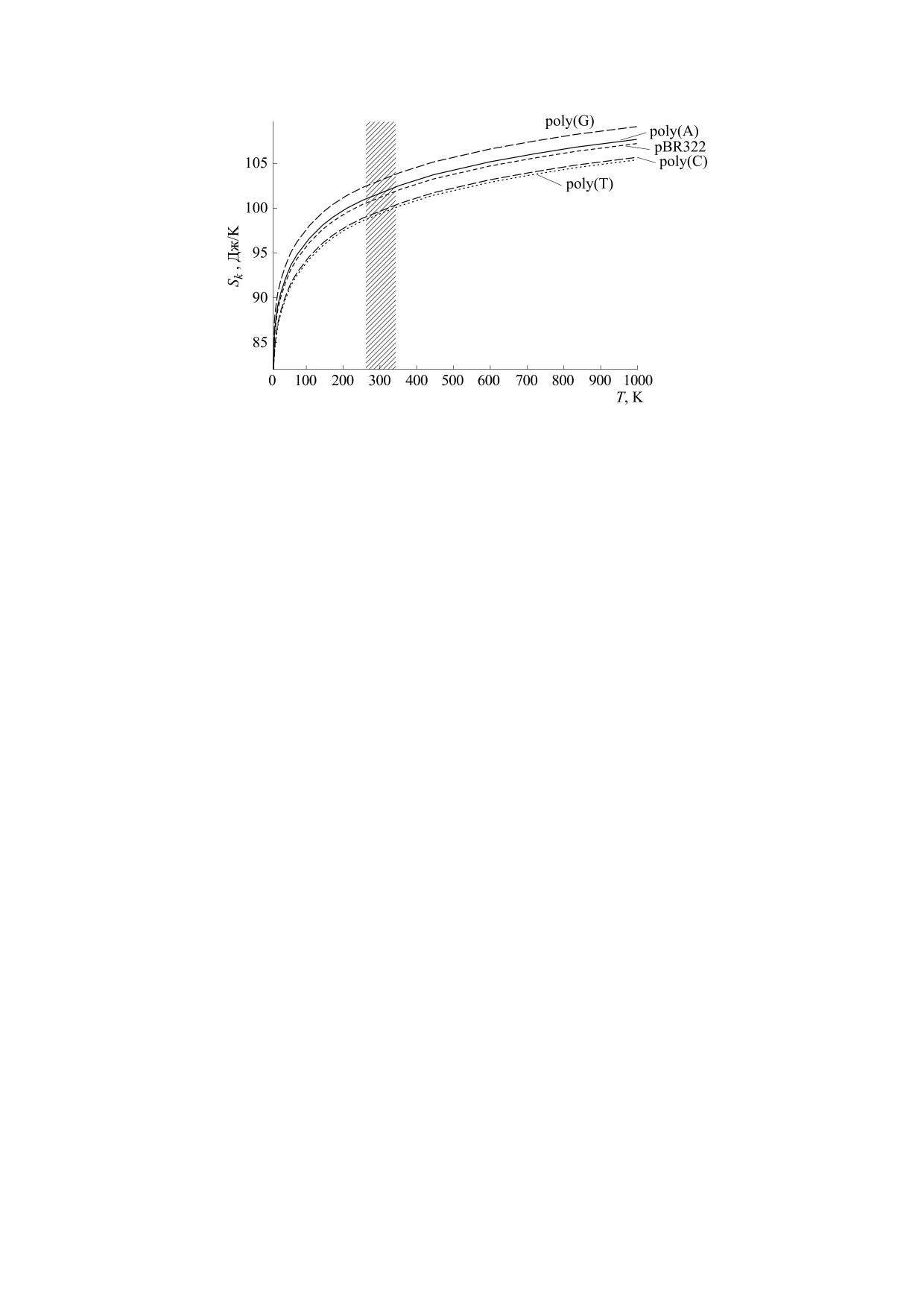
Графики температурной зависимости энтро-
Средняя энергия. Средняя энергия ансамбля из
пии ансамбля из N одинаковых кинков, активи-
N кинков ДНК определяется следующей форму-
рованных в различных последовательностях
лой [25]:
ДНК, а также в последовательности плазмиды
pBR322, представлены на рис. 4.
2
∂ln
Z
k
T
k
⎛
B
⎞
ε
=
(k
T)
=
N⎜E
+
(21)
k
B
0k
⎟
Из рис. 4 видно, что в отличие от двух преды-
∂(k
T)
⎝
2
⎠
B
дущих графиков, энтропия заметно растет с ро-
Мы построили графики температурной зави-
стом температуры для всех пяти последователь-
симости средней энергии ансамбля из N одинако-
ностей. Однако взаимное расположение кривых
вых кинков, активированных в различных после-
остается таким же, как и предыдущих случаях.
довательностях ДНК, а также в последовательно-
сти плазмиды pBR322 (см. рис. 3).
ОБСУЖДЕНИЕ И ВЫВОДЫ
В отличие от предыдущего случая средняя
энергия ансамбля растет с ростом температуры.
В данной работе были исследованы статисти-
Однако, как видно из рис. 3, в заданном интерва-
ческие свойства ансамбля кинков ДНК, получе-
ле значений температуры это повышение проис-
ны графики температурной зависимости свобод-
БИОФИЗИКА том 65
№ 1
2020
34
КРАСНОБАЕВА, ЯКУШЕВИЧ
Рис. 4. Энтропия ансамбля из N кинков ДНК. Заштрихованной полосой показан температурный интервал
(280…320 K), характерный для большинства живых систем. Расчеты сделаны для пяти видов последовательностей
ДНК.
ной энергии, средней энергии и энтропии, а так-
энергий покоя кинков, активированными в рас-
же функция распределения кинков ДНК по
сматриваемых последовательностях. Это говорит
скоростям. Эти характеристики были рассчитаны
об определяющей роли значений энергий покоя в
для случая фиксированного числа одинаковых
определении взаимного расположения кривых
кинков и при условии пренебрежимо малого вза-
статистических характеристик ансамблей кин-
имодействия между ними. В расчетах были ис-
ков, активированных в различных последова-
пользованы новые данные о динамических пара-
тельностях.
метрах кинков ДНК.
Следует отметить, однако, что все эти резуль-
Расчеты были проведены для четырех одно-
таты получены в рамках упрощенной модели
родных последовательностей: poly(A), poly(T),
ДНК, которая ограничена рассмотрением только
poly(G) и poly(С), а также для последовательно-
одного вида внутренних движений ДНК - угло-
сти плазмиды pBR322. Показано, что с ростом
вых колебаний азотистых оснований. Другие ви-
температуры свободная энергия ансамбля кинков
ды внутренних движений, такие, например, как
уменьшается, а средняя энергия и энтропия уве-
продольные и поперечные смещения оснований,
личиваются, причем на одном и том же заданном
а также движения сахаров и фосфатов, не учиты-
интервале температур увеличение энтропии про-
вались. Еще одно ограничение нашего подхода
исходит довольно заметно, а увеличение средней
связано с использованием квазиоднородного
энергии происходит очень медленно (практиче-
приближения. В этом приближении учитывается
ски незаметно). При любой фиксированной тем-
состав полинуклеотидных цепочек, но не учиты-
пературе T самые высокие значения этих трех ста-
вается расположение азотистых оснований в по-
тистических характеристик наблюдались в случае
следовательности ДНК. Тем не менее можно
poly(G), а самые низкие - в случае poly(Т). Кри-
предположить, что изложенные выше методы и
вые, относящиеся к плазмиде, располагались
подходы имеют более общее значение и могут
между кривыми, рассчитанными для poly(A) и
быть успешно использованы для исследования
poly(C). Расчеты показали, что в рассматривае-
статистических и динамических свойств молекул
мом приближении теплоемкость ансамбля кин-
в рамках более сложных и более реалистичных
ков не зависит ни от температуры, ни от вида по-
моделей ДНК.
следовательности, а функция распределения
кинков по скоростям имеет форму колокола, вы-
КОНФЛИКТ ИНТЕРЕСОВ
сота которого достигает максимума при υk = 0.
Авторы заявляют об отсутствии конфликта
Самое большое значение высоты наблюдалось в
интересов.
случае последовательности poly(G), а самое низ-
кое - в случае poly(T), причем кривая, относяща-
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
яся к плазмиде, всегда располагалась между кри-
выми, рассчитанными для poly(A) и poly(C). Та-
Настоящая работа не содержит описания ка-
кое расположение кривых на всех графиках
ких-либо исследований с использованием людей
согласуется с соотношениями между значениями
и животных в качестве объектов.
БИОФИЗИКА том 65
№ 1
2020
ДИНАМИЧЕСКИЕ И СТАТИСТИЧЕСКИЕ СВОЙСТВА КИНКОВ ДНК
35
СПИСОК ЛИТЕРАТУРЫ
14. Л. В. Якушевич и Л. А. Кpаcнобаева, Биофизика 61
(2), 236 (2016). DOI: 10.1134/S0006350908010041
1. Л. В. Якушевич, Методы теоретической физики в
15. M. Cadoni, R. De Leo, S. Demelio, and G. Gaeta, J.
исследованиях свойств биополимеров (ОНТИ НЦБИ
Nonlinear Math. Phys.
17
(4),
557
(2010).
АН СССР, Пущино, 1990).
DOI: 10.1142/S1402925110001069
2. M. Peyrard, Nonlinearity 17 (2), 1 (2004). DOI:
16. S. Vedad and A. Heidari, Progr. Appl. Math. 4 (2), 1
10.1088/0951-7715/17/2/R01
(2012). DOI: 10.3968/j.pam.19252502820120402.1520
3. Nonlinear Excitations in Biomolecules, Ed. by M. Pey-
17. S. Zdravković, M. V. Satarić, and M. Daniel, Int. J.
rard (Springer, Berlin, 1995).
Modern Phys. B
27
(31),
1350184
(2013). DOI:
4. A. Scott, Encyclopedia of Nonlinear Science (Frances
10.1142/S0217979213501841
and Taylor, New York, 2005).
18. A. Di Garbo, Biophys. Chem. 208 (1), 76 (2016). DOI:
5. M. Peyrard and A. R. Bishop, Phys. Rev. Lett. 62 (23),
10.1016/j.bpc.2015.09.006
2755 (1989).
19. L. Liu and Ch. Li, Adv. Math. Phys. 2018, 1-7 (2018).
6. T. Dauxois, M. Peyrard and A. R. Bishop, Phys. Rev. E
DOI: 10.1155/2018/4676281
47 (1), 684 (1993). DOI: 10.1103/PhysRevE.47.684
20. L. V. Yakushevich and L. A. Krasnobaeva, Math. Biol.
7. S. W. Englander, N. R. Kallenbach, A. J. Heeger, et al.,
Bioinform. 12 (1), 1 (2017). DOI: 10.17537/2017.12.1
Proc. Natl. Acad. Sci. USA 77 (12), 7222 (1980).
21. Л. А. Краснобаева и Л. В. Якушевич, Биофизика 63
8. A. A. Grinevich, A. A. Ryasik, and L. V. Yakushevich,
(1) 41 (2018). DOI: 10.1134/S0006350918010190
Chaos, Solitons & Fractals 75,
62
(2015). DOI:
10.1016/j.chaos.2015.02.009
22. F. Bolivar, R. L. Rodriguez, P. J. Greene, et al., Gene 2
(2), 95 (1977).
9. L. V. Yakushevich, J. Biol. Phys. 24 (2-4), 131 (1999).
DOI: 10.1023/A:1005143428994
23. Г. Ф. Караваев, Основные принципы статистиче-
ской физики (ТГУ, Томск, 1993).
10. G. A. Wildes, M. Marty-Roda, S. Cuesta-López, et al.,
J. Phys. Chem. B
122
(9),
2504
(2018). DOI:
24. И. А. Квасников, Термодинамика и статистиче-
10.1021/acs.jpcb.7b11608
ская физика (Эдиториал УРСС, М., 2010).
11. А. С Шигаев, О. А. Пономарёв и В. Д. Лахно, Мат.
25. И. П. Базаров, Термодинамика (Высш. шк., М.,
биология и биоинформатика 8 (2), 553 (2013). DOI:
1991).
10.17537/2018.13.t162
26. А. В. Леванов и Э. Е. Антипенко, Определение тер-
12. Л. В. Якушевич, Нелинейная физика ДНК (РХД,
модинамических свойств статистическими мето-
Москва-Ижевск, 2007).
дами. Классический идеальный газ (МГУ, М., 2006).
13. Л. В. Якушевич, Л. А. Кpаcнобаева, А. В. Шапова-
27. А. Зоммерфельд, Термодинамика и статистиче-
лов и Н. P. Кинтеpо, Биофизика 50 (3), 450 (2005).
ская физика (М., 1955).
Dynamic and Statistical Properties of DNA Kinks
L.A. Krasnobaeva*, ** and L.V. Yakushevich***
*Siberian State Medical University, Moskovsky trakt 2, Tomsk, 634050 Russia
**Tomsk State University, prosp. Lenina 36, Tomsk, 634050 Russia
***Institute of Cell Biophysics, Russian Academy of Sciences, Institutskaya ul. 3, Pushchino, Moscow Region, 142290 Russia
Conformational mobility is one of the most important properties of the DNA molecule. A striking example
of this mobility is the formation of regions of the locally unwound double helix. This motion is called the
formation of open states of DNA having an important role in the processes of transcription, replication, and
denaturation. In the “nonrelativistic” approximation, the open states of DNA are often modeled as quasipar-
ticles - kinks with a certain mass (mk), velocity (uk) and rest energy (E0k). If there is more than one open state
in the DNA molecule, the question may be raised as to the statistics of an ensemble of the N number of DNA
kinks Statistical properties of the ensemble still remain poorly studied. In this paper, we investigate these
properties based on recent data on the dynamic characteristics of the DNA kinks. It has been assumed that
the N number of kinks is fixed, the interaction between the kinks is small and all kinks are identical. We have
calculated the statistical sum (Zk), the free energy (Fk), the velocity distribution function (ρ1(υk), the average
energy (εk) , the heat capacity (Cv,k), the entropy (Sk) of ensemble of the N number of DNA kinks, as well as
obtained and compared the curves for temperature dependence relative to these characteristics for the four
homogeneous sequences: poly(A), poly(T), poly(G), poly(C), and for the sequence of plasmid pBR322.
Keywords: DNA kinks, statistics of an ensemble of kinks, statistical sum, free energy, entropy, plasmid pBR322
БИОФИЗИКА том 65
№ 1
2020