БИОФИЗИКА, 2020, том 65, № 2, с. 237-249
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.322.7,577.323.7,535.56,543.422.8
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ В СТРУКТУРНОЙ ОРГАНИЗАЦИИ
ХРОМАТИНА. 2. ВЗАИМОДЕЙСТВИЕ С ДНК И ЯДЕРНЫМИ БЕЛКАМИ
© 2020 г. Е.В. Чихиржина*, Т.Ю. Старкова*, А.М. Поляничко*, **
*Институт цитологии РАН, 194064, Санкт-Петербург, Тихорецкий просп., 4
E-mail: chikhir@gmail.com
**Санкт-Петербургский государственный университет,
199034, Санкт-Петербург, Университетская наб., 7/9
E-mail: a.polyanichko@spbu.ru
Поступила в редакцию 30.01.2019 г.
После доработки 02.10.2019 г.
Принята к публикации 24.01.2020 г.
В первой части обзора (Биофизика, 63, 1070 (2018)) были рассмотрены структура линкерных
гистонов семейства Н1, их посттрансляционные модификации, а также роль гистона Н1 в
формировании компактного транскрипционно неактивного хроматина. Вторая часть посвящена
роли линкерных гистонов семейства Н1 в структурной организации хроматина на различных
уровнях: от нуклеосом до метафазных хромосом. Обсуждаются механизмы взаимодействия гистона
Н1 с другими элементами хроматина, в том числе с ДНК и ядерными белками.
Ключевые слова: ДНК, коровые гистоны, линкерный гистон Н1, нуклеосома, структура хроматина,
HMG белки.
DOI: 10.31857/S0006302920020040
семейства Н1 в формировании различных уров-
В последние годы опубликовано большое ко-
ней структурной организации хроматина.
личество работ, посвященных исследованию
структурной организации хроматина и основан-
ных на сочетании разнообразных молекулярно-
НУКЛЕОСОМА
биологических, биохимических, физических и
Структура хроматина характеризуется доста-
математических подходов [1-11]. Среди них -
точно сложной многоуровневой организацией,
данные электронной и атомно-силовой микро-
которая обеспечивается разнообразными ДНК-
скопии [12-14], рентгеноструктурного анализа
белковыми и белок-белковыми взаимодействия-
[3, 15-18], методов молекулярной спектроскопии
ми (рис.1). Самыми многочисленными из ядер-
[19-24], в т.ч. кругового дихроизма [25-33], флу-
ных белков, принимающих участие в формирова-
оресцентной спектроскопии [34, 35], методов
нии структуры хроматина, являются белки гисто-
биоинформатики [11] и т. д. Согласно современ-
новой группы, характеризующиеся высоким
ным представлениям ключевую роль в структур-
содержанием положительно заряженных амино-
ной организации хроматина играют ДНК-белко-
кислотных остатков лизина и аргинина. Белки
вые и белок-белковые взаимодействия. В первой
данного типа подразделяются на две функцио-
части обзора [36] нами были рассмотрены вопро-
нально различные группы: так называемые кро-
сы, касающиеся структуры линкерных гистонов,
вые (Н2А, Н2В, Н3 и Н4) и линкерные гистоны
посттрансляционные модификации гистона Н1 и
семейства Н1 (в эритроцитах птиц существует ва-
их роль в функционировании хроматина. Особое
риант, названный Н5). Именно взаимодействие
внимание было уделено особенностям структуры
гистоновых белков с ядерной ДНК обеспечивает
линкерных гистонов Н1 в ядрах транскрипцион-
формирование первого и наиболее хорошо изу-
но неактивных клеток, отличающихся высокой
ченного уровня структурной организации хрома-
степенью компактизации ДНК. Во второй части
тина - нуклеосом, которые традиционно счита-
обзора мы предприняли попытку отразить наибо-
ются элементарными структурными единицами
лее интересные, с нашей точки зрения, результа-
хроматина.
ты, иллюстрирующие роль линкерных гистонов
Значительный прогресс в изучении механизма
При подготовке рукописи все
упаковки ДНК в хроматине был достигнут еще в
авторы внесли равный вклад.
237
238
ЧИХИРЖИНА и др.
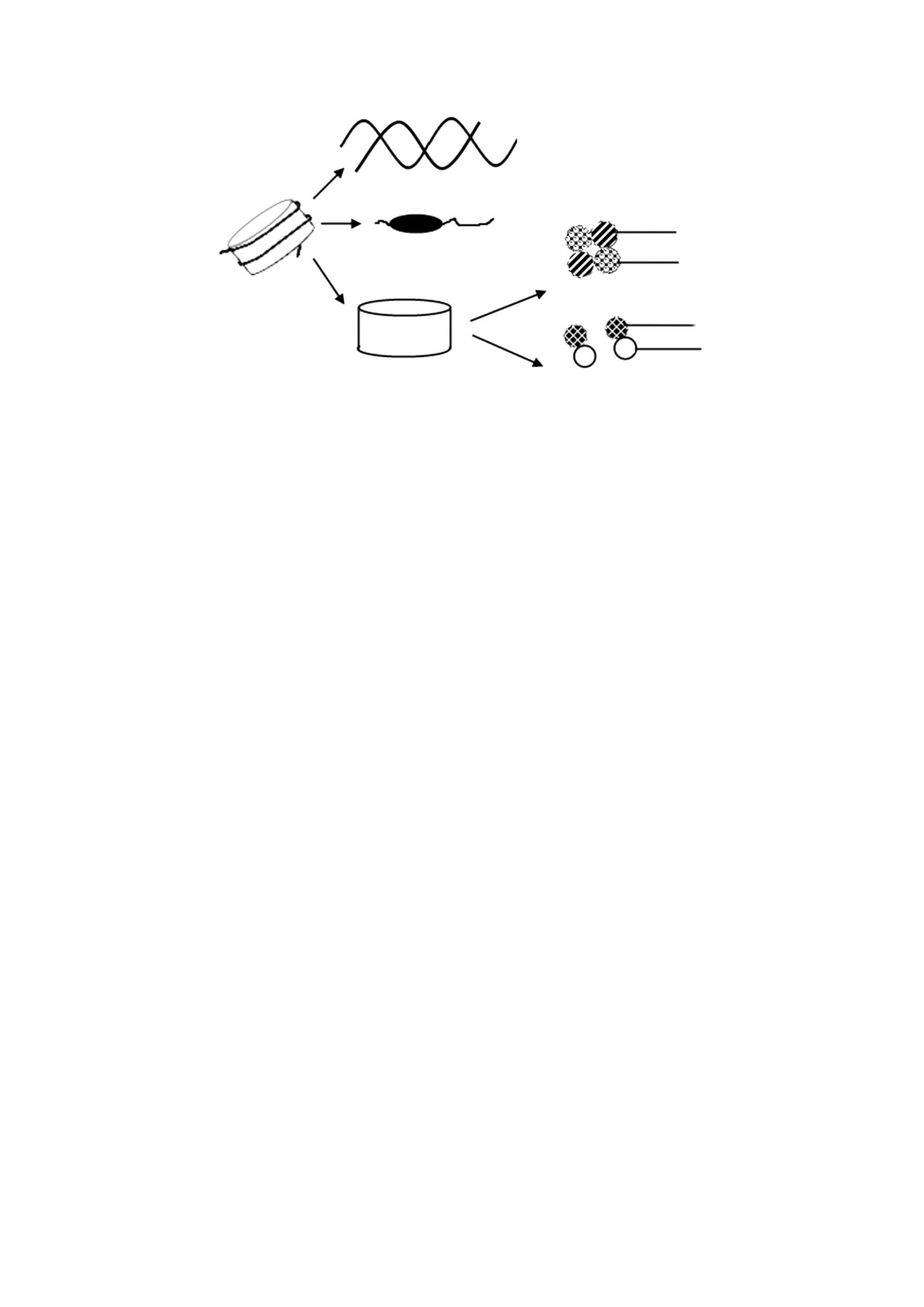
ДНК
Тетрамер (Н3/Н4)2
Н3
Линкерный гистон Н1
Н4
Нуклеосома
Н2А
Н2В
Гистоновый октамер
2 гетеродимера Н2А-Н2В
Рис. 1. Уровни структурной организации хроматина.
1974 г. после открытия субъединичной структуры
строек внутри коровой частицы [41-43], которые
хроматиновой фибриллы. С помощью метода
играют ключевую роль в процессах ремоделиро-
электронной микроскопии было установлено,
вания хроматина. В частности, для разворачива-
что у всех эукариот элементарная хроматиновая
ния молекулы ДНК при транскрипции и для ста-
фибрилла представляет собой линейно упорядо-
бильности самой нуклеосомы необходима неко-
ченную цепочку периодически повторяющихся
торая пластичность димера Н2А-Н2В. При
субъединиц - нуклеосом, которые соединяются
накручивании ДНК на поверхность гистонового
линкерной ДНК [12]. Затем методом дифракции
октамера 121 из 146 п.н., входящих в состав нукле-
нейтронов была показана локализация ДНК в
осомы, связываются с глобулярными доменами
нуклеосоме снаружи белковой частицы [37].
гистонов. Оставшиеся 25 п.н. взаимодействуют с
вытянутыми N-концевыми участками коровых
Нуклеосома состоит из белковой сердцевины
гистонов, которые выходят на поверхность нук-
(так называемый кор, от англ. «core»), по форме
леосомы сквозь витки ДНК приблизительно че-
напоминающей цилиндр диаметром 11 нм и вы-
рез каждые 20 п.н. [15, 38, 44-47]. Высокая эво-
сотой 5,7 нм, вокруг которого уложен по спирали
люционная консервативность аминокислотной
фрагмент ДНК длиной ~146 п.н. (рис. 1) [17].
последовательности коровых гистонов обеспечи-
ДНК на поверхности белковой частицы стабили-
вает, помимо прочего, эффективную самосборку
зирована многочисленными электростатически-
нуклеосомы, а многочисленные посттрансляци-
ми взаимодействиями и водородными связями
онные модификации коровых гистонов позволя-
[15]. Белковый кор формируется при взаимодей-
ют сделать этот процесс обратимым и управляе-
ствии друг с другом гистонов Н2А, Н2В, Н3 и Н4
мым [4, 10, 48, 49].
[3, 5, 6, 15, 38], откуда и происходит термин
«кровые гистоны». Коровые гистоны представ-
Во всех эукариотических клетках нуклеосомы
ляют собой небольшие (102-135 а.о.) высококон-
соединены между собой коротким фрагментом
сервативные глобулярные белки с молекулярной
ДНК, так называемым «линкерным» участком,
массой 11-16 кДа [3, 15, 38, 39]. Коровая частица
формируя структуру типа
«бусины-на-нити»
содержит по две молекулы каждого из четырех ги-
(beads-on-a-string) [38]. Нуклеосома вместе с
стонов: центральный тетрамер (Н3/Н4)2 и два ге-
линкерной ДНК образуют полную нуклеосому. В
отличие от инвариантной структуры нуклеосомы,
теродимера Н2А-Н2В, расположенные на пери-
одинаковой в хроматине всех эукариот, полные
ферии. Данные рентгеноструктурного анализа с
нуклеосомы в ядрах клеток различных видов мо-
разрешением 1.9 Å [40] и 2.8 Å [15] подтвердили,
гут существенно отличаться как за счет различий
что на белковую коровую частицу накручен уча-
в длине линкерной ДНК, так и по структуре свя-
сток двухцепочечной ДНК длиной 145-147 п.н. с
занного с ней гистона Н1. Длина фрагмента ДНК
образованием левозакрученной суперспирали
в составе полной нуклеосомы может варьировать
(80 п.н. на виток), при этом боковые группы ами-
в клетках различных тканей от 195-200 п.н. в ак-
нокислотных остатков коровых гистонов взаимо-
тивном хроматине до 230-270 п.н. в его тран-
действуют с ДНК.
скрипционно неактивных областях. Недавно бы-
Совсем недавно получено экспериментальное
ло обнаружено [50], что при обработке экзо-
подтверждение возможности структурных пере-
нуклеазой III можно получить две новые
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
239
промежуточные частицы, названные авторами
tion), которая наглядно демонстрирует связыва-
«протохроматосомы», содержащие по 7 п.н. лин-
ние гистона Н1 с ДНК на входе/выходе из нукле-
керной ДНК соответственно с одного или с обоих
осомы. Взаимодействие линкерного гистона с
концов нуклеосомы. Эти частицы, по мнению ав-
нуклеосомой происходит за счет связывания Lys
торов работы [50], могут определять свойства сай-
и Arg глобулярного домена белка с фосфатными
та связывания гистона H1, однако в деталях роль
группами основной цепи ДНК. При этом ДНК
прото-хроматосомной частицы в структурной ор-
остается в В-форме [25, 56]. Кроме того, в глобу-
ганизации хроматина пока не ясна.
лярном домене гистона Н5 располагаются пять
ключевых аминокислотных остатков Ile, которые
По сравнению с коровыми гистонами, струк-
отвечают за связывание этой области с нуклеосо-
турно-функциональные особенности взаимодей-
ствия Н1 с другими элементами хроматина, равно
мой [57]. Использование метода ядерного маг-
как и структурные характеристики этого белка в
нитного резонанса и криоэлектронной микро-
свободном состоянии, существенно менее изуче-
скопии позволило установить, что концевые
домены гистона Н1 не вовлечены в непосред-
ны [36]. Взаимодействие гистона Н1 с линкерной
ственное взаимодействие с нуклеосомой [57, 58].
ДНК на входе/выходе из нуклеосомы [47] приво-
дит к стабилизации нуклеосомного комплекса
В последние годы для исследования структуры
путем «стягивания» расположенных по соседству
нуклеосомы успешно применяются и такие мето-
нуклеосом [51, 52] и способствует формированию
ды, как численное моделирование и малоугловое
высших (наднуклеосомных) уровней структур-
рентгеновское и нейтронное рассеяние (SAXS и
ной организации хроматина, таких как 30-нано-
SANS). Метод малоуглового рассеяния является
метровая фибрилла [40].
мощным инструментом исследования простран-
ственной структуры хроматина и митотических
хромосом, который позволяет выявлять периоди-
ПОСТНУКЛЕОСОМНЫЙ УРОВЕНЬ
ческие структуры в биологических образцах в
СТРУКТУРНОЙ ОРГАНИЗАЦИИ
растворе. Использование нового инструментария
ХРОМАТИНА
поставило под вопрос гипотезу о наличии ста-
Следующий уровень структурной организации
бильных уровней структурной организации хро-
хроматина
- формирование 30-нанометровой
матина более высокого порядка, чем 30-наномет-
фибриллы
- обеспечивает дополнительное
ровая фибрилла. Совсем недавно с помощью это-
уменьшение линейных размеров ДНК в 40-
го метода было показано, что хроматин в
50 раз. Формирование 30-нанометровой фибрил-
интерфазных ядрах и митотических хромосомах в
лы происходит в два этапа: образование цепи нук-
основном состоит из нерегулярно сложенных во-
леосом диаметром 10 нм (10-нанометровая фиб-
локон без строгой структуры 30-нанометровой
рилла) вследствие их стягивания гистоном Н1 [6,
фибриллы [59]. Таким образом, авторы пришли к
7] и дальнейшее сворачивания этой 10-наномет-
выводу, что в интерфазном и митотичесом хрома-
ровой фибриллы в более компактные наднукле-
тине компактизация и нерегулярное сворачива-
осомные структуры с диаметром 30 нм [2, 5, 6, 53].
ние хроматиновой фибриллы может проходить
Вместе с тем процессы формирования 30-нано-
без образования структур диаметром 30 нм. Более
метровых фибрилл обратимы и по сути хроматин
подробно познакомиться с этими вопросами
представляет собой систему, находящуюся в ди-
можно в опубликованных в последнее время об-
намическом равновесии между «открытой» кон-
зорах по структуре хроматина (например, [1, 2, 9,
формацией типа «бусины-на-нити» и компакт-
11, 60-62]).
ной 30-нанометровой фибриллой [54]. На фор-
В настоящее время в литературе рассматрива-
мирование последней оказывают существенное
ются два типа моделей вторичной структуры хро-
влияние различные варианты (подтипы) гисто-
матина [13]: модель соленоида (спираль «one-
нов, их посттрансляционные модификации, а
start») и зигзагообразная модель (спираль «two-
также различные негистоновые белки хроматина.
start»). В первом случае нуклеосомы в фибрилле
Фундаментальной структурной единицей 30-на-
взаимодействуют с пятой и шестой соседними
нометровой фибриллы является хроматосома,
нуклеосомами, а линкерная ДНК изгибается, об-
которая содержит гистоновый октамер и одну мо-
разуя спираль [62, 64]. В зигзагообразной модели
лекулу гистона Н1, связанные со 165 парами нук-
нуклеосомы расположены в виде зигзага таким
леотидов ДНК [55].
образом, что линкерный участок ДНК остается
Несмотря на то что структура нуклеосомы бы-
прямым [2].
ла решена достаточно давно, вопрос о структуре
хроматосомы до сих пор остается открытым. В
В образовании 30-нанометровой хроматино-
2015 г. была получена первая кристаллическая
вой фибриллы первостепенную роль играет
структура комплекса нуклеосомы с глобулярным
именно длина линкерной ДНК, которая варьиру-
доменом гистона Н5 и 167 п.н. ДНК [56] с разре-
ет от 20 п.н. у дрожжей до 35-50 п.н. в соматиче-
шением, близким к атомному (near-atomic resolu-
ских клетках позвоночных [2, 5] и до 70-100 п.н.
БИОФИЗИКА том 65
№ 2
2020
240
ЧИХИРЖИНА и др.
в спермиях иглокожих [5, 6]. Вариация длины
рассеяния растворов подобных комплексов, что
линкерной ДНК обеспечивает разнообразие
проявляется в виде так называемого ψ-типа
структуры хроматина в клетках различного типа.
(PSI - Polymer and Salt Indused) спектров круго-
вого дихроизма ДНК [14, 19-24, 66]. Такое состо-
Сравнительный анализ хроматина в тран-
яние ДНК возникает при связывании не только с
скрипционно неактивных и активных клетках
позволяет заключить, что присутствие обогащен-
лизин-богатыми гистонами Н1, но и с катионны-
ных аргинином линкерных белков и большая
ми полипептидами и некоторыми другими ядер-
длина линкерной ДНК обеспечивают плотную
ными белками [5, 14, 24-26, 28-30, 32, 67]. В слу-
чае взаимодействия ДНК с ядерными белками
упаковку нуклеосом и суперкомпактное состоя-
появление ψ-типа спектров кругового дихроизма
ние хроматина в транскрипционно неактивных
клетках. Напротив, короткая линкерная ДНК от-
ДНК обусловлено формированием надмолеку-
ветственна за менее компактное расположение
лярных структур и эффектами рассеяния поляри-
ДНК в нуклеосомной цепочке, что поддерживает
зованного света на крупных конденсированных
частицах. При этом эксперименты по рентгенов-
хроматин нейронов в состоянии, способном к
скому рассеянию показали, что ДНК в этих слу-
транскрипции. Таким образом, длина линкерной
ДНК является важным параметром, который
чаях остается в В-форме [25, 68, 69].
оказывает существенное влияние на высшие
Гистоны Н1 и Н5 способны избирательно и с
уровни организации и функциональную актив-
высоким сродством взаимодействовать с участка-
ность хроматина. Вариация длины линкерной
ми ДНК, уже имеющими структурные наруше-
ДНК обеспечивает разнообразие структуры хро-
ния: разнообразные перекрестные структуры
матина в клетках различного типа.
[70], структуры типа хиазмы Холлидея [71], участ-
На сегодняшний день все еще нет четкого
ки связывания ДНК с лекарственными препара-
представления о структуре высших уровней орга-
тами [72, 73] и проч. Степень предпочтения ги-
низации хроматина. Роль 30-нанометровой хро-
стона Н1 к связыванию с нуклеосомной ДНК вы-
матиновой фибриллы в ядре не ограничивается
ше по сравнению с его сродством к свободной
ДНК по разным данным от двух-десяти [74] до
компактизацией ДНК. Она также может модули-
ровать возможность доступа специфических по-
150 [1] раз. Столь существенный разброс может
следовательностей ДНК для регуляторных фак-
объясняться тем, что разные авторы использова-
торов. Считается, что 30-нанометровая фибрилла
ли в своих исследованиях различные по длине
организована в петельные домены (50-150 тыс.
участки ДНК в качестве модельной мишени свя-
зывания. Так, сродство Н1 к перекрестным
п.н.), которые закреплены на ядерном матриксе.
При этом каждый домен представляет собой кла-
структурам ДНК типа 4Н/4WJ (Four Way Junction)
стер генов, функционально связанных между со-
примерно в 100 раз выше, чем к линейной
бой. Природа подобных доменов пока оконча-
ДНК [1].
тельно не ясна, однако, по всей видимости, они
Важную роль во взаимодействиях с ДНК игра-
построены по принципу суперспирали более вы-
ет положительно заряженный С-концевой уча-
сокого порядка.
сток гистона Н1 [1, 75]. Как уже было отмечено
ранее, основные функциональные различия меж-
ду подтипами белка Н1, по мнению ряда авторов
ОСОБЕННОСТИ ВЗАИМОДЕЙСТВИЯ
[1, 5, 6, 76-80], обусловлены вариативностью
ЛИНКЕРНОГО ГИСТОНА Н1 С ДНК
первичной структуры их С-концевого фрагмента.
Взаимодействие ДНК с гистонами семейства
Это предположение находит свое подтверждение,
Н1 происходит, прежде всего, между отрицатель-
в частности, на примере исследования взаимо-
но заряженным сахарофосфатным остовом и по-
действия ДНК со спермий-специфическими ги-
ложительно заряженными аминокислотными
стонами семейства Н1 [26-28, 32, 67]. Немало-
остатками белка [19, 20, 65]. Вместе с тем в неко-
важным представляется и то, что С-концевой
торых экспериментах наблюдали взаимодействие
участок сохраняется и в белках, родственных ги-
глобулярного домена гистона Н1 с основаниям
стону Н1, но лишенных канонического глобуляр-
ДНК в области большой бороздки [14, 20-22, 24].
ного домена [81]. Характерной чертой С-конце-
Было показано, что связывание гистона Н1 при-
вого фрагмента является большое количество до-
водит к некоторому нарушению конформации
статочно равномерно распределенных вдоль
двойной спирали ДНК [25, 26, 28, 32]. Однако
полипептидной цепи остатков лизина [81, 82], что
вплоть до настоящего времени нет прямых струк-
способствует стабилизации комплексов ДНК-
турных данных для характеристики комплексов
Н1 в хроматине. Согласно литературным дан-
гистона Н1 с линкерной ДНК. Непрямые струк-
ным, гистон Н1, лишенный N-концевой после-
турно-чувствительные методы, такие как круго-
довательности, конденсирует ДНК так же, как и
вой дихроизм в ультрафиолетовой области, ока-
интактная молекула [83]. Это служит косвенным
зываются неинформативными при изучении над-
подтверждением того, что в конденсации хрома-
молекулярных структур в силу высокого
тина принимает активное участие именно С-кон-
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
241
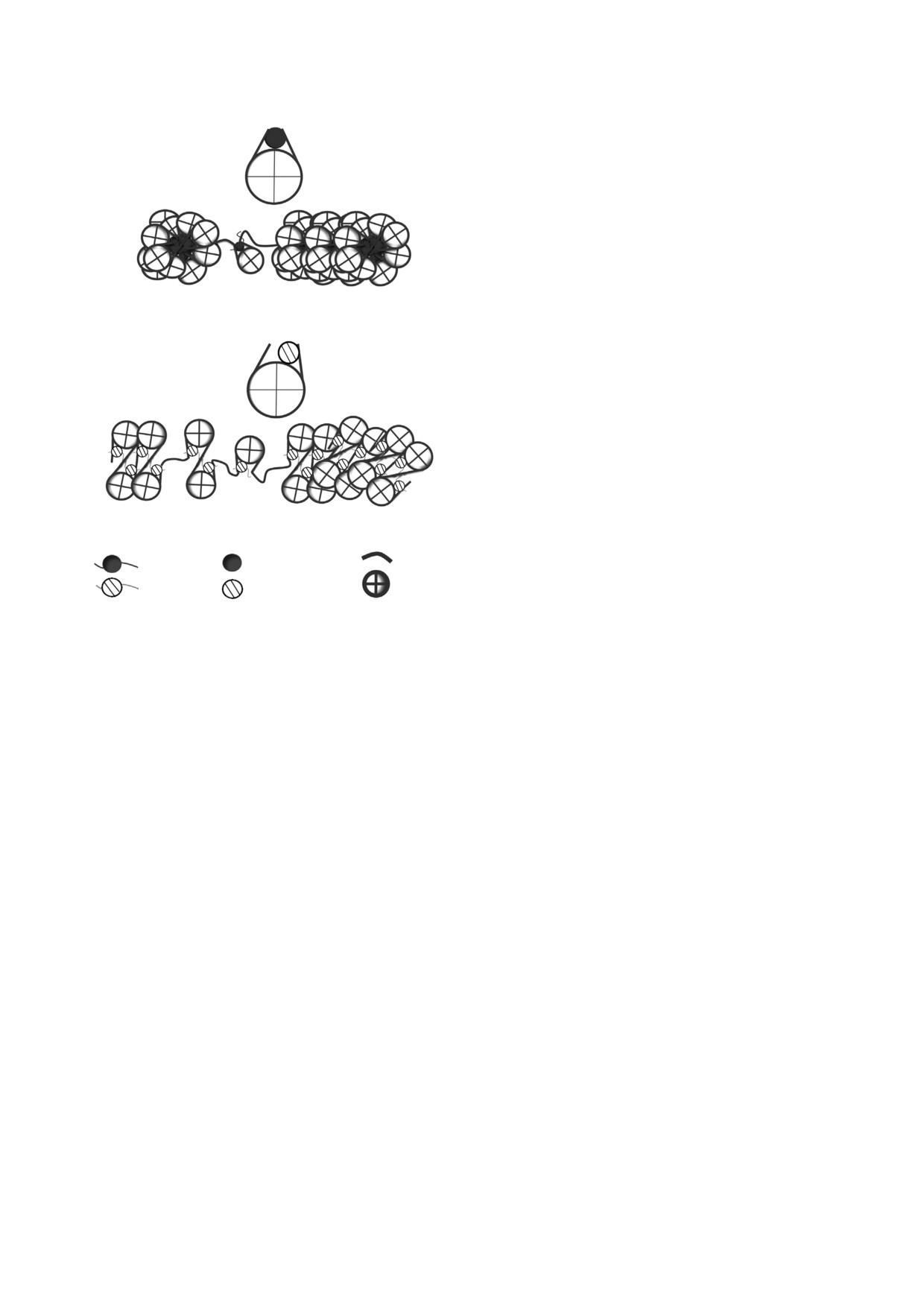
(а)
(б)
(в)
2
2
2
3
3
3
1
1
1
Симметричная
Асимметричные модели
модель
Рис. 2. Схематическое представление моделей возможной локализации глобулярного домена Н1 на нуклеосоме:
симметричная (а) и ассиметричные (б, в) модели; 1 - глобулярный домен гистона Н1; 2 - ДНК; 3 - гистоновый
октамер.
цевой участок Н1 [36]. Именно этот фрагмент
ной участка ДНК, который закрыт белком от воз-
белка взаимодействует с ДНК и необходим для
действия микрококковой нуклеазы. В симмет-
прочного связывания линкерных гистонов с нук-
ричной модели [89] глобулярный домен располо-
леосомой [84, 85]. При этом в пределах С-конце-
жен в центре нуклеосомы с внешней стороны, а
вого фрагмента молекулы Н1 выделяют две обла-
N- и С-концевые фрагменты белка взаимодей-
сти, разделенные коротким линкером [51, 78, 79]:
ствуют с нуклеосомной ДНК и защищают по
область I - с 97-го по 121-й а.о., расположенная
10 п.н. с каждой стороны от точки ее входа/выхо-
непосредственно за глобулярным доменом Н1, и
да из нуклеосомы (рис. 2). Согласно асимметрич-
область II - со 145-го по 169-й а.о. Интересно от-
ной модели [90, 91], ДНК защищена у оси сим-
метить, что удаление фрагментов 122-144 и 170-
метрии по 15 и 5 п.н. с каждой стороны в месте
196 а.о. не приводит к изменениям функции
входа/выхода ДНК из нуклеосомы. При этом гло-
С-концевого домена. Кроме того, исследование
була расположена на расстоянии 70 п.н. от оси
мутантных белков, в которых была проведена за-
симметрии таким образом, что она соединяет два
мена аминокислот со 122-го по 144-й и со 170-го
витка спирали ДНК (рис. 2а). Согласно альтерна-
по 196-й на последовательность 97-121, показало,
тивному варианту несимметричной модели [92,
что модифицированный белок ведет себя так же,
93] глобулярный домен расположен между ДНК и
как и белок дикого типа [51, 76].
нуклеосомой (рис. 2б). В этой модели 20 п.н. за-
щищены только с одной стороны оси симметрии.
Недавние исследования показали, что ДНК-
связывающие свойства гистона Н1 сохраняются и
Основываясь на последних структурных
при изменении очередности расположения ами-
исследованиях методами криоэлектронной мик-
нокислот в С-концевом домене [84, 86, 87]. Та-
роскопии и ядерного магнитного резонанса, уда-
ким образом, основными факторами в регуляции
лось подтвердить существование элементов
взаимодействия С-концевого домена Н1 с лин-
хроматина, удовлетворяющих асимметричной
керной ДНК являются специфические амино-
модели связывания [84]. В дополнение к ранее
кислотные остатки в этой области, т.е. опреде-
описанным вариантам модели несимметричного
ленный набор образующих его аминокислотных
связывания в этом случае глобулярный домен
остатков, а не какая-либо конкретная аминокис-
линкерного гистона, ДНК и коровая частица со-
лотная последовательность [51, 75-77, 84, 85, 88].
единены мостиками и также несимметричным
Это означает, что для компактизации ДНК важно
образом защищены 10 п.н. ДНК (так называемая
наличие определенных аминокислот, но не поря-
«off-dyad» модель, рис. 3б). В глобулярном доме-
док их расположения.
не выделены три сайта связывания белка с ДНК,
точное положение которых до сих пор не опреде-
Кроме С-концевого участка Н1 во взаимодей-
лено [84, 94]. Однако показано, что в каждом из
ствии с ДНК принимает участие и глобулярный
них есть Arg74 [95]. Кроме того, показано, что в
домен. На основании проведенных эксперимен-
образовании комплекса гистона Н5 с нуклеосо-
тов было предложено несколько моделей связы-
мой принимает участие и С-концевой домен ги-
вания линкерных гистонов с нуклеосомой
стона Н2А [84].
(рис. 2). На сегодняшний день все предложенные
модели можно разделить на модели симметрич-
Таким образом, многочисленные исследова-
ного и несимметричного связывания. Эти модели
ния, посвященные взаимодействию гистона Н1 с
отличаются друг от друга расположением глобулы
нуклеосомой, позволяют сделать вывод, что воз-
относительно оси симметрии нуклеосомы и дли-
можно существование различных альтернатив-
БИОФИЗИКА том 65
№ 2
2020
242
ЧИХИРЖИНА и др.
(а)
Структура гистонов Н1 и Н5 очень схожа (го-
мология Н5 курицы с Н1.0 мыши составляет
~88%), и можно ожидать, что и связывание этих
белков с ДНК будет проходить схожим образом.
Несмотря на то что концевые участки линкерного
гистона не играют важной роли в позициониро-
вании глобулы в хроматосоме, высокий положи-
тельный заряд N-концевого домена может стаби-
лизировать связывание белка с ДНК. Вероятнее
всего, существуют несколько различных меха-
низмов, подобных описанным выше, посред-
Конденсированный хроматин
ством которых линкерные гистоны формируют
структуры хроматина высшего порядка.
(б)
Несмотря на то что и глобулярный домен ги-
стона Н1, и его С-концевой участок отвечают за
связывание с ДНК, их функции при этом совер-
шенно различны. Глобулярный домен служит
якорем при взаимодействии гистона с линкерной
ДНК. Этот домен имеет по меньшей мере два сай-
та связывания с ДНК: основной сайт расположен
со стороны большой бороздки суперспирали
ДНК, а второй - с внутренней стороны малой
вблизи оси симметрии. Наличие таких разделен-
Менее конденсированный хроматин
ных в пространстве сайтов взаимодействия с
ДНК отчетливо проявляется при связывании ги-
ДНК
стона Н1 с перекрестными или деформирован-
Линкерный
Глобулярный
Коровая
ными участками ДНК [70-73], а также при связы-
гистон
домен гистона Н1
частица
вании линкерного гистона в точке входа/выхода
из нуклеосомы. Обнаруженные в С-концевой по-
Рис. 3. Связывание нуклеосомной ДНК с глобуляр-
следовательности S/TPKK-мотивы (Ser/Thr-
ным доменом гистона Н5, выделенного из эритроци-
Pro-Lys-Lys) отвечают за способность этого
тов курицы [56]. Рисунок представлен с любезного
участка изгибать ДНК при ее взаимодействии с
согласия авторов. (а) - Модель «on-dyad» связыва-
Н1 и тем самым модулировать геометрию двой-
ния, соответствующая более конденсированному со-
стоянию хроматина; (б) - модель «off-dyad» связыва-
ной спирали ДНК в хроматине. По всей видимо-
ния, соответствующая менее конденсированному со-
сти, глобулярный домен может рассматриваться
стоянию хроматина.
как независимый нуклеосома-связывающий до-
мен.
ных структур такого комплекса. В частности, эти
конфигурации отличаются положением и ориен-
ВЗАИМОДЕЙСТВИЕ ЛИНКЕРНОГО
тацией глобулярного домена линкерного гистона
ГИСТОНА Н1 С ДРУГИМИ БЕЛКАМИ
относительно самой нуклеосомы. Совсем недав-
В состав хроматина помимо гистонов входит
но с помощью рентгеноструктурного анализа и
большое количество разнообразных негистоно-
метода ядерного магнитного резонанса удалось
вых белков. Гистон Н1 иногда относят к природ-
описать взаимодействие 165 п.н. нуклеосомной
но неупорядоченным белкам [36, 69, 82], способ-
ДНК с глобулярным доменом гистона Н5, выде-
ным взаимодействовать со множеством разнооб-
ленного из эритроцитов курицы [56], с разреше-
разных партнеров. Взаимодействия Н1 с другими
нием 3.5 Å. Надо отметить, что эта модель не про-
белками оказываются существенными для фор-
тиворечит моделям, описанным ранее. Авторы
мирования высших уровней структурной органи-
показали, что глобула гистона Н5 располагается
зации хроматина [63, 96]. Белки, взаимодейству-
на оси нуклеосомы и перекрывает оба плеча лин-
ющие с гистоном Н1, можно условно разделить
керной ДНК, демонстрируя симметричное («on-
на несколько категорий. Среди них белки, при-
dyad») связывание (рис. 3а) [56].
нимающие участие в сплайсинге; в процессах,
связанных с распознаванием повреждений ДНК;
Авторы моделей симметричного «on-dyad»
в транскрипции и трансляции; выступающие в
(рис. 3а) и несимметричного «off-dyad» (рис. 3б)
связывания предполагают, что первая из них от-
качестве шаперонов для коровых гистонов при
носится к случаю разрыхленного состояния хро-
сборке/разборке нуклеосомной частицы [77].
матина, а вторая описывает конденсированный
Специфическое связывание Н1 с белками бы-
хроматин [56].
ло продемонстрировано c помощью различных
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
243
методов. Сочетание метода иммунопреципита-
участков белком HMGN сопровождается исто-
ции и масс-спектрометрического анализа позво-
щением Н1 [109, 110]. Белок HMGN снижает уро-
лило выявить белки, специфически взаимодей-
вень компактизации хроматина, вызванной ги-
ствующие с Н1 [97]. Среди них компоненты рибо-
стоном Н1, и стимулирует транскрипцию [111].
сомальных субъединиц
40S и
60S, а также
Совсем недавно было установлено, что белки
некоторые ядерные рибонуклеопротеиды. Было
группы HMGN оказывают непосредственное
показано, что взаимодействие гистона с этими
влияние на стабилизацию высокоупорядоченной
белками вызывает подавление транскрипции не-
структуры хроматина и на взаимодействие конце-
которых генов. На сегодняшний день с помощью
вых участков гистонов с ДНК [112].
масс-спектрометрии в ядре и ядрышке выявлено
более сотни белков, которые прямо или косвенно
Вторая группа HMG белков, взаимодействую-
взаимодействуют с Н1.0 [77]. Установлено, что
щих с линкерными гистонами, - это самые рас-
гистон Н1 является ключевым белком, необходи-
пространенные и наиболее изученные на сего-
мым для формирования канонической структуры
дняшний день негистоновые белки хроматина -
ядрышка и для его функционирования [98]. Кро-
HMGB-доменные белки (HMGB1/2) [113-115].
ме того, Н1 может быть участником более круп-
HMGB-белки имеют в своем составе гомологич-
ных макромолекулярных белок-белковых ком-
ные структурно-консервативные ДНК-связыва-
плексов.
ющие домены (так называемые HMGB-домены).
Хорошо известно, что линкерный гистон Н1
Представителями сайт-специфичных однодо-
может быть медиатором транскрипции [77, 99].
менных HMGB-белков являются транскрипци-
При этом он способен выступать как в качестве
онные факторы TCF и LEF-1 [116], белки, опре-
репрессора специфической генной транскрип-
деляющие пол SRY и SOX [117-120], факторы пе-
ции [96, 97, 100, 101], так и в качестве активатора
рестройки хроматина BAF57 и PB1 [121-123] и т.
транскрипционных процессов [77, 80, 102, 103].
д. Среди многодоменных белков данного семей-
Недавние исследования показали, что Н1 играет
ства можно выделить белки HMGB1-4 [113-115,
важную роль в эпигенетических процессах [104].
122, 124, 125], митохондриальные факторы mtTF1
Например, снижение уровня экспрессии Н1.2,
и ABF2 [122], транскрипционный фактор РНК-
Н1.3 и Н1.4 в клетках приводит к понижению
полимеразы I UBF [126] и т.д.
уровня метилирования ДНК [62, 104]. Согласно
Как и гистоны Н1, белки HMGB1 и HMGB2
литературным данным, Н1.0 связывается с рядом
взаимодействуют с линкерным участком ДНК
белков, принимающих непосредственное участие
[113, 127] в точке входа/выхода ее из нуклеосомы
в процессах репарации ДНК [62, 77]. К ним отно-
и могут выступать как в качестве конкурентов,
сятся белки Ku70 и Ku86, валозинсодержащий бе-
так и в качестве партнеров при связывании с ДНК
лок VCP и др. Взаимодействие гистона Н1.0 с эти-
ми белками приводит к сильному уплотнению
[128]. Имеющиеся экспериментальные данные
хроматина и тем самым способствует защите по-
указывают, что связывание одного из белков с
ДНК стимулирует связывание с ней и второго [14,
врежденных участков.
20, 22, 24, 66]. При их одновременном присут-
Одними из первых белков, для которых была
ствии в комплексе с ДНК положительно заря-
показана способность взаимодействовать с лин-
женный гистон Н1 взаимодействует преимуще-
керными гистонами, являются представители
ственно с фосфатными группами ДНК и С-кон-
большого семейства ядерных белков с высокой
цевым доменом HMGB1, экранируя их
электрофоретической подвижностью - HMG-
отрицательные заряды и стабилизируя образую-
белки (High Mobility Group). Первая группа -
щиеся надмолекулярные ДНК-белковые ком-
белки HMGN (ранее известные как HMG14/17).
плексы [14, 24]. Исследования структуры ком-
Эти белки, формируя гетеродимерный комплекс,
плекса, образованного ДНК с гистоном Н1 в при-
взаимодействуют с нуклеосомой и облегчают
транскрипцию, но при этом не являются частью
сутствии HMGB1 [14, 24, 66, 130], показали, что
наблюдается связывание индивидуальных белков
транскрипционного комплекса [105]. Взаимодей-
ствию линкерного гистона Н1 с белками данной
как с сахарофосфатным остовом, так и с основа-
группы в процессах ремоделирования структуры
ниями ДНК [14, 19, 66]. Методом атомной сило-
хроматина способствует близкое расположение
вой микроскопии было показано, что в комплек-
глобулярных доменов HMGN и Н1 [106]. Не ис-
се HMGB1/Н1 с ДНК наблюдается формирова-
ключено, что имеет место конкуренция между
ние фибрилло-подобных структур, каждая из
HMGN белками и Н1 за участок связывания с
которых образована несколькими молекулами
ДНК [107]. В частности, показано, что конкурен-
ДНК [14, 66, 131]. При этом белковые молекулы
ция между гистоном Н1 и HMGN белками за свя-
связывают друг с другом индивидуальные моле-
зывание с АТ-богатыми участками влияет на ком-
кулы ДНК в фибрилле. Множественные белок-
пактизацию хроматина [108]. В активно транс-
белковые и ДНК-белковые взаимодействия дела-
крибируемых генах обогащение некоторых
ют такие структуры весьма стабильными.
БИОФИЗИКА том 65
№ 2
2020
244
ЧИХИРЖИНА и др.
В других работах [130, 132, 133] было проде-
моделирования хроматина [137]. Кроме того, вре-
монстрировано взаимодействие Н1 с HMGB1 (в
мя связывания Н1 с нуклеосомой влияет и на воз-
отсутствие ДНК) in vitro. Прямое взаимодействие
можность доступа к ДНК транскрипционных
двух белков было также показано методами кру-
факторов и других ядерных белков [138]. HMGB-
гового дихроизма и динамического светорассея-
белки также мобильны, время пребывания на
ния [134], а также флуоресцентными методами
нуклеосоме этих белков, так же как и гистона Н1,
[34, 35]. Взаимодействие двух белков приводит к
непродолжительно [128]. Однако Н1 значительно
изменению структуры как минимум одного из
дольше находится на нуклеосоме по сравнению
них: увеличивается доля α-спиральных участков,
со всеми представителями белков HMG-семей-
что вызывает небольшие изменения третичной
ства [139-141]. Учитывая, что взаимодействие
структуры [35, 134]. Возможность взаимодей-
между H1 и HMG происходит в течение довольно
ствия между гистоном Н1 и белком HMGB1 по-
короткого времени, достаточно трудно устано-
средством их С-концевых участков также была
вить, какое именно биологическое значение име-
показана с использованием малоуглового рентге-
ет конкретное взаимодействие HMG-белков с ги-
новского рассеяния и метода ядерного магнитно-
стоном H1. Вполне вероятно, что эти взаимодей-
го резонанса [130]. В некоторых работах указыва-
ствия также оказывают определенное влияние на
ется на то, что результат взаимодействия двух
конкретный ген или ткань [139, 140]. Предполага-
белков может зависеть от редокс-состояния (от
ется, что ряд биологических эффектов напрямую
англ. redox - reduction/oxidation) остатков цисте-
связан с взаимодействиями между H1 и HMG-
ина белка HMGB1 [114, 135]. Восстановленная
белками [128]. Однако вопрос о том, как связыва-
форма HMGB1 легко вытесняет гистон Н1 с
ние между конкретными вариантами H1 и HMG-
ДНК, в то время как связывание белка в окислен-
белков влияет на структуру и биологические
ном состоянии заметно ограничивает мобиль-
функции генома, остается открытым по настоя-
ность обоих белков в хроматине [114].
щее время.
Имеющиеся на сегодняшний день экспери-
Не стоит забывать и о посттрансляционных
ментальные данные указывают, что взаимодей-
модификациях гистона Н1, которые также могут
ствие гистона Н1 с другими белками происходит
модулировать ДНК-белковые и белок-белковые
в основном за счет его С-концевого участка [60,
взаимодействия (подробнее см. в первой части
77, 97, 136]. В недавних экспериментах было пока-
обзора [36]). Кроме того, обратимые изменения в
зано, что удаление С-концевого участка приводит
состояниях модификаций гистонов влияют на
к потере примерно 25% обнаруженных в ядре
структуру хроматина в целом [142]. Например,
взаимодействий Н1.0 с другими белками [60]. По-
метилирование лизина 26 в Н1.4 способствует его
лагают, что, связываясь с другими белками, ги-
взаимодействию с гетерохроматиновым белком
стон Н1 экранирует отрицательный заряд моле-
HP1, что приводит к формированию областей
кулы-партнера и тем самым облегчает ее взаимо-
хроматина с пониженным уровнем транскрип-
действие с ДНК. Недавние исследования белка
ции [143]. Деметилирование лизина 26 в H1.4 спо-
Н1.0 показали возможность участия во взаимо-
собствует блокированию данного белок-белково-
действии с другими белками и его глобулярного
го взаимодействия и, следовательно, приводит к
фрагмента [60]. Авторы предполагают, что влия-
активации транскрипционной активности, а од-
ние каждого из трех функциональных доменов
новременное фосфорилирование соседнего
Н1 на его связывание с другими белками отвечает
остатка серина в 27-м положении сводит его на
за специфическое взаимодействие с большин-
нет [144].
ством из них. Структурная неупорядоченность N-
С точки зрения функциональной значимости
и С-концевых доменов может облегчать конкрет-
вопросы взаимодействия гистона Н1 с другими
ные белок-белковые взаимодействия. Однако в
белками остаются плохо изученными и требуют
целом вопросы о влиянии этих областей на взаи-
дальнейшего тщательного исследования. Множе-
модействие Н1 с различными белками остаются
ственные контакты с другими белками хроматина
открытыми.
позволяют предположить, что функции гистона
Связывание различных подтипов гистона Н1 с
Н1 в ядре гораздо шире, чем только компактиза-
хроматином носит динамический характер. Ос-
ция ДНК [60]. Сегодня можно с уверенностью го-
новным фактором, определяющим характер это-
ворить, что многофункциональность линкерного
го связывания, является длина С-концевого
гистона по крайней мере частично обусловлена
фрагмента белка: наименьшая продолжитель-
его взаимодействием с множеством ядерных и яд-
ность пребывания на нуклеосоме выявлена для
рышковых белков.
Н1 с самым коротким С-концевым участком [87].
Величина промежутка времени, когда Н1 связан с
ЗАКЛЮЧЕНИЕ И ПЕРСПЕКТИВЫ
нуклеосомой, регулирует степень конденсации
хроматина и его доступность для других белков, а
Несмотря на активные исследования структу-
также возможность прохождения процессов ре-
ры хроматина, проводимые на протяжении по-
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
245
следних десятилетий, можно сказать, что на сего-
гистоном [167]. В 2015 г. in vivo были проведены
дняшний день сформировано достаточно ясное
исследования хроматиновой фибриллы на уровне
представление лишь о структуре нуклеосомы -
единичной клетки с разрешением ~20 нм с помо-
первого уровня компактизации хроматина. Име-
щью метода STORM-микроскопии (Stochastic
ющиеся к настоящему времени сведения о струк-
Optical Reconstruction Microscopy) [167]. Авторы
туре хроматина на следующих, более высоких
показали, что нуклеосомы собираются в группы
уровнях структурной организации, зачастую до-
разного размера - «муфты нуклеосом». При этом
вольно противоречивы и до сих пор не позволяют
в дифференцированных фибробластах человека
сформировать однозначную картину. С появле-
обнаружено порядка восьми нуклеосом в «муф-
нием новых методов исследования биологиче-
те», в то время как в стволовых клетках - порядка
ских молекул (флуоресцентный резонансный
четырех нуклеосом. Вероятно, хроматиновые
перенос энергии (FRET) [145, 146], высокоско-
фибриллы толщиной 30 нм существуют в виде ко-
ростная атомная силовая микроскопия, спектро-
ротких фрагментов [62], что вполне согласуется с
скопия поглощения и оптической активности в
результатами исследований некоторых авторов,
инфракрасной области (VCD и ROA) [88, 131,
которые показали, что в природном хроматине
147-150], малоугловое рентгеновское и нейтрон-
нет регулярных 30-нанометровых фибрилл хро-
ное рассеяние (SAXS и SANS) [59, 145, 146, 150-
матина [168-171]. На организацию и динамику
152] и многие другие) быстро увеличивается и
таких групп нуклеосом оказывают существенное
объем доступной структурной информации. По-
влияние различные взаимодействия, в том числе
рой, некоторые высказанные ранее гипотезы,
и нуклеосом-нуклеосомные [8]. Интерфазные
требуют пересмотра и дополнительной экспери-
хромосомы могут быть организованы в глобуляр-
ментальной проверки. Наиболее ярко, на наш
ные структуры - топологически ассоциирован-
взгляд, это иллюстрируется недавними данными
ные домены (TAD) [41, 168], которые сохраняют-
о структуре 30-нанометровой фибриллы [3, 56,
ся в течение всего клеточного цикла [166]. Сего-
152-157].
дня в литературе рассматривается модель,
30-нанометровая фибрилла - одна из наибо-
согласно которой в метафазных хромосомах хро-
лее интенсивно изучавшихся форм компактного
матин находится в состоянии «жидкой капли»
хроматина in vitro [158]. Согласно общепринятым
[166]. Однако такая модель не в состоянии объяс-
представлениям, 10-нанометровая цепочка нук-
нить существование структурированной мета-
леосом складывается в спиральные структуры
фазной хромосомы. В качестве альтернативы су-
диаметром 30 нм, а затем уплотняется до размера
ществующим представлениям недавно была
от 120 до 300-700 нм и в результате формируются
предложена модель двухфазной фрактальной
митотические хромосомы. Авторы ряда работ [17,
структуры хроматина, которая позволяет описать
63, 159] указывают на то, что образование 30-на-
и компактизацию ДНК и динамику хроматина
нометровой фибриллы является необходимым
([172], более подробно в обзоре [11]).
условием для сборки и стабилизации конденси-
рованных интерфазных и митотических хромо-
На основе результатов, полученных с помо-
сом. В ядрах некоторых терминально дифферен-
щью метода EMANIC (Electron Microscopy-As-
цированных клеток выявлены хорошо различи-
sisted Nucleosome Interaction Capture), был сделан
мые 30-нанометровые фибриллы [160-162], что
вывод, что не существует единого типа спираль-
указывает на роль таких структур в транскрипци-
ной организации 30-нанометровой фибриллы в
онно неактивном хроматине. Однако следует от-
хроматине. Это может быть связано с тем, что
метить, что непосредственно in vivo во многих яд-
структура хроматиновой фибриллы в значитель-
рах эукариотических клеток [163-165], в частно-
ной степени зависит от длины линкерной ДНК
сти в интактных клетках млекопитающих [152,
[3, 173]. Регулярные зигзагообразные структуры
163, 164], такие фибриллы до сих пор не описаны.
(«two-start helix») образуются в случае коротких и
Нуклеосомы могут не складываться в фибриллу
средних длин линкерного участка ДНК (173-
со строгой структурой диаметром в 30 нм, а взаи-
209 п.н.). В то же время структуре 30-нанометро-
модействия между отдаленными нуклеосомами
вой фибриллы в хроматине с большей длиной
могут привести к образованию менее регулярной
линкера (218-226 п.н.) лучше соответствует мо-
полимерной структуры [41-43, 153, 165-168].
дель соленоида. В обсуждаемой в литературе мо-
Не исключено, что 30- и 120-нанометровые
дели гетероморфной 30-нанометровой фибриллы
структуры свойственны определенным типам
идет чередование участков со структурой солено-
клеток. В последнее время некоторыми авторами
ида с элементами зигзагообразной модели [3, 63].
было показано, что хроматин некоторых типов
Таким образом, задача адекватного описания
клеток существует преимущественно в виде воло-
высших уровней структурной организации хро-
кон диаметром 10 нм [169] или гетерогенных
матина до сих пор далека от своего решения и
групп нуклеосом, которые связаны с линкерным
требует дальнейшего тщательного исследования.
БИОФИЗИКА том 65
№ 2
2020
246
ЧИХИРЖИНА и др.
ФИНАНСИРОВАНИЕ РАБОТЫ
19.
A. Polyanichko and H. Wieser, Biopolymers 78, 329
(2005).
Работа выполнена при финансовой поддержке
20.
A. Polyanichko, E. Chikhirzhina, V. Andruschchenko,
Российского фонда фундаментальных исследова-
et al., Biopolymers 83, 182 (2006).
ний (грант № 18-08-01500 (структурные исследо-
вания ДНК-белковых взаимодействий) и грант
21.
A. Polyanichko and H. Wieser, in Methods in Protein
№ 18-04-01199 (исследование роли ядерных бел-
Structure and Stability Analysis: Vibrational Spectrosco-
ков HMGB1/2, Н1 в структурной организации
py, ed. by E. Permyakov and V. Uversky (Nova Science
хроматина)).
Publishers, Inc., N.-Y., 2007), pp. 267-302.
22.
A. Polyanichko and E. Chikhirzhina, Spectroscopy 27,
393 (2012).
КОНФЛИКТ ИНТЕРЕСОВ
23.
A. M. Polyanichko, V. V. Andrushchenko, P. Bour,
Авторы заявляют об отсутствии конфликта
et al., Circular Dichroism: Theory and Spectroscopy, ed.
интересов.
by D. S. Rodgers (Nova Science Publishers, N.-Y.,
2012), pp. 67-126.
24.
А. М. Поляничко, В. И. Воробьев и Е. В. Чихиржи-
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
на, Молекуляр. биология 47 (2), 338 (2013).
Настоящая работа не содержит описания ка-
25.
J. Zlatanova and J. Yaneva, DNA Cell Biol. 10, 239
ких-либо исследований с использованием людей
(1991).
и животных в качестве объектов.
26.
Е. В. Чихиржина, Т. Ю. Старкова, Е. И. Костылева
и др., Цитология 53 (10), 826 (2011).
СПИСОК ЛИТЕРАТУРЫ
27.
Е. И. Рамм, Е. В. Чихиржина, Е. И. Костылева
и др., Биохимия 60 (1), 150 (1995).
1. A. E. White, A. R. Hieb, and K. Luger, Sci. Rep. 6,
19122 (2016).
28.
Е. В. Чихиржина, Е. И. Костылева, Е. И. Рамм
и др., Цитология 40 (10), 883 (1998).
2. C. L. Woodcock, and R. P. Ghosh, Gold Spring Harb
Perspect. Biol., a000296 (2010).
29.
А. М. Поляничко, С. Г. Давыденко, Е. В. Чихир-
жина и др., Цитология 42 (8), 787 (2000).
3. K. Luger, M. L. Dechassa, and D. J. Tremethick, Nat.
30.
Е. В. Чихиржина, А. М. Поляничко, А. Н. Сквор-
Rev. Mol. Cell Biol. 13, 436 (2012).
цов и др., Молекуляр. биология 36 (3), 525 (2002).
4. T. L. Caterino, and J. J. Hayes, Biochem. Cell Biol. 89,
31.
A. M. Polyanichko, E. V. Chikhirzhina, A. N. Skvor-
35 (2011).
tsov, et al., J. Biomol. Struct. Dyn. 19, 1053 (2002).
5. E. Chikhirzhina, G. Chikhirzhina, and A. Polyanich-
32.
E. Chikhirzhina, T. Starkova, E. Kostyleva, et al.,
ko, Biomed. Spectr. Imag. 3, 345 (2014).
Spectroscopy: Int. J. 27, 433 (2012).
6. Е. В. Чихиржина и В. И. Воробьев, Цитология 44,
33.
Е. В. Чихиржина, А. М. Поляничко, Е. И. Косты-
721 (2002).
лева и др., Молекуляр. биология 45 (2), 356 (2011).
7. S. A. Grigoryev, G. Arya, S. Correll, et al., Proc. Natl.
34.
А. В. Фонин, О. В. Степаненко, К. К. Туроверов
Acad. Sci. USA 106, 13317 (2009).
и др., Цитология 52 (11), 946 (2010).
8. А. Kalashnikova, М. Porter-Goff, U. Muthurajan,
35.
A. Fonin, O. V. Stepanenko, I. M. Kuznetsova, et al.,
et al., J. Roy. Soc. Interface 10, 20121022 (2013).
Spectroscopy 24, 165 (2010).
9. J. Ausio, Bioessays 37, 46 (2015).
36.
Е. Чихиржина, Т. Старкова и А. Поляничко, Био-
10. A. Sadakierska-Chudy and M. Filip, Neurotox. Res.
физика 63, 1070 (2018).
27, 172 (2015).
37.
J. B. Baldwin, P. G. Boseley, E. M. Bradbury, et al.,
11. А. В. Илатовский, Д. В. Лебедев, М. В. Филатов
Nature 253, 245 (1975).
и др., Цитология 54 (4), 298 (2012)
38.
R. D. Kornberg, Science 184, 868 (1974).
12. A. L. Olins and D. E. Olins, Science 184, 868 (1974).
39.
C. L. Peterson and M. A. Laniel, Curr. Biol. 14, R546
13. H. Schiessel, J. Widom, R. F. Bruinsma, et al., Phys.
(2004).
Rev. Lett. 86, 4414 (2001).
40.
C. A. Davey, D. F. Sargent, K. Luger, et al., J. Mol.
14. A. M. Polyanichko and E. V. Chikhirzhina, J. Mol.
Biol. 319, 1097 (2002).
Struct. 1044, 167 (2013).
41.
S. Bilokapic, M. Strause, and M. Halic, Nat. Struct.
15. K. Luger, A. W. Mader, R. K. Richmond, et al., Nature
Mol. Biol. 25, 101 (2018).
389, 251 (1997).
42.
S. Bilokapic, M. Strause, and M. Halic, Nat. Commun.
16. R. K. Suto, R. S. Edayathumangalam, C. L. White,
9, 1330 (2018).
et al., J. Mol. Biol. 326, 371 (2003).
43.
S. Bilokapic, M. Strauss, and M. Halic, Sci. Rep. 8,
17. T. Schalch, S. Duda, D. F. Sargent, et al., Nature 436,
7046 (2018).
138 (2005).
44.
G. Arents, R. W. Burlingame, B. C. Wang, et al., Proc.
18. A. Klug, Annu. Rev. Biochem. 79, 1 (2010).
Natl. Acad. Sci. USA 88, 10148 (1991).
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
247
45. J. M. Harp, B. L. Hanson, D. E. Timm, et al., Acta
72.
J. N. Yaneva, E. G. Paneva, S. I. Zacharieva et al.,
Cryst. D: Biol. Cryst. 56, 1513 (2000).
Z. Naturforsch C 61, 879 (2006).
46. C. M. Wood, J. M. Nicholson, S. J. Lambert, et al., Ac-
73.
J. N. Yaneva, E. G. Paneva, S. I. Zacharieva et al.,
ta Cryst. Sect. F: Struct. Biol. Cryst. Comm. 61, 541
Z. Naturforsch C 62, 905 (2007).
(2005).
74.
K. P. Nightingale, D. Pruss, and A. P. Wolffe, J. Biol.
47. А. Jerzmanowski, in Chormatin Struture and Dynamics:
Chem. 271, 7090 (1996).
State-of-the-Art, ed. by J. Zlatanova and S. H. Leuba
75.
X. Lu and J. C. Hansen, Biochem. Cell Biol. 81, 173
(Elsevier, London, 2004), pp. 75-102.
(2003).
48. B. Sarg, W. Helliger, H. Talasz, et al., J. Biol. Chem.
76.
X. Lu, B. Hamkalo, M. H. Parseghian, et al., Biochem-
281, 6573 (2006).
istry 48, 164 (2009).
49. A. Kowalski and J. Palyga, Cell Biol. Int. 36, 981
77.
A. A. Kalashnikova, R. A. Rogge, and J. C. Hansen,
(2012).
Biochim. Biophys. Acta 1859, 455 (2016).
50. J. Ocampo, F. Cui, V. B. Zhurkin, and D. J. Clark, Nu-
78.
A. Roque, I. Ponte, and P. Suau, Chromosoma 126, 83
cleus 7, 382 (2016).
(2017).
51. X. Lu and J. C. Hansen, J. Biol. Chem. 279, 8701
79.
A. Roque, I. Ponte, and P. Suau, Biochim. Biophys.
(2004).
Acta 1859, 444 (2016).
52. C. Crane-Robinson, Biochim. Biophys. Acta 1859, 431
80.
A. Roque, I. Ponte, J. L. Arrondo, et al., Nucl. Acids
(2016).
Res. 36, 4719 (2008).
53. D. J. Tremethick, Cell 128, 651 (2007).
81.
H. E. Kasinsky, J. D. Lewis, J. B. Dacks, et al., FASEB
54. G. Li and D. Reinberg, Curr. Opin. Genet. Dev. 21, 175
J. 15, 34 (2001).
(2011).
82.
J. C. Hansen, X. Lu, E. D. Ross, et al., J. Biol. Chem.
55. D. Angelov, J. Vitolo, V. Mutskov, et al., Proc. Nutl.
281, 1853 (2006).
Acad. Sci. USA 98, 6599 (2001).
83.
J. Allan, D. Ram, N. Harborne, et al., J. Cell. Biol. 98,
56. B. R. Zhou, J. Jiang, H. Feng, et al., Mol. Cell 59, 628
1320 (1984).
(2015).
84.
B. R. Zhou, H. Feng, H. Kato, et al., Proc. Natl. Acad.
57. B. R. Zhou, H. Feng, R. Ghirlando, et al., J. Mol. Biol.
Sci. 110, 19390 (2013).
428, 3948 (2016).
85.
M. J. Hendzel, M. A. Lever, E. Crawford, et al., J. Biol.
58. J. Bednar, I. Garcia-Saez, R. Boopathi, et al., Mol.
Chem. 279, 20028 (2004).
Cell, 66, 384 (2017).
86.
N. Raghuram, G. Carrero, J. Th'ng et al., Biochem.
59. K. Maeshima, R. Imai, T. Hikima, et al., Methods 70,
Cell Biol. 87, 189 (2009).
154 (2014).
87.
J. P. Th’ng, R. Sung, M. Ye et al., J. Biol. Chem. 280,
60. A. A. Kalashnikova, D. D. Winkler, S. J. McBryant,
27809 (2005).
et al., Nucl. Acids Res. 41, 4026 (2013).
88.
H. Fang, D. J. Clark, and J. J. Hayes, Nucl. Acids Res.
61. P. Zhu and G. Li, IUBMB Life 68, 873 (2016).
40, 1475 (2012).
62. D. V. Fyodorov, B. R. Zhou, A. I. Skoultchi, et al., Nat.
89.
J. Allan, P. G. Hartman, C. Crane-Robinson, et al.,
Rev. Mol. Cell Biol. 19, 192 (2018).
Nature 288, 675 (1980).
63. P. J. Robinson and D. Rhodes, Curr. Opin. Struct. Bi-
90. Y. Zhou, S. Gershman, V. Ramakrishnan, et al., Na-
ol. 16, 336 (2006).
ture 395, 402 (1998).
64. P. J. Robinson, L. Fairall, A. T. Van Huynh, et al.,
91.
S. Lambert, S. Muyldermans, J. Baldwin, et al., Bio-
Proc. Natl. Acad. Sci. USA 103, 6506 (2006).
chem. Biophys. Res. Commun. 179, 810 (1991).
65. V. Andrushchenko, J. H. van de Sande, and H. Wieser,
92.
J. Hayes, Biochemistry 35, 11931 (1996).
Biopolymers 72, 374 (2003).
93.
D. Pruss, B. Bartholomew, J. Persinger, et al., Science
66. A. Polyanichko, and E. Chikhirzhina, Adv. Biomed.
274, 614 (1996).
Spect. 7, 185 (2013).
94. D. T. Brown, T. Izard, and T. Misteli, Nat. Struct. Mol.
67. E. Chikhirzhina, T. Starkova, E. Kostyleva et al., Adv.
Biol. 13, 250 (2006).
Biomed. Spectr. 7, 177 (2013).
95.
L. Fan and V. A. Roberts, Proc. Natl. Acad. Sci. USA
103, 8384 (2006).
68. T. Maniatis, J. H. Venable Jr., and L. S. Lerman, J.
Mol. Biol. 84, 37 (1974).
96.
S. J. McBryant, X. Lu, and J. C. Hansen, Cell Res. 20,
69. A. L. Turner, M. Watson, O. G. Wilkins, et al., Proc.
519 (2010).
Natl. Acad. Sci. USA, pii: 201805943 (2018).
97.
J.-Q. Ni, L.-P. Liu, D. Hess, et al., Genes Dev. 20,
70. F. Totsingan and A. J. Bell Jr., Prot. Sci. 22, 1552
1959 (2006).
(2013).
98.
H. J. Szerlong, J. A. Herman, C. M. Krause et al., J.
Mol. Biol. 427, 2056 (2015).
71. J. Zlatanova and K. van Holde, BioEssays 20, 584
(1998).
99.
J. Zlatanova, Trends Biochem. Sci. 15, 273 (1990).
БИОФИЗИКА том 65
№ 2
2020
248
ЧИХИРЖИНА и др.
100. H. Lee, R. Habas, and C. Abate-Shen, Science 304,
129. А. М. Поляничко, З. В. Леоненко, Д. Крамб и др.,
1675 (2004).
Биофизика 53 (3), 407 (2008).
101. P. J. Laybourn and J. T. Kadonaga, Science 254, 238
130. L. Cato, K. Stott, M. Watson, et al., Mol. Biol. 384,
(1991).
1262 (2008).
102. Q. Lin, A. Inselman, X. Han, et al., J. Biol. Chem. 279,
131. A. Polyanichko and H. Wieser, Spectroscopy 24, 239
23525 (2004).
(2010).
103. Y. Zheng, S. John, J. J. Pesavento, et al., J. Cell Biol.
132. L. A. Kohlstaedt, E. C. Sung, A. Fujishige, et al., J.
189, 407 (2010).
Biol. Chem. 262, 524 (1987).
104. S. Yang, B. J. Kim, L. N. Toro, et al., Proc. Natl. Acad.
133. L. A. Kohlstaedt and R. D. Cole, Biochemistry 33, 570
Sci. USA 110, 1708 (2013).
(1994).
105. Y. V. Postnikov, L. Trieschmann, A. Rickers, et al., J.
134. А. М. Поляничко, Б. А. Дрибинский, И. Б. Кипен-
Mol. Biol. 252, 423 (1995).
ко и др., Структура и динамика молекулярных си-
стем 8А, 3 (2010).
106. Y. Postnikov and M. Bustin, Biochim. Biophys. Acta
1799, 62 (2010).
135. E. Polanska, S. Pospisilova, and M. Stros, PLoS One
107. H. Kato, H. van Ingen, B. R. Zhou, et al., Proc. Natl.
9, e89070 (2014).
Acad. Sci. USA 108, 12283 (2011).
136. P. Widlak, M. Kalinowska, M. H. Parseghian, et al.,
108. R. D. Phair, P. Scaffidi, C. Elbi, et al., Mol. Cell. Biol.
Biochemistry 44, 7871 (2005).
24, 6393 (2004).
137. S. W. Harshman, N. L.Young, M. R. Parthun, et al.,
109. Y. V. Postnikov, V. V. Shick, A. V. Belyavsky, et al., Nu-
Nucl. Acids Res. 41, 9593 (2013).
cl. Acids Res. 19, 717 (1991).
138. E. Cheung, A. S. Zarifyan, and W. L. Kraus, Mol. Cell.
110. T. Deng, Z. I. Zhu, S. Zhang, et al., Mol. Cell. Biol.
Biol. 22, 2463 (2002).
33, 3377 (2013).
139. R. D. Phair and T. Misteli, Nature 404, 604 (2000).
111. H. F. Ding, M. Bustin, and U. Hansen, Mol. Cell. Bi-
140. M. Harrer, H. Luhrs, M. Bustin, et al., J. Cell Sci. 117,
ol. 17, 5843(1997).
3459 (2004).
112. K. J. Murphy, A. R. Cutter, H. Fang, et al., Nucl. Acids
141. F. Catez, H. Yang, K. J. Tracey, et al., Mol. Cell. Biol.
Res. 45, 9917 (2017).
24, 4321 (2004).
113. M. Stros, Biochim. Biophys. Acta 1799, 101 (2010).
142. A. Allahverdi, R. Yang, N. Korolev, et al., Nucl. Acids
114. M. Stros, E. Polanska, M. Kucirek, et al., PLoS One
Res. 39, 1680 (2011).
10, e0138774 (2015).
143. P. Trojer, J. Zhang, M. Yonezawa, et al., J. Biol. Chem.
115. R. Reeves, DNA Repair 36, 122 (2015).
284, 8395 (2009).
116. F. A. Atcha, A. Syed, B. Wu, et al., Mol Cell Biol. 27,
144. T. K. Hale, A. Contreras, A. J. Morrison, et al., Mol.
8352 (2007).
Cell 22, 693 (2006).
117. L. H. Pevny and S. K. Nicolis, Int. J. Biochem. Cell
145. M. Brehove, T. Wang, J. North, et al., J. Biol. Chem.
Biol. 42, 421 (2010).
290, 22612 (2015).
118. P. Bernard and V. R. Harley, Int. J. Biochem. Cell Biol.
146. R. Morra, T. Fessl, Y. Wang, et al., Methods Mol. Biol.
42, 400 (2010).
1431, 175 (2016).
119. F. Oppel, N. Müller, G. Schackert, et al., Mol Cancer.
147. I. H. McColl, E. W. Blanch, A. C. Gill, et al., J. Am.
10, 137(2011).
Chem. Soc. 125, 10019 (2003).
120. O. Leis, A. Eguiara, E. Lopez-Arribillaga, et al., Onco-
148. F. Zhu, N. W. Isaacs, L. Hecht, et al., Structure 13,
gene 31, 1354 (2012).
1409 (2005).
121. T. Chi, Nat. Rev. Immunol. 4, 965 (2004).
149. L. D. Barron, Biomed. Spectr. Imag. 4, 223 (2015).
122. M. Stros, D. Launholt, and K. D. Grasser, Cell. Mol.
150. M. L. Mello and B. C. Vidal, PLoS One 7, e43169
Life Sci. 64, 2590 (2007).
(2012).
123. D. Lai, M. Wan, J. Wu, et al., Proc. Natl. Acad. Sci.
151. H. B. Stuhrmann, Acta Crystallogr. A 64, 181 (2008).
USA 106, 1169 (2009).
152. Y. Joti, T. Hikima, Y. Nishino, et al., Nucleus 3, 404
124. R. Catena, E. Escoffier, C. Caron, et al., Biol. Reprod.
(2012).
80, 358 (2009).
153. I. Garcia-Saez, H. Menoni, R. Boopathi, et al., Mol.
125. S. Park and S. J. Lippard, Biochemistry 51, 6728
Cell 72, 1 (2018).
(2012).
154. M. A. Ozturk, V. Cojocaru, and R. C. Wade, Structure
126. G. J. Sullivan and B. McStay, Nucl. Acids Res. 26,
26, 1 (2018).
3555 (1998).
155. M. A. Ozturk, V. Cojocaru, and R. C. Wade, Biophys.
127. V. Ramakrishnan, Annu. Rev. Biophys. Biomol.
J. 114, 2363 (2018).
Struct. 26, 83 (1997).
156. B. R. Zhou, J. Jiang, R. Ghirlando, et al., J. Mol. Biol.
128. Y. V. Postnikov and M. Bustin, BBA 1859, 462 (2016).
430, 3093 (2018).
БИОФИЗИКА том 65
№ 2
2020
РОЛЬ ЛИНКЕРНЫХ ГИСТОНОВ
249
157. M. A. Ozturk, G. V. Pachov, R. C. Wade, et al., Nucl.
166. T. Nozaki, R. Imai, M. Tanbo, et al., Mol. Cell 67, 282
Acids Res. 44, 6599 (2016).
(2017).
158. J. C. Hansen, Ann. Rev. Biophys. Biomol. Struct. 31,
167. M. A. Ricci, C. Manzo, M. F. Garcı´a-Parajo, et al.,
361 (2002).
Cell 160, 1145 (2015).
159. F. Song, P. Chen, D. Sun, et al., Science 344, 376
168. K. Maeshima, R. Rogge, S. Tamura, et al., EMBO J.
(2014).
35, 1115 (2016).
160. C. L. Woodcock, J. Cell Biol. 125, 11 (1994).
169. K. Maeshima, R. Imai, S. Tamura, et al., Chromo-
161. C. Kizilyaprak, D. Spehner, D. Devys, et al., PLoS
soma 123, 225 (2014).
One 5, e11039 (2010).
162. M. P. Scheffer, M. Eltsov, and A. S. Frangakis, Proc.
170. S. A. Grigoryev, G. Bascom, J. M. Buckwalter, et al.,
Natl. Acad. Sci. USA 108, 16992 (2011).
Proc. Natl. Acad. Sci. USA 113, 1238 (2016).
163. M. Eltsov, K. M. Maclellan, K. Maeshima et al., Proc.
171. W. Li, P. Chen, J. Yu, et al., Mol Cell 64, 120 (2016).
Natl. Acad. Sci. USA 105, 19732 (2008).
172. A. Bancaud, S. Huet, N. Daigle, et al., EMBO J. 28,
164. E. Fussner, M. Strauss, U. Djuric, et al., EMBO Rep.
3785 (2009).
13, 992 (2012).
165. H. D. Ou, S. Phan, T. J. Deerinck, et al., Science 357,
173. A. Routh, S. Sandin, and D. Rhodes, Proc. Natl.
pii: eaag0025 (2017).
Acad. Sci. USA 105, 8872 (2008).
The Role of the Linker Histones in Chromatin Structural Organization.
2. The Interactions with DNA and Nuclear Proteins
E.V. Chikhirzhina*, Т.Yu. Starkova*, and А.M. Polyanichko*, **
*Institute of Cytology, Russian Academy of Sciences, Tikhoretsky prosp. 4, St. Petersburg, 194064 Russia
**St. Petersburg State University, Universitetskaya nab. 7/9, St. Petersburg, 199034 Russia
In the first part of our review (Biophysics, 63, 858 (2018)) we analyzed a structure of the linker histones of the
H1 family, their post-translational modifications, and the role of the histone H1 in the formation of compact
transcriptionally inactive chromatin. In the second part of the review, we describe the current understanding
of the role of the linker histones in structural organization of chromatin: from nucleosomes to metaphase
chromosomes. The various aspects of interactions of the linker histone H1 with other elements of chromatin,
including DNA and nuclear proteins, are discussed.
Keywords: DNA, core histones, linker histone H1, nucleosome, chromatin structure, HMG proteins
БИОФИЗИКА том 65
№ 2
2020