БИОФИЗИКА, 2020, том 65, № 2, с. 263-275
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.32
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ В СОРТЕ НУТА CDC
FRONTIER МЕТОДАМИ МАШИННОГО ОБУЧЕНИЯ
© 2020 г. Б.С. Подольный*, В.В. Гурский**, М.Г. Самсонова*
*Санкт-Петербургский политехнический университет Петра Великого,
195251, Санкт-Петербург, Политехническая ул., 29
**Физико-технический институт им. А.Ф. Иоффе, 194021, Санкт-Петербург, Политехническая ул., 26
E-mail: m.samsonova@spbstu.ru
Поступила в редакцию 16.12.2019 г.
После доработки 16.12.2019 г.
Принята к публикации 24.12.2019 г.
Представлены результаты анализа динамики экспрессии генов, контролирующих переход к цвете-
нию у сорта нута CDC Frontier. Построено несколько версий модели, предсказывающей динамику
экспрессии пяти генов цветения по данным экспрессии их регуляторов. Модели обучены с помо-
щью метода случайных лесов на опубликованных ранее данных экспрессии десяти генов цветения
для условий короткого и длинного дня выращивания растений. Полученные модели правильно
предсказывают динамику средних уровней экспрессии в условиях длинного дня. Показано, что мо-
дели для CDC Frontier в целом воспроизводят регуляторные взаимодействия между ключевыми ге-
нами, описанные для модельного растения Arabidopsis thaliana. На основе анализа моделей высказа-
но предположение о том, что данные короткого дня и данные длинного дня содержат качественно
разную информацию, что может определяться функционированием разных регуляторных модулей
в разных условиях. Среди регуляторов генов идентичности цветковых меристем AP1 и LFY модели
предсказывают лидирующую роль гена FTa3 как активатора и гена TFL1c как репрессора в условиях
длинного дня.
Ключевые слова: нут, CDC Frontier, генная сеть цветения, алгоритм случайного леса.
DOI: 10.31857/S0006302920020076
соцветия, включающая в себя вторичные мери-
Исследование процессов, лежащих в основе
стемы соцветия, что подразумевает наличие до-
принятия растением решения о переходе к цвете-
полнительных генов, контролирующих идентич-
нию, и, в частности, контроль времени инициа-
ность этих меристем [7, 8]. Другим отличием яв-
ции цветения сельскохозяйственных культур
ляется то, что геномы представителей семейства
имеет важное значение для разработки сценариев
бобовых содержат несколько гомологов ключе-
эффективной адаптации культур к вариабельно-
вых генов из Arabidopsis. В частности, активатор
сти сезонов выращивания [1-3]. Эта задача осо-
цветения FLOWERING LOCUS T (FT) и репрессор
бенно актуальна в условиях меняющегося клима-
цветения TERMINAL FLOWER 1 (TFL1) присут-
та [4, 5]. В число важных сельскохозяйственных
ствуют в нуте в виде соответственно пяти и двух
культур входит нут (Cicer arietinum), представи-
гомологов (гены FTa1, FTa2, FTa3, FTb, FTc и гены
тель семейства бобовых. К настоящему времени
TFL1a, TFL1c) [14].
собрано большое количество информации о
Согласно классической схеме регуляции пере-
сходствах и отличиях в механизмах зацветания у
хода к цветению в модельном растении Arabidopsis
бобовых и у модельного организма Arabidopsis
thaliana, продукты экспрессии FT-генов в листьях
thaliana [6-12]. Основные гены, контролирующие
доставляются в апикальные меристемы растения,
время перехода к цветению и наиболее полно
где они активируют экспрессию генов идентич-
описанные в Arabidopsis, консервативны и при-
ности цветковых меристем LEAFY (LFY) и
сутствуют во многих растениях [6, 13]. Одним из
APETALA1 (AP1) [15]. Экспрессия этих генов по-
существенных отличий бобовых от Arabidopsis
давляется белками TFL1-генов с целью поддер-
является более сложная (составная) архитектура
жания локального вегетативного состояния. Бел-
ки FT и TFL1 осуществляют свою регуляторную
Сокращения: LD - длинный световой день, SD - короткий
световой день, НСКО - нормализованная среднеквадра-
функцию, образуя комплекс с транскрипцион-
тичная ошибка.
ным фактором FD или его гомологом в случае бо-
263
264
ПОДОЛЬНЫЙ и др.
бовых [15, 16]. Высокий уровень AP1 можно счи-
Появление развивающихся цветковых бутонов
тать маркером инициации цветения. Были разра-
впервые было детектировано на 31-е сутки разви-
ботаны несколько динамических моделей этой
тия в условиях короткого дня и на 32-е сутки в
генной сети в Arabidopsis, предсказывающих вре-
условиях длинного дня [14].
мя инициации цветения по уровням экспрессии
Поскольку переход к цветению должен быть
ключевых генов [15, 17].
связан с ростом экспрессии AP1, несколько позд-
Экспрессия десяти генов (пять FT-генов, два
них наблюдений, соответствующих резкому па-
TFL1-гена, а также гены FD, LFY, AP1), вовлечен-
дению уровня экспрессии AP1, были исключены.
ных в процесс зацветания, была измерена в двух
Данное падение может быть связано с изменени-
сортах нута (ICCV 96029 и CDC Frontier) в про-
ем механизмов регуляции после начала цветения.
цессе выращивания растений в условиях корот-
Таким образом, для дальнейшего анализа из дан-
кого и длинного дня [14]. ICCV 96029 является
ных длинного дня был исключен один последний
слабо чувствительным к фотопериоду и самым
день наблюдений, а из данных короткого дня —
раннецветущим сортом нута. CDC Frontier пред-
три последних дня.
ставляет собой чувствительный к фотопериоду
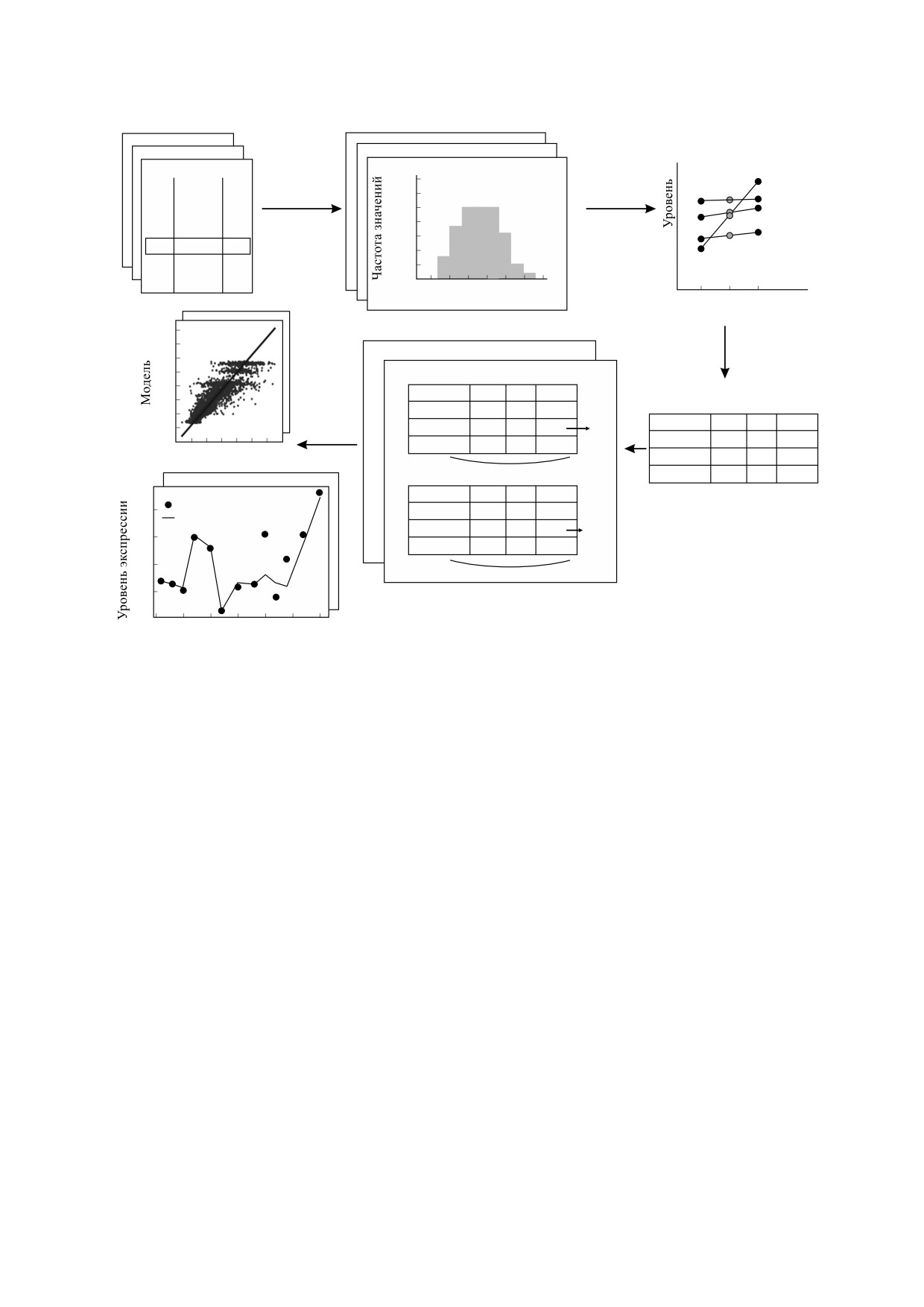
Общая схема моделирования описана на
сорт, для которого был построен референсный ге-
рис. 1. Далее детально описаны этапы моделиро-
ном [18]. На полученных данных по экспрессии
вания, проиллюстрированные на рисунке.
генов цветения в этих сортах была построена ди-
Для построения модели методами машинного
намическая модель на основе дифференциаль-
обучения необходимы обучающая и тестовые вы-
ных уравнений, которая в случае сорта ICCV
борки данных. Для этого на первом этапе исход-
96029 корректно воспроизводила динамику экс-
ные данные экспрессии мультиплицировались и
прессии всех генов и позволила протестировать
создавались искусственные выборки значений
несколько гипотез о функционировании гомоло-
уровней экспрессии всех генов для каждого дня
гов FT и TFL1 в этом сорте [19]. Однако эта мо-
развития растения. Такие выборки генерирова-
дель не смогла воспроизвести динамику экспрес-
лись множественным сэмплированием из усечен-
сии в сорте CDC Frontier, из-за чего оставалось не
ных нормальных распределений, определенных
ясным, является ли эта неудача в моделировании
на положительной вещественной полуоси,
следствием специфики модельного подхода или
где исходные средние значения и стандартные
же связана с особенностями данных для этого
отклонения уровней экспрессии генов были ис-
сорта нута.
пользованы в качестве параметров распреде-
В представленной работе используется другой
лений.
подход к моделированию динамики экспрессии
Были рассмотрены два типа моделей: модели
генов цветения в CDC Frontier, основанный на
первого типа обучались совместно по LD- и SD-
машинном обучении. Используя результаты обу-
данным, модели второго типа - только по LD-
чения методом случайного леса, предложен спо-
данным. Для моделей первого типа в каждый день
соб построения динамических предсказаний
всего было сгенерировано по 325 значений уров-
уровней экспрессии в каждый день на основе
ней экспрессии для LD- и SD-условий с целью
данных или предсказаний модели в предыдущий
обучения и по 125 значений для тестирования.
день. Полученная таким способом динамика экс-
Для обучения и тестирования моделей второго
прессии в модели исследуется при разных усло-
типа было сгенерировано соответственно по 750 и
виях обучения модели, а также при выключении
250 значений.
отдельных генов.
В исходных данных измерения для LD и SD не
всегда проводили в одинаковые дни, при этом для
ДАННЫЕ И МОДЕЛЬ ЭКСПРЕССИИ
построения моделей, учитывающих оба типа
условий, необходимо, чтобы обучающие и тесто-
Модель обучалась на опубликованных ранее
динамических данных экспрессии генов цвете-
вые выборки в каждый день содержали значения
для обоих условий выращивания. Для заполне-
ния в сорте нута CDC Frontier [14] (данные в виде
ния пропущенных значений была проведена ли-
csv-файлов
можно найти по адресу:
нейная интерполяция LD-значений на те дни,
когда измерения проводили только для SD, и на-
ренные гены цветения включают в себя пять го-
мологов гена FT (FTa1, FTa2, FTa3, FTb, FTc), два
оборот. Для интерполяции из выборки искус-
ственных данных случайным образом выбирали
гомолога гена TFL1 (TFL1a, TFL1c), ген FD, а так-
пары измерений из соседних дней.
же гены идентичности цветковых меристем LFY и
AP1. Данные представляют собой средние значе-
Уровни экспрессии FT-генов в данных соот-
ния и стандартные отклонения уровней экспрес-
ветствуют экспрессии этих генов в листьях, тогда
сии, измеренных с 9-х по 43-и сутки после посева
как уровни экспрессии остальных генов, включая
с двух- или трехдневным интервалом в условиях
потенциальные гены-мишени FT-генов (AP1 и
длинного (LD) или короткого (SD) светового дня.
LFY), соответствуют экспрессии в апикальной
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
265
Данные экспрессии (10 генов)
Мультиплицированные данные
Определение данных
в промежуточные дни
LFY, SD
День d
1
AP1,
LD
День d
0.233
ДеньУровеньСКО
Семплирование
0.200
0.167
0.133
Интерполяция
0.100
между
d
355
106
0.067
случайными
0.033
парами
соседних
0.000
100200300400500600700
точек
Уровень AP1, LD
d d
1d
1
День
160
FD
140
Тестовая выборка
120
100
Обучающая выборка
80
№ строки
FT
a1
TFL1c
60
Обучение
Генеральная совокупность
40
модели
День
y=F(Х)
x=
№ строки
FT
a1
TFL1c
20
k
d
0
150
Данные
10 переменных
Динамика, FD
№ строки
FT
a1
TFL1c
Данные
№ строки
{дни} {SD,LD}
100
Модель
y=
День
{число мульт. данных
}
k
d+
1
80
60
5 переменных
40
20
10
20
30
40
Дни
Рис. 1. Схема моделирования. Детальное описание отдельных этапов приведено в тексте.
меристеме. Поскольку модель описывает регуля-
но с учетом FT-генов, экспрессирующихся в ли-
цию в апикальной меристеме, значения экспрес-
стьях) в предыдущий день: y = F(X), где X - век-
сии FT-генов сдвигались с помощью линейной
тор, содержащий уровни экспрессии генов FTa1,
интерполяции на фиксированное время τ, необ-
FTa2, FTa3, FTb, FTc, AP1, LFY, FD, TFL1a, TFL1c
ходимое для транспорта белков FT из листьев в
в день d; y - вектор, содержащий уровни экспрес-
апекс. Исходя из полученной ранее оценки этого
сии генов AP1, LFY, FD, TFL1a, TFL1c в день d + 1.
времени для Arabidopsis, было принято значение
Таким образом, обучающая и тестовая выборки
τ = 0.5 суток [17]. Для сдвига первого дня измере-
представляли собой наборы факторов - десяти-
ний (9-е сутки после посева) был добавлен нуле-
мерных векторов уровней экспрессии генов в
вой день с нулевыми значениями уровней экс-
определенный день и откликов - пятимерных
прессии для всех генов и проведена интерполя-
векторов уровней экспрессии целевых генов на
ция между нулевым и первым днем наблюдений.
следующий день.
В результате такой обработки данных была со-
брана генеральная совокупность данных, содер-
АЛГОРИТМ И ПАРАМЕТРЫ
жащая уровни экспрессии десяти генов (в муль-
ОБУЧЕНИЯ МОДЕЛИ
типлицированной форме) для каждого дня и для
двух условий выращивания. Из этой совокупно-
Для построения и обучения модели использо-
сти случайным образом делали выборки для обу-
валась реализация алгоритма случайного леса
чения и тестирования модели (рис. 1). Модель
«RandomForestRegressor» из пакета sklearn [20]. Для
строилась как регрессионная модель, предсказы-
нахождения оптимальной модели производилась
вающая уровни экспрессии пяти целевых генов
настройка следующих гиперпараметров по соот-
(т. е. генов, экспрессирующихся в апикальной
ветствующим наборам значений:
меристеме) в текущий день по значениям уровней
экспрессии всех десяти генов (т. е. дополнитель-
- количество деревьев: {20, 50, 100, 300},
БИОФИЗИКА том 65
№ 2
2020
266
ПОДОЛЬНЫЙ и др.
Таблица 1. Взаимодействия, учтенные в усеченных моделях, согласно генной сети зацветания у Arabidopsis [15]
Регуляторы
FT-гены
AP1
LFY
TFL1-гены
FD
Мишени
AP1, LFY
LFY, TFL1-гены
AP1, FD
AP1, LFY
AP1, LFY
– минимальное число значений в листе дерева:
векторов X и y, которые соответствуют взаимо-
{10, 20, 50, 100},
действиям из генной сети у Arabidopsis (табл. 1),
- максимальная глубина дерева: {5, 8, 20, без
при этом характер взаимодействия (активация
ограничений}.
или репрессия) априори не постулируется и опре-
деляется в результате обучения модели.
Для каждого фиксированного значения каж-
дого гиперпараметра проводилось обучение мо-
Полная и усеченная модели обучались на дан-
дели на обучающей выборке данных с пятикрат-
ных двух типов - либо на полной выборке дан-
ной кроссвалидацией, в ходе которого миними-
ных, включающей в себя данные по экспрессии
зировалoсь среднеквадратичное отклонение
для двух условий (LD+SD), либо на данных толь-
(СКО) вектора откликов y в модели от данных. В
ко для длинного дня (LD). Таким образом, всего
результате перебора всех значений гиперпарамет-
было построено и обучено четыре модели: полная
ров выбиралась модель, дающая минимальное
модель на данных LD+SD, полная модель на дан-
СКО на валидационных выборках.
ных LD, усеченная модель на данных LD+SD и
усеченная модель на данных LD. В табл. 2 приве-
Были построены два типа моделей вида
дены значения гиперпараметров, найденные в
y = F(X). В моделях первого типа (далее — «пол-
ходе обучения каждой из моделей.
ные» модели) не делалось никаких предположе-
ний о характере регуляции между генами, являю-
щимися входными факторами в модели и входя-
СПОСОБЫ ВЫЧИСЛЕНИЯ
щими в вектор X, и целевыми генами, входящими
ДИНАМИЧЕСКИХ ПРЕДСКАЗАНИЙ
в вектор откликов y модели. Математически это
МОДЕЛИ
выражается в том, что отображение F априори (т.
е. до обучения) включает в себя влияние каждого
После обучения модели F на выборке, содер-
компонента вектора X на каждый компонент век-
жащей данные сразу для всех дней выращивания,
тора y. При построении моделей второго типа (да-
необходимо создать процедуру генерации пред-
лее - «усеченные» модели) использовалось пред-
сказаний модели в динамике. Были рассмотрены
положение о том, что исследуемая генная сеть по-
два способа генерации динамики экспрессии це-
добна соответствующей генной сети у Arabidopsis
левых генов в модели. В первом способе предска-
thaliana [15, 19]. В усеченных моделях отображе-
зание y(d) для экспрессии пяти целевых
ние F связывает только те пары фактор-отклик из
генов в любой день d вычисляли по данным экс-
Таблица 2. Значения гиперпараметров, соответствующие лучшим результатам обучения рассмотренных моделей
Количество
Минимальное число
Максимальная глубина
Модель
деревьев
значений в листе
дерева
Полная LD+SD
100
10
Без ограничений
Полная LD
300
10
Без ограничений
AP1
300
10
Без ограничений
FD
100
100
5
Усеченная LD + SD
LFY
300
10
Без ограничений
TFL1a
100
100
5
TFL1c
100
100
5
AP1
300
10
Без ограничений
FD
300
100
5
Усеченная LD
LFY
300
10
Без ограничений
TFL1a
300
100
5
TFL1c
100
100
5
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
267
Таблица 3. Результаты обучения и тестирования полной модели на выборке LD + SD
НСКО
Коэффициент корреляции
Обучающая выборка
Тестовая выборка
Обучающая выборка
Тестовая выборка
AP1
0.038
0.062
0.97
0.92
FD
0.062
0.074
0.92
0.89
LFY
0.058
0.084
0.89
0.76
TFL1a
0.051
0.062
0.94
0.91
TFL1c
0.064
0.073
0.91
0.88
Примечание. Для каждого гена отдельно приведены оценки качества предсказаний модели на обучающей и тестовой
выборках, в терминах нормализованной среднеквадратичной ошибки (НСКО) и коэффициента корреляции Пирсона.
НСКО вычисляли нормализацией СКО на максимальный уровень экспрессии соответствующего гена.
прессии X(d - 1) всех десяти генов в предыдущий
Для выяснения характера влияния отдельных
день d - 1: y(d) = F(X(d - 1)), d = 2, 3, … . Во вто-
генов на генную сеть с помощью модели симули-
ром способе динамика предсказаний y(d) строи-
ровались нокауты этих генов. При симуляции но-
лась согласно следующему алгоритму:
каутов вычислялись динамические предсказания
модели вторым способом, однако при этом уров-
1. По исходным данным X(1) первого дня (d = 1)
ни экспрессии нокаутированных генов во все дни
вычисляли предсказания экспрессии пяти целе-
фиксировали на нулевых значениях.
вых генов для второго дня (d = 2): y(2) = F(X(1)).
2. Во второй день (d = 2) строился вектор Xy(2),
равный вектору X(2) данных экспрессии для это-
РЕЗУЛЬТАТЫ ОБУЧЕНИЯ
го дня, в котором уровни экспрессии пяти целе-
ПОЛНОЙ МОДЕЛИ
вых генов заменялись на соответствующие уров-
На первом этапе была обучена полная модель
ни экспрессии из предсказания y(2) из предыду-
на совместной выборке, включающей данные для
щего пункта. Таким образом, в векторе Xy(d) из
условий длинного светового дня и короткого све-
данных берутся только уровни экспрессии пяти
тового дня (модель «Полная LD+SD» из табл. 2).
FT-генов.
Результирующая модель примерно одинаково хо-
3. Предсказания для третьего дня (d = 3) вы-
рошо описывает уровни экспрессии всех генов, и
числяли по вектору Xy(2) из предыдущего пункта.
качество предсказаний модели примерно одина-
Таким образом, для любого дня, начиная с тре-
ково на обучающей и тестовой выборках (табл. 3).
тьего, динамику экспрессии пяти целевых генов
Однако полученная таким образом модель
вычисляли по формуле: y(d) = F(Xy(d - 1)), d ≥ 3.
имеет ряд недостатков. Динамические предсказа-
Описанные два способа вычисления динами-
ния модели на усредненных входных данных, вы-
ческих предсказаний модели можно сравнить с
численные с учетом данных на предыдущем дне
решением модели на основе дифференциальных
(первый способ, описанный выше), хорошо соот-
уравнений. Первый из описанных способов гене-
ветствуют данным длинного дня (черные кривые
рации динамических предсказаний модели ана-
на рис. 2). Средний по генам коэффициент кор-
логичен использованному ранее подходу к моде-
реляции между предсказаниями модели и данны-
лированию динамики экспрессии генов цветения
ми в динамике равен r = 0.84 для LD и r = 0.74 для
в сорте нута ICCV 96029 [19]. В этом подходе ди-
SD. Однако динамика усредненной экспрессии в
намические предсказания модели получались пу-
модели при условиях LD значительно ухудшается
тем решения дифференциальных уравнений
при вычислении с учетом откликов модели на
модели, в правой части которых вместо динами-
предыдущем дне (второй способ, описанный вы-
ческих переменных (уровней экспрессии) под-
ше) (красные кривые на рис. 2; средний r = 0.23).
ставлялись экспериментальные данные. Такие
В частности, уровень экспрессии AP1 растет не-
решения должны рассматриваться лишь как ап-
достаточно сильно (рис. 2), и в целом падает кор-
проксимация истинного решения модельных
реляция между предсказаниями модели и данны-
уравнений. Второй способ генерации динамики в
ми. В условиях SD недостаток активации AP1
модели, описанный выше, соответствует такому
проявляется в модели при любом способе вычис-
истинному решению модели, сформулированной
ления динамики экспрессии, а для второго спосо-
в терминах дифференциальных уравнений.
ба для всех генов, кроме AP1, в модели наблюда-
БИОФИЗИКА том 65
№ 2
2020
268
ПОДОЛЬНЫЙ и др.
1400
Модель
300
AP1, LD
AP1, SD
1200
255
Данные
1000
200
800
150
600
100
400
50
200
5
0
0
10
15
20
25
30
35
40
10
15
20
25
30
35
140
80
FD, LD
FD, SD
120
70
100
60
80
50
60
40
40
30
20
10
15
20
25
30
35
40
10
15
20
25
30
35
400
175
LFY, LD
LFY, SD
350
150
300
125
250
100
200
75
150
50
100
50
25
0
0
10
15
20
25
30
35
40
10
15
20
25
30
35
TFL1a, LD
40
TFL1a, SD
60
35
50
30
40
25
30
20
20
15
10
10
10
15
20
25
30
35
40
10
15
20
25
30
35
50
70
TFL1c, LD
TFL1c, SD
45
60
40
50
35
40
30
30
25
20
20
15
10
10
15
20
25
30
35
40
10
15
20
25
30
35
День после посева
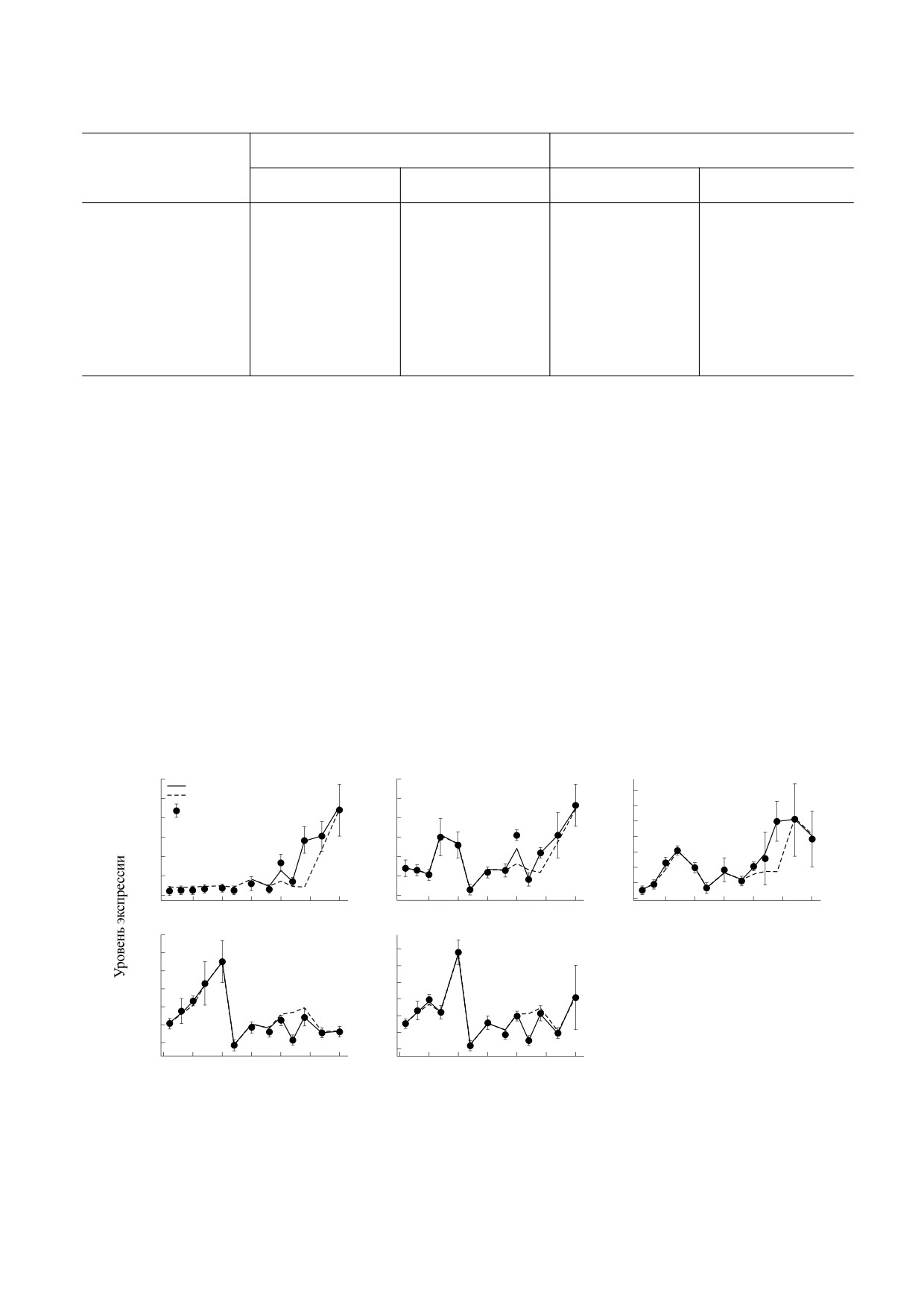
Рис. 2. Динамические предсказания полной модели, обученной на выборке LD+SD, в сравнении со средними
уровнями экспрессии в данных. Черная кривая описывает динамику экспрессии в модели, вычисленную первым
способом, описанным в тексте (предсказания в каждый день зависят от данных в предыдущий день). Пунктирная
кривая показывает динамику экспрессии в модели, вычисленную вторым способом (предсказания в каждый день
зависят от данных FT-генов и предсказаний модели для остальных генов в предыдущий день).
ется аномальное повышение экспрессии на 25-й
r = 0.24 для второго способа вычисления динами-
день (рис. 2); также для второго способа корреля-
ки в условиях SD).
ция между предсказаниями модели и данными
Из этого анализа можно сделать предположе-
низкая (средние r = 0.74 для первого способа и ние о том, что модель не восприимчива к данным
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
269
Таблица 4. Результаты обучения и тестирования полной модели на LD-выборке
НСКО
Коэффициент корреляции
Обучающая выборка
Тестовая выборка
Обучающая выборка
Тестовая выборка
AP1
0.042
0.073
0.96
0.90
FD
0.061
0.075
0.89
0.86
LFY
0.084
0.111
0.85
0.81
TFL1a
0.058
0.071
0.91
0.89
TFL1c
0.063
0.081
0.90
0.88
для условий короткого дня, и поэтому обучение
РЕЗУЛЬТАТЫ ОБУЧЕНИЯ
модели на данных для LD и SD одновременно
УСЕЧЕННОЙ МОДЕЛИ
привело к недостаточно качественной динамике
На следующем этапе была обучена усеченная
в модели и для условий длинного дня. В рамках
модель, в которой явно учтены (как описано вы-
такого предположения полная модель была обу-
ше) только те взаимодействия между генами, ко-
чена только на данных длинного дня (модель
торые присутствуют в генной сети зацветания у
«Полная LD» из табл. 2). Результирующая модель
Arabidopsis thaliana (табл. 1) [15, 19]. Такая задача
показала хорошую степень близости к данным на
обусловлена общим предположением о консерва-
обучающей и тестовой выборках (табл. 4). При
тивности этой генной сети у растений и, следова-
этом, в отличие от случая обучения на объединен-
тельно, направлена на проверку того, могут ли
ной выборке LD+SD, теперь модель хорошо
исследуемые данные экспрессии генов цветения
предсказывает динамику усредненных уровней
у сорта нута CDC Frontier быть результатом функ-
экспрессии всех генов для обоих способов вычис-
ционирования в точности такой генной сети. Как
ления такой динамики (рис. 3; средний r = 0.99
и полная модель, усеченная модель обучалась от-
для первого способа и r = 0.89 для второго способа
дельно на выборке LD+SD и только на LD-вы-
вычисления динамики).
борке.
140
140
AP1, LD
FD, LD
LFY, LD
Модель
175
120
120
150
Данные
100
100
125
80
80
100
75
60
60
50
40
40
25
20
20
10
15
20
25
30
35
40
10
15
20
25
30
35
40
010
15
20
25
30
35
40
70
TFL1a, LD
70
TFL1c, LD
60
60
50
50
40
40
30
30
20
20
10
10
10
15
20
25
30
35
40
10
15
20
25
30
35
40
День после посева
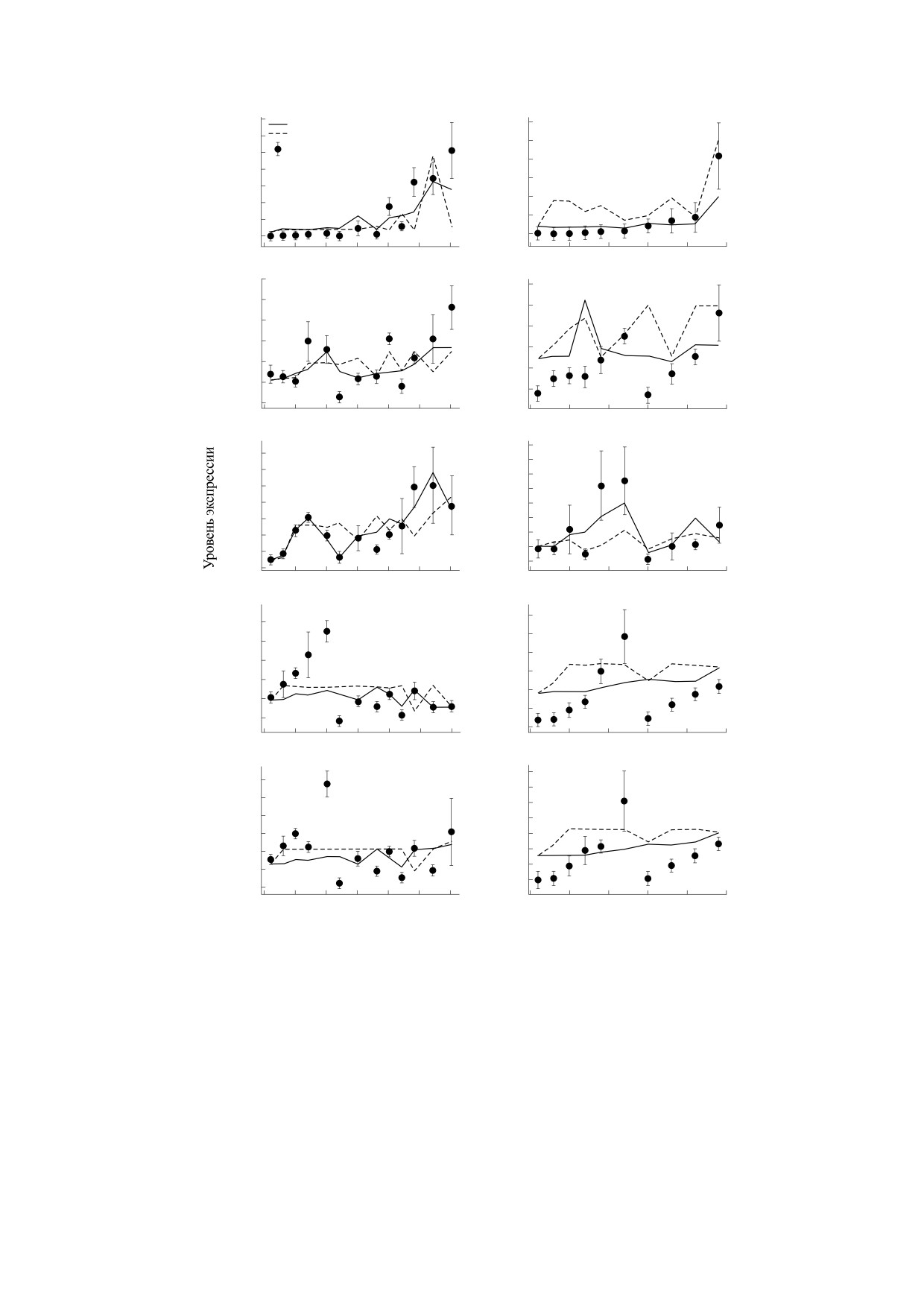
Рис. 3. Динамические предсказания полной модели, обученной на LD-выборке, в сравнении со средними уровнями
экспрессии в данных. Все обозначения соответствуют рис. 2.
БИОФИЗИКА том 65
№ 2
2020
270
ПОДОЛЬНЫЙ и др.
Таблица 5. Результаты обучения и тестирования усеченной модели на выборке LD+SD
НСКО
Коэффициент корреляции
Обучающая выборка
Тестовая выборка
Обучающая выборка
Тестовая выборка
AP1
0.050
0.066
0.94
0.91
FD
0.141
0.145
0.44
0.44
LFY
0.067
0.086
0.85
0.74
TFL1a
0.138
0.143
0.30
0.30
TFL1c
0.148
0.151
0.27
0.25
Усеченная модель, обученная на данных для
АНАЛИЗ НОКАУТОВ ГЕНОВ В МОДЕЛЯХ
условий длинного светового дня и короткого све-
тового дня (модель
«Усеченная LD+SD» из
Для определения характера влияния отдельно-
табл. 2) показала достаточно хорошие корреля-
го гена на экспрессию других в моделях вычисля-
ции на тестовой выборке для генов AP1 и LFY и
ли предсказания в условиях, когда этот ген вы-
плохие корреляции для остальных генов (табл. 5).
ключен («нокаутирован»). Для этого сравнива-
Анализ динамических предсказаний модели на
лись площади под графиком динамики
усредненных данных показал значительные де-
экспрессии гена-мишени в модели для дикого ти-
фекты в динамике экспрессии (рис. 4). В частно-
па и при нокауте. Увеличение этой площади при
сти, в отличие от данных, экспрессия AP1 в моде-
нокауте гена-регулятора соответствует репрессии
ли падает в последний день наблюдений при
(прямой или через другие регуляторные пути в
условиях LD и демонстрирует повышенный уро-
генной сети) со стороны гена-регулятора, а
вень при условиях SD. В случае гена LFY динами-
уменьшение соответствует активации. Визуали-
ка средней экспрессии достаточно хорошая в
зация результатов таких вычислений для всех ге-
обоих условиях роста, но только в случае первого
нов показывает, что модели воспроизводят боль-
способа вычисления динамики в модели, тогда
шую часть регуляторных взаимодействий из ген-
как для второго способа динамические предска-
ной сети для Arabidopsis (рис. 6).
зания в модели значительно ухудшаются (рис. 4).
Основной интерес представляет влияние по-
Остальные гены демонстрируют еще более значи-
тенциальных регуляторов на гены идентичности
тельные дефекты.
цветковых меристем AP1 и LFY, поскольку экс-
Гипотеза о плохой репрезентативности дан-
прессия этих генов является маркером инициа-
ных для условий SD была исследована также в
ции цветения. Во всех моделях TFL1-гены оказы-
контексте усеченной модели, по аналогии с тем,
вают репрессивное действие на эти гены в усло-
как это было сделано с полной моделью. Для это-
виях LD, в то время как в условиях SD их влияние
го усеченная модель была обучена только на LD-
не столь однозначно (рис. 6). Среди FT-генов
данных (модель «Усеченная LD» из табл. 2). Так
только FTa3 проявляет себя как активатор AP1 и
же, как и в случае обучения на выборке LD+SD, в
LFY во всех моделях и в обоих условиях роста (LD
результате были получены достаточно высокие
и SD). Кроме того, FTa3 является сильнейшим
коэффициенты корреляции между предсказани-
активатором для этих генов-мишеней среди всех
ями модели и данными на тестовой выборке толь-
FT-генов в условиях LD. Поскольку полная мо-
ко для экспрессии генов AP1 и LFY (табл. 6). Ана-
дель, обученная на LD-выборке, демонстрирует
лиз динамических предсказаний для средних
наиболее адекватную динамику средней экспрес-
уровней экспрессии в модели выявил дефекты в
сии (рис. 3), для этой модели на рис. 7 показаны
динамике, аналогичные описанным для усечен-
примеры динамики экспрессии гена AP1 при вы-
ной модели, обученной на выборке LD+SD
ключении некоторых генов. В частности, AP1 в
(рис. 5). Таким образом, в отличие от полной мо-
этой модели очень слабо меняется при выключе-
дели, в случае усеченной модели исключение
нии всех FT-генов, кроме FTa3, и практически пе-
данных короткого дня из обучающей выборки ка-
рестает экспрессироваться при выключении FTa3
чественно не улучшило результаты обучения.
(рис. 7а). Также из двух репрессоров (TFL1a и
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
271
1400
300
Модель
AP1, LD
AP1, SD
1200
255
Данные
1000
200
800
150
600
100
400
50
200
5
0
0
10
15
20
25
30
35
40
10
15
20
25
30
35
140
FD, LD
80
FD, SD
120
70
100
60
80
50
60
40
40
30
20
10
15
20
25
30
35
40
10
15
20
25
30
35
400
175
LFY, LD
LFY, SD
350
150
300
125
250
100
200
75
150
50
100
50
25
0
0
10
15
20
25
30
35
40
10
15
20
25
30
35
TFL1a, LD
40
TFL1a, SD
60
35
50
30
40
25
30
20
20
15
10
10
10
15
20
25
30
35
40
10
15
20
25
30
35
50
70
TFL1c, LD
TFL1c, SD
45
60
40
50
35
40
30
30
25
20
20
10
15
10
15
20
25
30
35
40
10
15
20
25
30
35
День после посева
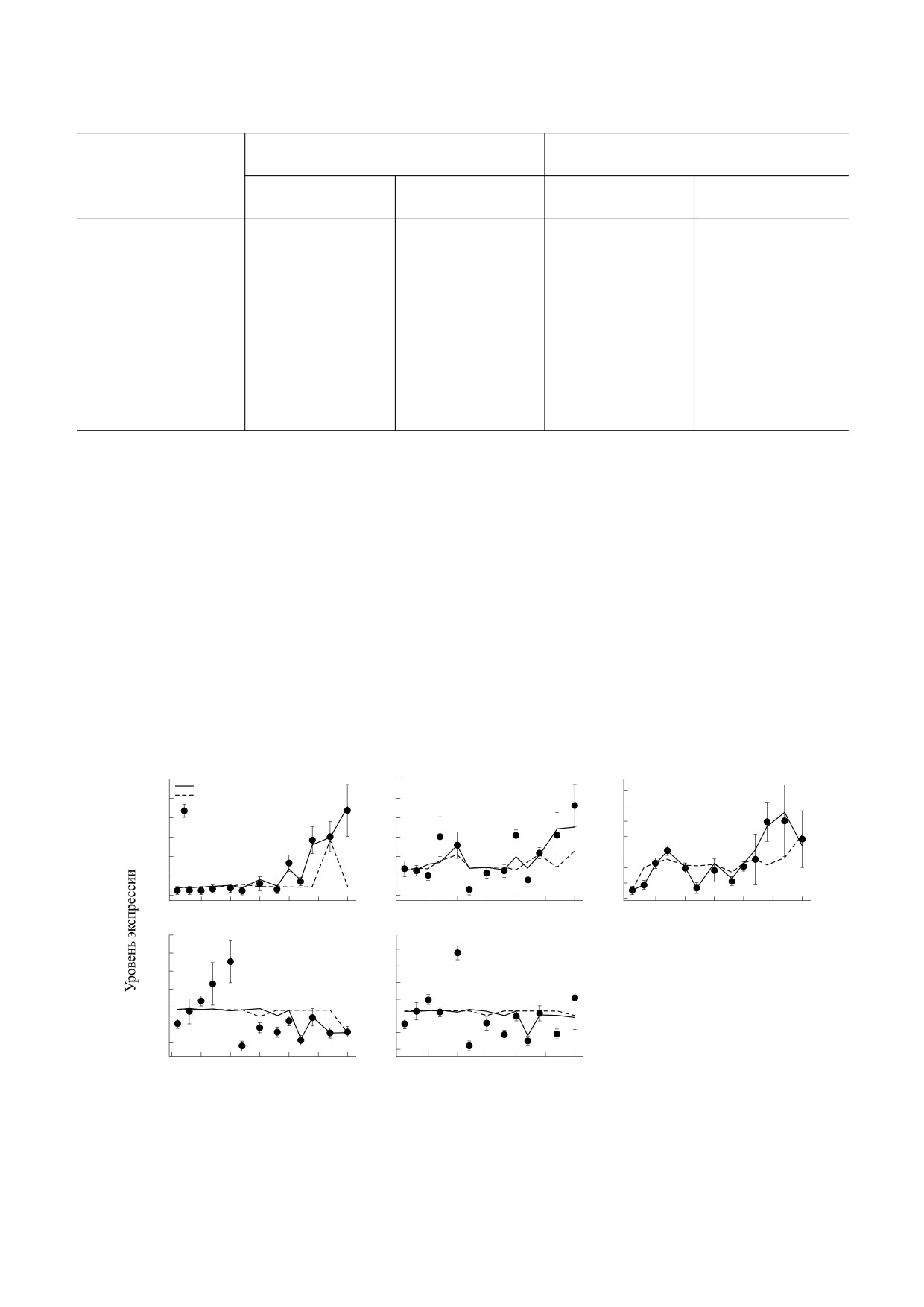
Рис. 4. Динамические предсказания усеченной модели, обученной на выборке LD+SD, в сравнении со средними
уровнями экспрессии в данных. Все обозначения соответствуют рис. 2.
TFL1c) наиболее сильное влияние на AP1 оказы-
в сорте CDC Frontier регуляторного модуля, со-
вает TFL1c (рис. 7в).
стоящего из взаимной активации между AP1 и
LFY. Например, в полной модели, обученной на
В соответствии с результатами, полученными
LD+SD выборке, в условиях LD AP1 слабо ре-
ранее в рамках моделирования экспрессии генов
цветения в сорте нута ICCV 96029 [19], представ-
прессирует LFY, а в условиях SD LFY является
ленные модели также предсказывают нарушение
сильным репрессором AP1 (рис. 6). В полной мо-
БИОФИЗИКА том 65
№ 2
2020
272
ПОДОЛЬНЫЙ и др.
Таблица 6. Результаты обучения и тестирования усеченной модели на LD-выборке
НСКО
Коэффициент корреляции
Обучающая выборка
Тестовая выборка
Обучающая выборка
Тестовая выборка
AP1
0.050
0.075
0.94
0.90
FD
0.108
0.117
0.59
0.60
LFY
0.077
0.113
0.88
0.80
TFL1a
0.125
0.141
0.41
0.39
TFL1c
0.139
0.163
0.29
0.26
дели, обученной на LD-выборке, LFY является
модели позволяют вычислять динамику средних
активатором AP1 (рис. 7б), однако AP1 слабо ре-
уровней экспрессии, которая хорошо соотносит-
прессирует LFY (рис. 6).
ся с наблюдаемой динамикой в данных [14] в слу-
чае, когда данные используются как входные зна-
чения для предсказаний уровня экспрессии на
ЗАКЛЮЧЕНИЕ
следующий день. Этот результат контрастирует с
плохими результатами предсказания такой дина-
Полученные результаты демонстрируют при-
мики для сорта CDC Frontier, полученными ранее
менимость методов машинного обучения для
с помощью модели на основе дифференциальных
анализа экспрессии генов цветения в нуте. Алго-
уравнений [19].
ритм случайного леса показывает хорошее каче-
ство обучения и тестирования на данных экс-
Из четырех построенных моделей только пол-
прессии в сорте нута CDC Frontier в условиях
ная модель, обученная на LD-выборке, позволяет
длинного дня. Построенные на этом обучении
корректно воспроизводить динамику средней
140
140
AP1, LD
FD, LD
LFY, LD
Модель
175
120
120
150
Данные
100
100
125
80
80
100
75
60
60
50
40
40
25
20
20
10
15
20
25
30
35
40
10
15
20
25
30
35
40
010
15
20
25
30
35
40
70
TFL1a, LD
70
TFL1c, LD
60
60
50
50
40
40
30
30
20
20
10
10
10
15
20
25
30
35
40
10
15
20
25
30
35
40
День после посева
Рис. 5. Динамические предсказания усеченной модели, обученной на LD-выборке, в сравнении со средними
уровнями экспрессии в данных. Все обозначения соответствуют рис. 2.
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
273
Полная модель LD+SD
Полная LD-модель
AP1_LD
AP1_LD
1.05
FD_LD
1.8
FD_LD
0.90
_LD
LFY
LFY_LD
0.75
TFL1a_LD
1.5
TFL1a_LD
0.60
_LD
TFL1c
AP1_SD
1.2
TFL1c_LD
0.45
FD_SD
LFY_SD
0.9
TFL1a_SD
TFL1c_SD
0.6
У
сеченная модель LD+SD
Усеченная LD-модель
1.05
AP1_LD
AP1_LD
FD_LD
1.05
0.90
FD_LD
LFY_LD
LFY_LD
0.75
TFL1a_LD
1.90
TFL1a_LD
TFL1c_LD
0.60
AP1_SD
0.75
TFL1c_LD
0.45
FD_SD
0.60
LFY_SD
TFL1a_SD
0.45
TFL1c_SD
Выключенный ген
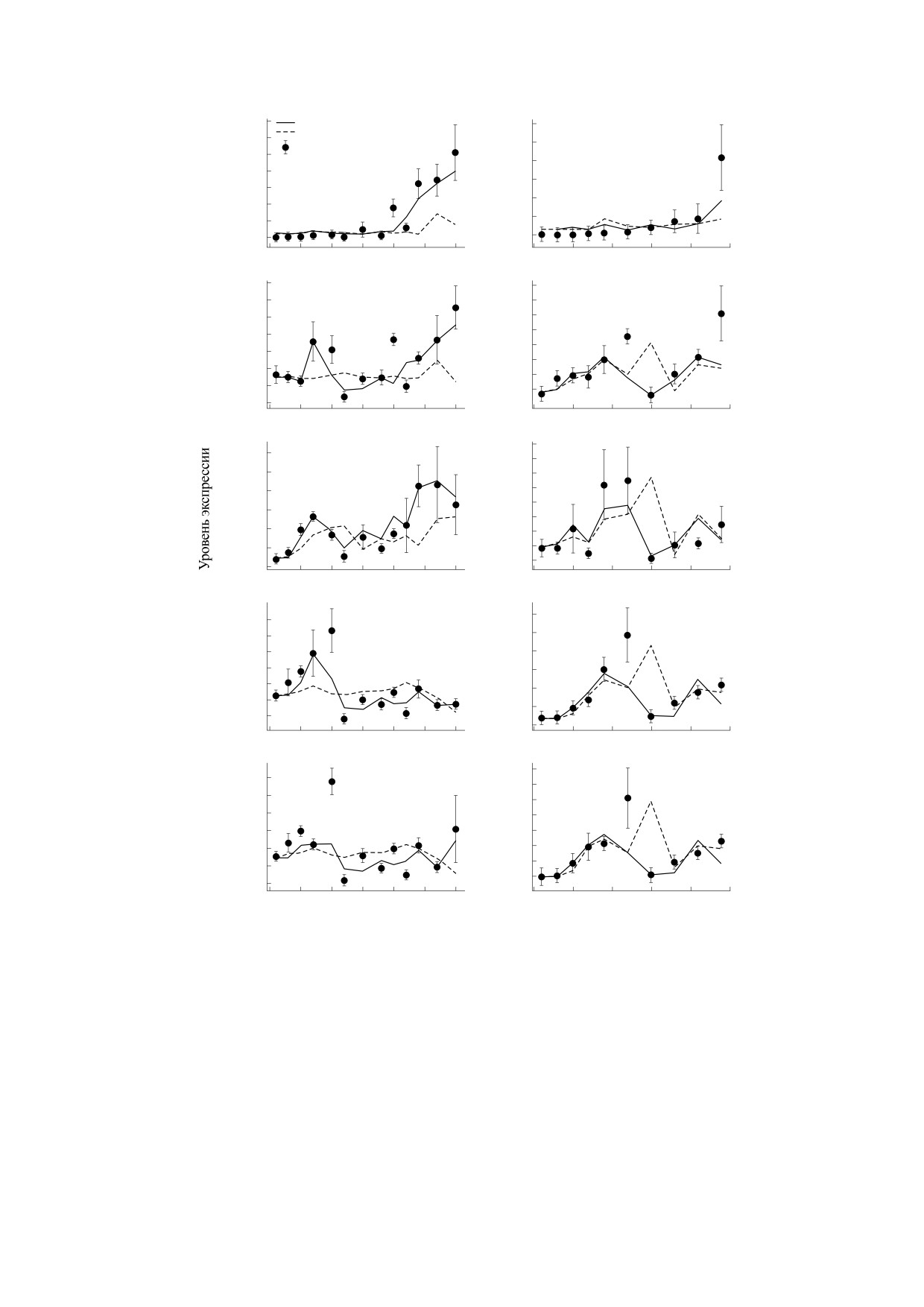
Рис. 6. Результаты моделирования нокаутов генов. Для каждой из четырех моделей показано отношение площади под
кривой динамики среднего уровня экспрессии гена-мишени в модели при одном выключенном гене-регуляторе
(AUC_KO) к такой же площади для условий дикого типа, т. е. когда все регуляторы присутствуют в модели (AUC_WT),
отдельно для условий SD и LD. Динамику экспрессии в модели вычисляли вторым способом, описанным в тексте.
Значение AUC_KO/AUC_WT > 1 при выключенном регуляторе соответствует репрессии гена-мишени этим
регулятором, а значения AUC_KO/AUC_WT < 1 - активации.
экспрессии в случае, когда такая динамика вы-
Наличие нескольких FT-генов и нескольких
числяется не на основе данных в предыдущий
TFL1-генов в нуте ставит вопрос о функциональ-
день, а используя выходы самой модели в преды-
ной роли отдельных гомологов и о характере по-
дущий день (кроме данных экспрессии FT-генов,
тенциальной активации и репрессии генов цвете-
являющихся внешними факторами в модели).
ния отдельными FT- и TFL1-генами соответ-
Этот результат свидетельствует, что использова-
ственно. Из представленных результатов
ние SD-данных в обучении скорее ухудшает каче-
моделирования можно сделать предположение о
ство предсказаний модели. Также модели, обу-
ведущей роли FTa3 в активации и TFL1c в репрес-
ченные с учетом и без учета данных короткого
сии генов идентичности цветковых меристем в
дня, предсказывают разный характер взаимодей-
условиях длинного дня. Как и для сорта нута
ствий между генами. Таким образом, моделиро-
ICCV 96029 [19], в случае CDC Frontier моделиро-
вание показывает, что SD- и LD-данные содер-
вание также выделяет взаимную активацию меж-
жат качественно разную информацию, что,
ду AP1 и LFY в качестве одного из регуляторных
возможно, связано с наличием разных регулятор-
модулей, нарушение которого отличает нут от
ных взаимодействий в условиях короткого и
Arabidopsis. Общая изменчивость характера взаи-
длинного дня.
модействий, наблюдающаяся между моделями,
БИОФИЗИКА том 65
№ 2
2020
274
ПОДОЛЬНЫЙ и др.
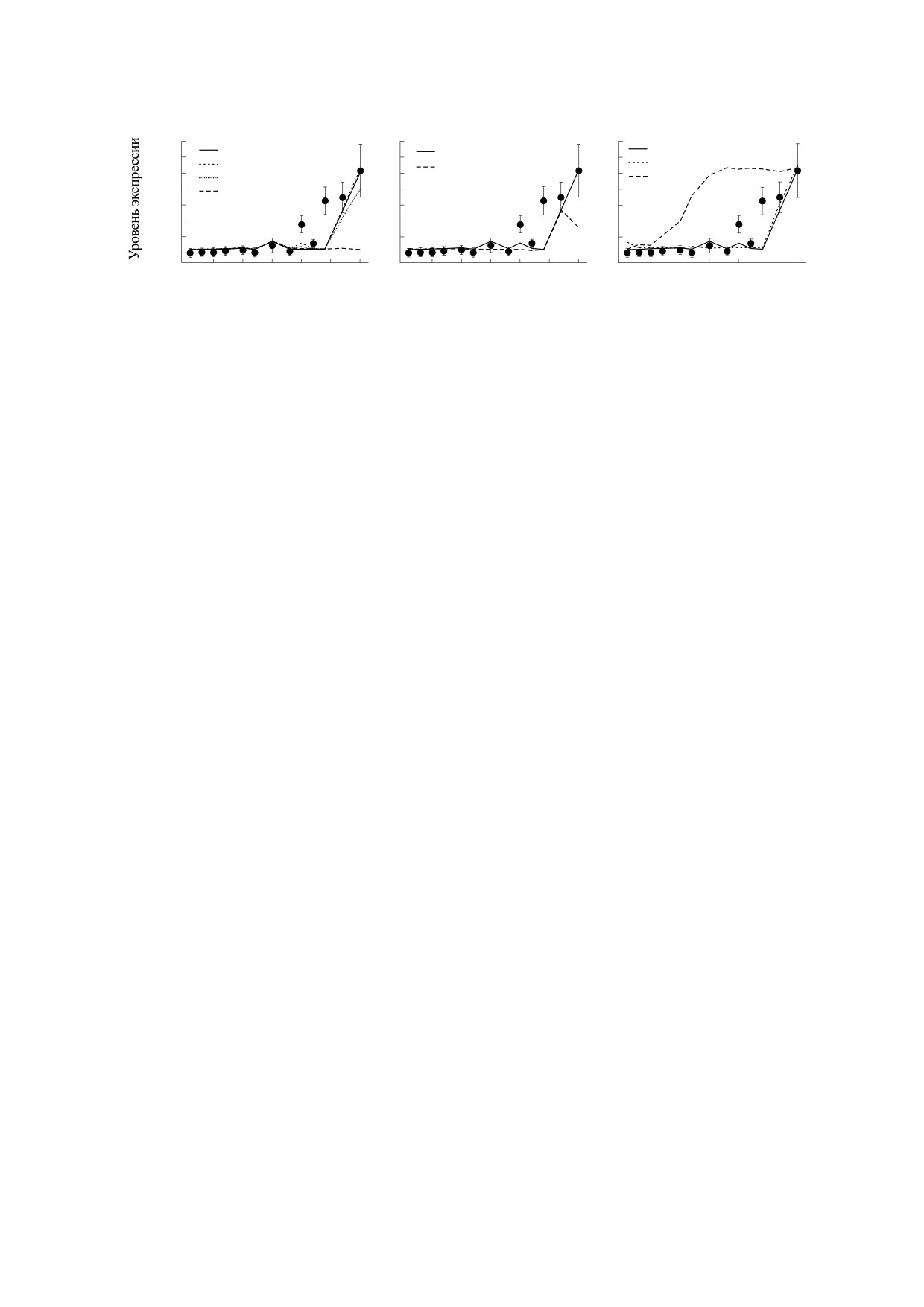
(а)
(б)
(в)
1400
Дикий тип
Дикий тип
Дикий тип
1200
FTa1, FTa1, FTc
LFY
TFL1a
1000
FTb
TFL1c
800
FTa3
600
400
200
0
10
15
20
25
30
35
40
10
15
20
25
30
35
40
10
15
20
25
30
35
40
День после посева
Рис. 7. Динамика экспрессии AP1 в полной модели, обученной на LD-выборке, в условиях, когда один из следующих
генов выключен: (а) - один из FT-генов, (б) - LFY-ген, (в) - один из TFL1-генов. Для сравнения на каждом графике
также показана динамика экспрессии в диком типе (черная кривая). Название выключенных генов приведены на
графиках рядом с цветом соответствующей кривой. Кривые, соответствующие нокауту каждого из генов FTa1, FTa2 и
FTc, показаны одной пунктирной линией в силу слабого различия между этими кривыми. Кривая дикого типа
сливается с этими кривыми на графике (а).
построенными с учетом и без учета генной сети из
8.
J. L. Weller and R. Ortega, Front. Plant Sci. 6, 207
Arabidopsis, вероятно, свидетельствует о недоста-
(2015). DOI: 10.3389/fpls.2015.00207
точности взаимодействий, лежащих в основе
9.
C.-H. Jung, С. E. Wong, M. B. Singh, and P. L. Bhalla,
этой сети, для адекватного описания данных экс-
прессии генов цветения в CDC Frontier.
PloS One 7, e38250 (2012). DOI: 10.1371/journal.
pone.0038250
ФИНАНСИРОВАНИЕ РАБОТЫ
10.
H. D. Upadhyaya, D. Bajaj, S. Das, et al., Plant Mol.
Biol. 89, 403 (2015). DOI: 10.1007/sl1103-015-0377-z
Работа выполнена при финансовой поддержке
Министерства науки и высшего образования Рос-
11.
J. L. Weller and R. C. Macknight, Methods Mol. Biol.
сийской Федерации, задание № 1.8697.2017/БЧ.
1822,
261
(2018). DOI: 10.1007/978-1-4939-8633-
0_17
КОНФЛИКТ ИНТЕРЕСОВ
12.
R. Ortega, V. F. Hecht, J. S. Freeman, et al., Front.
Автор заявляет об отсутствии конфликта инте-
Plant
Sci.
10,
824
(2019).
DOI:
ресов.
10.3389/fpls.2019.00824
13.
M. Blümel, N. Dally, and C. Jung, Curr. Opin. Bio-
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
tech. 32, 121 (2015). DOI: 10.1016/j.copbio.2014.11.023
Настоящая работа не содержит описания ка-
ких-либо исследований с использованием людей
14.
S. Ridge, A. Deokar, R. Lee, et al., Plant Physiol. 175,
и животных в качестве объектов.
802 (2017). DOI: 10.1104/pp.l7.00082
15.
К. E. Jaeger, N. Pullen, S. Lamzin, et al., Plant Cell.
СПИСОК ЛИТЕРАТУРЫ
25, 820 (2013). DOI: 10.1105/tpc. 113.109355
1. С. Jung and А. Е. Müller, Trends Plant Sci. 14, 563
16.
F. C. Sussmilch, A. Berbel, V. Hecht, et al., Plant Cell.
(2009). DOI: 10.1016/j.tplants.2009.07.005
27, 1046 (2015). DOI: 10.1105/tpc. 115.136150
2. A. Kanth and M. Schmid, Cell Mol. Life Sci. 68, 2013
(2011). DOI: 10.1007/s00018-011-0673-y
17.
F. Valentim, S. van Mourik, D. Posé, et al., PloS One
3. M. Khan, X Ai, and J. Zhang, Wiley Interdiscip. Rev.
10
(2), e0l16973
(2015). DOI:
10.1371/journal.
RNA 5, 347 (2014). DOI: 10.1002/wma.l215
pone.0116973
4. J. A. Banta, I. M. Ehrenreich, S. Gerard, et al., Ecol.
18.
R. K. Varshney, C. Song, R. K. Saxena, et al., Nat. Bio-
Lett.
15,
769
(2012). DOI:
10.1111/j.1461-0248.
2012.01796.x
technol. 31, 240 (2013). DOI: 10.1038/nbt.2491
5. F. Andrés and G. Coupland, Nat. Rev. Genet. 13, 627
19.
V. V. Gursky, K. N. Kozlov, S. V. Nuzhdin, and
(2012). DOI: 10.1038/nrg3291
M. G. Samsonova, Front. Gen. 9, 547 (2018). DOI:
6. V. Hecht, F. Foucher, C. Ferrándiz, et al., Plant
10.3389/fgene.2018.00547
Physiol. 137, 1420 (2005). DOI: 10.104/pp.l04.057018
7. R. Benlloch, A. Berbel, L. Ali, et al., Front. Plant Sci.
20.
F. Pedregosa, G. Varoquaux, A. Gramfort, et al., J.
6, 543 (2015). DOI: 10.3389/fpls.2015.00543
Machine Learning Res. 12, 2825 (2011).
БИОФИЗИКА том 65
№ 2
2020
АНАЛИЗ ЭКСПРЕССИИ ГЕНОВ ЦВЕТЕНИЯ
275
Machine-Learning Analysis of Flowering Gene Expression
in CDC Frontier Chickpea Cultivar
B.S. Podolny*, V.V. Gursky**, and M.G. Samsonova*
*Peter the Great St. Petersburg Polytechnic University, Polytekhnicheskaya ul. 29, St. Petersburg 195251 Russia
**Ioffe Physical Technical Institute, Polytekhnicheskaya ul. 26, St. Petersburg, 194021 Russia
We analyze gene expression dynamics in floral transition in the CDC Frontier chickpea cultivar. We provide
a model, in several versions, to predict the expression dynamics of five flowering genes taking the expression
of their regulators as the input. The models were trained using the random forest method on previously pub-
lished expression data for ten flowering genes under the short- and long-day growing conditions. The result-
ing models correctly predict the dynamics of the average expression levels under long days. We show that the
models for CDC Frontier mainly reproduce regulatory interactions between the key genes described for the
model plant Arabidopsis thaliana. Based on the analysis, we hypothesize that the short-day data and the long-
day data contain qualitatively different information, which can be related to different regulatory modules
functioning in different conditions. For the regulators of the flower meristem identity genes AP1 and LFY,
our models predict FTa3 as the main activator and TFL1c as the main repressor under long days.
Keywords: chickpea, CDC Frontier, flowering gene network, random forest algorithm
БИОФИЗИКА том 65
№ 2
2020