БИОФИЗИКА, 2021, том 66, № 3, с. 440-446
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.322.7, 577.323.7, 535.56, 543.422.8
СТРУКТУРНАЯ ОРГАНИЗАЦИЯ ЯДЕРНОГО БЕЛКА HMGB1
И ЕЕ ВЛИЯНИЕ НА ФОРМИРОВАНИЕ УПОРЯДОЧЕННЫХ
НАДМОЛЕКУЛЯРНЫХ КОМПЛЕКСОВ
© 2021 г. Е.В. Чихиржина*, Т.Ю. Старкова*, А.М. Поляничко*, **
*Институт цитологии РАН, 194064, Санкт-Петербург, Тихорецкий просп., 4
E-mail: e.chikhirzhina@incras.ru
**Санкт-Петербургский государственный университет, 199034, Санкт-Петербург, Университетская наб., 7/9
E-mail: a.polyanichko@spbu.ru
Поступила в редакцию 02.12.2019 г.
После доработки 02.12.2019 г.
Принята к публикации 04.03.2021 г.
Белок HMGB1 является одним из ключевых клеточных белков. Основные функции HMGB1 вы-
полняет в ядре, являясь компонентом сложных белок-белковых и ДНК-белковых комплексов.
Прежде всего HMGB1 играет важную роль в основных клеточных процессах: транскрипции, репли-
кации, репарации. Помимо ядра этот белок обнаружен в цитоплазматическом и внеклеточном про-
странстве. Несмотря на достаточно большое количество работ, посвященных исследованию струк-
туры и функций белка HMGB1, на сегодня нет четкого представления о молекулярных механизмах,
определяющих разнообразие выполняемых им функций. В работе рассматриваются особенности
структурной организации белка HMGB1 и ее влияние на взаимодействие белка с ДНК и другими
белками.
Ключевые слова: белок HMGB1, ДНК, гистон Н1, структура хроматина.
DOI: 10.31857/S0006302921030030
Хроматин эукариотических клеток представ-
скрипции, репликации, рекомбинации и репара-
ляет собой достаточно сложную и динамичную
ции ДНК [8, 9].
систему, в которой ДНК взаимодействует не
только с хорошо изученными гистонами, но и с
СТРУКТУРА БЕЛКА HMGB1
большим количеством негистоновых белков [1-
Белок HMGB1 (как и гомологичный ему
6]. Среди них наиболее интересными, на наш
HMGB2) состоит из двух гомологичных ДНК-
взгляд, являются широко распространенные в
связывающих HMGB-доменов, соединенных ко-
хроматине и эволюционно консервативные пред-
ротким линкером (пять-семь аминокислотных
ставители обширной группы белков с высокой
остатков), небольшого N-концевого фрагмента и
электрофоретической подвижностью (High Mo-
длинного С-концевого участка. Последний со-
bility Group). Наиболее многочисленными пред-
стоит из непрерывной последовательности трид-
ставителями этой группы являются белки семей-
цати дикарбоновых аминокислот [1].
ства HMGB, содержащие один или несколько
В составе ДНК-связывающего HMGB-домена
структурно-консервативных ДНК-связывающих
(~80 аминокислотных остатков) можно выделить
домена (так называемые HMGB-домены) [2, 7, 8].
три спиральных участка (I, II, и III), формирую-
Двухдоменный белок HMGB1, как и гистон Н1,
щих необычную Г-образную структуру, которая
взаимодействует с линкерной ДНК и формирует
стабилизируется сильными гидрофобными взаи-
наднуклеосомные уровни структурной организа-
модействиями в вершине угла между спиралями I
ции хроматина [2, 4]. Белок HMGB1 хорошо из-
и II [10]. Стоит отметить, что помимо HMGB1/2 к
вестен как «архитектурный» фактор транскрип-
белкам HMGB-семейства относится также и це-
ции, осуществляющий свои функции путем сбор-
лый ряд транскрипционных факторов [2, 11-17].
ки на ДНК транскрипционно активного
Относительно недавно было показано [18-20],
многокомпонентного белкового комплекса [3, 8].
что HMGB-белки способны изменять свою про-
Кроме того, он играет важную роль в функциони-
странственную структуру в зависимости от мише-
ровании генома, в том числе на этапах тран-
ни связывания, что позволило их сравнить с при-
440
СТРУКТУРНАЯ ОРГАНИЗАЦИЯ ЯДЕРНОГО БЕЛКА HMGB1
441
родно неупорядоченными белками [8, 21]. Кон-
Белки Н1 и HMGB1 взаимодействуют с меж-
формационная подвижность полипептидной
нуклеосомными участками ДНК [2, 5]. Исследо-
цепи HMGB-белков, по всей видимости, важна
вания взаимодействия этих белков с ДНК различ-
для сборки сложных ДНК-белковых и белок-бел-
ными физико-химическими методами показало,
ковых комплексов, активно участвующих в регу-
что связывание с ДНК одного из белков в значи-
ляции структуры и динамики хроматина [22-25].
тельной степени облегчает взаимодействие с ней
второго белка [4, 6, 39-42]. HMGB1 и Н1 взаимо-
действуют с ДНК по разным бороздкам двойной
ЛОКАЛИЗАЦИЯ БЕЛКА HMGB1
спирали, изменяя ее структуру сходным образом.
Разнообразие выполняемых белком HMGB1
В нескольких работах было показано прямое
функций отчасти связано с тем, что он может
взаимодействие между белками Н1 и HMGB1 [40,
иметь ядерную, цитоплазматическую и внекле-
42-45]. Следует отметить, что на взаимодействие
точную локализацию. Локализация белка зависит
между этими белками также влияет степень окис-
от характера посттрансляционных модификаций
ления цистеина в HMGB1 [7, 46], что хорошо со-
(в первую очередь ацетилирования в областях
гласуется с описанными в литературных источ-
ядерной локализации (nuclear localization se-
никах функциями белка. Кроме того, окисление
quences, NLS), фосфорилирования и метилиро-
цистеина влияет и на взаимодействие HMGB1 с
вания), а также окислительно-восстановительно-
рядом других мишеней. Было показано и непо-
го статуса остатков цистеина [2, 26-28]. Восста-
средственное взаимодействие HMGB1 с гисто-
новленная форма белка (восстановлены все
ном Н3, которое происходит с участием С-конце-
цистеины С23, С45 и С106) характерна для ядер-
вого участка HMGB1 и гибкого N-концевого
ной локализации HMGB1, где белок функциони-
фрагмента гистона [19].
рует как «шаперон», принимая активное участие
Как упоминалось выше, белок HMGB1 явля-
в процессах транскрипции, репарации, рекомби-
ется участником сложных функционально-зна-
нации, репликации, осуществляя посадку на нук-
чимых белковых комплексов. В литературе нако-
леосому хроматин-ремоделирующего комплекса,
пилось довольно много данных о прямом взаимо-
и связывание с ДНК транскрипционных факто-
действии HMGB1 с большим количеством других
ров [29, 30].
белков. Так, и in vivo, и in vitro показано прямое
Ацетилирование HMGB1 в областях ядерной
связывание HMGB1 с белком р53, который акти-
локализации приводит к транслокации белка в
вируется при различных повреждениях ДНК, а
цитоплазму, где он принимает участие в регули-
также регулирует множество клеточных процес-
ровании процессов аутофагии и апоптоза [31].
сов, таких как клеточный цикл, апоптоз, диффе-
В случае некроза или другого повреждения це-
ренцировка, старение, репарация ДНК
[8].
лостности клетки HMGB1 попадает во внекле-
HMGB1 стимулирует связывание р53 с конкрет-
точное пространство, где постепенно происходит
ными сайтами ДНК, в том числе с участками, мо-
окисление остатков цистеина в положениях С23,
дифицированными цисплатином [47]. В экспери-
С45 и С106. В зависимости от степени окисления
ментах in vitro было показано, что HMGB1 может
белок выступает в роли сигнальной молекулы,
выступать в качестве «шаперона» при взаимодей-
инициирующей миграцию клеток, реакцию им-
ствии р53 с ДНК [48]. На первом этапе происхо-
мунного ответа клетки, синтез противовоспали-
дит сильный изгиб ДНК, индуцированный
тельных цитокинов [26-28, 32-34]. Выполнение
HMGB1, а затем р53 прочно связывается с ДНК,
белком внеклеточных сигнальных функций дало
а HMGB1 уходит из комплекса. При этом с р53
предпосылки отнести HMGB1 к классу аларми-
связан А-домен HMGB1, а само взаимодействие
нов [28, 35-38]. Молекулярные механизмы, ле-
регулируется его С-концевым участком [49]. Та-
жащие в основе многообразия выполняемых
кой комплекс р53/HMGB1 регулирует аутофагию
HMGB1 функций, на сегодняшний день остают-
и апоптоз внутри живых клеток [50, 51].
ся до конца не выясненными.
Белок HMGB1 участвует во всех путях процес-
са рекомбинации. Он принимает участие в фор-
мировании мультибелкового комплекса при ре-
ВЗАИМОДЕЙСТВИЕ HMGB1 С ГИСТОНОМ
парации некомплементарных нуклеотидов при
Н1 И ДРУГИМИ БЕЛКАМИ
репликации ДНК (mismatch repair, MMR).
Наряду с линкерным гистоном Н1 белок
HMGB1 работает на начальных этапах распозна-
HMGB1 играет ключевую роль в функциониро-
вания повреждения/разрыва ДНК и взаимодей-
вании хроматина. Как уже отмечалось выше,
ствует с белками MMR - белком теплового шока
HMGB1 принимает активное участие в формиро-
р70, MSH2 и MLH1 [52-54]. Эксцизионная репа-
вании сложного транскрипционно активного
рация оснований и нуклеотидов (base excision re-
комплекса, в связи с чем его относят к «архитек-
pair, BER и nucleotide excision repair, NER) также
турным» факторам транскрипции.
связаны с взаимодействием между HMGB1 и раз-
БИОФИЗИКА том 66
№ 3
2021
442
ЧИХИРЖИНА и др.
личными ферментами [55-60]. При восстановле-
Характерной особенностью HMGB1 (так же,
нии двунитевых разрывов ДНК (double-strand
как и HMGB2) является его способность узнавать
break repair, DSBR) происходит связывание гете-
и взаимодействовать с участками ДНК, имею-
родимера Ku70/Ku80 с поврежденными концами
щими различные структурные нарушения: од-
ДНК, что активирует работу каталитической
нонитевые разрывы, 4Н ДНК [68-70], платино-
субъединицы ДНК-зависимой протеинкиназы
вые аддукты, образованные на ДНК в результате
(DNA-PKcs) [61]. Фосфорилирование этой про-
действия противоопухолевых препаратов (карбо-
теинкиназы приводит к конформационным из-
платин, цисплатин) и т.д. [2]. Связывание этих
менениям поврежденных концов ДНК, что об-
белков с такими поврежденными областями ДНК
легчает работу нуклеаз и лигаз при восстановле-
является сигналом для начала работы репараци-
нии разрывов. Белок HMGB1 стимулирует in vitro
онной системы клетки. В экспериментах in vitro
активность протеинкиназы DNA-PKcs [61, 62],
показано, что в присутствии HMGB-доменных
белков RAG1 и RAG2 [63] и усиливает активность
белков замедляется восстановление повреждений
ДНК-лигазы Т4 [64]. При рекомбинации именно
ДНК в месте образования платиновых аддуктов.
формирование комплекса из трех белков RAG1,
В результате увеличивается эффективность таких
RAG2 и HMGB1 приводит к образованию на
препаратов.
ДНК шпильки [65, 66]. Причем этот белковый
Сравнительно недавно было показано, что мо-
комплекс стабилен во время всего процесса ре-
лекула белка HMGB1 находится в динамическом
комбинации. Присутствие HMGB1 активирует
равновесии между закрытым (свернутым) и от-
лиазу dRP, участвующую в восстановлении одно-
крытым (развернутым) состояниями [18-20]. В
цепочечных разрывов ДНК. HMGB1 взаимодей-
неактивном состоянии отрицательно заряжен-
ствует с ДНК в месте образования платиновых ад-
ный С-концевой домен взаимодействует с линке-
дуктов, защищая от восстановления поврежден-
ром (остатки аргинина 72 и 162, лизина 81 и 164 и
ные цисплатином участки ДНК [57]. Важную
изолейцина 158 [71]), что обеспечивает его распо-
роль в этом процессе играют и посттрансляцион-
ложение в полости между положительно заря-
ные модификации HMGB1, в первую очередь
женными ДНК-связывающими доменами и ста-
ацетилирование и фосфорилирование [58, 60].
билизацию белковой молекулы в целом [19]. Пе-
реход в функционально-активное развернутое
состояние сопровождается нарушением данного
ВЗАИМОДЕЙСТВИЕ HMGB1 С ДНК
взаимодействия и, следовательно, разворачива-
нием белка HMGB1 [20]. Регуляция данного кон-
ДНК-связывающая функция белков HMGB1
формационного перехода может включать как
и HMGB2 на сегодняшний день наиболее изуче-
структурно-адаптивные механизмы, когда в зави-
на и подробно описана в литературе [2, 8, 67]. Вза-
симости от объекта связывания белок способен
имодействие HMGB1/2 с ДНК характеризуется
изменять свою структуру, так и наличие пост-
формированием изгибов двойной спирали ДНК
трансляционных модификаций [18].
при связывании с белком и/или способностью
белка узнавать и избирательно связываться с
HMGB1 не является сиквенс-специфичным
участками ДНК, имеющими различные наруше-
белком. Однако некоторыми авторами показано,
ния структуры.
что с ГЦ-богатыми участками ДНК в основном
взаимодействует В-домен HMGB1 [72]. В случае
Роль HMGB-доменов. При взаимодействии с
АТ-богатых последовательностей в связывании с
ДНК ароматические аминокислотные остатки в
ДНК принимают участие оба ДНК-связывающих
составе HMGB-доменов частично интеркалиру-
домена.
ют со стороны малой бороздки, изгибая ДНК в
Роль С-концевого домена. При сравнении ами-
сторону большой бороздки, при этом угол изгиба
нокислотной последовательности белков группы
может достигать 140° [1]. Совокупные исследова-
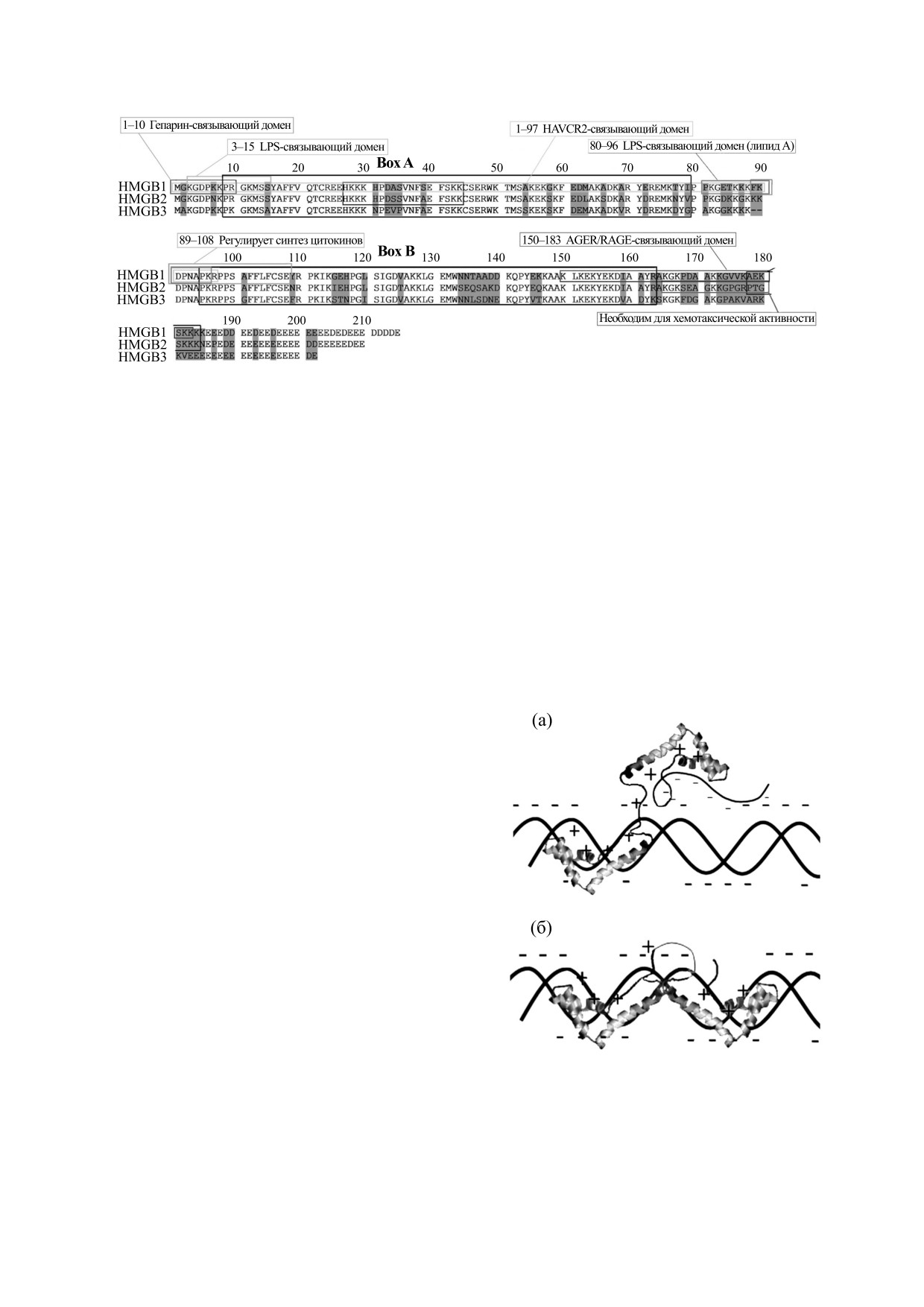
HMGB1-3 (рис. 1) можно заметить, что все три
ния процесса формирования ДНК-белковых
белка по первичной структуре очень близки. От-
комплексов методами кругового дихроизма, ана-
личие между ними проявляется в длине неупоря-
литического ультрацентрифугирования и ретар-
доченного С-концевого домена: у HMGB2 и
дации в агарозном геле показали, что при образо-
HMGB3 они короче, чем у HMGB1, на 5 и 15 а.к.
вании ДНК-белкового комплекса при большом
соответственно. Как уже отмечалось выше, имен-
содержании HMGВ1 в комплексе основную роль
но этот домен отвечает за взаимодействие белка с
играют белок-белковые взаимодействия между
другими молекулами, в том числе с ДНК и гисто-
молекулами HMGВ1. В любом случае при связы-
нами.
вании с ДНК проявляется специфичность не к
нуклеотидной последовательности, а к простран-
Основные работы, посвященные исследова-
ственной организации молекулы ДНК, и эта спе-
нию влияния сильно заряженного С-концевого
цифичность определяется ДНК-связывающими
участка HMGB1 на связывание белка с ДНК, вы-
доменами белка.
полнялись с использованием спектроскопиче-
БИОФИЗИКА том 66
№ 3
2021
СТРУКТУРНАЯ ОРГАНИЗАЦИЯ ЯДЕРНОГО БЕЛКА HMGB1
443
Рис. 1. Аминокислотная последовательность двудоменнх негистоновых белков HMGB1, HMGB2 и HMGB3. Рисунок
ских подходов (ультрафиолетовая и инфракрас-
HMGB1 (как и гомологичный ему белок
ная спектроскопия, круговой дихроизм) и атом-
HMGB2) может служить стимулирующим тран-
ной силовой микроскопии. Сравнительный
скрипцию фактором [4]. Существует гипотеза,
анализ взаимодействия ДНК с полноразмерным
согласно которой гистон Н1 и белок HMGB1 об-
HMGB1 и с укороченным белком HMGB1-
разуют единый комплекс с ДНК. С одной сторо-
(A+B), состоящим только из двух ДНК-связыва-
ны, оба белка взаимодействуют с линкерным
ющих доменов, выявил значительное отличие в
участком ДНК, с другой - Н1 располагается по
структуре формирующихся комплексов [73-75].
большой бороздке двойной спирали, а HMGB1 -
в малой [10], при одновременном связывании с
Было установлено, что рекомбинантный бе-
ДНК они сходным образом изменяют структуру
лок HMGB1-(A+B) образует с ДНК комплексы,
ДНК и тем самым облегчают друг другу связыва-
обладающие высокой степенью упорядоченности
ние с ней. Исследования взаимодействия гистона
и аномально высокой оптической активностью
Н1 и белка HMGB1 с ДНК различными физико-
[73, 76, 77]. С помощью метода атомной силовой
микроскопии было показано, что для данных си-
стем характерно формирование своего рода «жгу-
тов», сплетенных из нитей ДНК-белкового ком-
плекса [75]. Это говорит о доминирующей роли
межмолекулярных взаимодействий в такой си-
стеме. Образующиеся ассоциаты сформированы
сразу несколькими молекулами ДНК в составе
ДНК-белкового комплекса и состоят из
одинаково ориентированных «цилиндров», име-
ющих очень близкие размеры. Расположенные
вокруг нити ДНК содержат чрезвычайно большое
количество изломов, что указывает на высокое
число связанных с ней молекул белка. На основе
полученных данных была предложена модель
связывания белка HMGB1 с ДНК (рис. 2). Со-
гласно этой модели, при взаимодействии ДНК с
полноразмерным белком связывание происходит
посредством только одного ДНК-связывающего
домена (рис. 2а). В случае рекомбинантного белка
HMGB1-(А+В) во взаимодействии с ДНК участ-
вуют оба ДНК-связывающих домена (рис. 2б).
Связанные с межнуклеосомным участком
ДНК, белок HMGB1 и линкерный гистон Н1 яв-
Рис. 2. Модель связывания белка HMGB1 с ДНК: (а) -
взаимодействие ДНК с полноразмерным белком
ляются ключевыми белками в функционирова-
HMGB1; (б) - взаимодействие ДНК с рекомбинант-
нии хроматина [2,3,6]. В то время как гистон Н1
ным белком HMGB1-(А+В), который состоит только
является репрессором транскрипции, белок
из ДНК-связывающих доменов.
БИОФИЗИКА том 66
№ 3
2021
444
ЧИХИРЖИНА и др.
химическими методами (круговой дихроизм в
КОНФЛИКТ ИНТЕРЕСОВ
ультрафиолетовом и инфракрасном диапазоне,
Авторы заявляют об отсутствии конфликта
абсорбционная спектроскопия, спектрофотомет-
интересов.
рическое плавление, гель-ретардация) показыва-
ют, что связывание этих белков с ДНК скорее
всего не носит конкурентного характера, а обла-
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
дает признаками кооперативного взаимодей-
Настоящая работа не содержит описания ис-
ствия [4, 6, 39-42].
следований с использованием людей и животных
в качестве объектов.
Анализ структуры комплексов ДНК/HMGB1 в
присутствии гистона Н1 с помощью метода кру-
гового дихроизма в инфракрасном диапазоне
СПИСОК ЛИТЕРАТУРЫ
позволяет увидеть связывание отдельных белков
1.
M. Bustin and R. Reeves, Proc. Nat. Acad. Sci. USA
как с сахаро-фосфатным остовом, так и с основа-
54, 35 (1996).
ниями ДНК [39-41, 78]. При одновременном
2.
M. Stros, Biochim. Biophys. Acta 1799, 101 (2010).
присутствии в комплексе с ДНК обоих белков,
гистон Н1 взаимодействует преимущественно с
3.
E. Chikhirzhina, G. Chikhirzhina, and A. Polyanich-
фосфатными группами ДНК, частично экрани-
ko, Biomed. Spectr. Imaging 3, 345 (2014).
руя их заряд, чем облегчает связывание HMGB1 с
4.
Y. V. Postnikov and M. Bustin, Biochim. Biophys. Acta
основаниями ДНК в малой бороздке. Белок-бел-
1859, 462 (2016).
ковые взаимодействия стимулируют конденса-
5.
E. Chikhirzhina, T. Starkova, and A. Polyanichko, Bio-
цию ДНК с образованием крупных ДНК-белко-
physics 63, 858 (2018).
вых комплексов [79]. Методом АСМ показано,
6.
E. Chikhirzhina, T. Starkova, and A. Polyanichko, Bio-
что для комплексов ДНК/HMGB1 в присутствии
physics 65, 202 (2020).
гистона Н1 характерно формирование фибрил-
7.
M. Stros, E. Polanska, M. Kucirek, et al., PLoS One
лярных структур, сплетенных из нитей ДНК, ко-
торые удерживаются между собой молекулами
10, e0138774 (2015).
белков [40, 75]. Эти данные указывают на перво-
8.
R. Reeves, DNA Repair 36, 122 (2015).
степенную роль взаимодействий между HMGB1
9.
P. Mandke and K. M. Vasquez, DNA Repair (Amst.)
и H1 при формировании тройного комплекса.
83, 102701 (2019).
10.
A. M. Read, P. D. Cary, C. Crane-Robinson, et al.,
Nucl. Acids Res. 21, 3427 (1993).
ЗАКЛЮЧЕНИЕ
11.
T. Chi, Nat. Rev. Immunol. 4, 965 (2004).
Таким образом, мы видим доминирующую
12.
M. Stros, D. Launholt, and K. D. Grasser, Cell. Mol.
роль неупорядоченного отрицательно заряжен-
Life Sci. 64, 2590 (2007).
ного С-концевого фрагмента белка HMGB1 как
13.
L. H. Pevny and S. K. Nicolis, Int. J. Biochem. Cell
при связывании белка с ДНК, так и при его взаи-
модействии с другими белковыми молекулами
Biol. 42, 421 (2010).
[79, 80]. Этот участок снижает эффективность
14.
P. Bernard and V. R. Harley, Int. J. Biochem. Cell Biol.
связывания белка с ДНК. В его отсутствие во вза-
42, 400 (2010).
имодействии с ДНК принимают участие оба
15.
F. Oppel, N. Müller, G. Schackert, et al., Mol Cancer.
ДНК-связывающих домена. Связывание двух до-
10, 137 (2011).
менов белка с ДНК приводит к образованию меж-
16.
O. Leis, A. Eguiara, E. Lopez-Arribillaga, et al., Onco-
молекулярных «скрепок» на ДНК и стимулирует
gene 31, 1354 (2012).
формирование надмолекулярного порядка в си-
стеме, что влияет на прочность связывания белка
17.
A. Lai, M. Wan, J. Wu, et al., Proc. Natl. Acad. Sci.
с ДНК, а также на структуру и стабильность обра-
USA 106, 1169 (2009).
зующихся комплексов.
18.
J. O. Thomas and K. Stott, Biochem. Soc. Trans. 40,
341 (2012).
19.
M. Watson, K. Stott, and J. O. Thomas, J. Mol. Biol.
ФИНАНСИРОВАНИЕ РАБОТЫ
374, 1286 (2007).
Работа выполнена при финансовой поддержке
20.
K. Stott, M. Watson, F. S. Howe, et al., J. Mol. Biol.
Российского фонда фундаментальных исследова-
403, 706 (2010).
ний (гранты № 18-04-01199 (исследование струк-
21.
V. N. Uversky, Eur. J. Biochem. 269, 2 (2002).
туры HMGB1 в зависимости от мишени связыва-
22.
L. Breydo, J. M. Redington, and V. N. Uversky, Int.
ния и роли белка в процессах клеточной диффе-
Rev. Cell. Mol. Biol. 329, 145 (2017).
ренцировки) и
№ 18-08-01500 (изучение
структуры ДНК-белковых комплексов)).
23.
V. N. Uversky, Prot. Sci. 22, 693 (2013).
БИОФИЗИКА том 66
№ 3
2021
СТРУКТУРНАЯ ОРГАНИЗАЦИЯ ЯДЕРНОГО БЕЛКА HMGB1
445
24. V. N. Uversky, Cell. Mol. Life Sci. 74, 3065 (2017).
51. K. M. Livesey, R. Kang, P. Vernon, et al., Cancer Res.
72, 1996 (2012).
25. A. L. Darling and V. N. Uversky, Molecules 22, pii:
52. E. Y. Krynetski, N. F. Krynetskaia, M. E. Bianchi,
E2027 (2017).
et al., Cancer Res. 63, 100 (2003).
26. E. Venereau, M. Casalgrandi, M. Schiraldi, et al., J.
53. F. Yuan, L. Gu, S. Guo, C. Wang, et al., J. Biol. Chem.
Exp. Med. 209, 1519 (2012).
279, 20935 (2004).
27. H. Yang, P. Lundback, L. Ottosson, et al., Mol. Med.
54. Y. Zhang, F. Yuan, S. R. Presnell, et al., Cell 122, 693
(2005).
18, 250 (2012).
55. R. Prasad, Y. Liu, L. J. Deterding, et al., Mol. Cell. 27,
28. A. Raucci, S. Di Maggio, F. Scavello et al., Cell. Mol.
829 (2007).
Life Sci. 76, 211 (2019).
56.
Y. Liu, R. Prasad, and S. H. Wilson, Biochim. Biophys.
29. M. Stros, M. Kucirek, S.A. Sani, et al., Biochim. Bio-
Acta 1799, 119 (2010).
phys. - Acta Gen. Regul. Mech. 1861, 200 (2018).
57.
J. Malina, J. Kasparkova, G. Natile, et al., Chem. Biol.
9, 629 (2002).
30. G. Verrijdt, A. Haelens, E. Schoenmakers, et al., Bio-
58.
I. Ugrinova, I. G. Pashev, and E. A. Pasheva, Biochem-
chem. J. 361 (Pt 1), 97 (2002).
istry 48, 6502 (2009).
31. X. Zhu, J. S. Messer, Y. Wang, et al., J. Clin. Invest.
59.
I. Ugrinova, S. Zlateva, I. G. Pashev, et al., Int. J. Bio-
125, 1098 (2015).
chem. Cell Biol. 41, 1556 (2009).
32. H. Yang, H. Wang, Z. Ju, et al., J. Exp. Med. 212, 5
60.
I. Ugrinova, I. G. Pashev, and E. A. Pasheva, Mol. Biol.
Rep. 36, 1399 (2009).
(2015).
61.
D. J. Sawchuk, J. Mansilla-Soto, C. Alarcon, et al., J.
33. K. Stark, V. Philippi, S. Stockhausen, et al., Blood 128,
Biol. Chem. 279, 29821(2004).
2435 (2016).
62.
Y. Yumoto, H. Shirakawa, M. Yoshida, et al., J. Bio-
34. M. Tirone, N. L. Tran, C. Ceriotti, et al., J. Exp. Med.
chem. 124, 519 (1998).
215, 303 (2018).
63.
D. C. van Gent, K. Hiom, T. T. Paull, et al., EMBO J.
16, 2665 (1997).
35. E. Venereau, C. Ceriott, and M. E. Bianchi, Front. Im-
64. M. Stros, D. Cherny, and T. M. Jovin, Eur. J. Biochem.
munol. 6, 442 (2015).
267, 4088 (2000).
36. N. Lohani and M. R. Rajeswari, Curr. Prot. Pept. Sci.
65.
A. J. Little, E. Corbett, F. Ortega, et al., Nucl. Acids
17, 762 (2016).
Res. 41, 3289 (2013).
37. M. E. Bianchi, M. P. Crippa, A. A. Manfredi, et al.,
66.
R. B. West and M. R. Lieber, Mol.Cell Biol. 18, 6408
(1998).
Immunol. Rev. 280, 74 (2017).
67.
R. Reeves, Biochim. Biophys. Acta 1799, 3 (2010).
38. Б. И. Кузник, В. Х. Хавинсон, Н. С. Линькова
68.
T. C. Johnstone, J. J. Wilson, and S. J. Lippard, Inorg.
и др., Успехи физиол. наук 48, 40 (2017).
Chem. 52, 12234 (2013).
39. A. Polyanichko and E. Chikhirzhina, Spectroscopy 27,
69.
Q. Wang, M. Zeng, W. Wang, et al., Biochem. Biophys.
393 (2012).
Res. Commun. 360, 14 (2007).
40. A. M. Polyanichko and E.V. Chikhirzhina, J. Mol.
70.
F. Totsingan and A. J. Bell Jr., Prot. Sci. 22, 1552
Struct. 1044, 167 (2013).
(2013).
41. A. Polyanichko, E. Chikhirzhina, V. Andruschchenko,
71.
S. Knapp, S. Muller, G. Digilio, et al., Biochem. 43,
et al., Biopolymers 83, 182 (2006).
11992 (2004).
42. E. V. Chikhirzhina, V. I. Vorob’ev, and A. M. Polyanic-
72.
S. Muller, M. E. Bianchi, and S. Knapp, Biochem. 40,
hko, Mol. Biol. 47, 299 (2013).
10254 (2001).
43. L. Cato, K. Stott, M. Watson, et al., Mol. Biol. 384,
73.
E. Chikhirzhina, A. Polyanichko, Z. Leonenko, et al.,
1262 (2008).
Spectroscopy 24, 361 (2010).
44. L. A. Kohlstaedt, E. C. Sung, A. Fujishige, et al., J. Bi-
74.
А. М. Поляничко, С. Г. Давыденко, Е. В. Чихир-
ol. Chem. 262, 524 (1987).
жина и др., Цитология 42, 787 (2000).
45. L. A. Kohlstaedt and R. D. Cole, Biochemistry 33, 570
75.
A. M. Polyanichko, Z. V. Leonenko, D. Cramb, et al.,
(1994).
Biophysics 53, 202 (2008).
76.
E. V. Chikhirzhina, A. M. Polyanichko, A. N. Skvor-
46. E. Polanska, S. Pospisilova, and M. Stros, PLoS One 9,
tsov, et al., Mol. Biol. 36, (2002).
e89070 (2014).
77.
A. M. Polyanichko, E. V. Chikhirzhina, A. N. Skvort-
47. T. Imamura, H. Izumi, G. Nagatani, et al., J. Biol.
sov, et al., J. Biomol. Struct. Dyn. 19, 1053 (2002).
Chem. 276, 7534 (2001).
78.
A. Polyanichko and H. Wieser, Biopolymers 78, 329
48. K. McKinney and C. Prives, Mol. Cell Biol. 22, 6797
(2005).
(2002).
49. P. Rowell, K. L. Simpson, K. Stott, et al., Structure 20,
79.
E. V. Chikhirzhina, T. Yu. Starkova, A. Beljajev, et al.,
2014 (2012).
Inter. J. Mol. Sci. 21, 7948 (2020).
50. K. M. Livesey, R. Kang, H. J. Zeh 3rd, et al., Autophagy
80.
Е. В. Чихиржина, А. М. Поляничко и Т. Ю. Стар-
8, 846 (2012).
кова, Цитология 62, 716 (2020).
БИОФИЗИКА том 66
№ 3
2021
446
ЧИХИРЖИНА и др.
Structural Organization of the Nuclear Protein HMGB1 and Its Effect
on the Formation of the Ordered Supramolecular Complexes
E.V. Chikhirzhina*, Т.Yu. Starkova*, and А.M. Polyanichko*, **
*Institute of Cytology, Russian Academy of Sciences, Tikhoretsky prosp. 4, St. Petersburg, 194064 Russia
**St. Petersburg State University, Universitetskaya nab. 7/9, St. Petersburg, 199034 Russia
HMGB1 is one of the key proteins of the cell. HMGB1 performs its main functions predominantly in the cell
nucleus, being an essential component of DNA-protein and multiprotein complexes. It plays an important
role in such cellular processes as transcription, replication, and DNA repair. However, it was also found out-
side the nucleus: in cytoplasmic and extracellular space. Despite numerous papers on the structure and func-
tioning of HMGB1, the molecular mechanisms, which determine a vast variety of functions this protein per-
forms, still remain unclear. In this paper, we reported recent data on organization of the protein structure, its
effect on interactions of HMGB1 with DNA and other proteins.
Keywords: HMGB1 protein, DNA, histone Н1, chromatin structure
БИОФИЗИКА том 66
№ 3
2021