БИОФИЗИКА, 2022, том 67, № 2, с. 222-228
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.323.7
АНАЛИЗ СТРУКТУРЫ ЭЛОНГАЦИОННЫХ КОМПЛЕКСОВ
В ПОЛИАКРИЛАМИДНОМ ГЕЛЕ МЕТОДОМ ФЁРСТЕРОВСКОГО
РЕЗОНАНСНОГО ПЕРЕНОСА ЭНЕРГИИ
© 2022 г. Н.С. Герасимова*, А.Н. Коровина*, Д.А. Афонин*,
К.В. Шайтан*, А.В. Феофанов*, В.М. Студитский*, **
*Биологический факультет Московского государственного университета имени М.В. Ломоносова,
119234, Москва, Ленинские горы, 1/12
** Центр исследований рака Фокс Чейз, Филадельфия, PA 19111-2497, США
E-mail: shordome@gmail.com
Поступила в редакцию 10.12.2021 г.
После доработки 13.01.2022 г.
Принята к публикации 14.01.2022 г.
Для изучения структурных особенностей остановленных элонгационных комплексов, формирую-
щихся в ходе транскрипции нуклеосом РНК-полимеразой 2, предложено использовать подход, ос-
нованный на измерении эффективности фёрстеровского резонансного переноса энергии в поли-
акриламидном геле после разделения интермедиатов транскрипции методом электрофореза. Флуо-
ресцентно-меченые нуклеосомы были получены путем введения пары флуорофоров (донора и ак-
цептора) в соседние супервитки нуклеосомной ДНК. Остановленные элонгационные комплексы
формировали, проводя транскрипцию нуклеосом в присутствии ограниченного набора нуклеозид-
трифосфатов. Сравнительный анализ эффективности фёрстеровского резонансного переноса
энергии для свободных нуклеосом и полученных на их основе элонгационных комплексов с РНК-
полимеразой 2 выявил изменение структуры нуклеосом в составе элонгационных комплексов. По-
казано, что элонгационные комплексы с одинаковой длиной РНК могут существовать в различных
конформационных состояниях.
Ключевые слова: транскрипция, хроматин, нуклеосома, РНКП 2, FRET, элонгационный комплекс
DOI: 10.31857/S0006302922020028
Все матричные РНК и большинство клеточ-
Эукариотическая ядерная ДНК организована
ных некодирующих регуляторных РНК синтези-
с привлечением большого количества ядерных
руются РНК-полимеразой 2 (РНКП 2). Несмотря
белков в надмолекулярную нуклеопротеидную
на то, что хроматин удаляется при активации ге-
структуру - хроматин. Минимальной единицей
нов в живых клетках, кодирующие области уме-
хроматина является нуклеосома, которая состоит
ренно транскрибируемых генов остаются упако-
из участка ДНК длиной 145-147 п.н., накрученно-
ванными в нуклеосомы
[2], таким образом,
го на октамер белков-гистонов, включающий
РНКП 2 встречается с нуклеосомами во время
центральный тетрамер (H3-H4)2 и два димера
элонгации транскриптов. Нуклеосома представ-
H2A-H2B [1]. Нуклеосомная упаковка затрудняет
ляет собой барьер для РНКП 2 [3-6], для его пре-
доступ ферментов и белковых факторов к ДНК,
одоления транскрипция в хроматине происходит
поэтому существуют специальные механизмы
по сложному механизму, включающему форми-
рование внутринуклеосомных петель ДНК и
взаимодействия белков с нуклеосомой, обеспе-
обеспечивающему сохранение тетрамера гисто-
чивающие успешное протекание процессов тран-
нов (H3-H4)2 и одного из димеров H2A-H2B на
скрипции, репликации и репарации эукариоти-
ДНК во время транскрипции [7-9].
ческой ядерной ДНК.
Продвижение РНКП 2 вдоль протяженных
участков ДНК, организованных в нуклеосомы
Сокращения: РНКП 2 - РНК-полимераза 2, ЭК - элонга-
ционный комплекс, FRET - фёрстеровский резонансный (более сотен тысяч п.н.), происходит с высокой
перенос энергии (Förster resonance energy transfer), ПААГ -
скоростью (3-4 тысячи п.н. в минуту [10]). По-
полиакриламидный гель, ЭК-5 - элонгационный ком-
добная скорость транскрипции наблюдается
плекс с расположением активного центра РНК-полимера-
зы за 5 п.н. до входа в нуклеосомную ДНК.
in vitro на ДНК, свободной от гистонов [11, 12].
222
АНАЛИЗ СТРУКТУРЫ ЭЛОНГАЦИОННЫХ КОМПЛЕКСОВ
223
Это достигается за счет действия белковых фак-
ществляли 30 циклов повторения 93°С 10 с, 55°С
торов. Ключевые данные для понимания меха-
10 с, 72°С 15 с и дополнительно выдерживали 3 мин
низмов действия факторов позволяют получать
при 72°С в программируемом термостате Veriti Ther-
структурные исследования [13-16].
mal Cycler (Life Technologies, США). Продукты ам-
плификации очищали от компонентов реакции с
В настоящей работе для изучения структурных
использованием набора для очистки ДНК из агароз-
особенностей элонгационных комплексов (ЭК)
ного геля и реакционных смесей Cleanup Standard
РНКП 2 с нуклеосомой применен разработанный
(«Евроген», Россия).
ранее метод анализа нуклеосом в полиакрила-
мидном геле методом детекции фёрстеровского
Для получения «липкого» конца для лигирования
резонансного переноса энергии (FRET, Förster
элонгационного комплекса РНКП 2 полученную
resonance energy transfer) [17]. Основным инстру-
ДНК инкубировали с ферментом TspRI в буфере
ментом для детекции структурных перестроек в
CutSmart (NEB, США). Эффективность реакции
нуклеосомной ДНК в этих исследованиях были
определяли методом электрофореза в
8%-м
флуоресцентные метки, составляющие пару для
ПААГ (соотношение акриламида и бисакрилами-
наблюдения FRET. Эффективность такого пере-
да 19 : 1) в денатурирующих условиях в присут-
носа энергии зависит от расстояния между метка-
ствии мочевины в концентрации 8 М в 0.5× буфе-
ми. Ранее была показана успешность применения
ре ТВЕ (44.5 мМ триса, 44.5 мМ борной кислоты,
таких нуклеосом для изучения процесса тран-
1 мМ ЭДТА) в камере для вертикального электро-
скрипции с детекцией FRET [18], однако в неко-
фореза Mini-PROTEAN Tetra Cell (Bio-Rad Labo-
торых случаях анализ невозможен без предвари-
ratories, США). Определение длин одноцепочеч-
тельного разделения комплексов в полиакрила-
ных фрагментов ДНК проводили по сигналу флу-
мидном геле (ПААГ).
оресцентной метки с помощью лазерного сканера
Amersham Typhoon RGB (Cytiva, Швеция). ДНК-
фрагменты, расщепленные эндонуклеазой с эф-
МАТЕРИАЛЫ И МЕТОДЫ
фективностью не менее 95%, очищали методом
Матрица ДНК. Работу проводили на ДНК с
препаративного электрофореза. Полученные
позиционирующей нуклеосому последователь-
фрагменты ДНК разделяли в ходе электрофореза
ностью 603 [19]: 5'd(CAAGCGACACCGGCACTG-
(4-6 В/см, 2 ч) в 1.5%-м агарозном геле в 0.5× бу-
GG^CCCGGTTCGCGCTCCCTCCTTCCGTGT-
фере ТВЕ с добавкой 4 М мочевины в камере для
GTTGTCGTCTCTCGGGCGTCTAAGTACGCT-
горизонтального электрофореза Mini-Sub Cell GT
TTCACTCGGGCTTCTAAGTACGCTTAGCGC-
(«Bio-Rad Laboratories», США). Концентрацию
ACGGTAGAGCGCAATCCAAGGCTAACCACC-
ДНК измеряли спектрофотометрически с ис-
GTGCATCGATGTTGGAAGAGGCCCTCCGTC-
пользованием оборудования Nanodrop 2000c UV-
CTGAATTCTTCAAGTCCCTGGGGTACGGAT-
Vis CC (Thermo Fisher Scientific, США).
CCGACG)3' (последовательность 603 подчеркну-
Сборку нуклеосом на анализируемых фрагмен-
та, сайт узнавания TspRI выделен полужирным
тах ДНК проводили методом ступенчатого диа-
шрифтом, место разрезания сахарофосфатного
лиза против буферных растворов с понижающей-
остова указано знаком ^). Плазмида, содержащая
ся ионной силой. В качестве источника октаме-
последовательность
603
[19], была любезно
ров гистонов использовали хроматин без
предоставлена доктором Дж. Видомом.
линкерных гистонов, выделенный из эритроци-
Амплификацию фрагментов ДНК проводили ме-
тов цыплят согласно протоколу, подробно опи-
тодом полимеразной цепной реакции с использова-
санному в работе [20]. Так, в буфере, содержащем
нием флуоресцентно-меченых олигонуклеотидов
10 мМ трис-HCl, pH 7.5, 0.1% NP-40, 0.2 мМ
ProxFw40
(5'd(CAAGCGACACCGGCACTGGGC-
ЭДТА, 5 мМ бета-меркаптоэтанола и 1 M NaCl,
CCGGTTCGCGCT*CCCTCCTT-CCGTGTGTTG-
смешивали фрагменты ДНК и хроматин в соот-
TCGTCTCTCGGGCGTCTAAGTACGCTT)) и
ношении 1 : 4 по массе ДНК (например, 2 мкг
ProxRev
(5'd(ACCCCAGGGACTTGAAGTAATAA-
синтетических фрагментов и 8 мкг хроматина).
GGACGGAGGGCCTCTTTCAACATCGATGCAC-
Диализ проводили при температуре 4°С против
GGT#GGTTAG)) (по 0.5 мкМ каждый) (IDT, Гер-
буферных растворов, содержащих 10 мМ трис-
мания), где T* и T# - соответственно Су3- и Су5-
HCl, pH 7.5, 0.1% NP-40, 0.2 мМ ЭДТА, 5 мМ бе-
меченые нуклеотиды. В качестве матрицы исполь-
та-меркаптоэтанола и хлористый натрий в пони-
зована плазмида с модифицированной последова-
жающейся концентрации (1 М в первом буфере,
тельностью. Реакцию проводили в буфере для Taq-
инкубация 1 ч; 0.75 М - 1 ч; 0.5 М - 2.5 ч; 0.01 М - в
ДНК-полимеразы, содержащем 3 мМ MgCl2, дезок-
течение ночи). Анализ продуктов сборки прово-
сирибонуклеотиды (в концентрации 0.2 мМ каж-
дили методом электрофоретического разделения
дый) и Taq-ДНК-полимеразу (0.05 ед. акт./мкл). Все
в 4.5%-м ПААГ в 0.5× буфере ТВЕ в камере для
реактивы производства компании «Евроген» (Рос-
вертикального электрофореза Mini-PROTEAN
сия). Пробы инкубировали 3 мин при 94°С, осу-
Tetra Cell (Bio-Rad Laboratories, США).
БИОФИЗИКА том 67
№ 2
2022
224
ГЕРАСИМОВА и др.
Очистку РНК-полимеразы 2 Saccharomyces cere-
пользована так называемая
«пропускающая»
visiae проводили по методике, описанной в работе
транскрипционная ориентация, которая лучше
[21].
воспроизводит основные свойства транскрипции
хроматина РНКП 2 in vivo, такие как сохранение
Транскрипцию нуклеосом РНК-полимеразой 2
тетрамера гистонов H3/H4 на ДНК и потерю ча-
начинали со сборки элонгационных комплексов
РНКП 2 (200 нМ), дуплекса матричной ДНК и
сти димеров гистонов H2A/H2B [25, 26]. В ходе
РНК и нематричной цепи ДНК при 25°С в тече-
амплификации в ДНК в составе синтетических
олигонуклеотидов (затравок для ПЦР) внесены
ние 15 мин в транскрипционном буфере TB40
флуоресцентные метки, которые при сборке нук-
(20 мМ трис HCl, pH 7.9, 5 мМ MgCl2, 40 мМ KCl
леосом оказываются в соседних супервитках
и 1 мМ бета-меркаптоэтанола) (номер ТВ обозна-
ДНК и составляют FRET-пару (Су3 в качестве до-
чает концентрацию KCl, процесс сборки элонга-
нора энергии и Су5 - в качестве акцептора)
ционного комплекса с указанием используемых
(рис. 1а). Такие нуклеосомы позволяют наблю-
олигонуклеотидов подробно описан в работе
дать за конформационными изменениями в ДНК
[22]). Полученный ЭК лигировали с нуклеосом-
(рис. 1б), сопутствующими различным процес-
ным фрагментом (40 нМ) в присутствии 0.2 мг/мл
сам, например, транскрипции нуклеосомной
бычьего сывороточного альбумина, 1% полиэти-
ДНК.
ленгликоля-8000, 0.5 мМ АТФ и ДНК-лигазы
бактериофага Т4 в течение 16 ч при 4°С. Добавля-
Схема эксперимента по транскрипции пред-
ли ГТФ и АТФ до концентрации 10 мкМ, ЦТФ до
ставлена на рис. 1в. Элонгационный комплекс
концентрации 2 мкМ и инкубировали 15 мин при
собирали из РНКП 2, дуплекса матричной ДНК и
25°С. Таким образом получали ЭК с расположе-
РНК и нематричной цепи ДНК, лигировали с
нием активного центра РНКП 2 на расстоянии
нуклеосомным фрагментом, добавляли ограни-
5 нт до входа в нуклеосомную ДНК (ЭК-5). После
ченный набор рибонуклеозидтрифосфатов
добавления всех нуклеозидтрифосфатов в кон-
(АТФ, ЦТФ, ГТФ). Так получали ЭК с располо-
центрации 400 мкМ и KCl до 150 мМ элонгацию
жением активного центра РНКП 2 за 5 нт до входа
транскрипта продолжали 10 мин при 25°С. Реак-
в нуклеосомную ДНК (ЭК-5). После добавления
цию останавливали добавлением ЭДТА (рН 8.0)
всех НТФ в концентрации 400 мкМ элонгацию
до концентрации 20 мМ. Продукты анализирова-
транскрипта продолжали. ЭК разделяли методом
ли электрофоретическим разделением в 4.5%-м
электрофореза в ПААГ в нативных условиях и
ПААГ (соотношение акриламида и бисакрилами-
изучали методом «FRET-в-геле» [17].
да 39 : 1) в нативных условиях (в 8 М мочевине) в
FRET в свободных нуклеосомах. Согласно схе-
0.5× буфере ТВЕ в камере для вертикального
ме эксперимента методом ПЦР были получены
электрофореза Mini-PROTEAN Tetra Cell при
фрагменты ДНК, включающие небольшой уча-
4°С. Пробы растворяли в буфере, содержащем
сток ДНК для лигирования элонгационного ком-
40% сахарозы. Распределение продуктов в геле
плекса и модифицированную для получения
анализировали с помощью системы детекции
остановленных в определенном положении элон-
флуоресцентного сигнала лазерного сканера
гационных комплексов последовательность 603
Amersham Typhoon RGB (Cytiva, Швеция). Было
(рис. 1а, дорожка «ДНК»). На полученных фраг-
проведено по три независимых эксперимента.
ментах ДНК методом ступенчатого диализа про-
Количественный анализ проводили с использо-
тив растворов с понижающейся ионной силой в
ванием программного обеспечения Fiji ImageJ,
присутствии донорного хроматина без линкер-
как описано ранее [17], предел погрешностей
ных гистонов были собраны нуклеосомы (рис. 1а,
определяли как стандартное отклонение значе-
дорожка
«Нуклеосома»). Условия процедуры
ний в выборке.
подбирали так, чтобы минимизировать количе-
ство свободной ДНК. В дальнейшей работе ис-
пользовали только смеси, содержащие не менее
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
90% мононуклеосом.
Экспериментальная система транскрипции хро-
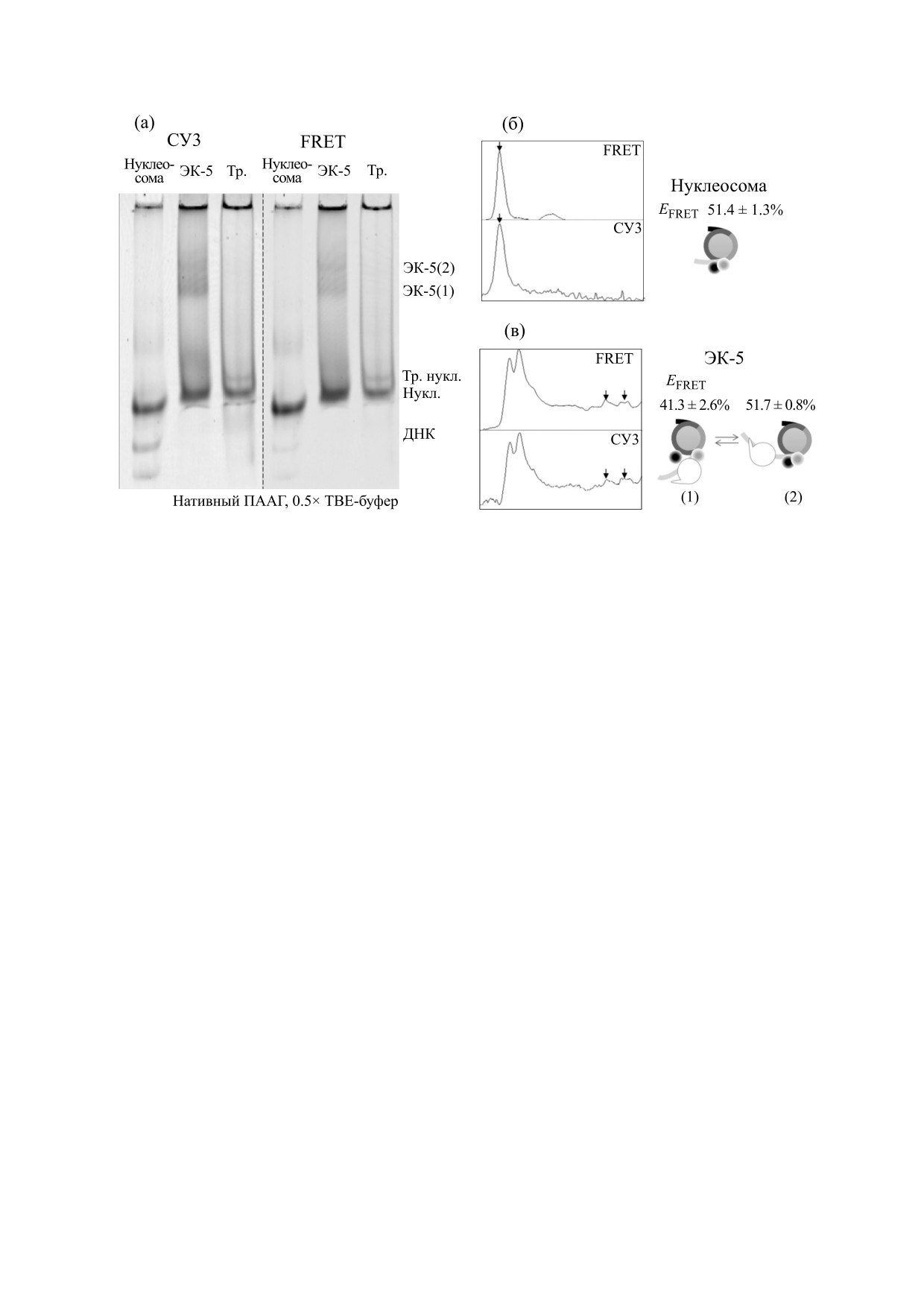
Эффективность FRET (EFRET) свободных
матина in vitro. В данной работе процесс тран-
нуклеосом (рис. 1а и 2а) была определена по ме-
скрипции хроматина изучали в эксперименталь-
тодике, описанной в работе [17], и составила
ной системе, состоящей из собранных на корот-
51.4 ± 1.3% (рис. 2б). Полученное значение близко
ких фрагментах ДНК одиночных нуклеосом и
к тому, которое ранее получали этим методом для
высокоочищенных белков [19]. Для точного по-
нуклеосом с тем же расположением флуоресцент-
зиционирования октамера гистонов в ДНК была
ных меток и другой структурой линкерной ДНК
внесена высокоаффинная последовательность
[17].
603 [23]. Такие нуклеосомы представляют собой
асимметричный барьер для РНКП 2 [24], то есть
Элонгационные комплексы с одинаковой длиной
его высота зависит от ориентации последователь-
РНК могут иметь различную конформацию. Была
ности относительно промотора. Здесь была ис-
проведена транскрипция полученных нуклеосом
БИОФИЗИКА том 67
№ 2
2022
АНАЛИЗ СТРУКТУРЫ ЭЛОНГАЦИОННЫХ КОМПЛЕКСОВ
225
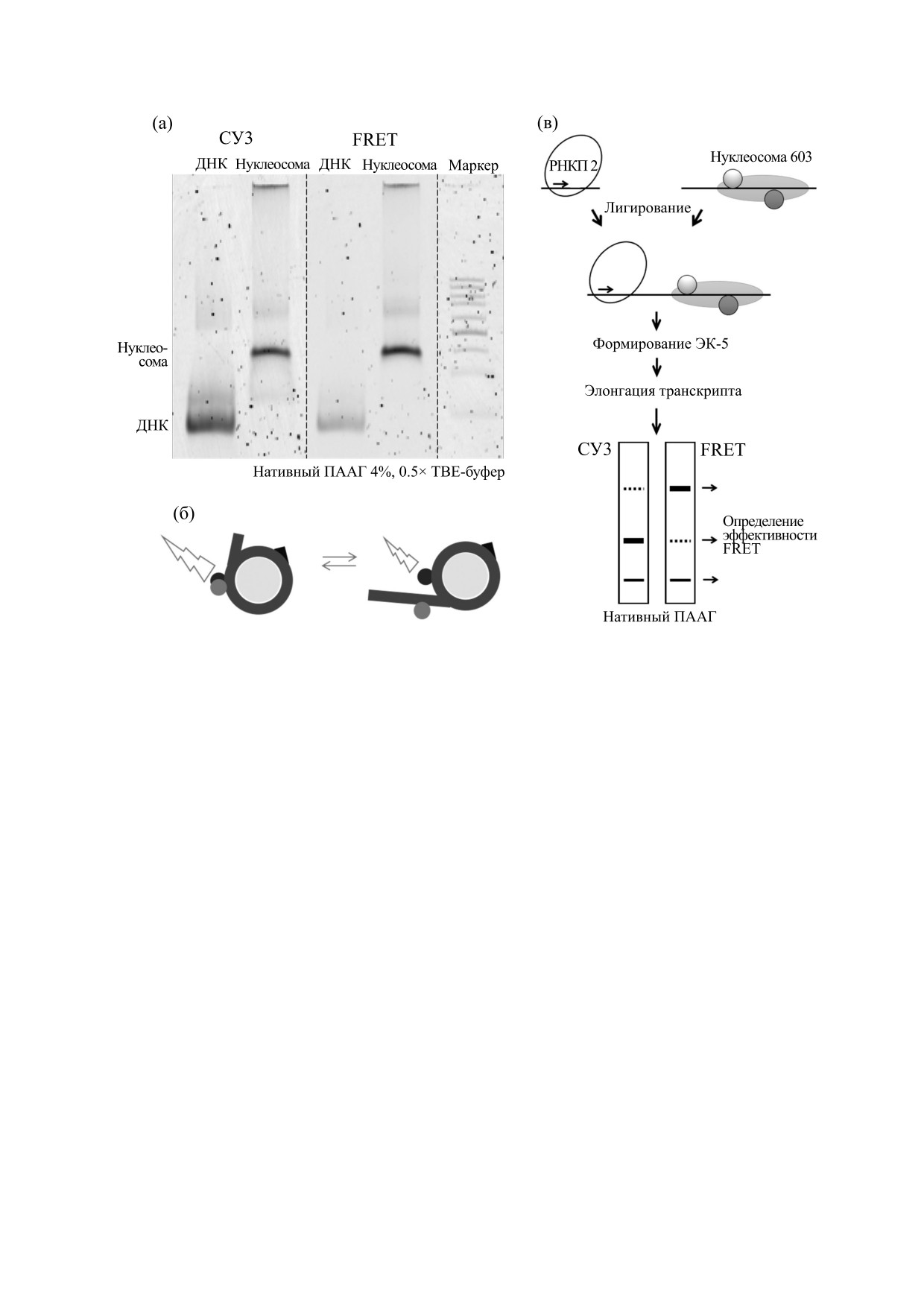
Рис. 1. Экспериментальная система для изучения структурных особенностей элонгационных комплексов, формирующих-
ся в ходе транскрипции нуклеосом РНКП 2. (а) - Получение флуоресцентно-меченых нуклеосом. В соседние супервитки
нуклеосомной ДНК введены флуоресцентные метки Су3 и Су5, представляющие собой донор-акцепторную пару при фёр-
стеровском резонансном переносе энергии. В свободной ДНК метки находятся далеко друг от друга, и FRET между ними
не происходит (слабый сигнал от ДНК при детекции FRET и высокий при детекции сигнала от Cy3). При формировании
нуклеосомы метки оказываются сближенными, и между ними происходит перенос энергии (интенсивности сигналов
близки при детекции FRET и Cy3 от нуклеосомы). (б) - Когда под действием какого-либо фактора нуклеосомная ДНК от-
ворачивается от октамера гистонов, происходит расхождение флуоресцентных меток в пространстве и наблюдаемый сиг-
нал FRET снижается. (в) - Схема транскрипционного эксперимента. Элонгационный комплекс собирается из РНКП 2,
ДНК-олигонуклеотидов, представляющих собой матричную и нематричную цепи дуплекса ДНК, и затравочного РНК-
олигонуклеотида, по последовательности комплементарного матричному ДНК-олигонуклеотиду. К полученному ЭК ли-
гируется нуклеосома. При добавлении ограниченного набора нуклеотидов происходит формирование ЭК-5, в котором ак-
тивный центр РНКП 2 расположен на расстоянии 5 п.н. до входа в нуклеосому. После добавления всех рибонуклеозидтри-
фосфатов транскрипция продолжается. На любом этапе реакции продукты могут быть проанализированы методом элек-
трофореза в полиакриламидном геле с последующим определением эффективности FRET.
РНКП 2 по схеме, приведенной на рис. 1в. После
отметить, что в связи с использованным в реак-
разделения методом ЭФ в 4%-м ПААГ в натив-
ции избытком мононуклеосом большое количе-
ных условиях продукты проанализированы мето-
ство их сохранялось в реакционной смеси
дом
«FRET-в-геле». Результаты эксперимента
(рис. 2в, высокие пики на графиках), однако в
для одной из повторностей приведены на рис. 2а.
данном случае за счет разделения ЭК в геле они
не влияли на интерпретацию полученных дан-
При добавлении ограниченного набора рибо-
ных. Это является преимуществом применяемого
нуклеотидов - АТФ, ГТФ и ЦТФ - РНКП 2 за-
метода.
нимает положение за 5 нт до входа в нуклеосому
(рис. 2а, дорожка «ЭК-5»). При формировании
Обнаружено, что один из двух комплексов не
ЭК-5 наблюдается два ЭК, обладающих разной
отличается по эффективности FRET от свобод-
электрофоретической подвижностью. Следует
ной нуклеосомы (51.7 ± 0.8%) и мигрирует с за-
БИОФИЗИКА том 67
№ 2
2022
226
ГЕРАСИМОВА и др.
Рис. 2. Изучение структуры элонгационных комплексов (ЭК) методом FRET в полиакриламидном геле. (а) - По схеме,
приведенной на рис. 1в, на флуоресцентно-меченых нуклеосомах получены ЭК и разделены методом электрофореза в
нативном ПААГ. (б, в) - Интенсивности флуоресцентных сигналов в геле, измеренные с помощью флуоресцентного
сканера, были проанализированы с помощью программы ImageJ. Проведен анализ интенсивностей флуоресценции Су3 и
Су5 при возбуждении флуорофора Сy3, рассчитана эффективность FRET в нуклеосомах и ЭК-5. Справа на рисунке
приведена интерпретация полученных данных: схематично показаны предполагаемые структуры двух комплексов ЭК-5
(комплекс 1 и комплекс 2) с указанием среднего значения FRET.
метно меньшей подвижностью, чем нуклеосома.
таты согласуются с полученными ранее данными
Другой комплекс отличается несколько большей
о характерных особенностях транскрипции хро-
подвижностью и сниженным FRET (41.3 ± 2.6%)
матина РНКП 2 in vitro [27].
(рис. 2, дорожка «ЭК-5»). Предположительно,
ЭК-5 может существовать в двух формах - с ком-
пактной нуклеосомой и отдаленным расположе-
ЗАКЛЮЧЕНИЕ
нием РНКП 2 (возможно, с откатом фермента на-
В работе для определения структурных осо-
зад по матрице ДНК) и с небольшим отворачива-
бенностей остановленных элонгационных ком-
нием проксимального к промотору участка ДНК.
плексов, формирующихся в ходе транскрипции
Так, достижение положения 5 относительно нук-
нуклеосом РНК-полимеразой 2 был использован
леосомы может быть связано с отворачиванием
подход, основанный на измерении эффективно-
ДНК.
сти FRET в полиакриламидном геле после разде-
ления интермедиатов транскрипции методом
При добавлении всех рибонуклеотидов
электрофореза. Такой подход может быть успеш-
РНКП 2 продолжает элонгацию через нукле-
но применен для анализа длительно живущих
осомную ДНК. Через 10 мин транскрипции про-
элонгационных комплексах, спонтанно образую-
исходит эффективное высвобождение нуклеосом
щихся в ходе транскрипции в течение ограничен-
из элонгационных комплексов. Процесс также
ного времени, а также для остановленных ком-
сопровождается образованием свободной ДНК. В
плексов, образуемых в ходе транскрипции специ-
таком случае флуоресцентные метки удалены
ально сконструированных матриц в присутствии
друг от друга, и эффективность FRET падает до
ограниченного набора рибонуклеозидтрифосфа-
исходной, наблюдаемой в свободной ДНК. Через
тов.
10 с после начала транскрипции из активных
элонгационных комплексов высвобождается 8%
Необходимо отметить, что полученные дан-
свободной ДНК, через 10 мин - 24%. Эти резуль- ные не могут быть использованы для точного
БИОФИЗИКА том 67
№ 2
2022
АНАЛИЗ СТРУКТУРЫ ЭЛОНГАЦИОННЫХ КОМПЛЕКСОВ
227
определения расстояния между флуоресцентны-
8.
N. A. Pestov, N. S. Gerasimova, O. I. Kulaeva, and
ми метками и построения молекулярных моде-
V. M. Studitsky, Sci. Adv. 1, e1500021 (2015). DOI:
лей, так как расчет эффективности FRET произ-
10.1126/sciadv.1500021
водится без учета квантовых выходов флуорес-
9.
N. S. Gerasimova, N. A. Pestov, O. I. Kulaeva, et al.,
ценции и влияния белков на эффективность
Transcription
7,
91
(2016).
DOI:
FRET. На основе полученных данных можно сде-
10.1080/21541264.2016.1182240
лать выводы относительно изменения конформа-
10.
J. Singh and R. A. Padgett, Nature Struct. Mol. Biol.
ции нуклеосомной ДНК и соответственно общей
16, 1128 (2009). DOI: 10.1038/nsmb.1666
структуры ЭК. Так, в настоящем исследовании
было обнаружено, что ЭК-5 может существовать
11.
M. G. Izban and D. S. Luse, J. Biol. Chem. 267, 13647
в различных конформационных состояниях.
(1992).
12.
B. Cheng and D. H. Price, J. Biol. Chem. 282, 21901
(2007). DOI: 10.1074/jbc.M702936200
БЛАГОДАРНОСТИ
13.
H. Ehara, T. Kujirai, Y. Fujino, et al., Science 363, 744
Исследование выполнено с использованием
(2019). DOI: 10.1126/science.aav8912
инфраструктурных возможностей Междисци-
14.
W. Kobayashi and H. Kurumizaka, Curr. Opin. Struct.
плинарной научно-образовательной школы Мос-
Biol. 59, 107 (2019). DOI: 10.1016/j.sbi.2019.07.011
ковского университета «Молекулярные техноло-
гии живых систем и синтетическая биология».
15.
T. Kujirai, H. Ehara, Y. Fujino, et al., Science 362, 595
(2018). DOI: 10.1126/science.aau9904
16.
D. A. Gaykalova, O. I. Kulaeva, O. Volokh, et al.,
ФИНАНСИРОВАНИЕ РАБОТЫ
Proc. Natl. Acad. Sci. USA 112, E5787 (2015). DOI:
Исследование выполнено при финансовой
10.1073/pnas.1508371112
поддержке Российского научного фонда (проект
17.
O. V. Chertkov, M. E. Valieva, N. V. Malyuchenko,
№ 19-44-02013).
and A. V. Feofanov, Moscow Univ. Biol. Sci. Bull. 72,
196 (2017). DOI: 10.3103/S0096392517040034.
КОНФЛИКТ ИНТЕРЕСОВ
18.
K. S. Kudryashova, O. V. Chertkov, D. V. Nikitin,
et al., in: Chromatin Protocols (Methods in Molecular
Авторы заявляют об отсутствии конфликта
Biology, vol. 1288), Ed. by S. Chellappan (Humana
интересов.
Press,
New York,
2015),
pp.
395-412.
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
19.
A. Thastrom, P. T. Lowary, H. R. Widlund, et al., J.
Mol. Biol. 288, 213 (1999).
Настоящая работа не содержит описания ка-
ких-либо исследований с использованием людей
20.
W. Walter, M. L. Kireeva, V. Tchernajenko, et al.,
Methods Enzymol.
371,
564
(2003). DOI:
и животных в качестве объектов.
10.1016/S0076-6879(03)71042-8
21.
M. L. Kireeva, N. Komissarova, D. S. Waugh, and M.
СПИСОК ЛИТЕРАТУРЫ
Kashlev, J. Biol. Chem. 275, 6530 (2000).
1.
K. Luger, A. W. Mäder, R. K. Richmond, et al., Na-
22.
H. W. Chang, F. K. Hsieh, S. S. Patel, and V. M. Stu-
ture 389, 251 (1997). DOI: 10.1038/38444
ditsky, Methods
159-160,
90
(2019). DOI:
2. G. A. Hartzog, J. L. Speer, and D. L. Lindstrom, Bio-
10.1016/j.ymeth.2019.01.016
chim. Biophys. Acta 1577, 276 (2002).
23.
P. T. Lowary and J. Widom, J. Mol. Biol. 276, 19
3. M. G. Izban and D. S. Luse, Genes Dev. 6, 1342
(1998). DOI: 10.1006/jmbi.1997.1494
(1992).
24.
V. A. Bondarenko, L. M. Steele, A. Ujvari, et al., Mol.
4. M. L. Kireeva, W. Walter, V. Tchernajenko, et al., Mol.
Cell
24,
469
(2006).
DOI:
10.1016/j.mol-
Cell
9,
541
(2002).
DOI:
10.1016/j.mol-
cel.2006.09.009
cel.2005.02.027.
25.
H. W. Chang, O. I. Kulaeva, A. K. Shaytan, et al., Nu-
5. L. S. Churchman and J. S. Weissman, Nature 469, 368
cl
Acids Res.
42,
1619
(2014).
DOI:
(2011). DOI: 10.1038/nature09652
10.1093/nar/gkt1120
6. C. M. Weber, S. Ramachandran, and S. Henikoff,
26.
O. I. Kulaeva, D. A. Gaykalova, N. A. Pestov, et al.,
Mol. Cell
53,
819
(2014). DOI:
10.1016/j.mol-
Nature Struct. Mol. Biol. 16, 1272 (2009). DOI:
cel.2014.02.014
10.1038/nsmb.1689
7. O. I. Kulaeva, D. A. Gaykalova, N. A. Pestov, et al.,
27.
D. A. Gaykalova, V. Nagarajavel, V. A. Bondarenko, et
Nature Struct. Mol. Biol. 16, 1272 (2009). DOI:
al., Nucl. Acids Res.
39,
3520
(2011). DOI:
10.1038/nsmb.1689
10.1093/nar/gkq1273
БИОФИЗИКА том 67
№ 2
2022
228
ГЕРАСИМОВА и др.
Analysis of Structure of Elongation Complexes in Polyacrylamide Gel
with Förster Resonance Energy Transfer Technique
N.S. Gerasimova*, A.N. Korovina*, D.A. Afonin*, K.V. Shaytan*,
A.V. Feofanov*, and V.M. Studitsky*, **
*Faculty of Biology, Lomonosov Moscow State University, Leninskie Gory 1/12, Moscow, 119991 Russia
**Fox Chase Cancer Center, Philadelphia, PA 19111-2497, USA
To study the structural features of elongation complexes formed during nucleosome transcription by RNA
polymerase 2, it was proposed to use an approach based on the measurement of the efficiency of Förster res-
onance energy transfer in polyacrylamide gel after separation of transcription intermediates by electrophore-
sis. Fluorescently labeled nucleosomes were obtained by introducing a pair of fluorophores (donor and ac-
ceptor) into adjacent gyres of nucleosomal DNA. Stalled elongation complexes were formed by transcription
of nucleosomes in the presence of a limited set of nucleoside triphosphates. Comparative analysis of the effi-
ciency of Förster resonance energy transfer for free nucleosomes and elongation complexes with RNA poly-
merase 2 obtained on their basis revealed changes in the structure of nucleosomes in elongation complexes.
It was found that elongation complexes with the same RNA length could exist in different conformational
states.
Keywords: transcription, chromatin, nucleosome, RNA polymerase 2, FRET, elongation complex
БИОФИЗИКА том 67
№ 2
2022