БИОФИЗИКА, 2022, том 67, № 5, с. 868-876
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 577.322
СТРУКТУРА И АФФИННОСТЬ КОМПЛЕКСОВ МЕЖДУ
ДНК-СВЯЗЫВАЮЩИМ ДОМЕНОМ БЕЛКА Swi4 И ДНК
© 2022 г. Д.А. Афонин*, #, О.В. Гераськина*, Т.В. Лосева*, М.П. Кирпичников*,
В.М. Студитский*, **, А.В. Феофанов*
*Биологический факультет Московского государственного университета имени М.В. Ломоносова,
Ленинские горы, 1/12, Москва, 119234, Россия
**Центр исследований рака Фокс Чейз, просп. Коттмана, 333, Филадельфия, Пенсильвания, 19111, США
#E-mail: afoninmsu@outlook.com
Поступила в редакцию 04.08.2022 г.
После доработки 04.08.2022 г.
Принята к публикации 10.08.2022 г.
Получен рекомбинантный ДНК-связывающий домен белка Swi4, входящего в состав транскрипци-
онного фактора SBF почкующихся дрожжей Saccharomyces cerevisiae. По данным спектроскопии
кругового дихроизма в структуре этого домена содержится 29% α-спиралей, 22% β-складчатых ли-
стов, 21% β-поворотов и 28% неупорядоченной структуры. Исследовано взаимодействие домена с
ДНК, содержащей сайт связывания SBF SCB (Swi4 Cell-cycle Box). Образование комплексов сопро-
вождается конформационной перестройкой в белке, которая затрагивает не менее десяти пептид-
ных связей и приводит к 6%-му увеличению содержания α-спиралей с сопутствующим снижением
содержания β-складчатых листов. По данным спектроскопии флуоресценции конформационные
перестройки и межмолекулярные взаимодействия не затрагивают области локализации остатков
Trp68 и Trp112, находящихся вблизи границ ДНК-связывающего мотива «спираль-поворот-спи-
раль». Установлено, что константа диссоциации комплекса ДНК-связывающего домена белка Swi4
с ДНК составляет 20 ± 3 нМ.
Ключевые слова: SBF, Swi4, SCB, константа диссоциации, круговой дихроизм, флуоресценция.
DOI: 10.31857/S0006302922050039, EDN: JIDJWB
ся компонентом сети активации транскрипции у
SBF - транскрипционный фактор, который
нескольких сложно регулируемых промоторов, в
вместе с паралогом MBF контролирует разные
частности промотора гена эндонуклеазы HO,
ветви транскрипционного ответа при G1-S-пере-
контролирующего переключение типа спарива-
ходе клеточного цикла у почкующихся дрожжей
ния [2].
Saccharomyces cerevisiae. SBF и MBF гетеродимер-
Несмотря на то, что SBF является объектом
ны, они содержат общую субъединицу Swi6 и раз-
интенсивных in vivo исследований [3-5], молеку-
личаются ДНК-связывающими субъединицами -
лярные механизмы, определяющие преимуще-
Swi4 и Mbp1, которые связывают промоторы, со-
ственное связывание SBF с сайтом SCB, изучены
держащие специфичные для них сайты - SCB
недостаточно [6]. Опыты по ограниченному про-
(Swi4 Cell-cycle Box) и MCB (MluI Cell-cycle Box)
теолизу N-концевого фрагмента Swi4 в комплек-
соответственно. SBF ответственен за экспрессию
се со специфичной ДНК позволили определить
генов, управляющих морфогенезом и почковани-
границы его ДНК-связывающего домена. Было
ем, в то время как MBF контролирует гены ре-
установлено, что минимальный ДНК-связываю-
пликации и репарации при переходе в S-фазу
щий домен Swi4 включает около 140 аминокис-
клеточного цикла [1]. Помимо этого, SBF являет-
лотных остатков и локализован в области с остат-
ка 32 по остаток 172 [7].
Сокращения: dSwi4 - ДНК-связывающий домен Swi4 с за-
меной С50А, слитый на N-конце с полипептидом GSSHH-
Структура комплекса Swi4 с ДНК неизвестна.
HHHHSSGENLYFQSHM; TCEP
- трис(2-карбокси-
Анализ кристаллической структуры комплекса
этил)фосфин; SCB_R
- олигонуклеотид gTTTTCgTgg- ДНК-связывающего домена белка PCG2 (орто-
CACC; SCB_F
- олигонуклеотид
[Cy5]ggTACCACg-
логичного ДНК-связывающему домену Mbp1) из
AAAAC; SCB-ДНК - двуцепочечный 14 п.н. олигонуклео-
тид, образованный SCB_R и SCB_F, КД - круговой ди-
фитопатогенных грибов Magnaporthe oryzae с
хроизм.
ДНК показал, что PCG2 использует для распо-
868
СТРУКТУРА И АФФИННОСТЬ КОМПЛЕКСОВ
869
знавания сайта связывания ДНК спирали αA и αB
Для сайт-направленного мутагенеза Сys50 на
структурного мотива
«спираль-поворот-спи-
аланин использовали измененный прямой прай-
раль», а также дополнительные неканонические
мер (gCAAACATATgTCAAACCATCCAgTgATTgA-
структуры полипептидной цепи [8]. Несмотря на
AATAgCTACgTATTCAgAAACCgATgTATACgAAg-
присутствие аналогичного мотива «спираль-по-
CgTATATACgTggg). Ампликон, содержащий му-
ворот-спираль» у Swi4, использование структуры
тацию, клонировали в вектор pET-15b, а получен-
dPCG2-ДНК для анализа взаимодействий Swi4 с
ную плазмиду проверили секвенированием в
SCB затруднено из-за палиндромности сайта свя-
ЦКП
«Геном» (pET-15b-2C-TEV-Swi4[DBD]:
зывания MCB (5'-ACGCGT-3’), которая приво-
C19A). Данная плазмида кодировала ДНК-связы-
дит к образованию двух типов комплекса PCG2-
вающий домен Swi4 с мутацией С50А, у которого
ДНК. Остается неясным, какой тип комплекса
на N-конце находилась полигистидиновая метка
следует ожидать в случае взаимодействия Swi4 с
(6×His), слитая с белком с помощью линкера
ассиметричным сайтом SCB (5’-CACGAAA-3).
(здесь и далее dSwi4, рис. 1).
Ключевые аминокислоты PCG2 (Q82 и Q89), от-
Плазмиду трансформировали в клетки E. coli
ветственные за распознавание сайта MCB, при-
экспрессионного штамма Rosetta
2(DE3)
сутствуют также и у Swi4, что ставит вопрос о ме-
(Novagen). Трансформированные клетки выра-
ханизме специфичного взаимодействия Swi4 с
щивали в среде LB, содержавшей 100 мкг/мл ам-
SCB.
пициллина и 24 мкг/мл хлорамфеникола, в тече-
Для исследования взаимодействия Swi4 с
ние 4 ч при 37°C до оптической плотности (на
ДНК, была разработана методика получения ре-
длине волны 600 нм) 0.6-0.8 опт. ед./см. После
комбинантного ДНК-связывающего домена Swi4
добавки изопропил-β-D-1-тиогалактопиранози-
(dSwi4). Изучено взаимодействие dSwi4 с фраг-
да (1 мМ) клетки инкубировали 4 ч при 37°C, оса-
ментом ДНК, содержащим SCB (SCB-ДНК) и
ждали при 3200 g (30 мин), промывали 50 мл фос-
определена константа диссоциации комплексов.
фатно-солевого буфера и осаждали 15 мин при
Методом спектроскопии кругового дихроизма
8000 g. Осадок замораживали и хранили при
проведен сравнительный анализ структуры dSwi4
-80°C.
в свободном и связанном состоянии. С помощью
Выделение белка. Клетки разрушали ультра-
спектроскопии флуоресценции изучено микро-
звуковой обработкой (Sonic Dismembrator FB120,
окружение Trp68 и Trp112, фланкирующих ДНК-
Fisher Scientific, США) в буфере А (20 мМ фос-
связывающий мотив
«спираль-поворот-спи-
фатно-солевого буфера, pH 7.0, 500 мМ NaCl,
раль», в свободном и связанном dSwi4.
0.3% Triton X-100,
1 мM трис(2-карбокси-
этил)фосфина (TCEP)) с добавкой 25 мМ имида-
МАТЕРИАЛЫ И МЕТОДЫ
зола,
2 мМ фенилметансульфонилфторида и
1 таблетки коктейля ингибиторов протеаз (cOm-
Клонирование и экспрессия ДНК-связывающе-
plete, EDTA-free Protease Inhibitor Cocktail, Roche,
го домена Swi4. Фрагмент гена Swi4 S. cerevisiae,
Швейцария).
кодирующий ДНК-связывающий домен (32-173)
[7], амплифицировали с помощью прямого
Лизат клеток центрифугировали при 18500 g
(gCAAACATATgTCAAACCATCCAgTgATTgAAAT-
(4°C) в течение 40 мин. Супернатант фильтровали
AgCTACg) и обратного (gCAAACTCgAgTTATgT-
через фильтр с диаметром пор 0.22 мкм и наноси-
TTTATTgTAgCTCgATggAgAAgTTATCTTCg) прай-
ли со скоростью 2 мл/мин на хроматографиче-
меров, в которых подчеркнуты вносимые сайты
ский картридж HiTrap Chelating HP (5 мл), пред-
рестрикции. Полученный ампликон клонирова-
варительно уравновешенный буфером А с 25 мМ
ли в вектор для бактериальной экспрессии pET-
имидазола. Картридж промывали 50 мл буфера А
15b (Novagen, Merck KGaA, Германия) обработ-
с 25 мМ имидазола и затем 50 мл буфера А с 80 мМ
кой эндонуклеазами рестрикции NdeI и XhoI с
имидазола. Белок элюировали 10 мл буфера А с
последующим лигированием. Вектор был предва-
400 мМ имидазола, разводили в 10 раз буфером Б
рительно модифицирован вставкой дополни-
(50 мМ буфера Бицин, pH 9.0, 1 мМ TCEP), нано-
тельного мини-цистрона для улучшения экспрес-
сили со скоростью 1 мл/мин на картридж HiTrap
сии [9]. Полученный конструкт (pET-15b-2C-
SP HP (1 мл), который затем промывали 10 мл бу-
TEV-Swi4[DBD]) был проверен секвенированием
фера Б. Белок элюировали c картриджа буфером
по Сэнгеру в ЦКП «Геном» (Россия).
Б, повышая концентрацию NaCl до 800 мМ с по-
Поскольку ДНК-связывающий домен Swi4
мощью хроматографа AKTA Purifier (GE, США).
склонен к димеризации в растворе из-за образо-
Фракции, содержавшие dSwi4 по данным элек-
вания межмолекулярной дисульфидной связи
(данные не показаны), было решено заменить
трофореза в денатурирующем полиакриламид-
Сys50 (здесь и далее используется нумерация
ном геле, объединяли, диализовали 14 ч при 4°C
аминокислотных остатков в полноразмерном
против буфера В (50 мМ HEPES, pH 7.8, 200 мМ
белке) на аланин.
KCl, 1 мМ TCEP) с помощью диализной мембра-
БИОФИЗИКА том 67
№ 5
2022
870
АФОНИН и др.
Рис. 1. Выравнивание аминокислотных последовательностей ДНК-связывающих доменов Swi4 и его гомолога PCG2
(82-219) из Magnaporthe oryzae, для которого установлена структура в комплексе с ДНК [8]. Кодируемый
экспрессионным вектором линкер, содержащий полигистидиновую метку (6xHis), выделен в начальной части
последовательности dSwi4 рамкой. Жирным шрифтом выделены последовательность ДНК-связывающего домена
Swi4 (32-172) [7] и фрагмент PCG2 (95-209), структура которого установлена с высоким разрешением [8]. На основе
разрешенной структуры комплекса PCG2 с ДНК (PDB:4UX5) рамками отмечены элементы вторичной структуры, а
также «крыло» и петля (20l), участвующие в связывании PCG2 с ДНК. Подчеркнут ДНК-связывающий мотив
«спираль-поворот-спираль». Подвергнутый мутагенезу Cys50 dSwi4 обозначен стрелкой. Триптофаны Trp68 и
Trp112 Swi4, а также гомологичные им Trp129 и Trp173 PCG2 выделены серым цветом и курсивом. Символами “*”, “:”
и
“.” отмечены соответственно совпадающие, слабо
и сильно отличающиеся аминокислотные остатки в
сравниваемых белках.
ны Spectra/Por (CE), отсекающей белки >10 кДа.
B_R (gTTTTCgTggCACC) и SCB_F ([Cy5]ggTAC-
При анализе методом электрофореза в полиакри-
CACgAAAAC), меченый по 5'-концу флуорес-
ламидном геле в качестве маркеров использовали
центной меткой Cy5.
реагенты PageRuler Prestained Protein Ladder 10-
В опытах по подбору условий формирования
180 kDa и PageRuler Unstained Low Range Protein
двухцепочечной ДНК (SCB-ДНК) SCB_F и
Ladder (ThermoFisher Scientific, США), смешан-
SCB_R смешивали в концентрации 10 мкМ в ди-
ные с интеркалирующим флуоресцентным кра-
стиллированной воде категории Milli-Q и либо
сителем SYBR Gold (Invitrogen, США).
инкубировали 1 ч при 4°C, либо подвергали двух-
Ретентант концентрировали до объема
минутному плавлению при 80°C и инкубировали
~500 мкл с помощью центрифужного концентра-
1 ч при 30, 37 или 42°C. Образцы подвергали элек-
тора Amicon Ultra-4 (10 кДа, Merck, Германия),
трофорезу и анализу, как описано ниже. В каче-
затем центрифугировали при 16100 g (4°C) 30 мин
стве маркера длин ДНК использовали реагент
и полученный супернатант очищали методом
GeneRuler Ultra Low Range DNA Ladder (Thermo-
гель-фильтрации с помощью хроматографа AK-
Fisher Scientific, США), смешанный с интеркали-
TA Purifier и колонки Superdex 75 10/300 GL (Cy-
рующим флуоресцентным красителем SYBR Gold
tiva, Швеция) при скорости потока 500 мкл/мин.
(Invitrogen, США).
Фракции, содержавшие чистый dSwi4 по дан-
Для измерения спектров кругового дихроизма
ным денатурирующего белкового электрофореза,
(КД) смешивали по 21 мкМ олигонуклеотидов
объединяли. Концентрацию белка (расчетная мо-
SCB_F и SCB_R и 20 мкМ dSwi4 в буфере, содер-
лекулярная масса - 18825.2 Да) измеряли спек-
жащем 12.5 мМ HEPES (pH 8.0), 50 мМ KCl,
трофотометрически, используя коэффициент
250 мкМ TCEP, и инкубировали 1 ч при 4°C. Для
молярной экстинкции ε280 нм = 21400 M-1см-1.
измерения спектров флуоресценции смешивали
по 200 нМ SCB_F и SCB_R с 190 нМ dSwi4 и ин-
Чистоту dSwi4 и отсутствие геномной ДНК E. coli
кубировали 1 ч при 4°C.
дополнительно подтверждали спектрофотомет-
рически по соотношению поглощений на длинах
Для измерения константы диссоциации Kd
волн 260 и 280 нм, которое составляло < 0.6.
комплекса dSwi4 с SCB-ДНК смешивали 5 нМ
Получение комплексов dSwi4 с ДНК. Для полу-
SCB_F и 6 нМ SCB_R без dSwi4 или с возрастаю-
чения комплексов dSwi4 с ДНК использовали ко-
щими концентрациями dSwi4 (10-80 нМ) в буфе-
роткие (14 п.н.) олигонуклеотиды, синтезирован-
ре, содержащем 20 мМ PIPES, 30 мМ KOH
ные компанией Lumiprobe (Москва, Россия): SC-
(pH 7.3), 120 мМ KCl, 100 мкМ TCEP, 100 мкг/мл
БИОФИЗИКА том 67
№ 5
2022
СТРУКТУРА И АФФИННОСТЬ КОМПЛЕКСОВ
871
бычьего сывороточного альбумина, 10% (мас-
Величины Kd определяли в двух независимых
са/объем) сахарозы. Полученные смеси инкуби-
экспериментах и усредняли.
ровали 1 ч при 4°C. Аналогично готовили образ-
Спектроскопия КД. Спектры КД измеряли с
цы dSwi4 (10-80 нМ) в смеси с 5 нМ SCB_F.
помощью спектрополяриметра Jasco-810 (Jasco,
Анализ комплексообразования методом электро-
Япония): оптическая ширина щели монохрома-
фореза. Смеси SCB_F, SCB_R и dSwi4 или dSwi4
тора - 1 нм, диапазон сканирования - 190-
и SCB_F, приготовленные как описано выше,
250 нм, шаг сканирования - 0.2 нм, скорость ска-
подвергали нативному электрофорезу в полиа-
нирования - 20 нм/мин. Спектры усредняли по
криламидном геле, содержащем 10 мМ HEPES-
четырем повторным сканам. Для измерений ис-
Na, pH 7.8, 0.2 мМ этилендиаминтетрауксусной
пользовали разборную кварцевую кювету с тол-
кислоты, 18% (масса/объем) полиакриаламида
щиной оптического слоя 0.1 мм (Hellma, Герма-
(19 частей акриламида на 1 часть бис-акрилами-
ния).
да) и 20% (объем/объем) глицерина. Электрофо-
Из спектров КД раствора dSwi4 или его ком-
рез проводили при постоянном токе 5 мА в тече-
плексов с SCB-ДНК вычитали базовую линию,
ние 3 ч. Электрофореграммы сканировали с по-
измеренную соответственно от буфера или от
мощью флуоресцентного сканера Amersham
раствора SCB-ДНК (21 мкМ). Спектры, получен-
Typhoon RGB (Cytiva, Швеция) с возбуждением
ные в двух независимых экспериментах, усредня-
632 нм и регистрацией флуоресценции в области
ли и анализировали с помощью пакета программ
670 нм (пространственное разрешение - 25 мкм).
CDPro, используя референсный набор белков
Полученные изображения обрабатывали с помо-
SP43 [10].
щью программного пакета ImageQuant TL 1D
Спектроскопия флуоресценции. Спектры флуо-
версия 8.1 (Cytiva, Швеция), оценивая яркость
ресценции измеряли с помощью спектрофлуори-
полос, соответствующих SCB-ДНК, свободным
метра Cary Eclipse (Varian, США) в кварцевой
олигонуклеотидам SCB_F и комплексам dSwi4 с
ультра-микрокювете 2 × 10 мм (Hellma, Герма-
SCB-ДНК.
ния). Флуоресценцию возбуждали на длине вол-
По измеренным яркостям полос рассчитывали
ны 280 нм и детектировали в диапазоне 295-
возрастание количества комплекса dSwi4 с SCB-
450 нм. Из спектров вычитали базовую линию,
ДНК, а также изменение количества свободных
измеренную от буфера или от раствора олигонук-
SCB-ДНК и SCB_F при образовании ком-
леотидов (200 нМ) в буфере (в случае комплексов
плексов.
Swi4 с SCB-ДНК). Спектры, полученные в двух
Для анализа образования комплекса строили
независимых экспериментах, усредняли.
зависимость яркости полосы комплекса I1(C) от
концентрации dSwi4 (С) и описывали эти зависи-
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
мости в приближении одного центра связывания
формулой:
Получение рекомбинантного dSwi4. С использо-
ванием созданной нами плазмиды pET-15b-2C-
I1(С) = Bm1C/(Kd + C),
(1)
TEV-Swi4[DBD]:C19A была изучена эффектив-
ность экспрессии белка dSwi4 в штаммах E. coli
где С - концентрация dSwi4, Bm1 - максимальное
BL21(DE3) и Rosetta 2(DE3) (Novagen). Значи-
значение, к которому стремится величина I1(С),
тельную экспрессию dSwi4 наблюдали в штамме
Kd - константа диссоциации комплекса.
Rosetta
2(DE3), что определило выбор этого
штамма для дальнейшей работы (рис. 2а). Повы-
Для анализа образования комплекса по умень-
шенный уровень экспрессии dSwi4 был достигнут
шению яркости полосы SCB-ДНК рассчитывали
при выращивании клеток после индукции при
параметр I0 - I2(C), где I0 - яркость контрольной
37°С. Методика выделения dSwi4 включала в себя
полосы SCB_ДНК в отсутствии dSwi4, I2(C) - яр-
стадии металл-аффинной и катион-обменной
кость анализируемой полосы при определенной
хроматографии, а также гель-фильтрацию
концентрации dSwi4. Строили зависимости пара-
(рис. 2б). Чистота полученного dSwi4 по данным
метра I0 - I2(C) от концентрации dSwi4 и описы-
хроматографического анализа превышает
99%
(данные не представлены), а его выход составил
вали эти зависимости в приближении одного
5 мг с 1 л культуры E. coli.
центра связывания формулой:
Подбор условий формирования SCB-ДНК и ее
I0 - I2(С) = Bm2C/(Kd + C),
(2)
комплексов с dSwi4. Для оптимизации условий
формирования комплексов dSwi4 с SCB-ДНК
где Bm2 - максимальное значение, к которому
было проведено сравнение несколько вариантов
стремится величина I0 - I2(С).
получения SCB-ДНК из одноцепочечных олиго-
БИОФИЗИКА том 67
№ 5
2022
872
АФОНИН и др.
Рис. 2. Результаты гель-электрофореза в денатурирующих условиях лизатов E. coli до индукции и после экспрессии
dSwi4 (А), а также гель-фильтрационных фракций dSwi4, объединенных для дальнейших опытов с dSwi4. М - маркер.
Рис. 3. Анализ формирования дуплекса SCB-ДНК из олигонуклеотидов SCB_F (10 мкМ) и SCB _R (10 мкМ) и образования
комплекса SCB-ДНК (10 мкМ) с dSwi4 (10 мкМ) при различных условиях по данным нативного электрофореза в
полиакриламидном геле. М - маркер.
нуклеотидов SCB_F и SCB_R. Для этого SCB_F и
нуклеотидами, и на это равновесие не оказывают
SCB_R смешивали в концентрации 10 мкМ каж-
влияние стадии плавления и отжига. Наблюдае-
дого и либо инкубировали при 4°С, либо подвер-
мая нестабильность двуцепочечной спирали мо-
гали плавлению (2 мин, 80°С) и отжигу (1 ч) при
жет быть связана с короткой длиной олигонук-
30, 37 или 42°C. Анализ проб методом электрофо-
леотидов и особенностями нуклеотидной после-
довательности. Учитывая полученные результаты
реза в полиакриламидном геле в нативных усло-
(рис. 3), было решено формировать SCB-ДНК в
виях показал, что эффективность образования
упрощенных условиях при 4°С без стадий плавле-
SCB-ДНК не зависит от наличия стадий плавле-
ния и отжига. Отметим, что SCB-ДНК соответ-
ния и отжига (рис. 3). Во всех случаях на электро-
ствует консенсусной последовательности сайта
фореграмме помимо полосы SCB-ДНК присут-
связывания SBF в геноме - RNNYCACgAAAA
ствовала полоса одноцепочечного олигонуклео-
[11].
тида, и яркость этих полос была сравнима при
разных условиях реакции. Можно предположить,
Исследована эффективность образования
что в смеси SCB_F и SCB_R устанавливается рав-
комплекса dSwi4 с SCB-ДНК при разных темпе-
новесие между двух- и одноцепочечными олиго-
ратурах инкубации смеси (рис. 3). Установлено,
БИОФИЗИКА том 67
№ 5
2022
СТРУКТУРА И АФФИННОСТЬ КОМПЛЕКСОВ
873
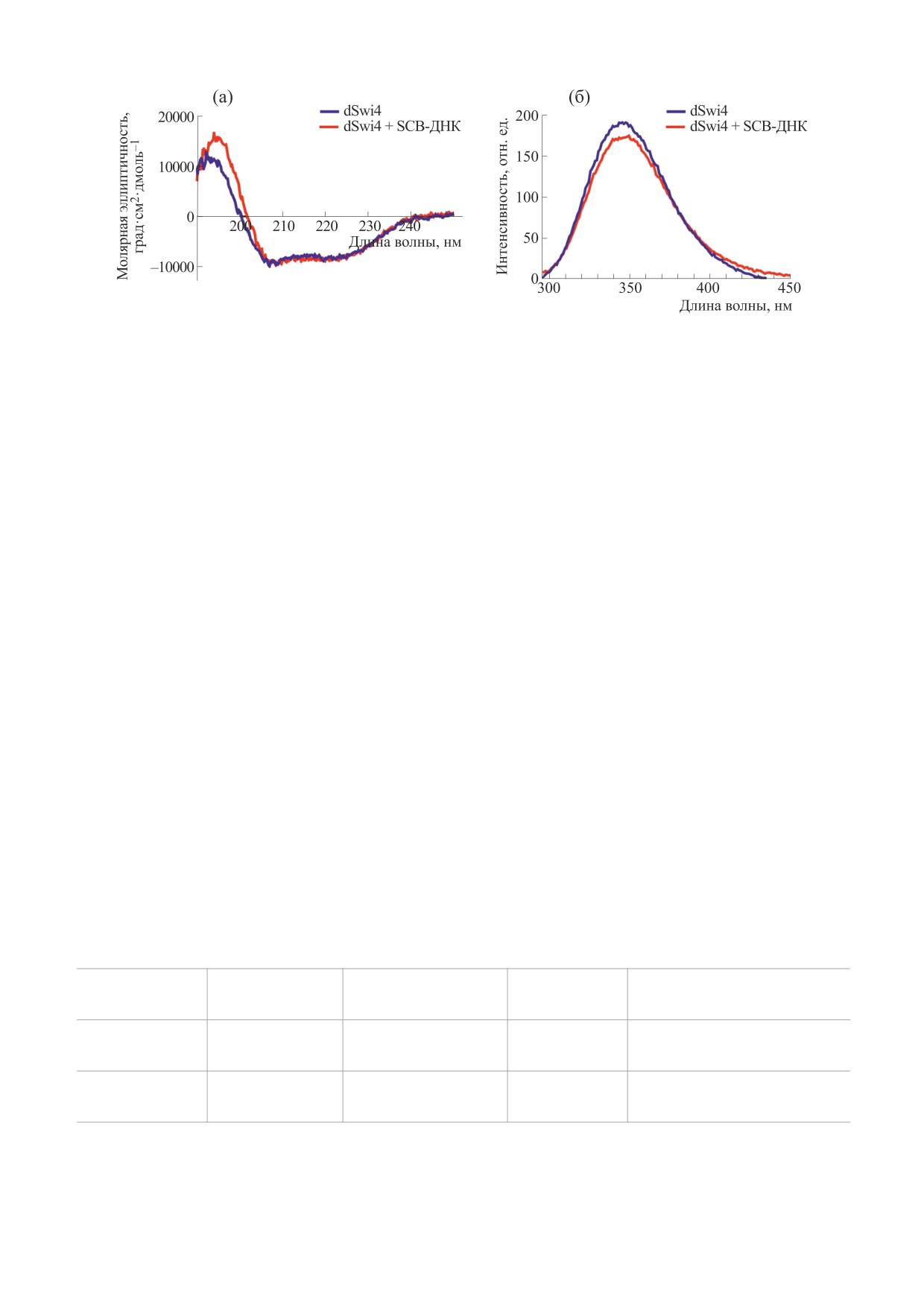
Рис. 4. Спектральный анализ dSwi4 и его комплекса с SCB-ДНК. Представлены спектры КД (а) и флуоресценции (б) dSwi4
и его комплекса с SCB-ДНК.
что эффективность образования комплекса не за-
этой области вследствие стабилизации и/или
висит от температуры в диапазоне 4-37°С, что
формирования α-спиральных участков, участву-
позволило проводить всю пробоподготовку при
ющих в комплексообразовании.
4°С.
По данным рентгеноструктурного анализа
Структурный анализ dSwi4 и его комплекса с
(структура PDB:4ux5) у dPCG2 в комплексе с
SCB-ДНК. Анализ спектра КД dSwi4 (рис. 4а) по-
ДНК, содержащей сайт MCB (мода А с большим
казал, что во вторичной структуре белка преобла-
интерфейсом взаимодействия), 26% аминокис-
дают элементы β-структур, включая β-складча-
лотных остатков формируют β-складчатые ли-
тые листы (22%) и β-повороты (21%, табл. 1). В
сты, а 33% остатков - α-спиральные структуры
α-спиральной конформации находятся 29% ами-
[8]. Хотя доля α-спиралей в структуре комплексов
нокислотных остатков, а 28% остатков располо-
dPCG2 и dSwi4 с ДНК сходна (табл. 1), комплек-
жены в участках с неупорядоченной структурой
сы заметно отличаются по содержанию β-склад-
(табл. 1). Формирование комплекса dSwi4 с SCB-
чатые листов (табл. 1). Структурные отличия ком-
ДНК приводит к изменениям в КД-спектре белка
плексов, по-видимому, во многом связаны с
(рис. 4а). Эти изменения соответствуют неболь-
ограниченной гомологией dPCG2 и dSwi4, кото-
шому увеличению доли аминокислотных остат-
рая составляет 41% (рис. 1), и указывают на необ-
ходимость детального изучения структуры ком-
ков, формирующих α-спирали, и сопутствующе-
плекса dSwi4 с ДНК, содержащей сайт SCB.
му снижению доли β-складчатых структур
(табл. 1). Изменения затрагивают не менее
Вблизи границ ДНК-связывающего мотива
10 пептидных связей и позволяют предположить,
dSwi4
«спираль-поворот-спираль» находятся
что образование комплекса сопровождается не-
два остатка Trp (W68 и W112, рис. 1), спектры флу-
большой структурной перестройкой белка. С уче-
оресценции которых, как известно, чувствитель-
том наличия в белке ДНК-связывающего мотива
ны к изменениям полярности микроокружения и
«спираль-поворот-спираль» (helix-turn-helix,
взаимодействий с соседними аминокислотными
остатки 71-92) можно предположить, что часть
остатками [12, 13]. Других остатков Trp в исследу-
структурных изменений происходит именно в
емом белке нет. Флуоресценция Trp свободного
Таблица 1. Содержание канонических типов вторичных структур (в %) в составе свободного dSwi4 и в комплексе
с SCB-ДНК (по данным КД-спектроскопии)
α-Спираль
β-Складчатый лист
β-Поворот
Неупорядоченная структура
dSwi4
29 ± 1
22 ± 1
21 ± 1
28 ± 1
dSwi4:SCB-ДНК
35 ± 3
17 ± 1
21 ± 1
27 ± 1
БИОФИЗИКА том 67
№ 5
2022
874
АФОНИН и др.
dSwi4 имеет максимум испускания при 345 нм
го олигонуклеотида яркость полосы SCB_F в диа-
(рис. 4б), что можно объяснить высокой враща-
пазоне концентраций dSwi4 10-100 нМ не меня-
тельной свободой боковых цепей Trp [14]. Срав-
лась (рис. 5б,е), и появление новой полосы, соот-
нение спектров флуоресценции Trp свободного
ветствующей комплексу, не обнаружено (рис.
dSwi4 и в комплексе dSwi4 с ДНК не выявило зна-
5б). С учетом этих данных dSwi4-зависимое
чимых отличий по форме и положению максиму-
уменьшение яркости полосы SCB_F в случае
ма (рис. 4б). Наблюдаемое небольшое снижение
присутствия в растворе комплементарного SC-
интенсивности флуоресценции может быть вы-
B_R можно объяснить сдвигом равновесия в сто-
звано эффектом так называемого внутреннего
рону образования SCB-ДНК в первой реакции из
фильтра, т.е. перепоглощения испускаемого све-
двух в следующей цепи:
та в кювете вследствие повышения оптической
SCB_F+ SCB_R SCB-ДНК + dSwi4
плотности раствора в присутствии ДНК. Отсут-
SCB-ДНК:dSwi4,
(3)
ствие сдвига максимума спектра позволяет пред-
положить, что взаимодействие с ДНК, а также
который, в свою очередь, происходит из-за ухода
конформационные перестройки в белке, вызван-
свободной SCB-ДНК в комплекс с dSwi4. Форми-
ные образованием комплекса, не распространя-
рование дополнительной SCB-ДНК в процессе
ются на области локализации остатков Trp68 и
титрования способно влиять на результаты ана-
Trp112 и не вызывают изменений во вращатель-
лиза комплексобразования, приводя к некоторо-
ной свободе боковых цепей Trp.
му завышению оцениваемой величины констан-
ты диссоциации Кd комплексов dSwi4 с SCB-
Сравнение полученных результатов с данны-
ДНК.
ми о микроокружении гомологичных остатков
Trp в комплексе dPCG2 c ДНК, содержащей сайт
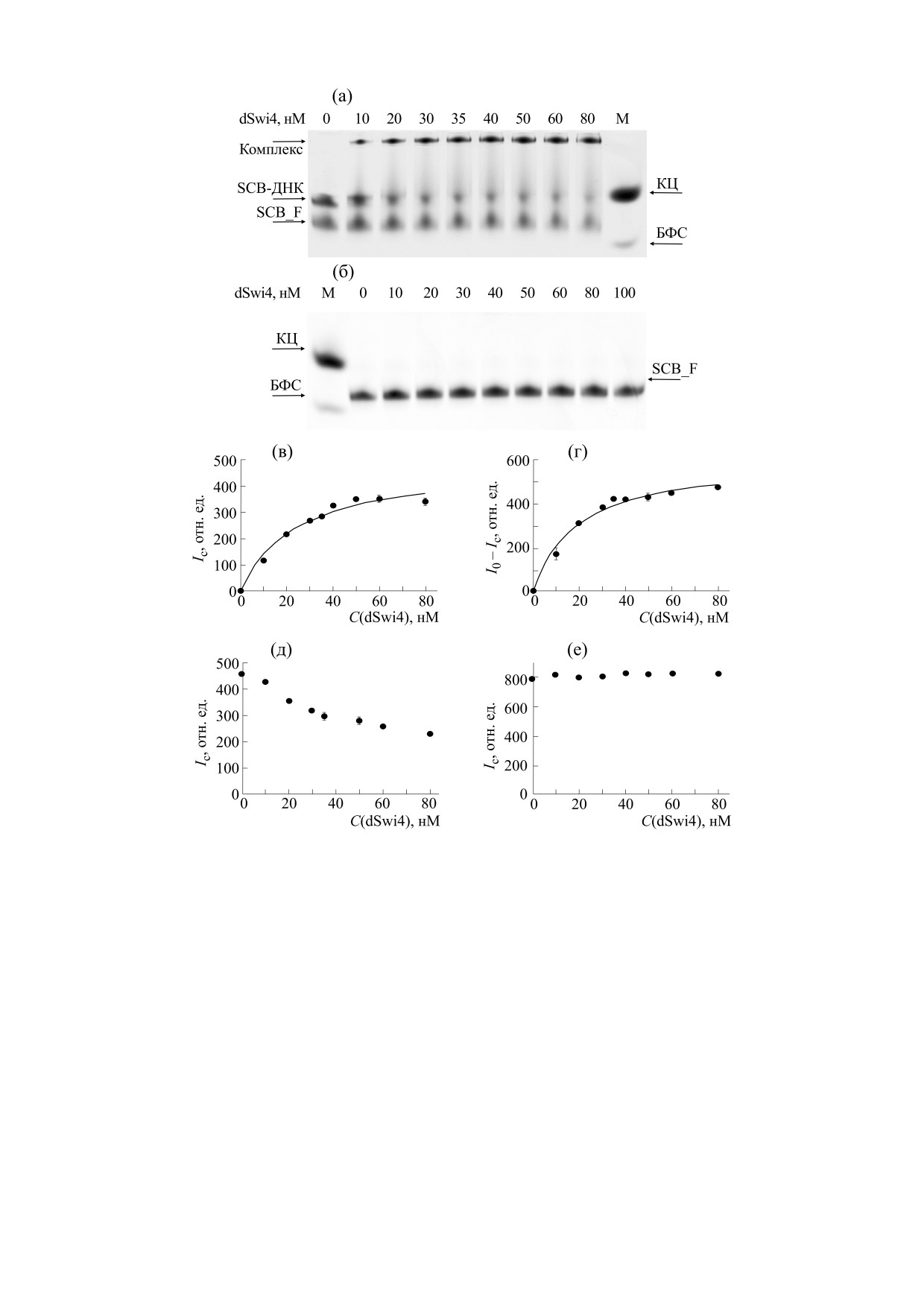
Расчеты на основе измеренных зависимостей
MCB (структура PDB:4ux5), показало, что Trp173
(рис. 5в,г) с использованием формул (1) и (2) да-
(гомологичен Trp112 у Swi4) образует водородную
ют согласующиеся величины Кd равные 24 ± 4 и
связь с Tyr169 в участке цепи, формирующем сайт
20 ± 3 нМ, которые являются верхней оценкой
специфического узнавания MCB. Этот участок,
константы диссоциации комплексов dSwi4 с
скорее всего, меняет свою конформацию при об-
SCB-ДНК. Полученные значения свидетельству-
разовании комплекса, что, вероятно, сопровож-
ют о существенно большей аффинности Swi4 к
дается изменением микроокружения Trp173. У
SCB, чем сообщалось ранее (Kd = 830 нМ) [7].
dSwi4 в гомологичном положении находится фе-
Различия в полученных данных могут быть вы-
нилаланин, с которым Trp112 водородную связь
званы отличиями в нуклеотидной последователь-
образовать не может. Остаток Trp129 (гомологи-
ности исследуемой ДНК. Мы исследовали ДНК,
чен Trp68 у dSwi4) находится на внешней, не свя-
имеющую консенсусную последовательность
занной с ДНК, поверхности dPCG2 и не образует
SCB [11], а в опубликованной ранее работе [7]
связей с другими аминокислотными остатками,
изучали ДНК, воспроизводящую сайт связыва-
что похоже на выявленное по спектрам флуорес-
ния SBF в одном из промоторов. Кроме того, от-
ценции состояние остатков Trp в dSwi4.
личия в константах могут быть частично связаны
Анализ аффинности dSwi4 к SCB-ДНК. Иссле-
со снижением потерь катионного dSwi4 из-за не-
дования методом электрофореза в полиакрила-
специфической сорбции, которое было достигну-
мидном геле показали, что комплексы dSwi4 с
то нами за счет введения в реакционную смесь
SCB-ДНК образуются в наномолярном диапазо-
бычьего сывороточного альбумина.
не концентраций белка (рис. 5а). Анализ электро-
фореграмм показал, что образование комплекса
ЗАКЛЮЧЕНИЕ
сопровождается характерным увеличением ярко-
Транскрипционный фактор S. cerevisiae SBF
сти полосы комплекса и сопутствующим сниже-
(димер Swi4 и Swi6), контролирует переход от G1-
нием яркости полосы SCB-ДНК (рис. 5в,г). Об-
к S-фазе клеточного цикла почкующихся дрож-
наружено зависящее от концентрации dSwi4 сни-
жей, регулируя экспрессию генов, управляющих
жение яркости полосы одноцепочечного
морфогенезом и почкованием. Связывание SBF с
олигонуклеотида SCB_F (рис. 5д), что поставило
ДНК в геноме дрожжей определяется его субъ-
вопрос о возможном формировании комплекса
единицей Swi4. Проведенные нами исследования
между dSwi4 и одноцепочечным олигонуклеоти-
выявили высокую аффинность ДНК-связываю-
дом. Методом электрофореза проведено исследо-
щего домена Swi4 (dSwi4) к консенсусной после-
вание предполагаемого образования комплекса
довательности сайта связывания SBF в геноме и
между dSwi4 и SCB_F, которое не подтвердило
позволили выяснить характер структурных изме-
данную гипотезу: в отсутствии комплементарно-
нений в белке при образовании комплекса с SCB-
БИОФИЗИКА том 67
№ 5
2022
СТРУКТУРА И АФФИННОСТЬ КОМПЛЕКСОВ
875
Рис. 5. Анализ взаимодействия dSwi4 с SCB-ДНК и SCB_F. (а), (б) - Нативный гель-электрофорез смесей (а) dSwi4 с
SCB_F (5 нМ) и SCB_R (5 нМ), (б) dSwi4 с SCB_F (5 нМ). В качестве маркеров (М) использовали смесь растворов
ксиленцианола (КЦ) и бромфенолового синего (БФС). (в), (г) - Результаты усредненного денситометрического анализа
электрофореграмм, пример которых приведен на рисунке (а), характеризирующие образование комплекса dSwi4:SCB-
ДНК по увеличению яркости полосы dSwi4:SCB-ДНК (в) и уменьшению яркости полосы SCB-ДНК (г). (д), (е) -
Денситометрический анализ изменения яркости полосы SCB_F на электрофореграммах, представленных соответственно
на рисунках (а) и (б).
ДНК. С учетом полученных ранее данных [7]
зависимой экспрессии генов. Таким образом, мо-
можно предположить, что аффинность dSwi4 к
лекулярные аспекты сайт-специфического узна-
сайту SCB существенно зависит от последова-
вания белком Swi4 промоторов, контролируемых
тельностей нуклеотидов, фланкирующих SСB в
фактором SBF, требуют дальнейшего детального
составе промоторов. Такая зависимость может
изучения и, в том числе, установления структур
являться инструментом тонкой регуляции Swi4-
высокого разрешения комплексов dSwi4 с ДНК.
БИОФИЗИКА том 67
№ 5
2022
876
АФОНИН и др.
БЛАГОДАРНОСТИ
2. D. J. Stillman, Trends Biochem. Sci., 38 (9), 467
(2013).
Исследование выполнено с использованием
инфраструктурных возможностей Междисци-
3. S. Takahata, Y. Yu, and D. J. Stillman, Mol. Cell, 34
плинарной научно-образовательной школы Мос-
(4), 405 (2009).
ковского университета «Молекулярные техноло-
4. S. Takahata, Y. Yu, and D. J. Stillman, EMBO J., 28
гии живых систем и синтетическая биология».
(21), 3378 (2009).
5. S. Takahata, Y. Yu, and D. J. Stillman, J. Biol. Chem.,
ФИНАНСИРОВАНИЕ РАБОТЫ
286 (40), 34809 (2011).
Работа выполнена при финансовой поддержке
6. J. M. Bean, E. D. Siggia, and F. R. Cross, Genetics,
Министерства науки и высшего образования
171 (1), 49 (2005).
Российской Федерации в рамках Соглашения
7. I. A. Taylor, P. B. McIntosh, P. Pala, et al., Biochemis-
№075-15-2021-1354 от 07.10.2021 г.
try, 39 (14), 3943 (2000).
8. J. Liu, J. Huang, Y. Zhao, et al., Nucl. Acids Res., 43
КОНФЛИКТ ИНТЕРЕСОВ
(2), 1231 (2015).
Авторы заявляют об отсутствии конфликта
9. B. E. Schoner, Methods Mol. Biol., 62, 89 (1997).
интересов.
10. N. W. Sreerama, Anal. Biochem., 287, 252 (2000).
11. D. J. Stillman, A. T. Bankier, A. Seddon, EMBO J., 7
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
(2), 485 (1988).
Настоящая статья не содержит каких-либо ис-
12. J. R. Lakowicz, Topics in fluorescence spectroscopy: bio-
следований с участием людей или животных в ка-
chemical applications (Kluwer Acad. Publ., New York.
честве объектов исследований.
2002).
13. J. T. Vivian and P. R. Callis, Biophys. J., 80 (5), 2093
СПИСОК ЛИТЕРАТУРЫ
(2001).
1. A. Hendler, E. M. Medina, N. E. Buchler, et al., Curr.
14. V. V. Khrustalev, V. V. Poboinev, A. N. Stojarov, and
Genetics, 64 (1), 81 (2018).
T. A. Khrustaleva, Eur. Biophys. J., 48 (6), 523 (2019).
Structure and Affinity of Complexes between DNA-Binding Domain of Swi4 and DNA
D.A. Afonin*, O.V. Geras’kina*, T.V. Loseva*, M.P. Kirpichnikov*,
V.M. Studitsky*, **, and A.V. Feofanov*
*Faculty of Biology, Lomonosov Moscow State University, Leninskie Gory 1/12, Moscow, 119991 Russia
**Fox Chase Cancer Center, Philadelphia, PA 19111-2497, USA
A recombinant DNA-binding domain of the Swi4 protein, which is the component of the transcription factor
SBF of the budding yeast Saccharomyces cerevisiae, was obtained. Based on the information derived by circu-
lar dichroism spectroscopy, a structure of the domain involves α-helices (29%), β-sheets (22%), β-turns
(21%) and a random coil (28%). The interaction of the domain with DNA containing the SCB (Swi4 Cell-
cycle Box) binding site for SBF was studied. The formation of complexes is accompanied by a conformational
rearrangement in the protein, which affects at least 10 peptide bonds and leads to a 6% increase in the α-he-
lical content with a concomitant decrease in the β-sheet content. Fluorescence spectroscopy revealed
that conformational rearrangements and intermolecular interactions do not affect the regions of Trp68 and
Trp112 residues located near the boundaries of the DNA-binding helix-turn-helix motif. It was found that the
dissociation constant of the complex composed of DNA-binding domain of the Swi4 protein and DNA is
20 ± 3 nM.
Keywords: SBF, Swi4, SCB, dissociation constant, circular dichroism, fluorescence
БИОФИЗИКА том 67
№ 5
2022