БИОФИЗИКА, 2022, том 67, № 6, с. 1109-1121
МОЛЕКУЛЯРНАЯ БИОФИЗИКА
УДК 578.23
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
КАК ПРОТИВОВИРУСНАЯ МИШЕНЬ ДЛЯ ИНГИБИТОРОВ АКТИВНОГО
ЦЕНТРА И БЕЛОК-БЕЛКОВЫХ ВЗАИМОДЕЙСТВИЙ
© 2022 г. П.В. Ершов*, #, Е.О. Яблоков*, Ю.В. Мезенцев*, Г.Н. Чуев**, М.В. Федотова***,
С.Е. Кручинин***, А.С. Иванов*
*Научно-исследовательский институт биомедицинской химии имени В.Н. Ореховича,
Погодинская ул., 10/8, Москва, 119121, Россия
**Институт теоретической и экспериментальной биофизики РАН,
Институтская ул., 3, Пущино Московской области, 142290, Россия
***Институт химии растворов им. Г. А. Крестова РАН, Иваново, Академическая ул., 1, 153045, Россия
#E-mail: pavel79@inbox.ru
Поступила в редакцию 05.06.2022 г.
После доработки 09.08.2022 г.
Принята к публикации 12.08.2022 г.
Папаиноподобная протеаза PLpro короновируса SARS-CoV-2 является мультифункциональным
ферментом, который катализирует протеолитический процессинг двух вирусных полипротеинов
pp1a и pp1ab. PLpro также расщепляет пептидные связи между белками клеток хозяина и
убиквитином (или убиквитино-подобными белками), что связывают с нарушением иммунных
процессов. Были приотеризированы девять структур наиболее действенных ингибиторов активного
центра PLpro по параметрам биохимических (IC50) и клеточных тестов для оценки подавления
вирусной репликации (EC50) и цитотоксичности (CC50). Литературный поиск показал, что PLpro
может взаимодействовать с не менее чем 60 потенциальными белковыми партнерами в клетках,
23 из которых являются мишенями для других вирусных белков (вирус папилломы человека, вирус
Эпштейна-Барр). Анализ белок-белковых взаимодействий показал, что белки USP3, UBE2J1,
RCHY1 и FAF2, вовлеченные в процессы де- и убиквитинилирования, содержат наибольшее число
связей с другими белками, а взаимодействие с ними вирусных белков может отражаться на
архитектуре всей сети белок-белковых взаимодействий. На примере пространственной модели
комплекса PLpro/убиквитин и набора из 154 соединений природного происхождения с известной
противовирусной активностью были предсказаны
13 соединений (молекулярные массы в
диапазоне 454-954 Да) в качестве потенциальных ингибиторов PLpro. Эти соединения связываются
с «горячими» аминокислотными остатками протеазы в положениях Gly163, Asp164, Arg166, Glu167
и Tyr264, участвующими во взаимодействии с убиквитином. Таким образом, фармакологическое
воздействие на периферические сайты PLpro, играющие важные роли в связывании белковых
субстратов, может быть дополнительной мишень-ориентированной противовирусной стратегией.
Ключевые слова: SARS-CoV-2, папаиноподобная протеаза, PLpro, белок-белковые взаимодействия,
вирус-хозяин, поверхностный плазмонный резонанс, ингибиторы.
DOI: 10.31857/S0006302922060084, EDN: LJZOBK
Всемирная организация здравоохранения
здравоохранения и привело к смерти нескольких
11 марта 2020 г. объявила о начале пандемии забо-
миллионов человек во всем мире [1]. Еще до на-
левания COVID-19, которое было вызвано коро-
чала волны штамма омикрон (B.1.1.529) к середи-
новирусом SARS-CoV-2. Заболевание оказало се-
не ноября 2021 года более 40% населения мира за-
рьезную нагрузку на национальные системы
разилось COVID-19 хотя бы один раз [2]. Появле-
ние и быстрое распространение новых штаммов
Сокращения: PLpro - папаиноподобная протеаза SARS-
CoV-2, Mpro - главная протеаза SARS-CoV-2, ББВ - SARS-CoV-2 стимулирует разработку лекарствен-
белок-белковые взаимодействия, SPR - поверхностный
ных средств, блокирующих разные стадии жиз-
плазмонный резонанс (Surface Plasmon Resonance), а.к.о. -
аминокислотные остатки.
ненного цикла вируса. Это достигается также че-
1109
1110
ЕРШОВ и др.
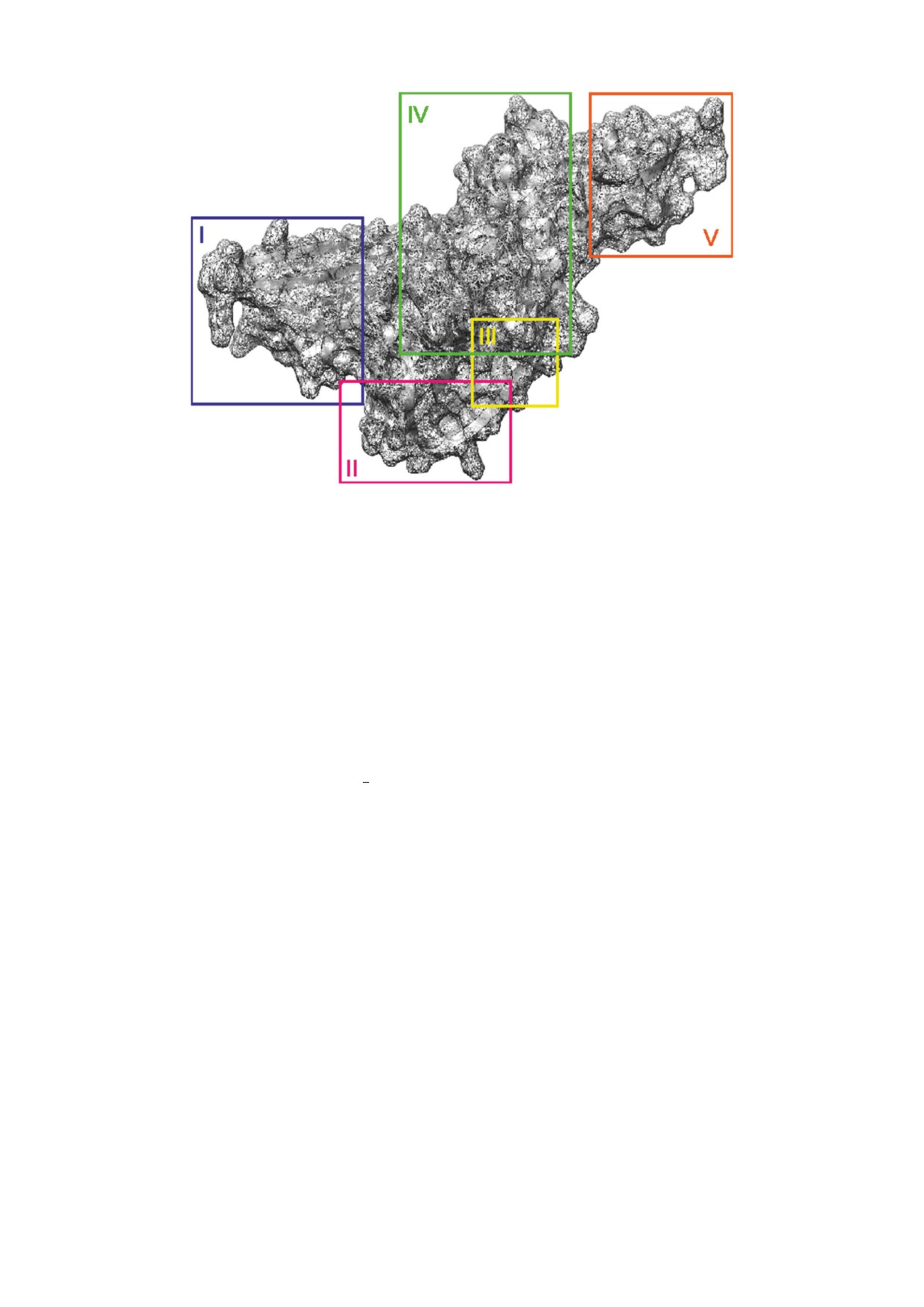
Рис. 1. Кристаллографическая структурная модель PLpro SARS-CoV-2 (PDB ID 7cmd [6]). Римскими цифрами на рисунке
обозначены: I - Zn-связывающий домен (один ион цинка координируют четыре остатка цистеина (Cys189, Cys192, Cys224,
Cys226)), II - домен Palm, III - каталитическая триада (Cys111-His272-Asp286), IV - домен «большой палец», V -
убиквитино-подобный домен.
рез ингибирование ключевых ферментов вирус-
превышать число экспериментально верифици-
ного аппарата репликации [3].
рованных ингибиторов. Данный факт порождает
очевидную проблему, состоящую в том, что дале-
Папаиноподобная протеаза (PLpro) и главная
ко не все гипотезы бывают продуктивны [12, 13].
протеаза (Mpro) SARS-CoV-2 осуществляют про-
В этом контексте использование мультиметоди-
теолитическое расщепление двух вирусных поли-
ческого инструментария (биохимические тесты,
протеинов pp1a и pp1ab, что важно для созрева-
оценка связывающей способности, клеточные
ния 16 белков, вовлеченных в репликацию и
тесты) является эталонным вариантом приорите-
сборку вирусных частиц [4]. PLpro является мо-
зации новых ингибиторов PLpro SARS-CoV-2
номерным белком с мультидоменной структурой
[14-16]. Однако уместно заметить, что фармако-
(рис. 1) и «каталитической триадой» (аминокис-
логическое воздействие на активный центр PLpro
лоты Cys112, His273, Asp287) в активном центре,
имеет некоторые биологические ограничения.
которая распознает и расщепляет мотив Leu-X-
Во-первых, возникновение аминокислотных за-
Gly-Gly полипротеина (где Х — любой аминокис-
мен в активном центре вследствие высокой мута-
лотный остаток) с образованием вирусных белков
ционной изменчивости разных штаммов вируса
nsp1, nsp2 и nsp3 [5].
может быстро формировать резистентность к
На сегодняшний день пока отсутствуют разре-
конкурентным ингибиторам. Во-вторых, широ-
шенные для клинического применения ингиби-
кая представленность остатков Cys в составе ак-
торы PLpro. По данным портала Clinical Trials
тивных центров клеточных ферментов увеличи-
проводятся клинические исследования двух кан-
вает вероятность неселективного действия инги-
дидатных ингибиторов PLpro - изотретиноина
биторов, образующих ковалентные связи с Cys.
(NCT04361422) и эбселена (NCT04484025). По-
давляющее большинство научных разработок по-
Особенность PLpro взаимодействовать с кле-
священо идентификации обратимых и необрати-
точными белками подчеркивает мультифункцио-
мых ингибиторов, нацеленных именно на актив-
нальный аспект фермента. Было установлено, что
ный центр вирусного фермента. Параллельно с
это явление сопровождается расщеплением пеп-
генерацией большого объема информации о но-
тидных связей между белками-метками (убикви-
вых ингибиторах PLpro нельзя не заметить, что
тин, ISG15 - интерферон-стимулируемый ген 15
число предсказанных in silico биоинформацион-
и убиквитино-подобный белок Nedd8) и целевы-
ных гипотез [7-11] примерно на порядок может
ми белками [16-18]. Так, PLpro, снижая уровень
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1111
убиквитинилирования белков TRAF3, TBK1,
ной активности был использован спектральный
IKKε, STING и IRF3, косвенно влияет на экс-
метод, основанный на флуоресцентном резо-
прессию гена интерферона (IFN) [19], тем самым
нансном переносе энергии (FRET, от англ. fluo-
нарушая протекание иммунных процессов, а воз-
rescence resonance energy transfer) с флуоресцент-
можность PLpro участвовать в белок-белковых
но меченными пептидами, мотивы которых соот-
взаимодействиях (ББВ) [20] позволяет в целом
ветствуют сайтам узнавания PLpro, например,
рассматривать ее как экзогенный модулятор кле-
Z-RLRGG-AMC (где AMC - это 7-амидо-4-ме-
точных сигнальных путей через изменение спек-
тилкумарин) или Ac-LRGG-ACC (где ACC - это
тра ББВ. Как будет показано далее, в ББВ PLpro и
7-амино-4-карбамоилметилкумарин). Так, при
клеточных белков, по-видимому, вовлечена кон-
протеолитическом расщеплении субстрата на-
сервативная область, смежная с активным цен-
блюдалось значительное возрастание интенсив-
тром вирусного фермента, поэтому альтернатив-
ности флуоресценции AMC (λex
= 360 nm;
ная стратегия снижения токсического действия
λem = 460 nm) или ACC (λex = 355 nm, λem = 460 nm),
PLpro на организм человека, могла бы реализо-
а начальная скорость реакции (V0) была пропор-
ваться в фармакологической блокаде ББВ в коор-
циональна активности PLpro. Для регистрации
динатах вирус-хозяин.
деубиквитиназной активности PLpro использо-
Одной из целей данной работы является обзор
вали флуоресцентно меченные белковые субстра-
наиболее перспективных и экспериментально ве-
ты на основе убиквитина и ISG-15. Буферные
рифицированных кандидатных ингибиторов
растворы содержали
20-50 мМ трис-HCl
PLpro SARS-CoV-2, обнаруженных к настоящему
(рН 6.8-8.0) с добавлением NaCl до финальной
времени. Второй целью являются систематиза-
концентрации 150 мМ. Время преинкубации ин-
ция данных по интерактомике PLpro и обсужде-
гибитора с PLpro перед добавлением субстрата
ние возможностей блокирования ББВ с участием
составляло от 10 до 30 мин при комнатной темпе-
PLpro низкомолекулярными соединениями с из-
ратуре или при 37°С. Финальные концентрации
вестной противовирусной активностью.
PLpro и субстрата в реакционной смеси составля-
ли 10-100 нМ и 10-50 мкМ соответственно, в
среднем молярное соотношение фермент : суб-
ЭКСПЕРИМЕНТАЛЬНАЯ ВЕРИФИКАЦИЯ
страт было равно 1:500. Содержание добавок для
ПРЕДСКАЗАННЫХ IN SILICO
улучшения растворимости низкомолекулярных
ИНГИБИТОРОВ
соединений в буферных растворах, таких как
Тритон X-100 и диметилсульфоксид, составляло
Поверхностный плазмонный резонанс (SPR,
0.01-0.05% (v/v) и 0-2% соответственно. Добав-
от англ. Surface Plasmon Resonance,) является од-
ление в реакционную смесь дитиотреитола до фи-
ним из способов экспериментальной верифика-
нальной концентрации 3-5 мМ необходимо для
ции предсказанных in silico потенциальных
ограничения неспецифического влияния низко-
ингибиторов PLpro. SPR-анализ позволяет
молекулярных соединений на остаток цистеина в
определить кинетические, равновесные и термо-
активном центре PLpro [25]. Так, шесть серосо-
динамические константы межмолекулярного
держащих соединений, не показавших ранее тор-
взаимодействия. В работах, где использовали
можения вирусной репликации, но ингибировав-
SPR-биосенсор, рекомбинантный полноразмер-
ших ряд цистеиновых протеаз в биохимическом
ный препарат PLpro был ковалентно иммобили-
тесте в отсутствии дитиотреитола, не снижали
зован на оптическом чипе CM5 за свободные
ферментативную активность протеаз в его присут-
аминогруппы белка [14] или за аффинный поли-
ствии. Так же, как и в SPR-анализе, в качестве по-
гистидиновый таг [15]. Целевой уровень иммоби-
зитивного контроля использовали контрольный
лизации PLpro составил 12-13 тысяч RU (reso-
ингибитор GRL0617 (IC50 = 0.6 и Ki = 0.5 мкМ). Для
nance unit). В качестве позитивного контроля свя-
зывания с PLpro был использован контрольный
изучения механизма ингибирования разные кон-
ингибитор GRL0617.
центрации целевого соединения инкубировали в
течение 30 мин в присутствии разных концентра-
В биохимических тестах изучают кинетику
ций PLpro (25-150 нМ) в объеме 90 мкл. Затем ре-
ферментативной реакции PLpro в отсутствии
акцию запускали добавлением 10 мкл субстрата
(контроль) и присутствии разных концентраций
PLpro до конечной концентрации 30 мкМ. Дан-
потенциальных ингибиторов для определения
ные в координатах начальных скоростей и кон-
значений IC50. Композиция параметров биохи-
центраций PLpro обрабатывали при помощи ре-
мического теста (рН и ионная сила буферного
грессионного анализа [21].
раствора, соотношение концентраций фермента
и ингибитора, время инкубации, температурный
Мы выполнили поиск соединений, которые
режим, присутствие добавок) существенно отли-
были описаны в литературе одновременно как
чалась, что следует из анализа литературных ис-
ингибиторы ферментативной активности PLpro
точников [15, 21-24]. Для регистрации протеаз-
по известным параметрам IC50, так и ингибиторы
БИОФИЗИКА том 67
№ 6
2022
1112
ЕРШОВ и др.
Рис. 2. Цифрами обозначены следующие соединения: (а) - XR8-23, (б) - YM155, (в) - проантоцианидин,
(г) - Jun9-75-4, (д) - соединение 29, (е) - соединение 6, (ж) - дигидротаншинон I, (з) - тропифексор,
(и) - соединение 7. Лекарствоподобные (от англ. drug-likeness) свойства были предсказаны для
соединений (б), (г), (д), (е), (ж) и (и), согласно данным ресурса SwissADME [36].
репликации SARS-CoV-2 по параметрам цито-
разных субстратов и композиции биохимических
токсичности (СС50) и полумаксимальной эффек-
тестов. На рис. 2 представлена выборка низкомо-
лекулярных соединений непептидной природы,
тивной концентрации (EC50). Определение
показавших наиболее благоприятный профиль
параметров СС50 и EC50 можно считать более
ингибирования по параметрам IC50, СС50, EC50:
«стандартизованными» ввиду использования
XR8-23 (рис. 1а) [15], YM155 (рис. 1б) [26], проан-
одинаковых методик и клеточных моделей (Vero
тоцианидин (рис. 1в) [27], Jun9-75-4 (рис. 1г) [28],
E6, A549, Caco2, Calu3). Что касается значений
соединение
29 (рис.
1д)
[23], соединение
6
IC50, то их достаточно сложно сравнивать между
(рис. 1е) [29], дигидротаншинон I (рис. 1ж) [30],
собой из разных работ вследствие применения тропифексор (рис. 1з) [31], соединение 7 (рис. 1и)
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1113
[32]. Важной характеристикой ингибиторов ви-
галось в концентрациях порядка 10-4-10-3 М [39].
русных ферментов является их побочное дей-
В работе [30] было установлено, что соединения с
ствие в отношении клеточных мишеней.
антипролиферативной
активностью
CAS
Предсказание спектра «нецелевых» клеточных
No. 331253-86-2, CAS No. 265312-55-8 и CAS
мишеней было выполнено для шести лекарство-
No. 37854-59-4 ингибировали PLpro cо значения-
подобных соединений (рис. 2б,г,д,е,ж,и) на веб-
ми IC50, равными 0.26, 0.39 и 0.53 мкМ соответ-
серверах SwissTargetPrediction
[33], BindingDB
ственно. Тем не менее, только соединение CAS
[34] и TargetNet (ECFP2, ECFP4, ECFP6, MACCS
No. 37854-59-4 показало приемлемый уровень
с AUC=0.95) [35] c отбором гипотез при значении
подавления вирусной репликации (EC50
=
параметра probability score 1. Так, гликогенфос-
= 20 мкМ) [30].
форилаза была предсказана как потенциальная
мишень для соединений, представленных на
рис. 2в,е,и; арахидонат-15-липоксигеназа - для
АМИНОКИСЛОТНЫЕ ОСТАТКИ
соединений, представленных на рис. 2г,е; кальпа-
ЦИСТЕИНА PLpro В КАЧЕСТВЕ МИШЕНЕЙ
ин-1 и неприлизин - для соединений, представ-
ДЛЯ СЕЛЕКТИВНЫХ ИНГИБИТОРОВ
ленных на рис. 2д,и; печеночная карбоксиэстраза
Цинк-связывающий домен PLpro SARS-CoV-2
1 - для соединений, представленных на рис. 2е,ж;
(рис.
1) представлен
“цистеиновой тетрадой”
форболин-1 - для соединений, представленных
(Cys189-X-X-Cys192-Xn-Cys224-X-Cys226), ко-
на рис. 2е,и. Другие белковые мишени были
ординирующей ион цинка. Принято считать, что се-
предсказаны в качестве уникальных для каждого
росодержащие лекарства активные в отношении
из шести соединений, а среднее число мишеней в
PLpro, такие как каптоприл или 6-тиогуанин, могут
расчете на одно соединение составило 12 (интер-
неселективно “выталкивать” ионы цинка не только
вал от 3 до 21). Только три «нецелевых» белковых
из PLpro, но и из клеточных белков [40]. Одно из
мишени были предсказаны для соединения,
удачных решений было реализовано в пептидоми-
представленного на рис. 2б.
метиках, нацеленных на остатки цистеина активно-
го центра PLpro. Примерами были необратимые ин-
гибиторы VIR250 и VIR251, которые образуют кова-
РЕПОЗИЦИОНИРОВАНИЕ
лентную тиоэфирную связь с остатком цистеина
ФАРМАКОЛОГИЧЕСКИ АКТИВНЫХ
PLpro в положении 111 [41].
СОЕДИНЕНИЙ В КАЧЕСТВЕ
ИНГИБИТОРОВ PLpro
ИНГИБИТОРЫ ДВОЙНОГО ДЕЙСТВИЯ,
Авторы работы [37] изучали ингибиторный
НАЦЕЛЕННЫЕ НА PLpro И Mpro
потенциал в отношении PLpro SARS-CoV-2 биб-
лиотеки из 70 известных ингибиторов деубикви-
Анализ работ в поисках соединений, способ-
тиназ и цистеиновых протеаз. Было установлено,
ных ингибировать сразу обе протеазы SARS-
что соединения SJB2-043 (ингибитор убикви-
CoV-2, выявил целый ряд кандидатных соедине-
тин-специфичной протеазы 1), TCID (ингибитор
ний с определенным фармакологическим потен-
убиквитин-С-терминальной гидролазы L3) и
циалом. Одно из производных халкона (соедине-
PR-619 (неселективный ингибитор деубиквити-
ние 6, рис. 2е) ингибировало Mpro и PLpro со
наз) характеризовались значениями IC50, равны-
значениями IC50, равными 11 и 1 мкМ соответ-
ми 0.6, 6.4 и 6.1 мкМ соответственно. При исполь-
ственно [22]. Гинкголовая и анакардиновая кис-
зовании меченного AMC убиквитина в качестве
лоты, подавляющие репликацию SARS-CoV-2,
субстрата PLpro значение IC50 составляло
были идентифицированы в качестве необрати-
0.09 мкМ в случае SJB2-043, который связывает-
мых ингибиторов Mpro и PLpro с IC50 2 и 16 мкМ
ся в периферическом сайте PLpro [37]. Противо-
соответственно, причем при добавлении дитио-
опухолевый препарат тарлокситиниб, необрати-
треитола значения IC50 не изменялись [21]. Со-
мый ингибитор рецепторных тирозинкиназ EG-
единение 29, подавляющее репликацию вируса
FR и HER2, продемонстрировал активность в
(EC50 около 1 мкМ), ингибировало обе протеазы
отношении PLpro SARS-CoV-2 (IC50 = 0.3 мкМ и
SARS-CoV-2 с IC50 0.67-1.72 мкМ и KD выше
Ki = 0.2 мкМ) по типу конкурентного ингибиро-
25 мкМ. Молекулярный докинг показал два воз-
вания. На клеточной модели гепатокарциномы
можных варианта связывания соединения 29 в
HUH7 тарлокситиниб снижал на 25% реплика-
активном центре: нековалентное и ковалентное,
цию SARS-CoV-2 в концентрации 10 мкМ, не
которое в последнем случае указывает на меха-
проявляя значимого цитотоксического эффекта
низм необратимого ингибирования [23]. Два-
[38]. Лозартан, антагонист рецептора ангиотензи-
дцать три производных эбселена, который обла-
на второго типа, подавлял на 50% репликацию
дает противовоспалительной, антиоксидантной и
SARS-CoV-2 (EC50 ~ 14 мкМ), однако, ингибиро-
цитопротекторной активностью, показали диф-
вание ферментативной активности PLpro дости-
ференциальный спектр ингибирования PLpro и
БИОФИЗИКА том 67
№ 6
2022
1114
ЕРШОВ и др.
который встречался в двух и более статьях. Функ-
Mpro SARS-Cov-2 в диапазоне от 10-8 до 10-6 М
циональный анализ всего спектра потенциаль-
[24].
ных белковых партнеров PLpro приведен в табл. 1.
Большинство белков расположено в цитоплазме
ИНГИБИТОРЫ PLpro ПЕПТИДНОЙ
и в мембранах эндоплазматического ретикулума.
ПРИРОДЫ
Они выполняют как структурные (например, свя-
зывание с микротрубочками, макромолекулами и
Фармакологически активные агенты пептид-
фосфолипидами), так и ферментативные функ-
ной природы, нацеленные на белки репликаци-
ции (например, проявление лигазной активно-
онного аппарата SARS-CoV-2, представляют не-
сти). Далее была проверена биоинформационная
малый интерес для разработки противовирусных
гипотеза о том, являются ли клеточные белки,
лекарств, прежде всего, с точки зрения удобства
связывающиеся с PLpro короновирусов SARS-
органического синтеза [42]. В литературе описа-
CoV-2 и SARS-CoV, мишенями для экзогенных
ны результаты доклинических исследований про-
белков, появляющихся в клетках при инфициро-
филактического вакцинного препарата для ин-
вании/репликации другими вирусами. Поиск
траназального введения с непродолжительным
таких интерактомных данных и визуализация се-
сроком действия на основе липопептида, меха-
ти ББВ были выполнены с использованием ре-
низм действия которого заключается в блокаде
взаимодействия SARS-CoV-2 с восприимчивыми
vard.edu/V_hostome/) [55] на примере вируса па-
клетками [43]. Стоит сказать, что сообщения по
пилломы человека, вируса Эпштейна-Барр и
пептидным ингибиторам PLpro SARS-CoV-2
аденовируса (рис. 3). Из рис. 3 следует, что по
встречаются значительно реже по сравнению с
крайней мере около трети клеточных белков
низкомолекулярными соединениями непептид-
(23 из 61 белка), взаимодействующих с PLpro, яв-
ной природы. Помимо чисто биоинформацион-
ляются мишенями для белков других вирусов, что
ного предсказания подобных пептидов [44, 45],
может указывать на «универсальность» выбора
есть работы с экспериментальной верификацией
клеточных мишеней среди этих трех вирусов и
компьютерных предсказаний. Так, авторы рабо-
короновирусов SARS-CoV-2 и SARS-CoV. Это
ты [46] создали конструкции на основе LRGG
следует из того, что несколько разных вирусных
мотива узнавания PLpro, сшитого посредством
белков, в том числе и PLpro, взаимодействуют с
химического линкера с ингибитором GRL0617
одной клеточной мишенью (например, MBOAT7,
(IC50 порядка 10-6 М). В работе [47] было показа-
рис. 3). С другой стороны, вирусный белок
но, что димерные модифицированные производ-
HPV6B-E5A взаимодействует с шестью разными
ные пептидов на основе мотива ботропс-токсина I
клеточными белками (FAF2, VMP1, AUP1,
(KKYRYHLKPFCKK) подавляли репликацию
SNX19, TMEM43, ARL6IP5) (рис. 3) Таким обра-
SARS-CoV-2 (EC50 = 28-65 мкM) и более специ-
зом, можно предположить, что если область кон-
фично ингибировали PLpro (IC50 = 1.0-3.5 мкM)
такта вирусного белка с клеточными белками
представлена одним и тем же структурным эле-
в сравнении с Mpro. Результаты компьютерного
ментом, то ее можно рассматривать как мишень
моделирования позволили создать модели связы-
для нацеливания фармакологически активных
вания пептидных ингибиторов с PLpro в области
ингибиторов ББВ.
BL2 петли (265-TGNYQCG-271), которая критич-
на для узнавания субстрата и связывания извест-
Одиннадцать потенциальных белковых парт-
ных низкомолекулярных ингибиторов [47].
неров (MKRN2, MKRN3, RCHY1, FAF2, AUP1,
TRIM13, UBE2J1, UFSP2, UFL1, USP3, ZER1),
взаимодействующих с PLpro SARS-CoV-2 и
БЕЛОК-БЕЛКОВЫЕ ВЗАИМОДЕЙСТВИЯ
SARS-CoV, вовлечены в процессы убиквитини-
С УЧАСТИЕМ PLpro В КАЧЕСТВЕ
лирования и деубиквитинилирования. Это хоро-
МИШЕНЕЙ ДЛЯ ФАРМАКОЛОГИЧЕСКОГО
шо согласуется с кристаллографическими данны-
ВОЗДЕЙСТВИЯ
ми о связывании PLpro c убиквитином и белком
Взаимодействие вирусных белков с клеточны-
ISG15 [56-58] и наличием деубиквитиназной ак-
ми белками играет значимую роль в патогенезе
тивности PLpro. Сеть ББВ с участием 61 вершин
заболевания, поскольку может перепрограмми-
(клеточных белковых мишеней PLpro) была смо-
ровать естественные процессы для поддержания
делирована в программе NetworkAnalyst v. 3.0 [59]
жизненного цикла вируса. На данный момент
имеется достаточно много результатов интерак-
ем в качестве источников данных IMEx (Interna-
томного профилирования потенциальных белко-
tional Molecular Exchange Consortium) и
вых партнеров вирусных белков SARS-CoV-2.
STRINGdb (Search Tool for the Retrieval of Interact-
Анализ восьми статей [20, 48-54] позволил нам
ing proteins database). Для каждой из 11 вершин
извлечь 472 клеточных белковых партнера PLpro.
(целевых белков) в сети ББВ (табл. 2) приведены
Однако в целевую группу мы включили 61 белок,
данные по числу связей конкретной вершины с
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1115
Таблица 1. Анализ групп клеточных потенциальных белковых мишеней PLpro SARS-CoV-2, представленных
терминами генной онтологии (от англ. gene ontology)
Группа генной онтологии, p < 0.05
%*
Белки представленные в группе
GO:0005783 ~ субклеточная
57
ANKLE2, ATL1, FAF2, STX18, UFSP2, AUP1, UBE2J1, UFL1,
локализация в эндоплазматическом
SMPD4, TMEM43, DDRGK1, ARL6IP5, CLCC1, SEC63, WLS,
ретикулуме
OSBPL8, CAMLG, MBOAT7, SURF4, CDKAL1, ACSL3, VRK2,
TEX2, VMP1, RINT1, LSG1, STIM1, TMEM214, SOAT1, STIM2,
REEP4, TRIM13, ESYT2, DHCR7, SLC27A4
GO:0005737 ~ субклеточная
85
VEZT, ANKLE2, FMR1, FAF2, UFSP2, AUP1, UFL1, SMPD4,
локализация в цитоплазме
TMEM43, GPBP1, ARL6IP5, CLCC1, SEC63, WLS, CAMLG,
AKTIP, USP3, CDKAL1, ACSL3, VRK2, FNDC3A, TEX2, RINT1,
LSG1, STIM1, TMEM214, ESPL1, SOAT1, STIM2, TRIM13,
SLC27A4, SNAP47, ATL1, STX18, RCHY1, UBE2J1, FXR1,
CCNB2, FXR2, DDRGK1, SNX25, OSBPL8, MBOAT7, SURF4,
HOOK3, VMP1, TMEM199, SNX19, REEP4, SNX14, ESYT2,
DHCR7
GO:0010256 ~ субклеточная
20
VMP1, OSBPL8, CCNB2, ANKLE2, TMEM43, REEP4, AKTIP,
локализация в мембранах органелл
ATL1, SURF4, STX18, ACSL3, HOOK3
GO:0016874 ~ лигазная активность
11
UFL1, MKRN2, TRIM13, MKRN3, ACSL3, RCHY1, SLC27A4
GO:0035091 ~ участие в связывании
8
OSBPL8, SNX19, SNX25, SNX14, ESYT2
с фосфатидилинозитолом
GO:0019787 ~ убиквитино-подобная
10
UFL1, ZER1, AKTIP, TRIM13, RCHY1, UBE2J1
трансферазная активность
GO:0005515 ~ участие в связывании
75
VEZT, SNAP47, ANKLE2, GRAMD1A, TEX264, FMR1, ATL1,
с белками
FAF2, STX18, UFSP2, AUP1, RCHY1, UBE2J1, FXR1, UFL1,
CCNB2, FXR2, TMEM43, DDRGK1, SNX25, GPBP1, ARL6IP5,
SEC63, WLS, CAMLG, AKTIP, MBOAT7, USP3, SURF4,
CDKAL1, ACSL3, VRK2, HOOK3, VMP1, TMEM199, RINT1,
SNX19, STIM1, ESPL1, SOAT1, STIM2, REEP4, TRIM13,
MKRN2, MKRN3, ESYT2
GO:0008289 ~ участие в связывании
11
OSBPL8, SNX19, SOAT1, SNX25, SNX14, ESYT2, TEX2
липидов
GO:0005543 ~ участие в связывании
8
OSBPL8, SNX19, SNX25, SNX14, ESYT2
фосфолипидов
GO:0008017 ~ участие в связывании
6
STIM1, REEP4, FMR1, HOOK3
с микротрубочками
Примечание. * - Процент белков в группе от общего числа потенциальных белковых мишеней PLpro (61 белок).
другими вершинами в сети и «степени посредни-
Пока отсутствуют кристаллографические мо-
чества» (от англ. betweenness centrality), который
дели взаимодействия PLpro с клеточными белка-
указывает на число кратчайших путей, проходя-
ми, за исключением убиквитина и белка ISG-15,
щих через вершину [60]. Из табл. 2 можно видеть,
поэтому концепцию модуляции ББВ с участием
что убиквитин-протеинлигазы RCHY1 и FAF2, а
PLpro низкомолекулярными соединениями мы
также деубиквитиназы USP3 и UBE2J1, являются
рассмотрим далее именно на этих двух белках.
белками с наибольшем числом связей в сети ББВ,
Хотя аминокислотные последовательности PLpro
следовательно, взаимодействие с ними вирусных
SARS-CoV и SARS-CoV-2 обладают высокой го-
белков может приводить к изменениям архитек-
мологичностью, анализ областей контакта PLpro
туры значительной части сети ББВ.
c убиквитином или ISG-15 выполняли для обеих
БИОФИЗИКА том 67
№ 6
2022
1116
ЕРШОВ и др.
Рис. 3. Репертуар перекрестных белок-белковых взаимодействий с участием клеточных белков, которые могут
взаимодействовать с PLpro SARS-CoV (голубой цвет), с белками вируса папилломы человека (HPV), вируса
Эпштейна-Барр (EBV) и аденовируса (ADENO) по данным ресурса Virhostome. В качестве поискового запроса
был использован список из 61 потенциальной белковой мишени PLpro.
вирусных протеаз. Из табл. 3 следует, что большая
составила
92% (TYR273),
69% (ASP164 и
часть водородных связей и солевых мостиков в
ARG166), 54% (LEU162 и LYS157) и 31% (GLY163,
области контакта образуется консервативными
GLU167, TYR264, GLY266 и TYR268). При этом
аминокислотными остатками (а.к.о.) 163-170 и
GLY163, ASP164, ARG166, GLU167 и TYR264
265-273 PLpro. Виртуальный скрининг потенци-
PLpro SARS-CoV-2 были вовлечены во взаимо-
альных ингибиторов ББВ, способных связывать-
действие с убиквитином и ISG-15 (табл. 3). Ре-
зультаты компьютерного моделирования в одной
ся в области контакта между PLpro и убиквити-
из работ указали на то, что соединение (-)-эпи-
ном (или ISG-15), был выполнен на платформе
галлокатехингалат образует водородные связи с
для компьютерного дизайна лекарств ezCADD
остатками ASP164 и TYR273 PLpro, что сопоста-
среди 154 низкомолекулярных соединений при-
вимо с данными докинга (табл.
3 и
4), а
родного происхождения с установленной проти-
при концентрации 100 мкМ это соединение не-
вовирусной активностью (Catalog No. BCL0032,
значительно (на
13%)
снижает активность
BioCrick Co., Ltd, КНР) [61]. Наилучшие модели
PLpro [62]. Однако было установлено, что (-)-
связывания PLpro c
13 соединениями
(455-
эпигаллокатехингалат связывается также и с
954 Да) были предсказаны в боксе 20×25×20 с ко-
Mpro (KD = 6 мкМ), при этом с более выражен-
ординатами центра (X = 0, Y = 72, Z = 40) и ото-
ным ингибиторным эффектом (IC50 = 0.8 мкМ)
браны по значениям «score» меньше -8.0. Водо-
[63].
родные связи между соединениями и а.к.о. PLpro
SARS-CoV-2 приведены в табл. 4. Частота встре-
Связывание низкомолекулярных соединений
чаемости а.к.о. PLpro, значимых для ее взаимо-
с а.к.о. PLpro в области контакта с убиквитином
действия с низкомолекулярными соединениями,
должно затруднять доступ субстрата в активный
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1117
Таблица 2. Положение потенциальных белковых партнеров PLpro SARS-CoV и SARS-CoV-2 в сети белок-
белковых взаимодействий
Источник
IMEX*
STRINGdb**
данных
«Степень
«Степень
Белки
Число связей
Число связей
посредничества»
посредничества»
MKRN2
3
436
1
0
UFSP2
3
1249
5
2
ZER1
5
1579
8
6055
TRIM13
13
4819
0
0
UFL1
13
3207
7
871
USP3
26
14776
25
20083
UBE2J1
28
10830
16
6028
AUP1
36
14442
13
2280
MKRN3
50
24427
0
0
RCHY1
60
31175
23
20688
FAF2
69
33358
49
38202
Примечание. * - International Molecular Exchange Consortium (www.imexconsortium.org); ** - Search Tool for the Retrieval of In-
центр и подавлять деубиквитинилирование кле-
вативными а.к.о. Gly163, Asp164 и Glu167 PLpro,
точных белков. Пример модели связывания с та-
которые вовлечены в связывание убиквитина. Та-
ким соединением (корилагином) представлен на
ким образом, данное низкомолекулярное соеди-
рис. 4. Как видно из рисунка, корилагин связыва-
нение может ингибировать деубиквитиназную
ется с активным центром PLpro непосредственно
активность PLpro путем создания стерических за-
в зоне контакта протеазы с убиквитином,
труднений для связывания убиквитина и доступа
при этом значение «score» равно -10.0 (по дан-
полипептидной цепи к активному центру PLpro.
ным ezCADD) и энергия взаимодействия равна -
Интересно отметить, что ранее корилагин был
7.58 ккал/моль (по данным SwissDock) [64]. Так-
идентифицирован как блокатор взаимодействия
же из табл. 3 и 4 можно видеть, что корилагин (со-
белка «шипа» SARS-CoV-2 с ACE2 рецептором на
гласно модели связывания) в этом положении
поверхности клеток [65]. Воздействие фармако-
может образовывать водородные связи с консер-
логически активными соединениями на область
Таблица 3. Параметры области контакта PLpro коронавирусов SARS-CoV и SARS-CoV-2 с клеточными белками
Параметр/PD
Солевые
А.к.о. PLpro, вовлеченные в
Площадь, Å2
Н+-связи
S-S-связи
B ID*
мостики
связывание с клеточными белками
4m0w
999
17
4
0
Leu163**, Gly164, Asp165, Glu168,
Tyr265, Gly272, Glu162, Arg167, Gln175,
5tl7
817
12
5
0
Tyr268
6xaa
945
17
7
0
Gly163, Asp164, Arg166, Glu167, Tyr264,
Gly271, Glu161, Leu162, Ser170, Glu203,
6xa9
804
11
4
0
Met208, Thr225, Tyr268, Tyr269
Примечание. * - Параметры области контакта кристаллографических моделей определяли на сервере PDBePISA
6xaa и 6xa9 - SARS-CoV-2 PLpro/убиквитин и PLpro/ISG-15 соответственно; ** - Консервативные а.к.о. выделены
подчеркиванием.
БИОФИЗИКА том 67
№ 6
2022
1118
ЕРШОВ и др.
Таблица 4. Предсказание аминокислотных остатков PLpro коронавирусов SARS-CoV-2, вовлеченных в
связывание соединений природного происхождения
№
Название соединения
Mr
CAS No
Аминокислотные остатки
1
(-)-Epigallocatechin
458
989515
ASP164, GLY266, TYR268, TYR273
gallate
2
Calceolarioside B
478
105471985
LYS157, LEU162, ASP164, ALA246, GLY266, TYR273
3
Chebulagic acid
954
23094715
GLY163, ASP164, ARG166, GLU167, GLY266, TYR268, TYR 273
4
Corilagin
634
23094691
LYS157, GLY163, ASP164, GLU167, TYR273
5
Forsythoside A
624
79916771
LYS157, ASP164, ARG166, GLU167, TYR264, GLY266, TYR273
6
Ganoderiol F
454
114567474
LYS157, ASP164, ARG166, TYR264, ASN267, ASP302
7
Glycyrrhizic acid
819
-
LEU162, ASP164, ARG166, GLU167, TYR264, TYR268, TYR273
8
Hinokiflavone
538
19202369
LEU162, ARG166, TYR273
9
Mulberrofuran G
562
87085005
LEU162, ARG166, TYR273
10
Mulberroside C
458
102841430
LYS157, LEU162, GLY163, ARG166, TYR273
11
Myriceric acid B
634
55497795
LYS157, ARG166, TYR273
12
Procyanidin B1
578
29106512
GLY163, ASP164, ARG166, GLY266, TYR264, TYR273
13
Sennoside A
862
81276
LYS157, LEU162, ASP164, TYR268, TYR273
контакта PLpro с убиквитином и ISG-15 может
ственную реакцию клетки на его присутствие.
быть дополнительной антивирусной стратегией,
Ингибиторы белкового комплексообразования,
блокируя возможность вируса нарушать есте- связывающиеся вне области активного центра
Рис. 4. (а) - Визуализация 3D-модели комплекса PLpro SARS-CoV-2 и убиквитина (PDB ID 6xaa [56],
разрешение 2.70 Å, 8.5 кДа). (б) - Модель докинга низкомолекулярного соединения корилагина (630 Да) в
область контакта PLpro SARS-CoV-2 и убиквитина (PDB ID: 6wrh [5], разрешение 1.60 Å). Сплошной и
пунктирной рамками выделены области активного центра и Zn-связывающего домена соответственно.
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1119
PLpro, опосредованно влияют на протеолитиче-
ным фармакокинетическим профилем и высо-
скую активность [66]. Последнее актуально также
ким потенциалом ингибирования согласно зна-
в свете работы [31], в которой на in silico моделях
чениям IC50 и EC50, которые могут быть исполь-
связывания PLpro SARS-CoV-2 c тремя соедине-
зованы в качестве базовых структур для создания
ниями EACC, KY-226 и тропифексором (IC50 по-
более специфичных и действенных кандидатных
рядка 10-5-10-6 М) в U-образном кармане проте-
противовирусных лекарственных средств. Но как
справедливо отмечают авторы работы [69], не-
азы (также сайт связывания контрольного инги-
смотря на обнадеживающий прогресс в иденти-
битора GRL0617) была показана вовлеченность
фикации по меньшей мере 70 структур ингибито-
а.к.о. Gln269, Asp164, Tyr268, Lys157 в комплексо-
ров PLpro за последние два года, предстоит еще
образование. Итак, PLpro SARS-CoV-2, может
долгий путь для внедрения ингибиторов в клини-
участвовать во взаимодействии как минимум c
ческую практику. До сих пор не было показано,
61 клеточными белками, что следует из система-
что рационально разработанные ингибиторы
тизации интерактомной информации по данным
PLpro обладают противовирусной эффективно-
нескольких публикаций, однако, на сегодняш-
стью in vivo против инфекции SARS-CoV-2 на жи-
ний день кристаллографические модели суще-
ствуют только для двух комплексов
—
вотных моделях.
PLpro/убиквитин и PLpro/ISG-15. Следователь-
Систематизация данных по интерактомному
но, пока остается неизученным вопрос, является
профилированию белок-белковых взаимодей-
ли сайт связывания этих двух белков на PLpro
ствий с участием PLpro и клеточных белков поз-
уникальным или характерным для позициониро-
волила выявить 11 клеточных белков, принимаю-
вания других потенциальных белковых партне-
щих участие в процессах убиквитинилирования и
ров. Далее, какое преимущество даст блокирова-
деубиквитинилирования. Мы полагаем, что се-
ние сайта связывания убиквитина низкомолеку-
лекция фармакологически активных агентов, на-
лярными соединениями с противовирусной
целенных на периферические сайты PLpro, таких
активностью широкого спектра, например, кори-
как область контакта PLpro и клеточных белков,
лагином, и подавление деубиквитиназной актив-
может быть альтернативным вариантом противо-
ности PLpro? Известно, что вирусные деубикви-
вирусного воздействия на SARS-CoV-2.
тиназы нацелены на несколько клеточных про-
цессов, в основном на различные молекулы,
вовлеченные в сигнальный путь врожденного им-
ФИНАНСИРОВАНИЕ РАБОТЫ
мунитета, тем самым опосредовано подавляя его
Работа была выполнена в рамках Программы
и стимулируя репликацию вирусов [67, 68]. В ос-
фундаментальных научных исследований в Рос-
нове этих событий лежат несколько причин: деу-
сийской Федерации на долгосрочный период
биквитинилирование белков TRAF3 и TRAF6
(2021-2030 годы), тема № 122030100168-2.
(факторы, ассоциированные с рецептором TNF)
с последующими нарушениями интерфероново-
го сигнального пути и выработкой провоспали-
КОНФЛИКТ ИНТЕРЕСОВ
тельных цитокинов; инактивация пути с участи-
Авторы заявляют об отсутствии конфликта
ем Toll-подобных рецепторов и универсального
интересов.
транскрипционного фактора NF-kB, который ре-
гулирует экспрессию генов иммунного ответа, в
том числе гена интерферона-бета; PCNA (ядер-
СОБЛЮДЕНИЕ ЭТИЧЕСКИХ СТАНДАРТОВ
ный антиген пролиферирующих клеток)-ассоци-
ированная блокада рекрутирования ДНК-поли-
Статья не содержит описания исследований,
меразы Nu к сайтам повреждения ДНК и некото-
выполненных кем-либо из авторов, с участием
рые другие причины [67, 68].
людей или использованием животных в качестве
объектов.
ЗАКЛЮЧЕНИЕ
СПИСОК ЛИТЕРАТУРЫ
Открытие новых специфичных ингибиторов
вирусных протеаз Mpro и PLpro SARS-CoV-2
1. R. Ganesh, K. Mahalingam, N. Kandaswamy, et al.,
является крайне динамичным и высококонку-
Ind. J. Public Health, 65 (4), 375 (2021).
рентным направлением разработок среди многих
2. R. M. Barber, R. J. D. Sorensen, D. M. Pigott, et al.,
научных групп в мире. Большинство экспери-
Lancet, S0140 (2022).
ментально верифицированных ингибиторов на-
3. M. Mei and X. Tan, Front. Mol. Biosci., 8, 671263
целены на активный центр протеаз SARS-CoV-2.
(2021).
В данной работе мы проанализировали ряд пер-
спективных ингибиторов PLpro и выделили, по
4. Y. M. Báez-Santos, S. E. St John, and A. D. Mesecar,
крайней мере, шесть соединений c благоприят-
Antiviral Res., 115, 21 (2015).
БИОФИЗИКА том 67
№ 6
2022
1120
ЕРШОВ и др.
5. J. Osipiuk, S.-A. Azizi, S. Dvorkin, et al., Nat. Com-
32. B. Sanders, S. Pohkrel, A. Labbe, et al., Res. Square
mun., 12 (1), 743 (2021).
(2021). DOI: 10.21203/rs.3.rs-906621/v1
6. X. Gao, B. Qin, P. Chen, et al., Acta Pharm. Sin. B, 11
33. D. Gfeller, A. Grosdidier, M. Wirth, et al., Nucl. Acids
(1), 237 (2021).
Res., 42 (Web Server issue), W32 (2014).
7. A. Stasiulewicz, A. W. Maksymiuk, M. L. Nguyen,
34. M. K. Gilson, T. Liu, M. Baitaluk, et al., Nucl. Acids
et al., Int. J. Mol. Sci., 22 (8), 3957 (2021).
Res., 44 (D1), D1045 (2016).
8. D. Li, J. Luan, and L. Zhang, Biochem. Biophys. Res.
35. Z.-J. Yao, J. Dong, Y.-J. Che, et al., J. Comput. Aided
Commun., 538, 72 (2021).
Mol. Des., 30 (5), 413 (2016).
9. S. Rajpoot, M. Alagumuthu, and M. S. Baig, Curr. Res.
36. A. Daina, O. Michielin, and V. Zoete, Sci. Rep., 7,
Struct. Biol., 3, 9 (2021).
42717 (2017).
10. C. Wu, Y. Liu, Y. Yang, et al., Acta Pharm. Sin. B, 10
37. C.-C. Cho, S. G. Li, T. J. Lalonde, et al., ChemMed-
(5), 766 (2020).
Chem, 17 (1), e202100455 (2022).
11. P. Delre, F. Caporuscio, M. Saviano, et al., Front.
38. M. A. Redhead, C. D. Owen, L. Brewitz, et al., Sci.
Chem., 8, 594009 (2020).
Rep., 11 (1), 13208 (2021).
12. M. Loffredo, H. Lucero, D.-Y. Chen, et al., Sci. Rep.,
39. R. Nejat, A. S. Sadr, B. Freitas, et al., J. Pharm. Pharm.
11 (1), 5433 (2021).
Sci., 24, 390 (2021).
13. C. Ma and J. Wang, ACS Pharmacol. Transl. Sci., 5 (2),
40. B. K. Maiti, ACS Pharmacol. Transl. Sci., 3 (5), 1017
102 (2022).
(2020).
14. H. Shan, J. Liu, J. Shen, et al., Cell Chem. Biol., 28 (6),
41. W. Rut, Z. Lv, M. Zmudzinski, et al., Sci. Adv., 6 (42),
855 (2021).
eabd4596 (2020).
15. Z. Shen, K. Ratia, L. Cooper, et al., J. Med. Chem., 65
42. A. S. Skwarecki, M. G. Nowak, and M. J. Milewska,
(4), 2940 (2022).
ChemMedChem, 16 (20), 3106 (2021).
16. D. Shin, R. Mukherjee, D. Grewe, et al., Nature, 587
43. R. D. de Vries, K. S. Schmitz, F. T. Bovier, et al., Sci-
(7835), 657 (2020).
ence, 371 (6536), 1379 (2021).
17. N. Barretto, D. Jukneliene, K. Ratia, et al., J. Virol., 79
44. M. Moradi, R. Golmohammadi, A. Najafi, et al., Int.
(24), 15189 (2005).
J. Pept. Res. Ther., 28 (1), 24 (2022).
18. S. G. Devaraj, N. Wang, Z. Chen, et al., J. Biol. Chem.,
45. S. Sasidharan, C. Selvaraj, S. K. Singh, et al., J. Bio-
282 (44), 32208 (2007).
mol. Struct. Dyn., 39 (15), 5706 (2021).
19. X. Chen, X. Yang, Y. Zheng, et al., Prot. Cell, 5 (5), 369
46. N. Liu, Y. Zhang, Y. Lei, et al., J. Med. Chem., 65 (1),
(2014).
876 (2022).
20. A. Stukalov, V. Girault, V. Grass, et al., Nature, 594
47. M. C. L. C. Freire, G. D. Noske, N. V. Bitencourt,
(7862), 246 (2021).
et al., Molecules, 26 (16), 4896 (2021).
21. Z. Chen, Q. Cui, L. Cooper, et al., Cell Biosci., 11 (1),
48. J. Li, M. Guo, X. Tian, et al., Med. (NY), 2 (1), 99
45 (2021).
(2021).
22. J.-Y. Park, J.-A. Ko, D. W. Kim, et al., J. Enzyme In-
49. E. M. N. Laurent, Y. Sofianatos, A. Komarova, et al.,
hib. Med. Chem., 31 (1), 23 (2016).
bioRxiv (2020). DOI:10.1101/2020.08.28.272955
23. V. Di Sarno, G. Lauro, S. Musella, et al., Eur. J. Med.
50. P. Samavarchi-Tehrani, H. Abdouni, J. D. R. Knight,
Chem., 226, 113863 (2021).
et al., bioRxiv (2020). DOI:10.1101/2020.09.03.282103
24. M. Zmudzinski, W. Rut, K. Olech, et al., bioRxiv
51. J. R. St-Germain, A. Astori, P. Samavarchi-Tehrani,
(2020). DOI:10.1101/2020.08.30.273979
et al., bioRxiv (2020). DOI:10.1101/2020.08.28.269175
25. C. Ma, Y. Hu, J. A. Townsend, et al., ACS Pharmacol.
52. D.-K. Kim, B. Weller, C.-W. Lin, et al., bioRxiv (2021).
Transl. Sci., 3 (6), 1265 (2020).
DOI:10.1101/2021.03.15.433877
26. Y. Zhao, X. Du, Y. Duan, et al., Prot. Cell, 12 (11), 877
53. S. Pfefferle, J. Schöpf, M. Kögl, et al., PLoS Pathog., 7
(2021).
(10), e1002331 (2011).
27. C.-J. Kuo, T.-L. Chao, H.-C. Kao, et al., Antimicrob.
54. Z. Chen, C. Wang, X. Feng, et al., EMBO J., 40 (17),
Agents Chemother., 65 (4), e02577 (2021).
e107776 (2021).
28. Z. Xia, M. D. Sacco, C. Ma, et al., BioRxiv (2021).
55. O. Rozenblatt-Rosen, R. C. Deo, M. Padi, et al., Na-
DOI:10.1101/2021.03.15.435551
ture, 487 (7408), 491 (2012).
29. B. T. Freitas, I. A. Durie, J. Murray, et al., ACS Infect.
56. T. Klemm, G. Ebert, D. J. Calleja, et al., EMBO J., 39
Dis., 6 (8), 2099 (2020).
(18), e106275 (2020).
30. C. T. Lim, K. W. Tan, M. Wu, et al., Biochem. J., 478
57. C.-Y. Chou, H.-Y. Lai, H.-Y. Chen, et al., Acta Crys-
(13), 2517 (2021).
tallogr. D. Biol. Crystallogr., 70 (Pt 2), 572 (2014).
31. C. Ma, Y. Hu, Y. Wang, et al., ACS Infect. Dis., 8 (5),
58. C. M. Daczkowski, J. V. Dzimianski, J. R. Clasman,
1022 (2022).
et al., J. Mol. Biol., 429 (11), 1661 (2017).
БИОФИЗИКА том 67
№ 6
2022
ПАПАИНОПОДОБНАЯ ПРОТЕАЗА PLpro КОРОНАВИРУСА SARS-COV-2
1121
59. G. Zhou, O. Soufan, J. Ewald, et al., Nucl. Acids Res.,
65. L. J. Yang, R. H. Chen, S. Hamdoun, et al., Phytomed-
47 (W1), W234 (2019).
icine, 87, 153591 (2021).
60. J. K. Das, S. Roy, and P. H. Guzzi, Infect. Genet.
66. H. Lee, H. Lei, B. D. Santarsiero, et al., ACS Chem.
Evol., 93, 104921 (2021).
Biol., 10 (6), 1456 (2015).
61. A. Tao, Y. Huang, Y. Shinohara, et al., J. Chem. Inf.
Model., 59 (1), 18 (2019).
67. Q. Zhang, Q. Jia, W. Gao, et al., Front. Microbiol., 13,
62. E. Pitsillou, J. Liang, K. Ververis, et al., J. Mol. Graph.
839624 (2022).
Model., 104, 107851 (2021).
68. S. M. Soh, Y.J. Kim, H.H. Kim, et al., Int. J. Mol. Sci.,
63. A. Du, R. Zheng, C. Disoma, et al., Int. J. Biol. Mac-
23 (1), 492 (2022).
romol., 176, 1 (2021).
64. A. Grosdidier, V. Zoete, and O. Michielin, Nucl. Acids
69. H. Tan, Y Hu, P. Jadhav, et al., J. Med. Chem., 65
Res., 39 (Web Server issue), W270 (2011).
(11), 7561 (2022).
SARS-COV-2 Coronavirus Papain-like Protease PLpro as an Antiviral Drug Target
for Inhibitors of Active Site and Protein-Protein Interactions
P.V. Ershov*, E.O. Yablokov*, Yu.V. Mezentsev*, G.N. Chuev**, M.V. Fedotova***,
S.E. Kruchinin***, and A.S. Ivanov*
*V.N. Orekhovich Research Institute of Biomedical Chemistry, Pogodinskaya ul. 10/8, Moscow, 119121 Russia
**Institute of Theoretical and Experimental Biophysics, Russian Academy of Sciences,
Institutskaya ul. 3, Pushchino, Moscow Region, 142290 Russia
***G.A. Krestov Institute of Solution Chemistry, Russian Academy of Sciences, Akademicheskaya ul. 1, Ivanovo, 153045 Russia
The SARS-CoV-2 papain-like protease PLpro is a multifunctional enzyme that catalyzes the proteolytic pro-
cessing of two viral polyproteins such as pp1a and pp1ab. PLpro also cleaves peptide bonds between host cell
proteins and ubiquitin (or ubiquitin-like proteins), probably because of an impaired immune system. Nine
structures of the most effective inhibitors for PLpro active site were prioritized based on biochemical (IC50)
and cellular data to estimate viral replication suppression (EC50) and cytotoxicity (CC50). The literature re-
view demonstrated that PLpro can interact with no less than 60 potential cellular protein partners, 23 of which
are targets for other viral proteins (human papillomaviruses and Epstein-Barr virus). Analysis of protein-pro-
tein interactions revealed that USP3, UBE2J1, RCHY1, and FAF2, involved in deubiquitination/ubiquitina-
tion process, have the greatest number of bonds with other proteins, and the interaction of viral proteins with
these enzymes can impact the architecture of the entire network. Thirteen compounds (molecular weights of
454-954 Da) were predicted using a spatial model of the PLpro/ubiquitin complex and a panel of 154 natu-
rally occurring compounds with known antiviral activity. These compounds bind to the “hot spot” amino ac-
id residues of protease at the positions (Gly163, Asp164, Arg166, Glu167, Tyr264) which are involved in the
interaction with ubiquitin. Thus, the pharmacological effect on the peripheral sites of PLpro which play im-
portant roles in the binding of protein substrates can be an additional target-based antiviral strategy.
Keywords: SARS-CoV-2, papain-like protease, PLpro, protein-protein interactions, host virus, surface plasmon
resonance, inhibitors
БИОФИЗИКА том 67
№ 6
2022