БИОХИМИЯ, 2019, том 84, вып. 2, с. 252 - 260
УДК 577.2
ПРОТЕОМ ГОЛОВНОГО МОЗГА Drosophila melanogaster:
ОБОГАЩЕНИЕ ЯДЕРНЫМИ БЕЛКАМИ*,**
© 2019 К.Г. Кузнецова1, М.В. Иванов2, М.А. Пятницкий1,3,
Л.И. Левицкий2, И.Ю. Ильина1, А.Л. Чернобровкин4,
Р.А. Зубарев4,5, М.В. Горшков2, С.А. Мошковский1,6***
1 НИИ биомедицинской химии имени В.Н. Ореховича,
119121 Москва, Россия; электронная почта: smosh@mail.ru
2 Институт энергетических проблем химической физики
им. В.Л. Тальрозе РАН, 119334 Москва, Россия
3 Национальный исследовательский университет
«Высшая школа экономики», 101000 Москва, Россия
4 Каролинский институт, SE<171 77, Стокгольм, Швеция
5 Первый Московский государственный медицинский университет
им. И.М. Сеченова Минздрава России (Сеченовский Университет),
119991 Москва, Россия
6 Российский национальный исследовательский медицинский
университет им. Н.И. Пирогова Минздрава России;
117997 Москва, Россия
Поступила в редакцию 30.08.18
После доработки 10.10.18
Принята к публикации 10.10.18
Методом хромато масс спектрометрии высокого разрешения впервые охарактеризован протеом выделен
ного головного мозга плодовой мушки, содержащий идентификации около четырех тысяч белков. Полу
ченные данные сравнивали с протеомами целых насекомых и их голов, опубликованными ранее. Для этого
доступные необработанные масс спектрометрические данные обсчитывали так же, как собственные дан
ные. Как и ожидалось, протеом головного мозга оказывался подмножеством протеомов целого организма и
головы насекомого. Тем не менее, после тщательной проверки удалось обнаружить 24 белка, отсутствующих
в более глубоких протеомах, причем 13 из них не были ранее обнаружены на белковом уровне. Примеча
тельно, что 14 белков из этой группы характеризовались ядерной локализацией. В соответствии с этим,
сравнение трех использованных в работе наборов данных с учетом количественной оценки белков в протео
мах без использования метки выявило статистически значимое обогащение протеома головного мозга дро
зофилы ядерными белками. Таким образом, для исследования белков этой клеточной локализации можно
рекомендовать выделение препаратов головного мозга плодовой мушки.
КЛЮЧЕВЫЕ СЛОВА: протеомика, масс спектрометрия, Drosophila melanogaster, головной мозг, ядерный
белок.
DOI: 10.1134/S0320972519020088
Плодовая мушка Drosophila melanogaster - ле
протеомные карты дрозофилы начали получать
гендарный модельный организм для молекуляр
с самого начала развития протеомики [2]. Такие
ной биологии и медицины. Полагают, что при
материалы служат отправной точкой для срав
мерно три четверти генов человека, связанных с
нения протеомов, например, в различных моде
заболеваниями, имеют функциональные гомо
лях патологии, старения или воздействия фак
логи в геноме этого насекомого [1]. Типовые
торов среды. За более чем двадцать лет сущест
вования протеомики меняются применяемые в
ней методы, и типовые, референсные протеом
* Первоначально английский вариант рукописи опубли
ные данные также должны отвечать этим изме
msu.ru/biokhimiya, в рубрике «Papers in Press», BM 18 246,
нениям. Важное нововведение, определившее
19.11.2018. Статья на английском языке опубликована в один из широко используемых сегодня подхо
томе 84, вып. 1, 2019.
дов, скорострельную или панорамную протео
** Приложение опубликовано на сайте «Biochemistry»
(Moscow), Vol. 84, issue 2, 2019.
мику, заключается в том, что в исследованиях
*** Адресат для корреспонденции.
стали чаще применяться масс спектрометри
252
ПРОТЕОМ ГОЛОВНОГО МОЗГА Drosophila melanogaster
253
ческие детекторы с высоким разрешением. Од
лого возраста, примерно в равных отношениях
ним из флагманов такого анализа служит ион
обоих полов. Животных фиксировали замора
ная ловушка Orbitrap, используемая в биологии
живанием при -80 °С.
с начала 2000 х годов [3]. Прогресс обеспечил
Выделение головного мозга. В течение всей
возможность так называемого «углубления»
процедуры предварительно замороженных мух
протеомного анализа, когда в одном биологи
держали на льду в чашке Петри. У каждого насе
ческом образце, в зависимости от динамическо
комого тело быстро отделяли иглой. Голову по
го диапазона, удавалось идентифицировать от
мещали в 0,01 М фосфатно солевой буфер (PBS),
двух-трех до десяти тысяч белков [4].
pH 7,4 («Sigma Aldrich», США). Затем головную
До недавнего времени в основном источнике
капсулу разрывали двумя пинцетами под визу
протеомных данных в свободном доступе, репо
альным контролем при помощи бинокуляра
зитории PRIDE [5], имел место некоторый де
(«Nikon SMZ645», Япония) при 10× увеличении.
фицит данных по протеомике плодовой мушки,
Изъятые мозги помещали в PBS, после чего
полученных с высоким разрешением. Примером
центрифугировали при 6000 g в течение 15 мин
могли служить данные протеома целого орга
при 4 °С на центрифуге 5415R, («Eppendorf»,
низма мух, полученные путем сочетания одно
Германия). Буферный раствор удаляли, а осадок,
мерного электрофореза с хромато масс спект
содержащий головной мозг мух, замораживали
рометрией [6], а также данные по протеомике
и хранили при -80 °С до получения образцов
голов мух, полученные без использования электро
для протеомного анализа.
форетического геля, путем анализа белковых
Подготовка образцов для протеомного анализа.
фракций [7]. Только в процессе подготовки ре
Осадок, содержащий головной мозг от 200 мух,
зультатов, описанных в этой статье, появились
ресуспендировали в 100 мкл раствора для лизиса,
более сложные протеомные данные, например,
содержащего 0,1 % (w/v) Protease MAX Surfactant
связанные с анализом стадий развития дрозо
(«Promega», США), 50 мМ бикарбоната аммония
фил [8]. В своем проекте, посвященном послед
и 10% (v/v) ацетонитрила (ACN). Клеточный ли
ствиям редактирования РНК на протеомном
зат встряхивали на шейкере («Eppendorf», Гер
уровне, мы сфокусировались на анализе голов
мания) при комнатной температуре в течение
ного мозга мух [9]. Именно в центральной нерв
60 мин при 550 об/мин. Затем смесь обрабатыва
ной системе происходит большинство событий
ли ультразвуком посредством устройства Bandelin
редактирования интересующего нас типа. В ра
Sonopuls HD2070 («Bandelin Electronic», Герма
боте Kuznetsova et al. [9], исследуя сайты редак
ния) в течение 5 мин при 30% ной амплитуде,
тирования, мы описали состав протеома голов
короткими импульсами. После центрифугиро
ного мозга плодовой мушки на уровне пептид
вания при 15 700 g («Eppendorf», Германия) в те
ных фрагментов, содержащих точечные амино
чение 10 мин при 20 °C и собирали надосадоч
кислотные замены, но не на уровне целых бел
ную жидкость. Общую концентрацию белка из
ков. Между тем, оказалось, что мы впервые по
меряли путем анализа с бихинхониновой кисло
лучили такой протеом. Таким образом, в этой
той, используя BCA Kit («Sigma Aldrich», США).
статье мы хотим сообщить об особенностях про
К образцам добавляли раствор, содержащий
теома головного мозга взрослых дрозофил по
0,5 М дитиотреита (DTT) в 50 мМ бикарбоната
сравнению с протеомами головы и целого орга
триэтиламмония (TEABC), до конечной концент
низма этого насекомого [6, 7].
рации DTT, равной 10 мМ, после чего раствор
инкубировали в течение 20 мин при 56 °C. Затем
в раствор добавляли 0,5 М йодацетамид (IAM) в
МЕТОДЫ ИССЛЕДОВАНИЯ
50 мМ TEABC до конечной концентрации алки
лирующего агента 10 мМ. Смесь инкубировали
Культура Drosophila melanogaster. Живые об
в темноте при комнатной температуре в течение
разцы Drosophila melanogaster линии Canton S
30 мин.
любезно предоставлены к.б.н. Н.И. Романовой
Далее белки расщепляли трипсином («Pro
(кафедра генетики биологического факультета
mega», США). Фермент добавляли в отношении
МГУ им. М.В. Ломоносова). Мух содержали при
1 : 40 (w/w) к общему количеству белка. Смесь
25 °C с использованием готовой среды для дро
инкубировали в течение ночи при 37 °C. Рас
зофил Formula 5-24 («Carolina Biological Supply
щепление останавливали добавлением 5% (v/v)
Company», США) в одноразовых пластиковых
раствора уксусной кислоты.
пробирках объемом 50 мл («Orange Scientific»,
Образец встряхивали на шейкере («Eppendorf»,
Бельгия). Мух переносили в новые пробирки по
Германия) в течение 30 мин при 500 об/мин
достижении взрослого возраста. Для получения
при 45 °C, после чего центрифугировали его при
одного образца использовали двести мух взрос
15 700 g в течение 10 мин при 20 °C. Затем надо
БИОХИМИЯ том 84 вып. 2 2019
254
КУЗНЕЦОВА и др.
садочную жидкость наносили на фильтр с отсе
перименты проведены на масс спектрометре
чением 10 кДа («Millipore», США) и центрифу
Orbitrap QExactive Plus («Thermo Fischer Scien
гировали при 13 400 g в течение 20 мин при 20 °C.
tific», США). В более ранних работах эти спектры
После этого на фильтр добавляли 100 мкл 50%
получали на гибридном приборе LTQ Orbitrap
(v/v) муравьиной кислоты, и образец центрифу
Velos («Thermo Fischer Scientific», США). Для
гировали в прежних условиях. Конечную кон
поиска использовали базу данных Uniprot Dro<
центрацию пептидов измеряли с помощью на
sophila melanogaster Reference Proteome, 03.2018,
бора Peptide Assay и спектрофотометра NanoDrop
число записей 42 524. Настройки поисковика для
(«Thermo Fisher Scientific», США). Образцы упа
собственных данных были следующими (приво
ривали на вакуумном концентраторе («Eppendorf»,
дятся на английском языке в соответствии с
Германия) при 45 °C в стеклянной микропробир
языком интерфейса): enzyme - trypsin; number of
ке. Высушенные пептиды хранили при -80 °C
missed cleavages - 1; precursor accuracy unit - ppm;
до анализа путем жидкостной хроматографии -
precursor accuracy left - 10; precursor accuracy
тандемной масс спектрометрии (LC MS/MS).
right - 10; precursor isotope mass error - 0; product
Протеомный анализ методом жидкостной хро>
accuracy, Da - 0,1; FDR 1,0; FDR type - psm;
матографии - тандемной масс>спектрометрии.
minimum charge - 2; maximum charge - 4; generate
Хроматографическое разделение пептидов осу
decoy db - yes; decoy method - reverse; decoy pre
ществляли на полученной в лаборатории окта
fix - DECOY_; dynamic range - 100; peptide mi
дециловой колонке C18 длиной 25 см (хромато
nimum length - 7: peptide maximum length - 30;
графический носитель Silica Tip 360 μm OD, 75 μm
peptide minimum mass - 300; peptide maximum
ID, («New Objective», США)), присоединенной к
mass - 10 000; fragments in spectra, min - 4; fragments
хроматографической системе Ultimate 3000 RSLC
in spectra, max - 50; product minimum m/z - 150;
nano («Thermo Fisher Scientific», США), без ис
maximum fragments charge - 1; matched fragments,
пользования предколонки. Пептиды элюирова
min - 1; use scoring function - RNHS (renormalized
ли со скоростью потока 300 нл/мин в течение
hyperscore); score threshold - 0; show unmached
240 мин в линейном градиенте 2-26% ACN с
spectra in results - no; report number of sequence
0,1% (v/v) муравьиной кислотой. Элюирован
candidates - 1; peptide mass shift - 0; deisotope - yes;
ные пептиды ионизировали электрораспылением
deisotoping mass tolerance - 0,3. Фиксированных
и анализировали на масс спектрометре Orbitrap
модификаций аминокислотных остатков не за
QExactive Plus («Thermo Fisher Scientific», США).
давали. В качестве вариабельных модификаций
Обзорный масс спектр получали с разрешением
вводили окисление остатков метионина и алки
60 000 в интервале m/z 200-2000. Данные MS/MS
лирование остатков цистеина йодацетамидом.
для 20 наиболее интенсивных ионов предшест
В разделе дополнительной валидации результа
венников с зарядом не менее 2 получали с ис
тов поиска (Post search validation) использовали
пользованием коллизионной диссоциации с вы
все параметры, кроме charge status. Для данных
сокой энергией (HCD) с разрешением 15 000.
протеомов головы и целых организмов исполь
Динамическое исключение не более 500 пред
зовали те же параметры поиска, за исключение
шественников в течение 60 с использовали во
следующего: product accuracy, Da - 0,3.
избежание повторного анализа одних и тех же
Файлы выдачи в виде списка идентифици
пептидов. Всю процедуру повторяли в трех тех
рованных трипсиновых пептидов и собранных
нических репликатах.
из них белков обрабатывали для сравнения про
Полученные протеомные данные поместили
теомов. Для выявления уникальных пептидов,
в доступный общественности репозиторий Pro
присущих протеому головного мозга, подгото
вили скрипт на языке программирования R
[10] под инвентарным номером PXD004949.
(файл 1 в Приложении). Для работы скрипта ис
Обработка данных протеомного анализа. По
пользовали списки идентифицированных пеп
иски для идентификации белков проводили с
тидов набора данных по головному мозгу, голо
использованием программного обеспечения с
ве и целому организму плодовой мушки (файлы
открытым кодом Identipy [11], разработанного
2-4 Приложения соответственно).
Levitsky et al. Эта новая поисковая машина ис
В файлах выдачи по белкам для сравнения
пользует упрощенный алгоритм X!Tandem [12] с
протеомов (табл. S1 в Приложении) и оценки
улучшениями в различных частях поискового
белков ядерной локализации использовали рас
конвейера. Параметры поиска несколько отли
считываемый поисковой машиной Identipy ко
чались для собственных данных по анализу го
личественный параметр, называемый нормали
ловного мозга и для данных по голове [7] (учет
зованным фактором спектрального обилия (nor
ная запись в репозитории PRIDE PXD001712) и
malized spectral abundance factor, NSAF) [13] и
целому организму [6] (PXD000455). Наши экс
доступные идентификаторы Gene Ontology [14].
БИОХИМИЯ том 84 вып. 2 2019
ПРОТЕОМ ГОЛОВНОГО МОЗГА Drosophila melanogaster
255
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
vitro дезамидирование боковой группы аспара
гина и глутамина с образованием аспарагиновой
Сравнение протеомов головного мозга, головы
и глутаминовой кислот нельзя отличить от ко
и целого организма плодовой мушки. Идентифи
дируемой в геноме замены, и это не единствен
кация белков посредством одной и той же поис
ный пример [17]. Поэтому для того, чтобы сооб
ковой машины привела к обнаружению продук
щить об уникальных для головного мозга бел
тов 4005 генов в собственных данных по протео
ках, мы использовали эвристическое правило
му головного мозга, 6905 - в данных по протео
идентификации как минимум двух уникальных
му головы [7] и 7652 - в данных целого организ
пептидов каждого белка (табл. S2 в Приложе
ма дрозофилы [6]. Теперь следовало выяснить,
нии). По этому принципу удалось найти 24 спе
насколько перекрываются полученные протео
цифичных для мозга белка, среди которых
мы. Однако, когда мы начали их сравнивать,
шесть были аннотированы в Uniprot как пред
выяснилось, что из за запутанной синонимики
сказанные (Protein predicted), то есть, не обнару
названий белков и генов в базе данных Uniprot
женные ранее ни на уровне транскрипта, ни на
якобы уникальные для каждого из субпротеомов
белковом уровне (таблица). При анализе списка
белковые продукты при ручной проверке оказы
белков в таблице обращает на себя внимание еще
вались общими. Следует отметить, что аннота
один факт - больше половины из них, а имен
ция генов и белков модельных организмов в ба
но, 13 из 24, аннотируется как локализованные в
зах данных не настолько тщательно выверена,
ядре. Среди них связывающие ДНК регуляторы
как для человека, протеому которого посвящен,
и другие белки хроматина. Статистически дос
например, отдельный ресурс NextProt [15]. В ито
товерно показать обогащение ядерными белка
ге, наиболее корректно оценить перекрывание
ми списка из двадцати четырех продуктов слож
интересующих нас протеомов удалось, оперируя
но, однако, вопрос можно было исследовать в
отдельными идентифицированными пептидами.
масштабе протеома.
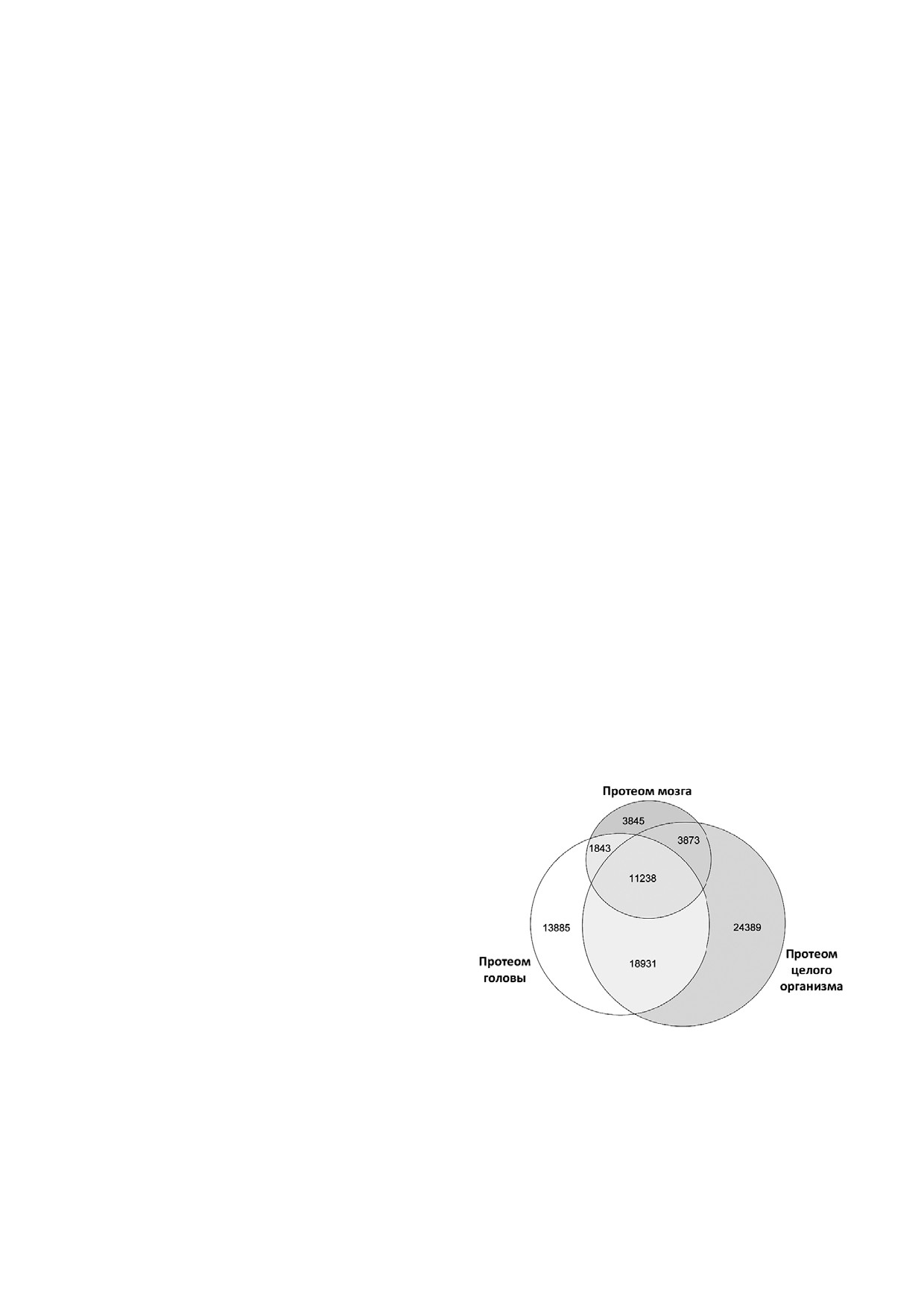
Как видно из диаграммы на рис. 1, лишь
Количественное обогащение протеома голов>
~18% пептидов из наших данных, полученных
ного мозга дрозофилы белками с ядерной локали>
из выделенного головного мозга, уникальны и
зацией. При небольшом количестве характер
не встречаются в более крупных наборах дан
ных для протеома мозга белков качественное
ных. Соответственно, для протеома головы и
обогащение белками ядерной локализации ос
целого насекомого соответствующие цифры
тается умозрительным наблюдением. Еще одно
составляют 30 и 41%. Сравнение протеомов на
наблюдение было сделано при просмотре дан
пептидном уровне, в общем, подтверждает оче
ных безметочного количественного анализа
видную мысль, что протеом головного мозга
всех трех исследуемых протеомов (табл. S1 в
представляет подмножество протеома головы и
Приложении). Оказалось, что в полученном в
целого организма. Однако, среди уникальных
этой работе протеоме, если сортировать белки
пептидов интересно было выявить те белки, из
которых они получились, и их характеристики,
чтобы понять, для каких исследований целесо
образно анализировать именно выделенный
мозг.
Белки, идентифицированные исключительно в
головном мозге. Поскольку анализ, а особенно
сравнение протеомов плодовой мушки по иден
тификаторам белков в автоматическом режиме
оказался затруднен из за запутанной синоними
ки этих идентификаторов, лучшим способом ус
тановить набор белков, найденных нами в го
ловном мозге, было заново собрать их из уни
кальных пептидов. Определение белка по одно
му уникальному пептиду, хотя и привлекатель
но, но, в соответствии с принятой практикой,
ненадежно: поисковые машины могут не разли
Рис. 1. Перекрывание протеома головного мозга Drosophila
чить два пептида с разной последовательностью
melanogaster с полученными ранее протеомами целого ор
ганизма [6] и головы насекомого [7], представленное в ви
по причине полного совпадения молекулярной
де диаграммы Венна. В связи с вырожденностью синони
массы одного неизмененного аминокислотного
мов в названиях белков плодовой мушки данные представ
остатка и другого с химической модификацией
лены на уровне уникальных пептидов, идентифицирован
[16]. Например, часто встречающееся in vivo и in
ных в сходных условиях поиска
БИОХИМИЯ том 84 вып. 2 2019
256
КУЗНЕЦОВА и др.
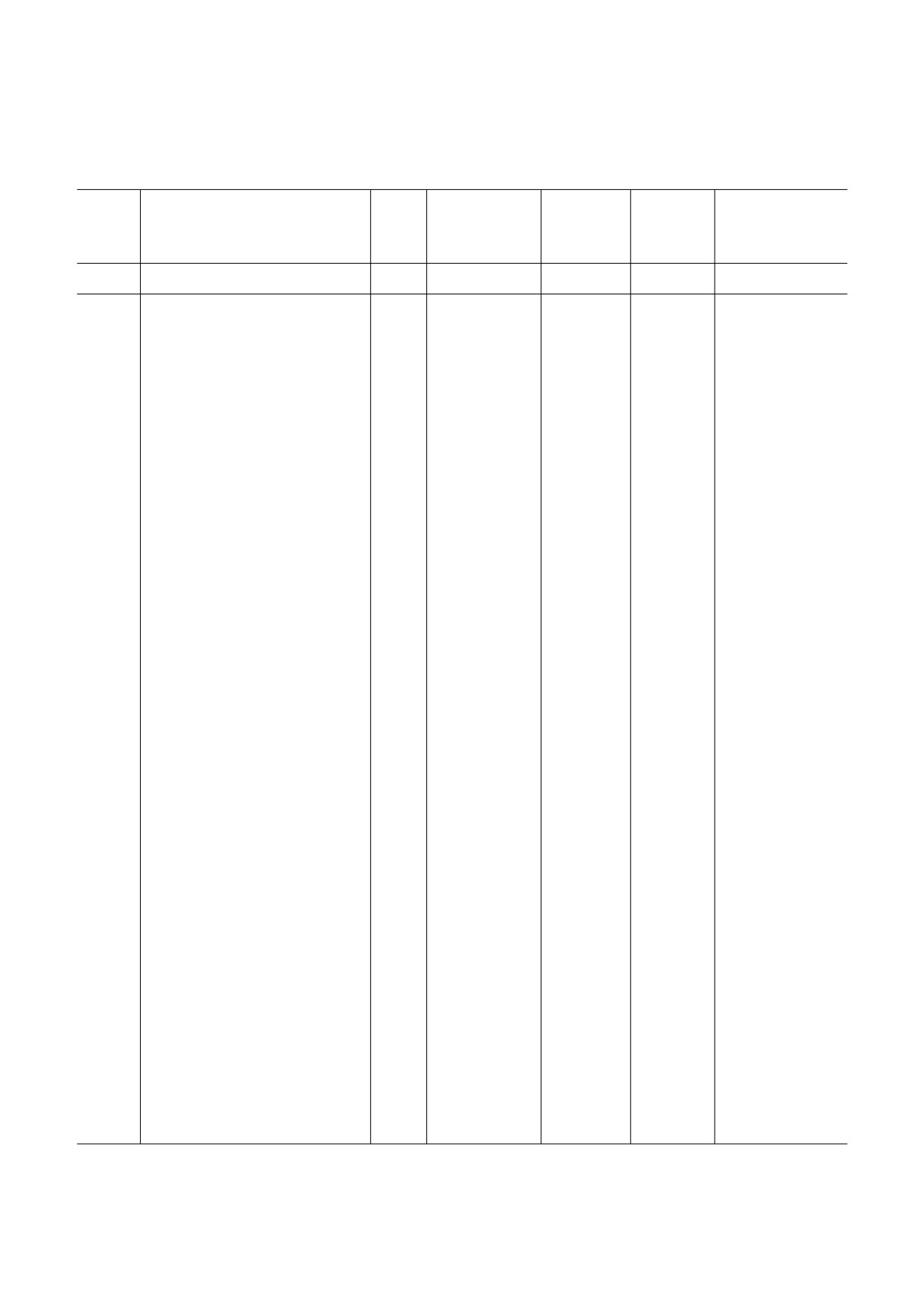
Белки, идентифицированные в протеоме головного мозга Drosophila melanogaster по сравнению с полученными ранее про
теомами головы [7] и целого организма насекомого [6]. Условия выбора белков - идентификация двух уникальных для
полученного нами набора данных пептидов. Названия генов, белков, внутриклеточная локализация и статус белка при
ведены по базе знаний Uniprot [18]
Последовательности идентифи
Число
Название
Клеточная
Uniprot
цированных пептидов, уникаль
пепти
Название гена
белка
локализа
Статус белка
ID
ных для набора данных по
дов
ция
головному мозгу
1
2
3
4
5
6
7
Q9VE88
GGAAEATNEIYPGDR;
7
Dmel\CG15803
неохаракте
цито
существование
GTHVPSVINTSQNPEAFETR;
ризованный
плазма
белка предсказано
LQLGDHLLQIGEVNLR;
белок,
LLQGLELDEAVSILK;
изоформа B
SILADGPVGR; LLSDPEELSR;
SLLPGGHQSLQNLLSK
Q9W263
SLALAQQLSAR;
6
Liprin<gamma
липрин
цито
существование
GNSLSAFSGSLSGGSGATAGDR;
гамма,
плазма
белка предсказано
SIDLGEYADNLK;
изоформа A
VQSETVDTDPIVWTNQR;
LQEQLQEAR; EASQLPIDR
Q9VXK9
SEQDSSLLVGNNAFDLEELGR;
4
anon<
CG9170,
неизвестно
доказано в экспери
LEALEDQFPENTLDAPYHLR;
WO0140519.214
изоформа A
менте на уровне
ISSSTASAELTK; ISGSGDTVSDK
транскрипта
Q9VC96
AALAQAAAGPSR; LGPLVDR;
4
BRWD3
BRWD3
ядро
доказано в экспери
VPDEVGGAVPVLPEEIR;
менте на уровне
FLAAGPLEETAK
белка
P29617
TPDDLLIAGDSELYR;
4
pros
белок с го
ядро
доказано в экспери
QADSVTAAAEQLNK;
меобоксом
менте на уровне
DLLLASQILDR; FVVESTLR
prospero
белка
Q9W2F9
SGVSWNQGDWGQIIR;
4
twz
RE04768p
ядро*
доказано в экспери
LFNGQIPIVLDSLK;
менте на уровне
YTAPVHIDVGGTIYTSSLETLTK;
транскрипта
ALLDELFPEASQATQSSR
Q59E33
AVITNKPEDGTLEDGQQPQK;
3
scaf6
LD21442p
ядро*
доказано в экспери
NQGIDTPISGGEVR;
менте на уровне
LPAPAPQSER
белка
Q7PL95
VLFTQAQVYELER;
3
scro
Scarecrow,
ядро
существование
NSASWYGSTANDPR; VAVPVLVK
изоформа A
белка предсказано
Q8IGK3
VQEVPFYFTLTPQQATEIASNR;
3
Su(var)2-10
RE73180p
ядро
доказано в экспери
ILSFLNISFAGR;
менте на уровне
QAVASSTSNGSGGGQR
белка
Q9W283
DLLQQEQTSSPLPQR;
2
a
Arc,
плазмати
доказано в экспери
LTIRDEEMAEVIR
изоформа A
ческая
менте на уровне
мембрана
транскрипта
Q9VZI2
TFDIGIFPR;
2
Ack
активируе
плазмати
доказано в экспери
LAHFDYVLPDDLER
мая Cdc42
ческая
менте на уровне
киназа
мембрана
транскрипта
Q9VEY2
QLPADYQLTPPTQESSTQDVTY
2
BcDNA:SD04753
CG6006,
плазмати
существование
LFR; IIEAAAK
изоформа B
ческая
белка предсказано
мембрана
Q9V9N4
NIIFTNAINGQPATIQYQTADG
2
Clamp
связанный с
ядро*
доказано в экспери
TILK;
хроматином
менте на уровне
IQIVNQNKPIAANTISNISFK
адаптер для
белка
БИОХИМИЯ том 84 вып. 2 2019
ПРОТЕОМ ГОЛОВНОГО МОЗГА Drosophila melanogaster
257
Окончание таблицы
1
2
3
4
5
6
7
белков MSL,
изоформа A
A1Z7A8
SPEASDQVENEPAPK;
2
coil
коилин
ядро
доказано в экспери
NQETSPDILSEK
менте на уровне
белка
Q24307
EALALGIDGGVVRNAIQR;
2
Diap2
ассоцииров
ядро
доказано в экспери
AASVPIPVADSIPAKPQAAEAVA
анный с
менте на уровне
NISK
гибелью
белка
ингибитор
апоптоза 2
Q9VWS5
IELIPNR; ILGISSPR
2
Dmel/CG15040
неохарактер
неизвестно
существование
изованный
белка предсказано
белок
Q9VJ77
SSFQDFTGGDVDYR;
2
Dmel/CG15160
FI24106p1
ядро
доказано в экспери
ASSTAAPIEYNELLHTNPAAL
менте на уровне
GAEDVDHR
транскрипта
Q9W0D6
ANDLGELFSLEHLR;
2
Dmel/CG7879
CG7879,
ядро
доказано в экспери
VVAITPQEIADR
изоформа A
менте на уровне
белка
O76861
NSDLASEAQVEK; FVPSTLR
2
EG:100G10.1
CG2685
ядро
доказано в экспери
менте на уровне
транскрипта
Q9VCU6
GLELAEIVGATDVTGDIK;
2
HP1c
белок гете
ядро
доказано в экспери
GVPEELR
рохромати
менте на уровне
на 1c
белка
D0IQL4
QPTSNENAQHSPK;
2
NimA
MIP14095p
плазмати
существование
NFHEPLPEPPVAFAVTPQSNE
ческая
белка предсказано
GITTR
мембрана
O96690
NSELINSLLSLPK;
2
Pdf
белок pdf
экскрети
доказано в экспери
YPLILENSLGPSVPIR
руется
менте на уровне
белка
Q9W2F3
FGIGASLAPR;
2
PTP<ER
протеиновая
цитоплазма
доказано в экспери
VLLESESSELTSLLGEIKR
тирозиновая
менте на уровне
фосфатаза
транскрипта
ERK
Q95TU2
RLETLEDTNLQLASDTYSAEE
2
SART1
CG6686,
ядро
доказано в
LAK; VEVEDDDLER
изоформа A
эксперименте на
уровне белка
* Клеточная локализация не указана в базе знаний Uniprot, предсказана нами по анализу содержания баз данных и науч
ной литературы.
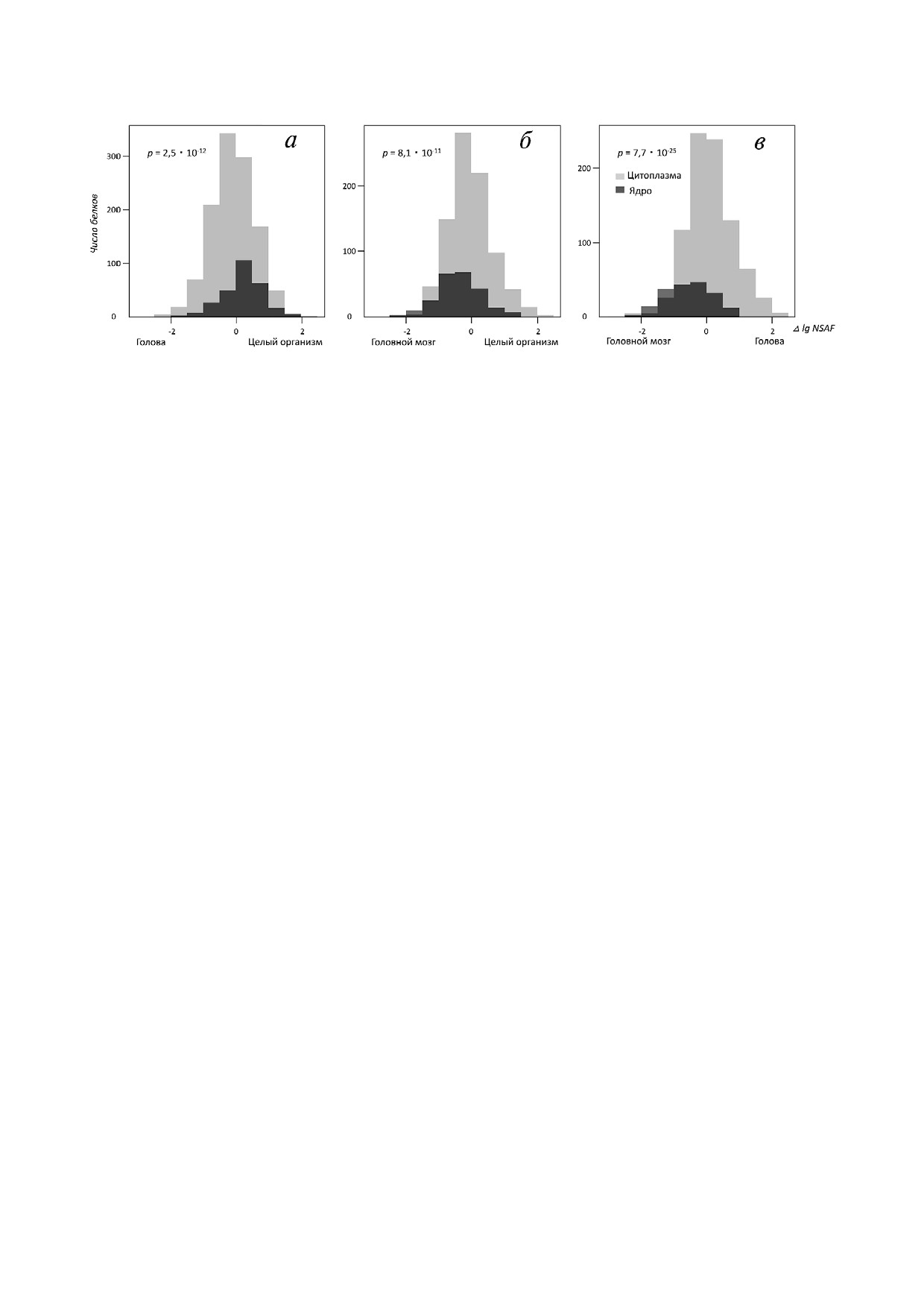
по относительному количеству, гистоны ока
ядерной и цитоплазматической локализацией
жутся значительно выше в списке, чем в других
по системе Gene Ontology [14]. Вопрос: изменя
двух протеомах. Это свидетельствует о количе
ются ли определенные в условных величинах,
ственном обогащении ядерными белками про
по масс спектрометрическому сигналу, преоб
теома головного мозга. Но как это доказать?
разованному в выбранный нами параметр NSAF
Мы выделили присутствующие во всех трех
[13], концентрации ядерных и цитоплазмати
исследуемых протеомах - головного мозга, го
ческих белков относительно друг друга? Для от
ловы и целого организма насекомого - белки с
вета на него преобразованные в десятичные ло
8 БИОХИМИЯ том 84 вып. 2 2019
258
КУЗНЕЦОВА и др.
Рис. 2. Количественное обогащение протеома головного мозга Drosophila melanogaster белками с ядерной локализацией.
Гистограммы представляют белки с аннотацией Gene Ontology [14] «цитоплазма» или «ядро», где по оси абсцисс предс
тавлены разности десятичных логарифмов количественного параметра NSAF [13] одних и тех же белков в разных проте
омах (Δlg NSAF), а по оси ординат - число белков в каждом классе гистограммы. Смещение гистограмм друг относитель
но друга в направлении того или иного протеома означает обогащение одной из клеточных локализаций. Вероятность
достоверного отличия между концентрациями белков ядра относительно белков цитоплазмы в разных протеомах рассчи
тана при помощи теста Крускала-Уоллиса. а - Сравнение протеомов головы и целого организма; б - cравнение протео
мов мозга и целого организма; в - cравнение протеомов мозга и головы
гарифмы значения нормализованного фактора
белками ядерной локализации. Поэтому при про
спектрального обилия белков в исследуемых
теомном анализе с особым интересом к ядерным
протеомах вычитали друг из друга. Значения
регуляторам, например, факторам транскрип
разности для цитоплазматических и ядерных
ции, можно рекомендовать исследовать именно
белков формировали гистограммы (рис. 2), сме
выделенные препараты головного мозга, а не го
щение которых относительно друг друга визуа
ловы насекомых, как это иногда делается.
лизировало различия, достоверность которых
можно было оценить тестом Крускала-Уоллиса
[19]. Как видно из рис. 2, ядерные белки в про
Финансирование
теоме целого организма находились в более вы
соких концентрациях, чем в протеоме головы,
Работа выполнена при финансовой поддержке
если в качестве группы сравнения использовать
Российского научного фонда (грант №17 15
цитоплазматические белки. В свою очередь, по
01229).
лученный нами протеом головного мозга, как и
ожидалось по умозрительной оценке, был коли
Благодарности
чественно обогащен белками ядерной локализа
ции, по сравнению с двумя другими протеома
Авторы признательны к.б.н. Н.И. Романовой
ми. Более низкая концентрация ядерных белков
(кафедра генетики биологического факультета
в протеоме головы может объясняться наличием
МГУ им. М.В. Ломоносова) за предоставление
большого количества ткани глаза с объемными
живых плодовых мушек.
клетками глазков омматидиев с меньшим коли
чеством вещества ядра.
Конфликт интересов
Впервые проанализированный протеом го
У авторов отсутствует конфликт интересов в
ловного мозга плодовой мушки позволил иден
финансовой и в какой либо иной сфере.
тифицировать небольшое количество генных
продуктов, ранее не известных на белковом
Соблюдение этических норм
уровне. В целом, как и ожидалось, он оказался
подмножеством опубликованных ранее протео
Все применимые международные, нацио
мов целого организма насекомого и его головы.
нальные и/или институциональные принципы
Примечательно, что в количественном отноше
ухода и использования животных были соблю
нии протеом головного мозга оказался обогащен
дены.
БИОХИМИЯ том 84 вып. 2 2019
ПРОТЕОМ ГОЛОВНОГО МОЗГА Drosophila melanogaster
259
СПИСОК ЛИТЕРАТУРЫ
1.
Pandey, U.B., and Nichols, C.D. (2011) Human disease
Washburn, M.P. (2006) Quantitative proteomic analysis of
models in Drosophila melanogaster and the role of the fly in
distinct mammalian Mediator complexes using normalized
therapeutic drug discovery, Pharmacol. Rev., 63, 411-436.
spectral abundance factors, Proc. Natl. Acad. Sci. USA,
2.
Vierstraete, E., Cerstiaens, A., Baggerman, G., van den
103, 18928-18933.
Bergh, G., De Loof, A., and Schoofs, L.
(2003)
14.
Blake, J.A., Dolan, M., Drabkin, H., Hill, D.P., Ni, L.,
Proteomics in Drosophila melanogaster: first 2D database of
Sitnikov, D., Bridges, S., Burgess, S., Buza, T., McCar
larval hemolymph proteins, Biochem. Biophys. Res.
thy, F., Peddinti, D., Pillai, L., Carbon, S., Dietze, H.,
Commun., 304, 831-838.
Ireland, A., Lewis, S.E., Mungall, C.J., Gaudet, P.,
3.
Scigelova, M., and Makarov, A. (2006) Orbitrap mass ana
Chisholm, R.L., Fey, P., Kibbe, W.A., Basu, S., Sie
lyzer—overview and applications in proteomics, Proteo<
gele, D.A., McIntosh, B.K., Renfro, D.P., Zweifel, A.E.,
mics, 6 Suppl. 2, 16-21.
Hu, J.C., Brown, N.H., Tweedie, S., Alam Faruque, Y.,
4.
Tyanova, S., Albrechtsen, R., Kronqvist, P., Cox, J.,
Apweiler, R., Auchinchloss, A., Axelsen, K., Bely, B.,
Mann, M., and Geiger, T. (2016) Proteomic maps of breast
Blatter, M.C., Bonilla, C., Bougueleret, L., Boutet, E.,
cancer subtypes, Nat. Commun., 7, 10259.
Breuza, L., Bridge, A., Chan, W.M., Chavali, G., Cou
5.
Vizcaino, J.A., Cote, R.G., Csordas, A., Dianes, J.A.,
dert, E., Dimmer, E., Estreicher, A., Famiglietti, L.,
Fabregat, A., Foster, J.M., Griss, J., Alpi, E., Birim, M.,
Feuermann, M., Gos, A., Gruaz Gumowski, N., Hieta, R.,
Contell, J., O’Kelly, G., Schoenegger, A., Ovelleiro, D.,
Hinz, U., Hulo, C., Huntley, R., James, J., Jungo, F.,
Perez Riverol, Y., Reisinger, F., Rios, D., Wang, R., and
Keller, G., Laiho, K., Legge, D., Lemercier, P., Lieber
Hermjakob, H. (2013) The PRoteomics IDEntifications
herr, D., Magrane, M., Martin, M. J., Masson, P.,
(PRIDE) database and associated tools: status in 2013,
Mutowo Muellenet, P., O’Donovan, C., Pedruzzi, I.,
Nucleic Acids Res., 41, D1063-D1069.
Pichler, K., Poggioli, D., Porras Millan, P., Poux, S.,
6.
Xing, X., Zhang, C., Li, N., Zhai, L., Zhu, Y., Yang, X.,
Rivoire, C., Roechert, B., Sawford, T., Schneider, M.,
and Xu, P. (2014) Qualitative and quantitative analysis of
Stutz, A., Sundaram, S., Tognolli, M., Xenarios, I.,
the adult Drosophila melanogaster proteome, Proteomics,
Foulger, R., Lomax, J., Roncaglia, P., Khodiyar, V.K.,
14, 286-290.
Lovering, R.C., Talmud, P.J., Chibucos, M., Gwinn
7.
Aradska, J., Bulat, T., Sialana, F.J., Birner Gruenber
Giglio, M., Chang, H.Y., Hunter, S., McAnulla, C.,
ger, R., Erich, B., and Lubec, G. (2015) Gel free mass
Mitchell, A., Sangrador, A., Stephan, R., Harris, M.A.,
spectrometry analysis of Drosophila melanogaster heads,
Oliver, S.G., Rutherford, K., Wood, V., Bahler, J., Lock, A.,
Proteomics, 15, 3356-3360.
Kersey, P.J., McDowall, M. D., Staines, D.M., Dwi
8.
Casas Vila, N., Bluhm, A., Sayols, S., Dinges, N., Dejung, M.,
nell, M., Shimoyama, M., Laulederkind, S., Hayman, T.,
Altenhein, T., Kappei, D., Altenhein, B., Roignant, J.Y.,
Wang, S.J., Petri, V., Lowry, T., D’Eustachio, P.,
and Butter, F. (2017) The developmental proteome of
Matthews, L., Balakrishnan, R., Binkley, G., Cherry, J.M.,
Drosophila melanogaster, Genome Res., 27, 1273-1285.
Costanzo, M.C., Dwight, S.S., Engel, S.R., Fisk, D.G.,
9.
Kuznetsova, K.G., Ilina, I.Y., Chernobrovkin, A.L.,
Hitz, B.C., Hong, E.L., Karra, K., Miyasato, S.R., Nash, R.S.,
Novikova, S.E., Farafonova, T.E., Karpov, D.S., Ivanov, M.V.,
Park, J., Skrzypek, M.S., Weng, S., Wong, E.D., Berar
Voronko, O.E., Ilgisonis, E.V, Kliuchnikova, A.A.,
dini, T.Z., Li, D., Huala, E., Mi, H., Thomas, P.D., Chan, J.,
Zgoda, V.G., Zubarev, R.A., Gorshkov, M.V, and
Kishore, R., Sternberg, P., Van Auken, K., Howe, D., and
Moshkovskii, S.A. (2018) Proteogenomics of adenosine
Westerfield, M. (2013) Gene ontology annotations and
to inosine RNA editing in fruit fly, J. Proteome Res., 17,
resources, Nucleic Acids Res., 41, D530-D535.
3889-3903.
15.
Gaudet, P., Michel, P.A., Zahn Zabal, M., Britan, A.,
10.
Vizcaino, J., Deutsch, E.E. W., Wang, R., Vizcaino, J.A.,
Cusin, I., Domagalski, M., Duek, P.D., Gateau, A.,
Deutsch, E.E. W., Wang, R., Csordas, A., Reisinger, F.,
Gleizes, A., Hinard, V., Rech de Laval, V., Lin, J., Nikitin, F.,
Rios, D., Dianes, J.A., Sun, Z., Farrah, T., Bandeira, N.,
Schaeffer, M., Teixeira, D., Lane, L., and Bairoch, A.
Binz, P.A., Xenarios, I., Eisenacher, M., Mayer, G., Gatto, L.,
(2017) The neXtProt knowledgebase on human proteins:
Campos, A., Chalkley, R.J., Kraus, H.J., Albar, J.P.,
2017 update, Nucleic Acids Res., 45, D177-D182.
Martinez Bartolome, S., Apweiler, R., Omenn, G.S.,
16.
Moshkovskii, S.A., Ivanov, M.V., Kuznetsova, K.G., and
Martens, L., Jones, A.R., and Hermjakob, H. (2014)
Gorshkov, M.V. (2018) Identification of single amino acid
ProteomeXchange provides globally coordinated pro
substitutions in proteogenomics, Biochemistry (Moscow),
teomics data submission and dissemination, Nat. Biotech.,
83, 250-258.
32, 223-226.
17.
Chernobrovkin, A.L., Kopylov, A.T., Zgoda, V.G., Moysa, AA.,
11.
Levitsky, L.I., Ivanov, M.V., Lobas, A.A., Bubis, J.A.,
Pyatnitskiy, M.A., Kuznetsova, K.G., Ilina, I.Y., Kar
Tarasova, I.A., Solovyeva, E.M., Pridatchenko, M.L., and
pova, M.A., Karpov, D.S., Veselovsky, A.V., Ivanov, M.V.,
Gorshkov, M.V. (2018) IdentiPy: an extensible search
Gorshkov, M.V., Archakov, A.I., and Moshkovskii, S.A.
engine for protein identification in shotgun proteomics,
(2015) Methionine to isothreonine conversion as a source
J. Proteome Res., 17, 2249-2255.
of false discovery identifications of genetically encoded
12.
Craig, R., and Beavis, R.C. (2003) A method for reducing
variants in proteogenomics, J. Proteomics, 120, 169-178.
the time required to match protein sequences with tandem
18.
UniProt Consortium (2015) UniProt: a hub for protein
mass spectra, Rapid Commun. Mass Spectrom.,
17,
information, Nucleic Acids Res., 43, D204-D212.
2310-2316.
19.
Kruskal, W., and Wallis, W.A. (1952) Use of ranks in one
13.
Paoletti, A.C., Parmely, T.J., Tomomori Sato, C., Sato, S.,
criterion variance analysis, J. Am. Stat. Assoc., 47,
Zhu, D., Conaway, R.C., Conaway, J.W., Florens, L., and
583-621.
БИОХИМИЯ том 84 вып. 2 2019
8*
260
КУЗНЕЦОВА и др.
BRAIN PROTEOME OF Drosophila melanogaster
IS ENRICHED WITH NUCLEAR PROTEINS
K. G. Kuznetsova1, M. V. Ivanov2, M. A. Pyatnitskiy1,3, L. I. Levitsky2,
I. Y. Ilina1, A. L. Chernobrovkin4, R. A. Zubarev4,5, M. V. Gorhskov2,
and S. A. Moshkovskii1,6*
1 Institute of Biomedical Chemistry, 119121 Moscow,
Russia; E<mail: smosh@mail.ru
2 Institute of Energy Problems of Chemical Physics, Russian
Academy of Sciences, 119334 Moscow, Russia
3 Higher School of Economics, 101000 Moscow, Russia
4 Karolinska Institutet, SE<171 77, Stockholm, Sweden
5 Sechenov First Moscow State Medical University
(Sechenov University), 119991 Moscow, Russia
6 Pirogov Russian National Research Medical University
(RNRMU), 117997 Moscow, Russia
Received August 30, 2018
Revision received October 10, 2018
Accepted October 10, 2018
The brain proteome of Drosophila melanogaster was characterized by liquid chromatography/high resolution mass
spectrometry and compared to the earlier characterized Drosophila whole body and head proteomes. Raw data for all
the proteomes were processed in a similar manner. Approximately 4000 proteins were identified in the brain proteome
that represented, as expected, the subsets of the head and body proteomes. However, after thorough data curation, we
reliably identified 24 proteins unique for the brain proteome; 13 of them have never been detected before at the pro
tein level. Fourteen of 24 identified proteins have been annotated as nuclear proteins. Comparison of three used
datasets by label free quantitation showed statistically significant enrichment of the brain proteome with nuclear pro
teins. Therefore, we recommend the use of isolated brain preparations in the studies of Drosophila nuclear proteins.
Keywords: proteomics, mass spectrometry, Drosophila melanogaster, brain, nuclear protein
БИОХИМИЯ том 84 вып. 2 2019