БИОХИМИЯ, 2019, том 84, вып. 3, с. 365 - 379
УДК 576.53
КЛОНАЛЬНЫЙ СОСТАВ МУЛЬТИПОТЕНТНЫХ
МЕЗЕНХИМНЫХ СТРОМАЛЬНЫХ КЛЕТОК ЧЕЛОВЕКА:
ПРИМЕНЕНИЕ ГЕНЕТИЧЕСКИХ ШТРИХ КОДОВ ДЛЯ
ИССЛЕДОВАНИЯ*
© 2019 А.Е. Бигильдеев1**, А.М. Пилунов2, Н.В. Сац1, В.Л. Сурин1,
И.Н. Шипунова1, Н.А. Петинати1, М.Д. Логачева3, А.В. Федотова3,
А.С. Касьянов4, А.С. Артюхов5, Э.Б. Дашинимаев6, Н.И. Дризе1
1 Национальный медицинский исследовательский центр
гематологии Минздрава России, 125167 Москва, Россия;
электронная почта: bigildeev.ae@gmail.com
2 Московский государственный университет им. М.В. Ломоносова,
119991 Москва, Россия
3 НИИ физико)химической биологии МГУ им. М.В. Ломоносова,
119992 Москва, Россия
4 Институт общей генетики им. Н.И. Вавилова РАН,
119991 Москва, Россия
5 РНИМУ им. Н.И. Пирогова Минздрава России,
117997 Москва, Россия
6 Институт биологии развития им. Н.К. Кольцова РАН,
119334 Москва, Россия
Поступила в редакцию 12.09.2018
После доработки 27.11.2018
Принята к публикации 27.11.2018
В работе изучали клональный состав мультипотентных мезенхимных стромальных клеток (ММСК) челове!
ка, маркированных с помощью лентивирусных векторов, несущих генетические штрих!коды. ММСК были
инфицированы клонированной библиотекой самоинактивирующихся лентивирусных векторов, несущих
667 неповторяющихся штрих!кодов. При каждом пассировании культуры 120 клеток клонировали в 96!лу!
ночных планшетах по одной клетке на лунку. Определяли эффективность клонирования и маркирования
клоногенных клеток. Из клеточных колоний!клонов выделяли ДНК и идентифицировали штрих!коды с
помощью секвенирования по методу Сэнгера. Из общей популяции ММСК каждого пассажа также выде!
ляли ДНК и определяли разнообразие, а также представленность штрих!кодов с помощью метода глубоко!
го секвенирования на платформе Illumina. Было показано, что доля маркированных лентивирусными век!
торами ММСК стабильна в пассажах. Процедура маркирования при высоких значениях множественности
заражения может приводить к снижению пролиферации ММСК. Анализ штрих!кодов в отдельных клеточ!
ных клонах подтвердил поликлональность популяции ММСК. При пассировании культуры ее клональный
состав значительно менялся вследствие истощения пролиферативного потенциала большинства клеток.
Большие клоны были обнаружены на первом пассаже ММСК, далее в популяции выявлялось множество
небольших клонов с ограниченным пролиферативным потенциалом. Результаты глубокого секвенирования
подтвердили данные об изменении клонального состава ММСК. В поликлональной популяции ММСК со!
держится лишь небольшое количество клеток с высоким пролиферативным потенциалом, среди которых
могут быть и стволовые. ММСК с высоким пролиферативным потенциалом чаще выявляются на самых
ранних пассажах. В связи с этим для использования ММСК в подходах регенеративной медицины, опира!
ющихся на пролиферацию клеток, рекомендуется применять ММСК ранних пассажей.
КЛЮЧЕВЫЕ СЛОВА: мультипотентные мезенхимные стромальные клетки, генетическое маркирование,
штрих!код, лентивирусный вектор.
DOI: 10.1134/S0320972519030072
Принятые сокращения: СКК - стволовая кроветворная клетка; МСК - мезенхимная стволовая клетка; ММСК -
мультипотентные мезенхимные стромальные клетки; eGFP - зеленый флуоресцентный белок (enhanced green fluorescent
protein); NGS - высокопроизводительное параллельное секвенирование (next generation sequencing); ППС - полная пита!
тельная среда; ПЦР - полимеразная цепная реакция; CNV - количество провирусов в расчете на один диплоидный геном
(copy number variation); MOI - множественность заражения (multiplicity of infection).
* Приложение к статье на английском языке опубликовано на сайте журнала «Biochemistry» (Moscow) и на сайте издатель!
ства Springer (Link.springer.com), том 84, вып. 3, 2019.
** Адресат для корреспонденции.
365
366
БИГИЛЬДЕЕВ и др.
В костном мозге присутствуют два типа
клеток, которые предполагается маркировать [12].
стволовых клеток: стволовые кроветворные
Определить маркеры можно как в отдельных
клетки (СКК) и мезенхимные стволовые клетки
клонах, так и в общей популяции маркированных
(МСК). Если СКК интенсивно изучают почти
клеток с помощью современных методов высо!
100 лет, то активные исследования МСК нача!
копроизводительного параллельного секвени!
лись в последней четверти ХХ века. Впервые на!
рования (next generation sequencing, NGS) [13]. Со!
личие МСК было описано А.Я. Фриденштей!
четание методов генетического штрих!кодиро!
ном [1], который называл их стромальными ме!
вания и NGS позволило решить многие иссле!
ханоцитами. Термин МСК был введен А. Капла!
довательские задачи, в частности, определить
ном для обозначения прилипающих к пластику
клональный состав и его динамику в кроветвор!
стромальных клеток человека, способных к
ной системе [11, 14-18], в популяции стволовых
дифференцировке in vitro в костную, хрящевую
клеток молочной железы [19] и других клеточ!
и жировую ткани [2]. В 2005 г. Общество по кле!
ных популяциях [20]. Однако маркирование ме!
точной терапии сделало заявление, что эти клет!
зенхимных стволовых клеток и ММСК костно!
ки не подходят под определение стволовых, т.к.
го мозга с помощью штрих!кодов до настоящего
обладают ограниченным пролиферативным по!
времени не проводилось. Целью данной работы
тенциалом [3]. Было предложено называть их
являлось установление клонального состава по!
мультипотентными мезенхимными стромаль!
пуляции ММСК человека с помощью маркиро!
ными клетками (ММСК), а также был разрабо!
вания клеток библиотекой лентивирусных век!
тан четкий критерий клеток, подходящих под
торов с генетическими штрих!кодами.
это определение [4]. Необходимость такого кри!
терия была связана с тем, что эти клетки стали
активно использовать для лечения различных
МЕТОДЫ ИССЛЕДОВАНИЯ
заболеваний, а также в регенеративной медици!
не [5, 6]. Применение этих клеток часто опира!
Получение клонированной библиотеки генети
ется на их способность к пролиферации и диф!
ческих штрих кодов. Библиотека плазмид LeGO!
ференцировке. Оказалось, что популяция ММСК
nude!BC32 была любезно предоставлена проф. Бо!
гетерогенна и представлена множеством клеточ!
рисом Фезе и Керстин Корнилс (University Me!
ных клонов, различающихся по своим свой!
dical Center Hamburg!Eppendorf, Гамбург, Герма!
ствам [7-9]. Результаты этих работ имеют об!
ния). Библиотека была получена на основе плаз!
щий недостаток: исследованные клетки не были
миды системы лентивирусных векторов 3!го по!
маркированы, что не позволяло сделать вывод о
коления LeGO [21], в которую вместо гена, ко!
клональном составе популяции ММСК. Нашей
дирующего флуоресцентный белок, были клони!
группой была предпринята попытка индивиду!
рованы последовательности генетических
ально маркировать ММСК с помощью лентиви!
штрих!кодов вида NNNACTNNCGANNCTTN!
русного вектора, содержащего зеленый флуо!
NCGANNCTTNNGGANNCTANNACTNNC!
ресцентный белок (eGFP). Была установлена ге!
GANNCTTNNCGANNCTTNNGGANNCTAN!
терогенность популяции по пролиферативному
NACTNNCGANNCTC, где N - вырожденные
потенциалу и ее поликлональность [10]. Однако
нуклеотиды. Штрих!коды содержат 35 вырож!
эта работа также имела недостатки: проанализи!
денных нуклеотидов и 48 константных. Теорети!
ровано небольшое число клонов, а также не бы!
ческое разнообразие комбинаций вырожденных
ло проведено исследование общей популяции
нуклеотидов в таких штрих!кодах составляет 435,
ММСК.
или ≈1019. Нуклеотидный состав и структура штрих!
Несколько лет назад было предложено мар!
кодов подробно охарактеризованы в работе Ara!
кировать СКК с помощью библиотеки ретрови!
nyossy et al. [22]. Компетентные клетки E. coli штам!
русных векторов, содержащей большое разно!
ма XL1 трансформировали библиотекой плаз!
образие коротких нуклеотидных последователь!
мид методом теплового шока в стандартных ус!
ностей, так называемых «генетических штрих!
ловиях согласно руководству Maniatis et al. [23].
кодов» [11]. После интеграции провируса каж!
Отдельные колонии трансформантов получали
дая клетка исследуемой популяции приобретает
на чашках Петри с 1,5%!ным LB!агаром и ампи!
уникальный, стабильный и наследуемый мар!
циллином («Синтез», Россия) в концентрации
кер, позволяющий проследить за судьбой потом!
100 мкг/мл после инкубации при 37 °С в течение
ства маркированной клетки и отличать его от
17 ч. Из индивидуальных колоний выделяли ДНК
потомства других меченых стволовых клеток.
и определяли трансформанты, несущие вставку.
Важный аспект такого маркирования - разно!
Из ночных культур трансформантов объемом
образие штрих!кодов в библиотеке должно мно!
2 мл выделяли плазмидную ДНК и определяли
гократно превосходить количество стволовых
нуклеотидную последовательность штрих!кода
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
367
с помощью секвенирования по Сэнгеру (прай!
ной среде с 1% бычьего сывороточного альбумина
мер для секвенирования - BC!PCR!Seq, табл. 1).
(БСА) пятой фракции («Sigma», США), получая
Для создания библиотеки использовали штрих!
вирусный супернатант. Параллельно, в тех же усло!
коды, соответствующие следующим критериям:
виях и с помощью тех же вспомогательных плаз!
1) штрих!коды, не содержащие мутаций в конс!
мид, получали супернатант лентивирусов, несущих
тантной области; 2) штрих!коды, имеющие в
ген eGFP вместо генетических штрих!кодов.
константной части только замены; 3) штрих!коды,
Определение вирусного титра. Часть объема
содержащие не более одной инсерции в констант!
вирусного супернатанта, содержавшего вирио!
ную или вариабельную часть, а также имеющие
ны с библиотекой штрих!кодов, отбирали и тит!
делецию, включающую не более четырех вариа!
ровали, разбавляя полной питательной средой
бельных нуклеотидов подряд; 4) штрих!коды,
(ППС), содержавшей αMEM с 10% эмбриональ!
вариабельная часть которых отличается по
ной телячьей сыворотки Hyclone («GE Life
меньшей мере по 10 позициям. Отобранные
Sciences», США), 2 мМ L!глутамина («ICN»,
плазмиды с уникальными штрих!кодами сме!
Швейцария), 100 ед/мл пенициллина («Ферейн»,
шивали в эквимолярном соотношении, что поз!
Россия), 50 мкг/мл стрептомицина («Ферейн»,
волило получить полностью охарактеризован!
Россия) с полибреном в концентрации 8 мкг/мл
ную библиотеку штрих!кодов. Далее произво!
(«Sigma», США). Было сделано шесть разведений
дили очистку смешанной библиотеки от бакте!
с шагом 1 : 10. Титрованный вирусный суперна!
риального эндотоксина на колонке EndoFree
тант добавляли к клеткам линии Phoenix!GP, из
DNA Purification Kit («Qiagen», США) согласно
которых выделяли ДНК. Эту ДНК использовали
инструкциям фирмы!производителя. Целевую
для определения вирусного титра методом ПЦР
ДНК после переосаждения в изопропаноле раст!
в реальном времени. Титр определяли по калиб!
воряли в стерильном TE!буфере без эндотокси!
ровочной кривой, которую строили по шести
на из набора и использовали для получения ви!
разведениям библиотеки плазмид со штрих!ко!
русного супернатанта.
дами в диапазоне концентраций (5 × 107)…(5 × 102)
Получение вирусного супернатанта. На клет!
копий/мкл. В каждую реакционную смесь до!
ках линии Phoenix!GP проводили трансфекцию
бавляли геномную ДНК неинфицированных
стандартным методом преципитации с хлоридом
клеток линии Phoenix!GP для того, чтобы урав!
кальция [24]. К клеткам добавляли смесь плаз!
нять эффективность реакции амплификации с
мид phCMVC!VSV!G (R861), pMDLg/pRRE,
плазмидами и ДНК, выделенной из инфициро!
pRSV!Rev, кодирующих структурные компонен!
ванных эукариотических клеток. Праймеры
ты лентивируса, а также полученную библиоте!
WPRE!F и WPRE!R и флуоресцентный зонд
ку со штрих!кодами. Через 24 ч среду отбирали,
WPRE!probe были комплементарны участку
центрифугировали 10 мин при 1500 об/мин и
WPRE, который содержали плазмиды, несущие
пропускали через фильтр с размером ячеек
штрих!коды (табл. 1). Реакцию проводили в
0,45 мкм. Затем его центрифугировали в течение
приборе Applied Biosystems ABI PRISM 7500
3,5 ч при 18 000 об/мин. Осадок, содержавший
(«Thermo Fischer Scientific», США). По калибро!
вирусные частицы, растворяли в бессывороточ!
вочной кривой определяли количество прови!
Таблица 1. Праймеры и зонды, использованные в работе
Название
Последовательности праймеров (5'-3')
BC!PCR!Seq
CTGAATGATACGGCGACCAC
WPRE!F
CCTTGTATAAATCCTGGTTGCTGTCT
WPRE!R
GGAAAGGAGCTGACAGGTGGT
WPRE!probe
R6G!TCAGGCAACGTGGCGTGGTGTG!BHQ1
ddPCR!BC!F
CATCGATACCGTCGACCTC
ddPCR!BC!R
GCGTCGTGTAGGGAAAGAG
ddPCR!BC!probe
R6G!TGCTGATTGTGCCTGGCTAGAAG!BHQ1
ddPCR!ACTB!F
GGCTTCTTGTCCTTTCCTTC
ddPCR!ACTB!R
CATGTCGTCCCAGTTGGTG
ddPCR!ACTB!probe
FAM!CCTGAAGTACCCCATCGAGCAC!RTQ1
BC!PCR!R
TCGTTGGGAGTGAATTAGCC
BC!PCR!F!nested
CACGACGCTCTTCCGATCTN
BC!PCR!R!nested
GCTCTTCCGATCTCTCGAGN
БИОХИМИЯ том 84 вып. 3 2019
368
БИГИЛЬДЕЕВ и др.
русов в зараженных клетках, вычисляли вирус!
Определение среднего количества провирусов
ный титр в инфекционных частицах на 1 мл ви!
на один диплоидный геном. CNV определяли с
русного супернатанта. Вирусный титр суперна!
помощью цифровой эмульсионной ПЦР. Для
танта с eGFP определяли с помощью проточной
этого собирали реакционную смесь, содержав!
цитофлуориметрии, заражая клетки Phoenix!GP
шую 3 мкл ДНК, разведенной до концентрации
лимитирующими разведениями супернатанта.
2-20 мкг/мл,
11 мкл
2× смеси реагентов
Получение и маркирование ММСК. ММСК бы!
ddPCR™ Supermix for Probes («Bio!Rad», США),
ли получены от шести здоровых доноров (4 муж!
450 нМ каждого из праймеров, 250 нМ каждого
чины и 2 женщины в возрасте 20-47 лет, медиа!
из зондов («Синтол», Россия), деионизованной
на 22 года). Костный мозг забирали во время
воды до 22 мкл. Каждая реакционная смесь со!
эксфузии в отделении высокодозной химиоте!
держала две пары праймеров и два зонда. Первая
рапии и трансплантации костного мозга ФГБУ
пара праймеров (ddPCR!BC!F и ddPCR!BC!R)
«НМИЦ гематологии» МЗ РФ после подписа!
и соответствующий ей зонд (ddPCR!BC!probe)
ния донорами информированного согласия.
были комплементарны последовательности век!
ММСК получали из 8-10 мл костного мозга,
тора (табл. 1). Вторая пара праймеров (ddPCR!
как описано в предыдущих работах [25]. По дос!
ACTB!F и ddPCR!ACTB!R) и зонд (ddPCR!
тижении ММСК конфлуентности их пассирова!
ACTB!probe) были комплементарны последова!
ли по 105 клеток во флакон Т25. Через сутки
тельности гена β!актина человека (табл. 1). Вод!
ППС заменяли на 5 мл свежей ППС, содержав!
но!капельная эмульсия была сгенерирована из
шей 100 мкл концентрата вирусного супернатан!
каждой реакционной смеси с помощью Automa!
та и 4 мкг/мл полибрена («Sigma», США). Инку!
ted Droplet Generator («Bio!Rad», США). После
бировали 6 ч, после чего полностью меняли сре!
этого следовала ПЦР на приборе C1000 Touch™
ду на свежую ППС. Эффективность маркирова!
Thermal Cycler («Bio!Rad», США) со следующими
ния, определенная в параллельном эксперимен!
параметрами: 95 °С - 5 мин; 40 циклов: 95 °С -
те с помощью лентивируса, содержавшего eGFP,
30 с, 60 °С - 1 мин; охлаждение и инкубация при
составила (27 ± 6)%.
4 °С - 5 мин; запекание капель при 90 °С - 5 мин.
Клонирование и пассирование ММСК. Марки!
Количество положительных капель в каждом
рованные ММСК растили до достижения кон!
образце после ПЦР было подсчитано с по!
флуентного монослоя. Клетки промывали раство!
мощью ридера капель QX200 AutoDG Droplet
ром Версена, а затем обрабатывали 0,025%!ным
Digital PCR System («Bio!Rad», США). CNV и
раствором трипсина («MP Biomedicals», США).
абсолютную концентрацию мишеней определяли
Концентрацию клеток в одноклеточной суспен!
с помощью программного обеспечения Quanta!
зии определяли в камере Горяева. На следую!
Soft v.1.7.4 («Bio!Rad», США).
щий пассаж переносили по 105 ММСК. Уровень
Анализ штрих кодов в клонах ММСК. Из лу!
удвоения популяции ММСК определяли по ме!
нок, содержавших клеточные колонии, выде!
тодике, описанной ранее [26]. На каждом пасса!
ляли ДНК, которую использовали как матрицу
же с помощью последовательных разведений
для ПЦР. В качестве праймеров применяли
получали суспензию, содержавшую ~1 клетку на
олигонуклеотиды ddPCR!BC!F и BC!PCR!R,
200 мкл среды. Суспензию разносили на 60 ячеек
комплементарные областям провируса, флан!
двух 96!луночных планшетов из расчета 1 клет!
кирующим штрих!код (табл. 1). Результаты
ка (200 мкл) на лунку (клонирование). Клетки
анализировали с помощью гель!электрофореза
культивировали в среде α!MEM с 20% эмбрио!
в 1,5%!ном агарозном геле. Полоску геля с
нальной телячьей сыворотки с основным фак!
ПЦР!продуктом расчетной длины вырезали
тором роста фибробластов (любезно предостав!
скальпелем и очищали на колонке Wizard SV
лен к.б.н. М.Э. Гаспарян, лаборатория инжене!
Gel and PCR Clean!Up System («Promega»,
рии белка, ИБХ им. академиков М.М. Шемяки!
США) согласно инструкции фирмы!произво!
на и Ю.А. Овчинникова РАН [27]) в концентра!
дителя. Нуклеотидную последовательность
ции 5 нг/мл в течение 14 дней. Эффективность
очищенного ПЦР!продукта определяли с по!
клонирования определяли с помощью распре!
мощью секвенирования по Сэнгеру. Если по!
деления Пуассона [28]. Из оставшихся после
лученная последовательность содержала не!
пассирования и клонирования клеток выделяли
сколько штрих!кодов, которые невозможно
ДНК. Ее использовали для определения среднего
было различить, то исходную ДНК клонов пов!
количества провирусов в расчете на один дипло!
торно анализировали с помощью двухстадий!
идный геном (copy number variation, CNV) с по!
ной ПЦР. В этом случае на первой стадии ис!
мощью цифровой эмульсионной ПЦР («Bio!Rad»,
пользовали те же праймеры, что и при односта!
США), а также для определения штрих!кодов в
дийной ПЦР, а на второй стадии один из прай!
общей клеточной популяции с помощью NGS.
меров был сдвинут внутрь вплотную к последо!
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
369
вательности штрих!кода («полугнездовая ПЦР»).
Reagent Kit v.3 (150 циклов) в режиме парных
Для второй стадии реакции продукт первой
чтений. Для прочтения индексов использовали
стадии разводили в 250 раз деионизованной во!
отдельное прочтение. Длина смыслового проч!
дой и брали 2 мкл на реакционную смесь. Гото!
тения составляла 80 нуклеотидов. Перевод ис!
вили четыре варианта реакционной смеси, со!
ходных данных в формат fastq проводили с по!
державшие прямой праймер BC!PCR!F!nested
мощью программы bcl2fastq2 («Illumina», США).
(табл. 1), где N - один из четырех нуклеотидов,
Число прочтений на каждый образец (глубина
комплементарный первому нуклеотиду штрих!
секвенирования) приведено в табл. S1 (см. При!
кода, а обратный праймер BC!PCR!R - тот же,
ложение).
что и в первой стадии ПЦР (или наоборот: пря!
Скрипты, использованные в ходе дальнейшей
мой праймер ddPCR!BC!F - тот же, что в пер!
биоинформатической обработки, доступны по
вой стадии, а обратный праймер BC!PCR!R!
адресу: http://makarich.fbb.msu.ru/kasianov/Bigildeev/.
nested сдвинут в сторону штрих!кода, послед!
На первом этапе производили соотнесение
ний нуклеотид на 3'!конце комплементарен
прочитанных индексов образцам ДНК. Далее
последнему вариабельному нуклеотиду штрих!
проводили кластеризацию смысловых чтений
кода). Результаты анализировали аналогично
по штрих!кодам. Для этих целей применяли
одностадийной ПЦР.
скрипт Cluster.perl. На вход он принимал набор
Анализ штрих кодов в суммарной популяции
чтений в файле формата fastq. Перечень исполь!
ММСК. Из общей клеточной популяции ММСК
зованных при кластеризации референсных
каждого пассажа выделяли ДНК и секвенирова!
штрих!кодов приведен в табл. S2 (см. Приложе!
ли совокупность штрих!кодов с помощью мето!
ние). Кластеризацию по штрих!кодам проводи!
да глубокого секвенирования на платформе
ли в трех режимах: точное совпадение подпосле!
Illumina. ДНК выделяли стандартным способом
довательности чтения с референсным штрих!
с осаждением этанолом [23]. По 50 нг выделен!
кодом, совпадение подпоследовательности чте!
ной ДНК было использовано для определения
ния с референсным штрих!кодом с учетом од!
последовательности штрих!кодов с помощью
ной замены, совпадение подпоследовательнос!
NGS на платформе Illumina MiSeq. Области
ти чтения с референсным штрих!кодом с учетом
ДНК, содержавшие штрих!код в составе инте!
шести замен. Количество прочтений всех клас!
грированного провируса, амплифицировали с
теризованных последовательностей суммирова!
помощью ПЦР. Для этого использовали готовую
ли для каждого референсного штрих!кода. Ре!
реакционную смесь NEBNext Ultra II («New
зультаты кластеризации собирали в одну табли!
England Biolabs, Inc.», США) с полимеразой Q5
цу для всех образцов с использованием скрипта
в своем составе (12,5 мкл на реакцию), универ!
ConcatBarco!desReadCountsFile.perl. Результаты
сальный (2,5 мкл на реакцию) и одиночные ин!
объединения приведены в табл. S3, S4 и S5 (см.
дексные праймеры (2,5 мкл на реакцию) из на!
Приложение). Для анализа представленности
боров NEBNext Multiplex Oligos for Illumina®
штрих!кодов в суммарной клеточной популя!
(«New England Biolabs, Inc.», США) с добавле!
ции ММСК использовались результаты класте!
нием раствора ДНК (7,5 мкл на реакцию). ПЦР
ризации с шестью заменами.
проводили на приборе MJ Mini («Bio!Rad»,
Зная количество клеток, ДНК которых была
США) по следующей программе: 98 °С - 30 с;
использована для секвенирования, и количест!
25 циклов: 98 °С - 10 с, 60 °С - 30 с, 72 °С - 30 с;
во классифицированных чтений в соответству!
72 °С - 5 мин. Затем продукты ПЦР очищали с
ющем образце, вычисляли долю чтений (вклад),
помощью магнитных частиц Ampure XP («Beck!
соответствующую одной клетке. Сравнивая с
man!Coulter», США) в соотношении 1,2 : 1 (объ!
этим значением вклад каждого штрих!кода, оп!
ем частиц к объему ДНК). Длину продуктов
ределяли, какому количеству клеток он соответ!
ПЦР определяли на приборе Bioanalyzer 2100
ствует. Условно все штрих!коды были разделены
(«Agilent», США) с помощью набора реагентов
на три категории: 1) вклад соответствует <8 клет!
DNA High Sensitivity Kit («Agilent», США). После
кам; 2) вклад соответствует 8-32 клеткам; 3) вклад
очистки концентрацию ДНК измеряли на флуо!
соответствует >32 клеткам.
риметре Qubit с использованием набора реаген!
Статистический анализ. Статистическую зна!
тов dsDNA HS Assay Kit («Thermo Fisher Scien!
чимость различий оценивали по критерию Ман!
tific», США) и объединяли в равных количест!
на-Уитни. Для сравнения кумулятивной кле!
вах. После объединения концентрацию образца
точной продукции маркированных и немарки!
измеряли, доводили до 2 нМ и далее проводили
рованных ММСК от тех же доноров использо!
подготовку образца к секвенированию согласно
вали тест Вилкоксона для сопряженных пар.
рекомендациям производителя прибора. Для
Различия между группами считали статистичес!
секвенирования использовали реагенты Miseq
ки значимыми при p < 0,05.
6 БИОХИМИЯ том 84 вып. 3 2019
370
БИГИЛЬДЕЕВ и др.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
равно 26 ± 3 при том, что общее количество ва!
риабельных нуклеотидов в штрих!коде состав!
Дизайн эксперимента. Общую схему экспери!
ляло 35. Такой набор гарантировал уникаль!
мента можно представить следующим образом
ность каждого штрих!кода и простоту его отли!
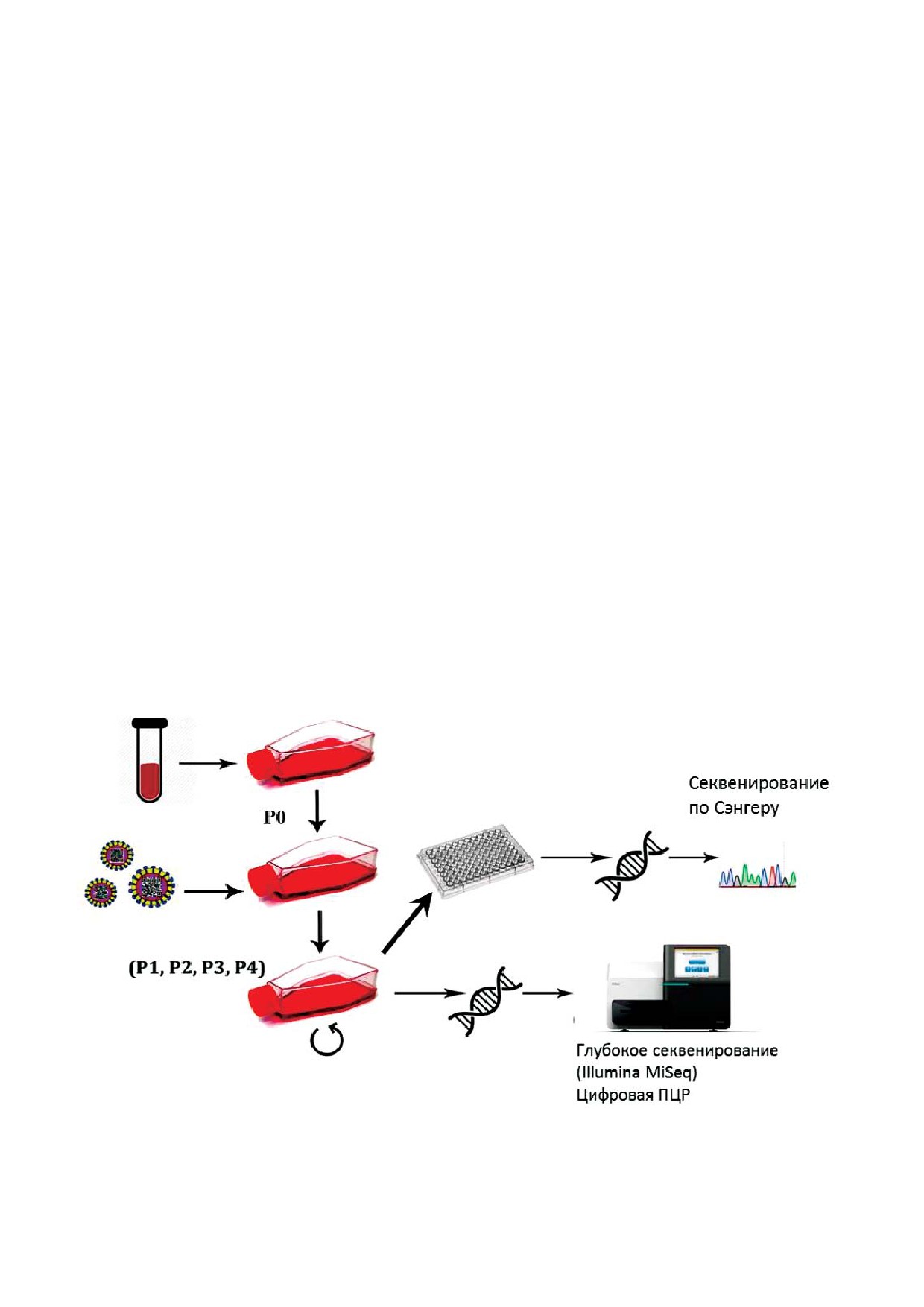
(рис. 1). В культуральный флакон добавляли ви!
чия от других при анализе. Плазмиды, содер!
русную библиотеку на следующие сутки после
жавшие отобранные штрих!коды, были смеша!
посадки 105 ММСК нулевого пассажа. Через 6 ч
ны в равной пропорции, так что каждый штрих!
среду полностью меняли и продолжали культи!
код в общей библиотеке имел одну и ту же час!
вировать ММСК до конфлуентности. При пас!
тоту встречаемости.
сировании культуры 120 клеток клонировали по
Определение вирусного титра и множествен
одной клетке на лунку 96!луночного планшета,
ности заражения. Титр вируса, оцененный с по!
105 клеток возвращали во флакон, из оставшихся
мощью ПЦР, составил (6,0 ± 2,7) × 106 инфекци!
клеток выделяли ДНК. Эту процедуру проводи!
онных частиц/мл. Параллельно в тех же услови!
ли на первом, втором, третьем и четвертом пас!
ях ММСК инфицировали тем же вектором, со!
сажах культуры каждого донора костного мозга.
державшим ген eGFP вместо штрих!кодов. В этом
Библиотека штрих кодов. Для построения
случае титр определяли методом проточной ци!
библиотеки плазмид со штрих!кодами было
тофлуориметрии. Титр вируса, определенный
проанализировано 1876 трансформантов E. coli.
этим способом, оказался равным (55 ± 11) × 106
Было отмечено, что 70% трансформантов содер!
инфекционных частиц/мл. Поскольку титр был
жали плазмиду со вставкой нужной длины.
определен двумя независимыми методами с раз!
Только такие колонии использовали для после!
личной чувствительностью, можно предпола!
дующих этапов создания библиотеки. У 867 кло!
гать, что истинный титр находился между двумя
нированных и выделенных плазмид была ус!
этими значениями.
пешно установлена нуклеотидная последова!
Опираясь на полученные значения вирус!
тельность штрих!кода. Таким образом, эффек!
ного титра, была вычислена множественность
тивность метода получения клонированных
заражения (multiplicity of infection, MOI). Для
плазмид из общей смеси составила 46%. На ос!
заражения культуры ММСК использовали
новании критериев качества и разнообразия,
100 мкл вирусного супернатанта с концентра!
описанных в разделе «Методы исследования»,
цией ~(6 × 106)…(55 × 106) инфекционных час!
было отобрано 667 плазмид для создания биб!
тиц/мл; следовательно, суммарно в культуру до!
лиотеки. Среднее расстояние Хэмминга между
бавили (6-55) × 105 инфекционных частиц. Ко!
штрих!кодами полученной библиотеки было
личество клеток в культуре составляло ~105.
Рис. 1. Схема эксперимента. Обозначения: P0-P4 - номера пассажей.
С цветным вариантом рис. 1 можно ознакомиться в электронной версии статьи на сайте: www.elibrary.ru
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
371
Следовательно, MOI находилась в диапазоне
40
6-55. Высокое значение MOI было подтвержде!
но цифровой капельной ПЦР, которую исполь!
35
зовали для определения среднего количества
провирусов в расчете на диплоидный геном
30
(CNV) в популяции маркированных ММСК на
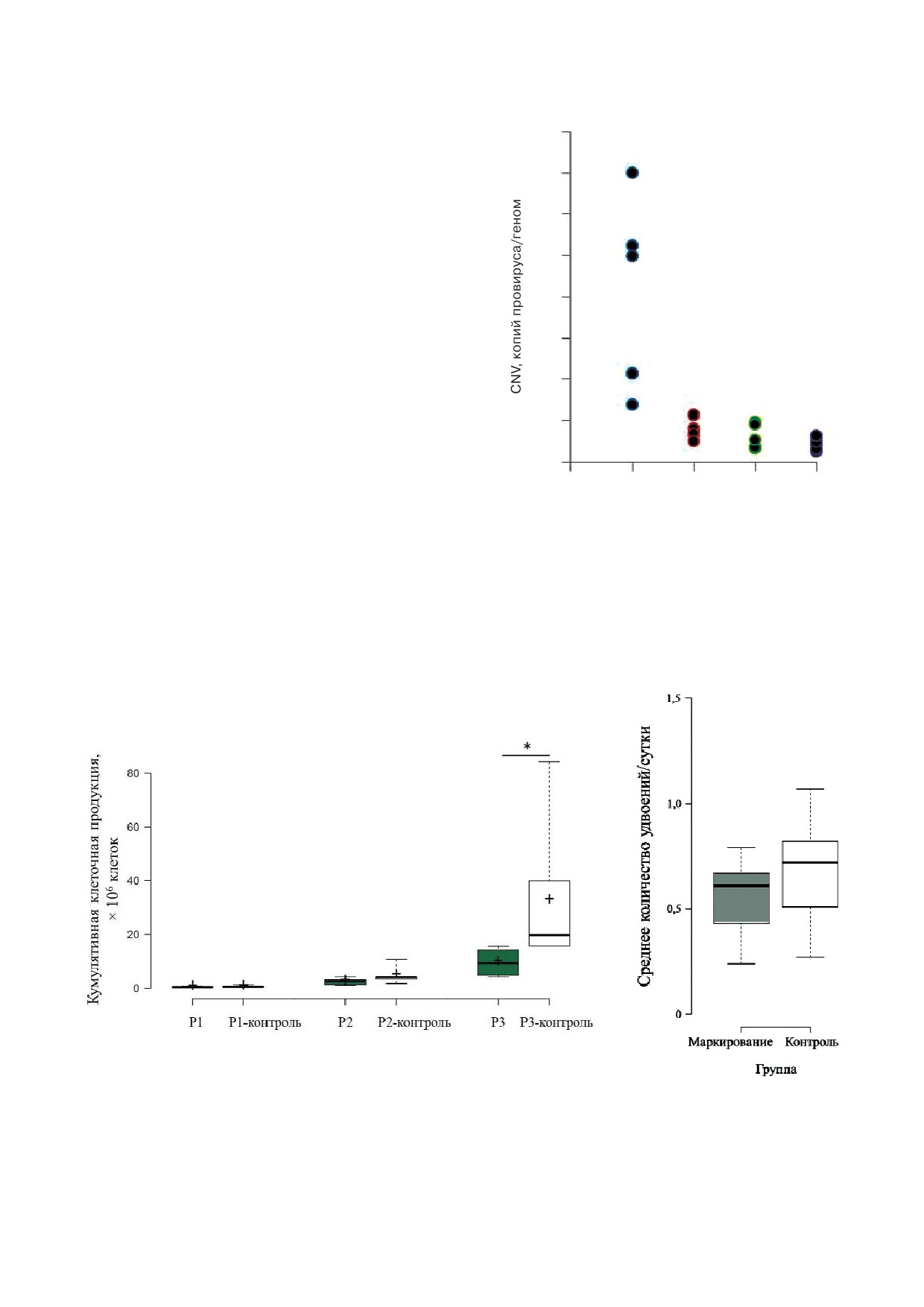
каждом пассаже культуры (рис. 2).
Было показано, что все исследованные в ра!
25
боте культуры содержат маркер. CNV умень!
шалось от первого пассажа (медиана 17,8) к чет!
20
вертому (медиана 2,2). По нашему предположе!
нию это обусловлено тем, что те клетки, в кото!
15
рых произошли множественные интеграции
провирусов, погибают или утрачивают способ!
10
ность к пролиферации и поэтому достаточно
быстро элиминируются из культуры, в отличие
от тех клеток, в которых количество интеграций
5
было небольшим.
Ростовые характеристики ММСК, эффектив
0
ность клонирования и маркирования. Процедура
1
2
3
4
маркирования ММСК оказывает влияние на их
способность к пролиферации, что выражается в
Пассаж
достоверном (p = 0,04) уменьшении суммарной
клеточной продукции к третьему пассажу по
Рис. 2. Динамика изменения среднего количества прови!
сравнению с контрольными ММСК (рис. 3, а).
русов в расчете на диплоидный геном (CNV).
Результат согласуется с ранее полученными дан!
С цветным вариантом рис. 2 можно ознакомиться в элект!
ными, указывающими на то, что стрессовое воз!
действие в виде вирусной трансдукции может
негативно сказываться на росте ММСК [29].
а
б
Рис. 3. Сравнение кумулятивной клеточной продукции (а) и среднего уровня пролиферации (б) маркированных и конт!
рольных ММСК на 1-3!ем пассажах. Указан разброс значений, медианы и межквартильный диапазон Q3-Q1. Плюсы
обозначают среднее значение. Звездочка указывает на статистически значимые различия по парному тесту Вилкоксона.
С цветным вариантом рис. 3 можно ознакомиться в электронной версии статьи на сайте: www.elibrary.ru
БИОХИМИЯ том 84 вып. 3 2019
6*
372
БИГИЛЬДЕЕВ и др.
При анализе среднего количества клеточных
удвоений отмечалась тенденция к снижению
этого параметра у маркированных ММСК, од!
нако различия были статистически незначимыми
в среднем для трех пассажей (рис. 3, б). Третий
пассаж был проанализирован отдельно, но здесь
также не наблюдали статистически значимых
различий, вероятно, в силу небольшой выборки
(данные не представлены). По мере пассирова!
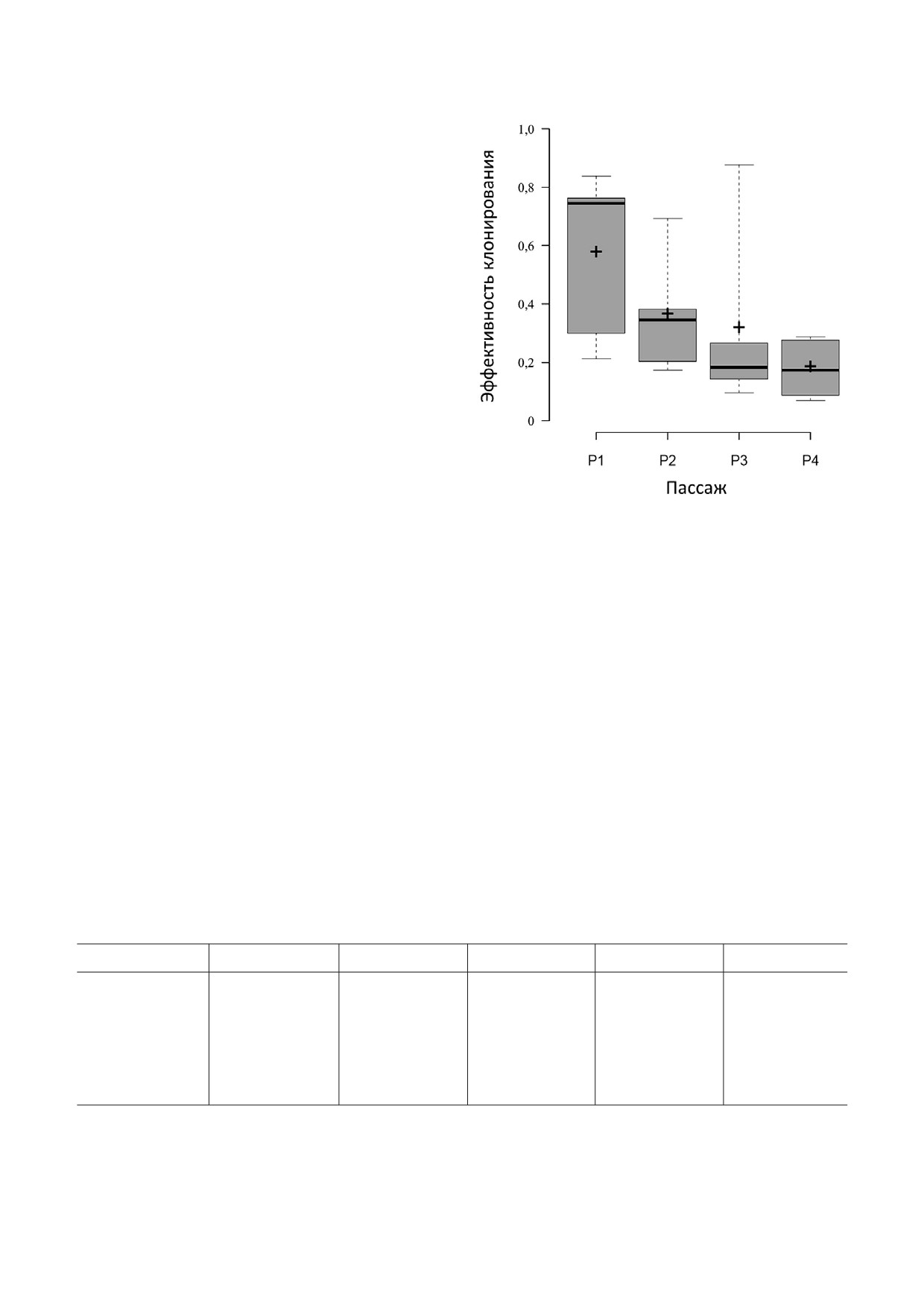
ния наблюдалось снижение эффективности
клонирования (рис. 4) маркированных ММСК
аналогично тому, как это происходило для не!
маркированных ММСК в предыдущих экспери!
ментах [29]. При этом пропорция маркирован!
ных клеток практически не изменялась по мере
пассирования (табл. 2).
Учитывая результаты, полученные по сред!
нему количеству провирусов в расчете на один
диплоидный геном, можно заключить, что про!
цедура переноса генетического маркера может
негативно сказываться на пролиферации кле!
точной культуры, но это влияние ограничивает!
Рис. 4. Эффективность клонирования маркированных
ся теми клетками, в ДНК которых интегрирова!
ММСК. Указан разброс значений, медианы и межквартиль!
ло значительное количество провирусов (более
ный диапазон Q3-Q1. Плюсы обозначают среднее значение
четырех).
Анализ штрих кодов в клонах ММСК. Про!
анализировано 458 маркированных клонов, в ко!
симость наблюдали при анализе штрих!кодов,
торых удалось выявить 381 уникальный штрих!
обнаруженных в ММСК каждого донора по от!
код из 667 штрих!кодов (57%), исходно присут!
дельности (данные не приведены).
ствовавших в библиотеке. Полученные резуль!
Три наиболее часто встречающихся штрих!
таты свидетельствуют о том, что разнообразие
кода (штрих!коды № 332, 611 и 968) были сум!
штрих!кодов в вирусном супернатанте меньше,
марно обнаружены по 13 раз среди всех исследо!
чем в исходной смеси плазмид. Вероятно, это
ванных ММСК. При анализе ММСК отдельных
объясняется стохастической природой процесса
доноров именно эти штрих!коды были наиболее
трансфекции клеток пакующей линии, процес!
часто встречающимися в ММСК каждого доно!
сов сборки вирионов и последующей трансдук!
ра. Такое распределение частоты встречаемости
ции ММСК. Более того, анализ частоты встреча!
может быть обусловлено несколькими причина!
емости штрих!кодов в клеточных колониях!кло!
ми. Например, оно может свидетельствовать о
нах ММСК от всех доноров показал, что неко!
том, что в вирусном супернатанте нарушена
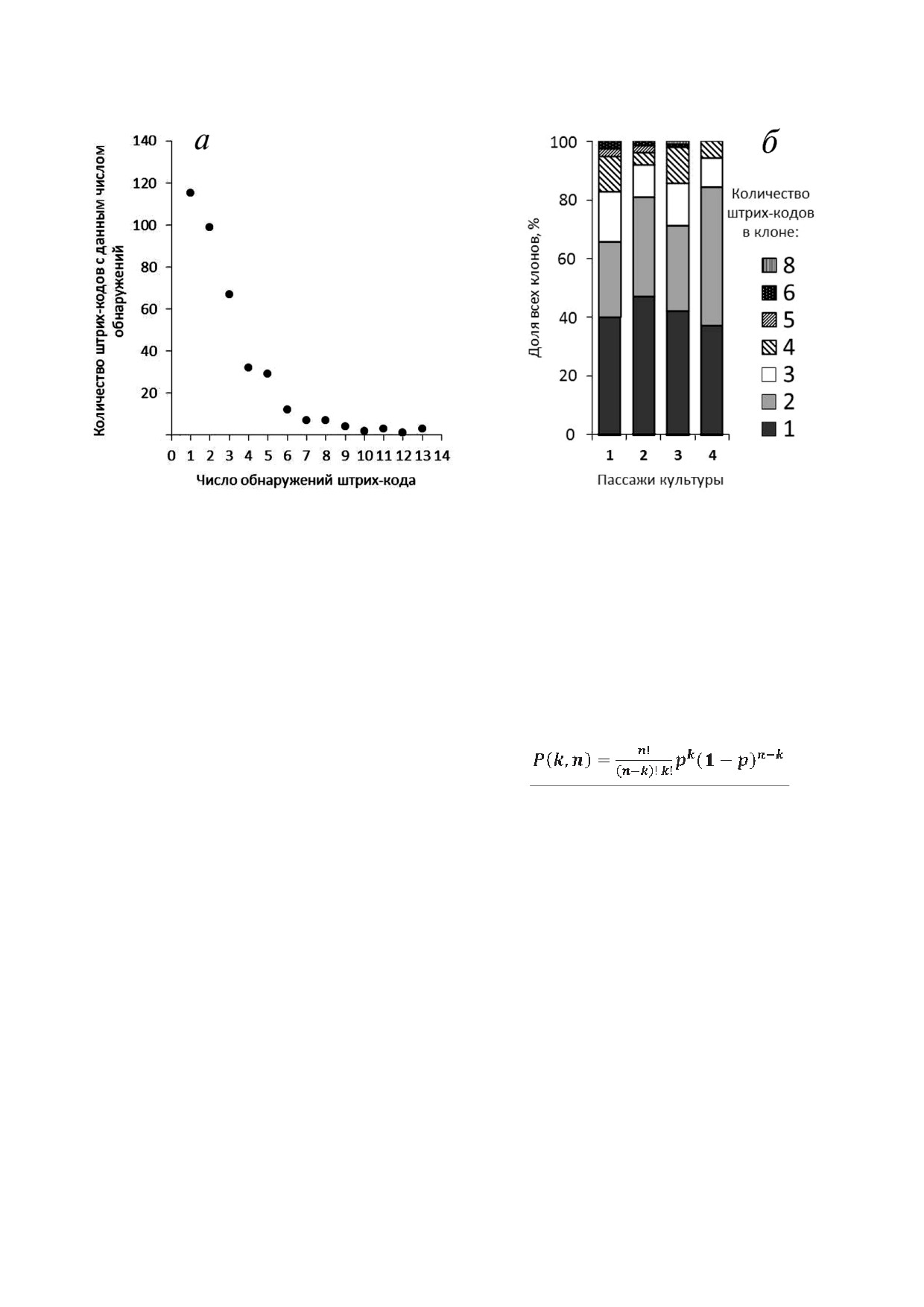
торые штрих!коды встречаются чаще, чем дру!
равномерность представленности штрих!кодов
гие (рис. 5, а). Несмотря на то что большинство
(вирионы с определенными штрих!кодами
штрих!кодов было обнаружено 1-5 раз, некото!
встречаются чаще, чем другие). Вероятно, это
рые из них встречались >10 раз. Подобную зави!
связано со стохастической природой процесса
Таблица 2. Доля маркированных клонов в ММСК
Донор
Пассаж 1
Пассаж 2
Пассаж 3
Пассаж 4
Всего для донора
250
12/22 (55%)
12/22 (55%)
15/28 (54%)
14/22 (64%)
53/94 (56%)
251
29/39 (74%)
28/46 (61%)
4/10 (40%)
11/19 (58%)
72/114 (63%)
252
45/61 (74%)
18/19 (95%)
8/15 (53%)
3/7 (43%)
74/102 (73%)
253
54/59 (92%)
18/21 (86%)
17/22 (77%)
8/9 (89%)
97/111 (87%)
254
49/62 (79%)
43/53 (81%)
52/65 (80%)
18/29 (62%)
162/209 (78%)
256*
N/A
N/A
N/A
N/A
N/A
* ММСК донора 256 не были клонированы. Для каждого донора на каждом пассаже культуры указано количество марки!
рованных клонов/общее количество клонов (процент маркированных клонов).
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
373
Рис. 5. а - Распределение штрих!кодов по частоте встречаемости. По горизонтали отложено, сколько раз штрих!код был
встречен в клонах ММСК (суммарно по всем культурам и пассажам). По вертикали отложено число штрих!кодов, кото!
рые были обнаружены соответствующее число раз; б - распределение клеточных клонов по количеству штрих!кодов (ин!
теграций провирусов)
трансфекции клеток!продуцентов и продукции
встроилось более четырех провирусов, утрачи!
ими вирусных частиц. Сужение разнообразия
вают способность к пролиферации и элимини!
штрих!кодов и искажение равномерности их
руются из культуры.
представленности в вирусном супернатанте по
Оценка уникальности маркирования. Степень
сравнению с библиотекой плазмид является не!
уникальности маркирования клеточной популя!
избежным недостатком применения лентиви!
ции штрих!кодами может быть предсказана с
русных векторов. Степень сужения репертуара
помощью биномиального распределения [12]:
штрих!кодов и искажения представленности
каждого из них остается открытым вопросом и
,
(1)
требует детального изучения, поскольку может
влиять на выводы исследователей, применяю!
где P - вероятность того, что все клетки будут
щих подобные методики. В этом исследовании
маркированы уникально, k - количество марки!
90% штрих!кодов было детектировано не более
рованных клоногенных клеток в тестируемой
5 раз. Данные результаты позволили заключить,
популяции, n - количество уникальных штрих!
что большинство штрих!кодов представлено
кодов в библиотеке, p - вероятность «успеха»,
равномерно, т.к. частота их встречаемости неве!
т.е. того, что клетка будет маркирована конкрет!
лика, а увеличенная частота встречаемости не!
ным штрих!кодом.
которых из них может объясняться наличием в
Оценим вероятность того, что каждая клетка
ММСК каждого донора нескольких клонов -
будет уникально маркирована для донора № 250
потомков одной маркированной клетки, облада!
(репрезентативный образец). Пусть в культуре
ющей высоким пролиферативным потенциалом.
на момент заражения было 100 000 клеток.
Интересно, что распределение клеточных
Эффективность клонирования ММСК донора
клонов по количеству интеграций провирусов
№ 250 на первом пассаже (наиболее близком к
было практически одинаковым на всех исследо!
моменту маркирования) равнялась 0,21 (табл. S6,
ванных пассажах (рис. 5, б). В подавляющем
Приложение), а средняя эффективность марки!
большинстве клонов было обнаружено от одно!
рования ММСК этого донора составляла 56%
го до четырех штрих!кодов. Доля клонов с боль!
(табл. 2). Таким образом, исходная культура долж!
шим количеством штрих!кодов была мала и
на содержать ~11 700 маркированных клоногенных
снижалась к поздним пассажам до 0, что еще раз
клеток (k) в популяции (100 000 × 0,21 × 0,56).
указывает на то, что те клетки, в ДНК которых
Разнообразие штрих!кодов в библиотеке n = 667,
БИОХИМИЯ том 84 вып. 3 2019
374
БИГИЛЬДЕЕВ и др.
а вероятность «успеха» равна 1/n = 1/667. Под!
клеток. Результаты анализа представлены на
ставив значения n и k в формулу (1), получим,
рис. 6. Большинство клонов встречается один
что вероятность того, что все клоны маркирова!
раз на протяжении всех четырех пассажей куль!
ны уникально при интеграции одного штрих!
туры. Полученные данные свидетельствуют о
кода, стремится к нулю.
том, что ММСК поддерживаются, в основном,
Следовательно, если были встречены два
за счет клонов с небольшим пролиферативным
клона, несущие каждый по одному одинаковому
потенциалом, который исчерпывается в течение
штрих!коду, то их считали потомками разных
одного пассажа культуры. В результате на каж!
маркированных клеток. Истинное разнообразие
дом следующем пассаже клональный состав
библиотеки увеличивается за счет того, что одна
ММСК существенно изменяется, в пролифера!
клетка может быть маркирована более чем од!
цию вступают те клоны, которые на предыду!
ним штрих!кодом. Предположим, что клетка
щем пассаже не делились и не вносили большого
маркирована двумя штрих!кодами. Число уни!
вклада в общую клеточную популяцию. В ММСК
кальных комбинаций можно оценить с по!
доноров 250 и 254 было обнаружено по одной
мощью формулы комбинаций m элементов из
паре одинаковых клонов на разных пассажах
множества n элементов с повторениями:
культуры (отмечены красным). Ранее было по!
казано, что ММСК могут содержать клоны, со!
(2)
храняющиеся в культуре на протяжении несколь!
ких пассажей [10]. Динамика их вклада в общую
Приняв n = 667 и m = 2, получим, что коли!
клеточную популяцию может быть различной.
чество комбинаций составит >225 тыс. Оценив
Этим можно объяснить то, что, например, у до!
уникальность маркирования по формуле бино!
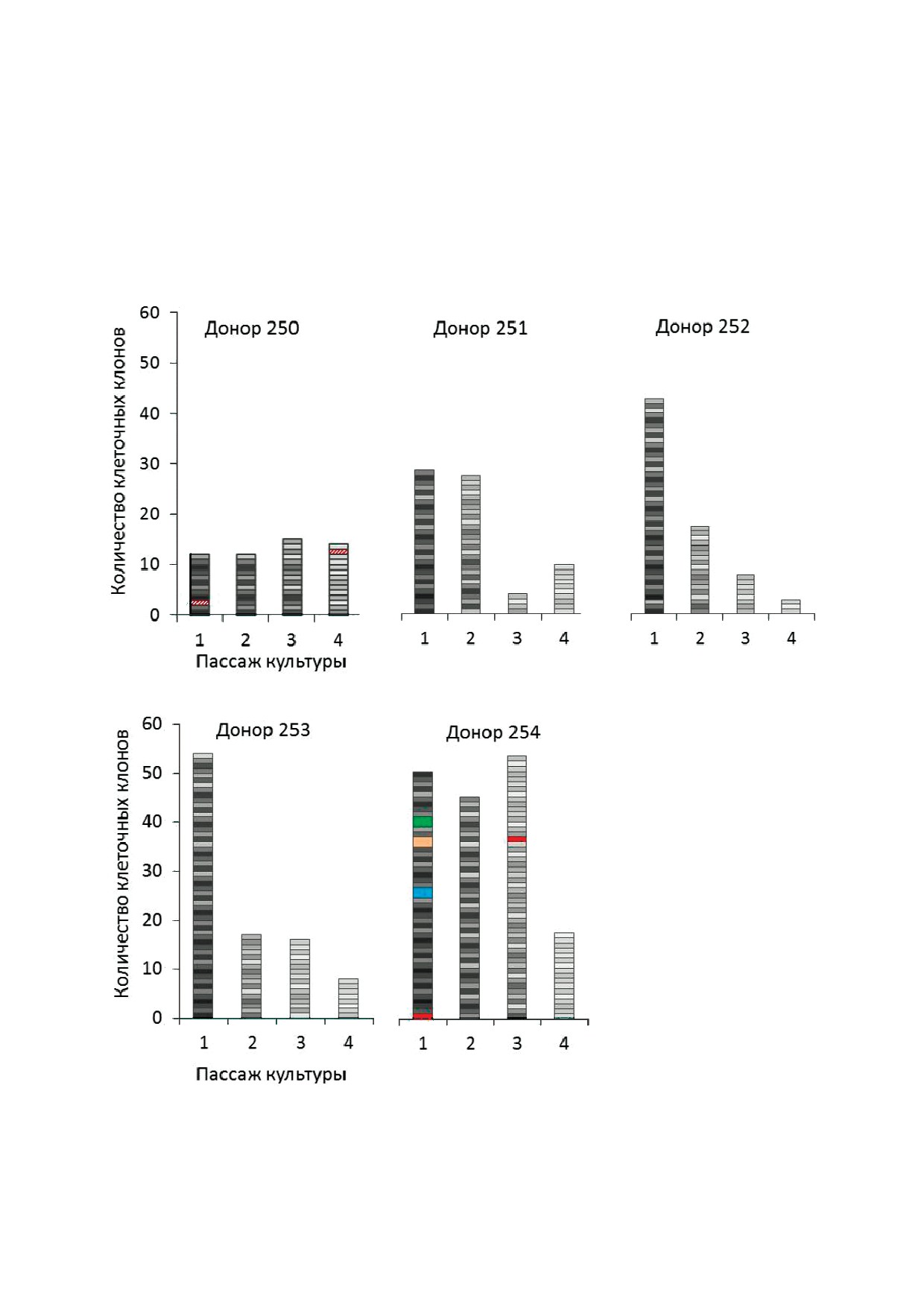
нора № 250 одинаковые клоны (таковыми счи!
миального распределения (1), получим, что ве!
тали клоны, совпадающие по двум и более
роятность того, что все клоны маркированы
штрих!кодам) были обнаружены только на пер!
уникально, стремится к 1. Таким образом, при!
вом и четвертом пассажах, а на втором и третьем
обретение клеткой двух штрих!кодов позволяет
их выявить не удалось.
обеспечить уникальность маркирования. Сле!
В ММСК донора 254 было обнаружено три
довательно, если в эксперименте выявлялись
пары одинаково маркированных клонов на пер!
два клеточных клона, совпадающих по двум (и
вом пассаже культуры. Они соответствуют та!
более) штрих!кодам, такие клеточные клоны
ким клонам, которые вносят существенный
считали произошедшими из одной исходно
вклад в общую клеточную популяцию. Только в
трансдуцированной клетки. Всего в работе было
этом случае вероятность попадания нескольких
обнаружено пять таких клонов (рис. 6).
их представителей в 120 клонированных клеток
Существует вероятность того, что в одну
могла быть высокой. Ранее было показано, что в
ячейку планшета при клонировании попадает
ММСК встречаются клоны, вклад которых в об!
более одной клоногенной клетки. В результате в
щую клеточную популяцию составляет 10-40%
ячейке образуется популяция из нескольких
[10]. Тот факт, что такие клоны были обнаруже!
маркированных клонов, что при анализе будет
ны только у одного из пяти исследованных до!
выглядеть как один клон, содержащий несколь!
норов, свидетельствует о том, что количество
ко штрих!кодов. В силу этого анализ ДНК, вы!
клонов с высоким пролиферативным потенциа!
деленной из двух ячеек, содержащих одинаково
лом невелико по сравнению с общим количест!
маркированные клоны плюс дополнительные
вом клонов на каждом пассаже культуры. Часто!
клеточные колонии, будет давать частично пе!
ту встречаемости таких клонов (например, для
рекрывающиеся наборы штрих!кодов. Если эти
донора № 250) можно оценить как отношение
наборы перекрывались по двум и более штрих!
маркированных клоногенных клеток к общему
кодам, то считали, что в той ячейке, где обнару!
количеству клоногенных клеток в пассаже, кото!
живались дополнительные штрих!коды, присут!
рое было оценено ранее, или 60/11 000 = 0,005.
ствует несколько клеточных клонов, один из ко!
Наличие и количество клонов с высоким проли!
торых несет общие штрих!коды с клоном из
феративным потенциалом может быть связано с
первой ячейки, а остальные несут любые другие
концентрацией МСК в исследуемом костном
комбинации.
мозге. Полученные данные еще раз подтвержда!
Клональный состав ММСК. В клеточных
ют то, что ММСК не идентичны МСК.
клонах, полученных на 1-4!м пассажах ММСК
Анализ штрих кодов в общей клеточной популя
каждого донора, было проведено сравнение
ции ММСК. На каждом пассаже культуры из сум!
штрих!кодов. С учетом соображений, описан!
марной популяции клеток, оставшихся после
ных в предыдущем разделе, были выявлены кло!
пассирования и клонирования, выделяли ДНК и
ны - потомки одних и тех же маркированных
анализировали вклад штрих!кодов. Репрезента!
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
375
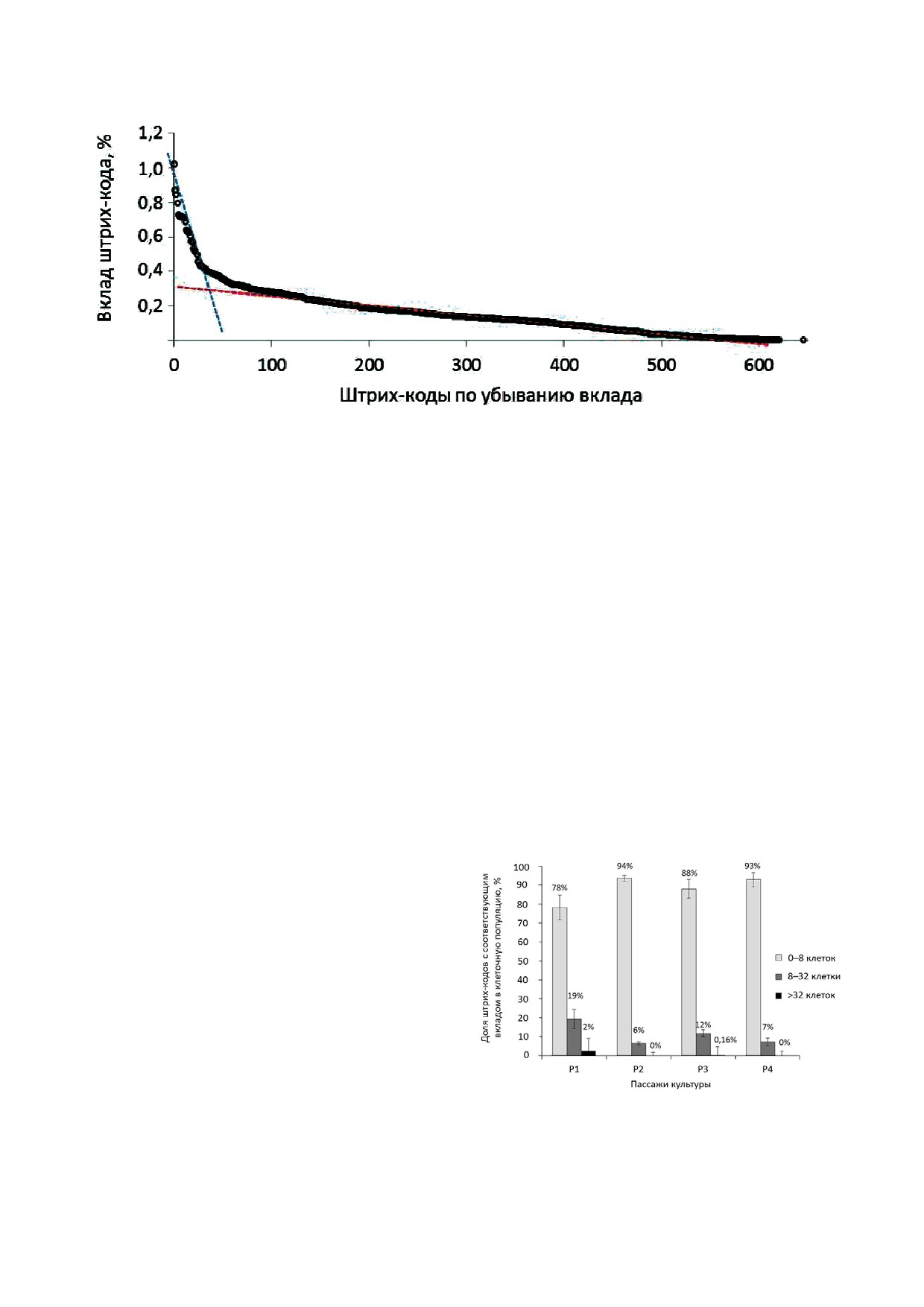
тивное распределение вклада штрих!кодов для
купности с данными цифровой капельной ПЦР
ММСК донора 254 на первом пассаже приведе!
по оценке количества провирусов в расчете на
но на рис. 7.
один диплоидный геном позволяют сделать од!
Из рис. 7 видно, что NGS выявило подавля!
нозначный вывод о поликлональности культу!
ющее большинство штрих!кодов исходной биб!
ры. Действительно, цифровая ПЦР продемон!
лиотеки. В каждом образце выявлялось >600 из
стрировала, что в среднем по всем пассажам и
исходных 667 штрих!кодов, при этом >550 штрих!
всем донорам на один геном приходится 9 про!
кодов имели
5 прочтений. Эти данные в сово! вирусов. Одновременно с этим, NGS показало,
Рис. 6. Клональный состав ММСК. Каждый прямоугольник - клеточный клон. Серые прямоугольники обозначают та!
кие клеточные клоны, которые у данного донора встретились всего один раз (независимо от пассажа). Цветные прямоу!
гольники обозначают клоны, встретившиеся более одного раза
БИОХИМИЯ том 84 вып. 3 2019
376
БИГИЛЬДЕЕВ и др.
Рис. 7. Распределение вклада штрих!кодов в общую клеточную популяцию ММСК донора 254 на первом пассаже культу!
ры. По вертикали - вклад штрих!кода, представленный как отношение количества прочтений данного штрих!кода к ко!
личеству классифицированных прочтений, выраженное в процентах. По горизонтали - штрих!коды, расположенные по
вкладу от бóльшего к меньшему.
С цветным вариантом рис. 7 можно ознакомиться в электронной версии статьи на сайте: www.elibrary.ru
что в любом образце идентифицируется не!
ладающих максимальным вкладом в ММСК до!
сколько сотен штрих!кодов. Такое соотношение
нора № 250 на первом пассаже, один штрих!код
неизбежно означает, что культура состоит из
(№ 967) входил в 10 наиболее представленных
множества одновременно функционирующих
на первом пассаже штрих!кодов у всех проана!
клонов.
лизированных доноров, еще пять штрих!кодов
Медиана вклада каждого отдельного штрих!
(№ 31, 42, 463, 669, 699) входили в топ!10 у четы!
кода составила 0,013%. Из рис. 7 видно, что сре!
рех из пяти доноров, а остальные четыре штрих!
ди всего множества выделяется группа из не!
кода также встречались среди топ!10 у более чем
скольких десятков штрих!кодов, вклад которых
одного донора. Из этого следует, что в исходном
значительно выбивается из общего распределе!
общем вирусном супернатанте, которым зара!
ния и превышает 0,4%. Наличие такого излома
жали ММСК всех доноров, вирусов с данными
можно объяснить несколькими способами. Эти
штрих!кодами было относительно больше, чем
штрих!коды могут соответствовать совокупнос!
всех остальных. По мере пассирования вклад
ти клеточных клонов, которые давали относи!
каждого отдельного штрих!кода меняется. Было
тельно большой вклад в общую клеточную по!
показано, что количество штрих!кодов, внося!
пуляцию ММСК на данном пассаже. В пользу
щих существенный вклад в общую популяцию,
этой версии говорит то, что все одинаковые кло!
невелико и уже ко второму пассажу определяет!
ны, обнаруженные на первом пассаже культуры
ся как десятые доли процента (рис. 8).
этого донора, содержали штрих!коды, входящие
в топ!35 по своему вкладу. Таким образом, ре!
зультаты, полученные при секвенировании штрих!
кодов в отдельных клонах, и данные NGS кор!
релируют между собой.
Возможно альтернативное объяснение. Наи!
более часто встречающимися могут быть штрих!
коды, чья представленность в исходном вирус!
ном супернатанте была больше. Следовательно,
они интегрировались в сравнительно бóльшее
количество клеток при маркировании культуры,
если предположить, что вероятность интегра!
ции провирусной ДНК не зависит от нуклеотид!
ной последовательности штрих!кода. В пользу
этой версии говорит то, что одни и те же штрих!
Рис. 8. Динамика распределения штрих!кодов, обладающих
коды входят в топ!10 штрих!кодов у нескольких
вкладом, соответствующим различному количеству клеток
доноров одновременно. Из 10 штрих!кодов, об!
популяции ММСК
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
377
Максимальный вклад штрих!кодов соответ!
нов может быть еще больше. Значительные из!
ствовал ~100 клеткам, такие штрих!коды были
менения клонального состава популяции от
выявлены у половины проанализированных до!
пассажа к пассажу подтверждают ограниченный
норов. У одного донора (Д250) их было 90, у ос!
пролиферативный потенциал большинства
тавшихся - шесть (Д254) и один (Д251). Учиты!
представителей ММСК. По!прежнему нельзя
вая, что для секвенирования было взято количе!
исключить, что в состав этой популяции не вхо!
ство ДНК, соответствующее ~8000 клеток, мож!
дят клетки, близкие по своим характеристикам
но оценить максимальная представленность
к стволовым. ММСК в основном представляют
клеточных клонов в культуре. В этом экспери!
собой гетерогенную популяцию стромальных
менте оно составило 100/8000 = 1,2%. Один
предшественников с ограниченной способ!
штрих!код может присутствовать в нескольких
ностью к пролиферации, но сохраняющих по!
клонах, поэтому реальная представленность
липотентность.
клонов в культуре ММСК может быть ниже, что
подтверждается полученными ранее данными
[10]. Подавляющее большинство штрих!кодов
Конфликт интересов
на всех пассажах выявлялось в
8 клетках.
Описанные в работе сложности, связанные с
Авторы заявляют об отсутствии конфликта
уникальностью маркирования и искажением
интересов.
представленности штрих!кодов в библиотеке,
указывают на то, что необходимо продолжать по!
Соблюдение этических норм
иск новых стратегий по маркированию ММСК.
В их основе должны лежать плазмидные библи!
Все процедуры, выполненные в исследова!
отеки, в которых достигается баланс между раз!
нии с участием людей, соответствуют этическим
нообразием и точностью характеризации биб!
стандартам институционального комитета по
лиотеки. Более того, желательно анализировать
исследовательской этике и Хельсинской декла!
лентивирусную библиотеку по разнообразию и
рации 1964 года и ее последующим изменениям
представленности штрих!кодов непосредствен!
и сопоставимым нормам этики. От каждого из
но перед ее использованием.
включенных в исследование участников было
Применение техники маркирования ММСК
получено информированное добровольное со!
с помощью генетических штрих!кодов и после!
гласие.
дующего глубокого секвенирования маркеров под!
твердило наличие поликлональности в ММСК
Благодарности
человека, которая была установлена ранее с по!
мощью ПЦР, опосредованной лигированием [10].
Авторы благодарят проф. Б. Фезе и К. Кор!
При этом в данной работе было проанализиро!
нилс (University Medical Center Hamburg!
вано существенно бóльшее количество клонов и
Eppendorf, Гамбург, Германия) за предоставлен!
выявлено пропорционально бóльшее их разно!
ную библиотеку плазмид, содержащих штрих!
образие. Полученные результаты означают, что
коды, и линию!продуцент для получения ленти!
в популяции ММСК реальное количество кло!
вирусного вектора.
СПИСОК ЛИТЕРАТУРЫ
1.
Friedenstein, A.J., Chailakhjan, R.K., and Lalykina, K.S.
Minimal criteria for defining multipotent mesenchymal
(1970) The development of fibroblast colonies in monolay!
stromal cells. The International Society for Cellular
er cultures of guinea!pig bone marrow and spleen cells, Cell
Therapy position statement, Cytotherapy, 8, 315-317,
Tissue Kinet., 3, 393-403, doi: 10.1111/j.1365!2184.
doi: 10.1080/14653240600855905.
1970.tb00347.
5.
Jones, B.J. and McTaggart, S.J. (2008) Immunosup!pres!
2.
Caplan, A.I. (1991) Mesenchymal stem cells, J. Orthop.
sion by mesenchymal stromal cells: from culture to clinic,
Res., 9, 641-650, doi: 10.1002/jor.1100090504.
Exp. Hematol., 36, 733-741, doi: 10.1016/j.exphem.
3.
Horwitz, E.M., Le Blanc, K., Dominici, M., Mueller, I.,
2008.03.006.
Slaper!Cortenbach, I., Marini, F.C., Deans, R.J., Krause, D.S.,
6.
Spees, J.L., Lee, R.H., and Gregory, C.A.
(2016)
Keating, A., and International Society for Cellular
Mechanisms of mesenchymal stem/stromal cell function,
Therapy. (2005) Clarification of the nomenclature for
Stem Cell Res. Ther., 7, 125, doi: 10.1186/s13287!016!
MSC: The International Society for Cellular Therapy posi!
0363!7.
tion statement, Cytotherapy, 7, 393-395, doi: 10.1080/
7.
Pittenger, M.F., Mackay, A.M., Beck, S.C., Jaiswal, R.K.,
14653240500319234.
Douglas, R., Mosca, J.D., Moorman, M.A., Simonetti, D.W.,
4.
Dominici, M., Le Blanc, K., Mueller, I., Slaper!
Craig, S., and Marshak, D.R. (1999) Multilineage poten!
Cortenbach, I., Marini, F., Krause, D., Deans, R.,
tial of adult human mesenchymal stem cells, Science, 284,
Keating, A., Prockop, Dj., and Horwitz, E.
(2006)
143-147, doi: 10.1126/science.284.5411.143.
БИОХИМИЯ том 84 вып. 3 2019
378
БИГИЛЬДЕЕВ и др.
8.
Palau, P., Dominguez, E., Lopez, L., Heredia, R.,
state reconstitution phases, Cell Stem Cell, 19, 107-119,
Gonzalez, J., Ramon, J.M., Serra, P., Santas, E., Bodi, V.,
doi: 10.1016/j.stem.2016.04.016.
Sanchis, J., Chorro, F.J., and Nunez, J. (2016) Inspiratory
19.
Nguyen, L.V, Pellacani, D., Lefort, S., Kannan, N.,
muscle training and functional electrical stimulation for
Osako, T., Makarem, M., Cox, C.L., Kennedy, W., Beer, P.,
treatment of heart failure with preserved ejection fraction:
Carles, A., Moksa, M., Bilenky, M., Balani, S., Babovic, S.,
rationale and study design of a prospective randomized
Sun, I., Rosin, M., Aparicio, S., Hirst, M., and Eaves, C.J.
controlled trial, Clin. Cardiol., 39, 433-439, doi: 10.1002/
(2015) Barcoding reveals complex clonal dynamics of de
clc.22555.
novo transformed human mammary cells, Nature, 528,
9.
Russell, K.C., Phinney, D.G., Lacey, M.R., Barrilleaux, B.L.,
267-271, doi: 10.1038/nature15742.
Meyertholen, K.E., and O’Connor, K.C. (2010) In vitro
20.
Cornils, K., Thielecke, L., Huser, S., Forgber, M.,
high!capacity assay to quantify the clonal heterogeneity in
Thomaschewski, M., Kleist, N., Hussein, K., Riecken, K.,
trilineage potential of mesenchymal stem cells reveals a
Volz, T., Gerdes, S., Glauche, I., Dahl, A., Dandri, M.,
complex hierarchy of lineage commitment, Stem Cells, 28,
Roeder, I., and Fehse, B. (2014) Multiplexing clonality:
788-798, doi: 10.1002/stem.312.
combining RGB marking and genetic barcoding, Nucleic
10.
Bigildeev, A.E., Zhironkina, O.A., Shipounova, I.N.,
Acids Res., 42, e56, doi: 10.1093/nar/gku081.
Sats, N.V, Kotyashova, S.Y., and Drize, N.J. (2012) Clonal
21.
Weber, K., Bartsch, U., Stocking, C., and Fehse, B. (2008)
composition of human multipotent mesenchymal stromal
A multicolor panel of novel lentiviral «gene ontology»
cells, Exp. Hematol., 40, 847-856, doi: 10.1016/j.exphem.
(LeGO) vectors for functional gene analysis, Mol. Ther.,
2012.06.006.
16, 698-706, doi: 10.1038/mt.2008.6.
11.
Gerrits, A., Dykstra, B., Kalmykowa, O.J., Klauke, K.,
22.
Aranyossy, T., Thielecke, L., Glauche, I., Fehse, B., and
Verovskaya, E., Broekhuis, M.J.C., de Haan, G., and
Cornils, K. (2017) Genetic barcodes facilitate competitive
Bystrykh, L.V. (2010) Cellular barcoding tool for clonal
clonal analyses in vivo, Hum. Gene Ther., 28, 926-937,
analysis in the hematopoietic system, Blood,
115,
doi: 10.1089/hum.2017.124.
2610-2618, doi: 10.1182/blood!2009!06!229757.
23.
Sambrook, J., Fritsch, E., and Maniatis, T.
(1989)
12.
Bystrykh, L.V., and Belderbos, M.E. (2016) Clonal analy!
Molecular cloning: a laboratory manual, Cold Spring
sis of cells with cellular barcoding: when numbers and sizes
Harbor, Cold Spring Harbor Laboratory Press, N.Y.
matter, Methods Mol. Biol., 1516, 57-89, doi: 10.1007/
24.
Ausubel, F.M., Brent, R., Kingston, R.E., Moore, D.D.,
7651_2016_343.
Seidman, J., Smith, J.A., and Struhl, K. (1991) Current
13.
Ребриков Д.В., Коростин Д.О., Шубина Е.С., Ильин!
protocols in molecular biology, New York, John Wiley &
ский В.В. (2015) NGS: высокопроизводительное секве)
Sons, Inc.
нирование, Москва, Лаборатория знаний.
25.
Kuzmina, L.A., Petinati, N.A., Parovichnikova, E.N.,
14.
Glimm, H., Ball, C.R., and von Kalle, C. (2011) You can
Lubimova, L.S., Gribanova, E.O., Gaponova, T.V,
count on this: barcoded hematopoietic stem cells, Cell Stem
Shipounova, I.N., Zhironkina, O.A., Bigildeev, A.E.,
Svinareva, D.A., Drize, N.J., and Savchenko, V.G. (2012)
2011.10.013.
Multipotent mesenchymal stromal cells for the prophylaxis
15.
Maetzig, T., Brugman, M.H., Bartels, S., Heinz, N.,
of acute graft!versus!host disease!a phase II study, Stem
Kustikova, O.S., Modlich, U., Li, Z., Galla, M.,
Cells Int., 2012, 1-8, doi: 10.1155/2012/968213.
Schiedlmeier, B., Schambach, A., and Baum, C. (2011)
26.
Todoerti, K., Lisignoli, G., Storti, P., Agnelli, L., Novara, F.,
Polyclonal fluctuation of lentiviral vector!transduced and
Manferdini, C., Codeluppi, K., Colla, S., Crugnola, M.,
expanded murine hematopoietic stem cells, Blood, 117,
Abeltino, M., Bolzoni, M., Sgobba, V., Facchini, A.,
3053-3064, doi: 10.1182/blood!2010!08!303222.
Lambertenghi!Deliliers, G., Zuffardi, O., Rizzoli, V.,
16.
Lu, R., Neff, N.F., Quake, S.R., and Weissman, I.L. (2011)
Neri, A., and Giuliani, N. (2010) Distinct transcriptional
Tracking single hematopoietic stem cells in vivo using high!
profiles characterize bone microenvironment mesenchymal
throughput sequencing in conjunction with viral genetic
cells rather than osteoblasts in relationship with multiple
barcoding, Nat. Biotechnol., 29, 928-933, doi: 10.1038/
myeloma bone disease, Exp. Hematol., 38, 141-153,
nbt.1977.
doi: 10.1016/j.exphem.2009.11.009.
17.
Verovskaya, E., Broekhuis, M.J.C., Zwart, E., Ritsema, M.,
27.
Gasparian, M.E., Elistratov, P.A., Drize, N.I., Nifontova, I.N.,
van Os, R., de Haan, G., and Bystrykh, L.V. (2013)
Dolgikh, D.A., and Kirpichnikov, M.P. (2009) Over!
Heterogeneity of young and aged murine hematopoietic
expression in Escherichia coli and purification of human
stem cells revealed by quantitative clonal analysis using cel!
fibroblast growth factor (FGF!2), Biochemistry (Moscow),
lular barcoding, Blood, 122, 523-532, doi: 10.1182/blood!
74, 221-225, doi: 10.1134/S000629790902014X.
2013!01!481135.
28.
Bailey, N.T.J. (1995) Statistical methods in biology, 3rd edi!
18.
Biasco, L., Pellin, D., Scala, S., Dionisio, F., Basso!Ricci, L.,
tion, Cambridge, Cambridge University Press.
Leonardelli, L., Scaramuzza, S., Baricordi, C., Ferrua, F.,
29.
Жиронкина О.А., Шипунова И.Н., Бигильдеев А.Е.,
Cicalese, M.P., Giannelli, S., Neduva, V., Dow, D.J.,
Сац Н.В., Петинати Н.А., Дризе Н.И. (2011) Проли!
Schmidt, M., Von Kalle, C., Roncarolo, M.G., Ciceri, F.,
феративный потенциал мультипотентных мезенхи!
Vicard, P., Wit, E., Di Serio, C., Naldini, L., and Aiuti, A.
мальных стромальных клеток, выделенных из костного
(2016) In vivo tracking of human hematopoiesis reveals
мозга человека, Клеточные технол. биол. мед.,
patterns of clonal dynamics during early and steady!
230-235.
БИОХИМИЯ том 84 вып. 3 2019
КЛОНАЛЬНЫЙ СОСТАВ ПОПУЛЯЦИИ ММСК
379
CLONAL COMPOSITION OF HUMAN MULTIPOTENT
MESENCHYMAL STROMAL CELLS: APPLICATION
OF GENETIC BARCODES FOR RESEARCH
A. E. Bigildeev1*, A. M. Pilunov2, N. V. Sats1, V. L. Surin1,
I. N. Shipounova1, N. A. Petinati1, M. D. Logacheva3, A. V. Fedotova3,
A. S. Kasyanov4, A. S. Artyukhov5, E. B. Dashinimaev6, and N. J. Drize1
1 National Research Center for Hematology, Ministry of Health
of the Russian Federation, 125167 Moscow, Russia;
E)mail: bigildeev.ae@gmail.com
2 Lomonosov Moscow State University, 119991 Moscow, Russia
3 Belozersky Institute of Physico)Chemical Biology, Lomonosov
Moscow State University, 119992 Moscow, Russia
4 Vavilov Institute of General Genetics, Russian Academy
of Sciences, 119991 Moscow, Russia
5 Pirogov Russian National Research Medical University (RNRMU),
Ministry of Health of the Russian Federation, 117997 Moscow, Russia
6 Koltzov Institute of Developmental Biology, Russian
Academy of Sciences, 119334 Moscow, Russia
Received September 12, 2018
Revised November 27, 2018
Accepted November 27, 2018
Clonal composition of human multipotent mesenchymal stromal cells (MMSCs), marked with lentiviral vectors car!
rying genetic barcodes, was studied. MMSCs were infected with a cloned library of self!inactivating lentiviral vectors
carrying 667 non!repeating barcodes. At each passage of the culture, 120 cells were cloned one cell per well in 96!well
plates. The effectiveness of cloning and labeling of clonogenic cells was determined. From the one cell!derived
colonies DNA was extracted, and barcodes were identified by Sanger sequencing. DNA was also extracted from the
total MMSC population at each passage, and the diversity and representation of barcodes was determined by the
Illumina!based deep sequencing. It was shown that the proportion of MMSCs marked with lentiviral vectors is stable
in passages. Given high multiplicity of infection, the labeling procedure can decrease MMSCs proliferative potential.
Barcode identification in individual cell clones confirmed the polyclonal character of the MMSC population. Clonal
composition of MMSCs changes with the passages significantly due to depletion of the proliferative potential of most
cells. Large clones were found at the first passage of MMSCs, and then many small clones with a limited proliferative
potential were detected in the population. The results of deep sequencing confirmed the changes in MMSCs clonal
composition. The polyclonal population of MMSC contains only a small number of cells with a high proliferative
potential, among which there may be stem cells. MMSCs with high proliferative potential are more often detected in
the earliest passages. In this regard, the MMSCs of early passages are recommended for use in regenerative medicine
based on cell proliferation.
Keywords: multipotent mesenchymal stromal cells, genetic marking, barcode, lentiviral vector
БИОХИМИЯ том 84 вып. 3 2019