БИОХИМИЯ, 2019, том 84, вып. 6, с. 786 - 794
УДК 577.123.1
ДИФФЕРЕНЦИАЛЬНЫЕ ПРОФИЛИ ЭКСПРЕССИИ
микроРНК МОГУТ ЯВЛЯТЬСЯ ПОТЕНЦИАЛЬНЫМИ
БИОМАРКЕРАМИ АДЕНОКАРЦИНОМЫ ПРОТОКОВ
ПОДЖЕЛУДОЧНОЙ ЖЕЛЕЗЫ*,**
© 2019
Y. Zhu1#, J. Wang2#, F. Wang3, Z. Yan3, G. Liu2,
Y. Ma4, W. Zhu5, Y. Li3, L. Xie3, A.V. Bazhin6***, X. Guo3***
1 Department of Oncology, International Joint Laboratory for Cell Medical Engineering
of Henan Province, Henan University Huaihe Hospital, No. 8 Baobei Road, Kaifeng,
Henan 475000, People’s Republic of China; E mail: celltransplant@163.com
2 Department of Oncology, the Second Affiliated Hospital of Zhengzhou University,
Zhengzhou, No. 2 Jingba Road, Henan 450014, People’s Republic of China;
E mail: wj68happy@hotmail.com
3 Department of Preventive Medicine, Cell Signal Transduction Laboratory,
Joint National Laboratory for Antibody Drug Engineering, Institute of Biomedical
Informatics, Medical School, Henan University, Kaifeng, Henan 475004,
People’s Republic of China; E mail: xqguo@henu.edu.cn
4 College of Pharmacy and Tianjin Key Laboratory of Molecular Drug Research,
Nankai University, Haihe Education Park, 38 Tongyan Road, Tianjin 300353,
People’s Republic of China; E mail: mayonggang@nankai.edu.cn
5 Department of Anesthesia, Stanford University, CA 94305, USA; E mail: wan.zhu@stanford.edu
6 Department of General, Visceral, and Transplantation Surgery, Ludwig Maximilians University
Munich, Munich 81377, Germany; E mail: alexandr.bazhin@med.uni muenchen.de
Поступила в редакцию 04.12.2018
После доработки 02.02.2019
Принята к публикации 04.02.2019
Лечение аденокарциномы протоков поджелудочной железы (PDAC) вызывает определенные трудности,
вызванные проблемами с ранней диагностикой данного заболевания. Возможными биологическими мар
керами для раннего диагностирования этого вида рака могут служить специфичные сывороточные мик
роРНК (miРНК). В представленной работе мы проанализировали образцы плазмы крови здоровых людей и
пациентов с PDAC для идентификации и оценки уровней экспрессии различных miРНК с помощью техно
логии секвенирования следующего поколения и биоинформационного анализа. Мы установили, что уров
ни экспрессии 165 зрелых miРНК претерпевали значительные изменения у больных раком, причем из них
экспрессия 75 miРНК регулировалась положительно, а экспрессия 90 miРНК - отрицательно у больных па
циентов по сравнению со здоровыми индивидами. Кроме того, мы идентифицировали 1029 новых miРНК.
Можно заключить, что профили экспрессии miРНК в плазме крови различались между собой у пациентов
с PDAC и здоровых людей. Полученные нами результаты могут свидетельствовать о возможности использо
вания miРНК в качестве диагностических и прогностических биомаркеров при PDAC.
КЛЮЧЕВЫЕ СЛОВА: аденокарцинома протоков поджелудочной железы (PDAC), miРНК плазмы крови,
профили экспрессии.
DOI: 10.1134/S0320972519060046
Аденокарцинома протоков поджелудочной же
ростью метастазирования опухоли и отсутстви
лезы (PDAC) - это заболевание с высокой смерт
ем ее ранних клинических проявлений; у ~80%
ностью, при котором в течение 5 лет выживает
пациентов опухоль диагностируется лишь на
<5% пациентов [1]. Это связано с высокой ско
поздних стадиях, когда уже потеряно время для
Принятые сокращения: PDAC - аденокарцинома протоков поджелудочной железы; miРНК, miR - микроРНК.
biokhimiya, в рубрике «Papers in Press», BM18 333, 08.04.2019.
** Приложение к статье на английском языке опубликовано на сайте журнала «Biochemistry» (Moscow) и на сайте издатель
ства Springer (Link.springer.com), том 84, вып. 5, 2019. Статья на английском языке опубликована в Biochemistry (Moscow),
том 84, вып. 5, 2019.
*** Адресат для корреспонденции.
# Авторы внесли равный вклад в работу.
786
ПРОФИЛЬ ЭКСПРЕССИИ miРНК В ПЛАЗМЕ КРОВИ ПРИ PDAC
787
успешного хирургического вмешательства [2].
Кроме того, было показано, что эти РНК могут
Поэтому чрезвычайно важна разработка новых
быть использованы в качестве возможных био
методов ранней диагностики PDAC.
маркеров для диагностики PDAC [19]. Несмотря
МикроРНК (miРНК) - это одноцепочечные
на то что некоторые профили miРНК в сыворот
РНК длиной 19-25 нуклеотидов, синтезирую
ке больных PDAC ранее уже исследовались
щиеся на шпилькообразных мРНК и открытые
[20-23], общий набор и уровни экспрессии спе
в 1993 г. [3]. miРНК не кодируют белки, но регу
цифичных miРНК для диагностики PDAC пока
лируют экспрессию ~50% генов, кодирующих
еще никем не были определены.
белки [4]. Эти регуляторные факторы сначала
Целью данной работы являлись идентифи
транскрибируются, а затем подвергаются про
кация и анализ профилей экспрессии miРНК в
цессингу при помощи рибонуклеаз Dicer и
образцах плазмы крови здоровых людей и боль
Drosha, превращаясь в зрелые miРНК длиной
ных PDAC с помощью секвенирования и био
21-23 нуклеотидов [5]. miРНК встраивается в
информатического анализа.
РНК индуцируемый молчащий комплекс
(RISC), содержащий Dicer и другие ассоцииро
ванные белки [6]. RISC регулирует посттрансля
МЕТОДЫ ИССЛЕДОВАНИЯ
ционную экспрессию мРНК обычно путем свя
зывания с комплементарной последователь
Пациенты. Для исследования было привле
ностью 3' нетранслируемого района (3' UTR)
чено пять пациентов с диагнозом PDAC (группа P)
мРНК, препятствуя узнаванию кэпа фактором
из отделения общей хирургии Второго филиала
инициации eIF4E и последующему связыванию
госпиталя университета в Чженчжоу с октября
с факторами трансляции [7]. Проведенные ра
2015 г. по март 2016 г. Диагноз PDAC был под
нее исследования показали, что действие
твержден при гистопатологическом обследова
miРНК коррелирует не только с образованием и
нии хирургического материала. Пятеро выбран
развитием опухоли [4, 8, 9], но также и с форми
ных случайно здоровых индивидов (группа Н)
рованием лекарственной устойчивости, метаста
были привлечены из отдела физических иссле
зированием рака, ангиогенезом, рецидивирова
дований или отдела пищеварительной медици
нием рака и т.д. [10-13]. miРНК могут быть
ны. Члены этой группы были здоровы и никогда
обнаружены с помощью полностью автоматизи
ранее не болели раком. Клинико эпидемиоло
рованной высокопроизводительной процедуры.
гические характеристики всех участников экс
miРНК, полученные из разрушенных клеток,
перимента приведены в табл. 1.
транспортируются в экзосомы и поступают в
Образцы для исследования. Образцы крови
кровь [14, 15]. Будучи защищенными в эндосо
собирали до проведения хирургической опера
мах, циркулирующие в крови miРНК являются
ции в полиэтиленовые контейнеры, предвари
стабильными биомаркерами и могут быть ис
тельно промытые ЭДТА. Кровь центрифугиро
пользованы в диагностике злокачественных но
вали при 2500 об/мин в течение 5 мин при 4 °С.
вообразований, прогностической и терапевти
Плазму немедленно отбирали и хранили при
ческой оценки [14, 16].
-80 °С.
Сообщалось о том, что у больных PDAC име
Выделение РНК и секвенирование. Фракцию
ются miРНК, отвечающие за апоптоз, пролифе
общей РНК экстрагировали с помощью мини
рацию, эпителиально мезенхимальное преобра
набора Norgen Plasma/Serum Circulating RNA
зование раковых клеток, их метастазирование,
Purification Mini Kit («Norgen», Канада) в соот
инвазию и лекарственную устойчивость [17, 18].
ветствии с прилагаемой инструкцией. Количест
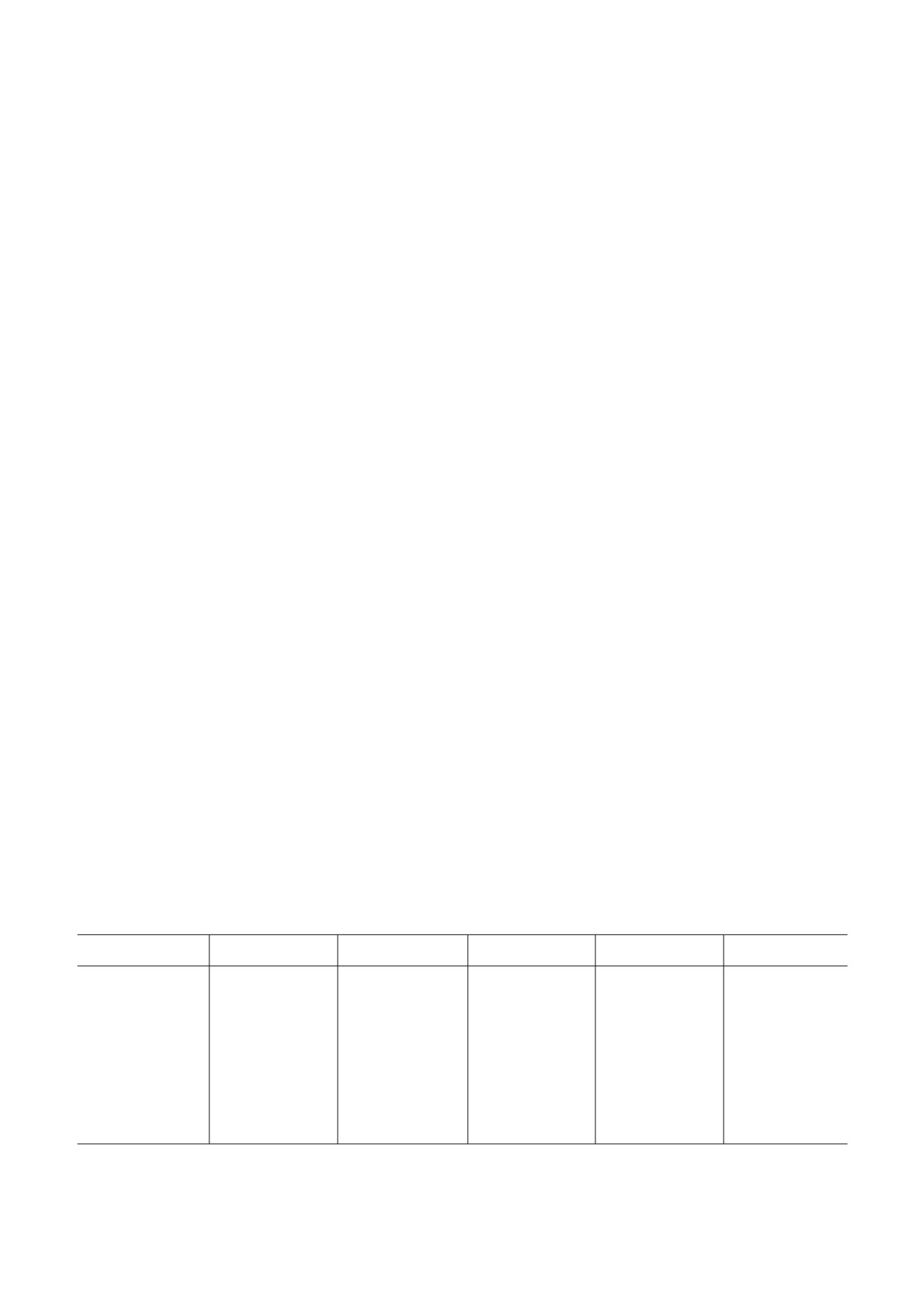
Таблица 1. Клинико эпидемиологические характеристики здоровых индивидов (группа Н) и пациентов с PDAC (группа Р)
Группа
Номер образца
Возраст, годы
Вес, кг
Пол
Стадия рака
ZYFR1
47
67,7
М
-
H
ZYFR2
77
63,5
М
-
ZYFR3
63
59,7
Ж
-
ZYFR4
52
57,2
Ж
-
ZYFR5
77
56,5
Ж
-
ZYFR11
77
62,7
М
IV
P
ZYFR12
49
67,8
М
III
ZYFR13
75
62,5
М
IV
ZYFR14
70
56,5
Ж
IV
ZYFR15
29
69,7
М
II
БИОХИМИЯ том 84 вып. 6 2019
788
ZHU и др.
во и чистоту фракции общей РНК определяли
пами здоровых людей и пациентов с PDAC оце
с использованием Bioanalyzer 2100 («Agilent»,
нивали путем анализа вариаций (ANOVA); post
США) и соответствующего набора реактивов
hoc анализ выполняли с использованием теста
RNA 6000 Nano LabChip Kit («Agilent», США).
Таки-Крамера. Значения р < 0,05 считали ста
Секвенирование проводили только для образ
тистически значимыми.
цов РНК, индекс целостности (RNA Integrity
Number, RIN) которых превышал 7,0. Около
0,2-0,7 мкг общей РНК было использовано для
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
создания библиотеки малых РНК в соответ
ствии с инструкцией к набору TruSeq Small RNA
Профили экспрессии микроРНК в плазме кроE
Sample Prep Kits («Illumina», США). Для созда
ви. Для доказательства возможности использо
ния библиотеки малых РНК общую РНК фрак
вания miРНК в качестве потенциальных био
ционировали в геле с целью выделения РНК
маркеров для диагностирования PDAC были
длиной 18-30 нуклеотидов; полосу на геле, со
сформированы две группы людей по пять инди
ответствующую этой фракции, вырезали; нук
видов в каждой: группа Р - больные с аденокар
леиновую кислоту экстрагировали из геля и
циномой, группа Н - здоровые индивиды. Об
очищали. Очищенные малые РНК были сшиты
разцы крови собирали, и содержащиеся в них
с адаптерными РНК по 5' и затем по 3' концам.
miРНК секвенировали с использованием при
Продукты лигирования очищали, подвергали
бора Hiseq2500 («Illumina», США) и программы
обратной транскрипции и амплифицировали
LC BIO. Количество секвенированных после
методом ПЦР. После очистки продукты ПЦР
довательностей, равное 130 162 923 и 163 059 214,
секвенировали с одного конца с использовани
было использовано для последующего анализа
ем прибора Hiseq2500 («Illumina», США) и про
образцов плазмы индивидов групп Р (образцы
граммы LC BIO (Ханчжоу, Китай) в соответ
ZYFR11-15) и Н (образцы ZYFR1-5) соответ
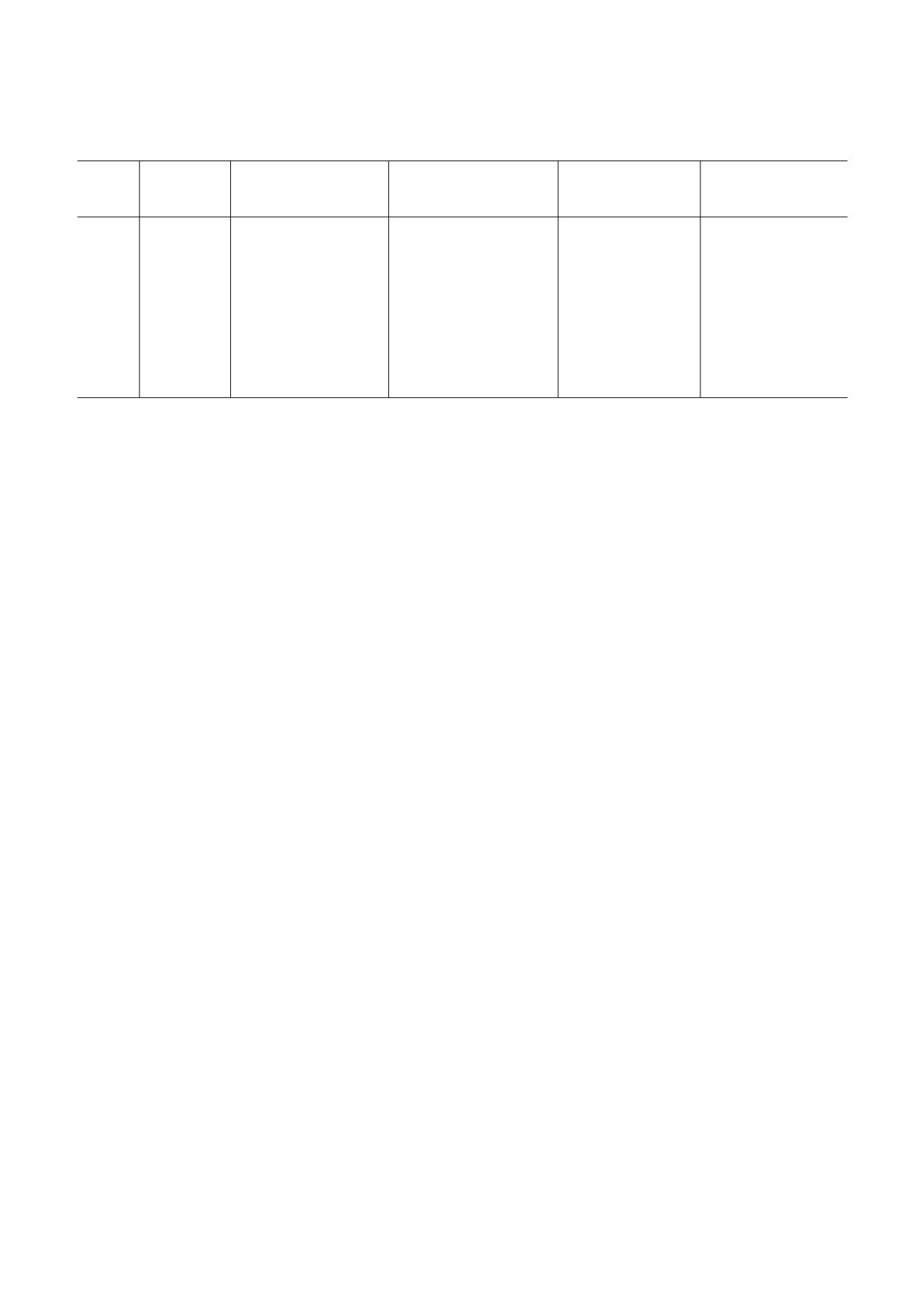
ствии с прилагаемой инструкцией.
ственно. Среди них были обнаружены 581 изве
Биоинформатический анализ. При проведе
стная зрелая miРНК (покрытие
5×) в образцах
нии биоинформатического анализа в представ
сыворотки индивидов группы Р и 558 miРНК -
ленном исследовании в целях всесторонней ха
в образцах сыворотки индивидов группы Н
рактеристики секвенированных последователь
(табл. 2).
ностей малых РНК был использован алгоритм
После этого профили экспрессии miРНК
CAP miRSeq для идентификации, аннотирова
были нормализованы (рис. 1, а, b). С этой целью
ния и количественного определения miРНК в со
была произведена неконтролируемая иерархи
ответствии с руководством пользователя с неко
ческая кластеризация данных по экспрессии
торыми модификациями [24]. Предварительную
miРНК, ее результаты представлены в форме
обработку данных по секвенированию выполня
тепловой карты на рис. 1, c. Значения р при
ли с использованием программы FastQC v. 0.10.1
сравнении различий в экспрессии между образ
с целью выявления низкокачественных резуль
цами от индивидов групп Н и Р были рассчита
татов секвенирования и удаления из анализа
ны и представлены на диаграмме рассеяния
последовательностей адаптерных РНК. Ото
данных (рис. 1, d).
бранные последовательности длиной > 17 нук
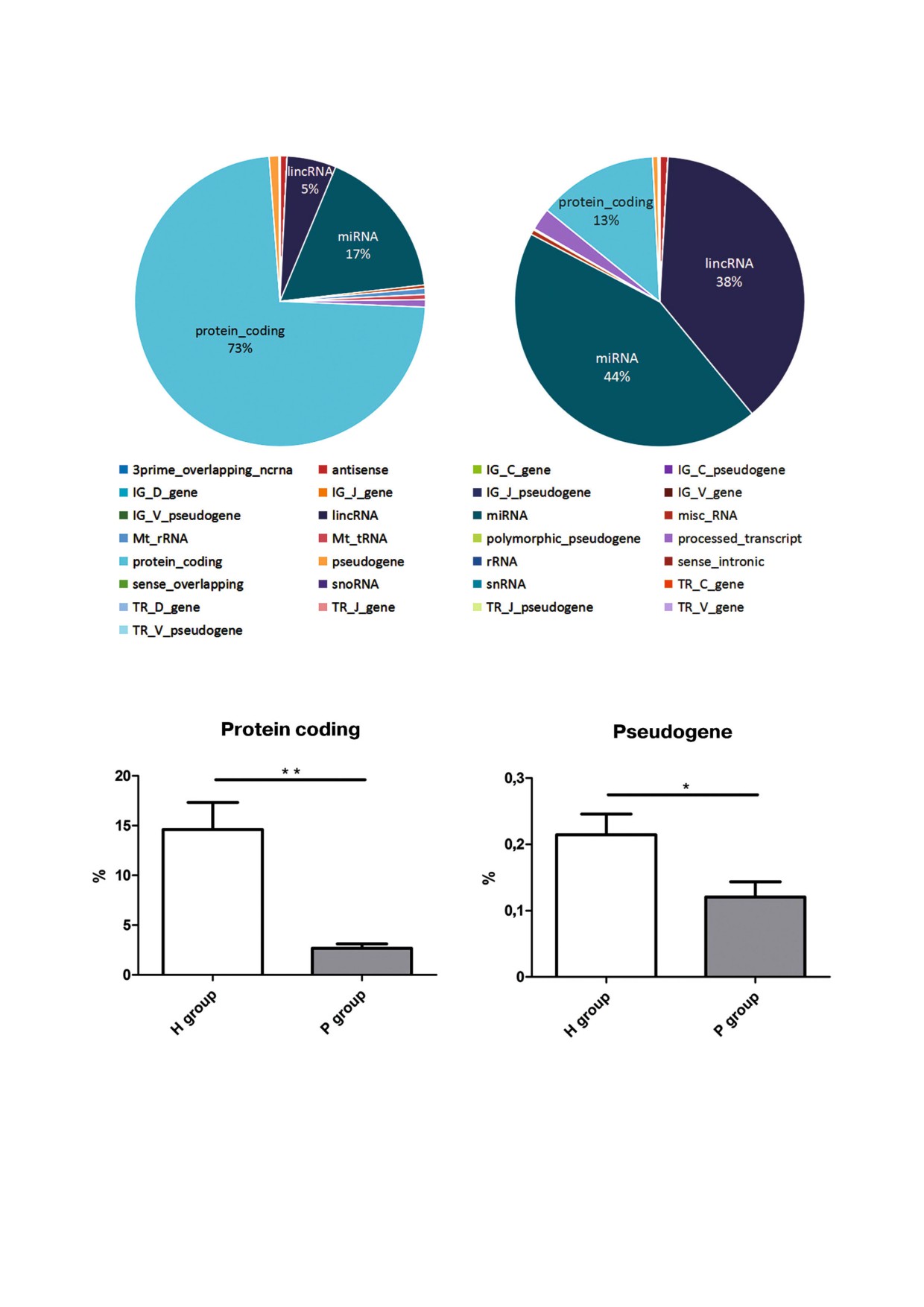
Распределение малых РНК в геноме. При со
леотидов были выравнены относительно генома
поставлении нуклеотидной структуры малых
человека (hg19) и последовательности miRBase 20
РНК с последовательностью генома человека
с использованием программы Bowtie v. 0.12.7 [25].
(hg19) было установлено, что малые РНК коди
Окончательную идентификацию и количест
руются в геномных локусах 27 типов, располо
венное определение известных и предсказанных
женных главным образом в районах кодирова
потенциальных miРНК выполняли с использо
ния белков и miРНК, районах длинных некоди
ванием программы miRDeep v. 2.0.0.5 [26, 27].
рующих РНК (lincРНК) и др. Процент малых
Существенные изменения в уровнях экспрессии
РНК, кодируемых в этих областях генома, сос
различных miРНК между группами здоровых
тавлял 73, 17 и 5% соответственно для индиви
людей (группа Н) и пациентов с PDAC (группа Р)
дов группы Н и 13, 44 и 38% соответственно для
были выявлены с использованием программы
индивидов группы Р (рис. 2, a, b). Среди 27 ге
edgeR [28]. Полученные в этом исследовании
номных локусов с картированными малыми
данные о профилях экспрессии малых РНК
РНК у индивидов группы Н их подавляющее
опубликованы в базе данных GEO (GSE44853).
большинство находится в области генов, коди
Статистическая обработка данных. Все полу
рующих белки (~15%), по сравнению с областя
ченные данные представлены как средние зна
ми некодирующих псевдогенов (~0,2%), тогда
чения ± s.d. Значимость различий между груп
как у индивидов группы Р это преобладание бы
БИОХИМИЯ том 84 вып. 6 2019
ПРОФИЛЬ ЭКСПРЕССИИ miРНК В ПЛАЗМЕ КРОВИ ПРИ PDAC
789
Таблица 2. Основная информация о секвенировании miРНК из образцов плазмы здоровых индивидов (группа Н) и паци
ентов с PDAC (группа Р)
Номер
Общее количество
Количество считанных
Количество
Известные miРНК
Группа
образца
считанных
предшественников
считанных зрелых
с покрытием
5×
последовательностей
miРНК
miРНК
H
ZYFR1
26 423 364
2058
1 376 892
637
ZYFR2
32 198 124
2541
2 273 633
701
ZYFR3
31 026 114
401
398 921
413
ZYFR4
38 297 930
1671
328 423
345
ZYFR5
35 113 682
4372
1 969 065
694
P
ZYFR11
26 084 797
19 447
1 633 708
527
ZYFR12
22 687 051
4655
572 342
291
ZYFR13
30 007 205
13 576
13 892 602
811
ZYFR14
20 235 930
8966
1 452 705
482
ZYFR15
31 147 940
7756
15 118 396
795
ло не столь значительным (~2 и 0,1% соответ
miРНК в плазме крови человека с целью даль
ственно); значения р составляли 0,0024 и 0,0419
нейшего применения полученных знаний в кли
для групп Н и Р соответственно (рис. 2, c, d).
нической практике. В данной работе мы проана
Полученные данные означают, что miРНК де
лизировали профили экспрессии miРНК в плаз
монстрируют дифференциальную экспрессию у
ме крови здоровых индивидов (группа Н) и па
пациентов с PDAC и здоровых индивидов.
циентов с PDAC (группа Р) среди жителей Ки
Дифференциальная экспрессия зрелых miРНК
тая путем анализа профилей экспрессии малых
и новых miРНК в группах Р и Н. С целью выясне
РНК, широко представленных в геноме челове
ния возможного диагностического значения
ка. Было установлено, что уровни экспрессии
идентифицированных нами малых РНК был
165 зрелых miРНК существенно различались у
выполнен дифференциальный анализ с исполь
индивидов групп Н и Р, причем уровни 75 из них
зованием программы edgeR для выявления раз
были повышены, а 95 - снижены у больных по
личий в экспрессии этих РНК у индивидов
сравнению со здоровыми людьми. Наиболее
групп Р и Н. Установлено, что экспрессия 165
значительно возрастали уровни экспрессии двух
miРНК значительно различалась (p < 0,05) у ин
miРНК у индивидов группы Р: hsa miR 182 5p
дивидов обеих групп. Уровни экспрессии 75 из
(logFold Change = 4,00, p = 1,77E 10) и hsa miR
этих зрелых miРНК были существенно повыше
4732 5p (logFold Change = 3,78, p = 4,76E 11),
ны, а уровни 90 miРНК - снижены у индивидов
тогда как наиболее значительно снижались уров
группы Р по сравнению с индивидами группы Н
ни экспрессии двух других miРНК: hsa miR 139 5p
(p < 0,05; табл. S1 Приложения).
(logFold Change = -4,07, p = 3,35E 15) и hsa miR
Кроме того, нами было выявлено 279 новых
23b 3p (logFold Change = -3,93, p = 4,31E 15).
miРНК у индивидов группы Н и 785 новых
Ранее сообщалось о том, что miR 182 5p
miРНК у индивидов группы Р (табл. S2 и S3
(предыдущие названия: miR 182, MiRBase) с из
Приложения), причем 35 из этих новых miРНК
бытком экспрессировалась в различных линиях
были общими у индивидов обеих групп.
клеток PDAC и тканях раковой опухоли и вы
ступала как активатор пролиферации и миграции
раковых клеток путем воздействия на сигналь
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
ный путь β TrCP/β катенин [29]. Кроме того,
сверхэкспрессия miR 182 5p сопровождалась
Раннее диагностирование PDAC является
подавлением экспрессии фактора транскрип
чрезвычайно важным для своевременного нача
ции FOXO3a (forkhead box O3), что влияло на
ла лечения пациентов. Сообщалось о том, что
BIM/Bax зависимый митохондриальный апоп
профиль экспрессии определенных miРНК в
тотический сигнальный путь [30, 31]. В соответ
плазме крови может являться надежным биомар
ствии с полученными нами данными, miR 182 5p
кером для раннего диагностирования PDAC -
присутствовала в плазме крови человека. Кро
чувствительным, специфичным и стабильным.
ме того, Chen et al. [32] показали, что уро
Однако в результате всех проведенных ранее ис
вень циркулирующей miR 182 у пациентов с
следований не было выявлено ни одного подхо
PDAC был значительно выше, чем у здоровых
дящего набора miРНК для использования в ка
людей. Поэтому miR 182 может рассматривать
честве воспроизводимого надежного биомарке
ся как возможный удобный маркер для неинва
ра [19-23]. Нами было проведено изучение
зивного диагностирования PDAC, однако тре
БИОХИМИЯ том 84 вып. 6 2019
790
ZHU и др.
Рис. 1. Ненормализованные (a) и нормализованные (b) статистические данные по экспрессии малых РНК в различных
образцах плазмы крови; c - тепловая карта количественной экспрессии miРНК в образцах плазмы крови индивидов
групп Н (ZYFR1-5) и Р (ZYFR11-15); d - диаграмма рассеяния данных.
biokhsm/
БИОХИМИЯ том 84 вып. 6 2019
ПРОФИЛЬ ЭКСПРЕССИИ miРНК В ПЛАЗМЕ КРОВИ ПРИ PDAC
791
a
b
с
d
Рис. 2. Локусы кодирования в геноме человека малых РНК у индивидов групп Н (a) и Р (b); c - пропорциональные раз
личия (p = 0,0024) между группами Н и Р в кодирующих малые РНК генных локусах, расположенных в районах кодиро
вания белков; d - пропорциональные различия (p = 0,0422) между группами Н и Р в кодирующих малые РНК генных ло
кусах, расположенных в районах псевдогенов.
biokhsm/
БИОХИМИЯ том 84 вып. 6 2019
792
ZHU и др.
буются дополнительные исследования на более
у пациентов с PDAC, что указывает на потенци
многочисленной группе индивидов.
альную диагностическую роль при выявлении
Как было установлено ранее, miR 4732 5p
PDAC.
экспрессируется в молочной железе человека
[33]. Эта miРНК может непосредственно взаи
Итак, нами были установлены профили
модействовать с 5' нетранслируемым районом
экспрессии широко представленных в геноме
мРНК Wrap53 в клетках рака молочной железы,
малых РНК с использованием в качестве источ
препятствуя связыванию мРНК p53 [34]. Это
ника РНК образцов плазмы крови пациентов с
свидетельствует о функциональной взаимосвязи
PDAC и здоровых индивидов. Было установле
между miR 4732 5p и белком р53, являющимся
но, что 165 зрелых miРНК по разному экспрес
супрессором опухолевого роста. Как было пока
сировались у индивидов групп Р и Н. Предпола
зано ранее, р53 играет важную роль на ранней
гается, что эти miРНК могут использоваться в
стадии развития PDAC, что указывает на то, что
качестве биомаркеров для ранней и неинвазив
miR 4732 5p, возможно, также может играть важ
ной диагностики PDAC, когда полученные дан
ную роль в возникновении этого вида рака [35].
ные будут подтверждены в исследованиях с
Результаты исследования функций miR 139 5p
участием большого количества людей.
(предыдущие названия: miR 139, MiRBase) при
раке во многом противоречивы. Li et al. [36] со
общили о сверхэкспрессии miR 139 в клетках
Финансирование. Данная работа была частич
эндотелия раковой опухоли поджелудочной же
но поддержана проектом Департамента науки и
лезы человека, что положительно коррелирова
технологии провинции Хэнань (№ 162102410006),
ло с миграцией раковых клеток и ангиогенезом
Комиссией по здравоохранению и планирова
в ткани опухоли. Эта miРНК могла также содей
нию семьи провинции Хэнань (№ 2017049), На
ствовать прогрессии колоректального рака и
циональным фондом по естественным наукам
плоскоклеточного рака полости рта [37, 38]. Од
Китая (No. 81602362), Программой по науке и
нако результаты некоторых исследований сви
техническому развитию провинции Хэнань
детельствуют о том, что miR 139 является суп
(№ 162102310391), Программой для юных пре
рессором злокачественных новообразований и
подавателей провинции Хэнань (2016GGJS 214),
претерпевает значительную отрицательную ре
вспомогательными грантами Хэнаньского уни
гуляцию в клетках немелкоклеточного рака лег
верситета (№ 2015YBZR048 и B2015151), Стипен
ких (NSCLC), гепатоцеллюлярной карциномы,
диальной программой реки Желтой (№ H2016012),
базальноклеточной карциномы и острой миело
Программой поддержки инновационных талан
идной лейкемии [39-43].
тов в области науки и техники провинции Хэнань
Другой miРНК, подверженной наиболее зна
(№ 18HASTIT048).
чительной отрицательной регуляции у пациен
Благодарности. Авторы горячо признательны
тов группы Р, является hsa miR 23b 3p (преды
проф. Deling Yin (Department of Internal Medicine
дущие названия: miR 23b, MiRBase), однако ее
and Biomedical Sciences, State University’s Quillen
функции все еще изучаются. Проведенные ра
College of Medicine, the East Tennessee) за инструк
нее исследования показали, что экспрессия
тирование и помощь.
miR 23b 3p негативно регулировалась в тканях
Конфликт интересов. Авторы заявляют об от
пациентов с NSCLC [44]. В то же время другие
сутствии конфликта интересов.
исследователи сообщали о том, что miR 23b 3p
Соблюдение этических норм. Исследование
являлась положительно регулируемым факто
было санкционировано комитетом по этике
ром в циркулирующих экзосомах у пациентов с
Второго филиала госпиталя университета
NSCLC [45, 46]. Wang et al. [47] обнаружили, что
Чжанчжоу, Китай (№ лицензии LL201508001).
miR 23b ингибировала аутофагию клеток рака
Все процедуры, выполненные в исследова
поджелудочной железы человека линий BxPC3
нии с участием людей, соответствуют этическим
и Panc 1 путем непосредственного воздействия
стандартам институционального и/или нацио
на ATG12 (autophagy related protein 12) и в даль
нального комитета по исследовательской этике
нейшем повышала устойчивость раковых кле
и Хельсинкской декларации 1964 г. и ее последу
ток к радиоактивному облучению [47]. Обратная
ющим изменениям или сопоставимым нормам
корреляция между экспрессией miR 23b и ATG12
этики.
была обнаружена у больных PDAC. В своей ра
От каждого из включенных в исследование
боте мы выявили, что уровень экспрессии miR
участников было получено информированное
23b 3p в плазме крови был значительно снижен
добровольное согласие.
БИОХИМИЯ том 84 вып. 6 2019
ПРОФИЛЬ ЭКСПРЕССИИ miРНК В ПЛАЗМЕ КРОВИ ПРИ PDAC
793
СПИСОК ЛИТЕРАТУРЫ
1.
Raimondi, S., Maisonneuve, P., and Lowenfels, A.B.
creatic adenocarcinoma: predictive/prognostic biomarkers
(2009) Epidemiology of pancreatic cancer: an overview,
or therapeutic targets? Oncotarget, 6, 23323-23341.
Nat. Rev. Gastroenterol. Hepatol., 6, 699-708.
19.
Liu, R., Chen, X., Du, Y., Yao, W., Shen, L., Wang, C.,
2.
Hartwig, W., Werner, J., Jager, D., Debus, J., and Buchler, M.W.
Hu, Z., Zhuang, R., Ning, G., Zhang, C., Yuan, Y., Li, Z.,
(2013) Improvement of surgical results for pancreatic can
Zen, K., Ba, Y., and Zhang, C.Y. (2012) Serum microRNA
cer, Lancet Oncol., 14, e476-e485.
expression profile as a biomarker in the diagnosis and prog
3.
Lee, R.C., Feinbaum, R.L., and Ambros, V. (1993) The C. ele
nosis of pancreatic cancer, Clin. Chem., 58, 610-618.
gans heterochronic gene lin 4 encodes small RNAs with
20.
Cote, G.A., Gore, A.J., McElyea, S.D., Heathers, L.E.,
antisense complementarity to lin 14, Cell, 75, 843-854.
Xu, H., Sherman, S., and Korc, M. (2014) A pilot study to
4.
Krol, J., Loedige, I., and Filipowicz, W. (2010) The wide
develop a diagnostic test for pancreatic ductal adenocarci
spread regulation of microRNA biogenesis, function and
noma based on differential expression of select miRNA in
decay, Nat. Rev. Genet., 11, 597-610.
plasma and bile, Am. J. Gastroenterol., 109, 1942-1952.
5.
Van Kouwenhove, M., Kedde, M., and Agami, R. (2011)
21.
Wang, J., Chen, J., Chang, P., LeBlanc, A., Li, D., Ab
MicroRNA regulation by RNA binding proteins and its
bruzzesse, J.L., Frazier, M.L., Killary, A.M., and Sen, S.
implications for cancer, Nat. Rev. Cancer, 11, 644-656.
(2009) MicroRNAs in plasma of pancreatic ductal adeno
6.
Rana, T.M. (2007) Illuminating the silence: understanding
carcinoma patients as novel blood based biomarkers of di
the structure and function of small RNAs, Nat. Rev. Mol.
sease, Cancer Prev. Res. (Phila), 2, 807-813.
Cell Biol., 8, 23-36.
22.
Liu, J., Gao, J., Du, Y., Li, Z., Ren, Y., Gu, J., Wang, X.,
7.
Stefani, G., and Slack, F.J. (2008) Small non coding RNAs
Gong, Y., Wang, W., and Kong, X. (2012) Combination of
in animal development, Nat. Rev. Mol. Cell Biol., 9, 219-230.
plasma microRNAs with serum CA19 9 for early detection
8.
Suzuki, H.I., Arase, M., Matsuyama, H., Choi, Y.L.,
of pancreatic cancer, Int. J. Cancer, 131, 683-691.
Ueno, T., Mano, H., Sugimoto, K., and Miyazono, K.
23.
Schultz, N.A., Dehlendorff, C., Jensen, B.V., Bjerregaard, J.K.,
(2011) MCPIP1 ribonuclease antagonizes dicer and termi
Nielsen, K.R., Bojesen, S.E., Calatayud, D., Nielsen, S.E.,
nates microRNA biogenesis through precursor microRNA
Yilmaz, M., Hollander, N.H., Andersen, K.K., and Johan
degradation, Mol. Cell, 44, 424-436.
sen, J.S. (2014) MicroRNA biomarkers in whole blood for
9.
Esquela Kerscher, A., and Slack, F.J. (2006) Oncomirs -
detection of pancreatic cancer, JAMA, 311, 392-404.
microRNAs with a role in cancer, Nat. Rev. Cancer, 6, 259-269.
24.
Sun, Z., Evans, J., Bhagwate, A., Middha, S., Bockol, M.,
10.
An, X., Sarmiento, C., Tan, T., and Zhu, H. (2017)
Yan, H., and Kocher, J.P. (2014) CAP miRSeq: a compre
Regulation of multidrug resistance by microRNAs in anti
hensive analysis pipeline for microRNA sequencing data,
cancer therapy, Acta Pharm. Sin. B, 7, 38-51.
BMC Genomics, 15, 423.
11.
Jafri, M.A., Al Qahtani, M.H., and Shay, J.W. (2017) Role
25.
Langmead, B., Trapnell, C., Pop, M., and Salzberg, S.L.
of miRNAs in human cancer metastasis: implications for
(2009) Ultrafast and memory efficient alignment of short
therapeutic intervention, Semin. Cancer Biol., 44, 117-131.
DNA sequences to the human genome, Genome Biol., 10,
12.
Fattore, L., Costantini, S., Malpicci, D., Ruggiero, C.F.,
R25.
Ascierto, P.A., Croce, C.M., Mancini, R., and Ciliberto, G.
26.
An, J., Lai, J., Lehman, M.L., and Nelson, C.C. (2013)
(2017) MicroRNAs in melanoma development and resis
miRDeep*: an integrated application tool for miRNA
tance to target therapy, Oncotarget, 8, 22262-22278.
identification from RNA sequencing data, Nucleic Acids
13.
Fabbri, M., Paone, A., Calore, F., Galli, R., Gaudio, E.,
Res., 41, 727-737.
Santhanam, R., Lovat, F., Fadda, P., Mao, C., Nuovo, G.J.,
27.
Friedlander, M.R., Mackowiak, S.D., Li, N., Chen, W.,
Zanesi, N., Crawford, M., Ozer, G.H., Wernicke, D.,
and Rajewsky, N. (2012) miRDeep2 accurately identifies
Alder, H., Caligiuri, M.A., Nana Sinkam, P., Perrotti, D.,
known and hundreds of novel microRNA genes in seven
and Croce, C.M. (2012) MicroRNAs bind to Toll like
animal clades, Nucleic Acids Res., 40, 37-52.
receptors to induce prometastatic inflammatory response,
28.
Robinson, M.D., McCarthy, D.J., and Smyth, G.K.
Proc. Natl. Acad. Sci. USA, 109, E2110-E2116.
(2010) edgeR: a bioconductor package for differential
14.
Cortez, M.A., Bueso Ramos, C., Ferdin, J., Lopez
expression analysis of digital gene expression data,
Berestein, G., Sood, A.K., and Calin, G.A.
(2011)
Bioinformatics, 26, 139-140.
MicroRNAs in body fluids - the mix of hormones and bio
29.
Chen, X., Su, Z., Wang, S., and Xu, H. (2016) Clinical and
markers, Nat. Rev. Clin. Oncol., 8, 467-477.
prognostic significance of Arl4c expression in colorectal
15.
Stroun, M., Lyautey, J., Lederrey, C., Olson Sand, A., and
cancer, Cancer Biomark., 16, 253-257.
Anker, P. (2001) About the possible origin and mechanism
30.
Li, Y., Li, A., Wu, J., He, Y., Yu, H., Chai, R., and Li, H.
of circulating DNA apoptosis and active DNA release,
(2016) MiR 182 5p protects inner ear hair cells from cis
Clin. Chim. Acta, 313, 139-142.
platin induced apoptosis by inhibiting FOXO3a, Cell Death
16.
Mitchell, P.S., Parkin, R.K., Kroh, E.M., Fritz, B.R.,
Dis., 7, e2362.
Wyman, S.K., Pogosova Agadjanyan, E.L., Peterson, A.,
31.
Czabotar, P.E., Colman, P.M., and Huang, D.C. (2009)
Noteboom, J., O’Briant, K.C., Allen, A., Lin, D.W.,
Bax activation by Bim? Cell Death Differ., 16, 1187-1191.
Urban, N., Drescher, C.W., Knudsen, B.S., Stirewalt, D.L.,
32.
Chen, Q., Yang, L., Xiao, Y., Zhu, J., and Li, Z. (2014)
Gentleman, R., Vessella, R.L., Nelson, P.S., Martin, D.B.,
Circulating microRNA 182 in plasma and its potential
and Tewari, M. (2008) Circulating microRNAs as stable
diagnostic and prognostic value for pancreatic cancer, Med.
blood based markers for cancer detection, Proc. Natl.
Oncol., 31, 225.
Acad. Sci. USA, 105, 10513-10518.
33.
Persson, H., Kvist, A., Rego, N., Staaf, J., Vallon Chris
17.
Karius, T., Schnekenburger, M., Dicato, M., and
tersson, J., Luts, L., Loman, N., Jonsson, G., Naya, H.,
Diederich, M. (2012) MicroRNAs in cancer management
Hoglund, M., Borg, A., and Rovira, C. (2011) Identifi
and their modulation by dietary agents, Biochem.
cation of new microRNAs in paired normal and tumor
Pharmacol., 83, 1591-1601.
breast tissue suggests a dual role for the ERBB2/Her2 gene,
18.
Brunetti, O., Russo, A., Scarpa, A., Santini, D., Reni, M.,
Cancer Res., 71, 78-86.
Bittoni, A., Azzariti, A., Aprile, G., Delcuratolo, S.,
34.
Pouladi, N., Kouhsari, S.M., Feizi, M.H., Gavgani, R.R.,
Signorile, M., Gnoni, A., Palermo, L., Lorusso, V.,
and Azarfam, P. (2013) Overlapping region of p53/wrap53
Cascinu, S., and Silvestris, N. (2015) MicroRNA in pan
transcripts: mutational analysis and sequence similarity
4 БИОХИМИЯ том 84 вып. 6 2019
794
ZHU и др.
with microRNA 4732 5p, Asian Pac. J. Cancer Prev., 14,
42. Xu, K., Shen, K., Liang, X., Li, Y., Nagao, N., Li, J., Liu, J.,
3503-3507.
and Yin, P. (2016) MiR 139 5p reverses CD44+/CD133+
35.
Maitra, A., Adsay, N.V., Argani, P., Iacobuzio Donahue, C.,
associated multidrug resistance by downregulating
De Marzo, A., Cameron, J.L., Yeo, C.J., and Hruban, R.H.
NOTCH1 in colorectal carcinoma cells, Oncotarget, 7,
(2003) Multicomponent analysis of the pancreatic adeno
75118-75129.
carcinoma progression model using a pancreatic intraepi
43. Chen, H., Xu, H., Meng, Y.G., Zhang, Y., Chen, J.Y., and
thelial neoplasia tissue microarray, Mod. Pathol., 16, 902-912.
Wei, X.N. (2016) miR 139 5p regulates proliferation,
36.
Li, L., Li, B., Chen, D., Liu, L., Huang, C., Lu, Z., Lu, N.,
apoptosis, and cell cycle of uterine leiomyoma cells by tar
and Wan, X. (2015) miR 139 and miR 200c regulate pan
geting TPD52, Onco Targets Ther., 9, 6151-6160.
creatic cancer endothelial cell migration and angiogenesis,
44. Janikova, M., Zizkova, V., Skarda, J., Kharaishvili, G.,
Oncol. Rep., 34, 51-58.
Radova, L., and Kolar, Z. (2016) Prognostic significance of
37.
Miyoshi, J., Toden, S., Yoshida, K., Toiyama, Y., Alberts, S.R.,
miR 23b in combination with P gp, MRP and LRP/MVP
Kusunoki, M., Sinicrope, F.A., and Goel, A. (2017) MiR
expression in non small cell lung cancer, Neoplasma, 63,
139 5p as a novel serum biomarker for recurrence and
576-587.
metastasis in colorectal cancer, Sci. Rep., 7, 43393.
45. Cinegaglia, N.C., Andrade, S.C., Tokar, T., Pinheiro, M.,
38.
Chen, Z., Yu, T., Cabay, R.J., Jin, Y., Mahjabeen, I., Luan, X.,
Severino, F.E., Oliveira, R.A., Hasimoto, E.N., Cataneo, D.C.,
Huang, L., Dai, Y., and Zhou, X. (2017) miR 486 3p,
Cataneo, A.J., Defaveri, J., Souza, C.P., Marques, M.M.,
miR 139 5p, and miR 21 as biomarkers for the detection
Carvalho, R.F., Coutinho, L.L., Gross, J.L., Rogatto, S.R.,
of oral tongue squamous cell carcinoma, Biomark. Cancer,
Lam, W.L., Jurisica, I., and Reis, P.P. (2016) Integrative
9, 1-8.
transcriptome analysis identifies deregulated microRNA
39.
Sun, C., Sang, M., Li, S., Sun, X., Yang, C., Xi, Y., Wang, L.,
transcription factor networks in lung adenocarcinoma,
Zhang, F., Bi, Y., Fu, Y., and Li, D. (2015) Hsa miR 139 5p
Oncotarget, 7, 28920-28934.
inhibits proliferation and causes apoptosis associated with
46. Begum, S., Hayashi, M., Ogawa, T., Jabboure, F.J., Brait, M.,
down regulation of c Met, Oncotarget, 6, 39756-39792.
Izumchenko, E., Tabak, S., Ahrendt, S.A., Westra, W.H.,
40.
Mo, Y., Lu, Y., Wang, P., Huang, S., He, L., Li, D., Li, F.,
Koch, W., Sidransky, D., and Hoque, M.O. (2015) An
Huang, J., Lin, X., Li, X., Che, S., and Chen, Q. (2017)
integrated genome wide approach to discover deregulated
Long non coding RNA XIST promotes cell growth by regu
microRNAs in non small cell lung cancer: clinical signifi
lating miR 139 5p/PDK1/AKT axis in hepatocellular car
cance of miR 23b 3p deregulation, Sci. Rep., 5, 13236.
cinoma, Tumour Biol., 39, 1010428317690999.
47. Wang, P., Zhang, J., Zhang, L., Zhu, Z., Fan, J., Chen, L.,
41.
Sand, M., Skrygan, M., Sand, D., Georgas, D., Hahn, S.A.,
Zhuang, L., Luo, J., Chen, H., Liu, L., Chen, Z., and
Gambichler, T., Altmeyer, P., and Bechara, F.G. (2012)
Meng, Z. (2013) MicroRNA 23b regulates autophagy asso
Expression of microRNAs in basal cell carcinoma, Br. J.
ciated with radioresistance of pancreatic cancer cells,
Dermatol., 167, 847-855.
Gastroenterology, 145, 1133-1143.e12.
DIFFERENTIAL microRNA EXPRESSION PROFILES AS POTENTIAL
BIOMARKERS FOR PANCREATIC DUCTAL ADENOCARCINOMA
Y. Zhu1#, J. Wang2#, F. Wang3, Z. Yan3, G. Liu2, Y. Ma4, W. Zhu5, Y. Li3,
L. Xie3, A. V. Bazhin6*, and X. Guo3*
1 Department of Oncology, International Joint Laboratory for Cell Medical Engineering of Henan Province, Henan
University Huaihe Hospital, Kaifeng, Henan 475000, People’s Republic of China; E mail: celltransplant@163.com
2 Department of Oncology, The Second Affiliated Hospital of Zhengzhou University, Zhengzhou, Henan 450014,
People’s Republic of China; E mail: wj68happy@hotmail.com
3 Department of Preventive Medicine, Cell Signal Transduction Laboratory, Joint National Laboratory for Antibody
Drug Engineering, Institute of Biomedical Informatics, Medical School, Henan University, Kaifeng, Henan 475004,
People’s Republic of China; E mail: xqguo@henu.edu.cn
4 College of Pharmacy and Tianjin Key Laboratory of Molecular Drug Research, Nankai University,
Haihe Education Park, Tianjin 300353, People’s Republic of China; E mail: mayonggang@nankai.edu.cn
5 Department of Anesthesia, Stanford University, CA 94305, USA; E mail: wan.zhu@stanford.edu
6 Department of General, Visceral, and Transplantation Surgery, Ludwig Maximilians University Munich,
Munich 81377, Germany; E mail: alexandr.bazhin@med.uni muenchen.de
Received December 4, 2018
Revised February 2, 2019
Accepted February 4, 2019
Pancreatic ductal adenocarcinoma (PDAC) remains a clinical challenge due to a deteriorated prognosis. Therefore,
the early diagnosis of PDAC is extremely important for achieving curation. MicroRNAs (miRNAs) could serve as a
potential biomarker for the early detection and prognosis of PDAC. In this work, we analyzed plasma samples from
healthy persons and PDAC patients to assess differential miRNA expression profiles by the next generation sequencing
technology and bioinformatics analysis. In this way, 165 mature miRNAs were found to be significantly deregulated
in the patient group, of which 75 and 90 mature miRNAs were up and down regulated compared with healthy per
sons, respectively. Furthermore, 1029 novel miRNAs were identified. In conclusion, plasma miRNA expression pro
files are different in healthy persons and PDAC patients. This data provides a possibility for usage of miRNA as diag
nostic and prognostic biomarkers of PDAC.
Keywords: рancreatic ductal adenocarcinoma, plasma miRNA, expression profiling
БИОХИМИЯ том 84 вып. 6 2019