БИОХИМИЯ, 2019, том 84, вып. 8, с. 1099 - 1114
УДК 575.852
РОЛЬ ОБРАТНОЙ ТРАНСКРИПТАЗЫ
В ВОЗНИКНОВЕНИИ ЖИЗНИ
Обзор
© 2019
Р.Н. Мустафин1*, Э.К. Хуснутдинова2
1 Башкирский государственный медицинский университет,
450008 Уфа, Россия; электронная почта: ruji79@mail.ru
2 Институт биохимии и генетики Уфимского федерального
исследовательского центра РАН, 450054 Уфа, Россия
Поступила в редакцию 08.03.2019
После доработки 14.05.2019
Принята к публикации 14.05.2019
Предполагается, что рибозим РНК полимераза, обладающая активностью обратной транскриптазы и ин
тегразы, сыграла ключевую роль в закономерностях возникновения жизни на Земле. Представлена гипоте
за, согласно которой, благодаря обратной транскриптазе в эволюции были сформированы универсальные
предковые единицы всего живого - ретроэлементы (РЭ). Их склонность к мутациям и способность к взаи
моинтеграции стала основой для образования комплексных структур ДНК, первичных геномов, от которых
произошли все археи, эукариоты, бактерии и вирусы. Консервативные свойства ретроэлементов сохрани
лись на протяжении всей эволюции - при модификациях их использования возникли новые способы взаи
модействий белков и нуклеиновых кислот. Жизнь эволюционировала благодаря инсерционному мутагене
зу и противоборству автономно реплицирующихся полинуклеотидов для сохранения структур с адаптивны
ми свойствами. Сделано предположение, что при отборе механизмов защиты от инсерций на основе рибо
нуклеазной способности рибозима обратной транскриптазы возникли все универсальные ферментативные
системы процессинга молекул РНК. Они стали ключевыми источниками дальнейших эволюционных пре
образований геномов и их регуляторных особенностей. Приведены данные, позволяющие предположить,
что система трансляции, объединившая мир РНК и ДНК с белками, возникла как модификация механиз
мов защиты от инсерций. Образуемые с ее помощью полипептиды стали потенцировать работу рибозимов
в составе рибонуклеопротеинов (РНП) и даже функционально замещать их при большей успешности ката
лиза биологических реакций. Проведен анализ механизмов использования ретроэлементов в структурных и
регуляторных преобразованиях геномов эукариот, которые могут отражать адаптивные принципы, сформи
рованные при зарождении жизни. Параллельно с эволюцией существующих белков, из РЭ возникают и из
меняются рибозимы, такие как длинные некодирующие РНК. Они могут функционировать в комплексе с
белками в составе РНП, способны к самостоятельной каталитической активности и трансляции. Их гены
обладают потенциалом к преобразованию в белок кодирующие. Т.е. консервативные принципы взаиморе
гуляции РНК, ДНК и белков, сформированные при возникновении жизни, используются на протяжении
всей эволюции.
КЛЮЧЕВЫЕ СЛОВА: обратная транскриптаза, полимераза, процессинг, рибозимы, ретроэлементы,
транспозоны, эволюция.
DOI: 10.1134/S0320972519080037
Гипотеза мира РНК при возникновении
щих самостоятельной каталитической актив
жизни на Земле является доминирующей в сов
ностью. Хотя все сохранившиеся в наши дни
ременной науке. Накоплены многочисленные
формы жизни зависят от белковых ферментов, в
данные о существовании рибозимов, обладаю
мире РНК репликацию генетической информа
Принятые сокращения: РНП - рибонуклеопротеины, нкРНК - некодирующие РНК, мяРНК - малые ядерные
РНК, мяоРНК - малые ядрышковые РНК, ТЕ - транспозоны (transposable elements), НП - нуклеотидные последователь
ности, РНКи - РНК интерференция, РЭ - ретроэлементы, CRISPR - короткие палиндромные повторы, регулярно рас
положенные группами (clustered regularly interspaced short palindromic repeats), LTR - длинные концевые повторы (long ter
minal repeats), LINE - длинные диспергированные элементы ядра (long interspersed nuclear element), nonLTR РЭ - ретро
элементы, не содержащие длинные концевые повторы, RISC - РНК индуцированный комплекс выключения гена
(RNA induced silencing complex), RT - обратная транскриптаза (reverse transcriptase), SINE - короткие диспергированные
элементы ядра (short interspersed nuclear elements).
* Адресат для корреспонденции.
1099
4*
1100
МУСТАФИН, ХУСНУТДИНОВА
ции и ее преобразование в функциональные
ное разбавление и разрешает проблему кон
последовательности обеспечивали только моле
центрации, связанную с возникновением жизни
кулы полирибонуклеотидов. В лабораторных ус
[4].
ловиях подобные механизмы могут быть рекон
Мир РНК без белков функционировал еще
струированы и воспроизведены при полном от
до образования клеточных форм жизни - пос
сутствии белков. И в экспериментах рибозимы
ледний универсальный общий предок сущест
оказались способными к саморепликации с экс
вовал ~3,9 миллиардов лет назад [5]. Несмотря
поненциальным ростом [1]. Организмам мира
на это, рибозимы остаются успешными и весьма
РНК требовался рибозим РНК полимераза для
распространенными структурно функциональ
наследования на основе РНК и для экспрессии
ными элементами. К ним относятся консерва
«генов РНК». Данный предковый фермент был
тивные (тРНК, рРНК, малые ядерные РНК
утрачен в ходе эволюции. Однако ключевые
(мяРНК), малые ядрышковые РНК (мяоРНК),
функциональные аспекты репликации РНК ка
рибозимы сплайсосомы) и изменчивые (интро
тализируемой РНК можно изучить «by proxy» с
ны группы I и группы II, некодирующие РНК
использованием генерируемых селекцией in
(нкРНК)) полирибонуклеотиды. Первый рибо
vitro современных рибозимов, таких как РНК
зим, интрон группы I, был открыт у инфузории
полимераза R18 [2].
Tetrahymena еще в 1982 году [6]. В 1983 году об
В 2016 году Horning и Joyce опубликовали
наружен рибозим рибонуклеаза Р, участвующая
данные об успешном использовании in vitro ри
в процессинге молекул тРНК [7]. В 1986 году
бозима РНК полимеразы. Она была способна
описаны рибозимы типа hammerhead (молоточ
реплицировать короткие последовательности
ковые) вирусов сателлитов растений [8]. Их
РНК без белка, синтезируя разнообразные
многочисленные вариации оказались распрост
функциональные молекулы, в том числе апта
раненными у представителей всех царств живо
меры и рибозимы. Продемонстрирована полная
го [9]. В последние годы опубликованы работы о
самодостаточность репликации и экспрессии
роли рибозимов длинных нкРНК в возникнове
молекул РНК. Модулирование активности ри
нии новых белок кодирующих генов [10-13],
бозимов in vitro позволило воспроизвести само
регуляции работы генома в онтогенезе и взаи
поддерживающуюся эволюцию РНК. Получен
мосвязи с мобильными генетическими элемен
ные результаты могут отражать процессы, сход
тами, транспозонами (ТЕ - transposable ele
ные с зарождением жизни на Земле [3] при под
ments) [14, 15]. Эти данные могут отражать древ
ходящих неравновесных пограничных условиях
ние эволюционные принципы, сформирован
преодоления эффектов термодинамического
ные при возникновении жизни во время перехо
равновесия, таких как разбавление и деградация
да мира РНК-ДНК в мир с участием белков.
олигонуклеотидов [4].
Важным условием для экспоненциального
Предполагается, что поток тепловой энергии
роста количества и разнообразия полинуклео
через открытые поры в подводных горных
тидов на Земле была возможность самовоспро
породах способствовал репликации олигонук
изведения более стойких, по сравнению с РНК,
леотидов с селекцией более длинных цепочек. В
биополимеров ДНК, сохраняющих необходи
эксперименте с использованием взаимодей
мые для выживания адаптивные последователь
ствия молекулярного термофореза и ламинар
ности. Ключевым событием для этого стало об
ной конвекции для обеспечения экспоненци
разование обратной транскриптазы (RT
-
альной репликации, цепочки из 75 нуклеотидов
reverse transcriptase), которая обеспечивает счи
выживали, тогда как более короткие разруша
тывание генетической информации с полимера
лись. Взаимодействие данных двух механизмов
РНК на ДНК. Сохранение RT в геномах всех ор
основано на уравновешивании энтропии сме
ганизмов говорит о ее глобальном значении на
шивания. При разнице температур термофорез
протяжении эволюции. При возникновении
направляет молекулы горизонтально от теплой
жизни RT могла быть рибозимом, обеспечиваю
стороны к холодной. В то же время жидкость
щим ДНК новым генетическим материалом да
движется вертикально путем ламинарной кон
же от одного источника. Это связано с аккуму
векции и уносит с собой молекулы. Конвекция
лированием молекулами РНК мутаций, вероят
отклоняет горизонтальное термофоретическое
ность которых значительно выше (10-3-10-4),
истощение и усиливает вертикальное накопле
чем у ДНК (10-5-10-8) [16]. Действительно, был
ние молекул. Это взаимодействие молекулярно
получен рибозим РНК полимераза, обладаю
го движения и потока жидкости приводит к эф
щая свойствами RT в качестве вторичной функ
фективному сетевому транспорту олигонуклео
ции [17]. Кроме того, выявлена взаимосвязь
тидов в нижнюю часть компартмента. В резуль
между структурами белковых доменов RT и ри
тате накопление уравновешивает диффузион
бозимами [18], что говорит об их преемствен
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1101
ности в эволюции и альтернативном использо
скрипты, которые создавали механизмы проти
вании в идентичных биологических процессах,
воборства инсерциям в наиболее важные адап
а также о древнем происхождении белка RT.
тивные области ДНК. Данные защитные меха
В связи с этим можно предположить, что свой
низмы стали основой для возникновения корот
ства белковых аналогов рибозимов могут отра
ких, регулярно чередующихся, палиндромных
жать сходные характеристики их предковых
повторов (систем CRISPR, clustered regularly
РНК предшественников.
interspaced short palindromic repeats) и РНК ин
Белок RT обладает активностью рибонукле
терференции (РНКи), систем сплайсосомного
азы Н [19]. В то же время, к аналогам данного
сплайсинга и трансляции. Условием для их об
фермента, относятся Argonaute, Dicer, Cas9,
разования стала рибонуклеазная активность ри
транспозазы, интегразы и системы сплайсинга
бозима RT. Наиболее вероятно, что рРНК и
[20]. Поэтому, вероятно, рибозимы, обладаю
тРНК, а также участвующие в их созревании ри
щими свойствами данных ферментов, могли
бозимы процессинга первично были направле
произойти от RT при ее модификациях под вли
ны на защиту от инсерций (рис. 1). Подтвержде
янием мутаций. Т.е. рибозим RT обладал потен
нием служит сохранение в эволюции взаимо
циалом обеспечивать не только обратную тран
связей рРНК, тРНК, мяРНК, мяоРНК с ретро
скрипцию, но и интеграцию образованных мо
элементами [21-25].
лекул полинуклеотидов, а также процессинг мо
В настоящее время доказано, что RT играет
лекул РНК. Это способствовало эволюционно
ключевую роль в эволюции всех форм жизни.
му успеху в связи удлинением комплексных
В геномах эукариот РЭ наиболее распростране
структур ДНК.
ны и занимают от 3 до 85% всех нуклеотидных
Высокая мутабельность РНК являлась при
последовательностей (НП) ДНК [26]. Согласно
чиной изменчивости первичных геномов, но
представляла угрозу для сохранения адаптивных
repbase/), РЭ относятся к I классу ТЕ [27]. Они
свойств полинуклеотидов, необходимых для их
могут быть автономными и неавтономными.
стабильного самовоспроизведения. Выживали
Среди последних, основную долю у эукариот за
такие комплексные молекулы ДНК и их тран
нимают короткие диспергированные элементы
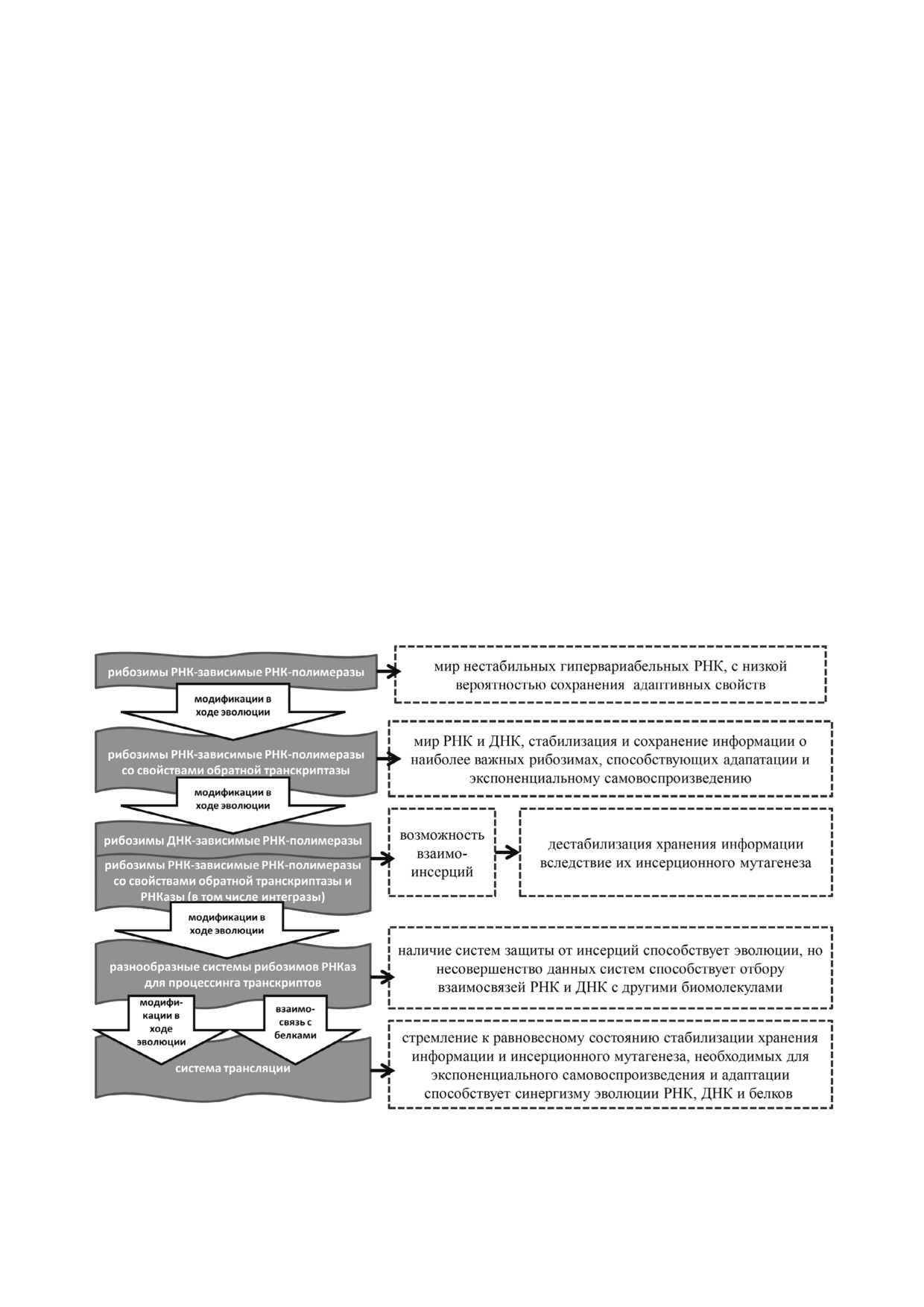
Рис. 1. Эволюция рибозимов при возникновении жизни на Земле
БИОХИМИЯ том 84 вып. 8 2019
1102
МУСТАФИН, ХУСНУТДИНОВА
ядра (SINE, short interspersed nuclear elements)
при модификации которых возникали новые
[28]. Они перемещаются из одного локуса гено
системы процессинга.
ма в другие путем «копирования и вставки» при
Предполагается, что решающим событием в
помощи ферментов длинных диспергирован
происхождении жизни было появление молеку
ных элементов ядра (LINE, long interspersed
лы РНК, способной реплицировать первобыт
nuclear elements). В то же время SINE имеют об
ный «геном» РНК. При этом самые ранние и
щие НП и свойства с ретроэлементами, не со
простые биологические процессы зависели от
держащими длинных концевых повторов (non
РНК как для наследственности, так и для мета
LTR РЭ) [29]. SINE являются производными
болизма. Доказательствами мира РНК на ран
тРНК, мяРНК, 5S рРНК [21, 24, 29-31], про
них этапах эволюции могут служить современ
дукты процессинга которых участвуют в РНКи
ные данные о центральной каталитической роли
транспозонов [23, 25]. Это свидетельствует о
РНК в сплайсинге, экспрессии генов и трансля
взаимосвязи РЭ с возникновением системы
ции, а также о многосторонних функциях РНК
трансляции на ранних этапах эволюции жизни.
в формировании специфических рецепторов и
Подтверждением данного предположения и
катализаторов [2]. При зарождении жизни в ка
эволюционного родства ретроэлементов, содер
честве универсальных единиц могли служить
жащих длинные концевые повторы (LTR РЭ) с
ТЕ подобные элементы. Их взаимодействие и
nonLTR РЭ, могут служить данные об использо
противоборство при формировании комплекс
вании молекул тРНК в качестве праймеров для
ных первичных геномов послужило основой для
обратной транскрипции LTR РЭ [28].
всех последующих эволюционных процессов.
Ко II классу ТЕ относятся ДНК транспозо
Однако из за высокого уровня мутабельности
ны (ДНК ТЕ), которые могут перемещаться по
НП транспозонов [16] их глобальную роль в
механизму
«катящегося кольца» (Helitron,
формировании геномов современных организ
Maverick) и «вырезания-вставки» (TIR, Crypton)
мов трудно идентифицировать. В работе de
[32]. Они кодируют фермент транспозазу, ката
Koning et al. при анализе ДНК человека с по
лизирующую вырезание ДНК ТЕ и ее интегра
мощью специфических олигонуклеотидов, бы
цию в новую область генома [28]. Филогенети
ло обнаружено, что ТЕ занимают >67% всех НП,
ческий анализ показал, что белки ДНК ТЕ име
а не 45%, как считалось ранее [37]. Современ
ют древнейшее происхождение и содержат
ные методы анализа позволяют выявлять «скры
складки (специфические структурные мотивы
тые» ТЕ в геномах разных видов животных для
белка), которые существовали еще в период об
определения их роли в формировании регуля
разования самых первых клеточных организмов
торных элементов и белок кодирующих генов в
[33]. При возникновении жизни основное зна
ходе эволюции. В результате обнаруживается
чение имели энзимы, необходимые для ампли
значительно большее количество НП, произо
фикации биополимеров и увеличения их длины.
шедших от ТЕ [38].
Механизм изменчивости путем
«выреза
Вероятно, вся имеющаяся в мире биомасса
ния-вставки» стал актуален на более поздних
была образована благодаря ТЕ в качестве уни
этапах эволюции, поэтому можно предполо
версальных единиц, начиная с зарождения жиз
жить, что ДНК ТЕ произошли от РЭ. Под
ни на Земле. В качестве подтверждения можно
тверждением является появление у RT в качест
привести работу Horning и Joyce [3], в которой
ве вторичной активности доменов рибонуклеа
была спроектирована искусственная эволюция
зы Н [19], которые характерны для сходных по
РНК in vitro путем уменьшения точности рабо
молекулярной структуре транспозазы и инте
ты рибозима в безбелковой среде с формирова
гразы [34, 35]. Рибонуклеаза Н относится к су
нием 10% ошибок при элонгации новой цепи
персемейству нуклеотидил трансфераз, в состав
РНК. Случайные мутации на каждый десятый
которого входят также транспозаза, ретровирус
нуклеотид по всем молекулам РНК привели к
ная интеграза, резольваза и нуклеаза РНК ин
образованию 1014 различных вариантов РНК.
дуцированного комплекса выключения гена
Это стало доказательством эффективности эво
(RISC, RNA induced silencing complex) [36].
люции при участии полимераз, способных об
Ферменты, подобные рибонуклеазе Н, являют
разовывать большое количество ошибок [3].
ся участниками универсальных консервативных
В результате поддерживалась эффективная из
для всего живого защитных систем против виру
менчивость при переходе из мира РНК в мир с
сов и ТЕ, включая RISC (Argonaute, Dicer) и
участием ДНК, РНК и белков при возникнове
CRISPR (Cas9) [20]. В связи с возможной преем
нии жизни на Земле. Это согласуется с харак
ственностью доменов РНК и белков в эволюции
терными свойствами большинства RT образо
[18], рибозимы RT также могли обладать свой
вывать ошибки из за отсутствия корректирую
ствами одновременно репликаз и рибонуклеаз,
щего домена (3'-5' экзонуклеазы). Эволюцион
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1103
ными предшественниками RT считаются РНК
послужила не только ключевым звеном для пе
зависимые РНК полимеразы, которые, как
рехода мира РНК в мир РНК-ДНК-белки, но и
правило, тоже не содержат 3'-5' экзонуклеазно
сохранила свои универсальные способности уско
го домена, ответственного за корректирующую
рять эволюцию на всех ее последующих этапах.
активность [39]. Вероятно, сохранение способ
Предшественниками RT считаются РНК зави
ности формировать ошибки говорит о сохране
симые РНК полимеразы [39], которые при воз
нии древних свойств RT, играющих глобальную
никновении жизни были представлены рибози
роль в эволюции всего живого. Сходство доме
мами [2, 3]. Поэтому древняя RT также могла
нов рибозимов и их белковых аналогов [18] поз
быть рибозимом, способным синтезировать
воляет предположить, что в мире РНК обратная
ДНК на основе матрицы РНК, что и было дока
транскриптаза также обладала данным свой
зано [17].
ством.
Использование нуклеозидтрифосфатов для
Согласно альтернативной гипотезе, предло
удлинения молекул РНК или ДНК является
женной в 1999 году Freeland et al. [40], ДНК в
фундаментальным свойством для всех живых
триаде РНК-ДНК-белок появилась последней.
организмов. Реакция при помощи РНК рибо
Преимуществом предлагаемой авторами гипо
зима сходна с функционированием теломеразы,
тезы является их утверждение, что биохимичес
специализированной ДНК полимеразы, в сос
кое восстановление рибонуклеотидов до дезок
тав которой входит короткая РНК, служащая
синуклеотидов выходит за рамки каталитичес
матрицей для синтеза ДНК теломерных повто
ких способностей РНК [40]. Однако авторы не
ров. Предполагается, что рибозимы, сходные по
учли возможности абиогенеза мономеров ДНК
активности с нуклеотидил трансферазой, ис
без промежуточной стадии рибонуклеотидов.
пользовались как теломеразы еще до появления
Кроме того, было доказано, что для механизма
белкового катализа. Хотя тРНК нуклеотидил
трансляции требуется большее количество нас
трансферазы прежде всего ответственны за до
ледственной информации, чем может поддер
бавление и поддержание CCA 3' хвоста акцеп
живаться РНК геномами [17]. Это обосновыва
торного стебля тРНК, данный клеточный фер
ет предлагаемую нами гипотезу, согласно кото
мент может функционировать в качестве «тело
рой белок в эволюции появляется последним.
меразы» для РНК вирусных геномов, которые
Наиболее вероятно, система трансляции воз
принимают тРНК подобные структуры на их
никла, как один из множества механизмов за
3' концах [46]. В этом отношении имеются дан
щиты первичных геномов ДНК от инсерций с
ные о тесной связи тРНК с РЭ у эукариот [21,
использованием каталитических доменов поли
25, 28], что может быть отражением их эволюци
пептидов в комплексе с рибозимами. В пользу
онного родства. В качестве универсальных мо
этого предположения говорят данные о роли
лекул посредников тРНК первоначально могли
продуктов процессинга тРНК, мяоРНК, рРНК
использоваться, как необходимое звено для по
[23, 25] в РНКи транспозонов.
лимеризации РНК и ДНК (возможно, еще до
появления рибозима интегразы и удлинения
ДНК при помощи инсерций). Дальнейший от
ОСОБЕННОСТИ ОБРАТНОЙ
бор определенных тРНК, обладающих свой
ТРАНСКРИПТАЗЫ
ством взаимодействия с аминокислотами при
участии различных рибозимов, позволил объе
Семейство RT имеет единое древнее эволю
динить мир полинуклеотидов с миром белков за
ционное происхождение, что было доказано на
счет универсального для всего живого триплет
основании гомологии аминокислот и наличия
ного кода. Подтверждением данного предполо
RT во всех доменах жизни [39]. Фермент RT от
жения служит тесная взаимосвязь тРНК c рет
ветственна за «превращение» РНК в кДНК и не
роэлементами, которые содержат комплемен
обходима для инвазии РЭ и их распространения
тарные им НП и используют тРНК в качестве
в геномах. RT впервые описана в 1970 году дву
праймеров [19]. В живой природе у инфузорий
мя исследовательскими группами у РНК содер
Tetrahymena thermophila был выделен естествен
жащего вируса лейкемии мышей и вируса сар
ный рибозим, катализирующий полимериза
комы Рауса [41, 42]. Бактериальные RT были
цию РНК за счет удлинения РНК праймера.
открыты у РЭ, известных как ретроны, в 1989
Это является одним из доказательств теории са
году на Myxococcus xanthus [43] и Escherichia coli B
морепликации пребиотической РНК [47]. В ла
[44]. Более 50% идентифицированных на сегод
бораторных условиях было показано, что неста
няшний день бактериальных RT кодируются
бильные нефункциональные комплексы, соб
интронами группы II, которые являются мо
ранные из более коротких 3' усеченных олиго
бильными РЭ со свойствами рибозимов [45]. RT
нуклеотидов, могут стабилизироваться и обре
БИОХИМИЯ том 84 вып. 8 2019
1104
МУСТАФИН, ХУСНУТДИНОВА
тать функциональность путем неферментатив
зим РНК полимераза способна функциониро
ного удлинения праймера [48].
вать в качестве RT. Данная RT активность име
Предполагается, что, в отличие от RT, у дру
ла решающее значение для перехода от РНК к
гих семейств ДНК полимераз в ходе эволюции
ДНК геномам при возникновении жизни на
развились корректирующие механизмы для по
Земле. Предполагается, что она могла возник
вышения точности синтеза ДНК при реплика
нуть как вторичная функция рибозима РНК за
ции генома. Чтобы определить, является ли от
висимой РНК полимеразы [17]. Хотя РЭ коди
сутствие корректирующей активности истори
руют и другие белки, повышающие эффектив
ческим совпадением или функциональным ог
ность обратной транскрипции, их RT обладает
раничением обратной транскрипции, была раз
двойной ферментативной активностью. В каче
работана термостабильная ДНК полимераза
стве ДНК полимеразы она может копировать
высокой надежности для эффективного исполь
как РНК, так и ДНК матрицу, а в качестве рибо
зования матричных РНК. В результате оказа
нуклеазы Н расщепляет РНК в составе гибрида
лось, что корректура совместима с обратной
РНК/ДНК [19].
транскрипцией [39]. Вероятно, полученные
Наличие у RT активности рибонуклеазы Н
данные свидетельствуют об эволюционном со
позволяет предположить ее глобальную роль в
хранении древнего типа RT, так как амплифика
возникновении ключевых систем процессинга
ция с ее помощью сопровождается выраженной
транскриптов живых систем. Это связано с при
мутабельностью, необходимой для изменчивос
надлежностью рибонуклеазы Н к суперсемей
ти и обретения новых адаптивных способнос
ству нуклеотидил трансфераз, к которому отно
тей. Это универсальное свойство, обеспечиваю
сятся транспозаза, ретровирусная интеграза, ре
щее амплификацию ДНК с ее изменчивостью,
зольваза и RISC нуклеаза Argonaute [36]. В эво
могло быть ключевым моментом для возникно
люции имеется преемственность строения
вения жизни.
пространственных доменов РНК рибозимов и
Сходство структур белковых доменов RT и
белковых ферментов, выполняющих аналогич
рибозимов [18] говорит о возможных эволюци
ную функцию [18]. Поэтому весьма вероятно,
онных путях преобразований каталитических
что при возникновении жизни модификации
РНК молекул в белковые, об их тесной взаи
рибозима RT могли обладать рибонуклеазной
мосвязи и взаимозаменяемости (рис. 2). Пред
активностью и стать основой для систем про
шественниками RT считаются РНК зависимые
цессинга.
РНК полимеразы [39], предковыми формами
При сравнении структур ретровирусных ин
которых были РНК рибозимы. Поэтому логич
теграз, транспозаз и сайтов связывания с ними
но предположить наличие рибозимов RT в жи
в ДНК, оказалось, что оба фермента могут об
вой природе. Действительно, получены доказа
ладать общими биохимическими и генетичес
тельства того, что высокоорганизованный рибо
кими особенностями. Это говорит о возможном
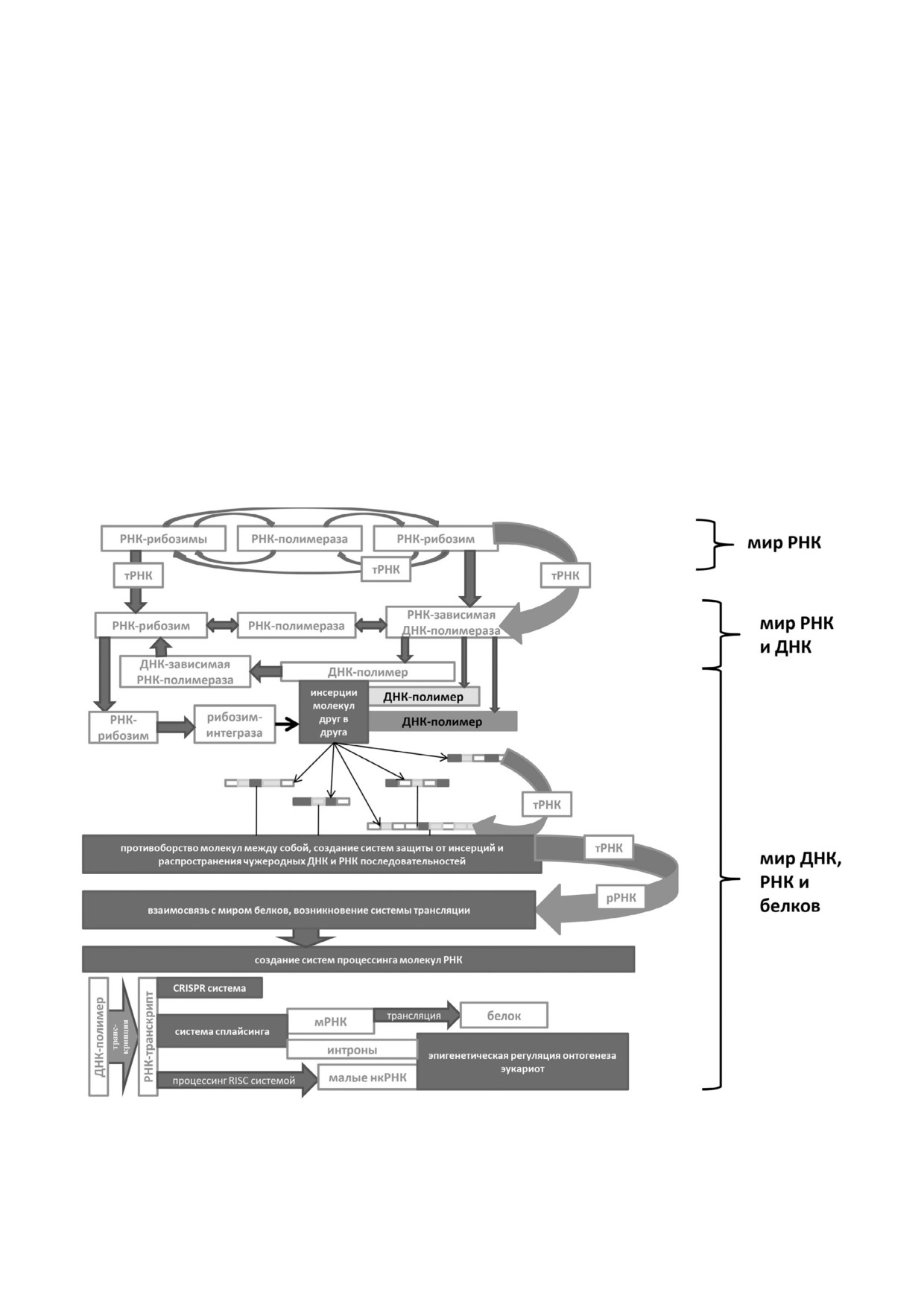
Рис. 2. Взаимосвязь рибозимов и белковых ферментов в эволюции
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1105
их эволюционном происхождении от единых
ВОЗНИКНОВЕНИЕ СИСТЕМЫ
предковых последовательностей [34]. Транспо
ТРАНСЛЯЦИИ И ПРЕЕМСТВЕННОСТЬ
заза и ретровирусная интеграза имеют общий
ФУНКЦИЙ РИБОЗИМОВ БЕЛКАМИ
домен, подобный рибонуклеазе Н, с каталити
ческой триадой DDE/D, которая координирует
Отбор и возникновение рибозимов RT из
двухвалентные катионы, необходимые для рас
РНК полимераз был необходимым этапом для
щепления ДНК и интеграции [35]. Описаны хи
сохранения и преумножения компонентов сис
мерные рибозимы, обладающие свойствами
тем автономно реплицирующихся РНК. Это
разных ферментов, что подтверждает возмож
обусловлено большей стабильностью молекул
ность многофункциональности древней RT.
ДНК с сохранением адаптивного генетического
Так, у бактерии Clostridium difficile был обнару
материала. При его считывании обратно в РНК
жен рибозим, имеющий черты интронов груп
при помощи рибозима ДНК зависимой РНК
пы I и инсерционных элементов [49]. РЭ коди
полимеразы образовывались успешные для амп
руют интегразу и RT как единый полипептид,
лификации молекулы полинуклеотидов. Подоб
который посттранскрипционно процессирует
но миру РНК, в котором при модификации ри
ся при помощи кодируемой этим же РЭ протеа
бозима RT возникла интегразная функция,
зой [50]. Приведенные данные позволяют пред
множественные инсерции способствовали эво
положить, что модификации рибозима RT мог
люции и преумножению систем автономно реп
ли обладать дополнительными свойствами ри
лицирующихся полинуклеотидов. Однако это
бонуклеаз и интеграз. Механистическое сход
стало угрозой для стабильных ДНК геномов
ство ферментов ДНК транспозазы и интегразы
вследствие накопления мутаций, когда вновь
РЭ подчеркивает их тесную эволюционную вза
возникшие адаптивные свойства нарушались
имосвязь. Считается, что ретровирусы про
новыми инсерциями. В результате, в ходе отбо
изошли от LTR РЭ Ty3/Gypsy. Копии их ДНК
ра стали возникать механизмы противоборства
можно найти в геномах большинства живых су
геномов в виде систем процессинга РНК моле
ществ [51]. Эволюционное родство и взаимос
кул. Основой для данных способов защиты яв
вязь различных ферментов, ответственных за
лялись модифицированные доменные структу
транспозиции, свидетельствуют о возможном
ры RT-рибонуклеазы. Наиболее вероятно, ри
сценарии возникновения жизни от РЭ в качест
бозимы РНК полимеразы и RT обладали спо
ве структурно функциональных единиц, от ко
собностью при их модификации вследствие му
торых произошли другие ТЕ. Их противобор
табельности становиться источниками различ
ство и взаимодействие, наряду со способностью
ных систем процессинга, направленных на за
к амплификации за счет RT, могло стать осно
щиту от инсерций. Отражением данной способ
вой для эволюции.
ности являются сходные процессы в мире бел
Взаимопаразитизм ТЕ привел к распростра
ков, так как белок RT обладает свойствами ри
нению первично не кодирующих необходимых
бонуклеазы Н [19]. Члены ее суперсемейства яв
для транспозиции ферментов неавтономных
ляются основными участниками систем процес
РЭ. Эти элементы содержат НП, играющие важ
синга транскриптов всего живого (РНК интер
ную роль в регуляторных процессах. Например,
ференции, CRISPR, сплайсинга [20, 36]).
Alu РЭ ассоциированы со стимуляцией экспрес
Одним из механизмов процессинга и защи
сии генов и обладают высоким потенциалом ре
ты в мире РНК-ДНК могла стать система
гуляции в зависимости от состояния хроматина
трансляции. Этим можно объяснить преем
[52]. Alu относятся к SINE и произошли в эво
ственность доменов рибозимов и белков в эво
люции от обратнотранскрибированных 7SL
люции, их взаимосвязь в регуляции единых био
РНК. Последние входят в состав РНП в качест
логических процессов. Возможность управле
ве рибозима и составной части сигнал распоз
ния при помощи белков реакциями, в которых
нающей частицы (SRP), необходимой для
не участвовали рибозимы, также подтверждает
транспорта рибосом через трансмембранные
данное предположение, так как отбор и консер
поры. Интересно, что вторичные структуры SRP
вативность полипептидов с целью усовершен
животных, растений, грибов, бактерий и архей
ствования защиты подразумевает преимущество
очень схожи [22], что говорит о древнем эволю
их использования перед рибозимами. Первона
ционном происхождении 7SL РНК. Это дает ос
чально рРНК, тРНК и процессирующие их ри
нование предположить возникновение SRP от
бозимы могли произойти от ТЕ и выполнять
ТЕ, самых первых универсальных источников
иные функции, необходимые для противобор
всех генов в эволюции. Alu и другие SINE, а так
ства первичных геномов, в том числе в составе
же 7SL РНК процессируются ферментом Dicer с
других систем защиты. Так, у РЭ и ретровирусов
образованием малых нкРНК [53].
тРНК используется в качестве праймеров [19],
БИОХИМИЯ том 84 вып. 8 2019
1106
МУСТАФИН, ХУСНУТДИНОВА
что может отражать древнее эволюционное
мер, R element, MITE, Pokey характеризуются
свойство тРНК. Кроме того, рРНК [23, 54],
инсерциями в мультигенные рДНК [60], а ретро
мяРНК [23], тРНК [55] и мяоРНК [56, 57] под
элемент R2 - в области генов 28S рДНК [61].
вергаются неслучайному процессингу с исполь
При рассмотрении других систем процес
зованием полученных продуктов для РНКи
синга РНК, обнаруживается та же закономер
транспозонов [23, 25, 55, 58]. Рибозимы могут
ность тесной связи и эволюционного родства
участвовать в разных процессах и выполнять са
всех их компонентов с ТЕ. К примеру, сплайсо
мые разнообразные функции. Например, тРНК
сомные интроны эволюционировали от интро
используется не только в качестве праймера, но
нов группы II. Последние являются ТЕ со свой
также для транспортировки аминокислот и
ствами рибозимов и RT активностью [62]. В то
РНКи. Белковые ферменты также могут участ
же время у эукариот ТЕ служат источниками
вовать в различных процессах и выполнять раз
сплайсосомных интронов [63, 64], сигналов
личные функции - RT теломеразы (TERT), по
сплайсинга [65, 66], энхансеров и сайленсеров
мимо удлинения теломер, используется для регу
сплайсинга [67, 68], структурно функциональ
ляции транскрипции генов [59]. Эволюционная
ных компонентов самой сплайсосомы [69]. Кро
взаимосвязь рибозимов системы трансляции с
ме того, подобно ТЕ, сплайсосомные интроны
транспозонами отражается также в сайт специ
подвергаются процессингу специфическими
фических особенностях интеграций ТЕ. Напри
рибонуклеазами с образованием нкРНК [70].
Рис. 3. Схема эволюции систем процессинга как защитных механизмов
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1107
Благодаря возникновению системы транс
ницы (28S) [31]. У насекомых описаны химер
ляции, живые организмы перешли на новый
ные SINE, HaSE3, состоящие из тРНК и 5S
уровень структурно функциональной органи
рРНК [30]. Кроме того, в геномах крокодилов,
зации. В результате, в противоборствующих ге
аллигаторов и гавиалов идентифицированы
номных системах белковые ферменты могли за
SINEU, происходящие от мяРНК U1 и U2 [24],
мещать функции рибозимов, действовать в
что может свидетельствовать об эволюционном
комплексе с ними в виде РНП и образовывать
родстве системы трансляции и сплайсинга. Это
домены с новыми не присущими рибозимам
согласуется с тем, что мяРНК также подверга
функциями. Несмотря на это, в эволюции со
ются неслучайному процессингу [23] для РНКи
хранились консервативные, специфичные ис
последовательностей ТЕ в геноме. Таким обра
ключительно для рибозимов способности, кото
зом, при возникновении жизни компоненты,
рые служат доказательствами их роли в возник
необходимые для взаимосвязи мира РНК и
новении системы трансляции. Так, в составе
ДНК с белками, были образованы благодаря RT
рибосомы синтезом белка управляют непосред
и ТЕ. Основные принципы данных закономер
ственно молекулы рРНК. Было показано, что
ностей сохраняются в эволюции (рис. 3).
каталитическая активность рибосом обусловле
на активными сайтами, состоящими только из
РНК. В отличие от рРНК, рибосомальные бел
ЗНАЧЕНИЕ ОБРАТНОЙ ТРАНСКРИПТАЗЫ
ки находятся на значительно большем расстоя
В ЭВОЛЮЦИИ ПРОКАРИОТ
нии от активных центров рибосом [71]. Не ис
ключено, что рибосомальные рибозимы прои
Хотя RT считаются ферментами эукариот,
зошли от модифицированных ТЕ в ходе форми
они весьма распространены у бактерий [72]. RT
рования систем противоборства с другими эго
обеспечивают обмен генами между плазмидами
истическими элементами. Подтверждением яв
и нуклеоидом, что необходимо для передачи
ляется участие нкРНК, образованных при про
адаптивных признаков между отдельными клет
цессинге рРНК, в РНКи транспозонов [23].
ками и их потомками [73]. РЭ (ретроны) в гено
Кроме того, наиболее вероятно, что эволюци
мах прокариот впервые описаны еще в 1989 году
онными предшественниками тРНК также явля
у E. coli [44] и у M. xanthus [43]. В 2002 году выяв
лись ТЕ. Доказательством служит как исполь
лен РЭ прокариот, названный DGR (diversity
зование процессированных транскриптов
generating retroelement) [74]. Филогенетический
тРНК в сайленсинге ТЕ (что говорит о наличии
анализ показал, что бактериальные RT могут
между ними комплементарности НП) [25, 55],
быть классифицированы на 17 групп: RT интро
так и использование тРНК [21] и рРНК [21,
нов группы II, ретронов и ретрон подобных
29-31] в геномах в качестве основы для неавто
элементов, DGR RT, Abi like RT, RT систем
номных ТЕ.
CRISPR Cas, G2L (group II like RT) и 11 других
Таким образом, взаимосвязь мира РНК-
групп RT с неизвестными функциями [75]. В ге
ДНК и белков возникла благодаря RT и содер
номах бактерий идентифицировано множество
жащим ее универсальным консервативным
не охарактеризованных RT и связанных с ними
структурам - РЭ и их производным (ДНК ТЕ).
последовательностей, которые демонстрируют
Успешное распространение и сохранение в эво
большое разнообразие доменных структур. За
люции обратнотранскрибированных рРНК и
исключением интронов группы II, содержащие
тРНК в качестве неавтономных ТЕ в геномах
RT элементы не проявляют признаков активной
различных эукариот подтверждает их эволюци
ретромобильности, но вовлечены в защиту ге
онное родство с другими ТЕ. Это обусловлено
номов от фагов [72]. Эти геномные структуры
общими свойствами с автономными ТЕ, а также
сохранили древнее эволюционное свойство,
наличием гомологичных НП в их составе, бла
направленное на противоборство эгоистических
годаря чему они активно распространяются и
геномов, в ходе которого образовывались новые
сохраняются в ряду поколений [23]. Интересно,
защитные системы. У бактерий >50% RT, кото
что
3' конец SINE3, произошедший от
5S
рые могут быть использованы для генерации
рРНК, имеет значительное сходство с CR1 по
комплементарной ДНК из матрицы РНК, коди
добными non LTR РЭ. Наряду с CR1 подобным
руются интронами группы II. Данные интроны
РЭ, копии SINE3 не фланкированы дупликаци
действуют как рибозимы и мобильные РЭ и
ями целевых сайтов, а их 3' конец состоит из
классифицируются на A, B, C, D, E, F, G, CL1/2
микросателлитов (ACATT)n и (ATT)n [29]. В ге
(chloroplast like), ML (mitochondrion like) груп
номах млекопитающих выявлены также тран
пы. Все они эволюционировали при помощи ас
скрипционно активные SINE28, происходящие
социации древней RT со структурной каталити
от 3' конца большой рибосомальной субъеди
ческой РНК [75].
БИОХИМИЯ том 84 вып. 8 2019
1108
МУСТАФИН, ХУСНУТДИНОВА
Интроны группы II кодируют собственную
мегабаз у S. cerevisiae до 148 852 мегабаз у Paris
RT, которая стабилизирует каталитически ак
japonica) [79]. Однако была обнаружена выра
тивную РНК для прямого и обратного сплай
женная гомология между системами CRISPR и
синга и конвертирует интегрированную интрон
РНКи с участием малых интерферирующих
ную РНК обратно в ДНК [76]. Впервые интроны
РНК [20]. Можно предположить, что у общего
группы II были выделены еще в 1986 году из
предка всех доменов живого существовал за
транскрипта митохондрии Saccharomyces cere*
щитный механизм, модификации которого дали
visiae [77]. Предполагается, что данные элемен
основу для развития CRISPR у прокариот и
ты являлись эволюционными предшественни
RISC эукариот. Специфика их функционирова
ками не только сплайсосомных интронов, но
ния повлияла на структурные особенности ге
также самих сплайсосом, РЭ и теломер эукариот
номов разных доменов. Несмотря на длитель
[76]. Структура каталитического домена их RT
ный период эволюции с момента возникнове
сильно напоминает теломеразу, а активный
ния жизни, в современных организмах сохраня
центр сплайсинга - белок Prp8 сплайсосомы.
ются процессы, отражающие преемственность
Это говорит о предковых взаимосвязях между
функций рибозимов белками на начальных эта
сплайсингом и ретромобильностью [18]. Кроме
пах. Наиболее ярким примером являются длин
того, показано, что белок Prp8 в эволюции про
ные нкРНК.
изошел от RT транспозонов [69].
Была обнаружена взаимосвязь RT системы
CRISPR Cas типа III с кодируемыми интронами
ЗНАЧЕНИЕ ОБРАТНОЙ ТРАНСКРИПТАЗЫ
группы II обратными транскриптазами. Ассоци
В ЭВОЛЮЦИИ ЭУКАРИОТ
ация RT с системой CRISPR Cas происходила
множество раз в ходе эволюции. При филогене
РЭ эукариот классифицируются на 2 основ
тическом анализе RT системы CRISPR были
ных класса по механизму их транспозиций и ор
распределены на 12 групп. При этом выявлена
ганизации ДНК. К первому классу относятся
коэволюция RT с белками Cas1, а также их гори
LTR РЭ, которые характеризуются прямыми
зонтальный перенос в геномы архей [45]. RT со
повторами в несколько сотен п.н. на их концах и
держит последовательности, обладающие свой
содержат гены pol и gag. Продукт гена pol содер
ствами рибонуклеазы Н, к суперсемейству нук
жит несколько ферментных доменов (RT, рибо
леотидил трансфераз которых относятся Cas9
нуклеаза Н, интеграза и протеиназа). Ген gag ко
[20]. Поэтому можно предположить, что в эво
дирует белки GAG (Group specific AntiGen).
люции формирование CRISPR происходило из
Представители второго класса, nonLTR РЭ, мо
защитных механизмов одних РЭ против других
гут содержать одну открытую рамку считывания
ТЕ. Прокариоты противоборствуют с вирусами
(ORF, Open Reading Frame), в которой закодиро
при помощи врожденных иммунных систем мо
ван белок с доменами RT и EN (эндонуклеазы)
дификации рецепторов, рестрикции модифи
или две ORF (первая проявляет сходство с геном
кации и абортивной инфекции. РНК опосредо
gag ретровирусов, вторая кодирует домены RT и
ванная адаптивная иммунная система CRISPR
EN). Предполагается, что LTR РЭ эволюциони
Cas содержится у большинства архей и ~40%
ровали от non LTR РЭ за счет возникновения
бактерий [45]. RT в данной системе играет важ
интегразы, а ретровирусы произошли от LTR
ную роль для обратной транскрипции чужерод
РЭ с помощью включения генов оболочки (env
ных РНК [78].
доменов) от других вирусов [45]. При секвени
У прокариот интеграция новых НП (спейсе
ровании геномов было обнаружено, что эукарио
ров) при помощи CRISPR сопровождается уда
ты могут содержать больше генов RT по сравне
лением других частей генома, состоящих из пов
нию с кодирующими любой другой белок. Око
торов, что позволяет сохранять относительно
ло 40% НП геномов млекопитающих представ
равноценные размеры ДНК их нуклеоидов.
лены РЭ, в основном LINE и SINE. У растений
В результате максимальная длина генома у
содержание ТЕ может достигать 91% их ДНК
M. xanthus больше минимальной у Micoplasma
(Asparagus officinalis), главным образом за счет
genitalium всего в 16 раз. Эволюционный успех,
LTR РЭ [80].
огромное разнообразие фенотипов и грандиоз
RT эукариот вовлечены в такие процессы,
ные различия в размерах геномов эукариот мо
как добавление теломер, репликация митохон
гут объясняться глобальной ролью инсерций в
дриальных плазмид, транскрипция и пролифе
их эволюции и функционировании, что может
рация ретровирусных геномов [39], дупликация
отражать механизмы, происходившие при воз
генов [80]. RT составляет основу РЭ, которые у
никновении жизни на Земле. Размеры геномов
эукариот служат ключевыми источниками воз
эукариот могут отличаться в 70 000 раз (от 2,3
никновения как регуляторных НП [65], так и бе
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1109
лок кодирующих генов путем доместикации са
ловлено способностью длинных нкРНК связы
мих РЭ [81, 82] и экзонизации их НП [66, 83, 84].
ваться с рибосомами и транслироваться в функ
РЭ оказались источниками важнейших консер
циональные пептиды [95].
вативных свойств геномов эукариот, отличаю
Благодаря RT в геномах эукариот активно
щих их от прокариотических. Так, от РЭ прои
распространяются РЭ, которые становятся ис
зошли центромерные сателлиты [85] и центро
точниками биохимически активных строго ре
мер связывающие белки CENP/CENH3 [86], с
гулируемых некодирующих элементов, таких
которыми взаимодействуют содержащиеся в
как сайты связывания с ТФ и нкРНК. В ходе
центромерах РЭ [87], теломеры [88], и теломера
эволюции это способствует перестройке генных
за [89], транскрипционные факторы [65] и сай
регуляторных сетей, за счет чего формируется
ты связывания с ними [90]. От РЭ и других ТЕ
основа для изменчивости и образования новых
происходят многие микроРНК [91], а также их
видов [96]. Кроме того, РЭ являются важными
целевые НП в составе белок кодирующих ге
источниками гибридного дисгенеза при видооб
нов, что связано с ролью ТЕ в их возникновении
разовании. Были выявлены вспышки транспо
[65, 66, 81-84]. Не исключено, что, подобно
зиций, наблюдаемые после гибридизации и
сплайсосоме, белки процессинга нкРНК, а так
способствующие генетической нестабильности.
же основные компоненты системы РНКи в эво
Связь гибридных транспозиций и деметилиро
люции также произошли от ТЕ. Таким образом,
вания наблюдается как у млекопитающих, так и
на примере механизмов эволюции геномов эу
у растений [97].
кариот можно предположить, что РЭ и входя
Подобно длинным нкРНК, молекулы пре
щая в их состав RT могли быть источниками
мРНК белок кодирующих генов также могут
происхождения всей жизни. Это связано с ис
формировать различные пространственные
пользованием РЭ в качестве универсальных
конфигурации. При этом их вторичные и тре
единиц, ставших консервативными на всех эта
тичные структуры играют важную роль в про
пах эволюции. Для эукариот характерно функ
цессинге и стабильности пре мРНК [98], что
ционирование длинных нкРНК. Они являются
позволяет предположить функциональную зна
наилучшим примером, отражающим принципы
чимость эволюционных предков этих РНК и их
участия ТЕ в возникновении саморегуляторных
древнее происхождение от ТЕ. Например, насы
систем и преемственности рибозимов белковы
щенность доменов длинных нкРНК последова
ми ферментами.
тельностями ТЕ может быть связана с их спо
Длинные нкРНК представляют собой рибо
собностью образовывать конформации, участ
зимы, способные функционировать как само
вующие в важных биологических реакциях [14].
стоятельно, так и в составе РНП. Они участвуют
Для предсказания вторичных структур РНК раз
в регуляции транскрипции, воздействуя на мо
рабатываются различные алгоритмы, в которых
дификацию гистонов и ДНК связывающие
используются минимальная свободная энергия
комплексы, в том числе транскрипционные
и максимально ожидаемая точность со сравни
факторы (ТФ) [92]. При этом НП многих про
тельными эволюционными методами. Однако
цессированных транскриптов длинных нкРНК
эти инструменты не идеальны. Вторичная
полностью совпадают с ТЕ [15], а >80% актив
структура пре мРНК может оказывать усилива
ных доменов всех длинных нкРНК происходят
ющее или подавляющее воздействие на сплай
от транспозонов [14]. РЭ способны функциони
синг собственной молекулы в зависимости от
ровать непосредственно в качестве генов длин
особенностей НП в интронах. После сплайсин
ных нкРНК [93, 94]. Т.е. процессированные
га вторичная структура РНК также может вли
транскрипты РЭ проявляют свойства рибози
ять на стабильность и регуляцию РНКи. На
мов, что может отражать их древнюю консерва
сплайсинг молекул пре мРНК оказывает воз
тивную характеристику, ставшую ключевым мо
действие также их третичная структура. Приме
ментом для возникновения жизни. Исследова
ром является G квадруплекс, который усилива
ние роли длинных нкРНК в образовании новых
ет или подавляет сплайсинг путем создания или
белок кодирующих генов может отображать
затенения участков связывания РНК с белком
сходные процессы на ранних этапах эволюции,
[98]. Сходными способностями обладают РЭ.
когда переход из мира РНК и ДНК в мир с учас
Они могут управлять распознаванием сайтов
тием белков характеризовался преемствен
сплайсинга за счет образования специфических
ностью функций рибозимов полипептидными
пространственных структур в виде шпилек [99].
ферментами. В независимых исследованиях вы
Характерен также процессинг интронов бе
явлено происхождение эволюционно новых
лок кодирующих генов в функциональные
белков эукариот от продуктов трансляции тран
нкРНК [70], что объединяет их по свойствам с
скриптов длинных нкРНК [10-13]. Это обус
ТЕ [91]. Сохранение в эволюции РНКи при по
БИОХИМИЯ том 84 вып. 8 2019
1110
МУСТАФИН, ХУСНУТДИНОВА
мощи процессированных транскриптов ТЕ го
Участие гистонов в регуляции работы генома от
ворит об универсальности этого механизма. Ак
ражает принцип использования белков при воз
тивное использование и консервативность ко
никновении жизни, как необходимых компо
дирования аминокислот нуклеотидами могли
нентов не только для преемственности функций
быть связаны с возможностью взаимодействий
рибозимов, но и для создания новых, оптималь
пептидов с первичной и вторичной структурами
но адаптивных свойств. Кроме того, от ТЕ про
ДНК [100] для обеспечения защитных механиз
изошли гены, принимающие непосредственное
мов. В различных биологических реакциях бо
участие в ремоделировании хроматина [106]. ТЕ
лее успешны или рибозимы или белки. В тех
оказались источниками возникновения ацетил
процессах, где рибозимы обладают уникальны
трансферазного комплекса HDP1/2 [107], фак
ми свойствами, они сохранили свои функции.
торов модификации хроматина BEAF 32 и
Однако вновь возникающие белковые фермен
HIM 17 [108], центромерных белков Abp1 [65] и
ты способны заместить эти молекулы РНК для
инсуляторов [109].
лучшей адаптации. Данное состояние неравно
весия является источником эволюционных пре
В литературе накоплены многочисленные
образований и создания новых белок кодирую
данные об универсальности RT в качестве клю
щих генов из областей ДНК, содержащих гены
чевого звена для взаимосвязи мира РНК с ДНК
нкРНК [10-13].
при возникновении жизни. В эволюции бакте
Считается, что большинство RT подобных
рий, архей и эукариот проявляется консерватив
ферментов принадлежат к ретроэлементам или
ность использования RT для формирования но
вирусам и не обладают определенной функцией
вых принципов саморегуляции и самовоспроиз
в клетке хозяине, за исключением теломеразы.
ведения, что может отражать сходные механиз
Однако был обнаружен уникальный класс RT
мы предковых доклеточных форм жизни. Моди
связанных клеточных генов, названных rvt. Они
фикации рибозима РНК полимеразы могли об
являются генами «хозяев» и уже не служат ком
ладать свойствами RT, рибонуклеазы и интегра
понентами РЭ. Гены rvt представлены един
зы для поддержания самовоспроизведения био
ственной копией и могут содержать интроны в
полимеров РНК и ДНК. Однако сохранение ин
эволюционно консервативных позициях, изме
формации об адаптивных свойствах, необходи
няясь под действием отбора. Эти гены обнару
мых для выживания биомолекул на базе ДНК,
живаются во всех основных таксономических
нуждалось в балансе между инсерциями (для из
группах, включая протисты, грибы, животные,
менчивости и увеличения длины первичных ге
растения и бактерии с неоднородным филогене
номов) и защитой важного генетического мате
тическим распределением [101]. RT использует
риала. В связи с этим происходил отбор меха
ся в создании генов с выраженной амплифика
низмов защиты от инсерций с формированием
цией, таких как MADS*box и цитохромы Р450
систем процессинга РНК, таких как RISC,
[102]. Гены гистонов также характеризуются
CRISPR, сплайсинг и система трансляции. Ос
кластерной организацией и не содержат интро
новой для их возникновения должны были быть
нов, что позволяет предположить их происхож
модификации рибозимов RT, обладающие раз
дение от ТЕ у общего предка всех эукариот.
личными рибонуклеазными активностями.
Подтверждением служит возникновение от ТЕ
Аминокислоты и пептиды могли использовать
гена CENP/CENH, продукт которого является
ся для оптимизации защитных механизмов в
вариантом гистона Н3 [86]. Малые нкРНК спо
противоборствующих эгоистических молекулах
собны оказывать свое эпигенетическое воздей
полинуклеотидов. В дальнейшем усовершен
ствие путем модификаций гистонов [103], в то
ствование системы трансляции стало основой
время как сами ТЕ служат источниками нкРНК
для тесной взаимосвязи мира ДНК и РНК с по
[91]. Для разных типов ТЕ характерны специфи
липептидами с преемственностью функций ри
ческие модификации гистонов в областях их
бозимов белковыми ферментами. Данный
расположения (LTR и LINE - метки H3K9me3,
принцип сохраняется у эукариот, так как рибо
другие ТЕ - H3K27me3) [104], а распределение
зимы длинных нкРНК способны транслиро
длины консервативных и неконсервативных об
ваться с образованием функциональных моле
ластей интронов имеют максимумы, близкие по
кул. В результате отбора гены длинных нкРНК
длине динуклеосомной и нуклеосомной ДНК
могут преобразовываться в белок кодирующие,
[105]. Приведенные данные свидетельствуют об
при этом их процессированные транскрипты
универсальных свойствах ТЕ в качестве консер
сохраняют свою ферментативную активность.
вативных единиц всего живого, способных со
В результате у эукариот РЭ стали источниками
здавать саморегуляторные комплексные струк
ТФ и сайтов связывания с ними, центромер и
туры, которые эффективно эволюционируют.
центромер связывающегося белка, теломер и
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1111
теломеразы, белок кодирующих генов и взаи
Конфликт интересов. Авторы заявляют, что у
модействующих с ними нкРНК, интронов и
них нет конфликтов интересов.
компонентов сплайсосомы. Это отражает уни
Соблюдение этических норм. Настоящая
версальный принцип саморегуляции РЭ и их
статья не содержит каких либо исследований с
производных, необходимый для возникновения
участием людей или использованием животных
жизни и ее эволюции.
в качестве объектов исследований.
СПИСОК ЛИТЕРАТУРЫ
1.
Lincoln, T.A., and Joyce, G.F. (2009) Self sustained repli
15.
Kapusta, A., and Feschotte, C. (2014) Volatile evolution of
cation of an RNA enzyme, Science, 323, 1229-1232,
long noncoding RNA repertoires: mechanisms and biolog
doi: 10.1126/science.1167856.
ical implications, Trends Genet.,
30,
439-452,
2.
Wochner, A., Attwater, J., Coulson, A., and Holliger, P.
doi: 10.1016/j.tig.2014.08.004.
(2011) Ribozyme catalyzed transcription of an active
16.
Lukash, L.L. (2007) Mutagenesis induced by integration
ribozyme, Science, 332, 209-212, doi: 10.1126/science.
processes and evolution of nuclear genome, Biopolym. Cell,
1200752.
23, 172-187.
3.
Horning, D.P., and Joyce, G.F. (2016) Amplification of
17.
Samanta, B., and Joyce G.F. (2017) A reverse transcriptase
RNA by an RNA polymerase ribozyme, Proc. Natl Acad.
ribozyme, Elife, 6, e31153, doi: 10.7554/eLife.31153.
Sci. USA, 113, 9786-9791, doi: 10.1073/pnas.1610103113.
18.
Qu, G., Kaushal, P.S., Wang, J., Shigematsu, H., Piazza, C.L.,
4.
Kreysing, M., Keil, L., Lanzmich, S., and Braun, D.
Agrawal, R.K., Belfort, M., and Wang H.W.
(2016)
(2015) Heat flux across an open pore enables the continu
Structure of a group II intron in complex with its reverse
ous replication and selection of oligonucleotides towards
transcriptase, Nat. Struct. Mol. Biol., 23, 549-557,
increasing length, Nat. Chem., 7, 203-208, doi: 10.1038/
doi: 10.1038/nsmb.3220.
nchem.2155.
19.
Hughes, S.H. (2015) Reverse transcription of retroviruses and
5.
Betts, H.C., Puttick, M.N., Clark, J.W., Williams T.A.,
LTR retrotransposons, Microbiol. Spectr., 3, MDNA3 0027
Donoghue P.C.J., and Pisani, D. (2018) Integrated genom
2014, doi: 10.1128/microbiolspec.MDNA3 0027 2014.
ic and fossil evidence illuminates life’s early evolution and
20.
Moelling, K., and Broecker, F. (2015) The reverse tran
eukaryote origin, Nat. Ecol. Evol.,
2,
1556-1562,
scriptase RNase H: from viruses to antiviral defense, Ann.
doi: 10.1038/s41559 018 0644 x.
N. Y. Acad. Sci., 1341, 126-135, doi: 10.1111/nyas.12668.
6.
Kruger, K., Grabowski, P.J., Zaug, A.J., Sands, J.,
21.
Gogolevsky, K.P., Vassetzky, N.S., and Kramerov, D.A.
Gottschling, D.E., and Cech, T.R. (1982) Self splicing
(2009) 5S rRNA derived and tRNA derived SINEs in fruit
RNA: autoexcision and autocyclization of the ribosomal
bats, Genomics,
93,
494-500, doi:
10.1016/j.ygeno.
RNA intervening sequence of Tetrahymena, Cell, 31,
2009.02.001.
147-157.
22.
Rosenbland, M.A., Larsen, N., Samuelsson, T., and
7.
Guerrier Takada, C., Gardiner, K., Marsh, T., Pace, N.,
Zwieb, C. (2009) Kinship in the SRP RNA family, RNA
and Altman, S. (1983) The RNA moiety of ribonuclease P
Biol., 6, 508-516.
is the catalytic subunit of the enzyme, Cell, 35, 849-857.
23.
Li, Z., Ender, C., Meister, G., Moore, P.S., Chang, Y., and
8.
Prody, G.A., Bakos, J.T., Buzayan, J.M., Schneider, I.R.,
John, B. (2012) Extensive terminal and asymmertric pro
and Bruening, G. (1986) Autolytic processing of dimeric
cessing of small RNAs from rRNAs, snoRNAs, snRNAs,
plant virus satellite RNA, Science, 231, 1577-1580.
and tRNAs, Nucleic Acids Res.,
40,
6787-6799,
9.
de la Pena, M., Garcia Robles, I., and Cervera, A. (2017)
doi: 10.1093/nar/gks307.
The Hammerhead Ribozyme: a long history for a short
24.
Kojima, K.K. (2015) A new class of SINEs with snRNA
RNA, Molecules,
22, pii: E78, doi:
10.3390/mole
gene derived heads, Genome Biol. Evol., 7, 1702-1712,
cules22010078.
doi: 10.1093/gbe/evv100.
10.
Levine, M.T., Jones, C.D., Kern, A.D., Lindfors, H.A.,
25.
Martinez, G., Choudury, S.G., and Slotkin, R.K. (2017)
and Begun, D.J. (2006) Novel genes derived from noncod
tRNA derived small RNAs target transposable element
ing DNA in Drosophila melanogaster are frequently
transcripts, Nucleic Acids Res.,
45,
5142-5152,
X linked and exhibit testis biased expression, Proc. Natl.
doi: 10.1093/nar/gkx103.
Acad. Sci. USA, 103, 9935-9939.
26.
Startek, M.P., Nogly, J., Gromadka, A., Grzebelus, D.,
11.
Cai, J., Zhao, R., Jiang, H. and Wang, W. (2008) De novo
and Gambin, A. (2017) Inferring transposons activity
origination of a new protein coding gene in Saccharomyces
chronology by TRANAcendence TEs database and de
cerevisiae, Genetics, 179, 487-496, doi: 10.1534/genetics.
novo mining tool, BMC Bioinformatics,
18,
422,
107.084491.
doi: 10.1186/s12859 017 1824 4.
12.
Xie, C., Zhang, Y.E., Chen, J.Y., Liu, C.J., Zhou, W.Z., Li, Y.,
27.
Alzohairy, A.M., Gyulai, G., Jansen, R.K., and Bahieldin, A.
Zhang, M., Zhang, R., Wei, L., and Li, C.Y. (2012)
(2013) Transposable elements domesticated and neofunc
Hominoid specific de novo protein coding genes originat
tionalized by eukaryotic genomes, Plasmid, 69, 1-15,
ing from long non coding RNAs, PLoS Genet., 8,
doi: 10.1016/j.plasmid.2012.08.001.
e1002942, doi: 10.1371/journal.pgen.1002942.
28.
Kramerov, D.A., and Vassetzky, N.S. (2011) Origin and
13.
Ruiz Orera, J., Messeguer, X., Subirana, J.A., and Alba, M.M.
evolution of SINEs in eukaryotic genomes, Heredity
(2014) Long non coding RNAs as a source of new peptide,
(Edinb.), 107, 487-495, doi: 10.1038/hdy.2011.43.
Elife, 3, e03523, doi: 10.7554/eLife.03523.
29.
Kapitonov, V.V., and Jurka, J. (2003) A novel class of SINE
14.
Johnson, R., and Guigo, R. (2014) The RIDL hypothesis:
elements derived from 5S rRNA, Mol. Biol. Evol., 20,
transposable elements as functional domains of long non
694-702.
coding RNAs, RNA, 20, 959-976, doi: 10.1261/rna.
30.
Wang, J., Wang, A., Han, Z., Zhang, Z., Li, F., and Li, X.
044560.114.
(2012) Characterization of three novel SINE families with
БИОХИМИЯ том 84 вып. 8 2019
1112
МУСТАФИН, ХУСНУТДИНОВА
unusual features in Helicoverpa armigera, PLoS One, 7,
tures of group I introns and insertion elements, Mol.
e31355, doi: 10.1371/journal.pone.0031355.
Microbiol., 36, 1447-1459.
31.
Longo, M.S., Brown, J.D., Zhang, C., O’Neill, M.J. and
50.
Gao, X., and Voytas, D.F. (2005) A eukaryotic gene family
O’Neill, R.J. (2015) Identification of a recently active
related to retroelements integrases, Trends Genet., 21,
mammalian SINE derived from ribosomal RNA, Genome
133-137.
Biol. Evol., 7, 775-788, doi: 10.1093/gbe/evv015.
51.
Skala, A.M. (2014) Retroviral DNA transposition: themes
32.
Wicker, T., Sabot, F., Hua Van, A., Bennetzen, J.L., Capy, P.,
and variations, Microbiol. Sperctr., 2, doi: 10.1128/micro
Chalhoub, B., Flavell, A., Leroy, P., Morgante, M.,
biolspec.MDNA3 0005 2014.
Panaud, O., Paux, E., SanMiguel, P., and Schulman, A.H.
52.
Zeng, L., Pederson, S.M., Cao, D., Qu, Z., Hu, Z.,
(2007) A unified classification system for eukaryotic trans
Adelson, D.L., and Wei, C. (2018) Genome wide analysis
posable elements, Nat. Rev. Genet., 8, 973-982.
of the association of transposable elements with gene regu
33.
Abrusan, G., Zhang, Y., and Szilagyi, A. (2013) Structure
lation suggests that alu elements have the largest overall
prediction and analysis of DNA transposon and LINE
regulatory impact, J. Comput. Biol.,
25,
551-562,
retrotransposons proteins, J. Biol. Chem., 288, 16127-
doi: 10.1089/cmb.2017.0228.
16138, doi: 10.1074/jbc.M113.451500.
53.
Ren, Y.F., Li, G., Wu, J., Xue, Y.F., Song, Y.J., Lv, L.,
34.
Rice, P.A., and Baker, T.A. (2001) Comparative architec
Zhang, X.J., and Tang, K.F. (2012) Dicer dependent bio
ture of transposase and integrase complexes, Nat. Struct.
genesis of small RNAs derived from 7SL RNA, PLoS One,
Biol., 8, 302-307.
7, e40705, doi: 10.1371/journal.pone.0040705.
35.
Wolkowicz, U.M., Morris, E.R., Robson, M., Trubitsyna, M.,
54.
Jacob, M.D., Audas, T.E., Mullineux, S.T., and Lee, S.
and Richardson, J.M. (2014) Structural basis of Mos1
(2012) Where no RNA polymerase has gone before: novel
transposase inhibition by the anti retroviral drug
functional transcripts derived from the ribosomal
Raltegravir, ACS Chem. Biol., 9, 743-751, doi: 10.1021/
Intergenic spacer, Nucleus, 3, 315-319.
cb400791u.
55.
Kumar, P., Anaya, J., Mudunuri, S.B., and Dutta, A.
36.
Nowotny, M., Gaidamakov, S.A., Crouch, R.J., and Yang, W.
(2014) Meta analysis of tRNA derived RNA fragments
(2005) Crystal structures of RNase H bound to an
reveals that they are evolutionarily conserved and associate
RNA/DNA hybrid: substrate specificity and metal depen
with AGO proteins to recognize specific RNA targets,
dent catalysis, Cell, 121, 1005-1016.
BMC Biol., 12, 78, doi: 10.1186/s12915 014 0078 0.
37.
De Koning, A.P., Gu, W., Castoe, T.A., Batzer, M.A., and
56.
Ender, C., Krek, A., Friedlander, M.R., Beitzinger, M.,
Polock, D.D. (2011) Repetitive elements may comprise
Weinmann, L., Chen, W., Pferffer, S., Rajewsky, N., and
over two thirds of the human genome, PloS Genet., 7,
Meister, G. (2008) A human snoRNA with microRNA
e1002384, doi: 10.1371/journal.pgen.1002384.
like functions, Mol. Cell., 32, 519-528, doi: 10.1016/j.mol
38.
Goerner Potvin, P., and Bourque, G. (2018) Computa
cel.2008.10.017.
tional tools to unmask transposable elements, Nat. Rev.
57.
Taft, R.J., Glazov, E.A., Lassmann, T., Hayashizaki, Y.,
Genet., 19, 688-704, doi: 10.1038/s41576 018 0050 x.
Carninci, P., and Mattick, J.S. (2009) Small RNAs derived
39.
Ellefson, J.W., Gollihar, J., Shoroff, R., Shivram, H., Lyer, V.R.,
from snoRNAs, RNA, 15, 1233-1240, doi: 10.1261/rna.
and Ellington, A.D. (2016) Synthetic evolutionary origin of
1528909.
a proofreading reverse transcriptase, Science,
352,
58.
Venkatesh, T., Suresh, P.S., and Tsutsumi, R. (2016) tRFs:
1590-1593, doi: 10.1126/science.aaf5409.
miRNAs in disguise, Gene, 579, 133-138, doi: 10.1016/
40.
Freeland S.J., Knight R.D., Landweber L.F. (1999) Do
j.gene.2015.12.058.
proteins predate DNA, Science, 286, 690-692.
59.
Zhou, J., Ding, D., Wang, M., and Cong, Y.S. (2014)
41.
Baltimore, D. (1970) RNA dependent DNA polymerase
Telomerase reverse transcriptase in the regulation of gene
in virions of RNA tumour, Nature, 226, 1209-1211.
expression, BMB Rep., 47, 8-14.
42.
Temin, H.M., and Mizutani, S. (1970) RNA dependent
60.
Elliott, T.A., Stage, D.E., Crease, T.J., and Eickbush, T.H.
DNA polymerase in virions of Rous sarcoma, Nature, 226,
(2013) In and out of the rRNA genes: characterization of
1211-1213.
Pokey elements in the sequenced Daphnia genome, Mob.
43.
Lampson, B.C., Inouye, M. and Inouye, S. (1989) Reverse
DNA, 4, 20, doi: 10.1186/1759 9753 4 20.
transcriptase with concomitant ribonuclease H activity in
61.
Jamburuthugoda, V.K., and Eickbush, T.H.
(2014)
the cell free synthesis of branched RNA linked msDNA of
Identification of RNA binding motifs in the R2 retrotrans
Myxococcus xanthus, Cell, 56, 701-707.
poson encoded reverse transcriptase, Nucleic Acids Res.,
44.
Lim, D., and Maas, W.K. (1989) Reverse transcriptase
42, 8405-8415, doi: 10.1093/nar/gku514.
dependent synthesis of a covalently linked, branched
62.
Novikova, O., and Belfort, M. (2017) Mobile group II
DNA RNA compound in E. coli B, Cell, 56, 891-904.
introns as ancestral eukaryotic elements, Trends Genet., 33,
45.
Toro, N., Martinez Abarca, F., and Gonzalez Delgado, A.
773-783, doi: 10.1016/j.tig.2017.07.009.
(2017) The reverse transcriptases associated with CRISPR
63.
Wang, D., Su, Y., Wang, X., Lei, H., and Yu, J. (2012)
Cas systems, Sci. Rep., 7, 7089, doi: 10.1038/s41598 017
Transposon derived and satellite derived repetitive
07828 y.
sequences play distinct functional roles in mammalian
46.
Ekland, E.H., and Bartel, D.P. (1996) RNA catalysed
intron size expansion, Evol. Bioinform. Online, 8, 301-319,
RNA polymerization using nucleoside triphosphates,
doi: 10.4137/EBO.S9758.
Nature, 382, 373-376.
64.
Yenerall, P., and Zhou, L. (2012) Identifying the mecha
47.
Been, M.D., and Cech, T.R. (1988) RNA as an RNA poly
nisms of intron gain: progress and trends, Biol. Direct., 7,
merase: net elongation of an RNA primer catalyzed by the
29, doi: 10.1186/1745 6150 7 29.
Tetrahymena ribozyme, Science, 239, 1412-1416.
65.
Feschotte, C. (2008) The contribution of transposable ele
48.
Adamala, K., Engelhart, A.E. and Szostak, J.W. (2015)
ments to the evolution of regulatory networks, Nat. Rev.
Generation of functional RNAs from inactive oligonu
Genet., 9, 397-405.
cleotide complexes by non enzymatic primer extension,
66.
Tajnik, M., Vigilante, A., Braun, S., Hanel, H., Luscombe, N.M.,
J. Am. Chem. Soc., 137, 483-489.
Ule, J., Zarnack, K., and Koning, J. (2015) Inergenic Alu
49.
Braun, V., Mehlig, M., Moos, M., Rupnik, M., Kalt, B.,
exonisation facilitates the evolution of tissue specific tran
Mahony, D.E., and von Eichel Streiber, C.
(2000)
script ends, Nucleic Acids Res.,
43,
10492-10505,
A chimeric ribozyme in clostridium difficile combines fea
doi: 10.1093/nar/gkv956.
БИОХИМИЯ том 84 вып. 8 2019
ОБРАТНАЯ ТРАНСКРИПТАЗА И ВОЗНИКНОВЕНИЕ ЖИЗНИ
1113
67.
Lei, H., and Vorechovsky, I. (2005) Identification of splic
85. Cheng, Z.J., and Murata, M. (2003) A centromeric tan
ing silencers and enhancers in sense Alus: a role for
dem repeat family originating from a part of Ty3/gypsy
pseudoacceptors in splice site repression, Mol. Cell. Biol.,
retroelement in wheat and its relatives, Genetics, 164,
25, 6912-6920.
665-672.
68.
Pastor, T., Talotti, G., Lewandowska, M.A., and Pagani, F.
86. Kipling, D., and Warburton, P.E. (1997) Centromeres,
(2009) An Alu derived intronic splicing enhancer facili
CENP B and Tigger too, Trends Genet., 13, 141-145.
tates intronic processing and modulates aberrant splicing in
87. Mestrovic, N., Mravinac, B., Pavlek, M., Vojvoda Zeljko, T.,
ATM, Nucleic Acids Res., 37, 7258-67, doi: 10.1093/
Satovic, E., and Plohl, M. (2015) Structural and function
nar/gkp778.
al liaisons between transposable elements and satellite
69.
Galej, W.P., Oubridge, C., Newman, A.J., and Nagai, K.
DNAs, Chromosome Res., 23, 583-596, doi: 10.1007/
(2013) Crystal structure of Prp8 reveals active site cavity of
s10577 015 9483 7.
the spliceosome, Nature, 493, 638-643, doi: 10.1038/
88. Arkhipova, I.R. (2018) Neutral theory, transposable ele
nature11843.
ments, and eukaryotic genome evolution, Mol. Biol. Evol.,
70.
Rearick, D., Prakash, A., McSweeny, A., Shepard S.S.,
35, 1332-1337, doi: 10.1093/molbev/msy083.
Fedorova, L., and Fedorov, A. (2011) Critical association
89. Garavis, M., Gonzalez, C., and Villasante, A. (2013) On
of ncRNA with introns, Nucleic Acids Res., 39, 2357-2366,
the origin of the eukaryotic chromosome: the role of non
doi: 10.1093/nar/gkq1080.
canonical DNA structures in telomere evolution, Genome
71.
Nissen, P., Hansen, J., Ban, N., Moore P.B., and Steitz, T.A.
Biol. Evol., 5, 1142-1150, doi: 10.1093/gbe/evt079.
(2000) The structural basis of ribosome activity in peptide
90. De Souza, F.S., Franchini, L.F., and Rubinstein, M.
bond synthesis, Science, 289, 920-930.
(2013) Exaptation of transposable elements into novel cis
72.
Zimmerly, S., and Wu, L. (2015) An unexplored diversity
regulatory elements: is the evidence always strong, Mol.
of reverse transcriptases in bacteria, Microbiol. Spectr., 3,
Biol. Evol., 30, 1239 -1251, doi: 10.1093/molbev/mst045.
MDNA3 0058 2014, doi: 10.1128/microbiolspec.MDNA3
91. Gim, J., Ha, H., Ahn, K., Kim, D.S., and Kim, H.S.
0058 2014.
(2014) Genome Wide Identification and Classification of
73.
Равин Н.В., Шестаков С.В. (2013) Геном прокариот,
microRNAs derived from repetitive elements, Genomic
Вавиловский журнал генетики и селекции, 17, 972-984.
Inform., 12, 261-267, doi: 10.5808/GI.2014.12.4.261.
74.
Liu, M., Deora, R., Doulatov, S.R. Gingery, M., Eiserling, F.A.,
92. Long, Y., Wang, X., Youmans, D.T., and Cech, T.R. (2017)
Preston, A., Maskell, D.J., Simons, R.W., Cotter, P.A.,
How do lncRNAs regulate transcription, Sci. Adv., 3,
Parkhill, J., and Miller, J.F. (2002) Reverse transcriptase
eaao2110, doi: 10.1126/sciadv.aao2110.
mediated tropism switching in Bordetella bacteriophage,
93. Lu, X., Sachs, F., Ramsay, L., Jacques, P.E., Goke, J.,
Science, 295, 2091-2094.
Bourque, G., and Ng, H.H. (2014) The retrovirus HERVH
75.
Toro, N., Martinez Abarca, F., Gonzalez Delgado, A.,
is a long noncoding RNA required for human embryonic
and Mestre, M.R. (2018) On the origin and evolutionary
stem cell identity, Nat. Struct. Mol. Biol., 21, 423-425,
relationships of the reverse transcriptases associated with
doi: 10.1038/nsmb.2799.
type III CRISPR-Cas Systems, Front. Mircrobiol., 9,
94. Honson, D.D., and Macfarlan, T.S. (2018) A lncRNA like
1317, doi: 10.3389/fmicb.2018.01317.
role for LINE1s in development, Dev. Cell, 46, 132-134,
76.
Lambowitz, A.M., and Zimmerly, S. (2011) Group II
doi: 10.1016/j.devcel.2018.06.022.
introns: mobile ribozymes that invade DNA, Cold Spring
95. Anderson, D.M., Anderson, K.M., Cang, C.L.,
Harb. Perspect. Biol., 3, a003616, doi: 10.1101/cshper
Makarewich, C.A., Nelson, B.R., McAnally, J.R.,
spect.a003616.
Kasaragod, P., Shelton, J.M., Liou, J., Bassel Duby, R.,
77.
Peebles, C.L., Perlman, P.C., Mecklenburg, K.L., Petrillo, M.L.,
and Olson, E.N. (2015) A micropeptide encoded by a puta
Tabor, J.H., Jarrell, K.A., and Cheng, H.L. (1986) A self
tive long noncoding RNA regulates muscle performance,
splicing RNA excises an intron lariat, Cell, 44, 213 223.
Cell, 160, 595-606, doi: 10.1016/j.cell.2015.01.009.
78.
Silas, S., Mohr, G., Sidote, D.J., Markham, L.M.,
96. Chuong, E.B., Elde, N.C., and Feschotte, C. (2017)
Sanchez Amat, A., Bhaya, D., Lambowitz, A.M., and
Regulatory activities of transposable elements: from con
Fire, A.Z. (2016) Direct CRISPR spacer acquisition from
flicts to benefits, Nat. Rev. Genet.,
18,
71-86,
RNA by natural reverse transcriptase Cas1 fusion protein,
doi: 10.1038/nrg.2016.139.
Science, 351, aad4234, doi: 10.1126/science.aad4234.
97. Fontdevila, A. (2005) Hybrid genome evolution by trans
79.
Elliott, T.A., and Greqory, T.R. (2015) Do larger genomes
position, Cytogenet. Genome Res., 110, 49-55.
contain more diverse transposable elements, BMC Evol.
98. Soemedi, R., Cygan, K.J., Rhine, C.L., Glidden, D.T.,
Biol., 15, 69-81, doi: 10.1186/s12862 015 0339 8.
Taggart, A.J., Lin, C.L., Fredericks, A.M., and
80.
Kubiak, M.R., and Makalowska, I. (2017) Protein coding
Fairbrother, W.G. (2017) The effects of structure on pre
genes’ retrocopies and their functions, Viruses, 9, pii: E80,
mRNA processing and stability, Methods, 125, 36-44,
doi: 10.3390/v9040080.
doi: 10.1016/j.ymeth.2017.06.001.
81.
Zdobnov, E.M., Campillos, M., Harrington, E.D.,
99. Kralovicova, J., Patel, A., Searle, M. and Vorechovsky, I.
Torrents, D., and Bork, P. (2005) Protein coding potential
(2015) The role of short RNA loops in recognition of a sin
of retroviruses and other transposable elements in verte
gle hairpin exon derived from a mammalian wide inter
brate genomes, Nucleic Acids Res., 33, 946-954.
spersed repeat, RNA Biol., 12, 54-69, doi: 10.1080/
82.
Campillos, M., Doerks, T., Shah, P.K., and Bork, P. (2006)
15476286.2015.1017207.
Computational characterization of multiple Gag like
100. Хавинсон В.Х., Соловьев А.Ю., Шатаева Л.К. (2006)
human protein, Trends Genet., 22, 585-589.
Молекулярный механизм взаимодействия олигопеп
83.
Sela, N., Kim, E., and Ast, G. (2010) The role of transpos
тидов и двойной спирали ДНК, Бюл. экспер. биол., 141,
able elements in the evolution of non mammalian verte
443-447.
brates and invertebrates, Genome Biol.,
11, R59,
101. Gladyshev, E.A., and Arkhipova, I.R. (2011) A widespread
doi: 10.1186/gb 2010 11 6 r59.
class of reverse transcriptase related cellular genes, Proc.
84.
Schmitz, J., and Brosius, J. (2011) Exonization of trans
Natl. Acad. Sci. USA, 108, 20311-20316, doi: 10.1073/
posed elements: a challenge and opportunity for evolution,
pnas.1100266108.
Biochimie, 93, 1928-1934, doi: 10.1016/j.biochi.2011.
102. Kim, S., and Choi, D. (2018) New role of LTR retrotrans
07.014.
posons for emergence and expansion of disease resistance
5 БИОХИМИЯ том 84 вып. 8 2019
1114
МУСТАФИН, ХУСНУТДИНОВА
genes and high copy gene families in plants, BMB Rep., 51,
107. Duan, C.G., Wang, X., Pan, L., Miki, D., Tang, K., Hsu, C.C.,
55-56.
Lei, M., Zhong, Y., Hou, Y.J., Wang, Z., Zhang, Z.,
103. Zhang, H., Tao, Z., Hong, H., Chen, Z., Wu, C., Li, X.,
Mangrauthia, S.K., Xu, H., Zhang, H., Dilkes, B., Tao,
Xiao, J., and Wang, S. (2016) Transposon derived small
W.A., and Zhu, J.K. (2017) A pair of transposon derived
RNA is responsible for modified function of WRKY45
proteins function in a histone acetyltransferase complex for
locus, Nat. Plants,
2,
16016-16023, doi:
10.1038/
active DNA demethylation, Cell Res., 27, 226-240,
nplants.2016.16.
doi: 10.1038/cr.2016.147.
104. Trizzino, M., Kapusta, A., and Brown, C.D.
(2018)
108. Sinzelle, L., Izsvak, Z., and Ivics, Z. (2009) Molecular
Transposable elements generate regulatory novelty in a tis
domestication of transposable elements: From detrimental
sue specific fashion, BMC Genomics, 19, 468, doi: 10.1186/
parasites to useful host genes, Cell. Mol. Life Sci., 66,
s12864 018 4850 3.
1073-1093, doi: 10.1007/s00018 009 8376 3.
105. Виноградов А.Е. (2011) Функциональное значение базо*
109. Wang, J., Vicente Garcia, C., Seruggia, D., Molto, E.,
вых свойств структуры генома эукариот. Дис. докт.
Fernandez Minan, A., Neto, A., Lee, E., Gomez
биол. наук, Санкт Петербург.
Skarmeta, J.L., Montoliu, L., Lunyak, V.V., and Jordan, I.K.
106. Joly Lopez, Z., and Bureau, T.E. (2018) Exaptation of
(2015) MIR retrotransposons sequences provide insulators
transposable element coding sequences, Curr. Opin. Genet.
to the human genome, Proc. Natl. Acad. Sci. USA, 112,
Dev., 49, 34-42, doi: 10.1016/j.gde.2018.02.011.
4428-4437, doi: 10.1073/pnas.1507253112.
THE ROLE OF REVERSE TRANSCRIPTASES IN THE ORIGIN OF LIFE
R. N. Mustafin1* and E. K. Khusnutdinova2
1 Bashkir State Medical University, 450008 Ufa, Russia; E*mail: ruji79@mail.ru
2 Institute of Biochemistry and Genetics, Ufa Federal Research Centre
of the Russian Academy of Sciences, 450054 Ufa, Russia
Received March 8, 2019
Revised May 14, 2019
Accepted May 14, 2019
We proposed that the ribozyme RNA polymerase, possessing reverse transcriptase and integration activity, played a
key role in the patterns of the origin of life on Earth. We presented the hypothesis that the universal ancestral units of
all living organisms were retroelements with reverse transcriptase and integrase activity. Their insertion capacity was
the basis for the formation of complex DNA structures, the primary genomes that gave the rise to all archaea, eukary
otes, bacteria and viruses. Conservative properties of retroelements are preserved throughout the evolution.
Modifications of their use have led to the emergence of new ways of interaction of proteins and nucleic acids. Life
evolved through insertional mutagenesis and competition of autonomously replicating polynucleotides thus preserv
ing the structures with adaptive properties. We assume that the natural selection of protection mechanisms against
insertions based on the ribonuclease ability of the reverse transcriptase ribozyme has led to the emergence of all uni
versal enzymatic systems for processing RNA molecules. These systems were and remain key sources of structural and
functional transformations of genomes in evolution. The data are presented enabling us to speculate that the transla
tion system, which united the world of RNA and DNA with proteins, arose as a modification of the protection mech
anisms against insertions. Polypeptides formed using this translation system began to potentiate the work of ribozymes
in the RNPs and even functionally replace them due to more efficient catalysis of biological reactions. We analyzed
the mechanisms of the use of retroelements in structural and regulatory transformations of eukaryotic genomes, which
may reflect the adaptive principles formed upon the origin of life. Simultaneously with the evolution of existing pro
teins, retroelements give rise to ribozymes, such as long non coding RNA. They can function in combination with
proteins in the RNPs and are also capable of independent catalytic activity and translation. Their genes have the
potential to transform into protein coding genes. That is, the conservative principles of the interactions of RNA,
DNA, and proteins, formed upon the origin of life on Earth, are used throughout the evolution.
Keywords: reverse transcriptase, polymerase, processing, ribozymes, retroelements, transposable elements, evolution
БИОХИМИЯ том 84 вып. 8 2019