БИОХИМИЯ, 2019, том 84, вып. 8, с. 1115 - 1128
УДК 577.21
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК ЧЕЛОВЕКА
Обзор
© 2019
Л.А. Зиновкина
Московский государственный университет им. М.В. Ломоносова,
факультет биоинженерии и биоинформатики, 119234 Москва, Россия;
электронная почта: luzinovkina@yandex.ru
Поступила в редакцию 26.03.2019
После доработки 26.03.2019
Принята к публикации 16.04.2019
Репликацию в митохондриях человека исследуют уже несколько десятилетий, однако ее механизм не поня
тен до сих пор. В последние 15 лет появилось много новых экспериментальных данных о митохондриаль
ной репликации, но они крайне противоречивы. Предложено три модели репликации: две асинхронных -
Strand displacement и RITOLS и одна синхронная - Strand(coupled. В асинхронных моделях ориджин Н цепи
начинает работать раньше, поэтому репликация цепей завершается не одновременно. Синхронная модель
более традиционна - в одном ориджине инициируются две вилки репликации с лидирующей и отстающей
цепями. Для каждой из трех моделей есть как подтверждающие, так и противоречащие ей эксперименталь
ные факты. Вероятно, единой модели митохондриальной репликации не существует. Можно предположить,
что уникальная репликативная машина митохондрий, возникшая в результате эндосимбиоза, успешно об
служивает митохондриальный геном, проявляя неожиданное разнообразие стратегий. Необычное сочета
ние ферментов, имеющих разное происхождение (фаговое, бактериальное, эукариотическое), а также уни
кальные особенности самого генома (разделение на тяжелую и легкую цепи, вставки рибонуклеотидов, раз
нообразие ориджинов), вероятно, дает возможность для репликации разными способами. В человеческих
митохондриях, по видимому, преобладает асинхронная репликация, но в определенных условиях реплика
ция может идти и синхронно. В сердце человека кольцевой митохондриальный геном перестраивается в
сеть из быстро воспроизводящихся линейных геномов, что говорит об очень широком спектре возможных
репликативных механизмов в митохондриях. В обзоре описаны основные стадии митохондриальной репли
кации и ее основные ферменты, а также обсуждаются перспективы исследований в этой области.
КЛЮЧЕВЫЕ СЛОВА: мтДНК, нуклеоид, D петля, репликация, strand displacement model, RITOLS, strand(
coupled model.
DOI: 10.1134/S0320972519080049
Митохондриальная ДНК (мтДНК) присут
кулу ДНК. Но встречаются линейные (у некото
ствует во всех описанных митохондриях. В про
рых кишечнополостных, водорослей, грибов и
цессе эволюции у эукариотических организмов
одноклеточных эукариот) митохондриальные
происходили значительные изменения предко
ДНК [1] и митохондриальные геномы, состоя
вого для всех митохондрий α протеобактери
щие из нескольких хромосом (у высших расте
ального генома. В связи с этим современные
ний и трипаносоматид) [2].
митохондриальные геномы весьма разнообраз
Самые крупные современные митохондри
ны и отличаются как по своей структуре, так и
альные геномы характерны для высших расте
по размеру. Чаще всего митохондриальный ге
ний, в среднем, 200-300 т.п.н. [3], рекорд со
ном представляет собой одну кольцевую моле
ставляет Silene conica - 11 млн п.н. [4]. У грибов
размеры митогенома почти на порядок меньше,
Принятые сокращения: мтДНК - митохондриаль
хотя и варьируют в широких пределах: 30-90
ная ДНК; Н цепь - тяжелая цепь, L цепь - легкая цепь; т.п.н. [5]. У позвоночных животных геномы го
POLRMT - РНК полимераза; TFAM - транскрипцион
ный фактор А митохондрий; SSB - single(strand binding pro(
раздо меньше, например, у млекопитающих -
tein; OH - ориджин репликации тяжелой цепи; OL - орид
16-18 т.п.н. [6].
жин репликации легкой цепи; ori b и ori z - ориджины b и z;
МтДНК млекопитающих - кольцевая двуце
NCR - non(coding region; CSB - conserved sequence blocks; почечная молекула, которая может, как показа
TAS - termination(associated sequence; TEFM - mitochondrial
ли электронно микроскопические исследова
transcription elongation factor; MGME1 - mitochondrial genome
maintenance exonuclease
1; RITOLS - RNA incorporated
ния, находиться в митохондриях в четырех раз
through out the lagging strand.
ных формах: релаксированное кольцо (open cir(
1115
5*
1116
ЗИНОВКИНА
cle), суперскрученное кольцо (supercoiled circle),
процессинг РНК и трансляцию. Кроме того, в
кольцевые мультимеры (в основном, димеры -
нуклеоиде должны содержаться белки, прик
head(to(tail circular dimer) и катенаны - кольца,
репляющие его к внутренней митохондриаль
замкнутые друг на друга [7]. Первые две формы
ной мембране. В настоящее время эту функцию
составляют большинство среди всех наблюдае
приписывают белку ATAD3 [16], но, возможно,
мых. Кольцевые мультимеры в значительных
у него есть партнеры.
количествах обнаружены в некоторых линиях
Идентификация компонентов нуклеоида
культивируемых клеток, а также в лейкоцитах
оказалась технически непростой задачей, в свя
человека при лейкемиях. Катенаноподобные
зи с чем разные научные коллективы получают
структуры в мтДНК млекопитающих составля
противоречивые данные [17]. Ниже приведен
ют 10-30% от всех форм ДНК и были описаны
список белков коровой части нуклеоида, кото
неоднократно. В человеческих клеточных лини
рые участвуют в репликации:
ях встречаются катенаны из 8 ми молекул ДНК,
1) РНК полимераза POLRMT (выполняет
и количество катенанов коррелирует с числом
функции праймазы в инициации репликации и
копий мтДНК [7].
осуществляет транскрипцию [18]);
Удивительно, что в противоположность че
2) транскрипционные факторы TFAM [19],
тырем перечисленным выше формам кольцевых
TFB1M/TFB2M, TEFM [20] (неoбходимы для
молекул, наблюдаемым в миокарде младенцев
работы РНК полимеразы POLRMT [21]);
или других видов млекопитающих (крыса,
3) ДНК полимераза γ (POLG) (осуществляет
мышь, кролик), в сердечной мышце взрослого
собственно репликацию мтДНК).
человека мтДНК образует многомерную сеть,
POLG состоит из одной каталитической
содержащую десятки линейных геномов [8]. По
(продукт гена polg размером 140 кДа) и двух до
хожим образом организована мтДНК у некото
полнительных (продукты гена polg2 размером по
рых растений, малярийного плазмодия и гри
55 кДа) субъединиц [22].
бов. В таких «переплетенных» геномах можно
Каталитическая субъединица имеет гомоло
наблюдать классические структуры Холлидея,
гию с ДНК полимеразой фага Т7 и, помимо по
образующиеся при гомологичной рекомбина
лимеразной, обладает тремя дополнительными
ции. В других тканях человека, за исключением
ферментативными активностями: 3’→5’ экзо
мозга (сеть в нем гораздо менее густая, но пе
нуклеазная активность позволяет полимеразе γ
реплетения также встречаются), такие структу
репарировать неправильно вставленные нуклео
ры мтДНК идентифицированы не были, что мо
тиды; за счет активности обратной транскрип
жет объясняться необходимостью интенсивной
тазы POLG может проходить часто встречаю
репликации, а, возможно, и рекомбинации, в
щиеся в мтДНК рибонуклеотидные вставки
тканях с наиболее функционально активными
[23]; и, наконец, 5’→3’ дезоксирибофосфатли
митохондриями [9].
азная активность необходима ДНК полимеразе γ
для участия в репарации BER [9, 24]. Каталити
ческая субъединица состоит из N концевого эк
СТРОЕНИЕ НУКЛЕОИДА
зонуклеазного и С концевого полимеразного
домена с характерной пространственной струк
МтДНК всегда находится в митохондриях в
турой, напоминающей ладонь, части этого до
форме комплексов с белками, эти комплексы
мена соответственно названы ладонью (palm),
принято называть «нуклеоидами» [10, 11]. Не
большим пальцем (thumb) и остальными пальца
смотря на то что исследования состава и струк
ми (fingers) [22]. Экзонуклеазный и полимераз
туры нуклеоидов важны для понимания всех
ный домены соединены спейсером, через кото
процессов, происходящих в митохондриях, и
рый к каталитической субъединице присоеди
ведутся не одно десятилетие, организация нук
няются дополнительные. Дополнительные
леоида и его точный белковый состав описаны
субъединицы необходимы для дополнительного
далеко не полностью [10].
связывания с ДНК и увеличения процессивнос
Митохондриальный нуклеоид млекопитаю
ти фермента. Мутации в генах polg и polg2 имеют
щих представляет собой структуру размером
сходное фенотипическое проявление, они при
~100 нм в диаметре [12, 13] с многоуровневой
водят к развитию хронической прогрессирую
организацией [14, 15]. Белки, физически взаи
щей офтальмоплегии (CPEO) [22];
модействующие с мтДНК, образуют коровую
4) ДНК хеликаза Twinkle;
часть нуклеоида и участвуют в процессах мито
5) топоизомеразы I снимают супервитки, об
хондриальной репликации, репарации и транс
разованные при движении репликативной вил
крипции. На внешнем уровне нуклеоида распо
ки и разъединяют катенаны (о них будет по
лагаются в основном белки, осуществляющие
дробнее написано ниже);
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1117
6) mtSSB - митохондриальный ортолог бак
рии и ЭПР. Вероятно, это необходимо для коор
териального белка SSB, связывающийся с одно
динации между делением митохондрий и репли
цепочечными участками ДНК, образующимися
кацией их ДНК, необходимой для распределе
в ходе репликации [25];
ния новообразованных нуклеоидов по мито
7) РНКаза H1 удаляет РНК затравки, обра
хондриальной сети [35].
зованные POLRMT;
Клетка обычно содержит несколько мито
8) ДНК лигаза 3 осуществляет сшивание
хондрий, а каждая митохондрия может содер
вновь синтезированной мтДНК в кольца, сши
жать десятки-сотни нуклеоидов [36], поэтому в
вает фрагменты Оказаки, участвует в репарации.
одной клетке обычно содержится множество
До сих пор нет единого мнения о числе ко
молекул мтДНК, для млекопитающих это, в
пий мтДНК в нуклеоиде. Ранние работы указы
среднем, 1000-10 000 копий на клетку [32].
вали на наличие в каждом нуклеоиде 1-2 моле
Мутации редко затрагивают все клеточные
кул ДНК [26], затем появилась серия публика
копии митохондриального генома. Поэтому
ций, демонстрирующих наличие в одном нукле
большинство мутаций в мтДНК находятся в ге
оиде, в среднем, 5-6 копий мтДНК [27-29]. Са
тероплазмическом состоянии, и фенотипиче
мая современная работа с применением слож
ское проявление таких мутаций зависит от уров
ных микроскопических методов подтвердила
ня гетероплазмии [9, 37]. Не исследован до кон
первоначальную точку зрения. [13]. Поэтому
ца вопрос о возможности обмена ДНК между
сейчас принято считать, что в большинстве нук
нуклеоидами. Это явление, сопровождавшееся
леоидов присутствует единичная молекула ми
рекомбинацией, было показано в единичной ра
тохондриальной ДНК. Вместе с тем высказыва
боте в 2004 г. [38]. Вероятно, такой обмен моле
ются предположения, что содержание ДНК в
кулами мтДНК может использоваться клеткой
нуклеоиде может быть разным в митохондриях
для отбора нормальных митогеномов в том слу
разных тканей организма и/или зависеть от ка
чае, когда в ней присутствуют как обычные, так
ких либо условий. Например, показано, что ко
и мутантные копии. В более поздних работах пе
личество молекул ДНК на один нуклеоид в со
реход молекул мтДНК из одного нуклеоида в
матических клетках выше, чем в половых [30].
другой не подтвердился [13, 29].
Кроме того, на состояние и количество
мтДНК в нуклеоиде большое влияние оказыва
ет белок TFAM. Он был описан как транскрип
РЕГУЛЯТОРНЫЕ УЧАСТКИ
ционный фактор: инициация транскрипции в
мтДНК И D'ПЕТЛЯ
митохондриях начинается с присоединения к
промоторной области TFAM, который изгибает
МтДНК млекопитающих - кольцевая двуце
ДНК, только после этого к ДНК может присое
почечная молекула. Ее цепи называются L (light)
диниться РНК полимераза [21]. Помимо этой,
и H (heavy) из за заметной разницы в плавучей
основной своей функции, TFAM способен свя
плотности в градиенте хлористого цезия.
зываться с мтДНК неспецифически по всей
Н цепь богата G, в L цепи G значительно мень
длине, изгибая и запетливая ее, что приводит к
ше, этому факту есть изящное объяснение: при
компактизации и, в конечном итоге, недоступ
асинхронной репликации (см. ниже) материн
ности ДНК для ферментов. В экспериментах in
ская Н цепь долгое время остается в одноцепо
vitro показано, что при увеличении концентра
чечном состоянии, и в ней происходит спонтан
ции TFAM резко возрастает число суперком
ное дезаминирование цитозина с образованием
пактизированных молекул ДНК [31, 32]. Поэто
тимина и аденина с образованием гуанина, что
му TFAM рассматривают как основной регулятор
постепенно «обогащает» Н цепь гуанином [39].
состояния ДНК в нуклеоидах: от его количества
Нужно отметить, что генетический код в мито
зависит количество молекул ДНК, способных к
хондриях имеет некоторые отличия от универ
экспрессии и/или репликации и репарации.
сального: триплеты AGA и AGG служат стоп
С помощью микроскопии с высоким разре
кодонами (в универсальном коде они кодируют
шением [33] показано, что существуют разные
Arg); AUA кодирует Met (Ile в универсальном
виды нуклеоидов. Можно предположить, что в
коде); UGA кодирует Trp (стоп кодон в универ
компактных нуклеоидах мтДНК неактивна, в то
сальном коде).
время как в более крупных нуклеоидах мтДНК
У человека и других млекопитающих в мито
вовлечена в транскрипцию/репликацию, а регу
геноме отсутствуют интроны и совсем мало не
лятором состояния ДНК в нуклеоиде служит
кодирующих участков. Наиболее протяженный
как раз TFAM [34]. Недавно показано, что нук
некодирующий фрагмент (NCR, non(coding
леоиды с активной репликацией мтДНК обыч
region) имеет длину ~ 700 п.н. и расположен
но расположены в местах контакта митохонд
между генами tRNAPro и tRNAPhe. NCR содержит
БИОХИМИЯ том 84 вып. 8 2019
1118
ЗИНОВКИНА
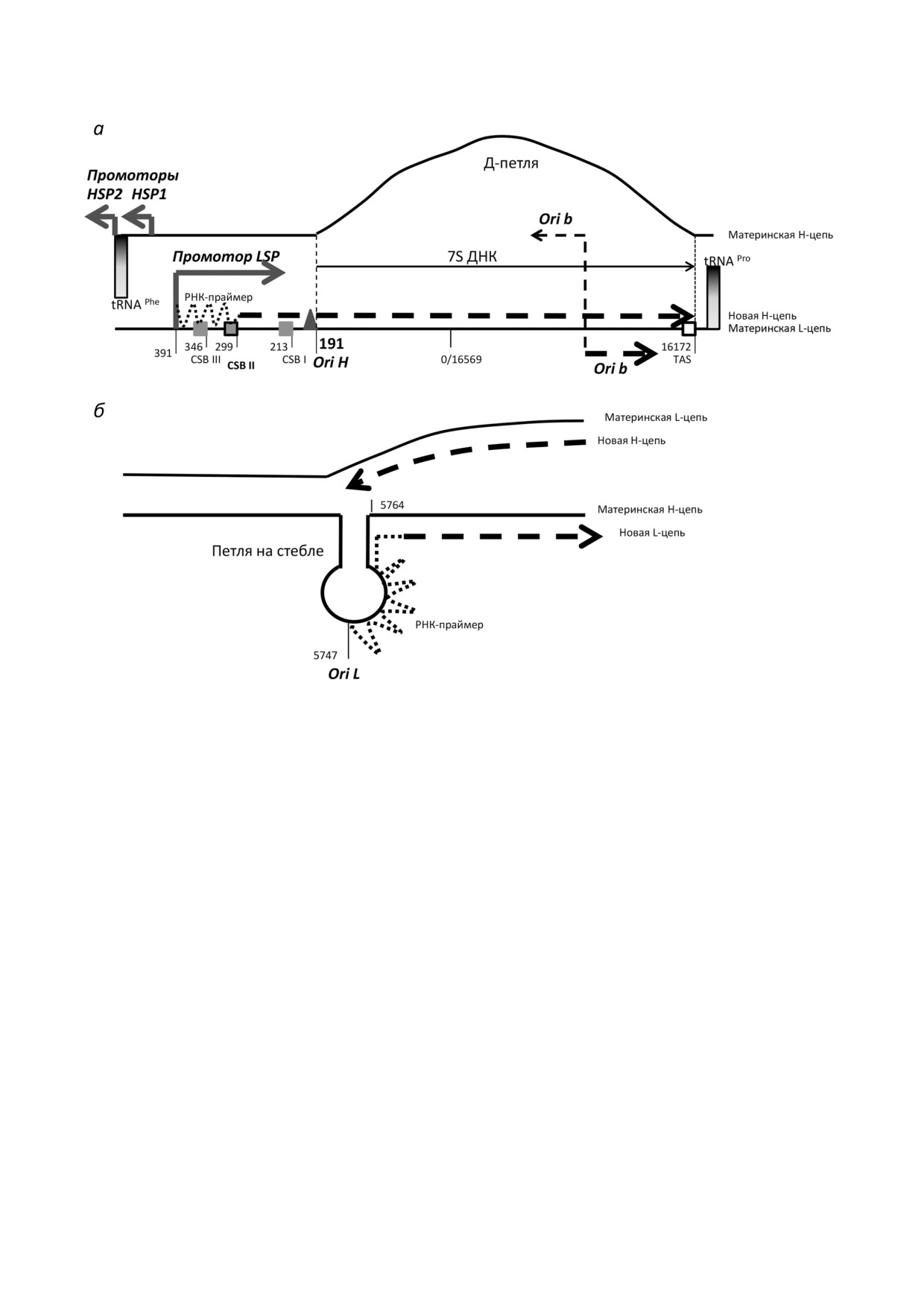
Рис. 1. Основные регуляторные элементы митохондриального генома человека. Числа обозначают положение элемента в
митогеноме. а - NCR, ограниченный генами tRNPhe и tRNAPro, содержит OH, двунаправленный ori b, промоторы HSP1 и
LSP. В участке CSBII предположительно происходит переключение с синтеза РНК праймера на синтез ДНК. При терми
нации репликации в TAS образуется 7S ДНК, а материнская Н цепь образует D петлю. Если терминации в TAS не про
исходит, происходит синтез полноразмерной Н цепи; б - инициация репликации в OL. Когда синтез новой Н цепи дохо
дит до OL, материнская Н цепь образует петлю на стебле, которая имеет одноцепочечный участок, где синтезируется
РНК праймер для образования новой L цепи
почти все регуляторные элементы митогенома,
nation(associated sequence) - здесь происходит
подробное описание которых будет дано в на
терминация репликации при образовании 7S
стоящем обзоре [40] (рис. 1, а): 1) ориджин ре
ДНК, образующей D петлю (см. ниже).
пликации OH и многочисленные дополнитель
Таким образом, большая часть регуляторных
ные ориджины (с них предположительно может
участков, в том числе и ответственных за репли
начинаться репликация согласно модели strand'
кацию (за исключением OL), компактно распо
coupled model (см. ниже), OL расположен в дру
ложена в NCR. Рассмотрим происходящие в
гом коротком некодирующем участке; 2) промо
нем процессы более подробно.
торы для обеих цепей LSP и HSP1 (третий про
Итак, митохондриальная репликация начи
мотор, HSP2, расположен совсем рядом - внут
нается в OH (или согласно некоторым моделям
ри гена tRNAPhe, но формально этот ген уже не
вблизи него, см. ниже). У существенной части
входит в NCR); 3) участки регуляции реплика
молекул (в разных работах и разных клетках
ции CSB (conserved sequence blocks) - три консер
процент молекул мтДНК с D петлей разный
вативных блока нуклеотидов, на CSB II при ре
[40]) репликация заканчивается на расстоянии
пликации происходит переключение с синтеза
650 п.н. в участке TAS (рис. 1, а). В этом случае
РНК праймера на синтез ДНК (см. ниже);
полноразмерная Н цепь не синтезируется, об
4) участок терминации репликации TAS (termi(
разуется только 7S ДНК. Это приводит к образо
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1119
ванию в NCR трицепочечной структуры, полу
ность в ее полноразмерных копиях), количество
чившей название D петля (от displacement loop):
Twinkle на ДНК резко возрастает, и уровень 7S
старая Н цепь выпетливается, что хорошо за
ДНК падает [42]. Таким образом, Twinkle каким
метно на электронных микрофотографиях [41].
то образом контролирует число копий мтДНК.
Несмотря на то что D петля описана более 40
Молекулярный механизм такой регуляции толь
лет назад, ее функции абсолютно неизвестны до
ко предстоит выяснить.
сих пор [40]. Совершенно непонятно, каков ме
D петля фланкирована двумя похожими
ханизм терминации репликации в TAS: почему в
между собой палиндромами длиной 15 п.н.
некоторых случаях терминация происходит, а в
(АTGN9CAT). Один расположен на 5’ конце 7S
некоторых нет - и тогда образуется полнораз
ДНК и входит в CSBI, а второй находится внут
мерная Н цепь. Очевидно, что терминация
ри терминатора TAS, чуть ниже 3’ конца 7S
должна регулироваться. Пока есть данные толь
ДНК. Есть предположение, что эти палиндромы
ко об участии в этой регуляции хеликазы
могут участвовать в регуляции синтеза 7S ДНК
Twinkle. Показано, что в нормальных условиях
[34]. Были предприняты попытки найти белки,
ДНК полимераза γ останавливается на 3’ конце
узнающие этот палиндром, но пока специфи
D петли, и Twinkle на ДНК в этот момент мало.
чески связывающиеся с этими последователь
Когда же самой мтДНК мало (т.е. есть потреб
ностями белки не идентифицированы.
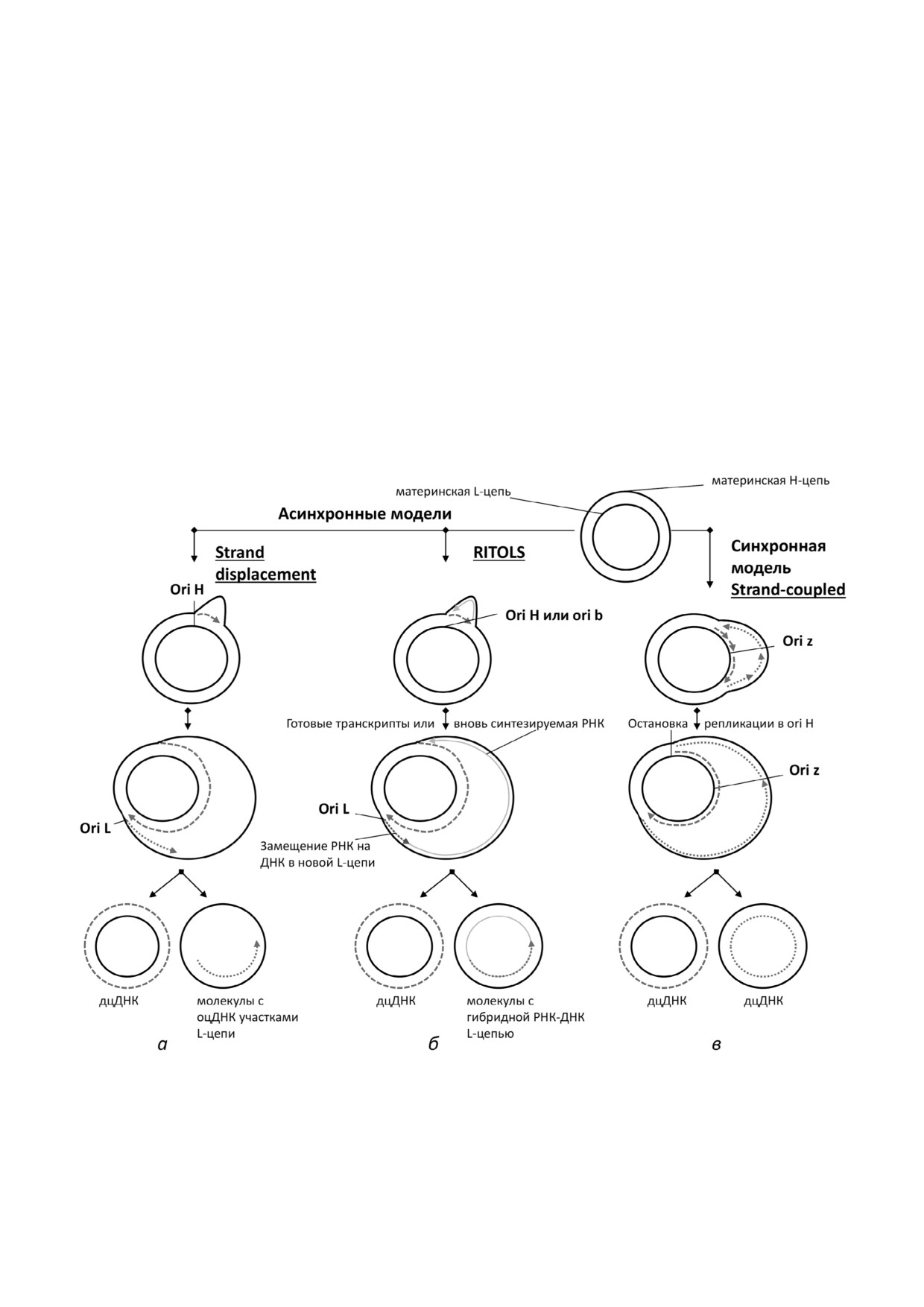
Рис. 2. Три основных модели репликации мтДНК млекопитающих. а и б - Асинхронные модели Strand displacement и
RITOLS соответственно; в - синхронная модель Strand coupled. Детальное описание моделей дано в тексте. Материнские
цепи мтДНК показаны черной сплошной линией, темно серым пунктиром с длинным штрихом показана новая Н цепь,
темно серым пунктиром с коротким штрихом показана новая L цепь, светло серой сплошной линией показана РНК в
составе L цепи
БИОХИМИЯ том 84 вып. 8 2019
1120
ЗИНОВКИНА
МОДЕЛИ РЕПЛИКАЦИИ мтДНК
для двунаправленной синхронной репликации
структуры [46]. Кроме того, в митогеноме между
В настоящее время рассматриваются три ос
ОH и последовательностью TAS были картиро
новных модели репликации двуцепочечного
ваны многочисленные дополнительные орид
кольцевого митогенома. Для каждой из трех есть
жины репликации [47]. На основе этих фактов
как подтверждающие, так и противоречащие
была предложена Strand'coupled model [46, 48],
наблюдения.
предполагающая двунаправленную репликацию
Исторически первой была предложена мо
из этих ориджинов с последующим синхронным
дель «вытеснения цепи» (Strand displacement
движением двух вилок по мтДНК [47] (рис. 2, в).
model) [43] (рис. 2, а). Она была предложена на
В каждой вилке есть лидирующая и отстающая
основе данных электронной микроскопии (на
цепи, последняя синтезируется в форме фраг
снимках наблюдали структуры с протяженными
ментов Оказаки. Вилка, двигающаяся в направ
одноцепочечными участками), а также обнару
лении NCR, быстро останавливается (в районе
жили чувствительность продуктов репликации к
ОH), и далее синтез ДНК происходит однона
нуклеазам, расщепляющим только оцДНК. Со
правленно. Таким образом, согласно второй мо
гласно этой модели, репликация начинается в
дели, репликация мтДНК происходит сначала
OH. Материнская Н цепь покрывается белком
двунаправленно, а затем однонаправленно, и
SSB, происходит синтез только новой Н цепи.
синхронно, что очень похоже на репликацию
Когда вилка репликации проходит примерно
бактериальных кольцевых геномов.
две трети генома, она достигает OL. Как только
В 2002 г. Hоlt et al. опубликовали данные о
ДНК в области OL становится одноцепочечной,
наличии среди промежуточных продуктов ре
она образует структуру stem(loop, что предотвра
пликации молекул РНК, комплементарно свя
щает связывание SSB в этой области. РНК по
занных с ДНК, причем рибонуклеотиды вклю
лимераза POLRMT, не способная связываться с
чались только в L цепь [49]. Это открытие стало
двуцепочечной матрицей, синтезирует праймер
сенсационным. Модель репликации с участием
длиной ~25 п.н. на одноцепочечном участке
фрагментов РНК назвали RITOLS (RNA incorpo(
петли [44] (рис. 1, б). Далее синтез продолжает
rated through out the lagging strand, ассоциация
ДНК полимераза γ. С этого момента обе цепи
РНК с отстающей цепью) [50] (рис. 2, б). Нали
синтезируются одновременно. Синтез Н цепи
чием таких РНК-ДНК гибридов можно легко
заканчивается раньше, поэтому при реализации
объяснить наблюдаемые многими исследовате
этой модели есть стадия, когда вторая молекула
лями протяженные одноцепочечные молекулы
ДНК (с недостроенной L цепью) имеет протя
ДНК, которые ранее связывали с асинхронной
женный одноцепочечный участок, что хорошо
репликацией по типу «вытеснения цепи»: РНК в
объясняет
экспериментальные
данные
гибридах при выделении митохондрий и раз
(рис. 2, а). Таким образом, в соответствии с дан
личных манипуляциях с препаратами легко де
ной моделью, митохондриальная репликация яв
градирует, поэтому легкая цепь остается одноце
ляется однонаправленной, асимметричной и
почечной. В таком случае можно было считать,
асинхронной. Помимо этого, данная модель ха
что репликация в митохондриях идет асинхрон
рактеризуется отсутствием фрагментов Оказаки
но, и при этом отстающая цепь сначала синте
(обязательных участников других систем реплика
зируется в форме РНК, которая затем заменяет
ции). Много лет эта модель была общепринятой.
ся на ДНК [49]. На этом этапе часть исследова
В 1990 е гг. для исследования механизмов
телей стали считать традиционную модель «вы
репликации ДНК в митохондриях группа Holt
теснения цепи» устаревшей и/или редкой, по
разработала метод двумерного электрофореза
скольку она никак не объясняет существование
молекул ДНК с разделением по размеру и кон
ДНК-РНК гибридов [7].
фигурации с последующим анализом продуктов
В 2006 г. с помощью комбинации описанно
с помощью электронной микроскопии. Кроме
го выше двумерного электрофоретического ана
того, промежуточные продукты репликации об
лиза и картирования 5’ концов ДНК в группе
рабатывали нуклеазами разной специфичности
Holt показали, что инициация репликации
[45]. С помощью такого подхода были получены
RITOLS у птиц и млекопитающих происходит в
новые экспериментальные данные, на основе
NCR - вблизи Он в ori b, который первоначаль
которых практически одновременно возникли
но был описан как ориджин для двунаправлен
две новых модели репликации мтДНК.
ной репликации [51], и в самом Он (рис. 1, а и
В своих экспериментах Holt et al. наряду с
рис. 2, б) репликация RITOLS тем не менее идет
наблюдаемыми и ранее одноцепочечными
асинхронно и однонаправленно: Н цепь синте
фрагментами ДНК, характерными для асин
зируется в виде ДНК, а отстающая L цепь сна
хронной репликации, обнаружили характерные
чала представляет собой РНК. Остается нере
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1121
шенным вопрос - в каком месте/местах генома
тавленные соответствовали зрелым транскрип
и в какое время начинает происходить замеще
там из разных участков мт генома [52].
ние РНК на ДНК в L цепи. По видимому, этот
Модель репликации RITOLS с использова
процесс может идти по разному: в некоторых
нием ранее синтезированных молекул РНК для
случаях вся отстающая цепь целиком синтези
синтеза отстающей цепи назвали моделью
руется как РНК и только после этого замещает
«шнурка» (Bootlace model, bootlace(SA replication
ся на РНК (печень цыпленка), в других молеку
(bootlace strand(asynchronous replication)) [52].
лах замещение РНК на ДНК начинается с нес
Согласно этой модели, предварительно сфор
кольких определенных локусов (печень цып
мированные транскрипты РНК на отстающей
ленка), во многих случаях замещение начинает
цепи последовательно включаются в промежу
ся с OL (печень мыши) [50].
точные продукты репликации по мере продви
Теоретически молекулы РНК, входящие в
жения вилки репликации мтДНК посредством
ДНК-РНК гибриды, образующиеся при реп
3’-5’ гибридизации с вытесненной материн
ликации, могут иметь разное происхождение:
ской H цепью, затем эти транскрипты заменя
1) либо для гибридизации с Н цепью использу
ются на отстающую цепь ДНК.
ются заранее синтезированные транскрипты,
Открытие в митохондриях промежуточных
2) либо эти транскрипты образуются одновре
репликативных комплексов в форме ДНК-РНК
менно с синтезом ДНК, т.е. РНК синтезируется
гибридов с самого начала вызвало острые дис
непосредственно в репликативной вилке.
куссии среди ученых, занимающихся реплика
В 2013 г. в группе Holt с помощью радиоактив
цией [53-55]. До сих пор часть исследователей с
ного мечения РНК и ДНК и введения в мито
недоверием относится к модели RITOLS [34].
хондрии ингибитора транскрипции было пока
С одной стороны, наличие РНК в репликатив
зано, что ингибирование синтеза РНК ожидае
ных комплексах доказано разными методами:
мо блокирует включение метки в образующиеся
эти комплексы выделены, они иммунопреципи
свободные транскрипты, а также ингибирует
тируются моноклональными антителами, спе
включение метки РНК в промежуточные ре
цифичными к РНК-ДНК гибридам [56], они
пликативные комплексы. При этом структура
образуют структуры с определенными, отлич
этих комплексов не изменялась, никаких изме
ными от других репликативных струкутур, ха
нений в репликации не происходило. Включе
рактеристиками при двумерном электрофорезе
ние метки ДНК в репликативные комплексы
[45]. С другой стороны, противники модели
при ингибировании транскрипции не снижа
репликации RITOLS высказывают два основ
лось, в то время как ингибитор синтеза ДНК
ных обоснованных аргумента: 1) белок SSB по
напротив быстро блокировал включение в нее
последним данным, полученным в эксперимен
метки. Эти данные свидетельствовали о том,
тах in vivo, плотно покрывает материнскую
что длинные участки РНК, входящие в проме
Н цепь во время репликации [57]. Такая карти
жуточные репликативные комплексы мтДНК,
на скорее соответствует модели «вытеснения це
не являются продуктами транскрипции, сопря
пи»;
2) в митохондриях показано наличие
женной с репликацией. Отсутствие эффекта
РНКазы Н1, которая эффективно работает
ингибитора синтеза РНК на репликацию
именно с РНК-ДНК гибридами. При этом ни
мтДНК исключает возможность того, что некая
какой системы регуляции активности РНКазы
праймаза отвечает за синтез РНК в отстающей
Н1, которая могла бы защитить от нее РНК в
цепи. Если бы такая праймаза была чувстви
промежуточных репликативных комплексах, в
тельна к ингибитору транскрипции, реплика
митохондриях пока не описано [34].
ция была бы задержана или сильно менялась бы
Поскольку, как видно из вышеописанного,
структура промежуточных репликативных
за последние годы накопилось множество до
комплексов. Напротив, праймаза, не чувстви
статочно сенсационных и противоречивых дан
тельная к ингибитору транскрипции, включала
ных о репликации в митохондриях, три описан
бы метку РНК в промежуточные комплексы,
ные выше схемы репликации (рис. 2) стали тре
чего не наблюдалось в экспериментах. Таким
бовать уточнений. Основываясь на конфигура
образом, РНК, обнаруженные в промежуточ
циях, наблюдаемых промежуточных реплика
ных продуктах репликации RITOLS, должны
тивных комплексов, а также учитывая тип орид
быть синтезированы до начала репликации
жина, с которого начинается репликация,
ДНК. Источником таких РНК могут быть зре
Yasukawa и Kang в своем последнем обзоре [45]
лые митохондриальные транскрипты. Действи
предложили выделять две, а не три возможных
тельно, в составе промежуточных репликатив
модели репликации:
ных комплексов, выделенных из гелей, были
1) асинхронная модель шнурка Bootlace объ
разные РНК, но самые длинные и часто предс
единяет модель вытеснения цепи и модель
БИОХИМИЯ том 84 вып. 8 2019
1122
ЗИНОВКИНА
RITOLS. Такое объединение стало возможным
жат для репликации только одной цепи, т.е. од
из за того, как уже отмечалось выше, реплика
нонаправленны.
тивные промежуточные комплексы для этих
Инициация на OН. Принято считать, что
двух моделей действительно могут выглядеть
асинхронная репликация мтДНК (модели
одинаково, если предположить, что в ряде случа
Bootlace SA, Strand displacement model и
ев, особенно в ранних работах, РНК в гибрид
RITOLS) начинается с OH или иногда с ori b, ко
ных молекулах деградировала (рис. 2, а и б). Ре
торый будет рассмотрен ниже. Синтез РНК
пликация начинается с синтеза H цепи в OH или
праймера для репликации начинается с промо
ori b. Синтез лидирующей цепи ДНК идет одно
тора LSP, вероятно, функции праймазы выпол
направленно с одновременным включением РНК
няет РНК полимераза POLRMT, по крайней
в отстающую цепь: митохондриальные транс
мере, никакой другой специализированной
крипты по мере прохождения вилки последова
праймазы в митохондриях пока не обнаружили.
тельно гибридизуются с родительской Н цепью.
РНК праймер имеет длину несколько десятков
Синтез отстающей цепи ДНК может иницииро
нуклеотидов, затем синтез ДНК продолжает
ваться в нескольких местах, не обязательно в OL.
ДНК полимераза γ (рис. 1, а). В 2015 г. [59] в
Молекулы РНК затем постепенно удаляются.
клетках мыши было показано присутствие кова
Стоит отметить, что такую асинхронную мо
лентных РНК-ДНК гибридов, у которых 5’ ко
дель подтверждают данные, полученные мето
нец РНК начинался в области LSP, а точка пере
дом Mito SMARD, позволяющим наблюдать
хода между РНК и ДНК была либо в OH , либо в
промежуточные продукты репликации с по
ori b. Не до конца понятно, как происходит пе
мощью флуоресцентных меток [58].
реключение с синтеза РНК на синтез ДНК, т.е.
2) Strand coupled DNA replication (SCD repli(
почему в некоторых случаях с LSP идет транс
cation) фактически с небольшими уточнениями
крипция с образованием полноразмерных моле
повторяет Strand coupled model (рис. 2, в). Ре
кул РНК, а в некоторых случаях образуется ко
пликация в рамках этого механизма начинается
роткий РНК праймер, и синтез продолжает
с широкой зоны в несколько килобаз, назван
ДНК полимераза. В 2015 г. [20] на роль «пере
ной ori z. Репликация здесь инициируется дву
ключателя» с транскрипции на репликацию был
направленно, и OН, по видимому, функциони
предложен TEFM. В опытах in vitro присутствие
рует как барьер для репликативной вилки. Син
TEFM препятствовало образованию G квад
тез лидирующей и отстающих цепей происходит
руплекса при транскрипции в участке CSBII, в
синхронно, обе они состоят из ДНК. Механизм
результате происходила транскрипция с образо
синтеза отстающей цепи пока непонятен до
ванием полноразмерной РНК. Если же TEFM
конца, видимо, она синтезируется отдельными
не было в системе, в участке CSBII РНК образо
фрагментами, напоминающими ядерные фраг
вывала G квадруплекс, транскрипция останав
менты Оказаки. Какой фермент исполняет роль
ливалась, и образовывался короткий праймер
праймазы, тоже не известно. Принято считать,
для репликации. К сожалению, переключение с
что РНК праймеры синтезирует POLRMT, но
синтеза РНК на синтез ДНК в CSBII не объяс
строго in vivo это не показано.
няет существования ковалентных РНК-ДНК
Обобщая данные о митохондриальных ре
гибридов, у которых точка перехода между РНК
пликативных моделях, можно заключить, что по
и ДНК находится либо в OH , либо в ori b, кото
ка полной ясности, по какому именно механизму
рые находятся на расстоянии от CSBII ~ 100 и
идет репликация в митохондриях, нет. Более то
500 нуклеотидов соответственно. Это противо
го, кажется все более вероятным, что единого ме
речие должно быть разрешено в будущем, когда
ханизма не существует, а в зависимости от усло
станут понятными механизмы регуляции транс
вий репликация в митохондриях может идти
крипции и репликации в митохондриях.
немного по разному - синхронно или асинхрон
Инициация на OL. Согласно моделям асин
но, с использованием РНК или без него.
хронной репликации, синтез L цепи начинается
в OL. Когда репликация Н цепи доходит до
участка WANCY, в котором расположен OL, ма
ОСНОВНЫЕ СТАДИИ РЕПЛИКАЦИИ
теринская Н цепь переходит в одноцепочечное
состояние и на ней образуется петля на стебле
Инициация репликации мтДНК. В мтДНК су
(рис. 1, б). Двуцепочечный стебель защищает од
ществуют два основных ориджина: OH (для син
ноцепочечную петлю от взаимодействия с SSB,
теза Н цепи), расположенный в NCR, и OL (для
и с одноцепочечным участком взаимодействует
синтеза L цепи), расположенный в коротком
РНК полимераза POLRMT, синтезирующая
некодирующем участке мтДНК внутри кластера
РНК праймер длиной ~ 25 нуклеотидов. Далее
генов пяти тРНК (WANCY). Оба ориджина слу
синтез продолжает ДНК полимераза γ. Иссле
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1123
дования OL in vivo и in vitro показали, что функ
[50], они могли образоваться и при асинхрон
ционировать как ориджин может структура с
ном однонаправленном синтезе.
двуцепочечным стеблем, богатым пиримидина
Таким образом, детали механизма работы ori b
ми, и одноцепочечной петлей длиной как мини
при инициации репликации еще предстоит вы
мум 10 нуклеотидов [60]. В связи с этим выска
яснить.
зывается предположение [61], что возможность
Двунаправленная инициация синтеза
образования дополнительных петель на стебле
мтДНК возможна в широкой области длиной в
на участке Н цепи между OL и OН может сни
несколько килобаз, получившей название ori z.
жать количество делеций, которые образуются
Начало репликации в ori z показано в тканях че
при репликации из за «залипания» прямых пов
ловека, крысы и цыпленка на фрагментах
торов в мтДНК друг на друга. Чем ближе нахо
мтДНК, вообще не содержащих NCR [47].
дятся OL и OН друг к другу, тем меньше пар пря
Это доказывает возможность инициации
мых повторов могут провзаимодействовать меж
репликации вне NCR. Видимо, c ori z начинает
ду собой, пока Н цепь находится в одноцепо
ся синхронная репликация, описываемая мо
чечном состоянии.
делью Strand'coupled model, поскольку промежу
Делеции в мтДНК вносят существенный
точные репликативные комплексы, образующи
вклад в продолжительность жизни. В работе [61]
еся при такой инициации, устойчивы к обработ
проведено любопытное теоретическое сравне
ке РНКазой Н, что свидетельствует об отсут
ние количества возможных ориджиноподобных
ствии в них РНК [45] (рис. 2, в).
структур на участке между OL и OН у трех видов
млекопитающих - мыши, человека и голого
землекопа. У долгоживущего землекопа насчи
ЭЛОНГАЦИЯ И ТЕРМИНАЦИЯ
тывается четыре дополнительные петли, теоре
тически способных служить ориджином, по
ДНК полимераза γ осуществляет реплика
сравнению с мышью, имеющей короткую про
цию, пока не пройдет полный круг. Лигирова
должительность жизни. У человека есть возмож
ние концов ДНК для образования кольцевой
ность образования одной дополнительной
молекулы в митохондриях осуществляет ДНК
ориджиноподобной структуры. К сожалению,
лигаза 3 [63, 64]. Для правильного лигирования
эта гипотеза пока не имеет экспериментальных
5’ и 3’ концы ДНК должны точно совпасть, по
подтверждений.
этому перед лигированием необходимо удале
Дополнительные ориджины в мтДНК. Описа
ние РНК праймеров РНКазой Н1 и процессинг
но также несколько дополнительных ориджи
концов молекулы ДНК. Нокаутные мыши без
нов. Оri b расположен вблизи гена tRNAPro [51] и
РНКазы Н1 не жизнеспособны, у них происхо
впервые описан как ориджин для двунаправлен
дит потеря мтДНК, а в мышиных эмбриональ
ной репликации (рис. 1, а). Однако по анализу
ных фибробластах, лишенных РНКазы Н1, в об
промежуточных репликативных структур, сог
ласти ориджина сохраняются РНК праймеры
ласно [50], с ori b начинается асинхронный од
[60, 65].
нонаправленный синтез Н цепи (рис. 2, б). Та
Процессинг концов мтДНК для лигирова
ким образом, непонятно как именно работает
ния пока не исследован детально. Пройдя пол
этот ориджин - как однонаправленный или как
ный круг, ДНК полимераза γ встречает 5’ конец
двунаправленный? Yasukawa и Kang [45] предла
только что синтезированной ею молекулы ДНК.
гают два возможных объяснения этому противо
В этом месте ДНК полимераза γ начинает рабо
речию (рис. 2): 1) ori b может функционировать
тать «вхолостую»: она осуществляет циклы по
в обеих моделях репликации - синхронной и
лимеризации деградации, благодаря своей
асинхронной. Поскольку широкая зона иници
3’-5’ экзонуклеазной активности. Такая «про
ации ori z включает в себя и ori b, можно предпо
буксовка» необходима для нормального лигиро
ложить, что в некоторых случаях область ини
вания: ДНК полимераза γ без экзонуклеазной
циации сужается; 2) при репликации по меха
активности не «буксует», а продолжает синтез с
низму Bootlace SA replication или RITOLS обра
образованием дцДНК, которая не может быть
зующие отстающую цепь фрагменты РНК так
субстратом для лигирования [65]. Вероятно,
быстро замещаются на ДНК, что при анализе
именно из за этого у мышей с дефектной ДНК
промежуточные структуры выглядят идентично
полимеразой γ без экзонуклеазной активности
тем, что образуются при репликации по меха
показано наличие одноцепочечных разрывов в
низму Strand'coupled model. В этом случае ori b
области ОН [65, 66].
работает только однонаправленно, и, хотя про
Вопрос точного картирования
5’ конца
межуточные продукты репликации выглядят
мтДНК перед лигированием не решен до конца.
как образованные при синхронной репликации
Принято считать, что в большинстве молекул
БИОХИМИЯ том 84 вып. 8 2019
1124
ЗИНОВКИНА
ДНК 5’ конец расположен в положении 191, это
ный механизм работы Top3α в митохондриях
часть Он, но каким именно образом образуется
еще предстоит описать, но уже сейчас можно
этот конец не совсем ясно [34]. Как уже отмеча
предполагать, что этот фермент работает в
лось выше, место перехода РНК праймера в
комплексе с несколькими другими белками, как
ДНК картируют в разных местах: или в участке
это показано для него в ядре. Но поскольку в
CSB II, или в Он (положение 191 или поблизос
митохондриях не показана локализация ни од
ти), или даже в ori b (еще дальше от CSB II)
ного из известных ядерных партнеров Top3α,
(рис. 1, а). Понятно, что, если точка перехода
скорее всего, механизм работы этой топоизоме
РНК/ДНК совпадает с местом лигирования,
разы в митохондриях немного отличается от
после удаления РНК праймера концы ДНК бу
описанного в ядре [72].
дут совпадать, и никакого дополнительного
процессинга не требуется. В случае же несовпа
Итак, в последние годы достигнут сущест
дения сайтов лигирования и перехода РНК
венный прогресс в исследованиях митохондри
праймера в ДНК, необходимо удалить не только
альной репликации. Некоторые исследователи
РНК праймер, но и 5’ концевой участок ДНК
рассматривают три модели репликации, некото
между точкой перехода РНК/ДНК и точкой ли
рые - две модели: синхронную и асинхронную.
гирования:
~100 нуклеотидов в случае
Но ни одна из моделей не описывает все имею
РНК/ДНК переключения в ОН и ~500 нуклеоти
щиеся экспериментальные данные. Поэтому
дов в случае РНК/ДНК перехода в ori b. В пос
единой модели репликации человеческого ми
леднем случае лигирование, вероятно, должно
тохондриального генома, видимо, просто не су
происходить не в положении 191, а в районе ло
ществует.
кализации ori b (рис.1, а).
Можно предполагать, что митохондрия, бу
В митохондриях охарактеризован фермент,
дучи потомком α протеобактерий, попавших в
который может удалять ДНК c 5’ конца - это
уникальные условия симбиоза с эукариотиче
5’-3’ экзонуклеаза MGME1 [67,68]. Вероятно,
ской клеткой, способна реплицировать свой ге
активность MGME1 необходима для нормаль
ном по разному в зависимости от своих внут
ного лигирования мтДНК: в человеческих клет
ренних потребностей и потребностей клетки в
ках с дефектной MGME1 нормального лигиро
целом. С одной стороны, репликация в митохон
вания не происходило, образовывались линей
дриях происходит автономно (независимо от
ные молекулы с делециями [67].
клеточного цикла), большинство ферментов
Таким образом, пока известно, что в процес
репликации отличаются от ядерных. С другой
сировании концов мтДНК для лигирования
стороны, митохондриальная репликация конт
ДНК лигазой 3 принимает участие экзонуклеа
ролируется ядром клетки: все репликативные
за MGME1 и РНКаза Н1. Скорее всего, в терми
ферменты закодированы в ядерном геноме,
нации репликации и лигировании участвуют и
экспрессия их генов контролируется клеткой на
другие дополнительные ферменты, которые бу
разных уровнях. Видимо, эндосимбиотическое
дут описаны в будущем.
происхождение и, как его следствие, - полуав
Необходимо отметить участие топоизомераз
тономность и позволяют митохондриям реали
в митохондриальной репликации. Митохондри
зовывать разные схемы репликации.
альная изоформа топоизомеразы 1 TOP1MT ра
Митохондриальную репликацию обеспечи
ботает в митохондриях совместно с реплисомой,
вает уникальный набор ферментов. ДНК поли
снимая суперспирализацию ДНК [69]. Нокаут
мераза γ, хеликаза TWINKLE и РНК полимера
ные мыши без гена Top1mt жизнеспособны, но у
за POLRMT имеют высокую степень гомологии
них увеличена суперспирализация мтДНК [70,
с соответствующими ферментами фага Т7 [73] и
71]. Кроме того, топоизомеразная активность
работают в человеческих клетках только в мито
необходима в митохондриях для разъединения
хондриях; транскрипционный фактор TFBM2,
катенанов, которые образуются при репликации
необходимый для инициации транскрипции, в
кольцевых геномов. Эту функцию, по видимо
том числе и для инициации синтеза РНК прай
му, выполняет митохондриальная изоформа
меров для репликации, произошел от рРНК ме
Top3α. Показано, что потеря Top3α приводит к
тилтрансферазы α протеобактерий [74]. Такое
уменьшению количества мтДНК и формирова
необычное сочетание ферментов, имеющих раз
нию сети из катенанов вместо единичных коль
ное происхождение и часто обладающими до
цевых молекул. У пациентов с мутациями в
полнительными ферментативными активностя
Top3α проявляются те же симптомы, что и при
ми, помимо основной, вероятно, может дать воз
мутациях в других ферментах митохондриаль
можность для репликации разными способами.
ной репликации - делеции в мтДНК в мышцах
Кроме того, сам митохондриальный геном
и хроническая офтальмоплегия [72]. Конкрет
тоже имеет уникальные черты. В одной из цепей
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1125
мтДНК значительно больше гуанина, что повы
OH. Этот факт также может быть объяснен
шает ее плавучую плотность, и отразилось в на
асинхронной репликацией - именно в одноце
звании - «тяжелая цепь». В составе обеих цепей
почечном участке материнской Н цепи между
мтДНК встречаются рибонуклеотидные вставки
OH и OL может происходить залипание прямых
[23], что необычно для двуцепочечных кольце
повторов друг на друга, что и приводит к деле
вых ДНК геномов. В человеческом митогеноме
циям [61].
большое количество ориджинов, среди которых
Тем не менее, экспериментальных данных,
встречаются однонаправленные и двунаправ
указывающих на синхронную репликацию, под
ленные. Не известно, являются ли эти особен
робно описанных в данном обзоре, также много.
ности следствием как раз уникальности фер
Можно предположить, что синхронная репли
ментативного аппарата, или же, наоборот, столь
кация необходима в каких то специфических
необычное сочетание ферментов потребовалось
условиях, например, когда нужно быстро полу
для воспроизведения не совсем обычного гено
чить большое число геномов. Вероятно, сущест
ма. Тем не менее, репликативная машина мито
вует некая регуляция, позволяющая митохонд
хондрий, возникшая в результате симбиоза, ус
риям выбирать репликативный механизм. Более
пешно обслуживает митохондриальный геном,
того, в сердце человека кольцевой митохондри
проявляя неожиданное разнообразие стратегий.
альный геном перестраивается в сеть из линей
По работам последних лет складывается впе
ных геномов [8] и тоже каким то образом ре
чатление, что в митохондриях преобладает
плицируется, что говорит об очень широком
асинхронная репликация. Это подтверждается
спектре возможных репликативных механизмов
большим количеством как прямых эксперимен
в митохондриях. Детали этих механизмов, а так
тальных исследований (см., например, [58]), так
же способы их регуляции, которые являются
и косвенными данными. Высокое содержание
очень интересными и необычными, предстоит
гуанина в тяжелой цепи легко объяснить асин
исследовать в будущем.
хронной репликацией: материнская Н цепь
долгое время остается в одноцепочечном состо
янии, и в ней происходит спонтанное дезамини
Финансирование. Работа выполнена при фи
рование цитозина с образованием тимина и аде
нансовой поддержке Российского фонда фунда
нина с образованием гуанина, что постепенно
ментальных исследований (проект 18 04 01110).
«обогащает» Н цепь гуанином [39]. Большин
Конфликт интересов. Авторы заявляют об от
ство детектируемых делеций, фланкированных
сутствии конфликта интересов.
прямыми повторами, в мтДНК человека проис
Соблюдение этических норм. Настоящая
ходит в «Major arc» - между OH и OL, несмотря
статья не содержит описания каких либо иссле
на то что число прямых повторов почти в два ра
дований с использованием людей или животных
за больше в «Minor arc» - участке между OL и
в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Burger, G., Gray, M.W., and Lang, B.F. (2003) Mito
6.
Anderson, S., Bankier, A.T., Barrell, B.G., de Bruijn, M.H.,
chondrial genomes: anything goes, Trends Genet., 19,
Coulson, A.R., Drouin, J., Eperon, I.C., Nierlich, D.P.,
709-716, doi: 10.1016/j.tig.2003.10.012.
Roe, B.A., Sanger, F., Schreier, P.H., Smith, A.J., Staden, R.,
2.
Kolesnikov, A.A., and Gerasimov, E.S. (2012) Diversity of
and Young, I.G. (1981) Sequence and organization of the
mitochondrial genome organization, Biochemistry (Moscow),
human mitochondrial genome, Nature, 290, 457-465, doi:
77, 1424-1435, doi: 10.1134/S0006297912130020.
10.1038/290457a0.
3.
Gualberto, J.M., Mileshina, D., Wallet, C., Niazi, A.K.,
7.
Pohjoismäki, J.L.O., and Goffart, S. (2011) Of circles,
Weber Lotfi, F., and Dietrich, A. (2014) The plant mito
forks and humanity: topological organisation and replica
chondrial genome: dynamics and maintenance, Biochimie,
tion of mammalian mitochondrial DNA, Bioessays, 33,
100, 107-120, doi: 10.1016/j.biochi.2013.09.016.
290-299, doi: 10.1002/bies.201000137.
4.
Sloan, D.B., Alverson, A.J., Chuckalovcak, J.P., Wu, M.,
8.
Pohjoismäki, J.L.O., Goffart, S., Tyynismaa, H., Willcox, S.,
McCauley, D.E., Palmer, J.D., and Taylor, D.R. (2012)
Ide, T., Kang, D., Suomalainen, A., Karhunen, P.J.,
Rapid evolution of enormous, multichromosomal genomes
Griffith, J.D., Holt, I.J., and Jacobs, H.T. (2009) Human
in flowering plant mitochondria with exceptionally high
heart mitochondrial DNA is organized in complex catenat
mutation rates, PLoS Biol., 10, e1001241, doi: 10.1371/
ed networks containing abundant four way junctions and
journal.pbio.1001241.
replication forks, J. Biol. Chem., 284, 21446-21457, doi:
5.
Foury, F., Roganti, T., Lecrenier, N., and Purnelle, B.
10.1074/jbc.M109.016600.
(1998) The complete sequence of the mitochondrial
9.
Zinovkina, L.A. (2018) Mechanisms of mitochondrial
genome of Saccharomyces cerevisiae, FEBS Lett., 440,
DNA repair in mammals, Biochemistry (Moscow), 83, 233-
325-331, doi: 10.1016/S0014 5793(98)01467 7.
249, doi: 10.1134/S0006297918030045.
БИОХИМИЯ том 84 вып. 8 2019
1126
ЗИНОВКИНА
10.
Spelbrink, J.N. (2010) Functional organization of mam
25.
McKinney, E.A., and Oliveira, M.T. (2013) Replicating
malian mitochondrial DNA in nucleoids: history, recent
animal mitochondrial DNA, Genet. Mol. Biol., 36,
developments, and future challenges, IUBMB Life, 62,
308-315, doi: 10.1590/S1415 47572013000300002.
19-32, doi: 10.1002/iub.282.
26.
Satoh, M., and Kuroiwa, T. (1991) Organization of multi
11.
Bogenhagen, D.F. (2012) Mitochondrial DNA nucleoid
ple nucleoids and DNA molecules in mitochondria of a
structure, Biochim. Biophys. Acta,
1819,
914-920,
human cell, Exp. Cell Res., 196, 137-140, doi: 10.1016/
doi: 10.1016/j.bbagrm.2011.11.005.
0014 4827(91)90467 9.
12.
Brown, T.A., Tkachuk, A.N., Shtengel, G., Kopek, B.G.,
27.
Iborra, F.J., Kimura, H., and Cook, P.R. (2004) The func
Bogenhagen, D.F., Hess, H.F., and Clayton, D.A. (2011)
tional organization of mitochondrial genomes in human
Superresolution fluorescence imaging of mitochondrial
cells, BMC Biol., 2, 9, doi: 10.1186/1741 7007 2 9.
nucleoids reveals their spatial range, limits, and membrane
28.
Legros, F., Malka, F., Frachon, P., Lombès, A., and Rojo, M.
interaction, Mol. Cell. Biol., 31, 4994-5010, doi: 10.1128/
(2004) Organization and dynamics of human mitochon
MCB.05694 11.
drial DNA, J. Cell Sci., 117, 2653-2662, doi: 10.1242/
13.
Kukat, C., Wurm, C.A., Spåhr, H., Falkenberg, M.,
jcs.01134.
Larsson, N. G., and Jakobs, S. (2011) Super resolution
29.
Gilkerson, R.W., Schon, E.A., Hernandez, E., and
microscopy reveals that mammalian mitochondrial
Davidson, M.M. (2008) Mitochondrial nucleoids maintain
nucleoids have a uniform size and frequently contain a sin
genetic autonomy but allow for functional complementa
gle copy of mtDNA, Proc. Natl. Acad. Sci. USA, 108,
tion, J. Cell. Biol., 181, 1117-1128, doi: 10.1083/jcb.
13534-12539, doi: 10.1073/pnas.1109263108.
200712101.
14.
Lee, S.R., and Han, J. (2017) Mitochondrial nucleoid:
30.
Van Blerkom, J. (2011) Mitochondrial function in the
shield and switch of the mitochondrial genome, Oxid.
human oocyte and embryo and their role in developmental
Med. Cell. Longev., 2017, 8060949, doi: 10.1155/2017/
competence, Mitochondrion, 11, 797-813, doi: 10.1016/
8060949.
j.mito.2010.09.012.
15.
Bogenhagen, D.F., Rousseau, D., and Burke, S. (2008)
31.
Kaufman, B.A., Durisic, N., Mativetsky, J.M., Costantino, S.,
The layered structure of human mitochondrial DNA
Hancock, M.A., Grutter, P., and Shoubridge, E.A. (2007)
nucleoids, J. Biol. Chem., 283, 3665-3675, doi: 10.1074/
The mitochondrial transcription factor TFAM coordinates
jbc.M708444200.
the assembly of multiple DNA molecules into nucleoid
16.
Holt, I.J., He, J., Mao, C. C., Boyd Kirkup, J.D.,
like structures, Mol. Biol. Cell, 18, 3225-3236, doi:
Martinsson, P., Sembongi, H., Reyes, A., and Spelbrink, J.N.
10.1091/mbc.e07 05 0404.
(2007) Mammalian mitochondrial nucleoids: organizing
32.
Farge, G., Mehmedovic, M., Baclayon, M., van den
an independently minded genome, Mitochondrion, 7,
Wildenberg, S.M.J.L., Roos, W.H., Gustafsson, C.M.,
311-321, doi: 10.1016/j.mito.2007.06.004.
Wuite, G.J.L., and Falkenberg, M. (2014) In vitro recon
17.
Hensen, F., Cansiz, S., Gerhold, J.M., and Spelbrink, J.N.
stituted nucleoids can block mitochondrial DNA replica
(2014) To be or not to be a nucleoid protein: a comparison
tion and transcription, Cell Rep., 8, 66-74, doi: 10.1016/
of mass spectrometry based approaches in the identifica
j.celrep.2014.05.046.
tion of potential mtDNA nucleoid associated proteins,
33.
Kukat, C., Davies, K.M., Wurm, C.A., Spåhr, H.,
Biochimie, 100, 219-226, doi: 10.1016/j.biochi.2013.
Bonekamp, N.A., Kühl, I., Joos, F., Polosa, P.L., Park, C.B.,
09.017.
Posse, V., Falkenberg, M., Jakobs, S., Kühlbrandt, W., and
18.
Arnold, J.J., Smidansky, E.D., Moustafa, I.M., and
Larsson, N. G. (2015) Cross strand binding of TFAM to a
Cameron, C.E. (2012) Human mitochondrial RNA poly
single mtDNA molecule forms the mitochondrial
merase: structure-function, mechanism and inhibition,
nucleoid, Proc. Natl. Acad. Sci. USA, 112, 11288- 11293,
Biochim. Biophys. Acta, 1819, 948-960, doi: 10.1016/
doi: 10.1073/pnas.1512131112.
j.bbagrm.2012.04.002.
34.
Falkenberg, M. (2018) Mitochondrial DNA replication in
19.
Ngo, H.B., Kaiser, J.T., and Chan, D.C. (2011) The mito
mammalian cells: overview of the pathway, Essays
chondrial transcription and packaging factor Tfam impos
Biochem., 62, 287-296, doi: 10.1042/EBC20170100.
es a U turn on mitochondrial DNA, Nat. Struct. Mol. Biol.,
35.
Lewis, S.C., Uchiyama, L.F., and Nunnari, J. (2016) ER
18, 1290-1296, doi: 10.1038/nsmb.2159.
mitochondria contacts couple mtDNA synthesis with
20.
Agaronyan, K., Morozov, Y.I., Anikin, M., and Temiakov, D.
mitochondrial division in human cells, Science, 353, 5549,
(2015) Mitochondrial biology. Replication transcription
doi: 10.1126/science.aaf5549.
switch in human mitochondria, Science, 347, 548-551,
36.
Malka, F., Lombès, A., and Rojo, M. (2006) Organization,
doi: 10.1126/science.aaa0986
dynamics and transmission of mitochondrial DNA: focus
21.
Morozov, Y.I., Parshin, A. V, Agaronyan, K., Cheung, A.C.M.,
on vertebrate nucleoids, Biochim. Biophys. Acta, 1763,
Anikin, M., Cramer, P., and Temiakov, D. (2015) A model
463-472, doi: 10.1016/j.bbamcr.2006.04.001.
for transcription initiation in human mitochondria,
37.
Wang, J., Schmitt, E.S., Landsverk, M.L., Zhang, V.W.,
Nucleic Acids Res., 43, 3726-3735, doi: 10.1093/nar/
Li, F. Y., Graham, B.H., Craigen, W.J., and Wong, L. J.C.
gkv235.
(2012) An integrated approach for classifying mitochondr
22.
Yin, Y.W. (2011) Structural insight on processivity, human
ial DNA variants: one clinical diagnostic laboratory’s expe
disease and antiviral drug toxicity, Curr. Opin. Struct. Biol.,
rience, Genet. Med., 14, 620-626, doi: 10.1038/gim.
21, 83-91, doi: 10.1016/j.sbi.2010.12.001.
2012.4.
23.
Berglund, A. K., Navarrete, C., Engqvist, M.K.M.,
38.
D’Aurelio, M., Gajewski, C.D., Lin, M.T., Mauck, W.M.,
Hoberg, E., Szilagyi, Z., Taylor, R.W., Gustafsson, C.M.,
Shao, L.Z., Lenaz, G., Moraes, C.T., and Manfredi, G.
Falkenberg, M., and Clausen, A.R. (2017) Nucleotide
(2004) Heterologous mitochondrial DNA recombination
pools dictate the identity and frequency of ribonucleotide
in human cells, Hum. Mol. Genet., 13, 3171-3179,
incorporation in mitochondrial DNA, PLoS Genet., 13,
doi: 10.1093/hmg/ddh326.
e1006628, doi: 10.1371/journal.pgen.1006628.
39.
Kennedy, S.R., Salk, J.J., Schmitt, M.W., and Loeb, L.A.
24.
Kazak, L., Reyes, A., and Holt, I.J. (2012) Minimizing the
(2013) Ultra sensitive sequencing reveals an age related
damage: repair pathways keep mitochondrial DNA intact,
increase in somatic mitochondrial mutations that are
Nat. Rev. Mol. Cell Biol., 13, 726-726, doi: 10.1038/
inconsistent with oxidative damage, PLoS Genet., 9,
nrm3439.
e1003794, doi: 10.1371/journal.pgen.1003794.
БИОХИМИЯ том 84 вып. 8 2019
РЕПЛИКАЦИЯ МИТОХОНДРИАЛЬНОЙ ДНК
1127
40.
Nicholls, T.J., and Minczuk, M. (2014) In D loop: 40
not burst, Trends Biochem. Sci., 28, 404-405, doi:
years of mitochondrial 7S DNA, Exp. Gerontol., 56,
10.1016/S0968 0004(03)00165 8.
175-181, doi: 10.1016/j.exger.2014.03.027.
55.
Holt, I.J., and Jacobs, H.T. (2003) Response: the mitochon
41.
Arnberg, A., van Bruggen, E.F., and Borst, P. (1971) The
drial DNA replication bubble has not burst., Trends Biochem.
presence of DNA molecules with a displacement loop in
Sci., 28, 355-6, doi: 10.1016/S0968 0004(03)00133 6.
standard mitochondrial DNA preparations, Biochim.
56.
Pohjoismäki, J.L.O., Holmes, J.B., Wood, S.R., Yang, M. Y.,
Biophys. Acta,
246,
353-357, doi:
10.1016/0005
Yasukawa, T., Reyes, A., Bailey, L.J., Cluett, T.J., Goffart,
2787(71)90147 X.
S., Willcox, S., Rigby, R.E., Jackson, A.P., Spelbrink, J.N.,
42.
Jemt, E., Persson, Ö., Shi, Y., Mehmedovic, M., Uhler, J.P.,
Griffith, J.D., Crouch, R.J., Jacobs, H.T., and Holt, I.J.
Dávila López, M., Freyer, C., Gustafsson, C.M.,
(2010) Mammalian mitochondrial DNA replication inter
Samuelsson, T., and Falkenberg, M. (2015) Regulation of
mediates are essentially duplex but contain extensive tracts
DNA replication at the end of the mitochondrial D loop
of RNA/DNA hybrid, J. Mol. Biol., 397, 1144-1155, doi:
involves the helicase TWINKLE and a conserved sequence
10.1016/j.jmb.2010.02.029.
element, Nucleic Acids Res.,
43,
9262-9275, doi:
57.
Miralles Fusté, J., Shi, Y., Wanrooij, S., Zhu, X., Jemt, E.,
10.1093/nar/gkv804.
Persson, Ö., Sabouri, N., Gustafsson, C.M., and
43.
Robberson, D.L., Kasamatsu, H., and Vinograd, J. (1972)
Falkenberg, M. (2014) In vivo occupancy of mitochondrial
Replication of mitochondrial DNA. Circular replicative
single stranded DNA binding protein supports the strand
intermediates in mouse L cells., Proc. Natl. Acad. Sci. USA,
displacement mode of DNA replication, PLoS Genet., 10,
69, 737-741.
e1004832, doi: 10.1371/journal.pgen.1004832.
44.
Fusté, J.M., Wanrooij, S., Jemt, E., Granycome, C.E.,
58.
Phillips, A.F., Millet, A.R., Tigano, M., Dubois, S.M.,
Cluett, T.J., Shi, Y., Atanassova, N., Holt, I.J., Gustafsson, C.M.,
Crimmins, H., Babin, L., Charpentier, M., Piganeau, M.,
and Falkenberg, M. (2010) Mitochondrial RNA poly
Brunet, E., and Sfeir, A. (2017) Single molecule analysis of
merase is needed for activation of the origin of light strand
mtDNA replication uncovers the basis of the common
DNA replication, Mol. Cell,
37,
67-78, doi:
deletion, Mol. Cell, 65, 527-538, doi: 10.1016/j.mol
10.1016/j.molcel.2009.12.021.
cel.2016.12.014.
45.
Yasukawa, T., and Kang, D. (2018) An overview of mam
59.
Holmes, J.B., Akman, G., Wood, S.R., Sakhuja, K.,
malian mitochondrial DNA replication mechanisms., J.
Cerritelli, S.M., Moss, C., Bowmaker, M.R., Jacobs, H.T.,
Biochem., 164, 183-193, doi: 10.1093/jb/mvy058.
Crouch, R.J., and Holt, I.J. (2015) Primer retention owing
46.
Holt, I.J., Lorimer, H.E., and Jacobs, H.T. (2000) Coupled
to the absence of RNase H1 is catastrophic for mitochon
leading and lagging strand synthesis of mammalian mito
drial DNA replication, Proc. Natl. Acad. Sci. USA, 112,
chondrial DNA, Cell, 100, 515-524, doi: 10.1016/S0092
9334-9339, doi: 10.1073/pnas.1503653112.
8674(00)80688 1.
60.
Wanrooij, S., Miralles Fusté, J., Stewart, J.B., Wanrooij, P.H.,
47.
Bowmaker, M., Yang, M.Y., Yasukawa, T., Reyes, A.,
Samuelsson, T., Larsson, N. G., Gustafsson, C.M., and
Jacobs, H.T., Huberman, J.A., and Holt, I.J.
(2003)
Falkenberg, M. (2012) In vivo mutagenesis reveals that OriL
Mammalian mitochondrial DNA replicates bidirectionally
is essential for mitochondrial DNA replication, EMBO Rep.,
from an initiation zone, J. Biol. Chem., 278, 50961-50969,
13, 1130-1137, doi: 10.1038/embor.2012. 161.
doi: 10.1074/jbc.M308028200.
61.
Khaidakov, M. (2016) Species specific lifespans: can it be a
48.
Holt, I.J., and Jacobs, H.T. (2014) Unique features of
lottery based on the mode of mitochondrial DNA replica
DNA replication in mitochondria: a functional and evolu
tion? Mech. Ageing Dev.,
155,
1-6, doi:
10.1016/
tionary perspective, Bioessays,
36,
1024-1031, doi:
j.mad.2016.02.012.
10.1002/bies.201400052.
62.
Lakshmipathy, U., and Campbell, C. (1999) The human
49.
Yang, M.Y., Bowmaker, M., Reyes, A., Vergani, L., Angeli, P.,
DNA ligase III gene encodes nuclear and mitochondrial
Gringeri, E., Jacobs, H.T., and Holt, I.J. (2002) Biased
proteins, Mol. Cell. Biol., 19, 3869-3876.
incorporation of ribonucleotides on the mitochondrial
63.
Puebla Osorio, N., Lacey, D.B., Alt, F.W., and Zhu, C.
L strand accounts for apparent strand asymmetric DNA
(2006) Early embryonic lethality due to targeted inactiva
replication, Cell, 111, 495-505, doi: 10.1016/S0092
tion of DNA ligase III, Mol. Cell. Biol., 26, 3935-3941,
8674(02)01075 9.
doi: 10.1128/MCB.26.10.3935 3941.2006.
50.
Yasukawa, T., Reyes, A., Cluett, T.J., Yang, M. Y.,
64.
Cerritelli, S.M., Frolova, E.G., Feng, C., Grinberg, A.,
Bowmaker, M., Jacobs, H.T., and Holt, I.J.
(2006)
Love, P.E., and Crouch, R.J. (2003) Failure to produce
Replication of vertebrate mitochondrial DNA entails tran
mitochondrial DNA results in embryonic lethality in
sient ribonucleotide incorporation throughout the lagging
Rnaseh1 null mice, Mol. Cell, 11, 807-815, doi: 10.1016/
strand, EMBO J., 25, 5358-5371, doi: 10.1038/sj.emboj.
S1097 2765(03)00088 1.
7601392.
65.
Macao, B., Uhler, J.P., Siibak, T., Zhu, X., Shi, Y., Sheng, W.,
51.
Yasukawa, T., Yang, M. Y., Jacobs, H.T., and Holt, I.J.
Olsson, M., Stewart, J.B., Gustafsson, C.M., and
(2005) A bidirectional origin of replication maps to the
Falkenberg, M. (2015) The exonuclease activity of DNA
major noncoding region of human mitochondrial
polymerase γ is required for ligation during mitochondrial
DNA, Mol. Cell, 18, 651-662, doi: 10.1016/j.molcel.2005.
DNA replication, Nat. Commun., 6, 7303, doi: 10.1038/
05.002.
ncomms8303.
52.
Reyes, A., Kazak, L., Wood, S.R., Yasukawa, T., Jacobs, H.T.,
66.
Trifunovic, A., Wredenberg, A., Falkenberg, M., Spelbrink, J.N.,
and Holt, I.J. (2013) Mitochondrial DNA replication pro
Rovio, A.T., Bruder, C.E., Bohlooly Y, M., Gidlöf, S.,
ceeds via a “bootlace” mechanism involving the incorpora
Oldfors, A., Wibom, R., Törnell, J., Jacobs, H.T., and
tion of processed transcripts, Nucleic Acids Res., 41,
Larsson, N. G. (2004) Premature ageing in mice express
5837-5850, doi: 10.1093/nar/gkt196.
ing defective mitochondrial DNA polymerase, Nature,
53.
Bogenhagen, D.F., and Clayton, D.A. (2003) The mito
429, 417-423, doi: 10.1038/nature02517.
chondrial DNA replication bubble has not burst, Trends
67.
Nicholls, T.J., Zsurka, G., Peeva, V., Schöler, S., Szczesny, R.J.,
Biochem. Sci.,
28,
357-360, doi:
10.1016/S0968
Cysewski, D., Reyes, A., Kornblum, C., Sciacco, M.,
0004(03)00132 4.
Moggio, M., Dziembowski, A., Kunz, W.S., and Minczuk, M.
54.
Bogenhagen, D.F., and Clayton, D.A. (2003) Concluding
(2014) Linear mtDNA fragments and unusual mtDNA
remarks: the mitochondrial DNA replication bubble has
rearrangements associated with pathological deficiency of
БИОХИМИЯ том 84 вып. 8 2019
1128
ЗИНОВКИНА
MGME1 exonuclease, Hum. Mol. Genet., 23, 6147-6162,
supercoiling of mtDNA in TOP1mt knockout mice and
doi: 10.1093/hmg/ddu336.
presence of topoisomerases IIα and IIβ in vertebrate mito
68. Kornblum, C., Nicholls, T.J., Haack, T.B., Schöler, S.,
chondria, Nucleic Acids Res., 42, 7259-7267, doi: 10.1093/
Peeva, V., Danhauser, K., Hallmann, K., Zsurka, G.,
nar/gku384.
Rorbach, J., Iuso, A., Wieland, T., Sciacco, M., Ronchi, D.,
72. Nicholls, T.J., Nadalutti, C.A., Motori, E., Sommerville, E.W.,
Comi, G.P., Moggio, M., Quinzii, C.M., DiMauro, S.,
Gorman, G.S., Basu, S., Hoberg, E., Turnbull, D.M.,
Calvo, S.E., Mootha, V.K. et al. (2013) Loss of function
Chinnery, P.F., Larsson, N. G., Larsson, E., Falkenberg, M.,
mutations in MGME1 impair mtDNA replication and
Taylor, R.W., Griffith, J.D., and Gustafsson, C.M. (2018)
cause multisystemic mitochondrial disease, Nat. Genet.,
Topoisomerase 3α is required for decatenation and segre
45, 214-219, doi: 10.1038/ng.2501.
gation of human mtDNA, Mol. Cell,
69,
9-23,
69. Stewart, L., Redinbo, M.R., Qiu, X., Hol, W.G., and
doi: 10.1016/j.molcel.2017.11.033.
Champoux, J.J. (1998) A model for the mechanism of
73. Huynen, M.A., Duarte, I., and Szklarczyk, R. (2013) Loss,
human topoisomerase I, Science,
279,
1534-1541,
replacement and gain of proteins at the origin of the mito
doi: 10.1126/science.279.5356.1534.
chondria, Biochim. Biophys. Acta, 1827, 224-231, doi:
70. Douarrure, C., Sobier, C., Dalla Rosa, I., Brata Das, B.,
10.1016/j.bbabio.2012.08.001.
Redon, C.E., Zhang, H., Neckers, L., and Pommier, Y.
74. Shutt, T.E., and Gray, M.W. (2006) Homologs of mito
(2012) Mitochondrial topoisomerase I is critical for mito
chondrial transcription factor B, sparsely distributed with
chondrial integrity and cellular energy metabolism, PLoS
in the eukaryotic radiation, are likely derived from the
One, 7, e41094, doi: 10.1371/journal.pone.0041094.
dimethyladenosine methyltransferase of the mitochondrial
71. Zhang, H., Zhang, Y. W., Yasukawa, T., Dalla Rosa, I.,
endosymbiont, Mol. Biol. Evol.,
23,
1169-1179,
Khiati, S., and Pommier, Y. (2014) Increased negative
doi: 10.1093/molbev/msk001.
DNA REPLICATION IN HUMAN MITOCHONDRIA
L. A. Zinovkina
Lomonosov Moscow State University, Faculty of Bioengeneering and Bioinformatics,
119234 Moscow, Russia; E(mail: luzinovkina@yandex.ru
Received March 26, 2019
Revised April 16, 2019
Accepted April 16, 2019
DNA replication in human mitochondria has been studied for several decades; however, its mechanism still remains
unclear. During the last 15 years, many new experimental data on the mitochondrial replication have appeared,
although extremely contradictory. Two asynchronous (strand displacement and RITOLS) and one synchronous
(strand coupled) replication models have been proposed. In the asynchronous models, replication from the origin in
the H chain starts earlier, so that the replication of the two chains ends at different times. The synchronous model is
more traditional and implies two replication forks with leading and lagging strands initiated at the same origin. For
each of the three models, both confirming and contradicting experimental data exist. Most likely, there is no single
model of mitochondrial replication. It is possible that the unique mitochondrial replication machinery that has orig
inated as a results of endosymbiosis has an unexpected variety of replication strategies to maintain the mitochondrial
genome. An unusual combination of enzymes of different origin (phage, bacterial, eukaryotic) and unique features of
the mitochondrial genome (existance of heavy and light chains, insertions of ribonucleotides, a variety of origins) can
allow replication through different mechanisms. In human mitochondria, asynchronous replication seems to domi
nate; however, synchronous replication is also possible under certain conditions. In the human heart mitochondria,
circular mitochondrial DNA (mtDNA) molecules can rearrange in a network of rapidly replicating linear genomes,
thereby suggesting possible existence of a wide range of replication mechanisms in the mitochondria. The review
describes the main stages of mtDNA replication and enzymes involved in this process, as well as discusses the
prospects of mitochondrial replication studies.
Keywords: mtDNA, nucleoid, D loop, replication, strand displacement model, RITOLS, strand coupled model
БИОХИМИЯ том 84 вып. 8 2019