БИОХИМИЯ, 2019, том 84, вып. 9, с. 1244 - 1258
УДК 577.24
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ БЕЛКОВ
ЭКСЦИЗИОННОЙ РЕПАРАЦИИ НУКЛЕОТИДОВ
И ИХ РОЛЬ В РЕГУЛЯЦИИ ПРОЦЕССА
Обзор
© 2019
Н.И. Речкунова1,2*, Е.А. Мальцева1, О.И. Лаврик1,2
1 Институт химической биологии и фундаментальной медицины СО РАН, 630090 Новосибирск, Россия;
электронная почта: nadyarec@niboch.nsc.ru
2 Новосибирский государственный университет, 630090 Новосибирск, Россия
Поступила в редакцию 21.03.2019
После доработки 26.04.2019
Принята к публикации 15.05.2019
Эксцизионная репарация нуклеотидов (NER) - один из путей репарации ДНК, направленных на поддер
жание стабильности генома. Исправление повреждений системой NER - сложный многостадийный про
цесс, протекающий с образованием множества промежуточных комплексов, сборка и функционирование
которых осуществляются за счет ДНК-белковых и белок-белковых взаимодействий, требующих четкой
координации и регуляции. Белки NER подвергаются посттрансляционным модификациям, таким как
убиквитинирование, сумоилирование, фосфорилирование, ацетилирование и поли(ADP рибозил)ирова
ние. Эти модификации влияют на взаимодействие белков с ДНК и другими белками и таким образом могут
регулировать привлечение факторов репарации в комплекс на определенной стадии процесса или диссоциа
цию их из комплекса, а также модулируют функциональную активность белков и процесса в целом. В на
стоящем обзоре рассмотрены основные известные к настоящему времени посттрансляционные модифика
ции белков NER и данные об их влиянии на процесс репарации. Наиболее детально проанализировано по
ли(ADP рибозил)ирование белков, катализируемое поли(ADP рибоза)полимеразой 1, и его влияние на ак
тивность процесса NER, поскольку ранее такой анализ не проводился.
КЛЮЧЕВЫЕ СЛОВА: факторы эксцизионной репарации нуклеотидов, посттрансляционные модифика
ции белков, регуляция активности.
DOI: 10.1134/S0320972519090033
Геномная ДНК подвержена повреждениям
личных патологий человека, включая рак и ней
вследствие внутренней неустойчивости, а также
родегенеративные заболевания. Для противо
под действием эндогенных факторов и экзоген
стояния этим неблагоприятным последствиям в
ных генотоксических агентов. Повреждения
ходе эволюции возникли разнообразные пути
ДНК могут приводить к нарушениям основных
репарации ДНК [1], среди которых важное мес
процессов ее метаболизма, включая реплика
то занимает эксцизионная репарация нуклеоти
цию и транскрипцию, тем самым вызывая мута
дов (nucleotide excision repair, NER). NER - наи
ции, хромосомные аберрации, а также останов
более универсальный путь репарации, с по
ку клеточного цикла и апоптоз. Последствия
мощью которого происходит удаление широко
повреждений ДНК могут быть причиной раз
го спектра повреждений, дестабилизирующих
дуплексную структуру ДНК. К субстратам NER
относятся индуцированные ультрафиолетовым
Принятые сокращения: NER - эксцизионная репа
рация нуклеотидов; GG NER - общегеномная репарация (УФ) светом фотопродукты - циклобутанпири
нуклеотидов; TC NER - репарация нуклеотидов, связан
мидиновые димеры (CPD) и пиримидин пири
ная с транскрипцией; RNAP - РНК полимеразы; XP -
пигментная ксеродерма (xeroderma pigmentosum); ХРС,
мидон (6 4) фотопродукты (6 4PP), а также
ХРA - факторы пигментной ксеродермы; RPA - реплика
внутрицепочечные сшивки и объемные аддукты
тивный белок А; PARP - поли(ADP рибоза)полимераза; оснований ДНК с реакционноспособными ме
CPD - циклобутанпиримидиновые димеры; CS - синдром таболитами некоторых химических канцероге
Коккейна; TFIIH - фактор транскрипции IIH; PAR - по
нов или химиотерапевтическими агентами [2].
лимер ADP рибозы; BPDE - бенз[a]пирен диолэпоксид;
SUMO - убиквитин подобный белок модификатор (small
Существуют два пути NER: общегеномная
ubiquitin like modifier).
репарация (global genome repair, GG NER), уда
* Адресат для корреспонденции.
ляющая повреждения во всей геномной ДНК, и
1244
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1245
репарация, связанная с транскрипцией (tran
ства промежуточных комплексов, сборка и
scription coupled repair, TC NER), с помощью
функционирование которых осуществляются за
которой происходит удаление повреждений в
счет ДНК-белковых и белок-белковых взаимо
транскрибируемой цепи ДНК. Эти пути отлича
действий, требующих четкой координации и ре
ются способом узнавания повреждения и набо
гуляции. Известно, что ряд белков NER подвер
ром белков, участвующих на начальных этапах
гается посттрансляционным модификациям,
процесса. В TC NER узнавание повреждения
таким как убиквитинирование, сумоилирова
сопряжено с остановкой на нем РНК полиме
ние, фосфорилирование, ацетилирование и по
разы II (RNAP II) [3], что служит сигналом для
ли(ADP рибозил)ирование. Эти модификации
сборки инициирующего репарацию комплекса,
влияют на взаимодействие белков с ДНК и дру
в состав которого входят белки CSA и CSB [4].
гими белками и таким образом могут регулиро
Мутации в генах этих белков вызывают синдром
вать привлечение факторов репарации в ком
Коккейна (Cockayne syndrome, CS). В случае
плекс на определенной стадии процесса или дис
GG NER узнавание повреждения осуществляет
социацию их из комплекса, а также модулируют
фактор пигментной ксеродермы С (ХРС) в
активность модифицированных белков и про
комплексе с белками RAD23B и центрин 2
цесса в целом. В настоящем обзоре рассмотрены
(Cen2). Комплекс ХРС/RAD23B/Cen2 (далее
известные к настоящему времени посттрансля
комплекс XPC) определяет нарушение компле
ционные модификации белков NER и данные
ментарности в парах оснований и/или их деста
об их влиянии на процесс репарации. Наиболее
билизацию, вызванную повреждением, а не са
детально проанализированы результаты иссле
мо повреждение ДНК [5, 6], и взаимодействует с
дований поли(ADP рибозил)ирования, катали
неповрежденным участком противоположной
зируемого поли(ADP рибоза)полимеразой
1
цепи [7-10]. Этот механизм обеспечивает ши
(PARP1), поскольку ранее такой анализ не про
рокую субстратную специфичность процесса
водился.
GG NER, однако не позволяет узнавать УФ ин
дуцированные фотопродукты, особенно CPD,
которые практически не дестабилизируют
УБИКВИТИНИРОВАНИЕ
ДНК дуплекс. Узнавание таких повреждений
И СУМОИЛИРОВАНИЕ
осуществляет специализированный белок UV
DDB (UV damaged DNA binding protein) - гете
Убиквитин (Ub), небольшой (76 а.о.) высо
родимер, состоящий из субъединиц DDB1 и
коконсервативный полипептид, обнаружен у
DDB2 с молекулярной массой 127 и 48 кДа соот
всех эукариот. Одна или несколько молекул Ub
ветственно, который специфически взаимодей
с помощью каскада ферментативных реакций
ствует с CPD и 6 4PP и привлекает к ним XPC
ковалентно присоединяются к белку акцептору.
[11, 12]. Далее комплекс XPC (либо комплекс
Различают моноубиквитинирование (присое
факторов TC NER) взаимодействует с мульти
динение одной молекулы Ub), полиубиквити
функциональным 10 субъединичным белком -
нирование (присоединение полиубиквитино
фактором транскрипции IIH (transcription factor
вых цепей к одному сайту белка акцептора) и
IIH, TFIIH), который осуществляет частичное
мультимоноубиквитинирование (присоедине
раскручивание ДНК дуплекса за счет геликаз
ние одиночных молекул Ub к разным сайтам
ной активности субъединицы XPD и одновре
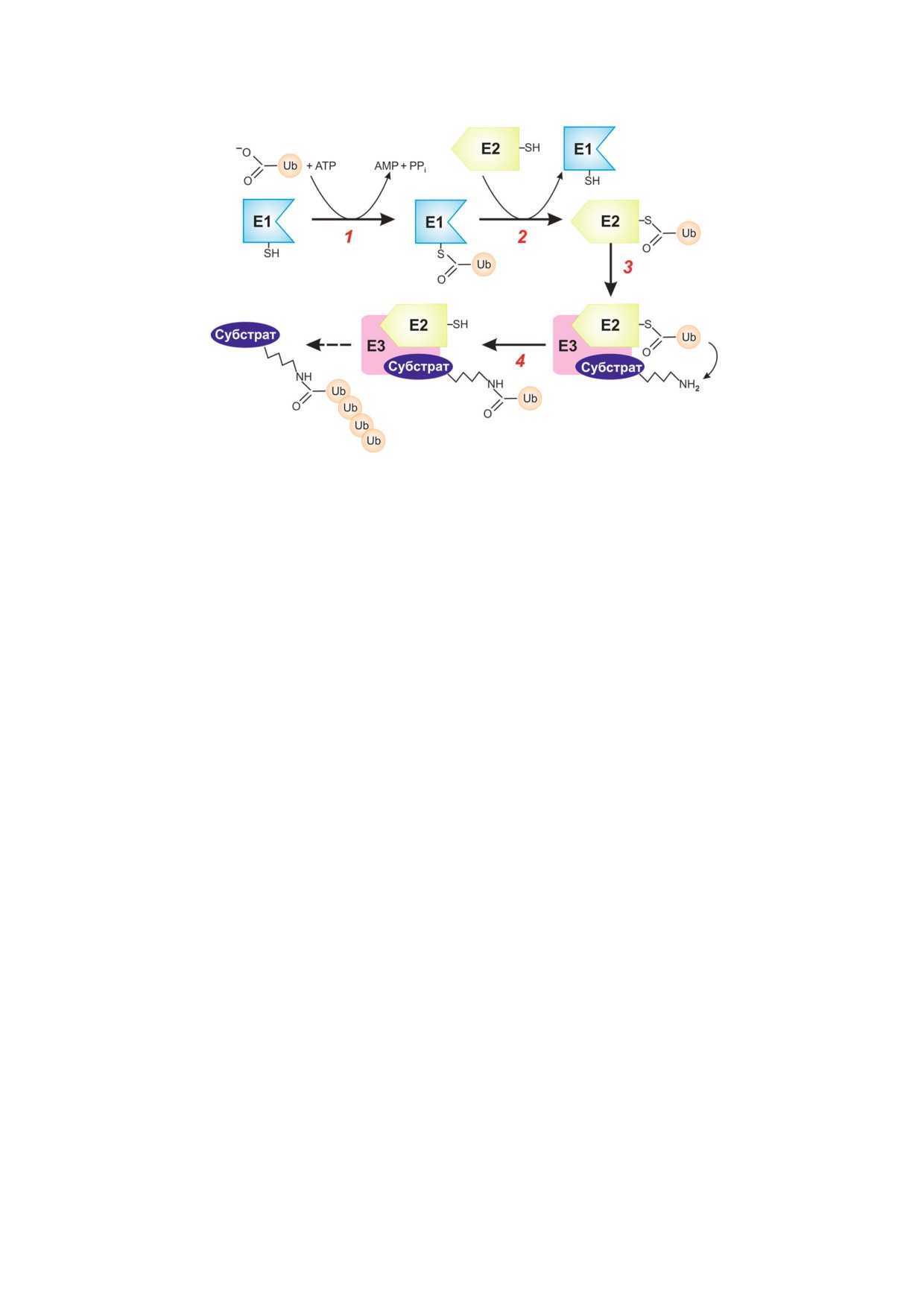
белка акцептора). Убиквитинирование включа
менно проверку повреждения ДНК, останавли
ет:
1) АТР зависимую активацию Ub под
ваясь на нем [13, 14]. На частично открытом
действием активирующего фермента Е1 с обра
дуплексе формируется так называемый «пред
зованием промежуточного тиоэфира Ub—Е1,
расщепляющий» комплекс, в состав которого
2) перенос активированного Ub на конъюгирую
входят репликативный белок А (RPA), фактор
щий фермент Е2, 3) образование комплекса
пигментной ксеродермы А (XPA), а также струк
между тиоэфиром Ub—E2, белком мишенью и
туроспецифичные эндонуклеазы XPF ERCC1 и
убиквитинлигазой Е3, 4) присоединение Ub к
XPG, расщепляющие поврежденную цепь ДНК
белку акцептору (рис. 1). Убиквитинирование
с 5′ и 3′ стороны от повреждения соответствен
влияет на функциональную активность белков,
но [14-16]. Ресинтез удаленного участка осуще
их клеточную локализацию и выполняет сиг
ствляет репликативный комплекс, лигирование
нальную и регуляторную функции во многих
ника - ДНК лигаза I или комплекс ДНК лига
процессах, таких как протеасомная деградация
зы III с XRCC1 [17].
и транслокация белков, транскрипция, кон
Таким образом, исправление повреждений
троль клеточного цикла, репарация ДНК и дру
системой NER - сложный многостадийный
гие [18, 19]. В образовании полиубиквитиновых
процесс, протекающий с образованием множе
цепей могут принимать участие разные остатки
БИОХИМИЯ том 84 вып. 9 2019
1246
РЕЧКУНОВА и др.
Рис. 1. Схема процесса убиквитинирования. 1 - АТР зависимая активация Ub под действием активирующего фермента Е1
с образованием тиоэфира Ub—Е1; 2 - перенос активированного Ub на конъюгирующий фермент Е2; 3 - образование ком
плекса между тиоэфиром Ub—E2, белком мишенью и убиквитинлигазой Е3; 4 - присоединение Ub к белку акцептору.
biokhsm/
лизина (К) в структуре убиквитина. Как прави
Полиубиквитинированный DDB2 диссоции
ло, модификация по 48 му остатку лизина (K48
рует из комплекса с поврежденной ДНК и под
тип) приводит к дальнейшей деградации белков
вергается протеасомной деградации, тогда как
протеасомой 26S [20].
модификация XPC обратима и не приводит к
Известно, что ряд факторов NER подверга
его диссоциации и последующей деградации, а,
ется полиубиквитинированию. В настоящий
напротив, увеличивает сродство к поврежден
момент наиболее подробно изучена модифика
ной ДНК [24]. Считалось, что таким образом
ция факторов, узнающих повреждение в ДНК в
осуществляется замена DDB2, узнающего УФ
процессе GG NER - DDB2 и XPC. Результаты
индуцированное повреждение в ДНК, на XPC,
систематизированы в ряде обзоров [21-23].
который инициирует процесс репарации [23].
Первоначально было открыто, что оба белка яв
Однако позже появились данные о том, что
ляются субстратами убиквитинлигазного ком
XPC, наоборот, становясь приоритетной ми
плекса CRL4ADDB2, в состав которого входят
шенью для убиквитинлигазы, защищает DDB2
куллин 4A (CUL4A), белок, содержащий RING
от полиубиквитинирования и последующей
домен, RBX1 (другое название ROC1) и димер
протеолитической деградации; если XPC недо
DDB1/DDB2 [24]. В общем случае CUL4A и
статочно, DDB2 протеолизуется и репарация не
RBX1 составляют каталитическое ядро убикви
идет [29]. Прямых данных о том, к какому типу
тинлигазы, DDB1 играет роль адаптера для при
относятся полиубиквитиновые цепи, синтези
соединения к убиквитинлигазному комплексу
руемые CRL4ADDB2 на XPC и DDB2, нет. Пред
субстратных рецепторов - белков, в частности
полагается, что это цепи K48 типа, поскольку
DDB2, которые, в свою очередь, отвечают за
полиубиквитинирование DDB2 приводит к по
привлечение белков субстратов [25]. DDB2 в
следующему его протеолизу. Более того, извест
данном случае является и субстратом, и частью
но, что оба белка, DDB2 и XPC, действительно
молекулярной машины, осуществляющей поли
могут модифицироваться полиубиквитиновыми
убиквитинирование как его самого, так и других
цепями K48 типа, после чего становятся
белков - ХРС и некоторых гистонов [26, 27].
субстратами для сегрегазы p97 - шаперона,
После УФ облучения клеток UV DDB оказыва
участвующего в том числе в реорганизации по
ется прочно связанным с хроматином, в резуль
лиубиквитинированных белков, способствуя их
тате чего от ассоциированной с ним убиквитин
последующей деградации [30]. Подавление син
лигазы диссоциирует сигналосома COP9, и
теза p97 в случае DDB2 приводит к подавлению
убиквитинлигаза активируется [25, 28].
деградации белка, а в случае XPC - к увеличе
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1247
нию количества убиквитинированного белка.
могут подвергаться сумоилированию, однако
Как следствие, увеличивается время задержки
роль этой модификации в NER до конца не яс
обоих белков на повреждении, что снижает эф
на. Так, известно, что центрин 2 модифициру
фективность репарации. Неизвестно, почему
ется SUMO2, что имеет значение для ядерной
убиквитинирование XPС K48 типа не приводит
локализации и взаимодействия с XPC, но не за
к его протеолитической деградации. Предпола
висит от УФ облучения [38]. DDB2 модифици
гается, что в стабилизации белка могут играть
руется преимущественно SUMO1, и эффектив
роль как RAD23B, так и деубиквитинирующие
ность модификации зависит от УФ облучения.
ферменты [31].
Сумоилирование DDB2 стимулирует репара
Как отмечалось выше, убиквитинлигаза
цию повреждений, вносящих незначительные
CRL4ADDB2 вовлечена также в УФ индуциро
искажения в двойную спираль ДНК, в частнос
ванное убиквитинирование гистонов H2A [26]
ти CPD, так как способствует привлечению XPC
и/или H3/H4 [27]. Кроме нее, в модификации
к месту повреждения [35].
гистонов в ответ на УФ облучение принимают
Ряд факторов TC NER также подвергается
участие CUL4B содержащие убиквитинлигазы
убиквитинированию. Инициация данного про
[27, 32]. Показано, что моноубиквитинирование
цесса начинается с остановки RNAP II на по
H3 и H4 in vitro приводит к диссоциации гисто
вреждении и последующего привлечения белков
новых октамеров от ДНК [27]. Следует заметить,
CSB и CSA. Последний является субъединицей,
что если повреждение ДНК расположено в коре
выполняющей функцию субстратного рецепто
нуклеосомы, его доступность для XPC и других
ра в комплексе убиквитинлигазы CSA/DDB1/
белков NER сильно затруднена, и в этом кон
CUL4A/RBX1 (CRLCSA), структурно похожей на
тексте роль UV DDB и ассоциированных с ним
CRLDDB2, участвующую в GG NER [25, 30, 33].
убиквитинлигаз может заключаться в пере
CSA, в отличие от DDB2, который служит од
стройке структуры хроматина вокруг повреж
новременно и субстратным рецептором, и бел
денного участка ДНК.
ком мишенью [25], практически не убиквити
В ответ на УФ облучение XPC подвергается
нируется. Автоубиквитинирование CSA показа
также сумоилированию - модификации белком
но только в реконструированной системе, но
SUMO (small ubiquitin like modifier) [33, 34].
роль этой модификации не установлена [25]. В
Также, как и убиквитинирование, эта модифи
неактивном состоянии CRLCSA находится в
кация регулирует процесс NER [22, 34, 35]. Су
комплексе с COP9. При связывании CRLCSA c
ществует несколько изоформ SUMO. Данные,
CSB, основной мишенью этой убиквитинлига
касающиеся модификации XPC, противоречи
зы, происходит диссоциация COP9, CRLCSA ак
вы. Вначале было показано, что в ответ на УФ
тивируется и полиубиквитинирует CSB, что
облучение XPC модифицируется SUMO1, и эта
приводит к протеолитической деградации CSB
модификация приводит к защите XPC от протео
на поздних стадиях репарации с участием сегре
литической деградации [33]. Однако в более
газы p97 [31]. Ингибирование протеосомной
поздних исследованиях показано полисумоили
деградации в целом препятствует высвобожде
рование XPC с помощью SUMO2 [36]. В резуль
нию CSB из репарационного комплекса, а также
тате такой модификации белок становился
негативно сказывается на восстановлении уров
субстратом для убиквитинлигазы RNF111 (дру
ня синтеза РНК после удаления повреждения. В
гое название Arkadia), синтезирующей поли
ответ на УФ облучение убиквитинируется и
убиквитиновые цепи K63 типа, что способство
RNAP II. Основные литературные данные си
вало удалению XPC с поврежденной ДНК и
стематизированы в обзоре Wilson et al. [39]. Из
привлечению к месту повреждения последую
начально предполагалось, что убиквитинирова
щих факторов NER - эндонуклеаз XPG и
ние с последующей деградацией RNAP II, оста
ERCC1 XPF. Полиубиквитинирование по K63
новившейся на повреждении, необходимы для
типу не служит сигналом к протеасомной дегра
того, чтобы освободить место для сборки репа
дации, а выполняет регуляторную функцию в
рационного комплекса. Однако позже выясни
ряде процессов, в том числе в репарации ДНК
лось, что это альтернативный репарации путь,
[36]. В работе Akita et al. [37] обнаружено, что
запускающийся в случае невозможности удале
XPC модифицируется SUMO даже в отсутствии
ния повреждения и восстановления транскрип
повреждения, причем полисумоилирование ма
ционного синтеза РНК [39].
ловероятно, и эта модификация не меняет
Убиквитинирование остальных факторов
ДНК связывающих свойств XPC, зато влияет на
NER изучено значительно хуже. Так, имеются
связывание XPC с UV DDB, что имеет значение
данные о том, что XPA модифицируется убикви
для вытеснения последнего с поврежденной
тинлигазой HERC2 с последующей протеолити
ДНК. Известно, что DDB2 и центрин 2 также
ческой деградацией, что наряду с ацетилирова
БИОХИМИЯ том 84 вып. 9 2019
1248
РЕЧКУНОВА и др.
нием XPA (см. ниже) играет роль в суточных ко
что каким то образом USP45 влияет на привле
лебаниях эффективности NER [40]. Известно,
чение ERCC1 XPF к месту повреждения, при
что при репликативном стрессе RPA убиквити
чем для этого важна каталитическая активность
нируется убиквитинлигазами PRP19 и RFWD3
USP45.
[41], а также подвергается сумоилированию
[42], однако данных о том, что эти модифика
ции играют роль в NER, в настоящий момент
ПОЛИ(ADP6РИБОЗИЛ)ИРОВАНИЕ
нет. Имеются также косвенные данные о том,
что убиквитинирование ERCC1 может влиять
В ответ на повреждение ДНК в клетках выс
на эффективность NER [43]. Так, было показа
ших эукариот происходит модификация ядер
но, что деубиквитиназа USP45 влияет на репа
ных белков с помощью полимера ADP рибозы
рацию УФ индуцированных повреждений. Од
(PAR). Поли(ADP рибозил)ирование (PAR
ним из наиболее вероятных субстратов USP45
илирование) белков катализируется нескольки
является ERCC1. Подавление деубиквитинирую
ми поли(ADP рибоза)полимеразами (PARP),
щей активности USP45 увеличивает количест
которые постоянно и в значительном количест
во полиубиквитинированной (по K48 и K63
ве присутствуют в клетке. Белки PARP образу
типу) формы ERCC1 в экстрактах клеток чело
ют суперсемейство, включающее 17 представи
века, но не влияет на устойчивость ERCC1 к
телей, объединенных по характерному призна
протеолитической деградации. Предполагается,
ку - наличию консервативного домена, содер
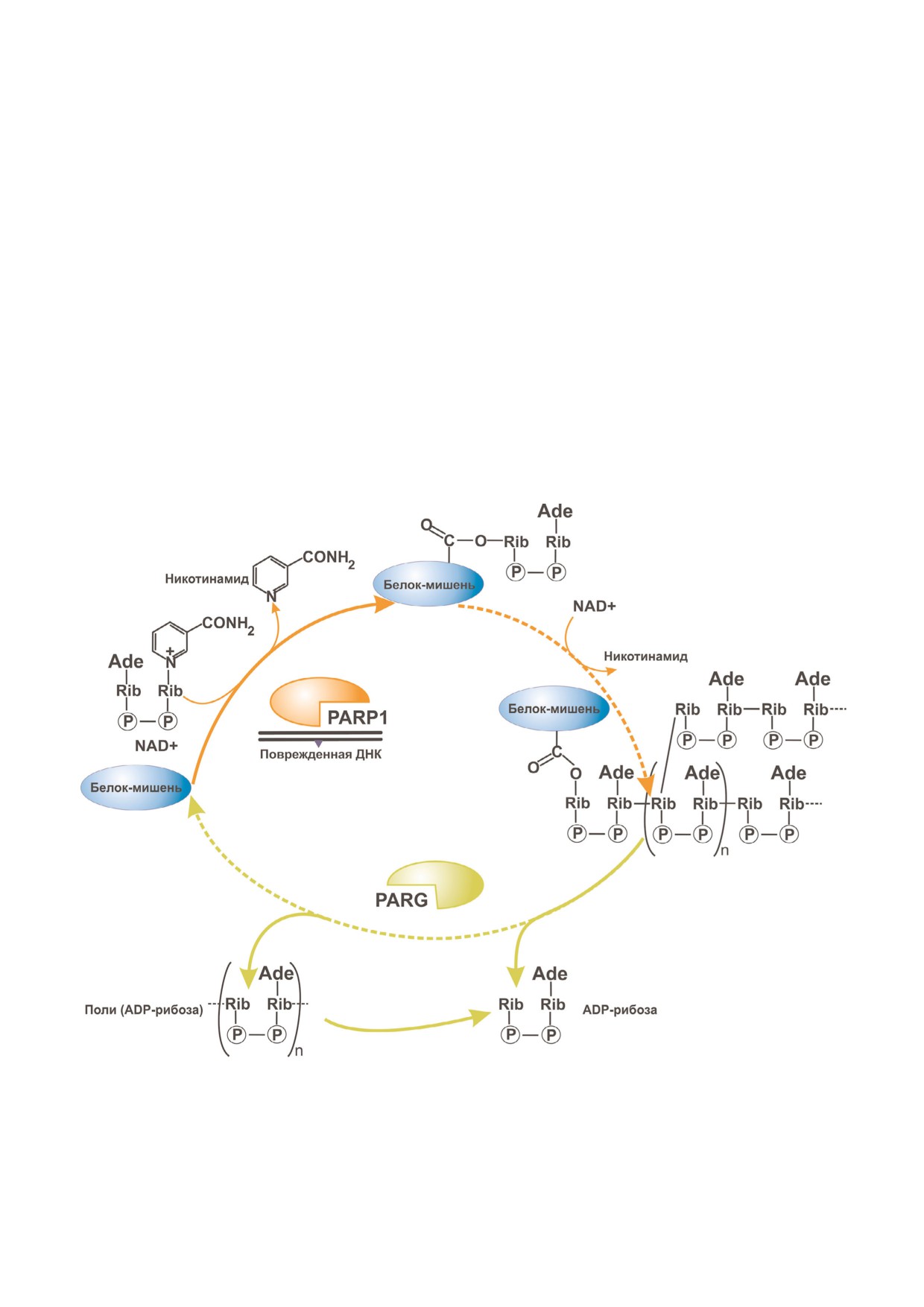
Рис. 2. Синтез и деградация поли(ADP рибозы). В ответ на повреждение ДНК PARP1 катализирует синтез поли(ADP ри
бозы), используя в качестве субстрата NAD+. Поли(ADP рибоза)гликогидролаза (PARG) расщепляет PAR, регулируя уро
вень PARилирования белков и синтеза PAR в клетке.
biokhsm/
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1249
жащего мотив «PARP signature» - высококон
массива белков, подвергающихся PAR илирова
сервативную последовательность, которая
нию в ответ на генотоксический стресс, в числе
участвует в формировании активного центра
которых также оказался ХРС [63].
[44]. В качестве источника ADP рибозы PARP
Предположение о связи синтеза PAR в ответ
используют NAD+ и переносят эту группу на бе
на УФ облучение с процессом NER было вы
лок акцептор.
сказано на основании результатов, демонстри
Из 17 белков семейства PARP в клеточном
рующих активацию PARP на повреждениях,
ответе на повреждение ДНК участвуют три -
возникающих в ДНК при УФ облучении, таких
PARP1, PARP2 и PARP3 [45, 46]. Их мишенями
как тиминовые димеры [64, 65]. Дальнейшие ис
преимущественно служат белки, участвующие в
следования показали прямое взаимодействие
укладке хроматина и метаболизме ДНК, вклю
PAPR1 с поврежденной ДНК в местах локально
чая гистоны, белки репарации ДНК и тран
го УФ облучения [66]. Активация PAPR1 и ис
скрипционные факторы, а также сами PARP. Из
тощение клеточного пула NAD+ наблюдались
трех ферментов PARP, активируемых повреж
также при воздействии бенз[a]пирена (B[a]P)
денной ДНК, наиболее хорошо изучен PARP1,
[67], метаболиты которого образуют аддукты с
на долю которого приходится до 90% синтезиру
основаниями ДНК, удаляемые преимуществен
емой в клетке поли(ADP рибозы) [46]. Реакция
но системой NER. В клетках, дефицитных по
PAR илирования обратима: PAR подвергается
PAPR1, снижался уровень репарации поврежде
расщеплению с помощью фермента поли(ADP
ний ДНК, возникающих под действием B[a]P в
рибоза)гликогидролазы (PARG) (рис. 2), что
первые часы после снятия повреждающего воз
обеспечивает дополнительную регуляцию уров
действия [68].
ня PAR илирования белков и синтеза PAR в
В опубликованной недавно работе Fischer et
клетке.
al. [69] попытались выявить механизм влияния
PARP1 считается одним из ключевых регуля
PAR илирования на генотоксический стресс,
торов процессов репарации ДНК и других кле
индуцируемый одним из наиболее активных ме
точных процессов [46-51]. Участие PARP1 в
таболитов B[a]P - диолэпоксидом (BPDE). С
процессе эксцизионной репарации оснований
использованием клеточных моделей с нокаутом
(base excision repair, BER) исследовано достаточ
гена parp1 или с добавлением фармакологичес
но подробно [52]. Показано, что PARP1 взаимо
кого ингибитора этого фермента на основе ола
действует с белками BER и модулирует их актив
париба исследованы краткосрочные (до 24 ч) и
ность [52-54]. Некоторые белки репарации спо
пролонгированные (7 дней после обработки) ге
собны связывать PAR, как свободный, так и
нотоксические эффекты B[a]P. Обнаружено, что
присоединенный к PARP1, что рассматривается
уровень синтеза PAR в клетках HeLa, обрабо
как один из механизмов вовлечения этих белков
танных BPDE, зависел от времени воздействия
в соответствующие процессы и их регуляцию
и дозы реагента, но динамика накопления PAR
[45]. Взаимодействие осуществляется через
при воздействии BPDE существенно отличалась
PAR связывающие домены, либо в нем участву
от динамики, наблюдаемой при обработке кле
ют РНК или ДНК связывающие домены бел
ток трет бутилгидроперитом, что может свиде
ков, в этом случае может наблюдаться конку
тельствовать о различии механизмов ответа на
ренция между нуклеиновыми кислотами (PAR -
повреждения ДНК, вызываемые этими агента
полимер нуклеотидной природы, «третья нуклеи
ми. Полученные данные демонстрировали раз
новая кислота») за связывание с белком.
носторонний вклад активности PARP1 в клеточ
Роль PARP1 и синтезируемого этим фермен
ный ответ на генотоксический стресс, вызван
том PAR в процессе NER долгое время остава
ный BPDE. С одной стороны, абляция PARP1
лась практически неисследованной, по крайней
предотвращала BPDE индуцированное истоще
мере систематически, хотя активация PARP1 в
ние клеточного NAD+ и обеспечивала кратко
ответ на УФ облучение и вызываемые им по
срочную защиту клетки от токсического воздей
вреждения ДНК была показана еще в 80 е годы
ствия BPDE. С другой стороны, наблюдались
прошлого столетия [55-57] и периодически
сильные долгосрочные эффекты сенсибилиза
подтверждалась в дальнейших работах [58, 59]. В
ции клеток к BPDE - через 7 дней после обра
числе фактов, указывающих на возможную связь
ботки BPDE число выживших колоний было
процесса NER и синтеза PAR, было обнаруже
значительно ниже в случае ингибирования
ние в ХРА PAR связывающего домена [60] и взаи
PARP или абляции PARP1, по сравнению с
модействие этого белка с PAR [60, 61]. Позднее
клетками с нормальным уровнем активности
PAR связывающий домен был идентифициро
PARP. Авторы также исследовали влияние ак
ван в структуре ХРС [62]. Развитие протеомных
тивности PARP на репарацию ДНК, содержа
исследований привело к обнаружению большого
щих аддукты B[a]P, однако полученные резуль
БИОХИМИЯ том 84 вып. 9 2019
1250
РЕЧКУНОВА и др.
таты не выявили связи между активностью
висимую от DDB2, роль в рекрутировании и
PARP и эффективностью процесса NER в отно
стабилизации XPC при УФ индуцированных
шении таких повреждений [69]. Наблюдаемые
повреждениях ДНК. PARP1 формирует стабиль
эффекты сенсибилизации клеток к генотокси
ный комплекс с ХРС в нуклеоплазме в стацио
ческому воздействию BPDE при снижении
нарных условиях до облучения и быстро сопро
уровня синтеза PAR связаны, скорее всего, с по
вождает его к поврежденной ДНК после УФ об
явлением двуцепочечных разрывов и, как след
лучения DDB2 независимым образом. Катали
ствие, развитием репликативного стресса.
тическая активность PARP1 не требуется для
PARP1 была идентифицирована в составе
первоначального комплексообразования с XPC
комплекса белков, ассоциированного с DDB2
в нуклеоплазме, но она усиливает рекрутирова
[70]. PARP1 участвует в индуцированной УФ
ние XPC к месту повреждения ДНК после облу
облучением деконденсации хроматина: в местах
чения. С использованием очищенных белков
повреждений ДНК было обнаружено снижение
показано, что комплекс PARP1/XPC облегчает
плотности коровых гистонов, строго зависящее
посадку XPC на поврежденный участок в присут
от АТР, активности DDB2 и PARP1 [71]. Кроме
ствии UV DDB. Таким образом, поиск повреж
того, PARP1 непосредственно взаимодействует
дений в геномном контексте комплексом XPC
с обеими субъединицами гетеродимера UV
контролируется самим XPC, DDB2 и PARP1.
DDB (DDB1 и DDB2) и модифицирует их в от
Прямое взаимодействие с PARP1 и PAR, а
вет на УФ облучение как in vivo, так и in vitro [70,
также PAR илирование обеих субъединиц гете
72]. В свою очередь, DDB2 стимулирует актив
родимера XPC/RAD23B, катализируемое
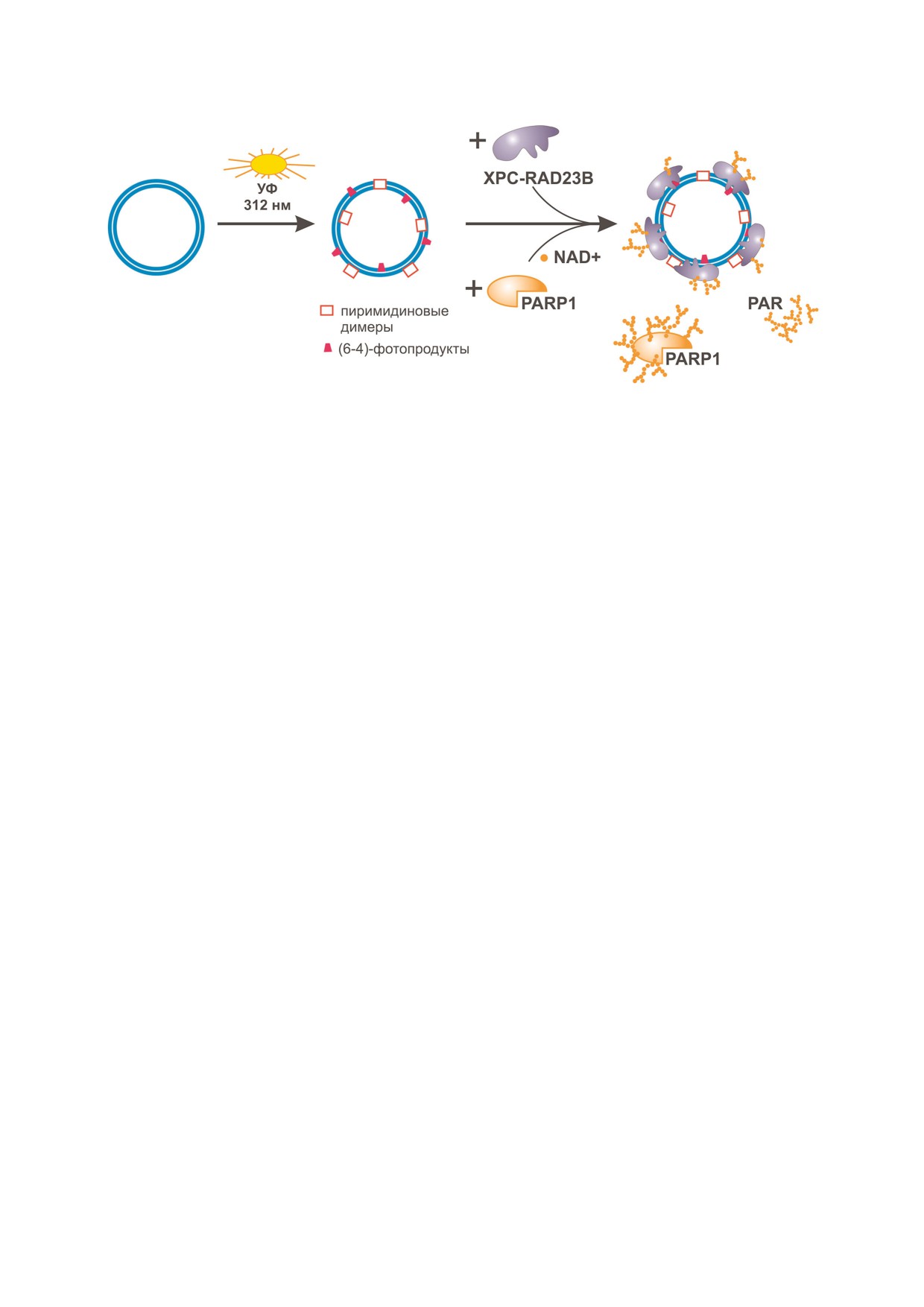
ность PARP1. Снижение уровня PAR под
PARP1, показаны с использованием биохими
действием ингибитора PARP приводило к сни
ческих методов [74]. Эффективность модифика
жению эффективности репарации УФ индуци
ции зависела от структуры ДНК, используемой
рованных повреждений. В работе Pines et al. [70]
для активации PARP1, и значительно возрастала
впервые установлена взаимосвязь двух типов
после облучения ДНК УФ светом (рис. 3).
посттрансляционной модификации белков
Сродство XPC RAD23B к PAR возрастало при
NER - убиквитинирования и PAR илирования.
увеличении длины полимера; взаимодействие с
Как PARP1, так и CUL4A/RBX1 взаимодейству
PAR ингибировало связывание белка с ДНК.
ют с DDB2, регулируя время и уровень связыва
Как упоминалось выше, ХРА был первым
ния его с поврежденной ДНК и конкурируя за
фактором NER, для которого показано взаимо
возможность модификации N концевого участ
действие с PAR. С помощью биохимических ис
ка: PAR илирование DDB2 подавляет его убикви
следований установлено, что связывание PAR
тинирование. Как отмечалось выше, в случае
происходит с С концевой частью XPA, в кото
повреждений ДНК, вызываемых производным
рой расположен специфический PAR связыва
B[a]P, не удалось установить влияние активнос
ющий мотив [60, 61]. Дальнейшие исследования
ти PARP на эффективность процесса NER [69].
были направлены на определение функцио
Авторы предположили, что различие влияния
нального значения этого взаимодействия. В ра
активности PARP на репарацию УФ и BPDE
боте King et al. [75] показана связь активации
индуцированных повреждений ДНК может
PARP в ответ на УФ облучение с ассоциацией
быть связано с различиями в узнавании этих
ХРА с поврежденным участком хроматина. УФ
повреждений: узнавание УФ индуцированных
облучение стимулировало активность PARP и
повреждений осуществляет DDB2 [11, 12], тогда
способствовало ассоциации XPA с PAR и
как в узнавании повреждений, вызываемых хи
PARP1. Подавление активности PARP с по
мическими канцерогенами, этот белок не участ
мощью ингибитора или нокдаун гена parp1 при
вует.
водили к накоплению УФ индуцированных
В ряде работ показано влияние активности
повреждений в ДНК кератиноцитов человека.
PARP1 на узнавание УФ индуцированных по
Ингибирование активности PARP уменьшало
вреждений фактором XPC [71-73]. Это влияние
взаимодействие XPA и PARP1 in vitro, в том чис
может быть опосредовано DDB2, взаимодей
ле в цельноклеточных экстрактах, затрудняло
ствие с которым необходимо для узнавания XPC
связывание XPA с PARP1 на хроматине и блоки
пиримидиновых димеров. Действительно, мето
ровало индуцированную УФ облучением ассо
дом коиммунопреципитации было показано,
циацию XPA с хроматином, что указывает на за
что обработка клеток ингибитором PARP при
висимость связывания XPA с хроматином от
водила к снижению взаимодействия DDB2 с
PAR.
XPC в ответ на УФ облучение [72]. В дальней
Аналогичные данные получены другой груп
шей работе этих исследователей [73] показано,
пой исследователей [76], которые подтвердили,
что PARP1 может играть дополнительную, неза
что фармакологическое ингибирование процес
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1251
Рис. 3. PAR илирование XPC RAD23B в присутствии УФ облученной ДНК. Уровень PAR илирования зависел от дозы
облучения [74].
biokhsm/
са PAR илирования в клетке снижает эффек
единиц гетеротримера [79], а также влияние
тивность NER. Авторы показали, что взаимо
RPA на активность PARP1 [80]. Однако на дан
действие с PAR опосредуется специфическими
ный момент нет никаких данных о связи этой
основными аминокислотами в высококонсерва
модификации с процессом NER. Следует отме
тивном PAR связывающем мотиве XPA, кото
тить, что PAR илированию в ответ на поврежде
рый перекрывается с участками связывания
ние ДНК также подвергается белок CSB, участ
DDB2 и TFIIH. Биохимические исследования
вующий в процессе TC NER [81], хотя влияние
выявили взаимную регуляцию функций PARP1
этой модификации на эффективность данного
и XPA - с одной стороны, взаимодействие XPA
процесса достоверно не установлено.
с PAR снижает сродство белка к ДНК, с другой
В пользу возможного участия PARP1 в про
стороны, XPA значительно стимулирует фер
цессе NER также свидетельствует идентифика
ментативную активность PARP1. Методом ла
ция этого белка в числе основных мишеней се
зерной сканирующей микроскопии обнаруже
лективного мечения фотореакционноспособ
но, что в клетках остеосаркомы человека,
ными ДНК аналогами субстратов NER в
экспрессирующих GFP XPA, ингибирование
экстракте клеток HeLa [82].
активности PARP приводило к снижению ско
Таким образом, накоплено достаточное ко
рости привлечения флуоресцентного белка на
личество данных, которые демонстрируют связь
поврежденные лазерным излучением участки
активности PARP с репарацией УФ индуциро
ДНК, что напрямую демонстрирует активную
ванных повреждений и подтверждают роль
роль PAR илирования в пространственно вре
PARP1 в NER. В целом, все рассмотренные дан
менном контроле клеточной локализации XPA в
ные подтверждают известную парадигму, что
ответ на повреждение ДНК в клетке. Таким об
взаимодействие различных белков с PARP1 и
разом, в данной работе показана взаимная регу
PAR может быть важным для функционирова
ляция XPA и PARP1, зависящая от PAR, кото
ния этих белков, а сам белок PARP1 и катализи
рую потенциально можно рассматривать как
руемый им процесс синтеза PAR можно рас
механизм тонкой пространственно временной
сматривать в качестве универсального механиз
регуляции функционирования этих белков в
ма регуляции множества клеточных процессов,
процессе NER.
в том числе репарации ДНК.
Один из ключевых партнеров ХРА в процес
се NER - RPA, гетеротримерный белок, состоя
щий из субъединиц с молекулярной массой 70,
АЦЕТИЛИРОВАНИЕ
32 и 14 кДа (р70, р32 и р14 соответственно) -
И ФОСФОРИЛИРОВАНИЕ
также взаимодействует с PARP1 и PAR и подвер
гается PAR илированию. Вначале разными ме
К числу распространенных модификаций
тодами было показано PAR илирование субъ
белков, выполняющих сигнальную и регулятор
единиц р70 и р32 [63, 77, 78], а затем и всех субъ
ную функции во многих клеточных процессах,
БИОХИМИЯ том 84 вып. 9 2019
1252
РЕЧКУНОВА и др.
включая репарацию ДНК, относятся также аце
ление SIRT1 значительно повышало чувстви
тилирование и фосфорилирование.
тельность клеток к УФ облучению. Установле
Роль ацетилирования в процессе NER опре
но, что SIRT1 взаимодействует с ХРА, и это взаи
деляется главным образом влиянием ацетили
модействие усиливается после УФ облучения.
рования гистонов на структуру хроматина и, как
SIRT1 деацетилирует XPA как in vitro, так и в
следствие, на доступность поврежденных участ
клетках. Важно отметить, что опосредованное
ков ДНК для факторов NER [83-85]. Достовер
SIRT1 деацетилирование XPA необходимо для
но показано взаимодействие гетеродимера UV
оптимизации процесса NER, поскольку ком
DDB с различными ацетилтрансферазами гис
плементация дефицитных по XPA клеток мутан
тонов: CBP/p300
[86,
87], GCN5
[88] и
том XPA K6367Q, имитирующим гиперацети
KAT7/HBO1/MYST2 [89], а также с гистон де
лированный XPA, приводила к значительно
ацетилазами HDAC1/2 [90, 91], что позволяет
большей УФ чувствительности этих клеток по
предположить участие этого белка в координа
сравнению с клетками, несущими XPA дикого
ции посттрансляционных модификаций гисто
типа. В частности, уменьшалась выживаемость
нов (и/или других белков) и создании локально
клеток и резко снижался уровень репарации
го окружения в хроматине для эффективного уз
CPD. Кроме того, SIRT1 опосредованное де
навания повреждения и протекания процесса
ацетилирование XPA усиливает его взаимодей
репарации.
ствие с субъединицей р32 RPA [94]. Таким обра
В недавней работе группы японских иссле
зом, SIRT1 может регулировать процесс NER
дователей, возглавляемой проф. K. Sugasawa,
посредством модуляции статуса ацетилирова
обнаружена зависимость связывания ХРС с
ния XPA.
ДНК в составе хроматина от уровня ацетилиро
Недавно обнаружено ацетилирование субъ
вания определенных гистонов [92]. Обработка
единицы р70 RPA по остатку К163, опосредован
клеток ингибиторами гистон деацетилазы за
ное ацетилтрансферазами PCAF и GCN5, и ис
медляла рекрутирование ХРС на участки УФ
следована роль этой модификации в процессе
индуцированного повреждения ДНК и последую
NER [95, 96]. Показано, что ацетилирование
щий процесс репарации. Биохимические иссле
RPA имеет решающее значение для устойчивого
дования показали взаимодействие XPC с гисто
накопления XPA на поврежденных участках
ном H3, которое значительно ослаблялось после
ДНК и активации NER и регулируется деацети
делеции N концевого «хвоста» гистона H3. Кро
лазами HDAC6 и SIRT1 [95]. УФ облучение кле
ме того, XPC также взаимодействовал с гисто
ток способствовало транслокации HDAC6 в ци
ном H1. Важно отметить, что ацетилирование
топлазму, тем самым нарушая взаимодействие
гистона H3 заметно ослабляло его взаимодей
HDAC6 с RPA, что приводило к увеличению
ствие с XPC in vitro, а местное УФ облучение
уровня ацетилирования RPA. Показано также,
клеток приводило к снижению уровня ацетили
что мутация в участке ацетилирования р70 при
рованного по остатку К27 гистона H3 в повреж
водит к специфическому ослаблению взаимо
денных областях хроматина. Таким образом, де
действия RPA с XPA, снижению уровня XPA в
ацетилирование гистонов может играть важную
местах УФ индуцированных повреждений ДНК,
роль в процессе узнавания повреждений ДНК
затруднению процесса NER и снижению выжи
комплексом XPC, поскольку состояние ацети
ваемости клеток в ответ на УФ облучение [96].
лирования гистонов регулирует локализацию и
Таким образом, статус ацетилирования гис
функции этого фактора (рис. 4).
тонов и ключевых факторов NER - ХРА и RPA -
Ацетилированию подвергаются и некоторые
существенным образом влияет на активность
факторы NER. Например, обнаружено, что аце
системы NER в восстановлении УФ индуциро
тилтрансферазы гистонов p300 и CBP ацетили
ванных повреждений ДНК. Следует отметить,
руют XPG - одну из эндонуклеаз NER [93]. На
что влияние этой модификации имеет разно
настоящий момент функциональная значи
направленный характер в случае ХРА и RPA -
мость ацетилирования XPG не изучена. Показа
повышение уровня ацетилирования ХРА сни
но также ацетилирование ХРА ацетилтрансфе
жает его функциональную активность в процес
разами p300 и CBP по остаткам К63 и К67, в ре
се NER, тогда как ацетилирование RPA прямо
гуляции которого участвует NAD+ зависимая
коррелирует с его активностью в NER.
гистон деацетилаза SIRT1 [94]. SIRT1 играет
Фосфорилирование - наиболее распростра
важную роль во многих биологических процес
ненная посттрансляционная модификация бел
сах, включая транскрипцию генов, клеточный
ков, определяющая их функциональную актив
метаболизм, реакцию на стресс и канцерогенез.
ность во всех ключевых клеточных процессах, в
В работе Fan и Luo показано, что SIRT1 вовле
том числе вовлеченных в ответ на повреждение
чена также в регуляцию процесса NER. Подав
ДНК [97, 98]. Фосфорилированию, катализируе
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1253
мому различными протеинкиназами, в основ
ном подвергаются остатки серина, в значительно
меньшей степени модифицируются треонин, ти
розин, гистидин, аргинин, лизин, а также аспар
тат и глутамат [99]. Использование протеомных
методов в сочетании с масс спектрометричес
ким анализом позволило выявить многочислен
ные сайты фосфорилирования во всех факторах
NER, однако лишь некоторые из них подтверж
дены альтернативными методами исследования.
Ацетилированный
«хвост» гистона H3
«хвост» гистона H3
Функциональная значимость модификации для
большинства сайтов остается неизученной.
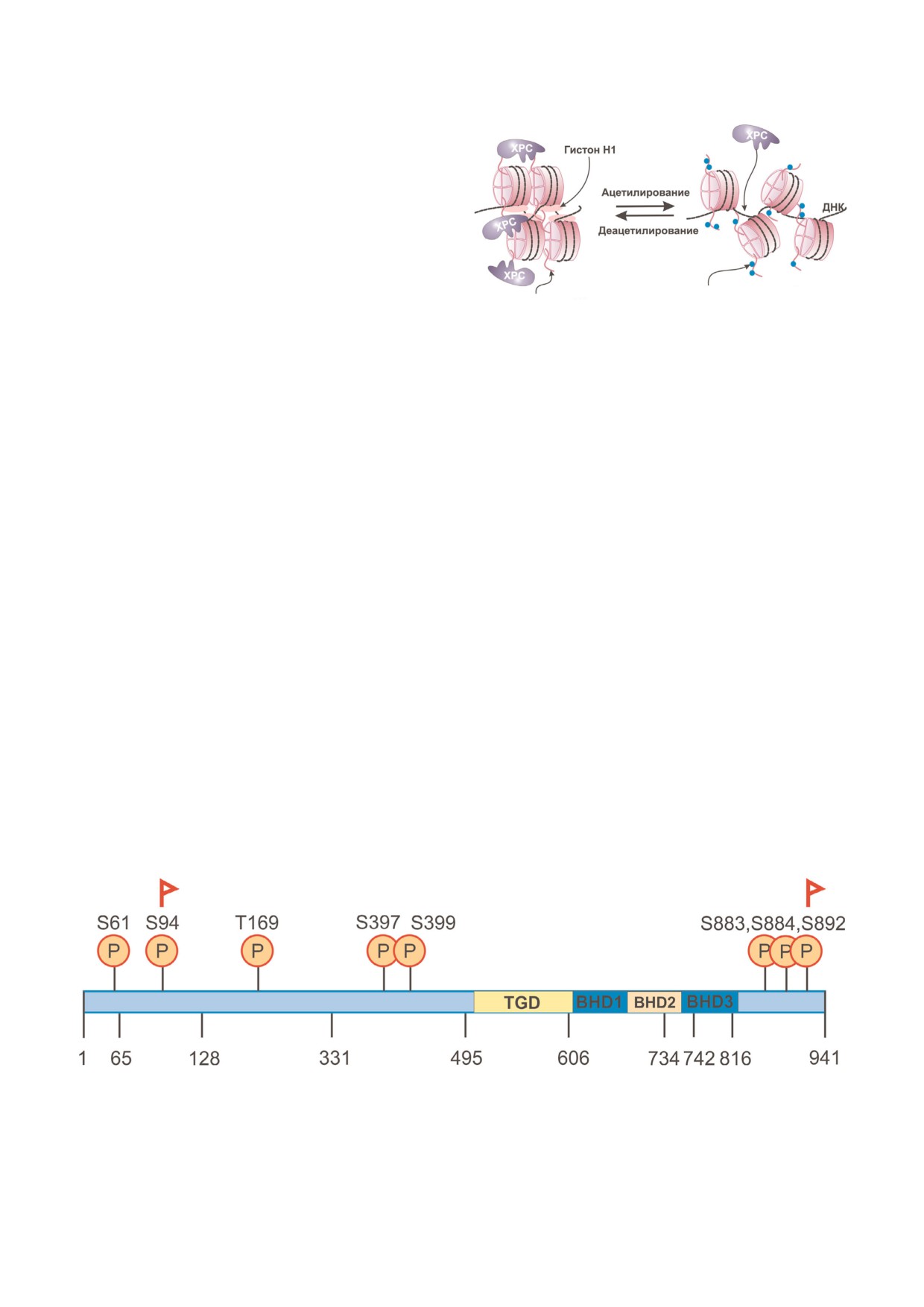
Рис. 4. Связывание ХРС с ДНК в составе хроматина регу
Методом высокопроизводительного скри
лируется уровнем ацетилирования гистона Н3 (адаптиро
нинга были установлены несколько мишеней
вано из [92] с модификациями). Ацетилирование гистона
H3 приводит к ослаблению взаимодействия ХРС с его
фосфорилирования в ХРС, а именно S61, S94,
N концевым участком и относительной деконденсации
S397, S399, S883, S884, S892 и T169 [97, 100]
хроматина, что облегчает связывание ХРС с ДНК вне нук
(рис. 5). Но лишь недавно появились данные,
леосомы. Таким образом, состояние ацетилирования гис
демонстрирующие связь некоторых из этих мо
тонов может играть важную роль в процессе узнавания
дификаций, а именно фосфорилирования S94 и
повреждений ДНК комплексом XPC.
С цветным вариантом рис. 4 можно ознакомиться в элек
S892, с активностью белка в NER [101]. Авторы
исследования показали, что фосфорилирование
journal/biokhsm/
S94 увеличивает активность ХРС в ответ на УФ
облучение, тогда как модификация S892 приво
дит к обратному эффекту. Кроме того, фосфори
его убиквитинирование и последующую дегра
лирование ХРС по S94 способствует убиквити
дацию, и тем самым активирует процесс NER
нированию XPC и рекрутированию этого и по
[102, 103]. Фосфорилирование геликазы XPB,
следующих факторов NER к поврежденным
входящей в комплекс TFIIH, по остатку S751,
участкам хроматина после УФ облучения. С ис
наоборот, ингибирует процесс репарации [104].
пользованием ингибиторов различных протеин
Выявлено и детально изучено фосфорилиро
киназ, потенциально способных фосфорилиро
вание средней субъединицы RPA, р32. Показа
вать XPC, установлено, что фосфорилирование
но, что в ответ на УФ облучение этот полипеп
XPC по S94 осуществляет казеинкиназа II
тид подвергается гиперфосфорилированию с
(CK2), ингибирование или нокдаун которой
участием ATM [105, 106]. Хотя эта модификация
приводили к снижению активности процесса
известна давно и ее роль в функционировании
NER. Таким образом, фосфорилирование XPC
RPA в различных клеточных процессах исследо
можно рассматривать как новый посттрансля
валась во многих работах, связь с процессом
ционный механизм регуляции NER.
NER не установлена [107].
Показано, что фосфорилирование ХРА по
Как и в случае других модификаций, влия
S196, осуществляемое протеинкиназой ATR [97,
ние на активность NER оказывает не только
102], повышает стабильность белка, ингибируя
фосфорилирование белков, непосредственно
Рис. 5. Сайты фосфорилирования в структуре ХРС. Флажками обозначены остатки S94 и S892, модификация которых
связана с активностью процесса NER [101].
biokhsm/
БИОХИМИЯ том 84 вып. 9 2019
1254
РЕЧКУНОВА и др.
участвующих в процессе, но и факторов, моду
обеспечивает дополнительную регуляцию уров
лирующих структуру хроматина. Так, фосфори
ня модификации и активности процесса в це
лирование гистонацетилтрансферазы HBO1 с
лом. Значительный вклад в регуляцию процесса
помощью ATM и/или ATR в ответ на УФ облу
NER вносят модификации гистонов, а также
чение облегчает ее связывание с DDB2 [89].
факторов модуляции хроматина, к числу кото
Кроме того, фосфорилированная HBO1 опосре
рых относится и PARP1, который служит ми
дует ацетилирование гистонов в сайтах CPD,
шенью как для авто PAR илирования, так и для
что облегчает рекрутирование XPC к повреж
остальных упомянутых модификаций [109]. Та
денным участкам ДНК [108]. Показано также,
ким образом, регуляция ответа на повреждения
что HBO1 способствует накоплению SNF2H
ДНК, исправляемые системой NER, с помощью
ACF1, АТФ зависимого комплекса ремоделиро
посттрансляционных модификаций - сложная
вания хроматина, в сайтах CPD. Истощение
цепь взаимозависимых событий, совокупность
HBO1 ингибировало репарацию CPD и усили
которых обеспечивает эффективное удаление
вало чувствительность клеток к УФ облучению.
повреждений.
Однако истощение HBO1 в клетках, получен
ных от пациентов с пигментной ксеродермой
комплементационных групп XP E, XP C и XP A,
Финансирование. Работа выполнена при под
не приводило к дополнительной УФ чувстви
держке Российского фонда фундаментальных
тельности. Авторы исследования предполагают,
исследований (гранты № 18 04 00596 и 19 04
что HBO1 совместно с SNF2H ACF1 изменяет
00481) и Программы фундаментальных научных
структуру хроматина, делая ее более доступной
исследований государственных академий наук
для факторов NER [108].
на 2013-2020 гг. (№ АААА А17 117020210022 4,
Данные о посттрансляционных модифика
для ОИЛ).
циях белков NER постоянно расширяются и до
Благодарности. Авторы благодарны Краси
полняются. Большинство рассмотренных моди
ковой Ю.С. за помощь в оформлении рукописи.
фикаций существенно влияют на активность
Конфликт интересов. Авторы заявляют, что у
белка мишени и всего процесса репарации.
них нет конфликта интересов.
Кроме факторов, непосредственно участвую
Соблюдение этических норм. Настоящая
щих в процессе репарации, модификации под
статья не содержит каких либо исследований с
вергаются также некоторые белки, осуществляю
участием людей или с использованием живот
щие эту или альтернативную модификацию, что ных в качестве объектов исследований.
СПИСОК ЛИТЕРАТУРЫ
1.
Friedberg, E.C. (2003) DNA damage and repair, Nature,
DNA containing photoreactive damages, Bioorg. Chem.,
421, 436-440, doi: 10.1038/nature01408.
36, 77-84, doi: 10.1016/j.bioorg.2007.11.004.
2.
Gillet, L.C., and Scharer, O.D. (2006) Molecular mecha
9. Rechkunova, N.I., and Lavrik, O.I. (2010) Nucleotide
nisms of mammalian global genome nucleotide excision
excision repair in higher eukaryotes: mechanism of prima
repair, Chem. Rev., 106, 253-276, doi: 10.1021/cr040483f.
ry damage recognition in global genome repair, Subcell.
3.
Tornaletti, S., and Hanawalt, P.C. (1999) Effect of DNA
Biochem., 50, 251-277, doi: 10.1007/978 90 481 3471
lesions on transcription elongation, Biochimie,
81,
7_13.
139-146.
10. Krasikova, Y.S., Rechkunova, N.I., Maltseva, E.A.,
4.
Fousteri, M., and Mullenders, L.H. (2008) Transcription
Pestryakov, P.E., Petruseva, I.O., Sugasawa, K., Chen, X.,
coupled nucleotide excision repair in mammalian cells:
Min, J.H., and Lavrik, O.I. (2013) Comparative analysis of
molecular mechanisms and biological effects, Cell Res., 18,
interaction of human and yeast DNA damage recognition
73-84, doi: 10.1038/cr.2008.6.
complexes with damaged DNA in nucleotide excision
5.
Sugasawa, K., Shimizu, Y., Iwai, S., and Hanaoka, F.
repair, J. Biol. Chem., 288, 10936-10947, doi: 10.1074/
(2002) A molecular mechanism for DNA damage recogni
jbc.M112.444026.
tion by the xeroderma pigmentosum group C protein com
11. Fitch, M.E., Nakajima, S., Yasui, A., and Ford, J.M.
plex, DNA Repair (Amst), 1, 95-107.
(2003) In vivo recruitment of XPC to UV induced cyclobu
6.
Maillard, O., Camenisch, U., Clement, F.C., Blagoev, K.B.,
tane pyrimidine dimers by the DDB2 gene product, J. Biol.
and Naegeli, H. (2007) DNA repair triggered by sensors of
Chem., 278, 46906-46910, doi: 10.1074/jbc.M307254200.
helical dynamics, Trends Biochem. Sci., 32, 494-499,
12. Moser, J., Volker, M., Kool, H., Alekseev, S., Vrieling, H.,
doi: 10.1016/j.tibs.2007.08.008.
Yasui, A., van Zeeland, A.A., and Mullenders, L.H. (2005)
7.
Min, J.H., and Pavletich, N.P. (2007) Recognition of DNA
The UV damaged DNA binding protein mediates efficient
damage by the Rad4 nucleotide excision repair protein,
targeting of the nucleotide excision repair complex to UV
Nature, 449, 570-575, doi: 10.1038/nature06155.
induced photo lesions, DNA Repair (Amst.), 4, 571-582,
8.
Maltseva, E.A., Rechkunova, N.I., Petruseva, I.O.,
doi: 10.1016/j.dnarep.2005.01.001.
Vermeulen, W., Scharer, O.D., and Lavrik, O.I. (2008)
13. Houten, B.V., Kuper, J., and Kisker, C. (2016) Role of
Crosslinking of nucleotide excision repair proteins with
XPD in cellular functions: to TFIIH and beyond, DNA
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1255
Repair (Amst.), 44, 136-142, doi: 10.1016/j.dnarep.
28.
Groisman, R., Polanowska, J., Kuraoka, I., Sawada, J.,
2016.05.019.
Saijo, M., Drapkin, R., Kisselev, A.F., Tanaka, K., and
14.
Evans, E., Moggs, J.G., Hwang, J.R., Egly, J.M., and
Nakatani, Y. (2003) The ubiquitin ligase activity in the
Wood, R.D. (1997) Mechanism of open complex and dual
DDB2 and CSA complexes is differentially regulated by
incision formation by human nucleotide excision repair
the COP9 signalosome in response to DNA damage, Cell,
factors, EMBO J., 16, 6559-6573, doi: 10.1093/emboj/
113, 357-367.
16.21.6559.
29.
Matsumoto, S., Fischer, E.S., Yasuda, T., Dohmae, N.,
15.
Staresincic, L., Fagbemi, A.F., Enzlin, J.H., Gourdin, A.M.,
Iwai, S., Mori, T., Nishi, R., Yoshino, K., Sakai, W.,
Wijgers, N., Dunand Sauthier, I., Giglia Mari, G.,
Hanaoka, F., Thoma, N.H., and Sugasawa, K. (2015)
Clarkson, S.G., Vermeulen, W., and Scharer, O.D. (2009)
Functional regulation of the DNA damage recognition
Coordination of dual incision and repair synthesis in
factor DDB2 by ubiquitination and interaction with xero
human nucleotide excision repair, EMBO J.,
28,
derma pigmentosum group C protein, Nucleic Acids Res.,
1111-1120, doi: 10.1038/emboj.2009.49.
43, 1700-1713, doi: 10.1093/nar/gkv038.
16.
Krasikova, Y.S., Rechkunova, N.I., Maltseva, E.A.,
30.
Puumalainen, M.R., Lessel, D., Ruthemann, P.,
Petruseva, I.O., and Lavrik, O.I. (2010) Localization of
Kaczmarek, N., Bachmann, K., Ramadan, K., and
xeroderma pigmentosum group A protein and replication
Naegeli, H. (2014) Chromatin retention of DNA damage
protein A on damaged DNA in nucleotide excision repair,
sensors DDB2 and XPC through loss of p97 segregase
Nucleic Acids Res., 38, 8083-8094, doi: 10.1093/nar/
causes genotoxicity, Nat. Commun., 5, 3695, doi: 10.1038/
gkq649.
ncomms4695.
17.
Kemp, M.G., Gaddameedhi, S., Choi, J.H., Hu, J., and
31.
He, J., Zhu, Q., Wani, G., Sharma, N., Han, C., Qian, J.,
Sancar, A. (2014) DNA repair synthesis and ligation affect
Pentz, K., Wang, Q.E., and Wani, A.A. (2014) Ubiquitin
the processing of excised oligonucleotides generated by
specific protease 7 regulates nucleotide excision repair
human nucleotide excision repair, J. Biol. Chem., 289,
through deubiquitinating XPC protein and preventing XPC
26574-26583, doi: 10.1074/jbc.M114.597088.
protein from undergoing ultraviolet light induced and
18.
Grabbe, C., Husnjak, K., and Dikic, I. (2011) The spatial
VCP/p97 protein regulated proteolysis, J. Biol. Chem.,
and temporal organization of ubiquitin networks, Nat. Rev.
289, 27278-27289, doi: 10.1074/jbc.M114.589812.
Mol. Cell Biol., 12, 295-307, doi: 10.1038/nrm3099.
32.
Guerrero Santoro, J., Kapetanaki, M.G., Hsieh, C.L.,
19.
Husnjak, K., and Dikic, I. (2012) Ubiquitin binding pro
Gorbachinsky, I., Levine, A.S., and Rapic Otrin, V. (2008)
teins: decoders of ubiquitin mediated cellular functions,
The cullin 4B based UV damaged DNA binding protein
Annu. Rev. Biochem., 81, 291-322, doi: 10.1146/annurev
ligase binds to UV damaged chromatin and ubiquitinates
biochem 051810 094654.
histone H2A, Cancer Res., 68, 5014-5022, doi: 10.1158/
20.
Komander, D., and Rape, M. (2012) The ubiquitin code,
0008 5472.CAN 07 6162.
Annu. Rev. Biochem., 81, 203-229, doi: 10.1146/annurev
33.
Wang, Q.E., Zhu, Q., Wani, G., El Mahdy, M.A., Li, J.,
biochem 060310 170328.
and Wani, A.A. (2005) DNA repair factor XPC is modified
21.
Van Cuijk, L., Vermeulen, W., and Marteijn, J.A. (2014)
by SUMO 1 and ubiquitin following UV irradiation,
Ubiquitin at work: the ubiquitous regulation of the damage
Nucleic Acids Res., 33, 4023-4034, doi: 10.1093/nar/
recognition step of NER, Exp. Cell Res., 329, 101-109,
gki684.
doi: 10.1016/j.yexcr.2014.07.018.
34.
Van Cuijk, L., van Belle, G.J., Turkyilmaz, Y., Poulsen, S.L.,
22.
Ruthemann, P., Balbo Pogliano, C., and Naegeli, H.
Janssens, R.C., Theil, A.F., Sabatella, M., Lans, H.,
(2016) Global genome nucleotide excision repair con
Mailand, N., Houtsmuller, A.B., Vermeulen, W., and
trolled by ubiquitin/sumo modifiers, Front. Genet., 7, 68,
Marteijn, J.A. (2015) SUMO and ubiquitin dependent
doi: 10.3389/fgene.2016.00068.
XPC exchange drives nucleotide excision repair, Nat.
23.
Chitale, S., and Richly, H. (2017) Timing of DNA lesion
Commun., 6, 7499, doi: 10.1038/ncomms8499.
recognition: ubiquitin signaling in the NER pathway, Cell
35.
Han, C., Zhao, R., Kroger, J., He, J., Wani, G., Wang, Q.E.,
Cycle, 16, 163-171, doi: 10.1080/15384101.2016.1261227.
and Wani, A.A. (2017) UV radiation induced SUMOylation
24.
Sugasawa, K., Okuda, Y., Saijo, M., Nishi, R., Matsuda, N.,
of DDB2 regulates nucleotide excision repair, Carcinogenesis,
Chu, G., Mori, T., Iwai, S., Tanaka, K., Tanaka, K., and
38, 976-985, doi: 10.1093/carcin/bgx076.
Hanaoka, F. (2005) UV Induced ubiquitylation of XPC
36.
Poulsen, S.L., Hansen, R.K., Wagner, S.A., van Cuijk, L.,
protein mediated by UV DDB ubiquitin ligase complex,
van Belle, G.J., Streicher, W., Wikstrom, M., Choudhary, C.,
Cell, 121, 387-400, doi: 10.1016/j.cell.2005.02.035.
Houtsmuller, A.B., Marteijn, J.A., Bekker Jensen, S., and
25.
Fischer, E.S., Scrima, A., Bohm, K., Matsumoto, S.,
Mailand, N. (2013) RNF111/Arkadia is a SUMO targeted
Lingaraju, G.M., Faty, M., Yasuda, T., Cavadini, S.,
ubiquitin ligase that facilitates the DNA damage response,
Wakasugi, M., Hanaoka, F., Iwai, S., Gut, H., Sugasawa, K.,
J. Cell. Biol., 201, 797-807, doi: 10.1083/jcb.201212075.
and Thoma, N.H.
(2011) The molecular basis of
37.
Akita, M., Tak, Y.S., Shimura, T., Matsumoto, S., Okuda
CRL4DDB2/CSA ubiquitin ligase architecture, targeting,
Shimizu, Y., Shimizu, Y., Nishi, R., Saitoh, H., Iwai, S.,
and activation, Cell, 147, 1024-1039, doi: 10.1016/j.cell.
Mori, T., Ikura, T., Sakai, W., Hanaoka, F., and Sugasawa, K.
2011.10.035.
(2015) SUMOylation of xeroderma pigmentosum group C
26.
Kapetanaki, M.G., Guerrero Santoro, J., Bisi, D.C.,
protein regulates DNA damage recognition during
Hsieh, C.L., Rapic Otrin, V., and Levine, A.S. (2006) The
nucleotide excision repair, Sci. Rep.,
5,
10984,
DDB1 CUL4A DDB2 ubiquitin ligase is deficient in
doi: 10.1038/srep10984.
xeroderma pigmentosum group E and targets histone H2A
38.
Klein, U.R., and Nigg, E.A. (2009) SUMO dependent
at UV damaged DNA sites, Proc. Natl. Acad. Sci. USA,
regulation of centrin 2, J. Cell. Sci., 122, 3312-3321,
103, 2588-2593, doi: 10.1073/pnas.0511160103.
doi: 10.1242/jcs.050245.
27.
Wang, H., Zhai, L., Xu, J., Joo, H.Y., Jackson, S.,
39.
Wilson, M.D., Harreman, M., and Svejstrup, J.Q. (2013)
Erdjument Bromage, H., Tempst, P., Xiong, Y., and
Ubiquitylation and degradation of elongating RNA poly
Zhang, Y. (2006) Histone H3 and H4 ubiquitylation by the
merase II: the last resort, Biochim. Biophys. Acta, 1829,
CUL4 DDB ROC1 ubiquitin ligase facilitates cellular
151-157, doi: 10.1016/j.bbagrm.2012.08.002.
response to DNA damage, Mol. Cell, 22, 383-394,
40.
Kang, T.H., Lindsey Boltz, L.A., Reardon, J.T., and
doi: 10.1016/j.molcel.2006.03.035.
Sancar, A. (2010) Circadian control of XPA and excision
БИОХИМИЯ том 84 вып. 9 2019
1256
РЕЧКУНОВА и др.
repair of cisplatin DNA damage by cryptochrome and
ultraviolet irradiated human fibroblasts, J. Biol. Chem.,
HERC2 ubiquitin ligase, Proc. Natl. Acad. Sci. USA, 107,
258, 103-107.
4890-4895, doi: 10.1073/pnas.0915085107.
58.
Yoon, Y.S., Kim, J.W., Kang, K.W., Kim, Y.S., Choi, K.H.,
41.
Yates, M., and Marechal, A. (2018) Ubiquitylation at the
and Joe, C.O. (1996) Poly(ADP ribosyl)ation of histone
fork: making and breaking chains to complete DNA repli
H1 correlates with internucleosomal DNA fragmentation
cation, Int. J. Mol. Sci., 19, E2909, doi: 10.3390/
during apoptosis, J. Biol. Chem., 271, 9129-9134.
ijms19102909.
59.
Chang, H., Sander, C.S., Muller, C.S., Elsner, P., and
42.
Dou, H., Huang, C., Singh, M., Carpenter, P.B., and
Thiele, J.J. (2002) Detection of poly(ADP ribose) by immuno
Yeh, E.T. (2010) Regulation of DNA repair through
cytochemistry: a sensitive new method for the early identifica
deSUMOylation and SUMOylation of replication protein
tion of UVB and H2O2 induced apoptosis in keratinocytes,
A complex, Mol. Cell, 39, 333-345, doi: 10.1016/j.molcel.
Biol. Chem., 383, 703-708, doi: 10.1515/BC.2002.072.
2010.07.021.
60.
Pleschke, J.M., Kleczkowska, H.E., Strohm, M., and
43.
Perez Oliva, A.B., Lachaud, C., Szyniarowski, P., Munoz, I.,
Althaus, F.R. (2000) Poly(ADP ribose) binds to specific
Macartney, T., Hickson, I., Rouse, J., and Alessi, D.R.
domains in DNA damage checkpoint proteins, J. Biol.
(2015) USP45 deubiquitylase controls ERCC1 XPF
Chem., 275, 40974-40980, doi: 10.1074/jbc.M006520200.
endonuclease mediated DNA damage responses, EMBO J.,
61.
Fahrer, J., Kranaster, R., Altmeyer, M., Marx, A., and
34, 326-343, doi: 10.15252/embj.201489184.
Bürkle, A. (2007) Quantitative analysis of the binding
44.
Ame, J.C., Spenlehauer, C., and de Murcia, G. (2004) The
affinity of poly(ADP ribose) to specific binding proteins as
PARP superfamily, Bioessays, 26, 882-893, doi: 10.1002/
a function of chain length, Nucleic Acids Res., 35, e143,
bies.20085.
doi: 10.1093/nar/gkm944.
45.
Schreiber, V., Dantzer, F., Ame, J.C., and de Murcia, G.
62.
Gagne, J.P., Isabell, M., Lo, K.S., Bourassa, S., Hendzel, M.J.,
(2006) Poly(ADP ribose): novel functions for an old mole
Dawson, V.L., Dawson, T.M., and Poirier, G.G. (2008)
cule, Nat. Rev. Mol. Cell Biol., 7, 517-528, doi: 10.1038/
Proteome wide identification of poly(ADP ribose) bind
nrm1963.
ing proteins and poly(ADP ribose) associated protein
46.
Shieh, W.M., Ame, J.C., Wilson, M.V., Wang, Z.Q.,
complexes, Nucleic Acids Res.,
36,
6959-6976,
Koh, D.W., Jacobson, M.K., and Jacobson, E.L. (1998)
doi: 10.1093/nar/gkn771.
Poly(ADP ribose) polymerase null mouse cells synthesize
63.
Jungmichel, S., Rosenthal, F., Altmeyer, M., Lukas, J.,
ADP ribose polymers. J. Biol. Chem., 273, 30069-30072.
Hottiger, M.O., and Nielsen, M.L. (2013) Proteome wide
47.
Virag, L., and Szabo, C. (2002) The therapeutic potential
identification of poly(ADP ribosyl)ation targets in differ
of poly(ADP ribose) polymerase inhibitors, Pharmacol.
ent genotoxic stress responses, Mol. Cell., 52, 272-285,
Rev., 54, 375-429.
doi: 10.1016/j.molcel.2013.08.026.
48.
Burkle, A., and Virag, L. (2013) Poly(ADP ribose):
64.
Flohr, C., Burkle, A., Radicella, J.P., and Epe, B. (2003)
PARadigms and PARadoxes, Mol. Aspects Med., 34,
Poly(ADP ribosyl)ation accelerates DNA repair in a path
1046-1065, doi: 10.1016/j.mam.2012.12.010.
way dependent on Cockayne syndrome B protein, Nucleic
49.
Kraus, W.L., and Hottiger, M.O. (2013) PARP 1 and gene
Acids Res., 31, 5332-5337.
regulation: progress and puzzles, Mol. Aspects Med., 34,
65.
Vodenicharov, M.D., Ghodgaonkar, M.M., Halappanavar, S.S.,
1109-1123, doi: 10.1016/j.mam.2013.01.005.
Shah, R.G., and Shah, G.M. (2005) Mechanism of early
50.
Bock, F.J., Todorova, T.T., and Chang, P. (2015) RNA reg
biphasic activation of poly(ADP ribose) polymerase 1 in
ulation by poly(ADP ribose) polymerases, Mol. Cell, 58,
response to ultraviolet B radiation, J. Cell. Sci., 118,
959-969, doi: 10.1016/j.molcel.2015.01.037.
589-599, doi: 10.1242/jcs.01636.
51.
Liu, C., Vyas, A., Kassab, M.A., Singh, A.K., and Yu, X.
66.
Purohit, N.K., Robu, M., Shah, R.G., Geacintov, N.E.,
(2017) The role of poly ADP ribosylation in the first wave
and Shah, G.M. (2016) Characterization of the interac
of DNA damage response, Nucleic Acids Res., 45,
tions of PARP 1 with UV damaged DNA in vivo and in
8129-8141, doi: 10.1093/nar/gkx565.
vitro, Sci. Rep., 6, 19020, doi: 10.1038/srep19020.
52.
Khodyreva, S.N., and Lavrik, O.I. (2016) Poly(ADP
67.
Lin, T., and Yang, M.S. (2008) Benzo[a]pyrene induced
ribose) polymerase 1 as a key regulator of DNA repair, Mol.
necrosis in the HepG(2) cells via PARP 1 activation and
Biol.
(Moskow),
50,
580-595, doi:
10.7868/
NAD(+) depletion, Toxicology, 245, 147-153, doi: 10.1016/
S0026898416040030.
j.tox.2007.12.020.
53.
Sukhanova, M.V., Khodyreva, S.N., Lebedeva, N.A.,
68.
Tao, G.H., Yang, L.Q., Gong, C.M., Huang, H.Y., Liu, J.D.,
Prasad, R., Wilson, S.H., and Lavrik, O.I. (2005) Human
Liu, J.J., Yuan, J.H., Chen, W., and Zhuang, Z.X. (2009)
base excision repair enzymes apurinic/apyrimidinic
Effect of PARP 1 deficiency on DNA damage and repair in
endonuclease 1 (APE1), DNA polymerase beta and
human bronchial epithelial cells exposed to
poly(ADP ribose) polymerase 1: interplay between strand
benzo(a)pyrene, Mol. Biol. Rep.,
36,
2413-2422,
displacement DNA synthesis and proofreading exonucle
doi: 10.1007/s11033 009 9472 z.
ase activity, Nucleic Acids Res.,
33,
1222-1229,
69.
Fischer, J.M.F., Zubel, T., Jander, K., Fix, J.,
doi: 10.1093/nar/gki266.
Trussina, I.R.E.A., Gebhard, D., Bergemann, J., Burkle, A.,
54.
Sukhanova, M.V., Khodyreva, S.N, and Lavrik, O.I.
and Mangerich, A.
(2018) PARP1 protects from
(2004) Poly(ADP ribose) polymerase 1 inhibits strand
benzo[a]pyrene diol epoxide induced replication stress
displacement synthesis of DNA catalyzed by DNA poly
and mutagenicity, Arch. Toxicol.,
92,
1323-1340,
merase beta, Biochemistry (Moscow), 69, 558-568.
doi: 10.1007/s00204 017 2115 6.
55.
Berger, N.A., Sikorski, G.W., Petzold, S.J., and Kurohara, K.K.
70.
Pines, A., Vrouwe, M.G., Marteijn, J.A., Typas, D.,
(1980) Defective poly(adenosine diphosphoribose) synthe
Luijsterburg, M.S., Cansoy, M., Hensbergen, P., Deelder, A.,
sis in xeroderma pigmentosum, Biochemistry, 19, 289-293.
de Groot, A., Matsumoto, S., Sugasawa, K., Thoma, N.,
56.
McCurry, L.S., and Jacobson, M.K. (1981) Poly(ADP
Vermeulen, W., Vrieling, H., and Mullenders, L. (2012)
ribose) synthesis following DNA damage in cells heterozy
PARP1 promotes nucleotide excision repair through
gous or homozygous for the xeroderma pigmentosum
DDB2 stabilization and recruitment of ALC1, J. Cell Biol.,
genotype, J. Biol. Chem., 256, 551-553.
199, 235-249, doi: 10.1083/jcb.201112132.
57.
Jacobson, E.L., Antol, K.M., Juarez Salinas, H., and
71.
Luijsterburg, M.S., Lindh, M., Acs, K., Vrouwe, M.G.,
Jacobson, M.K. (1983) Poly(ADP ribose) metabolism in
Pines, A., van Attikum, H., Mullenders, L.H., and
БИОХИМИЯ том 84 вып. 9 2019
ПОСТТРАНСЛЯЦИОННЫЕ МОДИФИКАЦИИ ФАКТОРОВ NER
1257
Dantuma, N.P. (2012) DDB2 promotes chromatin decon
84.
Waters, R., van Eijk, P., and Reed, S. (2015) Histone mod
densation at UV induced DNA damage, J. Cell Biol., 197,
ification and chromatin remodeling during NER, DNA
267-281, doi: 10.1083/jcb.201106074.
Repair (Amst.), 36, 105-113, doi: 10.1016/j.dnarep.
72.
Robu, M., Shah, R. G., Petitclerc, N., Brind’Amour, J.,
2015.09.013.
Kandan Kulangara, F., and Shah, G.M. (2013) Role of
85.
Yu, S., Evans, K., van Eijk, P., Bennett, M., Webster, R.M.,
poly(ADP ribose) polymerase 1 in the removal of UV
Leadbitter, M., Teng, Y., Waters, R., Jackson, S.P., and
induced DNA lesions by nucleotide excision repair, Proc.
Reed, S.H. (2016) Global genome nucleotide excision
Natl. Acad. Sci. USA, 110, 1658-1663, doi: 10.1073/
repair is organized into domains that promote efficient
pnas.1209507110.
DNA repair in chromatin, Genome Res., 26, 1376-1387,
73.
Robu, M., Shah, R.G., Purohit, N.K., Zhou, P., Naegeli, H.,
doi: 10.1101/gr.209106.116.
and Shah, G.M. (2017) Poly(ADP ribose) polymerase 1
86.
Datta, A., Bagchi, S., Nag, A., Shiyanov, P., Adami, G.R.,
escorts XPC to UV induced DNA lesions during
Yoon, T., and Raychaudhuri, P. (2001) The p48 subunit of
nucleotide excision repair, Proc. Natl. Acad. Sci. USA, 114,
the damaged DNA binding protein DDB associates with
6847-6856, doi: 10.1073/pnas.1706981114.
the CBP/p300 family of histone acetyltransferase, Mutat.
74.
Maltseva, E.A., Rechkunova, N.I., Sukhanova, M.V., and
Res., 486, 89-97.
Lavrik, O.I. (2015) Poly(ADP ribose) Polymerase 1 mod
87.
Rapić Otrin, V., McLenigan, M.P., Bisi, D.C., Gonzalez, M.,
ulates interaction of the nucleotide excision repair factor
and Levine, A.S. (2002) Sequential binding of UV DNA
XPC RAD23B with DNA via Poly(ADP ribosyl)ation,
damage binding factor and degradation of the p48 subunit
J. Biol. Chem., 290, 21811-21820, doi: 10.1074/jbc.M115.
as early events after UV irradiation, Nucleic Acids Res., 30,
646638.
2588-2598.
75.
King, B.S., Cooper, K.L., Liu, K.J., and Hudson, L.G.
88.
Martinez, E., Palhan, V.B., Tjernberg, A., Lymar, E.S.,
(2012) Poly(ADP ribose) contributes to an association
Gamper, A.M., Kundu, T.K., Chait, B.T., and Roeder, R.G.
between poly(ADP ribose) polymerase 1 and xeroderma
(2001) Human STAGA complex is a chromatin acetylating
pigmentosum complementation group A in nucleotide
transcription coactivator that interacts with pre mRNA
excision repair, J. Biol. Chem., 287, 39824-39833,
splicing and DNA damage binding factors in vivo, Mol.
doi: 10.1074/jbc.M112.393504.
Cell. Biol., 21, 6782-6795, doi: 10.1128/MCB.21.20.6782
76.
Fischer, J.M., Popp, O., Gebhard, D., Veith, S.,
6795.2001.
Fischbach, A., Beneke, S., Leitenstorfer, A., Bergemann, J.,
89.
Matsunuma, R., Niida, H., Ohhata, T., Kitagawa, K.,
Scheffner, M., Ferrando May, E., Mangerich, A., and
Sakai, S., Uchida, C., Shiotani, B., Matsumoto, M.,
Burkle, A. (2014) Poly(ADP ribose) mediated interplay of
Nakayama, K.I., Ogura, H., Shiiya, N., and Kitagawa, M.
XPA and PARP1 leads to reciprocal regulation of protein
(2015) UV damage induced phosphorylation of HBO1
function, FEBS J.,
281,
3625-3641, doi:
10.1111/
triggers CRL4DDB2 mediated degradation to regulate cell
febs.12885.
proliferation, Mol. Cell. Biol., 36, 394-406, doi: 10.1128/
77.
Eki, T., and Hurwitz, J. (1991) Influence of poly(ADP
MCB.00809 15.
ribose) polymerase on the enzymatic synthesis of SV40
90.
Zhao, R., Han, C., Eisenhauer, E., Kroger, J., Zhao, W.,
DNA, J. Biol. Chem., 266, 3087-3100.
Yu, J., Selvendiran, K., Liu, X., Wani, A.A., and Wang, Q.E.
78.
Gagne, J.P., Pic, E., Isabelle, M., Krietsch, J., Ethier, C.,
(2014) DNA damage binding complex recruits HDAC1 to
Paquet, E., Kelly, I., Boutin, M., Moon, K.M., Foster, L.J.,
repress Bcl 2 transcription in human ovarian cancer cells,
and Poirier, G.G. (2012) Quantitative proteomics profiling
Mol. Cancer Res., 12, 370-380, doi: 10.1158/1541
of the poly(ADP ribose) related response to genotoxic
7786.MCR 13 0281.
stress, Nucleic Acids Res., 40, 7788-7805, doi: 10.1093/
91.
Zhu, Q., Battu, A., Ray, A., Wani, G., Qian, J., He, J.,
nar/gks486.
Wang, Q.E., and Wani, A.A. (2015) Damaged DNA bind
79.
Illuzzi, G., Fouquerel, E., Ame, J.C., Noll, A., Rehmet, K.,
ing protein down regulates epigenetic mark H3K56Ac
Nasheuer, H.P., Dantzer, F., and Schreiber, V. (2014) PARG
through histone deacetylase 1 and 2, Mutat. Res., 776,
is dispensable for recovery from transient replicative stress
16-23, doi: 10.1016/j.mrfmmm.2015.01.005.
but required to prevent detrimental accumulation of
92.
Kakumu, E., Nakanishi, S., Shiratori, H.M., Kato, A.,
poly(ADP ribose) upon prolonged replicative stress, Nucleic
Kobayashi, W., Machida, S., Yasuda, T., Adachi, N., Saito, N.,
Acids Res., 42, 7776-7792, doi: 10.1093/nar/gku505.
Ikura, T., Kurumizaka, H., Kimura, H., Yokoi, M., Sakai, W.,
80.
Maltseva, E.A., Krasikova, Y.S., Sukhanova, M.V.,
and Sugasawa, K. (2017) Xeroderma pigmentosum group
Rechkunova, N.I., and Lavrik OI. (2018) Replication pro
C protein interacts with histones: regulation by acetylated
tein A as a modulator of the poly(ADP ribose)polymerase
states of histone H3, Genes Cells,
22,
310-327,
1 activity, DNA Repair (Amst.),
72,
28-38, doi:
doi: 10.1111/gtc.12479.
10.1016/j.dnarep.2018.09.010.
93.
Tillhon, M., Cazzalini, O., Nardo, T., Necchi, D.,
81.
Thorslund, T., von Kobbe, C., Harrigan, J.A., Indig, F.E.,
Sommatis, S., Stivala, L.A., Scovassi, A.I., and Prosperi, E.
Christiansen, M., Stevnsner, T., and Bohr, V.A. (2005)
(2012) p300/CBP acetyl transferases interact with and
Cooperation of the cockayne syndrome group B protein
acetylate the nucleotide excision repair factor XPG, DNA
and poly(ADP ribose) polymerase 1 in the response to
Repair (Amst.), 11, 844-852, doi: 10.1016/j.dnarep.2012.
oxidative stress, Mol. Cell. Biol.,
25,
7625-7636,
08.001.
doi: 10.1128/MCB.25.17.7625 7636.2005.
94.
Fan, W., and Luo, J. (2010) SIRT1 regulates UV induced
82.
Evdokimov, A.N., Petruseva, I.O., Pestryakov, P.E., and
DNA repair through deacetylating XPA, Mol. Cell, 39,
Lavrik, O.I. (2011) Photoactivated DNA analogs of sub
247-258, doi: 10.1016/j.molcel.2010.07.006.
strates of the nucleotide excision repair system and their
95.
Zhao, M., Geng, R., Guo, X., Yuan, R., Zhou, X., Zhong, Y.,
interaction with proteins of NER competent extract of
Huo, Y., Zhou, M., Shen, Q., Li, Y., Zhu, W., and Wang, J.
HeLa cells. Synthesis and application of long model DNA,
(2017) PCAF/GCN5 mediated acetylation of RPA1 pro
Biochemistry (Moscow), 76, 157-166.
motes nucleotide excision repair, Cell Rep.,
20,
83.
Yu, Y., and Waters, R. (2005) Histone acetylation, chro
1997-2009, doi: 10.1016/j.celrep.2017.08.015.
matin remodelling and nucleotide excision repair: hint
96.
He, H., Wang, J., and Liu, T. (2017) UV Induced RPA1
from the study on MFA2 in Saccharomyces cerevisiae, Cell
acetylation promotes nucleotide excision repair, Cell Rep.,
Cycle, 4, 1043-1045, doi: 10.4161/cc.4.8.1928.
20, 2010-2025, doi: 10.1016/j.celrep.2017.08.016.
4 БИОХИМИЯ том 84 вып. 9 2019
1258
РЕЧКУНОВА и др.
97. Matsuoka, S., Ballif, B.A., Smogorzewska, A.,
HERC2 during nucleotide excision repair, Oncogene, 33,
McDonald, E.R. 3rd, Hurov, K.E., Luo, J., Bakalarski, C.E.,
19-25, doi: 10.1038/onc.2012.539.
Zhao, Z., Solimini, N., Lerenthal, Y., Shiloh, Y., Gygi, S.P.,
104. Coin, F., Auriol, J., Tapias, A., Clivio, P., Vermeulen, W.,
and Elledge, S.J. (2007) ATM and ATR substrate analysis
and Egly, J.M. (2004) Phosphorylation of XPB helicase
reveals extensive protein networks responsive to DNA dam
regulates TFIIH nucleotide excision repair activity, EMBO
age, Science, 316, 1160-1166, doi: 10.1126/science.1140321.
J., 23, 4835-4846, doi: 10.1038/sj.emboj.7600480.
98. Zannini, L., Delia, D., and Buscemi, G. (2014) CHK2
105. Oakley, G.G., Loberg, L.I., Yao, J., Risinger, M.A.,
kinase in the DNA damage response and beyond, J. Mol.
Yunker, R.L., Zernik Kobak, M., Khanna, K.K., Lavin, M.F.,
Cell Biol., 6, 442-457, doi: 10.1093/jmcb/mju045.
Carty, M.P., and Dixon, K. (2001) UV induced hyper
99. Attwood, P.V., Besant, P.G., and Piggott, M.J. (2011)
phosphorylation of replication protein A depends on DNA
Focus on phosphoaspartate and phosphoglutamate, Amino
replication and expression of ATM protein, Mol. Biol. Cell,
Acids, 40, 1035-1051, doi: 10.1007/s00726 010 0738 5.
12, 1199-1213, doi: 10.1091/mbc.12.5.1199.
100. Hornbeck, P.V., Kornhauser, J.M., Tkachev, S., Zhang, B.,
106. Liu, V.F., and Weaver, D.T. (1993) The ionizing radiation
Skrzypek, E., Murray, B., Latham, V., and Sullivan, M.
induced replication protein A phosphorylation response
(2012) PhosphoSitePlus: a comprehensive resource for
differs between ataxia telangiectasia and normal human
investigating the structure and function of experimentally
cells, Mol. Cell. Biol., 13, 7222-7231.
determined post translational modifications in man and
107. Rodrigo, G., Roumagnac, S., Wold, M.S., Salles, B., and
mouse, Nucleic Acids Res., 40, D261-D270, doi: 10.1093/
Calsou, P. (2000) DNA replication but not nucleotide exci
nar/gkr1122.
sion repair is required for UVC induced replication protein
101. Shah, P., Zhao, B., Qiang, L., and He, Y.Y.
(2018)
A phosphorylation in mammalian cells, Mol. Cell. Biol., 20,
Phosphorylation of xeroderma pigmentosum group C reg
2696-2705.
ulates ultraviolet induced DNA damage repair, Nucleic
108. Niida, H., Matsunuma, R., Horiguchi, R., Uchida, C.,
Acids Res., 46, 5050-5060, doi: 10.1093/nar/gky239.
Nakazawa, Y., Motegi, A., Nishimoto, K., Sakai, S.,
102. Wu, X., Shell, S.M., Yang, Z., and Zou, Y.
(2006)
Ohhata, T., Kitagawa, K., Moriwaki, S., Nishitani, H., Ui, A.,
Phosphorylation of nucleotide excision repair factor xero
Ogi, T., and Kitagawa, M. (2017) Phosphorylated HBO1 at
derma pigmentosum group A by ataxia telangiectasia
UV irradiated sites is essential for nucleotide excision
mutated and Rad3 related dependent checkpoint pathway
repair, Nat. Commun.,
8,
16102, doi:
10.1038/
promotes cell survival in response to UV irradiation,
ncomms16102.
Cancer Res., 66, 2997-3005, doi: 10.1158/0008 5472.
109. Krishnakumar, R., and Kraus, W.L. (2010) The PARP side
CAN 05 3403.
of the nucleus: molecular actions, physiological outcomes,
103. Lee, T.H., Park, J.M., Leem, S.H., and Kang, T.H. (2014)
and clinical targets, Mol. Cell, 39, 8-24, doi: 10.1016/
Coordinated regulation of XPA stability by ATR and
j.molcel.2010.06.017.
POST6TRANSLATIONAL MODIFICATIONS OF NUCLEOTIDE EXCISION
REPAIR PROTEINS AND THEIR ROLE IN THE PROCESS REGULATION
N. I. Rechkunova1,2*, E. A. Maltseva1, and O. I. Lavrik1,2
1 Institute of Chemical Biology and Fundamental Medicine, Siberian Branch of the Russian Academy of Sciences,
630090 Novosibirsk, Russia; E-mail: nadyarec@niboch.nsc.ru
2 Novosibirsk State University, 630090 Novosibirsk, Russia
Received March 21, 2019
Revised April 26, 2019
Accepted May 15, 2019
Nucleotide excision repair (NER) is one of the major DNA repair pathways to maintain genome stability. Correction
of damage by the NER system is a complex multistage process that proceeds with the formation of a multitude of
intermediate complexes based on DNA-protein and protein-protein interactions, which require precise coordina
tion and regulation. NER proteins undergo post translational modifications, such as ubiquitination, sumoylation,
phosphorylation, acetylation, and poly(ADP ribosyl)ation. These modifications affect the interaction of the proteins
with DNA and other proteins and thus regulate the involvement of repair factors into the complex at a certain stage
of the process or their dissociation from the complex as well as modulate the functional activity of the proteins and
the process in total. The currently known post translational modifications of NER proteins and data on their effect
on the repair process are reviewed. Protein poly(ADP ribosyl)ation catalyzed by poly(ADP ribose)polymerase 1 and
its effect on the NER process were analyzed in detail, since such analysis has not been performed before.
Keywords: nucleotide excision repair factors, post translational modification of proteins, activity regulation
БИОХИМИЯ том 84 вып. 9 2019