БИОХИМИЯ, 2020, том 85, вып. 1, с. 116 - 125
УДК 577.151.02; 577.152.2
МОДЕЛИРОВАНИЕ ФЕРМЕНТ СУБСТРАТНЫХ КОМПЛЕКСОВ
ПОЛИ(ADP РИБОЗО)ПОЛИМЕРАЗЫ 1 ЧЕЛОВЕКА*,**
© 2020
Д.К. Нилов1#, С.В. Пушкарев2#, И.В. Гущина2, Г.А. Манасарян3,
К.И. Кирсанов4, В.К. Швядас1,2***
1 Московский государственный университет им. М.В. Ломоносова, НИИ физико"химической биологии
им. А.Н. Белозерского, 119991 Москва, Россия; электронная почта: vytas@belozersky.msu.ru
2 Московский государственный университет им. М.В. Ломоносова,
факультет биоинженерии и биоинформатики, 119991 Москва, Россия
3 Московский государственный университет им. М.В. Ломоносова,
факультет фундаментальной медицины, 119991 Москва, Россия
4 Национальный медицинский исследовательский центр онкологии им. Н.Н. Блохина,
Научно"исследовательский институт канцерогенеза, 115478 Москва, Россия
Поступила в редакцию 19.08.2019
После доработки 16.10.2019
Принята к публикации 16.10.2019
Поли(ADP рибозо)полимераза 1 (ПАРП 1) является ключевым ферментом репарации ДНК и важной ми
шенью для терапии онкологических заболеваний. Сложное строение субстратов ПАРП 1 ограничивает воз
можности экспериментального изучения механизма реакции, однако необходимые данные могут быть по
лучены путем молекулярного моделирования. В представленной работе впервые получена молекулярно ди
намическая модель фермент субстратного комплекса ПАРП 1, содержащего молекулу NAD+ и конец цепи
поли(ADP рибозы) в виде молекулы ADP. Охарактеризованы взаимодействия с остатками активного цент
ра, среди которых определяющую роль играют Gly863, Lys903, Glu988, а также предложен SN1 подобный
механизм для катализируемой реакции ADP рибозилирования. С помощью молекулярного докинга полу
чены модели комплексов ПАРП 1 с более сложными двухзвенными фрагментами растущей цепи полимера
и конкурентными ингибиторами 3 аминобензамидом и 7 метилгуанином.
КЛЮЧЕВЫЕ СЛОВА: репарация ДНК, молекулярная динамика, докинг, субстраты, ингибиторы.
DOI: 10.31857/S0320972520010091
Поли(ADP рибозо)полимераза 1 человека
ков глутаминовой и аспарагиновой кислоты, а
(ПАРП 1; КФ 2.4.2.30) является ферментом су
также лизина) [6, 7]. Далее происходит синтез
персемейства ADP рибозилтрансфераз, кото
полианиона ПАР путем последовательного при
рый обладает ДНК зависимой активностью и
соединения новых звеньев ADP рибозы с обра
катализирует синтез поли(ADP рибозы) (ПАР,
зованием гликозидных связей α(1→2) [8, 9].
рис. 1) из молекул NAD+ [1-5]. ПАРП 1 осуще
Связывание ПАРП 1 с разрывами ДНК при
ствляет перенос ADP рибозы на белок акцеп
водит к модификации других белков, вовлечен
тор с высвобождением никотинамида (модифи
ных в метаболизм ДНК, а также к автомодифи
кации могут подвергаться боковые цепи остат
кации [11, 12]. Результатом поли(ADP рибо
зил)ирования является реорганизация структу
Принятые сокращения: ПАРП 1 - поли(ADP ри
ры хроматина и мобилизация белков репарации
бозо)полимераза 1, ПАР - поли(ADP рибоза), МД - мо
для устранения повреждения [13-16]. В част
лекулярная динамика.
ности, автомодифицированная ПАРП 1 образу
* Первоначально английский вариант рукописи опубли
ет комплекс с белком эксцизионной репарации
XRCC1, который, в свою очередь, взаимодей
msu.ru/biokhimiya, в рубрике «Papers in Press», BM19 253,
ствует с ДНК полимеразой β и ДНК лигазой III
16.12.2019.
[17, 18]. Поскольку ПАРП 1 является ключевым
** Приложение к статье на английском языке опублико
вано на сайте журнала «Biochemistry» (Moscow) и на сайте
ферментом репарации ДНК в опухолевых клет
ках, большое внимание уделяется поиску его
10541), том 85, вып. 1, 2020.
ингибиторов, обладающих собственным анти
*** Адресат для корреспонденции.
пролиферативным эффектом или действующих
# Авторы внесли равный вклад в работу.
в сочетании с ДНК повреждающими препара
116
МОДЕЛИРОВАНИЕ КОМПЛЕКСОВ ПАРП 1
117
тами [19-22]. Недавно для лечения рака молоч
термедиатов, поэтому актуальной задачей явля
ной железы и яичников были одобрены три син
ется моделирование фермент субстратных
тетических ингибитора ПАРП 1: олапариб, ру
комплексов на основе доступных кристаллогра
капариб и нирапариб [23-25]. Клеточная функ
фических данных. Детальное исследование мо
ция ПАРП 1, а также способы ее подавления
лекулярных взаимодействий ПАРП 1 с субстра
подробно рассмотрены в большом числе обзо
тами представляет не только фундаментальный
ров [26-31].
интерес, но и может создать основу для рацио
О молекулярном механизме реакции ADP
нального дизайна эффективных конкурентных
рибозилирования известно не так много. В ак
ингибиторов. Успешное решение данной задачи
тивном центре каталитического домена ПАРП 1
в настоящее время становится возможным бла
можно выделить участок связывания донора
годаря следующим факторам: наличию много
(NAD+) и участок связывания акцептора (ПАР)
доменной структуры апоформы 4dqy [37, 38],
[32]. Молекула NAD+, по видимому, образует
структуры каталитического домена со связан
водородные связи с Gly863 и гидрофобный кон
ным аналогом субстрата акцептора 1a26 [35] и
такт с Tyr907 подобно миметикам никотинамид
недавнему появлению структуры каталитичес
ного фрагмента, для которых установлена
кого домена ПАРП 1 со связанным аналогом
структура фермент ингибиторных комплексов
субстрата донора 6bhv [39].
[33, 34]. Некоторые предположения относитель
но связывания субстрата акцептора можно сде
лать на основе кристаллической структуры
МЕТОДЫ ИССЛЕДОВАНИЯ
ПАРП 1 с неактивным структурным аналогом,
аденозиндифосфатный фрагмент которого об
Молекулярную модель ПАРП 1 человека
разует гидрофобный контакт с Met890 и водо
конструировали на основе кристаллической
родные связи с Lys903 и Glu988 [35]. Карбок
структуры 4dqy (цепи A, B, C, M и N). Координа
сильная группа остатка Glu988 находится вбли
ты неразрешенной петли 576-583 в домене WGR
зи расщепляемой N гликозидной связи NAD+
перенесли из структуры 2cr9. Координаты петли
и, предположительно, может выступать в роли
645-661 между WGR и каталитическим доменом
общего основания, активирующего нуклео
были предсказаны с помощью программы
фильную группу субстрата акцептора, и/или
Modeller 9.20 (рис. 2) [40]. Координаты аналога
участвовать в стабилизации переходного состо
NAD+ перенесли из структуры 6bhv, после чего
яния [33, 35, 36].
преобразовали бензамидный фрагмент в нико
На данный момент нет достоверной инфор
тинамидный путем замены соответствующего
мации о взаимной ориентации субстратов
атома углерода на азот. Координаты боковой це
ПАРП 1, необходимой для протекания реак
пи Arg878, взаимодействующей с адениновой
ции, и о структуре переходных состояний и ин
группой NAD+ и обладающей существенной
Рис. 1. Химическое строение ПАР. Синим цветом показано первое звено ADP рибозы, присоединенное к белку акцепто
ру. Ветвление полимера происходит, когда в реакцию нуклеофильного замещения вовлекается «никотинамидная» рибоза
ПАР. Присоединение ADP рибозы в результате реакций элонгации и ветвления происходит в соотношении 41/1 [10].
biokhsm/
БИОХИМИЯ том 85 вып. 1 2020
118
НИЛОВ и др.
Рис. 2. Многодоменное строение полученной модели ПАРП 1 человека со связанными молекулами ДНК, NAD+ и ADP.
biokhsm/
конформационной подвижностью, также заим
зовали силовое поле ff14SB [45], для описания
ствовали из 6bhv. Координаты ADP в качестве
молекул NAD+ и ADP - параметры из базы дан
структурного аналога субстрата акцептора пере
ных Amber Parameter Database [46-48].
несли из структуры 1a26. Для наложения струк
Среди кадров траектории равновесной симу
тур использовали программу Matt 1.00 [41].
ляции выбрали структуру, в которой взаимное
Далее структуру оптимизировали и исследо
расположение субстратов было близко к реак
вали методом молекулярной динамики (МД) с
ционноспособной конфигурации, и провели
помощью AmberTools 15 и пакета Amber 14 [42,
минимизацию ее энергии (5000 шагов наиско
43], установленного на суперкомпьютере МГУ
рейшего спуска + 5000 шагов по методу сопря
[44]. Атомы водорода добавляли с учетом иони
женных градиентов). Полученную структуру ис
зационных свойств остатков, в частности в ими
пользовали для ковалентного докинга в про
дазольном кольце остатка активного центра
грамме Lead Finder 1.1.15 [49, 50]: молекула ADP
His862 был протонирован Nδ1 атом. Структуру
была достроена до фрагментов ПАР, состоящих
окружали слоем (12 Å) воды TIP3P, для нейтра
из двух звеньев ADP рибозы. Недостающие
лизации отрицательного суммарного заряда до
группы атомов присоединяли к С5′ атому
бавляли ионы натрия. На первой стадии мини
(элонгация, ветвление) и С1′ атому (ветвление).
мизации энергии полученной системы (2500 ша
Кроме того, с помощью Lead Finder был осуще
гов по методу наискорейшего спуска + 2500 ша
ствлен стандартный докинг ингибиторов 3 ами
гов по методу сопряженных градиентов) коорди
нобензамида и 7 метилгуанина в активный
наты белка, ДНК и субстратов фиксировали по
центр (для этого из структуры ПАРП 1 были
зиционными ограничениями 2 ккал/(моль⋅Å2)
предварительно удалены молекулы субстратов).
на тяжелых атомах. Вторую стадию минимиза
Для визуализации структур использовали VMD
ции (5000 шагов наискорейшего спуска + 5000
1.9.2 [51].
шагов по методу сопряженных градиентов) про
водили без каких либо ограничений. Затем сис
тему разогревали от 0 до 300 К с использовани
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
ем позиционных ограничений 1 ккал/(моль⋅Å2)
на атомах белка, ДНК и субстратов (250 пс, пос
На основе доступного набора кристалличес
тоянный объем) и уравновешивали при 300 К
ких структур ПАРП 1 была впервые сконструи
(500 пс, постоянное давление). Достижение рав
рована МД модель фермент субстратного
новесной конформации субстратов подтвержда
комплекса c NAD+ и концевым фрагментом
ли путем анализа среднего квадратичного от
ПАР, представленным в виде молекулы ADP
клонения их атомов от начального положения.
(рис. S1 в Приложении). Полученная сольвати
В дальнейшем с использованием подготовлен
рованная система помимо субстратов включала
ной структуры рассчитывали и анализировали
703 аминокислотных остатка, 52 нуклеотида,
траекторию равновесной симуляции МД дли
2 иона Zn2+, 49 ионов Na+ и 73281 молекулу во
ною 5000 пс. Шаг интегрирования составлял
ды. В результате анализа траектории равновес
0,002 пс с учетом использования алгоритма
ной симуляции ПАРП 1 были обнаружены сле
SHAKE. Радиус отсечения невалентных взаимо
дующие важные межмолекулярные взаимодей
действий составлял 10 Å. Регуляцию температу
ствия. Никотинамидная группа NAD+ образует
ры осуществляли по методу Ланжевена, давле
две водородные связи с остатком Gly863 (табли
ния - по методу Берендсена. Для молекулярно
ца и рис. S2 в Приложении), а также π стэкинг с
механического описания белка и ДНК исполь
боковой цепью Tyr907, что согласуется с резуль
БИОХИМИЯ том 85 вып. 1 2020
МОДЕЛИРОВАНИЕ КОМПЛЕКСОВ ПАРП 1
119
татами гомологичного моделирования, которые
Ключевые взаимодействия в активном центре ПАРП 1,
выявленные с помощью равновесной МД симуляции.
были получены с использованием структур дру
Приведены средние значения расстояний вместе со стан
гих представителей суперсемейства (дифтерий
дартным отклонением
ного токсина и экзотоксина А) [34, 52]. Рибоза
адениновой части NAD+ образует водородную
Взаимодействие
Расстояние, Å
связь с Nε2 атомом имидазольного кольца
1,7 ± 0,1
His862. Адениновый заместитель концевой ри
Glu988:OE1 ⋅⋅⋅ NAD+:2′N OH:H
бозы ПАР формирует устойчивый гидрофобный
Glu988:OE2 ⋅⋅⋅ ADP:3′A OH:H
1,7 ± 0,2
контакт с боковой цепью Met890, а пирофос
2,0 ± 0,2
Gly863:H ⋅⋅⋅ NAD+:CONH2:O
фатная группа образует водородную связь с ами
2,4 ± 0,4
Gly863:O ⋅⋅⋅ NAD+:CONH2:H
ногруппой Lys903, которая, в свою очередь, ста
1,9 ± 0,3
Lys903:NH3:H* ⋅⋅⋅ ADP:P2O7:O
билизирует положение наиболее важного для
2,0 ± 0,2
катализа остатка Glu988. Карбоксильная группа
Lys903:NH3:H* ⋅⋅⋅ Glu988:OE2
Glu988 образует водородные связи с 2′N гидрок
1,9 ± 0,2
NAD+:CONH2:H ⋅⋅⋅ NAD+:P2O7:O**
сильной группой NAD+ и 3′A гидроксильной
группой фрагмента ПАР, обеспечивая требуемое
* Для каждого кадра траектории в расчет принимали ми
взаимное расположение субстрата донора и
нимальное из расстояний до атомов водорода аминогруп
пы Lys903, поскольку данная группа способна претерпе
субстрата акцептора (рис. 3, а; нижние индексы
вать вращение.
«N» и «A» обозначают принадлежность к нико
** Внутримолекулярная водородная связь NAD+.
тинамидной и адениновой рибозе).
Здесь следует вспомнить о поддерживаемом
некоторыми авторами предположении о том,
но предложить механизм и для реакции иници
что Glu988 образует также водородную связь с
ации синтеза ПАР, когда акцептором ADP ри
2′ гидроксильной группой ПАР и является ак
бозы служит остаток глутаминовой или аспара
цептором протона при нуклеофильной атаке по
гиновой кислоты на поверхности модифицируе
механизму SN2 [35, 36, 53, 54]. Однако в ходе си
мого белка. В этом случае один из атомов кисло
муляции мы не наблюдали ни образования упо
рода модифицируемой карбоксильной группы
мянутой водородной связи, ни реакционноспо
может занимать положение 2′ гидроксильной
собной линейной конфигурации атомов
группы ПАР вблизи расщепляемой гликозид
ADP:O2′A, NAD+:C1′N и NAD+:N1N, характерной
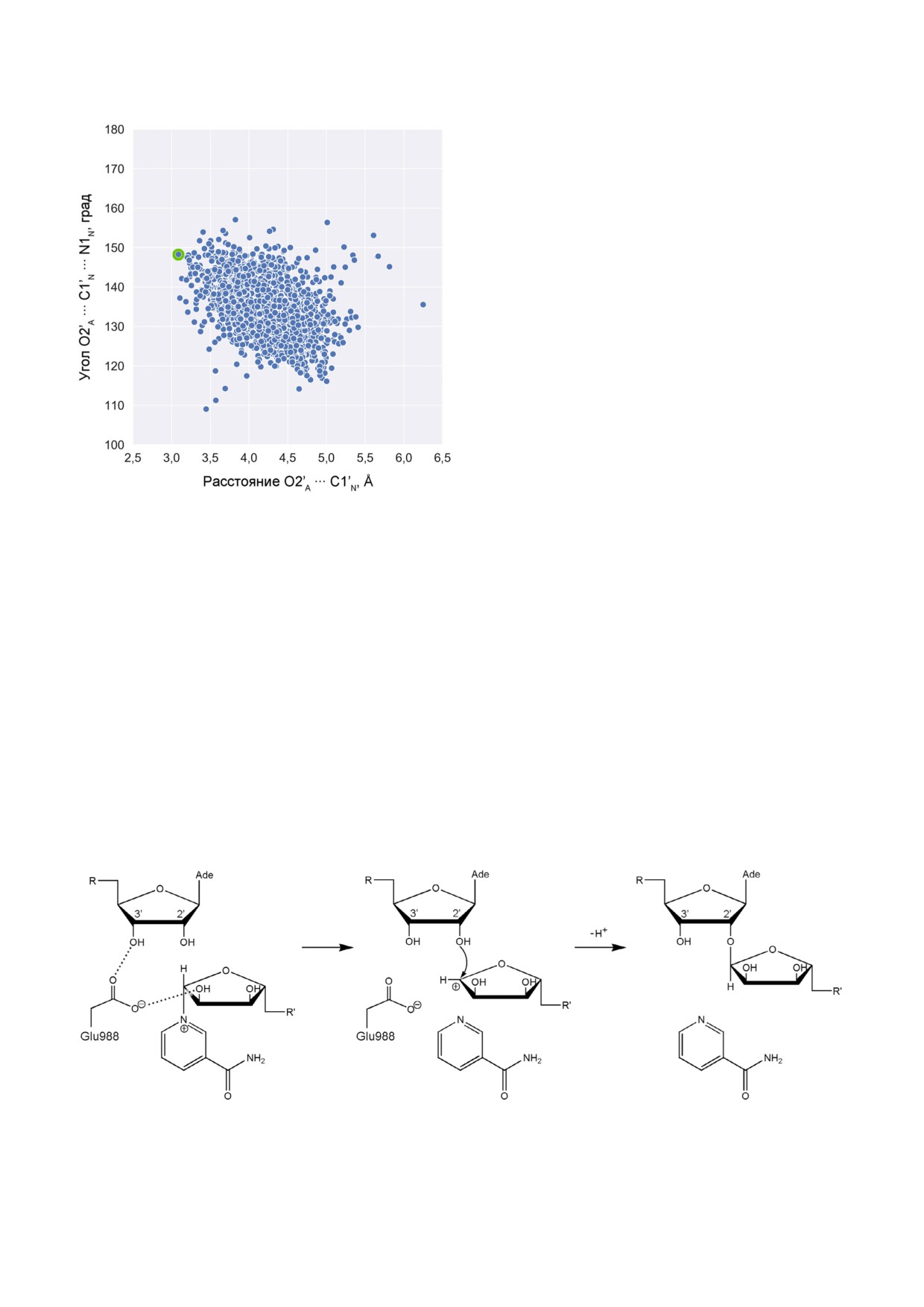
ной связи. Отрицательный заряд карбоксиль
для механизма SN2. Среднее значение расстоя
ной группы белка акцептора способствует рас
ния O2′A ⋅⋅⋅ C1′N составило 4,2 Å, угла O2′A ⋅⋅⋅ C1′N
щеплению NAD+ с образованием иона оксокар
⋅⋅⋅ N1N - 135° (рис. 4), в то время как необходи
бения, после чего происходит нуклеофильная
мое для нуклеофильной атаки расстояние со
атака. Данное предположение объясняет тот
ставляет ~3 Å, а угол не должен сильно откло
факт, что мутации E988Q и E988A существенно
няться от значения 180° [55]. Это дает основание
снижают способность ПАРП 1 катализировать
предположить, что катализируемая ПАРП 1 ре
реакцию элонгации, но мало влияют на стадию
акция ADP рибозилирования протекает по аль
инициации [36]. Glu988 обеспечивает реакци
тернативному, SN1 подобному механизму
онноспособную ориентацию субстратов (NAD+
(рис. 5). По видимому, сначала происходит об
и ПАР) и стабилизирует интермедиат при нара
разование интермедиата - иона оксокарбения,
щивании цепи ПАР. Однако на стадии инициа
который стабилизируется благодаря отрица
ции этот остаток не столь важен, поскольку его
тельному заряду карбоксильной группы Glu988.
стабилизирующую функцию выполняет карбок
Реакционный центр приобретает плоскую кон
сильная группа белка акцептора.
фигурацию, что облегчает последующую атаку
Полученная МД структура ПАРП 1 характе
2′ гидроксильной группой акцептора. Посколь
ризует взаимную ориентацию NAD+ и атакую
ку ПАР является отрицательно заряженным био
щей рибозы ПАР на примере модельной молеку
полимером, его связывание в активном центре
лы ADP, однако она не дает представления о по
ПАРП 1 в качестве субстрата акцептора может
ложении растущей цепи полимера. Конструиро
способствовать образованию иона оксокарбе
вание более сложных фрагментов ПАР, состоя
ния. Интересно, что похожий механизм был ус
щих из двух звеньев ADP рибозы, было осущест
тановлен для других представителей суперсе
влено методом ковалентного докинга. Для этого
мейства ADP рибозилтрансфераз: ПАРП 10
был выбран кадр траектории МД симуляции, в
[56], дифтерийного токсина [57, 58], экзотокси
котором взаимное расположение субстратов
на А [59, 60] и йота токсина [61, 62].
близко к реакционноспособной конфигурации
Допуская возможность образования иона
(рис. 4), а никотинамидная рибоза NAD+ пред
оксокарбения в активном центре ПАРП 1, мож
ставлена в конформации 3′ экзо. В данной кон
БИОХИМИЯ том 85 вып. 1 2020
120
НИЛОВ и др.
Рис. 3. Взаимное расположение NAD+ и ПАР в моделях фермент субстратных комплексов ПАРП 1, полученных метода
ми МД и докинга. а и б - Элонгация; в и г - ветвление. Можно видеть, что положение пирофосфатной группы, присое
диненной к атакующей рибозе ПАР, практически не отличается.
biokhsm/
формации атомы C2′N, C1′N, O4′N и С4′N находятся
вое звено переносится на концевую адениновую
в одной плоскости (как в ионе оксокарбения),
рибозу ПАР; реакционноспособная ориентация
что должно облегчать образование интермедиата
субстратов представлена на рис. 3, а и б. С опре
реакции. Далее к молекуле ADP присоединили
деленной периодичностью также происходит
недостающие группы атомов, получив коорди
ветвление полимера, когда новое звено присое
наты субстрата акцептора (ПАР) для реакций
диняется к «никотинамидной» рибозе (рис. 1)
элонгации и ветвления. В случае элонгации но
[10, 63, 64]. Считается, что для осуществления
БИОХИМИЯ том 85 вып. 1 2020
МОДЕЛИРОВАНИЕ КОМПЛЕКСОВ ПАРП 1
121
бозы с инвертированием конфигурации C1′
атома (рис. 3, в и г). На рис. 3, б и г, можно срав
нить ориентацию растущей цепи полимера в
случае элонгации и ветвления.
Адекватность полученной модели фермент
субстратного комплекса и важность установлен
ных взаимодействий в активном центре МД
структуры подтвердили результаты докинга двух
известных ингибиторов ПАРП 1 с предполагае
мым конкурентным механизмом действия. Пер
вое соединение, 3 аминобензамид, является хо
рошо изученным структурным аналогом нико
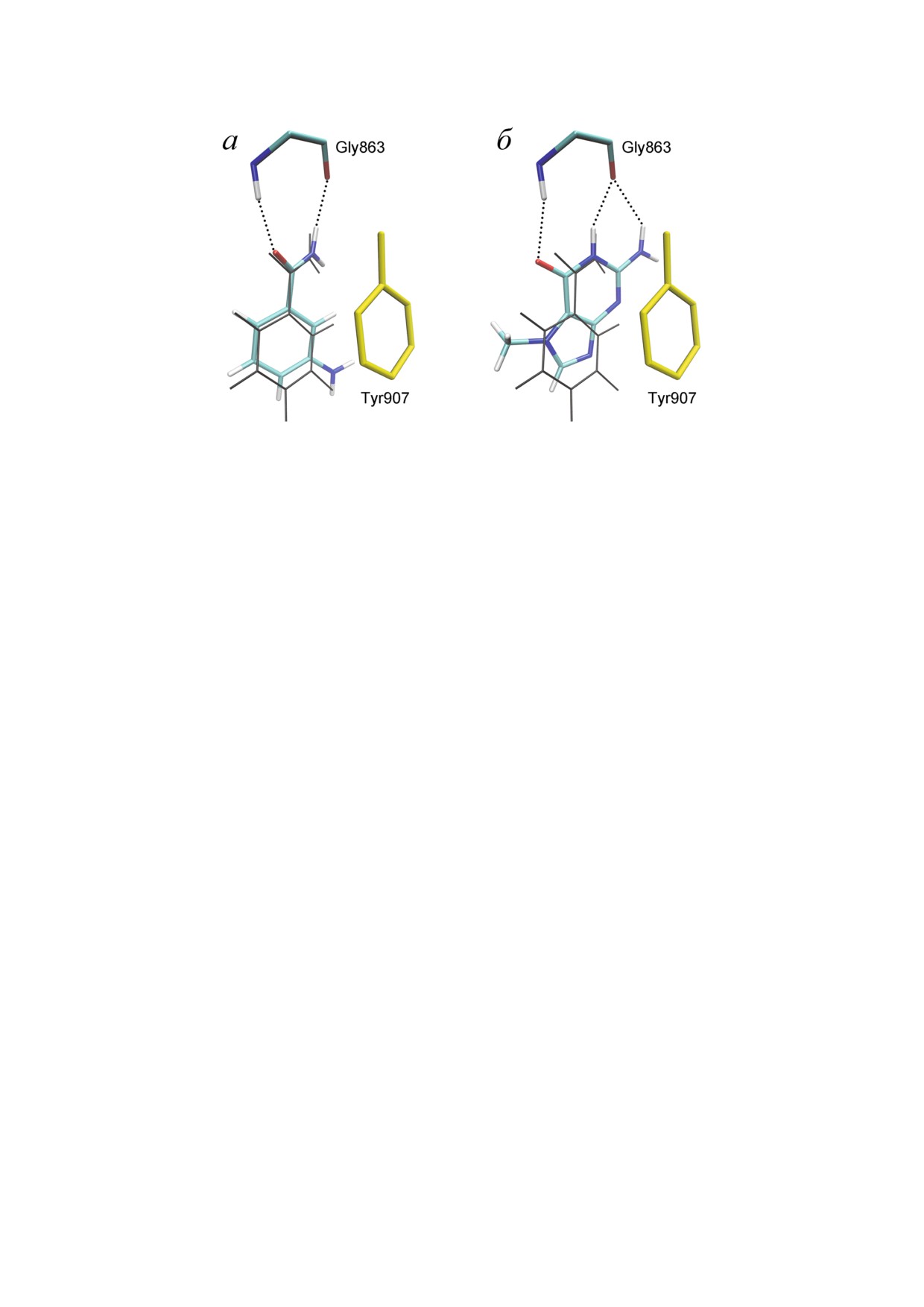
тинамидной группы NAD+ [65-67]. Второе со
единение, 7 метилгуанин, представляет собой
новый ингибитор ПАРП с перспективным про
филем фармакокинетики и токсичности. Дан
ный метаболит нуклеиновых кислот усиливает
апоптотическую гибель раковых клеток в комби
нации с цисплатином и доксорубицином in vitro,
не оказывая при этом существенного побочного
действия на организм в предварительных испы
Рис. 4. Распределение пар значений расстояние-угол для
таниях in vivo [68-70]. Молекулярный докинг
атомов в реакционном центре ПАРП 1, полученное в ре
показал, что 3 аминобензамид и 7 метилгуанин
зультате равновесной МД симуляции. Каждая точка соот
занимают участок никотинамидной группы
ветствует определенному кадру траектории, зеленым цве
NAD+, образуя свойственные субстрату взаимо
том отмечена структура (t = 3234 пс), которую использова
действия с остатками Gly863 и Tyr907 (рис. 6). В
ли для проведения докинга.
С цветным вариантом рис. 4 можно ознакомиться в элек
случае 7 метилгуанина функциональная амид
ная группа встроена в систему конденсирован
journal/biokhsm/
ных колец, однако это не мешает формированию
водородных связей с Gly863 (рис. 6, б).
В результате проведенных исследований со
данной реакции ПАР разворачивается на 180°,
здана модель фермент субстратного комплекса
что позволяет группе 2′N OH занять реакцион
ПАРП 1 человека. При анализе МД траектории
носпособное положение 2′A OH (рис. S3 в При
комплекса охарактеризованы взаимная ориента
ложении). Положение пирофосфатной группы,
ция субстрата донора (молекула NAD+) и
присоединенной к атакующей рибозе, не пре
субстрата акцептора (фрагмент ПАР), а также их
терпевает при этом существенных изменений
взаимодействия с остатками активного центра,
[35]. Для моделирования положения ПАР, соот
среди которых определяющую роль играют
ветствующего реакции ветвления, адениновая
Gly863, Lys903 и Glu988. Это позволило сделать
группа ADP была заменена на остаток ADP ри
предположение о том, что катализируемый
Рис. 5. Предполагаемый SN1 подобный механизм реакции ADP рибозилирования, катализируемой ПАРП 1. Отрица
тельный заряд карбоксильной группы Glu988 стабилизирует интермедиат (ион оксокарбения)
БИОХИМИЯ том 85 вып. 1 2020
122
НИЛОВ и др.
Рис. 6. Моделирование связывания 3 аминобензамида (а) и 7 метилгуанина (б) в активном центре ПАРП 1. Желтым цве
том показана фенильная группа Tyr907, образующая с ингибитором π стэкинг. Серым цветом показаны координаты ни
котинамидной группы NAD+ в МД структуре ПАРП 1.
biokhsm/
ПАРП 1 синтез ПАР происходит по SN1 подоб
Финансирование. Исследование выполнено
ному механизму через образование ионов оксо
при финансовой поддержке Российского фонда
карбения. Методом докинга показано, что учас
фундаментальных исследований (гранты № 18
ток связывания никотинамидной группы NAD+
315 00389 мол_а, и № 17 08 01614 А).
является мишенью для ингибиторов 3 амино
Конфликт интересов. Авторы заявляют об от
бензамида и 7 метилгуанина. В дальнейшем по
сутствии конфликта интересов.
лученная модель фермент субстратного комп
Соблюдение этических норм. Настоящая
лекса может быть использована при рациональ
статья не содержит описания выполненных ав
ном дизайне ингибиторов ПАРП нового поколе
торами исследований с участием людей или ис
ния. В частности, с помощью гибридных кванто
пользованием животных в качестве объектов.
во механических/молекулярно механических
Благодарности. Исследование выполнено с
методов на ее основе может быть получена струк
использованием оборудования Центра коллек
тура интермедиата ПАРП 1 для скрининга моле
тивного пользования сверхвысокопроизводи
кул, комплементарных соответствующему кон
тельными вычислительными ресурсами МГУ
формационному состоянию активного центра.
имени М.В. Ломоносова.
СПИСОК ЛИТЕРАТУРЫ
1.
Cohen, M.S., and Chang, P. (2018) Insights into the bio
6.
Tao, Z., Gao, P., and Liu, H.W. (2009) Identification of the
genesis, function, and regulation of ADP ribosylation,
ADP ribosylation sites in the PARP 1 automodification
Nat. Chem. Biol., 14, 236-243.
domain: analysis and implications, J. Am. Chem. Soc., 131,
2.
Taniguchi, T. (1987) Reaction mechanism for automodifi
14258-14260.
cation of poly(ADP ribose) synthetase, Biochem. Biophys.
7.
Altmeyer, M., Messner, S., Hassa, P.O., Fey, M., and
Res. Commun., 147, 1008-1012.
Hottiger, M.O.
(2009) Molecular mechanism of
3.
Lin, H. (2007) Nicotinamide adenine dinucleotide:
poly(ADP ribosyl)ation by PARP1 and identification of
beyond a redox coenzyme, Org. Biomol. Chem., 5,
lysine residues as ADP ribose acceptor sites, Nucleic Acids
2541-2554.
Res., 37, 3723-3738.
4.
Naegeli, H., Loetscher, P., and Althaus, F.R. (1989) Poly
8.
Drenichev, M.S., and Mikhailov, S.N. (2015) Poly(ADP
ADP ribosylation of proteins. Processivity of a post trans
ribose) - a unique natural polymer structural features, bio
lational modification, J. Biol. Chem., 264, 14382-14385.
logical role and approaches to the chemical synthesis,
5.
Ménard, L., Thibault, L., and Poirier, G.G.
(1990)
Nucleosides Nucleotides Nucleic Acids, 34, 258-276.
Reconstitution of an in vitro poly(ADP ribose) turnover
9.
Miwa, M., Ishihara, M., Takishima, S., Takasuka, N.,
system, Biochim. Biophys. Acta, 1049, 45-58.
Maeda, M., Yamaizumi, Z., Sugimura, T., Yokoyama, S.,
БИОХИМИЯ том 85 вып. 1 2020
МОДЕЛИРОВАНИЕ КОМПЛЕКСОВ ПАРП 1
123
and Miyazawa, T. (1981) The branching and linear portions
25.
Zimmer, A.S., Gillard, M., Lipkowitz, S., and Lee, J.M.
of poly(adenosine diphosphate ribose) have the same
(2018) Update on PARP inhibitors in breast cancer, Curr.
alpha(1 → 2) ribose ribose linkage, J. Biol. Chem., 256,
Treat. Options Oncol., 19, 21.
2916-2921.
26.
Ray Chaudhuri, A., and Nussenzweig, A. (2017) The mul
10.
Keith, G., Desgrès, J., and de Murcia, G. (1990) Use of
tifaceted roles of PARP1 in DNA repair and chromatin
two dimensional thin layer chromatography for the com
remodeling, Nat. Rev. Mol. Cell Biol., 18, 610-621.
ponents study of poly(adenosine diphosphate ribose), Anal.
27.
Ryu, K.W., Kim, D.S., and Kraus, W.L. (2015) New facets
Biochem., 191, 309-313.
in the regulation of gene expression by ADP ribosylation
11.
Mendoza Alvarez, H., and Alvarez Gonzalez, R. (1993)
and poly(ADP ribose) polymerases, Chem. Rev., 115,
Poly(ADP ribose) polymerase is a catalytic dimer and the
2453-2481.
automodification reaction is intermolecular, J. Biol. Chem.,
28.
Curtin, N.J., and Szabo, C. (2013) Therapeutic applica
268, 22575-22580.
tions of PARP inhibitors: anticancer therapy and beyond,
12.
Mendoza Alvarez, H., and Alvarez Gonzalez, R. (1999)
Mol. Aspects Med., 34, 1217-1256.
Biochemical characterization of mono(ADP ribosyl)ated
29.
Ferraris, D.V. (2010) Evolution of poly(ADP ribose) poly
poly(ADP ribose) polymerase, Biochemistry,
38,
merase 1 (PARP 1) inhibitors. From concept to clinic, J.
3948-3953.
Med. Chem., 53, 4561-4584.
13.
Hassler, M., and Ladurner, A.G. (2012) Towards a struc
30.
Virág, L., and Szabó, C. (2002) The therapeutic potential
tural understanding of PARP1 activation and related sig
of poly(ADP ribose) polymerase inhibitors, Pharmacol.
nalling ADP ribosyl transferases, Curr. Opin. Struct. Biol.,
Rev., 54, 375-429.
22, 721-729.
31.
Малюченко Н.В., Котова Е.Ю., Кулаева О.И., Кир
14.
Schreiber, V., Dantzer, F., Ame, J.C., and de Murcia, G.
пичников М.П., Студитский В.М. (2015) Ингибиторы
(2006) Poly(ADP ribose): novel functions for an old mole
PARP1: разработка противоопухолевых препаратов,
cule, Nat. Rev. Mol. Cell Biol., 7, 517-528.
Acta Naturae, 7, 30-41.
15.
Hassa, P.O., Haenni, S.S., Elser, M., and Hottiger, M.O.
32.
Barkauskaite, E., Jankevicius, G., and Ahel, I. (2015)
(2006) Nuclear ADP ribosylation reactions in mammalian
Structures and mechanisms of enzymes employed in the
cells: where are we today and where are we going?
synthesis and degradation of PARP dependent protein
Microbiol. Mol. Biol. Rev., 70, 789-829.
ADP ribosylation, Mol. Cell, 58, 935-946.
16.
Jagtap, P., and Szabó, C. (2005) Poly(ADP ribose) poly
33.
Ruf, A., Mennissier de Murcia, J., de Murcia, G., and
merase and the therapeutic effects of its inhibitors, Nat.
Schulz, G.E. (1996) Structure of the catalytic fragment of
Rev. Drug Discov., 4, 421-440.
poly(AD ribose) polymerase from chicken, Proc. Natl.
17.
Brem, R., and Hall, J. (2005) XRCC1 is required for DNA
Acad. Sci. USA, 93, 7481-7485.
single strand break repair in human cells, Nucleic Acids
34.
Ruf, A., de Murcia, G., and Schulz, G.E. (1998) Inhibitor
Res., 33, 2512-2520.
and NAD+ binding to poly(ADP ribose) polymerase as
18.
Masson, M., Niedergang, C., Schreiber, V., Muller, S.,
derived from crystal structures and homology modeling,
Menissier de Murcia, J., and de Murcia, G.
(1998)
Biochemistry, 37, 3893-3900.
XRCC1 is specifically associated with poly(ADP ribose)
35.
Ruf, A., Rolli, V., de Murcia, G., and Schulz, G.E. (1998)
polymerase and negatively regulates its activity following
The mechanism of the elongation and branching reaction
DNA damage, Mol. Cell. Biol., 18, 3563-3571.
of poly(ADP ribose) polymerase as derived from crystal
19.
Jain, P.G., and Patel, B.D. (2019) Medicinal chemistry
structures and mutagenesis, J. Mol. Biol., 278, 57-65.
approaches of poly ADP Ribose polymerase 1 (PARP1)
36.
Marsischky, G.T., Wilson, B.A., and Collier, R.J. (1995)
inhibitors as anticancer agents - a recent update, Eur. J.
Role of glutamic acid 988 of human poly ADP ribose
Med. Chem., 165, 198-215.
polymerase in polymer formation. Evidence for active site
20.
Martin, S.A., Lord, C.J., and Ashworth, A. (2008) DNA
similarities to the ADP ribosylating toxins, J. Biol. Chem.,
repair deficiency as a therapeutic target in cancer, Curr.
270, 3247-3254.
Opin. Genet. Dev., 18, 80-86.
37.
Langelier, M.F., Planck, J.L., Roy, S., and Pascal, J.M. (2012)
21.
Cepeda, V., Fuertes, M.A., Castilla, J., Alonso, C.,
Structural basis for DNA damage dependent poly(ADP
Quevedo, C., Soto, M., and Pérez, J.M. (2006) Poly(ADP
ribosyl)ation by human PARP 1, Science, 336, 728-732.
ribose) polymerase 1 (PARP 1) inhibitors in cancer
38.
Langelier, M.F., Eisemann, T., Riccio, A.A., and Pascal, J.M.
chemotherapy, Recent Pat. Anticancer Drug Discov., 1,
(2018) PARP family enzymes: regulation and catalysis of
39-53.
the poly(ADP ribose) posttranslational modification, Curr.
22.
Nilov, D.K., Yashina, K.I., Gushchina, I.V., Zakharenko, A.L.,
Opin. Struct. Biol., 53, 187-198.
Sukhanova, M.V., Lavrik, O.I., and Švedas, V.K. (2018)
39.
Langelier, M.F., Zandarashvili, L., Aguiar, P.M., Black, B.E.,
2,5 Diketopiperazines: a new class of poly(ADP
and Pascal, J.M. (2018) NAD+ analog reveals PARP 1 sub
ribose)polymerase inhibitors, Biochemistry (Moscow), 83,
strate blocking mechanism and allosteric communication
152-158.
from catalytic center to DNA binding domains, Nat.
23.
Frampton, J.E. (2015) Olaparib: a review of its use as
Commun., 9, 844.
maintenance therapy in patients with ovarian cancer,
40.
Sali, A., and Blundell, T.L. (1993) Comparative protein
BioDrugs, 29, 143-150.
modelling by satisfaction of spatial restraints, J. Mol. Biol.,
24.
Mittica, G., Ghisoni, E., Giannone, G., Genta, S.,
234, 779-815.
Aglietta, M., Sapino, A., and Valabrega, G. (2018) PARP
41.
Menke, M., Berger, B., and Cowen, L. (2008) Matt: local
inhibitors in ovarian cancer, Recent Pat. Anticancer Drug
flexibility aids protein multiple structure alignment, PLoS
Discov., 13, 392-410.
Comput. Biol., 4, e10.
БИОХИМИЯ том 85 вып. 1 2020
124
НИЛОВ и др.
42.
Case, D.A., Berryman, J.T., Betz, R.M., Cerutti, D.S.,
57.
Bell, C.E., and Eisenberg, D. (1996) Crystal structure of
Cheatham, T.E. 3rd, et al. (2015) AMBER 2015. University
diphtheria toxin bound to nicotinamide adenine dinu
of California, San Francisco.
cleotide, Biochemistry, 35, 1137-1149.
43.
Salomon Ferrer, R., Case, D.A., and Walker, R.C. (2013)
58.
Parikh, S.L., and Schramm, V.L. (2004) Transition state
An overview of the Amber biomolecular simulation pack
structure for ADP ribosylation of eukaryotic elongation
age, WIREs Comput. Mol. Sci., 3, 198-210.
factor 2 catalyzed by diphtheria toxin, Biochemistry, 43,
44.
Воеводин В.В., Жуматий С.А., Соболев С.И., Антонов А.С.,
1204-1212.
Брызгалов П.А., Никитенко Д.А., Стефанов К.С., Во
59.
Jørgensen, R., Merrill, A.R., Yates, S.P., Marquez, V.E.,
еводин В.В. (2012) Практика суперкомпьютера «Ло
Schwan, A.L., Boesen, T., and Andersen, G.R. (2005)
моносов», Открытые системы, 7, 36-39.
Exotoxin A-eEF2 complex structure indicates ADP ribo
45.
Maier, J.A., Martinez, C., Kasavajhala, K., Wickstrom, L.,
sylation by ribosome mimicry, Nature, 436, 979-984.
Hauser, K.E., and Simmerling, C.
(2015) ff14SB:
60.
Jørgensen, R., Wang, Y., Visschedyk, D., and Merrill, A.R.
Improving the accuracy of protein side chain and backbone
(2008) The nature and character of the transition state for the
parameters from ff99SB, J. Chem. Theory Comput., 11,
ADP ribosyltransferase reaction, EMBO Rep., 9, 802-809.
3696-3713.
61.
Tsuge, H., Nagahama, M., Oda, M., Iwamoto, S.,
46.
Walker, R.C., de Souza, M.M., Mercer, I.P., Gould, I.R.,
Utsunomiya, H., Marquez, V.E., Katunuma, N.,
and Klug, D.R. (2002) Large and fast relaxations inside a
Nishizawa, M., and Sakurai, J. (2008) Structural basis of
protein: calculation and measurement of reorganization
actin recognition and arginine ADP ribosylation by
energies in alcohol dehydrogenase, J. Phys. Chem. B, 106,
Clostridium perfringens iota toxin, Proc. Natl. Acad. Sci.
11658-11665.
USA, 105, 7399-7404.
47.
Pavelites, J.J., Gao, J., Bash, P.A., and MacKerell, A.D. Jr.
62.
Tsurumura, T., Tsumori, Y., Qiu, H., Oda, M., Sakurai, J.,
(1997) A molecular mechanics force field for NAD+,
Nagahama, M., and Tsuge, H. (2013) Arginine ADP ribo
NADH, and the pyrophosphate groups of nucleotides, J.
sylation mechanism based on structural snapshots of
Comput. Chem., 18, 221-239.
iota toxin and actin complex, Proc. Natl. Acad. Sci. USA,
48.
Meagher, K.L., Redman, L.T., and Carlson, H.A. (2003)
110, 4267-4272.
Development of polyphosphate parameters for use with the
63.
Rolli, V., O’Farrell, M., Ménissier de Murcia, J., and de
AMBER force field, J. Comput. Chem., 24, 1016-1025.
Murcia, G. (1997) Random mutagenesis of the poly(ADP
49.
Stroganov, O.V., Novikov, F.N., Stroylov, V.S., Kulkov, V.,
ribose) polymerase catalytic domain reveals amino acids
and Chilov, G.G. (2008) Lead finder: an approach to
involved in polymer branching, Biochemistry,
36,
improve accuracy of protein ligand docking, binding ener
12147-12154.
gy estimation, and virtual screening, J. Chem. Inf. Model.,
64.
Kistemaker, H.A., Overkleeft, H.S., van der Marel, G.A.,
48, 2371-2385.
and Filippov, D.V. (2015) Branching of poly(ADP ribose):
50.
Захаренко А.Л., Суханова М.В., Ходырева С.Н., Но
synthesis of the core motif, Org. Lett., 17, 4328-4331.
виков Ф.Н., Стройлов В.С., Нилов Д.К., Чилов Г.Г.,
65.
Banasik, M., and Ueda, K. (1994) Inhibitors and activators
Швядас В.К., Лаврик О.И. (2011) Усовершенствован
of ADP ribosylation reactions, Mol. Cell. Biochem., 138,
ная процедура поиска потенциальных ингибиторов
185-197.
поли(АДФ рибозо) полимеразы 1 с использованием
66.
Nguewa, P.A., Fuertes, M.A., Cepeda, V., Alonso, C.,
молекулярного докинга, Мол. биология, 45, 565-569.
Quevedo, C., Soto, M., and Pérez, J.M. (2006) Poly(ADP
51.
Humphrey, W., Dalke, A., and Schulten, K. (1996) VMD:
ribose) polymerase 1 inhibitor
3 aminobenzamide
visual molecular dynamics, J. Mol. Graph., 14, 33-38.
enhances apoptosis induction by platinum complexes in
52.
Иванисенко Н.В., Жечев Д.А., Иванисенко В.А.
cisplatin resistant tumor cells, Med. Chem., 2, 47-53.
(2016) Структурное моделирование мод связывания
67.
Zheng, Y.D., Xu, X.Q., Peng, F., Yu, J.Z., and Wu, H.
НАД+ с ПАРП 1, Вавиловский журнал генетики и се"
(2011) The poly(ADP ribose) polymerase 1 inhibitor 3
лекции, 20, 857-862.
aminobenzamide suppresses cell growth and migration,
53.
Bellocchi, D., Costantino, G., Pellicciari, R., Re, N.,
enhancing suppressive effects of cisplatin in osteosarcoma
Marrone, A., and Coletti, C. (2006) Poly(ADP ribose)
cells, Oncol. Rep., 25, 1399-1405.
polymerase catalyzed hydrolysis of NAD+: QM/MM sim
68.
Нилов Д.К., Тараров В.И., Куликов А.В., Захаренко А.Л.,
ulation of the enzyme reaction, ChemMedChem, 1,
Гущина И.В., Михайлов С.Н., Лаврик О.И., Швядас В.К.
533-539.
(2016) Ингибирование поли(ADP рибозо)полимера
54.
Alemasova, E.E., and Lavrik, O.I. (2019) Poly(ADP ribo
зы метаболитом нуклеиновых кислот 7 метилгуани
syl)ation by PARP1: reaction mechanism and regulatory
ном, Acta Naturae, 8, 120-128.
proteins, Nucleic Acids Res., 47, 3811-3827.
69.
Nilov, D., Kirsanov, K., Antoshina, E., Maluchenko, N.,
55.
Yang, S. Y., Fleurat Lessard, P., Hristov, I., and Ziegler, T.
Feofanov, A., Kurgina, T., Zakharenko, A., Khodyreva, S.,
(2004) Free energy profiles for the identity SN2 reactions
Gerasimova, N., Studitsky, V., Lavrik, O., and Švedas, V.
Cl- + CH3Cl and NH3 + H3BNH3: a constraint ab initio
(2018) 7 Methylguanine: a natural DNA repair inhibitor
molecular dynamics study, J. Phys. Chem. A, 108,
and a promising anticancer compound, FEBS Open Bio, 8,
9461-9468.
P.09 198 W.
56.
Kleine, H., Poreba, E., Lesniewicz, K., Hassa, P.O.,
70.
Maluchenko, N., Nilov, D., Feofanov, A., Lys, A.,
Hottiger, M.O., Litchfield, D.W., Shilton, B.H., and
Kutuzov, M., Gerasimova, N., and Studitsky, V. (2019) 7
Lüscher, B. (2008) Substrate assisted catalysis by PARP10
Methylguanine traps PARP 1 on nucleosomes: spFRET
limits its activity to mono ADP ribosylation, Mol. Cell, 32,
microscopy study, Microsc. Microanal.,
25(S2),
57-69.
1282-1283.
БИОХИМИЯ том 85 вып. 1 2020
МОДЕЛИРОВАНИЕ КОМПЛЕКСОВ ПАРП 1
125
MODELING OF THE ENZYME−SUBSTRATE COMPLEXES
OF HUMAN POLY(ADP RIBOSE) POLYMERASE 1*,**
D. K. Nilov1#, S. V. Pushkarev2#, I. V. Gushchina2,
G. A. Manasaryan3, K. I. Kirsanov4, and V. K. Švedas1,2*
1 Belozersky Institute of Physico"Chemical Biology, Lomonosov Moscow State University,
119991 Moscow, Russia; E"mail: vytas@belozersky.msu.ru
2 Lomonosov Moscow State University, Faculty of Bioengineering and Bioinformatics, 119991 Moscow, Russia
3 Lomonosov Moscow State University, Faculty of Fundamental Medicine, 119991 Moscow, Russia
4 Blokhin National Medical Research Center of Oncology, Institute of Carcinogenesis, 115478 Moscow, Russia
Received August 19, 2019
Revised October 16, 2019
Accepted October 16, 2019
Poly(ADP ribose) polymerase 1 (PARP 1) is a key DNA repair enzyme and an important target for cancer treatment.
Conventional methods of studying the PARP 1 reaction mechanism have limitations because of the complex struc
ture of PARP 1 substrates, however, the necessary data can be obtained by molecular modeling. In the present work,
a molecular dynamics model for the PARP 1 enzyme-substrate complex containing NAD+ molecule, and the end
of poly(ADP ribose) chain in the form of ADP molecule was obtained for the first time. Interactions with the active
site residues have been characterized where Gly863, Lys903, Glu988 play a crucial role, and an SN1 like mechanism
for the enzymatic ADP ribosylation reaction has been proposed. Models of PARP 1 complexes with more sophisti
cated two unit fragments of the growing polymer chain as well as with competitive inhibitors 3 aminobenzamide and
7 methylguanine have been obtained by molecular docking.
Keywords: DNA repair, molecular dynamics, docking, substrates, inhibitors
БИОХИМИЯ том 85 вып. 1 2020