БИОХИМИЯ, 2020, том 85, вып. 11, с. 1569 - 1586
УДК 577.21
МЕТИЛИРОВАНИЕ рРНК И УСТОЙЧИВОСТЬ К АНТИБИОТИКАМ
Обзор
© 2020
И.А. Остерман1,2, О.А. Донцова1,2,3, П.В. Сергиев1,2,4*
1 Сколковский институт науки и технологии, Центр наук о жизни, 143028 Сколково, Россия;
электронная почта: petya@genebee.msu.ru
2 Московский государственный университет имени М.В. Ломоносова,
химический факультет, 119991 Москва, Россия
3 Институт биоорганической химии имени акад. М.М. Шемякина
и Ю.А. Овчинникова РАН, 117997 Москва, Россия
4 Московский государственный университет имени М.В. Ломоносова,
Институт функциональной геномики, 119991 Москва, Россия
Поступила в редакцию 12.06.2020
После доработки 16.07.2020
Принята к публикации 16.07.2020
Метилирование нуклеотидных остатков рРНК - один из основных механизмов устойчивости бактерий к
действию ингибиторов синтеза белка. Гены необходимых для этого метилтрансфераз встречаются и у штам%
мов%продуцентов, и у клинических изолятов патогенных бактерий. В ряде случаев, напротив, метилирова%
ние рРНК ферментами «домашнего хозяйства» оказывается необходимым для активности антибиотиков.
Известно как о кооперативности, так и о взаимоисключающем характере модификаций рРНК, так или ина%
че связанных с эффективностью антибиотиков. Интересно понимание эволюционных взаимоотношений
систем модификации рРНК ферментами «домашнего хозяйства» и метилтрансфераз антибиотикорезистент%
ности. Обсуждению всего этого спектра вопросов посвящен данный обзор.
КЛЮЧЕВЫЕ СЛОВА: рибосома, метилирование, антибиотики, трансляция, макролиды, аминогликозиды.
DOI: 10.31857/S0320972520110056
ВВЕДЕНИЕ
ков рРНК, распространенных среди более ши%
роких групп бактерий и даже повсеместно, чье
Один из основных механизмов действия ан%
присутствие, на первый взгляд, не связано с ан%
тибиотиков - нарушение синтеза белка за счет
тибиотиками. Однако среди подобного рода
связывания в функциональных центрах рибосо%
консервативно модифицированных, как прави%
мы - пептидилтрансферазном (ПТЦ) и декоди%
ло, метилированных нуклеотидных остатков,
рующем (ДЦ) центрах и пептидном туннеле
встречаются те, что обеспечивают как умерен%
(ПТ) [1]. В этих же центрах располагается боль%
ный уровень устойчивости, так и, наоборот, оп%
шая часть модифицированных нуклеотидных
ределяют чувствительность к антибиотикам,
остатков рибосомной РНК (рРНК) [2]. Извест%
влияют на приспособленность к устойчивости
но, что метилирование участков связывания -
(так называемые fitness cost) и служат эволюци%
это один из основных механизмов устойчивости
онным резервуаром для возникновения генов
к антибиотикам, действующим на рибосому.
устойчивости. Данный обзор посвящен разным
Подобное метилирование распространено как
аспектам влияния метилирования рРНК на ус%
среди продуцентов этих антибактериальных
тойчивость к антибиотикам.
агентов, так и среди патогенных бактерий, вы%
зывающих инфекционные заболевания челове%
ка и животных. В то же время известно множе%
МЕТИЛИРОВАНИЕ НУКЛЕОТИДНЫХ
ство модифицированных нуклеотидных остат%
ОСТАТКОВ 23S рРНК, НАХОДЯЩИХСЯ
В ПЕПТИДНОМ ТУННЕЛЕ
Принятые сокращения: ДЦ
- декодирующий
центр; ПТЦ - пептидилтрансферазный центр; ПТ - пеп%
Пептидный туннель, через который проходят
тидный туннель; рРНК - рибосомная РНК.
синтезируемые рибосомой белки, пронизывает
* Адресат для корреспонденции.
большую субчастицу рибосомы от ПТЦ до про%
1569
1570
ОСТЕРМАН и др.
тивоположной, так называемой цитоплазмати%
рРНК располагается в ПТ на небольшом рассто%
ческой, стороны. С туннелем взаимодействуют
янии от ПТЦ и контактирует с антибиотиками
как регуляторные пептидные последовательнос%
этих трех классов. Диметилирование (а в случае
ти, так и несколько классов антибиотиков [3].
линкозамидов - монометилирование) при помо%
Самым известным, а также наиболее значимым с
щи TlrD и нескольких родственных ферментов
точки зрения практической медицины случаем,
[5] нарушает связывание антибиотиков за счет
когда метилирование вызывает устойчивость к
неспособности образовать водородную связь с
антибиотикам, является диметилирование рас%
A2058 и перекрывания с метильной группой
положенного в ПТ нуклеотидого остатка А2058
(рис. 1, в). Этот механизм устойчивости широко
23S рРНК (здесь и далее нумерация нуклеотид%
распространен среди продуцентов макролидов и
ных остатков рРНК Escherichia сoli) по N6 атому
патогенных штаммов, для борьбы с которыми
метилтрансферазами семейства Erm [4] (рис. 1,
используются эти антибиотики [6]. Предполага%
а-в; таблица). Эта модификация вызывает ус%
ется, что источником распространения генов се%
тойчивость к большой группе антибиотиков -
мейства erm среди бактерий, в том числе патоген%
макролидам, линкозамидам и стрептограмминам
ных, был горизонтальный перенос генов от
Б класса [4, 5]. Нуклеотидный остаток А2058 23S
штаммов%продуцентов [7]. В то же время остает%
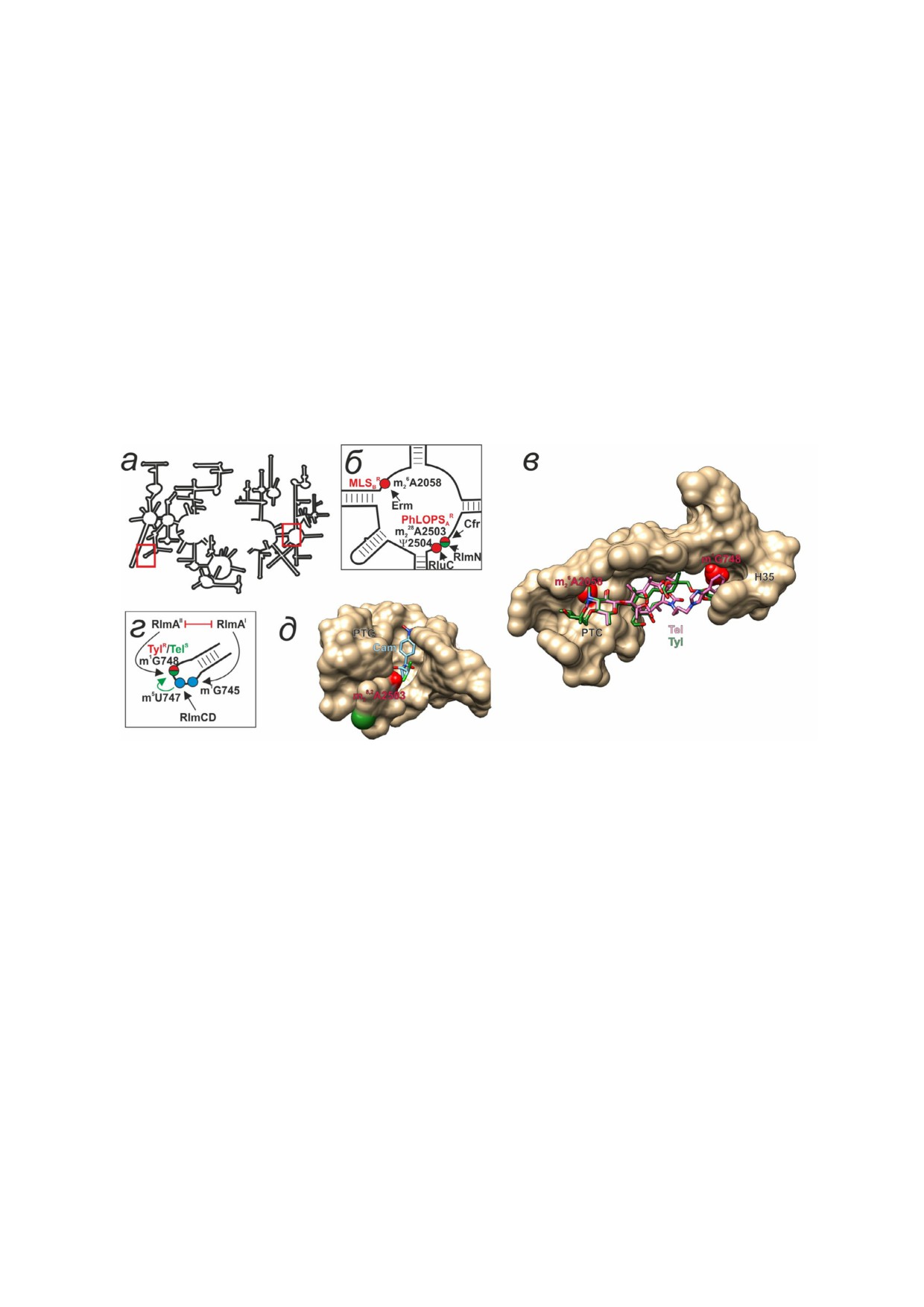
Рис. 1. Влияние модифицированных нуклеотидных остатков пептидного туннеля и ПТЦ на взаимодействие с антибиоти%
ками. а - Схема вторичной структуры 23S рРНК. Красными прямоугольниками обозначены области, рассмотрению ко%
торых посвящены другие панели рисунка. б - Вторичная структура участка V домена 23S рРНК, образующего часть ПТЦ
и ПТ. Модифицированные нуклеотидные остатки, обсуждаемые в тексте, обозначены цветными кружками и подписаны.
Красным выделены модификации устойчивости к антибиотикам (MLSBR - устойчивость к макролидам, линкозамидам и
стрептограмминам Б; PhLOPSAR - устойчивость к фениколам, линкозамидам, оксазалидинонам, плевромутилинам и
стрептограмминам А), зеленым - модификация, способствующая связыванию антибиотиков. Стрелками показана актив%
ность модифицирующих ферментов (подписаны), направленная на соответствующие нуклеотидные остатки. в - Прост%
ранственная структура участка рибосомы, образующего ПТ (показана в виде молекулярной поверхности), и связанных с
рибосомой антибиотиков тилозина (показан зеленой проволочной моделью и подписан) и телитромицина (показан ро%
зовой проволочной моделью и подписан). Красными Ван%дер%Ваальсовыми сферами показаны метильные группы нуклео%
тидных остатков m1G748 и m26A2058 23S рРНК, перекрывающиеся с участком связывания тилозина и, в случае m26A2058,
телитромицина. Подписаны некоторые элементы вторичной структуры 23S рРНК. г - Вторичная структура спирали 35
23S рРНК, образующей часть ПТ. Модифицированные нуклеотидные остатки, обсуждаемые в тексте, обозначены цвет%
ными кружками и подписаны. Красным и зеленым выделена и подписана модификация, определяющая повышенную ус%
тойчивость к тилозину и чувствительность к телитромицину. Синими кружками показаны модификации ферментами
«домашнего хозяйства». Черными стрелками показана активность модифицирующих ферментов (подписаны), направ%
ленная на соответствующие нуклеотидные остатки. Зеленая стрелка обозначает стимулирующее влияние одной модифи%
кации на другую, а красная линия, заканчивающаяся чертой - взаимоисключающие модификации. д - Пространствен%
ная структура участка рибосомы, образующего ПТЦ (обозначен PTC) и связанный с рибосомой антибиотик хлорамфени%
кол (показан голубой проволочной моделью и подписан). Красными и зелеными Ван%дер%Ваальсовыми сферами показа%
ны метильные группы, присоединенные к С2 и С8 атомам нуклеотидного остатка m28,2A2503 23S рРНК, препятствующие
и способствующие связыванию хлорамфеникола и ряда других антибиотиков соответственно. При создании рисунка,
включая наложение пространственных структур друг на друга и моделирование модифицированных нуклеотидных остат%
ков, использовалась программа UCSF Chimera [10]. В создании рисунка использовались вторичные структуры рРНК [11],
а также пространственные структуры 1K9M [12], 4V7Z [13], 6ND5 [14]. (С цветными вариантами рис. 1-4 можно
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1571
ся открытым вопрос о происхождении предко%
[23], при этом модификации А2058 или G748 по
вых генов erm в самих штаммах%продуцентах.
отдельности недостаточно для обеспечения ус%
Поиск аминокислотных последовательностей,
тойчивости. Формирование m1G748 23S рРНК
подобных белкам Erm, среди бактерий [8] одно%
осуществляет метилтрансфераза RlmAII (TlrD),
значно приводит к идентификации универсаль%
которая встречается у многих грамположитель%
но консервативной 16S рРНК метилтрансферазы
ных бактерий, у грамотрицательных обычно
RsmA(KsgA), как наиболее эволюционно близ%
присутствует ее гомолог RlmAI, модифицирую%
кой. Более того, специфичность RsmA(KsgA) мо%
щий расположенный рядом нуклеотидный оста%
жет быть изменена на способность модифициро%
ток G745 23S рРНК по N1 положению [24]
вать субстрат Erm всего лишь с помощью замены
(рис. 1, г; таблица) и относящийся к ферментам
двух петель [9]. Можно предположить, что
«домашнего хозяйства». Возможно, эти метил%
m26A1518/19 16S рРНК метилтрансфераза, также
трансферазы происходят от общего предка. Экс%
осуществляющая диметилирование остатков
прессия гена метилтрансферазы rlmAII Bacillus
аденозина по экзоциклическому N6 атому, была
subtilis регулируется за счёт аттенюации тран%
предковым ферментом для эволюции устойчи%
скрипции и трансляции, по всей видимости, что%
вости метилтрансфераз семейства Erm.
бы минимизировать вредное влияние метилиро%
Почему же, в отличие от RsmA(KsgA), гены
вания на приспособленность в отсутствие анти%
Erm%метилтрансфераз не распространились сре%
биотика [25], что делает спорным отнесение подоб%
ди других таксономических групп организмов?
ных ферментов к генам «домашнего хозяйства».
По%видимому, дело во вредном влиянии моди%
Любопытно, что для повышения устойчи%
фикации m26A2058 23S рРНК на приспособлен%
вости (MIC) Streptococcus pneumoniae с дважды
ность клеток бактерий в отсутствие антибиоти%
метилированным А2058 к синтетическому про%
ков. Негативный эффект от метилирования, су%
изводному эритромицина, телитромицину с 2 до
дя по всему, связан не с повышенными затрата%
32 мкг/мл нужна не экспрессия, как в случае с
ми на синтез метилтрансферазы, а с тем, что мо%
тилозином, а инактивация гена rlmAII и соответ%
дифицированные рибосомы синтезируют белки
ственно отсутствие метилирования G748 23S
в неоптимальных пропорциях за счет взаимо%
рРНК [26]. Эта особенность объясняется, по%
действия модифицированного А2058 с амино%
видимому, тем, что участок связывания телитро%
кислотными остатками растущего белка внутри
мицина не перекрывается с расположением ме%
ПТ [15]. Клетки, имеющие постоянно активную
тильной группы, а наоборот, расположен так,
метилтрансферазу ErmC и соответственно диме%
чтобы было возможно взаимодействие между
тилированный А2058, проигрывают конкурен%
модифицированным нуклеотидом и антибиоти%
цию тем, у которых модификации нет [15].
ком (рис. 1, в). Нуклеотидный остаток m5U747
Именно этим, по%видимому, объясняется подав%
23S рРНК, образующийся посредством консти%
ление экспрессии генов erm в отсутствие макро%
тутивной метилтрансферазы RlmCD [27] (рис. 1,
лидов не только у большинства устойчивых
г) и соседствующий с G748, способствует узна%
штаммов, но даже у штамма%продуцента [16].
ванию последнего метилтрансферазой RlmAII и,
Наиболее распространенный механизм регуля%
соответственно, косвенно способствует связы%
ции экспрессии генов erm связан с торможением
ванию телитромицина [28].
рибосомы на короткой рамке считывания, рас%
Итак, на примере макролидов мы видим, что
положенной перед геном метилтрансферазы
метилирование рРНК может приводить к устой%
[17-19], из%за чего инициаторный участок гена
чивости к антибиотикам за счёт перекрывания
устойчивости становится доступным для связы%
метильных групп с участком связывания анти%
вания с другой рибосомой. Альтернативными
биотика, а также к чувствительности к антибио%
регуляторными механизмами, позволяющими
тикам за счёт взаимодействия метильной группы
активировать гены семейства erm в присутствии
с антибиотиком. Также на данном примере мож%
антибиотиков, являются аттенюация транскрип%
но иллюстрировать взаимоисключающее фило%
ции [20], использование специфичного тран%
генетическое распространение рРНК метил%
скрипционного фактора [21] или индуцируемый
трансфераз (rlmAI и rlmAII) и кооперативность в
кетолидами сдвиг рамки считывания [22].
процессе модификации (RlmCD и RlmAII).
Метилирование нуклеотидого остатка G748
23S рРНК, также расположенного в туннеле
большой субчастицы рибосомы (рис. 1, а, в и г)
МЕТИЛИРОВАНИЕ НУКЛЕОТИДНЫХ
по N1 атому, в комбинации с монометилирова%
ОСТАТКОВ 23S рРНК, НАХОДЯЩИХСЯ В ПТЦ
нием А2058 обеспечивает устойчивость бакте%
рии%продуцента к 16%членному макролиду с объ%
Пептидилтрансферазный центр - это глав%
емными сахарными заместителями - тилозину
ный ферментативный центр рибосомы, функ%
БИОХИМИЯ том 85 вып. 11 2020
1572
ОСТЕРМАН и др.
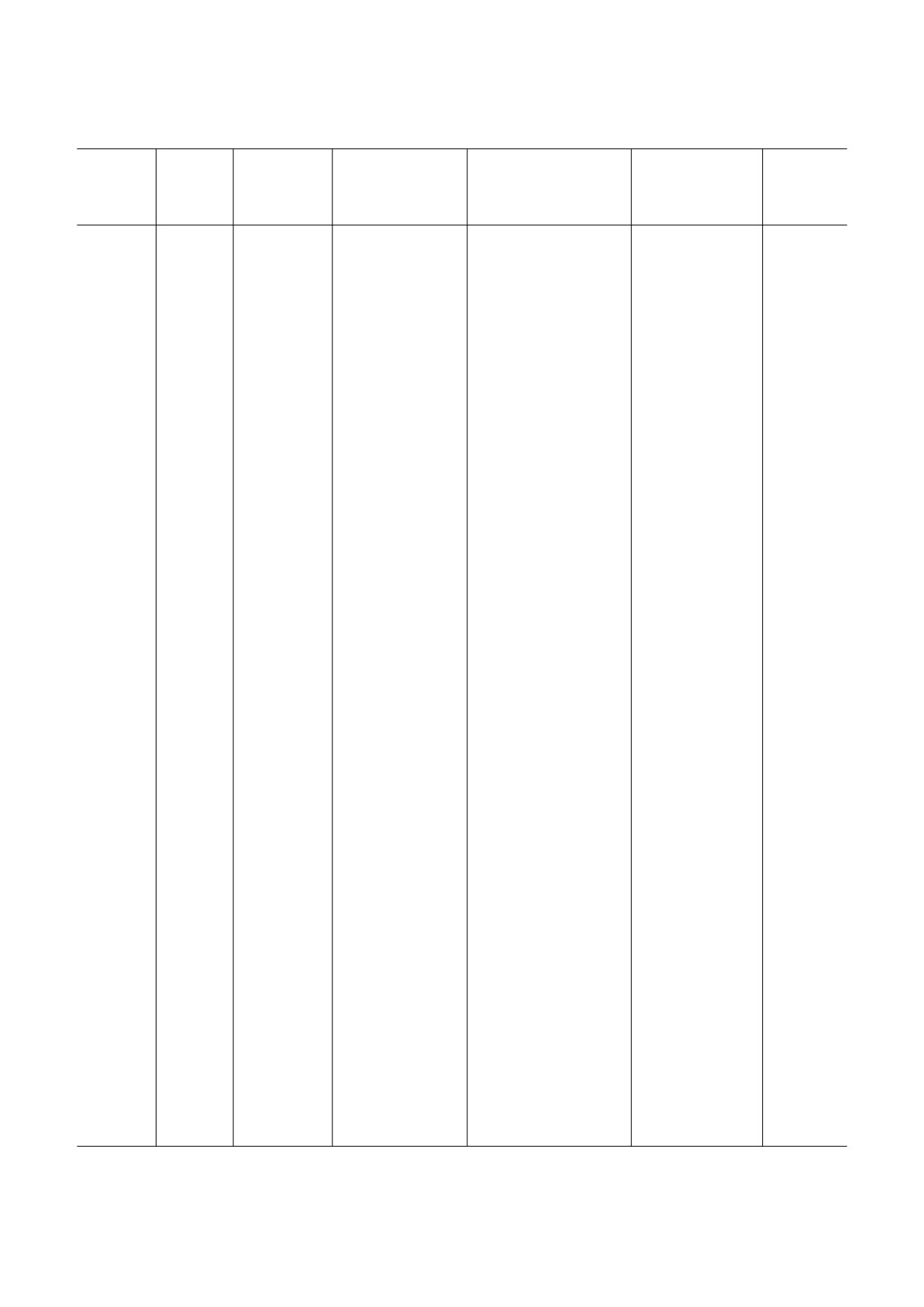
рРНК метилтрансферазы, активность которых влияет на устойчивость к антибиотикам
Отношение метилиро%
Механизм влияния
Предполо%
ID
Название
Продукт
Встречаемость
вания к антибиотико%
на активность
жительный
резистентности
антибиотика
предковый
фермент*
P36999
RlmAI
m1G745
грамотрицательные
чувствительность: вио%
предположительно
RlmAII**
бактерии
мицин (умеренно)
конформационное
изменение
Q9S1M6
RlmAII
m1G748
продуцент тилози%
устойчивость: тилозин
непосредственное
RlmAI**
на Streptomyces rose?
чувствительность: те%
стерическое пре%
oflavus, другие
литромицин
пятствие; гидро%
грамположитель%
фобный контакт
ные бактерии
P18644
TsnR
Am1067
продуцент тио%
устойчивость: тиостреп%
непосредственное
TrmH,
стрептона, Strepto?
тон
стерическое пре%
RlmB
myces azureus
пятствие
P9WJ63
TlyA
Cm1920 (а
патоген Mycobac?
чувствительность: кап%
предположительно
также Cm1409
terium tuberculosis и
реомицин, виомицин
гидрофобный кон%
16S рРНК)
некоторые другие
такт или конфор%
бактерии
мационное изме%
нение
P13956
Erm
m26A2058 23S
множество проду%
устойчивость: макроли%
непосредственное
RsmA(KsgA)
семейство
рРНК
центов и патогенов
ды, линкозамиды,
стерическое пре%
стрептограммины Б
пятствие
P97178
TlrD
m6A2058 23S
продуцент тилози%
устойчивость: линкоза%
непосредственное
RsmA(KsgA)
рРНК
на Streptomyces rose?
миды, макролиды (уме%
стерическое пре%
oflavus, другие про%
ренно)
пятствие
дуценты
Q9FBG4
Cfr
m8A2503
патоген Staphylo?
устойчивость: фенико%
непосредственное
RlmN
coccus sciuri и дру%
лы, линкозамиды, окса%
стерическое пре%
гие
залидиноны, плеврому%
пятствие
тилины, стрептограм%
мины А
P36979
RlmN
m2A2503
у всех бактерий
чувствительность: лине%
предположительно
залид, спарсомицин,
конформационное
тиамулин (умеренно)
изменение
Q9F5K6
AviRb
Um2479
продуцент авила%
устойчивость: авилами%
непосредственное
TrmH,
мицина
цин
стерическое пре%
RlmB
Streptomyces viri?
пятствие
dochromogenes
Q93CQ2
EmtA
mG2470
патоген Enterococ?
устойчивость: вилами%
непосредственное
THUMPD3,
cus faecium и другие
цин, эверниамицин
стерическое пре%
RlmKL,
пятствие
Trm14
Q9F5K5
AviRa
mG2535
продуцент авила%
устойчивость: авилами%
непосредственное
мицина, Strepto?
цин (умеренно)
стерическое пре%
myces viridochromo?
пятствие
genes
P0A6U5
RsmG
m7G527
у всех бактерий
чувствительность:
неизвестно
стрептомицин (умерен%
но)
неизвестно
Pct
m1A964
продуцент пакта%
устойчивость: пактами%
непосредственное
мицина Strepto?
цин
стерическое пре%
myces pactum
пятствие
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1573
Окончание таблицы
Отношение метилиро%
Механизм влияния
Предполо%
ID
Название
Продукт
Встречаемость
вания к антибиотико%
на активность
жительный
резистентности
антибиотика
предковый
фермент*
P0ADX9
RsmD
m2G966
у всех бактерий
устойчивость: тетрацик%
неизвестно
лин (умеренно)
чувствительность: тиге%
циклин (умеренно)
E9KIK3
EfmM
m5C1404
патоген E. faecium
устойчивость: канами%
RsmF
цин, тобрамицин (уме%
ренно)
Q6F5A0
ArmA
m7G1405
патоген Klebsiella
устойчивость: амино%
непосредственное
pneumoniae и другие
гликозиды с 4%6 заме%
стерическое пре%
щенным дезоксистреп%
пятствие
тамином
Q33DX5
RmtA%H
m7G1405
патоген Pseudo?
устойчивость: амино%
непосредственное
monas aeruginosa и
гликозиды с 4%6 заме%
стерическое пре%
другие
щенным дезоксистреп%
пятствие
тамином
Q53316
KgmB и
m7G1405
продуцент небра%
устойчивость: амино%
непосредственное
другие
мицина Strepto?
гликозиды с 4%6 заме%
стерическое пре%
myces tenebrarius и
щенным дезоксистреп%
пятствие
другие
тамином
A8C927
NpmA
m1A1408
патоген Escherichia
устойчивость: амино%
непосредственное
TrmB
coli
гликозиды с 4%6 и 4%5
стерическое пре%
замещенным дезокси%
пятствие
стрептамином
P25920
KamB
m1A1408
Продуцент тобра%
устойчивость: амино%
непосредственное
TrmB
мицина S. tenebrar?
гликозиды с 4%6 и 4%5
стерическое пре%
ius
замещенным дезокси%
пятствие
стрептамином
A6YEH1
CmnU
m1A1408
продуцент капрео%
устойчивость: капрео%
непосредственное
TrmB
мицина Strepto?
мицин
стерическое пре%
myces capreolus
пятствие
P9WJ63
TlyA
Cm1409 (а
патоген Mycobac?
чувствительность: кап%
предположительно
также Cm1920
terium tuberculosis и
реомицин, виомицин
гидрофобный кон%
23S рРНК)
некоторые другие
такт или конфор%
бактерии
мационное изме%
нение
P0AGL7
RsmE
m3U1498
у всех бактерий
чувствительность: ами%
предположительно
ногликозиды с 4%6 и 4%5
конформационное
замещенным дезокси%
изменение
стрептамином (умеренно)
P06992
RsmA
m26A1518/19
у всех организмов
чувствительность: касу%
конформационное
(KsgA)
гамицин
изменение
* Сходство аминокислотных последовательностей определяли с помощью программы Blast [8].
** RlmAI и RlmAII являются паралогами, присутствующими у грамотрицательных и грамположительных бактерий
соответственно.
ция которого заключается в полимеризации
зяйства» [2], а также нуклеотидные остатки, мо%
присоединенных к тРНК аминокислот. ПТЦ со%
дификация которых влияет на устойчивость к
держит множество нуклеотидных остатков, мо%
антибиотикам. Так, модификация нуклеотидно%
дифицированных ферментами «домашнего хо%
го остатка A2503 23S рРНК (рис. 1, б и д) блоки%
БИОХИМИЯ том 85 вып. 11 2020
1574
ОСТЕРМАН и др.
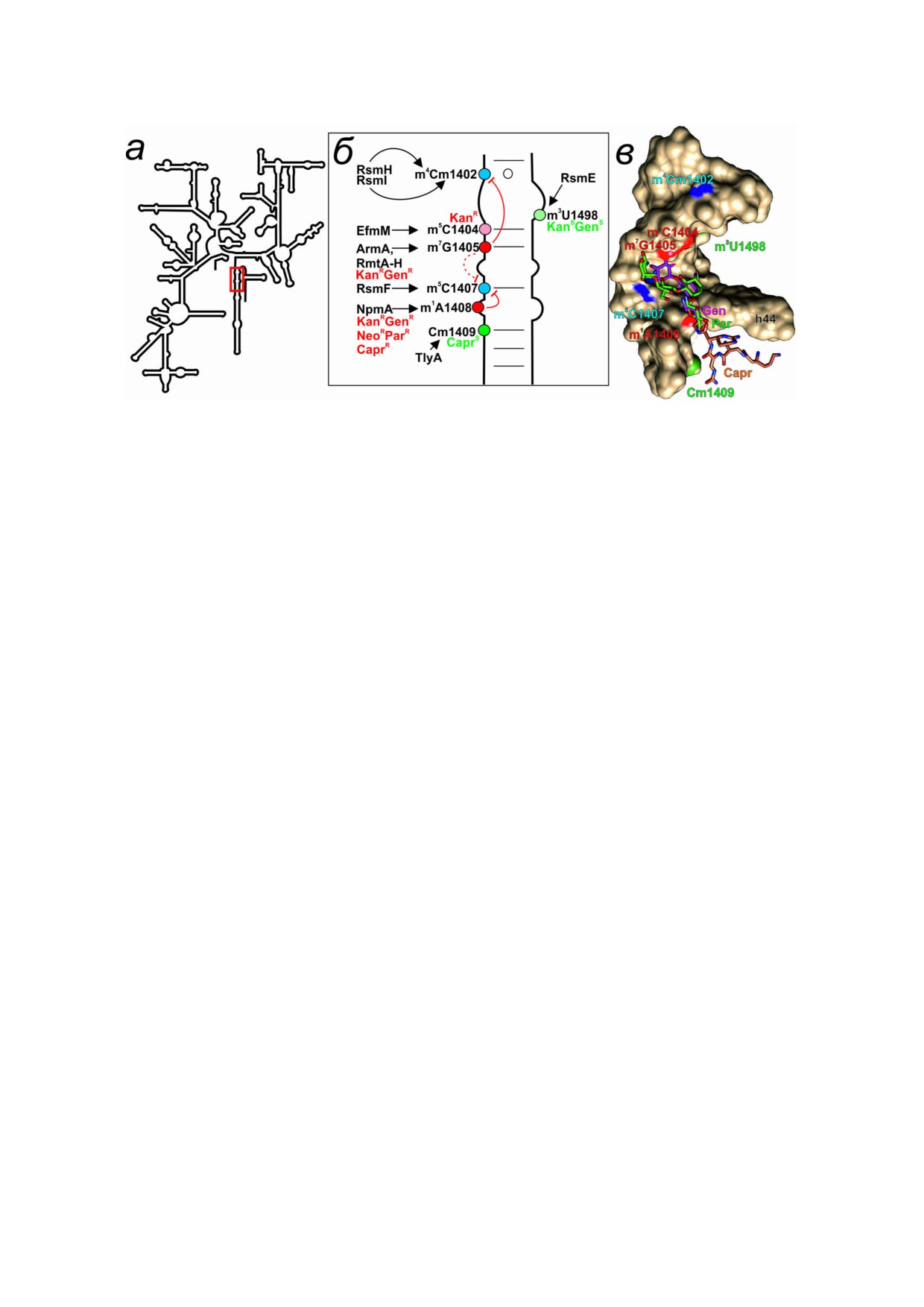
Рис. 2. Влияние модифицированных нуклеотидных остатков ДЦ 16S рРНК на взаимодействие с антибиотиками. а - Схе%
ма вторичной структуры 16S рРНК. Красным прямоугольником обозначена область, рассмотрению которой посвящены
другие панели рисунка. б - Вторичная структура участка спирали 44 16S рРНК, образующей часть ДЦ. Модифицирован%
ные нуклеотидные остатки, обсуждаемые в тексте обозначены цветными кружками и подписаны. Красным выделены мо%
дификации устойчивости к антибиотикам (KanRGenR - устойчивость к канамицину и гентамицину, аминогликозидам,
содержащим 4%6 замещенный остаток дезоксистрептамина; NeoRParR - устойчивость к неомицину и паромомицину, ами%
ногликозидам, содержащим 4%5 замещенный остаток дезоксистрептамина; CaprR - устойчивость к капреомицину), а зе%
леным - модификации, способствующие связыванию антибиотиков (подписи соответствуют антибиотикам). Синими
кружками показаны модификации ферментами «домашнего хозяйства». Стрелками показана активность модифицирую%
щих ферментов (подписаны), направленная на соответствующие нуклеотидные остатки. Красные линии, заканчивающа%
яся чертой - ингибирующее влияние модификаций друг на друга. Пунктирная красная черта - влияние, поставленное
под сомнение. в - Пространственная структура участка спирали 44 16S рРНК, образующей часть ДЦ (показана в виде мо%
лекулярной поверхности), и связанных с рибосомой антибиотиков гентамицина (4%6 замещенный аминогликозид, пока%
зан розовой проволочной моделью и подписан), паромомицина (4%5 замещенный аминогликозид, показан зеленой про%
волочной моделью и подписан) и капреомицина (показан оранжевой проволочной моделью и подписан). Красными, зе%
леными и синими Ван%дер%Ваальсовыми сферами показаны метильные группы нуклеотидных остатков, препятствую%
щих, способствующих, а также не влияющих на связывание антибиотиков. При создании рисунка, включая наложение
пространственных структур друг на друга и моделирование модифицированных нуклеотидных остатков, использовалась
программа UCSF Chimera [10]. В создании рисунка использовались вторичные структуры рРНК [11], а также простран%
ственные структуры 4V53 [51], 4V7M [52] и 4V51 [53]
рует связывание сразу нескольких классов анти%
эритромицином, что вызывает устойчивость
биотиков. Метилирование по С8 атому метил%
сразу к 7 классам антибиотиков [31].
трансферазой Cfr делает клетки устойчивыми к
Кроме Cfr, на нуклеотидный остаток A2503
действию фениколов, линкозамидов, оксазали%
23S рРНК также действует повсеместно распро%
динонов, плевромутилинов и стрептрограмминов
страненная метилтрансфераза «домашнего хо%
класса А [29]. Удивительно, что несмотря на ши%
зяйства» RlmN, присоединяющая метильную
рокое распространение гена cfr среди грамполо%
группу к атому С2 [32]. В отсутствие данного ме%
жительных и грамотрицательных бактерий, он
тилирования возникает не чувствительность, а
не встречается у продуцентов антибиотиков. Эта
небольшой уровень устойчивости к линезолиду
модификация не стала постоянной у всех бакте%
[33]. Тем не менее приобретение умеренной ус%
рий. Логично было бы предположить, что она
тойчивости к антибиотикам в отсутствие этого
оказывает отрицательный эффект на жизнедея%
метилирования, по%видимому, не перевешивает
тельность, однако на данный момент получены
его пользы для функционирования рибосом.
данные, свидетельствующие об очень неболь%
Клетки, лишенные метилтрансферазы RlmN, не
шом замедлении роста бактерий при экспрес%
способны поддерживать высокий уровень
сии cfr [30]. Экспрессия гена cfr изучена не на%
экспрессии рекомбинантных белков, а также
столько подробно, как генов erm, но в одном из
демонстрируют умеренное увеличение количе%
обнаруженных патогенных штаммов Staphylo?
ства интермедиатов сборки рибосомных субчас%
coccus aureus рамке cfr предшествует небольшая,
тиц в случае культивирования при 20 °С [34].
возможно, регуляторная рамка считывания. В
Любопытно, что в клетках, лишенных модифи%
другом случае гену cfr предшествует ген ermB, а
кации А2503 по С2 положению слабее происхо%
промотор их общего оперона индуцируется
дит индукция экспрессии гена метилтрансфера%
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1575
зы ermC, что указывает на возможное взаимо%
малой субчастице и образован, по большей час%
действие этого модифицированного нуклеотид%
ти, спиралью 44 16S рРНК. Множество антибио%
ного остатка с растущим пептидом и его участие
тиков, в основном принадлежащих классу ами%
в регуляции синтеза белка [35].
ногликозидов, взаимодействует с этим участком
Эволюционное происхождение гена cfr вы%
рибосомы и вызывает ошибки декодирования
зывает споры. Так, поиск родственных последо%
[44], а также препятствует транслокации [45].
вательностей позволяет говорить о родстве Cfr и
Устойчивость к аминогликозидам может воз%
RlmN, причем Cfr, как предполагается, произо%
никнуть за счет метилирования нуклеотидных
шел от RlmN, возможно, по эволюционной тра%
остатков ДЦ G1405 16S рРНК по N7 атому и
ектории, проходящей через гены РНК метил%
A1408 16S рРНК по атому N1 [46] (рис. 2; табли%
трансфераз пластид [36]. В пользу общности
ца). Ферменты, способные осуществлять эти
происхождения этих ферментов говорит как то,
модификации, встречаются и у штаммов%проду%
что они модифицируют один и тот же нуклео%
центов аминогликозидов, и у патогенных бакте%
тидный остаток, так и то, что они имеют одина%
рий, куда они попали, скорее всего, в результате
ковый и довольно малораспространенный ради%
горизонтального переноса генов
[46]. Так,
кальный механизм метилирования [37, 38]. Тем
m7G1405 образуется в рибосомах продуцентов
не менее в других работах [39, 40] высказывает%
аминогликозидов, содержащих 4%6 замещенный
ся идея, что Cfr произошла от метилтрансфера%
дезоксистрептамин, при помощи большого се%
зы клостридий, родственной RlmN, однако
мейства сходных по структуре метилтрансфераз
имеющей другой субстрат, нежели A2503 23S
KgmB, Sgm, FmrO, GrmA, GrmB, GrmO, Kmr,
рРНК. Учитывая, что RlmN проявляет двойную
NbrB и Srm1 [47]. Весьма широко распростране%
специфичность, будучи способной метилиро%
ны N7 метилтрансферазы ArmA и RmtA%H, при%
вать тРНК [41], можно предположить, что
водящие к образованию нуклеотидного остатка
родственные RlmN и Cfr метилтрансферазы
m7G1405 16S рРНК у патогенных бактерий [48]
клостридий могут иметь не рРНК, а исключи%
и вызывающие устойчивость к аминогликози%
тельно тРНК субстрат [39].
дам, содержащим 4%6 замещенный дезокси%
Только одна из довольно широко распрост%
стрептамин, таким как канамицин и гентами%
раненных модификаций нуклеотидных остат%
цин (рис. 2, б и в; см. расположение гентамици%
ков ПТЦ приводит к повышению устойчивости
на на панели в, показан розовым, и нуклеотид%
к антибиотикам, т.е. является своего рода конс%
ного остатка m7G1405 16S рРНК) [46]. Модифи%
титутивной модификацией устойчивости. Это
кация m1A1408 16S рРНК приводит к устойчи%
псевдоуридинилирование нуклеотидного остат%
вости к более широкому спектру аминогликози%
ка U2504 при помощи фермента RluC [42], кото%
дов, а именно содержащих как 4%6, так и 4%5 за%
рое повышает устойчивость к действию тиаму%
мещенный дезоксистрептамин, таким как нео%
лина, клиндамицина и линезолида и, в меньшей
мицин и паромомицин (рис. 2, б и в; сравните
степени, к действию хлорамфеникола, гигроми%
расположение гентамицина и паромомицина на
цина А и спарсомицина [43]. Несмотря на то что
панели в, показаны розовым и зеленым соответ%
RluC превращает сразу три уридина в псевдо%
ственно, и нуклеотидного остатка m1A1408 16S
уридины (955, 2504 и 2580), именно Ψ2504
рРНК). Эти метилтрансферазы встречаются у
(рис. 1, б) повышает устойчивость к перечислен%
продуцентов аминогликозидов (KamA, KamB и
ным ингибиторам пептидилтрансферазной ре%
KamC) и не родственного аминогликозидам
акции.
противотуберкулезного антибиотика капреоми%
Суммируя данный раздел, можно говорить
цина (CmnU) [47]. Долгое время считалось, что
как об эволюционировании рРНК метилтранс%
у патогенных бактерий такого рода устойчивос%
феразы устойчивости (Cfr) из конститутивной
ти нет, однако в 2007 г. у патогенной Escherichia
рРНК/тРНК метилтрансферазы (RlmN), так и о
coli была обнаружена подобная N1 A1408 метил%
возможном превращении псевдоуридинсинтазы
трансфераза NpmA [49]. Метилтрансферазы,
устойчивости в конститутивный модифицирую%
модифицирующие N1 атом нуклеотидного ос%
щий фермент «домашнего хозяйства» (RluC).
татка A1408, по%видимому, родственны между
собой, как можно понять из сходства их амино%
кислотных последовательностей. Эволюцион%
МЕТИЛИРОВАНИЕ НУКЛЕОТИДНЫХ
ное происхождение всего этого семейства фер%
ОСТАТКОВ 16S рРНК,
ментов не вполне ясно. Возможно, оно про%
НАХОДЯЩИХСЯ В ДЦ
изошло от тРНК%специфичной гуанин N7 метил%
трансферазы TrmB [50].
Второй важнейший функциональный центр
Еще один механизм устойчивости к амино%
рибосомы - декодирующий. Он расположен на
гликозидам (канамицину и тобрамицину, но не
БИОХИМИЯ том 85 вып. 11 2020
1576
ОСТЕРМАН и др.
гентамицину) связан с метилированием нуклео%
на панели в, показан оранжевым, и нуклеотид%
тидного остатка С1404 16S рРНК по С5 атому
ного остатка Cm1409 16S рРНК), повышенная
[54], осуществляемым метилтрансферазой
чувствительность к этому антибиотику рибосом,
EfmM, обнаруженной у патогенного штамма
модифицированных TlyA, может объясняться
Enterococcus faecium. Этот белок похож на метил%
изменением подвижности субчастиц друг отно%
трансферазу RsmF, от которой, возможно, про%
сительно друга во время транслокации, так как
исходит. Любопытно, что у Thermus thermophilus
туберактиномицины действуют именно на эту
метилтрансфераза RsmF модифицирует сразу
стадию работы рибосомы [60]. В ходе исследова%
три нуклеотидных остатка C1400, C1404 и C1407
ния ортологов гена tlyA оказалось, что есть два
[55], свидетельствуя о достаточно легком эволю%
варианта этого белка - TlyAI, который модифи%
ционировании субстратной специфичности по%
цирует только С1920 23S рРНК и TlyAII, метили%
добных ферментов. Модификация m5С1404 рас%
рующий нуклеотидные остатки обеих субчас%
полагается в непосредственной близости от сай%
тиц. Экспрессия гена, кодирующего TlyAII в
та связывания канамицина [56], однако не пе%
клетках E. coli, делает их более чувствительными
рекрывается с ним, исключая стерическое пре%
к туберактиномицинам, но при этом не дает ни%
пятствие, как механизм устойчивости. Об этом
какого преимущества при культивировании в
говорит также небольшая, 12%кратная, в случае
отсутствие антибиотика [61]. В чем причина на%
экспрессии efmM в E. coli, разница в минималь%
личия гена tlyA у патогенных бактерий, если он
ной ингибирующей концентрации в присут%
приводит к увеличению чувствительности к ан%
ствии и в отсутствие антибиотика. Удивительно,
тибиотикам? Возможно, белок TlyA обладает
что связывание гентамицина, имеющего более
дополнительной функцией, кроме модифика%
объемную метиламиногруппу вместо незаме%
ции рРНК. Было показано, что рекомбинант%
щенной аминогруппы канамицина в месте кон%
ный белок TlyA проявляет гемолитическую ак%
такта с метильной группой m5C1404, не наруша%
тивность in vitro, а при экспрессии его гена в
ется в результате этого метилирования 16S
E. coli присутствие этого белка было детектиро%
рРНК. В научной литературе устойчивость к ка%
вано в мембране клетки [62]. На примере C. jeju?
намицину и тобрамицину, вызванную метили%
ni было показано, что наличие гена tlyA обуслов%
рованием m5С1404 [54], связывают с вытеснени%
ливает вирулентность, причем мутации, подав%
ем воды, взаимодействующей с антибиотиком.
ляющие ферментативную активность TlyA, ока%
В случае гентамицина, наоборот, образование
зывают такой же эффект, как и удаление гена
m5C1404 16S рРНК приводит к возникновению
целиком. Значит именно метилирование вносит
дополнительного гидрофобного контакта
вклад в патогенность [58]. Несмотря на близость
(рис. 2, б и в; см. расположение гентамицина на
нуклеотидных остатков%мишеней TlyA к сайту
панели в, показан розовым, и нуклеотидного ос%
связывания аминогликозидов, наличие или от%
татка m5C1404 16S рРНК).
сутствие этой метилтрансферазы никак не ска%
Гидрофобным контактом может объясняться
зывается на эффективности действия аминогли%
и взаимодействие капреомицина, антибиотика,
козида амикацина. В то же время мутация рас%
используемого для борьбы с туберкулезом, с 2′%
положенного рядом с сайтом метилирования
OMe%группой Сm1409 [57]. У некоторых пато%
нуклеотидного остатка A1408G обеспечивает ус%
генных бактерий, включая Mycobacterium sp.,
тойчивость и к амикацину, и к капреомицину
Campylobacter jejuni и Brachyspira (Serpulina) hyo?
[63]. Интересно, что инактивация гена tlyA
dysenteriae, была обнаружена метилтрансфераза
практически не сказывается на скорости роста
TlyA, отвечающая за модификацию нуклеотид%
бактерий без мутаций в рРНК, в то время как
ных остатков С1409 16S и C1920 23S рРНК по 2′%
экспрессия tlyA повышает приспособленность
OH%группе [58, 59]. Делеция или мутация гена
бактерий с мутацией A1408G, что является весь%
tlyA у Mycobacterium sp. приводила к появлению
ма необычным типом взаимодействия метили%
устойчивости к капреомицину. При образова%
рования и антибиотикорезистентности [63]. В
нии мостика B2a между рибосомными субчасти%
данном случае можно говорить о том, что мети%
цами нуклеотидные остатки C1409 и C1920 ока%
лирование понижает фенотипическую стои%
зываются сближенными. Структурные исследо%
мость устойчивости к антибиотикам, вызванной
вания показали, что здесь же располагаются и
мутацией гена рРНК.
туберактиномицины - виомицин и капреоми%
У продуцента капреомицина Saccharothrix
цин [52]. Метилирование обеих субчастиц дела%
mutabilis subsp. Capreolus был обнаружен ген,
ет рибосому более чувствительной к этим анти%
обеспечивающий защиту от собственного ток%
биотикам [59]. Кроме предположительного гид%
сического продукта, cmnU, кодирующий гомо%
рофобного контакта Сm1409 с капреомицином
лог метилтрансфераз, модифицирующих А1408
(рис. 2, б и в; см. расположение капреомицина
[64]. В случае идентичности продуктов метили%
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1577
рования устойчивость к капреомицину можно
щала модификацию Сm1402 ферментом RsmI. В
объяснить стерическим препятствием к связы%
свою очередь, оказалось, что активность RsmF,
ванию (рис. 2, б и в; см. расположение капрео%
формирующая m5C1407 16S рРНК, подавляется
мицина на панели в, показан оранжевым, и нук%
ферментом NpmA, метилирующим A1408 по N1
леотидного остатка m1A1408 16S рРНК).
положению [69]. Экспрессия npmA не так значи%
В ДЦ есть еще одна модификация, отсут%
тельно, как armA, сказывается на приспособлен%
ствие которой у Mycobacterium smegmatis приво%
ности клеток [69]. Гены, модифицирующие
дит к устойчивости к аминогликозидам. Это ме%
G1405 и A1408, встречаются и у штаммов%про%
тилирование U1498 16S рРНК по N3 атому
дуцентов аминогликозидов, и у патогенов, од%
(рис. 2) [65], осуществляемое метилтрансфера%
нако их экспрессия имеет разные последствия
зой «домашнего хозяйства» RsmE [66]. Надо за%
для эффективности роста. В отсутствие антибио%
метить, что уровень устойчивости изменяется
тика штаммы патогенных бактерий с экспресси%
примерно в 2 раза, что может быть объяснено
ей генов метилтрансфераз устойчивости пол%
косвенными причинами. Отсутствие данной
ностью пропадают при конкурентом культиви%
модификации также не сильно сказывается на
ровании, а в случае продуцентов антибиотиков
жизнедеятельности клеток в отсутствие антибио%
экспрессия генов метилтрансфераз не оказыва%
тика [66].
ет столь отрицательного влияния на приспособ%
Чрезвычайно интересным является взаимо%
ленность [71].
исключающее образование некоторых метили%
Таким образом, из анализа метилирования
рованных нуклеотидных остатков, обеспечива%
ДЦ можно сделать несколько интересных выво%
ющих устойчивость к антибиотикам, и консти%
дов. Например, о крайне необычных взаимо%
тутивно метилируемых нуклеотидных остатков
исключающих взаимодействиях метилтрансфе%
(рис. 2, б). Метилирование G1405 по N7 атому
раз устойчивости и «домашнего хозяйства», а
метилтрансферазой RmtC, обнаруженной у па%
также о возможности ухудшать связывание ан%
тогенной E. coli, снижает активность метил%
тибиотика без перекрывания положений ме%
трансферазы «домашнего хозяйства» RsmF, мо%
тильной группы и самого соединения, влияя на
дифицирующей С1407 по С5 положению [67].
контакты, опосредованные водой. Также обра%
Фенотипическая «цена» метилирования G1405
щает на себя внимание положительная роль ме%
метилтрансферазой RmtC практически отсут%
тилирования Cm1409 в связывании капреоми%
ствует [68]. Инактивация гена rsmF немного за%
цина, по%видимому, за счёт гидрофобного кон%
медляет скорость роста бактерий, не приводя к
такта.
дефектам сборки рибосом [31], однако без этой
метилтрансферазы значительно снижается спо%
собность клеток конкурировать с клетками ди%
УСТОЙЧИВОСТЬ К ДРУГИМ
кого типа [67, 68], а устойчивость к аминоглико%
АНТИБИОТИКАМ ЗА СЧЁТ
зидам возрастает [68]. Позже это наблюдение
МЕТИЛИРОВАНИЯ, СТЕРИЧЕСКИ
было взято под сомнение, поскольку было пока%
ПРЕПЯТСТВУЮЩЕГО СВЯЗЫВАНИЮ
зано, что родственная метилтрансфераза ArmA,
модифицирующая тот же G1405 по N7 положе%
Помимо рассмотренных ранее примеров,
нию, блокирует активность не RsmF, а другой
когда метилирование рРНК вызывает устойчи%
метилтрансферазы
«домашнего хозяйства»,
вость к антибиотикам, связывающимся в пеп%
RsmI [69], модифицирующей 2′%OH нуклеотид%
тидном туннеле, пептидилтрансферазном цент%
ного остатка С1402 [70]. Отсутствие метилиро%
ре и декодирующем центре, имеется ряд случа%
вания Cm1402 может лишь частично объяснять
ев, когда метилирование рРНК дает устойчи%
значительное падение приспособленности бак%
вость к антибиотикам, связывающимся вне этих
терий, экспрессирующих armA в отсутствие ан%
функциональных центров. Это, как правило,
тибиотиков [69], поскольку известно, что инак%
модификации рибосом продуцентов, защищаю%
тивация RsmI приводит к небольшому возраста%
щие свои рибосомы от производимого самой
нию частоты +1 и -1 сдвигов рамки считывания
бактерией соединения.
без сильного влияния на скорость роста бакте%
Метилирование нуклеотидного остатка
рий [70].
A1067 23S рРНК по 2′%OH%группе рибозы (рис. 3,
Антагонизм модификации нуклеотидных
а и б; таблица) даёт устойчивость к тиострепто%
остатков Cm1402 и m7G1405 16S рРНК наблю%
ну и микрокоцину [72]. Ген устойчивости, tsnR, в
дался также у Pseudomonas aeruginosa, у которой
природе был обнаружен только у продуцентов
в норме отсутствует ген метилтрансферазы
тиострептона, например у штамма Streptomyces
RsmF. У этой бактерии метилтрансфераза
azureus. Оказалось, что метилтрансферза TsnR
RmtD, образующая m7G1405, также предотвра%
модифицирует свободную 23S рРНК, а в присут%
5 БИОХИМИЯ том 85 вып. 11 2020
1578
ОСТЕРМАН и др.
ствии тиострептона или рибосомного белка L11
что рибосомы с модификацией и без одинаково
модификация не происходит [73]. Структура
обеспечивают GTPазную активность фактора
комплекса тиострептона и
50S субчастицы
EF%G [75].
Deinococcus radiodurans (рис. 3, в) иллюстрирует
В кластере биосинтеза авиламицина, обнару%
механизм устойчивости - метилирование рибо%
женного в геноме Streptomyces viridochromogenes
зы А1067 препятствует сближению петли 1 анти%
Tü57, закодированы гены двух метилтрансфераз,
биотика и спирали H43 [74]. Основное преиму%
aviRa и aviRb, экспрессия которых обеспечивает
щество данного антибиотика заключается в том,
устойчивость к действию этого антибиотика,
что он связывается вдалеке от большинства дру%
при этом экспрессия aviRa в Streptomyces lividans
гих ингибиторов синтеза белка и не демонстри%
TK66 обеспечивает незначительный уровень ус%
рует перекрестной устойчивости с другими ан%
тойчивости (10 мкг/мл), а aviRb - значительный
тибиотиками. Данных о влиянии этой модифи%
(250 мкг/мл) [77]. Оказалось, что обе метилтра%
кации на приспособленность нет, но показано,
нсферазы модифицируют нуклеотидные остат%
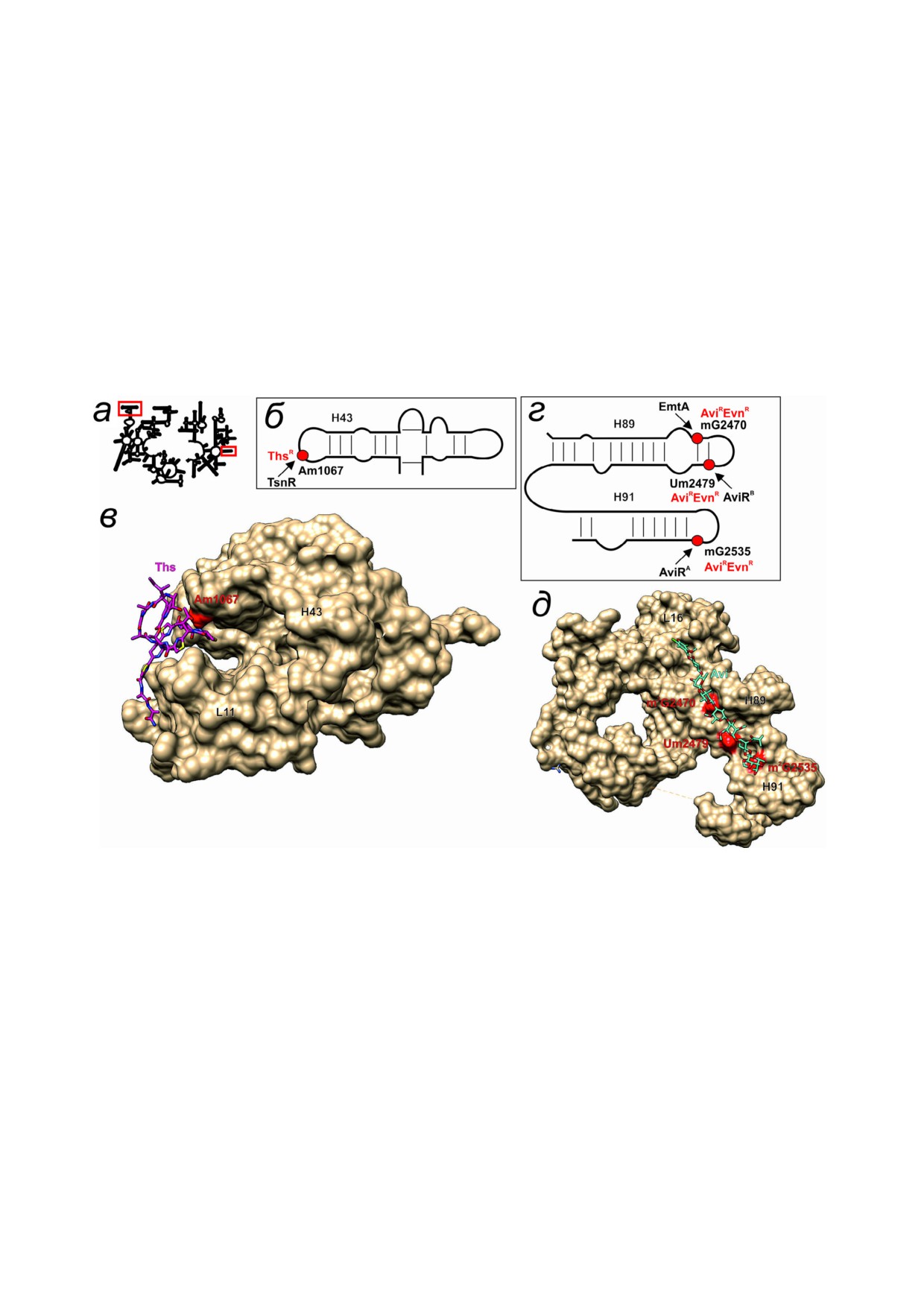
Рис. 3. Влияние модифицированных нуклеотидных остатков 23S рРНК на взаимодействие с тиострептоном и ортозоми%
цинами. а - Схема вторичной структуры 23S рРНК. Красными прямоугольниками обозначены области, рассмотрению
которых посвящены другие панели рисунка. б - Вторичная структура участка II домена 23S рРНК, взаимодействующего
с факторами трансляции GTPазы. Нуклеотидный остаток Am1067 23S рРНК, метилирование которого приводит к устой%
чивости к тиострептону (ThsR), выделен красным кружком. Стрелкой показана активность модифицирующего фермента
TsnR (подписан). в - Пространственная структура участка рибосомы (показана в виде молекулярной поверхности), обра%
зующего участок связывания тиострептона (показан розовой проволочной моделью и подписан). Красными Ван%дер%Ва%
альсовыми сферами показана метильная группа нуклеотидного остатка Am1067 23S рРНК, перекрывающаяся с участком
связывания тиострептона. Подписаны спираль 43 23S рРНК и белок L11. г - Вторичная структура спиралей 89 и 91 23S
рРНК, образующих участок связывания ортозомицинов. Модифицированные нуклеотидные остатки, вызывающие ус%
тойчивость к эвернимицину (EvnR) и авиламицину (AviR), обозначены красными кружками и подписаны. Черными стрел%
ками показана активность модифицирующих ферментов (подписаны), направленная на соответствующие нуклеотидные
остатки. д - Пространственная структура участка рибосомы (показана в виде молекулярной поверхности), образующего
участок связывания ортозомицинов (авиламицин показан голубой проволочной моделью и подписан). Красными Ван%
дер%Ваальсовыми сферами показаны метильные группы нуклеотидных остатков (подписаны), перекрывающиеся с участ%
ком связывания авиламицина. Подписаны спирали 89 и 91 23S рРНК и белок L16. При создании рисунка, включая нало%
жение пространственных структур друг на друга и моделирование модифицированных нуклеотидных остатков, использо%
валась программа UCSF Chimera [10]. В создании рисунка использовались вторичные структуры рРНК [11], а также
пространственные структуры 3CF5 [74] и 5KCR [76]
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1579
ки в рРНК: AviRa - G2535 по N1 или N2 положе%
часто встречаются метилированные нуклеотид%
нию (предположено исходя из данных по оста%
ные остатки, присоединенные ферментами «до%
новке обратной транскриптазы), а AviRb - U2479
машнего хозяйства», устойчивость к нему может
по 2′%OH%положению рибозы [78] (рис. 3, г; таб%
быть обеспечена метилированием рРНК.
лица). Устойчивость к авиламицину и родствен%
ному эверниамицину может быть также обеспе%
чена за счет модификации G2470 метилтрансфе%
УСТОЙЧИВОСТЬ К АНТИБИОТИКАМ
разой EmtA (минимальная ингибирующая кон%
ЗА СЧЕТ ОТСУТСТВИЯ МЕТИЛИРОВАНИЯ
центрация больше 256 мкг/мл) [79], обнаружен%
ной на плазмиде у патогенной бактерии E. faeci?
Помимо устойчивости к антибиотикам, выз%
um. Структура комплекса авиламицина и эвер%
ванной стерическими препятствиями из%за вне%
ниамицина с рибосомой E. coli, полученная ме%
сения в участок связывания дополнительной
тодом криоэлектронной микроскопии, объяс%
метильной группы рРНК метилтрансферазами,
няет эффекты метилирования (рис. 3, д). Ме%
существует несколько прямо противоположных
тильная группа, будучи присоединенной к 2′%
примеров (рис. 4; таблица). Наиболее извест%
OH U2479, перекрывается с участком связыва%
ный из них - это устойчивость к касугамицину
ния антибиотика. Точное положение метильных
при отсутствии диметилирования нуклеотидных
групп, присоединяемых к G2535 и G2470, не оп%
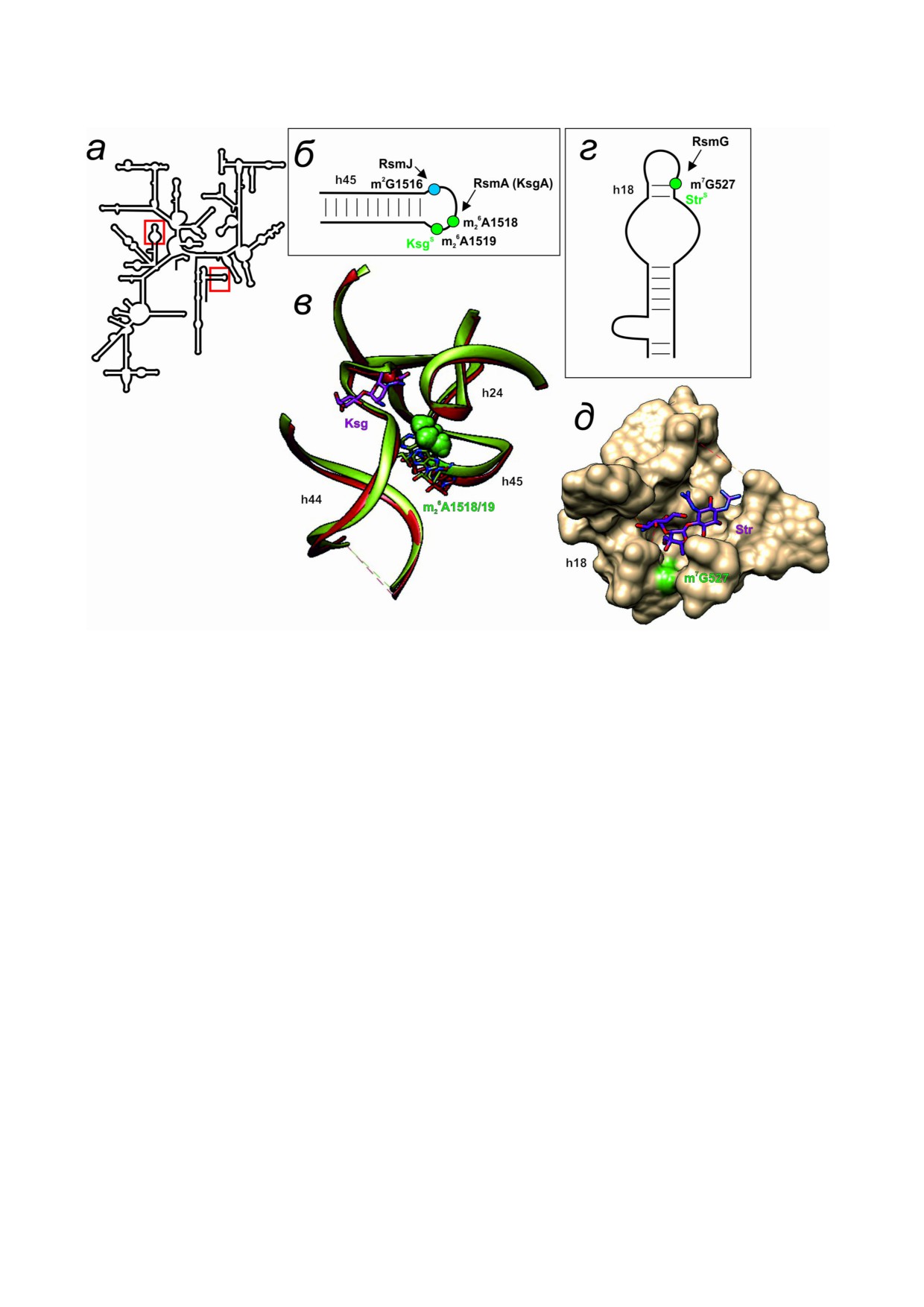
остатков A1518 и A1519 16S рРНК (рис. 4, а и б;
ределено, но исходя из того, что данные моди%
таблица) по N6 положению [87]. Структура
фикации обеспечивают устойчивость и блоки%
комплекса касугамицина с рибосомой (рис. 4, в)
руют обратную транскрипцию, можно предпо%
показала, что нуклеотидные остатки A1518/19
ложить, что модификация проходит по N1 или
не контактируют с антибиотиком, поэтому на%
N2 атомам. Исходя из структуры комплекса ави%
личие или отсутствие метильных групп вряд ли
ламицина с рибосомой [76] можно предполо%
может напрямую влиять на связывание антибио%
жить, что для перекрывания метильных групп,
тика, например, создавая гидрофобный контакт
присоединяемых как AviRa, так и EmtA, с анти%
[88]. Позже эксперименты подтвердили воз%
биотиками этой группы метилированию должна
можность взаимодействия касугамицина с ри%
подвергаться N2 аминогруппа. Об этом говорит
босомами, лишенными метилирования нуклео%
и то, что Blast%поиск ферментов, схожих по пос%
тидных остатков А1518/19 16S рРНК [89]. Нес%
ледовательности с EmtA, позволяет идентифи%
мотря на то что без метилирования A1518/19
цировать возможное родство этого белка с гуа%
клетки становятся устойчивыми к касугамицину
нин%N2 РНК метилтрансферазами. О влиянии
и таким образом имеют определенное преиму%
этих модификаций на эффективность роста бак%
щество хотя бы в специфических условиях нали%
терий в отсутствие антибиотиков на данный мо%
чия в среде этого антибиотика, практически все
мент нет информации.
про% и эукариоты имеют ортологичные метилт%
У продуцента пактомицина Streptomyces
рансферазы. Структура рибосомы с немодифи%
pactum метилирование А964 16S по N1 положе%
цированными A1518/19 показала, что метили%
нию (таблица) обеспечивает устойчивость за
рование необходимо для формирования струк%
счет ингибирования связывания этого антибио%
туры ДЦ малой субчастицы (рис. 4, в, сравните
тика [80]. При этом мутации устойчивости к пак%
структуру нескольких спиралей 16S рРНК в ри%
томицину A694G, C795U и C796U [81] распола%
босомах, содержащих (зеленый) и не содержа%
гаются в непосредственной близости от сайта
щих (красный) метильные группы, вносимые
связывания, определенного при помощи рентге%
RsmA(KsgA)) [90]. По всей видимости, именно
ноструктурного анализа [82]. О роли метилиро%
изменение структуры 30S субчастицы и являет%
вания А964 на эффективность роста на данный
ся причиной устойчивости к касугамицину при
момент нет данных, в непосредственной близос%
инактивации гена rsmA(ksgA). Помимо этого,
ти расположены нуклеотидные остатки G966 и
метилтрансфераза RsmA(KsgA) участвует в био%
С967, модифицируемые метилтрансферазами
генезе 30S субчастицы как белок%переключатель
«домашнего хозяйства» RsmD [83] и RsmB [84,
[91]. Бактерии, лишенные этой метилтрансфе%
85] соответственно, участвующие в контроле со%
разы, показывают сниженную скорость роста, а
отношения скорости транскрипции и трансля%
Yersinia pestis без RsmA(KsgA) проявляют мень%
ции [86]. Неизвестно пока и о возможных функ%
шую вирулентность [92].
циональных взаимосвязях между модификацией
В отличие от большинства аминогликози%
А964 и соседних нуклеотидных остатков.
дов, устойчивость к стрептомицину не может
Из данного раздела можно сделать самый
возникнуть за счет метилирования нуклеотид%
тривиальный вывод: где бы ни связывался анти%
ных остатков спирали 44 16S рРНК из%за отли%
биотик, даже вдали от тех участков, в которых
чающегося места связывания (рис. 4, д). Однако
БИОХИМИЯ том 85 вып. 11 2020
5*
1580
ОСТЕРМАН и др.
Рис. 4. Влияние модифицированных нуклеотидных остатков 16S рРНК на взаимодействие с касугамицином и стрептоми%
цином. а - Схема вторичной структуры 16S рРНК. Красными прямоугольниками обозначены области, рассмотрению ко%
торых посвящены другие панели рисунка. б - Вторичная структура спирали 45 16S рРНК. Модифицированные нуклео%
тидные остатки обозначены цветными кружками и подписаны. Зеленым выделены модификации, способствующие свя%
зыванию касугамицина, а синим - модификация ферментом «домашнего хозяйства», не влияющим на активность анти%
биотика. Стрелками показана активность модифицирующих ферментов (подписаны), направленная на соответствующие
нуклеотидные остатки. в - Пространственная структура участка рибосомы (показана в виде трубчатой модели, спирали
16S рРНК подписаны), конформация которого зависит от метилированных нуклеотидных остатков m26A1518/19. Зеле%
ным показана структура, соответствующая рибосомам, содержащим метилированные, а красным - неметилированные
нуклеотидные остатки A1518/19 (показаны проволочной моделью соответствующих цветов и подписаны, метильные
группы показаны зелеными Ван%дер%Ваальсовыми сферами). Касугамицин, взаимодействующий с рибосомой, содержа%
щей метилированные нуклеотидные остатки, показан розовой проволочной моделью и подписан. г - Вторичная структу%
ра спирали 18 16S рРНК, участвующей в образовании участка связывания стрептомицина. Нуклеотидный остаток
m7G527, модификация которого способствует активности стрептомицина, обозначен зеленым кружком и подписан. Чер%
ной стрелкой показана активность модифицирующего его фермента RsmG (подписан). д - Пространственная структура
участка рибосомы (показана в виде молекулярной поверхности), образующего участок связывания стрептомицина (пока%
зан сиреневой проволочной моделью и подписан). Зелеными Ван%дер%Ваальсовыми сферами показана метильная группа
нуклеотидного остатка m7G527 16S рРНК (подписана). Подписана спираль 18 16S рРНК. При создании рисунка, вклю%
чая наложение пространственных структур друг на друга и моделирование модифицированных нуклеотидных остатков,
использовалась программа UCSF Chimera [10]. В создании рисунка использовались вторичные структуры рРНК [11], а
также пространственные структуры 1J5E [93], 3OTO [90], 4V4H [94], 4NXM [95]
инактивация гена метилтрансферазы «домаш%
G527) у патогенных бактерий M. tuberculosis и
него хозяйства» RsmG, модифицирующей G527
Salmonella enterica также обеспечивает неболь%
16S рРНК по N7 положению (рис. 4, а и г; таб%
шую устойчивость к стрептомицину [97, 98].
лица), обеспечивает небольшой уровень устой%
Структура рибосомы в комплексе со стрептоми%
чивости к данному антибиотику у штамма
цином (рис. 4, д) показывает, что нуклеотидный
Streptomyces coelicolor [96]. В ходе дальнейших
остаток G527 находится в непосредственной
исследований было показано, что удаление гена
близости от места связывания антибиотика [99],
gidB (гомолога rsmG, также модифицирующего
но метильная группа не перекрывается напря%
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1581
мую с положением антибиотика. Предположе%
вости данных наблюдений это один из немногих
ние о том, что метилирование нуклеотидного
примеров, когда метилтрансфераза «домашнего
остатка G527 16S рРНК приводит к конформа%
хозяйства» обеспечивает некоторый уровень ус%
ционным изменениям не было подтверждено
тойчивости к антибиотикам. Мы также видим,
экспериментально [95]. Несмотря на отсутствие
что нередко для чувствительности к антибиоти%
значительного влияния делеции гена rsmG на
кам нужны модифицированные ферментами
рост бактерий [34], на примере Salmonella было
«домашнего хозяйства» нуклеотидные остатки.
показано, что такие мутанты проигрывают кон%
куренцию штаммам дикого типа [98]. Хотя
инактивация гена rsmG дает весьма небольшой
ЗАКЛЮЧЕНИЕ
уровень устойчивости к стрептомицину, по
сравнению с мутациями в гене рибосомного
Антибиотики - один из инструментов, ис%
белка S12, повреждение гена rsmG оказывается
пользуемый живыми организмами в конкурент%
важной предпосылкой для дальнейшего появле%
ной борьбе за ресурсы и территорию, при этом
ния этих мутаций. На примере Bacillus subtilis
механизмы устойчивости обеспечивают воз%
было показано, что делеция rsmG повышает точ%
можность продуцирования антибиотиков или
ность трансляции, и при этом в таких штаммах
выживания в их присутствии. Большинство из%
значительно (в 200 раз) возрастает частота мута%
вестных ингибиторов синтеза белка связывают%
ции устойчивости к стрептомицину в гене rpsL,
ся в функциональных центрах рибосомы. Эти
кодирующем белок S12 [96]. Аналогичный ре%
центры также обогащены модифицированны%
зультат был получен и для E. coli. На фоне деле%
ми, в основном метилированными, нуклеотид%
ции rsmG, обеспечивающей умеренную устойчи%
ными остатками [2]. Как видно, метилирование
вость к стрептомицину, повышается частота му%
рРНК является одним из основных механизмов
таций в rpsL (S12), еще сильнее увеличивающих
устойчивости к антибиотикам, причем высокий
устойчивость [100].
уровень устойчивости даёт присоединение ме%
Активность RlmAII у Bacillus subtilis, как уже
тильных групп так, что их положение начинает
обсуждалось ранее, обеспечивает устойчивость к
перекрываться с участком связывания антибио%
тилозину. В случае E. coli делеция гена его пара%
тика. Однако это не единственный механизм ус%
лога rlmAI, отвечающего за образование m1G745
тойчивости, вызванной метилированием. На
23S рРНК, приводит к устойчивости к виомици%
примере EfmM видно, что метилирование мо%
ну [101], несмотря на отсутствие контактов меж%
жет, предположительно, вытеснять молекулу во%
ду виомицином и этим модифицированным
ды, опосредующую дополнительный контакт
нуклеотидным остатком. Согласно результатам
антибиотика и рРНК.
экспериментов по футпринтингу, при делеции
Необычной, на первый взгляд, кажется ситу%
гена rlmAI скорее всего не происходит связыва%
ация, когда метилирование рРНК способствует
ние антибиотика. Как и в случае других делеций
связыванию антибиотика. Тем не менее это не
генов метилтрансфераз «домашнего хозяйства»,
редкость. В некоторых случаях можно предпо%
инактивация rlmAI имеет негативные послед%
ложить, что антибиотик напрямую контактиру%
ствия для приспособленности: снижается ско%
ет с метильной группой, формируя гидрофоб%
рость роста, способность рибосомных субчастиц
ные или Ван%дер%Ваальсовы взаимодействия.
к ассоциации, а также эффективность трансля%
Такой механизм можно предположить для взаи%
ции [101]. Возможно, структурные изменения,
модействия капреомицина с Cm1409 16S рРНК
снижающие эффективность взаимодействия
или телитромицина с m1G748. При отсутствии
субчастиц, также обусловливают устойчивость к
прямого контакта метильной группы рРНК с
виомицину, так как сайт связывания этого анти%
антибиотиком влияние на активность последне%
биотика расположен в непосредственной бли%
го может быть опосредовано конформацион%
зости от межсубчастичного мостика B2a.
ным изменением рРНК, зависящим от метили%
В сайте связывания тигециклина располо%
рования. Подобное явление хорошо документи%
жен нуклеотидный остаток m2G966 [102], моди%
ровано для влияния m26А1518/19 16S рРНК на
фицированный метилтрансферазой RsmD [83].
активность касугамицина. Подозревать подоб%
На примере S. pneumoniae было показано, что
ный механизм можно в случае m2A2503 23S
инактивация этой метилтрансферазы «домаш%
рРНК и линезолида, спарсомицина и тиамули%
него хозяйства» приводит к небольшому повы%
на, m3U1498 16S рРНК и аминогликозидов,
шению уровня устойчивости к тигециклину
m1G745 23S рРНК и виомицина. Сложнее объ%
[103] (таблица). И наоборот, при делеции rsmD в
яснить, как метильная группа рРНК может спо%
клетках E. coli чувствительность к тетрациклину
собствовать связыванию антибиотика, если не
возрастает [104]. При некоторой противоречи%
удается наблюдать ни контакт между ними, ни
БИОХИМИЯ том 85 вып. 11 2020
1582
ОСТЕРМАН и др.
изменение конформации, как например, в слу%
метилировании A2058 23S рРНК ферментами
чае m7G527 16S рРНК и стрептомицина. Тогда
Erm семейства, до практически нулевой, напри%
можно подозревать либо то, что взаимодействие
мер при метилировании A1408 белком NpmA.
или изменение конформации реализуется в ка%
Удивителен пример снижения «цены» устойчи%
ком%то другом функциональном, возможно,
вости к амикацину, вызванной мутацией
промежуточном состоянии транслирующей ри%
A1408G 16S рРНК при помощи метилтрансфе%
босомы, либо что метилирование влияет на свя%
разы TlyA, модифицирующей соседний нуклео%
зывание антибиотика опосредованно, напри%
тидный остаток.
мер, через вытеснение молекулы воды.
В научной литературе можно найти очень
Интересны эволюционные взаимоотноше%
мало примеров, не противоречащих возможно%
ния метилтрансфераз, вызывающих устойчи%
му эволюционному происхождению ферментов
вость к антибиотикам, и метилтрансфераз «до%
«домашнего хозяйства» от метилтрансфераз ан%
машнего хозяйства». Согласно общепринятому
тибиотикорезистентности. Модифицирующие
взгляду, последние распространяются «верти%
ферменты RsmD и RluC, принадлежащие к ко%
кально», т.е. эволюционируют вместе с соответ%
горте ферментов «домашнего хозяйства», при%
ствующими таксонами. Метилтрансферазы,
водят к некоторому уровню устойчивости к тет%
обеспечивающие устойчивость к антибиотикам,
рациклину и тиамулину, клиндамицину и лине%
характерны либо для узкой группы соответству%
золиду соответственно. Метилтрансфераза
ющих видов%продуцентов, либо распространя%
RlmAII, необходимая для образования m1G748
ются путем «горизонтального переноса генов»,
23S рРНК и весьма широко распространенная
при том что исходно также происходят от соот%
среди грамположительных бактерий, обеспечи%
ветствующих ферментов бактерий%продуцен%
вает некоторый уровень устойчивости к тилози%
тов. Если же задаваться вопросом, откуда по%
ну. Остается открытым вопрос: могут ли эти бел%
явились метилтрансферазы устойчивости у бак%
ки происходить от ферментов устойчивости к
терий, производящих антибиотики, и, возмож%
предковым антибиотикам, связывавшимся при%
но, у некоторых других, можно представить себе
мерно в тех же участках, что и вышеперечислен%
сценарий, в котором ферменты «домашнего хо%
ные современные?
зяйства» служат резервуаром для эволюции ан%
Хотя в прошлом исследование рРНК метил%
тибиотикорезистентности. Наиболее вероятен
трансфераз во многом ограничивалось модель%
такой ход событий для появления метилтранс%
ными организмами и патогенами, а в поиске
фераз семейства Erm из белков RsmA(KsgA).
продуцентов антибиотиков преобладал сугубо
Возможно ли обратное, т.е. превращение метил%
утилитарный подход, диктуемый медицинской
трансферазы устойчивости в метилтрансферазу
значимостью, будущее, на взгляд авторов дан%
«домашнего хозяйства»? Антибиотики и меха%
ного обзора, принадлежит комплексным иссле%
низмы устойчивости к ним эволюционируют
дованиям совместной эволюции как системы
давно, и биосинтез антибиотиков, теоретичес%
модификации рибосом, так и биосинтеза анти%
ки, может совершенствоваться таким образом,
бактериальных соединений.
чтобы избежать устойчивости. Возможно, что
повсеместное распространение генов устойчи%
вости может превратить последние в вариант
Финансирование. Работа поддержана Рос%
метилтрансфераз «домашнего хозяйства». Это
сийским фондом фундаментальных исследова%
возможно при условии, что метилирование не
ний (гранты №№ 20%04%00736 и 20%54%53014).
будет иметь негативных последствий, а возмож%
Конфликт интересов. Авторы заявляют об от%
но окажется полезным для приспособленности
сутствии конфликта интересов.
бактерий в отсутствие антибиотиков. «Цена»
Соблюдение этических норм. Настоящая
метилирования, вызывающего устойчивость к
статья не содержит описания выполненных ав%
антибиотикам, может варьировать от значитель%
торами исследований с участием людей или ис%
ной потери приспособленности, например при
пользованием животных в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Arenz, S., and Wilson, D. N. (2016) Bacterial protein syn%
evolutionary insights into ribosomal RNA methylation,
thesis as a target for antibiotic inhibition, Cold Spring Harb.
Nat. Chem. Biol., 14, 226%235.
Perspect. Med., 6, a025361, doi: 10.1101/cshperspect.
3.
Bogdanov, A. A., Sumbatyan, N. V., Shishkina, A. V.,
a025361.
Karpenko, V. V., and Korshunova, G. A. (2010) Ribosomal
2.
Sergiev, P. V., Aleksashin, N. A., Chugunova, A. A.,
tunnel and translation regulation, Biochemistry (Moscow),
Polikanov, Y. S., and Dontsova, O. A. (2018) Structural and
75, 1501%1516.
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1583
4.
Skinner, R., Cundliffe, E., and Schmidt, F. J. (1983) Site of
Pieters, J., Schoolnik, G., and Thompson, C. J. (2005)
action of a ribosomal RNA methylase responsible for resis%
Ancestral antibiotic resistance in Mycobacterium tuberculo?
tance to erythromycin and other antibiotics, J. Biol. Chem.,
sis, Proc. Natl. Acad. Sci. USA., 102, 12200%12205.
258, 12702%12706.
22.
Gupta, P., Kannan, K., Mankin, A. S., and Vázquez%
5.
Pernodet, J. L., Fish, S., Blondelet%Rouault, M. H., and
Laslop, N. (2013) Regulation of gene expression by
Cundliffe, E. (1996) The macrolide%lincosamide%strep%
macrolide%induced ribosomal frameshifting, Mol. Cell, 52,
togramin B resistance phenotypes characterized by using a
629%642.
specifically deleted, antibiotic%sensitive strain of
23.
Liu, M., and Douthwaite, S. (2002) Resistance to the
Streptomyces lividans, Antimicrob. Agents Chemother., 40,
macrolide antibiotic tylosin is conferred by single methyla%
581%585.
tions at 23S rRNA nucleotides G748 and A2058 acting in
6.
Roberts, M. C., Sutcliffe, J., Courvalin, P., Jensen, L. B.,
synergy, Proc. Natl. Acad. Sci. USA., 99, 14658%14663.
Rood, J., and Seppala, H. (1999) Nomenclature for macro%
24.
Liu, M., and Douthwaite, S. (2002) Methylation at
lide and macrolide%lincosamide%streptogramin B resistance
nucleotide G745 or G748 in 23S rRNA distinguishes
determinants, Antimicrob. Agents Chemother., 43, 2823%2830.
Gram%negative from Gram%positive bacteria, Mol.
7.
Arthur, M., Brisson%Noël, A., and Courvalin, P. (1987)
Microbiol., 44, 195%204.
Origin and evolution of genes specifying resistance to
25.
Yakhnin, H., Yakhnin, A. V., Mouery, B. L., Mandell, Z. F.,
macrolide, lincosamide and streptogramin antibiotics: data
Karbasiafshar, C., Kashlev, M., and Babitzke, P. (2019)
and hypotheses, J. Antimicrob. Chemother., 20, 783%802.
NusG%dependent RNA polymerase pausing and tylosin%
8.
Altschul, S. F., Gish, W., Miller, W., Myers, E. W., and
Dependent ribosome stalling are required for tylosin resis%
Lipman, D. J. (1990) Basic local alignment search tool, J.
tance by inducing 23S rRNA methylation in Bacillus sub?
Mol. Biol., 215, 403%410.
tilis, mBio, 10, e02665%19.
9.
Bhujbalrao, R., and Anand, R. (2019) Deciphering determi%
26.
Takaya, A., Sato, Y., Shoji, T., and Yamamoto, T. (2013)
nants in ribosomal methyltransferases that confer antimicro%
Methylation of 23S rRNA nucleotide G748 by RlmAII
bial resistance, J. Am. Chem. Soc., 141, 1425%1429.
methyltransferase renders Streptococcus pneumoniae
10.
Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S.,
telithromycin susceptible, Antimicrob. Agents Chemother.,
Greenblatt, D. M., Meng, E. C., and Ferrin, T. E. (2004)
57, 3789%3796.
UCSF Chimera - a visualization system for exploratory
27.
Desmolaize, B., Fabret, C., Brégeon, D., Rose, S.,
research and analysis, J. Comput. Chem., 25, 1605%1612.
Grosjean, H., and Douthwaite, S. (2011) A single methyl%
11.
The RNAcentral Consortium (2019) RNAcentral: a hub of
transferase YefA (RlmCD) catalyses both m5U747 and
information for non%coding RNA sequences, Nucleic Acids
m5U1939 modifications in Bacillus subtilis 23S rRNA,
Res., 47, D221%D229.
Nucleic Acids Res., 39, 9368%9375.
12.
Hansen, J. L., Ippolito, J. A., Ban, N., Nissen, P., Moore,
28.
Shoji, T., Takaya, A., Sato, Y., Kimura, S., Suzuki, T., and
P. B., and Steitz, T. A. (2002) The structures of four
Yamamoto, T. (2015) RlmCD%mediated U747 methylation
macrolide antibiotics bound to the large ribosomal subunit,
promotes efficient G748 methylation by methyltransferase
Mol. Cell, 10, 117%128.
RlmAII in 23S rRNA in Streptococcus pneumoniae; inter%
13.
Bulkley, D., Innis, C. A., Blaha, G., and Steitz, T. A.
play between two rRNA methylations responsible for
(2010) Revisiting the structures of several antibiotics bound
telithromycin susceptibility, Nucleic Acids Res., 43, 8964%
to the bacterial ribosome, Proc. Natl. Acad. Sci. USA., 107,
8972.
17158%17163.
29.
Long, K. S., Poehlsgaard, J., Kehrenberg, C., Schwarz, S.,
14.
Svetlov, M. S., Plessa, E., Chen, C.%W., Bougas, A.,
and Vester, B. (2006) The Cfr rRNA methyltransferase
Krokidis, M. G., Dinos, G. P., and Polikanov, Y. S. (2019)
confers resistance to Phenicols, Lincosamides,
High%resolution crystal structures of ribosome%bound
Oxazolidinones, Pleuromutilins, and Streptogramin A
chloramphenicol and erythromycin provide the ultimate
antibiotics, Antimicrob. Agents Chemother., 50, 2500%2505.
basis for their competition, RNA, 25, 600%606.
30.
LaMarre, J. M., Locke, J. B., Shaw, K. J., and Mankin, A. S.
15.
Gupta, P., Sothiselvam, S., Vázquez%Laslop, N., and
(2011) Low fitness cost of the multidrug resistance gene cfr,
Mankin, A. S. (2013) Deregulation of translation due to
Antimicrob. Agents Chemother., 55, 3714%3719.
post%transcriptional modification of rRNA explains why
31.
Smith, L. K., and Mankin, A. S. (2008) Transcriptional
erm genes are inducible, Nat. Commun., 4, 1984.
and translational control of the mlr operon, which confers
16.
Subramanian, S. L., Ramu, H., and Mankin, A. S. (2012)
resistance to seven classes of protein synthesis inhibitors,
Inducible resistance to macrolide antibiotics, in Antibiotic
Antimicrob. Agents Chemother., 52, 1703%1712.
Discovery and Development (Dougherty, T. J., and Pucci,
32.
Toh, S.%M., Xiong, L., Bae, T., and Mankin, A. S. (2008)
M. J., eds), Springer US, Boston, MA, pp. 455%484.
The methyltransferase YfgB/RlmN is responsible for mod%
17.
Vazquez%Laslop, N., Thum, C., and Mankin, A. S. (2008)
ification of adenosine 2503 in 23S rRNA, RNA, 14, 98%
Molecular mechanism of drug%dependent ribosome
106.
stalling, Mol. Cell, 30, 190%202.
33.
LaMarre, J. M., Howden, B. P., and Mankin, A. S. (2011)
18.
Arenz, S., Ramu, H., Gupta, P., Berninghausen, O.,
Inactivation of the indigenous methyltransferase RlmN in
Beckmann, R., Vázquez%Laslop, N., Mankin, A. S., and
Staphylococcus aureus increases linezolid resistance,
Wilson, D. N. (2014) Molecular basis for erythromycin%
Antimicrob. Agents Chemother., 55, 2989%2991.
dependent ribosome stalling during translation of the
34.
Pletnev, P., Guseva, E., Zanina, A., Evfratov, S., Dzama, M.,
ErmBL leader peptide, Nat. Commun., 5, 3501.
et al. (2020) Comprehensive functional analysis of Esche?
19.
Weisblum, B. (1995) Insights into erythromycin action
richia coli ribosomal RNA methyltransferases, Front.
from studies of its activity as inducer of resistance,
Genet., 11, 97.
Antimicrob. Agents Chemother., 39, 797%805.
35.
Vázquez%Laslop, N., Ramu, H., Klepacki, D., Kannan, K.,
20.
Kwak, J. H., Choi, E. C., and Weisblum, B. (1991)
and Mankin, A. S. (2010) The key function of a conserved
Transcriptional attenuation control of ermK, a macrolide%
and modified rRNA residue in the ribosomal response to
lincosamide%streptogramin B resistance determinant from
the nascent peptide, EMBO J., 29, 3108%3117.
Bacillus licheniformis, J. Bacteriol., 173, 4725%4735.
36.
Kaminska, K. H., Purta, E., Hansen, L. H., Bujnicki, J. M.,
21.
Morris, R. P., Nguyen, L., Gatfield, J., Visconti, K.,
Vester, B., and Long, K. S. (2010) Insights into the struc%
Nguyen, K., Schnappinger, D., Ehrt, S., Liu, Y., Heifets, L.,
ture, function and evolution of the radical%SAM 23S rRNA
БИОХИМИЯ том 85 вып. 11 2020
1584
ОСТЕРМАН и др.
methyltransferase Cfr that confers antibiotic resistance in
losis antibiotics viomycin and capreomycin bound to the
bacteria, Nucleic Acids Res., 38, 1652%1663.
70S ribosome, Nat. Struct. Mol. Biol., 17, 289%293.
37.
Giessing, A. M. B., Jensen, S. S., Rasmussen, A., Hansen,
53.
Selmer, M., Dunham, C. M., Murphy, F. V., Weixlbaumer, A.,
L. H., Gondela, A., Long, K., Vester, B., and Kirpekar, F.
Petry, S., Kelley, A. C., Weir, J. R., and Ramakrishnan, V.
(2009) Identification of 8%methyladenosine as the modifi%
(2006) Structure of the 70S ribosome complexed with
cation catalyzed by the radical SAM methyltransferase Cfr
mRNA and tRNA, Science, 313, 19351942.
that confers antibiotic resistance in bacteria, RNA, 15, 327%
54.
Galimand, M., Schmitt, E., Panvert, M., Desmolaize, B.,
336.
Douthwaite, S., Mechulam, Y., and Courvalin, P. (2011)
38.
Yan, F., LaMarre, J. M., Röhrich, R., Wiesner, J., Jomaa, H.,
Intrinsic resistance to aminoglycosides in Enterococcus fae?
Mankin, A. S., and Fujimori, D. G. (2010) RlmN and Cfr
cium is conferred by the 16S rRNA m5C1404%specific
are radical SAM enzymes involved in methylation of ribo%
methyltransferase EfmM, RNA, 17, 251%262.
somal RNA, J. Am. Chem. Soc., 132, 3953%3964.
55.
Demirci, H., Larsen, L. H. G., Hansen, T., Rasmussen, A.,
39.
Atkinson, G. C., Hansen, L. H., Tenson, T., Rasmussen, A.,
Cadambi, A., Gregory, S. T., Kirpekar, F., and Jogl, G.
Kirpekar, F., and Vester, B. (2013) Distinction between the
(2010) Multi%site%specific 16S rRNA methyltransferase
Cfr methyltransferase conferring antibiotic resistance and
RsmF from Thermus thermophilus, RNA, 16, 1584%1596.
the housekeeping RlmN methyltransferase, Antimicrob.
56.
François, B., Russell, R. J. M., Murray, J. B., Aboul%ela, F.,
Agents Chemother., 57, 4019%4026.
Masquida, B., Vicens, Q., and Westhof, E. (2005) Crystal
40.
Stojković, V., Noda%Garcia, L., Tawfik, D. S., and
structures of complexes between aminoglycosides and
Fujimori, D. G. (2016) Antibiotic resistance evolved via
decoding A site oligonucleotides: role of the number of
inactivation of a ribosomal RNA methylating enzyme,
rings and positive charges in the specific binding leading to
Nucleic Acids Res., 44, 8897%8907.
miscoding, Nucleic Acids Res., 33, 5677%5690.
41.
Ben tez Páez, A., Villarroya, M., and Armengod, M.%E.
57.
Maus, C. E., Plikaytis, B. B., and Shinnick, T. M. (2005)
(2012) The Escherichia coli RlmN methyltransferase is a
Mutation of tlyA confers capreomycin resistance in
dual%specificity enzyme that modifies both rRNA and
Mycobacterium tuberculosis, Antimicrob. Agents Chemother.,
tRNA and controls translational accuracy, RNA, 18, 1783%
49, 571%577.
1795.
58.
Sałamaszyńska Guz, A., Rose, S., Lykkebo, C. A., Taciak, B.,
42.
Conrad, J., Sun, D., Englund, N., and Ofengand, J. (1998)
B cal, P., Uśpieński, T., and Douthwaite, S. (2017) Biofilm
The rluC gene of Escherichia coli codes for a pseudouridine
formation and motility are promoted by Cj0588%directed
synthase that is solely responsible for synthesis of
methylation of rRNA in Campylobacter jejuni, Front. Cell.
pseudouridine at positions 955, 2504, and 2580 in 23 S
Infect. Microbiol., 7, 533.
ribosomal RNA, J. Biol. Chem., 273, 18562%18566.
59.
Johansen, S. K., Maus, C. E., Plikaytis, B. B., and
43.
Toh, S.%M., and Mankin, A. S. (2008) An indigenous post%
Douthwaite, S. (2006) Capreomycin binds across the
transcriptional modification in the ribosomal peptidyl
ribosomal subunit interface using tlyA%encoded 2’%O%
transferase center confers resistance to an array of protein
methylations in 16S and 23S rRNAs, Mol. Cell, 23, 173%
synthesis inhibitors, J. Mol. Biol., 380, 593%597.
182.
44.
Davies, J., Gorini, L., and Davis, B. D. (1965) Misreading
60.
Ermolenko, D. N., Spiegel, P. C., Majumdar, Z. K.,
of RNA codewords induced by aminoglycoside antibiotics,
Hickerson, R. P., Clegg, R. M., and Noller, H. F. (2007) The
Mol. Pharmacol., 1, 93%106.
antibiotic viomycin traps the ribosome in an intermediate
45.
Hausner, T. P., Geigenmüller, U., and Nierhaus, K. H.
state of translocation, Nat. Struct. Mol. Biol., 14, 493%497.
(1988) The allosteric three%site model for the ribosomal
61.
Monshupanee, T., Johansen, S. K., Dahlberg, A. E., and
elongation cycle. New insights into the inhibition mecha%
Douthwaite, S. (2012) Capreomycin susceptibility is
nisms of aminoglycosides, thiostrepton, and viomycin, J.
increased by TlyA%directed 2’%O%methylation on both
Biol. Chem., 263, 13103%13111.
ribosomal subunits, Mol. Microbiol., 85, 1194%1203.
46.
Doi, Y., and Arakawa, Y. (2007) 16S ribosomal RNA
62.
Rahman, A., Srivastava, S. S., Sneh, A., Ahmed, N., and
methylation: emerging resistance mechanism against
Krishnasastry, M. V. (2010) Molecular characterization of
aminoglycosides, Clin. Infect. Dis., 45, 88%94.
tlyA gene product, Rv1694 of Mycobacterium tuberculosis: a
47.
Grosjean, H. (2009) DNA and RNA modification enzymes:
non%conventional hemolysin and a ribosomal RNA methyl
structure, mechanism, function and evolution, Landes
transferase, BMC Biochem., 11, 35.
Bioscience, Austin, Tex.
63.
Freihofer, P., Akbergenov, R., Teo, Y., Juskeviciene, R.,
48.
Doi, Y., Wachino, J., and Arakawa, Y. (2016) Aminogly%
Andersson, D. I., and Böttger, E. C. (2016) Nonmutational
coside Resistance, Infect. Dis. Clin. North Am., 30, 523%
compensation of the fitness cost of antibiotic resistance in
537.
mycobacteria by overexpression of tlyA rRNA methylase,
49.
Wachino, J., Shibayama, K., Kurokawa, H., Kimura, K.,
RNA, 22, 1836%1843.
Yamane, K., et al. (2007) Novel plasmid%mediated 16S
64.
Felnagle, E. A., Rondon, M. R., Berti, A. D., Crosby, H. A.,
rRNA m1A1408 methyltransferase, NpmA, found in a
and Thomas, M. G. (2007) Identification of the biosyn%
clinically isolated Escherichia coli strain resistant to struc%
thetic gene cluster and an additional gene for resistance to
turally diverse aminoglycosides, Antimicrob. Agents
the antituberculosis drug capreomycin, Appl. Environ.
Chemother., 51, 4401%4409.
Microbiol., 73, 4162%4170.
50.
Macmaster, R., Zelinskaya, N., Savic, M., Rankin, C. R.,
65.
Bijpuria, S., Sharma, R., and Taneja, B. (2020) Deletion of
and Conn, G. L. (2010) Structural insights into the func%
RsmE 16S rRNA methyltransferase leads to low level
tion of aminoglycoside%resistance A1408 16S rRNA
increase in aminoglycoside resistance in Mycobacterium
methyltransferases from antibiotic%producing and human
smegmatis, bioRxiv, doi: 10.1101/2020.01.15.907279.
pathogenic bacteria, Nucleic Acids Res., 38, 7791%7799.
66.
Basturea, G. N., Rudd, K. E., and Deutscher, M. P. (2006)
51.
Borovinskaya, M. A., Pai, R. D., Zhang, W., Schuwirth, B. S.,
Identification and characterization of RsmE, the founding
Holton, J. M., et al. (2007) Structural basis for aminogly%
member of a new RNA base methyltransferase family,
coside inhibition of bacterial ribosome recycling, Nat.
RNA, 12, 426%434.
Struct. Mol. Biol., 14, 727%732.
67.
Andersen, N. M., and Douthwaite, S. (2006) YebU is a
52.
Stanley, R. E., Blaha, G., Grodzicki, R. L., Strickler, M. D.,
m5C methyltransferase specific for 16 S rRNA nucleotide
and Steitz, T. A. (2010) The structures of the anti%tubercu%
1407, J. Mol. Biol., 359, 777%786.
БИОХИМИЯ том 85 вып. 11 2020
АНТИБИОТИКИ И МЕТИЛИРОВАНИЕ
1585
68.
Gutierrez, B., Escudero, J. A., San Millan, A., Hidalgo, L.,
ifies guanine 966 of the 16 S rRNA: functional identifica%
Carrilero, L., Ovejero, C. M., Santos%Lopez, A., Thomas%
tion and tertiary structure, J. Biol. Chem., 282, 5880%5887.
Lopez, D., and Gonzalez%Zorn, B. (2012) Fitness cost and
84.
Gu, X. R., Gustafsson, C., Ku, J., Yu, M., and Santi, D. V.
interference of Arm/Rmt aminoglycoside resistance with
(1999) Identification of the 16S rRNA m5C967 methyl%
the RsmF housekeeping methyltransferases, Antimicrob.
transferase from Escherichia coli, Biochemistry, 38, 4053%
Agents Chemother., 56, 2335%2341.
4057.
69.
Lioy, V. S., Goussard, S., Guerineau, V., Yoon, E.%J.,
85.
Tscherne, J. S., Nurse, K., Popienick, P., Michel, H.,
Courvalin, P., Galimand, M., and Grillot%Courvalin, C.
Sochacki, M., and Ofengand, J. (1999) Purification,
(2014) Aminoglycoside resistance 16S rRNA methyltrans%
cloning, and characterization of the 16S RNA m5C967
ferases block endogenous methylation, affect translation
methyltransferase from Escherichia coli, Biochemistry, 38,
efficiency and fitness of the host, RNA, 20, 382%391.
1884%1892.
70.
Kimura, S., and Suzuki, T. (2010) Fine%tuning of the ribo%
86.
Prokhorova, I. V., Osterman, I. A., Burakovsky, D. E.,
somal decoding center by conserved methyl%modifications
Serebryakova, M. V., Galyamina, M. A., et al. (2013)
in the Escherichia coli 16S rRNA, Nucleic Acids Res., 38,
Modified nucleotides m(2)G966/m(5)C967 of Escherichia
1341%1352.
coli 16S rRNA are required for attenuation of tryptophan
71.
Vidučić, D., Obranić, S., Matovina, M., Babić, F., and
operon, Sci. Rep., 3, 3236.
Vlahoviček, G. M. (2014) Host fitness effects of aminogly%
87.
Helser, T. L., Davies, J. E., and Dahlberg, J. E. (1972)
coside resistance 16S rRNA G1405 and A1408 methyl%
Mechanism of kasugamycin resistance in Escherichia coli,
transferases from clinical pathogens and natural antibiotic
Nature New Biol., 235, 6%9.
producers, FEBS J., 281, Suppl. s1, 285.
88.
Schluenzen, F., Takemoto, C., Wilson, D. N., Kaminishi, T.,
72.
Thompson, J., Schmidt, F., and Cundliffe, E. (1982) Site
Harms, J. M., et al. (2006) The antibiotic kasugamycin
of action of a ribosomal RNA methylase conferring resis%
mimics mRNA nucleotides to destabilize tRNA binding
tance to thiostrepton, J. Biol. Chem., 257, 7915%7917.
and inhibit canonical translation initiation, Nat. Struct.
73.
Lentzen, G., Klinck, R., Matassova, N., Aboul%ela, F., and
Mol. Biol., 13, 871%878.
Murchie, A. I. H. (2003) Structural basis for contrasting
89.
Vila%Sanjurjo, A., Squires, C. L., and Dahlberg, A. E. (1999)
activities of ribosome binding thiazole antibiotics, Chem.
Isolation of kasugamycin resistant mutants in the 16 S ribo%
Biol., 10, 769%778.
somal RNA of Escherichia coli, J. Mol. Biol., 293, 1%8.
74.
Harms, J. M., Wilson, D. N., Schluenzen, F., Connell, S. R.,
90.
Demirci, H., Murphy, F., Belardinelli, R., Kelley, A. C.,
Stachelhaus, T., Zaborowska, Z., Spahn, C. M. T., and
Ramakrishnan, V., Gregory, S. T., Dahlberg, A. E., and
Fucini, P. (2008) Translational regulation via L11: molecu%
Jogl, G. (2010) Modification of 16S ribosomal RNA by the
lar switches on the ribosome turned on and off by
KsgA methyltransferase restructures the 30S subunit to
thiostrepton and micrococcin, Mol. Cell, 30, 26%38.
optimize ribosome function, RNA, 16, 2319%2324.
75.
Cundliffe, E., and Thompson, J. (1981) The mode of
91.
Connolly, K., Rife, J. P., and Culver, G.
(2008)
action of nosiheptide (multhiomycin) and the mechanism
Mechanistic insight into the ribosome biogenesis functions
of resistance in the producing organism, J. Gen. Microbiol.,
of the ancient protein KsgA, Mol. Microbiol., 70, 1062%
126, 185%192.
1075.
76.
Arenz, S., Juette, M. F., Graf, M., Nguyen, F., Huter, P.,
92.
Mecsas, J., Bilis, I., and Falkow, S. (2001) Identification of
Polikanov, Y. S., Blanchard, S. C., and Wilson, D.N.
attenuated Yersinia pseudotuberculosis strains and charac%
(2016) Structures of the orthosomycin antibiotics avila%
terization of an orogastric infection in BALB/c mice on
mycin and evernimicin in complex with the bacterial 70S
day 5 postinfection by signature%tagged mutagenesis,
ribosome, Proc. Natl. Acad. Sci. USA, 113, 7527%7532.
Infect. Immun., 69, 2779%2787.
77.
Weitnauer, G., Gaisser, S., Trefzer, A., Stockert, S.,
93.
Wimberly, B. T., Brodersen, D. E., Clemons, W. M.,
Westrich, L., Quiros, L. M., Mendez, C., Salas, J. A., and
Morgan%Warren, R. J., Carter, A. P., Vonrhein, C.,
Bechthold, A. (2001) An ATP%binding cassette transporter
Hartsch, T., and Ramakrishnan, V. (2000) Structure of the
and two rRNA methyltransferases are involved in resis%
30S ribosomal subunit, Nature, 407, 327%339.
tance to avilamycin in the producer organism Streptomyces
94.
Schuwirth, B. S., Day, J. M., Hau, C. W., Janssen, G. R.,
viridochromogenes Tь57, Antimicrob. Agents Chemother., 45,
Dahlberg, A. E., Cate, J. H. D., and Vila%Sanjurjo, A.
690%695.
(2006) Structural analysis of kasugamycin inhibition of
78.
Treede, I., Jakobsen, L., Kirpekar, F., Vester, B., Weitnauer, G.,
translation, Nat. Struct. Mol. Biol, 13, 879%886.
Bechthold, A., and Douthwaite, S. (2003) The avilamycin
95.
Demirci, H., Murphy, F. V., Murphy, E. L., Connetti, J. L.,
resistance determinants AviRa and AviRb methylate 23S
Dahlberg, A. E., Jogl, G., and Gregory, S. T. (2014)
rRNA at the guanosine 2535 base and the uridine 2479
Structural analysis of base substitutions in Thermus ther?
ribose, Mol. Microbiol., 49, 309%318.
mophilus 16S rRNA conferring streptomycin resistance,
79.
Mann, P. A., Xiong, L., Mankin, A. S., Chau, A. S.,
Antimicrob. Agents Chemother., 58, 4308%4317.
Mendrick, C. A., et al. (2001) EmtA, a rRNA methyltrans%
96.
Nishimura, K., Hosaka, T., Tokuyama, S., Okamoto, S.,
ferase conferring high%level evernimicin resistance, Mol.
and Ochi, K. (2007) Mutations in rsmG, encoding a 16S
Microbiol., 41, 1349%1356.
rRNA methyltransferase, result in low%level streptomycin
80.
Ballesta, J. P., and Cundliffe, E. (1991) Site%specific
resistance and antibiotic overproduction in Streptomyces
methylation of 16S rRNA caused by pct, a pactamycin
coelicolor A3(2), J. Bacteriol., 189, 3876%3883.
resistance determinant from the producing organism,
97.
Okamoto, S., Tamaru, A., Nakajima, C., Nishimura, K.,
Streptomyces pactum, J. Bacteriol., 173, 7213%7218.
Tanaka, Y., Tokuyama, S., Suzuki, Y., and Ochi, K. (2007)
81.
Mankin, A. S. (1997) Pactamycin resistance mutations in
Loss of a conserved 7%methylguanosine modification in
functional sites of 16 S rRNA, J. Mol. Biol., 274, 8%15.
16S rRNA confers low%level streptomycin resistance in
82.
Polikanov, Y. S., Osterman, I. A., Szal, T., Tashlitsky, V. N.,
bacteria, Mol. Microbiol., 63, 1096%1106.
Serebryakova, M. V., et al. (2014) Amicoumacin a inhibits
98.
Mikheil, D. M., Shippy, D. C., Eakley, N. M.,
translation by stabilizing mRNA interaction with the ribo%
Okwumabua, O. E., and Fadl, A. A. (2012) Deletion of
some, Mol. Cell, 56, 531%540.
gene encoding methyltransferase (gidB) confers high%level
83.
Lesnyak, D. V., Osipiuk, J., Skarina, T., Sergiev, P. V.,
antimicrobial resistance in Salmonella, J. Antibiot,, 65,
Bogdanov, A. A., et al. (2007) Methyltransferase that mod%
185%192.
БИОХИМИЯ том 85 вып. 11 2020
1586
ОСТЕРМАН и др.
99. Carter, A. P., Clemons, W. M., Brodersen, D. E., Morgan%
102. Jenner, L., Starosta, A. L., Terry, D. S., Mikolajka, A.,
Warren, R. J., Wimberly, B. T., and Ramakrishnan, V.
Filonava, L., Yusupov, M., Blanchard, S. C., Wilson, D. N.,
(2000) Functional insights from the structure of the 30S
and Yusupova, G. (2013) Structural basis for potent
ribosomal subunit and its interactions with antibiotics,
inhibitory activity of the antibiotic tigecycline during
Nature, 407, 340%348.
protein synthesis, Proc. Natl. Acad. Sci. U.A, 110, 3812%
100. Ben tez Páez, A., Cárdenas Brito, S., Corredor, M.,
3816.
Villarroya, M., and Armengod, M. E. (2013) Mutaciones
103. Lupien, A., Gingras, H., Leprohon, P., and Ouellette, M.
en genes modificadores de ARN ribosómico y la resistencia
(2015) Induced tigecycline resistance in Streptococcus
a aminoglucósidos: el caso del gen rsmG, Biomédica, 34,
pneumoniae mutants reveals mutations in ribosomal pro%
41, doi: 10.7705/biomedica.v34i0.1702.
teins and rRNA, J. Antimicrob. Chemother., 70, 2973%
101. Gustafsson, C., and Persson, B. C. (1998) Identification of
2980.
the rrmA gene encoding the 23S rRNA m1G745 methyl%
104. Nichols, R. J., Sen, S., Choo, Y. J., Beltrao, P., Zietek, M.,
transferase in Escherichia coli and characterization of an
et al. (2011) Phenotypic landscape of a bacterial cell, Cell,
m1G745%deficient mutant, J. Bacteriol., 180, 359%365.
144, 143%156.
rRNA METHYLATION AND ANTIBIOTIC RESISTANCE
Review
I. A. Osterman1,2, O. A. Dontsova1,2,3, and P. V. Sergiev1,2,4*
1 Center of Life Sciences, Skolkovo Institute of Science and Technology,
143028 Skolkovo, Russia; E?mail: petya@genebee.msu.ru
2 Faculty of Chemistry, Lomonosov Moscow State University, 119991 Moscow, Russia
3 Shemyakin and Ovchinnikov Institute of Bioorganic Chemistry, Russian Academy of Sciences, 117997 Moscow, Russia
4 Institute of Functional Genomics, Lomonosov Moscow State University, 119991 Moscow, Russia
Received June 12, 2020
Revised July 16, 2020
Accepted July 16, 2020
Methylation of nucleotides in rRNA is one of the basic mechanisms of bacterial resistance to protein synthesis
inhibitors. The genes for corresponding methyltransferases have been found in producer strains and clinical isolates
of pathogenic bacteria. In some cases, rRNA methylation by housekeeping enzymes is, on the contrary, required for
the action of antibiotics. The effects of rRNA modifications associated with antibiotic efficacy may be cooperative or
mutually exclusive. Evolutionary relationships between the systems of rRNA modification by housekeeping enzymes
and antibiotic resistance%related methyltransferases are of particular interest. In this review, we discuss the above top%
ics in detail.
Keywords: ribosome, methylation, antibiotics, macrolides, aminoglycosides
БИОХИМИЯ том 85 вып. 11 2020