БИОХИМИЯ, 2020, том 85, вып. 11, с. 1690 - 1700
УДК 577.322.9
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
Обзор
© 2020
К.С. Усачев1, М.М. Юсупов1,2*, Ш.З. Валидов1
1 Казанский (Приволжский) федеральный университет, 420008 Казань, Россия;
электронная почта: marat@igbmc.fr
2 Институт генетики, молекулярной и клеточной биологии (IGBMC),
67400 Илькирш+Граффенштаден, Франция
Поступила в редакцию 30.07.2020
После доработки 28.08.2020
Принята к публикации 28.08.2020
Эубактерии в ответ на стрессовые условия снижают уровень синтеза белка: разбирают рибосомы на 30S и
50S субъединицы или превращают их в трансляционно неактивные 70S и 100S комплексы. При этом реша!
ются две принципиально важные для клетки задачи: 1) снижение расходов на биосинтез белка в неблаго!
приятных условиях и 2) консервация функциональных рибосом для быстрого возобновления синтеза белка
при восстановлении внешних благоприятных условий. Все известные гены белков сайленсинга или гибер!
нации рибосом располагаются в оперонах, связанных с ответом на голодание, как одним из стрессов, что
очевидно помогает координировать замедление синтеза белка и общий ответ на стрессы. Вероятно, систе!
мы гибернации могут работать как регуляторы, которые позволяют привести скорость синтеза белка в со!
ответствие с энергетическим состоянием клетки. Учитывая лимитированное количество питательных ве!
ществ в естественных условиях и постоянное действие других стрессов на бактериальную клетку, бактери!
альная рибосома должна проводить большую часть времени в комплексе с белками сайленсинга/гиберна!
ции. Таким образом, между стадией рециркуляции и стадией инициации трансляции существует дополни!
тельная стадия, в которой рибосома поддерживается в «консервированном» состоянии в виде субъединиц,
нетранслирующих 70S частиц или 100S димеров. Механизм гибернации рибосом консервативен, прошел
долгий эволюционный путь и необходим для поддержания энергозатратного и ресурсоемкого процесса био!
синтеза белка в организмах, обреченных на жизнь в меняющихся условиях окружающей среды в условиях
стресса.
КЛЮЧЕВЫЕ СЛОВА: рибосома, трансляция, гибернация, сайленсинг.
DOI: 10.31857/S0320972520110111
СТРЕСС ЯВЛЯЕТСЯ ПОСТОЯННЫМ
ство доступных доноров и акцепторов электро!
СПУТНИКОМ ЖИВЫХ КЛЕТОК
нов, что затрудняет извлечение энергии из
субстратов [1]. Вероятно, в природе микроорга!
Неблагоприятные или стрессовые условия,
низмы постоянно находятся под воздействием
такие как экстремальные значения температуры
тех или иных стрессовых условий, что препят!
и pH, дефицит влаги, гипооксигенация и голода!
ствует их интенсивному размножению, напри!
ние приводят к замедлению роста микроорга!
мер такому, которое наблюдается в лаборатор!
низмов, поскольку в этих условиях ограничено
ных средах, где условия близки к идеальным.
количество питательных веществ и/или количе!
Однако даже в этих почти идеальных условиях
классическое представление о росте культуры
микроорганизмов выделяет 6 фаз: логарифми!
Принятые сокращения: EF!G - фактор элонгации; ческая фаза, фаза ускорения роста, экспоненци!
HPF - фактор гибернации рибосом, hibernation promoting
альная фаза, фаза замедления роста, стационар!
factor; RaiA - ассоциированный с рибосомами ингибитор
А, ribosome associated inhibitor A; RbfA - связывающий ри!
ная фаза и фаза отмирания клеток [2]. Стацио!
босому фактор A, ribosome binding factor A; RBS - сайт свя!
нарная фаза характеризуется остановкой роста
зывания рибосомы, ribosome binding site; RimM - фактор культуры и, как и в случае фазы замедления рос!
созревания рибосомы M, ribosome maturation factor M; та, связана, прежде всего, с истощением пита!
RimP - фактор созревания рибосомы P, ribosome matura!
тельных веществ в среде, то есть с голоданием
tion factor P; RMF - фактор, модулирующий рибосому,
ribosome modulating factor; RRF - фактор рециркуляции
клеток. В природе большинство микроорганиз!
рибосомы; RsfS - рибосомный сайленсинг!фактор.
мов находятся в стадии остановленного роста,
* Адресат для корреспонденции.
что близко к состоянию клеток в фазе замедле!
1690
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
1691
ния роста и в стационарной фазе и нуждается в
время которого осуществляется биосинтез бел!
значительном энерго! и ресурсосбережении, ко!
ка рибосомой, согласно прочтению генетичес!
торое реализуется как общее замедление метабо!
кого кода матричной РНК (мРНК) [11]. Рибо!
лизма живой клетки [1]. При этом снижается ин!
сома является крупнейшим рибонуклеопротеи!
тенсивность всех основных ресурсозатратных и
новым комплексом в клетке, который составля!
энергоемких процессов - репликация ДНК с ос!
ет 70S у прокариот [12] и 80S у эукариот [13] и
тановкой деления клеток [3], снижается уровень
состоит из двух субъединиц: большой (50S - у
транскрипции и изменяется спектр транскриби!
прокариот и 60S - у эукариот) и малой (30S - у
руемых генов [4], кроме того, значительно сни!
прокариот и 40S - у эукариот). Малая субъеди!
жается интенсивность трансляции и далее под!
ница отвечает за правильное прочтение кода
держивается на низком уровне синтез белка [5].
мРНК и кодон/антикодоновое взаимодействие
Хотя данное замедление метаболизма временное
с тРНК. Функция большой субъединицы зак!
и предполагает в будущем возобновление актив!
лючается в проведении пептидилтрасферазной
ности клетки при наступлении благоприятных
реакции (образование пептидной связи в расту!
условий, отдельные работы указывают на воз!
щей цепи белка). Основными этапами трансля!
можные длительные сроки нахождения в стадии
ции являются: инициация [14], элонгация [15],
остановленного роста без гибели клеток [6].
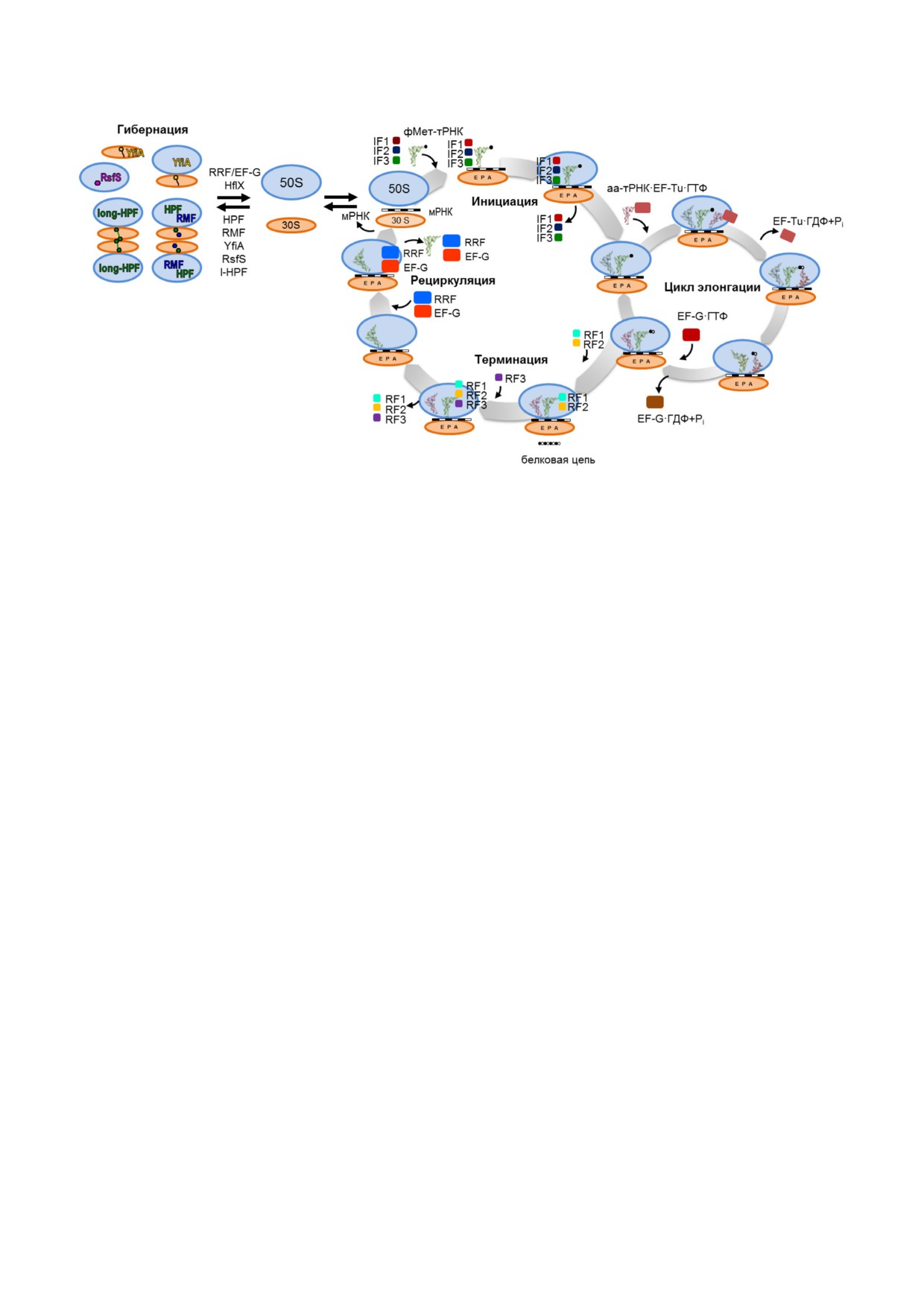
терминация [16] и рециркуляция [17] (рис. 1).
Наряду со стрессовыми условиями, которые
На стадии инициации происходит ассоциация
созданы окружающей средой, существует такое
субъединиц рибосомы на мРНК с позициони!
физиологическое состояние бактериальных
рованием на старт!кодоне формил!метионил!
культур, как биопленка, где снижение интенсив!
тРНК в P!сайте рибосомы. Этому процессу
ности биологических процессов необходимо для
способствуют факторы инициации IF1, IF2 и
поддержания жизнеспособных клеток при гипо!
IF3 [18]. На этапе элонгации с помощью факто!
оксигенации и меньшей доступности питатель!
ра элонгации EF!G и EF!Tu [19] происходит уд!
ных веществ внутри матрикса биопленок [7].
линение полипептидной цепи путем связыва!
Клетки со сниженной интенсивностью метабо!
ния амино!ацилированной тРНК (аа!тРНК) в
лизма, или покоящиеся клетки, часто обнаружи!
A!сайте рибосомы, согласно коду мРНК [11].
вают у патогенных микроорганизмов, находя!
На стадии элонгации функционирует еще один
щихся внутри организма хозяина [8]. Вероятно,
фактор - EF!P, отвечающий за облегчение син!
снижение интенсивности метаболизма необхо!
теза полипролиновых мотивов [20]. Делеция ге!
димо на отдельных стадиях паразитирования,
на efp не является летальной для большинства
поскольку покоящиеся клетки могут быть неза!
эубактерий, однако эукариотический аналог
метны для иммунной системы организма хозяи!
этого белка - eIF5α, абсолютно необходим для
на и даже могут переживать повышение темпе!
жизнеспособности эукариотической клетки
ратуры или воздействие антибиотиков, как это
[21]. Когда трансляционный комплекс доходит
показано для золотистого стафилококка, Sta+
до стоп!кодона мРНК, наступает этап термина!
phylococcus aureus [9]. Необходимость систем го!
ции трансляции, в результате которого с по!
лодания показана также для паразитирования в
мощью релиз!факторов первого (RF1, RF2) и
тканях растений: мутанты Erwinia amylovora по
второго (RF3) классов происходит высвобожде!
генам rpoN (сигма фактор голодания σ54) и yhbH
ние полипептида [16]. Следующая за этим ре!
(hpf, фактор гибернации рибосом) были неспо!
циркуляция заключается в диссоциации пост!
собны к размножению внутри плодов груши
терминационного комплекса
(70S, мРНК,
[10]. Таким образом, замедление метаболизма
тРНК) на субъединицы при совместном воз!
используется клетками бактерий для выживания
действии фактора рециркуляции рибосомы
в окружающей среде и для эффективного пара!
(RRF) и EF!G [22]. Считается, что фактор ини!
зитирования и заключается в значительном уг!
циации IF3, который связывается с 30S субъ!
нетении репликации ДНК, изменениях на уров!
единицей, препятствует ассоциации субъеди!
не транскрипции и снижении трансляционной
ниц рибосомы до следующего этапа инициации
активности, обеспечивая только небольшой уро!
[23]. Очевидно, часть 30S и 50S субъединиц
вень синтеза белка в покоящейся клетке.
могут реассоциировать и образовать вакант!
ную 70S рибосому, которая, впрочем, может
связываться с безлидерными РНК и осущес!
СТАДИИ РАБОТЫ РИБОСОМЫ
твлять синтез белка, минуя стадию инициации
И СИНТЕЗ БЕЛКА
[24]. Таким образом, в бактериальной клетке
после диссоциации пост!терминационного
Трансляция - важнейший процесс, являю!
комплекса рибосомы есть стадия, где присут!
щийся основой жизнедеятельности клетки, во
ствуют 30S и 50S субъединицы и может образо!
БИОХИМИЯ том 85 вып. 11 2020
12*
1692
УСАЧЕВ и др.
Рис. 1. Биосинтез белка бактерий. (С цветными вариантами рис. 1-3 можно ознакомиться в электронной версии статьи
ваться вакантная нетранслирующая 70S рибо!
та [32], что и объясняет одновременное появле!
сома, способная взаимодействовать с фактора!
ние димеров рибосом.
ми гибернации.
RMF обнаружен только у представителей γ!
протеобактерий, и, возможно, является частью
уникальной системы гибернации рибосом, ко!
ФАКТОРЫ ГИБЕРНАЦИИ
торая сохранила основные принципы действия
КАК ОТВЕТ НА СТРЕСС
гибернационных систем, но реализовала это в
виде белка, который изменяет конформацию
Голодание клеток, снижение уровня энерге!
самой рибосомы и стимулирует ее димериза!
тических молекул, которые могут быть вызваны
цию. Две молекулы RMF присоединяются к
внешними стрессами, требуют также замедле!
двум свободным 70S частицам, подготавливая
ния синтеза белка [1]. Снижение интенсивности
их к димеризации, после чего происходит обра!
синтеза белка производится специальными
зование 90S димеров. Далее в данный комплекс
факторами, которые блокируют работу рибосо!
встраиваются две молекулы фактора гиберна!
мы, непосредственно связываясь с ней [25] (рис.
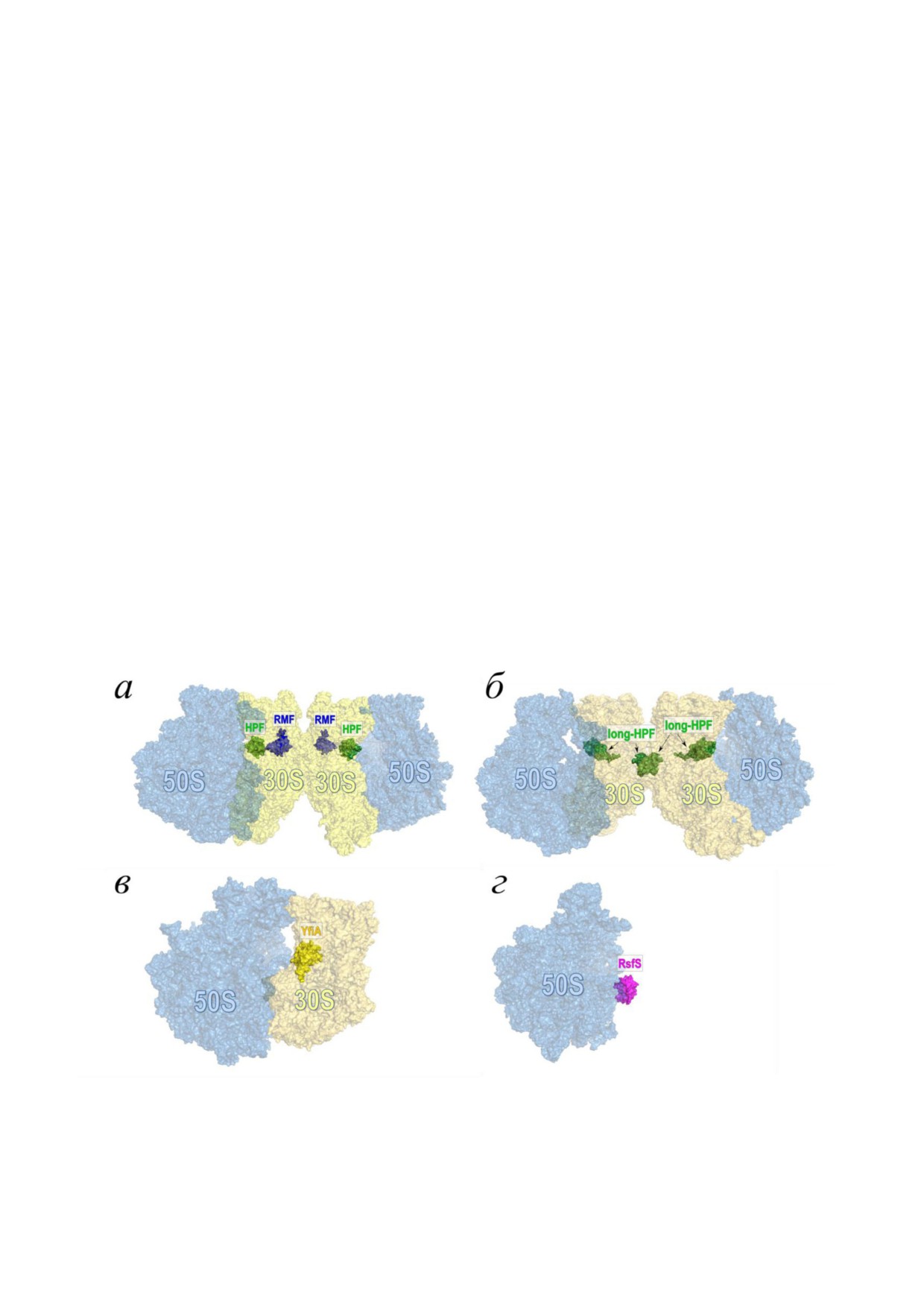
ции рибосом (HPF), завершая образование 100S
2). Внимание к системам гибернации было сти!
рибосомного комплекса, состоящего из двух 70S
мулировано фактом обнаружения димеров ри!
рибосом (рис. 2, а).
босом в 1960 году. На тот момент было высказа!
На экспрессию гена rmf оказывает влияние
но предположение, что димеры являются неак!
CRP (белок!рецептор цАМФ): в его отсутствие
тивными комплексами полностью собранных
синтез RMF значительно снижен [33]. Очевид!
рибосом, которые могут быть использованы в
но, на экспрессию rmf также влияет другой алар!
трансляции, что подтверждало появление диме!
мон - (p)ppGpp [34, 35].
ров рибосом в стационарной фазе и их быстрое
Структурные исследования в гетерологич!
исчезновение при возобновлении активного
ной системе показали, что RMF (E. coli) являет!
роста [26].
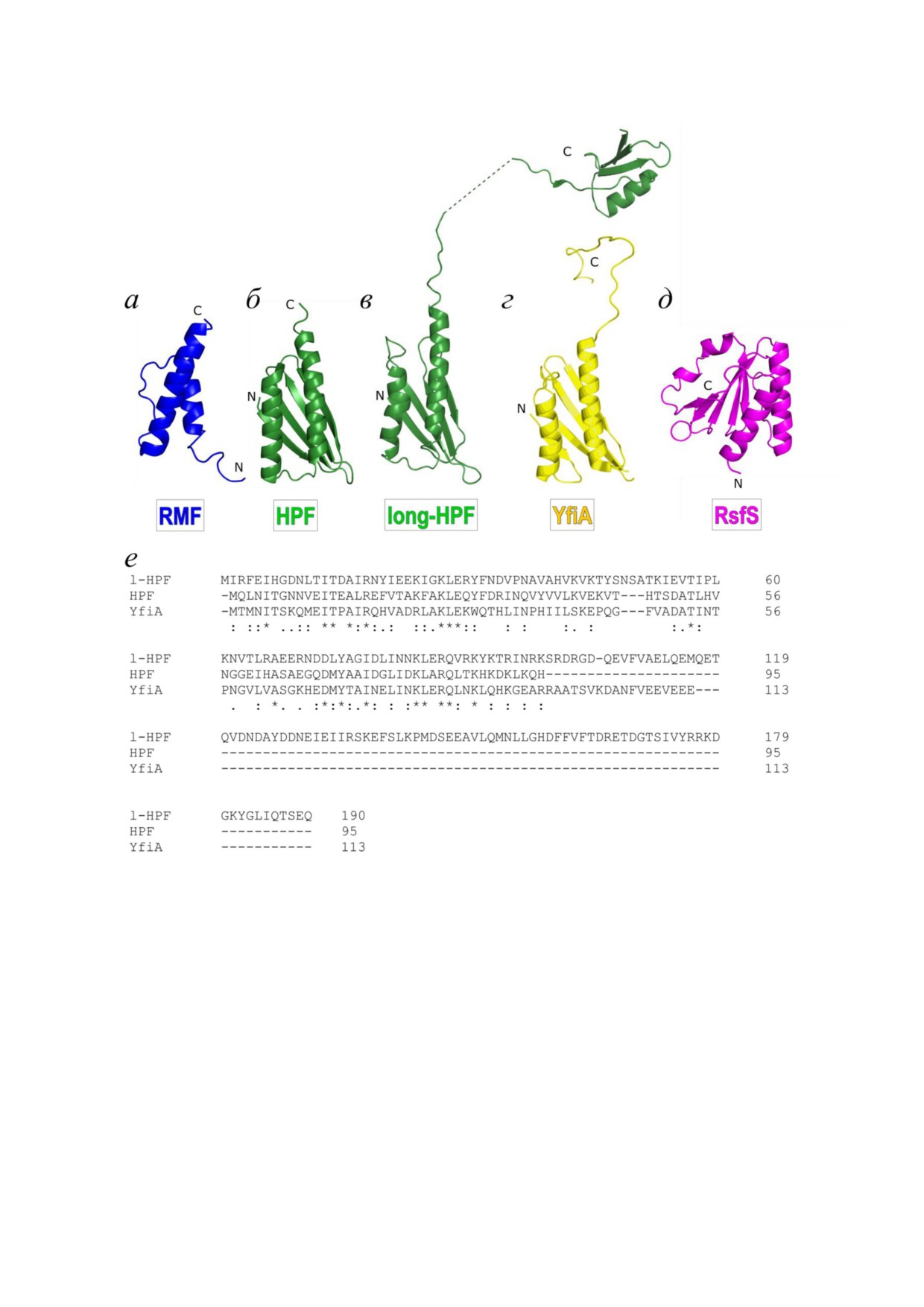
ся небольшим белком размером 55 а.о. (рис. 3),
RMF (ribosome modulating factor) фактор, мо8
и при реконструкции димеров рибосом in vitro
дулирующий рибосому. RMF был исторически
он связывается с малой субъединицей 70S рибо!
первым белком, на который обратили внима!
сомы Thermus thermophilus вблизи 3′!конца 16S
ние, поскольку он производил димеризацию ри!
рРНК в области анти!RBS (Ribosome Binding
босом в клетках Escherichia coli, а для регистра!
Site, сайт связывания рибосомы) и стерически
ции димеров рибосом инструментарий сущест!
препятствует взаимодействию с RBS мРНК [30].
вовал уже начиная с 1960 года [26]. RMF практи!
Эти данные были уточнены анализом 100S ди!
чески отсутствует в логарифмической и значи!
меров рибосом, выделенных из клеток E. coli в
тельно накапливается в стационарной фазе рос!
стационарной фазе. Сайт связывания RMF рас!
БИОХИМИЯ том 85 вып. 11 2020
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
1693
полагается в полости, образованной белками
ны по аминокислотным последовательностям и
uS2, uS7, uS9 и bS21, при этом α1!спираль RMF
проявляют значительную структурную гомоло!
располагается между спиралями h28 и h37/h40
гию, что может объясняться общностью проис!
16S рРНК. Взаимодействию с сахаро!фосфат!
хождения (рис. 3). Короткий HPF (95-100 а.о.)
ным остовом 16S рРНК способствует наличие
встречается у γ!протеобактерий и участвует в
положительно заряженных а.о. (Arg3, Lys5, Arg8,
образовании 100S димеров вместе с RMF [36].
Arg11 и Arg15). Также наблюдается взаимодей!
Длинный вариант HPF (l!HPF, 160-190 а.о.)
ствие N!концевой и С!концевой α!спиралей
намного более распространен и встречается у
RMF с белком uS7 [27]. RMF облегчает стабили!
эволюционно далёких видов эубактерий [28, 37,
зацию компактной конформации bS1, совмест!
38] и даже сохранился у пластид в растениях
ное действие белков препятствует взаимодей!
[39]. N!концевой домен длинного HPF гомоло!
ствию мРНК с анти!RBS, что предотвращает
гичен таковому у короткого HPF γ!протеобак!
инициацию трансляции. Кроме того, в интер!
терий, а C!концевой домен ответственен за ди!
фейсе димеризации С!концевой домен uS2 зак!
меризацию [40]. N!концевой домен YfiA также
рывает тоннель входа мРНК, препятствуя ее
гомологичен короткому HPF, однако YfiA со!
связыванию [27]. Можно утверждать, что RMF
держит еще и неструктурированную часть, ко!
не препятствует инициации непосредственно,
торая составляет 14-30 а.о. (рис. 3). У γ!протео!
поскольку нет сообщений о взаимодействии
бактерий присутствуют как короткий HPF, так
RMF с отдельными малыми 30S субъединицами
и YfiA, оба этих белка участвуют в гибернации
рибосомы и их димеризации. Поддержание 70S
рибосомы.
рибосом в комплексе, возможно, и является
Короткий HPF (YhbN). Структура короткого
препятствием для инициации. Закрытие тонне!
варианта белка HPF представлена двумя α!спи!
ля для мРНК может являться инструментом для
ралями и β!листом, состоящим из четырех β!тя!
предотвращения трансляции безлидерных
жей (рис 3). Встраивание HPF в 90S димер, ко!
мРНК, которые всё же могут происходить на ва!
торый был образован присоединением RMF,
кантных 70S рибосомах [24].
приводит к образованию 100S димеров рибосом
HPF и YfiA. Три гибернационных белка -
[41] (рис. 2, а). Короткая форма HPF не способ!
короткий и длинный HPF, а также YfiA - сход! на димеризовать рибосому, дополняющая функ!
Рис. 2. Структуры комплексов рибосом с факторами гибернации/сайленсинга. а - 100S димер рибосом со связанными
факторами RMF и коротким вариантом белка HPF (Escherichia coli) [27], б - 100S димер рибосом с длинным вариантом
HPF (S. aureus) [28, 29], в - комплекс 70S рибосомы с фактором YfiA (E. coli) [30], г - комплекс 50S рибосомной субчас!
тицы с фактором RsfS (S. aureus) [31]
БИОХИМИЯ том 85 вып. 11 2020
1694
УСАЧЕВ и др.
Рис. 3. Структура факторов гибернации/сайленсинга: а - RMF (E. coli), б - HPF (E. coli), в - long!HPF (S. aureus), г - YfiA
(E. coli), д - RsfS (S. aureus). е - Сравнение аминокислотных последовательностей белков l!HPF (S. aureus), HPF (E. coli)
и YfiA (E. coli)
ция этого белка может быть объяснена его кон!
комплексов 100S димеров рибосом, выделенных
куренцией с белком YfiA за P! и A!сайты на ма!
из клеток E. coli, отличались от данных, полу!
лой субъединице рибосомы. C!концевой нест!
ченных с помощью реконструкции димеров ри!
руктурированный фрагмент YfiA, предположи!
босом in vitro в гетерологичной системе. Инте!
тельно, препятствует взаимодействию рибосо!
ресным фактом является прямое взаимодей!
мы и RMF. Вероятно, этим фактом объясняется
ствие HPF с деацилированной тРНК, находя!
отсутствие димеров рибосом в мутантах E. coli
щейся в E!сайте, обнаруженное именно благо!
Δhpf in vivo [41]. HPF связывается в A и P сайтах
даря выделению димеров рибосом из клеток.
30S субъединиц близко к тРНК, располагаю!
При этом а.о. Arg33 и Lys93 белка HPF находят!
щейся в E!сайте между спиралями 16S рРНК
ся на расстоянии, сопоставимом с длиной водо!
h30 и h44. Данные, полученные при изучении
родных связей, которые могут возникать с нук!
БИОХИМИЯ том 85 вып. 11 2020
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
1695
леотидами антикодоновой петли тРНК, находя!
домена, который, так же как и короткий HPF, и
щейся в E!сайте. Авторы предполагают, что на!
N!концевой домен l!HPF, занимает в рибосоме
личие незагруженной тРНК в E!сайте рибосомы
область A! и P!сайтов, тогда как C!концевая
(что происходит в случае голодания) может сти!
неструктурированная часть, очевидно, распола!
мулировать образование димеров рибосом [27].
гается на поверхности 30S субъединицы, препя!
Длинный HPF (l HPF). Длинный вариант
тствуя взаимодействию рибосомы с RMF [30]
HPF, в отличие от всех других родственных бел!
(рис. 2, в, и рис. 3). В системе E. coli было пока!
ков, состоит из двух структурированных доме!
зано, что YfiA может взаимодействовать как с
нов (рис. 3). N!концевой домен занимает A! и P!
70S рибосомой, так и с малой (30S) субъедини!
сайты рибосомы, тогда как C!концевой домен
цей. Это взаимодействие препятствует инициа!
отвечает за димеризацию (рис. 2, б). Два домена
ции трансляции и способствует формированию
связаны между собой небольшим неструктури!
гибернирующей 70S рибосомы [49].
рованным фрагментом размером 30-35 а.о. По
Рибосомный сайленсинг8фактор RsfS (RsfA,
своей структуре и функциям N!концевой домен
YbeB). В отличие от белков HPF/RaiA и RMF
является аналогом короткого HPF, за счет взаи!
рибосомный сайленсинг!фактор RsfS широко
модействия этого домена l!HPF удерживается в
распространен у эубактерий [50] и даже обнару!
рибосоме, что было доказано в многочисленных
жен в геномах митохондрий [51]. RsfS является
экспериментах по реконструкции димеров ри!
белком стационарной фазы и связывается с бел!
босом в S. aureus [28, 29], Bacillus subtilis [42],
ком uL14 на большой (50S) субъединице рибо!
Lactococcus lactis [43] и T. thermophilus [38]. Как
сомы, препятствуя ассоциации субъединиц с
было показано Khusainov et al. [28] N!концевой
формированием 70S инициаторного комплекса,
домен располагается в A! и P!сайтах рибосомы,
что подавляет синтез белка в стрессовых услови!
перекрывая места связывания гигромицина В,
ях [52]. Делеционные мутанты E. coli по гену rsfS
тетрациклина, эдеина, пактамицина и касуго!
демонстрируют снижение адаптации при пере!
мицина и, возможно, защищает рибосому от
ходе с богатой среды на бедную и отличаются
взаимодействия с этими антибиотиками, что,
сниженной жизнеспособностью в стационар!
вероятно, объясняет повышенную устойчивость
ной фазе [50]. Детальную модель взаимодей!
покоящихся клеток эубактерий к ряду антибио!
ствия выявили последние исследования RsfS из
тиков [9, 44]. Также было показано участие C!
S. aureus (рис. 2, г, и рис. 3). Поскольку структу!
концевого домена белка l!HPF в димеризации
ра uL14 в рибосоме при взаимодействии RsfS с
[28, 29, 38, 42, 43] и выдвинуто предположение,
50S субъединицей, а также в изолированном ге!
что остаток фенилаланина в положении 160 иг!
теродимере uL14/RsfS не отличались, то данный
рает ключевую роль в образовании димера белка
гетеродимер был использован для изучения де!
HPF [42]. Более детальное исследование строе!
тального взаимодействия белков. В результате
ния димера и вклада отдельных а.о. было прове!
была определена структура с разрешением 2,3 Å,
дено на изолированном C!концевом домене
что позволило выявить взаимодействующие а.о.
HPF S. aureus. Было показано, что для димери!
белков RsfS и uL14. На основе данных рентгено!
зации рибосом in vitro важны по крайней мере
структурного анализа показано, что связывание
пять гидрофобных а.о.: Phe160, Val162, Ile173,
двух белков опосредуется серией контактов как
Tyr175 и Leu185 [40].
электростатических, так и гидрофобных. При!
Ассоциированный с рибосомой ингибитор YfiA
мечательно, что взаимодействие между амино!
(RaiA). Впервые нерибосомный белок (spot Y),
кислотами, такими как Arg97, Arg107, Lys113 из
расположенный в интерфейсе между субъеди!
uL14 и Glu70, Asp81, Tyr98 из RsfS, сохраняется
ницами рибосомы, был обнаружен в 1986 году
у RsfS из разных бактерий [31].
[45]. Первое сообщение о белке YfiA приводится
в работе Link et al. [46], где он упомянут как бе!
лок, ассоциированный с рибосомой. Позднее
ЗНАЧЕНИЕ МЕХАНИЗМА ГИБЕРНАЦИИ
этот же белок был локализован между субъеди!
ДЛЯ ВЫЖИВАНИЯ КЛЕТОК БАКТЕРИЙ
ницами рибосомы и обозначен pY [47]. Добавле!
ние pY в бесклеточную систему ингибировало
Делеция генов гибернации рибосом не явля!
синтез белка, что и послужило причиной пере!
ется летальной для бактериальной клетки и
именования данного белка в RaiA (ассоцииро!
практически не влияет на параметры роста
ванный с рибосомами ингибитор А, ribosome
культуры в логарифмической фазе. Делецион!
associated inhibitor A) [48]. Ввиду значительного
ные мутанты по генам сайленсинга/гибернации
сходства очень часто при аннотации геномов
уступают дикому типу в жизнеспособности в
название RaiA также используется для коротко!
стационарной фазе, а также при хранении или
го HPF и l!HPF, YfiA состоит из N!концевого
воздействии стресс!факторов [25]. Снижение
БИОХИМИЯ том 85 вып. 11 2020
1696
УСАЧЕВ и др.
жизнеспособности и более длительная логариф!
β!складчатой структурой, соединенных под!
мическая фаза у делеционных мутантов объяс!
вижной петлей. По своей структуре N!концевой
няются расходами энергии клетки на биосинтез
домен схож с тРНК!связывающим доменом EF!
белка, который не был снижен своевременно,
Tu и содержит высоко консервативный мотив
как это происходит в норме при функциониро!
GXXG, характерный для белков с KH!доменом
вании систем сайленсинга/гибернации.
и зачастую участвующий во взаимодействии с
Долгое время оставалась неясной роль диме!
РНК [64]. C!концевой домен RimM по своей
ризации рибосом, поскольку даже в отсутствие
структуре напоминает PRC!бочонок (Photosyn!
димеризующей активности l!HPF эффективно
thetic reaction center), однако в его составе отсут!
останавливает трансляцию. Недостающие дан!
ствует консервативный а.о., отвечающий за ре!
ные были получены при исследовании мутантов
гуляцию окислительно!восстановительного
B. subtilis, где было показано, что делеция гена
процесса во время фотосинтеза [63, 65]. Функ!
hpf или нарушение механизма димеризации уд!
ция C!концевого домена заключается в обеспе!
линенного варианта HPF приводит к потере
чении взаимодействия 3′!домена 16S рРНК с
белков uS2 и uS3 малой субъединицы рибосомы,
рибосомными белками (в частности uS19) в
которые располагаются в интерфейсе 100S ди!
процессе созревания 30S субъединицы [56, 62,
мера [53]. Данные белки наиболее подвержены
66]. Фактор созревания рибосомы P (RimP или
диссоциации, поскольку включаются в структу!
YhbC, Ribosome maturation factor P) - высоко!
ру рибосомы на конечной стадии созревания
консервативный среди прокариот белок с моле!
[54, 55], и их потеря приводит к деградации
кулярной массой 41 кДа, участвующий в про!
рРНК в рибосомах, не участвующих в трансля!
цессе сборки 30S субъединицы рибосомы и в
ции.
формировании центрального псевдоузла 16S
Для компенсации данного процесса проис!
рРНК. Согласно данным количественного
ходит экспрессия факторов созревания рибосо!
масс!спектрометрического анализа, в E. coli
мы RimM, RimP и RbfA, которые связываются с
фактор RimP способствует связыванию рибо!
малой субъединицей в A! и P!сайтах и, так же
сомных белков uS5, uS7 и uS12 с 5′!доменом р!
как и HPF, играют роль шаперонов в сворачива!
РНК in vitro [57]. Удаление гена rimP приводит к
нии рРНК, участвуют в формировании и стаби!
дестабилизации конформации центрального
лизации центрального псевдоузла 16S рРНК
псевдоузла рРНК и накоплению интермедиатов
[56-59]. Показано, что у 30S субъединиц рибо!
рибосом, у которых отсутствовали белки uS5 и
сом, выделенных из штаммов E. coli с делецией
uS12 на стадии созревания [57]. Поскольку в
генов rimM и rbfA, наблюдалась потеря белков
клетках млекопитающих не обнаружен гомолог
uS2, uS3 и bS21, это означает, что процесс созре!
RimP, данный белок может выступать потенци!
вания in vivo остановился до того, как данные
альной мишенью для разработки антибактери!
белки могут связаться с пре!30S субчастицей
альных препаратов. Интересно, что, согласно
[58]. Таким образом, повреждение рибосом в де!
последним данным, мутации в гене rimP приво!
леционных мутантах может быть отчасти ком!
дят к увеличению восприимчивости к даптоми!
пенсировано белками биогенеза рибосомы
цину метициллин!резистентных штаммов
RimM, RimP и RbfA.
S. aureus [67].
Связывающий рибосому фактор A (RbfA,
Ribosome binding factor A) - адаптивный белок
холодового шока с молекулярной массой
ГИБЕРНАЦИЯ И САЙЛЕНСИНГ
14,9 кДа, обеспечивающий рост бактерий при
КАК ОТДЕЛЬНАЯ СТАДИЯ
низких температурах. Он является представите!
ФУНКЦИОНИРОВАНИЯ РИБОСОМЫ
лем большого семейства белков, обозначенного
как RBFA, которые встречаются в протеомах
В условиях стресса клетка снижает уровень
большинства архебактерий и эубактерий [60,
синтеза белка до наступления благоприятных
61]. Ген rbfA необходим для нормального роста
условий. В это время избирательно транскри!
клеток при низких температурах (10-20 °C), и
бируются опероны, связанные с голоданием и
его делеция приводит к замедлению роста бак!
стресс!ответами вообще [1]. Таким образом, на
терий. Фактор созревания рибосомы M (RimM,
невысоком уровне, но все же поддерживается
ribosome maturation factor M) - консервативный
синтез белков, в том числе факторов гиберна!
у большинства эубактерий, отсутствующий у ар!
ции и сайленсинга, и их относительное количе!
хей и обнаруженный только у четырех видов эу!
ство ко всем остальным белкам может значи!
кариот белок с молекулярной массой 21 кДа,
тельно повышаться, так как это показано в ста!
участвующий в сборке 30S субъединицы рибо!
ционарной фазе для RMF [32], HPF [68], YfiA
сомы [62, 63]. Белок состоит из двух доменов с
[49] и RsfS [50]. Мы предполагаем, что данные
БИОХИМИЯ том 85 вып. 11 2020
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
1697
белки связываются с субъединицами или обра!
ре миллиард лет эволюции, поскольку встреча!
зовавшимися вакантными 70S рибосомами,
ются также в органеллах эукариотической клет!
превращая их в инактивированные комплексы
ки, что говорит о появлении систем гибернации
50S-RsfS [31], 30S-YfiA [49], 70S-YfiA [49] и
ещё до симбиотического образования митохонд!
100S-lHPF или 100S-RMF/HPF [69]. Далее
рий и пластид. Длительная инактивация рибосом
данные комплексы с помощью совместного
и их хранение белками гибернации/сайленсинга,
действия факторов RRF и EF!G или ГТФазы
вероятно, является самостоятельной стадией
теплового шока HflX могут быть вновь разобра!
функционирования рибосом, обеспечивающей
ны до 30S и 50S субъединиц [70]. Однако в при!
экономию энергии и ресурсов клетки в условиях
сутствии белков гибернации RMF, HPF, YfiA и
стрессов, а также сохраняющей синтезирующий
RsfS субъединицы рибосомы, вероятно, вновь
белок аппарат для быстрого возобновления син!
образуют неактивные комплексы (рис. 1). Учи!
теза белка при улучшении внешних условий. Без
тывая постоянное состояние голодания и вы!
возможности обратимой и контролируемой оста!
живания клетки в условиях стрессов [6], стадия
новки белок!синтезирующего аппарата поддер!
гибернации может быть протяженной во вре!
жание такого энерго! и ресурсоёмкого процесса,
мени, возможно, даже превышающей все ос!
как биосинтез белка, было бы невозможно в ме!
тальные стадии функционирования рибосом.
няющихся условиях окружающей среды.
При устранении давления стрессов профиль
транскрипции изменяется, в силу чего количе!
ство белков гибернации по сравнению с факто!
Финансирование. Исследование выполнено
рами инициации и элонгации резко снижается,
при финансовой поддержке Российского фонда
что, вероятно, позволяет рекрутировать боль!
фундаментальных исследований (грант № 20!
шинство субъединиц в инициацию трансля!
34!70021).
ции.
Конфликт интересов. Авторы заявляют об от!
сутствии конфликта интересов в финансовой
или какой!либо иной сфере.
ЗАКЛЮЧЕНИЕ
Соблюдение этических норм. Настоящая
статья не содержит исследований с участием
Системы гибернации широко представлены
людей или использованием животных в качест!
среди эубактерий и насчитывают по меньшей ме! ве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Bergkessel, M., Basta, D. W., and Newman, D. K. (2016)
stress response in the metabolically active population, J.
The physiology of growth arrest: uniting molecular and
Bacteriol., 194, 2062!2073, doi: 10.1128/JB.00022!12.
environmental microbiology, Nat. Rev. Microbiol., 14, 549!
8. Tan, N. C., Cooksley, C. M., Roscioli, E., Drilling, A. J.,
562, doi: 10.1038/nrmicro.2016.107.
Douglas, R., Wormald, P. J., and Vreugde, S. (2014) Small!
2.
Monod, J. (1947) Kinetics of bacterial growth, Nature,
colony variants and phenotype switching of intracellular
160, 105!106.
Staphylococcus aureus in chronic rhinosinusitis, Allergy, 69,
3.
Kolter, R., Siegele, D. A., and Tormo, A. (1993) The sta!
1364!1371, doi: 10.1111/all.12457.
tionary phase of the bacterial life cycle, Annu. Rev.
9. Tuchscherr, L., Medina, E., Hussain, M., Volker, W.,
Microbiol., 47, 855!874, doi: 10.1146/annurev.mi.47.
Heitmann, V., et al. (2011) Staphylococcus aureus pheno!
100193.004231.
type switching: an effective bacterial strategy to escape host
4.
Battesti, A., Majdalani, N., and Gottesman, S. (2011) The
immune response and establish a chronic infection, EMBO
RpoS!mediated general stress response in Escherichia coli,
Mol. Med., 3, 129!141, doi: 10.1002/emmm.201000115.
Annu. Rev. Microbiol., 65, 189!213, doi: 10.1146/annurev!
10. Ancona, V., Li, W., and Zhao, Y. (2014) Alternative sigma
micro!090110!102946.
factor RpoN and its modulation protein YhbH are indis!
5.
Gefen, O., Fridman, O., Ronin, I., and Balaban, N. Q.
pensable for Erwinia amylovora virulence, Mol. Plant.
(2014) Direct observation of single stationary!phase bacte!
Pathol., 15, 58!66, doi: 10.1111/mpp.12065.
ria reveals a surprisingly long period of constant protein
11. Aitken, C. E., Petrov, A., and Puglisi, J. D. (2010) Single
production activity, Proc. Natl. Acad. Sci. USA, 111, 556!
ribosome dynamics and the mechanism of translation,
561, doi: 10.1073/pnas.1314114111.
Annu. Rev. Biophys., 39, 491!513, doi: 10.1146/annurev.
6.
Finkel, S. E. (2006) Long!term survival during stationary
biophys.093008.131427.
phase: evolution and the GASP phenotype, Nat. Rev.
12. Yusupov, M. M., Yusupova, G. Z., Baucom, A.,
Microbiol., 4, 113!120, doi: 10.1038/nrmicro1340.
Lieberman, K., Earnest, T. N., Cate, J. H., and Noller, H. F.
7.
Williamson, K. S., Richards, L. A., Perez!Osorio, A. C.,
(2001) Crystal structure of the ribosome at 5.5 Å reso!
Pitts, B., McInnerney, K., Stewart, P. S., and Franklin, M. J.
lution, Science, 292, 883!896, doi: 10.1126/science.
(2012) Heterogeneity in Pseudomonas aeruginosa biofilms
1060089.
includes expression of ribosome hibernation factors in the
13. Ben!Shem, A., Garreau de Loubresse, N., Melnikov, S.,
antibiotic!tolerant subpopulation and hypoxia!induced
Jenner, L., Yusupova, G., and Yusupov, M. (2011) The
БИОХИМИЯ том 85 вып. 11 2020
1698
УСАЧЕВ и др.
structure of the eukaryotic ribosome at 3.0 Å resolution,
dimer from pathogenic Staphylococcus aureus, Nat. Comm.,
Science, 334, 1524!1529, doi: 10.1126/science.1212642.
8, 723, doi: 10.1038/s41467!017!00753!8.
14.
Laursen, B. S., Sorensen, H. P., Mortensen, K. K., and
30.
Polikanov, Y. S., Blaha, G. M., and Steitz, T. A. (2012)
Sperling!Petersen, H. U. (2005) Initiation of protein syn!
How hibernation factors RMF, HPF, and YfiA turn off pro!
thesis in bacteria, Microbiol. Mol. Biol. Rev., 69, 101!123,
tein synthesis, Science, 336, 915!918, doi: 10.1126/science.
doi: 10.1128/MMBR.69.1.101!123.2005.
1218538.
15.
Tollerson, R., 2nd, and Ibba, M. (2020) Translational reg!
31.
Khusainov, I., Fatkhullin, B., Pellegrino, S., Bikmullin, A.,
ulation of environmental adaptation in bacteria, J. Biol.
Liu, W. T., et al. (2020) Mechanism of ribosome shutdown
Chem., 295, 10434!10445, doi: 10.1074/jbc.REV120.
by RsfS in Staphylococcus aureus revealed by integrative
012742.
structural biology approach, Nat. Commun., 11, 1656,
16.
Korostelev, A. A. (2011) Structural aspects of translation
doi: 10.1038/s41467!020!15517!0.
termination on the ribosome, RNA, 17, 1409!1421,
32.
Yamagishi, M., Matsushima, H., Wada, A., Sakagami, M.,
doi: 10.1261/rna.2733411.
Fujita, N., and Ishihama, A. (1993) Regulation of the
17.
Pai, R. D., Zhang, W., Schuwirth, B. S., Hirokawa, G.,
Escherichia coli rmf gene encoding the ribosome modula!
Kaji, H., Kaji, A., and Cate, J. H. (2008) Structural
tion factor: growth phase! and growth rate!dependent con!
insights into ribosome recycling factor interactions with the
trol, EMBO J., 12, 625!630.
70S ribosome, J. Mol. Biol., 376, 1334!1347, doi: 10.1016/
33.
Shimada, T., Yoshida, H., and Ishihama, A.
(2013)
j.jmb.2007.12.048.
Involvement of cyclic AMP receptor protein in regulation
18.
Simonetti, A., Marzi, S., Jenner, L., Myasnikov, A.,
of the rmf gene encoding the ribosome modulation factor
Romby, P., Yusupova, G., Klaholz, B. P., and Yusupov, M.
in Escherichia coli, J. Bacteriol.,
195,
2212!2219,
(2009) A structural view of translation initiation in bacte!
doi: 10.1128/Jb.02279!12.
ria, Cell. Mol. Life Sci., 66, 423!436.
34.
Izutsu, K., Wada, A., and Wada, C. (2001) Expression of
19.
Nilsson, J., and Nissen, P. (2005) Elongation factors on the
ribosome modulation factor (RMF) in Escherichia coli
ribosome, Curr. Opin. Struc. Biol.,
15,
349!354,
requires ppGpp, Genes Cells, 6, 665!676, doi: 10.1046/
doi: 10.1016/j.sbi.2005.05.004.
j.1365!2443.2001.00457.x.
20.
Huter, P., Arenz, S., Bock, L. V., Graf, M., Frister, J. O., et
35.
Song, S., and Wood, T. K. (2020) ppGpp ribosome dimer!
al. (2017) Structural basis for polyproline!mediated ribo!
ization model for bacterial persister formation and resusci!
some stalling and rescue by the translation elongation fac!
tation, Biochem. Biophys. Res. Commun., 523, 281!286,
tor EF!P, Mol. Cell, 68, 515!527, doi: 10.1016/j.molcel.
doi: 10.1016/j.bbrc.2020.01.102.
2017.10.014.
36.
Sato, A., Watanabe, T., Maki, Y., Ueta, M., Yoshida, H.,
21.
Golubev, A. A., Validov, S. Z., Usachev, K. S., and Yusupov,
Ito, Y., Wada, A., and Mishima, M. (2009) Solution struc!
M. M. (2019) Elongation factor P: new mechanisms of
ture of the E. coli ribosome hibernation promoting factor
function and an evolutionary diversity of translation regu!
HPF: implications for the relationship between structure
lation, Mol. Biol.,
53,
501!512, doi:
10.1134/
and function, Biochem. Biophys. Res. Commun., 389, 580!
S0026893319040034.
585, doi: 10.1016/j.bbrc.2009.09.022.
22.
Zhou, D., Tanzawa, T., Lin, J., and Gagnon, M. G. (2020)
37.
Neira, J. L., Giudici, A. M., Hornos, F., Arbe, A., and
Structural basis for ribosome recycling by RRF and tRNA,
Rizzuti, B. (2018) The C terminus of the ribosomal!associ!
Nat. Struct. Mol. Biol., 27, 25!32, doi: 10.1038/s41594!
ated protein LrtA is an intrinsically disordered oligomer,
019!0350!7.
Int. J. Mol. Sci., 19, doi: 10.3390/ijms19123902.
23.
Seshadri, A., and Varshney, U. (2006) Mechanism of recy!
38.
Flygaard, R. K., Boegholm, N., Yusupov, M., and Jenner,
cling of post!termination ribosomal complexes in eubacte!
L. B. (2018) Cryo!EM structure of the hibernating
ria: a new role of initiation factor 3, J. Biosciences, 31, 281!
Thermus thermophilus 100S ribosome reveals a protein!
289, doi: 10.1007/Bf02703921.
mediated dimerization mechanism, Nat. Commun., 9,
24.
Udagawa, T., Shimizu, Y., and Ueda, T. (2004) Evidence
4179, doi: 10.1038/s41467!018!06724!x.
for the translation initiation of leaderless mRNAs by the
39.
Boerema, A. P., Aibara, S., Paul, B., Tobiasson, V.,
intact 70 S ribosome without its dissociation into subunits
Kimanius, D., et al. (2018) Structure of the chloro!
in eubacteria, J. Biol. Chem.,
279,
8539!8546,
plast ribosome with chl!RRF and hibernation!promoting
doi: 10.1074/jbc.M308784200.
factor, Nat. Plants, 4, 615!615, doi: 10.1038/s41477!018!
25.
Prossliner, T., Winther, K. S., Sorensen, M. A., and
0203!0.
Gerdes, K. (2018) Ribosome hibernation, Annu. Rev. Genet.,
40.
Usachev, K. S., Fatkhullin, B. F., Klochkova, E. A.,
52, 321!348, doi: 10.1146/annurev!genet!120215!035130.
Miftakhov, A. K., Golubev, A. A., et al.
(2020)
26.
McCarthy, B. J. (1960) Variation in bacterial ribosomes,
Dimerization of long hibernation promoting factor from
Biochim. Biophys. Acta, 39, 563!564.
Staphylococcus aureus: structural analysis and biochemical
27.
Beckert, B., Turk, M., Czech, A., Berninghausen, O.,
characterization, J. Struct. Biol., 209, 107408, doi: 10.1016/
Beckmann, R., et al. (2018) Structure of a hibernating
j.jsb.2019.107408.
100S ribosome reveals an inactive conformation of the
41.
Ueta, M., Yoshida, H., Wada, C., Baba, T., Mori, H., and
ribosomal protein S1, Nat. Microbiol., 3, 1115!1121,
Wada, A. (2005) Ribosome binding proteins YhbH and
doi: 10.1038/s41564!018!0237!0.
YfiA have opposite functions during 100S formation in the
28.
Khusainov, I., Vicens, Q., Ayupov, R., Usachev, K.,
stationary phase of Escherichia coli, Genes Cells, 10, 1103!
Myasnikov, A., et al. (2017) Structures and dynamics of
1112, doi: 10.1111/j.1365!2443.2005.00903.x.
hibernating ribosomes from Staphylococcus aureus mediat!
42.
Beckert, B., Abdelshahid, M., Schafer, H., Steinchen, W.,
ed by intermolecular interactions of HPF, EMBO J., 36,
Arenz, S., Berninghausen, O., Beckmann, R., Bange, G.,
2073!2087, doi: 10.15252/embj.201696105.
Turgay, K., and Wilson, D. N. (2017) Structure of the
29.
Matzov, D., Aibara, S., Basu, A., Zimmerman, E., Bashan,
Bacillus subtilis hibernating 100S ribosome reveals the basis
A., Yap, M. N. F., Amunts, A., and Yonath, A. E. (2017)
for
70S dimerization, Embo J.,
36,
2061!2072,
The cryo!EM structure of hibernating 100S ribosome
doi: 10.15252/embj.201696189.
БИОХИМИЯ том 85 вып. 11 2020
ГИБЕРНАЦИЯ - СТАДИЯ ФУНКЦИОНИРОВАНИЯ РИБОСОМ
1699
43.
Franken, L. E., Oostergetel, G. T., Pijning, T., Puri, P.,
quantitative mass spectrometry and electron microscopy
Arkhipova, V., Boekema, E. J., Poolman, B., and Guskov, A.
analysis of ribosomal 30S subunit assembly in E. coli, Elife,
(2017) A general mechanism of ribosome dimerization
3, e04491, doi: 10.7554/eLife.04491.
revealed by single!particle cryo!electron microscopy, Nat.
58.
Clatterbuck Soper, S. F., Dator, R. P., Limbach, P. A., and
Commun., 8, 722, doi: 10.1038/s41467!017!00718!x.
Woodson, S. A. (2013) In vivo X!ray footprinting of pre!
44.
McKay, S. L., and Portnoy, D. A. (2015) Ribosome hiber!
30S ribosomes reveals chaperone!dependent remodeling of
nation facilitates tolerance of stationary!phase bacteria to
late assembly intermediates, Mol. Cell, 52, 506!516,
aminoglycosides, Antimicrob. Agents Chemother., 59, 6992!
doi: 10.1016/j.molcel.2013.09.020.
6999, doi: 10.1128/AAC.01532!15.
59.
Wilson, D. N., and Nierhaus, K. H. (2007) The weird and
45.
Yusupov, M. M., and Spirin, A. S. (1986) Are there proteins
wonderful world of bacterial ribosome regulation, Crit. Rev.
between the ribosomal subunits? Hot tritium bombardment
Biochem. Mol. Biol.,
42,
187!219, doi:
10.1080/
experiments, FEBS Lett., 197, 229!233, doi: 10.1016/
10409230701360843.
0014!5793(86)80332!5.
60.
Huang, Y. J., Swapna, G. V., Rajan, P. K., Ke, H., Xia, B.,
46.
Link, A. J., Robison, K., and Church, G. M. (1997) Com!
Shukla, K., Inouye, M., and Montelione, G. T. (2003)
paring the predicted and observed properties of proteins
Solution NMR structure of ribosome!binding factor A
encoded in the genome of Escherichia coli K!12,
(RbfA), a cold!shock adaptation protein from Escherichia
Electrophoresis,
18,
1259!1313, doi:
10.1002/elps.
coli, J. Mol. Biol , 327, 521!536, doi: 10.1016/s0022!
1150180807.
2836(03)00061!5.
47.
Agafonov, D. E., Kolb, V. A., Nazimov, I. V., and Spirin, A. S.
61.
Rubin, S. M., Pelton, J. G., Yokota, H., Kim, R., and
(1999) A protein residing at the subunit interface of the
Wemmer, D. E. (2003) Solution structure of a putative
bacterial ribosome, Proc. Natl. Acad. Sci. USA, 96, 12345!
ribosome binding protein from Mycoplasma pneumoniae
12349, doi: 10.1073/pnas.96.22.12345.
and comparison to a distant homolog, J. Struct. Funct.
48.
Agafonov, D. E., Kolb, V. A., and Spirin, A. S. (2001)
Genom., 4, 235!243, doi: 10.1023/b:jsfg.0000016127.
Ribosome!associated protein that inhibits translation at
57320.82.
the aminoacyl!tRNA binding stage, EMBO Rep., 2, 399!
62.
Lovgren, J. M., Bylund, G. O., Srivastava, M. K.,
402, doi: 10.1093/embo!reports/kve091.
Lundberg, L. A., Persson, O. P., Wingsle, G., and
49.
Di Pietro, F., Brandi, A., Dzeladini, N., Fabbretti, A.,
Wikstrom, P. M. (2004) The PRC!barrel domain of the
Carzaniga, T., Piersimoni, L., Pon, C. L., and Giuliodori,
ribosome maturation protein RimM mediates binding to
A. M. (2013) Role of the ribosome!associated protein PY
ribosomal protein S19 in the 30S ribosomal subunits, RNA,
in the cold!shock response of Escherichia coli,
10, 1798!1812, doi: 10.1261/rna.7720204.
MicrobiologyOpen, 2, 293!307, doi: 10.1002/mbo3.68.
63.
Leong, V., Kent, M., Jomaa, A., and Ortega, J. (2013)
50.
Hauser, R., Pech, M., Kijek, J., Yamamoto, H., Titz, B., et
Escherichia coli rimM and yjeQ null strains accumulate
al. (2012) RsfA (YbeB) proteins are conserved ribosomal
immature 30S subunits of similar structure and protein
silencing factors, PLoS Genet.,
8, e1002815,
complement, RNA,
19,
789!802, doi:
10.1261/rna.
doi: 10.1371/journal.pgen.1002815.
037523.112.
51.
Wanschers, B. F. J., Szklarczyk, R., Pajak, A., van den
64.
Grishin, N. V. (2001) KH domain: one motif, two folds,
Brand, M. A. M., Gloerich, J., et al. (2012) C7orf30
Nucleic Acids Res., 29, 638!643, doi: 10.1093/nar/29.
specifically associates with the large subunit of the mito!
3.638.
chondrial ribosome and is involved in translation, Nucleic
65.
Anantharaman, V., and Aravind, L. (2002) The PRC!bar!
Acids Res., 40, 4040!4051, doi: 10.1093/nar/gkr1271.
rel: a widespread, conserved domain shared by photosyn!
52.
Jiang, M., Sullivan, S. M., Walker, A. K., Strahler, J. R.,
thetic reaction center subunits and proteins of RNA
Andrews, P. C., and Maddock, J. R. (2007) Identification
metabolism, Genome Biol.,
3, RESEARCH0061,
of novel Escherichia coli ribosome!associated proteins
doi: 10.1186/gb!2002!3!11!research0061.
using isobaric tags and multidimensional protein identifi!
66.
Guo, Q., Goto, S., Chen, Y., Feng, B., Xu, Y., Muto, A.,
cation techniques, J. Bacteriol.,
189,
3434!3444,
Himeno, H., Deng, H., Lei, J., and Gao, N. (2013)
doi: 10.1128/Jb.00090!07.
Dissecting the in vivo assembly of the 30S ribosomal sub!
53.
Feaga, H. A., Kopylov, M., Kim, J. K., Jovanovic, M., and
unit reveals the role of RimM and general features of the
Dworkin, J. (2020) Ribosome dimerization protects the
assembly process, Nucleic Acids Res., 41, 2609!2620,
small subunit, J. Bacteriol., 202, e00009!20, doi: 10.1128/
doi: 10.1093/nar/gks1256.
JB.00009!20.
67.
Berti, A. D., Shukla, N., Rottier, A. D., McCrone, J. S.,
54.
Traub, P., and Nomura, M. (1968) Structure and function
Turner, H. M., Monk, I. R., Baines, S. L., Howden, B. P.,
of E. coli ribosomes. V. Reconstitution of functionally active
Proctor, R. A., and Rose, W. E. (2018) Daptomycin selects
30S ribosomal particles from RNA and proteins, Proc. Natl.
for genetic and phenotypic adaptations leading to antibiot!
Acad. Sci. USA, 59, 777!784, doi: 10.1073/pnas.59.3.777.
ic tolerance in MRSA, J. Antimicrob. Chemother., 73, 2030!
55.
Mulder, A. M., Yoshioka, C., Beck, A. H., Bunner, A. E.,
2033, doi: 10.1093/jac/dky148.
Milligan, R. A., Potter, C. S., Carragher, B., and
68.
Yoshida, H., and Wada, A. (2014) The 100S ribosome:
Williamson, J. R. (2010) Visualizing ribosome biogenesis:
ribosomal hibernation induced by stress, Wiley Interdiscip.
parallel assembly pathways for the 30S subunit, Science,
Rev. RNA, 5, 723!732, doi: 10.1002/wrna.1242.
330, 673!677, doi: 10.1126/science.1193220.
69.
Ueta, M., Wada, C., Daifuku, T., Sako, Y., Bessho, Y., et al.
56.
Nord, S., Bhatt, M. J., Tukenmez, H., Farabaugh, P. J.,
(2013) Conservation of two distinct types of 100S ribosome in
and Wikstrom, P. M. (2015) Mutations of ribosomal pro!
bacteria, Genes Cells, 18, 554!574, doi: 10.1111/gtc.12057.
tein S5 suppress a defect in late!30S ribosomal subunit bio!
70.
Basu, A., Shields, K. E., and Yap, M. F. (2020) The hiber!
genesis caused by lack of the RbfA biogenesis factor, RNA,
nating 100S complex is a target of ribosome!recycling fac!
21, 1454!1468, doi: 10.1261/rna.051383.115.
tor and elongation factor G in Staphylococcus aureus, J.
57.
Sashital, D. G., Greeman, C. A., Lyumkis, D., Potter, C. S.,
Biol. Chem., 295, 6053!6063, doi: 10.1074/jbc.RA119.
Carragher, B., and Williamson, J. R. (2014) A combined
012307.
БИОХИМИЯ том 85 вып. 11 2020
1700
УСАЧЕВ и др.
HIBERNATION AS A STAGE OF RIBOSOME FUNCTIONING
Review
K. S. Usachev1, M. M. Yusupov1,2*, and Sh. Z. Validov1
1 Kazan Federal University, 420008 Kazan, Russia; E+mail: marat@igbmc.fr
2 Institute of Genetics and Molecular and Cellular Biology (IGBMC), 67400 Illkirch+Graffenstaden, France
Received July 30, 2020
Revised August 28, 2020
Accepted August 28, 2020
In response to stress, eubacteria reduce the level of protein synthesis and either disassemble ribosomes into the 30S
and 50S subunits or turn them into translationally inactive 70S and 100S complexes. This helps the cell to solve two
principal tasks: (i) to reduce the cost of protein biosynthesis under unfavorable conditions, and (ii) to preserve func!
tional ribosomes for rapid recovery of protein synthesis until favorable conditions are restored. All known genes for
ribosome silencing factors and hibernation proteins are located in the operons associated with the response to starva!
tion as one of the stress factors, which helps the cells to coordinate the slowdown of protein synthesis with the overall
stress response. It is possible that hibernation systems work as regulators that coordinate the intensity of protein syn!
thesis with the energy state of bacterial cell. Taking into account the limited amount of nutrients in natural conditions
and constant pressure of other stress factors, bacterial ribosome should remain most of time in a complex with the
silencing/hibernation proteins. Therefore, hibernation is an additional stage between the ribosome recycling and
translation initiation, at which the ribosome is maintained in a “preserved” state in the form of separate subunits, non!
translating 70S particles, or 100S dimers. The evolution of the ribosome hibernation has occurred within a very long
period of time; ribosome hibernation is a conserved mechanism that is essential for maintaining the energy! and
resource!consuming process of protein biosynthesis in organisms living in changing environment under stress condi!
tions.
Keywords: ribosome, translation, hibernation, silencing
БИОХИМИЯ том 85 вып. 11 2020