БИОХИМИЯ, 2020, том 85, вып. 11, с. 1701 - 1717
УДК 577.217.347;577.112.6;577.182.46;004.942
ВЗАИМОДЕЙСТВИЕ КАТИОННЫХ ПЕПТИДНЫХ АНАЛОГОВ
ХЛОРАМФЕНИКОЛА С РИБОСОМОЙ
© 2020
З.З. Хайруллина1#, А.Г. Терещенков2#, С.А. Завьялова3, Е.С. Комарова4,5,
Д.А. Лукьянов5, В.Н. Ташлицкий1, И.А. Остерман1,5, Н.В. Сумбатян1*
1 Московский государственный университет имени М.В. Ломоносова, химический факультет,
119991 Москва, Россия; электронная почта: sumbtyan@belozersky.msu.ru
2 НИИ физико)химической биологии имени А.Н. Белозерского, Московский государственный университет
имени М.В. Ломоносова, 119992 Москва, Россия
3 Институт биохимии имени А.Н. Баха, Федеральный исследовательский центр
«Фундаментальные основы биотехнологии» РАН, 119071 Москва, Россия
4 Московский государственный университет имени М.В. Ломоносова,
факультет биоинженерии и биоинформатики, 119992 Москва, Россия
5 Сколковский институт науки и технологий, 143025 Москва, Россия
Поступила в редакцию 13.07.2020
После доработки 07.09.2020
Принята к публикации 09.09.2020
На основе молекулярного докинга проведен виртуальный скрининг всех возможных трипептидных анало
гов хлорамфеникола по их аффинности к бактериальной рибосоме. Осуществлен синтез пептидных произ
водных хлорамфениколамина, показавших минимальные расчетные значения энергии взаимодействия с
рибосомой. Синтетическим путем получены производные хлорамфениколамина, содержащие в структуре
фрагменты антимикробных пролин богатых пептидов. По вытеснению флуоресцентно меченного эритро
мицина из его комплексов с рибосомами показано, что синтезированные пептидные аналоги хлорамфени
кола способны связываться с бактериальными рибосомами, все расчетные трипептидные аналоги хлорам
феникола и один из аналогов, содержащий характерный фрагмент из структуры пролин богатых антимик
робных пептидов, проявили аффинность к рибосомам Escherichia coli, значительно превышающую таковую
исходного антибиотика. Установлено наличие корреляции между рассчитанными и экспериментально по
лученными значениями эффективности лигандов. В экспериментах по ингибированию биосинтеза белка in
vitro показано, что аналог RAW CAM проявляет активность близкую к хлорамфениколу, что подтверждает
ся данными химического пробинга, указывающими на то, что по характеру связывания в рибосомном тун
неле этот аналог сходен с немодифицированным хлорамфениколом.
КЛЮЧЕВЫЕ СЛОВА: рибосома, хлорамфеникол, пептидные производные, молекулярный докинг, анти
микробные пептиды, рибосомный туннель.
DOI: 10.31857/S0320972520110123
ВВЕДЕНИЕ
Важными структурными элементами рибосомы
являются пептидилтрансферазный центр
В последние два десятилетия с появлением
(ПТЦ), катализирующий синтез полипептида, и
рентгеноструктурных данных и возможностью
рибосомный туннель (РТ), обеспечивающий
создания атомных моделей комплексов рибо
выход синтезируемой цепи из рибосомы [4-6].
сом с субстратами и белковыми факторами
Стенки РТ участвуют в мониторинге аминокис
трансляции начался новый этап в изучении ме
лотной последовательности перемещающегося
ханизмов трансляции и её регуляции [1-3].
вдоль них полипептида [4, 7, 8]. В РТ и ПТЦ
Принятые сокращения: Boc - трет бутилоксикарбонил; BODIPY - 5,7 диметил 4,4 дифтор 4 бора 3a,4a диаза s
индацен 3 пентановая кислота; CAM - хлорамфениколамин; CHL - хлорамфеникол; DIC - 1,3 диизопропилкарбоди
имид; DIPEA - диизопропилэтиламин; DMS - диметилсульфат; ERY - эритромицин; Fmoc - флуоренилметилоксикар
бонил; LC MS - хромато масс спектрометрия; MALDI TOF MS - времяпролетная масс спектрометрия с ионизацией
методом матричной десорбции; ONC - онкоцин112 (VDKPPYLPRPRPPRrIYNr NH2); Ρ - 2 хлортритильный полимер;
Pbf - 2,2,4,6,7 пентаметилдигидробензофуран 5 сульфонил; Pip - пиперидин; TFA - трифторуксусная кислота;
ДМФА - N,N диметилформамид; ПбАМП - пролин богатые антимикробные пептиды; ПТЦ - пептидилтрансферазный
центр; РТ - рибосомный туннель; Реагент К - TFA/фенол/вода/тиоанизол/β меркаптоэтанол = 82,5/5/5/5/2,5, v/v.
* Адресат для корреспонденции.
# Авторы внесли равный вклад в исследование.
1701
1702
ХАЙРУЛЛИНА и др.
расположены сайты связывания многих анти
Структуры пептидных аналогов хлорамфенико
биотиков, действие которых связано с подавле
ла были выбраны с использованием метода мо
нием процесса бактериальной трансляции, и,
лекулярного докинга с помощью двух подходов
как следствие этого, модификация нуклеотид
(рис. 1, а-д). Первый представлял виртуальный
ных остатков рРНК и аминокислотных остат
скрининг всех возможных трипептидных произ
ков белков, формирующих стенки РТ, приводит
водных хлорамфениколамина, моделирующих
к устойчивости бактерий к антибиотикам
3′ концевую область пептидил тРНК. Второй
[9-13]. РТ представляет собой динамическую
заключался в моделировании аналогов, в кото
систему, где осуществляются специфические
рых дихлорацетильный фрагмент хлорамфени
взаимодействия между пептидной цепочкой,
кола заменен на короткие последовательности,
антибиотиками и определенными структурны
характерные для ряда природных антимикроб
ми элементами рибосомы, воздействуя на её
ных пептидов: онкоцинов, металниковина, бак
функционирование в ряде случаев аллостери
теницина и их активных синтетических анало
чески [13-15].
гов [25, 26]. В работе измерены константы дис
Не так давно было показано, что элементы
социации комплексов полученных соединений
РТ способны взаимодействовать не только с
с рибосомами Escherichia coli; в экспериментах
вновь синтезируемой пептидной цепью, но и с
по ингибированию биосинтеза белка in vitro
некоторыми антимикробными пептидами, ко
найдены активные аналоги; методом химичес
торые попадают в него извне [16-20]. Эти анти
кого пробинга показано взаимодействие анало
бактериальные пептиды относятся к классу про
гов с нуклеотидами, образующими РТ; с по
лин богатых пептидов (ПбАМП), последова
мощью молекулярного докинга предсказаны
тельности которых обогащены содержанием ос
возможные модели взаимодействия новых со
татков аргинина, вследствие чего эти пептиды
единений с рибосомой.
также являются и катионными [21]. Механизм
действия ПбАМП определяется тем, что пепти
ды связываются в РТ бактерий, при этом, как
МАТЕРИАЛЫ И МЕТОДЫ
правило, их ориентация противоположна ори
ентации синтезируемой рибосомой полипеп
В работе были использованы следующие ре
тидной цепи - N конец располагается вблизи
агенты и растворители: хлорамфеникол
ПТЦ, а C конец направлен к выходу из РТ, пол
(«Sigma», Китай), производные L аминокислот
ностью его блокируя. Область связывания
и носитель для твердофазного синтеза, содержа
ПбАМП в РТ перекрывается с сайтами связыва
щий
2 хлортритилхлоридную группировку
ния большинства рибосомных антибиотиков, в
(2CTC Resin, «Iris Biotech», Германия), DCC
частности хлорамфеникола, нацеленных на
(1,3 дициклогексилкарбодиимид), DIPEA (ди
большую субъединицу рибосомы.
изопропилэтиламин), HBTU (O (бензотриазол
Необходимым условием проявления инги
1 ил) N,N,N′,N′ тетраметилуроний гексафтор
бирующих биосинтез белка свойств соединени
фосфат), HOBt (1 гидроксибензотриазол), N
ями, нацеленными на бактериальную рибосому,
гидроксисукцинимид, тиоанизол, 9 флуоренил
является их способность связываться с рибосо
метанол, янтарный ангидрид («Sigma Aldrich»,
мой в каком либо функциональном центре. Од
США, Швейцария, Япония), глутаровый ангид
нако не всегда высокая аффинность, например
рид («TCI», Япония), DIC (1,3 диизопропил
синтетических аналогов антибиотиков, приво
карбодиимид, «J&K Scientific», США), β мер
дит к их высокой ингибирующей активности
каптоэтанол («Ferak Berlin», Германия), TFA и
[22, 23]. Так, среди пептидных и аминокислот
абсолютные растворители: изопропанол, диок
ных аналогов хлорамфеникола были найдены
сан, ДМФА (N,N диметилформамид) и CH2Cl2
соединения, проявляющие сродство к рибосо
(«PanReac AppliChem», США), а также раство
мам на уровне исходного антибиотика, а в неко
рители российского производства фирм «Хим
торых случаях и значительно превышающее та
мед» и «Иреа2000». Флуоресцентное производ
ковое, и в то же время не воздействующих или
ное эритромицина BODIPY ERY было синтези
слабо воздействующих на процесс трансля
ровано по известной методике [27], Arg(Pbf)
ции [23, 24].
CAM [24] и N гидроксисукцинимидный эфир
В настоящей работе с целью выявления взаи
уксусной кислоты получали, как описано ранее
модействий и свойств, значимых для проявле
[28].
ния аффинности к рибосоме и ингибирующей
Хроматография. ТСХ проводили на пластин
трансляцию активности, осуществлен дизайн,
ках Kieselgel 60 F254 («Merck», Германия); для
синтез и изучение взаимодействий с рибосомой
колоночной хроматографии использовали Silica
новых пептидных аналогов хлорамфеникола.
gel
60
(0,063-0,200 мм и
0,04-0,063 мм,
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1703
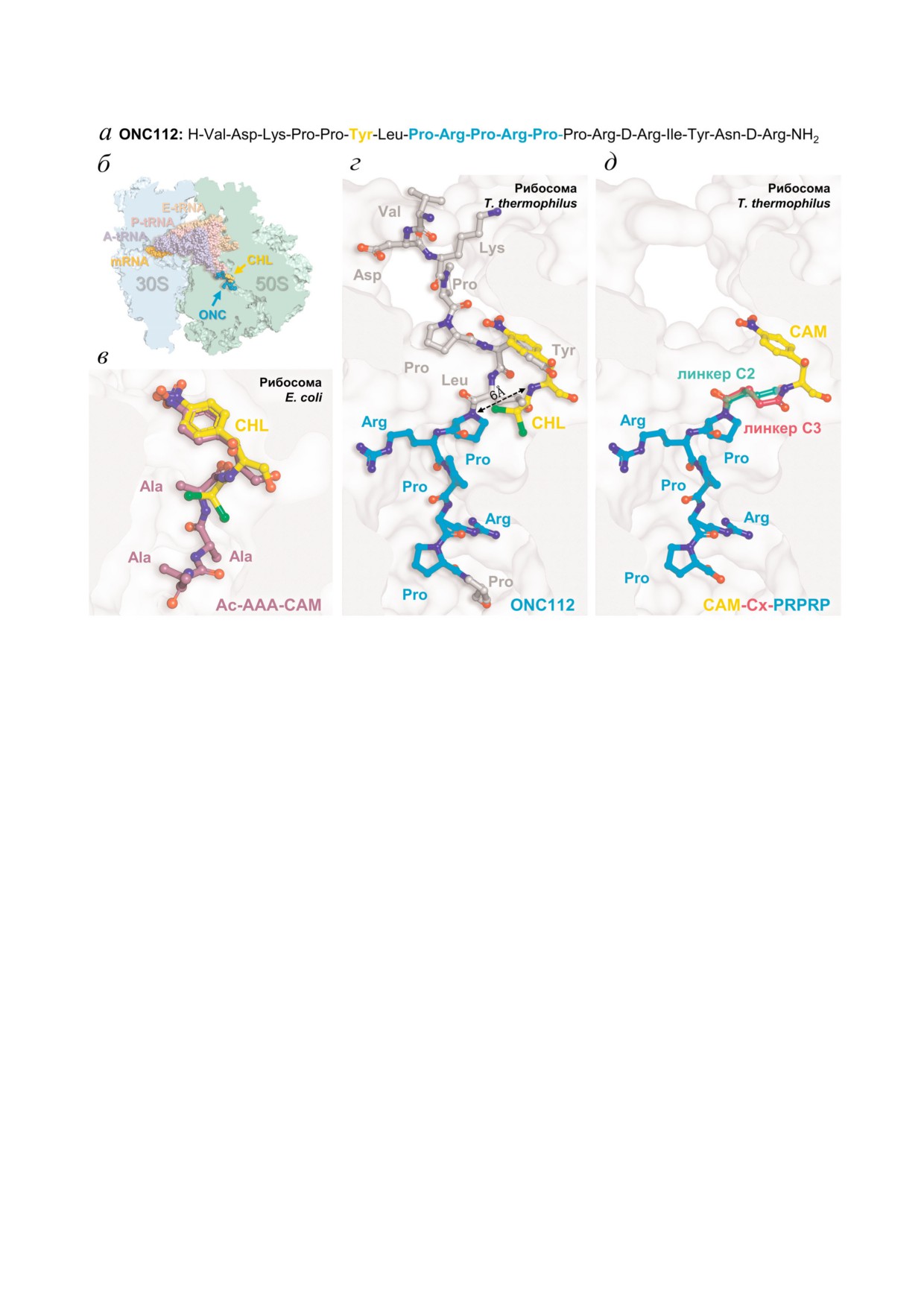
Рис. 1. Связывание хлорамфеникола, онкоцина112 и их производных с бактериальной рибосомой. а - Аминокислотная
последовательность антимикробного пептида онкоцина112. Желтым выделена аминокислота, попадающая в сайт связы
вания хлорамфеникола, голубым - пептидная последовательность, предложенная для модификации хлорамфеникола.
б - Схематичное отображение сайтов связывания хлорамфеникола (CHL, желтый) и онкоцина (ONC112, голубой) с ри
босомой Thermus thermophilus (PDB ID: 4Z8C) [16]. в - Наложение структуры комплекса хлорамфеникола (желтый) с ри
босомой E. coli (PDB ID: 4V7T) [15] и структуры пептидного аналога хлорамфеникола Ac AAA CAM (4a) (фиолетовый),
полученной методом молекулярного докинга. г - Наложение структуры комплекса хлорамфеникола (желтый, PDB ID:
6ND5 [38]) и онкоцина (серый и голубой, PDB ID: 4Z8C [16]) с рибосомой T. thermophilus. Голубым цветом отмечена пеп
тидная последовательность Pro8Arg9Pro10Arg11Pro12, предложенная для модификации хлорамфеникола. д - Модель конъ
югата хлорамфениколамина (CAM, желтый) с фрагментом онкоцина8-12 (голубой), соединенные линкерами разной дли
ны - бутандиовой (зеленый) и пентандиовой (розовый) кислотами в комплексе с рибосомой T. thermophilus (PDB
ID: 4Z8C) [16]
«Marcherey Nagel», Германия). Соединения, со
Препаративную ВЭЖХ проводили на хрома
держащие группы, поглощающие в УФ области,
тографе «Knauer», Германия, используя колонку
обнаруживали с помощью УФ кабинета
Nucleodur 100 5 C18 (5 мкм, 10 × 250 мм)
(«Camag», Англия); соединения, содержащие
(«Macherey Nagel», Германия) в градиенте
свободные либо трет бутилоксикарбонил за
0-60% ацетонитрила в 0,1 М ацетате аммония,
щищенные (Boc защищенные) аминогруппы,
скорость элюции - 5 мл/мин. Лиофилизацию
обнаруживали с помощью нингидринового ре
проводили на приборе «FreeZone 2.5 Liter Freeze
актива; для детекции соединений, содержащих
Dry System» («Labconco», Швейцария) при дав
остатки аргинина со свободной гуанидиновой
лении ~10-5 атм.
группой и триптофана, применяли реагенты Са
Спектры ЯМР соединений регистрировали на
кагучи и Эрлиха соответственно.
спектрометре Bruker Avance 400 («Bruker», Герма
Аминокислотный анализ осуществляли на
ния), рабочие частоты составляли 400 МГц - для
анализаторе «Hitachi 835», Япония. Кислотный
1H и 101 МГц - для 13C.
гидролиз проводили в запаянных ампулах
ХроматоBмассBспектрометрию (LCBMS) осу
смесью 6 М HCl и трифторуксусной кислот (2/1)
ществляли с помощью системы UPLC/MS/MS,
при 155 °C в течение 1 часа.
состоящей из хроматографа Acquity UPLC
БИОХИМИЯ том 85 вып. 11 2020
1704
ХАЙРУЛЛИНА и др.
System («Waters», США) и квадрупольного масс
са хлорамфеникола с рибосомой. Визуализацию
спектрометра TQD (регистрация сигналов с по
полученных данных проводили с помощью
мощью метода ESI MS; «Waters», США).
программы PyMol 2.4.
Спектры MALDIBTOF MS получали на
Синтез пептидных производных хлорамфеникоB
MALDI времяпролетном масс спектрометре
ламина. Синтез пептидов и их производных
«UltrafleXtreme», («Bruker Daltonik GmbH», Гер
1c-4c, 1d-4d, 1e-2e, 2f, флуоренилметилового эфи)
мания), оснащенном УФ лазером (Nd), в режи
ра бутандиовой (5d) и флуоренилметилового эфира
ме положительных ионов с использованием
пентандиовой кислот (5e), пептидил полимеров
рефлектрона.
ProArg(Pbf)Pro)P (5c), ProArg(Pbf)ProArg(Pbf)Pro)P
Поляризацию флуоресценции измеряли при
(6c), пентапептида Ac)ProArg(Pbf)ProArg(Pbf)Pro)
комнатной температуре с помощью планшетно
OH (7c) и модифицированного им β аланилхло
го мультиридера «VICTOR X5 Multilabel Plate
рамфениколамина Ac)ProArg(Pbf)ProArg(Pbf)Pro)
Reader» («Perkin Elmer», США) на 384 луночном
βAla)CAM (7a) описан в Приложении (http://
планшете. Длина волны поглощения составляла
485 нм, испускания - 535 нм.
Гидрохлорид (1R,2R))2)амино)1)(4)нитрофе)
Виртуальный скрининг и молекулярный доB
нил)пропан)1,3)диола (гидрохлорид хлорамфени)
кинг. Виртуальный скрининг проводили мето
коламина, CAM·HCl), 3)амино)N)[(1R,2R))1,3)
дом молекулярного докинга лигандов в жест
дигидрокси)1)(4)нитрофенил)пропан)2)ил]пропа)
кую структуру рецептора. Для этого структуры
намид (β)Ala)CAM, 7b), а также аргинин содер
пептидных аналогов хлорамфеникола были
жащее производное хлорамфениколамина
сгенерированы, исходя из SMILES формата
Arg(Pbf))CAM были синтезированы, согласно
записи молекул с помощью программы
описанным ранее в литературе методикам [33,
OpenBabel 2.3.2 [29]. Всего было рассмотрено
24].
10648 вариантов N ацетилированных трипеп
N)ацетил)L)фенилаланил)L)триптофил)L)
тидных аналогов хлорамфеникола, включаю
гистидил)[(1R,2R))2)гидрокси)2)(4)нитрофенил))
щих таутомерные формы гистидина и аргини
1)гидроксиметил)этил])амид (Ac)FWH)CAM, 1a).
на. Структуры лигандов, содержащие фрагмен
К холодному раствору 25,0 мг (48 мкмоль) Ac
ты онкоцина, дополнительно были оптимизи
PheTrpHis OH (1d) и 7,1 мг (62 мкмоль) HOSu
рованы полуэмпирическим методом в прог
(N гидроксисукцинимид) в 2 мл ДМФА (безв.)
рамме MOPAC2016 с использованием гамиль
добавляли 12,8 мг (62 мкмоль) DCC при 0 °C.
тониана PM7 [30]. В качестве рецептора ис
Смесь перемешивали в течение 2 ч при 0 °C, за
пользовали кристаллические структуры 70S
тем в течение ночи при 4 °C. Далее добавляли к
рибосомы E. coli в комплексе с хлорамфенико
раствору 11,9 мг (48 мкмоль) CAM·HCl и
лом (PDB ID: 4V7T) [15] и комплекс 70S рибо
8,3 мкл (48 мкмоль) DIPEA в 1 мл ДМФА. По
сомы Thermus thermophilus с онкоцином112
лученную смесь перемешивали в течение 7 ч
(PDB ID: 4Z8C) [16], из которых удаляли все
при комнатной температуре, затем в течение
неорганические ионы, низкомолекулярные ли
ночи при 4 °C. Реакционную смесь разбавляли
ганды и молекулы воды.
водой (15 мл) и экстрагировали этилацетатом
Молекулярный докинг выполняли с помощью
(3 × 5 мл). Органический слой сушили Na2SO4
программного обеспечения Autodock Vina [31] и
(безв.), а затем концентрировали на роторном
QuickVina 2 [32]. Область молекулярного докин
испарителе. Продукт очищали методом коло
га охватывала сайт связывания хлорамфеникола
ночной хроматографии на силикагеле в системе
и верхнюю часть РТ и описывалась кубом с дли
CHCl3/MeOH/19% NH4OH = 6/1/0,1. Выход:
ной ребра 30 Å (в случае трипептидных аналогов
4,7 мг (14%); ТСХ: Rf 0,24 (CHCl3/MeOH/19%
хлорамфеникола) и параллелепипедом величи
NH4OH = 6/1/0,1), Rf 0,51 (CHCl3/MeOH = 4/1);
ной 40 × 30 × 30 Å (в случае аналогов хлорамфе
LC MS, m/z вычислено для [C37H40N8O8 + H]+ -
никола, содержащих фрагменты онкоцина). Па
725,3; найдено 725,3; tR = 1,50 мин.
раметр «exhaustiveness», характеризующий точ
L)фенилаланил)L)триптофил)L)гистидил)
ность и полноту расчетов, был равен 100 при
[(1R,2R))2)гидрокси)2)(4)нитрофенил))1)гидрок)
скрининге трипептидных аналогов и 600 - для
симетил)этил])амид (FWH)CAM, 1b). Раствор,
соединений, содержащих фрагменты онкоцина.
состоящий из 825 мкл TFA, 50 мкл фенола,
Для каждого соединения были получены по 20
50 мкл воды, 50 мкл тиоанизола и 25 мкл β мер
возможных конформаций, характеризующихся
каптоэтанола (Реагент К), дегазировали с по
соответствующими энергиями взаимодействия.
мощью барботирования азота в течение нес
Дополнительно было рассчитано среднеквадра
кольких минут. Полученную смесь добавляли к
тичное отклонение амфеникольной части моле
10 мг (12,8 мкмоль) Boc PheTrpHis CAM (1c) и
кулы от рентгеноструктурных данных комплек
перемешивали в течение 4 ч при комнатной тем
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1705
пературе в атмосфере азота. Смесь упаривали на
16 мг
(97%); ТСХ (Al2O3): Rf 0,41 (CHCl3/
роторном испарителе, остаток растворяли в ми
MeOH/19% NH4OH = 6/1/0,1); LC MS, m/z вы
нимальном количестве воды и осаждали эфи
числено для [C29H42N8O7 + H]+ - 615,3; найдено
ром. Очистку проводили методом колоночной
615,2; tR = 0,80 мин.
хроматографии на силикагеле в системе
N)ацетил)L)аланил)L)аланил)L)аланил)
CHCl3/MeOH/19% NH4OH = 6/1/0,1. Выход:
[(1R,2R))2)гидрокси)2)(4)нитрофенил))1)гидрок)
6,4 мг (74%); ТСХ: Rf 0,26 (CHCl3/MeOH/19%
симетил)этил])амид (Ac)AAA)CAM, 4a) получали
NH4OH = 6/1/0,1); LC MS, m/z вычислено
подобно 3a, исходя из 7,5 мг (17,6 мкмоль) AAA
для [C35H38N8O7 + H]+ - 683,3; найдено 683,3;
CAM (4b), 3,0 мг (19,4 мкмоль) N гидроксисук
tR = 1,04 мин.
цинимидного эфира уксусной кислоты и 2,5 мкл
N)ацетил)L)аргинил)L)аланил)L)трипто)
(17,6 мкмоль) триэтиламина. Очистку проводи
фил)[(1R,2R))2)гидрокси)2)(4)нитрофенил))1)
ли методом колоночной хроматографии на си
гидроксиметил)этил])амид (Ac)RAW)CAM, 2a)
ликагеле в системе CH2Cl2/MeOH/19% NH4OH =
получали подобно
1b, исходя из
12,1 мг
= 65/25/4. Выход: 7,7 мг (94%); ТСХ: Rf 0,81
(13,2 мкмоль) Ac Arg(Pbf)AlaTrp CAM (2c) и
(CH2Cl2/MeOH/19% NH4OH
=
65/25/4);
500 мкл Реагента К. Очистку проводили мето
LC MS, m/z вычислено для [C20H29N5O8 + Na]+ -
дом колоночной хроматографии на силикагеле в
490,2; найдено 490,2; tR = 0,87 мин.
системе CHCl3/MeOH/19% NH4OH = 65/25/4.
L)аланил)L)аланил)L)аланил)[(1R,2R))2)гид)
Выход:
4,9 мг
(56%); ТСХ: Rf
0,09
рокси)2)(4)нитрофенил))1)гидроксиметил)
(CHCl3/MeOH/19% NH4OH = 65/25/4); LC MS,
этил])амид (AAA)CAM,
4b). К
19,5 мг
m/z вычислено для [C31H41N9O8 + H]+ - 668,3;
(37,1 мкмоль) Boc AlaAlaAla CAM (4c) добавля
найдено 668,4; tR = 1,00 мин.
ли 1 мл смеси TFA/CH2Cl2 (1/1) и перемешива
L)аргинил)L)аланил)L)триптофил)[(1R,2R))
ли 1 ч при комнатной температуре. Далее смесь
2)гидрокси)2)(4)нитрофенил))1)гидроксиметил)
упаривали на роторном испарителе, остаток
этил])амид (RAW)CAM, 2b) получали подобно
осаждали эфиром. Очистку проводили методом
1b, исходя из 15 мг (17,1 мкмоль) Arg(Pbf)AlaTrp
колоночной хроматографии на силикагеле в
CAM (2d) и 500 мкл Реагента К. Очистку прово
системе CHCl3/MeOH/19% NH4OH = 65/25/4.
дили методом колоночной хроматографии на
Выход:
13,1 мг
(83%); ТСХ: Rf
0,69
силикагеле в системе CHCl3/MeOH/19%
(CHCl3/MeOH/19% NH4OH = 65/25/4); ESI
NH4OH = 65/25/4. Выход: 4,6 мг (43%); ТСХ:
MS, m/z вычислено для [C18H27N5O7 + H]+ -
Rf 0,10 (CHCl3/MeOH/19% NH4OH = 65/25/4);
426,2; найдено 426,3.
LC MS, m/z вычислено для [C29H39N9O7 + H]+ -
N1)[(1R,2R))1,3)дигидрокси)1)(4)нитрофе)
626,3; найдено 626,3; tR = 0,80 мин.
нил)пропан)2)ил])N4)L)пролил)L)аргинил)L)про)
N)ацетил)L)валил)L)фенилаланил)L)арги)
лин)бутандиамид (CAM)C2)PRP, 5a). Флуоре
нил)[(1R,2R))2)гидрокси)2)(4)нитрофенил))1)
нилметиловый эфир бутандиовой (5d) кислоты
гидроксиметил)этил])амид (Ac)VFR)CAM, 3a). К
(3 экв.) и HBTU (3 экв.) перемешивали в ДМФА
раствору 9,5 мг (26,9 мкмоль) VFR CAM (3b) в
в течение 5 мин. Далее полученный активиро
1 мл ДМФА добавляли 4,2 мг (26,9 мкмоль) N
ванный эфир и DIPEA (3 экв.) добавляли к
гидроксисукцинимидного эфира уксусной кис
1 экв. пептидил полимера ProArg(Pbf)Pro Ρ (5c)
лоты и 4,7 мкл (26,9 мкмоль) DIPEA. Смесь пе
в сосуде Меррифилда. Перемешивали смесь
ремешивали 1 час при комнатной температуре, а
10 мин и добавляли DIPEA (0,6 экв.), затем про
затем ночь при 4 °C. Растворитель удаляли на
должали перемешивание в течение 5 ч. Далее
роторном испарителе. Очистку проводили мето
растворитель удаляли и промывали полимер,
дом колоночной хроматографии на оксиде алю
чередуя ДМФА (2 × 1 мин) и пропанол 2 (2 ×
миния в системе CHCl3/MeOH/19% NH4OH =
1 мин), после чего добавляли свежеприготов
= 65/25/4. Выход: 6,1 мг (35%); ТСХ (Al2O3):
ленный раствор для кэпирования (10 мл/г поли
Rf 0,40 (CHCl3/MeOH/19% NH4OH = 65/25/4);
мера): уксусный ангидрид/DIPEA/ДМФА =
LC MS, m/z вычислено для [C31H44N8O8 + H]+ -
= 5/6/89, v/v, выдерживали смесь в течение
657,3; найдено 657,2; tR = 1,44 мин.
5 мин, затем отфильтровывали полимер, зали
L)валил)L)фенилаланил)L)аргинил)[(1R,2R))
вали аналогичной порцией раствора для кэпи
2)гидрокси)2)(4)нитрофенил))1)гидроксиметил)
рования и выдерживали ещё 30 мин. Затем по
этил])амид (VFR)CAM, 3b) получали подобно 1b,
лимер отфильтровывали и промывали ДМФА
исходя из
26 мг
(26,9 мкмоль) Boc
(3 × 1 мин). Удаляли 9 флуоренилметильную
ValPheArg(Pbf)-CAM (3c) и 500 мкл Реагента К.
группу обработкой 20% ным раствором пипери
Очистку проводили методом колоночной хро
дина в ДМФА (2 × 15 мин), промывали полимер
матографии на оксиде алюминия в системе
ДМФА (3 × 1 мин). Далее добавляли раствор
CHCl3/MeOH/19% NH4OH = 6/1/0,1. Выход:
HOSu (3 экв.) и DIC (3 экв.) в ДМФА и переме
13 БИОХИМИЯ том 85 вып. 11 2020
1706
ХАЙРУЛЛИНА и др.
шивали 3 ч. Растворитель удаляли, добавляли к
но 900,6; аминокислотный анализ: Arg 2,00 (2),
полимеру раствор CAM·HCl (3 экв.) и DIPEA
Pro 3,22 (3).
(3 экв.) в ДМФА и перемешивали 3 ч. Далее син
N)ацетил)L)пролил)L)аргинил)L)пролил)L)ар)
тезированное производное хлорамфениколами
гинил)L)пролил)[(1R,2R))2)гидрокси)2)(4)нитрофе)
на отщепляли от полимерного носителя, соглас
нил))1)гидроксиметил)этил])амид (Ac)PRPRPβA)
но следующему протоколу: промывали последо
CAM, 7) получали аналогично 1b, исходя из 40 мг
вательно полимер ДМФА (3 × 1 мин) и CH2Cl2
(0,028 ммоль). Ac ProArg(Pbf)ProArg(Pbf)Pro β
(3 × 1 мин); добавляли Реагент К (10 мл/г поли
Ala CAM (7a) и 1 мл Реагента К. Выход: 21 мг
мера) и перемешивали в течение 4 ч; после чего
(80%); ТСХ: Rf 0,12 (CHCl3/MeOH/19% NH4OH =
полимер фильтровали и высаживали продукт из
=
65/25/5); LC MS, m/z вычислено для
фильтрата диэтиловым эфиром. Обработку по
[C41H64N14O11 + 2H]2+ - 465,3; найдено 465,7;
лимера Реагентом К и высаживание продукта
tR = 0,69 мин.
повторяли до полного отщепления продукта
Изучение связывания аналогов хлорамфениB
(контроль осуществляли по ТСХ). Выпавшие
кола с рибосомами E. coli. Аффинность соедине
осадки объединяли и подвергали очистке с по
ний к 70S рибосомам E. coli (штамм MRE 600)
мощью препаративной ВЭЖХ. Выход: 4,5 мг
определяли методом конкурентного связывания
(9%); ВЭЖХ: tR = 12,98 мин; MALDI TOF MS,
в присутствии флуоресцентного аналога эритро
m/z вычислено для [C29H43N8O10 - NO2 + NH
мицина - BODIPY ERY, как было описано ра
OH + H]+ - 649,3; найдено 649,5; m/z вычисле
нее [34, 24]. Концентрация BODIPY ERY сос
но для [C29H42N8O10 - NO2 + NH2 + H]+ - 633,3;
тавляла 4 нМ, рибосом - 30 нМ. Концентрации
найдено 633,5; аминокислотный анализ: Arg
исследуемых
веществ
варьировали
1,00 (1), Pro 1,51 (2).
(0,05-1000 мкМ). Смесь инкубировали 2 ч при
N1)[(1R,2R))1,3)дигидрокси)1)(4)нитрофе)
комнатной температуре, затем измеряли уро
нил)пропан)2)ил])N4)L)пролил)L)аргинил)L)про)
вень поляризации флуоресценции. Значения
лин)пентандиамид (CAM)C3)PRP, 5b) получали
поляризации флуоресценции были приведены к
аналогично 5a, исходя из пептидил полимера
относительной шкале, для чего за 100% был
ProArg(Pbf)Pro Ρ (5c) и флуоренилметилового
принят сигнал в отсутствие исследуемых соеди
эфира пентандиовой кислоты (5e). Выход: 4,1 мг
нений, а за 0% - при полном вытеснении BOD
(8%); ВЭЖХ: tR = 3,76 мин; LC MS, m/z вычис
IPY ERY избытком эритромицина. Для каждого
лено для [C30H44N8O10 + H]+ - 677,3; найдено
вещества проводили минимум по 2 повтора.
677,5; tR = 0,95 мин; аминокислотный анализ:
Расчет эффективных констант диссоциации
Arg 1,00 (1), Pro 2,11 (2).
проводили на основе стандартной модели, опи
N1)[(1R,2R))1,3)дигидрокси)1)(4)нитрофе)
сывающей равновесное конкурентное связыва
нил)пропан)2)ил])N4)L)пролил)L)аргинил)L)про)
ние двух лигандов в одном сайте [35].
лил)L)аргинил)L)пролин)бутандиамид (CAM)C2)
In vitro трансляция. Ингибирование синтеза
PRPRP, 6a) получали аналогично 5a, исходя из
белка в бесклеточной системе проводили при
пептидил полимера ProArg(Pbf)ProArg(Pbf)Pro
помощи набора PurExpress («NEB», США), сог
Ρ (6c) и флуоренилметилового эфира бутандио
ласно предложенному протоколу. В качестве
вой кислоты (5d). Выход: 3,8 мг (6%); ВЭЖХ:
матрицы для трансляции использовали 100 нг
tR = 16,24 мин; MALDI TOF MS, m/z вычислено
мРНК гена dhfR, предварительно полученной
для [C40H61N13O12 + H]+ - 916,5; найдено 916,6;
T7 транскрипцией (T7 Mega script, «Invitrogen»,
m/z вычислено для [C40H61N13O12 - NO2 + NH
США) с контрольной плазмиды набора
OH + H]+ - 902,5; найдено 902,6; m/z вычисле
PurExpress; реакцию проводили в 5 мкл в тече
но для [C40H61N13O12 - NO2 + NH2 + H]+ - 886,5;
ние часа. Для визуализации белка в геле добав
найдено 886,6; аминокислотный анализ: Arg
ляли в реакционную смесь BODIPY Met tRNA
2,00 (2), Pro 3,12 (3).
до 1 мкМ. Тестируемые антибиотики добавляли
N1)[(1R,2R))1,3)дигидрокси)1)(4)нитрофе)
в концентрации 30 мкМ. Реакции проводили в
нил)пропан)2)ил])N4)L)пролил)L)аргинил)L)про)
трех повторах и разделяли смеси в 12% ном
лил)L)аргинил)L)пролин)пентандиамид (CAM)
SDS геле, затем проводили детекцию сигнала
C3)PRPRP, 6b) получали аналогично 5a, исходя
BODIPY при помощи Typhoon phosphorimager
из пептидил полимера ProArgProArgPro P (6c) и
(«GE», США). Обсчёт сигнала проводили при
флуоренилметилового эфира пентандиовой
помощи ImageLab («Bio rad», США).
кислоты (5e). Выход: 3,2 мг (5%); ВЭЖХ:
Химический пробинг. Определение нуклеоти
tR = 19,67 мин; MALDI TOF MS, m/z вычислено
дов рРНК, взаимодействующих с антибиотика
для [C41H63N13O12 - NO2 + NH OH + H]+ -
ми, осуществляли при помощи анализа эффек
916,5; найдено
916,6; m/z вычислено для
тивности защиты от химической модификации.
[C41H63N13O12 NO2 + NH2 + H]+ - 900,5; найде
Образцы 70S рибосом E. coli в присутствии
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1707
30 мкМ тестируемых антибиотиков подвергали
Для дальнейшего синтеза были выбраны три
действию диметилсульфата (DMS), согласно
пептидные аналоги Ac FWH CAM (1a), Ac
классическому протоколу Moazed и Noller [36,
RAW CAM (2a), Ac VFR CAM (3a) (рис. 2), на
37], для обратной транскрипции был использо
ходящиеся среди 50 лучших производных, ран
ван олигонуклеотид, комплементарный участку
жированных по рассчитанной энергии взаимо
2,102-2,119 23S рРНК.
действия с рибосомой (табл. 1), а также их про
изводные со свободной N концевой аминогруп
пой: FWH CAM (1b), RAW CAM (2b), VFR
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
CAM (3b). Выбор данных соединений также был
обусловлен тем, что они содержат в своём соста
Виртуальный скрининг и молекулярный доB
ве либо положительно заряженную аминокис
кинг. Для отбора наиболее аффинных по отно
лоту Arg либо His, способный легко переходить
шению к рибосоме аналогов хлорамфеникола, в
в протонируемую форму при физиологических
структуре которых дихлорацетильная группа за
условиях. Триаланиновые аналоги Ac AAA
менена на трипептид, был проведен виртуаль
CAM (4a) и AAA CAM (4b) были предложены в
ный скрининг всех возможных комбинаций N
качестве контрольных соединений.
ацетилтрипептидных производных хлорамфе
Дизайн аналогов хлорамфеникола, содержа
николамина, исходя из 20 протеиногенных ами
щих фрагменты онкоцина, проводили на осно
нокислот. Ацетильная группа была введена для
вании наложения кристаллических структур
исключения возможного влияния концевой
хлорамфеникола (PDB ID: 6ND5) [38] и онко
аминогруппы и выявления взаимодействий,
цина112 (PDB ID: 4Z8C) [16] в комплексах с ри
специфичных для боковых радикалов амино
босомами T. thermophilus, ввиду отсутствия
кислот. Виртуальный скрининг проводили ме
структурных данных для комплекса рибосомы
тодом молекулярного докинга сгенерированных
E. coli с онкоцином. Принимая во внимание
лигандов в структуру рибосомы E. coli в комп
высокую консервативность фрагмента
23S
лексе с хлорамфениколом (PDB ID: 4V7T) [15].
рРНК, локализованного в области связывания
В результате было получено по 20 возможных
антибиотиков, а также сходство конформаций
конформаций для каждого из 8000 производных
хлорамфеникола, связанного с различными ри
и оценена их энергия взаимодействия с рибосо
босомами [15, 38], можно сделать предположе
мой. Отдельно была произведена оценка откло
ние об аналогичном взаимодействии моделиру
нения хлорамфеникольной части молекул от
емых аналогов как с рибосомами E. coli, так и с
рентгеноструктурных данных комплекса хло
T. thermophilus. Анализ наложения структур
рамфеникола с рибосомой. Во многих из полу
(рис. 1, г) показал, что аминогруппа хлорамфе
ченных конформаций нитрофенильный остаток
николамина находится на расстоянии 6 Å от α
мало смещается относительно соответствующей
иминогруппы Pro8 онкоцина. В качестве линке
кристаллической структуры хлорамфеникола, в
ров, соединяющих данные группы, были пред
то время как пептидная цепь направлена в сто
ложены линейные двухосновные кислоты, дли
рону РТ (рис. 1, в). Анализ результатов вирту
на которых была выбрана на основе результатов
ального скрининга (рис. S1 в Приложении) по
виртуального скрининга конъюгатов хлорамфе
казал, что наиболее выгодными аминокислота
николамина с фрагментами онкоцина (рис. S2,
ми в любом из положений по отношению к хло
a e в Приложении), в которых варьировалась
рамфениколамину являются содержащие аро
как длина цепи двухосновной кислоты (от про
матические группы Trp, Tyr, Phe, а также His.
пан до гександиовой кислоты), выступающей
в качестве линкера (рис. S2c в Приложении),
так и длина фрагментов онкоцина (рис. S2, d
Таблица 1. Полученные методом молекулярного докинга
в Приложении). В литературе имеются дан
энергии взаимодействия трипептидных аналогов хлорам
ные о том, что основной вклад в связывание
феникола с рибосомой E. coli
онкоцина с РТ вносят первые
12 а.о.
Соединение
Энергия связывания, ккал/моль
(Val1Asp2Lys3Pro4Pro5Tyr6Leu7Pro8Arg9Pro10Arg11Pro12)
[16, 17], поэтому рассматривались фрагменты
Ac FWH CAM (1a)
-14,1
онкоцина от одной (Pro8) до пяти
Ac RAW CAM (2a)
-12,3
(Pro8Arg9Pro10Arg11Pro12) аминокислот. Наиболь
шей по результатам молекулярного докинга аф
Ac VFR CAM (3a)
-12,3
финностью к рибосоме (рис. S2b в Приложе
Ac AAA CAM (4a)
-9,2
нии) обладали конъюгаты хлорамфениколами
Хлорамфеникол
-8,2
на с фрагментами онкоцина, в структуре кото
рых линкер содержал две (CAM C2 ONC8-12)
БИОХИМИЯ том 85 вып. 11 2020
13*
1708
ХАЙРУЛЛИНА и др.
или три метиленовые группы (CAM C3
которых были выбраны на основе виртуального
ONC8-12) (рис. 1, д). Для дальнейшего синтеза
скрининга: Ac FWH CAM (1a), Ac RAW CAM
были выбраны пептидные аналоги хлорамфени
(2a), Ac VFR CAM (3a) и Ac AAA CAM (4a), а
кола, в которых трипептид (Pro8Arg9Pro10) и пен
также их аналоги со свободной N концевой
тапептид (Pro8Arg9Pro10Arg11Pro12) соединены с
аминогруппой (FWH CAM (1b), RAW CAM
хлорамфениколамином через бутандиовую либо
(2b), VFR CAM (3b), AAA CAM (4b)). Схема
пентандиовую кислоты: CAM Cn PRP (n = 2,
синтеза соединений (1-4) включала гидролиз
5a, n = 3, 5b), CAM Cn PRPRP (n = 2, 6a, n = 3,
хлорамфеникола до хлорамфениколамина [33] и
6b) (рис. 2). Поскольку выбранные пептидные
ацилирование последнего с помощью защищен
последовательности являются палиндромными,
ных по боковым функциональным группам N
был также предложен аналог хлорамфеникола,
Boc , N Fmoc либо N ацетил пептидов с пос
содержащий в структуре пентапептидный фраг
ледующим удалением защитных групп (рис. 2).
мент с направлением цепи, обратным по отно
В случае соединений 2a, 3a и 4a ацетилирование
шению к последовательности онкоцина; в
трипептидных аналогов хлорамфеникола, со
структуре этого аналога аминогруппа хлорамфе
держащих свободную N концевую аминогруп
николамина соединена с карбоксильной груп
пу, проводили обработкой их N оксисукцини
пой С концевого пролина через остаток β ала
мидным эфиром уксусной кислоты. Соедине
нина: Ac PRPRPβA CAM (7) (рис. 2).
ния 3a и 3b получали присоединением к хлорам
Синтез. Были синтезированы новые пептид
фениколамину сукцинимидного эфира защи
ные аналоги хлорамфениколамина, структуры щенного по аминогруппе и боковой функции
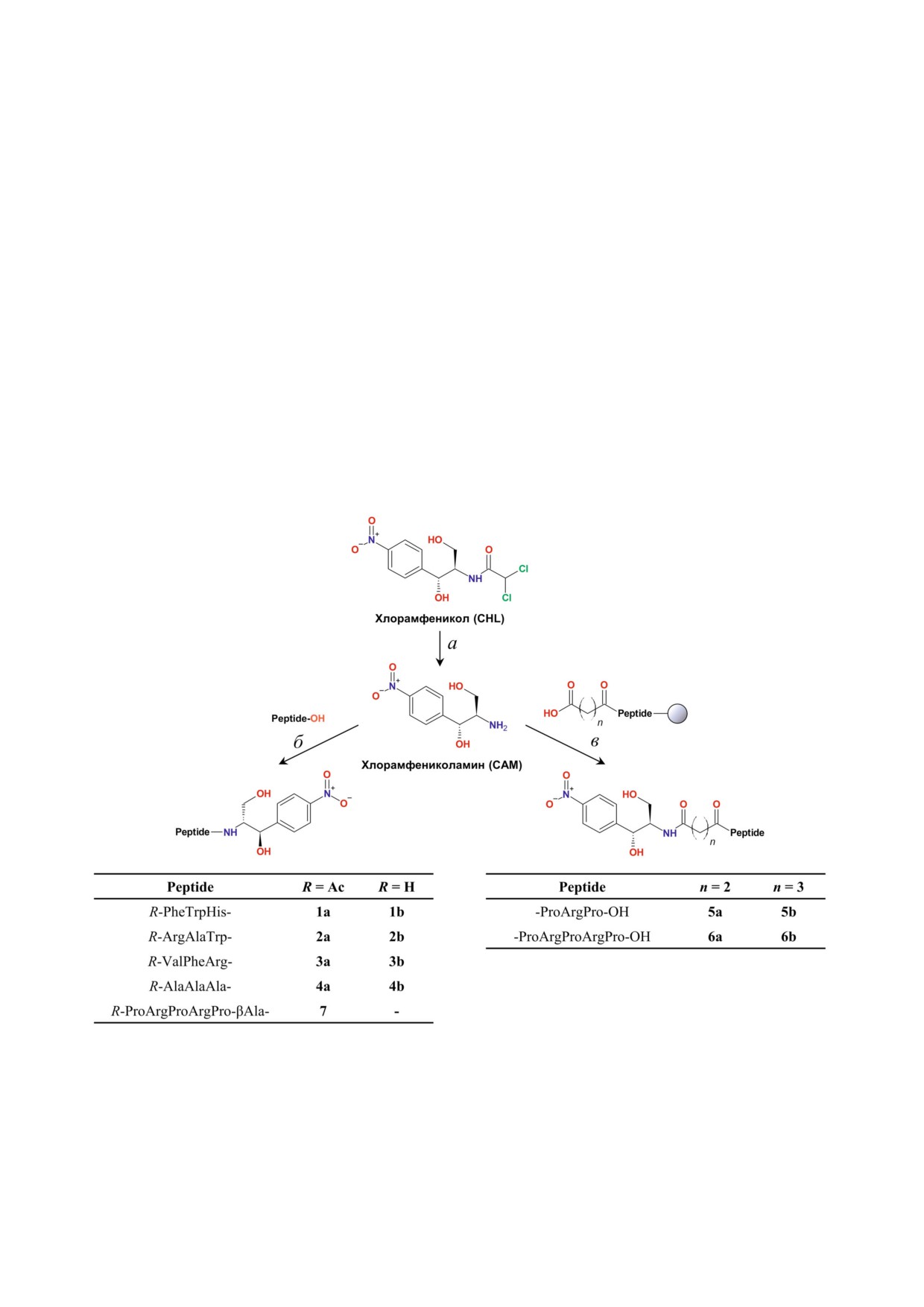
Рис. 2. Схема синтеза пептидных аналогов хлорамфеникола. а - 1M HCl, 100 °C; б - 1a: Ac PheTrpHis OSu; 1b: 1) Boc
PheTrpHis OSu, 2) Реагент К; 2a: 1) Fmoc Arg(Pbf)AlaTrp OSu, 2) Pip, 3) AcOSu, 4) Реагент К; 2b: 1) Fmoc Arg(Pbf)AlaTrp
OSu, 2) Pip, 3) Реагент К; 3a: 1) Fmoc Arg(Pbf) OSu, 2) Pip, 3) Boc ValPhe OSu, 4) Реагент К, 5) AcOSu; 3b: 1) Fmoc
Arg(Pbf) OSu, 2) Pip, 3) Boc ValPhe OSu, 4) Реагент К; 4a: 1) Boc AlaAlaAla OSu, 2) TFA, 3) AcOSu; 4b: 1) Boc AlaAlaAla
OSu, 2) TFA; в - 5a: 1) SuO C(O)(CH2)2C(O) ProArg(Pbf)Pro Ρ, 2) Реагент К; 5b: 1) SuO C(O)(CH2)3C(O) ProArg(Pbf)Pro
Ρ, 2) Реагент К; 6a: 1) SuO C(O)(CH2)2C(O) ProArg(Pbf)ProArg(Pbf)Pro Ρ, 2) Реагент К; 6b: 1) SuO C(O)(CH2)3C(O)
ProArg(Pbf)ProArg(Pbf)Pro Ρ, 2) Реагент К; 7: 1) Ac ProArg(Pbf)ProArg(Pbf)Pro OSu, 2) Реагент К
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1709
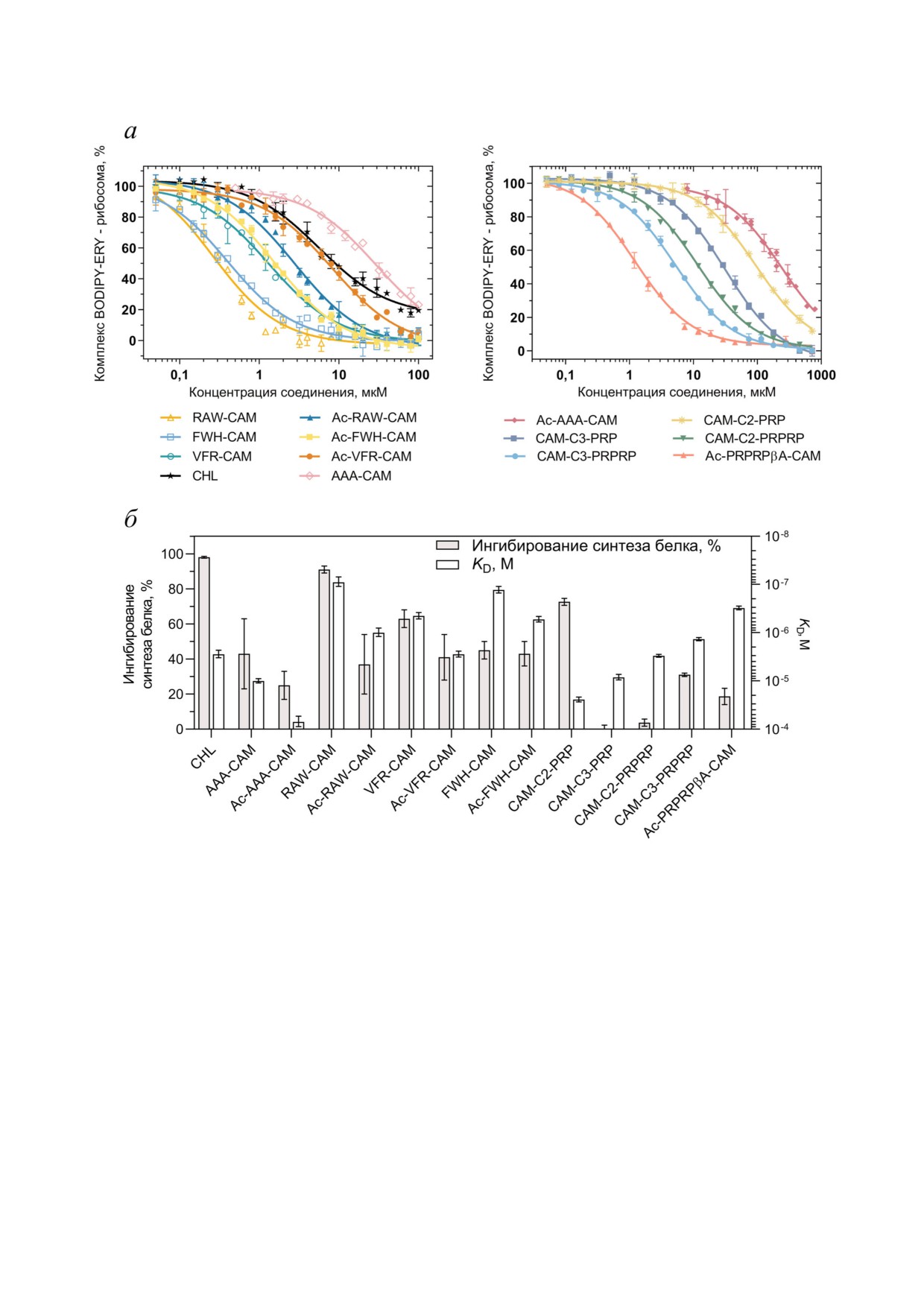
Рис. 3. Аффинность к рибосоме и ингибирующая активность пептидных аналогов хлорамфеникола. а - Конкурентное
связывание BODIPY ERY и пептидных аналогов хлорамфеникола с 70S рибосомами E. coli. Для каждой точки приведено
среднее значение со стандартным отклонением. б - Способность пептидных аналогов хлорамфеникола ингибировать
бактериальную трансляцию in vitro и эффективные константы диссоциации (KD) их комплексов с рибосомами. Приведе
ны средние значения с доверительным интервалом (α = 0,05)
аргинина, с последующим удалением Fmoc
не только пептидные фрагменты, но и пол
группы с его аминогруппы и присоединением к
ностью синтезировать конъюгаты хлорамфени
ней дипептида Boc ValPhe.
коламина с фрагментами онкоцина на твердо
Для получения производных хлорамфенико
фазном носителе (рис. 2 и рис. S3 в Приложе
ламина, содержащих в структуре характерные
нии). Синтез пептидов проводили согласно
последовательности из ПбАМП: CAM C2 PRP
стандартной Fmoc стратегии с использованием
(5a), CAM C3 PRP (5b), CAM C2 PRPRP (6a) и
Pbf группы для защиты боковой цепи аргинина.
CAM C3 PRPRP (6b), была разработана схема
После достижения необходимой длины пептид
твердофазного синтеза, позволяющая получить
ного фрагмента к нему присоединяли моно 9
БИОХИМИЯ том 85 вып. 11 2020
1710
ХАЙРУЛЛИНА и др.
Таблица 2. Эффективные константы диссоциации комплексов пептидных аналогов хлорамфеникола с рибосомами E. coli.
Приведены средние значения с доверительным интервалом (α = 0,05)
Соединение
KD, мкМ
Соединение
KD, мкМ
Ac FWH CAM (1a)
0,54 ± 0,06
FWH CAM (1b)
0,13 ± 0,02
Ac RAW CAM (2a)
1,0 ± 0,2
RAW CAM (2b)
0,09 ± 0,02
Ac VFR CAM (3a)
2,8 ± 0,4
VFR CAM (3b)
0,45 ± 0,07
Ac AAA CAM (4a)
80 ± 20
AAA CAM (4b)
10 ± 1
CAM C2 PRP (5a)
24 ± 3
CAM C3 PRP (5b)
8 ± 1
CAM C2 PRPRP (6a)
3,0 ± 0,2
CAM C3 PRPRP (6b)
1,4 ± 0,1
Ac PRPRPβA CAM (7)
0,31 ± 0,03
Хлорамфеникол
2,8 ± 0,5
флуоренилметиловый эфир бутандиовой (5d)
Согласно представленным результатам
либо пентандиовой кислоты (5e), которые полу
(табл. 2), все выбранные на основании вирту
чали заранее [39]. После удаления флуоренил
ального скрининга трипептидные аналоги хло
метиловой группировки с карбоксильной груп
рамфеникола проявляют бóльшую или сравни
пы последнюю активировали с помощью N
мую с хлорамфениколом аффинность к рибосо
гидроксисукцинимида и DIC, а затем присоеди
ме. Соединения со свободной N концевой ами
няли хлорамфениколамин [23], после чего де
ногруппой обладают в несколько раз большим
блокировали конъюгат с одновременным от
сродством к рибосоме по сравнению с их N аце
щеплением его от полимерного носителя.
тилированными производными; подобное явле
Для получения соединения Ac PRPRPβA
ние ранее наблюдалось для аминокислотных
CAM (7) хлорамфениколамин предварительно
аналогов хлорамфеникола [24]. Аффинность
модифицировали с помощью Boc β Ala, удаля
RAW CAM (2b) к рибосоме оказалась более чем
ли Boc группу [24, 40], а затем модифицировали
в 20 раз выше, чем у хлорамфеникола. В то же
по образовавшейся аминогруппе пептидом Ac
время трипептидные аналоги, выбранные в ка
ProArg(Pbf)ProArg(Pbf)Pro OH (7c) и удаляли
честве контроля - Ac AAA CAM (4a) и AAA
Pbf защиты с остатков аргинина (рис. 2). Синтез
CAM (4b), не показали значимого связывания в
пептида 7c был предварительно осуществлен на
области рибосомного туннеля.
твердой фазе, для сохранения защитных боко
Введение в структуру хлорамфениколамина
вых групп его отщепляли от 2 хлортритильного
фрагментов онкоцина в некоторых случаях так
полимера с применением гексафторизопропа
же привело к компенсации потери прочных взаи
нола (рис. S3 в Приложении) [41].
модействий, обусловленных дихлорацетильной
Изучение связывания аналогов хлорамфениB
группой [33, 38]. Производные хлорамфенико
кола с рибосомами E. coli. Аффинность получен
ламина, содержащие фрагменты онкоцина, свя
ных соединений к рибосоме была измерена с
занные через более длинный пентандиовый
помощью метода конкурентного связывания в
линкер (5b, 6b), связываются с рибосомой проч
присутствии флуоресцентно меченного произ
нее, чем соответствующие производные с бутан
водного эритромицина - BODIPY ERY, пос
диовым линкером (5a, 6a), а пентапептидные
кольку известно, что сайты связывания хлорам
аналоги (6a, 6b) прочнее, чем трипептидные (5a,
феникола и эритромицина перекрываются [38].
5b). Возможно, это объясняется тем, что более
Для всех изучаемых пептидных аналогов хло
длинный линкер обеспечивает бóльшую под
рамфеникола наблюдалось падение поляриза
вижность хлорамфеникольного и пептидного
ции флуоресценции при увеличении их концен
фрагментов в рибосомном туннеле, в то время
трации (рис. 3, а), что говорит о способности
как второй остаток аргинина в пентапептиде -
конъюгатов вытеснять BODIPY ERY из его сай
дополнительные взаимодействия с нуклеотида
та связывания, находящегося в рибосомном
ми РТ. Соединение 7, содержащее «переверну
туннеле. Нелинейный регрессионный анализ
тый» пептидный фрагмент, показало наиболь
полученных данных [35] позволил рассчитать
шую аффинность к бактериальным рибосомам
эффективные константы диссоциации (KD)
среди содержащих остатки онкоцина аналогов
комплексов аналогов хлорамфеникола с рибо
хлорамфеникола, в несколько раз превышаю
сомами (табл. 2).
щую таковую у хлорамфеникола.
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1711
Подтвержденное экспериментально пред
на немодифицированный хлорамфеникол, что
сказанное молекулярным моделированием вы
вместе с высокой аффинностью к рибосоме объ
сокое сродство полученных пептидных аналогов
ясняет наибольшую активность этого аналога. В
хлорамфеникола к бактериальным рибосомам
отличие от соединения 2b, другие трипептидные
рационализирует необходимость проверки их
аналоги хлорамфеникола (1b, 3b, 4b) вызывают
ингибирующей активности в отношении пепти
частичную защиту нуклеотида A2058. Связыва
дилтрансферазной реакции.
ние данных молекул, скорее всего, будет невоз
In vitro трансляция. Измерение ингибирую
можным по ходу трансляции, когда в туннеле
щей активности проводили в бесклеточной сис
будет располагаться растущий пептид, в отличие
теме трансляции мРНК ферментов дигидрофо
от хлорамфеникола, который может связывать
латредуктазы (DHFR) и люциферазы светлячка
ся в присутствии пептидил тРНК, вызывая тем
(Fluc). Оказалось, что, несмотря на прочное свя
самым арест трансляции [44].
зывание с бактериальной рибосомой, большин
В то же время их аффинность к соответству
ство производных ингибирует синтез белка сла
ющему сайту связывания, по видимому, недос
бее, чем хлорамфеникол (рис. 3, б). В то же вре
таточна, чтобы конкурировать с удлиняющейся
мя RAW CAM (2b) ингибирует сходным с хло
пептидной цепью в процессе элонгации, что
рамфениколом образом, а его ацетильное про
объясняет низкую эффективность этих произ
изводное (2a) уже такую активность не проявля
водных при подавлении трансляции в бескле
ет. Однако какая либо зависимость уровня ин
точной системе.
гибирования трансляции от наличия N конце
вой ацетильной группы либо от других особен
ностей структуры полученных соединений не
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
наблюдается. Как не обнаруживается и корреля
ции между значениями констант диссоциации
Ранее было показано, что трипептидные
комплексов аналогов хлорамфеникола с ри
аналоги хлорамфеникола, содержащие в струк
босомой и их ингибирующей активностью
туре последовательности известных «стоп» пеп
(рис. 3, б). Небольшая, но значимая корреляция
тидов, способны связываться с рибосомой E. coli
наблюдается лишь между уровнем ингибирова
на уровне близком к исходному антибиотику
ния синтеза белка исследуемыми соединениями
[23]. Было также показано, что аминокислотные
и их энергией взаимодействия с рибосомой,
аналоги хлорамфеникола, в которых дихлораце
приходящейся на один тяжелый (неводород
тильный фрагмент заменен на остаток амино
ный) атом (рис. S4a в Приложении) [42].
кислоты, способной протонироваться, также
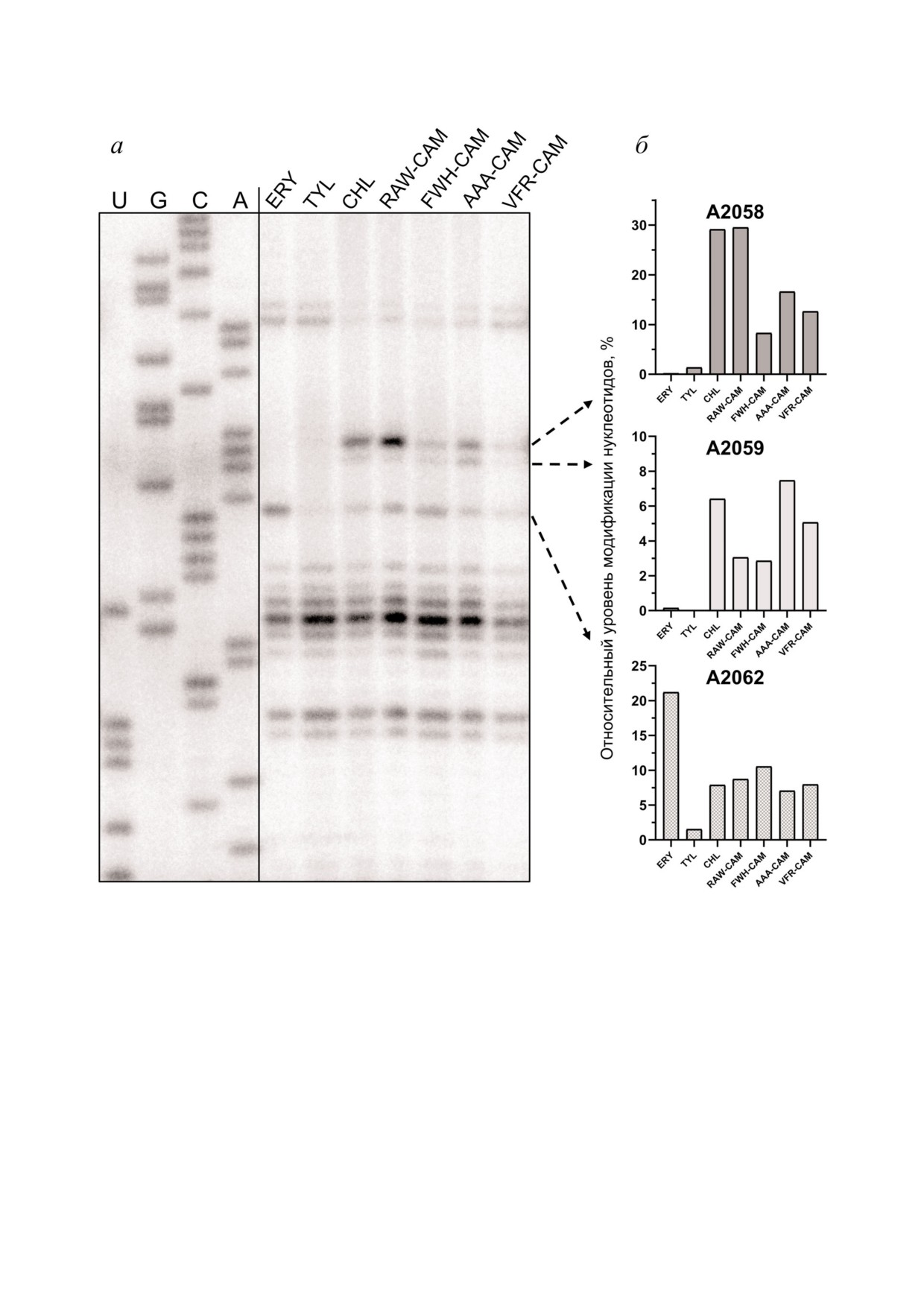
Химический пробинг. Трипептидные аналоги
проявляют высокую аффинность к бактериаль
хлорамфеникола со свободной N концевой ами
ным рибосомам, связываются с рибосомой в
ногруппой, проявившие значительную аффин
том же сайте, что и хлорамфеникол, при этом
ность к рибосоме, были исследованы на способ
заместитель при аминогруппе хлорамфеникола
ность непосредственного взаимодействия с нук
мина направлен в сторону выхода из РТ. Для
леотидами центральной петли домена V 23S
аминокислотных аналогов хлорамфеникола бы
рРНК рибосомы E. coli. Для этого комплексы
ло выявлено также отсутствие корреляций меж
рибосомы с аналогами хлорамфеникола, а также
ду способностью к связыванию с рибосомами и
с эритромицином, тилозином и хлорамфенико
их ингибирующей активностью in vitro [24]. Три
лом, взаимодействие которых с рибосомой хоро
пептидные аналоги хлорамфеникола, найден
шо изучено [43, 40], подвергали обработке DMS
ные в результате виртуального скрининга по их
и анализировали уровень модификации соответ
аффинности к бактериальной рибосоме и син
ствующих гетероциклических оснований, нор
тезированные в данном исследовании, все со
мировав его на степень модификации не вовле
держат в структуре а.о., способные заряжаться
ченных во взаимодействия с антибиотиками
положительно, а также гидрофобные а.о.
нуклеотидов (рис. 4, а, б). Все исследованные
(рис. 2 и 3, табл. 2).
трипептидные аналоги в той или иной мере вы
Подробный анализ результатов скрининга
зывают защиту нуклеотидов, выстилающих
показал преобладание ароматических амино
стенки рибосомного туннеля (A2058, A2059 и
кислот в структурах трипептидных аналогов
A2062), что согласуется со способностью данных
хлорамфеникола, обладающих наименьшей
производных вытеснять из него флуоресцент
рассчитанной энергией взаимодействия с рибо
ный аналог эритромицина BODIPY ERY.
сомой, независимо от модели их связывания
Уровень защит нуклеотидов РТ от модифи
(рис. S1 в Приложении). Несмотря на то, что ра
кации DMS указывает на то, что по характеру
нее была показана высокая аффинность гисти
связывания RAW CAM (2b) больше всего похож
динового аналога хлорамфеникола к бактери
БИОХИМИЯ том 85 вып. 11 2020
1712
ХАЙРУЛЛИНА и др.
Рис. 4. Модификация нуклеотидов центральной петли домена V 23S рРНК рибосомы E. coli с помощью DMS в присут
ствии антибиотиков эритромицина (ERY), тилозина (TYL), хлорамфеникола (CHL) и трипептидных аналогов хлорам
феникола: FWH CAM (1b), RAW CAM (2b), VFR CAM (3b), AAA CAM (4b). а - Электрофореграмма полученных в ре
зультате обратной транскрипции 23S рРНК олигонуклеотидов. б - Относительный уровень модификации нуклеотидов
A2058, A2059 и A2062, нормированный на степень модификации не вовлеченных во взаимодействия с антибиотиками
нуклеотидов
альной рибосоме [24], производные, содержа
жет также являться либо недоучет потери в кон
щие другие ароматические аминокислоты, не
формационной энтропии лигандов при связы
проявляли подобных свойств. Нельзя исклю
вании, либо переоценка вклада π π взаимодей
чать, что характер связывания пептидных ана
ствий в оценочной функции программного
логов отличен от аминокислотных производ
обеспечения, использующегося для молекуляр
ных, однако возможным объяснением наблюда
ного докинга. Поэтому для дальнейшего синте
емых результатов виртуального скрининга мо
тического получения были выбраны трипептид
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1713
ные аналоги, содержащие также положительно
плоскостей ароматических систем (рис. 5, б).
заряженные аминокислоты, конъюгаты кото
Кроме того, остатки фенилаланина и триптофа
рых с хлорамфениколамином показали ранее
на также образуют внутримолекулярные пер
высокое сродство к рибосоме [24]. В целом ре
пендикулярные π π взаимодействия. Остаток
зультаты виртуального скрининга для N ацетил
гистидина, находящийся около нуклеотидных
трипептидных производных хлорамфеникола
остатков A2058 и A2059, образует водородную
мина согласуются с экспериментально измерен
связь с O4′ G2505. В случае производного VFR
ной их аффинностью к рибосоме (табл. 1 и 2).
CAM (3b), кроме водородных связей с A2058,
Явный выпад энергии взаимодействия для не
A2062 и G2505, наблюдается наличие смещен
большой, по сравнению с трипептидными ана
ного стэкинга между гуанидиновой группой ар
логами, молекулы хлорамфеникола также мо
гинина и гетероциклическим основанием A2062
жет объясняться недостаточным негативным
(рис. 5, г), а также между бензольным кольцом
вкладом конформационной энтропии в оценоч
фенилаланина и основанием C2611. Несмотря
ную функцию программы, что приводит к силь
на отсутствие в структуре пептидного фрагмен
ному завышению результатов молекулярного
та соединения AAA CAM (4b) боковых радика
докинга с ростом размера молекулы. В то же
лов, содержащих полярные атомы, эта молекула
время при анализе энергий взаимодействия с
образует водородные связи с нуклеотидами РТ
рибосомой, приходящихся на один тяжелый
за счёт своего пептидного остова (рис. 5, в). Од
атом соединения (эффективностей лигандов)
нако отсутствие какого либо взаимодействия с
[42], рассчитанных для конформаций, средне
A2059, а также потенциально высокая подвиж
квадратичное отклонение хлорамфеникольной
ность молекулы, по видимому, приводят к сла
части молекулы в которых от соответствующих
бой его защите в случае химической модифика
рентгеноструктурных данных составляет менее
ции (рис. 4) и низкой аффинности соединения к
3 Å, наблюдается значимая корреляция с экспе
рибосоме. Защита от модификации A2062, наб
риментальными данными по эффективности
людаемая в химическом пробинге для всех сое
лигандов (рассчитанные из соответствующих
динений, видимо, в первую очередь обеспечива
KD) для всех полученных соединений (рис. S4b в
ется пептидным остовом молекулы. Во всех сое
Приложении).
динениях атомы кислорода амидной группы
Результаты молекулярного докинга (рис. 5)
пептидного фрагмента могут образовывать во
также согласуются с данными химического про
дородные связи с аминогруппой аденина A2062.
бинга трипептидных аналогов хлорамфеникола
Бóльшая аффинность пептидных аналогов со
(рис. 4) - пептидная часть молекул аналогов
свободной концевой аминогруппой, по види
1b-4b находится в непосредственной близости
мому, обеспечивается за счёт дополнительных
от нуклеотидов рибосомного туннеля A2058,
взаимодействий между этой группой и элемен
A2059 и A2062, что приводит к их частичной за
тами РТ. И если в случае 2b явно наблюдается
щите от химической модификации. Как видно
соответствующая водородная связь с U2506
из рис. 5, а, характер связывания RAW CAM
(рис. 5, а), то в других соединениях возможно
(2b) несколько отличается от остальных соеди
образование катион π взаимодействий с дан
нений. Подобно хлорамфениколу [15, 38], 2b не
ным нуклеотидом. Не исключено также, что,
взаимодействует с A2058, а образуя компактную
как и в случае гистидинового аналога хлорамфе
структуру, вступает в перпендикулярные π взаи
никола [24], реализуется каскад водородных
модействия с A2062 за счёт гуанидиновой груп
связей, образованных через молекулу воды.
пы аргинина, а также с G2505 за счёт остатка
Среди аналогов хлорамфеникола, содержа
триптофана. N Концевая аминогруппа данного
щих остатки антимикробного пептида онкоци
соединения, согласно результатам молекуляр
на (5a, 6a, 5b, 6b, 7), пентапептидное производ
ного докинга, образует водородную связь с
ное, содержащее линкер на основе пентандио
U2506. Судя по данным химического пробинга
вой кислоты, CAM C3 PRPRP (6b), и его палин
и молекулярного докинга, по механизму
дромный аналог Ac PRPRPβA CAM (7) прев
действия RAW CAM наиболее близок к хлорам
зошли по аффинности к рибосоме хлорамфени
фениколу, что, по видимому, и обуславливает
кол. Анализ результатов молекулярного докинга
его высокую ингибирующую активность
(рис. 6) показал, что структуры их комплексов с
(рис. 3, б).
рибосомой T. thermophilus, в которых амфени
Наличие ароматических аминокислот в
кольная часть молекулы находится в сайте свя
структуре FWH CAM (1b) приводит к их множе
зывания хлорамфеникола, отличаются от соот
ственному π π взаимодействию с гетероцикли
ветствующих рентгеноструктурных данных, по
ческими основаниями нуклеотидов U2585,
лученных для онкоцина [16]. Причинами этому
U2506, G2505, C2610 в различных ориентациях
могут быть как общие ограничения используе
БИОХИМИЯ том 85 вып. 11 2020
1714
ХАЙРУЛЛИНА и др.
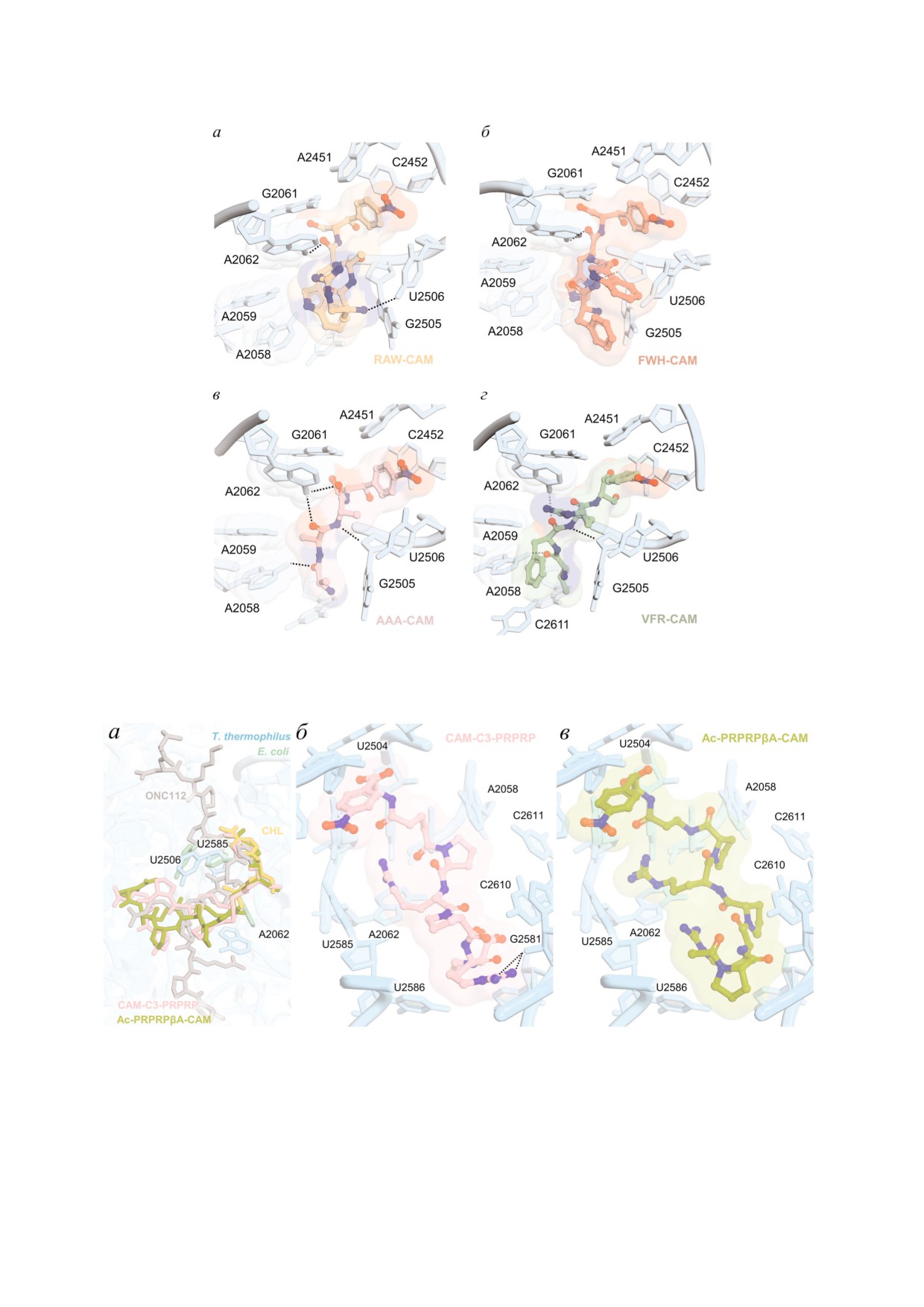
Рис. 5. Структуры комплексов трипептидных аналогов хлорамфеникола с рибосомой E. coli, полученные методом моле
кулярного докинга. а - RAW CAM (2b); б - FWH CAM (1b); в - AAA CAM (4b); г - VFR CAM (3b). Пунктирной линией
обозначены возможные водородные связи с нуклеотидами 23S рРНК
Рис. 6. Структуры комплексов пептидных аналогов хлорамфеникола, содержащих палиндромную последовательность
ProArgProArgPro, встречающуюся в антимикробном пептиде онкоцине, с рибосомой T. thermophilus (PDB ID: 4Z8C [16]),
полученные методом молекулярного докинга. Структуры хлорамфеникола (а - желтый цвет), онкоцина112 (а - серый),
аналога хлорамфеникола, содержащего фрагмент онкоцина Pro8Arg9Pro10Arg11Pro12, соединенный с хлорамфениколами
ном через пентандиовую кислоту: CAM C3 PRPRP (6b) (а и б - розовый) и аналога хлорамфеникола, содержащего «пе
ревернутый» фрагмент онкоцина Pro8Arg9Pro10Arg11Pro12, соединенный с хлорамфениколамином через β аланин: Ac
PRPRPβA CAM (7) (а и в - зеленый). Представлена нумерация нуклеотидов, характерная для рибосомы E. coli
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1715
мого метода молекулярного моделирования, так
аналогов, которая будет достаточна для конку
и тот факт, что нуклеотидные основания остат
ренции с растущей пептидной цепью в ходе
ков A2062, U2506 и U2585 по разному ориенти
трансляции и не будет связана с контекстной
рованы при связывании хлорамфеникола и он
специфичностью исходного антибиотика.
коцина.
В то же время, несмотря на противополож
Таким образом, в настоящей работе показа
ные направления пептидных цепей в аналогах
но, что виртуальный скрининг на основе моле
6b и 7, полученные для них конформации оказа
кулярного докинга позволяет предсказать отно
лись очень схожими. В обоих соединениях оста
сительную аффинность пептидных аналогов
ток аргинина, соответствующий положению 9 в
хлорамфеникола к бактериальной рибосоме,
последовательности онкоцина, взаимодействует
так же как и рациональный дизайн на основе из
с A2062, однако в отличие от антимикробного
вестных ингибиторов трансляции. Среди три
пептида его боковая цепь располагается с другой
пептидных аналогов хлорамфеникола катион
стороны относительно нуклеотида. В случае
ные аналоги способны связываться с рибосомой
производного 6b наблюдается возможность об
наиболее прочно, а экспериментально получен
разования водородной связи между остатком,
ные и рассчитанные значения эффективностей
моделирующим Arg11, и O2′ G2581. Предполага
лигандов коррелируют между собой. Аналог
емые взаимодействия могут быть причиной вы
RAW CAM в экспериментах по ингибированию
сокого сродства рассматриваемых аналогов к
биосинтеза белка in vitro проявляет активность,
рибосоме, но, как и в случае трипептидных ана
близкую к хлорамфениколу, что подтверждается
логов, не объясняют их низкую ингибирующую
данными химического пробинга, указывающи
активность в сравнении с хлорамфениколом.
ми на то, что по характеру связывания в РТ этот
Как следует из структур комплексов анало
аналог сходен с немодифицированным хлорам
гов хлорамфеникола 1b-4b (рис. 5) и 6b, 7
фениколом.
(рис. 6) с бактериальными рибосомами по ре
зультатам молекулярного докинга, а также, ис
ходя из данных химического пробинга, эти сое
Финансирование. Работа выполнена при фи
динения за счёт аминокислотных радикалов и
нансовой поддержке Российского фонда фунда
пептидного остова способны взаимодействовать
ментальных исследований (гранты № 20 04
с рядом нуклеотидных остатков РТ, важных для
00873 a (синтез аналогов, молекулярный до
регуляции и остановки трансляции [6, 7, 11, 16,
кинг, связывание с рибосомами) и № 19 34
17]. Несомненно, столь большое количество
51021 (in vitro трансляция, пробинг)). Работа вы
разнообразных взаимодействий аналогов с эле
полнена с использованием оборудования, при
ментами РТ может обеспечивать наблюдаемую
обретенного за счет средств Программы разви
высокую аффинность полученных соединений
тия Московского университета.
к рибосоме. Однако тот факт, что уровень инги
Благодарности. Мы благодарим А.А. Богда
бирования пептидилтрансферазной реакции
нова за инициирование и поддержку работ по
лишь для одного из них - RAW CAM (2b), дос
изучению пептидных производных рибосомных
тигает активности исходного антибиотика, хло
антибиотиков. Авторы также благодарят
рамфеникола, а также недавние исследования,
А.Л. Коневегу за предоставленные для работы
показавшие возможность связывания хлорам
рибосомы и BODIPY Met tRNA; М.В. Серебря
феникола с бактериальной рибосомой в присут
кову за масс спектрометрические анализы;
ствии растущей пептидной цепи [44-46], свиде
Ю.К. Гришина за ЯМР спектры.
тельствует о необходимости уточнения механиз
Конфликт интересов. Авторы заявляют об от
ма взаимодействия хлорамфеникола и его ана
сутствии конфликта интересов.
логов с функциональным комплексом рибосо
Соблюдение этических норм. Настоящая
мы в процессе трансляции.
статья не содержит описания выполненных ав
С другой стороны, проведение виртуального
торами исследований с участием людей или ис
скрининга производных хлорамфениколамина,
пользованием животных в качестве объектов.
основанного на имеющихся структурных дан
Дополнительные материалы. Приложение к
ных комплексов хлорамфеникола с рибосомой с
статье на английском языке опубликовано на
помощью их докинга в свободную рибосому, но
сайте журнала
«Biochemistry» (Moscow)
не ограниченного включениями в структуру
пептидных фрагментов, может позволить до
биться такой аффинности соответствующих
journal/10541), том 85, вып. 11, 2020.
БИОХИМИЯ том 85 вып. 11 2020
1716
ХАЙРУЛЛИНА и др.
СПИСОК ЛИТЕРАТУРЫ
1.
Ban, N., Nissen, P., Hansen, J., Moore, P. B., and Steitz, T. A.
the proline rich peptide oncocin, Nat. Struct. Mol. Biol.,
(2000) The complete atomic structure of the large riboso
22, 466 469, doi: 10.1038/nsmb.3031.
mal subunit at 2.4 Å resolution, Science, 289, 905 920,
17.
Seefeldt, A. C., Nguyen, F., Antunes, S., Pérébaskine, N.,
doi: 10.1126/science.289.5481.905.
Graf, M., et al. (2015) The proline rich antimicrobial pep
2.
Nissen, P., Hansen, J., Ban, N., Moore, P. B., and Steitz, T. A.
tide Onc112 inhibits translation by blocking and destabiliz
(2000) The structural basis of ribosome activity in peptide
ing the initiation complex, Nat. Struct. Mol. Biol., 22, 470
bond synthesis, Science, 289, 920 930, doi: 10.1126/science.
475, doi: 10.1038/nsmb.3034.
289.5481.920.
18.
Florin, T., Maracci, C., Graf, M., Karki, P., Klepacki, D.,
3.
Harms, J., Schluenzen, F., Zarivach, R., Bashan, A., Gat, S.,
et al. (2017) An antimicrobial peptide that inhibits trans
Agmon, I., Bartels, H., Franceschi, F., and Yonath, A.
lation by trapping release factors on the ribosome, Nat.
(2001) High resolution structure of the large ribosomal
Struct. Mol. Biol., 24, 752757, doi: 10.1038/nsmb.
subunit from a Mesophilic Eubacterium, Cell, 107, 679 688,
3439.
doi: 10.1016/s0092 8674(01)00546 3.
19.
Pérébaskine, N., Gambato, S., Mardirossian, M.,
4.
Bogdanov, A. A., Sumbatyan, N. V., Shishkina, A. V.,
Hofmann, S., Müller, C., et al. (2018) The dolphin pro
Karpenko, V. V., and Korshunova, G. A. (2010) Ribosomal
line rich antimicrobial peptide Tur1a inhibits protein syn
tunnel and translation regulation, Biochemistry (Moscow),
thesis by targeting the bacterial ribosome, Cell Chem. Biol.,
75, 1501 1516, doi: 10.1134/S0006297910130018.
25, 530 539, doi: 10.1016/j.chembiol.2018.02.004.
5.
Duc, K. D., Batr, S. S., Bhattacharya, N., Cate, J. H. D.,
20.
Gagnon, M. G., Roy, R. N., Lomakin, I. B., Florin, T.,
and Song, Y. S. (2019) Differences in the path to exit the
Mankin, A. S., and Steitz, T. A. (2016) Structures of pro
ribosome across the three domains of life, Nucleic Acids
line rich peptides bound to the ribosome reveal a common
Res., 47, 4198 4210, doi: 10.1093/nar/gkz106.
mechanism of protein synthesis inhibition, Nucleic Acids
6.
Gupta, P., Liu, B., Klepacki, D., Gupta, V., Sschulten, K.,
Res., 44, 2439 2450, doi: 10.1093/nar/gkw018.
Mankin, A., and Vázquez Laslop, N. (2016) Nascent pep
21.
Kumar, P., Kizhakkedathu, J., and Straus, S.
(2018)
tide assists the ribosome in recognizing chemically distinct
Antimicrobial peptides: diversity, mechanism of action and
small molecules, Nat. Chem. Biol.,
12,
153158,
strategies to improve the activity and biocompatibility in
doi: 10.1038/nchembio.1998.
vivo, Biomolecules, 8, 4 27, doi: 10.3390/biom8010004.
7.
Wilson, D. N., Arenz, S., and Beckmann, R. (2016)
22.
Mamos, P., Krokidis, M. G., Papadas, A., Karahalios, P.,
Translation regulation via nascent polypeptide mediated
Starosta, A. L., Wilson, D. N., Kalpaxis, D. L., and Dinos, G. P.
ribosome stalling, Curr. Opin. Struct. Biol., 37, 123 133,
(2013) On the use of the antibiotic chloramphenicol to tar
doi: 10.1016/j.sbi.2016.01.008.
get polypeptide chain mimics to the ribosomal exit tunnel,
8.
Gamerdinger, M., Kobayashi, K., Wallisch, A., Kreft, S. G.,
Biochimie, 95, 17651772, doi: 10.1016/j.biochi.2013.
Sailer, C., et al. (2019) Early scanning of nascent polypep
06.004.
tides inside the ribosomal tunnel by NAC, Mol. Cell, 75,
23.
Tereshchenkov, A. G., Shishkina, A. V., Tashlitsky, V. N.,
996 1006, doi: 10.1016/j.molcel.2019.06.030.
Korshunova, G. A., Bogdanov, A. A., Sumbatyan, N. V.
9.
Hansen, J. L., Moore, P. B., and Steitz, T. A. (2003)
(2016) Interaction of chloramphenicol tripeptide ana
Structures of five antibiotics bound at the peptidyl trans
logues with the ribosome, Biochemistry (Moscow), 81, 392
ferase center of the large ribosomal subunit, J. Mol. Biol.,
400, doi: 10.1134/S000629791604009X.
330, 1061 1075, doi: 10.1016/s0022 2836(03)00668 5.
24.
Tereshchenkov, A. G., Dobosz Bartoszek, M., Osterman,
10.
LaMarre, J., Mendes, R. E., Szal, T., Schwarz, S., Jones, R. N.,
I. A., Marks, J., Sergeeva, V. A., et al. (2018) Binding
and Mankin, A. S. (2013) The genetic environment of the
action of amino acid analogues of chloramphenicol upon
cfr gene and the presence of other mechanisms account for
the bacterial ribosome, J. Mol. Biol., 430, 842852,
the very high linezolid resistance of Staphylococcus epider)
doi: 10.1016/j.jmb.2018.01.016.
midis isolate 426 3147L, Antimicrob. Agents Chemother., 57,
25.
Graf, M., Mardirossian, M., Nguyen, F., Seefeldt, A. C.,
1173 1179, doi: 10.1128/AAC.02047 12.
Guichard, G., Scocchi, M., Innis, C. A., Wilson, D. N.
11.
Vázquez Laslop, N., Ramu, H., Klepacki, D., and
(2017) Proline rich antimicrobial peptides targeting pro
Mankin, A. S. (2010) The key function of a conserved and
tein synthesis, Nat. Prod. Rep., 34, 702 711, doi: 10.1039/
modified rRNA residue in the ribosomal response to the
C7NP00020K.
nascent peptide, EMBO J., 29, 3108 3117, doi: 10.1038/
26.
Knappe, D., Ruden, S., Langanke, S., Tikkoo, T., Ritzer, J.,
emboj.2010.180.
Mikut, R., Martin, L. L., Hoffmann, R., and Hilpert, K.
12.
Polikanov, Y. S., Aleksashin, N. A., Beckert, B., and
(2016) Optimization of oncocin for antibacterial activity
Wilson, D. N. (2018) The mechanisms of action of ribo
using a SPOT synthesis approach: extending the pathogen
some targeting peptide antibiotics, Front. Mol. Biosci., 5,
spectrum to Staphylococcus aureus, Amino Acids, 48, 269
48, doi: 10.3389/fmolb.2018.00048.
280, doi: 10.1007/s00726 015 2082 2.
13.
Arenz, S., Meydan, S., Starosta, A. L., Berninghausen, O.,
27.
Li, J., Kim, I. H., Roche, E. D., Beeman, D., Lynch, A. S.,
Beckmann, R., Vázquez Laslop, N., and Wilson, D. N.
Ding, C. Z., and Ma, Z. (2006) Design, synthesis, and bio
(2014) Drug sensing by the ribosome induces translational
logical evaluation of BODIPY®-erythromycin probes for
arrest via active site perturbation, Mol. Cell, 56, 446 452,
bacterial ribosomes, Bioorg. Med. Chem. Lett., 16, 794 797,
doi: 10.1016/j.molcel.2014.09.014.
doi: 10.1016/j.bmcl.2005.11.028.
14.
Arenz, S., Ramu, H., Gupta, P., Berninghausen, O.,
28.
Ji, J., Chakraborty, A., Geng, M., Zhang, X., Amini, A.,
Beckmann, R., Vázquez Laslop, N., Mankin, A. S., and
Bina, M., and Regnier, F. (2000) Strategy for qualitative
Wilson, D. N. (2014) Molecular basis for erythromycin
and quantitative analysis in proteomics based on signature
dependent ribosome stalling during translation of the
peptides, J. Chromatogr. B, 745, 197 210, doi: 10.1016/
ErmBL leader peptide, Nat. Commun., 5, 3501 3516,
s0378 4347(00)00192 4.
doi: 10.1038/ncomms4501.
29.
O’Boyle, N. M., Banck, M., James, C. A., Morley, C.,
15.
Dunkle, J. A., Xiong, L., Mankin, A. S., and Cate, J. H. D.
Vandermeersch, T., and Hutchison, G. R. (2011) Open
(2010) Structures of the Escherichia coli ribosome with
babel: an open chemical toolbox, J. Cheminformatics, 3, 33,
antibiotics bound near the peptidyl transferase center
doi: 10.1186/1758 2946 3 33.
explain spectra of drug action, Proc. Natl. Acad. Sci. USA,
30.
Stewart, J. J. P. (2012) Optimization of parameters for semi
107, 17152 17157, doi: 10.1073/pnas.1007988107.
empirical methods VI: more modifications to the NDDO
16.
Roy, R. N., Lomakin, I. B., Gagnon, M. G., and Steitz, T. A.
approximations and re optimization of parameters, J. Mol.
(2015) The mechanism of inhibition of protein synthesis by
Model., 19, 1 32, doi: 10.1007/s00894 012 1667 x.
БИОХИМИЯ том 85 вып. 11 2020
КАТИОННЫЕ ПЕПТИДНЫЕ АНАЛОГИ ХЛОРАМФЕНИКОЛА
1717
31.
Trott, O., and Olson, A. J. (2010) AutoDock Vina: improv
R. D. (2003) Process for preparing peptide intermediates,
ing the speed and accuracy of docking with a new scoring
European Patent Office, EP1226158A1.
function, efficient optimization, and multithreading, J.
40. Kostopoulou, O., Kourelis, T., Mamos, P., Magoulas, G.,
Comput. Chem., 31, 455 461, doi: 10.1002/jcc.21334.
and Kalpaxis, D. (2011) Insights into the chloramphenicol
32.
Alhossary, A., Handoko, S. D., Mu, Y., and Kwoh, C. K.
inhibition effect on peptidyl transferase activity, using two
(2015) Fast, accurate, and reliable molecular docking with
new analogs of the drug, Open Enzym. Inhib. J., 411, 1 10,
QuickVina 2, Bioinformatics, 31, 2214 2216, doi: 10.1093/
doi: 10.2174/1874940201104010001.
bioinformatics/btv082.
41. Bollhagen, R., Schmiedberger, M., Barlos, K., and Grell, E.
33.
Rebstock, M. C., Crooks, H. M., Controulis, J., and Bartz,
(1994) A new reagent for the cleavage of fully protected
Q. R. (1949) Chloramphenicol (chloromycetin). IV.
peptides synthesised on 2 chlorotrityl chloride resin, J.
Chemical studies, J. Am. Chem. Soc., 71, 2458 2462,
Chem. Soc. Chem. Commun., 22, 2559 2560, doi: 10.1039/
doi: 10.1021/ja01175a065.
C39940002559.
34.
Yan, K., Hunt, E., Berge, J., May, E., Copeland, R. A., and
42. Kuntz, I. D., Chen, K., Sharp, K. A., and Kollman, P. A.
Gontarek, R. R. (2005) Fluorescence polarization method
(1999) The maximal affinity of ligands, Proc. Natl. Acad.
to characterize macrolide ribosome interactions, Antimicrob.
Sci. USA, 96, 9997 10002, doi: 10.1073/pnas.96.18.9997.
Agents Chemother., 49, 3367 3372, doi: 10.1128/AAC.49.8.
43. Poulsen, S. M., Kofoed, C., and Vester, B.
(2000)
3367 3372.2005.
Inhibition of the ribosomal peptidyl transferase reaction by
35.
Wang, Z. X. (1995) An exact mathematical expression for
the mycarose moiety of the antibiotics carbomycin, spi
describing competitive binding of two different ligands to a
ramycin and tylosin, J. Mol. Biol.,
304,
471481,
protein molecule, FEBS Lett., 360, 111 114, doi: 10.1016/
doi: 10.1006/jmbi.2000.4229.
0014 5793(95)00062 e.
44. Choi, J., Marks, J., Zhang, J., Chen, D. H., Wang, J.,
36.
Moazed, D., and Noller, H. F. (1989) Interaction of tRNA
Vázquez Laslop, N., Mankin, A. S., and Puglisi, J. D.
with 23S rRNA in the ribosomal A, P, and E sites, Cell, 57,
(2019) Dynamics of the context specific translation arrest
585 597, doi: 10.1016/0092 8674(89)90128 1.
by chloramphenicol and linezolid, Nat. Chem. Biol., 16,
37.
Moazed, D., and Noller, H. F. (1989) Intermediate states
310 317, doi: 10.1038/s41589 019 0423 2.
in the movement of transfer RNA in the ribosome, Nature,
45. Marks, J., Kannan, K., Roncase, E. J., Klepacki, D., Kefi, A.,
342, 142 148, doi: 10.1038/342142a0.
Orelle, C., Vázquez Laslop, N., and Mankin, A. S. (2016)
38.
Svetlov, M. S., Plessa, E., Chen, C. W., Bougas, A.,
Context specific inhibition of translation by ribosomal
Krokidis, M. G., Dinos, G. P., and Polikanov, Y. S. (2019)
antibiotics targeting the peptidyl transferase center, Proc.
High resolution crystal structures of ribosome bound
Natl. Acad. Sci. USA, 113, 12150 12155, doi: 10.1073/
chloramphenicol and erythromycin provide the ultimate
pnas.1613055113.
basis for their competition, RNA,
25,
600606,
46. Makarov, G. I., and Makarova, T. M. (2018) A noncanon
doi: 10.1261/rna.069260.118.
ical binding site of chloramphenicol revealed via molecular
39.
Ashwood, M. S., Bishop, C. B., Cottrell, I. F., Emerson, K. M.,
dynamics simulations, Biochim. Biophys. Acta Gen. Subj.,
Hands, D., Ho, G. J., Lynch, J. E., Shi, Y. J., and Wilson,
1862, 2940 2947, doi: 10.1016/j.bbagen.2018.09.012.
INTERACTION OF CHLORAMPHENICOL CATIONIC PEPTIDE
ANALOGUES WITH THE RIBOSOME
Z. Z. Khairullina1#, A. G. Tereshchenkov2#, S. A. Zavyalova3, E. S. Komarova4,5,
D. A. Lukianov5, V. N. Tashlitsky1, I. A. Osterman1,5, and N. V. Sumbatyan1*
1 Department of Chemistry, Lomonosov Moscow State University, 119991 Moscow, Russia;
E)mail: sumbtyan@belozersky.msu.ru
2 Belozersky Institute of Physico)Chemical Biology, Lomonosov Moscow State University, 119992 Moscow, Russia
3 Bach Institute of Biochemistry, Federal Research Center of Biotechnology
of the Russian Academy of Sciences, 119071 Moscow, Russia
4 Faculty of Bioengineering and Bioinformatics, Lomonosov Moscow State University, 119992 Moscow, Russia
5 Skolkovo Institute of Science and Technology, 143025 Moscow, Russia
Received July 13, 2020
Revised September 7, 2020
Accepted September 9, 2020
Virtual screening of all possible tripeptide analogues of chloramphenicol was performed using molecular docking to
evaluate their affinity to bacterial ribosomes. Chloramphenicol analogues that demonstrated the lowest calculated
energy of interaction with ribosomes were synthesized. Chloramphenicol amine (CAM) derivatives, which contained
specific peptide fragments from the proline rich antimicrobial peptides were produced. It was demonstrated using dis
placement of the fluorescent erythromycin analogue from its complex with ribosomes that the novel peptide analogues
of chloramphenicol were able to bind bacterial ribosome; all the designed tripeptide analogues and one of the chlo
ramphenicol amine derivatives containing fragment of the proline rich antimicrobial peptides exhibited significantly
greater affinity to Escherichia coli ribosome than chloramphenicol. Correlation between the calculated and experi
mentally evaluated levels of the ligand efficiencies was observed. In vitro protein biosynthesis inhibition assay revealed,
that the RAW CAM analogue shows activity at the level of chloramphenicol. These data were confirmed by the chem
ical probing assay, according to which binding pattern of this analogue in the nascent peptide exit tunnel was similar
to chloramphenicol.
Keywords: ribosome, chloramphenicol, peptide derivatives, molecular docking, antimicrobial peptides, nascent pep
tide exit tunnel
БИОХИМИЯ том 85 вып. 11 2020