БИОХИМИЯ, 2020, том 85, вып. 11, с. 1718 - 1728
УДК 577.217.347;57.052.2;544.165;51 76
ИССЛЕДОВАНИЕ АЛЛОСТЕРИЧЕСКОГО ЭФФЕКТА
2,8 ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК E. coli МЕТОДОМ
МОЛЕКУЛЯРНО ДИНАМИЧЕСКОГО МОДЕЛИРОВАНИЯ
© 2020
Т.М. Макарова*, Г.И. Макаров
Южно Уральский государственный университет (национальный исследовательский университет),
454080 Челябинск, Россия; электронная почта: makarovatm@susu.ru
Поступила в редакцию 16.06.2020
После доработки 01.10.2020
Принята к публикации 02.10.2020
Рибосома - это молекулярная машина, синтезирующая все клеточные белки. Она также является мишенью
около половины клинически используемых антибиотиков. Один из способов защиты от антибиотиков зак"
лючается в адаптивной химической модификации остатков рибосомальных РНК. Любопытным примером
такой модификации служит 2,8"диметилирование А2503 23S рРНК, вызывающее устойчивость к фенико"
лам, линкозамидам, оксазолидинонам, плевромутилинам и некоторым макролидам. В настоящей работе
методом молекулярно"динамического моделирования исследовалось, как 2,8"диметилирование A2503 влия"
ет на конформации и подвижность нуклеотидных остатков 70S рибосом Escherichia coli. Значительные из"
менения были выявлены как в непосредственном окружении остатка А2503 23S рРНК, так и глубже в рибо"
сомном туннеле (РТ) среди остатков, о которых известно, что они вовлечены в процесс передачи сигнала от
антибиотиков, связывающихся в РТ, к пептидилтрансферазному центру. Эти изменения смещают рибосому
в сторону состояния А/А,Р/Р из конформационно иного состояния, в нашем примере - это состояние
Р/Р,Е/Е. Это позволяет заключить, что эффект модификации m2m8A2503 состоит в дополнительной стаби"
лизации состояния А/А,Р/Р, способствующего пептидилтрансферазной реакции (ПТР) в противополож"
ность действию антибиотиков - ингибиторов ПТР.
КЛЮЧЕВЫЕ СЛОВА: рибосома, устойчивость, антибиотики, A2503, молекулярная динамика.
DOI: 10.31857/S0320972520110135
ВВЕДЕНИЕ
ка на рибосоме. Тем не менее число патогенных
микроорганизмов, устойчивых к антибиотикам,
Рибосома - это крупный рибонуклеопроте"
связывающимся с рибосомой, растёт с каждым
идный комплекс, который синтезирует все кле"
годом [2, 3].
точные белки по программе, доставляемой мат"
Один из механизмов устойчивости бактери"
ричной РНК. Будучи одним из ключевых эле"
альной рибосомы к действующим на неё анти"
ментов живой клетки, эта древняя молекуляр"
биотикам состоит в химической модификации
ная машина содержит универсально консерва"
остатков рРНК, локализованных преимущест"
тивные структурные элементы, построенные из
венно в функциональных центрах рибосомы [4],
рибосомной РНК (рРНК), которые в природ"
и эти модификации, как правило, заключаются
ных условиях сравнительно редко подвергаются
в дополнительном неконститутивном метили"
мутациям [1]. Это обстоятельство, наряду с при"
ровании того или иного остатка рРНК [5]. При"
сутствием множества копий рибосомальной
мер этого - метилирование остатка A2503 (здесь
РНК в геномах живых организмов, сделало ри"
и ниже используется нумерация, принятая для
босому удобной мишенью для разнообразных
рРНК Escherichia coli) 23S рРНК большой субъ"
антибиотиков [2]. Примерно половина антибио"
единицы бактериальной рибосомы по положе"
тиков, используемых сегодня в клинической
нию С8, осуществляемое метилтрансферазой
практике, являются ингибиторами синтеза бел"
Cfr [6], впервые обнаруженной в Staphylococcus
sciuri [7], относящейся к семейству так называе"
мых радикальных SAM"метилтрансфераз
Принятые сокращения: МД - молекулярная дина" (SAM - S"аденозил"L"метионин) [8]. В рибосо"
мика; ПТР - пептидилтрансферазная реакция; ПТЦ -
ме бактерий дикого типа остаток A2503 подвер"
пептидилтрансферазный центр; РТ - рибосомный тун"
нель; рРНК - рибосомная РНК; RMSD - среднеквадрати"
жен конститутивному метилированию по поло"
ческое отклонение от заданной структуры.
жению С2, осуществляемому метилтрансфера"
* Адресат для корреспонденции.
зой RlmN; совместное действие этих метил"
1718
АЛЛОСТЕРИЧЕСКИЙ ЭФФЕКТ 2,8"ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК
1719
трансфераз приводит к диметилированию A2503
ному туннелю А751. Все эти остатки, согласно
и превращению его в m2m8A2503.
экспериментальным данным, участвуют в ряде
Дополнительная химическая модификация
случаев передачи сигнала из РТ в ПТЦ при оста"
приводит к устойчивости бактерий к антибиоти"
новке трансляции антибиотиками и так называ"
кам разных семейств (фениколам, линкозами"
емыми стоп"пептидами (stalling peptides), и они
дам, оксазолидинонам, плевромутилинам и
связаны с m2m8A2503 через сеть нековалентных
стрептограминам А), связывающимся с пепти"
взаимодействий.
дилтрансферазным центром (ПТЦ) рибосомы и
примыкающей к нему верхней части рибосом"
ного туннеля (РТ). Данный тип устойчивости
МАТЕРИАЛЫ И МЕТОДЫ
именуется PhLOPSa резистентностью по первым
буквам названий перечисленных семейств. По"
Моделируемая система. Исходным конструк"
явление у рибосомы столь ярко выраженной
том для моделируемых систем была выбрана
множественной лекарственной устойчивости,
структура рибосомы E. coli, полученная методом
как правило, объясняют тем, что дополнитель"
криоэлектронной микроскопии с разрешением
ная метильная группа в m2m8A2503 создаёт пря"
от 2,9 Å (код PDB: 5AFI) [10]. В эту структуру
мые стерические затруднения для связывания
комплекса рибосомы с деацилированной
того или иного антибиотика со своим сай"
E-тРНКfMet, fMet-P-tRNAfMet и Phe-A/T-тРНКPhe
том [10]. Впрочем, необходимо заметить, что
в составе тройного комплекса с EF"Tu, GTP и
пространственная структура рибосомы с диме"
кирромицином добавили недостающие моди"
тилированным остатком А2053 неизвестна, а де"
фицированные основания в соответствии со
тальный структурный анализ комплексов моди"
сведениями из банка данных [16].
фицированной таким образом рибосомы с анти"
Ранее нами уже был описан протокол полу"
биотиками до сих пор не проводился.
чения из структуры 5AFI комплексов рибосомы,
Консервативный нуклеотидный остаток
стабильно и воспроизводимо связывающих
m2A2503 соединяет два одноцепочечных тяжа
тРНК как в А/А" и Р/Р"сайтах, так и в Р/Р" и
рРНК, формирующих так называемые стенки
Е/Е"сайтах [17]. Упомянем лишь, что этот про"
туннеля. При этом остаток m2A2503 вклинива"
токол содержит минимум три коротких расчета
ется в цепочку нуклеотидов между A2059, с од"
МД с поэтапным снятием позиционных ограни"
ной стороны, и G2061 - с другой, в то время как
чений с различных частей системы. Системы с
A2060 выпетливается из стопки. Эта цепочка из
m2m8A2503 были сделаны на их основе, и расчё"
остатков 2058-2062 23S рРНК, как считается,
ту МД предшествовала минимизация энергии
образует путь передачи сигналов из внешних
сочетанием метода наискорейшего спуска с ал"
сайтов РТ в ПТЦ [11-13].
горитмом Бройдена-Флетчера-Гольдфарба-
Для изучения воздействия диметилирования
Шанно с ограниченным использованием памя"
A2503 на структуру рибосомы мы задействовали
ти [18].
метод моделирования молекулярной динамики
Условия моделирования. Все расчёты МД и
(МД), успешно применяемый для описания ди"
анализ полученных траекторий проводили с ис"
намических аспектов структуры и функциони"
пользованием пакета GROMACS [19, 20] версии
рования рибосомы [13]. Было исследовано влия"
5.1.4 и силового поля AMBER"14SB [21].
ние 8"метилирования m2A2503 как на А/А,Р/Р"
Топологии, оптимизация геометрии и моле"
состояние рибосомы, предшествующее пепти"
кулярных электростатических потенциалов не"
дилтрансферазной реакции (ПТР), так и на сос"
канонических остатков РНК и остатков амино"
тояние Р/Р,Е/Е, по ряду сведений, аллостери"
кислот, связанных с 3′"концом тРНК, были по"
чески антагонистическое первому состоя"
лучены с помощью квантово"химических расчё"
нию [14, 15].
тов. Предварительная оптимизация вновь пара"
В настоящей работе были смоделированы
метризуемых остатков осуществлялась с по"
комплексы целой 70S рибосомы E. coli в обоих
мощью программы ПРИРОДА [22, 23] методом
функциональных состояниях с двумя варианта"
DFT с функционалом mPBE [24] и базисным
ми профиля метилирования остатка А2503 для
набором L1 [25]. Оптимизация завершалась по
дальнейшего использования в расчётах МД. Бы"
выполнению 300 шагов или достижению гради"
ло изучено влияние модификации резистент"
ентом значения ниже 10-6 Хартри/Бор. Оконча"
ности на конформацию и нековалентные связи
тельная оптимизация геометрии остатка и рас"
остатков рРНК, и наибольшие конформацион"
чёт его электростатического потенциала выпол"
ные изменения были обнаружены у достаточно
нялся в пакете FIREFLY [26, 27] методом Харт"
удаленных от А2503 остатков U2609 и U1782, а
ри-Фока с базисом 6-31G*. Оптимизация так"
также расположенного еще глубже по рибосом"
же прекращалась по выполнению 300 шагов или
БИОХИМИЯ том 85 вып. 11 2020
1720
МАКАРОВА, МАКАРОВ
достижению градиентом значения ниже 10-6
• угол между плоскостью первого азотис"
Хартри/Бор.
того основания и отрезком, соединяющим гео"
Все моделирования МД выполнялись при
метрические центры, составлял как минимум
температуре 310 К (температурном оптимуме
45°; без этого условия компланарная пара осно"
E. coli) с периодом привязки 0,1 пс под управле"
ваний (например, уотсон"криковская) была бы
нием термостата масштабирования скоростей с
ошибочно распознана как пара стэкинг"взаимо"
добавочным стохастическим членом [28] и пе"
действия.
риодическими граничными условиями с изо"
тропным постоянным давлением, поддерживае"
мым баростатом Берендсена [29] с периодом
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
привязки 5 пс. Электростатические взаимодей"
ствия обрабатывались посредством сети частиц
Дополнительная метильная группа приводила
Эвальда [30] с шагом сетки 0,125 нм и четвертым
к усилению стэкинг взаимодействий m2m8A2503 с
порядком интерполяции. Моделируемая систе"
соседними основаниями. Остаток m2A2503
ма была центрирована в орторомбической ячей"
23S рРНК, располагаясь на тяже между спираль"
ке с рёбрами 24 × 27 × 25 нм, заполненной моле"
ными структурами Н89 и Н90, вклинивается в
кулами воды TIP4Pew так, что оконечности ри"
стопку стэкинг"взаимодействий аналогичного
босомы от границ ячейки отделял слой раство"
одноцепочечного тяжа между Н73 и Н74 с обра"
рителя минимум 1,5 нм. Отрицательный заряд
зованием неспаренной стопки С2063-G2061-
системы был нейтрализован ионами калия с оп"
m2A2503-А2059-А2058. При этом А2060 из ос"
тимизированными Ван"дер"Ваальсовыми пара"
новного тяжа тоже выпетливается и образует
метрами [31] вкупе с видимыми с помощью крио"
третичные стэкинг"взаимодействия с G2502 -
электронной микроскопии структурными иона"
основание"сосед исследуемого m2A2503.
ми магния. Нейтрализованная система содержа"
Мы воспользовались ранее полученными
ла 100 мМ KCl и 6 мМ MgCl2 в виде ионов.
системами и траекториями [17] следующих ти"
Временной шаг интегрирования во всех вы"
пов, чтобы внедрить в них модификацию
числениях составлял 2 фс, координаты записы"
m2m8A2503 и изучить её конформационное воз"
вались в файл траектории каждые 15 пс.
действие на своё окружение, каждая из которых
Методы анализа траекторий. Анализ некова"
в дальнейшем будет упоминаться в виде аббре"
летных взаимодействий и конформаций остат"
виатуры. Эти аббревиатуры обозначают следую"
ков РНК осуществлялся для отрезков траекто"
щее:
рий после 100 нс расчётов, когда среднеквадра"
• PE - комплекс рибосомы с fMet-P-
тическое отклонение от заданной структуры
тРНКfMet и деацилированной E-тРНКfMet;
(RMSD) и потенциальная энергия стабилизиро"
• AP - комплекс рибосомы с Ala-A-
вались.
тРНКPhe и fMet-P-тРНКfMet.
Водородные связи анализировались с по"
Получение исходных АР" и РЕ"систем под"
мощью встроенных возможностей GROMACS:
робно описано в нашей предыдущей работе [17].
в каждом кадре траектории наличие или отсут"
Тогда из этих систем было получено по 4 траек"
ствие конкретной связи определялось геометри"
тории длиной 200 нс для каждого функциональ"
ческими параметрами:
ного состояния рибосомы. После добавления
• атом"донор водорода и атом"акцептор
метильной группы в остаток m2A2503 23S рРНК
разделены расстоянием не более 3,5 Å;
по 8"му положению мы удостоверились, что
• угол водород"донор"акцептор меньше
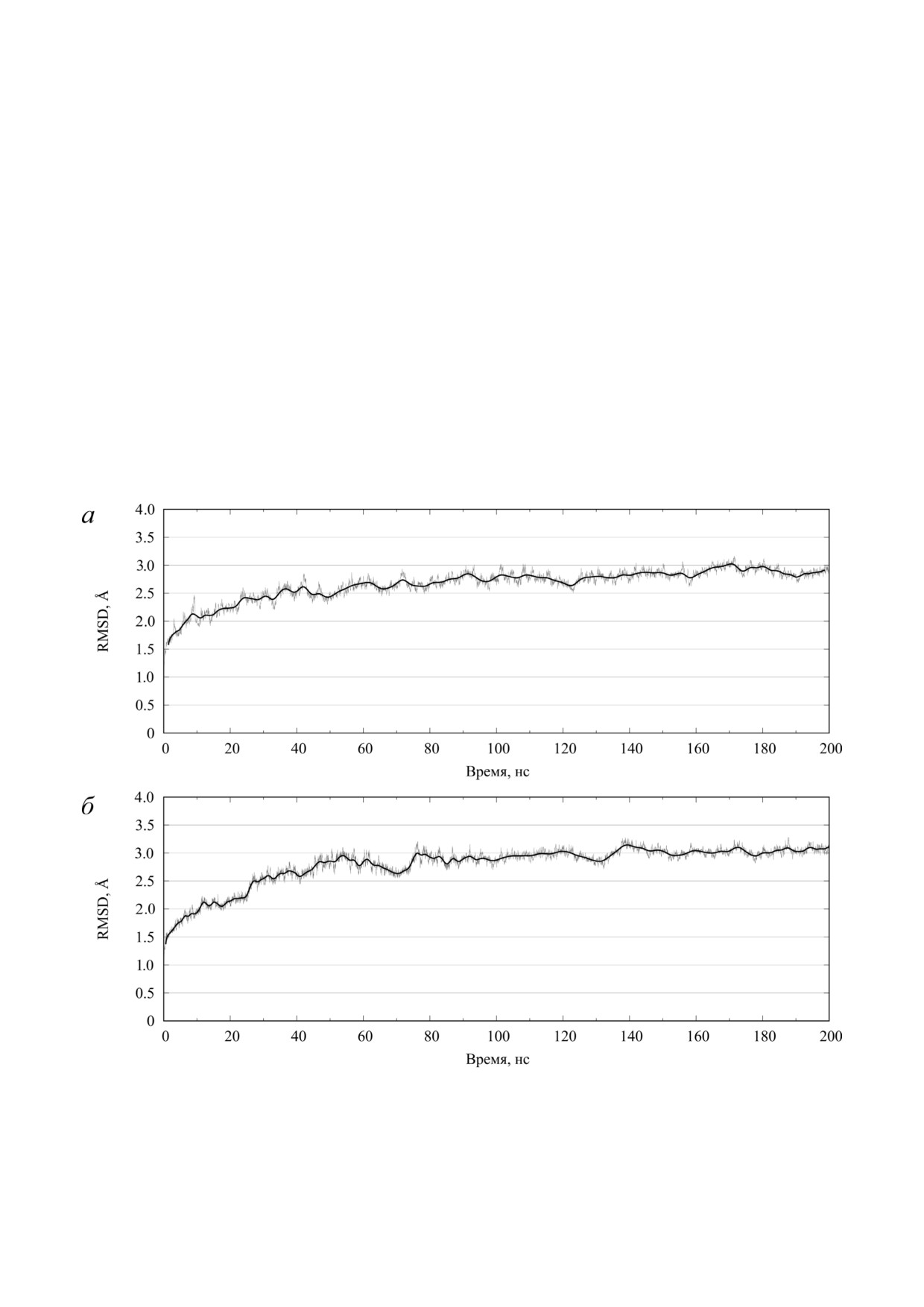
система быстро достигает стабильного состоя"
30°.
ния: рост RMSD прекращается до 60-80 нс
Также геометрические параметры были вве"
(рис. 1), а колебания потенциальной энергии
дены для распознавания стэкинг"взаимодей"
практически сразу исчерпывают какое"либо
ствий азотистых оснований остатков РНК, для
направленное движение (рис. 2).
чего вычислялся центр азотистого основания
Поэтому, как и в предыдущих расчётах систем
(среднее арифметическое координат атомов C2,
АР и РЕ с обычным m2A2503, для анализа были
C4 и C6 для пиримидинов и N1, N3 и C8 для пу"
взяты последние 100 нс каждой из траекторий.
ринов) и уравнение плоскости по трём этим точ"
Встречаемости водородных связей и стэкинг"
кам. Стэкинг"взаимодействие в кадре записы"
взаимодействий азотистых оснований остатков
валось, если:
рРНК во вновь полученных траекториях, как и
• расстояние между центрами не превы"
для канонических АР" и РЕ"состояний рибосом,
шало 5,5 Å;
были рассчитаны по последним 100 нс с вычис"
• угол между плоскостями азотистых ос"
лением среднего значения и стандартного откло"
нований не превышал 30°;
нения по 4 значениям для каждого типа системы.
БИОХИМИЯ том 85 вып. 11 2020
АЛЛОСТЕРИЧЕСКИЙ ЭФФЕКТ 2,8"ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК
1721
Наиболее непосредственным эффектом мо"
И все же перечисленные изменения касают"
дификации оказалось усиление способности к
ся только непосредственных соседей остатка
стэкинг"взаимодействию основания m2m8A2503
А2503 и не распространяются ниже в РТ или вы"
со своими соседями - G2061 и А2059 (рис. 3).
ше до ПТЦ. Таким образом, модификация
Это небольшое изменение в конформациях и
m2m8A2503 не вызывает существенных возму"
силе взаимодействий приводит к разрушению
щений AP"состояния рибосомы помимо непос"
стэкинг"взаимодействия соседнего остатка
редственной окрестности диметилированного
G2502 с А2060, из"за чего последний образует
остатка.
такое взаимодействие преимущественно с
Удаленные конформационные изменения в РТ
U1255 (таблица).
и других сайтах рибосомы, индуцированные вве
Кроме того, стабилизация положения остат"
дением m2m8A2503. Итак, дополнительное некон"
ка А2503 23S рРНК и перераспределение элект"
ститутивное метилирование остатка А2503
ронной плотности в нем способствуют значи"
23S рРНК приводит к перераспределению свя"
тельному увеличению встречаемости водород"
зей в области третичных контактов двух одноце"
ной связи его с аденином остатка А2062, совер"
почечных участков близ ПТЦ. Это воздействие,
шенно исключая встраивание А2062 между
как выяснилось, может передаваться глубже в РТ
G2061 и С2063, как это происходит во многих
- туда, где вновь синтезированная пептидная
кадрах траекторий рибосомы в состоянии AP.
цепь должна взаимодействовать с рибосомой.
Рис. 1. Изменение RMSD рибосомы при расчёте МД после добавления модифицированного остатка m2m8A2503: показа"
но на примере одной из 4"х траекторий длиной 200 нс для каждого типа системы. Непосредственные значения показаны
бледно"серым цветом на фоне скользящего среднего по 100 точкам, начерченного сплошной черной линией, сглаженной
методом кривых Безье. а - AР"состояние. б - РЕ"состояние
14 БИОХИМИЯ том 85 вып. 11 2020
1722
МАКАРОВА, МАКАРОВ
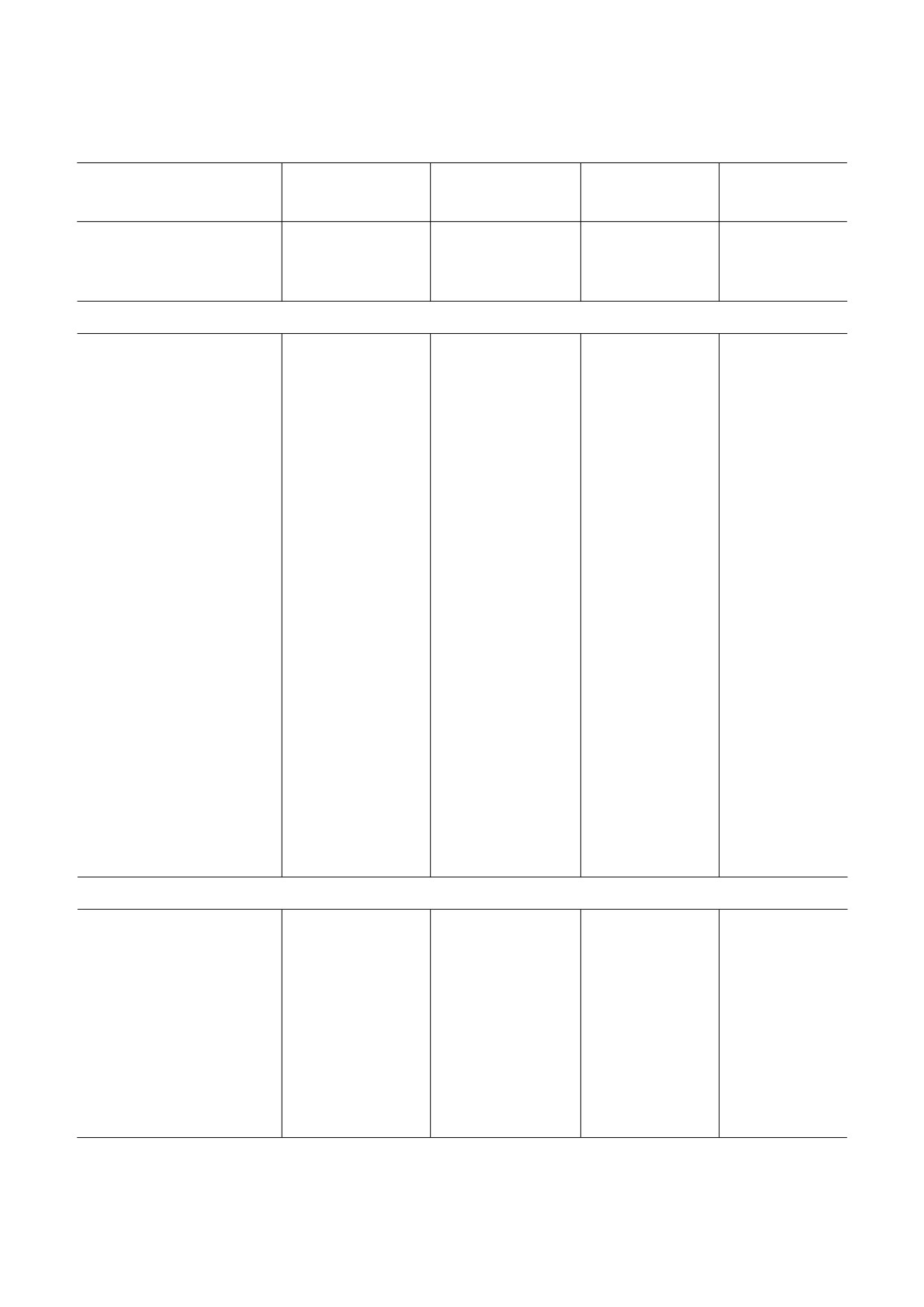
Связи, стабильно и воспроизводимо изменяющиеся в основном кластере 23S рРНК при добавлении 8"метильной группы
к m2A2503
Взаимодействие
AP дикий тип
АР m2m8A2503
РЕ дикий тип
РЕ m2m8A2503
G2061||m2A2503
33,8 ± 25,4
38,0 ± 33,5
15,4 ± 17,4
84,6 ± 3,8
A2062/N6-H...m2A2503/N7
8,3 ± 9,6
55,8 ± 40,5
37,3 ± 36,1
12,4 ± 24,9
Основные изменения в AP"состоянии
A2060||G2502
33,0 ± 23,0
7,3 ± 14,4
26,9 ± 33,9
0,1 ± 0,2
U1255||A2060
34,5 ± 30,8
55,4±19,5
23,1±28,1
21,6 ± 32,6
G830/N1-H...A2448/N7
44,6 ± 12,0
0,1 ± 0,3
0 ± 0
0 ± 0
C961||A2031
0,2 ± 0,3
38,8 ± 19,7
72,7 ± 2,2
61,1 ± 15,4
C2499/N4-H...G2454/O6
6,1 ± 7,1
70,5 ± 45,7
13,8 ± 11,4
22,0 ± 20,6
G2454/N2-H...C2499/O2
3,5 ± 7,1
67,6 ± 41,7
0 ± 0
0 ± 0
G2454/N1-H...C2499/N3
3,4 ± 6,7
68,9 ± 45,1
0 ± 0
0 ± 0
A2453/N6-H...U2500/O4
7,0 ± 14,1
89,8 ± 5,0
0 ± 0
1,9 ± 3,2
U2500/N3-H...A2453/N1
8,0 ± 15,9
62,6 ± 37,4
0 ± 0
12,5 ± 17,5
G2454||G2455
90,4 ± 11,9
19,2 ± 35,9
93,4 ± 2,6
89,1 ± 4,0
Ψ2504/N1-H...G2447/O6
14,8 ± 12,4
61,9 ± 31,4
0 ± 0
0 ± 0
Cm2498/N4-H...C2499/N3
0 ± 0
20,6 ± 3,6
0,8 ± 1,6
0 ± 0
Cm2498||C2499
37,4 ± 11,8
72,5 ± 19,5
84,5 ± 12,4
78,8 ± 23,4
A945/N6-H...A2448/N3
25,7 ± 13,5
0 ± 0
0 ± 0
0 ± 0
U2500||Ψ2504
13,3 ± 7,9
66,2 ± 22,6
1,4 ± 2,8
10,0 ± 18,0
G830||A945
70,0 ± 7,8
20,1 ± 34,8
0,1 ± 0,2
0,3 ± 0,3
G2446/N2-H...D2449/O2
12,6 ± 4,0
41,5 ± 23,8
84,5 ± 6,4
61,6 ± 42,6
C2452/N4-H...Ψ2504/O2
3,8 ± 7,5
56,1 ± 26,4
25,6 ± 48,2
48,2 ± 40,5
Изменения в PE"состоянии в РТ
A1783/N6-H...A2587/O2′
7,4 ± 14,8
5,1 ± 10,2
23,1 ± 32,2
68,0 ± 6,0
U1779/N3-H...G2588/O2′
13,0 ± 13,1
9,7 ± 9,8
64,1 ± 42,9
0,5 ± 0,6
A1785/N6-H...U1779/O4
0 ± 0
0 ± 0
81,3 ± 9,0
20,1 ± 22,1
A783/O2′-H...U1779/O2
39,8 ± 27,0
32,2 ± 22,2
62,0 ± 41,5
0,1 ± 0,1
A751||A789
0 ± 0
0,1 ± 0,3
33,0 ± 29,5
0 ± 0,1
U1782||U2609
94,8 ± 4,5
92,1 ± 9,0
84,4 ± 13,7
40,1 ± 30,0
A781/O2′-H...C1788/O2
3,3 ± 4,0
2,8 ± 2,6
1,1 ± 2,2
23,0 ± 17,1
БИОХИМИЯ том 85 вып. 11 2020
АЛЛОСТЕРИЧЕСКИЙ ЭФФЕКТ 2,8"ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК
1723
Окончание таблицы
Взаимодействие
AP дикий тип
АР m2m8A2503
РЕ дикий тип
РЕ m2m8A2503
Изменения в PE"состоянии в окрестности E"сайта
U2075/N3-H...A2435/N1
1,5 ± 3,0
0 ± 0
6,1 ± 5,6
52,5 ± 29,5
U2074/N3-H...G2597/N3
0 ± 0
0 ± 0
38,3 ± 11,2
61,4 ± 5,7
U200/N3-H...G248/O4′
8,3 ± 16,6
6,9 ± 10,2
69,0 ± 8,5
44,1 ± 9,9
U2076||U2596
79,3 ± 13,7
66,5 ± 19,7
60,7 ± 7,5
32,1 ± 4,6
A197/N6-H...A2430/O2′
57,9 ± 8,0
64,0 ± 19,1
52,8 ± 35,4
4,1 ± 4,3
U2431/O2′-H...A2433/N7
0 ± 0
0 ± 0
54,4 ± 20,9
8,1 ± 13,0
G2436/N2-H...U2074/O4
0 ± 0
0 ± 0
6,5 ± 7,8
48,7 ± 23,3
U2075/N3-H...A2077/N7
88,1 ± 12,2
95,2 ± 2,2
58,3 ± 9,1
21,5 ± 22,1
A2077/N6-H...U2075/O4
31,7 ± 47,0
0,1 ± 0,1
98,5 ± 0,4
54,0 ± 41,4
A2435/N6-H...U2075/O4
0 ± 0
0 ± 0
0 ± 0
38,6 ± 35,8
Примечание: АР и РЕ обозначают рибосому с А/А-тРНК и Р/Р-тРНК либо Р/Р-тРНК и Е/Е-тРНК соответственно.
Значения рассчитаны по 4"м независимым траекториям каждой из систем и представлены как среднее значение ± стан"
дартное отклонение. Троеточие обозначает водородную связь, двойная вертикальная черта || - стэкинг"взаимодействие.
Наиболее явные изменения были найдены в PE"
цию АР"состояния посредством данной мута"
состоянии рибосомы.
ции резистентности.
Последовательность азотистых оснований
Кроме того, остатки U2609 и U1782 и об"
С2063-G2061-m2A2503-А2059-А2058, образу"
ласть А751 участвуют в передаче аллостеричес"
ющих стэкинг"взаимодействия, переходит в
кого сигнала из РТ в ПТЦ и А"сайт, что являет"
спираль Н73, начинающуюся с пар G2057-
ся ключевым механизмом воздействия антибио"
C2611 и G2057-C2612. Они ковалентно связа"
тиков, связывающихся в рибосомном тунне"
ны с важными аллостерическими сенсорами в
ле [32].
РТ, С2610 и U2609 [32]. В АР"состоянии остат"
Что же касается остальной рибосомы, то из"
ки РТ сохраняют свою конформацию при лю"
менения в РТ в целом не вызывали масштабных
бой степени модификации А2503, однако в РЕ"
глобальных переконфигураций нековалентных
состоянии взаимодействие остатков U2609 и
связей в рРНК как, например, в случае мутаций,
U1782 расходится, и последний занимает поло"
явно нарушавших её функционирование [17].
жение, близкое к таковому в АР"состоянии
Стабильных и воспроизводимых изменений во
(рис. 4).
встречаемости нековалентных взаимодействий
Разворот U1782 передается на U1781 и свя"
РНК как минимум на 20% было суммарно обна"
занный с ним в стэкинг"взаимодействии А752.
ружено в 253 случаях для АР"состояния и в 296
Это смещает А751 в положение, воспроизводя"
случаях для РЕ"состояния (при наличии при"
щее АР"состояние (рис. 5), как это уже было от"
мерно 22 тыс. связей, воспроизводимых хотя бы
мечено с U1782, из"за чего полностью разруша"
в одном из состояний). Изменяющиеся связи
ется стэкинг"взаимодействие А751 и А789, что
были распределены по всей рибосоме, и в обоих
аналогично воспроизводит АР"состояние (таб"
случаях можно было выделить как минимум 2
лица). Таким образом, только в РЕ"состоянии,
крупных кластера, внутри которых остатки
функционально противоположном АР"состоя"
рРНК, образовавшие такую связь, отделены
нию, наблюдаются конформационные измене"
друг от друга не более чем тремя остатками, соеди"
ния в области РТ, воспроизводящие последнее
нёнными стабильной связью. Первый кластер
из аллостерически антагонистичных состояний.
содержал участки 23S и 5S рРНК центральной
Это может означать локальную гиперстабилиза"
области большой субъединицы рибосомы, вто"
БИОХИМИЯ том 85 вып. 11 2020
14*
1724
МАКАРОВА, МАКАРОВ
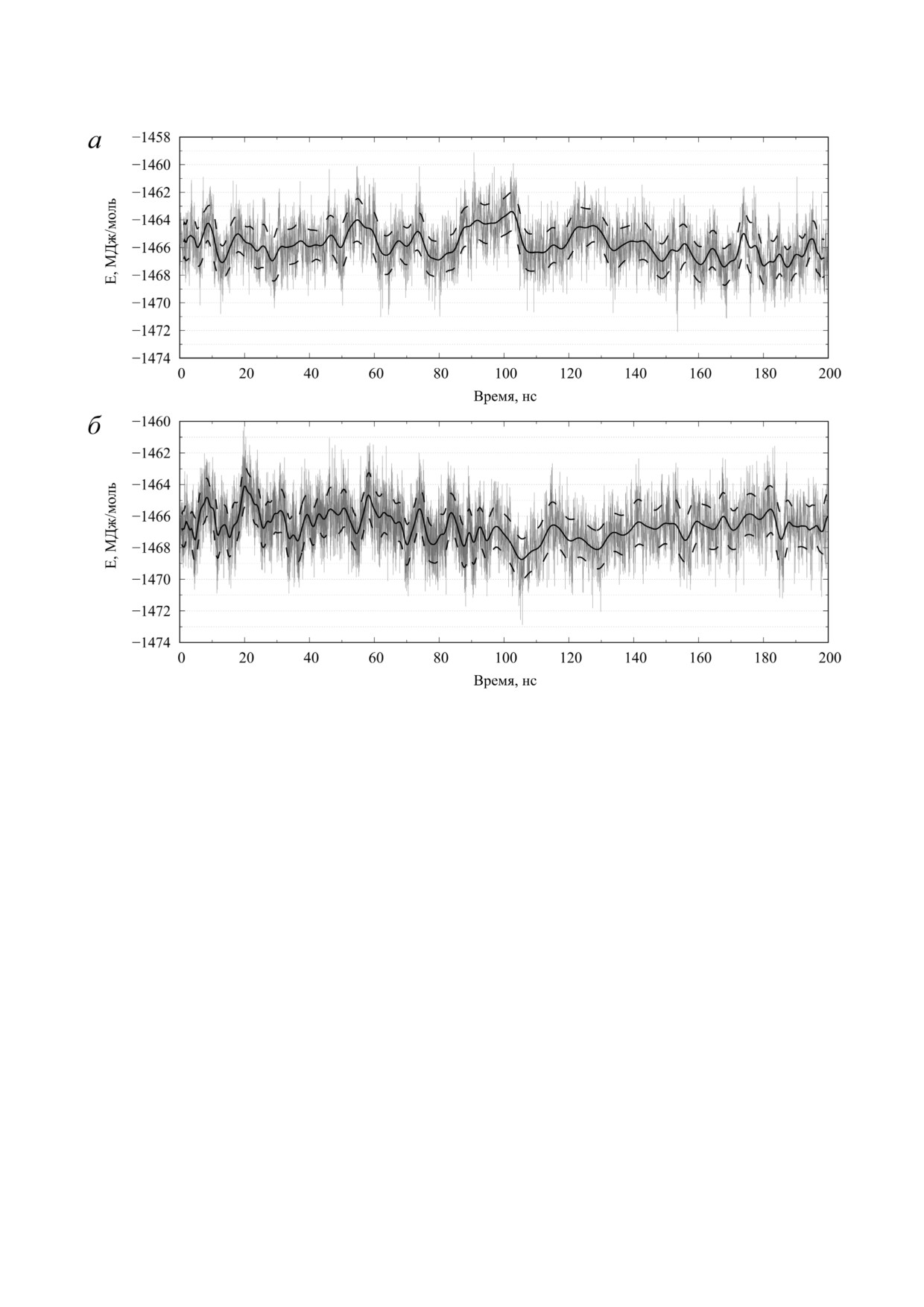
Рис. 2. Эволюция потенциальной энергии рибосомы во времени при расчёте МД после добавления модифицированного
остатка m2m8A2503: показано на примере одной из 4"х траекторий длиной 200 нс для каждого типа системы. Непосред"
ственные значения показаны бледно"серым цветом на фоне скользящего среднего по 100 точкам, начерченного сплош"
ной черной линией, сглаженной методом кривых Безье, диапазон значений в пределах скользящего стандартного откло"
нения ограничен пунктирными линиями. а - AР"состояние. б - РЕ"состояние
рой - фрагменты малой субъединицы и некото"
которое непосредственно примыкает к сайту
рых межсубъединичных мостов.
связывания остатка С75 Е-тРНК - предполо"
В самом крупном таком кластере остатков
жительным аллостерически чувствительным
рРНК наибольшее число изменений АР"состоя"
сайтом, связанным с Е-тРНК [17]. Шпилька
ния модификацией m2A2503 было обнаружено
Н93 из упомянутого сочленения спиралей наи"
вокруг ПТЦ в спиралях Н89 и Н74, а также об"
более непосредственно связывает Е"сайт и на"
ласти их стыка, и, кроме того, около области со"
чало РТ. Возможно, изменения на одном из её
единения спиралей Н37 и Н39. Обнаруженные
концов также влияют на разворот стопки из двух
изменения касались главным образом стабили"
оснований, U2609 и U1782, в положение, кото"
зации внутренних связей спиралей и межспи"
рое близко к таковому в АР"состоянии.
ральных элементов (таблица, раздел «Основные
Количественные данные встречаемостей об"
изменения в AP"состоянии»).
суждаемых выше связей указаны в таблице.
В противоположность этому, в РЕ"состоя"
нии перераспределения связей концентрируют"
Согласно данным молекулярно"динамичес"
ся вокруг универсально консервативного сочле"
кого моделирования систем рибосомы с консти"
нения спиралей Н74, Н75 и Н93 (таблица, раз"
тутивным и неконститутивным метилировани"
дел «Основные изменения в РЕ"состоянии»),
ем остатка A2503 23S рРНК, дополнительное
БИОХИМИЯ том 85 вып. 11 2020
АЛЛОСТЕРИЧЕСКИЙ ЭФФЕКТ 2,8"ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК
1725
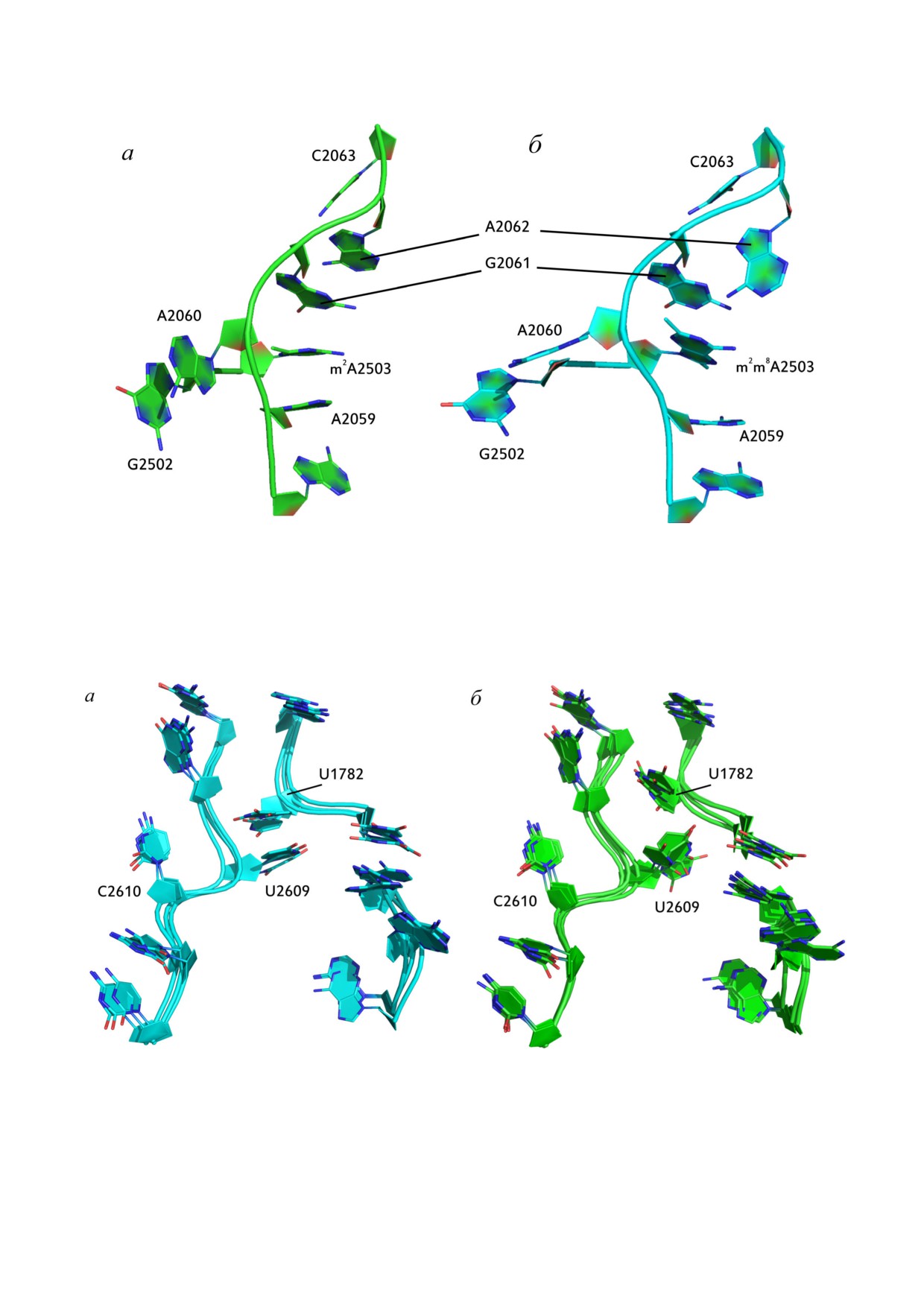
Рис. 3. Сравнение характерной конформации ближайшего окружения остатка 2503 23S рРНК при различных модифика"
циях последнего. Дополнительное 8"метилирование остатка m2A2503 усиливает стэкинг"взаимодействия этого остатка,
но вызывает разрушение аналогичного взаимодействия G2502 с A2060. Показан кадр траектории, наиболее близкий к
центроиду основного кластера изображенных остатков рРНК в 4"х конечных 100 нс участках из 200 нс для каждой систе"
мы. а - AР"состояние рибосомы дикого типа (с m2A2503). б - AР"состояние рибосомы с m2m8A2503. (С цветными
biokhsm/)
Рис. 4. Разрушение стэкинг"взаимодействия между остатками U2609 и U1782 23S рРНК в траекториях с 2,8"диметилиро"
ванным A2503. На рисунке изображены наиболее заселенные кластеры остатков РТ: 3 кластера рибосомы дикого типа, ко"
торые суммарно занимают 82,1% всех кадров из 4"х траекторий и 4 кластера рибосомы с m2m8A2503, которые охватывают
83,5% кадров соответственно. а - РЕ"состояние рибосомы дикого типа (с m2A2503). б - РЕ"состояние рибосомы с
m2m8A2503
БИОХИМИЯ том 85 вып. 11 2020
1726
МАКАРОВА, МАКАРОВ
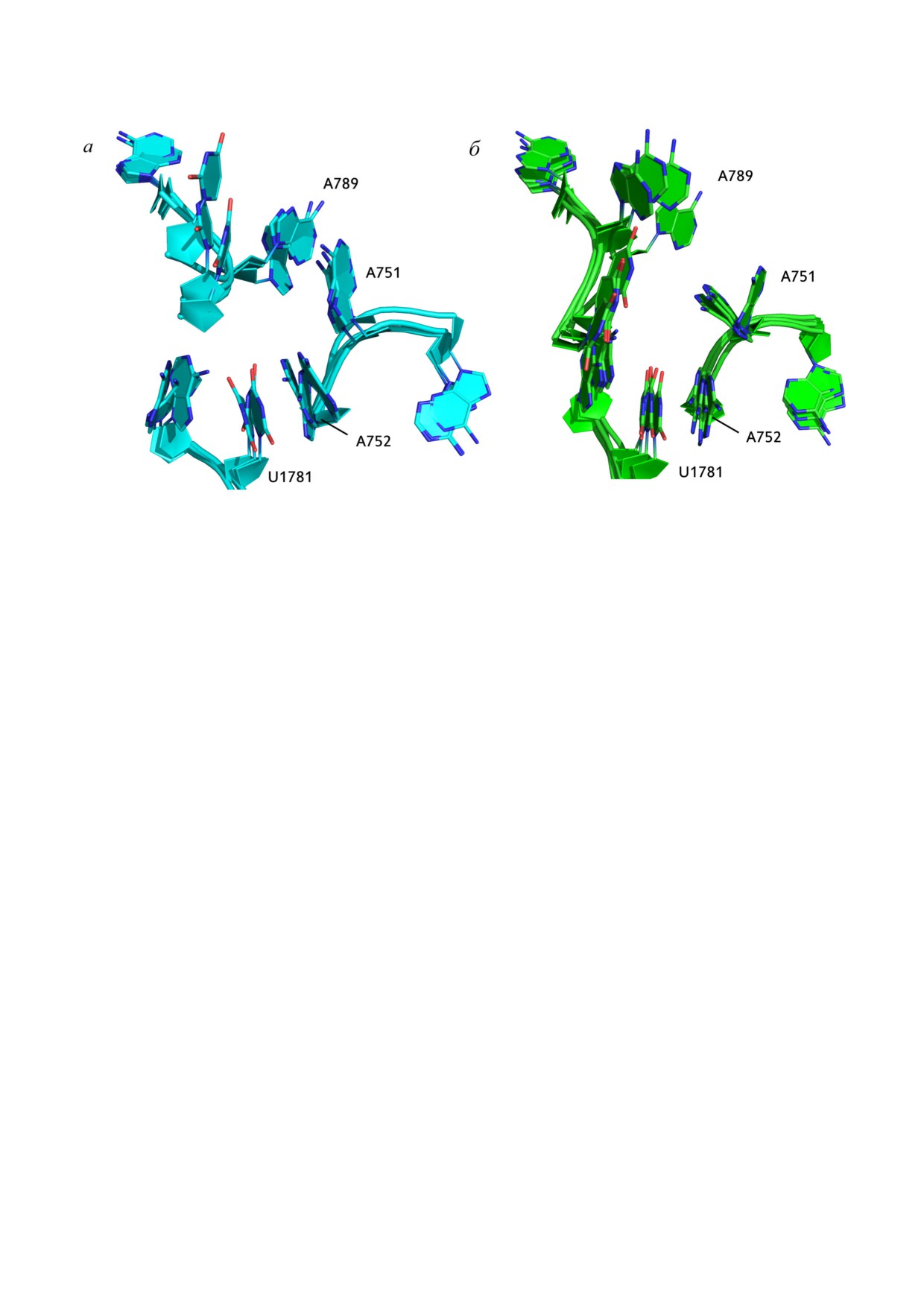
Рис. 5. Разрушение стэкинг"взаимодействия между остатками А751 и А789 23S рРНК в траекториях с 2,8"диметилирован"
ным A2503. На рисунке изображены наиболее заселенные кластеры остатков РТ: 3 кластера рибосомы дикого типа, кото"
рые суммарно занимают 82,1% всех кадров из 4"х траекторий и 4 кластера рибосомы с m2m8A2503, которые охватывают
83,5% кадров соответственно. а - РЕ"состояние рибосомы дикого типа (с m2A2503). б - РЕ"состояние рибосомы с
m2m8A2503
8"метилирование усиливает стэкинг"взаимо"
шем изучении данной модификации резис"
действия азотистого основания А2503. Это из"
тентности с антибиотиками и их стоп"пептида"
менение подтягивает остатки G2061 и A2059 к
ми.
A2503. В результате в АР"состоянии рибосомы,
предшествующем ПТР, обнаружилось лишь
Финансирование. Исследование было выпол"
несколько значительных конформационных из"
нено при финансовой поддержке Российского
менений и перегруппировок в непосредствен"
научного фонда (грант № 18"74"00022) и Прави"
ном сайте модификации. Напротив, в присут"
тельства Российской Федерации (резолюция
ствии тРНК в Е"сайте конформационные перег"
№ 1 от 16.03.2013, контракт № 02.A03.21.0011).
руппировки состоялись как в самом Е"сайте, так
Благодарность. Авторы выражают глубокую
и в РТ с участием остатков, известных как важ"
благодарность академику РАН Богданову Алек"
ные участники аллостерического взаимодей"
сею Алексеевичу за инициирование молекуляр"
ствия РТ и ПТЦ.
но"динамического исследования модификаций
Таким образом, модификация m2m8A2503
резистентности в рибосоме. Авторы благодарят
не нарушает AP"состояния рибосомы, необхо"
Вычислительный центр МГУ имени М.В. Ломо"
димого для протекания ПТР, но смещает анта"
носова за предоставленную возможность вести
гонистичное ему состояние обратно в сторону
расчеты МД на суперкомпьютере «Ломоно"
AP"подобной конформации некоторых остат"
сов-II».
ков. Отсюда следует, что объяснение широкому
Конфликт интересов. Авторы декларируют
спектру антибиотиков с разнообразными сай"
отсутствие конфликта интересов.
тами связывания, от которых данная модифи"
Соответствие этическим стандартам. Данная
кация защищает бактерии, может быть найде"
статья не содержит каких"либо исследований,
но в дополнительной стабилизации предшест"
содержащих эксперименты на людях или жи"
вующего ПТР состояния рибосомы. Этот факт
вотных, проводимых кем"либо из авторов
следует принимать во внимание при дальней"
статьи.
БИОХИМИЯ том 85 вып. 11 2020
АЛЛОСТЕРИЧЕСКИЙ ЭФФЕКТ 2,8"ДИМЕТИЛИРОВАНИЯ A2503 23S рРНК
1727
СПИСОК ЛИТЕРАТУРЫ
1.
Yonath, A. (2009) Ribosome: an ancient cellular nano"
16.
Cannone, J. J., Subramanian, S., Schnare, M. N., Collett,
machine for genetic code translation, in Biophysics and the
J. R., D’Souza, L. M., et al. (2002) The comparative RNA
challenges of emerging threats (Puglisi, J. D., ed.) Springer,
web (CRW) site: an online database of comparative
Netherlands, pp. 121"155, doi: 10.1007/978"90"481"2368"
sequence and structure information for ribosomal, intron,
1_8.
and other RNAs, BMC Bioinform., 3, 1"31, doi: 10.1186/
2.
Манькин А. (2001) Рибосомные антибиотики, Мо
1471"2105"3"2.
лекулярная биология,
35,
597"609, doi:
10.1023/
17.
Makarova, T., and Bogdanov, A. (2019) Allosteric regula"
A:1010510623805.
tion of the ribosomal A site revealed by molecular dynam"
3.
McCusker, K. P., and Fujimori, D. G. (2012) The chem"
ics simulations, Biochimie, 167, 179"186, doi: 10.1016/
istry of peptidyltransferase center"targeted antibiotics:
j.biochi.2019.09.019.
enzymatic resistance and approaches to countering resis"
18.
Byrd, R., Lu, P., and Nocedal, J. (1995) A limited memo"
tance, ACS Chem. Biol., 7, 64"72, doi: 10.1021/cb200418f.
ry algorithm for bound constrained optimization, SIAM J.
4.
Wilson, D. N. (2014) Ribosome"targeting antibiotics and
Sci. Comput., 16, 1190"1208.
mechanisms of bacterial resistance, Nat. Rev. Microbiol.,
19.
Van der Spoel, D., Lindahl, E., Hess, B., Groenhof, G.,
12, 35"48, doi: 10.1038/nrmicro3155.
Mark, A. E., and Berendsen, H. J. C. (2005) Gromacs:
5.
Vester, B., and Long, K. S. (2009) Antibiotic resistance in
fast, flexible, free, J. Comput. Chem., 26, 1701"1718,
bacteria caused by modified nucleosides in 23S ribosomal
doi: 10.1002/jcc.20291.
RNA, in DNA and RNA Modification Enzymes: Structure,
20.
Hess, B., and Kutzner, C., van der Spoel, D., and Lindahl,
Mechanism, Function and Evolution (Grosjean, H. ed.)
E. (2008) Gromacs 4: algorithms for highly efficient, load"
Landes Bioscience, Austin, USA.
balanced, and scalable molecular simulation, J. Chem.
6.
Giessing, A. M. B., Jensen, S. S., Rasmussen, A., Hansen,
Theory Comput., 4, 435"447, doi: 10.1021/ct700301q.
L. H., Gondela, A., Long, K., Vester, B., and Kirpekar, F.
21.
Maier, J. A., Martinez, C., Kasavajhala, K., Wickstrom, L.,
(2009) Identification of 8"methyladenosine as the modifi"
Hauser, K. E., and Simmerling, C. (2015) ff14SB: improv"
cation catalyzed by the radical SAM methyltransferase Cfr
ing the accuracy of protein side chain and backbone para"
that confers antibiotic resistance in bacteria, RNA, 15, 327"
meters from ff99SB, J. Chem. Theory Comput., 11, 3696"
336, doi: 10.1261/rna.1371409.
3713, doi: 10.1021/acs.jctc.5b00255.
7.
Schwarz, S., Werckenthin, C., and Kehrenberg, C. (2000)
22.
Лайков Д. Н., Устынюк Ю. А. (2005) Система кванто"
Identification of a plasmid"borne chloramphenicol"flor"
во"химических программ «ПРИРОДА"04». Новые
fenicol resistance gene in Staphylococcus sciuri, Antimicrob.
возможности исследования молекулярных систем с
Agents Chemother., 44, 2530"2533, doi: 10.1128/aac.44.9.
применением параллельных вычислений, Известия
2530"2533.2000.
Академии наук. Серия химическая, 3, 804"810.
8.
Yan, F., LaMarre, J. M., Rohrich, R., Wiesner, J., Jomaa, H.,
23.
Laikov, D. N. (2011) A new parametrizable model of mol"
Mankin, A. S., and Fujimori, D. G. (2010) RlmN and Cfr
ecular electronic structure, J. Chem. Phys., 135, 134120,
are radical SAM enzymes involved in methylation of ribo"
doi: 10.1063/1.3646498.
somal RNA, J. Am. Chem. Soc.,
132,
3953"3964,
24.
Adamo, C., and Barone, V. (2002) Physically motivated
doi: 10.1021/ja910850y.
density functionals with improved performances: the mod"
9.
Smith, L. K., and Mankin, A. S. (2008) Transcriptional
ified perdew-burke-ernzerhof model, J. Chem. Phys.,
and translational control of the mir operon, which confers
116, 5933"5940, doi: 10.1063/1.1458927.
resistance to seven classes of protein synthesis inhibitors,
25.
Laikov, D. N. (2005) A new class of atomic basis functions
Antimicrob. Agents Chemother., 52, 1703"1712, doi: 10.1128/
for accurate electronic structure calculations of molecules,
AAC.01583"07.
Chem. Phys. Lett., 416, 116"120, doi: 10.1016/j.cplett.
10.
Fischer, N., Neumann, P., Konevega, A. L., Bock, L. V.,
2005.09.046.
Ficner, R., Rodnina, M. V., and Stark, H. (2015) Structure
26.
Schmidt, M. W., Baldridge, K. K., Boatz, J. A., Elbert, S. T.,
of the E. coli ribosome-EF-Tu complex at < 3 Å resolu"
Gordon, M. S., et al. (1993) General atomic and molecu"
tion by Cs-corrected cryo-EM, Nature, 520, 567"570,
lar electronic structure system, J. Comput. Chem., 14, 1347"
doi: 10.1038/nature14275.
1363, doi: 10.1002/jcc.540141112.
11.
Makarov, G., Golovin, A., Sumbatyan, N., and Bogdanov, A.
27.
(2015) Molecular dynamics investigation of a mechanism of
28.
Bussi, G., Donadio, D., and Parrinello, M.
(2007)
allosteric signal transmission in ribosomes, Biochemistry
Canonical sampling through velocity rescaling, J. Chem.
(Moscow), 80, 1047"1056, doi: 10.1134/S0006297915080106.
Phys., 126, 014101, doi: 10.1063/1.2408420.
12.
Vázquez Laslop, N., Ramu, H., Klepacki, D., Kannan, K.,
29.
Berendsen, H., Postma, J., van Gunsteren, W., DiNola,
and Mankin, A. S. (2010) The key function of a conserved
A., and Haak, J. (1984) Molecular dynamics with coupling
and modified rRNA residue in the ribosomal response to
to an external bath, J. Chem. Phys., 81, 3684"3690,
the nascent peptide, EMBO J.,
29,
3108"3117,
doi: 10.1063/1.448118.
doi: 10.1038/emboj.2010.180.
30.
Darden, T., York, D., and Pedersen, L. (1993) Particle
13.
Sanbonmatsu, K. (2012) Computational studies of molec"
mesh Ewald: an nlog(n) method for Ewald sums in
ular machines: the ribosome, Curr. Opin. Struct. Biol., 22,
large systems, J. Chem. Phys., 98, 10089, doi: 10.1063/
168"174, doi: 10.1016/j.sbi.2012.01.008.
1.464397.
14.
Gnirke, A., Geigenmuller, U., Rheinberger, H. J., and
31.
Joung, I. S., and Cheatham, T. E. (2008) Determination of
Nierhaus, K. H. (1989) The allosteric three"site model for
alkali and halide monovalent ion parameters for use in
the ribosomal elongation cycle. analysis with a heteropoly"
explicitly solvated biomolecular simulations, J. Phys.
meric mRNA, J. Biol. Chem., 264, 7291"7301.
Chem. B, 112, 9020"9041, doi: 10.1021/jp8001614.
15.
Chen, C., Stevens, B., Kaur, J., Smilansky, Z., Cooperma,
32.
Vázquez Laslop, N., Ramu, H., and Mankin, A. (2011)
B. S., and Goldman, Y. E. (2011) Allosteric vs. sponta"
Nascent peptide"mediated ribosome stalling promoted by
neous exit"site (E"site) tRNA dissociation early in protein
antibiotics, in Ribosomes: Structure, Function, and
synthesis, Proc. Natl. Acad. Sci. USA, 108, 16980"16985,
Dynamics, (Rodnina, M. V., Wintermeyer, W., Green, R.,
doi: 10.1073/pnas.1106999108.
eds), published by Springer"Verlag, Wien, pp. 377"392.
БИОХИМИЯ том 85 вып. 11 2020
1728
МАКАРОВА, МАКАРОВ
INVESTIGATION OF ALLOSTERIC EFFECT OF 2,8 DIMETHYLATION
OF A2503 23S rRNA IN E. coli BY MOLECULAR DYNAMICS SIMULATION
T. M. Makarova* and G. I. Makarov
South Ural State University, 454080 Chelyabinsk, Russia; E mail: makarovatm@susu.ru
Received June 16, 2020
Revised October 1, 2020
Accepted October 2, 2020
Ribosome is a molecular machine that synthesizes all cellular proteins. It also is a target of about half of the clinical"
ly used antibiotics. Adaptive chemical modification of ribosomal RNAs residues is one of the ways to provide resis"
tance to certain antibiotics. A curious example of such modification is 2,8"dimethylation of A2503 in 23S rRNA,
which induces resistance to phenols, linkosamides, oxazolidinones, pleuromutilins, and certain macrolides. In this
article the effect of 2,8"dimethylation of A2503 on conformation and mobility of RNA residues of the 70S E. coli ribo"
some was investigated employing molecular dynamics simulations method. Significant alterations were detected both
in the immediate environment of the 2503 23S rRNA residue and in the nucleotides located deeper in the nascent
peptide exit tunnel (NPET), which are known to be involved in signal transmission from the antibiotics bound in the
NPET to the peptidyl transferase center. These alterations shift the ribosome towards the A/A, P/P"state from the
conformationally different state - P/P, E/E one in our case. The obtained results allow us to conclude that the effect
of m2m8A2503 modification involves additional stabilization of the A/A, P/P"state favoring the peptidyl transferase
reaction (PTR) contrary to antibiotics that inhibit PTR.
Keywords: ribosome, resistance, antibiotics, A2503, molecular dynamics
БИОХИМИЯ том 85 вып. 11 2020