БИОХИМИЯ, 2020, том 85, вып. 3, с. 449 - 455
ГИПОТЕЗА
УДК 578.832.1;579.22
УНИКАЛЬНАЯ БИПОЛЯРНАЯ АРХИТЕКТУРА ГЕНОВ
В РНК ГЕНОМЕ ВИРУСА ГРИППА А
© 2020
О.П. Жирнов1,2
1 Национальный исследовательский центр эпидемиологии и микробиологии им. Н.Ф. Гамалеи
Минздрава России, Институт вирусологии им. Д.И. Ивановского,
123098 Москва, Россия; электронная почта: zhirnov@inbox.ru
2 Русско)немецкая академия медицинских и биотехнологических наук,
121205 Москва, Сколково, Россия
Поступила в редакцию 06.11.2019
После доработки 22.12.2019
Принята к публикации 22.12.2019
Геном вируса гриппа А состоит из 8 сегментов одноцепочечной РНК негативной полярности. Посредством
классической негативно полярной стратегии восьмой сегмент NS кодирует анти интерфероновый белок
NS1 (27 кДа) и белок ядерного экспорта NEP (14 кДа). В сегменте NS обнаружена дополнительная откры
тая рамка, имеющая позитивную полярность прямой трансляции вирионной РНК для третьего белка
(NSP8 - negative strand protein; 18-25 кДа у различных штаммов). Обнаруженная локализация трех и более
разнополярных генов на одном участке одноцепочечной молекулы РНК представляется уникальным для
биосферы типом экономичной генной архитектуры. Анализ in silico геномов вирусов гриппа, циркулирую
щих среди людей и животных, показал, что ген NSP8 сформировался у вируса гриппа А ~100 лет назад
(«юный» ген) и имеет высокую эволюционную изменчивость. Получены данные, подтверждающие возмож
ность экспрессии гена NSP8 в организме инфицированных животных, что укрепляет концепцию о бипо
лярной (амбисенс) стратегии генома вируса гриппа А. Обнаруженная высокая изменчивость белка NSP8 на
водит на мысль о том, что этот «юный» ген находится в стадии оптимизации своей функции, и после накоп
ления мутаций и перехода их количества в новое функциональное качество зрелого белка NSP8 возникнет
полноценный биполярный вирус гриппа, который может приобрести новые непредсказуемые и угрожаю
щие для человека и животных свойства.
КЛЮЧЕВЫЕ СЛОВА: вирус гриппа, стратегия генома, ген NSP, амбисенс вирусы, структура генов.
DOI: 10.31857/S0320972520030148
Вирус гриппа А имеет липидную оболочку
вый белок (NS1), выполняющий функцию суп
(оболочечный вирус). Его геном состоит из 8
рессора интерферона, и белок ядерного экспер
сегментов одноцепочечной РНК с размерами
та (NEP), регулирующий ядерный экспорт ви
(0,9-3,2) × 103 нуклеотидных оснований. Сег
русных рибонуклеопротеидов в инфицирован
менты РНК имеют негативную полярность, так
ных клетках. мРНК для белка NEP претерпева
как не транслируются рибосомами, но тран
ет сплайсинг, приводящий к сдвигу рамки
скрибируются вирусной полимеразой с образо
трансляции относительно гена NS1 (рис. 1, а).
ванием матричных РНК, с которых транслиру
У подавляющего большинства штаммов ви
ются вирусные белки (рис. 1, а, б). У некоторых
руса гриппа А человека геномная РНК сегмен
вирусных сегментов транскрипция сопряжена
та NS имеет протяженную открытую рамку
со сплайсингом мРНК, а трансляция происхо
трансляции (ОРТ) в позитивной ориентации
дит с различных АUG кодонов и со сдвигом
(рис. 1, а) [3-9]. Эту рамку обозначили, как ген
трансляционной рамки. В результате геном ви
белка негативной цепи сегмента 8 (NSP8) (nega
руса гриппа А обеспечивает синтез 16 уникаль
tive strand protein; segment 8) [4, 5]. Наличие про
ных вирусных белков по негативно полярному
тяженной трансляционной рамки в геномной
пути [1, 2]. Наименьший сегмент NS вируса
NS РНК вируса, содержащей инициаторный
гриппа кодирует по негативному пути два вирус
AUG, терминирующий UGA, и структуриро
ных белка: неструктурный антиинтерфероно
ванную на 5′ конце зону, имеющую элементы,
характерные для IRES [10], предполагает, что ге
Принятые сокращения: HA - гемагглютинин; NA -
номная NS РНК может проявлять свойства мат
нейраминидаза; NS1
- антиинтерфероновый белок;
NEP - белок ядерного эксперта; NSP8 - белок негативной
ричной РНК и транслироваться с образованием
цепи сегмента 8; ОРТ - открытая рамка трансляции.
протяженного белка NSP8. Следует отметить,
449
450
ЖИРНОВ
а
б
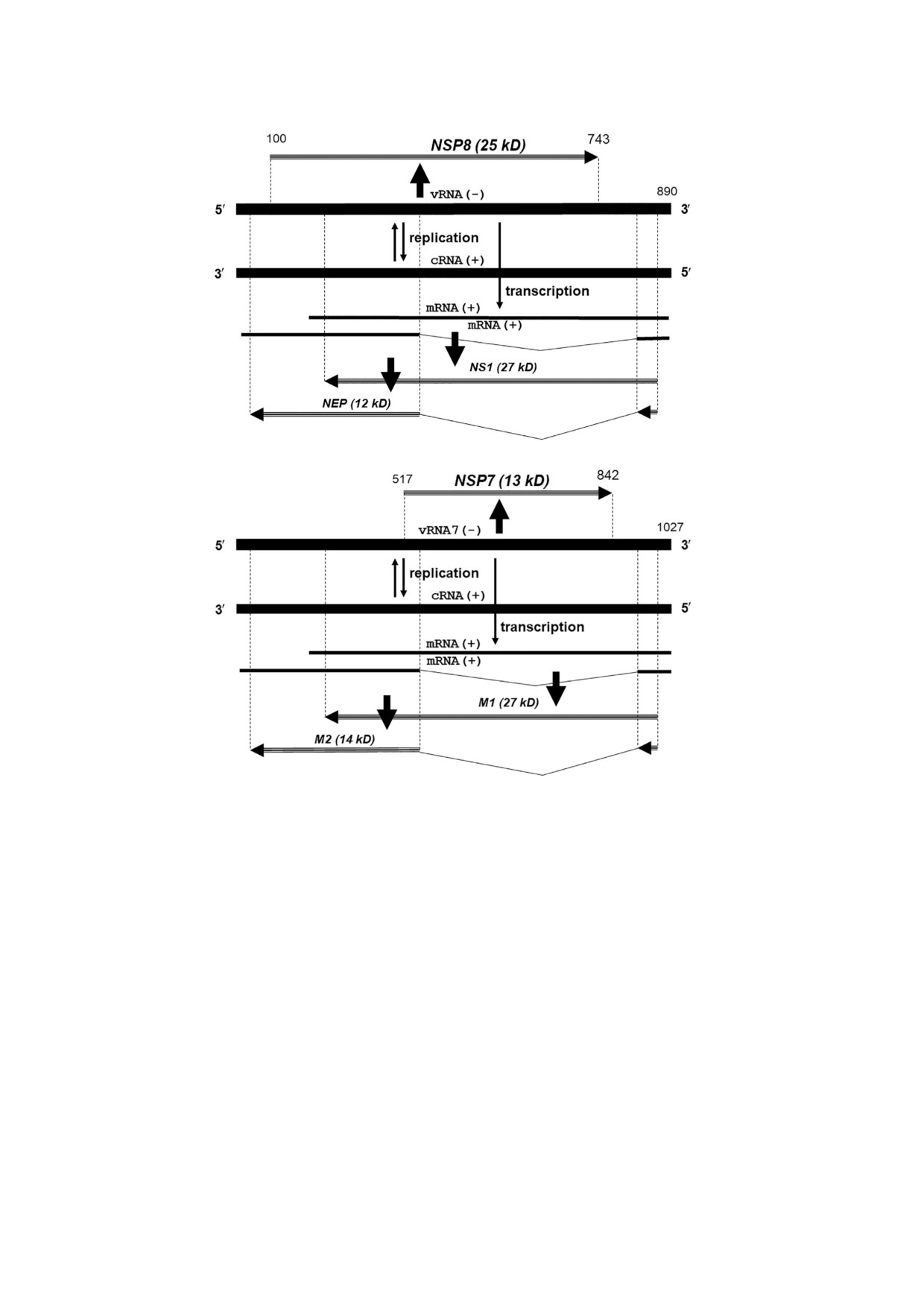
Рис. 1. Схема архитектуры генов в сегментах 7 (М) и 8 (NS) РНК вируса гриппа А. Показана локализация генов в сегмен
те NS (а) и М (б) (GenBank ac.n. CY121121.1 и CY121118.1 соответственно) вируса гриппа A/Aichi/2/68 (H3N2). Цифрами
обозначены позиции нуклеотидов от 5′ конца вирионной vRNA. vRNA - вирионная RNA негативной полярности;
cRNA - комплементарная RNA (репликативная форма) позитивной полярности. Экзоны генов для белков NS1, NEP и
NSP8 показаны стрелками. Ломаной линией показана зона сплайсинга мRNA гена NEP
что отсутствие стоп кодонов в гене NSP8 виру
донах рамок трансляции NS1 и NEP соответ
сов гриппа человека не обусловлено простым
ственно (рис. 1, а). Это дает свободу синони
структурным запретом их появления в результа
мичных замен в положении 3 гена NSP8 без по
те возможного сцепленного повреждения генов
вреждения гена NS1. Примечательно, что ОРТ
NS1 и/или NEP. На это указывает, в частности,
гена NSP8 у вирусов гриппа А человека сформи
наличие множественных стоп кодонов, не по
ровалась лишь ~100 лет назад, т.е. этот ген мож
вреждающих гены NS1 и NEP в теле гена NSP8,
но считать «юным» в плане его эволюционного
как у подавляющего большинства вирусов грип
развития. Это наблюдение было сделано на ос
па А птиц и других животных [5], так и присут
новании филогенетического сравнения нуклео
ствие стоп кодонов в гене NSP8 у пандемичес
тидных последовательностей РНК сегмента NS
кого вируса гриппа А человека H1N1pdm09 (см.
известных вирусов гриппа А человека и живот
ниже). Более того, колокализация генов в ви
ных методом мультиметрического алгоритма
русной РНК сегмента NS выстроена так, что
(см. Suppl. в статье Zhirnov et al. [5]).
третий нуклеотид кодона в рамке трансляции
Функция белка NSP8 пока не установлена, и
NSP8 совпадает с 3 м и 2 м нуклеотидами в ко получены лишь первые наблюдения о том, что
БИОХИМИЯ том 85 вып. 3 2020
АРХИТЕКТУРА ГЕНОВ У ВИРУСА ГРИППА
451
при экспрессии гена NSP8 в клетках насекомых
функции нового класса негативно полярных
посредством бакуловируса этот белок синтези
белков необходимы дальнейшие исследования.
ровался в форме полноразмерного продукта гена
Обнаружение нового гена у вирусов гриппа А
и имел цитоплазматическую локализацию по пе
поставило ряд важных вопросов относительно
риферии клеточного ядра [11]. Анализ in silico не
его происхождения, функции и эволюционной
выявляет полной гомологии белка NSP8 c извест
изменчивости. Один из принципиальных вопро
ными белками из доступных баз данных и пока
сов состоит в том, как мог возникнуть новый ген
зывает частичное сходство в первичной структу
NSP8 в позитивной ориентации на геномном
ре белка NSP8 с Q мотивом DEAD доменной
участке, кодирующем два других гена в негатив
РНК хеликазы гриба Ceraceosorus bombacis, пара
ной ориентации? Появление амбиполярного ге
зитирующего на растениях [12], участком проте
на наводит на мысль о существовании пока неиз
азного домена гидролаз класса С19, к которым
вестного закона соответствия (или правила об
относится убиквитин гидролаза шампиньона
ратного детерминирования) амбиполярных ге
Agaricus bisporus var. Burnettii [13], трансмембран
нов на одном участке молекулы РНК. Возможно,
ным доменом потенциал зависимого кальцие
закон состоит в том, что определенный предсу
вого ионного канала одноклеточного эукариота
ществующий ген может предопределять свойства
(протиста) Salpingoeca rosetta [14]. Параметры об
и механизм возникновения нового амбиполяр
наруженного сходства приведены в табл. 1 и по
ного гена. При отсутствии механизма детерми
казаны на рис. 2. Не исключено, что белок NSP8
нирования следует допустить хаотичное накопле
может обладать полифункциональными свой
ние мутаций, ведущее к появлению нового функ
ствами. Структурное сходство с рядом функцио
ционально смыслового гена с последующим от
нальных белков может указывать на наличие у
бором. Вероятность такого события мала, учиты
него как сходной функции, так и интерферирую
вая амбиполярное перекрывание предсуществу
щей ингибиторной активности против этих
ющих нескольких генов, когда изменения в од
функций в организме хозяине. Для раскрытия
ном гене ведут к изменению в сцепленных амби
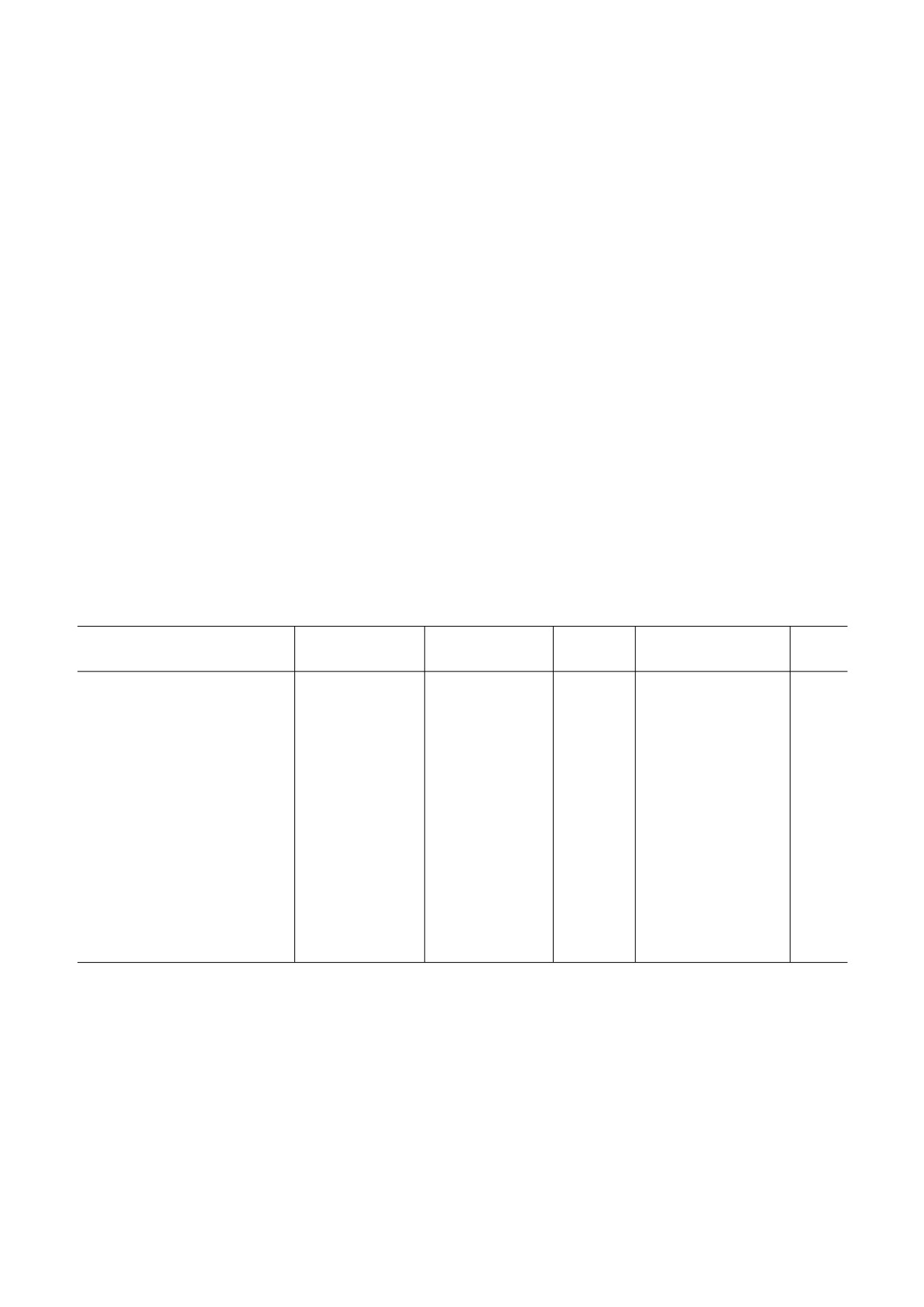
Таблица 1. Параметры сходства первичной структуры
Источник аминокислотной
Участок сравнения
Участок гомологии
Параметры
Функция домена в
Ссылки
последовательности белка NSP8
в белке NSP81)
в белке сравнения2)
сходства3)
белке сравнения3)
NS сегмент вирус гриппа
1-108 (121)
Убиквитин гидро
E: 6.1e 1,
спасение белков от
[14]
A/WSN/33 (H1N1) ac.n. U13683.1
лаза (UH) 479 600
ид.: 31%,
протеосомальной де
(1121)
поз.: 46%
градации в клетках
посредством отщепле
ния убиквитина
NS сегмент вирус гриппа
8-195 (216)
α1 субъединица
E: 4.6e 1,
трансмембранный
[15, 16]
А/Moscow/328/2003 (H3N2)
потенциал зависи
ид.: 26%,
транспорт ионов Ca2+,
ac.n. AAZ38747.1
мого кальциевого
поз.: 41%
участвуют в регуляции
канала G1 158 321
апоптоза и аутофагии
(2453)
клеток, а также в кан
церогенезе раковых
клеток
NS сегмент вирус гриппа
43-120 (216)
Q мотив DEAD
E: 2.4e 0,
отвечает за связыва
[13]
А/Moscow/328/2003 (H3N2)
РНК хеликазы
ид.: 33%,
ние и гидролиз АТР
ac.n. AAZ38747.1
306 383 (904)
поз.: 47%
при функционирова
нии РНК хеликазы
1) Указаны позиции аминокислот в NSP8 на участке гомологии. В скобках указано общее количество аминокислот в бел
ке NSP8.
2) Указаны позиции аминокислот в белке сравнения на участке гомологии. В скобках указано общее количество амино
кислот в белке сравнения.
3) Параметры сходства определены на основании попарного сравнения первичный структур белков в программе UniProt
(рис. 2). Указаны величины Е (мера количества ожидаемых совпадений в случайной базе данных), процент идентичных (ид.)
и позитивных (поз.) аминокислотных позиций в сравниваемых участках белков. Позитивные аминокислотные замены -
обозначают позиции, в которых идентифицированы замены на аминокислоты со сходными свойствами. Белки сравне
ния: трансмембранный домен субъединицы альфа 1/G1 потенциал зависимого кальциевого ионного канала типа Т
(α1/G1) одноклеточного эукариота Salpingoeca rosetta (ID: F2U5C1), участок протеазного домена убиквитин гидролаз (UH)
типа C19 шампиньона Agaricus bisporus var. Burnettii (ID: K5Y1E3), Q мотив DEAD РНК хеликазы растительного гриба
Ceraceosorus bombacis (ID: A0A0P1BHD7).
БИОХИМИЯ том 85 вып. 3 2020
452
ЖИРНОВ
а
б
Рис. 2. Частичное сходство первичной структуры белка NSP8 с белком кальциевого ионного канала (а) и убиквитин гид
ролазы (б). Первичную структуру белка NSP8 вируса гриппа A/Moscow/328/2003 (H3N2) (ac.n. AAZ38747.1) (а) и
BLOSUM 62 с брешами в режиме 250 обратных сравнений. Параметры сходства (Е - мера количества ожидаемых совпа
дений в случайной базе данных, процент идентичных (ид.) и позитивных (поз.) аминокислотных позиций в сравнивае
мых участках белков) приведены в табл.1. Цифры на рисунке показывают позиции аминокислот в соответствующих бел
полярных генах. В этом случае изменчивость и
ти вирионов и являющихся главной мишенью
отбор мутаций должны быть взаимосвязаны
для противовирусных факторов специфическо
сразу в трех вирусных генах: NS1, NEP и NSP8.
го иммунитета организма хозяина (табл. 2). При
Возможно, что амбисенс стратегия имеет
этом два других негативно полярных гена, NS1
место и в других сегментах РНК вируса гриппа А.
и NEP, в сегменте NS имеют более низкую сте
Об этом свидетельствуют протяженные ОРТ, ко
пень вариабельности [17]. Возможны два объяс
торые обнаружены в сегментах PB1, PB2, РА,
нения такого различия. Во первых, белок NSP8
NP и M (рис. 1, б). Белковые продукты перечис
может локализоваться на поверхности вирионов
ленных вирусных ОРТ пока не идентифициро
или инфицированных клеток, подобно НА и
ваны в биологических системах, однако данные
NA, и поэтому служить мишенью воздействия
in silico указывают на трансмембранные свой
факторов врожденного и специфического им
ства ионных каналов у выявленных гипотети
мунитета организма хозяина, провоцирующих
ческих продуктов указанных позитивно поляр
его повышенную изменчивость. Во вторых, бе
ных генов [4, 7]. Присутствие серии амбисенс
лок NSP, будучи молодым вирусным продуктом,
генов во многих сегментах РНК подразумевает
может находиться в стадии оптимизации уровня
наличие у вируса гриппа А человека и животных
экспрессии и своей функции по отношению к
универсального механизма, позволяющего реа
вирусным или клеточным партнерам, что и
лизовать в клетках мишенях как негативно , так
обусловливает его приспособительную измен
и позитивно полярную стратегию генома.
чивость к данным партнерам. Результаты срав
Второй важный вопрос связан с высокой ва
нительного анализа первичной структуры сег
риабельностью белка NSP у вирусов гриппа А
мента NS вирусов гриппа А субтипа H3N2, по
человека. Наши исследования показывают, что
казывающие высокое содержание несинони
белок NSP имеет высокую вариабельность, ко
мичных мутаций в гене NSP8 (dN/dS > 1,5) по
торая близка таковой у наиболее изменчивых
сравнению с генами NS1 и NEP (dN/dS ≤ 1)
гликопротеидов гемагглютинина (НА) и нейра
(табл. 3), полностью согласуются с объяснением
минидазы (NA), расположенных на поверхнос
о повышенной изменчивости гена NSP8 и нали
БИОХИМИЯ том 85 вып. 3 2020
АРХИТЕКТУРА ГЕНОВ У ВИРУСА ГРИППА
453
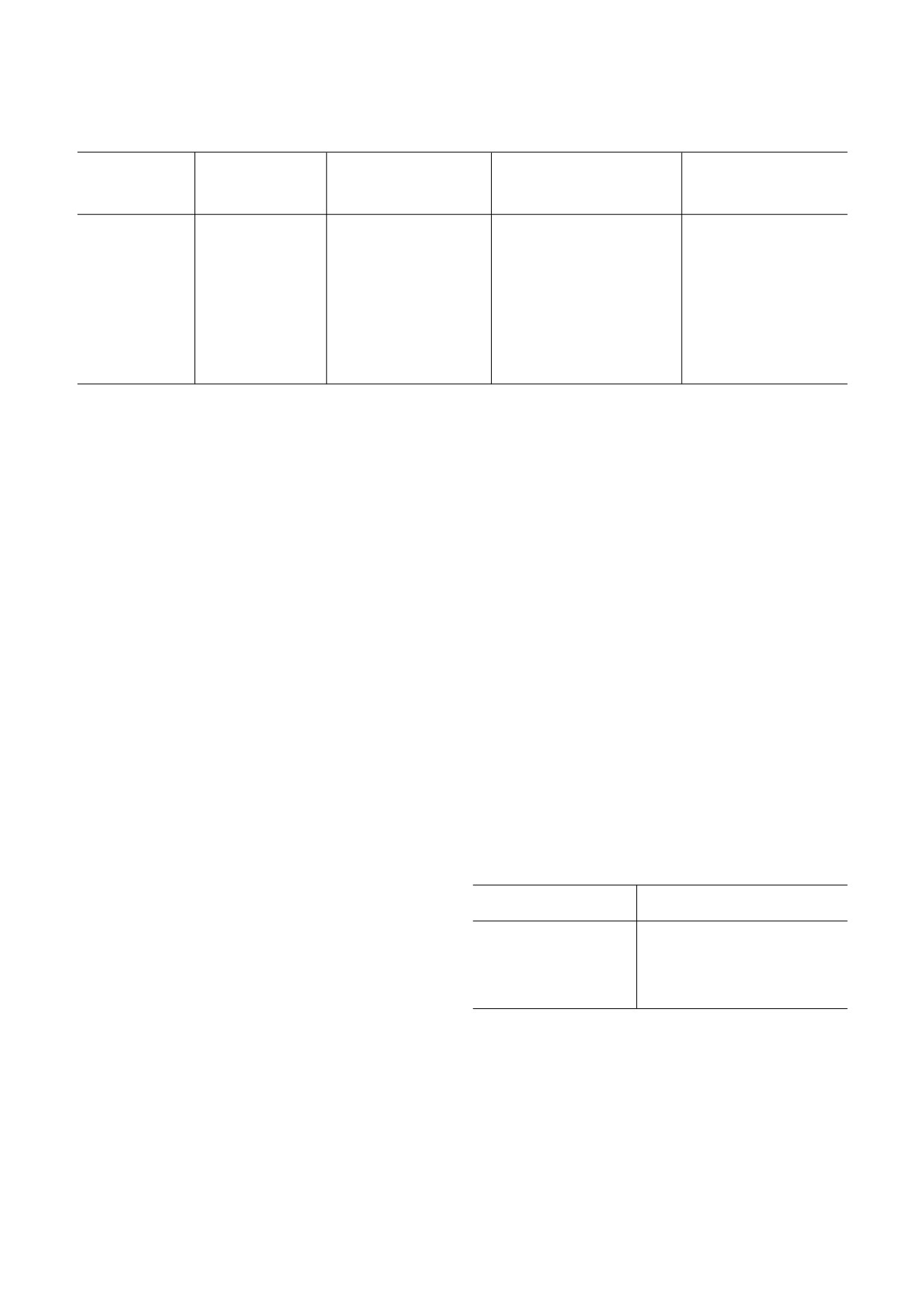
Таблица 2. Коэффициенты эволюционной изменчивости белков вируса гриппа А/H3N2 за период 1968-2018 гг.
Молекулярная
Общее количество
Количество выявленных
Коэффициент
Вирусные белки
масса белка (Да)
проанализированных
замен в белке
вариабельности (КВ)*
последовательностей
PB1
86 000
13 215
23
0,05
HA
75 000
19 639
139
0,4
NA
56 000
17 147
114
0,4
M1
27 000
16 145
12
0,09
NS1
27 000
13 847
34
0,25
NEP
12 000
13 739
5
0,08
NSP8
25 000
14 331
38
0,35
* Коэффициент вариабельности (КВ) определяли по количеству аминокислотных замен в отдельном вирусном белке у
штаммов вируса гриппа человека A/H3N2 за период 1968-2018 гг. КВ рассчитывали как общее количество аминокислот
ных замен за один год в расчете на 100 аминокислот в каждом белке [17]. Использовали данные GenBank. Вирусные бел
ки: PB1 субъединица вирусной полимеразы; HA - гемагглютинин; NA - нейраминидаза; M1 - внутренний матрикс ви
риона; NS1 - антиинтерфероновый белок; NEP - белок ядерного эксперта; NSP8 - белок негативной цепи сегмента 8.
чии фактора(ов) позитивной селекции (адапта
Эволюционная оптимизация гена NSP8 мо
ции) в его эволюции [18]. Количественное на
жет затрагивать регулирующие элементы гена.
копление мутаций в ходе эволюции вируса мо
Так, например, отмечается увеличение количе
жет привести к качественному скачку в его
ства AUG кодонов в стартовой зоне гена NSP8 в
функциональной активности. В этом случае мо
ходе 50 летней эволюции вируса гриппа А чело
жет появиться новая разновидность вируса
века субтипа H3N2: с одного в 1968 году до 4 х в
гриппа с неожиданными и непредсказуемыми
настоящее время [5]. Увеличение количества
свойствами, что таит угрозу для людей и живот
AUG кодонов в зоне трансляционного старта
ных. Изучение нового гена и раскрытие функ
может приводить к облегчению распознавания
ции возникшего белка NSP8 позволит нейтра
вирусной РНК факторами инициации трансля
лизовать возможность такой угрозы либо мето
ции и затем рибосомами и повышению уровня
дами вакцинопрофилактики против данного
ее трансляции в инфицированной клетке ми
белка, либо разработкой лекарственных инги
шени. Более того, такое накопление старт кодо
биторов данного белка или его гена.
нов в вирусной РНК матрице может способ
С предположением о приспособительной
ствовать увеличению дефектной трансляции ге
изменчивости коррелирует динамика эволюции
на NSP8, включая инициацию трансляции с
сегмента NS пандемического вируса гриппа че
ловека H1N1pdm09, который вызвал у людей
пандемию «мексиканского» гриппа в 2009 году
Таблица 3. Соотношение несинонимичных и синонимич
[19]. Этот штамм вируса гриппа А, представляю
ных замен в генах NSP8, NS1, NEP вирусов гриппа А суб
щий тройной реассортант вирусов гриппа чело
типа H3N2 за период 1968-2019 гг.
века, свиней и птиц, мигрировал к человеку из
Вирусный ген
Соотношение dN/dS1)
природного ареала и имел сегмент NS птичьего
типа, лишенного полноценного гена NSP, в теле
которого обнаруживалось 2 стоп кодона. Одна
NSP8
1,73
ко в ходе прошедшей 10 летней эволюции виру
NS1
1,25
са H1N1pdm09 в популяции людей этот ген ут
NEP
0,23
ратил 1 стоп кодон и сейчас увеличил непре
рывную протяженность гена NSP до 240 н.о. Ло
1) Соотношение несинонимичных и синонимичных мута
гично ожидать, что в ближайшее десятилетие
ций (dN/dS) рассчитывали на основании данных первич
при дальнейшей циркуляции и сохранении ви
ной структуры сегмента NS вирусов гриппа А/H3N2 чело
руса в популяции людей вирус утратит остав
века из базы данных GenBank за период 1968-2019 гг. При
расчете анализировали первичные последовательности со
шийся стоп кодон и сформирует полноценный
ответствующих мРНК для белков NSP8, NS1, NEP. Пред
ген NSP, что при его дальнейшей оптимизации
ставлены вирусные гены для белков: NS1 - антиинтерфе
может придать вирусу неожиданные и опасные
роновый белок; NEP - белок ядерного эксперта; NSP8 -
для человека свойства.
белок негативной цепи сегмента 8.
БИОХИМИЯ том 85 вып. 3 2020
454
ЖИРНОВ
альтернативных AUG кодонов, приводящей к
цифических иммунных лейкоцитов против дан
образованию так называемых дефектных пепти
ного белка или его компонентов [17, 22, 23].
дов (DRiPs), которые могут вовлекаться в регу
Свойства NSP8, как «белка невидимки», могут
ляцию MHC I -зависимого противовирусного
быть обусловлены низким уровнем его экспрес
иммунного ответа [20]. Однако принимая во
сии, лабильностью и коротким уровнем полу
внимание, что в бакуловирусной системе [11] и
жизни, а также возможной тканезависимой
в системе трансляции in vitro [10] синтезировал
экспрессией гена NSP8 лишь в определенных
ся полноразмерный и стабильный белок NSP8,
типах клеток мишеней, содержащих факторы,
идея о дефектной трансляции гена NSP8 не ка
необходимые для регуляции экспрессии пози
жется главным механизмом экспрессии.
тивного гена NSP. Дальнейшее изучение долж
Заслуживает внимания вопрос уникальности
но пролить свет на эти предположения.
обнаруженной архитектуры биполярных генов.
Заключительный пункт, связанный с обна
Локализация трех и более разнополярных генов
ружением гена NSP8, относится к возможному
на одном участке одноцепочечной молекулы
изменению классификации семейства ортомик
РНК представляется уникальным типом архи
совирусов (Orthomyxoviridae) и выделению от
тектуры генов среди разнообразных организмов
дельного рода амбиполярных альфа инфлюен
биосферы. Он показывает пример экономии ге
за вирусов («Ambisense Alfainfluenzavirus»). Пока
нетической информации на уровне РНК и ее
прямой продукт гена NSP8 точно не идентифи
эффективной реализации для синтеза разнооб
цирован в биологических системах вопрос о
разных белковых продуктов и создания макси
классификации остается открытым. Вместе с
мального белкового разнообразия на единицу
тем сохранение полноценного гена NSP8 в по
генетической информации в геноме РНК. Уни
пуляции вирусов гриппа человека в течение бо
кальность биполярной архитектуры генов в сег
лее 100 лет указывает на функциональную необ
менте NS вируса гриппа А имеет явное отличие
ходимость этого гена для пока гипотетической
от амбисенс локализации генов, обнаруженной
группы амбиполярных вирусов гриппа. Важно
в геноме РНК флебо , тоспо , аренавирусов жи
отметить, что при выраженной эволюционной
вотных и тениувирусов растений [21]. В отличие
изменчивости белка NSP8 не обнаруживалось
от перечисленных вирусов, у которых два амби
возникновения терминирующих стоп кодонов,
полярных гена не перекрываются и размещены
нарушающих ген NSP8, что также укрепляет
в разных участках молекулы РНК, у вирусов
идею о биологической детерминированности
гриппа А человека три амбиполярных гена ко
данного гена.
локализованы один над другим (так называемая
«стопка») на одном участке РНК (рис. 1, а, б).
При неперекрывающейся архитектуре биполяр
Финансирование. Работа выполнена при час
ных генов их изменчивость напрямую не сцеп
тичной поддержке научной программой SFB 293
лена между собой, и они могут изменяться неза
Немецкого научного общества (DFG).
висимо друг от друга.
Благодарности. Автор выражает благодар
Белковый продукт гена NSP пока не обнару
ность академику РАН Георгиеву Г.П. за поддер
жен в биологических системах, таких как инфи
жку работы и полезные дискуссии, а также д ру
цированные вирусом клеточные культуры или
D. Anhlan (Институт вирусологии Университета
организм животных, но имеются данные, под
г. Мюнстер, Германия) за помощь в анализе ва
тверждающие возможность экспрессии вирусно
риабельности гена NSP.
го РНК генома по позитивному пути в биологи
Конфликт интересов. Автор заявляет об отсут
ческих системах. Так, полноценный белок NSP8
ствии конфликта интересов в финансовой или
синтезируется в системе трансляции in vitro с ри
какой либо иной сфере.
босомами млекопитающих на матрице полно
Соблюдение этических норм. Настоящий об
размерной вирионной РНК вируса гриппа [10],
зор не содержит описания выполненных авто
а также в организме инфицированных живот
ром исследований с участием людей или ис
ных удается детектировать формирование спе
пользованием животных в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Dou, D., Revol, R., Ostbye, H., Wang, H., and Daniels, R.
es: an overview of recently discovered proteins, Virus Res.,
(2018) Influenza A virus cell entry, replication, virion
185, 53-63, doi: 10.1016/j.virusres.2014.03.015.
assembly and movement, Front. Immunol., 9, 1581,
3.
Baez, M., Zazra, J.J., Elliott, R.M., Young, J.F., and
doi: 10.3389/fimmu. 2018.01581.
Palese, P. (1981) Nucleotide sequence of the influenza
2.
Vasin, A.V., Temkina, O.A., Egorov, V.V., Klotchenko, S.A.,
A/duck/Alberta/60/76 virus NS RNA: conservation of the
Plotnikova, M.A., and Kiselev, O.I. (2014) Molecular
NS1/NS2 overlapping gene structure in a divergent influen
mechanisms enhancing the proteome of influenza A virus
za virus RNA segment, Virology, 113, 397-402.
БИОХИМИЯ том 85 вып. 3 2020
АРХИТЕКТУРА ГЕНОВ У ВИРУСА ГРИППА
455
4.
Zhirnov, O.P., Poyarkov, S.V., Vorob’eva, I.V., Safonova, O.A.,
14. Quesada, V., Dýěaz Perales, A., Gutiérrez Fernández, A.,
Malyshev, N.A., and Klenk, H.D. (2007) Segment NS of
Garabaya, C., Cal, S., and López Ot n, C. (2004) Cloning
influenza A virus contains an additional gene NSP in posi
and enzymatic analysis of 22 novel human ubiquitin specif
tive sense orientation, Dokl. Biochem. Biophys., 414,
ic proteases, Biochem. Biophys. Res. Commun., 314, 54-62,
127-33, doi: 10.1134/s1607672907030106.
doi: 10.1016/j.bbrc.2003.12.050.
5.
Zhirnov, O.P., Vorobjeva, I.V., Saphonova, O.A., Poyarkov, S.V.,
15. Vervliet, T., Clerix, E., Seitaj, B., Ivanova, H., Monaco, G.,
Ovcharenko, A.V., Anhlan, D., Malyshev, A.N., and Klenk, H.D.
and Bultynck, G. (2017) Modulation of Ca2+ signaling by
(2009) Structural and evolutionary characteristics of HA,
anti apoptotic B cell lymphoma 2 proteins at the endoplas
NA, NS and M genes of clinical influenza A/H3N2 viruses
mic reticulum mitochondrial interface, Front. Oncol., 7, 75,
passaged in human and canine cells, J. Clin. Virol., 45,
doi: 10.3389/fonc.2017.00075.
322-333, doi: 10.1016/j.jcv.2009.05.030.
16. Lauren, A., and Martin Caraballo, M. (2019) T type calci
6.
Clifford, M., Twigg, J., and Upton, C. (2009) Evidence for
um channels in cancer, Cancers, 11, 134, doi: 10.3390/
a novel gene associated with human influenza A viruses,
cancers11020134.
Virol. J., 6, 198, doi: 10.1186/1743 422X 6 198.
17. Zhirnov, O.P., Konakova, T.E., Anhlan, D., Ludwig, S.,
7.
Gong, Y.N., Chen, G.W., Chen, C.J., Kuo, R.L., and Shih, S.R.
and Isaeva, T.I. (2019) Cellular immune response in
(2014) Computational analysis and mapping of novel open
infected mice to NSP protein encoded by the negative
reading frames in influenza A viruses, PLoS One, 9,
strand NS RNA of influenza A virus, Microbiol. Indep.
e115016, doi: 10.1371/journal.pone.0115016.
Res. J., 6, 10-17, doi: 10.18527/2500 2236 2019 6 1 28
8.
Yang, C.W., and Chen, M.F. (2016) Uncovering the poten
36.
tial pan proteomes encoded by genomic strand RNAs of
18. Yang, Z., and Bielawski, J.P. (2000) Statistical methods for
influenza A viruses, PLoS One, 11, e0146936, doi: 10.1371/
detecting molecular adaptation, Trends Ecol. Evol., 15,
journal.pone. 0146936.
496-503, doi: 10.1016/s0169 5347(00)01994 7.
9.
Sabath, N., Morris, J.S., and Graur, D. (2011) Is there a
19. York, I., and Donis, R.O. (2013) The 2009 pandemic
twelfth protein coding gene in the genome of influenza A?
influenza virus: where did it come from, where is it now, and
A selection based approach to the detection of overlapping
where is it going? Curr. Top. Microbiol. Immunol., 370,
genes in closely related sequences, J. Mol. Evol., 73,
241-257, doi: 10.1007/82_2012_221.
305-315, doi: 10.1007/s00239 011 9477 9.
20. Wei, J., and Yewdell, J.W. (2018) Flu DRiPs in MHC class I
10.
Zhirnov, O.P., Akulich, K.A., Lipatova, A.V., and Usachev, E.V.
immunosurveillance, Virol. Sin., 34, 162-167, doi: 10.1007/
(2017) Negative sense virion RNA of segment 8 (NS) of
s12250 018 0061 y.
influenza a virus is able to translate in vitro a new viral pro
21. Nguyen, M., and Haenni, A.L. (2003) Expression strategies
tein, Dokl. Biochem. Biophys., 473, 122-127, doi: 10.1134/
of ambisense viruses, Virus Res., 93, 141-150, doi: 10.1016/
S1607672917020090.
s0168 1702(03)00094 7.
11.
Жирнов О.П., Кленк Х.Д. (2010) Интеграция гена NSP
22. Zhong, W., Reche, P.A., Lai, C.C., Reinhold, B., and
вируса гриппа А в геном бакуловируса и его экспрессия
Reinherz, E.L. (2003) Genome wide characterization of
в клетках насекомых, Вопросы вирусологии, 55, 4-8.
a viral cytotoxic T lymphocyte epitope repertoire,
12.
Cunningham, J.L., Bakshi, B.K., Lentz, P.L., and Gilliam, M.S.
J. Biol. Chem., 278, 45135-45144, doi: 10.1074/jbc.
(1976) Two new genera of leaf parasitic fungi (Basidio)
M307417200.
mycetidae: Brachybasidiaceae), Mycologia, 68, 640-654.
23. Hickman, H.D., Mays, J.W., Gibbs, J., Kosik. I., Magadan,
13.
Tanner, N.K., Cordin, O., Banroques, J., Doere, M., and
J.G., Takeda, K., and Yewdell, J.W. (2018) Influenza A virus
Linder, P. (2001) The Q motif: a newly identified motif in
negative strand RNA is translated for CD8+ T cell immuno
DEAD box helicases may regulate ATP binding and hydro
surveillance, J. Immunol., 201, 1222-1228, doi: 10.4049/
lysis, Mol. Cell., 11, 127-138.
jimmunol.1800586.
UNIQUE BIPOLAR GENE ARCHITECTURE
IN THE RNA GENOME OF INFLUENZA A VIRUS
O. P. Zhirnov
1 Ivanovsky Institute of Virology, Gamaleya Scientific Research Center of Epidemiology and Microbiology,
Russian Ministry of Health, 123098 Moscow, Russia; E)mail: zhirnov@inbox.ru
2 Russian)German Academy of Medical and Biotechnological Sciences, 121205 Moscow, Skolkovo, Russia
Received November 6, 2019
Revised December 22, 2019
Accepted December 22, 2019
The genome of influenza A virus consists of eight single stranded negative polarity RNA segments. The eighth seg
ment (NS) encodes the anti interferon protein NS1 (27 kDa) and the nuclear export protein NEP (14 kDa) via the
classic negative sense strategy. It also contains an additional positive sense open reading frame can be directly trans
lated into the negative strand protein 8 (NSP8; 18 25 kDa in different strains). The existence of three or more genes
of the opposite polarity in the same locus of a single stranded RNA appears to be a unique (“economical”) type of
gene architecture in living organisms. In silico analysis of genomes of human and animal influenza A viruses revealed
that the NSP8 gene had emerged in the influenza A virus population about 100 years ago (“young” gene) and is high
ly evolutionary variable. The obtained experimental data suggest that NSP8 gene is expressed in the infected animals,
which strengthens the concept of bipolar (ambisense) strategy of the influenza A virus genome. The high variability of
the NSP8 protein suggests that the “young” NSP8 gene is in the process of functional optimization. Further accu
mulation of mutations may alter the functions of mature NSP8 protein and lead to the emergence of mature bipolar
influenza A virus with some unexpected properties that would be threatening for humans and animals.
Keywords: influenza virus, genome strategy, NSP gene, ambisense viruses, gene structure
БИОХИМИЯ том 85 вып. 3 2020