БИОХИМИЯ, 2020, том 85, вып. 5, с. 695 - 705
УДК 577.15: 541.128
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239
В ПРОЦЕССЕ УЗНАВАНИЯ ПОВРЕЖДЕННОГО НУКЛЕОТИДА
УРАЦИЛ ДНК ГЛИКОЗИЛАЗОЙ ЧЕЛОВЕКА SMUG1*
© 2020
Д.А. Яковлев1,2, И.В. Алексеева1, Н.А. Кузнецов1**, О.С. Федорова1**
1 Институт химической биологии и фундаментальной медицины СО РАН, 630090 Новосибирск, Россия;
электронная почта: nikita.kuznetsov@niboch.nsc.ru, fedorova@niboch.nsc.ru
2 Новосибирский государственный университет, факультет естественных наук, 630090 Новосибирск, Россия
Поступила в редакцию 03.02.2020
После доработки 17.03.2020
Принята к публикации 19.03.2020
Урацил ДНК гликозилаза человека SMUG1 удаляет из ДНК остатки урацила и некоторые другие некано
нические или поврежденные основания. Несмотря на функциональную важность SMUG1, для нее до сих
пор не получены рентгеноструктурные данные. Ранее нами проведено гомологичное моделирование струк
туры SMUG1 и сделан вывод о роли некоторых аминокислотных остатков в процессе узнавания поврежден
ных нуклеотидов и их удаления из ДНК. В настоящей работе методом «остановленного потока» в режиме
реального времени по изменению интенсивности флуоресценции остатков Trp фермента и 2 аминопурина
и флуорофоров, обеспечивающих резонансный перенос энергии флуоресценции (FRET), в ДНК изучена
кинетика конформационных переходов в белке и ДНК субстратах в ходе ферментативного процесса. Уста
новлен кинетический механизм взаимодействия и рассчитаны кинетические параметры образования и рас
пада интермедиатов реакции. Полученные результаты позволили уточнить роль а.о. His239 и Arg243 в про
цессе узнавания поврежденного нуклеотида и удаления повреждения при катализе урацил ДНК гликози
лазой человека SMUG1.
КЛЮЧЕВЫЕ СЛОВА: репарация ДНК, урацил ДНК гликозилаза человека SMUG1, флуоресценция, кон
формационная динамика, метод «остановленного потока».
DOI: 10.31857/S0320972520050085
ВВЕДЕНИЕ
ДНК гликозилазой (SMUG1) и метил CpG
связывающим доменом 4 (MBD4) [7, 8]. Не
Урацил - одно из наиболее распространен
смотря на то, что все урацил ДНК гликозилазы
ных повреждений ДНК, которое образуется при
катализируют гидролиз N гликозидной связи с
спонтанном [1-3] или ферментативном [4, 5]
урацилом, они обладают индивидуальными от
дезаминировании цитозина, а также при оши
личиями, связанными со способностью фер
бочном включении остатка уридина во время
ментов узнавать одноцепочечные ДНК субстра
репликации [6]. Урацил в ДНК человека узнает
ты, мисматчи T/G [9-13] или некоторые окис
ся и удаляется несколькими ДНК гликозилаза
ленные основания, такие как 5 гидроксиметил
ми: урацил ДНК N гликозилазой (UNG2), ти
урацил, 5 формилурацил и 5 гидроксиурацил,
мин ДНК гликозилазой (TDG), специфичной
которые являются природными производными
к одноцепочечной ДНК монофункциональной
5 метилцитозина и образуются в процессе эпи
генетической регуляции [14].
Принятые сокращения: SMUG1 - специфичная к
Одной из актуальных проблем в области ре
одноцепочечной ДНК монофункциональная ДНК глико
парации ДНК остается выяснение механизмов,
зилаза; АР сайт - апуриновый/апиримидиновый сайт; F -
(2R,3S) 2 (гидроксиметил) 3 гидрокситетрагидрофуран;
обеспечивающих высокоточное узнавание по
aPu - 2 аминопурин, FAM - 6 карбоксифлуоресцеин;
врежденных оснований. В последние годы ак
BHQ1 - тушитель флуоресценции «black hole quencher»; тивно проводили исследования различных
FRET - резонансный перенос энергии флуоресценции;
ДНК гликозилаз, направленные на выяснение
PAGE- электрофорез в полиакриламидном геле, WT - ди
кий тип.
того, каким образом происходит поиск и узна
вание единичных повреждений гетероцикли
* Статья на английском языке опубликована в режиме от
ческих оснований ДНК среди огромного числа
springer.com/journal/10541), том 85, вып. 5, 2020.
немодифицированных оснований [15]. Анализ
** Адресат для корреспонденции.
литературных данных показывает [16], что раз
695
696
ЯКОВЛЕВ и др.
личные ДНК гликозилазы, имеющие совер
в двойную спираль ДНК между основаниями
шенно разную структуру ДНК связывающего
аминокислотный остаток - сенсор поврежде
центра, активного центра и аминокислотных
ния или «клин» (wedge amino acid) [17]. Эта мо
остатков, участвующих в специфическом узна
дель узнавания повреждений была значительно
вании поврежденного нуклеотида и катализе,
развита в исследованиях с применением одно
тем не менее обладают общими особенностями
молекулярных кинетических методов (single
при взаимодействии с субстратами. Так, боль
molecule kinetics) [18-21] и предстационарных
шая часть ДНК гликозилаз, для которых в нас
кинетических методов [22], которые позволили
тоящее время установлена структура, изгибают
в реальном времени визуализировать движение
ДНК и выворачивают поврежденный либо
фермента по молекуле ДНК при поиске повреж
комплементарный к нему нуклеотид из двойной
денного нуклеотида и изучить динамику кон
спирали ДНК. При этом, как правило, повреж
формационных перестроек фермента и ДНК
денный нуклеотид располагается в кармане ак
как на начальных, так и на последующих стади
тивного центра фермента, где происходит его
ях узнавания повреждения и их каталитического
окончательная верификация. В образовавшую
превращения.
ся полость в дуплексе ДНК встраиваются неко
В настоящее время структура ДНК гликози
торые аминокислотные остатки фермента, ко
лазы человека SMUG1 неизвестна. Ранее нами с
торые выталкивают поврежденный нуклеотид
помощью гомологичного моделирования [23] на
из спирали ДНК и стабилизируют внеспираль
основе структур с GmeSMUG1 из Geobacter met*
ное положение нуклеотида. Далее происходит
allireducens (Protein Data Bank [PDB] ID 5H98 и
гидролиз N гликозидной связи между повреж
5H9I) [24] и xSMUG1 из Xenopus laevis (PDB
денным гетероциклическим основанием и ос
1OE4 и 10OE5) [10] получена модель структуры
татком рибозы и образование апуринового/апи
фермента человека в свободном состоянии и в
римидинового сайта (АР сайта).
комплексе с ДНК, содержащей уридин (рис. 1).
В настоящее время наиболее обоснованным
Было показано, что 11 а.о. (239-249) SMUG1
механизмом узнавания поврежденных нуклео
человека формируют интеркалирующую петлю,
тидов ДНК гликозилазами на начальных этапах
которая может служить «клином», проникаю
взаимодействия является модель, в соответ
щим в двойную спираль ДНК в области повреж
ствии с которой фермент поочередно вставляет
денного нуклеотида [10, 25]. При этом функцио
нально важными а.о. интеркалирующей петли
могут являться Arg243, который занимает прост
ранство внутри спирали ДНК, образовавшееся
после выворачивания урацила, и His239, спо
собный образовывать контакты и с урацилом,
расположенным в активном центре, и с фосфат
ной группой ДНК. Мутационный анализ пока
зал [23, 25], что His239 необходим для протека
ния каталитического гидролиза N гликозидной
связи.
Кроме того, ранее [26] методом «останов
ленного потока» с регистрацией изменения ин
тенсивности флуоресценции остатков трипто
фана, входящих в состав фермента, и 2 амино
пурина (аPu) или FRET пары флуорофор/ту
шитель (6 карбоксифлуоресцеин (FAM) и ту
шитель флуоресценции «black hole quencher»
(BHQ1)), обеспечивающих резонансный перенос
энергии флуоресценции, расположенных в мо
дельных ДНК субстратах, был предложен ки
нетический механизм взаимодействия SMUG1
дикого типа (WT) с поврежденной ДНК (схе
ма 1). Показано, что взаимодействие SMUG1 с
ДНК включает, как минимум, две стадии (1 и 2)
связывания ДНК и узнавания поврежденного
нуклеотида, которые приводят к образованию
Рис. 1. Модель структуры SMUG1 человека в комплексе с
каталитически компетентного комплекса. В
ДНК, содержащей уридин
этом комплексе происходит необратимая ста
БИОХИМИЯ том 85 вып. 5 2020
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239 В SMUG1
697
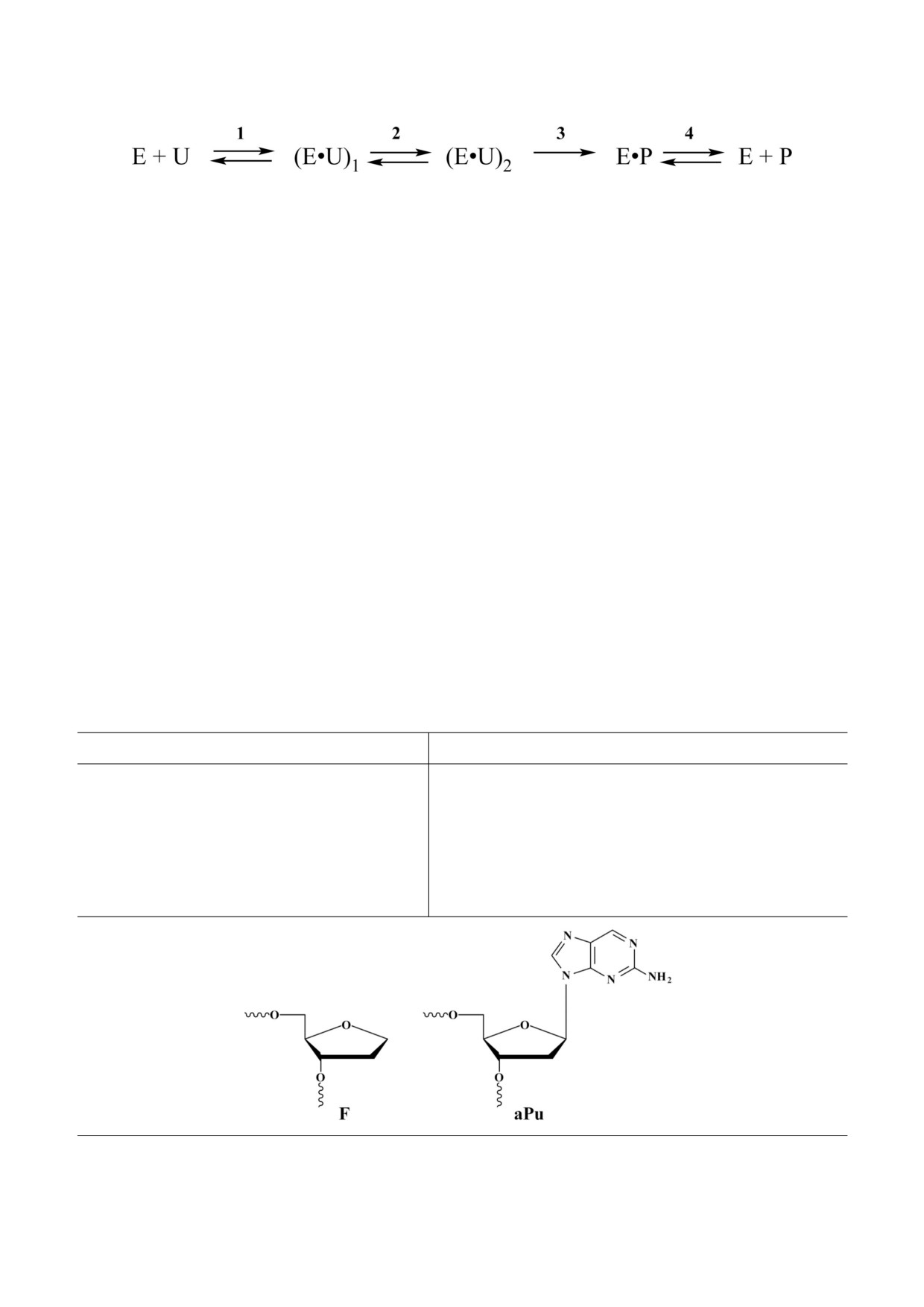
Схема 1. Кинетический механизм, характеризующий стадии взаимодействия SMUG1 WT человека с поврежденной ДНК
[26]. E - SMUG1; U - ДНК субстрат, содержащий уридин; (E•U)n - различные фермент субстратные комплексы, обра
зующиеся в ходе узнавания урацила; E•P - комплекс фермента с продуктом реакции Р
дия (3) каталитического гидролиза N гликозид
N гликозилазной реакции методом электрофо
ной связи с урацилом. Последняя стадия кине
реза в полиакриламидном геле (PAGE) нами бы
тического механизма (4) характеризует равно
ло показано, что мутантная форма SMUG1, со
весный процесс диссоциации комплекса фер
держащая замену H239A, каталитически неак
мента с продуктом реакции.
тивна, тогда как мутант R243A полностью сох
Анализ флуоресцентных данных, характери
раняет свою активность [23]. Для уточнения
зующих конформационные переходы как моле
функции остатков Arg243 и His239 в процессе
кулы SMUG1, так и ДНК в ходе каталитическо
специфического узнавания поврежденного нук
го цикла свидетельствует о том, что на первой
леотида и катализе в настоящем исследовании
стадии взаимодействия (1) происходит переме
был проведен комплексный предстационарный
щение интеркалирующей петли фермента, ко
кинетический анализ конформационных изме
торое сопровождается частичным плавлением
нений в мутантных формах SMUG1 (R243A и
ДНК дуплекса вблизи поврежденного нуклео
H239A) и в ДНК субстрате, а также в продукте
тида. По видимому, образование контактов на
его превращения. Для этого проводили регист
этой стадии взаимодействия также индуцирует
рацию изменений интенсивности флуоресцен
частичное выворачивание поврежденного осно
ции остатков Trp в молекуле фермента и аPu,
вания из ДНК дуплекса. Вторая стадия (2) ха
расположенного с 3′ стороны от апуриново
рактеризует процесс встраивания аминокислот
го/апиримидинового сайта в ДНК, или FRET
ных остатков, входящих в интеркалирующую
пары, введенной на 3′ и 5′ концы ДНК дуплек
петлю фермента. Ранее при исследовании кон
са. Эти данные позволили более детально прос
формационной динамики ДНК субстрата мето
ледить конформационные переходы белка и
дом FRET и кинетики накопления продукта
ДНК в процессе взаимодействия. Итогом
Таблица 1. Последовательности дезоксирибоолигонуклеотидов и структуры модифицированных нуклеотидов F и aPu
Сокращенное название
Последовательность
X субстрат/лиганд
5′ GCTCA(X)GTACAGAGCTG 3′
X = U/F
3′ CGAGT G CATGTCTCGAC 5′
X aPu субстрат/лиганд
5′ GCTCA(X)(aPu)TACAGAGCTG 3′
X = U/F
3′ CGAGT G C ATGTCTCGAC 5′
FRET X субстрат/лиганд
5′ FAM GCTCA(X)GTACAGAGCTG 3′
X = U/F
3′ CGAGT G CATGTCTCGAC BHQ1 5′
* F - стабильный аналог АР сайта, (2R,3S) 2 (гидроксиметил) 3 гидрокситетрагидрофуран; aPu - 2 аминопурин.
БИОХИМИЯ том 85 вып. 5 2020
698
ЯКОВЛЕВ и др.
а
представленной работы является уточнение ро
ли функционально значимых остатков His239 и
Arg243 в процессе узнавания поврежденного
основания и его удаления при катализе ДНК
гликозилазой человека SMUG1.
МАТЕРИАЛЫ И МЕТОДЫ
В работе были использованы реактивы: ак
риламид, N,N′ метиленбисакриламид, ДТТ, мо
чевина, ЭДТА, ацетонитрил, глицерин, Tris
(гидроксиметил) аминометан («Sigma Aldrich»,
США).
ДНК субстраты. Субстраты и лиганды фер
мента представляли собой 17 ти звенные дуп
лексы, состоящие из дезоксирибоолигонуклео
тидов, представленных в табл. 1. Олигонуклео
б
тиды очищали с помощью ВЭЖХ на ионно об
менной колонке PRP X500 (3,9 × 300 мм; размер
частиц 12-30 мкм; «Hamilton Company», США)
и последующей обращено фазовой хроматогра
фии (Nucleoprep 100-20 C18
10
× 250 мм,
«Macherey Nagel», Германия). Гомогенность
препаратов проверяли методом электрофореза в
20% ном (w/v) PAGE в денатурирующих условиях.
Фермент SMUG1. Мутантные формы фер
мента SMUG1, содержащие замену R243A или
H239A, были выделены из клеток Escherichia coli
штамма Rosetta 2, трансформированных плаз
мидой pET28c, несущей ген урацил ДНК гли
козилазы человека, как описано ранее [23, 26].
Все эксперименты по исследованию фермента
тивной реакции проводили в буферном раство
ре: 50 мМ Tris HCl, pH 7,5, 50 мМ KCl, 1 мМ
в
ЭДТА, 1 мМ ДТТ, 7% (v/v) глицерин при 25 °C.
Изучение кинетики процесса методом «оста
новленного потока». Флуоресцентные кине
тические кривые были получены на спектромет
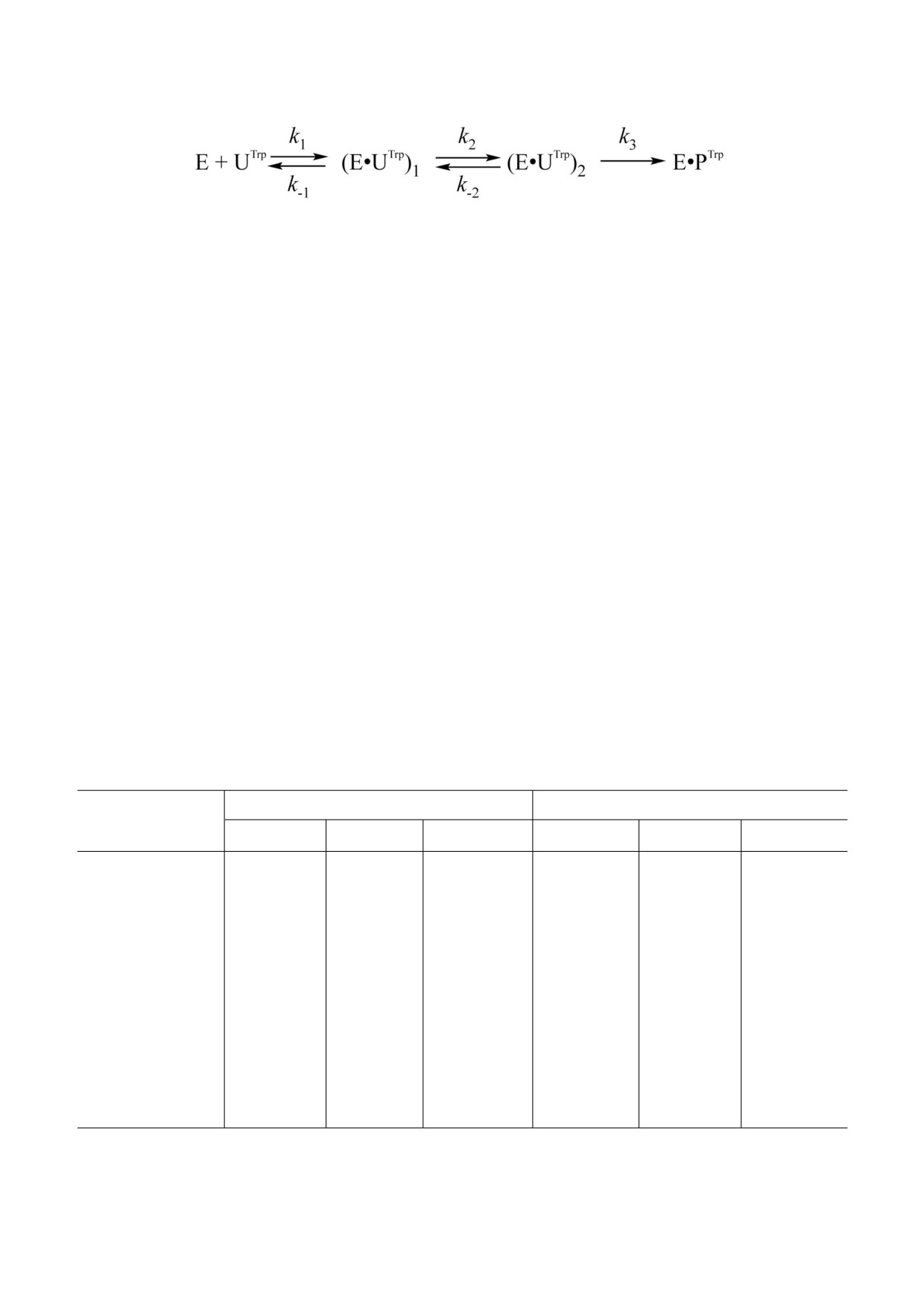
Таблица 2. Константы скорости и равновесия, соответству
ющие Схеме 2, описывающей взаимодействие SMUG1
R243A с ДНК, содержащей F сайт
Флуорофор
Константа
Trp
aPu
FRET
k1 × 10-6, M-1⋅с-1
35 ± 8
14 ± 4
6 ± 2
k-1, с-1
0,5 ± 0,3
5,4 ± 1,0
30 ± 7
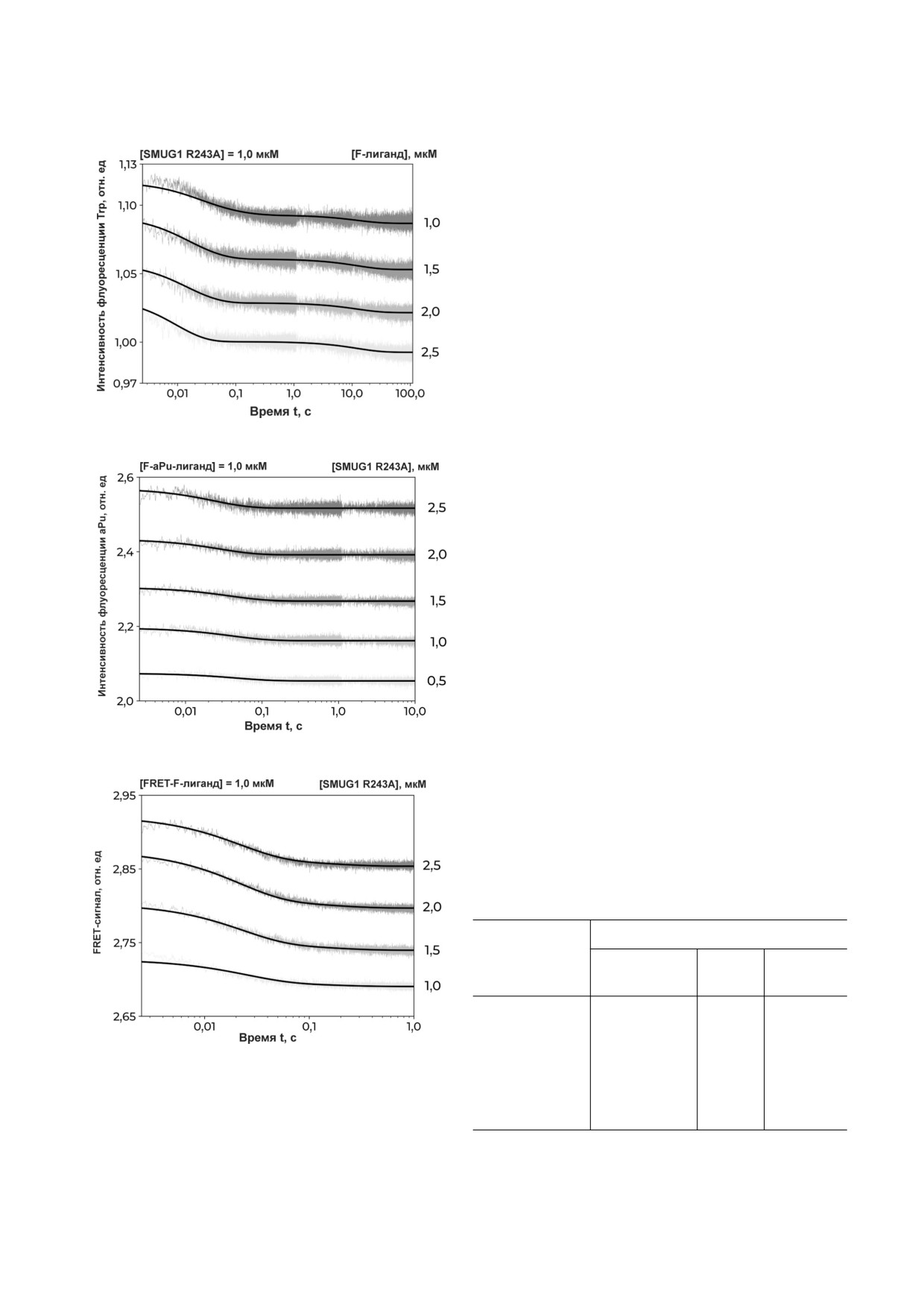
Рис. 2. Взаимодействие R243A SMUG1 с ДНК дуплексами,
k2, с-1
(1,1 ± 0,8) × 10-2
-
0,4 ± 0,1
содержащими F сайт. Изменения интенсивности флуо
ресценции остатков Trp белка (а), интенсивности флуорес
k-2, с-1
(7 ± 2) × 10-2
-
6 ± 2
ценции остатков aPu, расположенных с 3′ стороны от F
сайта (б) и FRET сигнала (в). Концентрации фермента и
ДНК указаны на рисунке
* Различия статистически достоверны при p < 0,05.
БИОХИМИЯ том 85 вып. 5 2020
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239 В SMUG1
699
Схема 2. Кинетический механизм, характеризующий образование комплекса SMUG1 R243A с продуктом реакции F, за
регистрированный по изменению интенсивности флуоресценции Trp и FRET сигнала
ре остановленной струи SX.20
(«Applied
аналогом продукта приводит к двухфазному
Photophysics», Великобритания), как описано
уменьшению интенсивности флуоресценции
ранее [27-30]. Белок SMUG1 содержит 4 остат
остатков Trp в течение времени t ≤ 100 с (рис. 2, а).
ка Trp и 4 остатка Tyr. Возбуждение флуоресцен
Падение сигнала на начальном участке кинети
ции SMUG1 проводили при 290 нм, регистра
ческих кривых, в течение 30-50 мс, по видимо
цию флуоресценции - при длинах волн >
му, соответствует образованию первичных нес
> 320 нм, фильтр WG 320 («Schott», Германия).
пецифических контактов с ДНК. Затем проис
В этих условиях основной вклад в флуоресцен
ходит небольшое снижение интенсивности флу
цию белка обеспечивается остатками Trp
оресценции во временном интервале t ≤ 10 с.
(>90%). При использовании субстратов, содер
Конформационные изменения фермента при
жащих остатки aPu, возбуждение флуоресцен
взаимодействии SMUG1 R243A с F лигандом
ции проводили при длине волны 310 нм, а реги
были описаны с помощью кинетической схе
страцию - при длинах волн > 370 нм, фильтр
мы 2, содержащей две обратимые стадии. Полу
LG 370 («Corion», США). Для анализа эффек
ченные константы скорости приведены в
тивности флуоресцентного резонансного пере
табл. 2.
носа энергии (FRET) пары FAM/BHQ1 флуо
Природа процессов, происходящих на этих
ресценцию красителя FAM возбуждали светом с
стадиях взаимодействия, была выяснена с по
длиной волны 494 нм. Регистрация флуоресцен
мощью анализа изменений флуоресценции ос
ции красителя FAM проводилась при длинах
татка aPu, расположенного с 3′ стороны от
волн > 515 нм с использованием светофильтра
F сайта (F aPu лиганд, табл. 1). Этот флуоро
OG 515 («Schott», Германия). Мертвое время
фор чувствителен к изменениям диэлектричес
прибора составляло 1,0 мс. Каждую кинетичес
кой проницаемости среды, и интенсивность его
кую кривую строили, как минимум, по результа
флуоресценции уменьшается при формирова
там трех независимых измерений.
нии гидрофобного микроокружения [35]. Сме
Анализ кинетических данных. Для определе
шивание фермента с F aPu лигандом также вы
ния минимальной кинетической схемы, описы
зывает падение интенсивности флуоресценции
вающей взаимодействие фермента с субстрата
остатка aPu на временах < 200 мс (рис. 2, б), что
ми, и расчета констант скорости конформаци
свидетельствует об образовании вокруг aPu гид
онных переходов в ходе всех элементарных ста
рофобного окружения, например, за счет взаи
дий данной схемы получали набор кинетичес
модействия ДНК с а.о. интеркалирующей пет
ких кривых для разных концентраций фермента
ли. Полученные кинетические кривые описыва
или субстрата. Количественную обработку ре
ли схемой 3, константы скорости приведены в
зультатов экспериментов проводили с помощью
табл. 2.
программы DynaFit («BioKin», США) [31] путем
При этом кинетические кривые, полученные
оптимизации значений параметров, входящих в
при регистрации сигнала FRET в процессе взаи
кинетические схемы, как описано ранее
модействия SMUG1 R243A с FRET F лиган
[32-34].
дом, характеризуются уменьшением сигнала в
начальной области до t ≅ 100-200 мс (рис. 2, в).
Падение сигнала FRET свидетельствует об
РЕЗУЛЬТАТЫ
Взаимодействие SMUG1 R243A со стабиль
ным аналогом продукта N гликозилазной реакции.
Для изучения конформационных изменений
SMUG1 R243A в процессе взаимодействия с
продуктом гидролиза N гликозидной связи был
Схема 3. Кинетический механизм, описывающий образо
использован модельный ДНК дуплекс, содер
вание комплекса SMUG1 R243A с продуктом N гликози
жащий тетрагидрофурановый аналог АР сайта
лазной реакции, зарегистрированный по изменению ин
(F лиганд, табл. 1). Взаимодействие фермента с
тенсивности флуоресценции aPu
БИОХИМИЯ том 85 вып. 5 2020
700
ЯКОВЛЕВ и др.
а
уменьшении расстояния между флуорофорами,
которое может происходить при изгибании мо
лекулы ДНК при связывании с ферментом. Ана
лиз кинетических кривых показал, что этот про
цесс можно описать двухстадийным равновес
ным механизмом (схема 2), константы скорости
приведены в табл. 2.
Таким образом, процесс взаимодействия
SMUG1 R243A с аналогом продукта реакции F
включает как минимум две обратимые стадии.
Анализ формы кинетических кривых (рис. 2) и
значений констант скорости (табл. 2) показывает,
что образование первичного комплекса со
провождается конформационными изменения
ми фермента (стадия 1, Trp, t ≤ 30-50 мс), затем
происходит изменение микроокружения в облас
ти остатка aPu (стадия 1, aPu, t < 100 мс). Такое
последовательное изменение конформации фер
б
мента и ДНК субстрата свидетельствует о том,
что этот процесс, вероятнее всего, связан с пере
мещением интеркалирующей петли и ее взаимо
действием с ДНК дуплексом в области повреж
денного нуклеотида и, возможно, сопровождает
ся выворачиванием F сайта в активный центр
фермента. Образование контактов между ДНК
связывающим центром и ДНК дуплексом также
индуцирует изгибание молекулы субстрата на
временах t < 100-200 мс (стадия 1, FRET). После
дующие более медленные процессы (стадия 2,
FRET и Trp), зарегистрированные по изменению
интенсивности флуоресценции Trp и FRET сиг
нала, по видимому, свидетельствует о дополни
тельных перестройках конформации фермента и
аналога продукта, содержащего остаток F.
Из полученных значений следует, что каж
в
дый из флуорофоров регистрирует разные этапы
конформационных перестроек, протекающих
во взаимодействующих молекулах. Наиболее
медленной стадией является стадия 2, наблюда
емая по флуоресценции Trp, которая, скорее
всего, относится к адаптации структуры
hSMUG1 к структуре F содержащего ДНК ли
ганда, моделирующего продукт N гликозилаз
ной реакции.
Взаимодействие SMUG1 R243A с ДНК, со
держащей уридин. Полный ферментативный
цикл был изучен с использованием модельных
ДНК субстратов, содержащих уридин (табл. 1).
В ходе ферментативной реакции происходит
связывание фермента с субстратом, формирова
ние каталитически компетентного комплекса,
гидролиз N гликозидной связи и диссоциация
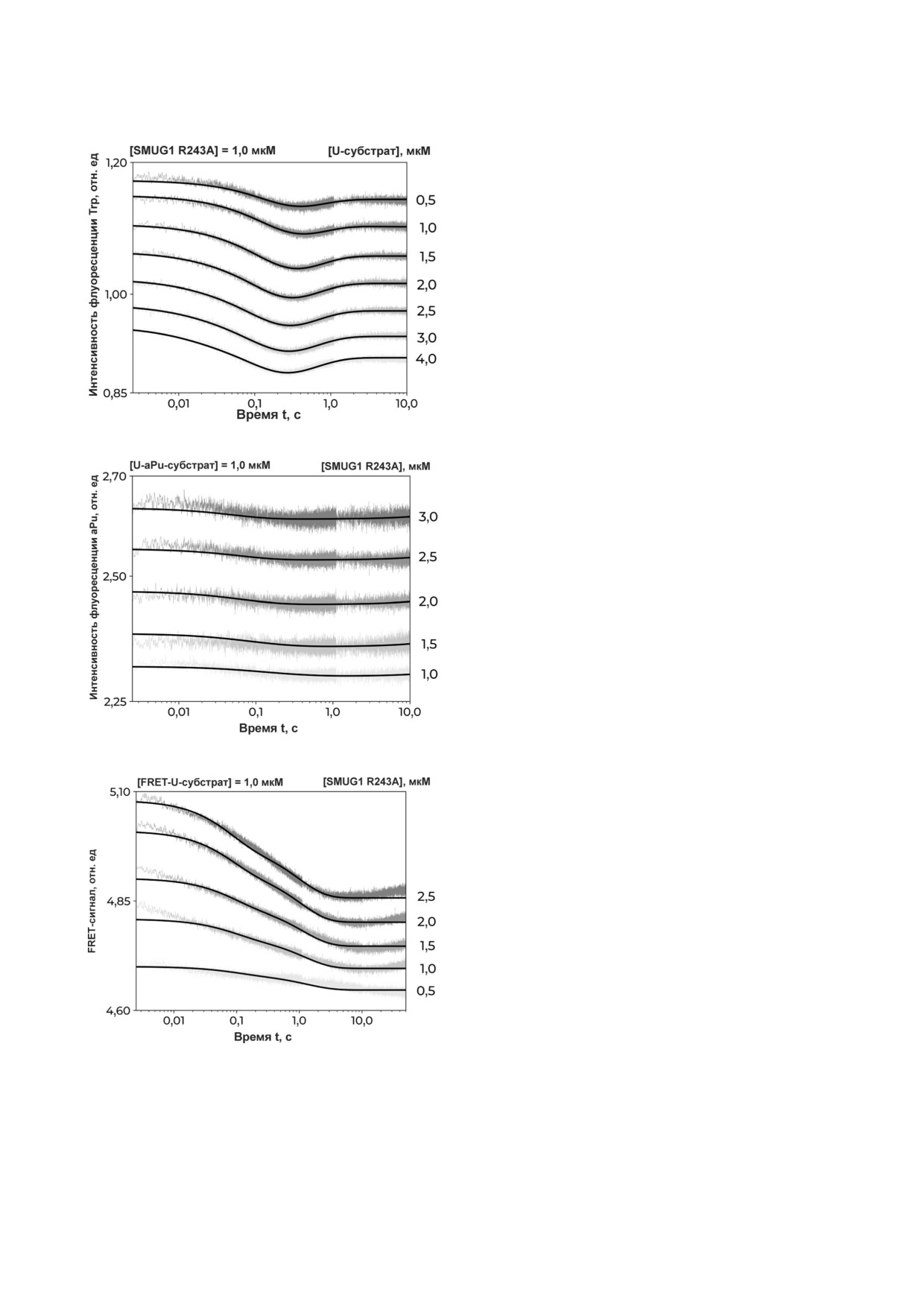
Рис. 3. Взаимодействие R243A SMUG1 с ДНК дуплекса
фермента с продуктом.
ми, содержащими уридин. Изменения интенсивности
Кинетические кривые, характеризующие из
флуоресценции остатков Trp белка (а), интенсивности
менение интенсивности флуоресценции Trp
флуоресценции остатков aPu, расположенных с 3′ сторо
ны от уридина (б) и FRET сигнала (в). Концентрации фер
(рис. 3, а), содержат фазу падения интенсивнос
мента и ДНК указаны на рисунке
ти флуоресценции при t ≈ 0,3 с и фазу роста с
БИОХИМИЯ том 85 вып. 5 2020
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239 В SMUG1
701
Схема 4. Кинетический механизм, характеризующий взаимодействие SMUG1 R243A с U субстратом
выходом на плато к t ≈ 5 с. Сравнение этих кине
Кинетика конформационных изменений в
тических кривых с кривыми, полученными для
ДНК, полученная при регистрации интенсив
процесса связывания с продуктом (рис. 2, а),
ности флуоресценции aPu в U aPu субстрате
показывает, что начальный участок при образо
(рис. 3, б), на начальном участке до 100 мс ана
вании каталитического комплекса с U субстра
логична кинетике взаимодействия SMUG1 с
том имеет похожие изменения в интенсивности
аналогом продукта (рис 2, б). Такая согласован
флуоресценции Trp. Поэтому фазу роста интен
ность конформационных изменений свидетель
сивности флуоресценции Trp на временах
ствует о том, что процесс гидролиза N глико
∼(0,5-10) с, отсутствующую в случае F лиганда,
зидной связи не требует серьезного изменения
можно отнести к процессу гидролиза N глико
геометрии субстрата. При этом в случае U aPu
зидной связи и диссоциации комплекса фер
субстрата происходит незначительное увеличе
мента с продуктом реакции. Ранее для фермента
ние интенсивности флуоресценции на более
SMUG1 WT было показано [26], что диссоциа
поздних временах ≤ 10 с, которое может характе
ция комплекса фермент-продукт является ско
ризовать медленный процесс диссоциации
рость лимитирующей стадией ферментативного
комплекса фермента с продуктом реакции. По
процесса. Поэтому в наблюдаемом временном
лученные кинетические кривые описывали схе
диапазоне (0,5-10 с) скорее всего не происходит
мой 5, что позволило оценить значение конс
процесс диссоциации комплекса фермента с
танты скорости распада комплекса фермента с
продуктом реакции. Действительно, анализ ки
продуктом реакции koff (табл. 3).
нетических кривых показал, что они описыва
При регистрации FRET сигнала в процессе
ются кинетической схемой 4, включающей две
взаимодействия SMUG1 R243A с FRET U
обратимые стадий связывания, за которыми
субстратом (рис. 3, в) были получены кинети
следует необратимая каталитическая стадия,
ческие кривые, содержащие снижение флуорес
приводящая к образованию комплекса фермен
ценции FAM на временах >10 с. При этом паде
та с продуктом (табл. 3).
ние сигнала FRET можно разделить на две фа
Таблица 3. Константы скорости и равновесия, соответствующие взаимодействию SMUG1 R243A с ДНК, содержащей ури
дин
SMUG1 R243A
SMUG1 WT*
Константа
Trp
aPu
FRET
Trp
aPu
FRET
k1 × 10-6, M-1⋅с-1
22 ± 5
8 ± 3
1,2 ± 0,2
115 ± 20
130 ± 60
140 ± 20
k-1, с-1
2,2 ± 0,6
0,19 ± 0,07
8 ± 1
760 ± 90
240 ± 30
410 ± 20
K1 × 10-6, M-1
10
41,6
0,16
0,15
0,54
0,34
k2, с-1
9 ± 3
-
1,7 ± 0,3
30 ± 8
15 ± 8
3,6 ± 0,8
k-2, с-1
0,8 ± 0,1
-
0,64 ± 0,06
11,5 ± 0,8
25 ± 6
9,7 ± 0,2
K2
11,1
-
2,66
2,6
0,6
0,37
k3, с-1
2,0 ± 0,3
-
-
1,7 ± 0,2
-
0,6 ± 0,1
k-3, с-1
-
-
-
-
-
0,11 ± 0,02
K3
-
-
-
-
-
5,4
koff, с-1
-
(8 ± 3) × 10-3
-
-
-
-
K1 × K2 × 10-6, M-1
111
-
0,43
0,39
0,32
-
K1 × K2 × K3
× 10-6, M-1
-
-
−
-
−
0,68
* Данные из работы Kuznetsova et al. [26].
БИОХИМИЯ том 85 вып. 5 2020
702
ЯКОВЛЕВ и др.
Схема 5. Кинетический механизм, характеризующий взаимодействие SMUG1 R243A с U aPu субстратом
Схема 6. Кинетический механизм, характеризующий взаимодействие SMUG1 R243A с FRET U субстратом
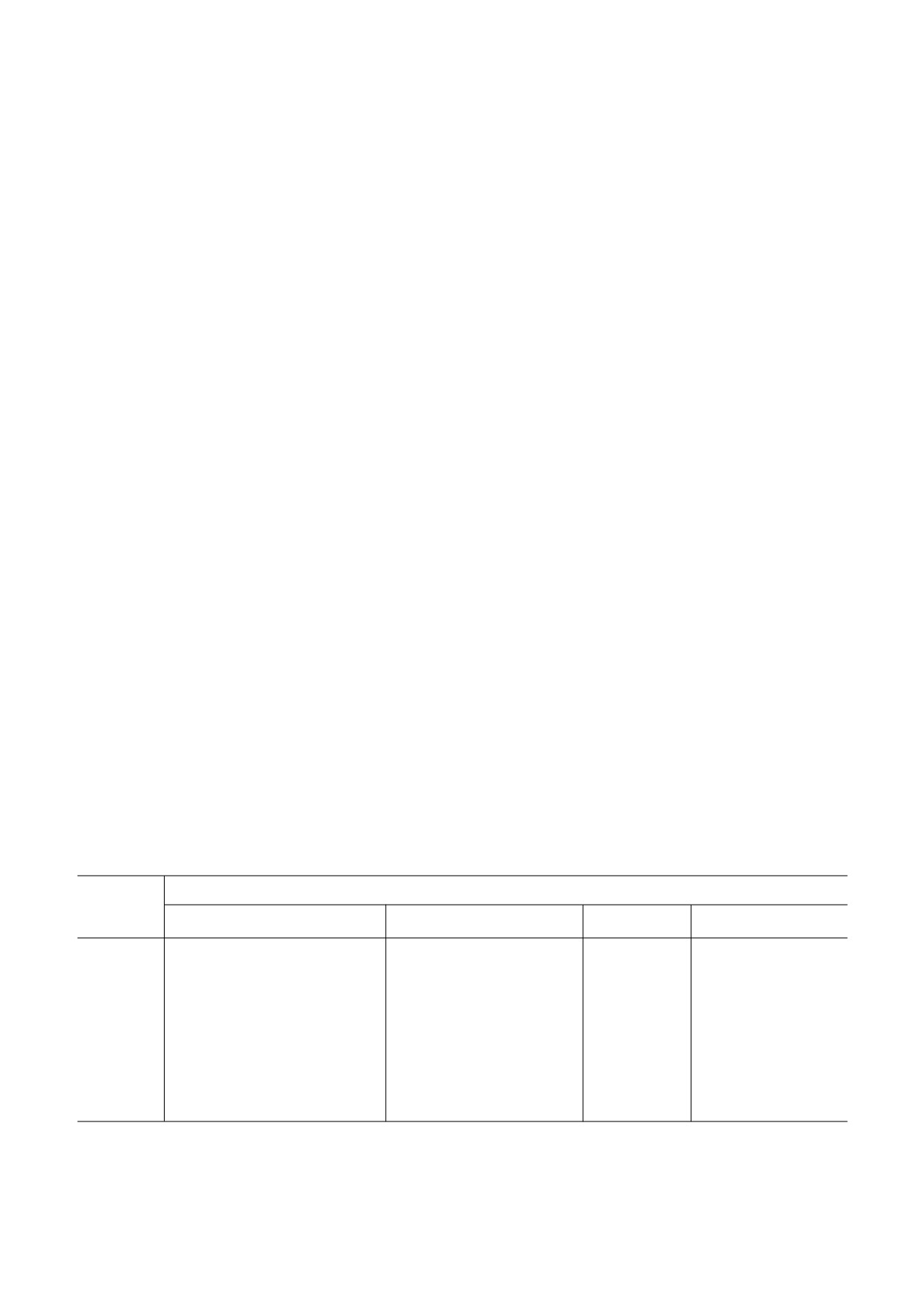
Схема 7. Кинетический механизм, характеризующий взаимодействие SMUG1 H239A с FRET U субстратом
зы: быстрое снижение до t ≈ 100 мс, как и в слу
субстратного комплекса включает идентичные
чае FRET F лиганда, и последующую медлен
стадии и не зависит от природы поврежденного
ную фазу до t ≈ 3 с. Можно предположить, что
нуклеотида.
кривые FRET не отражают каталитический про
Взаимодействие SMUG1 H239A с ДНК, со
цесс, так как кривые изменения интенсивности
держащей уридин. Как было показано ранее [23],
флуоресценции aPu показывают, что гидролиз
активность мутантной формы SMUG1 H239A по
N гликозидной связи не требует дополнитель
отношению к U содержащей ДНК значительно
ных конформационных изменений ДНК. Полу
ниже, чем активность фермента дикого типа или
ченные кинетические кривые описывали схе
мутантной формы R243A. Тем не менее процесс
мой 6, содержащей две равновесные стадии свя
образования фермент субстратного комплекса
зывания ДНК (табл. 3).
был зарегистрирован по изменению сигнала
Следует отметить, что начальные участки
FRET в ходе взаимодействия SMUG1 H239A с
кинетических кривых схожи в случае F лиган
FRET U субстратом в течение 100 с (рис. 4).
дов и U субстратов независимо от использован
Поскольку анализ активности методом электро
ного флуорофора (рис. 2 и 3). Этот факт указы
фореза в PAGE свидетельствует о накоплении
вает на то, что процесс образования фермент
лишь незначительного количества продукта за
Таблица 4. Константы скорости и равновесия, соответству
ющие взаимодействию SMUG1 H239A с ДНК, содержа
щей уридин
Константа
FRET
k1 × 10-6, M-1⋅с-1
1,2 ± 0,5
k-1, с-1
12 ± 5
K1 × 10-6, M-1
0,1
k2, с-1
2 ± 1
k-2, с-1
0,8 ± 0,7
K2
2,5
k3, с-1
0,04 ± 0,01
k-3, с-1
0,023 ± 0,007
K3
1,74
Рис. 4. Взаимодействие SMUG1 H239A с FRET U суб
стратом. Концентрации фермента и ДНК указаны на ри
K1 × K2 × K3
× 10-6, M-1
0,43
сунке
БИОХИМИЯ том 85 вып. 5 2020
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239 В SMUG1
703
этот период, то можно сделать вывод о том, что
продуктом, содержащим F сайт, было зарегист
процессы, наблюдаемые методом «остановлен
рировано по изменению интенсивности флуо
ного потока», соответствуют связыванию ДНК.
ресценции Trp белка, остатка 2 аминопурина
Как видно из рис. 4, кинетические кривые ха
или FRET пары FAM/BHQ1 (рис. 2 и 3). В то же
рактеризуются трехфазным уменьшением
время сравнительный анализ конформацион
FRET сигнала, которое было описано схемой 7,
ной динамики мутантной формы SMUG1
содержащей три обратимые стадии (табл. 4).
H239A с пониженной каталитической актив
ностью показал, что во время взаимодействия с
ДНК продуктом не происходит изменения ка
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
кого либо флуоресцентного сигнала (Trp, aPu
или FRET). При этом образование фермент
Из анализа модельных структур фермента
субстратного комплекса с ДНК, содержащей
SMUG1 WT и его комплекса с ДНК [23, 26] сле
уридин, удалось зарегистрировать только при
дует, что аминокислотные остатки His239 и
детектировании сигнала FRET (рис. 4).
Arg243 должны изменять свои конформации для
В совокупности, экспериментальные дан
встраивания в ДНК и образования контактов с
ные, характеризующие конформационные пе
вывернутым поврежденным основанием (рис. 1).
реходы мутантных форм SMUG1 R243A, H239A
Однако при этом мутация H239A не должна
и ДНК в ходе каталитического цикла, и данные,
приводить к значительным нарушениям сети
полученные для фермента дикого типа [26],
контактов белок ДНК, как и замена R243A,
позволили предложить схему поэтапного изме
несмотря на потерю возможности образования
нения конформаций белка и ДНК в ходе специ
нескольких водородных связей с ДНК. При
фического связывания ДНК и катализа (табл. 5).
этом мутантная форма SMUG1 R243A сохраня
Общий кинетический механизм включает две
ла каталитическую активность, а мутант H239A
обратимые стадии, характеризующие образова
почти полностью терял активность [23].
ние каталитически компетентного комплекса,
В настоящей работе функции аминокислот
которые были зарегистрированы с использова
ных остатков His239 и Arg243 были проанализи
нием всех трех типов флуорофоров, что свиде
рованы путем регистрации конформационных
тельствует о взаимных конформационных из
изменений в ферменте и модельных ДНК
менениях как в ферменте, так и в ДНК субстра
субстратов в процессе их взаимодействия. Для
те. При этом первичное связывание, по види
выявления конформационных изменений
мому, включает перемещение интеркалирую
SMUG1 R243A и H239A во время связывания
щей петли фермента, локальное плавление
ДНК использовали модельные ДНК дуплексы,
ДНК вблизи поврежденного нуклеотида и вы
содержащие уридин или F сайт.
ворачивание повреждения из ДНК дуплекса.
В случае SMUG1 R243A взаимодействие ка
Кроме того, движение интеркалирующей пет
талитически активной формы фермента с ДНК
ли, вероятно, связано с встраиванием в ДНК
субстратом, содержащим уридин, или с ДНК
дуплекс аминокислотного остатка - «сенсора
Таблица 5. Общий механизм ферментативного процесса, катализируемого SMUG1, сформулированный на основании
данных, полученных в настоящей работе путем регистрации флуоресценции Trp, aPu и FRET, а также с помощью метода
PAGE в работе Kuznetsova et al. [26]
Стадии взаимодействия SMUG1 WT с поврежденной ДНК (схема 1)
Тип
регистрации
стадия 1
стадия 2
стадия 3
стадия 4
Trp
конформационные изменения в
заполнение полости
каталитическая
нд*
области интеркалирующей петли
аминокислотными остатками
реакция
aPu
локальное плавление дуплекса
заполнение полости
нд
нд
и выворачивание уридина
аминокислотными остатками
FRET
образование первичного
изгибание ДНК
нд
перестройка комплекса
комплекса
PAGE
нд
нд
каталитическая
диссоциация комплекса
реакция
фермент-продукт
* нд - Стадия в кинетической схеме не может быть зарегистрирована с помощью данного типа регистрации.
БИОХИМИЯ том 85 вып. 5 2020
704
ЯКОВЛЕВ и др.
повреждения», который отвечает за начальные
рактеризующих связывание и узнавание
стадии узнавания поврежденного нуклеотида.
субстрата, в случае SMUG1 WT существенно
Этим остатком, по видимому, является Arg243,
больше, чем в случае мутантной формы R243A.
что не противоречит выводам, сделанным в ра
Аналогичный вывод можно сделать и по дан
боте Iakovlev et al. [23]. На второй стадии фор
ным, полученным для флуоресцентных меток
мируется каталитически активный комплекс, в
aPu и FRET, как в случае мутанта R243A, так и
котором а.о. интеркалирующей петли пол
Н239А. При этом сродство фермента к ДНК
ностью встраиваются в ДНК и образуют все
субстрату не меняется. Таким образом, полу
возможные контакты с вывернутым нуклеоти
ченные результаты позволяют заключить, что
дом, включая контакты His239 с фосфатными
а.о. Arg243 принимает участие в узнавании по
группами. Каталитическая реакция протекает
врежденного нуклеотида, но не важен для про
на третьей стадии взаимодействия. Ранее было
текания каталитической стадии, тогда как
показано, что для фермента дикого типа ско
His239 необходим как для узнавания повреж
рость лимитирующей стадией ферментативно
денного нуклеотида, так и для катализа.
го процесса выступает диссоциация комплекса
фермента с продуктом [26]. Так как замена
R243A уменьшает количество водородных свя
Финансирование. Работа выполнена при под
зей с ДНК, то это дестабилизирует как фер
держке Российского научного фонда (грант
мент субстратный комплекс, так и комплекс
№ 16 14 10038) и частичной поддержке бюджет
фермента с продуктом реакции, за счет чего
ного финансирования для обеспечения регла
увеличивается скорость оборота фермента.
ментных работ на использованном оборудова
Сравнение значений констант скорости отдель
нии № АААА А17 117020210022 4.
ных стадий, полученных ранее [26] по флуорес
Конфликт интересов. Авторы заявляют об от
ценции Trp в случае SMUG1 WT с данными для
сутствии конфликта интересов.
мутанта R243A (табл. 3) показывает, что их ка
Соблюдение этических норм. Настоящая
талитическая активность примерно одинакова:
статья не содержит каких либо исследований с
k3 = 1,7 с-1 и 2,0 с-1 для WT и R243A соответ
участием людей или использованием животных
ственно. Однако скорости 1 й и 2 й стадий, ха в качестве объектов исследований.
СПИСОК ЛИТЕРАТУРЫ
1.
Lari, S. U., Chen, C. Y., Vertéssy, B. G., Morré, J., and
es, and U in single stranded DNA, with hSMUG1 as a
Bennett, S. E. (2006) Quantitative determination of uracil
broad specificity backup, J. Biol. Chem., 277, 39926 39936.
residues in Escherichia coli DNA: contribution of ung, dug,
10. Wibley, J. E. A., Waters, T. R., Haushalter, K., Verdine, G. L.,
and dut genes to uracil avoidance, DNA Repair (Amst), 5,
and Pearl, L. H. (2003) Structure and specificity of the ver
1407 1420.
tebrate anti mutator uracil DNA glycosylase SMUG1,
2.
Lindahl, T. (1993) Instability and decay of the primary
Mol. Cell, 11, 1647 1659.
structure of DNA, Nature, 362, 709 715.
11. Hashimoto, H., Hong, S., Bhagwat, A. S., Zhang, X., and
3.
Lewis, C. A., Crayle, J., Zhou, S., Swanstrom, R.,
Cheng, X. (2012) Excision of 5 hydroxymethyluracil and
Wolfenden, R., and Wolfenden, R. (2016) Cytosine deam
5 carboxylcytosine by the thymine DNA glycosylase
ination and the precipitous decline of spontaneous muta
domain: its structural basis and implications for active
tion during Earth’s history, Proc. Natl. Acad. Sci. USA, 113,
DNA demethylation, Nucleic Acids Res., 40, 10203 10214.
8194 8199.
12. Hashimoto, H., Zhang, X., and Cheng, X. (2012) Excision
4.
Jaszczur, M., Bertram, J. G., Pham, P., Scharff, M. D.,
of thymine and 5 hydroxymethyluracil by the MBD4 DNA
and Goodman, M. F. (2013) AID and Apobec3G haphaz
glycosylase domain: structural basis and implications for
ard deamination and mutational diversity, Cell. Mol. Life
active DNA demethylation, Nucleic Acids Res., 40, 8276
Sci., 70, 3089 108.
8284.
5.
Rebhandl, S., Hümer, M., Greil, R., and Geisberger, R.
13. Sjolund, A. B., Senejani, A. G., and Sweasy, J. B. (2013)
(2015)
AID/APOBEC deaminases and cancer,
MBD4 and TDG: multifaceted DNA glycosylases with
Oncoscience, 2, 320.
ever expanding biological roles, Mutat. Res., 743 744, 12
6.
Ladner, R. D. (2001) The role of dUTPase and uracil
25.
DNA repair in cancer chemotherapy, Curr. Protein Pept.
14. Ito, S., and Kuraoka, I. (2015) Epigenetic modifications in
Sci., 2, 361 370.
DNA could mimic oxidative DNA damage: a double
7.
Jacobs, A. L., and Schar, P. (2012) DNA glycosylases: in
edged sword, DNA Repair (Amst), 32, 52 57.
DNA repair and beyond, Chromosoma, 121, 1 20.
15. Kuznetsov, N. A., and Fedorova, O. S. (2020) Kinetic
8.
Visnes, T., Doseth, B., Pettersen, H. S., Hagen, L.,
milestones of damage recognition by DNA glycosylases of
Sousa, M. M., Akbari, M., Otterlei, M., Kavli, B.,
the Helix hairpin Helix structural superfamily, Adv. Exp.
Slupphaug, G., and Krokan, H. E. (2009) Uracil in DNA
Biol. Med., (in press).
and its processing by different DNA glycosylases, Philos.
16. Brooks, S. C., Adhikary, S., Rubinson, E. H., and
Trans. R. Soc. Lond. B Biol. Sci., 364, 563 568.
Eichman, B. F. (2013) Recent advances in the structural
9.
Kavli, B., Sundheim, O., Akbari, M., Otterlei, M., Nilsen, H.,
mechanisms of DNA glycosylases, Biochim. Biophys. Acta,
Skorpen, F., Aas, P. A., Hagen, L., Krokan, H. E., and
1834, 247 271.
Slupphaug, G. (2002) hUNG2 is the major repair enzyme
17. Dunn, A. R., Kad, N. M., Nelson, S. R., Warshaw, D. M.,
for removal of uracil from U : A matches, U : G mismatch
and Wallace, S. S. (2011) Single Qdot labeled glycosylase
БИОХИМИЯ том 85 вып. 5 2020
РОЛЬ АМИНОКИСЛОТНЫХ ОСТАТКОВ Arg243 И His239 В SMUG1
705
molecules use a wedge amino acid to probe for lesions
monofunctional uracil DNA glycosylase SMUG1, Mol.
while scanning along DNA, Nucleic Acids Res., 39, 7487
Biosyst., 13, 2638 2649.
7498.
27. Kuznetsov, N. A., Faleev, N. G., Kuznetsova, A. A.,
18.
Lee, A. J., Warshaw, D. M., and Wallace, S. S. (2014)
Morozova, E. A., Revtovich, S. V., Anufrieva, N. V.,
Insights into the glycosylase search for damage from single
Nikulin ,A. D. D., Fedorova, O. S., and Demidkina, T. V.
molecule fluorescence microscopy, DNA Repair, 20, 23 31.
(2015) Pre steady state kinetic and structural analysis of
19.
Nelson, S. R., Dunn, A. R., Kathe, S. D., Warshaw, D. M.,
interaction of methionine gamma lyase from Citrobacter
and Wallace, S. S. (2014) Two glycosylase families diffu
freundii with inhibitors, J. Biol. Chem., 290, 671 681.
sively scan DNA using a wedge residue to probe for and
28. Kladova, O. A., Krasnoperov, L. N., Kuznetsov, N. A., and
identify oxidatively damaged bases, Proc. Natl. Acad. Sci.
Fedorova, O. S. (2018) Kinetics and thermodynamics of
USA., 111, E2091 E2099.
DNA processing by wild type DNA glycosylase endo III
20.
Lee, A. J., and Wallace, S. S. (2016) Visualizing the search
and its catalytically inactive mutant forms, Genes (Basel),
for radiation damaged DNA bases in real time, Radiat.
9, doi: 10.3390/genes9040190.
Phys. Chem. Oxf. Engl. 1993, 128, 126 133.
29. Kladova, O. A., Kuznetsova, A. A., Fedorova, O. S., and
21.
Lee, A. J., and Wallace, S. S. (2017) Hide and seek: how do
Kuznetsov, N. A. (2017) Mutational and kinetic analysis of
DNA glycosylases locate oxidatively damaged DNA bases
lesion recognition by Escherichia coli endonuclease VIII,
amidst a sea of undamaged bases? Free Radic. Biol. Med.,
Genes (Basel), 8, 1 13.
107, 170 178.
30. Yakovlev, D. A., Kuznetsova, A. A., Fedorova, O. S., and
22.
Kuznetsov, N. A., and Fedorova, O. S.
(2016)
Kuznetsov, N. A. (2017) Search for modified DNA sites
Thermodynamic analysis of fast stages of specific lesion
with the human methyl CpG binding enzyme MBD4,
recognition by DNA repair enzymes, Biochemistry
Acta Naturae, 9, 88 98.
(Moscow), 81, 1136 1152.
31. Kuzmic, P. (1996) Program DYNAFIT for the analysis of
23.
Iakovlev, D. A., Alekseeva, I. V., Vorobjev, Y. N.,
enzyme kinetic data: application to HIV proteinase, Anal.
Kuznetsov, N. A., and Fedorova, O. S. (2019) The role
Biochem., 237, 260 273.
of active site residues Phe98, HiS239, and Arg243 in
32. Alekseeva, I. V., Davletgildeeva, A. T., Arkova, O. V.,
DNA binding and in the catalysis of human uracil-DNA
Kuznetsov, N. A., and Fedorova, O. S. (2019) The impact
glycosylase SMUG1, Molecules,
24, doi:
10.3390/
of single nucleotide polymorphisms of human apurinic/
molecules24173133.
apyrimidinic endonuclease 1 on specific DNA binding and
24.
Zhang, Z., Shen, J., Yang, Y., Li, J., Cao, W., and Xie, W.
catalysis, Biochimie, 163, 73 83.
(2016) Structural basis of substrate specificity in geobacter
33. Kladova, O. A., Kuznetsov, N. A., and Fedorova, O. S.
metallireducens SMUG1, ACS Chem. Biol., 11, 1729 1736.
(2019) Thermodynamic parameters of endonuclease VIII
25.
Matsubara, M., Tanaka, T., Terato, H., Ohmae, E., Izumi, S.,
interactions with damaged DNA, Acta Naturae, 11, 29 37.
Katayanagi, K., and Ide, H. (2004) Mutational analysis of
34. Miroshnikova, A. D., Kuznetsova, A. A., Kuznetsov, N. A.,
the damage recognition and catalytic mechanism of
and Fedorova, O. S. (2016) Thermodynamics of damaged
human SMUG1 DNA glycosylase, Nucleic Acids Res., 32,
DNA binding and catalysis by human AP endonuclease 1,
5291 5302.
Acta Naturae, 8, 103 110.
26.
Kuznetsova, A. A., Iakovlev, D. A., Misovets, I. V.,
35. Rachofsky, E. L., Osman, R., and Ross, J. B. A. (2001)
Ishchenko, A. A., Saparbaev, M. K., Kuznetsov, N. A., and
Probing structure and dynamics of DNA with 2 aminop
Fedorova, O. S. (2017) Pre steady state kinetic analysis of
urine: effects of local environment on fluorescence,
damage recognition by human single strand selective
Biochemistry, 40, 946 956.
THE ROLE OF AMINO ACID RESIDUES Arg243 AND His239
IN THE PROCESS OF DAMAGE RECOGNITION
BY HUMAN URACIL DNA GLYCOSYLASE SMUG1*
D. A. Iakovlev1,2, I. V. Alekseeva1, N. A. Kuznetsov1** and O. S. Fedorova1**
1 Institute of Chemical Biology and Fundamental Medicine (ICBFM), Siberian Branch of Russian Academy of Sciences,
630090 Novosibirsk, Russia; E*mail: nikita.kuznetsov@niboch.nsc.ru; fedorova@niboch.nsc.ru
2 Novosibirsk State University, Department of Natural Sciences, 630090 Novosibirsk, Russia
Received February 3, 2020
Revised March 17, 2020
Accepted March 19, 2020
Human uracil DNA glycosylase SMUG1 removes uracil residues and some other noncanonical or damaged bases
from DNA. Despite the functional importance of this enzyme, its X ray structure is still unavailable. Previously, we
performed homology modeling of human SMUG1 structure and suggested the roles of some amino acid residues in
the recognition of damaged nucleotides and their removal from DNA. In this study, we investigated the kinetics of
conformational transitions in the protein and in various DNA substrates during enzymatic catalysis using the stopped
flow method based on changes in the fluorescence intensity of enzyme’s tryptophan residues and 2 aminopurine in
DNA or fluorescence resonance energy transfer (FRET) between fluorophores in DNA. The kinetic mechanism of
interactions between reaction intermediates was identified, and kinetic parameters of the intermediate formation and
dissociation were calculated. The obtained data help in elucidating the functions of His239 and Arg243 residues in the
recognition and removal of damaged nucleotides by SMUG1.
Keywords: DNA repair, human uracil DNA glycosylase SMUG1, fluorescence, conformational dynamics, stopped
flow kinetics
7 БИОХИМИЯ том 85 вып. 5 2020