БИОХИМИЯ, 2021, том 86, вып. 10, с. 1550 - 1562
УДК 577.2
СВЯЗЫВАНИЕ РНК in vitro СЕРПИНАМИ РАСТЕНИЙ
© 2021
Е.А. Толстыко1,2, Д.А. Чергинцев3, О.А. Толичева2, Д.С. Виноградова2,4,
А.Л. Коневега2,5,6, С.Ю. Морозов1,7, А.Г. Соловьев1,7*
1 Московский государственный университет имени М.В. Ломоносова,
биологический факультет, кафедра вирусологии, 119991 Москва, Россия
2 Петербургский институт ядерной физики имени Б.П. Константинова
Национального исследовательского центра «Курчатовский институт», 188300 Гатчина, Россия
3 Московский государственный университет имени М.В. Ломоносова,
биологический факультет, кафедра физиологии растений, 119991 Москва, Россия
4 «НаноТемпер Технолоджис рус», 191167 Санкт:Петербург, Россия
5 Санкт:Петербургский политехнический университет имени Петра Великого, 195251 Санкт:Петербург, Россия
6 Национальный исследовательский центр «Курчатовский институт», 123182 Москва, Россия
7 Научно:исследовательский институт физико:химической биологии имени А.Н. Белозерского,
Московский государственный университет имени М.В. Ломоносова,
119992 Москва, Россия; электронная почта: solovyev@belozersky.msu.ru
Поступила в редакцию 12.04.2021
После доработки 09.07.2021
Принята к публикации 09.07.2021
Серпины являются ингибиторами протеиназ, выполняющими регуляторные функции; они составляют об
ширное семейство белков и обнаружены у всех живых организмов. Большинство серпинов растений функ
ционально не охарактеризовано. Исключение составляет белок AtSerpin1 Arabidopsis thaliana, для которого
показано ингибирование проапоптотических протеиназ и предполагается участие в регулировании индук
ции программируемой клеточной смерти, и белок CmPS1 Cucurbita maxima, локализованный во флоэме и
предположительно ингибирующий пищеварительные протеиназы насекомых. Помимо этого, недавно бы
ло обнаружено, что белок CmPS1 способен связывать РНК, наиболее эффективно взаимодействуя с высо
коструктурированными РНК, в частности формируя специфический комплекс с тРНК. В настоящей рабо
те продемонстрировано, что белок AtSerpin1 также способен образовывать комплекс с тРНК. Анализ тРНК,
связанных белками AtSerpin1 и CmPS1 в условиях избытка тРНК, показал, что ни один из белков не прояв
ляет строгой избирательности в связывании индивидуальных тРНК, что указывает, вероятно, на специфи
ческое взаимодействие этих белков с элементами вторичной/третичной структуры, универсальными для
всех тРНК. При анализе взаимодействия белка CmPS1 с предшественником микроРНК 390 (pre miR390)
было обнаружено, что мутантный вариант pre miR390, стебель шпилечной структуры которого представлял
собой совершенный дуплекс, утрачивал способность формировать дискретный комплекс с CmPS1, тогда
как другой вариант pre miR390, содержащий природные неспаренные нуклеотидные остатки, такой спо
собностью обладал. Эти данные указывают на то, что специфическое взаимодействие серпинов растений со
структурированными РНК основано на узнавании структурно уникальных пространственных мотивов,
формируемых с участием неспаренных нуклеотидных остатков в дуплексах РНК.
КЛЮЧЕВЫЕ СЛОВА: серпин, связывание РНК, РНК связывающий белок, тРНК, микроРНК, структура
РНК, флоэма.
DOI: 10.31857/S0320972521100109
ВВЕДЕНИЕ
мов [1, 2]. Большинство белков семейства сер
пинов является ингибиторами сериновых про
Серпины составляют многочисленное се
теиназ, что и дало им название (serine protease
мейство белков ингибиторов протеиназ; они
inhibitor, serpin). Тем не менее среди серпинов
имеют характерную консервативную третичную
обнаружены белки, ингибирующие и другие ви
структуру и обнаружены у всех живых организ
ды протеиназ [3-5], а также неингибирующие
Принятые сокращения: AtMC1 - A. thaliana metacaspase 1 (метакаспаза 1 резуховидки Таля); AtMC9 - A. thaliana
metacaspase 9 (метакаспаза 9 резуховидки Таля); AtSerpin1 - A. thaliana serpin 1 (серпин 1 резуховидки Таля); CmPS1 -
Cucurbita maxima phloem serpin 1 (флоэмный серпин 1 тыквы гигантской); PSTVd - potato spindle tuber viroid (вироид вере
теновидности клубней картофеля); RCL - reactive center loop (ингибиторная петля); TCTP - translationally controlled tumor
protein (трансляционно контролируемый опухолевый белок).
* Адресат для корреспонденции.
1550
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1551
серпины [6-9], причисленные к данному семей
ной структуры серпинов разных видов растений
ству на основании характерной структуры бел
показал, что эти белки образуют несколько об
ковой молекулы.
особленных и достаточно удаленных друг от
Структура молекулы типичного серпина
друга кластеров, соответствующих порядкам
формируется приблизительно 350-400 амино
царства растений, что, как полагают, может
кислотными остатками и обычно включает три
свидетельствовать о давней дивергенции этих
β слоя и 8-9 α спиралей, окружающих цен
белков, происходившей после эволюционного
тральный β слой (т.н. β слой А), состоящий из
расхождения порядков, а также их функцио
пяти β тяжей. Сразу же за одним из двух цен
нальной специализации в отдаленных группах
тральных тяжей β слоя А расположен неструк
растений [19]. Исключение составляют серпины
турированный участок полипептидной цепи,
так называемой LR группы, названные по на
находящийся на поверхности молекулы серпина
личию инвариантных остатков лейцина (L) и
и носящий название «ингибиторная петля»
аргинина (R) в последовательности RCL и обна
(reactive center loop, RCL). Часть RCL служит
руженные у всех растений, включая однокле
«наживкой», узнаваемой протеиназой, ингиби
точные и колониальные зеленые водоросли.
руемой данным серпином, как субстрат для про
Предполагают, что серпины LR группы в силу
теолиза [10-12]. Ингибиторный механизм сер
консервативности первичной структуры могут
пина основан на резком изменении его конфор
выполнять сходные функции у эволюционно
мации, происходящем на промежуточной ста
удаленных растений [19].
дии протеолиза RCL протеиназой, которая, в
Серпины растений способны ингибировать
силу особенностей механизма катализа, в этот
протеиназы семейства химотрипсина, которые
момент ковалентно связана с RCL. При кон
являются наиболее распространенными мише
формационном переходе серпина ингибиторная
нями серпинов животных [1, 20]. Вместе с тем,
петля втягивается в пространство между двумя
поскольку геномы растений не кодируют проте
центральными тяжами β слоя А и образует до
иназы такого типа, выявление функций серпи
полнительный β тяж в его составе, при этом
нов растений не может основываться на сход
протеиназа теряет ферментативную активность
стве их последовательностей с белками других
в результате нарушения ее пространственной
организмов и требует экспериментальной рабо
структуры и оказывается неспособной завер
ты. В настоящее время с функциональной точки
шить расщепление ингибиторной петли
зрения охарактеризовано лишь несколько сер
[1, 12, 13]. Таким образом, при ингибировании
пинов растений.
протеиназы серпином происходит образование
Для белка Serpin1 Arabidopsis thaliana
ковалентной связи между одной молекулой про
(AtSerpin1), относящегося к LR группе, обнару
теиназы и одной молекулой серпина, которое
жено три растительных протеиназы мишени.
приводит к инактивации данной молекулы про
Во первых, AtSerpin1 способен образовывать
теиназы и является необратимым [2, 14].
ковалентные комплексы с папаин подобной
Помимо собственно ингибирования протеи
протеиназой RD21 (responsive to desiccation 21),
наз, известны и другие функции, выполняемые
которая локализуется в вакуоли [21]. Поскольку
серпинами, среди них - перенос гормонов
известно, что высвобождение вакуолярных про
[6, 7], участие в фолдинге белков [8], накопле
теиназ в цитоплазму в результате нарушения це
ние аминокислот [9]. Отдельно стоит упомянуть
лостности тонопласта вызывает клеточную
ингибитор ядерной протеазы MENT (myeloid
смерть при развитии гиперчувствительного от
and erythroid nuclear termination stage specific
вета у растений, была предложена модель, сог
protein), который способствует конденсации
ласно которой локализованный в цитоплазме
хроматина в терминально дифференцирован
AtSerpin1 контролирует активность протеиназы
ных клетках птиц [15, 16], а также ингибитор
RD21 при ее высвобождении в цитоплазму и та
лейкоцитарной эластазы (LEI), в определенных
ким образом модулирует индукцию клеточной
условиях приобретающий активность ДНКа
смерти [21]. В подтверждение этой модели пока
зы II и участвующий во фрагментации хромати
зано, что сверхэкспрессия белка AtSerpin1 в оп
на в процессе апоптоза [17, 18]. До недавнего
ределенных условиях способна подавлять разви
времени лишь для этих двух серпинов была по
тие клеточной смерти [22]. Во вторых, AtSerpin1
казана способность к взаимодействию с нуклеи
образует комплекс c протеиназой AtMC1
новыми кислотами.
(Arabidopsis thaliana metacaspase 1), относящейся
Анализ геномов нескольких десятков расте
к метакаспазам растений, отдаленно родствен
ний показал, что гены серпинов составляют не
ным каспазам животных, и ингибирует развитие
большие мультигенные семейства, включающие
клеточной смерти, индуцированное AtMC1 [23].
5-10 генов [19]. Сравнительный анализ первич
В третьих, AtSerpin1 способен взаимодейство
БИОХИМИЯ том 86 вып. 10 2021
1552
ТОЛСТЫКО и др.
вать с еще одной метакаспазой, AtMC9, однако
белка CmPS1 с предшественником микроРНК.
функциональное значение ингибирования ак
Полученные данные свидетельствуют о взаимо
тивности AtMC9 белком AtSerpin1 in vivo остает
действии серпинов с элементами третичной
ся неизвестным [24]. Таким образом, имеющие
структуры молекул РНК, такими как простран
ся данные свидетельствуют об участии белка
ственные мотивы в несовершенных дуплексах
AtSerpin1 в процессах, связанных с регулирова
РНК.
нием индукции программируемой клеточной
смерти.
Для флоэмного серпина тыквы (Cucurbita
МАТЕРИАЛЫ И МЕТОДЫ
maxima phloem serpin 1, CmPS1), также относя
щегося к LR группе, природная протеиназа ми
Генетические конструкции. Конструкции
шень неизвестна, однако показано, что он спо
pET33b(+)[CmPS1] для экспрессии белка
собен ингибировать эластазу - фермент млеко
CmPS1 в бактериальных клетках и
питающих, относящийся к сериновым протеи
pBSCII SK(+) miR390 для получения in vitro
назам [25]. Было обнаружено, что повышение
Т7 транскрипта pre miR390 описаны ранее
уровня CmPS1 коррелирует со снижением спо
[28,
32]. Для получения конструкции
собности тлей питаться и размножаться на рас
pET33b(+)[AtSeprin1] кодирующую последова
тениях, что дало основание предполагать защит
тельность белка AtSerpin1 амплифицировали с
ную роль CmPS1, которая может быть связана с
использованием праймеров 5′ GGCCATGGA
ингибированием пищеварительных протеиназ
CGTGCGTGAATCAATCTC и 5′ CGCTCGA
насекомых. Тем не менее в прямом эксперимен
GATGCAACGGATCAACAACTTGG на матрице
те добавление рекомбинантного белка CmPS1 к
кДНК A. thaliana. Полученный продукт ампли
корму тлей не имело какого либо эффекта на их
фикации расщепляли эндонуклеазами рестрик
выживание [25]. С другой стороны, питание с
ции NcoI и XhoI и лигировали с аналогичным
добавлением рекомбинантного белка AtSerpin1
образом разрезанным вектором pET33b(+). Для
приводило к замедлению роста и повышению
получения мутантных вариантов pre miR390,
смертности личинок тлей и египетской хлопко
Loop и 5М, проводили амплификацию на мат
вой совки Spodoptera littoralis [26]. Наличие за
рице pBSCII SK(+) miR390 с использованием
щитной функции, заключающейся в ингибиро
праймера 5′ GCGGTACCGAGAAATAGCTAT
вании пищеварительных протеаз насекомых,
AAAGCTCAGGAG в комбинации либо с прай
предполагается также у серпинов, которые в су
мером 5′ TCGGATCCAAGAAGAAGCTATGG
щественных количествах накапливаются в зер
AACTCAGGATGGATAGCGCCAAGCCATGG
нах злаковых [27].
CGCTATCCCTC, либо с праймером 5′ TCGGA
Недавно мы продемонстрировали способ
TCCAAGAAATAGCTATAAAACTCAGGA
ность CmPS1 к связыванию РНК. При этом в
GGGATAGCG соответственно. Полученные
условиях in vitro данный белок наиболее эффек
продукты разрезали с использованием рестрик
тивно взаимодействует с высокоструктуриро
таз KpnI и BamHI и лигировали с аналогичным
ванными РНК, в частности формируя специфи
образом разрезанным вектором pBSCII SK(+).
ческий комплекс с тРНК [28]. С учетом того, что
Для получения двойного мутанта последнюю
тРНК и тРНК подобные структуры, как полага
пару праймеров использовали для амплифика
ют, содержат сигналы, направляющие транс
ции на матрице мутантной конструкции
порт молекул РНК по флоэме [29], а также спо
pBSCII SK(+) miR390 Loop.
собности ортолога CmPS1 из огурца (Cucumis
Получение рекомбинантных белков. Для нара
sativus) к дальнему (системному) транспорту по
ботки рекомбинантных белков CmPS1 и AtSeprin1
флоэме [30], была предложена гипотеза, соглас
использовали конструкции pET33b(+)[CmPS1]
но которой белок CmPS1 обладает не связанны
и pET33b(+)[AtSeprin1], которыми трансформи
ми с ингибированием протеиназ дополнитель
ровали клетки E. coli штамма BL 21(DE3).
ными регуляторными функциями, которые мо
Полученные клоны продуценты выращивали в
гут заключаться в участии в системном сигна
течение ночи при 37 °С в среде 2YT, содержащей
линге, основанном на флоэмном транспорте
канамицин, при качании на скорости
РНК [31].
180 об./мин. Затем 30 мл ночной культуры ис
В настоящей статье продемонстрирована
пользовали для инокуляции 370 мл свежей сре
способность белка AtSerpin1 связывать РНК,
ды 2YT с канамицином. После инкубации куль
показано его эффективное взаимодействие с
туры в течение 1 ч при 37 °С при качании на ско
тРНК, а также представлены результаты иссле
рости 180 об./мин индуцировали экспрессию
дования специфичности связывания тРНК
рекомбинантных белков добавлением IPTG до
белками AtSerpin1 и CmPS1 и взаимодействия
конечной концентрации 1 мМ. Культуры инку
БИОХИМИЯ том 86 вып. 10 2021
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1553
бировали в течение 3 ч в тех же условиях, затем
ций, предварительно линеаризованных по сайту
собирали клетки центрифугированием при
BamHI. Транскрипт pre miR390 5M Loop был
4500 g в течение 10 мин и ресуспендировали в
получен с использованием продукта амплифи
10 мл буфера А (6 М гуанидина гидрохлорид,
кации двойного мутанта в качестве матрицы.
100 мМ NaH2PO4, 10 мМ Tris HCl, рН 8,0) с до
После завершения транскрипции РНК субстра
бавлением PMSF до конечной концентрации
ты были очищены из реакционной смеси с по
5 мМ, после чего инкубировали в течение часа
мощью экстракции смесью фенол/хлороформ и
при комнатной температуре и качании. Далее
осаждения этанолом.
лизат центрифугировали в течение 10 мин при
Анализ РНК9связывающих свойств белков ме9
20 200 g с отбором супернатанта в хроматогра
тодом сдвига в агарозном геле. Для связывания к
фическую колонку с Ni NTA агарозой «Protino»
РНК субстратам добавляли соответствующие
(«Macherey Nagel», Германия). Для связывания
различным молярным соотношениям рассчи
белков с Ni NTA агарозой колонку в течение ча
танные количества белка, после чего инкубиро
са инкубировали при комнатной температуре с
вали в течение 20 мин на ледяной бане в буфере
умеренным перемешиванием. Далее колонку
для связывания (50 мМ KCl, 0,1 мМ EDTA, 5%
промывали, последовательно добавляя по 10 мл
глицерин, 10 мМ HEPES HCl, рН 8,0). Концен
буфера А, буфера B (8 М мочевина, 100 мМ
трация РНК в смеси для связывания имела сле
NaH2PO4, 10 мМ Tris HCl, рН 8,0) и буфера С
дующие значения: тРНК - 171 нМ; PSTVd -
(8
М мочевина, 100 мМ NaH2PO4, 10 мМ
43
нМ; pre miR390 wt (дикий тип) и
Tris HCl, рН 6,3). Затем связанные белки элюи
pre miR390 5М - 105 нМ; pre miR390 Loop и
ровали буфером Е (8 М мочевина, 100 мМ
pre miR390 5М Loop - 148 нМ; TCTP - 66 нМ.
NaH2PO4, 10 мМ Tris HCl, рН 4,5) и ренатуриро
После инкубации образцы наносили на
вали путем диализа с использованием диализ
1,5% ный агарозный гель, содержащий бромис
ных мешков SERVAPOR® (MWCO 12000-14000,
тый этидий, для электрофореза. Результаты
диаметр 50 мм; «SERVA», Германия) против вы
фиксировали с помощью системы гель доку
сокоочищенной воды в случае CmPS1, либо
ментации ChemiDoc XRS+ («Bio Rad», США).
против буфера (200 мкМ Tris HCl, pH 7,5) - в
Микротермофорез. Анализ взаимодействия
случае диализа AtSerpin1.
тРНК с белком проводили в экспериментах тит
РНК9субстраты для связывания с белком. Для
рования методом микротермофореза [34, 35].
экспериментов по связыванию тРНК с белком
Комплексы формировали при постоянной кон
использовали коммерческий препарат дрожже
центрации 0,5 мкМ инициаторной tRNAfMet, не
вой тРНК («Sigma», США), а также обогащен
сущей флуоресцентную метку Bodipy FL
ную тРНК фракцию растительных РНК [28]. Для
(«Thermo Fisher Scientific») на α аминогруппе
ее получения с помощью реагента ExtractRNA
метионина, при концентрации белка, изменяю
(«Евроген», Россия) из листовой ткани растения
щейся от 0,0195 до 20 мкМ. Анализ проводили в
был выделен препарат тотальной РНК. Далее к
буферном растворе (50 мМ NaCl, 5% глицерин,
нему был добавлении хлорид лития до конечной
10 мМ HEPES HCl, рН 8,0). После смешивания
концентрации 2 М. Смесь инкубировали в тече
образцы инкубировали в течение 20 мин при
ние ночи при 4 °С с последующим центрифуги
температуре 0 °C, после чего центрифугировали
рованием при 20 000 g в течение 15 мин при 4 °С
при 11 000 об./мин в течение минуты. Анализ
для осаждения фракции длинных РНК. После
изменения флуоресценции проводили с по
этого к супернатанту был добавлен этанол для
мощью прибора Monolith NT.115 с использова
последующего осаждения центрифугированием
нием стандартных капилляров MO K022
обогащенной тРНК фракции, как было описано
(«NanoTemper Technologies», Германия). Интен
ранее [28]. Остальные РНК субстраты были по
сивность излучения ИК лазера составляла 40%.
лучены путем транскрипции in vitro с T7 РНК
Аппроксимация данных, полученных методом
полимеразой («Thermo Fisher Scientific», США)
термофореза, проводилась для трех измерений.
согласно протоколу производителя. Транскрип
Анализ селективности связывания тРНК бел9
ты РНК TCTP (translationally controlled tumor
ком. Для исследования селективности связыва
protein) и PSTVd (potato spindle tuber viroid) были
ния белка с тРНК была приготовлена смесь, со
получены на матрице ранее описанных
держащая РНК и белок в молярном соотноше
конструкций pBSCII SK(+) TCTP
[28] и
нии 1 : 10 в буфере для связывания (50 мМ KCl,
pBSCII SK(+) PSTV [33], предварительно лине
0,1 мМ EDTA, 5% глицерин, 10 мМ HEPES
аризованных по сайту XbaI. Транскрипты
HCl, рН 8,0). После инкубации в течение 20 мин
pre miR390 и двух ее мутантных вариантов
на ледяной бане смесь была нанесена на агароз
(pre miR390 Loop, pre miR390 5М) были полу
ный гель для проведения электрофореза. Затем
чены на матрицах соответствующих конструк
фрагмент геля, содержащий комплексы тРНК с
БИОХИМИЯ том 86 вып. 10 2021
1554
ТОЛСТЫКО и др.
белком, вырезали, помещали в спин колонку с
тах [28], при связывании тРНК белок CmPS1 да
фильтром Mini Bio Spin («Bio Rad») и центри
вал три типа комплексов, а именно: комплексы,
фугировали при 6000 g в течение 4 мин. Про
неспособные входить в гель; движущиеся в геле
шедшая через колонку жидкость была использо
комплексы, подвижность которых уменьшалась
вана для выделения РНК с помощью экстрак
с увеличением соотношения белок : РНК; и спе
ции смесью фенол/хлороформ и осаждения эта
цифический комплекс CmPS1-тРНК, видимый
нолом. После определения концентрации с
в геле как дискретная полоса (рис. 1). Комплек
помощью бескюветного спектрофотометра
сы двух первых типов возникают, как предпола
Nanodrop 2000 («Thermo Scientific») образцы
гается, ввиду наличия у белка CmPS1 способ
РНК секвенировали методом высокопроизво
ности к неспецифическому связыванию высо
дительного секвенирования (next generation
коструктурированных РНК, проявляющейся в
sequencing, NGS; ООО «Геноаналитика», Рос
образовании подобного рода комплексов с та
сия) параллельно с эквивалентным количеством
кими РНК, как РНК трансляционно контроли
исходных препаратов тРНК. Контроль качества
руемого опухолевого белка TCTP и вироида ве
секвенирования проводили с помощью про
ретеновидности клубней картофеля PSTVd, ко
граммы FastQC [36]. Для анализа селективности
торые имеют выраженную вторичную структуру
связывания тРНК белком AtSerpin1 чтения бы
и могут рассматриваться как несовершенные
ли выровнены на последовательности тРНК
дуплексы РНК [28]. При связывании белка
A. thaliana из базы данных tRNAdb [37] с по
AtSerpin1 с тРНК формировался дискретный
мощью алгоритма Bowtie2 [38, 39], а также на ге
комплекс, сходный с тем, который наблюдается
ном A. thaliana (идентификатор RefSeq:
в случае CmPS1, однако образующийся при не
GCF_000001735.4) с помощью HISAT2 [40]. Для
сколько большем соотношении белок : РНК.
анализа селективности связывания тРНК
При этом не способные входить в гель комплек
белком CmPS1 чтения были выровнены на ге
сы в случае белка AtSerpin1 не наблюдались
ном C. maxima (идентификатор RefSeq:
(рис. 1). Еще одно существенное отличие в ха
GCF_002738345.1) с помощью программы
рактере связывания тРНК белками AtSerpin1 и
STAR [41]. Подсчет количества выровненных
CmPS1 проявлялось в переходе РНК из состава
чтений проводили с помощью алгоритма
специфических комплексов в состав неспеци
HTSeq [42].
фических при увеличении соотношения бе
лок : РНК в случае CmPS1 и отсутствие такого
перехода в случае белка AtSerpin1 (рис. 1). Эти
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
наблюдения указывают на то, что способность
AtSerpin1 к неспецифическому связыванию
Способность белка AtSerpin1 к связыванию
РНК значительно ниже таковой белка CmPS1.
РНК. Способность связывать РНК была показа
Для проверки этого вывода было проведено свя
на для белка CmPS1 [28], но не изучалась для
зывание белком AtSerpin1 РНК TCTP и PSTVd,
других серпинов растений, и оставалось неяс
которые, как было показано ранее, образуют с
ным, является ли это свойство уникальной осо
белком CmPS1 комплексы за счет неспецифи
бенностью CmPS1. В настоящей работе прово
ческой РНК связывающей активности белка
дился анализ РНК связывающих свойств белка
CmPS1 [28]. В экспериментах по сдвигу в геле
A. thaliana AtSerpin1, относящегося, как и белок
комплексы этих РНК с белком AtSerpin1 прак
CmPS1, к LR группе серпинов. Кодирующая
тически не обнаруживались (рис. 1). Таким об
последовательность белка AtSerpin1 была ам
разом, можно заключить, что белок AtSerpin1,
плифицирована на матрице кДНК A. thaliana и
как и CmPS1, способен связывать тРНК, но об
клонирована в составе вектора pET33b(+). Ре
ладает сравнительно низкой неспецифической
комбинантный AtSerpin1, слитый с последова
РНК связывающей активностью.
тельностью из шести гистидиловых остатков,
Оценка аффинности AtSerpin1 к тРНК. Коли
был экспрессирован в клетках E. coli, аффинно
чественную оценку связывания тРНК с белком
очищен на Ni NTA агарозе и ренатурирован.
AtSerpin1 проводили при помощи микротермо
Анализ РНК связывающих свойств полученно
фореза, где в качестве репортера выступала мо
го белка проводили методом сдвига в геле. В
лекула инициаторной Bpy Met tRNAfMet [34, 35].
этих экспериментах РНК инкубировали с уве
В условиях увеличивающейся концентрации
личивающимися количествами белка и затем
белка подвижность флуоресцентно меченой
анализировали в агарозном геле. В качестве
тРНК изменялась в результате образования
контроля использовали аналогичным образом
комплекса. Аффинность такого взаимодействия
полученный препарат рекомбинантного белка
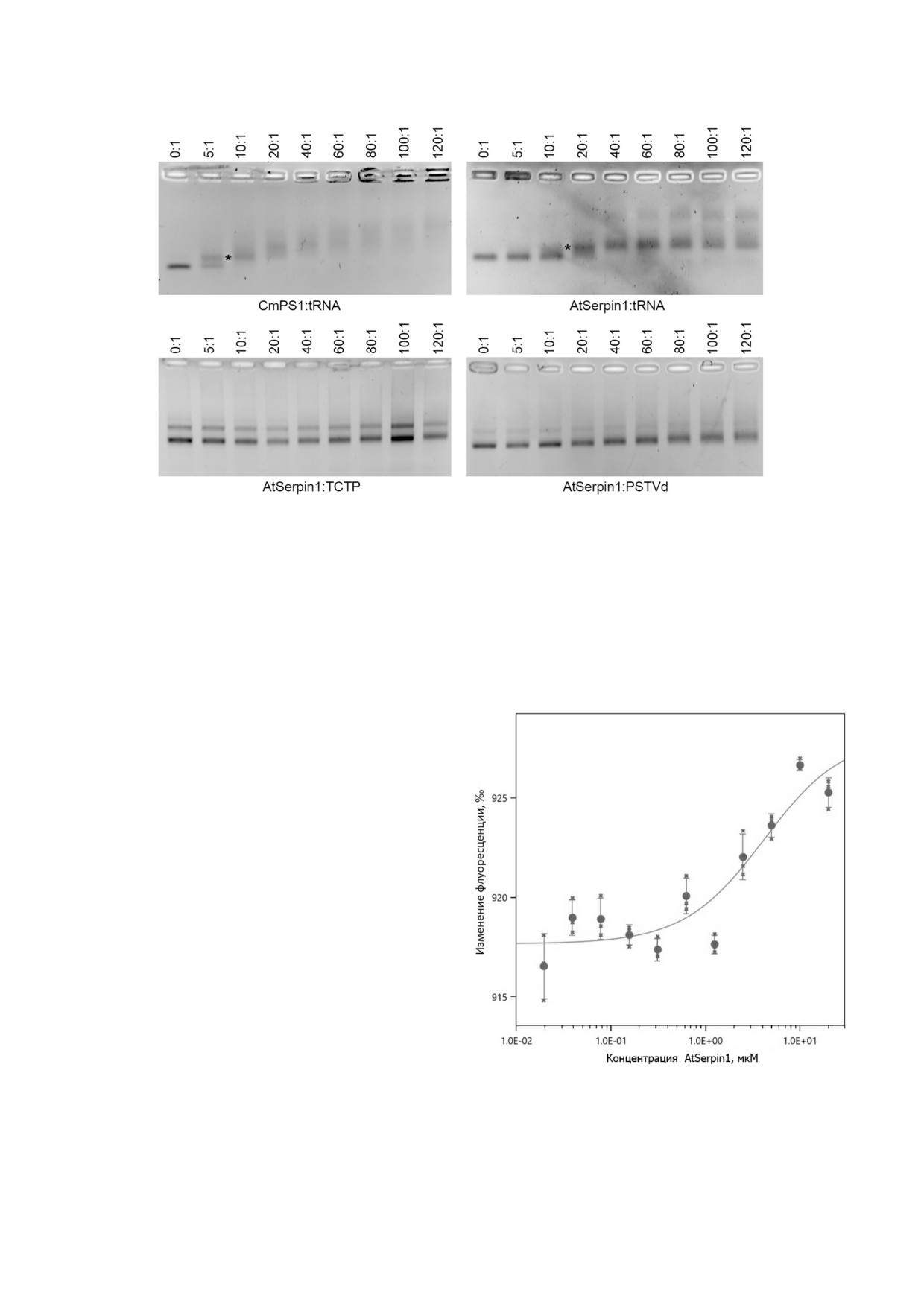
оценивалась в микромолярном диапазоне (Kd =
CmPS1. Как и в ранее описанных эксперимен
3,9 ± 2,9 мкМ) (рис. 2) и была соизмерима срод
БИОХИМИЯ том 86 вып. 10 2021
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1555
Рис. 1. Анализ РНК связывающих свойств белка AtSerpin1 методом сдвига в геле. Рекомбинантный белок инкубировали
с РНК и наносили на агарозный гель. Приведены эксперименты по связыванию белком AtSerpin1 тРНК, РНК TCTP и
PSTVd. В качестве контроля использовали связывание тРНК белком CmPS1. Молярное соотношение белок : РНК указа
но над каждой дорожкой. Звездочкой обозначено положение дискретного комплекса с тРНК
ству инициаторного фактора трансляции IF2 к
ходила в состав комплекса. Полоса, соответ
инициаторной Bpy Met tRNAfMet [35]. Получен
ствующая такому комплексу, была вырезана из
ное значение константы диссоциации комплек
агарозного геля и использована для выделения
са AtSerpin1 с инициаторной Bpy Met tRNAfMet
содержащейся в комплексе РНК, которую затем
мы рассматриваем как нижнюю оценку ввиду
недостаточного выхода кривой в насыщение от
носительно количества белка. Следует также за
метить, что данные микротермофореза являют
ся дополнительным подтверждением способ
ности белка AtSerpin1 связывать РНК.
Селективность связывания тРНК белком
AtSerpin1. При анализе РНК связывающей ак
тивности AtSerpin1 методом сдвига в геле ис
пользовали препарат тотальной тРНК дрожжей,
что не давало возможности оценить, способен
ли данный белок предпочтительно связывать
какие либо тРНК A. thaliana. Для проверки на
личия этой способности использовали фрак
цию, обогащенную тРНК, которая была выделе
на как описано ранее [28] из препарата тоталь
ной РНК, полученной из листьев A. thaliana, и
включала преимущественно тРНК, а также не
значительные количества других клеточных
РНК.
Для того, чтобы оценить возможность пред
почтительного связывания каких либо тРНК
Рис.
2. Образование комплекса белка AtSerpin1 c
белком AtSerpin1, анализировали состав РНК в
Bpy Met tRNAfMet, измеренное методом микротермофоре
комплексе тРНК-AtSerpin1, полученном при
за в условиях изменяющейся концентрации белка. Показа
соотношении белок : РНК = 10 : 1, при котором
ны значения, полученные в трех экспериментах, их сред
только часть РНК в инкубационной смеси пере
нее и стандартное отклонение
БИОХИМИЯ том 86 вып. 10 2021
1556
ТОЛСТЫКО и др.
подвергали высокопроизводительному секвени
сравнение экспериментальных и контрольных
рованию. В качестве контроля использовали
данных заведомо недостоверным. Полученные
РНК, инкубированную в отсутствие белка
данные (табл. 1) демонстрируют, что при обра
AtSerpin1 и, следовательно, имеющую исходную
зовании комплекса AtSerpin1 с тРНК происхо
электрофоретическую подвижность в агарозном
дит изменение относительных долей тРНК в
геле. Проводили секвенирование двух независи
сравнении с тотальной тРНК A. thaliana. Для ря
мо полученных опытных образцов и двух конт
да тРНК наблюдалось увеличение доли в срав
рольных образцов.
нении с контролем, тогда как для остальных -
После технической фильтрации данных NGS
уменьшение этой доли. В частности, в 3,68-4,19
для каждого образца определяли доли чтений,
раза по сравнению с контролем увеличивалась
соответствующих всем индивидуальным тРНК
доля tRNA Gly(GCC), tRNA Tyr(GUA) и
A. thaliana, имеющимся в базе данных тРНК дан
tRNA Ser(GCU), что может говорить об их
предпочтительном связывании. Вместе с тем,
лученные данные (таблица в Приложении) под
все виды тРНК A. thaliana обнаруживались в
вергались фильтрации по двум параметрам. Во
составе комплекса с белком AtSerpin1 в услови
первых, были отсеяны тРНК, для которых сум
ях, когда тРНК находилась в избытке и только
марное число чтений в двух опытных и двух
часть молекул тРНК в инкубационной смеси пе
контрольных образцах было меньше 200, что
реходила в состав комплекса. Эти данные гово
позволило исключить из рассмотрения последо
рят об отсутствии строгой избирательности свя
вательности с недостаточным для достоверного
зывания тРНК белком AtSerpin1 и его способ
анализа покрытием. Во вторых, были отсеяны
ности связывать все виды тРНК, наблюдаемая
тРНК, для которых разница в доле чтений меж
разница в эффективности связывания которых
ду двумя повторностями эксперимента и/или
является количественной, а не качественной.
контроля составляла более 30%, что делало
Поскольку AtSerpin1 формирует комплексы с
Таблица 1. Анализ предпочтительности связывания тРНК A. thaliana белком AtSerpin1 in vitro
тРНК
Идентификатор tRNAdb
Комплекс, %*
Контроль, %*
Отношение
tRNA Gly(GCC)
tdbD00005327
0,05 ± 0,01
0,013 ± 0,002
4,19
tRNA Tyr(GUA)
tdbD00011652
0,13 ± 0,01
0,033 ± 0,004
3,90
tRNA Ser(GCU)
tdbD00009863
0,27 ± 0,02
0,07 ± 0,01
3,68
tRNA Ala(UGC)
tdbD00003871
0,016 ± 0,001
0,0068 ± 0,0005
2,35
tRNA Ser(GGA)
tdbD00009864
3,1 ± 0,2
1,40 ± 0,06
2,20
tRNA Ala(AGC)
tdbD00003864
0,042 ± 0,004
0,019 ± 0,002
2,17
tRNA Asp(GUC)
tdbD00000505
0,270 ± 0,005
0,130 ± 0,005
2,08
tRNA Ala(AGC)
tdbD00003866
0,043 ± 0,005
0,0205 ± 0,0002
2,07
tRNA Thr(AGU)
tdbD00010496
0,1477 ± 0,0004
0,07 ± 0,01
2,07
tRNA Gln(UUG)
tdbD00008612
0,0335 ± 0,0005
0,016 ± 0,002
2,05
tRNA Asp(GUC)
tdbD00004461
0,272 ± 0,002
0,135 ± 0,005
2,02
tRNA Asp(GUC)
tdbD00004462
0,261 ± 0,005
0,132 ± 0,005
1,98
tRNA Thr(AGU)
tdbD00010494
0,14 ± 0,01
0,070 ± 0,007
1,96
tRNA Asp(GUC)
tdbD00000504
0,245 ± 0,006
0,127 ± 0,005
1,93
tRNA Gly(GCC)
tdbD00005326
0,11 ± 0,01
0,065 ± 0,001
1,65
tRNA Thr(AGU)
tdbD00010495
0,052 ± 0,002
0,032 ± 0,002
1,65
tRNA Gly(GCC)
tdbD00005330
0,073 ± 0,002
0,045 ± 0,002
1,62
tRNA Gly(GCC)
tdbD00005329
0,104 ± 0,009
0,068 ± 0,002
1,53
tRNA Pro(UGG)
tdbD00008227
0,045 ± 0,006
0,031 ± 0,002
1,42
tRNA His(GUG)
tdbD00005721
6,3 ± 0,2
4,8 ± 0,2
1,33
Примечание. В таблице показаны 20 тРНК, имеющих наибольшее отношение эксперимент : контроль и удовлетворяю
щих критериям фильтрации исходных данных (см. текст). Полные результаты анализа приведены в Приложении к статье.
* Среднее значение, полученное для двух независимых образцов.
БИОХИМИЯ том 86 вып. 10 2021
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1557
тРНК, но не с другими протестированными
Таблица 2. Анализ предпочтительности связывания тРНК
C. maxima белком CmPS1 in vitro
РНК (рис. 1), можно заключить, что данный бе
лок специфически связывает элементы вторич
тРНК
Комплекс, %
Контроль, %
Отношение
ной/третичной структуры, универсальные для
всех тРНК.
tRNA Ala(CGC)
2,73
0,94
2,92
Этот вывод согласуется с результатами экс
tRNA Cys(GCA)
5,79
2,54
2,28
периментов, в которых белок AtSerpin1 эффек
тивно связывал дрожжевые тРНК, которые не
tRNA Thr(GGU)
0,69
0,33
2,06
являются его природными лигандами (рис. 1).
tRNA Leu(AAG)
0,07
0,04
1,84
Помимо этого, выравнивание первичных чте
tRNA Ala(UGC)
4,60
2,59
1,77
ний, полученных с помощью NGS, с полной
tRNA Ala(AGC)
4,14
2,35
1,76
последовательностью генома A. thaliana показа
tRNA Pro(CGG)
0,03
0,02
1,72
ло, что часть чтений соответствует тРНК, коди
руемым геномами хлоропластов и митохондрий.
tRNA Ile(UAU)
0,03
0,02
1,71
Согласно полученным данным, доля таких
tRNA Thr(CGU)
0,16
0,10
1,68
тРНК, составлявшая около 60% в исходном пре
tRNA Ser(AGA)
0,45
0,27
1,67
парате, использованном для связывания, увели
tRNA Ile(AAU)
0,02
0,01
1,64
чивалась при формировании комплекса с
AtSerpin1 до 70% (рисунок в Приложении), что
tRNA Leu(UAA)
0,03
0,02
1,57
указывало на отсутствие преимущественного
tRNA Leu(UAG)
0,15
0,10
1,55
связывания этим белком тРНК, кодируемых
tRNA Val(CAC)
0,04
0,03
1,50
ядерным геномом. С учетом того, что AtSerpin1
tRNA Pro(UGG)
1,04
0,70
1,48
является цитоплазматическим белком [21] и не
tRNA Val(UAC)
0,02
0,01
1,47
может взаимодействовать in vivo с хлоропласт
ными и митохондриальными тРНК в силу ком
tRNA Leu(CAG)
0,03
0,02
1,47
партментализации, однако эффективно связы
tRNA Gln(UUG)
0,74
0,53
1,40
вает их in vitro, как и гетерологичные дрожжевые
tRNA Asn(AUU)
0,11
0,08
1,40
тРНК, можно предположить, что белок
tRNA Val(AAC)
0,12
0,08
1,39
AtSerpin1 взаимодействует с универсальными
элементами вторичной/третичной структуры
Примечание. В таблице показаны 20 тРНК, имеющих наи
тРНК.
большее отношение эксперимент : контроль.
Селективность связывания тРНК белком
CmPS1. Для того, чтобы оценить, присущ ли ха
рактер связывания тРНК, выявленный для
CmPS1, для которых наблюдалось наибольшее
AtSerpin1, белку CmPS1, был проведен экспери
увеличение доли в общем количестве тРНК в
мент по анализу селективности связывания,
сравнении с контролем (табл. 2), не соответ
сходный с описанным выше для AtSerpin1. Ре
ствовали тем тРНК, для которых было выявлено
комбинантный белок CmPS1 инкубировали с
наибольшее соотношение опыт : контроль в
обогащенной тРНК фракцией тотального пре
эксперименте с AtSerpin1 (табл. 1). В целом, эти
парата РНК C. maxima при соотношении бе
данные согласуются с выводом о способности
лок : РНК = 10 : 1, когда только часть тРНК в
белков AtSerpin1 и CmPS1, относящихся к кон
смеси переходила в состав комплексов, которые
сервативной LR группе серпинов, связывать
затем были отделены от несвязавшейся тРНК с
тРНК, узнавая характерные особенности вто
помощью электрофореза в агарозном геле. Да
ричной/третичной структуры, но не какую ли
лее полосу, соответствующую комплексу, выре
бо специфическую нуклеотидную последова
зали из агарозного геля и использовали для вы
тельность.
деления содержащейся в комплексе РНК. Полу
Связывание pre9miR390 белком CmPS1. Спе
ченный образец вместе с эквивалентным коли
цифическое взаимодействие белков и молекул
чеством исходного препарата обогащенной
РНК основано на узнавании белками простран
тРНК фракции секвенировали с помощью NGS.
ственных (3D) мотивов, которые создаются за
Полученные чтения выравнивали с последова
счет неспаренных нуклеотидных остатков в дуп
тельностью генома C. maxima. Результаты анали
лексах РНК, участвующих в неканонических
за данных NGS для CmPS1 (табл. 2) в целом
взаимодействиях и формирующих структурно
сходны с теми, которые были получены для
уникальные поверхности [43, 44]. Ранее нами
AtSerpin1. В условиях избытка тРНК белок
было высказано предположение, что связыва
CmPS1 в той или иной степени связывал все
ние тРНК белком CmPS1 может быть основано
тРНК. При этом тРНК в составе комплексов с
на узнавании такого рода 3D мотива [31]. В дан
БИОХИМИЯ том 86 вып. 10 2021
1558
ТОЛСТЫКО и др.
ной работе мы провели экспериментальную
РНК [28]. Далее был проведен анализ способ
проверку этого предположения с использовани
ности белка CmPS1 связывать мутантные вари
ем в качестве РНК лиганда белка CmPS1 пред
анты pre miR390, в одном из которых было вве
шественника микроРНК 390 (pre miR390), ко
дено пять точечных замен, устраняющих неком
торый представляет собой несовершенный дуп
плементарные нуклеотидные остатки в стебле
лекс, способный за счет присутствия в его соста
шпилечной структуры pre miR390 и превраща
ве неспаренных остатков формировать 3D мо
ющие ее в совершенный дуплекс (мутант 5М),
тивы (рис. 3). Связывание белка CmPS1 с полу
тогда как в другом был делетирован район, фор
ченным in vitro T7 транскриптом, соответствую
мирующий дистальную часть стебля и петлю
щим pre miR390, анализировали методом сдви
вторичной структуры pre miR390 (мутант Loop)
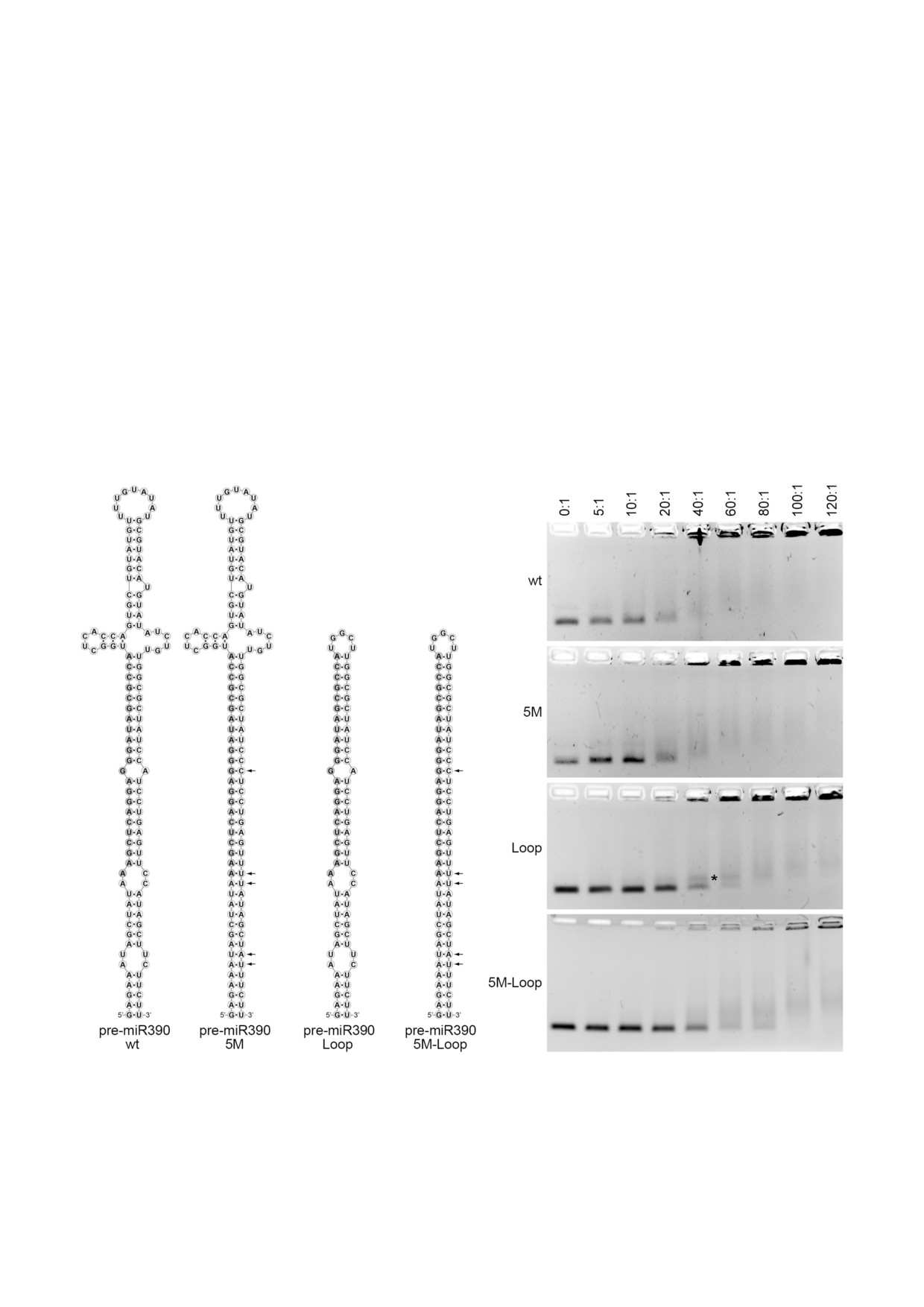
га в геле. Было обнаружено, что белок CmPS1
(рис. 3). В эксперименте по сдвигу в геле с
образует с pre miR390 два типа комплексов, а
Т7 транскриптами 5М и Loop было обнаруже
именно: комплексы, неспособные входить в
но, что оба мутанта сохраняют способность об
агарозный гель, и комплексы, мигрирующие в
разовывать два типа комплексов, характерные
геле (рис. 3), сходные с комплексами, наблю
для природной pre miR390 (рис. 3). При этом в
давшимися ранее при связывании CmPS1 с
случае мутанта Loop было обнаружено форми
РНК TCTP и PSTVd, представляющими собой,
рование дополнительного комплекса, наблюда
как и pre miR390, несовершенные дуплексы
емого как дискретная полоса в агарозном геле
Рис. 3. Связывание pre miR390 и ее мутантных вариантов белком CmPS1. Показаны модели вторичной структуры pre
miR390 дикого типа (wt) и мутантов 5М, Loop и 5M Loop. Нуклеотидные остатки, входящие в зрелую miR390, выделены
темно серым цветом. Стрелками отмечены остатки, мутированные в конструкциях 5М и 5M Loop. Анализ связывания
РНК белком CmPS1 проводили методом сдвига в геле. Указаны молярные соотношения белок : РНК для всех РНК
субстратов. Звездочкой обозначено положение дискретного комплекса
БИОХИМИЯ том 86 вып. 10 2021
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1559
(рис. 3) и сходного со специфическим комплек
C. sativus, способного к транспорту по флоэме в
сом, формируемым белком CmPS1 и тРНК.
удаленные части растения [30], было высказано
Можно предположить, что формирование тако
предположение о том, что связывание тРНК мо
го комплекса могло быть результатом взаимо
жет быть задействовано в двух различных меха
действия CmPS1 с 3D мотивами, формируемы
низмах сигналинга. Во первых, поскольку мно
ми некомплементарными остатками в стебле
гие мРНК, способные к транспорту по флоэме,
шпилечной структуры pre miR390. Вполне ве
несут в составе некодирующих областей после
роятно, что эти остатки маскированы в составе
довательности тРНК, которые служат сигнала
природной структуры за счет наличия ее дис
ми для транслокации таких мРНК по флоэ
тального района, включающего петлю, и стано
ме [29], белок CmPS1, взаимодействуя с этими
вятся доступными для взаимодействия с CmPS1
РНК, может служить белком транспортером,
в результате делеции этого района. Для того,
обеспечивающим их перенос к удаленным час
чтобы проверить это предположение, был скон
тям растения и/или повышающим их стабиль
струирован дополнительный мутант 5M Loop,
ность в ситовидных трубках флоэмы. Во вто
несущий обе мутации, присутствующие в
рых, с учетом того, что цитоплазма ситовидных
конструкциях 5М и Loop (рис. 3). Связывание
элементов содержит как полноразмерные
белка CmPS1 и транскрипта 5M Loop показало,
тРНК, так и фрагменты тРНК, которые, как из
что в этом случае дискретный комплекс не об
вестно, выполняют регуляторные функции при
разуется (рис. 3), что ясно свидетельствует о ро
ответах на стрессы и в первую очередь на вирус
ли неспаренных остатков стебля шпилечной
ную инфекцию, взаимодействие CmPS1 с тРНК
структуры pre miR390 в формировании этого
или их фрагментами может играть роль во фло
комплекса. Таким образом, специфическое взаи
эмном сигналинге [31]. Эта гипотеза согласуется
модействие белка CmPS1 с РНК может опреде
с фактом локализации белка CmPS1 в экстра
ляться 3D мотивами, существующими в РНК
фасцикулярной флоэме, содержащей белки, вы
за счет некомплементарных нуклеотидных ос
полняющие преимущественно защитные и сиг
татков.
нальные функции [45]. С учетом возможной ро
ли серпинов в системном сигналинге у высших
растений было бы интересно выяснить, облада
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
ют ли РНК связывающими свойствами серпи
ны LR типа, кодируемые эволюционно древни
Взаимодействие белка AtSerpin1 с тРНК,
ми организмами, не имеющими тканей и систем
продемонстрированное в данной работе, позво
межклеточной коммуникации, такими как, нап
ляет предполагать, что связывание РНК являет
ример, одноклеточная зеленая водоросль Chla:
ся общим свойством серпинов LR группы,
mydomonas reinhardtii и колониальная зеленая
включающей и AtSerpin1, и CmPS1 (флоэмный
водоросль Volvox carteri.
серпин тыквы), РНК связывающие свойства
С другой стороны, можно предположить на
которого были выявлены ранее [28]. С учетом
личие взаимосвязи между РНК связыванием и
того, что серпины LR группы являются эволю
ингибирующей активностью серпинов, что
ционно консервативными, предполагают, что
представляется весьма вероятным с учетом их
функции этих белков могут быть сходными у
структурных особенностей, молекулярного ме
широкого спектра организмов от одноклеточ
ханизма ингибирования протеиназ и ряда дан
ных зеленых водорослей до цветковых расте
ных, имеющихся для серпинов животных. В
ний [19]. На основании данных о том, что белок
частности, существенной особенностью серпи
AtSerpin1 может ингибировать по крайнем мере
нов является возможность регуляции ингиби
три протеиназы, задействованные в индукции
торной активности этих белков [1, 46]. Приме
клеточной смерти [21, 23, 24], можно полагать,
ром такого регулируемого ингибитора может
что одна из функций серпинов LR группы за
быть хорошо изученный антитромбин III чело
ключается в регулировании проапоптотических
века, ингибиторная петля которого в достаточ
механизмов. Вместе с тем роль связывания
ной степени экспонируется на поверхности бел
РНК, свойственного серпинам этой консерва
ковой молекулы и становится полностью дос
тивной группы, в настоящий момент остается
тупной для протеиназы только после связыва
неясной.
ния антитромбина с гепарином, который явля
Способность взаимодействовать с РНК мо
ется активатором белка [47-49]. Можно предпо
жет являться функцией, независимой от инги
лагать, что в отсутствие гепарина в растениях
бирующей активности серпинов. Принимая во
тРНК может играть роль регулятора ингибитор
внимание данные для CmPS1, который является
ной активности CmPS1 по аналогии с гепари
флоэмным белком [25], и его ортолога из
ном, участвующим в регуляции активности ан
БИОХИМИЯ том 86 вып. 10 2021
1560
ТОЛСТЫКО и др.
титромбина III, наиболее структурно близкого
уникальные пространственные структуры, с ко
белку CmPS1 серпина человека. Действительно,
торыми специфически связываются соответ
показано конкурентное ингибирование гепари
ствующие белки. С этой точки зрения можно
ном формирования комплекса CmPS1-тРНК
предположить, что исследованные серпины взаи
[28]. Кроме того, вполне вероятно, что ингиби
модействуют с консервативными элементами
рование протеиназы серпином, сопряженное с
пространственной укладки полинуклеотидной
кардинальным изменением его конформации,
цепи молекул тРНК. Способность серпинов уз
может существенным образом влиять на спо
навать 3D мотивы в молекулах РНК нашла
собность серпина связывать РНК [31].
подтверждение в экспериментах по связыванию
В настоящее время для белка AtSerpin1 не
pre miR390 белком CmPS1. Действительно, му
показана способность к транспорту по флоэме.
тантный вариант pre miR390, стебель шпилеч
Нельзя исключать, что AtSerpin1 обладает такой
ной структуры которого не включал неспарен
способностью, однако имеющиеся данные об
ные нуклеотидные остатки, утрачивал способ
ингибировании белком AtSerpin1 проапоптоти
ность формировать комплекс с CmPS1, види
ческих протеиназ, не имеющих флоэмной лока
мый в геле как дискретная полоса, тогда как
лизации, говорят в пользу того, что образование
другой вариант pre miR390, сохраняющий неком
комплекса AtSerpin1 с тРНК имеет регулятор
плементарные остатки, способные к образова
ную функцию, не связанную с флоэмным
нию 3D мотивов, такой способностью обладал.
транспортом. Можно предполагать, что связы
Мы предполагаем, что взаимодействие с такого
вание РНК белком AtSerpin1 представляет со
рода 3D мотивами является основой специфи
бой дополнительный уровень контроля про
ческого связывания серпинами растений неко
апоптотической активности протеиназ, являю
торых структурированных РНК, таких как
щихся мишенями AtSerpin1, за счет модулиро
тРНК, тогда как неспецифическое связывание
вания ингибирующей активности самого белка
РНК, наблюдаемое для CmPS1 и, в существенно
AtSerpin1. Для проверки этой гипотезы требуют
меньшей степени, для AtSerpin1, имеет под со
ся дополнительные эксперименты, которые ве
бой иную основу.
дутся в нашей лаборатории и ставят целью вы
Результаты, представленные в данной рабо
явление функциональной связи между двумя
те, выявляют новые особенности взаимодей
известными активностями серпинов LR груп
ствия белков AtSerpin1 и CmPS1 с РНК, но не
пы, а именно ингибированием протеиназ и
могут однозначно ответить на вопрос о функцио
РНК связыванием.
нальной значимости таких взаимодействий
В настоящей работе показано, что белок
in vivo. Исследование функциональной роли
AtSerpin1 обладает меньшей неспецифической
связывания РНК серпинами растений является
РНК связывающей активностью в сравнении с
задачей будущих исследований.
CmPS1. Вместе с тем, белки AtSerpin1 и CmPS1,
эффективно связывая тРНК, не проявляют вы
раженной специфичности в отношении каких
Финансирование. Работа Е.А. Толстыко,
либо видов тРНК растений, в той или иной сте
Д.А. Чергинцева, С.Ю. Морозова, А.Г. Соловье
пени связывая все тРНК в условиях проводив
ва выполнена при финансовой поддержке Рос
шихся экспериментов. Сочетание двух особен
сийского научного фонда (грант № 17 14
ностей исследованных серпинов, высокой аф
01032).
финности к тРНК по сравнению с другими вы
Конфликт интересов. А.Л. Коневега является
сокоструктурированными РНК и способности
учредителем компании «Нанотемпер Технолод
связывать все виды тРНК, может быть объясне
жис Рус», предоставляющей услуги по проведе
но взаимодействием этих белков с какими либо
нию исследований и представляющей техноло
элементами третичной структуры, общими для
гии «NanoTemper Technologies GmbH» в России.
всех молекул тРНК. Такая модель представляет
Соблюдение этических норм. Настоящая
ся вполне вероятной, поскольку специфическое
статья не содержит описания каких либо иссле
связывание белками молекул РНК основано на
дований с участием людей или животных в каче
узнавании пространственных (3D) мотивов, а не
стве объектов.
элементов первичной структуры [41, 42]. Такие
Дополнительные материалы. Приложение к
3D мотивы возникают в несовершенных дуп
статье на английском языке опубликовано на
лексах РНК за счет наличия в их составе неком
сайте журнала «Biochemistry» (Moscow) (http://
плементарных оснований, способных участво
protein.bio.msu.ru/biokhimiya/) и на сайте изда
вать в неканонических взаимодействиях с окру
жающими нуклеотидными остатками, создавая
10541), том 86, вып. 10, 2021.
БИОХИМИЯ том 86 вып. 10 2021
РНК СВЯЗЫВАЮЩИЕ СВОЙСТВА СЕРПИНОВ РАСТЕНИЙ
1561
СПИСОК ЛИТЕРАТУРЫ
1.
Gettins, P. G. W. (2002) Serpin structure, mechanism, and
blocks cell proliferation, J. Biol. Chem., 277, 13192 13201,
function, Chem. Rev., 102, 47514804, doi: 10.1021/
doi: 10.1074/jbc.M108460200.
cr010170+.
17.
Torriglia, A., Perani, P., Brossas, J. Y., Chaudun, E.,
2.
Law, R. H. P., Zhang, Q., McGowan, S., Buckle, A. M.,
Treton, J., et al. (1998) L DNase II, a molecule that links
Silverman, G. A., et al. (2006) An overview of the serpin
proteases and endonucleases in apoptosis, derives from the
superfamily, Genome Biol., 7, 1 11, doi: 10.1186/gb 2006
ubiquitous serpin leukocyte elastase inhibitor, Mol. Cell.
7 5 216.
Biol., 18, 3612 3619, doi: 10.1128/mcb.18.6.3612.
3.
Schick, C., Pemberton, P. A., Shi, G. P., Kamachi, Y.,
18.
Padron Barthe, L., Lepretre, C., Martin, E., Counis, M.
Çataltepe, S., et al. (1998) Cross class inhibition of the
F., and Torriglia, A. (2007) Conformational modification
cysteine proteinases cathepsins K, L, and S by the serpin
of serpins transforms leukocyte elastase inhibitor into an
squamous cell carcinoma antigen 1: a kinetic analysis,
endonuclease involved in apoptosis, Mol. Cell. Biol., 27,
Biochemistry, 37, 5258 5266, doi: 10.1021/bi972521d.
4028 4036, doi: 10.1128/mcb.01959 06.
4.
Irving, J. A., Pike, R. N., Dai, W., Brömme, D., Worrall,
19.
Cohen, M., Roberts, T. H., and Fluhr, R. (2015) Serpins in
D. M., et al. (2002) Evidence that serpin architecture intrin
plants, in Serpin Fam. Proteins with Mult. Funct. Heal. Dis.
sically supports papain like cysteine protease inhibition:
Cham, Springer International Publishing, pp. 15 28.
engineering α1 antitrypsin to inhibit cathepsin proteases,
20.
Roberts, T. H., and Hejgaard, J. (2008) Serpins in plants
Biochemistry, 41, 4998 5004, doi: 10.1021/bi0159985.
and green algae, Funct. Integr. Genomics,
8,
1 27,
5.
Ray, C. A., Black, R. A., Kronheim, S. R., Greenstreet,
doi: 10.1007/s10142 007 0059 2.
T. A., Sleath, P. R., et al. (1992) Viral inhibition of inflam
21.
Lampl, N., Alkan, N., Davydov, O., and Fluhr, R. (2013)
mation: Cowpox virus encodes an inhibitor of the inter
Set point control of RD21 protease activity by AtSerpin1
leukin 1β converting enzyme, Cell,
69,
597604,
controls cell death in Arabidopsis, Plant J., 74, 498 510,
doi: 10.1016/0092 8674(92)90223 Y.
doi: 10.1111/tpj.12141.
6.
Klieber, M. A., Underhill, C., Hammond, G. L., and
22.
Koh, E., Carmieli, R., Mor, A., and Fluhr, R. (2016)
Muller, Y. A. (2007) Corticosteroid binding globulin, a
Singlet oxygen induced membrane disruption and serpin
structural basis for steroid transport and proteinase trig
protease balance in vacuolar driven cell death, Plant
gered release, J. Biol. Chem.,
282,
2959429603,
Physiol., 171, 1616 1625, doi: 10.1104/pp.15.02026.
doi: 10.1074/jbc.M705014200.
23.
Lema Asqui, S., Vercammen, D., Serrano, I., Valls, M.,
7.
Zhou, A., Wei, Z., Read, R. J., and Carrell, R. W. (2006)
Rivas, S., et al. (2018) AtSERPIN1 is an inhibitor of the
Structural mechanism for the carriage and release of thy
metacaspase AtMC1 mediated cell death and autocatalyt
roxine in the blood, Proc. Natl. Acad. Sci. USA, 103, 13321
ic processing in planta, New Phytol., 218, 1156 1166,
13326, doi: 10.1073/pnas.0604080103.
doi: 10.1111/nph.14446.
8.
Mala, J. G. S., and Rose, C. (2010) Interactions of heat
24.
Vercammen, D., Belenghi, B., van de Cotte, B.,
shock protein 47 with collagen and the stress response: an
Beunens, T., Gavigan, J. A., et al. (2006) Serpin1 of
unconventional chaperone model? Life Sci., 87, 579 586,
Arabidopsis thaliana is a suicide inhibitor for metacaspase
doi: 10.1016/j.lfs.2010.09.024.
9, J. Mol. Biol., 364, 625 636, doi: 10.1016/j.jmb.2006.
9.
Huntington, J. A., and Stein, P. E. (2001) Structure and
09.010.
properties of ovalbumin, J. Chromatogr. B Biomed. Sci.
25.
Yoo, B. C., Aoki, K., Xiang, Y., Campbell, L. R., Hull,
Appl., 756, 189 198, doi: 10.1016/S0378 4347(01)00108 6.
R. J., et al. (2000) Characterization of Cucurbita maxima
10.
Kumar, A. (2015) Bayesian phylogeny analysis of verte
phloem serphin 1 (CmPS 1). A developmentally regulated
brate serpins illustrates evolutionary conservation of the
elastase inhibitor, J. Biol. Chem., 275, 3512235128,
intron and indels based six groups classification system
doi: 10.1074/jbc.M006060200.
fromlampreys for
∼500
MY, PeerJ,
2015, e1026,
26.
Alvarez Alfageme, F., Maharramov, J., Carrillo, L.,
doi: 10.7717/peerj.1026.
Vandenabeele, S., Vercammen, D., et al. (2011) Potential
11.
Marijanovic, E. M., Fodor, J., Riley, B. T., Porebski, B. T.,
use of a serpin from arabidopsis for pest control, PLoS One,
Costa, M. G. S., et al. (2019) Reactive centre loop dynam
6, e20278, doi: 10.1371/journal.pone.0020278.
ics and serpin specificity, Sci. Rep.,
9,
1 15,
27.
Cohen, M., Davydov, O., and Fluhr, R. (2019) Plant serpin
doi: 10.1038/s41598 019 40432 w.
protease inhibitors: Specificity and duality of function,
12.
Whisstock, J. C., and Bottomley, S. P. (2006) Molecular
J. Exp. Bot., 70, 2077 2085, doi: 10.1093/jxb/ery460.
gymnastics: serpin structure, folding and misfolding, Curr.
28.
Tolstyko, E. A., Lezzhov, A. A., Pankratenko, A. V.,
Opin. Struct. Biol., 16, 761 768, doi: 10.1016/j.sbi.2006.
Serebryakova, M. V., Solovyev, A. G., and Morozov, S. Y.
10.005.
(2020) Detection and in vitro studies of Cucurbita maxima
13.
Whisstock, J. C., Skinner, R., Carrell, R. W., and Lesk,
phloem serpin 1 RNA binding properties, Biochimie, 170,
A. M. (2000) Conformational changes in serpins: I. The
118 127, doi: 10.1016/j.biochi.2020.01.006.
native and cleaved conformations of α1 antitrypsin, J. Mol.
29.
Kehr, J., and Kragler, F. (2018) Long distance RNA move
Biol., 296, 685 699, doi: 10.1006/jmbi.1999.3520.
ment, New Phytol., 218, 29 40, doi: 10.1111/nph.15025.
14.
Rühlmann, A., Kukla, D., Schwager, P., Bartels, K., and
30.
Petersen, M. L. C., Hejgaard, J., Thompson, G. A., and
Huber, R. (1973) Structure of the complex formed by
Schulz, A. (2005) Cucurbit phloem serpins are graft trans
bovine trypsin and bovine pancreatic trypsin inhibitor.
missible and appear to be resistant to turnover in the sieve
Crystal structure determination and stereochemistry of the
element companion cell complex, J. Exp. Bot., 56, 3111
contact region, J. Mol. Biol., 77, 417 436, doi: 10.1016/
3120, doi: 10.1093/jxb/eri308.
0022 2836(73)90448 8.
31.
Tolstyko, E. A., Lezzhov, A. A., Morozov, S. Y., and
15.
Grigoryev, S. A., and Woodcock, C. L. (1998) Chromatin
Solovyev, A. G. (2020) Phloem transport of structured
structure in granulocytes. A link between tight compaction
RNAs: a widening repertoire of trafficking signals and pro
and accumulation of a heterochromatin associated protein
tein factors, Plant Sci., 299, 110602, doi: 10.1016/
(MENT), J. Biol. Chem., 273, 3082 3089, doi: 10.1074/
j.plantsci.2020.110602.
jbc.273.5.3082.
32.
Atabekova, A. K., Pankratenko, A. V., Makarova, S. S.,
16.
Irving, J. A., Shushanov, S. S., Pike, R. N., Popova, E. Y.,
Lazareva, E. A., Owens, R. A., et al. (2017) Phylogenetic
Brömme, D., et al. (2002) Inhibitory activity of a hete
and functional analyses of a plant protein related to human
rochromatin associated serpin (MENT) against papain
B cell receptor associated proteins, Biochimie, 132, 28 37,
like cysteine proteinases affects chromatin structure and
doi: 10.1016/j.biochi.2016.10.009.
11 БИОХИМИЯ том 86 вып. 10 2021
1562
ТОЛСТЫКО и др.
33.
Makarova, S. S., Solovyev, A. G., and Morozov, S. Y.
seq aligner, Bioinformatics, 29, 1521, doi: 10.1093/
(2014) RNA binding properties of the plant protein Nt
bioinformatics/bts635.
4/1, Biochemistry,
79,
717726, doi:
10.1134/
42. Anders, S., Pyl, P. T., and Huber, W. (2014) HTSeq - a
S000629791407013X.
Python framework to work with high throughput sequenc
34.
Asmari, M., Ratih, R., Alhazmi, H. A., El Deeb, S. (2018)
ing data, bioRxiv, 002824, doi: 10.1101/002824.
Thermophoresis for characterizing biomolecular interac
43. Chojnowski, G., Waleń, T., and Bujnicki, J. M. (2014)
tion, Methods, 146, 107 119, doi: 10.1016/j.ymeth.2018.
RNA Bricks - a database of RNA 3D motifs and their
02.003.
interactions, Nucleic Acids Res., 42, D123 31, doi: 10.1093/
35.
Vinogradova, D. S., Zegarra, V., Maksimova, E.,
nar/gkt1084.
Nakamoto, J. A., Kasatsky, P., et al. (2020) How the initi
44. Leontis, N. B., Lescoute, A., and Westhof, E. (2006) The
ating ribosome copes with ppGpp to translate mRNAs,
building blocks and motifs of RNA architecture, Curr.
PLoS Biol., 18, e3000593, doi: 10.1371/journal.pbio.
Opin. Struct. Biol., 16, 279 287, doi: 10.1016/j.sbi.2006.
3000593.
05.009.
36.
Andrews, S. (2010) FastQC: a quality control tool for
45. Gaupels, F., and Ghirardo, A. (2013) The extrafascicular
high throughput sequence data. Available online at:
phloem is made for fighting, Front. Plant Sci., 4, 187,
doi: 10.3389/fpls.2013.00187.
fastqc.
46. Gettins, P. G. W., and Olson, S. T. (2016) Inhibitory ser
37.
Jühling, F., Mörl, M., Hartmann, R. K., Sprinzl, M.,
pins. New insights into their folding, polymerization, regu
Stadler, P. F., and Pütz, J. (2009) tRNAdb 2009: compila
lation and clearance, Biochem. J., 473, 22732293,
tion of tRNA sequences and tRNA genes, Nucleic Acids
doi: 10.1042/BCJ20160014.
Res., 37, D159 D162, doi: 10.1093/nar/gkn772.
47. Whisstock, J. C., Pike, R. N., Jin, L., Skinner, R., Pei,
38.
Langmead, B., and Salzberg, S. L. (2012) Fast gapped read
X. Y., et al. (2000) Conformational changes in serpins: II.
alignment with Bowtie 2, Nat. Methods, 9, 357 359,
The mechanism of activation of antithrombin by heparin,
doi: 10.1038/nmeth.1923.
J. Mol. Biol., 301, 1287 1305, doi: 10.1006/jmbi.2000.3982.
39.
Langmead, B., Wilks, C., Antonescu, V., and Charles, R.
48. Rezaie, A. R. (1998) Calcium enhances heparin catalysis of
(2019) Scaling read aligners to hundreds of threads on gen
the antithrombin factor Xa reaction by a template mecha
eral purpose processors, Bioinformatics, 35, 421432,
nism: evidence that calcium alleviates Gla domain antago
doi: 10.1093/bioinformatics/bty648.
nism of heparin binding to factor Xa, J. Biol. Chem., 273,
40.
Kim, D., Paggi, J. M., Park, C., Bennett, C., and Salzberg,
16824 16827, doi: 10.1074/jbc.273.27.16824.
S. L. (2019) Graph based genome alignment and genotyp
49. Li, W., Johnson, D. J. D., Esmon, C. T., and Huntington,
ing with HISAT2 and HISAT genotype, Nat. Biotechnol.,
J. A. (2004) Structure of the antithrombin thrombin
37, 907 915, doi: 10.1038/s41587 019 0201 4.
heparin ternary complex reveals the antithrombotic mech
41.
Dobin, A., Davis, C. A., Schlesinger, F., Drenkow, J.,
anism of heparin, Nat. Struct. Mol. Biol., 11, 857 862,
Zaleski, C., et al. (2013) STAR: ultrafast universal RNA
doi: 10.1038/nsmb811.
RNA BINDING BY PLANT SERPINS in vitro
E. A. Tolstyko1,2, D. A. Chergintsev3, O. A. Tolicheva2, D. S. Vinogradova2,4,
A. L. Konevega2,5,6, S. Y. Morozov1,7, and A. G. Solovyev1,7*
1 Department of Virology, Biological Faculty, Lomonosov Moscow State University, 119234 Moscow, Russia
2 Konstantinov St. Petersburg Nuclear Physics Institute of National Research Center “Kurchatov Institute”,
188300 Gatchina, Russia
3 Department of Plant Physiology, Biological Faculty, Lomonosov Moscow State University, 119234 Moscow, Russia
4 NanoTemper Technologies Rus, 191167 Saint Petersburg, Russia
5 Peter the Great Saint Petersburg Polytechnic University, 195251 Saint Petersburg, Russia
6 National Research Center “Kurchatov Institute” 123182 Moscow, Russia
7 Belozersky Institute of Physico:Chemical Biology, Lomonosov Moscow State University,
119992 Moscow, Russia; e:mail: solovyev@belozersky.msu.ru
Serpins constitute a large family of protease inhibitors with regulatory functions found in all living organisms. Most
plant serpins have not been functionally characterized, with the exception of Arabidopsis thaliana AtSerpin1, an
inhibitor of pro apoptotic proteases, which is involved in the regulation of the programmed cell death induction, and
Cucurbita maxima CmPS1, a phloem protein, which presumably inhibits insect digestive proteases and binds RNA.
CmPS1 interacts most efficiently with highly structured RNA; in particular, it forms a specific complex with tRNA.
Here, we demonstrated that AtSerpin1 also forms a complex with tRNA. Analysis of tRNA species bound by
AtSerpin1 and CmPS1 in the presence of tRNA excess revealed that both proteins have no strict selectivity for indi
vidual tRNAs, suggesting specific interaction of AtSerpin1 and CmPS1 proteins with elements of the secondary/ter
tiary structure universal for all tRNAs. Analysis of CmPS1 binding of the microRNA precursor pre miR390 and its
mutants demonstrated that the pre miR390 mutant with a perfect duplex in the hairpin stem lost the ability to form
a discrete complex with CmPS1, whereas another variant of pre miR390 with the native unpaired nucleotide residues
in the stem retained this ability. These data indicate that specific interactions of plant serpins with structured RNA are
based on the recognition of structurally unique spatial motifs formed with the participation of unpaired nucleotide
residues in the RNA duplexes.
Keywords: serpin, RNA binding, RNA binding protein, tRNA, microRNA, RNA structure, phloem
БИОХИМИЯ том 86 вып. 10 2021