БИОХИМИЯ, 2021, том 86, вып. 3, с. 297 - 307
УДК 577.216.35
СТРУКТУРА И МЕХАНИЗМЫ ЭКСПРЕССИИ БОЛЬШИХ
(+)РНК ГЕНОМОВ ВИРУСОВ ВЫСШИХ ЭУКАРИОТ
Мини обзор
© 2021
А.А. Аграновский
Московский государственный университет имени М.В. Ломоносова, биологический факультет,
кафедра вирусологии, 119234 Москва, Россия; электронная почта: etdonas2@gmail.com
Поступила в редакцию 17.08.2020
После доработки 21.09.2020
Принята к публикации 29.09.2020
Быстрые темпы эволюции РНК геномов, которые обусловлены высоким уровнем мутаций и рекомбинаци
ей при копировании цепей РНК, позволяют вирусу изменять и приобретать последовательности для опти
мальной адаптации. Размеры РНК генома ограничены факторами, связанными с точностью РНК полиме
раз и упаковкой. В ходе эволюции (+)РНК геномы нидовирусов животных (артеривирусов, ронивирусов,
коронавирусов) и клостеровирусов растений преодолели барьер в 12 тыс. нуклеотидов. Коронавирусы и
клостеровирусы имеют общие свойства. Их РНК геномы содержат 5′ концевые гены, экспрессируемые с
помощью рибосомального сдвига рамки считывания и кодирующие домены папаин подобной протеиназы,
метилтрансферазы, мембраносвязывающих белков, хеликазы и РНК полимеразы. Коронавирусы, в допол
нение к этому, содержат домены экзонуклеазы с редактирующей активностью, гипотетической праймазы,
нуклеотидилтрансферазы и эндонуклеазы. РНК геном коронавирусов и клостеровирусов содержит на
3′ конце гены структурных и вспомогательных белков, для экспрессии которых используется набор котер
минальных субгеномных РНК. В ходе эволюции вирионов представители обеих вирусных групп приобрели
гибкие спирально симметричные нуклеокапсиды, что позволило снять ограничения на размер инкапсиди
руемых молекул РНК. Филогенетические реконструкции домена РНК полимеразы указывают лишь на от
даленное родство коронавирусов и клостеровирусов, и их общие свойства скорее всего возникли независи
мо при эволюции больших РНК геномов.
КЛЮЧЕВЫЕ СЛОВА: вирусные РНК геномы, нидовирусы, SARS CoV, клостеровирусы, эволюция,
экспрессия генов.
DOI: 10.31857/S0320972521030027
ВВЕДЕНИЕ
ство РНК содержащих вирусов имеет компакт
ные геномы размером 4-12 kb (1 kb - 1000 нук
Быстрые темпы эволюции вирусных РНК
леотидов) [1]. Размеры РНК генома ограничи
геномов, обусловленные высоким уровнем му
ваются рядом факторов, в первую очередь свя
таций и «перетасовкой генов» при репликации,
занных с точностью репликации РНК. Вирус
приводят к изменению и приобретению генов,
ные РНК зависимые РНК полимеразы вносят
необходимых для адаптации вируса. Большин
около 10-4 ошибок на копируемый нуклеотид
ный остаток, т.е. на несколько порядков боль
ше, чем ДНК полимеразы [2, 3]. Считается, что
Принятые сокращения: нт - нуклеотид; ОРТ - от
накопление неблагоприятных мутаций при ко
крытая рамка трансляции; сгРНК - субгеномные РНК;
пировании больших РНК геномов превышает
ЭР - эндоплазматический ретикулум; BYV - клостерови
рус желтухи свеклы (beet yellows virus); CP и CPm - мажор
критический уровень, при котором возможно
ный и минорный структурные белки; DMV - везикула, ок
воспроизводство жизнеспособных вариантов
руженная двухслойной мембраной (double membrane vesi
вируса [4]. Кроме того, размеры РНК влияют на
cle); HEL - РНК хеликаза; kb - kilobase, 1000 нуклеоти
ее стабильность и ограничивают возможность
дов; L - лидерная последовательность РНК; MHV - коро
упаковки в капсид определенного типа [5].
навирус мышиного гепатита; MP - (главная) сериновая
протеиназа; Mtr -N7 гуанин метилтрансфераза; MVC -
В ходе эволюции РНК геномы нидовирусов
мультивезикулярный комплекс; nsp - неструктурный бе
животных (сем. Arteriviridae, Coronaviridae,
лок; NU - нидовирусная уридилат специфичная эндори
Roniviridae и Mesoniviridae; порядок Nidovirales)
бонуклеаза; POL - РНК полимераза; pp - полипротеин;
и клостеровирусов растений (сем. Closteroviri
SARS CoV - коронавирусы тяжелого острого респиратор
ного синдрома; TRS - сигналы регуляции транскрипции
dae) преодолели барьер в 12 000 нуклеоти
нидовирусов (transcription regulatory signals).
дов (нт). Коронавирусы имеют самые крупные
297
298
АГРАНОВСКИЙ
неразделенные РНК геномы среди всех
КОРОНАВИРУСЫ
(+)РНК вирусов (до 41 kb у коронавируса пла
нарий [6]). Размеры генома клостеровирусов
Представители порядка Nidovirales - арте
варьируют от 14,5 до 19 kb [7, 8]. Рассмотрение
ривирусы, коронавирусы, мезонивирусы и ро
особенностей структуры, репликации и
нивирусы - весьма вариабельны с точки зрения
экспрессии больших РНК геномов является
размеров и структуры генома. Сравнение после
предметом настоящего обзора. Сравнение
довательностей аминокислот наиболее консер
свойств нидовирусов и клостеровирусов позво
вативного вирусного фермента, РНК полиме
ляет увидеть парадоксальные черты сходства
разы, указывает, что нидовирусы образуют ком
между этими эволюционно далекими друг от
пактный филогенетический кластер [9] в филю
друга вирусами животных и растений.
ме пикорна подобных вирусов [10]. В настоя
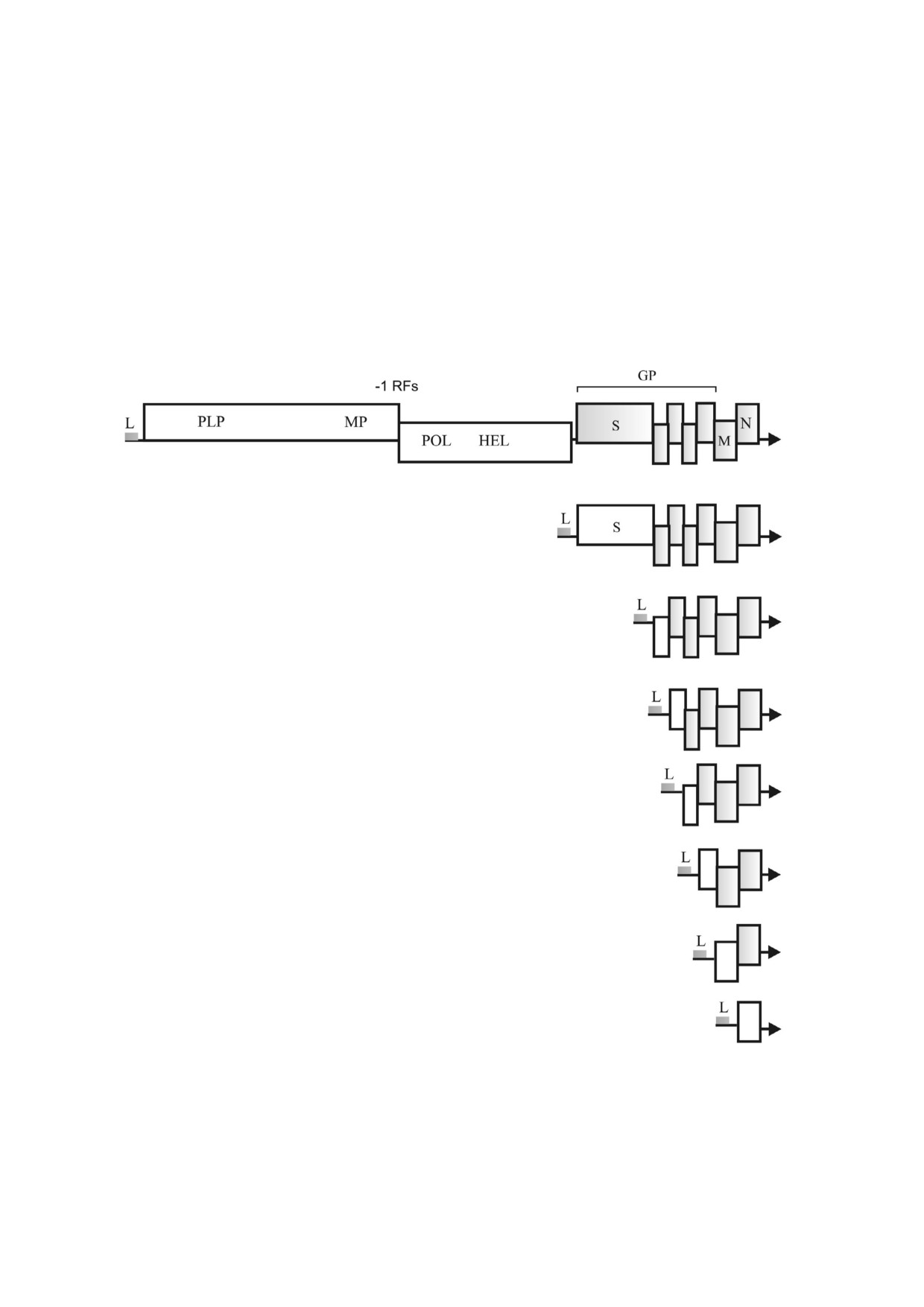
Рис. 1. Структура генома и субгеномных РНК коронавируса тяжелого острого респираторного синдрома (SARS CoV).
L - лидерная последовательность; -1 RFs - сигнал -1 рибосомального сдвига рамки считывания. Обозначения закоди
рованных белковых доменов: PLP - папаин подобная протеиназа; MP - (главная) сериновая протеиназа; POL - РНК поли
мераза; HEL - РНК хеликаза; S - белок шипа; GP - вспомогательные белки и гликопротеины наружной мембраны;
M - матриксный белок; N - белок нуклеокапсида. Гены, недоступные для трансляции в каждом типе мРНК, показаны
затененными прямоугольниками. 3′ конец РНК обозначен стрелкой
БИОХИМИЯ том 86 вып. 3 2021
РНК ГЕНОМЫ НИДОВИРУСОВ И КЛОСТЕРОВИРУСОВ
299
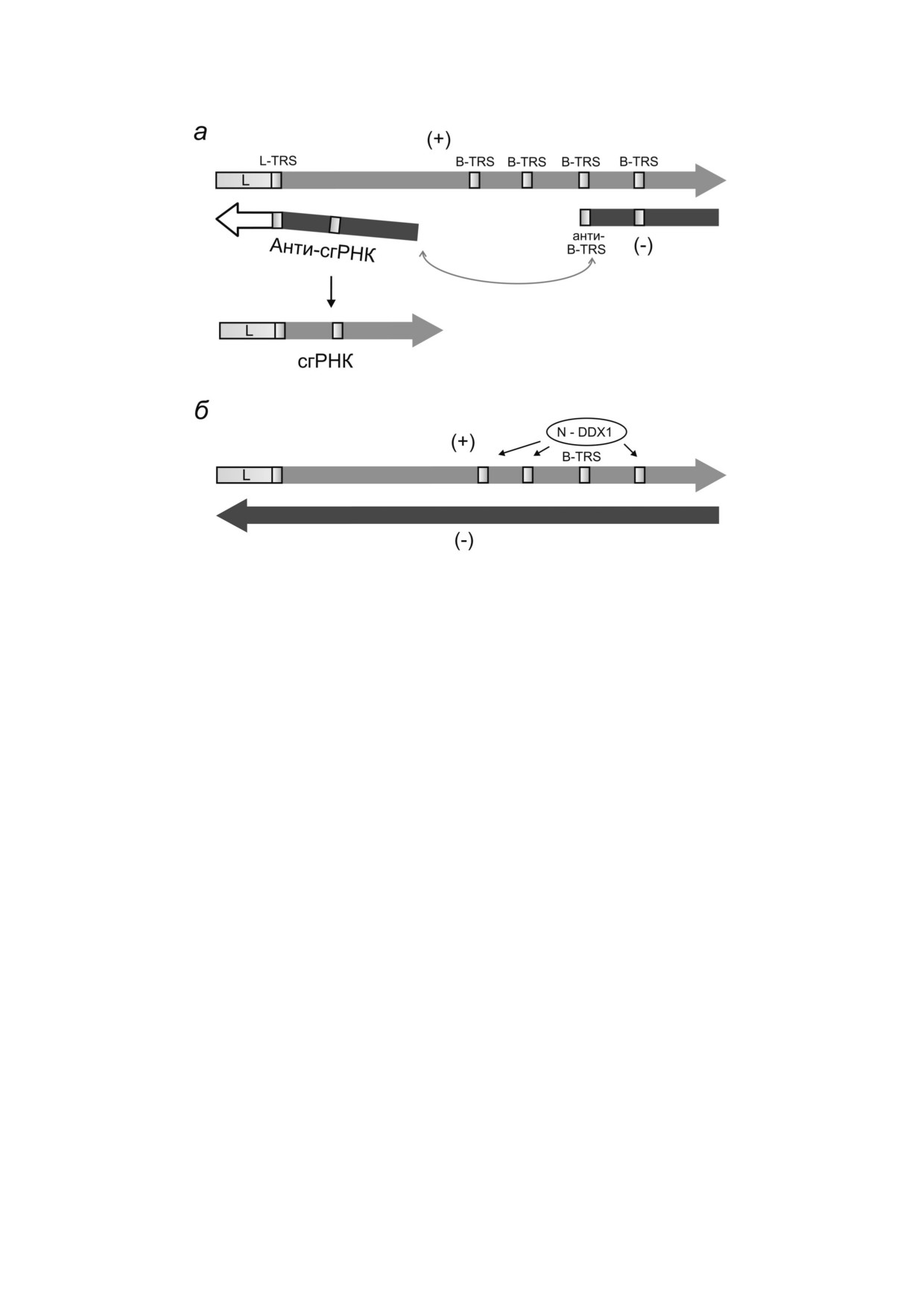
Рис. 2. Транскрипция и репликация РНК коронавирусов. а - Модель прерывистой транскрипции. При копировании ге
номной РНК может происходить остановка синтеза (-) цепи на сигнале регуляции транскрипции B TRS (показан пря
моугольником), «перескок» антисубгеномной (-) цепи РНК, отжиг анти B TRS на L TRS и достройка цепи на матрице
лидерной последовательности РНК (L). Кодирующие и некодирующие участки РНК изображены не в масштабе. б - Ви
русный белок нуклеокапсида N и хеликаза DDX1 присоединяются к B TRS, в результате чего стоп сигналы TRS игнори
руются и происходит процессивный синтез полноразмерной (-) цепи РНК, а на матрице (-) цепи - синтез дочерних
(+) цепей РНК (репликация). 3′ конец РНК обозначен стрелкой
щем обзоре мы ограничимся рассмотрением
ка (М) и нуклеокапсидного белка (N) - нахо
Coronaviridae, как наиболее эпидемиологически
дятся в 3′ концевой части генома и экспресси
значимого и хорошо изученного семейства по
руются с помощью 3′ котерминальных субге
рядка Nidovirales.
номных РНК (сгРНК) (рис. 1). Геномная и
Структура и экспрессия генома. Коронавиру
сгРНК несут на 5′ конце идентичные лидерные
сы имеют спирально симметричные нуклеокап
последовательности (L), длина которых состав
сиды, которые окружены липопротеидной обо
ляет 60-90 нт.
лочкой, снабженной белком шипа и другими
Экспрессия 3′ концевых генов структурных
вирусными гликопротеинами
[9]. Геномная
белков коронавирусов происходит с участием
РНК коронавирусов содержит 5′ концевую кеп
набора сгРНК, в каждой из которых для транс
структуру и 3′ концевую поли(А) последова
ляции доступен только
5′ проксимальный
тельность. Открытые рамки трансляции (ОРТ)
ген (рис. 1). Синтез нидовирусных сгРНК осу
1а и 1b, кодирующие компоненты репликазы,
ществляется с помощью уникального механиз
занимают 5′ концевую область генома (рис. 1).
ма прерывистой транскрипции [11, 12]. В цепи
Трансляция этих генов с помощью механиз
геномной РНК перед каждым геном (кроме
ма -1 рибосомального сдвига рамки считыва
ОРТ 1b) расположена консервативная последо
ния приводит к синтезу полипротеинов (pp) 1а
вательность длиной 6-8 нт - L TRS (лидерный
и 1аb в молярном соотношении 4/1. pp1а и pp1ab
транскрипционный регуляторный сайт, leader
коронавирусов содержат примерно 4 и 7 тыс.
transcription regulatory site, TRS) или B TRS
аминокислотных остатков (а.о.) соответствен
(body TRS) (рис. 2). Синтез антигеномной цепи
но. Процессинг рр1а и рр1ab вирусными проте
(-)РНК с некоторой вероятностью может задер
иназами приводит к образованию 16 неструк
живаться на B TRS в геномной РНК (рис. 2). За
турных белков (nsp). Гены структурных белков -
тем синтезированный фрагмент (-) цепи либо
мембранных гликопротеинов, матриксного бел
«перескакивает» на 5′ конец (+)РНК, либо
БИОХИМИЯ том 86 вып. 3 2021
300
АГРАНОВСКИЙ
участки сближаются в результате выпетливания
незначительно влияют на репликацию MHV в
(+)РНК. Происходит отжиг анти В TRS и L
культуре клеток [18].
TRS и достройка фрагмента (-)РНК на матрице
Многофункциональный белок nsp3 несет
(+)L (рис. 2, а). В результате получаются анти
домен протеиназы PLP и трансмембранный до
сгРНК, содержащие общую последовательность
мен [19]. TM белки nsp3, nsp4 и nsp6 индуциру
анти L, которые служат матрицами для образо
ют реорганизацию мембран клетки и создание
вания сгРНК. Фосфорилированный свободный
репликативных компартментов [20-22]. Проте
белок нуклеокапсида N связывается с B TRS и
иназа nsp5 осуществляет процессинг большин
привлекает клеточную хеликазу DDX1; эти вза
ства сайтов в pp1а и рр1ab (рис. 3). Зрелый белок
имодействия, осуществляемые на поздних ста
nsp5 входит в состав репликативного комплекса.
диях инфекционного цикла, позволяют репли
Функции малых белков nsp7-nsp11 связаны с
кативному комплексу игнорировать стоп сиг
синтезом РНК, и все они необходимы для реп
налы транскрипции и синтезировать полнораз
ликации. Белки nsp7 и nsp8 формируют цилинд
мерную антигеномную РНК, служащую матри
рический комплекс из 8 субъединиц каждого
цей для воспроизводства полноразмерных
белка, в котором положительно заряженные а.о.
(+)РНК (рис. 2, б) [13, 14].
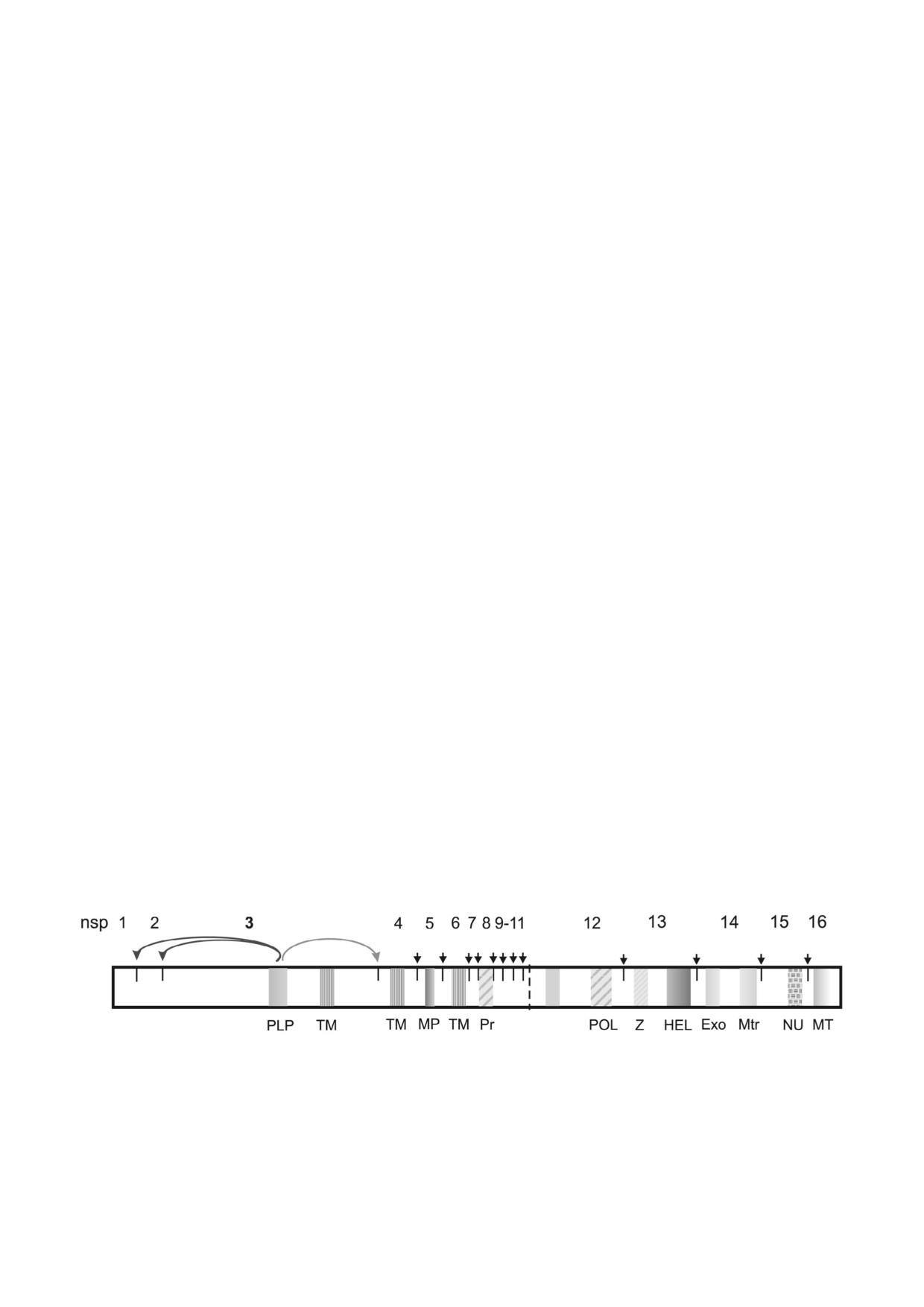
экспонированы в центральной полости. Уни
Репликативные белки. В состав репликатив
кальным свойством коронавирусов (впервые
ного комплекса коронавирусов входит 16 неструк
обнаруженным у SARS CoV 1) является участие
турных белков - продуктов процессинга pp1а и
в репликации белка nsp8 в качестве вспомога
pp1аb папаин подобной протеиназой (PLP) и се
тельной РНК полимеразы (гипотетической
риновой протеиназы (MP) (рис. 3). Интересно,
праймазы (Pr); рис. 3) [23], которая использует в
что родственные коронавирусы могут содержать
качестве матриц последовательности РНК с
один или два домена PLP [9]. В случае вируса
консенсусом 5′ (G/U)CC и синтезирует корот
мышиного гепатита (MHV) автокаталитическое
кие (до 6 нт) комплементарные РНК. Помимо
выщепление nsp1, nsp2, nsp3 и nsp4 происходит
низкой процессивности, nsp8 является самой
под действием двух доменов PLP, тогда как в слу
неточной РНК полимеразой из всех известных
чае SARS CoV 1, SARS CoV 2, MERS и вируса
ферментов данного типа (одна ошибка на 10 нт).
инфекционного бронхита (IBV) за эти реакции
Высказывались предположения, что Pr может
ответственна единственная PLP (рис. 1 и 3). Та
выполнять функции праймазы при репликации
ким образом, в процессе эволюции коронавиру
РНК коронавирусов или повышать процессив
сов, очевидно, произошла дупликация и дивер
ность РНК полимеразы nsp12 [23, 24].
генция доменов протеиназы. Процессинг боль
Белок nsp12 несет домены нуклеотидил
шинства сайтов в полипротеинах осуществляет
трансферазы и РНК полимеразы и является
ся in trans вирусной сериновой протеиназой, по
сердцевинным РНК синтезирующим фермен
лучившей название «главной протеиназы» (MP)
том коронавирусов [24]. Белок nsp13 содержит
[9]. Специфические функции nsp1 и nsp2 коро
цинк связывающий и хеликазный домены
навирусов, по видимому, связаны не с реплика
(рис. 3).
цией РНК, а с деградацией клеточных РНК и по
NSP14 содержит на N конце домен 3′ 5′ эк
давлением клеточной трансляции [15, 16], а так
зонуклеазы (Exo), а на С конце - домен N7 гуа
же с созреванием вирусных частиц [17]. Искус
нин метилтрансферазы (Mtr; рис. 3). Предска
ственные мутации, блокирующие разрезание
занные с помощью методов биоинформатики
сайта nsp1/nsp2, и даже делеции nsp1 и nsp2 лишь
активности экзонуклеазы и метилтрансферазы
Рис. 3. Структура репликативного полипротеина 1аb коронавируса SARS CoV 1. Вертикальный пунктирный отрезок -
граница между 1а и 1b в слитном белке. Домены неструктурных белков nsp1-nsp16 обозначены прямоугольниками. PLP -
папаин подобная протеиназа (сайты разрезания in cis показаны изогнутыми стрелками); MP - главная сериновая проте
иназа (сайты разрезания in trans показаны стрелками); ТМ - трансмембранные домены; Pr - гипотетическая праймаза;
POL - РНК полимераза; Z - цинк связывающий домен; HEL - РНК хеликаза; Exo - 3′ 5′ экзонуклеаза; Mtr - N7 гуа
нозин метилтрансфераза; NU - нидовирусная уридилат специфичная эндорибонуклеаза; МТ - рибозил 2′ О метил
трансфераза
БИОХИМИЯ том 86 вып. 3 2021
РНК ГЕНОМЫ НИДОВИРУСОВ И КЛОСТЕРОВИРУСОВ
301
были подтверждены экспериментально [25-27].
которые характеризуются несколькими отличи
Родственные коронавирусной Exo клеточные
тельными свойствами: (1) переносятся насеко
ферменты семейства DEDD участвуют в редак
мыми векторами полуперсистентно (т.е. вирус
тировании (proofreading), репарации и рекомби
задерживается в векторе в течение нескольких
нации нуклеиновых кислот. При репликации ко
часов); (2) обладают уникальной структурой ни
ронавирусных РНК nsp14 выполняет роль редак
тевидных частиц, построенных из нескольких
тирующего фермента, повышающего точность
структурных белков; (3) имеют большие РНК
копирования цепей; подобная функция уни
геномы (до 20 kb); (4) несут ген гомолога клеточ
кальна для РНК содержащих вирусов [25, 26].
ных шаперонов HSP70; (5) несут дуплицирован
NSP15 несет домен уридилат специфичной
ные гены или фрагменты генов (например, гены
эндонуклеазы (NU) (рис. 3) [28]. Коронавирус
основного и минорного структурных белков).
ная NU необходима для репликации и тран
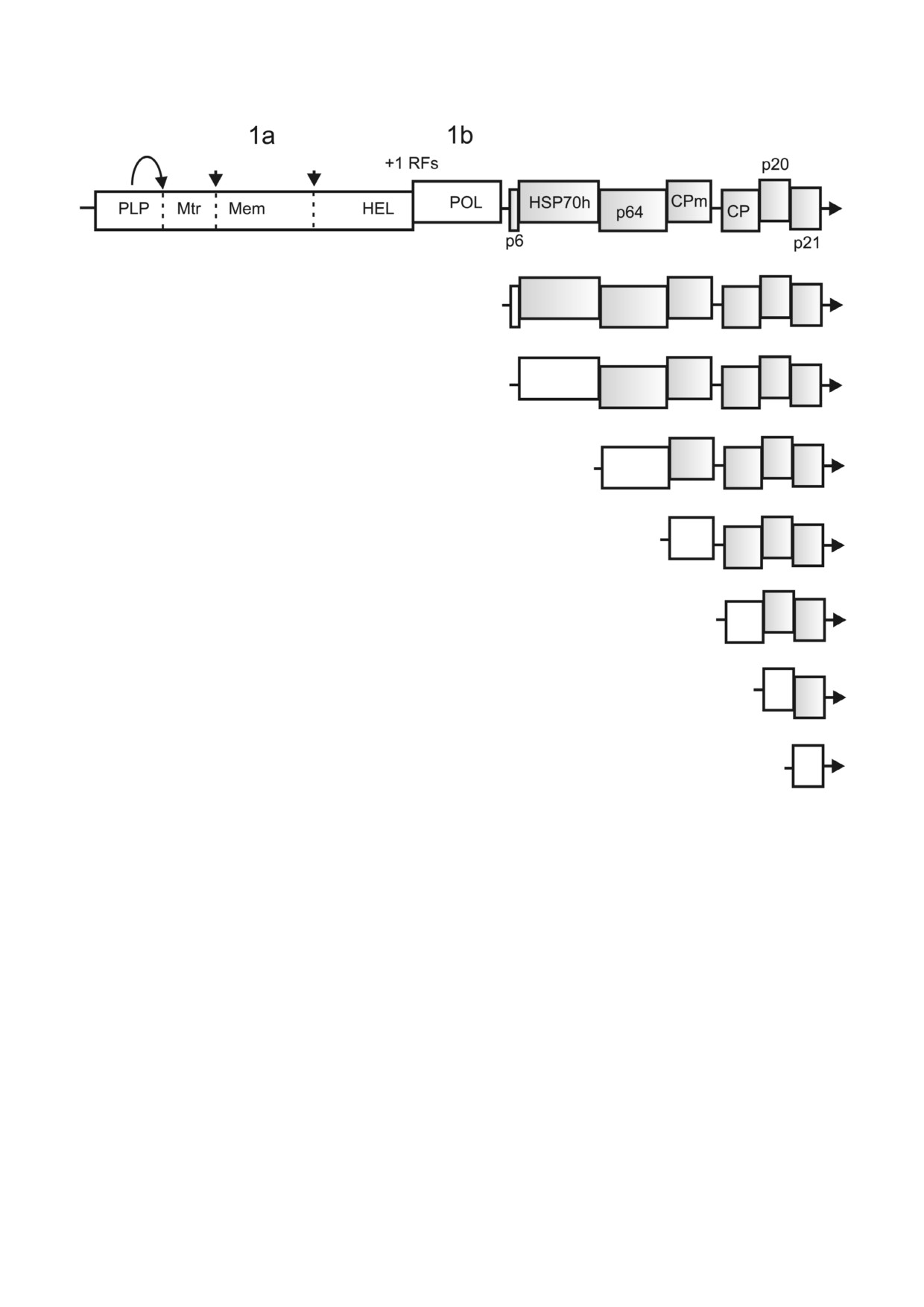
Структура и экспрессия генома. Геномная
скрипции, а также является антагонистом ин
РНК вируса желтухи свеклы (BYV; 14,5 kb) со
терферона. Белок NSP16 содержит домен рибо
держит 5′ кеп и не имеет 3′ поли(А) [7, 33, 35].
зил 2′ О метилтрансферазы (МТ), осуществля
В 5′ концевой области генома BYV и других
ющий метилирование второго остатка с 5′ кон
клостеровирусов находятся ОРТ 1а и 1b, кодиру
ца коронавирусных мРНК по 2′ О положению
ющие компоненты репликазы (рис.
4)
рибозы (m7GpppAm) [29]. 2′ О метилирование
[7, 33, 34]. Трансляция этих генов, расположен
кепа позволяет клетке дискриминировать
ных в конфигурации 0/+1, приводит к синте
«свои» и «чужие» мРНК с помощью интерфе
зу pp1а и рр1ab. Следует заметить, что механизм
рон индуцируемых IFIT белков, и метилирова
+1 сдвига рамки считывания у вирусов встреча
ние рибозного кольца второго остатка с 5′ кон
ется достаточно редко, в отличие от -1 сдвига
ца вирусных РНК позволяет обойти этот меха
рамки считывания, который используется при
низм рестрикции [30].
экспрессии РНК полимераз ретровирусов, ко
Цитология репликации. После попадания в
ронавирусов, астровирусов животных и лютео
клетку коронавирусный нуклеокапсид доставля
вирусов растений [7, 36], а также некоторых эу
ется в эндосомы, где происходит освобождение
кариотических генов и транспозонов [37]. Гены
РНК от белка N [31], свободная РНК выходит в
структурных и вспомогательных белков нахо
цитоплазму и направляется к рибосомам. В ре
дятся в 3′ концевой части генома и экспрес
зультате трансляции синтезируются pp1а и pp1b,
сируются с помощью
3′ котерминальных
разрезаемые вирусными протеиназами с образо
сгРНК [34] (рис. 4).
ванием репликативных белков. Гидрофобные
Папаин подобная цистеиновая протеиназа
белки nsp3, nsp4 и nsp6 связываются с мембрана
PLP BYV автокаталитически выщепляет лидер
ми аппарата Гольджи и эндоплазматического ре
ный белок, гидролизуя пептидную связь
тикулума (ЭР) и формируют (очевидно, привле
Gly588/589 [7]. В геномах некоторых клостеро
кая клеточные белки партнеры) репликативные
вирусов домен лидерной протеиназы дуплици
компартменты - двухслойные мембранные ве
рован, и выщепляется два лидерных белка [8].
зикулы (DMV) и мультивезикулярные комплек
Лидерный белок PLP BYV влияет на амплифи
сы (MVC) [20, 21]. Репликативные белки и ви
кацию вирусной РНК и транспорт инфекции по
русная РНК связаны с мембранами DMV
проводящим тканям растения (дальний транс
и MVC; в этих структурах («вирусных фабриках»)
порт) [38, 39]. Помимо выщепления PLP, pp1а
происходит синтез вирусных РНК (антиге
подвергается дополнительному процессингу не
номной
(-) цепи, дочерних
(+) цепей и
известной протеиназной активностью, приво
сгРНК) [22]. Геномные и сгРНК экспортируются
дящему к появлению in vivo репликативных бел
в цитоплазму, где транслируются или служат
ков с молекулярной массой 63 kDa (с доме
матрицами для репликации во вновь образуемых
ном Mtr) и 100 kDa (с доменом HEL) [40].
вирусных фабриках. DMV коронавирусов MHV
Мажорный структурный белок (CP; рис. 4)
и SARS CoV 2 имеют каналы в цитоплазму,
формирует «тело» нитевидной вирусной части
ключевым компонентом которых является белок
цы, одевая 95% геномной РНК; минорный
nsp3, служащие для импорта молекул субстрата
структурный белок (CPm) образует «хвост»,
rNTP и экспорта вирусных мРНК [32].
включающий 5′ концевой фрагмент РНК [41,
42]. Формирование вирионов BYV невозможно
без участия HSP70h и p64, и оба этих белка вхо
КЛОСТЕРОВИРУСЫ
дят в состав зрелых частиц (в количестве одной
или нескольких копий) [43-45]. Белок HSP70h
Семейство Closteroviridae объединяет около
имеет консервативный N концевой домен
40 РНК содержащих вирусов растений [33, 34],
ATPазы (гомологичный эквивалентным доме
БИОХИМИЯ том 86 вып. 3 2021
302
АГРАНОВСКИЙ
Рис. 4. Структура генома и субгеномных РНК клостеровируса желтухи свеклы (BYV). +1 RFs - сигнал +1 рибосомально
го сдвига рамки считывания при трансляции ОРТ 1а и 1b. Стрелки и пунктирные линии обозначают сайты разреза
ния pp1а. Обозначения закодированных белковых доменов: PLP - папаин подобная протеиназа; Mtr - N7 гуанин метил
трансфераза; Mem - мембраносвязывающий домен; HEL - РНК хеликаза; POL - РНК полимераза; p6 - малый гидро
фобный белок; HSP70h - гомолог белков теплового шока семейства HSP70; p64 - минорный структурный белок; CPm -
минорный структурный белок; CP - мажорный структурный белок. Гены, недоступные для трансляции в каждом типе
мРНК, показаны затененными прямоугольниками
нам клеточных HSP70) и вариабельный С кон
Нокаут каждого из генов, кодирующих эти бел
цевой домен [46]. Было показано, что N конце
ки, блокирует распространение инфекции BYV
вой домен HSP70h обладает Mg зависимой
по растению [50]. Функции продуктов 3′ конце
ATPазной активностью in vitro, однако, в отли
вых генов (рис. 4) связаны с дальним транспор
чие от клеточных шаперонов семейства HSP70,
том инфекции (p20) и подавлением пост тран
не способен взаимодействовать с развернутыми
скрипционного умолкания генов (p21) [51].
белковыми цепями [47]. Помимо вирусных час
Репликативные белки. Клостеровирусы от
тиц, HSP70h связывается с плазмодесмами зара
носят к супергруппе альфа подобных вирусов,
женных клеток и выполняет функции белка
включающей (+)РНК содержащие вирусы жи
межклеточного транспорта [48, 49]. Функция
вотных (вирус Синдбис, вирус краснухи и вирус
транспорта также зависит от p6, p64, CP и CPm.
гепатита E) и растений (вирус табачной мозаи
БИОХИМИЯ том 86 вып. 3 2021
РНК ГЕНОМЫ НИДОВИРУСОВ И КЛОСТЕРОВИРУСОВ
303
ки, вирус мозаики костра и другие). При всем
мембраной) [59], напоминающих репликатив
несходстве биологических свойств, структуры
ные ультраструктуры нидовирусов и флавивиру
вирионов и строения РНК генома все альфа
сов [60]. DMV и MVC формируются из мемб
подобные вирусы содержат консервативные до
ран ЭР. Репликативные белки BYV - PLP, Mtr и
мены Mtr, РНК хеликазы (Hel) и РНК полиме
HEL - ассоциированы с этими структурами, что
разы (POL) в составе репликазы [52, 53]. До
подтверждает их связь с репликацией РНК
мен Mtr обладает активностями гуанилилтранс
[61, 62]. В поисках белков BYV, способных мо
феразы и N7 гуанин метилтрансферазы и осу
дифицировать клеточные мембраны, были про
ществляет кепирование вирусных мРНК. До
ведены эксперименты по кратковременной
мен HEL расплетает цепи РНК при репликации
(транзиентной) экспрессии фрагментов цент
за счет энергии гидролиза NTP. Домен POL от
рального района белка 1а, слитых с репортер
ветственен за синтез комплементарных цепей
ным белком GFP, в растениях Nicotiana ben)
РНК; у альфа подобных вирусов синтез РНК
thamiana [60, 63]. Фрагмент длиной 198 а.о.
инициируется de novo и не требует затравки [54].
(Mem; рис. 4), включающий консервативный
Сравнение вирусных (+)РНК геномов поз
гидрофобный домен с вероятной амфипатичес
воляет увидеть простую закономерность: чем
кой альфа спиралью, вызывал в клетках расте
больше размеры геномной РНК, тем протяжен
ний трансформацию ЭР вокруг ядра и образова
нее ген РНК репликазы; иными словами, боль
ние скоплений глобул диаметром ~2 мкм. Глобу
шой геном реплицируется более сложно устро
лы обладали подвижностью, зарегистрирован
енным репликативным комплексом [33]. При
ной с помощью конфокальной микроскопии, и
эволюции альфа подобных вирусов, очевидно,
ассоциировались с актиновыми филамента
происходили вставки кодирующей последова
ми [63]. Высказывалось предположение, что ре
тельности между доменами Mtr и HEL при сох
организация мембран ЭР под действием гидро
ранении длины спейсера между HEL и POL [33].
фобного сегмента pp1а является одним из эта
Репликативный комплекс клостеровирусов уст
пов создания репликативных ультраструктур у
роен сложнее, чем у родственных вирусов расте
клостеровирусов [60, 63].
ний, и включает, по крайней мере, пять бел
ков (PLP, Mtr, центральный домен 1а, HEL и
HEL POL) [34]. В белке 1а не были предсказаны
ОБЩИЕ ПУТИ ЭВОЛЮЦИИ
домены, эквивалентные экзонуклеазе и прайма
БОЛЬШИХ РНК ГЕНОМОВ
зе нидовирусов, и остается неизвестным суще
ствуют ли у клостеровирусов ферментативные
В процессе эволюции нидовирусы живот
активности, способствующие процессивности и
ных и клостеровирусы растений преодолели
точности синтеза РНК [51]. Репликация генома
барьер размера (+)РНК генома в 12 kb. При
клостеровирусов происходит по общему для
этом с помощью ряда эволюционных «изобрете
альфа подобных вирусов механизму - реплика
ний» были решены проблемы упаковки и реп
ция инициируется после узнавания репликазой
ликации. По остроумной гипотезе Godeny et al.
цис сигнала на 3′ конце (+)РНК, синтезируется
[64], предок современных Nidovirales, скорее
полноразмерная комплементарная (-) цепь, на
всего, имел икосаэдрический нуклеокапсид.
матрице которой синтезируются дочерние
При последующей дивергенции «малые нидови
(+) цепи. До сих пор не были получены экспе
русы» (геном 14-16 kb) сохранили такой тип
риментальные данные в пользу одного из двух
нуклеокапсида, а «большие нидовирусы» (ге
возможных вариантов транскрипции - синтеза
ном 26-41 kb) в результате рекомбинации при
сгРНК на матрице полноразмерной (-)РНК
обрели новый белок N, образующий спирально
или на матрицах антисубгеномных (-)РНК [54].
симметричный нуклеокапсид и позволяющий
По видимому, механизм прерывистой тран
упаковывать РНК значительно большего разме
скрипции не используется в клостеровирусных
ра. Родственные клостеровирусам альфа подоб
системах, поскольку сгРНК клостеровирусов не
ные вирусы растений - бромовирус мозаики
имеют общей 5′ лидерной последовательности,
костра и тобамовирус табачной мозаики - име
а районы субгеномных промоторов не несут об
ют икосаэдрические и палочковидные частицы
щих элементов, напоминающих TRS нидовиру
соответственно. Размер РНК, который возмож
сов [55-58].
но упаковать в такие частицы, существенно ог
Цитология репликации. Инфекция клостеро
раничен; создание в процессе эволюции сверх
вирусов в растениях сопровождается образова
гибких спирально симметричных частиц клос
нием DMV (везикул диаметром ~100 нм, окру
теровирусов, состоящих из нескольких белков,
женных двойной мембраной) и MVC (скопле
позволило решить проблему упаковки РНК ге
ния везикул, окруженных общей однослойной
нома размером до 19 kb [5].
БИОХИМИЯ том 86 вып. 3 2021
304
АГРАНОВСКИЙ
Мутации, вносимые собственными РНК
ся пределом размеров вирусной РНК, копиро
полимеразами [2] и, возможно, клеточными ре
вание которой не требует дополнительной ре
дактирующими ферментами (например, дезами
дактирующей активности.
назами) [65], являются основой изменчивости
Сравнение структуры и экспрессии геномов
РНК вирусов. Другой движущей силой вирус
коронавирусов и клостеровирусов выявляет
ной эволюции является рекомбинация РНК [66].
удивительное сходство между этими группа
Анализ геномов коронавирусов и клостеровиру
ми (рис. 1 и 4). Геномы нидовирусов и клостеро
сов позволяет выявить явные следы рекомбина
вирусов содержат 5′ проксимальные гены реп
ционных событий: захвата гетерологичных пос
ликазы, экспрессируемые с помощью рибосо
ледовательностей, дупликации и дивергенции
мального сдвига рамки считывания с образова
генов. Так, эволюция «больших нидовирусов»
нием крупных pp1а и pp1ab. Полипротеины
сопровождалась приобретением как новых фер
процессируются одной или двумя папаин по
ментативных активностей (нуклеотидилтранс
добными протеиназами, высвобождающими
феразы, праймазы, 3′ 5′ экзонуклеазы, эндонук
лидерные белки, а также сериновой MP (нидо
леазы) [9], так и структурных генов (белка нуклео
вирусы) или неидентифированной протеина
капсида и белка внешней мембраны, родствен
зой (клостеровирусы), в результате чего образу
ного гемагглютинину вируса гриппа) [67]. В ходе
ются зрелые белки с активностями метилтранс
эволюции клостеровирусов и коронавирусов
феразы, хеликазы, РНК полимеразы и мембра
происходила дупликация и последующая дивер
носвязывающими доменами. Гены вспомога
генция последовательностей, кодирующих ли
тельных и структурных белков транслируются с
дерную протеиназу PLP. В геномах клостерови
матриц 3′ котерминальных сгРНК (рис. 1 и 4).
русов ген структурного белка был, вероятно,
Это сходство не должно создавать впечатление,
дуплицирован несколько раз, в результате чего
что эти вирусы животных и растений произош
возникли CP, CPm (некоторые представители
ли от общего предка, имевшего сходным обра
семейства несут два CPm подобных гена) и
зом устроенный геном и набор механизмов
p64 (рис. 4) [34, 51]. Кроме того, при эволюции
экспрессии генов - напротив, клостеровирусы
клостеровирусов произошел захват хозяйского
и нидовирусы представляют эволюционно уда
гена HSP70 (за счет вероятной рекомбинации с
ленные друг от друга группы [1, 10]. Более
клеточной мРНК) и его приспособление к вы
уместным представляется допущение, что сход
полнению определенных функций при сборке
ные черты геномной организации и экспрессии
частиц и межклеточном транспорте [33, 51].
нидовирусов и коронавирусов возникли в ходе
Синтез больших РНК «обслуживается» бо
эволюции предков этих вирусов конвергентно,
лее сложно устроенными РНК репликазами, и
как следствие увеличения размеров РНК гено
существенная часть увеличений размера РНК
ма, сделавшего необходимым приобретение
генома приходится на репликативные гены [33].
сходного набора функций и способов экспрес
Коронавирусы обладают ферментативными ак
сии вирусных генов [5, 7, 34, 51].
тивностями, позволяющими повысить точность
и процессивность РНК полимеразы (праймаза
и экзонуклеаза). Геномы клостеровирусов не ко
Благодарности. Автор выражает признатель
дируют эквивалентных белков. Возможно, это
ность проф. А.Г. Соловьеву и проф. С.Ю. Моро
отражается в более высоком уровне точечных
зову за обсуждение и критические замечания по
мутаций у клостеровируса тристецы цитрусо
тексту настоящего обзора.
вых (CTV) [68] в сравнении с SARS CoV и дру
Конфликт интересов. Автор заявляет об от
гими коронавирусами [65]. Среди представите
сутствии конфликта интересов.
лей Nidovirales коронавирусы и ронивирусы (ге
Соблюдение этических норм. Настоящая
ном 26-41 kb) кодируют домен экзонуклеазы, а
статья не содержит описания каких либо иссле
мезонивирусы и артеривирусы (геном 14-20 kb)
дований с участием людей или животных в каче
лишены этого домена. Возможно, 20 kb являет
стве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Koonin, E. V., and Dolja, V. V. (1993) Evolution and taxon
3.
Drake, J. W. (1993) Rates of spontaneous mutation among
omy of positive strand RNA viruses: implications of compar
RNA viruses, Proc. Natl. Acad. Sci. USA, 90, 4171 4175.
ative analysis of amino acid sequences, Crit. Rev. Biochem.
4.
Holmes, E. C. (2003) Error thresholds and the con
Mol. Biol., 28, 375 430, doi: 10.3109/10409239309078440.
straints to RNA virus evolution, Trends Microbiol., 11, 543
2.
Steinhauer, D. A., and Holland, J. (1987) Rapid evolution
546.
of RNA viruses, Annu. Rev. Microbiol., 41, 409 431,
5.
Dolja, V. V., Karasev, A. V., and Koonin, E. V. (1994)
doi: 10.1146/annurev.mi.41.100187.002205.
Molecular biology and evolution of closteroviruses: sophis
БИОХИМИЯ том 86 вып. 3 2021
РНК ГЕНОМЫ НИДОВИРУСОВ И КЛОСТЕРОВИРУСОВ
305
ticated build up of large RNA genomes, Annu. Rev.
induce double membrane vesicles, MBio, 4, e00524
Phytopathol., 32, 261 285.
e00513.
6.
Saberi, A., Gulyaeva, A. A., Brubacher, J. L., Newmark,
21.
de Wilde, A. H., Snijder, E. J., Kikkert, M., and van
P. A., and Gorbalenya, A. E. (2018) A planarian nidovirus
Hemert, M. J. (2018) Host factors in coronavirus replica
expands the limits of RNA genome size, PLoS Pathog., 14,
tion, Curr. Top. Microbiol. Immunol.,
419,
1 42,
e1007314, doi: 10.1371/journal.ppat.1007314.
doi: 10.1007/82_2017_25.
7.
Agranovsky, A. A., Koonin, E. V., Boyko, V. P., Maiss, E.,
22.
Knoops, K., Kikkert, M., Worm, S. H., Zevenhoven
Froetschl, R., Lunina, N. A., and Atabekov, J. G. (1994)
Dobbe, J. C., van der Meer, Y., et al. (2008) SARS corona
Beet yellows closterovirus: complete genome structure and
virus replication is supported by a reticulovesicular network
identification of a leader papain like thiol protease,
of modified endoplasmic reticulum, PLoS Biol., 6, e226,
Virology, 198, 311 324, doi: 10.1006/viro.1994.1034.
doi: 10.1371/journal.pbio.0060226.
8.
Karasev, A. V., Boyko, V. P., Gowda, S., Nikolaeva, O. V.,
23.
Te Velthuis, A. J., van den Worm, S. H., and Snijder, E. J.
Hilf, M. E., et al. (1995) Complete sequence of the citrus
(2012) The SARS coronavirus nsp7+nsp8 complex is a
tristeza virus RNA genome, Virology, 208, 511520,
unique multimeric RNA polymerase capable of both de
doi: 10.1006/viro.1995.1182.
novo initiation and primer extension, Nucleic Acids Res,
9.
Enjuanes, L., Gorbalenya, A. E., de Groot, R. J., Cowley,
40, 1737 1747, doi: 10.1093/nar/gkr893.
J. A., Ziebuhr, J., and Snijder, E. J. (2008) Nidovirales, in
24.
Kirchdoerfer, R. N., and Ward, A. B. (2019) Structure of
Encyclopedia of Virology (Mahy, B. W. J., and Van
the SARS CoV nsp12 polymerase bound to nsp7 and nsp8
Regenmortel, M. H. V., eds) Oxford, Elsevier, pp. 419 430.
co factors, Nat. Commun., 10, 2342. doi: 10.1038/s41467
10.
Koonin, E. V., Dolja, V. V., Krupovic, M., Varsani, A.,
019 10280 3.
Wolf, Y. I., et al. (2020) Global organization and proposed
25.
Bouvet, M., Imbert, I., Subissi, L., Gluais, L., Canard, B.,
megataxonomy of the virus world, Microbiol. Mol. Biol.
and Decroly, E. (2012) RNA 3′ end mismatch excision by
Rev., 84, e00061 19, doi: 10.1128/MMBR.00061 19.
the severe acute respiratory syndrome coronavirus non
11.
Sawicki, S. G., and Sawicki, D. L. (2005) Coronavirus
structural protein nsp10/nsp14 exoribonuclease complex,
transcription: a perspective, Curr. Top. Microbiol.
Proc. Natl. Acad. Sci. USA, 109, 9372 9377, doi: 10.1073/
Immunol., 287, 31 55, doi: 10.1007/3 540 26765 4_2.
pnas.1201130109.
12.
Sola, I., Almazán, F., Zúñiga, S., and Enjuanes, L. (2015)
26.
Subissi, L., Posthuma, C. C., Collet, A., Zevenhoven
Continuous and discontinuous RNA synthesis in coron
Dobbe, J. C., Gorbalenya, A. E., et al. (2014) One severe
aviruses,
Annu.
Rev.
Virol.,
2,
265288,
acute respiratory syndrome coronavirus protein complex
doi: 10.1146/annurev virology 100114 055218.
integrates processive RNA polymerase and exonuclease
13.
Zúñiga, S., Cruz, J. L., Sola, I., Mateos Gomez, P. A.,
activities, Proc. Natl. Acad. Sci. USA, 111, E3900 9,
Palacio, L., and Enjuanes, L. (2010) Coronavirus nucleo
doi: 10.1073/pnas.1323705111.
capsid protein facilitates template switching and is required
27.
Jin, X., Chen, Y., Sun, Y., Zeng, C., Wang, Y., et al. (2013)
for efficient transcription, J. Virol., 84, 2169 2175.
Characterization of the guanine N7 methyltransferase
14.
Wu, C. H., Chen, P. J., and Yeh, S. H.
(2014)
activity of coronavirus nsp14 on nucleotide GTP, Virus
Nucleocapsid phosphorylation and RNA helicase DDX1
Res., 176, 45 52, doi: 10.1016/j.virusres.2013.05.001.
recruitment enables coronavirus transition from discontin
28.
Ivanov, K. A., Hertzig, T., Rozanov, M., Bayer, S.,
uous to continuous transcription, Cell Host Microbe, 16,
Thiel, V., Gorbalenya, A. E., and Ziebuhr, J. (2004) Major
462 72.
genetic marker of nidoviruses encodes a replicative endori
15.
Terada, Y., Kawachi, K., Matsuura, Y., and Kamitani, W.
bonuclease, Proc. Natl. Acad. Sci. USA, 101,
12694
(2017) MERS coronavirus nsp1 participates in an efficient
12699, doi: 10.1073/pnas.0403127101.
propagation through a specific interaction with viral RNA,
29.
Chen, Y., Cai, H., Pan, J., Xiang, N., Tien, P., Ahola, T.,
Virology, 511, 95 105, doi: 10.1016/j.virol.2017.08.026.
and Guo, D. (2009) Functional screen reveals SARS coro
16.
Lokugamage, K. G., Narayanan, K., Huang, C., and
navirus nonstructural protein nsp14 as a novel cap N7
Makino, S. (2012) Severe acute respiratory syndrome
methyltransferase, Proc. Natl. Acad. Sci. USA, 106, 3484
coronavirus protein nsp1 is a novel eukaryotic translation
3489.
inhibitor that represses multiple steps of translation initia
30.
Daffis, S., Szretter, K. J., Schriewer, J., Li, J., Youn, S.,
tion, J. Virol., 86, 13598 13608, doi: 10.1128/JVI.01958
et al. (2010) 2′ O methylation of the viral mRNA cap
12.
evades host restriction by IFIT family members, Nature,
17.
Nakagawa, K., Narayanan, K., Wada, M., Popov, V. L.,
468, 452 456, doi: 10.1038/nature09489.
Cajimat, M., Baric, R. S., and Makino, S. (2018) The
31.
Kalicharran, K., Mohandas, D., Wilson, G., and Dales, S.
endonucleolytic RNA cleavage function of nsp1 of middle
(1996) Regulation of the initiation of coronavirus JHM
east respiratory syndrome coronavirus promotes the pro
infection in primary oligodendrocytes and L 2 fibroblasts,
duction of infectious virus particles in specific human cell
Virology, 225, 33 43, doi: 10.1006/viro.1996.0572.
lines, J. Virol., 92, e01157 18, doi: 10.1128/JVI.01157 18.
32.
Wolff, G., Ronald, W., Limpens R., Zevenhoven Dobbe,
18.
Graham, R. L., Sims, A. C., Brockway, S. M., Baric, R. S.,
J. C., Laugks, U., et al. (2020) A molecular pore spans the
and Denison, M. R. (2005) The nsp2 replicase proteins of
double membrane of the coronavirus replication organelle,
murine hepatitis virus and severe acute respiratory syn
Science, 369, 1395 1398, doi: 10.1126/science.abd3629.
drome coronavirus are dispensable for viral replication,
33.
Agranovsky, A. A. (1996) Principles of molecular organiza
J. Virol., 79, 13399 13411, doi: 10.1128/JVI.79.21.13399
tion, expression and evolution of closteroviruses: over the
13411.2005.
barriers, Adv. Virus Res., 47, 119 158.
19.
Sawicki, S. G., Sawicki, D. L., Younker, D., Meyer, Y.,
34.
Agranovsky A. A. (2016) Closteroviruses: molecular biolo
Thiel, V., Stokes, H., and Siddell, S. G. (2005) Functional
gy, evolution and interactions with cells, in Plant Viruses:
and genetic analysis of coronavirus replicase transcriptase
Evolution and Management (Gaur, R. K., Petrova, N., and
proteins, PLoS Pathog., 1, e39, doi: 10.1371/journal.ppat.
Stoyanova, M. I., eds) Springer Science+Business Media,
0010039.
Singapore, Chapt. 14, pp. 231 252, doi: 10.1007/978 981
20.
Angelini, M. M., Akhlaghpour, M., Neuman, B. W., and
10 1406 2_14.
Buchmeier, M. J. (2013) Severe acute respiratory syn
35.
Agranovsky, A. A., Boyko, V. P., Karasev, A. V., Lunina,
drome coronavirus nonstructural proteins 3, 4, and
6
N. A., Koonin, E. V., and Dolja, V. V. (1991) Nucleotide
2 БИОХИМИЯ том 86 вып. 3 2021
306
АГРАНОВСКИЙ
sequence of the 3′ terminal half of beet yellows clos
54.
Miller, W. A., and Koev, G. (2000) Synthesis of subgenom
terovirus RNA genome: unique arrangement of eight virus
ic RNAs by positive strand RNA viruses, Virology, 273,
genes, J. Gen. Virol., 72, 15 23.
1 8.
36.
Firth, A. E., Brierley, I. (2012) Non canonical translation
55.
Agranovsky, A. A., Koenig, R., Maiss, E., Boyko, V. P.,
in RNA viruses, J. Gen. Virol.,
93,
13851409,
Casper, R., and Atabekov, J. G. (1994) Expression of the
doi: 10.1099/vir.0.042499 0.
beet yellows closterovirus capsid protein and p24, a capsid
37.
Giedroc, D. P., and Cornish, P. V. (2009) Frameshifting
protein homologue, in vitro and in vivo, J. Gen. Virol., 75,
RNA pseudoknots: structure and mechanism, Virus Res.,
1431 1439.
139, 193 208.
56.
Karasev, A. V., Hilf, M. E., Garnsey, S. M., and Dawson,
38.
Peremyslov, V. V., Hagiwara, Y., and Dolja, V. V. (1998)
W. O. (1997) Transcriptional strategy of closteroviruses:
Genes required for replication of the 15.5 kilobase RNA
mapping the 5′ termini of the citrus tristeza virus subge
genome of a plant closterovirus, J. Virol., 72, 5870 5876.
nomic RNAs, J. Virol., 71, 6233 6236.
39.
Peng, C. W., Napuli, A. J., and Dolja, V.V. (2003) Leader
57.
Peremyslov, V. V., and Dolja, V. V. (2002) Identification of
proteinases of beet yellows virus functions in long distance
the subgenomic mRNAs that encode 6 kDa movement
transport, J. Virol., 77, 2843 2849.
protein and Hsp70 homolog of beet yellows virus, Virology,
40.
Erokhina, T. N., Zinovkin, R. A., Vitushkina, M. V.,
295, 299 306.
Jelkmann, W., and Agranovsky, A. A. (2000) Detection of
58.
Vitushkina, M. V., Rogozin, I. B., Jelkmann, W., Koonin,
beet yellows closterovirus methyltransferase like and heli
E. V., and Agranovsky, A. A. (2007) Completion of the
case like proteins in vivo using monoclonal antibodies,
mapping of transcription start sites for the five gene block
J. Gen. Virol., 81, 597 603.
subgenomic RNAs of beet yellows closterovirus and identi
41.
Agranovsky, A. A., Lesemann, D. E., Maiss, E., Hull, R.
fication of putative subgenomic promoters, Virus Res., 128,
and Atabekov, J. G. (1995) “Rattlesnake” structure of a fil
153 158.
amentous plant RNA virus built of two capsid proteins,
59.
Cronshaw, J., Hoefert, L., and Esau, K.
(1966)
Proc. Natl. Acad. Sci. USA, 92, 2470 2473.
Ultrastructural features of beta leaves infected with beet
42.
Zinovkin, R. A., Jelkmann, W., and Agranovsky, A. A.
yellows virus, J. Cell. Biol., 31, 429 443.
(1999) The minor coat protein of beet yellows closterovirus
60.
Gushchin, V. A., Solovyev, A. G., Erokhina, T. N.,
encapsidates the 5′ terminus of RNA in virions, J. Gen.
Morozov, S. Y., and Agranovsky, A. A. (2013) Beet yellows
Virol., 80, 269 272.
virus replicase and replicative compartments: parallels with
43.
Napuli, A. J., Alzhanova, D. V., Doneanu, C. E., Barofsky,
other RNA viruses, Front. Microbiol., 4, 38, doi: 10.3389/
D. F., Koonin, E. V., and Dolja, V. V. (2003) The 64 kDa
fmicb.2013.00038.
capsid protein homolog of beet yellows virus is required for
61.
Erokhina, T. N., Vitushkina, M. V., Zinovkin, R. A.,
assembly of virion tails, J. Virol., 77, 2377 2384.
Lesemann, D. E., Jelkmann, W., Koonin, E. V., and
44.
Napuli, A. J., Falk, B. W., and Dolja, V. V.
(2000).
Agranovsky, A. A. (2001) Ultrastructural localisation and
Interaction between HSP70 homolog and filamentous viri
epitope mapping of beet yellows closterovirus methyltrans
ons of the beet yellows virus, Virology, 274, 232 239.
ferase like and helicase like proteins, J. Gen. Virol., 82,
45.
Alzhanova, D. V., Napuli, A., Creamer, R., and Dolja, V. V.
1983 1994.
(2001) Cell to cell movement and assembly of a plant clos
62.
Zinovkin, R. A., Erokhina, T. N., Lesemann, D. E.,
terovirus: roles for the capsid proteins and Hsp70 homolog,
Jelkmann, W., and Agranovsky, A. A. (2003) Processing
EMBO J., 20, 6997 7007.
and subcellular localization of the leader papain like pro
46.
Agranovsky, A. A., Boyko, V. P., Karasev, A. V., Koonin,
teinase of beet yellows closterovirus, J. Gen. Virol., 84,
E. V., and Dolja, V. V. (1991) The putative 65K protein of
2265 2270.
beet yellows closterovirus is a homologue of HSP70 heat
63.
Gushchin V. A., Karlin, D. G., Makhotenko, A. V.,
shock proteins, J. Mol. Biol., 217, 603 610.
Khromov, A. V., Erokhina T. N., et al. (2017) A conserved
47.
Agranovsky, A. A., Folimonova, S. Y., Folimonov, A. S.,
region in the Closterovirus 1a polyprotein drives extensive
Denisenko, O. N., and Zinovkin, R. A. (1997) The beet
remodeling of endoplasmic reticulum membranes and
yellows closterovirus p65 homologue of HSP70 chaperones
induces motile globules in Nicotiana benthamiana cells,
has ATPase activity associated with its conserved N termi
Virology, 506, 106113, doi: 10.1016/j.virol.2016.12.
nal domain but does not interact with unfolded protein
006.
chains, J. Gen. Virol., 78, 535 542.
64.
Godeny, E. K., Chen, L., Kumar, S., Methven, S. L.,
48.
Medina, V., Peremyslov, V. V., Hagiwara, Y., and Dolja, V. V.
Koonin, E. V., and Brinton, M. A. (1993) Complete
(1999) Subcellular localization of the HSP70 homolog
genome sequence and phylogenie analysis of the lactate
encoded by beet yellows closterovirus, Virology, 260, 173 181.
dehydrogenase elevating virus (LDV), Virology, 194,
49.
Agranovsky, A. A., Folimonov, A. S., Folimonova, S. Y.,
585 96.
Morozov, S. Y., Schiemann, J., Lesemann, D. E., and
65.
Simmonds, P. (2020) Rampant C/U hypermutation in the
Atabekov, J. G. (1998) Beet yellows closterovirus HSP70
genomes of SARS CoV 2 and other coronaviruses: causes
like protein mediates the cell to cell movement of a
and consequences for their short and long term evolu
potexvirus transport deficient mutant and a hordeivirus
tionary trajectories, mSphere, 5, e00408 20, doi: 10.1128/
based chimeric virus, J. Gen. Virol., 79, 889 895.
mSphere.00408 20.
50.
Alzhanova, D. V., Hagiwara, Y., Peremyslov, V. V., and
66.
Bentley, K., and Evans, D. J. (2018) Mechanisms and con
Dolja, V. V. (2000) Genetic analysis of the cell to cell move
sequences of positive strand RNA virus recombination,
ment of beet yellows closterovirus, Virology, 268, 192 200.
J. Gen. Virol., 99, 1345 1356, doi: 10.1099/jgv.0.001142.
51.
Dolja, V. V., Kreuze, J. F., and Valkonen, J. P. (2006)
67.
Zeng, Q., Langereis, M. A., van Vliet A. L., Huizinga,
Comparative and functional genomics of closteroviruses,
E. G., and de Groot, R. J. (2008) Structure of coronavirus
Virus Res., 117, 38 51.
hemagglutinin esterase offers insight into corona and
52.
Goldbach, R., Le Gall, O., and Wellink, J. (1991) Alpha
influenza virus evolution, Proc. Natl. Acad. Sci. USA, 105,
like viruses of plants, Semin. Virol., 2,19 25.
9065 9069, doi: 10.1073/pnas.0800502105.
53.
Buck, K. W. (1996) Comparison of the replication of posi
68.
Harper, S. J. (2013) Citrus tristeza virus: evolution of com
tive stranded RNA viruses of plants and animals, Adv. Virus
plex and varied genotypic groups, Front. Microbiol., 4,
Res., 47, 159 251, doi: 10.1016/s0065 3527(08)60736 8.
9310, doi: 3389/fmicb.2013.00093.
БИОХИМИЯ том 86 вып. 3 2021
РНК ГЕНОМЫ НИДОВИРУСОВ И КЛОСТЕРОВИРУСОВ
307
STRUCTURE AND EXPRESSION OF LARGE POSITIVE SENSE
RNA GENOMES OF EUKARYOTIC VIRUSES
Mini Review
A. A. Agranovsky
Department of Virology, Faculty of Biology, Lomonosov Moscow State University,
119234 Moscow, Russia; E)mail: etdonas2@gmail.com
Viral positive sense RNA genomes evolve rapidly due to the high mutation rates during replication and RNA recom
bination, which allowing the viruses to acquire and modify genes for their adaptation. The size of RNA genome is lim
ited by several factors, including low fidelity of RNA polymerases and packaging constraints. However, the 12 kb size
limit is exceeded in the two groups of eukaryotic (+)RNA viruses - animal nidoviruses and plant closteroviruses.
These virus groups have several traits in common. Their genomes contain 5′ proximal genes that are expressed via
ribosomal frameshifting and encode one or two papain like protease domains, membrane binding domain(s),
methyltransferase, RNA helicase, and RNA polymerase. In addition, some nidoviruses (i.e., coronaviruses) contain
replication associated domains, such as proofreading exonuclease, putative primase, nucleotidyltransferase, and
endonuclease. In both nidoviruses and closteroviruses, the 3′ terminal part of the genome contains genes for struc
tural and accessory proteins expressed via a nested set of coterminal subgenomic RNAs. Coronaviruses and clos
teroviruses have evolved to form flexuous helically symmetrical nucleocapsids as a mean to resolve packaging con
straints. Since phylogenetic reconstructions of the RNA polymerase domains indicate only a marginal relationship
between the nidoviruses and closteroviruses, their similar properties likely have evolved convergently, along with the
increase in the genome size.
Keywords: viral positive sense RNA genomes, nidoviruses, SARS CoV, closteroviruses, evolution, gene expression
БИОХИМИЯ том 86 вып. 3 2021
2*