БИОХИМИЯ, 2021, том 86, вып. 3, с. 374 - 394
УДК 577.2
МИКРОБНЫЙ АРСЕНАЛ ПРОТИВОВИРУСНОЙ ЗАЩИТЫ. ГЛАВА I
Обзор
© 2021
А.Б. Исаев1*, О.С. Мушарова1,2, К.В. Северинов1,3*
1 Сколковский институт науки и технологий, 143028 Москва, Россия; электронная почта: tcft18@gmail.com
2 Институт молекулярной генетики РАН, 123182 Москва, Россия
3 Waksman Institute of Microbiology, Piscataway, NJ 08854, USA; E%mail: severik@waksman.rutgers.edu
Поступила в редакцию 10.08.2020
После доработки 18.08.2020
Принята к публикации 12.09.2020
Бактериофаги или фаги представляют собой вирусы, которые инфицируют бактериальные клетки (в рамках
этого обзора мы также рассмотрим вирусы, которые инфицируют архей). Постоянная угроза заражения фа!
гами является одной из основных движущих сил эволюции бактериальных геномов. Чтобы противостоять
инфекции, бактерии выработали многочисленные защитные стратегии, позволяющие избежать распозна!
вания фагами или прямо препятствующие размножению фагов внутри клетки. Исследования бактериофа!
гов и бактериальных систем защиты были исторически тесно переплетены с развитием методов классичес!
кой молекулярной биологии и генной инженерии. В настоящее время благодаря распространению фаговой
терапии, широкому применению технологий CRISPR!Cas и развитию биоинформатических подходов, ко!
торые облегчают задачу обнаружения новых систем, исследования в области биологии фагов переживают
возрождение. В настоящем обзоре описываются различные стратегии, используемые микробами для того,
чтобы противостоять фаговой инфекции. Особое внимание уделено новым защитным системам, открытым
в последние годы. Первая глава обзора посвящена защите связанной с поверхностью клетки, роли малых
молекул, а также системам врожденного иммунитета, зависящим от модификации ДНК.
КЛЮЧЕВЫЕ СЛОВА: бактериофаги, взаимодействия бактериофагов с клетками!хозяевами, антивирусная
защита, иммунные системы, CRISPR!Cas, рестрикция!модификация, BREX, DISARM, фосфоротиоат, сис!
темы Dnd.
DOI: 10.31857/S0320972521030076
ВВЕДЕНИЕ
индуцируемый фагами лизис клеток обеспечи!
вает поступление питательных веществ в пище!
Фаги встречаются в любой природной среде
вые цепи [7]. Фаги значительно ускоряют лате!
и считаются самыми распространенными био!
ральный перенос генов, и медицински значи!
логическими существами на Земле [1-3]. Рас!
мые бактериальные признаки часто ассоцииру!
четное количество фаговых частиц может дости!
ются с профагами [8]. Гены клетки!хозяина мо!
гать примерно 1010/литр в морской или пресной
гут быть включены в вирусный геном или упа!
воде и 109/грамм - в почве. Считается, что коли!
кованы в вирусный капсид при генерации
чество фагов превышает количество их микроб!
трансдуцирующих частиц [9]. Кроме того, бак!
ных хозяев в 10 или более раз [4, 5]. Фаги играют
терии могут продуцировать фагоподобные час!
важную экологическую роль, контролируя раз!
тицы (Gene Transferring Agents), несущие ДНК
мер и разнообразие микробных популяций [6], а
хозяина [10]. Распространение плазмид также
Принятые сокращения: МТаза - метилтрансфераза (MTase); ЭР - эндонуклеаза рестрикции, рестриктаза (REase,
Restriction Endonuclease); BREX - система исключения бактериофагов (Bacteriophage Exclusion); CRISPR!Cas - класте!
ризованные регулярно прерываемые повторы и ассоциированные с ними белки (Clustered Regularly Interspaced Repeats
and CRISPR!associated proteins); DISARM - система защитных островков, ассоциированная с рестрикцией!модификаци!
ей (Defence Island System Associated with Restriction!Modification); Dnd - фенотип, ассоциированный с деградацией
ДНК (DNA Degradation phenotype); DPD - 7!деазапурин в ДНК; ECM - внеклеточный матрикс (Extracellular Matrix);
LPS - липополисахарид; OMV - везикула на основе внешней мембраны (Outer Membrane Vesicle); Pgl - система ограни!
чения роста бактериофагов (Phage Growth Limitation); PLD - фосфолипаза D; PT - фосфоротиоат (phosphorothioate);
pVip - прокариотический белок вайперин (prokaryotic Viperin); QS - определение кворума (Quorum Sensing); RBP - ре!
цептор!связывающий белок (Receptor Binding Protein); R!M - рестрикция!модификация (Restriction!Modification);
SAM - S!аденозилметионин (S!Adenosyl Methionine); Sie - исключение суперинфекции (Superinfection Exclusion); TA -
токсин!антитоксин; TRD - домен, распознающий мишень (Target Recognition Domain).
* Адресат для корреспонденции.
374
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
375
облегчается при фаг!индуцированном лизисе
которые такие кластеры были исследованы экс!
клеток [11].
периментально. Это привело к валидации де!
Теоретические выкладки и математические
сятка новых типов противовирусных сис!
модели предполагают, что в любой системе реп!
тем [21, 22]. Несмотря на то что в последние го!
ликаторов неизбежно возникают паразитические
ды наше понимание обилия типов защитных
или эгоистичные элементы, поэтому для дости!
систем значительно расширилось, можно ожи!
жения их стабильного сосуществования необхо!
дать, что была раскрыта всего лишь небольшая
димы стратегии противодействия [12-14]. Таким
часть их реального разнообразия, поскольку
образом, наличие фагов и защитных систем
подавляющая часть геномных данных рассмат!
клеток!хозяев можно рассматривать как фунда!
ривается как «темная материя», а защитные
ментальное свойство прокариотических клеток.
островки, состоящие исключительно из генов с
Миллиарды лет совместной эволюции привели
неизвестной функцией, не обнаруживаются с
к развитию широкого круга стратегий нападе!
помощью современных методов [23]. Дальней!
ния и защиты, используемых вирусами и их хо!
шее совершенствование алгоритмов, использо!
зяевами. Гены, ассоциированные с защитой
вание более точной классификации белковых
клеток от фагов, могут составлять до ~10% ге!
доменов или применение методов глубокого
нома бактерии [15]. Традиционно, открытие
обучения, уже доказавших свою полезность в
систем защиты клеток от фагов было связано с
прогнозировании функций генов
[24-26],
отбором устойчивых к действию фага штаммов
должно способствовать дальнейшему продви!
и описанием их специфических свойств. Не!
жению в этой области. В то же время обнаруже!
давнее увеличение доступности геномных дан!
ние новых систем сопряжено с задачей характе!
ных и применение биоинформатических под!
ризации их биохимических функций, и в насто!
ходов значительно расширили эту область нау!
ящее время механизмы противовирусной защи!
ки и позволили провести систематическое
ты для большинства новых систем остаются не!
предсказание новых кластеров генов защиты от
выясненными.
фагов. Популярный подход «вины по ассоциа!
Общая схема защитных стратегий, действу!
ции» (guilt by association) основывается на фак!
ющих на различных стадиях жизненного цикла
те, что функционально связанные гены часто
вируса, представлена на рис. 1. Устойчивость
совместно локализованы в геноме [16]. Исполь!
микроорганизмов к действию фагов может быть
зуя ген с известной функцией в качестве «при!
связана с активностью специфических иммун!
манки», можно оценить вероятность обнаруже!
ных систем, основной функцией которых явля!
ния соседних генов вблизи от «приманки» в
ется ингибирование размножения чужеродного
различных геномах и предсказать их возмож!
генетического материала, или с мутациями и
ные функциональные связи [17]. Применение
фазовыми вариациями в генах хозяина, которые
этого подхода к известным системам защиты от
необходимы для продуктивной вирусной ин!
фагов позволило ввести важное понятие защит!
фекции. Устойчивость к действию фагов также
ных островков - геномных локусов, содержа!
может быть связана с малыми молекулами [27]
щих кластеры генов противовирусной защи!
или с активностью мобильных генетических
ты [18]. Отдельный геном может содержать нес!
элементов, препятствующих вирусной инфек!
колько защитных островков. Часто они ассоции!
ции (таких как индуцированные фагами хромо!
рованы с мобильными генетическими элемен!
сомные островки (PICI, phage!inducible chromo!
тами, которые вносят важный вклад в горизон!
somal islands) и индуцированные фагами эле!
тальный перенос генов защитных систем [19,
менты подобные хромосомным островкам
20]. Поскольку ~2/3 генов, обнаруженных в за!
(PLE, phage!inducible chromosomal island!like
щитных островках, не были связаны с извест!
element)), которые в некотором смысле можно
ными системами защиты, было предсказано су!
рассматривать как паразиты паразитов [28, 29].
ществование множества новых типов противо!
Иммунные системы часто полагаются на рас!
вирусных систем [18]. Недавно в результате
познавание специфических сайтов в чужерод!
проведения систематического анализа встреча!
ной нуклеиновой кислоте или иным образом
емости всех pfam белковых доменов в островках
чувствуют фаговую инфекцию, чтобы иниции!
защиты в микробном пангеноме были предска!
ровать ингибирующий ответ. Чтобы избежать
заны примерно 300 семейств генов, которые
токсического воздействия, связанного с несвое!
преимущественно встречаются в ассоциации с
временной активацией, используются различ!
известными защитными генами. Среди них ге!
ные механизмы дискриминации собственных и
ны, которые имели тенденцию образовывать
чужеродных молекул. Защитные системы мож!
консервативные кластеры, были предположены
но подразделить на врожденный иммунитет
в качестве кандидатных защитных систем, и не!
(включая различные типы рестрикции!модифи!
БИОХИМИЯ том 86 вып. 3 2021
376
ИСАЕВ и др.
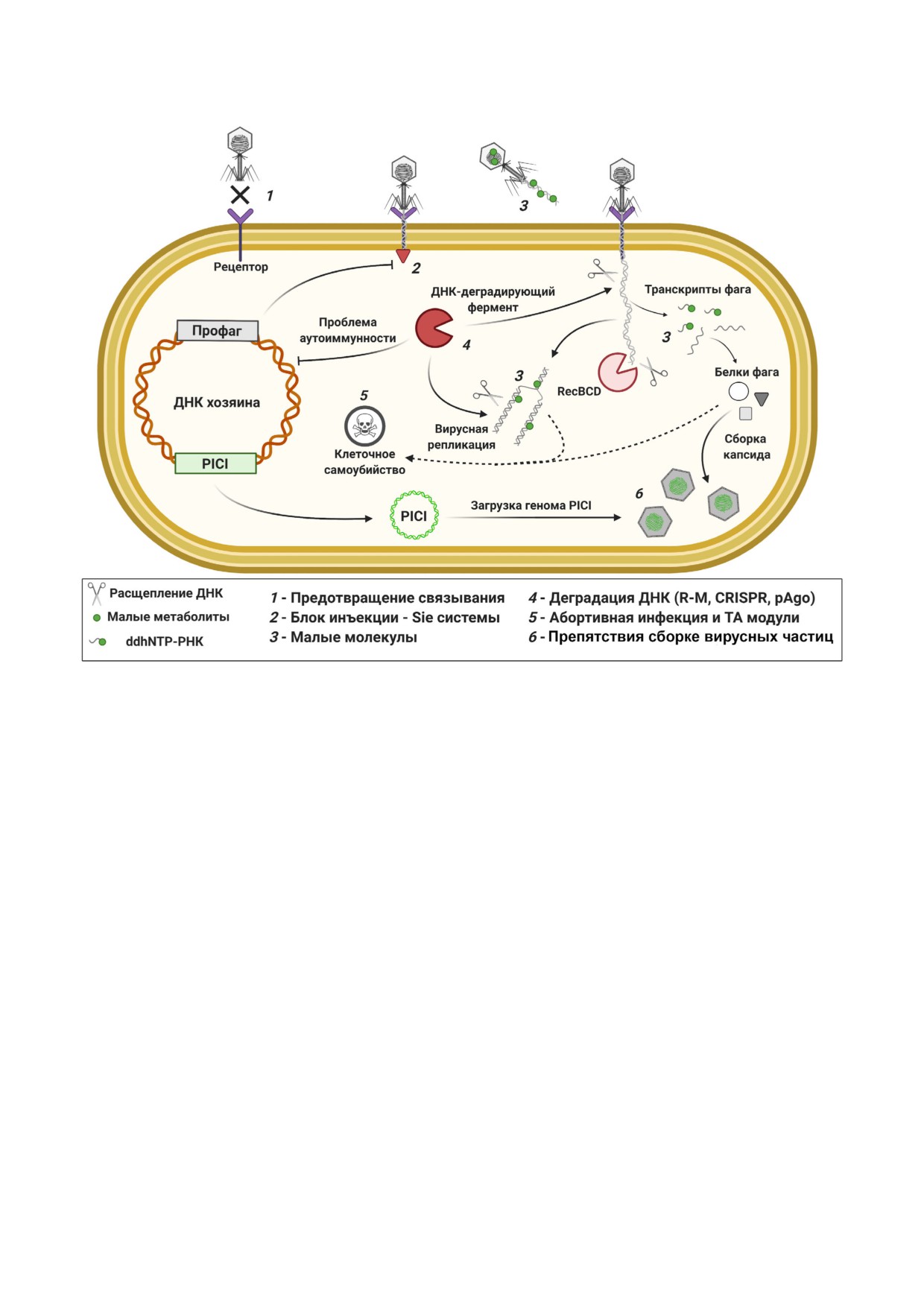
Рис. 1. Общая схема стратегий противовирусной защиты бактерий, действующих на различных стадиях жизненного
цикла вирусов. (С цветными вариантами рис. 1-6 можно ознакомиться в электронной версии статьи на сайте:
кации (R!M), систему исключения бактериофа!
случаев используют аппарат бактериальной
гов (BREX), систему защитных островков, ассо!
клетки для собственной транскрипции и транс!
циированных с рестрикцией!модификаци!
ляции [36] и часто полагаются на белки клетки!
ей (DISARM), токсин!антитоксин (TA), абор!
хозяина в качестве кофакторов, как в случае тио!
тивную инфекцию (Abi) и множество менее изу!
редоксина, который необходим для активности
ченных систем) и CRISPR!Cas!опосредован!
ДНК!полимеразы фага T7 [37]. Во многих ис!
ный адаптивный иммунитет (кластеризованные
следованиях была показана важность генов
регулярно прерываемые повторы и ассоцииро!
клетки!хозяина для эффективной вирусной ин!
ванные с ними белки). Онлайн базы данных,
фекции, включая работы с KEIO!коллекцией
посвященные прокариотическим иммунным
нокаутов отдельных генов Escherichia coli, спе!
системам, включают REBASE - собрание извест!
цифическим ингибированием конкретных ге!
ных систем R!M [30], TASmania, специализиру!
нов при помощи dCas9 или мутагенеза путем
ющуюся на системах TA [31], CRISPRminer и
вставки транспозонов [38-44]. Мутации в несу!
CRISPRCasdb для систем CRISPR!Cas [32, 33] и
щественных генах, необходимых для размноже!
PADS, содержащую аннотации генов, ассоции!
ния фагов, являются широко распространен!
рованных с различными типами защиты [34].
ным способом приобретения устойчивости,
Таксономическое распределение белковых до!
впервые описанным в классических экспери!
менов, связанных с противовирусной защитой,
ментах Luria и Delbruck [45].
также можно посмотреть в AnnoTree [35].
Практически для каждой известной страте!
Поскольку геномы фагов кодируют лишь
гии микробной защиты фаги выработали сред!
ограниченное число генов, они в большинстве ства преодоления, и описание кодируемых фа!
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
377
гами ингибиторов новых защитных систем оста!
ПРОСТЕЙШИЙ ПУТЬ ЗАЩИТЫ -
ется лишь вопросом времени [25, 26, 46]. Было
ОСТАТЬСЯ НЕЗАМЕЧЕННЫМ
высказано предположение, что, подобно защит!
ным островкам, анти!рестрикционные гены
Фаговая инфекция инициируется при рас!
имеют тенденцию образовывать кластеры в ге!
познавании специфических рецепторов на по!
номах фагов или в мобильных генетических эле!
верхности клетки фаговым рецептор!связываю!
ментах. Существование таких островков «про!
щим белком (RBP). В качестве рецепторов бак!
тиво!защиты» должно способствовать откры!
териофагов могут выступать различные типы
тию новых ингибиторов защиты хозяина [47].
молекул, которые располагаются на поверхнос!
Описание вирусных стратегий противодействия
ти клеток, в том числе пили и жгутики, белки,
бактериальной защите выходит за рамки насто!
липополисахариды (LPS) или углеводы. Взаи!
ящего текста, и с этой темой можно ознако!
модействие между RBP фага и рецептором клет!
миться в других обзорах [48-50].
ки!хозяина может рассматриваться как лимити!
Первая глава настоящего обзора будет пос!
рующая стадия инфекционного процесса, по
вящена микробным стратегиям, позволяющим
крайней мере, с точки зрения определения
избежать распознавания фагами, механизмам
спектра подходящих для инфицирования кле!
врожденного иммунитета, блокирующим ран!
ток!хозяев [51-53]. Для того, чтобы остаться не!
ние стадии инфекции, и системам, которые по!
замеченными для фагов, прокариоты использу!
лагаются на модификацию ДНК с целью дис!
ют маскировку рецепторов, их модификации и
криминации собственного и чужеродного гено!
мутации или продуцируют везикулы!ловуш!
мов. Вторая глава будет посвящена адаптивным
ки (рис. 2).
системам иммунитета и защитным механизмам,
Роль внеклеточного матрикса (ECM) и вези9
активирующимся на поздних стадиях инфек!
кул внешней мембраны (OMV). Многие бактерии
ции.
способны выделять высокомолекулярные поли!
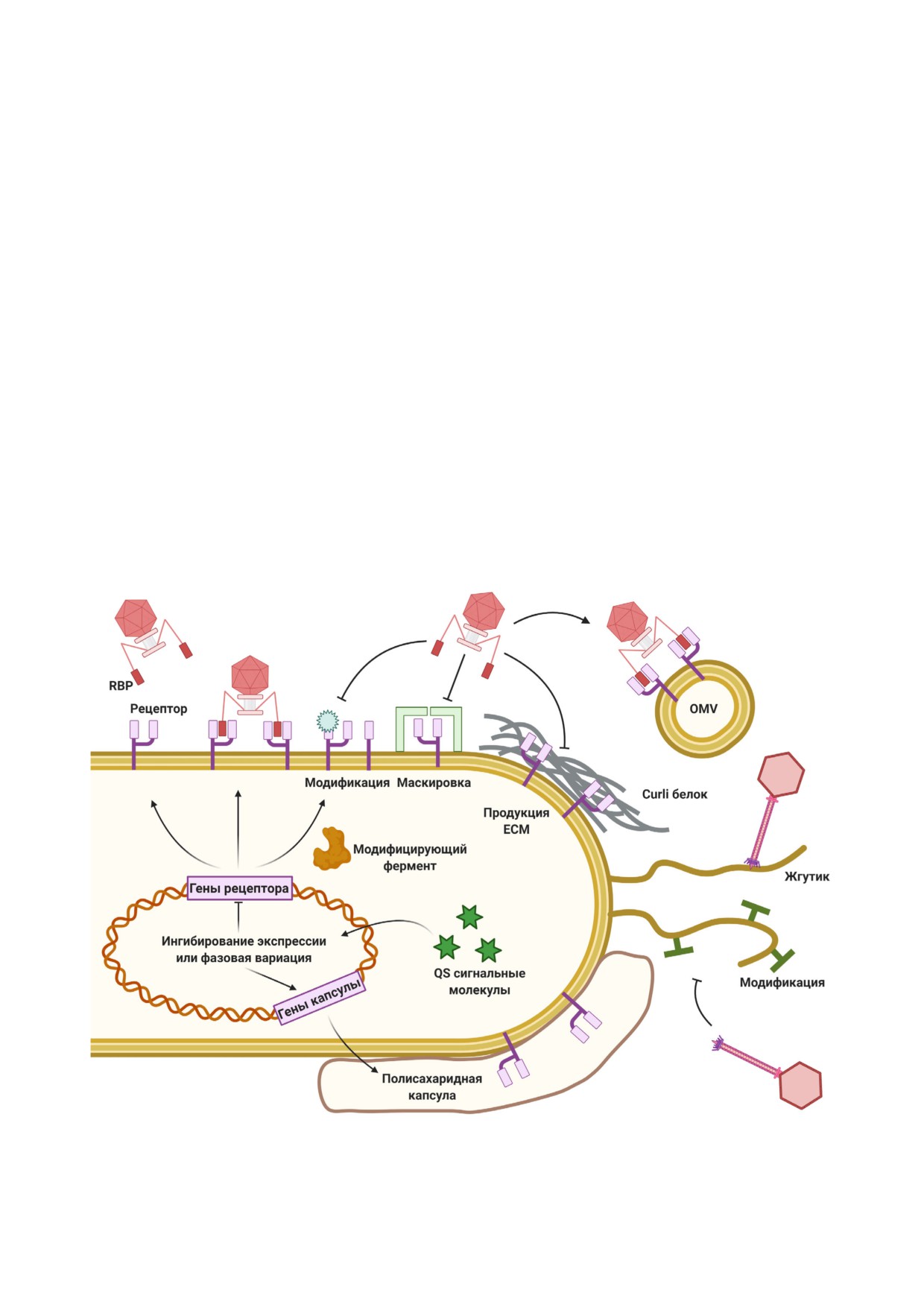
Рис. 2. Микробные стратегии, позволяющие предотвратить распознавание фагами
БИОХИМИЯ том 86 вып. 3 2021
378
ИСАЕВ и др.
меры, формирующие ECM, а пространственно!
FhuA или YueB в случае T5 или SSP1 соответ!
структурированные сообщества клеток, окру!
ственно) [51]. Изменения первичных и вторич!
женные ECM, образуют биопленки [54, 55]. Хо!
ных поверхностных рецепторов являются рас!
тя подробности взаимодействий фагов и клеток!
пространенным способом приобретения резис!
хозяев в биопленках полностью не выясне!
тентности к фагам [69]. Даже точечные мутации
ны [56], было показано, что микробные сообще!
в генах белков!рецепторов могут влиять на эф!
ства в биопленках более устойчивы к действию
фективность взаимодействия с фаговыми RBP,
вирусов. Повышенное давление со стороны фа!
как было показано, например в случае рецепто!
гов даже может приводить к усиленному образо!
ра FhuA фага Т5 или рецептора LamB фага λ [70,
ванию биопленок, в которых поверхностные
71]. Мутации, затрагивающие пути биосинтеза,
клеточные рецепторы становятся менее доступ!
которые ответственны за синтез компонентов
ными для вирусов [57, 58]. Другим преимущест!
клеточной стенки (LPS - у грамотрицательных
вом пространственной организации является то,
бактерий или тейхоевой кислоты - у грамполо!
что в окружающей среде фаги могут воздейство!
жительных бактерий), могут приводить к изме!
вать только на поверхностный слой клеток. Час!
нению структуры этих молекул и таким образом
то расположенные в этом слое клетки метаболи!
оказывать влияние на процесс распознавания
чески неактивны и таким образом не позволяют
клеток фагами, нацеленными на эти рецепто!
размножаться фагам, снижая при этом шансы
ры [72, 73]. Внеклеточные супраструктуры бак!
атаки на нижележащие клетки [59]. Компонен!
терий также могут служить в качестве рецепто!
ты ECM также могут выполнять функцию лову!
ров фагов, и известно, что мутации в генах,
шек или «поглотителей», которые адсорбируют
участвующих в образовании пилей или жгути!
и иммобилизуют фаги еще до того, как послед!
ков, могут обеспечить защиту от фагов [74, 75].
ние достигнут поверхности клеток [60, 61]. Было
Мутации рецепторов вряд ли можно рас!
показано, что белки curli, являющиеся компо!
сматривать непосредственно как стратегию за!
нентами ECM, ассоциированы с повышенной
щиты, поскольку фаговая инфекция лишь отби!
фагорезистентностью биопленок E. coli, в то
рает в популяции ранее существовавшие резис!
время как секреция экзополисахарида альгината
тентные клетки. В то же время специфические
защищает клетки Pseudomonas fluorescens [62, 63].
механизмы, контролирующие доступность ре!
Другим способом предотвращения адсорбции
цепторов, также существуют. Молекулы на по!
фагов является образование полисахаридной
верхности клетки вовлечены во многие важные
капсулы [64], например, повышенная продук!
для прокариот процессы, включая подвижность
ция колановой кислоты ассоциируется с устой!
или транспорт питательных веществ, и их мута!
чивостью клеток Escherichia к различным фагам,
ции могут быть связаны с издержками на под!
и в результате фаговой инфекции может прои!
держание жизнедеятельности клеток. Поэтому
зойти отбор мукоидных клеток с мутациями в
фазовые вариации (обратимое переключение
сигнальном пути RCS, контролирующем эту
экспрессии генов) или маскировка рецепторов
функцию [44, 65]. Роль ловушек также была по!
могут быть более безопасной долгосрочной
казана для внеклеточных везикул, образуемых
стратегией по сравнению с мутациями [76, 77].
клетками Vibrio и Escherichia. Такие везикулы,
Маскировка включает синтез молекул, которые
содержащие поверхностные рецепторы, могут
связываются с рецепторами хозяина и физичес!
адсорбировать фаги, тем самым понижая их титр
ки блокируют взаимодействие с фаговыми RBP,
в окружающей среде [66, 67]. С другой стороны,
как в случае связывания белка TraT с рецепто!
для Bacillus было показано, что везикулы, пере!
ром OmpA у E. coli [78]. Временные химические
носящие рецепторы, могут встраиваться в мемб!
модификации рецепторов также могут препят!
рану других невосприимчивых клеток, делая их
ствовать их распознаванию. В качестве приме!
чувствительными к инфекции [68].
ров можно привести гликозилирование пилей у
Изменения структуры рецепторов. Адсорб!
Pseudomonas aeruginosa или глюкозилирование
цию фагов часто можно рассматривать как двух!
O!антигена клеток Salmonella enterica [79, 80].
этапный процесс, в котором на первой стадии
Транскриптомные исследования показали, что
происходит обратимое связывание фага с экс!
маскирующие изменения рецепторов могут
понированными на поверхности клетки струк!
быть частью общего ответа на стресс как в слу!
турами (например, фаг T5 связывается с O!ан!
чае Lactococcus lactis, когда фаговая инфекция
тигеном LPS на поверхности клеток E. coli, в то
активирует гены, ответственные за D!аланили!
время как фаг SSP1 связывается с тейхоевой
рование клеточной стенки [81]. Снижение уров!
кислотой клеточной стенки Bacillus subtilis). За!
ня экспрессии рецепторов путем фазовой вари!
тем происходит необратимое прикрепление ко
ации может быть достигнуто рекомбинацией
вторичному рецептору (например, к белкам
или предрасположенными изменениями в про!
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
379
моторных областях [82, 83], в то время как мута!
соединений были выявлены молекулы, которые
ции в специфических «горячих точках» внутри
способны препятствовать инфицированию кле!
генов могут привести к сдвигу открытой рамки
ток E. coli фагом λ, при этом не влияя на рост
считывания и образованию укороченной фор!
бактерий. Противофаговая активность была так!
мы белка [84]. Системы, допускающие множест!
же показана в биологически значимом контекс!
венные инверсии, известны как шаффлоны, и
те: добавление отработанной культуральной сре!
также могут быть вовлечены в регуляцию
ды, собранной после роста штамма Streptomyces
экспрессии рецепторов [85], как в случае PilV,
peucetius, продуцирующего доксорубицин и дау!
кодируемого плазмидой IncI из E. coli, где для
норубицин, приводило к ингибированию про!
экспрессии белка может быть выбран один из
цесса инфицирования штамма Streptomyces coeli%
семи вариантов его C!концевого участка [86].
color, чувствительного к действию фага. Далее
Было показано, что распространенный симби!
авторы показали, что ~1/3 протестированных
онт кишечника человека Bacteroides thethaio%
экстрактов клеток Streptomyces обладают проти!
taamicron использует фазовую вариацию, по
вофаговой активностью в отношении природ!
крайней мере в 19 локусах, контролируя продук!
ных изолятов актинофагов, что позволяет пред!
цию различных типов капсул и экспрессию бел!
положить, что химическая защита является ши!
ков S!слоя [87]. Фазовые вариации допускают
роко распространенной защитной стратегией.
сосуществование микробных субпопуляций,
В качестве активных компонентов большинства
экспрессирующих различные варианты генов,
экстрактов были обнаружены антрациклины
что позволяет им найти баланс в защите от фа!
или другие ДНК!интеркалирующие агенты. От!
говых инфекций и воздействия факторов окру!
дельные ДНК!интеркалирующие агенты спо!
жающей среды.
собны инактивировать фаговые частицы до их
Структура клеточной стенки архей сильно
контакта с клеткой путем провоцирования некон!
отличается от бактериальной, и процесс при!
тролируемого выхода ДНК из капсида [98]. Од!
крепления вирусов к поверхности клеток архей
нако было показано, что в случае дауномицина
изучен довольно слабо [88]. Недавно была впер!
фаговая ДНК может проникнуть в клетки, хотя
вые предложена структурная модель связывания
ранние стадии инфекции при этом были подав!
археофага STIV с пили!подобной структурой
лены [27]. Результаты работы подняли ряд воп!
клеток Sulfolobus [89]. Было показано, что мута!
росов, касающихся механизмов ингибирования
ции в генах, ассоциированных с поверхностны!
малыми молекулами, их специфичности и избе!
ми молекулами клеток Sulfolobus, обеспечивают
гания токсичности для продуцирующих бакте!
резистентность клеток к SIRV2 [90]. Несмотря
рий, а также возможности использования проти!
на отсутствие данных, можно ожидать, что ме!
вовирусных метаболитов в качестве обществен!
ханизмы, подобные описанным для бактериаль!
ного ресурса в микробных популяциях.
ных клеток, также предотвращают адсорбцию
Вайперины и нуклеотиды, обрывающие рост
вирусов архей.
цепи. Интерферон!индуцированный противо!
вирусный ответ высших эукариот, в том числе и
человека, включает активацию фермента вайпе!
МАЛЫЕ МОЛЕКУЛЫ И ЗАЩИТА ОТ ФАГОВ
рина, ответственного за синтез рибонуклеоти!
да ddhCTP, обрывающего синтез новых цепей
Непосредственное участие в защите. В много!
РНК [99]. Предполагается, что обрыв цепи по!
численных ранних исследованиях изучалось
давляет вирусную транскрипцию и ингибирует
влияние различных химических соединений на
репликацию вирусов с РНК!геномами [100]. Ге!
эффективность вирусной инфекции [91-93].
ны вайперинов спорадически обнаруживались в
Было показано, что некоторые соединения,
геномах отдельных видов бактерий и архей. Не!
включая синтезируемые бактериями антибиоти!
давнее исследование показало, что прокариоти!
ки, могут влиять на производство фагового потом!
ческие вайперины (pVips) обеспечивают защиту
ства в концентрациях, субингибиторных для
от фаговой инфекции [101, 102]. В отличие от
роста бактерий [94-96]. Также было известно,
эукариотических гомологов, синтезирую!
что ДНК!специфичные красители и интеркали!
щих ddhCTP, pVip также продуцируют ddhUTP
рующие агенты (например, пропидиум иодид
и ddhGTP. Гетерологичная экспрессия различ!
или доксорубицин) могут инактивировать фаго!
ных pVip в E. сoli ингибировала инфекцию фа!
вые частицы [97]. Однако лишь недавно была
гом Т7 и транскрипцию, опосредованную РНК!
произведена переоценка роли малых молекул,
полимеразой Т7 [102]. Экспрессия pVip не влия!
продуцируемых бактериями, в их защите от фа!
ла на транскрипцию хозяина и не оказывала
гов [27]. В результате проведения высокопроиз!
токсичный эффект на клетки, что позволяет
водительного скрининга библиотек химических
предположить более высокую чувствительность
БИОХИМИЯ том 86 вып. 3 2021
380
ИСАЕВ и др.
вирусных РНК!полимераз к ингибированию с
ции бактерии способны перехватывать молеку!
участием ddhNTP. Интересно, что экспрес!
лы межвирусной коммуникации или вырабаты!
сия pVip обеспечивала более высокий уровень
вать собственные специфические сигналы для
защиты от фагов P1 и λ, которые используют хо!
мобилизации защитных барьеров в популяции.
зяйскую РНК!полимеразу для транскрипции
своих генов, подразумевая существование до!
полнительных защитных механизмов, ассоции!
ИНГИБИРОВАНИЕ ПРОНИКНОВЕНИЯ
рованных с вайперинами. Гены pVip в больших
ГЕНОМА ФАГА
количествах обнаруживаются внутри защитных
островков и часто находятся рядом с генами, ко!
Вслед за адсорбцией геном фага выходит из
дирующими нуклеотидкиназы, которые могут
капсида и транспортируется внутрь клетки!хо!
образовывать NTP из NMP и таким образом по!
зяина [115]. Существует несколько механизмов,
вышать уровень NTP как субстратов для pVip.
блокирующих эту стадию жизненного цикла ви!
Гены HicA!подобной РНКазы или белков, со!
руса [116]. Как правило, такие системы кодиру!
держащих домен анкириновых повторов, пред!
ются профагами и лежат в основе феномена
положительно участвующие в идентификации
исключения суперинфекции (Sie), т.е. предот!
фаговой инфекции [102], также обнаруживались
вращения вторичной инфекции гомоиммунны!
вблизи некоторых генов pVip.
ми фагами после установления первичной ин!
Регулирующая роль. Малые метаболиты так!
фекции (или лизогенизации) [117]. Мембранно!
же могут участвовать в защите бактерий от фа!
ассоциированные белки Sie могут блокировать
гов в качестве сигнальных молекул или в качест!
вход фаговой ДНК в клетку путем воздействия
ве кофакторов иммунных белков. Примеры пос!
на фаговый белок TMP (tape measure protein),
леднего будут описаны позднее, а здесь мы рас!
как это имеет место в случае экспрессии липо!
смотрим косвенную роль малых молекул в регу!
протеина профагом TP!J
34 в клетках
ляции экспрессии генов, обеспечивающих за!
Streptococcus thermophilus [118, 119] или бел!
щиту клеток. Феномен определения квору!
ка gp15 профагом HK97 в клетках E. coli [120,
ма (QS, Quorum Sensing) позволяет клеткам из!
121]. Известно, что белок Sp фага T4 ингибиру!
мерять плотность микробной популяции и мо!
ет лизоцим этого же фага, который необходим
жет рассматриваться как система коммуника!
для деградации пептидогликанового слоя кле!
ции, основанная на секреции малых моле!
точной стенки E. coli [122]. Предполагается, что
кул [103]. Популяции с высокой плотностью
клеточные белки хозяина, необходимые для
клеток более уязвимы к фаговым инфекциям, и
транслокации фаговой ДНК, также могут яв!
QS!опосредованная активация защитных барье!
ляться мишенями для Sie, как в случае защиты,
ров с увеличением плотности культуры полезна
опосредованной белком Imm фага T4 [122, 123]
для выживания. Например, в селекционных
или белком gp52 микобактериофага Fruitloop,
экспериментах было показано, что QS!профи!
который взаимодействует с белком Wag31 клет!
цитная культура P. aeruginosa достигает более
ки!хозяина и ингибирует инфицирование кле!
высокого уровня резистентности к фагам по
ток Wag31!зависимыми фагами [124]. Систе!
сравнению с QS!дефицитными клетками [104].
мы Sie с неизвестными мишенями были описа!
Кроме их роли в образовании биопленок [105],
ны в случае фага Tuc2009 Lactococcus lactis [125],
QS!сигналы могут регулировать экспрессию ге!
фага Р22 S. enterica [126], фага К139 Vibrio choler%
нов фаговых рецепторов и иммунных систем.
ae [127], фага Р1 E. coli [128] и В3!подобных фа!
Например, обработка лактоном N!ацилго!
гов клеток P. aeruginosa [129]. Несмотря на то что
моcерина приводит к снижению количества ре!
системы Sie рассматриваются в первую очередь
цепторов фага λ в клетках E. coli [106]. А в случае
как средства конкурентной борьбы между фага!
клеток Vibrio anguillarum эта обработка вызывает
ми [130], они способны принести пользу для
секрецию внеклеточных протеаз и снижает ко!
клетки!хозяина и в конечном итоге могут стать
личество рецепторов для фага KVP40 [107, 108].
неотъемлемой частью хромосомы, как в случае
Было показано, что QS!регуляция активирует
белка DicB, кодируемого криптическим профа!
экспрессию компонентов систем CRISPR!Cas в
гом Qin у E. coli [131].
клетках Serratia, Pseudomonas и других бакте!
рий [109, 110]. Для принятия решения о лизисе
или лизогении многие фаги используют бакте!
ИММУННЫЕ СИСТЕМЫ,
риальные QS!сигналы или кодируют собствен!
ЗАВИСЯЩИЕ ОТ МОДИФИКАЦИИ ДНК
ные сигнальные системы, как это недавно было
описано для системы arbitrium [111-114]. Мож!
Как только геном фага попадает внутрь
но предположить, что в случае фаговой инфек!
клетки, он становится мишенью для действия
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
381
различных ферментов, которые способны выз!
вать его деградацию. Например, комплекс нук!
леазы/хеликазы RecBCD, который также участ!
вует в репарации ДНК хозяина, распознает сво!
бодные концы ДНК, доступные на ранних ста!
диях инфицирования фагами с линейными
ДНК геномами [132, 133]. Наиболее часто дегра!
дацию ДНК, попадающей в клетку, осуществля!
ют системы врожденного и адаптивного имму!
нитета. Модификация ДНК клетки!хозяина,
которая необходима для того, чтобы дифферен!
цировать собственную ДНК и чужеродный ге!
нетический материал, является отличительным
признаком систем рестрикции!модификации.
Модификационный модуль этих систем ответ!
ственен за эпигенетическое мечение ДНК клет!
ки!хозяина, и поэтому немеченая фаговая ДНК
является объектом для эндонуклеазного рас!
щепления в результате действия рестрикцион!
ного модуля [134, 135]. Общий принцип меха!
низма действия R!M представлен на рис. 3.
Кроме классических систем R!M, множество
недавно описанных защитных систем кодируют
модификационный модуль, в то время как их
механизмы рестрикции чужеродного генетичес!
кого материала все еще не выяснены.
Классические системы R9M. Системы R!M
были открыты в начале 1950!х годов в процессе
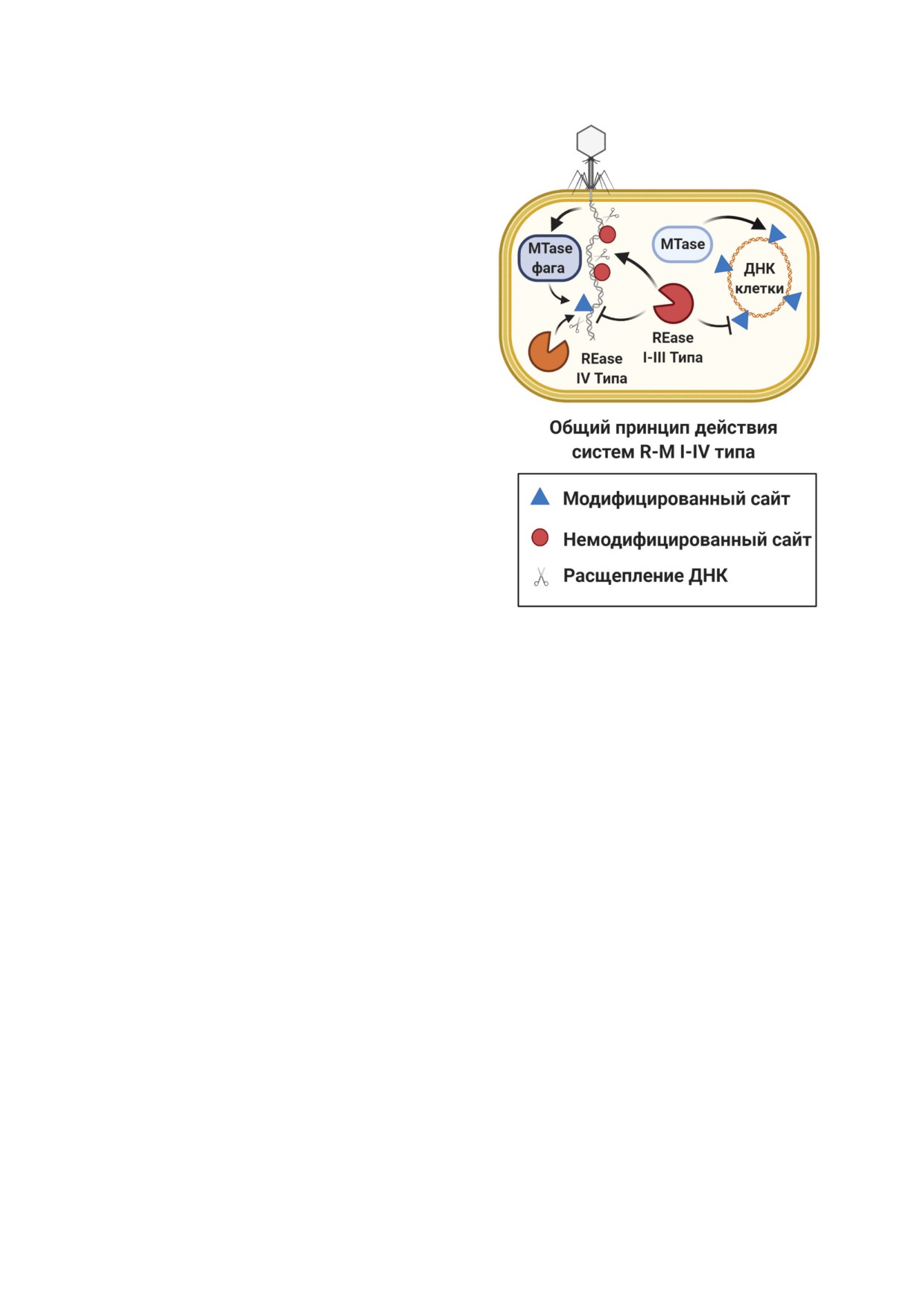
Рис. 3. Принцип действия классических систем рестрик!
изучения феномена модификации вирусов клет!
ции!модификации. Метилтрансфераза (МТаза, MTase) из
ками!хозяевами [136, 137]. Они были хорошо
систем типа I-III модифицирует специфические мотивы в
изучены в первые годы молекулярной биологии,
ДНК клетки!хозяина, в то время как неметилированные
что привело к их широкому применению и по!
участки в чужеродной ДНК подвергаются расщеплению
эндонуклеазой рестрикции (ЭР, REase). ЭР IV!го типа су!
явлению технологий рекомбинантной ДНК
ществует без сопряженной МТазы и расщепляет ДНК, мо!
[138]. Более 300 тысяч известных или предпола!
дифицированную вирусом
гаемых R!M!ферментов в настоящее время на!
ходятся в базе данных REBASE, а системы R!M
были обнаружены в ~90% секвенированных ге!
терны расщепления для систем R!M типов I-IV
номов бактерий и архей [30]. Функциональные
представлены на рис. 4.
субъединицы систем R!M состоят из метил!
R!M II!го типа наиболее изучена. Системы
трансферазы (МТазы, MTase), которая перено!
этого типа обычно состоят из отдельных белков
сит метильную группу от молекулы!донора
МТазы и ЭР. МТаза является мономером, в то
S!аденозилметионина (SAM) к цитозину или
время как ЭР действует как гомодимер. Как пра!
аденину в ДНК, и сопряженной эндонуклеазы
вило, оба фермента распознают один и тот же
рестрикции (ЭР, RЕase). Отдельные системы
палиндромный участок ДНК длиной 4-8 пар
также кодируют транслоказу, которая использу!
оснований. Расщепление ДНК происходит на
ет энергию гидролиза ATP для осуществления
обеих цепях ДНК в фиксированном положении
моторных функций, и субъединицу, которая со!
внутри или в непосредственной близости от не!
держит домены распознавания мишени (TRD),
метилированного сайта распознавания, и этот
определяющие специфичность ЭР и МТазы к
процесс зависит от наличия двухвалентных ка!
определенным последовательностям. В зависи!
тионов, в большинстве случаев от Mg2+ [141,
мости от состава субъединиц потребности в ко!
142]. МТаза эффективно метилирует неметили!
факторах и механизма действия системы R!M
рованные и полуметилированные сайты, кото!
подразделяются на 4 типа. Однако эта класси!
рые образуются после репликации полностью
фикация не отражает их эволюционного
метилированной ДНК, в то время как ЭР обла!
родства [139, 140]. Состав субъединиц для комп!
дает низким сродством к метилированным или
лексов, ответственных за модификацию и рест!
полуметилированным сайтам [143]. Помимо
рикцию, примеры сайтов распознавания и пат!
описанного простого способа действия, харак!
БИОХИМИЯ том 86 вып. 3 2021
382
ИСАЕВ и др.
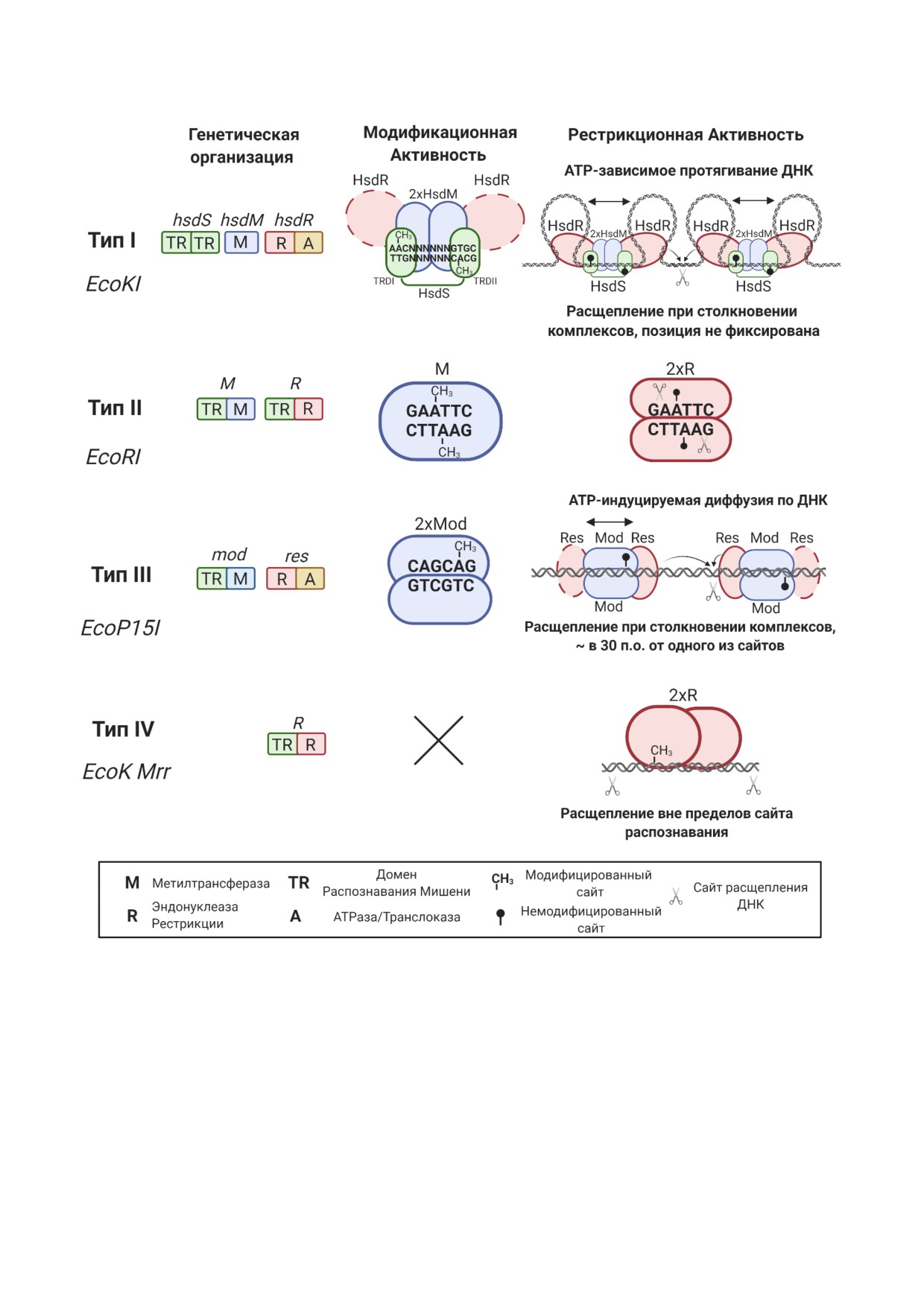
Рис. 4. Функциональные субъединицы, сайты распознавания, состав комплексов модификации и рестрикции для репре!
зентативных членов систем R!M типов I-IV. Системы R!M типа I модифицируют обе цепи двухчастных асимметричных
участков ДНК. Для рестрикции необходимо взаимодействие между двумя комплексами, связанными с неметилирован!
ными участками ДНК, которое достигается благодаря ATP!зависимой транслокации, при этом позиция сайта рестрикции
не фиксирована. Системы R!M типа II обычно распознают палиндромные сайты, и обе цепи ДНК внутри или в непосред!
ственной близости от неметилированных сайтов подвергаются расщеплению. Система R!M типа III модифицирует толь!
ко одну цепь в асимметричном сайте распознавания, и расщепление происходит в фиксированной позиции относитель!
но одного из сайтов, когда комплекс рестрикции, связанный с неметилированным сайтом, взаимодействует с другим
комплексом, активированным в результате распознавания близлежащего неметилированного сайта в обратной ориента!
ции. Система R!M типа IV не содержит метилтрансферазы и расщепляет молекулу ДНК после распознавания специфич!
ных модифицированных сайтов. Пунктирной линией показано, что данная субъединица может быть необязательной для
указанной активности
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
383
терного для подтипа IIP, ферменты, принадле!
зованных участках) [154]. Расщепление ДНК
жащие к другим подтипам, могут обладать не!
происходит в нефиксированной позиции между
обычными свойствами [139]. Например, фермен!
соседними участками распознавания [155]. Для
ты подтипов IIA, IIS и IIL распознают асиммет!
R!M I!го типа необходим SAM, причем не толь!
ричные сайты в ДНК, в подтипах IIC и IIL по!
ко в качестве донора метильных групп, но и как
липептиды ЭР и МТазы слиты в один белок; в то
кофактор комплекса рестрикции. Ферменты
время как ферменты подтипов IIE и IIF требуют
подтипа ISP представляют одиночные полипеп!
связывания с двумя сайтами для расщепления.
тиды, сочетающие в себе метилирующую и рест!
Ферменты подтипов IIL и IIG (например, MmeI
рикционную активности, при этом модифика!
и Eco571) распознают асимметричные сайты,
ции подвергается только одна цепь ДНК [156,
которые метилированы только по одной цепи
157]. Чтобы снизить риск повреждения ДНК
ДНК [144, 145]. Вторая цепь сайтов Eco571 под!
клетки!хозяина, может осуществляться допол!
вергается метилированию с участием дополни!
нительный контроль активности комплексов I!
тельной метилтрансферазы [145]. В то же время
го типа. Например, в результате ClpXP!опосре!
все еще не выяснено, как в системах подтипа IIL
дованного протеолитического расщепления
избегается расщепление неметилированных
субъединицы HsdR - феномен, известный как
сайтов, образующихся после репликации ДНК.
аллевиация рестрикции [158, 159]. Известно,
Избыточная экспрессия ЭР токсична для клет!
что благодаря фазовой вариации TRD!доменов
ки, в то время как избыток МТазы может при!
может происходить изменение специфичности
вести к метилированию геномов инфицирую!
ферментов I!го типа к сайтам распознава!
щих фагов и их защите от последующей рест!
ния [160].
рикции. Таким образом, для систем R!M необ!
Системы R!M типа III во многом похожи на
ходима координированная регуляция экспрес!
системы I!го типа [161]. Они функционируют
сии генов МТазы и ЭР, что достигается, напри!
как мультибелковые комплексы, состоящие из
мер, в результате активности контролирующего
субъединиц Mod и Res. Модификация ДНК про!
С!белка [146, 147] или при связывании МТазы
исходит в результате действия гомодимера Mod2,
со своим промотором и/или его метилировани!
а комплексы Res2-Mod2 или Res1-Mod2 высту!
ем [148].
пают в качестве ATP! и SAM!зависимой эндо!
Системы R!M типа I кодируют МТазу
нуклеазы рестрикции [162, 163]. Ферменты III!
(HsdM), ЭР (HsdR) и субъединицу, обеспечива!
го типа распознают короткие непалиндромные
ющую их специфичность (HsdS). Наиболее хо!
последовательности и метилируют только одну
рошо изученным примером является рестрикта!
цепь ДНК. Таким образом, подобно ситуации с
за EcoKI. Эти ферменты работают в виде комп!
ферментами подтипа ISP, половина из полуме!
лексов HsdM2-HsdS1-HsdR2, которые могут
тилированных участков после репликации ста!
осуществлять как рестрикцию, так и метилиро!
новится неметилированными. Чтобы предотв!
вание, в то время как метилирование также мо!
ратить атаку комплекса рестрикции на ДНК
жет осуществляться комплексами HsdM2-HsdS1
клетки!хозяина, для осуществления расщепле!
или HsdM2-HsdS1-HsdR1 [149-151]. Два участка
ния требуется наличие двух сайтов в противопо!
ДНК, разделенные вырожденной последова!
ложной ориентации (голова к голове или хвост к
тельностью (~AACNNNNNNGTGC для EcoKI),
хвосту), т.е. чтобы два неметилированных сосед!
распознаются двумя TRD!доменами на субъ!
них участка были локализованы на различных
единице HsdS, и обе цепи ДНК этих асиммет!
цепях ДНК, что обычно не встречается в ДНК
ричных участков подвергаются метилированию.
хозяина [164, 165]. Распознавание неметилиро!
Механистическая модель рестрикционной ак!
ванного участка ДНК приводит к инициации
тивности достаточно сложна. После распозна!
транслоказной активности субъединицы Res.
вания неметилированного сайта рестрикцион!
Однако, в отличие от ферментов I!го типа, при
ным комплексом активируется АTP!зависимая
этом расходуется меньше ATP, и вместо двунап!
моторная функция субъединицы HsdR, и комп!
равленного протягивания ДНК через комплекс
лекс протягивает через себя связанную молеку!
запускается одномерная диффузия вдоль цепи
лу ДНК в обоих направлениях, создавая пет!
ДНК [166, 167]. Расщепление ДНК происходит
ли [152]. Транслокация расходует ~3 молекулы
в фиксированной позиции от одного из сайтов
ATP на один нуклеотид [153]. Расщепление про!
распознавания, когда подвижный активирован!
исходит при столкновении двух комплексов
ный комплекс рестрикции взаимодействует с
рестрикции, закрепленных на разных сайтах,
другим комплексом, остающимся связанным с
или в случае, если дальнейшая транслокация
неметилированным сайтом. Экспрессия субъ!
ДНК осложнена (например, при столкновении
единицы Mod также может регулироваться пу!
с репликационной вилкой или в суперспирали!
тем фазовой вариации [168].
БИОХИМИЯ том 86 вып. 3 2021
384
ИСАЕВ и др.
Чтобы избежать расщепления рестриктаза!
Система Pgl была обнаружена только у
ми клетки!хозяина, фаги могут включать в свой
Actinomyces. Считается, что такое альтруистичес!
геном модифицированные основания [169].
кое поведение может быть обеспечено много!
В ответ на это у прокариот возникли системы R!
клеточными бактериями, которые способны по!
M IV!го типа, специфические к модифициро!
жертвовать одним компартментом ради защиты
ванным участкам ДНК [170, 171]. Тип IV - это
всего организма. В то же время Pgl!фенотип мо!
разнообразная и мало изученная группа одиноч!
жет обеспечить конкурентное преимущество в
ных эндонуклеаз рестрикции, у которых отсут!
борьбе с родственными Pgl!дефицитными клет!
ствуют сопряженные метилтрансферазы. Фер!
ками, так как клетки Pgl+ постоянно продуциру!
менты подтипа IIM также распознают метили!
ют модифицированные фаговые частицы, спо!
рованные основания, и некоторыми авторами
собные nоражать другие клетки, но не действую!
рассматриваются как ферменты типа IV [171].
щие на Pgl+ хозяина [186]. Фенотип Pgl имеет и
ЭР типа IV обычно обладают широкой субстрат!
дополнительное преимущество: в случае класси!
ной специфичностью, и их действие может быть
ческих систем R!M случайное метилирование
направлено на метилцитозины (McrA), метил!
генома фага часто приводит к появлению защи!
аденины (Mrr) или ДНК с фосфоротиоатными
щенного фагового потомства, которое будет спо!
связями (ScoMcrA) [172-174]. Некоторым фер!
собно уничтожить популяцию бактерий. Напро!
ментам из этой группы для расщепления требу!
тив, обратный механизм действия, характерный
ется гидролиз ATP или GTP и наличие более од!
для Pgl, гарантирует, что в ходе инфекционного
ного сайта рестрикции (McrBC или
процесса не появятся фаги, которым удастся из!
SauUSI) [175, 176]. Распространение этих бел!
бежать рестрикции (рис. 5).
ков и их экологическое значение, вероятно, все
Система Pgl кодирует 4 компонента: PglX -
еще остается недооцененным.
аденин!метилтрансферазу, PglY
- ATPазу,
Кроме их роли в защите от фагов и контро!
PglW - протеинкиназу и PglZ - щелочную фос!
ле горизонтального переноса генов, системы R!
фатазу (рис. 6, а) [187-189]. Для защиты необхо!
M оказывают влияние на другие биологические
димы все четыре белка, при этом активность
процессы [177]. Например, гены метилтрансфе!
первых трех компонентов была показана
раз часто обнаруживаются без сопряженных ге!
in vitro [190]. Делеция гена pglZ в присутствии
нов рестриктаз, и считается, что такие орфан!
функционального pglX невозможна. Поэтому
ные ферменты участвуют в регуляции экспрес!
было предположено, что белки, кодируемые
сии или репликации генов. Наиболее изученны!
этими генами, образуют пару токсин!антиток!
ми примерами являются Dam МТаза у E. coli и
син, а PglX играет ключевую роль в рестрикции,
CcrM - у Caulobacter crescentus [178, 179]. Неко!
когда его активность не сдерживается белком
торые системы R!M могут рассматриваться как
PlgZ [190]. До сих пор неизвестно, как система
эгоистичные элементы типа токсин!антиток!
Pgl распознает фаговую инфекцию и как работа!
син, поскольку потеря гена МТазы может при!
ет модуль рестрикции.
вести к постсегрегационной гибели клетки, свя!
Системы
исключения
бактериофагов
занной с повреждением ДНК в результате
(BacteRiophage EXclusion, BREX). Глобальный
действия рестриктазы [180]. Эволюционная и
анализ распространения гена pglZ в защитных
экологическая роль систем R!M также рассмот!
островках показал, что он обнаруживается не
рена в ряде других обзоров [76, 181-183].
только у Actinomyces, но также часто встречается
Система ограничения роста бактериофагов
в консервативных кластерах генов, отличных от
(Phage Growth Limitation, Pgl). Система Pgl может
Pgl!системы [18]. Было предположено, что pglZ!
представлять уникальный пример обратной
содержащие кластеры представляют новое су!
рестрикции!модификации, при которой моди!
персемейство систем защиты от фагов, которое
фицированная ДНК подвергается рестрикции,
было названо BREX [191]. Исходя из состава
но в отличие от систем R!M типа IV, модифика!
компонентов, системы BREX были классифи!
ция генома фага осуществляется самой системой
цированы на 6 типов, и Pgl был отнесен к BREX
защиты клетки!хозяина. Система Pgl была впер!
типа II (рис. 6, а). Кроме наличия PglZ, общей
вые описана при выделении фага ϕC31 на газоне
чертой всех систем BREX является присутствие
клеток Streptomyces coelicolor A(3)2 [184]. Позднее
ATPазы и метилтрансферазы. В системах IV!го
было показано, что потомство фага ϕC31 высво!
типа метилтрансфераза заменена на PAPS!ре!
бождается из клеток Pgl+ после первого раунда
дуктазу - фермент, участвующий в фосфоротио!
инфекции, но последующие раунды инфициро!
лировании ДНК [192]. Наиболее распростра!
вания клеток!хозяев были подавлены [185]. Бы!
ненным является BREX типа I, и системы этого
ло предположено, что вышедшие из клеток Pgl+
типа были экспериментально изучены в клетках
фаги несут Pgl!специфичные модификации.
B. subtilis, E. coli и V. Cholerae (где BREX встреча!
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
385
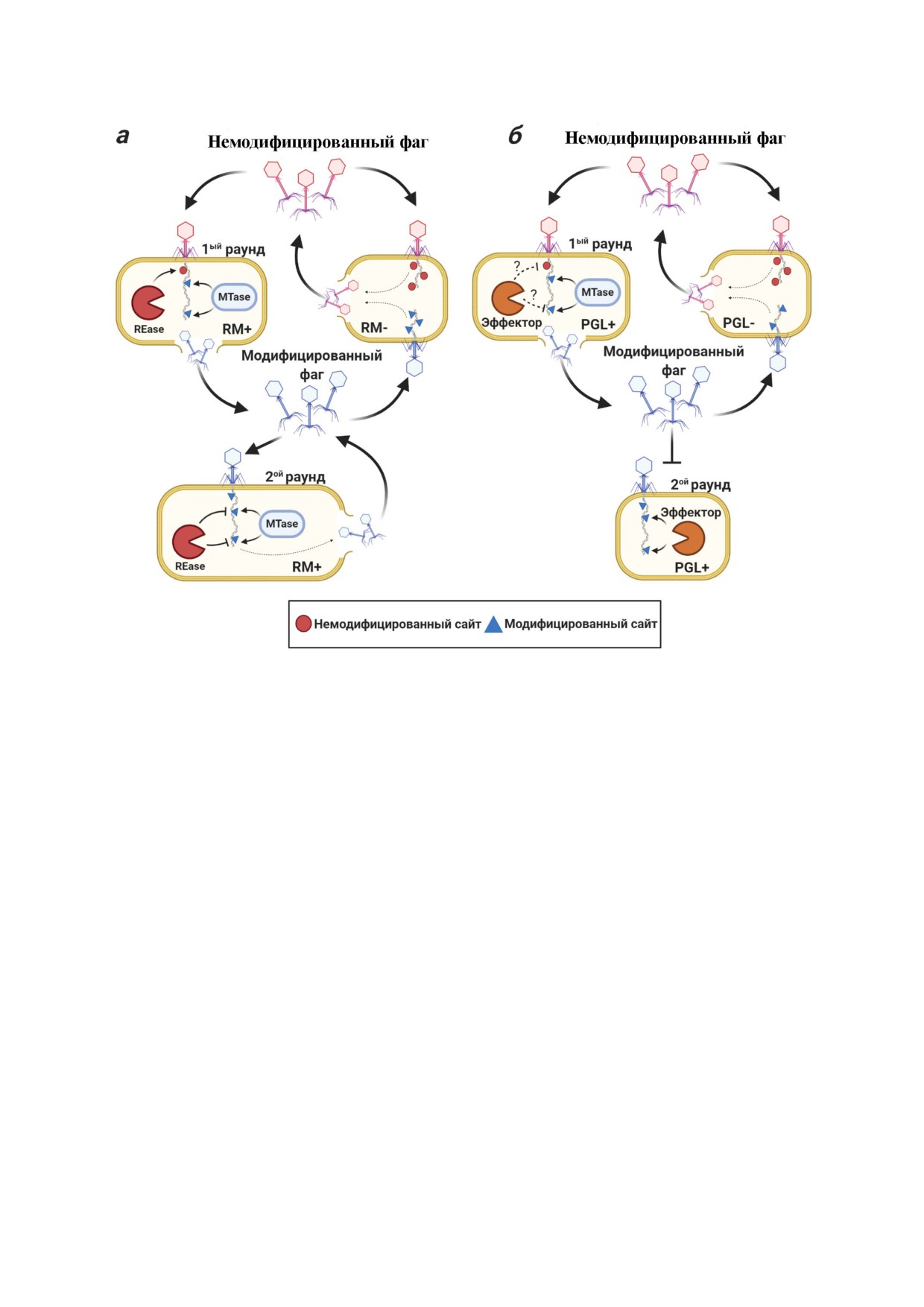
Рис. 5. а - Прямой механизм действия рестрикции!модификации: ошибочное метилирование фаговой ДНК во время ин!
фекции клеток, обладающих классической системой R!M (RM+), приведет к образованию потомства фага, которое бу!
дет способно продолжить эффективное инфицирование клеток RM+. Модификации могут быть утрачены только после
прохождения фага через R!M!дефицитные клетки (RM-). б - Обратный механизм действия системы рестрикции!моди!
фикации, который предполагается для системы Pgl: после первого раунда инфекции клетки Pgl+ продуцируют Pgl!моди!
фицированные фаговые частицы, которые подвергаются рестрикции в клетках Pgl+ во время второго раунда инфекции.
Pgl!модифированный фаг может эффективно инфицировать клетки Pgl-
ется в SXT конъюгативных элементах) [191, 193,
BREX!специфических модификаций в геноме
194]. Активность BREX метилтрансферазы так!
фага позволяет ему преодолеть защитную сис!
же была показана для клеток Lactobacillus
тему [193, 196]. Сайты системы BREX не явля!
casei [195]. Основными компонентами систем
ются палиндромами и метилируются только по
BREX типа I являются BrxX (PglX) - аденин!
одной цепи, что, подобно R!M системам ти!
специфичная метилтрансфераза, BrxZ (PglZ) -
па III и подтипа ISP, может подразумевать тре!
щелочная фосфатаза, BrxC - ATPаза, BrxL -
бование наличия нескольких сайтов и их спе!
Lon!подобная протеаза и небольшой белок BrxB
цифической ориентации для осуществления
с неизвестной функцией. Эти предсказанные
рестрикции. BREX действует на ранних стадиях
активности не были подтверждены in vitro, и
фаговой инфекции и препятствует накоплению
функция отдельных крупных доменов белков
вирусной ДНК внутри клеток. Тем не менее ме!
BREX все еще не определена. Также могут при!
ханизмы рестрикции остаются невыясненны!
сутствовать дополнительные небольшие белки,
ми. Защита BREX системы E. coli подавляется
которые, как предполагается, играют регулятор!
ДНК!мимикрирующим белком Ocr фа!
ную роль или необходимы для обеспечения за!
га Т7 [46], который является хорошо известным
щиты от определенных фагов (например,
ингибитором систем R!M типа I [151, 197]. Этот
BrxA) [191, 193].
результат, по!видимому, предполагает наличие
Было показано, что, подобно классическим
общих механистических особенностей для сис!
системам R!M, системы BREX метилируют
тем BREX и мультисубъединичных комплексов
собственную ДНК клетки!хозяина, и наличие
систем R!M.
7 БИОХИМИЯ том 86 вып. 3 2021
386
ИСАЕВ и др.
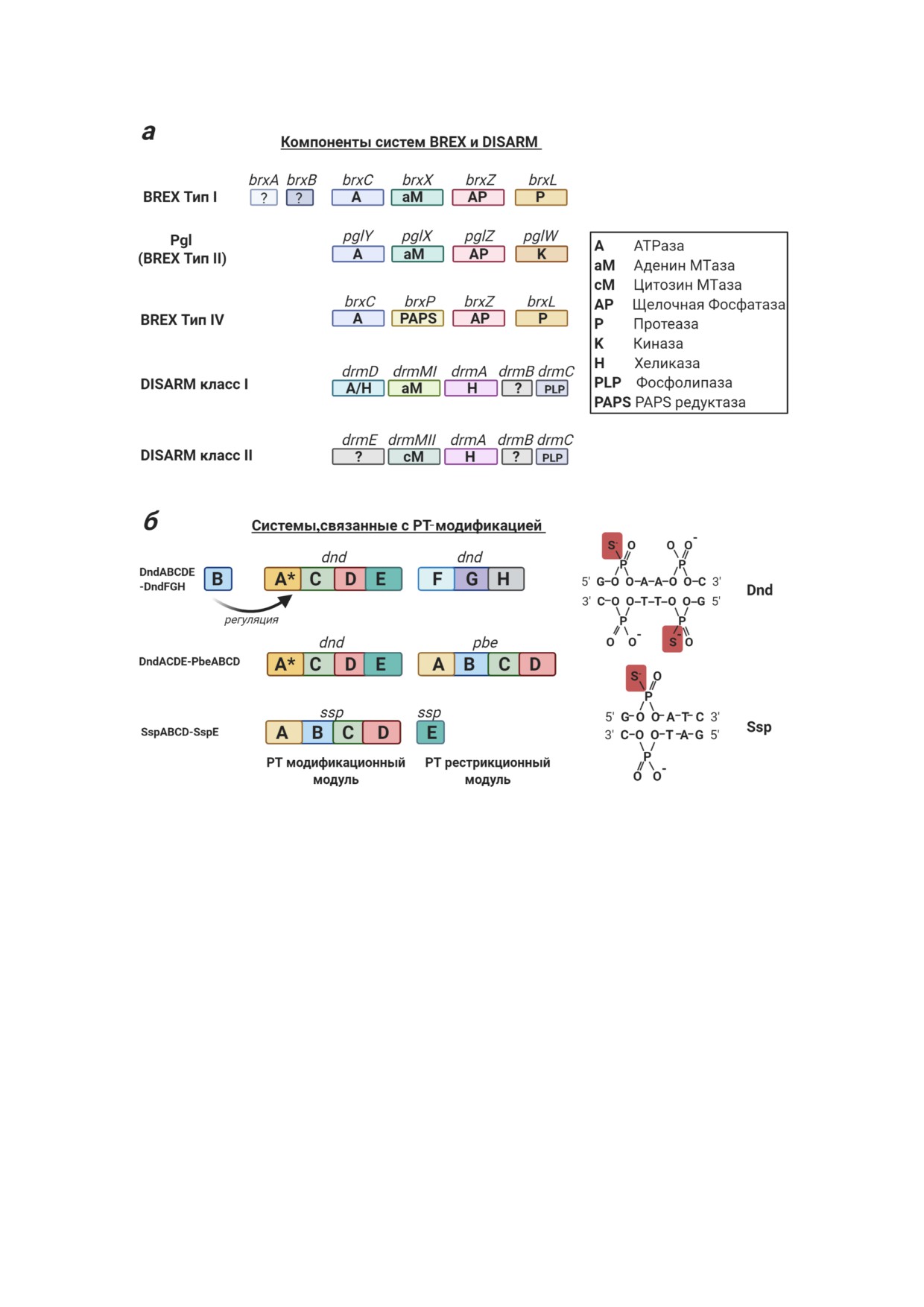
Рис. 6. а - Функциональные субъединицы, характерные для различных типов систем BREX и DISARM. Порядок распо!
ложения компонентов на схеме не всегда отражает действительную организацию генов в опероне. б - Системы, основан!
ные на фосфоротиолировании сахаро!фосфатного остова ДНК. Из!за транзиентного характера PT!модификации лишь
малая часть сайтов действительно является модифицированной в геноме, и мотивы Dnd систем могут оставаться полумо!
дифицированными
Системы DISARM. Вслед за открытием
феразы DrmMII [198]. Этот состав характерен
BREX подробное изучение консервативных
для DISARM класса 2, в то время как в более
кластеров генов в защитных островках привело
распространенных системах класса 1 белок
к предсказанию еще одной новой системы -
DrmMII заменен на аденин!метилтрансфера!
DISARM (Defence Island System Associated with
зу DrmMI, а белок DrmE - на SNF!2!подобную
Restriction!Modification) [198]. Противовирус!
хеликазу DrmD (рис. 6, а). Домены PLD могут
ная активность DISARM была исследована на
быть вовлечены в каталитическую активность
примере системы из Bacillus paralicheniformis.
нуклеаз [199]. Тем не менее DrmC не оказался
Она состоит из 5 компонентов: хеликазы DrmA,
необходимым для DISARM!опосредованной за!
белка DrmB, содержащего домен DUF1998 с не!
щиты от фагов. Экспрессии одного лишь бел!
установленной функцией, белка DrmC, содер!
ка DrmMII достаточно для метилирования сим!
жащего домен фосфолипазы D (PLD), бел!
метричных сайтов (CCWGG) в ДНК хозяина, а
ка DrmE и цитозин!специфичной метилтранс!
делеция гена метилтрансферазы в присутствии
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
387
полного кластера DISARM является токсичной
ATPазу/никирующую эндонуклеазу (DndD) и
для клеток. Но в отличие от классического фе!
небольшой белок DndE, который связывается с
нотипа R!M, фаг ϕ3T с DISARM!специфичным
никированной ДНК. При этом DndB регулиру!
метилированием был неспособен инфициро!
ет транскрипцию оперона dndBCDE и таким об!
вать клетки DISARM+, что позволяет предпо!
разом определяет долю PT!модифицированных
ложить, что метилирование необходимо, но не!
сайтов в геноме [204, 207-209]. Пока еще опре!
достаточно для того, чтобы избежать рестрик!
делены не все стадии биохимического пути, за!
ции [198]. Подобно BREX, DISARM не влияет
действованного в процесс фосфоротиолирова!
на адсорбцию фага и ингибирует ранние стадии
ния. Однако известно, что цистеин является до!
инфекции по неизвестному механизму.
нором атома серы, который переносится на
Системы DPD. Помимо метилирования
DndC и затем энергозависимым образом встраи!
ДНК, модификации на основе 7!деазагуанина
вается в ДНК, предварительно никированную
также могут быть связаны с R!M!подобными за!
DndD [192, 210, 211]. Недавно было показано,
щитными системами [200]. В синтезе 7!деазагуа!
что гены dnd также могут быть задействованы в
нина, который обычно выступает как предшест!
PT!модификации РНК [212]. Считается, что
венник модифицированных оснований в моле!
PT!модификация важна для поддержания ре!
куле транспортной РНК, участвуют несколько
докс!гомеостаза и контроля экспрессии ге!
ферментов. Некоторые прокариоты кодируют
нов [213], но у бактерий модификационный мо!
дополнительные кластеры генов биосинтеза,
дуль Dnd часто сопровождается кластером генов
которые ответственны за включение 7!деазагуа!
рестрикции dndFGH [214]. In vitro активность
нина в ДНК [200]. Такие системы DPD (7!деаза!
компонентов DndFGH не была изучена, однако
пурин в ДНК) могут содержать до 10 компонен!
in vivo присутствие кластера dndABCDE-FGH
тов (DpdA!K) [200, 201]. R!M!подобная актив!
ингибировало трансформацию немодифициро!
ность системы DPD была предположена на ос!
ванной ДНК, в то время как экспрессия
нове ингибирования трансформации немоди!
DndFGH в штамме, не содержащем DndABCE,
фицированных плазмид в клетки Salmonella
приводила к расщеплению собственной ДНК
montevideo, несущих кластер dpd [200]. Однако
клетки [214-216]. Наиболее заметной чертой
активность системы DPD против фаговой ин!
PT!модификации, которая довольно сильно от!
фекции до сих пор не показана, и возможный
личается от метилирования системами R!M, яв!
механизм рестрикции остается невыясненным.
ляется тот факт, что модифицируется лишь не!
Дополнительные компоненты DPD систем
большая доля доступных сайтов, и модифика!
включают хеликазы, ParB!подобную NTPазу и
ция каждого конкретного участка носит крат!
нуклеазу PLD, которые могут быть вовлечены в
ковременный характер. Это рождает вопросы о
рестрикцию немодифицированной ДНК. Инте!
механизмах избегания рестрикции собственной
ресно, что аналогичные кластеры модификации
ДНК [217, 218]. Наличие метилирования Dam
7!деазагуанина обнаружены в некоторых вирус!
влияет на распределение PT!модифицирован!
ных геномах (например, в фагах 9g или Cajan),
ных сайтов, но не влияет на их общую плот!
где было показано, что данная модификация за!
ность [219]. В связи с этим было предположено,
щищает фаговую ДНК от широкого спектра эн!
что специфичность РT!модификации определя!
донуклеаз рестрикции [202, 203].
ется общей геометрией участка ДНК, а не его
Системы, связанные с фосфоротиолировани9
последовательностью [219].
ем ДНК (PT). Рассмотренные ранее модифика!
PT!модификация также была показана для
ции затрагивали только азотистые основания,
архей, у которых функцию рестрикции вместо
но сахаро!фосфатный остов ДНК также может
dndFGH выполняет кластер генов pbeABCD [205].
подвергаться модификации. Замена атома кис!
Было показано, что dndCDEA-pbeABCD из кле!
лорода в гидроксильной группе фосфатного ос!
ток Haloterrigena jeotgali обеспечивает противо!
татка на атом серы, называемая фосфоротиоли!
вирусную защиту, и рестрикционная активность
рованием, приводит к появлению PT!свя!
зависит от функционально!активного модуля
зи (фосфоротиоат) в ДНК, которая может быть
PT!модификации, что отличается от поведения
ассоциирована с различными защитными сис!
dndFGH [205]. В инфицированных dndCDEA-
темами бактерий и архей [204-206]. Эти систе!
pbeABCD клетках не наблюдалось накопления
мы кратко представлены на рис. 6, б.
вирусной ДНК, хотя при этом не было показано
Фосфоротиолирование (PT!модификация)
и её расщепления. Гены pbeABCD могут также
происходит в результате активности ге!
обнаруживаться вне контеста dndCDEA или
нов dndABCDE (Dnd - фенотип, связанный с
вблизи генов метилтрансфераз, что подразуме!
деградацией ДНК), которые кодируют цистеин!
вает возможность обмена модулями между раз!
десульфуразу (DndA), PAPS!редуктазу (DndC),
личными защитными системами [205].
БИОХИМИЯ том 86 вып. 3 2021
7*
388
ИСАЕВ и др.
Недавно была открыта еще одна основан!
Финансирование. Выполнение данной рабо!
ная на PT!модификации защитная система -
ты происходило при финансовой поддержке
SspABCD!SspE [206]. Гены sspABCD не гомоло!
Российского фонда фундаментальных исследо!
гичны генам dndABCDE, но они кодируют сход!
ваний (грант № 19!14!50560). А.И. также под!
ные функциональные домены и выполняют
держан грантом Российского фонда фундамен!
PT!модификацию ДНК, в то время как SspE
тальных исследований (грант № 19!34!90160),
служит в качестве рестрикционного компонен!
О.М. поддержана грантом Российского научно!
та, ингибируя фаговую инфекцию. In vitro было
го фонда (грант № 19!74!00118).
показано, что SspE обладает активностью
Вклад авторов. А.И. подготовил текст обзор!
NTPазы, которая стимулируется наличием PT!
ной статьи, А.И. и О.М. подготовили рисунки,
модифицированных сайтов и неспецифи!
К.С. провел редактуру статьи.
ческой никазной эндонуклеазной актив!
Благодарности. Авторы выражают благодар!
ностью [206]. Отличительной особенностью
ность Анне Ершовой и Андрею Летарову за кри!
SspABCD является модификация только одной
тические замечания к отдельным разделам
цепи ДНК внутри непалиндромных участков
статьи. Иллюстрации были подготовлены при
узнавания.
помощи программы BioRender.
Конфликт интересов. Авторы заявляют об
Описание других стратегий защиты прока!
отсутствии конфликта интересов.
риотических клеток от вирусных инфекций, а
Соблюдение этических норм. Настоящая
также обсуждение взаимодействий между раз!
статья не содержит описания каких!либо иссле!
личными противовирусными системами будет
дований с участием людей или животных в каче!
продолжено во второй главе рукописи.
стве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Clokie, M. R. J., Millard, A. D., Letarov, A. V., and
parasites caused by evolutionary instability of parasite!free
Heaphy, S. (2011) Phages in nature, Bacteriophage, 1, 31!45.
states, Biol. Direct, 12, 1!12.
2. D az Muñoz, S. L., and Koskella, B. (2014) Bacteria!
13. Szathmáry, E., and Demeter, L. (1987) Group selection of
phage interactions in natural environments, in Advances in
early replicators and the origin of life, J. Theor. Biol., 128,
Applied Microbiology, Elsevier, Vol. 89, pp. 135!183.
463!486.
3. Batinovic, S., Wassef, F., Knowler, S. A., Rice, D. T. F.,
14. Krupovic, M., Dolja, V. V., and Koonin, E. V. (2019)
Stanton, C. R., et al. (2019) Bacteriophages in natural and
Origin of viruses: primordial replicators recruiting capsids
artificial environments, Pathogens, 8, 100.
from hosts, Nat. Rev. Microbiol., 17, 449!458.
4. Wommack, K. E., and Colwell, R. R. (2000) Virioplank!
15. Koonin, E. V., Makarova, K. S., and Wolf, Y. I. (2017)
ton: viruses in aquatic ecosystems, Microbiol. Mol. Biol.
Evolutionary Genomics of Defense Systems in Archaea
Rev., 64, 69!114.
and Bacteria, Annu. Rev. Microbiol., 71, 233!261.
5. Parikka, K. J., Le Romancer, M., Wauters, N., and
16. Galperin, M. Y., and Koonin, E. V (2000) Who’s your
Jacquet, S. (2017) Deciphering the virus!to!prokaryote
neighbor? New computational approaches for functional
ratio (VPR): insights into virus!host relationships in a vari!
genomics, Nat. Biotechnol., 18, 609!613.
ety of ecosystems, Biol. Rev., 92, 1081!1100.
17. Shmakov, S. A., Faure, G., Makarova, K. S., Wolf, Y. I.,
6. Rodriguez!Valera, F., Martin!Cuadrado, A.!B.,
Severinov, K. V., and Koonin, E. V. (2019) Systematic pre!
Rodriguez!Brito, B., Pasic, L., Thingstad, T. F., et al.
diction of functionally linked genes in bacterial and
(2009) Explaining microbial population genomics through
archaeal genomes, Nat. Protoc., 14, 3013!3031.
phage predation, Nat. Preced., doi: 10.1038/npre.2009.
18. Makarova, K. S., Wolf, Y. I., Snir, S., and Koonin, E. V.
3489.1.
(2011) Defense islands in bacterial and archaeal genomes
7. Breitbart, M., Bonnain, C., Malki, K., and Sawaya, N. A.
and prediction of novel defense systems, J. Bacteriol., 193,
(2018) Phage puppet masters of the marine microbial
6039!56.
realm, Nat. Microbiol., 3, 754!766.
19. Oliveira, P. H., Touchon, M., and Rocha, E. P. C. (2014)
8. Brüssow, H., Canchaya, C., and Hardt, W. D. (2004)
The interplay of restriction-modification systems with
Phages and the evolution of bacterial pathogens: from
mobile genetic elements and their prokaryotic hosts,
genomic rearrangements to lysogenic conversion,
Nucleic Acids Res., 42, 10618!10631.
Microbiol. Mol. Biol. Rev., 68, 560 602.
20. Koonin, E. V., Makarova, K. S., Wolf, Y. I., and
9. Chiang, Y. N., Penadés, J. R., and Chen, J. (2019) Genetic
Krupovic, M. (2020) Evolutionary entanglement of mobile
transduction by phages and chromosomal islands: the new
genetic elements and host defence systems: guns for hire,
and noncanonical, PLoS Pathog., 15, e1007878.
Nat. Rev. Genet., 21, 119!131.
10. Lang, A. S., Westbye, A. B., and Beatty, J. T. (2017) The
21. Doron, S., Melamed, S., Ofir, G., Leavitt, A., Lopatina, A.,
distribution, evolution, and roles of gene transfer agents in
et al. (2018) Systematic discovery of antiphage defense sys!
prokaryotic genetic exchange, Annu. Rev. Virol., 4, 87!104.
tems in the microbial pangenome, Science, 359, eaar4120.
11. Keen, E. C., Bliskovsky, V. V, Malagon, F., Baker, J. D.,
22. Gao, L., Altae!Tran, H., Böhning, F., Makarova, K. S.,
Prince, J. S., et al. (2017) Novel “superspreader” bacterio!
Segel, M., et al. (2020) Diverse enzymatic activities mediate
phages promote horizontal gene transfer by transforma!
antiviral immunity in prokaryotes, Science, 369, 1077!1084.
tion, MBio, 8, e02115!16.
23. Makarova, K. S., Wolf, Y. I., and Koonin, E. V. (2019)
12. Koonin, E. V., Wolf, Y. I., and Katsnelson, M. I. (2017)
Towards functional characterization of archaeal genomic
Inevitability of the emergence and persistence of genetic
dark matter, Biochem. Soc. Trans., 47, 389!398.
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
389
24.
Eraslan, G., Avsec, Ž., Gagneur, J., and Theis, F. J. (2019)
and single gene deletion screens, Mol. Microbiol., 108, 288!
Deep learning: new computational modelling techniques
305.
for genomics, Nat. Rev. Genet., 20, 389!403.
41.
Piya, D., Lessor, L., Koehler, B., Stonecipher, A.,
25.
Eitzinger, S., Asif, A., Watters, K. E., Iavarone, A. T.,
Cahill, J., and Gill, J. J. (2020) Genome!wide screens
Knott, G. J., et al. (2020) Machine learning predicts new
reveal Escherichia coli genes required for growth of T1!like
anti!CRISPR proteins, Nucleic Acids Res., 48, 4698!4708.
phage LL5 and V5!like phage LL12, Sci. Rep., 10, 1!9.
26.
Gussow, A. B., Park, A. E., Borges, A. L., Shmakov, S. A.,
42.
Rousset, F., Cui, L., Siouve, E., Becavin, C.,
Makarova, K. S., et al. (2020) Machine!learning approach
Depardieu, F., and Bikard, D. (2018) Genome!wide
expands the repertoire of anti!CRISPR protein families,
CRISPR!dCas9 screens in E. coli identify essential genes
Nat. Commun., 11, 1!12.
and phage host factors, PLoS Genet., 14, e1007749.
27.
Kronheim, S., Daniel!Ivad, M., Duan, Z., Hwang, S.,
43.
Kortright, K. E., Chan, B. K., and Turner, P. E. (2020)
Wong, A. I., et al. (2018) A chemical defence against phage
High!throughput discovery of phage receptors using trans!
infection, Nature, 564, 283!286.
poson insertion sequencing of bacteria, Proc. Natl. Acad.
28.
Fillol!Salom, A., Miguel!Romero, L., Marina, A.,
Sci. USA, 117, 18670!18679.
Chen, J., and Penadés, J. R. (2020) Beyond the CRISPR!
44.
Mutalik, V. K., Adler, B. A., Rishi, H. S., Piya, D.,
Cas safeguard: PICI!encoded innate immune systems pro!
Zhong, C., et al. (2020) High!throughput mapping of the
tect bacteria from bacteriophage predation, Curr. Opin.
phage resistance landscape in E. coli, PLoS Biol., 18,
Microbiol., 56, 52!58.
e3000877.
29.
O’Hara, B. J., Barth, Z. K., McKitterick, A. C., and Seed,
45.
Luria, S. E., and Delbrück, M. (1943) Mutations of bacte!
K. D. (2017) A highly specific phage defense system is a
ria from virus sensitivity to virus resistance, Genetics, 28,
conserved feature of the Vibrio cholerae mobilome, PLoS
491.
Genet., 13, 1!17.
46.
Isaev, A., Drobiazko, A., Sierro, N., Gordeeva, J.,
30.
Roberts, R. J., Vincze, T., Posfai, J., and Macelis, D.
Yosef, I., et al. (2020) Phage T7 DNA mimic protein Ocr is
(2015) REBASE - a database for DNA restriction and
a potent inhibitor of BREX defence, Nucleic Acids Res., 48,
modification: enzymes, genes and genomes, Nucleic Acids
5397!5406.
Res., 43, D298!D299.
47.
Pinilla!Redondo, R., Shehreen, S., Marino, N. D.,
31.
Akarsu, H., Bordes, P., Mansour, M., Bigot, D.!J.,
Fagerlund, R. D., Brown, C. M., et al. (2020) Discovery of
Genevaux, P., and Falquet, L. (2019) TASmania: a bacter!
multiple anti!CRISPRs highlights anti!defense gene clus!
ial toxin!antitoxin systems database, PLOS Comput. Biol.,
tering in mobile genetic elements, Nat. Commun., 11, 1!11.
15, e1006946.
48.
Samson, J. E., Magadán, A. H., Sabri, M., and
32.
Zhang, F., Zhao, S., Ren, C., Zhu, Y., Zhou, H., et al.
Moineau, S. (2013) Revenge of the phages: defeating bac!
(2018) CRISPRminer is a knowledge base for exploring
terial defences, Nat. Rev. Microbiol., 11, 675!87.
CRISPR!Cas systems in microbe and phage interactions,
49.
Hwang, S., and Maxwell, K. L. (2019) Meet the anti!
Commun. Biol., 1, 1!5.
CRISPRs: widespread protein inhibitors of CRISPR!Cas
33.
Pourcel, C., Touchon, M., Villeriot, N., Vernadet, J.!P.,
systems, Cris. J., 2, 23!30.
Couvin, D., et al. (2020) CRISPRCasdb a successor of
50.
Hampton, H. G., Watson, B. N. J., and Fineran, P. C.
CRISPRdb containing CRISPR arrays and cas genes from
(2020) The arms race between bacteria and their phage
complete genome sequences, and tools to download and
foes, Nature, 577, 327!336.
query lists of repeats and spacers, Nucleic Acids Res., 48,
51.
Bertozzi Silva, J., Storms, Z., and Sauvageau, D. (2016)
D535!D544.
Host receptors for bacteriophage adsorption, FEMS
34.
Zhang, Y., Zhang, Z., Zhang, H., Zhao, Y., Zhang, Z., and
Microbiol. Lett., 363, fnw002.
Xiao, J. (2020) PADS Arsenal: A database of prokaryotic
52.
Dowah, A. S. A., and Clokie, M. R. J. (2018) Review of the
defense systems related genes, Nucleic Acids Res., 48,
nature, diversity and structure of bacteriophage receptor
D590!D598.
binding proteins that target Gram!positive bacteria,
35.
Mendler, K., Chen, H., Parks, D. H., Lobb, B., Hug,
Biophys. Rev., 10, 535!542.
L. A., and Doxey, A. C. (2019) Annotree: visualization and
53.
Stone, E., Campbell, K., Grant, I., and McAuliffe, O.
exploration of a functionally annotated microbial tree of
(2019) Understanding and exploiting phage!host interac!
life, Nucleic Acids Res., 47, 4442!4448.
tions, Viruses, 11, 567.
36.
Howard!Varona, C., Lindback, M. M., Bastien, G. E.,
54.
Dragoš, A., and Kovács, Á. T. (2017) The peculiar func
Solonenko, N., Zayed, A. A., et al. (2020) Phage!specific
tions of the bacterial extracellular matrix, Trends
metabolic reprogramming of virocells, ISME J., 14, 881!
Microbiol., 25, 257 266.
895.
55.
Hansen, M. F., Svenningsen, S. Lo, Røder, H. L.,
37.
Ghosh, S., Hamdan, S. M., Cook, T. E., and Richardson,
Middelboe, M., and Burmølle, M. (2019) Big impact of
C. C. (2008) Interactions of Escherichia coli thiored!
the tiny: bacteriophage bacteria interactions in biofilms,
oxin, the processivity factor, with bacteriophage T7 DNA
Trends Microbiol., 27, 739 752.
polymerase and helicase, J. Biol. Chem., 283, 32077!
56.
Abedon, S. T. (2017) Phage “delay” towards enhancing
32084.
bacterial escape from biofilms: a more comprehensive way
38.
Qimron, U., Marintcheva, B., Tabor, S., and Richardson,
of viewing resistance to bacteriophages, AIMS Microbiol.,
C. C. (2006) Genomewide screens for Escherichia coli
3, 186.
genes affecting growth of T7 bacteriophage, Proc. Natl.
57.
Fernández, L., Rodr guez, A., and Garc a, P. (2018) Phage
Acad. Sci. USA, 103, 19039!19044.
or foe: an insight into the impact of viral predation on
39.
Maynard, N. D., Birch, E. W., Sanghvi, J. C., Chen, L.,
microbial communities, ISME J., 12, 1171 1179.
Gutschow, M. V., and Covert, M. W. (2010) A forward!
58.
Henriksen, K., Rørbo, N., Rybtke, M. L., Martinet, M.
genetic screen and dynamic analysis of lambda phage host!
G., Tolker!Nielsen, T., et al. (2019) P. aeruginosa flow!cell
dependencies reveals an extensive interaction network and
biofilms are enhanced by repeated phage treatments but
a new anti!viral strategy, PLoS Genet., 6, e1001017.
can be eradicated by phage!ciprofloxacin combination,
40.
Bohm, K., Porwollik, S., Chu, W., Dover, J. A., Gilcrease,
Pathog. Dis., 77, ftz011.
E. B., et al. (2018) Genes affecting progression of bacterio!
59.
Hosseinidoust, Z., Tufenkji, N., and van de Ven, T. G. M.
phage P22 infection in Salmonella identified by transposon
(2013) Formation of biofilms under phage predation: con!
БИОХИМИЯ том 86 вып. 3 2021
390
ИСАЕВ и др.
siderations concerning a biofilm increase, Biofouling, 29,
Pseudomonas aeruginosa defends against phages through
457!468.
type IV pilus glycosylation, Nat. Microbiol., 3, 47!52.
60.
Bull, J. J., Christensen, K. A., Scott, C., Jack, B. R., Crandall,
80.
Kim, M., and Ryu, S. (2012) Spontaneous and transient
C. J., and Krone, S. M. (2018) Phage!bacterial dynamics with
defence against bacteriophage by phase!variable glucosyla!
spatial structure: self organization around phage sinks can
tion of O!antigen in Salmonella enterica serovar
promote increased cell densities, Antibiotics, 7, 8.
Typhimurium, Mol. Microbiol., 86, 411!425.
61.
Tan, D., Dahl, A., and Middelboe, M. (2015) Vibriophages
81.
Fallico, V., Ross, R. P., Fitzgerald, G. F., and
differentially influence biofilm formation by Vibrio anguil%
McAuliffe, O. (2011) Genetic response to bacteriophage
larum strains, Appl. Environ. Microbiol., 81, 4489!4497.
infection in Lactococcus lactis reveals a four!strand
62.
Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D.,
approach involving induction of membrane stress proteins,
and Drescher, K. (2018) Dynamic biofilm architecture
D!alanylation of the cell wall, maintenance of proton
confers individual and collective mechanisms of viral pro!
motive force, and energy conservation, J. Virol., 85, 12032!
tection, Nat. Microbiol., 3, 26!31.
12042.
63.
Scanlan, P. D., and Buckling, A. (2012) Co!evolution with
82.
Power, P. M., Sweetman, W. A., Gallacher, N. J.,
lytic phage selects for the mucoid phenotype of
Woodhall, M. R., Kumar, G. A., et al. (2009) Simple
Pseudomonas fluorescens SBW25, ISME J., 6, 1148!1158.
sequence repeats in Haemophilus influenzae, Infect. Genet.
64.
Wilkinson, B. J., and Holmes, K. M. (1979) Staphylococcus
Evol., 9, 216!228.
aureus cell surface: capsule as a barrier to bacteriophage
83.
Turkington, C. J. R., Morozov, A., Clokie, M. R. J., and
adsorption, Infect. Immun., 23, 549!552.
Bayliss, C. D. (2019) Phage!resistant phase!variant sub!
65.
Kim, M. S., Kim, Y. D., Hong, S. S., Park, K., Ko, K. S.,
populations mediate herd immunity against bacteriophage
and Myung, H. (2015) Phage!encoded colanic acid!
invasion of bacterial meta!populations, Front. Microbiol.,
degrading enzyme permits lytic phage infection of a cap!
10, 1473.
sule!forming resistant mutant Escherichia coli strain, Appl.
84.
Seed, K. D., Faruque, S. M., Mekalanos, J. J.,
Environ. Microbiol., 81, 900!909.
Calderwood, S. B., Qadri, F., and Camilli, A. (2012) Phase
66.
Reyes!Robles, T., Dillard, R. S., Cairns, L. S., Silva!
variable O antigen biosynthetic genes control expression of
Valenzuela, C. A., Housman, M., et al. (2018) Vibrio
the major protective antigen and bacteriophage receptor in
cholerae outer membrane vesicles inhibit bacteriophage
Vibrio cholerae O1, PLoS Pathog, 8, e1002917.
infection, J. Bacteriol., 200, 1!9.
85.
Komano, T. (1999) Shufflons: multiple inversion systems
67.
Manning, A. J., and Kuehn, M. J. (2011) Contribution of
and integrons, Annu. Rev. Genet., 33, 171!191.
bacterial outer membrane vesicles to innate bacterial
86.
Brouwer, M. S. M., Jurburg, S. D., Harders, F., Kant, A.,
defense, BMC Microbiol., 11, 258.
Mevius, D. J., et al. (2019) The shufflon of IncI1 plasmids
68.
Tzipilevich, E., Habusha, M., and Ben!Yehuda, S. (2017)
is rearranged constantly during different growth condi!
Acquisition of phage sensitivity by bacteria through
tions, Plasmid, 102, 51!55.
exchange of phage receptors, Cell, 168, 186!199.e12.
87.
Porter, N. T., Hryckowian, A. J., Merrill, B. D., Fuentes,
69.
Betts, A., Gifford, D. R., MacLean, R. C., and King, K. C.
J. J., Gardner, J. O., et al. (2020) Phase!variable capsular
(2016) Parasite diversity drives rapid host dynamics and
polysaccharides and lipoproteins modify bacteriophage
evolution of resistance in a bacteria!phage system,
susceptibility in Bacteroides thetaiotaomicron, Nat.
Evolution (N.Y.), 70, 969!978.
Microbiol., 5, 1170!1181.
70.
Braun, V. (2009) FhuA (TonA), the career of a protein,
88.
Quemin, E. R. J., and Quax, T. E. F. (2015) Archaeal virus!
J. Bacteriol., 191, 3431!3436.
es at the cell envelope: entry and egress, Front. Microbiol.,
71.
Chatterjee, S., and Rothenberg, E. (2012) Interaction of
6, 552.
bacteriophage l with its E. coli receptor, LamB, Viruses, 4,
89.
Hartman, R., Eilers, B. J., Bollschweiler, D., Munson!
3162!3178.
McGee, J. H., Engelhardt, H., et al. (2019) The molecular
72.
Kulikov, E. E., Golomidova, A. K., Prokhorov, N. S.,
mechanism of cellular attachment for an archaeal virus,
Ivanov, P. A., and Letarov, A. V. (2019) High!throughput
Structure, 27, 1634!1646.
LPS profiling as a tool for revealing of bacteriophage infec!
90.
Deng, L., He, F., Bhoobalan!Chitty, Y., Martinez!
tion strategies, Sci. Rep., 9, 1!10.
Alvarez, L., Guo, Y., and Peng, X. (2014) Unveiling cell
73.
Moller, A. G., Lindsay, J. A., and Read, T. D. (2019)
surface and type IV secretion proteins responsible for
Determinants of phage host range in Staphylococcus
archaeal rudivirus entry, J. Virol., 88, 10264!10268.
species, Appl. Environ. Microbiol., 85, e00209!19.
91.
Asheshov, I. N., Strelitz, F., and Hall, E. A.
(1949)
74.
Wright, R. C. T., Friman, V.!P., Smith, M. C. M., and
Antibiotics active against bacterial viruses, Br. J. Exp.
Brockhurst, M. A. (2018) Cross!resistance is modular in
Pathol., 30, 175.
bacteria!phage interactions, PLoS Biol., 16, e2006057.
92.
Cohen, S. S. (1949) Growth requirements of bacterial
75.
Christen, M., Beusch, C., Bösch, Y., Cerletti, D., Flores!
viruses, Bacteriol. Rev., 13, 1.
Tinoco, C. E., et al. (2016) Quantitative selection analysis
93.
Putnam, F. W. (1953) Bacteriophages: nature and repro!
of bacteriophage ϕCbK susceptibility in Caulobacter cres%
duction, in Advances in Protein Chemistry, Elsevier, Vol. 8,
centus, J. Mol. Biol., 428, 419!430.
pp. 175!284.
76.
Bikard, D., and Marraffini, L. A. (2012) Innate and adap!
94.
Neter, E. (1942) Effects of tyrothricin and actinomycin a
tive immunity in bacteria: mechanisms of programmed
upon bacteriophage and bacterial toxins and toxin!like
genetic variation to fight bacteriophages, Curr. Opin.
substances, J. Bacteriol., 43.
Immunol., 24, 15!20.
95.
Jones, D. (1945) The effect of antibiotic substances upon
77.
Van Houte, S., Buckling, A., and Westra, E. R. (2016)
bacteriophage, J. Bacteriol., 50, 341.
Evolutionary ecology of prokaryotic immune mechanisms,
96.
Knezevic, P., and Sabo, V. A. (2019) Combining bacterio!
Microbiol. Mol. Biol. Rev., 80, 745!763.
phages with other antibacterial agents to combat bacteria, in
78.
Riede, I., and Eschbach, M. L. (1986) Evidence that TraT
Phage Therapy: A Practical Approach, Springer, pp. 257!293.
interacts with OmpA of Escherichia coli, FEBS Lett., 205,
97.
Morita, J., Tanaka, A., Komano, T., and Oki, T. (1979)
241!245.
Inactivation of phage φ X174 by anthracycline antibiotics,
79.
Harvey, H., Bondy!Denomy, J., Marquis, H., Sztanko, K.
aclacinomycin A, doxorubicin and daunorubicin, Agric.
M., Davidson, A. R., and Burrows, L. L.
(2018)
Biol. Chem., 43, 2629!2631.
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
391
98. Duan, Z. (2016) Identification and Characterization of
118. Bebeacua, C., Lorenzo Fajardo, J. C., Blangy, S., Spinelli,
Novel Anti%Phage Compounds Using a High Throughput
S., Bollmann, S., et al. (2013) X!ray structure of a superin!
Approach, Master Thesis, University of Toronto.
fection exclusion lipoprotein from phage TP!J34 and iden!
99. Rivera!Serrano, E. E., Gizzi, A. S., Arnold, J. J., Grove,
tification of the tape measure protein as its target, Mol.
T. L., Almo, S. C., and Cameron, C. E. (2020) Viperin
Microbiol., 89, 152!65.
reveals its true function, Annu. Rev. Virol., 7, 421!446.
119. Ali, Y., Koberg, S., Heßner, S., Sun, X., Rabe, B., et al.
100. Gizzi, A. S., Grove, T. L., Arnold, J. J., Jose, J., Jangra,
(2014) Temperate Streptococcus thermophilus phages
R. K., et al. (2018) A naturally occurring antiviral ribonu!
expressing superinfection exclusion proteins of the Ltp
cleotide encoded by the human genome, Nature, 558, 610!
type, Front. Microbiol., 5, 98.
614.
120. Cumby, N., Edwards, A. M., Davidson, A. R., and
101. Fenwick, M. K., Li, Y., Cresswell, P., Modis, Y., and
Maxwell, K. L. (2012) The bacteriophage HK97 gp15
Ealick, S. E. (2017) Structural studies of viperin, an antivi!
moron element encodes a novel superinfection exclusion
ral radical SAM enzyme, Proc. Natl. Acad. Sci. USA, 114,
protein, J. Bacteriol., 194, 5012!5019.
6806!6811.
121. Cumby, N., Reimer, K., Mengin!Lecreulx, D., Davidson,
102. Bernheim, A., Millman, A., Ofir, G., Meitav, G.,
A. R., and Maxwell, K. L. (2015) The phage tail tape mea!
Avraham, C., et al. (2021) Prokaryotic viperins produce
sure protein, an inner membrane protein and a periplasmic
diverse antiviral molecules, Nature, 589, 120!124.
chaperone play connected roles in the genome injection
103. Ng, W.!L., and Bassler, B. L. (2009) Bacterial quorum!
process of E. coli phage HK 97, Mol. Microbiol., 96, 437!
sensing network architectures, Annu. Rev. Genet., 43, 197!
447.
222.
122. Lu, M. J., and Henning, U. (1994) Superinfection exclu!
104. Moreau, P., Diggle, S. P., and Friman, V. (2017) Bacterial
sion by T!even!type coliphages, Trends Microbiol., 2, 137!9.
cell!to!cell signaling promotes the evolution of resistance
123. Lu, M. J., and Henning, U. (1989) The immunity (imm)
to parasitic bacteriophages, Ecol. Evol., 7, 1936!1941.
gene of Escherichia coli bacteriophage T4, J. Virol., 63,
105. Srivastava, D., and Waters, C. M. (2012) A tangled web:
3472!3478.
regulatory connections between quorum sensing and cyclic
124. Ko, C., and Hatfull, G. F. (2018) Mycobacteriophage
di!GMP, J. Bacteriol., 194, 4485!4493.
Fruitloop gp52 inactivates Wag31 (DivIVA) to prevent het!
106. Høyland-Kroghsbo, N. M., Mærkedahl, R. B., and
erotypic superinfection, Mol. Microbiol., 108, 443!460.
Svenningsen, S. Lo (2013) A quorum!sensing!induced
125. Mahony, J., McGrath, S., Fitzgerald, G. F., and van
bacteriophage defense mechanism, MBio, 4, e00362!12.
Sinderen, D. (2008) Identification and characterization of
107. Tan, D., Svenningsen, S. Lo, and Middelboe, M. (2015)
lactococcal!prophage!carried superinfection exclusion
Quorum sensing determines the choice of antiphage
genes, Appl. Environ. Microbiol., 74, 6206!6215.
defense strategy in Vibrio anguillarum, MBio, 6, 1!10.
126. Hofer, B., Ruge, M., and Dreiseikelmann, B. (1995) The
108. Castillo, D., Rørbo, N., Jørgensen, J., Lange, J., Tan, D.,
superinfection exclusion gene (sieA) of bacteriophage P22:
et al. (2019) Phage defense mechanisms and their genomic
identification and overexpression of the gene and localiza!
and phenotypic implications in the fish pathogen Vibrio
tion of the gene product, J. Bacteriol., 177, 3080!3086.
anguillarum, FEMS Microbiol. Ecol., 95, fiz004.
127. Nesper, J., Blaß, J., Fountoulakis, M., and Reidl, J. (1999)
109. Patterson, A. G., Jackson, S. A., Taylor, C., Evans, G. B.,
Characterization of the major control region of Vibrio
Salmond, G. P. C., et al. (2016) Quorum sensing controls
cholerae bacteriophage K139: immunity, exclusion, and
adaptive immunity through the regulation of multiple
integration, J. Bacteriol., 181, 2902!2913.
CRISPR!Cas systems, Mol. Cell, 64, 1102!1108.
128. Kliem, M., and Dreiseikelmann, B. (1989) The superim!
110. Høyland-Kroghsbo, N. M., Paczkowski, J., Mukherjee, S.,
munity gene sim of bacteriophage P1 causes superinfection
Broniewski, J., Westra, E., et al. (2017) Quorum sensing
exclusion, Virology, 171, 350!355.
controls the Pseudomonas aeruginosa CRISPR!Cas adap!
129. Carballo!Ontiveros, M. A., Cazares, A., Vinuesa, P.,
tive immune system, Proc. Natl. Acad. Sci. USA, 114, 131!
Kameyama, L., and Guarneros, G. (2020) The concerted
135.
action of two B3!like prophage genes exclude superinfect!
111.
Silpe, J. E., and Bassler, B. L. (2019) A host!produced
ing bacteriophages by blocking DNA entry into
quorum!sensing autoinducer controls a phage lysis!lysoge!
Pseudomonas aeruginosa, J. Virol.,
94, e00953!20,
ny decision, Cell, 176, 268!280.
doi: 10.1128/JVI.00953!20.
112. Erez, Z., Steinberger!Levy, I., Shamir, M., Doron, S.,
130. Domingo!Calap, P., Mora!Quilis, L., and Sanjuán, R.
Stokar!Avihail, A., et al. (2017) Communication between
(2020) Social bacteriophages, Microorganisms, 8, 1!10.
viruses guides lysis!lysogeny decisions, Nature, 541, 488!
131. Ragunathan, P. T., and Vanderpool, C. K. (2019) Cryptic!
493.
prophage!encoded small protein DicB protects Escherichia
113. Stokar!Avihail, A., Tal, N., Erez, Z., Lopatina, A., and
coli from phage infection by inhibiting inner membrane
Sorek, R. (2019) Widespread utilization of peptide com!
receptor proteins, J. Bacteriol., 201, e00475!19.
munication in phages infecting soil and pathogenic bacte!
132. Dillingham, M. S., and Kowalczykowski, S. C. (2008)
ria, Cell Host Microbe, 25, 746!755.
RecBCD enzyme and the repair of double!stranded DNA
114. Bernard, C., Li, Y., Lopez, P., and Bapteste, E. (2020)
breaks, Microbiol. Mol. Biol. Rev., 72, 642!671.
Beyond arbitrium: identification of a second communica!
133. Cheng, K., Wilkinson, M., Chaban, Y., and Wigley, D. B.
tion system in Bacillus phage phi3T that may regulate host
(2020) A conformational switch in response to Chi con!
defense
mechanisms, ISME J.,
15,
545!
verts RecBCD from phage destruction to DNA repair, Nat.
549, doi: 10.1038/s41396!020!00795!9.
Struct. Mol. Biol., 27, 71!77.
115. Grayson, P., and Molineux, I. J. (2007) Is phage DNA
134. Raleigh, E. A., and Brooks, J. E. (1998) Restriction modi!
‘injected’into cells - biologists and physicists can agree,
fication systems: where they are and what they do, in
Curr. Opin. Microbiol., 10, 401!409.
Bacterial Genomes, Springer, doi: 10.1007/978!1!4615!
116. Labrie, S. J., Samson, J. E., and Moineau, S. (2010)
6369!3_8.
Bacteriophage resistance mechanisms, Nat. Rev.
135. Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A.,
Microbiol., 8, 317!327.
Wilson, G. G., and Murray, N. E. (2014) Highlights of the
117. Abedon, S. T. (2015) Bacteriophage secondary infection,
DNA cutters: a short history of the restriction enzymes,
Virol. Sin., 30, 3!10.
Nucleic Acids Res., 42, 3!19.
БИОХИМИЯ том 86 вып. 3 2021
392
ИСАЕВ и др.
136. Bertani, G., and Weigle, J. J. (1953) Host controlled varia!
general mechanism of cleavage site selection by type I
tion in bacterial viruses, J. Bacteriol., 65, 113.
restriction enzymes, EMBO J., 18, 2638!2647.
137. Luria, S. E. (1953) Host!induced modifications of viruses,
155. Studier, F. W., and Bandyopadhyay, P. K. (1988) Model for
in Cold Spring Harbor Symposia on Quantitative Biology,
how type I restriction enzymes select cleavage sites in
Cold Spring Harbor Laboratory Press, Vol. 18, pp. 237!
DNA, Proc. Natl. Acad. Sci. USA, 85, 4677!4681.
244.
156. Smith, R. M., Diffin, F. M., Savery, N. J., Josephsen, J.,
138. Berg, P., and Mertz, J. E. (2010) Personal reflections on the
and Szczelkun, M. D. (2009) DNA cleavage and methyla!
origins and emergence of recombinant DNA technology,
tion specificity of the single polypeptide restriction-modi!
Genetics, 184, 9!17.
fication enzyme LlaGI, Nucleic Acids Res., 37, 7206!7218.
139. Roberts, R. J., Belfort, M., Bestor, T., Bhagwat, A. S.,
157. Chand, M. K., Carle, V., Anuvind, K. G., and
Bickle, T. A., et al. (2003) A nomenclature for restriction
Saikrishnan, K.
(2020) DNA!mediated coupling of
enzymes, DNA methyltransferases, homing endonucleases
ATPase, translocase and nuclease activities of a Type ISP
and their genes, Nucleic Acids Res., 31, 1805!12.
restriction-modification enzyme, Nucleic Acids Res., 48,
140. Bower, E. K. M., Cooper, L. P., Roberts, G. A., White,
2594!2603.
J. H., Luyten, Y., et al. (2018) A model for the evolution of
158. Makovets, S., Titheradge, A. J. B., and Murray, N. E.
prokaryotic DNA restriction-modification systems based
(1998) ClpX and ClpP are essential for the efficient acqui!
upon the structural malleability of Type I restriction-mod!
sition of genes specifying type IA and IB restriction sys!
ification enzymes, Nucleic Acids Res., 46, 9067!9080.
tems, Mol. Microbiol., 28, 25!35.
141. Pingoud, A., Fuxreiter, M., Pingoud, V., and Wende, W.
159. Simons, M., Diffin, F. M., and Szczelkun, M. D. (2014)
(2005) Type II restriction endonucleases: structure and
ClpXP protease targets long!lived DNA translocation
mechanism, Cell. Mol. Life Sci., 62, 685.
states of a helicase!like motor to cause restriction allevia!
142. Pingoud, A., Wilson, G. G., and Wende, W. (2014) Type II
tion, Nucleic Acids Res., 42, 12082!12091.
restriction endonucleases - a historical perspective and
160. Dybvig, K., Sitaraman, R., and French, C. T. (1998) A
more, Nucleic Acids Res., 42, 7489!7527.
family of phase!variable restriction enzymes with differing
143. Nelson, M., Raschke, E., and McClelland, M. (1993)
specificities generated by high!frequency gene rearrange!
Effect of site!specific methylation on restriction endonu!
ments, Proc. Natl. Acad. Sci. USA, 95, 13923!13928.
cleases and DNA modification methyltransferases, Nucleic
161. Rao, D. N., Dryden, D. T. F., and Bheemanaik, S. (2014)
Acids Res., 21, 3139.
Type III restriction-modification enzymes: a historical
144. Morgan, R. D., Bhatia, T. K., Lovasco, L., and Davis, T. B.
perspective, Nucleic Acids Res., 42, 45!55.
(2008) MmeI: a minimal Type II restriction-modification
162. Butterer, A., Pernstich, C., Smith, R. M., Sobott, F.,
system that only modifies one DNA strand for host protec!
Szczelkun, M. D., and Toth, J. (2014) Type III restriction
tion, Nucleic Acids Res., 36, 6558!6570.
endonucleases are heterotrimeric: comprising one helicase!
145. Janulaitis, A., Petrusyte, M., Maneliene, Z.,
nuclease subunit and a dimeric methyltransferase that binds
Klimasauskas, S., and Butkus, V. (1992) Purification and
only one specific DNA, Nucleic Acids Res., 42, 5139!5150.
properties of the Eco57l restriction endonuclease and
163. Gupta, Y. K., Chan, S.!H., Xu, S., and Aggarwal, A. K.
methylas - prototypes of a new class (type IV), Nucleic
(2015) Structural basis of asymmetric DNA methylation
Acids Res., 20, 6043!6049.
and ATP!triggered long!range diffusion by EcoP15I, Nat.
146. Sorokin, V., Severinov, K., and Gelfand, M. S. (2009)
Commun., 6, 1!10.
Systematic prediction of control proteins and their DNA
164. Janscak, P., Sandmeier, U., Szczelkun, M. D., and Bickle,
binding sites, Nucleic Acids Res., 37, 441!451.
T. A. (2001) Subunit assembly and mode of DNA cleavage
147. Klimuk, E., Bogdanova, E., Nagornykh, M., Rodic, A.,
of the type III restriction endonucleases EcoP1I and
Djordjevic, M., et al. (2018) Controller protein of restric!
EcoP15I, J. Mol. Biol., 306, 417!431.
tion-modification system Kpn2I affects transcription of its
165. Van Aelst, K., Tуth, J., Ramanathan, S. P., Schwarz, F. W.,
gene by acting as a transcription elongation roadblock,
Seidel, R., and Szczelkun, M. D. (2010) Type III restric!
Nucleic Acids Res., 46, 10810!10826.
tion enzymes cleave DNA by long!range interaction
148. Zakharova, M., Minakhin, L., Solonin, A., and
between sites in both head!to!head and tail!to!tail inverted
Severinov, K. (2004) Regulation of RNA polymerase pro!
repeat, Proc. Natl. Acad. Sci., 107, 9123!9128.
moter selectivity by covalent modification of DNA, J. Mol.
166. Schwarz, F. W., Tóth, J., van Aelst, K., Cui, G.,
Biol., 335, 103!111.
Clausing, S., et al. (2013) The helicase!like domains of
149. Murray, N. E. (2000) Type I restriction systems: sophisti!
type III restriction enzymes trigger long!range diffusion
cated molecular machines (a legacy of Bertani and Weigle),
along DNA, Science, 340, 353!356.
Microbiol. Mol. Biol. Rev., 64, 412!434.
167. Ahmad, I., Kulkarni, M., Gopinath, A., and
150. Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., and
Saikrishnan, K. (2018) Single!site DNA cleavage by Type
Wilson, G. G. (2014) Type I restriction enzymes and their
III restriction endonuclease requires a site!bound enzyme
relatives, Nucleic Acids Res., 42, 20!44.
and a trans!acting enzyme that are ATPase!activated,
151. Gao, Y., Cao, D., Zhu, J., Feng, H., Luo, X., et al. (2020)
Nucleic Acids Res., 46, 6229!6237.
Structural insights into assembly, operation and inhibition
168. Srikhanta, Y. N., Fox, K. L., and Jennings, M. P. (2010)
of a type I restriction-modification system, Nat.
The phasevarion: phase variation of type III DNA methyl!
Microbiol., 5, 1107!1118.
transferases controls coordinated switching in multiple
152. Neaves, K. J., Cooper, L. P., White, J. H., Carnally, S. M.,
genes, Nat. Rev. Microbiol., 8, 196!206.
Dryden, D. T. F., et al. (2009) Atomic force microscopy of
169. Weigele, P., and Raleigh, E. A. (2016) Biosynthesis and
the EcoKI Type I DNA restriction enzyme bound to DNA
function of modified bases in bacteria and their viruses,
shows enzyme dimerization and DNA looping, Nucleic
Chem. Rev., 116, 12655!12687.
Acids Res., 37, 2053!2063.
170. Tock, M. R., and Dryden, D. T. (2005) The biology of
153. Bianco, P. R., and Hurley, E. M. (2005) The type I restriction
restriction and anti!restriction, Curr. Opin. Microbiol., 8,
endonuclease EcoR124I, couples ATP hydrolysis to bidirec!
466!472.
tional DNA translocation, J. Mol. Biol., 352, 837!859.
171. Loenen, W. A. M., and Raleigh, E. A. (2014) The other
154. Janscak, P., MacWilliams, M. P., Sandmeier, U., Nagaraja,
face of restriction: modification!dependent enzymes,
V., and Bickle, T. A. (1999) DNA translocation blockage, a
Nucleic Acids Res., 42, 56!69.
БИОХИМИЯ том 86 вып. 3 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА I
393
172. Czapinska, H., Kowalska, M., Zagorskaitė, E., Manakova,
enzymes with DNA methyltransferase, protein kinase and
E., Slyvka, A., et al. (2018) Activity and structure of
ATPase activity, Virology, 477, 100!109.
EcoKMcrA, Nucleic Acids Res., 46, 9829!9841.
191. Goldfarb, T., Sberro, H., Weinstock, E., Cohen, O.,
173. Siwek, W., Czapinska, H., Bochtler, M., Bujnicki, J. M.,
Doron, S., et al. (2015) BREX is a novel phage resistance
and Skowronek, K. (2012) Crystal structure and mecha!
system widespread in microbial genomes, EMBO J., 34,
nism of action of the N6!methyladenine!dependent type
169!183.
IIM restriction endonuclease R. DpnI, Nucleic Acids Res.,
192. Pu, T., Mei, Z., Zhang, W., Liang, W., Zhou, X., et al.
40, 7563!7572.
(2020) An in vitro DNA phosphorothioate modification
174. Liu, G., Ou, H.!Y., Wang, T., Li, L., Tan, H., et al. (2010)
reaction, Mol. Microbiol., 113, 452!463.
Cleavage of phosphorothioated DNA and methylated
193. Gordeeva, J., Morozova, N., Sierro, N., Isaev, A.,
DNA by the type IV restriction endonuclease ScoMcrA,
Sinkunas, T., et al. (2019) BREX system of Escherichia coli
PLoS Genet., 6, e1001253.
distinguishes self from non!self by methylation of a specif!
175. Sutherland, E., Coe, L., and Raleigh, E. A. (1992) McrBC:
ic DNA site, Nucleic Acids Res., 47, 253!265.
a multisubunit GTP!dependent restriction endonuclease,
194. LeGault, K., Hays, S. G., Angermeyer, A., McKitterick, A.
J. Mol. Biol., 225, 327!348.
C., Johura, F., et al. (2020) Temporal shifts in antibiotic
176. Xu, S., Corvaglia, A. R., Chan, S.!H., Zheng, Y., and
resistance elements govern virus!pathogen conflicts,
Linder, P. (2011) A type IV modification!dependent
bioRxiv, doi: 10.1101/2020.12.16.423150.
restriction enzyme SauUSI from Staphylococcus aureus
195. Hui, W., Zhang, W., Kwok, L.!Y., Zhang, H., Kong, J., and
subsp. aureus USA300, Nucleic Acids Res., 39, 5597!5610.
Sun, T. (2019) Identification and functional analysis of the
177. Vasu, K., and Nagaraja, V. (2013) Diverse functions of
pglX gene of Lactobacillus casei Zhang bacteriophage exclu!
restriction-modification systems in addition to cellular
sive (BREX) system, Appl. Environ. Microbiol., AEM!01001.
defense, Microbiol. Mol. Biol. Rev., 77, 53!72.
196. Atanasiu, C., Su, T. J., Sturrock, S. S., and Dryden, D. T.
178. Murphy, J., Mahony, J., Ainsworth, S., Nauta, A., and van
F. (2002) Interaction of the ocr gene 0.3 protein of bacte!
Sinderen, D. (2013) Bacteriophage orphan DNA methyl!
riophage T7 with Eco KI restriction/modification enzyme,
transferases: insights from their bacterial origin, function,
Nucleic Acids Res., 30, 3936!3944.
and occurrence, Appl. Environ. Microbiol., 79, 7547!7555.
197. Bandyopadhyay, P. K., Studier, F. W., Hamilton, D. L., and
179. Adhikari, S., and Curtis, P. D. (2016) DNA methyltrans!
Yuan, R. (1985) Inhibition of the type I restriction-modi!
ferases and epigenetic regulation in bacteria, FEMS
fication enzymes EcoB and EcoK by the gene 0.3 protein
Microbiol. Rev., 40, 575!591.
of bacteriophage T7, J. Mol. Biol., 182, 567!578.
180. Kobayashi, I. (2001) Behavior of restriction-modification
198. Ofir, G., Melamed, S., Sberro, H., Mukamel, Z.,
systems as selfish mobile elements and their impact on
Silverman, S., et al. (2018) DISARM is a widespread bac!
genome evolution, Nucleic Acids Res., 29, 3742!3756.
terial defence system with broad anti!phage activities, Nat.
181. Ershova, A. S., Rusinov, I. S., Spirin, S. A., Karyagina, A.
Microbiol., 3, 90!98.
S., and Alexeevski, A. V. (2015) Role of restriction-modi!
199. Mahankali, M., Alter, G., and Gomez!Cambronero, J.
fication systems in prokaryotic evolution and ecology,
(2015) Mechanism of enzymatic reaction and protein!pro!
Biochemistry (Moscow), 80, 1373!1386.
tein interactions of PLD from a 3D structural model, Cell.
182. Seib, K. L., Srikhanta, Y. N., Atack, J. M., and Jennings,
Signal., 27, 69!81.
M. P. (2020) Epigenetic regulation of virulence and immu!
200. Thiaville, J. J., Kellner, S. M., Yuan, Y., Hutinet, G.,
noevasion by phase!variable restriction-modification sys!
Thiaville, P. C., et al. (2016) Novel genomic island modi!
tems in bacterial pathogens, Annu. Rev. Microbiol., 74, 655!
fies DNA with 7!deazaguanine derivatives, Proc. Natl.
671.
Acad. Sci. USA, 113, E1452!E1459.
183. Dimitriu, T., Szczelkun, M. D., and Westra, E. R. (2020)
201. Iyer, L. M., Zhang, D., Maxwell Burroughs, A., and
Evolutionary ecology and interplay of prokaryotic innate
Aravind, L. (2013) Computational identification of novel
and adaptive immune systems, Curr. Biol., 30, R1189!
biochemical systems involved in oxidation, glycosylation
R1202.
and other complex modifications of bases in DNA, Nucleic
184. Lomovskaia, N. D., Mkrtumian, N. M., and Gostimskaya,
Acids Res., 41, 7635!7655.
N. L. (1970) Isolation and characterization of the
202. Hutinet, G., Kot, W., Cui, L., Hillebrand, R.,
actinophage of Streptomyces coelicolor, Genetica, 6, 135.
Balamkundu, S., et al. (2019) 7!Deazaguanine modifica!
185. Chinenova, T. A., Mkrtumian, N. M., and Lomovskaia,
tions protect phage DNA from host restriction systems,
N. D. (1982) Genetic characteristics of a new phage resis!
Nat. Commun., 10, 1!12.
tance trait in Streptomyces coelicolor A3(2), Genetika, 18,
203. Kot, W., Olsen, N. S., Nielsen, T. K., Hutinet, G., de
1945!1952.
Crécy!Lagard, V., et al. (2020) Detection of preQ0 deaza!
186. Chater, K. F. (1986) Streptomyces phages and their applica!
guanine modifications in bacteriophage CAjan DNA using
tions to Streptomyces genetics, in The Bacteria, Academic
Nanopore sequencing reveals same hypermodification at two
Press Orlando, vol. 9, pp. 119!158.
distinct DNA motifs, Nucleic Acids Res., 48, 10383!10396.
187. Bedford, D. J., Laity, C., and Buttner, M. J. (1995) Two
204. Wang, L., Jiang, S., Deng, Z., Dedon, P. C., and Chen, S.
genes involved in the phase!variable phi C31 resistance
(2019) DNA phosphorothioate modification - a new
mechanism of Streptomyces coelicolor A3(2), J. Bacteriol.,
multi!functional epigenetic system in bacteria, FEMS
177, 4681!9.
Microbiol. Rev., 43, 109!122.
188. Laity, C., Chater, K. F., Lewis, C. G., and Buttner, M. J.
205. Xiong, L., Liu, S., Chen, S., Xiao, Y., Zhu, B., et al. (2019)
(1993) Genetic analysis of the phi C31!specific phage
A new type of DNA phosphorothioation!based antiviral
growth limitation (Pgl) system of Streptomyces
system in archaea, Nat. Commun., 10, 1!11.
coelicolor A3(2), Mol. Microbiol., 7, 329!36.
206. Xiong, X., Wu, G., Wei, Y., Liu, L., Zhang, Y., et al. (2020)
189. Sumby, P., and Smith, M. C. M. (2002) Genetics of the
SspABCD!SspE is a phosphorothioation!sensing bacterial
phage growth limitation (Pgl) system of Streptomyces coeli%
defence system with broad anti!phage activities, Nat.
color A3(2), Mol. Microbiol., 44, 489!500.
Microbiol., 5, 917!928.
190. Hoskisson, P. A., Sumby, P., and Smith, M. C. M. (2015)
207. Zhou, X., He, X., Liang, J., Li, A., Xu, T., et al. (2005) A
The phage growth limitation system in Streptomyces coeli%
novel DNA modification by sulphur, Mol. Microbiol., 57,
color A(3)2 is a toxin/antitoxin system, comprising
1428!1438.
БИОХИМИЯ том 86 вып. 3 2021
394
ИСАЕВ и др.
208. Wang, L., Chen, S., Xu, T., Taghizadeh, K., Wishnok, J. S.,
backbone S!modification in Salmonella, Nucleic Acids
et al. (2007) Phosphorothioation of DNA in bacteria by
Res., 38, 7133!41.
dnd genes, Nat. Chem. Biol., 3, 709!710.
215. Cao, B., Cheng, Q., Gu, C., Yao, F., DeMott, M. S., et al.
209. He, W., Huang, T., Tang, Y., Liu, Y., Wu, X., et al. (2015)
(2014) Pathological phenotypes and in vivo DNA cleavage
Regulation of DNA phosphorothioate modification in
by unrestrained activity of a phosphorothioate!based
Salmonella enterica by DndB, Sci. Rep., 5, 12368.
restriction system in Salmonella, Mol. Microbiol., 93, 776!
210. You, D., Wang, L., Yao, F., Zhou, X., and Deng, Z. (2007)
785.
A Novel DNA Modification by sulfur: DndA is a NifS!like
216. Gan, R., Wu, X., He, W., Liu, Z., Wu, S., et al. (2014)
cysteine desulfurase capable of assembling DndC as an
DNA phosphorothioate modifications influence the global
iron!sulfur cluster protein in Streptomyces lividans,
transcriptional response and protect DNA from double!
Biochemistry, 46, 6126!6133.
stranded breaks, Sci. Rep., 4, 6642.
211. Yao, F., Xu, T., Zhou, X., Deng, Z., and You, D. (2009)
217. Cao, B., Chen, C., DeMott, M. S., Cheng, Q., Clark,
Functional analysis of spfD gene involved in DNA phos!
T. A., et al. (2014) Genomic mapping of phosphorothioates
phorothioation in Pseudomonas fluorescens Pf0!1, FEBS
reveals partial modification of short consensus sequences,
Lett., 583, 729!733.
Nat. Commun., 5, 1!13.
212. Wu, Y., Tang, Y., Dong, X., Zheng, Y. Y.,
218. Li, J., Chen, Y., Zheng, T., Kong, L., Zhu, S., et al. (2019)
Haruehanroengra, P., et al. (2020) RNA phosphorothioate
Quantitative mapping of DNA phosphorothioatome
modification in prokaryotes and eukaryotes, ACS Chem.
reveals phosphorothioate heterogeneity of low modifica!
Biol., 15, 1301!1305.
tion frequency, PLoS Genet., 15, e1008026.
213. Tong, T., Chen, S., Wang, L., Tang, Y., Ryu, J. Y., et al.
219. Wu, X., Cao, B., Aquino, P., Chiu, T.!P., Chen, C., et al.
(2018) Occurrence, evolution, and functions of DNA
(2020) Epigenetic competition reveals density!dependent
phosphorothioate epigenetics in bacteria, Proc. Natl. Acad.
regulation and target site plasticity of phosphorothioate
Sci., 115, E2988!E2996.
epigenetics in bacteria, Proc. Natl. Acad. Sci. USA, 117,
214. Xu, T., Yao, F., Zhou, X., Deng, Z., and You, D. (2010) A
14322!14330, doi: 10.1073/pnas.2002933117.
novel host!specific restriction system associated with DNA
MICROBIAL ARSENAL OF ANTIVIRAL DEFENSES - PART I
Review
A. B. Isaev1*, O. S. Musharova1,2, and K. V. Severinov1,3*
1 Skolkovo Institute of Science and Technology, 143028 Moscow, Russia; E%mail: tcft18@gmail.com
2 Institute of Molecular Genetics, Russian Academy of Sciences, 123182 Moscow, Russia
3 Waksman Institute of Microbiology, Piscataway, NJ 08854, USA; E%mail: severik@waksman.rutgers.edu
Bacteriophages or phages are viruses that infect bacterial cells (for the scope of this review we will also consider virus!
es that infect Archaea). Constant threat of phage infection is a major force that shapes evolution of the microbial
genomes. To withstand infection, bacteria had evolved numerous strategies to avoid recognition by phages or to direct!
ly interfere with phage propagation inside the cell. Classical molecular biology and genetic engineering have been
deeply intertwined with the study of phages and host defenses. Nowadays, owing to the rise of phage therapy, broad
application of CRISPR!Cas technologies, and development of bioinformatics approaches that facilitate discovery of
new systems, phage biology experiences a revival. This review describes variety of strategies employed by microbes to
counter phage infection, with a focus on novel systems discovered in recent years. First chapter covers defense asso!
ciated with cell surface, role of small molecules, and innate immunity systems relying on DNA modification.
Keywords: bacteriophages, phage!host interactions, antiviral defence, immunity systems, CRISPR!Cas, restriction!
modification, BREX, DISARM, Phosphorothioate, Dnd systems
БИОХИМИЯ том 86 вып. 3 2021