БИОХИМИЯ, 2021, том 86, вып. 4, с. 469 - 482
УДК 577.218
ОСОБЕННОСТИ РЕГУЛЯЦИИ СИНТЕЗА БЕЛКОВ L11 ОПЕРОНА
ТЕРМОФИЛЬНЫХ БАКТЕРИЙ И L1 ОПЕРОНА АРХЕЙ
© 2021
А.О. Михайлина1*, Е.Ю. Никонова1, О.С. Костарева1,
В. Пиндл2, М. Эрлахер3, С.В. Тищенко1
1 Федеральное государственное бюджетное учреждение науки Институт белка Российской академии наук,
142290 Пущино, Россия; электронная почта: alisamikhaylina15@gmail.com
2 Медицинский университет Инсбрука, Институт медицинской химии и биохимии, 6020 Инсбрук, Австрия
3 Медицинский университет Инсбрука, Институт геномики и РНКомики, 6020 Инсбрук, Австрия
Поступила в редакцию 15.10.2020
После доработки 24.01.2021
Принята к публикации 24.01.2021
Рибосомный белок L1 - консервативный двухдоменный белок, участвующий в формировании L1 выступа
большой рибосомной субчастицы. В условиях дефицита 23S рибосомной РНК белок ингибирует собствен
ную трансляцию, связываясь со специфическим участком в последовательности мРНК своего оперона
(L11 оперона бактерий и L1 оперона архей). В работе продемонстрирована консервативность регулятор
ных свойств рибосомного белка L1 и его домена I в термофильных бактериях родов Thermus и Thermotoga и
в галофильной архее Haloarcula marismortui. Наряду с этим, выявлены особенности регуляции оперона в тер
мофильных бактериях, предполагающие наличие двух регуляторных участков.
КЛЮЧЕВЫЕ СЛОВА: регуляция экспрессии, L11 оперон, L1 оперон, р белки, мРНК.
DOI: 10.31857/S0320972521040023
ВВЕДЕНИЕ
Тем не менее чёткого подтверждения регуля
торных свойств рибосомного белка L1 (р белка)
Рибосомный белок L1 участвует в формиро
в других бактериях и археях на сегодняшний
вании L1 выступа большой рибосомной субчас
день не существует. Биоинформатический ана
тицы и является регулятором синтеза белков
лиз L11 оперонов продемонстрировал наличие
своего оперона. Белок L1 связывается со специ
потенциальных регуляторных сайтов перед ге
фическим сайтом на собственной мРНК и инги
ном белка L11 (у Proteobacteria, Spirochaetes,
бирует трансляцию всех цистронов рибосомных
Thermotoga и Tenericutes) и L1 (у Cyanobacteria,
белков оперона. Детально изучена регуляция
Actinobacteria и Chloroflexi); а в 40% геномов типа
L11 оперона бактерии Escherichia coli (включаю
Firmicutes обнаружены два потенциальных
щего гены рибосомных белков L1 и L11) [1] и
L1 связывающих участка - перед генами белков
L1 оперона архей рода Methanococcus (включаю
L11 и L1 [5]. Ранее мы локализовали участки
щего гены рибосомных белков L1, L10 и L12)
связывания белка L1 на мРНК L11 оперонов
[2]. Участок связывания L1 на мРНК E. coli рас
Thermus thermophilus и Thermotoga maritimа, опре
полагается в лидерной последовательности
делив константы диссоциации комплексов L1-
мРНК белка L11 [3], а у архей рода Methanococcus -
мРНК [6]. В данной статье мы демонстрируем
в начале кодирующей последовательности гена
наличие двух L1 связывающих участков на
белка L1 (между 29 м и 67 м нуклеотидами) [4].
мРНК L11 оперонов исследуемых термофиль
L1 связывающие участки мРНК имеют высо
ных бактерий, используя систему транскрип
кую гомологию с участком связывания этого
ции трансляции in vitro.
белка на 23S рРНК - как первичной, так и вто
В галофильных археях ген белка L1 котран
ричной структуры.
скрибируется с генами белков L10 и L12. Анализ
последовательностей L1 оперона выявил пред
положительный участок связывания белка L1 в
Принятые сокращения: 5′ НТО - 5′ нетранслируе
нетранслируемой области гена белка L1 [7], од
мая область; БСА - бычий сывороточный альбумин;
нако исследование возможности связывания
р белки - рибосомные белки; SD - последовательность
Шайна-Дальгарно; SPR - поверхностный плазмонный
этого участка с белком и регуляции синтеза бел
резонанс.
ков оперона не проводилось. В данной работе
* Адресат для корреспонденции.
мы определили L1 связывающий участок на
469
470
МИХАЙЛИНА и др.
мРНК L1 оперона Haloarcula marismortui и пока
Генетические конструкции TthL1, TthL1dI,
зали регуляторные свойства р белка L1 из этого
TmaL1 и TmaL1dI были получены в нашей лабо
организма.
ратории ранее [6, 8].
Структурно кинетический анализ комплек
Создание генетических конструкций, несущих
сов L1-РНК [8] показал, что ведущую роль в
фрагменты L11 оперона T. maritima, T. ther
этих взаимодействиях играет белковый домен I.
mophilus и L1 оперона H. marismortui. В качестве
Ранее было показано, что домен I белка L1
матриц в сопряженной системе транскрипции
T. thermophilus (TthL1dI) обладает регуляторны
трансляции E. coli были использованы плазми
ми свойствами полноразмерного белка, ингиби
ды на основе вектора pUC18, содержащие фраг
руя синтез архейного белка L1 Methanococcus
менты L11 оперона T. maritima, T. thermophilus и
vannielli in vitro [9]. В данной работе демонстри
L1 оперона H. marismortui под контролем
руется консервативность взаимодействий бел
Т7 промотора. Плазмиды pTmaL11/L1( 50) и
ка L1 с мРНК, поскольку домены I р белков L1
pTthL11/L1( 46) содержали гены белков L11 и
T. thermophilus, T. maritimа и H. marismortui обла
L1, а также 50 или 46 нуклеотидов (н.) 5′ нетранс
дают регуляторными свойствами полноразмер
лируемой области
(5′ НТО) L11 оперона
ных белков L1 в системе in vitro.
T. maritima и T. thermophilus соответственно.
Плазмида pHmaL1/L10( 74) содержала гены
белков HmaL1 и HmaL10 и 74 нуклеотида
МАТЕРИАЛЫ И МЕТОДЫ
5′ НТО L1 оперона H. marismortui (рис. 1).
Для отдельного исследования двух L1 регу
Получение генетических конструкций, несу
ляторных участков мРНК T. maritima и T. ther/
щих гены полноразмерного белка L1 и его домена I
mophilus были получены укороченные конструк
H. marismortui (HmaL1, HmaL1dI). Для получе
ции. Плазмиды pTthL1( 100) и pTmaL1( 25) со
ния белков HmaL1 и HmaL1dI были созданы
держали только ген белка L1 и последние 100
экспрессионные векторы на основе рЕТ 11a
или 25 нуклеотидов белка L11 (рис. 1). При ис
(pET 11a_HmaL1 и pET 11a_HmaL1dI). В соот
пользовании в качестве матрицы укороченной
ветствии с известной нуклеотидной последова
конструкции, содержащей ген белка TthL1 и
тельностью, кодирующей HmaL1, были синте
последние 100 н. гена белка TthL11, включая об
зированы праймеры
(«Евроген», Россия)
ласть перекрывания открытых рамок считыва
(табл. 1), содержащие последовательности для
ния генов белков TthL11 и TthL1 (pTthL1( 100)),
расщепления сайт специфическими эндонук
в сопряженной системе транскрипции трансля
леазами рестрикции FauNDI и BamHI, необхо
ции белок L1 не синтезировался (данные не
димые для вставки гена в экспрессионный век
представлены). По всей видимости, в T. ther/
тор рЕТ 11a. В качестве матрицы для получения
mophilus перед геном белка L1 нет собственной
полноразмерного белка была использована ге
последовательности SD, а его трансляция со
номная ДНК H. marismortui.
пряжена с трансляцией белка L11. Для того что
Домен II белка L1 является вставкой в до
бы оценить способность белка TthL1 регулиро
мен I. Ген домена I получен с помощью метода
вать свой синтез при взаимодействии с участком
перекрывающихся участков с использованием
мРНК в области перекрывания генов р белков
4 х праймеров и 3 х ПЦР (полимеразная цепная
L11 и L1, мы добавили к 5′ концу укороченной
реакция). Праймеры P1 и P3 (табл. 1) были ис
конструкции pTthL1( 100) участок, содержащий
пользованы для амплификации фрагмента ДНК,
последовательность SD (рис. 1). Плазмиды
содержащего участок до вырезаемого домена II и
pTmaL11( 50) и pTthL11( 46) содержали только
фрагмент после него. Пара праймеров P2 и P4
ген белка L11 и 50/46 нуклеотидов 5′ НТО
(табл. 1) была использована во второй ПЦР для
T. maritima и T. thermophilus соответственно
амплификации фрагмента ДНК, содержащего
(рис. 1).
участок после вырезаемого домена и фрагмента
Согласно известной нуклеотидной последо
до него. В качестве матрицы для ПЦР была ис
вательностью L11 оперона T. maritima, T. ther/
пользована плазмида pET 11a, несущая ген бел
mophilus и L1 оперона H. marismortui, были син
ка HmaL1. Таким образом, оба амплифициро
тезированы праймеры
(«Евроген», Россия)
ванных фрагмента содержали перекрывающиеся
(табл. 1), несущие сайты узнавания эндонуклеа
участки. Эти фрагменты смешивали, денатури
зами рестрикции, необходимые для вставки ге
ровали и отжигали друг на друга для получения
на в экспрессионный вектор рUC18. Прямой
гетеродуплекса, который затем использовали в
праймер содержал последовательность промо
третьей ПЦР для амплификации в полноразмер
тора для РНК полимеразы фага Т7 (табл. 1). На
ный фрагмент с помощью двух праймеров к гену
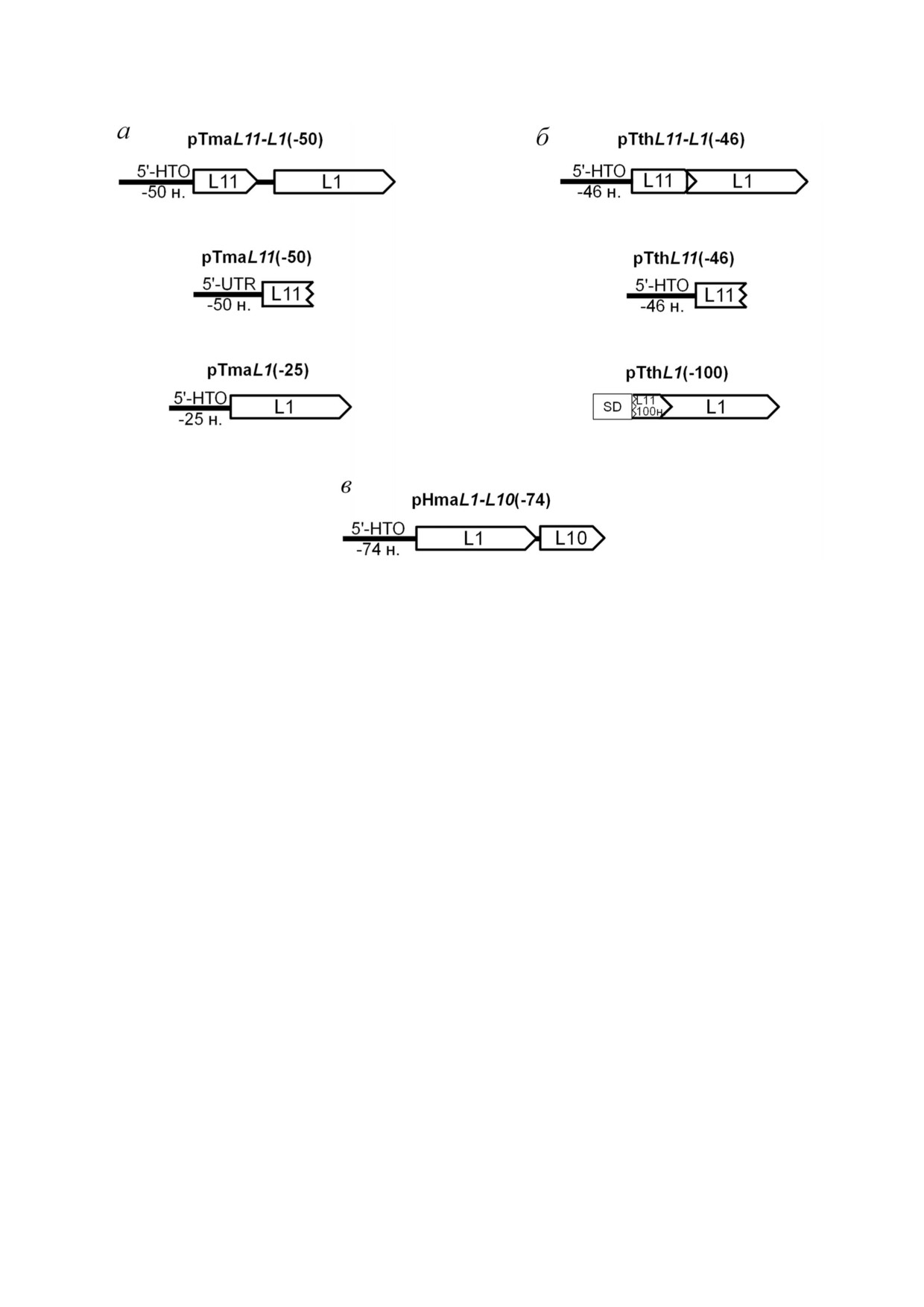
рисунке 1 представлены схемы полученных кон
rplA (прямого P1 и обратного P2).
струкций фрагментов L11 оперона и L1 оперона.
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
471
Таблица 1. Последовательности использованных праймеров
Фрагмент гена
Прямой праймер, 5′→3′
Обратный праймер, 5′→3′
HmaL1
P1
FauNDI
P2
BamHI
GGAATCCATATGGCAGATCAGGAA
CGCGGATCCTCAGGCAACCTCCATCGCA
HmaL1dI
P1
P3
CGGTACCGGTGCCAGCAGG
P4
CGGTGAGGTCATCGAAGGTCCGCACG
P2
pTthL11 L1( 46)
P5
XbaI
P6
EcoRI
AGGTCGACTCTAGAAATACGACTCACTATAG
ATGATTACGAATTCTTAGGAGTGGGGGTTGAT
AGGCCTAGAGGCGTTTGCA
GCGCA
pTthL11( 46)
P5
P7
EcoRI
ATCTAGGAATTCTTAGATCTCCAGGACCTGCTC
pTthL1( 100)
P8
XbaI
P7
AGGTCGACTCTAGAAATACGACTCACTATAG
GAAGGAGATATACATATGGTGGTGGGC
GCTCCGGAGGTGAAGG
pTmaL11 L1( 50)
P9
HindIII
P10
XmaI
CTACTGCAAAGCTTAATACGACTCACTATAG
ATCTAGCCCGGGTTACTCTTTCAACAGACTCT
TGAGAAAACGTGGGAGGAGGA
GAA
pTmaL11( 50)
P9
P10
XmaI
ATCTAGCCCGGGTTACAAGCTGTTTGCGTTC
AAATC
pTmaL1( 25)
P11
HindIII
P9
CTACTGCAAAGCTTAATACGACTCACTATAG
ATGCGGAAAGGAGGA
pHmaL1 L10( 74)
P12
HindIII
P13
EcoRI
CTACTGCAAAGCTTAATACGACTCACTATAG
ATGATTACGAATTCTTACTGAATGCGTGCGTC
CTACTCGCACGAGACAGGCATC
GGCACC
Примечание. Праймеры были использованы для клонирования генов белков HmaL1 и HmaL1dI и фрагментов L11 опе
рона T. maritima, T. thermophilus и L1 оперона H. marismortui. Сайты узнавания эндонуклеаз рестрикции подчеркнуты, се
рым выделена последовательность T7 промотора, рамкой выделен добавленный участок, содержащий последователь
ность Шайна-Дальгарно (SD).
Выделение белков L1 и L1dI. Получение бел
GGA (Gly)) [11]. Штамм E. coli BL21 (DE3)
ков TthL1, TthL1dI, TmaL1 и TmaL1dI описано
pRARE трансформировали плазмидой pET 11a,
ранее [6, 8]. Для получения штаммов суперпро
содержащей ген белка HmaL1 или HmaL1dI.
дуцентов белка L1 H. marismortui и его домена I
Несколько колоний высевали в жидкую среду
была использована система Штудиера [10]. По
LB (абб. от англ. lysogeny broth) (100 мкг/мкл ам
скольку ген белка HmaL1 содержит редкие в
пициллина и 35 мкг/мкл хлорамфеникола) и
E. coli кодоны для Gly, Arg и Ile, клетки штамма
культивировали в течение ночи при 37 °C. Затем
BL21(DE3) предварительно трансформировали
клетки пересевали в 500 мл среды LB и культи
плазмидой pRARE, которая несет гены тРНК,
вировали при температуре 37 °C до получения
узнающие редкие кодоны (AGG/AGA (Arg),
оптической плотности A590 ≈ 0,6 о.е., после чего
CGG (Arg), AUA (Ile), CUA (Leu), CCC (Pro),
к клеткам добавляли индуктор IPTG (изопро
БИОХИМИЯ том 86 вып. 4 2021
472
МИХАЙЛИНА и др.
Рис. 1. Схемы генетических конструкций фрагментов L11 оперона T. maritima (а), T. thermophilus (б) и L1 оперона
H. marismortui (в), использованных в качестве матриц в сопряженной системе транскрипции трансляции E. coli in vitro:
pTmaL11/L1( 50) и pTthL11/L1( 46) - гены белков L11 и L1, и 50/46 н. 5′ НТО перед геном белка L11; pTmaL11( 50) и
pTthL11( 46) - фрагмент гена белка L11 и 50/46 н. 5′ НТО перед ним; pTmaL1( 25) - ген белка TmaL1 и 25 н. 5′ НТО пе
ред ним; pTthL1( 100) - ген белка TthL1, последние 100 н. гена белка TthL11, включая область перекрывания открытых
рамок считывания генов белков TthL11 и TthL1, и последовательность SD; pHmaL1/L10( 74) - гены белков HmaL1 и
HmaL10 и 74 н. 5′ НТО перед геном белка HmaL1
пил β D 1 тиогалактопиранозид) до конечной
Препарат белка HmaL1 (или HmaL1dI) диа
концентрации 0,5 мМ. После добавления ин
лизовали против буфера (50 мМ Tris НСl, 3 М
дуктора клетки продолжали инкубировать в тех
KCl, pH 7,5 при 25 °C) и дополнительно очища
же условиях в течение 3 ч. Затем их осаждали
ли Superdex 75. Чистоту белковых препаратов
низкоскоростным центрифугированием (8 000 g,
оценивали при помощи Ds Na ПААГ электро
15 мин, 4 °C) и ресуспендировали в буферном
фореза [12].
растворе, содержащем 50 мМ Tris НСl, 2 М KCl,
Фрагменты мРНК T. thermophilus, T. maritima,
50 мМ MgCl2, 5 мМ β МЭ, 1 мМ ЭДТА Na2,
H. marismortui и РНК H. marismortui. Для кине
0,1 мМ PMSF, pH 7,5 при 25 °C. Клетки разру
тических исследований мы использовали полу
шали на ультразвуковом дезинтеграторе Sonic
ченные синтетически («Синтол», Россия) моди
Dismembrator 550 («Fisher Scientific», США). За
фицированные биотином фрагменты мРНК
тем центрифугированием последовательно
L11 оперона T. thermophilus и T. maritima,
осаждали клеточный дебрис (14 000 g, 30 мин,
L1 оперона H. marismortui, содержащие потен
4 °C) и рибосомы (90 000 g, 1 ч, 4 °C).
циальные регуляторные участки, а также специ
К безрибосомному лизату добавляли сульфат
фический фрагмент 23S рРНК H. marismortui,
аммония и KCl до конечной концентрации 1,5
содержащий три спирали (H76, H77, H78) [13].
и 3 M соответственно. Образец наносили на
Биотин располагался на 3′ конце фрагментов
колонку с носителем бутил сефароза
(«GE
мРНК.
Healthcare», США), уравновешенную со старто
Кинетический анализ взаимодействия бел
вым буфером (50 мМ Tris НСl, 3 М KCl, 1,5 М
ка L1 и его домена I T. thermophilus, T. maritima и
(NH4)2SO4, pH 7,5 при 25 °C). Элюирование бел
H. marismortui со специфическими фрагментами
ков проводили обратным линейным градиентом
мРНК и рРНК методом SPR. Кинетический ана
концентрации (NH4)2SO4 от 1,5 М до 0 М в стар
лиз взаимодействия белков со специфическими
товом буфере.
фрагментами РНК проводили с помощью поверх
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
473
ностного плазмонного резонанса (SPR - surface
кодирующие мРНК соответствующих оперо
plasmon resonance) [14] на системе ProteOn
нов, под контролем Т7 промотора. Анализ
XPR36 («Bio Rad», США). Биотинилированные
транскриптов проводили с помощью электро
фрагменты РНК наносили на сенсорные чипы
фореза в 5% ном ПААГ (19 : 1) в присутствии
NLC («Bio Rad», США) c иммобилизованным
8 М мочевины и электродного буфера (40 мМ
авидином [15].
Tris Ацетат, 0,2 мМ ЭДТА Na2, рН 8,0 при
Серии разведений из пяти концентраций
25 °C).
аналита (L1 или L1dI) готовили в буфере, содер
жащем 50 мМ Tris HCl, 175 мМ NaCl, 2 мМ
MgCl2, 0,05% Tween 20, pH 7,5 при 25 °C. Ско
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
рость протока составляла 30 мкл/мин, время ас
социации - 300 сек, время диссоциации -
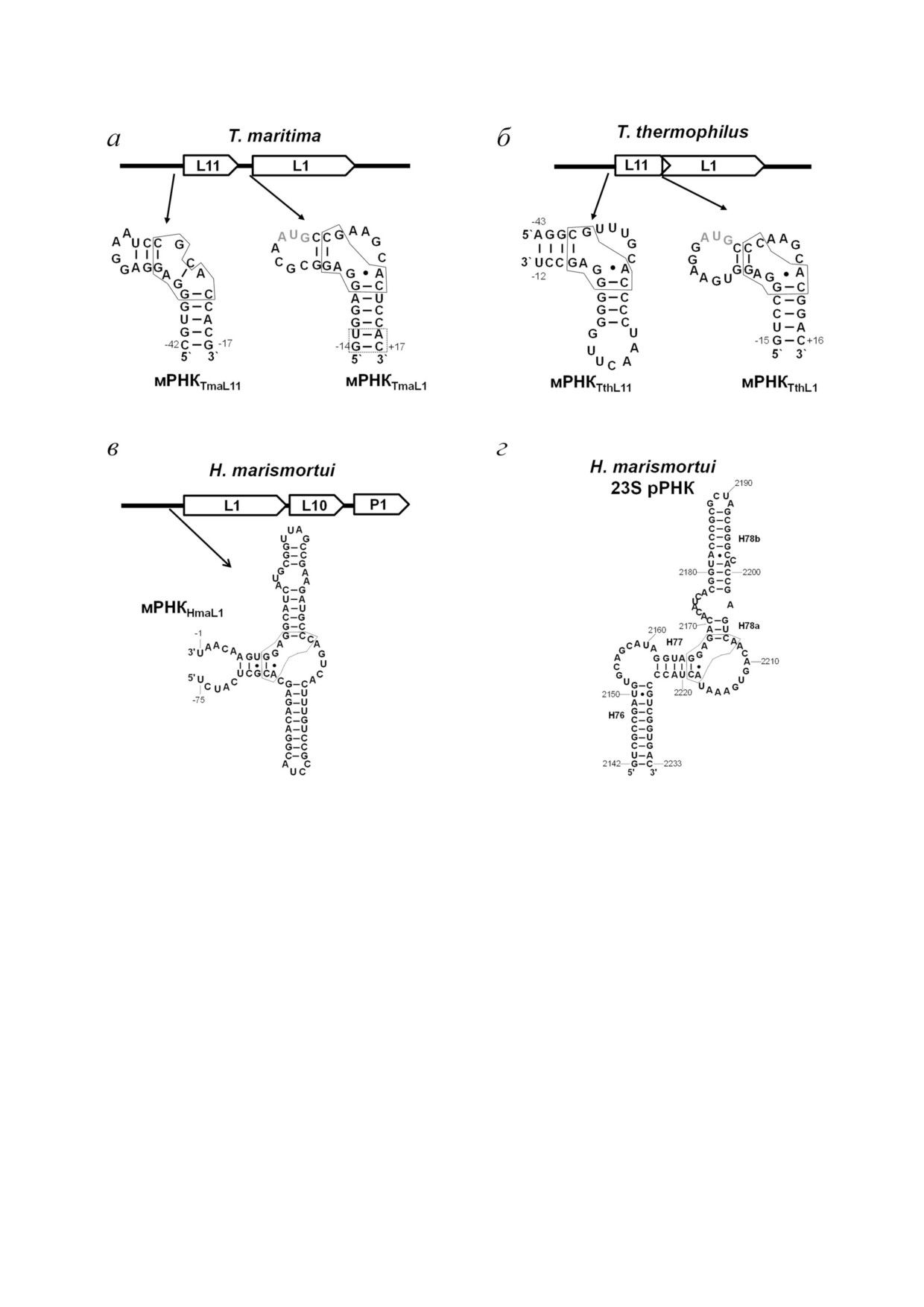
Фрагменты мРНК L11 оперона T. ther
700-1200 сек. Все эксперименты по связыванию
mophilus, T. maritima и L1 оперона H. marismortui,
проводили при температуре 25 °C.
содержащие L1 связывающие детерминанты. Ра
Набор из 3-5 сенсограмм обсчитывали в
нее мы определили, что в T. maritima имеется два
программе BIAEvaluation («Biacore», США) с
потенциальных участка связывания белка L1 на
использованием модели Ленгмюра (1 : 1) или
мРНК L11 оперона [6]. Первый участок распо
двухстадийной реакции.
ложен в лидерной последовательности мРНК
Анализ ингибирования синтеза белков
белка L11 (mRNATmaL11), второй участок включа
L11 оперона T. thermophilus, T. maritima и L1 опе
ет лидерный и кодирующий участки мРНК бел
рона H. marismortui в сопряженной системе тран
ка L1 (mRNATmaL1) (рис. 2, а). В T. thermophilus
скрипции трансляции in vitro. Для экспериментов
мы обнаружили лишь один потенциальный
в сопряженной системе транскрипции трансля
участок связывания белка L1, который располо
ции in vitro использовали набор PURExpress
жен между кодирующими частями мРНК бел
In Vitro Protein Synthesis Kit («New England
ков TthL11 и TthL1 (mRNATthL1) (рис. 2, б). Мы
Biolabs», Германия). Матрицами служили плаз
получили соответствующие фрагменты мРНК и
миды, несущие фрагменты L11 оперона T. ther/
определили сродство TthL1 и TmaL1 к этим
mophilus, T. maritima или L1 оперона H. maris/
фрагментам методом SPR [6]. Оказалось, что
mortui под контролем Т7 промотора (рис. 1). В
при взаимодействии белка L1 с этими фрагмен
качестве отрицательного контроля использова
тами мРНК, образуются стабильные комплексы
ли плазмиду, несущую ген р белка L3 из M. jan/
TthL1-mRNATthL1 и TmaL1-mRNATmaL11, однако
naschii (MjaL3). Реакцию проводили согласно
комплекс TmaL1-mRNATmaL1 не формируется.
рекомендациям производителя в присутствии
Несмотря на то, что все основные детерминан
L [35S]метионина в концентрации 11 мКи/мл
ты для РНК белкового взаимодействия были
(«PerkinElmer», США). Для ингибирования син
сохранены, фрагмент mRNATmaL1 не содержал
теза белков с плазмиды в реакционную смесь
нуклеотид С+17, который, согласно данным о
добавляли белок L1 или его домен I (0-20 мкМ).
структуре комплекса TthL1-mRNA, способен
В качестве контроля использовали бычий сыво
образовывать две закрытые от растворителя во
роточный альбумин (БСА) в тех же количествах.
дородные связи с белком L1, которые ранее счи
Для опытов по конкуренции в смесь добавляли
тались несущественными для РНК белкового
специфический для белка L1 фрагмент 23S
взаимодействия [16]. Мы получили удлинённый
рРНК до 20 мкМ. Реакцию проводили при 37 °C
на 2 пары нуклеотидов фрагмент mRNATmaL1
в течение 1 ч, после чего к смеси добавляли бу
(рис. 2, а) и показали, что он образует стабиль
фер для Ds Na ПААГ электрофореза и наноси
ный комплекс с белком L1.
ли на полиакриламидный гель.
Детальный анализ геномной последователь
Анализ способности белка L1 ингибировать
ности L11 оперона T. thermophilus на основе го
in vitro синтез мРНК L11 оперона T. thermophilus и
мологии со структурно консервативным
T. maritima, а также L1 оперона H. marismortui.
L1 связывающим модулем [17] позволил обна
Фрагменты мРНК были получены методом
ружить второй предположительный участок
транскрипции in vitro с использованием
связывания белка L1 на мРНК L11 оперона
РНК полимеразы фага Т7, как в отсутствие
T. thermophilus, который оказался расположен
р белка L1, так и в присутствии его 20 мкМ. В
перед геном белка L11 (mRNATthL11) (рис. 2, б).
качестве матрицы использовали плазмиды
Взаимодействие с этим фрагментом мРНК так
pTthL11( 46), pTmaL11( 50) и pHmaL1/L10( 74),
же было исследовано.
линеаризованные с помощью эндонуклеаз рест
Опероны, регулируемые белком L1 в археях,
рикции EcoRI, XmaI и EcoRI соответственно
отличаются от бактериальных. В H. marismortui
(табл. 1, рис. 1). Плазмиды содержали гены,
ген белка L1 котранскрибируется с генами бел
2 БИОХИМИЯ том 86 вып. 4 2021
474
МИХАЙЛИНА и др.
Рис. 2. а - Организация генов в L11 опероне T. maritima. Пунктирной рамкой на фрагменте mRNATmaL1 выделены добав
ленные пары нуклеотидов. Нуклеотиды 5′ НТО соответствующего гена обозначены знаком ( ), нуклеотиды смысловой
части мРНК обозначены знаком (+); б - организация генов в L11 опероне T. thermophilus; в - организация генов в L1 опе
роне H. marismortui; г - вторичная структура специфического фрагмента 23S рРНК H. marismortui. Также на рисунке пред
ставлены предположительные вторичные структуры фрагментов мРНК, с которыми может взаимодействовать р белок L1.
Стрелками показано положение L1 связывающих участков на соответствующей полицистронной мРНК. На фрагментах
мРНК в рамке выделены нуклеотиды, образующие консервативные контакты с белком L1, серым показан старт кодон
ков L10 и L12. В лидерной последовательности
деленными участками на мРНК соответствую
мРНК белка L1 H. marismortui находится учас
щих оперонов мы получили биотинилирован
ток, гомологичный консервативному сайту свя
ные фрагменты мРНК, содержащие эти предпо
зывания данного белка на мРНК, также схожий
ложительные L1 связывающие участки. Мето
с сайтом связывания белка HmaL1 на 23S рРНК
дом SPR были определены константы диссоци
H. marismortui [13] (рис. 2, в, г).
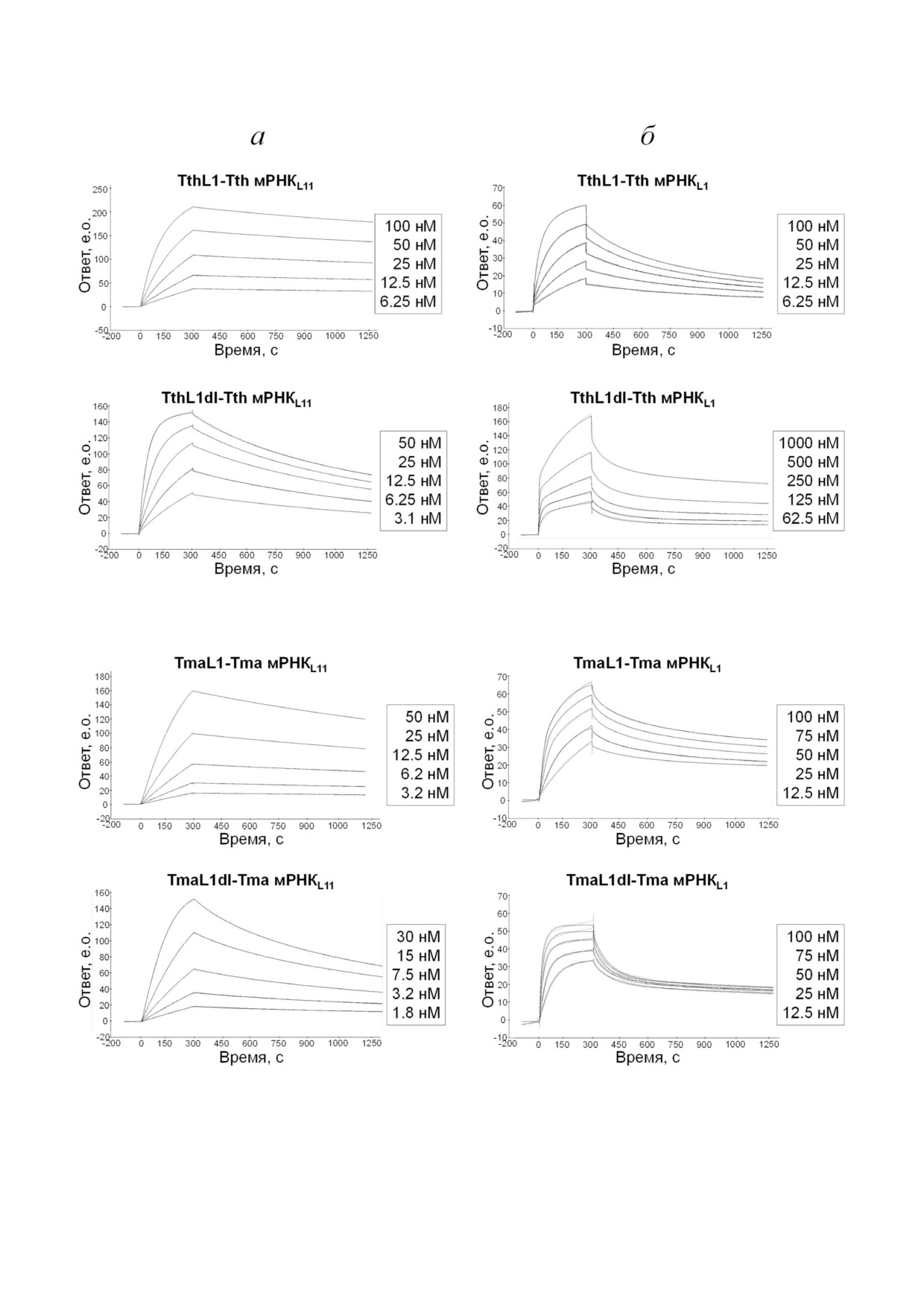
ации (KD) комплексов TthL1 (TthL1dI), TmaL1
Анализ взаимодействия белков L1 T. ther
(TmaL1dI), (рис. 3) и HmaL1 (HmaL1dI) (рис. 4)
mophilus, T. maritima и H. marismortui, а также их
со специфическими фрагментами мРНК
доменов I со специфическими фрагментами мРНК
(табл. 2).
тех же организмов. Для проверки способности
Оказалось, что сродство TthL1 и TthL1dI к
полноразмерных белков TthL1, TmaL1, HmaL1,
фрагменту мРНКTthL11 почти на порядок выше,
а также их доменов I взаимодействовать с опре
чем к фрагменту мРНКTthL1 (табл. 2). Константы
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
475
Рис. 3. Сенсограммы кинетического анализа взаимодействия белка L1 и его домена I T. thermophilus и T. maritima со спе
цифическими фрагментами мРНК. а - Соответствующие мРНКL11; б - соответствующие мРНКL1. Для каждого набора
сенсограмм указаны используемые концентрации аналита. Ровные линии соответствуют теоретической аппроксимации
полученных экспериментальных данных с использованием модели Ленгмюра (1 : 1) (а) или двухстадийной реакции (б)
БИОХИМИЯ том 86 вып. 4 2021
2*
476
МИХАЙЛИНА и др.
Рис. 4. Сенсограммы кинетического анализа взаимодействия белков HmaL1 и HmaL1dI со специфическими фрагмента
ми мРНК. Для каждого набора сенсограмм указаны используемые концентрации аналита. Ровные линии соответствуют
теоретической аппроксимации полученных экспериментальных данных с использованием модели Ленгмюра (1 : 1)
диссоциации (KD) комплексов белков TmaL1 и
Сродство доменов I к специфическим фраг
TmaL1dI с мРНКTmaL1 сравнимы с KD комплек
ментам мРНК сравнимо со сродством полно
сов белков с мРНКTmaL11 (табл. 2). Взаимодей
размерных белков, что позволяет предполо
ствие в комплексах TthL1(TthL1dI) - мРНКTthL11
жить, что как бактериальный, так и архейный
и TmaL1(TmaL1dI) - мРНКTmaL11 (рис. 3, а) опи
домен I белка L1 обладает регуляторными свой
сывается простой моделью Ленгмюра (1 : 1).
ствами наряду с полноразмерными белками.
Взаимодействие белков со специфическими
Исследование способности белка L1 регулиро
фрагментами мРНКL1 с лучшим приближением
вать синтез белков своего оперона на уровне тран
(χ2 < 10) описывается моделью двухстадийной
скрипции. Чтобы исключить влияние р белка L1
реакции, которая предполагает образование
исследуемых термофильных бактерий и археи
промежуточного комплекса (рис. 3, б).
на транскрипцию мРНК своего оперона, мы
HmaL1 и HmaL1dI связываются с фрагмен
проанализировали эффективность синтеза
том мРНКHmaL1, содержащим предполагаемый
мРНК методом транскрипции in vitro с по
регуляторный участок L1 оперона H. marismortui
мощью РНК полимеразы T7. Исследование
(рис. 4, табл. 2). Причем это взаимодействие
проводили как в присутствии, так и в отсутствие
слабее, чем со специфическим фрагментом 23S
р белка L1. Анализ мРНК транскриптов пока
рРНК (табл. 2), что согласуется с классическими
зал, что р белок L1 не оказывает влияния на
принципами регуляции синтеза р белков.
уровень транскрипции мРНК L11 оперона
Таблица 2. Кинетические параметры взаимодействия белка L1 и его домена I T. thermophilus, T. mari/
tima и H. marismortui со специфическими фрагментами мРНК
Комплекс
ka1, (×103 M-1s-1)
kd1, (×10-4 s-1)
ka2, (×10-6 s-1)
kd2, (×10-5 s-1)
KD, (нМ)
TmaL1-мРНКTmaL11
279
6,4
-
-
2,29
TmaL1dI-мРНКTmaL11
1190
23,5
-
-
1,97
TmaL1-мРНКTmaL1
272
0,1
738
10,8
3,78
TmaL1dI-мРНКTmaL1
986
22,3
456
78
2,26
TthL1-мРНКTthL11
137
2,67
-
-
1,93
TthL1dI-мРНКTthL11
810
13
-
-
1,60
TthL1-мРНКTthL1
33
4,7
1,14
9,65
14,24
TthL1dI-мРНКTthL1
5,88
3,87
43
248
65,82
HmaL1-мРНКHmaL1
0,98
4,09
-
-
417
HmaL1dI-мРНКHmaL1
0,64
5,44
-
-
850
HmaL1-рРНК
1,16
0,53
-
-
45,6
HmaL1dI-рРНК
2,5
1,7
-
–
71,2
Примечание. KD = kd1/ka1; ka1 - константа скорости ассоциации первой стадии реакции; kd1 - константа скорости дис
социации первой стадии реакции; KD - кажущаяся константа диссоциации; ka2 и kd2 - константы скорости ассоциации
и диссоциации второй стадии реакции соответственно.
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
477
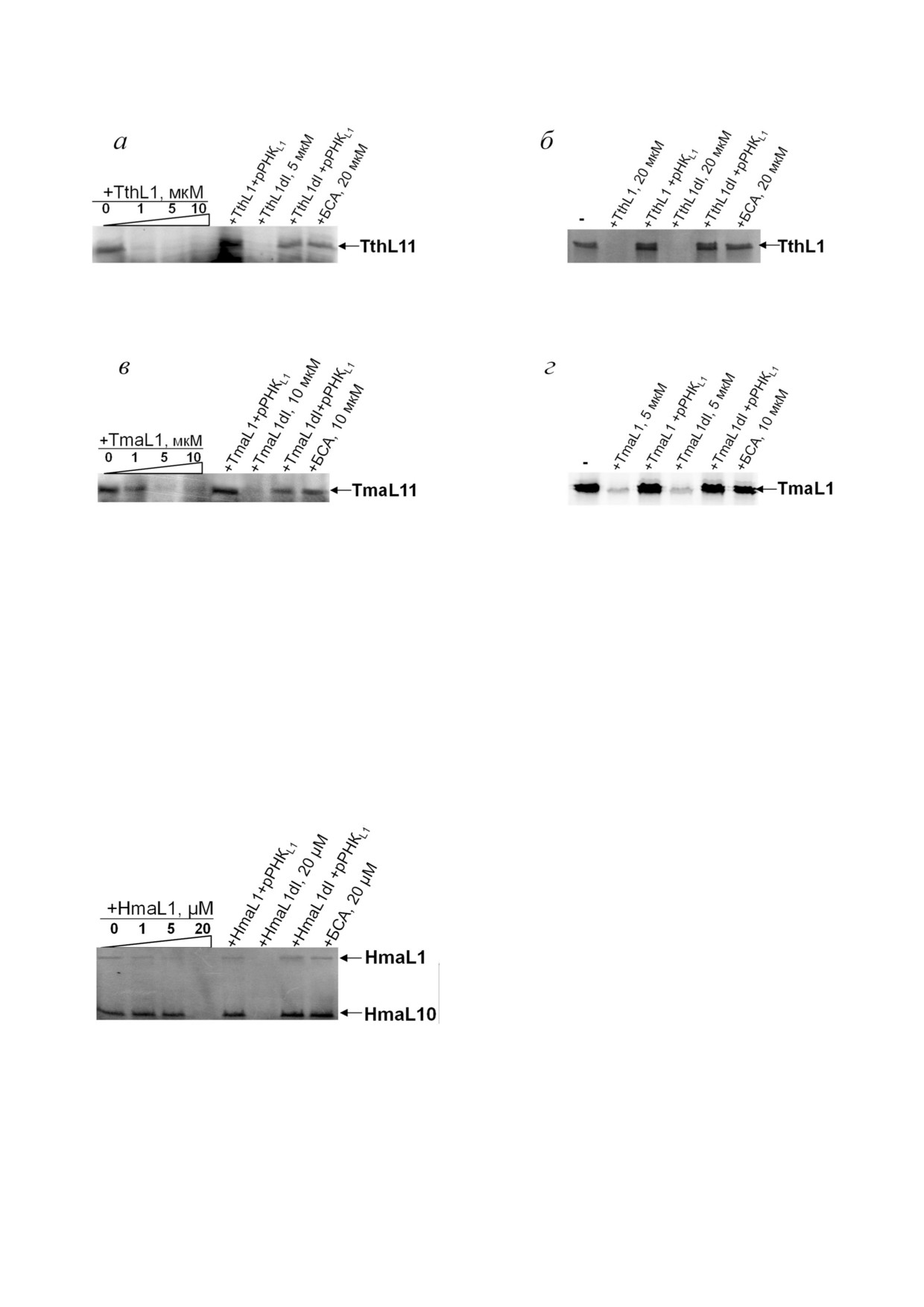
T. thermophilus и T. maritima, а также L1 оперона
Более того, при добавлении домена I белка L1
H. marismortui (рис. 5).
также происходит ингибирование синтеза обоих
Таким образом, мы показали, что исследуе
белков (рис. 6). В экспериментах по конкурент
мые белки не регулируют синтез белков своего
ному ингибированию добавление специфичес
оперона на уровне транскрипции.
кого фрагмента 23S рРНК T. thermophilus, содер
Анализ регуляторных свойств рибосомного
жащего L1 связывающий участок, ингибирова
белка L1 T. maritima и T. thermophilus. Последую
ния в обоих случаях (TthL1 и TmaL1) не проис
щие исследования мы проводили в сопряжен
ходит (рис. 6).
ной системе транскрипции-трансляции E. coli
Была проверена эффективность синтеза бел
in vitro. В наших экспериментах были использо
ков с укороченных конструкций, содержащих
ваны матрицы, несущие разные участки
только по одному участку связывания белка L1 и
L11 оперона T. thermophilus и T. maritima. Четы
одному гену L11 оперона T. thermophilus и
ре конструкции содержали один из генов белка,
T. maritima в присутствии белка L1. Показано,
L11 или L1, (pTmaL11( 50), pTthL11( 46),
что как полноразмерный белок, так и его до
pTmaL1( 25) и pTthL1( 100)) (рис.
1); две
мен I ингибируют синтез белков L11 и L1 с соот
конструкции содержали гены двух белков, L11 и
ветствующих конструкций (рис. 7).
L1, (pTmaL11 L1( 50) и pTthL11 L1( 46))
Анализ регуляторных свойств рибосомного
(рис. 1). В реакционную смесь добавляли раз
белка L1 археи H. marismortui. Регуляторные
личные количества L1/L1dI T. thermophilus или
свойства белка HmaL1 и его домена I были так
T. maritima. Как полноразмерные белки L1, так и
же исследованы в сопряженной системе тран
их домены I ингибировали синтез белков соот
скрипции трансляции in vitro (рис. 8). В качест
ветствующих конструкций (рис. 6, 7).
ве матрицы использовали конструкцию, несу
Было выявлено, что добавление в систему
щую два гена L1 оперона H. marismortui, кодиру
белка L1 ингибирует дозозависимым образом
ющие белки L1 и L10, и 5′ НТО (74 н.), предпо
синтез обоих белков L11 оперона (L11 и L1),
ложительно, включающую регуляторный учас
как на мРНК T. thermophilus, так и T. maritima.
ток (pHmaL1/L10( 74), рис. 1, в). При добавле
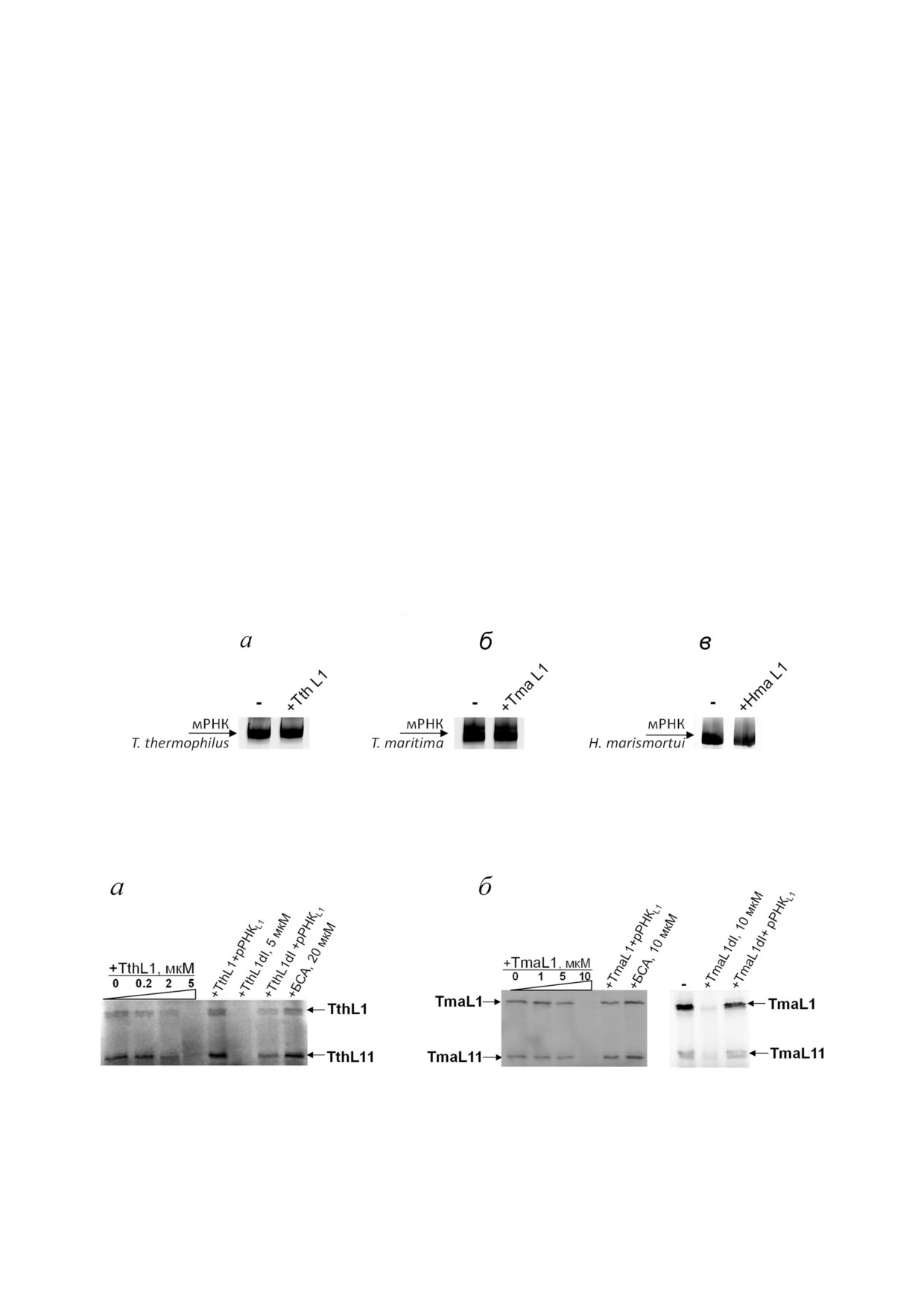
Рис. 5. Электрофоретический анализ транскрипции фрагментов мРНК. В качестве матрицы для транскрипции in vitro ис
пользовали конструкции, содержащие ген белка L11 и 5′ НТО перед ним термофильных бактерий: а - pTthL11( 46); б -
pTmaL11( 50); в - конструкция, содержащая гены белков HmaL1 и HmaL10 и 5′ НТО перед геном белка HmaL1 -
pHmaL1/L10( 74)
Рис. 6. Авторадиограмма белков L11 и L1 T. thermophilus (а) и T. maritima (б), синтезированных в присутствии L [35S]мети
онина, белка L1 (в различном количестве), его домена I, или БСА. В качестве матрицы в сопряженной системе транскрип
ции-трансляции использовали конструкции, несущие оба гена L11 оперона - rplK и rplA, кодирующие белки L11 и L1 со
ответственно, и 5′ НТО (pTmaL11 L1( 50) и pTthL11 L1( 46), рис. 1, а, б)
БИОХИМИЯ том 86 вып. 4 2021
478
МИХАЙЛИНА и др.
Рис. 7. Авторадиограммы белков L11 T. thermophilus и T. maritima (а, в) и белков L1 T. thermophilus и T. maritima (б, г), син
тезированных в присутствии L [35S]метионина и белка L1 (в различной концентрации), его домена I, БСА и фрагмента
23S рРНК T. thermophilus, содержащего L1 связывающий участок. В качестве матрицы в сопряженной системе транскрип
ции трансляции были использованы укороченные конструкции, содержащие только по одному L1 связывающему участ
ку и одному гену L11 оперона: а, в - ген белка L11 и участок 5′ НТО перед ним (pTmaL11( 50), pTthL11( 46), рис. 1);
б, г - ген белка L1 и участок перед ним (pTmaL1( 25) и pTthL1( 100), рис. 1)
нии в систему как белка HmaL1, так и HmaL1dI,
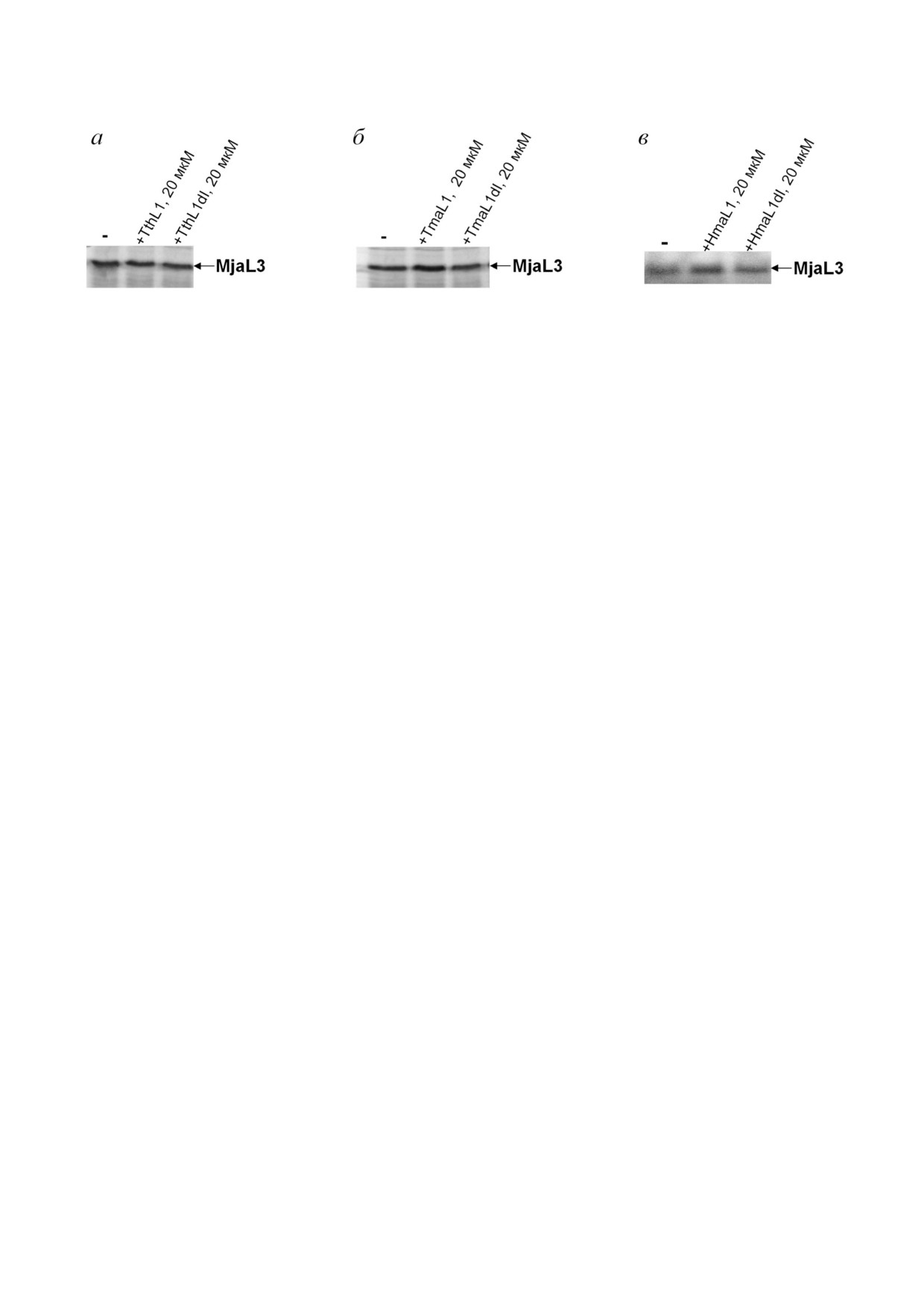
Для проверки специфичности взаимодей
дозозависимым образом ингибируется синтез
ствия белка L1 с мРНК были поставлены конт
обоих белков (L1 и L10) (рис. 8). В эксперимен
рольные эксперименты. В качестве отрицатель
тах по конкурентному ингибированию в обоих
ного контроля использовали плазмиду, несущую
случаях добавление специфического фрагмента
ген рибосомного белка L3 M. jannaschii (MjaL3)
23S рРНК H. marismortui (рис. 2, г), содержащего
[18] (рис. 9), где было показано, что L1 не инги
L1 связывающий участок, ингибирующий эф
бирует экспрессию гена белка другого оперона.
фект HmaL1 или HmaL1dI отсутствует (рис. 8).
Как контроль на специфичность взаимодей
ствия в экспериментах использовали белок
БСА. Синтез белков L11 оперона T. ther/
mophilus, T. maritima и L1 оперона H. marismortui
не зависел от добавления БСА в реакционную
систему (рис. 6-8).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Взаимодействие белка L1 с участками связы
вания мРНК L11 оперона и L1 оперона. В резуль
тате сравнительного структурного анализа
комплексов TthL1 со специфическими фраг
ментами мРНК и рРНК были определены нук
Рис. 8. Авторадиограмма белков L1 и L10 H. marismortui,
леотиды, формирующие структурно консерва
синтезированных в присутствии L [35S]метионина, белка
тивный модуль РНК, необходимый для образо
HmaL1 (в различных концентрациях), HmaL1dI или БСА. вания стабильного комплекса РНК-L1 [17].
В качестве матрицы в сопряженной системе транскрип
Проведённый нами анализ геномных последо
ции трансляции in vitro использовали плазмиду, несущую
гены двух белков L1 оперона H. marismortui - L1 и L10, а
вательностей T. thermophilus и T. maritimа в райо
также 5′ НТО (pHmaL1/L10( 74), рис. 1, г)
не L11 оперонов показал, что в мРНК этих бак
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
479
Рис. 9. Авторадиограмма белка MjaL3, синтезированного в присутствии L [35S]метионина при избытке белка L1 или его
домена I: а - T. thermophilus; б - T. maritima ; в - H. marismortui. В качестве матрицы в сопряженной системе транскрип
ции трансляции in vitro использована плазмида, несущая ген рибосомного белка MjaL3
терий имеется два потенциальных участка свя
нако удлинение фрагмента этого участка мРНК
зывания белка L1. Первый участок расположен,
на две пары нуклеотидов (рис. 2, а) привело к
как у E. coli, в лидерной последовательности
образованию комплекса с белками TmaL1 и
мРНК белка L11. Второй участок в T. maritima
TmaL1dI (табл. 2). Таким образом, вероятно,
включает нетранслируемый лидерный участок и
роль водородной связи между белком и нуклео
начало кодирующей части мРНК белка L1
тидом последних двух пар концевой спирали
(17
н.) (рис. 2, а). В T. thermophilus второй
фрагмента мРНК (U A
( 13/+16) и G C
L1 связывающий участок находится в области
( 14/+17) в мРНКTmaL1) не менее важна для обра
перекрывания генов белков L11 и L1 (рис. 2, б).
зования прочного комплекса, чем выявленные
Эти участки гомологичны L1 связывающему
ранее водородные связи между белком L1 и кон
сайту на рРНК [17].
сервативным L1 связывающим модулем (выде
Мы показали, что бактериальные рибосом
лен рамкой на рис. 2).
ные белки L1 T. thermophilus и T. maritima имеют
Таким образом, р белок L1 как в T. ther/
высокое сродство к двум участкам на мРНК
mophilus, так и в T. maritima, взаимодействует с
L11 оперона. Сродство р белка L1 к этим участ
двумя специфическими участками на мРНК
кам мРНК сравнимо (KD
= 10-9-10-8 M
своего оперона. По всей видимости, связываясь
(табл. 2)) с полученными ранее данными по вза
с этими участками мРНК, р белок L1 может ре
имодействию TthL1 с фрагментом мРНК
гулировать уровень синтеза белков L11 оперона
M. vannielii (KD = 8,6 × 10-9 М) [19].
в соответствующих организмах in vitro и in vivo.
Ранее нам не удавалось детектировать взаи
Однако характер взаимодействия белков L1 с
модействие белка TmaL1 c фрагментом мРНК
фрагментами мРНКL1 и мРНКL11 может разли
T. maritimа, содержащим лидерный участок
чаться. Анализ взаимодействия как полнораз
мРНК L1. В составе этого фрагмента были со
мерного р белка L1, так и его домена I со специ
хранены все основные детерминанты для РНК
фическими фрагментами мРНКL11 методом по
белкового взаимодействия [6], но длина конце
верхностного плазмонного резонанса показыва
вой спирали была короче на одну пару нуклео
ет, что наиболее подходящей является модель
тидов по сравнению с двумя другими фрагмен
Ленгмюра (1 : 1) (рис. 3, а, табл. 2). Взаимодей
тами мРНК (рис. 2, а, mRNATmaL1, пунктирная
ствие белков со специфическими фрагментами
рамка). Мы предположили, что отсутствие этих
мРНКL1 можно описать только моделью двух
нуклеотидов привело к потере двух водородных
стадийной реакции (рис. 3, б, табл. 2). Такая мо
связей, важных для ориентации белка на моле
дель была использована нами ранее для описа
куле РНК. В комплексе TthL1-мРНК M. van/
ния взаимодействий в комплексах мутантных
nielii Thr168, расположенный в домене I белка
форм белка TthL1 со специфическим фрагмен
TthL1, образует две закрытые от растворителя
том 23S рРНК [16]. В этой модели два компо
водородные связи с рибозой одного из нуклео
нента формируют сначала промежуточный
тидов последней пары фрагмента мРНК
комплекс с константами скорости ассоциации и
(рис. 2, а, mRNATmaL1, пунктирная рамка) [17]. В
диссоциации ka1 и kd1, далее образуя конечный
TmaL1 присутствует аналог этого аминокислот
комплекс с кинетическими константами ka2 и
ного остатка (Thr168). Ранее мы предполагали,
kd2. Промежуточный комплекс формируется
что данные контакты не играют определяющей
быстро, затем следует медленный конформаци
роли в формировании комплекса L1-РНК. Од
онный переход в более стабильный комплекс.
БИОХИМИЯ том 86 вып. 4 2021
480
МИХАЙЛИНА и др.
В мРНК L1 оперона архей H. marismortui мы
цию, содержащую ген белка TthL1 с добавлен
обнаружили только один участок, гомологич
ной последовательностью, содержащей SD,
ный консервативному участку связывания бел
поскольку природный SD подобный мотив вхо
ка L1 на 23S рРНК, - в лидерной последова
дил в состав L1 связывающего участка. Извест
тельности мРНК белка L1 (рис. 2, в, г). HmaL1 и
но, что положение предположительного L1 cвя
его домен I связываются с этим участком мРНК,
зывающего сайта в мРНК L11 оперона в раз
причем это взаимодействие слабее, чем с рибо
личных родах бактерий не строго консерватив
сомной РНК (KD(HmaL1-мРНКHma) = 417 нМ,
но. В мРНК E. coli имеется единственный
KD(HmaL1-рРНКHma) = 45,6 нМ, табл. 2). Такое
L1 связывающий участок, а в геномах некото
различие согласуется с принципом классичес
рых бактерий типа Firmicute обнаружены два по
кой регуляции синтеза рибосомных белков (ин
тенциальных участка связывания белка L1 [5],
гибирование по принципу обратной связи), ос
также как в исследованных нами бактериях
нованным на конкуренции между двумя участ
T. thermophilus и T. maritima. В отличие от данных
ками связывания.
литературы, наличие двух регуляторных участ
Сродство домена I как бактериальных, так и
ков L11 оперона в этих гипертермофильных
архейного белка L1 к фрагментам мРНК являет
бактериях подтверждено нами эксперименталь
ся примерно таким же, как сродство полнораз
но. Несомненно, в термофильных организмах
мерного белка (KD(HmaL1-мРНКHma) = 417 нМ,
имеются механизмы, поддерживающие рост
KD(HmaL1dI-мРНКHma) = 846 нМ, табл. 2), что
клеток при высоких температурах. Геномный
позволяет предположить, что L1dI может обладать
анализ промоторов и рибосом связывающих
такими же регуляторными свойствами, как бак
участков в T. maritima показал [23], что они ко
териальный TthL1dI [9] и архейный MjaL1dI [20].
дируют высококонсервативные жёсткие струк
Регуляция синтеза белков L11 оперона и
туры, которые могут обеспечить плотные кон
L1 оперона белком L1 и его доменом I. Для дока
такты, что очень важно в термодинамически
зательства регуляторных свойств белка L1 и его
неблагоприятных условиях (75-80 °C). Наличие
домена I мы исследовали их дозозависимое вли
двух L1 связывающих участков может являться
яние на уровень синтеза белков L11 оперона
примером усиленной регуляции синтеза белков
T. thermophilus, T. maritima и L1 оперона
L11 оперона у бактерий, живущих в гипертер
H. marismortui. Подобные эксперименты прово
мофильных условиях.
дились ранее для исследования ауторегуляции
В случае экстремального галофила H. maris/
синтеза белков L1 оперона архейным рибосом
mortui был обнаружен один регуляторный учас
ным белком L1 M. vannielii [2], а также для опре
ток, взаимодействие с которым р белка L1 мо
деления регуляторных свойств TthL1/TthL1dI и
жет ингибировать синтез белков своего L1 опе
MjaL1/MjaL1dI на мРНК M. vannielii [9, 20].
рона. Можно заметить, что в бесклеточной сис
Бесклеточная система транскрипции трансля
теме транскрипции-трансляции E. coli количе
ции E. coli может быть использована как для
ство синтезированного белка L1 археи H. maris/
экспрессии архейных, так и эукариотических
mortui было ниже, чем белка L11 (рис. 8). Ранее
белков [21].
при анализе регуляции трансляции мРНК
Как упоминалось выше, мы показали нали
L1 оперона другой археи M. vannielli в такой же
чие двух регуляторных участков в мРНК
in vitro системе E. coli наблюдалась аналогичная
L11 оперона бактерий T. thermophilus и T. mariti/
ситуация [2], причём в in vivo системе E. coli со
ma. Стоит отметить, что структурно консерва
отношение синтеза белков, кодируемых генами
тивный участок связывания белка L1 на РНК
этого архейного оперона, было другим. Такие
включает последовательность GGAG (рис. 2),
отличия могут быть связаны с особенностями
которая может быть принята за SD подобный
синтеза белков в системе in vitro.
мотив мРНК, будучи расположенной перед
В археях рода Methanococcus участок связы
AUG кодоном. Однако, согласно структурным
вания белка L1 находится в начале его кодиру
данным, 3 из 4 х нуклеотидов этой последова
ющей части мРНК (первые 30 нуклеотидов пос
тельности находятся в спиральных участках [17]
ле старт кодона) [2]. Мы показали, что в
и недоступны для РНК РНКовых взаимодей
H. marismortui регуляторный участок находится
ствий. В результате такой участок мРНК взаи
в 5′ НТО мРНК белка L1. При связывании бел
модействует с р белком L1, но не связывается с
ка HmaL1 ингибируется синтез белков L1, L11
анти SD на рибосоме. Ранее было показано на
и, предположительно, L12. Полученные нами
личие такой «маскированной» SD последова
данные подтверждают консервативность регу
тельности в мРНК белка L1 E. coli [22]. Для того
ляторных свойств р белка L1. При этом важно
чтобы в полной мере исследовать регуляторные
отметить, что ведущую роль в регуляторных
свойства белка TthL1, мы получили конструк
свойствах белка в исследуемых организмах иг
БИОХИМИЯ том 86 вып. 4 2021
РЕГУЛЯТОРНЫЕ УЧАСТКИ L11 И L1 ОПЕРОНОВ
481
рает его домен I, что свидетельствует о струк
Конфликт интересов. Авторы заявляют об от
турной консервативности РНК белковых взаи
сутствии конфликта интересов.
модействий. Более того, наличие двух регуля
Соблюдение этических норм. Настоящая
торных участков на мРНК L11 оперона гипер
статья не содержит описания каких либо иссле
термофильных бактерий экспериментально
дований с участием людей или животных в каче
подтверждено.
стве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Gourse, R. L., Thurlow, D. L., Gerbi, S. A., and
12.
Laemmli, U. (1970) Cleavage of structural proteins during
Zimmermenn, R. A. (1981) Specific binding of a procary
the assembly of the head of bacteriophage T4, Nature, 227,
otic ribosomal protein to an eukaryotic ribosomal RNA:
680 685.
implications for evolution and autoregulation, Proc. Natl.
13.
Gabdulkhakov, A., Tishchenko, S., Mikhaylina, A.,
Acad. Sci. USA, 78, 2722 2726, doi: 10.1073/pnas.78.5.
Garber, M., Nevskaya, N., and Nikonov, S. (2017) Crystal
2722.
structure of the 23S rRNA fragment specific to r protein
2.
Hanner, M., Mayer, C., Köhrer, C., Golderer, G.,
L1 and designed model of the ribosomal L1 stalk from
Gröbner, P., and Piendl, W. (1994) Autogenous transla
Haloarcula marismortui, Crystals, 7, 37, doi: 10.3390/
tional regulation of the ribosomal MvaL1 operon in the
cryst7020037.
archaebacterium Methanococcus vannielii, J. Bacteriol.,
14.
Katsamba, P. S., Park, S., and Laird Offringa, I. A. (2002)
176, 409 418, doi: 10.1128/jb.176.2.409 418.1994.
Kinetic studies of RNA-protein interactions using surface
3.
Baughman, G., and Nomura, M. (1984) Translational reg
plasmon resonance, Methods, 26, 95 104, doi: 10.1016/
ulation of the L11 ribosomal protein operon of Escherichia
S1046 2023(02)00012 9.
coli: analysis of the mRNA target site using oligonu
15.
Kostareva, O., Tishchenko, S., Nikonova, E.,
cleotide directed mutagenesis, Proc. Natl. Acad. Sci. USA,
Kljashtorny, V., Nevskaya, N., et al. (2011) Disruption of
81, 5389 5393, doi: 10.1073/pnas.81.17.5389.
shape complementarity in the ribosomal protein L1-RNA
4.
Mayer, C., Kohrer, C., Grobner, P., and Piendl, W. (1998)
contact region does not hinder specific recognition of the
MvaL1 autoregulates the synthesis of the three ribosomal
RNA target site, J. Mol. Recognit.,
24,
524532,
proteins encoded on the MvaL1 operon of the archaeon
doi: 10.1002/jmr.1063.
Methanococcus vannielii by inhibiting its own translation
16.
Tishchenko, S., Kostareva, O., Gabdulkhakov, A.,
before or at the formation of the first peptide bond, Mol.
Mikhaylina, A., Nikonova, E., et al. (2015) Protein-RNA
Microbiol., 27, 455 468, doi: 10.1046/j.1365 2958.1998.
affinity of ribosomal protein L1 mutants does not correlate
00693.x.
with the number of intermolecular interactions, Acta
5.
Fu, Y., Deiorio Haggar, K., Anthony, J., and Meyer,
Crystallogr. D Biol. Crystallogr., 71, 376 386, doi: 10.1107/
M. M. (2013) Most RNAs regulating ribosomal protein
S1399004714026248.
biosynthesis in Escherichia coli are narrowly distributed to
17.
Nevskaya, N., Tishchenko, S., Volchkov, S., Kljastorny, V.,
Gammaproteobacteria, Nucleic Acids Res., 41, 3491 3503,
Nikonova, E., et al. (2006) New insights into the interac
doi: 10.1093/nar/gkt055.
tion of ribosomal protein L1 with RNA, J. Mol. Biol., 355,
6.
Mikailina, A. O., Kostareva, O. S., Nikonova, E. Yu.,
747 759, doi: 10.1016/j.jmb.2005.10.084.
Garber, M. B., and Tischenko, S. V. (2018) Identification
18.
Mikhaylina, A., Kostareva, O., Sarskikh, A. V., Feodorov,
of binding sites for ribosomal protein L1 on mRNA of
R. V., Pindl, V., et al. (2014) Study of the regulatory prop
Thermus thermophilus and Thermotoga maritima, Mol. Biol.
erties of archaeal ribosomal protein L4, Biochemistry
(Mosk.), 52, 98 105.
(Moscow), 79, 87 95.
7.
Kraft, A., Lutz, C., Lingenhel, A., Gröbner, P., and
19.
Tishchenko, S., Kljashtorny, V., Kostareva, O.,
Piendl, W. (1999) Control of ribosomal protein L1 synthe
Nevskaya, N., Nikulin, A., et al. (2008) Domain II of
sis in mesophilic and thermophilic archaea, Genetics, 152,
Thermus thermophilus ribosomal protein L1 hinders recog
1363 1372.
nition of its Mrna, J. Mol. Biol.,
383,
301305,
8.
Tishchenko, S., Nikonova, E., Kljashtorny, V.,
doi: 10.1016/j.jmb.2008.08.058.
Kostareva, O., Nevskaya, N., and Piendl, W.
(2007)
20.
Mikhailina, A., Kostareva, O., Nikonova, E., and
Domain I of ribosomal protein L1 is sufficient for specific
Tischenko, S. (2016) Analysis of the interaction of domain
RNA binding, Nucleic Acids Res.,
35,
73897395,
I of archaeal ribosomal protein L1 with specific RNA frag
doi: 10.1093/nar/gkm898.
ments, Aktual. Vopr. Biol. Khim., 1, 239 243.
9.
Korepanov, A. P., Kostareva, O. S., Bazhenova, M. V.,
21.
Chen, F., Wang, J., Du, L., Zhang, X., Zhang, F., et al.
Bubunenko, M. G., Garber, M. B., and Tishchenko, S. V.
(2019) Functional expression of olfactory receptors using
(2015) Studying the properties of domain I of the riboso
cell free expression system for biomimetic sensors towards
mal protein L1: incorporation into ribosome and regula
odorant detection, Biosens. Bioelectron., 130, 382 388.
tion of the L1 operon expression, Protein J., 34, 103 110,
22.
Sor, F., Bolotin Fukuhara, M., and Nomura, M. (1987)
doi: 10.1007/s10930 015 9602 5.
Mutational alterations of translational coupling in the L11
10.
Studier, F., Rosenberg, A., Dunn, J., and Dubendorff, J.
ribosomal protein operon of Escherichia coli, J. Bacteriol.,
(1990) Use of T7 RNA polymerase to direct expression of
169, 3495 507.
cloned genes, J. Methods Enzymol., 185, 60 89.
23.
Latif, H., Lerman, J. A., Portnoy, V. A., Tarasova, Y.,
11.
Novy, R., Drott, D., Yaeger, K., and Mierendorf, R. (2001)
Nagarajan, H., et al. (2013) The genome organization of
Overcoming the codon bias of E. coli for enhanced protein
Thermotoga maritima reflects its lifestyle, PLoS Genet., 9,
expression, inNovations, 12, 1 3.
e1003485, doi: 10.1371/journal.pgen.1003485.
БИОХИМИЯ том 86 вып. 4 2021
482
МИХАЙЛИНА и др.
CHARACTERIZATION OF REGULATORY ELEMENTS
OF L11 AND L1 OPERONS IN THERMOPHILIC BACTERIA AND ARCHAEA
A. O. Mikhaylina1*, Е. Y. Nikonova1, O. S. Kostareva1,
W. Piendl2, M. Erlacher3, and S. V. Tishchenko1
1 Institute of Protein Research, Russian Academy of Sciences,
142290 Pushchino, Russia; E mail: alisamikhaylina15@gmail.com
2 Division of Medical Biochemistry, Biocenter, Medical University of Innsbruck,
6020 Innsbruck, Austria; E mail: wolfgang.piendl@i/med.ac.at
3 Division of Genomics and RNomics, Biocenter, Medical University of Innsbruck,
6020 Innsbruck, Austria; E mail: matthias.erlacher@i/med.ac.at
Ribosomal protein L1 is a conserved two domain protein that is involved in formation of the L1 stalk of the large ribo
somal subunit. When there are no free binding sites available on the ribosomal 23S RNA, the protein binds to the spe
cific site on the mRNA of its own operon (L11 operon in bacteria and L1 operon in archaea) preventing translation.
Here we show that the regulatory properties of the r protein L1 and its domain I are conserved in the thermophilic
bacteria Thermus and Thermotoga and in the halophilic archaeon Haloarcula marismortui. At the same time the
revealed features of the operon regulation in thermophilic bacteria suggest presence of two regulatory regions.
Keywords: regulation of expression, L11 operon, L1 operon, r proteins, mRNA
БИОХИМИЯ том 86 вып. 4 2021