БИОХИМИЯ, 2021, том 86, вып. 4, с. 529 - 553
УДК 577.2
МИКРОБНЫЙ АРСЕНАЛ ПРОТИВОВИРУСНОЙ ЗАЩИТЫ. ГЛАВА II
Обзор
© 2021
А.Б. Исаев1*, О.С. Мушарова1,2, К.В. Северинов1,3*
1 Сколковский институт науки и технологий, 143028 Москва, Россия; электронная почта: tcft18@gmail.com
2 Институт молекулярной генетики РАН, 119334 Москва, Россия
3 Waksman Institute of Microbiology, Piscataway, NJ 08854, USA; e%mail: severik@waksman.rutgers.edu
Поступила в редакцию 30.08.2020
После доработки 29.01.2021
Принята к публикации 29.01.2021
Бактериофаги, или фаги, представляют собой вирусы, которые инфицируют бактериальные клетки (в рамках
этого обзора мы также рассмотрим вирусы, которые инфицируют архей). Постоянная угроза заражения фа!
гами является одной из основных движущих сил эволюции бактериальных геномов. Чтобы противостоять
инфекции, бактерии выработали многочисленные защитные стратегии, позволяющие избежать распозна!
вания фагами или прямо препятствующие размножению фагов внутри клетки. Исследования бактериофа!
гов и бактериальных систем защиты были исторически тесно переплетены с развитием методов классичес!
кой молекулярной биологии и генной инженерии. В настоящее время благодаря распространению фаговой
терапии, широкому применению технологий CRISPR!Cas и развитию биоинформатических подходов, ко!
торые облегчают задачу обнаружения новых систем, исследования в области биологии фагов переживают
возрождение. В настоящем обзоре описываются различные стратегии, используемые микробами для того,
чтобы противостоять фаговой инфекции. Вторая глава посвящена системам адаптивного иммунитета, ме!
ханизмам абортивной инфекции, защитным системам, связанным с мобильными генетическими элемента!
ми, и новым системам, которые были открыты в последние годы с помощью метагеномного майнинга.
КЛЮЧЕВЫЕ СЛОВА: бактериофаги, иммунные системы, CRISPR!Cas, абортивная инфекция, токсин!ан!
титоксин, PICI, прокариотический белок Argonaute, CBASS.
DOI: 10.31857/S0320972521040060
ВВЕДЕНИЕ
разнообразия микробных противовирусных
систем.
В первой главе обзора были рассмотрены
стратегии, позволяющие клеткам!хозяевам из!
бегать распознавания фагами, механизмы
CRISPR&Cas СИСТЕМЫ
врождённого иммунитета, блокирующие ран!
АДАПТИВНОГО ИММУНИТЕТА
ние стадии инфекции, а также системы защиты,
использующие химические модификации для
В отличие от систем врождённого иммуните!
различения собственных и чужеродных моле!
та, основанных на модификации ДНК, в кото!
кул ДНК. В этой главе мы продолжим описание
рых распознавание мишени зависит от взаимо!
Принятые сокращения: CRISPR!Cas - кластеризованные регулярно прерываемые повторы и ассоциированные с
ними белки (Clustered Regularly Interspaced Repeats and CRISPR!associated proteins); PAM - мотив, смежный с протоспей!
сером (Protospacer Adjacent Motif); HEPN - нуклеотид!связывающий домен, характерный для высших эукариот и прока!
риот (Higher Eukaryotes and Prokaryotes Nucleotide!binding domain); R!M - рестрикция!модификация (Restriction!
Modification); MGE - мобильный генетический элемент; pAgo - прокариотический белок Argonaute (Prokaryotic
Argonaute); Abi - абортивная инфекция; TA - токсин!антитоксин; RTase - обратная транскриптаза; CBASS - антифаго!
вая сигнальная система на основе циклических олигонуклеотидов (Cyclic!Oligonucleotide!Based Anti!Phage Signalling
System); cGAMP - циклический гуанозинмонофосфат!аденозинмонофосфат (Cyclic Guanosine Monophosphate-
Adenosine Monophosphate); CD!NTase - cGAS/DncV!подобная нуклеотидил!трансфераза (cGAS/DncV!like Nucleotidyl
Transferase); Cap - белок, ассоциированный с CD!NTase (CD!NTase associated protein); DUF - домен с неизвестной функ!
цией (Domain of Unknown Function); PICI - индуцированные фагами хромосомные островки (Phage!Inducible
Chromosomal Islands); PLE - фаго!индуцируемый хромосомный островоподобный элемент (Phage!inducible Chromosomal
Island!like Element); SaPI - островок патогенности Staphylococcus aureus; DRT - обратные транскриптазы, ассоциирован!
ные с защитой (Defence!associated RTase); MOI - множественность заражения; STING - стимулятор генов интерферо!
на (Stimulator of Interferon Genes); TIR - Toll/интерлейкин!1 рецептор (Toll/interleukin!1 receptor).
* Адресат для корреспонденции.
529
530
ИСАЕВ и др.
действия защитных белков с заранее определен!
В ходе процесса CRISPR!адаптации происходит
ной последовательностью в геноме фага, у про!
удлинение кассеты на последовательность од!
кариот также имеются системы адаптивного им!
ного нового спейсера и одного повтора. Белки
мунитета CRISPR!Cas (кластеризованные регу!
адаптационого комплекса гомологичны во всех
лярно прерываемые повторы и ассоциирован!
системах CRISPR!Cas. Транскрипция CRISPR!
ные с ними белки (Clustered Regularly
кассет приводит к образованию пре!крРНК, ко!
Interspaced Repeats and CRISPR!associated pro!
торая затем процессируется с образованием ко!
teins)). В этом случае распознавание нуклеино!
ротких крРНК. В результате процессинга каж!
вой кислоты осуществляется за счёт отжига
дая крРНК содержит спейсер, окруженный час!
комплементарной молекулы РНК, и система
тичными повторами. Связанная с белками Cas,
может приобретать и хранить информацию для
крРНК образует эффекторный комплекс, спо!
интерференции с новыми последовательностя!
собный специфически распознавать протоспей!
ми. Способность сохранять информацию о пре!
сер, т.е. последовательность ДНК или РНК,
дыдущих инфекциях является общей чертой, ха!
комплементарную спейсерной части крРНК.
рактерной для систем CRISPR!Cas и иммунной
Вслед за распознаванием протоспейсера проис!
системы высших эукариот, в том числе и челове!
ходит деградация молекулы!мишени, содержа!
ка. В отличие от иммунитета млекопитающих,
щей протоспейсер. Процесс распознавания ми!
CRISPR!Cas!опосредованный адаптивный им!
шени и её деградации называется CRISPR!ин!
мунитет передаётся по наследству. Система
терференцией (рис. 1, а).
CRISPR!Cas состоит из CRISPR!кассет (коли!
Разнообразие механизмов CRISPR&интерфе&
чество кассет в геномах прокариот варьируется
ренции. Классификация систем CRISPR!Cas ос!
от одной до нескольких десятков) и ассоцииро!
нована на белковом составе эффекторных
ванных с ними генов cas [1-3]. CRISPR!кассета
комплексов. На настоящий момент системы
представляет собой кластер из коротких повто!
CRISPR!Cas можно разделить на 2 класса, 6 ти!
ряющихся фрагментов геномной ДНК, разде!
пов и 33 подтипа [4]. Системы класса 1 (типы I,
лённых уникальными спейсерными последова!
III и IV) используют мульти!субъединичные эф!
тельностями, по крайней мере некоторые из ко!
фекторы, в то время как эффекторы клас!
торых происходят из чужеродной ДНК. Перед
са 2 (типы II, V и VI) являются белками, образо!
CRISPR!кассетой находится АТ!богатая лидер!
ванными одной субъединицей (таблица). Раз!
ная область [1]. Гены cas кодируют белковые
личные типы систем CRISPR!Cas можно отли!
компоненты эффекторного комплекса систем
чить друг от друга по наличию специфических
CRISPR!Cas. Системы CRISPR!Cas участвуют в
«сигнатурных белков», участвующих в деграда!
двух различных процессах: адаптации и интер!
ции молекулы ДНК (белки Cas3, Csf1, Cas10,
ференции. CRISPR!адаптация - это процесс
Cas9, Cpf1 и C2c2 для типов I, IV, III, II, V и VI
интеграции новых спейсеров в CRISPR!кассету.
соответственно [4]).
Разнообразие механизмов интерференции и классификация систем CRISPR!Cas
Специфич!
Процессинг
Различение
Класс
Тип
Подтипы
ный белок
пре!крРНК
Мишень
собственной и
Эффекторы
чужеродной НК
Класс I
I
A, B, C, D, E, F1, F2, F3, G
Cas3
Cas6
ДНК
PAM
Cascade,
крРНК, Cas3
III
A, B, C, D, E, F
Cas10
Cas6, Cas10,
ДНК,
CRISPR повтор
Cmr/Csm,
Csm2, Csm5 (III!A)
РНК
крРНК, Cas10
IV
A1, A2, A3, B, C, D, E
Csf1
Csf5
ДНК?
?
Csf1, Csf3,
Csf5, Csf2,
крРНК
Класс II
II
A, B, C1, C2
Cas9
РНКаза III
ДНК
PAM
Cas9, крРНК,
traкрРНК
V
A, B1, B2, E, V!C, V!D, V!F1,
CpfI
CpfI
ДНК
V!F2, V!F3, V!G, V!H, V!I,
PAM
Cpf1, крРНК,
V!U1, V!U2, V!U3, V!U4, V!U5
traкрРНК
VI
A, B1, B2, C, D
C2c2
Cas13
РНК
?
C2c2, крРНК
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
531
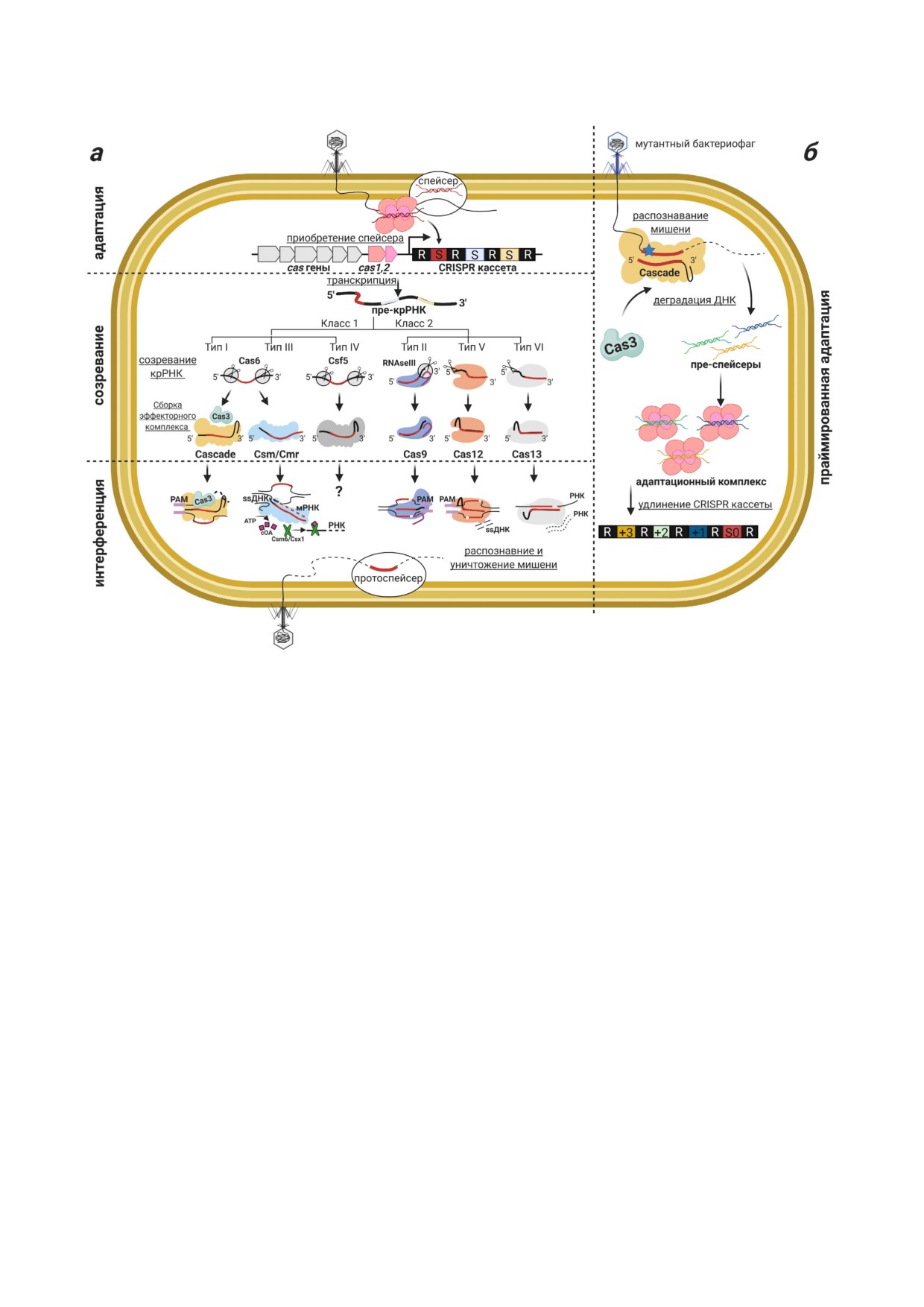
Рис. 1. Механизм адаптивного иммунитета CRISPR!Cas у прокариот. а - Фрагменты, происходящие из чужеродной ДНК,
могут быть встроены в CRISPR!кассету в процессе CRISPR!адаптации. CRISPR!кассета удлиняется на один новый спей!
сер и один повтор. Транскрипция CRISPR!кассет приводит к образованию пре!крРНК, которая затем расщепляется с об!
разованием коротких крРНК, и при этом каждая крРНК содержит спейсер, расположенный между частичными повтора!
ми. Гены cas кодируют белковые компоненты эффекторного и адаптационного комплексов. Эффекторный комплекс со!
стоит из крРНК, связанной с белками Cas, и взаимодействует с протоспейсером, т.е. последовательностью ДНК, компле!
ментарной последовательности спейсера крРНК. Распознавание протоспейсера эффекторным комплексом CRISPR при!
водит к деградации молекулы ДНК!мишени. Белковый состав модуля интерференции бывает различным, и он использу!
ется как основной критерий в классификации систем CRISPR!Cas. Системы CRISPR!Cas подразделяют на два класса,
шесть типов и несколько подтипов. Два класса отличаются друг от друга по составу интерференционного комплекса: сис!
темы CRISPR!Cas класса 1 являются мульти!субъединичными, а системы класса 2 содержат только один белок. б - В про!
цессе праймированной адаптации новые спейсеры предпочтительно отбираются из ДНК, таргетируемой эффекторным
комплексом. (С цветными вариантами рис. 1-7 можно ознакомиться в электронной версии статьи на сайте:
Системы CRISPR!Cas класса I включают
занной с Cascade, с комплементарным прото!
три типа: I, III и IV. Подробно изучены эффек!
спейсером приводит к локальному плавлению
торные комплексы систем типа I и III. Сходство
дцДНК!мишени и образованию R!петли - гете!
архитектуры эффекторных комплексов указы!
родуплекса между спейсером крРНК и таргет!
вает на общее происхождение этих систем [5].
ной цепью протоспейсера ДНК, в то время как
Эффектор типа I представлен большим мульти!
нетаргетная нить ДНК протоспейсера вытесня!
субъединичным белковым комплексом, называ!
ется и остаётся в однонитевой форме. Обяза!
емым Cascade, который содержит субъединицы
тельным условием распознавания мишени явля!
белка RAMP (Repeat!Associated Mysterious
ется наличие короткого, состоящего из двух!
Protein) в стехиометрии Cse11/Cse22/Cas76/
трех нуклеотидов мотива, прилегающего к про!
Cas51/Cas61. Cascade связывает процессирован!
тоспейсеру (PAM, Protospacer Adjacent Motif),
ную крРНК длиной 61 нуклеотид с 32!нуклео!
расположенному на 3′!конце цепи!мишени, то
тидным спейсером [6-9]. Отжиг крРНК, свя!
есть после протоспейсера. Наличие PAM приво!
БИОХИМИЯ том 86 вып. 4 2021
532
ИСАЕВ и др.
дит к тому, что системы CRISPR!Cas не атакуют
двумя нуклеазными доменами (RuvC и HNH) и
собственный геном, так как PAM никогда не на!
способен вносить двухцепочечные разрывы [29,
ходится вблизи спейсеров в локусах CRISPR.
30]. Он представляет систему с минимальным
После формирования R!петли нуклеаза/хелика!
интерференционным комплексом, и поэтому
за Cas3 рекрутируется к комплексу [10]. Сначала
стал предпочтительным инструментом при про!
Cas3 вносит одноцепочечный разрыв в нетар!
ведении работ по редактированию генома с ис!
гетную цепь протоспейсера на 11-15 нуклеоти!
пользованием CRISPR!Cas [31-33]. Системы
дов ниже РAM, а затем начинает раскручивать и
типа V характеризуются наличием эффекторно!
расщеплять ДНК в направлении 3′→5′.
го белка Cpf1. Cpf1 содержит домен нуклеа!
В системах типа III эффектор обладает сход!
зы RuvC, аналогичный Cas9, в то время как до!
ной с Cascade структурой [11, 12]. Однако эф!
мен HNH отсутствует [34]. Эффектор типа V
фектор типа III распознает не дцДНК, а после!
способен разрушать таргетную двуцепочечную
довательности РНК, комплементарные спейсе!
ДНК PAM!специфическим образом [34, 35], в
рам крРНК [13, 14]. Распознавание транскриби!
то время как связывание Cpf1 с мишенями так!
рованной РНК!мишени стимулирует неспеци!
же приводит к проявлению его неспецифичес!
фическую ДНК!азную активность сигнатурного
кой активности в отношении одноцепочечной
HD (гистидин!аспартат) домена нуклеазы
ДНК [36]. Большинство систем типа V содержат
Cas10, что приводит к in situ деградации ДНК в
эффектор Cpf1, тогда как в подтипе V!F он заме!
транскрипционном пузыре [15-20]. В то же вре!
нён на Cas14. На настоящий момент Cas14 явля!
мя активируется Palm!домен Cas10, ответствен!
ется самым маленьким по размеру из всех изве!
ный за синтез вторичных мессенджеров (вари!
стных CRISPR!эффекторов. Cas14a представля!
антов циклического олигоаденилата (cOA)), ко!
ет собой CRISPR!эндонуклеазу, таргетирующую
торый может активировать вспомогательные
оцДНК, и для его активации не требуется
рибонуклеазы (например, Csm6/Csx1), неспе!
РAM [37]. Некоторые эффекторы подтипа V!U
цифично разрушающие транскрипты клеточно!
демонстрируют филогенетическое сходство с
го и вирусного происхождения [21-23]. Систе!
транспозазами TnpB [37, 38]. Система CRISPR!
мы типа III не используют PAM для предотвра!
Cas типа VI была предсказана методами биоин!
щения аутоиммунной реакции, так как эффек!
форматического анализа в 2015 г. [39]. Вскоре
торный комплекс не способен таргетировать
был описан эффекторный белок C2c2 из подти!
CRISPR!кассету или крРНК. Однако, чтобы из!
па VI!A. У Leptotrichia shahii локус VI!A содержит
бежать риска расщепления собственной ДНК в
только 3 гена (cas1, cas2, c2c2) и CRISPR!кассе!
случае транскрипции CRISPR!кассеты с проти!
ту. Нуклеаза C2c2, связанная с крРНК, образует
воположной цепи, крРНК включает тэг (мет!
эффекторный комплекс, который способен рас!
ку) - последовательность из 8 нуклеотидов
щеплять молекулы одноцепочечной РНК. В от!
CRISPR!повтора, которая ингибирует актив!
личие от всех известных CRISPR!нуклеаз, C2c2
ность Cas10 [17]. Если последовательность ми!
расщепляет РНК за счёт активности доме!
шени комплементарна спейсеру и тэгу крРНК,
на HEPN (Higher Eukaryotes и Prokaryotes
то интерференция не происходит.
Nucleotide!binding, нуклеотид!связывающий
Точный механизм CRISPR!иммунитета в
домен высших эукариот и прокариот). Мутация
системах типа IV пока полностью не изучен.
в каталитическом центре домена HEPN приво!
Сигнатурным белком таких систем являет!
дит к потере активности эффекторного комп!
ся Csf1. CRISPR!Cas типа IV были обнаружены
лекса, хотя при этом сохраняется РНК!связыва!
локализованными на плазмидах или в геномах
ющая активность мутантного белка [40]. Благо!
профагов, что указывает на возможность пере!
даря способности связываться с молекулами
носа аппарата CRISPR!Cas на мобильные гене!
РНК заранее определенным образом, нуклеа!
тически элементы (MGE) и обратно [24, 25].
за C2c2 может быть использована как эффек!
Сигнатурные гены CRISPR!Cas типа IV не со!
тивный инструмент редактирования РНК и ре!
провождаются генами адаптационного моду!
гуляции экспрессии генов.
ля cas1, cas2 [26]. Это приводит к предположе!
Особый интерес представляет причина раз!
нию, что белки типа IV могут принимать учас!
нообразия эффекторов CRISPR и их филогене!
тие во внутриклеточных процессах, не связан!
тические связи. Структуры эффекторных комп!
ных с адаптивным иммунитетом [27, 28].
лексов систем типа I и III весьма сходны. При!
Системы CRISPR!Cas класса II включают
нято считать, что эффекторный комплекс сис!
три типа: тип II, тип V и тип VI. В системах ти!
темы типа III является более древним. При этом
па II мономерный белок Cas9 в комплексе с
гены cas не всегда ассоциированы с CRISPR!
крРНК отвечает как за распознавание, так и за
кассетой и модулем адаптации cas1-cas2 [41].
деградацию таргетной дцДНК. Cas9 обладает
В MGE, называемых каспозонами, были обна!
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
533
ружены автономные гомологи Cas1. Белки Cas9
сов CRISPR, известна как процесс наивной
и Cpf1, характерные для типов II и V соответ!
адаптации. Во время этого процесса новые
ственно, подобны белку, кодируемому транспо!
спейсеры могут быть получены как из внехро!
зоном TnpB, и содержат домен эндонуклеа!
мосомной ДНК, так и из генома клетки!хозяи!
зы RuvC [42]. У белка Cas13 (система типа VI)
на, и только 50% новых спейсеров содержат
есть РНК!азные домены HEPN. Таким образом,
консенсусный PAM. Наивная адаптация необ!
системы CRISPR!Cas могли эволюционировать
ходима для последующего распознавания и
в результате заимствования генов модулей ин!
уничтожения впервые проникшего в клетку ин!
терференции и адаптации от каспозонов, в то
фекционного агента, и, по!видимому, является
время как эффекторные нуклеазы могут проис!
универсальной особенностью всех систем
ходить из геномов клеток или мобильных гене!
CRISPR!Cas. Известно, что этот процесс хотя
тических элементов.
бы частично зависит от активности комплек!
CRISPR&адаптация. Наиболее консерватив!
сов RecBCD клетки!хозяина [63]. RecBCD осу!
ными белковыми компонентами систем
ществляет процессинг остановленных реплика!
CRISPR!Cas являются Cas1 и Cas2, чьё участие
ционных вилок, и предполагается, что образую!
необходимо на стадии приобретения спейсе!
щиеся при этом фрагменты ДНК могут быть ис!
ров [43]. Как правило, гены cas1 и cas2 локализу!
пользованы комплексом Cas1-Cas2 для вставки
ются вблизи друг от друга, а кодируемые ими
в CRISPR!кассету. Отсутствие RecBCD приво!
белки образуют стабильный комплекс [44, 45].
дит к снижению эффективности процесса наив!
Делеция cas1 и cas2 не влияет на CRISPR!интер!
ной адаптации, но не останавливает её. Следо!
ференцию и созревание крРНК в системах ти!
вательно, комплекс Cas1-Cas2 может использо!
пов I [46-49], II [50, 51] и III [52]. Cas1 является
вать другие источники спейсеров. Вопрос об
эндонуклеазой [53, 54], которая также способна
участии других белков клетки!хозяина в процес!
разрушать структуры Холлидея. In vitro Cas1 мо!
се CRISPR!адаптации и регуляции этого про!
жет способствовать интеграции и рекомбина!
цесса стал изучаться только недавно [64-67].
ции ДНК [55]. Также in vitro Cas2 проявляет нук!
Например, было показано, что ДНК!полимера!
леазную активность как в отношении РНК, так
за I необходима как для наивной, так и для
и ДНК [56, 57]. Однако для процесса CRISPR!
праймированной адаптации (предположитель!
адаптации in vivo требуется только нуклеазная
но, для заполнения одноцепочечных повторов,
активность белка Cas1 [44]. Для адаптации
возникающих при встраивании спейсеров) [68].
in vivo также важна способность собирать ста!
Dorman и Bhriain [65] предположили, что отри!
бильный комплекс Cas1-Cas2. Мутации, кото!
цательная суперспирализация может влиять на
рые нарушают образование комплекса in vitro,
различные стадии взаимодействия белков
препятствуют
приобретению спейсеров
CRISPR с ДНК, включая адаптацию, экспрес!
in vivo [44]. Во время процесса встраивания но!
сию генов cas и локусов CRISPR и фактически
вого спейсера комплекс Cas1-Cas2 вносит од!
интерференцию.
нонитевой разрыв точно в месте соединения ли!
Наличие РAM позволяет различать геном хо!
дерного участка и повтора в CRISPR!кассете,
зяина, содержащий спейсер в составе CRISPR!
катализируя нуклеофильную атаку 3′!OH!конца
кассеты, и протоспейсер в составе молекулы!
встраиваемого спейсера на 5′!конец первого
мишени. Однако мутации PAM или seed!после!
повтора. Точно также другая цепь надрезается
довательности могут защитить вирусы от их рас!
на стыке первого повтора и спейсера, и 5′!конец
познавания и деградации эффекторным комп!
повтора присоединяется к 3′!концу нового
лексом
[30,69-72]. Следовательно, система
спейсера. В результате встроенный спейсер ок!
CRISPR!Cas должна обновлять свою «память»,
ружен одноцепочечными повторяющимися
чтобы избежать заражения мутировавшими бак!
последовательностями, которые позднее до!
териофагами. Для достижения этой цели неко!
страиваются благодаря активности репарацион!
торые типы систем CRISPR!Cas используют
ных белков клетки!хозяина [58]. Подобные про!
праймированную адаптацию - высокоэффек!
межуточные структуры также обнаруживаются в
тивный процесс получения новых спейсеров из
процессе интеграции мобильных элементов с
уже известных ранее встречавшихся фагов,
участием транспозазы, и это позволяет предпо!
фрагменты генома которых были сохранены в
ложить, что реакции приобретения спейсера и
CRISPR!кассете в качестве иммунологической
интеграции транспозонов механически похожи
памяти. Праймированная адаптация была пока!
друг на друга [59-62].
зана для систем CRISPR!Cas I!E [48], I!F [49,
Способность адаптационного комплек!
73], I!B [74, 75], I!C [76], I!U [77] и II!A [78].
са Cas1-Cas2 приобретать новые спейсеры, не!
Праймированная адаптация приводит к высо!
зависимо от активности эффекторных комплек!
коэффективному накоплению новых спейсеров,
БИОХИМИЯ том 86 вып. 4 2021
534
ИСАЕВ и др.
локализованных в цис!положении к «праймиру!
и защиту от вирусов. Члены этого семейства бел!
ющему» протоспейсеру, распознаваемому
ков также широко распространены у бактерий и
крРНК в составе эффекторного комплекса [79].
архей [87, 88]. Функции прокариотических бел!
Наблюдаемая эффективность праймированной
ков Argonaute (pAgo) пока ещё до конца не уста!
адаптации (измеряемая как количество удлинен!
новлены, но эти белки также способны участво!
ных CRISPR!кассет в популяции) очень низка в
вать в подавлении чужеродного генетического
том случае, если праймирующий протоспейсер
материала [89, 90]. Чтобы распознать мишени в
полностью совпадает с крРНК и содержит PAM,
виде нуклеиновых кислот, белки pAgo использу!
способный к консенсусной интерферен!
ют молекулы!гиды, но, в отличие от CRISPR!
ции (AAG или ATG - в случае системы I!E из
Cas и эукариотических Ago, гидом часто служит
Escherichia coli) [48, 80]. Эффективность прайми!
короткая одноцепочечная ДНК [91], хотя также
рованной адаптации стимулируется наличием
есть примеры РНК!гидов [92]. 5′!Конец гида
мутаций PAM или протоспейсера, которые сни!
загружается в домен MID белка pAgo, в то время
жают эффективность интерференции [30, 70,
как
3′!конец взаимодействует с доменом
73]. Тем не менее для праймированной адапта!
PAZ [93]. В экспериментах in vitro с белками pAgo
ции необходим функциональный белок Cas3,
из различных организмов было показано, что
что позволяет предположить существование
распознавание комплементарной мишени при!
функциональной связь между CRISPR!интер!
водит к её нуклеолитическому расщеплению ка!
ференцией и праймированной адаптацией [71,
талитическим доменом PIWI. В основном pAgo
81]. Недавнее исследование in vitro предполагает,
таргетируют молекулы ДНК [94, 95], хотя in vitro
что Cascade, Cas1-Cas2 и Cas3 образуют единый
некоторые из них могут также расщеплять
праймирующий комплекс, активность которого
РНК [96-98]. Пока неясно, может ли таргетиро!
приводит к эффективному отбору новых спейсе!
вание РНК быть важным для активности pAgo
ров [82]. Чтобы объяснить такую связь, были
in vivo. Тем не менее потенциально существуют
предложены две альтернативные модели. Сог!
все возможные комбинации pAgo!опосредован!
ласно одной модели, эффекторы, связавшиеся с
ных взаимодействий ДНК/РНК гида/мише!
протоспейсерами с определенными PAM, при!
ни [89, 93, 99]. In vivo присутствие pAgo влияет на
нимают специфическую конформацию, что
поддержание плазмид и ингибирует трансфор!
приводит к привлечению адаптационного комп!
мацию [92, 100]. Хотя считается, что pAgo также
лекса Cas1-Cas2, а также белка Cas3 и последую!
участвуют в противовирусной защите, экспери!
щему направленному сканированию мишени, и
ментально было получено лишь одно доказа!
отбору новых спейсеров [83]. Напротив, комп!
тельство с pAgo из Clostridium butyricum, чья гете!
лексы, которые были сформированы на мише!
рологичная экспрессия в клетках E. coli приво!
нях с подходящими для интерференции PAM, не
дила к понижению титра фага M13 с хроничес!
задействуют Cas1-Cas2, приводя только к ин!
ким жизненным циклом и литического фа!
терференции [84]. Вторая модель постулирует,
га P1vir. Однако механизмы pAgo защиты опре!
что очевидная разница между эффективностью
делены не были [90]. Основываясь на архитекту!
процесса праймированной адаптации с различ!
ре доменов, белки pAgo делятся на классы, и, что
ными мишенями является следствием деграда!
неожиданно, некоторые белки содержат катали!
ции неоптимальных мишеней [81, 85]. Посколь!
тически неактивный домен PIWI [89, 101].
ку большинство MGE способны реплицировать!
Функции таких белков, если они существуют,
ся и имеют собственные механизмы поддержа!
ещё предстоит определить.
ния числа копий, конкуренция между ослаблен!
Одним из основных вопросов, связанных с
ной CRISPR!интерференцией и подобными
pAgo!интерференцией, является механизм об!
системами может привести к ситуации, когда
разования и источник молекул!гидов, а также
фрагменты геномов MGE присутствуют в клетке
вопрос, каким образом клетки избегают аутоим!
в течение достаточно длительного времени, что,
мунного ответа. Секвенирование ДНК!гидов,
предположительно, позволяет произойти более
связанных с pAgo in vivo, показало, что они в ос!
медленной реакции адаптации [86].
новном происходят из активно реплицирую!
щихся или многокопийных элементов, включая
плазмиды и транспозоны [90, 100]. Для различ!
ИНТЕРФЕРЕНЦИЯ
ных белков pAgo была показана гид!независи!
С УЧАСТИЕМ БЕЛКА Argonaute
мая нуклеазная активность, приводящая к рас!
щеплению ДНК [102, 103]. Неспецифичное по
У эукариот белки Argonaute (Ago) играют
отношению к нуклеотидной последовательнос!
ключевую роль в процессе РНК!интерферен!
ти измельчение плазмид может привести к обра!
ции, вовлечённом в регуляцию экспрессии генов
зованию пула фрагментов ДНК различного раз!
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
535
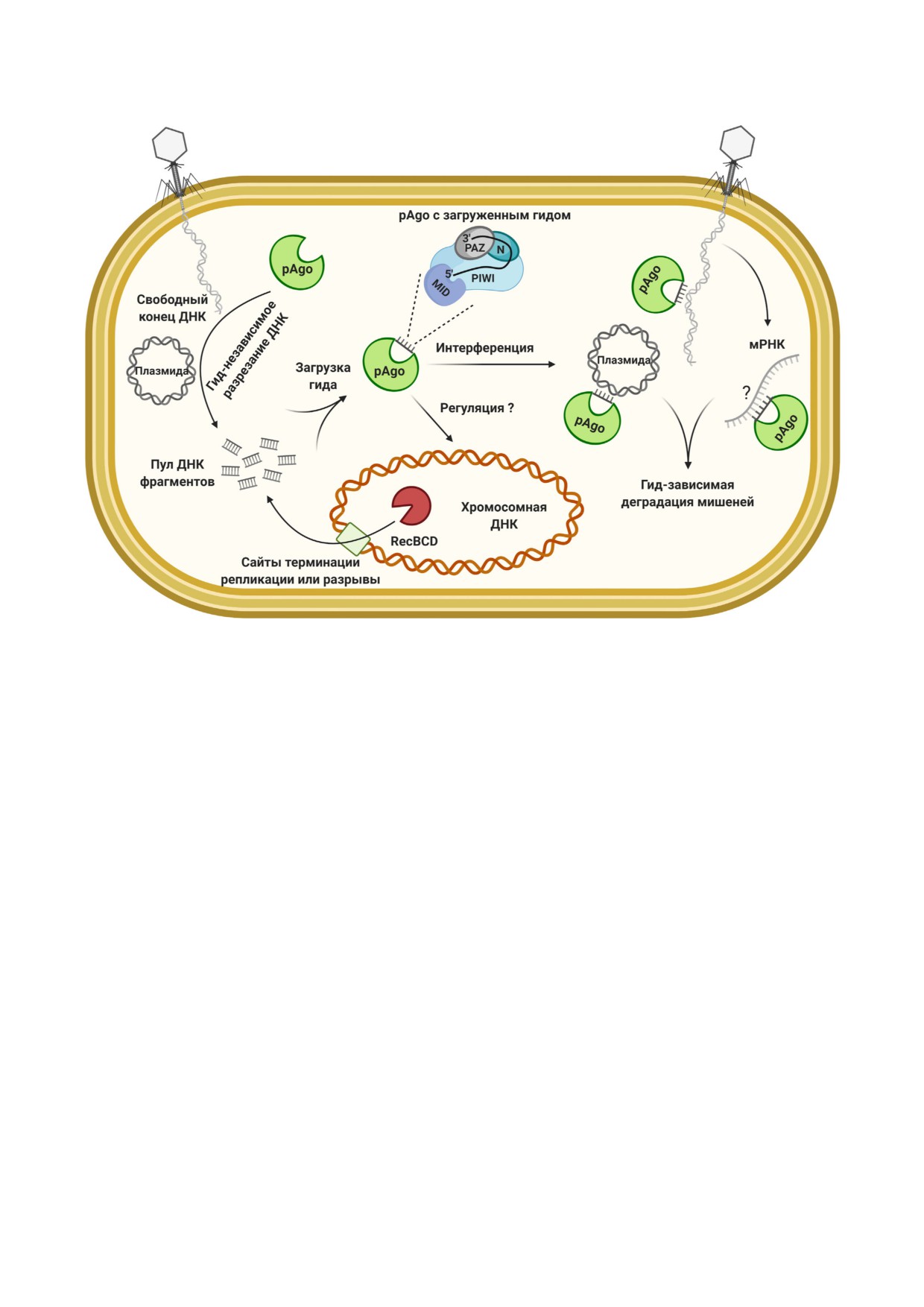
Рис. 2. Модель механизма действия pAgo и схематическое изображение белка с загруженным гидом
мера, и некоторые из них могут далее загружать!
родной ДНК [92]. Текущая модель механизма
ся в качестве молекул!гидов и активировать бо!
действия pAgo показана на рис. 2.
лее эффективную и зависимую от последова!
Описанный способ интерференции может
тельности деградацию комплементарных мише!
быть не очень эффективным против быстро
ней [103]. Для генерации гидов белки pAgo мо!
действующих литических фагов. Можно пред!
гут таргетировать свободные концы ДНК или
положить, что защитные механизмы с участием
интермедиаты процесса репликации, которые
pAgo приспособлены для контроля над менее
более часто присутствуют в экзогенной ДНК.
опасными мобильными элементами или могут
Подобно CRISPR!Cas, процесс генерации гидов
быть ассоциированы с другими защитными сис!
может зависеть от RecBCD комплексов [90].
темами, чтобы усилить защиту от вирусной ин!
Было показано, что, как и в случае C. butyricum,
фекции. В поддержку последнего предположе!
pAgo из Thermus thermophilus связывается с гида!
ния выступает тот факт, что гены pAgo внутри
ми, происходящими из областей терминации
защитных островков часто встречаются побли!
репликации, и, предположительно, вместе с
зости от генов нуклеаз или белков Cas [88, 101,
ДНК!гиразой участвует в контроле репликации
105].
клетки!хозяина путём разделения сцепленных
хромосом [104]. Компактизация ДНК, харак!
терная, например, для геномов архей, также мо!
ИНДУЦИРОВАННАЯ КЛЕТОЧНАЯ СМЕРТЬ
жет быть важна для различения собственного и
ИЛИ ОСТАНОВКА КЛЕТОЧНОГО ЦИКЛА -
чужеродного генетического материала белками
СИСТЕМЫ АБОРТИВНОЙ ИНФЕКЦИИ
pAgo [102]. Считается, что РНК!гиды, связан!
И ТОКСИН&АНТИТОКСИН
ные с pAgo из Rhodobacter sphaeroides, были нес!
пецифично отобраны из фрагментов деградиро!
В этом разделе мы рассмотрим абортивную
ванных транскриптов. Тем не менее белок сох!
инфекцию (Abi) в широком смысле - как кле!
раняет свою специфичность в отношении чуже!
точный ответ на инфекцию, который приводит
БИОХИМИЯ том 86 вып. 4 2021
536
ИСАЕВ и др.
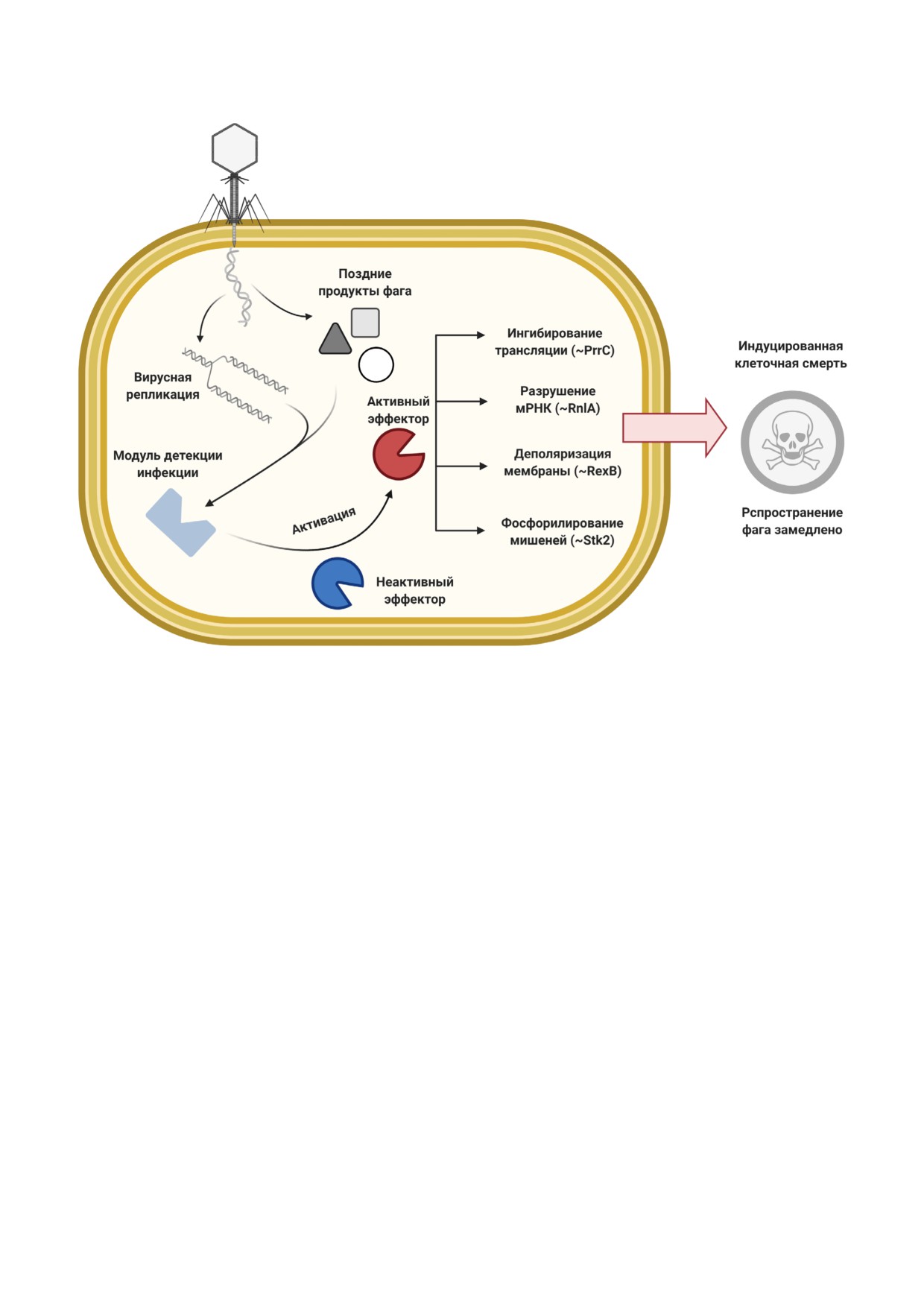
Рис. 3. Общий принцип абортивного ответа на инфекцию. Показаны примеры эффекторов с различными механизмами
действия
к прекращению метаболизма (бактериостати!
уровне сообщества и таким образом приносит
ческий эффект) или гибели клеток (бактерицид!
пользу клональной популяции [109, 110]. Неко!
ный эффект) до завершения жизненного цикла
торые системы, чьё действие фенотипически
вируса, что предотвращает образование актив!
напоминает Abi!ответ, могут непосредственно
ных фаговых частиц или снижает выход фагово!
интерферировать с вирусной инфекцией, но са!
го потомства [106, 107]. Чисто механически сис!
ми по себе не вызывают активную гибель кле!
темы Abi очень разнообразны. Как правило, они
ток. Однако их действие может сопровождаться
состоят из двух модулей (рис. 3). Один из них
лизисом клеток, вызванным произведёнными
обнаруживает фаговую инфекцию и передаёт
вирусом токсичными компонентами.
сигнал. При получении этого сигнала вто!
Системы Abi. Разнообразие кодируемых
рой (эффекторный) модуль останавливает мета!
плазмидами систем с механизмом Abi истори!
болизм клетки и/или вызывает её самоубий!
чески исследовалось на грамположительных
ство [107, 108]. Принято считать, что индуциро!
Lactococcus [106, 111]. Из 23 описанных систем,
ванное состояние подавленного метаболизма
обозначаемых как AbiA-AbiZ, механизм
даёт больше времени другим защитным меха!
действия был определен лишь в нескольких слу!
низмам для борьбы с инфекцией. Также счита!
чаях. Например, белок AbiZ взаимодействует с
ется, что некоторые системы Abi могут быть
холином и лизином фага ϕ31, вызывая прежде!
«последним средством защиты», т.е. они акти!
временный лизис клетки [112]; AbiK проявляет
вируют суицидальный ответ на поздних стадиях
безматричную ДНК!полимеризационную ак!
вирусной инфекции, в случае если фаг избежал
тивность [113]. Считается, что AbiA, AbiK,
действия других иммунных механизмов. Страте!
и AbiF ингибируют репликацию [106, 114], а ак!
гия самоуничтожения инфицированной клетки
тивность AbiB и AbiQ связана с деградацией
останавливает распространение инфекции на
мРНК [115, 116]; AbiD1 может вмешиваться в
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
537
процесс упаковки вирусной ДНК, ингибируя
биоплёнок и вносят вклад в антибиотикорезис!
нуклеазу, разрешающую конкатамеры вирусных
тентность (хотя последнее вызывает споры
геномов [117]. В то же время предполагается,
[129]), но также могут быть вовлечены в Abi про!
что AbiT и AbiV влияют на экспрессию поздних
тивовирусную защиту, поскольку фаговая ин!
фаговых белков [118, 119]. Для большинства
фекция часто влияет на метаболизм хозяина та!
этих систем неясно, как распознается фаговая
ким образом, что может вызывать потерю анти!
инфекция. В клетках Staphylococcus была обна!
токсина (рис. 4). Модули TA часто находятся
ружена система Abi с сенсорным модулем, осно!
внутри защитных островков, и между системами
ванным на фосфорилировании белков [120].
TA и Abi существует значительный обмен доме!
Фосфорилирование, как эффективный способ
нами [130]. Фактически нельзя провести чёткую
усиления сигнала, часто используется в эукари!
границу между Abi! и TA!системами, поскольку
отических противовирусных системах. Серин!
Abi - это стратегия защиты, а TA - организаци!
треониновая киназа Stk2 стафилококков акти!
онный/механистический принцип. Скорее, не!
вируется белком PacK фага ϕNM1 и фосфори!
которые системы Abi можно рассматривать как
лирует различные белки клетки!хозяина, вызы!
основанные на механизме TA, например, даже
вая ингибирование основных метаболических
некоторые системы Abi, которые обсуждались в
путей [120].
предыдущем разделе, можно рассматривать в
Множество механизмов Abi было также опи!
качестве отдельных токсинов, в то время как
сано на примере грамотрицательной бактерии
PrrC/EcoprrI можно рассматривать как настоя!
E. coli [121]. Так, белки Lit и PrrC, кодируемые
щую пару токсин!антитоксин. В зависимости от
криптическими профагами, действуют специ!
природы взаимодействия токсина и антитокси!
фично против фага T4. Протеаза Lit активирует!
на системы TA подразделяются на 6 типов. На!
ся в результате взаимодействия с консерватив!
пример, антитоксин может являться молекулой
ным пептидом Gol капсидного белка Т4 и оста!
РНК, которая напрямую ингибирует токсичный
навливает процесс трансляции, расщепляя фак!
белок (тип III) или регулирует уровень трансля!
тор элонгации трансляции EF!Tu [122]. РНКа!
ции мРНК токсина (тип I). В других типах сис!
за PrrC также ингибирует трансляцию, расщеп!
тем ТА антитоксин может быть белком, который
ляя tРНКLys. PrrC взаимодействует с комплексом
ингибирует токсин через белок!белковые взаи!
рестрикции Ecoprr системы рестрикции!моди!
модействия (тип II) или компенсирует воздей!
фикации (R!M) типа I, и активируется только
ствие токсина на другие мишени (тип IV)
при ингибировании комплекса рестрикции,
[129, 131].
вызванном кодируемым Т4 пептидом Stp [123].
Примером ответа Abi на основе модулей TA
В качестве другого интересного примера можно
могут служить системы ToxIN и RnlAB. ToxIN,
привести белок PifA, кодируемый F!плазмидой,
который исходно был идентифицирован как
который обеспечивает защиту клеток от фа!
AbiQ в клетках Lactococcus, широко распростра!
га T7 [124]. Этот мембраносвязанный белок ак!
нён в геномах бактерий и функционирует как
тивируется белками gp10 или gp1.2 фага T7 и,
система ТА типа III, в которой РНК!азная ак!
нарушая целостность мембраны, вызывает утеч!
тивность токсина ToxN блокируется в результа!
ку из инфицированной клетки ATP и других ма!
те взаимодействия с РНК!антитоксином ToxI
лых молекул [124, 125]. Система RexAB, кодиру!
[116, 132, 133]. Система RnlAB из клеток E. coli
емая профагом λ, также повышает проницае!
представляет ТА!модуль типа II, и она защища!
мость мембраны [126, 127]. Считается, что RexA
ет клетки от инфицирования фагом T4 [134].
распознает ДНК!белковые интермедиаты ви!
Токсин RnlA является стабильной РНКазой.
русных репликационных комплексов и стиму!
Антитоксин RnlB быстро разрушается протеаза!
лирует связанный с мембраной RexB, который
ми клетки!хозяина. Таким образом, если фаго!
образует ионный канал, что приводит к потере
вая инфекция препятствует непрерывной
мембранного потенциала и ингибированию
экспрессии генов, то предотвращение синте!
энергозависимых процессов [127].
за RnlB способствует проявлению токсичной
Защитные системы на основе взаимодействия
активности RnlA, что приводит к распаду внут!
токсин&антитоксин (TA). Системы токсин!анти!
риклеточной мРНК [134]. Гомологи RnlAB были
токсин представляют собой эгоистичные эле!
также обнаружены в плазмидах E. coli, и эта сис!
менты, состоящие из стабильной субъединицы
тема получила название LsoAB [135]. В качества
токсина и нестабильного антитоксина. В усло!
антитоксина для обеих систем может выступать
виях стресса деградация антитоксина приводит
белок Dmd, кодируемый фагом T4 [134, 135].
к повышению активности токсина и остановке
Многие системы ТА обладают обратимым
клеточного роста [128, 129]. Модули ТА прини!
действием и не вызывают гибели клеток. Тем не
мают участие в ответе на стресс, формировании
менее временное прекращение роста может
6 БИОХИМИЯ том 86 вып. 4 2021
538
ИСАЕВ и др.
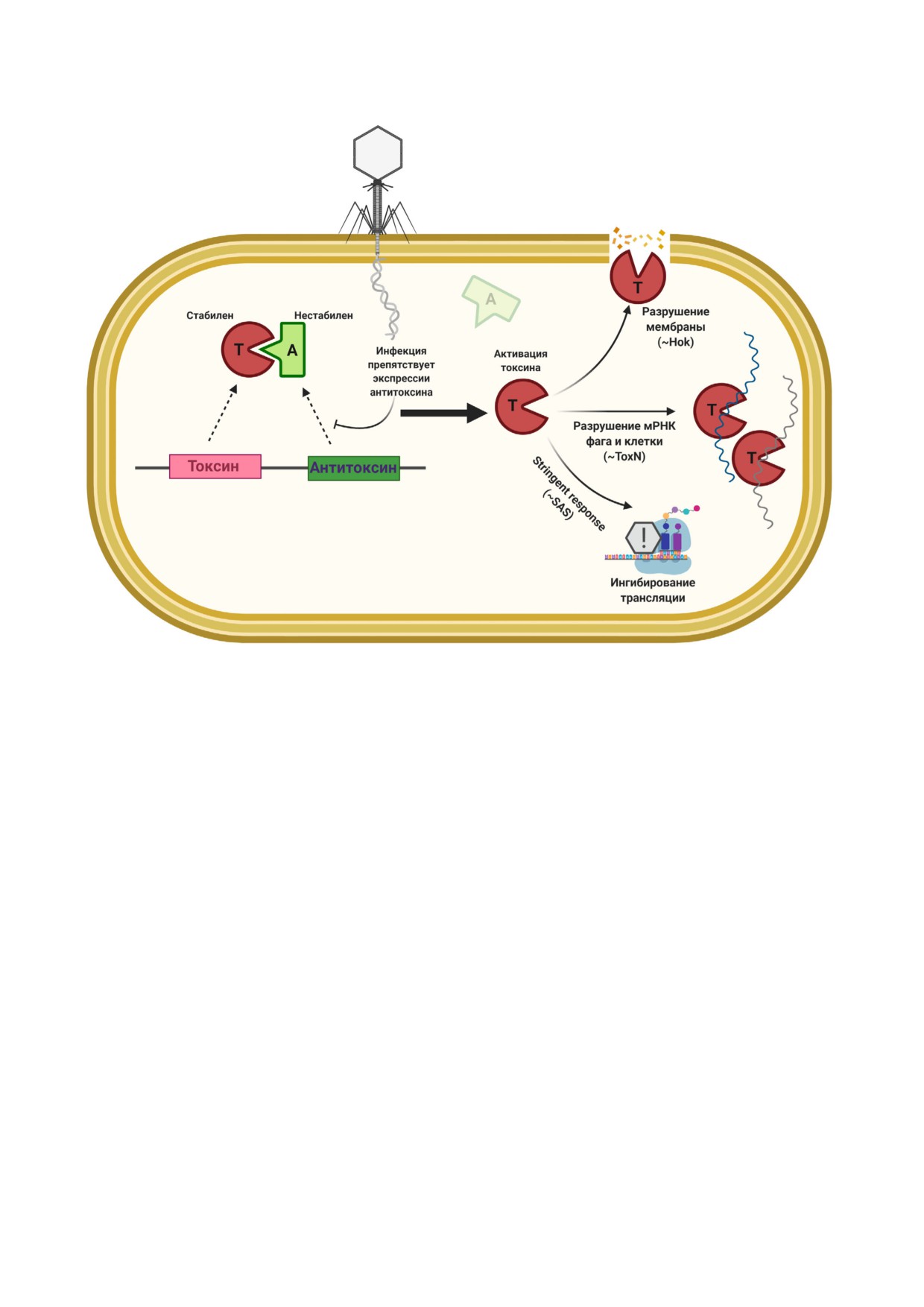
Рис. 4. Общий принцип абортивной инфекции, основанный на активности модулей TA. Приведено несколько примеров
эффекторов токсинов. (Stringent response - характерный клеточный ответ на стрессовые условия, связанный с синтезом
сигнальных молекул алармонов, SAS - Small Alarmone Synthetase)
обеспечить устойчивость к фагам. AbiE, систе!
син sok является антисмысловой РНК, вызыва!
ма ТА типа IV, является примером: ток!
ющей ингибирование синтеза Hok путём связы!
син AbiEii, транскрибируемый с промото!
вания с его мРНК [141]. В целом, роль TA!сис!
ра abiE, не взаимодействует напрямую с анти!
тем в защите от фагов плохо охарактеризована,
токсином AbiEi. Вместо этого AbiEi связывается
и этот вопрос остаётся противоречивым [142,
с промоторной областью и ингибирует тран!
143]. Однако, основываясь на обилии систем ТА
скрипцию всего TA!оперона
[136]. Токсин
и их участии в Abi!ответе у модельных бактерий,
AbiEii принадлежит к суперсемейству β!подоб!
можно ожидать, что защита от фагов на осно!
ных ДНК!полимераз и проявляет нуклеотидил!
ве ТА широко распространена среди бактерий
трансферазную активность [136]. Недавно было
[130, 142, 144].
показано, что MenT3, являющийся гомологом
Ретроны как защитные системы. Ретроны -
AbiEii в клетках Mycobacterium tuberculosis, может
это генетические элементы, которые кодируют
переносить пиримидины на акцепторную ветвь
обратную транскриптазу (RTase) и некодирую!
специфических тРНК [137]. В согласии с этим
щую РНК (нкРНК), которая используется об!
повышенная экспрессия токсина AbiE в клетках
ратной транскриптазой для образования кова!
Serratia вызывает прекращение роста клеток и
лентных гибридов РНК/ДНК [145]. До недав!
снижение уровня тРНК [138]. У E. coli систе!
него времени функциональная роль ретронов
ма MazEF типа II препятствует размножению
оставалась неизвестной, пока ряд работ не про!
фага P1 [139], а ТА!система hok/sok типа I сни!
демонстрировал, что ретроны могут являться
жает размножение фага T4 [140]. Последняя
частью трехкомпонентных систем ТА, участву!
система основана на холиноподобной актив!
ющих в защите от фагов посредством Abi
ности токсина Hok, в то время как антиток!
[146-149]. В нормальных условиях комплекс
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
539
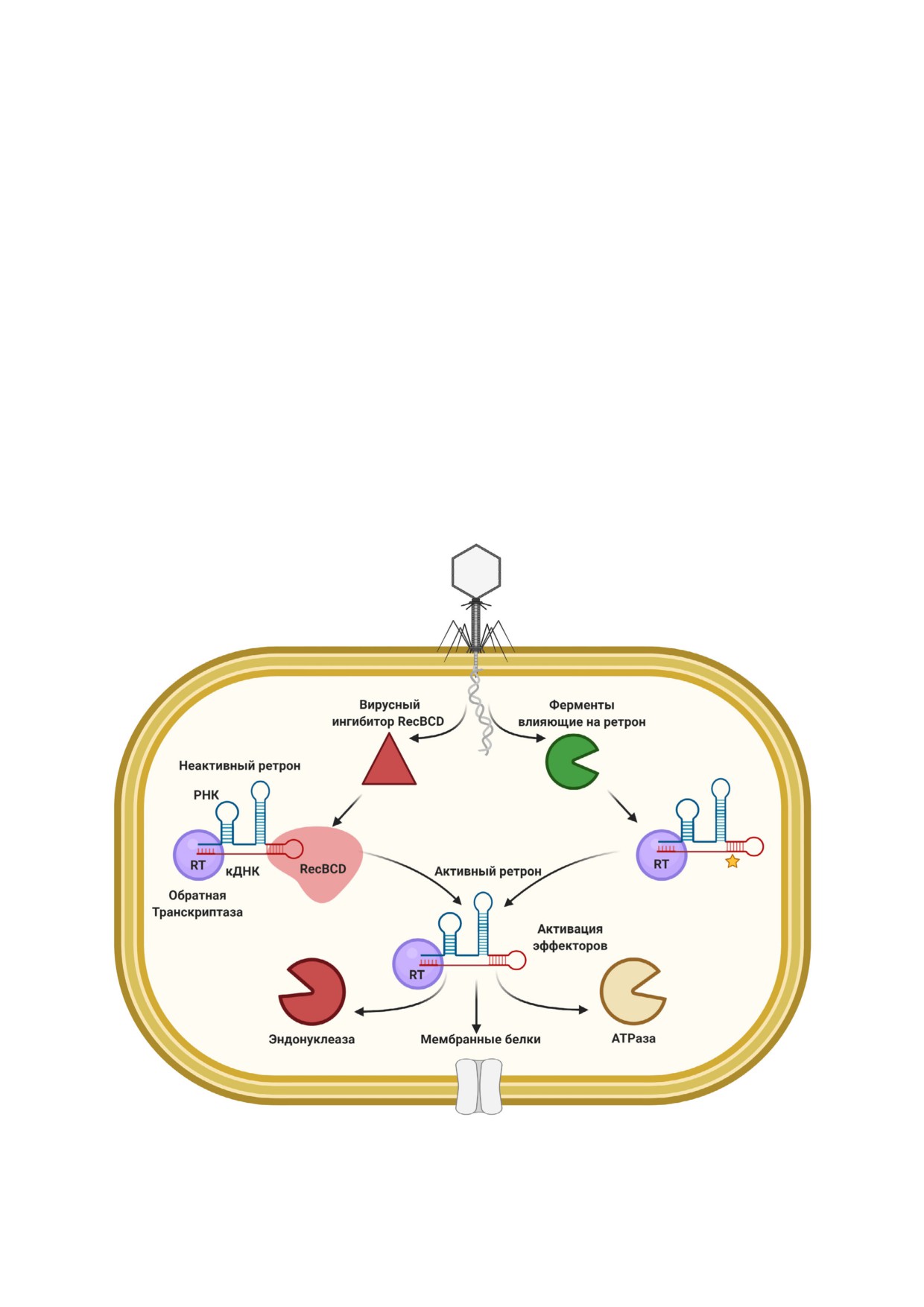
обратной транскриптазы с гибридом РНК/ДНК
тивность сопряжённого с системой заякорен!
неактивен, в то время как фаговая инфекция
ного в мембране эффектора, что вызывает
вызывает его активацию и передачу сигнала
преждевременный лизис клеток [146]. Для рет!
сопряженным эффекторам!токсинам (рис. 5).
рона Sen2 из клеток Salmonella enterica было по!
Антифаговая активность была продемонстри!
казано, что нарушение структуры ДНК части
рована для нескольких ретронов, при этом му!
ретрона вызывает активацию токсина RcaT
тации, влияющие на вторичную структу!
[147, 148]. Деградация или метилирование мо!
ру нкРНК и сайт ветвления или каталитичес!
лекул ДНК, ассоциированных с кодируемыми
кий мотив RTase, устраняли защиту [146, 149].
фагом эндонуклеазой RecE или метилтрансфе!
Внутри защитных островков было обнаружено
разой Dam, приводят к активации ответа, в то
~2000 систем, содержащих ретроны, при этом
время как отдельные белки, кодируемые про!
RTase могут быть слиты или находиться в не!
фагами, могут выступать в качестве блокаторов
посредственной близости от ATФаз, рибозил!
активации ретронов [147].
трансфераз и эндонуклеаз, выступающих в ка!
Антифаговые сигнальные системы на основе
честве эффекторных белков [146, 149]. Рет!
циклических олигонуклеотидов (CBASS). Недав!
рон Ec48 из клеток E. coli «охраняет» фер!
но было описано широко распространённое се!
мент RecBCD, который является одним из клю!
мейство систем, которые вызывают активацию
чевых барьеров на пути проникновения чуже!
Abi!ответа путём синтеза циклических олиго!
родной ДНК. Ингибирование RecB вирусными
нуклеотидов - CBASS (Cyclic!Oligonucleotide!
белками (например, Gam фага λ или gp5.9 фа!
Based Anti!Phage Signalling System) [107, 150,
га T7) активирует ретрон и высвобождает ак!
151]. CBASS!системы включают cGAS/DncV!
Рис. 5. Общий принцип действия абортивной инфекции на основе ретронов
БИОХИМИЯ том 86 вып. 4 2021
6*
540
ИСАЕВ и др.
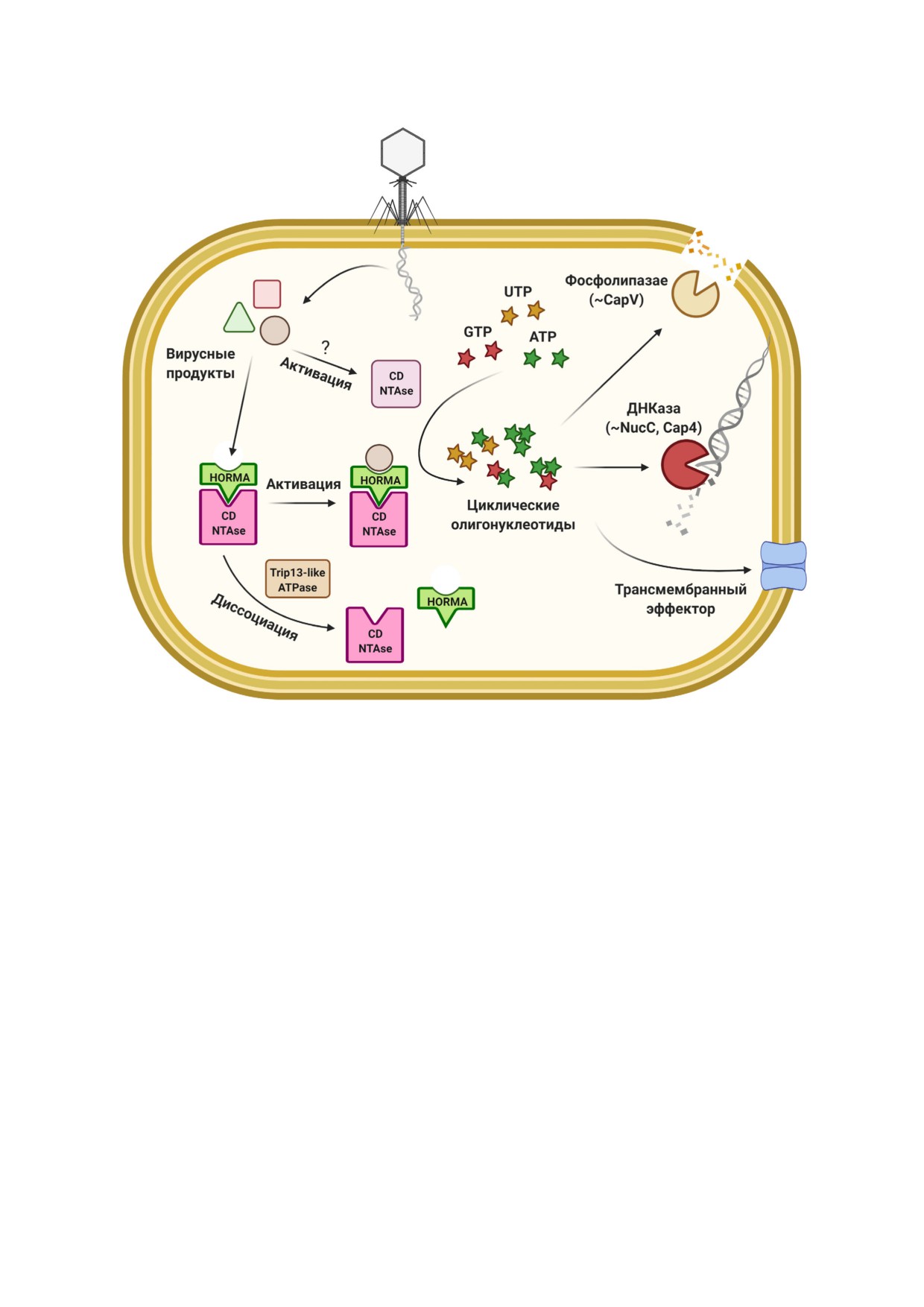
подобные нуклеотидил!трансферазы (CD!
структур STING!доменов многоклеточных жи!
NTase), и фаговая инфекция активирует их, за!
вотных и бактерий позволяет понять его переход
пуская синтез вторичного мессенджера (цикли!
от непосредственной эффекторной роли в
ческий GMP!AMP (cGAMP), циклический три!
CBASS к регуляторным функциям в иммуните!
аденилат (cAAA) и др.), который далее передаёт
те высших животных [158].
сигнал к различным эффекторам, индуцирую!
Многие бактериальные белки CD!NTase не!
щим программируемую гибель клетки (рис. 6)
активны в условиях in vitro [152]. Недавно было
[150-153]. Эти системы обеспечивают ещё одну
показано, что для их активности in vivo требуют!
связь между иммунной защитой эукариот и про!
ся вспомогательные белки [159]. У эукариот
кариот, поскольку у животных циклическая
белки c доменом HORMA связываются со спе!
GMP!AMP!синтаза (cGAS) участвует в проти!
цифическими закрывающими мотивами в бел!
вовирусном и воспалительном ответе через сиг!
ках!мишенях и собираются в сигнальные комп!
нальный путь cGAS!STING, активируемый
лексы [160]. В CBASS типа III из E. coli и
присутствием ДНК в цитозоле [154]. Гены оли!
Pseudomonas aeruginosa белки с доменом
гонуклеотид циклаз из семейства CD!NTAse об!
HORMA активируют CD!NTase, что приводит к
наруживаются примерно в 10% геномов прока!
продукции вторичного мессенджера цикличес!
риот, и более половины из них включены в сос!
кого триаденилата, который, в свою очередь, ак!
тав защитных островков. Разнообразие сис!
тивирует неспецифическую эндонуклеазную ак!
тем CBASS можно классифицировать в зависи!
тивность эффектора NucC, обеспечивая защиту
мости от состава оперона, типа эффектора или
от фагов по Abi!механизму [159, 161]. В отсут!
продуцируемой сигнальной молекулы [153].
ствии инфекции активность этой системы по!
Системы типа I содержат только CD!NTase и
давляется Trip13!подобной ATPазой, которая,
эффектор, в то время как другие системы также
предположительно, приводит к диссоциации
несут вспомогательные компоненты: гены с
комплекса CD!NTase с HORMA. Считается, что
убиквитин!ассоциированными доменами в ти!
распознавание специфических мотивов в фаго!
пе II, гены HORMA и Trip13!подобных доменов
вых белках вызывает конформационные изме!
в типе III или домены модификации нуклеоти!
нения в домене HORMA и активирует CD!
дов в редко встречающихся CBASS типа IV
NTase этого белкового комплекса. Интересно,
[153]. Первой экспериментально исследованной
что эффектор NucC может быть обнаружен как
системой была CBASS типа II из Vibrio choler%
вспомогательная эндонуклеаза в системах
ae [151, 155, 156]. Ядро этой системы состоит из
CRISPR!Cas типа III, которые также полагают!
двух компонентов: cGAS фермента DncV и фос!
ся на сигналинг, связанный с циклическим оли!
фолипазы CapV (Cap - белок, ассоциирован!
гоаденилатом [21, 22, 161]. Другим эффектором
ный с CD!NTase), реагирующей на cGAMP.
CBASS, который может реагировать на различ!
Этих двух компонентов вполне достаточно для
ные типы циклических олигонуклеотидов, яв!
обеспечения защиты от фага P1, однако для за!
ляется белок CapIV из Enterobacter cloacae [162],
щиты от других фагов требуются два вспомога!
который стал первым описанным членом цело!
тельных белка, несущих домены E1, E2 и JAB,
го семейства белков. Белки CapIV распознают
типичные для ферментов убиквитинирова!
вторичные мессенджеры через различные доме!
ния [151]. Сенсорный механизм до сих пор не
ны SAVED, состоящие из
2!х CRISPR!
определен, но было показано, что после зараже!
родственных субъединиц CARF, которые инду!
ния фагом клетки продуцируют cGAMP, кото!
цируют олигомеризацию и способствуют прояв!
рый вызывает активацию фосфолипазы CapV,
лению активности эффекторной эндонуклеазы,
разрушающей клеточную мембрану до заверше!
ранее известной как DUF4297 [162].
ния жизненного цикла вируса. Кроме фосфоли!
Несмотря на то, что в последние годы было
пазы, среди известных эффекторов систем
описано большое разнообразие бактериальных
СBASS присутствуют эндонуклеазы или белки,
CD!NTase и сопряжённых с ними эффекторов,
несущие трансмембранные домены [153, 157].
остаётся много вопросов. Как эти системы
Интересна группа эффекторов CBASS, содер!
чувствуют фаговую инфекцию? Каковы функ!
жащих домен, гомологичный эукариотическому
ции вспомогательных белков? Все ли CBASS!
домену STING (Stimulator of Interferon Genes;
системы участвуют в защите от фагов или неко!
стимулятор генов интерферона). У бактерий
торые из них могут выполнять другие функции?
распознавание циклических олигонуклеотидов
Каковы издержки экспрессии генов CBASS, и
с помощью STING приводит к активации со!
есть ли дополнительные механизмы, ограничи!
пряжённого домена TIR (Toll/interleukin!1
вающие их возможную токсичность в отноше!
receptor) в составе эффектора и последующей
нии самой клетки, как в случае негативной регу!
деградации NAD+ [158]. Сравнительный анализ
ляции Trip13!подобной ATPазы?
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
541
Рис. 6. Модель CBASS!опосредованного иммунного ответа
ЗАЩИТА, ОПОСРЕДОВАННАЯ
экспрессии литических генов профага, вторич!
ПРОФАГАМИ
ная литическая инфекция фагом, регулируемым
тем же или подобным репрессором, будет по!
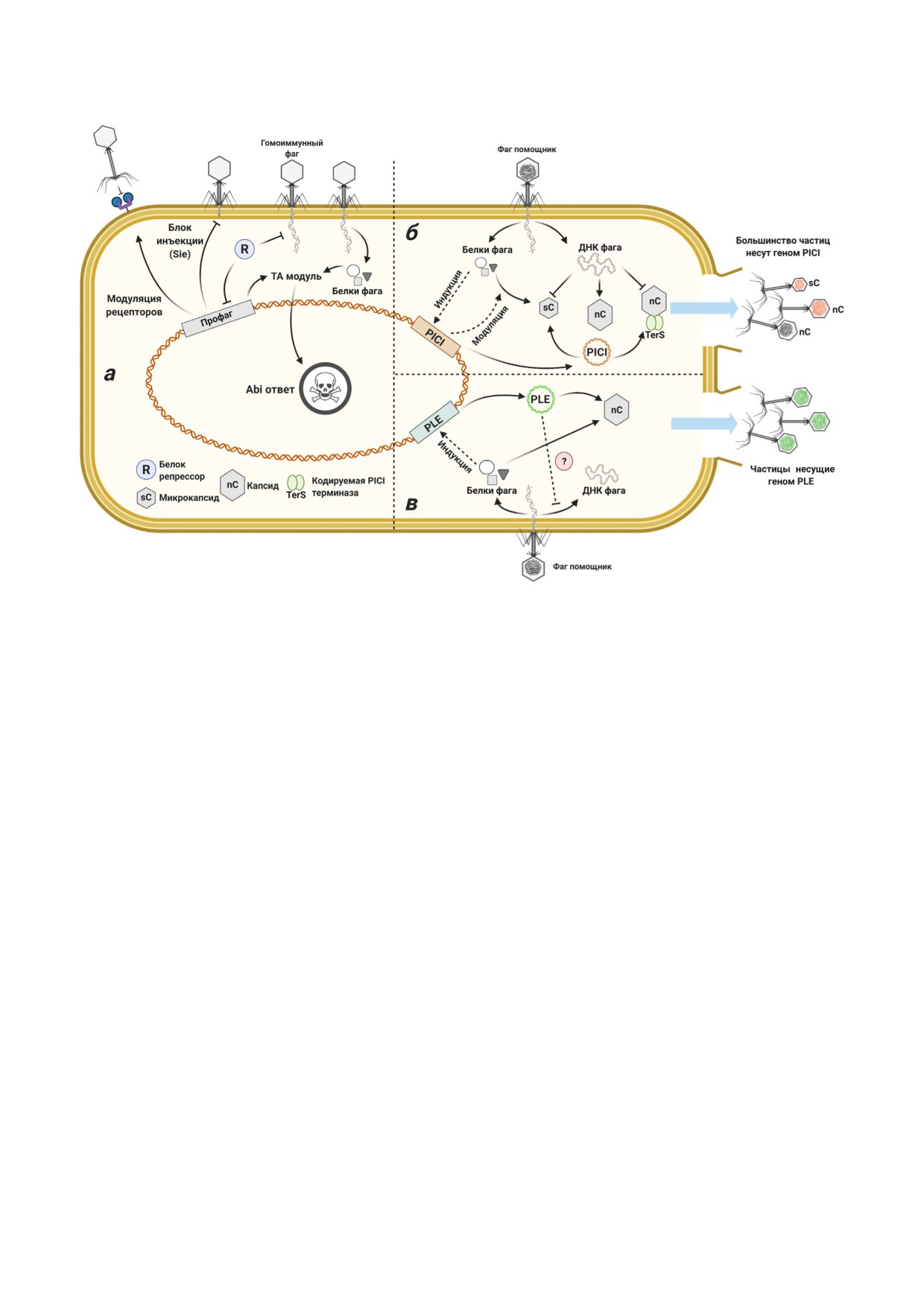
Умеренные фаги могут встраивать свои гено!
давлена [169]. Хотя защита, ассоциированная с
мы в хромосому клетки!хозяина с образованием
репрессорами, действует в узком диапазоне,
профагов [163]. Большинство известных бакте!
систематические исследования профагов в
рий несут профаги, и взаимодействие профага с
клетках P. aeruginosa и Mycobacterium smegmatis
хозяином можно рассматривать как мутуалис!
обнаружили многочисленные защитные гены,
тическое: поскольку выживание профагов зави!
которые обеспечивают гетеротипическую защи!
сит от клетки!хозяина, для профага выгодно
ту [165, 166].
исключить вторичное инфицирование лизоге!
Гены, которые не являются необходимыми
низированной клетки [164]. Действительно,
для выживания профага, но в то же время способ!
профаги часто несут гены, ассоциированные с
ствуют повышению устойчивости клетки!хозяи!
противовирусной защитой (рис. 7, а) [165-168].
на, были названы как «мороны» (т.е. добавляю!
Самый простой способ подавления инфициро!
щие больше («more on») к фенотипу). Мороны
вания гомоиммунными фагами - экспрессия
могут влиять на многие процессы внутри клет!
репрессорного белка - фактора транскрипции,
ки!хозяина, включая подвижность клеток, ус!
который регулирует переключение между лити!
тойчивость к антибиотикам, метаболизм и защи!
ческой и лизогенной жизненными стратегиями
ту от фагов [170, 171]. Некоторые системы ТА,
фага. Так как белки!репрессоры постоянно при!
кодируемые профагами, и белки Sie (исключе!
сутствуют в лизогенной клетке для подавления
ние суперинфекции), которые можно рассмат!
БИОХИМИЯ том 86 вып. 4 2021
542
ИСАЕВ и др.
,
Рис. 7. Защита, ассоциированная с профагами и мобильными генетическими элементами (MGE). а - Кодируемые про!
фагами защитные системы. б - Модель индукции PICI и интерференции. в - Модель индукции элементов, подобных
хромосомным островкам (PLE) и интерференции
ривать в качестве моронов, уже обсуждались.
даче сигнала молекулами алармонов [166, 179].
Мороны могут также оказывать влияние на кле!
Токсичный компонент - фермент SAS (Small
точную поверхность, препятствуя распознава!
Alarmone Synthetase), который синтезиру!
нию рецепторов. Например, фаги D3 и φ297 кле!
ет ppGpp или ppApp - сигнальные молекулы,
ток P. aeruginosa могут изменять конформацию
характерные для ответа клетки на стрессовые
субъединиц O!антигена в липополисахариде
условия, связанные с аминокислотным голода!
(LPS), кодируя собственную полимеразу O!ан!
нием (stringent response) - что вызывает прекра!
тигена [172-174], в то время как некоторые про!
щение роста [180]. Антитоксины напрямую свя!
фаги клеток Shigella и E. coli могут блокировать
зываются с синтазой или разрушают сигналь!
дальнейший рост цепи O!антигена за счёт ацети!
ный алармон [179]. Другая система Abi, широко
лирования или глюкозилирования [175, 176].
распространённая в профагах грамотрицатель!
Профаги микобактерий кодируют различ!
ных бактерий, состоит из эффектора BstA, кото!
ные системы исключения повторной инфекции.
рый локализуется совместно с реплицирующей!
Например, фаги Sbash и CarolAnn несут предпо!
ся ДНК фагов!мишеней и интерферирует с про!
лагаемые TA!модули с мембраносвязанными
цессом репликации по неопределённому меха!
эффекторами [177, 178]. Было показано, что
низму [181]. Эта система представляет собой ин!
другие микобактериофаги кодируют эндонукле!
тересный пример предотвращения автоиммун!
азы рестрикции, мембранные белки Sie или
ного ответа при активации профагов: BstA инак!
синтазы гуанозинпентафосфата (p)ppGpp, спо!
тивируется за счёт связывания со специфичес!
собные защитить клетку!хозяина [166]. Систе!
ким анти!BstA локусом (aba) в геноме фага и, та!
ма, кодирующая (p)ppGpp синтазу и её сопря!
ким образом, не предотвращает развитие лити!
жённый ингибитор, была первоначально найде!
ческой инфекции.
на у фага Phrann (белки gp29 и gp30), а позже
Мороны могут сильно отличаться друг от
было показано, что подобные системы широко
друга даже между близкородственными штам!
распространены у профагов и представляют но!
мами. Недавнее исследование P2! и P4!подоб!
вое семейство модулей TA, основанных на пере!
ных профагов выявило беспрецедентное разно!
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
543
образие компактных защитных систем в специ!
ные гены, включая токсины и факторы вирулент!
фических локусах их геномов [182]. Помимо из!
ности, и их присутствие может влиять на пато!
вестных противовирусных систем, в этих горя!
генность клетки!хозяина [185, 192]. Жизненный
чих точках были зарегистрированы множест!
цикл SaPI связан с инфицированием фагом!по!
венные кластеры генов, которые несут предска!
мощником, и обнаружение специфических бел!
занные защитные домены (TIR, SIR2, нуклеа!
ков фагов!помощников снимает репрессию,
зы, ATPазы) или домены с неустановленной
обусловленную действием Stl [193, 194]. После
функцией. Защитная активность была подтверж!
активации SaPI могут мешать репродукции фа!
дена для 14 новых систем. Например, предпола!
га!помощника при помощи нескольких различ!
гается, что система PARIS, состоящая из
ных механизмов (рис. 7, б). Они способствуют
ATPазы и белка DUF4435, обеспечивает абор!
сборке миниатюрных капсидов, способных заг!
тивный ответ на инфекцию, запускаемый анти!
ружать геном SaPI, но исключающих более
рестрикционным белком Ocr фага Т7, который
крупный геном фага!помощника [195]. SaPI
ингибирует защитные системы хозяина R!M и
также могут препятствовать упаковке вирусного
BREX [182-184]. Идентификация горячих то!
генома, ингибируя малую субъединицу терми!
чек разнообразия в других профагах может
назы TerS, в то время как загрузка собственного
представлять собой ценный и простой инстру!
генома SaPI обеспечивается SaPI!кодируемой
мент для обнаружения новых систем защиты от
терминазой [195]. Наконец, кодируемые SaPI
фагов.
белки могут связываться с вирусными фактора!
ми транскрипции, нарушая экспрессию позд!
них генов [196]. Хотя эти механизмы были опи!
ПАРАЗИТЫ ФАГОВ.
саны в случае стафилококковых SaPI, PICI ши!
МОЛЕКУЛЯРНОЕ ПИРАТСТВО PICI И PLE
роко распространены среди бактерий [190, 197],
и они могут использовать сходные механизмы
Мобильные генетические элементы могут
для поддержания их паразитического образа су!
случайным образом переноситься фагами в про!
ществования. Например, PmCI172 из клеток
цессе генерализованной трансдукции. Но неко!
Pasteurella multiocida способствует формирова!
торые MGE приобрели способность контроли!
нию капсидов небольшого размера, когда клет!
ровать механизмы упаковки фаговой ДНК в
ка инфицирована Mu!подобным фагом!помощ!
капсиды, чтобы использовать их для загрузки
ником, в то же время EcCICFT073 из кле!
собственных геномов - явление, которое иногда
ток E. coli кодирует белок Rpp, который вызыва!
называют молекулярным пиратством [185-188].
ет репрограммирование белка TerS фага λ для
Индуцируемые фагами хромосомные остров!
упаковки генома PICI [190, 198].
ки (PICI, Phage Inducible Chromosomal Islands)
PLE. Другой тип сателлитных MGE, специ!
широко распространены в геномах грамполо!
фичных для V. cholera и препятствующих рас!
жительных и грамотрицательных бактерий. Их
пространению фага ICP1, называется PLE - фа!
индукция из хромосомы зависит от инфициро!
го!индуцируемый элемент, подобный хромосом!
вания фагами!помощниками, и, с точки зрения
ным островкам (Phage!inducible Сhromosomal
хозяина, может рассматриваться как вариант
Island!like Element) [199]. Аналогично структу!
Abi!ответа [189, 190]. Заражённая клетка в ко!
рам PICI, PLE распознает белки фага!помощ!
нечном итоге лизирует, но выход потомства су!
ника ICP1, чтобы запустить процесс вырезания
перинфицирующего литического фага снижает!
из генома клетки, и использует ферменты и
ся, и большинство высвобождаемых частиц не!
структурные компоненты ICP1. Однако геном!
сут геном PICI вместо генома фага [191]. Пос!
ная организация PLE имеет свои особенности,
кольку элементы PICI не проходят литический
и, в отличие от PICI, которые лишь подавляют
цикл самостоятельно и ограничивают распрост!
процесс размножения фагов!помощников, при
ранение фагов!помощников, их присутствие
индукции PLE вовсе не происходит формирова!
может быть полезным для популяции бактерий.
ния инфекционных частиц ICP1 (рис. 7, в)
PICI. Наиболее изученной группой струк!
[199-202]. Было показано, что в клетках, несу!
тур PICI, которая была обнаружена у
щих PLE, репликация ICP1 в значительной сте!
Staphylococcus, является SaPI (островки патоген!
пени ингибируется. Также было показано, что
ности Staphylococcus aureus). Эти хромосомные
отдельные PLE модулируют экспрессию вирус!
островки имеют размер менее 15 т.п.н., они ко!
ных генов [202, 203]. Дополнительным механиз!
дируют интегразу, эксцизионный белок и ре!
мом, который может способствовать подавле!
пликационные компоненты. Экспрессия этих
нию ICP1, является продукция кодируемо!
генов находится под строгим контролем главно!
го PLE белка LidI, который нарушает систему
го репрессора Stl. SaPI часто несут дополнитель!
интерференции лизиса ICP1 и ускоряет лизис
БИОХИМИЯ том 86 вып. 4 2021
544
ИСАЕВ и др.
клеток [204]. Следует отметить, что ICP1, в свою
активности этого белка in vitro и потере устойчи!
очередь, кодирует систему CRISPR!Cas, наце!
вости клеток к фагу in vivo, проводя связь между
ленную на PLE [205].
защитными свойствами системы Thoeris и гид!
ролизом NAD+ [207, 211]. Домен TIR может слу!
жить передатчиком сигнала в иммунных путях
СИСТЕМЫ С ВЕРОЯТНЫМИ
эукариот [212], также он был выявлен в других
НОВЫМИ МЕХАНИЗМАМИ
прокариотических системах (например, CBASS).
В системе Thoeris фаговая инфекция активирует
Выявление кластеров консервативных ге!
домен TIR белка ThsA, что приводит к синтезу
нов, обнаруженных внутри защитных островков
изомера циклической ADP!рибозы. Далее сиг!
доступных прокариотических геномов, позво!
нал передаётся на белок ThsB, что приводит к
лило предсказать множество новых типов сис!
абортивному ответу на инфекцию, связанному с
тем защиты [149, 206-208]. Недавно была про!
расщеплением NAD+ [210]. Множественные ко!
ведена системная проверка этих предсказаний.
пии гена thsB могут быть ассоциированы с одним
Процесс включал клонирование 28 кандидат!
и тем же геном thsA, при этом диверсификация
ных систем из различных бактерий и их
доменов TIR обеспечивает защиту от более ши!
экспрессию в суррогатных хозяевах: грамотри!
рокого спектра инфицирующих фагов [210].
цательной E. coli или грамположительной
Было показано, что система Wadjet не актив!
Bacillus subtilis с последующим скринингом про!
на против фагов, но подавляет трансформацию
тив коллекции фагов различных семейств.
плазмид [207]. Система Wadjet состоит из четы!
В этой работе были подтверждены 10 новых сис!
рех компонентов: белков JetABC, гомологичных
тем, которые были названы в честь мифологи!
белкам поддержания структуры хромосомы
ческих божеств!покровителей [207]. Активность
(SMC), которые участвуют в сегрегации геномов
систем Druantia, Kiwa и Zorya была валидирова!
плазмид или клетки!хозяина, и белка JetD с
на в клетках E. coli, а активность систем Gabija,
предполагаемым доменом топоизомеразы VI
Hachiman, Lamassu, Thoeris, Septu, Shedu и
[207, 213]. Феномен подавления поддержания
Wadjet была продемонстрирована в клет!
плазмид, связанный с неканоническими белка!
ках B. subtilis. Было показано, что множество
ми SMC, был описан у M. smegmatis, где модуля!
белковых доменов, не типичных для уже изу!
ция статуса суперспирализации плазмиды нару!
ченных защитных систем, участвуют в противо!
шала её сегрегацию в дочерние клетки [214].
вирусной защите, и это предполагает новые ме!
Можно предположить, что Wadjet действует ана!
ханизмы действия для обнаруженных систем.
логичным образом для ограничения распрост!
Система Zorya активна против фагов как с
ранения чужеродных внехромосомных генети!
оцДНК, так и с дцДНК геномами. Эта система
ческих элементов.
кодирует белки ZorA и ZorB, гомологичные ком!
В другом недавнем исследовании применя!
понентам MotAB, формирующим протонные ка!
лась аналогичная логика экспериментальной
налы жгутикового мотора бактерий [209]. В сис!
проверки систем!кандидатов, но использовался
темах Zorya типа I белок ZorAB может сопровож!
другой алгоритме предсказания защитных ге!
даться предсказанной малой нуклеазой ZorE. В
нов. Вместо проведения оценки обилия белко!
системах типа II - хеликазой/ATPзой ZorC и
вых доменов все последовательности, встречаю!
Pfam00691!содержащим белком ZorD. Инфици!
щиеся на расстоянии 10 генов от известных за!
рование фагом культуры, несущей систе!
щитных систем, были проанализированы на
му Zorya, провоцирует преждевременный лизис
предмет частоты их локализации в пределах за!
клеток, предположительно, в результате Abi!от!
щитных островков. Всего было предсказано бо!
вета, опосредованного ZorAB эффектором, при!
лее 7000 генов!кандидатов, вовлечённых в защи!
водящим к деполяризации мембраны. Эта точка
ту от фагов, многие из которых содержали неан!
зрения была далее поддержана данными о том,
нотированные домены или домены с неизвест!
что мутации аминокислотных остатков, для ко!
ными функциями. В результате для дальнейших
торых было предсказано участие в протонном
исследований было отобрано 48 предсказанных
транспорте, приводят к снижению защитной
систем. Противовирусная активность 29 новых
функции системы Zorya [207].
систем была подтверждена с помощью гетеро!
Другая исследованная система (Thoeris) сос!
логичной экспрессии в клетках E. coli [149].
тоит из белка ThsA с доменами SIR2 и SLOG и
Исследование независимо обнаружило связь
белка ThsB, несущего домен TIR [207, 210]. Не!
ретронов с защитой от фагов. Кроме того, было
давно были опубликованы структуры обоих бел!
показано, что обратные транскриптазы, не свя!
ков [211]. Мутации в NAD!связывающем карма!
занные ретронами, также участвуют в защите, и
не домена SIR2 белка ThsA приводили к потере
6 групп RTase были объединены под общим наз!
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
545
ванием DRT (defence!associated RTase - обрат!
положено, что они действуют через меха!
ные транскриптазы, ассоциированные с защи!
низм Abi. Мутационный анализ подтвердил зна!
той). Некоторые DRT ассоциированы со вспо!
чимость ATPазы и предполагаемых эффекторов
могательными белками, тогда как DRT типа I
защитной системы AVAST [149].
сливаются с доменом нитрилазы, который часто
Функциональные домены других новых сис!
вовлечён в метаболизм малых молекул [215].
тем включают нуклеазы (например, было пока!
Мутации предсказанных каталитических ами!
зано, что DUF4297 из системы Lamassu являет!
нокислотных остатков RTase или нитрилазы
ся нуклеазой, участвующей также в CBASS), хе!
приводили к инактивации системы. Транскрип!
ликазы, SIR2, ДНК!связывающие белки, фос!
томный анализ определил, что DRT разных ти!
фатазы и ATPазы, а также множество неанноти!
пов не мешают экспрессии ранних вирусных ге!
рованных доменов [149, 207, 208, 217]. Напри!
нов, в то время как система типа I влияет на на!
мер, крупная система Druantia состоит в основ!
копление поздних вирусных транскриптов. Ме!
ном из генов с неизвестными функциями. Эти
ханизм защиты DRT остаётся невыясненным.
открытия существенно расширили наши пред!
Более детально была изучена система
ставления о многообразии биохимических ак!
RADAR (Restriction by Adenosine Deaminase
тивностей, которые могут быть задействованы
Acting on RNA; рестрикция с участием аденозин!
при защите клеток от вирусов, и должны проло!
деаминазы, воздействующей на РНК). Ядро этой
жить путь для дальнейших экспериментальных
системы образовано ATPазой RdrA и аденозин!
исследований новых механизмов защиты.
деаминазой RdrB, которые могут сопровождать!
ся вспомогательными белками (SLATT или
Csx27, которые также участвуют в защите
ВЫВОДЫ
CRISPR типа VI). Анализ транскриптома инфи!
цированных фагами клеток выявил замены A
Взаимодействие вирусов со своими хозяева!
на G в секвенированных ридах, в соответствии с
ми - это динамичный процесс, который приво!
предсказанным RdrB!опосредованным образо!
дит к созданию множества стратегий нападения
ванием инозина в РНК клетки!хозяина и вирус!
и защиты. Гены прокариот, связанные с устой!
ной РНК. Было показано, что предпочтительны!
чивостью к фагам, являются одними из наибо!
ми мишенями для редактирования являются
лее быстро эволюционирующих, и высокая ско!
вторичные структуры РНК типа шпилек. Инфи!
рость обновления прокариотических адапта!
цирование культуры клеток RADAR+ фагами
ций, сопровождаемая вирусными контрадапта!
при высокой множественности инфекции (MOI)
циями, часто описывается в терминах гипотезы
приводит к остановке клеточного цикла, что
Красной Королевы - каждая из сторон, прини!
предполагает защитный ответ через механизм
мающих участие в этой гонке вооружений, обя!
Abi. Такое поведение было приписано редакти!
зана приобретать новые адаптации лишь для
рованию транспортно!матричной РНК (тмРНК)
сохранения «статуса кво» [218, 219]. Долгосроч!
хозяина, которая высвобождает рибосомы, «за!
ные эволюционные исследования показывают,
стрявшие» на мРНК в процессе трансляции. Кро!
что скорость накопления мутаций выше в том
ме того, было показано, что экспрессия опреде!
случае, когда фаг эволюционирует вместе с
ленных ДНК!связывающих белков фага T2 за!
клеткой!хозяином, по сравнению с ситуацией,
пускает редактирование РНК в неинфицирован!
когда фаг эволюционирует, в то время как клет!
ной культуре клеток, что позволяет предполо!
ка!хозяин сохраняет свой генотип [220, 221].
жить механизм активации ответа RADAR [149].
Одним из примеров, на котором можно оценить
NTPase из суперсемейства STAND участвуют
динамику этих взаимодействий, является кон!
в путях передачи сигнала в процессе програм!
фронтация элементов PLE клеток V. cholerae с
мированной гибели клеток эукариот [216], од!
фагом ICP1. Используя образцы V. cholera, соб!
нако роль этих белков в клетках прокариот на
ранные начиная с 1940!х годов, можно просле!
протяжении долго времени оставалась невыяс!
дить историю этого соревнования: 5 различных
ненной. В обсуждаемой работе было показано,
типов PLE последовательно сменяли друг друга,
что несколько STAND NTPase проявляют ак!
предположительно, избегая защиты со стороны
тивность в противофаговой защите, и 5 типов
системы CRISPR!Cas, кодируемой фагом ICP1
таких систем были объединены под общим на!
[199, 205].
званием AVAST (AntiViral ATPаses/NTPases из
Лишь в последние годы мы начали осозна!
суперсемейства STAND). Было обнаружено, что
вать реальное обилие защитных систем и разно!
домены NTPase слиты с различными предпола!
образие их механизмов действия. Это сразу ста!
гаемыми эффекторными доменами (такими,
вит вопрос о том, как наличие нескольких за!
как нуклеаза, протеаза или SIR2), и было пред!
щитных систем у отдельной бактерии влияет на
БИОХИМИЯ том 86 вып. 4 2021
546
ИСАЕВ и др.
её выживаемость и устойчивость к фагам? Неко!
ми модулями, и сходные белковые домены мо!
торые защитные системы являются высоко спе!
гут быть вовлечены в различные типы защи!
цифичными, в то время как другие могут воз!
ты [208]. Например, модуль модификации сис!
действовать на несколько фагов как, например,
темы Dnd может быть ассоциирован с эффекто!
некоторые системы Abi, которые улавливают
рами DndFGH или PblABCDE, нуклеаза NucC
общие нарушения метаболизма хозяина, или
может быть ассоциирована с CBASS или
системы, основанные на распознавании ДНК,
CRISPR!Cas типа III, а домены TIR обнаружи!
которые могут адаптироваться или мутировать
ваются в CBASS и Thoeris и т.д. [161, 210, 228].
для взаимодействия с новыми последователь!
- Наличие защитных систем характерно не
ностями [3, 222]. Защитные системы оказывают
только для прокариотических геномов. Так,
действие на различные стадии жизненного цик!
MGE различного вида широко используют сис!
ла вирусов, и, в целом, можно обозначить три
темы защиты в конфликтах между собой или для
основных линии защиты: системы, которые
подавления клетки!хозяина [205, 226]. Напри!
влияют на поверхность клетки, чтобы предотв!
мер, недавно при метагеномном исследовании
ратить адсорбцию фага и проникновение его ге!
гигантских фагов было обнаружено множество
нома; системы, которые разрушают генетичес!
локусов CRISPR со спейсерами, нацеленными
кий материал фага и системы, которые индуци!
на другие фаги [229].
руют клеточную смерть или остановку клеточ!
- Было показано, что белки, характерные
ного цикла в том случае, если фаг избежал
для систем иммунитета эукариот, выполняют
действия первых двух линий защиты. Можно
аналогичные функции у прокариот, например,
предположить, что одновременное присутствие
cGAS, pAgo, STING, TIR, STAND [105, 149, 151,
различных защитных систем в одной клетке по!
158,
210]. Недавняя работа Burroughs и
вышает шансы клетки на выживание и расши!
Arravind [208] расширяет этот список, показы!
ряет круг таргетируемых паразитов. Известно
вая, что гомологи Wnt, YEATS, TPR!S и других
даже, что некоторые защитные системы
доменов встречаются в прокариотических ост!
действуют совместно. Например, деградация
ровках защиты. Филогенетический анализ этих
ДНК системами R!M типа I приводит к образо!
доменов указывает на прокариотическое проис!
ванию пула фрагментов ДНК, которые могут
хождение, и это позволяет предположить, что
быть использованы как предшественники спей!
некоторые иммунные механизмы возникли ещё
серов для системы CRISPR!Cas [64, 223, 224],
до ответвления эукариот и были унаследованы
или системы Abi, такие как PrrC и ретрон Ec48,
последними.
использующие «охранную» стратегию и активи!
руемые только в том случае, если фаг вмешива!
ется в функции других защитных систем [146].
Финансирование. Выполнение данной рабо!
В то же время наличие защитных систем требует
ты проходило при поддержке Российского фон!
от клетки!хозяина определенных расходов: при
да фундаментальных исследований (грант № 19!
отсутствии фаговой инфекции экспрессия за!
14!50560). АИ поддержан грантом Российского
щитных генов требует траты энергии, а их не!
фонда фундаментальных исследований (№ 19!
специфическая активность часто бывает опасна
34!90160), OM поддержана грантом Российско!
для самой клетки [110, 219, 225]. Таким образом,
го научного фонда (№ 19!74!00118). Оплата от!
баланс между преимуществами, которые дают
крытого доступа английской версии статьи:
системы противовирусной защиты, и сопутству!
Сколковский Институт Науки и Технологий.
ющими затратами определяет количество за!
Вклад авторов. ОМ и АИ написали раздел
щитных систем в геноме и распространенность
CRISPR!Cas, АИ подготовил остальную часть
конкретных систем защиты. В условиях низкого
статьи, АИ и ОМ подготовили рисунки, КС от!
давления со стороны фагов защитные системы
редактировал текст.
могут быть утрачены, и их сохранению в попу!
Благодарности. Авторы выражают благодар!
ляции способствуют активный горизонтальный
ность Андрею Кульбачинскому за критическое
перенос генов (HGT), подавление экспрессии и
прочтение раздела, посвящённого белку pAgo.
фазовые вариации [226, 227].
Конфликт интересов. Авторы заявляют об от!
Новые открытия позволили выделить допол!
сутствии конфликта интересов.
нительные особенности систем защиты, ранее
Соблюдение этических норм. Настоящая
не столь очевидные.
статья не содержит каких!либо исследований с
- Между различными защитными система!
участием людей или использованием животных
ми может происходить обмен функциональны!
в качестве объектов.
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
547
СПИСОК ЛИТЕРАТУРЫ
1.
Jansen, R., Embden, J. D. A., van Gaastra, W., and
19.
Liu, T., Pan, S., Li, Y., Peng, N., and She, Q. (2017) Type
Schouls, L. M. (2002) Identification of genes that are asso!
III CRISPR!Cas system: introduction and its application
ciated with DNA repeats in prokaryotes, Mol. Microbiol.,
for genetic manipulations, Curr. Issues Mol. Biol., 26,
43, 1565!1575.
1!14.
2.
Mojica, F. J. M., and Garrett, R. A. (2013) Discovery and
20.
Goldberg, G. W., Jiang, W., Bikard, D., and Marraffini,
seminal developments in the CRISPR field, In CRISPR%
L. A. (2014) Conditional tolerance of temperate phages via
Cas Systems, Springer, pp. 1!31.
transcription!dependent CRISPR!Cas targeting, Nature,
3.
Nussenzweig, P. M., and Marraffini, L. A.
(2020)
514, 633!637.
Molecular mechanisms of CRISPR!Cas immunity in bac!
21.
Kazlauskiene, M., Kostiuk, G., Venclovas, Č., Tamulaitis,
teria, Annu. Rev. Genet., 54, 93!120.
G., and Siksnys, V. (2017) A cyclic oligonucleotide signal!
4.
Makarova, K. S., Wolf, Y. I., Iranzo, J., Shmakov, S. A.,
ing pathway in type III CRISPR!Cas systems, Science,
Alkhnbashi, O. S., et al. (2020) Evolutionary classification
357, 605!609.
of CRISPR!Cas systems: a burst of class 2 and derived vari!
22.
Niewoehner, O., Garcia!Doval, C., Rostøl, J. T., Berk, C.,
ants, Nat. Rev. Microbiol., 18, 67!83.
Schwede, F., et al. (2017) Type III CRISPR!Cas systems
5.
Jackson, R. N., and Wiedenheft, B. (2015) A conserved
produce cyclic oligoadenylate second messengers, Nature,
structural chassis for mounting versatile CRISPR RNA!
548, 543!548.
guided immune responses, Mol. Cell, 58, 722!728.
23.
You, L., Ma, J., Wang, J., Artamonova, D., Wang, M.,
6.
Liu, T. Y., and Doudna, J. A. (2020) Chemistry of Class 1
et al. (2019) Structure studies of the CRISPR!Csm com!
CRISPR!Cas effectors: binding, editing, and regulation,
plex reveal mechanism of co!transcriptional interference,
J. Biol. Chem., 295, 14473!14487.
Cell, 176, 239!253.
7.
Reeks, J., Graham, S., Anderson, L., Liu, H., White,
24.
Koonin, E. V, and Makarova, K. S. (2017) Mobile genetic
M. F., and Naismith, J. H. (2013) Structure of the archaeal
elements and evolution of CRISPR!Cas systems: all the
Cascade subunit Csa5: relating the small subunits of
way there and back, Genome Biol. Evol., 9, 2812!2825.
CRISPR effector complexes, RNA Biol., 10, 762!769.
25.
Özcan, A., Pausch, P., Linden, A., Wulf, A., Schühle, K.,
8.
Jore, M. M., Lundgren, M., van Duijn, E., Bultema, J. B.,
et al. (2019) Type IV CRISPR RNA processing and effec!
Westra, E. R., et al. (2011) Structural basis for CRISPR
tor complex formation in Aromatoleum aromaticum, Nat.
RNA!guided DNA recognition by Cascade, Nat. Struct.
Microbiol., 4, 89!96.
Mol. Biol., 18, 529!536.
26.
Makarova, K. S., Wolf, Y. I., Alkhnbashi, O. S., Costa, F.,
9.
Wiedenheft, B., Lander, G. C., Zhou, K., Jore, M. M.,
Shah, S. A., et al. (2015) An updated evolutionary classifi!
Brouns, S.J. J., et al. (2011) Structures of the RNA!guided
cation of CRISPR!Cas systems, Nat. Rev. Microbiol., 13,
surveillance complex from a bacterial immune system,
722!736.
Nature, 477, 486!489.
27.
Koonin, E. V, and Makarova, K. S. (2019) Origins and evo!
10.
Sinkunas, T., Gasiunas, G., Waghmare, S. P., Dickman,
lution of CRISPR!Cas systems, Philos. Trans. R. Soc. B,
M. J., Barrangou, R., et al. (2013) In vitro reconstitution of
374, 20180087.
Cascade!mediated CRISPR immunity in Streptococcus
28.
Pinilla!Redondo, R., Mayo!Muñoz, D., Russel, J.,
thermophilus, EMBO J., 32, 385!394.
Garrett, R. A., Randau, L., et al. (2020) Type IV CRISPR!
11.
Benda, C., Ebert, J., Scheltema, R. A., Schiller, H. B.,
Cas systems are highly diverse and involved in competition
Baumgärtner, M., et al. (2014) Structural model of a
between plasmids, Nucleic Acids Res., 48, 2000!2012.
CRISPR RNA!silencing complex reveals the RNA!target
29.
Gasiunas, G., Barrangou, R., Horvath, P., and Siksnys, V.
cleavage activity in Cmr4, Mol. Cell, 56, 43!54.
(2012) Cas9!crRNA ribonucleoprotein complex mediates
12.
Rouillon, C., Zhou, M., Zhang, J., Politis, A., Beilsten!
specific DNA cleavage for adaptive immunity in bacteria,
Edmands, V., et al. (2013) Structure of the CRISPR inter!
Proc. Natl. Acad. Sci. USA, 109, E2579!E2586.
ference complex CSM reveals key similarities with cascade,
30.
Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna,
Mol. Cell, 52, 124!134.
J. A., and Charpentier, E. (2012) A programmable dual!
13.
Hale, C. R., Zhao, P., Olson, S., Duff, M. O., Graveley,
RNA!guided DNA endonuclease in adaptive bacterial
B. R., Wells, L., et al. (2009) RNA!guided RNA cleavage
immunity, Science, 337, 816!821.
by a CRISPR RNA-Cas protein complex, Cell, 139, 945!
31.
Hsu, P. D., Lander, E. S., and Zhang, F. (2014) Develop!
956.
ment and applications of CRISPR!Cas9 for genome engi!
14.
Zhang, J., Kasciukovic, T., and White, M. F. (2012) The
neering, Cell, 157, 1262!1278.
CRISPR associated protein Cas4 Is a 5′ to 3′ DNA exonu!
32.
Mali, P., Esvelt, K. M., and Church, G. M. (2013) Cas9 as
clease with an iron!sulfur cluster, PLoS One, 7, e47232.
a versatile tool for engineering biology, Nat. Methods, 10,
15.
Elmore, J. R., Sheppard, N. F., Ramia, N., Deighan, T.,
957!963.
Li, H., et al. (2016) Bipartite recognition of target RNAs
33.
Sander, J. D., and Joung, J. K. (2014) CRISPR!Cas sys!
activates DNA cleavage by the Type III!B CRISPR!Cas
tems for editing, regulating and targeting genomes, Nat.
system, Genes Dev., 30, 447!459.
Biotechnol., 32, 347!355.
16.
Estrella, M. A., Kuo, F.!T., and Bailey, S. (2016) RNA!
34.
Zetsche, B., Gootenberg, J. S., Abudayyeh, O. O.,
activated DNA cleavage by the type III!B CRISPR!Cas
Slaymaker, I. M., Makarova, K. S., et al. (2015) Cpf1 is a
effector complex, Genes Dev., 30, 460!470.
single RNA!guided endonuclease of a class 2 CRISPR!Cas
17.
Kazlauskiene, M., Tamulaitis, G., Kostiuk, G.,
system, Cell, 163, 759!771.
Venclovas, Č., and Siksnys, V. (2016) Spatiotemporal con
35.
Yamano, T., Zetsche, B., Ishitani, R., Zhang, F.,
trol of type III A CRISPR Cas immunity: coupling DNA
Nishimasu, H., and Nureki, O. (2017) Structural basis for
degradation with the target RNA recognition, Mol. Cell,
the canonical and non!canonical PAM recognition by
62, 295!306.
CRISPR!Cpf1, Mol. Cell, 67, 633!645.
18.
Samai, P., Pyenson, N., Jiang, W., Goldberg, G. W.,
36.
Chen, J. S., Ma, E., Harrington, L. B., Da Costa, M.,
Hatoum!Aslan, A., and Marraffini, L. A. (2015) Co!tran!
Tian, X., et al. (2018) CRISPR!Cas12a target binding
scriptional DNA and RNA cleavage during type III
unleashes indiscriminate single!stranded DNase activity,
CRISPR!Cas immunity, Cell, 161, 1164!1174.
Science, 360, 436!439.
БИОХИМИЯ том 86 вып. 4 2021
548
ИСАЕВ и др.
37.
Harrington, L. B., Burstein, D., Chen, J. S., Paez!
DNase activity of a conserved protein implicated in
Espino, D., Ma, E., et al. (2018) Programmed DNA
CRISPR!mediated genome defense, Structure, 17, 904!
destruction by miniature CRISPR!Cas14 enzymes,
912.
Science, 362, 839!842.
55.
Beloglazova, N., Lemak, S., Flick, R., and Yakunin, A. F.
38.
Chylinski, K., Makarova, K. S., Charpentier, E., and
(2015) Analysis of nuclease activity of Cas1 proteins
Koonin, E. V (2014) Classification and evolution of type II
against complex DNA substrates, in CRISPR, Springer,
CRISPR!Cas systems, Nucleic Acids Res., 42, 6091!6105.
pp. 251!264.
39.
Shmakov, S., Abudayyeh, O. O., Makarova, K. S., Wolf,
56.
Nam, K. H., Ding, F., Haitjema, C., Huang, Q., DeLisa,
Y. I., Gootenberg, J. S., et al. (2015) Discovery and func!
M. P., and Ke, A. (2012) Double!stranded endonuclease
tional characterization of diverse class 2 CRISPR!Cas sys!
activity in Bacillus halodurans clustered regularly inter!
tems, Mol. Cell, 60, 385!397.
spaced short palindromic repeats (CRISPR)!associated
40.
Abudayyeh, O. O., Gootenberg, J. S., Konermann, S.,
Cas2 protein, J. Biol. Chem., 287, 35943!35952.
Joung, J., Slaymaker, I. M., et al. (2016) C2c2 is a single!
57.
Dixit, B., Ghosh, K. K., Fernandes, G., Kumar, P.,
component programmable RNA!guided RNA!targeting
Gogoi, P., and Kumar, M. (2016) Dual nuclease activity of
CRISPR effector, Science, 353, aaf5573, doi: 10.1126/
a Cas2 protein in CRISPR!Cas subtype I!B of Leptospira
science.aaf5573.
interrogans, FEBS Lett., 590, 1002!1016.
41.
Koonin, E. V, and Krupovic, M. (2015) Evolution of adap!
58.
Savitskaya, E. E., Musharova, O. S., and Severinov, K. V
tive immunity from transposable elements combined with
(2016) Diversity of CRISPR!Cas!mediated mechanisms of
innate immune systems, Nat. Rev. Genet., 16, 184!192.
adaptive immunity in prokaryotes and their application in
42.
Koonin, E. V, Makarova, K. S., and Zhang, F. (2017)
biotechnology, Biochemistry (Moscow), 81, 653!661.
Diversity, classification and evolution of CRISPR!Cas sys!
59.
Mizuuchi, K., and Adzuma, K. (1991) Inversion of the
tems, Curr. Opin. Microbiol., 37, 67!78.
phosphate chirality at the target site of Mu DNA strand
43.
Makarova, K. S., and Koonin, E. V (2013) Evolution and
transfer: evidence for a one!step transesterification mecha!
classification of CRISPR!Cas systems and cas protein
nism, Cell, 66, 129!140.
families, in CRISPR%Cas System, Springer, pp. 61!91.
60.
Krupovic, M., Makarova, K. S., Forterre, P.,
44.
Nuñez, J. K., Kranzusch, P. J., Noeske, J., Wright, A. V.,
Prangishvili, D., and Koonin, E. V (2014) Casposons: a
Davies, C. W., and Doudna, J. A. (2014) Cas1-Cas2 com
new superfamily of self!synthesizing DNA transposons at
plex formation mediates spacer acquisition during
the origin of prokaryotic CRISPR!Cas immunity, BMC
CRISPR Cas adaptive immunity, Nat. Struct. Mol. Biol.,
Biol., 12, 36.
21, 528.
61.
Béguin, P., Charpin, N., Koonin, E. V., Forterre, P., and
45.
Nuñez, J. K., Harrington, L. B., Kranzusch, P. J.,
Krupovic, M. (2016) Casposon integration shows strong
Engelman, A. N., and Doudna, J. A. (2015) Foreign DNA
target site preference and recapitulates protospacer inte!
capture during CRISPR!Cas adaptive immunity, Nature,
gration by CRISPR!Cas systems, Nucleic Acids Res., 44,
527, 535!538.
10367!10376.
46.
Brouns, S. J. J., Jore, M. M., Lundgren, M., Westra, E. R.,
62.
Rollie, C., Graham, S., Rouillon, C., and White, M. F.
Slijkhuis, R. J. H., et al. (2008) Small CRISPR RNAs
(2018) Prespacer processing and specific integration in a
guide antiviral defense in prokaryotes, Science, 321, 960!
type IA CRISPR system, Nucleic Acids Res., 46, 1007!
964.
1020.
47.
Yosef, I., Goren, M. G., and Qimron, U. (2012) Proteins and
63.
Levy, A., Goren, M. G., Yosef, I., Auster, O., Manor, M.,
DNA elements essential for the CRISPR adaptation process
et al. (2015) CRISPR adaptation biases explain preference
in Escherichia coli, Nucleic Acids Res., 40, 5569!5576.
for acquisition of foreign DNA, Nature, 520, 505!510.
48.
Datsenko, K. A., Pougach, K., Tikhonov, A., Wanner,
64.
Weissman, J. L., Stoltzfus, A., Westra, E. R., and Johnson,
B. L., Severinov, K., and Semenova, E. (2012) Molecular
P. L. F. (2020) Avoidance of Self during CRISPR
memory of prior infections activates the CRISPR/Cas
Immunization,
Trends
Microbiol.,
28,
543!
adaptive bacterial immunity system, Nat. Commun., 3, 1!7.
553, doi: 10.1016/j.tim.2020.02.005.
49.
Vorontsova, D., Datsenko, K. A., Medvedeva, S., Bondy!
65.
Dorman, C. J., and Bhriain, N. N. (2020) CRISPR!Cas,
Denomy, J., Savitskaya, E. E., et al. (2015) Foreign DNA
DNA supercoiling, and nucleoid!associated proteins,
acquisition by the IF CRISPR!Cas system requires all
Trends Microbiol., 28, 19!27.
components of the interference machinery, Nucleic Acids
66.
Kurilovich, E., Shiriaeva, A., Metlitskaya, A.,
Res., 43, 10848!10860.
Morozova, N., Ivancic!Bace, I., et al. (2019) Genome
50.
Deltcheva, E., Chylinski, K., Sharma, C. M.,
maintenance proteins modulate autoimmunity mediated
Gonzales, K., Chao, Y., et al. (2011) CRISPR RNA mat!
primed adaptation by the Escherichia coli type IE
uration by trans!encoded small RNA and host factor
CRISPR!cas system, Genes (Basel), 10, 872.
RNase III, Nature, 471, 602!607.
67.
Radovčić, M., Killelea, T., Savitskaya, E., Wettstein, L.,
51.
Sapranauskas, R., Gasiunas, G., Fremaux, C.,
Bolt, E. L., and Ivančić Baće, I. (2018) CRISPR!Cas
Barrangou, R., Horvath, P., and Siksnys, V. (2011) The
adaptation in Escherichia coli requires RecBCD helicase
Streptococcus thermophilus CRISPR/Cas system provides
but not nuclease activity, is independent of homologous
immunity in Escherichia coli, Nucleic Acids Res., 39, 9275!
recombination, and is antagonized by 5′ ssDNA exonucle!
9282.
ases, Nucleic Acids Res., 46, 10173!10183.
52.
Hatoum!Aslan, A., Maniv, I., Samai, P., and Marraffini,
68.
Ivančić Baće, I., Cass, S. D., Wearne, S. J., and Bolt, E. L.
L. A. (2014) Genetic characterization of antiplasmid
(2015) Different genome stability proteins underpin
immunity through a type III!A CRISPR!Cas system,
primed and naive adaptation in E. coli CRISPR!Cas
J. Bacteriol., 196, 310!317.
immunity, Nucleic Acids Res., 43, 10821!10830.
53.
Babu, M., Beloglazova, N., Flick, R., Graham, C.,
69.
Deveau, H., Barrangou, R., Garneau, J. E., Labonté, J.,
Skarina, T., et al. (2011) A dual function of the CRISPR!
Fremaux, C., et al. (2008) Phage response to CRISPR!
Cas system in bacterial antivirus immunity and DNA
encoded resistance in Streptococcus thermophilus,
repair, Mol. Microbiol., 79, 484!502.
J. Bacteriol., 190, 1390!1400.
54.
Wiedenheft, B., Zhou, K., Jinek, M., Coyle, S. M.,
70.
Semenova, E., Jore, M. M., Datsenko, K. A.,
Ma, W., and Doudna, J. A. (2009) Structural basis for
Semenova, A., Westra, E. R., et al. (2011) Interference by
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
549
clustered regularly interspaced short palindromic repeat
87. Shabalina, S., and Koonin, E. (2008) Origins and evolu!
(CRISPR) RNA is governed by a seed sequence, Proc.
tion of eukaryotic RNA interference, Trends Ecol. Evol.,
Natl. Acad. Sci. USA, 108, 10098!10103.
23, 578!587.
71.
Fineran, P. C., Gerritzen, M. J. H., Suárez Diez, M.,
88. Makarova, K. S., Wolf, Y. I., van der Oost, J., and Koonin,
Künne, T., Boekhorst, J., et al. (2014) Degenerate target
E. V. (2009) Prokaryotic homologs of Argonaute proteins
sites mediate rapid primed CRISPR adaptation, Proc. Natl.
are predicted to function as key components of a novel sys!
Acad. Sci. USA, 111, E1629!E1638.
tem of defense against mobile genetic elements., Biol.
72.
Westra, E. R., Semenova, E., Datsenko, K. A., Jackson,
Direct, 4, 29.
R. N., Wiedenheft, B., et al. (2013) Type IE CRISPR!cas
89. Willkomm, S., Makarova, K. S., and Grohmann, D.
systems discriminate target from non!target DNA through
(2018) DNA silencing by prokaryotic Argonaute proteins
base pairing!independent PAM recognition, PLoS Genet.,
adds a new layer of defense against invading nucleic acids,
9, e1003742.
FEMS Microbiol. Rev., 42, 376!387.
73.
Richter, C., Dy, R. L., McKenzie, R. E., Watson, B. N. J.,
90. Kuzmenko, A., Oguienko, A., Esyunina, D., Yudin, D.,
Taylor, C., et al. (2014) Priming in the Type IF CRISPR!
Petrova, M., et al. (2020) DNA targeting and interference
Cas system triggers strand!independent spacer acquisition,
by a bacterial Argonaute nuclease, Nature, 587, 632!637,
bi!directionally from the primed protospacer, Nucleic Acids
doi: 10.1038/s41586!020!2605!1.
Res., 42, 8516!8526.
91. Wang, Y., Juranek, S., Li, H., Sheng, G., Tuschl, T., and
74.
Garrett, S., Shiimori, M., Watts, E. A., Clark, L., Graveley,
Patel, D. J. (2008) Structure of an argonaute silencing
B. R., and Terns, M. P. (2020) Primed CRISPR DNA
complex with a seed!containing guide DNA and target
uptake in Pyrococcus furiosus, Nucleic Acids Res., 48, 6120!
RNA duplex, Nature, 456, 921!926.
6135.
92. Olovnikov, I., Chan, K., Sachidanandam, R., Newman, D.
75.
Li, M., Wang, R., and Xiang, H. (2014) Haloarcula hispan%
K., and Aravin, A. A. (2013) Bacterial argonaute samples
ica CRISPR authenticates PAM of a target sequence to
the transcriptome to identify foreign DNA, Mol. Cell, 51,
prime discriminative adaptation, Nucleic Acids Res., 42,
594!605.
7226!7235.
93. Lisitskaya, L., Aravin, A. A., and Kulbachinskiy, A. (2018)
76.
Rao, C., Chin, D., and Ensminger, A. W. (2017) Priming
DNA interference and beyond: structure and functions of
in a permissive type IC CRISPR!Cas system reveals dis!
prokaryotic Argonaute proteins, Nat. Commun., 9, 1!12.
tinct dynamics of spacer acquisition and loss, RNA, 23,
94. Swarts, D. C., Hegge, J. W., Hinojo, I., Shiimori, M., Ellis,
1525!1538.
M. A., et al. (2015) Argonaute of the archaeon Pyrococcus
77.
Almendros, C., Nobrega, F. L., McKenzie, R. E., and
furiosus is a DNA!guided nuclease that targets cognate
Brouns, S. J. J. (2019) Cas4-Cas1 fusions drive efficient
DNA, Nucleic Acids Res., 43, 5120!5129.
PAM selection and control CRISPR adaptation, Nucleic
95. Kuzmenko, A., Yudin, D., Ryazansky, S.,
Acids Res., 47, 5223!5230.
Kulbachinskiy, A., and Aravin, A. A. (2019) Programmable
78.
Nussenzweig, P. M., McGinn, J., and Marraffini, L. A.
DNA cleavage by Ago nucleases from mesophilic bacteria
(2019) Cas9 cleavage of viral genomes primes the acquisi!
Clostridium butyricum and Limnothrix rosea, Nucleic Acids
tion of new immunological memories, Cell Host Microbe,
Res., 47, 5822!5836.
26, 515!526.
96. Yuan, Y.!R., Pei, Y., Ma, J.!B., Kuryavyi, V., Zhadina, M.,
79.
Savitskaya, E., Semenova, E., Dedkov, V., Metlitskaya, A.,
et al. (2005) Crystal structure of A. aeolicus argonaute, a
and Severinov, K. (2013) High!throughput analysis of type
site!specific DNA!guided endoribonuclease, provides
IE CRISPR/Cas spacer acquisition in E. coli, RNA Biol.,
insights into RISC!mediated mRNA cleavage, Mol. Cell,
10, 716!725.
19, 405!419.
80.
Xue, C., Seetharam, A. S., Musharova, O., Severinov, K.,
97. Wang, Y., Juranek, S., Li, H., Sheng, G., Wardle, G. S.,
Brouns, S. J., et al. (2015) CRISPR interference and prim!
et al. (2009) Nucleation, propagation and cleavage of target
ing varies with individual spacer sequences, Nucleic Acids
RNAs in Ago silencing complexes, Nature, 461, 754!761.
Res., 43, 10831!10847.
98. Kaya, E., Doxzen, K. W., Knoll, K. R., Wilson, R. C.,
81.
Künne, T., Kieper, S. N., Bannenberg, J. W., Vogel,
Strutt, S. C., et al. (2016) A bacterial Argonaute with non!
A. I. M., Miellet, W. R., et al. (2016) Cas3!derived target
canonical guide RNA specificity, Proc. Natl. Acad. Sci.
DNA degradation fragments fuel primed CRISPR adapta!
USA, 113, 4057!4062.
tion, Mol. Cell, 63, 852!864.
99. Willkomm, S., Zander, A., Gust, A., and Grohmann, D.
82.
Dillard, K. E., Brown, M. W., Johnson, N. V., Xiao, Y.,
(2015) A prokaryotic twist on argonaute function, Life, 5,
Dolan, A., et al. (2018) Assembly and translocation of a
538!553.
CRISPR!Cas primed acquisition complex, Cell, 175, 934!
100. Swarts, D. C., Jore, M. M., Westra, E. R., Zhu, Y.,
946.
Janssen, J. H., et al. (2014) DNA!guided DNA interfer!
83.
Redding, S., Sternberg, S. H., Marshall, M., Gibb, B.,
ence by a prokaryotic Argonaute, Nature, 507, 258!261.
Bhat, P., et al. (2015) Surveillance and processing of for!
101. Ryazansky, S., Kulbachinskiy, A., and Aravin, A. A. (2018)
eign DNA by the Escherichia coli CRISPR!Cas system,
The expanded universe of prokaryotic Argonaute proteins,
Cell, 163, 854!865.
MBio, 9, e01935!18, doi: 10.1128/mBio.01935!18.
84.
Sashital, D. G., Wiedenheft, B., and Doudna, J. A. (2012)
102. Zander, A., Willkomm, S., Ofer, S., Van Wolferen, M.,
Mechanism of foreign DNA selection in a bacterial adap!
Egert, L., et al. (2017) Guide!independent DNA cleavage
tive immune system, Mol. Cell, 46, 606!615.
by archaeal Argonaute from Methanocaldococcus jan%
85.
Semenova, E., Savitskaya, E., Musharova, O.,
naschii, Nat. Microbiol., 2, 1!10.
Strotskaya, A., Vorontsova, D., et al. (2016) Highly effi!
103. Swarts, D. C., Szczepaniak, M., Sheng, G., Chandradoss,
cient primed spacer acquisition from targets destroyed by
S. D., Zhu, Y., et al. (2017) Autonomous generation and
the Escherichia coli type IE CRISPR!Cas interfering com!
loading of DNA guides by bacterial Argonaute, Mol. Cell,
plex, Proc. Natl. Acad. Sci. USA, 113, 7626!7631.
65, 985!998.
86.
Severinov, K., Ispolatov, I., and Semenova, E. (2016) The
104. Jolly, S. M., Gainetdinov, I., Jouravleva, K., Zhang, H.,
influence of copy!number of targeted extrachromosomal
Strittmatter, L., et al. (2020) Thermus thermophilus
genetic elements on the outcome of CRISPR!Cas defense,
Argonaute functions in the completion of DNA replica!
Front. Mol. Biosci., 3, 45.
tion, Cell, 182, 1545!1559.
БИОХИМИЯ том 86 вып. 4 2021
550
ИСАЕВ и др.
105. Koonin, E. V. (2017) Evolution of RNA! and DNA!guid!
124. Cheng, X., Wang, W., and Molineux, I. J. (2004) F exclu!
ed antivirus defense systems in prokaryotes and eukaryotes:
sion of bacteriophage T7 occurs at the cell membrane,
common ancestry vs convergence, Biol. Direct, 12, 1!14.
Virology, 326, 340!352.
106. Chopin, M.!C., Chopin, A., and Bidnenko, E. (2005)
125. Schmitt, C. K., Kemp, P., and Molineux, I. J. (1991)
Phage abortive infection in lactococci: variations on a
Genes 1.2 and 10 of bacteriophages T3 and T7 determine
theme, Curr. Opin. Microbiol., 8, 473!9.
the permeability lesions observed in infected cells of
107. Lopatina, A., Tal, N., and Sorek, R. (2020) Abortive infec!
Escherichia coli expressing the F plasmid gene pifA,
tion: bacterial suicide as an antiviral immune strategy,
J. Bacteriol., 173, 6507!6514.
Annu. Rev. Virol., 7, 371!384.
126. Toothman, P., and Herskowitz, I. (1980) Rex!dependent
108. Labrie, S. J., Samson, J. E., and Moineau, S. (2010)
exclusion of lambdoid phages II. Determinants of sensitiv!
Bacteriophage resistance mechanisms, Nat. Rev.
ity to exclusion, Virology, 102, 147!160.
Microbiol., 8, 317!327.
127. Parma, D. H., Snyder, M., Sobolevski, S., Nawroz, M.,
109. Fukuyo, M., Sasaki, A., and Kobayashi, I. (2012) Success
Brody, E., and Gold, L. (1992) The Rex system of bacte!
of a suicidal defense strategy against infection in a struc!
riophage lambda: tolerance and altruistic cell death, Genes
tured habitat, Sci. Rep., 2, 238.
Dev., 6, 497!510.
110. Van Houte, S., Buckling, A., and Westra, E. R. (2016)
128. Yamaguchi, Y., Park, J.!H., and Inouye, M.
(2011)
Evolutionary ecology of prokaryotic immune mechanisms,
Toxin-antitoxin systems in bacteria and archaea, Annu.
Microbiol. Mol. Biol. Rev., 80, 745!763.
Rev. Genet., 45, 61!79.
111.
Barrangou, R., and Horvath, P. (2011) Lactic acid bacteria
129. Page, R., and Peti, W. (2016) Toxin-antitoxin systems in
defenses against phages, in Stress Responses of Lactic Acid
bacterial growth arrest and persistence, Nat. Chem. Biol.,
Bacteria, Springer, pp. 459!478.
12, 208!214.
112. Durmaz, E., and Klaenhammer, T. R. (2007) Abortive
130. Makarova, K. S., Wolf, Y. I., and Koonin, E. V. (2013)
phage resistance mechanism AbiZ speeds the lysis clock to
Comparative genomics of defense systems in archaea and
cause premature lysis of phage!infected Lactococcus lactis,
bacteria, Nucleic Acids Res., 41, 4360!4377.
J. Bacteriol., 189, 1417!1425.
131. Harms, A., Brodersen, D. E., Mitarai, N., and Gerdes, K.
113. Wang, C., Villion, M., Semper, C., Coros, C.,
(2018) Toxins, targets, and triggers: an overview of
Moineau, S., and Zimmerly, S. (2011) A reverse transcrip!
toxin-antitoxin biology, Mol. Cell, 70, 768!784.
tase!related protein mediates phage resistance and poly!
132. Fineran, P. C., Blower, T. R., Foulds, I. J., Humphreys,
merizes untemplated DNA in vitro, Nucleic Acids Res., 39,
D. P., Lilley, K. S., and Salmond, G. P. C. (2009) The
7620!7629.
phage abortive infection system, ToxIN, functions as a pro!
114. Tangney, M., and Fitzgerald, G. F. (2002) Effectiveness of
tein-RNA toxin-antitoxin pair, Proc. Natl. Acad. Sci.
the lactococcal abortive infection systems AbiA, AbiE,
USA, 106, 894!899.
AbiF and AbiG against P335 type phages, FEMS Microbiol.
133. Short, F. L., Akusobi, C., Broadhurst, W. R., and Salmond,
Lett., 210, 67!72.
G. P. C. (2018) The bacterial Type III toxin-antitoxin sys!
115. Parreira, R., Ehrlich, S. D., and Chopin, M.
(1996)
tem, ToxIN, is a dynamic protein-RNA complex with sta!
Dramatic decay of phage transcripts in lactococcal cells
bility!dependent antiviral abortive infection activity, Sci.
carrying the abortive infection determinant AbiB, Mol.
Rep., 8, 1!10.
Microbiol., 19, 221!230.
134. Koga, M., Otsuka, Y., Lemire, S., and Yonesaki, T. (2011)
116. Samson, J. E., Spinelli, S., Cambillau, C., and
Escherichia coli rnlA and rnlB compose a novel
Moineau, S. (2013) Structure and activity of AbiQ, a lacto!
toxin-antitoxin system, Genetics, 187, 123!130.
coccal endoribonuclease belonging to the type III
135. Otsuka, Y., and Yonesaki, T. (2012) Dmd of bacteriophage
toxin-antitoxin system, Mol. Microbiol., 87, 756!768.
T4 functions as an antitoxin against Escherichia coli LsoA
117. Bidnenko, E., Ehrlich, D., and Chopin, M.!C. (1995)
and RnlA toxins, Mol. Microbiol., 83, 669!681.
Phage operon involved in sensitivity to the Lactococcus lac%
136. Dy, R. L., Przybilski, R., Semeijn, K., Salmond, G. P. C., and
tis abortive infection mechanism AbiD1, J. Bacteriol., 177,
Fineran, P. C. (2014) A widespread bacteriophage abortive
3824!3829.
infection system functions through a type IV toxin-antitoxin
118. Bouchard, J. D., Dion, E., Bissonnette, F., and
mechanism, Nucleic Acids Res., 42, 4590!4605.
Moineau, S. (2002) Characterization of the two!compo!
137. Cai, Y., Usher, B., Gutierrez, C., Tolcan, A., Mansour, M.,
nent abortive phage infection mechanism AbiT from
Fineran, P. C., et al. (2020) A nucleotidyltransferase toxin
Lactococcus lactis, J. Bacteriol., 184, 6325!6332.
inhibits growth of Mycobacterium tuberculosis through inac!
119. Haaber, J., Samson, J. E., Labrie, S. J., Campanacci, V.,
tivation of tRNA acceptor stems, Sci. Adv., 6, eabb6651.
Cambillau, C., et al. (2010) Lactococcal abortive infection
138. Hampton, H. G., Smith, L. M., Ferguson, S., Meaden, S.,
protein AbiV interacts directly with the phage protein SaV
Jackson, S. A., and Fineran, P. C. (2020) Functional
and prevents translation of phage proteins, Appl. Environ.
genomics reveals the toxin-antitoxin repertoire and AbiE
Microbiol., 76, 7085!7092.
activity in Serratia, Microb. Genom., 6, mgen000458,
120. Depardieu, F., Didier, J.!P., Bernheim, A., Sherlock, A.,
doi: 10.1099/mgen.0.000458.
Molina, H., et al. (2016) A eukaryotic!like serine/threo!
139. Hazan, R., and Engelberg!Kulka, H. (2004) Escherichia
nine kinase protects staphylococci against phages, Cell Host
coli mazEF!mediated cell death as a defense mechanism
Microbe, 20, 471!481.
that inhibits the spread of phage P1, Mol. Genet. Genomics,
121. Snyder, L. (1995) Phage!exclusion enzymes: a bonanza of
272, 227!234.
biochemical and cell biology reagents? Mol. Microbiol., 15,
140. Pecota, D. C., and Wood, T. K. (1996) Exclusion of T4
415!20.
phage by the hok/sok killer locus from plasmid R1,
122. Yu, Y. T., and Snyder, L. (1994) Translation elongation fac!
J. Bacteriol., 178, 2044!2050.
tor Tu cleaved by a phage!exclusion system, Proc. Natl.
141. Hayes, F. (2003) Toxins!antitoxins: plasmid maintenance,
Acad. Sci. USA, 91, 802!806.
programmed cell death, and cell cycle arrest, Science, 301,
123. Levitz, R., Chapman, D., Amitsur, M., Green, R.,
1496!1499.
Snyder, L., and Kaufmann, G. (1990) The optional E. coli
142. Koonin, E. V., Makarova, K. S., and Wolf, Y. I. (2017)
prr locus encodes a latent form of phage T4!induced anti!
Evolutionary genomics of defense systems in archaea and
codon nuclease, EMBO J., 9, 1383!1389.
bacteria, Annu. Rev. Microbiol., 71, 233!261.
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
551
143. Song, S., and Wood, T. K. (2018) Post!segregational killing
161. Lau, R. K., Ye, Q., Birkholz, E. A., Berg, K. R., Patel, L.,
and phage inhibition are not mediated by cell death
et al. (2020) Structure and mechanism of a cyclic trinu!
through toxin/antitoxin systems, Front. Microbiol., 9, 814.
cleotide!activated bacterial endonuclease mediating bacte!
144. Sberro, H., Fremin, B. J., Zlitni, S., Edfors, F.,
riophage immunity, Mol. Cell, 77, 723!733.e6.
Greenfield, N., et al. (2019) Large!scale analyses of human
162. Lowey, B., Whiteley, A. T., Keszei, A. F. A., Morehouse,
microbiomes reveal thousands of small, novel genes, Cell,
B. R., Mathews, I. T., et al. (2020) CBASS immunity uses
178, 1245!1259.e14.
CARF!related effectors to sense 3′!5′!and 2′!5′!linked
145. Simon, A. J., Ellington, A. D., and Finkelstein, I. J. (2019)
cyclic oligonucleotide signals and protect bacteria from
Retrons and their applications in genome engineering,
phage infection, Cell, 182, 38!49.
Nucleic Acids Res., 47, 11007!11019.
163. Casjens, S. (2003) Prophages and bacterial genomics: what
146. Millman, A., Bernheim, A., Stokar!Avihail, A.,
have we learned so far? Mol. Microbiol., 49, 277!300.
Fedorenko, T., Voichek, M., et al. (2020) Bacterial retrons
164. Canchaya, C., Proux, C., Fournous, G., Bruttin, A., and
function in anti!phage defense, Cell, 183, 1551!1561.e12,
Brüssow, H. (2003) Prophage genomics, Microbiol. Mol.
doi: 10.1016/j.cell.2020.09.065.
Biol. Rev., 67, 238!276.
147. Bobonis, J., Mitosch, K., Mateus, A., Kritikos, G.,
165. Bondy!Denomy, J., Qian, J., Westra, E. R., Buckling, A.,
Elfenbein, J. R., et al. (2020) Phage proteins block and
Guttman, D. S., et al. (2016) Prophages mediate defense
trigger retron toxin/antitoxin systems, BioRxiv,
against phage infection through diverse mechanisms,
doi: 10.1101/2020.06.22.160242.
ISME J., 10, 2854!2866.
148. Bobonis, J., Mateus, A., Pfalz, B., Garcia!
166. Dedrick, R. M., Jacobs!Sera, D., Bustamante, C. A. G.,
Santamarina, S., Galardini, M., et al. (2020) Bacterial
Garlena, R. A., Mavrich, T. N., et al. (2017) Prophage!
retrons encode tripartite toxin/antitoxin systems, BioRxiv,
mediated defence against viral attack and viral counter!
doi: 10.1101/2020.06.22.160168.
defence, Nat. Microbiol., 2, 1!13.
149. Gao, L., Altae!Tran, H., Böhning, F., Makarova, K. S.,
167. Roux, S., Krupovic, M., Daly, R. A., Borges, A. L.,
Segel, M., et al. (2020) Diverse enzymatic activities medi
Nayfach, S., et al. (2019) Cryptic inoviruses revealed as
ate antiviral immunity in prokaryotes, Science, 369, 1077
pervasive in bacteria and archaea across Earth’s biomes,
1084.
Nat. Microbiol., 4, 1895!1906.
150. Burroughs, A. M., Zhang, D., Schäffer, D. E., Iyer, L. M.,
168. Benler, S., and Koonin, E. V (2020) Phage lysis!lysogeny
and Aravind, L. (2015) Comparative genomic analyses
switches and programmed cell death: danse macabre,
reveal a vast, novel network of nucleotide!centric systems
BioEssays, 42, e2000114, doi: 10.1002/bies.202000114.
in biological conflicts, immunity and signaling, Nucleic
169. Waldor, M. K., and Friedman, D. I. (2005) Phage regula!
Acids Res., 43, 10633!10654.
tory circuits and virulence gene expression, Curr. Opin.
151. Cohen, D., Melamed, S., Millman, A., Shulman, G.,
Microbiol., 8, 459!465.
Oppenheimer!Shaanan, Y., et al. (2019) Cyclic GMP!
170. Juhala, R. J., Ford, M. E., Duda, R. L., Youlton, A.,
AMP signalling protects bacteria against viral infection,
Hatfull, G. F., and Hendrix, R. W. (2000) Genomic
Nature, 574, 691!695.
sequences of bacteriophages HK97 and HK022: pervasive
152. Whiteley, A. T., Eaglesham, J. B., de Oliveira Mann, C. C.,
genetic mosaicism in the lambdoid bacteriophages, J. Mol.
Morehouse, B. R., Lowey, B., et al. (2019) Bacterial
Biol., 299, 27!51.
cGAS!like enzymes synthesize diverse nucleotide signals,
171. Taylor, V. L., Fitzpatrick, A. D., Islam, Z., and Maxwell,
Nature, 567, 194!199.
K. L. (2019) The diverse impacts of phage morons on bac!
153. Millman, A., Melamed, S., Amitai, G., and Sorek, R.
terial fitness and virulence, in Advances in Virus Research,
(2020) Diversity and classification of cyclic!oligonu!
Elsevier, Vol. 103, pp. 1!31.
cleotide!based anti!phage signalling systems, Nat.
172. Newton, G. J., Daniels, C., Burrows, L. L., Kropinski, A.
Microbiol., 5, 1608!1615, doi: 10.1038/s41564!020!0777!y.
M., Clarke, A. J., and Lam, J. S. (2001) Three!compo!
154. Ablasser, A., and Chen, Z. J. (2019) cGAS in action:
nent!mediated serotype conversion in Pseudomonas
expanding roles in immunity and inflammation, Science,
aeruginosa by bacteriophage D3, Mol. Microbiol., 39,
363, eaat8657, doi: 10.1126/science.aat8657.
1237!1247.
155. Davies, B. W., Bogard, R. W., Young, T. S., and
173. Krylov, S. V, Kropinski, A. M., Shaburova, O. V,
Mekalanos, J. J. (2012) Coordinated regulation of accesso!
Miroshnikov, K. A., Chesnokova, E. N., and Krylov, V. N.
ry genetic elements produces cyclic di!nucleotides for
(2013) New temperate Pseudomonas aeruginosa phage,
V. cholerae virulence, Cell, 149, 358!370.
phi297: specific features of genome structure, Russ. J.
156. Severin, G. B., Ramliden, M. S., Hawver, L. A., Wang, K.,
Genet., 49, 806!818.
Pell, M. E., et al. (2018) Direct activation of a phospholi!
174. Taylor, V. L., Hoage, J. F. J., Thrane, S. W., Huszczynski,
pase by cyclic GMP!AMP in El Tor Vibrio cholerae, Proc.
S. M., Jelsbak, L., and Lam, J. S. (2016) A bacteriophage!
Natl. Acad. Sci. USA, 115, E6048!E6055.
acquired O!antigen polymerase (Wzyβ) from P. aeruginosa
157. Yoon, S. H., and Waters, C. M. (2021) The ever!expanding
serotype O16 performs a varied mechanism compared to its
world of bacterial cyclic oligonucleotide second messen!
cognate Wzyα, Front. Microbiol., 7, 393.
gers, Curr. Opin. Microbiol., 60, 96!103.
175. Lehane, A. M., Korres, H., and Verma, N. K. (2005)
158. Morehouse, B. R., Govande, A. A., Millman, A., Keszei,
Bacteriophage!encoded glucosyltransferase GtrII of
A. F. A., Lowey, B., et al. (2020) STING cyclic dinu!
Shigella flexneri: membrane topology and identification of
cleotide sensing originated in bacteria, Nature, 586, 429!
critical residues, Biochem. J., 389, 137!143.
433.
176. Perry, L. L., SanMiguel, P., Minocha, U., Terekhov, A. I.,
159. Ye, Q., Lau, R. K., Mathews, I. T., Birkholz, E. A.,
Shroyer, M. L., et al. (2009) Sequence analysis of
Watrous, J. D., et al. (2020) HORMA domain proteins and
Escherichia coli O157: H7 bacteriophage ΦV10 and identi!
a Trip13!like ATPase regulate bacterial cGAS!like
fication of a phage!encoded immunity protein that modi!
enzymes to mediate bacteriophage immunity, Mol. Cell, 77,
fies the O157 antigen, FEMS Microbiol. Lett., 292, 182!
709!722.e7.
186.
160. Rosenberg, S. C., and Corbett, K. D. (2015) The multifac!
177. Gentile, G. M., Wetzel, K. S., Dedrick, R. M.,
eted roles of the HORMA domain in cellular signaling,
Montgomery, M. T., Garlena, R. A., et al. (2019) More evi!
J. Cell Biol., 211, 745!755.
dence of collusion: a new prophage!mediated viral defense
БИОХИМИЯ том 86 вып. 4 2021
552
ИСАЕВ и др.
system encoded by mycobacteriophage sbash, MBio, 10,
late phage gene transcription, Proc. Natl. Acad. Sci. USA,
e00196!19, doi: 10.1128/mBio.00196!19.
111, 14536!14541.
178. Montgomery, M. T., Guerrero Bustamante, C. A.,
197. Mart nez Rubio, R., Quiles Puchalt, N., Mart , M.,
Dedrick, R. M., Jacobs!Sera, D., and Hatfull, G. F. (2019)
Humphrey, S., Ram, G., et al. (2017) Phage inducible
Yet more evidence of collusion: a new viral defense system
islands in the Gram positive cocci, ISME J., 11, 1029
encoded by gordonia phage carolann, MBio, 10, 1!18.
1042.
179. Jimmy, S., Saha, C. K., Kurata, T., Stavropoulos, C.,
198. Fillol Salom, A., Bacarizo, J., Alqasmi, M., Ciges Tomas,
Oliveira, S. R. A., et al. (2020) A widespread toxin-anti!
J. R., Mart nez!Rubio, R., et al. (2019) Hijacking the
toxin system exploiting growth control via alarmone signal!
hijackers: Escherichia coli pathogenicity islands redirect
ing, Proc. Natl. Acad. Sci. USA, 117, 10500!10510.
helper phage packaging for their own benefit, Mol. Cell, 75,
180. Ferullo, D. J., and Lovett, S. T. (2008) The stringent
1020!1030.
response and cell cycle arrest in Escherichia coli, PLoS
199. O’Hara, B. J., Barth, Z. K., McKitterick, A. C., and Seed,
Genet., 4, e1000300.
K. D. (2017) A highly specific phage defense system is a
181. Owen, S. V, Wenner, N., Dulberger, C. L., Rodwell, E. V.,
conserved feature of the Vibrio cholerae mobilome, PLoS
Bowers!Barnard, A., et al. (2020) Prophage!encoded
Genet., 13, 1!17.
phage defence proteins with cognate self!immunity,
200. McKitterick, A. C., and Seed, K. D. (2018) Anti!phage
bioRxiv, doi: 10.1101/2020.07.13.199331.
islands force their target phage to directly mediate island
182. Rousset, F., Dowding, J., Bernheim, A., Rocha, E., and
excision and spread, Nat. Commun., 9, 1!8.
Bikard, D. (2021) Prophage!encoded hotspots of bacterial
201. McKitterick, A. C., Hays, S. G., Johura, F. T., Alam, M.,
immune systems, bioRxiv, doi:
10.1101/2021.01.21.
and Seed, K. D. (2019) Viral satellites exploit phage pro!
427644.
teins to escape degradation of the bacterial host chromo!
183. Atanasiu, C., Su, T. J., Sturrock, S. S., and Dryden,
some, Cell Host Microbe, 26, 504!514.e4.
D. T. F. (2002) Interaction of the ocr gene 0.3 protein of
202. Barth, Z. K., Silvas, T. V, Angermeyer, A., and Seed, K. D.
bacteriophage T7 with EcoKl restriction/modification
(2020) Genome replication dynamics of a bacteriophage
enzyme, Nucleic Acids Res., 30, 3936!3944.
and its satellite reveal strategies for parasitism and viral
184. Isaev, A., Drobiazko, A., Sierro, N., Gordeeva, J.,
restriction, Nucleic Acids Res., 48, 249!263.
Yosef, I., et al. (2020) Phage T7 DNA mimic protein Ocr is
203. Barth, Z. K., Netter, Z., Angermeyer, A., Bhardwaj, P., and
a potent inhibitor of BREX defence, Nucleic Acids Res., 48,
Seed, K. D. (2020) A family of viral satellites manipulates
5397!5406.
invading virus gene expression and can affect cholera toxin
185. Penadés, J. R., and Christie, G. E. (2015) The phage!
mobilization, mSystems, 5, e00358!20, doi: 10.1128/
inducible chromosomal islands: a family of highly evolved
mSystems.00358!20.
molecular parasites, Annu. Rev. Virol., 2, 181!201.
204. Hays, S. G., and Seed, K. D. (2020) Dominant Vibrio
186. Christie, G. E., and Dokland, T. (2012) Pirates of the
cholerae phage exhibits lysis inhibition sensitive to disrup!
Caudovirales, Virology, 434, 210!221.
tion by a defensive phage satellite, Elife, 9, e53200.
187. Dokland, T. (2019) Molecular piracy: redirection of bacte!
205. McKitterick, A. C., LeGault, K. N., Angermeyer, A.,
riophage capsid assembly by mobile genetic elements,
Alam, M., and Seed, K. D. (2019) Competition between
Viruses, 11, 1003.
mobile genetic elements drives optimization of a phage!
188. Fillol!Salom, A., Miguel!Romero, L., Marina, A., Chen,
encoded CRISPR!Cas system: insights from a natural arms
J., and Penadés, J. R. (2020) Beyond the CRISPR Cas
race, Philos. Trans. R. Soc. B, 374, 20180089.
safeguard: PICI encoded innate immune systems protect
206. Makarova, K. S., Wolf, Y. I., Snir, S., and Koonin, E. V.
bacteria from bacteriophage predation, Curr. Opin.
(2011) Defense islands in bacterial and archaeal genomes
Microbiol., 56, 52 58.
and prediction of novel defense systems, J. Bacteriol., 193,
189. Novick, R. P., Christie, G. E., and Penadés, J. R. (2010)
6039!6056.
The phage related chromosomal islands of Gram positive
207. Doron, S., Melamed, S., Ofir, G., Leavitt, A.,
bacteria, Nat. Rev. Microbiol., 8, 541 551.
Lopatina, A., et al. (2018) Systematic discovery of
190. Fillol Salom, A., Mart nez Rubio, R., Abdulrahman, R.
antiphage defense systems in the microbial pangenome,
F., Chen, J., Davies, R., and Penadés, J. R. (2018) Phage!
Science, 359, eaar4120, doi: 10.1126/science.aar4120.
inducible chromosomal islands are ubiquitous within the
208. Burroughs, A. M., and Aravind, L. (2020) Identification of
bacterial universe, ISME J., 12, 2114!2128.
uncharacterized components of prokaryotic immune sys!
191. Mitarai, N. (2020) How pirate phage interferes with helper
tems and their diverse eukaryotic reformulations,
phage: comparison of the two distinct strategies, J. Theor.
J. Bacteriol., 202, e00365!20, doi: 10.1128/JB.00365!20.
Biol., 486, 110096.
209. Santiveri, M., Roa!Eguiara, A., Kuhne, C., Wadhwa, N.,
192. Novick, R. P. (2019) Pathogenicity islands and their role in
Berg, H. C., et al. (2020) Structure and function of stator
staphylococcal
biology,
Microbiol.
Spectr.,
7,
units of the bacterial flagellar motor, Cell, 183, 244!257.
doi: 10.1128/microbiolspec.GPP3!0062!2019.
210. Ofir, G., Herbst, E., Baroz, M., Cohen, D., Millman, A.,
193. Tormo!Más, M. Á., Mir, I., Shrestha, A., Tallent, S. M.,
et al. (2021) Antiviral activity of bacterial TIR domains via
Campoy, S., Lasa, Í., et al. (2010) Moonlighting bacterio!
signaling molecules that trigger cell death, bioRxiv,
phage proteins derepress staphylococcal pathogenicity
doi: 10.1101/2021.01.06.425286.
islands, Nature, 465, 779!782.
211. Ka, D., Oh, H., Park, E., Kim, J., and Bae, E. (2020)
194. Mir!Sanchis, I., Mart nez!Rubio, R., Mart , M., Chen, J.,
Structural and functional evidence of bacterial antiphage
Lasa, Í., et al. (2012) Control of Staphylococcus aureus
protection by Thoeris defense system via NAD+ degrada!
pathogenicity island excision, Mol. Microbiol., 85, 833!
tion, Nat. Commun., 11, 2816, doi: 10.1038/s41467!020!
845.
16703!w.
195. Ram, G., Chen, J., Kumar, K., Ross, H. F., Ubeda, C.,
212. Bayless, A. M., and Nishimura, M. T. (2020) Enzymatic
et al. (2012) Staphylococcal pathogenicity island interfer!
functions for Toll/Interleukin!1 receptor domain proteins
ence with helper phage reproduction is a paradigm of mol!
in the plant immune system, Front. Genet., 11, 539.
ecular parasitism, Proc. Natl. Acad. Sci. USA, 109, 16300!5.
213. Badrinarayanan, A., Le, T. B. K., and Laub, M. T. (2015)
196. Ram, G., Chen, J., Ross, H. F., and Novick, R. P. (2014)
Bacterial chromosome organization and segregation, Annu.
Precisely modulated pathogenicity island interference with
Rev. Cell Dev. Biol., 31, 171!199.
БИОХИМИЯ том 86 вып. 4 2021
МИКРОБНЫЕ СРЕДСТВА ЗАЩИТЫ ОТ ВИРУСОВ. ГЛАВА II
553
214. Panas, M. W., Jain, P., Yang, H., Mitra, S., Biswas, D.,
tionary mechanism for protein diversification, PLoS One,
et al. (2014) Noncanonical SMC protein in Mycobacterium
6, e18819.
smegmatis restricts maintenance of Mycobacterium fortui%
223. Dupuis, M.!È., Villion, M., Magadán, A. H., and
tum plasmids, Proc. Natl. Acad. Sci. USA, 111, 13264!
Moineau, S. (2013) CRISPR!Cas and restriction!modifi!
13271.
cation systems are compatible and increase phage resis!
215. Pace, H. C., and Brenner, C. (2001) The nitrilase super!
tance, Nat. Commun., 4, 1!7.
family: classification, structure and function, Genome Biol.,
224. Hynes, A. P., Villion, M., and Moineau, S.
(2014)
2, 1!9.
Adaptation in bacterial CRISPR!Cas immunity can be dri!
216. Mekhedov, S. L., Makarova, K. S., and Koonin, E. V.
ven by defective phages, Nat. Commun., 5, 1!6.
(2017) The complex domain architecture of SAMD9 fam!
225. Bernheim, A., and Sorek, R. (2020) The pan!immune sys!
ily proteins, predicted STAND!like NTPases, suggests new
tem of bacteria: antiviral defence as a community resource,
links to inflammation and apoptosis, Biol. Direct, 12, 13.
Nat. Rev. Microbiol., 18, 113!119.
217. Forsberg, K. J., and Malik, H. S. (2018) Microbial
226. Koonin, E. V., Makarova, K. S., Wolf, Y. I., and
genomics: the expanding universe of bacterial defense sys!
Krupovic, M. (2020) Evolutionary entanglement of mobile
tems, Curr. Biol., 28, R361!R364.
genetic elements and host defence systems: guns for hire,
218. Van, V., and Van Valen, L. (1973) A New Evolutionary Law,
Nat. Rev. Genet., 21, 119!131.
The University of Chicago, Illinois.
227. Høyland!Kroghsbo, N. M., Paczkowski, J., Mukherjee, S.,
219. Stern, A., and Sorek, R. (2011) The phage!host arms race:
Broniewski, J., Westra, E., et al. (2017) Quorum sensing
shaping the evolution of microbes, Bioessays, 33, 43!51.
controls the Pseudomonas aeruginosa CRISPR!Cas adap!
220. Paterson, S., Vogwill, T., Buckling, A., Benmayor, R.,
tive immune system, Proc. Natl. Acad. Sci. USA, 114, 131!
Spiers, A. J., et al. (2010) Antagonistic coevolution accel!
135.
erates molecular evolution, Nature, 464, 275!278.
228. Xiong, L., Liu, S., Chen, S., Xiao, Y., Zhu, B., et al. (2019)
221. Ignacio!Espinoza, J. C., Ahlgren, N. A., and Fuhrman,
A new type of DNA phosphorothioation!based antiviral
J. A. (2020) Long!term stability and Red Queen!like strain
system in archaea, Nat. Commun., 10, 1!11.
dynamics in marine viruses, Nat. Microbiol., 5, 265!271.
229. Al!Shayeb, B., Sachdeva, R., Chen, L.!X., Ward, F.,
222. Furuta, Y., Kawai, M., Uchiyama, I., and Kobayashi, I.
Munk, P., et al. (2020) Clades of huge phages from across
(2011) Domain movement within a gene: a novel evolu!
Earth’s ecosystems, Nature, 578, 425!431.
MICROBIAL ARSENAL OF ANTIVIRAL DEFENSES. PART II
Review
A. B. Isaev1*, O. S. Musharova1,2, and K. V. Severinov1,3*
1 Skolkovo Institute of Science and Technology, 143028 Moscow, Russia; E%mail: tcft18@gmail.com
2 Institute of Molecular Genetics, Russian Academy of Sciences, 123182 Moscow, Russia
3 Waksman Institute of Microbiology, Piscataway, NJ 08854, USA; E%mail: severik@waksman.rutgers.edu
Bacteriophages or phages are viruses that infect bacterial cells (for the scope of this review we will also consider virus!
es that infect Archaea). The constant threat of phage infection is a major force that shapes evolution of microbial
genomes. To withstand infection, bacteria had evolved numerous strategies to avoid recognition by phages or to direct!
ly interfere with phage propagation inside the cell. Classical molecular biology and genetic engineering had been
deeply intertwined with the study of phages and host defenses. Nowadays, owing to the rise of phage therapy, broad
application of CRISPR!Cas technologies, and development of bioinformatics approaches that facilitate discovery of
new systems, phage biology experiences a revival. This review describes variety of strategies employed by microbes to
counter phage infection. In the first part defense associated with cell surface, roles of small molecules, and innate
immunity systems relying on DNA modification were discussed. The second part focuses on adaptive immunity sys!
tems, abortive infection mechanisms, defenses associated with mobile genetic elements, and novel systems discovered
in recent years through metagenomic mining.
Keywords: bacteriophages, immunity systems, CRISPR!Cas, Abortive infection, Toxin!Antitioxin, PICI, prokaryot!
ic Argonaute, CBASS
7 БИОХИМИЯ том 86 вып. 4 2021