БИОХИМИЯ, 2021, том 86, вып. 8, с. 1120 - 1137
УДК 577.12
ПУТЬ К РАСШИФРОВКЕ АТОМНЫХ СТРУКТУР
ПРОКАРИОТИЧЕСКОЙ И ЭУКАРИОТИЧЕСКОЙ РИБОСОМ
Обзор
© 2021
Г.Ж. Юсупова1, М.М. Юсупов1,2*
1 Институт генетики, молекулярной и клеточной биологии (IGBMC),
67400 Илькирш!Граффенштаден, Страсбург, Франция
2 Казанский (Приволжский) федеральный университет,
420008 Казань, Россия; электронная почта: marat@igbmc.fr
Поступила в редакцию 06.05.2021
После доработки 08.06.2021
Принята к публикации 08.06.2021
Первые кристаллические структуры прокариотической и эукариотической рибосом были решены нашей
группой, будучи при этом основаны на знаниях, накопленных за десятилетия исследований, начиная с по
лучения G. Palade в 1955 г. первых электронно микроскопических изображений рибосомы. В 1983 г. Алек
сандр Сергеевич Спирин, директор Института белка АН СССР, инициировал первые исследования, направ
ленные на решение структуры рибосом с помощью рентгеноструктурного анализа. В 1999 г. наша группа в
сотрудничестве с H. Noller опубликовала первую кристаллическую структуру целой бактериальной рибосо
мы в комплексе с основными функциональными лигандами, включая матричную РНК и три транспортные
РНК - в А , Р и Е сайтах. В 2011 г. наша лаборатория опубликовала первую атомарную структуру эукарио
тической рибосомы, решенную методом рентгеноструктурного анализа. Была подтверждена консерватив
ность структурной организации главных функциональных участков рибосомы, таких как декодирующий и
пептидилтрансферазный центры, и описаны эукариот специфичные элементы, определяющие различия с
прокариотическим протагонистом. С помощью рентгеноструктурного анализа мы исследовали общие
принципы ингибирования биосинтеза белка на эукариотических рибосомах, механизмы устойчивости к ан
тибиотикам, а также показали структурные детерминанты, определяющие разницу в ингибировании между
рибосомами бактерий и эукариот. Эти и последующие структуры функционирующей рибосомы впервые
продемонстрировали на атомарном уровне, как рибосома связывается с матричной и транспортными РНК,
представляя собой первый шаг к пониманию того, как структура рибосомы определяет ее функции.
КЛЮЧЕВЫЕ СЛОВА: рибосома, структура, рентгеноструктурный анализ, крио ЭМ.
DOI: 10.31857/S0320972521080054
ВВЕДЕНИЕ
белков и длинных цепей рибосомальных РНК
(рРНК). Несмотря на то что рибосома и ее ос
Синтез белка осуществляется с помощью
новные функции были описаны более 60 лет на
рибосом, универсальных рибонуклеопротеино
зад, большой размер и высокодинамичная при
вых комплексов, которые считывают генетичес
рода этой макромолекулы затрудняли ее струк
кую информацию, закодированную в матрич
турные исследования. В то же время было оче
ной РНК (мРНК), и катализируют формирова
видно, что теоретически только один метод мог
ние пептидных связей между аминокислотами в
обеспечить получение структурной информа
запрограммированном порядке, что приводит к
ции атомарного разрешения, которая в конеч
формированию новых пептидных цепей [1]. Эти
ном счете требовалась исследователям рибо
гигантские органеллы, имеющие молекулярную
сом - это рентгеноструктурный анализ.
массу ~2,5 МДа у бактерий и до 4,5 МДа - у выс
Наши знания о трехмерной структуре рибо
ших эукариот, состоят из множества различных сомы в 90 е гг. сводились в основном к фрагмен
тарной информации, полученной с помощью
ядерного магнитного резонанса (ЯМР) и рент
Принятые сокращения: крио ЭМ - криоэлектрон
геноструктурного анализа малых субдоменов
ная микроскопия; мРНК - матричная РНК; рРНК - ри
босомальная РНК; тРНК - транспортная РНК; ПТЦ -
рРНК и 10 индивидуальных рибосомных белков
пептидилтрансферазный центр.
[2-6]. Взаимосвязи между структурой и функ
* Адресат для корреспонденции.
циями рибосомы продолжали определять на ос
1120
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1121
нове моделей, полученных с помощью методов
лей рибосомных субчастиц, которые оказались
электронной микроскопии (ЭМ), нейтронной
важными стимуляторами научной активности,
дифракции и молекулярного моделирования,
включая поиск подтверждений указанных моде
базирующихся на результатах биофизических и
лей. Например, модель 30S субчастицы, предло
биохимических исследований [7-9]. Позднее
женная А. Спириным, В. Васильевым и И. Сер
были получены кристаллы и решены структуры
дюком, была основана на результатах ЭМ рибо
доменов рибосомы, содержащие фрагменты
сомных субчастиц, а также на данных исследо
рРНК и рибосомные белки [3, 5].
ваний иммунно электронной микроскопии и
Лаборатории Виктора Васильева и James
белок белковых сшивок. Модель проверялась
Lake впервые предложили модели несиммет
экспериментально методом нейтронного и
ричной структуры 30S субчастицы («V образ
рентгеновского рассеиваний в растворе [19].
ная» модель) и 50S субчастицы («коронообраз
Другой подход, молекулярное моделирование
ная» модель) бактериальной рибосомы [10-12].
малой рибосомной субчастицы, был основан на
Позднее был достигнут значительный прогресс
филогенетически предсказанной вторичной
в исследовании структуры рибосом и рибосом
структуре 16S РНК [20, 21].
ных комплексов низкого разрешения при ис
Одним из важнейших достижений в области
пользовании трехмерной реконструкции ЭМ
исследования структуры рибосом являлась
изображений рибосом, замороженных в тонком
опубликованная карта положения центров масс
слое жидкости (крио ЭМ) [13, 14]. К концу
21 рибосомного белка малой субъединицы, ре
1990 х гг. две исследовательские группы незави
шенная методом нейтронного рассеивания [22].
симо друг от друга использовали подход
Этот подход позволяет измерить расстояния
крио ЭМ для получения структуры бактериаль
между центрами масс двух белков в рибосоме.
ной рибосомы E. coli. Диаметр бактериальной
Предложенная на основе этих результатов мо
рибосомы, составляющий ~ 220 Å, и все описан
дель, хотя и оказалась позднее во многом оши
ные ранее классические особенности структу
бочной, тем не менее инициировала исследова
ры, в том числе «голова», «шея», «щели» и
ния по ее проверке и дискуссии в «рибосомном»
«платформа» малой субъединицы, а также три
сообществе.
выступа в верхней части большой субъединицы,
Эксперименты с поэтапной экстракцией
получили подтверждение при разрешении 25 Å
белков с рибосомных субчастиц путем обработ
[11, 15]. Таким образом, были описаны размеры
ки LiCl в повышающихся концентрациях с пос
и формы субъединиц, общая геометрия их ассо
ледующим ЭМ анализом полученных рибонук
циации друг с другом. При этом разрешении не
леиновых частиц показали, что рРНК определя
которые спирали РНК казались различимы, но
ет форму рибосомы [23, 24]. Результаты этих ис
в целом нельзя было четко отличить белки от
следований были использованы как при моде
РНК. Дальнейшее развитие метода и исследова
лировании малой рибосомной субчастицы, так
ние более сложных комплексов рибосомы поз
и третичной структуры субчастиц, исходя из
волило впервые визуализировать расположение
предсказанной вторичной структуры рРНК [25].
транспортных РНК (тРНК) на внутренней по
Для моделирования РНК в структуре
верхности рибосомных субчастиц. В этих рабо
30S субчастицы использовали интегрированный
тах было впервые в очевидном виде показано
подход на основе филогенетически определен
наличие Е участка связывания тРНК [7].
ной вторичной структуры 16S РНК [25, 26],
Взаиморасположение рибосомных белков,
нейтронной карты положения центров масс
выявление соседних белков, идентификация
белков 30S субчастицы [27], химического зонди
взаимодействующих с белками последователь
рования рРНК на разных этапах сборки субчас
ностей рРНК, а также участков рРНК и белков,
тицы [20, 28, 29] и результатов UV сшивок
участвующих в организации функциональных
рРНК [30].
центров рибосомы - все эти задачи решались с
В результате продолжительных исследова
помощью методов белок белковых или РНК
ний в конце 1990 х - начале 2000 х гг. были по
белковых сшивок, аффинных сшивок и моди
лучены первые рентгеновские структуры целых
фикаций [16-18]. К сожалению, эти подходы
бактериальных рибосом, 30S бактериальных и
для изучения структуры рибосомы не могли
50S архейных субчастиц, которые вызвали лави
предоставить однозначную информацию, т.к.
ну новых работ в области рибосомного биосин
они основаны на анализе малой доли рибосом
теза белка, значительно расширивших наше по
(иногда < 1%) исследуемого образца, который не
нимание функционирования этой молекуляр
может быть полностью структурно гомогенным.
ной машины [31-33].
Однако, основываясь на результатах таких экс
Значительные знания по эксперименталь
периментов, было предложено несколько моде
ной биохимии и гидродинамике рибосом, на
БИОХИМИЯ том 86 вып. 8 2021
1122
ЮСУПОВА, ЮСУПОВ
копленные во время работы в Институте белка в
ша группа, цель которой состояла в поиске под
лаборатории А. С. Спирина, позволили нашей
ходов для решения структуры рибосомы. В
группе проводить разработку новых методов вы
1983 г. был инициирован проект «Кристаллогра
деления и кристаллизации бактериальных, а за
фия рибосомы» в рамках сотрудничества между
тем и эукариотических рибосом. В настоящей
Институтом белка и Институтом кристаллогра
статье мы описываем стратегию и ключевые
фии АН СССР.
этапы исследований, проведенных в первую
Сегодня, по прошествии почти 40 лет после
очередь в нашей лаборатории, которые привели
начала проекта, можно с уверенностью сказать,
к определению первой кристаллической струк
что сложнейшей и наиболее длительной его
туры прокариотической рибосомы и её функцио
частью (что характерно для любого кристалло
нальных комплексов, а также к нашему недав
графического проекта) был поиск условий для
нему прогрессу в определении первой кристал
получения дифрагирующих кристаллов рибосо
лической структуры эукариотической рибосо
мы и ее субчастиц. Мы разработали методы
мы [33-38]. Решение первой кристаллической
очистки и кристаллизации рибосом из нового
структуры эукариотической
80S рибосомы
для лабораторных исследований организма,
представляет собой прорыв в структурно функ
бактерии Thermus thermophilus, которая растет в
циональных исследованиях эукариотических
экстремальных условиях - температура 75 °С яв
рибосом и позволяет рационализировать суще
ляется для нее оптимальной [42-44]. Позже эта
ствующую биохимическую и генетическую ин
экспериментальная модель была использована
формацию, что в конечном итоге стимулирует
для решения структуры малой 30S субчастицы
разработку будущих экспериментальных моде
[32, 45] и полной 70S рибосомы с функциональ
лей для изучения различных аспектов биосинте
ными лигандами, такими как тРНК и мРНК [33,
за белка.
46, 47], и по сей день остается полезной мо
делью в современных кристаллографических и
крио ЭМ исследованиях трансляции и механиз
КРИСТАЛЛИЗАЦИЯ
мов ее регуляции [48-50].
И РЕШЕНИЕ СТРУКТУРЫ
При разработке метода выделения и очистки
БАКТЕРИАЛЬНОЙ РИБОСОМЫ
рибосом T. thermophilus принципиальными ока
зались два этапа. Первый этап - это очистка ри
Бактериальные рибосомы, имеющие коэф
босом через высокоплотностную подушку, со
фициент седиментации 70S, состоят из малой и
держащую CsCl и сахарозу; данный вариант был
большой субчастиц (30S и 50S соответственно).
предложен сотрудником Института белка Зура
Каждая субчастица рибосомы состоит из рРНК,
бом Гогия. Второй этап - это гидрофобная хро
которая составляет две трети массы субчастицы,
матография рибосом на колонке TOYOPEARL
и рибосомных белков, составляющих одну
Butyl («Tosoh Bioscience», Германия), с помощью
треть. Большая субчастица 50S содержит две
которой получают рибосомы с прочно сцеплен
рРНК, 5S (120 нуклеотидов) и
23S рРНК
ными субчастицами, которые пригодны для
(~ 2900 нуклеотидов), в то время как малая суб
кристаллизации; эта часть протокола была
частица 30S содержит только одну 16S рРНК
предложена сотрудником Института кристал
(~ 1500 нуклеотидов). Белковая часть 30S суб
лографии Сергеем Трахановым.
частицы состоит из 21 индивидуального белка, а
Этот основной протокол из двух этапов был
50S - из 33 белков [1].
использован с незначительными изменениями
В 1980 х гг. были предприняты первые по
для кристаллизации трех типов кристаллов ри
пытки получить трехмерные кристаллы рибо
босом. Первая кристаллическая форма рибосом
сомных субчастиц, пригодные для исследования
была получена в Институте белка; ее создание
методом рентгеноструктурного анализа. В Инс
подтвердило принципиальную возможность ис
титуте им. Макса Планка (Max Planck) в Берли
пользования рентгеновского излучения для ис
не группа Ada Yonath в сотрудничестве с Heinz
следования структуры рибосом и ее функцио
Günter Wittman получила первые кристаллы
нальных комплексов [42-44]. Вторая кристал
50S рибосомных субчастиц, выделенных из
лическая форма была использована для первого
Bacillus stearothermophilus и Haloarcula marismortui
определения структуры рибосомы, содержащей
[39, 40]. Эти пионерские работы показали по
мРНК и тРНК, с разрешением 7,8 Å [46], а за
тенциальную возможность исследования рибо
тем - 5,5 Å [33, 47]. Это исследование было про
сом методом рентгеноструктурного анализа
ведено в сотрудничестве с Harry Noller в Центре
[41]. В это время по инициативе Александра
молекулярной биологии РНК в Калифорнийс
Сергеевича Спирина, директора Института бел
ком университете Санта Круз (UCSC). Третья
ка Академии наук СССР, была организована на
кристаллическая форма была получена в лабо
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1123
ратории Venki Ramakrishnan и использована для
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА
определения структуры рибосомы с разрешени
БАКТЕРИАЛЬНОЙ РИБОСОМЫ
ем 2,8 Å [51]. Ранее структура рибосомы E. coli
была определена с разрешением 3,5 Å в лабора
Трехмерные структуры рибосомы 70S и ее
тории Jamie Cate [52].
субчастиц были охарактеризованы при исполь
Наша группа также впервые получила крис
зовании различных методов электронной мик
таллы 30S субчастицы рибосомы T. thermophilus
роскопии в 1980 е гг. [10, 11]. В общей морфоло
[53, 54]. После оптимизации условий роста
гии малой субчастицы выделены такие основ
кристаллов и сбора рентгеновских данных эта
ные блоки как «голова» и «тело» субчастицы. Те
форма была использована для первого решения
ло образовано «плечом» и «платформой», струк
структуры 30S субчастицы в лабораториях Venki
турный элемент перехода «головы» в «тело» наз
Ramakrishnan и Ada Yonath [32, 45]. Таким же об
ван «шеей» малой субчастицы (рис. 1). Большая
разом кристаллы 50S субчастицы, ранее полу
субчастица представляет собой более компакт
ченные в лаборатории Ada Yonath [55], после оп
ную конструкцию, состоящую из округлого ос
тимизации роста кристаллов и сбора данных
нования с тремя выступами, названными
были использованы Peter Moore и Thomas Steitz
«L1 выступ», «центральный выступ» и «стебель
для решения структуры 50S субъединицы из
L7/L12». Значительное улучшение разрешения
H. marismortui [31, 56].
было достигнуто в 1990 е гг. с введением метода
Эти кристаллы отдельных рибосомных суб
реконструкции изображений, полученных с по
частиц (30S и 50S) были использованы для изу
мощью крио ЭМ [59].
чения функциональных комплексов с лиганда
Кристаллографическое исследование бакте
ми, мимикрирующими молекулы мРНК и
риальных рибосом показало, что выступ на «го
тРНК, а также для исследования декодирующе
лове» малой субъединицы состоит исключи
го и пептидилтрансферазного центров рибосо
тельно из спирали h33, а «центральный выступ»
мы [57, 58]. Структуры субчастиц высокого раз
(или «центральный протуберанец») большой
решения 30S T. thermophilus [32] и 50S H. maris!
рибосомной субчастицы состоит из 5S рРНК,
mortui [31] и экспериментальные карты элект
части 23S рРНК, а также рибосомных белков L5,
ронной плотности полной 70S рибосомы были
L18, L25 и L33. Более подробный анализ струк
использованы нашей группой для моделирова
тур субчастиц позволил выявить назначение до
ния полной рибосомы T. thermophilus [33, 46].
менов вторичной структуры рРНК. Например,
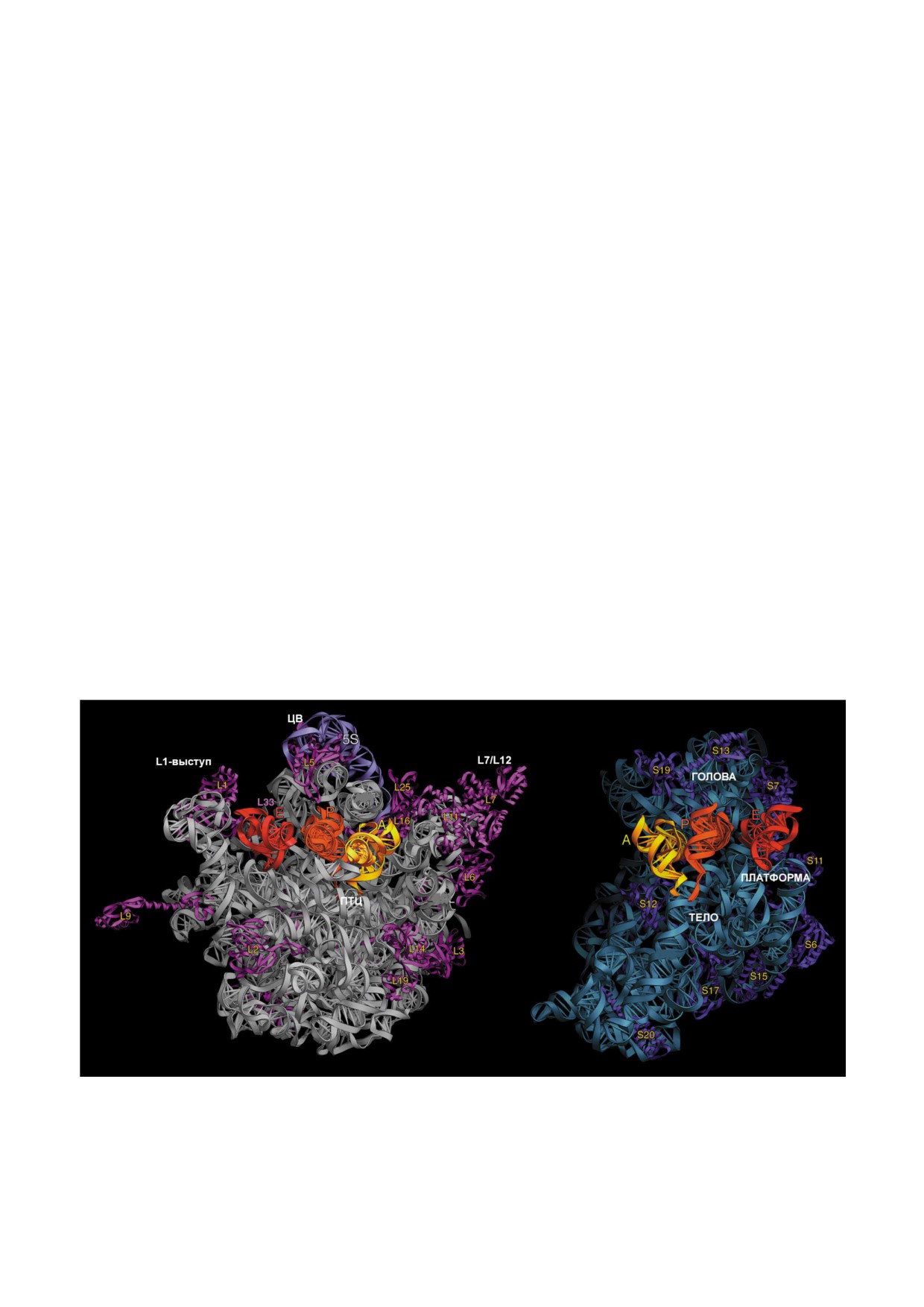
Рис. 1. Структура рибосомных субчастиц T. thermophilus по данным рентгеноструктурного анализа. Вид внутренней поверх
ности субчастиц с тРНК в А (желтый), Р (оранжевый) и Е сайтах (красный). На большой субчастице (слева) обозначе
ны «L1 выступ», «центральный выступ» (ЦВ), «стебель L7/L12» и положение пептидилтрансферазного центра (ПТЦ). На
малой субчастице (справа) обозначены «голова», «тело» и «платформа»
БИОХИМИЯ том 86 вып. 8 2021
1124
ЮСУПОВА, ЮСУПОВ
5′ домен 16S РНК образует «тело» малой рибо
Первая кристаллическая структура полной
сомной субчастицы, средняя часть 16S РНК -
70S рибосомы содержала три транспортные РНК
«платформу», а 3′ домен - «голову» субчастицы.
в А , Р и Е участках (сайтах) связывания и фраг
В целом кристаллическая структура рибосомы
мент матричной РНК длиной 24 нуклеотида, ста
подтвердила правильность предсказанной вто
билизированный в рибосоме между ее субчасти
ричной структуры рибосомальных РНК [33].
цами [33, 47]. На основании этих и последующих
Таким образом, решение кристаллических
кристаллографических исследований рибосомы,
структур бактериальных рибосомных субчастиц
осуществленных нашей группой, было описано
и полной 70S рибосомы привело к описанию
атомарное строение функциональных центров
более 50 структур индивидуальных рибосомных
рибосомы, таких как центры взаимодействия с
белков и точной их локализации. Особенностью
тРНК [33, 61, 62]. Сайты связывания мРНК и три
большинства рибосомных белков является на
сайта связывания тРНК (А, Р и Е) расположены
личие глобулярного домена, который обычно
на интерфейсе субчастиц, где находится и центр
находится на поверхности субчастицы, и длин
декодирования малой субчастицы, в котором
ной неструктурированной аминокислотной
происходит спаривание кодона и антикодона,
последовательности, которая проникает глубо
обеспечивающее точность декодирования
ко вовнутрь субчастицы в её так называемое
мРНК. Анализ полученных нами структур пока
«ядро», состоящее практически полностью из
зал, что мРНК проникает на интерфейс рибосо
рРНК. Анализ структур показал, что рибосом
мы через туннель, расположенный между «голо
ные белки, связываясь с разными спиралями
вой» и «плечом», обвиваясь вокруг «шеи» малой
рРНК, соединяют таким образом различные
субчастицы [47]; место выхода мРНК (5′ конец
структурные домены рибосомы между собой.
мРНК) расположено между «головой» и «плат
Пожалуй, одним из самых больших сюрпри
формой». В том же исследовании продемонстри
зов в ходе анализа кристаллической структуры
ровано, как мРНК образует изгиб между А и
полной рибосомы явилось отсутствие рибосом
Р кодонами, определяющий возможность од
ных белков на контактной поверхности субчас
новременного связывания двух тРНК. Кроме то
тиц, означающее, что функциональные центры
го, было описано, как последовательность Шай
рибосомы организованы только из рибосомаль
на-Дальгарно (ШД) на мРНК взаимодействует с
ной РНК. Ранее, по аналогии с клеточными
анти ШД участком рРНК, фиксируя таким об
ферментами, предполагалось, что энзиматичес
разом точное положение рамки считывания
кая активность рибосомы обеспечивается
мРНК (рис. 2), и как мРНК с этим новообразо
именно белками. Впервые отсутствие рибосом
ванным дуплексом перемещается во время пер
ных белков на контактной поверхности субчас
вых этапов синтеза белка [63-65].
тиц было показано нами в 1986 г. методом три
В последующем цикле работ мы с помощью
тиевой бомбардировки поверхности рибо
рентгеноструктурного анализа продемонстри
сом [60].
ровали молекулярные причины нарушения точ
Рис. 2. Расположение мРНК на малой субчастице. а - Срез субчастицы на уровне мРНК: последовательность Шай
на-Дальгарно (ШД) мРНК взаимодействует с анти ШД участком рРНК; б - проход мРНК вокруг «шеи» малой субчас
тицы между «головой» и «телом». Показана ориентация кодонов в А , Р и Е сайтах на внутренней поверхности субчас
тицы
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1125
ности декодирования на рибосоме, приводящие
лось невозможным получить кристаллы такой
к включению «неправильных» аминокислот в
гигантской макромолекулы (с молекулярным
синтезируемый белок, и предложили механизм
весом ~3,3 МДа у низших эукариот), дающие
ошибочного декодирования, «геометрической
дифракционную картину высокого разрешения.
мимикрии», основанный на комбинации прото
Первые свидетельства того, что эукариоти
нирования и редкой таутомеризации нуклеоти
ческая рибосома способна кристаллизоваться,
дов в кодон антикодоновых дуплексах [66-71].
были получены еще в 1966 г., когда в тканях ку
С использованием такой передовой технологии,
риных эмбрионов при гипотермии были обна
как длинноволновая макромолекулярная крис
ружены двумерные кристаллы рибосом [77]. Тем
таллография, мы впервые смогли напрямую
не менее попытки кристаллизовать эукариоти
точно локализовать сотни ионов калия в функ
ческие рибосомы до недавнего времени не были
ционирующей рибосоме [49]. Мы показали, что
успешными, несмотря на высокую конкурен
ионы калия участвуют не только в формирова
цию в области структурной биологии.
нии структуры комплекса рРНК и рибосомных
В 2010 г. нашей группе удалось закристалли
белков, но также играют важнейшую роль в
зовать полную эукариотическую 80S рибосому из
функциях рибосомы, таких как декодирование
Saccharomyces cerevisiae и получить первую рент
и пептидилтрансферазная реакция.
геновскую структуру эукариотической рибосо
Последующие структурные исследования
мы, сначала с разрешением 4,15 Å, а затем - 3,0 Å
новых функциональных комплексов продолжа
[34, 78]. Успешная кристаллизация рибосомы,
ют углублять наши знания о механизме связыва
выделенной из дрожжей, была основана на раз
ния тРНК в присутствии фактора элонгации Tu
работке нового метода очистки интактных эука
(EF Tu), о процессах декодирования мРНК и
риотических рибосом. Во первых, мы использо
образования пептидных связей, о механизме
вали известный экспериментальный факт, что
гидролиза GTP, транслокации, терминации и
глюкозное голодание растущих дрожжевых кле
рециклинга рибосом [37, 38, 72-75].
ток ингибирует инициацию трансляции и приво
дит к накоплению однородных рибосом, не со
держащих никаких лигандов. Таким образом, на
СТРУКТУРА ЭУКАРИОТИЧЕСКОЙ
начальном этапе клетки проходили стадию глю
РИБОСОМЫ
козного голодания для увеличения исходного ко
личества рибосомных мономеров. Во вторых,
Информация, полученная на основе реше
был разработан очень мягкий протокол очистки
ний кристаллических структур прокариотичес
рибосом, который гарантировал присутствие
ких рибосом высокого разрешения, несомненно
всех компонентов в составе рибосом в неповреж
расширила наши знания о синтезе белков в бак
денном виде. Adam Ben Shem ввел фракциони
териях, но в то же время понимание молекуляр
рование клеточного экстракта S30 полиэтилен
ных основ функционирования эукариотических
гликолем 20 000 с последующей очисткой 80S ри
рибосом оставалось сильно ограниченным. Бы
босом в градиенте сахарозы в недиссоциирую
ло известно, что в результате эволюции эукарио
щих условиях [78]. Таким образом были выделе
тические рибосомы имеют примерно на 40%
ны высокооднородные образцы рибосомного
большую массу (у низших эукариот) по сравне
препарата, кристаллизация которых привела к
нию с прокариотическими рибосомами [35, 76]
получению хорошо дифрагирующих кристаллов.
и, несмотря на присутствие консервативных
Кристаллические структуры эукариотичес
участков в рРНК и ряда общих белков, рибосо
кой рибосомы из S. cerevisiae значительно рас
мы про и эукариот имеют значительные разли
ширили понимание механизмов синтеза белка и
чия. Эти различия обусловлены присутствием
регуляции трансляции в клетке. Например, ана
примерно 30 дополнительных эукариот специ
лиз структуры рибосомы дрожжей, выделенной
фических белков и > 50 нуклеотидных последо
из подвергнутых глюкозному голоданию клеток,
вательностей, называемых сегментами расши
показал, что нерибосомный белок Stm1 оказал
рения РНК, которые входят в эволюционно
ся связанным с рибосомой 80S, создавая таким
консервативное ядро рРНК. До 2010 г. наши
образом дополнительный «мост» между двумя
знания о структурной организации эукариоти
рибосомными субчастицами и приводя к повы
ческой рибосомы базировались лишь на
шению стабильности рибосомы в условиях
ЭМ изображениях низкого разрешения. При
кристаллизации. Функциональная роль «стрес
этом использование рентгеноструктурного ана
сового» белка заключалась в полном блокирова
лиза, который мог обеспечить получение струк
нии связывания основных функциональных ли
турной информации на атомарном уровне, счи
гандов, таких как мРНК и тРНК, путем пере
талось экспериментально не достижимым: каза
крывания мест их связывания с рибосомой.
БИОХИМИЯ том 86 вып. 8 2021
1126
ЮСУПОВА, ЮСУПОВ
Кристаллографическое решение структур комп
Малая субчастица рибосомы 40S имеет те же
лексов эукариотической рибосомы с эукариот
структурные элементы, как и прокариотичес
специфическими ингибиторами, а также инги
кая 30S, известные как «голова», «тело» и «плат
биторами широкого спектра не только позволи
форма» (рис. 3). Информация о функциональ
ли исследователям определить общие принци
ных сайтах эукариотических рибосом получена
пы ингибирования белкового синтеза и резисте
путем их сопоставления с данными по кристал
нтности к нему, но и позволили улучшить каче
лической структуре функциональных комплек
ство кристаллов и сбор рентгеновских данных с
сов бактериальных рибосом [33, 62].
разрешением 2,8 Å [79-83].
Большая рибосомная субчастица имеет ко
Рибосомные субчастицы 40S и 60S эукарио
ронообразную форму, она включает «централь
тического организма Tetrahymena thermophila бы
ный выступ»,
«L1 выступ» и
«P стебель»
ли успешно кристаллизованы с белковыми фак
(рис. 3). На внутренней стороне большой рибо
торами в лаборатории Nenad Ban и определены с
сомной субчастицы расположены 3 сайта связы
разрешением 3,8 и 3,6 Å соответственно [84, 85].
вания тРНК (А, Р и Е) и пептидилтрансфераз
Рибосома 80S представляет собой асиммет
ный центр, где катализируется образование пеп
ричный комплекс из 80 различных белков и
тидных связей. Последний примыкает ко входу
4 цепей РНК (рис. 3). Каждый рибосомный
в туннель, по которому продвигается цепь за
компонент имеет одну копию, за исключением
рождающегося белка, прежде чем выйти из ри
белков Р стебля, которые имеют по четыре ко
босомы со стороны растворителя.
пии. Эксперименты также показали, что бакте
При сравнении рибосомных структур прока
риальные и эукариотические рибосомы имеют
риот и эукариот стало очевидным отсутствие
общее структурное ядро, состоящее из 34 кон
класс специфичных фрагментов на внутренних
сервативных белков (15 - в малой субчастице,
поверхностях субчастиц, что согласуется с кон
19 - в большой) и ~ 4400 нуклеотидных основа
сервативностью универсальных функций рибо
ний РНК, которые вместе образуют основные
сомы (рис. 4). Указанная консервативность
функциональные центры рибосом, включая
структуры наблюдается в декодирующем цент
сайт декодирования, пептидилтрансферазный
ре, пептидилтрансферазном центре и вокруг
центр и сайты связывания тРНК [76].
пептидного туннеля на стороне растворителя,
Эволюционно консервативное ядро рибосо
который используется для ассоциации рибосо
мы может быть структурно определено путем
мы с мембранами во время синтеза белка. Зна
сравнения модели 80S рибосомы дрожжей с ее
чительные различия в структурной организации
бактериальным аналогом (рис. 4) [34, 76]. По
малой и большой рибосомной субчастицы на
давляющее большинство ~1,35 МДа эукариот
стороне, обращенной к раствору, по видимому,
специфичных частей расположено на поверх
связаны со значительно более сложным путем
ности рибосомы, окружая эволюционно кон
инициации трансляции в эукариотических
сервативное ядро: 350 кДа составляют сегменты
клетках по сравнению с прокариотами.
экспансии (расширения) рРНК, 800 кДа белков
В целом можно отметить, что полученная
вообще отсутствует у бактерий, 200 кДа пред
нами модель первой и на сегодняшний день
ставляют эукариот специфичные домены, при
единственной кристаллографической структу
сутствующие в консервативных белках (рис. 4).
ры эукариотической рибосомы высокого разре
Рибосома дрожжей содержит 46 специфичных
шения включает ~90,5% из всех (~13 000) ами
для эукариот белков (18 - в 40S субчастице, 28 - в
нокислотных остатков рибосомных белков и
60S субчастице), а также эукариот специфичные
~95,5% нуклеотидов рРНК (из ~5500). Эта атом
«вставки» в большинстве основных белков. Рибо
ная модель продемонстрировала уникальную
сомальная РНК содержит несколько сегментов
топографию эукариот специфичных элементов
экспансии в своих консервативных цепях с об
и картину их взаимодействия с универсально
щей длиной более 900 оснований [34]. Большин
консервативным ядром, в том числе все эукари
ство из этих сегментов экспансии рРНК и «вста
от специфичные мосты между двумя рибосом
вок» белков окружают ядро со стороны раствори
ными субчастицами.
теля и доступны для потенциальных взаимодей
ствий с молекулярными партнерами, такими как
факторы трансляции и белки шапероны. В раз
НОВАЯ НОМЕНКЛАТУРА
личных организмах эукариот размер рибосо
РИБОСОМНЫХ БЕЛКОВ
мы 80S колеблется в пределах 0,5-1,0 МДа, что
в значительной степени объясняется размером
Для облегчения сравнения рибосом разных
вставок в сегменты экспансии ES7L, ES15L,
видов была принята номенклатурная система,
ES27L и ES39L в 25S-28S рРНК [76].
основанная на названиях семейств белков
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1127
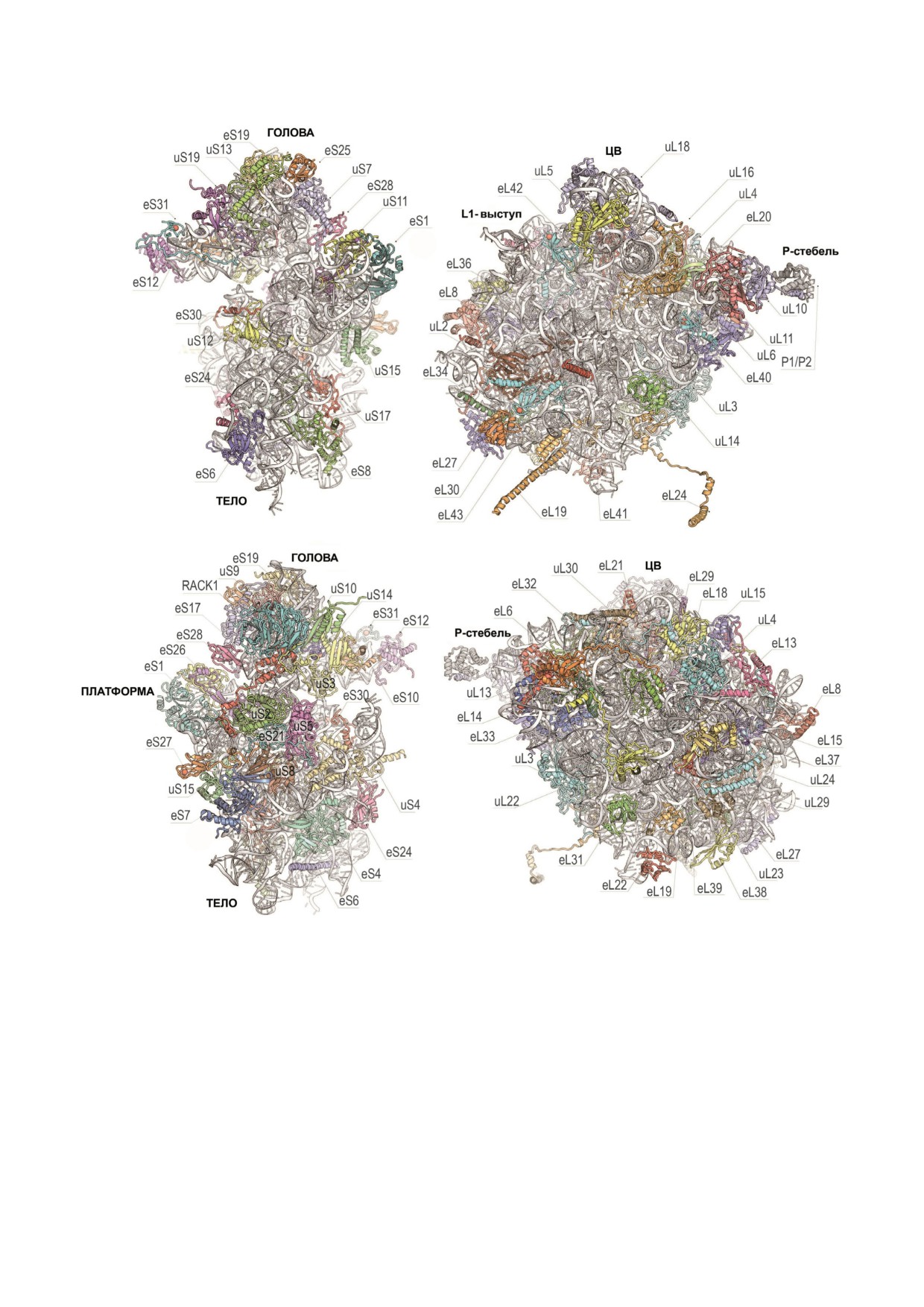
Рис. 3. Структура рибосомных субчастиц дрожжей (40S субчастица - слева, 60S субчастица - справа). Вверху показан вид
внутренней поверхности субчастиц, внизу - вид внешней поверхности субчастиц (с использованием новой номенклату
ры белков). Ориентиры включают «голову», «тело» и «платформу» 40S субчастицы, а также «центральный выступ» (ЦВ),
«L1 выступ» и «P стебель» 60S субчастицы
(рис. 3). Поскольку первыми были выделены и
имеют префикс «u» (universal), за которым сле
полностью секвенированы рибосомные белки
дуют их названия из E. coli. Бактериальные бел
из E. coli, их архейным и эукариотическим гомо
ки, не имеющие эукариотических гомологов,
логам были присвоены соответствующие назва
обозначаются префиксом «b» (bacterial); соответ
ния. Белки, обнаруженные в рибосомах всех
ственно эукариот специфические белки имеют
трех доменов (бактерии, археи и эукариоты),
префикс «e» (eukaryotic) [86].
БИОХИМИЯ том 86 вып. 8 2021
1128
ЮСУПОВА, ЮСУПОВ
СЕГМЕНТЫ ЭКСПАНСИИ
рибосомы (рис. 4, 5, а). Интерфейс между суб
РИБОСОМАЛЬНОЙ РНК
частицами рибосом, а также область входа
мРНК и туннель выхода полипептида очень
Сегменты экспансии рибосомальной РНК
консервативны и практически не содержат сег
расположены преимущественно на внешней по
ментов экспансии и эукариот специфичных
верхности обеих субчастиц эукариотической
белков. Субчастицы заметно различаются
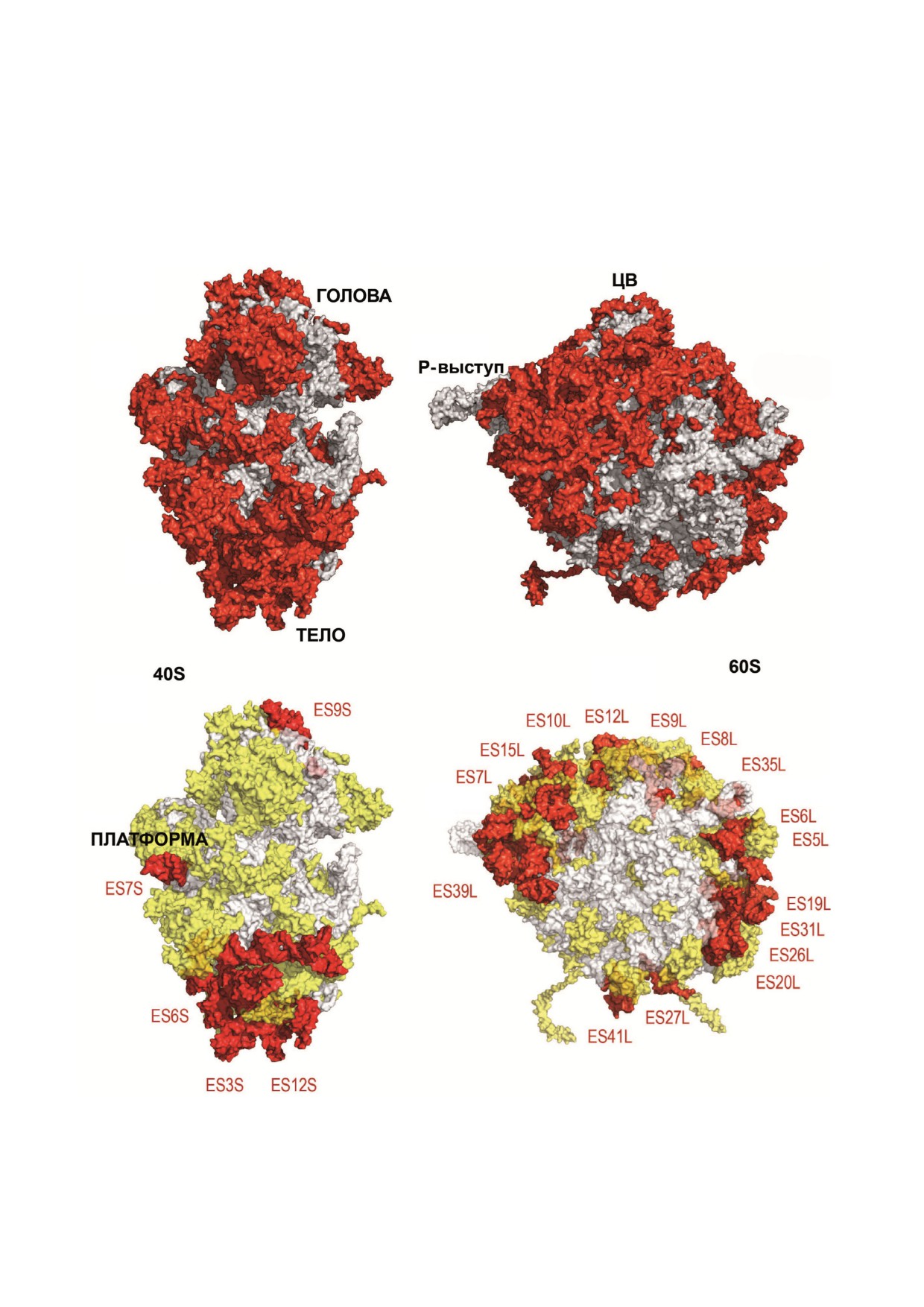
Рис. 4. Характеристика внешней поверхности рибосомных субчастиц дрожжей. Вверху - консервативное ядро субчасти
цы показано серым цветом; специфичные элементы рибосом эукариот показаны красным. Внизу - специфичные для эу
кариот белковые фрагменты (желтые), сегменты экспансии рРНК (красные; ES, expansion segments)
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1129
а
б
в
г
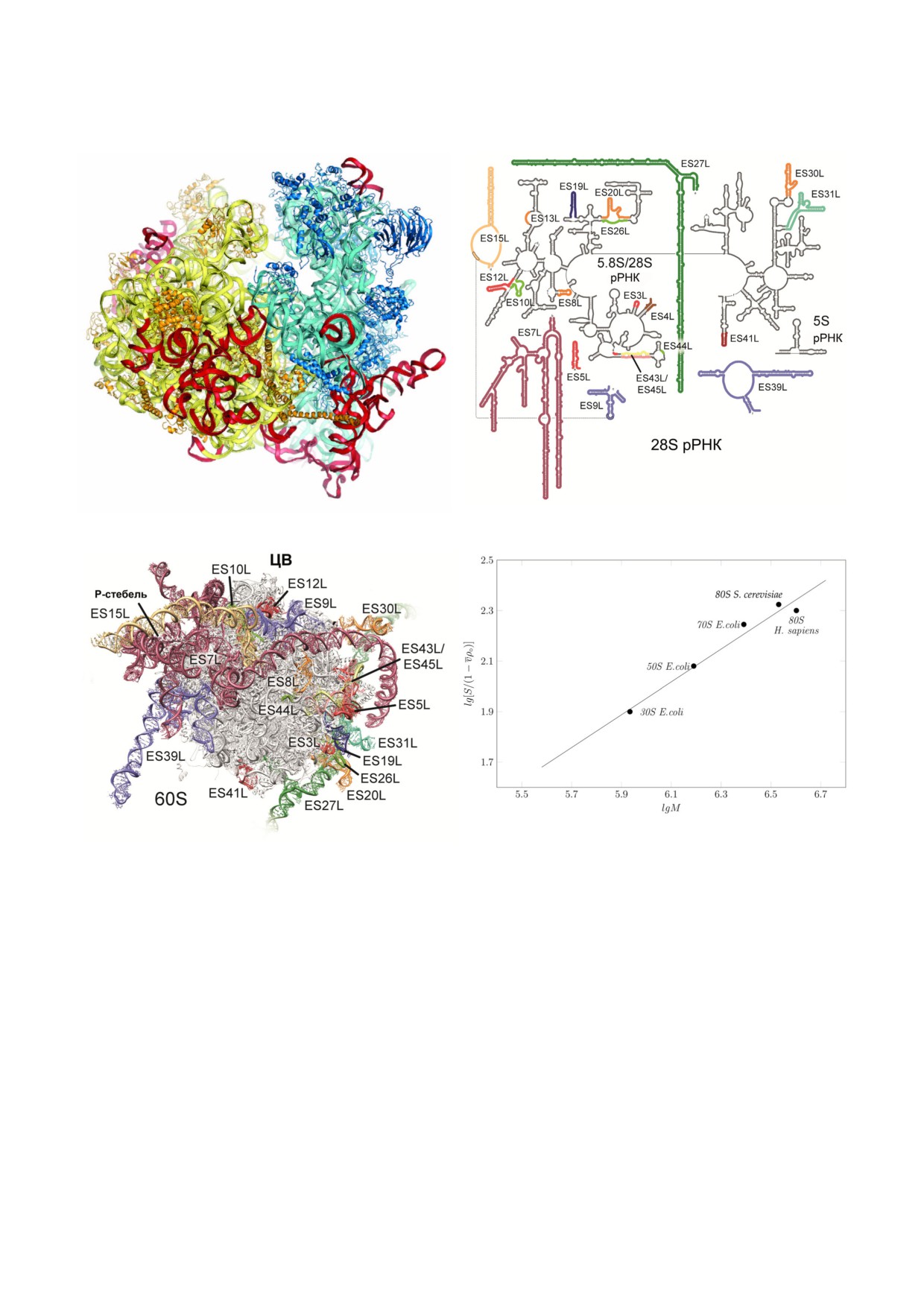
Рис. 5. Сегменты экспансии рибосомальной РНК. а - Структура рибосомы дрожжей, где сегменты экспансии показаны
красным; модель основана на данных по электронной плотности, полученных методом рентгеноструктурного анализа.
б - Вторичная структура 28S РНК человека, цветом показаны сегменты экспансии ES3S, ES6S, ES7L, ES15L, ES27L, ES30L
и ES39L, которые не могли быть интерпретированы в крио ЭМ реконструкциях из за их неструктурированной формы.
в - Пример молекулярной модели 60S субчастицы рибосомы человека с теоретически предсказанными сегментами экс
пансии рРНК, не имеющими экспериментального подтверждения [87]. Ориентиры включают центральный выступ (ЦВ),
L1 выступ и P стебель. Рисунок 60S субчастицы рибосомы человека любезно предоставлен D. Wilson. г - Зависимость ко
эффициента седиментации рибосомной частицы от молекулярной массы; график любезно предоставлен С. Агаларовым
пространственным распределением сегментов
липептида и тянется от P стебля по направле
экспансии рРНК. Например, в малой субчасти
нию к белку L1.
це большая часть эукариот специфичных рРНК
Информация о структуре сегментов экспан
сконцентрирована в нижней части, где происхо
сии была получены в последующих крио ЭМ
дит активное взаимодействие двух больших сег
экспериментах по анализу 80S рибосом дрозо
ментов экспансии ES3S и ES6S. Напротив, в
филы и человека [87, 88]. Были предложены тео
большой субчастице многочисленные сегменты
ретические модели всех сегментов экспансии,
экспансии образуют почти непрерывное коль
включающие 9 сегментов в 40S субчастице и
цо, которое охватывает туннель для выхода по
21 - в 60S субчастице (рис. 5, б, в). Сегменты
3 БИОХИМИЯ том 86 вып. 8 2021
1130
ЮСУПОВА, ЮСУПОВ
экспансии 28S РНК рибосомы человека по сос
решении 3,0 Å обеспечила более точное и де
таву подобны дрожжам, однако имеют значи
тальное представление о молекулярных компо
тельно бóльшую длину. Например, ES3S, ES7L,
нентах, опосредующих эти контакты между суб
ES9L, ES15L, ES27L и ES39L в дрожжах состав
частицами. Особенно примечательной является
ляют приблизительно 110, 200, 70, 20, 160 и 140
эволюционная консервативность межсубчас
нуклеотидов соответственно, а те же сегменты у
тичных мостов в ядре рибосомы. Обнаружено
человека длиннее на 50, 670, 40, 170, 550 и 100
семь «консервативных» мостов, а также не
нуклеотидов соответственно. При этом сегмен
сколько мостов, специфичных для бактерий и
ты экспансии рРНК человека ES3S, ES6S, ES7L,
эукариот [33, 34, 51, 52]; практически все ком
ES15L, ES27L, ES30L и ES39L не могут быть ин
поненты дополнительных мостов обеих субчас
терпретированы в крио ЭМ реконструкциях
тиц являются эукариот специфичными. Инте
из за их неструктурированной гибкости (формы).
ресно отметить, что в отличие от рибосом бакте
Таким образом, в модели рибосомы представле
рий, у эукариот именно белки играют домини
ны структуры рРНК, полученные вычислитель
рующую роль в формировании специфичных
ным методом определения вторичной и третич
межсубчастичных мостов [33]. Эукариот специ
ной структур нуклеиновых кислот, основываю
фичные мосты расположены на периферии ин
щимся на ее первичной структуре (рис. 5, б, в).
терфейса субчастиц, что значительно увеличи
Сравнение коэффициентов седиментации
вает поверхность их взаимодействия [34, 90, 91].
субчастиц бактериальной рибосомы 50S и 30S,
Существует только один специфичный для
полной бактериальной рибосомы 70S, дрожже
эукариот межсубчастичный мост, расположен
вой и человеческой рибосомы 80S показало, что
ный в центре рибосомы - мост eB14. Он образо
рибосома человека являлась частично разверну
ван самым маленьким белком дрожжевых кле
той (рис. 5, г): для рибосомы человека и дрож
ток (25 а.о.) eL41 (рис. 3), состоящим из одной
жей этот коэффициент был равен 78S и 80S со
альфа спирали. eL41 выступает из 60S субчасти
ответственно. Тем не менее молекулярная масса
цы и касается 40S субчастицы в непосредствен
рибосомы человека примерно на 500 000 Да
ной близости от центра декодирования и почти
больше по сравнению с рибосомой дрожжей по
полностью погружен в связывающий карман,
причине длинных сегментов экспансии
состоящий из спиралей h27, h45 и h44. В орга
28S РНК. Очевидно, что частично развернутая
низации этого моста примечательны два аспек
структура сегментов экспансии рибосомы чело
та. Во первых, связывающий карман eL41 в ма
века затрудняла как кристаллизацию этой рибо
лой субчастице высококонсервативен у эукари
сомы, так и другие структурно функциональ
от и бактерий. Во вторых, в контексте полной
ные исследования.
рибосомы eL41 гораздо сильнее связан с субчас
тицей 40S, чем с 60S. Тем не менее, несмотря на
лишь незначительные контакты eL41 с субчас
МЕЖСУБЧАСТИЧНЫЕ МОСТЫ
тицей 60S, он остается частью большой субчас
тицы при диссоциации рибосомы. У бактерий
Важность межсубчастичных мостов не вы
есть только один пример такого необычного
зывает вопросов, поскольку они поддерживают
моста, который образуется рибосомным белком
структурную целостность полной рибосомы, а
большой субчастицы и связывается с малой суб
также осуществляют коммуникационные сооб
частицей через функционально значимые части
щения между малой и большой субчастицами в
их структур [62]. Этот необычный мост образо
процессе синтеза белка. В ходе трансляции ри
ван белком bL31, который консервативен среди
босома подвергается глобальным конформаци
бактерий и соединяет центральный выступ
онным перестройкам, необходимым для транс
большой субчастицы с лабильным головным до
локации (передвижения) мРНК и тРНК, терми
меном малой субчастицы. Эукариот специфич
нации синтеза и других процессов. Эти измене
ной отличительной чертой являются два моста
ния включают в себя межсубчастичное враще
большой субчастицы, eB12 и eB13, образован
ние, например, поворот головного домена ма
ные длинными альфа спиралями белков eL19 и
лой субчастицы. Исследованная нами кристал
eL24 (рис. 3).
лографическая структура рибосомы 80S демон
стрирует эту органеллу именно в состоянии по
ворота [78].
ИНГИБИРОВАНИЕ ФУНКЦИОНИРОВАНИЯ
Первые несколько эукариот специфичных
ЭУКАРИОТИЧЕСКОЙ РИБОСОМЫ
мостов были обнаружены в крио ЭМ исследо
ваниях дрожжевой рибосомы с низким разреше
Десятилетия исследований антибактериаль
нием [89, 90]. Наша модель при атомарном раз
ных средств (антибиотиков) продемонстрирова
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1131
ли разнообразие молекулярных механизмов ин
рРНК и взаимодействует с eL42 способом, на
гибирования синтеза белка [92]. Атомарные
поминающим взаимодействие с CCA концом
структуры прокариотических рибосом стали ос
тРНК. Строгая селективность ингибиторов
новой для разработки новых ингибиторов рибо
Е сайта по отношению к эукариотам объясняет
сом, которые могут использоваться не только в
ся наличием двух специфичных остатков рРНК,
фармакологических целях, но и служить важны
которые формируют связывающий карман.
ми инструментами для изучения механизмов
Несмотря на то что циклогексимид и лактими
синтеза белка в бактериях. Аналогично эукарио
домицин связываются с одним и тем же сайтом
тическая рибосома является основной мишенью
и, вероятно, конкурируют с тРНК, они влияют
для эукариот специфических ингибиторов при
на трансляцию по разному. Лактимидомицин
родного происхождения. Несмотря на ограни
преимущественно ингибирует работу рибосомы
ченное понимание их молекулярных механиз
в процессе образования первой пептидной свя
мов, специфичные для эукариот ингибиторы
зи, тогда как циклогексимид останавливает ри
все чаще используются в исследованиях и по
босому во время продолжающейся трансля
тенциально могут функционировать в качестве
ции [99, 100].
новых терапевтических средств против широко
Пептидилтрансферазный центр состоит иск
го спектра инфекционных и онкологических за
лючительно из нуклеотидов рРНК, он располо
болеваний, генетических нарушений [93-96].
жен на большой субчастице. Образование пеп
Некоторые специфичные для эукариот ингиби
тидных связей требует, чтобы два субстрата,
торы были исследованы с использованием
пептидил тРНК и аминоацил тРНК, были пра
кристаллов из субчастицы 50S археи H. maris!
вильно выровнены в А и Р сайтах соответ
mortui ввиду ее близкого (хотя и не полного)
ственно. В то время как бластицидин S связыва
сходства с некоторыми частями эукариотичес
ется с Р участком большой субчастицы схожим
кой рибосомы [97, 98].
образом у эукариот, бактерий и архей, было об
В наших первых исследованиях механизмов
наружено, что многочисленные эукариот спе
ингибирования эукариотических рибосом с по
цифические ингибиторы связываются с
мощью рентгеноструктурного анализа было
А участком ПТЦ (рис. 4). При этом с точки зре
изучено 16 комплексов рибосомы S. cerevisiae с
ния химии ингибиторы имеют сходный способ
эукариот специфичными ингибиторами (рис. 6)
связывания внутри кармана. Все ингибиторы
[79]. Ингибиторы широкого спектра действия
А сайта при ассоциации с рибосомой индуциру
нацелены на пептидилтрансферазный центр на
ют сходные структурные перестройки в непос
большой субчастице (бластицидин S), декоди
редственной близости, а также на расстоянии до
рующий центр (генетицин/G418) и сайт связы
15 Å от ПТЦ. Показано, что нуклеотиды U2873
вания мРНК-тРНК на малой субчастице (пак
(U2504 в E .coli) и C2824 (C2452 в E. coli) участ
тамицин, эдеин). Впервые было показано, что
вуют во взаимодействии с ингибиторами в
существует принципиальное отличие механизма
дрожжах. Различные ориентации этих нуклео
ингибирования белкового синтеза у эукариот и
тидов в бактериях, обусловленные отличной ор
прокариот - по Е сайту связывания тРНК, пол
ганизацией во «вторичном» и, может быть, сле
ностью отсутствующему у бактерий. В итоге бы
дующих слоях связывающего «кармана», препят
ли описаны специфичные для эукариот ингиби
ствуют связыванию прокариотических рибосом
торы: а) циклогексимид, лактимидомицин и
с ингибиторами [79, 101].
филлантозид, взаимодействующие с Е сайтом
Декодирующий центр рибосомы локализо
связывания тРНК, б) токсин Т 2, дезоксинива
ван на малой субчастице и представляет собой
ленол, веррукарин, нарциклазин, ликорин, на
жестко зафиксированный «карман», в котором
гилактон С, анизомицин, гомогаррингтонин и
проходит точный отбор по антикодону амино
бластицидин, взаимодействующие с пептидил
ацил тРНК, комплементарному расположенно
трансферазным центром (рис. 6). Глутаримидные
му в А сайте кодону мРНК. У бактерий амино
ингибиторы циклогексимид и лактимидомицин
гликозидные антибиотики снижают точность
располагались в Е сайте, образованном высоко
трансляции и ингибируют транслокацию тРНК,
консервативными нуклеотидами 25S рРНК и
приводя к нарушению конформации нуклеоти
участком специфичного для эукариот бел
дов декодирующего центра. В дополнение к ис
ка eL42. Из них лактимидомицин несет допол
пользованию аминогликозидов против грамот
нительное лактонное кольцо, которое располо
рицательных бактерий их способность к подав
жено поверх eL42 и направлено к интерфейсу
лению преждевременной остановки трансля
субъединицы. Филлантозид, несмотря на разли
ции, приводящему к абортированию пептида,
чия с глутаримидами по химическим свойствам,
может найти применение в терапии наслед
вступает в контакт с теми же нуклеотидами
ственных болезней, вызванных нонсенс мута
БИОХИМИЯ том 86 вып. 8 2021
3*
1132
ЮСУПОВА, ЮСУПОВ
а
б
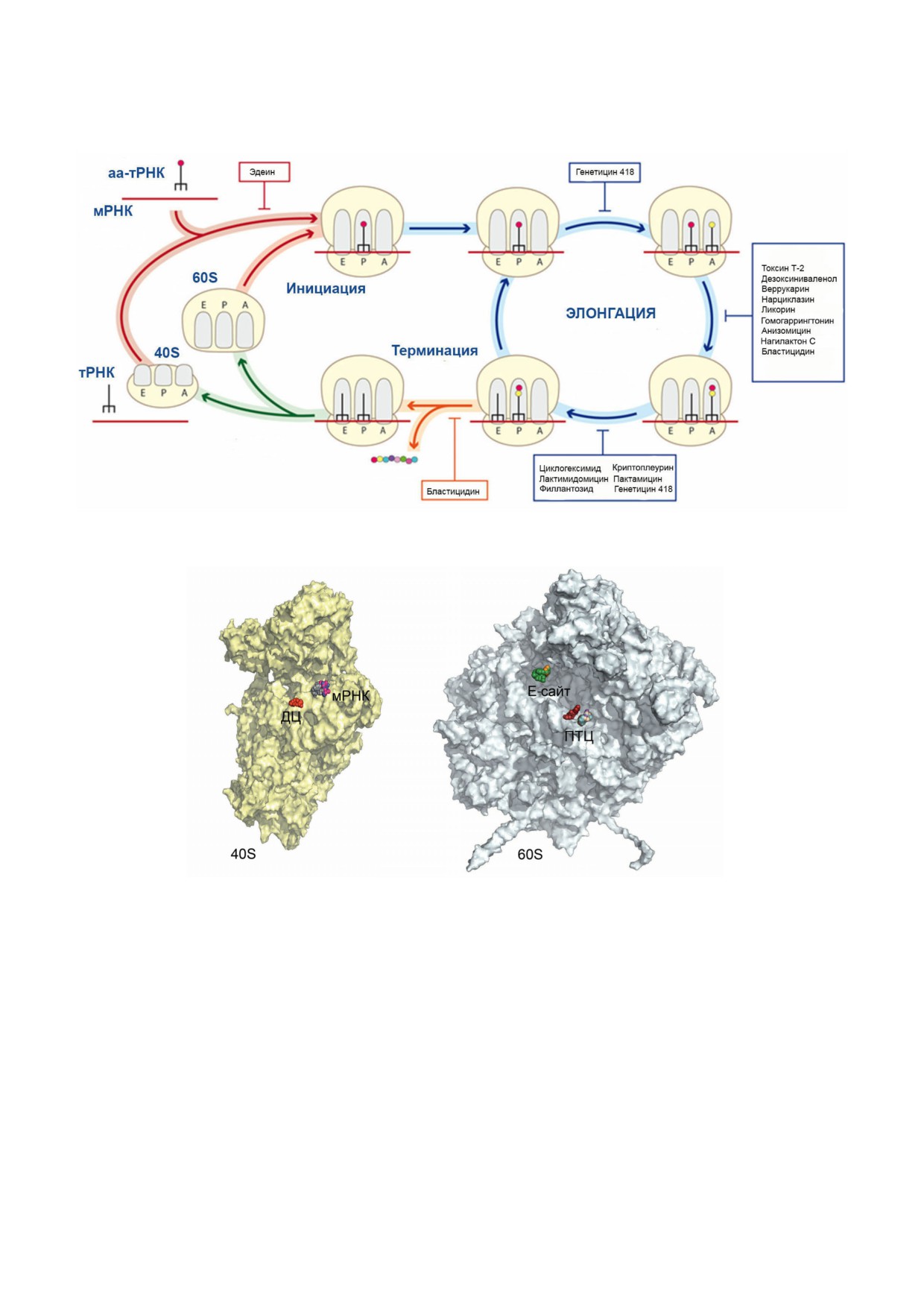
Рис. 6. Ингибирование функционирования эукариотической рибосомы. а - Схема стадий синтеза белка у эукариот с ука
занием участков связывания ряда ингибиторов; б - сайты связывания ингибиторов в функциональных центрах на внут
ренней поверхности малой (слева) и большой (справа) рибосомных субчастиц. ДЦ - декодирующий центр (генети
цин/G418), мРНК - проход матричной РНК (пактамицин, криптоплеурин, эдеин), Е сайт - участок связывания тРНК
(циклогексимид, лактимидомицин, филлантозид), ПТЦ - пептидилтрансферазный центр (бластицидин S, токсин Т 2,
дезоксиниваленол, веррукарин, нарциклазин, ликорин, нагилактон С, анизомицин, гомогаррингтонин)
циями [96, 102]. Канонический сайт связывания
риями и эукариотами, но идентичны в дрожжах
аминогликозидов находится в верхней части
и у людей: G1645 (A1408 в E. coli ) и A1754
спирали h44 18S рРНК и содержит консерватив
(G1491 в E. coli ). В наших работах было проде
ные нуклеотиды декодирующего центра A1755 и
монстрировано, как это различие нуклеотидов в
А1756 (A1492 и А1493 в E. coli соответственно).
декодирующем участке объясняет специфич
В непосредственной близости находятся два
ность ряда аминогликозидных антибиотиков,
нуклеотида, которые различаются между бакте
способных к ингибированию рибосом бактерий
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1133
в организме человека [79]. Мы также показали,
включая патогенные, и открывает новые воз
что аминогликозидные антибиотики, напри
можности для поиска путей борьбы с патогена
мер, паромомицин, G418, TC007 и гентамицин,
ми, такими как Candida albicans, Staphylococcus
обладают способностью связывания и с другими
aureus и другими.
участками на эукариотической рибосоме, поми
К сожалению, функционирование рибосом
мо декодирующего центра [103].
высокоразвитых эукариот, включая рибосомы
В исследованиях с использованием рентгено
человека, недостаточно изучено и требует до
структурного анализа мы продемонстрировали,
полнительных биохимических и гидродинами
что такие ингибиторы, как пактамицин, крип
ческих исследований, включая получение под
топлеурин и эдеин, связываются в канале мРНК
ходящих кристаллов для рентгеноструктурного
в Е сайте малой субчастицы и взаимодействуют
анализа. Основное отличие рибосом человека
только с 18S рРНК [79]. При этом пактамицин и
(молекулярный вес 4,5 МДа) от рибосом низ
эдеин являются ингибиторами широкого спект
ших эукариот (дрожжей, 3,3 МДа) связано с по
ра действия. У бактерий и эукариот описан кон
явлением сегментов расширения рибосомаль
сервативный сайт связывания пактамицина; в
ной РНК, локализованных в основном на поверх
случае эдеина связывание также идет в консерва
ности 60S субчастицы и обладающих особенной
тивном сайте, однако принимаемая ингибито
подвижностью. В недавних крио ЭМ исследо
ром конфигурация различается в дрожжах и бак
ваниях рибосом человека было показано, что
териальной малой субчастице. Криптоплеурин,
значительная часть этих сегментов расширения
напротив, был описан как специфичный для эу
рРНК (~1 МДа) оказалась не структурирована
кариот ингибитор [104]. Тем не менее получение
и, следовательно, не могла быть подвергнута
структуры комплекса криптоплеурина с рибосо
рентгеноструктурному анализу [87, 88, 105]. Та
мой дрожжей пока не смогло объяснить природу
ким образом, на сегодняшний день мы, к сожа
его специфичности. Положение эдеина, пакта
лению, не имеем полных данных об атомарном
мицина и криптоплеурина в ассоциации с рибо
строении рибосомы человека.
сомой позволяет предположить, что они влияют
Вопрос разработки методов стабилизации
на транслокацию деацилированной тРНК из
рибосом высших организмов для проведения
Р сайта в Е сайт и, предположительно, на ини
структурных исследований остается открытым.
циацию трансляции у эукариот. При использова
нии полученной ранее модели эукариотической
рибосомы мы продолжаем рентгеноструктурные
Благодарности. Мы благодарим Султана Ага
исследования новых ингибиторов, проявляющих
ларова (Институт белка АН РАН, Россия) и Да
антираковое действие [80-83].
ниэля Уилсона (Гамбургский университет, Гер
мания) за любезное предоставление рис. 3.
Финансирование. Исследование выполнено
ВЫВОДЫ
при финансовой поддержке Российского науч
ного фонда (грант № 20 65 47031).
Прогресс в развитии новых технологий в об
Конфликт интересов. Авторы заявляют об от
ласти рентгеноструктурной кристаллографии и
сутствии конфликта интересов.
крио ЭМ создал уникальную возможность для
Соблюдение этических норм. Настоящая
глубокого исследования механизмов функцио
статья не содержит описания каких либо иссле
нирования рибосом в различных организмах,
дований с участием людей в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Spirin, A. S. (2002) Ribosome as a molecular machine,
ery and evolution of the ribosome, Trends Biochem. Sci.,
FEBS Lett., 514, 2 10.
23, 208 212, doi: 10.1016/s0968 0004(98)01214 6.
2.
Fourmy, D., Recht, M. I., Blanchard, S. C., and Puglisi,
5.
Wimberly, B. T., White, S. W., and Ramakrishnan, V.
J. D. (1996) Structure of the A site of Escherichia coli 16S
(1997) The structure of ribosomal protein S7 at 1.9 Å reso
ribosomal RNA complexed with an aminoglycoside antibi
lution reveals a beta hairpin motif that binds double
otic, Science, 274, 13671371, doi: 10.1126/science.
stranded nucleic acids, Structure,
5,
11871198,
274.5291.1367.
doi: 10.1016/s0969 2126(97)00269 4.
3.
Agalarov, S. C., Sridhar Prasad, G., Funke, P. M., Stout,
6.
Bocharov, E. V., Gudkov, A. T., Budovskaya, E. V., and
C. D., and Williamson, J. R. (2000) Structure of the
Arseniev, A. S. (1998) Conformational independence of N
S15,S6,S18 rRNA complex: assembly of the 30S ribosome
and C domains in ribosomal protein L7/L12 and in the
central domain, Science, 288, 107 113, doi: 10.1126/sci
complex with protein L10, FEBS Lett., 423, 347 350,
ence.288.5463.107.
doi: 10.1016/s0014 5793(98)00121 5.
4.
Ramakrishnan, V., and White, S. W. (1998) Ribosomal
7.
Agrawal, R. K., Penczek, P., Grassucci, R. A., Li, Y.,
protein structures: insights into the architecture, machin
Leith, A., et al. (1996) Direct visualization of A , P , and
БИОХИМИЯ том 86 вып. 8 2021
1134
ЮСУПОВА, ЮСУПОВ
E site transfer RNAs in the Escherichia coli ribosome,
24.
Vasiliev, V. D., and Zalite, O. M. (1980) Specific compact
Science,
271,
10001002, doi:
10.1126/science.271.
selfpacking of the ribosomal 23S RNA, FEBS Lett., 121,
5251.1000.
101 104, doi: 10.1016/0014 5793(80)81275 0.
8.
Svergun, D. I., Burkhardt, N., Pedersen, J. S., Koch,
25.
Noller, H. F. (1984) Structure of ribosomal RNA, Annu.
M. H., Volkov, V. V., et al. (1997) Solution scattering struc
Rev. Biochem., 53, 119 162, doi: 10.1146/annurev.bi.53.
tural analysis of the 70 S Escherichia coli ribosome by con
070184.001003.
trast variation. I. Invariants and validation of electron
26.
Woese, C. R., Gutell, R., Gupta, R., and Noller, H. F.
microscopy models, J. Mol. Biol.,
271,
588601,
(1983) Detailed analysis of the higher order structure of
doi: 10.1006/jmbi.1997.1190.
16S like ribosomal ribonucleic acids, Microbiol. Rev., 47,
9.
Green, R., and Noller, H. F. (1997) Ribosomes and trans
621 669.
lation, Annu. Rev. Biochem., 66, 679 716, doi: 10.1146/
27.
Capel, M. S., Engelman, D. M., Freeborn, B. R.,
annurev.biochem.66.1.679.
Kjeldgaard, M., Langer, J. A., et al. (1987) A complete
10.
Vasiliev, V. D. (1974) Morphology of the ribosomal 30S
mapping of the proteins in the small ribosomal subunit of
subparticle according to electron microscopic data, Acta
Escherichia coli, Science, 238, 1403 1406, doi: 10.1126/sci
Biol. Med. Ger., 33, 779 793,
ence.3317832.
11.
Lake, J. A. (1976) Ribosome structure determined by elec
28.
Stern, S., Moazed, D., and Noller, H. F. (1988) Structural
tron microscopy of Escherichia coli small subunits, large
analysis of RNA using chemical and enzymatic probing
subunits and monomeric ribosomes, J. Mol. Biol., 105,
monitored by primer extension, Methods Enzymol., 164,
131 139.
481 489, doi: 10.1016/s0076 6879(88)64064 x.
12.
Vasiliev, V. D., Selivanova, O. M., Baranov, V. I., and
29.
Powers, T., and Noller, H. F. (1995) A temperature depen
Spirin, A. S. (1983) Structural study of translating 70S
dent conformational rearrangement in the ribosomal pro
ribosomes from Escherichia coli. I. Electron microscopy,
tein S4.16S rRNA complex, J. Biol. Chem., 270, 1238
FEBS Lett.,
155,
167172, doi:
10.1016/0014
1242, doi: 10.1074/jbc.270.3.1238.
5793(83)80232 4.
30.
Brimacombe, R., Gornicki, P., Greuer, B., Mitchell, P.,
13.
Frank, J. (1989) Image analysis of single macromolecules,
Osswald, M., et al. (1990) The three dimensional structure
Electron. Microsc. Rev., 2, 53 74, doi: 10.1016/0892
and function of Escherichia coli ribosomal RNA, as studied
0354(89)90010 5.
by cross linking techniques, Biochim. Biophys. Acta, 1050,
14.
Stark, H., Mueller, F., Orlova, E. V., Schatz, M., Dube, P.,
8 13, doi: 10.1016/0167 4781(90)90133 m.
et al. (1995) The 70S Escherichia coli ribosome at 23 Å res
31.
Ban, N., Nissen, P., Hansen, J., Moore, P. B., and Steitz,
olution: fitting the ribosomal RNA, Structure, 3, 815 821,
T. A. (2000) The complete atomic structure of the large
doi: 10.1016/s0969 2126(01)00216 7.
ribosomal subunit at 2.4 Å resolution, Science, 289, 905
15.
Vasiliev, V. D., and Kotliansky, V. E. (1977) The 30 S ribo
920, doi: 10.1126/science.289.5481.905.
somal subparticle retains its main morphological features
32.
Wimberly, B. T., Brodersen, D. E., Clemons, W. M., Jr.,
after removal of half the proteins, FEBS Lett., 76, 125
Morgan Warren, R. J., Carter, A. P., et al. (2000) Structure
128.
of the 30S ribosomal subunit, Nature, 407, 327 339,
16.
Sergiev, P., Leonov, A., Dokudovskaya, S., Shpanchenko,
doi: 10.1038/35030006.
O., Dontsova, O., et al. (2001) Correlating the X ray struc
33.
Yusupov, M. M., Yusupova, G. Z., Baucom, A.,
tures for halo and thermophilic ribosomal subunits with
Lieberman, K., Earnest, T. N., et al. (2001) Crystal struc
biochemical data for the Escherichia coli ribosome, Cold
ture of the ribosome at 5.5 Å resolution, Science, 292, 883
Spring Harb. Symp. Quant. Biol.,
66,
87100,
896, doi: 10.1126/science.1060089.
doi: 10.1101/sqb.2001.66.87.
34.
Ben Shem, A., Garreau de Loubresse, N., Melnikov, S.,
17.
Graifer, D., and Karpova, G. (2015) Interaction of tRNA
Jenner, L., Yusupova, G., and Yusupov, M. (2011) The
with eukaryotic ribosome, Int. J. Mol. Sci., 16, 7173 7194,
structure of the eukaryotic ribosome at 3.0 Å resolution,
doi: 10.3390/ijms16047173.
Science, 334, 1524 1529, doi: 10.1126/science.1212642.
18.
Graifer, D., and Karpova, G. (2015) Roles of ribosomal
35.
Yusupova, G., and Yusupov, M. (2014) High resolution
proteins in the functioning of translational machinery of
structure of the eukaryotic 80S ribosome, Annu. Rev.
eukaryotes, Biochimie, 109, 1 17, doi: 10.1016/j.biochi.
Biochem., 83, 467 486, doi: 10.1146/annurev biochem
2014.11.016.
060713 035445.
19.
Spirin, A. S., Serdyuk, I. N., Shpungin, J. L., and Vasiliev,
36.
Yusupova, G., and Yusupov, M. (2015) Ribosome bio
V. D. (1979) Quaternary structure of the ribosomal 30S
chemistry in crystal structure determination, RNA, 21,
subunit: model and its experimental testing, Proc. Natl.
771 773, doi: 10.1261/rna.050039.115.
Acad. Sci. USA, 76, 4867 4871, doi: 10.1073/pnas.76.
37.
Jenner, L., Melnikov, S., Garreau de Loubresse, N., Ben
10.4867.
Shem, A., Iskakova, M., et al. (2012) Crystal structure of
20.
Stern, S., Powers, T., Changchien, L. M., and Noller, H. F.
the 80S yeast ribosome, Curr. Opin. Struct. Biol., 22, 759
(1989) RNA protein interactions in 30S ribosomal sub
767, doi: 10.1016/j.sbi.2012.07.013.
units: folding and function of 16S rRNA, Science, 244,
38.
Demeshkina, N., Jenner, L., Yusupova, G., and
783 790, doi: 10.1126/science.2658053.
Yusupov, M. (2010) Interactions of the ribosome with
21.
Brimacombe, R. (1991) RNA protein interactions in the
mRNA and tRNA, Curr. Opin. Struct. Biol., 20, 325 332,
Escherichia coli ribosome, Biochimie,
73,
927936,
doi: 10.1016/j.sbi.2010.03.002.
doi: 10.1016/0300 9084(91)90134 m.
39.
Yonath, A., Mussig, J., and Wittmann, H. G. (1982)
22.
Moore, P. B., Engelman, D. M., Langer, J. A.,
Parameters for crystal growth of ribosomal subunits, J. Cell
Ramakrishnan, V. R., Schindler, D. G., et al. (1984)
Biochem., 19, 145 155, doi: 10.1002/jcb.240190205.
Neutron scattering and the 30S ribosomal subunit of E.
40.
Makowski, I., Frolow, F., Saper, M. A., Shoham, M.,
coli, Basic Life Sci, 27, 73 91, doi: 10.1007/978 1 4899
Wittmann, H. G., and Yonath, A. (1987) Single crystals of
0375 4_4.
large ribosomal particles from Halobacterium marismortui
23.
Vasiliev, V. D., Selivanova, O. M., and Koteliansky, V. E.
diffract to 6 Å, J. Mol. Biol., 193, 819 822, doi: 10.1016/
(1978) Specific selfpacking of the ribosomal 16S RNA,
0022 2836(87)90362 7.
FEBS Lett.,
95,
273276, doi:
10.1016/0014
41.
Von Bohlen, K., Makowski, I., Hansen, H. A., Bartels, H.,
5793(78)81009 6.
Berkovitch Yellin, Z., et al. (1991) Characterization and
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1135
preliminary attempts for derivatization of crystals of large
57.
Nissen, P., Hansen, J., Ban, N., Moore, P. B., and Steitz,
ribosomal subunits from Haloarcula marismortui diffract
T. A. (2000) The structural basis of ribosome activity in
ing to
3
Å resolution, J. Mol. Biol.,
222,
1115,
peptide bond synthesis, Science,
289,
920930,
doi: 10.1016/0022 2836(91)90730 t.
doi: 10.1126/science.289.5481.920.
42.
Trakhanov, S., Yusupov, M., Shirokov, V., Garber, M.,
58.
Ogle, J. M., Brodersen, D. E., Clemons, W. M., Jr., Tarry,
Mitschler, A., et al. (1989) Preliminary X ray investigation
M. J., Carter, A. P., and Ramakrishnan, V.
(2001)
of 70S ribosome crystals from Thermus thermophilus,
Recognition of cognate transfer RNA by the 30S ribosomal
J. Mol. Biol.,
209,
327328, doi:
10.1016/0022
subunit, Science, 292, 897902, doi: 10.1126/science.
2836(89)90282 9.
1060612.
43.
Yusupova, G., Yusupov, M., Spirin, A., Ebel, J. P.,
59.
Frank, J., Penczek, P., Grassucci, R., and Srivastava, S.
Moras, D., Ehresmann, C., and Ehresmann, B. (1991)
(1991) Three dimensional reconstruction of the 70S
Formation and crystallization of Thermus thermophilus 70S
Escherichia coli ribosome in ice: the distribution of riboso
ribosome/tRNA complexes, FEBS Lett., 290, 69 72,
mal RNA, J. Cell Biol., 115, 597 605.
doi: 10.1016/0014 5793(91)81228 z.
60.
Yusupov, M. M., and Spirin, A. S. (1986) Are there proteins
44.
Yusupov, M. M., Garber, M. B., Vasiliev, V. D., and Spirin,
between the ribosomal subunits? Hot tritium bombardment
A. S. (1991) Thermus thermophilus ribosomes for crystal
experiments, FEBS Lett., 197, 229 233, doi: 10.1016/
lographic studies, Biochimie, 73, 887 897, doi: 10.1016/
0014 5793(86)80332 5.
0300 9084(91)90130 s.
61.
Jenner, L., Demeshkina, N., Yusupova, G., and Yusupov,
45.
Schluenzen, F., Tocilj, A., Zarivach, R., Harms, J.,
M. (2010) Structural rearrangements of the ribosome at the
Gluehmann, M., et al. (2000) Structure of functionally acti
tRNA proofreading step, Nat. Struct. Mol. Biol., 17, 1072
vated small ribosomal subunit at 3.3 angstroms resolution,
1078, doi: 10.1038/nsmb.1880.
Cell, 102, 615 623, doi: 10.1016/s0092 8674(00)00084 2.
62.
Jenner, L. B., Demeshkina, N., Yusupova, G., and
46.
Cate, J. H., Yusupov, M. M., Yusupova, G. Z., Earnest,
Yusupov, M. (2010) Structural aspects of messenger RNA
T. N., and Noller, H. F. (1999) X ray crystal structures of
reading frame maintenance by the ribosome, Nat. Struct.
70S ribosome functional complexes, Science, 285, 2095
Mol. Biol., 17, 555 560, doi: 10.1038/nsmb.1790.
2104, doi: 10.1126/science.285.5436.2095.
63.
Yusupova, G., Jenner, L., Rees, B., Moras, D., and
47.
Yusupova, G. Z., Yusupov, M. M., Cate, J. H., and Noller,
Yusupov, M. (2006) Structural basis for messenger RNA
H. F. (2001) The path of messenger RNA through the ribo
movement on the ribosome, Nature, 444, 391394,
some, Cell,
106,
233241, doi:
10.1016/s0092
doi: 10.1038/nature05281.
8674(01)00435 4.
64.
Jenner, L., Romby, P., Rees, B., Schulze Briese, C.,
48.
Flygaard, R. K., Boegholm, N., Yusupov, M., and Jenner,
Springer, M., et al. (2005) Translational operator of mRNA
L. B. (2018) Cryo EM structure of the hibernating
on the ribosome: how repressor proteins exclude ribosome
Thermus thermophilus 100S ribosome reveals a protein
binding, Science, 308, 120 123, doi: 10.1126/science.
mediated dimerization mechanism, Nat. Commun., 9,
1105639.
4179, doi: 10.1038/s41467 018 06724 x.
65.
Jenner, L., Rees, B., Yusupov, M., and Yusupova, G.
49.
Rozov, A., Khusainov, I., El Omari, K., Duman, R.,
(2007) Messenger RNA conformations in the ribosomal
Mykhaylyk, V., et al. (2019) Importance of potassium ions
E site revealed by X ray crystallography, EMBO Rep., 8,
for ribosome structure and function revealed by long wave
846 850, doi: 10.1038/sj.embor.7401044.
length X ray diffraction, Nat. Commun.,
10,
2519,
66.
Demeshkina, N., Jenner, L., Westhof, E., Yusupov, M.,
doi: 10.1038/s41467 019 10409 4.
and Yusupova, G. (2012) A new understanding of the
50.
Hussain, T., Llacer, J. L., Wimberly, B. T., Kieft, J. S., and
decoding principle on the ribosome, Nature, 484, 256 259,
Ramakrishnan, V. (2016) Large scale movements of IF3
doi: 10.1038/nature10913.
and tRNA during bacterial translation initiation, Cell, 167,
67.
Demeshkina, N., Jenner, L., Westhof, E., Yusupov, M.,
133 144.e113, doi: 10.1016/j.cell.2016.08.074.
and Yusupova, G. (2013) New structural insights into the
51.
Selmer, M., Dunham, C. M., Murphy, F. V.,
decoding mechanism: translation infidelity via a G•U pair
Weixlbaumer, A., Petry, S., et al. (2006) Structure of the
with Watson-Crick geometry, FEBS Lett., 587, 1848 1857,
70S ribosome complexed with mRNA and tRNA, Science,
doi: 10.1016/j.febslet.2013.05.009.
313, 1935 1942, doi: 10.1126/science.1131127.
68.
Rozov, A., Demeshkina, N., Westhof, E., Yusupov, M., and
52.
Schuwirth, B. S., Borovinskaya, M. A., Hau, C. W.,
Yusupova, G. (2015) Structural insights into the transla
Zhang, W., Vila Sanjurjo, A., et al. (2005) Structures of the
tional infidelity mechanism, Nat. Commun., 6, 7251,
bacterial ribosome at 3.5 Å resolution, Science, 310, 827
doi: 10.1038/ncomms8251.
834, doi: 10.1126/science.1117230.
69.
Rozov, A., Westhof, E., Yusupov, M., and Yusupova, G.
53.
Yusupov, M. M., Trakhanov, S. D., Barinin, V. V.,
(2016) The ribosome prohibits the G•U wobble geometry
Boroviagin, B. D., Garber, M. B., et al.
(1987)
at the first position of the codon anticodon helix, Nucleic
Crystallization of the 30S subunits of Thermus thermophilus
Acids Res., 44, 6434 6441, doi: 10.1093/nar/gkw431.
ribosomes, Dokl. Akad. Nauk SSSR, 292, 1271 1274.
70.
Rozov, A., Demeshkina, N., Khusainov, I., Westhof, E.,
54.
Trakhanov, S. D., Yusupov, M., Agalarov, S., Garber M.,
Yusupov, M., and Yusupova, G. (2016) Novel base pairing
Ryazantzev, S., et al. (1987) Crystallization of 70S ribo
interactions at the tRNA wobble position crucial for accu
somes and 30S ribosomal subunits from Thermus ther!
rate reading of the genetic code, Nat. Commun., 7, 10457,
mophilus, FEBS Lett., 220, 319 322.
doi: 10.1038/ncomms10457.
55.
Harel, M., Shoham, M., Frolow, F., Eisenberg, H.,
71.
Rozov, A., Wolff, P., Grosjean, H., Yusupov, M.,
Mevarech, M., et al. (1988) Crystallization of halophilic
Yusupova, G., and Westhof, E. (2018) Tautomeric G•U
malate dehydrogenase from Halobacterium marismortui,
pairs within the molecular ribosomal grip and fidelity of
J. Mol. Biol.,
200,
609610, doi:
10.1016/0022
decoding in bacteria, Nucleic Acids Res., 46, 7425 7435,
2836(88)90547 5.
doi: 10.1093/nar/gky547.
56.
Ban, N., Freeborn, B., Nissen, P., Penczek, P., Grassucci,
72.
Voorhees, R. M., and Ramakrishnan, V. (2013) Structural
R. A., et al. (1998) A 9 Å resolution X ray crystallographic
basis of the translational elongation cycle, Annu. Rev.
map of the large ribosomal subunit, Cell, 93, 1105 1115,
Biochem., 82, 203 236, doi: 10.1146/annurev biochem
doi: 10.1016/s0092 8674(00)81455 5.
113009 092313.
БИОХИМИЯ том 86 вып. 8 2021
1136
ЮСУПОВА, ЮСУПОВ
73.
Schmeing, T. M., and Ramakrishnan, V. (2009) What
Saccharomyces cerevisiae-tRNA ribosome and subunit
recent ribosome structures have revealed about the mecha
subunit interactions, Cell, 107, 373 386, doi: 10.1016/
nism of translation, Nature, 461, 1234 1242, doi: 10.1038/
s0092 8674(01)00539 6.
nature08403.
90. Spahn, C. M., Gomez Lorenzo, M. G., Grassucci, R. A.,
74.
Rozov, A., Demeshkina, N., Westhof, E., Yusupov, M., and
Jorgensen, R., Andersen, G. R., et al. (2004) Domain
Yusupova, G. (2016) New structural insights into transla
movements of elongation factor eEF2 and the eukaryotic
tional miscoding, Trends Biochem. Sci., 41, 798 814,
80S ribosome facilitate tRNA translocation, EMBO J., 23,
doi: 10.1016/j.tibs.2016.06.001.
1008 1019, doi: 10.1038/sj.emboj.7600102.
75.
Noller, H. F., Yusupov, M. M., Yusupova, G. Z.,
91. Budkevich, T., Giesebrecht, J., Altman, R. B., Munro, J.
Baucom, A., and Cate, J. H. (2002) Translocation of tRNA
B., Mielke, T., et al. (2011) Structure and dynamics of the
during protein synthesis, FEBS Lett., 514, 11 16.
mammalian ribosomal pretranslocation complex, Mol.
76.
Melnikov, S., Ben Shem, A., Garreau de Loubresse, N.,
Cell, 44, 214 224, doi: 10.1016/j.molcel.2011.07.040.
Jenner, L., Yusupova, G., and Yusupov, M. (2012) One
92. Wilson, D. N. (2014) Ribosome targeting antibiotics and
core, two shells: bacterial and eukaryotic ribosomes, Nat.
mechanisms of bacterial resistance, Nat. Rev. Microbiol.,
Struct. Mol. Biol., 19, 560 567, doi: 10.1038/nsmb.2313.
12, 35 48, doi: 10.1038/nrmicro3155.
77.
Milligan, R. A., and Unwin, P. N. (1982) In vitro crystal
93. Hobbie, S. N., Kaiser, M., Schmidt, S., Shcherbakov, D.,
lization of ribosomes from chick embryos, J. Cell Biol., 95,
Janusic, T., et al. (2011) Genetic reconstruction of proto
648 653, doi: 10.1083/jcb.95.2.648.
zoan rRNA decoding sites provides a rationale for paro
78.
Ben Shem, A., Jenner, L., Yusupova, G., and Yusupov, M.
momycin activity against Leishmania and Trypanosoma,
(2010) Crystal structure of the eukaryotic ribosome,
PLoS Negl. Trop. Dis., 5, e1161, doi: 10.1371/journal.
Science, 330, 1203 1209, doi: 10.1126/science.1194294.
pntd.0001161.
79.
Garreau de Loubresse, N., Prokhorova, I., Holtkamp, W.,
94. Lu, W., Roongsawang, N., and Mahmud, T. (2011)
Rodnina, M. V., Yusupova, G., and Yusupov, M. (2014)
Biosynthetic studies and genetic engineering of pactamycin
Structural basis for the inhibition of the eukaryotic ribo
analogs with improved selectivity toward malarial parasites,
some, Nature, 513, 517 522, doi: 10.1038/nature13737.
Chem. Biol.,
18,
425431, doi:
10.1016/j.chembiol.
80.
Pellegrino, S., Meyer, M., Konst, Z. A., Holm, M., Voora,
2011.01.016.
V. K., et al. (2019) Understanding the role of intermolecu
95. Santagata, S., Mendillo, M. L., Tang, Y. C.,
lar interactions between lissoclimides and the eukaryotic
Subramanian, A., Perley, C. C., et al. (2013) Tight coordi
ribosome, Nucleic Acids Res.,47, 3223 3232, doi: 10.1093/
nation of protein translation and HSF1 activation supports
nar/gkz053.
the anabolic malignant state, Science, 341, 1238303,
81.
Pellegrino, S., Meyer, M., Zorbas, C., Bouchta, S. A.,
doi: 10.1126/science.1238303.
Saraf, K., et al. (2018) The Amaryllidaceae alkaloid hae
96. Bidou, L., Allamand, V., Rousset, J. P., and Namy, O.
manthamine binds the eukaryotic ribosome to repress can
(2012) Sense from nonsense: therapies for premature stop
cer cell growth, Structure, 26, 416 425.e414, doi: 10.1016/
codon diseases, Trends Mol. Med.,
18,
679688,
j.str.2018.01.009.
doi: 10.1016/j.molmed.2012.09.008.
82.
Konst, Z. A., Szklarski, A. R., Pellegrino, S., Michalak,
97. Gurel, G., Blaha, G., Steitz, T. A., and Moore, P. B. (2009)
S. E., Meyer, M., et al. (2017) Synthesis facilitates an
Structures of triacetyloleandomycin and mycalamide A
understanding of the structural basis for translation inhibi
bind to the large ribosomal subunit of Haloarcula maris!
tion by the lissoclimides, Nat. Chem., 9, 11401149,
mortui, Antimicrob. Agents Chemother., 53, 5010 5014,
doi: 10.1038/nchem.2800.
doi: 10.1128/AAC.00817 09.
83.
McClary, B., Zinshteyn, B., Meyer, M., Jouanneau, M.,
98. Gurel, G., Blaha, G., Moore, P. B., and Steitz, T. A. (2009)
Pellegrino, S., et al. (2017) Inhibition of eukaryotic trans
U2504 determines the species specificity of the A site cleft
lation by the antitumor natural product agelastatin A, Cell
antibiotics: the structures of tiamulin, homoharringtonine,
Chem. Biol., 24, 605 613 e605, doi: 10.1016/j.chembiol.
and bruceantin bound to the ribosome, J. Mol. Biol., 389,
2017.04.006.
146 156, doi: 10.1016/j.jmb.2009.04.005.
84.
Klinge, S., Voigts Hoffmann, F., Leibundgut, M.,
99. Schneider Poetsch, T., Ju, J., Eyler, D. E., Dang, Y.,
Arpagaus, S., and Ban, N. (2011) Crystal structure of the
Bhat, S., et al. (2010) Inhibition of eukaryotic translation
eukaryotic 60S ribosomal subunit in complex with initiation
elongation by cycloheximide and lactimidomycin, Nat.
factor 6, Science, 334, 941 948, doi: 10.1126/science.1211204.
Chem. Biol., 6, 209 217, doi: 10.1038/nchembio.304.
85.
Rabl, J., Leibundgut, M., Ataide, S. F., Haag, A., and
100. Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., and
Ban, N. (2011) Crystal structure of the eukaryotic 40S
Weissman, J. S. (2009) Genome wide analysis in vivo of
ribosomal subunit in complex with initiation factor 1,
translation with nucleotide resolution using ribosome pro
Science, 331, 730 736, doi: 10.1126/science.1198308.
filing, Science,
324,
218223, doi:
10.1126/science.
86.
Ban, N., Beckmann, R., Cate, J. H., Dinman, J. D.,
1168978.
Dragon, F., et al. (2014) A new system for naming riboso
101. Mailliot, J., Garreau de Loubresse, N., Yusupova, G.,
mal proteins, Curr. Opin. Struct. Biol., 24, 165169,
Meskauskas, A., Dinman, J. D., and Yusupov, M. (2016)
doi: 10.1016/j.sbi.2014.01.002.
Crystal structures of the uL3 mutant ribosome: illustration
87.
Armache, J. P., Jarasch, A., Anger, A. M., Villa, E.,
of the importance of ribosomal proteins for translation effi
Becker, T., et al. (2010) Cryo EM structure and rRNA
ciency, J. Mol. Biol., 428, 2195 2202, doi: 10.1016/j.jmb.
model of a translating eukaryotic 80S ribosome at 5.5 Å
2016.02.013.
resolution, Proc. Natl. Acad. Sci. USA, 107, 19748 19753,
102. Shulman, E., Belakhov, V., Wei, G., Kendall, A., Meyron
doi: 10.1073/pnas.1009999107.
Holtz, E. G., et al. (2014) Designer aminoglycosides that
88.
Armache, J. P., Jarasch, A., Anger, A. M., Villa, E.,
selectively inhibit cytoplasmic rather than mitochondrial
Becker, T., et al. (2010) Localization of eukaryote specific
ribosomes show decreased ototoxicity: a strategy for the
ribosomal proteins in a 5.5 Å cryo EM map of the 80S
treatment of genetic diseases, J. Biol. Chem., 289, 2318
eukaryotic ribosome, Proc. Natl. Acad. Sci. USA, 107,
2330, doi: 10.1074/jbc.M113.533588.
19754 19759, doi: 10.1073/pnas.1010005107.
103. Prokhorova, I., Altman, R. B., Djumagulov, M., Shrestha,
89.
Spahn, C. M., Beckmann, R., Eswar, N., Penczek, P. A.,
J. P., Urzhumtsev, A., et al. (2017) Aminoglycoside inter
Sali, A., et al. (2001) Structure of the 80S ribosome from
actions and impacts on the eukaryotic ribosome, Proc.
БИОХИМИЯ том 86 вып. 8 2021
КРИСТАЛЛИЧЕСКАЯ СТРУКТУРА РИБОСОМЫ
1137
Natl. Acad. Sci. USA, 114, E10899 E10908, doi: 10.1073/
Saccharomyces cerevisiae, Biochemistry, 21, 3181 3187,
pnas.1715501114.
doi: 10.1021/bi00256a023.
104. Dolz, H., Vazquez, D., and Jimenez, A.
(1982)
105. Khatter, H., Myasnikov, A. G., Natchiar, S. K., and
Quantitation of the specific interaction of [14a 3H]crypto
Klaholz, B. P. (2015) Structure of the human 80S ribo
pleurine with 80S and 40S ribosomal species from the yeast
some, Nature, 520, 640 645, doi: 10.1038/nature14427.
THE PATH FOR DETERMINATION OF THE ATOMIC STRUCTURES
OF PROKARYOTIC AND EUKARYOTIC RIBOSOMES
Review
G. Yusupova1 and M. Yusupov1,2*
1 Institut de Génétique et de Biologie Moléculaire et Cellulaire (IGBMC), INSERM U964,
CNRS UMR7104, Université de Strasbourg, 67404 Illkirch, France
2 Institute of Fundamental Medicine and Biology, Kazan Federal University,
420008 Kazan, Russia; e!mail: marat@igbmc.fr
The first crystal structures of prokaryotic and eukaryotic ribosomes were solved by our group and based on the knowl
edge accumulated over decades, starting with the first electron microscopic images of the ribosome obtained by
J. Pallade in 1955. In 1983, A. Spirin, director of the Protein Institute of the USSR Academy of Sciences, initiated
the first studies aimed at solving the structure of ribosomes using X ray structural analysis. In 1999 our group in col
laboration with H. Noller published the first crystal structure of an entire bacterial ribosome in complex with major
functional ligands, including messenger RNA and three transport RNAs at the A, P, and E sites. In 2011 our labora
tory published the first atomic structure of the eukaryotic ribosome solved by X ray diffraction analysis. The conser
vative structure of the main functional areas of ribosome, such as decoding and peptidyl transferase centers, was con
firmed to be true, while eukaryotic specific elements were also described. Using X ray structural analysis we investi
gated the general principles of protein biosynthesis inhibition on eukaryotic ribosomes along with the mechanisms of
antibiotic resistance. Structural differences between bacterial and eukaryotic ribosomes that determine functional dif
ferences in inhibition were shown. These and subsequent structures of the functioning ribosome demonstrated for the
first time at the atomic level the ribosome binding to the messenger and transport RNAs that deliver amino acids to
the growing peptide, representing a first step towards understanding how ribosome structure ultimately determines its
functions.
Keywords: ribosome, structure, X ray, cryo EM
БИОХИМИЯ том 86 вып. 8 2021