БИОХИМИЯ, 2021, том 86, вып. 8, с. 1138 - 1148
УДК 577.21
КОНТРОЛЬ КАЧЕСТВА У БАКТЕРИЙ,
АССОЦИИРОВАННЫЙ С РИБОСОМАМИ
Обзор
© 2021
М.С. Светлов
Center for Biomolecular Sciences, Department of Pharmaceutical Sciences,
University of Illinois at Chicago, 60607 Chicago, Illinois, USA; e mail: msvet2@uic.edu
Поступила в редакцию 14.05.2021
После доработки 14.05.2021
Принята к публикации 14.05.2021
Трансляция генетической информации в белки, выполняемая рибосомой, является ключевым процессом,
происходящим в клетках всех живых организмов. Трансляция обычно происходит без затруднений, но, к
сожалению, нежелательные события могут привести к остановке транслирующих рибосом. Чтобы спасти
эти дефектные остановившиеся рибосомы, бактериальные клетки используют три хорошо охарактеризо%
ванные системы контроля качества: тмРНК, ArfA и ArfB. Недавно в клетках Bacillus subtilis был обнаружен
дополнительный механизм спасения рибосом. В отличие от «канонических» систем, нацеленных на 70S%
рибосомы, этот механизм сначала разбирает полную рибосому на малую (30S) и большую (50S) субъедини%
цы, а затем очищает образовавшуюся большую субъединицу от неполного полипептида. В настоящей рабо%
те я расскажу о последних микробиологических, биохимических и структурных данных, касающихся функ%
ционирования этой новой системы спасения.
КЛЮЧЕВЫЕ СЛОВА: трансляция, остановка рибосом, контроль качества, образование полиаланинового
хвостового участка.
DOI: 10.31857/S0320972521080066
КАНОНИЧЕСКИЕ МЕХАНИЗМЫ
аминоацил%тРНК в A%сайт рибосомы, за кото%
СПАСЕНИЯ РИБОСОМ У БАКТЕРИЙ
рой следует её реакция с пептидил%тРНК, рас%
положенной в P%сайте, и заканчивается транс%
Рибосомы осуществляют синтез белка во
локацией вновь образованной пептидил%тРНК
всех живых клетках с помощью процесса, извест%
из A% в P%сайт. Полный цикл элонгации и его ос%
ного как трансляция (рис. 1). Рибосомы начина%
новные участники показаны на рис. 1, a и c.
ют синтез белка с распознавания стартового ко%
И наконец, рибосомы прекращают трансляцию
дона (обычно это кодон AUG и реже - кодо%
и высвобождают зрелый белок, когда они дости%
ны GUG или UUG) на молекуле информацион%
гают один из трёх стоп%кодонов (UAA, UAG
ной РНК (мРНК) (рис. 1, a). За этой стадией
или UGA) (рис. 1, a).
инициации следует несколько циклов элонга%
У активно растущих бактерий катализируе%
ции, в процессе которых рибосомы перемеща%
мая рибосомами полимеризация полипептид%
ются вдоль открытой рамки считывания
ных цепей происходит со скоростью око%
мРНК (сегмент мРНК, кодирующей специфи%
ло 15 аминокислот в секунду [1, 2]. Примеча%
ческий белок), считывая её нуклеотидную пос%
тельно, что эта скорость непостоянна, и быстрое
ледовательность и переводя (транслируя) её в
передвижение рибосомы вдоль мРНК может
аминокислотную последовательность синтези%
прерываться паузами. Во многих случаях паузы
руемого белка (рис. 1, c). Каждый цикл элонга%
рибосом во время процесса трансляции являют%
ции начинается с доставки соответствующей
ся нормальным и необходимым явлением, так
как они могут играть важную роль, например, в
Принятые сокращения: CAT%хвост - C%концевой
ко%трансляционном сворачивании и химичес%
хвостовой участок из остатков аланина и треонина; EF -
кой модификации белков или их сортировке по
фактор элонгации; IF - фактор инициации; NEMF - фак%
соответствующим компартментам [3, 4]. С дру%
тор%посредник ядерного экспорта; PTC - пептидил%транс% гой стороны, рибосомные паузы могут быть
феразный центр; RF - фактор терминации; RRF - фактор
вредными для клетки, если они вызваны транс%
рециклинга рибосомы; RQC - система контроля качества,
ассоциированная с рибосомами (Ribosome%associated
ляцией повреждённых мРНК или полимериза%
Quality Control); тмРНК - транспортная%матричная РНК.
цией проблемных аминокислотных последова%
1138
БАКТЕРИАЛЬНАЯ СИСТЕМА RQC
1139
тельностей в синтезируемых белках. Клетки ис%
ляционные паузы трудно или даже невозможно
пользуют несколько дополнительных факторов
устранить, и транслирующие рибосомы могут
трансляции, чтобы смягчить влияние этих не%
«застопориться» на специфическом месте в мо%
желательных пауз. Например, бактериальный
лекуле мРНК. Подобное явление особенно уси%
фактор элонгации P (EF%P) и его гомолог в
ливается при различных стрессах, таких как не%
клетках архей и эукариот, eIF5A, нивелируют
достаток аминокислот [9], изменение темпера%
трансляционные паузы, вызванные последова%
туры [10] и обработка клеток ингибиторами,
тельностями полипролина в синтезируемых по%
воздействующими на рибосомы [11]. В этих ус%
липептидах [5, 6]. С этой целью факторы стаби%
ловиях рибосомы могут остановиться на 3′%кон%
лизируют пептидил%тРНК в продуктивной кон%
це укороченных мРНК, лишённых стоп%кодона.
формации, способной вступать в реакцию с Pro%
Такие no%stop мРНК обычно появляются в клет%
тРНКPro, расположенной в А%сайте рибосо%
ке в результате преждевременного прекращения
мы [7]. Другой фактор элонгации трансляции,
транскрипции или расщепления полноразмер%
EF4 (LepA), как предполагается, способствует
ных транскриптов мРНК нуклеазой. Остановка
«смягчению» трансляциионных пауз, возника%
транслирующих рибосом может также происхо%
ющих при неблагоприятных условиях, таких как
дить и на интактных мРНК, приводя к накопле%
высокие концентрации магния или низкая тем%
нию no%stop или no%go комплексов рибосом.
пература [8]. Однако в некоторых случаях транс%
No%stop комплексы (которые по структуре иден%
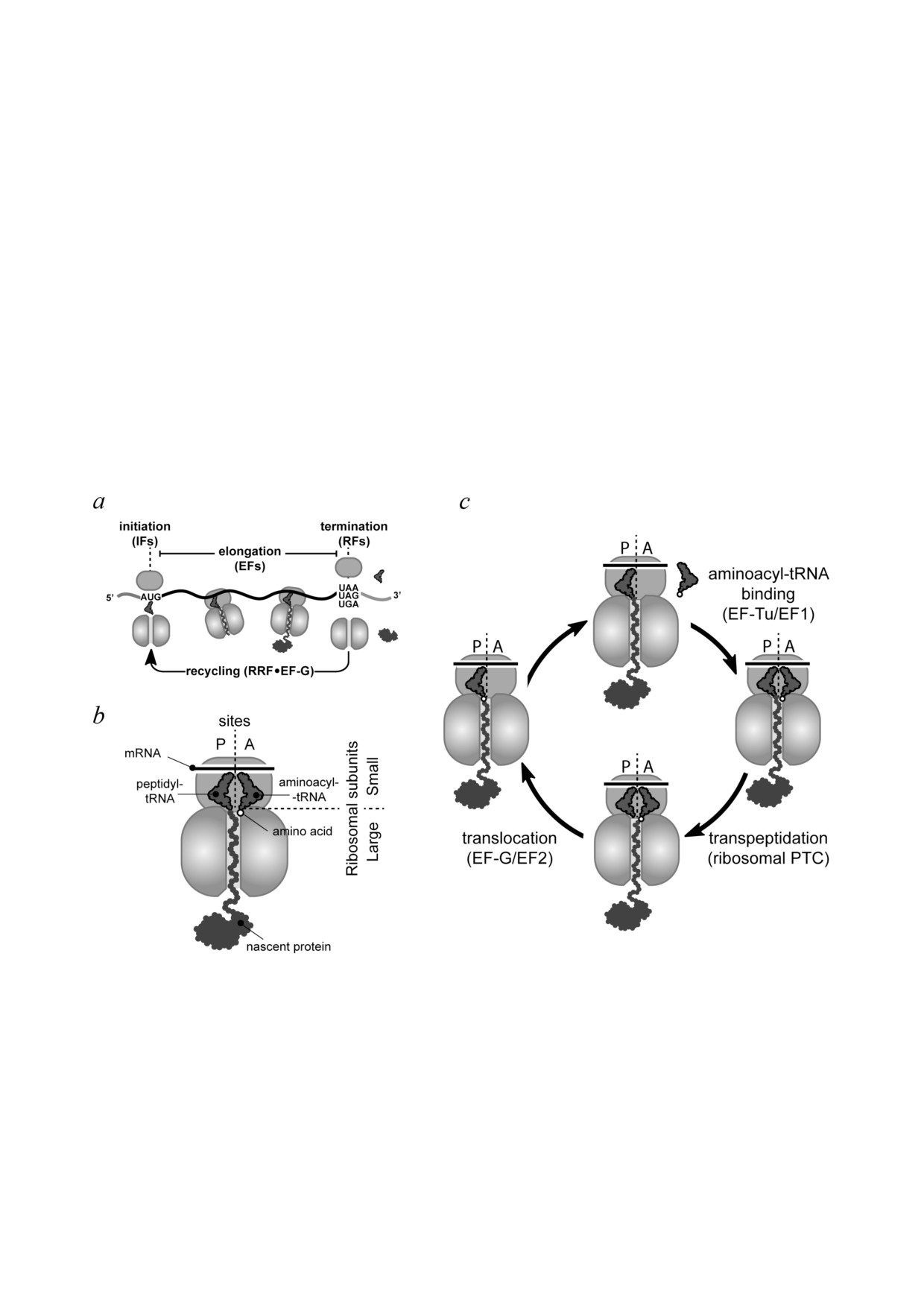
Рис. 1. Трансляционный цикл у бактерий. a - С помощью факторов инициации (IF) малая (30S) и большая (50S) субъ%
единицы соединяются с образованием полной 70S%рибосомы на стартовой позиции на мРНК (кодоны AUG, GUG
или UUG). Факторы элонгации (EF) помогают рибосоме полимеризовать аминокислоты в белок на стадии элонгации.
Полноразмерный белок с помощью факторов терминации (RF) высвобождается из рибосомы, когда она достигает стоп%
кодона (UAA, UAG или UGA). После терминации факторы рециклинга (RRF и EF%G у бактерий) разбирают рибосому на
субъединицы, чтобы она могла инициировать новый раунд трансляции. b - Схематическое изображение элонгирующей
рибосомы на стадии пре%транспептидации. Эта рибосома несёт транслируемую мРНК (расположена на малой субъеди%
нице), аминоацил%тРНК (находится в А%сайте рибосомы) и пептидил%тРНК (находится в P%сайте). c - Обзор элонгаци%
онного цикла. Цикл состоит из трёх последовательных этапов: 1) EF%Tu%опосредованное связывание аминоацил%тРНК,
комплементарной кодону мРНК, с А%сайтом рибосомы; 2) реакция аминоацил%тРНК с P%сайтовой пептидил%тРНК, ка%
тализируемая рибосомным пептидил%трансферазным центром (PTC) и 3) EF%G%опосредованная транслокация вновь об%
разованной пептидил%тРНК, удлинённой на один аминокислотный остаток, из A%сайта в P%сайт
БИОХИМИЯ том 86 вып. 8 2021
1140
СВЕТЛОВ
тичны комплексам, образующимся на укоро%
ния (рис. 2, a). Механизм действия этих систем,
ченных мРНК) накапливаются в том случае,
включающих SmpB•тмРНК%опосредованную
когда рибосомы ошибочно прочитывают стоп%
транс%трансляцию, ArfA и ArfB, были ранее под%
кодоны как значащие (то есть кодирующие ами%
робно рассмотрены [12-14], и я лишь кратко
нокислоту) и затем застревают на 3′%концах не%
опишу их ниже.
повреждённых мРНК. Отсутствие кодона в A%
Транс%трансляция - это основной механизм,
сайте рибосомы является отличительной чертой
который спасает no%stop рибосомы в бактери%
этих комплексов, распознаваемых «каноничес%
ях [15]. Этот механизм основан на действии двух
кими» бактериальными системами спасения.
внутриклеточных факторов: небольшого бел%
В отличие от no%stop комплексов no%go рибосо%
ка SmpB и транспортной%матричной РНК
мы останавливаются внутри кодирующих час%
(тмРНК), гибрида тРНК, содержащей остаток
тей транслируемых мРНК. «Канонические»
аланина, и мРНК, кодирующей специальный
системы не способны освободить такие рибосо%
короткий пептид, служащий сигналом для про%
мы из%за того, что в их мРНК%каналах находят%
теолитической деградации (дегрон) (рис. 2, b).
ся цепи мРНК. В качестве первого шага к раз%
SmpB и тмРНК образуют комплекс, который с
борке no%go комплексов, бактериальный ток%
помощью фактора EF%Tu доставляется на no%
син RelE (эндонуклеаза, активируемая амино%
stop рибосому [16]. После доставки на рибосому
кислотным голоданием) связывается с ними и
SmpB взаимодействует с малой субъединицей и
вносит разрыв в цепь мРНК, находящуюся в А%
«подтверждает», что канал для мРНК и A%сайт
сайте рибосомы. В результате расщепления эн%
пусты, в то время как тмРНК, имитируя амино%
донуклеазой RelE no%go комплексы превраща%
ацил%тРНК, связывается с рибосомой и разме%
ются в no%stop остановленные рибосомы, кото%
щает свой CCA%конец, аминоацилированный
рые могут служить мишенью для направленного
аланином, в пептидил%трансферазном центре
действия
«канонических» систем спасе% рибосомы (PTC), где она вступает в реакцию с
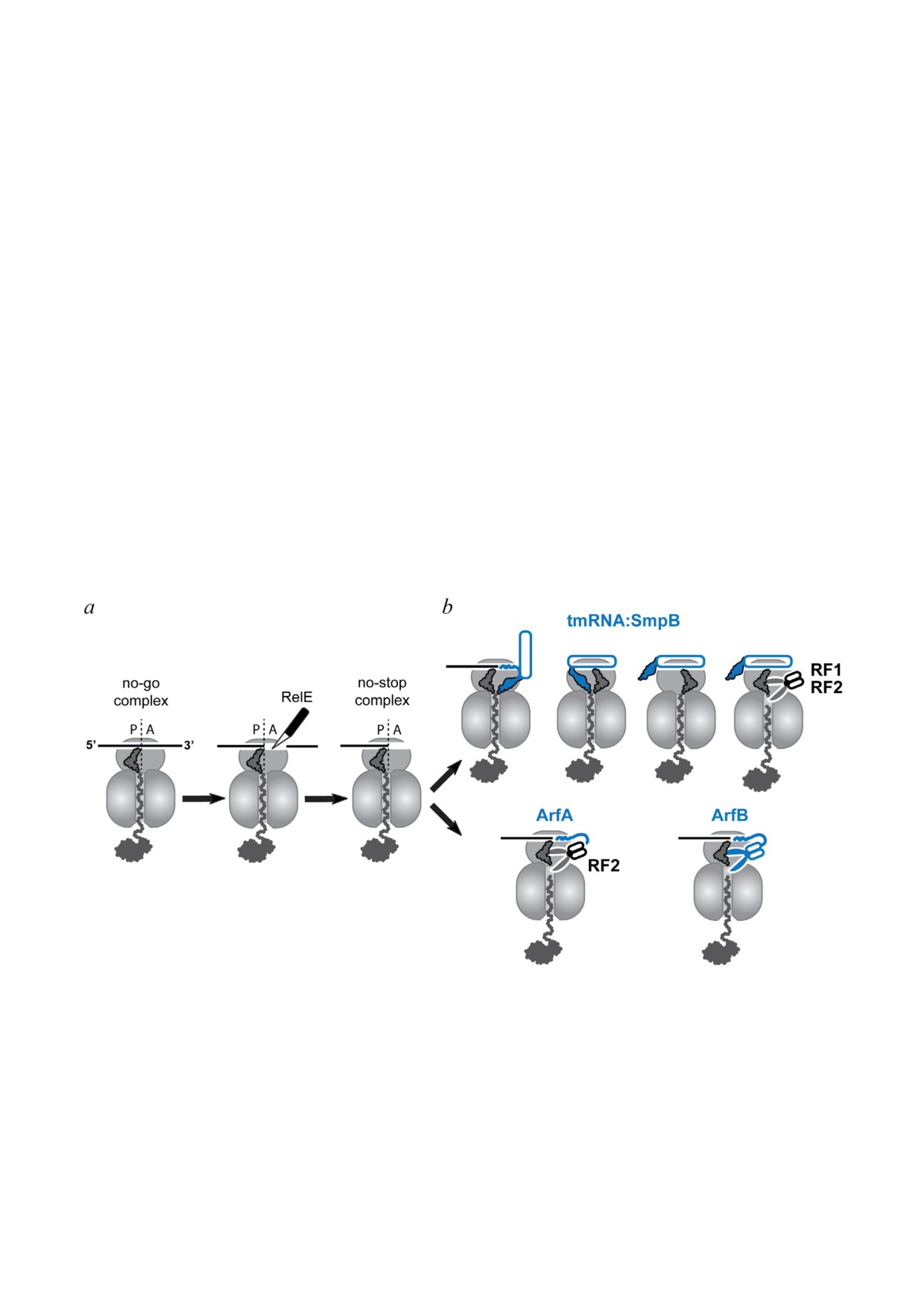
Рис. 2. Освобождение остановившихся рибосом каноническими бактериальными системами спасения. a - Остановка
движения рибосом может привести к образованию no%stop или no%go комплексов. Канонические системы спасения могут
непосредственно распознавать только no%stop рибосомы с вакантными (пустыми) мРНК%каналами, в то время как для ре%
циклизации no%go рибосом необходимо сперва их перевести в no%stop рибосомы с помощью эндонуклеазы RelE. b - Об%
разовавшиеся no%stop рибосомы высвобождаются в ходе процесса транс%трансляции, опосредованной транспортной%мат%
ричной РНК (тмРНК) в комплексе с белком SmpB. Трансляция мРНК%подобного сегмента тмРНК прекращается в ре%
зультате действия канонических факторов терминации, RF1 или RF2. Если действие этой системы контроля качества зат%
руднено, пептидил%тРНК может быть гидролизована фактором RF2, рекрутированным белком ArfA, или независимым от
стоп%кодонов фактором терминации ArfB. Все три канонические бактериальные системы спасения, SmpB•тмРНК, ArfA
и ArfB (показано синим цветом), распознают no%stop рибосомы, содержащие пустой или частично заполненный мРНК
канал в малой субъединице
БИОХИМИЯ том 86 вып. 8 2021
БАКТЕРИАЛЬНАЯ СИСТЕМА RQC
1141
пептидил%тРНК. После переноса пептида на
ОСВОБОЖДЕНИЕ ОСТАНОВИВШИХСЯ
тмРНК остановленная рибосома начинает
РИБОСОМ У ЭУКАРИОТ
трансляцию сегмента тмРНК, кодирующего
дегрон. Прекращение трансляции тмРНК на
В то время как вышеупомянутые бактериаль%
стоп%кодоне, расположенном в конце кодирую%
ные механизмы спасения нацелены на интакт%
щей последовательности, приводит к рецикли%
ные остановившиеся 70S%рибосомы, длитель%
зации рибосомы по стандартному механизму, а
ные трансляционные паузы у эукариот вызыва%
высвободившийся пептид, несущий дегрон на
ют разборку рибосомы 80S на составляющие её
C%конце, подвергается протеолитической дегра%
субъединицы (малая, 40S, и большая, 60S) с
дации.
последующей их независимой рециклизацией
Хотя компоненты системы транс%трансля%
для участия в следующих раундах трансля%
ции были обнаружены во всех секвенированных
ции [13]. Модель освобождения остановленной
видах бактерий, нарушение работы этой систе%
80S%рибосомы представлена на рис. 3, а. Раз%
мы обычно не является летальным для клет%
борка рибосомы требует согласованного
ки [17] благодаря существованию у некоторых
действия трёх факторов: Hbs1L (у дрож%
бактерий двух вспомогательных механизмов
жей Hbs1p), который связывается в непосред%
спасения, ArfA и ArfB. Эти системы поддержи%
ственной близости от незанятого мРНК канала,
вают рост бактерий, когда активность комплек%
eRF1 паралога, Pelota (у дрожжей Dom34p), ко%
са SmpB•тмРНК нарушена или недостаточна.
торый занимает пустой A%сайт «застопоренной»
ArfA - это небольшой белок, который связыва%
рибосомы, и АТФазы ABCE1 (у дрожжей Rli1p),
ется с A%сайтом в малой субъединице рибосомы,
которая рекрутируется на рибосому с по%
когда он не содержит кодона мРНК, а затем рек%
мощью Pelota после диссоциации Hbs1L, чтобы
рутирует фактор терминации 2 (RF2) на засто%
разобрать её на субъединицы (рис. 3, а, стадия
поренную рибосому для гидролиза пептидил%
разборки). После разборки рибосомы мРНК
тРНК [18, 19] (рис. 2, b). Интересно, что такая
высвобождается из 40S%субъединицы и подвер%
избирательность в отношении RF2 не присуща
гается деградации 5′-3′%экзонуклеазой Xrn1 и
всем бактериям. Так, у Francisella tularensis не%
3′-5′%экзосомой [23], а затем свободная от
большой аналогичный ArfA белок, ArfT, может
мРНК субъединица рециклизуется с участи%
рекрутировать как RF1, так и RF2 на no%stop ри%
ем ABCE1 [24]. Одновременно, поскольку у
босомы [20]. ArfB (ранее известный как YaeJ) яв%
Pelota/Dom34p отсутствует пептидил%тРНК гид%
ляется гомологом факторов терминации (RF),
ролазная активность, 60S%субъединица диссо%
чья пептидил%тРНК гидролазная активность не
циирует в виде комплекса с пептидил%РНК.
зависит от стоп%кодонов [21, 22] (рис. 2, b). По%
Дальнейшая судьба комплекса 60S•пептидил%
добно ArfA, ArfB связывается только с рибосо%
тРНК зависит от длины пептида, конъюгиро%
мой, расположенной на 3′%конце мРНК и при
ванного с тРНК. Если пептид относительно ко%
отсутствии кодона мРНК в A%сайте. Интересно,
роткий, то пептидил%тРНК может спонтанно
что обе резервные системы спасения отличают
диссоциировать с большой субъединицы [25].
остановленные от активно транслирующих
Напротив, если же конъюгированный с тРНК
рибосом, подобно тому как это делает комплекс
пептид достаточно длинный, чтобы установить
SmpB•тмРНК: они связываются с пустым кана%
стабильные взаимодействия внутри туннеля вы%
лом для входа мРНК на малой субъединице ри%
хода пептида или свернуться за пределами рибо%
босомы, в норме занятым транслируемой
сомы, пептидил%тРНК остаётся прочно связан%
мРНК, чтобы инициировать освобождение ри%
ной с 60S%субъединицей. В этой ситуации боль%
босомы. Учитывая существование этих резерв%
шая субъединица рибосомы, заблокированная
ных систем спасения у многих бактерий, недав%
пептидил%тРНК, распознаётся и «спасается»
нее открытие совершенно другого механизма,
белками специального пути, называемо%
освобождающего остановившиеся рибосомы у
го RQC [26] (рис. 3). Путь RQC запускается се%
Bacillus subtilis, стало неожиданным и приятным
лективным связыванием фактора%посредника
сюрпризом. Ещё более поразительным является
ядерного экспорта (NEMF/дрожжевой Rqc2) с
тот факт, что этот механизм спасения напоми%
интерфейсом между 60S%субъединицей и пепти%
нает систему контроля качества, связанную с
дил%тРНК, где этот белок играет по крайней ме%
рибосомами (RQC), которая существует у эука%
ре три роли: 1) он предотвращает соединение
риот. Чтобы понять сходство бактериальных и
40S%субъединицы с комплексом 60S•пептидил%
эукариотических систем RQC, я кратко опишу,
тРНК; 2) он рекрутирует E3 убиквитинлигазу
как застопорившиеся в процессе трансляции
Listerin (у дрожжей Ltn1p) для убиквитинилиро%
рибосомы освобождаются в эукариотических
вания синтезируемой полипептидной цепи;
клетках.
3) он рекрутирует Ala%tRNAAla и Thr%tRNAThr на
БИОХИМИЯ том 86 вып. 8 2021
1142
СВЕТЛОВ
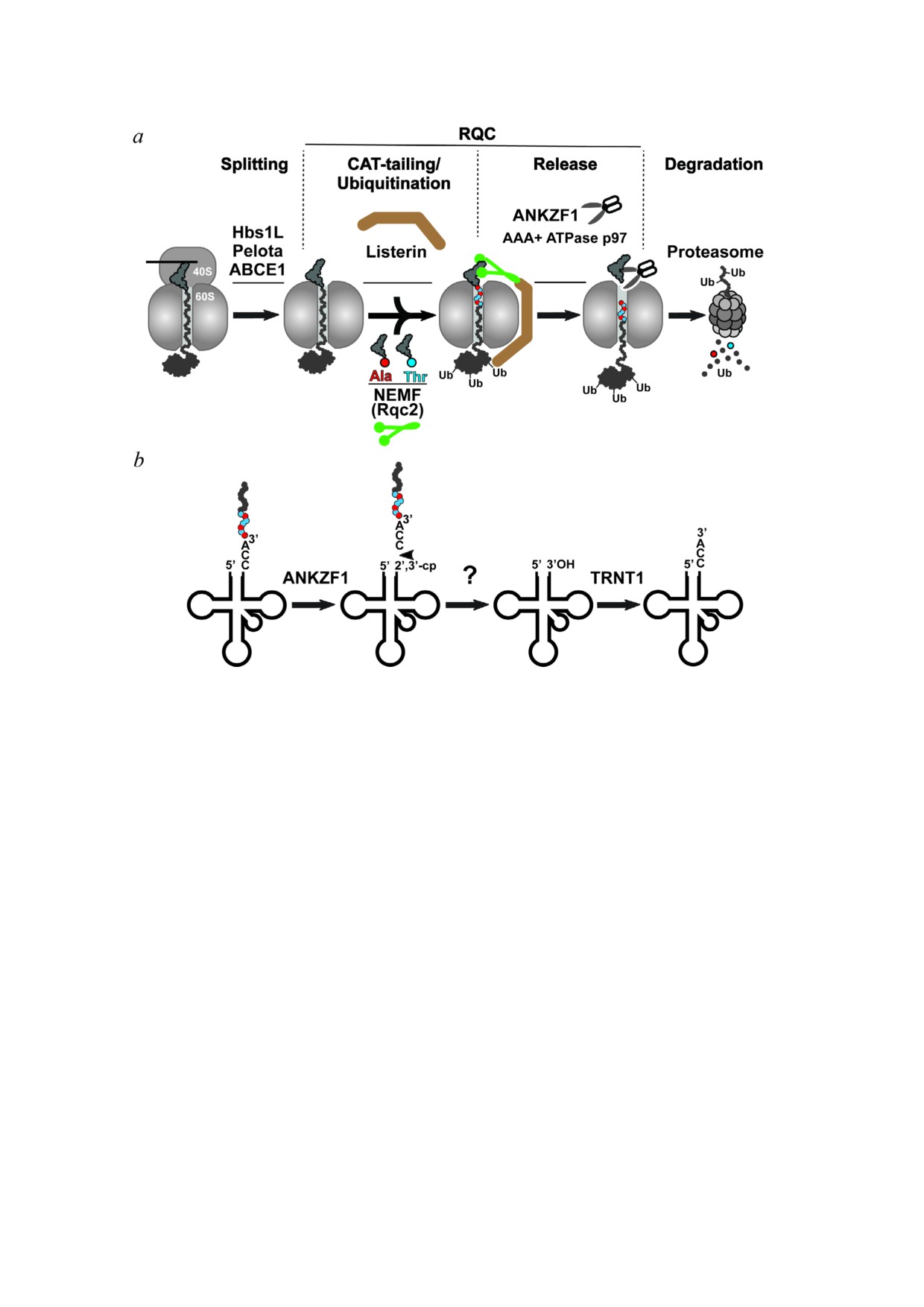
Рис. 3. Эукариотический путь контроля качества, связанный с эукариотическими рибосомами. a - Контролю качества,
связанному с рибосомами (RQC), предшествует разборка остановившейся 80S%рибосомы с помощью трёх белков: Pelota,
Hbs1L и ABCE1. Затем NEMF (зелёный) и Listerin (коричневый) связываются с образовавшимся комплексом 60S•пепти%
дил%тРНК и модифицируют неполный пептид. NEMF удлиняет C%конец образующегося пептида за счёт остатков алани%
на/треонина (CAT%хвост), чтобы экспонировать остатки лизина, которые, в противном случае, были бы скрыты туннелем
выхода из рибосомы, для катализируемого Listerin’ом убиквитинилирования. Образующийся пептид высвобождается из
60S%субъединицы с помощью эндонуклеазы ANKZF1, гидролизующей пептидил%тРНК, и доставляется с помощью
AAA+ АТФазы p97 в протеасому для последующей деградации. b - Модель рециклизации тРНК после ANKZF1%опосре%
дованного высвобождения образующегося пептида. ANKZF1 вырезает 3′%CCA%конец пептидил%тРНК, вызывая высво%
бождение конъюгата CCA-пептид, который подвергается деградации протеасомой, и образование укороченной тРНК,
несущей 2′,3′%циклический фосфат. После удаления циклического фосфата с помощью ELAC1 фермент TRNT1 рецикли%
зует молекулу тРНК, добавляя нуклеотид 3′%CCA на её 3′%конец
застопоренную рибосому и способствует мРНК%
(ANKZF1/дрожжевой Vms1p) [28] (рис. 3, а, ста%
независимому добавлению остатков аланина и
дия высвобождения). Следует отметить, что, в
треонина к C%концу неполного пептида (C%кон%
отличие от высвобождения пептида во время
цевое удлинение, называемое CAT%хвостом),
обычной терминации трансляции, когда кано%
приводящее, по%видимому, к экспозиции остат%
нические факторы терминации провоцируют
ков лизина в его цепи (которые, в противном
PTC рибосомы на осуществление гидролиза
случае, были бы скрыты туннелем выхода 60S%
сложноэфирной связи между тРНК и пептидом
субъединицы) для Listerin%опосредованного
в пептидил%тРНК, ANKZF1 избирательно от%
убиквитинирования (рис. 3, а, CAT%хвост/ста%
щепляет его концевой 3′%CCA%нуклеотид [29].
дия убиквитинилирования) [27]. Образующийся
Образующийся при этом конъюгат CCA и пеп%
убиквитин%модифицированный пептид затем
тида экстрагируется из комплексов RQC с по%
отщепляется от тРНК с помощью эндонукле%
мощью AAA+ АТФазы p97 (Cdc48p у дрожжей)
азы, называемой белок 1, содержащий анкири%
и её кофакторов, UFD1 и NPLOC4 (у дрожжей
новый повтор и домен цинкового пальца
это Ufd1p и Npl4p), и затем доставляется ими в
БИОХИМИЯ том 86 вып. 8 2021
БАКТЕРИАЛЬНАЯ СИСТЕМА RQC
1143
протеасому для последующей деградации
что RqcH может избирательно связывать и дос%
(рис. 3, стадия деградации). Одновременно уко%
тавлять Ala%тРНКAla (UGC) на 50S%субъединицу
роченная тРНК, генерируемая ANKZF1, диссо%
рибосомы, несущую пептидил%тРНК (разборка
циирует от 60S%субъединицы и рециклизуется
остановившейся 70S%рибосомы, вероятно, осу%
через двухэтапный процесс, который включает
ществляется неизвестным фактором спасения),
удаление 2′,3′%циклического фосфата гомоло%
для облегчения присоединения полиаланиново%
гом РНКазы Z млекопитающих, ELAC1 [30], с
го хвоста к C%концу неполного пептида. Эта мо%
последующим восстановлением 3′%CCA%конца
дификация пептида напоминает добавление
ферментом TRNT1, добавляющим CCA [29]
CAT%хвоста, опосредованное NEMF/Rqc2 у эу%
(рис. 3, b). В завершение, большая субъединица
кариот. Однако в отличие от eRQC, где CAT%
рибосомы, свободная от тРНК и пептида, может
хвост косвенно вовлечён в деградацию пептида,
принять участие в новых раундах трансляции.
способствуя его убиквитинилированию, C%кон%
цевая модификация полиаланинами у бактерий
непосредственно действует как сигнал деграда%
RQC)ПОДОБНАЯ СИСТЕМА
ции пептида, распознаваемый протеазой ClpXP
СПАСЕНИЯ У БАКТЕРИЙ
(рис. 4, a).
Структурные исследования проливают свет
До недавнего времени считалось, что меха%
на некоторые аспекты механизма модификации
низм RQC действует исключительно у эукари%
неполных пептидов, связанных с 50S%субчасти%
от (eRQC), в то время как освобождение остано%
цей, полиаланиновым дегроном у бактерий [36,
вившихся рибосом у бактерий достигается ис%
37] (рис. 4, b). Полученные с помощью криоэлек%
ключительно с помощью «канонических» спе%
тронной микроскопии (Крио%ЭМ) структуры
цифичных для бактерий систем, таких как
комплексов RQC B. subtilis показали, что прихо%
SmpB•тмРНК, ArfA/ArfT и ArfB. Этот вывод
дящая Ala%тРНК формирует многочисленные
был основан на нескольких фактах, включая
взаимодействия со связанным с рибосомой бел%
следующие:
ком RqcH. В то время как D% и T%петли Ala%
1. У многих бактерий комбинированное
тРНКAla взаимодействуют с положительно заря%
удаление «канонических» систем спасения в це%
женным участком на поверхности RqcH, два
лом является летальным [31, 32], что предпола%
нуклеотида антикодона (G35 и C36) участвуют в
гает отсутствие каких%либо других резервных
формировании специфической сети взаимодей%
систем спасения у изученных видов.
ствий с несколькими остатками RqcH (рис. 4, b).
2. У бактерий нет очевидных гомологов
Такое RqcH%опосредованное декодирование
ключевых факторов eRQC, таких как разбираю%
тРНК определяет высокую избирательность
щая триада Pelota%Hbs1%ABCE1, убиквитинли%
комплексов RQC в отношении Ala%тРНКAla.
газа E3 Ltn1 и 60S%зависимая пептидил%тРНК
Структурные, биохимические и генетические
эндонуклеаза ANKZF1/Vms1p.
данные дополнительно идентифицировали го%
3. Активность системы eRQC тесно связана
молог Hsp15, YabO, в качестве важного партне%
с убиквитинированием неполного пептида на
ра RqcH в процессе модификации пептида по%
застопоренной 60S%субъединице - модифика%
лиаланином (рис. 4, b и c). Этот небольшой бе%
цией, которая не распространена среди бакте%
лок стабилизирует колеблющуюся пептидил%
рий.
РНК в P%сайте 50S%субъединицы (и поэтому бе%
Тем не менее, хотя большинство факто%
лок YabO был переименован в RqcP) в «продук%
ров RQC являются специфическими для эука%
тивной» конформации, необходимой для её ре%
риот, гомологи NEMF/Rqc2 обнаружены у ар%
акции с Ala%тРНКAla, доставляемой бел%
хей, а также у некоторых видов бактерий, где
ком RqcH в A%сайт рибосомы. Наконец, струк%
они известны как члены семейства факторов ви%
туры RQC%комплексов, полученные на различ%
рулентности фибронектин%связывающего бел%
ных стадиях прикрепления полиаланинового
ка A (FbpA) [33]. Было высказано предположе%
дегрона, позволили предложить модель того,
ние, что белки FbpA, продуцируемые патоген%
как эта реакция протекает на рибосоме
ными бактериями, связываются с фибронекти%
(рис. 4, c). Согласно этой модели, после расщеп%
ном, присутствующим во внеклеточном мат%
ления застопоренной рибосомы синтезируемый
риксе, что способствует их адгезии на поверх%
пептид RqcP%стабилизированной пептидил%
ности клеток организма%хозяина [34]. Однако
тРНК переносится на Ala%тРНКAla, рекрутируе%
Lytvynenko et al. [35] недавно показали, что го%
мую фактором RqcH, в A%сайт, удлиняя C%конец
молог NEMF/Rqc2 из B. subtilis, RqcH, прини%
пептида на один остаток аланина. Диссоциа%
мает непосредственное участие в освобождении
ция RqcP из комплекса RQC позволяет деаци%
остановившихся рибосом. Было обнаружено,
лированной тРНК, находящейся в P%сайте, пе%
БИОХИМИЯ том 86 вып. 8 2021
1144
СВЕТЛОВ
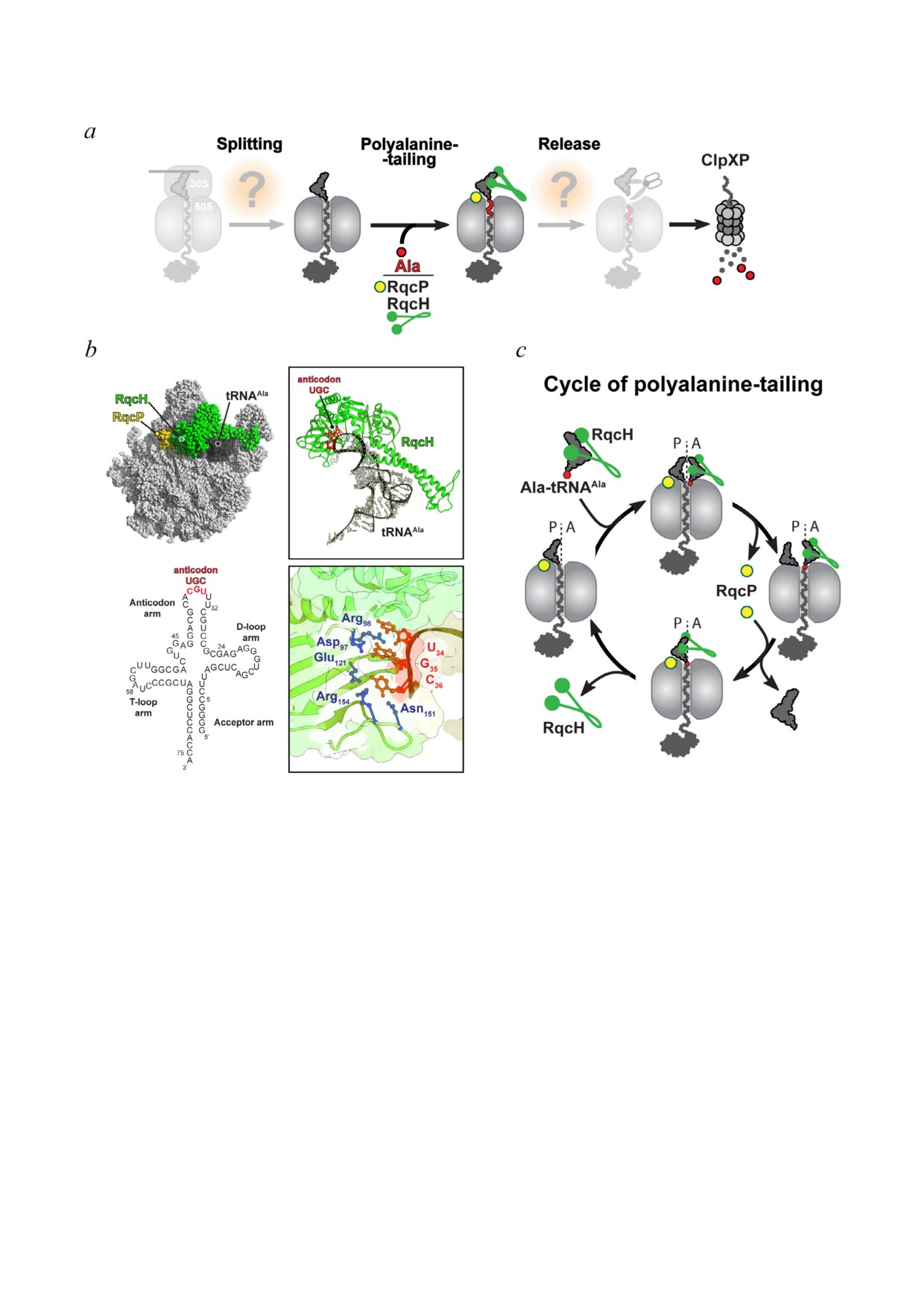
Рис. 4. Бактериальный RQC%подобный путь. a - В целом, бактериальный RQC%подобный путь состоит из тех же основ%
ных стадий, что и аналогичный путь у эукариот (см. рис. 3). Однако единственной хорошо изученной стадией этого меха%
низма является добавление полиаланинового дегрона к С%концу неполного пептида, связанного с 50S%субъединицей.
У B. subtilis эта стадия осуществляется при помощи RqcH, являющегося гомологом NEMF/Rqc2, и небольшого бел%
ка RqcP. b - Крио%ЭМ%структура бактериального комплекса RQC из B. subtilis (PDB: 7AS8). Комплекс RQC состоит из
50S%субъединицы (светло%серый), тРНКAla (темно%серый, её вторичная структура показана под крио%ЭМ%структурой),
RqcH (зелёный) и RqcP (жёлтый). В этом комплексе остатки Asp97, Arg98, Glu121, Asn151 и Arg154 (показаны си%
ним) RqcH распознают нуклеотиды G35 и C36 (показаны красным) антикодона UGC в тРНКAla. c - Цикл прикрепления
полиаланинового дегрона у B. subtilis (см. подробности в тексте статьи)
реместиться в E%сайт, и вновь образованная
нинового дегрона к С%концу неполного пептида.
пептидил%тРНК принимает A/P%подобную гиб%
Наконец, модифицированный пептид высво%
ридную конформацию. Последующая спонтан%
бождается из 50S%субъединицы другим ещё не%
ная диссоциация деацилированной тРНК из E%
известным фактором в цитозоль, где он расщеп%
сайта позволяет RqcP снова связаться с комп%
ляется протеазой ClpXP (рис. 4, a).
лексом, после чего происходит перемещение
Открытие RqcH% и RqcP%опосредованной
пептидил%Ala%тРНКAla из A/P%сайта в P%сайт и
модификации синтезируемых пептидов поли%
завершение цикла элонгации. Этот цикл элон%
аланином выявило существование RQC%подоб%
гации с добавлением аланина повторяется не%
ной системы спасения рибосом по крайней ме%
сколько раз, приводя к присоединению полиала%
ре у некоторых бактерий. Тем не менее многие
БИОХИМИЯ том 86 вып. 8 2021
БАКТЕРИАЛЬНАЯ СИСТЕМА RQC
1145
аспекты функционирования этой системы оста%
тидил%тРНК, так как в экспериментах in vitro
ются неизвестными. Неясно, какие условия
было четко показано, что способность комплек%
способствуют разборке остановившихся рибо%
са RRF•EF%G диссоциировать рибосомы огра%
сом на субъединицы и какой фактор(%ы) в бак%
ничена исключительно рибосомами, прошед%
териальных клетках стимулирует эту разборку.
шими стадию терминации и несущими деаци%
Также неизвестно, каким образом «спасение»
лированную тРНК [40].
большой субъединицы, несущей пептидил%
Стоит отметить, что увеличение случаев дли%
тРНК, осуществляется у гамма%протеобактерий
тельных трансляционных пауз происходит при
и актинобактерий, где отсутствует ключевой
определенных экстремальных условиях, таких
фактор С%концевой модификации полиалани%
как тепловой шок. Показано, что бактериальная
ном, RqcH. Также ещё предстоит установить,
ГТФаза теплового шока, HflX, способна разби%
что служит сигналом для прекращения присое%
рать на субъединицы свободные и связанные с
динения остатков аланина к неполному пептиду
мРНК рибосомы, несущие деацилированную
и какой клеточный фактор осуществляет гидро%
тРНК в P%сайте [41]. Основываясь на этих дан%
лиз образовавшейся пептидил%тРНК. Некото%
ных, было высказано предположение, что
рые важные вопросы, касающиеся механизмов
HflX может служить альтернативным фактором
RQC%подобного освобождения остановившихся
разборки, который рециклизует рибосомы пос%
рибосом у бактерий, на которые пока нет отве%
ле терминации трансляции (генерируемые пос%
тов, обсуждаются ниже.
редством регулярного или вызванного одной из
систем спасения высвобождения пептида) при
условиях, таких как тепловой шок, когда актив%
КАК ПРОИСХОДИТ РАЗБОРКА
ность рециклизующего комплекса RRF•EF%G
ОСТАНОВИВШИХСЯ РИБОСОМ
снижена. Хотя диссоциирующая актив%
У БАКТЕРИЙ?
ность HflX установлена, ещё предстоит прове%
рить его способность разбирать остановившиеся
Разборка остановившихся рибосом на субъ%
рибосомы, несущие пептидил%тРНК. Гомоло%
единицы является шагом, который запускает
ги HflX, рибосомо%зависимая АТФаза YchF и
RQC%опосредованный механизм спасения. Ус%
ГТФаза ObgE, также могут быть потенциальны%
ловия, вызывающие такую разборку у бактерий,
ми кандидатами на наличие способности разби%
и внутриклеточные факторы, участвующие в
рать рибосомы у бактерий [42, 43].
этом процессе, пока неизвестны. Интересно,
что было показано, что включение в синтезиру%
емый пептид нескольких остатков аспартата и
КАК ПРОИСХОДИТ ОСВОБОЖДЕНИЕ
глутамата подряд может дестабилизировать
БОЛЬШОЙ СУБЪЕДИНИЦЫ РИБОСОМЫ
транслирующую рибосому и индуцировать её
ОТ НЕПОЛНОГО ПЕПТИДА В БАКТЕРИЯХ,
фактор%независимую диссоциацию на субъеди%
У КОТОРЫХ ОТСУТСТВУЕТ RqcH?
ницы [38].
Несмотря на то что этот пример иллюстри%
У B. subtilis совместное действие RqcH и
рует возможность спонтанной разборки транс%
RqcP способствует модификации С%конца не%
лирующей рибосомы на малую и большую субъ%
полного пептида полиаланиновой последова%
единицы, подобные события, по%видимому, не
тельностью. Примечательно, что способность
являются распространёнными в клетке, так как
Hsp15 E. coli (гомолога Rqcp) связываться с 50S%
они провоцируются специфическими и, следо%
субъединицей, несущей пептидил%тРНК, хоро%
вательно, очень редкими аминокислотными мо%
шо задокументирована [44]. Тем не менее фило%
тивами в синтезируемых полипептидах, кото%
генетический анализ не выявил наличие гомо%
рые должны вытесняться эволюционным отбо%
лога RqcH у актинобактерий и гамма%протео%
ром из существующих белков [39]. Следователь%
бактерий (таких как E. coli), что ставит вопрос о
но, существование фактора(ов), специализиру%
том, каким образом у этих видов бактерий про%
ющихся на распознавании и активной разборке
исходит освобождение большой субъединицы
остановившихся рибосом в бактериальных
рибосомы от неполного пептида. Было высказа%
клетках, остаётся вполне вероятным.
но предположение, что Hsp15 E. coli может ста%
При нормально протекающей трансляции у
билизировать такую конформацию пептидил%
бактерий факторы рециклинга рибосом RRF и
тРНК в Р%сайте 50S%субъединицы, которая де%
EF%G разбирают рибосомы после завершения
лает возможным её гидролиз некоторым факто%
трансляции и высвобождения зрелого белка.
ром [45, 46]. Если это так, то механизм освобож%
Однако маловероятно, что эти белки диссоции%
дения остановившихся рибосом у актинобакте%
руют остановившиеся рибосомы, несущие пеп%
рий и гамма%протеобактерий может, в принци%
4 БИОХИМИЯ том 86 вып. 8 2021
1146
СВЕТЛОВ
пе, происходить минуя стадию добавления по%
скольку RQC%подобный механизм не критичес%
лиаланина. Альтернативно, добавление поли%
ки важен для жизнеспособности B. subtilis [35],
аланинового дегрона у этих видов может быть
может показаться, что он не может служить дос%
обусловлено согласованным действием Hsp15 и
таточно хорошей мишенью для антибиотиков.
некоторого не родственного RqcH белка. Оста%
Однако известно, что гомологи RqcH необходи%
ется выяснить, по какому из двух сценариев у
мы для вирулентности некоторых патогенов,
этих видов происходит разборка комплек%
включая Streptococcus pneumoniae, Staphylococcus
сов 50S•пептидил%тРНК.
aureus, Enterococcus faecalis и Listeria monocyto
genes [51-54]. Этот факт делает бактериальные
RQC привлекательной мишенью для разработки
КАК НЕПОЛНЫЙ ПЕПТИД
принципиально нового класса лекарственных
ВЫСВОБОЖДАЕТСЯ
средств. В отличие от традиционных антибиоти%
ИЗ 50S)СУБЪЕДИНИЦЫ РИБОСОМЫ?
ков, которые подавляют рост или убивают бакте%
рии, эти препараты должны сдерживать патоге%
Независимо от модификации неполного
ны, нейтрализуя их вирулентность. Теоретичес%
пептида полиаланиновой последовательностью
ки, эта стратегия должна значительно снизить
сложноэфирная связь в молекуле пептидил%
склонность патогенов к развитию устойчивости
тРНК, ассоциированной с большой субъедини%
к действию лекарств. Дальнейшие исследования
цей, должна быть гидролизована, а образовав%
систем спасения рибосом у бактерий, включая
шийся пептид должен быть высвобожден. «Ка%
RQC путь, могут выявить новые молекулярные
нонические» системы спасения бакте%
мишени внутри клеток для разрабатываемых
рий (SmpB•тмРНК, ArfA/ArfT и ArfB) запуска%
противоинфекционных средств. Перспектив%
ют гидролитическую активность PTC рибосо%
ность этой стратегии подтверждается недавним
мы, приводящую к гидролизу пептидил%тРНК.
открытием ациламинооксадиазолов (включая
Однако, так как эти механизмы работают иск%
так называемое соединение KKL%35), как инги%
лючительно с целой 70S%рибосомой, они вряд
биторов транс%трансляции с ярко выраженными
ли используются при высвобождении пептидов
антибактериальными свойствами [55, 56].
в процессе RQC%опосредованного спасения.
Пептидил%тРНК гидролаза, важный белок, ко%
торый гидролизует свободную от рибосом пеп%
Финансирование. Выполнение данной рабо%
тидил%тРНК [47, 48], и RF%гомолог, PrfH, функ%
ты было поддержано Национальным Институ%
ция которого в клетке остаётся невыяснен%
том Здоровья (грант № R21%AI137584).
ной [49], могут быть кандидатами на искомую
Благодарности. Я хочу выразить глубочай%
релиз%активность. Наконец, нельзя исключить
шую благодарность моему учителю, профессору
наличие в бактериях некоторой тРНК%специ%
Александру Сергеевичу Спирину, и сотрудни%
фической эндонуклеазы с Vms1/ANKZF1%по%
кам Института Белка в Пущино, где я получил
добной активностью.
бесценный опыт в области биосинтеза белка и
изучения рибосом. Я благодарен Александру
Ричардсону, доктору Юрию Поликанову, докто%
МЕДИЦИНСКОЕ ЗНАЧЕНИЕ
ру Норе Васкез%Ласлоп и доктору Александру
БАКТЕРИАЛЬНОЙ СИСТЕМЫ RQC
Манкину за тщательное прочтение моей статьи
и её плодотворное обсуждение.
Инфекционные заболевания, вызываемые
Конфликт интересов. Автор заявляет об отсут%
патогенными бактериями, обычно лечат антибио%
ствии у него конфликта интересов.
тиками, которые представляют собой неболь%
Соответствие этическим стандартам. В насто%
шие молекулы, подавляющие жизненно важные
ящей статье не содержатся данные каких%либо
процессы в бактериальных клетках и тем самым
работ с участием людей или животных, которые
предотвращающие их размножение [50]. По%
были выполнены автором.
СПИСОК ЛИТЕРАТУРЫ
1.
Dai, X., Zhu, M., Warren, M., Balakrishnan, R.,
tion speed based on LacZα complementation system,
Patsalo, V., et al. (2016) Reduction of translating ribosomes
Nucleic Acid Res., 44, e155.
enables Escherichia coli to maintain elongation rates during
3.
Jha, S., and Komar, A. A. (2011) Birth, life and death of
slow growth, Nat. Microbiol., 2, 16231.
nascent polypeptide chains, Biotechnol. J., 6, 623%640.
2.
Zhu, M., Dai, X., and Wang, Y.%P. (2016) Real time deter%
4.
Samatova, E., Daberger, J., Liutkute, M., and Rodnina,
mination of bacterial in vivo ribosome translation elonga%
M. V. (2021) Translational control by ribosome pausing in
БИОХИМИЯ том 86 вып. 8 2021
БАКТЕРИАЛЬНАЯ СИСТЕМА RQC
1147
bacteria: how a non%uniform pace of translation affects
24.
Pisarev, A. V., Skabkin, M. A., Pisareva, V. P., Skabkina,
protein production and folding, Front. Microbiol., 11,
O. V., Rakotondrafara, A. M., et al. (2010) The role of
619430.
ABCE1 in eukaryotic post%termination ribosomal recy%
5.
Doerfel, L. K., Wohlgemuth, I., Kothe, C., Peske, F.,
cling, Mol. Cell, 37, 196%210.
Urlaub, H., and Rodnina, M. V. (2013) EF%P is essential
25.
Shoemaker, C. J., Eyler, D. E., and Green, R. (2010)
for rapid synthesis of proteins containing consecutive pro%
Dom34:Hbs1 promotes subunit dissociation and peptidyl%
line residues, Science, 339, 85%88.
tRNA drop%off to initiate no%go decay, Science, 330, 369%
6.
Ude, S., Lassak, J., Starosta, A. L., Kraxenberger, T.,
372.
Wilson, D. N., and Jung, K. (2013) Translation elongation
26.
Joazeiro, C. A. P. (2019) Mechanisms and functions of
factor EF%P alleviates ribosome stalling at polyproline
ribosome%associated protein quality control, Nat. Rev. Mol.
stretches, Science, 339, 82%85.
Cell Biol., 20, 368%383.
7.
Huter, P., Arenz, S., Bock, L. V., Graf, M., Frister, J. O.,
27.
Shen, P. S., Park, J., Qin, Y., Li, X., Parsawar, K., et al.
et al. (2017) Structural basis for polyproline%mediated
(2015) Rqc2p and 60S ribosomal subunits mediate mRNA%
ribosome stalling and rescue by the translation elongation
independent elongation of nascent chains, Science, 347,
factor EF%P, Mol. Cell, 68, 515%527.
75%78.
8.
Pech, M., Karim, Z., Yamamoto, H., Kitakawa, M.,
28.
Verma, R., Reichermeier, K. M., Burroughs, A. M., Oania,
Qin, Y., and Nierhaus, K. H. (2011) Elongation factor 4
R. S., Reitsma, J. M., et al. (2018) Vms1/ANKZF1 pep%
(EF4/LepA) accelerates protein synthesis at increased
tidyl%tRNA hydrolases releases nascent chains from stalled
Mg2+ concentrations, Proc. Natl. Acad. Sci. USA, 108,
ribosomes, Nature, 557, 446%451.
3199%3203.
29.
Yip, M. C. J., Keszei, A. F. A., Feng, Q., Chu, V.,
9.
Betney, R., de Silva, E., Krishnan, J., and Stansfield, I.
McKenna, M. J., and Shao, S. (2019) Mechanism for
(2010) Autoregulatory systems controlling translation fac%
recycling tRNAs on stalled ribosomes, Nat. Struct. Mol.
tor expression: thermostat%like control of translational
Biol., 26, 343%349.
accuracy, RNA, 16, 655%663.
30.
Yip, M. C. J., Savickas, S., Gygi, S. P., and Shao, S. (2020)
10.
Shalgi, R., Hurt, J. A., Krykbaeva, I., Taipale, M.,
ELAC1 repairs tRNA cleaved during ribosome%associated
Lindquist, S., and Burge, C. B. (2013) Widespread regula%
quality control, Cell Rep., 30, 2106%2114.
tion of translation by elongation pausing in heat shock,
31.
Chadani, Y., Ono, K., Kutsukake, K., and Abo, T. (2011)
Mol. Cell, 49, 439%452.
Escherichia coli YaeJ protein mediates a novel ribosome%
11.
Vazquez%Laslop, N., and Mankin, A. S. (2018) How
rescue pathway distinct from SsrA% and ArfA%mediated
macrolide antibiotics work, Trends Biochem. Sci., 43, 668%
pathways, Mol. Microbiol., 80, 772%785.
684.
32.
Keiler, K. C., and Feaga, H. A. (2014) Resolving nonstop
12.
Muller, C., Crowe%McAuliffe, C., and Wilson, D. N.
translation complexes is a matter of life or death,
(2021) Ribosome rescue pathways in bacteria, Front.
J. Bacteriol., 196, 2123%2130.
Microbiol., 12, 652980.
33.
Burroughs, A. M., and Aravind, L. (2014) Analysis of two
13.
Buskirk, A. R., and Green, R. (2017) Ribosome pausing,
domains with novel RNA%processing activities throws light
arrest and rescue in bacteria and eukaryotes, Philos. Trans.
on the complex evolution of ribosomal RNA biogenesis,
R. Soc. Lond. B. Biol. Sci., 372, 20160183.
Front. Genet., 5, 424.
14.
Keiler, K. C. (2015) Mechanisms of ribosome rescue in
34.
Henderson, B., Nair, S., Pallas, J., and Williams, M. A.
bacteria, Nat. Rev. Microbiol., 13, 285%297.
(2011) Fibronectin: a multidomain host adhesin targeted
15.
Keiler, K. C., Waller, P. R., and Sauer, R. T. (1996) Role of
by bacterial fibronectin%binding proteins, FEMS Microbiol.
a peptide tagging system in degradation of proteins synthe%
Rev., 35, 147%200.
sized from damaged messenger RNA, Science, 271, 990%
35.
Lytvynenko, I., Paternoga, H., Thrun, A., Balke, A.,
993.
Muller, T. A., et al. (2019) Alanine tails signal proteolysis in
16.
Karzai, A. W., Susskind, M. M., and Sauer, R. T. (1999)
bacterial ribosome%associated quality control, Cell, 178,
SmpB, a unique RNA%binding protein essential for the
76%90.
peptide%tagging activity of SsrA (tmRNA), EMBO J., 18,
36.
Filbeck, S., Cerullo, F., Paternoga, H., Tsaprailis, G.,
3793%3799.
Joazeiro, C. A. P., and Pfeffer, S. (2021) Mimicry of
17.
Himeno, H., Nameki, N., Kurita, D., Muto, A., and
canonical translation elongation underlies alanine tail syn%
Abo, T. (2015) Ribosome rescue systems in bacteria,
thesis in RQC, Mol. Cell, 81, 104%114.
Biochimie, 114, 102%112.
37.
Crowe%McAuliffe, C., Takada, H., Murina, V., Polte, C.,
18.
Chadani. Y., Ito, K., Kutsukake, K., and Abo, T. (2012)
Kasvandik, S., et al. (2021) Structural basis for bacterial
ArfA recruits release factor 2 to rescue stalled ribosomes by
ribosome%associated quality control by RqcH and RqcP,
peptidyl%tRNA hydrolysis in Escherichia coli, Mol.
Mol. Cell, 81, 115%126.
Microbiol., 86, 37%50.
38.
Chadani, Y., Niwa, T., Izumi, T., Sugata, N., Nagao, A.,
19.
Shimizu, Y. (2012) ArfA recruits RF2 into stalled ribo%
et al. (2017) Intrinsic ribosome destabilization underlies
somes, J. Mol. Biol., 423, 624%631.
translation and provides an organism with a strategy of
20.
Goralski, T. D. P., Kirimanjeswara, G. S., and Keiler, K. C.
environmental sensing, Mol. Cell, 68, 528%539.
(2018) A new mechanism for ribosome rescue can recruit
39.
Chadani, Y., Sugata, N., Niwa, T., Ito, Y., Iwasaki, S., and
RF1 or RF2 to nonstop ribosomes, mBio, 9, e02436%18.
Taguchi, H. (2021) Nascent polypeptide within the exit
21.
Handa, Y., Inaho, N., and Nameki, N. (2011) YaeJ is a
tunnel ensures continuous translation elongation by stabi%
novel ribosome%associated protein in Escherichia coli that
lizing the translating ribosome, bioRxiv, doi: 10.1101/
can hydrolyze peptidyl%tRNA on stalled ribosomes,
2021.02.02.429294.
Nucleic Acids Res., 39, 1739%1748.
40.
Peske, F., Rodnina, M. V., and Wintermeyer, W. (2005)
22.
Gagnon, M. G., Seetharaman, S. V., Bulkley, D., and
Sequence of steps in ribosome recycling as defined by
Steitz, T. A. (2012) Structural basis for the rescue of stalled
kinetic analysis, Mol. Cell, 18, 403%412.
ribosomes: structure of YaeJ bound to the ribosome,
41.
Zhang, Y., Mandava, C. S., Cao, W., Li, X., Zhang, D.,
Science, 335, 1370%1372.
et al. (2015) HflX is a ribosome%splitting factor rescuing
23.
Shoemaker, C. J., and Green, R. (2012) Translation drives
stalled ribosomes under stress conditions, Nat. Struct. Mol.
mRNA quality control, Nat. Struct. Mol. Biol., 19, 594%601.
Biol., 22, 906%913.
БИОХИМИЯ том 86 вып. 8 2021
4*
1148
СВЕТЛОВ
42.
Tomar, S. K., Kumar, P., and Prakash, B. (2011) Decipher%
50. Lewis, K. (2020) The science of antibiotic discovery, Cell,
ing the catalytic machinery in a universally conserved ribo%
181, 29%45.
some binding ATPase YchF, Biochem. Biophys. Res.
51. Holmes, A. R., McNab, R., Millsap, K. W., Rohde, M.,
Commun., 408, 459%464.
Hammerschmidt, S., et al. (2001) The pavA gene of
43.
Feng, B., Mandava, C. S., Guo, Q., Wang, J., Cao, W.,
Streptococcus pneumoniae encodes a fibronectin%binding
et al. (2014) Structural and functional insights into the
protein that is essential for virulence, Mol. Microbiol., 41,
mode of action of a universally conserved Obg GTPase,
1395%1408.
PLos Biol., 12, e1001866.
52. Pracht, D., Elm, C., Gerber, J., Bergmann, S., Rohde, M.,
44.
Korber, P., Stahl, J. M., Nierhaus, K. H., and Bardwell,
et al. (2005) PavA of Streptococcus pneumoniae modulates
J. C. (2000) Hsp15: a ribosome%associated heat shock pro%
adherence, invasion, and meningeal inflammation, Infect.
tein, EMBO J., 19, 741%748.
Immun., 73, 2680%2689.
45.
Jiang, L., Schaffitzel, C., Bingel%Erlenmeyer, R., Ban, N.,
53. Singh, K. V., La Rossa, S. L., Somarajan, S. R., Roh, J. H.,
Korber, P., et al. (2009) Recycling of aborted ribosomal 50S
and Murray, B. E. (2015) The fibronectin%binding protein
subunit%nascent chain%tRNA complexes by the heat shock
EfbA contributes to pathogenesis and protects against
protein Hsp15, J. Mol. Biol., 386, 1357%1367.
infective endocarditis caused by Enterococcus faecalis,
46.
Starosta, A. L., Lassak, J., Jung, K., and Wilson, D. N.
Infect. Immun., 83, 4487%4494.
(2014) The bacterial translation stress response, FEMS
54. Dramsi, S., Bourdichon, F., Cabanes, D., Lecuit, M.,
Microbiol. Rev., 38, 1172%1201.
Fsihi, H., and Cossart, P. (2004) FbpA, a novel multifunc%
47.
Das, G., and Varshney, U. (2006) Peptidyl%tRNA hydro%
tional Listeria monocytogenes virulence factor, Mol.
lase and its critical role in protein biosynthesis,
Microbiol., 53, 639%649.
Microbiology, 152, 2191%2195.
55. Ramadoss, N. S., Alumasa, J. N., Cheng, L., Wang, Y.,
48.
Sharma, S., Kaushik, S., Sinha, M., Kushwaha, G. S.,
Li, S., et al. (2013) Small molecule inhibitors of trans%
Singh, A., et al. (2014) Structural and functional insights
translation have broad%spectrum antibiotic activity, Proc.
into peptidyl%tRNA hydrolase, Biochim. Biophys. Acta,
Natl. Acad. Sci. USA, 110, 10282%10287.
1844, 1279%1288.
56. Aron, Z. D., Mehrani, A., Hoffer, E. D., Connolly, K. L.,
49.
Burroughs, A. M., and Aravind, L. (2019) The origin and
Srinivas, P., et al. (2021) trans%Translation inhibitors bind
evolution of release factors: implications for translation
to a novel site on the ribosome and clear Neisseria gonor
termination, ribosome rescue, and quality control path%
rhoeae in vivo, Nat. Commun., 12, 1799.
ways, Int. J. Mol. Sci., 20, 1981.
RIBOSOME)ASSOCIATED QUALITY CONTROL IN BACTERIA
Review
M. S. Svetlov
Center for Biomolecular Sciences, Department of Pharmaceutical Sciences, University of Illinois at Chicago,
60607 Chicago, Illinois, USA; e mail: msvet2@uic.edu
Translation of the genetic information into proteins, performed by the ribosome, is a key cellular process in all organ%
isms. Translation usually proceeds smoothly, but, unfortunately, undesirable events can lead to stalling of translating
ribosomes. To rescue these faulty arrested ribosomes, bacterial cells possess three well%characterized quality control
systems, tmRNA, ArfA, and ArfB. Recently, an additional ribosome rescue mechanism has been discovered in
Bacillus subtilis. In contrast to the “canonical” systems targeting the 70S bacterial ribosome, this latter mechanism
operates by first splitting the ribosome into the small (30S) and large (50S) subunits to then clearing the resultant
jammed large subunit from the incomplete nascent polypeptide. Here, I will discuss the recent microbiological, bio%
chemical, and structural data regarding functioning of this novel rescue system.
Keywords: translation, ribosome stalling, quality control, polyalanine%tailing
БИОХИМИЯ том 86 вып. 8 2021