БИОХИМИЯ, 2021, том 86, вып. 8, с. 1149 - 1159
УДК 577.323.55
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР,
НАИБОЛЕЕ РАСПРОСТРАНЕННОГО МОТИВА СТРУКТУРЫ РНК
Обзор
© 2021
Е.Ф. Баулин1,2
1 Институт математических проблем биологии РАН - филиал ИПМ им. М.В. Келдыша РАН,
142290 Пущино, Московская обл., Россия; электронная почта: baulin@lpm.org.ru
2 Московский физико-технический институт (национальный исследовательский университет),
141701 Долгопрудный, Московская обл., Россия
Поступила в редакцию 01.05.2021
После доработки 13.06.2021
Принята к публикации 13.06.2021
Мотивы А минор - это мотивы третичной структуры РНК, как правило состоящие из канонического спа
ривания оснований и основания аденина, образующего водородные связи с нуклеотидами спаривания со
стороны малой бороздки. Мотивы А минор являются одним из самых распространенных типов третичных
взаимодействий в известных структурах РНК, сравнимым по частоте с неканоническими спариваниями ос
нований. Они часто обнаруживаются в функционально важных участках молекул некодирующих РНК и в
том числе играют одну из центральных ролей в процессе синтеза белка. В данном обзоре рассматриваются
локальные особенности геометрии А миноров, трудности их разметки в известных пространственных
структурах РНК, различия их структурного окружения и часто встречающиеся комотивы, а также разнооб
разные функции, выполняемые А минорами во множестве внутриклеточных процессов.
КЛЮЧЕВЫЕ СЛОВА: структура РНК, третичный мотив, А минор взаимодействие, мотив А минор,
А патч.
DOI: 10.31857/S0320972521080078
ВВЕДЕНИЕ
ческих структурах интрона группы I [5, 6] и ри
бозима типа hammerhead [7]. В этих ранних ра
Сегодня известно, что молекулы некодирую
ботах авторы сообщали о высококонсерватив
щих РНК играют важнейшую роль во множест
ных аденинах, образующих водородные связи с
ве молекулярных процессов, протекающих в
удаленными каноническими спариваниями ос
живых организмах [1]. Функция, выполняемая
нований со стороны малой бороздки двойной
молекулой РНК, напрямую зависит от её прост
спирали РНК.
ранственной структуры, образованной элемен
В 2001 г., благодаря только что определен
тами вторичной структуры (стемами, т.е. корот
ным структурам рибосомных РНК (рРНК) бак
кими участками двойной спирали, состоящими
терий, объём данных о структурах РНК увели
из последовательных канонических спариваний
чился практически на порядок, что открыло
оснований, и расположенными между ними
возможность выявления многочисленных пов
петлями) и третичными взаимодействиями
торяющихся взаимодействий [8]. Было показа
между ними [2]. Последние зачастую формиру
но, что структуры рРНК содержат необычно
ют повторяющиеся модули, называемые третич
много неспаренных аденинов, заметная часть
ными мотивами; они сохраняют свою конфигу
которых участвует в третичных взаимодействиях
рацию в разнообразных структурных окружени
и консервативна среди всех царств живых орга
ях [3]. Среди наиболее часто встречающихся и
низмов [4, 8]. Nissen et al. [4] ввели термин «мо
функционально важных третичных мотивов
тив А минор» (A minor motif) и выявили 186 та
РНК выделяют мотивы типа А минор [4].
ких мотивов в структуре рибосомной субъеди
Первые случаи обнаружения мотивов А ми
ницы 50S. Они показали, что в структуре 23S
нор были описаны в 1994-1996 гг. в кристалли рРНК А миноры формируются и между спира
лями, и между петлями, и между петлёй и спи
Принятые сокращения: H ребро - Хугстиновское
ралью [4]. В том же году в работе [9] был сделан
ребро (Hoogsteen edge); S ребро - Сахарное ребро (Sugar
edge); WC ребро - Уотсон Криковское ребро (Watson
вывод о том, что А миноры являются универ
Crick edge).
сальным способом укладки спиралей, который
1149
1150
БАУЛИН
встречается в рибозиме вируса гепатита дельта,
pers) [30, 34], петли lonepair triloop [35] и мотивы
23S рРНК, интроне группы I и рибозиме типа
adenosine wedge (A wedge) [36].
hammerhead.
В настоящем обзоре мы обсуждаем разные
В 2002 г. были разработаны биохимические
определения А минор взаимодействия и мотива
методы идентификации А минор взаимодей
А минор, а также перечисляем их геометричес
ствий с использованием аналогов аденози
кие типы; рассматриваем структурные свойства,
на [10]. Данный подход, в частности, позволил
базы данных и программы аннотации А мино
продемонстрировать участие А миноров в пози
ров; указываем наиболее важные классы А ми
ционировании 5′ экзона внутри каталитическо
норов, в частности вовлеченные в функциониро
го ядра интрона группы I [10].
вание рибосомы. Мы также разбираем характер
Позднее А минор взаимодействия были об
ные черты А миноров и указываем на сохраняю
наружены во множестве типов молекул некоди
щиеся проблемы их автоматической разметки.
рующих РНК, включая рибонуклеазу P [11, 12],
SRP РНК (РНК комплекса signal recognition
particle) [13], вирусные РНК [14, 15], рибозим
ЧТО ТАКОЕ А МИНОР?
типа pistol [16], прекурсор транспортной РНК
(пре тРНК) [17], разнообразные рибопереклю
Прежде чем приступать к обсуждению опре
чатели [18-20] и длинную некодирующую РНК
делений А минора, следует привести общепри
MALAT1 [21].
нятую номенклатуру спариваний рибонуклео
А миноры также бывают регулярно обнару
тидов, известную как классификация Леонти
жены как составной элемент более сложных мо
са-Вестхофа (LW классификация спариваний)
тивов, таких как мотивы kink turn [22, 23], взаи
[37]. Согласно данной классификации, каждое
модействия GNRA tetraloop/receptor [24-26],
основание спаривания образует водородные
псевдоузлы [14, 27, 28], коаксиальный стэкинг
связи одним из трех своих ребер: Хугстиновским
[29-33], рибозные застёжки/молнии (ribose zip
ребром (Hoogsteen edge, H ребро), Уотсон Кри
Рис. 1. а - Три ребра основания аденина: Хугстиновское, Уотсон Криковское и Сахарное (также включает O2′ атом ри
бозы). б - Четыре типа А минор взаимодействий, выделенные в работе [4]; А миноры типов I и II называют каноничес
кими. в - Пример неканонического WC/H А минора, составленного основаниями U4 и A6 цепи A и A6 - цепи B; адени
ны в WC/H А минорах образуют водородные связи со спариванием своим WC либо H ребром (PDB код: 1LPW)
БИОХИМИЯ том 86 вып. 8 2021
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР
1151
ковским ребром (Watson Crick edge, WC ребро)
торы также отмечают, что цепи аденина и бли
или Сахарным ребром (Sugar edge, S ребро)
жайшего к нему основания спаривания обяза
(рис. 1, а). S ребро, помимо атомов основания,
тельно антипараллельны. А миноры типов I и II
включает также и O2′ атом рибозы. Каждому
высокоспецифичны к основанию аденина бла
спариванию ставится в соответствие трехбук
годаря его гладкому S ребру с доступными для
венный LW тип, где две буквы обозначают взаи
образования водородных связей атомами N1,
модействующие ребра (H/S/W), а ещё одна - от
N3 и O2′. А миноры типов III и 0 менее спе
носительную ориентацию гликозидных связей
цифичны, но также «предпочитают» адени
[c/t, т.е. цис (cis) или транс (trans)]. Так, напри
ны [4].
мер, канонические Уотсон Криковские спари
В недавней работе [38] Torabi et al. определя
вания A U и G C и «плавающие» (wobble) спа
ют подкласс WC/H А миноров, в которых аде
ривания G U относят к типу cWW, т.к. они обра
нин взаимодействует с малой бороздкой спари
зованы с участием WC рёбер оснований с глико
вания либо WC ребром, либо H ребром - вмес
зидными связями в цис ориентации. В РНК в
то S ребра в случае классических А миноров
участках двойной спирали, образованных после
(рис. 1, в). Примеры таких неканонических взаи
довательными каноническими спариваниями,
модействий были описаны и раньше [16, 39], од
S рёбра оснований выстилают малую бороздку
нако не были отнесены к отдельному подклассу
спирали, а H рёбра - большую бороздку.
А миноров.
В 2001 г. Nissen et al. ввели термин «мотив
Мотив А минор. А миноры часто находятся в
А минор» для описания повторяющихся взаи
стэкинге с другими А минорами, образуя стоп
модействий в рРНК, образованных вставкой
ки, называемые А патчами (A patch) [4]. А пат
гладкого N1 C2 N3 конца основания адени
чи редко состоят более чем из трех А миноров и,
на (А) в малую бороздку двойной спирали
как правило, номер типа А миноров уменьша
(minor groove), преимущественно в области C G
ется в направлении от 5′ к 3′, т.е. обычно А пат
спариваний (рис. 1, б) [4]. Позднее вводились
чи имеют тип II,I или III,II,I. А патчи большего
другие определения «мотива А минор» и «А ми
размера формируются с участием двух нитей
нор взаимодействия», которые будут рассмотре
последовательных аденинов, образующих меж
ны ниже. Здесь и далее мы будем называть взаи
нитевой стэк; типы А миноров в таких А патчах
модействие трех оснований того или иного типа
увеличиваются от середины к концам, напри
«А минор взаимодействием» или просто «А ми
мер, II,I,I,II. А патчи, образованные двумя пос
нором», а «мотивом А минор» будем называть
ледовательными аденинами и двумя последова
стопку из нескольких А миноров, находящихся
тельными спариваниями, были определены в
в стэкинге.
работе [40] как мотивы типа sextuple - третич
А минор взаимодействие. В работе [4] Nissen
ные мотивы РНК, состоящие из шести основа
et al. применили следующий критерий для раз
ний, образующих сеть водородных связей.
метки А минор взаимодействий: (а) атом C2
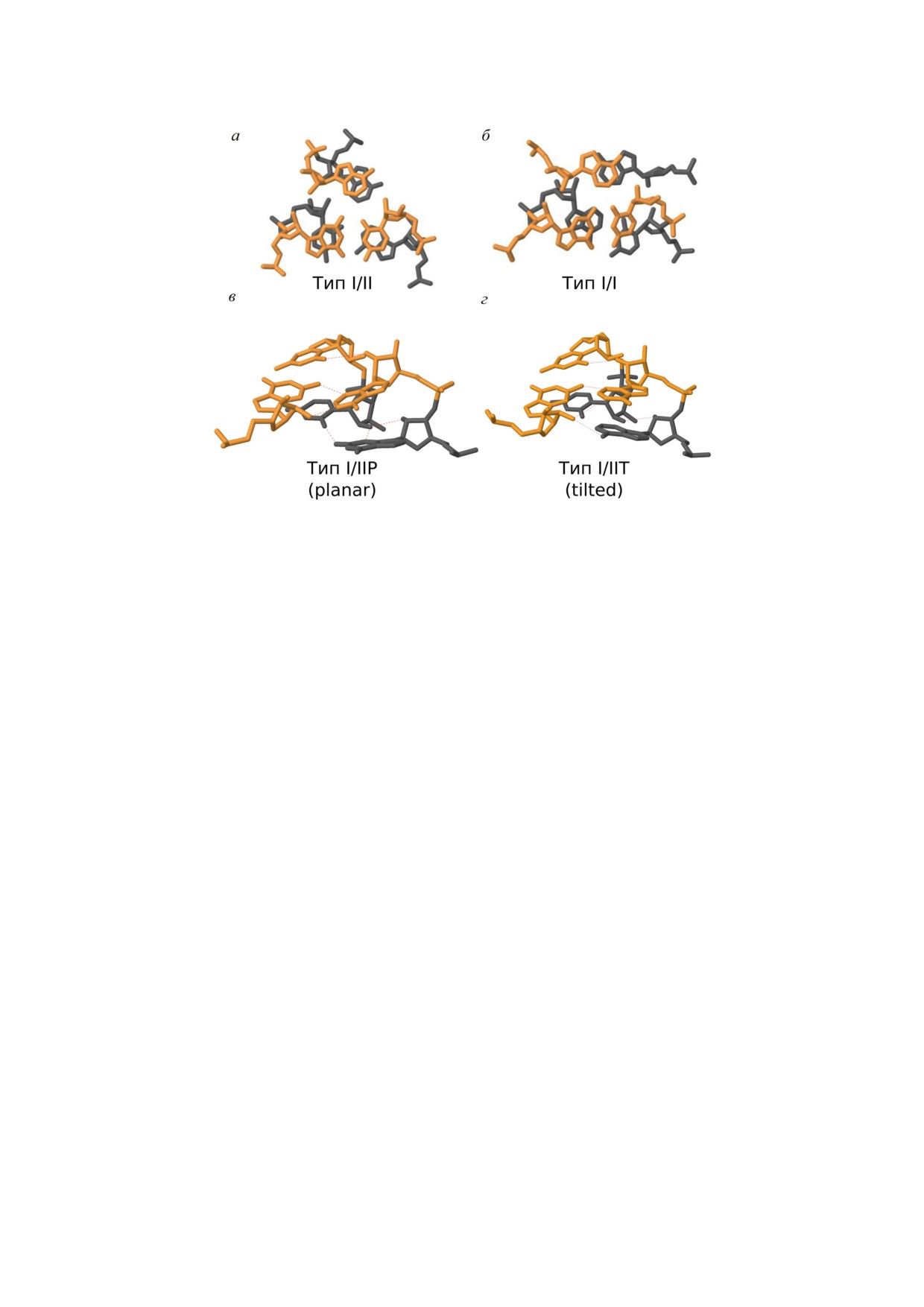
В работе [41] Lescoute et al. определяют мо
аденина располагается со стороны малой борозд
тив А минор типа I/II как мотив, включающий
ки спаривания; (б) хотя бы один атом хотя бы
два последовательных спаривания и два после
одного основания спаривания лежит в радиусе
довательных аденина, образующих А минор ти
3,7 Å от атома C2 аденина; (в) ближайший к
па I и А минор типа II (рис. 2, а). Отмечается,
атому C2 аденина атом спаривания находится
что в таком мотиве А минор типа II образован
под углом не более 45° от плоскости основания
ближайшим к 5′ концу цепи РНК аденином, а
аденина. Четыре предложенных геометричес
ближайший к 3′ концу аденин взаимодействует
ких типа А миноров определяются расположе
с обоими основаниями спаривания, образуя
нием атомов O2′ и N3 основания аденина отно
А минор типа I. Более редкий мотив А минор
сительно канонического спаривания (рис. 1, б).
типа I/I [42] состоит из двух А миноров типа I,
В А минорах типа I оба этих атома находятся
которые включают два аденина, образующие
между атомами O2′ оснований спаривания (т.е.
межнитевой стэк (рис. 2, б).
внутри малой бороздки); в случае типа II
В работах [24, 33] Geary et al. выделяют два
атом N3 аденина лежит внутри малой бороздки,
подкласса мотивов А минор типа I/II: тип I/IIP
а атом O2′ аденина располагается снаружи; в
(planar, плоский, рис. 2, в) и тип I/IIT (tilted,
А минорах типа III аденин целиком находится
смещенный, рис. 2, г). Тип I/IIP преимущест
вне малой бороздки спаривания так, что его
венно включает взаимодействия петля спираль,
атом N3 направлен в сторону атома O2′ ближай
в то время как тип I/IIT представляет взаимо
шего основания спаривания; в случае типа 0
действия петля рецептор, в которых последний
только атомы рибозы аденина располагаются
обеспечивает формирование дополнительных
между атомами O2′ оснований спаривания. Ав
стабилизирующих связей.
БИОХИМИЯ том 86 вып. 8 2021
1152
БАУЛИН
Рис. 2. а - Мотив А минор типа I/II, включающий стэк двух последовательных аденинов (PDB код: 6QZP): основания
G673, C674, A996, A997, (A2M)1031, C1032 цепи S2. б - Мотив А минор типа I/I, включающий межнитевой стэк адени
нов (PDB код: 5TBW): основания U2629, C2630, G2648, A2649, A2696, A2758 цепи 1. в - Мотив А минор типа I/IIP (pla
nar, плоский; PDB код: 2GCV): основания G31, G116, A117 цепи B и C10, U11 цепи A. г - Мотив А минор типа I/IIT (tilt
ed, смещенный; PDB код: 1HR2): основания A152, A153, C223, U224, G250 цепи B
РАЗМЕТКА А МИНОРОВ И ИХ СВОЙСТВА
В работе [44] авторы выделили 11 геометри
ческих параметров, которые могут быть исполь
В 2008 г. было показано [30], что А миноры
зованы для аннотации А миноров типов I, II, 0
являются самым распространенным типом тре
и III. Параметры были определены с использо
тичных мотивов РНК (37%), на втором месте
ванием комбинированного набора из 260 А ми
оказался коаксиальный стэкинг (32%), на треть
норов, анализируемых в работах [30, 45].
ем - рибозные застёжки/молнии (20%). Также
На данный момент существует несколько баз
было отмечено, что 73% рибозных застёжек
данных, в которых хранится информация об
включает хотя бы один аденин, образующий
А минор взаимодействиях, аннотированных в
А минор взаимодействие. Используемый в ра
известных пространственных структурах РНК
боте [30] набор данных состоял из 229 А мино
из банка PDB (Protein Data Bank [46]). База дан
ров, аннотированных с помощью программы
ных CaRNAval [47] содержит информацию о
FR3D [43] в 54 кристаллических структурах не
дальнодействующих модулях взаимодействий в
кодирующих РНК высокого разрешения. Набор
РНК (RIN, RNA interaction network), включая
данных включал 52% А миноров типа I, 31% -
А миноры типа I (194 модуля) и мотивы А ми
А миноров типа II, 10% и 7% А миноров типа 0
нор типа I/II (102 модуля). Одним из преиму
и III соответственно. Среди 229 А миноров 18%
ществ используемого в базе CaRNAval подхода
содержали спаривание cWW G C, 62% - спари
является его независимость от типов основа
вание cWW C G, 5% - cWW A U, 9% - cWW
ний, т.е. модули определяются исключительно
U A. Xin et al. показали [30], что спаривание
на основе геометрических типов спариваний.
А минора как правило располагается на конце
Так, А минор типа I определяется присутствием
участка двойной спирали РНК, примыкая к со
основания, вовлеченного в два спаривания типа
седней шпильке, внутренней петле или мульти
cSS и tSS c основаниями канонического
петле. В исследуемом наборе данных также при
cWW спаривания, в то время как А минор ти
сутствовали примеры неканонических А мино
па II, входящий в состав мотива А минор ти
ров, например, А минор типа I, образованный
па I/II, требует наличия одного cSS спаривания.
спариванием G C и основанием U, и А минор
Такой подход позволил Reinharz et al. [47] анно
типа I, образованный основанием А и некано
тировать один случай G минора типа I, 21 слу
ническим спариванием cWW A A.
чай G минора типа II и один случай U минора
БИОХИМИЯ том 86 вып. 8 2021
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР
1153
типа II. Еще одним хранилищем информации
ных пространственных структурах РНК, ~ 40%
об А минорах является база данных InterRNA
составляют локальные, а ~ 60% - дальнодей
[48]: в ней перечислены А миноры, аннотиро
ствующие А миноры [55].
ванные с помощью программ NASSAM [49] и
А миноры и мотивы kink turn. Более полови
COGNAC [50], включая А миноры типов I и II,
ны всех локальных А миноров во внутренних
а также G миноры типа II.
петлях известных структур РНК являются
Среди наиболее популярных программ, ис
частью мотива kink turn [55], представляющего
пользуемых для разметки А миноров, стоит от
собой асимметричную внутреннюю петлю с рез
метить FR3D [43] и DSSR [51]. Программа
ким изгибом сахарофосфатного остова [22]. Мо
FR3D [43] размечает в 3D структурах РНК тре
тив kink turn включает А минор типа I, образо
тичные мотивы, используя для поиска либо об
ванный аденином одной стороны изгиба и внут
разец мотива, либо символьный шаблон типов
ренним C G спариванием стема другой стороны
взаимодействий. В частности, FR3D успешно
изгиба [22]. В последовательности наблюдается
использовалась для разметки А миноров ти
сильное предпочтение к паре оснований C G,
пов I, II, 0 и III в работе [30] и А миноров ти
необходимой для оптимальной конфигурации
пов I и II в работе [47]. Программа FR3D позво
А минора [22, 23]. В работе [56] авторы показа
ляет осуществлять разметку А миноров, сфор
ли, что угол между стемами мотива kink turn ре
мированных другими типами оснований вместо
гулируется локальной геометрией А минора ти
аденина, но ограничена немодифицированны
па I. Изменение угла обусловлено вставкой мо
ми нуклеотидами без пропущенных (missing)
лекулы воды между аденином и ближним к его
атомов. Программа DSSR [51] среди широкого
атому O2′ основанием спаривания, что приво
спектра третичных мотивов и взаимодействий в
дит к образованию неканонической вариации
РНК также аннотирует А миноры типов I, II и
А минора, включающей контакты через моле
X. Тип X (eXtended, расширенный) определяется
кулы воды [56, 57].
как неканонический тип А миноров, в которых
А минорные мульти петли. Другим примером
аденин контактирует с малой бороздкой спари
частого комотива локальных А миноров явля
вания своим WC ребром или H ребром, не за
ются А минорные мульти петли (A minor junc
действуя атом O2′. По умолчанию множество
tions) [29, 31, 33]. А минорные мульти петли как
А миноров, размечаемых программой DSSR,
правило замыкают три или четыре стема и вклю
ограничено образованными каноническим спа
чают коаксиальный стэкинг, который стабили
риванием и основанием аденина, однако DSSR
зируется как минимум одним А минор взаимо
также предоставляет дополнительную опцию
действием. Нить, предоставляющая аденины для
«—a minor=BASE», где BASE может быть лю
взаимодействия, может образовывать как мотив
бым символом из строки ACGTURYN (Xiang
U turn, замкнутый хотя бы одним спариванием,
Jun Lu, личное общение). Для разметки А ми
так и более сложные и специфичные мотивы,
норов некоторых типов можно также восполь
обеспечивающие бóльшую стабильность [33].
зоваться одной из программ разметки спарива
Мульти петли, замыкающие пять и более сте
ний оснований в РНК [52-54] с последующей
мов, также могут содержать структурные эле
дополнительной обработкой результатов.
менты, характерные для А минорных мульти
петель [45]. Возможность образования А мино
ров успешно используется в качестве одного из
ЛОКАЛЬНЫЕ А МИНОРЫ
признаков, позволяющих предсказывать нали
чие коаксиального стэкинга в мульти петлях
Среди всех А миноров можно выделить две
РНК [58]. Интересно, что согласно работе [59],
большие группы - локальные и дальнодейству
А минорные мульти петли так же, как и мотивы
ющие А миноры. Будем считать А минор ло
kink turn, подвержены шарнирным колебаниям
кальным взаимодействием, если все три его
структуры, но, в отличие от мотивов kink turn,
нуклеотида находятся внутри одной петли лю
для А минорных мульти петель не удалось вы
бого типа - шпилька (hairpin), внутренняя пет
явить конкретные локальные вариации, обеспе
ля (internal loop), выпячивание (bulge), мульти
чивающие их подвижность.
петля (multiple junction) - или если аденин внут
Кроме того, в мульти петлях функциональ
ри петли контактирует со спариванием соседне
ных ядер A и G рибопереключателей были об
го для этой петли стема. В противном случае, т.е.
наружены А патчи, один из А миноров которых
при взаимодействии аденина и спаривания из
образован с участием связываемого пурина
двух удаленных элементов вторичной структуры
[18, 60]. Помимо этого, в работе [22] Klein et al.
РНК, будем называть А минор дальнодействую
упоминают обнаруженный в молекуле 23S рРНК
щим. Среди А миноров, обнаруженных в извест
H. marismortui редкий вариант трёхнитевого мо
БИОХИМИЯ том 86 вып. 8 2021
1154
БАУЛИН
тива kink turn, который формирует часть муль
жая по количеству даже комплементарные спа
ти петли и включает А минор взаимодействие.
ривания оснований, не являющиеся частью сте
А миноры и псевдоузлы. ABAB псевдоузлы,
мов вторичной структуры [24]. Множественные
также известные как H узлы, обычно сопровож
дальнодействующие А миноры наряду с други
даются стабилизирующими А минорами, обра
ми третичными взаимодействиями способству
зованными ближайшей к 3′ концу нитью псев
ют компактной укладке спиральных элементов
доузла и его АА стемом [14, 27, 28]. В работе [27]
больших структурированных РНК [30, 65]. В ра
авторы объясняют такую асимметрию H узлов
ботах [66, 67] Šponer et al. демонстрируют тот
правозакрученностью спирали РНК А формы,
факт, что А минор взаимодействия являются
которая в случае H узла подводит ближайшую к
более гидрофобными, чем канонические спари
3′ концу нить к малой бороздке АА стема, а
вания оснований, что делает их особенно при
ближайшую к 5′ концу - к большой бороздке
годными для укладки спиралей (helix packing).
BB стема. А миноры могут входить в состав и
А миноры и шпильки. Мотив GNRA
псевдоузлов других типов, например, «целую
tetraloop/receptor - самый распространенный
щихся петель» (kissing loops, ABACBC псевдоуз
комотив дальнодействующих А миноров [55],
лы) [55, 61].
обнаруживаемый в широком спектре молекул
А миноры и выпячивания. А миноры в выпя
некодирующих РНК [24-26]. Мотив включает
чиваниях не так распространены, но представля
одно или два А минор взаимодействия между
ют несколько функционально важных случаев.
аденинами шпильки GNRA (где N - любой
Например, А минор типа WC/H обнаруживается
нуклеотид, R - пурин) и удаленным участком
в состоящем из одного аденина выпячивании,
спирали или специфичным рецептором. Наибо
которое образовано интроном незрелой мРНК и
лее распространенным и стабильным видом мо
малой ядерной РНК U2 (мяРНК U2) [39]. Это
тива GNRA tetraloop/receptor является мотив
выпячивание называют мотивом узнавания точ
GAAA/11nt, чей рецептор представляет из себя
ки сплайсосомного ветвления (spliceosomal
консервативную внутреннюю петлю, состоя
branch site recognition motif), поскольку он вызы
щую из 11 нуклеотидов (nucleotides, nt) -
вает нуклеофильную атаку на 5′ сайт сплайсинга
CCUAAG UAUGG [68]. Мотив А минор ти
незрелой мРНК на первом этапе сплайсин
па I/II в мотивах GNRA tetraloop/receptor обра
га [39]. Другой пример - два симметричных
зован с участием пуринов в третьей и четвертой
А минора типа I (a15 G9 c12 и A15 g9 C12) в
позиций последовательности GNRA. Как пра
мотиве BHB (bulge helix bulge), структура кото
вило, последовательности GNGA формируют
рого была определена в комплексе с эндонукле
мотив А минор типа I/IIP с участием нуклеоти
азой, осуществляющей сплайсинг [17]. Мо
дов GA, а последовательности GNAA более
тив BHB является частью структуры незрелых
склонны к образованию мотива А минор ти
тРНК и распознается специфической эндонук
па I/IIT, для которого обязательно наличие аде
леазой, которая осуществляет сплайсинг тРНК в
нина в третьей позиции последовательности
ядре, а также всех РНК архей [62]. Интересно,
GNRA [24].
что оба А минора в мотиве BHB сопровождают
А миноры и внутренние петли. Вторым наибо
ся стэкинг взаимодействием с остатками гисти
лее распространенным структурным контекс
дина эндонуклеазы [17].
том дальнодействующих А миноров являются
Редким примером дальнодействующих
внутренние петли с межнитевым стэком адени
А миноров, которые включают аденин из выпя
нов, например, мотив UAA/GAN, обнаруживае
чивания, является мотив А минор типа I/IIP,
мый в структурах 23S рРНК, РНК рибонуклеа
образованный консервативным А богатым вы
зы P и интронах групп I и II [69]. Межнитевой
пячиванием (A rich bulge) и спиралью P4
стэк аденинов в мотиве UAA/GAN образован
(P4 helix) интрона группы I [5, 6, 63]. Данный
тремя основаниями: двумя аденинами нити
мотив образован основаниями A183 и A184 вы
UAA и центральным аденином нити GAN. Та
пячивания и спариваниями G110 C211 и
кой ААА стэк часто взаимодействует с удален
C109 G212 и, предположительно, отвечает за
ными элементами вторичной структуры РНК с
активацию интрона группы I [9, 64].
образованием А патча типа I,II,I. Отличитель
ной чертой таких А патчей является тот факт,
что три аденина образуют связи всего с двумя
ДАЛЬНОДЕЙСТВУЮЩИЕ А МИНОРЫ
последовательными спариваниями, в то время
как обычно число аденинов в А патчах совпада
Среди всех типов дальнодействующих кон
ет с числом спариваний [4, 55].
тактов в известных структурах РНК А миноры
В работе [15] был обнаружен А патч ти
являются наиболее распространенным, опере
па III,II,I, примыкающий к тройной спирали,
БИОХИМИЯ том 86 вып. 8 2021
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР
1155
образованной поли(А) хвостом вирусной поли
контролирующего образование третьего спари
аденилированной ядерной РНК (вирусная
вания спирали кодон антикодон, не существует,
PAN РНК, viral polyadenylated nuclear RNA) и
что объясняет избыточность генетического ко
U богатой внутренней петлей (U rich internal
да, т.е. допущение «плавающих» G U (wobble
loop) элемента ENE (expression and nuclear reten
G U) спариваний наряду с Уотсон Криковски
tion element) данной РНК. В работе [21] был
ми [71, 72]. Также стоит упомянуть, что основа
описан мотив А минор типа I/II, образованный
ния A1492 и A1493 бактериальной 16S рРНК со
между элементом ENE и поли(А) хвостом длин
ответствуют основаниям A1755 и A1756 эукари
ной некодирующей РНК MALAT1. Недавно в
отической 18S рРНК [74].
структуре двудоменного элемента ENE (double
domain ENE, dENE), закристаллизованной в
комплексе с поли(А) последовательностью из
А МИНОРЫ И РАБОТА РИБОСОМЫ
28
аденинов, были обнаружены одиночные
А миноры типа WC/H, также вблизи тройной
Центральная роль мотива А минор при де
спирали, образованной последовательностью
кодировании в процессе синтеза белка отвечает
поли(А) и U богатыми внутренними петлями
принципам гипотезы «мира РНК», не только
элемента dENE [38]. Так, А минор взаимодей
позволяя сделать вывод о том, что «рибосома яв
ствия различных типов принимают участие в за
ляется рибозимом» [4, 41, 75], но и поддерживая
щитной функции поли(А) хвоста, описанной
предположение о том, что рибосома эволюцио
для многих функциональных клеточных и ви
нировала из комплекса, образованного только
русных некодирующих РНК.
молекулами РНК [41]. В работе [76] авторы об
Одним из наиболее удивительных примеров
суждают возможность того, что способность
функционально важного дальнодействующего
А миноров «отличать» комплементарные спа
мотива А минор является взаимодействие меж
ривания от неканонических является реликтом
ду А сайтом 16S рРНК и короткой спиралью ко
некого механизма репликации РНК, имеющего
дон антикодон, образованной между мРНК и
место в мире РНК.
правильной/соответствующей (cognate) тРНК
Помимо А миноров, осуществляющих «мо
[41]. Существование данного взаимодействия
ниторинг» формирования спирали кодон анти
впервые было предсказано в ходе биохимичес
кодон, в рибосомных РНК присутствуют и мно
ких экспериментов [70], а затем эксперимен
гие другие функционально важные А минор
тально подтверждено с получением кристалли
взаимодействия. Перечислим некоторые из них.
ческой структуры малой субъединицы рибосо
В процессе инициации трансляции инициа
мы 30S [71]. Мотив А минор типа I/II образует
торная формилметионин тРНК связывается
ся с участием универсально консервативных ос
напрямую с P сайтом, причём особая стабиль
татков A1492 и A1493 внутренней петли А сайта
ность такого связывания отчасти объясняется
(положения соответствуют бактериальной 16S
образованием мотива А минор типа I/II между
рРНК) [71]. Аденины A1492 и A1493 либо распо
универсально консервативными основаниями
лагаются внутри петли, либо выпячиваются на
G1338 и A1339 16S рРНК и G C спариваниями
ружу для образования взаимодействия. Основа
3040 и 2941 антикодонового стема тРНК
ние A1493 контактирует и с первым основанием
[77, 78]. Другие тРНК могут содержать в указан
кодона, и с третьим основанием антикодона,
ных позициях спаривания A U вместо G C и не
формируя А минор типа I, в то время как осно
иметь способности к формированию указанной
вание A1492 взаимодействует только со вторым
выше связи. Кроме того, основания G1338 и
основанием кодона и образует А минор типа II.
A1339 разделяют тРНК P и E сайта, предотвра
В большинстве мотивов А минор типа I/II аде
щая нежелательное перемещение из первого во
нин, образующий А минор типа II, формирует
второй [78].
водородные связи с молекулами воды, которые
А миноры также участвуют в функциониро
играют роль моста к основанию спаривания
вании пептидилтрансферазного центра рибосо
дальней от аденина нити, однако в случае моти
мы. Так, универсально консервативные остатки
ва, образованного основаниями A1492 и A1493,
A76 тРНК в А и P сайтах формируют А мино
вместо молекул воды часть малой бороздки спи
ры типа I со спариваниями домена V 23S
рали кодон антикодон занимает основание
рРНК [4, 79, 80].
G530 [72]. Известно, что аденины A1492 и A1493
Единственным консервативным третичным
«отличают» Уотсон Криковские спаривания от
контактом между 5S и 23S рРНК является сим
неканонических спариваний, обеспечивая узна
метричный А патч, образованный с участием
вание правильной/соответствующей тРНК [41,
стэка трех аденинов петли E (E loop) 5S рРНК и
73]. Стоит отметить, что отдельного аденина,
стэка трех аденинов спирали 38 23S рРНК [4, 81].
БИОХИМИЯ том 86 вып. 8 2021
1156
БАУЛИН
Данный А патч является еще одним примером
ров [41]. Взаимодействие GNRA tetraloop/helix
тенденции внутренних петель, содержащих меж
осуществляется между стабильными сформиро
нитевой стэк аденинов, к контактированию с
ванными мотивами, в то время как GAAA/
удалёнными элементами вторичной структуры.
11nt - только между частично сформированны
Рибосомная РНК субъединицы рибосомы
ми мотивами. Основания A1492, A1493 А сайта
50S содержит не менее 7 мотивов kink turn [22],
рРНК (номенклатура бактериальной 16S рРНК)
подверженных шарнирным (hinge like) колеба
демонстрируют наибольшие структурные раз
ниям, обусловленным локальными изменения
личия между связанным и свободным состояни
ми геометрии А миноров типа I. Такие «локте
ями, что позволяет им выполнять роль молеку
подобные» (elbow like) свойства мотивов kink
лярного переключателя [41].
turn могут обеспечивать важные функциональ
А миноры также являются распространен
ные изменения в относительном расположении
ным типом межмолекулярных РНК РНК взаи
удалённых элементов РНК, например, элемента
модействий. Помимо множественных межмоле
пальца A сайта (спираль 38) и фактор связыва
кулярных А миноров в рРНК, такие взаимодей
ющего сайта L11 (спирали 42-44) [56].
ствия обнаруживаются, например, в междомен
В работе [82] авторы показывают, что спи
ной области кристаллической структуры глици
раль 68 23S рРНК осуществляет постоянный
нового рибопереключателя бактерии Vibrio
контакт со спиралью 75 посредством четырех
cholerae, который является гомодимером [20, 83].
консервативных аденинов (A1853, A1854, A1889,
Более того, А минор взаимодействия использо
A1890), образующих А патч типа II,I,I,II - и та
вались для формирования синтетических гомо
кой контакт, вероятно, опосредует подвижность
и гетеродимеров тектоРНК (tectoRNA) [24].
элемента L1 stalk и вращение субъединиц рибо
Поскольку типы I и II А миноров преоблада
сомы друг относительно друга.
ют над другими типами, в настоящее время явля
А миноры также образуют сайты связыва
ется общепринятым ограничивать определение
ния рибосомной РНК с белками, например,
А минора триплексом оснований, содержащим
А патчи в спиралях 95 и 97 23S рРНК участвуют
каноническое cWW спаривание и основание
в связывании с рибосомным белком L6 [4].
(обычно аденин), образующее cSS и/или
tSS спаривания с основаниями cWW спарива
ния [84]. К сожалению, такой подход приводит к
ОБСУЖДЕНИЕ
исключению из рассмотрения многих неканони
ческих А миноров, например, подкласса WC/H.
В данном обзоре нами рассмотрены различ
Более того, существует проблема разметки пог
ные определения А миноров, приведены при
раничных случаев А миноров, геометрические
меры функциональных мотивов А минор, а так
параметры которых немного превышают допус
же систематически перечислены наиболее рас
каемые пороги значений. Множество таких пог
пространенные типы структурного окружения
раничных А миноров присутствует в рибосомах
А миноров.
и, предположительно, обеспечивает общую гиб
В целом можно резюмировать, что мотивы
кость структуры рибосомы, в частности, под
А минор, которые эволюционировали для раз
вижность субъединиц друг относительно друга
личения комплементарных и неканонических
[80, 85]. На принципиальную необходимость та
спариваний, например, контакт между спи
кой подвижности для осуществления процесса
ралью домена P4 и А богатым выпячиванием
трансляции было указано в работе А.С. Спирина
домена P6 интрона группы I и взаимодействие
ещё в 1968 г. [86]. Хороший пример - межмоле
А сайт/кодон антикодон, принадлежат ти
кулярный А патч между 5S и 23S рРНК. Нали
пу I/IIP и являются аденин специфичными
чие такого А патча в структуре с PDB кодом
[24, 64]. В то же время мотивы, отличающие
4V9F (аденины A80, A103, A104 5S рРНК и A955,
комплементарные A U спаривания от компле
A1013, A1014 23S рРНК) не аннотируется прог
ментарных G C спариваний, такие как мотив
раммами DSSR и FR3D. DSSR размечает только
GAAA/11nt и А миноры между остатками GA
отдельные составляющие водородные связи и
16S рРНК и антикодоновым стемом инициатор
рибозные застёжки, в то время как программа
ной тРНК P сайта, либо включают основание G
FR3D улавливает только часть спариваний осно
вместо А, либо образуют конформацию ти
ваний и контактов типа основание рибоза с по
па I/IIT, которая требует дополнительных стаби
меткой «near» (рядом) - как не удовлетворяю
лизирующих взаимодействий [33, 78].
щие стандартным порогам значений, но включа
Отдельного упоминания заслуживают осо
емые при использовании расширенных порогов.
бенности динамики составляющих частей моти
Без сомнений, с ускорением темпов опреде
вов, отличные для разных комотивов А мино
ления новых 3D структур различных молекул
БИОХИМИЯ том 86 вып. 8 2021
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР
1157
РНК список функционально важных А минор
Финансирование. Работа не была финанси
взаимодействий будет быстро расти. Соответ
рована какими либо фондами или проектами.
ственно, существует необходимость как в усо
Конфликт интересов. Авторы заявляют об от
вершенствовании программ разметки разнооб
сутствии конфликта интересов.
разных типов А миноров, так и в разработке
Соблюдение этических норм. Настоящий об
возможности установления связей между А ми
зор не содержит описания каких либо исследо
норами и другими третичными мотивами РНК
ваний с участием людей или животных в качест
для улучшения их функциональной аннотации. ве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Lekka, E., and Hall, J. (2018) Noncoding RNAs in dis
16.
Nguyen, L. A., Wang, J., and Steitz, T. A. (2017) Crystal
ease, FEBS Lett., 592, 2884 2900, doi: 10.1002/1873
structure of Pistol, a class of self cleaving ribozyme,
3468.13182.
Proc. Natl. Acad. Sci. USA, 114, 1021 1026, doi: 10.1073/
2.
Novikova, I. V., Hennelly, S. P., Tung, C. S., and
pnas.1611191114.
Sanbonmatsu, K. Y. (2013) Rise of the RNA machines:
17.
Xue, S., Calvin, K., and Li, H. (2006) RNA recognition
exploring the structure of long non coding RNAs, J. Mol.
and cleavage by a splicing endonuclease, Science, 312, 906
Biol., 425, 3731 3746, doi: 10.1016/j.jmb.2013.02.030.
910, doi: 10.1126/science.1126629.
3.
Leontis, N. B., Lescoute, A., and Westhof, E. (2006) The
18.
Serganov, A., Yuan, Y. R., Pikovskaya, O., Polonskaia, A.,
building blocks and motifs of RNA architecture, Curr.
Malinina, L., et al. (2004) Structural basis for discrimina
Opin. Struct. Biol., 16, 279 287, doi: 10.1016/j.sbi.2006.
tive regulation of gene expression by adenine and guanine
05.009.
sensing mRNAs, Chem. Biol., 11, 1729 1741, doi: 10.1016/
4.
Nissen, P., Ippolito, J. A., Ban, N., Moore, P. B., and
j.chembiol.2004.11.018.
Steitz, T. A. (2001) RNA tertiary interactions in the large
19.
Dann, C. E. 3rd, Wakeman, C. A., Sieling, C. L., Baker,
ribosomal subunit: the A minor motif, Proc. Natl. Acad.
S. C., Irnov, I., and Winkler, W. C. (2007) Structure and
Sci. USA, 98, 4899 4903, doi: 10.1073/pnas.081082398.
mechanism of a metal sensing regulatory RNA, Cell, 130,
5.
Murphy, F. L., and Cech, T. R. (1994) GAAA tetraloop and
878 892, doi: 10.1016/j.cell.2007.06.051.
conserved bulge stabilize tertiary structure of a group I
20.
Jones, C. P., and Ferré D’Amaré, A. R. (2015) RNA qua
intron domain, J. Mol. Biol., 236, 49 63, doi: 10.1006/
ternary structure and global symmetry, Trends Biochem.
jmbi.1994.1117.
Sci., 40, 211 220, doi: 10.1016/j.tibs.2015.02.004.
6.
Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden,
21.
Brown, J. A., Bulkley, D., Wang, J., Valenstein, M. L.,
B. L., et al. (1996) Crystal structure of a group I ribozyme
Yario, T. A., et al. (2014) Structural insights into the stabi
domain: principles of RNA packing, Science, 273, 1678
lization of MALAT1 noncoding RNA by a bipartite triple
1685, doi: 10.1126/science.273.5282.1678.
helix, Nat. Struct. Mol. Biol., 21, 633, doi: 10.1038/
7.
Scott, W. G., Finch, J. T., and Klug, A. (1995) The crystal
nsmb.2844.
structure of an all RNA hammerhead ribozyme: a pro
22.
Klein, D. J., Schmeing, T. M., Moore, P. B., and Steitz,
posed mechanism for RNA catalytic cleavage, Cell, 81,
T. A. (2001) The kink turn: a new RNA secondary struc
991 1002, doi: 10.1016/S0092 8674(05)80004 2.
ture motif, EMBO J., 20, 4214 4221, doi: 10.1093/emboj/
8.
Nissen, P. (2020) The a minor motif, in Structural Insights
20.15.4214.
into Gene Expression and Protein Synthesis, pp. 461 463,
23.
Réblová, K., Šponer, J. E., Špačková, N., Beššeová, I., and
doi: 10.1142/9789811215865_0055.
Šponer, J. (2011) A minor tertiary interactions in RNA
9.
Doherty, E. A., Batey, R. T., Masquida, B., and Doudna,
kink turns. Molecular dynamics and quantum chemical
J. A. (2001) A universal mode of helix packing in RNA,
analysis, J. Phys. Chem. B,
115,
1389713910,
Nat. Struct. Biol., 8, 339 343, doi: 10.1038/86221.
doi: 10.1021/jp2065584.
10.
Strobel, S. A. (2002) Biochemical identification of A
24.
Geary, C., Baudrey, S., and Jaeger, L.
(2008)
minor motifs within RNA tertiary structure by interference
Comprehensive features of natural and in vitro selected
analysis, Biochem. Soc. Transact.,
30,
11261131,
GNRA tetraloop binding receptors, Nucleic Acids Res., 36,
doi: 10.1042/bst0301126.
1138 1152 10.1093/nar/gkm1048.
11.
Krasilnikov, A. S., Yang, X., Pan, T., and Mondragón, A.
25.
Wu, L., Chai, D., Fraser, M. E., and Zimmerly, S. (2012)
(2003) Crystal structure of the specificity domain of
Structural variation and uniformity among tetraloop
ribonuclease P, Nature, 421, 760764, doi: 10.1038/
receptor interactions and other loop helix interactions in
nature01386.
RNA crystal structures, PLoS One,
7, e49225,
12.
Krasilnikov, A. S., Xiao, Y., Pan, T., and Mondragón, A.
doi: 10.1371/journal.pone.0049225.
(2004) Basis for structural diversity in homologous RNAs,
26.
Fiore, J. L., and Nesbitt, D. J. (2013) An RNA folding
Science, 306, 104 107, doi: 10.1126/science.1101489.
motif: GNRA tetraloop-receptor interactions, Quart. Rev.
13.
Nagai, K., Oubridge, C., Kuglstatter, A., Menichelli, E.,
Biophys., 46, doi: 10.1017/S0033583513000048.
Isel, C., and Jovine, L. (2003) Structure, function and evo
27.
Aalberts, D. P., and Hodas, N. O. (2005) Asymmetry in
lution of the signal recognition particle, EMBO J., 22,
RNA pseudoknots: observation and theory, Nucleic Acids
3479 3485, doi: 10.1093/emboj/cdg337.
Res., 33, 2210 2214, doi: 10.1093/nar/gki508.
14.
Schüler, M., Connell, S. R., Lescoute, A., Giesebrecht, J.,
28.
Giedroc, D. P., and Cornish, P. V. (2009) Frameshifting
Dabrowski, M., et al. (2006) Structure of the ribosome
RNA pseudoknots: structure and mechanism, Virus Res.,
bound cricket paralysis virus IRES RNA. Nat. Struct. Mol.
139, 193 208, doi: 10.1016/j.virusres.2008.06.008.
Biol., 13, 1092 1096, doi: 10.1038/nsmb1177.
29.
Lescoute, A., and Westhof, E. (2006) Topology of three
15.
Mitton Fry, R. M., DeGregorio, S. J., Wang, J., Steitz,
way junctions in folded RNAs, RNA,
12,
8393,
T. A., and Steitz, J. A. (2010) Poly (A) tail recognition by a
doi: 10.1261/rna.2208106.
viral RNA element through assembly of a triple helix,
30.
Xin, Y., Laing, C., Leontis, N. B., and Schlick, T. (2008)
Science, 330, 1244 1247, doi: 10.1126/science.1195858.
Annotation of tertiary interactions in RNA structures
БИОХИМИЯ том 86 вып. 8 2021
1158
БАУЛИН
reveals variations and correlations, RNA, 14, 2465 2477,
49.
Hamdani, H. Y., Appasamy, S. D., Willett, P., Artymiuk,
doi: 10.1261/rna.1249208.
P. J., and Firdaus Raih, M. (2012) NASSAM: a server to
31.
Laing, C., and Schlick, T. (2009) Analysis of four way
search for and annotate tertiary interactions and motifs in
junctions in RNA structures, J. Mol. Biol., 390, 547 559,
three dimensional structures of complex RNA molecules,
doi: 10.1016/j.jmb.2009.04.084.
Nucleic Acids Res., 40, W35 W41, doi: 10.1093/nar/gks513.
32.
Cruz, J. A., and Westhof, E. (2009) The dynamic land
50.
Firdaus Raih, M., Hamdani, H. Y., Nadzirin, N.,
scapes of RNA architecture, Cell,
136,
604609,
Ramlan, E. I., Willett, P., and Artymiuk, P. J. (2014)
doi: 10.1016/j.cell.2009.02.003.
COGNAC: a web server for searching and annotating
33.
Geary, C., Chworos, A., and Jaeger, L. (2011) Promoting
hydrogen bonded base interactions in RNA three dimen
RNA helical stacking via A minor junctions, Nucleic Acids
sional structures, Nucleic Acids Res., 42, W382 W388,
Res., 39, 1066 1080, doi: 10.1093/nar/gkq748.
doi: 10.1093/nar/gku438.
34.
Tamura, M., and Holbrook, S. R. (2002) Sequence and
51.
Lu, X. J., Bussemaker, H. J., and Olson, W. K. (2015)
structural conservation in RNA ribose zippers, J. Mol.
DSSR: an integrated software tool for dissecting the spatial
Biol., 320, 455 474, doi: 10.1016/S0022 2836(02)00515 6.
structure of RNA, Nucleic Acids Res., 43, e142 e142,
35.
Lee, J. C., Cannone, J. J., and Gutell, R. R. (2003) The
doi: 10.1093/nar/gkv716.
lonepair triloop: a new motif in RNA structure, J. Mol.
52.
Yang, H., Jossinet, F., Leontis, N., Chen, L.,
Biol., 325, 65 83, doi: 10.1016/S0022 2836(02)01106 3.
Westbrook, J., et al. (2003) Tools for the automatic identi
36.
Gagnon, M. G., and Steinberg, S. V. (2010) The adenosine
fication and classification of RNA base pairs, Nucleic Acids
wedge: A new structural motif in ribosomal RNA, RNA, 16,
Res., 31, 3450 3460, doi: 10.1093/nar/gkg529.
375 381, doi: 10.1261/rna.1550310.
53.
Gendron, P., Lemieux, S., and Major, F. (2001) Quantit
37.
Leontis, N. B., and Westhof, E. (2001) Geometric nomen
ative analysis of nucleic acid three dimensional structures,
clature and classification of RNA base pairs, RNA, 7, 499
J. Mol. Biol., 308, 919 936, doi: 10.1006/jmbi.2001.4626.
512, doi: 10.1017/s1355838201002515.
54.
Lu, X. J., and Olson, W. K. (2008) 3DNA: a versatile, inte
38.
Torabi, S. F., Vaidya, A. T., Tycowski, K. T., DeGregorio,
grated software system for the analysis, rebuilding and visu
S. J., Wang, J., et al. (2021) RNA stabilization by a poly (A)
alization of three dimensional nucleic acid structures,
tail 3′ end binding pocket and other modes of poly (A)
Nat. Protoc., 3, 1213, doi: 10.1038/nprot.2008.104.
RNA interaction, Science, 371, doi: 10.1126/science.
55.
Shalybkova, A. A., Mikhailova, D. S., Kulakovskiy, I. V.,
abe6523.
Fakhranurova, L. I., and Baulin, E. F. (2021) Annotation
39.
Newby, M. I., and Greenbaum, N. L. (2002) Sculpting of
of the local context of the RNA secondary structure
the spliceosomal branch site recognition motif by a con
improves the classification and prediction of A minors,
served pseudouridine, Nat. Struct. Biol., 9, 958965,
RNA, rna 078535, doi: 10.1261/rna.078535.120.
doi: 10.1038/nsb873.
56.
Rázga, F., Koča, J., Šponer, J., and Leontis, N. B. (2005)
40.
Hamdani, H. Y., and Firdaus Raih, M.
(2019)
Hinge like motions in RNA kink turns: the role of the sec
Identification of structural motifs using networks of hydro
ond A minor motif and nominally unpaired bases,
gen bonded base interactions in RNA crystallographic
Biophys. J., 88, 3466 3485, doi: 10.1529/biophysj.104.
structures, Crystals, 9, 550, doi: 10.3390/cryst9110550.
054916.
41.
Lescoute, A., and Westhof, E. (2006) The A minor motifs
57.
Sponer, J., Bussi, G., Krepl, M., Banaš, P., Bottaro, S.,
in the decoding recognition process, Biochimie, 88, 993
et al. (2018) RNA structural dynamics as captured by mol
999, doi: 10.1016/j.biochi.2006.05.018.
ecular simulations: a comprehensive overview, Chem. Rev.,
42.
Lescoute, A., and Westhof, E. (2006) The interaction net
118, 4177 4338, doi: 10.1021/acs.chemrev.7b00427.
works of structured RNAs, Nucleic Acids Res., 34, 6587
58.
Laing, C., Wen, D., Wang, J. T., and Schlick, T. (2012)
6604, doi: 10.1093/nar/gkl963.
Predicting coaxial helical stacking in RNA junctions,
43.
Petrov, A. I., Zirbel, C. L., and Leontis, N. B. (2011)
Nucleic Acids Res., 40, 487 498, doi: 10.1093/nar/gkr629.
WebFR3D - a server for finding, aligning and analyzing
59.
Beššeová, I., Reblova, K., Leontis, N. B., and Šponer, J.
recurrent RNA 3D motifs, Nucleic Acids Res., 39, W50
(2010) Molecular dynamics simulations suggest that RNA
W55, doi: 10.1093/nar/gkr249.
three way junctions can act as flexible RNA structural ele
44.
Sheth, P., Cervantes Cervantes, M., Nagula, A., Laing, C.,
ments in the ribosome, Nucleic Acids Res., 38, 6247 6264,
and Wang, J. T. (2013) Novel features for identifying A
doi: 10.1093/nar/gkq414.
minors in three dimensional RNA molecules, Computat.
60.
Lescoute, A., and Westhof, E. (2005) Riboswitch struc
Biol. Chem., 47, 240 245, doi: 10.1016/j.compbiolchem.
tures: purine ligands replace tertiary contacts, Chem. Biol.,
2013.10.004.
12, 10 13, doi: 10.1016/j.chembiol.2005.01.002.
45.
Laing, C., Jung, S., Iqbal, A., and Schlick, T. (2009)
61.
Baulin, E., Yacovlev, V., Khachko, D., Spirin, S., and
Tertiary motifs revealed in analyses of higher order RNA
Roytberg, M. (2016) URS DataBase: universe of RNA
junctions, J. Mol. Biol., 393, 67 82, doi: 10.1016/j.jmb.
structures and their motifs, Database, 2016, doi: 10.1093/
2009.07.089.
database/baw085.
46.
Burley, S. K., Berman, H. M., Kleywegt, G. J., Markley,
62.
Calvin, K., and Li, H. (2008) RNA splicing endonuclease
J. L., Nakamura, H., and Velankar, S. (2017) Protein Data
structure and function, Cell. Mol. Life Sci., 65, 1176 1185,
Bank (PDB): the single global macromolecular structure
doi: 10.1007/s00018 008 7393 y.
archive, Protein Crystallogr., 1607, 627 641, doi: 10.1007/
63.
Ikawa, Y., Yoshimura, T., Hara, H., Shiraishi, H., and
978 1 4939 7000 1_26.
Inoue, T. (2002) Two conserved structural components, A
47.
Reinharz, V., Soulé, A., Westhof, E., Waldispühl, J., and
rich bulge and P4 XJ6/7 base triples, in activating the
Denise, A. (2018) Mining for recurrent long range interac
group I ribozymes, Genes Cells,
7,
12051215,
tions in RNA structures reveals embedded hierarchies in
doi: 10.1046/j.1365 2443.2002.00601.x.
network families, Nucleic Acids Res., 46, 38413851,
64.
Battle, D. J., and Doudna, J. A. (2002) Specificity of
doi: 10.1093/nar/gky197.
RNA-RNA helix recognition, Proc. Natl. Acad. Sci. USA,
48.
Appasamy, S. D., Hamdani, H. Y., Ramlan, E. I., and
99, 11676 11681, doi: 10.1073/pnas.182221799.
Firdaus Raih, M. (2016) InterRNA: a database of base
65.
Schwalbe, H., Buck, J., Fürtig, B., Noeske, J., and
interactions in RNA structures, Nucleic Acids Res., 44,
Wöhnert, J. (2007) Structures of RNA switches: insight
D266 D271, doi: 10.1093/nar/gkv1186.
into molecular recognition and tertiary structure,
БИОХИМИЯ том 86 вып. 8 2021
СВОЙСТВА И ФУНКЦИИ МОТИВА А МИНОР
1159
Angewandte Chemie Int. Edn., 46, 1212 1219, doi: 10.1002/
75. Steitz, T. A., and Moore, P. B. (2003) RNA, the first
anie.200604163.
macromolecular catalyst: the ribosome is a ribozyme,
66.
Šponer, J. E., Leszczynski, J., Sychrovský, V., and
Trends Biochem. Sci., 28, 411 418, doi: 10.1016/S0968
Šponer, J. (2005) Sugar edge/sugar edge base pairs in
0004(03)00169 5.
RNA: stabilities and structures from quantum chemical
76. Noller, H. F. (2012) Evolution of protein synthesis from an
calculations, J. Phys. Chem. B,
109,
1868018689,
RNA world, Cold Spring Harb. Perspect. Biol., 4, a003681,
doi: 10.1021/jp053379q.
doi: 10.1101/cshperspect.a003681.
67.
Šponer, J. E., Reblova, K., Mokdad, A., Sychrovský, V.,
77. Lancaster, L., and Noller, H. F. (2005) Involvement of 16S
Leszczynski, J., and Šponer, J. (2007) Leading RNA ter
rRNA nucleotides G1338 and A1339 in discrimination of
tiary interactions: structures, energies, and water insertion
initiator tRNA, Mol. Cell, 20, 623 632, doi: 10.1016/j.mol
of A minor and P interactions. A quantum chemical view,
cel.2005.10.006.
J. Phys. Chem. B, 111, 9153 9164, doi: 10.1021/jp0704261.
78. Steitz, T. A. (2008) A structural understanding of the
68.
Costa, M., and Michel, F. (1995) Frequent use of the same
dynamic ribosome machine, Nat. Rev., Mol. Cell Biol., 9,
tertiary motif by self folding RNAs, EMBO J., 14, 1276
242 253, doi: 10.1038/nrm2352.
1285, doi: 10.1002/j.1460 2075.1995.tb07111.x.
79. Hansen, J. L., Schmeing, T. M., Moore, P. B., and Steitz,
69.
Lee, J. C., Gutell, R. R., and Russell, R. (2006) The
T. A. (2002) Structural insights into peptide bond forma
UAA/GAN internal loop motif: a new RNA structural ele
tion, Proc. Natl. Acad. Sci. USA, 99, 1167011675,
ment that forms a cross strand AAA stack and long range
doi: 10.1073/pnas.172404099.
tertiary interactions, J. Mol. Biol.,
360,
978988,
80. Noller, H. F. (2005) RNA structure: reading the ribosome,
doi: 10.1016/j.jmb.2006.05.066.
Science, 309, 1508 1514, doi: 10.1126/science.1111771.
70.
Yoshizawa, S., Fourmy, D., and Puglisi, J. D. (1999) Re
81. Szymański, M., Barciszewska, M. Z., Erdmann, V. A., and
cognition of the codon anticodon helix by ribosomal
Barciszewski, J. (2003) 5S rRNA: structure and interac
RNA, Science, 285, 1722 1725, doi: 10.1126/science.
tions, Biochem. J., 371, 641 651, doi: 10.1042/bj20020872.
285.5434.1722.
82. Mohan, S., and Noller, H. F. (2017) Recurring RNA struc
71.
Ogle, J. M., Brodersen, D. E., Clemons, W. M., Tarry, M. J.,
tural motifs underlie the mechanics of L1 stalk movement,
Carter, A. P., and Ramakrishnan, V. (2001) Recognition of
Nat. Commun., 8, 1 11, doi: 10.1038/ncomms14285.
cognate transfer RNA by the 30S ribosomal subunit,
83. Bou Nader, C., and Zhang, J. (2020) Structural insights
Science, 292, 897 902, doi: 10.1126/science.1060612.
into RNA dimerization: Motifs, interfaces and functions,
72.
Ogle, J. M., and Ramakrishnan, V. (2005) Structural
Molecules, 25, 2881, doi: 10.3390/molecules25122881.
insights into translational fidelity, Annu. Rev. Biochem., 74,
84. Leontis, N. B., and Westhof, E. (2003) Analysis of RNA
129 177, doi: 10.1146/annurev.biochem.74.061903.155440.
motifs, Curr. Opin. Struct. Biol.,
13,
300308,
73.
Gromadski, K. B., Daviter, T., and Rodnina, M. V. (2006)
doi: 10.1016/S0959 440X(03)00076 9.
A uniform response to mismatches in codon anticodon
85. Frank, J., Gao, H., Sengupta, J., Gao, N., and Taylor,
complexes ensures ribosomal fidelity, Mol. Cell, 21, 369
D. J. (2007) The process of mRNA-tRNA translocation,
377, doi: 10.1016/j.molcel.2005.12.018.
Proc.Natl. Acad. Sci. USA, 104, 19671 19678, doi: 10.1073/
74.
Prokhorova, I., Altman, R. B., Djumagulov, M., Shrestha,
pnas.0708517104.
J. P., Urzhumtsev, A., et al. (2017) Aminoglycoside inter
86. Spirin, A. S. (1968) How does the ribosome work? A
actions and impacts on the eukaryotic ribosome, Proc.
hypothesis based on the two subunit construction of the
Natl. Acad. Sci. USA, 114, E10899 E10908, doi: 10.1073/
ribosome, Curr. Mod. Biol., 2, 115 127, doi: 10.1016/0303
pnas.1715501114.
2647(68)90017 8.
FEATURES AND FUNCTIONS OF THE A MINOR MOTIF,
THE MOST COMMON MOTIF OF RNA STRUCTURE
Review
E. F. Baulin1,2
1 Institute of Mathematical Problems of Biology RAS - the Branch of Keldysh Institute of Applied Mathematics
of Russian Academy of Sciences, 142290 Pushchino, Moscow Region, Russia; e mail: baulin@lpm.org.ru
2 Moscow Institute of Physics and Technology, 141701 Dolgoprudny, Moscow Region, Russia
A minor motifs are RNA tertiary motifs generally involving a canonical base pair and an adenine base forming hydro
gen bonds with the minor groove of the base pair. Such motifs are among the most numerous tertiary interactions in
known RNA structures, comparable in number with non canonical base pairs. They are often found within function
ally important regions of non coding RNA molecules and, in particular, play a central role in protein synthesis. Here,
we review local variations of A minor geometry, present annotation difficulties, various structural contexts and com
mon A minor co motifs, and the diverse functions that A minors perform in a wide range of processes in a living cell.
Keywords: RNA structure, tertiary motif, A minor interaction, A minor motif, A patch
БИОХИМИЯ том 86 вып. 8 2021