БИОХИМИЯ, 2021, том 86, вып. 8, с. 1160 - 1175
УДК 577.218
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ:
НОВЫЕ ТРЮКИ ДЛЯ СТАРОЙ СОБАКИ
Обзор
© 2021
A. Ariza Mateos, A. Nuthanakanti, Alexander Serganov*
Department of Biochemistry and Molecular Pharmacology, New York University Grossman School of Medicine,
New York, New York 10016, USA; e mail: alexander.serganov@nyulangone.org
Поступила в редакцию 01.05.2021
После доработки 01.05.2021
Принята к публикации 19.05.2021
Обнаруженные почти двадцать лет назад рибопереключатели оказались одной из самых распространенных
регуляторных систем у бактерий, они встречаются как у эукариот, так и у архей. В отличие от многих других
регуляторных элементов, рибопереключатели полностью состоят из РНК и способны модулировать
экспрессию генов в клетке путём прямого связывания небольших молекул. Первоначально считалось, что
бактериальные рибопереключатели через регуляторные цепи обратной связи контролируют синтез только
тех ферментов и транспортёров, которые связаны с небольшими органическими молекулами. Однако более
поздние открытия выявили рибопереключатели, управляющие экспрессией широкого спектра генов и от)
вечающие на различные классы молекул, включая ионы, сигнальные молекулы и другие. Подавляющее
большинство рибопереключателей находятся в 5′)нетранслируемой области мРНК. Рибопереключатели
модулируют транскрипцию или трансляцию нижестоящих генов посредством конформационных перестро)
ек в лиганд)чувствительных доменах и примыкающих к ним платформах, контролирующих экспрессию.
С годами набор изученных регуляторных механизмов, используемых рибопереключателями, значительно
расширился; самые последние исследования подчеркнули важность альтернативных механизмов, таких как
деградация РНК, для генетических цепей, управляемых рибопереключателями. В этом обзоре обсуждается
множество бактериальных механизмов рибопереключения и показано, каким образом рибопереключатели
используют характерные особенности и способы для получения различных регуляторных ответов.
КЛЮЧЕВЫЕ СЛОВА: рибопереключатель, транскрипция, трансляция, мРНК, метаболит.
DOI: 10.31857/S032097252108008X
ВВЕДЕНИЕ
шающей пороговое значение, ген может быть
отключён благодаря чувствительности к про)
Большинство бактерий живут в постоянно
дукту этого гена. Этот принцип регуляции, ког)
меняющихся условиях окружающей среды и
да молекула контролирует свое собственное
должны быстро корректировать свой метабо)
производство, получил название саморегуля)
лизм в соответствии с доступностью питатель)
ция. Саморегуляция не обязательно требует из)
ных веществ и другими внешними факторами.
менений в окружающей среде. Гомеостатичес)
Бактерии активируют экспрессию генов для
кое поддержание определенных уровней генно)
производства белков и РНК, необходимых для
го продукта и предотвращение накопления не)
максимальной адаптации к окружающей среде.
нужных молекул могут быть одинаково важны
Чтобы сэкономить ресурсы, бактерии также
для нормальной работы клетки в обычных усло)
отключают гены, продукты которых в данный
виях. Саморегуляция может быть достигнута с
момент не требуются. Контроль генов часто дос)
помощью механизмов на разных уровнях, вклю)
тигается за счёт регулирования конечным про)
чая прямое подавление транскрипции или
дуктом по механизму обратной связи: когда мо)
трансляции и многие посттранскрипционные и
лекула синтезируется в концентрации, превы)
посттрансляционные механизмы, такие как ко)
валентные модификации, ингибирование фер)
ментативной активности и другие.
Принятые сокращения: SD - последовательность
Шайн-Дальгарно; SAM - S)аденозилметионин; PRPP -
Помимо механизмов регуляции с помощью
фосфорибозилпирофосфат; c)di)GMP - циклический ди)
белков, являющихся конечными продуктами
гуанилат.
работы генов, многочисленные примеры регу)
* Адресат для корреспонденции.
ляции по принципу механизма обратной связи
1160
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1161
включают небольшие метаболиты, продуцируе)
большинство рибопереключателей обеспечива)
мые белками в качестве конечных продуктов.
ют регуляцию по принципу механизма обратной
В этом случае небольшие молекулы восприни)
связи, контролируя уровни транскрипции или
маются и связываются с белками, а образующи)
трансляции. Рибопереключатели обычно состо)
еся комплексы модулируют, например, тран)
ят из двух частей: эволюционно консервативно)
скрипцию генов посредством связывания с опе)
го «аптамерного» домена, воспринимающего
раторами транскрипции [1]. Чтобы реагировать
метаболит, и вариабельной «платформы экс)
на множество клеточных метаболитов и контро)
прессии», содержащей сигналы экспрессии ге)
лировать экспрессию генов, ассоциированных с
нов (рис. 1). Различные платформы экспрессии
этими метаболитами, клеткам пришлось бы вы)
могут сопровождать один и тот же тип сенсор)
рабатывать десятки белковых репрессоров и ак)
ного домена, но с разными результатами
тиваторов, по одному для каждого гена или опе)
экспрессии генов.
рона. Хотя такой подход возможен, он приведёт
Способность рибопереключателей прини)
к неэффективной трате ресурсов клетки. Может
мать две взаимоисключающие конформации
ли регулирование с помощью небольшой моле)
РНК определяет основу генетического ответа.
кулы происходить без посредника, чувствитель)
Специфичное связывание метаболитов вызыва)
ного к нему белка?
ет изменение конформации сенсора метаболита
Небольшая молекула ppGpp может напря)
и платформы экспрессии, что вызывает один ге)
мую связываться с РНК)полимеразой и рибосо)
нетический ответ; в то же время адаптация к аль)
мой, мРНК и другими структурами, влияющи)
тернативной конформации приводит к противо)
ми на синтез белков, и модулировать их актив)
положному эффекту (рис. 1). Рибопереключате)
ность во время адаптации к нехватке аминокис)
ли включают более 40 классов, которые распоз)
лот [2]. Однако (p)ppGpp - это вторичный мес)
нают около 30 различных небольших молекул,
сенджер, синтезируемый специальными фер)
от ионов до витаминов [8]. Молекулярной осно)
ментами, а не конечный продукт работы фер)
вой специфичности сенсоров является форми)
ментов, продуцирующих аминокислоты. Хотя
рование сложных трёхмерных структур, настро)
прямое связывание с полимеразой и рибосомой
енных на точное распознавание лиганда или ли)
представляет собой удобный и эффективный
гандов и отторжение похожих метаболитов и
способ контроля экспрессии генов, эти клеточ)
предшественников [9]. Поскольку размер апта)
ные механизмы сами по себе не могут воспри)
меров (т.е. его лиганд)узнающих частей) состав)
нимать десятки различных клеточных метабо)
ляет примерно 30-250 нуклеотидов, их структу)
литов. Может ли небольшая молекула связы)
ра и особенности распознавания метаболитов
ваться с ДНК или мРНК, чтобы модулировать
сильно различаются. Напротив, платформы
транскрипцию или трансляцию? В то время как
экспрессии чаще всего сворачиваются в шпиль)
двуцепочечная геномная ДНК не обладает дос)
ки, которые функционируют как терминаторы
таточной сложностью для специфического рас)
транскрипции, антитерминаторы или структу)
познавания небольшой молекулы, РНК может
ры, препятствующие связыванию рибосомы.
сворачиваться в сложные трёхмерные структуры
Недавние исследования определили или
для специфического связывания низкомолеку)
предложили концептуально новые механизмы
лярных соединений [3]. Следовательно, модуля)
рибопереключателей и обнаружили множество
ция экспрессии гена с помощью небольшой мо)
вариаций общих механизмов, тем самым значи)
лекулы кажется возможной, если молекула свя)
тельно расширив репертуар функционирования
зывается с мРНК и аллостерически изменяет её
структуру, чтобы скрыть или выявить генные ре)
гуляторные сигналы, встроенные в последова)
тельность мРНК. Именно эта идея почти
20 лет назад привела к открытию рибопереклю)
чателей.
Термин «рибопереключатели» был придуман
для регуляторных участков РНК, способных мо)
дулировать экспрессию генов посредством пря)
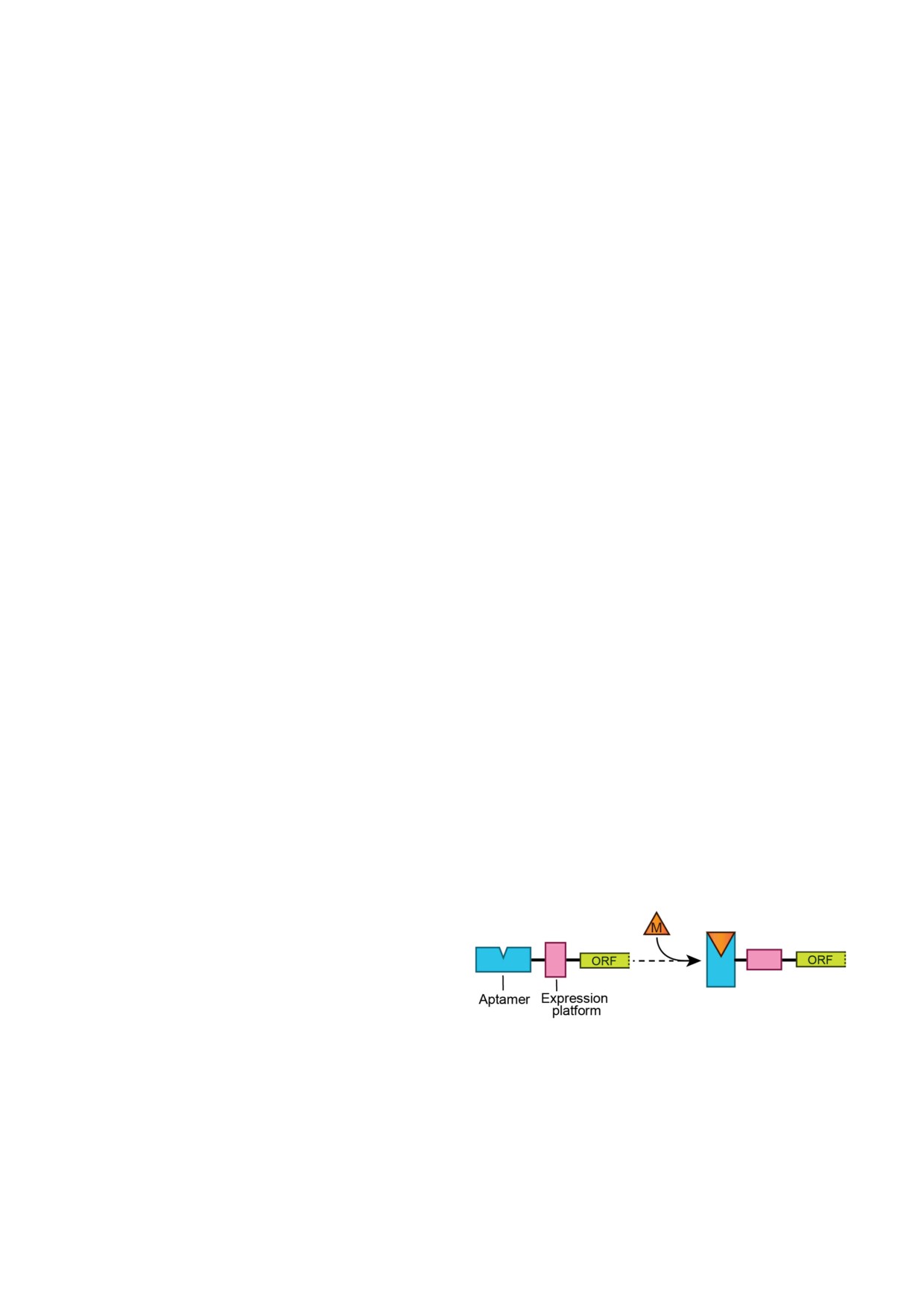
Рис. 1. Домены типичного рибопереключателя. Чувстви)
мого восприятия клеточных метаболитов [4-6].
тельный к метаболитам аптамерный домен, платформа
Рибопереключатели обычно располагаются в 5′)
экспрессии и открытая рамка считывания (ORF) гена по)
казаны прямоугольниками. Связывание метаболита (M)
нетранслируемых областях (5′)НТО) мРНК ге)
стабилизирует альтернативную конформацию доменов ри)
нов, участвующих в метаболизме или транспор)
бопереключателя. Две конформации рибопереключателя
те лигандов рибопереключателей или родствен)
вызывают два противоположных регуляторных ответа
ных клеточных метаболитов [7]. Подавляющее
(не показаны)
5 БИОХИМИЯ том 86 вып. 8 2021
1162
ARIZA)MATEOS et al.
рибопереключателей. Чтобы проиллюстриро)
дениями, что тиамин, рибофлавин и кобаламин
вать универсальность рибопереключателей в ге)
ингибируют гены биосинтеза витаминов B1, B2
нетическом контроле, в этом обзоре рассматри)
и B12 соответственно. мРНК этих генов содер)
ваются типичные механизмы рибопереключате)
жит консервативные регуляторные последова)
лей и обсуждаются несколько новых открытий и
тельности или «боксы»; однако не было обнару)
гипотез.
жено специфичного для этих боксов белкового
репрессора [17, 18], что свидетельствует о пря)
мом связывании витаминов с мРНК [17, 19-21].
ДОРОГА, ВЕДУЩАЯ К ОТКРЫТИЮ
После неудачных попыток [22, 23] было убеди)
РИБОПЕРЕКЛЮЧАТЕЛЕЙ
тельно показано, что три производных витами)
нов, аденозилкобаламин (AdoCbl) [4], тиамин)
Хотя открытие рибопереключателей стало
пирофосфат (TPP) [5, 6] и флавинмононуклео)
неожиданностью для многих исследователей,
тид (FMN) [6], связывают соответствующие
ряд экспериментов подготовил почву для их об)
мРНК, модулируют конформацию РНК и регу)
наружения. Одним из важных факторов было
лируют экспрессию генов. После обнаружения
открытие обратной связи с помощью белковых
рибопереключателей в бактериях последовали
авторепрессоров, таких как рибосомные бел)
примеры из других форм жизни, хотя активные
ки [10]. Чтобы сбалансировать биосинтез рРНК
эукариотические рибопереключатели почти не
и рибосомных белков для сборки рибосомы,
распространялись и представлены только связу)
несколько рибосомных белков, синтезируемых
ющими TPP [24-26]. Напротив, многие виды
в избытке по сравнению с рРНК, могут связы)
бактерий обладают рибопереключателями, мно)
ваться с 5′)НТО своей собственной мРНК и
гие бактерии содержат различные классы рибо)
предотвращать инициацию трансляции за счёт
переключателей, а некоторые виды имеют не)
секвестирования сайта связывания рибосомы и
сколько рибопереключателей одного и того же
кодона инициации. Мишени мРНК и рРНК
класса, участвующих в контроле различных ге)
этих белков имеют некоторое сходство либо на
нов и оперонов [8].
уровне последовательности, либо на структур)
ном уровне, но сродство связывания рРНК всег)
да превышает сродство связывания мРНК, что)
КАНОНИЧЕСКИЕ МЕХАНИЗМЫ
бы гарантировать надлежащий контроль [11].
РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
Контроль с помощью механизма обратной связи
не ограничивается одними белками. Так назы)
Первые сообщения об идентификации ри)
ваемые «Т)боксы» в мРНК специфически свя)
бопереключателей, которые содержат опреде)
зывают тРНК и, в зависимости от присутствия
ленный сенсор метаболита и платформу экс)
на тРНК ковалентно присоединенной амино)
прессии, сразу предложили четыре основных ме)
кислоты, способствуют сворачиванию нижеле)
ханизма контроля экспрессии генов (рис. 2) [7].
жащих областей в терминаторы транскрипции
Связывание метаболитов стабилизирует общую
или антитерминаторы, напоминающие контро)
структуру сенсорного домена и облегчает обра)
лирующие элементы транскрипционных рибо)
зование и стабилизацию спирали, которая
переключателей [12]. Специфическое связыва)
обычно блокирует сенсорный домен. Эта регу)
ние небольшой молекулы с РНК также не было
ляторная спираль, обозначенная как «Спарива)
новым наблюдением. Связывание гуанозиново)
ние 1» (P1), включает область, обычно находя)
го кофактора самосплайсирующимся интроном
щуюся на 3′)конце, способную к альтернатив)
группы I было описано почти за два десятилетия
ному спариванию оснований с комплементар)
до открытия рибопереключателей [12]. Селек)
ной последовательностью, которая располагает)
ция in vitro РНК)аптамеров, специфичных для
ся ниже, когда метаболит не связан с доменом)
различных малых молекул, укрепила мнение о
сенсором. Альтернативное спаривание основа)
том, что РНК может избирательно распознавать
ний спирали P1 является квинтэссенцией регу)
небольшие молекулы с различными характерис)
ляции для большинства рибопереключателей;
тиками [13-15]. Связывание аминокислот с
однако клеточные процессы контроля и задей)
мРНК и их участие в регуляции также наблюда)
ствованные механизмы сильно различаются.
ли в случаях аттенуации, когда белки и неболь)
У бактерий канонические механизмы рибопе)
шие молекулы коллективно перестраивают
реключения основаны на терминации тран)
структуру мРНК и вызывают терминацию тран)
скрипции и инициации трансляции.
скрипции [16].
Первые два механизма рассчитаны на спо)
Идентификация первых рибопереключате)
собность РНК)полимеразы «оглядываться» на
лей была в первую очередь обусловлена наблю)
транскрибируемую мРНК и прекращать тран)
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1163
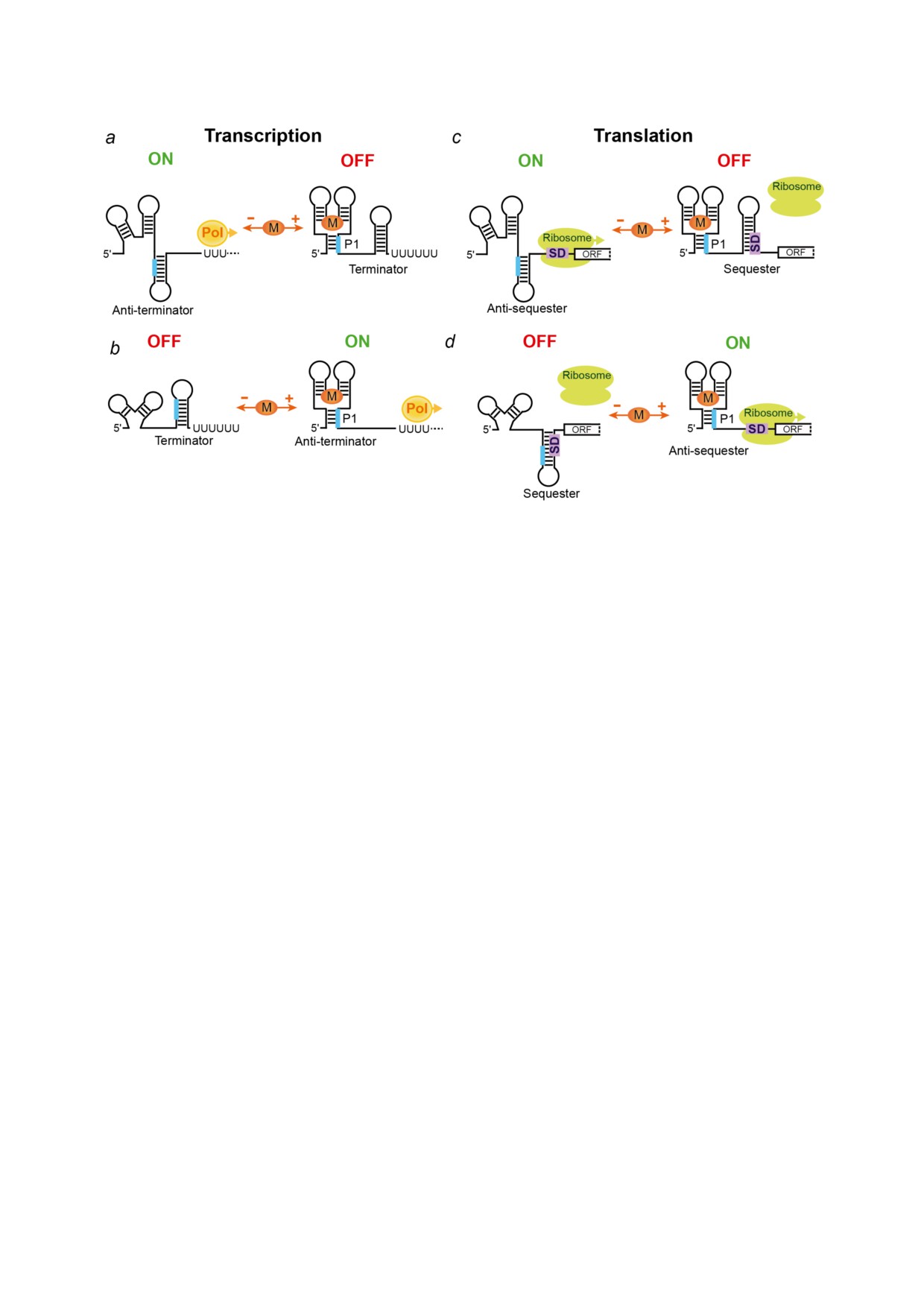
Рис. 2. Канонические механизмы рибопереключателей. а - Ингибирование экспрессии генов через механизм термина)
ции транскрипции. При отсутствии метаболита домен аптамера не сворачивается, а рибопереключатель образует шпиль)
ку)антитерминатор. РНК)полимераза (Pol) транскрибирует весь ген, включая его экспрессируемую часть. Связывание
метаболита (M) стабилизирует связанную с лигандом форму, закрытую спиралью P1, и способствует образованию внут)
реннего терминатора транскрипции, тем самым выключая экспрессию гена. Фрагмент рибопереключателя (жирная ли)
ния) участвует в двух альтернативных соединениях оснований в безлигандном и связанном с лигандом состояниях рибо)
переключателя. b - Активация экспрессии гена через механизм терминации транскрипции. При отсутствии метаболита
рибопереключатель образует терминатор транскрипции. В присутствии метаболита фрагмент, участвующий в образова)
нии терминатора, образует альтернативное спаривание оснований внутри P1, которое блокирует связанный с метаболи)
том аптамер. с - Подавление трансляции связыванием метаболитов. Состояние без метаболитов позволяет рибосомам
связываться с последовательностью Шайн-Дальгарно (SD) (прямоугольник с надписью «SD») и инициировать трансля)
цию. После связывания лиганда последовательности SD изолируются в структуре шпильки, и трансляция не может ини)
циироваться. d - Активация трансляции. Связывание метаболитов приводит к регуляторному ответу, противоположному
ответу, показанному на панели c
скрипцию после синтеза внутреннего термина)
цесс регуляции, этот процесс не может быть от)
тора - самокомплементарной области РНК, ко)
менён. РНК)полимераза не может удлинить
торая сворачивается в шпильку, за которой сле)
терминированную РНК и не может остановить
дует отрезок из остатков уридиловой кисло)
транскрипцию после прохождения через после)
ты (рис. 2, а и b). Такие внутренние терминато)
довательность терминатора, если шпилька тер)
ры транскрипции могут образовываться в при)
минатора не была образована.
сутствии или в отсутствие метаболита, что при)
Два других классических механизма рибопе)
водит к репрессии или активации транскрипции
реключения основаны на том факте, что рибо)
соответственно. Преждевременное прекраще)
сомная субъединица 30S требует спаривания ос)
ние транскрипции предотвращает производство
нований между сайтом связывания 16S рРНК
мРНК для трансляции и, следовательно, эконо)
рибосомы (последовательность Шайн-Даль)
мит нуклеотиды и другие ресурсы, необходимые
гарно или последовательности SD) и неспарен)
для создания мРНК. Неудивительно, что терми)
ным инициирующим кодоном для эффектив)
нация транскрипции является преобладающим
ной инициации трансляции (рис. 2, c и d). Спа)
механизмом работы рибопереключателей и час)
ривание оснований SD)последовательности и
то используется для контроля по принципу от)
кодона инициации трансляции внутри шпильки
рицательной обратной связи. Интересно, что
может предотвратить загрузку рибосомы и
механизм транскрипции, опосредованный ри)
трансляцию мРНК. Освобождение последова)
бопереключателем, не включает истинное «пе)
тельности SD и кодона инициации позволит ри)
реключение» рибопереключателя и может быть
босоме связываться и транслировать мРНК. Ре)
лучше описан как «молекулярное слияние» [27].
гулирование уровня трансляции требует тран)
В самом деле, как только РНК вступает в про)
скрипции по крайней мере 5′)конца мРНК и ка)
БИОХИМИЯ том 86 вып. 8 2021
5*
1164
ARIZA)MATEOS et al.
жется менее экономичным, чем контроль тран)
ность SD. В этих рибопереключателях не суще)
скрипции. Однако регулирование трансляции
ствует платформы экспрессии, а последователь)
имеет свои преимущества. Во)первых, это мо)
ность SD можно рассматривать как неотъемле)
жет позволить контролировать отдельный ген в
мую часть связанного с лигандом аптамерного
полицистронной мРНК, если ген транслируется
домена. Например, в рибопереключателе SAM)III
с помощью собственной последовательнос)
[29] связывание лиганда происходит с участием
ти SD. Во)вторых, время отклика для контроля
SD)последовательности при спаривании осно)
трансляции может быть очень быстрым, пос)
ваний в сочленении спиралей [30]. Последова)
кольку мРНК уже синтезирована. В случае акти)
тельность SD становится недоступной для свя)
вации трансляция может начаться сразу после
зывания с рибосомой, и трансляция не может
освобождения последовательности SD. Нако)
инициироваться.
нец, трансляционные рибопереключатели могут
Участие SD)последовательности в сворачи)
быть настоящими переключателями [28], связы)
вании связанного с лигандом рибопереключате)
вающими и высвобождающими метаболит, а
ля особенно важно для аптамеров, принимаю)
также активирующими или подавляющими
щих так называемую структуру псевдоузла.
трансляцию одной и той же молекулы мРНК в
В нескольких рибопереключателях, таких как
зависимости от колебаний концентрации мета)
SAM)II, биохимические и структурные исследо)
болита. Однако рибопереключатели обычно не
вания подтвердили биоинформатически предс)
используют эту опцию, потому что большин)
казанное удалённое спаривание оснований
ство рибопереключателей управляются кинети)
между удалёнными участками последователь)
чески, а не термодинамически; другими слова)
ности SD и внутренней петлёй в середине апта)
ми, для активации им требуется концентрация
мера [31, 32]. Это третичное спаривание приво)
метаболита выше, чем KD [27]. Кинетически уп)
дит к формированию структуры псевдоузла, ко)
равляемые трансляционные рибопереключате)
торая включает две коаксиально уложенные
ли могут выбирать регуляторный путь сразу пос)
друг на друга спирали, соединённые двумя пет)
ле того, как 5′)конец мРНК синтезирован. Нап)
лями (рис. 3, а). Предполагают, что и другие
ротив, достижение термодинамического равно)
репрессоры трансляции, которые, по прогно)
весия может быть длительным процессом,
зам, формируют сходные спаривания между
приближающимся ко времени жизни мРНК или
удалёнными участками, также образуют струк)
превышающим его, и делающим ответ «устарев)
туры псевдоузлов при связывании лиганда [33].
шим». Поскольку транскрипция и трансляция у
В отличие от трансляционных рибопереклю)
бактерий связаны, трансляция начинается вско)
чателей количество примеров оптимизирован)
ре после инициации транскрипции, до заверше)
ных транскрипционных рибопереключателей
ния транскрипции.
ограничено [34, 35]. Рибопереключатель гуани)
дин)IV использует петлю шпильки терминатора
для формирования структуры аптамера за счёт
ПЕРЕКЛЮЧЕНИЯ В КАНОНИЧЕСКИХ
спаривания оснований с внутренней петлёй в
МЕХАНИЗМАХ РАБОТЫ
теле аптамера (рис. 3, b) [34]. Следовательно,
РИБОПЕРЕКЛЮЧАТЕЛЯ
связывание лиганда нарушает структуру терми)
натора и включает транскрипцию. В отсутствие
Многие рибопереключатели, использующие
гуанидина образование терминатора тран)
канонические регуляторные механизмы, содер)
скрипции не затруднено, и транскрипция преж)
жат отдельный аптамерный домен и платформу
девременно прерывается.
экспрессии, каждый из которых способен к сво)
Рибопереключатель, отвечающий на предшест)
рачиванию альтернативно в зависимости от дос)
венник тиаминпирофосфата (TPP), HMP)PP,
тупности лиганда рибопереключателя. Для эф)
демонстрирует ещё более необычную архитекту)
фективной регуляции такое расположение двух
ру, в которой очень маленький лиганд)чувстви)
доменов не является абсолютно необходимым
тельный аптамер почти полностью встроен в
требованием, и некоторые рибопереключатели
классический терминатор внутренней тран)
объединяют оба элемента, сохраняя при этом ре)
скрипции (рис. 3, c) [35]. Рибопереключатель
гуляторный ответ «двух состояний» нетронутым.
использует одну и ту же последовательность
Поскольку для инициации трансляции не
РНК для сворачивания в две взаимоисключаю)
требуется структура, присутствующая в мРНК,
щие структуры: конфигурацию терминатора без
многие репрессоры трансляции представляют
лиганда, преждевременно прерывающую тран)
собой оптимизированные рибопереключатели,
скрипцию, и конфигурацию связанного с ли)
у которых отсутствует выделенная шпилька,
гандом аптамера, предотвращая раннюю терми)
изолирующая или открывающая последователь)
нацию транскрипции.
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1165
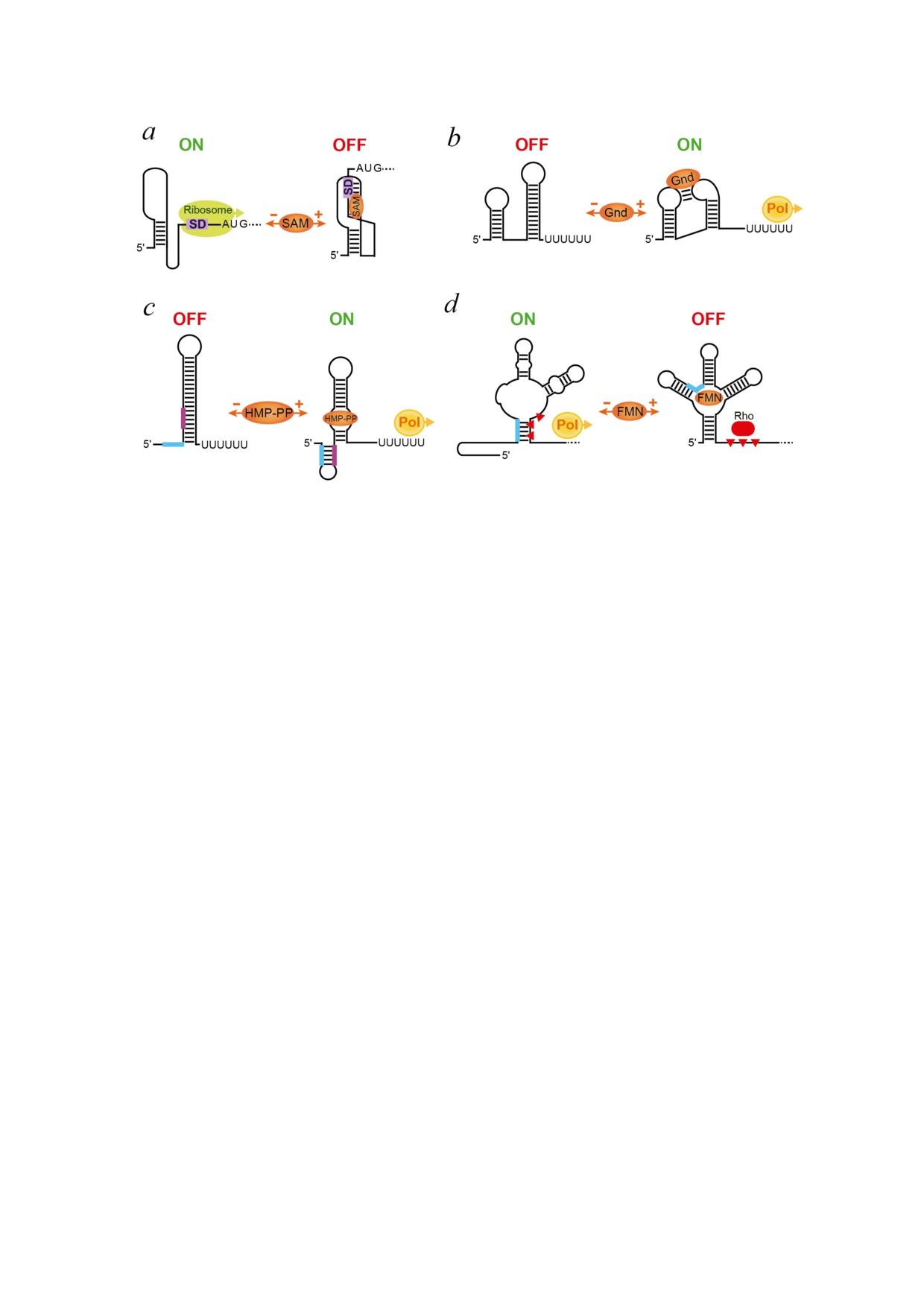
Рис. 3. Оптимизированные и неканонические механизмы работы рибопереключателей. а - Регуляция инициации транс)
ляции рибопереключателем SAM)II [31, 101]. Связывание с SAM стабилизирует фолдинг псевдоузла аптамера. Последо)
вательность SD участвует в формировании псевдоузла и не может быть доступна для рибосомы. При отсутствии SAM
псевдоузел не образуется, рибосома может связываться с последовательностью SD и инициировать трансляцию мРНК.
b - Предполагаемый механизм активации транскрипции рибопереключателя гуанидин)IV, включающий третичные взаи)
модействия между аптамерным доменом и платформой экспрессии [34]. При связывании лиганда структура терминатора
транскрипции нарушается за счёт разъединения оснований на расстояние больше необходимого для образования струк)
туры «аптамера». Петли содержат консервативные нуклеотиды, участвующие в третичных взаимодействиях. Терминатор
транскрипции обычно формируется в отсутствие гуанидина. c - Активация транскрипции рибопереключателем HMP)PP [35].
В отсутствие лиганда рибопереключатель сворачивается в канонический терминатор транскрипции. Связывание лиганда
способствует образованию альтернативной конформации, разрушающей терминатор. Толстыми линиями обозначены
ключевые фрагменты, участвующие в формировании альтернативных структур. d - Предполагаемый механизм Rho)зави)
симой репрессии транскрипции рибопереключателя FMN из Escherichia coli [36]. В конформации, связанной с FMN, фак)
тор Rho способствует прекращению транскрипции С)богатой области, расположенной ниже аптамера. При отсутствии
лиганда фрагмент в 5′)половине аптамера соединяется с С)богатой областью, тем самым препятствуя терминации тран)
скрипции
Некоторые рибопереключатели, обнаружен)
рая устраняет Rho)зависимую терминацию и
ные у грамотрицательных бактерий, в отличие
позволяет транскрипции продолжаться до кон)
от их аналогов в Bacillus subtilis, по)видимому, не
ца гена.
имеют канонических терминаторов. Было пока)
зано, что такие рибопереключатели контроли)
руют экспрессию генов на уровне транскрип)
МУЛЬТИЛИГАНДНАЯ
ции, но с помощью фактора терминации
ЧУВСТВИТЕЛЬНОСТЬ
Rho [36]. Rho - это общий фактор терминации,
РИБОПЕРЕКЛЮЧАТЕЛЕЙ
который перемещается с РНК)полимеразой и
отвечает за терминацию примерно 20% тран)
Восприятие более чем одной молекулы лиганда
скрипционных единиц в E. coli [37]. Rho преры)
одним сенсором. Многие наблюдения того, что
вает транскрипцию незащищённых РНК на
сенсорный домен рибопереключателя связывает
сайтах, которые обычно обогащены С)остатка)
одну молекулу метаболита, никогда не исключа)
ми и лишены G)остатков. В рибопереключателе
ли возможности того, что рибопереключатель
флавинмононуклеотида (FMN) E. coli стабили)
может воспринимать две или более молекулы
зация FMN)связанного аптамера индуцирует
лиганда.
Rho)зависимую терминацию транскрипции ни)
Одно из первых доказательств появилось
же аптамера и выше кодирующей белок области
после биохимической и структурной характе)
(рис. 3. d) [36]. Нарушение структуры аптамера
ристики рибопереключателя M)бокс (Mg)I), от)
при отсутствии FMN вызывает альтернативную
вечающего на катионы Mg2+ [38, 39]. Эта боль)
конформацию нетранслируемой области, кото)
шая РНК связывает несколько катионов Mg2+
БИОХИМИЯ том 86 вып. 8 2021
1166
ARIZA)MATEOS et al.
для сворачивания и генетического ответа, но
связанными с лигандом и опосредуют его взаи)
другие катионы также могут заменять катионы
модействия с РНК [46-48]. Три катиона Mg2+
Mg2+, чтобы стимулировать сворачивание РНК.
инкапсулируют фторид)анион и помогают это)
Более селективный рибопереключатель NiCo
му маленькому лиганду осуществлять прямое
реагирует на катионы Ni2+или Co2+ [40] и, воз)
сворачивание фторидного рибопереключате)
можно, на Fe2+ и Mn2+ [41]. Структура и биохи)
ля [49]. Катионы K+ и Mg2+ способствуют связы)
мические данные показывают, что рибопере)
ванию карбоксилатной части лизина и глицина
ключатель одновременно связывает четыре ка)
соответственно с родственными им рибопере)
тиона Ni2+ или Co2+, хотя неясно, все ли сайты
ключателями [50, 51].
активны при физиологических значениях кон)
Хотя приведённые выше примеры соответ)
центраций этих катионов [40]. Отдельный сен)
ствуют сигнальной модели с одним входом, су)
сор иона металла, рибопереключатель Mn2+, со)
ществуют рибопереключатели, управление ко)
держит два соседних сайта связывания метал)
торыми зависит или включает в себя двойной
ла - один для Mn2+, а другой для Mg2+ [42]. Хо)
вход. Недавнее исследование показало, что ре)
тя катион Mn2+ в миллимолярных концентраци)
акция лизинового рибопереключателя lysC
ях может заменять Mg2+ в его сайте связывания
B. subtilis на лизин сильно зависит от незначи)
in vitro, эта ситуация, вероятно, не является био)
тельных изменений внутриклеточных уровней
логически значимой, поскольку E. coli поддер)
катионов Mg2+ [52]. В нижней части физиологи)
живает внутриклеточную концентрацию Mn2+
ческого диапазона концентраций Mg2+ (<1 мМ)
на низких микромолярных уровнях, чтобы избе)
аптамер остаётся неструктурированным и требу)
жать токсичности.
ет более высокой концентрации лизина для от)
Связывание с несколькими лигандами не ог)
вета. Увеличение концентрации Mg2+ на 1 мМ
раничивается катионами металлов, которые из)за
заранее организует структуру для связывания
небольшого размера могут иметь ограничения в
лизина, так что лиганд может взаимодействовать
управлении фолдингом РНК, если они связыва)
с рибопереключателем при концентрации на два
ются с ней в единственном числе. Рибоперек)
порядка ниже. Таким образом, изменения уров)
лючатель THF)I связывает две молекулы тетра)
ней концентрации Mg2+, например в нормаль)
гидрофолата (ТГФ) [43]. Рибопереключатель
ных условиях роста и в стрессовой среде, могут
стратегически размещает одну молекулу ТГФ в
определять активность рибопереключателя.
шарнирной области, чтобы гарантировать па)
Специфичность рибопереключателя часто
раллельное выравнивание основного стержня с
означает, что сродство связи рибопереключате)
отпочкованной шпилькой, в то время как вто)
ля с родственным метаболитом выше, чем с ана)
рая молекула ТГФ связывается в конце шпильки
логичными молекулами. Считается, что в клет)
и обеспечивает дальнодействующие контакты с
ках подобные неродственные лиганды отверга)
основным стержнем. Эти третичные взаимодей)
ются рибопереключателем, и регуляторный от)
ствия стабилизируют спираль P1 для регулятор)
вет приписывается тому лиганду, который луч)
ного ответа.
ше связывается. Тем не менее при высоких кон)
Рибопереключатель циклического диадено)
центрациях неродственные лиганды могут кон)
зинмонофосфата (c)di)AMP) взаимодействует с
курировать с родственными лигандами за свя)
двумя молекулами c)di)AMP в двух сайтах свя)
зывание с рибопереключателем. При связыва)
зывания, расположенных поперёк псевдосим)
нии неродственный лиганд может не продвигать
метричного аптамера четырёхугольной фор)
сворачивание рибопереключателя до связанно)
мы [44, 45] (рис. 4, a). Несмотря на сходство,
го с лигандом состояния и вызывать соответ)
сайты не равноценны для связывания лиганда,
ствующий регуляторный ответ. Исследование
и, хотя нарушение в работе одного сайта устра)
in vivo показало, что активность рибопереклю)
няет способность связываться с обоими сайта)
чателя, рибозима glmS B. subtilis, модулируется
ми, мутация второго сайта только снижает спо)
как родственным лигандом глюкозамин)6)фос)
собность связываться с первым сайтом [44].
фатом (GlcN6P), активирующим ответ, так и его
В зависимости от платформы экспрессии рибо)
несколькими предшественниками, ингибирую)
переключатель подавляет транскрипцию или
щими ответ [53]. Таким образом, в клетках даже
трансляцию.
рибопереключатели с одним входом могут ин)
Ряду лигандов требуется помощь других не)
тегрировать информацию из массива химичес)
больших молекул, обычно катионов металлов,
ких сигналов и реагировать на основе концент)
для эффективного связывания с рибопереклю)
раций нескольких соединений.
чателями. Практически все метаболиты, содер)
Связывание одних и тех же лигандов несколь
жащие фосфатную группу, нейтрализуют эти
кими сенсорами. В то время как большинство ри)
фрагменты катионами Mg2+, которые остаются
бопереключателей используют один аптамер
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1167
Рис. 4. Механизмы мультилигандного зондирования и их регуляция. а - Предполагаемый механизм контроля транскрип)
ции с помощью рибопереключателя c)di)AMP [44, 102]. Рибопереключатель требует связывания двух молекул c)di)AMP
для адаптации структуры аптамера, связанного с лигандом, и терминации транскрипции [44, 45]. b - Механизм актива)
ции трансляции рибопереключателя гуанидин)II [60-62]. Связывание двух лигандов с консервативными апикальными
петлями способствует образованию димера «голова к голове», который высвобождает последовательность SD для связы)
вания с рибосомой. c - Предполагаемый ступенчатый механизм трансляционной репрессии с помощью рибопереключа)
теля NAD+ [66]. Поскольку NAD+ всегда присутствует в клетках, одна молекула NAD+ может связываться с формирую)
щимся транскриптом, как только аптамер 1 транскрибируется. Высокие концентрации NAD+ будут стимулировать свя)
зывание NAD+ с аптамером 2, который имеет более низкое сродство к лиганду, и изолирование последовательности SD в
структуре аптамера. d - Гипотетический механизм рибопереключателя SAM)I-tRNA из Xanthomonas campestris [67]. При
обнаружении различных концентраций SAM и tRNAfMet рибопереключатель может принимать четыре различных состоя)
ния, которые соответствуют четырём регуляторным ответам. Фрагменты РНК, участвующие в наиболее обширных кон)
формационных перестройках, показаны более светлым цветом. e - Регуляторные ответы на тандемный рибопереключа)
тель гуанин)PRPP [68]. В безлигандной форме рибопереключатель активирует транскрипцию. Связывание гуанина при
отсутствии PRPP разрушает аптамер PRPP и вызывает преждевременное прекращение транскрипции. Связывание PRPP
отдельно или в присутствии гуанина стабилизирует аптамер PRPP и предотвращает связывание гуанина, что приводит к
непрерывной транскрипции. f - Регуляторные ответы системы рибопереключателей SAM)I и AdoCbl [73]. Система вклю)
чает транскрипцию при отсутствии обоих лигандов. Связывание одного лиганда или обоих лигандов одновременно пре)
рывает транскрипцию и выключает экспрессию гена. g - Регуляторные ответы системы рибопереключателей TPP и
HMP)PP [35]. Рибопереключатель TPP подавляет транскрипцию при связывании лиганда, в то время как рибопереклю)
чатель HMP)PP активирует транскрипцию в связанной с лигандом форме. Следовательно, ген экспрессируется только
при отсутствии TPP и в присутствии HMP)PP. Состояния без лиганда, TPP)связанный, TPP)связанный/HMP)PP)свя)
занный выключают экспрессию гена
БИОХИМИЯ том 86 вып. 8 2021
1168
ARIZA)MATEOS et al.
для связывания лиганда, несколько классов ри)
может объяснить кооперативное связывание гу)
бопереключателей регулируют экспрессию ге)
анидина, наблюдаемое в биохимических экспе)
нов с помощью нескольких смежных аптамеров,
риментах с полноразмерным рибопереключате)
каждый из которых имеет сайт связывания ли)
лем [60], и предполагает регуляторный меха)
ганда, за которым следует единственная плат)
низм, основанный на димеризации двух аптаме)
форма экспрессии. Несколько аптамеров позво)
ров внутри одного рибопереключателя [62, 63].
ляют добиться более чётких количественных ха)
Гуанидин)II действует как трансляционный ри)
рактеристик ответов на изменение концентра)
бопереключатель «ON». При отсутствии гуани)
ции лиганда. Первый пример таких рибопере)
дина последовательность SD изолируется спари)
ключателей относится к началу 2000)х годов [54].
ванием оснований с линкерной областью и нук)
Это исследование обнаружило глицин)специ)
леотидами на 5′)конце первого аптамера, тем са)
фические рибопереключатели, состоящие из
мым предотвращая инициацию трансляции.
двух похожих сенсорных доменов тандемно)
Связывание гуанидина способствует димериза)
расположенных друг относительно друга; это
ции двух аптамеров и обеспечивает простран)
исследование указывало на кооперативное свя)
ственное разделение последовательности SD от
зывание двух молекул глицина, по одной моле)
комплементарной последовательности анти)SD.
куле на сенсор. Структурные и биохимические
Неспаренная последовательность SD может свя)
исследования выявили межмолекулярные взаи)
зывать рибосому и инициировать трансляцию.
модействия между двумя модулями [51, 55]. Хо)
Так же как рибопереключатель гуанидин)II,
тя такая кооперативная связь была поставлена
рибопереключатель никотинамид)аденинди)
под сомнение [56], недавнее исследование под)
нуклеотид (NAD+) класса I, образован двумя
твердило существование кооперативности, ко)
шпильками; однако подобные консервативные
торая может быть замаскирована разницей в
области расположены в выпуклых петлях
сродстве связывания двух сенсоров [57].
(рис. 4, c) [64]. Структурные исследования по)
Ещё несколько рибопереключателей содер)
казали, что NAD+ действительно связывает
жат несколько аптамеров, за которыми следует
РНК в этом выступе [65, 66]. Биоинформатика
одна платформа экспрессии. Более половины
выявила небольшие различия между двумя ап)
рибопереключателей глутамина glnA (глутамин)I)
тамерами в центральных областях, а исходное
расположены в тандемной ориентации, где два
исследование не сообщало о связывании NAD+
или три аптамера разделены небольшими лин)
со вторым аптамером [64]. В более поздней ра)
керными областями [58]. Многие рибопереклю)
боте было обнаружено связывание со вторым
чатели глутамин)I имеют сходные линкерные
аптамером, но с гораздо более низким срод)
последовательности, которые, как предполага)
ством, чем с первым аптамером [66]. Исследо)
ется, образуют пару оснований с последователь)
вание структуры показало, что связывание ли)
ностью, расположенной ниже 3′)домена. Рент)
ганда стабилизирует третичный контакт между
геновская кристаллическая структура показала
лиганд)связывающим сайтом и средней нерегу)
связывание лиганда в стыке, соединяющем
лярной областью РНК. У второго аптамера этот
стебли рибопереключателя, и их большую пе)
контакт отсутствует. Интересно, что оба апта)
регруппировку при связывании лиганда [59].
мера распознают только аденозиновый фраг)
Биохимические эксперименты не обнаружили
мент лиганда, а не никотинамидный фрагмент,
доказательств кооперативности [58] и, хотя
что позволяет предположить, что структуры от)
структурные исследования предсказывают сход)
дельных доменов, возможно, не охватывают все
ное связывание лиганда со всеми аптамерами
взаимодействия с лигандом. Вероятно, никоти)
тандемных рибопереключателей, преимущества
намидный фрагмент лиганда, связанный с ап)
наличия более чем одного аптамера не ясны.
тамером 1, может распознаваться аптамером 2.
Рибопереключатель гуанидин)II состоит из
Поскольку NAD+ всегда присутствует в клет)
двух шпилек, содержащих сходные и высоко)
ках, существует интригующая вероятность того,
консервативные апикальные петли, которые ко)
что рибопереключатель действует как двухкон)
оперативно связывают свободный гуани)
центрационный сенсор [66]. При низких кон)
дин [60] (рис. 4, b). Структурные исследования
центрациях NAD+ связывается с первым апта)
отдельных аптамеров показали, что каждая пет)
мером, оставляя аптамер 2 несвязанным и нест)
ля содержит сайт связывания гуанидина и что
руктурированным, SD)последовательность от)
связывание гуанидина облегчает гомодимериза)
крытой для трансляции. В более высоких кон)
цию двух аптамерных доменов посредством тре)
центрациях NAD+ также связывается и с апта)
тичного спаривания оснований между петля)
мером 2, способствуя спариванию оснований
ми [61, 62]. Образование этого лиганд)зависи)
SD)последовательности и запуская репрессию
мого димера с конфигурацией «голова к голове»
трансляции.
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1169
Восприятие различных лигандов несколькими
ческий дигуанилат, c)di)GMP), который распо)
сенсорами. Среди наиболее интересных откры)
ложен перед самосплайсирующимся интроном
тий в области рибопереключателей - открытие
группы I [71]. Этот РНК)регулятор сочетает в
рибопереключателей с двумя входами, которые
себе самосплайсинг и активацию трансляции
обладают отдельными сайтами связывания для
для регулирования последующих генов. Связы)
разных лигандов, но работают согласованно че)
вание c)di)GMP с его аптамером делает возмож)
рез единую платформу экспрессии. Эти откры)
ным фолдинг ядра интрона, связывание GTP с
тия особенно интересны с эволюционной точки
сайтом сплайсинга 5′)интрона и самовырезание
зрения. Хотя дупликация генов может распола)
интрона. Удаление интрона объединяет две уда)
гать похожие аптамеры рядом друг с другом,
лённо расположенные части последовательнос)
труднее всего представить, как сенсоры с разной
ти SD с образованием полной последователь)
специфичностью эволюционировали, чтобы
ности SD для инициации трансляции [71, 72].
объединиться и контролировать одну и ту же
При отсутствии c)di)GMP GTP связывается в
единицу экспрессии.
альтернативном сайте, и новое расщепление
Недавно был предложен интересный меха)
приводит к укорачиванию последовательнос)
низм двойного входа для рибопереключателя
ти SD, которая не может эффективно связать
SAM)I, который регулирует синтез метионина у
рибосому и не инициирует трансляцию.
X. campestris (рис. 4, d) [67]. Этот рибопереклю)
чатель в первую очередь отвечает на клеточный
S)аденозилметионин (SAM) на уровне трансля)
«СОТРУДНИЧЕСТВО» НЕЗАВИСИМЫХ
ции, однако платформа экспрессии также со)
РИБОПЕРЕКЛЮЧАТЕЛЕЙ
держит сайт связывания для незаряженной ини)
циаторной Met)тРНК. Связывание тРНК и
Хотя большинство рибопереключателей
SAM, по)видимому, способствует образованию
функционируют как единая регуляторная еди)
нескольких альтернативных пар оснований и
ница, которая включает один или несколько ап)
вызывает ряд регуляторных ответов. В то время
тамеров и единую платформу экспрессии или
как связывание SAM изолирует последователь)
эквивалентную область мРНК, некоторые рибо)
ность SD и отменяет трансляцию, одновремен)
переключатели содержат две независимые регу)
ное связывание тРНК изолирует последователь)
ляторные единицы в тандемном расположе)
ность анти)SD и частично высвобождает после)
нии [35, 73-75]. Чаще всего эти рибопереключа)
довательность SD для связывания рибосомы.
тели содержат сенсоры, отвечающие на один и
Частичная экспрессия генов также может наб)
тот же метаболит, например TPP, за которым
людаться при отсутствии обоих лигандов, тогда
следует тот же тип платформы экспрессии, как у
как полная дерепрессия, по)видимому, требует
терминатора транскрипции [73]. Вероятно, ком)
связывания тРНК при отсутствии SAM.
позитные переключатели, напоминающие
Сложный регуляторный результат наблю)
двойной TPP)рибопереключатель, обеспечива)
дался также в рибопереключателе, включающем
ют более выраженный количественный ответ
сенсор гуанина, за которым следует сенсор фос)
или большую чувствительность к изменениям
форибозилпирофосфата (PRPP) [68] (рис. 4, e).
концентрации метаболитов по сравнению с
Последовательность между этими аптамерами
одиночными рибопереключателями. В некото)
короткая, и 5′)область сенсора PRPP перекры)
рых случаях сенсоры реагируют на разные ли)
вается с 3′)областью предшествующего гуани)
ганды.
нового сенсора. Следовательно, связывание гуа)
В тандемной системе рибопереключателя
нина препятствует образованию сенсора PRPP и
SAM)I и AdoCbl связывание SAM или AdoCbl с
облегчает образование терминатора транскрип)
их соответствующими сенсорами вызывает тер)
ции, тем самым останавливая транскрипцию.
минацию транскрипции в отдельном рибопе)
С другой стороны, аптамер PRPP «преформиро)
реключателе, что отменяет транскрипцию гена.
ван» при отсутствии как гуанина, так
Соответственно, ген включается только тогда,
и PRPP [69, 70]. Эта конформация аптамера
когда оба лиганда присутствуют в клетках в кон)
препятствует образованию терминатора и поз)
центрациях ниже пороговых (рис. 4, f).
воляет транскрипции идти до конца. В присут)
Тандемная система рибопереключателей
ствии обоих лигандов при связывании с рибопе)
TPP и HMP)PP содержит репрессирующий ри)
реключателем PRPP доминирует и предотвра)
бопереключатель TPP, за которым следует акти)
щает связывание гуанина, обеспечивая непре)
вирующий рибопереключатель HMP)PP. Эта
рывную транскрипцию гена [68].
система контролирует экспрессию гена, кото)
Особый механизм был продемонстрирован
рый кодирует фермент, необходимый для синте)
для рибопереключателя цикло)di)GMP (цикли)
за другого предшественника на пути образова)
БИОХИМИЯ том 86 вып. 8 2021
1170
ARIZA)MATEOS et al.
ния TPP [35]. Бактериям не нужно производить
личие от других рибопереключателей, GlcN6P
TPP, когда его много; следовательно, связыва)
связывается с предварительно сформирован)
ние TPP с его аптамером останавливает экспрес)
ным аптамером [82, 83] и вместо аллостеричес)
сию нижележащего гена независимо от концен)
кой модуляции его структуры катализирует ре)
трации HMP)PP (рис. 4, g). Когда TPP отсут)
акцию расщепления мРНК перед аптаме)
ствует, ответ может следовать двум сценариям.
ром [80, 84]. Полученный РНК)продукт больше
Если в клетках также отсутствует HMP)PP, ген
не защищён трифосфатом на 5′)конце и подвер)
не экспрессируется. Высокие концентрации
гается быстрой деградации экзонуклеазой
HMP)PP активируют экспрессию гена.
РНКазой J [81].
Существование систем рибопереключате)
Следующий пример показывает другое учас)
лей, в которых отдельные регуляторные едини)
тие РНКазы в механизме рибопереключе)
цы с различной специфичностью объединены в
ния [85]. После связывания лизина с сенсорным
тандемные структуры, открывает возможность
доменом лизинового рибопереключателя lysC
интеграции различных сигналов из окружаю)
E. coli платформа экспрессии сворачивается в
щей и внутриклеточной среды. Каждый рибопе)
шпильку, изолирующую SD)последователь)
реключатель воспринимает один сигнал, однако
ность, которая выключает трансляцию и откры)
регуляторный ответ носит комбинаторный ха)
вает два сайта расщепления РНКазой E, распо)
рактер. Такие сложные системы могут позво)
ложенные на платформе экспрессии (рис. 5, b).
лить точное регулирование, зависящее от при)
Эти расщепления приводят к образованию РНК
сутствия конечных продуктов, а также от мета)
с монофосфорилированным 5′)концом, кото)
болического состояния клетки или присутствия
рая, по)видимому, запускает ускоренную дегра)
молекул)предшественников.
дацию кодирующей области lysC РНКазой E.
В отсутствие лизина рибопереключатель образу)
ет антисеквестрирующую SD)шпильку, которая
ДЕГРАДАЦИЯ РНК
также изолирует сайты расщепления РНКа)
КАК ДОПОЛНИТЕЛЬНЫЙ МЕХАНИЗМ
зой E, позволяя инициации трансляции проис)
РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
ходить без деградации мРНК. Таким образом,
лизиновый рибопереключатель является регу)
Бесспорно, контроль транскрипции и транс)
лятором двойного действия: два разных меха)
ляции играет очень важную роль в регуляции ге)
низма, подавление трансляции и деграда)
нов. Кроме того, изменение времени жизни
ция РНК, ингибируют экспрессию гена lysC.
мРНК может сильно повлиять на продукцию
Подобное участие РНКазы может вносить вклад
белков. У бактерий стабильность мРНК регули)
в генетические ответы других рибопереключате)
руется несколькими рибонуклеазами и связан)
лей [86].
ными с ними факторами, которые варьируются
В другой системе связывание метаболитов
в зависимости от вида [76]. У E. coli и других грам)
оказывает противоположный эффект на ста)
отрицательных бактерий деградация РНК обыч)
бильность РНК [87, 88]. Цикло)ди)GMP)зави)
но начинается с внутреннего расщепления
симый (c)di)GMP) рибопереключатель Vc2, по)
РНКазой E, за которым следует расщепление 3′)
видимому, подавляет экспрессию гена Vibrio
экзонуклеазой. У B. subtilis и других Firmicutes в
cholerae tfoY в ответ на связывание лиганда путём
деградации мРНК участвует другая эндонуклеа)
изолирования SD)последовательности и инги)
за - РНКаза Y и 5′)экзонуклеаза, РНКаза J.
бирования трансляции [88-90]. Связывание
Вклад рибонуклеаз в механизмы рибопереклю)
c)di)GMP с Vc2 также ведёт к накоплению вы)
чения отмечен давно, но только недавние иссле)
шележащих нетранслируемых РНК, которые,
дования показали, что деградация РНК может
вероятно, функционируют как малые РНК
быть основным механизмом действия рибопе)
(sRNA), контролирующие подвижность кле)
реключателя [77, 78], а не только средством уда)
ток [87]. Хотя молекулярная основа стабильнос)
ления РНК после завершения регуляторного от)
ти предполагаемых малых РНК неясна, стаби)
вета [79].
лизация аптамера связанным c)di)GMP, вероят)
Первая взаимосвязь работы рибопереключа)
но, задействует 3′)конец при спаривании осно)
теля и аппарата деградации РНК была установ)
ваний и предотвращает доступ к нему 3′)экзо)
лена после открытия рибопереключателя рибо)
нуклеаз.
зим glmS (рис. 5, а) [80, 81]. У B. subtilis мРНК
Недавнее исследование элегантно продемон)
glmS кодирует синтазу GlcN6P. При высоких
стрировало совершенно другой механизм, ис)
концентрациях конечный продукт реак)
пользуемый рибопереключателем для регуля)
ции, GlcN6P, инициирует регуляцию по прин)
ции экспрессии генов [78] (рис. 5, c). Этот меха)
ципу отрицательной обратной связи [80]. В от)
низм основан на нескольких открытиях, связан)
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1171
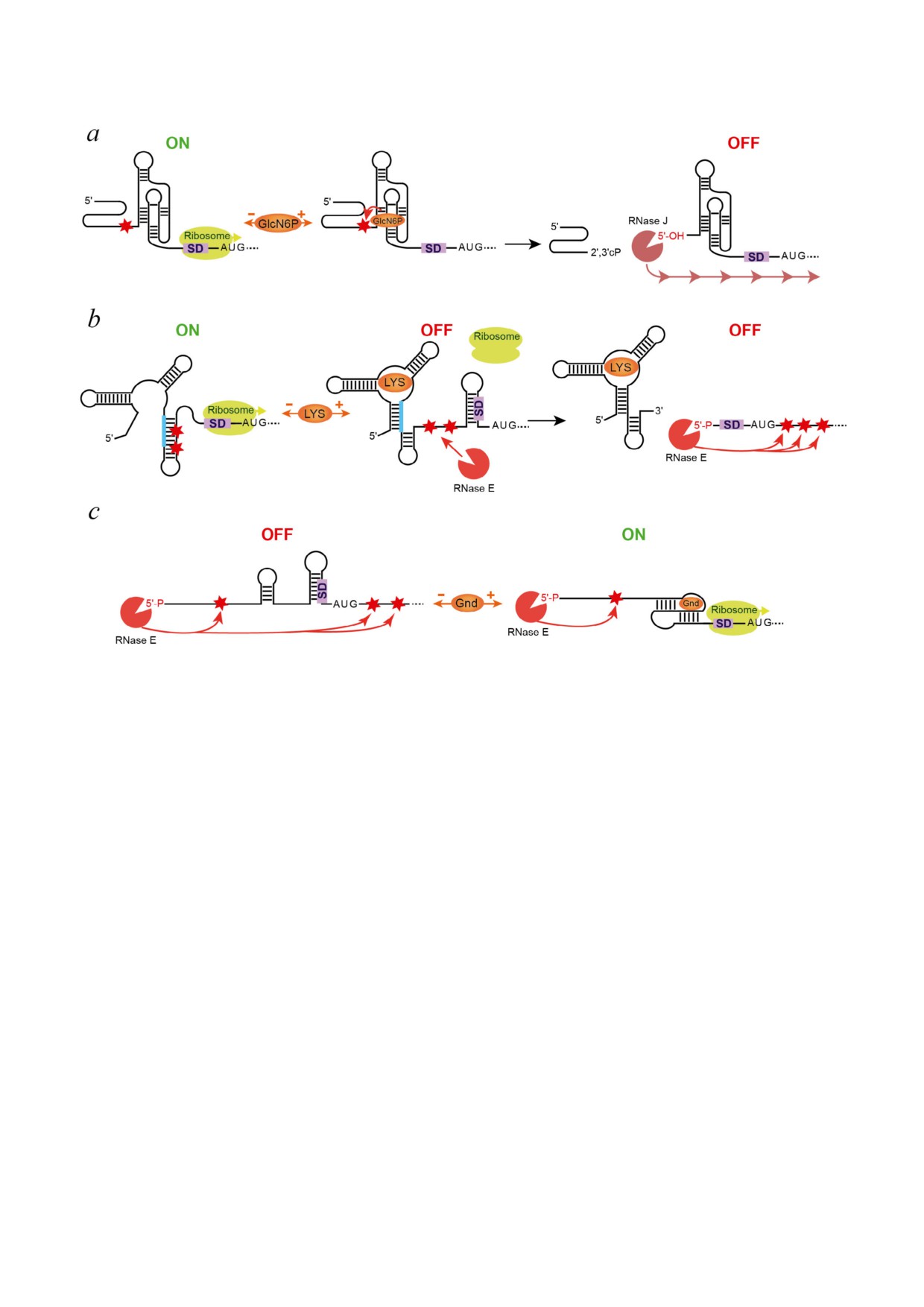
Рис. 5. Участие деградации РНК в ответе рибопереключателя. а - Механизм действия рибопереключателя рибозим
glmS [80, 81]. В отсутствие GlcN6P ген транскрибируется и транслируется. Связывание GlcN6P вызывает расщепле)
ние (стрелка) в сайте (звездочка) перед аптамером. Экзонуклеаза РНКаза J связывается с дефосфорилированным 5′)кон)
цом и разрушает мРНК (линия со стрелками), выключая экспрессию гена. b - Двойной механизм лизинового рибопере)
ключателя lysC E. coli, контролирующего инициацию трансляции и разрушение мРНК [85]. В отсутствие лизина рибопе)
реключатель сворачивается в состояние «ON», которое открывает SD)последовательность для инициации трансляции и
изолирует сайт расщепления РНКазой E. После связывания лиганда домен аптамера принимает состояние «OFF», кото)
рое изолирует SD)последовательность и открывает сайт расщепления РНКазой E. РНКаза E начинает связываться с мо)
нофосфорилированным 5′)концом РНК и производить дальнейшие эндонуклеолитические расщепления (длинные
стрелки) с последующим 3′)экзонуклеолитическим расщеплением (не показано). с - Участие деградации РНК в механиз)
ме рибопереключателя гуанидин)III sugE Legionella pneumophila. В отсутствие лиганда связанная с 5′)концом РНКаза E
может производить эндонуклеолитические расщепления как перед аптамером, так и после него, поскольку свободный от
лиганда рибопереключатель не останавливает поиск сайтов расщепления по направлению от 5′) к 3′)концу. мРНК рас)
щепляется, и белок не продуцируется. Образование связанного с лигандом псевдоузла в домене аптамера высвобождает
SD)последовательность для трансляции (не показана) и препятствует сканированию с помощью РНКазы E, предотвра)
щая расщепление ниже аптамерного домена. Следовательно, связанный с лигандом аптамер защищает транслирующую
рибосому от деградации, обеспечивая состояние «ON» экспрессии гена
ных с активностью РНКазы E. Эта эндорибо)
ствия, такие как стебли)петли с ортогональны)
нуклеаза разрезает РНК в определенных местах
ми парами оснований, но останавливается на
в одноцепочечных областях [91], и её актив)
более крупных препятствиях, таких как связан)
ность значительно ускоряется после связывания
ные белки или структуры, содержащие элемен)
с 5′)монофосфорилированной РНК через кар)
ты с парами оснований, коаксиальные (имею)
ман, специфичный для 5′)конца монофосфори)
щие общую ось) с направлением сканирова)
лированной РНК [92, 93]. После связывания
ния [78, 94]. Трансляция транскрипта sugE
РНКаза E ищет сайты расщепления путём ска)
Legionella pneumophila контролируется рибопе)
нирования мРНК по направлению от 5′) к 3′)
реключателем гуанидин)III. После связывания
концу. Фермент обходит небольшие препят)
лиганда домен аптамера принимает компактную
БИОХИМИЯ том 86 вып. 8 2021
1172
ARIZA)MATEOS et al.
структуру псевдоузла, в то время как SD)секве)
рибопереключения. Могут ли эти новые меха)
стрирующая шпилька в платформе экспрессии
низмы включать Rho)зависимую терминацию
разворачивается и освобождает SD)последова)
транскрипции, деградацию РНК или другие
тельность для связывания с рибосомами [78].
способы? Новые методологии, такие как RNA)
Кроме того, связанный с лигандом псевдоузел в
Seq, уже идентифицировали множественные
аптамере представляет собой значительное пре)
малые РНК, потенциально связанные с рибопе)
пятствие для сканирования РНКазой E и защи)
реключателями [98-100]; следовательно, можно
щает расположенные ниже сайты расщепления
ожидать поток исследований, расшифровываю)
от доступа нуклеаз. В результате повышается
щих связанные с малыми РНК механизмы ри)
стабильность мРНК sugE. Поскольку актив)
бопереключателей. Идентификация родствен)
ность РНКазы E не зависит от контекста после)
ных лигандов для многих «сиротских» рибопе)
довательности, этот механизм может быть об)
реключателей и открытие новых классов рибо)
щим для многих систем, использующих рибопе)
переключателей также обогатит репертуар меха)
реключатели, чьи связанные с лигандом аптаме)
низмов работы рибопереключателей. Эти иссле)
ры мешают сканированию.
дования дополнительно помогут нам понять ре)
гуляторные ответы и определить основных
участников метаболических путей, а также соз)
ПЕРСПЕКТИВЫ
дать молекулярную основу вмешательства в наи)
более важные клеточные процессы патогенных
Относительно простые механизмы работы
бактерий.
рибопереключателей, обсуждаемые в этом обзо)
ре, не дают полного представления обо всём
многообразии этих механизмов. Опосредован)
Финансирование. Работа поддержана гран)
ный рибопереключателями контроль экспрес)
том Фонда Рамона Аресеса (Ramon Areces
сии генов может включать дополнительные
Foundation) (A.A.)M.) и грантом Национального
уровни восприятия внешних факторов [95] или
института здравоохранения
2R01GM112940
генерировать более сложные многокомпонент)
(A.S.).
ные регуляторные ответы с участием некодиру)
Конфликт интересов. Авторы заявляют об от)
ющих РНК и белков [96, 97]. Учитывая, что био)
сутствии конфликта интересов в финансовой
информационные поиски не выявили платфор)
или иной сфере.
мы экспрессии для многих предполагаемых ри)
Соблюдение этических норм. Эта статья не
бопереключателей, мы можем ожидать, что в бу)
содержит исследований с участием людей или
дущем будет обнаружено больше механизмов животных, выполненных кем)либо из авторов.
СПИСОК ЛИТЕРАТУРЫ
1.
McNerney, M. P., and Styczynski, M. P. (2018) Small mol)
7. Nudler, E., and Mironov, A. S. (2004) The riboswitch con)
ecule signaling, regulation, and potential applications in
trol of bacterial metabolism, Trends Biochem. Sci., 29, 11)
cellular therapeutics, Wiley Interdiscip. Rev. Syst. Biol.
17, doi: 10.1016/j.tibs.2003.11.004.
Med., 10, doi: 10.1002/wsbm.1405.
8. Pavlova, N., Kaloudas, D., and Penchovsky, R. (2019)
2.
Hauryliuk, V., Atkinson, G. C., Murakami, K. S.,
Riboswitch distribution, structure, and function in bacte)
Tenson, T., and Gerdes, K. (2015) Recent functional
ria, Gene, 708, 38)48, doi: 10.1016/j.gene.2019.05.036.
insights into the role of (p)ppGpp in bacterial physiology,
9. Serganov, A., and Patel, D. J. (2012) Metabolite recogni)
Nat. Rev. Microbiol.,
13,
298)309, doi:
10.1038/
tion principles and molecular mechanisms underlying
nrmicro3448.
riboswitch function, Annu. Rev. Biophys., 41, 343)370,
3.
Zimmermann, G. R., Jenison, R. D., Wick, C. L.,
doi: 10.1146/annurev)biophys)101211)113224.
Simorre, J. P., and Pardi, A. (1997) Interlocking structural
10. Nomura, M., Gourse, R., and Baughman, G. (1984) Re)
motifs mediate molecular discrimination by a theo)
gulation of the synthesis of ribosomes and ribosomal com)
phylline)binding RNA, Nat. Struct. Biol., 4, 644)649,
ponents, Annu. Rev. Biochem., 53, 75)117, doi: 10.1146/
doi: 10.1038/nsb0897)644.
annurev.bi.53.070184.000451.
4.
Nahvi, A., Sudarsan, N., Ebert, M. S., Zou, X., Brown,
11. Serganov, A., Ennifar, E., Portier, C., Ehresmann, B., and
K. L., and Breaker, R. R. (2002) Genetic control by a
Ehresmann, C. (2002) Do mRNA and rRNA binding sites
metabolite binding mRNA, Chem. Biol.,
9,
1043,
of E. coli ribosomal protein S15 share common structural
doi: 10.1016/s1074)5521(02)00224)7.
determinants? J. Mol. Biol., 320, 963)978, doi: 10.1016/
5.
Winkler, W., Nahvi, A., and Breaker, R. R.
(2002)
s0022)2836(02)00553)3.
Thiamine derivatives bind messenger RNAs directly to reg)
12. Cech, T. R., Zaug, A. J., and Grabowski, P. J. (1981)
ulate bacterial gene expression, Nature, 419, 952)956,
In vitro splicing of the ribosomal RNA precursor of
doi: 10.1038/nature01145.
Tetrahymena: involvement of a guanosine nucleotide in the
6.
Mironov, A. S., Gusarov, I., Rafikov, R., Lopez, L. E.,
excision of the intervening sequence, Cell, 27, 487)496,
Shatalin, K., et al. (2002) Sensing small molecules by
doi: 10.1016/0092)8674(81)90390)1.
nascent RNA: a mechanism to control transcription in
13. Ellington, A. D., and Szostak, J. W. (1990) In vitro selec)
bacteria, Cell,
111,
747)756, doi:
10.1016/s0092)
tion of RNA molecules that bind specific ligands, Nature,
8674(02)01134)0.
346, 818)822, doi: 10.1038/346818a0.
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1173
14.
Robertson, D. L., and Joyce, G. F. (1990) Selection in vitro
S)adenosylmethionine, Nat. Struct. Mol. Biol., 15, 177)
of an RNA enzyme that specifically cleaves single)stranded
182, doi: 10.1038/nsmb.1371.
DNA, Nature, 344, 467)468, doi: 10.1038/344467a0.
32.
Corbino, K. A., Barrick, J. E., Lim, J., Welz, R., Tucker,
15.
Tuerk, C., and Gold, L. (1990) Systematic evolution of lig)
B. J., et al. (2005) Evidence for a second class of S)adeno)
ands by exponential enrichment: RNA ligands to bacterio)
sylmethionine riboswitches and other regulatory RNA
phage T4 DNA polymerase, Science, 249, 505)510,
motifs in alpha)proteobacteria, Genome Biol., 6, R70,
doi: 10.1126/science.2200121.
doi: 10.1186/gb)2005)6)8)r70.
16.
Yanofsky, C. (1981) Attenuation in the control of expres)
33.
Panchapakesan, S. S. S., Corey, L., Malkowski, S. N.,
sion of bacterial operons, Nature,
289,
751)758,
Higgs, G., and Breaker, R. R. (2021) A second riboswitch
doi: 10.1038/289751a0.
class for the enzyme cofactor NAD+, RNA, 27, 99)105,
17.
Miranda)Rios, J., Navarro, M., and Soberon, M. (2001) A
doi: 10.1261/rna.077891.120.
conserved RNA structure (thi box) is involved in regulation
34.
Salvail, H., Balaji, A., Yu, D., Roth, A., and Breaker, R. R.
of thiamin biosynthetic gene expression in bacteria, Proc.
(2020) Biochemical validation of a fourth guanidine
Natl. Acad. Sci. USA, 98, 9736)9741, doi: 10.1073/pnas.
riboswitch class in Bacteria, Biochemistry, 59, 4654)4662,
161168098.
doi: 10.1021/acs.biochem.0c00793.
18.
Nou, X., and Kadner, R. J. (1998) Coupled changes in
35.
Atilho, R. M., Mirihana Arachchilage, G., Greenlee,
translation and transcription during cobalamin)dependent
E. B., Knecht, K. M., and Breaker, R. R. (2019) A bacter)
regulation of btuB expression in Escherichia coli,
ial riboswitch class for the thiamin precursor HMP)PP
J. Bacteriol., 180, 6719)6728, doi: 10.1128/JB.180.24.
employs a terminator)embedded aptamer, Elife,
8,
6719)6728.1998.
doi: 10.7554/eLife.45210.
19.
Gelfand, M. S., Mironov, A. A., Jomantas, J., Kozlov, Y. I.,
36.
Hollands, K., Proshkin, S., Sklyarova, S., Epshtein, V.,
and Perumov, D. A. (1999) A conserved RNA structure ele)
Mironov, A., et al. (2012) Riboswitch control of Rho)
ment involved in the regulation of bacterial riboflavin syn)
dependent transcription termination, Proc. Natl. Acad. Sci.
thesis genes, Trends Genet., 15, 439)442, doi: 10.1016/
USA, 109, 5376)5381, doi: 10.1073/pnas.1112211109.
s0168)9525(99)01856)9.
37.
Peters, J. M., Mooney, R. A., Kuan, P. F., Rowland, J. L.,
20.
Perkins, J. B., and Pero, J. (2002) Biosynthesis of
Keles, S., and Landick, R. (2009) Rho directs widespread
riboflavin, biotin, folic acid, and cobalamin, in Bacillus
termination of intragenic and stable RNA transcription,
subtillis and Its Closest Relatives: from Genes to Cells
Proc. Natl. Acad. Sci. USA,
106,
15406)15411,
(Sonenshein, A. L., Hoch, J. A., and Losick, R., eds.)
doi: 10.1073/pnas.0903846106.
ASM Press, Washington, pp. 271)286
38.
Dann, C. E., 3rd, Wakeman, C. A., Sieling, C. L., Baker,
21.
Stormo, G. D., and Ji, Y. (2001) Do mRNAs act as direct
S. C., Irnov, I., and Winkler, W. C. (2007) Structure and
sensors of small molecules to control their expression?
mechanism of a metal)sensing regulatory RNA, Cell, 130,
Proc. Natl. Acad. Sci. USA, 98, 9465)9467, doi: 10.1073/
878)892, doi: 10.1016/j.cell.2007.06.051.
pnas.181334498.
39.
Wakeman, C. A., Ramesh, A., and Winkler, W. C. (2009)
22.
Ravnum, S., and Andersson, D. I. (2001) An adenosyl)
Multiple metal)binding cores are required for metalloregu)
cobalamin (coenzyme)B12))repressed translational
lation by M)box riboswitch RNAs, J. Mol. Biol., 392, 723)
enhancer in the cob mRNA of Salmonella typhimurium,
735, doi: 10.1016/j.jmb.2009.07.033.
Mol. Microbiol., 39, 1585)1594, doi: 10.1046/j.1365)
40.
Furukawa, K., Ramesh, A., Zhou, Z., Weinberg, Z.,
2958.2001.02346.x.
Vallery, T., et al. (2015) Bacterial riboswitches cooperative)
23.
Nou, X., and Kadner, R. J. (2000) Adenosylcobalamin
ly bind Ni2+ or Co2+ ions and control expression of heavy
inhibits ribosome binding to btuB RNA, Proc. Natl. Acad.
metal transporters, Mol. Cell, 57, 1088)1098, doi: 10.1016/
Sci. USA, 97, 7190)7195, doi: 10.1073/pnas.130013897.
j.molcel.2015.02.009.
24.
Sudarsan, N., Barrick, J. E., and Breaker, R. R. (2003)
41.
Xu, J., and Cotruvo, J. A., Jr. (2020) The czcD (NiCo)
Metabolite)binding RNA domains are present in the genes
riboswitch responds to Iron(II), Biochemistry, 59, 1508)
of eukaryotes, RNA, 9, 644)647, doi: 10.1261/rna.
1516, doi: 10.1021/acs.biochem.0c00074.
5090103.
42.
Price, I. R., Gaballa, A., Ding, F., Helmann, J. D., and
25.
Cheah, M. T., Wachter, A., Sudarsan, N., and Breaker,
Ke, A. (2015) Mn2+)sensing mechanisms of yybP)ykoY
R. R. (2007) Control of alternative RNA splicing and gene
orphan riboswitches, Mol. Cell,
57,
1110)1123,
expression by eukaryotic riboswitches, Nature, 447, 497)
doi: 10.1016/j.molcel.2015.02.016.
500, doi: 10.1038/nature05769.
43.
Trausch, J. J., Ceres, P., Reyes, F. E., and Batey, R. T.
26.
Bocobza, S. E., and Aharoni, A. (2008) Switching the light
(2011) The structure of a tetrahydrofolate)sensing
on plant riboswitches, Trends Plant Sci., 13, 526)533,
riboswitch reveals two ligand binding sites in a single
doi: 10.1016/j.tplants.2008.07.004.
aptamer, Structure, 19, 1413)1423, doi: 10.1016/j.str.2011.
27.
Wickiser, J. K., Winkler, W. C., Breaker, R. R., and
06.019.
Crothers, D. M. (2005) The speed of RNA transcription
44.
Gao, A., and Serganov, A. (2014) Structural insights into
and metabolite binding kinetics operate an FMN
recognition of c)di)AMP by the ydaO riboswitch, Nat.
riboswitch, Mol. Cell., 18, 49)60, doi: 10.1016/j.molcel.
Chem. Biol., 10, 787)792, doi: 10.1038/nchembio.1607.
2005.02.032.
45.
Ren, A., and Patel, D. J. (2014) c)di)AMP binds the ydaO
28.
Smith, A. M., Fuchs, R. T., Grundy, F. J., and Henkin,
riboswitch in two pseudo)symmetry)related pockets, Nat.
T. M. (2010) The SAM)responsive SMK box is a reversible
Chem. Biol., 10, 780)786, doi: 10.1038/nchembio.1606.
riboswitch, Mol. Microbiol., 78, 1393)1402, doi: 10.1111/
46.
Serganov, A., Polonskaia, A., Phan, A. T., Breaker, R. R.,
j.1365)2958.2010.07410.x.
and Patel, D. J. (2006) Structural basis for gene regulation
29.
Fuchs, R. T., Grundy, F. J., and Henkin, T. M. (2006) The
by a thiamine pyrophosphate)sensing riboswitch, Nature,
S(MK) box is a new SAM)binding RNA for translational
441, 1167)1171, doi: 10.1038/nature04740.
regulation of SAM synthetase, Nat. Struct. Mol. Biol., 13,
47.
Serganov, A., Huang, L., and Patel, D. J. (2009) Coenzyme
226)233, doi: 10.1038/nsmb1059.
recognition and gene regulation by a flavin mononu)
30.
Lu, C., Smith, A. M., Fuchs, R. T., Ding, F.,
cleotide riboswitch, Nature, 458, 233)237, doi: 10.1038/
Rajashankar, K., et al. (2008) Crystal structures of the
nature07642.
SAM)III/SMK riboswitch reveal the SAM)dependent
48.
Edwards, T. E., and Ferre)D’Amare, A. R. (2006) Crystal
translation inhibition mechanism, Nat. Struct. Mol. Biol.,
structures of the thi)box riboswitch bound to thiamine
15, 1076)1083, doi: 10.1038/nsmb.1494.
pyrophosphate analogs reveal adaptive RNA)small mole)
31.
Gilbert, S. D., Rambo, R. P., Van Tyne, D., and Batey,
cule recognition, Structure, 14, 1459)1468, doi: 10.1016/
R. T. (2008) Structure of the SAM)II riboswitch bound to
j.str.2006.07.008.
БИОХИМИЯ том 86 вып. 8 2021
1174
ARIZA)MATEOS et al.
49.
Ren, A., Rajashankar, K. R., and Patel, D. J. (2012)
67.
Tang, D. J., Du, X., Shi, Q., Zhang, J. L., He, Y. P., et al.
Fluoride ion encapsulation by Mg2+ ions and phosphates in
(2020) A SAM)I riboswitch with the ability to sense and
a fluoride riboswitch, Nature, 486, 85)89, doi: 10.1038/
respond to uncharged initiator tRNA, Nat. Commun., 11,
nature11152.
2794, doi: 10.1038/s41467)020)16417)z.
50.
Serganov, A., Huang, L., and Patel, D. J. (2008) Structural
68.
Sherlock, M. E., Sudarsan, N., Stav, S., and Breaker, R. R.
insights into amino acid binding and gene control by a
(2018) Tandem riboswitches form a natural Boolean logic
lysine riboswitch, Nature, 455, 1263)1267, doi: 10.1038/
gate to control purine metabolism in bacteria, Elife, 7,
nature07326.
doi: 10.7554/eLife.33908.
51.
Huang, L., Serganov, A., and Patel, D. J. (2010) Structural
69.
Peselis, A., and Serganov, A. (2018) ykkC riboswitches
insights into ligand recognition by a sensing domain of the
employ an add)on helix to adjust specificity for polyanion)
cooperative glycine riboswitch, Mol. Cell, 40, 774)786,
ic ligands, Nat. Chem. Biol., 14, 887)894, doi: 10.1038/
doi: 10.1016/j.molcel.2010.11.026.
s41589)018)0114)4.
52.
McCluskey, K., Boudreault, J., St)Pierre, P., Perez)
70.
Knappenberger, A. J., Reiss, C. W., and Strobel, S. A.
Gonzalez, C., Chauvier, A., et al. (2019) Unprecedented
(2018) Structures of two aptamers with differing ligand
tunability of riboswitch structure and regulatory function
specificity reveal ruggedness in the functional landscape of
by sub)millimolar variations in physiological Mg2+, Nucleic
RNA, Elife, 7, doi: 10.7554/eLife.36381.
Acids Res., 47, 6478)6487, doi: 10.1093/nar/gkz316.
71.
Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., and
53.
Watson, P. Y., and Fedor, M. J. (2011) The glmS riboswitch
Breaker, R. R. (2010) An allosteric self)splicing ribozyme
integrates signals from activating and inhibitory metabo)
triggered by a bacterial second messenger, Science, 329,
lites in vivo, Nat. Struct. Mol. Biol., 18, 359)363,
845)848, doi: 10.1126/science.1190713.
doi: 10.1038/nsmb.1989.
72.
Chen, A. G., Sudarsan, N., and Breaker, R. R. (2011)
54.
Mandal, M., Lee, M., Barrick, J. E., Weinberg, Z.,
Mechanism for gene control by a natural allosteric group I
Emilsson, G. M., et al. (2004) A glycine)dependent
ribozyme, RNA, 17, 1967)1972, doi: 10.1261/rna.2757311.
riboswitch that uses cooperative binding to control gene
73.
Sudarsan, N., Hammond, M. C., Block, K. F., Welz, R.,
expression, Science, 306, 275)279, doi: 10.1126/science.
Barrick, J. E., et al. (2006) Tandem riboswitch architec)
1100829.
tures exhibit complex gene control functions, Science, 314,
55.
Butler, E. B., Xiong, Y., Wang, J., and Strobel, S. A. (2011)
300)304, doi: 10.1126/science.1130716.
Structural basis of cooperative ligand binding by the glycine
74.
Stoddard, C. D., and Batey, R. T. (2006) Mix)and)match
riboswitch, Chem. Biol., 18, 293)298, doi: 10.1016/j.chembiol.
riboswitches, ACS Chem. Biol., 1, 751)754, doi: 10.1021/
2011.01.013.
cb600458w.
56.
Sherman, E. M., Esquiaqui, J., Elsayed, G., and Ye, J. D.
75.
Stav, S., Atilho, R. M., Mirihana Arachchilage, G.,
(2012) An energetically beneficial leader)linker interaction
Nguyen, G., Higgs, G., and Breaker, R. R.
(2019)
abolishes ligand)binding cooperativity in glycine ribo)
Genome)wide discovery of structured noncoding RNAs in
switches, RNA, 18, 496)507, doi: 10.1261/rna.031286.111.
bacteria, BMC Microbiol., 19, 66, doi: 10.1186/s12866)
57.
Torgerson, C. D., Hiller, D. A., and Strobel, S. A. (2020)
019)1433)7.
The asymmetry and cooperativity of tandem glycine
76.
Hui, M. P., Foley, P. L., and Belasco, J. G. (2014) Messen)
riboswitch aptamers, RNA, 26, 564)580, doi: 10.1261/rna.
ger RNA degradation in bacterial cells, Annu. Rev. Genet.,
073577.119.
48, 537)559, doi: 10.1146/annurev)genet)120213)092340.
58.
Ames, T. D., and Breaker, R. R. (2011) Bacterial aptamers
77.
Richards, J., and Belasco, J. G. (2021) Riboswitch control
that selectively bind glutamine, RNA Biol., 8, 82)89,
of bacterial RNA stability, Mol. Microbiol., doi: 10.1111/
doi: 10.4161/rna.8.1.13864.
mmi.14723.
59.
Ren, A., Xue, Y., Peselis, A., Serganov, A., Al)Hashimi,
78.
Richards, J., and Belasco, J. G. (2021) Widespread protec)
H. M., and Patel, D. J. (2015) Structural and dynamic basis
tion of RNA cleavage sites by a riboswitch aptamer that
for low)affinity, high)selectivity binding of L)glutamine by
folds as a compact obstacle to scanning by RNase E, Mol.
the glutamine riboswitch, Cell Rep., 13, 1800)1813,
Cell, 81, 127)138.e124, doi: 10.1016/j.molcel.2020.10.025.
doi: 10.1016/j.celrep.2015.10.062.
79.
Shahbabian, K., Jamalli, A., Zig, L., and Putzer, H. (2009)
60.
Sherlock, M. E., Malkowski, S. N., and Breaker, R. R.
RNase Y, a novel endoribonuclease, initiates riboswitch
(2017) Biochemical validation of a second guanidine
turnover in Bacillus subtilis, EMBO J., 28, 3523)3533,
riboswitch class in Bacteria, Biochemistry, 56, 352)358,
doi: 10.1038/emboj.2009.283.
doi: 10.1021/acs.biochem.6b01270.
80.
Winkler, W. C., Nahvi, A., Roth, A., Collins, J. A., and
61.
Huang, L., Wang, J., and Lilley, D. M. J. (2017) The struc)
Breaker, R. R. (2004) Control of gene expression by a nat)
ture of the guanidine)II riboswitch, Cell. Chem. Biol., 24,
ural metabolite)responsive ribozyme, Nature, 428, 281)
695)702.e2, doi: 10.1016/j.chembiol.2017.05.014.
286, doi: 10.1038/nature02362.
62.
Reiss, C. W., and Strobel, S. A. (2017) Structural basis for
81.
Collins, J. A., Irnov, I., Baker, S., and Winkler, W. C. (2007)
ligand binding to the guanidine)II riboswitch, RNA, 23,
Mechanism of mRNA destabilization by the glmS ribozyme,
1338)1343, doi: 10.1261/rna. 061804.117.
Genes Dev., 21, 3356)3368, doi: 10.1101/gad.1605307.
63.
Wuebben, C., Vicino, M. F., Mueller, M., and
82.
Klein, D. J., and Ferre)D’Amare, A. R. (2006) Structural
Schiemann, O. (2020) Do the P1 and P2 hairpins of the
basis of glmS ribozyme activation by glucosamine)6)phos)
Guanidine)II riboswitch interact? Nucleic Acids Res., 48,
phate, Science, 313, 1752)1756, doi: 10.1126/science.
10518)10526, doi: 10.1093/nar/gkaa703.
1129666.
64.
Malkowski, S. N., Spencer, T. C. J., and Breaker, R. R.
83.
Cochrane, J. C., Lipchock, S. V., and Strobel, S. A. (2007)
(2019) Evidence that the nadA motif is a bacterial
Structural investigation of the GlmS ribozyme bound to its
riboswitch for the ubiquitous enzyme cofactor NAD+,
catalytic cofactor, Chem. Biol., 14, 97)105, doi: 10.1016/
RNA, 25, 1616)1627, doi: 10.1261/rna.072538.119.
j.chembiol.2006.12.005.
65.
Huang, L., Wang, J., and Lilley, D. M. J. (2020) Structure
84.
Davis, J. H., Dunican, B. F., and Strobel, S. A. (2011)
and ligand binding of the ADP)binding domain of the
glmS Riboswitch binding to the glucosamine)6)phosphate
NAD+ riboswitch, RNA, 26, 878)887, doi: 10.1261/rna.
alpha)anomer shifts the pKa toward neutrality,
074898.120.
Biochemistry, 50, 7236)7242, doi: 10.1021/bi200471c.
66.
Chen, H., Egger, M., Xu, X., Flemmich, L.,
85.
Caron, M. P., Bastet, L., Lussier, A., Simoneau)Roy, M.,
Krasheninina, O., et al. (2020) Structural distinctions
Masse, E., and Lafontaine, D. A. (2012) Dual)acting
between NAD+ riboswitch domains 1 and 2 determine dif)
riboswitch control of translation initiation and mRNA
ferential folding and ligand binding, Nucleic Acids Res., 48,
decay, Proc. Natl. Acad. Sci. USA, 109, E3444)3453,
12394)12406, doi: 10.1093/nar/gkaa1029.
doi: 10.1073/pnas.1214024109.
БИОХИМИЯ том 86 вып. 8 2021
МЕХАНИЗМЫ РАБОТЫ РИБОПЕРЕКЛЮЧАТЕЛЕЙ
1175
86.
Takemoto, N., Tanaka, Y., and Inui, M. (2015) Rho and
dering access to distal cleavage sites, Mol. Cell, 74, 284)
RNase play a central role in FMN riboswitch regulation in
295.e5, doi: 10.1016/j.molcel.2019.01.044.
Corynebacterium glutamicum, Nucleic Acids Res., 43, 520)
95. Reining, A., Nozinovic, S., Schlepckow, K., Buhr, F.,
529, doi: 10.1093/nar/gku1281.
Furtig, B., and Schwalbe, H. (2013) Three)state mecha)
87.
Pursley, B. R., Fernandez, N. L., Severin, G. B., and
nism couples ligand and temperature sensing in riboswitch)
Waters, C. M. (2019) The Vc2 cyclic di)GMP)dependent
es, Nature, 499, 355)359, doi: 10.1038/nature12378.
riboswitch of Vibrio cholerae regulates expression of an
96. Mellin, J. R., Koutero, M., Dar, D., Nahori, M. A.,
upstream putative small RNA by controlling RNA stability,
Sorek, R., and Cossart, P. (2014) Sequestration of a two)
J. Bacteriol., 201, doi: 10.1128/JB.00293)19.
component response regulator by a riboswitch)regulated
88.
Pursley, B. R., Maiden, M. M., Hsieh, M. L., Fernandez,
noncoding RNA, Science, 345, 940)943, doi: 10.1126/
N. L., Severin, G. B., and Waters, C. M. (2018) Cyclic di)
science.1255083.
GMP regulates TfoY in Vibrio cholerae to control motility
97. DebRoy, S., Gebbie, M., Ramesh, A., Goodson, J. R.,
by both transcriptional and posttranscriptional mecha)
Cruz, M. R., et al. (2014) A riboswitch)containing sRNA
nisms, J. Bacteriol., 200, doi: 10.1128/JB.00578)17.
controls gene expression by sequestration of a response reg)
89.
Inuzuka, S., Nishimura, K., Kakizawa, H., Fujita, Y.,
ulator, Science, 345, 937)940, doi: 10.1126/science.
Furuta, H., et al. (2016) Mutational analysis of structural
1255091.
elements in a class)I cyclic di)GMP riboswitch to elucidate
98. Karunker, I., Rotem, O., Dori)Bachash, M.,
its regulatory mechanism, J. Biochem., 160, 153)162,
Jurkevitch, E., and Sorek, R. (2013) A global transcrip)
doi: 10.1093/jb/mvw026.
tional switch between the attack and growth forms of
90.
Inuzuka, S., Kakizawa, H., Nishimura, K. I., Naito, T.,
Bdellovibrio bacteriovorus, PLoS One,
8, e61850,
Miyazaki, K., et al. (2018) Recognition of cyclic)di)GMP
doi: 10.1371/journal.pone.0061850.
by a riboswitch conducts translational repression through
99. Weber, L., Thoelken, C., Volk, M., Remes, B.,
masking the ribosome)binding site distant from the
Lechner, M., and Klug, G. (2016) The conserved Dcw
aptamer domain, Genes Cells, 23, 435)447, doi: 10.1111/
gene cluster of R. sphaeroides is preceded by an uncom)
gtc.12586.
monly extended 5′ leader featuring the sRNA UpsM, PLoS
91.
Kaberdin, V. R. (2003) Probing the substrate specificity of
One, 11, e0165694, doi: 10.1371/journal.pone.0165694.
Escherichia coli RNase E using a novel oligonucleotide)
100. Soutourina, O. A., Monot, M., Boudry, P., Saujet, L.,
based assay, Nucleic Acids Res.,
31,
4710)4716,
Pichon, C., et al. (2013) Genome)wide identification of
doi: 10.1093/nar/gkg690.
regulatory RNAs in the human pathogen Clostridium diffi
92.
Mackie, G. A. (1998) Ribonuclease E is a 5′)end)depen)
cile, PLoS Genet., 9, e1003493, doi: 10.1371/journal. pgen.
dent endonuclease, Nature, 395, 720)723, doi: 10.1038/
1003493.
27246.
101. Haller, A., Rieder, U., Aigner, M., Blanchard, S. C., and
93.
Callaghan, A. J., Marcaida, M. J., Stead, J. A., McDowall,
Micura, R. (2011) Conformational capture of the SAM)II
K. J., Scott, W. G., and Luisi, B. F. (2005) Structure of
riboswitch, Nat. Chem. Biol., 7, 393)400, doi: 10.1038/
Escherichia coli RNase E catalytic domain and implica)
nchembio.562.
tions for RNA turnover, Nature,
437,
1187)1191,
102. Nelson, J. W., Sudarsan, N., Furukawa, K., Weinberg, Z.,
doi: 10.1038/nature04084.
Wang, J. X., and Breaker, R. R. (2013) Riboswitches in
94.
Richards, J., and Belasco, J. G. (2019) Obstacles to scan)
eubacteria sense the second messenger c)di)AMP, Nat.
ning by RNase E govern bacterial mRNA lifetimes by hin)
Chem. Biol., 9, 834)839, doi: 10.1038/nchembio.1363.
RIBOSWITCH MECHANISMS: NEW TRICKS FOR AN OLD DOG
Review
A. Ariza Mateos, A. Nuthanakanti, and A. Serganov*
Department of Biochemistry and Molecular Pharmacology, New York University Grossman School of Medicine,
New York, New York 10016, USA; e mail: alexander.serganov@nyulangone.org
Discovered almost twenty years ago, riboswitches turned out to be one of the most broadly spread regulatory systems
in bacteria, with representatives found in eukaryotes and archaea. Unlike many other regulatory elements, riboswitch)
es are entirely composed of RNA and capable of modulating expression of genes by direct binding of small cellular
molecules. While bacterial riboswitches were initially thought to control production of enzymes and transporters asso)
ciated with small organic molecules via feedback regulatory circuits, later findings identified riboswitches directing
expression of a wide range of genes and responding to various classes of molecules, including ions, signaling mole)
cules, and others. The 5′)untranslated mRNA regions host the vast majority of riboswitches, which modulate tran)
scription or translation of downstream genes through conformational rearrangements in the ligand)sensing domains
and adjacent expression)controlling platforms. Over years, the repertoire of the regulatory mechanisms employed by
riboswitches has greatly expanded and most recent studies highlighted the importance of alternative mechanisms,
such as RNA degradation, for riboswitch)mediated genetic circuits. This review discusses the plethora of bacterial
riboswitch mechanisms and illustrates how riboswitches utilize different features and approaches to elicit various reg)
ulatory responses.
Keywords: riboswitch, transcription, translation, mRNA, metabolite
БИОХИМИЯ том 86 вып. 8 2021