БИОХИМИЯ, 2021, том 86, вып. 8, с. 1176 - 1194
УДК 577.21
КОД ВНУТРИ КОДА: КАК КОДОНЫ ОСУЩЕСТВЛЯЮТ
ТОНКУЮ НАСТРОЙКУ СВОРАЧИВАНИЯ БЕЛКА В КЛЕТКЕ
Обзор
© 2021
А.А. Комар1,2,3,4
1 Center for Gene Regulation in Health and Disease and Department of Biological, Geological
and Environmental Sciences, Cleveland State University, Cleveland, Ohio 44115, USA; e mail: a.komar@csuohio.edu
2 Department of Biochemistry and Center for RNA Science and Therapeutics,
Case Western Reserve University, Cleveland, Ohio 44106, USA
3 Genomic Medicine Institute, Lerner Research Institute, Cleveland Clinic, Cleveland, Ohio 44195, USA
4 DAPCEL, Inc., Cleveland, Ohio 44106, USA
Поступила в редакцию 11.04.2021
После доработки 11.04.2021
Принята к публикации 28.04.2021
Генетический код устанавливает соответствие между последовательностью определенного триплета нуклео
тидов в молекуле мРНК, называемого кодоном, и аминокислотой, добавляемой к растущей полипептидной
цепи в процессе синтеза белка. Комбинации четырех азотистых оснований (A, G, U и C) приводят к обра
зованию 64 триплетных кодонов, из которых 61 являются смысловыми (кодирующими аминокислоты), а
3 - стоп кодонами (определяющими терминацию трансляции). У большинства организмов для синтеза
белка используется 20 стандартных аминокислот. Таким образом, генетический код является вырожден
ным, так как большинство аминокислот (за исключением метионина и триптофана) кодируется более, чем
одним (синонимическим) кодоном. Изначально предполагалось, что синонимические кодоны обладают
полностью эквивалентными функциями, однако обнаружение того факта, что синонимические кодоны ис
пользуются в мРНК с неодинаковой частотой, позволило предположить, что выбор положения определен
ных кодонов в мРНК может быть не случаен и может нести дополнительную функциональную нагрузку по
мимо кодирования аминокислотных остатков. Установление факта неэквивалентного использования кодо
нов в мРНК поставило вопрос о возможности наличия в генетическом коде дополнительной информации.
Действительно, было обнаружено, что генетический код имеет «несколько слоёв», несущих такую дополни
тельную информацию, и что синонимические кодоны расположены в мРНК не случайно, а стратегически -
чтобы гарантировать определенную кинетику трансляции, которая, в свою очередь, обеспечивает тонкую
настройку процесса сворачивания белка в клетке и способствует упорядоченному котрансляционному сво
рачиванию, которое происходит в ходе поэтапного и последовательного структурирования отдельных
участков полипептидной цепи, выходящих из рибосомы в различные моменты времени. В настоящем обзо
ре приведены основные данные, полученные в этой области, которые способствовали установлению роли
синонимических кодонов (и их использования) в процессе сворачивания белков в клетке.
КЛЮЧЕВЫЕ СЛОВА: генетический код, использование/предпочтение кодонов, синонимические кодоны,
кинетика трансляции, насцентные пептиды, котрансляционное сворачивание белка.
DOI: 10.31857/S0320972521080091
ВВЕДЕНИЕ
дирует одну аминокислоту, что код является не
перекрывающимся, что последовательность ос
Шестьдесят лет назад Crick, Barnett, Brenner
нований считывается с фиксированной началь
и Watts Tobin постулировали основные свойства
ной точки, что код не содержит каких либо осо
генетического кода [1]. Они предположили, что
бых «запятых», которые могли бы указывать на
группа из трех азотистых оснований (кодон) ко
выбор правильных триплетов, и что код является
Принятые сокращения: мРНК - информационная/матричная РНК; тРНК - транспортная РНК, РНКаза A - ри
бонуклеаза A; ЯМР - ядерный магнитный резонанс (nuclear magnetic resonance); ADAMTS13 - 13 й член семейства диз
интегринов и металлопротеиназ с тромбоспондиновым мотивом 1 го типа (a disintegrin and metalloproteinase with a throm
bospondin type 1 motif, member 13); CAT - хлорамфениколацетилтрансфераза (chloramphenicol acetyltransferase);
ExAC - консорциум агрегации экзомов (the Exome Aggregation Consortium); FRET - резонансный перенос энергии флу
оресценции (fluorescence resonance energy transfer); MDR1 - ген множественной устойчивости к лекарствам 1 (multidrug
resistance 1); PGK - фосфоглицераткиназа (phosphoglycerate kinase); P gp - Р гликопротеин (P glycoprotein); VWF - фак
тор фон Виллебранда (von Willebrand factor).
1176
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1177
вырожденным, т.е., как правило, одна конкрет
тивностью синтеза белка [32-36]. В рамках этой
ная аминокислота может кодироваться несколь
теории также было сделано предположение о
кими (синонимическими) кодонами [1]. После
том, что естественный отбор благоприятствует
дующие работы по изучению связывания амино
выбору оптимальных и часто используемых ко
ацил тРНК с короткими тринуклеотидными
донов (в противовес редким) для обеспечения
матрицами позволили установить общие законо
эффективной продукции белка [32-36]. Однако
мерности кодирования и выявить точную при
позднее стало понятно, что редко используемые
роду вырожденности генетического кода [2-8].
кодоны также подвержены направленному от
В дальнейшем было установлено, что сино
бору и обладают важной функцией, особенно в
нимические кодоны, кодирующие одну и ту же
гомеостазе мРНК и регуляции её стабильности
аминокислоту, могут быть не полностью эквива
внутри клетки [37-44], а также при сворачива
лентными [9-19]. Было показано, что декодиро
нии белка в клетке (см. ниже).
вание синонимических кодонов происходит с
Параллельно с попытками определить при
различными скоростями и эффективностью (и
роду генетического кода и процесса декодиро
точностью), и что частота использования сино
вания было установлено, что аминокислотная
нимических кодонов во многом определяет ско
последовательность белка содержит код свора
рость их декодирования [20-22]. В этой связи
чивания белка, т.е. информацию, необходимую
следует отметить, что скорость и эффективность
для определения его уникальной трехмерной
считывания кодона в значительной степени оп
структуры [45]. Хотя точная природа кода свора
ределяется
доступностью соответствую
чивания белка не была установлена (и остается
щих/родственных (cognate) тРНК, в то время
не полностью разгаданной до настоящего вре
как конкуренция с другими/неродственными
мени), тем не менее был сделан вывод о том, что
тРНК оказывает влияние на точность трансля
нативное свернутое состояние белка достигает
ции [23-27]. Так как было обнаружено, что час
ся не путем случайного перебора всех возмож
тоты использования кодонов обычно коррели
ных конформаций на пути к нативной структуре
руют с доступностью и концентрацией соответ
(что потребовало бы огромного количества вре
ствующих тРНК [12, 13, 16], то было сделано
мени для его осуществления), но, скорее всего,
предположение о том, что выбор кодона влияет
достигается через ограниченный набор четко
на скорость элонгации [20-22] (действительно,
определенных путей с ограниченным числом
как оказалось, концентрация тРНК является од
доступных конформационных пространств (см.
ним из основных факторов, определяющих вре
обзоры [46-51]).
мя декодирования кодонов [28, 29]). Во многих
Примерно в то же время (в начале 60 х и в
последующих экспериментах было показано,
70 е гг. прошлого столетия) было также обнару
что часто используемые кодоны подвергаются
жено, что сворачивание белка in vivo, по крайней
более быстрой трансляции по сравнению с те
мере, в случае некоторых белков, является кот
ми, что используются реже. Это обусловлено
рансляционным процессом, (т.е. протекает во
большей доступностью (во время процесса счи
время синтеза белка на рибосоме) [52-58]. Что в
тывания мРНК) соответствующих часто встре
то время было неясно, так это то, могут ли рибо
чающихся родственных тРНК [20-22, 30, 31].
сома и/или процесс трансляции оказывать влия
Кроме того, было обнаружено, что гены с высо
ние на процесс сворачивания белка, и до какой
ким уровнем экспрессии содержат в своей по
степени конкретный путь сворачивания белка в
следовательности более предпочтительные/час
клетке (если он существует) может обеспечивать
то используемые кодоны, в то время как низко
высокую эффективность процесса сворачивания
экспрессируемые гены оказались обогащены
белка in vivo. Ответы на эти вопросы стали мед
редко используемыми синонимическими кодо
ленно возникать в течение последних 50 лет и
нами [17, 18]. И несмотря на то что набор опти
продолжают появляться в наши дни.
мальных/часто используемых кодонов различа
Декодирование или трансляция молекул
ется у разных биологических видов (эти измене
мРНК осуществляется рибосомами с добавле
ния тем не менее оказались скоординированны
нием каждой новой аминокислоты к растущей
ми с изменениями состава генов тРНК [13, 16]),
полипептидной цепи. Можно предположить,
был сделан вывод о том, что мРНК не только ко
что природа процесса декодирования, или сама
дирует аминокислотную последовательность
рибосома (как большой макромолекулярный
белка, но и определяет уровень его экспрессии
комплекс), и/или скорость полимеризации по
[32-36]. Как следствие, была выдвинута теория
липептидной цепи могут влиять на конечную
совместной эволюции кодонов и тРНК, которая
конформацию белка, воздействуя на конформа
объясняла необходимость существования опре
ционное пространство растущей полипептид
деленного баланса между точностью и эффек
ной цепи и/или на кинетику процесса сворачи
6 БИОХИМИЯ том 86 вып. 8 2021
1178
КОМАР
вания белка в процессе синтеза полипептидной
КОД СВОРАЧИВАНИЯ БЕЛКА
цепи, или на то и другое. Необходимо отметить,
что это предположение нашло свое подтвержде
Основополагающие эксперименты по обра
ние в ходе многочисленных недавних экспери
тимой денатурации рибонуклеазы, выполнен
ментов, в которых было показано, что механизм
ные Christian Anfinsen и его коллегами в
декодирования, а также сама рибосома и, что
1950-1960 х гг. [45], а также последующие иссле
важно, кинетика трансляции, которая опреде
дования других групп с использованием большо
ляет скорость полимеризации полипептидной
го числа других белков показали, что аминокис
цепи, могут оказывать влияние на процесс сво
лотная последовательность белка содержит всю
рачивания белка и определять его эффектив
необходимую информацию для образования его
ность in vivo (см. ниже). Однако таких данных не
уникальной трехмерной структуры
[46-51].
существовало до 1985 г., когда стереохимичес
Многие последующие теоретические исследова
кий анализ осуществляемой рибосомой реакции
ния, а также работы по компьютерному модели
транспептидации, выполненный Лимом и Спи
рованию сворачивания белка, включая недавние
риным, позволил предположить, что процесс
прорывные исследования в этой области, пока
декодирования может влиять на сворачивание
завшие возможность точного предсказания
белка, и что рибосома может способствовать
структуры белка на основе его аминокислотной
формированию альфа спирали в растущей по
последовательности [62, 63], свидетельствуют о
липептидной цепи [59, 60]. Таким образом, по
том, что ключевой постулат «проблемы сворачи
лученные данные позволили утверждать, что
вания белка» является верным. В связи с этим
трансляция - это не просто процесс последова
необходимо отметить недавние исследования по
тельного добавления аминокислот к растущему
in silico моделированию и предсказанию белко
полипептиду, но и процесс, который может ак
вых структур, которые в ряде случаев оказались
тивно влиять на сворачивание белка.
практически неотличимыми от структур, опре
Впоследствии и мы, и другие авторы показа
деленных с помощью рентгеновской кристал
ли, что различия в скоростях трансляции в раз
лографии [64], что является значительным дос
личных участках мРНК, которые в основном
тижением в области и важным доказательством
обусловлены неравномерным и неслучайным
постулата (догмы) Анфинсена.
использованием синонимических кодонов, мо
Для объяснения процесса сворачивания бел
гут влиять на сворачивание белка, обусловливая
ка была разработана концепция «энергетичес
упорядоченное и последовательное образование
кой воронки», предполагающая существование
дискретных структур во вновь синтезированных
множества различных путей, которые приводят
участках насцентной полипептидной цепи, и
процесс сворачивания белка к нативной кон
что кинетика синтеза белка может служить в ка
формации, характеризующейся минимумом
честве вторичного кода для тонкой настройки
свободной энергии (см. обзоры [46-51]). В даль
процесса сворачивания белка в клетке (см. об
нейшем эта концепция была дополнена появле
зор [61]). Более того, анализ распределения ко
нием новой модели, описывающей процесс сво
донов в молекуле мРНК позволил нам и другим
рачивания белка через образование отдельных
авторам найти «отпечатки» структуры белка, за
кооперативных единиц сворачивания, так назы
кодированные в последовательности мРНК
ваемых фолдонов (см. обзоры [65-67]). Действи
[61]. Стало ясно, что естественный отбор не
тельно, для нескольких белков было показано,
только направлен на поддержание эффективной
что их сворачивание происходит через такие
экспрессии белка, но и также благоприятствует
последовательные промежуточные состояния, а
определенному и неслучайному распределению
также высказано предположение о том, что ко
кодонов в матричной РНК, и что мутационное
оперативная по своему существу природа фолдо
давление и генетический дрейф способствуют
нов определяет быстрое формирование натив
расположению редко используемых кодонов в
ной структуры [65-67]. Гипотеза фолдонов так
определенных местах, опосредуя, таким обра
же смогла объяснить предпочтительный выбор
зом, выполнение ими важных функций, в част
определенных энергетических состояний в обра
ности в процессе сворачивания белка [61].
зующихся кооперативных структурах, направ
Настоящий обзор посвящен рассмотрению
ленный в сторону образования нативных взаи
ряда ключевых результатов, свидетельствующих
модействий. Ограниченное количество и не
в пользу существования внутри генетического
большой размер таких фолдонов (∼15-35 ами
кода вторичного кода, который способствует
нокислотных остатков) обеспечивали также ре
тонкой регуляции сворачивания белка в клетке,
шение парадокса Левинталя [68].
а также рассмотрению роли редких кодонов в
Для многих, в особенности небольших, бел
этом процессе.
ков теоретические исследования, эксперименты
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1179
по компьютерному моделированию белковой
этапы сворачивания белка и предотвращая не
структуры, а также эксперименты in vitro по раз
правильное сворачивание [73-78]. Тем не менее
ворачиванию и сворачиванию полипептидной
предполагается, что шапероны и катализаторы
цепи пришли в полное согласие друг с другом
фолдинга в основном принимают участие в ки
(работа [69] и указанные в ней ссылки). Эти ис
нетическом разделении между правильным сво
следования дали общие ответы на ряд ключевых
рачиванием и агрегацией белка. Таким образом,
вопросов, а именно: как сворачиваются белки и
считается, что они в основном влияют на выход
почему они сворачиваются таким образом?
правильно свернутого белка, а не на механизм
Тем не менее во многих случаях (и особенно
его сворачивания [73-78]. Необходимо также
в случае больших белков) эти эксперименты не
отметить, что котрансляционное сворачивание
позволяли описать весь процесс целиком [70].
белка начинается на самых ранних этапах синте
Кроме того, многие попытки добиться сворачи
за полипептидной цепи на рибосоме, внутри ри
вания (рефолдинга) в пробирке очищенных де
босомного канала, с образования отдельных эле
натурированных белков имели лишь частичный
ментов вторичной структуры (таких как, напри
успех. К тому же восстановление трехмерной
мер, альфа спирали) [89-92] в верхних отделах
структуры в пробирке для некоторых белков
канала, а также с образования ряда небольших
оказалось чрезвычайно медленным процессом
элементов третичной структуры - в «вестибюле»
[46], занимавшим часы или даже дни [71, 72]. В
(нижнем/расширенном участке) рибосомного
то же время было хорошо известно, что эта
канала [93-96] (рис. 1). Важно отметить, что ко
«проблема несоответствия» скорости сворачива
трансляционное сворачивание является «век
ния белка в пробирке и в клетке легко решается
торным» (направленным) процессом, происхо
in vivo. Таким образом, стало понятно, что котранс
дящим от N к C концу растущего полипептида
ляционная природа сворачивания белка в клетке
и, как правило, заключается в последовательном
может служить ключом к пониманию природно
структурировании отдельных участков полипеп
го кода сворачивания белков. Как результат, ис
тидной цепи, выходящих из рибосомного кана
следователи в области теории сворачивания бел
ла [61, 79-84].
ка, компьютерного моделирования процесса
Несмотря на выявление ряда исключений из
сворачивания, а также исследователи, непосред
этого общего правила, когда было показано, что
ственно вовлеченные в лабораторные экспери
сворачивание определенного небольшого доме
менты по изучению фолдинга, переключили
на белка может протекать в направлении, про
свое внимание на изучение процесса фолдинга
тивоположном по отношению к синтезу поли
in vivo, для того чтобы лучше понять сходства и
пептидной цепи на рибосоме ([97] и указанные в
различия путей и механизмов сворачивания бел
ней ссылки), все же был сделан вывод о том, что
ка in vitro и in vivo (см. обзоры [73-78]).
для большинства белков синтез и сворачивание
В этой связи следует отметить, что котранс
происходят параллельно и однонаправленно
ляционное сворачивание белка представляет со
бой процесс, который существенно отличается
от процесса сворачивания (рефолдинга) белка
in vitro [79-84]. Это процесс, который характери
зуется тем, что насцентная полипептидная цепь
в процессе её синтеза остается прикреплённой
(через её C концевой участок) к рибосоме, что
приводит к ограничению её конформационного
пространства и уменьшению числа возможных
степеней свободы, тем самым существенно огра
Рис. 1. Котрансляционное сворачивание белка представ
ничивая число возможных интермедиатов фол
ляет собой поэтапный векторный процесс, протекающий в
динга и, следовательно, снижая число возмож
большинстве случаев от N к C концу растущей полипеп
ных путей сворачивания белка [79-84]. Также
тидной цепи. Появление отдельных промежуточных ин
термедиатов сворачивания связано с локальными и общи
необходимо отметить, что рибосома служит в ка
ми скоростями трансляции. Процесс сворачивания начи
честве хаба [85] для многих молекулярных шапе
нается на самых ранних этапах синтеза полипептидной це
ронов. Кроме того, было показано, что самой
пи на рибосоме с образованием некоторых элементов вто
рибосоме присуща «шаперон подобная» актив
ричной структуры (таких, как альфа спирали) внутри ри
ность ([86-88] и указанные в них ссылки). Мо
босомного канала (в его верхних участках), а также неко
торых элементов третичной структуры, формирующихся
лекулярные шапероны и катализаторы фолдинга
внутри «вестибюля» (нижнего/расширенного участка ка
могут взаимодействовать с растущей насцентной
нала) на выходе из него. Третичная структура белка оказы
полипептидной цепью, выходящей из рибосом
вается практически полностью сформированной к концу
ного канала, таким образом ускоряя медленные
его синтеза
БИОХИМИЯ том 86 вып. 8 2021
6*
1180
КОМАР
[61, 79]. Наконец, котрансляционное сворачи
1) существование легко измеряемых свойств
вание белков тесным образом связано с элонга
(присущих свернутому белку), которые позво
цией трансляции, которая не является однород
ляли бы проводить оценку правильного свора
ным/непрерывным процессом ([61, 79, 98] и
чивания насцентных полипептидных цепей на
указанные в них ссылки), происходящим с по
рибосоме;
стоянной/одной скоростью. Было высказано
2) наличие уверенности в том, что конкрет
предположение о том, что локальные скорости
ные исследуемые структурные особенности
биосинтеза белка эволюционно адаптированы
действительно связаны с насцентными цепями,
таким образом, чтобы оптимизировать процесс
а не с полипептидными цепями, связанными с
фолдинга, позволяя обходить глубокие кинети
рибосомами/полирибосомами неспецифически;
ческие ловушки в процессе котрансляционного
3) наличие уверенности в том, что изучаемые
сворачивания ([61, 79, 98-107] и указанные в
полипептидные цепи были синтезированы
них ссылки). Таким образом, было постулирова
de novo для гарантии того, что измеряемые спе
но, что самосборка белка in vivo скоординирова
цифические свойства белка (отражающие его
на с синтезом белка и эволюционно оптимизи
фолдинг) действительно являются результатом
рована для быстрого высокоэффективного дос
котрансляционного процесса.
тижения правильно свернутой конформации
Большая часть этих ранних экспериментов
полипептидной цепи ([61, 79, 98-107] и ссылки
заключалась в выделении/фракционировании
в них). Мы и другие авторы предположили
(из клеточных экстрактов) комплексов, состоя
(см. ниже), что редкие кодоны (которые транс
щих из насцентных цепей, связанных с рибосо
лируются замедленно) могут находиться под
мами, при помощи градиента плотности сахаро
давлением эволюционного отбора для обеспече
зы. Затем проводили оценку структурных
ния эффективного котрансляционного свора
свойств насцентных полипептидных цепей по
чивания белка, а места локализации трансляци
средством определения ряда их параметров:
онных пауз могут определять то временнóе ок
1) специфической ферментативной активности
но, в котором происходит локальное сворачива
[52-54], 2) распознавания определенных струк
ние фрагментов белковой структуры, особенно
тур специфическими/конформационными ан
в тех участках, где процесс далёк от равновесия
тителами [55] или 3) определения образования
([61, 79, 98-107] и ссылки в них). Тем не менее
правильных дисульфидных связей внутри и/или
следует отметить, что многие детали процесса
между насцентными цепями [56-58]. В этих
котрансляционного сворачивания остаются
первых экспериментах было принципиально
лишь частично понятыми, а его механизм и путь
показано существование котрансляционного
остаются предметом интенсивных исследова
сворачивания белков, однако понимание меха
ний в наши дни.
низмов и масштабов этого явления было в то
время весьма ограничено.
Интерес к изучению котрансляционного
КОТРАНСЛЯЦИОННОЕ СВОРАЧИВАНИЕ
сворачивания затем в значительной мере ослаб,
БЕЛКА: ПРОГРЕСС В ПОНИМАНИИ
в связи с доминированием экспериментов по де
ЭТОГО ПРОЦЕССА
натурации/ренатурации белков in vitro. Возоб
новление интереса произошло в конце 1980 х и в
Прогресс, достигнутый в понимании котранс
середине 1990 х гг. с осознанием того, что ко
ляционного сворачивания белков неразрывно
трансляционное сворачивание, вероятно, харак
связан с разработкой новых методов и подходов
терно не только для нескольких отдельных слу
для его изучения (см. обзор [82]).
чаев, но может быть присуще многим белкам в
Работы по изучению котрансляционного
клетке (см. обзор [82]). Исследованиям в этой
сворачивания белков можно разделить на три
области способствовало массовое применение
основных периода. В начале 1960 х и в 1970 е гг.
бесклеточных систем трансляции in vitro, таких
были получены первые данные, которые позво
как, например, лизат из ретикулоцитов кролика
лили предположить, что сворачивание полипе
(RRL - rabbit reticulocyte lysate), который был
птидов in vivo, по крайней мере, в случае некото
разработан Pelham и Jackson в 1976 г. [108]; ис
рых белков, является котрансляционным про
пользование новых модельных белков, в част
цессом [52-58]. Это в основном были экспери
ности, хемилюминесцентных и флуоресцентных;
менты, проводившиеся в равновесных условиях,
разработка новых кинетических подходов, кото
для которых были сформулированы и определе
рые проложили дорогу к изучению котрансляци
ны основные требования к методам, используе
онного сворачивания белков в реальном времени.
мым для изучения котрансляционного сворачи
В этом плане работа Колба, Макеева и Спи
вания белков, такие как:
рина представляет особый интерес, т.к. является
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1181
одной из первых попыток изучения котрансля
вило неоспоримые доказательства в пользу ко
ционного сворачивания белка в реальном вре
трансляционного сворачивания белков (см. об
мени. Эти авторы разработали методику, позво
зоры [61, 82]).
ляющую наблюдать ферментативную актив
Начало третьего периода исследований ко
ность вновь синтезированной люциферазы
трансляционного сворачивания можно отнести
светлячка в бесклеточной системе в люмино
примерно к началу 2000 х годов. Этот период
метрической кювете в реальном времени [109].
продолжается и до сих пор и характеризуется
Люциферазная активность, указывающая на
взрывным ростом большого количества новых
правильное сворачивание белка, регистрирова
методов и подходов для изучения котрансляци
лась при образовании полноцепочечной моле
онного сворачивания белков, таких как одномо
кулы белка в реакции трансляции [109]. Следует
лекулярная флуоресцентная спектроскопия и
отметить, что добавление РНКазы A нарушало и
флуоресценция с временным разрешением [96],
трансляцию, и накопление активной люцифе
позволяющих изучать процесс сворачивания
разы, указывая на то, что синтез и сворачивание
белка в реальном времени [96] и на уровне еди
люциферазы светлячка происходят одновре
ничных молекул; ЯМР спектроскопия и крио
менно. Несмотря на то что авторы не смогли об
электронная микроскопия, которые, в свою
наружить люциферазную активность в поли
очередь, позволяют определить структуру свя
пептидных цепях, связанных с рибосомой (было
занных с рибосомой насцентных цепей с ато
показано, что 12 C концевых аминокислотных
марным разрешением. Разработка множества
остатков имеют важное значение для активнос
других подходов, сочетающих эксперименты
ти фермента ([109] и ссылки в ней), тем не менее
in vivo и in vitro (бесклеточная трансляция), и су
они обнаружили, что люцифераза светлячка
щественный прогресс в разработке компьютер
становится активной сразу после высвобожде
ных подходов для моделирования фолдинга так
ния насцентных цепей [109]. Важно отметить,
же приближают нас к более глубокому понима
что столь быстрое проявление ферментативной
нию процесса котрансляционного сворачива
активности было несовместимо со сценарием
ния белков (см. обзор [82]).
посттрансляционного сворачивания, поскольку
Стало очевидно, что котрансляционное сво
рефолдинг фермента из денатурированного со
рачивание характерно почти для всех белков как
стояния проходил намного медленнее [109].
прокариотических, так и эукариотических кле
В то же время наша группа на кафедре моле
ток, как однодоменных, так и мультидоменных,
кулярной биологии биологического факультета
а также состоящих из одной или нескольких
МГУ им. М.В. Ломоносова совместно с лабора
субъединиц, цитоплазматических, секретируе
торией А.С. Спирина в Институте белка в Пу
мых или мембраносвязанных, и что без понима
щино использовала метод связывания гема с
ния механизма и пути котрансляционного сво
глобиновыми цепями для изучения котрансля
рачивания белков всестороннее понимание ко
ционного сворачивания цепей альфа глобина
да сворачивания белка будет невозможным.
[110, 111]. Способность кофакторов и лигандов,
таких как гем, специфически связываться с рас
тущими полипептидными цепями, может рас
КОД ИСПОЛЬЗОВАНИЯ КОДОНОВ
сматривается как показатель того, что конфор
ДЛЯ СВОРАЧИВАНИЯ БЕЛКА В КЛЕТКЕ:
мация, необходимая для их связывания, уже по
ЕГО ОСНОВНЫЕ ЧЕРТЫ
является на рибосоме [82, 110, 111]. Изучая ре
акции трансляции in vitro, в присутствии
Изучение процесса котрансляционного сво
[3H] гемина и [14C] лейцина или [35S] метиони
рачивания белка и особенно эксперименты, по
на с помощью центрифугирования в градиенте
казывающие, что рефолдинг белков из денату
плотности сахарозы, в условиях наличия или от
рированного состояния происходит намного
сутствия пуромицина, мы показали, что связан
медленнее по сравнению с их котрансляцион
ные с рибосомой цепи глобина способны эф
ным сворачиванием (даже в условиях, когда та
фективно связывать гем [110, 111]. Кроме того,
кое сравнение проводится в одинаковых усло
мы обнаружили, что неполные насцентные це
виях, например, таких как клеточные экстрак
пи глобина, прикрепленные к рибосоме, спо
ты, используемые для трансляции in vitro) [109],
собны котрансляционно связывать гем, что ука
привели к предположению о том, что векторная
зывало на то, что структура, позволяющая нас
природа котрансляционного процесса, а также
центной полипептидной цепи связывать гем,
дополнительная информация, которую несет
возникает еще до завершения синтеза альфа
молекула мРНК (помимо непосредственно ко
глобина [111]. Множество других исследований,
дирования аминокислотной последовательнос
выполненных в тот период времени, предоста
ти), могут объяснить высокую эффективность
БИОХИМИЯ том 86 вып. 8 2021
1182
КОМАР
котрансляционного фолдинга in vivo [112-115].
ванию) остатков цистеинов, расположенных в
Тем не менее стало ясно, что направленность
разных участках насцентной полипептидной це
котрансляционного сворачивания скорее всего
пи (постулировалось, что длина участка расту
играет роль лишь тонкой настройки (при этом
щего насцентного пептида, расположенного
очень важной, как будет видно из обсуждения
внутри рибосомного канала (покрываемого ка
ниже) в общем процессе фолдинга, поскольку
налом) изменяется (увеличивается) в зависи
некоторые белки, например, циркулярно пер
мости от формирования им альфа спиральной
мутированные белки или белки, полученные в
структуры и, таким образом, изменяет момент
ходе химического синтеза по Меррифилду, ока
доступности определенного цистеина для пеги
зываются свернутыми правильным образом и
лирования вне канала) [90]; а также с помощью
после изменения направления синтеза [46, 116].
прямого определения структуры пептида в кана
Работы Лима и Спирина [59, 60], о которых
ле (см. обзор [91]).
упоминалось выше, позволили предположить,
Следует также отметить, что проведенные в
что считывание мРНК на рибосоме может об
середине 1970 х и в 1980 х гг. исследования по
легчить формирование альфа спиральной кон
казали, что элонгация трансляции - это нерав
формации в растущей насцентной полипептид
номерный процесс [117-125]. Несмотря на вы
ной цепи. В связи с этим предположением воз
явление нескольких причин неравномерности
никло сразу несколько вопросов:
трансляции (в частности, таких как структура
1) если рибосомы способствуют образова
мРНК [118], которая может препятствовать дви
нию альфа спиральной конформации - то как
жению рибосомы), стало ясно, что неравномер
тогда образуются другие вторичные структуры
ное распределение синонимических кодонов
(другие типы спиралей, повороты, бета структу
является одним из ключевых факторов, контро
ры и др.)?
лирующих скорость элонгации трансляции
2) альфа спирали, как известно, сворачива
[12-22, 124]. Как было отмечено ранее, было об
ются быстро [46]; может ли тогда формирование
наружено, что частые/оптимальные кодоны,
быстро сворачивающихся элементов структуры
как правило, транслировались быстрее по срав
нуждаться в более высоких скоростях трансля
нению с редкими [12-22].
ции, а формирование медленно сворачиваю
Используя эти знания как отправную точку
щихся элементов - требовать более медленных
наших исследований, мы поставили перед со
скоростей?
бой задачу проанализировать выбор кодонов в
3) может ли существовать и быть необходи
контексте их распределения вдоль последова
мым определенный период времени для превра
тельности мРНК и изучить взаимосвязи между
щения альфа спирали в другую (другие) вторич
участками локализации определенных кодонов
ную структуру в процессе синтеза белка на ри
и кодируемыми ими структурами в белках
босоме?
[113-115]. В частности, нам было интересно вы
4) могут ли сайты трансляционных пауз раз
яснить, могут ли редкие кодоны обнаруживать
делять образование различных структурных эле
ся в пограничных участках между различными
ментов (таких как вторичные структуры одного
элементами вторичной структуры или на грани
или другого типа) во время синтеза белка на ри
цах между доменами. Мы постулировали, что
босоме?
последовательные события процесса котрансля
5) может ли потребоваться более длительная
ционного сворачивания (такие как формирова
задержка трансляции (трансляционная пауза)
ние определенных вторичных и/или третичных
для формирования больших единиц сворачива
структур, например, доменов) могут быть разде
ния (например, доменов в мультидоменных бел
лены редкими кодонами и сайтами трансляци
ках)?
онных пауз [113-115]. Независимо от нас груп
6) наконец, как изменения кинетики транс
па Alistair Brown из Института генетики универ
ляции могут отразиться на сворачивании белков
ситета Глазго (Великобритания) высказала
в клетке?
предположение, что сворачивание доменов бел
Примечательно, что возможность образова
ков на рибосоме может быть разделено трансля
ния компактной (альфа спиральной) структуры
ционными паузами, и что такие участки замед
внутри рибосомного канала была эксперимен
ленной трансляции могут служить в качестве
тально продемонстрирована через 30 лет после
«знаков препинания» в процессе котрансляци
исходного теоретического предположения, выд
онного фолдинга белков [112].
винутого Лимом и Спириным. Для этого были
Следует признать, что эти ранние исследова
применены несколько подходов, включая FRET
ния не были достаточно полными (частично
(перенос энергии флуоресцентного резонан
ввиду отсутствия достаточной информации о
са) [89]; определение доступности (к пегилиро
структурах и последовательностях белков). Тем
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1183
не менее мы пришли к заключению, что распре
турой белка путем сравнения профилей исполь
деление редких кодонов (и их кластеров) вдоль
зования кодонов структурно гомологичных бел
молекул мРНК носит эволюционно консерва
ков. Мы предположили, что если такая связь су
тивный характер, и что эти кодоны действитель
ществует, то структурно гомологичные белки из
но могут разделять формирование определен
различных организмов (независимо от различий
ных элементов вторичной структуры, таких как,
между предпочтениями кодонов у этих организ
например, альфа спирали (анализ был проведен
мов) должны иметь сходные профили использо
на нескольких однодоменных белках, таких как
вания кодонов [115]. Мы обнаружили, что это
цитохромы c, миоглобины и глобины), а также
действительно так [115]. Одно и мультидомен
элементов третичной структуры, таких как, на
ные структурно гомологичные белки, такие как
пример, домены (как было показано на примере
цитохромы c, миоглобины, гамма кристалли
мультидоменных белков, таких как кристалли
ны, фосфоглицераткиназы (рис. 2), как оказа
ны гамма B и бета B2, фосфоглицераткиназы
лось, имели очень схожие профили использова
(PGKs
- phosphoglycerate kinases) и др.)
ния кодонов. В этих исследованиях нами было
[113-115, 126]. Многие последующие углублён
также продемонстрировано сохранение пози
ные исследования [127-136] выявили сходные
ций кластеров наиболее редких кодонов, напри
тенденции как на границах перехода между раз
мер, на границах доменов [79, 114, 115].
личными элементами вторичной структуры
В нескольких последующих in silico исследо
(например, бета тяж → спираль; спираль → бе
ваниях, в частности, по анализу хлорамфеникол
та тяж) [134], так и между доменами [127,
ацетилтрансфераз [131] и лакритинов глаза [136]
131-133]. Также стало очевидно, что в распреде
из различных организмов, были получены сход
лении кластеров редких кодонов вдоль мРНК
ные результаты. Следует также отметить, что
существует определенная иерархия [61, 79, 98,
совсем недавно было показано, что рибосомные
113-115, 127, 128]. Как оказалось, наиболее ред
профили (отражающие распределение трансли
кие кодоны (и их кластеры) часто кодировали
рующих рибосом на мРНК) оказались высоко
границы между относительно большими струк
консервативны у структурно и эволюционно
турными единицами (например, доменами), в
родственных белков и белковых доменов, что
то время как менее редкие кодоны - границы
дополнительно свидетельствует в пользу того
между более мелкими единицами структуры
факта, что кинетика трансляции мРНК у струк
(например, мотивами, субдоменами). Было
турно родственных белков является высококон
высказано предположение, что это различие от
сервативным процессом, необходимым для
ражает потребность в обеспечении более значи
обеспечения их правильного котрансляционно
тельной задержки трансляции для независимого
го сворачивания [137].
сворачивания больших единиц структуры по
Тем не менее, несмотря на широкое призна
сравнению с небольшими структурными эле
ние того факта, что часто используемые кодоны
ментами [61, 79, 98, 113-115]. Мы также пред
ускоряют трансляцию, а редкие кодоны вызыва
положили, что ускорение трансляции в опреде
ют трансляционные паузы, получить экспери
ленных участках мРНК является не менее важ
ментальные доказательства того, что конкрет
ным фактором, поскольку быстро формирую
ный редко используемый кодон (или кластер
щиеся фрагменты структуры могут нуждаться в
редких кодонов) определяет соответствующую
более высоких скоростях трансляции [61, 79, 98,
трансляционную паузу на конкретном участке
113-115]. Таким образом, было предложено рас
молекулы мРНК, оказалось достаточно затруд
сматривать котрансляционное сворачивание
нительным. Тем не менее в начале и середине
как волновой процесс, протекающий через по
1990 х гг. мы смогли показать, используя одно и
следовательные смены медленных и быстрых
мультидоменные белки, что участки замедлен
фаз, сопряженных с медленными и быстрыми
ной трансляции могут действительно разделять
фазами трансляции. Мы также предположили,
этапы формирования отдельных структурных
что так называемый профиль частот использо
элементов, таких как альфа спирали у глобинов
вания кодонов, отражающий специфический
[126] и/или домены у кристаллина гамма В
характер распределения кодонов вдоль мРНК,
[138]. Интересно, что проведенное в то же время
может выступать в качестве кинетического пу
изучение кинетики индуцированного мочеви
теводителя котрансляционного сворачивания
ной равновесного разворачивания и сворачива
белка [61, 79, 98, 113-115].
ния кристаллина гамма В быка, показало, что
Основываясь на этих знаниях, мы задались
сворачивание этого белка не только происходит
вопросом о том, можно ли визуализировать
через последовательное структурирование его
связь между конкретной картиной распределе
доменов, но и также характеризуется дифферен
ния синонимических кодонов в мРНК и струк
циальной кинетикой их сворачивания [139].
БИОХИМИЯ том 86 вып. 8 2021
1184
КОМАР
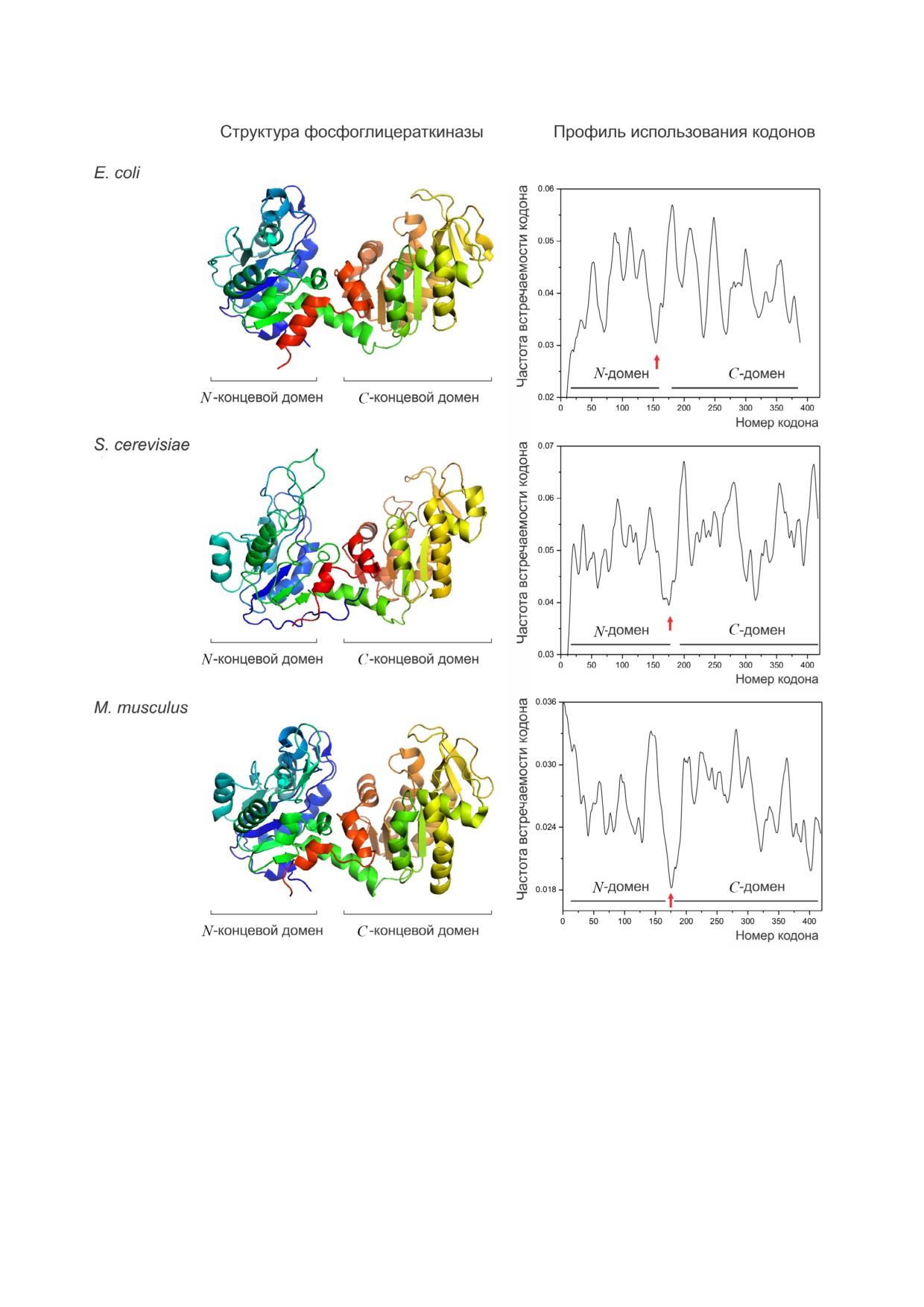
Рис. 2. Консерватизм структуры белка находит отражение в сходстве профилей использования кодонов. Модели третич
ной структуры фосфоглицераткиназ (PGK) из кишечной палочки Escherichia coli (PDB ID: 1ZMR) (вверху), дрожжей
Saccharomyces cerevisiae (PDB ID: 3PGK) (посередине) и мыши Mus musculus (PDB ID: 2P9T) (внизу). Соответствующие
профили использования кодонов указаны справа (обозначены границы между N и C концевыми доменами). Красными
стрелками показан консервативный кластер редких кодонов в области границ доменов
Так, было показано, что сворачивание N конце
факт неравномерности трансляции мРНК этого
вого домена происходит гораздо быстрее, чем
белка с характерными паузами, но также позво
C концевого [139]. Проведенный нами впослед
лил продемонстрировать (путем сравнения ки
ствии анализ процесса биосинтеза бычьего
нетики трансляции природного кристаллина
кристаллина гамма В не только подтвердил
гамма В быка и его циркулярно пермутирован
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1185
ного варианта с измененным порядком распо
мРНК) по сравнению с C концевым доме
ложения N и C концевых доменов), что его
ном [135] (рис. 3). Таким образом, мы пришли к
N концевой домен (в результате неравномерно
заключению, что скорости трансляции могут
го распределения частых и редких кодонов в мо
быть действительно оптимизированы для наст
лекуле мРНК) подвергается более быстрой
ройки синтеза и сворачивания насцентных по
трансляции (о чем свидетельствовало меньшее
липептидных цепей на рибосоме, и что для
число трансляционных пауз на этом участке
быстро сворачивающихся фрагментов белка
Рис. 3. Модульная (двухдоменная) структура кристаллина гамма В и дифференциальная кинетика сворачивания его доме
нов отражены в различиях в использовании кодонов в его доменах и неравномерной трансляции белка. a - Модель третич
ной структуры кристаллина гамма В быка (Bos taurus, PDB ID: 4GCR); N концевой домен (быстро сворачивающийся) по
казан синим цветом, C концевой (медленно сворачивающийся) - желтым; линкерный участок, связывающий два домена,
показан красным цветом. b - Гистограмма использования/частоты кодонов: N концевой домен (в среднем) кодируется бо
лее часто используемыми кодонами по сравнению с C концевым; между двумя доменами есть четкая граница. c - [35S] Ав
торадиограмма продуктов трансляции кристаллина гамма В in vitro (из [138]), где a - кристаллин гамма В спустя 15 мин
трансляции; b-d - насцентные пептиды, выделенные из фракций полирибосом спустя 5, 10 и 15 мин трансляции белка
гамма B соответственно; e - отдельно транслированный N концевой домен гамма B; f - циркулярно пермутированный
белок гамма (гамма CP) спустя 15 мин трансляции; g-i - насцентные пептиды, выделенные из фракций полирибосом
спустя 5, 10 и 15 мин трансляции гамма CP. Основные пептиды обозначены цифрами в соответствии с их длиной (рассчи
танной на основе молекулярного веса пептидов). Стрелками обозначена пауза в междоменном (линкерном) участке. d -
Схематическое изображение ожидаемого распределения положения рибосом (пауз в трансляции) в случае кристаллина
гамма В дикого типа и циркулярно пермутированного белка. Увеличение времени нахождения рибосомы в определенных
положениях на молекуле мРНК ведет к увеличению накопления насцентных цепей соответствующих размеров
БИОХИМИЯ том 86 вып. 8 2021
1186
КОМАР
действительно может быть необходима более
удельной активности белков и/или их функций
быстрая трансляция соответствующих участков
(см. обзоры [61, 79]). Тем не менее прямые дока
мРНК [139].
зательства в поддержку этого постулата было по
Следующим логичным вопросом был вопрос
лучены лишь относительно недавно [154]. Для
о том, насколько изменения в использовании
изучения того, как различное распределение си
синонимических кодонов в мРНК могут влиять
нонимических кодонов влияет на кинетику
на сворачивание белка в клетке?
трансляции, ко и посттрансляционное сворачи
В 1999 г. мы представили одно из первых до
вание, а также конформацию белка и его ста
казательств того, что замены синонимических
бильность, мы использовали кристаллин гам
кодонов могут действительно влиять на движе
ма В быка, анализируя экспрессию этого белка
ние рибосомы по мРНК, а также на сворачива
in vivo в клетках E. coli и in vitro в полностью ре
ние модельного белка CAT (chloramphenicol
конструированной системе бесклеточной транс
acetyltransferase, хлорамфениколацетилтрансфе
ляции. Мы сравнили экспрессию двух вариантов
разы) в процессе его синтеза [140]. Мы показа
кристаллина гамма B: один вариант характери
ли, что замена 16 ти последовательно располо
зовался оптимальным использованием кодонов
женных редких кодонов в гене CAT на частые
для трансляции белка в клетках E. coli (с профи
кодоны приводит к ускорению движения рибо
лем использования кодонов, сходным с таковым
сомы (в системе трансляции in vitro) на участке
в клетках B. taurus, что, как ожидалось, должно
мРНК, содержащем замены этих кодонов, а так
приводить к более естественной кинетике транс
же влияет на удельную активность фермента (в
ляции), другой вариант - с неизменным соста
сравнении с белком дикого типа) [140]. По
вом кодонов, который не являлся оптимальным
скольку удельную активность белка можно рас
для трансляции белка в клетках E. coli [154]. По
сматривать как меру его правильного сворачи
лученные нами данные о влиянии предпочтения
вания, то мы пришли к выводу о том, что заме
синонимических кодонов на трансляцию мРНК
ны кодонов оказали влияние на процесс свора
кристаллина гамма В показали, что выбор кодо
чивания белка CAT [140].
нов изменяет скорости трансляции (как общую
Во многих последующих экспериментах бы
скорость, так и локальную скорость считывания
ла подтверждена значимость использования си
в определенных участках мРНК) и вызывает
нонимических кодонов для сворачивания бел
формирование альтернативных конформаций
ков и показано, что замены кодонов могут вли
белка как в прикрепленных насцентных цепях,
ять на степень подверженности белков ограни
так и после высвобождения белка из рибосомы
ченному протеолизу [141-143], изменять спект
[154]. Важно подчеркнуть, что с помощью изме
ральные характеристики белков [144], изменять
рений в реальном времени на основе флуорес
их подверженность агрегации [144-146], а также
ценции и FRET мы показали, что кинетика син
менять их удельную активность [141, 147], вли
теза и котрансляционного сворачивания крис
ять на их внутриклеточные характеристики
таллина гамма В действительно изменяется в ре
[148] и в конечном итоге приводить к возникно
зультате замен синонимических кодонов. Более
вению заболеваний [149-152].
того, нами впервые была выявлена значительная
Кроме того, было высказано предположе
структурная гетерогенность очищенных полно
ние, что выбор синонимичных кодонов может
размерных препаратов синонимических вариан
влиять на эффективность взаимодействия нас
тов кристаллина гамма B, что было продемон
центных полипептидов с сигнал распознающей
стрировано с использованием прямого опреде
частицей (SRP) [153], таким образом влияя на
ления структуры методом 2D NMR (двумерной
секрецию белка.
ЯМР спектроскопии), что также влияло на под
Следует отметить, что в гипотезе «кода пред
верженность вариантов этих белков к агрега
почтения кодонов» [112-115] также было посту
ции [154]. Мы пришли к выводу, что синоними
лировано, что изменения кинетики трансляции
ческие замены в молекуле мРНК изменили
должны в первую очередь приводить к измене
распределение различных состояний в ланд
нию конформации прикрепленных к рибосоме
шафте доступных конформаций белка - как на
насцентных цепей, что затем может приводить к
рибосоме, так и после высвобождения полипеп
изменению конечной конформации высвобож
тидной цепи [154] (рис. 4).
даемого полипептида и/или вызывать сдвиг рав
Эти эксперименты были проведены в сотруд
новесия между конформерами белка (правильно
ничестве с Harald Schwalbe из университета Ио
и неправильно свернутыми), что, в свою оче
ганна Вольфганга Гёте (Франкфурт, Германия) и
редь, может привести, например, к повышенной
Мариной Родниной из Института биофизичес
агрегации белков и/или их деградации (ко или
кой химии имени Макса Планка (Гёттинген,
посттрансляционной) или вызвать изменения
Германия), представив одно из самых убедитель
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1187
Рис. 4. Генетический код управляет сворачиванием белка в клетке. Возникновение отдельных промежуточных интерме
диатов сворачивания связано как с общей, так и с локальной скоростью трансляции, определяемой использованием си
нонимических кодонов. Изменение набора используемых кодонов вызывает изменение кинетики трансляции и процес
са сворачивания белка как на рибосоме, так и после высвобождения полипептидной цепи, что также может приводить к
изменению конечной конформации белка и отличному от нативного минимуму свободной энергии
ных доказательств гипотезы, согласно которой
ничная синонимическая мутация может нару
специфический характер распределения сино
шать сворачивание белка и его функционирова
нимических кодонов в молекуле мРНК может
ние [141, 152, 158].
действительно служить в качестве вторичного
В 2007
г. Chava Kimchi Sarfaty, Michael
кода для сворачивания белка в клетке [154].
Gottesman и их коллеги показали, что одна
единственная синонимическая мутация в гене
MDR1 (multi drug resistance 1; ген множествен
КОД ПРЕДПОЧТЕНИЯ КОДОНОВ ДЛЯ
ной лекарственной резистентности 1), кодиру
СВОРАЧИВАНИЯ БЕЛКА В КЛЕТКЕ:
ющем белок P gp (P glycoprotein, Р гликопроте
ЕГО ЗНАЧЕНИЕ
ин) может вызывать изменение активности это
го белка, таким образом влияя на чувствитель
Открытие того факта, что замены синоними
ность пациентов к действию различных лекар
ческих кодонов могут не только влиять на уров
ственных препаратов [141]. P гликопротеин яв
ни экспрессии белков, но также на их сворачи
ляется трансмембранным белком, который вы
вание, привело к углублению понимания миро
качивает из клеток различные лекарства
вым научным сообществом механизма влияния
(см. [141] и ссылки в ней). В нормальных усло
распределения синонимических кодонов на
виях он обеспечивает устойчивость клеток к эк
функции белков. Стало ясно, что замены сино
зогенным лекарственным веществам и/или эн
нимических кодонов могут играть роль в разви
догенным метаболитам [141, 149], однако у
тии заболеваний человека [149-154], а также
больных с онкологическими заболеваниями из
потенциально влиять на безопасность и эффек
менение активности этого белка может приво
тивность использования рекомбинантных/тера
дить к снижению эффективности химиотера
певтических белков [155-157]. К тому же, и что
пии, поэтому он представляет важную мишень в
удивительно, было обнаружено, что даже еди
работе над доставкой лекарств [149]. Chava
БИОХИМИЯ том 86 вып. 8 2021
1188
КОМАР
Kimchi Sarfaty и коллеги показали, что единич
боспондина 1 го типа, 13 й член семейства), ко
ная синонимическая замена в гене MDR1
дирующем большой мультидоменный секрети
(c.3435C>T, ATC>ATT, p.Ile1145Ile) может при
руемый белок, регулирующий тромбогенез пу
водить к изменению субстратной специфичнос
тем расщепления адгезивного гликопротеина
ти P гликопротеина [141]. Ранее было обнару
крови, фактора фон Виллебранда (VWF - von
жено, что концентрация в плазме и сыворотке
Willebrand factor), может придать этому фермен
крови, например, сердечного гликозида дигок
ту более высокую специфическую активность
сина, антигистаминового лекарства фексофена
[158]. Эта замена является естественным вари
дина, который является агонистом μ опиоидно
антом, который присутствует в человеческой
го рецептора лоперамида, и некоторых других
популяции (частота аллеля: 0,026 согласно 1000
лекарств у людей с однонуклеотидным поли
Genomes, 0,0627 согласно ExAC) [158], и до нас
морфизмом c.3435C>T отличается (по сравне
тоящего момента она не была ассоциирована с
нию с нормальными индивидами) после еди
патологическим фенотипом [158, 164]. На се
ничного или многократного (орального) приема
годняшний день вариант c.354G>A гена
этих лекарств, однако точная причина этих раз
ADAMTS13 представляет действительно уни
личий исходно не была выяснена [149]. В своей
кальный пример, т.к. эта замена приводит к уве
работе Kimchi Sarfaty и коллеги объяснили при
личению специфической активности бел
чину изменения фармакокинетики P gp у лю
ка [158]. Еще предстоит установить, дает ли эта
дей, несущих этот полиморфизм, и впервые по
мутация какое либо преимущество её носите
казали, что возникающие естественным обра
лям.
зом вариации синонимических кодонов в опре
Обнаружение того факта, что замены сино
деленном гене могут дать начало белковому
нимических кодонов могут являться фактором
продукту с той же аминокислотной последова
риска для правильной функции белка (и влиять
тельностью, но другими структурными и функ
на структуру белка) добавляет дополнительные
циональными свойствами [141].
сложности в процесс производства рекомбинант
Впоследствии в сотрудничестве с Chava
ных белков, включая белковые терапевтические
Kimchi Sarfaty мы показали, что единичная си
средства. В настоящее время биофармацевти
нонимическая замена c.459G>A (GTG>GTA,
ческая промышленность широко использует ряд
p.Val153Val или p.Val107Val (107 - позиция ами
доступных компьютерных программ и методов,
нокислоты после отщепления препропептида
которые учитывают предпочтение использова
при секреции)) в гене F9 (кодирующем фактор
ния синонимических кодонов для направленно
коагуляции крови FIX), которая была связана со
го изменения генов с целью достижения их по
снижением уровня свертывания крови и отно
вышенной экспрессии (см. обзор [156]). Следу
сительно легкой формой гемофилии B у ее но
ет, однако, отметить, что эти инструменты
сителей, влияет на синтез FIX и его конформа
обычно нацелены на увеличение эффектив
цию, приводя к снижению уровня внеклеточно
ности трансляции (и обычно включают массо
го белка за счет его повышенной деградации
вые замены большинства нечасто используемых
[152]. Таким образом, мы смогли определить па
кодонов на синонимические часто используе
тогенетическую основу однонуклеотидной си
мые) и обладают ограниченной (если вообще
нонимической замены в гене F9, ассоциирован
обладают) способностью оценки эффекта замен
ной с гемофилией B [152]. Этот случай озадачи
конкретных синонимических кодонов на свора
вал исследователей, обнаруживших замену еще
чивание белка [156]. Тем не менее, как упомина
в 2008 г. [159], поскольку его нельзя было объяс
лось выше, максимальное увеличение скорости
нить измененными свойствами мРНК (уровнем
и выхода белка при трансляции может вступать
мРНК, сплайсингом/пропуском экзонов, удер
в определенные противоречия с процессом его
жанием интронов или измененной стабиль
синтеза и сворачивания, потенциально приводя
ностью мРНК), что обычно является причиной
к неправильному сворачиванию. Кроме того,
[150, 151, 160-163] возникновения многих забо
все большее число работ показывает, что даже
леваний, ассоциированных с синонимическими
незначительные нарушения структуры белка
мутациями, до тех пор, пока не стало ясно, что
могут вызвать изменения иммуногенных
синонимические мутации могут влиять на био
свойств белковых терапевтических средств и
генез белка.
привести к образованию нейтрализующих анти
Кроме того, также в сотрудничестве с Chava
тел [155, 165]. Эти данные представляют собой
Kimchi Sarfaty мы недавно показали, что еди
дополнительный фактор риска при производ
ничная синонимическая замена c.354G>A
стве терапевтических средств на основе белков.
(CCG>CCA, p.Pro118Pro) в гене ADAMTS13 (диз
В настоящее время оценка иммуногенного по
интегрин и металлопротеиназа с мотивом тром
тенциала белковых терапевтических средств
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1189
представляет собой еще одну неудовлетворен
кафедре в качестве дипломника и аспиранта в
ную потребность в биотехнологической индуст
группе профессора Владимира Владимировича
рии и в клинической практике [165].
Юркевича и затем в группе профессора Игоря
Таким образом, становится очевидной необ
Александровича Крашенинникова, проводя в
ходимость разработки более совершенных стра
том числе многие работы в тесном сотрудниче
тегий оптимизации использования кодонов.
стве с профессором Спириным. Я также имел
Ряд компаний, включая, например, DAPCEL,
возможность провести довольно много времени
Inc. [156], сейчас применяет вышеизложенные
в лаборатории Спирина в Институте белка в Пу
знания для разработки новых стратегий, на
щино (в начале 1990 х гг.), проводя исследова
правленных на продукцию (в любом желаемом
ния по котрансляционному сворачиванию гло
организме хозяине) правильно свернутых рас
бина. Этот опыт был бесценен для формирова
творимых белков.
ния моих научных взглядов и отношения к науч
Тем не менее предстоит еще много работы.
ной работе.
Необходимо отметить, что пока ещё не сущест
вует полного понимания точного влияния как
единичных, так и множественных синоними
Благодарности. Настоящая работа была бы
ческих мутаций на сворачивание белка и его
невозможна без исходного участия и вклада
функционирование. Тем не менее, несмотря на
Ивана Аджубея и Игоря А. Крашенинникова, а
отсутствие понимания многих деталей этого
также поддержки со стороны Славы Колба, Ай
процесса, стало очевидным, что использование
гара Коммера, Валентина М. Степанова, Льва
синонимических кодонов лежит в основе вто
П. Овчинникова и Александра С. Спирина и
ричного кода для сворачивания белков in vivo и
последующих совместных исследований, прове
осуществляет тонкую настройку процесса ко
денных с Райнером Янике, Клодом Райсом, а
трансляционного сворачивания, позволяя бел
также в самое ближайшее время с Хавой Кимчи
кам обходить глубокие кинетические ловушки в
Сарфати, Харальдом Швальбе и Мариной
процессе синтеза, обеспечивая быстрое и эф
В. Родниной.
фективное формирование белковой структуры в
Я бесконечно признателен всем моим учите
клетке. Таким образом, очевидно, что естест
лям, коллегам и сотрудникам за их чрезвычайно
венный отбор использует одновременно множе
щедрые и вдохновляющие обсуждения и неоце
ство различных возможностей для оптимизации
нимый вклад. Я также приношу извинения тем
экспрессии генетической информации.
авторам, чьи работы или оригинальные публи
кации не были процитированы в этой короткой
обзорной статье.
ЗАКЛЮЧЕНИЕ
Финансирование. В последние годы работа
моей лаборатории была поддержана грантами
Настоящий обзор посвящен памяти Алекса
Human Frontier Science Program Organization
ндра Сергеевича Спирина и его 90 летнему
[HFSP, грант №RGP0024/2010], the American
юбилею, а также частично отражает его вклад в
Heart Association [AHA, грант 13GRNT17070025],
развитие области котрансляционного сворачи
the National Institutes of Health [NIH, гранты
вания белка.
HL121779; HL151392 и GM128981], the Center for
Мой интерес к биосинтезу белка и, как я по
Gene Regulation in Health and Disease (GRHD) at
лагаю, интерес многих студентов и научных со
CSU и биотехнологической компании DAPCEL,
трудников кафедры молекулярной биологии
Inc.
биологического факультета МГУ им. М.В. Ло
Конфликт интересов. Я являюсь одним из ос
моносова, которую профессор Спирин возглав
нователей, а также вице президентом по науке
лял с 1972 по 2012 г., был в значительной степе
DAPCEL, Inc., биотехнологической компании,
ни вызван его лекциями по структуре рибосомы
которая разрабатывает инновационные подхо
и механизму биосинтеза белка. Мне довелось
ды для редизайна генов с целью производства
посещать эти лекции в 1984 г., когда я был сту
правильно свернутых белков в любом желаемом
дентом кафедры молекулярной биологии. Они
организме хозяине.
были поистине вдохновляющими, стимулирую
Соблюдение этических норм. Настоящая
щими и вызывали множество вопросов. Мне
статья не содержит описания каких либо иссле
посчастливилось продолжить мою учебу на этой
дований с участием людей в качестве объектов.
БИОХИМИЯ том 86 вып. 8 2021
1190
КОМАР
СПИСОК ЛИТЕРАТУРЫ
1.
Crick, F. H., Barnett, L., Brenner, S., and Watts Tobin,
bias, and its potential applications, Nucleic Acids Res., 15,
R. J. (1961) General nature of the genetic code for pro
1281 1295.
teins, Nature, 192, 1227 1232.
19.
Sharp, P. M., Cowe, E., Higgins, D. G., Shields, D. C.,
2.
Nirenberg, M., and Leder, P. (1964) RNA codewords and
Wolfe, K. H., and Wright, F. (1988) Codon usage patterns
protein synthesis. The effect of trinucleotides upon the
in Escherichia coli, Bacillus subtilis, Saccharomyces cerevisi
binding of sRNA to ribosomes, Science, 145, 1399 1407.
ae, Schizosaccharomyces pombe, Drosophila melanogaster
3.
Leder, P., and Nirenberg, M. (1964) RNA codewords and
and Homo sapiens; a review of the considerable within
protein synthesis. II. nucleotide sequence of a valine RNA
species diversity, Nucleic Acids Res., 16, 8207 8211.
codeword, Proc. Natl. Acad. Sci. USA, 52, 420 427.
20.
Sørensen, M.A., Kurland, C. G., and Pedersen, S. (1989)
4.
Bernfield, M. R., and Nirenberg, M. W. (1965) RNA code
Codon usage determines translation rate in Escherichia
words and protein synthesis. The nucleotide sequences of
coli, J. Mol. Biol., 207, 365 377.
multiple codewords for phenylalanine, serine, leucine, and
21.
Andersson, S. G., and Kurland, C. G. (1990) Codon pref
proline, Science, 147, 479 484.
erences in free living microorganisms, Microbiol. Rev., 54,
5.
Trupin, J. S., Rottman, F. M., Brimacombe, R. L.,
198 210.
Leder, P., Bernfield, M. R., and Nirenberg, M. W. (1965)
22.
Kurland, C. G. (1991) Codon bias and gene expression,
RNA codewords and protein synthesis, VI. On the
FEBS Lett., 285, 165 169.
nucleotide sequences of degenerate codeword sets for
23.
Kramer, E. B., and Farabaugh, P. J. (2007) The frequency
isoleucine, tyrosine, asparagine, and lysine, Proc. Natl.
of translational misreading errors in E. coli is largely deter
Acad. Sci. USA, 53, 807 811.
mined by tRNA competition, RNA, 13, 87 96.
6.
Nirenberg, M., Leder, P., Bernfield, M., Brimacombe, R.,
24.
Zaher, H. S., and Green, R. (2009) Fidelity at the molecu
Trupin, J., et al. (1965) RNA codewords and protein syn
lar level: lessons from protein synthesis, Cell, 136, 746 762.
thesis, VII. On the general nature of the RNA code, Proc.
25.
Kramer, E. B., Vallabhaneni, H., Mayer, L. M., and
Natl. Acad. Sci. USA, 53, 1161 1168.
Farabaugh, P. J. (2010) A comprehensive analysis of trans
7.
Brimacombe, R., Trupin, J., Nirenberg, M., Leder, P.,
lational missense errors in the yeast Saccharomyces cere
Bernfield, M., and Jaouni, T. (1965) RNA codewords and
visiae, RNA, 16, 1797 1808.
protein synthesis, 8. Nucleotide sequences of synonym
26.
Ribas de Pouplana, L., Santos, M. A., Zhu, J. H.,
codons for arginine, valine, cysteine, and alanine, Proc.
Farabaugh, P. J., and Javid, B. (2014) Protein mistransla
Natl. Acad. Sci. USA, 54, 954 960.
tion: friend or foe? Trends. Biochem. Sci., 39, 355 362.
8.
Söll, D., Ohtsuka, E., Jones, D. S., Lohrmann, R.,
27.
Huang, Y., Koonin, E. V., Lipman, D. J., and Przytycka,
Hayatsu, H., et al. (1965) Studies on polynucleotides,
T. M. (2009) Selection for minimization of translational
XLIX. Stimulation of the binding of aminoacyl sRNA’s to
frameshifting errors as a factor in the evolution of codon
ribosomes by ribotrinucleotides and a survey of codon
usage, Nucleic Acids Res., 37, 6799 6810.
assignments for 20 amino acids, Proc. Natl. Acad. Sci. USA,
28.
Savir, Y., and Tlusty, T. (2013) The ribosome as an optimal
54, 1378 1385.
decoder: a lesson in molecular recognition. Cell, 153, 471 479.
9.
Goel, N. S., Rao, G. S., Ycas, M., Bremermann, H. J., and
29.
Dana, A., and Tuller, T. (2014) The effect of tRNA levels
King, L. (1972) A method for calculating codon frequen
on decoding times of mRNA codons, Nucleic Acids Res.,
cies in DNA, J. Theor. Biol., 35, 399 457.
42, 9171 9181.
10.
Grantham, R., Gautier, C., Gouy, M., Mercier, R., and
30.
Bonekamp, F., Andersen, H. D., Christensen, T., and
Pavé, A. (1980) Codon catalog usage and the genome
Jensen, K. F. (1985) Codon defined ribosomal pausing in
hypothesis, Nucleic Acids Res., 8, r49 r62.
Escherichia coli detected by using the pyrE attenuator to
11.
Grantham, R., Gautier, C., and Gouy, M. (1980) Codon
probe the coupling between transcription and translation,
frequencies in 119 individual genes confirm consistent
Nucleic Acids Res., 13, 4113 4123.
choices of degenerate bases according to genome type,
31.
Curran, J. F., and Yarus, M. (1989) Rates of aminoacyl
Nucleic Acids Res., 8, 1893 1912.
tRNA selection at 29 sense codons in vivo, J. Mol. Biol.,
12.
Ikemura, T. (1981) Correlation between the abundance of
209, 65 77.
Escherichia coli transfer RNAs and the occurrence of the
32.
Bulmer, M. (1987) Coevolution of codon usage and trans
respective codons in its protein genes, J. Mol. Biol., 146, 1 21.
fer RNA abundance, Nature, 325, 728 730.
13.
Ikemura, T. (1982) Correlation between the abundance of
33.
Duret, L. (2000) tRNA gene number and codon usage in
yeast transfer RNAs and the occurrence of the respective
the C. elegans genome are co adapted for optimal transla
codons in protein genes. Differences in synonymous codon
tion of highly expressed genes, Trends Genet., 16, 287
choice patterns of yeast and Escherichia coli with reference
289.
to the abundance of isoaccepting transfer RNAs, J. Mol.
34.
Li, W. H. (1987) Models of nearly neutral mutation with
Biol., 158, 573 597.
particular implications for nonrandom usage of synony
14.
Bennetzen, J. L., and Hall, B. D. (1982) Codon selection
mous codons, J. Mol. Evol., 24, 337 345.
in yeast, J. Biol. Chem., 257, 3026 3031.
35.
Shields, D. C. (1990) Switches in species specific codon
15.
Hastings, K. E. M., and Emerson, C. P. Jr. (1983) Codon
preferences: the influence of mutation biases, J. Mol. Evol.,
usage in muscle genes and liver genes, J. Mol. Evol., 19,
31, 71 80.
214 218.
36.
Bulmer, M. (1991) The selection mutation drift theory of
16.
Ikemura, T. (1985) Codon usage and tRNA content in uni
synonymous codon usage, Genetics, 129, 897 907.
cellular and multicellular organisms, Mol. Biol. Evol., 2,
37.
Presnyak, V., Alhusaini, N., Chen, Y. H., Martin, S.,
13 34.
Morris, N., et al. (2015) Codon optimality is a major deter
17.
Sharp, P. M., Tuohy, T. M., and Mosurski, K. R. (1986)
minant of mRNA stability. Cell,
160,
11111124,
Codon usage in yeast: cluster analysis clearly differentiates
doi: 10.1016/j.cell.2015.02.029.
highly and lowly expressed genes, Nucleic Acids Res., 14,
38.
Boël, G., Letso, R., Neely, H., Price, W. N., Wong,
5125 5143.
K. H., et al. (2016) Codon influence on protein expres
18.
Sharp, P. M., and Li, W. H. (1987) The Codon Adaptation
sion in E. coli correlates with mRNA levels, Nature, 529,
Index - a measure of directional synonymous codon usage
358 363.
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1191
39.
Mishima, Y., and Tomari, Y. (2016) Codon usage and 3′
60.
Lim, V. I., and Spirin, A. S. (1986) Stereochemical analy
UTR length determine maternal mRNA stability in
sis of ribosomal transpeptidation. Conformation of nascent
zebrafish, Mol. Cell, 61, 874 885.
peptide, J. Mol. Biol., 188, 565 574.
40.
Bazzini, A. A., Del Viso, F., Moreno Mateos, M. A.,
61.
Komar, A. A. (2019) Synonymous codon usage a guide for
Johnstone, T. G., Vejnar, C. E., et al. (2016) Codon identi
co translational protein folding in the cell, Mol. Biol.
ty regulates mRNA stability and translation efficiency dur
(Mosk), 53, 883 898.
ing the maternal to zygotic transition, EMBO J., 35, 2087
62.
Noé, F., De Fabritiis, G., and Clementi, C.
(2020)
2103
Machine learning for protein folding and dynamics, Curr.
41.
Radhakrishnan, A., Chen, Y. H., Martin, S.,
Opin. Struct. Biol., 60, 77 84.
Alhusaini, N., Green, R., and Coller, J. (2016) The
63.
Gao, W., Mahajan, S. P., Sulam, J., and Gray, J. J. (2020)
DEAD box protein Dhh1p couples mRNA decay and
Deep learning in protein structural modeling and design,
translation by monitoring codon optimality, Cell, 167, 122
Patterns (N Y), 1, 100142.
132.
64.
Senior, A. W., Evans, R., Jumper, J., Kirkpatrick, J.,
42.
Hia, F., Yang, S. F., Shichino, Y., Yoshinaga, M.,
Sifre, L., et al. (2020) Improved protein structure prediction
Murakawa, Y., et al. (2019) Codon bias confers stability to
using potentials from deep learning, Nature, 577, 706 710.
human mRNAs. EMBO Rep., 20, e48220.
65.
Lindberg, M. O., and Oliveberg, M. (2007) Malleability of
43.
Wu, Q., Medina, S. G., Kushawah, G., DeVore, M. L.,
protein folding pathways: a simple reason for complex
Castellano, L. A., et al. (2019) Translation affects mRNA
behaviour, Curr. Opin. Struct. Biol., 17, 21 29.
stability in a codon dependent manner in human cells,
66.
Englander, S. W., and Mayne, L. (2014) The nature of pro
Elife, 8, e45396.
tein folding pathways, Proc. Natl. Acad. Sci. USA, 111,
44.
Medina Muñoz, S. G., Kushawah, G., Castellano, L. A.,
15873 15880.
Diez, M., DeVore, M. L., et al. (2021) Crosstalk between
67.
Englander, S. W., Mayne, L., Kan, Z. Y., and Hu, W.
codon optimality and cis regulatory elements dictates
(2016) Protein folding how and why: by hydrogen
mRNA stability, Genome Biol., 22, 14.
exchange, fragment separation, and mass spectrometry,
45.
Anfinsen, C. B. (1973) Principles that govern the folding of
Annu. Rev. Biophys., 45, 135 152.
protein chains, Science, 181, 223 230.
68.
Levinthal, C. (1969). How to fold graciously, in Mossbauer
46.
Jaenicke, R. (1991) Protein folding: local structures,
Spectroscopy in Biological Systems, Proceedings of a
domains, subunits, and assemblies, Biochemistry, 30, 3147
Meeting held at Allerton House, Monticello, Illinois
3161.
(Debrunner, P., Tsibris, J. C. M., and Münck, E., eds.)
47.
Fersht, A. R. (2008) From the first protein structures to our
University of Illinois Press, Urbana, p. 22.
current knowledge of protein folding: delights and skepti
69.
Gopan, G., Gruebele, M., and Rickard, M. (2020) In cell
cisms, Nat. Rev. Mol. Cell Biol., 9, 650 654.
protein landscapes: making the match between theory, sim
48.
Bartlett, A. I., and Radford, S. E. (2009) An expanding
ulation and experiment, Curr. Opin. Struct. Biol., 66, 163
arsenal of experimental methods yields an explosion of
169.
insights into protein folding mechanisms, Nat. Struct. Mol.
70.
Gershenson, A., Gosavi, S., Faccioli, P., and Wintrode,
Biol., 16, 582 588.
P. L. (2020) Successes and challenges in simulating the
49.
Finkelstein, A. V. (2018) 50+ years of protein folding,
folding of large proteins, J. Biol. Chem., 295, 15 33.
Biochemistry (Moscow), 83 (Suppl 1), S3 S18.
71.
Chow, M. K., Amin, A. A., Fulton, K. F., Fernando, T.,
50.
Abaskharon, R. M., and Gai, F. (2016) Meandering down
Kamau, L., et al. (2006) The REFOLD database: a tool for
the energy landscape of protein folding: are we there yet?
the optimization of protein expression and refolding,
Biophys. J., 110, 1924 1932.
Nucleic Acids Res., 34 (Database issue), D207 212.
51.
Ferina, J., and Daggett, V. (2019) Visualizing protein fold
72.
Mizutani, H., Sugawara, H., Buckle, A. M., Sangawa, T.,
ing and unfolding, J. Mol. Biol., 431, 1540 1564.
Miyazono, K. I., et al. (2017) REFOLDdb: a new and sus
52.
Cowie, D. B., Spiegelman, S., Roberts, R. B., and
tainable gateway to experimental protocols for protein
Duerksen, J. D. (1961) Ribosome bound β galactosidase,
refolding, BMC Struct. Biol., 17, 4.
Proc. Natl. Acad. Sci. USA, 47, 114 122
73.
Hartl, F. U., and Hayer Hartl, M. (2009) Converging con
53.
Zipser, D., and Perrin, D. (1963) Complementation on
cepts of protein folding in vitro and in vivo, Nat. Struct. Mol.
ribosomes, Cold Spring Harb. Symp. Quant. Biol., 28, 533
Biol., 16, 574 581.
537.
74.
Hingorani, K. S., and Gierasch, L. M. (2014) Comparing
54.
Kiho, Y., and Rich, A. (1964) Induced enzyme formed on
protein folding in vitro and in vivo: foldability meets the fit
bacterial polyribosomes, Proc. Natl. Acad. Sci. USA, 51,
ness challenge, Curr. Opin. Struct. Biol., 24, 81 90.
111 118.
75.
Balchin, D., Hayer Hartl, M., and Hartl, F. U. (2016) In
55.
Hamlin, J., and Zabin, I.
(1972) β Galactosidase:
vivo aspects of protein folding and quality control, Science,
immunological activity of ribosome bound, growing
353, aac4354.
polypeptide chains, Proc. Natl. Acad. Sci. USA, 69, 412
76.
Gruebele, M., Dave, K., and Sukenik, S. (2016) Globular
416.
protein folding in vitro and in vivo, Annu. Rev. Biophys., 45,
56.
Bergman, L. W., and Kuehl, W. M. (1979) Formation of
233 251.
intermolecular disulfide bonds on nascent immunoglobu
77.
Dahiya, V., and Buchner, J. (2019) Functional principles
lin polypeptides, J. Biol. Chem., 254, 5690 5694.
and regulation of molecular chaperones, Adv. Protein.
57.
Bergman, L. W., and Kuehl, W. M. (1979) Formation of an
Chem. Struct. Biol., 114, 1 60.
intrachain disulfide bond on nascent immunoglobulin light
78.
Jayaraj, G. G., Hipp, M. S., and Hartl, F. U. (2019)
chains, J. Biol. Chem., 254, 8869 8876.
Functional modules of the proteostasis network, Cold
58.
Bergman, L. W., and Kuehl, W. M. (1979) Co translation
Spring Harb. Perspect. Biol., 12, a033951.
al modification of nascent immunoglobulin heavy and light
79.
Komar, A. A. (2009) A pause for thought along the co
chains, J. Supramol. Struct., 11, 9 24.
translational folding pathway, Trends Biochem. Sci., 34, 16
59.
Lim, V. I., and Spirin, A. S. (1985) Stereochemistry of the
24.
transpeptidation reaction in the ribosome. The ribosome
80.
Gloge, F., Becker, A. H., Kramer, G., and Bukau, B.
generates an alpha helix in the synthesis of the protein
(2014) Co translational mechanisms of protein matura
polypeptide chain, Dokl. Akad. Nauk. SSSR, 280, 235 239.
tion, Curr. Opin. Struct. Biol., 24, 24 33.
БИОХИМИЯ том 86 вып. 8 2021
1192
КОМАР
81. Thommen, M., Holtkamp, W., and Rodnina, M. V. (2017)
103. Chaney, J. L., and Clark, P. L. (2015) Roles for synony
Co translational protein folding: progress and methods,
mous codon usage in protein biogenesis, Annu. Rev.
Curr. Opin. Struct. Biol., 42, 83 89.
Biophys., 44, 143 166.
82. Komar, A. A. (2018) Unraveling co translational protein
104. Jacobson, G. N., and Clark, P. L. (2016) Quality over
folding: concepts and methods, Methods, 137, 71 81.
quantity: optimizing co translational protein folding with
83. Williams, N. K., and Dichtl, B. (2018) Co translational
non “optimal” synonymous codons, Curr. Opin. Struct.
control of protein complex formation: a fundamental path
Biol., 38, 102 110.
way of cellular organization? Biochem. Soc. Trans., 46,
105. Sharma, A. K., and O’Brien, E. P. (2018) Non equilibrium
197 206.
coupling of protein structure and function to translation
84. Waudby, C. A., Dobson, C. M., and Christodoulou, J.
elongation kinetics, Curr. Opin. Struct. Biol., 49, 94 103.
(2019) Nature and regulation of protein folding on the
106. Samatova, E., Daberger, J., Liutkute, M., and Rodnina,
ribosome, Trends Biochem. Sci., 44, 914 926.
M. V. (2021) Translational control by ribosome pausing in
85. Jha, S., and Komar, A. A. (2011) Birth, life and death of
bacteria: how a non uniform pace of translation affects
nascent polypeptide chains, Biotechnol. J., 6, 623 640.
protein production and folding, Front. Microbiol., 11,
86. Das, D., Das, A., Samanta, D., Ghosh, J., Dasgupta, S.,
619430.
et al. (2008) Role of the ribosome in protein folding,
107. Liu, Y., Yang, Q., and Zhao, F. (2021) Synonymous but not
Biotechnol. J., 3, 999 1009.
silent: the codon usage code for gene expression and pro
87. Voisset, C., Saupe, S. J., and Blondel, M. (2011) The vari
tein folding, Annu. Rev. Biochem., doi: 10.1146/annurev
ous facets of the protein folding activity of the ribosome,
biochem 071320 112701.
Biotechnol. J., 6, 668 673.
108. Pelham, H. R., and Jackson, R. J. (1976) An efficient
88. Banerjee, D., and Sanyal, S. (2014) Protein folding activi
mRNA dependent translation system from reticulocyte
ty of the ribosome (PFAR) - a target for antiprion com
lysates, Eur. J. Biochem., 67, 247 256.
pounds, Viruses, 6, 3907 3924.
109. Kolb, V. A., Makeyev, E. V., and Spirin, A. S. (1994)
89. Woolhead, C. A., McCormick, P. J., and Johnson, A. E.
Folding of firefly luciferase during translation in a cell free
(2004) Nascent membrane and secretory proteins differ in
system, EMBO J., 13, 3631 3637.
FRET detected folding far inside the ribosome and in their
110. Komar, A. A., Kommer, A., Krasheninnikov, I. A., and
exposure to ribosomal proteins, Cell, 116, 725 736.
Spirin, A. S. (1993) Cotranslational heme binding to
90. Lu, J., and Deutsch, C. (2005) Folding zones inside the ribo
nascent globin chains, FEBS Lett., 326, 261 263.
somal exit tunnel, Nat. Struct. Mol. Biol., 12, 1123 1129.
111.
Komar, A. A., Kommer, A., Krasheninnikov, I. A., and
91. Wilson, D. N., and Beckmann, R. (2011) The ribosomal
Spirin A. S. (1997) Cotranslational folding of globin,
tunnel as a functional environment for nascent polypeptide
J. Biol. Chem., 272, 10646 10651.
folding and translational stalling, Curr. Opin. Struct. Biol.,
112. Purvis, I. J., Bettany, A. J., Santiago, T. C., Coggins, J. R.,
21, 274 282.
Duncan, K., et al. (1987) The efficiency of folding of some
92. Tu., L., and Deutsch, C. (2017) Determinants of helix for
proteins is increased by controlled rates of translation
mation for a Kv1.3 transmembrane segment inside the
in vivo. A hypothesis, J. Mol. Biol., 193, 413 417.
ribosome exit tunnel, J. Mol. Biol., 429, 1722 1732.
113. Krasheninnikov, I. A., Komar, A. A., and Adzhubei, I. A.
93. Gilbert. R. J., Fucini, P., Connell, S., Fuller, S. D.,
(1988) Role of the rare codon clusters in defining the
Nierhaus, K. H., et al. (2004) Three dimensional struc
boundaries of polypeptide chain regions with identical sec
tures of translating ribosomes by Cryo EM, Mol. Cell, 14,
ondary structures in the process of co translational folding
57 66.
of proteins [in Russian], Dokl. Akad. Nauk SSSR, 303,
94. Kosolapov, A., and Deutsch, C. (2009) Tertiary interac
995 999.
tions within the ribosomal exit tunnel, Nat. Struct. Mol.
114. Krasheninnikov, I. A., Komar, A. A., and Adzhubei, I. A.
Biol., 16, 405 411.
(1989) Frequency of using codons in mRNA and coding of
95. Tu, L., Khanna, P., and Deutsch, C. (2014) Transmem
the domain structure of proteins [in Russian], Dokl. Akad.
brane segments form tertiary hairpins in the folding
Nauk. SSSR, 305, 1006 1012.
vestibule of the ribosome, J. Mol. Biol., 426, 185 198.
115. Krasheninnikov, I. A., Komar, A. A., and Adzhubei, I. A.
96. Holtkamp, W., Kokic, G., Jäger, M., Mittelstaet, J.,
(1989) Role of the code redundancy determining cotrans
Komar, A. A., and Rodnina, M. V. (2015) Cotranslational
lational protein folding, Biokhimiia, 5, 187 200.
protein folding on the ribosome monitored in real time,
116. Heinemann, U., and Hahn, M. (1995) Circular permuta
Science, 350, 1104 1107.
tion of polypeptide chains: implications for protein folding
97. Chen, X., Rajasekaran, N., Liu, K., and Kaiser, C. M.
and stability, Prog. Biophys. Mol. Biol., 64, 121 143.
(2020) Synthesis runs counter to directional folding of a
117. Protzel, A., and Morris, A. J. (1974) Gel chromatographic
nascent protein domain, Nat. Commun., 11, 5096.
analysis of nascent globin chains. Evidence of nonuniform
98. Komar, A. A. (2018) The Yin and Yang of codon usage,
size distribution, J. Biol. Chem., 249, 4594 4600.
Hum. Mol. Genet., 25(R2), R77 R85.
118. Chaney, W. G., and Morris, A. J. (1978) Nonuniform size
99. Komar, A. A. (2007) SNPs, silent but not invisible, Science,
distribution of nascent peptides: the role of messenger
315, 466 467.
RNA, Arch. Biochem. Biophys., 191, 734 741.
100. Tsai, C. J., Sauna, Z. E., Kimchi Sarfaty, C., Ambudkar,
119. Lizardi, P. M., Mahdavi, V., Shields, D., and Candelas, G.
S. V., Gottesman, M. M., and Nussinov, R. (2008)
(1979) Discontinuous translation of silk fibroin in a reticu
Synonymous mutations and ribosome stalling can lead to
locyte cell free system and in intact silk gland cells, Proc.
altered folding pathways and distinct minima, J. Mol. Biol.,
Natl. Acad. Sci. USA, 76, 6211 6215.
383, 281 291.
120. Randall, L. L., Josefsson, L. G., and Hardy, S. J. (1980)
101. Zhang, G., and Ignatova, Z. (2011) Folding at the birth of
Novel intermediates in the synthesis of maltose binding
the nascent chain: coordinating translation with co trans
protein in Escherichia coli, Eur. J. Biochem., 107, 375 379.
lational folding, Curr. Opin. Struct. Biol., 21, 25 31.
121. Abraham, A. K., and Pihl, A. (1980) Variable rate of
102. O’Brien, E. P., Ciryam, P., Vendruscolo, M., and Dobson,
polypeptide chain elongation in vitro. Effect of spermidine,
C. M. (2014) Understanding the influence of codon trans
Eur. J. Biochem., 106, 257 262.
lation rates on cotranslational protein folding, Acc. Chem.
122. Varenne, S., Knibiehler, M., Cavard, D., Morlon, J., and
Res., 47, 1536 1544.
Lazdunski, C. (1982) Variable rate of polypeptide chain
БИОХИМИЯ том 86 вып. 8 2021
КОД ТОНКОЙ НАСТРОЙКИ СВОРАЧИВАНИЯ БЕЛКА
1193
elongation for colicins A, E2 and E3, J. Mol. Biol., 159, 57
141. Kimchi Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E.,
70.
Calcagno, A. M., et al. (2007) A “silent” polymorphism in
123. Candelas, G., Candelas, T., Ortiz, A., and Rodr guez, O.
the MDR1 gene changes substrate specificity, Science, 315,
(1983) Translational pauses during a spider fibroin synthe
525 528.
sis, Biochem. Biophys. Res. Commun., 116, 1033 1038.
142. Zhang, G., Hubalewska, M., and Ignatova, Z. (2009)
124. Varenne, S., Buc, J., Lloubes, R., and Lazdunski, C.
Transient ribosomal attenuation coordinates protein syn
(1984) Translation is a non uniform process. Effect of
thesis and co translational folding, Nat. Struct. Mol. Biol.,
tRNA availability on the rate of elongation of nascent
16, 274 280.
polypeptide chains, J. Mol. Biol., 180, 549 576.
143. Zhou, M., Guo, J., Cha, J., Chae, M., Chen, S., et al.
125. Wolin, S. L., and Walter, P. (1988) Ribosome pausing and
(2013) Non optimal codon usage affects expression, struc
stacking during translation of a eukaryotic mRNA,
ture and function of clock protein FRQ, Nature, 495, 111
EMBO J., 7, 3559 3569.
115.
126. Krasheninnikov, I. A., Komar, A. A., and Adzhubei, I. A.
144. Sander, I. M., Chaney, J. L., and Clark, P. L. (2014)
(1991) Nonuniform size distribution of nascent globin pep
Expanding Anfinsen’s principle: contributions of synony
tides, evidence for pause localization sites, and a cotransla
mous codon selection to rational protein design,
tional protein folding model, J. Protein. Chem., 10, 445
J. Am. Chem. Soc., 136, 858 861.
454.
145. Hu, S., Wang, M., Cai, G., and He, M. (2013) Genetic
127. Thanaraj, T. A., and Argos, P. (1996) Ribosome mediated
code guided protein synthesis and folding in Escherichia
translational pause and protein domain organization,
coli, J. Biol. Chem., 288, 30855 30861.
Protein Sci., 5, 1594 1612.
146. Kim, S. J., Yoon, J. S., Shishido, H., Yang, Z., Rooney,
128. Thanaraj, T. A., and Argos, P. (1996) Protein secondary
L. A., et al. (2015) Protein folding. Translational tuning
structural types are differentially coded on messenger
optimizes nascent protein folding in cells, Science, 348,
RNA, Protein Sci., 5, 1973 1983.
444 448.
129. Adzhubei, A. A., Adzhubei, I. A., Krasheninnikov, I. A.,
147. Yu, C. H., Dang, Y., Zhou, Z., Wu, C., Zhao, F., et al.
and Neidle, S. (1996) Non random usage of “degenerate”
(2015) Codon usage influences the local rate of translation
codons is related to protein three dimensional structure,
elongation to regulate co translational protein folding,
FEBS Lett., 399, 78 82.
Mol. Cell, 59, 744 754.
130. Oresic, M., and Shalloway, D. (1998) Specific correlations
148. Walsh, I. M., Bowman, M. A., Soto Santarriaga, I. F.,
between relative synonymous codon usage and protein sec
Rodriguez, A., and Clark, P. L. (2020) Synonymous codon
ondary structure, J. Mol. Biol., 281, 31 48.
substitutions perturb cotranslational protein folding in vivo
131. Widmann, M., Clairo, M., Dippon, J., and Pleiss, J. (2008)
and impair cell fitness, Proc. Natl. Acad. Sci. USA, 117,
Analysis of the distribution of functionally relevant rare
3528 3534.
codons, BMC Genomics, 9, 207.
149. Komar, A. A. (2007) Silent SNPs: impact on gene function
132. Clarke, T. F. 4th, and Clark, P. L. (2008) Rare codons clus
and phenotype, Pharmacogenomics, 8, 1075 1080.
ter, PLoS One, 3, e3412.
150. Sauna, Z. E., and Kimchi Sarfaty, C.
(2011)
133. Chartier, M., Gaudreault, F., and Najmanovich, R. (2012)
Understanding the contribution of synonymous mutations
Large scale analysis of conserved rare codon clusters sug
to human disease, Nat. Rev. Genet., 12, 683 691.
gests an involvement in co translational molecular recog
151. Hunt, R. C., Simhadri, V. L., Iandoli, M., Sauna, Z. E.,
nition events, Bioinformatics, 28, 1438 1445.
and Kimchi Sarfaty, C. (2014) Exposing synonymous
134. Ma, X. X., Wang, Y. N., Cao, X. A., Li, X. R., Liu, Y. S.,
mutations, Trends Genet., 30, 308 321.
et al. (2018) The effects of codon usage on the formation of
152. Simhadri, V. L., Hamasaki Katagiri, N., Lin, B. C.,
secondary structures of nucleocapsid protein of peste des
Hunt, R., Jha, S., et al. (2017) Single synonymous muta
petits ruminants virus, Genes Genomics, 40, 905 912.
tion in factor IX alters protein properties and underlies
135. Newaz, K., Wright, G., Piland, J., Li, J., Clark, P. L., et al.
haemophilia B, J. Med. Genet., 54, 338 345.
(2020) Network analysis of synonymous codon usage,
153. Pechmann, S., Chartron, J. W., and Frydman, J. (2014)
Bioinformatics, 36, 4876 4884.
Local slowdown of translation by nonoptimal codons pro
136. McKown, R. L., Raab, R. W., Kachelries, P., Caldwell, S.,
motes nascent chain recognition by SRP in vivo, Nat.
and Laurie, G. W. (2013) Conserved regional 3′ grouping of
Struct. Mol. Biol., 21, 1100 1105.
rare codons in the coding sequence of ocular prosecretory
154. Buhr, F., Jha, S., Thommen, M., Mittelstaet, J., Kutz, F.,
mitogen lacritin, Invest. Ophthalmol. Vis. Sci., 54, 1979
et al. (2016) Synonymous codons direct cotranslational
1987.
folding toward different protein conformations, Mol. Cell,
137. Nissley, D. A., Carbery, A., Chonofsky, M., and Deane,
61, 341 351.
C. M. (2021) Ribosome occupancy profiles are conserved
155. Kimchi Sarfaty, C., Schiller, T., Hamasaki Katagiri, N.,
between structurally and evolutionarily related yeast
Khan, M. A., Yanover, C., and Sauna, Z. E. (2013)
domains, Bioinformatics, doi: 10.1093/bioinformatics/
Building better drugs: developing and regulating engi
btab020.
neered therapeutic proteins, Trends Pharmacol. Sci., 34,
138. Komar, A. A., and Jaenicke, R. (1995) Kinetics of transla
534 548.
tion of gamma B crystallin and its circularly permutated
156. Komar, A. A. (2016) The art of gene redesign and recombi
variant in an in vitro cell free system: possible relations to
nant protein production: approaches and perspectives, Top.
codon distribution and protein folding, FEBS Lett., 376,
Med. Chem., 2, 1 17.
195 198.
157. Alexaki, A., Hettiarachchi, G. K., Athey, J. C., Katneni,
139. Rudolph, R., Siebendritt, R., Nesslauer, G., Sharma,
U. K., Simhadri, V., et al. (2019) Effects of codon opti
A. K., and Jaenicke, R. (1990) Folding of an all beta pro
mization on coagulation factor IX translation and struc
tein: independent domain folding in gamma II crystallin
ture: implications for protein and gene therapies, Sci. Rep.,
from calf eye lens, Proc. Natl. Acad. Sci. USA, 87, 4625 4629.
9, 15449.
140. Komar, A. A., Lesnik, T., and Reiss, C.
(1999)
158. Hunt, R., Hettiarachchi, G., Katneni, U., Hernandez, N.,
Synonymous codon substitutions affect ribosome traffic
Holcomb, D., et al. (2019) A single synonymous variant
and protein folding during in vitro translation. FEBS Lett.,
(c.354G>A [p.P118P]) in ADAMTS13 confers enhanced
462, 387 391.
specific activity, Int. J. Mol. Sci., 20, 5734.
7 БИОХИМИЯ том 86 вып. 8 2021
1194
КОМАР
159. Knobe, K. E., Sjorin, E., and Ljung, R. C. (2008) Why
structure in determining the pathogenicity of synonymous
does the mutation G17736A/Val107Val (silent) in the F9
and non synonymous mutations in haemophilia,
gene cause mild haemophilia B in five Swedish families?
Haemophilia, 23, e8 e17.
Haemophilia, 14, 723 728.
163. Katneni, U. K., Liss, A., Holcomb, D., Katagiri, N. H.,
160. Gartner, J. J., Parker, S. C., Prickett, T. D., Dutton
Hunt, R., et al. (2019) Splicing dysregulation contributes
Regester, K., Stitzel, M. L., et al. (2013) Whole genome
to the pathogenicity of several F9 exonic point variants,
sequencing identifies a recurrent functional synonymous
Mol. Genet. Genomic Med., 7, e840.
mutation in melanoma, Proc. Natl. Acad. Sci. USA, 110,
164. Edwards, N. C., Hing, Z. A., Perry, A., Blaisdell, A.,
13481 13486.
Kopelman, D. B., F et al. (2012) Characterization of cod
161. Hamasaki Katagiri, N., Salari, R., Wu, A., Qi, Y.,
ing synonymous and non synonymous variants in
Schiller, T., et al. (2013) A gene specific method for pre
ADAMTS13 using ex vivo and in silico approaches, PLoS
dicting hemophilia causing point mutations, J. Mol. Biol.,
One, 7, e38864.
425, 4023 4033.
165. Sauna, Z. E., Richards, S. M., Maillere, B., Jury, E. C.,
162. Hamasaki Katagiri, N., Lin, B. C., Simon, J., Hunt,
and Rosenberg, A. S. (2020) Editorial: immunogenicity of
R. C., Schiller, T., et al. (2017) The importance of mRNA
proteins used as therapeutics, Front. Immunol., 11, 614856.
A CODE WITHIN A CODE: HOW CODONS FINEJTUNE
PROTEIN FOLDING IN THE CELL
Review
A. A. Komar1,2,3,4
1 Center for Gene Regulation in Health and Disease and Department of Biological, Geological and Environmental
Sciences, Cleveland State University, Cleveland, Ohio 44115, USA; e mail: a.komar@csuohio.edu
2 Department of Biochemistry and Center for RNA Science and Therapeutics,
Case Western Reserve University, Cleveland, Ohio 44106, USA
3 Genomic Medicine Institute, Lerner Research Institute, Cleveland Clinic, Cleveland, Ohio 44195, USA
4 DAPCEL, Inc., Cleveland, Ohio 44106, USA
The genetic code sets the correspondence between the sequence of a given nucleotide triplet in an mRNA molecule,
called a codon, and the amino acid that it directs to be added to the growing polypeptide chain during protein syn
thesis. With four bases (A, G, U, and C), there are 64 possible triplet codons; 61 sense (encoding amino acids) and
3 nonsense (so called, stop codons that direct termination of translation). In most organisms, there are 20 com
mon/standard amino acids used in protein synthesis; thus, the genetic code is redundant with most amino acids (with
the exception of Met and Trp) are being encoded by more than one (synonymous) codon. Synonymous codons were
initially presumed to have entirely equivalent functions, however, the finding that synonymous codons are not present
at equal frequencies in mRNA suggested that specific codon choice might have functional implications beyond amino
acid coding. Observation of nonequivalent use of codons in mRNAs raised a possibility of the existence of auxiliary
information in the genetic code. Indeed, it has been found that genetic code contains several layers of such addition
al information and that synonymous codons are strategically placed within mRNAs to ensure a particular translation
kinetics facilitating and fine tuning co translational protein folding in the cell via a step wise/sequential structuring
of distinct regions of the polypeptide chain emerging from the ribosome at different points in time. This review sum
marizes key findings in the field that have identified the role of synonymous codons and their usage in protein folding
in the cell.
Keywords: genetic code, codon usage, synonymous codons, translation kinetics, nascent peptides, co translational
protein folding
БИОХИМИЯ том 86 вып. 8 2021