БИОХИМИЯ, 2021, том 86, вып. 8, с. 1195 - 1206
УДК 577.217.348
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
Обзор
© 2021
Ч. Бао, Д.Н. Ермоленко*
Department of Biochemistry & Biophysics, School of Medicine and Dentistry
and Center for RNA Biology, University of Rochester, Rochester, NY, USA;
e mail: Chen_Bao@urmc.rochester.edu; dmitri_ermolenko@urmc.rochester.edu
Поступила в редакцию 10.05.2021
После доработки 21.05.2021
Принята к публикации 21.05.2021
В процессе синтеза белка рибосома передвигается вдоль молекулы мРНК, считывая один кодон за другим.
Транслокация рибосомы индуцируется консервативным белком, фактором элонгации G (EF#G) у бактерий
и фактором элонгации 2 (EF#2) у эукариот. Индуцируемая фактором элонгации EF#G транслокация приво#
дит к раскручиванию внутримолекулярных вторичных структур мРНК на три пары оснований одновремен#
но, что превращает транслирующую рибосому в процессивную хеликазу. Профессор Александр Сергеевич
Спирин внёс значительный вклад в понимание молекулярного механизма транслокации. В настоящем об#
зоре рассмотрены идей Спирина о транслокации рибосом и последние достижения в этой области, которые
стали возможны благодаря новаторской работе Спирина. Мы также обсудим нерешённые проблемы, каса#
ющиеся транслоказной и хеликазной активности рибосомы.
КЛЮЧЕВЫЕ СЛОВА: рибосома, транслокация, хеликаза, рабочий ход, броуновский храповик.
DOI: 10.31857/S0320972521080108
ВВЕДЕНИЕ
рии Александра Спирина на протяжении более
50 лет [1-6]. Лаборатория Спирина открыла ряд
На стадии элонгации биосинтеза белка ри#
важных аспектов механизма транслокации
босома производит декодирование нуклеотид#
[7-11]. Спиринские модели рибосомной транс#
ной последовательностей кодонов, связываю#
локации со «смыканием-размыканием» [2, 6] и
щихся с антикодоном молекулы тРНК, несущей
«броуновским храповиком» [4, 12, 13] оказали
аминокислотные остатки. И малые (30S - у бак#
большое влияние и заложили основу для иссле#
терий), и большие (50S - у бактерий) субъеди#
дований транслокации на протяжении десяти#
ницы содержат три сайта связывания тРНК:
летий. Ниже мы рассмотрим ключевой вклад ла#
сайты A (аминоацил), P (пептидил) и E (выход).
боратории Александра Спирина и недавний
В начале каждого цикла элонгации вновь посту#
прогресс в исследованиях рибосомной трансло#
пившая аминоацил#тРНК связывается с А#сай#
кации, основанный на идеях Спирина. Более
том рибосомы. После реакции переноса пепти#
подробные обзоры современного состояния по#
дила образующиеся пептидил#тРНК и деацили#
нимания механизма транслокации можно найти
рованная тРНК вместе с ассоциированными ко#
в других источниках [14-16].
донами мРНК транслоцируются из сайтов А и Р
в сайты Р и Е соответственно (рис. 1, a-f). Этот
процесс катализируется универсально консер#
РОЛЬ EF!G И МОЛЕКУЛ тРНК
вативным белковым фактором EF#G (EF#2 - у
В ТРАНСЛОКАЦИИ РИБОСОМЫ
эукариот). Молекулярный механизм транслока#
ВДОЛЬ МОЛЕКУЛЫ мРНК
ции интересовал учёных с момента зарождения
области синтеза белков и оставался одним из ос#
Универсально консервативная ГТФаза EF#G
новных направлений исследований в лаборато# ускоряет транслокацию рибосом примерно на
4 порядка по сравнению с некатализируемой ре#
акцией [18-20]. Гидролиз ГТФ под действием
Принятые сокращения: ASL - антикодоны «сте#
EF#G активируется в результате взаимодействия
бель-петля»; EF#G - фактор элонгации G; EF#2 - фактор
элонгации 2; R, NR и CH - повёрнутое, неповёрнутое и
G#домена этого белка с сарцин#рициновой пет#
химерное состояния 50S# и 30S#субъединиц рибосомы.
лей (SRL) 23S рРНК [21, 22]. Роль гидролиза
* Адресат для корреспонденции.
ГТФ в транслокации была установлена в работах
1195
7*
1196
БАО, ЕРМОЛЕНКО
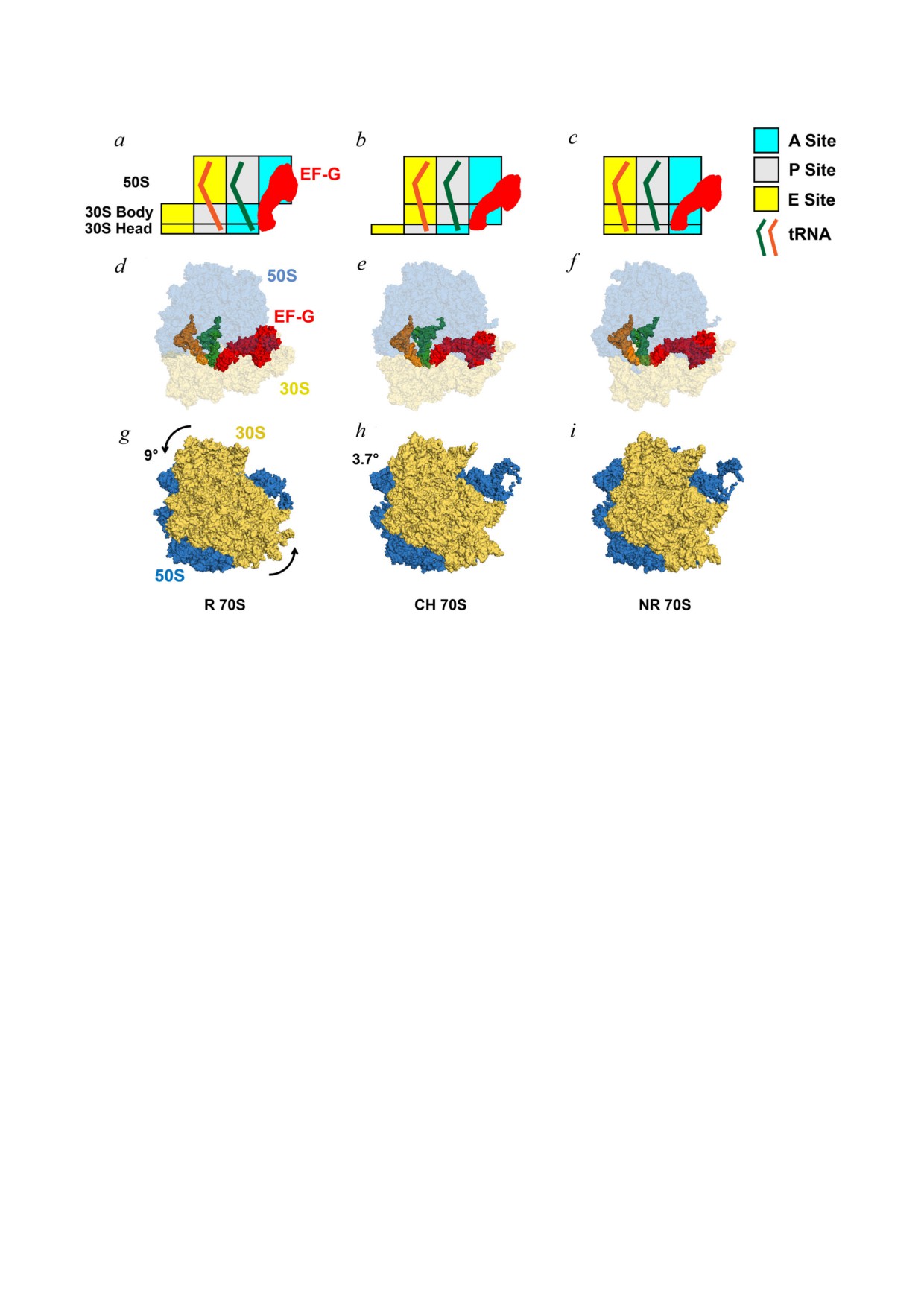
Рис. 1. Движение тРНК и конформационные перестройки рибосомы в составе комплекса EF#G-рибосома. а-c - Блок#
диаграммы, показывающие положения пептидил#тРНК (зелёный) и деацилированной (оранжевый) тРНК относительно
сайтов A (бирюзовый), P (серый) и E (жёлтый) на 50S#субъединице и головке и теле 30S#субъединицы. d-f - Вид струк#
туры со стороны интерфейса 70S#субъединицы рибосомы, связанной с тРНК и EF#G, на котором 50S# и 30S#субъедини#
цы показаны светло#голубым и светло#жёлтым цветом соответственно, а две тРНК - темно#оранжевым и темно#зелёным,
EF#G - темно#красным. g-i - Межсубъединичное вращение, сопровождающее транслокацию и наблюдаемое со сторо#
ны 30S#субъединицы, обращённой к растворителю. 50S# и 30S#субъединицы показаны синим и жёлтым цветом соответ#
ственно. Стрелками обозначено вращение против часовой стрелки 30S#субъединицы относительно 50S#субъединицы.
Значения углов межсубъединичного вращения (согласно статье Mohan et al. [17]), измеренные относительно структуры
невращающейся рибосомы (PDBID 4V51). a, d и g - Повёрнутая (R) рибосома перед транслокацией связана с EF#G и
тРНК в гибридных состояниях A/P и P/E (PDBID 4V7D). b, e и h - Частично повёрнутая рибосома, содержащая тРНК в
химерных (CH) состояниях ap/P и pe/E молекул тРНК (PDBID 4W29). c, f и i - Рибосома, связанная с EF#G после транс#
локации, в неповёрнутой (NR) конформации (PDBID 4V5F), которая содержит тРНК, связанные в классических P/P
и E/E состояниях
сотрудников лабораторий Казиро и Спирина [9,
бождения EF#G. Результаты экспериментов
23-25]. Они продемонстрировали, что EF#G
Спирина и Казиро были подтверждены более
индуцирует эффективную транслокацию в
поздними кинетическими исследованиями, по#
ГТФ#связанной форме. В присутствии ГДФ или
казавшими, что негидролизуемые аналоги ГТФ
при отсутствии нуклеотидов EF#G не способ#
не изменяют путь транслокации [26, 27]. Более
ствует транслокации. Замены ГТФ на негидро#
того, замена ГТФ на негидролизуемые аналоги
лизуемые аналоги сохраняли способность EF#G
лишь умеренно влияла на скорость одного раун#
индуцировать транслокацию, оцениваемую по
да транслокации, снижая её в 2-50 раз в зависи#
повышению чувствительности пептидил#тРНК
мости от условий эксперимента [19, 26, 28, 29].
к пуромицину или по высвобождению деацили#
Ещё один фундаментальный аспект меха#
рованной тРНК из рибосомы. Однако негидро#
низма транслокации был обнаружен в лаборато#
лизуемые аналоги ГТФ удерживают EF#G на ри#
рии Спирина в ходе экспериментов, показав#
босоме. Эти результаты позволили сделать пред#
ших, что молекулы тРНК могут перемещаться в
положение о том, что гидролиз GTP не требует#
рибосоме при отсутствии мРНК [10, 11]. На ос#
ся для транслокации, но необходим для высво#
новании этих результатов было сделано предпо#
БИОХИМИЯ том 86 вып. 8 2021
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
1197
ложение о том, что мРНК приводится в движе#
Через двадцать лет после появления модели
ние транслокацией антикодонов «стебель-пет#
«смыкания-размыкания» Данеш Моазед и Гарри
ля» (ASL) в тРНК, находящихся на A# и P#сай#
Ноллер использовали химическое зондирова#
тах. Более поздние исследования показали, что
ние для картирования сайтов связывания тРНК,
для транслокации мРНК необходимо присут#
чтобы показать, что реакция транспептидации
ствие ASL в сайте A и полноразмерной тРНК -
запускает спонтанную транслокацию акцептор#
на сайте P пре#транслокационной рибосо#
ных ветвей пептидил#тРНК и деацилированной
мы [30]. Идея о транслокации мРНК, движимой
тРНК из сайтов A и P в сайты P и E большой
тРНК, была подтверждена с помощью методов
субъединицы соответственно, тогда как
измерения одиночных молекул, показавших,
ASL тРНК остаются в исходных сайтах A и P ма#
что мРНК за один раз перемещает три нуклео#
лой субъединицы [36]. Таким образом, были об#
тида без каких#либо промежуточных эта#
наружены промежуточные гибридные состоя#
пов [31]. Поскольку тРНК взаимодействует как
ния связывания тРНК, A/P и P/E (рис. 1, а и d).
с 30S#, так и с 50S#субъединицей (рис. 1, a-f),
Было показано, что для завершения транслока#
движимый молекулами тРНК механизм транс#
ции тРНК в малую субъединицу требуются EF#G
локации означает, что в этом процессе участву#
и ГТФ (рис. 1, а-g).
ют обе субъединицы рибосомы.
Прошло ещё десять лет, прежде чем было по#
лучено подтверждение другого предсказания мо#
дели «смыкания-размыкания». Иоахим Франк
ПЕРЕСТРОЙКА СТРУКТУРЫ РИБОСОМЫ:
и Раджендра Агравал изучили комплекс EF#G и
ГИПОТЕЗА СМЫКАНИЯ-РАЗМЫКАНИЯ
рибосомы с помощью cryo#EM [37]. В этих экс#
периментах было показано, что связывание
В 1968-1969 гг., основываясь на субъединич#
EF#G вызывает вращение малой 30S#субъеди#
ной структуре рибосомы, Спирин предложил
ницы относительно большой 50S#субъединицы
модель транслокации, основанную на смыка#
параллельно плоскости межсубъединичного ин#
нии-размыкании [1, 2, 6]. Эта модель предпола#
терфейса (рис. 1, g-i; рис. 2, a-b). За открытия#
гала, что: 1) транслокация тРНК включает пере#
ми гибридного промежуточного состояния и
ход рибосомы из «сомкнутой» конформации в
межсубъединичного вращения последовали
«разомкнутую», что способствует диффузии
многочисленные структурные и одномолеку#
тРНК через рибосому; 2) реакция с пептидил#
лярные исследования, которые принесли бес#
трансферазой запускает образование промежу#
прецедентное понимание структурных перест#
точного соединения транслокации, при кото#
роек рибосом, тРНК и EF#G, сопровождающих
ром тРНК сдвигаются в большой субъединице,
транслокацию [38, 39].
но ещё не перемещаются в малой субъединице;
Эксперименты с использованием методов
3) транслокация включает перемещение рибо#
сryo#EM и smFRET показали, что межсубъеди#
сомных субъединиц друг относительно друга.
ничное вращение на 6-10 ° связано с переходом
Сходные идеи были независимо от Спирина
пептидил#тРНК и деацилированной тРНК в
представлены М.С. Бретчером [32]. Как будет
гибридные состояния A/P и P/E (рис. 2, a-b)
обсуждаться ниже, многие аспекты этой модели
[38, 40-43]. Эти данные установили тождествен#
оказались пророческими. По сей день этап, ог#
ность исходной (неповёрнутой) и повёрнутой
раничивающий скорость транслокации, часто
конформаций с классическим и гибридным сос#
называют «размыкание».
тояниями связывания тРНК соответственно.
С помощью метода нейтронного рассеива#
Использование методов сryo#EM и smFRET
ния Спирин, Сердюк и Мэй получили первые
также позволило показать, что образование по#
косвенные доказательства межсубъединичных
вёрнутого гибридного состояния рибосомы соп#
перестроек, сопровождающих транслока#
ровождается движением внутрь подвижного до#
цию [33, 34]. Тем не менее дальнейшая проверка
мена большой субъединицы рибосомы, называ#
ключевых предсказаний, вытекающих из моде#
емого L1#выступом (рис. 2, c), который включа#
ли «смыкания-размыкания», заняла несколько
ет рибосомный белок uL1 и спиральные струк#
десятилетий и потребовала разработки новых
туры 76, 77 и 78 23S рРНК [35, 39]. При перехо#
экспериментальных подходов, таких как хими#
де от открытой к закрытой конформации конец
ческое зондирование структуры РНК, криоэлек#
L1#выступа перемещается на целых 60 Å и ста#
тронная микроскопия (cryo#EM) и микроско#
новится связанным с локтем тРНК в сайте P/E.
пия, основанная на Фёрстеровском индуктив#
Исследования smFRET также показали, что
ном резонансном переносе энергии электрон#
при отсутствии EF#G пре#транслокационная
ного возбуждения одиночных макромоле#
рибосома спонтанно совершает непроизволь#
кул (smFRET) [35].
ные переходы между неповёрнутой (классичес#
БИОХИМИЯ том 86 вып. 8 2021
1198
БАО, ЕРМОЛЕНКО
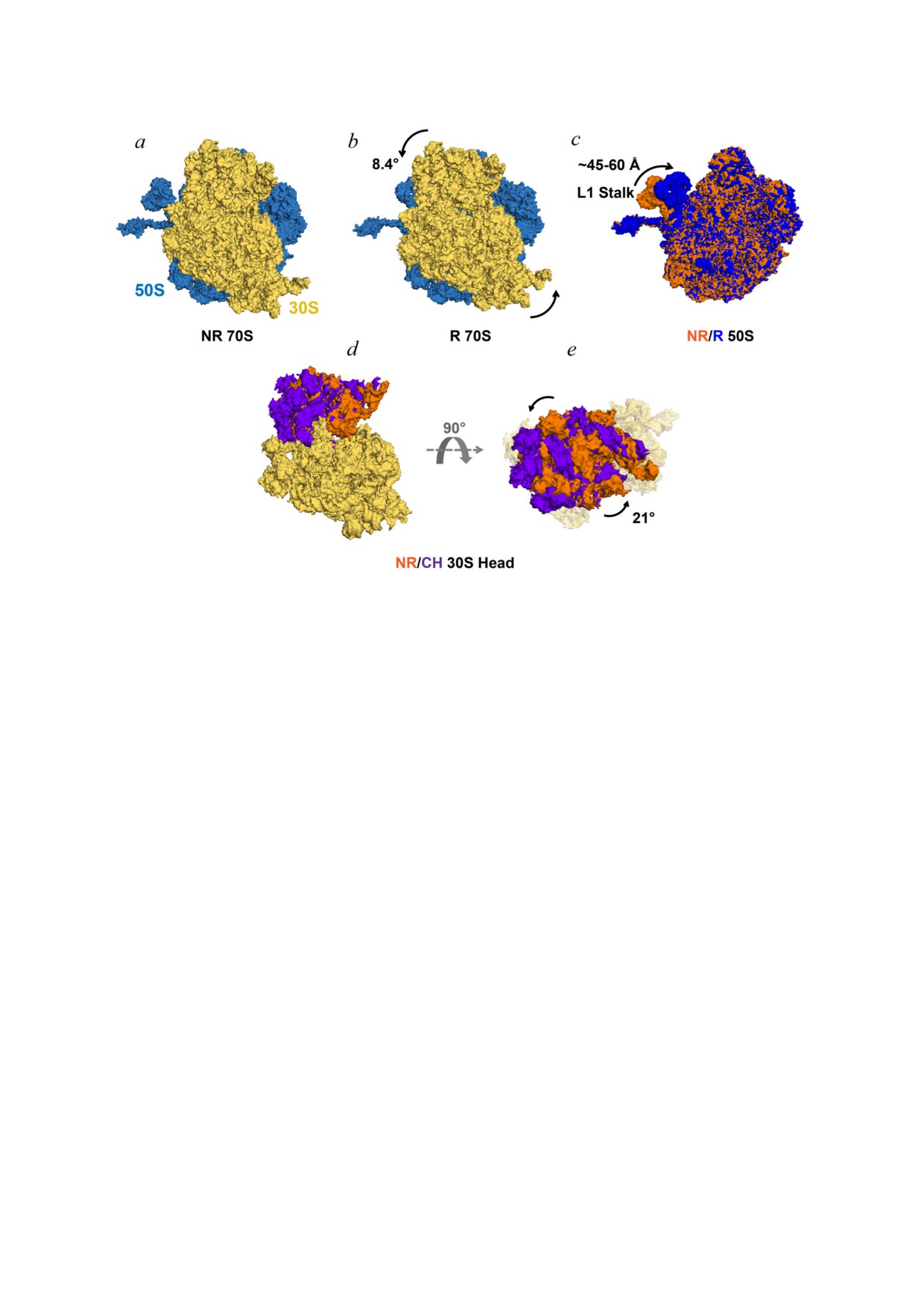
Рис. 2. Межсубъединичное вращение, движение L1#стебля и поворот головки 30S#субъединицы, наблюдаемые в рибосо#
мах, свободных от EF#G. Структуры комплекса 70S#рибосомы и тРНК (PDBID 4V9D) в неповёрнутом (NR) классичес#
ком состоянии (a) и в повёрнутом (R) гибридном состоянии (b) 50S# и 30S#субъединицы рибосомы показаны синим и
желтым цветом соответственно. Обе структуры рибосомы показаны со стороны, обращённой к растворителю 30S#субъ#
единицы. Вращение против часовой стрелки 30S#субъединицы относительно 50S#субъединицы показано изогнутыми
стрелками. c - L1#выступ в 50S#субъединицах NR (оранжевый) и R (синий) рибосом (PDBID 4V9D) наложенный путём
структурного выравнивания 23S рРНК. d и e - Поворот домена головки 30S#субъединицы показан структурным вырав#
ниванием доменов тела и платформы (жёлтый) 16S рРНК рибосом NR (PDBID 4V51) и химерного состоя#
ния (CH) (PDBID 4W29). 30S#субъединица показана со стороны растворителя (d) и со стороны «вершины» головки 30S#
субъединицы (e). Домены головки 30S#субъединицы рибосомы в NR# и CH#состояниях показаны оранжевым и фиолето#
вым цветом соответственно. Поворот головки 30S#субъединицы в состоянии CH и межсубъединичное вращение в состо#
янии R (согласно статье Mohan et al. [17]) были измерены относительно 70S#рибосомы в состоянии NR (PDBID 4V51)
кой) и повёрнутой (гибридное состояние) кон#
с EF#G промежуточном состоянии транслока#
формациями (рис. 2, a-b) [38, 44-46]. Связыва#
ции, визуализируемом с помощью рентгенов#
ние EF#G•ГТФ временно стабилизирует повёр#
ской кристаллографии и cryo#EM, в котором
нутую конформацию в гибридном состоянии
30S#головка наблюдается в повёрнутой конфор#
(рис. 1, a, d и g). Транслокация мРНК и тРНК на
мации, две тРНК перемещаются вдоль 50S#
малой субъединице сопряжена с обратным пе#
субъединицы и 30S#платформы/тела, но ещё не
реходом в конформацию неповёрнутого, клас#
перемещаются относительно
30S#головки
сического состояния (рис. 1, c, f и i) [26, 40, 47].
(рис. 1, b, e и h) [50, 51]. В этих положениях, ко#
Кроме межсубъединичных перестроек и
торые обозначаются как химерные состоя#
движения L1#выступа, транслокация сопровож#
ния ap/P и pe/E, тРНК находятся на полпути
дается большими структурными изменениями
между гибридными (состояния A/P и P/E) и
внутри малой субъединицы рибосомы. Малая
пост#транслокационными классическими (сос#
субъединица состоит их трёх структурных доме#
тояния P/P и E/E) состояниями и, вероятно,
нов: головка, тело и платформа. Структурные
представляют собой позднее промежуточное
исследования показали, что головка 30S#субъ#
состояние транслокации.
единицы поворачивается на угол до 20 ° относи#
Конец домена IV фактора элонгации EF#G
тельно остальной части малой субъединицы
играет ключевую роль в транслокационной ак#
вокруг ортогональной оси межсубъединичного
тивности EF#G и поддержании рамки считыва#
вращения (рис 2, d-e) [48, 49]. В связанном
ния [19, 52-54]. Когда EF#G связан с пре#транс#
БИОХИМИЯ том 86 вып. 8 2021
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
1199
локационной рибосомой в конформации повер#
локации, возможно, за счёт ослабления взаимо#
нутого гибридного состоянии, домен IV EF#G
действия тРНК с сайтами А и Р на 30S#субъеди#
располагается рядом с ASL тРНК A/P (рис. 1, a
нице соответственно [65]. Кроме того, сообща#
и d) [55]. После транслокации домен IV EF#G
лось, что однократная транслокация без факто#
стыкуется с сайтом A малой субъединицы, осво#
ров элонгации может быть вызвана антибиоти#
бождённой от пептидил#тРНК (рис. 1, c и f)
ками спарсомицином, линкомицином и хло#
[38, 56]. Следовательно, при обратном межсубъ#
рамфениколом, которые связываются с сай#
единичном вращении и обратном повороте 30S#
том А на 50S#субъединице и таким образом де#
субъединицы домен IV EF#G вытесняет ASL
стабилизируют связывание тРНК на А#сай#
пептидил#тРНК и предотвращает его обратное
те [20, 66].
движение.
Основываясь на наблюдениях бесфакторной
Не совсем понятно, какая из вышеперечис#
транслокации, Спирин постулировал, что этот
ленных конформационных перестроек лимити#
процесс является неотъемлемым свойством ри#
рует скорость транслокации, «размыкает» рибо#
босомы и что энергии пептидил#трансферазных
сому (по терминологии Спирина) и облегчает
реакций вполне достаточно для того, чтобы спо#
транслокацию тРНК. Реакция транспептидации
собствовать движению тРНК [3]. Однако низкая
«размыкает» рибосому в том смысле, что она де#
скорость спонтанной транслокации и наблюде#
лает возможным спонтанное межсубъединич#
ние спонтанной обратной транслокации в неко#
ное вращение и колебания тРНК между класси#
торых контекстах тРНК/мРНК позволяют пред#
ческим и гибридным состояниями [44-46]. Од#
положить, что реакция транспептидации - не
нако при отсутствии EF#G эти колебания бес#
единственный источник энергии для трансло#
полезны и не приводят к классической трансло#
кации, и ей также способствует энергия, запа#
кации тРНК и мРНК [44-46]. Согласно нес#
сённая в виде EF#G•ГТФ. Действительно, было
кольким линиям доказательств, домен IV EF#G
подсчитано, что движимая транспептидацией
дестабилизирует взаимодействия A#сайта тРНК
транслокация будет нуждаться в пример#
с 16S рРНК [57-61]. Следовательно, вызванные
но 80% эффективности конверсии химической
EF#G изменения в сайте A могут «разомкнуть»
энергии в механическое движение [67]. Такая
рибосому. Наконец, другой вероятной «размы#
высокая эффективность нетипична для макро#
кающей» перестройкой является поворот голов#
молекулярных моторов [67].
ки 30S#субъединицы, который открывает путь
Две альтернативные идеализированные мо#
для передвижения тРНК из Р#сайта в Е#сайт,
дели, рабочий ход и броуновский храповик, ис#
которое в противном случае ограничено [49].
пользуются для описания преобразования хи#
Необходимы дальнейшие исследования, чтобы
мической энергии в механическую работу мак#
установить полную последовательность струк#
ромолекулярными двигателями [68, 69]. Хими#
турных перестроек, сопровождающих трансло#
ческая энергия может быть преобразована в уп#
кацию, и определить лимитирующую стадию в
ругую энергию или в конформационный пере#
этом процессе.
ход, который вызывает большое конформаци#
онное изменение макромолекулы, т.е. рабочий
ход. В качестве альтернативы можно использо#
ЭНЕРГЕТИКА ТРАНСЛОКАЦИИ
вать энергию химической реакции для смеще#
И МОДЕЛЬ БРОУНОВСКОГО ХРАПОВИКА
ния случайных движений макромолекулы под
действием тепла в однонаправленное движение.
Хотя EF#G значительно ускоряет процесс
Таким образом, химическая реакция играет роль
трансляции, в лабораториях Спирина и Пестки
собачки, управляющей движением колеса меха#
было показано, что транслокация может также
нической трещотки. Химическое изменение ли#
происходить спонтанно при отсутствии белко#
бо строго предшествует изменению конформа#
вых факторов [7, 8, 62]. В лаборатории Спирина
ции (рабочий ход), либо следуют за ним (броу#
было обнаружено, что спонтанная транслока#
новский храповик) [68]. Эти два механизма
ция стимулируется модификацией универсаль#
можно различить, исследуя зависимость моле#
но консервативного рибосомного белка uS12
кулярного моторного движения от нагруз#
30S#субъединицы тиол#специфичными реаген#
ки [68].
тами, которые при добавлении инактивируют
Несколько групп исследователей предполо#
фактор элонгации EF#G и таким образом ис#
жили, что транслокация тРНК опосредована ра#
ключают присутствие следовых количеств этого
бочим ходом домена IV EF#G, управляемым
белка [7, 63, 64]. В более поздних исследованиях
гидролизом ГТФ [19, 70, 71]. Эта гипотеза под#
было показано, что удаление 30S#белков uS12
тверждается кинетическими данными, предпо#
и uS13 вызывает усиление бесфакторной транс#
лагающими, что гидролиз ГТФ фактором элон#
БИОХИМИЯ том 86 вып. 8 2021
1200
БАО, ЕРМОЛЕНКО
гации EF#G предшествует транслокации [19].
делает движение тРНК необратимым (рис. 1,
Однако эксперименты, выполненные в лабора#
c и f).
ториях Спирина и Казиры, и более поздние ки#
нетические измерения показывают, что транс#
локация происходит быстро и эффективно при
РОЛЬ ТРАНСЛОКАЦИИ РИБОСОМЫ
отсутствии гидролиза ГТФ, если заменить ГТФ
В РЕГУЛЯЦИИ ТРАНСЛЯЦИИ:
на его негидролизуемые аналоги [9, 19, 23-26,
РИБОСОМА В РОЛИ ХЕЛИКАЗЫ
28, 29]. Эти данные предполагают, что гидро#
лиз ГТФ с помощью EF#G не связан с трансло#
В то время как в результате проведения
кацией, и противоречат механизму рабочего хо#
структурных исследований и биофизических
да, который реализовывался бы за счёт гидроли#
измерений одиночных молекул были получены
за ГТФ.
беспрецедентные данные о механизме трансло#
Основываясь на наблюдениях спонтанной
кации, остаётся не до конца ясным, как ско#
транслокации при отсутствии факторов элонга#
рость транслокации модулируется в живых
ции, Спирин пришёл к выводу, что броуновское
клетках, чтобы регулировать удлинение транс#
движение тРНК является достаточным для объ#
ляции. Было показано, что эукариотическая
яснения транслокации без индукции рабочего
транслоказа EF#2 подавляется в стрессовых ус#
хода с помощью EF#G [3, 12, 13]. Эта гипотеза
ловиях в результате фосфорилирования [73-75].
была дополнительно подтверждена данными
Кроме того, EF#2 может быть инактивирована
smFRET, продемонстрировавшими спонтанное
АДФ#рибозилированием, катализируемым ди#
межсубъединичное вращение и колебания
фтерийным токсином [76]. Ряд антибиотиков
тРНК между классическим и гибридным состо#
препятствуют росту клеток, препятствуя транс#
яниями [44-46]. Идеи Спирина, в конечном
локации в бактериях [14]. По#видимому, наиме#
итоге, были подкреплены измерениями с по#
нее понятым и наиболее интересным аспектом
мощью одномолекулярного оптического твизе#
регуляции транслокации является модуляция
ра транслокации рибосомы против приложен#
скорости транслокации вторичной структу#
ной силы, показавшими, что катализируемая
рой мРНК.
EF#G транслокация также лучше всего описы#
Вычислительный анализ показывает, что
вается моделью броуновского храповика [67].
большинство (если не все) молекул мРНК
В броуновском храповом механизме транс#
склонны к образованию по всей последователь#
локации, катализируемой EF#G, EF#G•ГТФ,
ности обширных внутримолекулярных вторич#
по#видимому, действует как собачка храпового
ных структур, включая и открытую рамку счи#
механизма, который смещает диффузию тРНК
тывания (ORF) [77]. Сворачивание мРНК при#
через рибосому и связывает транслокацию с ди#
водит к образованию компактных структур с
намикой рибосомы [14, 15]. Полученная струк#
близко расположенными концевыми участка#
тура повёрнутой пре#трансляционной рибосо#
ми [78]. Работы по зондированию структуры
мы с EF#G (рис. 1, a и d) продемонстрировала
РНК in vivo на уровне транскриптома [79-84]
основу для сопряжения межсубъединичного
показали, что по крайней мере до некоторой
вращения и транслокации тРНК/мРНК [55].
степени молекулы мРНК сворачиваются в жи#
Непродуктивные спонтанные колебания рибо#
вых клетках, несмотря на присутствие РНК#хе#
сомы между повёрнутой и неповёрнутой кон#
ликаз и других РНК#связывающих белков, ко#
формациями приводят к возвращению пепти#
торые могут нарушать вторичную структуру
дил#тРНК из гибридного состояния A/P в клас#
РНК. В соответствии с идеей о том, что мРНК
сическое состояние A/A. Однако, когда EF#G
обладает развитой вторичной структурой in vivo,
связывается с повёрнутой пре#транслокацион#
было показано, что отдельные элементы струк#
ной рибосомой, домен IV фактора EF#G создаёт
туры мРНК регулируют инициацию трансля#
стерические помехи для возвращения пепти#
ции, в том числе бактериальные рибопереклю#
дил#тРНК из гибридного состояния A/P в клас#
чатели [85], индуцирующие сдвиг рамки считы#
сическое состояние A/A [55]. Сходным образом
вания «шпильки», и псевдоузлы эукариотичес#
домен IV фактора EF#G стерически препятству#
ких вирусов
[86], а также IRES (Internal
ет возвращению пептидил#тРНК из химерного
Ribosome Entry Sites ) [87] и IRE (Iron Response
состояния ap/P в классическое состояние A/A
Elements), расположенные на 5′#UTR#тран#
после обратного поворота головки 30S#субъеди#
скриптов, кодирующих белки, участвующие в
ницы [72]. Кроме того, после транслокации
метаболизме железа
[88] и CITE (Cap#
пептидил#тРНК из сайта A на сайт P малой
Independent Translational Enhancers) [89]. Кроме
субъединицы домен фактора элонгации EF#G
того, было обнаружено, что связывание белков
связывается с А#сайтом 30S#субъединицы, что
и миРНК с мРНК регулируется структурой
БИОХИМИЯ том 86 вып. 8 2021
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
1201
РНК [90-95], что даёт дополнительные доказа#
няет белки комплекса сращивания экзо#
тельства важности вторичной структуры мРНК.
нов (EJC) и другие белки, располагающиеся на
С помощью биохимических методов и мето#
мРНК внутри ядра [106], может способствовать
дов исследования одиночных макромолекул бы#
сворачиванию мРНК в компактные структуры
ло показано, что транслирующая рибосома яв#
после завершения синтеза белка. В самом деле,
ляется очень эффективной хеликазой [96, 97],
исследования флуоресцентной гибридизации
которая расплетает три пары оснований за один
in situ с мономолекулярным разрешени#
шаг транслокации (рис. 3). Транслирующая ри#
ем (smFISH) и исследования лигирования прок#
босома разрушает вторичную структуру мРНК,
симальных групп показывают отсутствие взаи#
так как узкий канал для мРНК на малой субъ#
модействий между отдалёнными сегментами
единице рибосомы может принять только одно#
ядерных мРНК, связанных с белками комплек#
цепочечную мРНК [96, 98-101]. Наряду с демон#
са сращивания экзонов (EJC) [104, 107]. Напро#
страцией хеликазной активности рибосом, ана#
тив, для 5′# и 3′#концевых участков цитоплазма#
лиз профиля рибосомы по всему транскриптому
тических мРНК, которые не подвергаются ак#
демонстрирует, что большинство элементов вто#
тивной трансляции, характерна совместная ло#
ричной структуры в кодирующих областях
кализация благодаря образованию внутримоле#
мРНК не влияют на скорость элонгации транс#
кулярной вторичной структуры [104, 105], кото#
ляции [102]. Большинство структурированных
рая сближает концы мРНК [78, 108].
элементов мРНК, которые регулируют трансля#
Как это ни парадоксально, несмотря на хе#
цию, находятся в 5′# или 3′#UTR.
ликазную активность рибосомы, определенные
Хеликазная активность рибосомы, по#види#
структуры РНК, такие как «стебель-петля», мо#
мому, играет основную роль в ремоделлинге
гут индуцировать торможение движения рибо#
структуры мРНК и в регулировании взаимодей#
сомы, приводящее к накоплению укороченных
ствия мРНК с РНК#связывающими белка#
полипептидов [109] и разрушению остановив#
ми [103]. Например, при трансляции мРНК по#
шихся мРНК (No#Go) (рис. 3) [110]. Более того,
лирибосомами благодаря хеликазной активнос#
эволюционно консервативные структуры «сте#
ти рибосомы происходит разрушение вторич#
бель-петля» молекул мРНК и псевдоузлы за#
ной структуры открытой рамки считыва#
пускают запрограммированные трансляцион#
ния (ORF) мРНК [103-105]. Первый этап
ные паузы [111] и стимулируют -1 запрограм#
трансляции мРНК рибосомой, которая вытес#
мированный сдвиг рамки считывания рибо#
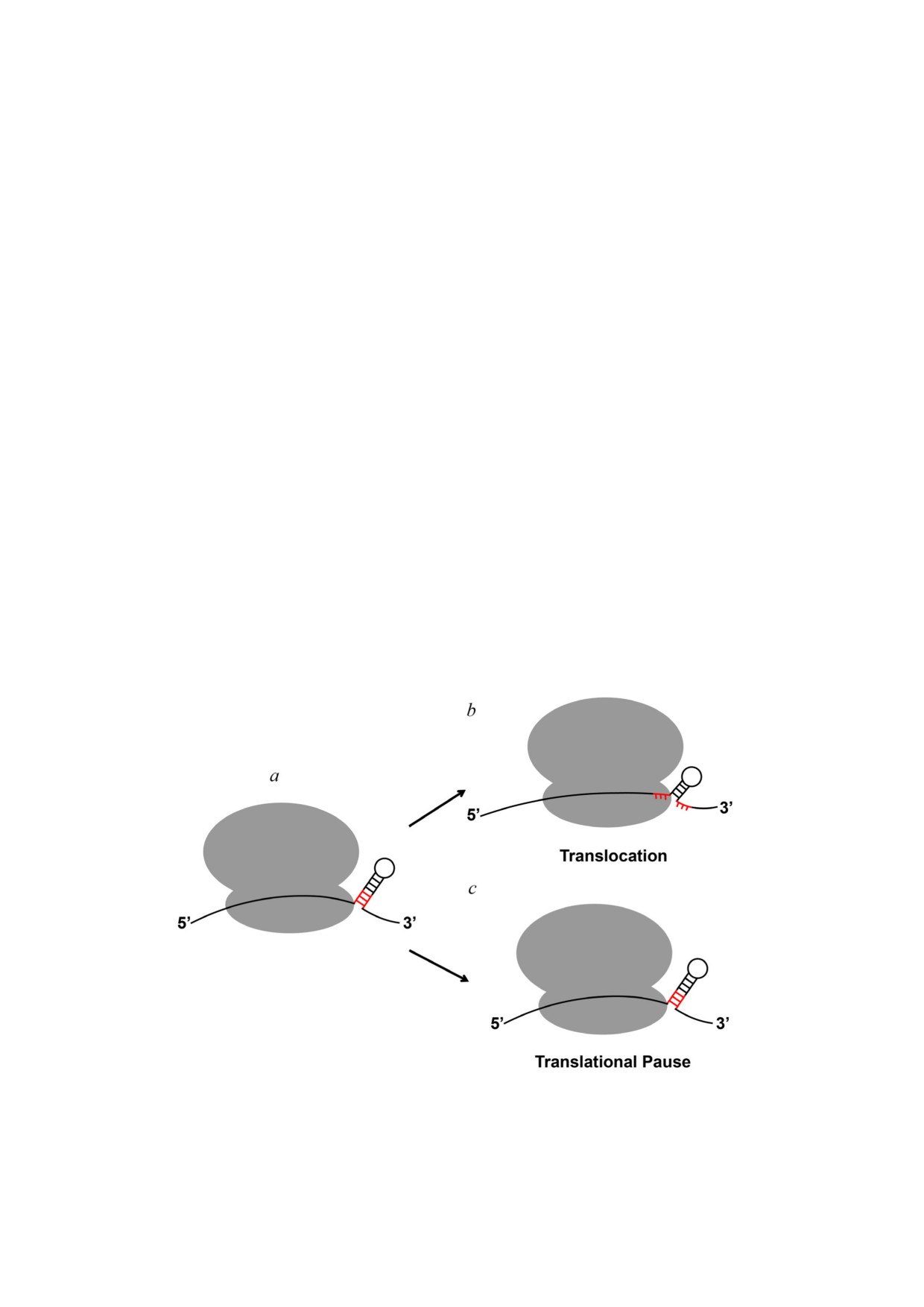
Рис. 3. Структуры «стебель-петля» в молекуле мРНК могут либо быстро разворачиваться рибосомой, либо вызывать па#
узу в трансляции. Находясь в канале входа мРНК, определённые вторичные регуляторные структуры мРНК вызывают па#
узу в её продвижении по рибосоме, несмотря на хеликазную активность рибосомы. Три пары оснований структуры «сте#
бель-петля», примыкающие к каналу входа мРНК, которые разворачиваются на этапе транслокации, показаны красным
БИОХИМИЯ том 86 вып. 8 2021
1202
БАО, ЕРМОЛЕНКО
сом (PRF), который контролирует экспрессию
показано, что при встрече с элементами вторич#
различных белков бактерий, вирусов и эукари#
ной структуры мРНК рибосома перемещается
от [112]. В частности, -1 PRF регулирует синтез
по двум альтернативным (быстрому и медленно#
ДНК#полимеразы III у бактерий [113], рецепто#
му) путям [31]. Взаимодействия структур «сте#
ра цитокинов вируса иммунодефицита челове#
бель-петля», индуцирующих сдвиг рамки счи#
ка ccr5 у высших эукариот [114], белков gag#pol
тывания, или псевдоузлов с каналом входа
ретровирусов, включая вирус иммунодефицита
мРНК могут увеличивать поток через медлен#
человека (HIV) [115], и удлинённого на С#конце
ный путь и, таким образом, снижать среднюю
полипротеина коронавирусов, в том числе
скорость транслокации рибосом [31].
SARS#CoV#2, который вызвал пандемию
Многие гениальные идеи Спирина о меха#
COVID#19 [116, 117].
низме транслокации рибосомы были подтверж#
Механизм остановки рибосомы, индуцируе#
дены за последнюю пару десятилетий успехами,
мый вторичной структурой мРНК, до конца не#
достигнутыми благодаря структурам рибосомы
ясен. Ряд опубликованных исследований, вы#
с высоким разрешением и биофизическим экс#
полненных на уровне единичных молекул,
периментам с одиночными молекулами. Тем не
предполагает, что медленное раскручивание
менее транслокация рибосомы вдоль молекулы
вторичной структуры, которое часто связывают
мРНК по#прежнему остается одним из наиболее
с трансляционной паузой, вряд ли можно свя#
интересных аспектов биосинтеза белка. Полная
зать со степенью ингибирования трансляции,
картина, воссоздающая структурные перестрой#
вызванного определенными структурами типа
ки рибосомы, EF#G и тРНК во время трансло#
«стебель-петля» молекулы мРНК [31, 118, 119].
кации, пока еще не создана. Лишь только начи#
Транслокация через три пары оснований GC
нает формироваться более полное представле#
происходит лишь в 2-3 раза медленнее, чем
ние о том, как транслокация рибосомы ремоде#
транслокация вдоль одноцепочечного кодо#
лирует вторичную структуру мРНК и модулиру#
на [31, 118, 119]. Это указывает на то, что ста#
ет взаимодействия мРНК с различными регуля#
бильность трех пар оснований, прилегающих к
торными белками. Механизмы регуляции
каналу для входа мРНК, оказывает относитель#
транслокации рибосомы с помощью вариаций
но умеренное влияние на скорость транслока#
во вторичной структуре мРНК ждут дальнейше#
ции. Напротив, было показано, что индуцирую#
го изучения.
щие сдвиг рамки считывания структуры «сте#
бель-петля» и псевдоузлы вызывают длитель#
ные паузы в рибосомах [120-127].
Финансирование. Работа лаборатории Ермо#
Похоже, что вместо того, чтобы создавать
ленко была поддержана Национальным Инсти#
простую механическую преграду на пути движе#
тутом Здоровья (гранты №№ R01GM099719 и
ния рибосомы, структуры
«стебель-петля»
R01GM132041).
мРНК индуцируют запрограммированные пау#
Благодарности. Эта статья посвящена памяти
зы движения рибосомы, специфически взаимо#
профессора Спирина, который был руководите#
действуя с ней. Недавние исследования показы#
лем дипломной работы одного из авторов статьи
вают, что структуры «стебель-петля» мРНК, ин#
(Д.Н. Ермоленко). Д.Н. Ермоленко выражает
дуцирующие сдвиг рамки считывания могут на#
благодарность Александру Сергеевичу Спирину
рушать элонгацию трансляции путём докинга
за его бесценные уроки критического мышле#
А#сайта 30S#субъединицы, препятствующего
ния и строгого подхода к проведению научных
связыванию тРНК [128]. Более того, при распо#
исследований. Авторы благодарят Грегори Бал#
ложении у входа в канал мРНК 30S#субъедини#
ларда за его комментарии, касающиеся настоя#
цы «стебли-петли» и псевдоузлы, индуцирую#
щей статьи.
щие сдвиг рамки считывания, ингибируют ско#
Конфликт интересов. Авторы заявляют об от#
рость рибосомной транслокации более чем на
сутствии конфликта интересов.
один порядок, что показано в ряде эксперимен#
Соблюдение этических норм. В статье не содер#
тов с кинетическим ансамблем и с одиночными
жатся материалы работ, выполненных кем#либо
молекулами [120, 121, 126-129]. Недавно было
из авторов, с участием людей или животных.
СПИСОК ЛИТЕРАТУРЫ
1.
Spirin, A. S. (1968) How does the ribosome work? A
2.
Spirin, A. S. (1969) A model of the functioning ribosome:
hypothesis based on the two subunit construction of the
locking and unlocking of the ribosome subparticles, Cold
ribosome, Curr. Mod. Biol., 2, 115#127, doi: 10.1016/0303#
Spring Harb. Symp. Quant. Biol., 34, 197#207, doi: 10.1101/
2647(68)90017#8.
sqb.1969.034.01.026.
БИОХИМИЯ том 86 вып. 8 2021
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
1203
3.
Spirin, A. S. (1985) Ribosomal translocation: facts and
23.
Inoue#Yokosawa, N., Ishikawa, C., and Kaziro, Y. (1974)
models, Prog. Nucleic Acid. Res. Mol. Biol., 32, 75#114,
The role of guanosine triphosphate in translocation reac#
doi: 10.1016/s0079#6603(08)60346#3.
tion catalyzed by elongation factor G, J. Biol. Chem., 249,
4.
Spirin, A. S. (2009) The ribosome as a conveying thermal
4321#4323, doi: 10.1016/S0021#9258(19)42519#2.
ratchet machine, J. Biol. Chem., 284, 21103#21119,
24.
Belitsina, N. V., Glukhova, M. A., and Spirin, A. S. (1976)
doi: 10.1074/jbc.X109.001552.
Stepwise elongation factor G#promoted elongation of poly#
5.
Finkelstein, A. V., Razin, S. V., and Spirin, A. S. (2018)
peptides on the ribosome without GTP cleavage, J. Mol.
Intersubunit mobility of the ribosome, Mol. Biol. (Mosk.),
Biol., 108, 609#613, doi: 10.1016/s0022#2836(76)80140#4.
52, 921#934, doi: 10.1134/S0026898418060083.
25.
Belitsina, N. V., Glukhova, M. A., and Spirin, A. S. (1979)
6.
Spirin, A. S. (1968) On the mechanism of ribosome func#
Elongation factor G#promoted translocation and polypep#
tion. The hypothesis of locking#unlocking of subparticles
tide elongation in ribosomes without GTP cleavage: use of
[in Russsian], Dokl. Akad. Nauk SSSR, 179, 1467#1470.
columns with matrix#bound polyuridylic acid, Methods
7.
Gavrilova, L. P., and Spirin, A. S. (1971) Stimulation of
Enzymol., 60, 761#779, doi: 10.1016/s0076#6879(79)60070#8.
“non#enzymic” translocation in ribosomes by p#
26.
Ermolenko, D. N., and Noller, H. F. (2011) mRNA
chloromercuribenzoate, FEBS Lett.,
17,
324#326,
translocation occurs during the second step of ribosomal
doi: 10.1016/0014#5793(71)80177#1.
intersubunit rotation, Nat. Struct. Mol. Biol., 18, 457#462,
8.
Gavrilova, L. P., and Spirin, A. S. (1974) “Nonenzymatic”
doi: 10.1038/nsmb.2011.
translation, Methods Enzymol., 30, 452#462, doi: 10.1016/
27.
Flis, J., Holm, M., Rundlet, E. J., Loerke, J., Hilal, T.,
0076#6879(74)30045#6.
et al. (2018) tRNA Translocation by the eukaryotic 80S
9.
Belitsina, N. V., Glukhova, M. A., and Spirin, A. S. (1975)
ribosome and the impact of GTP hydrolysis, Cell Rep., 25,
Translocation in ribosomes by attachment#detachment of
2676#2688.e7, doi: 10.1016/j.celrep.2018.11.040.
elongation factor G without GTP cleavage: evidence from
28.
Pan, D., Kirillov, S. V., and Cooperman, B. S. (2007)
a column#bound ribosome system, FEBS Lett., 54, 35#38,
Kinetically competent intermediates in the translocation
doi: 10.1016/0014#5793(75)81062#3.
step of protein synthesis, Mol. Cell,
25,
519#529,
10.
Belitsina, N. V., Tnalina, G. Z., and Spirin, A. S. (1981)
doi: 10.1016/j.molcel.2007.01.014.
Template#free ribosomal synthesis of polylysine from lysyl#
29.
Salsi, E., Farah, E., and Ermolenko, D. N. (2016) EF#G
tRNA, FEBS Lett., 131, 289#292, doi: 10.1016/0014#
activation by phosphate analogs, J. Mol. Biol., 428, 2248#
5793(81)80387#0.
2258, doi: 10.1016/j.jmb.2016.03.032.
11.
Belitsina, N. V., Tnalina, G. Z., and Spirin, A. S. (1982)
30.
Joseph, S., and Noller, H. F. (1998) EF#G#catalyzed
Template#free ribosomal synthesis of polypeptides from
translocation of anticodon stem#loop analogs of transfer
aminoacyl#tRNAs, Biosystems, 15, 233#241, doi: 10.1016/
RNA in the ribosome, EMBO J.,
17,
3478#3483,
0303#2647(82)90008#9.
doi: 10.1093/emboj/17.12.3478.
12.
Spirin, A. S. (2002) Ribosome as a molecular machine,
31.
Desai, V. P., Frank, F., Lee, A., Righini, M., Lancaster, L.,
FEBS Lett., 514, 2#10, doi: 10.1016/s0014#5793(02)02309#8.
et al. (2019) Co#temporal force and fluorescence measure#
13.
Spirin, A. S. (2004) The ribosome as an RNA#based mole#
ments reveal a ribosomal gear shift mechanism of transla#
cular machine, RNA Biol., 1, 3#9, doi: 10.4161/rna.1.1.889.
tion regulation by structured mRNAs, Mol. Cell, 75, 1007#
14.
Ling, C., and Ermolenko, D. N. (2016) Structural insights
1019.e5, doi: 10.1016/j.molcel.2019.07.024.
into ribosome translocation, Wiley Interdiscip. Rev. RNA, 7,
32.
Bretscher, M. S. (1968) Translocation in protein synthesis:
620#636, doi: 10.1002/wrna.1354.
a hybrid structure model, Nature,
218,
675#677,
15.
Noller, H. F., Lancaster, L., Zhou, J., and Mohan, S.
doi: 10.1038/218675a0.
(2017) The ribosome moves: RNA mechanics and translo#
33.
Spirin, A. S., Baranov, V. I., Polubesov, G. S., Serdyuk,
cation, Nat. Struct. Mol. Biol.,
24,
1021#1027,
I. N., and May, R. P. (1987) Translocation makes the ribo#
doi: 10.1038/nsmb.3505.
some less compact, J. Mol. Biol.,
194,
119#126,
16.
Rodnina, M. V., Peske, F., Peng, B. Z., Belardinelli, R.,
doi: 10.1016/0022#2836(87)90720#0.
and Wintermeyer, W. (2019) Converting GTP hydrolysis
34.
Serdyuk, I., Baranov, V., Tsalkova, T., Gulyamova, D.,
into motion: versatile translational elongation factor G,
Pavlov, M., et al. (1992) Structural dynamics of translating
Biol. Chem., 401, 131#142, doi: 10.1515/hsz#2019#0313.
ribosomes, Biochimie, 74, 299#306, doi: 10.1016/0300#
17.
Mohan, S., Donohue, J. P., and Noller, H. F. (2014)
9084(92)90107#p.
Molecular mechanics of 30S subunit head rotation, Proc.
35.
Frank, J., and Gonzalez, R. L., Jr. (2010) Structure and
Natl. Acad. Sci. USA, 111, 13325#13330, doi: 10.1073/
dynamics of a processive Brownian motor: the translating
pnas.1413731111.
ribosome, Annu. Rev. Biochem., 79, 381#412, doi: 10.1146/
18.
Gavrilova, L. P., Kostiashkina, O. E., Koteliansky, V. E.,
annurev#biochem#060408#173330.
Rutkevitch, N. M., and Spirin, A. S. (1976) Factor#free
36.
Moazed, D., and Noller, H. F. (1989) Intermediate states
(“non#enzymic”) and factor#dependent systems of transla#
in the movement of transfer RNA in the ribosome, Nature,
tion of polyuridylic acid by Escherichia coli ribosomes,
342, 142#148, doi: 10.1038/342142a0.
J. Mol. Biol.,
101,
537#552, doi:
10.1016/0022#
37.
Frank, J., and Agrawal, R. K. (2000) A ratchet#like inter#
2836(76)90243#6.
subunit reorganization of the ribosome during transloca#
19.
Rodnina, M. V., Savelsbergh, A., Katunin, V. I., and
tion, Nature, 406, 318#322, doi: 10.1038/35018597.
Wintermeyer, W. (1997) Hydrolysis of GTP by elongation
38.
Valle, M., Zavialov, A., Sengupta, J., Rawat, U.,
factor G drives tRNA movement on the ribosome, Nature,
Ehrenberg, M., and Frank, J. (2003) Locking and unlock#
385, 37#41, doi: 10.1038/385037a0.
ing of ribosomal motions, Cell,
114,
123#134,
20.
Fredrick, K., and Noller, H. F. (2003) Catalysis of riboso#
doi: 10.1016/S0092#8674(03)00476#8.
mal translocation by sparsomycin, Science, 300, 1159#
39.
Korostelev, A., Ermolenko, D. N., and Noller, H. F. (2008)
1162, doi: 10.1126/science.1084571.
Structural dynamics of the ribosome, Curr. Opin. Chem.
21.
Parmeggiani, A., and Sander, G. (1981) Properties and
Biol., 12, 674#683, doi: 10.1016/j.cbpa.2008.08.037.
regulation of the GTPase activities of elongation factors Tu
40.
Ermolenko, D. N., Majumdar, Z. K., Hickerson, R. P.,
and G, and of initiation factor 2, Mol. Cell. Biochem., 35,
Spiegel, P. C., Clegg, R. M., and Noller, H. F. (2007)
129#158, doi: 10.1007/BF02357085.
Observation of intersubunit movement of the ribosome in
22.
Moazed, D., Robertson, J. M., and Noller, H. F. (1988)
solution using FRET, J. Mol. Biol., 370, 530#540,
Interaction of elongation factors EF#G and EF#Tu with a
doi: 10.1016/j.jmb.2007.04.042.
conserved loop in 23S RNA, Nature, 334, 362#364,
41.
Ermolenko, D. N., Spiegel, P. C., Majumdar, Z. K.,
doi: 10.1038/334362a0.
Hickerson, R. P., Clegg, R. M., and Noller, H. F. (2007)
БИОХИМИЯ том 86 вып. 8 2021
1204
БАО, ЕРМОЛЕНКО
The antibiotic viomycin traps the ribosome in an interme#
tor G trapped in the posttranslocational state, Science, 326,
diate state of translocation, Nat. Struct. Mol. Biol., 14, 493#
694#699, doi: 10.1126/science.1179709.
497, doi: 10.1038/nsmb1243.
58.
Khade, P. K., and Joseph, S. (2011) Messenger RNA inter#
42.
Agirrezabala, X., Lei, J., Brunelle, J. L., Ortiz#Meoz, R. F.,
actions in the decoding center control the rate of translo#
Green, R., and Frank, J. (2008) Visualization of the hybrid
cation, Nat. Struct. Mol. Biol.,
18,
1300#1302,
state of tRNA binding promoted by spontaneous ratcheting
doi: 10.1038/nsmb.2140.
of the ribosome, Mol. Cell, 32, 190#197, doi: 10.1016/
59.
Liu, G., Song, G., Zhang, D., Zhang, D., Li, Z., et al.
j.molcel.2008.10.001.
(2014) EF#G catalyzes tRNA translocation by disrupting
43.
Julian, P., Konevega, A. L., Scheres, S. H., Lazaro, M.,
interactions between decoding center and codon#anti#
Gil, D., et al. (2008) Structure of ratcheted ribosomes with
codon duplex, Nat. Struct. Mol. Biol., 21, 817#824,
tRNAs in hybrid states, Proc. Natl. Acad. Sci. USA, 105,
doi: 10.1038/nsmb.2869.
16924#16927, doi: 10.1073/pnas.0809587105.
60.
Abeyrathne, P. D., Koh, C. S., Grant, T., Grigorieff, N.,
44.
Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Jr., Puglisi,
and Korostelev, A. A. (2016) Ensemble cryo#EM uncovers
J. D., and Chu, S. (2004) tRNA dynamics on the ribosome
inchworm#like translocation of a viral IRES through the
during translation, Proc. Natl. Acad. Sci. USA, 101, 12893#
ribosome, Elife, 5, e14874, doi: 10.7554/eLife.14874.
12898, doi: 10.1073/pnas.0403884101.
61.
Taylor, D. J., Nilsson, J., Merrill, A. R., Andersen, G. R.,
45.
Cornish, P. V., Ermolenko, D. N., Noller, H. F., and
Nissen, P., and Frank, J. (2007) Structures of modified
Ha, T. (2008) Spontaneous intersubunit rotation in single
eEF2 80S ribosome complexes reveal the role of GTP
ribosomes, Mol. Cell, 30, 578#588, doi: 10.1016/j.molcel.
hydrolysis in translocation, EMBO J., 26, 2421#2431,
2008.05.004.
doi: 10.1038/sj.emboj.7601677.
46.
Fei, J., Kosuri, P., MacDougall, D. D., and Gonzalez,
62.
Pestka, S. (1968) Studies on the formation of transfer
R. L., Jr. (2008) Coupling of ribosomal L1 stalk and tRNA
ribonucleic acid#ribosome complexes. 3. The formation of
dynamics during translation elongation, Mol. Cell, 30, 348#
peptide bonds by ribosomes in the absence of supernatant
359, doi: 10.1016/j.molcel.2008.03.012.
enzymes, J. Biol. Chem., 243, 2810#2820, doi: 10.1016/
47.
Marshall, R. A., Aitken, C. E., and Puglisi, J. D. (2009)
S0021#9258(18)93445#9.
GTP hydrolysis by IF2 guides progression of the ribosome
63.
Gavrilova, L. P., Koteliansky, V. E., and Spirin, A. S. (1974)
into elongation, Mol. Cell, 35, 37#47, doi: 10.1016/j.molcel.
Ribosomal protein S12 and ‘non#enzymatic’ translocation,
2009.06.008.
FEBS Lett., 45, 324#328, doi: 10.1016/0014#5793(74)80872#0.
48.
Spahn, C. M., Gomez#Lorenzo, M. G., Grassucci, R. A.,
64.
Gavrilova, L. P., and Spirin, A. S. (1974) Interaction of
Jorgensen, R., Andersen, G. R., et al. (2004) Domain
SH#reagents with the ribosomal 30 S subparticle and ‘non#
movements of elongation factor eEF2 and the eukaryotic
enzymatic’ translocation, FEBS Lett.,
39,
13#16,
80S ribosome facilitate tRNA translocation, EMBO J., 23,
doi: 10.1016/0014#5793(74)80005#0.
1008#1019, doi: 10.1038/sj.emboj.7600102.
65.
Cukras, A. R., Southworth, D. R., Brunelle, J. L., Culver,
49.
Schuwirth, B. S., Borovinskaya, M. A., Hau, C. W.,
G. M., and Green, R. (2003) Ribosomal proteins S12 and
Zhang, W., Vila#Sanjurjo, A., et al. (2005) Structures of the
S13 function as control elements for translocation of the
bacterial ribosome at 3.5 Å resolution, Science, 310, 827#
mRNA:tRNA complex, Mol. Cell,
12,
321#328,
834, doi: 10.1126/science.1117230.
doi: 10.1016/s1097#2765(03)00275#2.
50.
Ramrath, D. J., Lancaster, L., Sprink, T., Mielke, T.,
66.
Ermolenko, D. N., Cornish, P. V., Ha, T., and Noller, H. F.
Loerke, J., et al. (2013) Visualization of two transfer RNAs
(2013) Antibiotics that bind to the A site of the large ribo#
trapped in transit during elongation factor G#mediated
somal subunit can induce mRNA translocation, RNA, 19,
translocation, Proc. Natl. Acad. Sci. USA, 110, 20964#
158#166, doi: 10.1261/rna.035964.112.
20969, doi: 10.1073/pnas.1320387110.
67.
Liu, T., Kaplan, A., Alexander, L., Yan, S., Wen, J. D.,
51.
Zhou, J., Lancaster, L., Donohue, J. P., and Noller, H. F.
et al. (2014) Direct measurement of the mechanical work
(2014) How the ribosome hands the A#site tRNA to the P
during translocation by the ribosome, Elife, 3, e03406,
site during EF#G#catalyzed translocation, Science, 345,
doi: 10.7554/eLife.03406.
1188#1191, doi: 10.1126/science.1255030.
68.
Howard, J. (2006) Protein power strokes, Curr. Biol., 16,
52.
Martemyanov, K. A., and Gudkov, A. T. (1999) Domain IV
R517#519, doi: 10.1016/j.cub.2006.06.045.
of elongation factor G from Thermus thermophilus is
69.
Hwang, W., and Karplus, M. (2019) Structural basis for
strictly required for translocation, FEBS Lett., 452, 155#
power stroke vs. Brownian ratchet mechanisms of motor
159, doi: 10.1016/S0014#5793(99)00635#3.
proteins, Proc. Natl. Acad. Sci. USA, 116, 19777#19785,
53.
Niblett, D., Nelson, C., Leung, C. S., Rexroad, G.,
doi: 10.1073/pnas.1818589116.
Cozy, J., et al. (2021) Mutations in domain IV of elonga#
70.
Peske, F., Matassova, N. B., Savelsbergh, A., Rodnina,
tion factor EF#G confer#1 frameshifting, RNA, 27, 40#53,
M. V., and Wintermeyer, W. (2000) Conformationally
doi: 10.1261/rna.077339.120.
restricted elongation factor G retains GTPase activity but is
54.
Peng, B. Z., Bock, L. V., Belardinelli, R., Peske, F.,
inactive in translocation on the ribosome, Mol. Cell, 6, 501#
Grubmuller, H., and Rodnina, M. V. (2019) Active role of
505, doi: 10.1016/S1097#2765(00)00049#6.
elongation factor G in maintaining the mRNA reading
71.
Chen, C., Cui, X., Beausang, J. F., Zhang, H., Farrell, I.,
frame during translation, Sci. Adv.,
5, eaax8030,
et al. (2016) Elongation factor G initiates translocation
doi: 10.1126/sciadv.aax8030.
through a power stroke, Proc. Natl. Acad. Sci. USA, 113,
55.
Brilot, A. F., Korostelev, A. A., Ermolenko, D. N., and
7515#7520, doi: 10.1073/pnas.1602668113.
Grigorieff, N. (2013) Structure of the ribosome with elon#
72.
Ratje, A. H., Loerke, J., Mikolajka, A., Brunner, M.,
gation factor G trapped in the pretranslocation state, Proc.
Hildebrand, P. W., et al. (2010) Head swivel on the ribo#
Natl. Acad. Sci. USA, 110, 20994#20999, doi: 10.1073/
some facilitates translocation by means of intra#subunit
pnas.1311423110.
tRNA hybrid sites, Nature, 468, 713#716, doi: 10.1038/
56.
Salsi, E., Farah, E., Dann, J., and Ermolenko, D. N.
nature09547.
(2014) Following movement of domain IV of elongation
73.
Ryazanov, A. G., Natapov, P. G., Shestakova, E. A.,
factor G during ribosomal translocation, Proc. Natl. Acad.
Severin, F. F., and Spirin, A. S. (1988) Phosphorylation of
Sci. USA,
111,
15060#15065, doi:
10.1073/pnas.
the elongation factor 2: the fifth Ca2+/calmodulin#depen#
1410873111.
dent system of protein phosphorylation, Biochimie, 70,
57.
Gao, Y. G., Selmer, M., Dunham, C. M.,
619#626, doi: 10.1016/0300#9084(88)90245#3.
Weixlbaumer, A., Kelley, A. C., and Ramakrishnan, V.
74.
Ryazanov, A. G., Shestakova, E. A., and Natapov, P. G.
(2009) The structure of the ribosome with elongation fac#
(1988) Phosphorylation of elongation factor 2 by EF#2
БИОХИМИЯ том 86 вып. 8 2021
РИБОСОМА КАК ТРАНСЛОКАЗА И ХЕЛИКАЗА
1205
kinase affects rate of translation, Nature, 334, 170#173,
91. Lu, Z. J., and Mathews, D. H. (2008) Efficient siRNA
doi: 10.1038/334170a0.
selection using hybridization thermodynamics, Nucleic
75.
Ryazanov, A. G., Rudkin, B. B., and Spirin, A. S. (1991)
Acids Res., 36, 640#647, doi: 10.1093/nar/gkm920.
Regulation of protein synthesis at the elongation stage.
92. Lu, Z. J., and Mathews, D. H. (2008) Fundamental differ#
New insights into the control of gene expression in eukary#
ences in the equilibrium considerations for siRNA and
otes, FEBS Lett., 285, 170#175, doi: 10.1016/0014#
antisense oligodeoxynucleotide design, Nucleic Acids Res.,
5793(91)80798#8.
36, 3738#3745, doi: 10.1093/nar/gkn266.
76.
Davydova, E. K., and Ovchinnikov, L. P. (1990) ADP#ribo#
93. Tafer, H., Ameres, S. L., Obernosterer, G., Gebeshuber,
sylated elongation factor 2 (ADP#ribosyl#EF#2) is unable
C. A., Schroeder, R., et al. (2008) The impact of target site
to promote translocation within the ribosome, FEBS Lett.,
accessibility on the design of effective siRNAs,
261, 350#352, doi: 10.1016/0014#5793(90)80589#B.
Nat. Biotechnol., 26, 578#583, doi: 10.1038/nbt1404.
77.
Ermolenko, D. N., and Mathews, D. H. (2021) Making
94. Li, X., Quon, G., Lipshitz, H. D., and Morris, Q. (2010)
ends meet: new functions of mRNA secondary structure,
Predicting in vivo binding sites of RNA#binding proteins
Wiley Interdiscip. Rev. RNA, 12, e1611, doi: 10.1002/
using mRNA secondary structure, RNA, 16, 1096#1107,
wrna.1611.
doi: 10.1261/rna.2017210.
78.
Lai, W. C., Kayedkhordeh, M., Cornell, E. V., Farah, E.,
95. Li, X., Kazan, H., Lipshitz, H. D., and Morris, Q. D.
Bellaousov, S., et al. (2018) mRNAs and lncRNAs intrinsi#
(2014) Finding the target sites of RNA#binding proteins,
cally form secondary structures with short end#to#end dis#
Wiley Interdiscip. Rev. RNA, 5, 111#130, doi: 10.1002/
tances, Nat. Commun., 9, 4328, doi: 10.1038/s41467#018#
wrna.1201.
06792#z.
96. Takyar, S., Hickerson, R. P., and Noller, H. F. (2005)
79.
Ding, Y., Tang, Y., Kwok, C. K., Zhang, Y., Bevilacqua,
mRNA helicase activity of the ribosome, Cell, 120, 49#58,
P. C., and Assmann, S. M. (2014) In vivo genome#wide
doi: 10.1016/j.cell.2004.11.042.
profiling of RNA secondary structure reveals novel regula#
97. Wen, J. D., Lancaster, L., Hodges, C., Zeri, A. C.,
tory features, Nature,
505,
696#700, doi:
10.1038/
Yoshimura, S. H., et al. (2008) Following translation by
nature12756.
single ribosomes one codon at a time, Nature, 452, 598#
80.
Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., and
603, doi: 10.1038/nature06716.
Weissman, J. S. (2014) Genome#wide probing of RNA
98. Yusupova, G. Z., Yusupov, M. M., Cate, J. H., and Noller,
structure reveals active unfolding of mRNA structures
H. F. (2001) The path of messenger RNA through the ribo#
in vivo, Nature,
505,
701#705, doi:
10.1038/
some, Cell,
106,
233#241, doi:
10.1016/s0092#
nature12894.
8674(01)00435#4.
81.
Aw, J. G., Shen, Y., Wilm, A., Sun, M., Lim, X. N., et al.
99. Balakin, A., Skripkin, E., Shatsky, I., and Bogdanov, A.
(2016) In vivo mapping of eukaryotic RNA interactomes
(1990) Transition of the mRNA sequence downstream
reveals principles of higher#order organization and regula#
from the initiation codon into a single#stranded conforma#
tion, Mol. Cell, 62, 603#617, doi: 10.1016/j.molcel.
tion is strongly promoted by binding of the initiator tRNA,
2016.04.028.
Biochim. Biophys. Acta, 1050, 119#123, doi: 10.1016/0167#
82.
Lu, Z., Zhang, Q. C., Lee, B., Flynn, R. A., Smith, M. A.,
4781(90)90151#q.
et al. (2016) RNA Duplex map in living cells reveals high#
100. De Smit, M. H., and van Duin, J. (1990) Secondary struc#
er#order transcriptome structure, Cell, 165, 1267#1279,
ture of the ribosome binding site determines translational
doi: 10.1016/j.cell.2016.04.028.
efficiency: a quantitative analysis, Proc. Natl. Acad. Sci.
83.
Sharma, E., Sterne#Weiler, T., O’Hanlon, D., and
USA, 87, 7668#7672, doi: 10.1073/pnas.87.19.7668.
Blencowe, B. J. (2016) Global mapping of human
101. Poot, R. A., Tsareva, N. V., Boni, I. V., and van Duin, J.
RNA-RNA interactions, Mol. Cell,
62,
618#626,
(1997) RNA folding kinetics regulates translation of phage
doi: 10.1016/j.molcel.2016.04.030.
MS2 maturation gene, Proc. Natl. Acad. Sci. USA, 94,
84.
Ziv, O., Gabryelska, M. M., Lun, A. T. L., Gebert, L. F. R.,
10110#10115, doi: 10.1073/pnas.94.19.10110.
Sheu#Gruttadauria, J., et al. (2018) COMRADES deter#
102. Del Campo, C., Bartholomaus, A., Fedyunin, I., and
mines in vivo RNA structures and interactions,
Ignatova, Z. (2015) Secondary Structure across the bacter#
Nat. Methods, 15, 785#788, doi: 10.1038/s41592#018#
ial transcriptome reveals versatile roles in mRNA regula#
0121#0.
tion and function, PLoS Genet.,
11, e1005613,
85.
Roth, A., and Breaker, R. R. (2009) The structural and
doi: 10.1371/journal.pgen.1005613.
functional diversity of metabolite#binding riboswitches,
103. Khong, A., and Parker, R. (2020) The landscape of eukary#
Annu. Rev. Biochem., 78, 305#334, doi: 10.1146/annurev.
otic mRNPs, RNA, 26, 229#239, doi: 10.1261/rna.
biochem.78.070507.135656.
073601.119.
86.
Giedroc, D. P., and Cornish, P. V. (2009) Frameshifting
104. Adivarahan, S., Livingston, N., Nicholson, B.,
RNA pseudoknots: structure and mechanism, Virus Res.,
Rahman, S., Wu, B., Rissland, O. S., and Zenklusen, D.
139, 193#208, doi: 10.1016/j.virusres.2008.06.008.
(2018) Spatial organization of single mRNPs at different
87.
Mauger, D. M., Siegfried, N. A., and Weeks, K. M. (2013)
stages of the gene expression pathway, Mol. Cell, 72, 727#
The genetic code as expressed through relationships
738.e5, doi: 10.1016/j.molcel.2018.10.010.
between mRNA structure and protein function,
105. Khong, A., and Parker, R. (2018) mRNP architecture in
FEBS Lett.,
587,
1180#1188, doi:
10.1016/j.febslet.
translating and stress conditions reveals an ordered path#
2013.03.002.
way of mRNP compaction, J. Cell Biol., 217, 4124#4140,
88.
Leipuviene, R., and Theil, E. C. (2007) The family of iron
doi: 10.1083/jcb.201806183.
responsive RNA structures regulated by changes in cellular
106. Maquat, L. E., Tarn, W. Y., and Isken, O. (2010) The pio#
iron and oxygen, Cell. Mol. Life Sci., 64, 2945#2955,
neer round of translation: features and functions, Cell, 142,
doi: 10.1007/s00018#007#7198#4.
368#374, doi: 10.1016/j.cell.2010.07.022.
89.
Simon, A. E., and Miller, W. A. (2013) 3′ cap#independent
107. Metkar, M., Ozadam, H., Lajoie, B. R., Imakaev, M.,
translation enhancers of plant viruses, Annu. Rev.
Mirny, L. A., et al. (2018) Higher#order organization prin#
Microbiol.,
67,
21#42, doi:
10.1146/annurev#micro#
ciples of pre#translational mRNPs, Mol. Cell, 72, 715#
092412#155609.
726.3, doi: 10.1016/j.molcel.2018.09.012.
90.
Shao, Y., Chan, C. Y., Maliyekkel, A., Lawrence, C. E.,
108. Cetin, B., Song, G. J., and O’Leary, S. E. (2020) Hetero#
Roninson, I. B., and Ding, Y. (2007) Effect of target sec#
geneous dynamics of protein-RNA interactions across tran#
ondary structure on RNAi efficiency, RNA, 13, 1631#1640,
scriptome#derived messenger RNA populations, J. Am. Chem.
doi: 10.1261/rna.546207.
Soc., 142, 21249#21253, doi: 10.1021/jacs.0c09841.
БИОХИМИЯ том 86 вып. 8 2021
1206
БАО, ЕРМОЛЕНКО
109. Yan, S., Wen, J. D., Bustamante, C., and Tinoco, I., Jr.
ribosomes through mRNA secondary structures, Nat.
(2015) Ribosome excursions during mRNA translocation
Struct. Mol. Biol., 20, 582#588, doi: 10.1038/nsmb.2544.
mediate broad branching of frameshift pathways, Cell, 160,
120. Chen, J., Petrov, A., Johansson, M., Tsai, A., O’Leary,
870#881, doi: 10.1016/j.cell.2015.02.003.
S. E., and Puglisi, J. D. (2014) Dynamic pathways of #1
110. Doma, M. K., and Parker, R. (2006) Endonucleolytic cleav#
translational frameshifting, Nature,
512,
328#332,
age of eukaryotic mRNAs with stalls in translation elonga#
doi: 10.1038/nature13428.
tion, Nature, 440, 561#564, doi: 10.1038/nature04530.
121. Kim, H. K., Liu, F., Fei, J., Bustamante, C., Gonzalez,
111.
Young, J. C., and Andrews, D. W. (1996) The signal recog#
R. L., Jr., and Tinoco, I., Jr. (2014) A frameshifting stimu#
nition particle receptor alpha subunit assembles co#transla#
latory stem loop destabilizes the hybrid state and impedes
tionally on the endoplasmic reticulum membrane during
ribosomal translocation, Proc. Natl. Acad. Sci. USA, 111,
an mRNA#encoded translation pause in vitro, EMBO J.,
5538#5543, doi: 10.1073/pnas.1403457111.
15, 172#181.
122. Tu, C., Tzeng, T. H., and Bruenn, J. A. (1992) Ribosomal
112. Caliskan, N., Peske, F., and Rodnina, M. V.
(2015)
movement impeded at a pseudoknot required for
Changed in translation: mRNA recoding by #1 pro#
frameshifting, Proc. Natl. Acad. Sci. USA, 89, 8636#8640,
grammed ribosomal frameshifting, Trends Biochem. Sci.,
doi: 10.1073/pnas.89.18.8636.
40, 265#274, doi: 10.1016/j.tibs.2015.03.006.
123. Somogyi, P., Jenner, A. J., Brierley, I., and Inglis, S. C.
113. Tsuchihashi, Z., and Kornberg, A. (1990) Translational
(1993) Ribosomal pausing during translation of an RNA
frameshifting generates the gamma subunit of DNA poly#
pseudoknot, Mol. Cell. Biol., 13, 6931#6940, doi: 10.1128/
merase III holoenzyme, Proc. Natl. Acad. Sci. USA, 87,
mcb.13.11.6931#6940.1993.
2516#2520, doi: 10.1073/pnas.87.7.2516.
124. Lopinski, J. D., Dinman, J. D., and Bruenn, J. A. (2000)
114. Belew, A. T., Meskauskas, A., Musalgaonkar, S., Advani,
Kinetics of ribosomal pausing during programmed #1
V. M., Sulima, S. O., et al. (2014) Ribosomal frameshifting
translational frameshifting, Mol. Cell. Biol., 20, 1095#1103,
in the CCR5 mRNA is regulated by miRNAs and the
doi: 10.1128/mcb.20.4.1095#1103.2000.
NMD pathway, Nature, 512, 265#269, doi: 10.1038/
125. Kontos, H., Napthine, S., and Brierley, I.
(2001)
nature13429.
Ribosomal pausing at a frameshifter RNA pseudoknot is
115. Jacks, T., Power, M. D., Masiarz, F. R., Luciw, P. A., Barr,
sensitive to reading phase but shows little correlation with
P. J., and Varmus, H. E. (1988) Characterization of riboso#
frameshift efficiency, Mol. Cell. Biol., 21, 8657#8670,
mal frameshifting in HIV#1 gag#pol expression, Nature,
doi: 10.1128/MCB.21.24.8657#8670.2001.
331, 280#283, doi: 10.1038/331280a0.
126. Caliskan, N., Katunin, V. I., Belardinelli, R., Peske, F.,
116. Kelly, J. A., Olson, A. N., Neupane, K., Munshi, S., San
and Rodnina, M. V. (2014) Programmed #1 frameshifting
Emeterio, J., et al. (2020) Structural and functional con#
by kinetic partitioning during impeded translocation, Cell,
servation of the programmed #1 ribosomal frameshift signal
157, 1619#1631, doi: 10.1016/j.cell.2014.04.041.
of SARS coronavirus 2 (SARS#CoV#2), J. Biol. Chem.,
127. Caliskan, N., Wohlgemuth, I., Korniy, N., Pearson, M.,
295, 10741#10748, doi: 10.1074/jbc.AC120.013449.
Peske, F., and Rodnina, M. V. (2017) Conditional switch
117. Kelly, J. A., Woodside, M. T., and Dinman, J. D. (2021)
between frameshifting regimes upon translation of dnaX
Programmed #1 ribosomal frameshifting in coronaviruses:
mRNA, Mol. Cell, 66, 558#567.e554, doi: 10.1016/
a therapeutic target, Virology, 554, 75#82, doi: 10.1016/
j.molcel.2017.04.023.
j.virol.2020.12.010.
128. Bao, C., Loerch, S., Ling, C., Korostelev, A. A.,
118. Qu, X., Wen, J. D., Lancaster, L., Noller, H. F.,
Grigorieff, N., and Ermolenko, D. N. (2020) mRNA
Bustamante, C., and Tinoco, I., Jr. (2011) The ribosome
stem#loops can pause the ribosome by hindering A#site
uses two active mechanisms to unwind messenger RNA
tRNA binding, Elife, 9, e55799, doi: 10.7554/eLife.
during translation, Nature, 475, 118#121, doi: 10.1038/
55799.
nature10126.
129. Choi, J., O’Loughlin, S., Atkins, J. F., and Puglisi, J. D.
119. Chen, C., Zhang, H., Broitman, S. L., Reiche, M.,
(2020) The energy landscape of #1 ribosomal frameshifting,
Farrell, I., et al. (2013) Dynamics of translation by single
Sci. Adv., 6, eaax6969, doi: 10.1126/sciadv.aax6969.
RIBOSOME AS A TRANSLOCASE AND HELICASE
Review
C. Bao and D. N. Ermolenko*
Department of Biochemistry & Biophysics, School of Medicine and Dentistry
and Center for RNA Biology, University of Rochester, Rochester, NY, USA;
e mail: Chen_Bao@URMC.Rochester.edu; dmitri_ermolenko@urmc.rochester.edu
During protein synthesis, ribosome moves along mRNA to decode one codon after the other. Ribosome translocation
is induced by a universally conserved protein, elongation factor G (EF#G) in bacteria and elongation factor 2 (EF#2)
in eukaryotes. EF#G#induced translocation results in unwinding of the intramolecular secondary structures of mRNA
by three base pairs at a time that renders the translating ribosome a processive helicase. Professor Alexander
Sergeevich Spirin has made numerous seminal contributions to understanding the molecular mechanism of translo#
cation. Here, we review Spirin’s insights into the ribosomal translocation and recent advances in the field that
stemmed from Spirin’s pioneering work. We also discuss key remaining challenges in studies of translocase and heli#
case activities of the ribosome.
Keywords: ribosome, translocation, helicase, power stroke, Brownian ratchet
БИОХИМИЯ том 86 вып. 8 2021