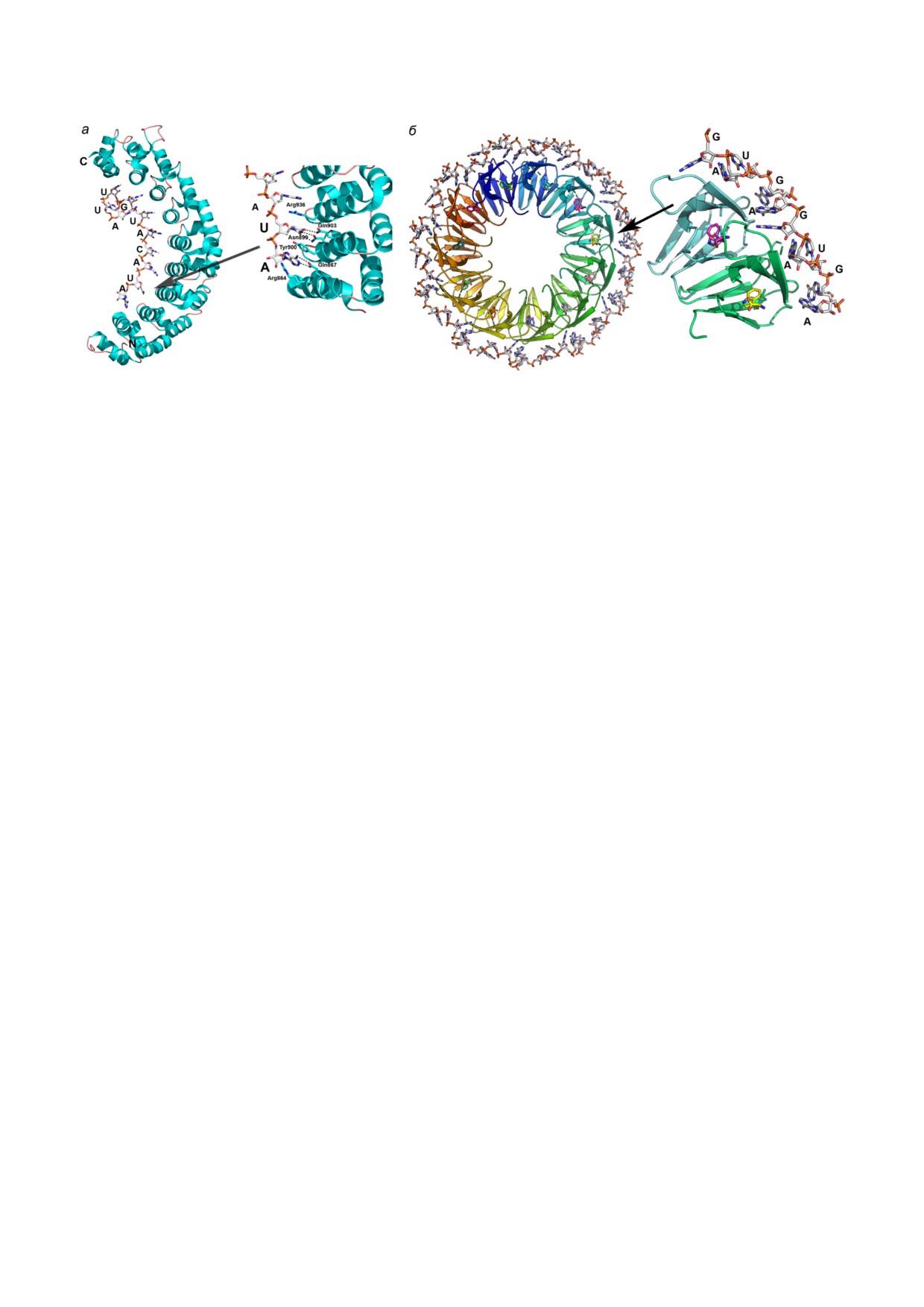БИОХИМИЯ, 2021, том 86, вып. 8, с. 1231 - 1248
УДК 548.73
ОСОБЕННОСТИ ВЗАИМОДЕЙСТВИЯ БЕЛКОВ
С ОДНОЦЕПОЧЕЧНЫМИ И ДВУЦЕПОЧЕЧНЫМИ РНК
Обзор
© 2021
А.Д. Никулин
Институт белка Российской академии наук, 142290 Пущино,
Московская обл., Россия; электронная почта: nikulin@vega.protres.ru
Поступила в редакцию 29.04.2021
После доработки 01.06.2021
Принята к публикации 01.06.2021
В обзоре на примерах ряда структур РНК белковых комплексов обсуждается отличие специфического взаи
модействия (узнавания) белков с одноцепочечными и двуцепочечными РНК. Показано, что в случае одно
цепочечных РНК белки контактируют с основаниями РНК, что позволяет узнавать определенную последо
вательность нуклеотидов. В случае двуцепочечных РНК формирование таких контактов затруднено, и бел
ки узнают уникальную конформацию пространственной структуры РНК, взаимодействуя преимуществен
но с сахарофосфатным остовом РНК.
КЛЮЧЕВЫЕ СЛОВА: РНК белковые взаимодействия, одноцепочечная РНК, двуцепочечная РНК, РНП
мотив, рибосомные белки.
DOI: 10.31857/S0320972521080133
ВВЕДЕНИЕ
зором РНК белковых комплексов Corley et al.
[9]. Специфически узнаваемые белками участки
РНК связывающие белки участвуют в целом
РНК можно разделить на два типа - одноцепо
ряде важных для жизни клеток процессов, таких
чечные участки, в которых большая часть нук
как регуляция трансляции, созревание, транс
леотидов не образуют пары с комплементарны
порт и локализация РНК, сплайсинг и деграда
ми им основаниями и открыта для контактов с
ция мРНК и др. Исследование структурных осо
атомами белка, и двуцепочечные участки, в ко
бенностей узнаваемых белками элементов РНК
торых такие пары формируются, и атомы осно
и определение структур специфических РНК
ваний малодоступны для внешних контактов.
белковых комплексов позволяет пролить свет на
Одноцепочечные РНК (оцРНК), как правило,
то, как эти процессы реализуются, а впослед
представляют собой функционально активные
ствии - целенаправленно и осознанно воздей
участки молекул РНК, например шпильки с
ствовать на них. В Институте белка РАН струк
петлёй от 3 до 8 нуклеотидов (stem loop). Кроме
турно функциональные исследования РНК
экспонированных одноцепочечных участков,
белковых комплексов ведутся уже более 30 лет.
молекулы РНК содержат большое количество
Начало этим работам положили исследования
сложных пространственных элементов, таких
сотрудниками Института целого ряда рибосом
как сочленение трёх спиралей РНК (three way
ных белков в комплексах с фрагментами рибо
junction), единичные выпетливания нуклеоти
сомных РНК при исследовании структуры бак
дов (bulges), псевдоузлы (pseudoknots) и др. Они
териальных рибосом [1-7].
имеют сложную трёхмерную укладку, но пос
Вопрос классификации РНК белковых взаи
кольку большая часть нуклеотидов в них нахо
модействий неоднократно рассматривался в ли
дится в спаренном состоянии, то часто такие об
тературе, начиная с классического обзора
ласти РНК называют двуцепочечной РНК
Draper [8] в 1999 году и заканчивая недавним об
(дцРНК). Разнообразие укладок пространствен
ных элементов дцРНК приводит к тому, что бел
ки, узнающие такие участки, подстраиваются
Принятые сокращения: оцРНК - одноцепочечные
РНК, дцРНК - двуцепочечные РНК, dsRBD - двуцепо
под них и, как правило, имеют уникальную
чечно РНК связывающий домен.
структуру.
1231
1232
НИКУЛИН
ОСНОВНЫЕ СТРУКТУРНЫЕ МОТИВЫ
лот, а общая длина РНП домена составляет
БЕЛКОВ, УЗНАЮЩИХ
80-90 а.о. и включают в себя четырёхтяжевый
ОДНОЦЕПОЧЕЧНЫЕ УЧАСТКИ РНК
антипараллельный β лист с двумя α спиралями,
расположенными с одной стороны β листа.
Белки с РНП'доменом. Белки с РНП доме
Аминокислотные остатки мотива РНП1 распо
ном (рибонуклеопротеиновый домен, синони
лагаются на тяже β3, а РНП2 - на тяже β1.
мы: РНК связывающий домен, РНК узнающий
Определение структур более чем 30 белков,
мотив; англ. ribonucleoprotein domain, RNP
содержащих РНП домен (РНП белки), в комп
domain, RNA binding domain, RBD, RNA recog
лексах с фрагментами РНК позволило детально
nition motif, RRM) являются, пожалуй, наиболее
проанализировать возможные нюансы взаимо
распространенными белками, узнающими
действия представителей этого класса белков с
оцРНК. Имеются данные, что не менее
РНК и детализировать принципы идентифика
0,5-1,0% генов человека содержат нуклеотид
ции близких по последовательности РНК
ные последовательности, соответствующие
[13-15]. Классический пример такого белка -
аминокислотной последовательности РНП до
белок U1A, компонент малой ядерной РНП U1
мена [10]. Впервые РНП домен был идентифи
(мяРНП U1) - одной из пяти мяРНП, формиру
цирован при сравнении последовательностей
ющих сплайсосому [16, 17] (рис. 1). РНП белки
белков, входящих в состав малых ядерных
способны связывать различные по длине одно
РНП (мяРНП), в середине 1980 х годов [11, 12].
цепочечные участки РНК - от двух (CBP20
Он имел две консервативные последовательнос
[18, 19], нуклеолин [20, 21]) до восьми нуклеоти
ти: мотив РНП1 из восьми преимущественно
дов (U2B′′ [22]). В большинстве случаев три кон
положительно заряженных и ароматических
сервативных а.о. фенилаланина или тирозина в
аминокислот
[K/R] G [F/Y] [G/A] [F/Y]
положениях
3 и
5
РНП1 и в положе
[I/L/V] X [F/Y] и мотив РНП2 в N концевой
нии 2 РНП2 (рис. 1, в) контактируют с двумя ос
части белков из шести а.о.
[I/L/V] [F/Y]
нованиями РНК таким образом, что все вместе
[I/L/V] X N L [13]. Эти два участка разделены
формируют непрерывную область стэкинг взаи
между собой вставкой примерно в 40 аминокис
модействий. Анализ структур показывает, что
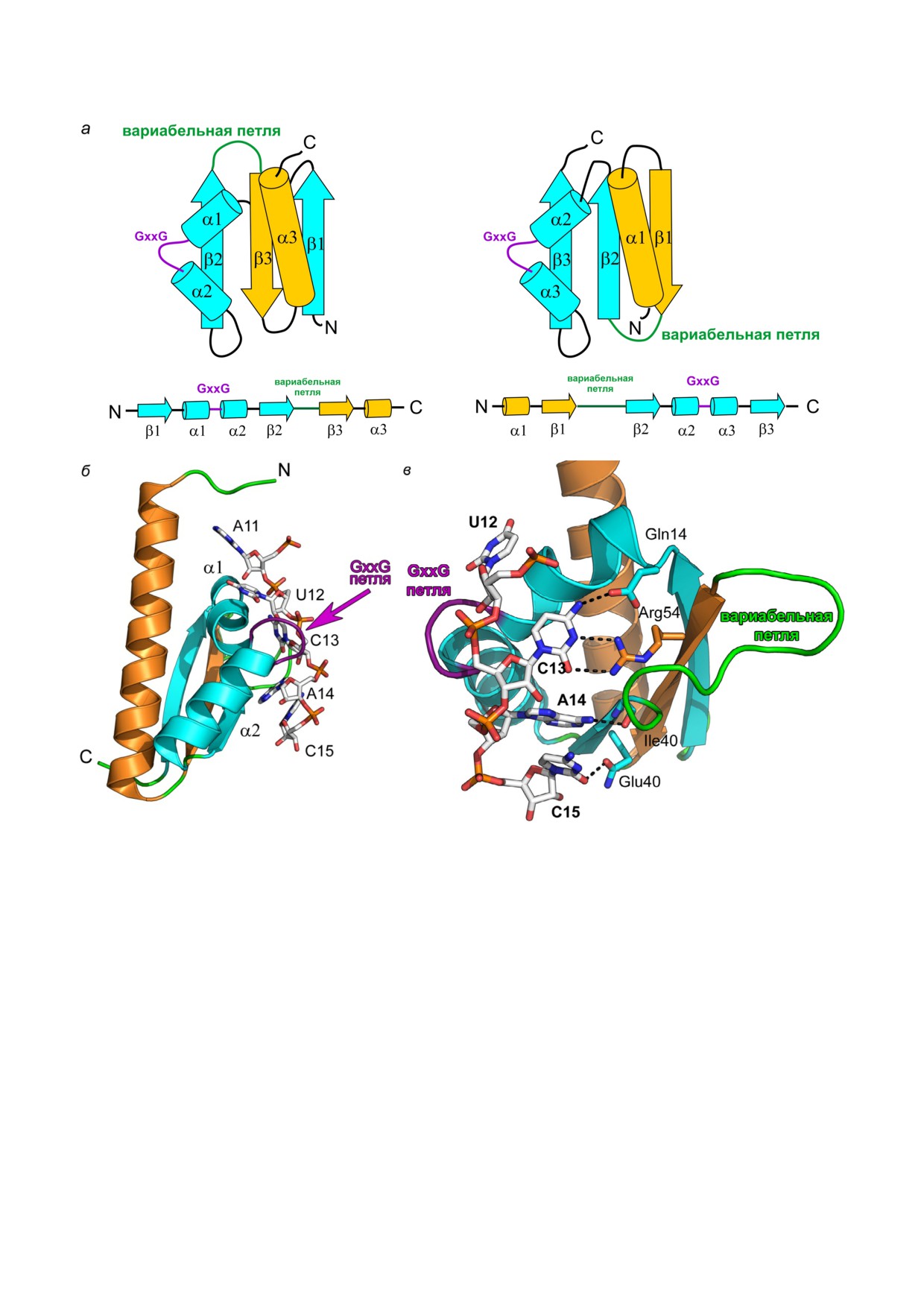
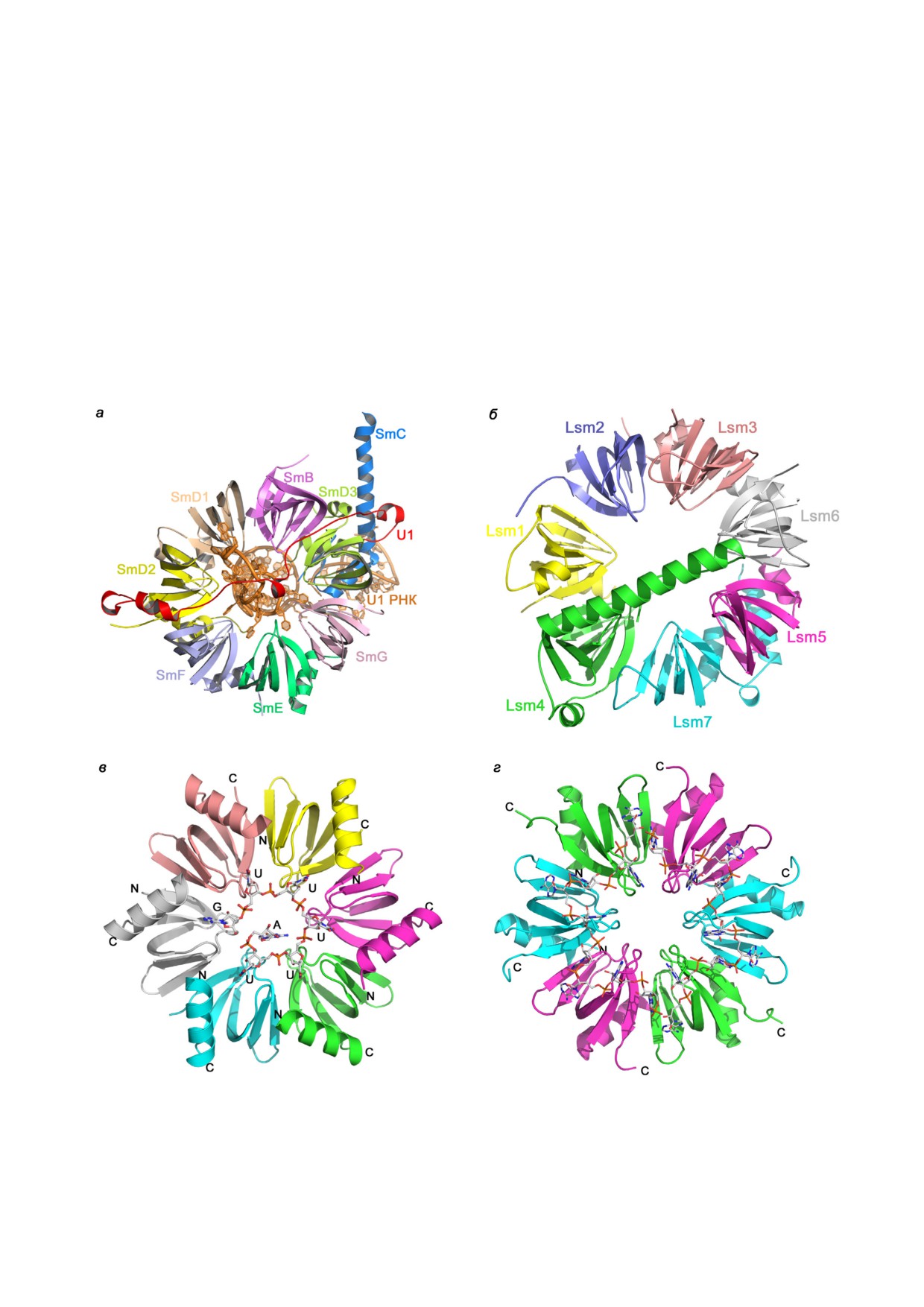
Рис. 1. Особенности пространственной организации белков с РНП доменом. а - Пространственная структура белка U1A
в комплексе со шпилькой II U1 мяРНК (PDB 1URN). Обозначены элементы вторичной структуры белка и концы фраг
мента РНК, использованного при кристаллизации комплекса. Здесь и далее изображения трёхмерных структур построе
ны на основе координат атомов структур в программе PyMol. б - Область взаимодействия белка U1A с U1 мяРНК. Обоз
начены наиболее важные аминокислотные остатки, взаимодействующие боковыми цепями с атомами оснований РНК.
в - Расположение консенсусов РНП1 и РНП2 на β листе РНК связывающего домена. Выделены ароматические остатки
консенсусов
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1233
РНП белки сохраняют в большинстве случаев
зываемый «КН домен» (heterogeneous nuclear
консервативный консенсус РНП мотивов, од
ribonucleoprotein K (hnRNP K) homology
нако имеются исключения из этого прави
domain). Он имеет размер порядка 70 а.о. и ха
ла [13]. Тем не менее во всех случаях β лист вы
рактеризуется аминокислотным мотивом [ILV]
полняет роль «подложки», которая определяет
I G X X G X X [ILV] центральной части доме
положение связываемой молекулы РНК, а бо
на [26]. Структурно КН домен представляет со
ковые цепи а.о., направленные к РНК, -
бой двухслойный белок с трёхтяжевым β лис
роль «крючков», обеспечивающих взаимодей
том и тремя α спиралями с одной из его сторон
ствие белка с РНК [23].
и имеет два разных типа пространственной ук
Важной особенностью РНП белков являет
ладки полипептидной цепи. Эукариотические
ся возможность объединения нескольких доме
белки (КН домен типа I) и бактериальные бел
нов для кооперативного узнавания РНК так на
ки (тип II) имеют общий
«минимальный
зываемыми
«тандемными доменами»
[24].
кор» βααβ, который дополняется одной α спи
В этом случае два РНП домена, связанных меж
ралью и одним β тяжем с С конца белка (тип I)
ду собой короткой перетяжкой линкером, взаи
или с N конца (тип II) относительно централь
модействуют с двумя последовательными пос
ной части белка. В результате эукариотический
ледовательностями РНК. Такая структурная ор
КН домен типа I имеет топологию βααββα с
ганизация белка позволяет значительно увели
антипараллельным β листом, а бактериальный
чить область РНК белкового интерфейса и по
КН домен типа II имеет топологию αββααβ
высить сродство белка к оцРНК. Впервые ко
(рис. 2, а).
оперативное узнавание оцРНК тандемными
Две α спирали центральной части белка сое
РНП белками было обнаружено в белке Sxl, ко
динены консервативной петлей GxxG, входя
торый специфически взаимодействует с после
щей в состав мотива КН домена. Показано, что
довательностью UGU8 [24]. Начало последова
белки с КН подобной пространственной струк
тельности РНК - UGU - узнается первым до
турой без консервативного мотива GxxG обла
меном белка, а дальнейшая олиго(U) последо
дают низким сродством к РНК, но могут моду
вательность - вторым доменом Sxl. В этом слу
лировать РНК связывающую активность других
чае происходит узнавание полностью разверну
белков [27]. Считается, что глицины этой петли
той одноцепочечной РНК, что отличает этот бе
располагаются очень близко к атомам сахаро
лок от большинства РНП белков, где происхо
фосфатного остова РНК, и их замена на дру
дит связывание шпилечной структуры РНК с
гие а.о. может привести к стерическим препят
небольшим одноцепоченым участком.
ствиям контакта белка с РНК. Особенность
Анализ последовательностей оцРНК пока
пространственной организации белков с КН
зывает относительно низкую специфичность их
доменом приводит к тому, что основания четы
узнавания РНП белками. Например, бе
рёх последовательно расположенных оснований
лок SRSF2 RRM может связывать последова
РНК направлены к поверхности белка и форми
тельности UCCAGU и UGGAGU, причём па
руют сеть контактов с его аминокислотными ос
ра GG взаимодействует с белком в том же участ
татками (рис. 2, б и в). Специфически узнавае
ке на поверхности β листа, что и пара СС [24].
мый тетрануклеотид имеет, как правило, в пер
Таким образом, РНП1 и РНП2 мотивы обеспе
вом и четвёртом положении пиримидины, а во
чивают некую платформу для взаимодействия
втором и третьем - аденины или цитозины [28].
белков с одноцепочечными участками РНК, а за
Узнавание цитозина во втором положении реа
узнавание определенных последовательностей
лизуется формированием двух водородных свя
РНК отвечают либо концевые участки белка,
зей атомов О2 и N3 основания с атомами боко
либо его партнеры - другие белки. В настоящее
вой цепи аргинина центрального β тяжа. Если
время считается, что РНП1/2 являются «LEGO
вместо цитозина в данном положении находит
блоками» для постройки оцРНК связывающих
ся аденин, то в белке происходит замена арги
белков, работающих одновременно с другими
нина на более подвижный лизин. В третьем по
белками [13, 15].
ложении основание нуклеотида специфически
Для ознакомления с особенностями струк
узнается благодаря формированию двух водо
тур белков с РНП доменами можно рекомендо
родных связей с амидной и карбонильной груп
вать обзоры (например, [25]) и базы данных по
пами главной цепи одного из остатков второго
классификации структур (Pfam PF00076,
(тип I КН домена) или третьего (тип II) β тяжа.
InterPro IPR000504).
Как аденин, так и цитидин могут формировать
Белки с КН'доменом. Вторым по распрост
по две водородных связи в этом положении, а их
раненности оцРНК связывающим мотивом
дискриминация обеспечивается дополнитель
после РНП домена, является, пожалуй, так на
ной водородной связью с атомом боковой цепи
БИОХИМИЯ том 86 вып. 8 2021
1234
НИКУЛИН
Рис. 2. Особенности пространственной организации белков с КН доменами. а - Схема укладки элементов вторичной
структуры в домене I типа (слева) и в домене II типа (справа). «Коровая» часть белка выделена голубым цветом, допол
нительные элементы - желтым. б - Структура комплекса белка Nova 2 KH3 (третий домен антигена Nova млекопитаю
щих, важного семейства регуляторов метаболизма РНК в нейронах) со шпилечной РНК (PDB 1EC6). Показан фрагмент
РНК с А11 по С15. Малиновым цветом выделена область GxxG петли белка. в - Область взаимодействия тетрануклеоти
да U12C13A14C15 РНК с белком Nova 2 KH3. Водородные связи показаны пунктирными линиями
еще одного аминокислотного остатка из спира
комплексов [33]. Для ознакомления с особен
ли α2 [29, 30].
ностями структур белков с KH доменами мож
Следует отметить, что для белков с КН до
но рекомендовать обзоры [29, 34] и базы данных
менами также имеются примеры с дополнитель
по классификации структур (Pfam PF00013,
ными структурными элементами (например, бе
InterPro IPR004088).
лок STAR - Signal Transduction and Activation of
Белки с небольшими повторяющимися доме'
RNA fold [31]) или с тандемными доменами
нами. Белки с доменом «цинковые пальцы». Инте
(например, белок NusA [32]). Это повышает
ресным примером РНК связывающих мотивов
сродство белков к РНК с микромолярных до на
являются домены типа «цинковых пальцев».
номолярных величин константы диссоциации
Впервые такие домены были описаны, как не
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1235
большие ДНК связывающие домены размером
ких, водородных связей между а.о. и основания
примерно 30 а.о. с консенсусом [YF] X C X C
ми, но важную роль играет стэкинг ароматичес
X(2,4) C X(3) F X(5) L X(2) H X(3,4) H
ких боковых цепей белка и оснований РНК. Для
X(5) [35-37]. Они имеют топологию ββα, при
увеличения сродства белок-РНК и повышения
чём две консервативные пары остатков цистеи
специфичности белки этого типа также имеют
на и гистидина связывают ион цинка, стабили
несколько повторов «цинковых пальцев», одна
зирующего структуру домена (рис. 3). Эти доме
ко в них нет выраженной специфичности к
ны укладываются в большой желобок двуцепо
ДНК или к РНК, что является особенностью
чечной спирали ДНК, при этом атомы боковых
этого типа белков.
цепей заряженных а.о. взаимодействуют с ато
Белки с Pum доменом (PUF повторы). Ещё
мами оснований c образованием водородных
одним примером небольших повторяющихся
связей. Ион цинка не задействован во взаимо
доменов, связывающих протяжённый оцРНК
действии белка с ДНК, и его основная роль зак
тракт, является так называемый PUM
лючается в стабилизации структуры такого не
HD (Pumilio homology domain). Своё название
большого домена.
он получил от найденного в дрозофиле белка
Как было найдено позже, ряд РНК связыва
Pumilio, выполняющего важную роль в регуля
ющих белков также содержат домены типа
ции созревания [42]. Установлено, что белки
«цинковые пальцы». Так, белок Tis11d (вовлечён
этого типа взаимодействуют с некодирующим
в регуляцию стабильности мРНК, связывается с
участком мРНК, располагающимся на её 3′ кон
AU богатым элементом класса II (ARE) в 3′ не
це
(3′ НТО мРНК), тем самым влияя на
кодирующем участке мРНК мишени и способ
экспрессию многих генов
[43,
44]. Белок
ствует их деаденилированию и деградации [38,
Pumilio1 PUM HD человека содержит восемь
39]) содержит два домена «цинковый палец» с
мотивом C X(8) C X(5) C X(3) H (или СССН
тип), а нуклеокапсидный белок MMLV
(Moloney murine leukaemia virus) содержит один
такой домен (рис. 3) [40, 41]. В белке Tis11d каж
дый домен специфически узнаёт одноцепочеч
ную последовательность UAUU в AU богатом
элементе (ARE) класса II 3′ некодирующем
участке мРНК и способствует их деаденилиро
ванию и деградации. В этом комплексе четыре
основания РНК попадают в карман каждого до
мена и формируют стэкинг с боковыми цепями
фенилаланина (петля между третьим цистеином
и гистидином «цинкового пальца») и тирозина
(петля между вторым и третьим цистеинами
«цинкового пальца»). Специфичность узнава
ния обеспечивают контакты атомов оснований с
атомами главной цепи «цинковых пальцев» за
единственным исключением - контакт боковой
цепи глутамина Glu157.
Нуклеокапсидный белок вируса мышиной
лейкемии MMLV особо интересен тем, что со
держит минимальный домен «цинковый палец»
(по английски его даже назвали knuckle - фа
ланга пальца) типа C X(2) C X(3) H X(4) C
(или ССНС) (рис. 3, б). Взаимодействие с РНК
происходит аналогично белку Tis11d за счёт стэ
кинга оснований между тирозинами и трипто
фаном «цинковых пальцев», но в данном случае
большинство контактов с атомами основания
осуществляют боковые цепи аминокислотных
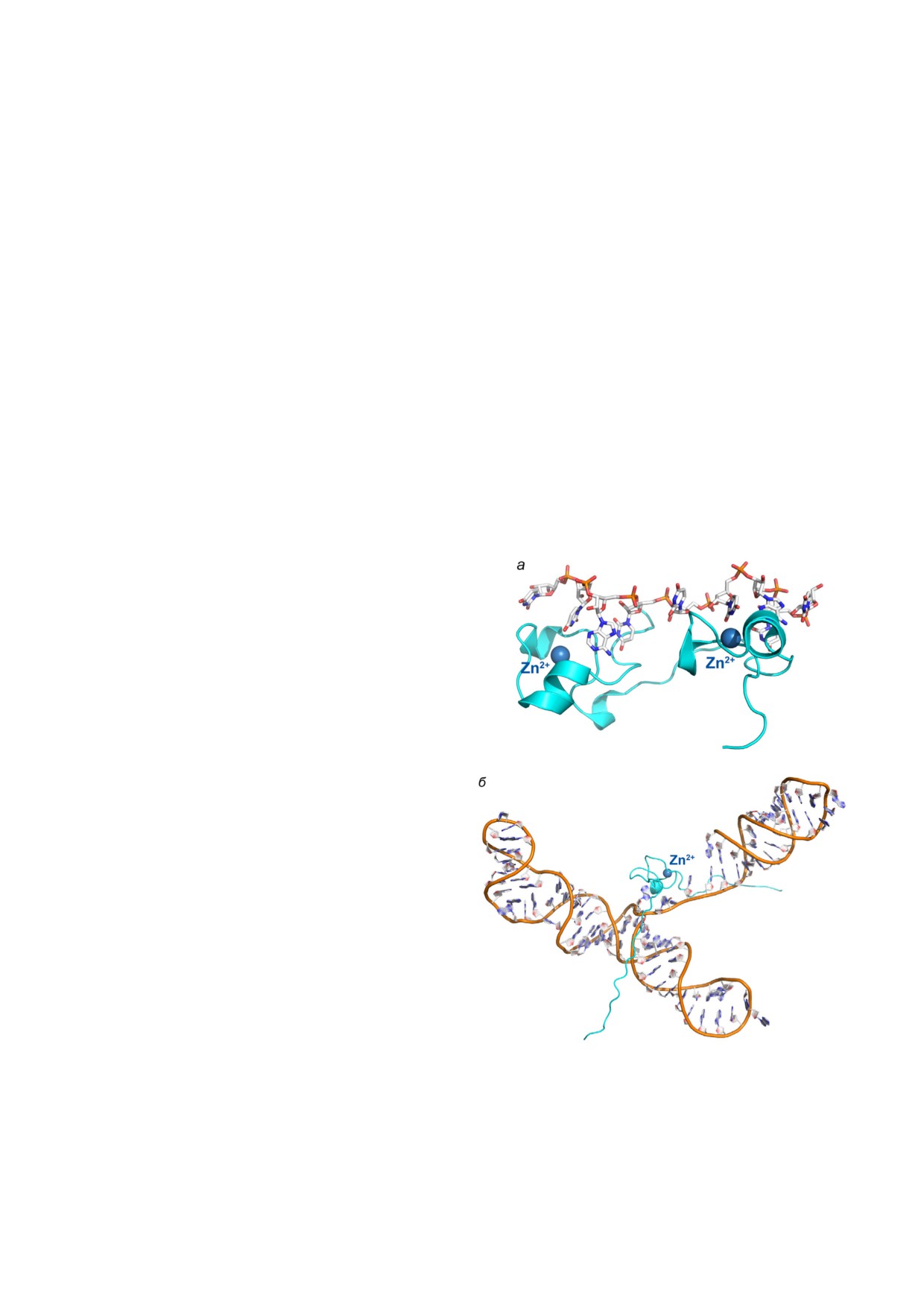
Рис. 3. Особенности пространственной организации бел
ков с доменами типа «цинковые пальцы». а - Структура
остатков.
белка Tis11d в комплексе с
5′ UUAUUUAUU 3′
Суммируя имеющиеся данные, можно ска
РНК (PDB 1RGO). б - Структура белка MMLV в комплек
зать, что белки с доменами «цинковые пальцы»
се с сигнальной РНК (PDB 1U6P). Обозначены ионы
узнают РНК благодаря формированию несколь
цинка
БИОХИМИЯ том 86 вып. 8 2021
1236
НИКУЛИН
Рис. 4. Особенности пространственной организации белков с Pum доменами и белка TRAP. а - Структура белка
Pumilio1 PUM HD человека в комплексе с десяти нуклеотидной оцРНК (PDB 1M8Y). На врезке - детали взаимодей
ствия РНК с белком в области контактов одного из повторов. б - Структура белка TRAP в комплексе с 53 нуклеотидной
оцРНК, содержащей 11 GAG триплетов, разделённых между собой AU динуклеотидами (показаны по окружности бел
ка) (PDB 1C9S). На поверхности β листа мономеров белка расположены молекулы связанного триптофана. Справа - уве
личенная область взаимодействия белок-РНК. Обозначены узнаваемые триплеты GAG и «спейсерные» AU
PUF повторов длиной 37 а.о. каждый, а также
ных молекул триптофана он взаимодействует с
N и С концевые последовательности, похожие
некодирующим участком мРНК, располагаю
по структуре и последовательности на PUF пов
щимся на 5′ конце мРНК, и формирует «терми
торы (рис. 4, а) [42].
наторную петлю» на ней, что приводит к терми
Каждый PUF повтор состоит из трёх α спи
нации трансляции [47, 48].
ралей, формирующих изогнутый сегмент.
Белок TRAP представляет собой полностью
оцРНК взаимодействует с вогнутой внутренней
β белок (двухслойный антипараллельный β
частью белка таким образом, что каждое осно
сэндвич), который формирует четвертичную
вание располагается между двумя соседними
структуру в виде симметричного кольца из
повторами, а сахарофосфатный остов РНК не
11 мономеров (рис. 4, в). Триптофан связывает
имеет контактов с белком. Основания нуклео
ся белком в области между двумя β слоями.
тидов находятся в стэкинге с боковыми цепями
В структуре комплекса с
53 нуклеотидной
тирозинов и аргининов, а атомы Уотсон-Кри
оцРНК каждый из 11 повторов GAGAU связы
ковского края основания формируют водород
вается с одним из мономеров белка [47]. Трипле
ные связи с боковыми цепями полярных и заря
ты GAG располагаются между двумя соседними
женных остатков спирали α2 каждого повто
мономерами и взаимодействует с ними, а ди
ра [42]. Интересно, что если в качестве таких ос
нуклеотид AU служит «спейсером» между ними.
татков выступают пара глутамин и аспарагин, то
Система водородных связей приводит к специ
с повтором связывается урацил, если глутамин и
фическому узнаванию оснований А и третьего G
цистеин - то аденин, если глутамин и серин -
каждого триплета GAG. Между атомами белка и
то гуанин, причём их заданные замены меняют
сахарофосфатного остова РНК контактов нет, за
специфичность белка к последовательности
исключением водородной связи 2′ гидроксила
оцРНК [42, 45, 46]. В данном случае наблюдает
рибозы основания G3 с амидной группой глав
ся экстремальный случай принципа повтора до
ной цепи остатка Phe32. По всей видимости, та
менов для повышения сродства белка к РНК,
кой контакт важен для избирательного связыва
поскольку каждый отдельный домен представ
ния РНК, а не ДНК, поскольку последняя при
ляет собой PUF повтор, узнающий индивиду
той же самой последовательности нуклеотидов
альный нуклеотид.
имеет сродство к TRAP в 10 000 раз меньше [49].
Белок TRAP. Ещё одним представителем бел
Белки с доменом «ОВ'фолд». Домен с уклад
ков с большим количеством малых доменов, ко
кой «ОВ фолд» назван так из за вхождения в
торые контактируют с оцРНК, является бе
состав белков со способностью связывать оли
лок TRAP (tryptophan RNA binding attenuation
гонуклеотиды и олигосахариды (oligonucleotide/
protein). Белок TRAP регулирует в ряде бацилл
oligosaccharide binding). Это структура в виде β
экспрессию генов белков, отвечающих за синтез
бочонка из пяти β тяжей. На практике ОВ
L триптофана. При связывании белком свобод
фолд домены имеются в совершенно различаю
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1237
щихся по функции белках размером 70-150 а.о.
позволяют говорить о специфичности белка к
[50]. По структурной классификации SCOP, ОВ
цитидинам. Одно из оснований расположено в
фолд подразделяется на 16 суперсемейств, из
гидрофобном кармане между двумя тирозина
которых только одно имеет название «nucleic
ми, а второе - образует водородные связи с бо
acid binding proteins» (суперсемейство SCOP 50249).
ковыми цепями аргинина, аспарагина и гидрок
ОВ фолд не является определяющим для воз
силом боковой цепи тирозина. Контактов 2′ ОН
никновения у белка РНК связывающих свойств,
группы рибозы с белком не наблюдается, что со
а служит удобной платформой для построения
ответствует данным о возможности белка Rho
белков с такими свойствами, поскольку пред
связывать как РНК, так и ДНК [52].
ставляет собой весьма жёсткую и стабильную
Имеется ещё целый ряд белков с доменами
структуру из за плотной и стабильной упаков
типа ОВ фолд, которые проявляют ДНК/РНК
ки β листа.
связывающие свойства, например, семейство
Характерным примером РНК связывающе
белков холодового шока с доменом CSD
го белка этого семейства является фактор тер
(Pfam PF00313, InterPro IPR002059) [53-55],
минации транскрипции Rho из E. coli. В раство
структурированный домен эукариотических
ре он формирует гексамеры в виде не полностью
белков YB 1 (относится к белкам с CSD доме
замкнутого кольца (рис. 5) [51]. Мономер белка
ном) [56], домены бактериальных рибосомных
состоит из двух доменов: меньший по размеру
белков S1 (InterPro IPR000110) и другие. Неко
N концевой РНК связывающий домен и боль
торую структурную гомологию можно обнару
ший по размеру С концевой АТФазный домен.
жить и между ОВ фолдом и Sm фолдом, кото
N концевой домен белка Rho, кроме β бочонка
рый будет рассматриваться дальше.
ОВ фолда, включает в себя 47 а.о., формирую
Белки с доменом «Sm'фолд». Своё название
щих три α спирали на N конце домена. В струк
Sm фолд получил благодаря так называемым
туре комплекса этого домена с олиго(С) РНК
«Sm белкам», входящим в состав эукариотичес
два цитидина связываются с областью тяжей β2
ких малых ядерных РНП (мяРНП) [57]. Семь го
и β3, прикрываясь с одной стороны петлей меж
мологичных, но различающихся по последова
ду тяжами β1 β2 и спиралью α4, а с другой сто
тельности Sm белков (B/B′, D3, D2, D1, E, F и
роны - петлей β2 β3 [52]. Контакты между ами
G), вместе с несколькими уридин богатыми
нокислотными остатками белка и нуклеотидами
мяРНК формируют консервативную часть
Рис. 5. Пространственная структура гексамерного фактора терминации транскрипции Rho из Escherichia coli в комплексе
с короткой РНК и нуклеотидом ADPPNP (PDB 1PVO). Справа показан мономер фактора Rho со связанным динуклеоти
дом UC и молекулой ADPPNP. Овалом выделен домен «ОВ фолд»
БИОХИМИЯ том 86 вып. 8 2021
1238
НИКУЛИН
сплайсосомной мяРНП [58-60]. Sm белки име
Lsm - «Like Sm» [61]. Они входят в состав РНП,
ют длину около 80 а.о., организованных в струк
участвующих в созревании и процессинге
турно гомологичный ОВ фолду пятитяжевый β
мРНК и тРНК, декэпировании мРНК и ряде
бочонок с N концевой α спиралью (рис. 6).
других процессов [62]. Lsm белки также форми
В составе мяРНП они формируют кольцевой ге
руют гетерогептамеры, однако для этого не тре
терогептамер, чем напоминают ранее рассмот
буется присутствие РНК [63].
ренный белок Rho, однако это полностью замк
Гомологи эукариотических Sm/Lsm белков
нутое кольцо, и контакты между соседними мо
найдены в археях и в бактериях. В бактериаль
номерами белка организованы крайними тяжа
ных клетках Lsm белки называются Hfq и игра
ми β листа, а не дополнительными С концевы
ют роль глобального регулятора экспрессии ге
ми доменами. Следует отметить, что Sm белки
нов [64, 65]. Архейные Lsm белки называ
собираются в гептамеры только в присутствии
ют SmAP (Sm Archaeal Protein); их функция в
мяРНК [58]. В эукариотах имеются белки, гомо
клетках до сих пор не очень ясна [61, 65]. Как
логичные перечисленным Sm; их называют
архейные, так и бактериальные Lsm белки фор
Рис. 6. Особенности пространственной организации Sm/Lsm белков. а - Структура минимального U1 мяРНП челове
ка (PDB 4PJO). В центре гептамера располагается U1 мяРНК. Белки комплекса обозначены разным цветом и подписаны.
б - Структура гетерогептамера комплекса Lsm1 7 из дрожжей (PDB 4C92). в - Структура комплекса Hfq из Staphylococcus
aureus с АU5G РНК (PDB 1KQ2). г - Структура комплекса Hfq из E. coli с олиго(А) РНК (PDB 3GIB)
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1239
мируют стабильные кольцевые мультимеры,
ляют единственную консервативную структуру,
причём архейные SmAP образуют гомогептаме
называемую dsRBD (double stranded RNA
ры, а бактериальные Hfq - гомогексамеры.
Binding Domain - дцРНК связывающий домен)
Sm/Lsm белки связывают олиго(U) РНК во
[70,
71]. Среди РНК связывающих белков
внутренней полости олигомера со стороны α
dsRBD находится на втором месте по встречае
спиралей мономеров (рис. 6, в) [66]. Каждый
мости после РНП домена, отвечающего за узна
уридин располагается в кармане, организован
вание оцРНК. Он представляет собой неболь
ном двумя соседними мономерами, урацилы на
шой домен 65-70 а.о. из трёхтяжевого антипа
ходятся в стэкинге с ароматическими боковыми
раллельного β листа с расположенными на од
цепями аминокислотных остатков петель β2 β3
ной из его плоскостей N и С концевыми спира
и β4 β5, а атомы оснований РНК формируют
лями. Впервые dsRBD был идентифицирован
водородные связи с атомами заряженных боко
при сравнении первичных структур ряда эука
вых цепей белка. Поскольку размер внутренней
риотических белков, имеющих высокую специ
полости достаточно мал, то не все мономеры
фичность к дцРНК, но низкое сродство к
белка имеют одинаковые контакты с основани
оцРНК и к любой ДНК, причём изменения в
ями РНК из за необходимости «входа» и «выхо
нуклеотидной последовательности дцРНК не
да» оцРНК.
влияли на сродство к ней dsRBD белков [72].
Бактериальные белки Hfq имеют сродство
Один из таких белков - второй домен РНК
также к олиго(А) РНК [67, 68]. Такая РНК свя
связывающего белка A из Xenopus laevis (Xlrbpa 2),
зывается с противоположной от участка связы
взаимодействует с участком дцРНК длиной
вания олиго(U) РНК стороне гексамера
10 пар оснований (GGCGCGCGCC)2 в трёх об
Hfq (рис. 6, г). Аденины располагаются в гидро
ластях (рис. 7) [73]. Первая (N концевая α спи
фобном кармане белка, формируют стэкинг с
раль) и вторая (петля между тяжами β1 и β2) об
ароматическими боковыми цепями белка и ряд
ласти белка контактируют с двумя удаленными
водородных связей с боковыми цепями амино
на виток спирали участками малого желобка
кислотных остатков. Расстояние между специ
РНК, а третья (С концевая спираль) - с участ
фически связываемыми аденинами здесь боль
ком большого желобка двойной спирали. Ами
ше, чем в случае олиго(U) РНК. В промежутке
нокислотные остатки белка во всех трёх участ
между ними расположены другие нуклеотиды,
ках контактируют с атомами сахарофосфатного
которые могут как взаимодействовать с амино
остова РНК, многие из них - с 2′ ОН группами
кислотными остатками белка [67], так и распо
рибоз. Регулярная структура малого желобка А
лагаться на поверхности белка, не имея специ
формы дцРНК нарушается в области петли β1
фических контактов с атомами белка [69].
β2 Xlrbpa (область 2), причём с этим изменён
В Sm/Lsm белках реализуется принцип пов
ным участком контактирует консервативный
торения доменов для повышения сродства белка
гистидин, важный для взаимодействия белка с
к РНК, точнее - к единичному основанию, ури
дцРНК. Интересно, что для взаимодействия с
дину или аденину. Пространственная укладка
дцРНК оказались важны не только определен
элементов вторичной структуры здесь является
ные аминокислотные остатки, имеющие кон
фундаментом для создания стабильных мульти
такты с РНК, но и целый ряд гидрофобных ос
меров белка, которые работают как единое целое,
татков, формирующих правильную упаковку α
а поверхность β листа не используется в качестве
спиралей в структуре белка и определяющих не
«рабочей поверхности» для взаимодействия с
обходимую ориентацию контактирующих с
РНК. Области узнавания расположены в карма
РНК заряженных остатков [70].
нах белка, организованных аминокислотными
Рассматривая известные структуры dsRBD
остатками петель между элементами вторичной
белков в комплексах с дцРНК можно заметить,
структуры. Они включают гидрофобные а.о., ор
что эти белки имеют очень малое число контак
ганизующие стэкинг с основаниями нуклеоти
тов с основаниями РНК, а почти все контакты
дов, а также несколько заряженных/полярных
формируются с атомами сахарофосфатного ос
аминокислотных остатков, формирующих систе
това и 2′ гидроксилом рибозы. Это наиболее су
му водородных связей с атомами оснований.
щественное отличие dsRBD белков от оцРНК
узнающих белков.
Для повышения сродства к дцРНК dsRBD
БЕЛКИ, УЗНАЮЩИЕ
белки могут использовать дополнительные эле
ДВУЦЕПОЧЕЧНЫЕ УЧАСТКИ РНК
менты вторичной структуры, такие как увели
ченную N концевую α спираль или повторяю
Двуцепочечно'РНК'связывающий домен dsRBD.
щиеся домены [70, 71, 74]. Тем не менее, незави
В белках, взаимодействующих с дцРНК, выде
симо от этого, dsRBD белки формируют кон
БИОХИМИЯ том 86 вып. 8 2021
1240
НИКУЛИН
Рис. 7. Структура комплекса белка Xlrbpa 2 и дцРНК (PDB 1DI2). Показаны два соседних додекамерных фрагмента
дцРНК (GGCGCGCGCC)2. Обозначены элементы вторичной структуры белка и области его контактов с дцДНК. Внизу
показаны три области РНК белковых контактов в приближении
такты с атомами сахарофосфатного остова, а не
ную структуру [77, 78]. Эти РНК белковые
с атомами оснований нуклеотидов дцРНК.
комплексы можно рассматривать, как комплек
Рибосомные белки. Интересными примерами
сы белков с дцРНК. Важно отметить, что ряд
белков, узнающих сложные пространственные
первично связывающихся рибосомных белков
структуры РНК, являются рибосомные белки.
регулируют уровень своего синтеза, взаимодей
Рибосома - макромолекулярный комплекс из
ствуя с мРНК, которые их кодируют, таким об
нескольких рибосомных РНК (рРНК) и более
разом, специфически узнавая две разные РНК
50 белков. рРНК имеет сложную простран
мишени. Изучение процессов авторегуляции
ственную структуру, способную самоорганизо
синтеза рибосомных белков представляет очень
ваться in vitro. Сборка рибосомных субъеди
интересную задачу для исследования принци
ниц - это кооперативный процесс со строгой
пов РНК белкового узнавания и дискримина
очередностью этапов, инициируемый взаимо
ции двух разных РНК мишеней одним белком.
действием первично связывающихся рибосом
Ниже будут рассмотрены три бактериальных
ных белков со специфически узнаваемыми
рибосомных белка, которые являются одновре
участками рРНК. Их присоединение к рРНК
менно первично связывающимися рибосомны
приводит к значительной компактизации фор
ми белками и регуляторами трансляции бакте
мируемых рибонуклеопротеиновых частиц и
риальных оперонов. Для эукариотических рибо
обеспечивает последующее присоединение ос
сомных белков аналогичных исследований не
тальных рибосомных белков [75, 76]. Первично
проводилось. Следует упомянуть, что в 2014 го
связывающиеся рибосомные белки взаимодей
ду была введена новая номенклатура наимено
ствуют с рРНК независимо от других компонен
ваний рибосомных белков для устранения несо
тов рибосомы, причём участки связывания бел
ответствия наименований рибосомных белков
ков на рРНК имеют сложную пространствен
из бактерий, архей и эукариот [79]. Согласно
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1241
этой номенклатуры универсально консерватив
фатным остовом рРНК. Консервативные нуклео
ные рибосомные белки, которые имеются в ри
тиды 595-598/640-644 отвечают за нарушения
босомах из организмов всех трёх царств, имеют
А формы спирали дцРНК, образуя два триплета
префикс «u», характерные только для бактери
нуклеотидов, взаимосвязанных пересекающи
альных рибосом - «b», а только для эукариоти
мися водородными связями. Эти же нуклеотиды
ческих - «e».
формируют узнаваемую белком S8 поверхность
Рибосомный белок uS8. Универсально кон
на 16S рРНК [4].
сервативный рибосомный белок uS8 (далее бу
Структура белка S8 из E. coli в комплексе с
дет именоваться как S8) входит в число рибо
фрагментом узнаваемого участка мРНК spc опе
сомных белков, формирующих платформу ма
рона показала незначительные отличия в после
лой рибосомной субчастицы [80-84]. Он важен
довательностях и пространственной структуре
для правильной сборки центрального домена
связываемых участков на рРНК и мРНК [94].
16S рРНК [81, 83, 85], а ряд мутаций в белке
Контакты между аминокислотными остатками
приводит к дефектам при ассоциации рибосом
белка и мРНК распределены аналогично тако
ных субчастиц в рибосому [86]. Белок S8 связы
вым в рибосомном комплексе. Несмотря на
вается с участком спирали h21 16S рРНК в об
сходство структур узнаваемых белком участков
ласти нуклеотидов 595-598/640-644 [87-89].
РНК, белок S8 имеет существенную разницу в
Одновременно белок S8 является регулятором
сродстве к мРНК. На данный момент разница в
транскрипции spc оперона E. coli по механизму
сродстве белка S8 к рРНК и мРНК не нашла
обратной связи [90, 91]. Область связывания на
строгого объяснения.
spc мРНК очень схожа со структурой области
Сравнивая комплексы белка S8 с рРНК/
связывания белка на спирали h21 16S рРНК, что
мРНК и комплекс белка Xlrbpa 2 с дцРНК,
изначально приводило к идее узнавания схожих
нельзя не заметить внешнее сходство этих
по структуре элементов двух РНК [90, 92, 93].
структур. Действительно, областью взаимодей
Структура комплекса белка S8 с фрагментом
ствия дцРНК с белком в обоих случаях стано
16S рРНК показала, что два домена белка кон
вится нарушенная регулярная структура малого
тактируют с двумя соседними участками малого
желобка РНК, которая является местом контак
желобка спирали РНК, причём белок образует
та единичных аминокислотных остатков с осно
«мостик» над большим желобком РНК
ваниями РНК. Этот участок окружён областями
(рис. 8, а) [4]. Основная часть контактирующих
контактов заряженных и полярных аминокис
с рРНК аминокислотных остатков сосредоточе
лотных остатков с сахарофосфатным остовом
на в С концевом домене белка, а превалирую
РНК, что обеспечивает закрытие области узна
щая часть контактов реализуется с сахарофос
вания от воды и ионов и увеличивает выигрыш в
Рис. 8. Структуры комплексов рибосомных белков с РНК. а - Структура белка S8 из Methanococcus jannaschii в комплек
се с фрагментом 16S рРНК (PDB 1I6U). Показаны контактирующие аминокислотные остатки белка S8. б - Структура
комплекса белка S15 из Thermus thermophilus со специфическим фрагментом 16S рРНК (PDB 1DK1). Указаны спирали
16S рРНК (h21, h22 и h23) и спирали белка S15. Слева - вид со стороны белка S15, справа - вид повернут на 90° по вер
тикальной оси относительно левого рисунка
10 БИОХИМИЯ том 86 вып. 8 2021
1242
НИКУЛИН
энергии при формировании РНК белкового
структурных и биохимических данных о взаимо
контакта. Однако в случае 16S рРНК нарушение
действии белка S15 с 16S рРНК предполагают,
структуры спирали РНК значительное и вызва
что именно область консервативных нуклеоти
но формированием двух нуклеотидных трипле
дов GU/GC является специфически узнаваемой
тов, а в случае Xlrbpa 2 нарушение меньше и
аминокислотными остатками белка, а контакты
вызвано выпетливанием единичного нуклеоти
белка с областью соединения трёх спиралей
да. Таким образом, несмотря на внешнее сход
16S рРНК позволяют зафиксировать их взаим
ство, участки связывания белка на РНК сущест
ное расположение при сборке малой рибосом
венно отличаются по своей пространственной
ной субчастицы [5].
структуре.
Моделирование структуры комплекса бел
Рибосомный белок uS15. Универсально кон
ка S15 со специфически узнаваемым участком
сервативный рибосомный белок uS15 (далее бу
мРНК показало, что этот участок имеет прост
дет именоваться S15) также участвует в форми
ранственную структуру типа
«псевдоузла»
ровании платформы малой рибосомной субчас
(англ.: pseudo&knot) с дополнительным вторым
тицы рибосомы, однако именно он первым при
участком связывания [102-106]. В целом струк
соединяется к центральному домену 16S рРНК и
тура узнаваемых участков на рРНК и мРНК ока
обеспечивает дальнейшее связывание с ним ри
залась в некоторой степени похожа, что и объяс
босомных белков S6, S18, S11 и S21 [80, 81, 95,
няет возможность узнавания одним белком двух
96]. Как и белок S8, он регулирует трансляцию
разных РНК. Такая схожесть пространственной
своего оперона, связываясь с мРНК при избыт
структурной организации участков узнавания
ке в клетке и подавляя его продукцию [97-100].
двух различных РНК была названа авторами
Рибосомный белок S15 - небольшой белок,
«молекулярной мимикрией» [106]. Однако раз
состоящий из четырёх α спиралей, упакован
ница в структурах мРНК и рРНК довольно за
ных в один домен. Белок S15 связывается с
метная: в первом случае - это псевдоузел, а во
16S рРНК в области трёх спиралей - h20, h21
втором - соединение трёх спиралей, что и явля
и h22 (рис. 8, б) [83, 101]. Из почти двух десятков
ется предполагаемой причиной отличия срод
контактирующих с рРНК аминокислотных ос
ства белка S15 к двум молекулам РНК.
татков только 4 являются положительно заря
Рибосомный белок uL1. Универсально кон
женными, а большая часть контактов формиру
сервативный рибосомный белок uL1 (далее бу
ется полярными боковыми цепями аминокис
дет именоваться как L1) вместе со спираля
лот с атомами сахарофосфатного остова РНК
ми Н76, Н77 и Н78 23S рРНК формирует под
[5, 95]. Белок контактирует с 16S рРНК в двух
вижный функционально важный участок боль
областях: непосредственно в области сочлене
шой рибосомной субчастицы, называемый L1
ния трёх спиралей 16S рРНК (англ.: three&way
выступом [107]. В E. coli белок L1 является регу
junction) и с GU/GC мотивом малого желобка
лятором трансляции L11 оперона, в состав ко
двойной спирали рРНК, отстоящего на один ви
торого входят гены рибосомных белков L1 и
ток от первого участка. Важной особенностью
L11 [108, 109].
первого участка связывания является формиро
Белок L1 состоит из двух доменов, причём
вание триплета оснований, который служит
первый домен включает N концевую и С конце
ключевым элементом в фиксации конформации
вую части белка, а второй домен является встав
места соединения трёх спиралей 16S рРНК.
кой среднего участка полипептидной це
С этим триплетом напрямую или через ион маг
пи [107]. Первый домен имеет двухслойную
ния взаимодействуют боковые остатки амино
структуру «расщеплённой abc/d единицы» или
кислот спирали α3. С соседними нуклеотидами
«split β α β», а второй домен - уникальную для
контактируют аминокислотные остатки петли
рибосомных белков трёхслойную укладку по
между спиралями α1 и α2. Большинство этих
Россману. Домены I и II соединены гибкой пе
контактов реализуются посредством сахарофос
ретяжкой и могут несколько менять взаимную
фатного остова малого желобка РНК.
ориентацию в растворе [110].
Вторая область РНК белковых контактов
Белок L1 в комплексах с фрагментами
располагается в верхней части спирали h22, где
23S рРНК имеет две области контактов с
белок S15 специфически связывается с высоко
РНК (рис. 9, а) [3]. Одна из этих областей кон
консервативными нуклеотидами малого желоб
тактов располагается на поверхности домена I.
ка GU/GC. Консервативные аминокислотные
Три тяжа β листа этого домена формируют нес
остатки района петли α2 α3, направленные в
колько вогнутую поверхность контакта со спи
сторону РНК, образуют непосредственно или
ралью Н77 23S рРНК. Можно отметить, что та
через воду водородные связи со всеми четырьмя
кая архитектура белка несколько напоминает
перечисленными нуклеотидами. На основании
структуру РНП домена, который также включа
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1243
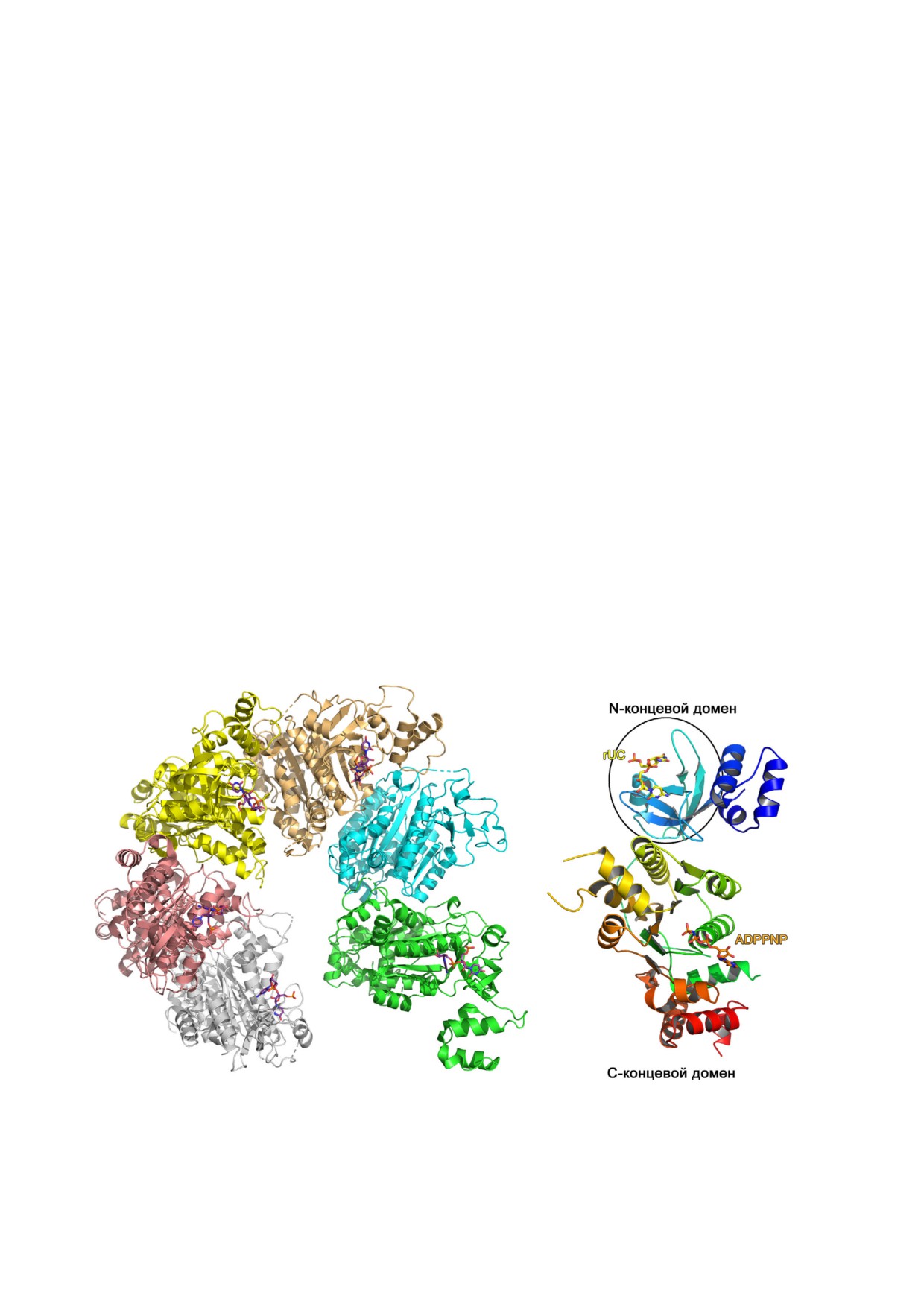
Рис. 9. Структуры комплексов рибосомных белков L1 с РНК. а - Структура комплекса рибосомного белка L1 из Sulfolobus
solfataricus с фрагментом 23S рРНК из T. thermophilus (PDB 1MZP). Указаны спирали Н76, Н77 и Н78 23S рРНК. б - Струк
тура комплекса рибосомного белка L1 из T. thermophilus с фрагментом мРНК из Methanococcus vannielii (PDB 2HW8).
Структуры ориентированы одинаково относительно домена I белка L1
ет контактирующую с РНК поверхность β лис
ваемые белком L1, имеют схожую структуру и
та, однако последовательность белка L1 не име
последовательность нуклеотидов, однако в их
ет РНП консенсусов, да и аминокислотные ос
составе отсутствует одна из петель РНК, поэто
татки белка L1 взаимодействуют не с оцРНК, а с
му вторая область РНК белкового контакта яв
малым желобком дцРНК. Большая часть кон
ляется усечённой (рис. 9, б). Это приводит к
тактов между белком и РНК в области первого
практически полному отсутствию контактов во
домена реализуется посредством водородных
второй области и к значительному уменьшению
связей с сахарофосфатным остовом РНК, одна
сродства белка L1 с мРНК. Позднее было пока
ко имеется значимое количество водородных
зано, что изолированный домен I рибосомного
связей и с основаниями.
белка L1 имеет более низкое сродство к мРНК
Вторая область РНК белкового взаимодей
по сравнению с рРНК и играет определяющую
ствия включает аминокислотные остатки спи
роль при дифференциации между разными
рали α4 и петли α5 β6 домена II белка L1, кото
РНК [114, 115].
рые контактируют со сложной структурой
23S рРНК, сформированной двумя длинными
петлями РНК. В этой области безоговорочно
ЗАКЛЮЧЕНИЕ
доминируют контакты боковых цепей заряжен
ных аминокислотных остатков лизина и арги
Рассматривая взаимодействие белков с од
нина с атомами сахарофосфатного остова РНК.
ноцепочечными участками РНК, можно отме
Число контактирующих с РНК аминокислот
тить разнообразие белковых мотивов, узнающих
ных остатков во второй области меньше, чем в
РНК. В ряде случаев важным элементом мотива
первой, поэтому было высказано предположе
является β лист, который предоставляет «под
ние, что именно контакты аминокислотных ос
ложку» для РНК. Однако почти во всех случаях
татков домена I белка L1 с 23S рРНК являются
эта поверхность хоть и является местом РНК
определяющими в связывании, а контакты до
белкового контакта, но не играет существенной
мена II с РНК стабилизируют рРНК белковый
роли при дискриминации последовательности
комплекс.
РНК. Её выполняют области, сформированные
Пространственная структура комплексов
близкорасположенными гидрофобными боко
белка L1 с фрагментами мРНК показала причи
выми цепями белка, обеспечивающие позицио
ну существенной разницы в сродстве белка с
нирование определенных оснований. Положи
рРНК и мРНК [111-113]. Участки мРНК, узна
тельно заряженные или полярные боковые цепи
БИОХИМИЯ том 86 вып. 8 2021
10*
1244
НИКУЛИН
белка, выходящие в эти карманы, формируют
короткие участки РНК - от 4 до 6 нуклеотидов.
ионные пары и водородные связи с атомами ос
Такая небольшая область контактов не всегда
нований, тем самым отвечая за специфичность
достаточна для прочного связывания РНК, по
узнавания нуклеотидов. Зачастую такие карма
этому в оцРНК связывающих белках часто ис
ны формируют дополнительные, не входящие в
пользуется повторение (дупликация) РНК свя
состав консервативного мотива, структурные
зывающих доменов. Это позволяет значительно
элементы белка. Специфические карманы могут
увеличить область контакта и повысить ста
формироваться и в отсутствие ββ листа, но
бильность образуемых РНК белковых комплек
принцип позиционирования оснований РНК за
сов, а также разнообразить узнаваемые нуклео
счёт стэкинга с ароматическими остатками бел
тидные последовательности.
ка и узнавания за счёт контактов с полярны
В случае дцРНК связывающих белков про
ми/заряженными боковыми цепями остатков в
исходит узнавание уникальных стабильных
них сохраняется.
пространственных структур РНК. По имею
Рассматривая имеющиеся структуры белков
щимся данным, такие белки имеют заранее
в комплексах со сложными пространственными
сформированную комплементарную область
структурами РНК, можно заметить, что белки
РНК на своей поверхности [116]. Требование
имеют небольшое число контактов с основани
комплементарности областей РНК белковых
ями РНК, а почти все контакты образованы ато
контактов приводит к уникальности дцРНК
мами сахарофосфатного остова. Связано это с
связывающих структур у белков. Исключением
особенностью конформации А спирали двой
являются белки с dsRBD доменом, которые взаи
ной спирали РНК. Она имеет глубокий и закры
модействуют с дцРНК с «выпетливаниями» оди
тый от внешних контактов большой желобок,
ночных оснований. Остальные известные на те
что затрудняет прямые контакты с основаниями
кущий момент времени дцРНК связывающие
дцРНК. Малый желобок А спирали РНК, в
белки «узнают» уникальные пространственные
свою очередь, мелкий и широкий, что благо
структуры РНК, что обеспечивает необходимую
приятно сказывается на доступности нуклеоти
специфичность и сродство между взаимодей
дов, однако это одновременно повышает дос
ствующими молекулами.
тупность потенциальных РНК белковых кон
тактов к внешним воздействиям (воде и ионам)
и не позволяет получить значительный выиг
Благодарности. Автор выражает благодар
рыш в энергии при формировании комплексов.
ность С.В. Тищенко за полезные и конструктив
В результате доминирующими становятся кон
ные обсуждения в ходе создания этой работы, а
такты атомов белка с атомами сахарофосфатно
также С.В. Никонову, М.Б. Гарбер и Н.А. Невс
го остова, однако в таком случае затруднено или
кой за заложение основ этой работы.
невозможно узнавание последовательности
Конфликт интересов. Автор заявляет об отсут
нуклеотидов, а происходит узнавание простран
ствии конфликта интересов в финансовой или
ственной структуры РНК. Это наиболее сущест
какой либо иной сфере.
венное отличие взаимодействия РНК связыва
Соблюдение этических норм. Настоящий об
ющих белков с оцРНК и с дцРНК.
зор не содержит описания выполненных авто
Ещё одной особенностью оцРНК связыва
ром исследований с участием людей или ис
ющих белков является то, что они связывают
пользованием животных в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Fedorov, R., Meshcheryakov, V., Gongadze, G.,
4.
Tishchenko, S., Nikulin, A., Fomenkova, N., Nevskaya, N.,
Fomenkova, N., Nevskaya, N., et al. (2001) Structure of
Nikonov, O., et al. (2001) Detailed analysis of RNA-protein
ribosomal protein TL5 complexed with RNA provides new
interactions within the ribosomal protein S8-rRNA com
insights into the CTC family of stress proteins, Acta
plex from the Archaeon Methanococcus jannaschii, J. Mol.
Crystallogr. Sect. D Biol. Crystallogr.,
57,
968976,
Biol., 311, 311 324, doi: 10.1006/jmbi.2001.4877.
doi: 10.1107/S0907444901006291.
5.
Nikulin, A., Serganov, A., Ennifar, E., Tishchenko, S.,
2.
Perederina, A., Nevskaya, N., Nikonov, O., Nikulin, A.,
Nevskaya, N., et al. (2000) Crystal structure of the
Dumas, P., et al. (2002) Detailed analysis of RNA-protein
S15-rRNA complex, Nat. Struct. Biol., 7, 273277,
interactions within the bacterial ribosomal protein L5/5S
doi: 10.1038/74028.
rRNA complex, RNA, 8, 15481557, doi: 10.1017/
6.
Gabdulkhakov, A., Mitroshin, I., and Garber, M. (2020)
s1355838202029953.
Structure of the ribosomal P stalk base in archaean
3.
Nikulin, A., Eliseikina, I., Tishchenko, S., Nevskaya, N.,
Methanococcus jannaschii, J. Struct. Biol., 211, 107559,
Davydova, N., et al. (2003) Structure of the L1 protuber
doi: 10.1016/j.jsb.2020.107559.
ance in the ribosome, Nat. Struct. Biol., 10, 104 108,
7.
Kostareva, O. S., Nevskaya, N. A., Tishchenko, S. V.,
doi: 10.1038/nsb886.
Gabdulkhakov, A. G., Garber, M. B., and Nikonov, S. V.
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1245
(2018) Influence of nonconserved regions of L1 protuber
the RNA binding domain of the U1A spliceosomal protein
ance of Thermus thermophilus ribosome on the affinity of
complexed with an RNA hairpin, Nature, 372, 432 438,
L1 protein to
23s rRNA, Mol. Biol.,
52,
9195,
doi: 10.1038/372432a0.
doi: 10.1134/S0026893318010090.
24.
Handa, N., Nureki, O., Kurimoto, K., Kim, I.,
8.
Draper, D. E. (1999) Themes in RNA-protein recogni
Sakamoto, H., et al. (1999) Structural basis for recognition
tion, J. Mol. Biol.,
293,
255270, doi:
10.1006/
of the Tra mRNA precursor by the sex lethal protein,
jmbi.1999.2991.
Nature, 398, 579 584, doi: 10.1038/19242.
9.
Corley, M., Burns, M. C., and Yeo, G. W. (2020) How
25.
Afroz, T., Cienikova, Z., Cléry, A., and Allain, F. H. T.
RNA binding proteins interact with RNA: molecules and
(2015) One, two, three, four! How multiple RRMs read the
mechanisms, Mol. Cell, 9 29, doi: 10.1016/j.molcel.
genome sequence, Methods Enzymol.,
235278,
2020.03.011.
doi: 10.1016/bs.mie.2015.01.015.
10.
Iacobuzio Donahue, C. A., Ashfaq, R., Maitra, A., Adsay,
26.
Grishin, N. V. (2001) KH domain: one motif, two folds,
N. V., Shen Ong, G. L., et al. (2003) Highly expressed
Nucleic Acids Res., 29, 638643, doi: 10.1093/nar/
genes in pancreatic ductal adenocarcinomas: a compre
29.3.638.
hensive characterization and comparison of the transcrip
27.
Oddone, A., Lorentzen, E., Basquin, J., Gasch, A.,
tion profiles obtained from three major technologies,
Rybin, V., et al. (2007) Structural and biochemical charac
Cancer Res., 63, 8614 8622, doi: 10.1126/science.1058040.
terization of the yeast exosome component Rrp40, EMBO
11.
Swanson, M. S., Nakagawa, T. Y., LeVan, K., and
Rep., 8, 63 69, doi: 10.1038/sj.embor.7400856.
Dreyfuss, G. (1987) Primary structure of human nuclear
28.
Auweter, S. D., Oberstrass, F. C., and Allain, F. H. T.
ribonucleoprotein particle C proteins: conservation of
(2006) Sequence specific binding of single stranded RNA:
sequence and domain structures in heterogeneous nuclear
is there a code for recognition? Nucleic Acids Res., 34,
RNA, mRNA, and pre rRNA binding proteins, Mol. Cell.
4943 4959, doi: 10.1093/nar/gkl620.
Biol., 7, 1731 1739, doi: 10.1128/MCB.7.5.1731.
29.
Nicastro, G., Taylor, I. A., and Ramos, A. (2015) KH
12.
Sillekens, P. T. G., Habets, W. J., Beijer, R. P., and van
RNA interactions: back in the groove, Curr. Opin. Struct.
Venrooij, W. J. (1987) CDNA cloning of the human U1
Biol., 30, 63 70, doi: 10.1016/j.sbi.2015.01.002.
SnRNA associated A protein: extensive homology between
30.
Lewis, H. A., Musunuru, K., Jensen, K. B., Edo, C.,
U1 and U2 SnRNP specific proteins, EMBO J., 6, 3841
Chen, H., et al. (2000) Sequence specific RNA binding by
3848.
a nova KH domain, Cell, 100, 323 332, doi: 10.1016/
13.
Muto, Y., and Yokoyama, S. (2012) Structural insight into
S0092 8674(00)80668 6.
RNA recognition motifs: versatile molecular lego building
31.
Ryder, S. P., and Massi, F. (2010) Insights into the struc
blocks for biological systems, Wiley Interdisc. Rev. RNA, 3,
tural basis of RNA recognition by star domain proteins,
229 246, doi: 10.1002/wrna.1107.
Adv. Exp. Med. Biol., 693, 37 53, doi: 10.1007/978 1
14.
Messias, A. C., and Sattler, M. (2004) Structural basis of
4419 7005 3_3.
single stranded RNA recognition, Accounts Chem. Res.,
32.
Beuth, B., Pennell, S., Arnvig, K. B., Martin, S. R., and
37, 279 287, doi: 10.1021/ar030034m.
Taylor, I. A. (2005) Structure of a Mycobacterium tubercu&
15.
Cléry, A., Blatter, M., and Allain, F. H. T. (2008) RNA
losis NusA-RNA complex, EMBO J., 24, 3576 3587,
recognition motifs: boring? Not quite, Curr. Opin. Struct.
doi: 10.1038/sj.emboj.7600829.
Biol., 18, 290 298, doi: 10.1016/j.sbi.2008.04.002.
33.
Lunde, B. M., Moore, C., and Varani, G. (2007) RNA
16.
León, B., Kashyap, M. K., Chan, W. C., Krug, K. A.,
binding proteins: modular design for efficient function,
Castro, J. E., et al. (2017) A challenging pie to splice: drug
Nat. Rev. Mol. Cell Biol., 8, 479490, doi: 10.1038/
ging the spliceosome, Angewandte Chemie Int. Edn., 56,
nrm2178.
12052 12063, doi: 10.1002/anie.201701065.
34.
Valverde, R., Edwards, L., and Regan, L. (2008) Structure
17.
Van der Feltz, C., Anthony, K., Brilot, A., and Pomeranz
and function of KH domains, FEBS J., 275, 2712 2726,
Krummel, D. A. (2012) Architecture of the spliceosome,
doi: 10.1111/j.1742 4658.2008.06411.x.
Biochemistry, 51, 3321 3333, doi: 10.1021/bi201215r.
35.
Diakun, G. P., Fairall, L., and Klug, A. (1986) EXAFS
18.
Mazza, C., Segref, A., Mattaj, I. W., and Cusack, S. (2002)
study of the zinc binding sites in the protein transcription
Large scale induced fit recognition of an m(7)GpppG Cap
factor IIIA, Nature, 324, 698 699, doi: 10.1038/324698a0.
analogue by the human nuclear Cap binding complex,
36.
Ginsberg, A. M., King, B. O., and Roeder, R. G. (1984)
EMBO J., 21, 5548 5557, doi: 10.1093/emboj/cdf538.
Xenopus 5S gene transcription factor, TFIIIA: characteri
19.
Calero, G., Wilson, K. F., Ly, T., Rios Steiner, J. L.,
zation of a CDNA clone and measurement of RNA levels
Clardy, J. C., and Cerione, R. A. (2002) Structural basis of
throughout development, Cell,
39,
479489,
M7GpppG Binding to the nuclear Cap binding protein
doi: 10.1016/0092 8674(84)90455 0.
complex, Nat. Struct. Biol., 9, 912 917, doi: 10.1038/
37.
Miller, J., McLachlan, A. D., and Klug, A.
(1985)
nsb874.
Repetitive zinc binding domains in the protein transcrip
20.
Johansson, C., Finger, L. D., Trantirek, L., Mueller, T. D.,
tion factor IIIA from Xenopus oocytes, EMBO J., 4, 1609
Kim, S., et al. (2004) Solution structure of the complex
1614.
formed by the two N terminal RNA binding domains of
38.
Hudson, B. P., Martinez Yamout, M. A., Dyson, H. J.,
nucleolin and a pre rRNA target, J. Mol. Biol., 337, 799
and Wright, P. E. (2004) Recognition of the mRNA au rich
816, doi: 10.1016/j.jmb.2004.01.056.
element by the zinc finger domain of TIS11d, Nat. Struct.
21.
Allain, F. H., Bouvet, P., Dieckmann, T., and Feigon, J.
Mol. Biol., 11, 257 264, doi: 10.1038/nsmb738.
(2000) Molecular basis of sequence specific recognition of
39.
Iwanaga, E., Nanri, T., Mitsuya, H., and Asou, N. (2011)
pre ribosomal RNA by nucleolin, EMBO J., 19, 6870
Mutation in the RNA binding protein TIS11D/ZFP36L2
6881, doi: 10.1093/emboj/19.24.6870.
is associated with the pathogenesis of acute leukemia,
22.
Price, S. R., Evans, P. R., and Nagai, K. (1998) Crystal
Int. J. Oncology, 38, 25 31, doi: 10.3892/ijo_00000820.
structure of the spliceosomal U2B” U2A’ protein complex
40.
Dey, A., York, D., Smalls Mantey, A., and Summers, M. F.
bound to a fragment of U2 small nuclear RNA, Nature,
(2005) Composition and sequence dependent binding of
394, 645 650, doi: 10.1038/29234.
RNA to the nucleocapsid protein of Moloney murine
23.
Oubridge, C., Ito, N., Evans, P. R., Teo, C. H., and
leukemia virus, Biochemistry, 44, 3735 3744, doi: 10.1021/
Nagai, K. (1994) Crystal structure at 1.92 Å resolution of
bi047639q.
БИОХИМИЯ том 86 вып. 8 2021
1246
НИКУЛИН
41.
D’Souza, V., and Summers, M. F. (2004) Structural basis
human spliceosomal U1 SnRNP at 5.5 Å resolution,
for packaging the dimeric genome of Moloney murine
Nature, 458, 475 480, doi: 10.1038/nature07851.
leukaemia virus, Nature, 431, 586 590, doi: 10.1038/
59.
Yong, J., Golembe, T. J., Battle, D. J., Pellizzoni, L., and
nature02944.
Dreyfuss, G. (2004) SnRNAs contain specific SMN bind
42.
Wang, X., McLachlan, J., Zamore, P. D., and Hall,
ing domains that are essential for SnRNP assembly, Mol.
T. M. T. (2002) Modular recognition of RNA by a human
Cell. Biol., 24, 2747 2756, doi: 10.1128/MCB.24.7.2747
pumilio homology domain, Cell,
110,
501512,
2756.2004.
doi: 10.1016/s0092 8674(02)00873 5.
60.
Kondo, Y., Oubridge, C., van Roon, A. M. M., and
43.
De Moor, C. H., Meijer, H., and Lissenden, S. (2005)
Nagai, K. (2015) Crystal structure of human U1 SnRNP, a
Mechanisms of translational control by the 3′ UTR in
small nuclear ribonucleoprotein particle, reveals the mech
development and differentiation, Semin. Cell Dev. Biol, 16,
anism of 5′ splice site recognition, eLife, 4, doi: 10.7554/
49 58, doi: 10.1016/j.semcdb.2004.11.007.
eLife.04986
44.
Wickens, M., Bernstein, D. S., Kimble, J., and Parker, R.
61.
Mura, C., Randolph, P. S., Patterson, J., and Cozen, A. E.
(2002) A PUF family portrait: 3′UTR regulation as a way
(2013) Archaeal and eukaryotic homologs of Hfq, RNA
of life, Trends Genet. TIG, 18, 150 157, doi: 10.1016/
Biol., 10, 636 651, doi: 10.4161/rna.24538.
s0168 9525(01)02616 6.
62.
He, W., and Parker, R. (2000) Functions of Lsm proteins in
45.
Mackay, J. P., Font, J., and Segal, D. J. (2011) The prospects
mRNA degradation and splicing, Curr. Opin. Cell Biol., 12,
for designer single stranded RNA binding proteins, Nat.
346 350, doi: 10.1016/S0955 0674(00)00098 3.
Struct. Mol. Biol., 18, 256 261, doi: 10.1038/nsmb.2005.
63.
Sharif, H., and Conti, E. (2013) Architecture of the Lsm1
46.
Cheong, C. G., and Hall, T. M. T. (2006) Engineering
7 Pat1 complex: a conserved assembly in eukaryotic
RNA sequence specificity of pumilio repeats, Proc. Natl.
mRNA turnover, Cell Rep., 5, 283 291, doi: 10.1016/j.celrep.
Acad. Sci. USA, 103, 13635 13639, doi: 10.1073/pnas.
2013.10.004.
0606294103
64.
Sauer, E. (2013) Structure and RNA binding properties of
47.
Antson, A. A., Dodson, E. J., Dodson, G., Greaves, R. B.,
the bacterial LSm protein Hfq, RNA Biol., 10, 610 618,
Chen, X., and Gollnick, P. (1999) Structure of the Trp
doi: 10.4161/rna.24201.
RNA binding attenuation protein, TRAP, bound to RNA,
65.
Мурина В. Н., Никулин А. Д. (2011) РНК связываю
Nature, 401, 235 242, doi: 10.1038/45730..
щие Sm подобные белки бактерий и архей: сходство и
48.
Babitzke, P. (1997) Regulation of tryptophan biosynthesis:
различие структур и функций, Усп. Биол. Химии, 51,
Trp Ing the TRAP or how Bacillus subtilis reinvented the
133 164.
wheel, Mol. Microbiol., 26, 1 9, doi: 10.1046/j.1365
66.
Schumacher, M. A., Pearson, R. F., Møller, T., Valentin
2958.1997.5541915.x.
Hansen, P., and Brennan, R. G. (2002) Structures of the
49.
Elliott, M. B., Gottlieb, P. A., and Gollnick, P. (1999) Probing
pleiotropic translational regulator Hfq and an Hfq-RNA
the TRAP RNA interaction with nucleoside analogs, RNA, 5,
complex: a bacterial sm like protein, EMBO J., 21, 3546
1277 1289, doi: 10.1017/s1355838299991057.
3556, doi: 10.1093/emboj/cdf322.
50.
Theobald, D. L., Mitton Fry, R. M., and Wuttke, D. S.
67.
Link, T. M., Valentin Hansen, P., and Brennan, R. G.
(2003) Nucleic acid recognition by OB fold proteins,
(2009) Structure of Escherichia coli Hfq bound to polyri
Annu. Rev. Biophys. Biomol. Struct.,
32,
115133,
boadenylate RNA, Proc. Natl. Acad. Sci. USA, 106, 19292
doi: 10.1146/annurev.biophys.32.110601.142506.
19297, doi: 10.1073/pnas.0908744106.
51.
Skordalakes, E., and Berger, J. M. (2003) Structure of the
68.
Vogel, J., and Luisi, B. F. (2011) Hfq and its constellation
Rho transcription terminator, Cell,
114,
135146,
of RNA, Nat. Rev. Microbiol., 9, 578 589, doi: 10.1038/
doi: 10.1016/S0092 8674(03)00512 9.
nrmicro2615.
52.
Bogden, C. E., Fass, D., Bergman, N., Nichols, M. D.,
69.
Someya, T., Baba, S., Fujimoto, M., Kawai, G.,
and Berger, J. M. (1999) The structural basis for terminator
Kumasaka, T., and Nakamura, K. (2012) Crystal structure
recognition by the Rho transcription termination factor,
of Hfq from Bacillus subtilis in complex with SELEX
Mol. Cell, 3, 487 493, doi: 10.1016/S1097 2765(00)80476 1.
derived RNA aptamer: insight into RNA binding proper
53.
Keto Timonen, R., Hietala, N., Palonen, E.,
ties of bacterial Hfq, Nucleic Acids Res., 40, 1856 1867,
Hakakorpi, A., Lindström, M., and Korkeala, H. (2016)
doi: 10.1093/nar/gkr892.
Cold shock proteins: a minireview with special emphasis
70.
Masliah, G., Barraud, P., and Allain, F. H. T. (2012) RNA
on Csp family of enteropathogenic yersinia, Front.
recognition by double stranded RNA binding domains: a
Microbiol., 7, doi: 10.3389/fmicb.2016.01151.
matter of shape and sequence, Cell. Mol. Life Sci., 70,
54.
Horn, G., Hofweber, R., Kremer, W., and Kalbitzer, H. R.
doi: 10.1007/s00018 012 1119 x.
(2007) Structure and function of bacterial cold shock pro
71.
Gleghorn, M. L., and Maquat, L. E. (2014) ‘Black sheep’
teins, Cell. Mol. Life Sci., 64, 1457 1470, doi: 10.1007/
that don’t leave the double stranded RNA binding domain
s00018 007 6388 4.
fold, Trends Biochem. Sci., 39, 328 340, doi: 10.1016/
55.
Amir, M., Kumar, V., Dohare, R., Islam, A., Ahmad, F.,
j.tibs.2014.05.003.
and Hassan, M. I. (2018) Sequence, structure and evolu
72.
St Johnston, D., Brown, N. H., Gall, J. G., and Jantsch,
tionary analysis of cold shock domain proteins, a member
M. (1992) A conserved double stranded RNA binding
of OB Fold family, J. Evol. Biol.,
31,
19031917,
domain, Proc. Natl. Acad. Sci. USA, 89, 10979 10983,
doi: 10.1111/jeb.13382.
doi: 10.1073/pnas.89.22.10979.
56.
Lyabin, D. N., Eliseeva, I. A., and Ovchinnikov, L. P.
73.
Ryter, J. M., and Schultz, S. C. (1998) Molecular basis of
(2014) YB 1 protein: functions and regulation, RNA, 5, 95
double stranded RNA-protein interactions: structure of a
110, doi: 10.1002/wrna.1200.
DsRNA binding domain complexed with DsRNA,
57.
Hermann, H., Fabrizio, P., Raker, V. A., Foulaki, K.,
EMBO J., 17, 7505 7513, doi: 10.1093/emboj/17.24.7505.
Hornig, H., et al. (1995) SnRNP Sm proteins share two
74.
Stefl, R., Xu, M., Skrisovska, L., Emeson, R. B., and
evolutionarily conserved sequence motifs which are
Allain, F. H. T. (2006) Structure and specific RNA binding
involved in sm protein-protein interactions, EMBO J., 14,
of ADAR2 double stranded RNA binding motifs,
2076 2088.
Structure, 14, 345 355, doi: 10.1016/j.str.2005.11.013.
58.
Pomeranz Krummel, D. A., Oubridge, C., Leung,
75.
Spillmann, S., Dohme, F., and Nierhaus, K. H. (1977)
A. K. W., Li, J., and Nagai, K. (2009) Crystal structure of
Assembly in vitro of the 50 S subunit from Escherichia coli
БИОХИМИЯ том 86 вып. 8 2021
ПРИНЦИПЫ РНК БЕЛКОВОГО УЗНАВАНИЯ
1247
ribosomes: proteins essential for the first heat dependent
91. Yates, J. L., Arfsten, A. E., and Nomura, M. (1980) In vitro
conformational change, J. Mol. Biol., 115, 513523,
expression of Escherichia coli ribosomal protein genes:
doi: 10.1016/0022 2836(77)90168 1.
autogenous inhibition of translation, Proc. Natl. Acad. Sci.
76.
Held, W. A., and Nomura, M. (1973) Structure and func
USA, 77, 1837 1841, doi: 10.1073/pnas.77.4.1837.
tion of bacterial ribosomes. XX. Rate determining step in
92. Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L.,
the reconstitution of Escherichia coli 30S ribosomal sub
and Nomura, M. (1988) Translational regulation of the spc
units, Biochemistry,
12,
32733281, doi:
10.1021/
operon in Escherichia coli, J. Mol. Biol., 204, 309 325,
bi00741a020.
doi: 10.1016/0022 2836(88)90578 5.
77.
Никулин А. Д. (2002) Изучение взаимодействий рибо
93. Olins, P. O., and Nomura, M. (1981) Translational regula
сомных белков с рибосомными РНК, Усп. Биол. Хи&
tion by ribosomal protein S8 in Escherichia coli: structural
мии, 42, 61 88.
homology between rRNA binding site and feedback target
78.
Никулин, А. Д. (2018) Структурные особенности узна
on mRNA, Nucleic Acids Res., 9, 1757 1764, doi: 10.1093/
вания рибосомных РНК рибосомными белками, Усп.
nar/9.7.1757.
Биол. Химии, 58, 241 284.
94. Merianos, H. J., Wang, J., and Moore, P. B. (2004) The
79.
Ban, N., Beckmann, R., Cate, J. H., Dinman, J. D.,
structure of a ribosomal protein S8/Spc operon mRNA
Dragon, F., et al. (2014) A new system for naming riboso
complex, RNA, 10, 954 964, doi: 10.1261/rna.7030704.
mal proteins, Curr. Opin. Struct. Biol., 24, 165169,
95. Agalarov, S. C., Sridhar Prasad, G., Funke, P M., Stout,
doi: 10.1016/j.sbi.2014.01.002.
C. D., and Williamson, J. R. (2000) Structure of the
80.
Held, W. A., Ballou, B., Mizushima, S., and Nomura, M.
S15,S6,S18 rRNA complex: assembly of the 30S ribosome
(1974) Assembly mapping of 30 S ribosomal proteins from
central domain, Science, 288, 107 113, doi: 10.1126/
Escherichia coli. Further studies, J. Biol. Chem., 249, 3103
science.288.5463.107.
3111.
96. Duss, O., Stepanyuk, G. A., Grot, A., O’Leary, S. E.,
81.
Gregory, R. J., Zeller, M. L., Thurlow, D. L., Gourse,
Puglisi, J. D., and Williamson, J. R. (2018) Real time
R. L., Stark, M. J. R., et al. (1984) Interaction of ribosomal
assembly of ribonucleoprotein complexes on nascent RNA
proteins S6, S8, S15 and S18 with the central domain of 16
transcripts, Nat. Commun., 9, 5087, doi: 10.1038/s41467
S ribosomal RNA from Escherichia coli, J. Mol. Biol., 178,
018 07423 3.
287 302, doi: 10.1016/0022 2836(84)90145 1.
97. Portier, C., Dondon, L., and Grunberg Manago, M.
82.
Mougel, M., Eyermann, F., Westhof, E., Romby, P.,
(1990) Translational autocontrol of the Escherichia coli
Expert Bezançon, A., et al. (1987) Binding of Escherichia
ribosomal protein S15, J. Mol. Biol., 211, 407414,
coli ribosomal protein S8 to 16 S rRNA. A model for the
doi: 10.1016/0022 2836(90)90361 O.
interaction and the tertiary structure of the RNA binding
98. Portier, C., Philippe, C., Dondon, L., Grunberg
site, J. Mol. Biol., 198, 91107, doi: 10.1016/0022
Manago, M., Ebel, J. P., et al. (1990) Translational control
2836(87)90460 8.
of ribosomal protein S15, Biochim. Biophys. Acta Gene Struct.
83.
Svensson, P., Changchien, L. M., Craven, G. R., and
Express., 1050, 328 336, doi: 10.1016/0167 4781(90)90190 D.
Noller, H. F. (1988) Interaction of ribosomal proteins, S6,
99. Philippe, C., Eyermann, F., Benard, L., Portier, C.,
S8, S15 and S18 with the central domain of 16 S ribosomal
Ehresmann, B., and Ehresmann, C. (1993) Ribosomal
RNA, J. Mol. Biol., 200, 301 308, doi: 10.1016/0022
protein S15 from Escherichia coli modulates its Own trans
2836(88)90242 2.
lation by trapping the ribosome on the mRNA initiation
84.
Chen, S. S., Sperling, E., Silverman, J. M., Davis, J. H.,
loading site, Proc. Natl. Acad. Sci. USA, 90, 4394 4398,
and Williamson, J. R. (2012) Measuring the dynamics of
doi: 10.1073/pnas.90.10.4394.
E. coli ribosome biogenesis using pulse labeling and quan
100. Philippe, C., Bénard, L., Eyermann, F., Cachia, C.,
titative mass spectrometry, Mol. bioSystems, 8, 3325 3334,
Kirillov, S. V, et al. (1994) Structural elements of RpsO
doi: 10.1039/c2mb25310k.
mRNA involved in the modulation of translational initia
85.
Menichelli, E., Edgcomb, S. P., Recht, M. I., and
tion and regulation of E. coli ribosomal protein S15,
Williamson, J. R. (2012) The structure of aquifex aeolicus
Nucleic Acids Res., 22, 2538 2546, doi: 10.1093/nar/
ribosomal protein S8 reveals a unique subdomain that con
22.13.2538.
tributes to an extremely tight association with 16S rRNA,
101. Mougel, M., Philippe, C., Ebel, J. P., Ehresmann, B., and
J. Mol. Biol., 415, 489 502, doi: 10.1016/j.jmb.2011.10.046.
Ehresmann, C. (1988) The E. coli 16S rRNA binding site of
86.
Geyl, D., Böck, A., and Wittmann, H. G. (1977) Cold
ribosomal protein S15: higher order structure in the
sensitive growth of a mutants of Escherichia coli with an
absence and in the presence of the protein, Nucleic Acids
altered ribosomal protein S8: analysis of revertants, Mol.
Res., 16, 2825 2839.
Gen. Genet., 152, 331 336, doi: 10.1007/BF00693088.
102. Serganov, A., Ennifar, E., Portier, C., Ehresmann, B., and
87.
Powers, T., and Noller, H. F. (1995) Hydroxyl radical foot
Ehresmann, C. (2002) Do mRNA and rRNA binding sites
printing of ribosomal proteins on 16S rRNA, RNA, 1, 194
of E. coli ribosomal protein S15 share common structural
209.
determinants? J. Mol. Biol., 320, 963 978, doi: 10.1016/
88.
Moine, H., Cachia, C., Westhof, E., Ehresmann, B., and
S0022 2836(02)00553 3.
Ehresmann, C. (1997) The RNA binding site of S8 riboso
103. Serganov, A., Polonskaia, A., Ehresmann, B.,
mal protein of Escherichia coli: selex and hydroxyl radical
Ehresmann, C., and Patel, D. J. (2003) Ribosomal protein
probing studies, RNA, 3, 255 268.
S15 represses its own translation via adaptation of an
89.
Kalurachchi, K., Uma, K., Zimmermann, R. A., and
rRNA like fold within its mRNA, EMBO J., 22, 1898
Nikonowicz, E. P. (1997) Structural features of the binding
1908, doi: 10.1093/emboj/cdg170.
site for ribosomal protein S8 in Escherichia coli 16S rRNA
104. Springer, M., and Portier, C. (2003) More than one way to
defined using NMR spectroscopy, Proc. Natl. Acad. Sci.
skin a cat: translational autoregulation by ribosomal pro
USA, 94, 2139 2144, doi: 10.1073/pnas.94.6.2139.
tein S15, Nat. Struct. Biol., 10, 420 422, doi: 10.1038/
90.
Gregory, R. J., Cahill, P. B. F., Thurlow, D. L., and
nsb0603 420.
Zimmermann, R. A. (1988) Interaction of Escherichia coli
105. Mathy, N., Pellegrini, O., Serganov, A., Patel, D. J.,
ribosomal protein S8 with its binding sites in ribosomal
Ehresmann, C., and Portier, C. (2004) Specific recognition
RNA and messenger RNA, J. Mol. Biol., 204, 295 307,
of RpsO mRNA and 16S rRNA by Escherichia coli riboso
doi: 10.1016/0022 2836(88)90577 3.
mal protein S15 relies on both mimicry and site differenti
БИОХИМИЯ том 86 вып. 8 2021
1248
НИКУЛИН
ation, Mol. Microbiol., 52, 661 675, doi: 10.1111/j.1365
112. Nevskaya, N., Tishchenko, S., Volchkov, S.,
2958.2004.04005.x.
Kljashtorny, V., Nikonova, E., et al. (2006) New insights
106. Ehresmann, C., Ehresmann, B., Ennifar, E., Dumas, P.,
into the interaction of ribosomal protein L1 with RNA,
Garber, M., et al. (2004) Molecular mimicry in transla
J. Mol. Biol., 355, 747 759, doi: 10.1016/j.jmb.2005.10.
tional regulation: the case of ribosomal protein S15, RNA
084.
Biol., 1, 66 73, doi: 10.4161/rna.1.1.958.
113. Tishchenko, S., Nikonova, E., Nikulin, A., Nevskaya, N.,
107. Nikonov, S., Nevskaya, N., Eliseikina, I., Fomenkova, N.,
Volchkov, S., et al. (2006) Structure of the ribosomal pro
Nikulin, A., et al. (1996) Crystal structure of the RNA
tein L1-mRNA complex at 2.1 Å resolution: common fea
binding ribosomal protein L1 from Thermus thermophilus,
tures of crystal packing of L1-RNA complexes, Acta
EMBO J., 15, 1350 1359.
Crystallogr. Sect. D Biol. Crystallogr., 62, 15451554,
108. Brot, N., Caldwell, P., and Weissbach, H.
(1981)
doi: 10.1107/S0907444906041655.
Regulation of synthesis of Escherichia coli ribosomal pro
114. Tishchenko, S., Nikonova, E., Kljashtorny, V.,
teins L1 and L11, Arch. Biochem. Biophys., 206, 51 53,
Kostareva, O., Nevskaya, N., et al. (2007) Domain I of
doi: 10.1016/0003 9861(81)90064 3.
ribosomal protein L1 is sufficient for specific RNA bind
109. Yates, J. (1981) Feedback regulation of ribosomal protein
ing, Nucleic Acids Res., 35, 738995, doi: 10.1093/
synthesis in E. coli: localization of the mRNA target sites
nar/gkm898.
for repressor action of ribosomal protein L1, Cell, 24, 243
115. Tishchenko, S., Kostareva, O., Gabdulkhakov, A.,
249, doi: 10.1016/0092 8674(81)90520 1.
Mikhaylina, A., Nikonova, E., et al. (2015) Protein-RNA
110. Tishchenko, S., Nikonova, E., Kostareva, O.,
affinity of ribosomal protein L1 mutants does not correlate
Gabdulkhakov, A., Piendl, W., et al. (2011) Structural
with the number of intermolecular interactions, Acta
analysis of interdomain mobility in ribosomal L1 proteins,
Crystallogr. Sect. D Biol. Crystallogr.,
71,
376386,
Acta Crystallograp.
Sect.
D,
67,
10231027,
doi: 10.1107/S1399004714026248.
doi: 10.1107/S0907444911043435.
116. Nevskaya, N. A., Nikonov, O. S., Revtovich, S. V., Garber,
111.
Nevskaya, N. (2005) Ribosomal protein L1 recognizes the
M. B., and Nikonov, S. V. (2004) Identification of RNA
same specific structural motif in its target sites on the
recognizing modules on the surface of ribosomal proteins,
autoregulatory mRNA and 23S rRNA, Nucleic Acids Res.,
Mol. Biol.,
38,
789798, doi:
10.1023/B:MBIL.
33, 478 485, doi: 10.1093/nar/gki194.
0000043948.74962.05.
CHARACTERISTIC FEATURES OF PROTEIN INTERACTIONS
WITH SINGLE' AND DOUBLE'STRANDED RNA
Review
A. D. Nikulin
Institute of Protein Research Russian Academy of Sciences,
142290 Pushchino, Russia; E&mail: nikulin@vega.protres.ru
The review discusses the difference between the specific interaction (recognition) of proteins with single stranded and
double stranded RNA based on the structures of the RNA protein complexes. It has been shown that in the case of
single stranded RNAs, proteins are in contact with RNA bases, which makes it possible to recognize a specific
nucleotide sequence. In the case of double stranded RNAs, the formation of the contacts is difficult and proteins rec
ognize the unique conformation of the RNA spatial structure, interacting mainly with the RNA sugar phosphate
backbone.
Keywords: RNA-protein interaction, single stranded RNA, double stranded RNA, RNP, ribosomal proteins
БИОХИМИЯ том 86 вып. 8 2021