БИОХИМИЯ, 2021, том 86, вып. 9, с. 1328 - 1344
УДК 577.21
РАЗЛИЧИЯ И СХОДСТВО ПРОЦЕССОВ ТЕРМИНАЦИИ
ТРАНСЛЯЦИИ И СПАСЕНИЯ РИБОСОМЫ В БАКТЕРИАЛЬНЫХ
КЛЕТКАХ И В МИТОХОНДРИЯХ И ЦИТОПЛАЗМЕ
ЭУКАРИОТИЧЕСКИХ КЛЕТОК
Обзор
© 2021
А.A. Коростелев
RNA Therapeutics Institute, Department of Biochemistry and Molecular Pharmacology, UMass Medical School,
Worcester, MA, USA; e mail: Andrei.Korostelev@umassmed.edu
Поступила в редакцию 24.05.2021
После доработки 24.05.2021
Принята к публикации 17.06.2021
Когда рибосома встречает стоп кодон на молекуле мРНК, она прекращает процесс трансляции, высвобож
дает вновь синтезированный белок и разбирается на субъединицы, чтобы инициировать процесс трансля
ции на новой мРНК. Терминация трансляции является высоко динамичным процессом, в котором рили
зинг факторы (RF1 и RF2 у бактерий и eRF1•eRF3•ГТФ у эукариот) координируют высвобождение пеп
тида с крупномасштабными молекулярными перестройками рибосомы. Рибосомы, остановившиеся на
аберрантных мРНК, освобождаются и рециклизуются различными бактериальными, митохондриальными
или цитоплазматическими механизмами контроля качества. Эти механизмы работают при участии факто
ров спасения, обладающих пептидил тРНК гидролазной активностью (бактериальные факторы ArfA•RF2
и ArfB, митохондриальные факторы ICT1 и mtRF R и цитоплазматический фактор Vms1), которые отлича
ются друг от друга и от рилизинг факторов. Тем не менее в результате проведённых недавно структурных ис
следований было показано замечательное сходство между механизмами терминации трансляции и спасе
ния рибосомы. В этом обзоре описывается, как в этих механизмах используется внутренняя динамика ри
босомы, подчёркивая активную роль рибосомы на всех этапах процесса трансляции.
КЛЮЧЕВЫЕ СЛОВА: трансляция, терминация, рибосома, спасение.
DOI: 10.31857/S0320972521090062
ВВЕДЕНИЕ
заменимые факторы aRF3/eRF3 у архей/эука
риот. Показано, что они выполняют различные
Трансляция мРНК в белок начинается со
функции в этих доменах жизни. Понимание
стартового кодона (обычно кодона AUG) и за
подробных структурных механизмов термина
канчивается одним из трёх стоп кодонов: UAA,
ции трансляции имеет большое значение в свя
UAG или UGA [1, 2]. Для своевременного био
зи с фундаментальной ролью процесса термина
синтеза белков, рециклизации рибосомы и ини
ции в биосинтезе белка, проиллюстрированной
циации трансляции необходим точный и эф
катастрофическими последствиями мутаций
фективный процесс терминации [3, 4]. В отли
или других изменений структуры мРНК, кото
чие от смысловых кодонов, которые распозна
рые приводят к аберрантному высвобождению.
ются различными аминоацил тРНК, стоп ко
Например, большая часть генетических наруше
доны распознаются рилизинг факторами: RF1
ний вызвана преждевременными стоп кодона
или RF2 у бактерий и aRF1/eRF1 у архей/эука
ми, приводящими к образованию укороченных
риот [5-9]. Кроме того, в процессе терминации
белков и деградации мРНК [10].
принимают участие рилизинг факторы с
Кроме рилизинг факторов, в бактериальных
ГТФазной активностью, такие как несущест
и эукариотических митохондриальных и цито
венный фактор RF3 у некоторых бактерий и не
плазматических системах трансляции недавно
были идентифицированы различные RF подоб
ные белки, которые могут смягчать трансляци
Принятые сокращения: крио ЭМ - криогенная
онный стресс. В их число входят механизмы
электронная микроскопия; DC - центр декодирования;
H69 - 23S спираль 69; PTC - пептидил трансферазный
спасения рибосом и контроля качества, которые
центр; RF - рилизинг фактор.
способствуют рециклизации рибосом, остано
1328
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1329
вившихся на аберрантных молекулах мРНК.
ТЕРМИНАЦИЯ ТРАНСЛЯЦИИ
Последние включают укороченные мРНК и
У БАКТЕРИЙ И МИТОХОНДРИЙ
мРНК с неоптимальными (редкими) кодонами
или структурными особенностями (например,
Рилизинг факторы являются бифункцио
вторичной структурой мРНК или сайтами свя
нальными белками: они распознают стоп кодон
зывания с белками), которые предотвращают
и катализируют гидролиз сложноэфирной связи
процесс элонгации рибосом. Распознавание,
в пептидил тРНК. Бактериальные рилизинг
высвобождение пептида и рециклинг останов
факторы RF1/RF2 независимо распознают ко
ленных рибосом достигается независимыми пу
доны UAA, UAG/UAA и UGA соответственно.
тями, включая участие ArfA и ArfB у бактерий,
Эти белки длиной ~350-380 а.о. включают су
mtRF1a и mtRF R в митохондриях и Dom34 и
пердомен распознавания кодонов (домены 2
Vms1 в цитоплазме эукариот. Недавние биофи
и 4), который связывается с малой 30S субъеди
зические и структурные исследования продемон
ницей рибосомы, и каталитический домен 3, ко
стрировали ключевую роль динамики рибосомы
торый связывается с большой 50S субъедини
в процессах трансляции, терминации и спасе
цей рибосомы (рис. 1, a-c). Подвижный домен 1
ния самой рибосомы. Механика перестройки
взаимодействует с периферическим L11 высту
рибосом перекликается с моделями, предло
пом на 50S субъединице (рис. 1, b-c), способ
женными Александром Спириным для трансло
ствуя связыванию RF с рибосомой [13, 14]. Су
кации рибосом вдоль мРНК, включая крупно
пердомен распознавания кодонов содержит
масштабные перестройки субъединиц рибосо
консервативный мотив P184XT186 (где X часто
мы [11, 12]. Эти данные подчёркивают, что ри
представлен A, V или другой короткой амино
босома играет основную механическую роль на
кислотой) в RF1 или мотив S206PF208 в RF2 [15,
всех этапах трансляции, включая терминацию и
16], которые взаимодействуют со стоп кодона
спасение рибосом.
ми (для бактериальных белков и РНК приводит
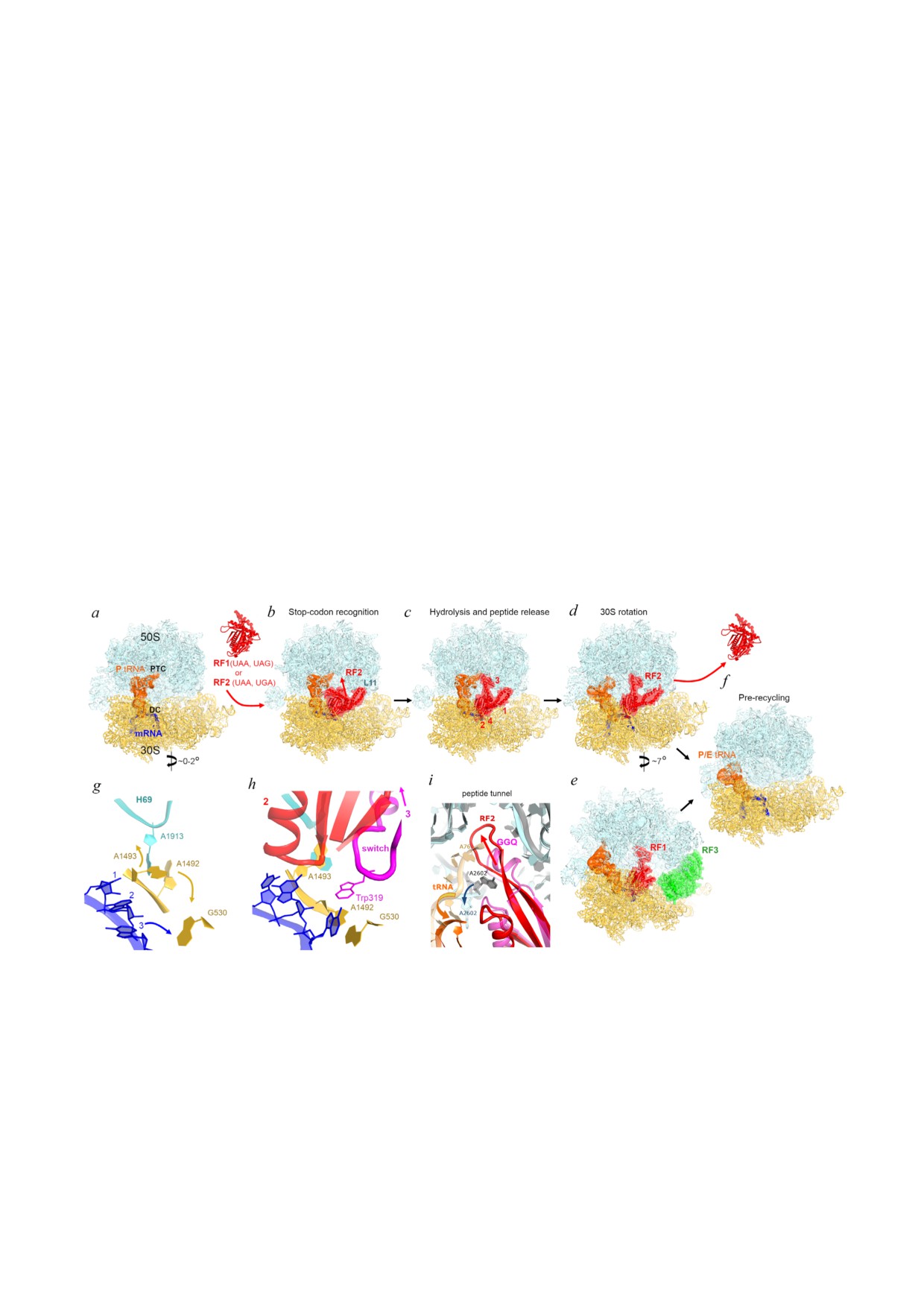
Рис. 1. Терминация трансляции у бактерий происходит вследствие крупномасштабных конформационных изменений.
a-d - Крио ЭМ структуры, демонстрирующие перестройки рилизинг факторов и межсубъединичное вращение бакте
риальной 70S рибосомы после распознавания стоп кодона и высвобождения пептида. Показан белок RF2 E. coli: пане
ли (b и c) [51] и d [61]. Центр декодирования (DC) и пептидил трансферазный центр (PTC) отмечены на панели (a). Крис
таллическая структура свободного RF2 показана между панелями (a) и (b) [40]. Доменная организация RF1/RF2 показа
на арабскими цифрами на панели (c). e - Крио ЭМ структура RF1 и RF3 на рибосоме E. coli с P/E тРНК и повёрнутой
30S субъединицей [58]. f - Крио ЭМ структура рибосомы E. coli до рециклинга с P/E тРНК и повёрнутой 30S субъеди
ницей после диссоциации факторов высвобождения [61]. g и h - Перемещение кодона и DC после связывания
RF2 (Структура II в [61]). Стоп кодон и DC на панели g были смоделированы на основе Структуры I в [54]. i - Переме
щение RF2 в PTC [61]. Каталитическая конформация RF2 с GGQ несущей α спиралью показана розовым цветом, и ри
босома окрашена в серый цвет. Конформация β шпильки RF2 (красный) совпадает с перемещением A2602 и отходом
тРНК (оранжевый) из PTC (сине зеленый)
6 БИОХИМИЯ том 86 вып. 9 2021
1330
КОРОСТЕЛЕВ
ся цифровое обозначение остатков, характерное
на (рис. 1, a и b). Однако в ранних структурных
для Escherichia coli, если не указано иное)
исследованиях было показано, что в рибосоме
[17-20]. В биохимических исследованиях и с
RF находятся в раздвинутой («открытой») кон
помощью мутационного анализа были установ
формации (рис. 1, c), в которой RF1 или RF2 в
лены другие аминокислотные остатки домена 2,
A сайте рибосомы соединяют 30S и 50S субъ
имеющие важное значение для обеспечения
единицы [17-20 43, 44]. На основании этих ре
специфичности связывания рилизинг факторов
зультатов была предложена привлекательная ги
со стоп кодонами [21, 22].
потеза о том, что рилизинг факторы выбирают
Каталитический домен 3 содержит длинную
А сайт рибосомы, находясь в компактной кон
α спираль, заканчивающуюся универсально
формации, при которой мотив GGQ располага
консервативным мотивом GGQ (рис. 1, c и 2, c),
ется далеко от PTC. Было высказано предполо
который, как было показано в ранних исследо
жение, что «открытие» рилизинг фактора про
ваниях, играет ключевую роль в катализе [23,
исходит после распознавания стоп кодона [17,
24]. Замены глицина в бактериальных и эукарио
43, 45-47]. Структурные доказательства такого
тических рилизинг факторах вызывали их
перехода были получены лишь недавно. Крис
инактивацию [24-26]. Напротив, замены остат
таллографические исследования со сверхточ
ков глутамина RF не влияли на каталитическую
ным мутантом RF1 и антибиотиком бластици
активность [25, 27, 28], за исключением мутант
дином S (BlaS) зафиксировали кодон распозна
ных форм с мотивом GGP, которые были неак
ющий супердомен в А сайте, в то время как ка
тивными [18, 29]. Эти исследования показыва
талитический домен был в неупорядоченном
ют, что уникальная конформация GGQ очень
состоянии в межсубъединичном пространстве в
важна для проявления каталитической актив
связи с динамическими перестройками рили
ности рилизинг факторов.
зинг факторов на рибосоме [48]. Последующие
Кристаллографические исследования высо
крио ЭМ исследования визуализировали ком
кого разрешения позволили получить подроб
пактный RF2 на рибосоме, связанный с альтер
ную картину процесса распознавания стоп ко
нативным рилизинг фактором A (ArfA; [49]), и
донов и каталитического механизма действия
мутантом ArfA [50], что подчёркивает сродство
рилизинг факторов на все три стоп кодо
компактизованных рилизинг факторов к рибо
на [17-20] (см. обзоры [30-34]). Последние дос
соме (рис. 2, a). ArfA помогает RF2 распознавать
тижения в области криогенной электронной
рибосомы без стоп кодонов (обсуждается ни
микроскопии (крио ЭМ) позволяют определять
же), поэтому структурная динамика RF на тер
ранее недостижимые переходные состояния
минирующей рибосоме оставалась неясной до
макромолекул с разрешением, близким к атом
недавнего исследования крио ЭМ в реальном
ному. Вместе с результатами биохимических и
времени [51]. В этом исследовании удалось по
биофизических исследований [35-38], недав
лучить как RF1, так и RF2 в компактной неак
ние структурные исследования позволяют поч
тивной конформации в ранние моменты реак
ти полностью реконструировать динамический
ции терминации (рис. 1, b). Они перестроились
механизм терминации - от начального распоз
в каталитически активную открытую конформа
навания стоп кодона до гидролиза пептидил
цию в более поздние моменты времени (рис. 1, c;
тРНК и диссоциации RF.
[51]). Открытие RF сопряжено с локальной пе
Динамичные рилизинг-факторы имеют важное
рестройкой DC рибосомы (т.е., А сайта 30S
значение для правильной терминации трансляции.
субъединицы) и PTC на 50S субъединице, что
В клетке RF могут стохастически связываться с
совместно обеспечивает высокую точность тер
А сайтом рибосомы, содержащим смысловой
минации трансляции.
или стоп кодон. Для точности терминации гид
Локальные перестройки в центре декодирова-
ролазная функция должна быть строго коорди
ния и пептидил-трансферазном центре. DC обра
нирована с распознаванием стоп кодона. Эта
зован универсально консервативными остатка
координация достигается путем предотвраще
ми малой рибосомной РНК (G530, A1493 и
ния вставки каталитического домена 3 в пепти
A1493 16S рРНК E. coli), которые определяют
дил трансферазный центр (PTC), если только
точность декодирования мРНК. Во время элон
супердомен распознавания кодонов не разме
гации эти нуклеотиды стабилизируют спарива
щён в A сайте, содержащем стоп кодон. Крис
ние оснований Уотсона-Крика между кодоном
таллические структуры и исследования в раст
мРНК и антикодоном тРНК и обеспечивают
ворах показали, что свободные рилизинг фак
точность элонгации [52-54]. Эти нуклеотиды
торы принимают компактную конформа
также необходимы во время терминации, по
цию [39-42], при которой каталитический до
скольку они способствуют открытию рилизинг
мен упакован в домен распознавания кодо
фактора для катализа высвобождения пепти
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1331
Рис. 2. Механизмы спасения и высвобождения рибосом с участием бактериальных факторов ArfA и ArfB и митохондри
альных факторов mtRF1a, ICT1 и mtRF R. a и b - Крио ЭМ структуры рибосом E. coli с укороченной мРНК, ArfA и ком
пактным (a) или развёрнутым (b) RF2 [49]. c - Крупный план взаимодействий между ArfA, RF2, мРНК и тРНК в Р сайте
рибосомы. d-g - Крио ЭМ структуры рибосом E. coli с укороченной мРНК (с выступом длиной 0 или 9 нуклеотидов пос
ле кодона P сайта) и ArfB [110, 111]. h - Крио ЭМ структура терминирующего комплекса 55S митохондрий свиньи с
mtRF1a, связанным со стоп кодоном [87], очень напоминает бактериальные терминальные комплексы с RF1. i - Крио
ЭМ структура митохондриального комплекса спасения 55S с ICT1 [87] очень напоминает бактериальный комплекс 70S
с ArfB [сравните с панелью (e)]. j - Крио ЭМ структура большой митохондриальной субъединицы, связанной с фактором
спасения человека mtRF R и вспомогательным фактором MTRES1 [98]
да (рис. 1, g и h; [17]). В частности, «терминиру
ключения (с Trp319 в RF2) против A1492 и A1493
ющая» конформация DC может вмещать гибкий
направляет домен 3 в PTC, приводя к активации
линкерный участок между кодон распознаю
развёрнутого RF (рис. 1, h). В соответствии с ос
щим и каталитическим доменами RF (рис. 1, h),
новной ролью переключающей петли, её мута
который называется петлёй переключения
ции нарушают динамику открытия RF и изме
(а.о. ∼290-305 в RF1 и а.о. ∼310-325 в RF2) и об
няют точность процесса терминации [19, 48].
ладает гибкостью в компактизованном рили
В пептидил трансферазном центре мо
зинг факторе [51]. Распознавание стоп кодо
тив GGQ располагается таким образом, чтобы
на RF приводит к стекингу третьего нуклеотида
катализировать гидролиз сложноэфирной свя
стоп кодона на G530 и перемещению A1492
зи, связывающей образующийся пептид с тРНК
и A1493 в специфичную для терминации кон
в Р сайте [17]. Кристаллические структуры, вы
формацию (рис. 1, g и h). Упаковка петли пере
полненные при высоком разрешении, показали,
БИОХИМИЯ том 86 вып. 9 2021
6*
1332
КОРОСТЕЛЕВ
что A2602, необходимый для эффективности
в пост реакционных состояниях [17-20]. Одна
терминации [55-57], содействует докингу моти
ко недавние биофизические наблюдения пока
ва GGQ в PTC за счёт стекинга на консерватив
зали, что взаимодействие рилизинг факторов с
ном остатке Arg256 (RF1). Мотив GGQ образует
рибосомой ассоциировано с крупномасштаб
короткую α спираль (рис. 1, i), в которой амид
ной динамикой рибосомы [35]. Последующие
основной цепи глутамина расположен рядом с
крио ЭМ исследования визуализировали RF1
рибозой концевого нуклеотида P тРНК A76.
[58] и RF2 [61], связанные с рибосомой в раз
NH группа расположена таким образом, чтобы
личных вращательных состояниях, отличных от
стабилизировать промежуточное переходное
ранее определенных кристаллических структур.
состояние и уходящую группу [17], в соответ
Были получены изображения того, как пере
ствии с основной каталитической ролью глута
стройки рибосом облегчают диссоциацию RF
минового остова [29].
из рибосомы.
После гидролиза пептидил тРНК и пептид,
После гидролиза пептидил тРНК рибосома
и рилизинг фактор должны диссоциировать из
содержит деацилированную тРНК в Р сайте и
рибосомы, чтобы позволить ей разделиться на
рилизинг фактор в А сайте (рис. 1, c). Благода
субъединицы и начать новый раунд трансляции.
ря пониженному сродству деацил тРНК к Р
Два крио ЭМ исследования сообщили о не
сайту на 50S субъединице (в сравнении со срод
скольких структурах, которые предполагают
ством пептидил тРНК) и повышенному срод
конформационные перестройки, приводящие к
ства к Е сайту на 50S субъединице, тРНК может
диссоциации RF1 и RF2. Был визуализирован
принимать гибридное состояние P/E, в котором
комплекс рибосомы со связанным RF1 с ис
акцепторное плечо переносится на большую
пользованием Api137 [58], который связывается
субъединицу. Исследования FRET и крио ЭМ
с рибосомным туннелем и задерживает RF1 на
показали, что деацил тРНК может спонтанно
рибосоме [59]. Этот комплекс образовывался в
колебаться на развёрнутой рибосоме между
присутствии ГТФазы RF3, которая стимулирует
классическим P/P и гибридным P/E состояни
диссоциацию RF1 [35, 60]. В другом исследова
ями, совпадая с не повёрнутой и повёрнутой
нии были получены структуры комплекса со
конформациями рибосомы соответственно [37,
связанным RF2 и без RF3, что соответствует не
69-74]. Подобные колебания могут происхо
зависимой от RF3 диссоциацией RF2 [35].
дить в присутствии RF [58, 61]. Постепенное
Структуры визуализировали ретракцию деаци
вращение малой субъединицы связано с движе
лированного 3′ конца тРНК CCA из PTC, свя
нием каталитического домена рилизинг факто
занную с поворотом A2602 из его GGQ коорди
ра из PTC, в то время как домен распознавания
нирующего положения (рис. 1, i). GGQ несу
кодонов остаётся прикреплённым к DC [61].
щий мотив RF2 был перемещён в длинную β
При повороте малой субъединицы примерно
шпильку, которая простиралась в рибосомный
на 7° наблюдается равновесие структур со свя
туннель, как если бы закупоривала PTC
занным RF2 и без него, это позволяет предполо
(рис. 1, i). Это говорит о том, что верхушка ката
жить, что RF2 диссоциирует в основном из по
литического домена может перестраиваться та
вёрнутой конформации рибосомы (рис. 1, d и f).
ким образом, чтобы смещать диффузию вновь
Точно так же RF1 диссоциирует в присутствии
формируемого белка в направлении выхода из
ГТФазы RF3 (рис. 1, e), которая стабилизирует
туннеля, облегчая высвобождение белка из ри
конформацию повёрнутой рибосомы
[58,
босомы.
75-78]. Примечательно, что RF3 не консервати
Межсубъединичное вращение после высво-
вен у бактерий [79], и он незаменим для роста
бождения пептида сопряжено с диссоциацией RF.
E. coli [80, 81]. Это означает, что RF1 может осу
Во время трансляции рибосома подвергается
ществлять терминацию и диссоциировать
ряду межсубъединичных перестроек. На стадии
без RF3, и что диссоциация как RF1, так и RF2
элонгации тРНК и мРНК перемещаются внут
управляется перестройками рибосом. Дестаби
ри рибосомы, поскольку малая субъединица
лизация RF на малой субъединице сопряжена с
спонтанно поворачивается на ∼10° и связывает
разборкой центрального межсубъединичного
ГТФазную транслоказу EF G в бактериях или
мостика, образованного 23S спиралью 69 (H69)
eEF2 у эукариот [33, 62-66]. Напротив, крупно
и декодирующим центром (рис. 1, h). В повёрну
масштабные перестройки рибосомы не счита
той рибосоме без RF2 H69 отделяется от DC и
лись частью механизма терминации. Действи
прикрепляется к
50S субъединице вблизи
тельно, исследования FRET и рентгеновская
A2602, указывая тем самым, что реструктуриза
кристаллография показали, что RF связывают и
ция рибосомы совпадает с локальными перест
стабилизируют не повёрнутую рибосому
ройками PTC после высвобождения пепти
[35-37, 67] как в пред реакционных [68], так и
да [61]. В этом положении H69 будет конфлик
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1333
товать с RF2 в соответствии с состоянием после
mtRF1a [94]. Поскольку mtRF1 несёт консерва
диссоциации RF2. В случае отделения H69 от
тивный каталитический домен с мотивом GGQ,
малой субъединицы рибосома становится суб
mtRF1 может участвовать в механизме контроля
стратом, с которым связывается фактор рецик
качества, который распознает специфическую
линга рибосомы RRF [82, 83], расщепляющий
конформацию А сайта рибосомы [92] и/или
рибосому на субъединицы с помощью EF G.
требует дополнительных факторов, которые ещё
Митохондрии кодируют два RF-подобных
предстоит определить.
белка, mtRF1a и mtRF1, но только для mtRF1a
было показано, что он катализирует процесс
терминации трансляции [84]. Как и бактериаль
СПАСЕНИЕ ОСТАНОВИВШИХСЯ
ный RF1, mtRF1a человека (HMRF1L) содер
РИБОСОМ С ПОМОЩЬЮ МЕХАНИЗМОВ
жит мотив P206KT208 и катализирует высвобожде
ТЕРМИНАЦИИ У БАКТЕРИЙ
ние пептида на стоп кодонах UAA и UAG [85,
И МИТОХОНДРИЙ
86] (для эукариотических белков используются
номера аминокислотных остатков в белках че
Рибосомы останавливаются на молекулах
ловека, если не указано иное). Недавняя крио
мРНК, которые либо укорочены, не содержат
ЭМ работа визуализировала комплекс термина
стоп кодон, либо же останавливают рибосомы с
ции митохондрий свиньи с mtRF1a (рис. 2, h;
помощью других механизмов [95]. Недавние ис
[87]). Взаимодействия домена распознавания
следования выявили системы спасения рибосо
кодонов, петли переключения и домена 3 с ри
мы, которые высвобождают пептиды из остано
босомой демонстрируют, что структурные меха
вившихся рибосом. У бактерий альтернативные
низмы распознавания кодонов и терминации с
факторы спасения A (ArfA) и B (ArfB) представ
помощью mtRF1a сходны с механизмами
ляют собой независимые молекулярные меха
действия RF1. Хотя диссоциацию mtRF1a ещё
низмы, которые дополняют транс трансляци
предстоит визуализировать, сохранение меж
онную систему спасения [95-97]. Недавно в
субъединичных перестроек в миторибосо
структурных исследованиях были визуализиро
мах [88, 89] указывает на то, что диссоциа
ваны структурные механизмы этих факторов
ция mtRF1a при отсутствии митохондриального
спасения. В митохондриях две системы спасе
ортолога RF3 может быть сходна с диссоциаци
ния рибосомы включают RF подобные белки,
ей RF2.
которые недавно были визуализированы с по
Напротив, остаётся неясной функция
мощью крио EM: ICT1, который напоминает
mtRF1 [84]. Этот биоинформатически иденти
бактериальный ArfB и действует на миторибосо
фицированный гомолог RF1, состоящий из
мы, несущие укороченные молекулы мРНК
∼445 а.о. [90], имеет N концевое удлинение до
[87], и mtRF R (кодируемый c12orf65), который
мена 1 (∼70 а.о.), которое может влиять на свя
кооперирует с MTRES1 (кодируемый c6orf203) в
зывание с рибосомой. Более того, супердомен
распознавании большой митохондриальной
распознавания кодонов существенно отличает
субъединицы с остановившейся пептидил
ся от такового в RF1 и содержит мо
тРНК [98].
тив P264EVGLS269 и другие расширения, кото
Бактериальный альтернативный фактор спасе-
рые, вероятно, несовместимы с кодонами
ния A (ArfA). ArfA (ранее называвшийся YhdL) -
мРНК в А сайте [91, 92]. Было высказано пред
это небольшой белок, состоящий из 70 а.о., ко
положение, что mtRF1 распознает кодоны арги
торый рекрутирует RF2 для высвобождения из
нина (AGA и AGG), которые, как полагали, бы
рибосом пептидов, остановившихся на укоро
ли переназначены как кодоны терминации в
ченных мРНК, не содержащих стоп кодон
митохондриях [93]. Однако не удалось обнару
(рис. 2, a-c; [99-101]). Пять крио ЭМ исследо
жить связывающую или каталитическую актив
ваний продемонстрировали, что положительно
ность mtRF1 на рибосомах с мРНК, содержа
заряженный C концевой хвост ArfA связывается
щих стоп кодоны или кодоны аргинина. В ре
с мРНК туннелем, образованным преимущест
зультате этот белок не может компенсировать
венно рибосомной 16S РНК, таким образом
делецию гомолога функционального RF у дрож
«чувствуя» рибосомы, туннель которых свобо
жей [85-87]. В последующей работе было пока
ден из за укороченной молекулы мРНК [49, 50,
зано, что терминация на кодонах аргинина в
102-104]. N Концевая часть белка сворачивает
митохондриях человека происходит вследствие
ся с образованием компактного домена вблизи
отсутствия тРНК, способных декодировать эти
декодирующего центра и взаимодействует с до
кодоны, приводящие к сдвигу рамки -1, кото
меном распознавания кодонов RF2, хотя для
рый позиционирует кодон UAG в А сайте для
этого взаимодействия не требуется консерва
канонической терминации с помошью
тивный мотив SPF белка RF2 (рис. 2, c). Во всех
БИОХИМИЯ том 86 вып. 9 2021
1334
КОРОСТЕЛЕВ
этих исследованиях ArfA был связан с RF2 в от
рибосомы и позволяют предположить, что дру
крытой, каталитически компетентной конфор
гие пути спасения, такие как ArfA у бактерий и
мации, сходной с конформацией рилизинг
Dom34 у эукариот (обсуждается ниже), могут
факторов на стоп кодоне (рис. 2, b). В этой кон
обнаруживать рибосомы с более широким диа
формации петля переключения RF2 стабилизи
пазоном выступов и/или последовательнос
руется гидрофобными взаимодействиями остат
тей мРНК.
ка Trp319 с ArfA. Кроме того, в двух исследова
В комплексах с мРНК, выступающей за пре
ниях был обнаружен компактный RF2
делы кодона Р сайта, α спиральный хвост ArfB
(рис. 2, a), домен распознавания кодонов кото
связывается в туннеле с мРНК, в то время как
рого находится примерно на 5 Å дальше от А
+9 выступ мРНК удалён из туннеля и либо не
сайта [49, 50]. Хотя взаимодействия RF2 с ArfA
упорядочен в межсубъединичном простран
отличаются от взаимодействий RF2 со стоп ко
стве [110], либо стабилизируется второй копией
доном, полученные структуры подчёркивают
белка ArfB (рис. 2, e и f) [111]. Эти наблюдения
сходство механизма активации RF через откры
указывают на то, что ArfB может обнаруживать
тие RF2 после распознавания специфического
остановившиеся рибосомы разными путями в
сигнала в DC. Список ArfA подобных факторов
зависимости от последовательности мРНК или
спасения продолжает расти, выявляя родовые
структурной динамики, допускающей экскурсы
особенности механизмов контроля качест
мРНК за пределы туннеля. Кроме того, класси
ва [105]. Например, если BrfA Bacillus subtilis за
фикация данных крио ЭМ выявила набор
висит от RF2 и подобен ArfA [106], то ArfT
структур с каталитическим N концевым доме
Francisella tularensis может рекрутировать как
ном ArfB внутри или вне PTC (рис. 2, d и e) [111].
RF1, так и RF2 для высвобождения пептидов из
Эта динамика согласуется с биохимическими
остановленных рибосом [107, 108].
наблюдениями и указывает на то, что механизм
Бактериальный альтернативный фактор спасе-
распознавания субстрата напоминает механизм
ния B (ArfB). ArfB (ранее называвшийся YaeJ) -
канонических рилизинг факторов, которые
это рилизинг фактор (~140 а.о.), который со
сначала распознают DC на малой субъединице,
держит как С концевой хвост, связывающийся с
а затем перегруппировываются, чтобы закре
рибосомным туннелем, так и RF подобный ка
пить каталитический домен рядом с расщепляе
талитический домен, несущий мотив GGQ
мой связью в пептидил тРНК.
(рис. 2, d-g). Биохимические исследования по
С помощью крио ЭМ на рибосомах были
казали, что ArfB может спасать не только рибо
идентифицированы ArfB с различной степенью
сомы с мРНК, укороченной сразу после кодона
межсубъединичного вращения в сочетании с об
Р сайта, но также и рибосомы с большими уд
разованием гибридного состояния P/E деацили
линениями мРНК [109], хотя каталитическая
рованной тРНК (рис. 2, f-g). Было показано, что
активность ArfB снижается по мере увеличения
степень связывания димера ArfB понижается по
длины мРНК и может зависеть от последова
мере увеличения вращения 30S субъединицы.
тельности выступа мРНК [110, 111]. Кристалло
Сильно повёрнутые рибосомы обнаруживают
графическая структура показала, что положи
равновесие между частицами с мономерным
тельно заряженный хвост образует α спираль в
ArfB связанным и вакантным A сайтом
свободном туннеле, тогда как каталитический
(рис. 2, g) [111]. Спираль 69 свободных рибосом
N концевой домен стыкуется с PTC подобно то
диссоциирует из DC и, скорее всего, будет ме
му, как это происходит у канонических RF [112].
шать связыванию ArfB с большой субъединицей.
Связывание ArfB с не повёрнутой рибосомой
Эти результаты подтверждают, что ArfB, подоб
напоминает связывание рилизинг факторов,
но каноническим RF, диссоциирует после спон
что соответствует распознаванию субстрата с ос
танного межсубъединичного вращения и нару
тановленной пептидил тРНК. Поскольку тун
шения мостика H69-DC. Хотя компоненты па
нель для мРНК не может быть занят одновре
раллельного пути спасения с участием ArfA и
менно ArfB и более длинным выступом мРНК,
RF2 были визуализированы только на не повёр
при исследованиях крио ЭМ была получена
нутой рибосоме, консервативная рибосомная
структура 70S рибосомы с ArfB и мРНК, высту
динамика комплексов, связанных с RF и ArfB,
пающей на 2 или 9 нуклеотидов за пределы P
предполагает, что аналогичный механизм может
сайта [110, 111]. Показано, что нуклеотиды де
быть использован для высвобождения ArfA и
кодирующего центра (A1492 и A1493) взаимо
RF2 и подготовки рибосомы к рециклингу.
действуют с ArfB и мРНК, обеспечивая аккомо
Митохондриальный фактор спасения ICT1
дацию различных выступов мРНК посредством
(∼200 a.о.), близкий гомолог бактериально
различных конформаций [111]. Эти результаты
го ArfB, катализирует высвобождение пептидов
подчёркивают структурную пластичность DC
из остановившихся рибосом [113]. ICT1 («imma
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1335
ture colon carcinoma transcript 1», также обозна
ся интактной, вероятно, из за уникальной кон
чаемый как MRPL58) необходим для роста кле
формации концевого остатка лизина образую
ток [113-115], и нарушение регуляции его син
щегося пептида, который, как было показано,
теза связано с туморогенезом [116]. Интересно,
является сигнатурой остановившихся рибо
что одна молекула ICT1 связывается с централь
сом [129, 130]. Хотя mtRF R вместе с MTRES1
ным выступом митохондриальной рибосо
может гидролизовать пептидил тРНК в модель
мы [117], но эта позиция далека от А сайта, ука
ных 50S комплексах E. coli in vitro [98], ещё
зывая на то, что эта молекула не катализирует
предстоит показать, спасает ли и как mtRF R
спасение рибосомы (рис. 2, i). Центральный
остановленные митохондриальные субъедини
выступ большой субъединицы взаимодействует
цы с лизил тРНК. Диссоциация mtRF R и
с малой субъединицей и с молекулами тРНК,
MTRES1 из большой субъединицы, необходи
что делает его структуру критичной для пра
мая для рециклинга рибосомы, вероятно, соп
вильной сборки рибосом и их функционирова
ряжена с диссоциацией деацил тРНК после
ния во всех царствах жизни [118-122]. Хотя ар
гидролиза пептидил тРНК. Не исключено, что в
хитектурная функция ICT1 может частично
разборке этого спасательного комплекса помо
вносить вклад в регуляцию митохондриальной
гают дополнительные факторы. Будущие иссле
трансляции, именно каталитическая функция
дования определят субстратную специфичность
другой - временно связанной - молекулы ICT1
mtRF R и механизм диссоциации mtRF R/
важна для митохондриальной трансляции. Зна
MTRES1.
чимость каталитической функции была проде
монстрирована с использованием мутационно
го анализа, показавшего прекращение роста
ЦИТОПЛАЗМАТИЧЕСКАЯ ТЕРМИНАЦИЯ
клеток после изменения мотива GGQ в
И СПАСЕНИЕ РИБОСОМЫ У ЭУКАРИОТ
ICT1 [113].
В недавно созданной крио ЭМ структуре
Терминация трансляции с участием eRF1•eRF3.
связанного с ICT1 спасательного комплекса [87]
Эукариотический фактор eRF1
(~440
а.о.)
положительно заряженная C концевая спираль
структурно отличается от бактериальных рили
ICT1 обнаруживается в мРНК туннеле
зинг факторов, и его каталитическая функция
(рис. 2, i). Каталитический N концевой домен
зависит от ГТФазной активности eRF3 [23,
стыкуется с PTC аналогично ArfB. Положение
131-133]. Домен распознавания кодонов eRF1
мотива GGQ поддерживает каталитическую
(домен N) сложен иначе, чем у бактериаль
роль остова глутамина, аналогичную роли RF.
ных RF, и несёт консервативные участки ами
Крио ЭМ исследование было выполнено с ис
нокислот, такие как мотив T58ASNIKS64, кото
пользованием укороченной мРНК в P сайте.
рый необходим для распознавания всех трёх
Остаётся визуализировать: если/как ICT1 рас
стоп кодонов [134-136]. Кроме того, в отличие
познает рибосомы с более длинными выступами
от бактериальных рилизинг факторов, eRF1
мРНК [123] и отражает ли склонность изолиро
распознает тетрануклеотид, включающий
ванного ICT1 к димеризации [124] механисти
3 нуклеотида стоп кодона и последующий нук
ческий сценарий, аналогичный сценарию ди
леотид [137-139]. Профилирование рибосом и
мерного ArfB, который может стабилизировать
другие исследования показали, что стоп кодон,
длинные выступы мРНК [111].
за которым следует пиримидин, приводит к ин
Митохондриальный рилизинг-фактор mtRF-R
тенсивному считыванию аннотированного
(∼170 а.о.) представляет собой митохондриаль
стоп кодона [140] в соответствии с более низ
ную пептидил тРНК гидролазу, которая необхо
кой каталитической активностью eRF1 на тет
дима для жизнеспособности клеток [125]. Мута
рануклеотиде с концевым пиримидином [138].
ции или дисрегуляция этого белка нарушают
Эта зависимость способствует переназначению
трансляцию в митохондриях и ассоциируются с
стоп кодонов в смысловые кодоны у инфузо
заболеваниями [126-128]. Недавнее исследова
рий, хотя «четвёртый» нуклеотид не является
ние крио ЭМ визуализировало человеческий
исключительной детерминантой переназначе
mtRF R с пептидил тРНК на большой митохон
ния [141-143]. Дополнительные характеристи
дриальной субъединице рибосомы [98]. Свя
ки мРНК, такие как последовательность и
зыванию mtRF R в А сайте способствует
структура вокруг стоп кодона и ниже него, иг
MTRES1, закреплённый со стеблем антикодона
рают роль в эффективности терминации эука
тРНК (рис. 2, j). Как ожидалось, каталитичес
риот [144-147].
кий домен mtRF R входит в PTC и выдвигает
Несмотря на различия между эукариотичес
мотив GGQ вблизи концевого нуклеотида P
кими и бактериальными рилизинг факторами,
тРНК. Удивительно, но пептидил тРНК остает
эукариотическая терминация сходна с бактери
БИОХИМИЯ том 86 вып. 9 2021
1336
КОРОСТЕЛЕВ
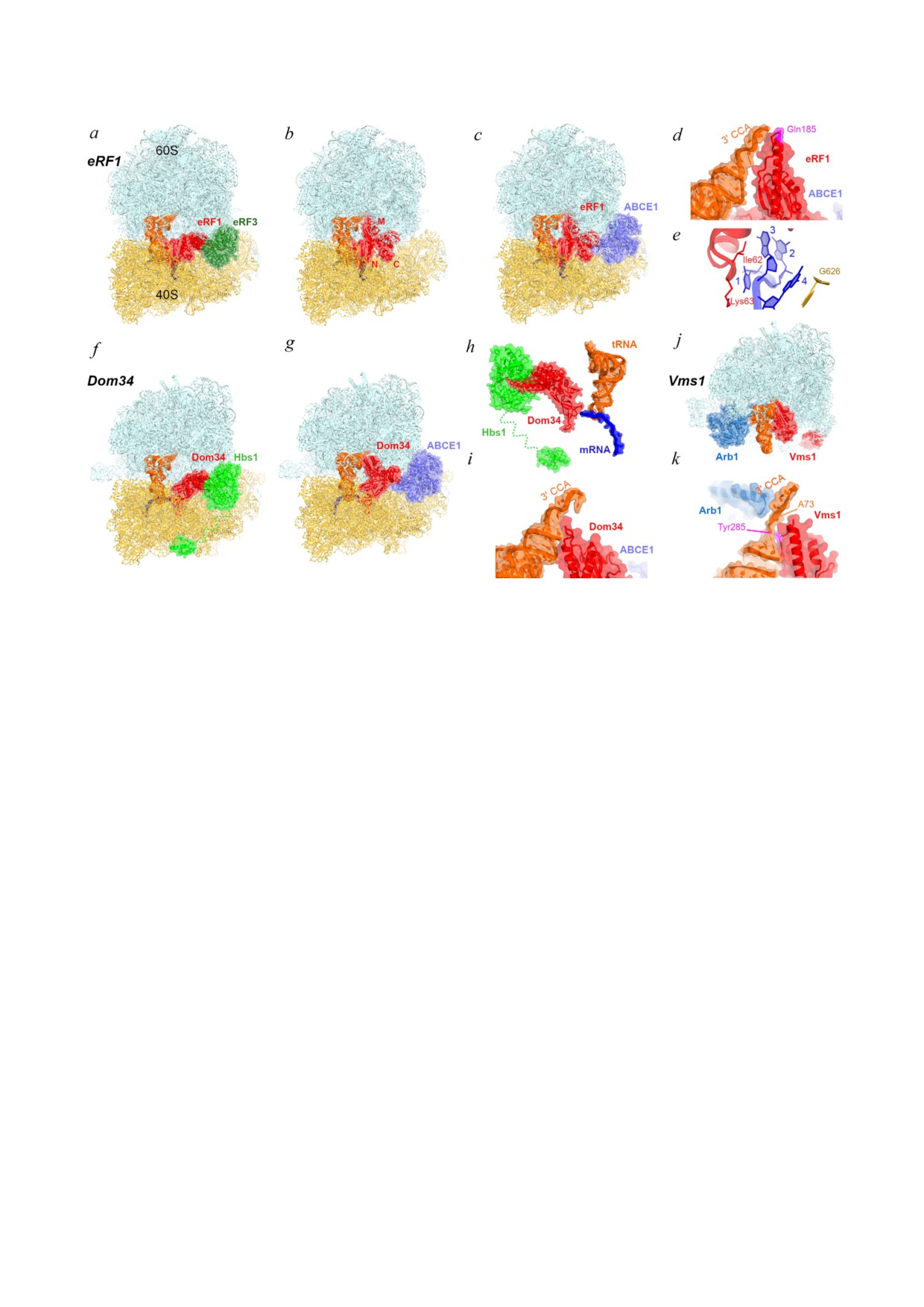
Рис. 3. Терминация трансляции эукариот и механизмы спасения цитоплазматических рибосом. a-c - Перестройка рили
зинг фактора eRF1 на 80S рибосоме со стоп кодоном после гидролиза ГТФ на eRF3 (a-b), и взаимодействие с фактором
рециклинга ABCE1 (c; Rli1 у дрожжей). d - Взаимодействие каталитической конформации eRF1 с тРНК в Р сайте [137].
e - Распознавание 4 нуклеотидного стоп сигнала eRF1 происходит при участии аминокислотных остатков моти
ва TASNIKS (показаны Ile62 и Lys63) и G626 18S рРНК [137]. f и g - Перестройка Pelota (Dom34 у дрожжей) на 80S рибо
соме с укороченной мРНК после гидролиза ГТФ на Hbs1 [163] и связывания ABCE1 [164]. h - Распознавание укорочен
ной мРНК белком Dom34 [163]. i - Взаимодействие открытой конформации Dom34 с тРНК в Р сайте [164]. j - Спаса
тельный 60S комплекс с пептидил тРНК гидролазой Vms1 и Arb1 [165]. k - Взаимодействие Vms1 и Arb1 с тРНК
альной тем, что её точность контролируется
связывается аналогично другим трансляцион
крупномасштабными конформационными из
ным ГТФазам, таким как факторы элонгации
менениями белка eRF1. Крио ЭМ исследова
EF Tu и eEF1A. Аминокислотные остатки рас
ния показали eRF1 на рибосоме в трёх функцио
познавания кодонов eRF1 взаимодействуют со
нальных состояниях (рис. 3, a-c): с eRF3 (с ком
всеми четырьмя нуклеотидами сигнала терми
пактным eRF1, неактивным в высвобождении
нации на мРНК, которая принимает конформа
пептида) [148-150], без eRF3 (с удлинённым
цию U поворота (рис. 3, e), приводящую к пере
eRF1, активированным для высвобождения
мещению мРНК в рибосомный туннель [157].
пептида) [139, 151] и с АТФ связывающим кас
Интересно, что несмотря на дивергенцию
сетным белком ABCE1 (Rli1 в дрожжах) [137,
структуры и последовательности от бактериаль
148, 152], фактором рециклинга, который раз
ной терминации (рис. 1, c), терминация эукари
бирает рибосомы на субъединицы [153-155].
от и бактерий имеет сходные аспекты распозна
Эти структуры в сочетании с биохимическими и
вания стоп кодона, такие как распознавание
биофизическими данными позволяют рекон
пурина в положении 3 (R3) консервативными
струировать большинство этапов терминации у
остатками Thr58 и Ile62 TASNIKS мотива
эукариот. После связывания eRF1•eRF3•ГТФ
eRF1 (рис. 3, e; [137]) и Thr198 и Ile196 белка
со стоп кодоном А сайта рибосомы
[156]
RF1 E. coli [158]. Кроме того, последний пурин в
(рис. 3, a) гидролиз ГТФ высвобождает eRF3 и
сигнале терминации (R4 у эукариот или R3 у
приводит к встраиванию каталитического доме
бактерий) стэкируется с нуклеотидом G626
на M eRF1 с мотивом GGQ в PTC (рис. 3, b и d).
(G530 у E. coli) декодирующего центра в соответ
Гидролиз ГТФ катализируется сарцин рицино
ствии с предпочтением пурина перед пирими
вой петлёй большой субъединицы, где eRF3
дином в этом положении (рис. 1, h и 3, e).
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1337
В открытой каталитической конформации
терминации, разборка связанных с Dom34 ри
C домен eRF1, который связывает N и M до
босом происходит с участием ABCE1 [172], ко
мены, может взаимодействовать с Fe S доме
торый взаимодействует с Dom34 после диссоци
ном ABCE1 и инициировать рециклинг рибосо
ации Hbs1 (рис. 3, g; [148, 164]). Будущие иссле
мы (рис. 3, c; [148, 152]). ABCE1, конформаци
дования приведут к пониманию подробностей
онная динамика которого контролируется дву
механизмов разборки рибосомы после канони
мя сайтами связывания АТФ [159] и который
ческой терминации и связывания Dom34, кото
обладает сродством как к 80S рибосоме, так и к
рые, вероятно, происходят в результате меж и
40S субъединице [160-162] помогает в расщеп
внутрисубъединичных структурных перестроек.
лении посттерминационной рибосомы через
Пептидил тРНК, связанная с большой 60S
промежуточные состояния диссоциации eRF1.
субъединицей в результате действия механизмов
Эти состояния необходимо будет визуализиро
контроля качества рибосомы, разделяется таки
вать.
ми механизмами, как добавление С концевых
Спасение остановившихся рибосом с помощью
аланил треониновых повторов (CAT хвост) к
Dom34•Hbs1 и Vms1. Хотя эукариотических
пептиду с помощью Rqc2, и его убиквитиниро
аналогов ArfA или ArfB пока не выявлено, было
вание с помощью Ltn1 (Листерин у млекопита
обнаружено несколько механизмов контроля
ющих [173]) и расщепление пептидил тРНК.
качества, которые напоминают эукариотичес
Белок Vms1 (ANKZF1 у млекопитающих) пред
кую систему терминации [166]. Dom34 (Pelota у
ставляет собой большую (~600-750 а.о.) пепти
млекопитающих) и ГТФаза Hbs1 образуют гете
дил тРНК гидролазу, которая катализирует дис
родимер, который спасает рибосомы, остано
социацию
комплексов
60S•пептидил
вившиеся на укороченных мРНК, структуриро
тРНК [174, 175]. Последовательность его ката
ванные мРНК или 3′ UTR мРНК [167-170].
литического домена напоминает последователь
Dom34 и Hbs1 очень похожи на eRF1 и eRF3 со
ность eRF1 и содержит мотив GXQ (где X -
ответственно (рис. 3, f и g). Однако у Dom34 от
это Ser294 у Saccharomyces cerevisiae) с функцио
сутствует каталитический мотив GGQ, поэтому
нально важным остатком глутамина [165, 174,
он не гидролизует пептидил тРНК, а вместо
175]. Несмотря на это сходство и на некоторое
этого способствует диссоциации субъеди
сходство с митохондриальным mtRF R [98],
ниц [170]. Крио ЭМ структуры показали, что
Vms1 не гидролизует сложноэфирную связь в
связывание Dom34 и Hbs1 с рибосомой анало
пептидил тРНК. Вместо этого Vms1 расщепляет
гично связыванию eRF1 и eRF3 [148, 163, 171].
фосфодиэфирную связь между нуклеотидами 73
ГТФазный домен Hbs1 стыкуется с сарцин ри
и 74 в тРНК, отделяя CCA пептид от остальной
циновой петлёй, в то время как длинный N кон
тРНК [176, 177]. Фрагмент тРНК подвергается
цевой хвост Hbs1 достигает туннеля входа
репарации с помощью ELAC1 и CCA добавляю
мРНК (рис. 3, f и h), принимая участие в распоз
щего фермента TRNT1 [176, 178]. Крио ЭМ ви
навании остановившихся мРНК субстра
зуализировала Vms1, связанный с 60S субъеди
тов [163, 171]. β Шпилька Dom34 входит в деко
ницей, с АТФазой Arb1 ABCF типа вблизи
дирующий центр, отдаленно напоминая бакте
тРНК (рис. 3, j) [165]. Arb1, вероятно, способ
риальные факторы спасения, которые связыва
ствует позиционированию субстрата пептидил
ются в свободном мРНК туннеле (рис. 3, h).
тРНК, тем самым повышая эффективность ре
Сравнение структуры рибосомы, связанной с
акции [165]. Каталитический домен Vms1 нахо
тРНК, eRF1 и Dom34, выявило различную кон
дится рядом с тРНК (рис. 3, k), подобно M до
формацию DC [148], подчёркивая пластичность
мену Dom34 (рис. 3, i). Петля GSQ не упорядо
эукариотического DC, напоминающую плас
чена вблизи ССА конца тРНК. Консерватив
тичность бактериальных рибосом [111]. Более
ный соседний остаток Tyr285 образует стэкинг
того, Dom34 может быть связан в присутствии
взаимодействие с нуклеотидом 72 молекулы
более длинных мРНК, включая поли A хвост,
тРНК, вытесняя нуклеотид 73 и экспонируя
благодаря перенаправлению выступа мРНК в
подлежащую расщеплению фосфодиэфирную
межсубъединичное пространство [148]. Это на
связь для гидролиза (рис. 3, k). Vms1 охватывает
поминает комплексы спасения бактерий с
большую площадь на межсубъединичном ин
ArfB [110, 111] и объясняет, как Dom34 может
терфейсе 60S субъединицы в соответствии с
распознавать ряд субстратов, не ограничиваясь
ролью Vms1 в координации гидролиза пепти
мРНК, укороченными после кодона Р сайта.
дил тРНК и деградации аномального пептида.
После открытия Dom34 (рис. 3, g) M домен
Домены типа анкириновый повтор и спирали
связывает ножку акцепторного плеча тРНК,
зованная спираль (coiled coil) направлены на
чтобы способствовать вытеснению тРНК из ри
выход из рибосомного туннеля, где VIM домен,
босомы (рис. 3, i). Аналогично канонической
вероятно, способствует Cdc48 опосредованной
БИОХИМИЯ том 86 вып. 9 2021
1338
КОРОСТЕЛЕВ
экстракции и клиренсу пептида протеасомой
сильно зависит от её конформации и структур
[179-181]. Будущие исследования позволят по
ных перестроек, превращающих рибосому в ак
лучить подробности того, как Vms1 кооперирует
тивного участника всех этапов процесса транс
с другими белками в диссоциации CCA пептида
ляции и контроля качества образующейся моле
и фрагмента тРНК из 60S субъединицы, позво
кулы белка.
ляя рециркулировать последнюю для трансля
ции новой мРНК.
Благодарности. Я благодарен Анне Лавлэнд и
Даррилу Конте мл. за комментарии к данной
ВЫВОДЫ
статье.
Финансирование. Данная работа выполнена
Недавние исследования раскрыли многие
при финансовой поддержке Фонда кистозного
детали механизмов терминации трансляции и
фиброза (грант № 670773) и Национального
спасения рибосомы во всех царствах жизни.
Института Здоровья (грант № R35 GM127094).
Они продемонстрировали разнообразие сцена
Конфликт интересов. Автор заявляет об отсут
риев высвобождения пептидов, катализируемых
ствии конфликта интересов в финансовой или
белками с различной архитектурой. Некоторые
иной сфере.
аспекты этих сценариев ещё предстоит визуали
Соблюдение этических норм. В настоящей
зировать, однако ясно, что распознавание и
статье не содержится описание исследований с
принятие решения относительно терминации и
участием людей или животных, выполненных
судьбы остановившихся комплексов рибосомы автором.
СПИСОК ЛИТЕРАТУРЫ
1. Brenner, S., Barnett, L., Katz, E. R., and Crick, F. H.
(nonenzymic) translocation in ribosomes of Escherichia
(1967) UGA: a third nonsense triplet in the genetic code,
coli by p chloromercuribenzoate, Mol. Biol., 6, 248 254.
Nature, 213, 449 450.
13. Van Dyke, N., and Murgola, E. J. (2003) Site of function
2. Brenner, S., Stretton, A. O., and Kaplan, S.
(1965)
al interaction of release factor 1 with the ribosome, J. Mol.
Genetic code: the “nonsense” triplets for chain termina
Biol., 330, 9 13.
tion and their suppression, Nature, 206, 994 998.
14. Bouakaz, L., Bouakaz, E., Murgola, E. J., Ehrenberg, M.,
3. Spirin, A. S. (1999) Termination of translation, in
and Sanyal, S. (2006) The role of ribosomal protein L11 in
Ribosomes. Cellular Organelles, Springer, Boston, MA,
class I release factor mediated translation termination and
pp. 261 270.
translational accuracy, J. Biol. Chem., 281, 4548 4556.
4. Sogorin, E. A., Agalarov, S., and Spirin, A. S. (2016) Inter
15. Ito, K., Uno, M., and Nakamura, Y. (2000) A tripeptide
polysomal coupling of termination and initiation during
“anticodon” deciphers stop codons in messenger RNA,
translation in eukaryotic cell free system, Sci. Rep., 6,
Nature, 403, 680 684.
24518, doi: 10.1038/srep24518.
16. Oparina, N. J., Kalinina, O. V., Gelfand, M. S., and
5. Tompkins, R. K., Scolnick, E. M., and Caskey, C. T. (1970)
Kisselev, L. L. (2005) Common and specific amino acid
Peptide chain termination. VII. The ribosomal and release
residues in the prokaryotic polypeptide release factors RF1
factor requirements for peptide release, Proc. Natl. Acad.
and RF2: possible functional implications, Nucleic Acids
Sci. USA, 65, 702 708.
Res., 33, 5226 5234, doi: 10.1093/nar/gki841.
6. Scolnick, E., Tompkins, R., Caskey, T., and Nirenberg, M.
17. Laurberg, M., Asahara, H., Korostelev, A., Zhu, J.,
(1968) Release factors differing in specificity for terminator
Trakhanov, S., and Noller, H. F. (2008) Structural basis for
codons, Proc. Natl. Acad. Sci. USA, 61, 768 774.
translation termination on the 70S ribosome, Nature, 454,
7. Capecchi, M. R. (1967) Polypeptide chain termination in
852 857.
vitro: isolation of a release factor, Proc. Natl. Acad. Sci.
18. Korostelev, A., Asahara, H., Lancaster, L., Laurberg, M.,
USA, 58, 1144 1151.
Hirschi, A., et al. (2008) Crystal structure of a translation
8. Capecchi, M. R. (1967) A rapid assay for polypeptide chain
termination complex formed with release factor RF2, Proc.
termination, Biochem. Biophys. Res. Commun., 28, 773
Natl. Acad. Sci. USA, 105, 19684 19689.
778.
19. Korostelev, A., Zhu, J., Asahara, H., and Noller, H. F.
9. Nakamura, Y., Ito, K., Matsumura, K., Kawazu, Y., and
(2010) Recognition of the amber UAG stop codon by
Ebihara, K. (1995) Regulation of translation termination:
release factor RF1, EMBO J., 29, 2577 2585, doi: 10.1038/
conserved structural motifs in bacterial and eukaryotic
emboj.2010.139.
polypeptide release factors, Biochem. Cell. Biol., 73, 1113
20. Weixlbaumer, A., Jin, H., Neubauer, C., Voorhees, R. M.,
1122.
Petry, S., et al. (2008) Insights into translational termina
10. Frischmeyer, P. A., and Dietz, H. C. (1999) Nonsense
tion from the structure of RF2 bound to the ribosome,
mediated mRNA decay in health and disease, Hum. Mol.
Science, 322, 953 956.
Genet., 8, 1893 1900.
21. Korkmaz, G., and Sanyal, S. (2017) R213I mutation in
11. Spirin, A. S. (1969) A model of the functioning ribosome:
release facto 2 (RF2) is one step forward for engineering an
locking and unlocking of the ribosome subparticles, Cold
omnipotent release factor in bacteria Escherichia coli, J. Biol.
Spring Harb. Symp. Quant. Biol., 34, 197 207.
Chem., 292, 15134 15142, doi: 10.1074/jbc.M117. 785238.
12. Gavrilova, L. P., and Spirin, A. S. (1972) Mechanism of
22. Field, A., Hetrick, B., Mathew, M., and Joseph, S. (2010)
translocation in ribosomes. II. Activation of spontaneous
Histidine 197 in release factor 1 is essential for a site bind
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1339
ing and peptide release, Biochemistry, 49, 9385 9390,
tide release factor 1 from Thermotoga maritima reveal
doi: 10.1021/bi1012047.
domain flexibility required for its interaction with the ribo
23.
Frolova, L., Le Goff, X., Rasmussen, H. H.,
some, J. Mol. Biol., 341, 227 239.
Cheperegin, S., Drugeon, G., et al. (1994) A highly con
40.
Vestergaard, B., Van, L. B., Andersen, G. R., Nyborg, J.,
served eukaryotic protein family possessing properties of
Buckingham, R. H., and Kjeldgaard, M. (2001) Bacterial
polypeptide chain release factor, Nature, 372, 701 703.
polypeptide release factor RF2 is structurally distinct from
24.
Frolova, L. Y., Tsivkovskii, R. Y., Sivolobova, G. F.,
eukaryotic eRF1, Mol. Cell, 8, 1375 1382.
Oparina, N. Y., Serpinsky, O. I., et al. (1999) Mutations in
41.
Zoldak, G., Redecke, L., Svergun, D. I., Konarev, P. V.,
the highly conserved GGQ motif of class 1 polypeptide
Voertler, C. S., et al. (2007) Release factors 2 from
release factors abolish ability of human eRF1 to trigger
Escherichia coli and Thermus thermophilus: structural, spec
peptidyl tRNA hydrolysis, RNA, 5, 1014 1020.
troscopic and microcalorimetric studies, Nucleic Acids
25.
Shaw, J. J., and Green, R. (2007) Two distinct components
Res., 35, 1343 1353.
of release factor function uncovered by nucleophile parti
42.
JCSG (2005) Crystal structure of Peptide chain release factor
tioning analysis, Mol. Cell, 28, 458 467.
1 (RF 1) (SMU.1085) from Streptococcus mutans at 2.34 A
26.
Zavialov, A. V., Mora, L., Buckingham, R. H., and
resolution, Protein Data Bank.
Ehrenberg, M. (2002) Release of peptide promoted by the
43.
Rawat, U. B., Zavialov, A. V., Sengupta, J., Valle, M.,
GGQ motif of class 1 release factors regulates the GTPase
Grassucci, R. A., et al. (2003) A cryo electron microscop
activity of RF3, Mol. Cell, 10, 789 798.
ic study of ribosome bound termination factor RF2,
27.
Seit Nebi, A., Frolova, L., Ivanova, N., Poltaraus, A., and
Nature, 421, 87 90.
Kiselev, L. (2000) Mutation of a glutamine residue in the
44.
Petry, S., Brodersen, D. E., Murphy, F. V., Dunham,
universal tripeptide GGQ in human eRF1 termination fac
C. M., Selmer, M., et al. (2005) Crystal structures of the
tor does not cause complete loss of its activity, Mol. Biol.
ribosome in complex with release factors RF1 and RF2
(Mosk.), 34, 899 900.
bound to a cognate stop codon, Cell, 123, 1255 1266.
28.
Seit Nebi, A., Frolova, L., Justesen, J., and Kisselev, L.
45.
He, S. L., and Green, R. (2010) Visualization of codon
(2001) Class 1 translation termination factors: invariant
dependent conformational rearrangements during transla
GGQ minidomain is essential for release activity and ribo
tion termination, Nat. Struct. Mol. Biol., 17, 465 470.
some binding but not for stop codon recognition, Nucleic
46.
Hetrick, B., Lee, K., and Joseph, S. (2009) Kinetics of stop
Acids Res., 29, 3982 3987.
codon recognition by release factor 1, Biochemistry, 48,
29.
Santos, N., Zhu, J., Donohue, J. P., Korostelev, A. A., and
11178 11184.
Noller, H. F. (2013) Crystal structure of the 70S ribosome
47.
Trappl, K., and Joseph, S. (2016) Ribosome induces a
bound with the Q253P mutant form of release factor RF2,
closed to open conformational change in release factor 1,
Structure, 21, 1258 1263, doi: 10.1016/j.str.2013.04.028.
J. Mol. Biol., 428, 1333 1344, doi: 10.1016/j.jmb.2016.
30.
Korostelev, A. A. (2011) Structural aspects of translation
01.021.
termination on the ribosome, RNA, 17, 1409 1421.
48.
Svidritskiy, E., and Korostelev, A. A.
(2018)
31.
Dunkle, J. A., and Cate, J. H. (2010) Ribosome structure
Conformational control of translation termination on the
and dynamics during translocation and termination, Annu.
70S ribosome, Structure, 26, 821 828.
Rev. Biophys., 39, 227 244, doi: 10.1146/annurev.biophys.
49.
Demo, G., Svidritskiy, E., Madireddy, R., Diaz Avalos, R.,
37.032807.125954.
Grant, T., et al. (2017) Mechanism of ribosome rescue by
32.
Ramakrishnan, V. (2011) Structural studies on decoding,
ArfA and RF2, Elife, 6, doi: 10.7554/eLife.23687.
termination and translocation in the bacterial ribosome, in
50.
James, N. R., Brown, A., Gordiyenko, Y., and
Ribosomes: Structure, Function, and Dynamics (Rodnina,
Ramakrishnan, V. (2016) Translational termination with
M., Wintermeyer, W., and Green, R., eds.) Springer,
out a stop codon, Science, 354, 1437 1440, doi: 10.1126/
pp. 19 30.
science.aai9127.
33.
Rodnina, M. V. (2018) Translation in prokaryotes, Cold
51.
Fu, Z., Indrisiunaite, G., Kaledhonkar, S., Shah, B.,
Spring Harb. Perspect. Biol., 10, doi: 10.1101/cshperspect.
Sun, M., et al. (2019) The structural basis for release fac
a032664.
tor activation during translation termination revealed by
34.
Youngman, E. M., McDonald, M. E., and Green, R.
time resolved cryogenic electron microscopy, Nat.
(2008) Peptide release on the ribosome: mechanism and
Commun., 10, 2579, doi: 10.1038/s41467 019 10608 z.
implications for translational control, Annu. Rev.
52.
Fislage, M., Zhang, J., Brown, Z. P., Mandava, C. S.,
Microbiol., 62, 353 373.
Sanyal, S., et al. (2018) Cryo EM shows stages of initial
35.
Adio, S., Sharma, H., Senyushkina, T., Karki, P.,
codon selection on the ribosome by aa tRNA in ternary
Maracci, C., et al. (2018) Dynamics of ribosomes and
complex with GTP and the GTPase deficient EF
release factors during translation termination in E. coli,
TuH84A, Nucleic Acids Res., 46, 5861 5874, doi: 10.1093/
Elife, 7, doi: 10.7554/eLife.34252.
nar/gky346.
36.
Prabhakar, A., Capece, M. C., Petrov, A., Choi, J., and
53.
Ogle, J. M., Brodersen, D. E., Clemons, W. M., Jr., Tarry,
Puglisi, J. D. (2017) Post termination ribosome intermedi
M. J., Carter, A. P., and Ramakrishnan, V.
(2001)
ate acts as the gateway to ribosome recycling, Cell Rep., 20,
Recognition of cognate transfer RNA by the 30S ribosomal
161 172, doi: 10.1016/j.celrep.2017.06.028.
subunit, Science, 292, 897 902.
37.
Sternberg, S. H., Fei, J., Prywes, N., McGrath, K. A., and
54.
Loveland, A. B., Demo, G., Grigorieff, N., and
Gonzalez, R. L., Jr. (2009) Translation factors direct
Korostelev, A. A. (2017) Ensemble cryo EM elucidates the
intrinsic ribosome dynamics during translation termination
mechanism of translation fidelity, Nature, 546, 113 117,
and ribosome recycling, Nat. Struct. Mol. Biol., 16, 861
doi: 10.1038/nature22397.
868, doi: 10.1038/nsmb.1622.
55.
Amort, M., Wotzel, B., Bakowska Zywicka, K., Erlacher,
38.
Indrisiunaite, G., Pavlov, M. Y., Heurgue Hamard, V., and
M. D., Micura, R., and Polacek, N. (2007) An intact ribose
Ehrenberg, M. (2015) On the pH dependence of class 1
moiety at A2602 of 23S rRNA is key to trigger peptidyl
RF dependent termination of mRNA translation, J. Mol.
tRNA hydrolysis during translation termination, Nucleic
Biol., 427, 1848 1860, doi: 10.1016/j.jmb.2015.01.007.
Acids Res., 35, 5130 5140, doi: 10.1093/nar/gkm539.
39.
Shin, D. H., Brandsen, J., Jancarik, J., Yokota, H.,
56.
Polacek, N., Gomez, M. J., Ito, K., Xiong, L.,
Kim, R., and Kim, S. H. (2004) Structural analyses of pep
Nakamura, Y., and Mankin, A. (2003) The critical role of
БИОХИМИЯ том 86 вып. 9 2021
1340
КОРОСТЕЛЕВ
the universally conserved A2602 of 23S ribosomal RNA in
72.
Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Jr., Puglisi,
the release of the nascent peptide during translation termi
J. D., and Chu, S. (2004) tRNA dynamics on the ribosome
nation, Mol. Cell, 11, 103 112.
during translation, Proc. Natl. Acad. Sci. USA, 101, 12893
57.
Youngman, E. M., Brunelle, J. L., Kochaniak, A. B., and
12898, doi: 10.1073/pnas.0403884101.
Green, R. (2004) The active site of the ribosome is com
73.
Agirrezabala, X., Lei, J., Brunelle, J. L., Ortiz Meoz, R. F.,
posed of two layers of conserved nucleotides with distinct
Green, R., and Frank J. (2008) Visualization of the hybrid
roles in peptide bond formation and peptide release, Cell,
state of tRNA binding promoted by spontaneous ratcheting
117, 589 599.
of the ribosome, Mol. Cell, 32, 190 197, doi: 10.1016/
58.
Graf, M., Huter, P., Maracci, C., Peterek, M., Rodnina,
j.molcel.2008.10.001.
M. V., and Wilson, D. N. (2018) Visualization of transla
74.
Julian, P., Konevega, A. L., Scheres, S. H., Lazaro, M.,
tion termination intermediates trapped by the Apidaecin
Gil, D., et al. (2008) Structure of ratcheted ribosomes with
137 peptide during RF3 mediated recycling of RF1,
tRNAs in hybrid states, Proc. Natl. Acad. Sci. USA, 105,
Nat. Commun., 9, 3053, doi: 10.1038/s41467 018 05465 1.
16924 16927, doi: 10.1073/pnas.0809587105.
59.
Florin, T., Maracci, C., Graf, M., Karki, P., Klepacki, D.,
75.
Zhou, J., Lancaster, L., Trakhanov, S., and Noller, H. F.
et al. (2017) An antimicrobial peptide that inhibits trans
(2012) Crystal structure of release factor RF3 trapped in
lation by trapping release factors on the ribosome, Nat.
the GTP state on a rotated conformation of the ribosome,
Struct. Mol. Biol., 24, 752757, doi: 10.1038/nsmb.
RNA, 18, 230 240, doi: 10.1261/rna.031187.111.
3439.
76.
Ermolenko, D. N., Majumdar, Z. K., Hickerson, R. P.,
60.
Freistroffer, D. V., Pavlov, M. Y., MacDougall, J.,
Spiegel, P. C., Clegg, R. M., and Noller, H. F. (2007)
Buckingham, R. H., and Ehrenberg, M. (1997) Release
Observation of intersubunit movement of the ribosome in
factor RF3 in E.coli accelerates the dissociation of release
solution using FRET, J. Mol. Biol., 370, 530 540.
factors RF1 and RF2 from the ribosome in a GTP depen
77.
Gao, H., Zhou, Z., Rawat, U., Huang, C., Bouakaz, L.,
dent manner, EMBO J., 16, 4126 4133, doi: 10.1093/
et al. (2007) RF3 induces ribosomal conformational
emboj/16.13.4126.
changes responsible for dissociation of class I release fac
61.
Svidritskiy, E., Demo, G., Loveland, A. B., Xu, C., and
tors, Cell, 129, 929 941, doi: 10.1016/j.cell.2007.03.050.
Korostelev, A. A. (2019) Extensive ribosome and RF2
78.
Jin, H., Kelley, A. C., and Ramakrishnan, V. (2011) Crystal
rearrangements during translation termination, Elife, 8,
structure of the hybrid state of ribosome in complex with
e46850, doi: 10.7554/eLife.46850.
the guanosine triphosphatase release factor 3, Proc. Natl.
62.
Ling, C., and Ermolenko, D. N. (2016) Structural insights
Acad. Sci. USA, 108, 15798 15803, doi: 10.1073/pnas.
into ribosome translocation, WIREs RNA, 7, 620 636,
1112185108.
doi: 10.1002/wrna.1354.
79.
Leipe, D. D., Wolf, Y. I., Koonin, E. V., and Aravind, L.
63.
Noller, H. F., Lancaster, L., Zhou, J., and Mohan, S.
(2002) Classification and evolution of P loop GTPases and
(2017) The ribosome moves: RNA mechanics and translo
related ATPases, J. Mol. Biol., 317, 41 72, doi: 10.1006/
cation, Nat. Struct. Mol. Biol.,
24,
10211027,
jmbi.2001.5378.
doi: 10.1038/nsmb.3505.
80.
Mikuni, O., Ito, K., Moffat, J., Matsumura, K.,
64.
Voorhees, R. M., and Ramakrishnan, V. (2013) Structural
McCaughan, K., et al. (1994) Identification of the prfC
basis of the translational elongation cycle, Annu. Rev.
gene, which encodes peptide chain release factor 3 of
Biochem., 82, 203 236, doi: 10.1146/annurev biochem
Escherichia coli, Proc. Natl. Acad. Sci. USA, 91, 5798 5802.
113009 092313.
81.
Grentzmann, G., Brechemier Baey, D., Heurgue, V.,
65.
Chen, J., Tsai, A., O’Leary, S. E., Petrov, A., and Puglisi,
Mora, L., and Buckingham, R. H. (1994) Localization and
J. D. (2012) Unraveling the dynamics of ribosome translo
characterization of the gene encoding release factor RF3 in
cation, Curr. Opin. Struct. Biol., 22, 804 814, doi: 10.1016/
Escherichia coli, Proc. Natl. Acad. Sci. USA, 91, 5848 5852.
j.sbi.2012.09.004.
82.
Pai, R. D., Zhang, W., Schuwirth, B. S., Hirokawa, G.,
66.
Frank, J., and Gonzalez, R. L., Jr. (2010) Structure and
Kaji, H., et al. (2008) Structural insights into ribosome
dynamics of a processive Brownian motor: the translating
recycling factor interactions with the 70S ribosome, J. Mol.
ribosome, Annu. Rev. Biochem., 79, 381 412.
Biol., 376, 1334 1347, doi: 10.1016/j.jmb.2007.12.048.
67.
Casy, W., Prater, A. R., and Cornish, P. V. (2018) Operative
83.
Borovinskaya, M. A., Pai, R. D., Zhang, W., Schuwirth,
binding of Class I release factors and YaeJ stabilizes the
B. S., Holton, J. M., et al. (2007) Structural basis for
ribosome in the nonrotated state, Biochemistry, 57, 1954
aminoglycoside inhibition of bacterial ribosome recycling,
1966, doi: 10.1021/acs.biochem.7b00824.
Nat. Struct. Mol. Biol., 14, 727732, doi: 10.1038/
68.
Jin, H., Kelley, A. C., Loakes, D., and Ramakrishnan, V.
nsmb1271.
(2010) Structure of the 70S ribosome bound to release fac
84.
Ayyub, S. A., Gao, F., Lightowlers, R. N., and
tor 2 and a substrate analog provides insights into catalysis
Chrzanowska Lightowlers, Z. M. (2020) Rescuing stalled
of peptide release, Proc. Natl. Acad. Sci. USA, 107, 8593
mammalian mitoribosomes - what can we learn from bac
8598.
teria? J. Cell Sci., 133, doi: 10.1242/jcs.231811.
69.
Wasserman, M. R., Alejo, J. L., Altman, R. B., and
85.
Soleimanpour Lichaei, H. R., Kuhl, I., Gaisne, M.,
Blanchard, S. C. (2016) Multiperspective smFRET reveals
Passos, J. F., Wydro, M., et al. (2007) mtRF1a is a human
rate determining late intermediates of ribosomal translo
mitochondrial translation release factor decoding the
cation, Nat. Struct. Mol. Biol., 23, 333 341, doi: 10.1038/
major termination codons UAA and UAG, Mol. Cell, 27,
nsmb.3177.
745 757.
70.
Cornish, P. V., Ermolenko, D. N., Noller, H. F., and Ha, T.
86.
Nozaki, Y., Matsunaga, N., Ishizawa, T., Ueda, T., and
(2008) Spontaneous intersubunit rotation in single ribo
Takeuchi, N. (2008) HMRF1L is a human mitochondrial
somes, Mol. Cell, 30, 578 588, doi: 10.1016/j.molcel.
translation release factor involved in the decoding of the
2008.05.004.
termination codons UAA and UAG, Genes Cells, 13, 429
71.
Sharma, H., Adio, S., Senyushkina, T., Belardinelli, R.,
438.
Peske, F., and Rodnina, M. V. (2016) Kinetics of sponta
87.
Kummer, E., Schubert, K. N., Schoenhut, T., Scaiola, A.,
neous and EF G accelerated rotation of ribosomal sub
and Ban, N. (2021) Structural basis of translation termina
units, Cell Rep., 16, 2187 2196, doi: 10.1016/j.celrep.
tion, rescue, and recycling in mammalian mitochondria,
2016.07.051.
Mol. Cell, doi: 10.1016/j.molcel.2021.03.042.
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1341
88. Aibara, S., Singh, V., Modelska, A., and Amunts, A. (2020)
106. Shimokawa Chiba, N., Muller, C., Fujiwara, K.,
Structural basis of mitochondrial translation, Elife, 9,
Beckert, B., Ito, K., et al. (2019) Release factor dependent
doi: 10.7554/eLife.58362.
ribosome rescue by BrfA in the Gram positive bacterium
89. Kummer, E., and Ban, N. (2020) Structural insights into
Bacillus subtilis, Nat. Commun., 10, 5397, doi: 10.1038/
mammalian mitochondrial translation elongation cat
s41467 019 13408 7.
alyzed by mtEFG1, EMBO J., 39, e104820, doi: 10.15252/
107. Goralski, T. D. P., Kirimanjeswara, G. S., and Keiler, K. C.
embj.2020104820.
(2018) A new mechanism for ribosome rescue can recruit
90. Zhang, Y., and Spremulli, L. L. (1998) Identification and
RF1 or RF2 to nonstop ribosomes, mBio, 9, doi: 10.1128/
cloning of human mitochondrial translational release fac
mBio.02436 18.
tor 1 and the ribosome recycling factor, Biochim. Biophys.
108. Burroughs, A. M., and Aravind, L. (2019) The origin and
Acta, 1443, 245 250, doi: 10.1016/s0167 4781(98)00223 1.
evolution of release factors: implications for translation
91. Lind, C., Sund, J., and Aqvist, J. (2013) Codon reading
termination, ribosome rescue, and quality control path
specificities of mitochondrial release factors and transla
ways, Int. J. Mol. Sci., 20, doi: 10.3390/ijms20081981.
tion termination at non standard stop codons, Nat.
109. Handa, Y., Inaho, N., and Nameki, N. (2011) YaeJ is a
Commun., 4, 2940, doi: 10.1038/ncomms3940.
novel ribosome associated protein in Escherichia coli that
92. Huynen, M. A., Duarte, I., Chrzanowska Lightowlers,
can hydrolyze peptidyl tRNA on stalled ribosomes,
Z. M., and Nabuurs, S. B. (2012) Structure based hypoth
Nucleic Acids Res., 39, 17391748, doi: 10.1093/nar/
esis of a mitochondrial ribosome rescue mechanism,
gkq1097.
Biol. Direct., 7, 14, doi: 10.1186/1745 6150 7 14.
110. Chan, K. H., Petrychenko, V., Mueller, C., Maracci, C.,
93. Young, D. J., Edgar, C. D., Murphy, J., Fredebohm, J.,
Holtkamp, W., et al. (2020) Mechanism of ribosome rescue
Poole, E. S., and Tate, W. P. (2010) Bioinformatic, struc
by alternative ribosome rescue factor B, Nat. Commun., 11,
tural, and functional analyses support release factor like
4106, doi: 10.1038/s41467 020 17853 7.
MTRF1 as a protein able to decode nonstandard stop
111.
Carbone, C. E., Demo, G., Madireddy, R., Svidritskiy, E.,
codons beginning with adenine in vertebrate mitochondria,
and Korostelev, A. A. (2020) ArfB can displace mRNA to
RNA, 16, 1146 1155.
rescue stalled ribosomes, Nat. Commun., 11, 5552,
94. Temperley, R., Richter, R., Dennerlein, S., Lightowlers,
doi: 10.1038/s41467 020 19370 z.
R. N., and Chrzanowska Lightowlers, Z. M. (2010)
112. Gagnon, M. G., Seetharaman, S. V., Bulkley, D., and
Hungry codons promote frameshifting in human mito
Steitz, T. A. (2012) Structural basis for the rescue of stalled
chondrial ribosomes, Science, 327, 301.
ribosomes: structure of YaeJ bound to the ribosome,
95. Ito, K., Chadani, Y., Nakamori, K., Chiba, S.,
Science, 335, 1370 1372, doi: 10.1126/science.1217443.
Akiyama, Y., and Abo, T. (2011) Nascentome analysis
113. Richter, R., Rorbach, J., Pajak, A., Smith, P. M., Wessels,
uncovers futile protein synthesis in Escherichia coli, PLoS
H. J., et al. (2010) A functional peptidyl tRNA hydrolase,
One, 6, e28413, doi: 10.1371/journal.pone.0028413.
ICT1, has been recruited into the human mitochondrial
96. Hayes, C. S., and Keiler, K. C. (2010) Beyond ribosome
ribosome, EMBO J., 29, 1116 1125.
rescue: tmRNA and co translational processes, FEBS
114. Handa, Y., Hikawa, Y., Tochio, N., Kogure, H.,
Lett., 584, 413 419, doi: 10.1016/j.febslet.2009.11.023.
Inoue, M., et al. (2010) Solution structure of the catalytic
97. Keiler, K. C. (2015) Mechanisms of ribosome rescue in
domain of the mitochondrial protein ICT1 that is essential
bacteria, Nat. Rev. Microbiol., 13, 285 297, doi: 10.1038/
for cell vitality, J. Mol. Biol., 404, 260 273.
nrmicro3438.
115. Feaga, H. A., Quickel, M. D., Hankey Giblin, P. A., and
98. Desai, N., Yang, H., Chandrasekaran, V., Kazi, R.,
Keiler, K. C. (2016) Human cells require non stop ribo
Minczuk, M., and Ramakrishnan, V. (2020) Elongational
some rescue activity in mitochondria, PLoS Genet., 12,
stalling activates mitoribosome associated quality control,
e1005964, doi: 10.1371/journal.pgen.1005964.
Science, 370, 1105 1110, doi: 10.1126/science.abc7782.
116. Pan, X., Tan, J., Weng, X., Du, R., Jiang, Y., et al. (2021)
99. Chadani, Y., Ito, K., Kutsukake, K., and Abo, T. (2012) ArfA
ICT1 Promotes osteosarcoma cell proliferation and
recruits release factor 2 to rescue stalled ribosomes by pep
inhibits apoptosis via STAT3/BCL 2 pathway, Biomed. Res.
tidyl tRNA hydrolysis in Escherichia coli, Mol. Microbiol.,
Int., 2021, 8971728, doi: 10.1155/2021/8971728.
86, 37 50, doi: 10.1111/j.1365 2958.2012.08190.x.
117. Greber, B. J., Boehringer, D., Leitner, A., Bieri, P., Voigts
100. Shimizu, Y. (2012) ArfA recruits RF2 into stalled ribosomes,
Hoffmann, F., et al. (2014) Architecture of the large sub
J. Mol. Biol., 423, 624 631, doi: 10.1016/j.jmb.2012.08.007.
unit of the mammalian mitochondrial ribosome, Nature,
101. Zeng, F., and Jin, H. (2016) Peptide release promoted by
505, 515 519, doi: 10.1038/nature12890.
methylated RF2 and ArfA in nonstop translation is
118. Kater, L., Mitterer, V., Thoms, M., Cheng, J.,
achieved by an induced fit mechanism, RNA, 22, 49 60,
Berninghausen, O., et al. (2020) Construction of the cen
doi: 10.1261/rna.053082.115.
tral protuberance and L1 stalk during 60S subunit biogene
102. Huter, P., Muller, C., Beckert, B., Arenz, S.,
sis, Mol. Cell, 79, 615 628.e615, doi: 10.1016/j.molcel.
Berninghausen, O., et al. (2017) Structural basis for ArfA
2020.06.032.
RF2 mediated translation termination on mRNAs lacking
119. Nikolay, R., Hilal, T., Qin, B., Mielke, T., Burger, J., et al.
stop codons, Nature,
541,
546549, doi:
10.1038/
(2018) Structural visualization of the formation and activa
nature20821.
tion of the 50S ribosomal subunit during in vitro reconstitu
103. Ma, C., Kurita, D., Li, N., Chen, Y., Himeno, H., and
tion, Mol. Cell, 70, 881 893.e883, doi: 10.1016/j.molcel.
Gao, N. (2017) Mechanistic insights into the alternative
2018.05.003.
translation termination by ArfA and RF2, Nature, 541,
120. Davis, J. H., Tan, Y. Z., Carragher, B., Potter, C. S.,
550 553, doi: 10.1038/nature20822.
Lyumkis, D., and Williamson, J. R. (2016) Modular
104. Zeng, F., Chen, Y., Remis, J., Shekhar, M., Phillips, J. C.,
assembly of the bacterial large ribosomal subunit, Cell, 167,
et al. (2017) Structural basis of co translational quality
1610 1622.e1615, doi: 10.1016/j.cell.2016.11.020.
control by ArfA and RF2 bound to ribosome, Nature, 541,
121. Huang, S., Aleksashin, N. A., Loveland, A. B.,
554 557, doi: 10.1038/nature21053.
Klepacki, D., Reier, K., et al. (2020) Ribosome engineer
105. Muller, C., Crowe McAuliffe, C., and Wilson, D. N.
ing reveals the importance of 5S rRNA autonomy for ribo
(2021) Ribosome rescue pathways in bacteria, Front.
some assembly, Nat. Commun., 11, 2900, doi: 10.1038/
Microbiol., 12, 652980, doi: 10.3389/fmicb.2021.652980.
s41467 020 16694 8.
БИОХИМИЯ том 86 вып. 9 2021
1342
КОРОСТЕЛЕВ
122. Waltz, F., Soufari, H., Bochler, A., Giege, P., and
close to the stop codon in the A site of the 80S ribosome,
Hashem, Y. (2020) Cryo EM structure of the RNA rich
Mol. Biol., 41, 781 789.
plant mitochondrial ribosome, Nat. Plants, 6, 377 383,
137. Brown, A., Shao, S., Murray, J., Hegde, R. S., and
doi: 10.1038/s41477 020 0631 5.
Ramakrishnan, V. (2015) Structural basis for stop codon
123. Akabane, S., Ueda, T., Nierhaus, K. H., and Takeuchi, N.
recognition in eukaryotes, Nature,
524,
493496,
(2014) Ribosome rescue and translation termination at
doi: 10.1038/nature14896.
non standard stop codons by ICT1 in mammalian mito
138. Brown, C. M., Stockwell, P. A., Trotman, C. N., and Tate,
chondria, PLoS Genet., 10, e1004616, doi: 10.1371/journal.
W. P. (1990) Sequence analysis suggests that tetra
pgen.1004616.
nucleotides signal the termination of protein synthesis in
124. Vaishya, S., Kumar, V., Gupta, A., Siddiqi, M. I., and
eukaryotes, Nucleic Acids Res., 18, 6339 6345.
Habib, S. (2016) Polypeptide release factors and stop
139. Matheisl, S., Berninghausen, O., Becker, T., and
codon recognition in the apicoplast and mitochondrion of
Beckmann, R. (2015) Structure of a human translation ter
Plasmodium falciparum, Mol. Microbiol., 100, 1080 1095,
mination complex, Nucleic Acids Res., 43, 8615 8626,
doi: 10.1111/mmi.13369.
doi: 10.1093/nar/gkv909.
125. Kogure, H., Hikawa, Y., Hagihara, M., Tochio, N.,
140. Jungreis, I., Lin, M. F., Spokony, R., Chan, C. S.,
Koshiba, S., et al. (2012) Solution structure and siRNA
Negre, N., et al. (2011) Evidence of abundant stop codon
mediated knockdown analysis of the mitochondrial dis
readthrough in Drosophila and other metazoa, Genome
ease related protein C12orf65, Proteins, 80, 2629 2642,
Res., 21, 2096 2113, doi: 10.1101/gr.119974.110.
doi: 10.1002/prot.24152.
141. Swart, E. C., Serra, V., Petroni, G., and Nowacki, M.
126. Antonicka, H., Ostergaard, E., Sasarman, F.,
(2016) Genetic codes with no dedicated stop codon: con
Weraarpachai, W., Wibrand, F., et al. (2010) Mutations in
text dependent translation termination, Cell, 166, 691
C12orf65 in patients with encephalomyopathy and a mito
702, doi: 10.1016/j.cell.2016.06.020.
chondrial translation defect, Am. J. Hum. Genet., 87, 115
142. Heaphy, S. M., Mariotti, M., Gladyshev, V. N., Atkins,
122, doi: 10.1016/j.ajhg.2010.06.004.
J. F., and Baranov, P. V. (2016) Novel ciliate genetic code
127. Wesolowska, M., Gorman, G. S., Alston, C. L., Pajak, A.,
variants including the reassignment of all three stop codons
Pyle, A., et al. (2015) Adult onset leigh syndrome in the
to sense codons in Condylostoma magnum, Mol. Biol. Evol.,
intensive care setting: a novel presentation of a C12orf65
33, 2885 2889, doi: 10.1093/molbev/msw166.
related mitochondrial disease, J. Neuromuscul. Dis., 2,
143. Zahonova, K., Kostygov, A. Y., Sevcikova, T.,
409 419, doi: 10.3233/JND 150121.
Yurchenko, V., and Elias, M. (2016) An unprecedented
128. Zorkau, M., Albus, C. A., Berlinguer Palmini, R.,
non canonical nuclear genetic code with all three termina
Chrzanowska Lightowlers, Z. M. A., and Lightowlers,
tion codons reassigned as sense codons, Curr. Biol., 26,
R. N. (2021) High resolution imaging reveals compart
2364 2369, doi: 10.1016/j.cub.2016.06.064.
mentalization of mitochondrial protein synthesis in cul
144. Anzalone, A. V., Zairis, S., Lin, A. J., Rabadan, R., and
tured human cells, Proc. Natl. Acad. Sci. USA, 118,
Cornish, V. W. (2019) Interrogation of eukaryotic stop
doi: 10.1073/pnas.2008778118.
codon readthrough signals by in vitro RNA selection,
129. Chandrasekaran, V., Juszkiewicz, S., Choi, J., Puglisi,
Biochemistry, 58, 1167 1178, doi: 10.1021/acs.biochem.
J. D., Brown, A., et al. (2019) Mechanism of ribosome
8b01280.
stalling during translation of a poly(A) tail, Nat. Struct.
145. Firth, A. E., Wills, N. M., Gesteland, R. F., and Atkins, J. F.
Mol. Biol., 26, 11321140, doi: 10.1038/s41594 019
(2011) Stimulation of stop codon readthrough: frequent
0331 x.
presence of an extended 3′ RNA structural element, Nucleic
130. Tesina, P., Lessen, L. N., Buschauer, R., Cheng, J., Wu,
Acids Res., 39, 6679 6691, doi: 10.1093/nar/gkr224.
C. C., et al. (2020) Molecular mechanism of translational
146. Bonetti, B., Fu, L., Moon, J., and Bedwell, D. M. (1995)
stalling by inhibitory codon combinations and poly(A)
The efficiency of translation termination is determined by
tracts, EMBO J., 39, e103365, doi: 10.15252/embj.
a synergistic interplay between upstream and downstream
2019103365.
sequences in Saccharomyces cerevisiae, J. Mol. Biol., 251,
131. Frolova, L., Le Goff, X., Zhouravleva, G., Davydova, E.,
334 345.
Philippe, M., and Kisselev, L. (1996) Eukaryotic polypep
147. Harrell, L., Melcher, U., and Atkins, J. F.
(2002)
tide chain release factor eRF3 is an eRF1 and ribosome
Predominance of six different hexanucleotide recoding sig
dependent guanosine triphosphatase, RNA, 2, 334 341.
nals 3′ of read through stop codons, Nucleic Acids Res., 30,
132. Alkalaeva, E. Z., Pisarev, A. V., Frolova, L. Y., Kisselev,
2011 2017, doi: 10.1093/nar/30.9.2011.
L. L., and Pestova, T. V. (2006) In vitro reconstitution of
148. Shao, S., Murray, J., Brown, A., Taunton, J.,
eukaryotic translation reveals cooperativity between release
Ramakrishnan, V., and Hegde, R. S. (2016) Decoding
factors eRF1 and eRF3, Cell, 125, 1125 1136.
mammalian ribosome mRNA states by translational
133. Salas Marco, J., and Bedwell, D. M. (2004) GTP hydrol
GTPase complexes, Cell,
167,
12291240.e1215,
ysis by eRF3 facilitates stop codon decoding during
doi: 10.1016/j.cell.2016.10.046.
eukaryotic translation termination, Mol. Cell Biol., 24,
149. Muhs, M., Hilal, T., Mielke, T., Skabkin, M. A.,
7769 7778.
Sanbonmatsu, K. Y., et al. (2015) Cryo EM of ribosomal
134. Frolova, L., Seit Nebi, A., and Kisselev, L. (2002) Highly
80S complexes with termination factors reveals the translo
conserved NIKS tetrapeptide is functionally essential in
cated cricket paralysis virus IRES, Mol. Cell, 57, 422 432,
eukaryotic translation termination factor eRF1, RNA, 8,
doi: 10.1016/j.molcel.2014.12.016.
129 136.
150. Des Georges, A., Hashem, Y., Unbehaun, A., Grassucci,
135. Muramatsu, T., Heckmann, K., Kitanaka, C., and
R. A., Taylor, D., et al. (2014) Structure of the mammalian
Kuchino, Y. (2001) Molecular mechanism of stop codon
ribosomal pre termination complex associated with
recognition by eRF1: a wobble hypothesis for peptide anti
eRF1.eRF3.GDPNP, Nucleic Acids Res., 42, 3409 3418,
codons, FEBS Lett., 488, 105 109, doi: 10.1016/s0014
doi: 10.1093/nar/gkt1279.
5793(00)02391 7.
151. Li, W., Chang, S. T., Ward, F. R., and Cate, J. H. D. (2020)
136. Bulygin, K. N., Popugaeva, E. A., Repkova, M. N.,
Selective inhibition of human translation termination by a
Meschaninova, M. I., Ven’yaminova, A. G., et al. (2007)
drug like compound, Nat. Commun., 11, 4941, doi: 10.1038/
The C domain of translation termination factor eRF1 is
s41467 020 18765 2.
БИОХИМИЯ том 86 вып. 9 2021
ТЕРМИНАЦИЯ И СПАСЕНИЕ РИБОСОМЫ
1343
152. Preis, A., Heuer, A., Barrio Garcia, C., Hauser, A., Eyler,
167. Van den Elzen, A. M., Schuller, A., Green, R., and
D. E., et al. (2014) Cryoelectron microscopic structures of
Seraphin, B. (2014) Dom34 Hbs1 mediated dissociation of
eukaryotic translation termination complexes containing
inactive 80S ribosomes promotes restart of translation after
eRF1 eRF3 or eRF1 ABCE1, Cell Rep.,
8,
5965,
stress, EMBO J.,
33,
265276, doi:
10.1002/embj.
doi: 10.1016/j.celrep.2014.04.058.
201386123.
153. Pisarev, A. V., Skabkin, M. A., Pisareva, V. P., Skabkina,
168. Guydosh, N. R., and Green, R. (2014) Dom34 rescues
O. V., Rakotondrafara, A. M., et al. (2010) The role of
ribosomes in 3′ untranslated regions, Cell, 156, 950 962,
ABCE1 in eukaryotic posttermination ribosomal recycling,
doi: 10.1016/j.cell.2014.02.006.
Mol. Cell, 37, 196 210.
169. Passos, D. O., Doma, M. K., Shoemaker, C. J.,
154. Annibaldis, G., Domanski, M., Dreos, R., Contu, L.,
Muhlrad, D., Green, R., et al. (2009) Analysis of Dom34
Carl, S., et al. (2020) Readthrough of stop codons under
and its function in no go decay, Mol. Biol. Cell, 20, 3025
limiting ABCE1 concentration involves frameshifting and
3032, doi: 10.1091/mbc.E09 01 0028.
inhibits nonsense mediated mRNA decay, Nucleic Acids
170. Shoemaker, C. J., Eyler, D. E., and Green, R. (2010)
Res., 48, 10259 10279, doi: 10.1093/nar/gkaa758.
Dom34:Hbs1 promotes subunit dissociation and peptidyl
155. Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A.
tRNA drop off to initiate no go decay, Science, 330, 369
G., and Green, R. (2015) Rli1/ABCE1 recycles terminat
372, doi: 10.1126/science.1192430.
ing ribosomes and controls translation reinitiation in 3′
171. Becker, T., Armache, J. P., Jarasch, A., Anger, A. M.,
UTRs in vivo, Cell, 162, 872 884, doi: 10.1016/j.cell.
Villa, E., et al. (2011) Structure of the no go mRNA decay
2015.07.041.
complex Dom34 Hbs1 bound to a stalled 80S ribosome,
156. Lawson, M. R., Lessen, L. N., Wang, J., Prabhakar, A.,
Nat. Struct. Mol. Biol., 18, 715720, doi: 10.1038/
Corsepius, N. C., et al. (2021) Mechanisms that ensure
nsmb.2057.
speed and fidelity in eukaryotic translation termination,
172. Pisareva, V. P., Skabkin, M. A., Hellen, C. U., Pestova,
bioRxiv, doi: 10.1101/2021.04.01.438116.
T. V., and Pisarev, A. V. (2011) Dissociation by Pelota,
157. Kryuchkova, P., Grishin, A., Eliseev, B., Karyagina, A.,
Hbs1 and ABCE1 of mammalian vacant 80S ribosomes
Frolova, L., and Alkalaeva, E. (2013) Two step model of
and stalled elongation complexes, EMBO J., 30, 1804
stop codon recognition by eukaryotic release factor eRF1,
1817, doi: 10.1038/emboj.2011.93.
Nucleic Acids Res., 41, 4573 4586, doi: 10.1093/nar/
173. Kostova, K. K., Hickey, K. L., Osuna, B. A., Hussmann,
gkt113.
J. A., Frost, A., et al. (2017) CAT tailing as a fail safe
158. Svidritskiy, E., Demo, G., and Korostelev, A. A. (2018)
mechanism for efficient degradation of stalled nascent
Mechanism of premature translation termination on a
polypeptides, Science, 357, 414 417, doi: 10.1126/science.
sense codon, J. Biol. Chem.,
293,
1247212479,
aam7787.
doi: 10.1074/jbc.AW118.003232.
174. Verma, R., Reichermeier, K. M., Burroughs, A. M., Oania,
159. Gouridis, G., Hetzert, B., Kiosze Becker, K., de Boer, M.,
R. S., Reitsma, J. M., et al. (2018) Vms1 and ANKZF1
Heinemann, H., et al. (2019) ABCE1 controls ribosome
peptidyl tRNA hydrolases release nascent chains from
recycling by an asymmetric dynamic conformational equi
stalled ribosomes, Nature, 557, 446 451, doi: 10.1038/
librium, Cell Rep., 28, 723 734.e726, doi: 10.1016/j.celrep.
s41586 018 0022 5.
2019.06.052.
175. Zurita Rendon, O., Fredrickson, E. K., Howard, C. J., Van
160. Heuer, A., Gerovac, M., Schmidt, C., Trowitzsch, S.,
Vranken, J., Fogarty, S., et al. (2018) Vms1p is a release fac
Preis, A., et al. (2017) Structure of the 40S ABCE1 post
tor for the ribosome associated quality control complex,
splitting complex in ribosome recycling and translation ini
Nat. Commun., 9, 2197, doi: 10.1038/s41467 018 04564 3.
tiation, Nat. Struct. Mol. Biol., 24, 453 460, doi: 10.1038/
176. Yip, M. C. J., Keszei, A. F. A., Feng, Q., Chu, V.,
nsmb.3396.
McKenna, M. J., and Shao, S. (2019) Mechanism for
161. Mancera Martinez, E., Brito Querido, J., Valasek, L. S.,
recycling tRNAs on stalled ribosomes, Nat. Struct. Mol.
Simonetti, A., and Hashem, Y. (2017) ABCE1: a special
Biol., 26, 343 349, doi: 10.1038/s41594 019 0211 4.
factor that orchestrates translation at the crossroad
177. Kuroha, K., Zinoviev, A., Hellen, C. U. T., and Pestova,
between recycling and initiation, RNA Biol., 14, 1279
T. V. (2018) Release of ubiquitinated and non ubiquitinat
1285, doi: 10.1080/15476286.2016.1269993.
ed nascent chains from stalled mammalian ribosomal com
162. Kratzat, H., Mackens Kiani, T., Ameismeier, M.,
plexes by ANKZF1 and Ptrh1, Mol. Cell, 72,
286
Potocnjak, M., Cheng, J., et al. (2021) A structural inven
302.e288, doi: 10.1016/j.molcel.2018.08.022.
tory of native ribosomal ABCE1 43S pre initiation com
178. Yip, M. C. J., Savickas, S., Gygi, S. P., and Shao, S. (2020)
plexes, EMBO J., 40, e105179, doi: 10.15252/embj.
ELAC1 repairs tRNAs cleaved during ribosome associated
2020105179.
quality control, Cell Rep.,
30,
21062114.e2105,
163. Hilal, T., Yamamoto, H., Loerke, J., Burger, J., Mielke, T.,
doi: 10.1016/j.celrep.2020.01.082.
and Spahn, C. M. (2016) Structural insights into ribosomal
179. Defenouillere, Q., Yao, Y., Mouaikel, J., Namane, A.,
rescue by Dom34 and Hbs1 at near atomic resolution,
Galopier, A., et al. (2013) Cdc48 associated complex
Nat. Commun., 7, 13521, doi: 10.1038/ncomms13521.
bound to 60S particles is required for the clearance of aber
164. Becker, T., Franckenberg, S., Wickles, S., Shoemaker,
rant translation products, Proc. Natl. Acad. Sci. USA, 110,
C. J., Anger, A. M., et al. (2012) Structural basis of highly
5046 5051, doi: 10.1073/pnas.1221724110.
conserved ribosome recycling in eukaryotes and archaea,
180. Verma, R., Oania, R. S., Kolawa, N. J., and Deshaies, R. J.
Nature, 482, 501 506, doi: 10.1038/nature10829.
(2013) Cdc48/p97 promotes degradation of aberrant
165. Su, T., Izawa, T., Thoms, M., Yamashita, Y., Cheng, J.,
nascent polypeptides bound to the ribosome, Elife, 2,
et al. (2019) Structure and function of Vms1 and Arb1 in
e00308, doi: 10.7554/eLife.00308.
RQC and mitochondrial proteome homeostasis, Nature,
181. Brandman, O., Stewart Ornstein, J., Wong, D.,
570, 538 542, doi: 10.1038/s41586 019 1307 z.
Larson, A., Williams, C. C., et al. (2012) A ribosome
166. Inada, T. (2020) Quality controls induced by aberrant
bound quality control complex triggers degradation of
translation, Nucleic Acids Res.,
48,
10841096,
nascent peptides and signals translation stress, Cell, 151,
doi: 10.1093/nar/gkz1201.
1042 1054, doi: 10.1016/j.cell.2012.10.044.
БИОХИМИЯ том 86 вып. 9 2021
1344
КОРОСТЕЛЕВ
DIVERSITY AND SIMILARITY OF TERMINATION AND RIBOSOME
RESCUE IN BACTERIAL, MITOCHONDRIAL,
AND CYTOPLASMIC TRANSLATION
Review
A. A. Korostelev
RNA Therapeutics Institute, Department of Biochemistry and Molecular Pharmacology, UMass Medical School,
Worcester, MA, USA; E mail: Andrei.Korostelev@umassmed.edu
When a ribosome encounters the stop codon of an mRNA, it terminates translation, releases the newly made protein,
and is recycled to initiate translation on a new mRNA. Termination is a highly dynamic process in which release fac
tors (RF1 and RF2 in bacteria; eRF1•eRF3•GTP in eukaryotes) coordinate peptide release with large scale mole
cular rearrangements of the ribosome. Ribosomes stalled on aberrant mRNAs are rescued and recycled by diverse bac
terial, mitochondrial, or cytoplasmic quality control mechanisms. These are catalyzed by rescue factors with peptidyl
tRNA hydrolase activity (bacterial ArfA•RF2 and ArfB, mitochondrial ICT1 and mtRF R, and cytoplasmic Vms1),
that are distinct from each other and from release factors. Nevertheless, recent structural studies demonstrate a
remarkable similarity between translation termination and ribosome rescue mechanisms. This review describes how
these pathways rely on inherent ribosome dynamics, emphasizing the active role of the ribosome in all translation
steps.
Keywords: translation, termination, ribosome, rescue
БИОХИМИЯ том 86 вып. 9 2021