БИОХИМИЯ, 2022, том 87, вып. 3, с. 321 - 336
УДК 577.29
ИЗУЧЕНИЕ ВЗАИМОДЕЙСТВИЯ ДНК ЗАВИСИМЫХ БЕЛКОВ
СЕМЕЙСТВА PARP С НУКЛЕОСОМНЫМИ ЧАСТИЦАМИ,
СОДЕРЖАЩИМИ ИНТЕРМЕДИАТЫ НАЧАЛЬНЫХ СТАДИЙ
ПРОЦЕССА BER
© 2022
А.А. Украинцев1#, Е.А. Белоусова1#, М.М. Кутузов1, О.И. Лаврик1,2*
1 Институт химической биологии и фундаментальной медицины СО РАН, 630090 Новосибирск, Россия;
электронная почта: lavrik@niboch.nsc.ru
2 Новосибирский государственный университет, 630090 Новосибирск, Россия
Поступила в редакцию 05.10.2021
После доработки 20.01.2022
Принята к публикации 22.02.2022
Клеточным сигналом в ответ на повреждение ДНК является реакция (ADP рибозил)ирования, катализиру
емая ДНК зависимыми белками семейства поли(ADP рибозо)полимераз (PARP) - PARP1, PARP2 и
PARP3. Эти белки являются участниками процесса эксцизионной репарации оснований (BER). Несмотря
на обширные исследования, остаётся неизвестным, каким образом PARP участвуют в регуляции процес
са BER и как распределяются роли между ДНК зависимыми членами семейства. В настоящей работе мы
исследовали взаимодействие ДНК зависимых белков семейства PARP с нуклеосомными частицами, содер
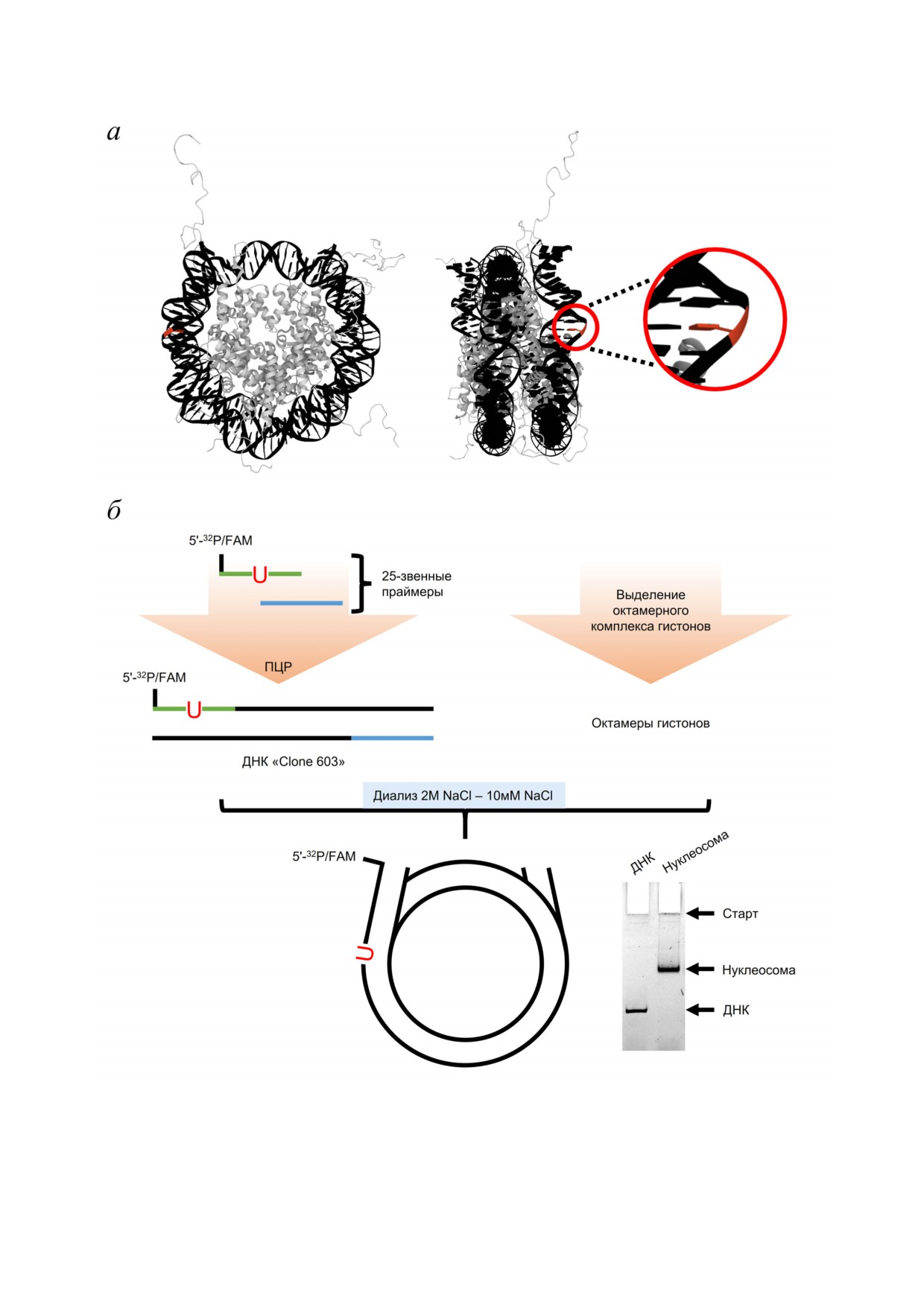
жащими в своем составе ДНК интермедиаты начальных стадий процесса BER. Для этого были реконструи
рованы нуклеосомы на основе последовательности ДНК Widom «Clone 603», содержащие повреждение
вблизи одного из тупых концов ДНК дуплекса. Были определены константы диссоциации комплексов
PARP c нуклеосомами, ДНК которых содержали урацил (Native), апуриновый/апиримидиновый сайт (AP
сайт) или однонуклеотидную брешь c 5′ dRp фрагментом (Gap). Показано, что сродство белков к нуклео
соме увеличивается в ряду PARP3 << PARP2 < PARP1, а сродство каждого белка к определённому типу пов
реждений увеличивается в ряду Native = AP сайт < Gap для PARP1 и PARP2 и Gap <<< Native = AP сайт -
для PARP3. Также были определены области взаимодействия белков PARP с нуклеосомой методами боргид
ридной сшивки и футпринтинга. На основании полученных и литературных данных обсуждены закономер
ности вовлечения PARP1, PARP2 и PARP3 во взаимодействие с нуклеосомными частицами, содержащими
ДНК интермедиаты процесса BER.
КЛЮЧЕВЫЕ СЛОВА: нуклеосома, PARP1, PARP2, PARP3, BER.
DOI: 10.31857/S0320972522030022
ВВЕДЕНИЕ
ты (АР сайты), алкилированные или окислен
ные азотистые основания [1-3]. Процесс BER
BER (base excision repair) - система эксцизи
проходит в несколько этапов и требует актив
онной репарации оснований ДНК, действие ко
ности целого ряда ферментов и белковых факто
торой направлено на удаление небольших пов
ров. На первом этапе происходит распознавание
реждений, возникающих в процессе нормаль
повреждённого азотистого основания с после
ной жизнедеятельности клетки и не вызываю
дующим его удалением; на втором этапе проис
щих значительных искажений структуры двой
ходит гидролиз сахарно фосфатного остова по
ной спирали ДНК. К таким повреждениям от
остатку АР сайта; на третьем - реализуется про
носятся апуриновые/апиримидиновые сай
цессинг разрыва, включающий коррекцию кон
цов разрыва и включение комплементарно
го dNMP; на последнем этапе происходит лиги
Принятые сокращения: пнт - пара нуклеотидов;
AP сайт - апуриновый/апиримидиновый сайт; AP клас
рование цепи ДНК [4].
тер - кластер AP сайтов; APE1 - апуриновая/апиримиди
Несмотря на то что сам процесс BER извес
новая эндонуклеаза 1; BER - эксцизионная репарация ос
тен достаточно давно, механизмы его регуляции
нований; DNase I - дезоксирибонуклеаза I; 5′ dRP - 5′ де
являются основным предметом исследований
зоксирибозофосфат; Gap - 5′ dRp фрагмент; NCP - нук
многих лабораторий. В ответ на повреждение
леосома, нуклеосомная частица; PARилирование - по
ли(ADP рибозил)ирование; PARP - поли(ADP рибо
молекулы ДНК регуляция процессов репарации
зо)полимераза; UDG - урацил ДНК гликозилаза.
осуществляется в том числе с помощью клеточ
* Адресат для корреспонденции.
ного сигнала поли(ADP рибозил)ирова
# Авторы внесли равный вклад в работу.
ния (PARилирования), катализируемого белка
2
321
322
УКРАИНЦЕВ и др.
ми семейства поли(ADP рибозо)полимераз
например репарационных ДНК связывающих
(PARP) [5]. Из 17 представителей семейства
белков. Нуклеотиды в составе ДНК дуплекса
PARP три белка (PARP1, PARP2 и PARP3) явля
располагаются несимметрично относительно
ются ДНК зависимыми, активируются и участ
гистонового октамера и могут быть обращены
вуют в формировании клеточного ответа на по
внутрь, наружу или занимать промежуточное
вреждение ДНК [6-8]. Белки PARP1 и PARP2 ка
положение относительно октамера гисто
тализируют присоединение полимера ADP ри
нов [32]. Различная ориентация нуклеотидов
бозы, а PARP3 - перенос одного остатка ADP
накладывает отпечаток на функциональную ак
рибозы на белок акцептор [7-9]. Их мишенями
тивность ферментов при их взаимодействии с
преимущественно являются белки, обеспечива
нуклеосомой [33, 34]. Наиболее доступными для
ющие динамику процесса укладки хроматина и
ферментов являются ориентированные наружу
метаболизм ДНК, включая гистоны, транскрип
нуклеотиды, наименее
- ориентированные
ционные факторы, белки репарации ДНК, а
внутрь.
также сами PARP1, PARP2 и PARP3 [8, 10-14].
На сегодняшний день известно, что при взаи
Белок PARP1 способен распознавать различ
модействии с нуклеосомной частицей PARP1
ные повреждения ДНК, в том числе интермеди
связывается как с нуклеосомной ДНК [35], так и
аты процесса BER, активируется при взаимо
с октамером гистонов [36, 37]. PARP2 тоже спо
действии с ними и катализирует реак
собен эффективно взаимодействовать с ДНК в
цию PARилирования в ответ на их появление [9,
составе нуклеосомы [38]. Группа Caldecott [11]
15-22]. PARP2 активируется при взаимодей
показала, что в нуклеосомном контексте PARP3
ствии с одноцепочечными разрывами, частич
может являться сенсором одноцепочечных раз
ными ДНК дуплексами с одно и двунуклеотид
рывов с 5′ P и 3′ OH группами, но количест
ными брешами и флэп структурами [9, 21-26].
венные характеристики такого взаимодействия
Было показано, что PARP3 также может высту
определены не были. Кроме того, PARP3 спосо
пать в роли сенсора одноцепочечных разрывов,
бен моно(АДФ рибозил)ировать гистон H2B по
но, в отличие от PARP1, PARP3 активируется
аминокислотному остатку Glu2 in vitro. Таким
in vitro только в том случае, если 5′ конец разры
образом, совокупность полученных данных го
ва содержит фосфатную группу [24, 11]. Кроме
ворит о способности ДНК зависимых белков
того, было показано, что PARP1, PARP2
семейства PARP взаимодействовать с разными
и PARP3 катализируют ADP рибозилирова
компонентами NCP. Однако характер взаимо
ние ДНК, и реакция идёт наиболее эффективно
действий PARP1, PARP2 и PARP3 с нуклеосом
при взаимодействии с ДНК дуплексами, содер
ной частицей, содержащей в своём составе ин
жащими однонуклеотидную брешь с канони
термедиаты начальных стадий процесса BER,
ческими 5′ P и 3′ OH группами [27-29].
остаётся неизвестным.
На сегодняшний день множество работ по
Настоящая работа посвящена исследованию
исследованию регуляции процесса BER с по
взаимодействия ДНК зависимых белков семей
мощью ДНК зависимых белков PARP были
ства PARP, а именно: PARP1, PARP2 и PARP3, c
проведены в контексте свободных ДНК
нуклеосомными частицами, имитирующими
субстратов, тогда как в клетке ДНК компактизо
интермедиаты начальных стадий процесса экс
вана в хроматин. Базовой структурной единицей
цизионной репарации оснований. В качестве
хроматина является нуклеосома (NCP). Нуклео
субстрата были использованы NCP, реконструи
сома представляет собой белок нуклеиновый
рованные в присутствии октамера гистонов до
комплекс, в котором вокруг белкового ядра на
машних кур Gallus gallus и 147 пнт ДНК последо
мотана ДНК длиной 147 пнт [30, 31] (рис. 1, а).
вательности Widom «Clone 603», содержащей
Ядро нуклеосомы состоит из четырёх гетероди
изотоп 32P или остаток FAM на одном из 5′ кон
меров гистонов: двух H3-H4 и двух H2A-H2B.
цов ДНК дуплекса и имеющей в своём составе
Гистоны являются высококонсервативными
урацил в положении 12 от 5′ меченого кон
белками среди эукариот и имеют молекулярную
ца (рис. 1, б). Интермедиаты начальных стадий
массу ~11-15 кДа. Гистоновый октамер связы
процесса BER, т.е. NCP, содержащие в своём
вается с ДНК длиной ~121 пнт, ещё по 13 пнт с
составе ДНК с AP сайтом или одноцепочечным
концов ДНК поддерживается за счёт взаимодей
разрывом с 5′ дезоксирибозофосфатом (5′ dRp)
ствий с N концевыми α спиралями гисто
и 3′ P группами, получали при обработке рекон
нов H3. Кроме того, 8 оставшихся N и C «хвос
струированных частиц урацил ДНК гликозила
тов» гистонов, имеющих большую подвиж
зой (UDG) и/или апуриновой/апиримидиновой
ность, выступают за пределы нуклеосомного яд
эндонуклеазой 1 (APE1). С помощью методов
ра. Потенциально эти участки могут влиять на
EMSA, боргидридной сшивки и ферментатив
белок нуклеиновые взаимодействия других,
ного футпринтинга с использованием дезокси
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
323
Рис. 1. Структура нуклеосомы (PDB: ID 1KX5); красным выделен нуклеотид в позиции 12 ДНК дуплекса, гетероцикли
ческое основание которого обращено наружу относительно октамера гистонов (а). б - Схема реконструирования нуклео
сомы (согласно [31]) и автограф 4% ного нативного ПААГ после разделения продуктов реакции реконструирования
нуклеосом
БИОХИМИЯ том 87 вып. 3 2022
2*
324
УКРАИНЦЕВ и др.
рибонуклеазы I (DNase I) были охарактеризова
[γ32P] АТР полинуклеотидкиназы Т4, как опи
ны взаимодействия белков PARP1, PARP2 и
сано в работе Belousova et al. [28], либо мечен
PARP3 с NCP. На основании полученных ре
ный флуоресцеином при химическом синтезе.
зультатов было выдвинуто предположение о раз
Амплификацию ДНК с использованием
ном характере взаимодействия ДНК зависимых
5′ [32P] или 5′ FAM олигодезоксирибонуклео
белков семейства PARP с повреждениями ДНК,
тидных праймеров для последующего рекон
имитирующими интермедиаты начальных эта
струирования NCP проводили в соответствии с
пов процесса BER в нуклеосомном контексте.
протоколом, описанным ранее [31]. При нара
ботке ДНК субстрата, содержащего статисти
чески распределённый dUMP для получения
МАТЕРИАЛЫ И МЕТОДЫ
кластера AP сайтов (AP кластера), реакционная
смесь для амплификации содержала смесь
Для наработки целевой ДНК была использо
dNTP в соотношении dATP/dGTP/dCTP/
вана плазмида pGEM 3z/603
(«AddGene»,
dUTP/dTTP = 1/1/1/0,9/0,1. Чистоту препарата
США), содержащая последовательность ДНК
реконструированных NCP оценивали по элект
Widom «Clone 603»: 5′ ACCCCAGGGACTTG
рофоретической подвижности в 4% ном ПААГ
AAGTAATAAGGACGGAGGGCCTCTTTCAA
в неденатурирующих условиях. При использо
CATCGATGCACGGTGGTTAGCCTTGGA
вании флуоресцентных компонентов в смеси
TTGCGCTCTACCGTGCGCTAAGCGTACTTA
после разделения продуктов реакции в геле де
GAAGCCCGAGTGACGACTTCACACGGTAG
текцию проводили с помощью системы
GTGGGCGCGCGAACTGGG 3′, 147 нт.
«Typhoon FLA
9500»
(«GE Healthcare Life
Синтетические олигодезоксирибонуклеоти
Science», США) с использованием лазера с дли
ды, использованные в качестве праймеров при
ной волны возбуждения 473 нм. При использо
амплификации ДНК «Clone 603», в том числе
вании радиоактивных компонентов детекцию
олигодезоксирибонуклеотиды, меченные флуо
проводили с помощью радиолюминесцентного
ресцеином по 5′ концу (5′ FAM), были синтези
сканирования экрана Image Screen K («Kodak»,
рованы в Лаборатории биомедицинской химии
США) с помощью системы «Typhoon FLA 9500»
ИХБФМ СО РАН (Россия): 5′ ACCCCAGGGA
с использованием лазера с длиной волны воз
CUTGAAGTAATAAGG 3′; 5′ FAM ACCCCA
буждения 635 нм. Полученные электрофоре
GGGACUTGAAGTAATAAGG 3′; 5′ CCCAG
граммы анализировали с использованием про
TTCGCGCGCCCACC 3′.
граммы
«Quantity One»
(«Bio Rad», США).
В работе использовали следующие реакти
Во всех случаях АР сайт получали in situ с по
вы: диализные мембраны 3,5 кДа («Spectrum
мощью активности UDG. Для этого реакцион
Laboratories Inc.», США); бромфеноловый си
ную смесь, содержащую 0,1 мкМ ДНК или
ний и ксиленцианол («Fluka», Швейцария);
NCP с одиночным или статистически распреде
[γ32P] ATP
с
удельной
активностью
лённым по ДНК остатком урацила и
185 ТБк/ммоль и [α32P] ATP с удельной актив
4 мкМ UDG в буфере следующего состава:
ностью 111 ТБк/ммоль (Лаборатория биотехно
10
мМ Tris HCl (pH
7,5),
0,1% NP 40,
логии ИХБФМ СО РАН); Taq полимера
0,2 мМ EDTA, 5 мМ β ME, 10 мM NaCl; инку
за
(«БИОСАН», Россия); DNase I
(«New
бировали при 37 °C в течение 30 мин, после че
England Biolabs», США). Основные реактивы,
го использовали для дальнейших превращений.
использованные в работе, были производства
Получение субстратов, содержащих брешь, осу
«Sigma» (США).
ществляли ферментативным гидролизом AP
Рекомбинантные белки PARP1 человека,
сайта с помощью APE1. Реакционная смесь со
PARP2 Mus musculus, PARP3 человека, гистоно
держала 0,1 мкМ ДНК или NCP с одиночным
вые октамеры H2A, H2B, H3 и H4 G. gallus были
остатком урацила, 4 мкМ UDG и 10 нМ APE1 в
выделены согласно опубликованным ранее ме
буфере следующего состава: 10 мМ Tris HCl
тодикам [28, 31, 39] соответственно. Рекомби
(pH
7,5),
0,1% NP 40,
0,2
мМ EDTA,
нантная APE1 человека и урацил ДНК глико
5 мМ β ME, 10 мM NaCl и 5 мM MgCl2. Смесь
зилаза E. coli были любезно предоставлены Хо
инкубировали при 37 °C в течение 30 мин, пос
дыревой С.Н. (ИХБФМ СО РАН), полинуклео
ле чего использовали для дальнейших превра
тидкиназа фага Т4 - Петрусевой И.О. (ИХБФМ
щений.
СО РАН).
Определение значений Kd. Значения констант
Получение субстратов ДНК и NCP. В работе
диссоциации определяли методом EMSA, сог
использовали олигодезоксирибонуклеотидный
ласно материалам работы Kutuzov et al. [38]. Ре
праймер, содержащий в положении 12 оста
акционная смесь объёмом 10 мкл содержала
ток dUMP и меченный по 5′ концу с помощью
0,1 мкM 5′ [32P] меченой ДНК или NCP и 0,001,
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
325
0,005, 0,01, 0,05, 0,1, 0,5, 1 мкМ PARP1, PARP2
смесь обрабатывали метоксиамином в конечной
или PARP3 или 0,05, 0,1, 0,3, 0,5, 0,7, 0,85, 1,
концентрации 30 мМ во льду в течение 30 мин.
1,5 мкМ PARP3 для структуры с 5′ dRp фраг
Реакцию останавливали добавлением 1/5 объё
ментом (Gap) Gap ДНК и 0,2, 0,5, 1, 1,5, 3, 5,
ма раствора, содержащего 90% формамида,
7,5, 10 мкМ PARP3 - для структуры Gap NCP в
50 мМ EDTA, 0,025% бромфенолового синего и
буфере следующего состава: 10 мМ Tris HCl
0,025% ксиленцианола. Пробы разделяли в
(pH
7,5),
0,1% NP 40,
0,2
мМ EDTA,
20% ном ПААГ в денатурирующих условиях.
5 мМ β ME, 10 мM NaCl и 5 мM MgCl2. Реакци
Детекцию и обработку результатов проводили,
онную смесь инкубировали при 37 °C в течение
как описано выше.
20 мин. Компоненты реакционной смеси разде
ляли в 4% ном ПААГ в неденатурирующих ус
ловиях. Детекцию продуктов электрофорети
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
ческого разделения в геле проводили, как опи
сано выше. Расчёт значений констант диссоци
Определение значений Kd комплексов
ации проводили с использованием программно
ДНК/NCP-PARP1/PARP2/PARP3 методом за
го обеспечения OriginPro
7.5
(«OriginLab
держки в геле. Для оценки вклада взаимодей
Corporation», США).
ствий ДНК зависимых белков семейства PARP
Изучение белок нуклеиновых взаимодействий
с повреждением в составе ДНК дуплекса или
методом боргидридной сшивки. Реакционную
гистоновыми компонентами NCP был исполь
смесь объёмом 10 мкл, содержащую 0,2 мкМ
зован метод EMSA [41]. В работе были исполь
5′ [FAM] меченую АР ДНК или АP NCP и
зованы ДНК субстраты, свободный ДНК дуп
0,005, 0,01, 0,05, 0,1, 0,5, 1 мкМ PARP1, PARP2
лекс и намотанный на октамер гистонов, ими
или PARP3 в буфере следующего состава:
тирующие интермедиаты начальных стадий
10
мМ Tris HCl (pH
7,5),
0,1% NP 40,
процесса BER - 147 пнт ДНК c AP сайтом и
0,2 мМ EDTA, 5 мМ β ME, 10 мM NaCl; инку
147 пнт ДНК, содержащая однонуклеотидную
бировали при 37 °C в течение 20 мин. Далее к
брешь с 5′ dRР фрагментом - Gap (рис. 2, а).
смеси добавляли NaBH4 до конечной концентра
Для контрольных экспериментов была исполь
ции 20 мМ и инкубировали во льду в течение
зована нативная форма 147 пнт ДНК. Наличие
30 мин, после чего реакционную смесь прогрева
на радиоавтографе полос, обладающих меньшей
ли при 65 °C в течение 5 мин для удаления остат
электрофоретической подвижностью по сравне
ков непрореагировавшего NaBH4. Для деграда
нию с контрольным препаратом, говорит об об
ции несвязанной белками ДНК в реакционную
разовании продуктов комплексообразования
смесь добавляли 1 Ед DNase I и инкубировали
белков PARP со свободной ДНК и ДНК/гисто
при 37 °C в течение 15 мин. Реакцию останавли
нами в составе NCP (рис. 2, б). По результатам
вали добавлением 1/5 объёма раствора буфера
EMSA были рассчитаны значения констант дис
Лэммли. Анализ компонентов реакционной сме
социации для всех исследованных типов комп
си проводили с помощью гель электрофореза в
лексов (таблица).
10% ном ПААГ по Лэммли [40]. Детекцию про
Оказалось, что сродство ДНК связывающих
дуктов реакции проводили, как описано выше.
белков PARP к использованным субстратам из
Изучение белок нуклеиновых взаимодействий
меняется в ряду PARP3 << PARP2 < PARP1. При
методом ферментативного футпринтинга. Реакци
появлении повреждения в составе ДНК дуплек
онная смесь объёмом 10 мкл содержала 0,2 мкМ
са белки PARP1 и PARP2 проявляют следующую
5′ [32P] меченой ДНК или NCP и 0,1 мкМ
закономерность как при взаимодействии с ДНК
PARP1, PARP2 или PARP3 в буфере следующего
в свободной форме, так и при комплексообразо
состава: 10 мМ Tris HCl (pH 7,5), 0,1% NP 40,
вании с NCP: Native = AP < Gap. Повышенное
0,2 мМ EDTA, 5 мМ β ME, 10 мM NaCl,
сродство PARP1 к Gap субстратам проявляется
5 мM MgCl2. Реакционную смесь инкубировали
как при использовании свободной ДНК, так и
при 37 °С в течение 15 мин. Для реакции
при использовании нуклеосомы. При формиро
PARирования реакционную смесь дополняли
вании комплексов PARP2 с NCP этот эффект
400 мкМ NAD+. Далее пробы с ДНК обрабаты
становится особенно выраженным. Следует от
вали
0,002
Ед DNase I, пробы с NCP
-
метить, что количественные данные, получен
0,007 Ед DNase I при 37 °С в течение 15 мин.
ные методом измерения анизотропии флуорес
Концентрации всех белковых компонентов бы
ценции для комплексов PARP1 и PARP2 с раз
ли подобраны экспериментально для получения
личными типами NCP, в общем случае коррели
оптимальной картины при анализе продуктов
руют с данными метода EMSA [38].
расщепления белок нуклеиновых комплексов.
Для PARP3 в целом наблюдается более низ
Для стабилизации AP сайтов реакционную
кое сродство к использованным субстратам по
БИОХИМИЯ том 87 вып. 3 2022
326
УКРАИНЦЕВ и др.
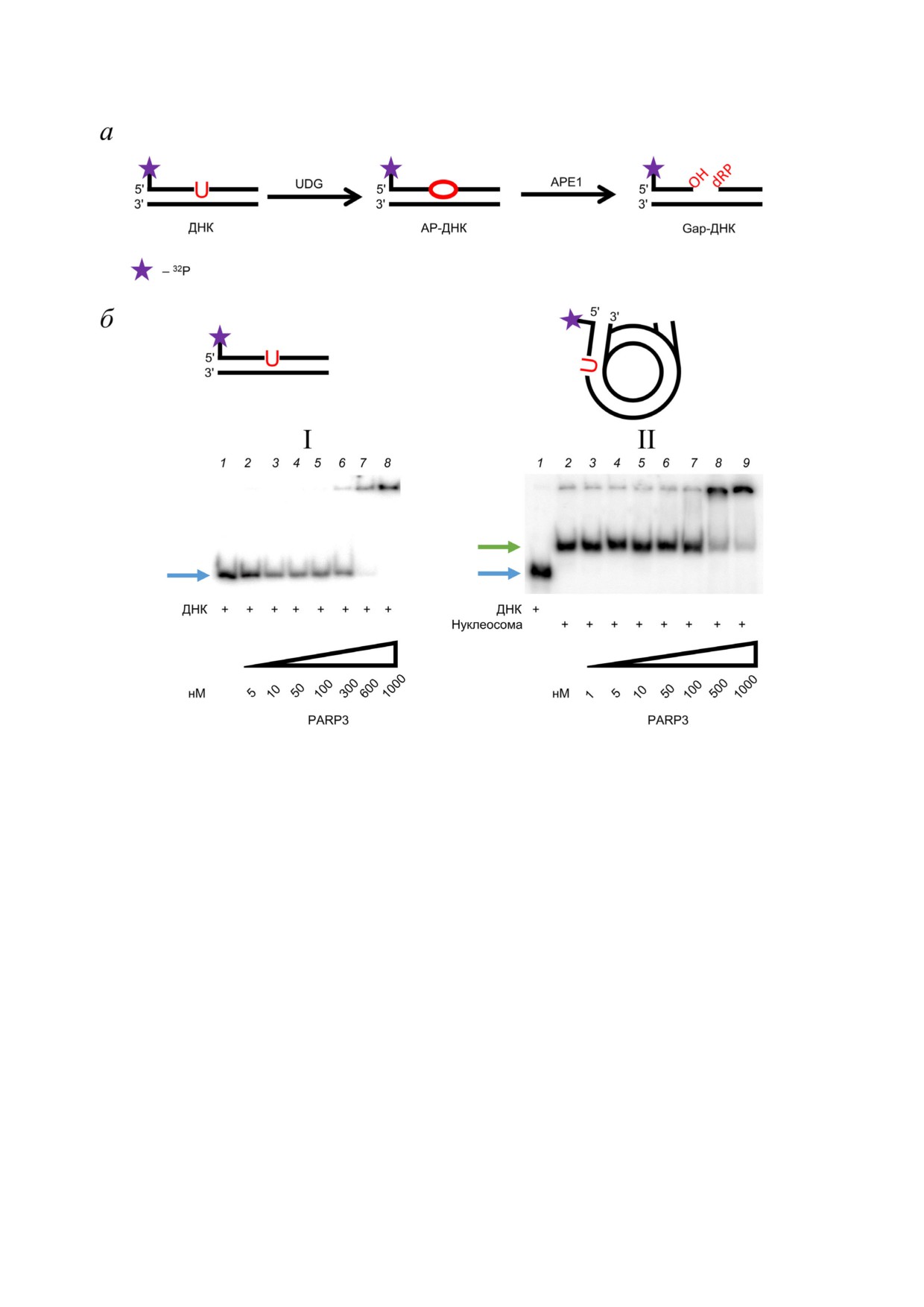
Рис. 2. Схема получения субстратов начальных стадий процесса BER (а). б - Радиоавтограф 4% ного ПААГ после разде
ления продуктов реакции комплексообразования PARP3 с нативной ДНК в свободном (I) и нуклеосомном (II) состоя
ниях в нативных условиях. I: дорожка 1 - свободная нативная ДНК, дорожки 2-8 - ДНК с PARP3; II: дорожка 1 - сво
бодная нативная ДНК, дорожка 2 - NCP, дорожки 3-9 - NCP с PARP3. Стрелками обозначена подвижность: синей -
свободной ДНК, зеленой - NCP. Концентрация ДНК/NCP составляла 100 нМ; концентрация PARP3 - 1-1000 нМ
сравнению с PARP1 и PARP2, а закономерность
С другой стороны, следствием формирования
в изменении прочности комплекса трансфор
основания Шиффа между белком и цепью ДНК
мируется к Gap <<< Native = AP независимо от
может быть расщепление последней по АР сай
формы ДНК.
ту с образованием одноцепочечного разры
Изучение взаимодействия ДНК зависимых
ва [43]. Соответственно, если АР сайты образу
белков семейства PARP с нуклеосомами методом
ются одновременно в двух цепях ДНК дуплек
боргидридной сшивки. В работах Zhou et al. [42],
са, то возможно появление двуцепочечного раз
Sczepanski et al. [43] и Yang et al. [44] показано,
рыва наиболее токсичной формы поврежде
что в составе нуклеосомной частицы октамеры
ний [44]. Изучение взаимодействия гистонов и
гистонов способны образовывать основания
белков семейства PARP с ДНК, содержащей
Шиффа с AP сайтами ДНК. С одной стороны, в
АР сайты в составе нуклеосомы, может помочь
зависимости от расположения АР сайта взаи
в понимании механизма репарации множест
модействие гистонов с таким типом поврежде
венных повреждений в контексте хроматина,
ния может влиять и на взаимодействие ДНК
особенно учитывая тот факт, что PARP1 и
связывающих белков, например PARP1, PARP2
PARP2 обладают АР лиазной активностью
и PARP3 с повреждённой нуклеосомной ДНК.
[16, 45].
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
327
Остатки дезоксирибозы AP сайтов в моле
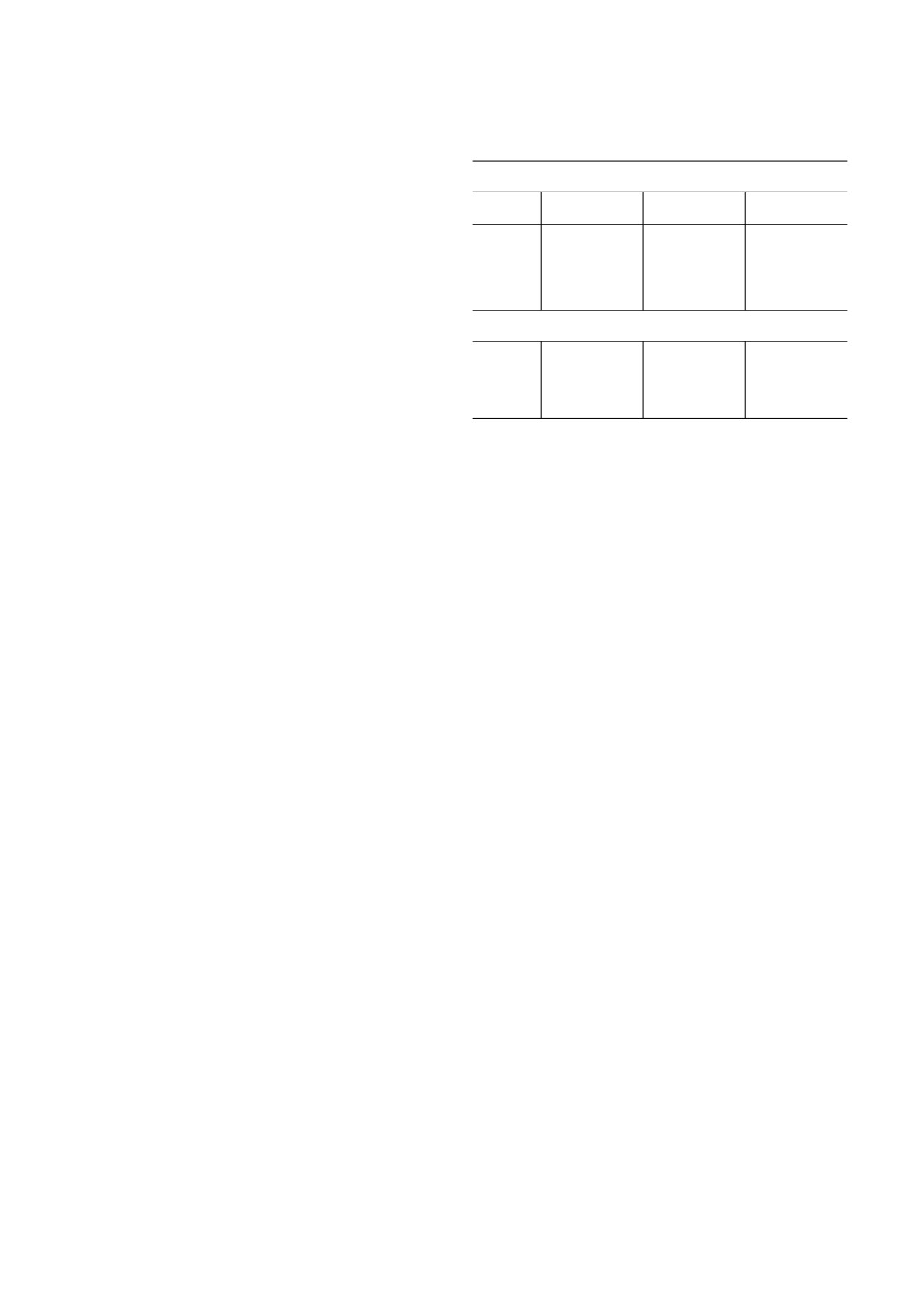
Константы диссоциации комплексов белков PARP1,
PARP2 и PARP3 со свободной ДНК и нуклеосомой*
куле ДНК представлены в нескольких равно
весных формах [46]. Одной из них является
ДНК
ациклическая альдегидная форма AP сайта, ко
торая может образовывать основания Шиффа с
Структура
Kd, нM (PARP3)
Kd, нM (PARP2)
Kd, нM (PARP1)
аминогруппами ДНК связывающих белков
[47], в частности с различными участника
Native
113 ± 5
300 ± 100
461 ± 9
ми BER [2, 16, 45, 48, 49]. Основание Шиффа
AP
92 ± 3
330 ± 80
426 ± 8
формируется как интермедиат многих фермен
Gap
48 ± 3
240 ± 80
750 ± 20
тативных реакций, и его распад по механизму
β элиминирования приводит к разрыву сахаро
Нуклеосома
фосфатного остова ДНК [42]. Тем не менее
in vitro этот интермедиат можно стабилизиро
Native
65 ± 3**
192 ± 10**
287 ± 5
вать восстановлением боргидридом натрия [50]
AP
61 ± 5**
130 ± 4**
250 ± 30
(рис. 3, а). Таким образом, при дальнейшем
анализе продуктов реакции с помощью гель
Gap
38 ± 4**
57 ± 8**
4400 ± 300
электрофореза по Лэммли появление белок
* Данные представлены как среднее минимум из трёх не
нуклеиновых аддуктов будет свидетельствовать
зависимых экспериментов.
о формировании комплекса между исследуе
** Данные опубликованы в работе Kutuzov et al. [38].
мым белком и локализованным АР сайтом
ДНК. Следует отметить, что при анализе про
дуктов реакции могут наблюдаться аддукты, со
одиночным АР сайтом, так и с AP кластером
ответствующие пришивке белка к целой цепи
свободной или нуклеосомной ДНК. Формиро
ДНК, а также к участку цепи ДНК, расщеплён
вание оснований Шиффа между PARP1, PARP2
ному по положению АР сайта. Кроме того, ис
и AP сайтом в составе свободной ДНК было по
пользование денатурирующих условий при
казано ранее [15, 16, 45]. Формирование основа
проведении эксперимента не всегда гарантиру
ний Шиффа между PARP3 и AP сайтом в соста
ет полную денатурацию двух цепей ДНК дуп
ве свободной ДНК, а также PARP1, PARP2,
лекса, особенно в составе белок нуклеинового
PARP3 и AP сайтом в составе нуклеосомы пока
аддукта (рис. 3, б). Все эти факты будут влиять
зано впервые.
на электрофоретическую подвижность аддук
При добавлении PARP1 в реакционную
тов. Метод боргидридной сшивки можно при
смесь наблюдается образование ковалентных
менять как универсальный подход при исследо
аддуктов с AP сайтом, расположенным вблизи
вании белок нуклеиновых взаимодействий с
тупого конца свободной или нуклеосомной
использованием AP сайт содержащих ДНК
ДНК, с молекулярной массой выше 95 кДа
различной структуры [51].
(рис. 4, а и 5, а, дорожки 4-15). Кроме того, наб
Для установления характера взаимодействий
людается формирование аддуктов с большей
белков PARP1, PARP2 и PARP3 с АР содержа
молекулярной массой, которые можно отнести
щими нуклеосомными частицами методом бор
к продуктам ковалентного присоединения
гидридной сшивки были использованы 5′ FAM
PARP1 к нерасщеплённой по АР сайту це
меченые ДНК структуры, содержащие как оди
пи ДНК, а также к белок нуклеиновым аддук
ночный АР сайт, так и кластер АР сайтов, в ко
там, содержащим неденатурированную двуце
тором повреждения были распределены статис
почечную форму ДНК. Однако при взаимодей
тически по всей длине дуплекса. Статистичес
ствии PARP1 с AP кластером как в составе сво
кое распределение АР сайтов по ДНК субстра
бодной ДНК, так и в нуклеосомном контексте
ту позволит выявить потенциальные участки
также происходило образование высокомолеку
взаимодействия белков семейства PARP по всей
лярных аддуктов, вероятно, являющихся след
поверхности нуклеосомной частицы независи
ствием посадки нескольких молекул PARP1 на
мо от положения АР сайта, как одиночного
AP кластер. В целом, наличие AP кластера су
повреждения. Для контрольных экспериментов
щественно не повлияло на характер взаимодей
были использованы свободные АР ДНК анало
ствия PARP1 с нуклеосомой, что может быть
гичной структуры.
следствием преимущественного взаимодей
На рис. 4 и 5 представлены автографы гелей
ствия PARP1 с тупыми концами ДНК дуплекса
после разделения продуктов реакции боргид
или АР сайтом, расположенным в непосред
ридной сшивки с помощью гель электрофореза
ственной близости от тупого конца.
по Лэммли. Оказалось, что все три белка семей
При формировании оснований Шиффа
ства PARP способны взаимодействовать как с
между PARP2 и AP сайтом происходит перера
БИОХИМИЯ том 87 вып. 3 2022
328
УКРАИНЦЕВ и др.
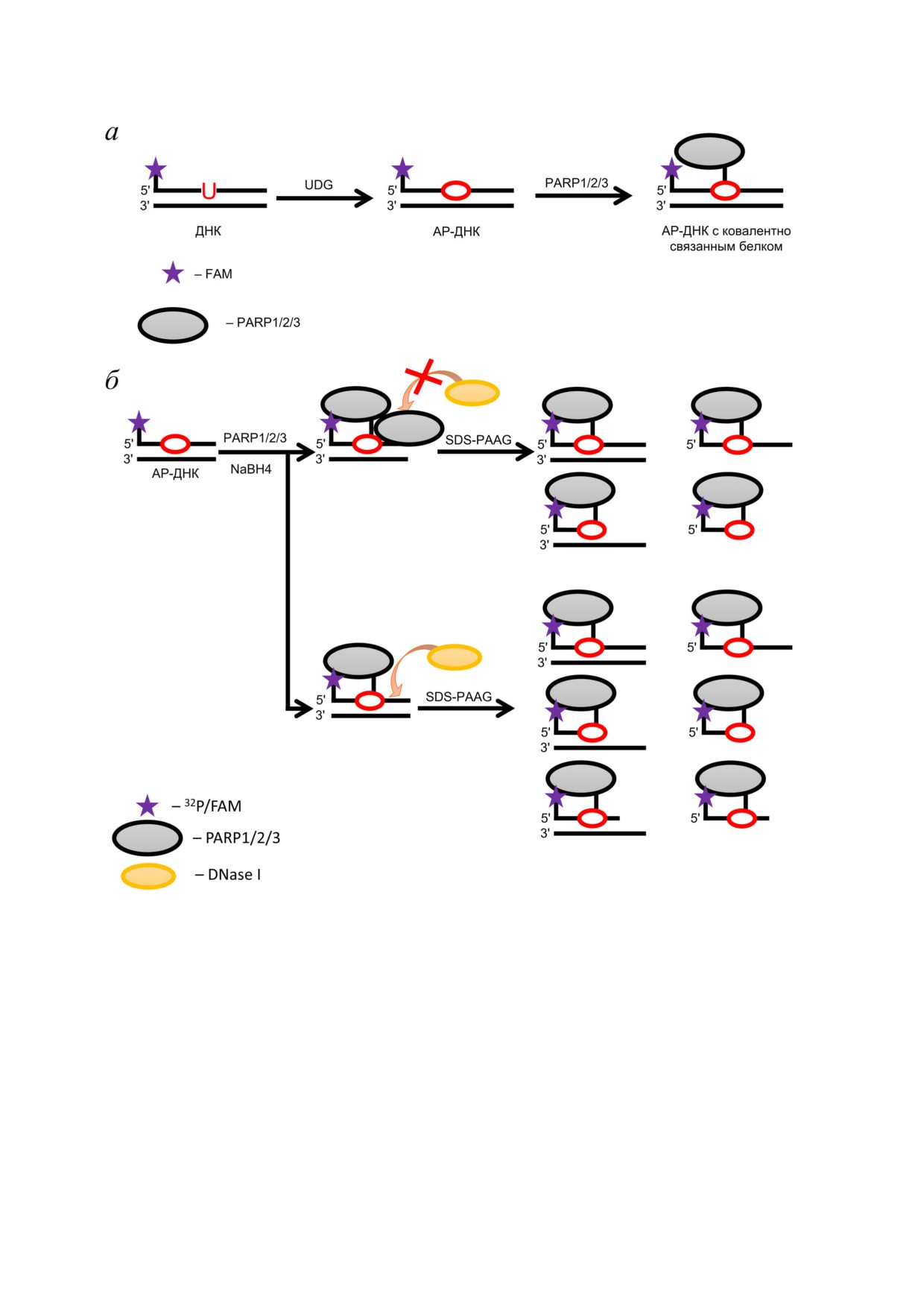
Рис. 3. Получение белок нуклеиновых аддуктов методом боргидридной сшивки. а - Схема получения ковалентного ад
дукта ДНК-белок; б - типы возможных белок нуклеиновых аддуктов при разделении продуктов реакции гель элек
трофорезом по Лэммли
спределение доли аддуктов с низкой и высокой
модействия молекулы PARP2 с разными участ
молекулярными массами: при увеличении ко
ками NCP.
личества AP сайтов доля высокомолекулярных
Сродство PARP3 к АР содержащим субстра
продуктов увеличивается с 45 до 59% (дорож
там существенно ниже, чем PARP1 и PARP2, что
ка 15 на рис. 4, бI и 5, бI) при использовании
выражается в том числе в количестве непрореа
свободной ДНК и с 53 до 64% (дорожка 15 на
гировавшей с PARP3 АР содержащей ДНК (от
рис. 4, бII и 5, бII) при использовании ДНК в
мечено синей стрелкой на рис. 4, в, ср. дорож
составе NCP (рис. 4, б и 5, б, дорожки 4-15). По
ки 1 и 10-15 в обеих частях). Интересно, что ин
добные изменения могут быть следствием взаи
тенсивность сшивки между PARP3 и NCP
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
329
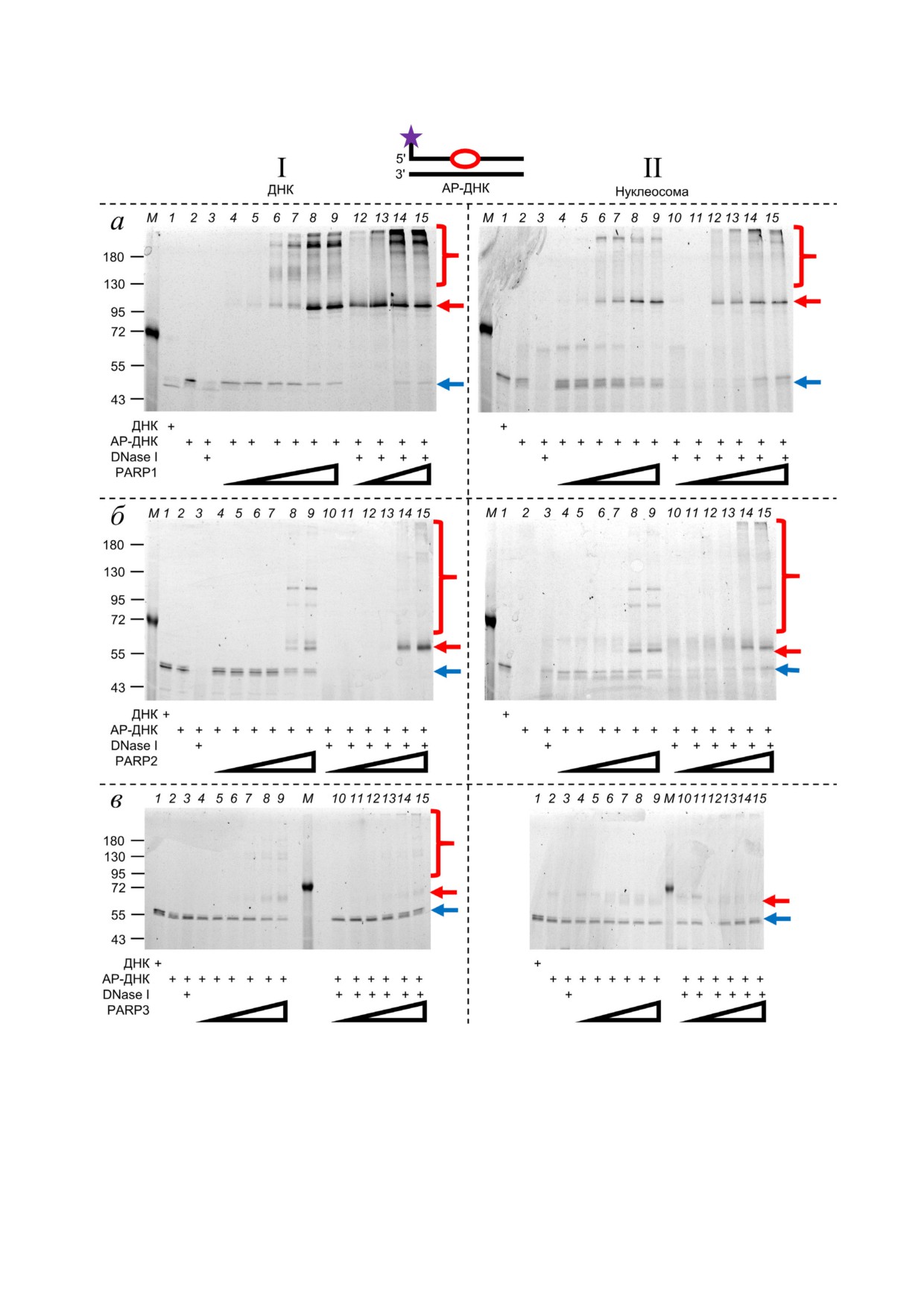
Рис. 4. Взаимодейсвтие белков PARP1 (а), PARP2 (б) и PARP3 (в) с AP сайтом в составе свободной (I) и нуклеосом
ной (II) ДНК (см. «Материалы и методы»). Автографы гелей после разделения продуктов реакции в 10% ном ПААГ по
Лэммли. Слева указаны положения маркёров молекулярных масс (кДа). М - маркёры молекулярных масс; дорожка 1 -
нативная ДНК/NCP; дорожка 2 - ДНК/NCP, обработанная UDG; дорожка 3 - ДНК/NCP, обработанная UDG и DNase I.
Концентрация PARP в дорожках 4-9 и 10-15 составляла 5, 10, 50, 100, 500, 1000 нМ соответственно. Синими стрелками
обозначена электрофоретическая подвижность нерасщеплённой цепи АР ДНК, красными стрелками и скобками -
белок нуклеиновые аддукты
БИОХИМИЯ том 87 вып. 3 2022
330
УКРАИНЦЕВ и др.
субстратом возрастает при переходе от одиноч
размерной ДНК в присутствии PARP1 свиде
ного к кластерному AP сайту - доля комплекса
тельствует о преимущественной локализации
с молекулярной массой выше 55 кДа по отноше
белка вблизи 5′ концевого участка дуплек
нию к непрореагировавшей ДНК увеличивается
са ДНК в составе NCP (дорожки 10, 11).
с 22% до 38% (ср. дорожки 15 на рис. 4, вII
Формирование комплекса между PARP1 и
и 5, вII). Однако образования аддуктов, соответ
АР содержащей свободной ДНК практически
ствующих одновременному взаимодействию
полностью подавляет реакцию ферментативно
нескольких молекул PARP3 с несколькими AP
го расщепления (рис. 6, ср. дорожку 5 с 6 и 7),
сайтами в составе NCP, не наблюдается
что говорит о равномерном распределении мо
(рис. 5, вII), что может быть обусловлено взаи
лекул белка по всей длине ДНК субстрата. Од
модействием PARP3 с АР сайтом, расположен
нако появление продуктов β элиминирования
ным вблизи тупого конца ДНК дуплекса или
говорит о специфическом взаимодействии
наличием структурных изменений АР NCP в
PARP1 с АР сайтом (рис. 6, ср. дорожку 5 с 6 и 7;
присутствии большого количества AP сайтов.
продукт β элиминирования отмечен синей звёз
Следует отметить, что сшивок, соответству
дочкой) [16].
ющих белок нуклеиновым аддуктам октамера
При взаимодействии PARP1 и АР содержа
гистонов при использовании описанных АР со
щей NCP на электрофореграмме пропадает один
держащих субстратов, не наблюдалось. Этот
из сигналов, соответствующий полноразмерной
факт позволяет исключить вклад конкретного
5′ [32P] ДНК в составе нативной NCP (рис. 6,
типа повреждения в формирование одноцепо
ср. дорожки 10, 11 с 13, 14). Этот факт говорит о
чечных разрывов в нуклеосомной ДНК за счёт
формировании разного типа комплексов
октамера гистонов.
PARP1-NCP с 5′ концевым участком ДНК дуп
Изучение локализации PARP1, PARP2 и
лекса при появлении АР сайта. Кроме того, по
PARP3 на нуклеосоме методом ферментативного
является продукт реакции β элиминирования,
футпринтинга. Выявить белок белковые контак
т.е. специфического взаимодействия PARP1 c
ты членов семейства PARP, а также их специфи
АР сайтом в составе нуклеосомной ДНК.
ческих взаимодействий с повреждением ДНК в
Равномерный характер распределения по
составе NCP позволяет метод футпринтинга с
лос, а также наличие интенсивного сигнала, со
использованием фермента DNase I [52]. В пред
ответствующего полноразмерной ДНК, при раз
ставленной работе для изучения локализации
делении продуктов расщепления комплексов
PARP1, PARP2 и PARP3 на свободной ДНК и
свободной ДНК с PARP2 и PARP3 указывает на
нуклеосоме использовали ДНК субстраты как в
преимущественное связывание белков с 5′ кон
нативной форме, так и с одиночным АР сайтом.
цами ДНК субстрата (рис. 6, дорожка 15 -
Как видно из представленных данных, при
для PARP2, дорожка 23 - для PARP3). Присут
сутствие PARP1 практически полностью подав
ствие NAD+ в реакционной смеси существенно
ляет реакцию ферментативного расщепления
не влияет на изменение распределения полос
нативной формы свободной ДНК (рис. 6, до
после реакции расщепления (рис. 6, ср. дорож
рожка 3). Такая ситуация возможна только в том
ки 15 и 16 - для PARP2, дорожки 25 и 26 -
случае, когда молекулы белка в составе комп
для PARP3). В случае PARP2 этот факт можно
лекса равномерно распределены по всей длине
объяснить пониженным сродством фермента и
ДНК субстрата, включая 5′ концевой участок
его более низкой каталитической активностью
дуплекса. Добавление NAD+ в реакционную
по сравнению с PARP1, что может выражаться в
смесь запускает реакцию PARилирования
отсутствии драматических изменений в концен
(рис. 6, дорожка 4). Из представленных на
трациях белок нуклеинового комплекса в усло
электрофореграмме данных видно, что характер
виях проведения эксперимента. PARP3 имеет
распределения полос ферментативного расщеп
ещё более низкое сродство к ДНК и катализиру
ления после PARилирования совпадает с харак
ет перенос лишь одного остатка ADP рибозы,
тером распределения полос свободной ДНК, но
что может не приводить к существенному изме
не комплекса PARP1-ДНК. Следовательно,
нению концентрации белок нуклеинового
после PARилирования одноимённый заряд
комплекса за счёт электростатического отталки
ДНК и поли(ADP рибозы) способствует диссо
вания компонентов при PARилировании.
циации комплекса поли(ADP рибозил)ирован
Наличие АР сайта в составе свободной ДНК
ный PARP-ДНК.
приводит к изменению типа взаимодействия
Из представленных данных видно, что дос
белков PARP2 и PARP3 с 5′ концевой частью
туп к ДНК в составе нуклеосомы ограничен по
ДНК субстрата (рис. 6, ср. дорожки 15 с 17 - для
сравнению со свободной ДНК (рис. 6, ср. до
PARP2, дорожки 23 с 25 - для PARP3). Наличие
рожки 2 и 9, 5 и 12). Сохранение сигнала полно
продуктов β,δ элиминирования говорит о спон
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
331
Рис. 5. Взаимодействие белков PARP1 (а), PARP2 (б) и PARP3 (в) с AP кластером в составе свободной (I) и нуклеосом
ной (II) ДНК (см. «Материалы и методы»). Автографы гелей после разделения продуктов реакции в 10% ном ПААГ по
Лэммли. Слева указаны положения маркёров молекулярных масс (кДа). М - маркёры молекулярных масс; дорожка 1 -
нативная ДНК/NCP; дорожка 2 - ДНК/NCP, обработанная UDG; дорожка 3 - ДНК/NCP, обработанная UDG и DNase I.
Концентрация PARP1 и PARP2 в дорожках 4-9 и 10-15 составляла 5, 10, 50, 100, 500, 1000 нМ соответственно. Концент
рация PARP3 в дорожках 5-9 вI и 11-15 вI составляла 10, 50, 100, 500, 1000 нМ соответственно; концентрация PARP3 в
дорожках 5-9 вII и 12-15 вII составляла 10, 50, 100, 500, 1000 нМ и 50, 100, 500, 1000 нМ соответственно. Синими стрел
ками обозначена электрофоретическая подвижность нерасщеплённой цепи АР ДНК, красными стрелками и скобка
ми - белок нуклеиновые аддукты
БИОХИМИЯ том 87 вып. 3 2022
332
УКРАИНЦЕВ и др.
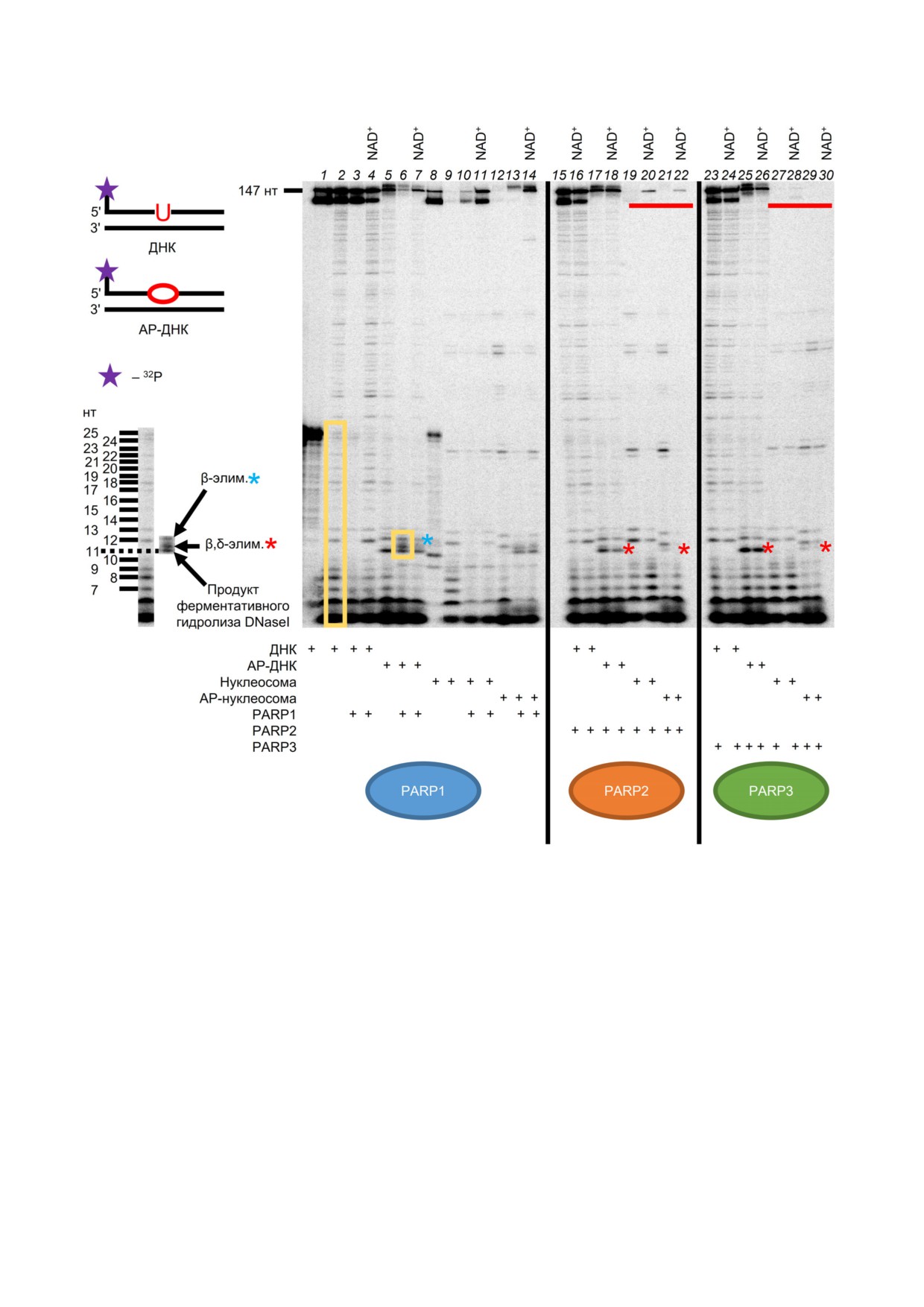
Рис. 6. Исследование структуры белок нуклеиновых комплексов PARP1, PARP2 и PARP3 со свободной ДНК и с нуклео
сомой. Дорожка 1 - смесь 5′ [32P] меченых компонентов системы - 147 пнт ампликон и 25 нт праймер; дорожка 8 - смесь
5′ [32P] меченых компонентов системы - NCP и 25 нт праймер с урацилом в положении 12; дорожки 2, 9 - контрольная
ДНК/NCP после обработки DNase I; дорожки 5, 12 - контрольная AP ДНК/AP NCP после обработки DNase I; дорож
ки 3, 4, 6, 7, 10, 11, 13, 14 - продукты расщепления комплексов PARP1 с ДНК, АР ДНК, NCP и АР NCP соответственно;
дорожки 15-22 - продукты расщепления комплексов PARP2 с ДНК, АР ДНК, NCP и АР NCP; дорожки 23-30 - про
дукты расщепления комплексов PARP3 с ДНК, АР ДНК, NCP и АР NCP. Синей звёздочкой отмечены продукты β эли
минирования, красной звёздочкой - продукты β,δ элиминирования, красной линией отмечено положение полноразмер
ной 5′ [32P] ДНК. Зоны в жёлтой рамке вынесены за пределы электрофореграммы
танном разрушении АР сайта в процессе гель
чем PARP1, что выражается в появлении про
электрофореза, т.е. об отсутствии АР лиазной
дукта реакции β элиминирования (рис.
6,
активности PARP3 (рис. 6, ср. дорожки
5
ср. дорожки 5 с 17, 18; продукт β,δ элиминиро
с 25, 26; продукт β,δ элиминирования отмечен
вания отмечен красной звёздочкой) [45].
красной звёздочкой). PARP2 способен прояв
Характер продуктов расщепления белок
лять АР лиазную активность в отношении АР
нуклеиновых комплексов указывает на отсут
сайта, хотя и в существенно меньшей степени,
ствие взаимодействий белков PARP2 и PARP3 с
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
333
5′ концевой частью ДНК субстрата в составе
следствие, влиять на белок белковые взаимо
нуклеосомы независимо от наличия АР сайта
действия в процессе репарации.
(рис. 6, ср. дорожки 9, 12 с 19-22 (для PARP2)
Ранее с использованием модельных ДНК бы
и с 27-30 (для PARP3); отмечено красной ли
ло установлено, что сродство PARP2 к тупым
нией). Тем не менее продукт спонтанного рас
концам ДНК дуплекса ниже, чем к одноцепо
пада АР сайта также отсутствует, но наблюдает
чечному разрыву [21]. В работе Gaullier et al. [54]
ся продукт β элиминирования, что говорит о
с использованием криоэлектронной микроско
специфическом взаимодействии PARP2 и
пии было показано, что PARP2 взаимодействует
PARP3 с АР сайтом и/или гистонами вблизи
с NCP через связывание с тупыми концами
повреждения ДНК в составе NCP (рис. 6,
ДНК дуплекса. Стоит отметить, что такое взаи
ср. дорожки 19 с 23 - для PARP2 и 27 с 31 -
модействие было продемонстрировано с исполь
для PARP3).
зованием реконструированной NCP с двумя
линкерными участками длиной 9 пнт. На основе
значений Kd, полученных в настоящей работе,
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
можно предположить, что наличие одноцепочеч
ного разрыва ДНК обеспечивает эффективность
В представленной работе был изучен харак
взаимодействия PARP2 с нуклеосомой. Методом
тер взаимодействия белков PARP1, PARP2 и
футпринтинга было показано, что PARP2 не взаи
PARP3 c нуклеосомными частицами, содержа
модействует с тупыми концами нуклеосомной
щими в своём составе ДНК интермедиаты на
ДНК, но способен взаимодействовать с AP сай
чальных стадий процесса BER. Использован
том вблизи тупого конца. Таким образом, на ос
ный ДНК субстрат содержал повреждение на
новании полученных нами данных, а также лите
расстоянии чуть более одного витка спирали от
ратурных данных с использованием свобод
одного из тупых концов ДНК дуплекса. Опира
ной ДНК и ДНК в составе NCP [21, 22, 38] мож
ясь на полученные результаты, можно сделать
но говорить об эффективном взаимодействии
ряд последовательных выводов, которые важны
PARP2 как с АР сайтом, так и с разрывом в ДНК
для понимания механизма действия ДНК зави
в контексте нуклеосомы, содержащей поврежде
симых белков семейства PARP и регуляции на
ние вблизи конца ДНК дуплекса.
чальных стадий процесса BER.
С использованием модельных структур ДНК
Известно, что PARP1 с высокой эффектив
и NCP было установлено, что PARP3 взаимо
ностью связывается с тупыми концами модель
действует и активируется ДНК с одноцепочеч
ных ДНК дуплексов, проявляет АР лиазную ак
ным разрывом, содержащим 5′ концевую фос
тивность при взаимодействии с АР сайтами, а
фатную группу, - классическим интермедиатом
также эффективно взаимодействует с разрывом
поздних этапов BER [11, 24]. Тем не менее ока
ДНК после действия APE1 [6, 21, 22]. Отсут
залось, что PARP3 взаимодействует с использо
ствие существенных различий в значениях
ванными в работе Gap структурами менее эф
констант диссоциации PARP1 с использован
фективно, чем с нативной и AP содержащей
ными в представленной работе структура
ДНК. Вероятно, такое различие в данных мож
ми ДНК и NCP может быть связано с предпоч
но объяснить тем, что используемый в представ
тительным взаимодействием PARP1 с тупыми
ленной работе классический интермедиат более
концами ДНК дуплекса, являющимися анало
ранних стадий BER - ДНК дуплекс с одноцепо
гом двуцепочечного разрыва [35, 53]. Стоит об
чечной брешью с 5′ dRP фрагментом - не явля
ратить внимание, что сродство PARP1 к свобод
ется каноническим субстратом для PARP3. Кро
ной ДНК оказалось несколько ниже, чем к нук
ме того, наличие одноцепочечного разрыва мо
леосоме. Это может свидетельствовать о влия
жет приводить к структурным изменениям NCP,
нии дополнительных белок белковых взаимо
что, вероятно, является критическим фактором
действий PARP1 с гистонами при формирова
при взаимодействии PARP3 с нуклеосомой.
нии комплекса PARP1-NCP. Тем не менее, по
Следовательно, аномально высокое значение
данным метода боргидридной сшивки, характер
константы диссоциации комплекса PARP3-
взаимодействия PARP1 с ДНК и ДНК в составе
Gap NCP, вероятно, можно объяснить сущест
NCP существенно не изменяется (см. рис. 3, а
вованием пространственной структуры нуклео
и 4, а) либо эти изменения не выявляются с по
сомной частицы, не позволяющей PARP3 эф
мощью использованного подхода. Можно пред
фективно взаимодействовать с повреждением в
положить, что в контексте таких структур близ
цепи ДНК. Таким образом, аффинность PARP3
кое расположение тупого конца дуплекса ДНК
к ДНК с разрывом в составе NCP может опреде
будет маскировать взаимодействие PARP1 с АР
ляться как размером бреши, так и модификаци
сайтом или одноцепочечным разрывом и, как
ей 5′ концевого фрагмента.
БИОХИМИЯ том 87 вып. 3 2022
334
УКРАИНЦЕВ и др.
Методом футпринтинга было показано, что
характере взаимодействия ДНК зависимых бел
PARP3 взаимодействует с AP сайтом в соста
ков семейства PARP c повреждёнными ДНК в
ве NCP. Тем не менее при использовании NCP с
нуклеосомном контексте: PARP1 взаимодей
AP кластером не было зафиксировано высокомо
ствует преимущественно с тупыми концами
лекулярных белок нуклеиновых аддуктов, кото
ДНК дуплекса; PARP2 взаимодействует с пов
рые обнаруживались при взаимодействии PARP3
реждениями в структуре NCP и/или октамерами
со свободной ДНК аналогичной структуры. Воз
гистонов; PARP3 способен распознавать AP
можно, PARP3 предпочтительно взаимодействует
сайты ДНК в нуклеосомном контексте и/или
только с AP сайтом вблизи тупых концов ДНК
октамеры гистонов.
дуплекса. Альтернативно такой результат может
быть связан с наличием структурных изменений
в NCP, содержащей ДНК с кластером AP сайтов.
Финансирование. Работа выполнена при фи
Таким образом, совокупность полученных дан
нансовой поддержке Российского фонда фунда
ных позволяет предположить, что PARP3 может
ментальных исследований (грант № 20 04
взаимодействовать с АР сайтами и/или октаме
00674) и Проекта государственного задания
ром гистонов NCP, но вряд ли способен направ
ИХБФМ СО РАН № 121031300041 4.
ленно конкурировать с белками PARP1 и PARP2
Конфликт интересов. Авторы заявляют об от
за взаимодействие с интермедиатами начальных
сутствии конфликта интересов.
стадий BER после действия АРE1.
Соблюдение этических норм. Настоящая
статья не содержит описания каких либо иссле
Таким образом, совокупность всех результа
дований с участием людей или животных в каче
тов, представленных в работе, говорит о разном стве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Lindahl, T. (1993) Instability and decay of the primary
trometry, Proteome Sci., 8, 22 32, doi: 10.1186/1477 5956
structure of DNA, Nature, 362, 709 715, doi: 10.1038/
8 22.
362709a0.
11. Grundy, G. J., Polo, L. M., Zeng, Z., Rulten, S. L., Hoch,
2. Wilson, D. M., and Barsky, D. (2001) The major human
N. C., et al. (2016) PARP3 is a sensor of nicked nucleo
abasic endonuclease: formation, consequences and repair
somes and monoribosylates histone H2B(Glu2), Nat.
of abasic lesions in DNA, Mutat. Res., 485, 283 307,
Commun., 7, 12404 12415, doi: 10.1038/ncomms12404.
doi: 10.1016/s0921 8777(01)00063 5.
12. Van Beek, L., McClay, E., Patel, S., Schimpl, M.,
3. Schärer, O. D. (2003) Chemistry and biology of DNA
Spagnolo, L., et al. (2021) A PARP power: Structural per
repair, Angew. Chem. Int. Ed. Engl., 42, 29462974,
spective on PARP1, PARP2, and PARP3 in DNA damage
doi: 10.1002/anie.200200523.
repair and nucleosome remodelling, Int. J. Mol. Sci., 22,
4. Kim, Y. J., and Wilson, D. M. (2012) Overview of base
5112, doi: 10.3390/ijms22105112.
excision repair biochemistry, Curr. Mol. Pharmacol., 5, 3
13. Huang, X., LeDuc, R. D., Fornelli, L., Schunter, A. J.,
13, doi: 10.2174/1874467211205010003.
Bennett, R. L., et al. (2019) Defining the NSD2 interac
5. Shall, S. S., and de Murcia, G. (2000) Poly(ADP ribose)
tome: PARP1 PARylation reduces NSD2 histone methyl
polymerase 1: what have we learned from the deficient
transferase activity and impedes chromatin binding, J. Biol.
mouse model? Mutat. Res., 460, 1 15, doi: 10.1016/s0921
Chem.,
294,
1245912471, doi:
10.1074/jbc.RA118.
8777(00)00016 1.
006159.
6. Khodyreva, S. N., and Lavrik, O. I. (2016) Poly(ADP
14. Boehler, C., Gauthier, L. R., Mortusewicz, O., Biard,
Ribose) polymerase 1 as a key regulator of DNA repair, Mol.
D. S., Saliou, J. M., et al. (2011) Poly(ADP ribose) poly
Biol.
(Mosk),
50,
580595,
doi:
10.7868/
merase 3 (PARP3), a newcomer in cellular response to
S0026898416040030.
DNA damage and mitotic progression, Proc. Natl. Acad.
7. Amé, J. C., Rolli, V., Schreiber, V., Niedergang, C.,
Sci. USA, 108, 2783 2788, doi: 10.1073/pnas.1016574108.
Apiou, F., et al. (1999) PARP 2, a novel mammalian DNA
15. Cistulli, C., Lavrik, O. I., Prasad, R., Hou, E., and Wilson,
damage dependent poly(ADP ribose) polymerase, J. Biol.
S. H. (2004) AP endonuclease and poly(ADP ribose) poly
Chem., 274, 17860 17868, doi: 10.1074/jbc.274.25.17860.
merase 1 interact with the same base excision repair inter
8. Rouleau, M., McDonald, D., Gagné, P., Ouellet, M. E.,
mediate,
DNA Repair (Amst),
3,
581591,
Droit, A., et al. (2007) PARP 3 associates with polycomb
doi: 10.1016/j.dnarep.2003.09.012.
group bodies and with components of the DNA damage
16. Khodyreva, S. N., Prasad, R., Ilina, E. S., Sukhanova,
repair machinery, J. Cell. Biochem.,
100,
385401,
M. V., Kutuzov, M. M., et al. (2010) Apurinic/apyrimidinic
doi: 10.1002/jcb.21051.
(AP) site recognition by the 5′ dRP/AP lyase in poly(ADP
9. Beck, C., Robert, I., Reina San Martin, B., Schreiber, V.,
ribose) polymerase 1 (PARP 1), Proc. Natl. Acad. Sci. USA,
and Dantzer, F. (2014) Poly(ADP ribose) polymerases in
107, 22090 22095, doi: 10.1073/pnas.1009182107.
double strand break repair: focus on PARP1, PARP2 and
17. Sukhanova, M., Khodyreva, S., and Lavrik, O. (2010)
PARP3, Exp. Cell Res., 329, 18 25, doi: 10.1016/j.yexcr.
Poly(ADP ribose) polymerase 1 regulates activity of DNA
2014.07.003.
polymerase β in long patch base excision repair, Mutat. Res.,
10. Isabelle, M., Moreel, X., Gagné, J. P., Rouleau, M.,
685, 80 89, doi: 10.1016/j.mrfmmm.2009.08.009.
Ethier, C., et al. (2010) Investigation of PARP 1, PARP 2,
18. Lavrik, O. I., Prasad, R., Sobol, R. W., Horton, J. K.,
and PARG interactomes by affinity purification mass spec
Ackerman, E. J., et al. (2001) Photoaffinity labeling of
БИОХИМИЯ том 87 вып. 3 2022
ВЗАИМОДЕЙСТВИЕ PARP С ИНТЕРМЕДИАТАМИ ПРОЦЕССА BER
335
mouse fibroblast enzymes by a base excision repair interme
33.
Cole, H. A., Tabor Godwin, J. M., and Hayes, J. J. (2010)
diate. Evidence for the role of poly(ADP ribose) poly
Uracil DNA glycosylase activity on nucleosomal DNA
merase 1 in DNA repair, J. Biol. Chem., 276, 25541 25548,
depends on rotational orientation of targets, J. Biol. Chem.,
doi: 10.1074/jbc.M102125200.
285, 2876 2885, doi: 10.1074/jbc.M109.073544.
19.
Hanzlikova, H., Kalasova, I., Demin, A. A., Pennicott,
34.
Hinz, J. M. (2014) Impact of abasic site orientation within
L. E., Cihlarova, Z., et al. (2018) The importance of
nucleosomes on human APE1 endonuclease activity, Mutat.
poly(ADP ribose) polymerase as a sensor of unligated
Res., 766, 19 24, doi: 10.1016/j.mrfmmm.2014.05.008.
Okazaki fragments during DNA replication, Mol. Cell, 71,
35.
Clark, N. J., Kramer, M., Muthurajan, U. M., and
319 331, doi: 10.1016/j.molcel.2018.06.004.
Luger, K.
(2012) Alternative modes of binding of
20.
Hakmé, A., Wong, H. K., Dantzer, F., and Schreiber, V.
poly(ADP ribose) polymerase 1 to free DNA and nucleo
(2008) The expanding field of poly(ADP ribosyl)ation
somes, J. Biol. Chem., 287, 32430 32439, doi: 10.1074/jbc.
reactions. “Protein Modifications: Beyond the Usual
M112.397067.
Suspects” Review Series, EMBO Rep., 9, 10941100,
36.
D’Amours, D., Desnoyers, S., D’Silva, I., and Poirier,
doi: 10.1038/embor.2008.191.
G. G. (1999) Poly(ADP ribosyl)ation reactions in the regu
21.
Sukhanova, M. V., Abrakhi, S., Joshi, V., Pastre, D.,
lation of nuclear functions, Biochem. J., 342, 249 268.
Kutuzov, M. M., et al. (2016) Single molecule detection of
37.
Messner, S., Altmeyer, M., Zhao, H., Pozivil, A.,
PARP1 and PARP2 interaction with DNA strand breaks
Roschitzki, B., et al. (2010) PARP1 ADP ribosylates lysine
and their poly(ADP ribosyl)ation using high resolution
residues of the core histone tails, Nucleic Acids Res., 38,
AFM imaging, Nucleic Acids Res.,
44,
1 12,
6350 6362, doi: 10.1093/nar/gkq463.
doi: 10.1093/nar/gkv1476.
38.
Kutuzov, M. M., Belousova, E. A., Kurgina, T. A.,
22.
Sukhanova, M. V., Hamon, L., Kutuzov, M. M., Joshi, V.,
Ukraintsev, A. A., Vasil’eva, I. A., et al. (2021) The contri
Dobra, I., et al. (2019) A single molecule atomic force
bution of PARP1, PARP2 and poly(ADP ribosyl)ation to
microscopy study of PARP1 and PARP2 recognition of base
base excision repair in the nucleosomal context, Sci. Rep.,
excision repair DNA intermediates, J. Mol. Biol., 431,
11, 4849 4865, doi: 10.1038/s41598 021 84351 1.
2655 2673, doi: 10.1016/j.jmb.2019.05.028.
39.
Amé, J. C., Kalisch, T., Dantzer, F., and Schreiber, V.
23.
Kutuzov, M. M., Khodyreva, S. N., Amé, J. C., Ilina, E. S.,
(2011) Purification of recombinant poly(ADP ribose) poly
Sukhanova, M. V., et al. (2013) Interaction of PARP 2 with
merases, Methods Mol. Biol.,
780,
135152,
DNA structures mimicking DNA repair intermediates and
doi: 10.1007/978 1 61779 270 0_9.
consequences on activity of base excision repair proteins,
40.
Laemmli, U. K. (1970) Cleavage of structural proteins dur
Biochimie, 95, 12081215, doi: 10.1016/j.biochi.2013.
ing the assembly of the head of bacteriophage T4, Nature,
01.007.
227, 680 685, doi: 10.1038/227680a0.
24.
Langelier, M. F., Riccio, A. A., and Pascal, J. M. (2014)
41.
Dey, B., Thukral, S., Krishnan, S., Chakrobarty, M.,
PARP 2 and PARP 3 are selectively activated by 5′ phos
Gupta, S., et al. (2012) DNA-protein interactions:
phorylated DNA breaks through an allosteric regulatory
Methods for detection and analysis, Mol. Cell. Biochem.,
mechanism shared with PARP 1, Nucleic Acids Res., 42,
365, 279 299, doi: 10.1007/s11010 012 1269 z.
7762 7775, doi: 10.1093/nar/gku474.
42.
Zhou, C., Sczepanski, J. T., and Greenberg, M. M. (2012)
25.
Yélamos, J., Schreiber, V., and Dantzer, F. (2008) Toward
Mechanistic studies on histone catalyzed cleavage of
specific functions of poly(ADP ribose)polymerase 2,
apyrimidinic/apurinic sites in nucleosome core particles,
Trends Mol. Med., 14, 169 178, doi: 10.1016/j.molmed.
J. Am. Chem. Soc., 134, 1673416741, doi: 10.1021/
2008.02.003.
ja306858m.
26.
Kutuzov, M. M., Khodyreva, S. N., Schreiber, V., and Lavrik,
43.
Sczepanski, J. T., Zhou, C., and Greenberg, M. M. (2013)
O. I. (2014) The role of PARP2 in DNA repair, Mol. Biol.
Nucleosome core particle - catalyzed strand scission at
(Mosk), 48, 561 572, doi: 10.1134/S0026893314040062.
abasic sites, Biochemistry, 52, 2157 2164, doi: 10.1021/
27.
Talhaoui, I., Lebedeva, N. A., Zarkovic, G., Saint
bi3010076.
Pierre, C., Kutuzov, M. M., et al. (2016) Poly(ADP ribose)
44.
Yang, K., and Greenberg, M. M. (2018) Enhanced cleavage
polymerases covalently modify strand break termini in
at abasic sites within clustered lesions in nucleosome core
DNA fragments in vitro, Nucleic Acids Res., 44, 9279 9295,
particles, Chembiochem, 19, 2061 2065, doi: 10.1002/cbic.
doi: 10.1093/nar/gkw67523.
201800338.
28.
Belousova, E. A., Ishchenko, A. A., and Lavrik, O. I. (2018)
45.
Kutuzov, M. M., Khodyreva, S. N., Ilina, E. S., Sukhanova,
DNA is a new target of Parp3, Sci. Rep., 8, 4176 4187,
M. V., Amé, J. C., et al. (2015) Interaction of PARP 2 with
doi: 10.1038/s41598 018 22673 3.
AP site containing DNA, Biochimie,
112,
1019,
29.
Zarkovic, G., Belousova, E. A., Talhaoui, I., Saint
doi: 10.1016/j.biochi.2015.02.010.
Pierre, C., Kutuzov, M. M., et al. (2018) Characterization
46.
Wiederhold, L., Leppard, J. B., Kedar, P., Karimi
of DNA ADP ribosyltransferase activities of PARP2 and
Busheri, F., Rasouli Nia, A., et al. (2004) AP endonucle
PARP3: new insights into DNA ADP ribosylation, Nucleic
ase independent DNA base excision repair in human cells,
Acids Res., 46, 2417 2431, doi: 10.1093/nar/gkx1318.
Mol. Cell, 15, 209 220, doi: 10.1016/j.molcel.2004.06.003.
30.
McGinty, R. K., and Tan, S. (2015) Nucleosome structure
47.
Khodyreva, S. N., and Lavrik, O. I. (2020) Non canonical
and function, Chem. Rev., 115, 2255 2273, doi: 10.1021/
interaction of DNA repair proteins with intact and cleaved
cr500373h.
AP sites, DNA Repair (Amst),
90,
102847102861,
31.
Kutuzov, M. M., Kurgina, T. A., Belousova, E. A.,
doi: 10.1016/j.dnarep.2020.102847.
Khodyreva, S. N., and Lavrik, O. I. (2019) Optimization of
48.
Fromme, J. C., and Verdine, G. L. (2004) Base excision
nucleosome assembly from histones and model DNAs and
repair, Adv. Protein Chem., 69, 1 41, doi: 10.1016/S0065
estimation of the reconstitution efficiency, Biopolym. Cell,
3233(04)69001 2.
35, 91 98, doi: 10.7124/bc.00099A.
49.
Krokeide, S. Z., Bolstad, N., Laerdahl, J. K., Bjørås, M.,
32.
Beard, B. C., Wilson, S. H., and Smerdon, M. J. (2003)
and Luna, L. (2009) Expression and purification of NEIL3,
Suppressed catalytic activity of base excision repair enzymes
a human DNA glycosylase homolog, Protein Expr. Purif.,
on rotationally positioned uracil in nucleosomes, Proc.
65, 160 164, doi: 10.1016/j.pep.2008.11.014.
Natl. Acad. Sci. USA, 100, 7465 7470, doi: 10.1073/pnas.
50.
Piersen, C. E., McCullough, A. K., and Lloyd, R. S. (2000)
1330328100.
AP lyases and dRPases: Commonality of mechanism,
БИОХИМИЯ том 87 вып. 3 2022
336
УКРАИНЦЕВ и др.
Mutat. Res.,
459,
4353, doi:
10.1016/s0921
53. Maluchenko, N. V., Nilov, D. K., Pushkarev, S. V., Kotova,
8777(99)00054 3.
E. Y., Gerasimova, N. S., et al. (2021) Mechanisms of
51. Khodyreva, S. N., and Lavrik, O. I. (2011) Affinity modifica
nucleosome reorganization by PARP1, Int. J. Mol. Sci., 22,
tion in a proteomic study of DNA repair ensembles, Bioorg.
12127, doi: 10.3390/ijms222212127.
Khim., 37, 91 107, doi: 10.1134/s1068162011010109.
54. Gaullier, G., Roberts, G., Muthurajan, U. M.,
52. Galas, D. J., and Schmitz, A. (1978) DNAse footprinting: a
Bowerman, S., Rudolph, J., et al. (2020) Bridging of nucle
simple method for the detection of protein DNA binding
osome proximal DNA double strand breaks by PARP2
specificity, Nucleic Acids Res., 5, 3157 3170, doi: 10.1093/
enhances its interaction with HPF1, PLoS One, 15,
nar/5.9.3157.
doi: 10.1371/journal.pone.0240932.
THE STUDY OF THE INTERACTION PARP FAMILY DNA DEPENDENT
PROTEINS WITH NUCLEOSOMES CONTAINING DNA INTERMEDIATES
OF THE INITIAL STAGES OF THE BER PROCESS
A. A. Ukraintsev1#, E. A. Belousova1#, M. M. Kutuzov1, and O. I. Lavrik1,2*
1 Institute of Chemical Biology and Fundamental Medicine Siberian Branch of the Russian Academy of Sciences,
630090 Novosibirsk, Russia; e-mail: lavrik@niboch.nsc.ru
2 Novosibirsk State University, 630090 Novosibirsk, Russia
The cellular signal at the DNA damage response is the reaction of (ADP ribosyl)ation catalyzed by PARP’s family
DNA dependent proteins - PARP1, PARP2, and PARP3. These proteins are involved in the base excision repair
process, BER. Despite of the extensive research, it remains unknown how PARPs are involved in BER regulation and
how roles are distributed between DNA dependent family members. Here, we investigated the interaction of PARP’s
family DNA dependent proteins with nucleosome core particles containing DNA intermediates of the BER initial
stages. To do that, the nucleosomes containing damage in the vicinity of the duplex blunt end were reconstituted based
on the Widom “Clone 603” DNA sequence. The dissociation constants of PARP complexes with nucleosomes bear
ing DNA contained uracil (Native), AP site, or single nucleotide gap (Gap), were determined. It was shown that the
affinity of proteins for the nucleosomes was increased in row PARP3<<PARP2<PARP1; and the affinity of each pro
tein for the specific damage type was increased in the row Native = AP site < Gap for PARP1 and PARP2,
Gap <<< Native = AP site for PARP3. The interaction regions of each PARP protein with the nucleosome were also
determined by sodium borohydride cross linking and footprinting assay. Based on the obtained and published data,
the involvement pattern of PARP1, PARP2, and PARP3 into the interaction with NCPs containing DNA intermedi
ates of the BER process was discussed.
Keywords: nucleosome, PARP1, PARP2, PARP3, BER
БИОХИМИЯ том 87 вып. 3 2022