БИОХИМИЯ, 2022, том 87, вып. 7, с. 897 - 917
УДК 577.217.347;577.112.6;577.182.62;544.165;004.942
КОНЪЮГАТЫ ДЕСМИКОЗИНА С ФРАГМЕНТАМИ
АНТИМИКРОБНОГО ПЕПТИДА ОНКОЦИНА:
СИНТЕЗ, АНТИБАКТЕРИАЛЬНАЯ АКТИВНОСТЬ,
ВЗАИМОДЕЙСТВИЕ С РИБОСОМОЙ
© 2022 З.З. Хайруллина1, Г.И. Макаров2, А.Г. Терещенков3, В.С. Буев4,
Д.А. Лукьянов1,5, В.И. Польшаков6, В.Н. Ташлицкий1, И.А. Остерман1,5, Н.В. Сумбатян1*
1 Московский государственный университет имени М.В. Ломоносова, химический факультет,
119991 Москва, Россия; электронная почта: sumbtyan@belozersky.msu.ru
2 Южно-Уральский государственный университет (национальный исследовательский университет),
454080 Челябинск, Россия
3 НИИ физико-химической биологии имени А.Н. Белозерского,
Московский государственный университет имени М.В. Ломоносова, 119992 Москва, Россия
4 Московский государственный университет имени М.В. Ломоносова,
факультет биоинженерии и биоинформатики, 119992 Москва, Россия
5 Сколковский институт науки и технологий, 143025 Сколково, Россия
6 Московский государственный университет имени М.В. Ломоносова,
факультет фундаментальной медицины, 119991 Москва, Россия
Поступила в редакцию 20.05.2022
После доработки 17.06.2022
Принята к публикации 18.06.2022
С целью развития подходов к созданию новых антимикробных соединений осуществлены дизайн
и синтез конъюгатов, сочетающих в структуре макролидный антибиотик десмикозин и фрагмен-
ты антимикробного пептида онкоцина. Показано, что новые соединения способны связываться с
70S-рибосомами Escherichia coli подобно десмикозину и онкоцину, ингибировать бактериальную
трансляцию in vitro, а также подавлять рост бактериальных штаммов. Конъюгаты гекса- и тетра-
пептидных N-концевых фрагментов онкоцина с 3,2′,4′′-триацетилдесмикозином оказались активны-
ми в отношении штаммов, резистентных к макролидам. Методом молекулярной динамики найдены
структурные особенности взаимодействий этих производных с бактериальными рибосомами, в том
числе содержащими мутацию A2059G в 23S РНК.
КЛЮЧЕВЫЕ СЛОВА: макролиды, пептидные производные, антимикробные пептиды, рибосома, молеку-
лярная динамика, рибосомный туннель.
DOI: 10.31857/S0320972522070065, EDN: AVTKLC
ВВЕДЕНИЕ
разработка новых эффективных и безопасных
антибиотиков. Одним из подходов к созданию
Устойчивость патогенных микроорганиз-
новых антимикробных соединений является
мов к антибиотикам представляет серьёзную
рациональный дизайн, основанный на знаниях
проблему при их применении на практике, и
о механизмах действия известных антибиоти-
способом решения этой проблемы является
ков и их молекулярной мишени [1-4].
Принятые сокращения: МИК - минимальная ингибирующая концентрация; ПТЦ - пептидил-трансферазный
центр; РТ - рибосомный туннель; Boc - трет-бутилоксикарбонил; DCC - 1,3-дициклогексилкарбодиимид; DES -
десмикозин; DIPEA - диизопропилэтиламин; DMAP - диметиламинопиридин; DMF - N,N-диметилформамид; Fmoc -
флуоренилметилоксикарбонил; ERY - эритромицин; HBTU - O-(бензотриазол-1-ил)-N,N,N′,N′-тетраметилуроний
гексафторфосфат; HFIP - 1,1,1,3,3,3-гексафторизопропанол; GABA - 4-аминобутановая кислота; MALDI-TOF MS -
времяпролетная масс-спектрометрия с ионизацией методом матричной десорбции; MBS - сайт связывания макролидов
(Macrolide Binding Site); ONC - онкоцин 112 (VDKPPYLPRPRPPRrIYNr-NH2); Pip - пиперидин; RMSF - среднеквадра-
тичная флуктуация атомов (Root-Mean-Square Fluctuation); TFA - трифторуксусная кислота; TYL - тилозин.
* Адресат для корреспонденции.
897
898
ХАЙРУЛЛИНА и др.
Макролиды представляют собой класс
зинового ряда оказались способны действо-
антибиотиков, структура которых включает
вать на некоторые «необычные» рибосомы, в
лактонное кольцо с присоединёнными к нему
частности рибосомы апикопластов малярий-
углеводными остатками; действуя на бакте-
ных плазмодиев [19-21] или бактерии воль-
риальные рибосомы, макролиды проявляют
бахии, внутриклеточного симбионта круглых
активность в отношении многих патогенов,
червей филярий [22], и проявлять активность
вследствие чего находят широкое применение
в отношении возбудителей некоторых трудно
в медицинской и ветеринарной практике [5-7].
поддающихся лечению тропических болезней.
Механизм действия макролидов заключается в
В первые десятилетия после того, как были
том, что антибиотики связываются в начальном
найдены TYL, DES и другие антибиотики этого
участке рибосомного туннеля (РТ) бактерий в
ряда, было синтезировано множество их про-
непосредственной близости к пептидил-транс-
изводных по разным функциональным груп-
феразному центру (ПТЦ) в так называемом
пам лактонного кольца и углеводных остатков,
сайте связывания макролидов (MBS), при
многие из этих аналогов оказались активны в
этом макролиды избирательно ингибируют
отношении клинически значимых бактериаль-
трансляцию бактериальных белков, причём их
ных штаммов [23-25]. В том числе было пока-
действие зависит не только от структуры ан-
зано, что введение заместителей по 4′- и 4′′-гид-
тибиотика, но и от последовательности расту-
роксилам микаминозы и микарозы приводит
щей пептидной цепи, т.е. является контекст-
к аналогам, способным действовать против
но-специфичным [8-10]. Известны различные
устойчивых штаммов бактерий [26, 27]. С тех
механизмы формирования резистентности у
пор как были расшифрованы структуры бак-
бактерий к макролидным антибиотикам, среди
териальных рибосом и их комплексов с анти-
которых основную роль играет модификация
биотиками
[28-31], появилась возможность
мишени их действия, т.е. нуклеотидов 23S РНК
не только детального объяснения механизма
и белков
50S-субъединицы бактериальной
действия макролидов, механизмов возникно-
рибосомы в области MBS [7, 11, 12]. У боль-
вения устойчивости бактерий к ним и причин
шинства бактерий устойчивость возникает в
активности многих синтетических аналогов
результате моно- и диметилирования N6-ами-
антибиотиков, но и рационального дизайна
ногруппы нуклеотида А2058* под действием
аналогов макролидных антибиотиков [32-35].
метилтрансфераз семейства Erm (Erythromycin
С целью изучения взаимодействий пептид-
resistance methyltransferases), метилирования
ной цепи с элементами РТ был синтезирован
G748, а также некоторых других пуриновых
ряд пептидных аналогов TYL, DES и О-ми-
нуклеотидов доменов II и V 23S рРНК [13, 14].
каминозилтилонолида, в которых антибио-
Регуляция экспрессии генов устойчивости, в
тик служил якорем для закрепления пептида
частности тех или иных метилтрансфераз, свя-
в РТ [12, 36-38], эти аналоги связывались с
зана с образованием тройного комплекса рибо-
бактериальными рибосомами, ингибировали
сома-макролид-растущий пептид [8]. У ряда
бактериальную трансляцию in vitro и прояв-
микроорганизмов устойчивость к макролидам
ляли антибиотическую активность, однако не
возникает в результате мутаций в домене V
действовали в отношении устойчивых штам-
23S рРНК, чаще всего встречаются замена нук-
мов бактерий.
леотида A2062 на C (реже - на другие нуклео-
Около 20 лет назад были описаны анти-
тиды), мутации А2058G, A2059G и некоторых
микробные пептиды онкоцины, а сравни-
других нуклеотидных остатков, а также мута-
тельно недавно было обнаружено, что их дей-
ции и делеции в белках L4 и L22.
ствие направлено на рибосомы бактерий [39,
Макролиды на основе 16-членного лак-
40]. Онкоцины связываются в РТ, практиче-
тона, основными представителями которых
ски полностью его блокируя, и препятствуют
являются антибиотики тилозинового ряда: ти-
встраиванию аминоацил-тРНК, что позволя-
лозин (TYL) и десмикозин (DES) [15-17], об-
ет образоваться инициаторному комплексу,
ладают некоторыми преимуществами по отно-
но препятствует следующему шагу трансля-
шению к другим представителям этого класса
ции [41, 42]. Ориентация онкоцинов при связы-
как за счёт благоприятных фармакологических
вании противоположна ориентации синтези-
свойств, так и благодаря их активности в от-
руемой пептидной цепи - N-конец направлен
ношении определённых устойчивых штаммов
в сторону ПТЦ, а С-конец - в сторону выхода
бактерий [18]. Кроме того, антибиотики тило-
из РТ. Область связывания этих пептидов пе-
рекрывается с сайтами связывания большин-
ства антибиотиков, целью которых является
* Здесь и далее номера н.о. приводятся по последователь-
ности 23S РНК рибосомы Escherichia coli.
рибосома, в частности, макролидов [41, 43, 44].
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
899
В настоящей работе с целью развития под-
ласти, обнаруживали с помощью УФ-кабинета
ходов к созданию новых антимикробных сое-
(«Camag», Англия); соединения, содержащие
динений осуществлены дизайн и синтез, про-
свободные либо защищенные трет-бутилок-
тестированы антибактериальные свойства и
сикарбонилом (Boc) аминогруппы - нингид-
изучены взаимодействия с рибосомой новых
риновым реактивом.
конъюгатов, сочетающих в структуре макро-
Аминокислотный анализ осуществляли на
лидный антибиотик десмикозин и фрагменты
анализаторе «Hitachi 835» («Hitachi», Япония).
антимикробного пептида онкоцина. Дизайн
Кислотный гидролиз проводили в запаянных
соединений осуществлён методом статиче-
ампулах смесью 6 М HCl и TFA (2/1) при 155 °C
ского моделирования; аффинность соедине-
в течение 1 ч.
ний к рибосомам оценена по вытеснению ими
Хромато-масс-спектрометрию осуществля-
флуоресцентно меченного аналога эритроми-
ли с помощью системы UPLC/MS/MS, состо-
цина (ERY) из комплексов с 70S-рибосома-
ящей из хроматографа Acquity UPLC («Waters»,
ми E. coli; способность новых соединений ин-
США) и квадрупольного масс-спектрометра
гибировать биосинтез белка и подавлять рост
TQD («Waters») с регистрацией позитивных
бактерий протестирована in vitro и на ряде
ионов с помощью метода ESI MS, с исполь-
бактериальных штаммов; с помощью метода
зованием колонки Acquity BEH C18, 1,7 мкм,
молекулярной динамики проведён поиск воз-
50 × 2,1 мм («Waters»), 0.5 мл/мин, при 35°C С
можных взаимодействий этих соединений в
с градиентом 5-100% CH3CN в 20 мМ HCOOH
комплексах с бактериальными рибосомами.
за 4 мин.
Спектры MALDI-TOF MS получали на
MALDI-времяпролетном масс-спектрометре
МАТЕРИАЛЫ И МЕТОДЫ
«Ultrafle Xtreme Bruker Daltonics»
(«Bruker»,
Германия), оснащённом УФ-лазером (Nd), в
В работе были использованы реаген-
режиме положительных ионов с использовани-
ты и растворители следующего производ-
ем рефлектрона.
ства: 1,1,1,3,3,3-гексафторизопропанол (HFIP,
Спектры ЯМР соединений регистрирова-
«Acros Organics», Бельгия); носитель для твер-
ли на спектрометре «Bruker Avance» («Bruker»),
дофазного синтеза, содержащий
2-хлортри-
рабочие частоты составляли 600 МГц для 1H и
тилхлоридную группировку
(2CTC Resin) и
151 МГц - для 13C при 298 К в ДМСО-d6 с ис-
производные L-аминокислот («Iris Biotech», Гер-
пользованием тетраметилсилана в качестве
мания); 1,3-дициклогексилкарбодиимид (DCC),
внутреннего стандарта. Отнесение сигна-
O-(бензотриазол-1-ил)-N,N,N′,N′-тетраметил-
лов 1H и 13С было осуществлено с использова-
уроний гексафторфосфат (HBTU), диизо-
нием одномерных спектров 1H и 13С и двумер-
пропилэтиламин (DIPEA)
(«Sigma-Aldrich»,
ных 1H-1H ROESY (время смешивания 320 мс),
США, Швейцария); диметиламинопиридин
1H-1H DQF-COSY, 1H-1H TOCSY, 13C-1H HSQC
(DMAP,
«Serva», США);
4-аминобутановая
и 13C-1H HMBC. Спектры обрабатывали с помо-
кислота (GABA, «Reanal», Венгрия); трифтор-
щью программного обеспечения NMRPipe [46]
уксусная кислота (TFA, «PanReac AppliChem»,
с использованием стандартного протокола,
Германия) и абсолютные растворители: мета-
включающего взвешивающую функцию Ло-
нол
(«LiChrosolv», Германия); изопропанол,
ренца-Гаусса, метод прямого-обратного ли-
диоксан, пиридин, N,N-диметилформамид
нейного предсказания и метод полиноми-
(DMF), CH2Cl2, ацетонитрил («PanReac Appli-
альной коррекции базовой линии. Спектры
Chem», США), а также растворители российско-
одномерного ЯМР обрабатывали и анализиро-
го производства фирм «Химмед» и «Иреа2000».
вали с помощью программы Mnova («Mestrelab
TYL (4) был выделен из препарата «Тилозин 200»
Research», Испания). Двумерные спектры ана-
(ЗАО «Нита-Фарм», Россия) с помощью мето-
лизировали с помощью программного обеспе-
да колоночной хроматографии на силикагеле в
чения NMRFAM-Sparky [47].
системе CHCl3/MeOH = 15/1. Флуоресцентное
Анизотропию флуоресценции измеряли при
производное эритромицина BODIPY-ERY было
комнатной температуре с помощью планшет-
синтезировано по известной методике [45].
ного мультиридера «VICTOR X5 Multilabel Plate
Хроматография. ТСХ проводили на пла-
Reader» («Perkin Elmer», США) на 384-луноч-
стинках Kieselgel 60 F254 («Merck», Германия);
ном планшете. Длина волны поглощения со-
для колоночной хроматографии использовали
ставляла 485 нм, испускания - 535 нм.
Silica gel 60 (0,063-0,200 мм и 0,04-0,063 мм;
Дизайн молекул выполняли с помощью ме-
«Marcherey Nagel», Германия). Соединения,
тода статического моделирования с использова-
содержащие группы, поглощающие в УФ-об-
нием программного обеспечения Avogadro [48]
БИОХИМИЯ том 87 вып. 7 2022
900
ХАЙРУЛЛИНА и др.
и PyMOL 2.6 (The PyMOL Molecular Graphics
1083,6; найдено - 1083,6. 1H ЯМР, COSY, TOCSY,
System, Version 2.6 Schrödinger, LLC).
ROESY, HMBC, HSQC (CDCl3; 600 MHz) δ (ppm):
Синтез пептидных производных десмикози-
0,84 (t, J = 7,1 Hz, 3H, H17); 0,85-0,88 (m, 3H, H18);
на. Синтез пептидов Fmoc-Lys(Boc)-Pro-Pro-
1,04-1,07 (m, 3H, H6′); 1,10 (d, J = 6,3 Hz, 3H, H6′′);
Tyr(tBu)-OH (1a),Fmoc-Asp(tBu)-Lys(Boc)-Pro-Pro-
1,15 (d, J = 6,7 Hz, 3H, H21); 1,36 (s, 9H, H4′h); 1,45-
Tyr(tBu)-OH (2a), Boc-Val-Asp(tBu)-Lys(Boc)-Pro-
1,48 (m, 1H, H7α); 1,50-1,57 (m, 3H, H4, H7β, H16α);
Pro-Tyr(tBu)-OH (3a) описан в «Приложении»,
1,73-1,75 (m, 5H, H22, H4′c); 1,77-1,81 (m, 1H, H16β);
схема синтеза пептидов представлена на рис. S1
1,85 (br d, J = 3,8 Hz, 1H, H2α); 1,98 (s, 3H, H2′b);
в Приложении.
2,04 (s, 3H, H4′′b); 2,08 (s, 3H, H3b); 2,25-2,28 (m, 8H,
3,2′,4′′,4′′′-О-тетраацетил-тилозин (5). TYL (4)
H4′b, H3′a); 2,31-2,40 (m, 1H, H6); 2,40-2,56 (m, 3H,
(512 мг; 0,56 ммоль; 1 экв.) растворяли в пири-
H8, H19α, H2β); 2,64-2,73 (m, 2H, H19β, H3′); 2,94-
дине (5 мл) и добавляли уксусный ангидрид
2,99 (m, 2H, H14, H2′′); 3,07-3,11 (m, 2H, H4d); 3,30-
(290 мкл; 3,1 ммоль; 5,5 экв.). Раствор пере-
3,36 (m, 1H, H5′); 3,38 (s, 3H, H2′′a); 3,45 (s, 3H, H3′′a);
мешивали сутки при комнатной температуре.
3,46-3,52 (m, 1H, H23α); 3,55 (br d, J = 10,1 Hz, 1H, H5);
Пиридин удаляли на роторном вакуумном ис-
3,74 (br d, J = 10,1 Hz, 1H, H3); 3,80-3,86 (m, 2H,
парителе в смеси с бензолом. Продукт очищали
H3′′, H5′′); 3,90 (dd, J = 9,7, 4,5 Hz, 1H, H23β); 4,24 (d,
на силикагеле в системе CHCl3/MeOH = 20/1.
J = 7,9 Hz, 1H, H1′); 4,37 (t, J = 8,1 Hz, 1H, H4′′);
Выход: 479 мг (79%; 0,44 ммоль); ТСХ: Rf 0,43
4,55 (d, J = 7,5 Hz, 1H, H1′′); 4,64 (td, J = 9,9, 3,7 Hz,
(CHCl3/MeOH
=
20/1); MALDI-TOF MS,
1H, H4′); 4,70-4,76 (m, 1H, H15); 4,81-4,86 (m, 2H,
m/z вычислено для [C54H85NO21 + H]+ - 1084,6;
H2′, H4′e); 5,84 (d, J = 10,4 Hz, 1H, H13); 6,23 (d,
найдено - 1084,6.
J = 15,1 Hz, 1H, H10); 7,32 (d, J = 15,1 Hz, 1H, H11);
3,2′,4′′-О-триацетил-десмикозин (6). Ацети-
9,61 (d, J = 2,4 Hz, 1H, H20). 13C ЯМР, HMBC, HSQC
лированное производное тилозина (5) (359 мг;
(CDCl3, 151 MHz) δ (ppm): 7,55 (C18); 8,41 (С17);
0,33 ммоль; 1 экв.) растворяли в небольшом
12,12 (C22); 16,35 (C6′′); 16,36 (C6′); 16,42 (C21);
количестве ацетонитрила и добавляли 5,92 мл
19,65 (C3b); 19,87 (C4′′b); 20,29 (C2′b); 24,13 (C4′c);
0,2 N HCl (3,6 экв.). Смесь перемешивали
24,61 (С16); 27,29 (3C, C4′h); 30,81 (C4′b); 30,96 (C7);
при комнатной температуре несколько часов
31,53 (C6); 38,25 (C2); 38,70 (C4′d); 39,72 (C4);
до достижения полного гидролиза, который
40,30 (2C, C3′a); 42,71 (C19); 43,27 (C14); 44,01 (C8);
контролировали по ТСХ в системе CHCl3/
58,39 (C2′′a); 60,48 (C3′′a); 65,69 (C3); 66,07 (C3′);
MeOH = 20/1. По окончании реакции смесь
66,30 (C5′′); 68,72 (C23); 69,42 (C2′); 69,99 (C5′);
нейтрализовали с помощью 5%-ного раствора
70,29 (C4′); 73,67 (C4′′); 74,02 (С15); 76,64 (C3′′);
NaHCO3, экстрагировали хлороформом, промы-
78,07 (C4′g); 79,52 (C2′′); 79,69 (C5); 100,05 (C1′′);
вали насыщенным раствором NaCl и сушили
100,73 (C1′); 117,55 (C10); 133,85 (C12); 140,45 (C13);
над б/в Na2SO4. Очистку продукта проводили
146,95 (C11); 154,84 (C4′f); 168,06 (C2′a); 169,02 (C4′′a);
с использованием колоночной хроматографии
169,84 C3a); 171,02 (C4′a); 172,62 (C1); 201,54 (C20);
в системе CHCl3/MeOH = 20/1. Выход: 236 мг
202,31 (C9).
(80%; 0,26 ммоль); ТСХ: Rf 0,29 (CHCl3/MeOH =
3,2′,4′′-О-триацетил-4′-О-(4-аминобутаноил)-
= 20/1); Rf 0,31 (CHCl3/MeOH/19% NH4OH =
десмикозин (8). Соединение 7 (76 мг; 0,070 ммоль)
= 16/1/0,1); MALDI-TOF MS, m/z вычислено
растворяли в 3 мл 50%-ной TFA в CH2Cl2 при
для [C45H71NO17 + H]+ - 898,5; найдено - 898,6.
0 °С и перемешивали в течение 1 ч. Летучие ком-
3,2′,4′′-О-триацетил-4′-О-(4-[(трет-буток-
поненты смеси удаляли на роторном вакуум-
сикарбонил)амино]бутаноил)-десмикозин (7). Про-
ном испарителе, остаток растворяли в неболь-
изводное 6 (208 мг; 0,23 ммоль; 1 экв.) и Boc-
шом объёме CH2Cl2 и осаждали диэтиловым
GABA (94 мг; 0,46 ммоль; 2 экв.) растворяли в
эфиром, осадок центрифугировали, переоса-
хлористом метилене (абс.) при 0 °С. Далее после-
ждали продукт таким образом ещё несколько
довательно добавляли DCC (143 мг; 0,69 ммоль;
раз, сушили на воздухе. Продукт использова-
3 экв.) и DMAP (42 мг; 0,34 ммоль; 1,5 экв.).
ли на следующей стадии без дополнительной
Смесь оставляли перемешиваться при комнат-
очистки. Выход: 16 мг (23%; 0,016 ммоль); ТСХ:
ной температуре на ночь. Растворитель удаляли
Rf 0,17 (CHCl3/MeOH = 20/1); MALDI-TOF MS,
на роторном вакуумном испарителе, образовав-
m/z вычислено для [C49H78N2O18 + H]+ - 983,5;
шийся осадок растворяли в ацетонитриле и отде-
найдено - 983,7.
ляли от осадка дициклогексилмочевины филь-
3,2′,4′′-О-триацетил-4′-О-[[(N2-флуоре-
трованием. Очистку продукта проводили
нилметоксикарбонил)-N6-трет-бутоксикарбо-
методом колоночной хроматографии в системе
нил-L-лизил]-L-пролил-L-пролил-(О-трет-бу-
CHCl3/MeOH = 20/1. Выход: 137 мг (57%; 0,13 ммоль);
тил-L-тирозил)-4-аминобутаноил]-десмико-
ТСХ: Rf 0,51 (CHCl3/MeOH = 20/1); MALDI-
зин (9a). Защищённый тетрапептид 1a (54 мг;
TOF MS, m/z вычислено для [C54H86N2O20 + H]+ -
61 мкмоль; 1,5 экв.) растворяли в DMF и добав-
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
901
ляли HBTU (31 мг; 82 мкмоль; 2 экв.), DIPEA
стом метилене при 0 °С, и смесь перемешивали
(21 мкл; 123 мкмоль; 3 экв.) и перемешивали в
в течение 1 ч при 0 °С. Далее TFA и раствори-
течение 15 мин. Далее в смесь добавляли про-
тель удаляли в вакууме. Получившийся сухой
изводное 8 (40 мг; 41 мкмоль; 1 экв.). Затем по
остаток растворяли в небольшом количестве
истечении 20 мин добавляли 20% DIPEA от ис-
хлористого метилена и осаждали диэтиловым
ходного количества (4,2 мкл). Смесь оставля-
эфиром, осадок отделяли центрифугировани-
ли перемешиваться на ночь, затем разбавляли
ем, затем полученный продукт переосаждали
водой в несколько раз и экстрагировали сме-
таким образом несколько раз. Выход: 1 мг (8%;
сью гексан/этилацетат = (1/1). Органическую
0,68 мкмоль); MALDI-TOF MS, m/z вычис-
фазу последовательно промывали небольшими
лено для [C74H113N7O23 + H]+ - 1468,8; найде-
объёмами 0,1 М HCl, воды и 5%-ного раство-
но - 1468,9.
ра NaHCO3. Очистку целевого соединения про-
3,2′,4′′-О-триацетил-4′-О-(L-аспартил-L-ли-
водили в системе CHCl3/MeOH = 20/1. Выход:
зил-L-пролил-L-пролил-L-тирозил-4-амино-
23 мг (29%; 12 мкмоль); ТСХ: Rf 0,42 (CHCl3/
бутаноил)-десмикозин (2) получали аналогично 1
MeOH = 20/1); MALDI-TOF MS, m/z вычис-
исходя из 9b (9 мг; 4,5 мкмоль). Выход: 1 мг
лено для [C98H139N7O27 + H]+ - 1847,0; найде-
(14%; 0,63 мкмоль); MALDI-TOF MS, m/z вы-
но - 1846,8.
числено для [C78H118N8O26 + H]+ - 1583,8; най-
3,2′,4′′-О-триацетил-4′-О-[[(N-флуоренил-
дено - 1583,9.
метоксикарбонил)-О-трет-бутил-L-аспартил]-
3,2′,4′′-О-триацетил-4′-О-(L-валил-L-аспар-
(N6-трет-бутоксикарбонил-L-лизил)-L-про-
тил-L-лизил-L-пролил-L-пролил-L-тирозил-4-
лил-L-пролил-(О-трет-бутил-L-тирозил)-4-
аминобутаноил)-десмикозин (3). Соединение 9c
аминобутаноил]-десмикозин (9b). Получали ана-
(30 мг;
15 мкмоль) растворяли в
500 мкл
логично 9a, исходя из 41 мг (39 мкмоль; 1,3 экв.)
50%-ной TFA в хлористом метилене при 0 °С
защищённого пентапептида 2a, HBTU (23 мг;
и перемешивали в течение 1 ч при 0 °С. Далее
60 мкмоль; 2 экв.), DIPEA (19 мкл; 107 мкмоль;
TFA и растворитель удаляли в вакууме. Полу-
3,6 экв.), производного 8 (29 мг; 30 мкмоль;
чившийся сухой остаток растворяли в неболь-
1 экв.). Очистку целевого соединения прово-
шом количестве хлористого метилена и оса-
дили в системе CHCl3/MeOH = 20/1. Выход:
ждали диэтиловым эфиром, осадок отделяли
9 мг (15%; 4,5 мкмоль); ТСХ: Rf 0,35 (CHCl3/
центрифугированием, затем полученный про-
MeOH = 20/1); MALDI-TOF MS, m/z вычис-
дукт переосаждали несколько раз. Выход: 5 мг
лено для [C106H152N8O30 + H]+ - 2018,1; найде-
(20%; 3,0 мкмоль); MALDI-TOF MS, m/z вы-
но - 2017,8.
числено для [C83H127N9O27 + H]+ - 1682,9; най-
3,2′,4′′-О-триацетил-4′-О-[(N-трет-бутил-
дено - 1683,0.
оксикарбонил-L-валил)-(О-трет-бутил-L-аспар-
Изучение связывания аналогов десмикози-
тил)-(N6-трет-бутоксикарбонил-L-лизил)-L-
на с рибосомами E. coli. Аффинность соедине-
пролил-L-пролил-(О-трет-бутил-L-тирозил)-
ний к 70S-рибосомам E. coli (штамм MRE-600
4-аминобутаноил]-десмикозин (9c). Получали ана-
(70S-рибосомы E. coli были любезно предо-
логично 9a, исходя из 80 мг защищённого гекса-
ставлены лабораторией А.Л. Коневеги)) опре-
пептида (3a) (78 мкмоль; 1,5 экв.), HBTU (39 мг;
деляли методом конкурентного связывания в
103 мкмоль; 2 экв.), DIPEA (32 мкл; 185 мкмоль;
присутствии флуоресцентного аналога эри-
3,6 экв.), производного 8 (51 мг; 52 мкмоль;
тромицина - BODIPY-ERY, как было описано
1 экв.). Очистку целевого соединения прово-
ранее [49, 50]. Концентрации исследуемых ве-
дили в системе CHCl3/MeOH = 20/1. Выход:
ществ варьировались от 1 нМ до 10 мкМ. Кон-
30 мг (29%; 15 мкмоль); ТСХ: Rf 0,3 (CHCl3/
центрация 70S-рибосом E. coli и BODIPY-ERY
MeOH = 20/1); MALDI-TOF MS, m/z вычислено
составляла 50 и 16 нМ соответственно. Смесь
для [C101H159N9O31 + H]+ - 1995,1; найдено - 1994,9.
инкубировали 2 ч при комнатной температуре,
3,2′,4′′-О-триацетил-4′-О-(L-лизил-L-пролил-
затем измеряли уровень анизотропии флуорес-
L-пролил-L-тирозил-4-аминобутаноил)-десмико-
ценции. Для каждого соединения было выпол-
зин (1). Соединение 9a массой 16 мг (8,7 мкмоль)
нено минимум 2 повтора. Расчёт эффективных
растворяли в 500 мкл 20%-ного раствора пипе-
констант диссоциации был проведён на основе
ридина (Pip) в DMF. Смесь перемешивали в те-
стандартной модели, которая описывает равно-
чение 40 мин при комнатной температуре. Да-
весное конкурентное связывание двух лиган-
лее продукт осаждали из раствора петролейным
дов в одном сайте [51].
эфиром, осадок отделяли центрифугировани-
In vitro трансляция. Ингибирование синте-
ем, и вещество переосаждали таким образом
за белка в бесклеточной системе проводили
несколько раз. Полученный твёрдый остаток
на основе системы трансляции люцифера-
добавляли к 500 мкл 50%-ной TFA в хлори-
зы светлячка, как было описано ранее [52] -
БИОХИМИЯ том 87 вып. 7 2022
902
ХАЙРУЛЛИНА и др.
T7-транскрибируемая мРНК (Fluc) люцифера-
плотности при 600 нм. Измерение проводи-
зы светлячка (Photinus pyralis) транслировалась
ли на планшетном мультиридере «VICTOR X5
в бесклеточной S30-системе E. coli согласно
Multilabel Plate Reader». В качестве минималь-
известной методике [53]. Концентрация ис-
ной ингибирующей концентрации (МИК)
следуемых соединений составляла
20 мкМ,
принимали наименьшую концентрацию, при
уровень трансляции in vitro после инкубиро-
которой исследуемое вещество полностью по-
вания измеряли с помощью системы Bright-
давляло рост бактерий.
Glo® Luciferase Assay System («Promega», США)
Молекулярная динамика*. Моделируемая
на планшетном мультиридере
«VICTOR X5
система. Структура рибосомы E. coli была по-
Multilabel Plate Reader».
лучена исходя из данных рентгеноструктур-
Тестирование на антибиотическую актив-
ного анализа структуры с разрешением 3,1 Å
ность проводили на следующих штаммах: E. coli
(PDB ID: 4V7U) [58] с добавлением в 23S рРНК
JW5503 ΔtolC, модифицированном репортёр-
модифицированных оснований в соответ-
ной последовательностью pDualrep2 [54, 55], в
ствии со сведениями из банка данных [59].
котором остановка процесса трансляции при-
Положения модифицированных оснований
водит к экспрессии красного флуоресцентного
были оптимизированы минимизацией энер-
белка Katushka2S; E. coli JW5503 ΔtolC KanR, ре-
гии сочетанием метода наискорейшего спуска
зистентный к канамицину; E. coli JW5503 ΔtolC,
с алгоритмом Бройдена-Флетчера-Гольдфар-
модифицированном плазмидой pKH80
[56],
ба-Шанно с ограниченным использованием
обеспечивающей устойчивость к эритроми-
памяти [60], за которой следовал расчёт ко-
цину за счёт экспрессии метилтрансферазы
роткой молекулярной динамики. При этом все
ErmC; E. coli SQ171 ΔtolC, трансформирован-
немодифицированные основания были пол-
ном плазмидой pAM552 [57]; E. coli SQ171 ΔtolC,
ностью зафиксированы, тогда как модифици-
модифицированном pAM552 с заменой A2058G
рованные основания, ионы и вода двигались,
в 23S рРНК; E. coli SQ171 ΔtolC, модифициро-
а эритромицин, содержавшийся в исходной
ванном pAM552 с заменой A2059G в 23S рРНК
рентгеновской структуре, был изъят. После оп-
(плазмиды pAM552, содержащие замены
тимизации были выделены все остатки, у кото-
A2058G и A2059G, были любезно предостав-
рых хотя бы один атом попадал в кубическую
лены А.С. Манькиным (Иллинойсский уни-
область с ребром длиной 7 нм, включающую в
верситет в Чикаго)). Тестируемые соединения
себя весь РТ и ПТЦ, так что центр этой обла-
в количестве 10 нмоль, а также эритромицин в
сти находился в туннеле, а сам туннель проле-
количестве 6,8 нмоль наносили на чашки с ага-
гал вдоль мысленной оси аппликат. Подобный
ром, которые уже содержали соответствующий
подход использовался в работах ранее [61, 62].
штамм. Чашки инкубировали при 37 °С в тече-
На полученное выделение накладывали струк-
ние ночи, уровень экспрессии флуоресцент-
туру рибосомы Haloarcula marismortui, имею-
ных белков в штамме, содержащем плазмиду
щую разрешение 3,0 Å (PDB ID: 1K9M) [28],
pDualrep2, и зоны ингибирования бактериаль-
устанавливая в этот фрагмент рибосомы TYL.
ного роста в остальных штаммах оценивали с
Затем остаток микарозы удаляли, а конфор-
помощью системы визуализации ChemiDoc
мацию остатка мицинозы в сайте связывания
(«Bio-Rad», США).
оптимизировали докингом, применяя пакет
Измерение
минимальной
ингибирую-
rDock [63], сохраняя при этом положение, ори-
щей концентрации проводили на следующих
ентацию и конформацию лактонного коль-
штаммах: E. coli JW5503 ΔtolC KanR, E. coli
ца и остатка микаминозы неизменными. При
JW5503 ΔtolC pKH80, E. coli SQ171 ΔtolC pAM552,
моделировании комплекса онкоцина с рибо-
E. coli SQ171 ΔtolC pAM552 A2058G, E. coli SQ171
сомой E. coli на то же выделение накладыва-
ΔtolC pAM552 A2059G. Для проведения экспе-
ли структуру комплекса онкоцина 112 (ONC,
римента
96-луночный планшет заполняли
VDKPPYLPRPRPPRrIYNr-NH2) с рибосомой
суспензией клеток, полученной путём разбав-
Thermus thermophilus, имеющую разрешение
ления ночной культуры в 200 раз в среде LB
2,9 Å (PDB ID: 4Z8C) [43], устанавливая в этот
(«Amresco», США). В ячейки первого ряда
фрагмент ONC.
добавляли
4 мкл тестируемого соединения
При моделировании конъюгатов десмико-
(в концентрации 4-68 мМ в ДМСО), после
зина с фрагментами онкоцина (1-3) исходные
чего готовили серию двукратных разведений
состояния структуры их комплексов с рибосомой
в последующих рядах до десятого. После этого
E. coli получали, накладывая на описанную выше
планшеты инкубировали при 37 °С в течение
ночи с аэрацией 200 об./мин. О концентра-
* Детализированное описание методов молекулярно-ди-
ции клеток судили по значению оптической
намического моделирования приведено в Приложении.
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
903
структуру комплекса десмикозина N-концевые
но заряженных групп [75] (программный код
пептиды онкоцина Val1Asp2Lys3Pro4Pro5Tyr6 или
был любезно предоставлен А.О. Залевским).
Lys3Pro4Pro5Tyr6 из структуры комплекса ONC
Кроме того, часть молекул воды была случай-
с рибосомой T. thermophilus. При этом остаток
ным образом заменена ионами калия, магния
4-аминобутановой кислоты, соединяющий
и хлорид-анионами с тем, чтобы препятство-
десмикозиновую и онкоциновую части конъю-
вать вымыванию противоионов магния и ка-
гатов, а также ацетильные группы в 3,2′,4′′-по-
лия в водную фазу; при этом в водной фазе
ложениях были смоделированы с помощью
устанавливались концентрации 7 мМ MgCl2 и
программы PyMOL.
100 мМ KCl. Остатки, хотя бы один атом кото-
В ходе молекулярно-динамического мо-
рых находится в пределах 0,1 нм от грани моде-
делирования этилальдегидная группа каждого
лируемого участка рибосомы, были позицион-
рассматриваемого производного десмикозина
но ограничены, при этом все остальные могли
была ковалентно связана с экзоциклической
свободно двигаться.
аминогруппой основания A2062 с образова-
При моделировании структуры комплекса
нием карбиноламина. При изучении эффекта
соединения 3 с рибосомой E. coli выполнялись
мутации A2059G в построенных таким обра-
расчёты уравновешенной метадинамики [76] с
зом структурах заменялось соответствующее
обменом реплик [77] для введённых ацетиль-
азотистое основание.
ных групп. Расчёт выполняли при помощи па-
Общие условия моделирования. Все расчёты
кета PLUMED версия 2.3 [78], работающего в
молекулярной динамики и анализ полученных
комплексе с пакетом GROMACS версия 5.1.4.
траекторий проводили с использованием паке-
Взвешивающий потенциал подстраивали,
та GROMACS версия 5.1.4 [64, 65]. Канониче-
добавляя к нему одно- и двумерные гауссо-
ские и модифицированные аминокислотные
вы функции высотой 3 кДж/моль и шириной
и нуклеотидные остатки моделировали с ис-
10 градусов с периодичностью 2500 шагов (5 пс)
пользованием силового поля Amber14sb [66], а
до тех пор, пока высота гауссиан не достигла
DES и его производные - с помощью силово-
порядка 0,1 кДж/моль. Взвешивающий коэф-
го поля GAFF [67]. При этом оптимизирован-
фициент составлял 8, использовалось 6 реплик.
ные трёхмерные структуры и молекулярные
В ходе этих расчётов подвижность атомов саха-
электростатические потенциалы вновь пара-
рофосфатного остова 23S рРНК была ограни-
метризуемых остатков и соединений получа-
чена параболическим потенциалом с силовой
ли квантово-химическими расчётами методом
константой 1000 кДж/нм2, подвижность атомов
Хартри-Фока с базисом 6-31G*. Точечные за-
пептидной части конъюгата была ограничена
ряды вычисляли в модели RESP [68]. Времен-
параболическим потенциалом с силовой кон-
ной шаг интегрирования во всех вычислениях
стантой 10 кДж/нм2, а подвижность десмико-
составлял 2 фс, координаты записывались в
зиновой части конъюгата, воды, ионов, а также
файл траектории каждые 25 пс. Для ограниче-
аминокислотных остатков белков рибосомы и
ния длин связей с атомами водорода использо-
азотистых оснований не ограничивалась.
вали алгоритм LINCS [69]. Моделирование вы-
Методы анализа траекторий. Анализ ме-
полнялось при температуре 310 К с периодом
стонахождения и частоты встречаемости во-
привязки 0,1 пс под управлением термостата
дородных связей и стэкинг-взаимодействий
масштабирования скоростей с добавочным
производили по описанной ранее методи-
стохастическим членом [70] и периодических
ке
[79]. Частота встречаемости водородной
граничных условиях с изотропным постоян-
связи или стэкинг-взаимодействия рассчи-
ным давлением, поддерживаемым баростатом
тывалась как отношение количества кадров
Берендсена [71] с периодом привязки 5 пс.
траектории, в которых обнаружено соответ-
Электростатические взаимодействия обраба-
ствующее взаимодействие, к общему коли-
тывались посредством сети частиц Эвальда [72]
честву кадров в траектории. Кластеризацию
с шагом сетки 0,125 нм и четвёртым порядком
проводили методом GROMOS
[80]. Так-
интерполяции. Моделируемые системы цен-
же вычисляли значения среднеквадратич-
трировали в тетрагональной ячейке размером
ной флуктуации атомов (RMSF). RMSF
9,5 × 9,5 × 9,4 нм, заполненной молекулами
i-атома с координатой ri на протяжении T кад-
воды TIP4PEW [73] так, что грани системы были
ров траектории рассчитывали по формуле:
покрыты слоем растворителя в 0,9 нм. Оста-
точный отрицательный заряд системы был
компенсирован ионами калия с оптимизиро-
ванными параметрами [74], причём добавля-
При анализе траекторий значения RMSF ато-
емые ионы располагались вблизи отрицатель-
мов усредняли по рассматриваемым молекулам.
БИОХИМИЯ том 87 вып. 7 2022
904
ХАЙРУЛЛИНА и др.
Визуализацию полученных данных прово-
альных рибосом с TYL и ONC (рис. 1, г) по-
дили с помощью программного обеспечения
казало, что 4′-O-атом микаминозы TYL на-
PyMOL 2.6 (The PyMOL Molecular Graphics
ходится на расстоянии 5,5 Å от атома азота
System, Version 2.6 Schrödinger, LLC).
амидной группы Tyr6 ONC. Поэтому в каче-
стве фрагментов онкоцина для присоединения
к DES были выбраны тетра-(Lys3Pro4Pro5Tyr6),
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
пента-(Asp2Lys3Pro4Pro5Tyr6) и гексапептид
(Val1Asp2Lys3Pro4Pro5Tyr6). Подходящим по
Статическое моделирование. С помощью
длине линкером для соединения 4′-OH-группы
статического моделирования был выполнен ди-
микаминозы и карбоксильной группы Tyr6 ока-
зайн конъюгатов десмикозина с фрагментами
зался остаток
4-аминобутановой кисло-
онкоцина, целью которого было определение
ты (рис. 1, д).
оптимальной аминокислотной последователь-
Синтез. Синтез сконструированных в со-
ности в структуре онкоцина (рис. 1, а), с кото-
ответствии с данными статического моде-
рой может быть осуществлена конъюгация, а
лирования конъюгатов десмикозина с фраг-
также нахождение наиболее подходящего лин-
ментами онкоцина
(1-3) был осуществлён
кера для соединения макролидной и пептид-
по схеме (рис. 2). На первом этапе проводили
ной частей молекулы. Для этого использовали
ацетилирование TYL с последующим кис-
кристаллические структуры TYL в комплексе с
лотным гидролизом с получением производ-
рибосомой H. marismortui (PDB ID: 1K9M) [28]
ного десмикозина 6, в структуре которого все
и ONC в комплексе с рибосомой T. thermo-
гидроксильные группы, кроме 4′-OH, были
philus (PDB ID: 4Z8C) [43], полученные мето-
ацетилированы. Производное модифициро-
дом рентгеноструктурного анализа (рис. 1, б-г).
вали с помощью Boc-4-аминобутановой кис-
Наложение структур комплексов бактери-
лоты по 4′-OH-группе, получая таким обра-
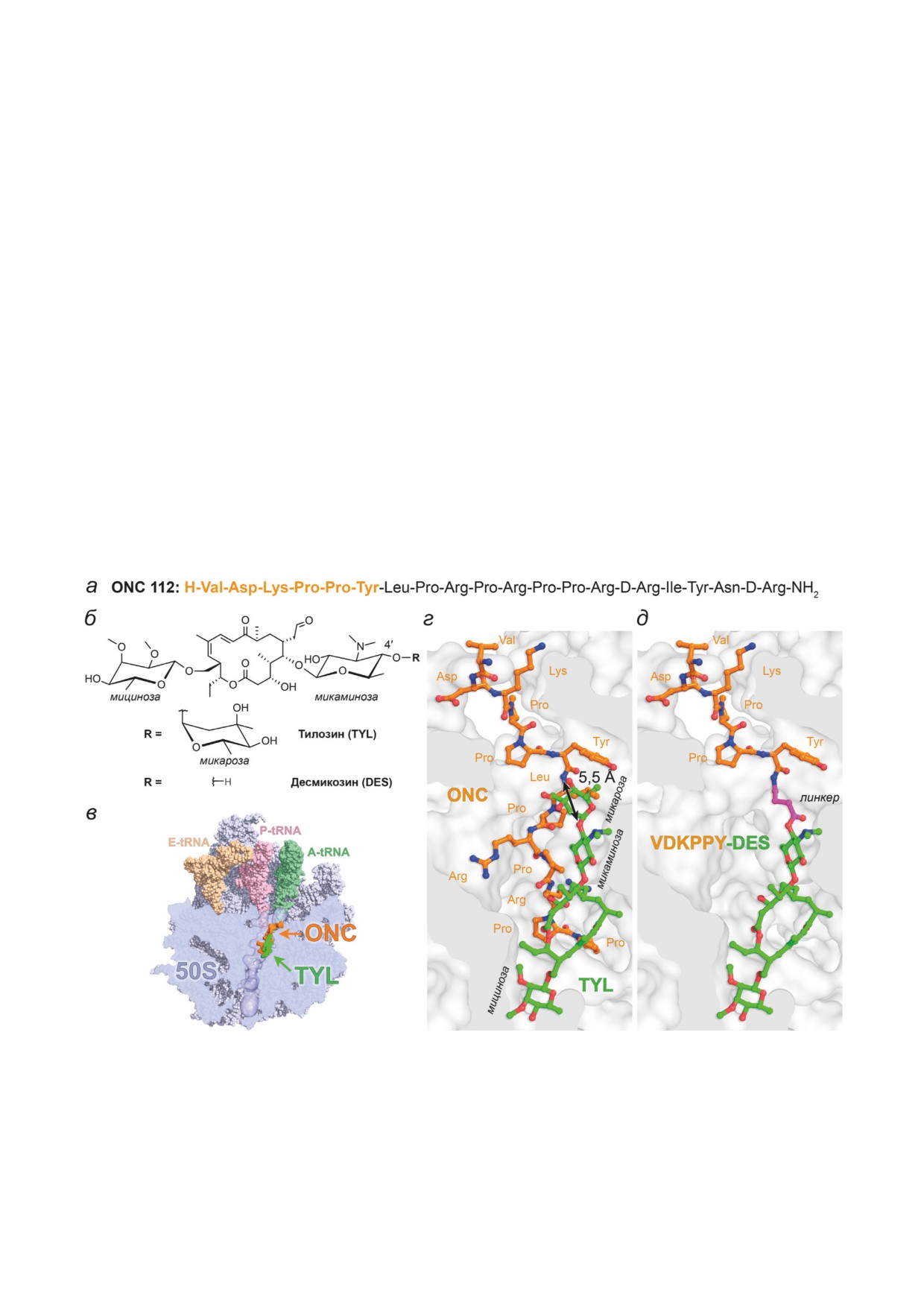
Рис. 1. Структуры и комплексы с рибосомой онкоцина 112 (ONC), тилозина (TYL), десмикозина (DES) и их производ-
ного VDKPPY-DES. а - Аминокислотная последовательность антимикробного пептида ONC. Оранжевым выделена
последовательность, предложенная для модификации DES. б - Структура антибиотиков TYL и DES. в - Схематичное
отображение сайтов связывания TYL (зелёный) и ONC (оранжевый) с 50S-субъединицей рибосомы T. thermophilus.
г - Наложение структуры комплекса TYL (зёленый) с рибосомой H. marismortui (PDB ID: 1K9M) и структуры комплек-
са ONC (оранжевый) с рибосомой T. thermophilus (PDB ID: 4Z8C). Показано расстояние между атомом азота амидной
группы Tyr6 онкоцина и 4′-кислородом микаминозы. д - Модель конъюгата десмикозина (зелёный) с фрагментом он-
коцина1-6 (Val1Asp2Lys3Pro4Pro5Tyr6, оранжевый), соединённые через остаток 4-аминобутановой кислоты (розовый), в
комплексе с рибосомой T. thermophilus (PDB ID: 4Z8C)
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
905
зом соединение 7, после чего деблокировали
пептидов от носителя с сохранением защитных
аминогруппу. К полученному производному
групп применяли HFIP (рис. S1 в Приложении).
десмикозина
(8) присоединяли N-концевые
Конденсацию пептидов 1a-3a с аминогруппой
пептидные фрагменты онкоцина (1a-3a), за-
производного десмикозина 8 осуществляли с
щищённые флуоренилметилоксикарбонильной
помощью HBTU. Удаление защитных групп
группой (Fmoc-группой) по α-аминофункции,
пептидных фрагментов соединений 9a-9c при-
Boc-группой - по ε-аминогруппе лизина и
водило к получению конъюгатов 3,2′,4′′-триа-
tBu-группами - по боковым функциональным
цетилдесмикозина с фрагментами онкоцина
группам аспарагиновой кислоты и тирозина.
Lys3-Tyr6 (1), Asp2-Tyr6 (2) и Val1-Tyr6 (3).
Пептиды 1a-3a были предварительно синте-
Очистка промежуточных и целевых соеди-
зированы методом твёрдофазного синтеза на
нений проводилась с помощью метода колоноч-
2-хлортритильном полимере; для отщепления
ной хроматографии на силикагеле. Соединения
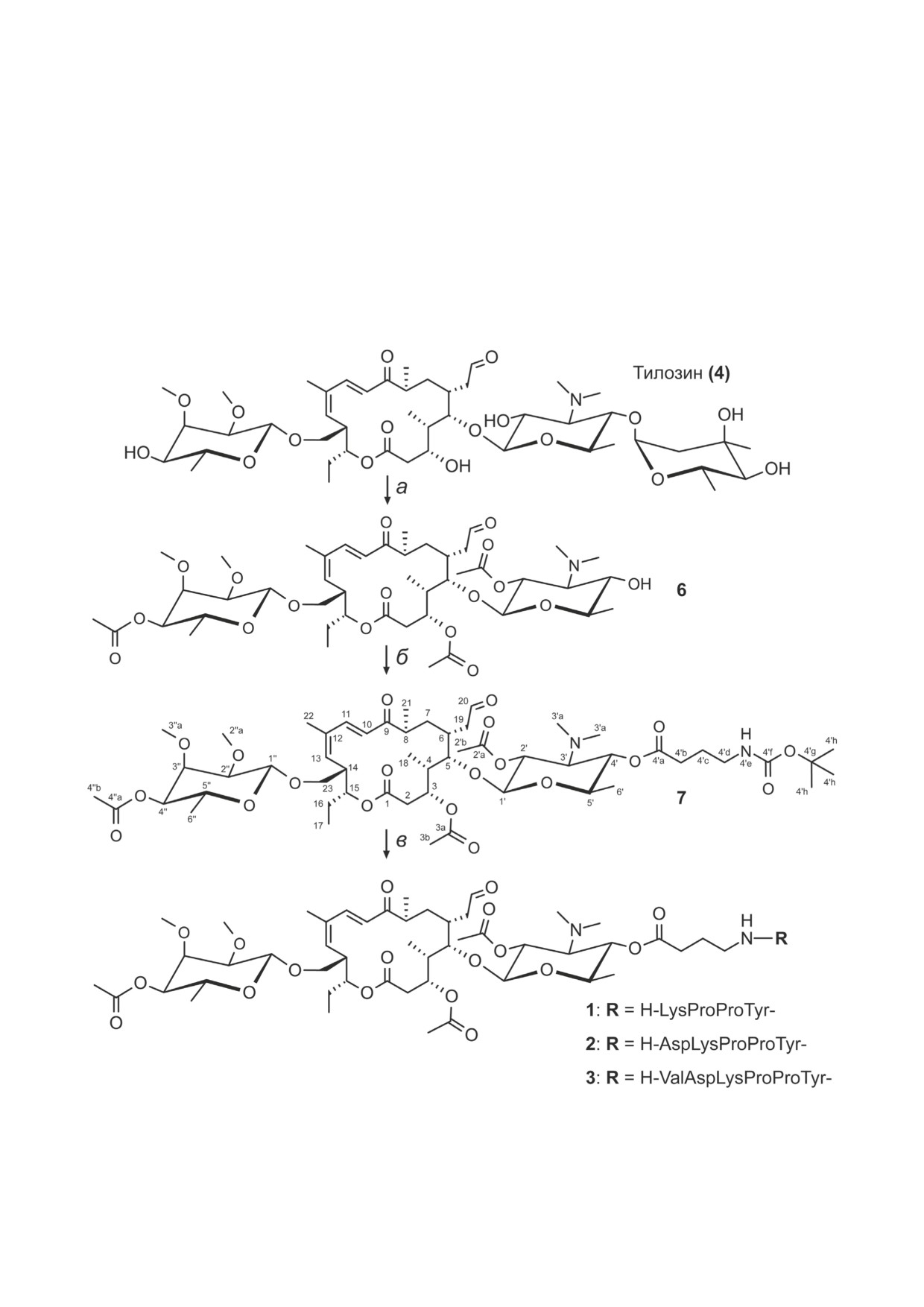
Рис. 2. Схема синтеза конъюгатов десмикозина с фрагментами пептида онкоцина. а - 1) Ac2O, Py (5), 2) 0,2 M HCl (6);
б - Boc-GABA, DMAP, DCC (7); в - 1: 1) 50% TFA/CH2Cl2, 0 °C (8), 2) Fmoc-Lys(Boc)ProProTyr(tBu)-OH (1а), HBTU,
DIPEA (9a), 3) 20% Pip/DMF, 4) 50% TFA/CH2Cl2, 0 °C (1); 2: 1) 50% TFA/CH2Cl2, 0 °C (8), 2) Fmoc-Asp(tBu)Lys(Boc)
ProProTyr(tBu)-OH (2а), HBTU, DIPEA (9b), 3) 20% Pip/DMF, 4) 50% TFA/CH2Cl2, 0 °C (2); 3: 1) 50% TFA/CH2Cl2,
0 °C (8), 2) Boc-ValAsp(tBu)Lys(Boc)ProProTyr(tBu)-OH (3а), HBTU, DIPEA (9c), 3) 50% TFA/CH2Cl2, 0 °C (3)
5
БИОХИМИЯ том 87 вып. 7 2022
906
ХАЙРУЛЛИНА и др.
были охарактеризованы методами хроматогра-
пазоне, что характерно как для класса макро-
фии и масс-спектрометрии. Положение замес-
лидов [49], так и для взаимодействующих с
тителя по 4′-гидроксильной группе DES было
рибосомой пролин-богатых антимикробных
доказано для промежуточного соединения 7 с
пептидов (ПбАМП), в том числе ONC (6-
помощью методов двумерного ЯМР: COSY,
93 нМ) [81-84], они всё же на несколько поряд-
TOCSY, ROESY, 13C-HMBC, 13C-HSQC (рис. S2
ков превышают значение KD для DES. При этом
в Приложении). Так, на спектре 13C-HMBC на-
наблюдается уменьшение степени сродства
блюдается кросс-пик, соответствующий взаи-
полученных конъюгатов к РТ с ростом длины
модействию атома H4′ микаминозы с углеро-
пептидного фрагмента. Тем не менее аффин-
дом С4′a карбонильной группы Boc-GABA.
ность производных десмикозина, содержащих
Эти атомы, в свою очередь, однозначно иден-
фрагменты онкоцина, к бактериальной рибо-
тифицируются по наличию характеристичных
соме превышает таковую для многих антибио-
корреляций в спектрах 13C-HMBC и ROESY.
тиков, связывающихся в ПТЦ и РТ [85], в том
Изучение связывания производных десмико-
числе некоторых ПбАМП [81-84], эффективно
зина с рибосомами E. coli. Способность соедине-
подавляющих рост бактерий, и значительно
ний связываться с бактериальными рибосомами
превосходит аффинность фрагмента ONC1-
была оценена методом конкурентного связы-
9 (KD = 2,8 ± 4,3 мкМ) [84].
вания в присутствии флуоресцентного аналога
In vitro трансляция. Поскольку известно,
эритромицина - BODIPY-ERY, как было опи-
что для многих макролидов ингибирование
сано ранее [49, 50]. С помощью данного метода
пептидил-трансферазной реакции происхо-
удалось определить эффективные константы
дит в процессе трансляции с участием синте-
диссоциации (KD) комплексов полученных со-
зирующейся полипептидной цепи [86-88] и
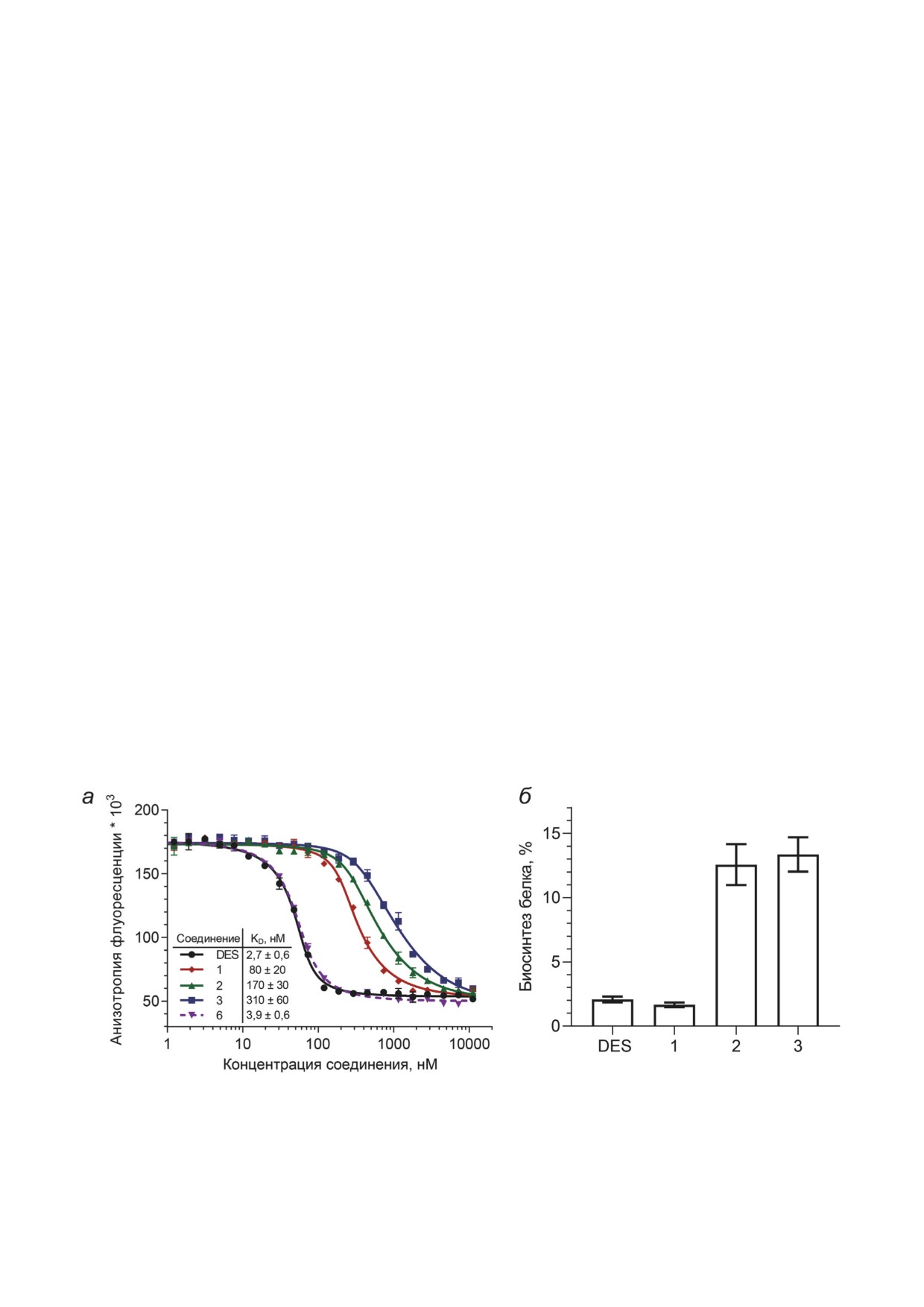
единений с рибосомами E. coli (рис. 3, а). Ока-
может не коррелировать с их аффинностью к
залось, что ацетилирование десмикозина хотя
«пустой» рибосоме [9, 10], следующим этапом
и снижает эффективность связывания с РТ,
была оценка эффективности ингибирования
однако этот эффект выражен слабо - значе-
полученными соединениями 1-3 трансляции
ние KD для триацетилдесмикозина, соедине-
in vitro. Измерение ингибирующей активности
ние 6 (3,9 ± 0,6 нМ) мало отличается от такового
проводили в бесклеточной системе трансля-
для исходного антибиотика DES (2,7 ± 0,6 нМ).
ции мРНК люциферазы светлячка (Fluc). Все
В то же время аффинность к рибосоме про-
конъюгаты десмикозина с фрагментами онко-
изводных десмикозина, содержащих фраг-
цина ингибируют синтез белка (рис. 3, б). Зна-
менты онкоцина, существенно снизи-
чительный уровень подавления трансляции,
лась. Несмотря на то что значения KD для
близкий к исходному антибиотику DES, про-
соединений 1 (80 ± 20 нМ), 2 (170 ± 30 нМ)
демонстрировало соединение 1. Несмотря на
и 3 (310 ± 60 нМ) лежат в наномолярном диа-
то что среди полученных конъюгатов это сое-
Рис. 3. Аффинность к рибосоме и ингибирующая активность конъюгатов десмикозина с фрагментами онкоци-
на 1-3. а - Конкурентное связывание BODIPY-ERY и соединений 1-3 с 70S-рибосомами E. coli. Для каждой точ-
ки приведено среднее значение со стандартным отклонением. Эффективные константы диссоциации (KD) ком-
плексов исследуемых соединений с рибосомами представлены как средние значения с доверительным интервалом
(α = 0,05). б - Способность конъюгатов десмикозина с фрагментами онкоцина ингибировать бактериальную трансля-
цию in vitro. Приведены средние значения со стандартным отклонением
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
907
динение также имело наибольшую аффинность
тибиотиков к рибосоме и потере ими анти-
к РТ (рис. 3, а), корреляции между ингибирую-
бактериальной активности [90, 91]. Мутация
щей активностью и сродством к рибосоме для
A2058G - одна из наиболее частых в клиниче-
всего ряда производных не наблюдается.
ски выделенных резистентных к макролидам
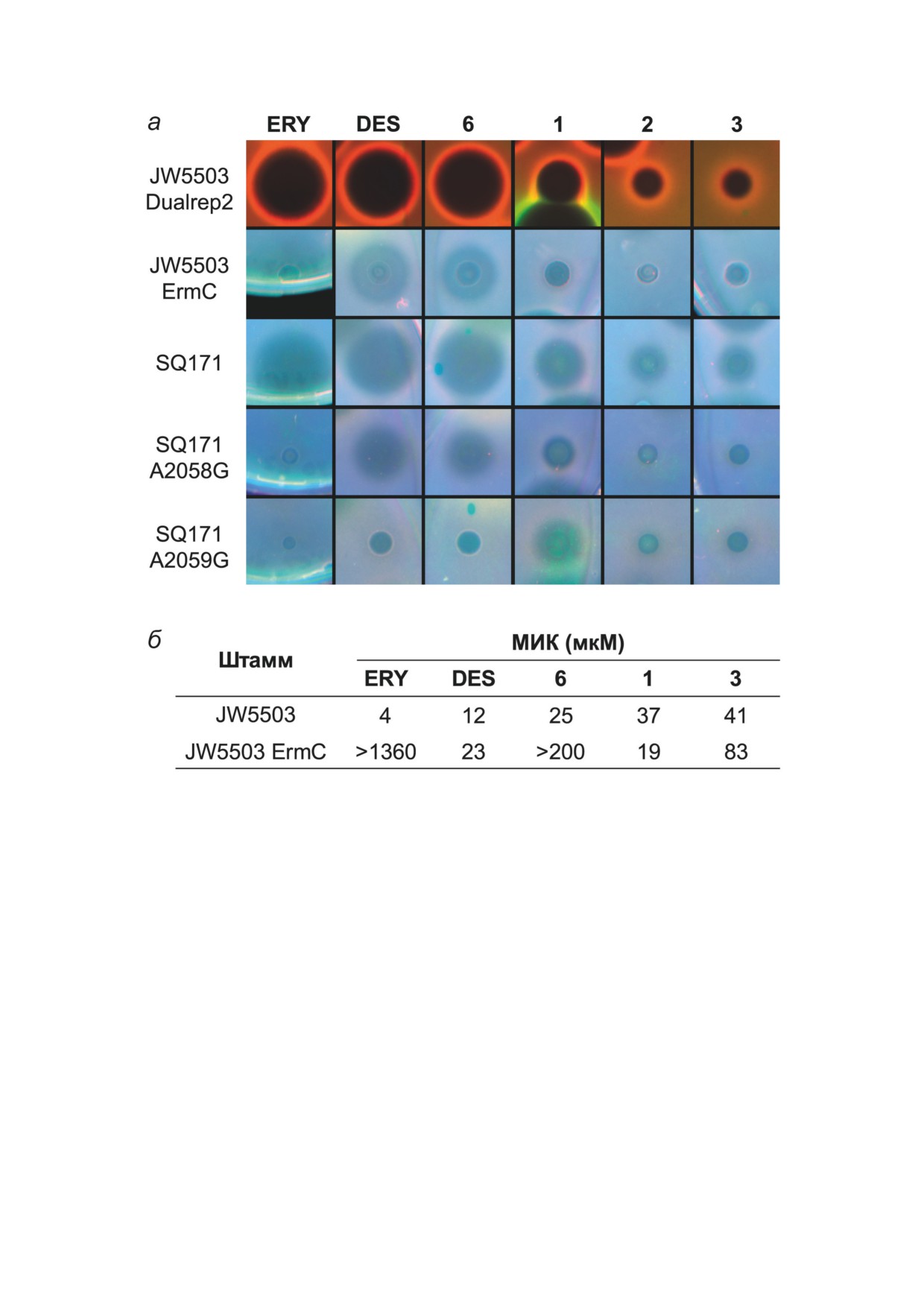
Антибактериальная активность. Способ-
штаммах. ERY практически не проявляет ан-
ность синтезированных конъюгатов 1-3 к по-
тибактериального действия по отношению к
давлению роста бактерий была проверена на
данным штаммам, в то время как активность
ряде штаммов E. coli, в том числе резистентных
DES и его ацетилированного аналога 6 значи-
к макролидам (рис. 4 и рис. S3 в Приложении).
тельно снижена, особенно в случае мутации
Штамм E. coli JW5503 ΔtolC pDualrep2 со-
A2059G (рис. 4, а и табл. S1 в Приложении). В то
держит двойную репортёрную систему с плаз-
же время производное 1 оказалось активным
мидой pDualrep2. Действие соединений, вызы-
как в отношении штамма с заменой A2058G,
вающих остановку рибосомы, приводит в такой
так и в случае мутации A2059G (рис. 4, а). Этот
системе к экспрессии красного флуоресцент-
факт может свидетельствовать об ином, уни-
ного белка Katushka2S, что позволяет быстро
кальном по сравнению с исходным антибио-
и точно определить механизм действия веще-
тиком, паттерне взаимодействий полученной
ства [54]. Результаты эксперимента (рис. 4, а)
молекулы с элементами РТ.
показали, что соединения в разной степени
Молекулярная динамика. Методами моле-
проявляют антимикробную активность, причём
кулярной динамики были исследованы ком-
их действие на бактерии связано с подавлением
плексы конъюгатов десмикозина с N-концевы-
бактериальной трансляции, о чём свидетель-
ми пептидными фрагментами онкоцина (1 и 3)
ствует появление красного окрашивания вокруг
с рибосомами E. coli дикого типа, а также содер-
зон ингибирования штамма JW5503 pDualrep2,
жащими мутацию A2059G в сравнении с ана-
содержащего репортёрную систему. Актив-
логичными комплексами DES и ONC (табли-
ность триацетилированного производного 6 в
ца и рис. 5). Производные десмикозина были
отношении этого штамма близка к исходному
ковалентно связаны с экзоциклической ами-
антибиотику DES, а антибактериальное дей-
ногруппой основания A2062 через карбинола-
ствие конъюгатов 1-3 несколько снижено.
минную группу (рис. S4 в Приложении), как это
Устойчивость штамма E. coli JW5503 ΔtolC
было показано для TYL [28]. Количество и про-
pKH80 к макролидам возникает за счёт транс-
тяжённость траекторий приведены в табл. S2
ляции метилтрансферазы ErmC, которая мети-
в Приложении. В результате было выявлено,
лирует А2058 23S рРНК бактерий, что приво-
что DES в комплексе с рибосомой E. coli, по-
дит к понижению эффективности связывания
добно другим 16-членным макролидам [79],
макролидов и рибосомы [89]. Тестирование
удерживается в сайте связывания прочными
конъюгатов 1-3 на этом резистентном штам-
водородными связями между гидроксильными
ме (рис. 4, а и б) показало, что все соедине-
группами мицинозы и нуклеотидными остат-
ния ингибируют его рост. Несмотря на то что
ками G748 и A751 и между 2′-гидроксильной
по действию на немодифицированный штамм
группой микаминозы и остатком A2058. При
производное 1 уступает DES и ERY, на резис-
введении мутации A2059G DES почти полно-
тентном штамме, содержащем метилтрансфе-
стью теряет водородные связи с мицинозой,
разу ErmC, оно проявляет активность, в от-
а встречаемость водородной связи 2′-гидрок-
личие от полностью неактивного ERY, и эта
сильной группы микаминозы с A2058 умень-
активность оказывается более значительной,
шается более чем вдвое. При этом водородная
чем в случае DES (рис. 4, б). Несколько мень-
связь, образованная протонированной 3′-ами-
шая активность отмечена для гексапептидного
ногруппой, переключается с
4′-гидроксиль-
производного 3. Следует отметить, что триаце-
ной группы остатка G2505 на N7 его гетеро-
тилированный аналог десмикозина 6 проявил
циклического основания. Причиной служит
слабое антибактериальное действие в отноше-
возникающее стерическое противоречие меж-
нии ErmC-устойчивого штамма (рис. 4, б).
ду остатком микаминозы и экзоциклической
Устойчивость штаммов E. coli SQ171 ΔtolC
аминогруппой G2059, вследствие которого
A2058G и E. coli SQ171 ΔtolC A2059G к макро-
DES поворачивается в сайте связывания, раз-
лидам обеспечена за счёт соответствующих
рывая водородные связи с мицинозой (рис. S5
замен нуклеотидов в консервативном участке
в Приложении и таблица).
23S рРНК. A2058 и A2059 являются ключевыми
При введении в структуру конъюгатов аце-
нуклеотидами во взаимодействии макролидов
тильных групп по 4′′-гидроксильной группе
с рибосомой, замена которых на G приводит
остатка мицинозы, 2′-гидроксильной группе
к значительному снижению аффинности ан-
остатка микаминозы и 3-гидроксильной груп-
БИОХИМИЯ том 87 вып. 7 2022
5*
908
ХАЙРУЛЛИНА и др.
Рис. 4. Тестирование конъюгатов десмикозина с фрагментами онкоцина на антибиотическую активность. а - Зоны
ингибирования исследуемыми соединениями роста различных штаммов бактерий E. coli. JW5503 pDualrep2 - штамм
E. coli JW5503 ΔtolC, модифицированный репортёрной системой pDualrep2, красное свечение вокруг зон ингибирования
свидетельствует об аресте трансляции; JW5503 ErmC - штамм E. coli JW5503 ΔtolC, трансформированный pKH80 (резис-
тентный к эритромицину за счёт экспрессии метилтрансферазы ErmC); SQ171 - штамм E. coli SQ171 ΔtolC, трансформи-
рованный плазмидой pAM552; SQ171 A2058G - штамм E. coli SQ171 ΔtolC, трансформированный плазмидой pAM552 с
заменой A2058G в 23S рРНК; SQ171 A2059G - штамм E. coli SQ171 ΔtolC, трансформированный плазмидой pAM552 с
заменой A2059G в 23S рРНК. DES, а также соединения 1-3 и 6 наносили в количестве 10 нмоль, ERY - в количестве
6,8 нмоль. Полная версия отсканированных чашек представлена на рис. S3 в Приложении. б - Значения МИК, полу-
ченные для штаммов JW5503 (E. coli JW5503 ΔtolC KanR) и JW5503 ErmC (E. coli JW5503 ΔtolC pKH80, резистентный к
эритромицину за счёт экспрессии метилтрансферазы ErmC)
пе тилонолида в комплексах конъюгатов 1 и 3 с
лентной связи с основанием A2062, наблюда-
рибосомой E. coli возникают стерические про-
емой для TYL [28] (рис. 5, а-г).
тиворечия между ацетильными группами и
Для конъюгатов 1 и 3, ацетилированных
остатками 23S рРНК при связывании десми-
по 3-, 2′- и 4′′-гидроксильным группам, не со-
козиновой части конъюгатов в MBS, соответ-
храняются водородные связи DES, в образо-
ствующем рентгеноструктурным данным [28].
вании которых участвовали соответствующие
Поэтому мы методами молекулярно-динами-
гидроксильные группы (таблица и табл. S3-S5
ческого моделирования нашли такие конфор-
в Приложении). Кроме того, как указывалось
мации конъюгатов (1 и 3), которые обеспечи-
выше, введённые ацетильные группы созда-
вали бы их стабильное взаимодействие с MBS
ют стерические противоречия, приводящие
при условии возможности образования кова-
к изменению положения десмикозинового
БИОХИМИЯ том 87 вып. 7 2022
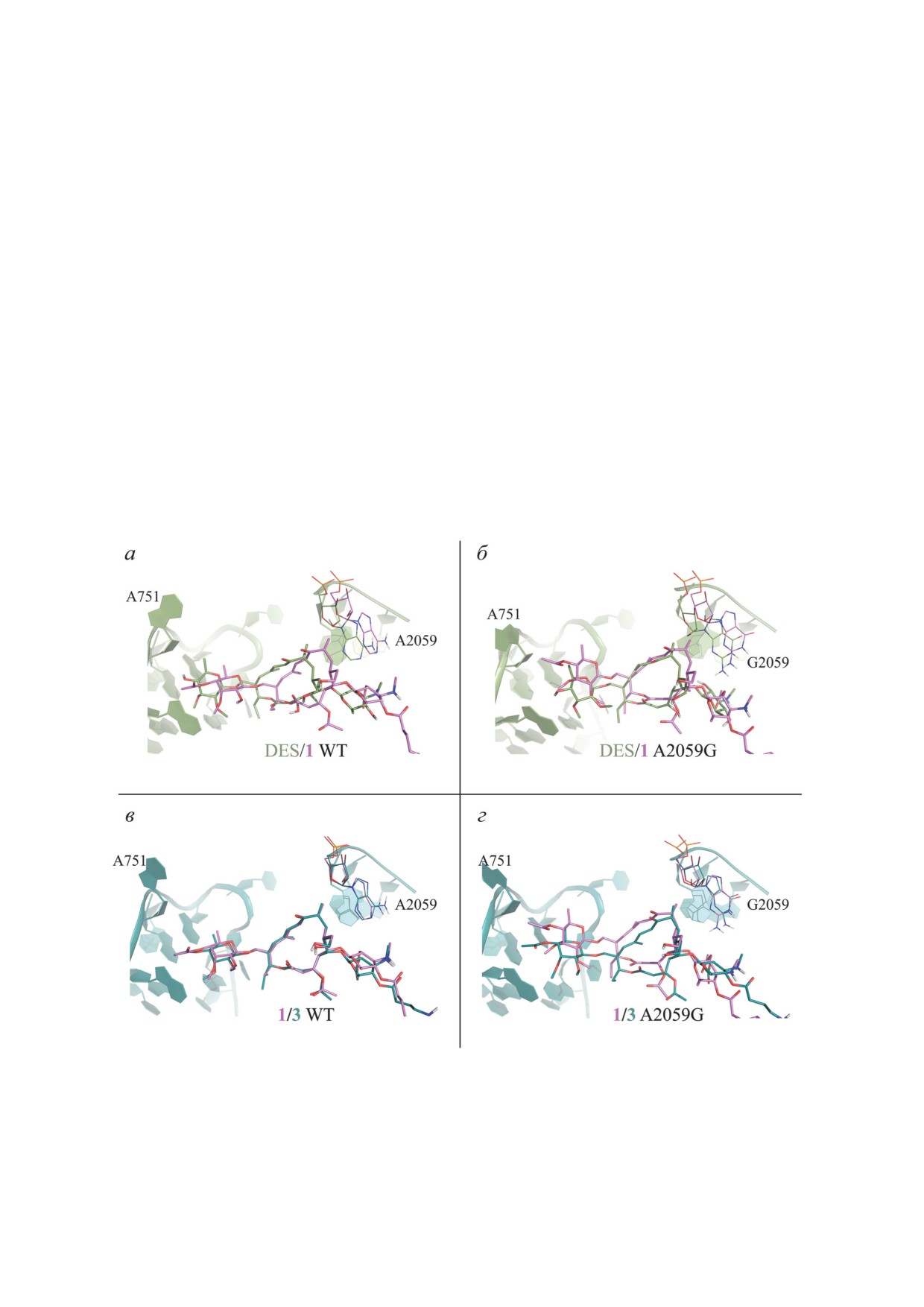
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
909
Встречаемость водородных связей, образованных DES, ONC и их фрагментами в составе конъюгатов десмикози-
на с N-концевыми пептидами онкоцина в комплексах с рибосомами E. coli дикого типа (WT) и содержащей мута-
цию A2059G
DES
DES
ONC
ONC
1
1
3
3
Донор
Акцептор
WT
A2059G
WT
A2059G
WT
A2059G
WT
A2059G
Фрагмент десмикозина
Мициноза/O4′′-H
A751/O2′
74
2
-
-
-
-
-
-
Мициноза/O4′′-H
G748/O6
88
-
-
-
-
-
-
-
Микаминоза/N3′-H
G2505/N7
-
60
-
-
-
-
-
-
Микаминоза/N3′-H
G2505/O4′
73
2
-
-
-
1
-
3
Микаминоза/O2′-H
A2058/N1
94
35
-
-
-
-
-
-
A752/N6-H
Мициноза/O9′′
-
-
-
-
-
18
-
35
G748/N1-H
Мициноза/O3′′
96
4
-
-
1
4
-
-
G748/N1-H
Мициноза/O4′′
-
-
-
-
52
7
23
9
Фрагмент онкоцина
Val1/N-H
C2507/O2′
-
-
-
2
-
-
82
53
Lys3/Nε-H
C2452/O2′
-
-
-
28
3
3
20
10
Lys3/Nε-H
C2507/Oфосфат
-
-
-
-
95
96
89
89
Lys3/Nε-H
C2573/Oфосфат
-
-
-
-
85
95
98
97
Tyr6/N-H
U2506/O2′
-
-
82
-
-
-
-
-
Arg9/Nδ-H
C2610/Oфосфат
-
-
9
28
-
-
-
-
Arg9/Nω-H
C2610/O1фосфат
-
-
7
32
-
-
-
-
Arg9/Nω-H
C2610/O2фосфат
-
-
14
50
-
-
-
-
Примечание. Таблица составлена по данным молекулярно-динамических расчётов. Встречаемость состояний дана в %
от кадров траекторий. Нумерация атомов представлена в соответствии с рис. S4 в Приложении.
фрагмента конъюгата в комплексе с рибосо-
ного пептида ONC (таблица и рис. S6 в При-
мой по сравнению с исходным десмикозином.
ложении).
Тем не менее в смоделированных структу-
Введение мутации A2059G приводит к
рах комплексов конъюгатов 1 и 3 с рибосо-
некоторому ослаблению водородных связей
мой (рис. 5, в) имеется водородная связь меж-
онкоциновой части конъюгатов 1 и 3 и суще-
ду N1-H основания G748 и сложноэфирным
ственному ослаблению водородных связей аце-
кислородом 4′′-ацетокси-группы остатка ми-
тилированной мицинозы с основанием G748
цинозы, эта связь более стабильна в комплек-
(табл. S1 и S6 в Приложении и рис. 5, б и г). При
сах, образованных конъюгатом 1 (таблица).
этом конформация конъюгатов 1 и 3 меняется
Разрушение водородных связей, типичных
относительно дикого типа слабо, поскольку их
для 16-членных макролидов, и одновремен-
связывание в целом зависит в первую очередь
ная стабилизация взаимодействий онкоцино-
от связывания фрагментов онкоцина и во вто-
вой части конъюгатов с 23S рРНК приводят к
рую очередь - от связывания ацетилированного
смещению десмикозиновой части конъюга-
остатка мицинозы. Ацетилированный остаток
тов в сторону ПТЦ (рис. 5, а). В этой новой
микаминозы обеспечивает лишь неспецифи-
конформации соединений 1 и 3 их пептидные
ческое кулоновское притягивание к 23S рРНК,
фрагменты образуют довольно стабильные во-
которое оказывается нечувствительным к виду
дородные связи, но не характерные для исход-
пуринового основания нуклеотида 2059.
БИОХИМИЯ том 87 вып. 7 2022
910
ХАЙРУЛЛИНА и др.
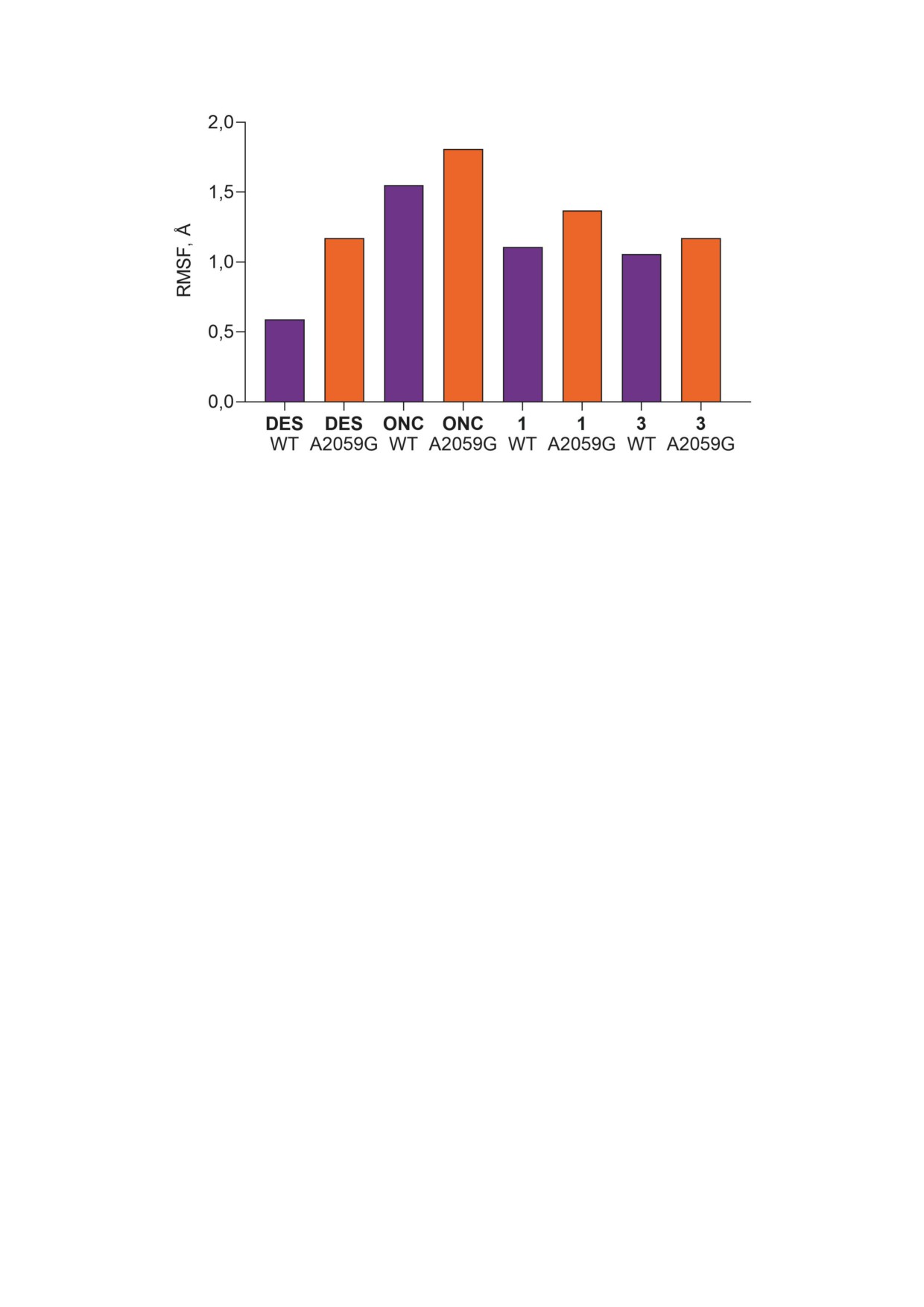
Проведённый анализ значений RMSF
конъюгатов 1 и 3 при переходе к резистентному
для DES, ONC, а также конъюгатов 1 и 3 по-
штамму (рис. 4, а).
казал, что замена остатка A2059 на G в MBS
рибосомы приводит к росту значений RMSF
для всех исследуемых соединений (рис. 6). Наи-
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
более выраженный эффект наблюдается для
DES - подвижность его атомов увеличивается
В работе осуществлён дизайн и синтез серии
в 2 раза при связывании с мутантной рибосо-
конъюгатов N-концевых фрагментов онкоцина
мой, что согласуется с его низкой антибакте-
(Val1-Tyr6, Asp2-Tyr6, Lys4-Tyr6) и 3,2′,4′′-три-
риальной активностью на соответствующем
ацетилдесмикозина, в которых пептидные фраг-
резистентном штамме (рис. 4, а). Введение в
менты присоединены по 4′-положению мици-
структуру DES фрагментов онкоцина ожидаемо
нозы через линкер на основе 4-аминобутановой
приводит к росту RMSF за счёт большей под-
кислоты (1-3). Конъюгаты представляли инте-
вижности пептидной части молекулы. Влияние
рес как потенциальные антимикробные аген-
мутации A2059G на флуктуации атомов ONC, а
ты, а также как лиганды, взаимодействующие
также соединений 1 и 3 заметно слабее. По-ви-
с функционально важными центрами бактери-
димому, компенсация потерянных водородных
альной рибосомы - MBS, с которым связыва-
связей тилонолида за счёт остатков онкоцина
ется десмикозин, и ПТЦ, с которым взаимодей-
приводит к стабилизации конформации анти-
ствуют N-концевые фрагменты онкоцина.
биотиков в мутированном MBS, что проявляет-
В синтезированных аналогах десмикозина
ся в сохранении антибактериальной активности по 3, 2′ и 4′′-гидроксилам макролида содержат-
Рис. 5. Структуры соединений 1, 3 и DES в комплексе с рибосомами E. coli дикого типа (WT) и содержащей мута-
цию A2059G, полученные методом молекулярной динамики. Изображены только десмикозиновые части молекул.
Альдегидная группа в положении 20 тилонолида показана в форме карбиноламина, образованного с участием экзо-
циклической аминогруппы основания A2062 (само основание А2062 на рисунке не показано). а - Наложение струк-
туры DES (зелёный) и соединения 1 (фиолетовый) в комплексе с рибосомой дикого типа. б - Наложение структуры
DES (зелёный) и соединения 1 (фиолетовый) в комплексе с рибосомой, содержащей мутацию A2059G. в - Наложение
структуры соединения 1 (фиолетовый) и 3 (голубой) в комплексе с рибосомой дикого типа. г - Наложение структуры
соединения 1 (фиолетовый) и 3 (голубой) в комплексе с рибосомой, содержащей мутацию A2059G
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
911
Рис. 6. Среднеквадратичные флуктуации атомов (RMSF) для структур конъюгатов десмикозина с N-концевыми фраг-
ментами онкоцина 1 и 3 в сравнении с DES и ONC в рибосоме E. coli дикого типа (WT, фиолетовый цвет) и E. coli, со-
держащей мутацию A2059G (оранжевый цвет) по данным молекулярной динамики
ся ацетильные группы (рис. 2), которые были
способными связываться с бактериальными
введены по следующим соображениям. Ацети-
рибосомами, однако степень их аффинно-
лирование антибиотиков обычно упоминается
сти снижена по сравнению с исходным анти-
в литературе в контексте возникновения устой-
биотиком десмикозином и 3,2′,4′′-триацетил-
чивости у бактерий по отношению к ним [92,
десмикозином (рис. 3, а). Тем не менее все
93], однако в случае макролидов существуют
конъюгаты подавляют бактериальную транс-
природные антибиотики, ацетилированные
ляцию in vitro (рис. 3, б), причём конъюгат с
или ацилированные по гидроксильным груп-
тетрапептидным заместителем (1) по уровню
пам лактона и углеводных остатков (напри-
ингибирования подобен DES. Эти результаты
мер, 14-членный макролид ланкамицин [94,
согласуются с данными тестирования в двойной
95], 16-членный макролид карбомицин А [28,
репортёрной системе, где наибольшим ингиби-
96] или триацетилолеандомицин [97, 98]). Вве-
рующим действием среди полученных конъю-
дение ацильных и ацетильных заместителей в
гатов также обладает соединение 1 (рис. 4, а).
структуру макролидов улучшает фармакоки-
Конъюгаты 1 и 3 проявили активность в
нетические свойства антибиотиков [99, 100], а
отношении устойчивых к макролидам штам-
в некоторых случаях приводит к активным ан-
мов (рис. 4), один из которых содержит ген ме-
тибактериальным соединениям, действующим
тилазы ErmC, метилирующей А2058 [56], а два
в отношении резистентных штаммов [27, 101,
других содержат замены в 23S рРНК A2058G
102]. Кроме того, при конструировании конъю-
либо А2059G [103, 104]. Поскольку ацетили-
гатов десмикозина с фрагментами онкоцина мы
рованное производное десмикозина
(6) не
полагали, что введение ацетильных групп по 3,
проявляет значительной активности на резис-
2′ и 4′′-гидроксилам десмикозинового фрагмен-
тентных штаммах (рис. 4 и табл. S1 в Прило-
та должно ослабить взаимодействия макролид-
жении), по-видимому, такое действие конъю-
ной части молекул с РТ вследствие нарушения
гатов 1 и 3 определяется наличием пептидных
водородных связей, образованных с участием
фрагментов в структуре этих соединений.
этих гидроксилов [28, 98], и привести к боль-
Для выяснения роли различных структур-
шей подвижности десмикозинового фрагмента
ных элементов в молекулах конъюгатов десми-
в комплексе конъюгата с рибосомой. Ослаб-
козина с N-концевыми фрагментами онкоцина
ление взаимодействий десмикозиновой части
в их взаимодействии с бактериальными рибосо-
конъюгата с нуклеотидами РТ может способ-
мами были выполнены расчёты молекулярной
ствовать тому, что влияние пептидных фраг-
динамики комплексов десмикозина, онкоцина
ментов онкоцина на связывание всей молекулы
и конъюгатов 1 и 3 с рибосомой E. coli дикого
в целом проявится более отчётливо.
типа, а также с содержащей мутацию A2059G в
Сконструированные и синтезированные
23S рРНК, приводящую к устойчивости соот-
в данной работе конъюгаты 1-3 оказались
ветствующего штамма к макролидам.
БИОХИМИЯ том 87 вып. 7 2022
912
ХАЙРУЛЛИНА и др.
По-видимому, как и для других 16-член-
проявляет наибольшую активность в отноше-
ных макролидов, для взаимодействия с ри-
нии резистентного штамма, содержащего му-
босомой конъюгатов десмикозина с фраг-
тацию A2059G в 23S РНК (рис. 4, а).
ментами онкоцина (1, 3) большое значение
Таким образом, в настоящей работе по-
имеет возможность расположения тилоно-
казано, что модификация десмикозина фраг-
лида в комплексе с рибосомой оптимальным
ментами антимикробного пептида онкоцина
образом для образования ковалентной связи
приводит к соединениям, способным связы-
с н.о. A2062 23S рРНК. При взаимодействии
ваться с бактериальными рибосомами подобно
конъюгатов 1 и 3 с рибосомой E. coli интен-
десмикозину и онкоцину и подавлять транс-
сивные взаимодействия онкоциновых фраг-
ляцию in vitro. Конъюгаты гекса- и тетра-
ментов и наличие ацетильных группировок в
пептидных N-концевых фрагментов онкоцина
десмикозиновой части соединений способ-
с
3,2′,4′′-триацетилдесмикозином оказались
ствуют смещению тилонолида от оптимально-
активными в отношении штаммов, резистент-
го расположения в MBS (рис. 5, а). Возможно
ных к макролидам. Методом молекулярной
также, что длина линкера на основе 4-ами-
динамики найдены структурные особенности
нобутановой кислоты, соединяющего макро-
взаимодействий этих производных с бактери-
лидный и пептидные фрагменты конъюгатов,
альными рибосомами, в том числе содержащи-
недостаточна, чтобы эти фрагменты могли в
ми мутацию A2059G в 23S РНК, позволяющие
полной мере, синергически, взаимодейство-
объяснить полученные экспериментальные ре-
вать со своими участками РТ, не перетягивая
зультаты.
друг друга. Вероятно, более длинный лин-
кер, например, на основе δ-аминовалериано-
вой или ε-аминокапроновой кислот, мог бы
Вклад авторов. З.З. Хайруллина - проведе-
обеспечить синергическое действие онкоци-
ние экспериментов (синтез, связывание с ри-
новой и десмикозиновой частей конъюгата.
босомами), обсуждение результатов исследова-
Изменение расположения тилонолида в ком-
ния, написание текста, редактирование текста;
плексах конъюгатов 1 и 3 с рибосомой под-
Г.И. Макаров - моделирование молекулярной
тверждается результатами расчёта среднеква-
динамики, обсуждение результатов исследо-
дратичных флуктуаций атомов исследуемых
вания, написание и редактирование текста;
соединений (RMSF), значения которых для
А.Г. Терещенков - проведение экспериментов
соединений 1 и 3 выше, чем для исходного
(связывание с рибосомами), обсуждение ре-
антибиотика DES (рис. 6). Этим может объяс-
зультатов, написание и редактирование текста,
няться как уменьшение аффинности конъю-
оформление рисунков; В.С. Буев - проведение
гатов 1 и 3 к рибосоме, так и некоторое сни-
экспериментов (in vitro трансляция, антибакте-
жение их антибактериальной активности по
риальная активность); Д.А. Лукьянов - про-
сравнению с DES на рибосомах дикого типа
ведение экспериментов (in vitro транс-
(рис. 3, а и рис. 4).
ляция,
антибактериальная
активность);
В случае рибосомы E. coli, содержащей му-
В.И. Польшаков
- проведение экспериментов
тацию A2059G, происходит увеличение под-
(ЯМР), анализ данных; В.Н. Ташлицкий - про-
вижности DES и соединений 1 и 3 в MBS по
ведение экспериментов (хроматография,
сравнению с рибосомой дикого типа (рис. 5
масс-спектрометрия); И.А. Остерман - ру-
и 6), что свидетельствует о дестабилизации
ководство работой, обсуждение результатов
взаимодействий десмикозинового фрагмента
исследования; Н.В. Сумбатян - концепция и
с элементами РТ. Однако, если для DES та-
руководство работой, обсуждение результатов,
кое изменение значительно и проявляется в
написание и редактирование текста.
снижении его антибактериальной активности
Финансирование. Работа выполнена при
по отношению к резистентному штамму, то
финансовой поддержке Российского фон-
флуктуация производных 1 и 3 увеличивается
да фундаментальных исследований (проекты
слабо, по-видимому, вследствие значительно-
№ 20-04-00873-a (синтез аналогов, молеку-
го вклада онкоцинового фрагмента во взаимо-
лярное моделирование, связывание с рибосо-
действие с рибосомой. Соединение 1 оказалось
мами), № 19-14-00115 (ЯМР исследования) и
наиболее устойчивым к мутации в MBS: не-
№ 21-64-00006 (in vitro трансляция, антибакте-
смотря на ослабление взаимодействия десми-
риальная активность)).
козиновой части конъюгата из-за влияния
Благодарности. Мы благодарим А.А. Бог-
мутации, онкоциновый фрагмент «удержива-
данова за инициирование и поддержку работ
ет» десмикозиновую часть от покидания MBS.
по изучению пептидных производных рибосо-
Это согласуется с тем, что данное производное
мных антибиотиков. Авторы также благодарят
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
913
А.Л. Коневегу за предоставленные для работы
М.В. Ломоносова за предоставленную возмож-
рибосомы; М.В. Серебрякову за масс-спек-
ность вести расчёты молекулярной динамики
трометрические анализы; А.Л. Ксенофонто-
на суперкомпьютере «Ломоносов-II».
ва за проведение аминокислотного анализа;
Конфликт интересов. Авторы заявляют об
О.Ю. Савельева за экспертную техническую
отсутствии конфликта интересов.
помощь в измерениях ЯМР. Работа выполне-
Соблюдение этических норм. Настоящая
на с использованием оборудования, приоб-
статья не содержит описания каких-либо ис-
ретённого за счёт средств Программы развития
следований с участием людей или животных в
Московского университета. Авторы выражают
качестве объектов.
благодарность МГУ имени М.В. Ломоносова
Дополнительные материалы. Приложение
за возможность использовать установки ЯМР,
к статье опубликовано на сайте журнала
а также Вычислительному центру МГУ имени
СПИСОК ЛИТЕРАТУРЫ
1.
König, G., Sokkar, P., Pryk, N., Heinrich, S.,
12.
Bogdanov, A. A., Sumbatyan, N. V., Shishkina, A.
Möller, D., et al. (2021) Rational prioritization strat-
V., Karpenko, V. V., and Korshunova, G. A. (2010)
egy allows the design of macrolide derivatives that
Ribosomal tunnel and translation regulation, Bio-
overcome antibiotic resistance, Proc. Natl. Acad. Sci.
chemistry (Moscow),
75,
1501-1516, doi: 10.1134/
USA, 118, e2113632118, doi: 10.1073/pnas.2113632118.
s0006297910130018.
2.
Llano-Sotelo, B., Dunkle, J., Klepacki, D., Zhang,
13.
Svetlov, M. S., Syroegin, E. A., Aleksandrova,
W., Fernandes, P., et al. (2010) Binding and action of
E. V., Atkinson, G. C., Gregory, S. T., et al. (2021)
CEM-101, a new fluoroketolide antibiotic that inhibits
Structure of Erm-modified 70S ribosome reveals
protein synthesis, Antimicrob. Agents Chemother., 54,
the mechanism of macrolide resistance, Nat. Chem.
4961-4970, doi: 10.1128/AAC.00860-10.
Biol., 17, 412-420, doi: 10.1038/s41589-020-00715-0.
3.
Jelić, D., and Antolović, R.
(2016) From
14.
Breiner-Goldstein, E., Eyal, Z., Matzov, D.,
erythromycin to azithromycin and new potential
Halfon, Y., Cimicata, G., et al. (2021) Ribosome-
ribosome-binding antimicrobials, Antibiotics (Basel,
binding and anti-microbial studies of the
Switzerland), 5, 29, doi: 10.3390/antibiotics5030029.
mycinamicins, 16-membered macrolide antibiotics
4.
Skripkin, E., McConnell, T. S., DeVito, J.,
from Micromonospora griseorubida, Nucleic Acids
Lawrence, L., Ippolito, J. A., et al. (2008) R chi-
Res., 49, 9560-9573, doi: 10.1093/nar/gkab684.
01, a new family of oxazolidinones that overcome
15.
Mcguire, J. M., Boniece, W. S., Higgens, C.
ribosome-based linezolid resistance, Antimicrob.
E., Hoehn, M. M., Stark, W. M., et al.
(1961)
Agents Chemother.,
52,
3550-3557, doi: 10.1128/
Tylosin, a new antibiotic: I. Microbiological studies,
AAC.01193-07.
Antibiot. Chemother., 11, 320-327.
5.
Lenz, K. D., Klosterman, K. E., Mukundan, H., and
16.
Baltz, R. H., Seno, E. T., Stonesifer, J., and Wild, G. M.
Kubicek-Sutherland, J. Z. (2021) Macrolides: from
(1983) Biosynthesis of the macrolide antibiotic tylosin.
toxins to therapeutics, Toxins, 13, 347, doi: 10.3390/
A preferred pathway from tylactone to tylosin, J. Antibiot.,
toxins13050347.
36, 131-141, doi: 10.7164/antibiotics.36.131.
6.
Fernandes, P., Martens, E., and Pereira, D. (2017)
17.
Hamill, R. L., Haney, M. E., and McGuire, J. M.
Nature nurtures the design of new semi-synthetic
(1965) Stamper MC: Antibiotics tylosin and des-
macrolide antibiotics, J. Antibiot.,
70,
527-533,
mycosin and derivatives thereof., Apr.13, U.S. Pat.
doi: 10.1038/ja.2016.137.
№ 3178341.
7.
Dinos, G. P.
(2017) The macrolide antibiotic
18.
Arsic, B., Barber, J., Čikoš, A., Mladenovic, M.,
renaissance, Br. J. Pharmacol., 174, 2967-2983, doi:
Stankovic, N., et al. (2018) 16-membered macrolide
10.1111/bph.13936.
antibiotics: a review, Int. J. Antimicrob. Agents, 51,
8.
Vázquez-Laslop, N., and Mankin, A. S. (2018) How
283-298, doi: 10.1016/j.ij antimicag.2017.05.020.
macrolide antibiotics work, Trends Biochem. Sci., 43,
19.
McColm, A. A., and McHardy, N. (1984) Evaluation of
668-684, doi: 10.1016/j.tibs.2018.06.011.
a range of antimicrobial agents against the parasitic proto-
9.
Mankin, A. S. (2008) Macrolide myths, Curr. Opin.
zoa, Plasmodium falciparum, Babesia rodhaini and Theile-
Microbiol., 11, 414-421, doi: 10.1016/j.mib.2008.08.003.
ria parva in vitro, Ann. Trop. Med. Parasitol., 78, 345-354,
10.
Kannan, K., and Mankin, A. S. (2011) Macrolide
doi: 10.1080/00034983.1984.11811831.
antibiotics in the ribosome exit tunnel: species-
20.
Goodman, C. D., Useglio, M., Peirú, S., Labadie, G.
specific binding and action, Ann. N.Y. Acad. Sci.,
R., McFadden, G. I., et al. (2013) Chemobiosynthesis
1241, 33-47, doi: 10.1111/j.1749-6632.2011.06315.x.
of new antimalarial macrolides. Antimicrob. Agents
11.
Wilson, D. N. (2014) Ribosome-targeting antibiotics
Chemother., 57, 907-913, doi: 10.1128/AAC.01825-12.
and mechanisms of bacterial resistance, Nat. Rev.
21.
Dahl, E. L., and Rosenthal, P. J. (2007) Multiple an-
Microbiol., 12, 35-48, doi: 10.1038/nrmicro3155.
tibiotics exert delayed effects against the Plasmodium
БИОХИМИЯ том 87 вып. 7 2022
914
ХАЙРУЛЛИНА и др.
falciparum apicoplast, Antimicrob. Agents Chemother.,
of 16-membered 9-O-arylalkyloxime macrolides, Bio-
51, 3485-3490, doi: 10.1128/AAC.00527-07.
org. Med. Chem. Lett., 16, 1259-1266, doi: 10.1016/
22.
Von Geldern, T. W., Morton, H. E., Clark, R. F.,
j.bmcl.2005.11.061.
Brown, B. S., Johnston, K. L., et al. (2019) Discovery
35.
Karahalios, P., Kalpaxis, D. L., Fu, H., Katz, L., Wil-
of ABBV-4083, a novel analog of tylosin A that has
son, D. N., et al. (2006) On the mechanism of action
potent anti-Wolbachia and anti-filarial activity, PLoS
of 9-O-arylalkyloxime derivatives of 6-O-mycami-
Negl. Trop. Dis., 13, e0007159, doi: 10.1371/journal.
nosyltylonolide, a new class of 16-membered mac-
pntd.0007159.
rolide antibiotics, Mol. Pharmacol., 70, 1271-1280,
23.
Debono, M., Willard, K. E., Kirst, H. A., Wind, J. A.,
doi: 10.1124/mol.106.026567.
Crouse, G. D., et al. (1989) Synthesis and antimicro-
36.
Sumbatyan, N. V., Korshunova, G. A., and
bial evaluation of 20-deoxo-20-(3,5-dimethylpiperi-
Bogdanov, A. A. (2003) Peptide derivatives of an-
din-1-yl)desmycosin (tilmicosin, EL-870) and related
tibiotics tylosin and desmycosin, protein synthesis
cyclic amino derivatives, J. Antibiot., 42, 1253-1267,
inhibitors, Biochemistry (Moscow),
68,
1156-1158,
doi: 10.7164/antibiotics.42.1253.
doi: 10.1023/a:1026318914546.
24.
Miyake, T., Takita, M., Hamada, M., Takeuchi, T.,
37.
Starosta, A. L., Karpenko, V. V., Shishkina,
and Umezawa, S. (2001) Macrolide antibiotics and
A. V., Mikolajka, A., Sumbatyan, N. V., et al.
treatment of pasteurellosis, Patent WO2001016148A1.
(2010) Interplay between the ribosomal tunnel,
25.
Kleefeld, G., Froyman, R., Ludwig, C., Omura, S.,
nascent chain, and macrolides influences drug
Sunazuka, T., Tomoyasu, H., Akihiro, S., Kazuro, S.
inhibition, Chem. Biol., 17, 504-514, doi: 10.1016/
(2014) WO2014/187957 Tylosin derivatives and meth-
j.chembiol.2010.04.008.
od for preparation thereof.
38.
Shishkina, A., Makarov, G., Tereshchenkov, A.,
26.
Phan, L. T., Jian, T., Chen, Z., Qiu, Y. L., Wang, Z.,
Korshunova, G., Sumbatyan, N., et al.
(2013)
et al. (2004) Synthesis and antibacterial activity of
Conjugates of amino acids and peptides with
a novel class of 4’-substituted 16-membered ring
5-O-mycaminosyltylonolide and their interaction with
macrolides derived from tylosin, J. Med. Chem., 47,
the ribosomal exit tunnel, Bioconjugate Chem., 24,
2965-2968, doi: 10.1021/jm034233n.
1861-1869, doi: 10.1021/bc400236n.
27.
Kiyoshima, K., Sakamoto, M., Nomura, H.,
39.
Schneider, M., and Dorn, A. (2001) Differential
Yoshioka, T., Okamoto, R., et al. (1989) Structure-
infectivity of two Pseudomonas species and the
activity relationship studies on
4′′-O-acyltylosin
immune response in the milkweed bug, Oncopeltus
derivatives: significance of their 23-O-mycinosyl and
fasciatus (Insecta: Hemiptera), J. Invertebr. Pathol., 78,
4′′-O-acyl moieties in antimicrobial activity against
135-140, doi: 10.1006/jipa.2001.5054.
macrolide-resistant microbes, J. Antibiot., 42, 1661-
40.
Knappe, D., Piantavigna, S., Hansen, A.,
1672, doi: 10.7164/antibiotics.42.1661.
Mechler, A., Binas, A., et al.
(2010) Oncocin
28.
Hansen, J. L., Ippolito, J. A., Ban, N., Nissen, P.,
(VDKPPYLPRPRPPRRIYNR-NH2): a novel anti-
Moore, P. B., et al. (2002) The structures of four
bacterial peptide optimized against gram-negative
macrolide antibiotics bound to the large ribosom-
human pathogens, J. Med. Chem., 53, 5240-5247,
al subunit, Mol. Cell,
10,
117-128, doi: 10.1016/
doi: 10.1021/jm100378b.
s1097-2765(02)00570-1.
41.
Seefeldt, A. C., Nguyen, F., Antunes, S.,
29.
Ban, N., Nissen, P., Hansen, J., Moore, P. B., and
Pérébaskine, N., Graf, M., et al. (2015) The proline-
Steitz, T. A. (2000) The complete atomic structure of
rich antimicrobial peptide Onc112 inhibits translation
the large ribosomal subunit at 2.4 Å resolution, Sci-
by blocking and destabilizing the initiation complex,
ence, 289, 905-920, doi: 10.1126/science.289.5481.905.
Nat. Struct. Mol. Biol., 22, 470-475, doi:10.1038/
30.
Nissen, P., Hansen, J., Ban, N., Moore, P. B., and
nsmb.3034.
Steitz, T. A. (2000) The structural basis of ribosome
42.
Polikanov, Y. S., Aleksashin, N. A., Beckert, B., and
activity in peptide bond synthesis, Science, 289, 920-
Wilson, D. N. (2018) The mechanisms of action of
930, doi: 10.1126/science.289.5481.920.
ribosome-targeting peptide antibiotics, Front. Mol.
31.
Harms, J., Schluenzen, F., Zarivach, R., Bashan,
Bosci., 5, 48, doi: 10.3389/fmolb.2018.00048.
A., Gat, S., et al. (2001) High resolution structure
43.
Roy, R. N., Lomakin, I. B., Gagnon, M. G., and
of the large ribosomal subunit from a Mesophilic
Steitz, T. A. (2015) The mechanism of inhibition of
Eubacterium, Cell, 107, 679-688, doi: 10.1016/s0092-
protein synthesis by the proline-rich peptide oncocin,
8674(01)00546-3.
Nat. Struct. Mol. Biol., 22, 466-469, doi: 10.1038/
32.
Pavlova, A., Parks, J. M., Oyelere, A. K., and
nsmb.3031.
Gumbart, J. C. (2017) Toward the rational design of
44.
Florin, T., Maracci, C., Graf, M., Karki, P.,
macrolide antibiotics to combat resistance, Chem.
Klepacki, D., et al. (2017) An antimicrobial peptide
Biol. Drug Des., 90, 641-652, doi: 10.1111/cbdd.13004.
that inhibits translation by trapping release factors
33.
Sugawara, A., Maruyama, H., Shibusawa, S., Matsui,
on the ribosome, Nat. Struct. Mol. Biol., 24, 752-757,
H., Hirose, T., et al. (2017) 5-O-Mycaminosyltylo-
doi: 10.1038/nsmb.3439.
nolide antibacterial derivatives: design, synthesis and
45.
Li, J., Kim, I. H., Roche, E. D., Beeman, D., Lynch,
bioactivity, J. Antibiot.,
70,
878-887, doi: 10.1038/
A. S., et al. (2006) Design, synthesis, and biological
ja.2017.61.
evaluation of BODIPY®-erythromycin probes for
34.
Fu, H., Marquez, S., Gu, X., Katz, L., and Myles,
bacterial ribosomes, Bioorg. Med. Chem. Lett., 16,
D. C. (2006) Synthesis and in vitro antibiotic activity
794-797, doi: 10.1016/j.bmcl.2005.11.028.
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
915
46.
Delaglio, F., Grzesiek, S., Vuister, G. W., Zhu, G.,
Natl. Acad. Sci. USA, 107, 17152-17157, doi: 10.1073/
Pfeifer, J., et al. (1995) NMRPipe: a multidimensional
pnas.1007988107.
spectral processing system based on UNIX pipes, J. Bio-
59.
Cannone, J. J., Subramanian, S., Schnare, M.
mol. NMR, 6, 277-293, doi: 10.1007/BF00197809.
N., Collett, J. R., D’Souza, L. M., et al.
(2002)
47.
Lee, W., Tonelli, M., and Markley, J. L.
(2015)
The comparative RNA web (CRW) site: an
NMRFAM-SPARKY: enhanced software for
online database of comparative sequence and
biomolecular NMR spectroscopy, Bioinformatics, 31,
structure information for ribosomal, intron, and
1325-1327, doi: 10.1093/bioinformatics/btu830.
other RNAs, BMC Bioinform., 3, 2, doi: 10.1186/
48.
Hanwell, M. D., Curtis, D. E., Lonie, D. C.,
1471-2105-3-2.
Vandermeersch, T., Zurek, E., et al. (2012) Avogadro:
60.
Byrd, R., Lu, P., and Nocedal, J.
(1995) A
an advanced semantic chemical editor, visualization,
limited memory algorithm for bound constrained
and analysis platform, J. Cheminformatics,
4,
17,
optimization, SIAM J. Scientif. Statistic. Comput., 16,
doi: 10.1186/1758-2946-4-17.
1190-1208, doi: 10.1137/0916069.
49.
Yan, K., Hunt, E., Berge, J., May, E., Copeland,
61.
Petrone, P. M., Snow, C. D., Lucent, D., and Pande,
R. A., et al. (2005) Fluorescence polarization method
V. S. (2008) Side-chain recognition and gating in the
to characterize macrolide-ribosome interactions,
ribosome exit tunnel, Proc. Natl. Acad. Sci. USA, 105,
Antimicrob. Agents Chemother.,
49,
3367-3372,
16549-16554, doi: 10.1073/pnas.0801795105.
doi: 10.1128/AAC.49.8.3367-3372.2005.
62.
Lucent, D., Snow, C. D., Aitken, C. E., and Pande,
50.
Tereshchenkov, A. G., Dobosz-Bartoszek, M.,
V. S.
(2010) Non-bulk-like solvent behavior in
Osterman, I. A., Marks, J., Sergeeva, V. A., et al.
the ribosome exit tunnel, PLoS Comput. Biol., 6,
(2018) Binding and action of amino acid analogs of
e1000963, doi: 10.1371/journal.pcbi.1000963.
chloramphenicol upon the bacterial ribosome, J. Mol.
63.
Ruiz-Carmona, S., Alvarez-Garcia, D., Foloppe, N.,
Biol., 430, 842-852, doi: 10.1016/j.jmb.2018.01.016.
Garmendia-Doval, A. B., Juhos, S., et al.
(2014)
51.
Wang, Z. X. (1995) An exact mathematical expression
rDock: a fast, versatile and open source program for
for describing competitive binding of two different
docking ligands to proteins and nucleic acids, PLoS
ligands to a protein molecule, FEBS Lett., 360, 111-
Comput. Biol.,
10, e1003571, doi: 10.1371/journal.
114, doi: 10.1016/0014-5793(95)00062-e.
pcbi.1003571.
52.
Polikanov, Y. S., Osterman, I. A., Szal, T.,
64.
Van Der Spoel, D., Lindahl, E., Hess, B., Groenhof,
Tashlitsky, V. N., Serebryakova, M. V., et al. (2014)
G., Mark, A. E., et al. (2005) GROMACS: fast,
Amicoumacin a inhibits translation by stabilizing
flexible, free, J. Comput. Chem.,
26,
1701-1718,
mRNA interaction with the ribosome, Mol. Cell, 56,
doi: 10.1002/jcc.20291.
531-540, doi: 10.1016/j.molcel.2014.09.020.
65.
Hess, B., Kutzner, C., van der Spoel, D., and
53.
Svetlov, M. S., Kommer, A., Kolb, V. A., and Spirin,
Lindahl, E.
(2008) GROMACS 4: algorithms for
A. S. (2006) Effective cotranslational folding of firefly
highly efficient, load-balanced, and scalable molecular
luciferase without chaperones of the Hsp70 family,
simulation, J. Chem. Theory Comput., 4, 435-447,
Protein Sci., 15, 242-247, doi: 10.1110/ps.051752506.
doi: 10.1021/ct700301q.
54.
Osterman, I. A., Komarova, E. S., Shiryaev, D. I.,
66.
Maier, J. A., Martinez, C., Kasavajhala, K.,
Korniltsev, I. A., Khven, I. M., et al. (2016) Sorting
Wickstrom, L., Hauser, K. E., et al. (2015) ff14SB:
out antibiotics’ mechanisms of action: a double
improving the accuracy of protein side chain and
fluorescent protein reporter for high-throughput
backbone parameters from ff99SB, J. Chem. Theory
screening of ribosome and DNA biosynthesis
Comput., 11, 3696-3713, doi: 10.1021/acs.jctc.5b00255.
inhibitors, Antimicrob. Agents Chemother., 60, 7481-
67.
Wang, J., Wolf, R. M., Caldwell, J. W., Kollman,
7489, doi: 10.1128/AAC.02117-16.
P. A., and Case, D. A. (2004) Development and
55.
Zakalyukina, Y. V., Birykov, M. V., Lukianov, D.
testing of a general amber force field, J. Comput.
A., Shiriaev, D. I., Komarova, E. S., et al.
(2019)
Chem., 25, 1157-1174, doi: 10.1002/jcc.20035.
Nybomycin-producing Streptomyces isolated from
68.
Bayly, C. I., Cieplak, P., Cornell, W., and Kollman,
carpenter ant Camponotus vagus, Biochimie, 160, 93-
P. A.
(1993) A well-behaved electrostatic potential
99, doi: 10.1016/j.biochi.2019.02.010.
based method using charge restraints for deriving
56.
Hardy, K., and Haefeli, C.
(1982) Expression
atomic charges: the RESP model, J. Phys. Chem., 97,
in Escherichia coli of a staphylococcal gene for resis-
10269-10280, doi: 10.1021/j100142a004.
tance to macrolide, lincosamide, and streptogramin
69.
Hess, B., Bekker, H., Berendsen, H. J. C., and
type B antibiotics, J. Bacteriol. Res., 152, 524-526,
Fraaij e, J. G. E. M. (1997) LINCS: a linear con-
doi: 10.1128/jb.152.1.524-526.1982.
straint solver for molecular simulations, J. Comput.
57.
Huang, S., Aleksashin, N. A., Loveland, A. B.,
Chem.,
18,
1463-1472, doi: 10.1002/(SICI)1096-
Klepacki, D., Reier, K., et al.
(2020) Ribosome
987X(199709)18:12<1463::AID-JCC4>3.0.CO;2-H.
engineering reveals the importance of
5S rRNA
70.
Bussi, G., Donadio, D., and Parrinello, M. (2007)
autonomy for ribosome assembly, Nat. Commun., 11,
Canonical sampling through velocity rescaling,
2900, doi: 10.1038/s41467-020-16694-8.
J. Chem. Phys., 126, 014101, doi: 10.1063/1.2408420.
58.
Dunkle, J. A., Xiong, L., Mankin, A. S., and
71.
Berendsen, H., Postma, J., van Gunsteren, W.,
Cate, J. H. (2010) Structures of the Escherichia
DiNola, A., and Haak, J. (1984) Molecular dynamics
coli ribosome with antibiotics bound near the peptidyl
with coupling to an external bath, J. Chem. Phys., 81,
transferase center explain spectra of drug action, Proc.
3684-3690, doi: 10.1063/1.448118.
БИОХИМИЯ том 87 вып. 7 2022
916
ХАЙРУЛЛИНА и др.
72.
Darden, T., York, D., and Pedersen, L. (1993) Particle
antimicrobial peptides in Escherichia coli, Anal.
mesh Ewald: an N∙log(N) method for Ewald sums
Bioanal. Chem.,
409,
5581-5592, doi: 10.1007/
in large systems, J. Chem. Phys., 98, 10089-10092,
s00216-017-0496-2.
doi: 10.1063/1.464397.
85.
Tereshchenkov, A. G., Shishkina, A. V., Karpenko,
73.
Horn, H. W., Swope, W. C., Pitera, J. W., Madura,
V. V., Chertkov, V. A., Konevega, A. L., et al. (2016)
J. D., Dick, T. J., et al. (2004) Development of an
New fluorescent macrolide derivatives for studying
improved four-site water model for biomolecular
interactions of antibiotics and their analogs with the
simulations: TIP4P-Ew, J. Chem. Phys., 120, 9665-
ribosomal exit tunnel, Biochemistry (Moscow), 81,
9678, doi: 10.1063/1.1683075.
1163-1172, doi: 10.1134/S0006297916100138.
74.
Joung, I. S., and Cheatham, T. E.,
3rd
(2008)
86.
Kannan, K., Vázquez-Laslop, N., and Mankin, A. S.
Determination of alkali and halide monovalent ion
(2012) Selective protein synthesis by ribosomes with
parameters for use in explicitly solvated biomolecular
a drug-obstructed exit tunnel, Cell, 151, 508-520,
simulations, J. Phys. Chem. B,
112,
9020-9041,
doi: 10.1016/j.cell.2012.09.018.
doi: 10.1021/jp8001614.
87.
Vázquez-Laslop, N., and Mankin, A. S.
(2018)
75.
Athavale, S. S., Petrov, A. S., Hsiao, C., Watkins, D.,
Context-specific action of ribosomal antibiotics,
Prickett, C. D., et al.
(2012) RNA folding and
Annu. Rev. Microbiol.,
72,
185-207, doi: 10.1146/
catalysis mediated by iron (II), PLoS One, 7, e38024,
annurev-micro-090817-062329.
doi: 10.1371/journal.pone.0038024.
88.
Beckert, B., Leroy, E. C., Sothiselvam, S., Bock, L. V.,
76.
Barducci, A., Bussi, G., and Parrinello, M. (2008)
Svetlov, M. S., et al. (2021) Structural and mechanistic
Well-tempered metadynamics: a smoothly converging
basis for translation inhibition by macrolide and
and tunable free-energy method, Phys. Rev. Lett., 100,
ketolide antibiotics, Nat. Commun.,
12,
4466,
020603, doi: 10.1103/PhysRevLett.100.020603.
doi: 10.1038/s41467-021-24674-9.
77.
Domene, C., Barbini, P., and Furini, S. (2015) Bias-
89.
Dzyubak, E., and Yap, M. N. (2016) The expression
exchange metadynamics simulations: an efficient
of antibiotic resistance methyltransferase correlates
strategy for the analysis of conduction and selectivity
with mRNA stability independently of ribosome
in ion channels, J. Chem. Theory Comput., 11, 1896-
stalling, Antimicrob. Agents Chemother., 60, 7178-
1906, doi: 10.1021/ct501053x.
7188, doi: 10.1128/AAC.01806-16.
78.
Tribello, G. A., Bonomi, M., Branduardi, D.,
90.
Vester, B., and Douthwaite, S. (2001) Macrolide
Camilloni, C., and Bussi, G. (2014) PLUMED 2: new
resistance conferred by base substitutions in 23S
feathers for an old bird, Comput. Phys. Commun., 185,
rRNA, Antimicrob. Agents Chemother., 45, 1-12, doi:
604-613, doi: 10.1016/j.cpc.2013.09.018.
10.1128/AAC.45.1.1-12.2001.
79.
Makarov, G. I., Sumbatyan, N. V., and Bogdanov,
91.
Tu, D., Blaha, G., Moore, P. B., and Steitz, T. A.
A. A.
(2017) Structural insight into interaction
(2005) Structures of MLSBK antibiotics bound to
between C20 phenylalanyl derivative of tylosin and
mutated large ribosomal subunits provide a structural
ribosomal tunnel, Biochemistry (Moscow), 82, 925-
explanation for resistance, Cell, 121, 257-270, doi:
932, doi: 10.1134/S0006297917080077.
10.1016/j.cell.2005.02.005.
80.
Daura, X., Gademann, K., Jaun, B., Seebach,
92.
Jeong, C. S., Hwang, J., Do, H., Cha, S. S., Oh,
D., van Gunsteren, W. F., et al.
(1999) Peptide
T. J., et al.
(2020) Structural and biochemical
folding: when simulation meets experiment, An-
analyses of an aminoglycoside 2’-N-acetyltransferase
gew. Chem. Int. Ed., 38, 236-240, doi: 10.1002/(SICI)
from Mycolicibacterium smegmatis, Sci. Rep.,
10,
1521-3773(19990115)38:1/2<236::AID-ANIE236>
21503, doi: 10.1038/s41598-020-78699-z.
3.0.CO;2-M.
93.
Shaw, W. V. (1984) Bacterial resistance to chloram-
81.
Ludwig, T., Krizsan, A., Mohammed, G. K., and
phenicol, Br. Med. Bull., 40, 36-41, doi: 10.1093/
Hoffmann, R. (2022) Antimicrobial activity and 70S
oxfordjournals.bmb.a071945.
ribosome binding of apidaecin-derived Api805 with
94.
Gäumann, E., Hütter, R., Keller-Schierlein, W.,
increased bacterial uptake rate, Antibiotics, 11, 430,
Neipp, L., Prelog, V., et al.
(1960) Lankamycin
doi: 10.3390/antibiotics11040430.
und lankacidin, Helv. Chim. Acta,
80,
601-606,
82.
Kolano, L., Knappe, D., Volke, D., Sträter, N., and
doi: 10.1002/hlca.19600430221.
Hoffmann, R.
(2020) Ribosomal target-binding
95.
Belousoff, M. J., Shapira, T., Bashan, A.,
sites of antimicrobial peptides Api137 and Onc112
Zimmerman, E., Rozenberg, H., et al. (2011) Crystal
are conserved among pathogens indicating new lead
structure of the synergistic antibiotic pair, lankamycin
structures to develop novel broad-spectrum antibi-
and lankacidin, in complex with the large ribosomal
otics, Chembiochem., 21, 2628-2634, doi: 10.1002/
subunit, Proc. Natl. Acad. Sci. USA, 108, 2717-2722,
cbic.202000109.
doi: 10.1073/pnas.1019406108.
83.
Krizsan, A., Volke, D., Weinert, S., Sträter, N.,
96.
Wagner, R. L., Hochstein, F. A., Murai, K., Messina, N.,
Knappe, D., et al. (2014) Insect-derived proline-
and Regna, P. P. (1953) Magnamycin. A new antibiotic,
rich antimicrobial peptides kill bacteria by inhibiting
J. Am. Chem. Soc., 75, 4684-4687, doi: 10.1021/ja01115a019.
bacterial protein translation at the 70S ribosome,
97.
Ross, S. (1968) Erythromycin, oleandomycin and
Angew. Chem. Int. Ed. Engl., 53, 12236-12239, doi:
triacetyloleandomycin, Pediatr. Clin. N. Am., 15, 119-
10.1002/anie.201407145.
129, doi: 10.1016/s0031-3955(16)32093-4.
84.
Holfeld, L., Hoffmann, R., and Knappe, D. (2017)
98.
Gürel, G., Blaha, G., Steitz, T. A., and Moore, P. B.
Correlating uptake and activity of proline-rich
(2009) Structures of triacetyloleandomycin and
БИОХИМИЯ том 87 вып. 7 2022
ОНКОЦИНОВЫЕ АНАЛОГИ ДЕСМИКОЗИНА
917
mycalamide A bind to the large ribosomal subunit
chemical properties of new acyl derivatives of tylosin
of Haloarcula marismortui, Antimicrob. Agents Chemo-
produced by microbial transformation, J. Antibiot., 33,
ther., 53, 5010-5014, doi: 10.1128/AAC.00817-09.
1300-1308, doi: 10.7164/antibiotics.33.1300.
99. Sano, M., Sunazuka, T., Tanaka, H., Yamashita,
102. Huang, G., Okabe, M., Kahar, P., Tsunekawa, H.,
K., Okachi, R., et al. (1985) Chemical modifica-
and Park, Y.
(2001) Optimization of tylosin
tion of spiramycins. VI. Synthesis and antibacte-
feeding rate profile in production of acetyl-
rial activities of
3,3′′-di-O-acyl-4′′-O-sulfonyl and
isovaleryl tylosin (AIV) from tylosin by Streptomyces
3,3′′-di-O-acyl-4′′-O-alkyl derivatives of spira-
thermotolerans YN554, J. Biosci. Bioeng., 91, 504-508,
mycin I, J. Antibiot., 38, 1350-1358, doi: 10.7164/
doi: 10.1263/jbb.91.504.
antibiotics.38.1350.
103. Ettayebi, M., Prasad, S. M., and Morgan, E. A. (1985)
100. Omura, S., Katagiri, M., Umezawa, I.,
Chloramphenicol-erythromycin resistance mutations
Komiyama, K., and Maekawa, T. (1968) Structure-bi-
in a 23S rRNA gene of Escherichia coli, J. Bacteriol.,
ological activities relationships among the leucomy-
162, 551-557, doi: 10.1128/jb.162.2.551-557.1985.
cins and their derivatives, J. Antibiot., 21, 532-538,
104. Sander, P., Prammananan, T., Meier, A., Frischkorn, K.,
doi: 10.7164/antibiotics.21.532.
and Böttger, E. C. (1997) The role of ribosomal RNAs
101. Okamoto, R., Fukumoto, T., Nomura, H.,
in macrolide resistance, Mol. Microbiol., 26, 469-480,
Kiyoshima, K., Nakamura, K., et al. (1980) Physico-
doi: 10.1046/j.1365-2958.1997.5811946.x.
CONJUGATES OF DESMYCOSIN WITH FRAGMENTS
OF ANTIMICROBIAL PEPTIDE ONCOCIN: SYNTHESIS,
ANTIBACTERIAL ACTIVITY, INTERACTION WITH RIBOSOME
Z. Z. Khairullina1, G. I. Makarov2, A. G. Tereshchenkov3, V. S. Buev4, D. A. Lukianov1,5,
V. I. Polshakov6, V. N. Tashlitsky1, I. A. Osterman1,5, and N. V. Sumbatyan1*
1 Faculty of Chemistry, Lomonosov Moscow State University,
119991 Moscow, Russia; e-mail: sumbtyan@belozersky.msu.ru
2South Ural State University, 454080 Chelyabinsk, Russia
3 Belozersky Institute of Physico-Chemical Biology, Lomonosov Moscow State University,119992 Moscow, Russia
4 Faculty of Bioengineering and Bioinformatics, Lomonosov Moscow State University, 119992 Moscow, Russia
5 Skolkovo Institute of Science and Technology, 143025 Skolkovo, Russia
6 Faculty of Fundamental Medicine, Lomonosov Moscow State University, 119991 Moscow, Russia
Design and synthesis of conjugates consisting of the macrolide antibiotic desmycosin and fragments of the
antibacterial peptide oncocin were perfomed to develop approaches to the creation of new antimicrobial
compounds. New compounds were shown to bind to the E. coli 70S ribosomes, to inhibit bacterial protein
synthesis in vitro as well as to suppress bacterial growth. The conjugates of N-terminus hexa- and tripeptide
fragments of oncocin and 3,2′,4′′-triacetyldesmycosin were found to be active against some strains of macrolide-
resistant bacteria. By simulating the dynamics of the complexes of these compounds with wild-type bacterial
ribosomes and with ribosomes, containing A2059G 23S RNA mutation, the specific structural features of their
interactions, were revealed.
Keywords: macrolides, peptide derivatives, antimicrobial peptides, ribosome, molecular dynamics simulations,
nascent peptide exit tunnel
БИОХИМИЯ том 87 вып. 7 2022