БИОХИМИЯ, 2023, том 88, вып. 11, с. 2235 - 2250
УДК 577.24
ЭКСЦИЗИОННАЯ РЕПАРАЦИЯ НУКЛЕОТИДОВ.
МЕТОДЫ ОПРЕДЕЛЕНИЯ
ЭФФЕКТИВНОСТИ ФУНКЦИОНИРОВАНИЯ
Обзор
© 2023 А.А. Попов1, И.О. Петрусева1, Н.В. Науменко1, О.И. Лаврик1,2*
1 Институт химической биологии и фундаментальной медицины СО РАН,
630090 Новосибирск, Россия; электронная почта: lavrik@niboch.nsc.ru
2 Новосибирский национальный исследовательский государственный университет,
630090 Новосибирск, Россия
Поступила в редакцию 29.03.2023
После доработки 30.08.2023
Принята к публикации 31.08.2023
Cистема эксцизионной репарации нуклеотидов (NER) ответственна за удаление из ДНК широ-
кого набора объемных повреждений, что вносит существенный вклад в поддержание стабильности
генома. Эффективность, с которой белки системы NER распознают и удаляют объемные повреж-
дения, зависит от многих факторов и имеет важное клинико-диагностическое значение. В обзоре
рассмотрены современные представления о молекулярных основах функционирования системы
NER в клетках эукариот, а также проанализированы методы и подходы, которые применяются
для определения эффективности функционирования данной системы репарации ДНК как in vitro,
так и ex vivo.
КЛЮЧЕВЫЕ СЛОВА: эксцизионная репарация нуклеотидов, повреждения ДНК, методы определения
активности NER.
DOI: 10.31857/S0320972523110155, EDN: MMRCFF
ВВЕДЕНИЕ
Эффективность удаления объемных пов-
реждений из ДНК зависит от функционального
Системы репарации ДНК - важнейшая
состояния и активности системы NER в клет-
группа молекулярных механизмов клетки, дей-
ках. Одним из примеров наследственных син-
ствие которых обеспечивает поддержание ста-
дромов, связанных с дефектами в работе NER у
бильности генома [1]. Белки системы эксци-
человека, является пигментная ксеродерма, от
зионной репарации нуклеотидов (Nucleotide
названия которой произошли обозначения це-
Excision Repair, NER) удаляют из ДНК объем-
лого ряда белков-участников NER (Xeroderma
ные повреждения - ковалентные аддукты раз-
Pigmentosum complementation group (XP-белки)
личной природы, которые формируются, как
A, B, C, D, E, F, G) [2]. Пониженная активность
правило, с гетероциклическими основаниями
NER наблюдается в клетках больных нейро-
нуклеотидных звеньев под действием экзоген-
дегенеративными заболеваниями и ассоцииро-
ного стресса, а именно УФ-облучения и вред-
вана со старением [3-5]. Данные об эффектив-
ных соединений из окружающей среды. Эти
ности удаления репаративным аппаратом клет-
повреждения вызывают значительные нару-
ки индуцированных терапией объемных пов-
шения регулярной двухцепочечной структуры
реждений ДНК необходимы при разработке
ДНК [1-3].
химиопрепаратов, действие которых основано
Принятые сокращения: CAT - хлорамфеникол ацетилтрансфераза; CPD - циклобутан-пиримидиновый димер;
HCR - реактивация клеток-хозяев; NER - эксцизионная репарация нуклеотидов; GG-NER - общегеномная эксци-
зионная репарация нуклеотидов; TC-NER - эксцизионная репарация нуклеотидов, ассоциированная с транскрипци-
ей; nAnt - N-[6-(9-антраценилкарбамоил)гексаноил]-3-амино-1,2-пропандиол; nFlu - N-[6-(5(6)-флуоресцеинилкар-
бамоил)гексаноил]-3-амино-1,2-пропандиол; TFIIH - фактор транскрипции IIH; XP-белки - Xeroderma Pigmentosum
complementation group; XPC - белок, участвующий в распознавании объемных аддуктов ДНК.
* Адресат для корреспонденции.
2235
2236
ПОПОВ и др.
на способности формировать такие поврежде-
жение участков ДНК, которые могут содержать
ния ДНК, поэтому рассматриваемая проблема
повреждения; 2) проверка наличия объемного
имеет как фундаментальное, так и прикладное
повреждения и сборка «предрасщепляющего»
(клинико-диагностическое) значение.
(предынцизионного) комплекса; 3) формиро-
Репаративный статус клетки в условиях
вание готового «расщепляющего» (инцизион-
генотоксического стресса может быть оценен
ного) комплекса, удаление одноцепочечного
по уровню транскрипции генов, связанных с
фрагмента ДНК с повреждением и репаратив-
репарацией ДНК, в том числе генов, кодирую-
ный синтез ДНК в образованной бреши.
щих ключевые белки системы NER, а также
Существуют две ветви NER - общегеном-
по уровням экспрессии белков репарации
ная (Global Genome, GG-NER) и NER, ас-
ДНК [6, 7]. Однако серьезным ограничением
социированная с транскрипцией (Transcrip-
при применении геномных, транскриптомных
tion-Coupled NER, TC-NER) [10]. В процессе
и протеомных подходов является то, что пост-
GG-NER поиск и обнаружение участков ДНК,
транскрипционная и посттрансляционная ре-
содержащих повреждение, осуществляет спе-
гуляция экспрессии и собственно активность
циализированный гетеротримерный комплекс
функционирования белков системы NER не
XPC-RAD23B-CETN2, в котором роль сенсо-
могут быть оценены с помощью этих мето-
ра, чувствительного к нарушениям регулярной
дов. Кроме того, корреляция между уровня-
двуцепочечной структуры ДНК, принадле-
ми транскрипции и активностью ферментов
жит белку XPC (белок, участвующий в распо-
репарации часто оказывается незначитель-
знавании объемных аддуктов ДНК) [11, 12].
ной [8]. В то же время измерение функцио-
Субъединица RAD23B (RADiation sensitivity ab-
нальной активности систем репарации ДНК
normal protein 23 homolog B) защищает XPC от
является более адекватным способом оценки
протеасомной деградации и обеспечивает ста-
репаративного статуса клетки, то есть ее спо-
бильность всего комплекса, но не контактиру-
собности удалять повреждения и восстанавли-
ет с поврежденной ДНК и высвобождается из
вать нативную структуру ДНК.
комплекса при связывании XPC с ДНК [13-15].
Целью данного обзора является рассмо-
Centrin 2 (CETN2) участвует в формировании
трение ряда существующих методов определе-
и стабилизации гетеротримерного комплекса
ния эффективности функционирования си-
и, наряду с XPC, может взаимодействовать
стемы NER, их преимуществ и недостатков.
с ДНК [15-17].
Первая часть обзора содержит краткое описа-
При обнаружении поврежденного участ-
ние механизмов NER c указанием ссылок на
ка ДНК XPC располагается на неповреж-
недавно опубликованные обзоры [3, 9], кото-
денной цепи ДНК напротив повреждения,
рые посвящены детальному описанию этих
происходит перегиб углеводофосфатного ос-
механизмов. Во второй части рассматриваются
това ДНК с формированием угла 140°, при
методы определения эффективности функ-
этом поврежденное основание выворачива-
ционирования NER, в том числе разработан-
ется из дуплекса [18]. Для обнаружения бел-
ные нами. Обзор актуален и может быть инте-
ком XPC участков, содержащих УФ-повреж-
ресен для исследователей, изучающих NER,
дения, присутствие которых не приводит к
поскольку рассмотрение спектра применяе-
заметному искажению регулярной структуры
мых методов определения активности этой си-
ДНК, требуется участие специализированного
стемы репарации ДНК ранее не проводилось
сенсорного белка - DDB2 (Damage specific
в других обзорах. Представленная в обзоре
DNA Binding protein 2), а также белков DDB1,
информация может быть полезна и применима
PARP1 (Poly(ADP-ribose) Polymerase 1), CUL4a
не только при выборе подходящего для иссле-
(Cullin 4a) и RBX1 (Ring-Box 1), работа кото-
дования метода определения активности NER,
рых делает УФ-повреждение «видимым» для
но и для совершенствования существующих,
XPC в контексте хроматина [19]. Формирова-
а также разработки новых эффективных под-
ние комплекса XPC-ДНК с характерной гео-
ходов к оценке репаративного статуса клетки,
метрией служит сигналом для привлечения
необходимых для развития медицины.
к месту повреждения фактора транскрипции
TFIIH (Transcription Factor IIH), который про-
водит проверку повреждения.
СИСТЕМА ЭКСЦИЗИОННОЙ
Инициация TC-NER происходит при бло-
РЕПАРАЦИИ НУКЛЕОТИДОВ
кировании продвижения РНК-полимеразы II
объемным повреждением, находящимся в тран-
В работе системы NER выделяют несколько
скрибируемой цепи [20, 21]. С блокированной
последовательных этапов: 1) поиск и обнару-
повреждением РНК-полимеразой II связыва-
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2237
ется белок CSB (Cockayne Syndrome B), кото-
ного синтеза цепи ДНК, в котором участвуют
рый изгибает углеводофосфатный остов мо-
белки репликации: ДНК-полимеразы δ или ε,
лекулы ДНК на 80° и осуществляет попытку
репликативные факторы RPA и RFC (Replica-
АТР-зависимой транслокации РНК-полиме-
tion Factor C), PCNA (Proliferating Cell Nuclear
разы II через вызвавший ее остановку «барь-
Antigen) [35]. В белковом комплексе, который
ер» [22-24]. Поскольку транслокация РНК-
связан с одноцепочечным ДНК-фрагментом
полимеразы II через объемное повреждение
с повреждением, происходят конформацион-
невозможна, CSB оттесняет РНК-полимера-
ные перестройки, в результате которых ката-
зу II и привлекает факторы репарации CSA
литический домен XPG активируется и вносит
(Cockayne Syndrome A) и UVSSA-USP7 (UV
разрыв с 3′-стороны от повреждения [36, 37];
Stimulated Scaffold protein A; Ubiquitin Specific
затем вместе с удаляемым ДНК-фрагментом,
Peptidase 7). CSA связывается с комплексом
содержащим повреждение, высвобождаются
белков CSB-РНК-полимераза II и осущест-
XPA, XPG и TFIIH [38]. Восстановление це-
вляет их убиквитинирование [25]. К сформи-
лостности вновь синтезированной цепи ДНК
рованному комплексу CSA-CSB-РНК-поли-
катализируют ДНК-лигазы (I или IIIα).
мераза II присоединяется гетеродимер UVSSA-
USP7, с которым через p62-субъединицу взаи-
модействует фактор TFIIH, ответственный за
МЕТОДЫ ОПРЕДЕЛЕНИЯ
следующую стадию узнавания повреждения.
ЭФФЕКТИВНОСТИ
Последующие стадии узнавания и удале-
ФУНКЦИОНИРОВАНИЯ
ния повреждения осуществляются комплек-
СИСТЕМЫ ЭКСЦИЗИОННОЙ
сами белков, которые формируются оди-
РЕПАРАЦИИ НУКЛЕОТИДОВ
наковым по составу для обеих ветвей NER
набором ферментов и факторов. В состав фак-
Широкая специфичность, большое коли-
тора TFIIH входят две ATP-зависимые хели-
чество белков-участников, многоэтапный ме-
казы XPB и XPD, а также белки p8, p34, p44,
ханизм процесса, а также существование двух
p52, p62 и субкомплекс CAK (CDK-Activating
ветвей эукариотической системы NER опре-
Kinase), выполняющие структурные и регу-
деляют разнообразие применяемых подходов
ляторные функции [26]. При высвобождении
оценки эффективности функционирования
CAK-субкомплекса XPB связывается с непов-
этой системы репарации и используемых для
режденной цепью ДНК с 3′-стороны от пов-
этого модельных белковых систем и ДНК-суб-
реждения и раскручивает ДНК примерно на 5
стратов.
п.н. в направлении 3′→5′ поврежденной цепи.
Модельные белковые системы и ДНК-суб-
Это позволяет XPD, выполняющей в NER
страты. Использование модельных систем
роль верифицирующей хеликазы, связаться
различной степени сложности, в том числе
с поврежденной цепью ДНК с 5′-стороны от
реконструированных из предварительно очи-
повреждения [27-29]. XPD продвигается по
щенных, как правило, рекомбинантных бел-
поврежденной цепи ДНК в направлении 5′→3′,
ков, позволило выяснить многие детали меха-
постепенно формируя асимметричный «пузырь»
низма репарации объемных повреждений в
и останавливаясь при встрече с объемным
клетках эукариот [39-41], а также сравнить
аддуктом [27, 30]. В сформированный на ДНК
субстратные свойства ДНК, содержащих пов-
фактором TFIIH комплекс вовлекаются бел-
реждения различной структуры [34]. Наибо-
ки RPA (Replication Protein A) и фактор пиг-
лее часто измеряется активность эндогенных
ментной ксеродермы А (XPA), которые обеспе-
белков NER, содержащихся во фракциониро-
чивают правильное расположение структурно
ванных по стандартной методике цельнокле-
специфичных эндонуклеаз XPG и XPF в ком-
точных экстрактах (NER-компетентных экс-
плексе с помогающим ее работе белком ERCC1
трактах) млекопитающих [42].
(Excision Repair Cross-Complementation group 1)
Агенты, повреждающие ДНК, часто гене-
на поврежденной ДНК и участвуют в форми-
рируют гетерогенные смеси повреждений, од-
ровании предынцизионного комплекса [31-34].
нако для сравнительной оценки репаративного
В присутствии XPG, связавшегося с пов-
статуса клеток и определения эффективно-
режденной ДНК с 3′-стороны от повреждения,
сти удаления повреждений конкретного типа
ERCC1-XPF вносит в ДНК разрыв с 5′-сто-
нужны ДНК-субстраты с определенной струк-
роны от повреждения, затем высвобождается
турой. Модельные ДНК, которые использу-
из комплекса. Появление в результате рас-
ются для определения эффективности функ-
щепления цепи ДНК свободной 3′-ОН-группы
ционирования системы NER in vitro и ex vivo,
служит сигналом для инициации репаратив-
представляют собой протяженные линейные
БИОХИМИЯ том 88 вып. 11 2023
2238
ПОПОВ и др.
ДНК-дуплексы и плазмидные ДНК, содержа-
кольцевых, так и линейных ДНК [48]. На эф-
щие в заданной позиции одной из цепей объ-
фективность эксцизии влияет не только струк-
емное повреждение
[43-48]. Минимальная
тура повреждения, но и нуклеотидная после-
длина линейного ДНК-дуплекса, необходимая
довательность окружающего его участка ДНК,
для протекания реакции специфической экс-
которая влияет на характер индуцируемых
цизии, катализируемой белками NER, состав-
присутствием повреждения нарушений струк-
ляет 100 п.н. [48]. Значительная часть работ
туры ДНК [55]. Таким образом, выбор модель-
по определению эффективности функциони-
ного повреждения и нуклеотидной последова-
рования NER in vitro была выполнена с ис-
тельности ДНК является важнейшим этапом
пользованием модельных кольцевых ДНК,
дизайна эксперимента с применением любо-
содержащих циклобутан-пиримидиновый ди-
го подхода для определения эффективности
мер (CPD), пиримидин-пиримидон-(6-4)-фо-
функционирования NER и до сих пор не явля-
топродукты и производные бенз[a]пирена [43,
ется абсолютно тривиальной задачей.
49], а также ацетиламинофлуорена [50]. Осно-
Определение эксцизионной активности NER
вой для создания модельных ДНК могут быть
in vitro. Оценка специфической эксцизионной
полученные методом твердофазного синтеза
активности системы NER in vitro дает инфор-
олигодезоксирибонуклеотиды, содержащие мо-
мацию об эффективности удаления из ДНК
дифицированное ненуклеотидное звено. Та-
участка с повреждением и позволяет про-
кие фрагменты могу быть встроены в более
водить сравнительные биохимические ис-
протяженные структуры, включая кольцевые
следования, характеризующие как свойства
плазмидные ДНК, с помощью ферментатив-
белковых систем, так и свойства модельных
ного лигирования либо при использовании в
ДНК [45, 56].
качестве праймера в катализируемой ДНК-по-
При прямой детекции продуктов эксци-
лимеразой реакции и последующего лигирова-
зии модельные ДНК содержат радиоактивную
ния [45, 48]. Возможно также введение в ДНК
метку вблизи повреждения, в результате чего
соответствующего дезоксинуклеозидмонофос-
эту метку содержат и продукты эксцизии [57].
фата с помощью ДНК-полимеразной и лигаз-
В процессе ферментативного синтеза про-
ной реакций [51, 52].
тяженных ДНК для прямой детекции 5′-ко-
Эффективность удаления повреждений из
нец олигодезоксирибонуклеотидного фраг-
ДНК зависит от многих факторов. Для ли-
мента фосфорилируется с использованием
нейных ДНК-дуплексов различной длины и
[γ-32P]АТР с удельной радиоактивностью
линеаризованной плазмиды, которые содер-
6000 Ки/ммоль. Это позволяет при невысоких
жали бенз[a]пиреновое производное дезокси-
уровнях эксцизии, которые часто наблюдают-
гуанина, эффективность эксцизии не пре-
ся in vitro, визуализировать продукты реакции
вышала 5-10%, в то время как эффективность
эксцизии и оценить их количество. Однако
эксцизии этого повреждения из той же плаз-
необходимость использования больших коли-
миды, находящейся в кольцевой форме, была
честв радиоактивной метки для синтеза суб-
в 6 раз выше [53]. Различия в эффективности
стратов, а также трудности, возникающие при
удаления системой NER одного и того же ад-
хранении высокорадиоактивных протяжен-
дукта из ДНК с одинаковым нуклеотидным
ных ДНК, значительно ограничивают приме-
контекстом могут быть обусловлены возмож-
нение этого метода. Недавно был разработан
ностью взаимодействия с линейными дцДНК
аналог данного метода, не требующий исполь-
белков PARP1 и комплекса Ku 70/80 с ДНК-
зования радиоактивной метки. В этом случае
зависимой протеинкиназой, которые при-
образовавшиеся продукты эксцизии (олиго-
сутствуют в клеточных экстрактах и обладают
дезоксирибонуклеотидов длиной ~20-30 нук-
повышенным сродством к двухцепочечным
леотидов) извлекали из лизата предваритель-
концам, что обеспечивает их конкуренцию с
но облученных УФ-светом клеток путем вклю-
белками NER [54]. Интересно, что эксцизия
чения в
3′-конец биотина и последующей
поврежденного участка, содержащего N-(дез-
конъюгации со стрептавидином, с которым
оксигуанозин-8-ил)-2-ацетиламинофлуорен,
ковалентно связана пероксидаза хрена, необ-
при инкубации с белками NER-компетентно-
ходимая для хемилюминесцентной визуали-
го экстракта клеток HeLa наблюдалась только
зации [58]. Ограничение предложенного под-
при использовании кольцевой ДНК [50]. В то
хода заключается в быстрой деградации обра-
же время эффективность удаления белками
зующихся продуктов эксцизии, что приводит
NER-компетентного экстракта клеток HeLa
к значительному снижению сигнала.
фрагментов, содержащих CPD, была прак-
Метод оценки эффективности удаления
тически одинаковой при использовании как
объемных повреждений белками NER in vitro
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2239
и характер ее изменения в кластерах различ-
ной структуры и состава, что позволило углу-
бить понимание взаимосвязи структуры ряда
повреждений с эффективностью формирова-
ния продуктивных комплексов на первом эта-
пе их узнавания белком XPC [52, 59]. Кроме
того, выполненный с использованием данного
метода сравнительный анализ репаративного
статуса клеток показал, что белки клеточных
экстрактов долгоживущего голого землекопа
(Heterocephalus glaber) более эффективно рас-
познают и удаляют из ДНК объемные повреж-
дения, чем белки экстрактов, полученных из
клеток мыши (Mus musculus) [56]. Полученный
результат является одним из немногих экспе-
риментальных подтверждений взаимосвязи
эффективно функционирующих систем репа-
рации ДНК с долголетием.
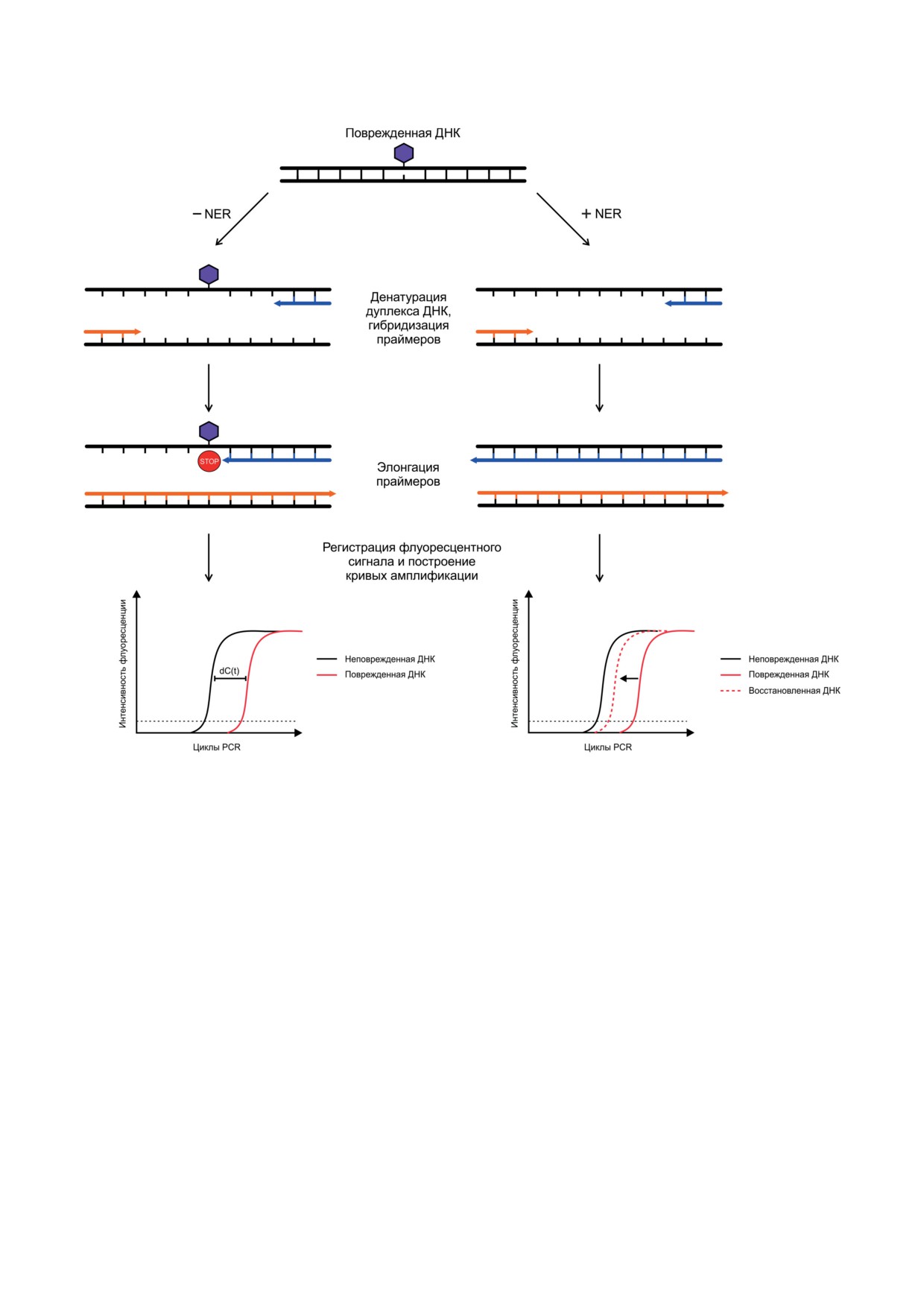
Оценка эффективности функционирования
NER методом количественной PCR. Главным
преимуществом подходов, использующих
количественную PCR (quantitative Polymerase
Chain Reaction, qPCR), является возможность
оценки эффективности репарации повреж-
дений при минимальных количествах ДНК-
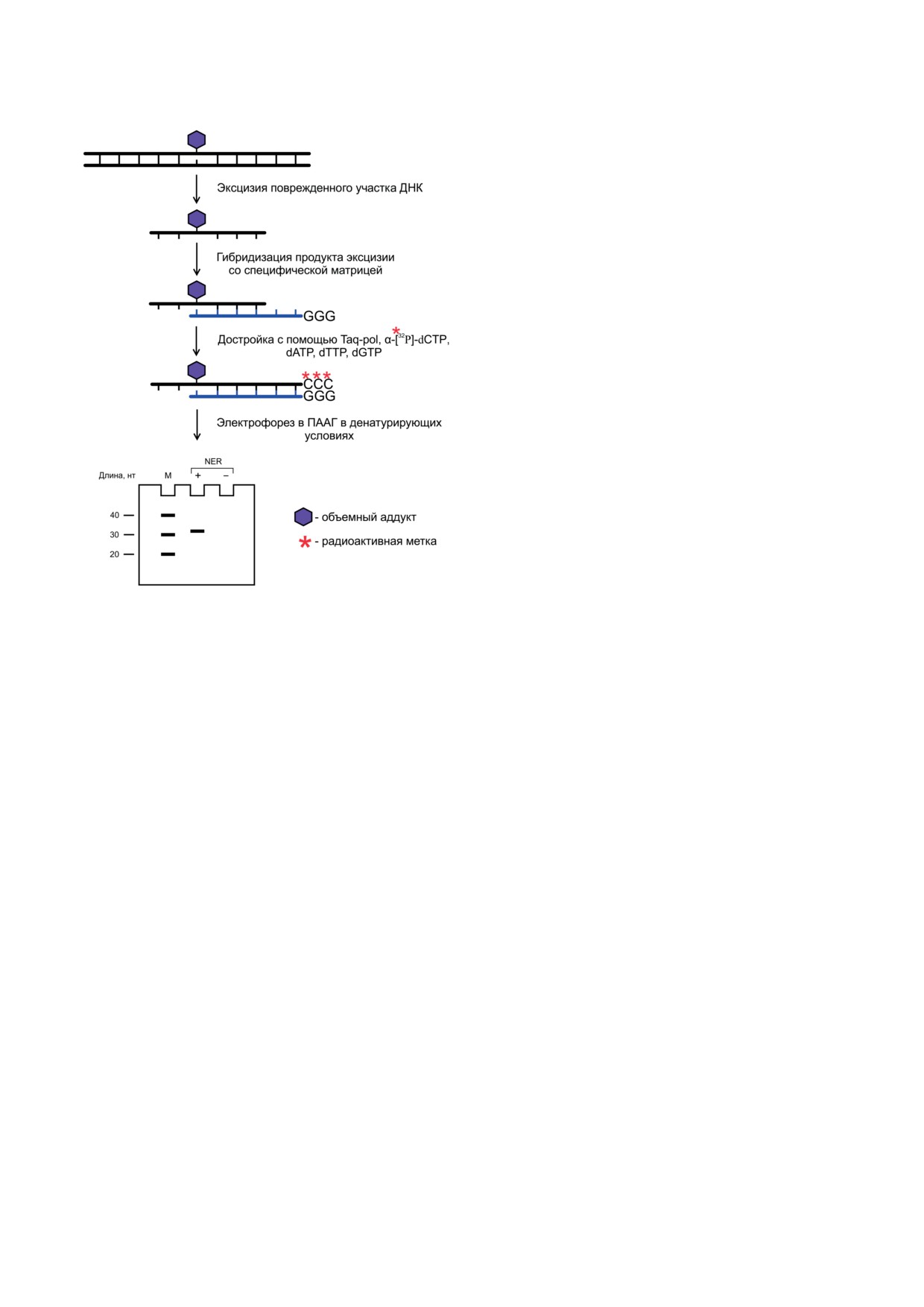
Рис. 1. Оценка эксцизионной активности NER in vitro
матрицы. Принцип определения активности
путем постэксцизионного мечения продуктов реакции
NER с помощью qPCR основан на разнице в
эксцизии, адаптирован из [56]
эффективности копирования неповрежденной
модельной ДНК и ДНК того же размера и по-
следовательности, содержащей в одной из це-
основан на постэксцизионном радиоактив-
пей объемное повреждение, способное блоки-
ном мечении 3′-концов продуктов специфиче-
ровать удлинение праймера ДНК-полимера-
ской эксцизии в процессе их достройки ДНК-
зой. В результате репарации структура ДНК-
полимеразой (например, Taq-полимеразой).
матрицы восстанавливается, что обеспечивает
В качестве матрицы используется олигодез-
полноценное удлинение обоих праймеров и
оксирибонуклеотид, последовательность кото-
эффективное копирование ДНК-матрицы в
рого частично комплементарна вырезаемому
ходе PCR [60]. Эффективность репарации опре-
фрагменту с повреждением (рис. 1). Оптими-
деляется по разнице в получаемых в ходе PCR
зированная структура матрицы обеспечивает
значениях порогового цикла C(t) между вос-
высокий уровень включения радиоактивно-
становленной ДНК и контрольной неповреж-
меченных нуклеотидов, что существенно сни-
денной ДНК (так называемая dC(t)). Умень-
жает расход радиоактивной метки, а также
шение значения dC(t) говорит о протекании
исключает возможность достройки самой ма-
репарации и позволяет определить концен-
трицы. Метод позволяет сравнивать как экс-
трацию (количество) восстановленной (про-
цизионную активность образцов, содержащих
шедшей полный цикл репарации) ДНК в ре-
белки NER, так и субстратные свойства ДНК
акционной смеси после проведения реакции
с разными по структуре повреждениями [45, 52].
NER (рис. 2).
Использование данного метода в соче-
Одной из главных методических проблем
тании с ДНК-субстратами, которые содер-
применения PCR для оценки эффективности
жали синтетические объемные повреждения,
удаления объемных повреждений является то,
позволило существенно продвинуться в пони-
что в результате достройки праймера на не-
мании влияния структуры повреждения на
поврежденной цепи ДНК-матрицы, содержа-
различные стадии репарационного процес-
щей объемное повреждение, в первом цикле
са [40, 52, 59], а также оценить эксцизионную
реакции образуется укороченная копия пов-
активность белков NER в отношении линей-
режденной цепи ДНК, дальнейшая амплифи-
ных ДНК-дуплексов, содержащих кластерные
кация которой происходить не будет. В то же
повреждения в различных положениях цепи,
время полноразмерный продукт копирования
БИОХИМИЯ том 88 вып. 11 2023
2240
ПОПОВ и др.
Рис. 2. Схематическое изображение способа оценки эффективности удаления объемных повреждений белками NER
c помощью qPCR
неповрежденной цепи, также образующийся
режденной цепи плазмидной ДНК. Клетки
при протекании первого цикла ДНК-полиме-
млекопитающих трансфицировали плазмид-
разной реакции, будет амплифицироваться.
ной ДНК, содержащей ДНК-белковую сшивку
Это приведет к уменьшению диапазона воз-
либо холестериновый аддукт, и некоторое вре-
можных значений dC(t) до одного цикла.
мя культивировали. Затем плазмидную ДНК
В работе Chesner и Campbell [61] предложен
выделяли из клеток и использовали в качестве
вариант методики оценки эффективности
матрицы для PCR. После многократного по-
удаления объемных повреждений с использо-
вторения циклов удлинения праймера 1 в реак-
ванием qPCR, который позволил обойти эту
ционную смесь добавляли праймер 2, компле-
проблему и тем самым расширить диапазон
ментарный второй цепи, и проводили qPCR.
возможных значений dC(t) до трех циклов.
Эффективность репарации объемного пов-
Авторы данной работы использовали прием
реждения рассчитывали на основе разницы
предварительной многократной элонгации
значений порогового цикла C(t) контроль-
праймера 1. Полноразмерный продукт элон-
ной неповрежденной и процессированной в
гации образуется, только если поврежденная
ходе репарации плазмид. С использованием
цепь плазмиды была восстановлена в ходе
такого подхода было показано, что репарация
репарации, поскольку присутствие объемно-
плазмидной ДНК, содержащей ДНК-белковую
го повреждения в цепи блокирует элонгацию
сшивку, проходит в 3 раза более эффективно
праймера 1, комплементарного находящемуся
в клетках человека, чем в клетках китайского
с 3′-стороны от повреждения участку пов- хомячка (CHO), тогда как значимых различий
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2241
в эффективности репарации плазмидной
клетки обрабатывают повреждающим агентом,
ДНК, содержащей холестериновый аддукт, в
затем помещают в гель из легкоплавкой ага-
сравниваемых клетках выявлено не было [61].
розы, приготовленный на предметном стекле
Предложенный в работе Shen et al. [62]
для микроскопии, и лизируют, чтобы удалить
подход, основанный на использовании 5′-био-
мембрану и растворимые клеточные компо-
тинилированного CPD-содержащего ДНК-дуп-
ненты [66]. ДНК, прикрепленную к ядерному
лекса шпилечной структуры в качестве суб-
матриксу, подвергают щелочной обработке и
страта, позволил оценить эффективность ра-
анализируют методом электрофореза в щелоч-
боты NER в различных типах клеток человека.
ных условиях с окраской интеркалирующим
Трансфицированные CPD-содержащей ДНК
флуоресцентным красителем. При движении в
клетки после культивирования подвергали
электрическом поле нуклеоид принимает вид
лизису, из лизата извлекали целевую ДНК и
«комет» с лидирующей при движении в геле
с использованием qPCR проводили оценку
«головой», сформированной сверхскрученной
эффективности ее репарации. Спустя 24 часа
ДНК, и характерным отстающим «хвостом»,
после трансфекции эффективность репарации
представляющим собой фрагменты ДНК с од-
CPD-содержащей ДНК системой NER клеток
ноцепочечными разрывами (релаксированные
линии HEK 293T составила 43,7%, тогда как
формы) [66, 67]. Оценку степени повреждения
в случае клеток линии SW480 этот показатель
ДНК ведут с помощью специализированного
не превышал 1% [62].
программного обеспечения (рис. 3).
Применение qPCR позволяет оценивать
Несмотря на свою популярность, первый
количество объемных повреждений, возни-
вариант метода ДНК-комет, разработанный в
кающих в ДНК в результате различных гено-
основном для анализа генотоксичности лекар-
токсических воздействий, включая химио-
ственных препаратов и загрязнителей окружаю-
терапию, а также длительность сохранения ин-
щей среды, имел ряд недостатков. Проведе-
дуцированных повреждений в ДНК клеток
ние экспериментов с применением данного
живого организма
[63, 64]. В работе Kali-
метода было продолжительным по времени и
nowski et al. [65] проведено сравнение количе-
трудоемким, а также не давало возможности
ства повреждений, образуемых воздействием
одновременного анализа большого числа об-
УФ-света и цисплатина на митохондриальную
разцов. Впоследствии развитие данного метода
и геномную ДНК. Согласно данным, получен-
позволило обойти некоторые ограничения,
ным в этой работе, количество УФ-повреж-
связанные с продолжительностью экспери-
дений во фрагменте митохондриальной ДНК
мента и его стоимостью, что позволило адап-
мышиных клеток линии L1210 было в 1,5 раза
тировать его для высокопроизводительного
выше, чем во фрагменте геномной ДНК, в то
скрининга [68].
время как при обработке цисплатином коли-
Поскольку разрывы в ДНК могут возни-
чество образуемых внутрицепочечных сшивок
кать не только при генотоксическом воздей-
в митохондриальной ДНК оказалось в 2,5 раза
ствии, но и в процессе удаления повреждений,
ниже, чем в геномном фрагменте.
это позволяет использовать метод ДНК-комет
Использование и дальнейшее развитие ме-
для изучения систем репарации ДНК, в том
тодов определения эффективности функцио-
числе и системы NER [69-72]. Однако селек-
нирования NER c помощью qPCR имеет
тивная оценка количества разрывов, форми-
важное прикладное значение. Высокая чув-
руемых при процессинге объемного повреж-
ствительность этого метода в сочетании с не-
дения, в данном случае была бы невозможна.
сложной процедурой проведения эксперимен-
Более избирательная, хотя и косвенная, оцен-
та открывает перспективы к использованию
ка эффективности репарации повреждений
qPCR-анализа для диагностики нарушений
стала возможной после разработки метода
работы системы NER и оперативной оценки
ДНК-комет с применением ингибиторов.
репаративного статуса больных тяжелыми
В работе Cipollini et al. [73] было показано, что
нейродегенеративными и раковыми заболева-
воздействие УФ-света на лимфоциты чело-
ниями.
века приводит к значительному увеличению
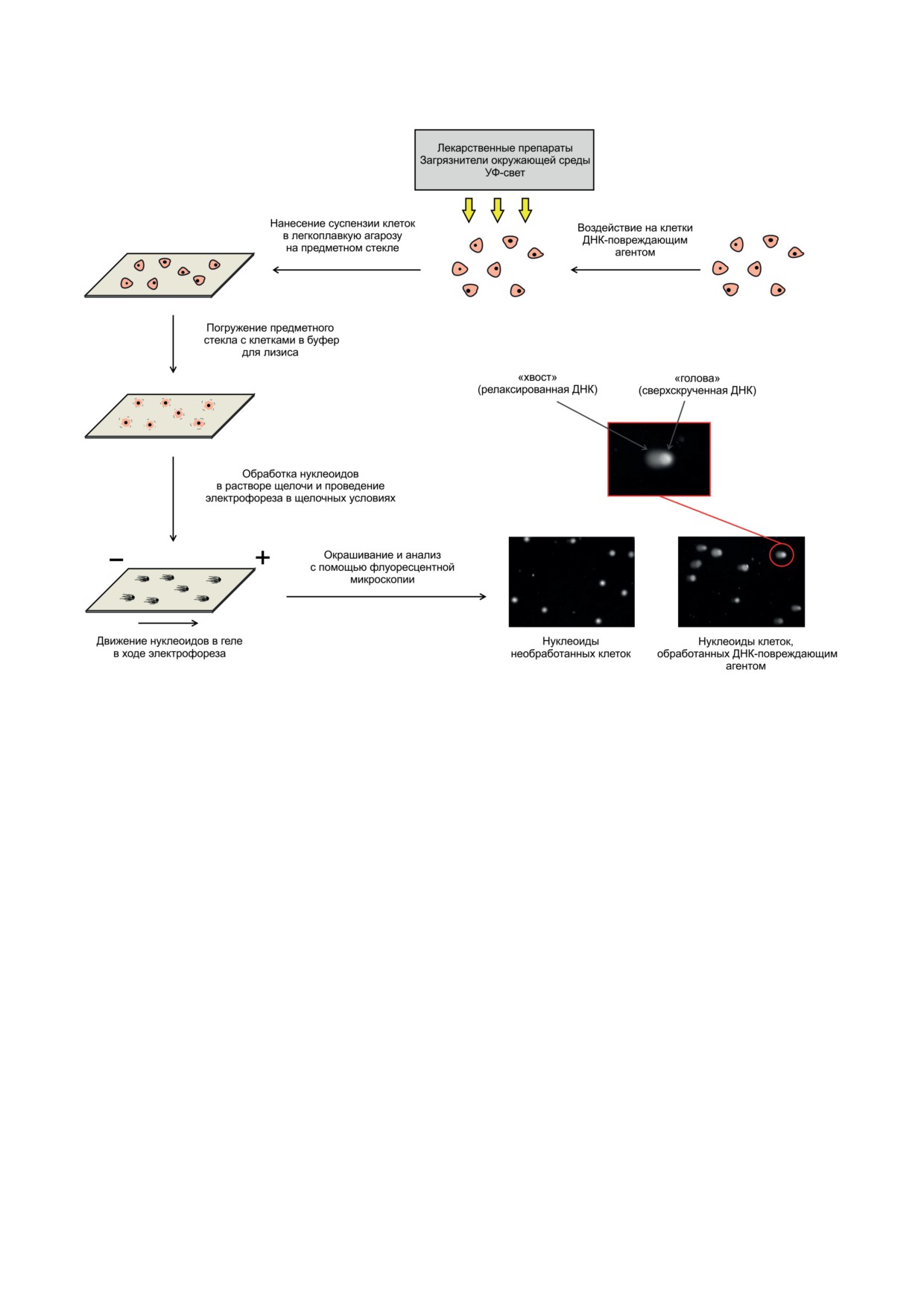
Применение метода ДНК-комет для оценки
интенсивности флуоресценции мигрирующего
эффективности функционирования NER ex vivo.
«хвоста» нуклеоида через 20-90 минут после
В качестве субстрата при оценке уровня обра-
облучения. Спустя 120-240 минут после облу-
зования разрывов в ДНК методом ДНК-комет
чения интенсивность флуоресценции падает,
используются нуклеоидные структуры (сверх-
что сигнализирует о восстановлении целост-
скрученная ДНК, прикрепленная к ядерному
ности ДНК. Обработка лимфоцитов человека
матриксу) отдельных клеток. Исследуемые
новобиоцином - ингибитором, блокирующим
14
БИОХИМИЯ том 88 вып. 11 2023
2242
ПОПОВ и др.
Рис. 3. Схема проведения эксперимента по оценке степени повреждения ДНК в клетке методом ДНК-комет
образование разрывов в ДНК, - приводила
такая модификация метода ДНК-комет позво-
к снижению количества ДНК во фракции,
лила повысить специфичность измерения экс-
формирующей «хвост» кометы. Присутствие
цизионной активности системы NER [74].
афидиколина
- ингибитора ДНК-полиме-
Анализ ДНК-комет концептуально прост
разы
- блокировало репаративный синтез,
и является ценным дополнительным подходом
в результате чего количество мигрирующей
к оценке репаративного статуса, однако трудо-
ДНК не менялось со временем. Таким обра-
емок и малопроизводителен; существуют так-
зом, применение модифицированного метода
же проблемы с воспроизводимостью резуль-
ДНК-комет позволяет следить за двумя про-
татов. К настоящему времени разработаны
цессами: интенсивностью образования в ДНК
усовершенствованные варианты комет-ана-
разрывов и эффективностью репаративного
лиза, которые значительно увеличивают про-
синтеза [73]. Проведенная с использованием
изводительность, воспроизводимость и специ-
данного варианта метода сравнительная оцен-
фичность оценки эффективности репарации
ка уровня активности NER в человеческих
определенных классов повреждений ДНК с
фибробластах дикого типа и XPA-/--фибро-
использованием различных ферментов репа-
бластах подтвердила его применимость для
рации ДНК [75-77]. Однако даже в усовер-
таких оценок. Эффективность репарации ин-
шенствованном виде комет-анализ обычно де-
дуцируемых процессингом бенз[a]пиреновых
тектирует репарацию различных повреждений
аддуктов одноцепочечных разрывов в клетках
ДНК и требует нескольких этапов обработки
дикого типа была выше, чем в XPA-/--клетках.
образцов, для того чтобы выявить поврежде-
Было также показано, что количество одно-
ния, специфичные для каждого из путей [78].
цепочечных разрывов, образующихся при воз-
Метод оценки эффективности функциони-
действии бенз[a]пирен-диол-эпоксида на де-
рования NER ex vivo путем реактивации экс-
фектные клетки, было в 1,3 раза больше, чем
прессии белков-репортеров в клетках хозяина.
в клетках дикого типа. Можно заключить, что
В настоящее время востребованным является
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2243
метод определения эффективности функцио-
держащей ген флуоресцентного белка GFP,
нирования системы NER, основанный на ре-
удалось провести количественную оценку эф-
активации экспрессии гена того или иного ре-
фективности репарации УФ-повреждений в
портерного белка в клетках хозяина (Host Cell
клетках HEK 293. Было показано, что система
Reactivation, HCR). С использованием данного
NER клеток HEK 293 способна провести пол-
подхода можно проводить оценку активности
ную репарацию плазмидной ДНК, содержа-
NER в культуре клеток (ex vivo). Для этого в
щей 1,4 УФ-повреждения на каждые 1000 нук-
качестве субстрата используется плазмидная
леотидов, в течение 12 часов после трансфек-
ДНК, содержащая объемное повреждение, ко-
ции [82].
торое блокирует экспрессию гена репортерного
Сравнительная оценка эффективности ре-
белка в исследуемых клетках [79]. Внедрение
парации плазмиды pEGFP-N1, предваритель-
плазмиды в клетку происходит путем транс-
но подвергшейся облучению УФ-светом, в раз-
фекции или электропорации. Способ оценки
личных типах клеток человека была проведена
уровня восстановления экспрессии репортер-
также с применением активируемой флуорес-
ного белка определяется его свойствами.
ценцией сортировки клеток (Fluorescent Ac-
С применением метода HCR была оцене-
tivated Cell Sorting, FACS). Репарация модель-
на эффективность репарации системой NER
ной плазмиды в кератиноцитах происходила
лимфоцитов человека плазмидных ДНК,
более эффективно, чем в фибробластах, а наи-
содержащих УФ-повреждение, либо бенз[a]пи-
меньшая эффективность репарации была от-
реновый аддукт в области промотора гена
мечена в меланоцитах [83]. Комбинация мето-
хлорамфеникол-ацетилтрансферазы (Chloram-
дов HCR и FACS позволила определить роль
phenicol Acetyltransferase, CAT) [79]. Лимфо-
GG-NER и TC-NER в репарации апуриновых/
циты человека с нормальным фенотипом
апиримидиновых сайтов (AP-сайтов) в клет-
и лимфоциты, дефектные по генам различ-
ках MRC-5 [84]. Было показано, что эффек-
ных XP-белков, трансфицировали плазмидой.
тивность репарации плазмиды pEGFP-Q205,
Доказательством восстановления экспрессии
содержащей в качестве повреждения тиотетра-
белка служило восстановление ацетилиро-
гидрофуран (S-THF) - труднорепарируемый
вания хлорамфеникола в присутствии радио-
белками эксцизионной репарации оснований
активномеченного субстрата CAT, [3H]ацетил-
(Base Excision Repair, BER) аналог AP-сайта,
КоА. По кинетике накопления [3H]ацетил-
содержащий фосфотиоатную группу, - снижа-
хлорамфеникола судили об эффективности
лась в 2 раза при отсутствии функционально
удаления объемного повреждения, блокирую-
активной TC-NER и в 4 раза - при отсутствии
щего экспрессию CAT. Это исследование по-
функционально активной GG-NER. Полу-
казало, что эффективность репарации ДНК
ченные результаты позволили рассматривать
в XP-дефектных клетках была в 10-20 раз
GG-NER как преобладающий механизм репа-
ниже, чем в нормальных [79]. Ранее примене-
рации таких повреждений [84].
ние метода HCR позволило также показать,
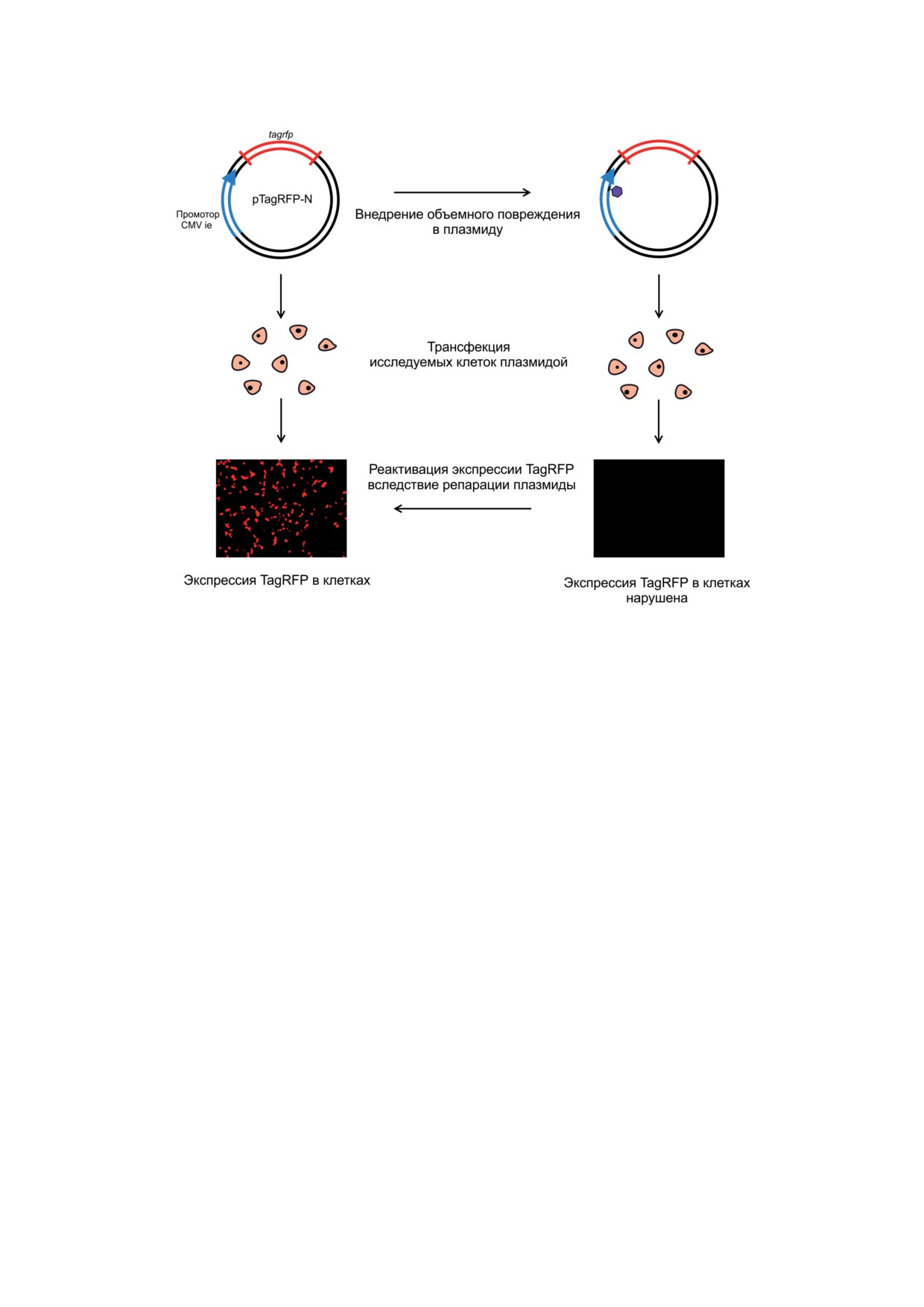
Недавно был предложен вариант метода
что репарация плазмидных ДНК, содержащих
определения эффективности функционирова-
индуцируемые воздействием цисплатина меж-
ния NER ex vivo с использованием рекомби-
цепочечные сшивки ДНК, в дефектных по
нантной плазмидной ДНК, несущей ген флуо-
XP-генам клетках человека происходит значи-
ресцентного белка TagRFP, и полученных на
тельно менее эффективно, чем в клетках дико-
ее основе ДНК с объемными повреждения-
го типа, что свидетельствует об участии белков
ми [85]. Блокирующие экспрессию гена флуо-
NER в репарации одного из самых токсичных
ресцентного белка синтетические объемные
повреждений ДНК [80].
повреждения в этом случае вводятся в тран-
В качестве репортерных чаще использу-
скрибируемую цепь ДНК в область промотора
ются флуоресцентные белки либо ферменты
гена tagrfp. Плазмидой с повреждением либо
люцифераза и β-галактозидаза, применение
контрольной плазмидой трансфицируют ис-
которых позволило упростить детекцию репа-
следуемые клетки, затем, инкубируя клетки в
рации. Исследование репарации плазмидных
соответствующих условиях, с помощью флуо-
ДНК, кодирующих ген репортерного белка
ресцентной микроскопии следят за изменени-
люциферазы (luc) и содержащих УФ-повреж-
ем уровня экспрессии TagRFP (рис. 4).
дение в промоторном участке, в нормальных
Поскольку присутствие объемного пов-
и XP-дефектных клетках продемонстриро-
реждения в транскрибируемой цепи в обла-
вало возможность применения этого метода
сти промотора блокирует транскрипцию гена
для сравнения репаративного статуса разных
tagrfp, флуоресцентный сигнал в клетках, транс-
клеток [81]. С использованием плазмиды, со-
фицированных плазмидой с повреждением,
БИОХИМИЯ том 88 вып. 11 2023
14*
2244
ПОПОВ и др.
Рис. 4. Схематическое изображение способа определения эффективности функционирования NER ex vivo, основан-
ного на реактивации экспрессии гена репортерного флуоресцентного белка в клетках хозяина. Адаптирован из [85]
не регистрируется. При удалении объемного
щей ДНК, а также эффективность их взаимо-
повреждения происходит восстановление экс-
действия с XPD на этапе верификации [40,
прессии TagRFP и появляется флуоресцентный
45, 86]. Эффективность этапа первичного уз-
сигнал.
навания поврежденного участка, выраженная
Была проведена сравнительная оценка
в уровне сродства XPC-ДНК, и эффектив-
эффективности репарации плазмидных ДНК,
ность специфической эксцизии nFlu и nAnt
содержащих синтетические объемные повреж-
были практически одинаковы. Согласно дан-
дения N-{2-[N-(4-азидо-2,5-дифторо-3-хлорпи-
ным моделирования, присутствие nFlu, в от-
ридин-6-ил)-3-аминопропионил]аминоэтил},
личие от nAnt, не обеспечивало уровень деста-
N-[6-(5(6)-флуоресцеинилкарбамоил)гекса-
билизации ДНК-дуплекса, который был бы
ноил]-3-амино-1,2-пропандиол (nFlu) и N-[6-
достаточен для формирования продуктивных
(9-антраценилкарбамоил)гексаноил]-3-амино-
для реакции NER комплексов XPC-ДНК [86].
1,2-пропандиол (nAnt), системой NER кле-
Размер и расположение nFlu относительно
ток HEK 293T. Появление флуоресцирующих
повреждения допускали формирование как
клеток HEK 293T, трансфицированных nAnt-
продуктивных, так и непродуктивных для NER
содержащей плазмидой, наступало спустя
комплексов. Однако сродство XPD к nFlu-
10 часов после трансфекции, в то время как в
ДНК было в 60 раз выше, чем сродство XPD к
случае клеток, трансфицированных плазми-
nAnt-ДНК [40]. Это, вероятно, увеличивало
дой с nFlu, это время составило 8 часов. Таким
эффективность процессирования nFlu-ДНК
образом, репарация плазмидной ДНК, содер-
и приводило к выравниванию уровня специ-
жащей nFlu, системой NER клеток HEK 293T
фической эксцизии этих повреждений в про-
проходила быстрее, чем репарация плазмид-
цессе GG-NER.
ной ДНК, содержащей повреждение nAnt [85].
Основанные на HCR подходы позволяют
Ранее были исследованы субстратные свой-
без использования радиоактивных меток оце-
ства ДНК, содержащих эти объемные повреж-
нивать в клетках эффективность процесса ре-
дения, в реакции специфической эксцизии,
парации в целом. Их развитие и применение
катализируемой белками NER-компетентных
позволит проводить мониторинг генотоксичес-
экстрактов, их влияние на структуру прилежа-
кого воздействия лекарственных препаратов,
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2245
а также оценивать эффективность удале-
основанных на знании деталей механизма
ния не только объемных повреждений, но и
данного процесса, позволит с применением
повреждений, удаляемых белками системы
разных модельных систем провести исследо-
BER [87, 88].
вания, которые обеспечат прогресс в оценке
персонального репаративного статуса, а также
мониторинга и прогнозирования эффектов
ЗАКЛЮЧЕНИЕ
химиотерапевтического воздействия при про-
ведении противоопухолевой терапии.
Эффективная репарация ДНК рассматри-
вается как один из факторов, определяющих
Вклад авторов. А.А. Попов, И.О. Петру-
высокую продолжительность жизни и устой-
сева, Н.В. Науменко - написание текста ста-
чивость млекопитающих к онкологическим
тьи, О.И. Лаврик - редактирование текста
и нейродегенеративным заболеваниям
[7,
статьи, Н.В. Науменко, А.А. Попов - графи-
89]. Широкая субстратная специфичность
ческое оформление статьи, И.О. Петрусева,
системы NER, большое количество белковых
О.И. Лаврик - руководство работой.
факторов и ферментов, разветвленный и мно-
Финансирование. Работа выполнена при
гоэтапный механизм делают оценку эффек-
финансовой поддержке Российского научно-
тивности функционирования этой системы
го фонда (грант № 19-74-10056-П) и Проекта
достаточно сложной задачей. Проведение
государственного задания ИХБФМ СО РАН
сравнительных исследований эффективности
(№ 121031300041-4, глава «Система эксцизион-
протекания NER в клетках долгоживущих и
ной репарации нуклеотидов»).
короткоживущих млекопитающих, а также в
Конфликт интересов. Авторы заявляют об
нормальных и раковых клетках поможет про-
отсутствии конфликта интересов.
двинуться в понимании молекулярных основ
Соблюдение этических норм. Настоящая
клеточного старения и онкоустойчивости. Раз-
статья не содержит описания каких-либо ис-
работка усовершенствованных методов оцен-
следований с участием людей или животных
ки эффективности функционирования NER,
в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Chatterjee, N., and Walker, G. C. (2017) Mechanisms
6. Christmann, M., and Kaina, B. (2013) Transcriptional
of DNA damage, repair, and mutagenesis, Environ.
regulation of human DNA repair genes following
Mol. Mutagen., 58, 235-263, doi: 10.1002/em.22087.
genotoxic stress: trigger mechanisms, inducible
2. Martens, M. C., Emmert, S., and Boeckmann, L.
responses and genotoxic adaptation, Nucleic Acids
(2021) Xeroderma pigmentosum: gene variants
Res., 41, 8403-8420, doi: 10.1093/nar/gkt635.
and splice variants, Genes, 12, 1173, doi: 10.3390/
7. MacRae, S. L., Croken, M. M., Calder, R. B.,
genes12081173.
Aliper, A., Milholland, B., White, R. R., Zhavoron-
3. Krasikova, Y., Rechkunova, N., and Lavrik, O. (2021)
kov, A., Gladyshev, V. N., Seluanov, A., Gorbuno-
Nucleotide excision repair: from molecular defects
va, V., Zhang, Z. D., and Vijg, J. (2015) DNA repair
to neurological abnormalities, Int. J. Mol. Sci., 22,
in species with extreme lifespan differences, Aging
6220, doi: 10.3390/ijms22126220.
(Albany NY), 7, 1171-1184, doi: 10.18632/aging.100866.
4. Paccosi, E., Balajee, A. S., and Proietti-De-Santis, L.
8. Figueroa-Gonzalez, G., and Perez-Plasencia, C.
(2022) A matter of delicate balance: loss and gain of
(2017) Strategies for the evaluation of DNA damage
Cockayne syndrome proteins in premature aging
and repair mechanisms in cancer, Oncol. Lett.,
and cancer, Front. Aging, 3, 960662, doi: 10.3389/
13, 3982-3988, doi: 10.3892/ol.2017.6002.
fragi.2022.960662.
9. Van den Heuvel, D., van der Weegen, Y., Boer,
5. Ataabadi, E. A., Golshiri, K., Van Der Linden, J.,
D. E. C., Ogi, T., and Luijsterburg, M. S.
(2021)
De Boer, M., Duncker, D. J., Jüttner, A., De Vries, R.,
Transcription-coupled DNA repair: from mechanism
Van Veghel, R., Van Der Pluijm, I., Dutheil, S.,
to human disorder, Trends Cell Biol., 31, 359-371,
Chalgeri, S., Zhang, L., Lin, A., Davis, R. E.,
doi: 10.1016/j.tcb.2021.02.007.
Gretchen, S. L., Danser, J. H. A., and Roks, A. J. M.
10. Kobaisi, F., Fayyad, N., Rezvani, H. R., Fayyad-
(2021) Vascular ageing features caused by selec-
Kazan, M., Sulpice, E., Badran, B., Fayyad-
tive DNA damage in smooth muscle cell, Oxid.
Kazan, H., Gidrol, X., and Rachidi, W. (2019) Sig-
Med. Cell Longev.,
2021,
2308317, doi:
10.1155/
naling pathways, chemical and biological modulators
2021/2308317.
of nucleotide excision repair: the faithful shield against
БИОХИМИЯ том 88 вып. 11 2023
2246
ПОПОВ и др.
UV genotoxicity, Oxid. Med. Cell. Longev., 2019,
M. S. (2020) The cooperative action of CSB, CSA,
4654206, doi: 10.1155/2019/4654206.
and UVSSA target TFIIH to DNA damage-stalled
11.
Le, J., and Min, J. H. (2023) Structural modeling
RNA polymerase II, Nat. Commun.,
11,
2104,
and analyses of genetic variations in the human XPC
doi: 10.1038/s41467-020-15903-8.
nucleotide excision repair protein, J. Biomol. Struct.
23.
Xu, J., Lahiri, I., Wang, W., Wier, A., Cianfrocco,
Dyn., 1-28, doi: 10.1080/07391102.2023.2177349.
M. A., Chong, J., Hare, A. A., Dervan, P. B.,
12.
Zebian, A., Shaito, A., Mazurier, F., Rezvani, H. R.,
DiMaio, F., Leschziner, A. E., and Wang, D. (2017)
and Zibara, K. (2019) XPC beyond nucleotide excision
Structural basis for eukaryotic transcription-coupled
repair and skin cancers, Mutat. Res. Rev. Mutat. Res.,
DNA repair initiation, Nature,
551,
653-657,
782, 108286, doi: 10.1016/j.mrrev.2019.108286.
doi: 10.1038/nature24658.
13.
Yokoi, M., and Hanaoka, F. (2017) Two mammali-
24.
Kokic, G., Wagner, F. R., Chernev, A., Urlaub, H.,
an homologs of yeast Rad23, HR23A and HR23B, as
and Cramer, P. (2021) Structural basis of human
multifunctional proteins, Gene, 597, 1-9, doi: 10.1016/
transcription-DNA repair coupling, Nature,
598,
j.gene.2016.10.027.
368-372, doi: 10.1038/s41586-021-03906-4.
14.
Grønbæk-Thygesen, M., Kampmeyer, C., Hof-
25.
Fischer, E. S., Scrima, A., Böhm, K., Matsumoto, S.,
mann, K., and Hartmann-Petersen, R.
(2023)
Lingaraju, G. M., Faty, M., Yasuda, T., Cavadini, S.,
The moonlighting of RAD23 in DNA repair
Wakasugi, M., Hanaoka, F., Iwai, S., Gut, H.,
and protein degradation, Biochim. Biophys. Acta
Sugasawa, K., and Thomä, N. H. (2011) The mo-
Gene Regul. Mech.,
1866,
194925, doi:
10.1016/
lecular basis of CRL4DDB2/CSA ubiquitin ligase
j.bbagrm.2023.194925.
architecture, targeting, and activation, Cell,
147,
15.
Rechkunova, N. I., Krasikova, Y. S., and Lavrik, O. I.
1024-1039, doi: 10.1016/j.cell.2011.10.035.
(2021) Interactome of base and nucleotide excision
26.
Kim, J., Li, C. L., Chen, X., Cui, Y., Golebiowski,
DNA repair systems, Mol. Biol. (Moscow), 55, 155-166,
F. M., Wang, H., Hanaoka, F., Sugasawa, K., and
doi: 10.1134/S0026893321020126.
Yang, W. (2023) Lesion recognition by XPC, TFIIH
16.
Feltes, B. C. (2021) Every protagonist has a sidekick:
and XPA in DNA excision repair, Nature,
617,
structural aspects of human xeroderma pigmentosum-
170-175, doi: 10.1038/s41586-023-05959-z.
binding proteins in nucleotide excision repair, Protein
27.
Kuper, J., and Kisker, C. (2021) Three targets in
Sci., 30, 2187-2205, doi: 10.1002/pro.4173.
one complex: A molecular perspective of TFIIH in
17.
Zhang, W., Shi, E., Zhao, Y., and Yang, B. (2018)
cancer therapy, DNA Repair (Amst), 105, 103143,
Modulation effect of double strand DNA on the
doi: 10.1016/j.dnarep.2021.103143.
self-assembly of N-terminal domain of Euplotes
28.
Tsutakawa, S. E., Tsai, C. L., Yan, C., Bralić, A.,
octocarinatus centrin, J. Inorg. Biochem., 180, 15-25,
Chazin, W. J., Hamdan, S. M., Schärer, O. D.,
doi: 10.1016/j.jinorgbio.2017.12.001.
Ivanov, I., and Tainer, J. A. (2020) Envisioning how
18.
Paul, D., Mu, H., Zhao, H., Ouerfelli, O., Jeffrey,
the prototypic molecular machine TFIIH functions
P. D., Broyde, S., and Min, J. H. (2019) Structure
in transcription initiation and DNA repair, DNA
and mechanism of pyrimidine-pyrimidone
(6-4)
Repair (Amst),
96,
102972, doi:
10.1016/j.dnarep.
photoproduct recognition by the Rad4/XPC nucle-
2020.102972.
otide excision repair complex, Nucleic Acids Res.,
29.
Greber, B. J., Toso, D. B., Fang, J., and Nogales,
47, 6015-6028, doi: 10.1093/nar/gkz359.
E. (2019) The complete structure of the human
19.
Apelt, K., Lans, H., Schärer, O. D., and Luijsterburg,
TFIIH core complex, Elife, 12, e44771, doi: 10.7554/
M. S. (2021) Nucleotide excision repair leaves a mark
eLife.44771.
on chromatin: DNA damage detection in nucleosomes,
30.
Kokic, G., Chernev, A., Tegunov, D., Dienemann, C.,
Cell. Mol. Life Sci., 78, 7925-7942, doi: 10.1007/
Urlaub, H., and Cramer, P. (2019) Structural ba-
s00018-021-03984-7.
sis of TFIIH activation for nucleotide excision re-
20.
Boetefuer, E. L., Lake, R. J., and Fan, H. Y.
pair, Nat. Commun., 10, 2885, doi: 10.1038/s41467-
(2018) Mechanistic insights into the regulation of
019-10745-5.
transcription and transcription-coupled DNA repair
31.
Kim, M., Kim, H. S., D’Souza, A., Gallagher, K.,
by Cockayne syndrome protein B, Nucleic Acids Res.,
Jeong, E., Topolska-Wós, A., Ogorodnik Le Meur, K.,
46, 7471-7479, doi: 10.1093/nar/gky660.
Tsai, C. L., Tsai, M. S., Kee, M., Tainer, J. A., Yeo,
21.
Lans, H., Hoeijmakers, J. H. J., Vermeulen, W., and
J. E., Chazin, W. J., and Schärer, O. D. (2022) Two
Marteijn, J. A. (2019) The DNA damage response
interaction surfaces between XPA and RPA organize
to transcription stress, Nat. Rev. Mol. Cell Biol., 20,
the preincision complex in nucleotide excision
766-784, doi: 10.1038/s41580-019-0169-4.
repair, Proc. Natl. Acad. Sci. USA, 119, e2207408119,
22.
Van der Weegen, Y., Golan-Berman, H., Mevissen,
doi: 10.1073/pnas.2207408119.
T. E. T., Apelt, K., González-Prieto, R., Goed-
32.
Krasikova, Y. S., Lavrik, O. I., and Rechkunova, N. I.
hart, J., Heilbrun, E. E., Vertegaal, A. C. O., van den
(2022) The XPA protein-life under precise control,
Heuvel, D., Walter, J. C., Adar, S., and Luijsterburg,
Cells, 11, 3723, doi: 10.3390/cells11233723.
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2247
33.
Van den Heuvel, D., Kim, M., Wondergem, A. P., van
stituted with purified protein components, Cell, 80,
der Meer, P. J., Witkamp, M., Lambregtse, F., Kim,
859-868, doi: 10.1016/0092-8674(95)90289-9.
H. S., Kan, F., Apelt, K., Kragten, A., González-
43.
Szeltner, Z., Póti, A., Harami, G. M., Kovács, M.,
Prieto, R., Vertegaal, A. C. O., Yeo, J. E., Kim, B. G.,
and Szüts, D. (2021) Evaluation and modulation
van Doorn, R., Schärer, O. D., and Luijsterburg, M. S.
of DNA lesion bypass in an SV40 large T antigen-
(2023) A disease-associated XPA allele interferes with
based in vitro replication system, FEBS Open Bio, 11,
TFIIH binding and primarily affects transcription-
1054-1075, doi: 10.1002/2211-5463.13099.
coupled nucleotide excision repair, Proc. Natl.
44.
Du, H., Wang, P., Li, L., and Wang, Y. (2019) Repair
Acad. Sci. USA, 120, e2208860120, doi: 10.1073/
and translesion synthesis of O6-alkylguanine DNA
pnas.2208860120.
lesions in human cells, J. Biol. Chem., 294, 11144-
34.
Li, C. L., Golebiowski, F. M., Onishi, Y., Samara,
11153, doi: 10.1074/jbc.RA119.009054.
N. L., Sugasawa, K., and Yang, W. (2015) Tripartite
45.
Evdokimov, A., Petruseva, I., Tsidulko, A., Koroleva, L.,
DNA lesion recognition and verification by XPC,
Serpokrylova, I., Silnikov, V., and Lavrik, O. (2013)
TFIIH, and XPA in nucleotide excision repair, Mol.
New synthetic substrates of mammalian nucleotide
Cell., 59, 1025-1034, doi: 10.1016/j.molcel.2015.08.012.
excision repair system, Nucleic Acids Res., 41, e123,
35.
Kemp, M. G., and Hu, J. (2017) Postexcision events
doi: 10.1093/nar/gkt301.
in human nucleotide excision repair, Photochem.
46.
Hilton, B., Gopal, S., Xu, L., Mazumder, S., Musich,
Photobiol., 93, 178-191, doi: 10.1111/php.12641.
P. R., Cho, B. P., and Zou, Y. Z. (2016) Dissociation
36.
González-Corrochano, R., Ruiz, F. M., Taylor, N. M. I.,
dynamics of XPC-RAD23B from damaged DNA is
Huecas, S., Drakulic, S., Spínola-Amilibia, M., and
a determining factor of NER efficiency, PLoS One,
Fernández-Tornero, C. (2020) The crystal structure
11, e0157784, doi: 10.1371/journal.pone.0157784.
of human XPG, the xeroderma pigmentosum group G
47.
Naumenko, N., Petruseva, I., Lomzov, A., and
endonuclease, provides insight into nucleotide
Lavrik, O.
(2021) Recognition and removal of
excision DNA repair, Nucleic Acids Res., 48, 9943-
clustered DNA lesions via nucleotide excision repair,
9958, doi: 10.1093/nar/gkaa688.
DNA Repair (Amst),
108,
103225, doi:
10.1016/
37.
Muniesa-Vargas, A., Theil, A. F., Ribeiro-Silva, C.,
j.dnarep.2021.103225.
Vermeulen, W., and Lans, H. (2022) XPG: a multi-
48.
Huang, J. C., and Sancar, A. (1994) Determination
tasking genome caretaker, Cell. Mol. Life Sci., 79, 166,
of minimum substrate size for human excinuclease,
doi: 10.1007/s00018-022-04194-5.
J. Biol. Chem.,
269,
19034-19040, doi:
10.1016/
38.
Hu, J., Choi, J. H., Gaddameedhi, S., Kemp, M. G.,
S0021-9258(17)32270-6.
Reardon, J. T., and Sancar, A. (2013) Nucleotide
49.
Hess, M. T., Gunz, D., Luneva, N., Geacintov, N. E.,
excision repair in human cells: fate of the excised
and Naegeli, H. (1997) Base pair conformation-
oligonucleotide carrying DNA damage in vivo,
dependent excision of benzo[a]pyrene diol epoxide-
J. Biol. Chem.,
288,
20918-20926, doi:
10.1074/
guanine adducts by human nucleotide excision repair
jbc.M113.482257.
enzymes, Mol. Cell Biol., 17, 7069-7076, doi: 10.1128/
39.
Krasikova, Y. S., Rechkunova, N. I., Maltseva, E. A.,
MCB.17.12.7069.
Pestryakov, P. E., Petruseva, I. O., Sugasawa, K.,
50.
Gillet, L. C., Alzeer, J., and Schärer, O. D. (2005) Site-
Chen, X., Min, J. H., and Lavrik, O. I.
(2013)
specific incorporation of N-(deoxyguanosin-8-yl)-2-
Comparative analysis of interaction of human and yeast
acetylaminofluorene (dG-AAF) into oligonucleotides
DNA damage recognition complexes with damaged
using modified ‘ultra-mild’ DNA synthesis, Nucleic
DNA in nucleotide excision repair, J. Biol. Chem.,
Acids Res., 33, 1961-1969, doi: 10.1093/nar/gki335.
288, 10936-10947, doi: 10.1074/jbc.M112.444026.
51.
Dezhurov, S. V., Khodyreva, S. N., Plekhanova, E. S.,
40.
Petruseva, I., Naumenko, N., Kuper, J., Anarbaev, R.,
and Lavrik, O. I. (2005) A new highly efficient pho-
Kappenberger, J., Kisker, C., and Lavrik, O. (2021)
toreactive analogue of dCTP. Synthesis, characteriza-
The interaction efficiency of XPD-p44 with bulky
tion, and application in photoaffinity modification of
DNA damages depends on the structure of the
DNA binding proteins, Bioconjug. Chem., 16, 215-222,
damage, Front. Cell Dev. Biol., 9, 617160, doi: 10.3389/
doi: 10.1021/bc0497867.
fcell.2021.617160.
52.
Lukyanchikova, N. V., Petruseva, I. O., Evdokimov,
41.
Barnett, J., Kuper, J., Koelmel, W., Kisker, C., and
A. N., Silnikov, V. N., and Lavrik, O. I. (2016) DNA
Kad, N. (2019) The TFIIH subunits p44/p62 act as
with damage in both strands as affinity probes and nu-
a damage sensor during nucleotide excision repair,
cleotide excision repair substrates, Biochemistry (Mos-
Nucleic Acids Res., 48, 12689-12696, doi: 10.1093/
cow), 81, 263-274, doi: 10.1134/S0006297916030093.
nar/gkaa973.
53.
Kolbanovskiy, M., Aharonoff, A., Sales, A. H.,
42.
Aboussekhra, A., Biggerstaff, M., Shivji, M. K. K.,
Geacintov, N. E., and Shafirovich, V. (2020) Remark-
Vilpo, J. A., Moncollin, V., Podust, V. N., Protić, M.,
able enhancement of nucleotide excision repair of a
Hübscher, U., Egly, J. M., and Wood, R. D. (1995)
bulky guanine lesion in a covalently closed circular
Mammalian DNA nucleotide excision repair recon-
DNA plasmid relative to the same linearized plasmid,
БИОХИМИЯ том 88 вып. 11 2023
2248
ПОПОВ и др.
Biochemistry,
59,
2842-2848, doi:
10.1021/acs.
64.
Jennerwein, M. M., and Eastman, A. (1991) A poly-
biochem.0c00441.
merase chain reaction-based method to detect cispla-
54.
Emerson, C. H., Lopez, C. R., Ribes-Zamora, A.,
tin adducts in specific genes, Nucleic Acids Res., 19,
Polleys, E. J., Williams, C. L., Yeo, L., Zaneveld, J. E.,
6209-6214, doi: 10.1093/nar/19.22.6209.
Chen, R., and Bertuch, A. A. (2018) Ku DNA end-
65.
Kalinowski, D. P., Illenye, S., and Van Houten, B.
binding activity promotes repair fidelity and influences
(1992) Analysis of DNA damage and repair in murine
end-processing during nonhomologous end-joining
leukemia L1210 cells using a quantitative polymerase
in Saccharomyces cerevisiae, Genetics, 209, 115-128,
chain reaction assay, Nucleic Acids Res.,
20,
doi: 10.1534/genetics.117.300672.
3485-3494, doi: 10.1093/nar/20.13.3485.
55.
Geacintov, N. E., and Broyde, S. (2017) Repair-
66.
Azqueta, A., Langie, S. A. S., Boutet-Robinet, E.,
resistant DNA lesions, Chem. Res. Toxicol.,
30,
Duthie, S., Ladeira, C., Møller, P., Collins, A. R., and
1517-1548, doi: 10.1021/acs.chemrestox.7b00128.
Godschalk, R. W. L. (2019) DNA repair as a human
56.
Evdokimov, A., Kutuzov, M., Petruseva, I.,
biomonitoring tool: Comet assay approaches, Mutat.
Lukjanchikova, N., Kashina, E., Kolova, E.,
Res., 781, 71-87, doi: 10.1016/j.mrrev.2019.03.002.
Zemerova, T., Romanenko, S., Perelman, P.,
67.
Collins, A. R. (2004) The comet assay for DNA
Prokopov, D., Seluanov, A., Gorbunova, V.,
damage and repair: principles, applications, and
Graphodatsky, A., Trifonov, V., Khodyreva, S., and
limitations, Mol. Biotechnol., 26, 249-261, doi: 10.1385/
Lavrik, O. (2018) Naked mole rat cells display more
MB:26:3:249.
efficient excision repair than mouse cells, Aging (Albany
68.
Vodenkova, S., Azqueta, A., Collins, A., Dusinska, M.,
NY), 10, 1454-1473, doi: 10.18632/aging.101482.
Gaivão, I., Møller, P., Opattova, A., Vodicka, P.,
57.
Liu, Z., Ding, S., Kropachev, K., Lei, J., Amin, S.,
Godschalk, R. W. L., and Langie, S. A. S. (2020)
Broyde, S., and Geacintov, N. E. (2015) Resis-
An optimized comet-based in vitro DNA repair assay
tance to nucleotide excision repair of bulky gua-
to assess base and nucleotide excision repair activity,
nine adducts opposite abasic sites in DNA duplexes
Nat. Protoc., 15, 3844-3878, doi: 10.1038/s41596-
and relationships between structure and function,
020-0401-x.
PLoS One,
10, e0142068, doi:
10.1371/journal.
69.
Vodicka, P., Vodenkova, S., Opattova, A., and
pone.0137124.
Vodickova, L.
(2019) DNA damage and repair
58.
Song, J., Kemp, M. G., and Choi, J. H. (2017) De-
measured by comet assay in cancer patients, Mutat.
tection of the excised, damage-containing oligonucle-
Res. Genet. Toxicol. Environ. Mutagen., 843, 95-110,
otide products of nucleotide excision repair in human
doi: 10.1016/j.mrgentox.2019.05.009.
cells, Photochem. Photobiol., 93, 192-198, doi: 10.1111/
70.
Speit, G., Leibiger, C., Kuehner, S., and Högel, J.
php.12638.
(2013) Further investigations on the modified comet
59.
Lukyanchikova, N. V., Petruseva, I. O., Evdokimov,
assay for measuring aphidicolin-block nucleotide
A. N., Koroleva, L. S., and Lavrik, O. I. (2018) DNA
excision repair, Mutagenesis, 28, 145-151, doi: 10.1093/
bearing bulky fluorescent and photoreactive damage
mutage/ges063.
in both strands as substrates of the nucleotide excision
71.
Ge, J., Ngo, L. P., Kaushal, S., Tay, I. J., Thadhani, E.,
repair system, Mol. Biol. (Moscow), 52, 237-246,
Kay, J. E., Mazzucato, P., Chow, D. N., Fessler, J. L.,
doi: 10.1134/S0026893318020061.
Weingeist, D. M., Sobol, R. W., Samson, L. D., Floyd,
60.
Govan, H. L., Valles-Ayoub, Y., and Braun, J. (1990)
S. R., and Engelward, B. P. (2021) CometChip enables
Fine-mapping of DNA damage and repair in specific
parallel analysis of multiple DNA repair activities,
genomic segments, Nucleic Acids Res., 18, 3823-3830,
DNA Repair (Amst),
106,
103176, doi:
10.1016/
doi: 10.1093/nar/18.13.3823.
j.dnarep.2021.103176.
61.
Chesner, L. N., and Campbell, C. (2018) A quanti-
72.
Valdiglesias, V., Sánchez-Flores, M., Fernández-
tative PCR-based assay reveals that nucleotide exci-
Bertólez, N., Au, W., Pásaro, E., and Laffon, B.
sion repair plays a predominant role in the removal
(2020) Expanded usage of the Challenge-Comet assay
of DNA-protein crosslinks from plasmids transfected
as a DNA repair biomarker in human populations:
into mammalian cells, DNA Repair (Amst), 62, 18-27,
protocols for fresh and cryopreserved blood samples,
doi: 10.1016/j.dnarep.2018.01.004.
and for different challenge agents, Arch. Toxicol.,
62.
Shen, J. C., Fox, E. J., Ahn, E. H., and Loeb, L. A.
94, 4219-4228, doi: 10.1007/s00204-020-02881-5.
(2014) A rapid assay for measuring nucleotide excision
73.
Cipollini, M., He, J., Rossi, P., Baronti, F.,
repair by oligonucleotide retrieval, Sci. Rep., 4, 4894,
Micheli, A., Rossi, A. M., and Barale, R. (2006)
doi: 10.1038/srep04894.
Can individual repair kinetics of UVC-induced DNA
63.
Aloisi, C. M. N., Nilforoushan, A., Ziegler, N., and
damage in human lymphocytes be assessed through the
Sturla, S. J. (2020) Sequence-specific quantitation of
comet assay? Mutat. Res., 601, 150-161, doi: 10.1016/
mutagenic DNA damage via polymerase amplification
j.mrfmmm.2006.06.004.
with an artificial nucleotide, J. Am. Chem. Soc., 142,
74.
Vande Loock, K., Decordier, I., Ciardelli, R.,
6962-6969, doi: 10.1021/jacs.9b11746.
Haumont, D., and Kirsch-Volders, M.
(2010)
БИОХИМИЯ том 88 вып. 11 2023
МЕТОДЫ ОПРЕДЕЛЕНИЯ АКТИВНОСТИ NER
2249
An aphidicolin-block nucleotide excision repair
lymphocytes, Mutat. Res., 509, 165-174, doi: 10.1016/
assay measuring DNA incision and repair capacity,
s0027-5107(02)00219-1.
Mutagenesis, 25, 25-32, doi: 10.1093/mutage/gep039.
82.
Roguev, A., and Russev, G. (2000) Two-wavelength
75.
Muruzabal, D., Sanz-Serrano, J., Sauvaigo, S.,
fluorescence assay for DNA repair, Anal. Biochem.,
Gützkow, K. B., López de Cerain, A., Vettorazzi, A.,
287, 313-318, doi: 10.1006/abio.2000.4865.
and Azqueta, A. (2020) Novel approach for the
83.
Burger, K., Matt, K., Kieser, N., Gebhard, D., and
detection of alkylated bases using the enzyme-
Bergemann, J. (2010) A modified fluorimetric host cell
modified comet assay, Toxicol. Lett., 330, 108-117,
reactivation assay to determine the repair capacity of
doi: 10.1016/j.toxlet.2020.04.021.
primary keratinocytes, melanocytes and fibroblasts,
76.
Ngo, L. P., Owiti, N. A., Swartz, C., Winters, J., Su, Y.,
BMC Biotechnol., 10, 46, doi: 10.1186/1472-6750-10-46.
Ge, J., Xiong, A., Han, J., Recio, L., Samson, L. D.,
84.
Kitsera, N., Rodriguez-Alvarez, M., Emmert, S.,
and Engelward, B. P. (2020) Sensitive CometChip
Carell, T., and Khobta, A. (2019) Nucleotide excision
assay for screening potentially carcinogenic DNA
repair of abasic DNA lesions, Nucleic Acids Res., 47,
adducts by trapping DNA repair intermediates, Nucleic
8537-8547, doi: 10.1093/nar/gkz558.
Acids Res., 48, e13, doi: 10.1093/nar/gkz1077.
85.
Popov, A. A., Orishchenko, K. E., Naumenko, K. N.,
77.
Ge, J., Prasongtanakij, S., Wood, D. K., Weingeist,
Evdokimov, A. N., Petruseva, I. O., and Lavrik, O. I.
D. M., Fessler, J., Navasummrit, P., Ruchirawat,
(2021) A method for assessing the efficiency of the
M., and Engelward, B. P. (2014) CometChip: a high-
nucleotide excision repair system ex vivo, Acta Naturae,
throughput
96-well platform for measuring DNA
13, 122-125, doi: 10.32607/actanaturae.11430.
damage in microarrayed human cells, J. Vis. Exp.,
86.
Evdokimov, A. N., Tsidulko, A. N., Popov, A. V.,
92, e50607, doi: 10.3791/50607.
Vorobiev, Y. N., Lomzov, A. A., Koroleva, L. S.,
78.
Azqueta, A., Ladeira, C., Giovannelli, L., Boutet-
Silnikov, V. N., Petruseva, I. O., and Lavrik, O. I.
Robinet, E., Bonassi, S., Neri, M., Gajski, G., Duthie, S.,
(2018) Structural basis for the recognition and pro-
Del Bo’, C., Riso, P., Koppen, G., Basaran, N.,
cessing of DNA containing bulky lesions by the
Collins, A., and Møller, P. (2020) Application of the
mammalian nucleotide excision repair system, DNA
comet assay in human biomonitoring: An hCOMET
Repair (Amst),
61,
86-98, doi:
10.1016/j.dnarep.
perspective, Mutat. Res. Rev. Mutat. Res., 783, 108288,
2017.10.010.
doi: 10.1016/j.mrrev.2019.108288.
87.
Piett, C. G., Pecen, T. J., Laverty, D. J., and Nagel,
79.
Athas, W. F., Hedayati, M. A., Matanoski, G. M.,
Z. D. (2021) Large-scale preparation of fluorescence
Farmer, E. R., and Grossman, L. (1991) Development
multiplex host cell reactivation (FM-HCR) reporters,
and field-test validation of an assay for DNA repair
Nat. Protoc., 16, 4265-4298, doi: 10.1038/s41596-
in circulating human lymphocytes, Cancer Res., 51,
021-00577-3.
5786-5793.
88.
Kitsera, N., Stathis, D., Lühnsdorf, B., Müller, H.,
80.
Chu, G., and Chang, E. (1988) Xeroderma pigmento-
Carell, T., Epe, B., and Khobta, A. (2011) 8-Oxo-7,8-
sum group E cells lack a nuclear factor that binds to
dihydroguanine in DNA does not constitute a barrier
damaged DNA, Science, 242, 564-567, doi: 10.1126/
to transcription, but is converted into transcription-
science.3175673.
blocking damage by OGG1, Nucleic Acids Res.,
81.
Qiao, Y., Spitz, M. R., Guo, Z., Hadeyati, M.,
39, 5926-5934, doi: 10.1093/nar/gkr163.
Grossman, L., Kraemer, K. H., and Wei, Q. (2002)
89.
Kogan, V., Molodtsov, I., Menshikov, L. I., Reis, R.
Rapid assessment of repair of ultraviolet DNA
J. S., and Fedichev, P. (2015) Stability analysis of a
damage with a modified host-cell reactivation assay
model gene network links aging, stress resistance, and
using a luciferase reporter gene and correlation with
negligible senescence, Sci. Rep., 5, 13589, doi: 10.1038/
polymorphisms of DNA repair genes in normal human
srep13589.
NUCLEOTIDE EXCISION REPAIR.
METHODS OF DETERMINATION THE EFFICIENCY FUNCTIONING
Review
A. A. Popov1, I. O. Petruseva1, N. V. Naumenko1, and O. I. Lavrik1,2*
1 Institute of Chemical Biology and Fundamental Medicine Siberian Branch Russian Academy of Sciences,
630090 Novosibirsk, Russia; e-mail: lavrik@niboch.nsc.ru
2 Novosibirsk National Research State University, 630090 Novosibirsk, Russia
БИОХИМИЯ том 88 вып. 11 2023
2250
ПОПОВ и др.
The nucleotide excision repair (NER) system is responsible for removing a wide range of bulky damages
from DNA, making a significant contribution to maintaining the stability of the genome. The efficiency
with which the proteins of the NER system recognize and remove bulky damages depends on many fac-
tors and has important clinical and diagnostic significance. The review examines the current understanding
of the molecular basis of the functioning of the NER system in eukaryotic cells, as well as analyzes the
methods and approaches that are used to study the efficiency functioning of this DNA repair system both
in vitro and ex vivo.
Keywords: nucleotide excision repair, DNA damages, methods for determining NER activity
БИОХИМИЯ том 88 вып. 11 2023