БИОХИМИЯ, 2023, том 88, вып. 12, с. 2446 - 2456
УДК 577.12
ТОПОЛОГИЯ ПОЛИУБИКВИТИНОВЫХ ЦЕПЕЙ
В ХРОМАТОСОМНОМ ОКРУЖЕНИИ
УБИКВИТИН-ЛИГАЗЫ E3 RNF168
© 2023 А.А. Кудряева*, Л.А. Якубова, Г.А. Саратов, В.И. Владимиров,
В.М. Липкин, А.А. Белогуров
Институт биоорганической химии имени академиков М.М. Шемякина и Ю.А. Овчинникова РАН,
117997 Москва, Россия; электронная почта: anna.kudriaeva@gmail.com
Поступила в редакцию 03.05.2023
После доработки 19.07.2023
Принята к публикации 20.09.2023
Убиквитин-лигаза E3 RNF168 суперсемейства RING является важнейшим компонентом комплек-
са, осуществляющего убиквитинирование гистонов H2A/H2AX вблизи двуцепочечных разрывов
ДНК, что является ключевой стадией привлечения факторов репарации к месту повреждения.
В настоящей работе нами однозначно показано, что RNF168 не имеет природной способности не-
посредственно различать архитектуру полиубиквитиновых цепей, за исключением тропности двух
её убиквитин-связывающих доменов UDM1/2 к убиквитинам, соединённым через аминокислот-
ный остаток К63. Анализ внутриклеточного хроматосомного окружения полноразмерной RNF168
и её доменов методом лиганд-индуцированного резонансного переноса биолюминесценции вы-
явил, что C-концевая часть UDM1 ассоциирована с К63-полиубиквитиновыми цепями; RING
и N-концевая часть UDM2 стерически сближены с K63- и К48-убиквитиновыми цепями, в то вре-
мя как C-концевая часть UDM1 колокализована со всеми возможными вариантами убиквитина.
Полученные результаты совместно с имеющимися структурными данными позволяют выдви-
нуть гипотезу, что C-концевая часть UDM1 связывает К63-полиубиквитиновые цепи на линкер-
ном гистоне Н1; RING и N-концевая часть UDM2 находятся в центральной части нуклеосомы
и сближены с H1 и К48-убиквитинированными альтернативными субстратами RNF168, возможно,
деметилазами JMJD2A/В, в то время как C-концевая часть UDM1, вероятно, находится в обла-
сти активированного остатка убиквитина, связанного с убиквитин-лигазой Е2 в составе комплекса
с RNF168. Резюмируя, наши данные могут объяснить наличие большого количества протяжён-
ных неструктурированных участков в RNF168, которые потенциально необходимы этой Е3-лигазе
для корректного позиционирования доменов UDM1/2 с целью многоточечного взаимодействия
со множеством партнёров в её динамическом хроматосомном окружении.
КЛЮЧЕВЫЕ СЛОВА: убиквитин, убиквитиновые цепи, убиквитин-лигаза, гистоны, репарация ДНК,
двуцепочечные разрывы ДНК, посттрансляционные модификации, NanoBRET.
DOI: 10.31857/S0320972523120084, EDN: NKVSRR
ВВЕДЕНИЕ
фрагментации хромосом и неминуемой гибели
клеток [3]. Существует два основных пути вос-
В каждой клетке высокоорганизованных
становления DSB: негомологичное соедине-
многоклеточных организмов ежесекундно про-
ние концов (NHEJ) и гомологичная рекомби-
исходят десятки событий, повреждающих ДНК.
нация (HR). Независимо от пути репарации,
Отсутствие репарации этих повреждений может
хроматин, окружающий место разрыва, должен
привести к геномной нестабильности и зло-
быть ремоделирован, чтобы обеспечить доступ
качественной трансформации [1, 2]. Двуцепо-
для белков репарации ДНК [4]. Ремоделиро-
чечные разрывы ДНК (DSB) являются одними
вание хроматина играет важную роль в функ-
из наиболее опасных повреждений, поскольку
ционировании всех клеточных процессов с
их неправильная репарация может привести к участием ДНК, включая транскрипцию, репли-
Принятые сокращения: ППР - поверхностный плазмонный резонанс; DSB - двуцепочечные разрывы ДНК; HR -
гомологичная рекомбинация; LR - мотив, содержащий дипептид лейцин-аргинин; NanoBRET - биолюминесцентный
резонансный перенос энергии с использованием люциферазы NanoLuc; NHEJ - негомологичное соединение концов.
* Адресат для корреспонденции.
2446
УБИКВИТИНОВЫЕ ЦЕПИ В ХРОМАТОСОМНОМ ОКРУЖЕНИИ RNF168
2447
кацию ДНК и репарацию ДНК, и состоит в со-
чению белка
53BP1, который способствует
гласованной передаче сигналов, обусловленной
дальнейшей репарации DSB по пути NHEJ, в
посттрансляционными модификациями гисто-
то время как опосредованное комплексом бел-
нов, такими как фосфорилирование, метили-
ков BRCA1-BARD1 моноубиквитинирование
рование, ацетилирование и убиквитинирова-
H2A по K125/K127 или K129 способствует за-
ние [5, 6].
мещению 53BP1 на BRCA1 и последующей ре-
Убиквитинирование - ковалентное при-
зекции концов ДНК, что является первым эта-
соединение убиквитина к субстратам - осу-
пом HR [14]. Поскольку все процессы требуют
ществляется при помощи системы, состоящей
тонкой временной и пространственной на-
из трёх ферментов: E1 (убиквитин-активи-
стройки, множество убиквитин-лигаз и DUB
рующий фермент), E2 (убиквитин-конъюги-
задействовано в передаче сигналов о повреж-
рующий фермент) и убиквитин-лигазы E3.
дении ДНК для обеспечения эффективной и
ε-Аминогруппы семи аминокислотных остат-
точной репарации DSB [15].
ков лизина (K6, K11, K27, K29, K33, K48, K63),
Убиквитин-лигаза E3 RNF168 - белок с
входящих в состав убиквитина, позволяют
ядерной локализацией длиной 571 а.о., явля-
ему образовывать изопептидные связи [7, 8].
ется одним из основных участников в пере-
Как правило, первый убиквитин присоеди-
даче сигнала при DDR (DNA damage response).
няется к остатку лизина, входящему в состав
Уровень RNF168 строго контролируется бел-
субстрата, с помощью C-концевого остатка
ками TRIP12 и UBR5, чтобы предотвратить
глицина (G76). Дальнейший рост цепи об-
чрезмерное усиление сигнала убиквитиниро-
условлен образованием изопептидных связей
вания [16]. Считается, что RNF168 способству-
между внутренними остатками лизинов уже
ет сборке K27-, K48- и K63-цепей, а также
встроенного убиквитина и C-концевым остат-
моноубиквитинированию [14, 17, 18]. Функцио-
ком глицина нового убиквитина. Также воз-
нальные домены RNF168 достаточно подроб-
можно образование цепи через остаток M1
но охарактеризованы: в N-концевой части
убиквитина. Параметры цепей крайне разно-
располагается домен RING, ответственный за
образны: они могут быть как гомогенными, то
убиквитин-лигазную активность; в середине
есть образовывать связи через остатки лизи-
располагается домен UDM1 (Ub-dependent DSB
нов в строго определённом положении, так и
recruitment module) и в C-концевой части -
гетерогенными - комбинировать разные типы
UDM2 (рис. 1). Считается, что домены UDM1
связей. Последние, в свою очередь, могут раз-
и UDM2 отвечают за распознавание убикви-
ветвляться посредством убиквитинирования
тиновых цепей, в первую очередь на гисто-
сразу по нескольким сайтам. Считается, что
нах вблизи двуцепочечных разрывов ДНК, и
значение сигнала убиквитинирования зави-
таким образом способствуют привлечению
сит от типа связи и длины убиквитиновой
RNF168 к месту повреждения [6]. Функцио-
цепи [9]. Удаление этих сигналов осуществля-
нальные домены RNF168 весьма консерватив-
ется с помощью деубиквитинирующих фер-
ны среди позвоночных, гомология составляет
ментов (DUB). Функциональные роли убик-
от 35 до 95% [19].
витиновых цепей, образованных через остатки
На настоящий момент имеются довольно
лизина K48 и K63, достаточно хорошо изуче-
противоречивые данные о структуре убиквити-
ны. K48-Убиквитинирование считается сиг-
новых цепей, способных распознаваться пол-
налом протеасомной деградации, тогда как
норазмерной RNF168 и ее функциональными
K63-убиквитинирование обеспечивает белок-
доменами. В состав домена UDM1 входят три
белковые взаимодействия в различных про-
мотива: LRM1 (мотив LR, содержащий дипеп-
цессах, в том числе при репарации ДНК [10, 11].
тид лейцин-аргинин), UMI (Ub-interacting
Помимо важности K11 и M1 убиквитиновых
motif [UIM] and MIU-related UBD) и MIU1
связей в регуляции клеточного цикла и акти-
(motif interacting with Ub). UDM2 содержит
вации NF-κB [12, 13], мало что известно об
UAD (ubiquitin-associated domain), MIU2 и
убиквитин-лигазах, рецепторах убиквитина и
мотив LRM2 (рис. 1). Считается, что убикви-
функциях других неканонических цепей убик-
тин-лигаза E3 RNF168 распознает убиквити-
витина (K6, K27, K29, K33).
нированные белки с помощью трёх различ-
Одной из основных функций убиквити-
ных убиквитин-связывающих мотивов: MIU1,
нирования в репарации DSB является под-
MIU2 и UMI. Мотив UMI домена UDM1 пред-
держание баланса между NHEJ и HR. Моно-
почтительно связывается с K63 убиквитино-
убиквитинирование гистонов H2A и H2AX
выми цепями. В то же время мутация моти-
по аминокислотным остаткам K13/K15 убик-
ва MIU1 резко снижает связывание RNF168
витин-лигазой RNF168 содействует привле-
с K48-цепями убиквитина [20]. Другое иссле-
БИОХИМИЯ том 88 вып. 12 2023
2448
КУДРЯЕВА и др.
Рис. 1. Структурная организация и функциональные домены убиквитин-лигазы Е3 RNF168
дование показало, что мотив MIU1 сам по
боте данная информация не была подтвер-
себе проявляет сходную аффинность связы-
ждена [21].
вания с моноубиквитином, K48- и K63-цепя-
Имеющиеся на настоящий момент литера-
ми убиквитина [21]. Есть предположение, что
турные данные по характеру взаимодействия
более высокой аффинности связывания UMI
RNF168 и убиквитиновых цепей ограничены
с К63-цепями, по сравнению с MIU2, способ-
экспериментами in vitro или с использованием
ствует мотив LRM1, несмотря на то что ин-
клеточных лизатов [20, 21, 23, 24]. В большин-
формация о способности его взаимодействия
стве случаев в анализе используется ограни-
с убиквитиновыми цепями отсутствует [21].
ченный набор вариантов убиквитина. Более
Считается, что консервативный мотив LR
того, анализ делетированных доменов и изо-
необходим для привлечения лигаз RNF168
лированных небольших фрагментов RNF168
и RNF169 к убиквитинированному H2A и,
может иметь лишь отдалённое сходство с фи-
возможно, способен связываться с H2A на-
зиологическими процессами в клетке. В дан-
прямую [22]. Интересно, что делеция моти-
ной работе нами проведён исчерпывающий
ва LRM2 домена UDM2, наоборот, способ-
анализ параметров внутриклеточного связы-
ствует повышению сродства данного домена
вания убиквитиновых цепей различного типа
к K63 убиквитиновым цепям [21]. В настоящее
ветвления с убиквитин-лигазой RNF168 и её
время считается, что UDM1 и UDM2 взаимо-
функциональными доменами с помощью ме-
действуют с различными K63-убиквитиниро-
тода лиганд-индуцированного резонансного
ванными мишенями: домен UDM1 способен
переноса биолюминесценции с использовани-
распознавать убиквитинированные мише-
ем люциферазы NanoLuc (NanoBRET).
ни, модифицированные убиквитин-лигазой
RNF8, тогда как домен UDM2 распознает
RNF168-опосредованные убиквитинированные
МАТЕРИАЛЫ И МЕТОДЫ
мишени [19]. Функциональное различие ме-
жду UDM1 и UDM2 может быть связано как
Получение плазмид. Генетические конструк-
с разными MIU-мотивами входящих в состав
ции, кодирующие функциональные домены
UDM, так и с консервативными LR-мотива-
убиквитин-лигазы RNF168 RING (1-110 а.о.),
ми в их составе (LRM1 - в UDM1 и LRM2 -
UDM1 (110-188 а.о.) и UDM2 (419-487 а.о.),
в UDM2) [21]. Есть данные, что полнораз-
слитные с Fc-доменом антитела IgG, а также
мерный белок RNF168 способен связываться
содержащие лидерный пептид, направляю-
K27-диубиквитином [17], однако в другой ра-
щий синтезируемый белок во внеклеточную
БИОХИМИЯ том 88 вып. 12 2023
УБИКВИТИНОВЫЕ ЦЕПИ В ХРОМАТОСОМНОМ ОКРУЖЕНИИ RNF168
2449
среду для наработки белков в клетках мле-
комплексов поверхность чипа обрабатывали
копитающих HEK293F, были получены на
регенерирующим раствором (10 мМ Gly-HCl,
основе вектора pFUSE (по сайтам рестрик-
pH 2,5) в течение 30 с при скорости потока
ции EcoRI и NheI). Генетические конструк-
30 мкл/мин. Анализ выполняли с использова-
ции, кодирующие полноразмерную убикви-
нием SPR-биосенсора Biacore 8K («Cytiva»).
тин-лигазу RNF168 и её функциональные
Для определения динамических констант дис-
домены RING, UDM1 и UDM2, слитные с
социации (Kd) диаграммы ППР анализирова-
белком HaloTag, и варианты убиквитина K0R
ли, применяя модель адсорбции Ленгмюра
К6/11/27/29/33/48/63, слитные с люциферазой
(одна молекула аналита взаимодействует с
NanoLuc и содержащие в своём составе един-
одной молекулой лиганда). Данные сенсо-
ственный функциональный остаток лизина
грамм взаимодействий обрабатывали в про-
(для использования в методе NanoBRET),
граммном комплексе Biacore Insight Evaluation
были получены на основе вектора pсDNA3
Version 3.0.11.15423.
(по сайтам рестрикции HindIII и XhoI). По-
Иммуноблоттинг. Проводили гель-электро-
следовательности RNF168 RING (1-110 а.о.),
форез белков в денатурирующих условиях (гра-
UDM1 (110-188 а.о.) и UDM2 (419-487 а.о.) и
диентный гель 4-20%) по Лэммли с использо-
полноразмерной RNF168 были амплифициро-
ванием предокрашенного белкового маркера
ваны с кДНК, полученной из клеток HEK293T.
(«Bio-Rad», США). Далее проводили перенос
Последовательности NanoLuc и HaloTag были
на нитроцеллюлозную мембрану («Bio-Rad») в
амплифицированы с плазмид pENTR4-HaloTag
течение 1 ч при силе тока 0,8 мА/см2. По окон-
(Addgene, #29644) и pcDNA3.1 NL (Addgene;
чании процесса мембрану помещали в блоки-
#113442) соответственно.
рующий раствор, содержащий 5% сухого моло-
Культивирование клеток млекопитающих.
ка в PBS, и инкубировали 1 ч. Гибридизацию
Клетки почки эмбриона человека HEK293T
с антителами anti-Flag-HRP («Sigma», США)
(ЦКП «Коллекция культур клеток позвоноч-
или anti-Strep-HRP
(«Bio-Rad») проводили
ных», Санкт-Петербург, Россия) выращивали
в конъюгатном буфере в течение 1 ч. Далее,
во флаконах для культивирования в среде
после тщательной отмывки наносили прояв-
DMEM («ПанЭко», Россия) с добавлением
ляющий раствор Clarity Western ECL Substrate
10% эмбриональной телячьей сыворотки
(«Bio-Rad») и проявляли на ChemiDoc MP
(«Hyclone, Cytiva», США) и смеси антибио-
Imaging System («Bio-Rad»).
тиков (пенициллин - 100 мкг/мл, стрептоми-
NanoBRET. Для проведения анализа ме-
цин - 100 мкг/мл, гентамицин - 50 мкг/мл)
тодом NanoBRET использовали набор Nano-
при 37 °C в атмосфере 5% CO2. Клетки HEK293F
BRET Nano-Glo («Promega», США), в соответ-
выращивали в колбах при постоянном пе-
ствии с инструкциями производителя. Клет-
ремешивании в среде Freestyle
(«Thermo
ки HEK293T при плотности ~ 3 × 105 клеток
Fisher Scientific», США) при 37 °C в атмосфе-
на лунку котрансфицировали
200 нг плаз-
ре 5% CO2.
мид NanoLuc-UbK0 K6/11/27/29/33/48/63 и
Экспрессия RNF168 и её функциональных
HaloTag-RNF168/RING/UDM1/2 или RNF168/
доменов в клетках млекопитающих HEK293F.
RING/UDM1/2-HaloTag или интактный HaloTag
Полученными плазмидами трансфицировали
в соотношении 1 : 100 с использованием ре-
линию клеток HEK293F с использованием
агента трансфекции GenJect-39
(«Molecta»,
реагента PEI MAX («Polysciences, Inc.», США)
Россия) в белых
96-луночных планшетах
и культивировали 96 ч. Очистку белков прово-
(«Greiner», Германия). Через 24 ч после транс-
дили с помощью магнитных частиц с иммоби-
фекции к клеткам добавляли лиганд HaloTag
лизованным на них белком G («Thermo Fisher
NanoBRET 618 до конечной концентрации
Scientific»).
100 нМ в среде DMEM Fluorobrite («Thermo
Поверхностный плазмонный резонанс (ППР).
Fisher Scientific»). Еще через 16 ч сигнал Nano-
На оптическом чипе СМ5 («Cytiva», США) с
BRET измеряли через 5 мин после добавления
применением протокола NHS-EDC иммоби-
субстрата NanoLuc фуримазина в концентра-
лизовали диубиквитины, связанные через раз-
ции 10 мкМ с использованием микроплан-
личные аминокислотные остатки лизина - K6,
шетного ридера Tecan Spark Сyto 600 («Tecan»,
K11, K27, K29, K33, K48, K63 («UbiQ», Нидер-
Германия) при 450/480 нм BP (люминесцен-
ланды). Производили инжекцию серии образ-
ция донора) и
610 нм LP (флуоресценция
цов белка-аналита (домены убиквитин-лига-
акцептора). Соотношения NanoBRET рас-
зы RNF168) в возрастающей концентрации
считывали как частное
610 нм LP (испус-
(1-15 мкМ) на скорости 10 мкл/мин. После
кание)/(450/480) нм BP (испускание) × 1000.
регистрации диссоциации межмолекулярных
Скорректированное соотношение NanoBRET
7
БИОХИМИЯ том 88 вып. 12 2023
2450
КУДРЯЕВА и др.
получали путём вычитания сигнала контро-
вратной заменой R→K), слитные на N-конце
ля, не содержащего акцепторного лиганда, а
с люциферазой NanoLuc. Модифицированный
затем дополнительно проводили нормировку
таким образом убиквитин имеет аналогичные
на сигнал NanoBRET в присутствии интакт-
способности образовывать полиубиквитино-
ного HaloTag.
вые цепи по сравнению с убиквитином, флан-
кированным N-концевым FLAG-эпитопом
(рис. 3, а). Дополнительно были созданы гене-
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
тические конструкции, кодирующие полно-
размерную RNF168, а также её функциональ-
Домены RNF168 обладают ограниченной
ные домены UDM1/2 и RING, слитные либо с
способностью распознавать диубиквитины раз-
N-, либо с C-конца с белком HaloTag и содер-
личной топологии in vitro. Рекомбинантные до-
жащие Strep-эпитоп (рис. 3, б).
мены UDM1/2 и RING, слитные с констант-
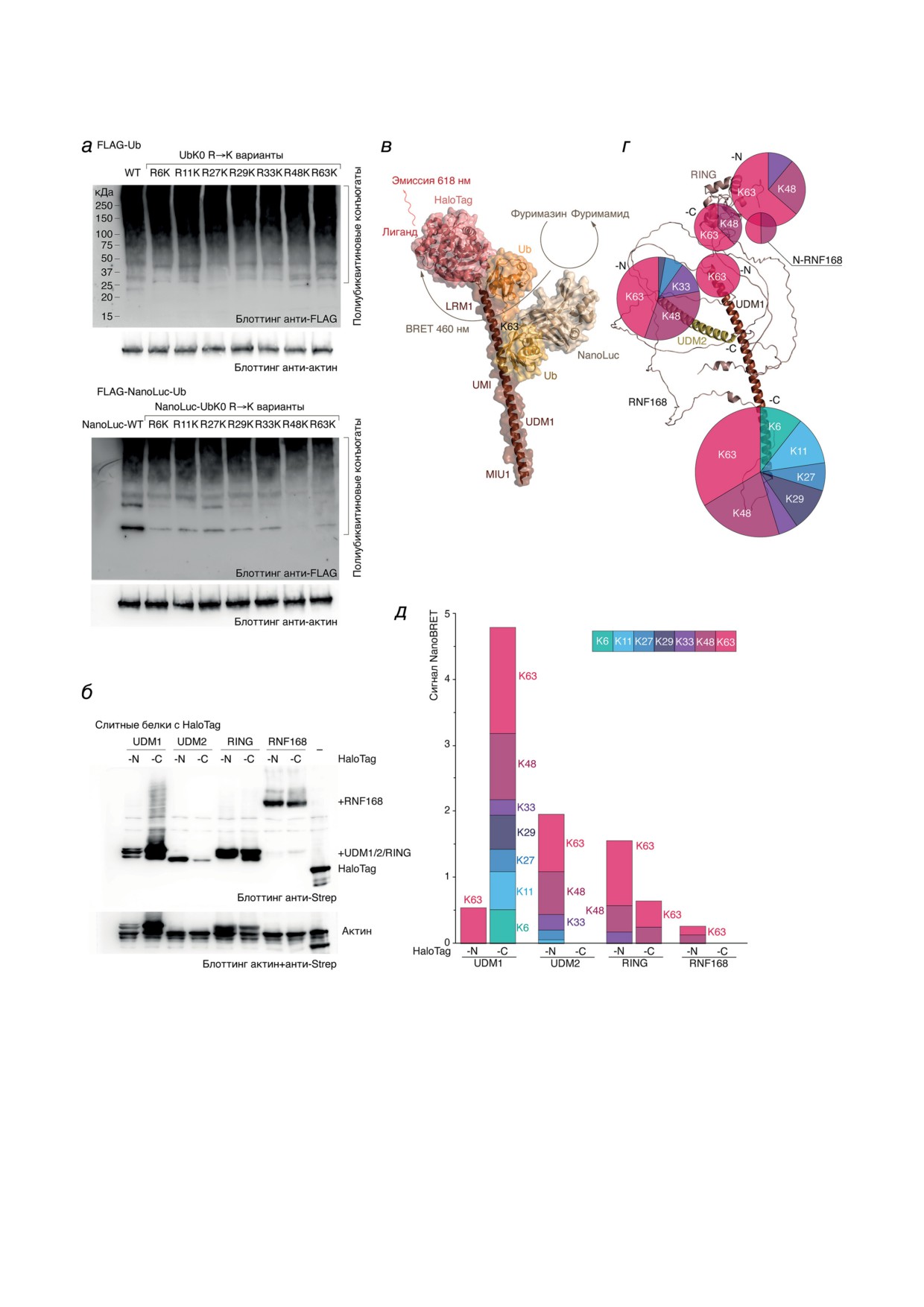
Наличие резонансного переноса биолюми-
ным фрагментом антитела человека, были
несценции в случае коэкспрессии соответству-
получены в клетках млекопитающих HEK293F.
ющих пар означает стерическую сближенность
Анализ взаимодействия очищенных функцио-
определённых доменов RNF168 и полиубикви-
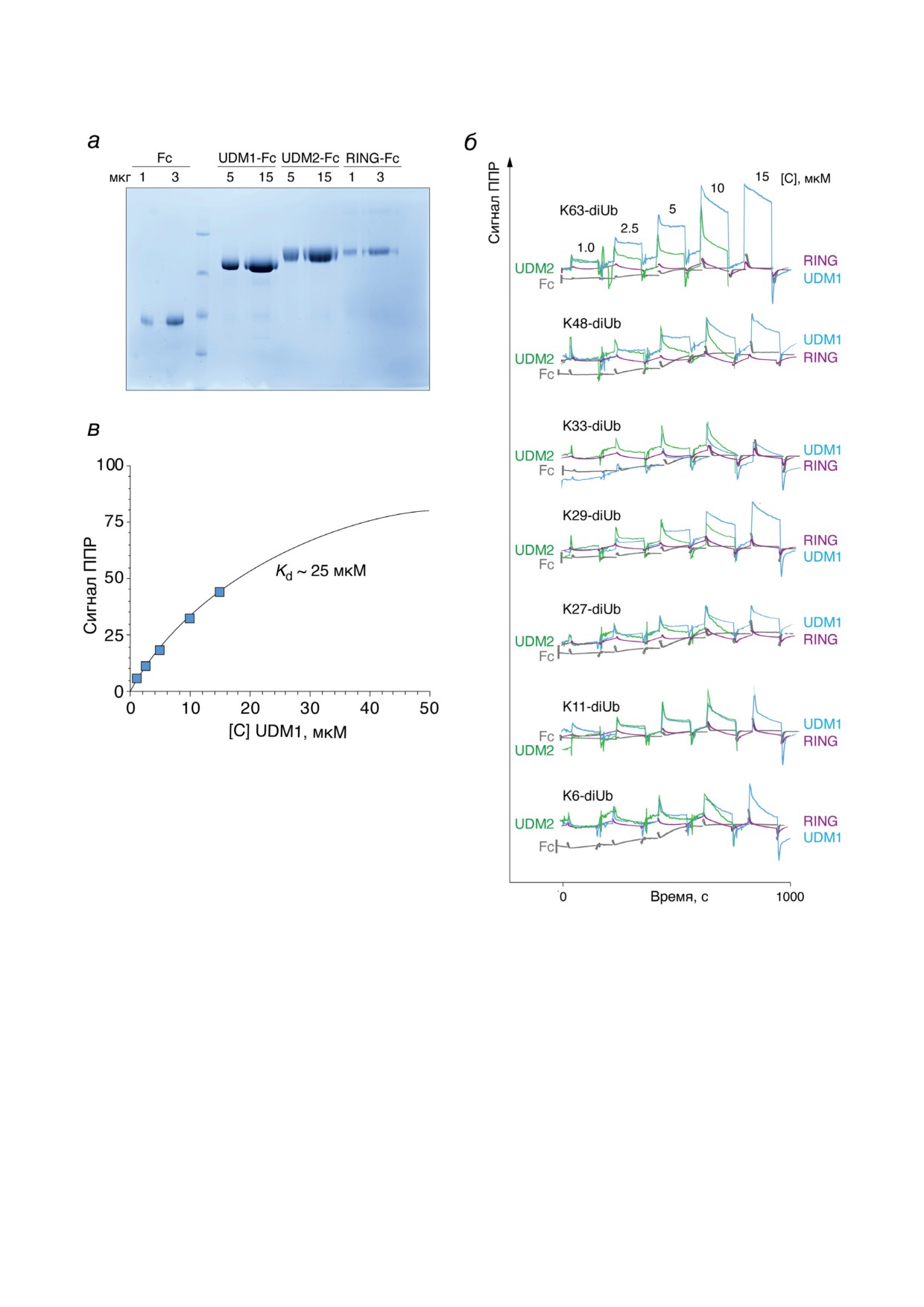
нальных доменов RNF168 (рис. 2, а) мето-
тиновых цепей определённого типа ветвле-
дом ППР, представленный на рис. 2, б, сви-
ния (рис. 3, в). Анализ уровня внутриклеточной
детельствует об отсутствии значимого уровня
флуоресценции котрансфецированных клеток
их связывания с диубиквитинами с различ-
(рис. 3, г и д) выявил повышенную ассоциа-
ным типом изопептидной связи за исклю-
цию N-концевой части домена UDM1 с К63-
чением домена UDM1 и диубиквитина, свя-
убиквитиновым цепям. Полноразмерная ли-
занного через аминокислотный остаток K63
газа RNF168, слитная с HaloTag с N-конца,
(Kd ~ 25 мкМ) (рис. 2, в). Данное наблюдение
а также RING-домен продемонстрировали
коррелирует с имеющимися литературными
преимущественную сближенность с убик-
данными [20], где было продемонстрировано
витиновыми цепями, образованными через
связывание diUb63 с транкированным вари-
аминокислотные остатки К63 и К48. Анализ эф-
антом домена UDM1 - LRM1-UMI, слит-
фективности NanoBRET показал, что C-кон-
ным с GST (Kd ~ 45 мкМ). Полученные сенсо-
цевая часть домена UDM1 взаимодействует со
граммы однозначно свидетельствуют о низкой
всеми возможными убиквитиновыми цепями с
аффинности (Kd >> 100 мкМ) всех остальных
некоторой повышенной тропностью к К48- и
проанализированных пар, в том числе к ди-
К63-цепям. N-Концевая часть UDM2-домена
убиквитинам, образованным через аминокис-
также имела повышенное сродство к поли-
лотные остатки К27 и K48.
убиквитиновым цепям, образованным через
Хроматосомное окружение Е3-лигазы
аминокислотные остатки К63 и К48, и менее
RNF168, выявленное методом лиганд-индуциро-
выраженное - к К33-убиквитиновым цепям.
ванного резонансного переноса биолюминесцен-
C-Концевые части домена UDM2 и полнораз-
ции. Относительно недавно была разработана
мерной RNF168 не показали какого-либо до-
новая система анализа внутриклеточных взаи-
стоверного сродства к убиквитиновым цепям.
модействий на основе биолюминесцентного
В случае UDM2-HaloTag это, возможно, свя-
резонансного переноса энергии - BRET-пара
зано с низким уровнем экспрессии данного
NanoLuc (460 нм) и HaloTag (618 нм) с расстоя-
слитного белка, выявленного по результатам
нием между пиками эмиссии и поглощения
иммуноблоттинга (рис. 3, б). Дополнительным
в 175 нм [25], названная авторами NanoBRET.
фактором отсутствия сигнала может быть на-
Такой разрыв в максимумах свечения дает
личие слитного белка именно на C-конце
высокую чувствительность и низкий фоновый
UDM2, радикально влияющего на функцио-
сигнал. Кроме того, данная система позволяет
нал этого домена RNF168, как это было нами
анализировать мультисубъединичные комплек-
показано в случае in vitro анализа (рис. 2, б).
сы и взаимодействие крупных белков благо-
даря относительно большому для таких систем
Фёрстеровскому радиусу, равному 6,97 нм, а
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
экспериментально установленные рабочие ди-
станции находятся в диапазоне 3-10 нм [26].
Полноразмерный домен UDM1 (LRM-
В этой связи нами были созданы генетические
UMI-MIU1) показал достоверное сродство
конструкции, кодирующие варианты убикви-
к K63-убиквитиновым цепям в in vitro систе-
тина только с одним функциональным остат-
ме (Kd ~ 25 мкМ), в отличие от домена UDM2
ком лизина (так называемые UbK0 с одной воз-
(UAD-MIU2-LRM2), для полноразмерного
БИОХИМИЯ том 88 вып. 12 2023
УБИКВИТИНОВЫЕ ЦЕПИ В ХРОМАТОСОМНОМ ОКРУЖЕНИИ RNF168
2451
Рис. 2. Анализ взаимодействия доменов RNF168 c диубиквитинами методом поверхностного плазмонного резонан-
са (ППР). а - Электрофореграмма очищенных белковых препаратов Fc и доменов RNF168 - UDM1/2 и RING, слит-
ных с Fc. б - Анализ взаимодействия различных функциональных доменов RNF168 (UDM1, UDM2, RING) в различ-
ной концентрации (1-15 мкМ) с диубиквитинами с разным вариантом изопептидной связи (K6/K11/K27/K29/K33/
K48/K63) методом ППР. в - Кривая титрования, отражающая взаимодействие между иммобилизованным на чипе
K63-диубиквитином с UDM1-Fc
и транкированного варианта (UAD-MIU2)
вой части UDM2 приводит к уменьшению со-
которого, слитного с N-конца c GST, также
ответствующего Kd [21].
ранее было показано связывание с K63-цепя-
Метод NanoBRET широко используется
ми [21]. Возможно, присоединение Fc-фраг-
в биохимических исследованиях, так как он
мента к C-концевой части домена UDM2 дра-
позволяет изучать взаимодействия белков в
матическим образом повлияло на способность
более естественных условиях в сравнении с
UDM2 распознавать K63-цепи, учитывая тот
искусственными in vitro системами. Это делает
факт, что удаление мотива LRM2 в C-конце-
возможным выявить наиболее реалистичный
БИОХИМИЯ том 88 вып. 12 2023
7*
2452
КУДРЯЕВА и др.
Рис. 3. Анализ убиквитинового окружения убиквитин-лигазы Е3 RNF168 методом NanoBRET. а - Блоттинг
лизатов клеток HEK293, трансфицированных кДНК, кодирующей FLAG-Ub и варианты FLAG-UbK0 R→K
6/11/27/29/33/48/63, а также NanoLuc-Ub и варианты NanoLuc-UbK0 R→K 6/11/27/29/33/48/63, с использованием
антител против FLAG-эпитопа и актина. б - Блоттинг клеток HEK293, трансфицированных кДНК, кодирующей
полноразмерную RNF168, а также её функциональные домены UDM1/2 и RING, слитные либо с N-, либо с C-конца
с белком HaloTag. в - Схематическое представление принципа анализа убиквитинового окружения убиквитин-лига-
зы RNF168 методом NanoBRET. Использованы cтруктуры домена UDM1 с K63 diUb (PDB 5XIS), люциферазы nLuc
(PDB 7SNS) и белка HaloTag, связанного с лигандом тетраметилродамином (PDB 6U32). г и д - Диаграмма сближен-
ности убиквитиновых цепей различной топологии с функциональными доменами убиквитин-лигазы RNF168 (пред-
положительная структура на основе предсказания нейросети AlphaFold; идентификатор AF-Q8IYW5-F1), построен-
ной по результатам NanoBRET. Площадь круга на панели (г) соответствует сумме всех сигналов NanoBRET (высоте
столбика) на панели (д). Значения сигнала NanoBRET на панели (д) нормализовано на сигнал белка HaloTag без
доменов RNF168, коэкспрессированного с соответствующим вариантом убиквитина
БИОХИМИЯ том 88 вып. 12 2023
УБИКВИТИНОВЫЕ ЦЕПИ В ХРОМАТОСОМНОМ ОКРУЖЕНИИ RNF168
2453
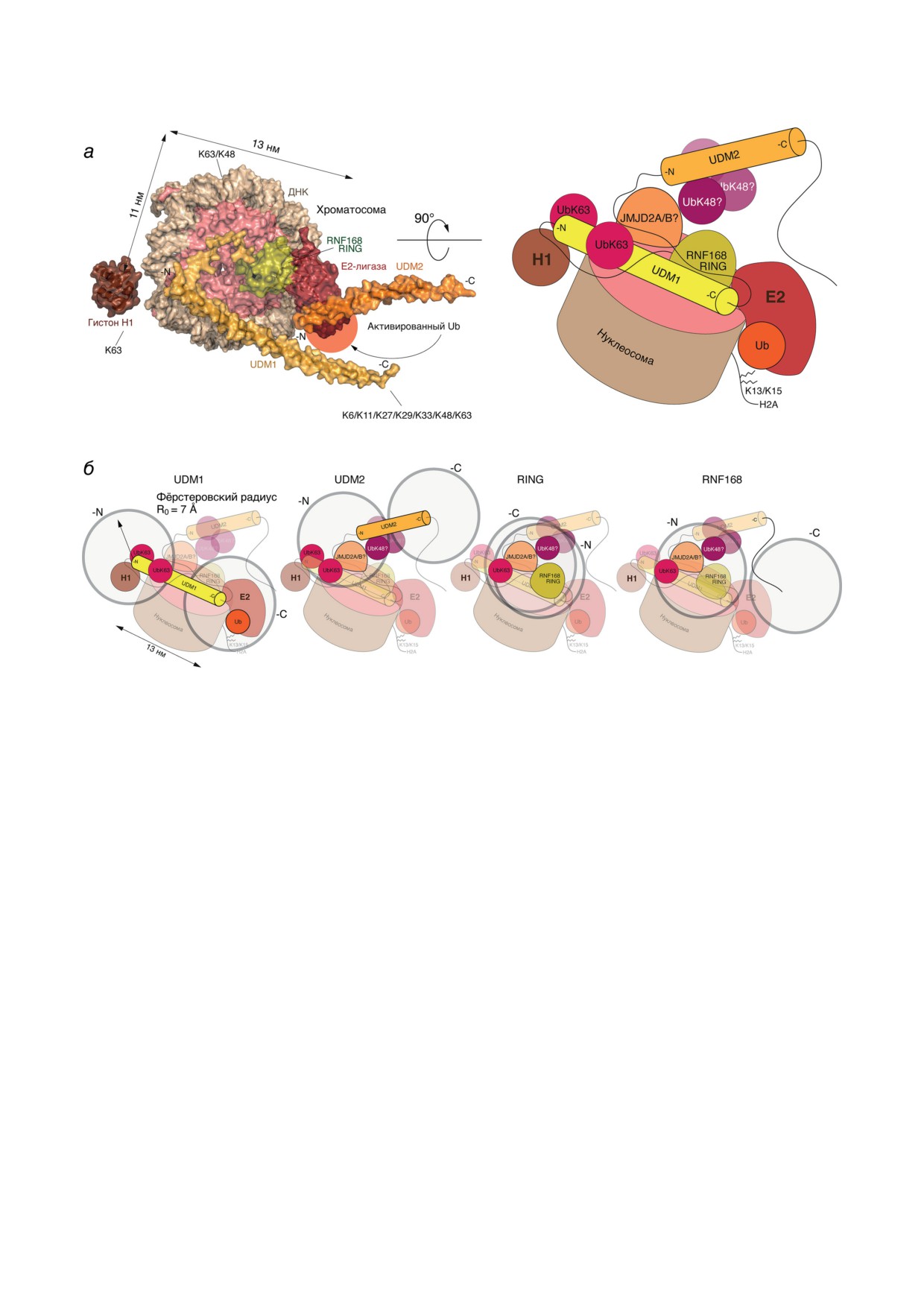
Рис. 4. Предположительная конфигурация убиквитинового окружения лигазы RNF168 в контексте хроматосомы.
а - Левая модель выполнена на основе предложенной структуры комплекса RING-домена убиквитин-лигазы RNF168
(оливковый) с нуклеосомой (розовый и серый) и убиквитин-лигазой UbcH5c (красный) (PDBDEV_00000029, [24]).
В структуру дополнительно добавлены домены UDM1 (желтый) и UDM2 (золотой) лигазы RNF168, а также глобу-
лярный домен гистона H1B из структуры 5NL0 [27]. Указаны положения активированного убиквитина (тёмно-оран-
жевый) и зоны локализации вариантов убиквитинов по результатам NanoBRET. Правая модель схематически допол-
нена полиубиквитиновыми цепями, а также деметилазой JMJD2A/B. б - Возможные конформации расположения
полноразмерной RNF168, а также её функциональных доменов UDM1/2 и RING, слитных либо с N-, либо с C-конца
с белком HaloTag в контексте хроматосомы с учетом Фёрстеровского радиуса сигнала NanoBRET
сценарий взаимодействия белков, что улучшает
ния системы NanoBRET (3-10 нм), можно го-
понимание многих биологических процессов.
ворить о том, что наблюдаемые нами сигналы
Данный метод также может использоваться
являются достоверными.
для ex vivo скрининга потенциальных лекар-
На основании наших данных и предложен-
ственных препаратов, которые могут влиять
ной на настоящий момент структуры комплек-
на белок-белковые взаимодействия. В целом,
са нуклеосомы и RING-домена RNF168 [23],
на настоящий момент метод NanoBRET явля-
можно предположить, что UDM1-домен RNF168
ется одним из наиболее мощных инструмен-
располагается параллельно спиралям цепи
тов для изучения белковых взаимодействий
ДНК вокруг нуклеосомы, пересекая её от од-
в живых клетках. В этой связи мы решили
ного полюса до другого (рис. 4, а и б).
использовать метод NanoBRET для изучения
N-Концевая часть домена UDM1 содер-
внутриклеточного убиквитинового окружения
жит мотивы LRM1 и UMI, в C-концевой ча-
убиквитин-лигазы E3 RNF168. Полученные
сти расположен мотив MIU1. Исходно счита-
данные потенциально могли бы свидетель-
лось, что LRM1 распознает RNF8-опосредо-
ствовать о способности RNF168 распознавать
ванно убиквитинированные белки [28]. Даль-
те или иные цепи, а также о топологии окружа-
нейшие исследования показали, что LRM1 в
ющих её цепей. Учитывая линейные размеры
UDM1 может взаимодействовать с линкер-
нуклеосомы (рис. 4, а) [27] и рабочие расстоя-
ным гистоном H1 [29]. Вместе с тем показано,
БИОХИМИЯ том 88 вып. 12 2023
2454
КУДРЯЕВА и др.
что лигаза RNF169, содержащая данный мотив,
и К48-содержащие полиубиквитиновые цепи),
с H1 не взаимодействует, что свидетельствует
что соотносится с N-концевой позицией это-
о неполном понимании функционала LRМ1.
го домена в структуре RNF168. C-Концевая
Существуют данные, что домен UDM1 ли-
часть, по-видимому, сильно удалена от убик-
газы RNF168, содержащий как LRM1, так
витиновых цепей, так как сигнал NanoBRET
и UMI, проявляет более сильную аффинность
в этом случае отсутствует.
к убиквитиновым цепям, образованным через
остаток K63, чем только UMI [21]. Данное на-
блюдение указывает на то, что LRM1 может
ЗАКЛЮЧЕНИЕ
иметь вспомогательную роль при взаимодей-
ствии RNF168 с K63-цепями, это полностью
Использование метода NanoBRET по-
согласуется с полученными нами данными
зволило впервые изучить внутриклеточное
о высокой специфичности N-концевой ча-
убиквитиновое окружение убиквитин-лигазы
сти UDM1 именно к K63-цепям, вероятно,
E3 RNF168, одного из ключевых участников
расположенным на гистоне H1 [20] (рис. 4).
репарации двуцепочечных разрывов ДНК.
C-Концевая часть UDM1, которая, по нашим
Предполагается, что с гистоном H1, модифи-
данным, распознает все виды убиквитино-
цированным К63-убиквитиновыми цепями,
вых цепей, скорее всего, расположена вблизи
связывается N-концевая часть домена UDM1,
E2-лигазы, удерживаемой RNF168, которая
в то время как его C-концевая часть находится
физически переносит убиквитин на N-конце-
диаметрально противоположно вблизи активи-
вую часть Н2А/Н2АХ (рис. 4). Вероятнее всего,
рованного остатка моноубиквитина, связан-
сигнал NanoBRET, который мы фиксируем с
ного тиоэфирной связью с убиквитин-лига-
конструкцией UDM1-HaloTag, - это тиоэфир-
зой Е2, удерживаемой RNF168. RING-домен,
ный конъюгат любого из вариантов убиквити-
как и N-концевая часть UDM2, находятся в
на, слитного с NanoLuc, и Е2.
центральной части нуклеосомы и, предпо-
N-Концевая часть домена UDM2, исходя
ложительно, стерически сближены с K48- и
из наших данных, более подвижна и может
К63-убиквитиновыми цепями, находящими-
быть расположена как поблизости от N-кон-
ся на линкерном гистоне Н1 и деметилазах
цевой части RING, рядом с К63-убиквитини-
JMJD2A/B. Полученные данные могут объяс-
рованным Н1, так и находиться ортогонально
нить наличие большого количества протяжён-
и выше UDM1 вблизи K48-цепей, связанных с
ных неструктурированных участков в RNF168,
альтернативными субстратами RNF168. В слу-
которые потенциально необходимы этой
чае C-концевой локализации HaloTag нам не
Е3-лигазе для корректного позиционирова-
удалось зафиксировать достоверного сигнала
ния доменов UDM1/2 с возможностью много-
NanoBRET, что, возможно, связно с удалённо-
точечного взаимодействия со множеством её
стью C-концевой части UDM2 от убиквитино-
хроматосомных партнёров и динамического
вых цепей. Также это наблюдение может быть
переключения между ними.
объяснено низким уровнем экспрессии дан-
ного слитного белка или нарушением функ-
Вклад авторов. А.А. Кудряева, А.А. Бело-
ционала UDM2 в целом вследствие наличия
гуров - концепция и руководство работой;
HaloTag на C-конце.
А.А. Кудряева, Л.А. Якубова, Г.А. Саратов,
RING-домен, обусловливающий сближе-
В.И. Владимиров - проведение эксперимен-
ние субстрата и E2-лигазы, предположитель-
тов; А.А. Кудряева, В.М. Липкин, А.А. Бело-
но, расположен в центральной части нуклео-
гуров - обсуждение результатов исследования;
сомы. Он напрямую взаимодействует с основ-
А.А. Кудряева, А.А. Белогуров
- написание
ным субстратом убиквитин-лигазы RNF168 -
текста; А.А. Кудряева, А.А. Белогуров - ре-
гистоном H2A [30], поэтому в относитель-
дактирование текста статьи.
ной близости от RING могут находиться
Финансирование. Работа выполнена при
K63-убиквитиновые цепи, расположенные
финансовой поддержке Российского научного
на гистонах H1 или H2A, либо, предположи-
фонда (грант № 21-74-10154).
тельно, K48-цепи на деметилазах JMJD2A
Конфликт интересов. Авторы заявляют об
и JMJD2B, которые также являются субстра-
отсутствии конфликта интересов.
тами RNF168 [31].
Соблюдение этических норм. Настоящая
Характер убиквитинового окружения
статья не содержит описания каких-либо ис-
N-концевой части полноразмерной RNF168
следований с участием людей или животных
схож с RING-доменом (преимущественно К63-
в качестве объектов.
БИОХИМИЯ том 88 вып. 12 2023
УБИКВИТИНОВЫЕ ЦЕПИ В ХРОМАТОСОМНОМ ОКРУЖЕНИИ RNF168
2455
СПИСОК ЛИТЕРАТУРЫ
1.
Khanna, K. K., and Jackson, S. P. (2001) DNA
14.
Uckelmann, M., and Sixma, T. K. (2017) Histone
double-strand breaks: Signaling, repair and the
ubiquitination in the DNA damage response,
cancer connection, Nat. Genet.,
27,
247-254,
DNA Repair (Amst),
56,
92-101, doi:
10.1016/
doi: 10.1038/85798.
j.dnarep.2017.06.011.
2.
Jackson, S. P., and Bartek, J. (2009) The DNA-
15.
Nishi, R. (2017) Balancing act: To be, or not to
damage response in human biology and disease,
be ubiquitylated, Mutat. Res.,
803-805,
43-50,
Nature, 461, 1071-1078, doi: 10.1038/nature08467.
doi: 10.1016/j.mrfmmm.2017.07.006.
3.
Ceccaldi, R., Rondinelli, B., and D’Andrea, A. D.
16.
Gudjonsson, T., Altmeyer, M., Savic, V., Toledo, L.,
(2016) Repair pathway choices and consequences at
Dinant, C., Grøfte, M., Bartkova, J., Poulsen, M.,
the double-strand break, Trends Cell Biol., 26, 52-64,
Oka, Y., Bekker-Jensen, S., Mailand, N., Neumann, B.,
doi: 10.1016/j.tcb.2015.07.009.
Heriche, J. K., Shearer, R., Saunders, D., Bartek, J.,
4.
Price, B. D., and D’Andrea, A. D. (2013) Chromatin
Lukas, J., and Lukas, C. (2012) TRIP12 and UBR5
remodeling at DNA double-strand breaks, Cell, 152,
suppress spreading of chromatin ubiquitylation at dam-
1344-1354, doi: 10.1016/j.cell.2013.02.011.
aged chromosomes, Cell, 150, 697-709, doi: 10.1016/
5.
Kim, J. J., Lee, S. Y., and Miller, K. M. (2019) Pre-
j.cell.2012.06.039.
serving genome integrity and function: the DNA dam-
17.
Gatti, M., Pinato, S., Maiolica, A., Rocchio, F.,
age response and histone modifications, Crit. Rev. Bio-
Prato, M. G., Aebersold, R., and Penengo, L. (2015)
chem. Mol. Biol., 54, 208-241, doi: 10.1080/10409238.
RNF168 promotes noncanonical K27 ubiquitination
2019.1620676.
to signal DNA damage, Cell Rep., 10, 226-238,
6.
Bacheva, A. V., Gotmanova, N. N., Belogurov, A. A.,
doi: 10.1016/j.celrep.2014.12.021.
and Kudriaeva, A. A. (2021) Control of genome
18.
Kudriaeva, A. A., Lipkin, V. M., and Belogurov,
through variative nature of histone-modifying
A. A. (2020) Topological features of histone H2A
ubiquitin ligases, Biochemistry (Moscow), 86, S71-S95,
monoubiquitination, Dokl. Biochem. Biophys., 493,
doi: 10.1134/S0006297921140066.
193-197, doi: 10.1134/S1607672920040079.
7.
Ciechanover, A.
(2015) The unravelling of the
19.
Kelliher, J., Ghosal, G., and Leung, J. W. C. (2022)
ubiquitin system, Nat. Rev. Mol. Cell. Biol., 16, 322-
New answers to the old RIDDLE: RNF168 and
324, doi: 10.1038/nrm3982.
the DNA damage response pathway, FEBS J., 289,
8.
Kudriaeva, A. A., and Belogurov, A. A. (2019) Pro-
2467-2480, doi: 10.1111/febs.15857.
teasome: a nanomachinery of creative destruction,
20.
Pinato, S., Gatti, M., Scandiuzzi, C., Confalonieri, S.,
Biochemistry (Moscow), 84, 159-192, doi: 10.1134/
and Penengo, L.
(2011) UMI, a novel RNF168
S0006297919140104.
ubiquitin binding domain involved in the DNA
9.
Kudriaeva, A. A., Livneh, I., Baranov, M. S.,
damage signaling pathway, Mol. Cell. Biol., 31, 118-
Ziganshin, R. H., Tupikin, A. E., Zaitseva, S. O.,
126, doi: 10.1128/mcb.00818-10.
Kabilov, M. R., Ciechanover, A., and Belogurov,
21.
Takahashi, T. S., Hirade, Y., Toma, A., Sato, Y.,
A. A. Jr. (2021) In-depth characterization of ubiquitin
Yamagata, A., Goto-Ito, S., Tomito, A., Nakada, S.,
turnover in mammalian cells by fluorescence track-
and Fukai, S.
(2018) Structural insights into
ing, Cell. Chem. Biol., 28, 1192-1205, doi: 10.1016/
two distinct binding modules for Lys63-linked
j.chembiol.2021.02.009.
polyubiquitin chains in RNF168, Nat. Commun., 9,
10.
Chen, Z. J., and Sun, L. J. (2009) Nonproteolytic
170, doi: 10.1038/s41467-017-02345-y.
functions of ubiquitin in cell signaling, Mol. Cell, 33,
22.
Kitevski-LeBlanc, J., Fradet-Turcotte, A., Kukic, P.,
275-286, doi: 10.1016/j.molcel.2009.01.014.
Wilson, M. D., Portella, G., Yuwen, T., Panier, S.,
11.
Jackson, S. P., and Durocher, D. (2013) Regulation of
Duan, S., Canny, M. D., van Ingen, H., Arrowsmith,
DNA damage responses by ubiquitin and SUMO, Mol.
C. H., Rubinstein, J. L., Vendruscolo, M.,
Cell, 49, 795-807, doi: 10.1016/j.molcel.2013.01.017.
Durocher, D., and Kay, L. E. (2017) The RNF168
12.
Iwai, K., and Tokunaga, F. (2009) Linear poly-
paralog RNF169 defines a new class of ubiquitylated
ubiquitination: a new regulator of NF-κB activa-
histone reader involved in the response to DNA
tion, EMBO Rep., 10, 706-713, doi: 10.1038/embor.
damage, Elife, 6, e23872, doi: 10.7554/eLife.23872.
2009.144.
23.
Pinato, S., Scandiuzzi, C., Arnaudo, N., Citterio, E.,
13.
Matsumoto, M. L., Wickliffe, K. E., Dong, K. C.,
Gaudino, G., and Penengo, L. (2009) RNF168, a
Yu, C., Bosanac, I., Bustos, D., Phu, L., Kirkpatrick,
new RING finger, MIU-containing protein that
D. S., Hymowitz, S. G., Rape, M., Kelley, R. F., and
modifies chromatin by ubiquitination of histones H2A
Dixit, V. M. (2010) K11-linked polyubiquitination in
and H2AX, BMC Mol. Biol., 10, 55, doi: 10.1186/
cell cycle control revealed by a K11 linkage-specific
1471-2199-10-55.
antibody, Mol. Cell,
39,
477-484, doi:
10.1016/
24.
Horn, V., Uckelmann, M., Zhang, H., Eerland, J.,
j.molcel.2010.07.001.
Aarsman, I., le Paige, U. B., Davidovich, C., Sixma, T. K.,
БИОХИМИЯ том 88 вып. 12 2023
2456
КУДРЯЕВА и др.
and van Ingen, H. (2019) Structural basis of spe-
histone H1, Mol. Cell, 66, 384-397.e8, doi: 10.1016/
cific H2A K13/K15 ubiquitination by RNF168,
j.molcel.2017.04.012.
Nat. Commun., 10, 1751, doi: 10.1038/s41467-019-
28. Panier, S., Ichijima, Y., Fradet-Turcotte, A., Leung,
09756-z.
C. C. Y., Kaustov, L., Arrowsmith, C. H., and
25. Machleidt, T., Woodroofe, C. C., Schwinn, M. K.,
Durocher, D.
(2012) Tandem protein interaction
Méndez, J., Robers, M. B., Zimmerman, K.,
modules organize the ubiquitin-dependent response
Otto, P., Daniels, D. L., Kirkland, T. A., and Wood,
to DNA double-strand breaks, Mol. Cell, 47, 383-395,
K. V (2015) NanoBRET - a novel BRET platform
doi: 10.1016/j.molcel.2012.05.045.
for the analysis of protein-protein interactions, ACS
29. Thorslund, T., Ripplinger, A., Hoffmann, S.,
Chem. Biol., 10, 1797-1804, doi: 10.1021/acschembio.
Wild, T., Uckelmann, M., Villumsen, B., Narita, T.,
5b00143.
Sixma, T. K., Choudhary, C., Bekker-Jensen, S., and
26. Weihs, F., Wang, J., Pfleger, K. D. G., and Dacres, H.
Mailand, N. (2015) Histone H1 couples initiation
(2020) Experimental determination of the biolumi-
and amplification of ubiquitin signalling after
nescence resonance energy transfer (BRET) Först-
DNA damage, Nature 527, 389-393, doi: 10.1038/
er distances of NanoBRET and red-shifted BRET
nature15401.
pairs, Anal. Chim. Acta X., 6, 100059, doi: 10.1016/
30. Mattiroli, F., Uckelmann, M., Sahtoe, D. D., van Dijk,
j.acax.2020.100059.
W. J., and Sixma, T. K. (2014) The nucleosome acidic
27. Bednar, J., Garcia-Saez, I., Boopathi, R., Cutter,
patch plays a critical role in RNF168-dependent
A. R., Papai, G., Reymer, A., Syed, S. H., Lone, I. N.,
ubiquitination of histone H2A, Nat. Commun., 5,
Tonchev, O., Crucifix, C., Menoni, H., Papin, C.,
3291, doi: 10.1038/ncomms4291.
Skoufias, D. A., Kurumizaka, H., Lavery, R.,
31. Mallette, F. A., and Richard, S. (2012) K48-linked
Hamiche, A., Hayes, J. J., Schultz, P., Angelov, D.,
ubiquitination and protein degradation regulate 53BP1
Petosa, C., and Dimitrov, S. (2017) Structure and dy-
recruitment at DNA damage sites, Cell Res., 22,
namics of a 197 bp nucleosome in complex with linker
1221-1223, doi: 10.1038/cr.2012.58.
TOPOLOGY OF UBIQUITIN CHAINS IN THE E3 UBIQUITIN
LIGASE RNF168 CHROMATOSOME ENTOURAGE
A. A. Kudriaeva*, L. A. Yakubova, G. A. Saratov, V. I. Vladimirov,
V. M. Lipkin, and A. A. Belogurov, Jr.
Shemyakin & Ovchinnikov Institute of Bioorganic Chemistry, Russian Academy of Sciences,
117997 Moscow, Russia; e-mail: anna.kudriaeva@gmail.com
Genome stability is critical for normal functioning of cells and depends on accuracy of DNA replication,
chromosome segregation, and DNA repair. Cellular defense mechanisms against DNA damage are im-
portant for preventing the development of cancer and aging. The E3 ubiquitin ligase RNF168 of the RING
superfamily is an essential component of the complex responsible for the ubiquitination of H2A/H2A.X
histones near DNA double-strand breaks, which is a key step in attracting repair factors to the injury site.
In this study, we unequivocally showed that RNF168 does not have ability to directly distinguish the archi-
tecture of polyubiquitin chains, except for tropism of its two ubiquitin-binding domains UDM1/2 to K63
ubiquitin chains. Analysis of the intracellular chromatosomal environment of full-length RNF168 and its
domains by ligand-induced bioluminescence resonance energy transfer (BRET) revealed that the C-ter-
minal part of UDM1 is associated with K63 ubiquitin chains; RING and the N-terminal part of UDM2
are sterically close to K63- and K48- ubiquitin chains, while the C-terminal part of UDM1 is colocalized
with all possible ubiquitin variants. Our observations together with the available structural data suggest
that the C-terminal part of UDM1 binds K63 polyubiquitin chains on linker histone H1; RING and the
N-terminal part of UDM2 are located in the central part of the nucleosome and sterically close to H1 and
K48-ubiquitinated alternative substrates of RNF168, such as JMJD2A/B demethylases, while the C-ter-
minal part of UDM1 is in the region of an activated ubiquitin residue associated with E2 ubiquitin ligase,
engaged by RNF168.
Keywords: ubiquitin, ubiquitin chains, ubiquitin ligase, histones, DNA repair, double-strand breaks, post-transla-
tional modifications, NanoBRET
БИОХИМИЯ том 88 вып. 12 2023