БИОХИМИЯ, 2023, том 88, вып. 5, с. 785 - 802
УДК 578.32;578.865;539.26
НУКЛЕОИД-АССОЦИИРОВАННЫЕ БЕЛКИ HU И IHF:
ОЛИГОМЕРИЗАЦИЯ В РАСТВОРЕ
И ГИДРОДИНАМИЧЕСКИЕ СВОЙСТВА
© 2023 Л.А. Дадинова1, М.В. Петухов1, А.М. Гордиенко1, В.А. Манувера2,3,
В.Н. Лазарев2,3, Т.В. Ракитина4,5, А.А. Можаев1,4, Г.С. Петерс5, Э.В. Штыкова1*
1 ФНИЦ «Кристаллография и фотоника» РАН,
Институт кристаллографии им. А.В. Шубникова,
119333 Москва, Россия; электронная почта: shtykova@ns.crys.ras.ru
2 Федеральный научно-клинический центр
физико-химической медицины им. Ю.М. Лопухина ФМБА России, 119435 Москва, Россия
3 Московский физико-технический институт (национальный исследовательский университет),
141701 Долгопрудный, Московская обл., Россия
4 Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН,
117997 Москва, Россия
5 НИЦ «Курчатовский институт», 123182 Москва, Россия
Поступила в редакцию 28.12.2022
После доработки 10.03.2023
Принята к публикации 23.03.2023
Структура и функция бактериального нуклеоида контролируются нуклеоид-ассоциированными
белками NAP. В любой фазе роста различные NAP, действуя последовательно, уплотняют нуклеоид
и обеспечивают его транскрипционно активную структуру. Однако в поздней стационарной фазе
происходит мощная экспрессия только одного из NAP, белка Dps, и формируются ДНК-белковые
кристаллы, трансформирующие нуклеоид в статическую структуру, эффективно защищённую
от внешних воздействий. Обнаружение кристаллических структур в живых клетках и связь этого
феномена с бактериальной резистентностью к антибиотикам вызвало огромный интерес к изуче-
нию этого явления. Целью настоящей работы является получение и сравнительное исследова-
ние структур двух родственных NAP (HU и IHF), поскольку именно они накапливаются в клет-
ке на поздней стационарной стадии роста, предшествующей началу формирования защитного
ДНК-Dps кристаллического комплекса. Для структурных исследований в работе применялись
два взаимодополняющих метода: малоугловое рентгеновское рассеяние (МУРР) в качестве основ-
ного метода изучения структуры белков в растворе и динамическое рассеяние света - в качестве
дополнительного. Для интерпретации данных МУРР использовались различные подходы и ком-
пьютерные программы (в частности, использовался анализ структурных инвариантов, метод мо-
лекулярной тектоники и анализ олигомерных смесей в терминах объёмных долей компонентов),
что позволило определить макромолекулярные характеристики и получить структурные 3D-моде-
ли различных олигомерных форм белков HU и IHF с типичным для МУРР разрешением ~2 нм.
Было показано, что эти белки олигомеризуются в растворе в разной степени, и для IHF характер-
но присутствие крупных олигомеров, состоящих из исходных димеров, выстроенных в цепочку.
Анализ экспериментальных и литературных данных позволил высказать гипотезу, что именно этот
белок непосредственно перед экспрессией Dps формирует наблюдавшиеся ранее in vivo тороидаль-
ные структуры и подготавливает платформу для образования кристаллов ДНК-Dps. Полученные
результаты необходимы для дальнейшего исследования феномена формирования биокристаллов
в бактериальных клетках и нахождения путей преодоления резистентности различных патогенов
к внешнему воздействию.
КЛЮЧЕВЫЕ СЛОВА: нуклеоид-ассоциированные белки, гистоноподобные белки HU и IHF, олигомери-
зация, малоугловое рентгеновское рассеяние, динамическое светорассеяние.
DOI: 10.31857/S032097252305007X, EDN: AXWAAH
Принятые сокращения: ДРС - динамическое светорассеяние; МУРР - малоугловое рентгеновское рассеяние; Dps -
ДНК-связывающий белок голодающих клеток; Fis - фактор инверсионной стимуляции; H-NS - гистоноподобный
белок, структурирующий нуклеоид; Lrp - регуляторный, чувствительный к лейцину белок; NAP - нуклеоид-ассоции-
рованные белки.
* Адресат для корреспонденции.
785
7
786
ДАДИНОВА и др.
ВВЕДЕНИЕ
ные участки ДНК, в результате чего проис-
ходит компактизация нуклеоида [17]. Харак-
Хранение и реализация генетической про-
терной особенностью H-NS является наличие
граммы функционирования живой клетки про-
двух ДНК-связывающих доменов, которые
исходит в хромосомах, которые представляют
могут взаимодействовать одновременно с дву-
собой комплексы нуклеиновых кислот с бел-
мя дуплексами ДНК, соединяя их наподобие
ками. В отличие от эукариотических клеток,
застёжки-молнии [18].
где различные ДНК-содержащие структуры
Другим важным представителем пер-
в компактном виде находятся в ядре, хромо-
вой группы является регуляторный, чувстви-
сомы безъядерных прокариот, в том числе бак-
тельный к лейцину белок (Leucine-responsive
терий, в большинстве случаев содержат только
regulatory protein, Lrp). Lrp обладает молеку-
одну кольцевую макромолекулу ДНК (нуклео-
лярной массой 15 кДа и функционально реа-
ид) и находятся в цитоплазме в свободном
гирует на изменение концентрации лейцина.
состоянии. Нуклеоид содержит от 1 до 5 млн
Основной функциональной единицей Lrp яв-
пар оснований (п.о.) и имеет длину порядка
ляется октамер, но могут существовать и более
1-2 мм, что вызывает необходимость его ком-
крупные олигомеры [19]. Октамеры Lrp обра-
пактной упаковки, не превышающей объём
зуют «дисковидные» структуры, которые на-
клетки. Компактизация ДНК в нуклеоиде
матывают на себя ДНК как нить на катушку,
достигается за счёт молекулярного краудинга
сближая удалённые друг от друга участки ДНК
и суперспирализации ДНК, происходящих
и тем самым эффективно её уплотняя [20].
при участии нуклеоид-ассоциированных бел-
Присутствие лейцина вызывает диссоциацию
ков (Nucleoid-Associated Proteins, NAP) [1-7].
Lrp и, соответственно, влияет на его ДНК-свя-
Эти архитектурные белки взаимодействуют
зывающие свойства [21, 22].
с ДНК нуклеоида, заставляя её складываться
К первой группе NAP также относят белок
в компактную структуру. Большая часть NAP
SMC (Structural Maintenance of Chromosomes),
представляет собой небольшие, основные, ди-
являющийся высококонсервативным во всём
или мультимерные белки с мономерами, со-
живом царстве от бактерий до человека и
стоящими примерно из 100 аминокислотных
обладающий высокой молекулярной массой
остатков. Эти белки часто называют гисто-
(выше 150-200 кДа) [23, 24]. Димер SMC об-
ноподобными белками по аналогии с гисто-
разует V-образную структуру с двумя длин-
нами эукариот. Помимо участия в организа-
ными двуспиральными плечами, которые
ции и компактизации хроматина, NAP играют
могут взаимодействовать сразу с несколькими
значительную роль в процессах, связанных с
регионами ДНК, что позволяет ему участво-
функционированием ДНК: рекомбинации, ре-
вать в сепарации вновь реплицированных хро-
парации, репликации и транскрипции [8-11],
мосом [25].
а также выполняют ДНК-защитные функ-
Важнейшими представителями NAP 2-й
ции [12]. Такое сочетание структурных и ре-
группы (белков, изгибающих ДНК) являются
гуляторных ролей NAP оказывает серьёзное
белки IHF (Integration Host Factor) и HU [26].
влияние как на функционирование нуклеоида,
Название HU означает гистоноподобный (H)
так и на жизнеспособность и вирулентность
и (U), где U93 - штамм Escherichia coli, от-
бактерий.
куда белок был выделен, а не heat unstable,
Условно NAP можно разделить на три ос-
что до сих пор встречается в литературе [27].
новные группы [13, 14]: 1) белки, соединяющие
HU и IHF имеют сходные первичные и про-
отдельные участки ДНК; 2) белки, изгибаю-
странственные структуры, но отличаются рас-
щие ДНК; 3) белки, участвующие в альтерна-
пространённостью в бактериальном царстве и
тивных механизмах организации и компакти-
особенностями ДНК-связывания.
зации ДНК.
HU - это наиболее широко распростра-
К первой группе относится гистонопо-
нённый и высокопредставленный в делящейся
добный белок, структурирующий нуклеоид
клетке NAP бактерий [28, 29]. У большинства
(Histone-like Nucleoid Structuring protein, H-NS).
бактерий HU является гомодимером с массой
H-NS имеет молекулярную массу 15,6 кДа и уча-
мономеров около 10 кДа. HU относят к α- или
ствует в регуляции бактериальной транскрип-
β-типу в зависимости от сходства с α- и β-субъ-
ции путём репрессии определённых генов [15,
единицами HU-белка E. coli, который является
16]. Основными функциональными единица-
гетеродимером HU (αβ), как и все HU из энте-
ми H-NS являются димеры, которые, в свою
робактерий. Субъединицы гетеродимерных HU
очередь, олигомеризуются, формируя сложные
гомологичны на 70%, и их пространственные
структуры, связывающие между собой различ-
структуры исключительно консервативны [28].
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
787
В них можно выделить димеризационный до-
тивного воздействия, в том числе от антибио-
мен, представляющий собой стабильное ядро
тиков [37-40]. Обнаружение кристаллических
из α-спиралей, переходящих в β-лист, и ДНК-
структур в живых клетках и связь этого фено-
связывающий домен, представляющий собой
мена с бактериальной резистентностью к ле-
подвижные β-тяжи («руки»), обхватывающие
карственным препаратам вызвало огромный
двойную спираль ДНК. Расположенные на
интерес к структуре самого Dps и его комплек-
концах «рук» остатки пролина интеркалиру-
са с ДНК [41-51].
ют в малую бороздку ДНК и изгибают её [26].
Dps представляет собой додекамер, моно-
HU не имеет специфических сайтов связывания
мер которого состоит из четырёх α-спиралей и
с ДНК, но имеет повышенное сродство к ДНК
имеет молекулярную массу 18,7 кДа [52]. Dps
со структурными искажениями, такими как
может также существовать в виде тримера,
разрывы одной или двух цепей, а также репли-
который, однако, не образует с ДНК кристал-
кативные вилки [10, 27]. Профили специфич-
лических форм [53]. Предполагается, что Dps
ности к ДНК-структурам у разных HU-белков
связывает ДНК за счёт электростатических
заметно отличаются, другие различия связаны
связей богатых лизином N-концевых доменов
с длиной сайта связывания, углом изгиба ДНК
с отрицательно заряженным сахарофосфат-
и степенью кооперативности [28, 30]. Предпо-
ным остовом ДНК [36, 37, 54].
лагается также, что взаимодействия HU-ДНК
Изменчивость качественного и количе-
играют критическую функцию в ремодели-
ственного состава NAP у разных бактерий и
ровании нуклеоидов, которое может служить
на разных стадиях жизненного цикла [4, 7], их
общим микробным механизмом регуляции
способность компенсировать отсутствие друг
транскрипции и адаптации к изменяющимся
друга и менять функциональные свойства в
условиям [31].
ответ на изменение клеточной среды препят-
IHF встречается только в энтеробактериях
ствуют изучению вклада индивидуальных NAP
и является гомологом HU (30-40% идентич-
в поддержание как структуры нуклеоида, так
ности) [28]. В отличие от HU, IHF представ-
и жизнеспособности и вирулентности бакте-
ляет собой облигатный гетеродимер, субъеди-
риальной клетки. В этой ситуации, когда ими-
ницы которого гомологичны примерно на 25%,
тировать сложные процессы, происходящие в
при этом α-субъединица имеет молекулярную
бактериальном нуклеоиде, in vitro не представ-
массу около 11 кДа, а β-субъединица - порядка
ляется возможным, традиционным решением
9,5 кДа. Как и HU, IHF использует интеркаля-
является последовательное изучение структу-
цию двух консервативных остатков пролина
ры и свойств отдельных белков, а затем на базе
в малую бороздку ДНК на расстоянии 9 п.о.
полученных данных - результата их взаимо-
друг от друга, индуцируя или стабилизируя из-
действия.
гиб ДНК [26]. В отличие от HU, IHF специфиче-
Ранее методом малоуглового рентгеновского
ски распознаёт последовательности из 13 п.о. с
рассеяния (МУРР) и криоэлектронной микро-
консенсусом 5′-(А/Т)ATCAANNNNTT(A/G)-3′,
скопии нами были определены типы кристал-
где N - это любой нуклеотид [32].
лической упаковки сокристаллов Dps-ДНК в
Ко второй группе NAP относится также
растворе в зависимости от состава буфера [44,
семейство Fis (Factor for inversion stimulation).
45], а также проведено изучение влияния кон-
Белки этого семейства представляют собой го-
формационной подвижности N-концевых об-
модимеры, каждая субъединица которых состоит
ластей Dps из E. coli на характер взаимодей-
из 98 аминокислот [33]. In vitro в высоких кон-
ствия белка с ДНК в разных условиях [46, 47].
центрациях Fis связывается с ДНК неспеци-
Целью настоящей работы является полу-
фически, однако in vivo белок выбирает опре-
чение и сравнительное исследование структур
делённые последовательности длиной 15 п.о.:
двух родственных NAP (HU и IHF), которые
(G/T)NN(C/T)(A/G)NN(A/T)NN(C/T)(A/G)
накапливаются в клетке на поздней стацио-
NN (C/A), где N - любой из нуклеотидов [34-36].
нарной стадии роста, предшествующей началу
К третьей группе NAP относится ДНК-
формирования защитного кристаллического
связывающий белок голодающих клеток (DNA-
комплекса Dps-ДНК. Для достижения этой
binding protein from starved cells, Dps), который,
цели, во-первых, были использованы два раз-
связываясь с ДНК, обеспечивает наиболее
ных метода получения рекомбинантных бел-
упорядоченный уровень её организации, так
ков HU и IHF. Белок HU представляет собой
как при этом образуется не просто компакт-
гомодимер HUa E. coli и был получен по ме-
ная структура, a устойчивый кристаллический
тоду, описанному в работе Agapova et al. [55].
комплекс Dps-ДНК, защищающий дезокси-
IHF является гетеродимером, и для его полу-
рибонуклеиновую кислоту от внешнего нега-
чения использовался оригинальный метод,
БИОХИМИЯ том 88 вып. 5 2023
7*
788
ДАДИНОВА и др.
описанный в данной работе ниже и основан-
МАТЕРИАЛЫ И МЕТОДЫ
ный на экспрессии бицистронных оперонов.
Во-вторых, для структурных исследований
Получение рекомбинантного белка HUa
применялись два независимых, взаимодопол-
E. coli. Использовали плазмиду pBAD-hupA
няющих метода: малоугловое рентгеновское
и клетки E. coli с делетированными генами
рассеяние в качестве основного метода изуче-
HU-белка. Плазмида и клетки были любезно
ния структуры белков в растворе и динамиче-
предоставлены J. Oberto (Институт физико-
ское рассеяние света (ДРС) - в качестве до-
химической биологии, Париж, Франция).
полнительного.
Предварительно в плазмиду по сайтам ре-
МУРР представляет собой упругое рассея-
стрикции BamHI и NdeI был встроен олиго-
ние рентгеновского излучения на неоднород-
нуклеотидный дуплекс, кодирующий по-
ностях электронной плотности вещества [56].
следовательности
6-гистидинового тага и
Пространственное распределение электрон-
сайта протеазы вируса гравировки табака
ной плотности, в свою очередь, зависит от
(MGSDKIHHHHHHENLYFQ*GH, * - сайт
структуры рассеивающего объекта и опреде-
расщепления). Полученной плазмидой pBad-
ляет профиль экспериментальной кривой рас-
HisTEVhupA трансформировали клетки E. coli
сеяния I(s), анализ которой даёт возможность
штамма С600 (hupA:Cm, hupB:Km). Клетки
вычислить радиус инерции Rg и максимальный
штамма-продуцента культивировали в среде LB,
размер Dmax рассеивающих частиц. Современ-
содержащей ампициллин (100 мкг/мл), канами-
ные методы интерпретации данных МУРР и
цин (25 мкг/мл) и хлорамфеникол (25 мкг/мл)
созданное для них программное обеспечение
при 37 °С в течение ночи. Ночную культуру раз-
позволяют определить не только упомяну-
водили в 100 раз средой LB, содержащей ампи-
тые инварианты малоуглового рассеяния, но
циллин (100 мкг/мл), и подращивали при 37 °С
также построить 3D структурные модели рас-
до оптической плотности ОД600 = 0,8. Экс-
сеивающих объектов с разрешением порядка
прессию индуцировали добавлением араби-
1-2 нм [56].
нозы до 0,2% (v/v) и проводили в течение ночи
ДРС основано на эффекте Доплера (из-
(16 ч) при 25 °С. Клетки осаждали центрифуги-
менение частоты падающего света) и броу-
рованием (6000 g, 10 °С, 20 мин). Выделение ре-
новского движения частиц в растворе, и с его
комбинантного белка проводили, как описа-
помощью измеряется флуктуация рассеяния
но ранее [55]. Схема выделения включала две
света, непосредственно связанная с диф-
стадии металлохелатной аффинной хромато-
фузией частиц в растворе, что даёт возмож-
графии (МХАХ), разделённые обработкой TEV-
ность определить гидродинамический ради-
протеазой, и финальную очистку белка с помо-
ус Rh, через уравнение Стокса-Эйнштейна.
щью ионообменной хроматографии. Все хро-
Важно отметить, что МУРР и ДРС позво-
матографии проводили с использованием хро-
ляют проводить изучение биологических объ-
матографической системы AKTA Prime («GE
ектов в среде, максимально приближенной
Healthcare», США).
к естественной, т.е. в растворе при заданных
Биомассу (около
10 г), полученную из
температуре, рН, солевом составе и других
1 литра бактериальной культуры, лизировали
параметрах среды [56]. В отличие от клас-
в 25 мл охлаждённого буферного раствора А
сических биохимических техник разделения
(50 мM Tris-HCl (pH 8,0), 500 мM NaCl ), кото-
белков (таких как хроматография или элек-
рый содержал 5% глицерина, 0,2% Triton X-100
трофорез), оба метода могут детектировать
и 1 мM PMSF, и обрабатывали ультразвуком
и изучать нестабильные (слабые и коротко-
с помощью ультразвукового дезинтегратора
живущие) комплексы. Необходимость со-
Ultrasonic Processor («Cole Parmer», США) в
вместного использования данных методов
режиме (5 × 30 с), охлаждая во льду. Нераство-
состоит в том, что ДРС позволяет определить
римые компоненты осаждали центрифугиро-
степень олигомеризации белков, которую
ванием (20 000 g, 4 °С, 20 мин). Осветлённый
необходимо учитывать при построении про-
лизат наносили на колонку с металлоафин-
странственных структурных моделей по дан-
ной смолой Ni-NTA Superflow («Quigen», Гер-
ным МУРР.
мания), уравновешенную буфером А. Для
Результаты настоящей работы будут слу-
удаления неспецифически-связавшихся бел-
жить основой для дальнейшего исследования
ков и фрагментов ДНК колонку промывали
феномена формирования биокристаллов в
буфером А, содержащим 1 М NaCl и 40 мМ
бактериальных клетках и нахождения путей
имидазола. Элюцию целевого белка прово-
преодоления резистентности различных пато-
дили буфером А, содержащим 300 мМ имида-
генов к внешнему воздействию.
зола. Фракции, содержащие целевой белок,
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
789
объединяли и инкубировали с протеазой TEV
по методу PIPE [58]. В результате была полу-
при комнатной температуре в течение 2 ч с
чена плазмида pET-IHFab, содержащая дву-
последующим диализом против буфера А в
цистронный оперон под контролем промото-
течение 16 ч при 4 °С. Для освобождения от
ра T7, кодирующий обе цепи IHF. В короткую
6-гистидинового тага и TEV-протеазы прово-
нетранслируемую область между участками
дили повторную МХАХ, собирая фракции, не
ДНК, кодирующими альфа- и бета-цепи, был
связавшиеся со смолой. Финальную очистку
введён сайт связывания рибосомы (RBS) для
проводили с помощью ионообменной хрома-
трансляции второго цистрона.
тографии, проводимой на колонке ResourceQ
Для ПЦР-амплификации участков ДНК,
(«GE Healthcare»), уравновешенной буфером
кодирующих альфа- и бета-цепи IHF, исполь-
(20 мМ Tris-HCl (pH 7,8), 50 мМ NaCl) с ис-
зовали пары праймеров IHFA-F и IHFA-R,
пользованием градиента NaCl (50 мМ-1 M).
IHFB-F и IHFB-R соответственно и ДНК
Для обессоливания и концентрирования пре-
E. coli K-12 MG1655 в качестве матрицы. Для
парата до и после ионообменной хроматогра-
амплификации плазмиды использовались
фии использовали колонки PD10 («GE Health-
праймеры PETPIPE-1 и PETPIPE-2, а также
care») и центрифужные концентраторы Amicon
плазмида pET-22b («Novagen», США) в каче-
Ultra 3kDa, («Millipore», США). Все белко-
стве матрицы. ДНК-фрагменты были очи-
вые фракции, полученные в процессе выде-
щены с помощью препаративного электрофо-
ления белка, анализировали с помощью элек-
реза в агарозном геле и выделены с помощью
трофореза в 15%-ном ПААГ в денатурирую-
набора diaGene («Диаэм», Россия). Далее три
щих условиях с последующим окрашиванием
ПЦР-фрагмента объединяли в экспрессион-
Кумасси G-250.
ную плазмиду по методу PIPE [58]. Для этого
Получение рекомбинантного белка IHF.
их смешивали в эквимолярных количествах и
Белок IHF, в отличие от HU, представляет
без лигирования трансформировали смесью
собой гетеродимер. В литературе описано,
компетентные клетки E. coli TOP10. За счёт на-
что для получения IHF требуется экспресси-
личия у фрагментов ДНК перекрывающихся
ровать гены обеих его цепей в одной клетке,
частей в клетке E. coli происходило восстанов-
иначе белок не образует правильной струк-
ление полной плазмиды. Клоны, содержащие
туры [57]. Для решения этой задачи мы полу-
рекомбинантные плазмиды, отбирали с помо-
чили генетическую конструкцию, содержащую
щью ПЦР клеточной суспензии с использова-
двуцистронный оперон, на базе плазмиды се-
нием праймеров T7 и T7t. Последовательности
мейства pET. С помощью ПЦР нарабатывали
праймеров, использованных в работе («Литех»,
три фрагмента ДНК, два из них кодировали
Россия), представлены в табл. 1. Из отобран-
альфа- и бета-цепи IHF, а третий представ-
ных клонов выделяли плазмидную ДНК и
лял собой практически полноразмерную плаз-
подтверждали соответствие её структуры ожи-
миду pET-22b без участка между промоторно-
даемой с помощью автоматического секвени-
операторной областью и Т7-терминатором.
рования по Сэнгеру на секвенаторе ABI Prism
Три фрагмента объединяли в одну плазмиду
(«Applied biosystems», США).
Таблица 1. Последовательности праймеров, используемых в работе
Название
Последовательность 5′→3′
IHFA-F
GAAGGAGATATACATATGGCGCTTACAAAAGCTGA
IHFA-R
CATTATTTACCTCCTTTATTTACTCGTCTTTGGGCGAAG
IHFB-F
GTAAATAAAGGAGGTAAATAATGACCAAGTCAGAATTGATAG
IHFB-R
GTGGTGGTGCTCGAGTTAACCGTAAATATTGGCGCGA
PETPIPE-1
CATATGTATATCTCCTTCTTAAAG
PETPIPE-2
CTCGAGCACCACCACCACCACCACTGA
T7
TAATACGACTCACTATAGGG
T7t
GCTAGTTATTGCTCAGCGG
БИОХИМИЯ том 88 вып. 5 2023
790
ДАДИНОВА и др.
Для наработки белка IHF плазми-
Для регистрации рентгенограмм использо-
дой pET-IHFab трансформировали клетки
вали двухкоординатный детектор DECTRIS
E. coli BL21(DE3) gold
(«Novagen», США).
Pilatus3 1M c площадью рабочей поверхности
Далее в колбу, содержащую 50 мл среды LB
168,7 × 179,4 мм, разрешением 981 × 1043 то-
и 150 мг/литр ампициллина, помещали отдель-
чек и размером пикселя - 172 мкм, установ-
ную колонию E. coli BL21-gold(DE3)/pET-
ленный на расстоянии около 750 мм от об-
IHFab. Колбу инкубировали в течение ночи
разца. Интенсивность рассеяния I(s) была
при 30 °С в шейкере-инкубаторе. Затем 50 мл
измерена в области значений векторов рас-
ночной культуры переносили в 1 литр свежей
сеяния 0,09 < s <4 нм-1, где s = (4πsinθ)/λ, 2θ -
среды LB (ампициллин 100 мг/литр) и под-
угол рассеяния и λ = 0,1445 нм - длина волны
ращивали в шейкере-инкубаторе при 37 °С до
рассеяния. Для каждого образца было снято
момента достижения культурой OD600 = 0,8.
по 12 экспериментальных кривых рассеяния со
Далее к культуре добавляли IPTG до конеч-
временем экспозиции 300 с каждая (суммарное
ной концентрации 0,5 мМ и инкубировали
время облучения 60 мин) с целью контроля
4 ч при температуре 37 °С в шейкере-инкуба-
возможных радиационных повреждений. Точ-
торе. Клетки осаждали центрифугированием.
ная калибровка расстояния образец-детек-
Клеточную массу ресуспендировали в 50 мл
тор осуществлялась в программе Fit2D [62]
20 мМ Tris-HCl (pH 7,5) и разрушали клетки
по образцу бегената серебра («Sigma-Aldrich»,
с помощью ультразвукового дезинтегратора.
Германия).
Клеточный дебрис осаждали центрифугирова-
Первичная обработка данных, включаю-
нием в течение 20 мин при ускорении 15 000 g.
щая усреднение кривых рассеяния и вычет
Полученный раствор фильтровали через фильтр
сигнала от буфера, а также определение струк-
с порами 0,22 мкм и наносили на хромато-
турных инвариантов МУРР, проводилась с по-
графическую колонку, содержащую 5 мл сор-
мощью программы PRIMUS [63]. Дальнейшая
бента DEAE Sepharose FF («GE Healthcare»)
обработка полученных данных проводилась
и уравновешанную 20 мМ Tris-HCl (pH 7,5).
с помощью программ специального пакета
Собирали фракцию белков, не связавшихся с
ATSAS [64].
колонкой. Большая часть белков из клеточного
Для определения максимального размера
лизата в этих условиях связывалась с сорбен-
рассеивающих частиц в растворе и построения
том, в то время как IHF проходил колонку без
функций распределения по расстояниям p(r)
связывания. После первичной очистки с помо-
использовалась программа GNOM [65].
щью DEAE-сефарозы IHF специфично очища-
Сравнение экспериментальных данных и
ли с помощью хроматографии на колонке с ге-
кривых МУРР, полученных от моделей, осу-
парин-сефарозой [59]. Для этого использовали
ществлялось программой CRYSOL [66]. Про-
колонку HiTrap Heparin HP 1ml («GE Health-
грамма использует мультипольное разложение
care») в соответствии с рекомендациями про-
амплитуд рассеяния для расчёта сферически
изводителя. В результате был получен электро-
усреднённой картины рассеяния и учитывает
форетически гомогенный препарат белка IHF.
гидратную оболочку. Экспериментальные дан-
Подготовка образцов для анализа. Замена
ные МУРР приближаются путём оптимизации
буфера и концентрирование белковых раство-
расчётной кривой рассеяния, минимизируя
ров проводили с помощью центрифужных кон-
расхождение между ними. Критерием сходи-
центраторов Amicon Ultra-4 с порогом отсе-
мости является значение невязки χ2:
чения - 3 кДа. Образцы переводили в бу-
фер (20 мМ Tris-HCl (pH 7,5), 50 мМ KCl,
1
2,
2 мМ MgCl2) и концентрировали до 5 мг/мл.
χ2 =
(1)
j
Σ [Iexp(sj) - cIcalc(sj)σ(s
]
N - 1
j)
Также образцы перед измерениями центри-
фугировали в течение часа при 11 000 g в охлаж-
даемой настольной центрифуге.
где N - число экспериментальных точек, Iexp(sj)
Эксперимент и анализ данных МУРР. Экс-
и σ(sj) - экспериментальные интенсивности и
перимент по малоугловому рентгеновскому
их ошибки, Icalc(sj) - интенсивность, вычислен-
рассеянию проводился на станции «БиоМУР»
ная от модели, c - шкалирующий множитель.
[60,
61] Курчатовского источника синхро-
Для моделирования структуры олигомеров
тронного излучения (НИЦ
«Курчатовский
HU и IHF нами была также разработана про-
институт», Москва, Россия) в геометрии на
грамма HEMIX. Данная программа позволяет
пропускание. Растворы с образцами поме-
строить мультичастичные образования, в ко-
щали в тонкостенные кварцевые капилляры
торых каждый следующий строительный блок
диаметром 2 мм и толщиной стенок 0,01 мм.
получается из предыдущего путём одинаковой
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
791
трансформации первого блока для получения
Эксперимент и анализ результатов динами-
второго из первого, третьего из второго и т.д.
ческого светорассеяния. Анализ проводили с
Затем программа рассматривает все возмож-
использованием прибора Zetasizer™ NanoS
ные конфигурации таких строительных блоков
(«Malvern Panalytical», Великобритания). Тех-
внутри олигомера и с помощью их комбина-
нические характеристики - 4 мВт He-Ne-лазер,
ции приближает данные МУРР от олигомер-
λ0 = 633 нм, θ = 173°.
ной смеси, содержащей как полный олигомер,
Настройки прибора оптимизировались
так и его составные части. В данной работе
автоматически с помощью программного
при моделировании строительным блоком для
обеспечения ZS XPLORER («Malvern Panalyt-
IHF был гетеродимер (PDB ID: 1ihf) и гомо-
ical»). Для каждого образца проведено пять
димер (PDB ID: 5l8z) - для HU, а в качестве
повторных измерений при 25 °С с термоста-
компонентов смеси, соответственно, исполь-
билизацией 120 с. Динамическое рассеяние
зовали один (димер), два (тетрамер, как часть
света измеряет автокорреляционную функ-
олигомера) и три (полный олигомер) строи-
цию, т.е. корреляцию флуктуации света, рас-
тельных блока.
сеянного наночастицами, находящимися в
Количественный состав равновесных сме-
броуновском движении в растворе. Коэффи-
сей, состоящих из компонентов, для каждого
циент диффузии наночастиц рассчитывается
из которых имеется кривая рассеяния (либо
путём подбора автокорреляционной функции
измеренная экспериментально, либо посчи-
и используется для определения диаметра (или
танная от модели программой CRYSOL [66]),
радиуса) частиц через уравнение Стокса-
определялся программой OLIGOMER
[63].
Эйнштейна:
OLIGOMER с помощью линейной комбина-
kβT
,
ции кривых МУРР от компонентов прибли-
d(H) =
(3)
6πηRh
жает экспериментальные данные от смеси.
При этом весовые коэффициенты компонен-
где kβ - константа Больцмана, Т - абсолют-
тов определяют их объёмные доли νk в такой
ная температура, η - вязкость жидкости, Rh -
смеси:
гидродинамический радиус частицы. Анализ
полученных данных проводится в предпо-
ложении сферической аппроксимации иссле-
(2)
I(s) = ΣK νkIk(s),k = 1
дуемого объекта. Распределение по гидро-
динамическим размерам белков и индекс
где Ik(s) - интенсивность рассеяния компо-
полидисперсности растворов получены с по-
нентой.
мощью программного обеспечения Zetasizer
Следует заострить внимание, что в фор-
(«Malvern Panalytical»). Вязкость воды была
мулу (2) входят объёмные доли компонентов, а
установлена на уровне 0,08872 мПа·с с ре-
не их молярные фракции, из чего следует, что
фракционным индексом 1,33. Ошибка изме-
вклад больших олигомеров будет более суще-
рений определена как стандартное отклонение
ственным, чем, например, димеров. Таким об-
пяти повторных измерений.
разом, при одних и тех же объёмных долях ко-
личество олигомеров в единицах будет обратно
пропорционально их молекулярному весу.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Моделирование методом молекулярной
тектоники по данным от олигомерной смеси
Анализ гидродинамического поведения бел-
проводилось программой SASREFMX
[67].
ков HU и IHF с помощью динамического свето-
Данная программа восстанавливает четвер-
рассеяния. Имеющиеся литературные данные
тичную структуру комплекса путём враще-
указывают на способность гистоноподобных
ния и сдвига относительно друг друга атом-
белков олигомеризоваться в растворе [17, 31,
ной структуры субъединиц, формирующих
68-70]. Поэтому степень олигомеризации,
комплекс. При этом экспериментальная кри-
т.е. распределение по гидродинамическим раз-
вая МУРР приближается смесью кривых рас-
мерам и объёмные доли отдельных компонент,
сеяния от целого комплекса и от его компонен-
белков HU и IHF были определены с помо-
тов, а в качестве дополнительных параметров
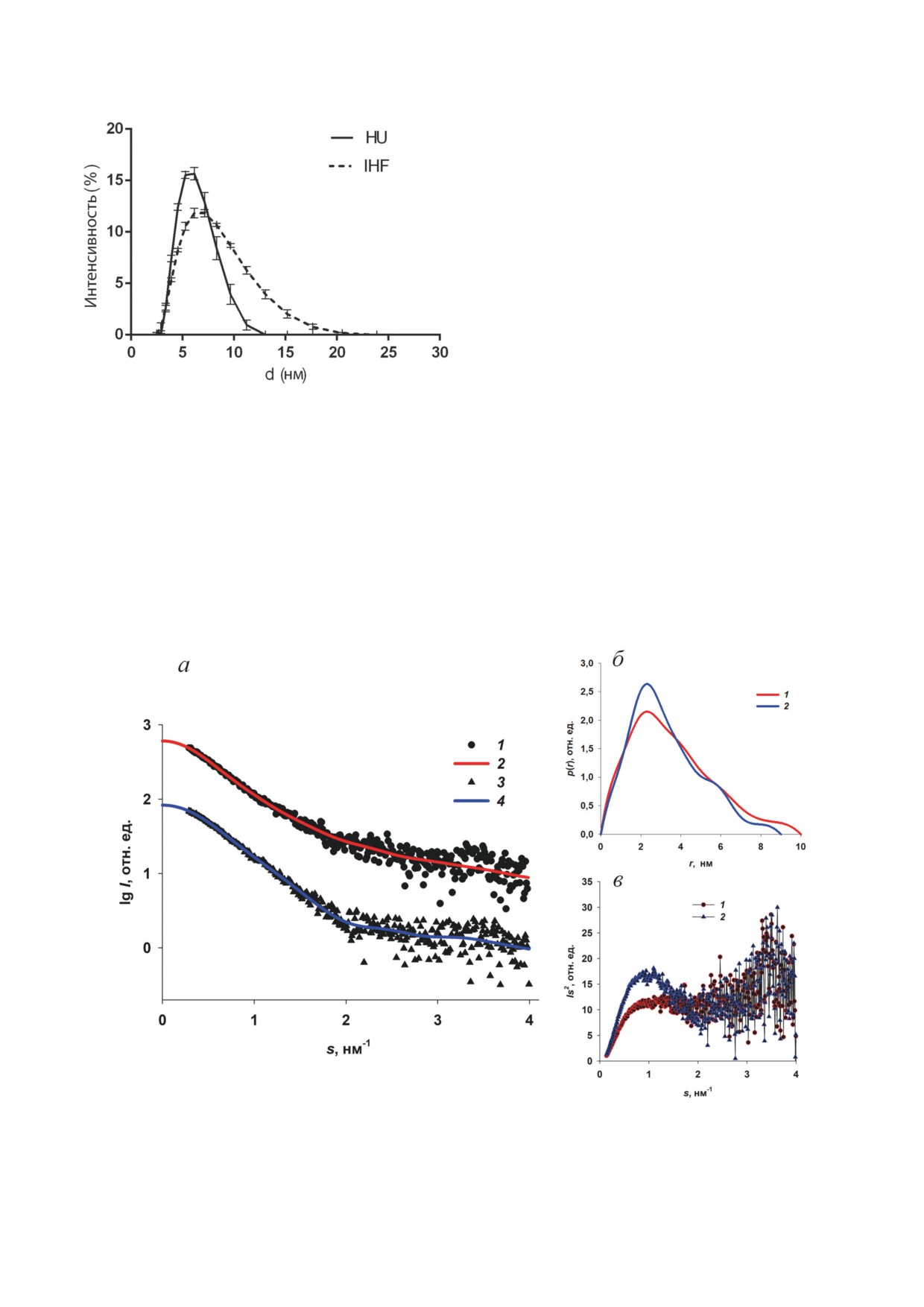
щью динамического рассеяния света (рис. 1).
моделирования используются объёмные доли
Полученные распределения по гидродина-
компонентов. В данной работе комплексом
мическим размерам демонстрируют асиммет-
являлся высокий олигомер белка, а в качестве
ричные профили для обоих белков, что сви-
субъединиц использовались составляющие его
детельствует о наличии в растворе не только
димеры.
димерных форм HU и IHF, но и более крупных
БИОХИМИЯ том 88 вып. 5 2023
792
ДАДИНОВА и др.
ло 17% (v/v) более крупных частиц. Для этого
белка профиль распределения по размерам
сравнительно узок (индекс полидисперсности
равен 0,32), в то время как для IHF степень
полидисперсности высокая (0,75): в растворе
присутствуют 45% (v/v) димеров, 35% (v/v)
более высоких олигомеров и порядка 20% (v/v)
ещё более крупных частиц.
Интегральные структурные характеристи-
ки гистоноподобных белков HU и IHF. Кривые
малоуглового рассеяния гистоноподобных
белков HU и IHF были измерены в буферном
растворe, содержащем катионы Mg2+. Выбор
солевого состава, содержащего бивалентные
катионы магния, был основан на результатах
Рис. 1. Распределение по гидродинамическим размерам
для гистоноподобных белков HU и IHF
ранее проведённых экспериментов по опти-
мизации условий формирования ДНК-бел-
ковых комплексов белка Dps, способного об-
образований. Тем не менее основной фракци-
разовывать сокристаллы Dps-ДНК [46, 47].
ей рассеивающих частиц являются частицы с
Из литературных данных также известно, что
z-средними гидродинамическими размерами
присутствие бивалентных катионов магния
порядка 8-10 нм, что хорошо соответствует
имеет решающее значение для образования
размерам димеров исследуемых белков с учё-
сокристаллов Dps-ДНК [46, 47, 71] и, следо-
том гидратной оболочки. Расчёты с помощью
вательно, может оказать влияние на структуру
программы Zetasizer показывают, что в раство-
белков HU и IHF, которые предшествуют Dps
ре присутствует 83% (v/v) димеров HU и око-
на поздних фазах роста клеток [13, 29].
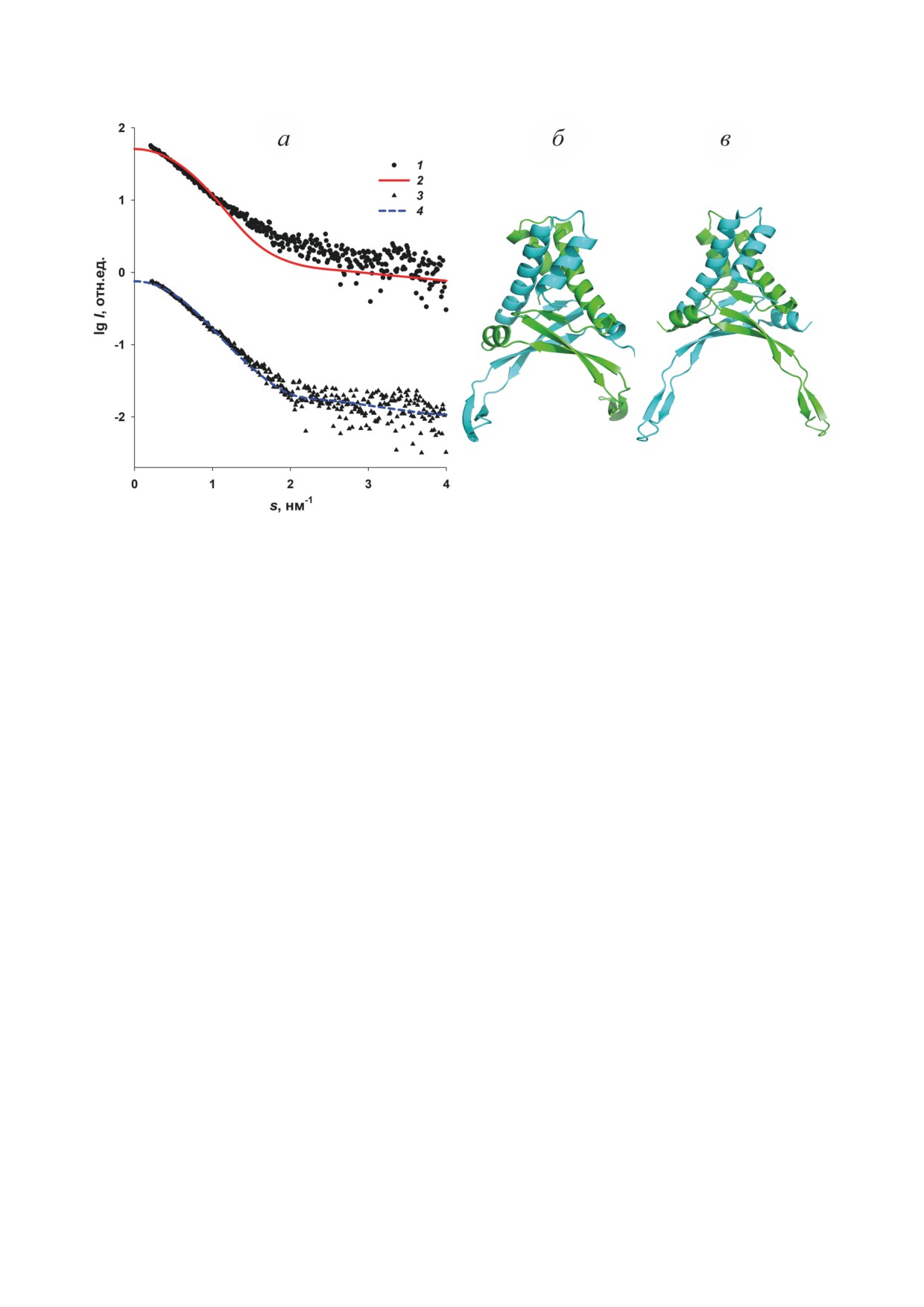
Рис. 2. Анализ кривых МУРР от гистоноподобных белков HU и IHF. а - Экспериментальные кривые малоуглового
рассеяния от IHF (1) и HU (3) и кривые, рассчитанные от функций распределения по расстояниям p(r) для IHF (2)
и HU (4). Кривые разнесены попарно по вертикали на один логарифмический порядок для лучшей визуализации;
б - функции распределения по расстояниям p(r), рассчитанные для IHF (1) и HU (2); в - графики в координатах
Кратки для IHF (1) и HU (2)
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
793
Таблица 2. Интегральные структурные характеристики гистоноподобных белков HU и IHF в растворе
Образец
Vp, нм3
Mэксп, кДа
Mтеор, кДа
Rg, нм
Rg*, нм
Dmax, нм
HU
45 ± 5
27 ± 3
20,5
2,62 ± 0,02
2,2
9,0
IHF
55 ± 5
33 ± 3
21,5
2,89 ± 0,03
2,1
10,0
Примечание. Rg* - теоретические значения радиуса инерции Rg.
Белки HU и IHF были измерены при кон-
случае такое восстановление невозможно
центрациях 2 мг/мл и 4,8 мг/мл. Поскольку
из-за присутствия разных олигомерных форм
концентрационной зависимости в этом ин-
белков HU и IHF в растворе. Однако с помо-
тервале концентраций не наблюдалось, для
щью функции p(r) можно определить усред-
дальнейших расчётов и структурного модели-
нённые максимальный размер Dmax и радиус
рования использовались кривые с концентра-
инерции Rg. В этом случае из-за полидисперс-
цией 4,8 мг/мл, как наиболее информативные
ности образцов Rg определяется не с помощью
и с меньшими экспериментальными шумами
графика Guinier [74], а как усреднённая вели-
(рис. 2, а; кривые 1 и 3).
чина, рассчитанная программой GNOM из
Поскольку кривые МУРР для обоих бел-
функции распределения по расстояниям p(r).
ков имеют тенденцию увеличения интенсив-
Как видно из табл. 2, усреднённые значения Rg
ности рассеяния в самых малых углах (боль-
для HU и IHF заметно превышают получен-
ше - для IHF и в меньшей степени - для HU),
ные с помощью программы CRYSOL [66] тео-
можно предположить, что в растворах могут
ретические значения Rg*, особенно для белка
присутствовать олигомеры более высоких сте-
IHF. Усреднённый максимальный размер
пеней - тетрамеры, гексамеры и другие. Рас-
Dmax IHF больше такового для белка HU, хотя
считанные с помощью программы PRIMUS
геометрические размеры их кристаллических
такие инварианты, как породовский объём Vp
структур (IHF PDB ID: 1ihf и HU PDB ID:
и молекулярные массы (табл. 2), явно ука-
5l8z) равны соответственно для IHF - 6,6 нм
зывают на наличие некоторого количества
и для HU - 7,3 нм. Для моделирования ис-
олигомеров выше, чем димер, в обоих рас-
пользовали структуру HU-белка Spiroplasma
творах: средние молекулярные массы Mэксп,
melliferum, имеющую одно из максимальных
определённые по кривым МУРР из соотноше-
разрешений, полученных для белков данного
ния Vp/1,65 [72], существенно превышают тео-
класса [75], так как в доступных кристалличе-
ретические (Mтеор).
ских структурах HUa (PDB ID: 1MUL, 2O97)
Важнейшей структурной характеристикой
не разрешены подвижные β-листовые «руки»,
белков является функция распределения по
а сравнительный анализ, проведённый в рабо-
расстояниям p(r) (рис. 2, б), связанная с ин-
те Kamashev et al. [28], показал исключительно
тенсивностью рассеяния I(s) преобразованием
высокую консервативность пространственных
Фурье и необходимая для анализа структуры
структур HU-белков.
рассеивающих объектов:
Представленные на рис. 2, б функции рас-
∞
пределения по расстояниям p(r) характерны для
1
p(r) =
∫ s r I(s) sin(sr)ds.
(4)
сильно вытянутых рассеивающих объектов -
2π2
0
максимум смещён влево, и отношение D
max
Функция p(r) представляет собой сово-
к размеру поперечного сечения, равного 2,3 нм
купность расстояний между каждыми двумя
и примерно соответствующего размеру бел-
точками в интервале между r и r + dr и распро-
кового ядра, равно 3,9 для белка HU и 4,4 -
страняется на весь интервал [0, Dmax], где Dmax -
для IHF. Максимумы на спаде функции p(r)
максимальное расстояние между двумя точ-
справа могут соответствовать рассеянию от по-
ками внутри рассеивающей частицы. Таким
вторяющихся в структуре форм, предположи-
образом, эта функция содержит информацию
тельно, димерам.
о форме, размере и структуре образца и позво-
На рис. 2, в представлены также графики в
ляет оценивать его максимальный размер Dmax
координатах Кратки для обоих белков, позво-
из условия p(r) = 0 при r > Dmax. Профиль p(r)
ляющие оценить степень свернутости/упоря-
отражает основные особенности рассеиваю-
доченности рассеивающих частиц. Колоколо-
щего объекта и для монодисперсных соедине-
образная форма максимума на графиках в
ний используется для ab initio восстановления
целом свидетельствует об упорядоченных рас-
структуры по данным МУРР [73]. В данном
сеивающих структурах, но для белка IHF этот
БИОХИМИЯ том 88 вып. 5 2023
794
ДАДИНОВА и др.
Рис. 3. Сравнение рассеяния от структур высокого разрешения белков IHF и HU с экспериментальными кривыми
МУРР. а - Экспериментальные данные от IHF (1) и HU (3), приближение атомной моделью димера IHF (IHF PDB ID:
1ihf) (2) и HU (HU PDB ID: 5l8z) (4); б - структура высокого разрешения димера IHF; в - структура высокого разреше-
ния димера HU. Мономеры белков IHF и HU показаны разными цветами
максимум менее выражен и характерен для бо-
ближает экспериментальные данные, получен-
лее развёрнутой и вытянутой структуры [76].
ные от раствора HU (рис. 3, кривая 3). В част-
Структурное моделирование белков HU
ности, в районе самых малых углов расчётная
и IHF по данным МУРР. Полученные нами
кривая систематически отличается от экспе-
интегральные структурные характеристики
риментальной, что, по всей видимости, также
HU и IHF ясно свидетельствуют об олиго-
связано с частичной мультимеризацией HU в
меризации белков в растворе. Хотя основным
растворе. Если в образце HU наряду с диме-
компонентом для обоих белков является ди-
рами предположить наличие определённого
мер, в растворе явно присутствуют олигомеры
количества тетрамеров (использующих димер
более высокого порядка, и особенно ярко это
как функциональную строительную единицу),
выражено для белка IHF. Ещё одним доказа-
то возможно приблизить данные МУРР. Ис-
тельством мультимеризации HU и IHF явля-
ходно был опробован тетрамер, образованный
ется сравнение теоретического рассеяния от
двумя соседними димерами в элементарной
структур высокого разрешения с эксперимен-
ячейке кристаллической структуры, получен-
тальными кривыми МУРР (рис. 3).
ный с помощью сервера PISA [77]. С учётом
Для белка IHF сравнение эксперименталь-
наличия в растворе определённого количества
ной (рис. 3, кривая 1) и теоретической, рассчи-
такого тетрамера экспериментальные данные
танной программой CRYSOL [66] из атомных
удалось приблизить во всём диапазоне углов
координат димера IHF (PDB ID: 1ihf) (рис. 3,
рассеяния (рис. 4, а; кривая 2). Полученные
кривая 2), кривых рассеяния свидетельствует
при этом программой OLIGOMER [63] объём-
об их несовпадении на всех диапазонах угло-
ные доли димера и тетрамера в их равновесной
вых векторов, что подтверждает сделанный
смеси составили 75% и 25% соответственно.
выше вывод о наличии в растворе этого белка
Использование тетрамера из кристалли-
более высоких, чем димер, олигомеров, причём
ческой структуры позволяет ограничить коли-
в достаточно больших количествах.
чество степеней свободы при моделировании
Для белка HU такое несовпадение не столь
четвертичной структуры мультимера в рас-
очевидно. Однако теоретическая кривая МУРР
творе, но в то же время такая конфигурация
для этого белка (использовались атомные ко-
может быть артефактом. Стоит отметить, что
ординаты его димера PDB ID: 5l8z) (рис. 3,
в действительности в растворе может присут-
кривая 4) не вполне удовлетворительно при-
ствовать тетрамер и с другой четвертичной
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
795
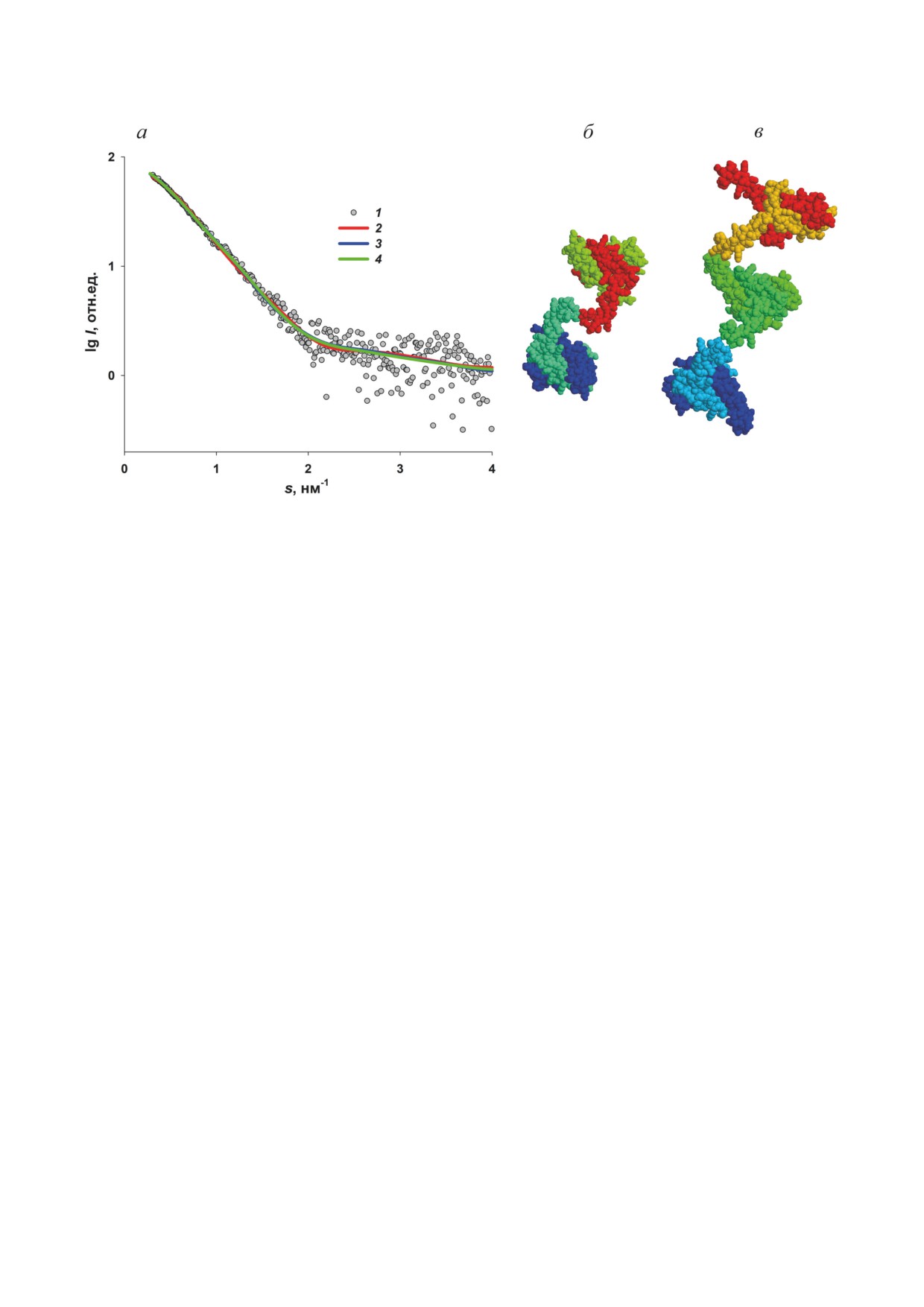
Рис. 4. Приближение экспериментальных данных МУРР от HU равновесными смесями (a): 1 - экспериментальные
данные; 2 - кривая рассеяния от смеси димеров и тетрамеров, полученных с помощью PISA; 3 - кривая рассеяния от
смеси димеров и тетрамеров, полученных с помощью SASREFMX; 4 - кривая рассеяния от смеси димеров, тетрамеров
и гексамеров, полученных в HEMIX. б - Модель тетрамера, построенная из двух димеров программой SASREFMX.
Отдельные мономеры показаны разным цветом. в - Модель гексамера, построенная HEMIX из трёх кристаллографи-
ческих димеров
структурой, имеющий интерфейс, отличный
тип контакта, полученный в модели тетрамера
от кристаллической упаковки. С использова-
с помощью SASREFMX (рис. 4, б и в). Одна-
нием программы расчёта четвертичной струк-
ко следует учесть, что полученные объёмные
туры с помощью метода молекулярной тек-
доли тетрамеров и гексамеров соответствуют
тоники для приближения данных МУРР от
меньшим молярным долям этих олигомеров.
полидисперсного раствора SASREFMX [67],
По сравнению с димером, молярные фракции
использовавшей димер в качестве твёрдого
снижаются в 2 и в 3 раза для тетрамера и гекса-
тела, были построены несколько различных
мера соответственно. Таким образом, в моляр-
тетрамеров с разными интерфейсами димер-
ном выражении присутствие высоких олиго-
димер. Одна из полученных моделей, показан-
меров достаточно ограничено.
ная на рис. 4, б, также приближает всю кривую
Предположительное присутствие в раство-
МУРР (рис. 4, а; кривая 3). Несмотря на разли-
ре высоких олигомеров IHF (например, гекса-
чия в конфигурации тетрамеров, полученные
меров) не позволяет провести структурное мо-
в отдельных восстановлениях, объёмные доли
делирование этого белка по той же схеме, как
тем не менее варьировались в узком диапазоне
это было сделано для белка HU. Например,
20-23%. Такой вклад тетрамера хорошо согла-
анализ с помощью PISA не мог быть прове-
суется с данными ДРС, предсказывающими на-
дён, т.к. в элементарной ячейке кристаллогра-
личие 17% частиц больших, чем димер, в рас-
фической модели (PDB ID: 1ihf) отсутствуют
творе HU. Были также предприняты попытки
связные фрагменты, состоящие из 6 мономе-
построить более длинные олигомеры (гекса-
ров белка.
меры) с помощью программы HEMIX, пред-
Для моделирования гексамера IHF так-
ставлявшую раствор как равновесную смесь
же не могла быть использована программа
димеров, тетрамеров и гексамеров. В этом слу-
SASREFMX, поскольку её алгоритм предпо-
чае приближение экспериментальных данных
лагает возможное наличие только двух фрак-
было также хорошим (рис. 4, а; кривая 4) при
ций в смеси (полная структура и один продукт
объёмных долях димера, тетрамера и гексаме-
диссоциации). В данном случае необходимо
ра 83%, 10% и 7% соответственно, что ещё луч-
было принимать во внимание три фракции в
ше согласуется с данными ДРС. Получивший-
растворе: димеры, тетрамеры и гексамеры, по-
ся интерфейс между отдельными димерами в
этому для структурного моделирования этого
гексамере (рис. 4, в) хорошо воспроизводит
белка была использована программа HEMIX.
БИОХИМИЯ том 88 вып. 5 2023
796
ДАДИНОВА и др.
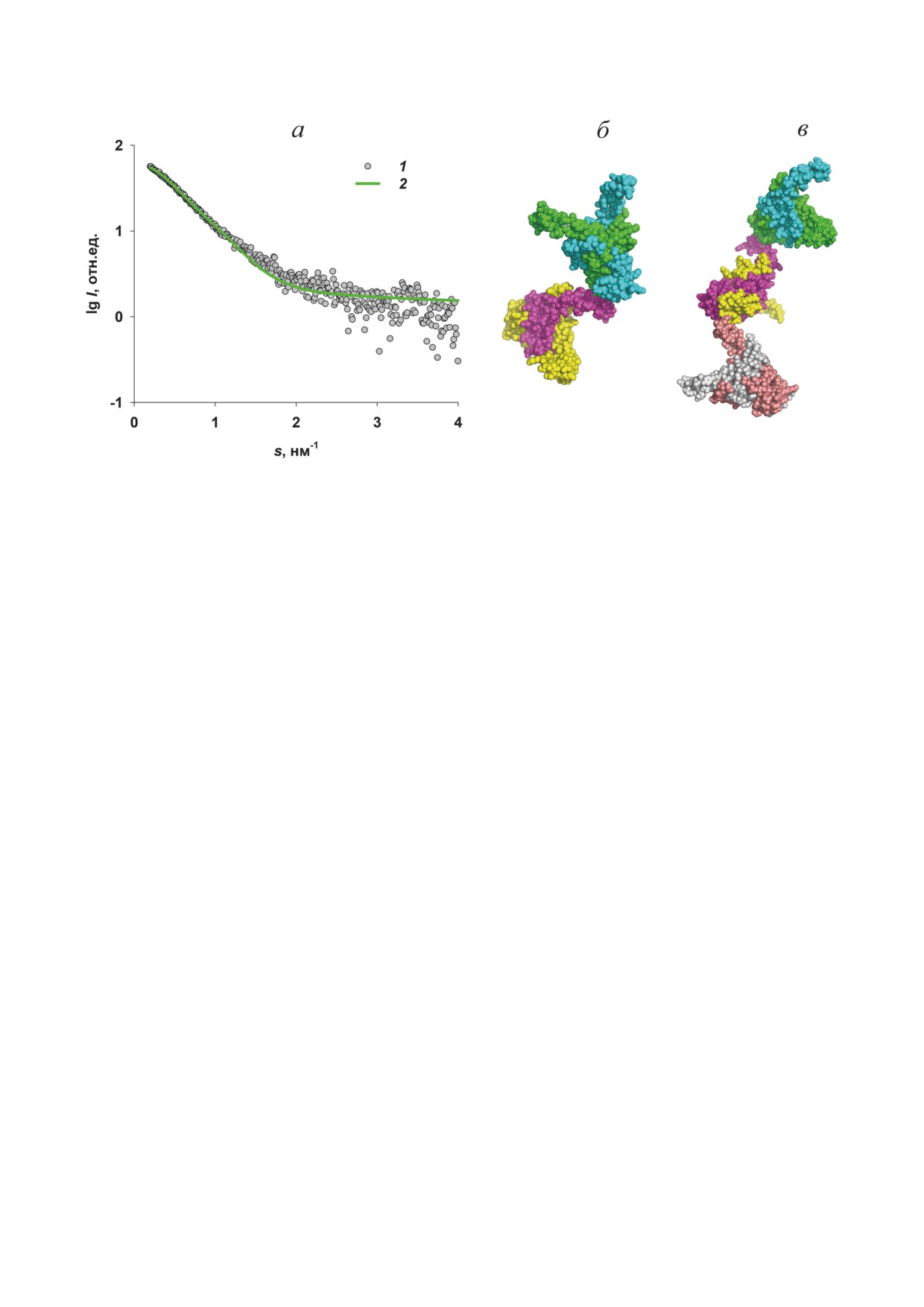
Рис. 5. Приближение экспериментальных данных МУРР от IHF равновесными смесями (a): 1 - экспериментальные
данные; 2 - кривая рассеяния от смеси димеров, тетрамеров и гексамеров, полученных в HEMIX. б - Модель, состоя-
щая из 2 димеров. в - Модель, состоящая из 3 димеров
С помощью программы HEMIX были по-
ковые комплексы, или, наоборот, проявляют
строены тетрамер и гексамер, где в качестве
антагонизм, противодействуя друг другу. Раз-
строительного блока использовался димер.
нообразие функций, контролируемых NAP на
Программа перебирает все возможные конфи-
разных этапах жизненного цикла клетки, при-
гурации таких строительных блоков. Модели,
водит к изменению их экспрессии в разные
построенные таким образом, представлены
периоды роста клеточной популяции [4, 7].
на рис. 5, б и в.
При этом в каждой фазе роста присутствует по
Для оценки объёмной доли компонентов
крайней мере один из белков NAP, который
в растворе белка IHF, полученных програм-
экспрессируется максимально
[13]. В соот-
мой HEMIX (димера, тетрамера и гексамера),
ветствии с данными, приведёнными в работе
использовалась программа OLIGOMER. Хоро-
Luijsterburg et al. [13] для 5 архитектурных бел-
шее приближение к экспериментальным дан-
ков E. coli (H-NS, HU, IHF, Fis и Dps), на ран-
ным для IHF (рис. 5, а; кривая 2) получилось
ней экспоненциальной стадии доминирует Fis,
при объёмных долях димера, тетрамера и гек-
на экспоненциальной - HU, в стационарной -
самера 45%, 33% и 22%. Молекулярная масса
IHF и поздней стационарной фазе - Dps.
такой смеси оказалась равной 38 кДа, что со-
Белок H-NS присутствует всегда, но никогда
впадает со значением этой величины, получен-
не доминирует, и его экспрессия резко умень-
ным по экспериментальным данным (табл. 2).
шается в стационарной и поздней стационар-
ной фазах. Существенно, что в любой фазе
роста в значительном количестве присутству-
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
ют белки, изгибающие ДНК (HU, IHF, Fis) и
противодействующие влиянию соединяющих
Организация и уплотнение ДНК служит
белков 1-й группы (H-NS). Эти NAP модули-
нескольким целям. С одной стороны, умень-
руют петлевую структуру нуклеоида, тем са-
шение полезного объёма бактериальной хро-
мым обеспечивая транскрипционно активную
мосоме требуется, чтобы поместиться в клетке.
структуру нуклеоида во время фаз относитель-
С другой стороны, становится всё более оче-
но быстрого роста [8-11]. И только в поздней
видным, что ответ на стимулы окружающей
стационарной фазе Dps образует кристаллы
среды в значительной степени опосредуется
с ДНК и в результате трансформирует динами-
действием архитектурных белков - NAP. Эти
ческий нуклеоид в статическую, и, как пред-
белки, весьма разнообразные по своим струк-
полагалось ранее, транскрипционно неактив-
турным и функциональным особенностям
ную структуру, эффективно защищённую от
(см. «Введение»), либо действуют однонаправ-
внешних воздействий [13, 78]. Однако недав-
ленно, формируя репрессивные мультибел-
ние исследования in vitro продемонстрирова-
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
797
ли, что Dps блокирует эндонуклеазы рестрик-
зависимости от состава буфера in vitro кри-
ции, но не РНК-полимеразы от связывания
сталлическая упаковка комплекса ДНК-Dps
с ДНК [79].
в растворе может меняться от гексагональной
Для выяснения сложного механизма функ-
до кубической с межслоевыми расстояниями
циональных взаимоотношений среди белков
соответственно 9,97 и 9,81 нм [44, 45], и ДНК
NAP необходимо сначала детально разобраться
в этих структурах располагается во впадинах
в молекулярных особенностях функциониро-
между сферическими молекулами белка.
вания индивидуальных белков. На настоящий
На основании анализа приведённых выше
момент основная структурная информация о
литературных данных и результатов нашей ра-
белках NAP получена с помощью рентгено-
боты мы можем предложить гипотезу, которая
структурного анализа (РСА) и не отражает их
несколько иначе представляет процесс фор-
поведения в растворе, о чём свидетельствуют
мирования комплексов ДНК-Dps, где вместо
структурные исследования методом ЯМР, про-
крупных сферических молекул Dps в ранней
ведённые, к сожалению, для незначительного
стационарной фазе присутствует более ком-
числа белков. В этой ситуации именно мало-
пактный белок IHF, который доминирует в
угловые исследования могут выявить важные
этой фазе и предшествует появлению Dps в
особенности поведения индивидуальных NAP
доминантных количествах. Как мы показали,
в нативном (не кристаллическом) состоянии.
IHF образует олигомеры в виде цепочек, что
Результаты проведённых в данной работе
предполагает возможность слоевой укладки
исследований с помощью МУРР показывают,
ДНК, изгибая её в тороидальную структуру в
что белки HU и IHF олигомеризуются в рас-
соответствии с функциональной особенностью
творе, причём в разной степени: для IHF ха-
белка этого класса. Кроме того, геометриче-
рактерно присутствие крупных олигомеров,
ские размеры и форма IHF могут объяснить
состоящих из исходных димеров, выстроен-
формирование тороидов в ранней стацио-
ных в цепочку. Это наблюдение очень важно
нарной фазе с межплоскостным расстоянием
с точки зрения взаимодействия этих бел-
6,8-7,5 нм, как это было определено Frenkiel-
ков с ДНК на поздних стадиях роста клеток.
Krispin et al. [43]. В этом случае можно предпо-
В работе Frenkiel-Krispin et al. [43] с помощью
ложить, что в поздней стационарной фазе мо-
просвечивающей электронной микроскопии
лекулы IHF заменяются на Dps, который даже
(ПЭМ) и томографической реконструкции
без ДНК образует мультислоевую структуру с
в эксперименте in vivo было показано, что в
гексагональной упаковкой додекамера [80] и
начале стационарной фазы роста бактериаль-
тем самым определяет структуру возникающе-
ный хроматин претерпевает массивную реор-
го кристаллического комплекса ДНК-Dps.
ганизацию и формируется в упорядоченные
Мы предполагаем, что именно таким об-
тороидальные структуры. Авторами был сде-
разом белки-предшественники HU и IHF на
лан вывод, что основным фактором такой ре-
разных стадиях жизненного цикла бактери-
организации является белок Dps. Тороидаль-
альной клетки подготавливают формирова-
ная форма хроматина характерна для периода
ние защитного кристаллического комплекса
24-часового голодания бактерий E. coli. Далее,
ДНК-Dps. Будучи гомологами, схожими по
по мере голодания тороидальная морфология,
форме, HU и IHF могут действовать и после-
по мнению авторов, действуя как структурный
довательно, и вместе. В целом, можно заклю-
шаблон, способствует формированию гексаго-
чить, что переход от активного роста бактерий
нальных кристаллических решёток ДНК-Dps
к стационарной фазе влечёт за собой скоор-
посредством эпитаксиального роста, и после
динированный процесс, в котором энерго-
48-часового голодания тороидальные струк-
зависимый динамический порядок хромати-
туры полностью исчезают. Интерпретация,
на последовательно заменяется равновесным
предложенная Frenkiel-Krispin et al. [43], не-
кристаллическим порядком. Тем не менее
сколько противоречива, поскольку в получен-
возможен и обратный переход. Так, в работе
ных тороидальных формах расстояние между
Lee et al. [81] показано, что изменение фак-
чередующимися слоями, предположительно
торов окружающей среды в физиологическом
ДНК-Dps, оценивалось в 6,8-7,5 нм, что не-
диапазоне, например, повышение pH и кон-
достаточно для наличия в этих слоях белка Dps
центрации MgCl2, приводит к переключению
с размерами 9 нм в присутствии нитей ДНК
связывания ДНК с Dps на IHF-связывание,
толщиной порядка
2,4 нм, учитывая даже
т.е. к возвращению к транскрипционно актив-
20%-ную возможную усадку под электронным
ной структуре нуклеоида, когда клетки могут
лучом. Нами с помощью МУРР и криоэлек-
перейти к экспоненциальной фазе и способны
тронной микроскопии было показано, что в
продолжать свою метаболическую активность.
БИОХИМИЯ том 88 вып. 5 2023
798
ДАДИНОВА и др.
Важно отметить, что подобные процессы
поведения индивидуальных NAP в нативном
в бактериальных клетках происходят посред-
(не кристаллическом) состоянии.
ством мультимеризации NAP. Эти процессы, а
также формирование комплексов NAP с ДНК
Вклад авторов. Л.А. Дадинова, Э.В. Шты-
являются обратимыми (см. выше), так как
кова
- концепция и руководство работой;
бактерии нуждаются в динамической модуля-
Л.А. Дадинова, М.В. Петухов, А.М. Горди-
ции своих нуклеоидных структур на разных
енко, Г.С. Петерс - проведение эксперимен-
фазах роста, а также в ответ на изменения
тов; В.А. Манувера, В.Н. Лазарев, Т.В. Раки-
окружающей среды. При этом большая часть
тина, А.А. Можаев
- получение образцов;
физиологически значимых белок-белковых
Л.А. Дадинова, М.В. Петухов, А.М. Гордиенко,
и белок-геномных взаимодействий являются
В.А. Манувера, В.Н. Лазарев, Т.В. Ракитина,
транзиентными, что позволяет белкам легко
А.А. Можаев, Г.С. Петерс, Э.В. Штыкова - на-
переходить из одного комплекса в другой при
писание текста; М.В. Петухов, Т.В. Ракитина,
изменении условий среды.
Э.В. Штыкова - редактирование текста статьи.
Олигомеризация NAP вызывает интерес
Финансирование. Работа выполнена при
многих научных групп, и эти процессы ещё
поддержке Российского научного фонда (грант
далеко не полностью исследованы. С помо-
№ 18-74-10071) в части исследований белка
щью малоуглового рентгеновского рассеяния
IHF и при поддержке Министерства науки и
нам удалось не только детектировать образую-
высшего образования в рамках выполнения
щиеся при сравнительно низких концентраци-
работ по Государственному заданию ФНИЦ
ях белка (2-5 мг/мл) мультимеры HU и IHF,
«Кристаллография и фотоника» РАН в части
но также определить их пространственную
исследований белка HU.
структуру. Полученные нами результаты ста-
Конфликт интересов. Авторы заявляют об
нут основой для дальнейшего изучения про-
отсутствии конфликта интересов.
цессов биокристаллизации и помогут выявить
Соблюдение этических норм. Настоящая
важные в научном (понимание механизма дей-
статья не содержит описания каких-либо ис-
ствия) и практическом (разработка фармако-
следований с участием людей или использова-
логических ингибиторов) плане особенности
нием животных в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Dame, R. T. (2005) The role of nucleoid-associated
strand segregation and the generation of phenotypic
proteins in the organization and compaction of
heterogeneity in bacteria, FEMS Microbiol. Rev.,
bacterial chromatin, Mol. Microbiol., 56, 858-870,
fuac049, doi: 10.1093/femsre/fuac049.
doi: 10.1111/j.1365-2958.2005.04598.x.
7. Amemiya, H. M., Schroeder, J., and Freddolino, P. L.
2. Johnson, R. C., Johnson, L. M., Schmidt, J. W.,
(2021) Nucleoid-associated proteins shape chromatin
and Gardner, J. F. (2005) Major Nucleoid Proteins
structure and transcriptional regulation across
in the Structure and Function of the Escherichia coli
the bacterial kingdom, Transcription, 12, 182-218,
Chromosome, in The Bacterial Chromosome (Patrick
doi: 10.1080/21541264.2021.1973865.
Higgins, N., ed.) ACM Press, Washington, DC,
8. Wold, S., Crooke, E., and Skarstad, K.
(1996)
pp. 65-131.
The Escherichia coli Fis protein prevents initiation of
3. Travers, A., and Muskhelishvili, G. (2005) Bacterial
DNA replication from oriC in vitro, Nucleic Acids Res.,
chromatin, Curr. Opin. Genet. Dev., 15, 507-514,
24, 3527-3532, doi: 10.1093/nar/24.18.3527.
doi: 10.1016/j.gde.2005.08.006.
9. Atlung, T., and Ingmer, H. (1997) H-NS: a modulator
4. Dillon, S. C., and Dorman, C. J. (2010) Bacterial
of environmentally regulated gene expression,
nucleoid-associated proteins, nucleoid structure and
Mol. Microbiol., 24, 7-17, doi: 10.1046/j.1365-2958.
gene expression, Nat. Rev. Microbiol., 8, 185-195,
1997.3151679.x.
doi: 10.1038/nrmicro2261.
10. Kamashev, D., and Rouviere-Yaniv, J. (2000) The his-
5. Glinkowska, M., Waldminghaus, T., and Riber, L.
tone-like protein HU binds specifically to DNA re-
(2021) Editorial: bacterial chromosomes under chang-
combination and repair intermediates, EMBO J., 19,
ing environmental conditions, Front. Microbiol., 12,
6527-6535, doi: 10.1093/emboj/19.23.6527.
633466, doi: 10.3389/fmicb.2021.633466.
11. Shahul Hameed, U. F., Liao, C., Radhakrishnan,
6. Norris, V., Kayser, C., Muskhelishvili, G., and Konto-
A. K., Huser, F., Aljedani, S. S., Zhao, X., Momin,
Ghiorghi, Y. (2022) The roles of nucleoid-associated
A. A., Melo, F. A., Guo, X., Brooks, C., Li, Y.,
proteins and topoisomerases in chromosome structure,
Cui, X., Gao, X., Ladbury, J. E., Jaremko, L.,
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
799
Jaremko, M., Li, J., and Arold, S. T. (2019) H-NS
23.
Losada, A., and Hirano, T. (2005) Dynamic molecular
uses an autoinhibitory conformational switch for
linkers of the genome: the first decade of SMC
environment-controlled gene silencing, Nucleic Acids
proteins, Genes Dev., 19, 1269-1287, doi: 10.1101/
Res., 47, 2666-2680, doi: 10.1093/nar/gky1299.
gad.1320505.
12.
Holowka, J., and Zakrzewska-Czerwinska, J. (2020)
24.
Nasmyth, K., and Haering, C. H. (2005) The structure
Nucleoid associated proteins: the small organizers that
and function of SMC and kleisin complexes, Annu.
help to cope with stress, Front. Microbiol., 11, 590,
Rev. Biochem., 74, 595-648, doi: 10.1146/annurev.
doi: 10.3389/fmicb.2020.00590.
biochem.74.082803.133219.
13.
Luijsterburg, M. S., Noom, M. C., Wuite, G. J., and
25.
Strunnikov, A. V. (2006) SMC complexes in bacterial
Dame, R. T. (2006) The architectural role of nucleoid-
chromosome condensation and segregation, Plasmid,
associated proteins in the organization of bacterial
55, 135-144, doi: 10.1016/j.plasmid.2005.08.004.
chromatin: a molecular perspective, J. Struct. Biol.,
26.
Swinger, K. K., and Rice, P. A. (2004) IHF and HU:
156, 262-272, doi: 10.1016/j.jsb.2006.05.006.
flexible architects of bent DNA, Curr. Opin. Struct.
14.
Wang, W., Li, G. W., Chen, C., Xie, X. S., and
Biol., 14, 28-35, doi: 10.1016/j.sbi.2003.12.003.
Zhuang, X. (2011) Chromosome organization by a
27.
Kamashev, D. E., Rakitina, T. V., Matyushkina, D. S.,
nucleoid-associated protein in live bacteria, Science,
Evsyutina, D. V., Vanyushkina, A. A., Agapova, Y. K.,
333, 1445-1449, doi: 10.1126/science.1204697.
Anisimova, V. E., Drobyshev, A. L., Butenko, I. O.,
15.
Hommais, F., Krin, E., Laurent-Winter, C.,
Pobeguts, O. V., and Fisunov, G. Y. (2019) Proteome
Soutourina, O., Malpertuy, A., Le Caer, J. P.,
of HU-Lacking E. coli studied by means of 2D Gel
Danchin, A., and Bertin, P.
(2001) Large-scale
electrophoresis, Russ. J. Bioorg. Chem., 45, 366-373,
monitoring of pleiotropic regulation of gene expression
doi: 10.1134/S1068162019050029.
by the prokaryotic nucleoid-associated protein, H-NS,
28.
Kamashev, D., Agapova, Y., Rastorguev, S., Talyzina,
Mol. Microbiol., 40, 20-36, doi: 10.1046/j.1365-2958.
A. A., Boyko, K. M., Korzhenevskiy, D. A., Vlaskina, A.,
2001.02358.x.
Vasilov, R., Timofeev, V. I., and Rakitina, T. V. (2017)
16.
Tendeng, C., and Bertin, P. N. (2003) H-NS in
Comparison of histone-like HU protein DNA-binding
Gram-negative bacteria: a family of multifaceted pro-
properties and HU/IHF protein sequence alignment,
teins, Trends Microbiol., 11, 511-518, doi: 10.1016/
PLoS One, 12, e0188037, doi: 10.1371/journal.pone.
j.tim.2003.09.005.
0188037.
17.
Dorman, C. J., Hinton, J. C., and Free, A. (1999)
29.
Stojkova, P., Spidlova, P., and Stulik, J.
(2019)
Domain organization and oligomerization among
Nucleoid-associated protein HU: A lilliputian in gene
H-NS-like nucleoid-associated proteins in bacteria,
regulation of bacterial virulence, Front. Cell. Infect.
Trends Microbiol.,
7,
124-128, doi:
10.1016/
Microbiol., 9, 159, doi: 10.3389/fcimb.2019.00159.
s0966-842x(99)01455-9.
30.
Prieto, A. I., Kahramanoglou, C., Ali, R. M., Fraser,
18.
Thanbichler, M., Wang, S. C., and Shapiro, L.
G. M., Seshasayee, A. S., and Luscombe, N. M. (2012)
(2005) The bacterial nucleoid: a highly organized and
Genomic analysis of DNA binding and gene regulation
dynamic structure, J. Cell. Biochem., 96, 506-521,
by homologous nucleoid-associated proteins IHF and
doi: 10.1002/jcb.20519.
HU in Escherichia coli K12, Nucleic Acids Res., 40,
19.
Leonard, P. M., Smits, S. H., Sedelnikova, S. E.,
3524-3537, doi: 10.1093/nar/gkr1236.
Brinkman, A. B., de Vos, W. M., van der Oost, J.,
31.
Remesh, S. G., Verma, S. C., Chen, J. H., Ekman,
Rice, D. W., and Rafferty, J. B. (2001) Crystal
A. A., Larabell, C. A., Adhya, S., and Hammel, M.
structure of the Lrp-like transcriptional regulator
(2020) Nucleoid remodeling during environmental
from the archaeon Pyrococcus furiosus, EMBO J., 20,
adaptation is regulated by HU-dependent DNA
990-997, doi: 10.1093/emboj/20.5.990.
bundling, Nat. Commun., 11, 2905, doi: 10.1038/
20.
Beloin, C., Jeusset, J., Revet, B., Mirambeau, G., Le
s41467-020-16724-5.
Hegarat, F., and Le Cam, E. (2003) Contribution of
32.
Hales, L. M., Gumport, R. I., and Gardner, J. F.
DNA conformation and topology in right-handed
(1994) Determining the DNA sequence elements
DNA wrapping by the Bacillus subtilis LrpC protein,
required for binding integration host factor to two
J. Biol. Chem., 278, 5333-5342, doi: 10.1074/jbc.
different target sites, J. Bacteriol., 176, 2999-3006,
M207489200.
doi: 10.1128/jb.176.10.2999-3006.1994.
21.
Calvo, J. M., and Matthews, R. G. (1994) The leucine-
33.
Kostrewa, D., Granzin, J., Koch, C., Choe, H. W.,
responsive regulatory protein, a global regulator of
Raghunathan, S., Wolf, W., Labahn, J., Kahmann, R.,
metabolism in Escherichia coli, Microbiol. Rev., 58,
and Saenger, W. (1991) Three-dimensional structure
466-490, doi: 10.1128/mr.58.3.466-490.1994.
of the E. coli DNA-binding protein FIS, Nature, 349,
22.
Brinkman, A. B., Ettema, T. J., de Vos, W. M., and
178-180, doi: 10.1038/349178a0.
van der Oost, J. (2003) The Lrp family of tran-
34.
Pan, C. Q., Finkel, S. E., Cramton, S. E., Feng, J. A.,
scriptional regulators, Mol. Microbiol., 48, 287-294,
Sigman, D. S., and Johnson, R. C. (1996) Variable
doi: 10.1046/j.1365-2958.2003.03442.x.
structures of Fis-DNA complexes determined by
БИОХИМИЯ том 88 вып. 5 2023
800
ДАДИНОВА и др.
flanking DNA-protein contacts, J. Mol. Biol., 264,
and Shtykova, E. (2021) Structural rearrangement
675-695, doi: 10.1006/jmbi.1996.0669.
of Dps-DNA complex caused by divalent Mg and
35.
Shao, Y., Feldman-Cohen, L. S., and Osuna, R. (2008)
Fe cations, Int. J. Mol. Sci., 22, 6056, doi: 10.3390/
Biochemical identification of base and phosphate
ijms22116056.
contacts between Fis and a high-affinity DNA
47.
Сошинская Е. Ю., Дадинова Л. А., Можаев А. А.,
binding site, J. Mol. Biol., 380, 327-339, doi: 10.1016/
Штыкова Э. В. (2020) Влияние состава буфера
j.jmb.2008.04.075.
на конформационную подвижность N-конце-
36.
Azam, T. A., and Ishihama, A. (1999) Twelve species
вых фрагментов Dps и характер взаимодействия
of the nucleoid-associated protein from Escherichia
с ДНК. Исследование методом малоуглового
coli. Sequence recognition specificity and DNA
рентгеновского рассеяния, Кристаллография, 65,
binding affinity, J. Biol. Chem., 274, 33105-33113,
886-895, doi: 10.31857/S0023476120060338.
doi: 10.1074/jbc.274.46.33105.
48.
Dubrovin, E. V., Dadinova, L. A., Petoukhov, M. V.,
37.
Almiron, M., Link, A. J., Furlong, D., and Kolter, R.
Soshinskaya, E. Y., Mozhaev, A. A., Klinov, D. V.,
(1992) A novel DNA-binding protein with regulatory
Schaffer, T. E., Shtykova, E. V., and Batishchev, O. V.
and protective roles in starved Escherichia coli, Genes
(2021) Spatial organization of Dps and DNA-Dps
Dev., 6, 2646-2654, doi: 10.1101/gad.6.12b.2646.
complexes, J. Mol. Biol., 433, 166930, doi: 10.1016/
38.
Nair, S., and Finkel, S. E. (2004) Dps protects
j.jmb.2021.166930.
cells against multiple stresses during stationary
49.
Shtykova, E. V., Petoukhov, M. V., and Mozhaev, A. A.
phase, J. Bacteriol., 186, 4192-4198, doi: 10.1128/
(2022) Formation of iron oxide nanoparticles in the
JB.186.13.4192-4198.2004.
internal cavity of ferritin-like Dps protein: studies by
39.
Frenkiel-Krispin, D., and Minsky, A. (2006) Nucleoid
anomalous X-ray scattering, Biochemistry (Moscow),
organization and the maintenance of DNA integrity in
87, 511-523, doi: 10.1134/S0006297922060037.
E. coli, B. subtilis and D. radiodurans, J. Struct. Biol.,
50.
Loiko, N., Danilova, Y., Moiseenko, A., Kovalenko, V.,
156, 311-319, doi: 10.1016/j.jsb.2006.05.014.
Tereshkina, K., Tutukina, M., El-Registan, G.,
40.
Martinez, A., and Kolter, R. (1997) Protection of
Sokolova, O., and Krupyanskii, Y. (2020) Morpho-
DNA during oxidative stress by the nonspecific DNA-
logical peculiarities of the DNA-protein complexes in
binding protein Dps, J. Bacteriol., 179, 5188-5194,
starved Escherichia coli cells, PLoS One, 15, e0231562,
doi: 10.1128/jb.179.16.5188-5194.1997.
doi: 10.1371/journal.pone.0231562.
41.
Reich, Z., Wachtel, E. J., and Minsky, A. (1994)
51.
Krupyanskii, Y. F., Kovalenko, V. V., Loiko, N. G.,
Liquid-crystalline mesophases of plasmid DNA
Generalova, A. A., Moiseenko, A. V., Tereshkin, E. V.,
in bacteria, Science, 264, 1460-1463, doi: 10.1126/
Sokolova, O. S., Tereshkina, K. B., El’-Registan,
science.8197460.
G. I., and Popov, A. N. (2022) Architecture of con-
42.
Wolf, S. G., Frenkiel, D., Arad, T., Finkel, S. E.,
densed DNA in the nucleoid of Escherichia coli
Kolter, R., and Minsky, A. (1999) DNA protection by
bacterium, Biophysics,
67,
506-517, doi:
10.1134/
stress-induced biocrystallization, Nature, 400, 83-85,
S0006350922040133.
doi: 10.1038/21918.
52.
Grant, R. A., Filman, D. J., Finkel, S. E., Kolter, R.,
43.
Frenkiel-Krispin, D., Ben-Avraham, I., Englander, J.,
and Hogle, J. M. (1998) The crystal structure of
Shimoni, E., Wolf, S. G., and Minsky, A. (2004)
Dps, a ferritin homolog that binds and protects
Nucleoid restructuring in stationary-state bacteria,
DNA, Nat. Struct. Biol., 5, 294-303, doi: 10.1038/
Mol. Microbiol.,
51,
395-405, doi:
10.1046/
nsb0498-294.
j.1365-2958.2003.03855.x.
53.
Gupta, S., and Chatterji, D. (2003) Bimodal protec-
44.
Dadinova, L. A., Chesnokov, Y. M., Kamyshinsky,
tion of DNA by Mycobacterium smegmatis DNA-bind-
R. A., Orlov, I. A., Petoukhov, M. V., Mozhaev, A. A.,
ing protein from stationary phase cells, J. Biol. Chem.,
Soshinskaya, E. Y., Lazarev, V. N., Manuvera, V. A.,
278, 5235-5241, doi: 10.1074/jbc.M208825200.
Orekhov, A. S., Vasiliev, A. L., and Shtykova, E. V.
54.
Ceci, P., Cellai, S., Falvo, E., Rivetti, C., Rossi,
(2019) Protective Dps-DNA co-crystallization in
G. L., and Chiancone, E. (2004) DNA condensation
stressed cells: an in vitro structural study by small-angle
and self-aggregation of Escherichia coli Dps are
X-ray scattering and cryo-electron tomography, FEBS
coupled phenomena related to the properties of the
Lett., 593, 1360-1371, doi: 10.1002/1873-3468.13439.
N-terminus, Nucleic Acids Res.,
32,
5935-5944,
45.
Kamyshinsky, R., Chesnokov, Y., Dadinova, L.,
doi: 10.1093/nar/gkh915.
Mozhaev, A., Orlov, I., Petoukhov, M., Orekhov, A.,
55.
Agapova, Y. K., Altukhov, D. A., Timofeev, V. I.,
Shtykova, E., and Vasiliev, A. (2019) Polymorphic
Stroylov, V. S., Mityanov, V. S., Korzhenevskiy, D. A.,
protective Dps-DNA co-crystals by cryo electron
Vlaskina, A. V., Smirnova, E. V., Bocharov, E. V.,
tomography and small Angle X-Ray scattering,
and Rakitina, T. V. (2020) Structure-based inhibitors
Biomolecules, 10, 39, doi: 10.3390/biom10010039.
targeting the alpha-helical domain of the Spiroplasma
46.
Dadinova, L., Kamyshinsky, R., Chesnokov, Y.,
melliferum histone-like HU protein, Sci. Rep., 10,
Mozhaev, A., Matveev, V., Gruzinov, A., Vasiliev, A.,
15128, doi: 10.1038/s41598-020-72113-4.
БИОХИМИЯ том 88 вып. 5 2023
ПОВЕДЕНИЕ В РАСТВОРЕ РОДСТВЕННЫХ NAP
801
56.
Svergun, D. I., Koch, M. H., Timmins, P. A., and
66.
Svergun, D. I., Barberato, C., and Koch, M. H. J.
May, R. P. (2013) Small Angle X-ray and Neutron
(1995) CRYSOL - a program to evaluate X-ray
Scattering from Solutions of Biological Macromolecules,
solution scattering of biological macromolecules
Oxford University Press, London.
from atomic coordinates, J. Appl. Cryst., 28, 768-773,
57.
Nash, H. A., Robertson, C. A., Flamm, E., Weisberg,
doi: 10.1107/S0021889895007047.
R. A., and Miller, H. I. (1987) Overproduction of
67.
Petoukhov, M. V., Billas, I. M., Takacs, M.,
Escherichia coli integration host factor, a protein with
Graewert, M. A., Moras, D., and Svergun, D. I.
nonidentical subunits, J. Bacteriol., 169, 4124-4127,
(2013) Reconstruction of quaternary structure from
doi: 10.1128/jb.169.9.4124-4127.1987.
X-ray scattering by equilibrium mixtures of biological
58.
Klock, H. E., and Lesley, S. A. (2009) The Poly-
macromolecules, Biochemistry,
52,
6844-6855,
merase Incomplete Primer Extension (PIPE) method
doi: 10.1021/bi400731u.
applied to high-throughput cloning and site-directed
68.
Winardhi, R. S., Yan, J., and Kenney, L. J. (2015)
mutagenesis, Methods Mol. Biol.,
498,
91-103,
H-NS regulates gene expression and compacts the
doi: 10.1007/978-1-59745-196-3_6.
nucleoid: insights from single-molecule experiments,
59.
Vorgias, C. E., and Wilson, K. S. (1991) A rapid
Biophys. J.,
109,
1321-1329, doi:
10.1016/j.bpj.
purification procedure of recombinant integration host
2015.08.016.
factor from Escherichia coli, Protein Expr. Purif., 2,
69.
Hammel, M., Amlanjyoti, D., Reyes, F. E., Chen,
317-320, doi: 10.1016/1046-5928(91)90089-2.
J. H., Parpana, R., Tang, H. Y., Larabell, C. A., Tainer,
60.
Peters, G. S., Zakharchenko, O. A., Konarev, P. V.,
J. A., and Adhya, S. (2016) HU multimerization shift
Karmazikov, Y. V., Smirnov, M. A., Zabelin, A. V.,
controls nucleoid compaction, Sci. Adv., 2, e1600650,
Mukhamedzhanov, E. H., Veligzhanin, A. A., Blagov,
doi: 10.1126/sciadv.1600650.
A. E., and Kovalchuk, M. V. (2019) The small-
70.
Huang, L., Zhang, Z., and McMacken, R. (2021)
angle X-ray scattering beamline BioMUR at the
Interaction of the Escherichia coli HU protein with
Kurchatov synchrotron radiation source, Nuclear
various topological forms of DNA, Biomolecules, 11,
Instr. Methods Phys. Res. Sec. A Accelerat. Spectrom.
1724, doi: 10.3390/biom11111724.
Detect. Assoc. Equip., 945, 162616, doi: 10.1016/j.nima.
71.
Frenkiel-Krispin, D., Levin-Zaidman, S., Shimoni, E.,
2019.162616.
Wolf, S. G., Wachtel, E. J., Arad, T., Finkel, S. E.,
61.
Peters, G. S., Gaponov, Y. A., Konarev, P. V.,
Kolter, R., and Minsky, A. (2001) Regulated phase
Marchenkova, M. A., Ilina, K. B., Volkov, V. V.,
transitions of bacterial chromatin: a non-enzymatic
Pisarevskiy, Y. V., and Kovalchuk, M. V.
(2022)
pathway for generic DNA protection, EMBO J., 20,
Upgrade of the BioMUR beamline at the Kurchatov
1184-1191, doi: 10.1093/emboj/20.5.1184.
synchrotron radiation source for serial small-angle
72.
Petoukhov, M. V., Franke, D., Shkumatov, A. V.,
X-ray scattering experiments in solutions, Nuclear
Tria, G., Kikhney, A. G., Gajda, M., Gorba, C.,
Instr. Methods Phys. Res. Sec. A Accelerat. Spectrom.
Mertens, H. D. T., Konarev, P. V., and Svergun, D. I.
Detect. Assoc. Equip., 1025, 166170, doi: 10.1016/
(2012) New developments in the ATSAS program
j.nima.2021.166170.
package for small-angle scattering data analysis,
62.
Hammersley, A. P.
(2016) FIT2D: a multi-
J. Appl. Crystallogr.,
45,
342-350, doi:
10.1107/
purpose data reduction, analysis and visualization
S0021889812007662.
program, J. Appl. Cryst., 49, 646-652, doi: 10.1107/
73.
Svergun, D. I.
(1999) Restoring low resolution
S1600576716000455.
structure of biological macromolecules from solution
63.
Konarev, P. V., Volkov, V. V., Sokolova, A. V., Koch,
scattering using simulated annealing, Biophys. J., 76,
M. H. J., and Svergun, D. I. (2003) PRIMUS - a
2879-2886, doi: 10.1016/S0006-3495(99)77443-6.
Windows-PC based system for small-angle scattering
74.
Guinier, A. (1939) La diffraction des rayons X aux
data analysis, J. Appl. Cryst.,
36,
1277-1282,
tres petits angles; application a l’etude de phenomenes
doi: 10.1107/S0021889803012779.
ultramicroscopiques, Ann. Phys. (Paris), 12, 161-237,
64.
Manalastas-Cantos, K., Konarev, P. V., Hajizadeh,
doi: 10.1051/anphys/193911120161.
N. R., Kikhney, A. G., Petoukhov, M. V., Molo-
75.
Boyko, K. M., Rakitina, T. V., Korzhenevskiy, D. A.,
denskiy, D. S., Panjkovich, A., Mertens, H. D. T.,
Vlaskina, A. V., Agapova, Y. K., Kamashev, D. E.,
Gruzinov, A., Borges, C., Jeffries, C. M., Svergun,
Kleymenov, S. Y., and Popov, V. O. (2016) Structural
D. I., and Franke, D. (2021) ATSAS 3.0: expanded
basis of the high thermal stability of the histone-like
functionality and new tools for small-angle scattering
HU protein from the mollicute Spiroplasma melliferum
data analysis, J. Appl. Cryst., 54, 343-355, doi: 10.1107/
KC3, Sci. Rep., 6, 36366, doi: 10.1038/srep36366.
S1600576720013412.
76.
Jacques, D. A., Guss, J. M., Svergun, D. I., and
65.
Svergun, D. I. (1992) Determination of the regular-
Trewhella, J. (2012) Publication guidelines for struc-
ization parameter in indirect-transform methods us-
tural modelling of small-angle scattering data from
ing perceptual criteria, J. Appl. Cryst., 25, 495-503,
biomolecules in solution, Acta Crystallogr. Sec. D, 68,
doi: 10.1107/S0021889892001663.
620-626, doi:10.1107/S0907444912012073.
8
БИОХИМИЯ том 88 вып. 5 2023
802
ДАДИНОВА и др.
77. Krissinel, E., and Henrick, K. (2007) Inference of
Global DNA compaction in stationary-phase bacteria
macromolecular assemblies from crystalline state,
does not affect transcription, Cell, 174, 1188-1199.
J. Mol. Biol.,
372,
774-797, doi:
10.1016/j.jmb.
e1114, doi: 10.1016/j.cell.2018.06.049.
2007.05.022.
80. Ren, B., Tibbelin, G., Kajino, T., Asami, O., and
78. Stephani, K., Weichart, D., and Hengge, R. (2003)
Ladenstein, R. (2003) The multi-layered structure
Dynamic control of Dps protein levels by ClpXP and
of Dps with a novel di-nuclear ferroxidase center,
ClpAP proteases in Escherichia coli, Mol. Microbiol.,
J. Mol. Biol., 329, 467-477, doi: 10.1016/s0022-2836
49, 1605-1614, doi: 10.1046/j.1365-2958.2003.03644.x.
(03)00466-2.
79. Janissen, R., Arens, M. M. A., Vtyurina, N. N., Rivai, Z.,
81. Lee, S. Y., Lim, C. J., Droge, P., and Yan, J. (2016)
Sunday, N. D., Eslami-Mossallam, B., Gritsenko, A. A.,
Regulation of bacterial DNA packaging in early
Laan, L., de Ridder, D., Artsimovitch, I., Dekker,
stationary phase by competitive DNA binding of Dps
N. H., Abbondanzieri, E. A., and Meyer, A. S. (2018)
and IHF, Sci. Rep., 5, 18146, doi: 10.1038/srep18146.
NUCLEOID-ASSOCIATED PROTEINS HU AND IHF:
OLIGOMERIZATION IN SOLUTION
AND HYDRODYNAMIC PROPERTIES
L. A. Dadinova1, M. V. Petoukhov1, A. M. Gordienko1, V. A. Manuvera2,3, V. N. Lazarev2,3,
T. V. Rakitina4,5, A. A. Mozhaev1,4, G. S. Peters5, and E. V. Shtykova1*
1 Shubnikov Institute of Crystallography of Federal Scientific Research Centre “Crystallography and Photonics”,
Russian Academy of Sciences, 119333 Moscow, Russia; shtykova@ns.crys.ras.ru
2 Lopukhin Federal Research and Clinical Center of Physical-Chemical Medicine,
Federal Medical Biological Agency, 119435 Moscow, Russia
3 Moscow Institute of Physics and Technology (State University),
141701 Dolgoprudny, Moscow Region, Russia
4 Shemyakin-Ovchinnikov Institute of Bioorganic Chemistry, Russian Academy of Sciences,
117997 Moscow, Russia
5 NRC “Kurchatov Institute”, 123182 Moscow, Russia
The structure and function of the bacterial nucleoid is controlled by nucleoid-associated NAP proteins.
In any phase of growth, various NAPs, acting sequentially, condense the nucleoid and provide its tran-
scriptionally active structure. However, in the late stationary phase, only one of the NAPs, the Dps protein,
is strongly expressed, and DNA-protein crystals are formed that transform the nucleoid into a static, tran-
scriptionally inactive structure, effectively protected from external influences. The discovery of crystal
structures in living cells and the association of this phenomenon with bacterial resistance to antibiotics has
aroused great interest in studying this phenomenon. The aim of this work is to obtain and compare the
structures of two related NAPs (HU and IHF), since they are the ones that accumulate in the cell at the late
stationary stage of growth, which precedes the formation of the protective DNA-Dps crystalline complex.
For structural studies, two complementary methods were used in the work: small-angle X-ray scattering
(SAXS) as the main method for studying the structure of proteins in solution and dynamic light scattering as
an additional one. Various approaches and computer programs were used to interpret the SAXS data, which
made it possible to determine the macromolecular characteristics and obtain reliable structural 3D models
of various oligomeric forms of the HU and IHF proteins. It was shown that these proteins oligomerize in
solution to varying degrees, and IHF is characterized by the presence of large oligomers consisting of initial
dimers arranged in a chain. It was suggested that just before Dps expression, it is this protein that forms the
toroidal structures previously observed in vivo and prepares the platform for the formation of DNA-Dps
crystals. The results obtained are necessary for further study of the phenomenon of biocrystal formation
in bacterial cells and finding ways to overcome the resistance of various pathogens to external conditions.
Keywords: nucleoid-associated proteins, histone-like proteins HU and IHF, oligomerization, small-angle X-ray
scattering, dynamic light scattering
БИОХИМИЯ том 88 вып. 5 2023