БИОХИМИЯ, 2023, том 88, вып. 6, с. 891 - 899
УДК 577.152
РЕЗИСТОМ Streptomyces rimosus - РЕЗЕРВУАР ГЕНОВ УСТОЙЧИВОСТИ
К АМИНОГЛИКОЗИДНЫМ АНТИБИОТИКАМ
© 2023 М.Г. Алексеева*, Н.Н. Рудакова, А.В. Ратькин,
Д.А. Мавлетова, В.Н. Даниленко
Институт общей генетики им. Н.И. Вавилова РАН,
119991 Москва, Россия; электронная почта: alekseevamg@mail.ru
Поступила в редакцию 16.03.2023
После доработки 20.04.2023
Принята к публикации 20.04.2023
Изучение аминогликозидацетилтрансфераз у актинобактерий рода Streptomyces является составной
частью исследования почвенных бактерий как основного резервуара и возможного источника генов
лекарственной устойчивости. Ранее нами в штамме Streptomyces rimosus subsp. rimosus ATCC 10970
(продуценте окситетрациклина), устойчивом к большинству природных аминогликозидных анти-
биотиков (АГ), были идентифицированы и биохимически охарактеризованы 3 аминогликозидфос-
фотрансферазы, которые обусловливают устойчивость к канамицину, неомицину, паромомицину,
стрептомицину и гигромицину В. В представленной работе было показано, что устойчивость к
остальным АГ у данного штамма связана с наличием фермента аминогликозидацетилтрансферазы,
относящейся к подсемейству AAC(2′). Индуцирование экспрессии гена, обозначенного нами как
aac(2′)-If, в клетках Escherichia coli определяет устойчивость к широкому спектру природных АГ
(неомицину, гентамицину, тобрамицину, сизомицину и паромомицину) и к повышению мини-
мальных ингибирующих концентраций данных антибиотиков.
КЛЮЧЕВЫЕ СЛОВА: множественная лекарственная устойчивость, Streptomyces rimosus, аминогликозид-
ацетилтрансферазы, МИК аминогликозидных антибиотиков.
DOI: 10.31857/S0320972523060015, EDN: EDCRBD
ВВЕДЕНИЕ
сящихся к роду Streptomyces, которые являются
продуцентами широкого спектра антибиоти-
Устойчивость к антибиотикам является
ков и значительным резервуаром генов устой-
одной из самых больших проблем в совре-
чивости к антибиотикам в почвах [3, 4].
менной медицине и глобальной угрозой для
Множественная лекарственная устойчи-
здравоохранения, обусловленных катастрофи-
вость к аминогликозидным антибиотикам (АГ)
ческим распространением клинических штам-
обусловлена наличием в геномах бактерий
мов бактерий с множественной лекарственной
модифицирующих ферментов, относящихся к
устойчивостью. Окружающая среда является
трем подклассам: аминогликозидфосфотранс-
крупнейшим источником и резервуаром рези-
феразам (APH), аминогликозид-3-N-ацетил-
стентности - почвенные, водные, атмосфер-
трансферазам (AAC) и аминогликозиднуклео-
ные, связанные с животными и искусственные
тидилтрансферазам (АНТ). Индуцирование
экосистемы содержат элементы устойчивости
синтеза ферментов, модифицирующих ами-
к антибиотикам [1, 2]. Возможность передачи
ногликозиды, представляет собой стратегию
генетических элементов устойчивости между
выживания устойчивых к антибиотикам бак-
бактериями в смешанных популяциях при-
терий [4, 5]. Ферменты APH и AAC идентифи-
водит к увеличению горизонтального пере-
цированы и охарактеризованы в основном у
носа, появлению и селекции устойчивых форм
штаммов-продуцентов аминогликозидов, таких
бактерий. Гены устойчивости к антибиотикам
как Streptomyces griseus (продуцент стрептоми-
гипотетически берут начало в бактериях, отно- цина), Streptomyces kanamyceticus (продуцент
Принятые сокращения: АГ - аминогликозидные антибиотики; МИК - минимальная ингибирующая концентрация;
ААС - аминогликозидацетилтрансферазы; APH - аминогликозидфосфотрансферазы.
* Адресат для корреспонденции.
891
892
АЛЕКСЕЕВА и др.
канамицина), Streptomyces fradiae (продуцент
США) [15], E. coli BL21(DE3) (F-, dcm, ompT,
неомицина). Данные штаммы устойчивы и к
hsdS(rB-mB-), gal λ (DE3)) («Novagen», США)
другим АГ, продуцентами которых они не явля-
[16] и E. coli NiCo21(DE3) (can::CBD fhuA2 [lon]
ются. Также было показано широкое распро-
ompT gal (λ DE3) [dcm] arnA::CBD slyD::CBD
странение множественной устойчивости к АГ
glmS6Ala ΔhsdS λ DE3 = λ sBamHIo ΔEcoRI-B
у АГ-непродуцирующих актиномицетов [6, 7].
int:: i21 Δnin5) («New England Biolabs», США) [17].
В то же время первичный биоинформа-
Для клонирования использовали экспрессион-
тический анализ показал, что гены, анноти-
ные векторы pET16b и pET32a («Novagen») [16],
рованные как aph и aac, широко представ-
содержащие His-Tag в N-концевой области для
лены во многих геномах актинобактерий рода
выделения и очистки рекомбинантных белков.
Streptomyces. При этом ферменты, обусловли-
Штамм S. rimosus ATCC 10970 выращивали
вающие устойчивость к АГ, в настоящее вре-
на жидкой среде YEME, содержащей 25% са-
мя мало изучены - некоторые из них связаны
харозы [18]. Для выращивания клеток E. coli
с устойчивостью к антибиотикам, остальные
использовали среду Лурия (L-бульон), твер-
имеют другие функции, в том числе могут уча-
дые среды содержали 2% (w/v) агара [19]. Для
ствовать в коммуникациях с другими почвен-
обеспечения селективного роста клеток, со-
ными организмами. Поэтому важной задачей
держащих плазмиды, добавляли ампициллин
является выявление и характеристика новых
(100 мкг/мл).
ферментов, участвующих в устойчивости к ами-
Манипуляции с ДНК. Геномную ДНК штам-
ногликозидным антибиотикам [6, 8].
ма S. rimosus ATCC 10970 выделяли методом,
Ранее было установлено, что штамм Strepto-
изложенным в руководстве Kieser et al. [18].
myces rimosus subsp. rimosus АТСС 10970 (проду-
Выделение плазмидной ДНК, получение ком-
цент окситетрациклина) обладает устойчиво-
петентных клеток E. coli, трансформацию и
стью к большинству природных АГ. В штамме
анализ рекомбинантных плазмид проводили
S. rimosus ATCC 10970 нами были идентифи-
стандартными методами [19]. Фрагмент ДНК,
цированы и биохимически охарактеризованы
кодирующий AAC(2′)-If S. rimosus, амплифи-
аминогликозидфосфотрансферазы: APH(3′)-VIII,
цировали с геномной ДНК с использованием
APH(3′′)-Id и AphSR2, обусловливающие устой-
набора Phusion High-Fidelity PCR Master Mix
чивость к канамицину, неомицину, паромоми-
(«Thermo Fisher Scientific», Литва) на мини-
цину, стрептомицину и гигромицину В. Получе-
циклере PTC-0150 («MJ Research Inc.», США).
ны пространственные структуры APH(3′)-VIII
Для амплификации были сконструированы
и APH(3′′)-Id (в апоформе и в комплексе со
олигонуклеотиды: AAC-SrN1 и AAC-SrC1 -
стрептомицином и ADP) [9-13].
для клонирования гена aac(2′)-If в плазми-
В настоящей работе проводилась иденти-
ду pET16b по сайтам эндонуклеаз рестрик-
фикация в геноме S. rimosus ATCC 10970 ами-
ции NdeI и BamHI и олигонуклеотиды:
ногликозидацетилтрансфераз, которые потен-
AAC-SrN2 и AAC-SrC2 - для клонирования
циально могут обусловливать устойчивость
гена aac(2′)-If в плазмиду pET32a по сайтам
штамма к АГ. Мы идентифицировали фер-
эндонуклеаз рестрикции BamHI и HindIII.
мент, относящийся к описанному ранее под-
Нуклеотидные последовательности прайме-
семейству, обозначенный нами как AAC(2′)-If.
ров, использованных в работе («Синтол», Рос-
При клонировании гена, кодирующего дан-
сия), представлены в табл. 1.
ный фермент, в клетках Escherichia coli уста-
новлено, что он определяет устойчивость
Таблица 1. Последовательности праймеров,
к широкому спектру природных АГ; были
использованных в работе
определены минимальные ингибирующие
концентрации (МИК) аминогликозидных
Название
Последовательность праймеров 5′→3′*
праймера
антибиотиков для клеток E. coli, экспрессиру-
ющих aac(2′)-If.
AAC-SrN1
tcgtcatatgatgaccgacgcacaccccct
AAC-SrC1
agccggatccctaccagacgtccccact
МАТЕРИАЛЫ И МЕТОДЫ
AAC-SrN2
tcgcggatccatgaccgacgcacaccccct
Бактериальные штаммы, векторы, среды
и условия культивирования. В работе исполь-
AAC-SrC2
ccgcaagcttctaccagacgtccccac
зовали штаммы: S. rimosus subsp. rimosus
ATCC 10970 [14], E. coli DH5a (F-, Ф
80
* Сайты для эндонуклеаз рестрикции выделены подчер-
ΔlacZΔM15, Δ(lacZYA-argF), U169) («Promega»,
киванием.
БИОХИМИЯ том 88 вып. 6 2023
РЕЗИСТОМ Streptomyces rimosus
893
Экспрессия гена aac(2′)-If штамма S. rimo-
добавляли по 100 мкл культуры клеток. Для ин-
sus ATCC
10970 в Е. coli. Клетки E. coli, со-
дуцирования экспрессии AAC(2′)-If в пробирки
держащие плазмиды pET16b:aac(2′)-If и
добавляли IPTG (100 мкМ). После культиви-
pET32a:aac(2′)-If, выращивали на качалке в
рования в течение 18 ч при 25 °С определяли
жидкой среде (L-бульон) с ампициллином при
значения МИК как самые низкие концентра-
37 °С до оптической плотности 0,6 при 625 нм
ции АГ, которые приводили к полному ингиби-
(~2 ч), затем индуцировали экспрессию добав-
рованию роста (что определялось спектрофото-
лением изопропил-D-тиогалактозида (ИПТГ)
метрически при 625 нм).
до финальной концентрации 0,5 и 1,0 мМ.
Биоинформатический анализ. Последователь-
Далее проводили культивирование при 28 °С в
ности аминогликозидацетилтрансфераз (ААС)
течение 18 ч, после чего клетки осаждали цен-
штамма S. rimosus ATCC 10970 были полу-
трифугированием (3000 g, 10 мин, 4 °C) и хра-
чены из базы данных NCBI. Для сравнения
нили в морозильной камере при -20 °C. Клет-
их с известными по литературе ААС исполь-
ки размораживали и суспендировали в буфере
зовали программы Blastp [22]; для сопостав-
для образцов, содержащем 62,5 мМ Tris-HCl
ления с последовательностями белков, пред-
(pH 6,8), 5% глицерина, 2% 2-меркаптоэта-
ставленных в базе данных PDB, с известными
нола,
0,1% SDS и 0,001% бромфенолового
3D-структурами - SAS [23]; для множествен-
синего, затем нагревали при 95 °C в течение
ных выравниваний аминокислотных по-
10 мин. Растворимую фракцию белков ана-
следовательностей использовали алгоритм
лизировали с помощью электрофореза в
ClustalW [24].
12,5%-ном Ds-Na-ПААГ с использованием
Статистический анализ проводили с исполь-
маркера молекулярной массы белков SM0441
зованием t-критерия Стьюдента.
(«Fermentas», Литва). В качестве контроля
анализировали растворимые фракции белков
штаммов E. coli BL21(DE3) и NiCo21(DE3),
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
содержащих плазмиды pET16b и pET32а
без вставки.
Биоинформатический анализ аминогликозид-
Анализ устойчивости к аминогликозидным
ацетилтрансфераз штамма S. rimosus ATCC 10970.
антибиотикам. Антимикробная активность для
В геноме штамма ATCC 10970 [14] аннотировано
клеток E. coli BL21(DE3), экспрессирующих
13 генов N-ацетилтранфераз семейства GNAT
AAC(2′)-If, определялась как МИК с исполь-
с номерами локусов генов: SRIM_009260;
зованием двух методов: метода диффузии с
SRIM_011135; SRIM_012310; SRIM_018100;
использованием стандартных дисков и метода
SRIM_020380; SRIM_020760; SRIM_025200;
линейных разведений. В качестве контроля
SRIM_029930; SRIM_030810; SRIM_033455;
анализировали клети E. coli BL21(DE3), содер-
SRIM_038510; SRIM_040130; SRIM_040160.
жащие плазмиду pET32а.
Все выявленные N-ацетилтранферазы содер-
Для диско-диффузионного метода [20] чаш-
жат домены «NAT_SF» и сайты связывания с
ки с LB-агаром, содержащим 10 мкг/мл ИПТГ,
коэнзимом А.
инокулировали путем равномерного покрытия
Сравнение аминокислотных последова-
их бактериальными культурами в концентра-
тельностей всех выявленных в S. rimosus фер-
ции 105-106 КОЕ/мл. Бумажные диски, содер-
ментов N-ацетилтранфераз с последователь-
жащие АГ, помещали на чашки с LB-агаром,
ностями известных ранее аминогликозидаце-
инкубировали при 37 °С в течение 18 ч, затем
тилтрансфераз, относящихся к подсемействам
измеряли диаметры зон вокруг дисков, в кото-
AAC(1), AAC(3), AAC(2′) и AAC(6′), с исполь-
рых отсутствовал рост клеток.
зованием программ BLAST и SAS показало,
МИК для аминогликозидных антибиоти-
что только один из них (SRIM_030810) имеет
ков определяли методом разведения в буль-
степень идентичности 52,1% с AAC(2′)-Ic My-
оне в соответствии с рекомендациями Инсти-
cobacterium tuberculosis (GenBank: CCP42991.1,
тута клинических и лабораторных стандартов
Код PDB: 1m44) и 50,0% - с AAC(2′)-Id My-
(CLSI) [21]. Клетки E. coli выращивали в 2 мл
colicibacterium smegmatis (GenBank: AIU12332.1,
бульона Мюллера-Хинтона (MHB) до плотно-
Код PDB: 7crm).
сти 0,3 при 625 нм для адаптации к стандарту
Для подсемейства AAC(2′) в настоящее
0,5 McFarland (мера плотности культуры; он
время идентифицирован только один под-
соответствует значению 1,5 × 108 КОЕ/мл), а
класс AAC(2′)-I. Ген, кодирующий AAC(2′)-Ia,
затем разбавляли до конечной плотности 105-
был обнаружен в хромосоме условно-патоген-
106 КОЕ/мл. В пробирки, содержащие 2-крат-
ной бактерии Providencia stuartii, AAC(2′)-Ib
ные разведения антибиотиков в 2 мл MHB,
идентифицирован у Mycolicibacterium fortuitum,
БИОХИМИЯ том 88 вып. 6 2023
894
АЛЕКСЕЕВА и др.
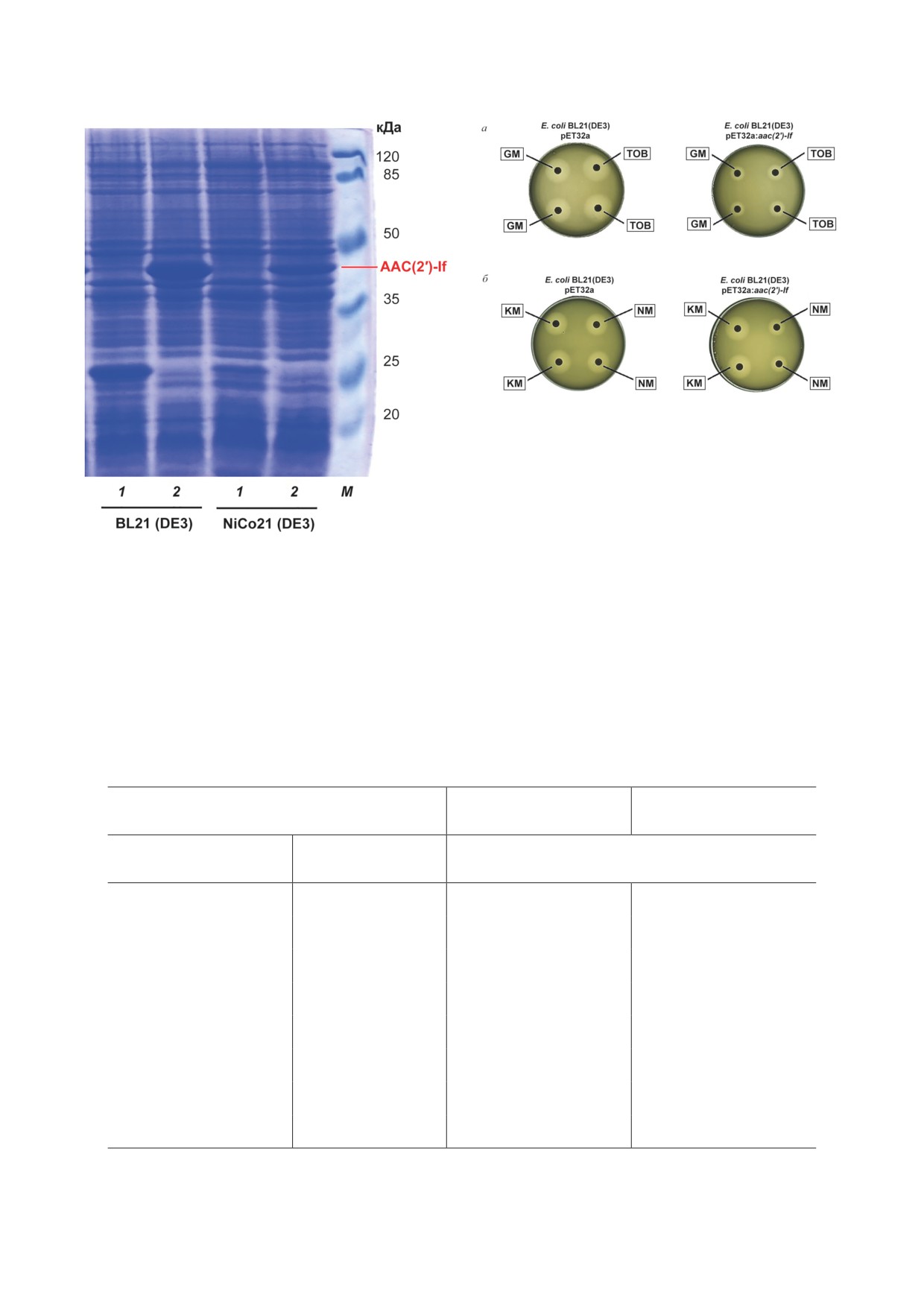
Рис. 1. Выравнивание последовательностей AAC(2′)-If (AAC(2′)-Sr) c AAC(2′)-Ia P. stuartii, AAC(2′)-Ib M. fortuitum,
AAC(2′)-Ic M. tuberculosis, AAC(2′)-Id M. smegmatis и AAC(2′)-Ie M. leprae. Консервативные аминокислоты, характер-
ные для AAC(2′)-I, выделены серым цветом
AAC(2′)-Ic - у M. tuberculosis и Mycobacterium
водили выращивание штаммов BL21(DE3) и
bovis, AAC(2′)-Id - у M. smegmatis, AAC(2′)-Ie -
NiCo21(DE3), содержащих рекомбинантную
у Mycobacterium leprae. Ферменты подсемейства
плазмиду pET16b:aac(2′)-If, в жидкой среде LB
AAC(2′) способны как к N-, так и к O-ацетили-
с индукцией ИПТГ, затем клетки осаждали
рованию многих аминогликозидных субстра-
центрифугированием и анализировали с по-
тов: канамицина, паромомицина, гентамицина,
мощью электрофореза в 12,5%-ном Ds-Na-
амикацина и тобрамицина [25-27].
ПААГ. Анализ электрофореграммы показал,
Степень идентичности (сходства) амино-
что ген aac(2′)-If не экспрессируется в E. coli в
кислотных последовательностей между SRIM_
составе экспрессионного вектора pET16b.
030810 (AAC(2′)-Sr) и AAC(2′)-Ia, AAC(2′)-Ib,
В связи с этим проводили клонирова-
AAC(2′)-Ic, AAC(2′)-Id, AAC(2′)-Ie составляет
ние гена aac(2′)-If в составе экспрессионного
36,5 (64,9)%; 51,7 (80,1)%; 51,2 (78,5)%; 48,2 (72,8)%
вектора pET32a, содержащего последователь-
и 48,6 (74,6)% соответственно. Выравнивание
ность гена тиоредоксина в N-концевой обла-
последовательностей (рис. 1) показывает, что
сти. Полученные результаты (рис. 2) показа-
AAC(2′)-Sr содержит все консервативные ами-
ли, что в клетках штаммов E. coli BL21(DE3)
нокислоты [28], характерные для последователь-
и NiCo21(DE3) наблюдалась дополнительная
ностей ферментов подсемейства AAC(2′), поэто-
фракция белка с молекулярной массой около
му мы обозначили этот фермент как AAC(2′)-If.
37 кДа, что соответствует расчетной моле-
Клонирование гена aac(2′)-If в Е. coli и ана-
кулярной массе белка AAC(2′)-If в сумме с
лиз экспрессии. На первом этапе проводили
молекулярной массой белка линкера плазми-
клонирование гена aac(2′)-If в составе экс-
ды pET32a, содержащей тиоредоксин. Макси-
прессионного вектора pET16b. Для изуче-
мальная экспрессия гена aac(2′)-If была уста-
ния экспрессии гена aac(2′)-If в E. coli про-
новлена в штамме E. coli BL21(DE3).
БИОХИМИЯ том 88 вып. 6 2023
РЕЗИСТОМ Streptomyces rimosus
895
Рис.
3. Анализ спектра устойчивости клеток E. coli
BL21(DE3), экспрессирующих ген aac(2′)-If, к АГ мето-
дом стандартных дисков: а - к гентамицину и тобрами-
цину; б - к канамицину и неомицину. KM - канамицин;
NM - неомицин; GM - гентамицин; TOB - тобрамицин
рующих ген aac(2′)-If, к АГ мы использовали
метод стандартных дисков. Установлено, что
Рис. 2. Электрофореграмма растворимой фракции бел-
ков AAC(2′)-If штаммов E. coli BL21(DE3) и NiCo21(DE3),
экспрессия гена aac(2′)-If обеспечивает устой-
содержащих плазмиды: 1 - pET32a, 2 - pET32a:aac(2′)-If;
чивость клеток E. coli BL21(DE3) к широкому
М - маркер молекулярной массы белков SM0441
спектру природных АГ: неомицину, гентами-
цину, тобрамицину, сизомицину и паромоми-
цину (табл. 2, рис. 3). Полученные результаты
Исследование спектра и уровня устойчиво-
согласуются с биоинформатическим анали-
сти E. coli BL21(DE3), содержащего плазмиду
зом, согласно которому данный ген отнесен
pET32a:aac(2′)-If, к аминогликозидным анти-
к подсемейству AAC(2′).
биотикам. Для определения спектра устойчи-
Мы определили МИК АГ с использованием
вости клеток E. coli BL21(DE3), экспресси- метода
2-кратных разведений антибиотиков
Таблица 2. Исследование спектра устойчивости E. coli BL21(DE3), содержащего плазмиду pET32a:aac(2′)-If,
к аминогликозидным антибиотикам
BL21(DE3)
BL21(DE3)
Антибиотики
pET32a
pET32a:aac(2′)-If
концентрация,
Название
диаметр зоны, мм*
мкг/диск
Канамицин
30
21 ± 0,5
21 ± 0,5
Неомицин
30
20 ± 0,5
# 15 ± 0,5
Паромомицин
10
17 ± 0,5
# 7 ± 0,5
Стрептомицин
10
22 ± 0,5
22 ± 0,5
Гентамицин
10
22 ± 0,5
# 15 ± 0,5
Тобрамицин
10
26 ± 0,5
# 17 ± 0,5
Сизомицин
10
25 ± 0,5
# 19 ± 0,5
Амикацин
30
24 ± 0,5
24 ± 0,5
Примечание. * Приведены усредненные результаты четырех независимых измерений ± стандартные отклонения.
# Достоверное отличие от контроля (BL21(DE3) pET32a), р < 0,01.
БИОХИМИЯ том 88 вып. 6 2023
896
АЛЕКСЕЕВА и др.
Таблица
3. Минимальные ингибирующие концентра-
чивость к АГ: канамицину, неомицину, паро-
ции (МИК) аминогликозидных антибиотиков
момицину, стрептомицину и гигромицину В.
Штаммы E. coli; МИК, мкг/мл*
Однако ранее было показано, что штамм
S. rimosus ATCC 10970 устойчив ко всем при-
АГ-антибиотик
BL21(DE3)
BL21(DE3)
родным АГ. В связи с этим фактом было сде-
pET32a
pET32a:aac(2′)-If
лано предположение, что устойчивость к АГ у
данного штамма может быть обусловлена не
Гентамицин
4 ± 1
80 ± 2#
только АРН, но и другими ферментами, таки-
ми как ААС.
Сизомицин
4 ± 1
64 ± 2#
Анализ генома S. rimosus ATCC 10970 [14]
позволил нам выявить 13 генов, кодирующих
Паромомицин
16 ± 2
250 ± 5#
N-ацетитранферазы семейства GNAT, содер-
жащих домены «NAT_SF» и сайты связывания
Тобрамицин
8 ± 2
120 ± 4#
с коэнзимом А. На основании сходства амино-
кислотных последовательностей выявленных
Неомицин
16 ± 2
120 ± 4#
N-ацетитранфераз с последовательностями опи-
санных ранее аминогликозидацетилтрансфераз
Канамицин
16 ± 2
16 ± 2
только одна из них была отнесена к известно-
му подсемейству AAC(2′).
Стрептомицин
16 ± 2
16 ± 2
Ферменты подсемейства AAC(2′) ранее были
идентифицированы в хромосоме условно-па-
Примечание. * Приведены усредненные результаты четы-
рех независимых измерений ± стандартные отклонения.
тогенной бактерии P. stuartii и у микобакте-
рий [25-27]. Множественное выравнивание
# Достоверное отличие от контроля (BL21(DE3) pET32a),
р < 0,01.
последовательностей показало, что AAC(2′)-Sr
содержит все консервативные аминокислоты,
характерные для последовательностей фер-
в жидкой среде. Полученные результаты (табл. 3)
ментов подсемейства AAC(2′) [28], поэтому
показывают, что индукция экспрессии гена
мы обозначили этот фермент как AAC(2′)-If.
aac(2′)-If приводит к 20-кратному увеличению
Мы клонировали ген aac(2′)-If в E. coli. Оцен-
МИК гентамицина - с 4 до 80 мкг/мл. Анало-
ка устойчивости к АГ показала, что индукция
гичные изменения наблюдались для сизоми-
синтеза фермента AAC(2′)-If обусловливает
цина, паромомицина и тобрамицина (15-16-
устойчивость E. coli к широкому спектру при-
кратное увеличение МИК) и неомицина (7,5-
родных АГ: неомицину, гентамицину, тобра-
кратное увеличение МИК). Таким образом,
мицину, сизомицину и паромомицину, что
идентифицированный нами фермент AAC(2′)-If,
согласуется с биоинформатическим анализом,
индуцируемый в клетках E. coli, активен in vivo
согласно которому данный фермент отнесен к
и эффективно защищает клетки от данных ан-
подсемейству AAC(2′).
тибиотиков.
Происхождение AAC можно проследить до
Таким образом, оба исследования показы-
видов микробов из окружающей среды, пред-
вают, что идентифицированный нами у S. ri-
ставляющих собой обширный резервуар для
mosus ATCC 10970 фермент AAC(2′)-If придает
новых и появляющихся ферментов резистент-
бактериям устойчивость к АГ: гентамицину,
ности, которые в настоящее время недоста-
тобрамицину, неомицину, паромомицину и
точно изучены. Индукция синтеза ферментов,
сизомицину, и не оказывает влияния на устой-
модифицирующих АГ, представляет собой стра-
чивость к канамицину и стрептомицину.
тегию выживания устойчивых к антибиоти-
На основании полученных результатов мы
кам бактерий. ААС относятся к moonlighting-
можем предполагать, что гентамицин, тобра-
белкам, т.е. проявляют полифункциональные
мицин, неомицин, паромомицин и сизомицин
свойства. Так, у микобактерий ААС вносят
являются субстратами для идентифицирован-
частичный вклад в устойчивость к АГ и могут
ного нами фермента AAC(2′)-If.
выполнять другие функции, в частности, спо-
собствовать ацетилированию белков клеточ-
ной стенки и пептидогликана, что объясняет
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
широкую субстратную специфичность ААС-
ферментов [26].
В результате предыдущих исследований
Впервые понятие «резистом» было введено
нами выявлены 3 фермента АРН (APH(3′)-VIII,
D’Costa et al. [29] в 2006 г. при исследовании
APH(3′′)-Id и AphSR2), обусловливающих устой-
генов устойчивости к антибиотикам почвенных
БИОХИМИЯ том 88 вып. 6 2023
РЕЗИСТОМ Streptomyces rimosus
897
бактерий рода Streptomyces
- продуцентов
ЗАКЛЮЧЕНИЕ
большинства антибиотиков. Значительно ра-
нее один из авторов данной статьи проводил
Обобщая данные, полученные авторами
исследования распространения и механизмов
в ранее опубликованных [9-13] и представ-
устойчивости к антибиотикам среди коллек-
ленной работах, нами в штамме S. rimosus
ции различных видов актинобактерий рода
АТСС 10970, не являющемся продуцентом АГ,
Streptomyces [30]. Штамм S. rimosus ATCC 10970
идентифицировано 3 фермента АРН и фер-
(продуцент окситетрациклина) обратил на
мент AAC(2′)-If, обусловливающие устойчи-
себя внимание как яркий представитель, об-
вость штамма ко всем природным АГ.
ладающий тотальной устойчивостью ко всем
Представленные исследования открыва-
природным АГ [31]. В то время активно об-
ют новые возможности для изучения распро-
суждалась гипотеза происхождения и эво-
странения и особенностей экспрессии генов,
люции генов устойчивости к антибиотикам,
определяющих природную устойчивость к АГ
обобщенная в более поздней работе Davies
у актинобактерии рода Streptomyces.
и Davies [32].
В рамках дальнейшей работы предпо-
Позднее понятие
«резистом» стали ис-
лагается выделение рекомбинантного белка
пользовать для характеристики микробиомов
AAC(2′)-If, получение пространственных струк-
различных экологических систем: почв, вод-
тур для выявления каталитически важных
ных резервуаров, воздуха, растений, сельско-
структурных мотивов, сравнительный ана-
хозяйственных животных, продуктов питания
лиз полученных пространственных структур с
и человека. Резистом кишечника человека
известными структурами аминогликозидаце-
представляется наиболее уязвимым для здоро-
тилтрансфераз других видов бактерий (в том
вья источником генов лекарственной устойчи-
числе патогенных).
вости [33, 34].
Известно, что гены устойчивости к анти-
Вклад авторов. В.Н. Даниленко - концеп-
биотикам легко передаются между группами
ция и руководство работой; М.Г. Алексеева и
бактерий различных микробиомов с помощью
Н.Н. Рудакова - проведение экспериментов;
мобильных генетических элементов и других
М.Г. Алексеева, А.В. Ратькин и Д.А. Мавле-
систем переноса генетического материала,
това - обсуждение результатов исследования;
включая внеклеточные везикулы [2, 35, 36].
М.Г. Алексеева - написание текста; Д.А. Мав-
В представленной работе в штамме
летова - редактирование текста статьи.
S. rimosus ATCC 10970 нами идентифицирован
Финансирование. Работа выполнена в рам-
новый ААС-фермент, имеющий сходство с ми-
ках Государственного задания № 0092-2022-
кобактериальными ферментами, относящими-
003; тема «Механизмы генетических процес-
ся к подсемейству AAC(2′). При этом важные
сов у микроорганизмов, растений, животных
вопросы взаимодействия резистомов между
и человека»: «Микробиом кишечника чело-
собой, приводящие к горизонтальному пере-
века: иммуномодулирующий и антиоксидант-
носу генов антибиотикоустойчивости, требу-
ный потенциал», подтема «Распространение
ют дальнейших исследований. Обмен генами
и функции генов аминогликозид ацетилтранс-
устойчивости к антибиотикам между почвен-
фераз в микробиоме почв и человека».
ными бактериями и клиническими патогенами
Конфликт интересов. Авторы заявляют об
возможен путем прямого обмена между поч-
отсутствии конфликта интересов.
венными микробами и патогенами человека
Соблюдение этических норм. Настоящая
или путем непрямой передачи через микробио-
статья не содержит описания каких-либо ис-
ту кишечника человека, что подчеркивает кли-
следований с участием людей или животных в
ническое значение почвенного резистома [37].
качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Darby, E. M., Trampari, E., Siasat, P., Gaya, M. S.,
antibiotic resistome of soil bacteria and human
Alav, I., Webber, M. A., and Blair, J. M. A. (2022)
pathogens, Science,
337,
1107-1111, doi:
10.1126/
Molecular mechanisms of antibiotic resistance revis-
science.1220761.
ited, Nat. Rev. Microbiol., 21, 280-295, doi: 10.1038/
3. Surette, M. D., and Wright, G. D. (2017) Lessons
s41579-022-00820-y.
from the environmental antibiotic resistome, Annu.
2. Forsberg, K. J., Reyes, A., Wang, B., Selleck, E. M.,
Rev. Microbiol., 71, 309-329, doi: 10.1146/annurev-
Sommer, M. O., and Dantas, G. (2012) The shared
micro-090816-093420.
БИОХИМИЯ том 88 вып. 6 2023
898
АЛЕКСЕЕВА и др.
4.
Ogawara, H. (2019) Comparison of antibiotic resis-
14.
Algora-Gallardo, L., Schniete, J. K., Mark, D. R.,
tance mechanisms in antibiotic-producing and patho-
Hunter, I. S., and Herron, P. R. (2021) Bilateral sym-
genic bacteria, Molecules, 24, 3430, doi: 10.3390/
metry of linear streptomycete chromosomes, Microb.
molecules24193430.
Genom., 7, 000692, doi: 10.1099/mgen.0.000692.
5.
Ramirez, M. S., and Tolmasky, M. E.
(2010)
15.
Inoue, H., Nojima, H., and Okayama, H. (1990)
Aminoglycoside modifying enzymes, Drug. Resist.
High efficiency transformation of Escherichia coli
Updat., 13, 151-171, doi: 10.1016/j.drup.2010.08.003.
with plasmids, Gene, 96, 23-28, doi: 10.1016/0378-
6.
Hotta, K. (2021) Basic and applied research on
1119(90)90336-p.
multiple aminoglycoside antibiotic resistance of
16.
Mierendorf, R., Yeager, K., and Novy, R.
(1994)
actinomycetes: an old-timer’s recollection, J. Ind.
Innovations, Newslett. Novagen, 1, 1-3.
Microbiol. Biotechnol., 48, kuab059, doi: 10.1093/
17.
Bolanos-Garcia, V. M., and Davies, O. R. (2006)
jimb/kuab059.
Structural analysis and classification of native proteins
7.
Heinzel, P., Werbitzky, O., Distler, J., and Piepers-
from E. coli commonly co-purified by immobilised
berg, W. (1988) A second streptomycin resistance gene
metal affinity chromatography, Biochim. Biophys. Acta,
from Streptomyces griseus codes for streptomycin-3′′-
1760, 1304-1313, doi: 10.1016/j.bbagen.2006.03.027.
phosphotransferase. Relationships between antibiotic
18.
Kieser, T., Bibb, M. J., Buttner, M. J., Chater, K. F., and
and protein kinases, Arch. Microbiol., 150, 184-192,
Hopwood, D. A. (2000) Practical Streptomyces Genetics,
doi: 10.1007/BF00425160.
The John Innes Foundation, Norwich UK, 613 pp.
8.
Perry, J. A., Westman, E. L., and Wright, G. D. (2014)
19.
Sambrook, J., Fritsch, E. E., and Maniatis, T. (1989)
The antibiotic resistome: what’s new? Curr. Opin.
Molecular Cloning: A Laboratory Manual, Cold Spring
Microbiol., 21, 45-50, doi: 10.1016/j.mib.2014.09.002.
Harbor Laboratory Press, 479 pp.
9.
Елизаров С. М., Алексеева M. Г., Новиков Ф. Н.,
20.
Barry, A. L., and Thornsberry, C. (1993) Susceptibility
Чилов Г. Г., Маслов Д. А., Штиль А. А., Даниленко
Tests: Diffusion Test Procedures (Murray, P., ed.) Wash-
В. Н. (2012) Идентификация сайтов фосфорили-
ington D.C, ASM Press, pp. 112-137.
рования аминогликозидфосфотрансферазы VIII
21.
Wiekler, M. A. (2015) CLSI. Methods for Dilution
Streptomyces rimosus, Биохимия, 77, 1504-1512.
Antimicrobial Susceptibility Tests for Bacteria That
10.
Boyko, K. M., Gorbacheva, M. A., Korzhenevskiy,
Grow Aerobically. Approved Standard - Tenth Edi-
D. A., Alekseeva, M. G., Mavletova, D. A.,
tion. CLSI document M07-A10, Wayne, PA: Clinical
Zakharevich, N. V., Elizarov, S. M., Rudakova, N. N.,
and Laboratory Standards Institute.
Danilenko, V. N., and Popov, V. O. (2016) Struc-
22.
Altschul, S. F., Gish, W., Miller, W., Myers, E. W.,
tural characterization of the novel aminoglycoside
and Lipman, D. J. (1990) Basic local alignment
phosphotransferase AphVIII from Streptomyces rimosus
search tool, J. Mol. Biol., 215, 403-410, doi: 10.1016/
with enzymatic activity modulated by phosphoryla-
S0022-2836(05)80360-2.
tion, Biochem. Biophys. Res. Commun., 477, 595-601,
23.
Milburn, D., Laskowski, R. A., and Thornton, J. M.
doi: 10.1016/j.bbrc.2016.06.097.
(1998) Sequences annotated by structure: a tool to
11.
Алексеева М. Г., Рудакова Н. Н., Захаревич Н. В.,
facilitate the use of structural information in sequence
Мавлетова Д. А., Бойко К. М., Николаева А. Ю.,
analysis, Protein Eng., 11, 855-859, doi: 10.1093/
Корженевский Д. А., Даниленко В. Н.
(2018)
protein/11.10.855.
Новый ген аминогликозидфосфотрансферазы
24.
Larkin, M. A., Blackshields, G., Brown, N. P.,
aph(3′′)-Id из Streptomyces rimosus АТСС10970, коди-
Chenna, R., McGettigan, P. A., McWilliam, H.,
рующий устойчивость к стрептомицину, Генети-
Valentin, F., Wallace, I. M., Wilm, A., Lopez, R.,
ка, 54, 1228-1232, doi: 10.1134/S001667581810003X.
Thompson, J. D., Gibson, T. J., and Higgins,
12.
Alekseeva, M. G., Boyko, K. M., Nikolaeva, A. Y.,
D. G. (2007) Clustal W and Clustal X version 2.0,
Mavletova, D. A., Rudakova, N. N., Zakharevich,
Bioinformatics,
23,
2947-2948, doi:
10.1093/
N. V., Korzhenevskiy, D. A., Ziganshin, R. H., Popov,
bioinformatics/btm404.
V. O., and Danilenko, V. N. (2019) Identification,
25.
Hegde, S. S., Javid-Majd, F., and Blanchard, J. S.
functional and structural characterization of novel
(2001) Overexpression and mechanistic analysis of
aminoglycoside phosphotransferase APH(3″)-Id from
chromosomally encoded aminoglycoside 2′-N-acetyl-
Streptomyces rimosus subsp. rimosus ATCC 10970,
transferase (AAC(2′)-Ic) from Mycobacterium tubercu-
Arch. Biochem. Biophys., 671, 111-122, doi: 10.1016/
losis, J. Biol. Chem., 276, 45876-45881, doi: 10.1074/
j.abb.2019.06.008.
jbc.M108810200.
13.
Рудакова Н. Н., Алексеева М. Г., Захаревич Н. В.,
26.
Sanz-García, F., Anoz-Carbonell, E., Pérez-Herrán, E.,
Мавлетова Д. А., Даниленко В. Н. (2020) Амино-
Martín, C., Lucía, A., Rodrigues, L., and Aínsa, J. A.
гликозидфосфотрансфераза AphSR2 Streptomyces
(2019) Mycobacterial aminoglycoside acetyltrans-
rimosus ATCC 10970: зависимость устойчивости к
ferases: a little of drug resistance, and a lot of oth-
антибиотикам от серин-треониновых протеин-
er roles, Front. Microbiol., 10, 46, doi: 10.3389/
киназ PkSR1 и PkSR2, Генетика, 56, 119-124.
fmicb.2019.00046.
БИОХИМИЯ том 88 вып. 6 2023
РЕЗИСТОМ Streptomyces rimosus
899
27. Bassenden, A. V., Dumalo, L., Park, J., Blanchet, J.,
Biol. Rev.,
74,
417-433, doi:
10.1128/MMBR.
Maiti, K., Arya, D. P., and Berghuis, A. M. (2021)
00016-10.
Structural and phylogenetic analyses of resistance
33. Crits-Christoph, A., Hallowell, H. A., Koutouvalis, K.,
to next-generation aminoglycosides conferred by
and Suez, J. (2022) Good microbes, bad genes?
AAC(2′) enzymes, Sci. Rep., 11, 11614, doi: 10.1038/
The dissemination of antimicrobial resistance in the
s41598-021-89446-3.
human microbiome, Gut Microbes,
14,
2055944,
28. Aínsa, J. A., Pérez, E., Pelicic, V., Berthet, F. X.,
doi: 10.1080/19490976.2022.2055944.
Gicquel, B., and Martín, C. (1997) Aminoglycoside
34. Lee, K., Raguideau, S., Sirén, K., Asnicar, F.,
2′-N-acetyltransferase genes are universally present
Cumbo, F., Hildebrand, F., Segata, N., Cha, C. J.,
in mycobacteria: characterization of the aac(2′)-Ic
and Quince, C.
(2023) Population-level impacts
gene from Mycobacterium tuberculosis and the
of antibiotic usage on the human gut microbiome,
aac(2′)-Id gene from Mycobacterium smegmatis, Mol.
Nat. Commun.,
14,
1191, doi:
10.1038/s41467-
Microbiol.,
24,
431-441, doi:
10.1046/j.1365-2958.
023-36633-7.
1997.3471717.x.
35. Ellabaan, M. M. H., Munck, C., Porse, A., Imamov-
29. D’Costa, V. M., McGrann, K. M., Hughes, D. W.,
ic, L., and Sommer, M. O. A. (2021) Forecasting the
and Wright, G. D. (2006) Sampling the antibiotic
dissemination of antibiotic resistance genes across bac-
resistome, Science,
311,
374-377, doi:
10.1126/
terial genomes, Nat. Commun., 12, 2435, doi: 10.1038/
science.1120800.
s41467-021-22757-1.
30. Даниленко В. Н., Пузынина Г. Г., Ломовская Н. Д.
36. MacNair, C. R., and Tan, M. W. (2023) The role
(1977) Множественная антибиотикорезистентность
of bacterial membrane vesicles in antibiotic resis-
актиномицетов, Генетика, 13, 1831-1842.
tance, Ann. N. Y. Acad. Sci., 1519, 63-73, doi: 10.1111/
31. Потехин И. А., Даниленко В. Н. (1985) Детер-
nyas.14932.
минант устойчивости к канамицину Streptomyces
37. Jeong, C. S., Hwang, J., Do, H., Cha, S. S., Oh,
rimosus: амплификация в составе хромосомы и
T. J., Kim, H. J., Park, H. H., and Lee, J. H.
обратимая генетическая нестабильность, Молеку-
(2020) Structural and biochemical analyses of an
лярная биология, 19, 805-817.
aminoglycoside 2′-N-acetyltransferase from Mycolici-
32. Davies, J., and Davies, D.
(2010) Origins and
bacterium smegmatis, Sci. Rep., 10, 21503, doi: 10.1038/
evolution of antibiotic resistance, Microbiol. Mol.
s41598-020-78699-z.
RESISTOM Streptomyces rimosus - A RESERVOIR
OF RESISTANCE GENES TO AMINOGLYCOSIDE ANTIBIOTICS
M. G. Alekseeva*, N. N. Rudakova, A. V. Ratkin, D. A. Mavletova, and V. N. Danilenko
Vavilov Institute of General Genetics, Russian Academy of Sciences,
119991 Moscow, Russia; e-mail: alekseevamg@mail.ru
The study of aminoglycoside acetyltransferases in actinobacteria of the genus Streptomyces is an integral part
of the study of soil bacteria as the main reservoir and possible source of drug resistance genes. Previously,
in the strain Streptomyces rimosus ATCC 10970 (producing oxyteteracycline), which is resistant to most
natural aminoglycoside antibiotics, we have identified and biochemically characterized 3 aminoglycoside
phosphotransferases, which cause resistance to kanamycin, neomycin, paromomycin, streptomycin, and
hygromycin B. In the presented work, it was shown that resistance to other AGs in this strain is associat-
ed with the presence of the enzyme aminoglycoside acetyltransferase, belonging to the AAC(2′) subfamily.
Induction of the expression of the gene, designated by us as aac(2′)-If, in Escherichia coli cells determines re-
sistance to a wide range of natural aminoglycoside antibiotics (neomycin, gentamicin, tobramycin, sisomycin,
and paromomycin) and to an increase in the minimum inhibitory concentrations of these antibiotics.
Keywords: multidrug resistance, Streptomyces rimosus, aminoglycoside acetyltransferase, MICs for aminoglycoside
antibiotics
БИОХИМИЯ том 88 вып. 6 2023