БИОХИМИЯ, 2023, том 88, вып. 6, с. 973 - 983
УДК 577.22; 577.214.5
МНОЖЕСТВЕННЫЕ НЕКАНОНИЧЕСКИЕ
СТЭКИНГ-ВЗАИМОДЕЙСТВИЯ ГЕТЕРОЦИКЛИЧЕСКИХ
ОСНОВАНИЙ КАК ОДИН ИЗ ГЛАВНЫХ ФАКТОРОВ
ОРГАНИЗАЦИИ ТРЕТИЧНОЙ СТРУКТУРЫ РНК
© 2023 В.Г. Метелев1, Е.Ф. Баулин2, А.А. Богданов1,3,4*
1 Московский государственный университет имени М.В. Ломоносова,
химический факультет, 119991 Москва, Россия
2 Институт математических проблем биологии РАН - филиал ИПМ имени М.В. Келдыша РАН,
142290 Пущино, Россия
3 НИИ физико-химической биологии имени А.Н. Белозерского,
Московский государственный университет имени М.В. Ломоносова,
119992 Москва, Россия; электронная почта: bogdanov@belozersky.msu.ru
4 Государственный научный центр Федеральное государственное бюджетное учреждение науки
Институт биоорганической химии имени академиков М.М. Шемякина и Ю.А. Овчинникова РАН,
117997 Москва, Россия
Поступила в редакцию 21.03.2023
После доработки 18.04.2023
Принята к публикации 20.04.2023
Межплоскостные (стэкинг) взаимодействия гетероциклических оснований нуклеотидных остат-
ков (н.о.) РНК являются одним из важнейших факторов организации её вторичной и третичной
структуры. Большая часть таких (канонических) взаимодействий осуществляется между непо-
средственными соседями в полинуклеотидных цепях РНК. Однако по мере накопления данных
об атомных третичных структурах самых разнообразных РНК и их комплексов с белками стало
ясно, что с помощью стэкинга оснований (неканонического) могут взаимодействовать н.о. РНК,
не являющиеся соседями в их полинуклеотидных цепях и иногда разделённые в первичной струк-
туре РНК десятками или сотнями н.о. В настоящей работе представлена исчерпывающая база дан-
ных о таких элементах и их окружении в макромолекулах природных и синтетических РНК.
Они названы NA-BSE (nonadjacent base-stacking elements). Анализ этих данных показал, что фор-
мирующие NA-BSE н.о. составляют в среднем около четверти всех н.о. той или иной РНК и дол-
жны рассматриваться как полноценные мотивы в их третичной структуре. В работе проведена
классификация NA-BSE по типам локализации в макромолекулах РНК. Показано, что структуро-
образующая роль NA-BSE состоит в компактном сворачивании однотяжевых петель РНК, в пре-
вращении двутяжевых и более сложноорганизованных петель в несовершенные спирали, а также
в связывании удалённых в первичной и вторичной структуре районов РНК.
КЛЮЧЕВЫЕ СЛОВА: стэкинг-взаимодействия, третичная структура РНК, рибосомные РНК, рибо-
нуклеаза Р, РНК-мотивы.
DOI: 10.31857/S0320972523060076, EDN: EFLFMJ
ВВЕДЕНИЕ
всё ещё не можем, располагая только нуклео-
тидной последовательностью некой достаточно
Известно, что макромолекулярная струк-
протяжённой РНК, достоверно предсказать её
тура РНК определяет её функциональные свой-
пространственную структуру.
ства. И хотя общие принципы организации вто-
За последние 20-25 лет в изучении макро-
ричной и третичной структуры одноцепочечных
молекулярной структуры РНК произошла на-
РНК были сформулированы Fresco et. al. [1] и
стоящая революция: в руках исследователей
Спириным [2] более 60 лет назад, сегодня мы оказались тысячи третичных структур разно-
Принятые сокращения: н.о. - нуклеотидные остатки; РНКаза Р - рибонуклеаза Р; BIE - base-intercalated element;
BWE - base-waged element; DC - double-crossing; NA-BSE - nonadjacent base-stacking element.
* Адресат для корреспонденции.
973
974
МЕТЕЛЕВ и др.
образных природных РНК и их синтетиче-
их под сокращённым обозначением NA-BSE
ских аналогов, полученных с атомным раз-
(nonadjacent base-stacking elements). Соответ-
решением с помощью рентгеноструктурного
ственно, межплоскостные взаимодействия ну-
анализа, криоэлектронной микроскопии или
клеиновых оснований в NA-BSE мы называем
ЯМР-спектроскопии. Следует подчеркнуть, что
здесь неканоническими, чтобы отличить их от
многие из этих структур описывают детали
канонических стэкинг-взаимодействий сосед-
пространственной организации макромолекул
них н.о. в РНК и полинуклеотидах.
РНК в комплексе со специфическими белка-
В этой статье мы представляем базу дан-
ми, то есть в состоянии, близком к тому, в ко-
ных, включающую все разновидности мотивов
тором они находятся в клетке. Это особенно
этого семейства, и рассматриваем их структур-
важно, поскольку пространственная структура
ную роль. Они обнаруживаются во всех при-
свободной РНК, за редкими исключениями,
родных и многих синтетических РНК, для
существенно отличается от таковой в природ-
которых на сегодняшний день установлена
ных РНК-белковых комплексах. В настоящее
третичная структура с высоким разрешением.
время общедоступные банки данных (PDB,
Следует отметить, что существование NA-BSE
NDB) содержат более 5000 таких структур.
некоторых типов ранее эпизодически фикси-
Анализ этих данных постоянно приносит ин-
ровалось в третичных структурах ряда РНК
формацию об общих структурных элементах,
и рибонуклеопротеинов (РНП), см., напри-
т.н. мотивах, которые повторяются с разной
мер, работы Dallas et al., Cate et al. и Teplova
степенью регулярности в третичной структу-
et al. [8-10]. Однако их никогда не рассматри-
ре широкого спектра РНК [3]. Так, например,
вали как повторяющиеся элементы (мотивы) в
уже при анализе первых атомных структур в
пространственной структуре РНК, их не клас-
рибосомах (рибосомных РНК, рРНК) были
сифицировали по типам локализации в макро-
идентифицированы такие элементы, как A-ми-
молекулах РНК, а также не анализировали
норы [4] и K-повороты [5], и продемонстри-
их структурообразующие и функциональные
рована их общность для РНК других классов.
свойства. Такой систематический анализ про-
Следует подчеркнуть, что открытие новых моти-
водится впервые в данной работе. Он показы-
вов постоянно продолжается. Например, отно-
вает, что NA-BSE не только широко распро-
сительно недавно был обнаружен и охаракте-
странены во всех классах РНК, но и играют
ризован широко распространённый мотив, в
значительную роль в организации их третич-
котором свободная пара электронов O4′-атома
ной структуры.
остатка рибозы одного нуклеотида взаимо-
действует с плоскостью гетероциклического
основания другого нуклеотида [6]. Мы, в свою
МАТЕРИАЛЫ И МЕТОДЫ
очередь, описали мотивы, которые мы на-
звали BIE и BWE (base-intercalated element и
Мы определили NA-BSE как максималь-
base-waged element соответственно) [7]. В этих
ный набор из k нуклеотидов N1, ..., Nk, такой,
структурных элементах гетероциклическое осно-
что для каждого i < k Ni и Ni+1 образуют не-
вание удалённого нуклеотидного остатка (н.о.)
каноническое стэкинг-взамодействие основа-
либо интеркалирует между двумя соседними
ний, т.е. они не являются соседями по по-
основаниями определённого динуклеотидного
следовательности, и их основания уложены
сегмента РНК (BIE), либо вытесняет среднее
в стопку.
основание из тринуклеотидного сегмента, за-
Для построения набора данных NA-BSE
нимая его место (BWE). BIE и BWE участвуют
мы использовали неизбыточный набор РНК-
не только в сворачивании полинуклеотидных
содержащих структур из PDB с ограничением
цепей РНК в уникальные пространственные
среднего разрешения 3,0 Å (версия 3.166 [11]).
структуры, но также важны для организации
Для аннотирования NA-BSE использовалась
функциональных центров РНК и РНК-бел-
программа DSSR (версия 2.0 [12]). Результирую-
ковых комплексов. В ходе анализа BIE и BWE
щий набор данных включал только NA-BSE
мы заметили, что эти структурные элементы
из цепей РНК длиной ≥ 25 нуклеотидов, при
являются представителями гораздо более об-
этом анализировались стэкинг-взаимодействия
ширного семейства мотивов. Общим для это-
оснований, для которых угол основание-осно-
го семейства является стэкинг-взаимодействие
вание был <30°, и расстояние между бли-
между основаниями н.о., которые располо-
жайшими атомами двух оснований составля-
жены в первичной структуре РНК на разных
ло <4,0 Å. Другой набор данных был создан с
расстояниях друг от друга, но не являются не-
использованием неизбыточного набора струк-
посредственными соседями. Мы объединили
тур с ограничением разрешения 3,5 Å.
БИОХИМИЯ том 88 вып. 6 2023
НОВЫЕ МОТИВЫ В ТРЕТИЧНОЙ СТРУКТУРЕ РНК
975
Столбцы подготовленных таблиц были та-
взаимодействием основания с фосфатными груп-
кими же, как в работе Baulin et al. [7], и вклю-
пами, 22,7% мотивов стабилизированы взаимо-
чали параметры стэкинга, параметры спарива-
действием основания одного н.о. с O4′-атомом
ний, в которых участвуют основания, и другие
рибозы другого н.о. В большинстве NA-BSE
(см. вкладку readme в табл. S1 Приложения).
(56,1%) их основания находятся в стэкинге с
Мы также аннотировали третичные мотивы
основаниями фланкирующих н.о., располо-
РНК, имеющие общие нуклеотиды с NA-BSE,
женных с обеих сторон мотива; у 35,1% моти-
включая коаксиальный стэкинг, A-миноры,
вов взаимодействующий с ними фланкирую-
К-повороты, рибозные молнии, BIE, BWE, ди-
щий нуклеотид расположен только с одной
нуклеотидные платформы, внутренние петли с
стороны, а 8,8% мотивов вообще не имеют
тандемными GA-спариваниями и внутренние
фланкирующих н.о. Кроме того, гетероцикли-
петли типа UAA/GAN (см. табл. S2 Приложе-
ческие основания в большинстве NA-BSE об-
ния). Внутренние петли были аннотированы
разуют с основаниями соседних районов РНК
с использованием пакета URSLIB [13], а все
как уотсон-криковские, так и (гораздо чаще)
остальные мотивы были аннотированы с ис-
неуотсон-криковские пары (см. табл. S1 При-
пользованием DSSR.
ложения и рассматриваемые ниже примеры).
6903 NA-BSE (58,8%) не участвуют в фор-
мировании каких-либо других известных мо-
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
тивов, обнаруженных в третичных структурах
РНК; в то же время 4830 NA-BSE (41,2%), вхо-
Базы данных и статистика. В качестве ос-
дящих в базу данных «3,0», имели общие ну-
новной в этой работе использовалась база
клеотиды по крайней мере с одним из таких
данных, разработанная на основании анализа
мотивов.
третичных структур РНК, полученных с раз-
Типы NA-BSE. Структурные мотивы, опи-
решением не ниже 3,0 Å (база данных «3,0»).
сываемые в этой работе, можно подразделить
Она включает в себя 11 733 NA-BSE различ-
на три типа.
ных размеров, образованных за счёт стэкинг-
NA-BSE первого типа (NA-BSE I) образо-
взаимодействий между гетероциклическими
ваны н.о. одного и того же одноцепочечного
основаниями 14 862 н.о., не являющихся не-
участка или (крайне редко) остатками одной
посредственными соседями в полинуклео-
из цепей двухцепочечного участка РНК. В пер-
тидных цепях РНК (табл. S1 Приложения).
вичной структуре РНК они разделены всего
При этом 9525 NA-BSE состоят из двух н.о.,
несколькими (часто одним) н.о., а образова-
1500 мотивов - из трёх, 512 мотивов - из четы-
ние мотива сопровождается выходом из петли
рёх, 179 мотивов - из пяти и 17 мотивов - из
или из двойной спирали одного или несколь-
шести н.о. В основной базе данных NA-BSE,
ких остатков (рис. 2, а). Эти остатки часто иг-
образованные н.о., принадлежащими разным
рают важную роль в узнавании РНК белками
молекулам РНК, составляют всего 3,3%.
(см., например, работы Yogesh et al. и Laura
Из 11 733 NA-BSE
60,5% представлены
et al. [14, 15]).
только пуриновыми н.о., 32,4% - пуриновыми
Ко второму типу (NA-BSE II) мотивов это-
и пиримидиновыми и 5,9% - только пирими-
го семейства относятся структурные элементы,
диновыми н.о.; 1,2% NA-BSE содержат моди-
в которых стэкинг-взаимодействия осущест-
фицированные основания.
вляются между гетероциклическими основа-
Мотивы, описываемые в этой работе, об-
ниями н.о., находящихся в разных цепях одной
наруживаются во всех элементах вторичной
двойной спирали (как, например, на рис. 2, б)
структуры РНК. Это хорошо иллюстрирует
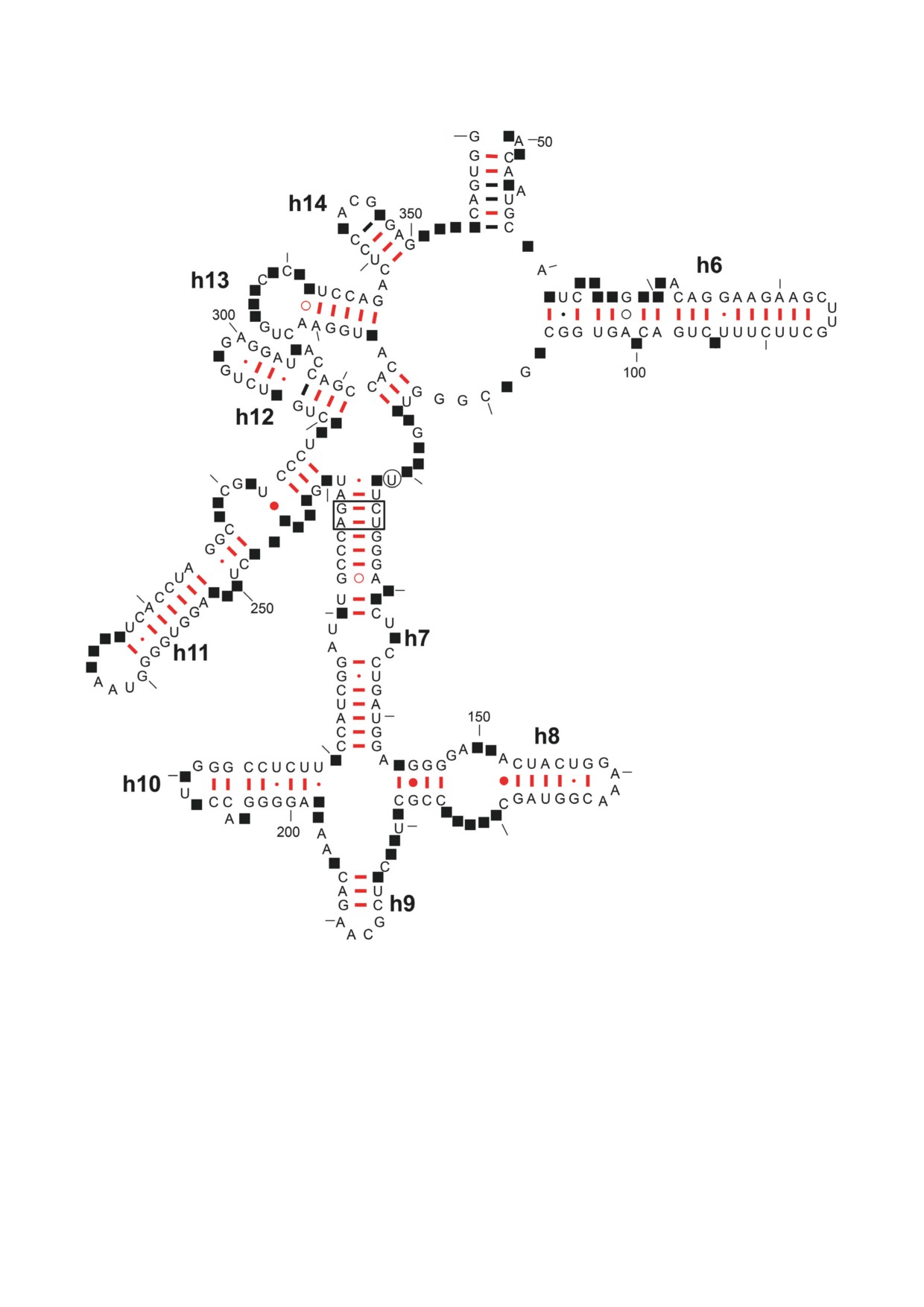
или более сложноорганизованной петли, а
рис. 1, на котором в классической модели вто-
также в участках противоположных полину-
ричной структуры сегмента (49-360) рРНК ма-
клеотидных цепей, связанных друг с другом, в
лой субъединицы рибосом E. coli отмечены н.о.,
частности, благодаря образованию неканони-
образующие NA-BSE в этом сегменте рРНК в
ческих (неуотсон-криковских) пар оснований.
составе малой субъединицы рибосомы. Они со-
(Следует заметить, что в традиционных схемах
ставляют примерно четверть (25,6%) от общего
вторичных структурах РНК они часто пред-
числа н.о. в этом сегменте РНК. Похожая кар-
ставлены в виде петель.)
тина наблюдается и для NA-BSE 23S рРНК этих
К третьему типу NA-BSE (NA-BSE LR, long-
рибосом, доля которых равна примерно 26%
range) относятся мотивы, образованные за счёт
от их общего числа н.о. в её макромолекуле.
стэкинг-взаимодействий нуклеотидов, распо-
Более 98% NA-BSE, входящих в основную
ложенных в разных петлях РНК, в разных це-
базу данных, дополнительно стабилизированы
пях одной и той же РНК (при этом расстояние
БИОХИМИЯ том 88 вып. 6 2023
976
МЕТЕЛЕВ и др.
motifs/2ds/escherichia_coli_16s). Чёрными квадратами отмечены н.о., образующие NA-BSE в этом сегменте рРНК в
составе малой субъединицы рибосомы
между взаимодействующими нуклеотидами в
Следует подчеркнуть, что BIE и BWE, по-
первичной структуре РНК может достигать не-
дробно описанные ранее [7], могут относить-
скольких сотен н.о. (рис. 3)) или в разных мо-
ся к любому из трёх типов NA-BSE. В основ-
лекулах РНК. Так, в частности, в связывании
ной базе данных, представленной в настоя-
5,8S рРНК с остальной частью рРНК большой
щей работе, они, как правило, входят в состав
субъединицы рибосом эукариот участвуют
NA-BSE, состоящих из четырёх н.о.
семь NA-BSE третьего типа (см., например,
Наиболее распространённым типом NA-BSE
файл 6ZMI в табл. S1 Приложения).
оказался тип II, на долю которого приходится
БИОХИМИЯ том 88 вып. 6 2023
НОВЫЕ МОТИВЫ В ТРЕТИЧНОЙ СТРУКТУРЕ РНК
977
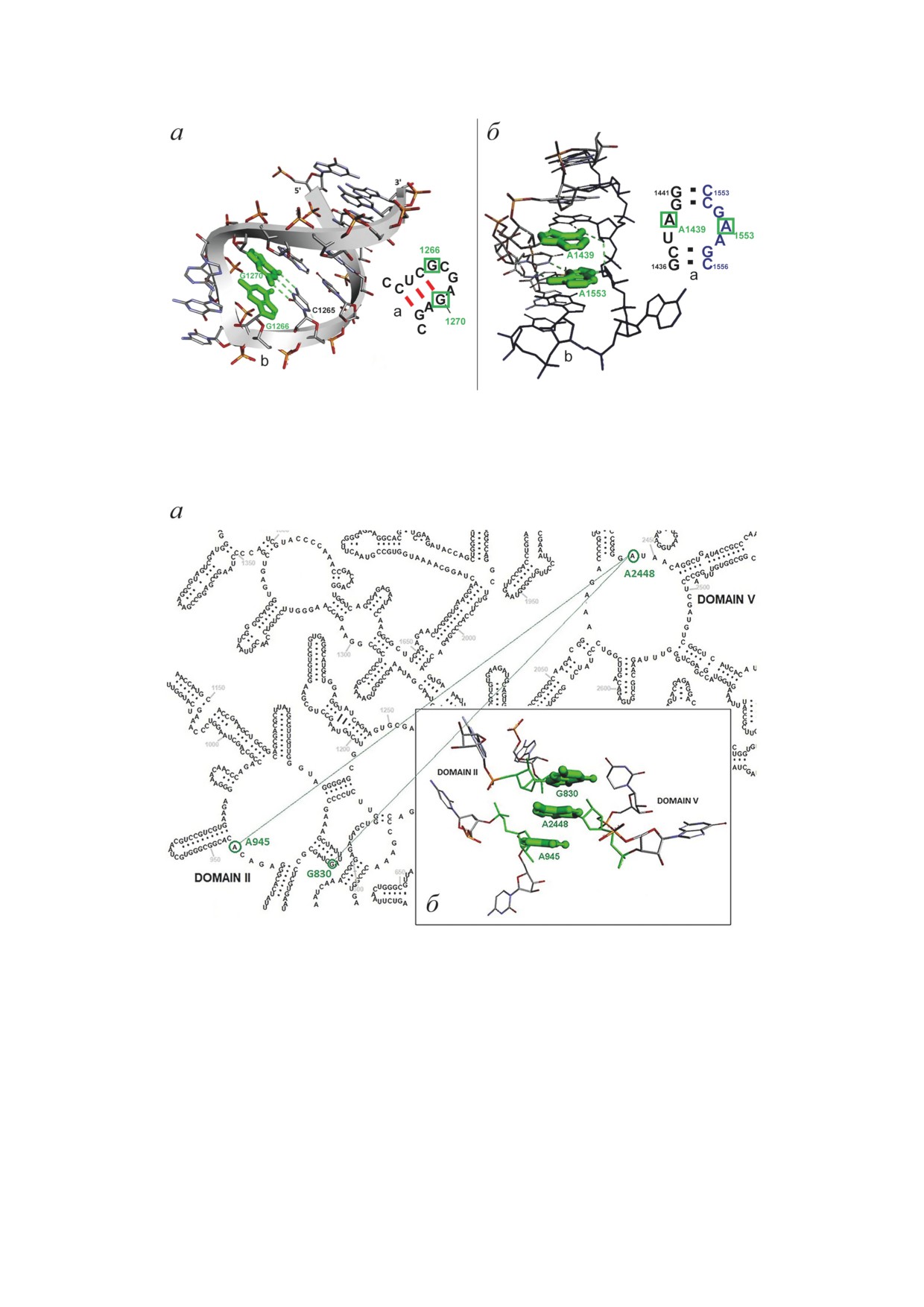
Рис. 2. Локализация н.о., образующих NA-BSE первого и второго типа. а - Локализация н.о., образующих NA-BSE
первого типа, во фрагменте 16S рРНК в составе рибосом E. coli. б - Локализация н.о., образующих NA-BSE второго
типа, во фрагменте 23S рРНК в составе рибосом Thermus thermоphilus. Панели (а) - локализация н.о. во вторичной
структуре, панели (b) - пространственная организация этих мотивов в третичной структуре рРНК (PDB ID 7K00
и 4Y4O соответственно)
Рис. 3. Локализация н.о., образующих NA-BSE третьего типа, в структуре 23S рРНК рибосом E. coli. а - Локализация
н.о. во вторичной структуре. б - Пространственная организация этого мотива во внутририбосомной третичной струк-
туре рРНК (PDB ID 7K00)
45,5% мотивов, при этом доли мотивов типа I
5′-концевого района которых они осуществля-
и типа LR в основной базе данных составили
ют, была детально изучена (для обзора см. ра-
31,8% и 22,7% соответственно.
боту Mondragón et al. [16]). Макромолекула
Структурообразующие функции NA-BSE.
РНКазы Р состоит из двух доменов: S-домена,
Разнообразие структурообразующих функций
специфически узнающего её субстрат, пред-
NA-BSE мы продемонстрируем здесь на при-
шественник тРНК, и С-домена, обладающего
мере рибонуклеазы Р (РНКазы Р). Структура
каталитической активностью. В настоящей ра-
рибозимов этого класса из различных источни-
боте была проанализирована атомная структу-
ков, а также их комплексов с тРНК, процессинг
ра S-домена РНКазы Р из T. thermоphilus, кото-
БИОХИМИЯ том 88 вып. 6 2023
978
МЕТЕЛЕВ и др.
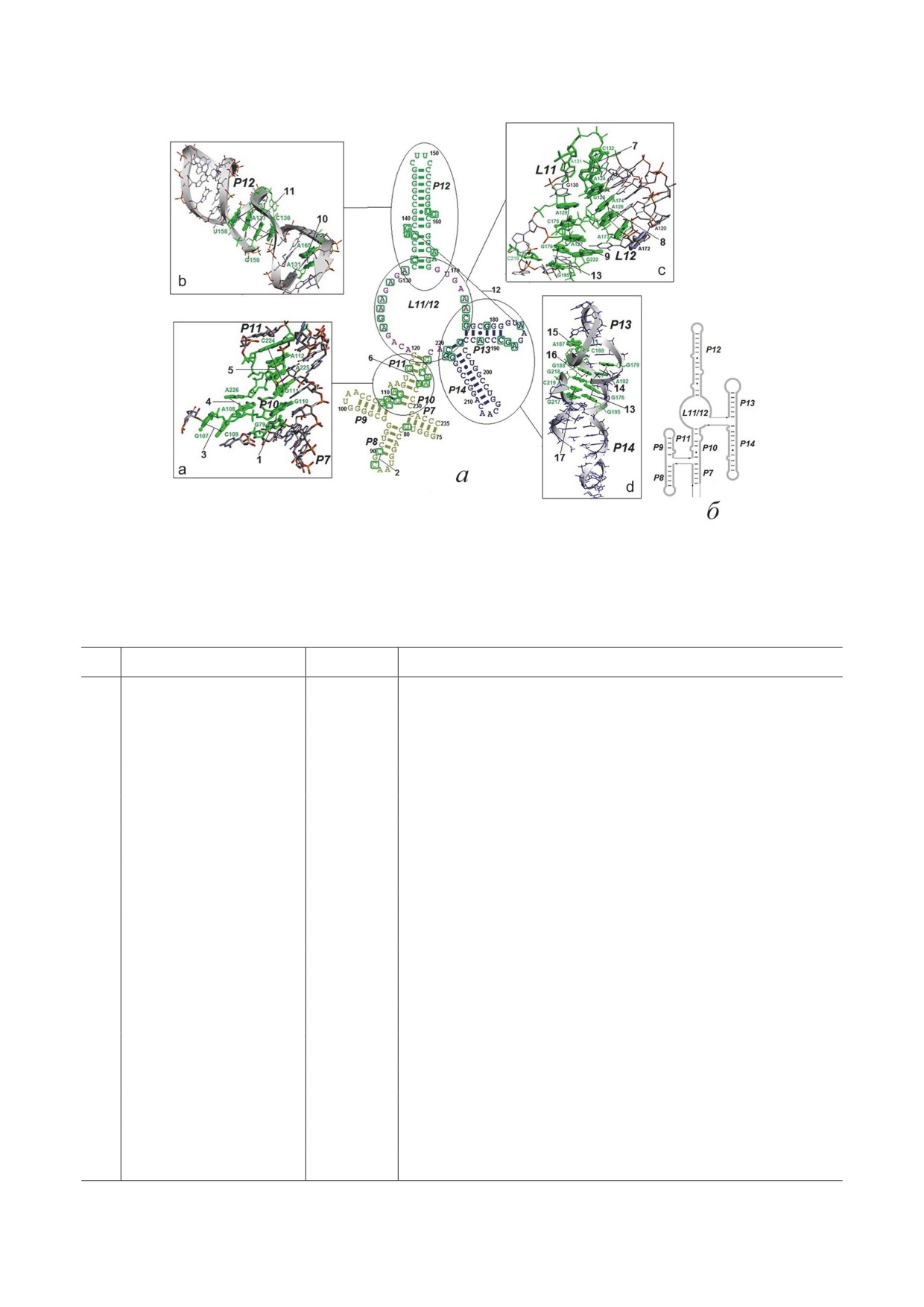
Рис. 4. Участие NA-BSE в организации третичной структуры РНКазы Р T. thermophilus. а - В центре - схема кано-
нической вторичной структуры S-домена рибозима [17]; нуклеотиды, образующие NA-BSE, помещены в квадраты;
во вставках справа и слева (a, b, c, d) - третичная структура фрагментов рибозима. б - Схема взаимного расположения
спиральных участков РНК S-домена РНКазы Р в его третичной структуре [19]
NA-BSE, участвующие в формировании третичной структуры S-домена РНКазы Р T. thermophilus [17, 18]
№
NA-BSE
Тип
Роль в формировании третичной структуры РНК
1
G79.G110
II
коаксиальный стэкинг спиралей Р7 и Р10
2
С88.С90
I
компактная укладка петли с экспонированием А89
3
A107.C109
I
внутриспиральный стэкинг с экспонированием А108
4
A108.A226
LR
межспиральный контакт спиралей Р9 и Р10
5
G111.A225.A112.C224
LR-LR-LR
формирование межпетлевого NA-BSE-ZIP мотива
6
G117.A227
II
коаксиальный стэкинг спиралей Р10 и Р11
7
А124.С132
I
компактная укладка петли L11
8
G125.A174.A126.A.173
II-II-II
формирование внутрипетлевого (L11/L12) NA-BSE-ZIP мотива
9
A129.C175.A127.G.222
II-II-II
формирование внутрипетлевого (L11/L12) NA-BSE-ZIP мотива
10
A131.A165
LR
формирование компактной структуры основания спирали Р12
11
С136.G159.A137.U158
LR-LR-LR
формирование NA-BSE-ZIP мотива в составе спирали Р12
12
G138.A184
LR
формирование контакта между спиралями Р12 и Р13
13
G176.G195
II
коаксиальный стэкинг спиралей Р13 и Р14
14
G179.A192
II
сшивание двух цепей в «дефектном» участке спирали Р13
15
А187.C189
I
компактная укладка петли в шпильке с экспонированием G188
16
G188.G218
LR
формирование межпетлевого контакта
17
G217.C219
I
внутриспиральный стэкинг с экспонированием G218
БИОХИМИЯ том 88 вып. 6 2023
НОВЫЕ МОТИВЫ В ТРЕТИЧНОЙ СТРУКТУРЕ РНК
979
рую Krasilnikov et al. [17, 18] удалось получить в
Другой интересный пример участия NA-
изолированном виде с максимально высоким
BSE в организации протяжённого спирального
(2,9 Å) разрешением для РНК этого класса
участка РНК - это образование блока из двух
(PDB ID 1U9S). Кроме того, S-домен в составе
пиримидиновых и двух пуриновых нуклеоти-
полноценной РНКазы Р, в отличие от С-доме-
дов в спирали Р12 (рис. 4, а, вставка «b», эле-
на, не связан ни с какими белками, модули-
мент 11; № 11 в таблице). Центральная ось
рующими структуру этого рибозима, и можно
этого блока фактически параллельна основной
считать, что его третичная структура в изоли-
оси спирали Р12. Важно, что в центре блока
рованном состоянии максимально приближена
находятся «петлевые» (в терминах вторичной
к внутриклеточной.
структуры) остатки А137 и G159, а на его кон-
На рис. 4, а приведена вторичная струк-
цах - остатки С136 и U158, дополнительно
тура S-домена РНКазы Р в канонической фор-
образующие в спирали Р12 уотсон-криков-
ме. Этот домен состоит из 161 н.о., ровно 100
скую CG- и неканоническую UG-пары соот-
из которых находятся в спиральных участках,
ветственно. При этом «петлевые» пуриновые
образуя уотсон-криковские пары. Из остав-
остатки занимают место в центре блока с мак-
шихся 61 н.о. 42 (т.е. примерно четверть от
симальным перекрыванием плоскостей всех
общего числа н.о. в этой РНК) входят в со-
четырёх оснований. Расположенный по сосед-
став NA-BSE. Их нуклеотидный состав, типы
ству G138 в формировании блока не участвует,
структуры и возможная роль в формировании
но, образуя NA-BSE с А184 (№ 12 в таблице),
третичной структуры РНК приведены в табли-
стабилизирует контакт между спиралями Р12
це. Рассмотрим эти данные подробнее.
и Р13. Сходным образом организованы блок,
Как видно из рис. 4, б, восемь двуспи-
формирующий протяжённую структуру спи-
ральных элементов канонической вторичной
ралей Р10-Р11 (№ 5 в таблице; рис. 4, а, встав-
структуры S-домена РНКазы Р в её третич-
ка «a»), а также два блока, ответственных за до-
ной структуре в действительности представ-
статочно компактную конформацию крупного
лены четырьмя достаточно протяжёнными
петлевого участка S-домена L11/L12 (№№ 8
спиралями. При этом в трёх случаях (спирали
и 9 в таблице; рис. 4, а, вставка «с»). В целом,
Р8-Р9, Р7-Р10 и Р13-Р14) это осуществляется с
структура каждого такого блока напоминает
помощью коаксиального стэкинга, одного из
застёжку «молния», и эти мотивы мы в даль-
самых детально изученных способов организа-
нейшем обозначаем здесь как NA-BSE-ZIP
ции макромолекулярной структуры РНК (для
(от англ. zipper).
обзора см. работу Butcher et al. [3]). В этом
Контакт между спиралями Р9 и Р10 ста-
процессе принимают участие три NA-BSE
билизируется также NA-BSE A108.A126 (№ 4 в
(№№ 1, 6 и 13 в таблице). Следует заметить,
таблице). При этом образование данного мо-
что участие этих мотивов в коаксиальном стэ-
тива становится возможным благодаря внутри-
кинге спиралей вполне ожидаемо, т.к. он, как
спиральному стэкингу оснований н.о. А107 и
правило, осуществляется с помощью уотсон-
С109, который происходит с экспонированием
криковских пар оснований, находящихся на
А108 (№ 3 в таблице; рис. 4, а, вставка «a»).
концах спиральных элементов (см., например,
Как уже отмечалось выше, такие стэкинг-взаи-
стэкинг-контакт нуклеотидов G176 и G195,
модействия важны не только для образования
входящих в состав концевых уотсон-криков-
третичной структуры РНК (как в данном слу-
ских пар в спиралях Р13 и Р14 соответственно;
чае), но и их контактов с белками.
рис. 4, а, вставка «d»).
Спираль Р12 не образует коаксиально-
Далее, роль мотива № 14 (G179.A192 в
го стэкинг-контакта ни с одной из спиралей
таблице) в стабилизации спирали Р13 также
S-домена. Однако её сочленение с петлёй L11
вполне очевидна: известно, что стэкинг пури-
организовано при помощи NA-BSE A131.A165,
новых оснований н.о., расположенных в раз-
который расположен в основании этой спи-
ных цепях несовершенного участка спирали,
рали (№ 10 в таблице; рис. 4, а, вставка «b»).
повышает устойчивость неуотсон-криковских
При этом он оказывается сближенным (но
пар оснований [20], в данном случае, пары
прямо не контактирует) с NA-BSE A124.C132,
А192-С178. Его следует назвать межцепочеч-
который делает более компактным один из
ным поперечным стэкингом (cross-strand stack-
участков большой «петли» L11 (№ 7 в таблице;
ing). Образующийся здесь NA-BSE и подоб-
рис. 4, а, вставка «c»).
ные ему мотивы в подавляющем большинстве
NA-BSE (№№ 15-17 в таблице) образуют
случаев входит в состав Е-мотивов, первый
целый ансамбль, выполняющий разнообраз-
из которых был обнаружен в т.н. Е-«петле» 5S
ные функции в организации третичной струк-
рРНК [8].
туры S-домена РНКазы Р. Действительно, об-
БИОХИМИЯ том 88 вып. 6 2023
980
МЕТЕЛЕВ и др.
разование NA-BSE первого типа (А187.С189,
№ 15 в таблице) в петле классической шпильки
Р13 сопровождается экспонированием G188,
который вступает в стэкинг-взаимодействие с
G218 в основании шпильки Р14 и создаёт кон-
такт между этими двумя спиральными участка-
ми, отчётливо видный на рис. 4, а (вставка «d»).
В то же время у G218 появилась возможность
образовать этот контакт благодаря стэкингу
оснований в н.о. G217 и С219 (NA-BSE перво-
го типа, № 17 в таблице).
Таким образом, анализируя третичную
структуру сравнительно небольшой РНК, мож-
но проследить разнообразие участия всех типов
NA-BSE в формировании её макромолекулы.
Они способствуют компактному сворачиванию
однотяжевых петель РНК (а иногда и определя-
Рис. 5. Пример наиболее распространённого варианта
ют его), участвуют в превращении двутяжевых
NA-BSE-DС. а - Мотив-образующие GA-последователь-
ности во вторичной структуре 23S рРНК E. coli (отмече-
петель в несовершенные спирали, играют важ-
ны кружками). б - Пространственная структура мотива
ную роль в связывании удалённых в первичной
[PDB ID 7K00]. в - Положение мотива в третичной струк-
и вторичной структуре районов РНК.
туре фрагмента 23S рРНК в составе рибосомы
Мотивы типа NA-BSE-ZIP, столь широко
представленные в S-домене РНКазы Р, встре-
чаются также в тРНК, в рРНК в составе рибо-
имеем дело с двойным поперечным межцепо-
сом и в некоторых других РНК (см. табл. S1
чечным стэкингом оснований. Мы обозначаем
Приложения). Более того, при анализе струк-
далее эти мотивы как NA-BSE-DC (double-
тур этих РНК видно, что они входят в доволь-
crossing). Впервые NA-ВSE-DC был замечен в
но обширное подсемейство NA-BSE с харак-
кристаллической структуре другого рибозима,
терным внедрением соседних в первичной
а именно в Р4-Р6 домене интрона группы I [9].
структуре РНК н.о. в противоположные по на-
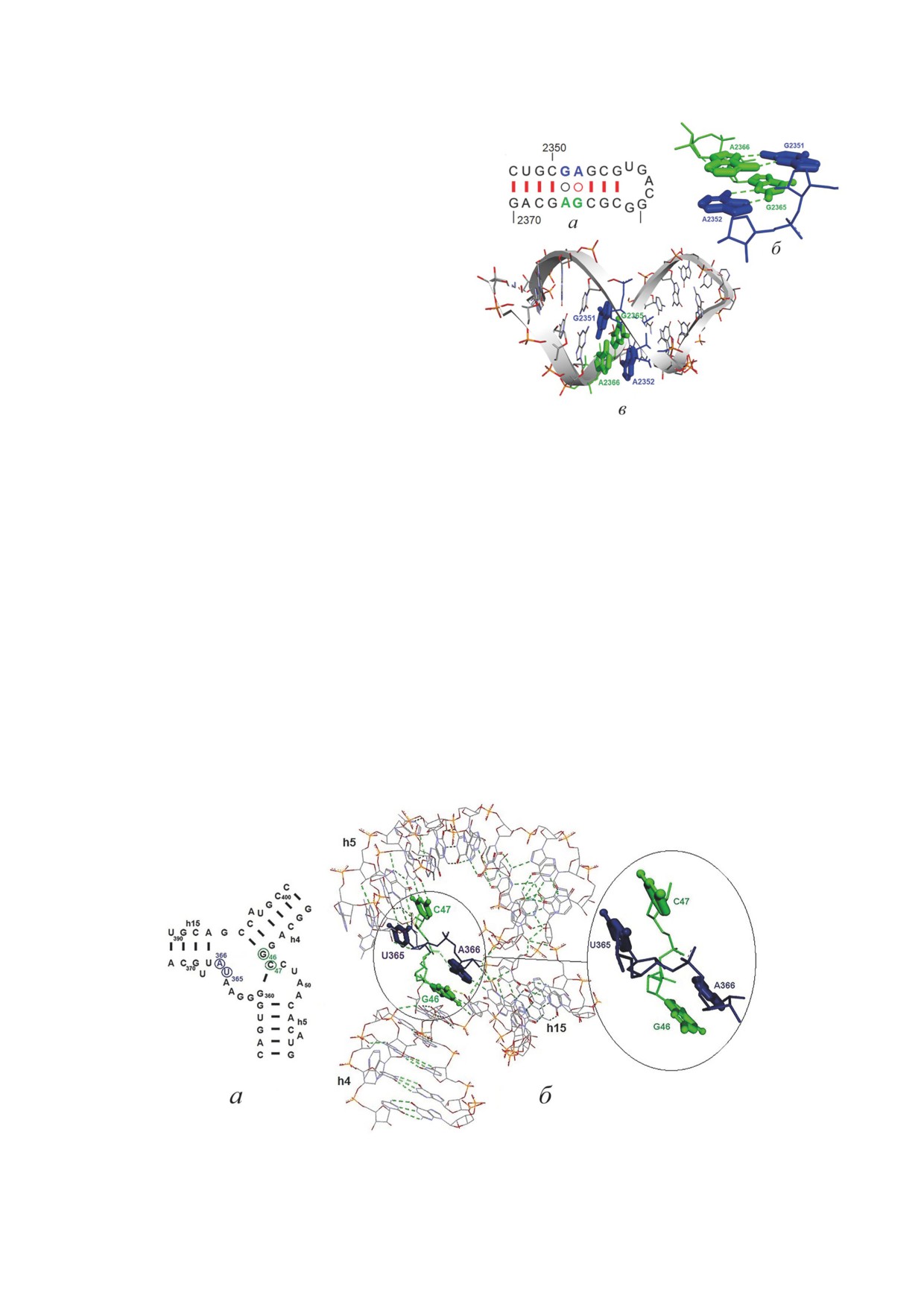
На рис. 5 приведён типичный пример NA-
правлению фосфодиэфирных связей участки
BSE-DC. В спирали Н86 23S рРНК большой
её полинуклеотидной цепи. При этом основа-
субъединицы рибосом E. coli он образован
ния каждого из этих остатков находятся в стэ-
двумя динуклеотидными GA-сегментами этой
кинге c н.о., которые также являются соседями
рРНК, расположенными друг над другом в её
в первичной структуре РНК. То есть здесь мы
канонической вторичной структуре (рис. 5, а).
Рис. 6. Пример NA-BSE-DC c максимальным расстоянием между соседними основаниями в первичной структуре РНК.
Показано расположение мотив-образующих н.о. во вторичной структуре (а) и в третичной структуре (б) 16S рРНК
в составе рибосомы E. coli (PDB ID 7K00)
БИОХИМИЯ том 88 вып. 6 2023
НОВЫЕ МОТИВЫ В ТРЕТИЧНОЙ СТРУКТУРЕ РНК
981
Поперечный стэкинг одноимённых пури-
оставшихся 58 нуклеотидах этой РНК неблаго-
новых оснований позволяет им образовать
приятны, остаётся не ясным.
полноценные хугстиновские GA-пары (trans-
Сопоставляя результаты работы Noller
Hoogsteen/SugarEdge пары оснований по но-
et al. с данными, полученными в настоящей
менклатуре Леонтиса-Вестхофа
[21]). При
работе, можно высказать следующие сооб-
этом общая форма спирали не искажается, и
ражения. Из 140 абсолютно консервативных
она лишь несколько изгибается в районе рас-
остатков, выявленных этими авторами, 41 уча-
положения NA-BSE-DC (рис. 5, б и в).
ствует в образовании NA-BSE. При этом
Другой пример NA-BSE-DC, иллюстри-
23 таким н.о. можно приписать определённую
рующий участие мотивов этого подсемейства
функциональную роль. Поиск ответа на во-
в формировании третичной структуры РНК
прос, почему основания в оставшихся 18 н.о.
(рис. 6), заметно отличается от предыдущего.
не могут быть заменены на другие, с нашей
Здесь непосредственные соседи в первичной
точки зрения, принципиально важен. Пред-
структуре РНК располагаются в достаточно
варительный анализ показывает, что NA-BSE,
удалённых друг от друга спиральных участках,
построенные из этих н.о., находятся внутри
и их фосфодиэфирные связи служат для не-
«головки» и «тела» малой субъединицы рибо-
обычного соединения этих спиралей. При этом
сомы, т.е. располагаются вне её функциональ-
основания н.о., образующих NA-BSE-DC, спа-
ных центров. С другой стороны, в литературе
рены не друг с другом (как в предыдущем слу-
накоплено большое число фактов, которые
чае), а с функциональными группами н.о. спи-
говорят о том, что и в рибосомах, и в других
ралей, в которые они включены.
РНК-содержащих макромолекулярных ком-
плексах происходит передача функциональ-
ных (конформационных) сигналов на большие
ЗАКЛЮЧЕНИЕ
расстояния [23-25]. Вполне логично предпо-
ложить, что NA-BSE в рРНК, а также во мно-
Итак, NA-BSE способствуют компактно-
гих других РНК участвуют в построении си-
му сворачиванию однотяжевых петель РНК
стемы передачи таких сигналов.
(а иногда и определяют его), участвуют в пре-
вращении двутяжевых петель в несовершен-
Вклад авторов. Е.Б. выполнил вычислитель-
ные спирали, играют важную роль в связыва-
ную работу и конформационный анализ. В.М.
нии удалённых в первичной или вторичной
проанализировал данные и подготовил все ри-
структуре районов РНК (например, отдельных
сунки. А.Б. разработал проект. Все авторы внес-
доменов рРНК или её сегментов, если рРНК
ли значительный вклад в написание рукописи.
транскрибируется в виде фрагментов).
Финансирование. В.М. и А.Б. благодарят за
В заключение отметим ещё одну потен-
поддержку Московский государственный уни-
циально важную черту рассмотренных здесь
верситет имени М.В. Ломоносова и Минобрна-
РНК-мотивов. Недавно Noller et al. [22], про-
уки (Соглашение 1075-15-2021-1949 от 28.09.21).
анализировав 2000 нуклеотидных последова-
Благодарности. Авторы благодарят рецен-
тельностей рРНК малой субъединицы рибо-
зентов за внимательное прочтение рукописи и
сом прокариот и цитоплазматических рибосом
конструктивные предложения.
эукариот, установили, что примерно деся-
Конфликт интересов. Авторы заявляют об
тая часть н.о. этих РНК (140 н.о. в 16S рРНК
отсутствии конфликта интересов.
E. coli) являются абсолютно консервативными.
Соблюдение этических норм. Настоящая
Анализируя все известные на сегодня экспери-
статья не содержит описания каких-либо ис-
ментальные данные, авторы смогли приписать
следований с участием людей или животных в
с некоторой долей вероятности прямую или
качестве объектов.
косвенную роль в функционировании рибо-
Дополнительные материалы. Приложение к
сомы 82 консервативным н.о. 16S рРНК. По-
статье опубликовано на сайте журнала «Био-
чему мутации гетероциклических оснований в
СПИСОК ЛИТЕРАТУРЫ
1. Fresco, J. R., Alberts, B. M., and Doty, P. (1960) Some
2. Spirin, A. S. (1960) On macromolecular structure of na-
molecular details of the secondary structure of ribonu-
tive high-polymer ribonucleic acid in solution, J. Mol.
cleic acids, Nature, 188, 98-101, doi: 10.1038/188098a0.
Biol., 2, 436-446, doi: 10.1016/S0022-2836(60)80054-X.
БИОХИМИЯ том 88 вып. 6 2023
982
МЕТЕЛЕВ и др.
3.
Butcher, S. E., and Pyle, A. M. (2011) The molecular
structure improves the classification and prediction
interactions that stabilize RNA tertiary structure: RNA
of A-minors, RNA,
27,
907-919, doi:
10.1261/
motifs, patterns, and networks, Acc. Chem. Res., 44,
rna.078535.120.
1302-1311, doi: 10.1021/ar200098t.
14.
Yogesh, K., Gupta, Y. K., Nair, D. T., Wharton, R. P.,
4.
Nissen, P., Ippolito, J. A., Ban, N., Moore, P. B.,
Aggarwal, A. K. (2008) Structures of human Pumilio
and Steitz, T. A. (2001) RNA tertiary interactions in
with noncognate RNAs reveal molecular mechanisms
the large ribosomal subunit: the A-minor motif, Proc.
for binding promiscuity, Structure,
16,
549-557,
Natl. Acad. Sci. USA, 98, 4899-4903, doi: 10.1073/
doi: 10.1016/j.str.2008.01.006.
pnas.081082398.
15.
Guogas, L. M., Filman, D. J., Hogle, J. M., and
5.
Klein, D. J., Schmeing, T. M., Moore, P. B., and
Gehrke, L. (2004) Cofolding organizes alfalfa mosaic
Steitz, T. A. (2001) The kink-turn, EMBO J., 20, 4214-
virus RNA and coat protein for replication, Science,
4221, doi: 10.1093/emboj/20.15.4214.
306, 2108-2111, doi: 10.1126/science.1103399.
6.
Chawla, M., Chermak, E., Zhang, O., Bujnicki,
16.
Mondragón, A. (2013) Structural studies of RNase P,
J. M., Oliva, R., and Cavallo, L. (2017) Occurrence
Annu. Rev. Biophys.,
42,
537-557, doi:
10.1146/
and stability of lone pair-stacking interactions be-
annurev-biophys-083012-130406.
tween ribose and nucleobases in functional RNAs,
17.
Krasilnikov, A. S., Xiao, Y., Pan, T., and
Nucleic Acids Res., 45, 11019-11032, doi: 10.1093/
Mondragón, A. (2004) Basis for structural diver-
nar/gkx757.
sity in homologous RNAs, Science, 306, 104-107,
7.
Baulin, E., Metelev, V., and Bogdanov, A. (2020)
doi: 10.1126/science.1101489.
Base-intercalated and base-wedged stacking elements
18.
Krasilnikov, A. S., Yang, X., Pan, T., and
in 3D-structure of RNA and RNA-protein complex-
Mondragón, A. (2003) Crystal structure of the speci-
es, Nucleic Acids Res., 48, 8675-8685, doi: 10.1093/
ficity domain of ribonuclease P, Nature, 421, 760-764,
nar/gkaa610.
doi: 10.1038/nature01386.
8.
Dallas, A., and Moore, P. B. (1997) The loop E-
19.
Reiter, N. J., Osterman, A., Torres-Larios, A.,
loop D region of Escherichia coli 5S rRNA: the
Swinger, K. K., Pan, T., and Mondragón, A. (2010)
solution structure reveals an unusual loop that may be
Structure of a bacterial ribonuclease P holoenzyme
important for binding ribosomal proteins, Structure, 5,
in complex with tRNA, Nature,
468,
784-789,
1639-1653, doi: 10.1016/s0969-2126(97)00311-0.
doi: 10.1038/nature09516.
9.
Cate, J. H., Gooding, A. R., Podell, E., Zhou, K.,
20.
Mignon, P., Loverix, S., Steyaert, J., and Geerlings, P.
Golden, B. L., Kundrot, C. E., Cech, T. R., and
(2005) Influence of the π-π interaction on the
Doudna, J. A. (1996) Crystal structure of a group I
hydrogen bonding capacity of stacked DNA/RNA
ribozyme domain: principles of RNA packing, Science,
bases, Nucleic Acids Res., 33, 1779-1789, doi: 10.1093/
273, 1678-1685, doi: 10.1126/science.273.5282.1678.
nar/gki317.
10.
Teplova, M., Malinina, L., Darnell, J. C., Song, J.,
21.
Leontis, N. B., and Westhof, E. (2001) Geometric
Lu, M., Abagyan, R., Musunuru, K., Teplov, A.,
nomenclature and classification of RNA base pairs,
Burley, S. K., Darnell, R. B., and Patel, D. J. (2011)
RNA, 7, 499-512, doi: 10.1017/s1355838201002515.
Protein-RNA and protein-protein recognition by
22.
Noller, H. F., Donohue, J. P., and Gutell, R. R.
DualKH1/2 domains of the neuronal splicing fac-
(2022) The universally conserved nucleotides of the
tor Nova-1, Structure, 19, 930-944, doi: 10.1016/j.str.
small subunit ribosomal RNAs, RNA, 28, 623-644,
2011.05.002.
doi: 10.1261/rna.079019.121.
11.
Leontis, N. B., and Zirbel, C. L. (2012) Nonredun-
23.
Sergiev, P. V., Kiparisov, S. V., Burakovsky, D. E.,
dant 3D structure datasets for RNA knowledge ex-
Lesnyak, D. V., Leonov, A. A., Bogdanov, A. A., and
traction and benchmarking, RNA 3D Structure Analy-
Dontsova, O. A. (2005) The conserved A-site finger
sis and Prediction (Leontis, N., and Westhof, E., eds),
of the 23S rRNA: just one of the intersubunit bridg-
Nucleic Acids and Molecular Biology, 27, 282-298,
es or a part of the allosteric communication path-
Springer-Verlag Berlin Heidelberg, doi:
10.1007/
way? J. Mol. Biol., 353, 116-123, doi: 10.1016/j.jmb.
978-3-642-25740-7_13.
2005.08.006.
12.
Lu, X.-J., Bussemaker, H. J., and Olson, W. K. (2015)
24.
Walkera, A. S., Russ, W. P., Ranganathanc, R., and
DSSR: an integrated software tool for dissecting the
Schepartza, A. (2020) RNA sectors and allosteric
spatial structure of RNA, Nucleic Acids Res., 43, e142,
function within the ribosome, Proc. Natl. Acad. Sci.
doi: 10.1093/nar/gkv716.
USA, 117, 19879-19887, doi: 10.1073/pnas.1909634117.
13.
Shalybkova, A. A., Mikhailova, D. S., Kulakovskiy,
25.
Peselis, A., and Serganov, A. (2021) Cooperativity and
I. V., Fakhranurova, L. I., and Baulin, E. F. (2021)
allostery in RNA systems, Methods Mol. Biol., 2253,
Annotation of the local context of RNA secondary
255-271, doi: 10.1007/978-1-0716-1154-8_15.
БИОХИМИЯ том 88 вып. 6 2023
НОВЫЕ МОТИВЫ В ТРЕТИЧНОЙ СТРУКТУРЕ РНК
983
MULTIPLE NON-CANONICAL BASE-STACKING INTERACTIONS
AS ONE OF THE MAJOR DETERMINANTS
OF RNA TERTIARY STRUCTURE ORGANIZATION
V. G. Metelev1, E. F. Baulin2, and A. A. Bogdanov1,3,4*
1 Faculty of Chemistry, Lomonosov Moscow State University, 119991 Moscow, Russia
2 Institute of Mathematical Problems of Biology of the Russian Academy of Sciences -
the Branch of Keldysh Institute of Applied Mathematics of Russian Academy of Sciences,
142290 Pushchino, Moscow Region, Russia
3 A. N. Belozersky Institute of Physico-Chemical Biology, Lomonosov Moscow State University,
119992 Moscow, Russia; e-mail: bogdanov@belozersky.msu.ru
4 Shemyakin-Ovchinnikov Institute of Bioorganic Chemistry, 117997 Moscow, Russia
Interplane (stacking) interactions of heterocyclic bases of nucleotide residues (n.t.) of RNA are one of the
most important factors in the organization of its secondary and tertiary structure. Most of these (canoni-
cal) interactions are carried out between neighbors in the polynucleotide chains of RNA. However, with
the accumulation of data on the atomic tertiary structures of a wide variety of RNAs and their complexes
with proteins, it became clear that RNA nucleotide residues that are not neighbors in their polynucleotide
chains and are sometimes separated in the RNA primary structure by tens or hundreds of n.t. can interact
with the help of base stacking (non-canonical). This paper presents an exhaustive database of such elements
and their environment in the macromolecules of natural and synthetic RNAs. They were called nonadjacent
base-stacking elements (NA-BSE). The analysis of these data showed that the NA-BSE forming nucleo-
tides, on average, account for about a quarter of all nucleotides of a particular RNA, therefore, they should
be considered as real motifs in their tertiary structure. The classification of NA-BSE by types of localization
in RNA macromolecules is carried out. It is shown that the structure-forming role of NA-BSE consists
in the compact folding of single-stranded RNA loops, in the transformation of double-stranded bulges
into imperfect helices, as well as in the binding of RNA regions removed in their primary and secondary
structure.
Keywords: stacking interactions, tertiary structure of RNA, ribosomal RNA, ribonuclease P, RNA motifs
БИОХИМИЯ том 88 вып. 6 2023