БИОХИМИЯ, 2023, том 88, вып. 9, с. 1620 - 1630
УДК 579.6;579.25
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
С ИСПОЛЬЗОВАНИЕМ СИСТЕМЫ CRISPR/Cas9 ДЛЯ ПОЛУЧЕНИЯ
БЕЗМАРКЕРНОГО ШТАММА-ПРОДУЦЕНТА ФИТАЗЫ
© 2023 А.А. Ткаченко*, Л.Н. Борщевская, С.П. Синеокий, Т.Л. Гордеева
НИЦ «Курчатовский институт»,
117545 Москва, Россия; электронная почта: artur.tka4enko10@gmail.com
Поступила в редакцию 19.04.2023
После доработки 19.04.2023
Принята к публикации 19.05.2023
На основе штамма Komagataella phaffii ВКПМ Y-4287 с высоким экспрессионным потенциа-
лом с использованием системы CRISPR/Cas9 были разработаны штаммы-реципиенты K. phaffii
ВКПМ Y-5013 (фенотип His-) и K. phaffii ВКПМ Y-5014 (фенотип Leu-), позволяющие на их ос-
нове получать безмаркерные штаммы-продуценты гетерологичных белков. Эффективность инак-
тивации генов с использованием различных вариантов гидовых РНК (sgРНК) составила 65-98%
для гена HIS4 и 15-72% - для гена LEU2. Было показано, что полученные штаммы-реципиенты
сохранили ростовые характеристики, присущие родительскому штамму K. Phaffii ВКПМ Y-4287,
и высокий экспрессионный потенциал, оцененный по уровню продукции гетерологичного фер-
мента фитазы из Citrobacter gillenii. Средняя продуктивность трансформантов, полученных на ос-
нове штаммов K. phaffii ВКПМ Y-5013 и K. phaffii ВКПМ Y-5014, была в 2,1 и 2,0 раза выше про-
дуктивности трансформантов, полученных на основе коммерческого штамма K. phaffii GS115
соответственно. Предложен способ последовательной интеграции экспрессионной кассеты в геном
штамма-реципиента K. phaffii ВКПМ Y-5013, и получен высокоэффективный многокопийный без-
маркерный штамм-продуцент фитазы C. gillenii.
КЛЮЧЕВЫЕ СЛОВА: CRISPR/Cas9, Komagataella phaffii, геномное редактирование, фитаза, Citrobacter
gillenii.
DOI: 10.31857/S0320972523090130, EDN: WUVMUH
ВВЕДЕНИЕ
рекомбинантных белков, способности дости-
гать высоких плотностей клеток при культиви-
Фитазы (мио-инозитол-1,2,3,4,5,6-гекса-
ровании в минимальных средах [1]. В K. phaffii
кисфосфат фосфогидролазы, EC
3.1.3.8 и
были экспрессированы фитазы таких микро-
3.1.3.26) - ценные промышленные ферменты,
организмов, как Escherichia coli, Yersinia inter-
широко применяющиеся в качестве кормовых
media, Peniophora lycii, Shigella sp. CD2, Citro-
добавок к комбикормам сельскохозяйствен-
bacter freundii и др. [2-6]. Однако применение
ных животных и рыб.
в России зарубежных штаммов-реципиентов
Создание современных ферментных пре-
K. phaffii в коммерческих целях ограничено
паратов, содержащих фитазы, основано на ис-
политикой распространения материала [7].
пользовании промышленных рекомбинантных
Для получения штаммов-продуцентов ге-
штаммов-продуцентов. Наиболее привлека-
терологичных белков на основе прототрофных
тельным объектом для создания штаммов-про-
штаммов необходимо использование экспрес-
дуцентов рекомбинантных белков являются
сионных кассет, содержащих гены устойчиво-
метилотрофные дрожжи Komagataella phaffii
сти к антибиотикам в качестве селективных
благодаря их биобезопасности, легкости мо-
маркеров. Отбор целевых антибиотикорези-
лекулярно-генетических манипуляций, мощ-
стентных штаммов требует добавления этих
ным системам экспрессии генов и секреции токсичных соединений в питательные среды,
Принятые сокращения: КЖ - культуральная жидкость; sgРНК - single guide РНК (единая направляющая, или гидо-
вая РНК); PAM - protospacer adjacent motif (мотив, расположенный рядом с протоспейсером).
* Адресат для корреспонденции.
1620
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
1621
что может негативно влиять на их клеточную
нуклеаз, такими как ZNF (Zink Finger Nucle-
функцию [8]. Кроме того, в промышленных
ases) и TALEN (Transcription Activator-Like Ef-
биотехнологических производствах суще-
fector Nucleases), система CRISPR/Cas9 име-
ствуют ограничения на использование генов
ет ряд преимуществ, а также является более
устойчивости к антибиотикам в штаммах-про-
эффективной, простой и удобной платфор-
дуцентах с целью предотвращения их возмож-
мой для прицельного геномного редактирова-
ного нежелательного распространения среди
ния [15, 16].
микроорганизмов окружающей среды.
Ранее нами был описан прототрофный
Таким образом, получение и использова-
штамм K. phaffii ВКПМ Y-4287 с высоким экс-
ние в биотехнологическом производстве без-
прессионным потенциалом [17] и фитаза Citro-
маркерных штаммов-продуцентов гетероло-
bacter gillenii, обладающая промышленно-цен-
гичных белков является актуальным и практи-
ными характеристиками [18].
чески значимым.
Целью настоящей работы явилась раз-
Одним из подходов к получению много-
работка штаммов-реципиентов на основе
копийных безмаркерных дрожжевых штаммов
K. phaffii ВКПМ Y-4287 с неизмененными ро-
является использование систем сайт-специ-
стовыми характеристиками и экспрессионным
фической рекомбинации [9]. Существуют спо-
потенциалом с использованием высокоспеци-
собы удаления селективных маркеров с ис-
фичной системы CRISPR/Cas9, а также воз-
пользованием различных систем, например,
можность осуществления последовательного
Cre/loxP, Flp/FRT и др. Однако удаление
введения генетического материала в хромо-
фланкированных специфическими сайтами
сому реципиента для получения многокопий-
генетических элементов (селективных марке-
ного штамма-продуцента фитазы C. gillenii.
ров) с помощью таких систем в значительной
мере является трудоемким и времязатратным
процессом.
МАТЕРИАЛЫ И МЕТОДЫ
Еще одним подходом является получение
штаммов-продуцентов на основе ауксотроф-
Реактивы. Триптон, пептон, дрожжевой экс-
ных штаммов с использованием экспрес-
тракт, глюкоза, L-лейцин, L-гистидин были
сионных кассет, содержащих селективные
получены от компании
«Диа-М» (Россия);
маркеры, способные восстанавливать прото-
все ферменты для молекулярных работ - от
трофность. Однако при таком подходе исклю-
фирмы
«Fermentas» (Литва); фитат натрия
чается возможность повторной многократной
(«Sigma-Aldrich», США); соли и другие реаген-
интеграции. Ауксотрофные штаммы возмож-
ты (все реактивы отечественного производства
но получить либо с помощью неспецифиче-
марки «х.ч.» или «ч.д.а.») - от фирмы «Хим-
ского мутагенеза, как в случае коммерческого
мед» (Россия).
штамма-реципиента K. phaffii GS115 [10], либо
Штаммы и среды. Для стандартных генно-
путем делеции целевого гена с использовани-
инженерных работ (конструирование плазмид,
ем механизма гомологичной рекомбинации.
наработка плазмидной ДНК) использовали
Применение неспецифического мутагенеза
штамм E. coli XL-1 Blue (recA1 endA1 gyrA96
часто негативно сказывается на физиологи-
thi-1 hsdR17 supE44 relA1 lac
[F′ proAB
ческих характеристиках штамма-реципиента,
lacIqZΔM15 Tn10 (Tetr)]) ВКПМ В-5667. Куль-
а процесс скрининга целевых мутантов явля-
туру растили при 37 °С в среде LB, г/литр: трип-
ется весьма трудоемким [11]. Использование
тон - 10; дрожжевой экстракт - 5; NaCl - 10;
же механизма гомологичной рекомбинации
если необходимо, добавляли ампициллин или
для получения ауксотрофных штаммов ослож-
канамицин («Sigma-Aldrich») в концентрации
няется низкой вероятностью данного события
100 мкг/мл или 50 мкг/мл соответственно.
в дрожжах K. phaffi [12, 13].
Штаммы K. phaffii GS115 (his4-) ВКПМ
В настоящее время для редактирова-
Y-2837 и K. phaffii ВКПМ Y-4287 были полу-
ния генома K. phaffii и других организмов
чены из Биоресурсного Центра Всероссийской
успешно применяется система CRISPR/Cas9,
Коллекции Промышленных Микроорганиз-
основанная на РНК-направляемых ДНК-
мов (БРЦ ВКПМ). Культуру дрожжей K. phaffii
нуклеазах [14]. Несмотря на ее относительно
растили при 30 °С на среде YPD, г/литр: пеп-
недавнее появление, эта система уже зареко-
тон - 10; дрожжевой экстракт - 10; глюко-
мендовала себя как точный и высокоэффек-
за - 20.
тивный инструмент геномной инженерии.
Селекцию трансформантов с инактивиро-
По сравнению с таргетными системами ге-
ванным геном HIS4 или LEU2 проводили по
номного редактирования на основе химерных
способности к росту на минимальной среде
БИОХИМИЯ том 88 вып. 9 2023
1622
ТКАЧЕНКО и др.
без содержания аминокислот YNB («Himedia»,
Для исследования были выбраны по
Индия) с добавлением и без добавления в
3 спейсерные последовательности для нацели-
среду гистидина или лейцина в концентрации
вания sgРНК на гены HIS4 и LEU2 (табл. 1).
50 мкг/мл. В качестве источника углерода в
Фрагменты ДНК, кодирующие sgРНК,
среду добавляли глюкозу в количестве 2 мас. %.
были синтезированы в компании «Евроген»
Отбор трансформантов, несущих в составе
(Россия). На 5′- и 3′-концах нуклеотидных по-
хромосомы ген фитазы из бактерий C. gillenii,
следовательностей были предусмотрены сайты
осуществляли с использованием среды YNB.
рестрикции BpiI.
Селекцию трансформантов, содержащих
Конструирование плазмид с sgРНК, и полу-
автономно реплицирующиеся плазмиды с раз-
чение ауксотрофных штаммов K. phaffii ВКПМ
личными вариантами гидовых РНК (sgРНК),
Y-5013 и K. phaffii ВКПМ Y-5014. Автономно
проводили на агаризованной среде YPD с до-
реплицирующиеся экспрессионные плазмиды
бавлением селективного агента генетици-
с sgРНК для инактивации генов HIS4 и LEU2
на (G418) («Thermo Scientific», США) в коли-
конструировали на основе экспрессионного
честве 600 мкг/мл.
вектора BB3cK_pGAP_23*_pTEF_Cas9 («Add-
Все плотные среды содержали агар в кон-
gene», США) [21]. Фрагменты ДНК, кодирую-
центрации 20 г/литр.
щие sgРНК, клонировали в вектор BB3cK_
Конструирование sgРНК. Дизайн sgРНК,
pGAP_23*_pTEF_Cas9 по сайтам BpiI.
состоящих из последовательностей, кодирую-
В результате были получены следующие
щих рибозим Hammerhead (HH), транс-акти-
плазмиды:
вирующей РНК (tracrРНК), рибозим Hepatitis
pGAP-sgRNA1_his4-pTEF-Cas9-Km,
Delta Virus (HDV) и вариабельных спейсер-
pGAP-sgRNA2_his4-pTEF-Cas9-Km,
ных последовательностей, осуществляли как
pGAP-sgRNA3_his4-pTEF-Cas9-Km - для
описано в работе Gao и Zhao [19]. Последо-
инактивации гена HIS4 и
вательности НН, tracrРНК и HDV приведены
pGAP-sgRNA1_leu2-pTEF-Cas9-Km,
в работе Gao и Zhao [19]. Подбор спейсерных
pGAP-sgRNA2_leu2-pTEF-Cas9-Km,
последовательностей и мотивов, расположен-
pGAP-sgRNA3_leu2-pTEF-Cas9-Km - для
ных рядом с протоспейсерами (PAM), осущес-
инактивации гена LEU2. Корректность плаз-
твляли с использованием онлайн-инструмента
мид была подтверждена с помощью рестрик-
E-CRISPR [20]. Чтобы исключить внесение
ционного анализа и секвенированием (данные
возможных нецелевых мутаций («off-target-эф-
не приведены).
фектов»), проводили гомологичное выравни-
Плазмиды трансформировали в штамм
вание подобранных нуклеотидных последова-
K. phaffii ВКПМ Y-4287 методом электропора-
тельностей спейсеров с геномом Pichia pastoris
ции, как описано ранее [22]. Селекцию транс-
(K. phaffii) CBS7435 с использованием про-
формантов проводили на агаризованной сре-
де YPD с добавлением генетицина в течение
Blast.cgi). В случае если последовательность
3-х суток при температуре 30 °С. Для инакти-
спейсера встречалась в геноме более одного
вации генов HIS4 и LEU2 отобранные транс-
раза (при 100% гомологии c целевым локусом),
форманты пересевали 2 раза на среде YPD
она не использовалась.
с добавлением генетицина.
Таблица 1. Нуклеотидный состав спейсерных и PAM-последовательностей
Спейсерные
PAM-последовательности
sgРНК
последовательности (5′→3′)
(5′→3′)
sgРНК1_his4 (1074-1093 п.н.)
GTACGGTGTGACGTTGGACG
AGG
sgРНК2_his4 (1591-1610 п.н.)
GTTGGCCTCTATATTCCTGG
TGG
sgРНК3_his4 (1690-1709 п.н.)
GCATCTCCACCTAAGAAGGA
TGG
sgРНК1_leu2 (430-449 п.н.)
GTTGTTCGTGAGCTTGTAGG
CGG
sgРНК2_leu2 (283-302 п.н.)
GGTGATGTCAGACCAGAACA
AGG
sgРНК3_leu2 (872-891 п.н.)
GCAAGGCTCGTACAGTCCAA
AGG
БИОХИМИЯ том 88 вып. 9 2023
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
1623
Селекцию трансформантов с инактивиро-
Таблица 2. Праймеры и зонды, использованные в работе
ванным геном HIS4 или LEU2 проводили по
Название
Последовательность (5′→3′)
способности к росту на среде YNB с добавле-
нием и без добавления в среду гистидина или
LEU2-F
gattgtagtttacctctgcca
лейцина в концентрации 50 мкг/мл. Отбирали
штаммы, являющиеся ауксотрофными по ги-
LEU2-R
ctagtttttcaaaatg
стидину или лейцину.
Выщепление автономно реплицирующих-
PhyCg-op-F
gacgaacaatctggtatgcaatt
ся плазмид из дрожжевых клеток проводили
путем культивирования штаммов в жидкой пи-
PhyCg-op-R
ttacttttcagcacattcgct
тательной среде YPD при 30 °С и 250 об./мин
в течение 48 ч. Далее клетки высевали на ага-
His-F
ttaaataagtcccagtttc
ризованную среду YPD и инкубировали в
течение 48 ч при 30 °С. Полученные колонии
His-R
acttattttattttgcattag
реплицировали на чашки со средой YPD с до-
бавлением и без добавления генетицина. От-
LEU2-R1
ctttcaatggggagagctt
бирали клоны, не способные к росту на среде
с генетицином.
GAPref-F
tttccagagctgacatcaaggt
Эффективность инактивации генов HIS4
и LEU2 с использованием различных sgРНК
GAPref-R
cttgtaagccttgtgggtagagt
определяли по отношению количества транс-
(ROX)-atcaacgacccattcattgctccaga-
формантов, не способных к росту на среде
GAP-X
(BHQ2)
YNB без добавления в среду гистидина или
лейцина, к общему количеству трансформантов.
PHYref-F
ccaaagaactagaaagactgg
Был получен штамм K. phaffii ВКПМ
Y-5013, ауксотрофный по гистидину, и штамм
PHYref-R
tgggtcagacttagacttgt
K. phaffii ВКПМ Y-5014, ауксотрофный по лей-
цину.
(FAM)-tggtttggctccaaagtgtaaggt-
PHY-X
Секвенирование области гена HIS4 в
(RTQ1)
штамме K. phaffii ВКПМ Y-5013 и гена LEU2
в штамме K. phaffii ВКПМ Y-5014 осущес-
твляли с использованием праймеров His-F и
нуклеотидной последовательности были пред-
His-R (для фрагмента гена HIS4), LEU2-F и
усмотрены сайты рестрикции PaeI и NheI для
LEU2-R1 (для фрагмента гена LEU2). По-
клонирования в вектор pPIC9-PhyCg-op.
следовательности праймеров представлены
В результате была получена экспрессион-
в табл. 2.
ная плазмида pAOX-PhyCg-op-LEU2. Рестрик-
Конструирование экспрессионной интегра-
ционный анализ и секвенирование показали,
тивной плазмиды pAOX-PhyCg-op-LEU2. Для
что плазмида сконструирована корректно
конструирования плазмиды pAOX-PhyCg-op-
(данные не приведены).
LEU2 использовали полученную ранее плаз-
Выделение геномной ДНК проводили с
миду pPIC9-PhyCg-op [18], содержащую в сво-
использованием комплекта реагентов для экс-
ем составе ген phyCg-op, кодирующий фитазу
пресс-выделения ДНК «ДНК-экспресс» («Син-
C. gillenii, встроенный в единую рамку считы-
тол», Россия). Выделение и очистку ПЦР-про-
вания с нуклеотидной последовательностью
дуктов проводили с использованием набора
сигнального пептида α-фактора Saccharomyces
GeneJET Gel Extraction Kit #K0692 («Thermo
cerevisiae, под контролем индуцибельного про-
Scientific»). Все стандартные генно-инженер-
мотора AOX1, терминатор транскрипции
ные манипуляции (обработка ДНК фермен-
TTAOX1 и дрожжевой селективный мар-
тами, лигирование, трансформация клеток
кер PpHIS4. В плазмиде pPIC9-PhyCg-op се-
E. coli) проводили в соответствии со сборни-
лективный маркер PpHIS4 заменяли на селек-
ком методик [23].
тивный маркер PpLEU2, комплементирующий
Получение трансформантов штаммов K. phaf-
у дрожжей K. phaffii мутацию в гене LEU2.
fii ВКПМ Y-5013 и K. phaffii ВКПМ Y-5014,
Ген LEU2 с его регуляторной областью синте-
продуцирующих фитазу C. gillenii. Плазмиды
зировали методом ПЦР, используя в качестве
pPIC9-PhyCg-op и pAOX-PhyCg-op-LEU2 лиа-
матрицы геномную ДНК дрожжей K. phaffii
неризовали эндонуклеазой рестрикции BglII,
ВКПМ Y-4287 и специфические праймеры
получали экспрессионные кассеты pCIT-His
LEU2-F и LEU2-R (табл. 2). На 5′- и 3′-концах
и pCIT-Leu, которые трансформировали в
БИОХИМИЯ том 88 вып. 9 2023
1624
ТКАЧЕНКО и др.
клетки штаммов K. phaffii ВКПМ Y-5013 и
Инактивацию гена HIS4 в штамме K. phaffii
K. phaffii ВКПМ Y-5014 методом электро-
Y-5013/PHF1 осуществляли, как описано
порации, как описано ранее
[22]. Транс-
выше. Полученные ауксотрофные трансфор-
форманты отбирали по способности расти
манты культивировали, измеряли фитазную
на среде YNB без добавления гистидина
активность в КЖ и отбирали трансформант, в
или лейцина.
котором не произошло изменение уровня про-
Наличие интегративной кассеты в составе
дукции фермента по сравнению с родитель-
хромосомы трансформантов штаммов K. phaffii
ским штаммом K. phaffii Y-5013/PHF1.
ВКПМ Y-5013 и K. phaffii ВКПМ Y-5014 опре-
В клетки штамма Y-5013/PHF1Δhis4 по-
деляли методом ПЦР с использованием прай-
вторно трансформировали экспрессионную
меров PhyCg-op-F и PhyCg-op-R (табл. 2).
кассету pCIT-His и отбирали трансформанты
Условия ферментации дрожжевых штам-
на среде YNB без добавления гистидина. Фер-
мов. Трансформанты культивировали следую-
ментацию отобранных трансформантов про-
щим образом. Посевную культуру (инокулят)
водили, как описано выше, оценивали уро-
получали выращиванием трансформантов в
вень продукции фитазы и отбирали наиболее
течение 24 ч при 30 °С и 250 об./мин в жидкой
продуктивный трансформант. Таким образом,
питательной среде YPD. Затем ауксотрофные
был получен многокопийный безмаркерный
штаммы засевали инокулятом в соотноше-
штамм K. phaffii Y-5013/PHF2X.
нии 1/10 в среду YPD, а восстановившие про-
Определение относительного числа копий
тотрофность трансформанты засевали ино-
гена phyCg-op. Относительное количество
кулятом в том же соотношении в среду YNB
копий гена phyCg-op, кодирующего фитазу
и выращивали при 30 °С в течение 24 ч при
C. gillenii, в геномах дрожжевых штаммов опре-
250 об./мин. Далее проводили культивиро-
деляли при помощи ПЦР в реальном времени
вание в течение 48 ч, добавляя метанол в ко-
(ПЦР-РВ) на приборе АНК-32 (Институт ана-
личестве 1% от объема культуральной жид-
литического приборостроения РАН, Москва)
кости (КЖ) каждые 24 ч. После ферментации
методом ΔСt [25]. Рассчитывали разницу между
отбирали аликвоту, КЖ центрифугировали
значениями Сt референсного гена GAP, пред-
при 14 000 g в течение 5 мин на центрифуге
ставленного в геноме в одной копии [26], и ис-
MiniSpin («Eppendorf», Германия) и проводили
следуемого гена. Для референсного гена были
анализ супернатанта на наличие фитазной ак-
синтезированы праймеры GAPref-F, GAPref-R
тивности.
и зонд GAP-X, меченный флуорофором ROX
Определение фитазной активности прово-
и гасителем флуоресценции BHQ-2; для гена
дили с помощью модифицированного метода
phyCg-op - PHYref-F, PHYref-R, а также зонд
Фиске-Субарроу [24], как описано в работе
PHY-X, меченный флуорофором FAM и гаси-
Tkachenko et al. [18].
телем флуоресценции RTQ-1. Последователь-
При исследовании ростовых характери-
ности праймеров приведены в табл. 2. Для
стик ночные культуры исследуемых штаммов
проведения ПЦР-РВ использовали набор ре-
инокулировали с начальным поглощением
активов ПЦР-Микс («Синтол»). В реакцион-
A600 0,1 в жидкую среду YPD и растили в тече-
ную смесь вносили по 5 пМ каждого праймера
ние 24 ч. Поглощение КЖ измеряли на спек-
и зонда, 2 мкл матрицы; общий объем реак-
трофотометре VersaMax Microplate Reader
ционной смеси доводили до 25 мкл ddH2O.
(«Molecular Devices», США). Все измерения
В качестве матрицы использовали хромосом-
проводили в трех независимых повторах.
ную ДНК штаммов K. phaffii Y-5013/PHF1
Последовательное введение экспрессион-
и K. phaffii Y-5013/PHF2Х. ПЦР-РВ проводи-
ной кассеты в хромосому штамма K. phaffii
ли по следующей программе: первоначаль-
ВКПМ Y-5013. В ауксотрофный штамм мето-
ное плавление цепей ДНК при 95 °C в тече-
дом электропорации трансформировали экс-
ние 300 с; далее 37 циклов: 95 °C - 15 с, 60 °C -
прессионную кассету pCIT-His. Трансформан-
40 с. ПЦР проводили в трех независимых по-
ты отбирали по способности расти на среде
вторах. Относительное количество продукта,
YNB без добавления гистидина. Ферментацию
образовавшегося в процессе ПЦР, рассчитыва-
отобранных трансформантов проводили, как
ли по формуле: P ~ (1 + E)n, где P - отно-
описано выше. После ферментации клетки
сительное количество продукта, Е
- сред-
осаждали центрифугированием, супернатанты
няя эффективность цикла, n - число ци-
анализировали на наличие фитазной актив-
клов. Специфичность ампликонов была под-
ности. По результатам ферментации отби-
тверждена путем анализа кривой плавления
рали наиболее продуктивный трансформант
после 30 циклов и электрофореза в агароз-
K. phaffii Y-5013/PHF1.
ном геле.
БИОХИМИЯ том 88 вып. 9 2023
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
1625
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
pGAP-sgRNA2_his4-pTEF-Cas9-Km, pGAP-
sgRNA3_his4-pTEF-Cas9-Km, pGAP-sgRNA1_
Получение ауксотрофных штаммов K. phaf-
leu2-pTEF-Cas9-Km, pGAP-sgRNA2_ leu2-
fii ВКПМ Y-5013 и K. phaffii ВКПМ Y-5014.
pTEF-Cas9-Km и pGAP-sgRNA3_ leu2-pTEF-
Ауксотрофные штаммы получали путем ин-
Cas9-Km, содержащие различные варианты
активации гена HIS4 или гена LEU2 в хромо-
sgРНК под контролем GAP-промотора, а так-
соме штамма K. phaffii Y-4287 с помощью си-
же оптимизированную последовательность
стемы CRISPR/Cas9.
гена cas9 из Streptococcus pyogenes, встроенную
Для успешного геномного редактирования
в единую рамку считывания с нуклеотидной
исследуемого штамма и исключения нецеле-
последовательностью сигнала ядерной локали-
вых мутаций необходим рациональный ди-
зации большого Т-антигена SV40, под контро-
зайн sgРНК. Кроме того, эффективная sgРНК
лем дрожжевого TEF-промотора и CYC1-тер-
должна одновременно демонстрировать наи-
минатора.
высшую целевую эффективность и наимень-
Плазмиды трансформировали в штамм
шую нецелевую активность. Поскольку фак-
K. phaffii ВКПМ Y-4287. Для определения эф-
тическая эффективность sgРНК может раз-
фективности инактивации генов HIS4 и LEU2
личаться, для исследования были разработаны
было проанализировано по 100 трансформан-
по 3 варианта sgРНК с различными спейсер-
тов, полученных с использованием каждого
ными последовательностями, нацеленные на
варианта sgРНК (рис. 1).
разные участки генов HIS4 и LEU2. Содержа-
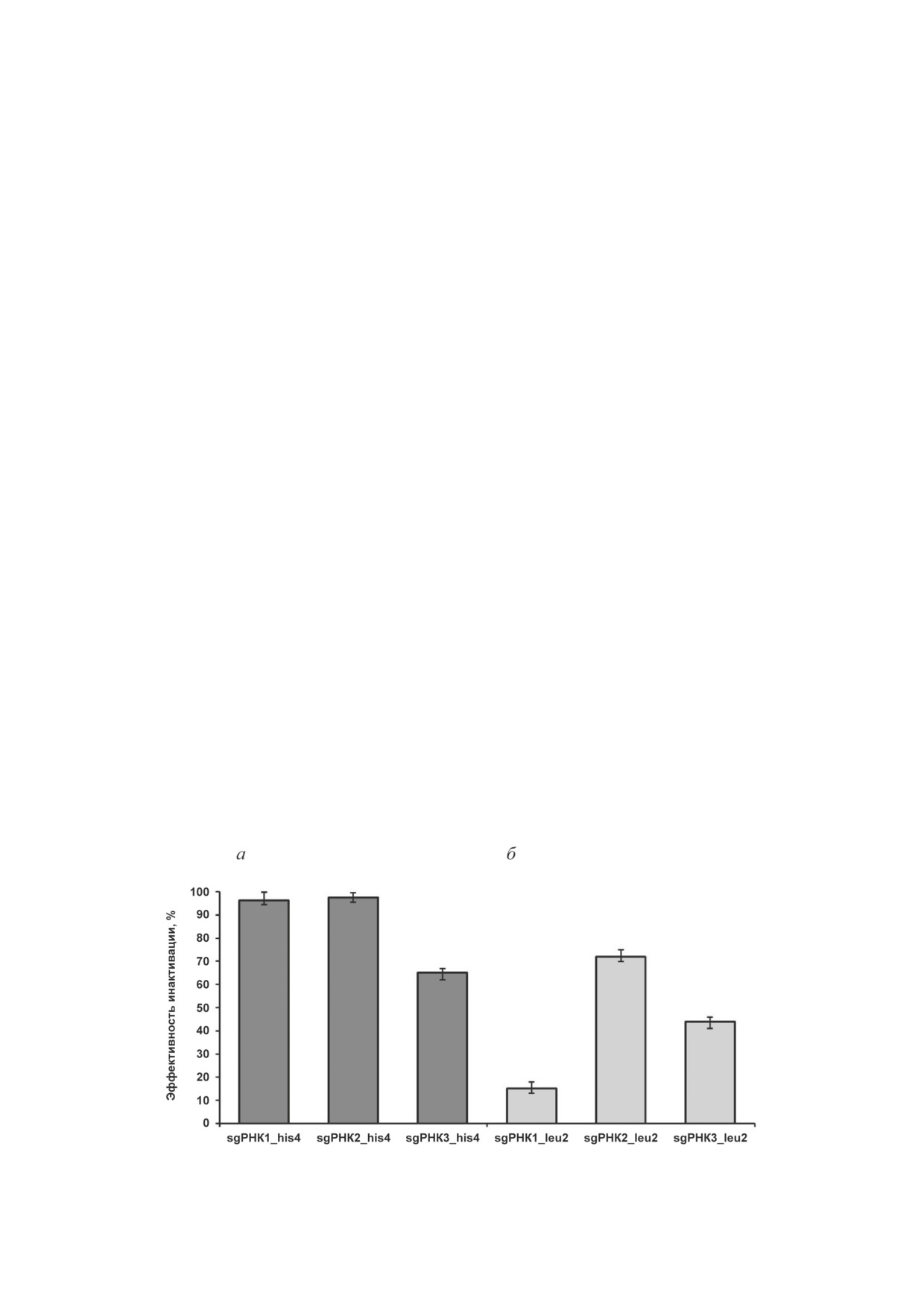
Исследования показали, что эффектив-
ние GC-пар в спейсперных последователь-
ность инактивации целевых генов различалась
ностях составляло 50-60%, так как в работе
в зависимости от используемой конструкции.
Wong et al. [27] было показано, что высокое и
Так, эффективность инактивации гена HIS4
низкое содержание GC-пар в спейсерной по-
с использованием различных sgРНК состави-
следовательности характерно для нефункцио-
ла 65-98%. Самыми эффективными оказались
нальных sgРНК. Последовательности спейсе-
sgРНК1_his4 и sgРНК2_his4, показав сравни-
ров приведены в табл. 1.
тельно одинаковую эффективность (97% - для
Преимуществом нацеливания sgРНК на
sgРНК1_his4 и 98% - для sgРНК2_his4).
последовательности генов, кодирующих фер-
Эффективность инактивации гена LEU2 со-
менты биосинтеза аминокислот, является про-
ставила 15-72%. Cамую низкую эффективность
стое и надежное подтверждение инактива-
инактивации показала sgРНК1_leu2
(15%),
ции этих генов, основанное на способности
а самой эффективной оказалась sgРНК2_leu2
полученных штаммов к росту на минималь-
с эффективностью 72%.
ной среде.
Известно, что эффективность системы
Для инактивации генов HIS4 и LEU2 были
CRISPR/Cas9 зависит от нуклеотидного со-
сконструированы автономно реплицирующиеся
става спейсерных последовательностей sgРНК
плазмиды pGAP-sgRNA1_his4-pTEF-Cas9-Km,
и PAM-сайта [28]. Так, в работе Yang et al. [29]
Рис. 1. Эффективность инактивации генов HIS4 и LEU2 с использованием различных вариантов sgРНК. a - Инакти-
вация гена HIS4; б - инактивация гена LEU2. Приведены средние значения и стандартные отклонения
БИОХИМИЯ том 88 вып. 9 2023
1626
ТКАЧЕНКО и др.
утверждалось, что предпочтительным PAM-
сайтом, примыкающим к спейсеру sgРНК, яв-
ляется CGG, однако в нашей работе высокую
эффективность инактивации показали sgРНК,
для которых сайтами PAM были AGG и TGG.
Опираясь на результаты исследований в рабо-
те Doench et al. [30], это можно, предположи-
тельно, объяснить различным нуклеотидным
составом PAM-проксимальных областей спей-
серов sgРНК.
Для дальнейшей работы были отобра-
ны штаммы K. phaffii TGBF7 (фенотип His-,
получен с использованием sgРНК3_his4) и
K. phaffii YLM9 (фенотип Leu-, получен с ис-
пользованием sgРНК2_leu2), не способные к
росту на среде YNB без добавления гистидина
или лейцина. Выщепление автономно репли-
цирующихся плазмид проводили путем дву-
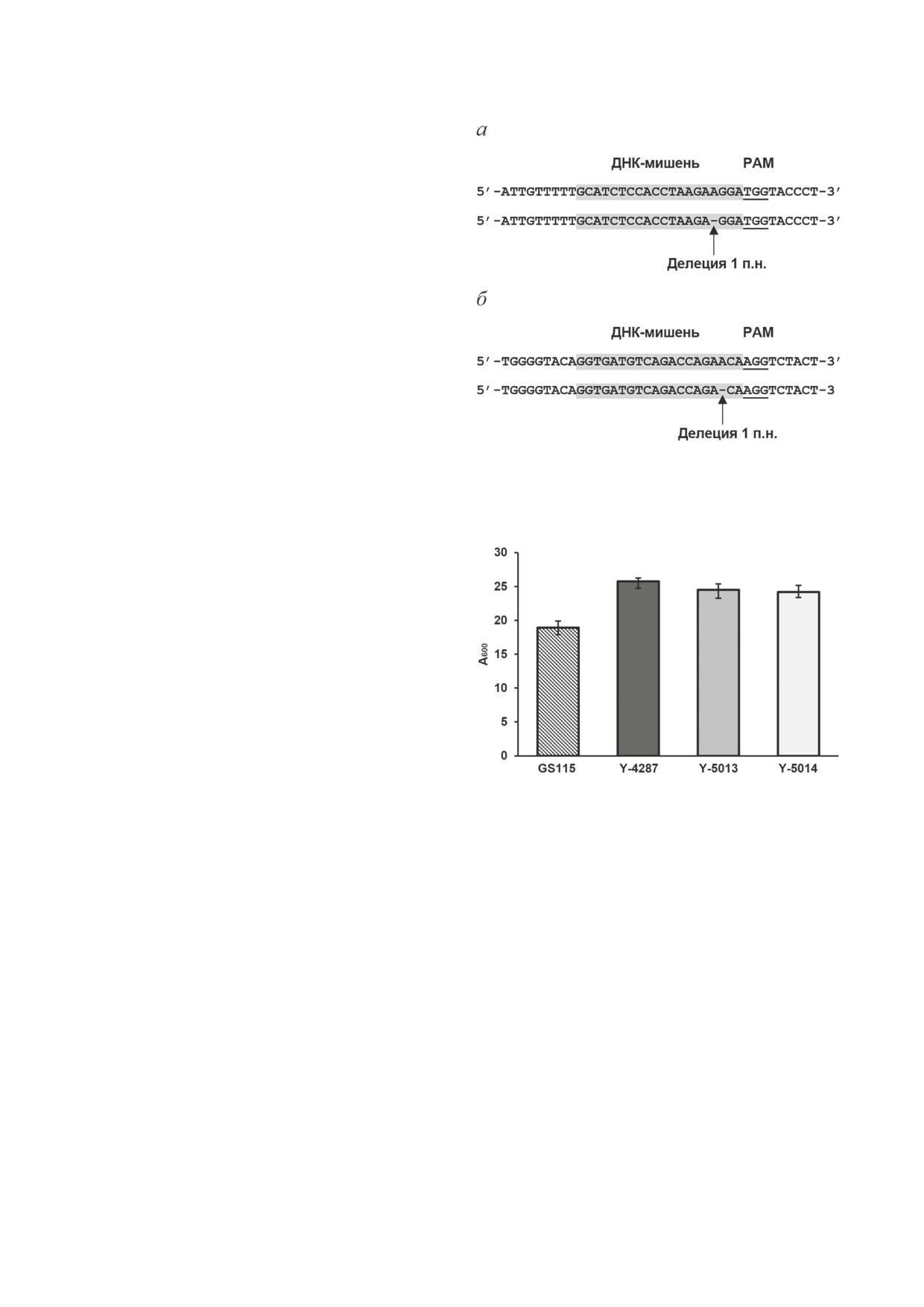
Рис. 2. Результаты секвенирования фрагментов гена HIS4
кратных пересевов на среде YPD без селектив-
штамма K. phaffii TGBF7 (a) и гена LEU2 штамма
ного давления.
K. phaffii YLM9 (б). Последовательности ДНК-мишеней
выделены серым цветом, сайты PAM - подчеркиванием
Для подтверждения инактивации гена HIS4
в штамме K. phaffii TGBF7 и гена LEU2 в штам-
ме K. phaffii YLM9 было проведено секвени-
рование областей, в которых при воздействии
нуклеазы Cas9 должны были произойти мута-
ции. Результаты секвенирования представле-
ны на рис. 2.
В результате действия комплекса sgРНК3_
his4-Cas9 в последовательности гена HIS4 про-
изошла делеция размером 1 п.н. на расстоянии
3 п.н. от сайта PAM, а в результате действия
комплекса sgРНК2_leu2-Cas9 в последователь-
ности гена LEU2 произошла делеция размером
1 п.н. на расстоянии 4 п.н. от сайта PAM. По-
лученные результаты объясняются тем, что у
дрожжей K. phaffii двухцепочечные разрывы ге-
Рис. 3. Накопление биомассы штаммами за 24 ч при 30 °C
номной ДНК, вносимые нуклеазой Cas9, вос-
станавливаются в основном клеточным меха-
низмом негомологичного соединения концов,
ВКПМ Y-5014. Ростовые характеристики
в результате чего в месте разрыва, как правило,
штаммов K. phaffii ВКПМ Y-5013 и K. phaffii
происходят делеции или вставки нуклеотидов,
ВКПМ Y-5014 изучались в сравнении с ха-
что приводит к сдвигу рамки считывания ко-
рактеристиками
родительского
штамма
дирующей последовательности [31].
K. phaffii Y-4287 и коммерческого
штамма
Сдвиг рамок считывания и образование
K. phaffii GS115 Y-2837. Было проведено срав-
преждевременных стоп-кодонов в последова-
нение уровня накопления биомассы за 24 ч в
тельностях генов HIS4 и LEU2 привело к не-
среде YPD путем измерения поглощения КЖ
возможности синтеза гистидинолдегидроге-
исследуемых штаммов. Результаты измерений
назы и β-изопропилмалатдегидрогеназы, уча-
представлены на рис. 3.
ствующих в биосинтезе гистидина и лейцина,
Исследования показали, что количество
в результате чего штаммы приобрели ауксо-
накопленной биомассы штаммами K. phaffii
трофность по этим аминокислотам.
ВКПМ Y-5013 и K. phaffii ВКПМ Y-5014 пре-
Ауксотрофные штаммы K. phaffii TGBF7 и
вышало таковое для контрольного штамма
K. phaffii YLM9 были депонированы в БРЦ
K. phaffii GS115 Y-2837 более чем на
20%.
ВКПМ под номерами ВКПМ Y-5013 и ВКПМ
Также наблюдалось незначительное сниже-
Y-5014 соответственно [32, 33].
ние накопленной биомассы ауксотрофных
Изучение ростовых характеристик штам-
штаммов относительно штамма дикого типа
мов K. phaffii ВКПМ Y-5013 и K. phaffii
K. phaffii Y-4287.
БИОХИМИЯ том 88 вып. 9 2023
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
1627
Рис. 4. Строение интегративных экспрессионных кассет. a - Кассета pCIT-His; б - кассета pCIT-Leu
Широко известно, что система CRISPR/
В качестве контроля получали трансфор-
Cas9 обеспечивает высокоэффективное редак-
манты штамма K. phaffii GS115 ВКПМ Y-2837.
тирование генома у широкого спектра организ-
Затем случайным образом отбирали по
10
мов [34], однако, несмотря на свою высокую
ПЦР-положительных трансформантов каждо-
специфичность, может проявлять нецелевую
го штамма и проводили их ферментацию. По-
активность [35], что может негативно сказы-
сле ферментации измеряли фитазную актив-
ваться на их физиологических характеристи-
ность трансформантов в КЖ.
ках. Несущественное изменение ростовых ха-
На основании полученных данных опре-
рактеристик штаммов K. phaffii ВКПМ Y-5013
деляли среднюю продуктивность трансфор-
и K. phaffii ВКПМ Y-5014 относительно роди-
мантов исследуемых штаммов. Результаты
тельского штамма K. phaffii Y-4287 свидетель-
сравнения этих величин со средней продуктив-
ствует о высокой специфичности и низкой
ностью трансформантов контрольного штам-
нецелевой активности разработанных sgРНК,
ма K. phaffii GS115 ВКПМ Y-2837, принятой
с использованием которых были получены
за 100%, приведены в табл. 3.
ауксотрофные штаммы.
Из данных, приведенных в табл. 3, следует,
Изучение экспрессионного потенциала штам-
что среднее значение продуктивности транс-
мов-реципиентов. Экспрессионный потенциал
формантов штаммов K. phaffii ВКПМ Y-5013
штаммов K. phaffii ВКПМ Y-5013 и K. phaffii
и K. phaffii ВКПМ Y-5014 превышает таковое
ВКПМ Y-5014 изучали по продуктивности
для трансформантов коммерческого штамма
получаемых на их основе трансформантов,
K. phaffii GS115 ВКПМ Y-2837 более чем в 2,1
секретирующих фитазу C. gillenii. Ранее было
и 2,0 раза соответственно. Таким образом,
показано, что ген phyCg-op, кодирующий дан-
было показано, что полученные штаммы-ре-
ную фитазу, эффективно экспрессируется в
ципиенты обладают высоким экспрессионным
дрожжах K. phaffii [18].
потенциалом, что позволяет использовать их
Плазмиды pPIC9-PhyCg-op и pAOX-PhyCg-
для получения высокопродуктивных штам-
op-LEU2 лианеризовали эндонуклеазой ре-
мов-продуцентов гетерологичных белков.
стрикции BglII, и получали экспрессионные
По результатам ферментации был ото-
кассеты pCIT-His (рис. 4, а) и pCIT-Leu
бран наиболее продуктивный штамм K. phaffii
(рис. 4, б), которые были трансформированы
Y-5013/PHF1, секретирующий фитазу C. gillenii
в клетки штаммов K. phaffii ВКПМ Y-5013
в количестве 286 ед./мл. Определение количе-
и K. phaffii ВКПМ Y-5014.
ства копий гена phyCg-op в хромосоме отобран-
ного штамма проводили методом ПЦР-РВ.
Было выявлено наличие 2-х копий гена, коди-
Таблица 3. Средняя продуктивность трансформантов аук-
рующего фитазу C. gillenii.
сотрофных штаммов
Повторная интеграция экспрессионной кас-
Средняя
сеты pCIT-His. Количество копий целевого
Относительная
продуктивность
гена является важным фактором для продук-
Штамм
продуктивность
трансформантов,
трансформантов, %
ции целевого белка в дрожжах K. phaffii [36].
ед./мл
Введение в состав хромосомы штаммов-ре-
ципиентов множественных копий целевых
Y-2837
126,2 ± 3,1
100
генов является одним из подходов к получе-
нию высокоактивных штаммов-продуцентов
Y-5013
265,4 ± 1,9
210
гетерологичных белков. В связи с этим был
предложен способ последовательной интегра-
Y-5014
253,6 ± 2,6
201
ции генетического материала в геном дрожжей
БИОХИМИЯ том 88 вып. 9 2023
1628
ТКАЧЕНКО и др.
K. phaffii с использованием системы CRISPR/
струированы ауксотрофные штаммы-реципи-
Cas9.
енты K. phaffii ВКПМ Y-5013 (фенотип His-) и
В штамме K. phaffii Y-5013/PHF1 была про-
K. phaffii ВКПМ Y-5014 (фенотип Leu-), обла-
ведена инактивация маркерного гена HIS4.
дающие ростовыми характеристиками, срав-
Для этого в клетки штамма трансформи-
нимыми с родительским штаммом, и высоким
ровали плазмиду pGAP-sgRNA3_his4-pTEF-
экспрессионным потенциалом, что было про-
Cas9-Km, получали трансформанты и про-
демонстрировано на примере экспрессии гена
водили их селекцию на предмет приобретения
фитазы C. gillenii.
ауксотрофности по гистидину, как описано
Был продемонстрирован способ последо-
выше. Проверка продуктивности 10 случай-
вательной интеграции генетического материа-
но отобранных ауксотрофных трансформан-
ла в хромосому дрожжей K. phaffii с использо-
тов показала отсутствие влияния введенной
ванием системы CRISPR/Cas9. Применение
мутации на продукцию фитазы. Был выбран
данного способа позволило получить многоко-
штамм Y-5013/PHF1Δhis4, в который повторно
пийный безмаркерный штамм-продуцент фи-
трансформировали экспрессионную кассету
тазы C. gillenii с активностью 480,5 ед./мл КЖ.
pCIT-His. Продуктивность трансформантов
определяли путем измерения фитазной актив-
Вклад авторов. Т.Л. Гордеева, С.П. Сине-
ности в КЖ. В результате был отобран наи-
окий - концепция и руководство работой;
более продуктивный штамм K. phaffii Y-5013/
А.А. Ткаченко - проведение экспериментов;
PHF2X, секретирующий фитазу C. gillenii в ко-
Т.Л. Гордеева, А.А. Ткаченко, Л.Н. Борщев-
личестве 480,5 ед./мл. Штамм был депониро-
ская - обсуждение результатов исследования;
ван в БРЦ ВКПМ под номером ВКПМ Y-5127.
А.А. Ткаченко, Т.Л. Гордеева - написание тек-
Методом ПЦР-РВ было определено, что в хро-
ста; Т.Л. Гордеева, Л.Н. Борщевская, А.А. Тка-
мосоме штамма K. phaffii ВКПМ Y-5127 содер-
ченко - редактирование текста статьи.
жится 5 копий гена phyCg-op. Таким образом,
Финансирование. Работа выполнена при
была показана возможность последовательной
финансовой поддержке Министерства науки и
интеграции генетического материала в хромо-
высшего образования Российской Федерации:
сому штамма-реципиента с использованием
грант для Курчатовского центра геномных ис-
системы CRISPR/Cas9.
следований (Соглашение № 075-15-2019-1659).
Конфликт интересов. Авторы заявляют об
отсутствии конфликта интересов.
ЗАКЛЮЧЕНИЕ
Соблюдение этических норм. Настоящая
статья не содержит описания выполненных
С использованием системы геномного
авторами исследований с участием людей или
редактирования CRISPR/Cas9 были скон-
животных в качестве объектов.
СПИСОК ЛИТЕРАТУРЫ
1. Ahmad, M., Hirz, M., Pichler, H., and Schwab, H.
High level expression of a synthetic gene encoding
(2014) Protein expression in Pichia pastoris: recent
Peniophora lycii phytase in methylotrophic yeast Pichia
achievements and perspectives for heterologous
pastoris, App. Microbiol. Biotechnol., 72, 1039-1047,
protein production, Appl. Microbiol. Biotechnol., 98,
doi: 10.1007/s00253-006-0384-8.
5301-5317, doi: 10.1007/s00253-014-5732-5.
5. Pal Roy, M., Mazumdar, D., Dutta, S., Saha,
2. Luo, H. Y., Yao, B., Yuan, T. Z., Wang, Y. R., Shi,
S. P., and Ghosh, S. (2016) Cloning and expression
X. Y., Wu, N. F., and Fan, Y. L. (2004) Overexpression
of phytase appA gene from Shigella sp. CD2 in
of Escherchia coli phytase with high specific activity,
Pichia pastoris and comparison of properties with
Sheng Wu Gong Cheng Xue Bao=Chin. J. Biotechnol.,
recombinant enzyme expressed in E. coli, PLoS One,
20, 78-84.
11, e0145745, doi: 10.1371/journal.pone.0145745.
3. Huang, H., Luo, H., Yang, P., Meng, K., Wang, Y.,
6. Zhao, W., Xiong, A., Fu, X., Gao, F., Tian, Y., and
Yuan, T., Bai, Y., and Yao, B. (2006) A novel
Peng, R. (2010) High level expression of an acid-
phytase with preferable characteristics from Yersinia
stable phytase from Citrobacter freundii in Pichia
intermedia, Biochem. Biophys. Res. Commun., 350,
pastoris, Appl. Biochem. Biotechnol., 162, 2157-2165,
884-889, doi: 10.1016/j.bbrc.2006.09.118.
doi: 10.1007/s12010-010-8990-4.
4. Xiong, A. S., Yao, Q. H., Peng, R. H., Zhang, Z.,
7. Розанов А. С., Першина Е. Г., Богачева Н. В.,
Xu, F., Liu, J. G., Han, P. L., and Chen, J. M. (2006)
Шляхтун В., Сычев А. А., Пельтек С. Е. (2020)
БИОХИМИЯ том 88 вып. 9 2023
РЕДАКТИРОВАНИЕ ГЕНОМА Komagataella phaffii
1629
Разнообразие и распространение метилотроф-
(Komagataella pastoris), FEMS Microbiol. Lett., 368,
ных дрожжей, используемых в генной инженерии,
fnaa217, doi: 10.1093/femsle/fnaa217.
Вавиловский журнал генетики и селекции, 24, 149-
19.
Gao, Y., and Zhao, Y. (2014) Self-processing of
157, doi: 10.18699/VJ20.602.
ribozyme-flanked RNAs into guide RNAs in vitro
8.
Pronk, J. T. (2002) Auxotrophic yeast strains in
and in vivo for CRISPR-mediated genome editing,
fundamental and applied research, App. Environ.
J. Integr. Plant. Biol., 56, 343-349, doi: 10.1111/
Microbiol., 68, 2095-2100, doi: 10.1128/AEM.68.5.
jipb.12152.
2095-2100.2002.
20.
Heigwer, F., Kerr, G., and Boutros, M.
(2014)
9.
Wang, Y., Yau, Y. Y., Perkins-Balding, D., and
E-CRISP: fast CRISPR target site identification, Nat.
Thomson, J. G. (2011) Recombinase technology:
Methods, 11, 122-123, doi: 10.1038/nmeth.2812.
applications and possibilities, Plant Cell Rep., 30, 267-
21.
Gassler, T., Heistinger, L., Mattanovich, D.,
285, doi: 10.1007/s00299-010-0938-1.
Gasser, B., and Prielhofer, R. (2019) CRISPR/Cas9-
10.
Cregg, J. M., Barringer, K. J., Hessler, A. Y., and
mediated homology-directed genome editing in Pichia
Madden, K. R. (1985) Pichia pastoris as a host system
pastoris, Recombinant protein production in yeast,
for transformations, Mol. Cell. Biol., 5, 3376-3385,
Methods in Molecular Biology, Humana Press, N.Y.,
doi: 10.1128/mcb.5.12.3376-3385.1985.
pp. 211-225, doi: 10.1007/978-1-4939-9024-5_9.
11.
Theodorakis, C. W.
(2018) Mutagenesis, in
22.
Gasser, B., Prielhofer, R., Marx, H., Maurer, M.,
Encyclopedia Ecology (S. E. Jørgensen, B. D. Fath,
Nocon, J., Steiger, M., Puxbaum, V., Sauer, M.,
eds.) Academic Press, pp. 2475-2484, doi: 10.1016/
and Mattanovich, D. (2013) Pichia pastoris: protein
b978-008045405-4.00408-0.
production host and model organism for biomedical
12.
Näätsaari, L., Mistlberger, B., Ruth, C., Hajek, T.,
research, Fut. Microbiol., 8, 191-208, doi: 10.2217/
Hartner, F. S., and Glieder, A. (2012) Deletion of the
fmb.12.133.
Pichia pastoris KU70 homologue facilitates platform
23.
Sambrook, J., Fritsch, E. F., and Maniatis, T. (1989)
strain generation for gene expression and synthetic
Molecular Cloning: A Laboratory Manual, 2nd Edn.,
biology, PLoS One, 7, e39720, doi: 10.1371/journal.
Cold Spring Harbor, Cold Spring Harbor Laboratory
pone.0039720.
Press, N.Y.
13.
Weninger, A., Fischer, J. E., Raschmanova, H.,
24.
Chen, C. C., Wu, P. H., Huang, C. T., and Cheng, K.
Vogl, T., and Glieder, A. (2018) Expanding the
(2004) A Pichia pastoris fermentation strategy for
CRISPR/Cas9 toolkit for Pichia pastoris with efficient
enhancing the heterologous expression of an Esche-
donor integration and alternative resistance markers,
richia coli phytase, Enzyme Microb. Technol., 35, 315-
J. Cell. Biochem.,
119,
3183-3198, doi:
10.1002/
320, doi: 10.1016/j.enzmictec.2004.05.007.
jcb.26474.
25.
Ребриков Д. В. (2011) ПЦР в реальном времени,
14.
Weninger A., Hatzl A. M., Schmid C., Vogl, T., and
БИНОМ, Москва.
Glieder, A. (2016) Combinatorial optimization of
26.
Waterham, H. R., Digan, M. E., Koutz, P. J.,
CRISPR/Cas9 expression enables precision genome
Lair, S. V., and Cregg, J. M. (1997) Isolation of
engineering in the methylotrophic yeast Pichia
the Pichia pastoris glyceraldehyde-3-phosphate
pastoris, J. Biotechnol., 235, 139-149, doi: 10.1016/
dehydrogenase gene and regulation and use of its
j.jbiotec.2016.03.027.
promoter, Gene, 186, 37-44, doi: 10.1016/S0378-1119
15.
Немудрый А. А., Валетдинова К. Р., Медведев
(96)00675-0.
С. П., Закиян С. М. (2014) Системы редакти-
27.
Wong, N., Liu, W., and Wang, X.
(2015)
рования геномов TALEN и CRISPR/Cas ин-
WU-CRISPR: characteristics of functional guide
струменты открытий, Acta Naturae,
6,
20-42,
RNAs for the CRISPR/Cas9 system, Genome Biol.,
doi: 10.32607/20758251-2014-6-3-19-40.
16, 218, doi: 10.1186/s13059-015-0784-0.
16.
Mohammadhassan, R., Tutunchi, S., Nasehi, N,
28.
Cho, S. W., Kim, S., Kim, Y., Kweon, J., Kim,
Goudarziasl, F., and Mahya, L. (2023) The prominent
H. S., Bae, S., and Kim, J. S. (2014) Analysis of
characteristics of the effective sgRNA for a precise
off-target effects of CRISPR/Cas-derived RNA-
CRISPR genome editing, CRISPR Technol. Recent
guided endonucleases and nickases, Genome Res., 24,
Adv., IntechOpen, doi: 10.5772/intechopen.106711.
132-141, doi: 10.1101/gr.162339.113.
17.
Гордеева Т. Л., Борщевская Л. Н., Федай Т. В.,
29.
Yang, Y., Liu, G., Chen, X., Liu, M., Zhan, C.,
Ткаченко А. А., Синеокий С. П. (2021) Изучение
Liu, X., and Bai, Z. (2020) High efficiency CRISPR/
экспрессионного потенциала новых штаммов
Cas9 genome editing system with an eliminable
дрожжей рода Komagataella, Биотехнология, 37,
episomal sgRNA plasmid in Pichia pastoris, Enzyme
5-13, doi: 10.21519/0234-2758-2021-37-4-5-13.
Microb. Technol.,
138,
109556, doi:
10.1016/
18.
Tkachenko, A. A., Kalinina, A. N., Borshchevskaya,
j.enzmictec.2020.109556.
L. N., Sineoky, S. P., and Gordeeva, T. L.
30.
Doench, J. G., Hartenian, E., Graham, D. B.,
(2021) A novel phytase from Citrobacter gillenii:
Tothova, Z., Hegde, M., Smith, I., Sullender, M.,
characterization and expression in Pichia pastoris
Ebert, B. L., Xavier, R. J., and Root, D. E. (2014)
БИОХИМИЯ том 88 вып. 9 2023
1630
ТКАЧЕНКО и др.
Rational design of highly active sgRNAs for CRISPR-
геном LEU2 - реципиент для конструирования
Cas9-mediated gene inactivation, Nat. Biotechnol., 32,
штаммов-продуцентов гетерологичных белков:
1262-1267, doi: 10.1038/nbt.3026.
№ 2022127025: заявл. 18.10.2022: опубл. 23.01.2023/
31. Liu, Q., Shi, X., Song, L., Liu, H., Zhou, X.,
Ткаченко А. А., Гордеева Т. Л., Синеокий С. П.,
Wang, Q., Zhang, Y., and Cai, M. (2019) CRISPR-
Борщевская Л. Н.; заявитель Федеральное госу-
Cas9-mediated genomic multiloci integration in Pichia
дарственное бюджетное учреждение «Националь-
pastoris, Microb. Cell Factories, 18, 144, doi: 10.1186/
ный исследовательский центр “Курчатовский ин-
s12934-019-1194-x.
ститут”» - 13 с.
32. Патент
№ 2787584С1 Российская Федерация,
34. Hsu, P. D., Lander, E. S., and Zhang, F. (2014) De-
МПК C12N1/19, C12N15/81, C12N9/24. Штамм
velopment and applications of CRISPR-Cas9 for ge-
дрожжей Komagataella phaffii с инактивирован-
nome engineering, Cell, 157, 1262-1278, doi: 10.1016/
ным геном HIS4 - реципиент для конструирова-
j.cell.2014.05.010.
ния безмаркерных штаммов-продуцентов гетеро-
35. Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S.,
логичных белков: №2022127026: заявл. 18.10.2022:
and Yang, S. H. (2015) Off-target effects in CRISPR/
опубл. 11.01.2023/Ткаченко А. А., Гордеева Т. Л.,
Cas9-mediated genome engineering, Mol. Ther.
Синеокий С. П., Борщевская Л. Н., Федай Т. Д.;
Nucleic. Acids, 4, E264, doi: 10.1038/mtna.2015.37.
заявитель Федеральное государственное бюджет-
36. Zhu, T., Guo, M., Tang, Z., Zhang, M., Zhuang, Y.,
ное учреждение «Национальный исследователь-
Chu, J., and Zhang, S. (2009) Efficient generation of
ский центр “Курчатовский институт”» - 11 с.
multi-copy strains for optimizing secretory expression
33. Патент
№ 2788528 Российская Федерация,
of porcine insulin precursor in yeast Pichia pastoris,
МПК C12N 1/19, C12N 15/63. Штамм дрож-
J. Appl. Microbiol.,
107,
954-963, doi:
10.1111/
жей Komagataella phaffii с инактивированным
j.1365-2672.2009.04279.x.
CRISPR/Cas9-MEDIATED GENOME EDITING
OF THE Komagataella phaffii TO OBTAIN A PHYTASE-PRODUCER
MARKERLESS STRAIN
A. A. Tkachenko*, L. N. Borshchevskaya, S. P. Sineoky, and T. L. Gordeeva
NRC “Kurchatov Institute”, 117545 Moscow, Russia; e-mail: artur.tka4enko10@gmail.com
Using the CRISPR/Cas9 system, the recipient strains K. phaffii VKPM Y-5013 (His- phenotype) and
K. phaffii VKPM Y-5014 (Leu- phenotype) were derived from the K. phaffii VKPM Y-4287 strain, which
has a high expression potential. Based on developed recipients, markerless producers can be obtained.
The gene inactivation efficiency with different variants of sgRNA ranged from 65 to 98% and from 15
to 72% for HIS4 and LEU2, respectively. The recipient strains retained the growth characteristics of the
parent strain and have a high expression potential, as estimated by the production of heterologous phytase
from Citrobacter gillenii. The average productivity of the transformants based on K. phaffii VKPM Y-5013
and K. phaffii VKPM Y-5014 strains was 2.1 and 2.0 times higher than the productivity of the transformants
of the commercial K. phaffii GS115 strain. Sequential integration of genetic material into the genome
of the K. phaffii VKPM Y-5013 strain was proposed. A highly effective multicopy markerless strain produc-
ing C. gillenii phytase was obtained.
Keywords: CRISPR/Cas9, Komagataella phaffii, genome editing, phytase, Citrobacter gillenii
БИОХИМИЯ том 88 вып. 9 2023