ПАРАЗИТОЛОГИЯ, 2023, том 57, № 1, с. 38-43.
УДК 576.895.121.56 / 599.742.11
МОЛЕКУЛЯРНАЯ ХАРАКТЕРИСТИКА
ECHINOCOCCUS CANADENSIS (CESTODA, TAENIIDAE)
ОТ ВОЛКА ИЗ КИРОВСКОЙ ОБЛАСТИ
© 2023 г. О. А. Логиноваa,*, С. Э. Спиридоновa,
О. В. Масленниковаb, Л. А. Букинаb
aФГБУН «Институт проблем экологии и эволюции им. А.Н. Северцова РАН»,
Ленинский просп., 33, Москва, 119071 Россия
bФГБОУ ВО «Вятский государственный агротехнологический университет»,
Октябрьский просп., 133, Киров, 610017 Россия
*e-mail: loginova_spb@bk.ru
Поступила в редакцию 13.12.2022 г.
После доработки 19.01.2023 г.
Принята к публикации 21.01.2023 г.
Представлены результаты изучения молекулярной характеристики паразитических ленточ-
ных червей Echinococcus canadensis. Гельминты были обнаружены при вскрытии волка (Ca-
nis lupus Linnaeus, 1758), добытого охотниками в Кировской области в 2021 г. Молекулярно-
филогенетическое исследование проводили путем анализа последовательности фрагмента
первой субъединицы митохондриального гена цитохромоксидазы (CoxI). Установили, что об-
наруженные эхинококки относятся к генотипу G10 E. canadensis, обычному для волков на
северных территориях Голарктики. Обнаружены четыре позиции, по которым выявляются ха-
рактерные только для этого генотипа замены. Обнаружена также замена по одной из позиций,
которая характерна исключительно для представителей генотипа G10, обнаруженных в России
и Финляндии.
Ключевые слова: эхинококк, Echinococcus canadensis, G10, волк, Кировская область
DOI: 10.31857/S0031184723010039; EDN: FJCPFX
Паразитические ленточные черви рода Echinococcus Rudolphi, 1801 известны че-
ловечеству еще со времен Гиппократа, Аретея и Галена (Beaver et al., 1984). Однако
даже в XXI столетии в изучении эхинококков остается много нерешенных вопро-
сов. Например, все еще нет полного понимания и единства мнений относительно
систематического положения видов этого рода. Известен список из девяти валид-
ных видов, включая E. granulosus s. str., E. canadensis (он же - E. g. canadensis или
E. g. borealis), а также генотипы E. canadensis - G6/G7, G8 и G10. Т.е. E. canadensis,
38
с одной стороны, входит в группу E. granulosus s. lato, а с другой - подразделяется
на генетически различные субъединицы. Важно, что генотипы E. canadensis имеют
некоторые различия по областям распространения, дефинитивным и промежуточным
хозяевам (Nakao et al., 2013a, 2013b). Было высказано предложение закрепить видовое
название E. canadensis за генотипом G10, генотип G6/G7 называть E. intermedius,
а G8 - E. borealis (Lymbery et al., 2015). В этой связи должна быть использована
любая возможность расширить наши представления о молекулярных характеристиках
эхинококков.
МАТЕРИАЛ И МЕТОДИКА
Отбор проб
10.10.2021 на границе Свечинского и Даровского районов Кировской области (58°65'13.4 N,
47°69'88.2 E) охотники добыли волка (Canis lupus Linnaeus, 1758). В тонком отделе кишечника
были обнаружены цестоды цвета слоновой кости длиной 3-6 мм. Методом многократного про-
мывания (Скрябин, 1928; Ивашкин и др., 1971) их извлекли и произвели подсчет по сколексам
(2920 экз.). Морфометрические характеристики собранных цестод соответствуют диагнозу вида
E. granulosus (Петров, Черткова, 1959 по: Абуладзе, 1964). Эхинококков поместили в пластико-
вую пробирку, которую наполнили дистиллированной водой и заморозили для последующего
хранения и транспортировки.
Молекулярно-филогенетическое исследование
Фрагменты стробилы трех эхинококков трижды ополоснули в стерильной воде, разделили
на части (размером не более 1-2 мм3) и поместили в пробирки Эппендорфа объемом 1.5 мл.
Выделение ДНК проводили с помощью колонок из набора Wizard Genomic DNA Purification Kit
(Promega, США) в соответствии с протоколом производителя. От выделенных проб эхинококков
использовали от 1.0 до 1.6 мкл экстрагированной ДНК для проведения полимеразной цепной
реакции (ПЦР). Таксономическую принадлежность цестод устанавливали путем анализа по-
следовательности фрагмента первой субъединицы митохондриального гена цитохромоксидазы
(CoxI). Для этого осуществляли постановку ПЦР с помощью праймеров Thg452 (TGCATT-
TAGCTGGTGCGTCAAGTA) и Thg1326R (ACAAACACGCCGGGGTAACC) и соответствующего
протокола (Littlewood et al., 2008) на апмлификаторе T100 Thermal Cycler (Bio-Rad, США). Для
визуальной оценки амплификации прибегали к электрофоретическому разделению продукта
ПЦР в 1%-ном агарозном геле в течение 30 мин при напряжении 100 В в системе для электро-
фореза Mini-Sub Cell GT + PowerPac Basic (Bio-Rad, США) с последующим просмотром геля
в трансиллюминаторе EXC-F15.M (Vilber Lourmat, Германия). Получив продукт ожидаемого
размера (что определяли на основании оригинальных публикаций или собственного предше-
ствующего опыта), оценивали его количество. В случае получения лишь слабых полос продукта
проводили дополнительную амплификацию. Достаточное количество продукта объединяли
в одну пробу и проводили ее очистку в 0.8% агарозном геле с помощью продолжительного
(70-90 мин) электрофореза. Полосу с нужным размером продукта вырезали из геля пласти-
ковым ножом. Из вырезанного фрагмента геля с амплифицированной ДНК выделяли продукт
ПЦР с помощью набора SV Gel and PCR Clean-Up (Promega, США). Очищенный с помощью
этих колонок продукт подвергали дополнительной очистке с помощью преципитации спиртом
в присутствии ацетата аммония. Очищенный осадок разводили в автоклавированной воде вы-
сокой очистки - Molecular Grade Water (G-Biosciences, США). Количество добавляемой воды
39
меняли от 10 до 40 мкл, в зависимости от выраженности полос в геле. С помощью спектро-
фотометра NanoDrop 2000 (Thermo Fisher Scientific, США) определяли концентрацию ДНК
в полученной пробе. Пробы с высокой концентрацией ДНК разводили до концентрации
15-30 нг на 1 мкл. В дальнейшем на базе центра коллективного пользования «Генотех» (Ин-
ститут молекулярной биологии РАН) проводили прямое секвенирование (т.е. секвенирование
с теми же праймерами, которые были использованы для получения первичного ПЦР продукта).
Полученные хроматограммы в формате ab1 для прямого и обратного прочтений объединя-
ли и анализировали с помощью программы Chromas 2.6.6 (Technelisium Pty Ltd, Австралия).
В ГенБанке (GenBank NCBI) проводили поиск филогенетически близких последовательностей
с помощью алгоритма BLASTN 2.13.0+ (Zhang et al., 2000; Morgulis et al., 2008). В качестве
внешней группы сравнения выбирали представителя другого семейства, рода или группы видов.
Собственные и полученные из ГенБанка данные использовали для построения выравниваний
в программе Clustal X (UCD, Ирландия). Полученные выравнивания в формате aln переводили
в формат msf для обработки в программе GeneDoc 2.6.002 - удаления несовпадающих по длине
частей последовательностей (необходимость этого определяется тем, что в ГенБанке депони-
рованы последовательности разной длины). Полученную прямоугольную матрицу с данными
анализировали в программе MEGA 7.0.14 (Kumar et al., 2016). Использовали три основных
алгоритма анализа: 1) максимальной экономии (maximum parsimony, MP); 2) присоединения
ближайшего соседа (neighbor-joining, NJ); 3) максимального правдоподобия (maximum likelihood,
ML). Для последнего варианта анализа определяли наиболее правдоподобную модель эволюции
с помощью встроенной подпрограммы в MEGA7.0.14. Конечная длина выравнивания по гену
CoxI составила 539 п.н. для всех последовательностей.
Ваучерный образец (№ 14308), содержащий 10 особей эхинококка в 80% этаноле, передали
на хранение в Музей гельминтологических коллекций Центра Паразитологии ФГБУН «Инсти-
тут проблем экологии и эволюции им. А.Н. Северцова Российской академии наук» (Москва).
Выделенная нуклеотидная последовательность депонирована в ГенБанк NCBI под номером
OP897742.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
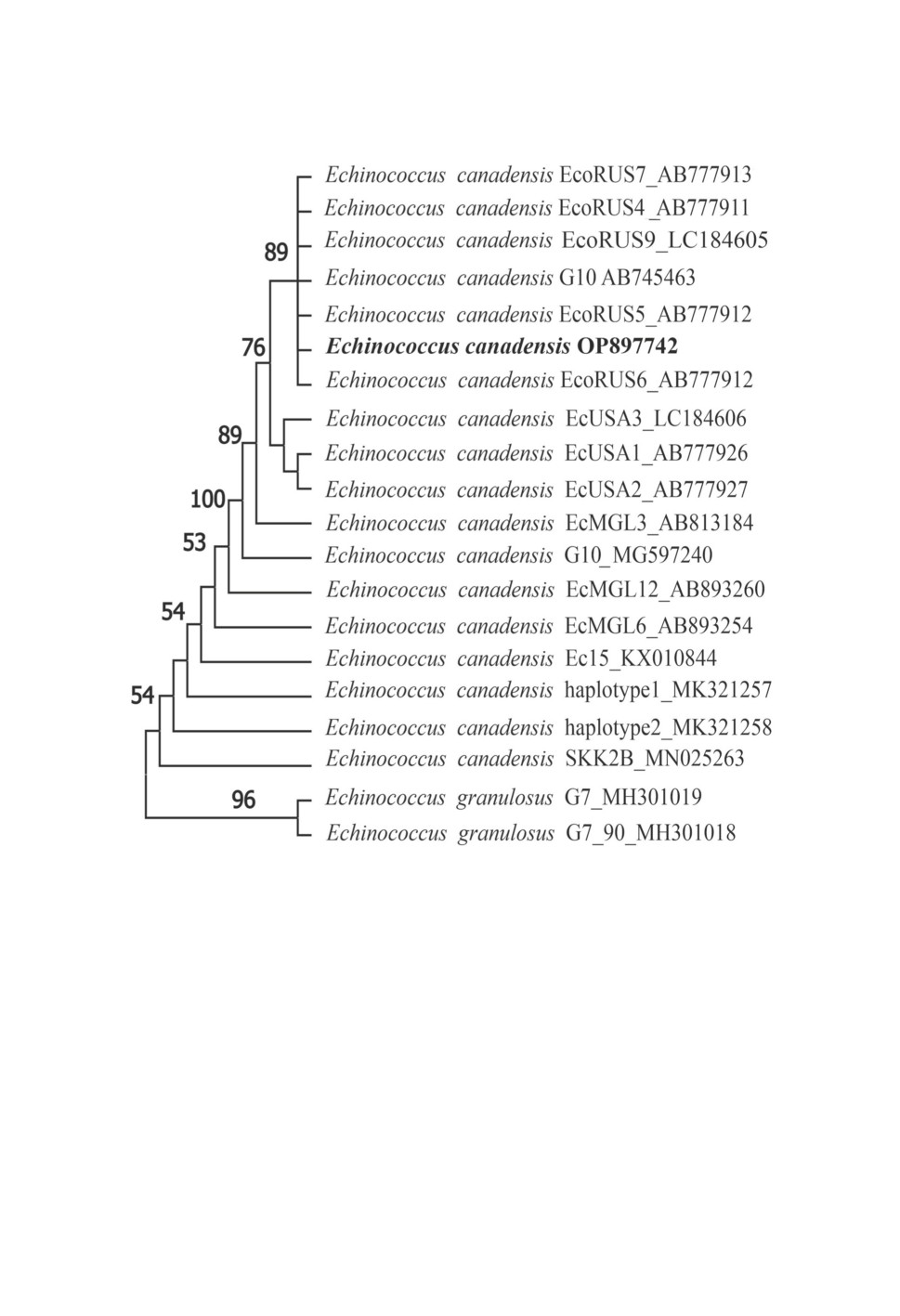
Филогенетический анализ изученных образцов эхинококков представлен на рис. 1.
Полученные нами нуклеотидные последовательности гена CoxI имагинальных эхи-
нококков от волка из Кировской области оказались на 100% идентичны таковым:
1) из личинок эхинокков от лося (Alces alces) из России, депонированных Yanagida,
Chinchuluun и Nakao (2017) под номером LC184605 (Yanagida et al., 2017); 2) из
личинок эхинококков от северного оленя (Rangifer tarandus) из России, депониро-
ванных Yanagida и Nakao (2013) под номером AB777913 (Konyaev et al., 2013); 3) от
взрослых эхинококков от волка из России, депонированных Yanagida и Nakao (2013)
под номером AB777912 (Konyaev et al., 2013); 4) из личинок эхинококков от лося из
России, депонированных Yanagida и Nakao (2013) под номером AB777911 (Konyaev
et al., 2013); 5) из личинок эхинококков от лося из Финляндии, депонированных Nakao
(2013) под номером AB745463 (Nakao et al., 2013a, 2013b).
Изученный нами образец попадает в группу (филогенетическую кладу) форм со
средним уровнем поддержки при анализе методом максимальной экономии (MP) и
40
Рисунок 1. Филогенетические связи образца половозрелого Echinococcus canadensis,
выявленного у волка (Canis lupus) в Кировской области, по результатам анализа частичной
последовательности митохондриального гена цитохромоксидазы I.
Значения bootstrap-поддержки даны в формате MP/NJ/ML (если данная филогенетическая
клада при данном виде анализа не выявляется, это отмечено знаком «-»).
Для ML-анализа использовали модель HKY. Для всех видов анализа для определения
значения bootstrap-поддержки применяли анализ при 500 псевдо-повторах.
Figure 1. Phylogenetic relationships of a sample of adult Echinococcus canadensis,
found in a wolf (Canis lupus) in the Kirov Region, according to the results of analysis
of a partial sequence of the mitochondrial cytochrome c oxidase subunit I gene.
Bootstrap support values are given in MP/NJ/ML format (noted with “-“ if this phylogenetic
clade is not detected in this type of analysis).
Maximum Likelihood (ML) method was used according HKY model with 500 bootstrap replicates.
41
присоединения ближайшего соседа (NJ). При анализе методом максимального прав-
доподобия (ML) эта группа не выявляется. При всех трех методах анализа самую
высокую поддержку (99-100%) показывает группа, объединяющая, как нам представ-
ляется, исключительно изученные формы E. canadensis генотипа G10. Визуальный
анализ полученного выравнивания позволил обнаружить четыре позиции, по кото-
рым выявляются характерные только для этого генотипа замены. Примечательно, что
в выравнивании можно обнаружить и замену по одной из позиций, которая характерна
исключительно для представителей генотипа G10, обнаруженных в России и в Фин-
ляндии (Nakao et al., 2013a, 2013b). Сам по себе генотип G10 приурочен к северным
территориям и таким дефинитивным хозяевам, как волки и собаки.
Несмотря на неразрешенный вопрос о таксономическом статусе E. canadensis,
важно понимать, что исследованные нами эхинококки не относятся к виду E. mul-
tilocularis (син. Alveococcus multilocularis (Leukart, 1863) Abuladse, 1960). Мы особо
подчеркиваем этот момент, поскольку известны случаи смешанной инвазии эхино-
кокков разных видов у волков (Schurer et al., 2014).
ЗАКЛЮЧЕНИЕ
Таким образом, эхинококки, обнаруженные при вскрытии волка в Кировской об-
ласти, относятся к генотипу G10 E. canadensis. А нуклеотидная последовательность
гена CoxI у изученных образцов обладает уникальной для G10 заменой, что позволяет
рассматривать ее как биогеографический маркер.
СПИСОК ЛИТЕРАТУРЫ
Абуладзе К.И. 1964. Основы цестодологии: Тениаты - ленточные гельминты животных и человека и вы-
зываемые ими заболевания (том IV). М., Наука, 530 с. [Abuladze K.I. 1964. Principles of cestodol-
ogy: Taeniata of Animals and Man and Diseases Caused by Them (Vol. IV). Moscow, Nauka, 530 pp.
(In Russian)] (English translation, Israel Program for Scientific Translations, 1970).
Ивашкин В.М., Контримавичус В.Н., Назарова Н.С. 1971. Методы сбора и изучения гельминтов наземных
млекопитающих. М., Наука, 123 с. [Ivashkin V.M., Kontrimavichus V.N., Nazarova N.S. 1971. Methods
for collecting and studying helminths of terrestrial mammals. Moscow, Nauka, 123 pp. (In Russian)]
Скрябин К.И. 1928. Метод полных гельминтологических вскрытий позвоночных, включая человека. М.,
Изд-во МГУ, 45 с. [Skryabin K.I. 1928. Method of complete helminthological dissections of vertebrates,
including humans. Moscow, Publishing House of Moscow State University, 45 pp. (In Russian)]
Beaver P.C., Jung R.C., Cupp E.W. 1984. Clinical Parasitology, 9th ed. Philadelphia, Pennsylvania, Lea & Fe-
biger, 825 pp.
Konyaev S.V., Yanagida T., Nakao M., Ingovatova G.M., Shoykhet Y.N., Bondarev A.Y., Odnokurtsev V.A.,
Loskutova K. S., Lukmanova G. I., Dokuchaev N. E., Spiridonov S., Alshinecky M. V., Sivkova T.N.,
Andreyanov O. N., Abramov S. A., Krivopalov A. V., Karpenko S. V., Lopatina N. V., Dupal T.A., Sako
Y., Ito A. 2013. Genetic diversity of Echinococcus spp. in Russia. Parasitology 140 (13): 1637-1647.
Kumar S., Stecher G., Tamura K. 2016. MEGA7: Molecular evolutionary genetics analysis. Version 7.0 for big-
Littlewood D.T. J., Waeschenbach A., Nikolov P.N. 2008. In search of mitochondrial markers for resolving the
42
phylogeny of cyclophyllidean tapeworms (Platyhelminthes, Cestoda) - A test study with Davaineidae. Acta
Lymbery A.J., Jenkins E.J., Schurer J.M., Thompson R.C.A. 2015. Echinococcus canadensis, E. borealis, and E. in-
Morgulis A., Coulouris G., Raytselis Y., Madden T.L., Agarwala R., Schäffer A.A. 2008. Da-
tabase Indexing for Production MegaBLAST Searches. Bioinformatics 24: 1757-1764.
Nakao M., Lavikainen A., Yanagida T., Ito A. 2013a. Phylogenetic systematics of the genus Echi-
nococcus (Cestoda: Taeniidae). International Journal for Parasitology 43 (12-13): 1017-1029.
Nakao M., Yanagida T., Konyaev S., Lavikainen A., Odnokurtsev V.A., Zaikov V.A., Ito A. 2013b. Mi-
tochondrial phylogeny of the genus Echinococcus (Cestoda: Taeniidae) with emphasis on re-
lationships among Echinococcus canadensis genotypes. Parasitology 140 (13): 1625-1636.
Schurer J., Gesy K., Elkin B., Jenkins E. 2014. Echinococcus multilocularis and Echinococcus canadensis in
wolves from western Canada. Parasitology 141 (2): 159-163. https://doi. org/10.1017/S0031182013001716
Yanagida T., Lavikainen A., Hoberg E.P., Konyaev S., Ito A., Sato M.O., Zaikov V.A., Beckmen K., Na-
kao M. 2017. Specific status of Echinococcus canadensis (Cestoda: Taeniidae) inferred from nu-
clear and mitochondrial gene sequences. International Journal of Parasitology 47 (14): 971-979.
Zhang Z., Schwartz S., Wagner L., Miller W. 2000. A greedy algorithm for aligning DNA sequences. Journal of
MOLECULAR CHARACTERIZATION OF ECHINOCOCCUS CANADENSIS
(CESTODA, TAENIIDAE) IN WOLF FROM THE KIROV REGION
O. A. Loginova, S. E. Spiridonov, O. V. Maslennikova, L. A. Bukina
Keywords: Echinococcus canadensis, G10, wolf, Kirov region
SUMMARY
The molecular characteristics of parasitic tapeworms of the genus Echinococcus were studied.
Helminths were discovered during the autopsy of a wolf (Canis lupus Linnaeus, 1758), hunted in
the Kirov region in October of 2021. Molecular phylogenetic study was performed by analyzing the
sequence of a fragment of the mitochondrial cytochrome c oxidase subunit 1 (COI) gene. It was found
that the obtained helminths belong to the G10 E. canadensis genotype, which is common in wolves
in the northern territories of the Holarctic. Four positions were found for which substitutions charac-
teristic only for this genotype are identified. A substitution was also found for one of the positions,
which is characteristic only for representatives of the G10 genotype found in Russia and Finland.
43