Почвоведение, 2023, № 5, стр. 594-602
Оценка хитинолитической и антибиотической активности актиномицетов Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468
Г. А. Терегулова a, О. Н. Синёва b, Н. Н. Маркелова b, В. С. Садыкова b, Г. В. Уваров a, М. А. Коваленко a, Н. А. Манучарова a, *
a МГУ им. М.В. Ломоносова
119991 Москва, Ленинские горы, 1, Россия
b Научно-исследовательский институт по изысканию новых антибиотиков им. Г.Ф. Гаузе
119021 Москва, ул. Большая Пироговская, 11, стр. 1, Россия
* E-mail: manucharova@mail.ru
Поступила в редакцию 29.09.2022
После доработки 04.01.2023
Принята к публикации 05.01.2023
- EDN: IEEWMZ
- DOI: 10.31857/S0032180X22601207
Аннотация
Проведена оценка антибиотической и хитинолитической активности у актиномицетов, выделенных из дерново-подзолистой почвы (Albic Retisol (Loamic)) Московской области (56°07′15″ N, 37°30′54″ E). На основании культурально-морфологических признаков и анализа гена 16S рРНК выделенные штаммы отнесены к Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468. Оценка антибиотической активности выделенных культур актиномицетов показала, что штаммы проявляют как антибактериальную активность в отношении грамположительных бактерий, так и антифунгальную активность в отношении коллекционных штаммов грибов Sac. cerevisiae ИНА 01042, C. albicans ATCC 14053, F. oxysporum ВКПМ F-148. Способность к синтезу противогрибковых соединений увеличивалась при росте на среде с хитином у штамма Streptomyces avidinii INA 01467, в том числе в отношении фитопатогенного штамма Fusarium oxysporum ВКПМ F-148. С помощью метода ПЦР в реальном времени (Real-Time-PCR) было установлено наличие функциональных генов сhitA, отвечающих за синтез ферментов хитиназ группы А, у исследуемых бактериальных штаммов Streptomyces и Micromonospora. Наибольшее количество гена обнаружено при росте на хитине у штамма Streptomyces avidinii INA 01467, оно достигает порядка 15 × 103 копий/мл. Результаты показали наличие у исследуемых штаммов хитиназной и антибиотической активности, в том числе в отношении фитопатогенных грибов, что позволяет использовать их в технологиях защиты растений и биоремедиации почв.
ВВЕДЕНИЕ
Хитин, являясь структурным полисахаридом членистоногих, кишечнополостных и грибов, широко распространен в наземных и водных экосистемах. По химической структуре хитин – это линейный аминополисахарид, построенный из N-ацетил-2-амино-2-дезокси-D-гликопиранозных звеньев, связанных β-(1-4)-гликозидными связями. Хитин имеет широкий спектр применений в биохимической, пищевой и химической промышленности. Однако вопрос об утилизации отходов промышленных предприятий, связанных с переработкой хитиносодержащих продуктов, по-прежнему актуален. Биоконвертация отходов с помощью микроорганизмов-хитинолитиков является одним из путей решения проблемы сохранения баланса экосистем. Деградация хитина возможна с помощью ферментов – хитиназ, продуцентами которых являются бактерии и грибы. Стоит отметить, что производство хитина в морской биосфере составляет миллиарды тонн, но благодаря процессам биоконверсии, которые осуществляют морские хитинолитические бактерии, содержание хитина в морских отложениях низкое [1, 5, 9, 10, 13]. В почвах активно происходит разложение хитина и других труднодоступных полисахаридов микробными сообществами бактерий и грибов, состав которых зависит от климатических зон. Показано, что роль бактерий в разложении хитина больше при высоких температурах, а грибов – при низких. Важную роль в разложении хитина а почвенных экосистемах играют актинобактерии, у которых обнаружено большое количество генов хитиназы [3, 6, 18].
Почвенные актиномицеты, одновременно со способностью к синтезу антибиотиков, часто содержат гены, кодирующие синтез ферментов, разрушающих труднодоступные соединения, например, азотсодержащий биополимер хитин [6, 18, 17 ]. Хитиназы обнаружены у актиномицетов родов Streptomyces, Nocardia, Actinoplanes, Micromonospora, Microbispora. Разложение актиномицетами природных полимеров, в том числе хитина, связано с функционированием эндо-, экзогидролаз и литических полисахаридных монооксигеназ [1, 6, 17, 19, 20]. Продуценты хитинолитических ферментов часто используются для борьбы с фитопатогенами. В связи с тем, что применение химических фунгицидов наносит серьезный ущерб окружающей среде, биофунгициды рассматриваются как экологически безопасный способ борьбы с фитопатогенами. Кроме того, известно, что хитиназы не только способны к разрушению клеточных стенок фитопатогенных грибов, но и стимулируют рост, развитие растений, повышают их иммунитет [6, 10, 16, 17].
Цель работы – поиск и выделение актиномицетов с полифункциональной активностью (способностью к синтезу антибиотиков и хитинолитических ферментов) из дерново-подзолистой почвы.
ОБЪЕКТЫ И МЕТОДЫ
Образцы дерново-подзолистой почвы (Albic Retisol (Loamic)) отбирали в Московской области (56°07′15″ N, 37°30′54″ E). Для выделения актиномицетов почву высушивали на воздухе при комнатной температуре в течение 14 сут, к 0.1 г образца почвы добавляли 9.9 мл стерильной воды, затем суспензию обрабатывали лимонным соком в концентрации 50% от объема суспензии (рН 2) и инкубировали в течение 30 мин при комнатной температуре. После чего суспензию встряхивали на вортексе в течение 5 мин. Аликвоты почвенных суспензий высевали на модифицированную среду Гаузе 2 (триптон – 3.0 г/л, пептон – 5.0 г/л, глюкоза – 10.0 г/л, NaCl – 5.0 г/л, агар – 20.0 г/л) и культивировали в термостате при температуре 28°С в течение 14 сут. Лимонный сок стерилизовали с использованием бактериальных фильтров d = 0.22 мкм. Выделенные культуры актиномицетов поддерживали на средах Гаузе 2 и овсяном агаре (овсяная мука – 20.0 г/л, агар – 20.0 г/л).
Антибиотическую активность определяли стандартным методом агаровых лунок при глубинном культивировании актиномицетов на средах Гаузе 1, среде с хитином следующего состава:
1. Среда с хитином, г/л: коллоидный хитин – 4, KH2PO4 – 0.3, K2HPO4 – 0.7, MgSO4·7H2O – 0.5, FeSO4·7H2O – 0.01, ZnSO4·7H2O – 0.01, MnC-l2·4H2O – 0.01.
2. Минеральный агар Гаузе 1, г/л: крахмал растворимый – 20.0, К2НРО4 – 0.5, MgSO4 – 0.5, NaC-l – 0.5, KNO3 – 1.0, FeSO4 – 0.01.
В качестве тест-культур использовали следующие микроорганизмы: Staphylococcus aureus INA 00985, Staphylococcus aureus INA 00761 (MRSA), Micrococcus luteus ATCC 9341, Bacillus subtilis ATCC 6633, Escherichia coli ATCC 25922, Pseudomonas aeruginosa ATCC 27853, Saccharomyces cerevisiae ИНА 01042, Candida albicans ATCC 14053, Fusarium oxysporum ВКПМ F-148. Тест-культуры: S. aureus INA 00985, S. aureus INA 00761 (MRSA), M. luteus ATCC 9341, B. subtilis ATCC 6633, P. aeruginosa ATCC 27853 поддерживали на МПА, E. coli ATCC 25922 – на LB-агаре, Sac. cerevisiae ИНА 01042, C. albicans ATCC 14053, F. oxysporum ВКПМ F-148 – на среде Сабуро.
В качестве параметров, подтверждающих разложение хитина исследуемыми штаммами микроорганизмов, использовали показатели активности дыхания и накопление биомассы исследуемыми культурами микроорганизмов. Опыт проводили следующим образом: чистую культуру микроорганизма помещали в пенициллиновые флаконы (объем инокулята составлял 10% от объема среды) с 5 мл стерильной среды и герметично закрывали стерильными резиновыми пробками. Чистые культуры актиномицетов выращивали в жидкой среде, в которой в качестве единственного источника углерода и азота был хитин. Инкубацию проводили динамически – на качалке (180 об./мин) при температуре 28°С. Контролем во всех экспериментах служили культуры во флаконах с голодной минеральной средой, отличающейся от опытной среды отсутствием хитина. Эксперимент проводили в течение 14 сут.
Эмиссию диоксида углерода культурами актиномицетов определяли на газовом хроматографе с детектором по теплопроводности (модель 3700). Длина колонки 3 м, наполнитель Полисорб-1. Скорость потока газа-носителя (Не) 25 мл/мин. Накопление диоксида углерода над образцами оценивали спустя сутки инкубации [4, 10, 15]. Мониторинг интенсивности дыхания проводили в течение 14 сут.
Длину мицелия актиномицетов оценивали с помощью люминесцентно-микроскопического метода. Суспензию обрабатывали на ультразвуковой установке УЗДН-1 (2 мин, сила тока 0.40 А, частота 22 кГц). Суспензию наносили пипеткой на обезжиренное предметное стекло (0.01 мл для определения бактерий) и равномерно распределяли петлей на площади 4 см2 (квадрат 2 × 2). Препараты высушивали на воздухе при комнатной температуре. После фиксации легким нагреванием на пламени газовой горелки препараты окрашивали водным раствором акридина оранжевого, разведение 1 : 10 000; 15 мин. Окрашенные препараты высушивали при комнатной температуре и просматривали на люминесцентном микроскопе Zeizz Mikroskop Axioskop 2 plus (Германия) (светофильтр Filterset 09). Количество микробных клеток, содержащихся в 1 г почвы, вычисляли по формуле:
(1)
$N = {{{{S}_{1}}an} \mathord{\left/ {\vphantom {{{{S}_{1}}an} {v{{S}_{2}}c}}} \right. \kern-0em} {v{{S}_{2}}c}},$Биомассу учитывали расчетным методом. Удельную массу микроорганизмов принимали равной 1 г/см3, а содержание воды в клетках – 80%. Показатель сухой биомассы для 1 м мицелия актиномицетов диаметром 0.5 мкм – 3.9 × 10–8 г [2].
Методы идентификации чистых культур. Культуральные признаки определяли при росте культур на плотных питательных средах Гаузе 1, Гаузе 2, овсяном агаре, используя определитель Bergey’s Manual of Systematic Bacteriology [7].
Морфологию воздушного и субстратного мицелия определяли с использованием микроскопа Olimpus BX-41 (Shibuya-ku Tokyo, Япония).
Для изучения геносистематических признаков проводили сравнительный анализ нуклеотидных последовательностей гена 16S rRNA. Для выделения ДНК актиномицетов использовали набор Power Soil DNA Isolation Kit (MO BIO Laboratories Inc., Carlsbad, CA, США), согласно инструкции производителя. Амплификацию проводили на автоматическом амплификаторе 2720 Thermal Cycler (Applied Biosystems, США), с набором реагентов фирмы Thermo Fisher Scientific (США) и праймеров: 27F (5'-AGAGTTTGATCMTGGCTCAG-3'), 1492R (5'-TACGGYTACCTTGTTACGACTT-3') фирмы Синтол (Россия). Секвенирование проводили на автоматическом капиллярном секвенаторе 3500 Genetic Analyser (Applied Biosystems, Beverly, MA, США) с использованием реагентов BigDye Terminator v3 Cycle Sequencing Kit (Applied Biosystems, США).
Филогенетический анализ последовательностей гена 16S rRNA выделенных штаммов проводили с использованием программы BLAST сервера NCBI (www.ncbi.nlm.nih.gov/blast/).
Определение наличия генов chitA. Фрагмент бактериального хитиназного гена chitA (около 400 п.н.) амплифицировали с помощью “вложенного” ПЦР согласно протоколу [21]: на первом этапе реакции применяли пару CODEHOP-праймеров GA1F-GAIR, на втором этапе – пару консенсусных праймеров GASQF-GASQR с GC-клампом на 5'-конце:
GA1F 5' – CGTCGACATCGACTGGGARTDBCC – 3' GA1R 5' – ACGCCGGTCCAGCCNCKNCCRTA – 3' GASQF 5' – CGTCGACATCGACTGGGA – 3'GASQR 5' – ACGCCGGTCCAGCC – 3' [21].
Температурно-временной профиль ПЦР: первый цикл: 94°С – 5 мин; последующие 35 циклов: 94°С – 1 мин, 57°С – 10 с и 72°С – 1 мин; После окончания последнего цикла смеси дополнительно выдерживали 10 мин при 72°С для достройки незавершенных цепей.
Метод ПЦР в реальном времени применяли для количественного анализа числа копий ДНК представителей бактерий, содержащих функциональные гены chitA, кодирующие синтез хитиназ. Измерение проводили на детектирующем амплификаторе DТLite4 ДНК-технология в динамике на определенные сутки роста культур. Полученные результаты обрабатывали с использованием пакета программы Real time_PCR. Данный детектирующий амплификатор DТLite4 совмещает в себе функции программируемого термоциклера и оптической системы, позволяющей регистрировать флуоресценцию реакционной смеси в пробирках в ходе полимеразной цепной реакции. Использовали интеркалирующий краситель SYBR® Green и красители, связанные с праймерами (Light cyaler – два зонда, связывающие ДНК-мишень на небольшом расстоянии друг от друга). Реакционную смесь готовили из препарата Super Mix Eva Green Biorad (концентрированный буфер с дезоксирибонуклеотидами, полимеразой Sso7d-fusion, MgCl2, красителем EvaGreen и стабилизаторами). Прибор калибровали по зависимости интенсивности флуоресценции от логарифма концентрации ДНК стандартных растворов. Количество исследуемой ДНК выражали в абсолютных или относительных единицах. Каждое количественное определение матрицы ДНК требует трех стандартов и отрицательного контроля (образца без матрицы ДНК).
В качестве стандарта использовали культуру Streptomyces violaceus [sec. Roseus: ser. Roseoviolaceus], способную использовать хитин в качестве единственного источника углерода и азота в среде [21]. Последовательности праймеров, используемых для выявления представителей, обладающих функциональным геном chitA, представлены в табл. 1.
Таблица 1.
Последовательности праймеров для функционального ген chitA
| Ген | Фермент | Нуклеотидная последовательность праймера (5'-3') | Размер целевого участка (bp) | Ссылка |
|---|---|---|---|---|
| chitA | Хитиназа семейства 18 гликозил гидролаз | ChiA_F2 (50-CGT GGA CAT CGA CTG GGA RTW YCC-30) 50 end labeled with 50 6-FAM and Reverse primer ChiA_R2 (50-CCC AGG CGC CGT AGA RRT CRT ARS WCA-30) | 400 | [20] |
Применяли следующий протокол для амплификации: 1 цикл: 94.0°C – 5 мин; 30 циклов: 94.0°C 1 мин; 60°C – 1 мин; 72°C – 1 мин; 1 цикл: 72°C – 3 мин; 1 цикл – 4°C–охлаждение [14].
Статистическую обработку данных проводили с помощью программы Statistica 6.0. Образцы анализировали в 5-кратной повторности.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
Изучение филогенетического положения выделенных штаммов актиномицетов на основании культурально-морфологических признаков и анализа генов 16S рРНК показало, что исследуемые штаммы относятся к Streptomyces avidinii (процент совпадения 100%) и Micromonospora aurantiaca (доля совпадения 97.99%). Культуры были депонированы в коллекцию микроорганизмов ФГБНУ “Научно-исследовательского института по изысканию новых антибиотиков имени Г.Ф. Гаузе” под следующими номерами: Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468.
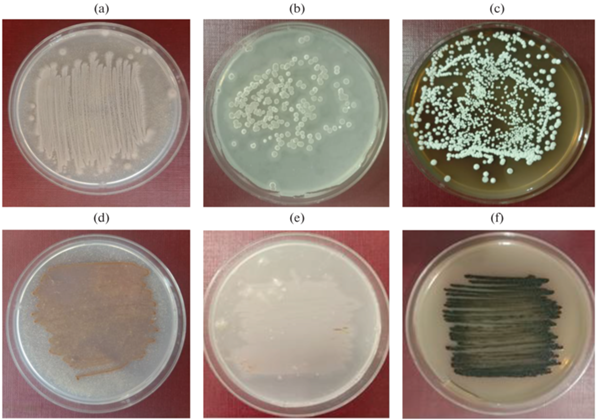
Предварительно была проведена оценка способности роста культур актиномицетов на плотных средах с хитином, Гаузе 1 и Гаузе 2 (рис. 1), которая показала, что штаммы способны к активному росту на данных средах.
Рис. 1.
Рост культур Streptomyces avidinii INA 01467 (а, b, c) и Micromonospora aurantiaca INA 01468 (d, e, f) на плотных питательных средах: с хитином (a, d) Гаузе 1 (b, e), Гаузе 2 (c, f).
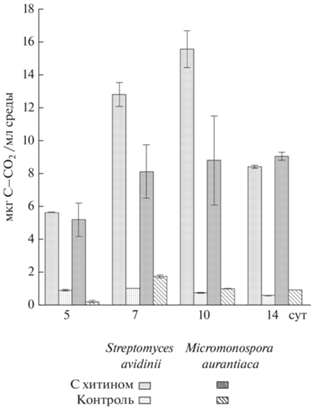
В качестве параметров, подтверждающих разложение хитина исследуемыми штаммами микроорганизмов, использовали показатели активности дыхания и накопление биомассы исследуемыми культурами микроорганизмов при их росте на селективной среде, в качестве единственного источника углерода и азота в которой был хитин.
Динамика эмиссии СО2 штаммами Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468, растущими на среде с хитином. При рассмотрении динамики эмиссии СО2 штаммами Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468, растущими на среде с хитином, отмечены следующие особенности. Эмиссия диоксида углерода на среде с хитином была больше по сравнению с контролем для обоих штаммов (рис. 2). Для штамма Streptomyces avidinii INA 01467 эмиссия СО2 на среде с хитином была максимальной на 10 сут эксперимента и достигала 15.57 мкг/мл среды за сутки, что на порядок превышало контрольные варианты. Динамика эмиссии диоксида углерода актиномицетом рода Micromonospora имела схожие закономерности, однако максимальные значения эмиссии на среде с хитином были меньше и достигали 8.80 мкг/мл среды за сутки, что в 4–5 раз превышало контрольные варианты (1.77 мкг/мл среды за сутки). Способность микромоноспор к деградации хитина отмечалась и другими авторами [11, 21].
Рис. 2.
Динамика эмиссии СО2 штаммов Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468 на среде с хитином и контроль.
Динамика накопления биомассы штаммами Micromonospora aurantiaca INA 01468 и Streptomyces avidinii INA 01467 на среде с хитином. Исследуемые штаммы активно накапливали биомассу при росте на хитине (рис. 3). Максимальные значения для Micromonospora aurantiaca INA 01468 составили 10.9 мкг/мл на 7 сут культивирования, что в 3 раза больше по сравнению с контролем.
Рис. 3.
Динамика накопления биомассы штаммами Streptomyces avidinii INA 01467 (99 л) и Micromonospora aurantiaca INA 01468 (69 л) на разных питательных средах.
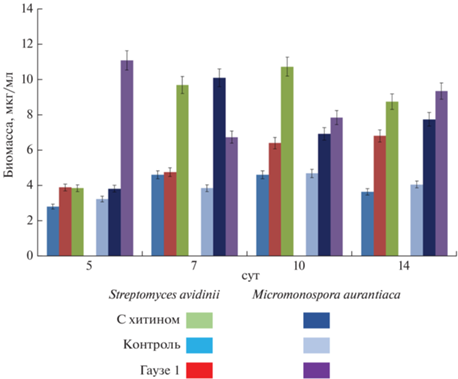
Streptomyces avidinii INA 01467 показал максимальную продуктивность на среде с хитином на 14 сут культивирования и по сравнению с ростом на контрольной среде превысил ее значения в 2 раза.
Исследуемые штаммы актиномицетов активно накапливали биомассу на специализированной для них среде Гаузе 1. При инкубировании на этой среде наблюдали максимальный выход биомассы для обоих штаммов. Таким образом, исследуемые штаммы Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468 при росте на среде с хитином активно накапливали биомассу и выделяли CО2.
Таблица 2.
Антибиотическая активность Micromonospora aurantiaca INA 01468 при культивировании на среде с хитином и Гаузе 1 (n = 5)
| Тест-организм | Зоны подавления роста, мм | |||
|---|---|---|---|---|
| сутки роста | ||||
| 5 | 7 | 10 | 14 | |
| Среда с хитином | ||||
| Staphylococcus aureus INA 00985 | 0 | 0 | 0 | 0 |
| Staphylococcus aureus INA 00761 (MRSA) | 0 | 0 | 0 | 0 |
| Micrococcus luteus ATCC 9341 | 0 | 0 | 0 | 0 |
| Bacillus subtilis ATCC 6633 | 0 | 0 | 0 | 0 |
| Escherichia coli ATCC 25922 | 0 | 0 | 0 | 0 |
| Pseudomonas aeruginosa ATCC 27853 | 0 | 0 | 0 | 0 |
| Saccharomyces cerevisiae ИНА 01042 | 20.4 ± 0.3 | 15.1 ± 0.4 | 15.2 ± 0.4 | 15.2 ± 0.4 |
| Candida albicans ATCC 14053 | 0 | 0 | 0 | 0 |
| Fusarium oxysporum ВКПМ F0148 | 12 ± 0.4 | 12.1 ± 0.2 | 12.1 ± 0.2 | 12 ± 0.4 |
| Гаузе 1 | ||||
| Staphylococcus aureus INA 00985 | 0 | 0 | 0 | 0 |
| Staphylococcus aureus INA 00761 (MRSA) | 0 | 0 | 0 | 0 |
| Micrococcus luteus ATCC 9341 | 0 | 0 | 0 | 0 |
| Bacillus subtilis ATCC 6633 | 0 | 0 | 0 | 0 |
| Escherichia coli ATCC 25922 | 0 | 0 | 0 | 0 |
| Pseudomonas aeruginosa ATCC 27853 | 0 | 0 | 0 | 0 |
| Saccharomyces cerevisiae ИНА 01042 | 20.3 ± 0.2 | 15.1 ± 0.4 | 15.1 ± 0.4 | 14.9 ± 0.4 |
| Candida albicans ATCC 14053 | 0 | 0 | 0 | 0 |
| Fusarium oxysporum ВКПМ F0148 | 12 ± 0.4 | 12 ± 0.4 | 12.1 ± 0.2 | 12.1 ± 0.2 |
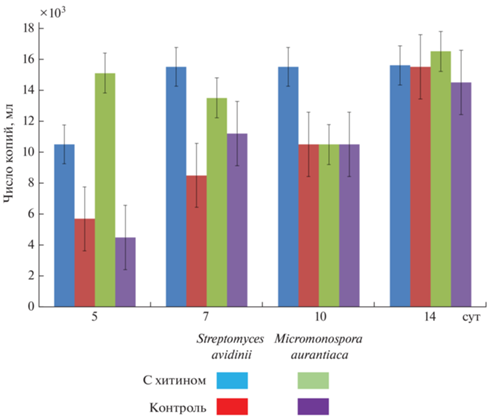
Детекция хитиназного гена сhitA. По результатам определения наличия функциональных генов сhitA, отвечающих за синтез ферментов хитиназ группы А, у исследуемых бактериальных штаммов Streptomyces и Micromonospora выявлены следующие закономерности. Наибольшее количество данного гена обнаруживается на 7, 10 и 14 сут при росте на хитине у штамма Streptomyces avidinii INA 01467, порядка 15 × 103 копий в мл (рис. 4). На среде Гаузе 1 на 5, 7 и 10 сут тем же штаммом образовывалось значительно меньшее количество числа копий гена сhitA, но к 14 сут число копий возрастало и достигло значений, схожих с показателями при росте на хитине.
Рис. 4.
Динамика численности хитиназных генов (ChitA), отвечающих за синтез ферментов хитиназ группы А штаммами Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468.
Количественное определение генов сhitA, кодирующих хитиназу семейства 18 гликозил гидролаз, в природных экосистемах впервые было определено с применением QC-PCR в озерных осадках о. Ардли (Антарктика) [22], где оно составило от 2.5 × 103 до 4.2 × 107 копий chitA/г в зависимости от типа исследуемого образца. Относительно недавно с помощью ПЦР в реальном времени (Real-Time-PCR) было оценено количество генов chitA в ризосфере разных сортов картофеля, выращиваемых в теплице и на поле [12].
Штамм Micromonospora aurantiaca INA 01468 продуцировал наибольшее количество копий chitA на 5 и 14 сут. Показатели этого штамма на среде Гаузе 1 были меньше и достигали 14 × 103 копий/мл среды.
Таблица 3.
Антибиотическая активность Streptomyces avidinii INA 01467 при культивировании на среде с хитином и Гаузе 1 (n = 5)
| Тест-организм | Зоны подавления роста, мм | |||
|---|---|---|---|---|
| сутки роста | ||||
| 5 | 7 | 10 | 14 | |
| Среда с хитином | ||||
| Staphylococcus aureus INA 00985 | 13.25 ± 0.6 | 15.2 ± 0.4 | 18.1 ± 0.2 | 0 |
| Staphylococcus aureus INA 00761 (MRSA) | 16.3 ± 0.3 | 20.4 ± 0.3 | 18.1 ± 0.2 | 0 |
| Micrococcus luteus ATCC 9341 | 20.3 ± 0.2 | 20.2 ± 0.3 | 23.9 ± 0.3 | 30.1 ± 2 |
| Bacillus subtilis ATCC 6633 | 0 | 20.4 ± 0.3 | 0 | 0 |
| Escherichia coli ATCC 25922 | 0 | 0 | 0 | 0 |
| Pseudomonas aeruginosa ATCC 27853 | 0 | 0 | 0 | 0 |
| Saccharomyces cerevisiae ИНА 01042 | 15.1 ± 0.4 | 14.5 ± 0.3 | 12 ± 0.4 | 12.1 ± 0.2 |
| Candida albicans ATCC 14053 | 10.3 ± 0.3 | 10.3 ± 0.3 | 11.2 ± 0.3 | 11.1 ± 0.3 |
| Fusarium oxysporum ВКПМ F0148 | 15.1 ± 0.4 | 15.1 ± 0.4 | 13.25 ± 0.6 | 13.35 ± 0.4 |
| Гаузе 1 | ||||
| Staphylococcus aureus INA 00985 | 0 | 13.1 ± 0.8 | 13.5 ± 0.6 | 12.1 ± 0.2 |
| Staphylococcus aureus INA 00761 (MRSA) | 0 | 13.5 ± 0.4 | 13.6 ± 0.6 | 14.5 ± 0.3 |
| Micrococcus luteus ATCC 9341 | 0 | 10.3 ± 0.3 | 15.2 ± 0.4 | 20.4 ± 0.3 |
| Bacillus subtilis ATCC 6633 | 0 | 0 | 0 | 15.1 ± 0.4 |
| Escherichia coli ATCC 25922 | 0 | 0 | 0 | 0 |
| Pseudomonas aeruginosa ATCC 27853 | 0 | 0 | 0 | 0 |
| Saccharomyces cerevisiae ИНА 01042 | 0 | 15.1 ± 0.4 | 0 | 0 |
| Candida albicans ATCC 14053 | 0 | 10.5 ± 0.3 | 11.2 ± 0.3 | 13.3 ± 0.6 |
| Fusarium oxysporum ВКПМ F0148 | 0 | 10.5 ± 0.3 | 12 ± 0.4 | 15.1 ± 0.4 |
ЗАКЛЮЧЕНИЕ
Выделенные штаммы актиномицетов Streptomyces avidinii INA 01467 и Micromonospora aurantiaca INA 01468 обладают одновременно хитинолитической и антибиотической активностью. Оценка антибиотической активности выделенных культур актиномицетов показала, что штаммы проявляют антибактериальную активность в отношении грамположительных бактерий, так и антифунгальную активность в отношении коллекционных штаммов грибов Sac. cerevisiae ИНА 01042, C. albicans ATCC 14053, F. oxysporum ВКПМ F-148. Способность к синтезу противогрибковых соединений увеличивалась при росте на среде с хитином у штамма Streptomyces avidinii INA 01467, в том числе в отношении фитопатогенного штамма F. oxysporum ВКПМ F-148. Выделенные штаммы могут быть использованы в технологиях защиты растений и биоремедиации почв. Способность актиномицетов-хитинолитиков к образованию антибиотиков подтверждает экологическую значимость антибиотиков для самих продуцентов.
Список литературы
Варламов В.П., Ильина А.В., Шагдарова Б.Ц., Луньков А.П., Мысякина И.С. Хитин/хитозан и его производные // Успехи биологической химии. 2020. Т. 60. С. 317–368. https://doi.org/10.1134/S0006297920140084
Кожевин П.А. Микробные популяции в природе. М.: Изд-во Моск. ун-та, 1989. 175 с.
Манучарова Н.А., Власенко А.Н., Менько Е.В., Звягинцев Д.Г. Специфика хитинолитического микробного комплекса в почвах, инкубируемых при различных температурах // Микробиология. 2011. Т. 80. № 2. С. 219–229. https://doi.org/10.1134/S002626171102010X
Поздняков Л.А., Степанов А.Л., Гасанов М.Э., Семенов М.В., Якименко О.С., Суада А.К., Рай А.Н., Щеголькова Н.М. Влияние лигногумата на биологическую активность почвы о. Бали, Индонезия // Почвоведение. 2020. № 5. С. 601–609. https://doi.org/10.31857/S0032180X20050111
Aly M.M., Sediq A.N., Baghdadi A.M., Amasha R.H. Chitin and chitinases, production, characterization and applications // IOSR J. Pharm. Biol. Sci. 2019. V. 14. P. 36043. https://doi.org/10.9790/3008-1402013643
Bai Y., Eijsink V.G., Kielak A.M., van Veen J.A., de Boer W. Genomic comparison of chitinolytic enzyme systems from terrestrial and aquatic bacteria // Environ. Microbiol. 2016. V. 18. P. 38–49. https://doi.org/10.1111/1462-2920.12545
Bergey’s Manual of Determinative Bacteriology / Eds. J.A. Holt et al. Baltimore etc: Williams and Wilkins, 2012. V. 5. The Actinobacteria. https://doi.org/10.1002/9781118960608
Bhattacharya D., Nagpure A., Gupta R.K. Bacterial chitinases: properties and potential // Crit. Rev. Biotechnol. 2007. V. 27. P. 21028. https://doi.org/10.1080/07388550601168223
Brzezinska M.S., Jankiewicz U., Burkowska A., Walczak M. Chitinolytic microorganisms and their possible application in environmental protection // Curr. Microbiol. 2014. V. 68. P. 71–81. https://doi.org/10.1007/s00284-013-0440-4
Dahiya N., Tewari R., Hoondal G.S. Biotechnological aspects of chitinolytic enzymes: a review // Appl. Microbiol. Biotechnol. 2006. V. 71. P. 7730782. https://doi.org/10.1007/s00253-005-0183-7
Golovchenko A.V., Gracheva T.A., Lypcan V.A., Dobrovol’skaya T.G., Manucharova N.A. Actinomycete Complex es in Eutrophic Peatlands // Eurasian Soil Science. 2022. T. 55. № 8. P. 1064–1073. https://doi.org/10.1134/S1064229322080026
Gschwendtner S., Reichmann M., Müller M., Radl V., Munch J.C., Schloter M. Abundance of bacterial genes encoding for proteases and chitinases in the rhizosphere of three different potato cultivars // Biol. Fertil. Soils. 2010. V. 46. P. 6490652. https://doi.org/10.1007/s00374-010-0460-1
Han Y., Yang B., Zhang F., Miao X., Li Z. Characterization of antifungal chitinase from marine Streptomyces sp. DA11 associated with South China sea sponge Craniella Australiensis // Mar. Biotechnol. 2009. V. 11. P. 132–140. https://doi.org/10.1007/s10126-008-9126-5
Hobel C.F.V., Hreggvidsson G.O., Marteinsson V.T., Bahrani Mougeot F., Einarsson J.M., Kristjansson J.K. Cloning, expression, and characterization of a highly thermostable family 18 chitinase from Rhodothermus marinus // Extremophiles. 2005. V 9. № 1. P. 53064. https://doi.org/10.1007/s00792-004-0422-3
Kashyap D.R., Vohra P.K., Chopra S., Tewari R. Application of pectinases in the commercial sector: a review// Bioresour. Technol. 2001. V. 77. P. 2150237. https://doi.org/10.1016/S0960-8524(00)00118-8
Kumar M., Amandeep Brar A., Yadav M., Chawade A., Vivekanand V., Nidhi Pareek N. Chitinases-potential candidates for enhanced plant resistance towards fungal pathogens // Agricult. 2018. V. 8. № 88. https://doi.org/10.3390/agriculture8070088
Lacombe-Harvey M.E., Brzezinski R., Beaulieu C. Chitinolytic functions in actinobacteria: ecology, enzymes, and evolution // Appl. Microbiol. Biotechnol. 2018. V. 102. P. 7219–7230. https://doi.org/10.1007/s00253-018-9149-4
Manucharova N.A. The microbial destruction of chitin, pectin, and cellulose in soils // Eurasian Soil Science. 2009. V. 42. № 13. P. 1526–1532. https://doi.org/10.1134/S1064229309130146
Nawani N.N., Kapadnis B.D., Das A.D., Rao A.S., Mahajan S.K. Purification and characterization of a thermophilic and acidophilic chitinase from Microbispora sp. V2 // J. Appl. Microbiol. 2002. V. 93. P. 965–975. https://doi.org/10.1046/j.1365-2672.2002.01766.x
Wang Q., Duan B., Yang R., Zhao Y., Zhang L. Screening and identification of chitinolytic actinomycetes and study on the inhibitory activity against Turfgrass Root rot disease fungi // J. Biosci. Medic. 2015. V. 3. P. 56065. https://doi.org/10.4236/jbm.2015.33009
Williamson N., Brian, P., Wellington E.M. Molecular detection of bacterial and streptomycete chitinase in the environment // Antonie Van Leeuwenhoek. 2000. V. 78. P. 315–321. https://doi.org/10.1023/A:1010225909148
Xiao X., Yin X., Lin J., Sun L., You Z., Wang P., Wang F. Chitinase Genes in Lake Sediments of Ardley Island, Antarctica // Appl. Environ. Microbiol. 2005. V. 71. P. 790407909. https://doi.org/10.1128/AEM.71.12.7904-7909.2005
Дополнительные материалы отсутствуют.


