Биология моря, 2021, T. 47, № 3, стр. 184-195
Генетический анализ морского блюдечка Lottia cf. borealis (Patellogastropoda: Lottiidae) из дальневосточных морей России
С. Н. Шарина 1, *, В. В. Маляр 1, А. В. Чернышев 1
1 Национальный научный центр морской биологии им. А.В. Жирмунского ДВО РАН
690041 Владивосток, Россия
* E-mail: sharina.svetlana@gmail.com
Поступила в редакцию 30.06.2020
После доработки 17.12.2020
Принята к публикации 18.12.2020
Аннотация
Проведен генетический анализ широко распространенной в дальневосточных морях России гастроподы морское блюдечко Lottia cf. borealis. На основе трех генетических маркеров (COI, 16S, 12S) показано, что этот вид не обладает выраженной генетической структурой в исследованной части ареала (южное побережье Приморского края, побережья о-ва Сахалин и южных Курильских островов). Филогенетический анализ показал, что Lottia cf. borealis является сестринским таксоном по отношению к L. paradigitalis (Fritchman, 1960).
Одна из наиболее древних групп ныне живущих гастропод – морские блюдечки подкласса Patellogastropoda (Lindberg, 1988; Harasewych, McArthur, 2000; Nakano, Ozawa, 2007; Uribe et al., 2019) широко распространены в Мировом океане и обитают преимущественно на мелководье. Морские блюдечки заселяют разные субстраты и играют важную роль в морской экосистеме (Lindberg, 2008; Nakano et al., 2009; González-Wevar et al., 2019). Пателлогастроподы широко используются как модельные организмы в эволюционных исследованиях (Giesel, 1970; Hockey et al., 1987; Byers, 1989; Espoz, Castilla, 2000), при изучении экологии, эмбриологии, популяционной генетики и биогеографии (Abbott et al., 1968; Hodgson et al., 1996; Koufopanou et al., 1999).
Lottia Gray, 1833 (= Collisella Dall, 1871) – самый крупный по числу видов род пателлогастропод, который включает 66 валидных видов (http://www.marinespecies.org/), распространенных главным образом в бореальных и субтропических широтах Тихого океана. Первая ревизия видов рода Lottia из дальневосточных морей России была проведена Москалевым (1970). В таксономической ревизии комплекса видов “Collisella heroldi” Японии (Sasaki, Okutani, 1994) были использованы признаки раковины (прежде всего ее скульптуры), радулы и челюстей, что было принято и при ревизии видов рода Lottia из дальневосточных морей России (Чернышев, Чернова, 2004, 2005, 2007). Однако у морских блюдечек многие признаки раковины (например, форма и окраска) крайне вариабельны, а строение радулы не может быть использовано для идентификации близких видов. Вероятно, поэтому применение традиционных морфологических подходов часто приводит к таксономическим ошибкам (Harasewych, McArthur, 2000; Nakano, Spencer, 2007; Johnson et al., 2008; Nakano, Sasaki, 2011; Yu et al., 2014; Sharina et al., 2016). Вместе с тем относительно простая по морфологическим признакам анатомия морских блюдечек может быть причиной переоценки фактического разнообразия группы (Lindberg, 2008; González-Wevar et al., 2019) или, наоборот, невозможности разграничения криптических видов (Simison, Lindberg, 2003; Crummett, Eernisse, 2007; Nakano, Ozawa, 2007; Evans, Paulay, 2012; Nakayama et al., 2017).
В последние два десятилетия для выяснения филогенетических отношений (Koufopanou et al., 1999; Nakano, Ozawa, 2004, 2007; Goldstien et al., 2006), идентификации видов (Hebert et al., 2003; Yu et al., 2014), выявления криптических видов (Simison, Lindberg, 1999, 2003; Nakano, Ozawa, 2005; Nakano, Spencer, 2007; Johnson et al., 2008; Nakano et al., 2009), установления границ (Nieukerken et al., 2012), а также для изучения популяционной структуры видов активно используются молекулярно-генетические методы. К тому же молекулярные маркеры позволяют отличить межвидовые морфологические различия от высокой внутривидовой изменчивости и фенотипической пластичности (Simison, Lindberg, 1999; Harasewych, McArthur, 2000; Nakano, Sasaki, 2011; Sharina et al., 2016; González-Wevar et al., 2019).
Однако, несмотря на широкое использование молекулярных маркеров, в настоящее время отсутствуют данные, позволяющие оценить генетическое разнообразие и определить границы видов рода Lottia в дальневосточных морях России. Цель нашего исследования – анализ генетического разнообразия и родственных связей широко распространенного и вариабельного вида Lottia cf. borealis с помощью молекулярных маркеров мтДНК (COI, 16S, 12S). Выбор вида обусловлен тем, что его таксономический статус до сих пор не ясен. В литературе вид известен под названиями Collisella radiata (Москалев, 1970; Голиков, Кусакин, 1978), Collisella borealis (см.: Lindberg, 1982), Lottia borealis (см.: Lindberg, 1986), Lottia cf. borealis (см.: Sasaki, 1999), Lottia paradigitalis (см.: Simison, Lindberg, 2003) и Lottia persona (Чернышев, 2004; Чернышев, Чернова, 2005, 2007). Установить приоритетное название лишь с помощью морфологических методов не представляется возможным.
МАТЕРИАЛ И МЕТОДИКА
Для изучения биоразнообразия морского блюдечка Lottia cf. borealis использовали 52 особи из 13 географических локаций Дальнего Востока России (рис. 1, табл. 1). Раковины и ткани образцов хранятся в Лаборатории генетики Национального научного центра морской биологии ДВО РАН. Препараты ДНК получены из фиксированных 96% этанолом тканей головы и ноги животных с использованием набора Genomic DNA Purification Kit (Thermo Fisher Scientific, США).
Рис. 1.
Карта-схема мест сбора морского блюдечка Lottia cf. borealis в Охотском море (а) и на материковом побережье Японского моря (б). Условные обозначения: 1 – юго-восточное побережье о-ва Сахалин (Soh); 2 – зал. Анива (An1); 3 – о-в Итуруп (It); 4 – мыс Глушевского, о-в Зеленый (KurGl); 5 – б. Горобец, о-в Шикотан (KurGor); 6 – б. Широкая, о-в Юрий (KurSh); 7 – о-в Кунашир (KurVal); 8 – Амурский залив (Am2); 9, 10 – Уссурийский залив (T, Uss); 11 – о-в Карамзина (Kar); 12 – зал. Восток (V); 13 – б. Киевка (K). В скобках указано название выборки.
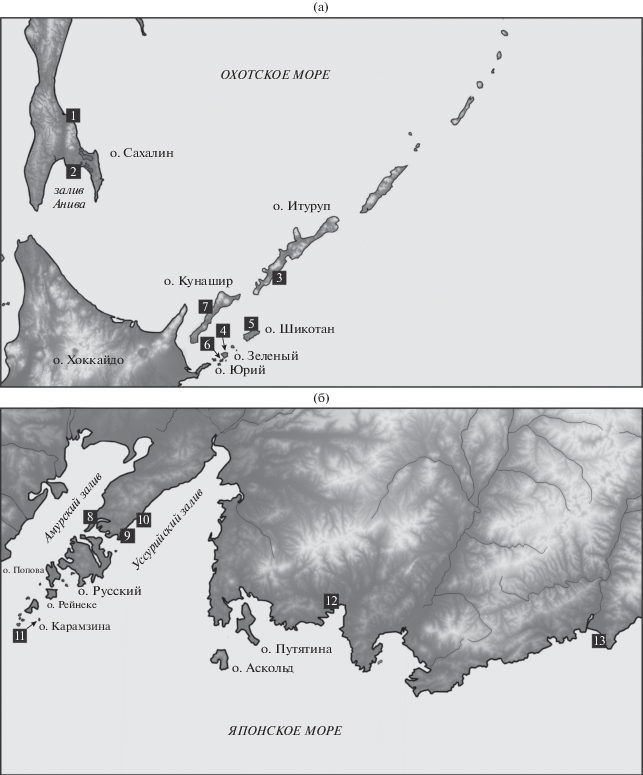
Таблица 1.
Места сбора и номера доступа GenBank NCBI для особей L. cf. borealis, использованных в данном анализе
| Номер пробы | Место сбора | COI | 16S | 12S |
|---|---|---|---|---|
| 17−15.1 Soh | О-в Сахалин, Охотское море | MT678937 | MT677798 | MT677751 |
| 17−15.2 Soh | О-в Сахалин, Охотское море | MT678938 | MT677799 | MT677752 |
| 16−15.1 Soh | О-в Сахалин, Охотское море | MT678939 | MT677800 | MT677753 |
| 16−15.3 Soh | О-в Сахалин, Охотское море | MT678940 | MT677801 | MT677754 |
| 16−15.4 Soh | О-в Сахалин, Охотское море | MT678941 | MT677802 | MT677755 |
| 16−15.5 Soh | О-в Сахалин, Охотское море | MT678942 | MT677803 | MT677756 |
| 17−15.3 Soh | О-в Сахалин, Охотское море | MT678943 | MT677804 | − |
| 18−15 Soh | О-в Сахалин, Охотское море | MT678944 | MT677805 | MT677757 |
| 19−15 Soh | О-в Сахалин, Охотское море | MT678945 | MT677806 | MT677758 |
| 20−15 Soh | О-в Сахалин, Охотское море | MT678946 | MT677807 | − |
| 16−15.2 Soh | О-в Сахалин, Охотское море | MT678947 | MT677808 | MT677759 |
| 17−15.4 Soh | О-в Сахалин, Охотское море | MT678948 | MT677809 | MT677760 |
| 17−15.5 Soh | О-в Сахалин, Охотское море | MT678949 | MT677810 | MT677761 |
| Lottia An1 | Мыс Юнона, зал. Анива, о-в Сахалин, Охотское море | MT678950 | MT677811 | MT677762 |
| 25−28.01.12 It | О-в Итуруп, Курильские острова, Охотское море | MT678951 | MT677812 | MT677763 |
| 17 KurGl | Мыс Глушевского, о-в Зеленый, Курильские острова, Охотское море | MT678952 | MT677813 | MT677764 |
| 30 KurGor | Б. Горобец, о-в Шикотан, Курильские острова, Охотское море | MT678953 | MT677814 | MT677765 |
| 10 KurGor | Б. Горобец, о-в Шикотан, Курильские острова, Охотское море | MT678954 | MT677815 | MT677766 |
| 11 KurGor | Б. Горобец, о-в Шикотан, Курильские острова, Охотское море | MT678955 | MT677816 | MT677767 |
| 27 KurGor | Б. Горобец, о-в Шикотан, Курильские острова, Охотское море | MT678956 | MT677817 | MT677768 |
| 28 KurGor | Б. Горобец, о-в Шикотан, Курильские острова, Охотское море | MT678957 | MT677818 | MT677769 |
| 7 KurSh | Б. Широкая, о-в Юрий, Курильские острова, Охотское море | MT678958 | MT677819 | MT677770 |
| 23 KurVal | О-в Кунашир, Курильские острова, Охотское море | MT678959 | MT677820 | MT677771 |
| 31 KurVal | О-в Кунашир, Курильские острова, Охотское море | MT678960 | MT677821 | MT677772 |
| 24 KurVal | О-в Кунашир, Курильские острова, Охотское море | MT678961 | MT677822 | MT677773 |
| II−17 Am2 | Спортивная гавань, Амурский залив, зал. Петра Великого, Японское море | MT678962 | MT677823 | MT677774 |
| III−28 Am2 | Спортивная гавань, Амурский залив, зал. Петра Великого, Японское море | MT678963 | MT677824 | MT677775 |
| II−18 Am2 | Спортивная гавань, Амурский залив, зал. Петра Великого, Японское море | MT678964 | MT677825 | MT677776 |
| 3−19 T | Б. Сухопутная, Уссурийский залив, зал. Петра Великого, Японское море | MT678965 | MT677826 | − |
| 2−19 T | Б. Сухопутная, Уссурийский залив, зал. Петра Великого, Японское море | MT678966 | MT677827 | − |
| 3-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678967 | MT677828 | MT677777 |
| 22-17.1 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678968 | MT677829 | MT677778 |
| II-23 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678969 | MT677830 | MT677779 |
| 2-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678970 | MT677831 | MT677780 |
| 6-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678971 | MT677832 | MT677781 |
| III-25 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678972 | MT677833 | MT677782 |
| 4-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678973 | MT677834 | MT677783 |
| III−26 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678974 | MT677835 | MT677784 |
| V−12 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678975 | MT677836 | MT677785 |
| 22-17.2 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678976 | MT677837 | MT677786 |
| 24-28.01.12 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678977 | MT677838 | − |
| II-24 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678978 | MT677839 | MT677787 |
| 5-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678979 | MT677840 | MT677788 |
| 7-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678980 | MT677841 | MT677789 |
| 8-XXII-09 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678981 | MT677842 | MT677790 |
| 23-28.01.12 Uss | Уссурийский залив, зал. Петра Великого, Японское море | MT678982 | MT677843 | MT677791 |
| 23-15.3 Kar | Северо-восточная часть о-ва Карамзина | MT678983 | MT677844 | MT677792 |
| 23-15.1 Kar | О-в Карамзина, зал. Петра Великого, Японское море | MT678984 | MT677845 | MT677793 |
| 23-15.5 Kar | О-в Карамзина, зал. Петра Великого, Японское море | MT678985 | MT677846 | MT677794 |
| 23-15.2 Kar | О-в Карамзина, зал. Петра Великого, Японское море | MT678986 | MT677847 | MT677795 |
| 21-28.01.12 V | Зал. Восток, зал. Петра Великого, Японское море | MT678987 | MT677848 | MT677796 |
| 443-09 K | Б. Киевка, Японское море | MT678988 | MT677849 | MT677797 |
При выборе молекулярных маркеров для данного исследования мы руководствовались следующим: во-первых, гены мтДНК эволюционируют быстрее, чем ядерные гены (Vawter, Brown, 1986; Corse et al., 2013), и это позволяет получить деревья с наилучшим разрешением для оценки филогенетических отношений на уровне рода; во-вторых, в генном банке для видов рода Lottia имеется сравнительный материал.
Для анализа использовали фрагменты генов митохондриальной ДНК (мтДНК): субъединицы I цитохромоксидазы с (COI), а также малой (12S) и большой (16S) субъединиц рибосомальной РНК (рРНК). Фрагменты генов амплифицировали, применяя пары праймеров HCO2198/COI30L (COI) (Nakano, Ozawa, 2005), 16SarL/16SbrH (16S) (Kappner, Bieler, 2006) и 12S97L/12Smb (12S) (Koufopanou et al., 1999) при соответствующих условиях полимеразной цепной реакции. Последовательности секвенировали, используя набор BrilliantDye Terminator v3.1 Cycle Sequencing Kit (Nimagen, Нидерланды), на генетическом анализаторе ABI Prism 3500 (Applied Biosystems, США) согласно протоколу производителя. Последовательности фрагментов гена морского блюдечка Lottia cf. borealis депонировали в GenBank под номерами: MT678937–MT678989 (COI), MT677798–MT677849 (16S) и MT677751–MT677797 (12S) (табл. 1).
Множественное выравнивание и анализ полученных нуклеотидных последовательностей исследуемых фрагментов генов осуществляли в программе MEGA7 (Kumar et al., 2016) по алгоритму CLUSTAL W (Thompson et al., 1994). Для сравнения полученных данных в анализе использовали все видовое разнообразие рода Lottia из базы данных GenBank, NCBI (https://www.ncbi.nlm.nih.gov/), доступное для исследуемых маркеров. Генетическую дивергенцию оценивали на основании значений внутригрупповых и межгрупповых нескорректированных p-дистанций с помощью пакета MEGA7. Оптимальную модель нуклеотидного замещения подбирали в программе MrModeltest 2.3 (Nylander, 2004), используя информационный критерий Акаике (AIC). Для визуализации разных филогрупп мтДНК по каждому генетическому маркеру в программе popArt 1.7.2 (Leigh, Bryant, 2015) строили медианные сети гаплотипов.
Байесовские филогенетические деревья (BI) реконструировали в программе MrBayes 3.2.7a (Ronquist et al., 2012) на базе высокопроизводительного сервера CIPRES (Miller et al., 2010) при следующих параметрах: 30 млн. итераций, модель нуклеотидного замещения SYM+G, отбор каждого тысячного из генерированных деревьев, значение burnin 25%. В качестве внешней группы для построений использовали последовательности Notoacmea sturnus (Hombron, Jacquinot, 1841) (AB353900) по COI и Notoacmea pileopsis (Quoy, Gaimard, 1834) (AB238373, AB238262) по 16S и 12S маркерам.
РЕЗУЛЬТАТЫ
Длина последовательностей после выравнивания и приведения к одной длине составила 500 пар нуклеотидов (п.н.) для фрагмента гена COI, 286 п.н. для 16S рРНК и 228 п.н. для 12S рРНК.
На филогенетических деревьях, построенных на основании трех маркеров, все последовательности, принадлежащие одному виду, кластеризуются вместе. Топологии деревьев, построенных с использованием разных маркеров, в целом совпадают. В качестве основного мы рассматриваем дерево, построенное на основании последовательностей гена COI, поскольку количество последовательностей, доступных для анализа в генном банке для данного маркера, было наибольшим (рис. 2). Практически все узлы филограммы разрешены, однако не во всех случаях имеются достаточно высокие поддержки ветвей (PP < 0.95). Дерево, построенное на основании гена большой субъединицы рРНК (16S), в целом имеет сходную топологию, однако на нем присутствуют неразрешенные ветви. Филограмма, реконструированная на основании гена малой субъединицы рРНК (12S), имеет большое количество неразрешенных узлов и не может считаться достоверной. На всех филогенетических деревьях последовательности видов рода Notoacmea Iredale, 1915 занимают базальное положение по отношению к таковым исследованных таксонов.
Рис. 2.
Байесовское консенсусное филогенетическое дерево, построенное на основании нуклеотидных последовательностей фрагмента гена COI, для видов рода Lottia. В узлах дерева указаны апостеорные вероятности. Последовательности, полученные нами, выделены жирным шрифтом, данные из NCBI – курсивом. Названия локаций, как на рис. 1.

Клада Lottia cf. borealis включает все нуклеотидные последовательности, полученные нами, а также две последовательности Lottia sp. (LC416608 и LC416607) с о-ва Хоккайдо (Япония) из базы данных NCBI (поддержка клады PP = 1 по всем маркерам). Сестринским таксоном для этой клады на филограмме COI является L. paradigitalis (Fritchman, 1960) (PP = 0.9) (рис. 2). На филограмме по данным маркеров 16S и 12S ветвь L. paradigitalis не разрешена. Значения генетических дистанций между этими видами соответствуют межвидовому уровню (табл. 2).
Таблица 2.
Среднее генетическое расстояние (p) и стандартная ошибка (SE) внутри и между кладами филогенетических деревьев по данным последовательностей мтДНК
| COI | 16S | 12S | |
|---|---|---|---|
| L. cf. borealis – L. paradigitalis | 0.173 ± 0.017 | 0.0631 ± 0.0136 | − |
| L. cf. borealis | 0.0018 ± 0.0005 | 0.0031 ± 0.0013 | 0.0071 ± 0.0035 |
| L. paradigitalis* | 0.00054 ± 0.00036 | − | − |
Наибольшее количество гаплотипов обнаружено при анализе данных гена COI. Из 18 гаплотипов центральное положение занимает наиболее часто встречающийся и широко распространенный гаплотип H4. Гаплотип включает особей из каждого крупного географического региона и формирует звездообразную структуру из 12 гаплотипов (18 особей), которые отличаются от центрального гаплотипа всего одной заменой. Гаплотипы H1, H2, H3 и H14 (6 особей) удалены на расстояние двух нуклеотидных замен; они включают по одному образцу из зал. Восток, с побережья о-ва Карамзина и о-ва Итуруп и 3 особи из Уссурийского залива. Наиболее удаленный гаплотип H13 представлен одной особью из Уссурийского залива и отличается от центрального гаплотипа тремя нуклеотидными заменами (рис. 3). Наибольшее количество (9) гаплотипов обнаружено в выборках из Уссурийского залива, а также с побережья о-ва Сахалин (4 гаплотипа) и Курильских островов (3 гаплотипа).
Рис. 3.
Медианная гаплотипическая сеть, полученная на основании анализа нуклеотидных последовательностей генов COI (а), 16S (б) и 12S (в) Lottia cf. borealis.

При анализе генов рРНК выявлено значительно меньшее количество гаплотипов. По гену 16S рРНК обнаружено всего 7 гаплотипов, из которых гаплотип H1 является центральным (рис. 3). Характерная звёздообразная структура образована меньшим количеством гаплотипов, чем по данным гена COI, при этом количество нуклеотидных замен относительно больше. Так, гаплотип H5 отличается от центрального гаплотипа тремя нуклеотидными заменами и включает по две особи с побережья Курильских островов и о-ва Сахалин, в то время как гаплотип H4 (одна особь из б. Киевка) удален на 4 нуклеотидные замены. Гаплотип H3 (7 особей с побережья Курильских островов и одна особь из б. Сухопутная) удален от центрального гаплотипа на две замены. Остальные гаплотипы, объединяющие по одной особи из Амурского залива (гаплотип H2), с побережья о-ва Итуруп (H6) и о-ва Сахалин (H7), равноудалены на одну нуклеотидную замену. Наибольшее число гаплотипов характерно для выборок с побережья Курильских островов.
Анализ медианных сетей по данным фрагмента гена 12S выявил 11 гаплотипов. Все обнаруженные гаплотипы, кроме центрального H2, включают по одной особи Lottia persona. Наибольшее генетическое разнообразие наблюдалось в выборках с побережья Курильских островов (7 гаплотипов) и из Уссурийского залива (4 гаплотипа). Для гаплотипической сети по данному генетическому маркеру характерно также наличие альтернативных связей.
ОБСУЖДЕНИЕ
Морские блюдечки из дальневосточных морей, которые в настоящей работе приводятся как Lottia cf. borealis, в предыдущих работах назывались Lottia borealis (см.: Lindberg, 1986; Гульбин, 2004), Lottia cf. borealis (см.: Sasaki, 1999), Lottia paradigitalis (см.: Simison, Lindberg, 2003) и Lottia persona (Чернышев, 2004; Чернышев, Чернова, 2005, 2007). Проведенный нами анализ показал обособленность дальневосточных особей от Lottia persona (Rathke, 1833) с тихоокеанского побережья Северной Америки, штат Вашингтон, США (AB238477) (Nakano, Ozawa, 2007) и Британская Колумбия, Канада (MK091859, KX069642) (Castelin et al., 2016). Следует отметить, что распространенный у берегов Северной Америки вид, называемый Lottia (= Tectura) persona sensu Lindberg, по раковине отличается от типового экземпляра Acmaea persona Rathke, 1833, поэтому под названием “persona”, по-видимому, скрываются два вида (Чернышев, 2004). По результатам анализа Lottia cf. borealis и L. paradigitalis являются сестринскими таксонами (рис. 2), дистанция между которыми соответствует межвидовому уровню (табл. 2). Образцы L. paradigitalis (KF643296 и KF643300, NCBI), включенные в наш анализ, собраны вдоль тихоокеанского побережья США и Канады (Simison, Lindberg, 2003; Kelly, Palumbi, 2010; Layton et al., 2014). По форме и окраске раковины особи с Дальнего Востока больше похожи на вид Lottia borealis, описанный с побережья Алеутских островов, поэтому Линдберг (Lindberg, 1982) считал их конспецифичными. Позже на основе сравнения фрагментов 16S вид L. borealis был синонимизирован с L. paradigitalis (см.: Simison, Lindberg, 2003). Однако авторы не привели фотографии отсеквенированных образцов с Алеутских островов, поэтому невозможно судить, насколько эти особи соответствуют первоописанию L. borealis. Какими бы не оказались раковины, изученные данными авторами, очевидно, что для синонимии L. paradigitalis и L. borealis необходимы привлечение большего числа особей с Алеутских островов и их генетический анализ на основе маркера COI. Очевидно, поэтому в WoRMS L. borealis приведен как валидный вид (http://www.marinespecies.org/). На данном основании до проведения более тщательного генетического анализа морских блюдечек с Алеутских островов, мы предлагаем называть особей из дальневосточных морей как Lottia cf. borealis.
От других дальневосточных морских блюдечек Lottia cf. borealis отличается скульптурой раковины, представленной очень тонкими широко расставленными и часто сдвоенными радиальными ребрышками (рис. 4д) (Чернышев, Чернова, 2007). Однако у большинства собранных особей скульптура раковины отсутствовала, и отнесение их к Lottia cf. borealis основывалось на форме сдвинутой в переднюю четверть слегка клювовидной вершины раковины, что не является надежным признаком. При этом окраска раковины сильно варьировала (рис. 4а–4г) и обычно состояла из прерывистых радиальных полос или расположенных в шахматном порядке пятен белого, серого или желтого цвета на темно-коричневом, темно-сером или оливковом фоне; белые радиальные пятна часто раздваивались по краю раковины, создавая многочисленные короткие радиальные параллельные линии вдоль устья раковины (рис. 4а–4в); иногда наружная поверхность раковины была почти однотонной: темно-серой, бурой или почти черной (рис. 4г) с единичными светлыми радиальными пятнышками. Многие определения были под вопросом или значились как Lottia sp. из-за сильно стертой поверхности раковины (рис. 4б, 4г). Однако генетический анализ показал, что все изученные особи относятся к одному виду. Это еще раз подтвердило, что окраска раковины у морских блюдечек – вариабельный признак, зависящий от окраски субстрата, на котором питаются виды (Giesel, 1970; Hockey et al., 1987; Byers, 1989; Lindberg, Pearse, 1990). Раковина морских блюдечек просто устроена и чрезвычайно изменчива; ее высота может варьировать, например, при пересыхании места обитания морских блюдечек (Segal, 1956; Vermeij, 1973; Branch, 1975) или от турбулентности воды (Simpson, 1985). Морфология радулы зависит от диеты; у неродственных видов со сходным питанием она может приобретать черты значительного конвергентного сходства (Steneck, 1982; Lindberg, Vermeij, 1985; Sasaki, Okutani, 1993). Из-за различий в условиях обитания наблюдается значительная внутривидовая изменчивость морфологических признаков, что часто приводит к расхождению результатов морфологических и молекулярно-генетических исследований (Nakano, Ozawa, 2005).
Рис. 4.
Вариации окраски (а–г) и скульптура раковины (д) Lottia cf. borealis. Масштаб: а–г – 2 мм, д – 0.1 мм.

При анализе гаплотипических сетей внутри клады Lottia cf. borealis по всем генетическим маркерам наблюдалась звездообразная структура с выраженным центральным гаплотипом, который имеет широкое распространение. Для остальных гаплогрупп характерно снижение уровня генетической дифференциации вдоль дальневосточного побережья России. Центральный гаплотип, вероятно, является предковым, а остальные гаплотипы дивергировали от него (Posada, Crandall, 2001). Низкий уровень генетического разнообразия и наличие широко распространенных доминантных гаплотипов согласуются с гипотезой о недавнем расширении ареала (Slatkin, Hudson, 1991; Rogers, Harpending, 1992) и высоком уровне миграции (Marko, 2004; Hoffman et al., 2011). Подобное отсутствие генетической структуры отмечено и у других морских организмов с непрямым развитием, например, у Mytilus edulis Linnaeus, 1758 (см.: Toro et al., 2004) и у морского блюдечка Nacella magellanica (Gmelin, 1791) (см.: González-Wevar et al., 2012).
Генетический обмен внутри популяций и между ними у большинства морских бентосных видов происходит благодаря пелагической стадии личинки (Scheltema, 1971), а расстояние, на которое могут распространиться личинки, зависит от гидрографических условий (Avise et al., 1986) и длительности личиночного развития (Nakano et al., 2010). Лецитотрофная личинка Lottia cf. borealis при температуре воды 20°С оседает на третьи сутки после оплодотворения (Колбин, Куликова, 2011). Cтолько же времени находятся в планктоне личинки Lottia goshimai Sasaki et Nakano, 2017 (Колбин, 2010: как Lottia kogamogai Sasaki & Okutani, 1994) и Nipponacmea moskalevi Chernyshev et Chernova, 2002 (см.: Колбин, Куликова, 2002), а у Lottia versicolor (Moskalev in Golikov & Scarlato, 1967) личинки оседают на седьмые сутки (Колбин, Куликова, 2002). При этом ареал у всех трех видов значительно меньше, чем у вида Lottia cf. borealis, который распространен от зал. Петра Великого до Командорских островов. Следует отметить, что при низкой температуре время развития личинки может значительно увеличиваться (Wanninger et al., 1999; Wanninger et al., 2000; Kay, Emlet, 2002; Nakano, Sasaki, 2011), и в популяциях Lottia cf. borealis у о-ва Сахалин и Курильских островов время пребывания личинок в планктоне может быть более длительным.
По-видимому, Lottia cf. borealis относится к видам с низкой специфичностью к среде, населяя средний и нижний горизонты литорали открытых и полузакрытых побережий (Голиков, Кусакин, 1978; Чернышев, Чернова, 2005). Такие виды не имеют строгой генетической дифференциации, а протяженные участки, на которых отсутствуют подходящие условия среды, могут стать барьером для потока генов, даже несмотря на существование планктонной стадии, в течение которой личинки пателлогастропод способны преодолеть расстояние в 100–200 км (Nakano, Sasaki, 2011).
Таким образом, можно заключить, что вид Lottia cf. borealis широко распространен вдоль материкового побережья и островов Дальнего Востока России, а также встречается в Японии у о-ва Хоккайдо (Sasaki, 1999). На исследованном ареале для Lottia cf. borealis характерны относительно низкое генетическое разнообразие и отсутствие внутривидовой подразделенности, что может свидетельствовать о сравнительно недавней экспансии этого вида и высоком уровне миграции.
Список литературы
Голиков А.Н., Кусакин О.Г. Раковинные брюхоногие моллюски литорали морей СССР // Определители по фауне СССР. Л.: Наука. 1978. Т. 116. 292 с.
Гульбин В.В. Фауна брюхоногих переднежаберных моллюсков залива Петра Великого Японского моря и ее биогеографический состав // Биол. моря. 2004. Т. 30. № 1. С. 20−29.
Колбин К.Г. Размножение и развитие некоторых массовых видов переднежаберных брюхоногих моллюсков залива Петра Великого Японского моря: Автореф. дис. … канд. биол. наук. Владивосток. 2010. 22 с.
Колбин К.Г., Куликова В.А. Размножение и развитие Lottia versicolor и Nipponacmea moskalevi (Gastropoda, Lottiidae) // V Регион. конф. по актуальным проблемам экологии, морской биологии и биотехнологии: Материалы конф. студентов, аспирантов и молодых ученых ДВГУ (академия экологии, морской биологии и биотехнологии), ИБМ ДВО РАН. Владивосток, 21–24 ноября 2002. Владивосток: Изд-во ДВГУ. 2002. С. 61–62.
Колбин К.Г., Куликова В.А. Размножение и личиночное развитие морского блюдечка Lottia persona (Rathke, 1833) (Gastropoda: Lottiidae) // Биол. моря. 2011. Т. 37. № 3. С. 229−232.
Москалев Л.И. Брюхоногие моллюски рода Collisella (Prosobranchia, Acmaeidae) окраинных азиатских морей Тихого океана // Тр. ИО АН СССР. 1970. Т. 88. С. 174−212.
Чернышев А.В. О типовом экземпляре Acmaea persona Rathke, 1833 (Gastropoda) // Ruthenica (Рус. малакол. журн.). 2004. Т. 14. № 1. С. 5–8.
Чернышев А.В., Чернова Т.В. Систематическое положение Lottia angusta (Moskalev in Golikov et Scarlato, 1967) (Gastropoda, Lottiidae) // Ruthenica (Рус. малакол. журн.). 2004. Т. 14. № 1. С. 1−4.
Чернышев А.В., Чернова Т.В. Пателлогастроподы (Patellogastropoda) дальневосточных морей России // Бюл. Дальневост. малакол. о-ва. 2005. Вып. 9. С. 7–26.
Чернышев А.В., Чернова Т.В. Ключи для определения пателлогастропод (Patellogastropoda) дальневосточных морей России // Бюл. Дальневост. малакол. о-ва. 2007. Вып. 11. С. 65–74.
Abbott D.P., Epel D., Phillips J.H., Abbot I.A. Undergraduate research and the biology of Acmaea // Veliger. 1968. V. 11 (suppl.). P. 1–5.
Avise J.C., Helfman G.S., Saunders N.C., Hales L.S. Mitochondrial DNA differentiation in North Atlantic eels: population genetic consequences of an unusual life history pattern // Proc. Natl. Acad. Sci. USA. 1986. V. 83. № 12. P. 4350–4354.
Branch G.M. Ecology of Patella species from the Cape Peninsula, South Africa. IV. Desiccation // Mar. Biol. 1975. V. 32. P. 179–188.
Byers B.A. Habitat-choice polymorphism associated with cryptic shell-color polymorphism in the limpet Lottia digitalis // Veliger. 1989. V. 32. P. 394–402.
Castelin M., Van Steenkiste N., Pante E et al. A new integrative framework for large-scale assessments of biodiversity and community dynamics, using littoral gastropods and crabs of British Columbia, Canada // Mol. Ecol. Resour. 2016. V. 16. № 6. P. 1322−1339.
Corse E., Rampal J., Cuoc C. et al. Phylogenetic analysis of Thecosomata Blainville, 1824 (Holoplanktonic Opisthobranchia) using morphological and molecular data // PLoS One. 2013. V. 8. № 4. Art. ID e59439. https://doi.org/10.1371/journal.pone.0059439
Crummett L.T., Eernisse D.J. Genetic evidence for the cryptic species pair, Lottia digitalis and Lottia austrodigitalis and microhabitat partitioning in sympatry // Mar. Biol. 2007. V. 152. P. 1–13.
Espoz C., Castilla J.C. Escape responses of four Chilean intertidal limpets to seastars // Mar. Biol. 2000. V. 137. № 5. P. 887–892.
Evans N., Paulay G. DNA Barcoding Methods for Invertebrates // DNA Barcodes: Methods and Protocols // Methods in molecular biology. V. 858. Totowa, N.J.: Humana Press. 2012. P. 44–77.
Giesel J.T. On the maintenance of a shell pattern and behavior polymorphism in Acmaea digitalis, a limpet // Evolution. 1970. V. 24. P. 98–119.
Goldstien S.J., Gemmell N.J., Schiel D.R. Molecular phylogenetics and biogeography of the nacellid limpets of New Zealand (Mollusca: Patellogastropoda) // Mol. Phylogenet. Evol. 2006. V. 38. P. 261–265.
González-Wevar C.A., Hüne M., Cañete J.I. et al. Towards a model of postglacial biogeography in shallow marine species along the Patagonian Province: lessons from the limpet Nacella magellanica (Gmelin, 1791) // BMC Evol. Biol. 2012. V. 12. Art. ID 139. https://doi.org/10.1186/1471-2148-12-139
González-Wevar C.A., Hüne M., Rosenfeld S. et al. Systematic revision of Nacella (Patellogastropoda: Nacellidae) based on a complete phylogeny of the genus, with the description of a new species from the southern tip of South America // Zool. J. Linn. Soc. 2019. V. 186. P. 303–336.
Harasewych M.G., McArthur A.G. A molecular phylogeny of the Patellogastropoda (Mollusca: Gastropoda) // Mar. Biol. 2000. V. 137. P. 183–194.
Hebert P.D.N., Cywinska A., Ball S.L., deWaard J.R. Biological identifications through DNA barcodes // Proc. R. Soc. London, Ser. B. 2003. V. 270. P. 313–321.
Hockey P.A.R., Bosman A.L., Ryan P.G. The maintenance of polymorphism and cryptic mimesis in the limpet Scurria variabilis by two species of Cinclodes (Aves: Furnariinae) in central Chile // Veliger. 1987. V. 30. P. 5–10.
Hodgson A.N., Ridgway S., Branch G.M., Hawkins S.J. Spermatozoan morphology of 19 species of prosobranch limpets (Patellogastropoda) with a discussion of patellid relationships // Philos. Trans. R. Soc. London, Ser. B. 1996. V. 351. P. 339–347.
Hoffman J.I., Clarke A., Linse K., Peck L.S. Effects of brooding and broadcasting reproductive modes on the population genetic structure of two Antarctic gastropod molluscs // Mar. Biol. 2011. V. 158. № 2. P. 287–296.
Johnson S.B., Warén N.A., Vrijenhoek R.C. DNA barcoding of Lepetodrilus limpets reveals cryptic species // J. Shellfish Res. 2008. V. 27. P. 43–51.
Kappner I., Bieler R. Phylogeny of venus clams (Bivalvia: Venerinae) as inferred from nuclear and mitochondrial gene sequences // Mol. Phylogenet. Evol. 2006. V. 40. Iss. 2. P. 317–331.
Kay M.C., Emlet R.B. Laboratory spawning, larval development, and metamorphosis of the limpets Lottia digitalis and Lottia asmi (Patellogastropoda, Lottiidae) // Invertebr. Biol. 2002. V. 121. P. 11–24.
Kelly R.P., Palumbi S.R. Genetic structure among 50 species of the northeastern Pacific rocky intertidal community // PLoS One. 2010. V. 5. № 1, art. ID e8594. https://doi.org/10.1371/journal.pone.0008594
Koufopanou V., Reid D.G., Ridgway S.A., Thomas R.H. A molecular phylogeny of the patellid limpets (Gastropoda: Patellidae) and its implications for the origins of their antitropical distribution // Mol. Phylogenet. Evol. 1999. V. 11. P. 138–156.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874.
Layton K.K.S., Martel A.L., Hebert P.D.N. Patterns of DNA barcode variation in Canadian marine molluscs // PLoS One. 2014. V. 9. № 4, art. ID e95003. https://doi.org/10.1371/journal.pone.0095003
Leigh J., Bryant D. POPART: Full-feature software for haplotype network construction // Methods Ecol. Evol. 2015. V. 6. № 9. P. 1110–1116.
Lindberg D.R. Taxonomic notes on members of the genus Collisella from the North Pacific Ocean including a description of a new species from Alaska (Gastropoda: Acmaeidae) // Wasmann J. Biol. 1982. V. 40. Nos. 1–2. P. 48–58.
Lindberg D.R. Name changes in the “Acmaeidae” // Veliger. 1986. V. 29. № 2. P. 142–148.
Lindberg D.R. Systematics of the Scurriini (new tribe) of the northern Pacific Ocean (Patellogastropoda: Lottiidae) // Veliger. 1988. V. 30. № 4. P. 387–394.
Lindberg D.R. Patellogastropoda, Neritimorpha and Cocculinoidea: The low-diversity gastropods clades // Phylogeny and evolution of the Mollusca. Berkeley, Calif.: Univ. of California Press. 2008. P. 271–296.
Lindberg D.R., Vermeij G.J. Patelloida chamorrorum spec. nov.: a new member of the Tethyan Patelloida profunda group (Gastropoda: Acmaeidae) // Veliger. 1985. V. 27. P. 411–417.
Lindberg D.R., Pearse J.S. Experimental manipulation of shell color and morphology of the limpets Lottia asmi (Middendorff) and Lottia digitalis (Rathke) (Mollusca: Patellogastropoda) // J. Exp. Mar. Biol. Ecol. 1990. V. 140. P. 173–185.
Marko P.B. ‘What’s larvae got to do with it?’ Disparate patterns of post-glacial population structure in two benthic marine gastropods with identical dispersal potential // Mol. Ecol. 2004. V. 13. P. 597–611.
Miller M.A., Pfeiffer W., Schwartz T. Creating the CIPRES science gateway for inference of large phylogenetic trees, in 2010 Gateway Computing Environments Workshop (GCE). New Orleans, LA, USA. 2010. P. 1–8.
Nakano T., Ozawa T. Phylogeny and historical biogeography of limpets or the order Patellogastropoda based on mitochondrial DNA sequences // J. Molluscan Stud. 2004. V. 70. P. 31–41.
Nakano T., Ozawa T. Systematic revision of Patelloida pygmaea (Dunker, 1860) (Gastropoda: Lottiidae), with a description of a new species // J. Molluscan Stud. 2005. V. 71. P. 357–370.
Nakano T., Ozawa T. Worldwide phylogeography of limpets of the order Patellogastropoda: molecular, morphological and palaeontological evidence // J. Molluscan Stud. 2007. V. 73. P. 79–99.
Nakano T., Spencer H.G. Simultaneous polyphenism and cryptic species in an intertidal limpet from New Zealand // Mol. Phylogenet. Evol. 2007. V. 45. P. 470–479.
Nakano T., Marshall B.A., Kennedy M., Spencer H.G. The phylogeny and taxonomy of New Zealand Notoacmea and Patelloida species (Mollusca: Patellogastropoda: Lottiidae) inferred from DNA sequences // Molluscan Res. 2009. V. 29. P. 33–59.
Nakano T., Sasaki T., Kase T. Color polymorphism and historical biogeography in the Japanese patellogastropod limpet Cellana nigrolineata (Reeve) (Patellogastropoda: Nacellidae) // Zool. Sci. 2010. V. 27. P. 811–820.
Nakano T., Sasaki T. Recent advances in molecular phylogeny, systematics and evolution of patellogastropod limpets // J. Molluscan Stud. 2011. V. 77. P. 203–217.
Nakayama R., Sasaki T., Nakano T. Molecular analysis reveals a new cryptic species in a limpet Lottia kogamogai (Patellogastropoda: Lottiidae) from Japan // Zootaxa. 2017. V. 4277. № 2. P. 237–251.
Nieukerken E.J.V., Mutanen M., Doorenweerd C. DNA barcoding resolves species complexes in Stigmella salicis and S. aurella species groups and shows additional cryptic speciation in S. salicis (Lepidoptera: Nepticulidae) // Entomologisk Tidskrift. 2012. V. 132. P. 235–255.
Nylander J.A.A. MrModeltest v2. Program distributed by the author. Uppsala, Sweden: Uppsala Univ., Evolutionary Biology Centre. 2004.
Posada D., Crandall K.A. Intraspecific gene genealogies: trees grafting into networks // Trends Ecol. Evol. 2001. V. 16. P. 37–45.
Rogers A., Harpending H. Population growth makes waves in the distribution of pairwise genetic differences // Mol. Biol. Evol. 1992. V. 9. № 3. P. 552–569.
Ronquist F., Teslenko M., van der Mark P. et al. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model choice across a large model space // Syst. Biol. 2012. V. 61. P. 539–542.
Sasaki T. The present state and problems of the taxonomy of Japanese patellogastropod limpets. Part II: Lottiidae // Newsl. Malacological Soc. Jpn. 1999. V. 29. P. 37–46.
Sasaki T., Okutani T. Anatomyand systematic position of Yayoiacmea, a new genus for Japanese tiny limpet “Collisella” oyamai Habe, 1955 (Gastropoda: Lottiidae) // Venus. 1993. V. 52. P. 193–209.
Sasaki T., Okutani T. An analysis on “Collisella heroldi” complex (Gastropoda: Lottiidae), with description of three new species // Venus. 1994. V. 53. № 4. P. 251–285.
Scheltema R.S. Larval dispersal as a means of genetic exchange between geographically separated populations of shallow-water benthic marine gastropods // Biol. Bul. 1971. V. 140. № 2. P. 284–322.
Segal E. Adaptive differences in water-holding capacity in an intertidal gastropod // Ecology. 1956. V. 37. P. 174–178.
Sharina S.N., Chernyshev A.V., Zaslavskaya N.I. Genetic diversity and phylogeny of limpets of the genus Nipponacmea (Patellogastropoda: Lottiidae) based on mitochondrial DNA sequences // Mitochondrial DNA, Part A. 2016. V. 28. № 5. P. 703–710.
Simison W.B., Lindberg D.R. Morphological and molecular resolution of a putative cryptic species complex: a case study of Notoacmea fascicularis (Menke, 1851) (Gastropoda: Patellogastropoda) // J. Molluscan Stud. 1999. V. 65. P. 99–109.
Simison W.B., Lindberg D.R. On the identity of Lottia strigatella (Carpenter, 1864) (Patellogastropoda: Lottiidae) // Veliger. 2003. V. 46. P. 1–19.
Simpson R.D. Relationship between allometric growth, with respect to shell height and habitats for two patellid limpets, Nacella (Patinigera) macquariensis Finlay, 1927 and Cellana tramoserica (Holten, 1802) // Veliger. 1985. V. 28. P. 18–27.
Slatkin M., Hudson R.R. Pairwise comparisons of mitochondrial DNA sequences in stable and exponentially growing populations // Genetics. 1991. V. 129. P. 555–562.
Steneck R.S. A limpet-coralline alga association: adaptations and defenses between a selective herbivore and its prey // Ecology. 1982. V. 63. P. 507–522.
Toro J.E., Ojeda J.A., Vergara A.M. The genetic structure of Mytilus chilensis (Hupé 1854) populations along the Chilean coast based on RAPDs analysis // Aquacult. Res. 2004. V. 35. P. 1466–1471.
Thompson J.D., Higgins D.G., Gibson T.J. Clustal W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties, and weight matrix choice // Nucleic Acids Res. 1994. V. 22. № 22. P. 4673–4680.
Vawter L., Brown W.M. Nuclear and mitochondrial DNA comparisons reveal extreme rate variation in the molecular clock // Science. 1986. V. 234. P. 194–196.
Vermeij G.J. Morphological patterns in high-intertidal gastropods: Adaptive strategies and their limitations // Mar. Biol. 1973. V. 20. P. 319–346.
Wanninger A., Ruthensteiner B., Lobenwein S. et al. Development of the musculature in the limpet Patella (Mollusca, Patellogastropoda) // Dev. Genes Evol. 1999. V. 209. P. 226–238.
Wanninger A., Ruthensteiner B., Haszprunar G. Torsion in Patella caerulea (Mollusca, Patellogastropoda): ontogenetic process, timing, and mechanisms // Invertebr. Biol. 2000. V. 119. P. 177–187.
Uribe J.E., Irisarri I., Templado J., Zardoya R. New patellogastropod mitogenomes help counteracting long-branch attraction in the deep phylogeny of gastropod mollusks // Mol. Phylogenet. Evol. 2019. V. 133. P. 12–23.
Yu S.-S., Wang J., Wang Q.-L. et al. DNA barcoding and phylogeographic analysis of Nipponacmea limpets (Gastropoda: Lottiidae) in China // J. Molluscan Stud. 2014. V. 80. P. 420–429.
Дополнительные материалы отсутствуют.


