Биология моря, 2022, T. 48, № 5, стр. 328-337
Изменчивость ядерных локусов у дальневосточного трепанга Apostichopus japonicus (Selenka, 1867) (Echinodermata: Holothuroidea) в выборках из залива Петра Великого Японского моря
В. Д. Ягодина 1, *, Н. М. Батищева 1, В. А. Брыков 1
1 Национальный научный центр морской биологии им. А.В. Жирмунского ДВО РАН
690041 Владивосток, Россия
* E-mail: iagodinavd@gmail.com
Поступила в редакцию 18.01.2022
После доработки 25.04.2022
Принята к публикации 02.06.2022
- EDN: RNSWFG
- DOI: 10.31857/S0134347522050102
Аннотация
На 122 особях из пяти выборок (зал. Петра Великого, Японское море) с применением пяти микросателлитных маркеров исследовано генетическое разнообразие дальневосточного трепанга Apostichopus japonicus. Все локусы полиморфны; среднее значение наблюдаемой гетерозиготности для всех выборок и локусов составило 0.461 ± 0.027, ожидаемой гетерозиготности – 0.575 ± 0.031. Значения коэффициента инбридинга в среднем были выше 0, что обусловлено дефицитом гетерозигот. Для некоторых микросателлитных локусов выявлены нуль-аллели. После их обнаружения исправлены ошибки генотипирования и проведена корректировка статистических данных, которая показала, что присутствие нулевых аллелей в 1.5–2 раза снижало значение наблюдаемой гетерозиготности и вело к отклонению от равновесия Харди−Вайнберга.
Дальневосточный трепанг Apostichopus japonicus (Selenka, 1867) широко распространен в прибрежных водах Восточной Азии, включая Японию, Китай, Северную и Южную Корею, а также Дальний Восток России (Chang et al., 2009). Этот вид имеет значительную коммерческую ценность и является одним из важнейших объектов аквакультуры, поскольку обладает высоким содержанием ценных питательных веществ, витаминов и минералов, а также рядом характерных биологически и фармакологически биоактивных соединений; способность A. japonicus к регенерации делает его важным объектом с медицинской точки зрения (Oh et al., 2017).
Для описания генетической изменчивости и структуры популяций дальневосточного трепанга применяются и разрабатываются в основном микросателлитные маркеры (Kanno et al., 2006; Chen et al., 2013) и однонуклеотидные полиморфизмы (Du et al., 2012; Dong et al., 2016). К основным преимуществам микросателлитных маркеров относят высокий уровень изменчивости как следствие высокой скорости накопления мутаций, кодоминантный тип наследования и легкую пробоподготовку. Недостатками микросателлитов считают появление “теневых полос”, или “заиканий” (Галинская и др., 2019), когда каждый аллель представляет собой серию фрагментов, кратных длине повторяющейся единицы микросателлита вследствие “проскальзывания” при реп-ликации в полимеразной цепной реакции (ПЦР), а также гомоплазию и наличие нулевых аллелей (Abdul-Muneer, 2014).
Нулевой аллель – любой аллель в микросателлитном локусе, который постоянно не амплифицируется с помощью полимеразной цепной реакции (Dakin, Avise, 2004). Исследования показали (Grimaldi, Crouau-Roy, 1997), что во фланкирующих локус областях происходят мутации, препятствующие отжигу праймеров с матричной ДНК во время ПЦР и приводящие к нулевым аллелям. Другие возможные причины возникновения нуль-аллелей − предпочтительная амплификация коротких аллелей из-за непостоянства качества или количества матрицы ДНК (Chapius, Estoup, 2007), а также проскальзывание ДНК-полимеразы во время амплификации (Ellegren, 2004). Наличие нулевых аллелей приводит к появлению ложных гомозигот, которые влияют на отклонение от равновесия Харди−Вайнберга и ведут, как следствие, к некорректной интерпретации результатов (Carlsson, 2008).
С использованием микросателлитных маркеров проведены исследования генетической структуры и разнообразия A. japonicus из зал. Тояма (Япония) (Soliman et al., 2012). Изучение данной голотурии из разных локальностей побережья Южной Кореи показало применимость выбранных авторами микросателлитных локусов для генетического анализа (Kim et al., 2008). При исследовании популяций дальневосточного трепанга только в работе Канно с коллегами (Kanno et al., 2006) были определены нулевые аллели и проведена работа по корректировке этих ошибок генотипирования.
Большая часть работ в российской литературе посвящена изучению биологии и образа жизни дальневосточного трепанга, а также процессов, связанных со снижением его численности (Селин, 2001; Гаврилова, 2013; Лысенко и др., 2018). Исследования по определению популяционной структуры и уровней генетического разнообразия A. japonicus на Дальнем Востоке России не проводились.
Цель настоящей работы – оценка генетической изменчивости у трепанга A. japonicus с помощью микросателлитных локусов в выборках из зал. Петра Великого Японского моря, а также изучение распределения выявленных нуль-аллелей и оценка их влияния на интерпретацию популяционных данных с использованием разных популяционно-генетических подходов.
МАТЕРИАЛ И МЕТОДИКА
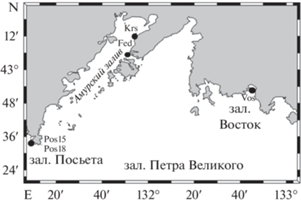
Исследовано пять выборок трепанга Apostichopus japonicus, собранных водолазным способом в зал. Петра Великого Японского моря в 2015 и 2018 гг.: зал. Восток (Vos, n = 28, июль 2015 г.), мыс Красный, Амурский залив (Krs, n = 20, июнь 2015 г.), зал. Посьета (Pos15, n = 34, июль 2015 г.), б. Федорова, Амурский залив (Fed, n = 26, июнь 2015 г.) и зал. Посьета (Pos18, n = 22, июль 2018 г.). Две выборки из зал. Посьета (3 и 5) были взяты с интервалом в три года в одной и той же локальности (рис. 1).
Геномную ДНК из фиксированной в 96% этаноле ткани выделяли методом щелочного лизиса (Truett, 2006). Выборки трепанга были проанализированы по пяти микросателлитным локусам. В полимеразной цепной реакции использовали праймеры, представленные в табл. 1. К 5'-концу каждого прямого праймера был пришит хвост M13 (в табл. 1 указан мелким шрифтом), который был помечен одним из четырех флуоресцентных красителей: ROX, 6FAM, R6G или TAMRA.
Таблица 1.
Использованные для анализа выборок трепанга Apostichopus japonicus локусы, праймеры и исследуемый повтор
| № | Локус | Последовательности праймеров (5'-3') | Повтор | Размер без праймера (пн) |
|---|---|---|---|---|
| 1 | AJ10675 | F: TGGAAACAGCTATGACCATGAGATGTCAGCCACATGCAAC R: GGTAAGCTTGTGGGAATGGA |
(TAG)5 | 185–190 |
| 2 | AJ13361 | F: TGGAAACAGCTATGACCATGTGGAAGACGAAGATGAGCAA R: GGAATGACCCTACGTCCAAA |
(CAG)5 | 165–205 |
| 3 | AJ20333 | F: TGGAAACAGCTATGACCATGCGTCGACCAAAGAGAGCAAT R: TCCCTGGACTGGCACTAATC |
(AGT)7 | 181–198 |
| 4 | AJ20385 | F: TGGAAACAGCTATGACCATGAGCAAACCACCGAGTACACC R:CTCCACCACTCTCCGATTCT |
(CAA)5 | 187–205 |
| 5 | AJ21199 | F: TGGAAACAGCTATGACCATGTACGCCTTTTGTCCGTTTTC R: TGCAAGGCACAATTCTAAAAGA |
(CTA)6 | 162–216 |
ПЦР проводили в реакционной смеси, содержавшей дистиллированную воду, 10Х буфер (Евроген), dNTP (содержание каждого дезоксинуклеотида 5 мМ), прямой праймер (5 мМ), обратный праймер (5 мМ), флуоресцентный краситель (5 мМ), Taq ДНК-полимеразу (5000 ед., Евроген) – 0.1 мкл и ДНК – 40−60 нг. Конечный объем реакции составлял 13 мкл. Условия для ПЦР были следующими: начальная денатурация при 94° в течение 3 мин; далее для 30 циклов денатурация при 94° – 45 с, отжиг (60°) – 30 с, элонгация (72°) – 2 мин; завершающая элонгация (72°) – 5 мин.
Продукт ПЦР проверяли с помощью гель-электрофореза в 1.5% агарозном геле; 1 мкл ПЦР-смеси добавляли в смесь для генотипирования, содержавшую формамид и размерный стандарт (S450) (COrDIS), затем подвергали электрофорезу на секвенаторе ABI3130 (Applied Biosystems). Длину фрагментов в полученных продуктах ПЦР визуализировали с помощью программы GeneMapper ver. 5.0 (Applied Biosystems).
Для генетической характеристики локусов определяли частоту аллелей в каждом локусе с использованием пакета GenAIEx версии 6.5.1 (Peakall, Smouse, 2012). При помощи программы “Arlequin” версии 3.5 (Excoffier, Lischer, 2010) рассчитывали наблюдаемую (Ho) и ожидаемую (He) гетерозиготность, коэффициент инбридинга (FIS), а также проводили тест на соответствие ожиданиям Харди−Вайнберга (P) с использованием метода цепей Маркова. Коэффициент инбридинга с корректировкой данных по нулевым аллелям рассчитывали при помощи IIM-модели (байесовский подход) с 50 000 итерациями, значением burn-in в 10 000 итераций в программе INEST 2.2 (Chybicki, Burczyk, 2009).
Частоты нулевых аллелей оценивали в следующих программах: FreeNA (Chapius, Estoup, 2007) в соответствии с алгоритмом максимизации ожиданий (EM-алгоритм – Expectation-maximization algorithm) Демпстера, Лэрда и Рубина (Dempster et al., 1977); INEST 2.2 – при помощи IIM подхода (байесовский подход) с количеством циклов в 50 000 и значением burn-in в 10 000; модель выбора включала нуль-аллели, инбридинг и ошибки генотипирования; MICRO-CHECKER (Van Oosterhout et al., 2004) – исходя из пропорций Харди−Вайнберга, частоты нулевых аллелей оценены и скорректированы в соответствии с методами, разработанными Ван Остерхоутом (Van Oosterhout et al., 2004).
Если частота нулевого аллеля меньше или равна 0.1, некоторые оценки можно использовать напрямую без корректировки; если она больше или равна 0.5, эффективность оценки слишком мала и такой локус следует исключить (Huang et al., 2016).
РЕЗУЛЬТАТЫ
Генетическая изменчивость трепанга
В исследованных выборках все локусы оказались полиморфными, частоты аллелей характеризовались значительной изменчивостью. Размер аллелей варьировал от 172 до 219 п. н. Всего обнаружено 36 аллелей по всем локусам, среднее число аллелей на локус составило 7.2.
Для пяти выборок трепанга по каждому локусу были рассчитаны основные статистические показатели, необходимые для интерпретации полученных результатов. Наибольшие различия в значениях Ho и He отмечены в двух локусах трех выборок: в группе особей из зал. Восток в локусе AJ20333; в выборках из зал. Посьета, отобранных в 2015 и 2018 гг., показатели также различались по локусу AJ13361. Средние для всех выборок и локусов значения наблюдаемой и ожидаемой гетерозиготности составили 0.439 ± 0.032 и 0.563 ± 0.030 соответственно (табл. 2).
Таблица 2.
Характеристика генетической изменчивости по пяти микросателлитным локусам в пяти исследованных выборках трепанга Apostichopus japonicus
| Выборка | Локус | Ho | He | FIS | P |
|---|---|---|---|---|---|
| Vos | AJ20333 | 0.250 | 0.627 | 0.612 | 0.006** |
| AJ20385 | 0.500 | 0.642 | 0.232 | 0.849 | |
| AJ10675 | 0.571 | 0.595 | 0.040 | 0.000*** | |
| AJ13361 | 0.333 | 0.503 | 0.343 | 0.011* | |
| AJ21199 | 0.571 | 0.780 | 0.275 | 0.037* | |
| Krs | AJ20333 | 0.538 | 0.440 | –0.235 | 0.623 |
| AJ20385 | 0.538 | 0.714 | 0.253 | 0.647 | |
| AJ10675 | 0.438 | 0.353 | –0.250 | 0.263 | |
| AJ13361 | 0.455 | 0.593 | 0.242 | 0.072 | |
| AJ21199 | 0.500 | 0.700 | 0.300 | 0.009** | |
| Pos15 | AJ20333 | 0.607 | 0.571 | –0.064 | 0.907 |
| AJ20385 | 0.375 | 0.492 | 0.242 | 0.030* | |
| AJ10675 | 0.355 | 0.337 | –0.054 | 0.694 | |
| AJ13361 | 0.065 | 0.567 | 0.888 | 0.000*** | |
| AJ21199 | 0.536 | 0.682 | 0.218 | 0.067 | |
| Fed | AJ20333 | 0.467 | 0.671 | 0.312 | 0.123 |
| AJ20385 | 0.750 | 0.633 | –0.200 | 0.436 | |
| AJ10675 | 0.522 | 0.464 | –0.128 | 0.472 | |
| AJ13361 | 0.417 | 0.543 | 0.237 | 0.002** | |
| AJ21199 | 0.600 | 0.747 | 0.206 | 0.044* | |
| Pos18 | AJ20333 | 0.526 | 0.595 | 0.118 | 0.000*** |
| AJ20385 | 0.300 | 0.514 | 0.423 | 0.001** | |
| AJ10675 | 0.133 | 0.129 | –0.037 | 0.782 | |
| AJ13361 | 0.182 | 0.723 | 0.758 | 0.003** | |
| AJ21199 | 0.444 | 0.464 | 0.045 | 0.601 | |
| Среднее для всех выборок и локусов | 0.439 ± 0.032 | 0.56 3 ± 0.030 | 0.175 ± 0.228 | ||
Примечание. Здесь и в таблицах 3–5: Ho – наблюдаемая гетерозиготность, He – ожидаемая гетерозиготность, FIS – коэффициент инбридинга, P – значение отклонения от равновесия Харди−Вайнберга (*P < 0.05; **P < 0.01; ***P < 0.001).
Средние значения генетической изменчивости по пяти микросателлитным локусам представлены в табл. 3. Для всех выборок наблюдаемая гетерозиготность была меньше ожидаемой, значение FIS для популяций Vos, Pos15 и Pos18 было больше 0.2.
Влияние нулевых аллелей на генетическое разнообразие трепанга
Частота нулевых аллелей по отдельным локусам в индивидуальных выборках, рассчитанная в программе MICRO-CHECKER, варьировала от −0.2957 до 0.7928. Частота нуль-аллелей в программе FreeNA изменялась от 0 до 0.3207; результаты программы INEST 2.2 по определению частоты нулевых аллелей варьировали от 0.0250 до 0.4580.
В программе MICRO-CHECKER обнаружены нулевые аллели в пяти локусах для всех выборок трепанга, которые нуждались в корректировке генотипов (см. приложение). После корректировки генотипов по методу Ван Остерхоута получены значения Ho, He и FIS для локусов с нуль-аллелями (табл. 4).
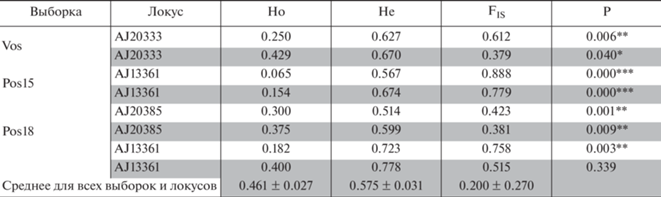
Сравнение данных без коррекции и с коррекцией генотипов показывает сильное изменение коэффициента инбридинга. Так, для особей из зал. Восток FIS по локусу AJ20333 без учета нулевых аллелей он составил 0.612, а с их учетом – 0.379; для особей из зал. Посьета, собранных в 2018 г., FIS был равен соответственно 0.758 и 0.515.
Средние значения генетической изменчивости по пяти микросателлитным локусам с учетом нуль-аллелей представлены в табл. 5. Значения коэффициента инбридинга, полученные в программе INEST 2.2, скорректированы исходя из частоты нулевых аллелей.
Таблица 5.
Средние значения генетической изменчивости по всем локусам в пяти исследованных выборках трепанга Apostichopus japonicus с учетом нуль-аллелей
| Выборка | N | Ho | He | FIS | FISIIM |
|---|---|---|---|---|---|
| Vos | 15.2 | 0.481 | 0.638 | 0.254 | 0.1132 (0–0.2919) |
| Krs | 12.2 | 0.494 | 0.560 | 0.107 | 0.0794 (0–0.2162) |
| Pos15 | 28.4 | 0.405 | 0.551 | 0.224 | 0.0860 (0–0.2097) |
| Fed | 16 | 0.551 | 0.612 | 0.085 | 0.1056 (0–0.2359) |
| Pos18 | 14.8 | 0.376 | 0.513 | 0.204 | 0.1001 (0–0.2833) |
Средние значения Ho и He до и после корректировки значительно различались, наблюдаемая гетерозиготность была меньше ожидаемой. Коэффициент инбридинга, полученный после корректировки генотипов в программе MICRO-CHECKER и с учетом нуль-аллелей в программе INEST 2.2, различался для некоторых выборок более чем на 0.1.
ОБСУЖДЕНИЕ
Генетическая изменчивость дальневосточного трепанга
При использовании микросателлитных локусов для исследования популяционно-генетической структуры видов возникает вопрос о необходимом и достаточном числе локусов. Некоторые исследователи считают, что, чем их больше, тем лучше. Однако показано, что для изучения размера популяции дикого кабана (Kolodziej et al., 2012) число маркёров можно сократить до четырех без изменения результата; в работе по определению минимального количества микросателлитных локусов, необходимых для оценки генетической структуры популяции (Arthofer et al., 2018), генетическая дифференциация групп особей определяется даже при использовании всего двух локусов. Наше исследование является предварительной оценкой генетического разнообразия дальневосточного трепанга в зал. Петра Великого (Японское море) с изучением влияния нулевых аллелей на полученные данные, поэтому использование пяти локусов можно считать достаточным.
Согласно полученным результатам, среднее значение Ho для всех выборок Apostichopus japonicus из зал. Петра Великого и локусов составило 0.439 ± 0.032, He = 0.563 ± 0.030. В других работах наблюдаемая гетерозиготность была также меньше ожидаемой: Ho = 0.378, He = 0.850 (Chang et al., 2009) и Ho = 0.537, He = 0.634 (Kanno et al., 2006), при этом особи были взяты из разных районов Японского моря и побережья Тихого океана. В работе Кима с коллегами (Kim et al., 2008) указано, что Ho = 0.576, He = 0.761; авторы связывают это с недостатком гетерозигот. В целом можно говорить о дефиците гетерозигот для изучаемых групп особей A. japonicus.
Во всех исследованных нами выборках значения FIS были выше нуля, что согласуется со значениями Ho и He. В работах других авторов этот коэффициент также имел положительные значения: для нескольких особей из зал. Петра Великого, собранных у г. Владивостока, FIS = 0.523 (Chang et al., 2009); для популяций трепанга из б. Тояма FIS = 0.347 (Soliman et al., 2012), а для трепанга из прибрежья Южной Кореи FIS = 0.260 (Kim et al., 2008).
В нашем случае отклонение от равновесия Харди−Вайнберга выявлено в каждой выборке A. japonicus: для Krs минимально – в локусе AJ21199, а для Vos – во всех локусах, кроме AJ20385. В работе Кима с коллегами (Kim et al., 2008) по исследованию популяционной структуры дальневосточного трепанга у берегов Южной Кореи 8 из 9 локусов демонстрировали отклонение от равновесия Харди−Вайнберга. При изучении A. japonicus, отловленного в водах Японии, Южной Кореи, Китая и Дальнего Востока России, Чанг с соавторами (Chang et al., 2009) отмечали, что для выбранных локусов в 85% случаев наблюдалось отклонение от равновесия. В работе южнокорейских ученых (An et al., 2013) для дикой популяции трепанга показана такая же ситуация с отклонением от равновесия Харди−Вайнберга по выбранным маркёрам.
Возможными причинами различия в значениях наблюдаемой и ожидаемой гетерозиготности по отклоняемым от равновесия Харди−Вайнберга локусам называют эффект Валунда, наличие нулевых аллелей и выпуск в акватории искусственно выращенного трепанга (Chang et al., 2009). Дефицит гетерозигот для дикой популяции трепанга Ан с соавторами (An et al., 2013) объясняют наличием нераспознанных нулевых аллелей, естественным отбором, действующим на генетические маркеры, близкородственным скрещиванием и возможным эффектом Валунда, а также сочетанием этих причин. Авторы другой работы (см.: Chen et al., 2008) значительные отклонения от равновесия Харди−Вайнберга для дикой популяции трепанга связывают с большим количеством нулевых аллелей, что частично объясняет недостаток гетерозигот.
Дефицит гетерозигот в нашем случае может быть обусловлен антропогенными факторами, когда при искусственном разведении для воспроизводства используется ограниченное количество особей. Однако хозяйств марикультуры в Приморье немного и их влияние на генетические параметры естественных популяций крайне незначительно.
Более существенной причиной может быть незаконный (браконьерский) лов трепанга, который в последние десятилетия сказывается на естественных популяциях. В акватории Приморья ведется наблюдение за изменением численности этой голотурии. В частности, в 2014 и 2018 гг. состояние его популяции оценено в Дальневосточном морском заповеднике (Лысенко и др., 2015, 2018). Было показано, что численность A. japonicus после запрещения браконьерского промысла стабилизировалась и дальневосточный трепанг не находится под угрозой исчезновения. Однако в настоящее время средняя плотность поселений этой голотурии приблизительно в 20 раз ниже, чем до начала незаконного вылова (Лысенко и др., 2018). При снижении численности особей в акваториях снижается и эффективный размер популяции, затрудняется поиск партнеров для размножения, а это приводит к повышению уровня инбридинга.
Другой причиной отклонения от равновесия Харди−Вайнберга может быть присутствие нулевых аллелей, влияние которых обсуждается ниже.
Влияние нуль-аллелей на генетические данные
Все использованные программы рассчитывают частоты нулевых аллелей и корректируют один или несколько статистических показателей. Рассчитанные частоты нуль-аллелей во всех программах были приблизительно одинаковы.
В программе FreeNA оценка частоты нулевых аллелей для каждого локуса и популяции проводится в соответствии с EM-алгоритмом (Dempster et al., 1977). С помощью этой программы можно не только оценивать частоту нуль-аллелей, но и корректировать долю изменчивости в субпопуляции по отношению к общей генетической изменчивости, так как показано, что присутствие нулевых аллелей влияет на генетическую дистанцию между популяциями (Chapius, Estoup, 2007).
Основное назначение INEST 2.2 – оценка коэффициента инбридинга, исходя из оценок наблюдаемой и ожидаемой гетерозиготности с поправкой на нулевые аллели. Программа предлагает два подхода (Chybicki, Burczyk, 2009): PIM (модель популяционного инбридинга, или максимальная вероятность) и IIM (модель индивидуального инбридинга, или байесовский подход). Подсчет коэффициента инбридинга возможен только для всех локусов в рамках одной выборки, вычисление FIS для отдельного локуса не проводится.
MICRO-CHECKER рассчитывает частоту нулевых аллелей по методам, описанным Чакраборти с соавторами (Chakraborty et al., 1992) и Брукфилдом (Brookfield, 1996) с использованием двух уравнений, а также Ван Остерхоутом с соавторами (Van Oosterhout et al., 2004). Особенностью программы является корректировка генотипов после обнаружения нулевых аллелей. Причем пользователь самостоятельно может выбрать метод оценки нуль-аллелей, с помощью которого будет произведено исправление генотипов. На основе наших данных в MICRO-CHECKER нулевые аллели определены в случаях, когда величины наблюдаемой и ожидаемой гетерозиготности значительно различались (для локусов AJ20333 выборки Vos, AJ13361 выборок Vos, Pos15 и Pos18, AJ20385 группы особей выборки Pos18).
При исследовании популяций дальневосточного трепанга Канно с коллегами (Kanno et al., 2006) также столкнулись с наличием нулевых аллелей в разных микросателлитных локусах. Они скорректировали частоты аллелей во избежание статистических ошибок, однако до и после коррекции большинство результатов не различалось из-за большого количества локусов, использованных в этом исследовании.
В нашей работе нулевые аллели выявлены не во всех локусах и не во всех исследованных выборках трепанга. Из пяти микросателлитных локусов три локуса (AJ13361, AJ2033, AJ20385) показали наличие нулевых аллелей в разных группах. В целом присутствие нулевых аллелей в 1.5–2 раза снижало значение наблюдаемой гетерозиготности и вело к отклонению от равновесия Харди−Вайнберга. Для локуса AJ13361 в популяции Pos18 после корректировки генотипов отклонение от равновесия не отмечено. Следовательно, не во всех случаях равновесие Харди−Вайнберга нарушено из-за дефицита гетерозигот, на него влияют также ошибки генотипирования.
При сравнении коэффициентов инбридинга для средних значений генетической изменчивости с корректировкой по нуль-аллелям и без нее данные с исправленными генотипами демонстрировали меньшие величины, более близкие к 0. Рассчитанный в двух разных программах (MICRO-CHECKER и INEST 2.2)FIS с учетом нуль-аллелей по всем локусам для некоторых выборок различался более чем на 0.2. Различие значений связано с тем, что INEST 2.2 корректирует коэффициент инбридинга даже при минимальных частотах нуль-аллелей, в то время как MICRO-CHECKER корректирует генотипы только для тех локусов, для которых частота нулевых аллелей превышает допустимый уровень с учетом методов подсчета.
Проверка наличия и эффекта нуль-аллелей на разные генетические параметры с использованием определенных аналитических инструментов проведена в некоторых исследованиях (Kalinowski, Taper, 2006; Chapius, Estoup, 2007; Girard, 2011; De Meeûs, 2018), однако лишь в немногих из них эмпирически проверено это влияние (Girard, Angers, 2008; Dąbrowski et al., 2015; Rico et al., 2017). Опубликованы работы, оценивающие воздействие нулевых аллелей путем изменения дизайна праймеров и сравнения исходных результатов с результатами “новых” праймеров, свободных от нулевых аллелей (Lemer et al., 2011).
Таким образом, в настоящее время проблема нулевых аллелей решается разными способами: меняется дизайн праймеров, корректируются частоты аллелей и генотипов на основе предполагаемых частот нулевых аллелей; иногда определенные локусы исключают из популяционного анализа. Последний метод коррекции не всегда применим при масштабных исследованиях, так как при увеличении выборок возрастает вероятность появления нулевых аллелей на каждый локус, что может привести к исключению большого числа локусов из популяционного анализа и отрицательно сказаться на качестве исследования.
На основе полученных результатов для дальнейшей работы с применением данного типа маркеров мы планируем использовать MICRO-CHECKER с возможностью выбора метода оценки нуль-аллелей и их корректировки. Данный подход поможет избежать исключения локусов с наиболее часто встречаемой ошибкой генотипирования, что особенно актуально для видов, не являющихся промысловыми объектами. Для подсчета коэффициента инбридинга для всех локусов в рамках одной выборки мы рекомендуем программу INEST 2.2; при использовании программы FreeNA можно не только оценивать частоту, но и скорректировать генетическую дистанцию между популяциями с учетом нуль-аллелей.
Применение статистических подходов при изучении популяционной генетики A. japonicus, в частности генетического разнообразия и генетической дифференциации данной голотурии, мы считаем необходимым шагом на пути к оценке состояния диких популяций дальневосточного трепанга, который является важным промысловым объектом.
Список литературы
Гаврилова Г.С. Товарное выращивание дальневосточного трепанга // Владивосток: ТИНРО-Центр. 2013. 99 с.
Галинская Т.В., Щепетов Д.М., Лысенков С.Н. Предубеждения о микросателлитных исследованиях и как им противостоять // Генетика. 2019. Т. 55. № 6. С. 617–632. https://doi.org/10.1134/S0016675819060043
Лысенко В.Н., Жариков В.В., Лебедев А.М. Численность и распределение дальневосточного трепанга Apostichopus japonicus (Selenka, 1867) (Echinodermata: Stichopodidae) в прибрежной зоне южного участка Дальневосточного морского заповедника ДВО РАН // Биол. моря. 2015. Т. 41. № 2. С. 146–149.
Лысенко В.Н., Жариков В.В., Лебедев А.М., Долганов С.М. Современное состояние популяции дальневосточного трепанга Аpostichopus japonicus в Дальневосточном морском заповеднике // Биол. моря. 2018. Т. 44. № 2. С. 133–139.
Селин Н.И. Вертикальное распределение дальневосточного трепанга Apostichopus japonicus в заливе Восток Японского моря // Биол. моря. 2001. Т. 27. № 4. С. 297–299.
Abdul-Muneer P.M. Application of microsatellite markers in conservation and fisheries management: recent advances in population structure analysis and conservation strategies // Genet. Res. Int. 2014. V. 2014. Art. ID 691759. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3997932. https://doi.org/10.1155/2014/691759
An H.S., Lee J.W., Hong S.W. et al. Genetic differences between wild and hatchery populations of red sea cucumber (Stichopus japonicus) inferred from microsatellite markers: implications for production and stocking programs design // Genes Genomics. 2013. V. 35. P. 709–717. https://doi.org/10.1007/s13258-013-0139-8
Arthofer W., Heussler C., Krapf P. et al. Identifying the mi-nimum number of microsatellite loci needed to assess population genetic structure: a case study in fly cultu-ring // Fly. 2018. V. 12. № 1. P. 13–22. https://doi.org/10.1080/19336934.2017.1396400
Brookfield J.F.Y. A simple new method for estimating null allele frequency from heterozygote deficiency // Mol. Ecol. 1996. V. 5. P. 453−455. https://doi.org/10.1111/j.1365-294X.1996.tb00336.x
Carlsson J. Effects of microsatellite null alleles on assignment testing // J. Hered. 2008. V. 99. № 6. P. 616–623. https://doi.org/10.1093/jhered/esn048
Chakraborty R., de Andrade M., Daiger S.P., Budowle B. Apparent heterozygote deficiencies observed in DNA typing data and their implications in forensic applications // Ann. Hum. Genet. 1992. V. 56. P. 45–57. https://doi.org/10.1111/j.1469-1809.1992.tb01128.x
Chang Y., Feng Z., Yu J., Ding J. Genetic variability analysis in five populations of the sea cucumber Stichopus (Apostichopus) japonicus from China, Russia, South Korea and Japan as revealed by microsatellite markers // Mar. Ecol. 2009. V. 30. P. 455–461. https://doi.org/10.1111/j.1439-0485.2009.00292.x
Chapuis M.-P., Estoup A. Microsatellite null alleles and estimation of population differentiation // Mol. Biol. Evol. 2007. V. 24. P. 621–631. https://doi.org/10.1093/molbev/msl191
Chen L., Li Q., Yang J. Microsatellite genetic variation in wild and hatchery populations of the sea cucumber (Apostichopus japonicus Selenka) from northern China // Aquacult. Res. 2008. V. 39. P. 1541–1549. https://doi.org/10.1111/j.1365-2109.2008.02027.x
Chen M., Gao L., Zhang W. et al. Identification of forty-five gene-derived polymorphic microsatellite loci for the sea cucumber, Apostichopus japonicus [Электронный ресурс] // J. Genet. 2013. V. 92. Art. e31–35. http://www.ias.as.in/jgenet/OnlineResources/92/e31.pdf. https://doi.org/10.1007/s12041-013-0234-2
Chybicki I., Burczyk J. Simultaneous estimation of null alleles and inbreeding coefficients // J. Hered. 2009. V. 100. № 1. P. 106–113. https://doi.org/10.1093/jhered/esn088
Dąbrowski M.J., Bornelöv S., Kruczyk M. et al. 'True' null allele detection in microsatellite loci: a comparison of methods, assessment of difficulties and survey of possible improvements // Mol. Ecol. Resour. 2015. V. 15. № 3. P. 477–488.
Dakin E.E., Avise J.C. Microsatellite null alleles in paren-tage analysis // Heredity. 2004. V. 93. № 5. P. 504–509. https://doi.org/10.1038/sj.hdy.6800545
De Meeûs T. Revisiting Fis, Fst, Wahlund effects, and null alleles // J. Hered. 2018. V. 109. № 4. P. 446–456. https://doi.org/10.1093/jhered/esx106
Dempster A.P., Laird N.M., Rubin D.B. Maximum likelihood from incomplete data via the EM algorithm // J. R. Stat. Soc. Ser. B. 1977. V. 39. № 1. P. 1–38. https://doi.org/10.1111/j.2517-6161.1977.tb01600.x
Dong Y., Li Q., Zhong X., Kong L. Development of gene-derived SNP markers and their application for the assessment of genetic diversity in wild and cultured populations in sea cucumber, Apostichopus japonicus // J. World Aquacult. Soc. 2016. V. 47. № 6. P. 873–888. https://doi.org/10.1007/s12686-013-9858-z
Du H., Bao Z., Yan J. et al. Development of 101 gene-based single nucleotide polymorphism markers in sea cucumber, Apostichopus japonicus // Int. J. Mol. Sci. 2012. V. 13. P. 7080–7097. https://doi.org/10.3390/ijms13067080
Ellegren H. Microsatellites: simple sequences with complex evolution // Nat. Rev. Genet. 2004. V. 5. P. 435–445. https://doi.org/10.1038/nrg1348
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Girard P. A robust statistical method to detect null alleles in microsatellite and SNP datasets in both panmictic and inbred populations // Stat. Appl. Genet. Mol. Biol. 2011. V. 10. Art. 9. https://pubmed.ncbi.nlm.nih.gov/21381434/.https://doi.org/10.2202/1544-6115.1620
Girard P., Angers B. Assessment of power and accuracy of methods for detection and frequency-estimation of null alleles // Genetica. 2008. V. 134. № 2. P. 187–197. https://doi.org/10.1007/s10709-007-9224-8
Grimaldi M.-C., Crouau-Roy B. Microsatellite allelic homoplasy due to variable flanking sequences // J. Mol. Evol. 1997. V. 44. P. 336–340. https://doi.org/10.1007/PL00006151
Huang K., Ritland K., Dunn D.W. et al. Estimating relatedness in the presence of null alleles // Genetics. 2016. V. 202. P. 247–260. https://doi.org/10.1534/genetics.114.163956
Kalinowski S.T., Taper M.L. Maximum likelihood estimation of the frequency of null alleles at microsatellite loci // Conserv. Genet. 2006. V. 7. P. 991–995. https://doi.org/10.1007/s10592-006-9134-9
Kanno M., Suyama J., Li Q., Kijima A. Microsatellite ana-lysis of Japanese sea cucumber, Stichopus (Apostichopus) japonicus, supports reproductive isolation in color variants // Mar. Biotechnol. 2006. V. 8. P. 672–685. https://doi.org/10.1007/s10126-006-6014-8
Kim M., Choi T., An H.S. Population genetic structure of sea cucumber, Stichopus japonicus in Korea using microsa-tellite markers // Aquacult. Res. 2008. V. 39. P. 1038–1045. https://doi.org/10.1111/j.1365-2109.2008.01962.x
Kolodziej K., Theissinger K., Brün J. et al. Determination of the minimum number of microsatellite markers for individual genotyping in wild boar (Sus scrofa) using a test with close relatives // Eur. J. Wildl. Research. 2012. V. 58. P. 621–628. https://doi.org/10.1007/s10344-011-0588-9
Lemer S., Rochel E., Planes S. Correction method for null alleles in species with variable microsatellite flanking regions, a case study of the black-lipped pearl oyster Pinctada margaritifera // J. Hered. 2011. V. 102. № 2. P. 243–246. https://doi.org/10.1093/jhered/esq123
Oh G.-W., Ko S.-C., Lee D.H. et al. Biological activities and biomedical potential of sea cucumber (Stichopus japo-nicus): a review // Fish. Aquat. Sci. 2017. V. 20. Art. 28. https://doi.org/10.1186/s41240-017-0071-y
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Exel. Population genetic software for teaching and research − an update // Bioinformatics. 2012. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Rico C., Cuesta J.A., Drake P. et al. Null alleles are ubiquitous at microsatellite loci in the Wedge Clam (Donax trunculus) // PeerJ. 2017. V. 5. Art. ID e3188. https://peerj.com/articles/3188/.https://doi.org/10.7717/peerj.3188
Selenka E. Beiträge zur Anatomie und Systematik der Holothurien // Z. Wiss. Zool. 1867. V. 17. P. 291–374.
Soliman T., Kanno M., Kijima A., Yamazaki Y. Population genetic structure and gene flow in the Japanese sea cucumber Apostichopus japonicus across Toyama Bay, Japan // Fish. Sci. 2012. V. 78. P. 775–783. https://doi.org/10.1007/s12562-012-0509-1
Truett G.E. Preparation of genomic DNA from animal tissues // DNA sequencing II: Optimizing preparation and cleanup. Boston: Jones and Bartlett Publishers. 2006. Ch. 3. P. 33–46.
Van Oosterhout C., Hutchinson W.F., Wills D.P.M., Shipley P. MICRO-CHECKER: software for identifying and correcting genotyping errors in microsatellite data // Mol. Ecol. Notes. 2004. V. 4. P. 535–538. https://doi.org/10.1111/j.1471-8286.2004.00684.x
Дополнительные материалы отсутствуют.