Цитология, 2020, T. 62, № 1, стр. 56-63
Детекция микоплазм в клеточных линиях эукариот методом ПЦР в реальном времени с использованием различных способов концентрирования образца
А. А. Чапленко 1, *, О. В. Меркулова 1, И. С. Семенова 1, А. Р. Сайфутдинова 1, Е. В. Мельникова 1, В. А. Меркулов 1
1 Научный центр экспертизы средств медицинского применения Минздрава РФ
127051 Москва, Россия
* E-mail: a.a.chaplenko@yandex.ru
Поступила в редакцию 05.09.2019
После доработки 10.10.2019
Принята к публикации 15.10.2019
Аннотация
Детекция микоплазменной контаминации – важный этап контроля качества клеточных линий эукариот, как используемых при производстве биотехнологических лекарственных препаратов, так и входящих в состав биомедицинских клеточных продуктов. Предложенные в Государственной Фармакопее методы анализа: микробиологический (культуральный) и цитохимический (индикаторной клеточной культуры) выполняются в течение нескольких суток, сложно стандартизуются, а их результат субъективно интерпретируется оператором. Альтернативные молекулярно-генетические методы анализа, наиболее широко используемым из которых является ПЦР, отличаются экспрессностью, однако менее чувствительны. Для решения данной проблемы используют предварительное концентрирование образца клеточной линии. В настоящем исследовании мы сравнили эффективность различных способов пробоподготовки образца, оценили влияние его состава на параметры процесса концентрирования, а также провели валидацию комбинированной методики детекции на примере двух видов микоплазм: Mycoplasma arginini и Acholeplasma laidlawii.
Контаминация клеточных линий бактериями класса Mollicutes, в том числе микоплазмами, ахолеплазмами и уреаплазмами, является одной из наиболее часто встречающихся проблем при культивировании клеток эукариот. Согласно литературным данным, доля контаминированных линий варьируется от 15 до 80% в зависимости от типа клеток, источника их получения и методов культивирования (Langdon, 2004). Продукты на основе соматических клеток человека перед введением пациенту обязательно должны быть протестированы на присутствие микоплазм.
В ГФ XIV (ОФС.1.7.2.0031.15 “Испытание на присутствие микоплазм”), Европейской Фармакопее (раздел 2.6.7.) и Фармакопее США (раздел 63) описаны методы детекции данного контаминанта: микробиологический (культуральный) и цитохимический (индикаторной клеточной культуры). Также заявлены требования к чувствительности методов детекции на уровне 10–100 КОЕ/мл.
Ключевое преимущество традиционного культурального метода детекции микоплазм – высокая чувствительность, возможность обнаружить единичные жизнеспособные клетки микроба. Однако сложность стандартизации, субъективная интерпретация результатов, а также необходимость длительной инкубации сводят к минимуму удобство использования метода и достоверность получаемых данных.
В качестве альтернативы исследователями предложены методики детекции микоплазм, основанные на использовании полимеразной цепной реакции (ПЦР), как с электрофоретической детекцией (van Kuppeveld et al., 1992, 1994; Ossewaarde et al., 1996; Kong et al., 2000, 2001; Bruchmüller et al., 2006), так и с детекцией в реальном времени (Jurstrand et al., 2005; Ishikawa et al., 2006; Dumke et al., 2007; Störmer et al., 2009). Разработано большое количество коммерческих наборов для ПЦР детекции микоплазм, как российского (“АмплиСенС”, “МИК-КОМ”), так и зарубежного (“Microsart ATMP Mycoplasma”, “E-myco”, “LookOut Mycoplasma PCR Detection Kit” и др.) производства. Заявленный предел обнаружения ПЦР-методик составляет 1–10 копий генов микоплазмы в 1 мкл образца, однако неясным остается соотношение числа копий ДНК в пробе и КОЕ микоплазмы в 1 мл исходного образца. Также остается без внимания влияние на чувствительность анализа матрицы пробы – компонентов питательных сред и самих клеток, в том числе погибших.
Для достижения в ПЦР методиках высокой чувствительности анализа целесообразно концентрирование образцов и предварительное выделение ДНК из пробы. Потери аналита на каждом из этапов анализа вносят свой вклад в общую правильность и воспроизводимость методики.
Целью настоящей работы является оценка чувствительности детекции микоплазм в клеточных линиях методом ПЦР при использовании различных способов пробоподготовки, а также валидация предложенных методик.
МАТЕРИАЛ И МЕТОДИКА
Образцы контаминантов. В работе использовали лиофилизированный стандартный образец Mycoplasma arginini (107 КОЕ/мл, концентрация определена до лиофилизации), восстановленный в стандартном фосфатно-солевом буфере, а также образец штамма Acholeplasma laidlawii PG8 в среде Каган жидкой с концентрацией 107 КОЕ/мл. Образцы микоплазм предоставлены лабораторией бактериологических питательных сред и культур клеток ФГБУ “НЦ ЭСМП” МЗ РФ. Для приготовления разведений использовали фосфатно-солевой буфер.
Клеточные линии. Для создания моделей образцов, зараженных микоплазмами, использовали суспензии клеточных линий DF-2 (получена из коллекции клеточных культур Института цитологии РАН), Jurkat-PD1-NFAT, Vero (получены из коллекции клеточных культур ATCC®). Клеточные линии культивировали в течение 5 сут в соответствии с рекомендациями производителя. Для приготовления суспензий с различными концентрациями клеток исходные образцы центрифугировали (2000 g, 5 мин), удаляли надосадочную жидкость, осадок ресуспендировали в фосфатно-солевом буфере.
Приготовление образцов. К суспензиям клеточных линий добавляли разведения M. arginini и A.laidlawii до достижения содержаний микоплазм в диапазоне 1–1000 КОЕ/мл. Затем образцы подвергали термической инактивации (95°С, 10 мин) с целью предотвращения риска распространения инфекции в помещениях лаборатории, а также для поддержания постоянной концентрации контаминанта.
Концентрирование образцов и выделение ДНК. Концентрирование (25×) производили двумя способами: центрифугированием 5 мл испытуемого образца (12000 g, 10 мин) для осаждения отдельных клеток микоплазмы или центрифугальной ультрафильтрацией с использованием концентратора Vivaspin 6100 кДа (Sartorius, Германия) (5000 g, 3 мин). 200 мкл приосадочной жидкости (включая осадок) использовали для выделения ДНК набором реагентов Microsart AMP Extraction (Sartorius, Германия) в соответствии с инструкцией производителя. В процессе экстракции получали 50 мкл раствора, содержащего ДНК испытуемых образцов. Таким образом, в ходе пробоподготовки испытуемый образец концентрировали в 100 раз (5 мл → 50 мкл).
Измерение концентрации ДНК. ДНК в пробах определяли спектрофотометрически (A260) на приборе NanoDrop 2000 (ThermoFisherScientific, США).
Проведение ПЦР анализа. ПЦР-анализ проб на присутствие микоплазмы проводили с использованием тест-системы “Microsart ATMP Mycoplasma” (Sartorius, Германия) (детекция в реальном времени) в соответствии с инструкцией производителя (объем пробы 10 мкл). Амплификацию проб и детекцию флуоресцентного сигнала проводили на приборе Bio-Rad CFX96 (Eppendorf, США).
Статистическая обработка результатов. Статистическую обработку результатов проводили с использованием MS Office Excel 2007. Для оценки статистической значимости различий независимых выборок количественных данных использовали двухвыборочный t-критерий Стьюдента для независимых выборок. Для оценки статистической значимости различий независимых выборок биномиальных данных использовали точный критерий Фишера.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Оценка соотношения числа копий генома и КОЕ микоплазмы. Результаты использования фармакопейных методов детекции микоплазмы представлены в единицах КОЕ, в то время как альтернативные молекулярно-биологические методы (к числу которых относится ПЦР) основаны на детекции копий генома микоплазм, как живых, так и убитых. Согласно данным исследований (Ёлшин, Петров, 2017; Peredeltchouk et al., 2011; Volokhov et al., 2011), 1 КОЕ/мл микоплазм соответствует содержанию в 1 мл от нескольких единиц до нескольких тысяч их копий генома, данное соотношение варьирует в зависимости от конкретного вида микроба, способа выделения нуклеиновых кислот и используемых стандартных образцов. Таким образом, актуально проведение оценки вышеназванного соотношения для образцов и наборов, используемых в настоящем исследовании.
На первом этапе готовили разведения стандартных образцов M. arginini и A. laidlawii, проводили концентрирование центрифугированием с использованием концентратора Vivaspin 6 100 kDa и выделение ДНК из образцов, после чего определяли содержание ДНК в пробах. Согласно литературным данным, геном A. laidlawii содержит ~1.5 млн пар оснований (Lazarev et al., 2011), что соответствует массе 1 копии генома, равной 2 × 10–15 г; геном M. arginini содержит ~600 тыс. пар оснований (Adeniji et al., 2018), масса 1 копии равна 8 × 10–16 г.
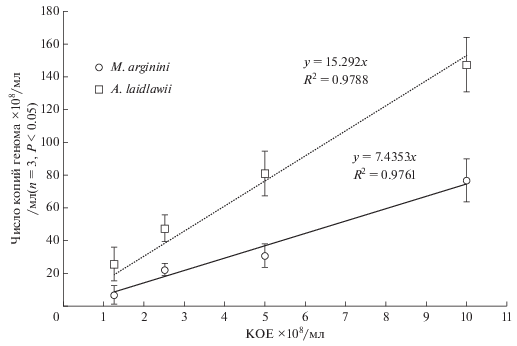
Соотношение рассчитанных (с учетом выделения и концентрирования) усредненных чисел КОЕ и чисел копий генома/мл пробы для обоих видов исследуемых микроорганизмов представлено на рис. 1.
Рис. 1.
Оценка соотношения числа КОЕ и копий генома М. arginini и A. laidlawii в анализируемых образцах. Линейная аппроксимация экспериментальных значений получена методом наименьших квадратов, значение тангенса угла наклона равно среднему числу геномных копий микроба, соответствующих 1 КОЕ: 7.4 – для М. arginini, 15.3 – для A. laidlawii.
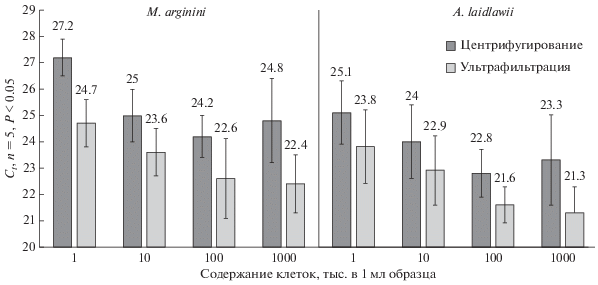
Сравнение эффективности способов концентрирования образца. Современные наборы для ПЦР-детекции микоплазм позволяют обнаружить единичные копии генома микоплазм в пробе, однако объем пробы, как правило, не превышает 10 мкл. С учетом случайной вариации (распределение Пуассона) количества копий, отбираемых из образца и вносимых в реакцию, достигаемый предел обнаружения составляет ~500–1000 копий генома/мл. Принимая во внимание рассчитанное выше соотношение копий генома и КОЕ, можно с большой долей уверенности заключить, что без использования дополнительного концентрирования образца чувствительность ПЦР-анализа ниже требуемой Фармакопеями.
Для достижения предела обнаружения микоплазмы, соответствующего фармакопейным требованиям, в настоящей работе проводили предварительное концентрирование испытуемых образцов – суспензий клеточной линии DF-2, 3 × 105 клеток/мл (соответствует примерному содержанию клеток в продуктах клеточной терапии), в которые внесены микроколичества микоплазм, соответствующие содержаниям 1–10 КОЕ/мл. Концентрирование проводили одним из двух способов: центрифугированием при 12 000 g (для осаждения клеток микоплазм) с последующим отбрасыванием супернатанта или центрифугальной ультрафильтрацией с использованием концентратора VivaSpin 6 100 kDa. При использовании концентратора в пробе сохраняются все вещества и частицы с молекулярной массой выше 100 кДа, однако некоторые потери возможны в случае сорбции частиц образца на материале фильтра.
После выделения ДНК из концентратов образцов проводили ПЦР в реальном времени детекцию М. arginini и A. laidlawii, результаты которой представлены на рис. 2.
Рис. 2.
Результаты ПЦР в реальном времени анализа концентрированных с помощью ультрафильтрации и центрифугирования образцов клеточных линий, содержащих M. arginini и A. laidlawii в концентрации 101–103 КОЕ/мл.
Согласно полученным данным, каждый из предложенных способов концентрирования образцов обеспечивает надежную детекцию M. arginini и A. laidlawii при их содержании, равном 10 КОЕ/мл. Средние значения порогового цикла (Сt) проб, содержащих A. laidlawii, статистически значимо меньше величин Сt соответствующих образцов с M. arginini, что связано с бо́льшим количеством копий генома, соответствующих 1 КОЕ для данного вида микоплазм.
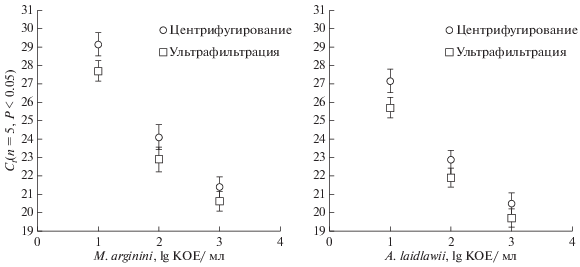
Эффективность концентрирования каждым из способов сравнивали косвенно по значению Сt. Показано, что в области высоких концентраций контаминанта (100–1000 КОЕ/мл) как для M. arginini, так и для A. laidlawii, ультрафильтрация и центрифугирование примерно одинаково эффективны, в то время как при минимальном содержании микоплазм в образце (10 КОЕ/мл) использование концентратора VivaSpin позволяет минимизировать потери аналита. Особенно заметным данное преимущество становится при анализе образцов, содержащих ультранизкие концентрации микоплазм (менее 10 КОЕ/мл) (рис. 3).
Рис. 3.
Результаты анализа ПЦР в реальном времени образцов клеточных линий, концентрированных с помощью ультрафильтрации и центрифугирования, содержащих M. arginini и A. laidlawii в концентрациях 1–5.0 КОЕ/мл.
Данные результаты, несмотря на малый размер выборки, свидетельствуют о статистически значимом превосходстве способа ультрафильтрации для отдельных видов и концентраций микоплазмы в образце (Р < 0.1).
Расчет валидационных параметров методики. В качестве ключевых валидационных параметров аналитических методик детекции посторонних примесей, нормируемых по пределу содержания (к которым относятся, в т. ч. микоплазмы), в соответствии с ГФ XIV и EP 9.0, рассматривают “Специфичность” и “Предел обнаружения”. Специфичность методик ПЦР-детекции микоплазм определяется селективностью связывания праймеров, входящих в тест-системы и валидируется на стадии разработки таких наборов. Для используемой в настоящем исследовании тест-системы производителем подтверждена специфичность в отношении отдельного набора видов микоплазм, поэтому дополнительно данный параметр не оценивали.
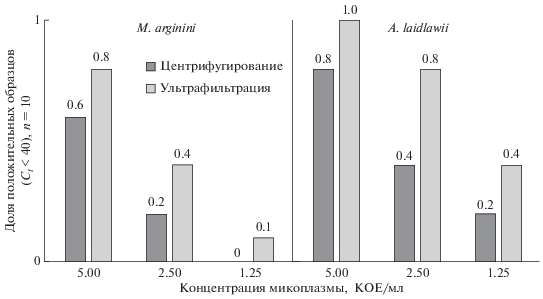
Предел обнаружения (limit of detection, LOD) методик ПЦР-анализа невозможно оценить по соотношению “сигнал–шум”, поскольку сигнал холостого образца отсутствует. Подходом, позволяющим избежать подобного выбора, является вероятностный способ оценки предела обнаружения. Для метода ПЦР существует область ненадежной реакции, где вероятность обнаружения аналита меняется от 0 до 100%. Как было показано ранее, вероятность детекции аналита в образце методом ПЦР в концентрациях, близких к пределу обнаружения описывается функцией распределения Пуассона (Sykes et al., 1992). Таким образом, можно оценить предел обнаружения, равный концентрации, обнаруживаемой с заданной статистически значимой вероятностью (95%). Оценку предела обнаружения проводили для ПЦР в реальном времени – методики обнаружения микоплазм с предварительным концентрированием ультрафильтрацией (рис. 4).
Рис. 4.
Оценка вероятности детекции микоплазмы в образцах в области ненадежной реакции (1–10 КОЕ/мл).
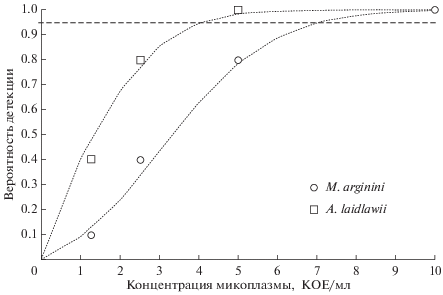
Предел обнаружения M. arginini, таким образом, составляет 7 КОЕ/мл, A. laidlawii − 4 КОЕ/мл. Оба значения удовлетворяют требованиям EP, USP и ГФ.
Влияние состава испытуемых образцов на чувствительность методики детекции контаминанта. Используемая питательная среда, концентрация клеток, состав их секретома и другие параметры образца могут влиять как на процессы концентрирования и выделения микоплазменной ДНК, так и на эффективность еe амплификации (Morozova et al., 2017).
Для оценки влияния концентрации клеток в образце на эффективность детекции микоплазмы были подготовлены и проанализированы суспензии клеток DF-2 (103–106 клеток/мл) с внесенными M. arginini и A. laidlawii (100 КОЕ/мл) (рис. 5).
Экспериментально было установлено, что концентрирование микоплазм из образца наиболее эффективно происходит при содержании клеток в образце порядка 105–106 клеток/мл. Плотный клеточный осадок, образующийся при центрифугировании или ультрафильтрации, надежно удерживает на своей поверхности частицы контаминанта, благодаря чему потери микоплазмы с отделяемой надосадочной жидкостью минимальны. При меньшем содержании клеток в образце риск потери микроба с супернатантом выше, поэтому чувствительность анализа несколько снижается.
Можно предположить, что при дальнейшем увеличении концентрации клеток чувствительность детекции будет снижаться в связи со снижением эффективности выделения и амплификации генетического материала микоплазмы.
Анализ результатов детекции микоплазм в образцах различных клеточных линий с предварительным концентрированием ультрафильтрацией не выявил статистически значимых различий в чувствительности методики (табл. 1).
Таблица 1.
Результаты детекции микоплазм в образцах различных клеточных линий
| Образец клеточной линии, 10 000 клеток/мл | Содержание микоплазмы, КОЕ/мл | Значение Сt (n = 5, Р < 0.05) |
|---|---|---|
| M. arginini | ||
| Jurkat-PD1-NFAT | 10 | 27.4 ± 0.9 |
| Vero | 26.6 ± 1.2 | |
| DF-2 | 27.1 ± 1.4 | |
| Jurkat-PD1-NFAT | 100 | 22.6 ± 0.9 |
| Vero | 21.8 ± 1.3 | |
| DF-2 | 22.6 ± 0.7 | |
| Jurkat-PD1-NFAT | 1000 | 20.1 ± 1.0 |
| Vero | 20.4 ± 0.8 | |
| DF-2 | 20.6 ± 1.0 | |
| A. laidlawii | ||
| Jurkat-PD1-NFAT | 10 | 25.2 ± 1.3 |
| Vero | 24.4 ± 1.3 | |
| DF-2 | 25.8 ± 2.0 | |
| Jurkat-PD1-NFAT | 100 | 22.4 ± 0.8 |
| Vero | 21.6 ± 0.9 | |
| DF-2 | 21.6 ± 0.7 | |
| Jurkat-PD1-NFAT | 1000 | 20.3 ± 0.8 |
| Vero | 20.9 ± 1.1 | |
| DF-2 | 19.9 ± 0.8 | |
Таким образом, в настоящей работе предложена и валидирована в соответствии с фармакопейными требованиями методика обнаружения M. arginini и A. laidlawii в клеточных линиях эукариот. Проведена оценка соотношения числа КОЕ к числу копий генома для представленных образцов микоплазм. Подобраны оптимальные условия пробоподготовки, наивысшая чувствительность анализа достигается в случае предварительного концентрирования образца методом центрифугальной ультрафильтрации. Оптимальное содержание клеток в анализируемой суспензии составляет порядка 105–106 клеток/мл. Показана пригодность методики для анализа нескольких наименований клеточных линий эукариот.
Список литературы
Ёлшин Н.Д., Петров А.В. 2017. Изучение возможности использования метода qPCR для контроля отсутствия микоплазменной контаминации в клеточных культурах. БИОпрепараты. Профилактика, диагностика, лечение. Т. 63. С. 173. (Yolshin N.D., Petrov A.V. 2017. Possibilities of qPCR control of mycoplasma contamination of cell cultures. BIOpreparations. V. 63. P. 173.)
Adeniji J.A., Faleye T.O.C., Adewumi O.M., Olayinka O.A., Donbraye E., Oluremi B., George U.E., Arowolo O.A., Omoruyi E.C., Ifeorah M.I., Akande A. 2018. Draft genome sequence of Mycoplasma arginini strain NGR_2017. Genome Announcements. V. 6. P. 26. https://doi.org/10.1128/genomeA.00577-18
Bruchmüller I., Pirkl E., Herrmann R., Stoermer M., Eichler H., Klüter H., Bugert P. 2006. Introduction of a validation concept for a PCR-based Mycoplasma detection assay. Cytotherapy. V. 8. P. 62.
Dumke R., Schurwanz N., Lenz M., Schuppler M., Lück C., Jacobs E. 2007. Sensitive detection of Mycoplasma pneumoniae in human respiratory tract samples by optimized real-time PCR approach. J. Clin. Microbiology. V.45. P. 2726.
Ishikawa Y., Takaharu K., Morita H., Saida K., Oka S., Masuo Y. 2006. Rapid detection of Mycoplasma contamination in cell cultures using SYBR Greenbased real-time polymerase chain reaction. In Vitro Cell Dev. Biol. Animal. V. 42. P. 63.
Jurstrand M., Jensen J.S., Fredlund H., Falk L., Mölling P. 2005. Detection of Mycoplasma genitalium in urogenital specimens by real-time PCR and conventional PCR assay. J. Med. Microbiology. V. 54. P. 23.
Kong F., Gordon S., Gilbert G.L. 2000. Rapid-cycle PCR for detection and typing of Mycoplasma pneumonia in clinical specimens. J. Clin. Microbiology. V. 38. P. 4256.
Kong F., James G., Gordon S., Zelynski A., Gilbert G.L. 2001. Species-specific PCR for identification of common contaminant Mollicutes in cell culture. Appl. Environ. Microbiology. V. 67. P. 3195.
Langdon S.P. 2004. Cell culture contamination. In: Cancer cell culture. Humana Press. P. 309.
Lazarev V.N., Levitskii S.A., Basovskii Y.I., Chukin M.M., Akopian T.A., Vereshchagin V.V., Kostrjukova E.S., Kovaleva G.Y., Kazanov M.D., Malko D.B., Vitreschak A.G., Sernova N.V., Gelfand M.S., Demina I.A., Serebryakova M.V. et al. 2011. Complete genome and proteome of Acholeplasma laidlawii. J. Bacteriology. V. 193. P. 4943.
Morozova A.V., Borchsenius S.N., Vishnyakov I.E., Malinin A.Y. 2017. Testing the purity of cell cultures using clinical diagnostic PCR kits. Cell Tiss. Biol. V. 11. P. 250.
Ossewaarde J.M., de Vries A., Bestebroer T., Angulo A.F. 1996. Application of a Mycoplasma group-specific PCR for monitoring decontamination of Mycoplasma infected Chlamy-dia sp. strains. Appl. Environ. Microbiology. V. 62. P. 328.
Peredeltchouk M., David S.W., Bhattacharya B., Volokhov D.V., Chizhikov V. 2011. Detection of mycoplasma contamination in cell substrates using reverse transcription-PCR assays. J. Appl. Microbiol. V. 110. P. 54.
Störmer M., Vollmer T., Henrich B., Kleesiek K., Dreier J. 2009. Broad-range real-time PCR assay for the rapid identification of cell-line contaminants and clinically important mollicute species. Int. J. Med. Microbiology. V. 299. P. 291.
Sykes P.J., Neoh S.H., Brisco M.J., Hughes E., Condon J., Morley A.A. 1992. Quantitation of targets for PCR by use of limiting dilution. Biotechniques. V. 13. P. 444.
van Kuppeveld F.J., Johansson K.E., Galama J.M., Kissing J., Bölske G., van der Logt J.T., Melchers W.J. 1994. Detection of mycoplasma contamination in cell cultures by a Mycoplasma group-specific PCR. Appl. Environ. Microbiology. V. 60. P. 149.
van Kuppeveld F.J., van der Logt J.T., Angulo A.F. Van Zoest M.J., Quint W.G., Niesters H.G., Galama J.M., Melchers W.J. 1992. Genus-species-specific identification of mycoplasmas by 16S rRNA amplification. Appl. Environ. Microbiology. V. 58. P. 2606.
Volokhov D.V., Graham L.J., Brorson K.A., Chizhikov V.E. 2011. mycoplasma testing of cell substrates and biologics: review of alternative non-microbiological techniques. Mol. Cell Probes. V. 25. P. 69.
Дополнительные материалы отсутствуют.