Цитология, 2020, T. 62, № 12, стр. 839-850
Хромотрипсис – форма реорганизации клеточного генома
М. А. Шилина 1, *, Т. М. Гринчук 1
1 Институт цитологии РАН
194064 Санкт-Петербург, Россия
* E-mail: shili-mariya@yandex.ru
Поступила в редакцию 27.08.2020
После доработки 03.09.2020
Принята к публикации 03.09.2020
Аннотация
В 2011 г. было открыто явление, изменившее представления об изменчивости генома, процессе онкогенной трансформации и механизмах наследственных заболеваний. Это явление – хромотрипсис, которое представляет из себя массивные хромосомные перестройки, возникающие одномоментно. В Международной системе цитогенетической номенклатуры человека хромотрипсис с 2013 г. определяется как сложные паттерны чередующихся изменений числа копий генов (нормального, увеличенного или с потерей) вдоль хромосомы или хромосомного сегмента. Механизмы и проявления этого феномена в полной мере еще не ясны и с каждым годом интерес исследователей к нему растет. В представленном обзоре мы постарались собрать всю доступную в настоящее время информацию о хромотрипсисе и дать представление об этом явлении. В обзоре описана история обнаружения хромотрипсиса, его распространенность в геномах, причины и механизмы, лежащие в основе этого явления, а также методы его детекции.
Геном, в частности кариотип, является сложнейшей системой, отлаженная работа которой обеспечивает корректное функционирование всего организма. Нарушения в кариотипе, как структурные, так и количественные могут привести к неправильному функционированию генома и стать причиной опухолевой трансформации клеток. Онкогенная клеточная трансформация как правило сопровождается структурными имениями кариотипа (аберрациями, хроматидными разрывами, кольцевыми хромосомами), нарушениями плоидности и наличием других особенностей – появлением дополнительного генетического материала в виде гомогенно окрашиваемых областей на хромосомах, неконденсированных целых хромосом (Heng, Chen, 1985; Heng et al., 1988) или хромосом с нарушенной конденсацией перицентрического гетерохроматина (Vig, Willcourt 1998; Heng et al., 2013), “липких” хромосом, хромосом в виде единичных двунитчатых волокон, ошибок сегрегации, микроядр (Bak et al., 1979). Эти обширные изменения, обнаруженные в геномах раковых клеток, являются результатом поэтапного процесса, в которых мутации накапливаются постепенно с течением времени (Vogelstein et al., 2013).
Одна из форм кариотипической нестабильности связана с фрагментацией хромосом, являющихся следствием разнотипных клеточных дефектов. Усиленная кариотипическая нестабильность клеток, связанная с фрагментацией хромосом, получила название “геномный хаос”. Хаос генома (хаос хромосом или хаос кариотипа) – это феномен быстрой и массовой перестройки клеточного генома с целью выживания генетической системы в условиях кризиса (Ye et al., 2019). Суть геномного хаоса сводится к тому, что хромосомы при определенных условиях сначала разрываются на мелкие и супермелкие фрагменты, а затем эти фрагменты случайным образом соединяются, создавая новую генетическую систему. Часть генетического материала при этом может быть утеряна. Вновь сформированные кариотипы, возникшие путем перестановки исходного генома, демонстрируют новый потенциал для выживания. Хаотичная сборка фрагментированного хромосомного материала сопряжена с изменением положения многих генов и их функций, что может в корне изменить генетический статус клетки. Возникшие изменения могут быть как благоприятными (что случается крайне редко), так и неблагоприятными, следствием чего может стать отбор популяции с клональными свойствами трансформированных злокачественных клеток.
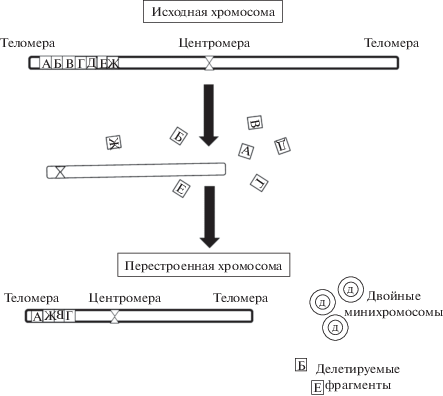
Реорганизация генома с участием одной (или нескольких) хромосом в пределах генома получила называние хромотрипсис. Tripsis в переводе с греческого – разрушение на мелкие части. Хромотрипсис был впервые описан 2011 году Стефансом с коллегами (Stephens et al., 2011), которые обнаружили массивные перестройки генома у пациентов с лимфолейкозом. Авторы столкнулись с необычным случаем, когда у одного из пациентов обнаружили 42 геномные перестройки, локализованные в длинном плече хромосомы 4. Было сделано предложение (Stephens et al., 2011), что эти перестроенные хромосомы возникали в результате одного “катастрофического события”, приводящего к хромосомным разрывам в нескольких точках с последующей случайной повторной сборкой (рис. 1). Огромное число и группировка этих перестроек были неожиданными и противоречили классической картине онкогенеза.
Рис. 1.
Схема хромосомных перестроек при хромотрипсисе. Во время хромотрипсиса хромосома или плечо хромосомы разрушаются с последующим неполным и случайным восстановлением фрагментов (А–Ж). При восстановлении хромосомы ориентация фрагментов может измениться (фрагмент В), часть фрагментов удаляется (фрагменты Б, Е), а часть образует внехромосомные двойные минихромосомы (фрагменты Д). Это приводит к перестроенной хромосоме или фрагменту хромосомы.
До определeнного времени существовала и доминировала мутационная концепция онкогенеза. В ее основе лежит отбор клеточных вариантов, характеризующихся определенными типами кариотипических изменений мутационной природы с последующим селективным отбором клеток с мутантными генами. В настоящее время на модели спонтанной клеточной иммортализации выстроено представление о том, что онкогенная клеточная трансформация проходит 2 стадии – стадию не клональных (случайных) изменений, которые могут исчезнуть стохастически, и стадию клональных (не случайных) изменений, возникающих ступенчато и поддерживаемых селективным клеточным отбором (Heng et al., 2013). Новая теория подчеркивает важность анализа кариотипа как целостной системы. Согласно этому представлению огромную важность в эволюционных преобразованиях генома играют случайные, неспецифические изменения. Наиболее современным является представление о том, что в основе злокачественной трансформации клеток лежат динамические отношения между случайными изменениями в кариотипе и закономерными (Heng et al., 2006). Открытие хромотрипсиса позволило выявить важный “взрывной” механизм быстрой дестабилизации клеточного генома, направленный на реконструирование генетического аппарата и создание новой трансформированной клетки.
Сначала было много дебатов относительно идеи, что подмножество раковых геномов, возможно, не связано с постепенным накоплением мутаций в геноме (Righolt, Mai, 2012; Kinsella et al., 2014), но в настоящее время общепризнанно, что хромотрипсис является широко распространенным мутационным явлением. В 2013 году Корбель и Кэмпбелл (Korbel, Campbell, 2013) предложили следующие параметры хромотриптических изменений в геноме, позволяющие различить перестройки, возникающих в результате хромотрипсиса от ступенчатых изменений ДНК.
1) Профили хромотриптического генома демонстрируют выраженную кластеризацию разрывов ДНК. Эта кластеризация определяется специфическими хромосомными областями, имеющими множественные разрывы в непосредственной близости, окруженные большими участками интактной хромосомной последовательности.
2) В хромотриптических профилях в определенных участках геномной ДНК число копий генов варьирует, но обнаруживаются только два (иногда три) разных состояния числа копий генов.
3) Хромотрипсис может привести к массивной потере фрагментов хромосом. Полученная хромотриптическая хромосома демонстрирует чередующийся паттерн сохранения и потери гетерозиготности между сегментами.
4) Хромотрипсис, как правило, происходит на одной родительской копии (гаплотип) хромосом.
5) Фрагменты хромосом объединяются в случайном порядке и случайно ориентированы. Число различных типов внутри- и межхромосомных структурных перестроек в основном одинаково, и положение фрагментов в хромотриптической области случайно.
6) После хромотрипсиса каждый фрагмент либо сохраняется, либо теряется в новой перестроенной хромосоме.
Как предположили сами авторы (Korbel, Campbell, 2013), эти параметры не являются исчерпывающими, а скорее предназначены для разработки руководящих принципов и подлежат обсуждению. Хромотрипсис был обнаружен лишь сравнительно недавно, и с развитием этой области новые исследования, вероятно, будут генерировать новые идеи, которые потребуют переоценки и корректировки определений. В Международной системе цитогенетической номенклатуры человека хромотрипсис с 2013 г. определяется как сложные паттерны чередующихся изменений числа копий генов (нормального, увеличенного или с потерей) вдоль хромосомы или хромосомного сегмента (Shaffer et al., 2013).
С каждым годом появляется все больше информации о природе хромотрипсиса. Но знания об этом явлении еще не полные и механизмы хромотрипсиса еще до конца не ясны. В представленном обзоре мы опишем распространенность хромотрипсиса в геномах, обсудим его происхождение и механизмы и отдельно остановимся на методах его детекции.
РАСПРОСТРАНЕННОСТЬ ХРОМОТРИПСИСА В ГЕНОМЕ
Исходно хромотрипсис был обнаружен у пациента с лимфолейкозом (Stephens et al., 2011). В связи с этим, начальное изучение хромотрипсиса было сконцентрировано на исследовании раковых больных. Считалось, что хромотрипсис – явление редкое, частота его встречаемости в раковых опухолях составляла 3–5%.
К настоящему времени хромотрипсис обнаружен при раке крови, раке центральной нервной системы, опухолях мягких тканей и карциномах (Rode et al., 2016; Мамаев и др., 2017). При остеосаркоме и глиобластоме его распространенность довольно значительна (Stephens et al., 2011; Malhotra et al., 2013; Kloosterman et al., 2014; Behjati et al., 2017; Erson-Omay et al., 2017). В разных типах рака частота встречаемости хромотрипсиса различна. Однако показазано, что частота встречаемости хромотрипсиса в раковых опухолях значительно выше, чем предполагалось ранее, и у некоторых типов рака достигает 50% (Cortés-Ciriano et al., 2020). Установлено, что встречаемость хромотрипсиса при раке значительно выше у пациентов с наследственными генетическими нарушениями, которые связаны с мутациями генов клеточного цикла и репарации ДНК (Rausch et al., 2012; Ratnaparkhe et al., 2017).
Хромотрипсис обнаружен и в доброкачественных опухолях. Так, было описано явление хромотрипсиса в клетках лейомиомы матки – доброкачественной опухоли миометрия матки, которая характеризуется высокой частотой хромосомных нарушений (Mehine et al., 2013; Holzmann et al., 2014; Mehine et al., 2014), и в менингиоме (Baltus et al., 2019). Интересно, что в образце миомы матки был найден хромотрипсис с делециями (от 43 до 13647 тыс. пар нуклеотидов.). Однако в процессе культивирования клетки этого образца миомы матки характеризовались нормальным кариотипом (Holzmann et al., 2014).
В 2015 г. при изучении геномов пациентов с тяжелыми врожденными аномалиями, у которых наблюдали хромотрипсис (de Pagter et al., 2015), было выявлено, что каждый из пациентов унаследовал хромотрипсис от своей матери. При этом удивляло то, что сами матери были здоровы, хотя имели в значительной степени сбалансированный хромотрипсис в своих геномах. Часть дефектных хромосом от матерей была передана их детям, что и привело к дисбалансу, который, скорее всего, явился причиной их заболевания. Хромотрипсис регулярно наблюдается среди носителей видимых кариотипически сбалансированных хромосомных перестроек. Присутствие хромотрипсиса в геномах здоровых людей, вероятно, очень редкое.
Явление хромотрипсиса было обнаружено и в зародышевых клетках. Интересно отметить, что обнаружены существенные различия между зародышевым хромотрипсисом и хромотрипсисом при раке (Kloosterman, Cuppen, 2013). У пациентов с врожденным заболеванием часто наблюдается более сбалансированная форма хромотрипсиса (Chiang et al., 2012). Число отклонений в зародышевой форме хромотрипсиса обычно ниже, чем при раке (Collins et al., 2017). Одним из объяснений этому является предположение о том, что большая часть отклонений отсеиваются во время развития плода.
Катастрофические комплексные перестройки не ограничиваются видом Homo sapiens. Они были зарегистрированы у тасманского дьявола (Sarcophilus harrisii) (Deakin et al., 2012), нематоды (Caenorhabditis elegans) (Itani et al., 2015), резуховидки Таля (Arabidopsis thaliana) (Tan et al., 2015) и винограда (Vitis vinifera) (Carbonell-Bejerano et al., 2017). В связи с этим можно предположить, что хромотрипсис может служить естественным явлением генетической изменчивости.
Интересно отметить, что вероятность вовлечения в хромотрипсис различных хромосом набора различается. У человека хромотрипто-подобные паттерны чаще выявлялись на хромосомах 8, 11, 12 и 17 при скрининге 22 347 раковых геномов (Cai et al., 2014). При остром миелобластном лейкозе хромотрипсис наблюдается почти во всех хромосомах, при этом чаще всего поражается 7-я хромосома (Rucker et al., 2018). Среди случаев хромотрипсиса, обнаруженных при раке поджелудочной железы, 11% произошли в 18-й хромосоме, а 8% – в 12-й хромосоме (Notta et al., 2017). При остеосаркомах хромотрипсис затрагивал в основном хромосомы 5, 12 и 17 (Behjati et al., 2017). События хромотрипсиса были сконцентрированы в хромосоме 17 в случаях всех подтипов рака молочной железы (Przybytkowski et al., 2014; Chen et al., 2017) и в хромосомах 9 и 12 при глиобластомах (Zack et al., 2013). Более половины (54%) вариантов хромотрипсиса (81 из 150), выявленного при раке мочевого пузыря, были связаны с хромосомами 4, 5 и 6 (Morrison et al., 2014). Хромотрипсис хромосомы 13 является рецидивирующим нарушением миелодиспластических синдромов высокого риска (Abaigar et al., 2016).
С одной стороны, преимущественное вовлечение определенных хромосом в хромотрипсис может быть обусловлено набором генов, локализованных в данных хромосомах. С другой стороны, повышенная распространенность хромотрипсиса в определенных хромосомных областях может быть обусловлена тем, что эти области являются структурно более хрупкими.
ПРИЧИНЫ И МЕХАНИЗМЫ ХРОМОТРИПСИСА
При обсуждении причин и механизмов возникновения хромотрипсиса изначально предполагали, что хромотрипсис является результатом воздействия жестких экзогенных стрессовых факторов (Stevens et al., 2011; Morishita et al., 2016). Однако повреждения ДНК, создаваемые такими триггерами, затрагивают весь геном, а не только одну хромосому или хромосомные области, что наблюдается при хромотрипсисе. Очевидно, что механизмы возникновения хромотрипсиса иные. Механизмы, связывающие клеточные процессы и молекулярные пути с хромотрипсисом, были выявлены недавно. В последние годы было предложено несколько альтернативных механизмов хромотрипсиса, которые, впрочем, не являются взаимоисключающими.
Фрагментация хромосом в микроядрах. Наиболее распространенной гипотезой возникновения хромотрипсиса в настоящее время является фрагментация хромосом в микроядрах. Хромосомы и их ацентрические фрагменты, которые отстают во время сегрегации при митозе, могут быть включены в ядерную оболочку вне основного ядра, что приводит к образованию микроядер (Leibowitz et al., 2015). Таким образом, микроядра представляют собой небольшие круглые внеядерные структуры, состоящие из ДНК, окруженной билипидным слоем (Fenech et al., 2011). Микроядра могут содержать как целую хромосому (так называемые отстающие хромосомы), так и часть хромосомы (Heddle, Carrano, 1977; Thompson, Compton, 2011).
Было продемонстрировано, что некоторые процессы, происходящие в главном ядре, являются дисфункциональными в микроядрах, включая транскрипцию и репликацию ДНК (Crasta et al., 2012; Terzoudi et al., 2015; Terradas et al., 2016). Это может приводить к разрывам двухцепочечной ДНК из-за остановленных или замедленных вилок репликации (Forment et al. 2012). Кроме того, было обнаружено, что репликация ДНК в микроядре асинхронна относительно основного ядра (Crasta et al., 2012; Terzoudi et al., 2015). Если реплицирующая ДНК внутри микроядра подвергается воздействию цитоплазматических “митотических сигналов” (когда основное ядро находится в митозе), микроядерные хромосомы могут подвергаться преждевременной компактизации, что в свою очередь приводит к пульверизации хромосом (Johnson, Rao, 1970).
Появились убедительные экспериментальные доказательства, связывающие хромотрипсис с отстающими хромосомами и инкапсуляцией микроядер (Zhang et al., 2015). Авторы этой работы использовали комбинацию визуализации живых клеток для отслеживания их после индукции микроядер с помощью нокодазол-опосредованной деполимеризации микротрубочек (Luijten et al., 2018) с последующим секвенированием отдельных дочерних клеток (Zhang et al., 2015). Генетический материал внутри микроядра был недостаточно реплицирован и поэтому мог быть идентифицирован по его асимметричному распределению в дочерних клетках. Геномный анализ этих клеток выявил хромотриптические перестройки de novo на хромосомах, которые ранее были инкапсулированы в микроядре (Zhang et al., 2015).
Укорочение теломер и формирование дицентрических хромосом. Другой возможный механизм хромотрипсиса связывают с циклами разрыв–слияние–мост в дицентрических хромосомах (Stephens et al., 2011; Sorzano et al., 2013). Хромотриптические точки разрыва часто расположены в пределах теломерных областей хромосомы, что указывает на возможную роль повреждения теломер в кластеризации точек разрыва (Fukami et al., 2017). Теломеры человека состоят из n-тандемных повторов -TTAGGG-, ограниченных белками шелтерина. Во время нормального деления соматических клеток теломеры укорачиваются из-за инактивации фермента теломеразы, необходимого для репликации теломер. После нескольких циклов пролиферации теломеры достигают критической длины, и клеточное старение активируется зависимым от p53 и pRB способом, чтобы предотвратить нежелательную клеточную пролиферацию. Потеря генов TP53 и pRB предотвращает активацию остановки клеточного цикла в ответ на укорочение теломер. Это приводит к продолжению пролиферации клеток и укорочению теломер, что в конечном итоге приводит к дисфункциональным и незащищенным теломерам. Когда защитные эффекты теломер теряются, механизмы репарации ДНК могут генерировать сквозные слияния теломер, приводящие к дицентрическим хромосомам.
Образование дицентрических хромосом во время митоза сопровождается образованием хроматиновых мостиков, которые подвергаются последующему разрыву (McClintock, 1939), т.е. они вступают в циклы разрыв–слияние–мост. Во время анафазы центромеры дицентрической хромосомы могут тянуться в разных направлениях делящейся клетки. Концы хромосом затем остаются соединенными между двумя дочерними клетками, предотвращая завершение сегрегации во время митоза и создавая хроматиновый мостик. В связи с этим явлением было высказано предположение, что связанные с укорочением теломер циклы разрыв–слияние–мост могут приводить к хромотрипсу (Stephens et al., 2011; Sorzano et al., 2013). Индуцировав образование оболочки, хроматиновый мостик разрушается цитоплазматической 3'-экзонуклеазой TREX1 (Maciejowski et al., 2015; Maciejowski, de Lange, 2017). Это может привести не только к множественным потерям и инверсиям хромосомных сегментов, но также к образованию двойных минихромосом. Цикл разрыв–слияние–мост может происходить одновременно с амплификацией фрагментов (Li et al., 2014). Эти циклы, однако, могут быть частью образования хромосом нового типа (неохромосом) и являться следствием, а не причиной хромотрипсиса (Garsed et al., 2014).
Прерванный апоптоз. В качестве третьего механизма возникновения хромострипсиса в клетках рассматривается прерывание апоптоза (Tubio, Estivill, 2011; Tang et al., 2012). Ключевой регулятор апоптоза – белок p53, который транскрибируется геном TP53. В 2012 г. были получены данные о том, что при мутации TP53 механизм апоптоза нарушается, клетка остается жизнеспособной при накоплении хромосмоных мутаций, что в результате дает картину хромотрипсиса. Так, при остром и хроническом лимфолейкозе мутации ТР53 могут сочетаться с хромотрипсисом (Pei et al., 2012). Наблюдение более высокой частоты хромотрипсиса при гиперплоидных медуллобластомах, по сравнению с диплоидными, позволили установить связь между хромотрипсом и гиперплоидизацией клеток. В связи с этим было высказано предположение, что гиперплоидизация может служить фактором развития хромотрипсиса (Mardin et al., 2015).
Названные выше пути рассматриваются как механизмы образования хромотрипсиса. И хотя пока предполагаются три пути образования хромотрипсиса, можно предположить, что существуют и другие механизмы его возникновения, в том числе и реализация нескольких механизмов одновременно.
КЛАССЫ МНОЖЕСТВЕННЫХ СЛОЖНЫХ ХРОМОСОМНЫХ ПЕРЕСТРОЕК
По мере накопления данных о множественных хромосомных перестройках стало понятно, что хромотрипис является не единственным типом сложных структурных перестроек. Был предложен термин “хромоанагенез” для всех катастрофических событий, приводящих к сложным хромосомным перестройкам с участием одной или нескольких хромосом (Holland, Cleveland, 2012). Хромоанагенез – явление, при котором большое количество сложных перестроек происходит в одном или нескольких хромосомных локусах в процессе одного катастрофического события. Хромотрипсис является одним из составляющих хромоанагенеза.
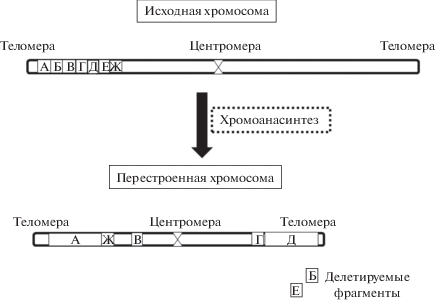
Помимо хромотрипсиса было выявлено событие, названное хромоанасинтезом. Хромоанасинтез заключается в том, что локально дефектная репликация ДНК инициирует последовательное гомологически обусловленное переключение последовательностей, которое вызывает локальные перестройки с изменением числа копий генов (рис. 2). Ключевым отличием между хромоанасинтезом и хромотрипсисом является наличие копий генов (две или три копии) в дополнение к делециям и нормально копируемым областям хромосомы.
Рис. 2.
Схема множественных сложных хромосомных перестроек при хромоанасинтезе. При хромоанасинтезе в результате аббрентной репликации ДНК часть хромосомы (фрагменты А–Ж) подвергается сложным перестройкам. В перестроенной хромосоме помимо изменения последовательности участка хромосомы (фрагменты Ж, В, Г), наблюдаются амплифицированнные участки (фрагменты А и Д), а часть фрагментов удаляется (фрагменты Б, Е).
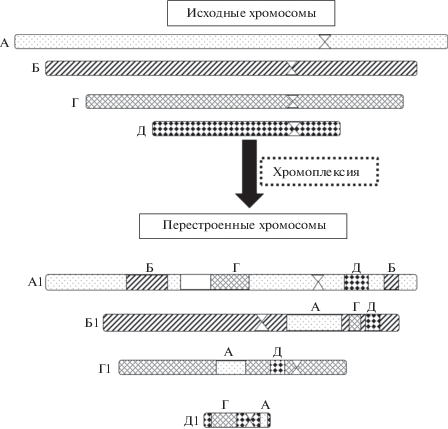
Секвенирование клеток рака предстательной железы привело к открытию хромоплексии – другого особого типа сложной перестройки хромосом, характеризующегося вовлечением множества (нескольких) хромосом (Shen, 2013). Термин хромоплексия происходит от греческого слова pleko, что означает плести или заплетать. Хромоплексия определяется как скопление связанных транслокаций с участием нескольких хромосом (до сих пор было обнаружено максимум 6 хромосом одновременно) (рис. 3). Получающиеся в результате хромосомные перестройки показывают незначительные изменения числа копий (или их отсутствие), в отличие от хромотрипсиса. Явление хромотрипсиса обычно ограничено одной или двумя хромосомами, при этом наблюдается колебания между двумя состояниями с различным числом копий.
Рис. 3.
Схема множественных сложных хромосомных перестроек при хромоплексии, которая включает связанные транслокации нескольких хромосом. Показано, что в результате хромоплексии из хромосомы А образовалась перестроенная хромосома А1, включающая фрагменты хромосом Б (2 фрагмента), Г и Д; из хромосомы Б образовалась перестроенная хромосома Б1, включающая фрагменты хромосом А, Г и Д; из хромосомы Г образовалась перестроенная хромосома Г1, включающая фрагменты хромосом А и Д; из хромосомы Д образовалась перестроенная хромосома Д1, включающая фрагменты хромосом Г и А.
Участвующие в хромоплексии хромосомы часто имеют меньшее количество перестроек, чем наблюдается при хромотрипсисе. Другое различие между хромотрипсисом и хромоплексией заключается в паттернах точек разрыва (соединения). При хромоплексии перестройки часто встречаются в виде замкнутых цепей с (почти) точными контактами и почти без делеций (Shen, 2013; Wang et al., 2013). Вследствие этого для хромоплексии не обнаружено характерного колебания числа копий, наблюдаемого на хромосомах после хромотрипсиса (Baca et al., 2013). В связи с этим, при обнаружении множественных хромосомных изменений важно понимать, с каким именно типом перестроек столкнулся исследователь, т.к. картины изменений, их механизмы и последствия в разных вариантах отличаются. С большей вероятностью можно сказать, что по мере накопления знаний о множественных хромосомных перестройках будут выявляться новые их типы.
МЕТОДЫ ВЫЯВЛЕНИЯ ХРОМОТРИПСИСА
Выявление и описание особенностей хромотрипсиса стало возможным за счет набора современных методов хромосомного анализа.
Методы полногеномного секвенирования. Они позволяют обнаружить комплексные хромосомные перестройки и определенные последовательности генов (Miller et al., 2010). В случае хромотрипсиса анализ требует последующей проверки секвенированием Сэнгера, что позволяет не только определить точную локализацию точек разрыва, но также получить данные о последовательностях нуклеиновых кислот в точках соединения (Gao, Smith, 2017). Несмотря на высокую стоимость и методические сложности, секвенирование широко используют в исследованиях хромотрипсиса.
Геномная гибридизация на основе микрочипов (CGH, aCGH). Это другой эффективный метод (сравнительный), который часто называют молекулярным кариотипированием (CGH) или анализом хромосом на микрочипах (aCGH). Анализ числа копий позволяет обнаружить делеции и дупликации, а также определять их точную локализацию и размер в геноме. Разрешение этого метода достаточно для обнаружения субмикроскопических аберраций. Для более высокого разрешения и информационной емкости, метод aCGH был объединен с методом определения однонуклеотидного полиморфизма на чипах (SNP) (Keren, 2014). Однако метод aCGH имеет ряд значительных ограничений: он не может обнаружить сбалансированные структурные хромосомные аберрации или определить порядок и ориентацию производных сегментов хромосомы (Balajee, Hande, 2018).
Флуоресцентная гибридизация in situ (FISH). Этот метод часто используют для обнаружения и локализации специфической последовательности ДНК или РНК. В исследованиях хромотрипсиса используются различные методики FISH, каждая из которых направлена на конкретную идентификацию структуры производной хромосомы. Спектральное кариотипирование (SKY) и многоцветный анализ FISH (M-FISH) с использованием зондов на целую хромосому, конъюгированных с различными флуорохромами, позволяют идентифицировать хромосомы, участвующие в перестройке. Метод многоцветного бандирования FISH (MCB-FISH) представляет собой сегментоспецифичный вариант хромосомного бэндинга, который позволяет определить структуру аберрантной хромосомы (Balajee, Hande, 2018). Комбинация SKY и гибридизации in situ с флуоресцентными локус-специфичными зондами используется для определения точной структуры не только производных хромосом, но и двойных минихромосом (Stephens et al., 2011). Кроме того, методы SKY и FISH (имеющие одноклеточное разрешение) позволяют обнаруживать варианты перестройки в одной клетке и таким образом обеспечивают обнаружение перестроек с низкой частотой в популяции, что важно при внутриопухолевой неоднородности.
Для обнаружения хромострипсиса в клинике используют метод обычного кариотипирования метафазных пластинок (G-бэндинга) периферических лимфоцитов. Этот метод позволяет идентифицировать числовые и структурные хромосомные аномалии, включая транслокации и инверсии, которые часто наблюдаются у пациентов с отклонениями. Однако сложная природа хромотрипсиса затрудняет их интерпретацию, поэтому для точного определения структуры перестроек при хромотрипсисе необходимо использовать комплексный подход, который включает классические методы цитогенетического анализа, визуализацию аберраций на метафазных хромосомах с помощью FISH и молекулярно-генетические методы.
ЗАКЛЮЧЕНИЕ
Из представленных в обзоре данных становится очевидным, что феномен хромотрипсиса вызывает большой интерес у исследователей. Неординарность этого явления очевидна. Открытие хромотрипсиса в геноме раковых клеток не только поставило под сомнение давнюю концепцию прогрессирующих генетических событий, ведущих к канцерогенезу, но и позволило пересмотреть научные взгляды, лежащие в основе его клеточных механизмов. Хромотрипсис обладает потенциалом для генерации массивных опухолевых мутационных изменений за относительно короткий период времени. Мы полагаем, что детальное изучение хромотрипсиса позволит более глубоко рассмотреть природу рака и врожденных генетических заболеваний.
Список литературы
Мамаев Н.Н., Гиндина Т.Л., Бойченко Э.Т. 2017. Хромотрипсис в онкологии: обзор литературы и собственные наблюдения. Клиническая онкогематология. Т. 10. №. 2. С. 191. (Mamaev N.N., Gindina T.L., Boychenko E.T. 2017. Chromothripsis in Oncology: Literature Review and Own Observations. Clinical Hematooncol. (Russ.). V. 10. № 2. P. 191.)
Abaigar M., Robledo C.,Benito R., Ramos F., Díez-Campelo M., Hermosin L., Sánchez-del-Real J., Alonso J.M., Cuello R., Megido M, Rodríguez J.N., Martín-Núñez G., Aguilar C., Vargas M., Martin A.A., García J.L., Kohlmann A., del Cañizo M.C., Hernández-Rivas J.M. 2016. Chromothripsis is a recurrent genomic abnormality in high-risk myelodysplastic syndromes. PLoS One. V. 11. e0164370. https://doi.org/10.1371/journal.pone.0164370
Baca S.C., Prandi D., Lawrence M.S., Mosquera J.M., Romanel A., Drier Y., Park K., Kitabayashi N., MacDonald T.Y., Ghandi M., Van Allen E., Kryukov G.V., Sboner A., Theurillat J.P., Soong T.D. et al. 2013. Punctuated evolution of prostate cancer genomes. Cell. V. 153. P. 666. https://doi.org/10.1016/j.cell.2013.03.021
Bak A.L., Bak P., Zeuthen J. 1979. Higher levels of organization in chromosomes. J. Theor. Biol. V. 76. P. 205.
Balajee A.S., Hande M.P. 2018. History and evolution of cytogenetic techniques: current and future applications in basic and clinical research. Mutat. Res. V. 836. P. 3.
Baltus C., Toffoli S., London F., Delrée P., Gilliard C., Gustin T. 2019. Chromothripsis in an early recurrent chordoid meningioma. World Neurosurg. V. 130. P. 380.
Behjati S., Tarpey P.S., Haase K., Ye H., Young M.D., Alexandrov L.B., Farndon S.J., Collord G., Wedge D.C., Martincorena I., Cooke S.L., Davies H., Mifsud W., Lidgren M., Martin S. et al. 2017. Recurrent mutation of IGF signalling genes and distinct patterns of genomic rearrangement in osteosarcoma. Nat. Commun. V. 8 P. 15936. https://doi.org/10.1038/ncomms15936
Cai H., Kumar N., Bagheri H.C., von Mering C., Robinson M.D., Baudis M. 2014. Chromothripsis-like patterns are recurring but heterogeneously distributed features in a survey of 22.347 cancer genome screens. BMC Genomics. V. 15. P. 82.
Carbonell-Bejerano P., Royo C., Torres-Pérez R., Grimplet J., Fernandez L., Franco-Zorrilla J.M., Lijavetzky D., Baroja E., Martínez J., García-Escudero E., Ibáñez J., Martínez-Zapater J.M. 2017. Catastrophic unbalanced genome rearrangements cause somatic loss of berry color in grapevine. Plant Physiol. V. 175. P. 786. https://doi.org/10.1104/pp.17.00715
Chen H., Singh R.R., Lu X., Huo L., Yao H., Aldape K., Abraham R., Virani S., Mehrotra M., Mishra B.M., Bousamra A., Albarracin C., Wu Y., Roy-Chowdhuri S., Kanagal-Shamanna R., Routbort M.J., Medeiros L.J., Patel K.P., Broaddus R., Sahin A., Luthra R. 2017. Genome-wide copy number aberrations and HER2 and FGFR1 alterations in primary breast cancer by molecular inversion probe microarray. Oncotarget. V. 8, 10 845. https://doi.org/10.18632/oncotarget.14802
Chiang C., Jacobsen J.C., Ernst C., Hanscom C., Heilbut A., Blumenthal I., Mills R.E., Kirby A., Lindgren A.M., Rudiger S.R., McLaughlan C.J., Bawden C.S., Reid S.J., Faull R.L., Snell R.G. et al. 2012. Complex reorganization and predominant non-homologous repair following chromosomal breakage in karyotypically balanced germline rearrangements and transgenic integration. Nat. Genet. V. 44. P. 390. https://doi.org/10.1038/ng.2202
Collins R.L., Brand H., Redin C.E., Hanscom C., Antolik C., Stone M.R., Glessner J.T., Mason T., Pregno G., Dorrani N., Mandrile G., Giachino D., Perrin D., Walsh C., Cipicchio M. et al. 2017. Defining the diverse spectrum of inversions, complex structural variation, and chromothripsis in the morbid human genome. Genome Biol. V. 18. P. 36.
Cortés-Ciriano I., Lee J.J., Xi R., Jain D., Jung Y.L., Yang L., Gordenin D., Klimczak L.J., Zhang C.Z., Pellman D.S. 2020. Comprehensive analysis of chromothripsis in 2.658 human cancers using whole-genome sequencing. Nat. Genet. V. 2. P. 331. https://doi.org/10.1038/s41588-019-0576-7
Crasta K., Ganem N.J., Dagher R., Lantermann A.B., Ivanova E.V., Pan Y., Nezi L., Protopopov A., Chowdhury D., Pellman D. 2012. DNA breaks and chromosome pulverization from errors in mitosis. Nature. V. 482. P. 53.
De Pagter M.S., van Roosmalen M.J., Baas A.F., Renkens I., Duran K.J., van Binsbergen E., Tavakoli-Yaraki M., Hochstenbach R., van der Veken L.T., Cuppen E., Kloosterman W.P. 2015. Chromothripsis in healthy individuals affects multiple protein-coding genes and can result in severe congenital abnormalities in offspring. Am. J. Hum. Genet. V. 96. P. 651. https://doi.org/10.1016/j.ajhg.2015.02.005
Deakin J.E., Bender H.S., Pearse A.M., Rens W., O’Brien P.C., Ferguson-Smith M.A., Cheng Y., Morris K., Taylor R., Stuart A., Belov K., Amemiya C.T., Murchison E.P., Papenfuss A.T., Graves J.A. 2012. Genomic restructuring in the tasmanian devil facial tumour: chromosome painting and gene mapping provide clues to evolution of a transmissible tumour. PLoS Genet. V. 8, e1002483. https://doi.org/10.1371/journal.pgen.1002483
Erson-Omay E.Z., Henegariu O., Omay S.B., Harmanci A.S., Youngblood M.W., Mishra-Gorur K., Li J., Özduman K., Carrión-Grant G., Clark V.E., Çağlar C., Bakırcıoğlu M., Pamir M.N., Tabar V., Vortmeyer A.O., Bilguvar K. et al. 2017. Longitudinal analysis of treatment-induced genomic alterations in gliomas. Genome Med. V. 9. P. 12.
Fenech M., Kirsch-Volders M., Natarajan A.T., Surralles J., Crott J.W., Parry J., Norppa H., Eastmond D.A., Tucker J.D., Thomas P. 2011. Molecular mechanisms of micronucleus, nucleoplasmic bridge and nuclear bud formation in mammalian and human cells. Mutagenesis. V. 26. P. 125.
Forment J.V., Kaidi A., Jackson S.P. 2012. Chromothripsis and cancer: Causes and consequences of chromosome shattering. Nat. Rev. Cancer. V. 12. P. 663. https://doi.org/10.1038/nrc3352
Fukami M., Shima H., Suzuki E., Ogata T., Matsubara K., Kamimaki T. 2017. Catastrophic cellular events leading to complex chromosomal rearrangements in the germline. Clin. Genet. V. 91. P. 653. https://doi.org/10.1111/cge.12928
Gao G., Smith D.I. 2017. How can mate-pair sequencing be utilized for cancer patients? Expert Rev. Mol. Diagn. V. 17. P. 1.
Garsed D.W., Marshall O.J., Corbin V.D., Hsu A., Di Stefano L., Schröder J., Li J., Feng Z.P., Kim B.W., Kowarsky M., Lansdell B., Brookwell R., Myklebost O., Meza-Zepeda L., Holloway A.J., Pedeutour F. et al. 2014. The architecture and evolution of cancer neochromosomes. Cancer Cell. V.26. P. 653. https://doi.org/10.1016/j.ccell.2014.09.010
Heddle J.A., Carrano A.V. 1977. The DNA content of micronuclei induced in mouse bone marrow by gamma-irradiation: evidence that micronuclei arise from acentric chromosomal fragments. Mutat. Res. V. 44. P. 63.
Heng H.H.Q., Liu G., Stevens J.B., Abdallah B.Y., Horne S.D., Ye K.J., Bremer S.W., Chowdhury S.K., Ye C.J. 2013. Karyotype heterogeneity and unclassified chromosomal abnormalities. Cytogenet. Genome Res. V. 139. P. 144.
Heng H.H., Liu G., Bremer S., Ye K.J., Stevens J., Ye C.J. 2006. Clonal and non-clonal chromosome aberrations and genome variation and aberration. Genome. V. 49. P. 195.
Heng H.Q., Chen W.Y. 1985. The study of the chromatin and the chromosome structure for Bufo gargarizans by the light microscope. J. Sichuan Univ. Nat. Sci. V. 2. P. 105.
Heng H.Q., Lin R., Zhao X.L., Chen W.Y. 1988. Structure of the chromosome and its formation. II. Studies on the sister unit fibers. Nucleus. V. 30. P. 2.
Holland A.J., Cleveland D.W. 2012. Chromoanagenesis and cancer: Mechanisms and consequences of localized, complex chromosomal rearrangements. Nat. Med. V. 18. P. 1630. https://doi.org/10.1038/nm.2988
Holzmann C., Markowski D.N., Koczan D., Küpker W., Helmke B.M., Bullerdiek J. 2014. Cytogenetically normal uterine leiomyomas without MED12-mutations – a source to identify unknown mechanisms of the development of uterine smooth muscle tumors. Mol. Cytogenet. V. 7. P. 88.
Itani O.A., Flibotte S., Dumas K.J., Moerman D.G., Hu P.J. 2015. Chromoanasynthetic genomic rearrangement identified in a N-ethyl-N-nitrosourea (ENU) mutagenesis screen in Caenorhabditis elegans. G3: Genes, Genomes, Genetics. V. 6. P. 351.
Johnson R.T., Rao P.N. 1970. Mammalian cell fusion: Induction of premature chromosome condensation in interphase nuclei. Nature. V. 226. P. 717.
Keren B. 2014. The advantages of SNP arrays over CGH arrays. Mol. Cytogenet. V. 7 (Suppl. 1). P. I31.
Kinsella M., Patel A., Bafna V. 2014. The elusive evidence for chromothripsis. Nucleic Acids Res. V. 42. P. 8231. https://doi.org/10.1093/nar/gku525
Kloosterman W.P., Cuppen E. 2013. Chromothripsis in congenital disorders and cancer: Similarities and differences. Curr. Opin. Cell Biol. V. 25. P. 341. https://doi.org/10.1016/j.ceb.2013.02.008
Kloosterman W.P., Koster J., Molenaar J.J. 2014. Prevalence and clinical implications of chromothripsis in cancer genomes. Curr. Opin. Oncol. V. 26. P. 64.
Korbel J.O., Campbell P.J. 2013. Criteria for inference of chromothripsis in cancer genomes. Cell. V. 152. P. 1226. https://doi.org/10.1016/j.cell.2013.02.023
Leibowitz M.L., Zhang C.Z., Pellman D. 2015. Chromothripsis: A new mechanism for rapid karyotype evolution. Annu. Rev. Genet. V. 49. P. 183.
Li Y., Schwab C., Ryan S., Papaemmanuil E., Robinson H.M., Jacobs P., Moorman A.V., Dyer S., Borrow J., Griffiths M., Heerema N.A., Carroll A.J., Talley P., Bown N., Telford N., Ross F.M. et al. 2014. Constitutional and somatic rearrangement of chromosome 21 in acute lymphoblastic leukaemia. Nature. V. 508. P. 98.
Luijten M.N.H., Lee J.X.T., Chen S., Crasta K.C. 2018. Generation of micronuclei and detection of chromosome pulverization. Methods Mol. Biol. V. 1769. P. 183.
Maciejowski J., de Lange T. 2017. Telomeres in cancer: Tumour suppression and genome instability. Nat. Rev. Mol. Cell Biol. V. 18. P. 175.
Maciejowski J., Li Y., Bosco N., Campbell P.J., de Lange T. 2015. Chromothripsis and kataegis induced by telomere crisis. Cell. V. 163. P. 1641. https://doi.org/10.1016/j.cell.2015.11.054
Malhotra A., Lindberg M., Faust G.G., Leibowitz M.L., Clark R.A., Layer R.M., Quinlan A.R., Hall I.M. 2013. Breakpoint profiling of 64 cancer genomes reveals numerous complex rearrangements spawned by homology-independent mechanisms. Genome Res. V. 5. P. 762.
Mardin B.R., Drainas A.P., Waszak S.M., Weischenfeldt J., Isokane M., Stütz A.M., Raeder B., Efthymiopoulos T., Buccitelli C., Segura-Wang M., Northcott P., Pfister S.M., Lichter P., Ellenberg J., Korbel J.O. 2015. A cell-based model system links chromothripsis with hyperploidy. Mol. Syst. Biol. V. 11. P. 828. https://doi.org/10.15252/msb.20156505
McClintock B. 1939. The behavior in successive nuclear divisions of a chromosome broken at meiosis. Proc. Natl. Acad. Sci. USA. V. 25. P. 405.
Mehine M., Kaasinen E., Mäkinen N., Katainen R., Kämpjärvi K., Pitkänen E., Heinonen H.R., Bützow R., Kilpivaara O., Kuosmanen A., Ristolainen H., Gentile M., Sjöberg J., Vahteristo P., Aaltonen L.A. 2013. Characterization of uterine leiomyomas by whole-genome sequencing. N. Engl. J. Med. V. 369. P. 43.
Mehine M., Mäkinen N., Heinonen H.R., Aaltonen L.A., Vahteristo P. 2014. Genomics of uterine leiomyomas: insights from high-throughput sequencing. Fertil. Steril. V. 102. P. 621.
Miller J.R., Koren S., Sutton G. 2010. Assembly algorithms for next-generation sequencing data. Genomics. V. 95. P. 315. https://doi.org/10.1016/j.ygeno.2010.03.001
Morishita M., Muramatsu T., Suto Y., Hirai M., Konishi T., Hayashi S., Shigemizu D., Tsunoda T., Moriyama K., Inazawa J. 2016. Chromothripsis-like chromosomal rearrangements induced by ionizing radiation using proton microbeam irradiation system. Oncotarget. V. 7. 10182. https://doi.org/10.18632/oncotarget.7186
Morrison C.D., Liu P., Woloszynska-Read A., Zhang J., Luo W., Qin M., Bshara W., Conroy J.M., Sabatini L., Vedell P., Xiong D., Liu S., Wang J., Shen H., Li Y., Omilian A.R., Hill A. et al. 2014. Whole-genome sequencing identifies genomic heterogeneity at a nucleotide and chromosomal level in bladder cancer. Proc. Natl. Acad. Sci. USA. V. 111. P. 672.
Notta F., Chan-Seng-Yue M., Lemire M., Li Y., Wilson G.W., Connor A.A., Denroche R.E., Liang S.B., Brown A.M., Kim J.C., Wang T., Simpson J.T., Beck T., Borgida A., Buchner N., Chadwick D. et al. 2017. A renewed model of pancreatic cancer evolution based on genomic rearrangement patterns. Nature. V. 538. P. 378. https://doi.org/10.1038/nature19823
Pei J., Jhanwar S.C., Testa J.R. 2012. Chromothripsis in a case of TP53-deficient chronic lymphocytic leukemia. Leuk. Res. Rep. V. 1. P. 4.
Przybytkowski E., Lenkiewicz E., Barrett M.T., Klein K., Nabavi S., Greenwood C.M., Basik M. 2014. Chromosome-breakage genomic instability and chromothripsis in breast cancer. BMC Genomics. V. 15. P. 579. https://doi.org/10.1186/1471-2164-15-579
Ratnaparkhe M., Hlevnjak M., Kolb T., Jauch A., Maass K.K., Devens F., Rode A., Hovestadt V., Korshunov A., Pastorczak A., Mlynarski W., Sungalee S., Korbel J., Hoell J., Fischer U. et al. 2017. Genomic profiling of Acute lymphoblastic leukemia in ataxia telangiectasia patients reveals tight link between ATM mutations and chromothripsis. Leukemia. V. 10. P. 2048. https://doi.org/10.1038/leu.2017.55
Rausch T., Jones D.T., Zapatka M., Stütz A.M., Zichner T., Weischenfeldt J., Jäger N., Remke M., Shih D., Northcott P.A., Pfaff E., Tica J., Wang Q., Massimi L., Witt H. et al. 2012. Genome sequencing of pediatric medulloblastoma links catastrophic DNA rearrangements with TP53 mutations. Cell. V. 148. P. 59.
Righolt C., Mai S. 2012. Shattered and stitched chromosomes-chromothripsis and chromoanasynthesis-manifestations of a new chromosome crisis? Genes Chromosomes Cancer. V. 51. P. 975. https://doi.org/10.1002/gcc.21981
Rode A., Maass K.K., Willmund K.V., Lichter P., Ernst A. 2016. Chromothripsis in cancer cells: An update. Int. J. Cancer. V. 138. P. 2322. https://doi.org/10.1002/ijc.29888
Rucker F.G., Dolnik A., Blatte T.J., Teleanu V., Ernst A., Thol F., Heuser M., Ganser A., Dohner H., Dohner K., Bullinger L. 2018. Chromothripsis is linked to TP53 alteration, cell cycle impairment, and dismal outcome in acute myeloid leukemia with complex karyotype. Haematologica. V. 3. e17. https://doi.org/10.3324/haematol.2017.180497
Shaffer L.G., McGowan-Jordan J., Schmid M. (Eds.). 2013. International system for human cytogenetic nomenclature. Basel: S. Karger. 139 pp.
Shen M.M. 2013. Chromoplexy: A new category of complex rearrangements in the cancer genome. Cancer Cell. V. 23. P. 567. https://doi.org/10.1016/j.ccr.2013.04.025
Sorzano C.O., Pascual-Montano A., Sanchez de Diego A., Martinez A.C., van Wely K.H. 2013. Chromothripsis: breakage-fusion-bridge over and over again. Cell Cycle. V.12, 2016. https://doi.org/10.4161/cc.25266
Stephens P.J., Greenman C.D., Fu B., Yang F., Bignell G.R., Mudie L.J., Pleasance E.D., Lau K.W., Beare D., Stebbings L.A., McLaren S., Lin M.L., McBride D.J., Varela I., Nik-Zainal S. et al. 2011. Massive genomic rearrangement acquired in a single catastrophic event during cancer development. Cell. V. 144. P. 27.
Stevens J.B., Abdallah B.Y., Liu G., Ye C.J., Horne S.D., Wang G., Savasan S., Shekhar M., Krawetz S.A., Hüttemann M., Tainsky M.A., Wu G.S., Xie Y., Zhang K., Heng H.H. 2011. Diverse system stresses: common mechanisms of chromosome fragmentation. Cell Death Dis. V. 2, e178. https://doi.org/10.10.1038/cddis.2011.60
Tan E.H., Henry I.M., Ravi M., Bradnam K.R., Mandakova T., Marimuthu M.P., Korf I., Lysak M.A., Comai L., Chan S.W. 2015. Catastrophic chromosomal restructuring during genome elimination in plants. Elife. V. 4, e06516. https://doi.org/10.7554/eLife.06516
Tang H.L., Tang H.M., Mak K.H., Hu S., Wang S.S., Wong K.M., Wong C.S., Wu H.Y., Law H.T., Liu K., Talbot C.C. Jr., Lau W.K., Montell D.J., Fung M.C. 2012. Cell survival, DNA damage, and oncogenic transformation after a transient and reversible apoptotic response. Mol. Biol. Cell. V.12. P. 2240. https://doi.org/10.1091/mbc.E11-11-0926
Terradas M., Martín M., Genesca A. 2016. Impaired nuclear functions in micronuclei results in genome instability and chromothripsis. Arch. Toxicol. V. 90. P. 2657. https://doi.org/10.1007/s00204-016-1818-4
Terzoudi G.I., Karakosta M., Pantelias A., Hatz V.I., Karachristou I., Pantelias G. 2015. Stress induced by premature chromatin condensation triggers chromosome shattering replicating in micronuclei or multinucleate cells when primary nuclei enter mitosis. Mutat. Res. Genet. Toxicol. Environ. Mutagen. V. 793. P. 185.
Thompson S.L., Compton D.A. 2011. Chromosome missegregation in human cells arises through specific types of kinetochore-microtubule attachment errors. Proc. Natl. Acad. Sci. USA. V. 108. P. 17974. https://doi.org/10.1073/pnas.1109720108
Tubio J.M., Estivill X. 2011. Cancer: When catastrophe strikes a cell. Nature. V. 470. P. 476.
Vig B.K., Willcourt M. 1998. Decondensation of pericentric heterochromatin alters the sequence of centromere separation in mouse cells. Chromosoma. V. 107. P. 417. https://doi.org/10.1007/s004120050325
Vogelstein B., Papadopoulos N., Velculescu V.E., Zhou S., Diaz L.A., Kinzler K.W.Jr. 2013. Cancer genome landscapes. Science. V. 339. P. 1546. https://doi.org/10.1126/science.1235122
Wang K., Wang Y., Collins C.C. 2013. Chromoplexy: a new paradigm in genome remodeling and evolution. Asian J. Androl. V. 15. 711. https://doi.org/10.1038/aja.2013.109
Ye C.J., Sharpe Z., Alemara S., Mackenzie S., Liu G., Abdallah B., Horne S., Regan S., Heng H.H. 2019. Micronuclei and genome chaos: Changing the system inheritance. Genes (Basel). V. 10. P. 366. https://doi.org/10.3390/genes10050366
Zack T.I., Schumacher S.E., Carter S.L., Cherniack A.D., Saksena G., Tabak B., Lawrence M.S., Zhsng C.Z., Wala J., Mermel C.H., Sougnez C., Gabriel S.B., Hernandez B., Shen H., Laird P.W. et al. 2013. Pan-cancer patterns of somatic copy number alteration. Nat. Genet. V. 45. P. 1134. https://doi.org/10.1038/ng.2760
Zhang C.Z., Spektor A., Cornils H., Francis J.M., Jackson E.K., Liu S., Meyerson M., Pellman D. 2015. Chromothripsis from DNA damage in micronuclei. Nature. V. 522. P. 179.
Дополнительные материалы отсутствуют.
Инструменты
Цитология


