Цитология, 2020, T. 62, № 5, стр. 316-321
Хромосомы индийского мунтжака (Muntiacus muntjak). Возвращение
Л. С. Билтуева 1, *, П. Л. Перельман 1, А. А. Проскурякова 1, Н. А. Лемская 1, Н. А. Сердюкова 1, А. С. Графодатский 1, **
1 Институт молекулярной и клеточной биологии СО РАН
630090 Новосибирск, Россия
* E-mail: bilar@mcb.nsc.ru
** E-mail: graf@mcb.nsc.ru
Поступила в редакцию 11.02.2020
После доработки 13.02.2020
Принята к публикации 14.02.2020
Аннотация
С помощью методов G-, C-, CDAG-, AgNOR-окрасок хромосом и in situ локализации 18S, 5.8S и 28S рРНК генов описаны хромосомы индийского мунтжака (Muntiacus muntjak, 2n = 6 у самок и 2n = 7 у самцов).
Абсолютно все обзорные работы по цитогенетике животных, позвоночных и млекопитающих в частности (Matthey, 1973; Ferguson-Smith, Trifonov, 2007; Graphodatsky et al., 2011), учебники по генетике и зоологии, статьи в энциклопедиях указывают на то, что наименьшее число хромосом среди млекопитающих (2n = 6 у самок и 2n = 7 у самцов) характерно для одного из вида оленей – индийского мунтжака (Muntiacus muntjak). С момента описания хромосом мунтжака Дорис Вюрстер и Куртом Бениршке в 1970 г. (Wurster, Benirschke, 1970) опубликовано несколько десятков работ по различным аспектам цитогенетики и геномики этого знакового вида. Описаны варианты дифференциальных окрасок хромосом, особенности локализации повторяющихся последовательностей ДНК, феномен образования огромных аутосом за счет тандемных слияний, получены данные по картированию хромосом индийского мунтжака с помощью пэйнтинг-проб китайского мунтжака (Muntiacus reevesi, 2n = 46), коровы и человека, и набора BAC-клонов, и, наконец, проведено полномасштабное секвенирование генома этого вида. Укажем лишь на часть из этих работ (Comings, 1971; Patterson, Petricciani, 1973; Kato et all., 1974; Pardue, Hsu, 1975; Green, Bahr, 1975; Carrano et all., 1975; Verma et al., 1979; Yamaguchi, Huh, 1979; Brat et al., 1979; Shi, Pathak, 1981; Johnston et al., 1982; Bogenberger et all., 1982, 1985, 1987; Babu, Verma, 1986; Elder, Hsu, 1988; Scherthan, 1990; Fontana, Rubini, 1990; Lin et al., 1991, 2008; Saitoh, Laemmli, 1994; Lee et al., 1993, 1997; Levy et al., 1993; Yang et al., 1995, 1997a,b; Fronicke, Scherthan, 1997; Fronicke et al., 1997; Li et al., 2000, 2002; Hartmann, Scherthan, 2004; Chi et al., 2005; Zhou et al, 2006; Murmann et al., 2008; Tsipouri et al., 2008; Schmid et al., 2016; Mudd et al., 2019). Таким образом, совершенно очевидно, что индийский мунтжак – один из самых изученных в цитогенетическом плане вид животных. Тем не менее, в настоящей работе мы возвращаемся к описанию хромосом этого вида с использованием самых традиционных методов цитогенетики. В марте 2020 г. выходит из печати Атлас хромосом млекопитающих (Graphodatsky et al., 2020) в котором представлены данные по дифференциальным окраскам хромосом более 1200 видов всех таксонов и около 400 карт хромосом, полученных с помощью хромосомной живописи. Собирая самые выдающиеся иллюстрации для каждого вида, мы, к своему изумлению, столкнулись с ситуацией, что для индийского мунтжака таковых нет. Авторы предыдущих работ чаще всего не озадачивались получением бэндинга сколько-нибудь высокого уровня разрешения. Это понятно, так как идентификация хромосом здесь не требовалась, пары хромосом мунтжака легко идентифицируются на любом уровне. Тем не менее, в свете дальнейших исследований генома этого вида новыми методами в рамках проектов VGP (Vertebrate Genome Project, https://vertebrategenomesproject.org/) и EBG (Earth BioGenome Project) (Lewin et al., 2019a, b), необходимости сборки геномов до отдельных хромосом (от теломеры до теломеры), и анализа эволюционно значимых участков хромосом, некое переописание хромосом одного из самых выдающихся животных представляется нам целесообразным.
МАТЕРИАЛ И МЕТОДИКА
В работе использована клеточная линия самца индийского мунтжака MMV1 из Куньминского клеточного банка АН КНР (Yang et al., 1996).
Для получения хромосомных препаратов и их дифференциальных окрасок использованы стандартные протоколы с модификациями, подробно описанные Графодатским и Раджабли (1988). GTG-окраска хромосом выполнена по методу Сибрайт (Seabright, 1971). С-окраска хромосомных препаратов проведена согласно протоколу Самнера (Sumner, 1972). Метод CDAG, позволяющий выявить блоки структурного гетерохроматина после GTG-окраски, детально описан Лемской с соавторами (Lemskaya et al., 2018). Для выявления функционально активных ядрышкообразующих районов был использован метод Ховелла и Блейка (Howell, Black, 1980). Флуоресцентная гибридизация in situ пробы, содержащей полный ген рибосомной РНК, проведена в соответствии с протоколом Янга и Графодатского (Yang, Graphodatsky, 2017). Препараты анализировали с помощью флуоресцентного микроскопа Olympus BX53 с использованием цифровых систем визуализации ВидеоТест-Карио и ВидеоТест-FISH (ВидеоТест, Санкт-Петербург, Россия).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
GTG-окраска хромосом. Кариотип индийского мунтжака состоит из 2-х пар аутосом и сложных половых хромосом, образовавшихся в результате транслокации Х-хромосомы на аутосому. Хромосомный набор самки состоит из 6-ти хромосом, включая половые хромосомы ХХ, а самца из 7-ми хромосом с половыми хромосомами XY1Y2, где Y1 – гомолог аутосомы, транслоцированной на Х-хромосому, а Y2 – истинный Y (рис. 1a). Хромосома 1 представлена крупным метацентриком с почти равными по размеру плечами. Хромосома 2 представлена субметацентриком, чье длинное плечо сопоставимо с плечами первой хромосомы, а короткое плечо столь мало, что Вюрстер и Бениршке (Wurster, Benirschke, 1970) пренебрегли им, посчитав эту хромосому акроцентриком. Позже Ши с соавторами (Shi et al., 1980) описывали ее как субтелоцентрика, а проведенные Брат с соавторами (Brat et al, 1979) измерения содержания ДНК и размеров хромосомных плеч показали, что короткое плечо хромосомы 2 сопоставимо по размеру с Y-хромосомой. Длинное плечо Х-хромосомы, как и гомологичная ей акроцентрическая аутосома, близки по размеру к длинным плечам остальных аутосом, тогда как ее короткое плечо, представленное истинной Х-хромосомой, вполовину меньше. Y-хромосома самый крошечный элемент набора. На рисунке 1а представлен кариотип индийского мунтжака на трех разных уровнях конденсации хромосом.
Рис. 1.
Хромосомы индийского мунтжака (Muntiacus muntjak vaginalis), 2n = 7. а – GTG-окрашенный кариотип на разных уровнях конденсации хромосом, б – C-окрашенная метафазная пластинка, стрелками отмечены гомологи хромосомы 2, полиморфные по содержанию гетерохроматина, в – метафазная пластинка, последовательно окрашенная с помощью методов GTG (слева) и CDAG (справа), помимо прицентромерных гетерохроматиновых блоков на половых хромосомах Х, Y1 и 1 визуализируются ЯОР-районы (указаны стрелками), г – Ag-окрашенная метафазная пластинка, стрелками показаны ЯОР-районы, д – флуоресцентная in situ гибридизация с ДНК пробой, содержащей 18S, 5.8S и 28S рРНК гены (справа) на GTG-окрашенных хромосомах (слева), стрелками отмечены сайты локализации рибосомных генов на хромосомах Х, Y1 и 1.
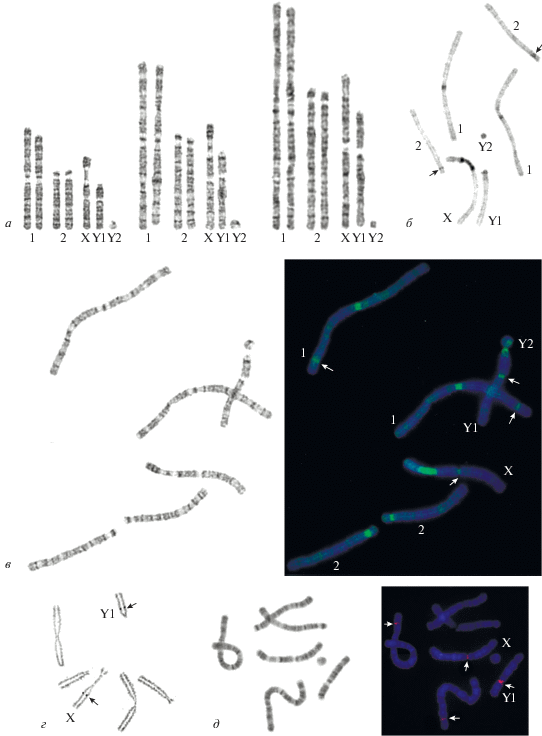
С- и CDAG-окраски гетерохроматина. C помощью стандартного метода C-окраски показано наличие крупного блока структурного гетерохроматина в Х-хромосоме, мелких прицентромерных С-блоков в аутосомах и целиком гетерохроматической Y-хромосомы (рис. 1б). Наши данные согласуются с представленными ранее результатами (Kato et al., 1974; Sharma, Dhaliwal, 1974), полученными с помощью этого же метода C-окраски, предложенного Самнером (Sumner, 1972) и названного им BSG-окраской (barium hydroxid/saline/Giemsa). Разработанный в нашей лаборатории Лемской с соавторами (Lemskaya et al., 2018) новый метод окраски гетерохроматина, именуемый CDAG (Chromomycin A3-DAPI-after G-banding), позволяет не только визуализировать блоки структурного гетерохроматина, выявляемые классическим методом, но одновременно оценить их состав. Метод CDAG, основанный на тепловой денатурации хромосом в присутствии формамида и последующей окраски AT- или GC-специфичными флуорохромами, позволяет идентифицировать хромосомы с помощью GTG-окраски, которая предваряет окрашивание гетерохроматиновых блоков. С помощью CDAG мы отчетливо видим GC-обогащенность гетерохроматиновых блоков и наличие интерстициальных GC-обогащенных районов хромосом индийского мунтжака (рис. 1в). Достаточно отчетливо визуализируются сигналы в районе вторичной перетяжки на хромосомах Х и Y1 и на дистальном конце длинного плеча хромосомы 1. Оба варианта окраски четко выявляют полиморфизм по содержанию гетерохроматина на хромосоме 2 (рис. 1б, в).
Локализация ядрышкообразующих районов (ЯОР) хромосом и сайтов рибосомной ДНК. Для выявления функционально активных рибосомных генов была проведена окраска хромосом индийского мунтжака азотнокислым серебром. Показано наличие двух ядрышкообразующих районов в проксимальной части хромосом Х и Y1 (рис. 1г). Следует отметить, что в этих же зонах вторичных перетяжек расположены CMA3 позитивные блоки. Почти не визуализируется ЯОР на дистальной части хромосомы 1, что может быть связано с размером и активностью сайта, хотя ранее показано присутствие ЯОР на одном или обоих гомологах хромосомы 1 (Goodpasture, Bloom, 1975). Следует отметить, что оба сайта локализации ЯОР на половых хромосомах Х и Y, и дистальном конце длинного плеча хромосомы 1 визуализируются с помощью CDAG, где расположены CMA3 позитивные блоки (рис. 1в).
Как и ожидалось, оба сайта локализации рибосомных генов индийского мунтжака на дистальном конце длинного плеча хромосомы 1 и на хромосомах Х и Y1 выявляются с помощью ДНК пробы, содержащей 18S, 5.8S и 28S рРНК гены (рис. 1д). Наиболее интенсивное свечение сигнала наблюдается на хромосоме Y1, гомологе аутосомы, транслоцированной на Х-хромосому. Наши данные согласуются с данными полученными ранее с помощью меченого трития (Pardue, Hsu, 1975), и позже подтвержденные Мурманном с соавторами (Murmann et al., 2008), которые картировали 38 генов, включая рибосомные гены, на хромосомы индийского мунтжака.
Полученное нами детальное описание хромосом индийского мунтжака мы планируем использовать в рамках продолжающегося международного исследования по описанию эволюционно значимых районов хромосом китопарнокопытных (Proskuryakova et al., 2017, 2018, 2019, Farré et al., 2019 a, b), перестройки в которых приводят к изменению функциональной активности сопряженных генов и изменению многих круциально важных анатомических и физиологических признаков. В этом плане планируемое изучение хромосом и генома индийского мунтжака, ключевого вида, в плане перестроенности кариотипа, представляется чрезвычайно интересным.
Список литературы
Графодатский А.С., Раджабли С.И. 1988. Хромосомы сельскохозяйственных и лабораторных млекопитающих. Новосибирск. Наука. Сибирское отделение. 127 с.
Babu A., Verma R.S. 1986. Expression of heterochromatin by restriction endonuclease treatment and distamycin A/DAPI staining of indian muntjac (Muntiacus muntjak) chromosomes. Cytogenet. Genome Res. V. 41. P. 96.
Bogenberger J., Schnell H., Fittler F. 1982. Characterization of X-chromosome specific satellite DNA of Muntiacus muntjak vaginalis. Chromosoma. V. 87. P. 9.
Bogenberger J.M., Neumaier P.S., Fittler F. 1985. The muntjac satellite IA sequence is composed of 31-base-pair internal repeats that are highly homologous to the 31-base-pair subrepeats of the bovine satellite 1.715. Eur. J. Biochem. V. 148. P. 55.
Bogenberger J.M., Neitzel H., Fittler F. 1987. A highly repetitive DNA component common to all cervidae: its organization and chromosomal distribution during evolution. Chromosoma. V. 95. P. 154.
Brat S.V., Verma R.S., Dosik H. 1979. Structural organization of chromosomes of the indian muntjac (Muntiacus muntjak). Cytogenet. Cell Genet. V. 24. P. 201.
Carrano A.V., Gray J.W., Moore I.I., Minkler J.L., Mayall B.H., van Dilla M.A., Mendelsohn M.L. 1976. Purification of the chromosomes of the Indian muntjac by flow sorting. J. Histochem. Cytochem. V. 24. P. 348.
Chi J.X., Huang L., Nie W., Wang J., Su B., Yang F. 2005. Defining the orientation of the tandem fusions that occurred during the evolution of Indian muntjac chromosomes by BAC mapping. Chromosoma. V. 114. P. 167.
Comings D.E. 1971. Heterochromatin of the Indian muntjac. Replication, condensation, DNA ultracentrifugation, fluorescent and heterochromatin staining. Exp. Cell Res. V. 67. P. 441.
Ferguson-Smith M.A., Trifonov V. 2007. Mammalian karyotype evolution. Nat. Rev. Gen. V. 8. P. 950.
Elder F.F.B., Hsu T.C. 1988. Tandem fusions in the evolution of mammalian chromosomes. In: Sandberg AA (Ed.) The cytogenetics of mammalian autochromosomal rearrangements. Alan R Liss, N.Y. P. 481.
Farré M., Kim J., Proskuryakova A.A., Zhang Y., Kulemzina A.I., Li Q., Zhou Y., Xiong Y., Johnson J.L., Perelman P., Johnson W.E., Warren W.C., Kukekova A.V., Zhang G., O’Brien S.J. et al. 2019. Evolution of gene regulation in ruminants differs between evolutionary breakpoint regions and homologous synteny blocks. Genome Res. V. 29. P. 576.
Farré M., Li Q., Darolti I., Zhou Y., Damas J., Proskuryakova A.A., Kulemzina A.I., Chemnick L.G., Kim J., Ryder O.A., Ma J., Graphodatsky A.S., Zhang G., Larkin D.M., Lewin H.A. 2019. An integrated chromosome-scale genome assembly of the Masai giraffe (Giraffa camelopardalis tippelskirchi). GigaScience. V. 8. giz090.
Fontana F., Rubini M. 1990. Chromosomal evolution in Cervidae. Biosystems. V. 24. P. 157.
Fronicke L., Scherthan H. 1997. Zoo-fluorescence in situ hybridization analysis of human and Indian muntjac karyotypes (Muntiacus muntjak vaginalis) reveals satellite DNA clusters at the margins of conserved syntenic segments. Chromosom. Res. V. 5. P. 254.
Fronicke L., Chowdhary B.P., Scherthan H. 1997. Segmental homology among cattle (Bos taurus), Indian muntjac (Muntiacus muntjak vaginalis), and Chinese muntjac (M. reevesi) karyotypes. Cytogenet. Cell Genet. V. 77. P. 223.
Goodpasture C., Bloom S.E. 1975. Visualization of nucleolar organizer regions in mammalian chromosomes using silver staining. Chromosoma. V. 53. P. 37.
Graphodatsky A.S., Trifonov V.A., Stanyon R. 2011. The genome diversity and karyotype evolution of mammals. Mol. Cytogenet. V. 4 : 22.
Graphodatsky A., Perelman P., O’Brien S.J. Ed’s. 2020. An Atlas of mammalian chromosomes. 2nd Ed. New York, N.Y.: John Wiley & Sons, 976 p.
Green R., Bahr G. 1975. Comparison of G-, Q-, and EM-banding patterns exhibited by the chromosome complement of the Indian muntjac, Muntiacus muntjak, with reference to nuclear DNA content and chromatin ultrastructure. Chromosoma. V. 50. P. 53.
Hartmann N., Scherthan H. 2004. Characterization of ancestral chromosome fusion points in the Indian muntjac deer. Chromosoma. V. 112. P. 213.
Howell W.M., Black D.A. 1980. Controlled silver-staining of nucleolus organizer regions with a protective colloidal developer: a 1-step method. Experientia. V. 36. P. 1014.
Johnston F.P., Church R.B., Lin C.C. 1982. Chromosome rearrangement between the Indian muntjac and Chinese muntjac is accompanied by a deletion of middle repetitive DNA. Can. J. Biochem. V. 60. P. 497.
Kato H., Tsuchiya K., Yosida T.H. 1974. Constitutive heterochromatin of Indian muntjac chromosomes revealed by DNAse treatment and a C-banding technique. Canadian J. Genet. Cytol. V. 16. P. 273.
Lee C., Sasi R., Lin C.C. 1993. Interstitial localization of telomeric DNA sequences in the Indian muntjac chromosomes: further evidence fortandem chromosome fusions in the karyotypic evolution of the Asian muntjacs. Cytogenet. Cell Genet. V. 63. P. 156.
Lee C., Court D.R., Cho C., Haslett J.L., Lin C.C. 1997. Higher-order organization of subrepeats and the evolution of cervid satellite I DNA. J. Mol. Evol. V. 44. P. 327.
Lemskaya N.A., Kulemzina A.I., Beklemisheva V.R., Biltueva L.S., Proskuryakova A.A., Perelman P.L., Graphodatsky A.S. 2018. The combined method of heterogeneous heterochromatin detection (CDAG) in different mammalian species. Chromosoma. V. 26. P. 307.
Levy H.P., Schultz R.A., Ordonez J.V., Cohen M.M. 1993. DNA content measurements and an improved idiogram for the Indian muntjac. Cytometry. V. 14. P. 362.
Lewin H.A., Robinson G.E., Kress W.J., Baker W.J., Coddington J., Crandall K.A., Durbin R., Edwards S.V., Forest F., Gilbert M.T.P., Goldstein M.M., Grigoriev I.V., Hackett K.J., Haussler D., Jarvis E.D. et al. 2018. Earth BioGenome project: Sequencing life for the future of life. Proc. Natl. Acad. Sci. USA. V. 115. P. 4325.
Lewin H.A., Graves J.A.M., Ryder O.A., Graphodatsky A.S., O’Brien S.J. 2019. Precision nomenclature for the new genomics. Gigascience. V. 8 : pii: giz086.
Li Y-C., Lee C., Sanoudou D., Hseu T-H., Li S-Y., Lin C.C. 2000. Interstitial colocalization of two cervid satellite DNAs involved in the genesis of the Indian muntjac karyotype. Chromosome Res. V. 8. P. 363.
Li Y.C., Lee C., Chang W.S., Li S.Y., Lin C.C. 2002. Isolation and identification of a novel satellite DNA family highly conserved in several Cervidae species. Chromosoma. V. 111. P. 176.
Lin C.C., Sasi R., Fan Y.S., Chen Z.Q. 1991. New evidence for tandem chromosome fusions in the karyotypic evolution of Asian muntjacs. Chromosoma. V. 101. P. 19.
Lin C.-C., Hsu P.-C., Li T.-S., Liao S.-J., Cheng Y.-M., Hsieh L.-J., Li Y.-S. 2008. Construction of an Indian muntjac BAC library and production of the most highly dense FISH map of the species. Zool. Stud. V. 47. P. 283.
Matthey R. 1973. Chromosome formulae of eutherian mammals. In: Chiarelli A.B., Capanna E. (eds) Cytotaxonomy and vertebrate evolution. Academic Press, London. P. 531.
Mudd A.B., Bredeson J.V., Baum R., Hockemeyer D., Rokhsar D.S. 2019. Muntjac chromosome evolution and architecture. BioRxiv. https://doi.org/10.1101/772343
Murmann A.E., Mincheva A., Scheuermann M.O., Gautier M., Yang F., Buitkamp J., Strissel P.L., Strick R., Rowley J.D., Lichter P. 2008. Comparative gene mapping in cattle, indian muntjac, and chinese muntjac by fluorescence in situ hybridization. Genetica. V. 134. P. 345.
Pardue M.L., Hsu T.C. 1975. Locations of 18S and 28S ribosomal genes on the chromosomes of the indian muntjac. J. Cell Biol. V. 64. P. 251.
Patterson R.M., Petricciani J.C. 1973. A comparison of prophase and metaphase G-bands in the muntjak. J. Hered. V. 64. P. 80.
Proskuryakova A.A., Kulemzina A.I., Perelman P.L., Makunin A.I., Larkin D.M., Farré M., Kukekova A.V., Johnson J.L., Lemskaya N.A., Beklemisheva V.R., Roelke-Parker M.E., Bellizzi J., Ryder O.A., O’Brien S.J., Graphodatsky A.S. 2017. X chromosome evolution in Cetartiodactyla. Genes. V. 8. P. 216.
Proskuryakova A.A., Kulemzina A.I., Perelman P.L., Serdukova N.A., Ryder O.A., Graphodatsky A.S. 2018. The case of X and Y localization of nucleolus organizer regions (NORs) in Tragulus javanicus (Cetartiodactyla, Mammalia). Genes. V. 9. P. 312.
Proskuryakova A.A, Kulemzina A.I., Perelman P.L., Yudkin D.V., Lemskaya N.A., Okhlopkov I.M., Kirillin E.V., Farré M., Larkin D.M., Roelke-Parker M.E., O’Brien S.J., Bush M., Graphodatsky A.S. 2019. Comparative chromosome mapping of musk ox and the X chromosome among some Bovidae species. Genes. V. 10. P. 857.
Saitoh Y. Laemmli U.K. 1994. Metaphase chromosome structure: Bands arise from a differential folding path of the highly AT-rich scaffold. Cell. V. 76. P. 609.
Sharma T., Dhaliwal M.K. 1974. Relationship between patterns of late S DNA synthesis and C- and G-banding in muntjac chromosomes. Exp. Cell Res. V. 87. P. 394.
Seabright M. 1971. A rapid banding technique for human chromosomes. Lancet. V. 298. P. 971.
Scherthan H. 1990. The localization of the repetitive telomeric sequence (TTAGGG) n in two muntjac species and implications for their karyotypic evolution. Cytogenet. Cell Genet. V. 53. P. 115.
Schmid M., Steinlein C., Lomb C., Sperling K., Neitzel H. 2016. 5-methylcytosine-rich heterochromatin in the Indian muntjac. Cytogenet. Genome Res. V. 147. P. 240.
Shi L., Ye Y., Duan X.S. 1980. Comparative cytogenetic studies on the red muntjac, Chinese muntjac and their F1 hybrids. Cytogenet. Cell Genet. V. 26. P. 22.
Shi L., Pathak S. 1981. Gametogenesis in a male Indian muntjac × Chinese muntjac hybrid. Cytogenet. Cell Genet. V. 30. P. 152.
Sumner A.T. 1972. A simple technique for demonstrating centromeric heterochromatin. Exp. Cell Res. V. 75. P. 304.
Tsipouri V., Schueler M.G., Hu S., Dutra A., Pak E., Riethman H., Green ED. 2008. Comparative sequence analyses reveal sites of ancestral chromosomal fusions in the indian muntjac genome. Genome Biol. V. 9. P. R155.
Verma R.S., Ved Brat S., Dosik H. 1979. Heterochromatin of chromosomes of indian muntjac as revealed by fluorescent banding techniques. J. Hered. V. 70. P. 438.
Wurster D.H., Benirschke K. 1970. Indian muntjac, Muntiacus muntjak: a deer with a low diploid chromosome number. Science. V. 168. P. 1364.
Yamaguchi N., Huh N. 1979. Establishment and characterization of Indian muntjak cell lines transformed with simian virus 40. J. Gen. Virol. V. 42. P. 289.
Yang F., Graphodatsky A.S. 2017. Animal probes and ZOO-FISH. In: Fluorescence in situ hybridization (FISH). Application Guide. 2nd edition (ed. Liehr T) Springer-Verlag, Germany. P. 395.
Yang F., Carter N.P., Shi L., Ferguson-Smith M.A. 1995. A comparative study of karyotypes of muntjacs by chromosome painting. Chromosoma. V. 103. P. 642.
Yang F., O’Brien P.C., Wienberg J., Ferguson-Smith M.A. 1997a. A reappraisal of the tandem fusion theory of karyotype evolution in Indian muntjac using chromosome painting. Chromosome Res. V. 5. P. 109.
Yang F., Muller S., Just R., Ferguson-Smith M.A., Wienberg J. 1997b. Comparative chromosome painting in mammals: human and the Indian muntjac (Muntiacus muntjak vaginalis). Genomics. V. 39. P. 396.
Zhou Q., Huang L., Zhang J., Zhao X., Zhang Q., Song F., Chi J., Yang F., Wang W. 2006. Comparative genomic analysis links karyotypic evolution with genomic evolution in the Indian muntjac (Muntiacus muntjak vaginalis). Chromosoma. V. 115. P. 427.
Дополнительные материалы отсутствуют.


