Физиология растений, 2021, T. 68, № 5, стр. 451-468
Транскрипционный фактор RIPENING INHIBITOR и его гомологи в регуляции созревания сочного плода различных видов растений
М. А. Слугина *
a Институт биоинженерии Федерального исследовательского центра
“Фундаментальные основы биотехнологии” Российской академии наук
Москва, Россия
* E-mail: mashinmail@mail.ru
Поступила в редакцию 28.10.2020
После доработки 13.01.2021
Принята к публикации 15.01.2021
Аннотация
MADS-доменный транскрипционный фактор RIPENING INHIBITOR (RIN) является глобальным регулятором процесса созревания плода томата Solanum lycopersicum L. RIN контролирует каскад реакций созревания сочного плода одновременно на нескольких уровнях: активируя экспрессию генов других регуляторных белков; активируя гены биосинтеза этилена (инициируя этилен-зависимый сигнальный путь); непосредственно запуская транскрипцию генов биосинтеза каротиноидов, ароматических соединений, генов метаболизма углеводов и модификации клеточной стенки (регулируя этилен-независимый путь); а также участвует в некоторых эпигенетических модификациях, сопровождающих процесс созревания сочного плода. Будучи открыты первоначально у томата, на сегодняшний день гомологи RIN описаны для многих культур с сочным плодом. В обзоре рассмотрены молекулярные особенности транскрипционного фактора RIN и его гомологов – MADS-факторов транскрипции различных видов растений, выполняющих схожую с RIN функцию контроля процессов созревания сочного плода и имеющих схожую эволюционную историю. Охарактеризованы структура, пространственно-временная экспрессия, гены-мишени данных регуляторов. На примере однодольных и двудольных растений, формирующих сочный плод, таких как томат, банан, перец, виноград, земляника показано, что MADS-белки подсемейства SEPALLATA играют центральную роль в чрезвычайно сложной транскрипционной регуляции каскада реакций созревания как климактерических, так и неклимактерических плодов.
ВВЕДЕНИЕ
ФИЗИОЛОГО-БИОХИМИЧЕСКИЕ ОСОБЕННОСТИ СОЗРЕВАНИЯ СОЧНОГО ПЛОДА
Плод – специализированный орган растений, необходимый для распространения семян. Исходя из морфологических и анатомических особенностей, существует множество признаков для классификации плодов. По наличию сочной мякоти плоды принято разделять на сухие и сочные. Сухие и сочные плоды образованы схожими тканями, однако различаются уровнем лигнификации, числом и размером клеток [1, 2].
В процессе эволюции сочный плод появился позднее, и его возникновение представляет эволюционное преимущество для растений, так как сочные плоды более привлекательны для животных, что позволяет растительному виду расширить ареал распространения семян [1, 2].
Развитие сочного плода включает несколько этапов: закладка, рост, созревание (maturation) и стадия биологической спелости (ripening). Созревание сочного плода – сложный процесс, сопровождающийся изменением физиологических и биохимических характеристик, включая содержание и состав фитогормонов, сахаров, пигментов, ароматических соединений, а также модификацию структуры клеточных стенок.
Исходя из физиологических особенностей, процесс созревания сочного плода может быть климактерическим и неклимактерическим [3, 4]. Климактерические плоды имеют характерный паттерн изменения интенсивности дыхания: у развивающегося плода дыхание сначала медленно снижается до нижней точки, известной как доклимактерический минимум (на данной стадии плод остается неспелым). Затем интенсивность дыхания начинает динамично расти, достигая климактерического максимума. В этот момент у большинства растений (или несколькими часами или днями позже) плод достигает биологической спелости. После стадии климактерического максимума интенсивность дыхания снижается, что свидетельствует о начале стадии старения плода [3]. Росту интенсивности дыхания у климактерических плодов сопутствует резкий скачок синтеза этилена [5]. Примерами климактерических плодов служат плоды томата, банана, яблока, авокадо, груши [4]. У неклимактерических плодов процесс созревания не сопровождается усилением интенсивности дыхания, и концентрация этилена при этом остается на низком уровне [4, 5]. Неклимактерические плоды – у земляники, винограда, цитрусовых, ананаса и др. [4].
Наличие этилена необходимо для нормального протекания процесса созревания климактерических плодов, так как он служит регулятором транскрипции и трансляции многих генов созревания [5]. Подтверждением этому является замедление или отсутствие созревания у трансгенных растений с заблокированным биосинтезом этилена [5].
Долгое время считалось, что роль этилена в неклимактерических плодах крайне ограничена, но многие современные исследования свидетельствуют об обратном. Например, у плодов винограда (Vitis vinifera L.) показано наличие функционального сигнального пути этилена, запуск которого сопровождается ростом плода и синтезом антоцианов [6]. У ананаса (Ananas comosus L. Merr.) процесс созревания сопровождается активацией генов биосинтеза этилена [7]. Однако механизмы этилен-опосредованной регуляции созревания неклимактерических плодов изучены недостаточно [8], поэтому данной проблеме посвящено множество современных исследований.
Несмотря на то, что этилен считается главным триггером процесса созревания, кроме этилен-зависимых существуют также и этилен-независимые пути активации генов созревания [5]. Помимо этилена в регуляции созревания сочного плода могут принимать участие и другие фитогормоны, однако, в настоящий момент, такие данные неоднозначны. Ранее считалось, что ауксин оказывает существенное влияние на процессы роста и созревания плода земляники (Fragaria × ananassa Duch.), так как рост цветоложа зависит от поступления в него ауксина из семянок (или орешков) [9]. В то же время анализ транскриптомов развивающегося плода земляники (многоорешка) выявил высокий уровень экспрессии генов биосинтеза и сигнального пути ауксина и в самом цветоложе в процессе созревания ложной ягоды [9]. Кроме того, в развитии плода земляники принимают участие и другие фитогормоны: гиббереллины [10], абсцизовая кислота [11, 12], жасмоновая кислота [13] и брассиностероиды [14]. При этом этилен не играет определяющей роли в процессе созревания земляники [15], хотя оказывает важное влияние на инициацию некоторых отдельных аспектов [16], но не всего процесса созревания, как в случае климактерических плодов [9].
Как в случае этилен-зависимого, так и в случае этилен-независимого пути созревания пространственно-временную координацию работы комплекса генов созревания осуществляют транскрипционные факторы (ТФ), связываясь с их промоторами и контролируя уровень их транскрипции. Транскрипционные факторы могут контролировать экспрессию генов как индивидуально, так и в комплексе с другими ТФ и белками.
Среди всех ТФ, регулирующих созревание сочного плода, особенно важную роль играет семейство MADS-доменных белков [17]. Представители данного семейства обнаружены в геномах всех эукариот и характеризуются присутствием N-концевого высоко консервативного ДНК-связывающего домена MADS. Наиболее полно охарактеризованные MADS-ТФ растений принадлежат к типу II (MIKC-тип) и играют важнейшую роль во всех процессах развития, контролируя не только идентичность цветковой меристемы и органов цветка, но и различные аспекты развития семяпочек и плодов [18, 19].
На сегодняшний день основным модельным растением с сочным плодом является томат обыкновенный (томат овощной; Solanum lycopersicum L.). Изучение спонтанных мутаций с ярким фенотипическим проявлением позволило выявить три регуляторных белка, без которых невозможно созревание сочного плода томата. К ним относятся MADS-ТФ RIPENING INHIBITOR (RIN, или SlMADS-RIN), NAC-ТФ NO-RIPENING (NOR), а также SPL-ТФ COLORLESS NON-RIPENING (CNR) [20–22]. Будучи открыты 20–50 лет назад, в настоящее время кодирующие их гены считаются ключевыми регуляторами созревания плода томата [23–25].
ТФ RIN запускает каскад реакций созревания, активируя экспрессию генов двух других регуляторов, NOR и CNR, тем самым контролируя наиболее ранние этапы созревания плода [23]. RIN также активирует гены биосинтеза этилена, инициируя этилен-зависимый сигнальный путь [4]. RIN может непосредственно запускать и транскрипцию генов биосинтеза каротиноидов и ароматических соединений, а также генов модификации клеточной стенки [26, 27], тем самым регулируя этилен-независимый путь созревания плода. Кроме того, RIN участвует в некоторых эпигенетических модификациях, например, метилировании ДНК [23], являясь, таким образом, глобальным регулятором процесса созревания плода томата (рис. 1) [28].
Рис. 1.
Молекулярные механизмы созревания сочного плода, контролируемые транскрипционным фактором RIN.
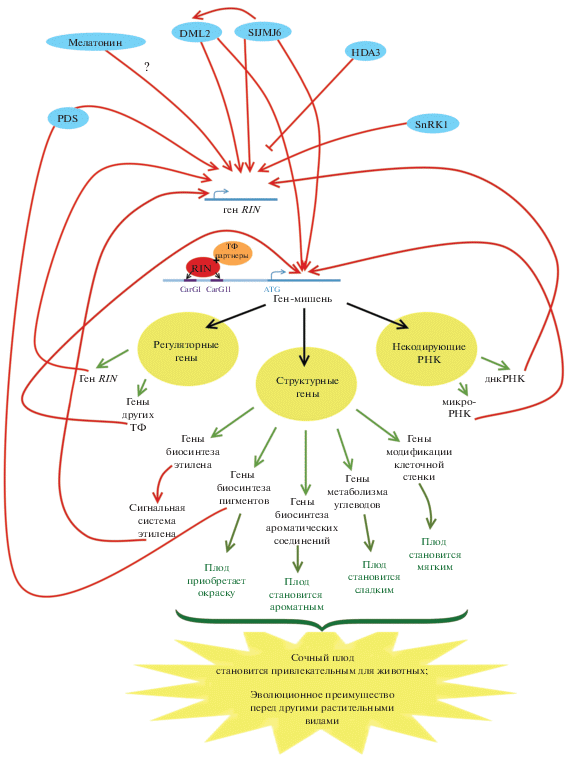
Исследования ТФ, определяющих сроки созревания сочных плодов, помимо теоретического, приобрели и большое практическое значение. Это подтверждается растущим количеством обзоров, посвященных контролю созревания сочного плода и участию в нем гена RIN [24, 29–31, 36]. Накопленные знания уже становятся предметом для экспериментов в области биотехнологии растений с использованием современных методов редактирования генома. Плоды с дефицитом активности RIN, полученные с помощью CRISPR/Cas9, созревают одновременно с плодами дикого типа (WT), но в сравнении с WT, синтезируют лишь 10% каротиноидов и этилена, а также значительно пониженное количество летучих веществ [32–34]. Это подтверждает участие RIN в созревании сочного плода и свидетельствует о его неучастии в инициации созревания [33]. То, что плоды растений с CRISPR/Cas9-нокаутом RIN демонстрируют избыточное смягчение и ускоренную деградацию клеточной стенки по сравнению с диким типом, позволило предположить, что RIN подавляет перезревание плода [35]. Кроме того, было показано, что усеченный ТФ RIN, лишенный C-концевого домена, кодируемый другим аллелем локуса RIN (rinG2), не активирует транскрипцию, но, подобно RIN дикого типа, образует комплексы ТФ, которые связываются с целевыми областями генома. Плоды, экспрессирующие RING2, имеют увеличенный срок хранения, но в отличие от плодов rin, накапливают ликопин и выглядят оранжевыми [35].
Большинство таких исследований и обзоров сосредоточено на модельном объекте – томате обыкновенном, однако эти данные не всегда возможно экстраполировать на другие культуры. С другой стороны, в последние годы накапливается все больше несистематизированных данных о гомологах ТФ RIN у видов растений как с климактерическим, так и с неклимактерическим типом созревания плода. Поэтому в данном обзоре особое внимание уделено сравнительной характеристике гомологов ТФ RIN, включая их роль в регуляции созревания сочного плода различных видов растений.
Гомологи MADS-ТФ RIN описаны для многих культур с сочным плодом. При этом у некоторых культур найдены полные функциональные гомологи RIN, а у других выявлено несколько функционально избыточных белков, сходных с RIN по структуре и/или функции. Объединяющим признаком является общее эволюционное происхождение данных белков, входящих в MADS-подсемейство SEPALLATA (SEP), но представляющих различные клады. Так, гены томата RIN и перца CaMADS-RIN гомологичны гену SEP4 Arabidopsis thaliana L. и относятся к кладе SEP4 [37]. Гены винограда, кодирующие функциональные гомологи RIN, относятся сразу к двум различным кладам: SEP3 (VviSEP3) и SEP4 (VviSEP4) [38]. Наиболее близкий по структуре гену RIN, ген земляники FaSEP4 в плодах экспрессируется крайне низко, в отличие от гена FaMADS9 (клада SEP1/2), гомология которого с RIN ниже, но который при этом высоко транскрибируется в плоде [4]. Подобные различия предполагают, что дупликация и диверсификация SEP-генов может лежать в основе современного многообразия форм и качеств климактерических и неклимактерических сочных плодов [37].
СТРУКТУРА ТРАНСКРИПЦИОННОГО ФАКТОРА RIN И ЕГО ГОМОЛОГОВ
RIN и его гомологи у других видов растений относятся к MIKC-типу MADS-ТФ и имеют схожую структуру: последовательно расположенные высоко консервативный N-концевой MADS-домен, междоменная I-область, средне консервативный кератин-подобный домен K и высоко вариабельная C-концевая область (рис. 2).
Рис. 2.
Основные домены и консервативные мотивы белков транскрипционных факторов подсемейства SEP, определяющих созревание сочного плода у различных видов растений. RIN (SlRIN; GenBank: AAM15775.1) – S. lycopersicum; CaMADS-RIN (CaRIN; GenBank: ABJ98752.1) – C. annuum, MaMADS2 (GenBank: ACJ64678.1) и MaMADS1 (GenBank: ACJ64679.1) – M. acuminata; FaMADS9 (GenBank: AAO49380.1) – F. ananassa; VviSEP3 (GenBank: AAM21344.1) и VviSEP4 (GenBank: XP_002263410.2) – V. vinifera; SEP1 (GenBank: NP_001119230.1), SEP2 (GenBank: NP_186880.1), SEP3 (GenBank: AAB67832.1) и SEP4 (GenBank: AAB38975.1) – A. thaliana. Консенсусные последовательности консервативных мотивов, выявленные с помощью программы MEME Suite 5.1.1 (http://meme-suite.org/index.html): 1 – MGRGRVEL, 2 – RIENKINRQVTFAKRRNGLLKKAYELSVLCDAEVALIIFSNRGKLYEFCS, 3 – SSSMLKTLEREQKCSYGALEVNQPAKEEQ, 4 – YQEYLKLKARVEALQ, 5 – QRNLLGEDLGPLNTKELEQLERQLDSSLKQIRSTKTQYMLDQLSDLQRKE, 6 – QMLLEANKALRRKLEESSQAN, 7 – WEPGEQNVQY, 8 – QPQSEGFFQPLECNPTLQIGY, 9 ‒ GYDNVPP, 10 – AASGQSVNGYIPGWM, 11 – PDPIAI, 12 – WENYGHP, 13 – WEHGGDHQH, 14 – HHIGGGW, 15 – NPVCSEQIAVT.

Домен MADS у MIKC-ТФ, в том числе у представителей клады SEP, относится к доменам типа MEF2-like (IPR033896; myocyte enhancer factor 2-like) [39]. Одной из функций данного домена является связывание с ДНК, а также участие в гомо- и гетеродимеризации [39, 40]. Функция К-домена заключается в образовании межбелковых связей [18]. С-область белка предположительно участвует в активации транскрипции и в образовании комплексов высокого порядка [18, 41, 42].
Белки ТФ, гомологичных RIN, определяющих процесс созревания сочного плода, а также родственные белки клады SEP A. thaliana имеют сходные размеры (235–251 а.о.), молекулярную массу (от 26.9 до 29.5 кДа) и протяженность доменов и междоменных областей (рис. 2). Структура данных белков консервативна у различных видов однодольных и двудольных растений, независимо от типа формируемого плода.
ПРОСТРАНСТВЕННО-ВРЕМЕННАЯ ЭКСПРЕССИЯ ГЕНА RIN И ЕГО ГОМОЛОГОВ
В геноме A. thaliana имеется четыре SEP-гена, которые представляют отдельные клады SEP1, SEP2, SEP3 и SEP4, различающиеся, в первую очередь, паттернами экспрессии [43, 44]. Согласно общепринятой генетической модели развития цветка ABCDE, гены SEP1 (AGL2), SEP2 (AGL4) и SEP3 (AGL9) транскрибируются в области закладки цветковых органов трех внутренних мутовок и, далее, в развивающихся лепестках, тычинках и плодолистиках до стадии терминации цветка [4, 37]. Профили экспрессии генов-гомологов SEP4 (AGL3), в том числе RIN, гораздо более разнообразны. Так, ген SEP4 (A. thaliana) транскрибируется во всех кругах цветка и других надземных органах растения, ген петунии PhFBP4 – в чашелистиках, лепестках, пестике и плоде, а ген яблони MdMADS4 – во всех четырех кругах цветка и плодах [37].
Среди растений с сочным плодом экспрессия SEP4-подобного гена RIN впервые была описана у томата обыкновенного [4]. RIN экспрессируется, главным образом, в плодах, достигая максимума во время инициации созревания (стадия Breaker) и полного покраснения плода (стадия Red Ripe) [45]. В отличие от многих других генов созревания плода томата, экспрессия RIN практически не зависит от воздействия этилена [4].
Интересно, что структурный гомолог гена RIN, ген CaMADS-RIN перца Capsicum annuum L. экспрессируется не только в плодах, но и в корнях, стеблях, листьях и цветках, но на низком уровне. Тем не менее, на конечных стадиях созревания плода транскрипция гена резко увеличивается, достигая максимума [8].
Два структурных гомолога RIN, гены MaMADS1 и MaMADS2 банана Musa acuminata L. по-разному экспрессируются в мякоти и кожуре плода, что предполагает наличие независимых механизмов созревания для различных частей плода. Так же, как и RIN, ген MaMADS2 транскрибируется независимо от воздействия этилена [44, 46].
У земляники F. × ananassa Duch. ген-гомолог RIN, FaMADS9, слабо экспрессируется в цветоложе и высоко в орешках, имея два пика резкого роста экспрессии – на стадиях зеленого и красного плода [47].
Структурные гомологи RIN винограда V. vinifera L., гены VviSEP4 и VviSEP3, транскрибируются в период созревания плодов [38, 48–51]. Но, в отличие от RIN, их экспрессионный профиль не имеет такой же четкой взаимосвязи с началом периода смены окраски (стадия Véraison) [52].
В мезокарпе плодов масличной пальмы (Elaeis guineensis Jacq.) экспрессируется пять SEP-генов, два из которых (CL1Contig8010, CL1Contig3848) активируются в период всплеска синтеза этилена, но являются структурными гомологами SEP3, а не RIN [53].
Структурный гомолог RIN идентифицирован и у ананаса A. comosus – AcMADS1; подобно RIN, данный ген экспрессируется в период созревания плода [54].
Таким образом, несмотря на существенные различия в морфологии и физиологии плодов, как климактерических, так и неклимактерических, как однодольных, так и двудольных, как настоящих, так и ложных (земляника, ананас), у всех описанных видов растений процесс созревания сочного плода сопровождается экспрессией одного или нескольких MADS-генов подсемейства SEP.
ГЕНЫ-МИШЕНИ ТРАНСКРИПЦИОННОГО ФАКТОРА RIN И ЕГО ГОМОЛОГОВ
MADS-белки регулируют экспрессию, связываясь со специфичным CArG-box-мотивом в промоторе гена-мишени. Данный мотив имеет каноническую последовательность CC(A/T)6GG. Последовательность CArG-box, предпочтительно узнаваемая ТФ RIN (консенсус CCA(A/T)(A/t)(A/T)ATAG), имеет высокую степень сходства с таковыми у ТФ SEP4 и SEP1 A. thaliana и содержит ‘CCA’ в начале и ‘TAG’ в конце мотива, а в центре исключительно A или T [55]. Многие гены, экспрессия которых регулируется ТФ RIN, содержат в составе промотора данный консенсус. Однако в промоторах некоторых генов-мишеней найдены нетипичные CArG-box-мотивы. К примеру, промотор гена экспансина томата LeEXP1 содержит неканонический CArG-box (CAATTTAAAG), а в промоторе гена 1-аминоциклопропан-1-карбоксилатсинтазы LeACS4 найдено три неканонических мотива (CAAATATAAG, CAATTTTAAG и CTAGTTAAAG). Методом ChIP-seq (метод анализа ДНК-белковых взаимодействий, основанный на иммунопреципитации хроматина (ChIP) и высокоэффективном секвенировании ДНК) показано, что сила связывания RIN с данными неканоническими мотивами больше, чем в случае CArG-box-консенсуса, что предполагает наличие более широкого спектра CArG-box мотивов, с которыми может связываться ТФ RIN in vivo [56]. Кроме того, связывание RIN исключительно с промоторами генов созревания может регулироваться составом белковых комплексов, в которые входит ТФ RIN [56]. Также селективность связывания может зависеть от структуры хроматина в промоторе целевого гена (например, от модификации гистонов, или степени метилирования) [56].
Согласно модели “квартет” (quartet model), ТФ RIN формирует тетрамерные комплексы с другими MADS-белками [57]. В частности, RIN предположительно способствует сборке мультимерных комплексов, как это показано для SEP-белков A. thaliana [56], и образует гомо- и гетеродимеры с MADS-белками подсемейств APETALA1 (AP1)/ FRUITFULL (FUL) (TM4 = TDR4 = FUL1 и SLMBP7 = FUL2), SEP (LeMADS1 = ENHANCER-OF-JOINTLESS-2) и AGAMOUS (TAGL1 и TAG1) [55, 58]. С учетом известных взаимодействий RIN, предполагается сборка комплексов RIN-FUL1-FUL2-TAGL1, RIN-FUL1-FUL2-TAG1 и других, каждый из которых специфичен к своему набору генов-мишеней и, таким образом, позволяет ТФ RIN контролировать определенный пул генов в зависимости от регулируемого процесса [55, 59]. Связывание комплекса, включающего ТФ RIN, с промоторами некоторых генов-мишеней в таком случае задействует сразу два CArG-box мотива [56] (рис. 1). ТФ RIN, в отличие от MADS-ТФ других подсемейств, имеет активаторный домен, что может использоваться тетрамерным комплексом для активации транскрипции генов-мишеней [56].
Для выявления генов-мишеней ТФ RIN применялись различные подходы: биоинформатический поиск CArG-box сайтов в промоторах потенциальных мишеней; ChIP-seq; сравнение транскриптомов томата дикого типа и с rin-мутацией. Результатом множества исследований стало достаточно подробное описание генов-мишеней RIN у томата обыкновенного [23, 55, 56, 60, 61]. Сравнение транскриптомов томата дикого типа и rin-мутанта обнаружило 342 гена созревания, экспрессия которых активируется ТФ RIN, и 473 гена, на экспрессию которых RIN оказывает негативное влияние [60]. Методом ChIP-seq было выявлено 292 гена, которые могут являться мишенями RIN [23]. Более того, с помощью RNA-seq показано, что RIN является именно активатором для своих генов-мишеней, а негативное воздействие на транскрипцию может объясняться метилированием сайтов связывания с RIN. В процессе созревания плода данные сайты деметилируются, в результате чего RIN может с ними связаться, что приводит к созреванию плода [23].
Ранее фенотипические проявления мутации rin объясняли отсутствием в клетке функционального белка RIN и, как следствие, нарушением профиля экспрессии контролируемых им генов. Однако детальный анализ показал, что у rin-растений вместо ТФ RIN образуется химерный ТФ RIN-MACROCALYX (MC, подробнее см. ниже), который связывается со своими специфическими мишенями [61]. Таким образом, на сегодня можно выделить три группы генов-мишеней, регулируемых белком RIN (1), совместно RIN и химерным ТФ (2) или только химерным белком (3). Так как настоящий обзор посвящен именно ТФ RIN, далее будут рассмотрены именно его гены-мишени.
Предполагается, что ТФ RIN может регулировать созревание за счет: (1) непосредственного связывания с промоторами генов созревания, что приводит к активации их экспрессии; (2) активации генов биосинтеза этилена, который в свою очередь запускает этилен-зависимые реакции созревания; (3) активации генов других ТФ, продукты которых, в свою очередь, запускают новые каскады реакций созревания.
Предположительно три описанных каскада реакций работают в клетке параллельно друг другу [56, 60] (рис. 1). Функционально гены-мишени RIN можно разделить на несколько групп (рис. 1, табл. 1) [23, 56, 61].
Таблица 1.
Мишени ТФ RIN.
| Функциональная группа | Гены-мишени ТФ RIN |
|---|---|
| Гены транскрипционных факторов | AP2-EREBP (Solyc03g044300, ERF2); GRAS (GRAS1, GRAS9, Solyc02g085600, Solyc01g008910); MYB (Solyc06g076770), MADS (TDR4); C2H2 (Solyc04g077980); bZIP (abz1, Opaque 2); HSF (Solyc12g007070); NAC (NAC-NOR); auxin-responsive protein (Solyc06g053840); Histone-Lys N-methyltransferase NSD3 (Solyc01g103250); C2C2-CO-like (Solyc12g096500); SBP (LeSPL-CNR); ULT (Solyc07g054450); WRKY (Solyc02g021680) |
| Гены транскрипционных регуляторов | auxin-responsive protein (Solyc03g120390), transcription elongation factor A protein2 (Solyc10g080930), DNA-repair protein (Solyc06g050510). |
| Гены биосинтеза и сигнального пути этилена | 1-aminocyclopropane-1-carboxylate synthases (ACS2, ACS4), 1-aminocyclopropane-1-carboxylate oxidases (ACO1, ACO3, ACO5), ethylene receptors (ETR3/NR, ETR4, ETR6, EIN3, EBF1, EBF2, EIL4, E4) |
| Гены метаболизма каротиноидов | phytoene synthase (PSY1), 15-cis-zeta-carotene-isomerase (ZISO), β-carotene hydroxylase (CHY), geranyl diphosphate synthase (GPPS2), lycopene beta-cyclase (LCY-b), isopentenyl-diphosphate delta-isomerase (IPP2), zeta-carotene desaturase (ZDS), carotenoid isomerase (CrtISO), plastid terminal oxidase (PTOX), protein LUTEIN DEFICIENT (CYP97A3) |
| Гены метаболизма флавоноидов и антоцианов | 4-coumarate: CoA ligase (4CL), phenylalanine ammonia-lyase (PAL) |
| Гены модификации клеточной стенки и гены метаболизма сахаров | endo-1,4-β-glucanases (Cel2, Cel3, Cel6), expansin-like protein (EXLA1), LeXYL1, xyloglucan endotransglucosylase hydrolase (XTH3), extensin (tegII), L-arabinokinase (ARA-1), expansins (EXP, EXPA5, EXPA6), beta-fructofuranosidases (TAI, LIN), polygalacturonases (PS-2, PGcat), arabinogalactan protein (AGP-1c), pectinesterase (PME2.1), xyloglucan endotransglycosylase/hydrolase protein 8 (XTH8), beta-galactosidases (TBG1,TBG4, TBG7), mannan endo-1,4-beta-mannosidase 4 (LeMAN4) |
| Другие группы генов | SGN-U571769 |
| Самоактивация | RIN |
| МикроРНК | MIR172a |
| Длинные некодирующие РНК | lncRNA2155, lncRNA1780, lncRNA3197, lncRNA2250, lncRNA609, lncRNA443. |
Регуляторные гены (гены транскрипционных факторов и транскрипционных регуляторов). Одним из подтверждений ключевой роли ТФ RIN в созревании сочного плода является тот факт, что он способен регулировать не только гены ферментов созревания, но и гены других ТФ, которые в свою очередь запускают подчиненные им каскады реакций. Белок RIN регулирует экспрессию генов ТФ различных семейств: AP2-EREBP, GRAS, MYB, MADS, C2H2, bZIP, HSF, NAC, WRKY и др. (рис. 1, табл. 1).
Структурные гены. Среди них можно выделить гены биосинтеза этилена, его рецепторов и гены ответа на воздействие этилена; гены метаболизма сахаров, каротиноидов, флавоноидов и антоцианов; гены модификации клеточной стенки (рис. 1, табл. 1).
Отдельно стоит отметить возможность самоактивации. Достоверно показано, что белок RIN in vivo способен активировать транскрипцию собственного гена, связываясь с CArG-box сайтами в собственном промоторе, что указывает на наличие механизма авторегуляции и объясняет резкое возрастание экспрессии гена RIN при созревании (рис. 1, табл. 1) [56].
МикроРНК. При изучении профилей экспрессии микроРНК у томата дикого типа и у rin-мутанта была показана дифференциальная экспрессия микроРНК 14 семейств; в промоторах генов многих из этих микроРНК найдены CArG-box мотивы. Достоверно показано, что ТФ RIN связывается с промотором гена микроРНК MIR172a (рис. 1, табл. 1) [63].
Длинные некодирующие РНК. Большинство исследований, посвященных поиску мишеней ТФ RIN, сконцентрировано на функциональных генах, однако помимо кодирующих последовательностей, показано, что RIN способен связываться и с длинными некодирующими РНК (днкРНК; long non-coding RNAs, lncRNAs) [64]. днкРНК представляют собой последовательности длиной более 200 п.н. [65], которые не кодируют белок, однако, играют важную роль в транскрипционной и пост-транскрипционной регуляции генов, эпигенетических модификациях в процессах дифференциации и развития клеток [64].
С целью поиска днкРНК-мишеней RIN было проведено широкоформатное исследование генома томата с помощью ChIP-seq в сочетании с транскриптомным анализом [64]. В результате найдено 627 днкРНК, содержащих в промоторах один или несколько сайтов связывания с RIN. 187 из них дифференциально экспрессировались в растениях томата с мутацией rin в сравнении с диким типом. Дальнейший in vivo и in vitro анализ подтвердил непосредственное связывание (разной силы) RIN с промоторами шести найденных генов днкРНК (lncRNA2155, lncRNA1780, lncRNA3197, l-ncRNA2250, lncRNA609 и lncRNA443) [64]. Более того, CRISPR/Cas9-нокаут генов данных шести днкРНК показал изменение фенотипа плода (задержка стадий покраснения (pink stage) и созревания) при замалчивании lncRNA2155 [64]. Это коррелировало с паттерном экспрессии lncRNA2155, чей экспрессионный максимум приходится на стадию покраснения плода. При этом снижался уровень экспрессии генов метаболизма этилена (ACS2 и ACS4) и биосинтеза каротиноидов (PSY1 и ZDS), а также генов ключевых ТФ созревания (NOR, CNR) и, что интересно, самого гена RIN, что, среди прочего, может указывать на наличие обратной регуляторной петли lncRNA2155-RIN (рис. 1) [64].
Таким образом, мишенями ТФ RIN могут быть как регуляторные и структурные гены, так и некодирующие регуляторные РНК (микроРНК, днкРНК), и, в совокупности, все они образуют сложную регуляторную сеть, обеспечивающую созревание сочного плода (рис. 1).
РОЛЬ ТФ RIN И ЕГО ГОМОЛОГОВ В КОНТРОЛЕ СОЗРЕВАНИЯ СОЧНОГО ПЛОДА РАЗЛИЧНЫХ ВИДОВ РАСТЕНИЙ
Растения, формирующие климактерические плоды
Томат. Плоды томата обыкновенного (S. lycopersicum L.) являются примером наиболее изученных климактерических плодов [30, 66]. Полногеномная последовательность томата известна и достаточно хорошо охарактеризована [67], для томата получено множество рекомбинантных линий, и он относительно легко поддается трансформации (что важно при изучении функций генов).
На сегодняшний день для томата обыкновенного доступна самая полная информация о структуре, регуляции и партнерах RIN, а также о каскаде запускаемых им реакций [31]. Впервые существование данного гена было предположено именно у томата в 1968 году при анализе растений с rin-мутацией, характеризующихся отсутствием созревания плода [20]. В 2002 году была детально изучена генетика и биохимия плода rin-томата на примере сорта Ailsa Craig [4].
Растения томата, гомозиготные по мутации rin, образуют недетерминантные соцветия, увеличенные чашелистики, и, главное, их плоды не созревают, имеют зеленую окраску и твердую текстуру. На физиологическом уровне для таких плодов характерны потеря стадий роста, синтеза этилена и климактерического усиления интенсивности дыхания, а также резкое снижение накопления пигментов и ароматических соединений [4, 21]. Однако в целом rin-растения сохраняют чувствительность к воздействию этилена, о чем свидетельствует старение лепестков и листьев и опадание цветков; тем не менее, в ответ на экзогенный этилен rin-плоды не созревают [4, 68, 69].
В гетерозиготном состоянии мутация rin широко используется в селекции сортов томата с повышенной лежкостью [70]. Такие томаты остаются твердыми и не портятся длительный период после сбора урожая, что увеличивает сроки их доставки потребителю и продажи, но при этом нарушается их питательная и вкусовая ценность [4]. Как говорилось выше, мутации гена RIN были получены также методом геномного редактирования CRISPR/Cas9 [32, 33].
Ген RIN расположен на хромосоме 5, состоит из восьми экзонов и имеет протяженность более 5 т.п.н. За геном RIN, отделенным межгенным спейсером (~3 т.п.н.), в той же ориентации следует ген, кодирующий еще один MADS-ТФ – MACROCALYX (MC, подсемейство AP1/FUL). Ген MC имеет сходную с RIN структуру и размеры (8 экзонов, более 5 т.п.н.). Ранее было предположено, что мутация rin представляет собой делецию размером 1.7 т.п.н., захватывающую часть последнего интрона 7 и весь экзон 8 гена RIN, и также часть межгенной (RIN-MC) последовательности [71]. В результате вместо транскриптов RIN и MC накапливается химерный транскрипт RIN-MC (GenBank NCBI ID – NM_001247047.2) [61]. Таким образом, вышеупомянутая измененная структура чашелистиков и соцветия у растения томата с мутацией rin, предположительно, является следствием нарушения структуры не только гена RIN, но и гена MC, который, подобно другим представителям AP1/FUL, участвует в определении идентичности меристемы цветка, чашелистиков, лепестков и плодолистиков [4, 61].
Особый интерес вызывает тот факт, что rin-фенотип может быть связан не только с отсутствием в клетке функционального ТФ RIN, но и с присутствием химерного белка RIN-MC, также являющимся ТФ. Так, химерный транскрипт RIN-MC экспрессируется в процессе развития rin-плодов на более высоком уровне, чем транскрипты RIN и MC в плодах дикого типа [61]. Гибридный белок (372 а.о.) содержит MADS-домен, I-область, К-домен и большую часть С-области белка RIN (215 из 242 а.о.), и далее – большую часть I-области, К-домен и С-область белка MC (157 из 219 а.о.) [24, 61]. Показано, что ТФ RIN-MC связывается c известными белками-партнерами ТФ RIN (MADS-ТФ FUL1, FUL2, MADS2 и TAGL-1) и, следовательно, потенциально способен in planta формировать мультимерные MADS-комплексы, регуляторные эффекты которых, однако, отличаются от эффекта комплексов с включением ТФ RIN [61]. Об этом косвенно свидетельствует то, что у rin-растений изменяется паттерн экспрессии множества генов созревания сочного плода [17, 26, 27]. Заметим также, что помимо нарушений транскрипции генов-мишеней ТФ RIN, выявляются новые гены, с промоторами которых способен связываться уже химерный белок RIN-MC [61]. Кроме участия в процессе регуляции созревания, гены-мишени RIN-MC могут определять и другие аспекты развития плода, к примеру, метаболизм аскорбиновой кислоты, структуру клеточных стенок, устойчивость к стрессу и др. [61]. Одно из подтверждений этому является то, что гены метаболизма аскорбиновой кислоты по-разному регулируются RIN и RIN-MC. В частности, при наличии химерного белка активируются гены дегидроаскорбатредуктазы (DHAR) и GDP-L-галактозофосфорилазы (GGP2), на транскрипцию которых RIN не влияет [61].
Таким образом, в растениях томата дикого типа ТФ RIN является главным регулятором созревания, который контролирует сотни генов, определяющих окраску, текстуру, аромат и вкусовые/ питательные качества плода. Делеция фрагмента участка хромосомы V, содержащего тандемно расположенные гены RIN и MC, приводит к резким морфофизиологическим изменениям в растениях, самым ярким из которых является несозревание плода.
Идентификация и характеристика гена RIN у томата вызвали большой интерес у исследователей и послужили началом для поиска ТФ-гомологов RIN, запускающих созревание, и у других видов растений, формирующих сочный плод. Однако, как показал дальнейший опыт, наиболее гомологичный по структуре ген не всегда имеет схожие с RIN паттерн экспрессии и функцию (как, например, ген FaMADS4 F. × ananassa). В связи с этим, другой стратегией стал поиск генов MADS-ТФ, которые высоко экспрессируются в зреющем плоде и сверхэкспрессия которых в растениях томата способна комплементировать эффект rin-мутации. При соблюдении обоих условий можно считать, что найденный ген выполняет функцию, сходную с функцией RIN.
Далее в обзоре приведены примеры таких ТФ, структурная гомология которых с RIN не всегда максимально высока, но которые выполняют схожие с RIN функции у различных видов растений.
Банан. Банан (M. acuminata L.), подобно томату, образует климактерический плод. Однако, в отличие от томата, это однодольное растение, поэтому механизмы созревания у этих двух культур могут различаться. Плод банана состоит из двух анатомически разных структур – кожуры и мякоти, имеющих разные паттерны биосинтеза этилена [44, 72]. Синтез этилена начинается в мякоти, и в дальнейшем приводит к инициации его синтеза в кожуре [44, 73]. Таким образом, в мякоти начинается процесс созревания плода банана.
У образца M. acuminata (AAA Cavendish subgroup Grand Nain) были клонированы и описаны шесть кодирующих последовательностей генов-кандидатов на роль гена, определяющего созревание (MaMADS1 – MaMADS6) [44]. Наибольшую гомологию с RIN показали белки MaMADS2 и MaMADS1 – 62% и 56% идентичности соответственно [46].
Пространственно-временные паттерны экспрессии идентифицированных генов MaMADS1–6 различаются в мякоти и кожуре плода, предполагая существование двух независимых механизмов созревания. Кроме того, различался уровень ответа генов MaMADS1–6 на воздействие этилена и его синтетического заменителя (1-метилциклопропен, 1-MCP); только транскрипция гена MaMADS2 не индуцировалась ни этиленом, ни 1-MCP. Это позволило сделать вывод, что транскрипция именно MaMADS2 активируется в мякоти раньше, чем инициируется биосинтез этилена, аналогично тому, как это происходит с геном RIN в плодах томата [44].
Однако, сверхэкспрессия двух генов MaMADS2 и MaMADS1 (последний был выбран из-за высокой экспрессии и в мякоти, и в кожуре) в rin-мутантном растении томата не компенсировала отсутствующей функции RIN. А именно, не наблюдалось ожидаемого увеличения синтеза этилена, размягчения плода и накопления каротиноидов; также отсутствовали видимые изменения в развитии цветка и плода [44]. Тем не менее, учитывая, что банан, в отличие от томата, является однодольным, и данные виды эволюционно очень далеки, отсутствие комплементации rin-мутации не является корректным доказательством того, что MaMADS1 и MaMADS2 не могут играть роль в созревании плода банана, схожую с ролью RIN у томата. Невысокий уровень идентичности белковых последовательностей (особенно, в С-области) данных ТФ с RIN предполагает, что MaMADS1 и MaMADS2 могут оказаться неспособными взаимодействовать с белковыми партнерами RIN и, следовательно, регулировать экспрессию генов-мишеней RIN в плодах томата.
С использованием РНК-интерференции и антисмысловой РНК были получены трансгенные растения банана с ограниченной транскрипцией генов MaMADS2 и MaMADS1 [46]. Было обнаружено, что у таких растений созревание плодов наступало позже, чем у растений дикого типа. При созревании плоды не становились мягче и не изменяли окраску, при этом увеличивалась их лежкость. Подавление транскрипции генов MaMADS2 и MaMADS1 приводило к изменению интенсивности дыхания и уровня синтеза этилена в плодах, а также изменяла экспрессию других MaMADS генов [46].
Результаты свидетельствуют, что в отличие от плодов томата, в плодах банана как минимум два MADS-ТФ могут играть роль, аналогичную RIN [46].
Интересно, что подобно гену RIN томата, гены MaMADS2 и MaMADS1 получили прикладное использование: у трансгенных растений банана с замалчиванием данных генов замедляется созревание и увеличивается лежкость плодов при хранении [74].
Растения, формирующие неклимактерические плоды
Перец. Перец (род Capsicum) относится к семейству Solanaceae и, следовательно, являются родственными томату растениями. Интересно, что из шести культивируемых видов перца (C. annuum L., C. chinense L, C. frutescens L, C. pubescens Ruiz and Pav, C. baccatum L. и C. assamicum Purkayastha and L.Singh.) только перец овощной C. annuum образует неклимактерические плоды, тогда как для плодов некоторых острых перцев был показан климактерический путь созревания [75–78].
У образца перца C. annuum сорта Bukang была клонирована и описана кодирующая последовательность гомолога гена RIN–CaMADS-RIN, содержащая открытую рамку считывания (732 п.н.) и нетранслируемые области 5′-НТО и 3′-НТО длиной 74 п.н. и 236 п.н., соответственно. Данный ген кодирует белок 243 а.о., в составе которого имеются все характерные для MADS-ТФ MIKC-типа консервативные структурные домены (MADS, I и K) и вариабельная C-область [8]. Максимум экспрессии CaMADS-RIN перца детектирован в начинающем краснеть и полностью покрасневшем плоде [8].
Для оценки роли ТФ CaMADS-RIN в процессе созревания плода, растения томата с rin-мутацией были трансформированы кассетой экспрессии транскрипта CaMADS-RIN под контролем конститутивного промотора 35S-CaMV. Полученные растения томата rin 35S-CaMADS-RIN сравнили с растением томата дикого типа и с rin-томатом. Было показано, что сверхэкспрессия CaMADS-RIN частично восстанавливала дикий фенотип плода томата, что выражалось в появлении желто-оранжевой окраски плода при созревании и подтверждало участие CaMADS-RIN в активации транскрипции генов биосинтеза каротиноидов. Содержание каротиноидов в таких плодах оказалось частично восстановленным и составило 30% от содержания каротиноидов в плодах дикого типа. Скорость созревания плодов rin 35S-CaMADS-RIN также возросла почти до уровня дикого типа, значительно превысив такие сроки у rin-плодов [8].
Анализ экспрессии основных генов каротиногенеза, которые являются мишенями RIN, показал, что у растений rin 35S-CaMADS-RIN повысился уровень транскрипции генов фитоиндесатуразы (PDS) и ζ-каротиндесатуразы (ZDS) по сравнению не только с rin-растением, но и с томатом дикого типа. Однако при этом экспрессия гена ключевого фермента метаболизма каротиноидов в плоде – фитоинсинтазы-1 (PSY1) снизилась по сравнению и с диким типом, и с rin-томатом [8].
Плоды томата со сверхэкспрессией CaMADS-RIN по мере созревания приобретали ту же мягкость, что и плоды дикого типа, чем существенно отличались от rin-плодов с твердой текстурой. В соответствии с этим изменялась экспрессия генов модификации клеточной стенки. Так, ген полигалактоуроназы (PG) экспрессировался в плодах растений rin 35S-CaMADS-RIN гораздо сильнее, чем в плодах с rin-мутацией, хотя и ниже, чем в плодах дикого типа. Также росла транскрипция генов β-галактозидазы-4 (TBG4) и α-экспансина-1 (EXP1), практически достигая уровня дикого типа [8].
Как было сказано выше, триггером процесса созревания в климактерических плодах является этилен, в то время как в неклимактерических плодах его роль не до конца ясна. Несмотря на то, что перец овощной образует неклимактерические плоды, сверхэкспрессия CaMADS-RIN в rin-плодах томата способствовала росту концентрации этилена в 1–3 раза по сравнению с rin-плодами, но при этом содержание этилена оставалось ниже, чем в плодах дикого типа. Это происходило за счет увеличения экспрессии генов ACS2, ACO1, E4 и E8 [8]. Таким образом, был сделан вывод о том, что ген CaMADS-RIN может запускать процессы биосинтеза этилена аналогично RIN томата, тем самым регулируя этилен-зависимые пути созревания сочного плода [8].
Чтобы определить, способен ли CaMADS-RIN перца регулировать этилен-независимые пути созревания плодов, была измерена экспрессия гена HDC, который у томата регулируется транскрипционным фактором RIN и нечувствителен к воздействию этилена. В плодах трансгенных растений rin 35S-CaMADS-RIN уровень транскрипции гена HDC был повышен по сравнению с rin-плодами, однако был существенно ниже, чем в плодах дикого типа. Также, сильно росла экспрессия гена ТФ NOR, с промотором которого в норме связывается RIN томата, причем не только в сравнении с rin-плодами, но и с плодами дикого типа [8]. Полученные данные указывают на то, что ТФ CaMADS-RIN способен регулировать как этилен-зависимые, так и этилен-независимые пути созревания плода.
Помимо перца овощного, ортологи RIN были выявлены и охарактеризованы у дикорастущих видов перца C. chinense и C. frutescens. У всех трех видов ген CaMADS-RIN экспрессируется максимально в плодах на стадии начала покраснения и в спелых красных плодах. Учитывая, что C. annuum образует неклимактерические плоды, а C. chinense и C. frutescens – климактерические, при том, что паттерны экспрессии CaMADS-RIN у всех видов схожи, был сделан вывод о том, что ТФ CaMADS-RIN регулирует созревание обоих типов плодов аналогичным образом [78].
Виноград. В геноме винограда (V. vinifera L. сорт Pinot noir) найдено два гена-кандидата на роль ортологов гена RIN томата, согласно их структуре, филогении и паттерну экспрессии [52]. Ген VviSEP-4 относится к кладе LOFSEP SEP4 (куда входит и RIN) [37, 38], а VviSEP3 – к кладе SEP3 [79].
Аналогично CaMADS-RIN перца овощного, кодирующие последовательности генов VviSEP3 и VviSEP4 были сверхэкспрессированы в трансгенном растении томата, несущем rin-мутацию, и в растениях дикого типа. Это привело к изменениям фенотипа спелого плода, подобным тем, как в случае сверхэкспрессии CaMADS-RIN [52].
Созревший плод томата с эктопической сверхэкспрессией VviSEP3 имел более желтую окраску, чем исходный rin-плод, что свидетельствовало о распаде хлорофилла-b при созревании плода. Однако желтая окраска плода в процессе созревания не становилась красной, как в случае дикого типа, что указывало на отсутствие биосинтеза ликопина, и было подтверждено биохимическим анализом [52]. В свою очередь сверхэкспрессия другого гомолога – гена VviSEP4 более радикально изменила фенотип спелого плода томата, который приобрел оранжево-красную окраску, но не ярко-красную, как плоды дикого типа, хотя содержание как ликопина, так и бета-каротина в плодах со сверхэкспрессией VviSEP4 оказалось сопоставимым с таковым в плодах дикого типа [52].
Таким образом, была продемонстрирована способность гена VviSEP4 максимально компенсировать функцию RIN в rin-плодах томата, в большей степени, нежели это может делать ген VviSEP3, несмотря на то, что последний структурно ближе гену RIN.
Земляника. Земляника (F. × ananassa Duch.) является модельной системой для изучения биологии неклимактерических плодов. Плоды земляники – ложные ягоды (многоорешки), состоящие из разросшегося сочного цветоложа и орешков на его поверхности. Характерно, что ген земляники FvMADS9 был идентифицирован с использованием гена RIN томата в качестве зонда. Так же, как и у гена RIN, экспрессия FvMADS9 специфична для плодов [4].
Сведения о гене FvMADS9 обширны, но иногда противоречивы [9, 80]. Выключение FaMADS9 у образца F. × anannasa сорт Calypso нарушало развитие лепестков и плодов, а также процесс самоопыления [80]. Цветки таких растений формировали лепестки с зеленой окраской, опадение которых либо задерживалось, либо не происходило вовсе, приводя к тому, что завязавшийся плод не развивался. У трансгенных линий, в которых плодам удалось достичь стадии созревания, данный процесс происходил с изменениями, как в цветоложе, так и в орешках. В зреющих семенах трансгенных линий с нокаутированным геном FaMADS9 хлорофилл не разрушался, и плоды были значительно тверже, чем у растений дикого типа. Так, накопление антоцианов в цветоложе задерживалось на 10 дней и происходило неравномерно, а их содержание всегда было меньше по сравнению с растениями дикого типа. Чем ниже был уровень экспрессии гена FaMADS9, тем больше фенотипических отклонений детектировалось [80].
Интересно, что замалчивание транскрипции FaMADS9 у растений земляники другого сорта (Camarosa) не приводило к существенному изменению фенотипа. Плоды оставались такими же красными и мягкими, как и плоды дикого типа, однако имели гораздо более низкое содержание растворимых сухих веществ [9]. Тем не менее, при сравнении данных транскриптомного анализа плодов сорта Camarosa с выключенной экспрессией FaMADS9 с плодами дикого типа было выявлено 2670 дифференциально экспрессирующихся генов на стадии белого плода, и 1943 – на стадии красного плода [9]. Среди найденных генов выделено несколько функциональных групп. Первую (82%) составили ТФ, в частности ТФ ауксинового ответа. Следующая большая группа генов связана с процессами роста клеток и энергетическим метаболизмом, что указывает на важную роль FaMADS9 в процессах роста цветоложа на ранних этапах его развития. Третья группа генов оказалась связана с транспортом. Также у трансгенных линий изменялась экспрессия генов β-амилаз и изоамилазы (что приводило к нарушениям в метаболизме крахмала), полигалактоуроназ и генов биосинтеза жирных кислот, а также снижалась экспрессия генов биосинтеза аскорбиновой кислоты GME и VTC1 (из-за чего уменьшалось содержание аскорбата) [9]. Все эти данные могут помочь составить представление не только о роли FaMADS9 в процессе созревания плода, но и о соответствующих регуляторных механизмах.
В отличие от гена RIN томата, мутация в котором приводит только к изменению созревания плода, замалчивание или снижение экспрессии гена FaMADS9 приводило к нарушению и более ранних этапов развития плода [80]. Следует отметить, что земляника является октоплоидом, и чем больше аллелей затрагивал нокаут, тем в большей степени были нарушены процессы развития плода, вплоть до полного отсутствия его завязывания [80]. Допустимо поэтому, что ген земляники FaMADS9, в отличие от RIN томата, имеет более широкий функциональный спектр. К примеру, способность регулировать транскрипцию измененного, расширенного набора генов-мишеней, взаимодействовать с другими и/или дополнительными белковыми партнерами и иметь специфичные GArG-консенсусы в промоторах регулируемых генов.
Фенотип лепестков земляники с выключенным FaMADS9 (зеленая окраска, отсутствие опадения) соотносится с фенотипом лепестков томата с замалчиванием гена ТФ TM29 клады SEP1/2 [80]. С другой стороны, данные об отсутствии выраженного фенотипа, полученные в исследовании [9], могут указывать на неспецифичность сайленсинга гена FaMADS9 в исследовании [80]: поскольку MADS-гены имеют консервативную структуру, могло произойти дополнительное замалчивание какого-либо другого MADS-гена (генов), помимо FaMADS9 [9].
АКТУАЛЬНЫЕ ВОПРОСЫ И БУДУЩИЕ ПЕРСПЕКТИВЫ
Идентификация генов гомологов RIN у новых видов растений. На сегодняшний день подавляющее большинство исследований, предметом которых являются ТФ RIN или кодирующий его ген, сфокусированы только на культивируемых видах широко используемых сельскохозяйственных растений (а именно плодах экономически значимых овощей и фруктов). Однако, чтобы лучше понять эволюцию, структуру и функции данного белка, необходима идентификация гомологичных генов у дикорастущих родственных видов. Сравнительная структурная и функциональная характеристика генов-гомологов RIN и кодируемых ими ТФ RIN культивируемых растений (для которых имеется детальное описание признаков сочного плода) и гомологичных генов дикорастущих родственных видов (плоды которых характеризуются нарушением отдельных аспектов созревания, в силу чего они несъедобны) может расширять наши представления о природе ТФ RIN и регулируемых им генах.
Помимо томата обыкновенного (S. lycopersicum) к секции Lycopersicon относят 12 дикорастущих родственных видов томата, которые также образуют сочный плод. Процесс созревания у наиболее древних видов томата отличается от такового у томата обыкновенного: не накапливаются каротиноиды и моносахара, сроки развития плодов существенно увеличены [81, 82]. По многим морфологическим и биохимическим характеристикам плоды дикорастущих видов томата напоминают плоды rin-мутанта культивируемого томата. Идентификация генов RIN дикорастущих видов, сравнение их структуры и аллельной вариабельности с геном RIN томата обыкновенного и rin-мутантных растений S. lycopersicum даст возможность понять эволюцию данного транскрипционного фактора и его участие в доместикации растений, а также определит дальнейшие перспективы для биотехнологического применения. Проведенный ранее анализ межвидовой вариабельности и экспрессии некоторых структурных генов созревания, которые являются мишенями ТФ RIN у дикорастущих видов и культивируемых сортов томата, показал наличие дифференциальной экспрессии у образцов с различными характеристиками созревания плода [83–86]. Подобные результаты могут быть объяснены не только структурной вариабельностью самих генов биосинтеза каротиноидов и метаболизма сахаров и промоторов этих генов, но и различиями в структуре и активности контролирующего их ТФ RIN, что в дальнейшем предстоит определить.
Поиск факторов, регулирующих активность RIN. Среди имеющихся на сегодня данных оказалось крайне сложно найти информацию о факторах, регулирующих работу самого RIN. Очевидно, что ТФ RIN является одним из ключевых регуляторов созревания, но полная картина о том, какие внешние или внутренние факторы задействованы в транскрипционной регуляции кодирующего его гена, только формируется. Исследование механизмов, лежащих в основе регуляции транскрипции RIN, позволит управлять процессом созревания сочного плода, что, несомненно, имеет важное прикладное значение.
Несмотря на то, что направленные исследования вышестоящих (upstream) регуляторов RIN не проводились, за последние несколько лет при описании белков различных семейств, экспрессирующихся в плодах, среди прочих функций была показана возможность их непосредственного или опосредованного влияния на транскрипцию гена RIN и/или активность ТФ RIN.
Найденные факторы можно разделить на две группы: (1) белки, связанные с эпигенетическими модификациями ДНК и гистонов; (2) – другие белки, связанные с синтезом отдельных метаболитов, и данные метаболиты (сахара, каротиноиды, гормоны, мелатонин).
В процессе созревания и старения растительных плодов могут происходить эпигенетические модификации [87, 88]. К примеру, у томата ингибирование активности ДНК-метилтрансфераз приводит к преждевременному созреванию плода [88]. Важным фактором, определяющим структурную организацию хроматина и экспрессию генов, является метилирование и деметилирование гистонов, которое осуществляется гистоновыми лизинметилтрансферазами и деметилазами [88].
В геноме томата найдено 25 гистоновых деметилаз (HDMs); экспрессия многих из них коррелирует с запуском процесса созревания плода [88]. На сегодняшний день получены данные, свидетельствующие о достоверном влиянии одной из таких гистоновых деметилаз – SlJMJ6, на процессы созревания плода томата [88]. SlJMJ6 ускоряет созревание плодов в результате деметилирования H3K27m3, что приводит к активации экспрессии 32 генов, контролирующих, в том числе, созревание [88]. Наибольший интерес представляет то, что SlJMJ6 может деметилировать участок хроматина, содержащий RIN, что активирует его экспрессию. Важным представляется и тот факт, что помимо этого, гистоновая деметилаза SlJMJ6 способна активировать и экспрессию генов-мишеней ТФ RIN (рис. 1) [88].
На примере плодов томата также показано, что на скорость созревания может влиять и ацетилирование гистонов. Снижение экспрессии гена гистоновой деацетилазы SlHDA3 ускоряет созревание плодов за счет увеличения экспрессии генов биосинтеза и сигнальных путей этилена, некоторых каротиноидов, генов модификации клеточной стенки, а также генов ТФ RIN, CNR и TAGL1 [89]. Однако обращает на себя внимание то, что среди генов созревания, чья экспрессия изменилась после ограничения работы SlHDA3, большинство являются мишенями ТФ RIN. Таким образом, можно предположить, что полученные результаты являются не следствием непосредственной регуляции экспрессии этих генов за счет ацетилирования/деацетилирования гистонов, а скорее следствием активации или деактивации экспрессии гена RIN при изменении активности гистоновой ацетилазы SlHDA3, которая является негативным регулятором RIN. Однако остается неясным, могут ли другие гистоновые деацетилазы и ацетилтранферазы оказывать на экспрессию RIN аналогичное действие (то есть всегда ли ацетилирование гистонов приводит к активации RIN), либо этот ген регулируется специфично за счет работы SlHDA3 (рис. 1).
Не менее важно отметить, что подобно модификации гистоновых белков, фактором активации гена RIN также является деметилирование ДНК. В частности, показано, что специфичная для плодов ДНК-деметилаза DML2 активирует экспрессию генов главных регуляторов созревания – ТФ RIN, NOR и CNR, за счет уменьшения метилирования ДНК [90, 91]. Интересно, что экспрессия гена DML2 была сильно увеличена в плодах растений томата со сверхэкспрессией гена гистоновой деметилазы SlJMJ6 по сравнению с плодами дикого типа. Более того, показано, что SlJMJ6 напрямую связывается с локусом DML2, снимает метилирование H3K27me3, в результате чего происходит активация транскрипции DML2, а кодируемый белок DML2, в свою очередь, снимает метилирование с ДНК-последовательности RIN, что приводит к активации его экспрессии (рис. 1) [88].
Таким образом, можно сделать вывод, что эпигенетические модификации ДНК и гистоновых белков являются одним из факторов, запускающих экспрессию гена RIN. Однако неясным остается, какие еще гистоновые деметилазы (помимо SlJMJ6), которых у томата описано 25 и подавляющее большинство которых активируется именно в процессе развития и созревания плодов, участвуют в активации гена RIN.
Что касается других видов растений, данные о конкретных регуляторах экспрессии гомологов RIN не найдены, однако для них доступны общие данные по изменению динамики метилирования ДНК в процессе созревания. Принято считать, что у климактерических плодов (показано на примере томата), процесс созревания сопровождается снятием метилирования ДНК за счет увеличения экспрессии генов ДНК-деметилаз [92]. У неклимактерических плодов (показано на примере земляники) процесс созревания также сопровождается деметилированием ДНК, однако у них не наблюдается увеличения экспрессии генов ДНК-деметилаз, но подавляется экспрессия генов, участвующих в РНК-направленном метилировании ДНК [92]. В отличие от томата и земляники, у другого неклимактерического растения – апельсина, процесс созревания плода принципиально иной и сопровождался глобальным метилированием ДНК, предположительно, за счет уменьшения экспрессии генов ДНК деметилаз [93].
Помимо эпигенетических факторов на активацию RIN и созревание плодов могут оказывать влияние некоторые отдельные метаболиты. Прежде всего, это простые сахара, которые выступают не только, как энергетический субстрат клетки, но и как сигнальные молекулы, а также каротиноиды, фитогормоны и другие метаболиты.
Известно, что в регуляции внутриклеточного сигналинга и метаболизма сахаров при созревании плода участвует протеинкиназа SnRK1 [94]. Так, гетерологичная сверхэкспрессия генов MhSnRK1 (яблоня) и PpSnRK1α (персик) в растениях томата приводила к изменению характеристик спелого плода, увеличению количества растворимых сахаров и крахмала и уменьшению сроков созревания [94–96]. Наличие прямого взаимодействия между PpSnRK1α и ТФ RIN было подтверждено in vivo и in vitro. Более того, сверхэкспрессия гена PpSnRK1α также вызывала увеличение уровня экспрессии не только гена RIN, но и его генов-мишеней (NOR, FUL1, ACS2, ACS4, E8) [94]. Исходя из этого, следует, что протеинкиназа PpSnRK1α взаимодействует с ТФ RIN, увеличивая экспрессию кодирующего гена RIN, и, следовательно, генов-мишеней ТФ RIN, тем самым участвуя в контроле созревания. Таким образом, SnRK1α может выступать регулятором активности RIN как на белковом, так и на транскрипционном уровне (рис. 1). Возможно, SnRK1 способна фосфорилировать белок RIN, однако, остается вопрос о том, влияет ли SnRK1 напрямую на гены-мишени ТФ RIN, или же это происходит опосредованно. Кроме того, активность SnRK1 в клетке приводит к увеличению интенсивности фотосинтеза и, как следствие, накоплению сахаров и других важных метаболитов. Могут ли они в свою очередь также послужить активатором RIN – еще предстоит выяснить.
Другой фермент, фитоиндесатураза PDS участвует в биосинтезе каротиноидов; снижение экспрессии гена PDS в плодах томата помимо сокращения содержания каротиноидов, приводило к уменьшению экспрессии гена RIN, а также ряда других генов созревания (большинство из которых являются мишенями ТФ RIN) [97]. Была показана корреляция между экспрессией генов PDS и RIN, но не ясно, за счет каких молекулярных механизмов это происходит (рис. 1). Можно предположить, что имеет место либо непосредственное взаимодействие между белками PDS и ТФ RIN (или PDS и геном RIN), либо отсутствие каротиноидов приводит к изменению концентраций других метаболитов и белков, которые в свою очередь репрессируют экспрессию гена RIN. Например, каротиноиды могут быть предшественниками для биосинтеза абсцизовой кислоты, которая потенциально может служить регулятором экспрессии RIN. Также можно предположить наличие обратной взаимосвязи между конечными продуктами реакций, которые осуществляют структурные гены созревания (мишени ТФ RIN) и активацией/репрессией самого гена RIN.
Помимо широко изученных метаболитов, связанных с созреванием плода, обращают на себя внимание новые мало описанные соединения, например, мелатонин (N-цетил-5-метокситриптамин). У томата обработка экзогенным мелатонином приводила к ускоренному созреванию плода и улучшению питательных характеристик [98]. Сравнение протеомов растений томата, обработанных экзогенным мелатонином, и контрольных растений показало, что среди других белков, концентрации которых существенно отличаются у двух групп образцов, есть и белок ТФ RIN, количество которого у образца, обработанного мелатонином, было выше, чем у контрольного растения [98]. Таким образом, на уровне белка показана корреляция количества мелатонина и ТФ RIN, но неясными остаются молекулярные механизмы данного явления: оказывает мелатонин прямое или опосредованное через другие метаболиты, белки или эпигенетические факторы действие на активацию экспрессии гена RIN (рис. 1).
ЗАКЛЮЧЕНИЕ
В настоящем обзоре изложены сведения о функциях транскрипционного фактора RIN томата и его гомологах у растений различных видов и семейств. Приведенные данные свидетельствуют о том, что MADS-белки подсемейства SEP играют центральную роль в чрезвычайно сложной транскрипционной регуляции каскада реакций созревания как климактерических, так и неклимактерических плодов однодольных и двудольных растений. Генная сеть, контролируемая SEP-белками, включает в себя не только регуляторные и структурные гены различных семейств, но и некодирующие РНК-последовательности – микроРНК и днкРНК. С помощью данной сети происходит регуляция путей биосинтеза фитогормонов, витаминов, пигментов и других вторичных метаболитов и биологически активных соединений, что, в свою очередь, определяет время созревания, текстуру, окраску и биохимию плода. Важно понимать, что в основе морфологического многообразия сочного плода различных видов высших растений может лежать эволюционная дупликация и диверсификация MADS-генов подсемейства SEP. При этом разные виды растений могли задействовать SEP-гены различных клад, а не только SEP4-гены, к которым принадлежит ген томата RIN. Кроме того, также возможно существование видов растений, где RIN-функцию выполняет не один, а несколько SEP-паралогов. Наконец, события дупликации и диверсификации происходили не только с SEP-генами, но и другими MADS-генами, поэтому всегда необходимо учитывать функции и структурные особенности MADS-партнеров SEP-ТФ, совместно с которыми осуществляется регуляция процессов, происходящих при созревании плода.
Работа выполнена при поддержке Российского научного фонда (грант № 19-76-00006) и, частично, Министерства науки и высшего образования Российской Федерации.
Автор заявляет об отсутствии конфликта интересов. Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследования.
Список литературы
Karlova R., Chapman N., David K., Angenent G.C., Seymour G.B., de Maagd R.A. Transcriptional control of fleshy fruit development and ripening // J. Exp. Bot. 2014. V. 65. P. 4527. https://doi.org/10.1093/jxb/eru316
Valenta K., Kalbitzer U., Razafimandimby D., Omeja P., Ayasse M., Chapman C.A., Nevo O. The evolution of fruit colour: phylogeny, abiotic factors and the role of mutualists // Sci. Rep. 2018. V. 8: 14302. https://doi.org/10.1038/s41598-018-32604-x
Hewitt S., Dhingra A. Beyond Ethylene: New Insights Regarding the Role of Alternative Oxidase in the Respiratory Climacteric // Front. Plant Sci. 2020. V. 11: 543958. https://doi.org/10.3389/fpls.2020.543958
Vrebalov J., Ruezinsky D., Padmanabhan V., White R., Medrano D., Drake R., Schuch W., Giovannoni J. A MADS-box gene necessary for fruit ripening at the tomato ripening-inhibitor (rin) locus // Science. 2002. V. 296. P. 343.
Alexander L., Grierson D. Ethylene biosynthesis and action in tomato: a model for climacteric fruit ripening // J. Exp. Bot. 2002. V. 53. P. 2039. https://doi.org/10.1093/jxb/erf072
Chervin C., El-Kereamy A., Roustan J.P., Latche A., Lamon J., Bouzayen M. Ethylene seems required for the berry development and ripening in grape, a non-climacteric fruit // Plant Science. 2004. V. 167. P. 1301.
Cazzonelli C.I., Cavallaro A.S. Botella J.R. Cloning and characterisation of ripening-induced ethylene biosynthetic genes from non-climacteric pineapple (Ananas comosus) fruits // Funct. Plant Biol. 1998. V. 25. P. 513. https://doi.org/10.1071/PP98013
Dong T., Chen G., Tian S., Xie Q., Yin W., Zhang Y., Hu Z. A non-climacteric fruit gene CaMADS-RIN regulates fruit ripening and ethylene biosynthesis in climacteric fruit // PloS One. 2014. V. 9: e95559. https://doi.org/10.1371/journal.pone.0095559
Vallarino J.G., Merchante C., Sánchez-Sevilla J.F., de Luis Balaguer M.A., Pott D.M., Ariza M.T., Casañal A., Posé D., Vioque A., Amaya I., Willmitzer L., Solano R., Sozzani R., Fernie A.R., Botella M.A., Giovannoni J.J., Valpuesta V., Osorio S. Characterizing the involvement of FaMADS9 in the regulation of strawberry fruit receptacle development // Plant Biotechnol. J. 2020. V. 18. P. 929. https://doi.org/10.1111/pbi.13257.20
Csukasi F., Osorio S., Gutierrez J.R., Kitamura J., Giavalisco P., Nakajima M., Fernie A.R., Rathjen J.P., Botella M.A., Valpuesta V., Medina-Escobar N. Gibberellin biosynthesis and signalling during development of the strawberry receptacle // New Phytol. 2011. V. 191. P. 376. https://doi.org/10.1111/j.1469-8137.2011.03700.x
Chai Y.M., Jia H.F., Li C.L., Dong Q.H., Shen Y.Y. FaPYR1 is involved in strawberry fruit ripening // J. Exp. Bot. 2011. V. 62. P. 5079.
Jia H., Chai Y., Li C., Lu D., Luo J., Qin L., Shen Y. Abscisic acid plays an important role in the regulation of strawberry fruit ripening // Plant Physiol. 2011. V. 157. P. 188.
Concha C.M., Figueroa N.E., Poblete L.A., Oñate F.A., Schwab W., Figueroa C.R. Methyl jasmonate treatment induces changes in fruit ripening by modifying the expression of several ripening genes in Fragaria chiloensis fruit // Plant Physiol. Biochem. 2013. V. 70. P. 433.
Chai Y.M., Zhang Q., Tian L., Li C.L., Xing Y., Qin L., Shen Y.Y. Brassinosteroid is involved in strawberry fruit ripening // Plant Growth Regul. 2013. V. 69. P. 63.
Perkins-Veazie P. Growth and ripening of strawberry fruit // Hortic. Rev. 1995. V. 17. P. 267.
Merchante C., Vallarino J.G., Osorio S., Aragüez I., Villarreal N., Ariza M.T., Martínez G.A., Medina-Escobar N., Civello M.P., Fernie A.R., Botella M.A., Valpuesta V. Ethylene is involved in strawberry fruit ripening in an organ-specific manner // J. Exp. Bot. 2013. V. 64. P. 4421. https://doi.org/10.1093/jxb/ert257
Fujisawa M., Shima Y., Nakagawa H., Kitagawa M., Kimbara J., Nakano T., Kasumi T., Ito Y. Transcriptional regulation of fruit ripening by tomato FRUITFULL homologs and associated MADS box proteins // Plant Cell. 2014. V. 26. P. 89.
Alvarez-Buylla E.R., Pelaz S., Liljegren S.J., Gold S.E., Burgeff C., Ditta G.S., Ribas de Pouplana L., Martinez-Castilla L., Yanofsky M.F. An ancestral MADS-box gene duplication occurred before the divergence of plants and animals // PNAS. 2000. V. 97. P. 5328.
Parenicová L., de Folter S., Kieffer M., Horner D.S., Favalli C., Busscher J., Cook H.E., Ingram R.M., Kater M.M., Davies B., Angenent G.C., Colombo L. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis: new openings to the MADS world // Plant Cell. 2003. V. 15. P. 1538. https://doi.org/10.1105/tpc.011544
Robinson R., Tomes M. Ripening inhibitor: a gene with multiple effect on ripening Tomato // Genet. Coop. 1968. V. 18. P. 36.
Tigchelaar E., Tomes M., Kerr E., Barman R. A new fruit ripening mutant, non-ripening (nor) // Rep. Tomato Genet. Coop. 1973. V. 23. P. 33.
Thompson A.J., Tor M., Barry C.S., Vrebalov J., Orfila C., Jarvis M.C., Giovannoni J.J., Grierson D., Seymour G.B. Molecular and genetic characterization of a novel pleiotropic tomato-ripening mutant // Plant Physiol. 1999. V. 120. P. 383.
Zhong S., Fei Z., Chen Y.R., Zheng Y., Huang M., Vrebalov J., McQuinn R., Gapper N., Liu B., Xiang J., Shao Y., Giovannoni J.J. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening // Nat. Biotechnol. 2013. V. 31. P. 154.
Wang R., Angenent G.C., Seymour G., de Maagd R.A. Revisiting the Role of Master Regulators in Tomato Ripening // Trends Plant Sci. 2020. V. 25. P. 291. https://doi.org/10.1016/j.tplants.2019.11.005
Wang R., Lammers M., Tikunov Y., Bovy A.G., Angenent G.C., de Maagd R.A. The rin, nor and Cnr spontaneous mutations inhibit tomato fruit ripening in additive and epistatic manners // Plant Sci. 2020. V. 294: 110436. https://doi.org/10.1016/j.plantsci.2020.110436
Fujisawa M., Nakano T., Shima Y., Ito Y. A large-scale identification of direct targets of the tomato MADS box transcription factor RIPENING INHIBITOR reveals the regulation of fruit ripening // Plant Cell. 2013. V. 25. P. 371.
Qin G., Wang Y., Cao B., Wang W., Tian S. Unraveling the regulatory network of the MADS box transcription factor RIN in fruit ripening // Plant J. 2012. V. 70. P. 243.
Osorio S., Alba R., Damasceno C.M., Lopez-Casado G., Lohse M., Zanor M.I., Tohge T., Usadel B., Rose J.K., Fei Z., Giovannoni J.J., Fernie A.R. Systems biology of tomato fruit development: combined transcript, protein, and metabolite analysis of tomato transcription factor (nor, rin) and ethylene receptor (Nr) mutants reveals novel regulatory interactions // Plant Physiol. 2011. V. 157. P. 405.
Ito Y. Regulation of Tomato Fruit Ripening by MADS-Box Transcription Factors // Horticulture. 2016. V. 50 P. 33.
Azzi L., Deluche C., Gévaudant F., Frangne N., Delmas F., Hernould M., Chevalier C. Fruit growth-related genes in tomato // J. Exp. Bot. 2015. V. 66. P. 1075. https://doi.org/10.1093/jxb/eru527
Li S., Chen K., Grierson D. A critical evaluation of the role of ethylene and MADS transcription factors in the network controlling fleshy fruit ripening // New Phytol. 2019. V. 221. P. 1724. https://doi.org/10.1111/nph.15545
Ito Y., Nishizawa-Yokoi A., Endo M., Mikami M., Toki S. CRISPR/ Cas9-mediated mutagenesis of the RIN locus that regulates tomato fruit ripening // Biochem. Biophys. Res. Commun. 2015. V. 467. P. 76. https://doi.org/10.1016/j. bbrc.2015.09.117
Ito Y., Nishizawa-Yokoi A., Endo M., Mikami M., Shima Y., Nakamura N., Kotake-Nara E., Kawasaki S., Toki S. Re-evaluation of the rin mutation and the role of RIN in the induction of tomato ripening // Nature Plants. 2017. V. 3. P. 866. https://doi.org/10.1038/s41477- 017-0041-5
Li S., Zhu B., Pirrello J., Xu C., Zhang B., Bouzayen M., Chen K., Grierson D. Roles of RIN and ethylene in tomato fruit ripening and ripening-associated traits // New Phytol. 2020. V. 226. P. 460. https://doi.org/10.1111/nph.16362
Ito Y., Sekiyama Y., Nakayama H., Nishizawa-Yokoi A., Endo M., Shima Y., Nakamura N., Kotake-Nara E., Kawasaki S., Hirose S., Toki S. Allelic Mutations in the Ripening-Inhibitor Locus Generate Extensive Variation in Tomato Ripening // Plant Physiol. 2020. V. 183. P. 80. https://doi.org/10.1104/pp.20.00020
Gao Y., Zhu N., Zhu X., Wu M., Jiang C.Z., Grierson D., Luo Y., Shen W., Zhong S., Fu D.Q., Qu G. Diversity and redundancy of the ripening regulatory networks revealed by the fruitENCODE and the new CRISPR/Cas9 CNR and NOR mutants // Hortic Res. 2019. V. 6: 39. https://doi.org/10.1038/s41438-019-0122-x
Malcomber S.T., Kellogg E.A. SEPALLATA gene diversification: brave new whorls // Trends Plant Sci. 2005. V. 10. P. 427. https://doi.org/10.1016/j.tplants.2005.07.008
Díaz-Riquelme J., Lijavetzky D., Martínez-Zapater J.M., Carmona M.J. Genome-wide analysis of MIKCC -type MADS box genes in grapevine. Plant Physiol. 2009. V. 149. P. 354. https://doi.org/10.1104/pp.108.131052
Shore P., Sharrocks A.D. The MADS-box family of transcription factors // Eur. J. Biochem. 1995. V. 229. P. 1.
Huang K., Louis J.M., Donaldson L., Lim F.L., Sharrocks A.D., Clore G.M. Solution structure of the MEF2A-DNA complex: structural basis for the modulation of DNA bending and specificity by MADS-box transcription factors // EMBO J. 2000. V. 19. P. 2615.
Egea-Cortines M., Saedler H., Sommer H. Ternary complex formation between the MADS-box proteins SQUAMOSA, DEFICIENS and GLOBOSA is involved in the control of floral architecture in Antirrhinum majus // EMBO J. 1999. V. 19. P. 5370. https://doi.org/10.1093/emboj/18.19.5370
Garcia-Maroto F., Carmona M.-J., Garrido J.-A., Vilches-Ferron M., Rodriguez-Ruiz J., Alonso L.D. New roles for MADS-box genes in higher plants // Biologia Plantarum. 2003. V. 46. P. 321.
De Bodt S., Raes J., Florquin K., Rombauts S., Rouzé P., Theissen G., Van de Peer Y. Genomewide structural annotation and evolutionary analysis of the type I MADS-box genes in plants // J. Mol. Evol. 2003. V. 56. P. 573.
Elitzur T., Vrebalov J., Giovannoni J.J., Goldschmidt E.E., Friedman H. The regulation of MADS-box gene expression during ripening of banana and their regulatory interaction with ethylene // J. Exp. Bot. 2010. V. 61. P. 1523. https://doi.org/10.1093/jxb/erq017
Slugina M.A., Dyachenko E.A., Kochieva E.Z., Shchennikova A.V. Structural and Functional Diversification of SEPALLATA Genes TM5 and RIN in Tomato Species (Section Lycopersicon) // Dokl. Biochem. Biophys. 2020. V. 492. P. 152. https://doi.org/10.1134/S1607672920030102
Elitzur T., Yakir E., Quansah L., Zhangjun F., Vrebalov J., Khayat E., Giovannoni J.J., Friedman H. Banana MaMADS Transcription Factors Are Necessary for Fruit Ripening and Molecular Tools to Promote Shelf-Life and Food Security // Plant Physiol. 2016. V. 171. P. 380. https://doi.org/10.1104/pp.15.01866
Sánchez-Sevilla J.F., Vallarino J.G., Osorio S., Bombarely A., Posé D., Merchante C., Botella M.A., Amaya I., Valpuesta V. Gene expression atlas of fruit ripening and transcriptome assembly from RNA-seq data in octoploid strawberry (Fragaria × ananassa) // Sci. Rep. 2017. V. 7: 13737.
Boss P.K., Sensi E., Hua C., Davies C., Thomas M.R. Cloning and characterisation of grapevine (Vitis vinifera L.) MADS-box genes expressed during inflorescence and berry development // Plant Sci. 2002. V. 162. P. 887.
Deluc L.G., Grimplet J., Wheatley M.D., Tillett R.L., Quilici D.R., Osborne C., Schooley D.A., Schlauch K.A., Cushman J.C., Cramer G.R. Transcriptomic and metabolite analyses of Cabernet Sauvignon grape berry development // BMC Genomics. 2007. V. 8: 429
Pilati S., Perazzolli M., Malossini A., Cestaro A., Demattè L., Fontana P., Dal Ri A., Viola R., Velasco R., Moser C. Genome-wide transcriptional analysis of grapevine berry ripening reveals a set of genes similarly modulated during three seasons and the occurrence of an oxidative burst at vèraison // BMC Genomics. 2007. V. 8: 428. https://doi.org/10.1186/1471-2164-8-428
Zenoni S., Ferrarini A., Giacomelli E., Xumerle L., Fasoli M., Malerba G., Bellin D., Pezzotti M., Delledonne M. Characterization of transcriptional complexity during berry development in Vitis vinifera using RNA-Seq // Plant Physiol. 2010. V. 152. P. 1787. https://doi.org/10.1104/pp.109.149716
Mellway R.D., Lund S.T. Interaction analysis of grapevine MIKCc-type MADS transcription factors and heterologous expression of putative véraison regulators in tomato // J. Plant Physiol. 2013. V. 16. P. 1424.
Tranbarger T.J., Dussert S., Joët T., Argout X., Summo M., Champion A., Cros D., Omore A., Nouy B., Morcillo F. Regulatory mechanisms underlying oil palm fruit mesocarp maturation, ripening, and functional specialization in lipid and carotenoid metabolism // Plant Physiol. 2011. V.156. P. 564.
Moyle R.L., Koia J.H., Vrebalov J., Giovannoni J., Botella J.R. The pineapple AcMADS1 promoter confers high level expression in tomato and Arabidopsis flowering and fruiting tissues, but AcMADS1 does not complement the tomato LeMADS-RIN (rin) mutant // Plant Mol. Biol. 2014. V. 86. P. 395. https://doi.org/10.1007/s11103-014-0236-3
Ito Y., Kitagawa M., Ihashi N., Yabe K., Kimbara J., Yasuda J., Ito H., Inakuma T., Hiroi S., Kasumi T. DNA-binding specificity, transcriptional activation potential, and the rin mutation effect for the tomato fruit-ripening regulator RIN // Plant J. 2008. V. 26. P. 212. https://doi.org/10.1111/j.1365-313X.2008.03491.x
Fujisawa M., Nakano T., Ito Y. Identification of potential target genes for the tomato fruit-ripening regulator RIN by chromatin immunoprecipitation // BMC Plant Biol. 2011. V. 11: 26. https://doi.org/10.1186/1471-2229-11-26
Robles P., Pelaz S. Flower and fruit development in Arabidopsis thaliana // Int. J. Dev. Biol. 2005. V. 49. P. 633. https://doi.org/10.1387/ijdb.052020pr
Leseberg C.H., Eissler C.L., Wang X., Johns M.A., Duvall M.R., Mao L. Interaction study of MADS-domain proteins in tomato // J. Exp. Bot. 2008. V. 59. P. 2253. https://doi.org/10.1093/jxb/ern094
Shima Y., Fujisawa M., Kitagawa M., Nakano T., Kimbara J., Nakamura N., Shiina T., Sugiyama J., Nakamura T., Kasumi T., Ito Y. Tomato FRUITFULL homologs regulate fruit ripening via ethylene biosynthesis // Biosci., Biotechnol., Biochem. 2014. V. 72. P. 231. https://doi.org/10.1080/09168451.2014.878221
Fujisawa M., Shima Y., Higuchi N., Nakano T., Koyama Y., Kasumi T., Ito Y. Direct targets of the tomato-ripening regulator RIN identified by transcriptome and chromatin immunoprecipitation analyses // Planta. 2012. V. 235. P. 1107.
Li S., Xu H., Ju Z., Cao D., Zhu H., Fu D., Grierson D., Qin G., Luo Y., Zhu B. The RIN-MC Fusion of MADS-Box Transcription Factors Has Transcriptional Activity and Modulates Expression of Many Ripening Genes // Plant Physiol. 2018. V. 176. P. 891. https://doi.org/10.1104/pp.17.01449
Collin V., Lamkemeyer P., Miginiac-Maslow M., Hirasawa M., Knaff D.B., Dietz K.J., Issakidis-Bourguet E. Characterization of plastidial thioredoxins from Arabidopsis belonging to the new y-type // Plant Physiol. 2004. V. 136. P. 4088. https://doi.org/10.1104/pp.104.052233
Gao C., Ju Z., Cao D., Zhai B., Qin G., Zhu H., Fu D., Luo Y., Zhu B. MicroRNA profiling analysis throughout tomato fruit development and ripening reveals potential regulatory role of RIN on microRNAs accumulation // Plant Biotechnol. J. 2015. V. 13. P. 370.
Yu T., Tzeng D.T.W., Li R., Chen J., Zhong S., Fu D., Zhu B., Luo Y., Zhu H. Genome-wide identification of long non-coding RNA targets of the tomato MADS box transcription factor RIN and function analysis // Ann. Bot. 2019. V. 123. P. 469. https://doi.org/10.1093/aob/mcy178
Jin J., Liu J., Wang H., Wong L., Chua N.-H. PLncDB: plant long non-coding RNA database // Bioinformatics. 2013. V. 29. P. 1068.
Quinet M., Angosto T., Yuste-Lisbona F.J., Blanchard-Gros R., Bigot S., Martinez J.P., Lutts S. Tomato Fruit Development and Metabolism // Front. Plant Sci. 2019. V. 10:1554. https://doi.org/10.3389/fpls.2019.01554
Tomato Genome Consortium. The tomato genome sequence provides insights into fleshy fruit evolution // Nature. 2012. V. 485. P. 635. PMID: ; PMCID: PMC3378239.https://doi.org/10.1038/nature1111922660326
Lincoln J.E., Fischer R.L. Regulation of Gene Expression by Ethylene in Wild-Type and rin Tomato (Lycopersicon esculentum) Fruit // Plant Physiol. 1988. V. 88. P. 370. https://doi.org/10.1104/pp.88.2.370
Yen H.-C., Lee S., Tanksley S.D., Lanahan M.B., Klee H.J., Giovannoni J.J. The tomato Never-ripe locus regulates ethylene-inducible gene expression and is linked to a homolog of the Arabidopsis ETR1 gene // Plant Physiol. 1995. V. 107. P. 1343.
Kuzmina Y. Methods of genome editing for increasing the shelf life of tomato fruit // Plant Biotechnology and Breeding. 2020. V. 3. P. 31. https://doi.org/10.30901/2658-6266-2020-1-o6
Giovannoni J.J. Genetic regulation of fruit development and ripening // The Plant Cell. 2004. V. 16. P. 170.
Clendennen S.K., Kipp P.B., May G.D. The role of ethylene in banana fruit ripening // Biology and biotechnology of the plant hormone ethylene / Ed. Kanellis A.K. Kluwer Academic Publishers, 1997. P. 141.
Dominguez M., Vendrell M. Ethylene biosynthesis in banana fruit: evolution of EFE activity and ACC levels in peel and pulp during ripening // J. Hortic. Sci. 1993. V. 68. P. 63.
Giovannoni J., Friedman H., Vrebalov J., Elitzur T. US Patent № US20130036515A1, 2013.
Villavicencio L., Blankenship S., Sanders D., Swallow W. Ethylene and carbon dioxide production in detached fruit of selected pepper cultivars // J. Am. Soc. Hortic. Sci. 1999. V. 124. P. 402.
Paul V., Pandey R., Srivastava G.C. The fading distinctions between classical patterns of ripening in climacteric and non-climacteric fruit and the ubiquity of ethylene // J. Food Sci. Technol. 2012. V. 49. P. 1.
Hou B.-Z., Li C.-L., Han Y.-Y., Shen Y.-Y. Characterization of the hot pepper (Capsicum frutescens) fruit ripening regulated by ethylene and ABA // BMC Plant Biol. 2018. V. 18: 162.
Dubey M., Jaiswal V., Rawoof A., Kumar A., Nitin M., Chhapekar S.S., Kumar N., Ahmad I., Islam K., Brahma V., Ramchiary N. Identification of genes involved in fruit development/ripening in Capsicum and development of functional markers // Genomics. 2019. V. 111. P. 1913. https://doi.org/10.1016/j.ygeno.2019.01.002
Fei Z., Tang X., Alba R.M., White J.A., Ronning C.M., Martin G.B., Tanksley S.D., Giovannoni J.J. Comprehensive EST analysis of tomato and comparative genomics of fruit ripening // Plant J. 2004. V. 40. P. 47.
Seymour G.B., Ryder C.D., Cevik V., Hammond J.P., Popovich A., King G.J., Vrebalov J., Giovannoni J.J., Manning K. A SEPALLATA gene is involved in the development and ripening of strawberry (Fragaria × ananassa Duch.) fruit, a non-climacteric tissue // J. Exp. Bot. 2011. V. 62. P. 1179. https://doi.org/10.1093/jxb/erq360
Slugina M.A., Shchennikova A.V., Pishnaya O.N., Kochieva E.Z. Assessment of the fruit-ripening-related FUL2 gene diversity in morphophysiologically contrasted cultivated and wild tomato species // Molecular Breeding. 2018. V. 38: 82 https://doi.org/10.1007/s11032-018-0842-x
Slugina M.A., Shchennikova A.V., Kochieva E.Z. The expression pattern of the Pho1a genes encoding plastidic starch phosphorylase correlates with the degradation of starch during fruit ripening in green-fruited and red-fruited tomato species // Funct. Plant Biol. 2019. V. 46. P. 1146. https://doi.org/10.1071/FP18317
Efremov G.I., Slugina M.A., Shchennikova A.V. Kochieva E.Z. Differential Regulation of Phytoene Synthase PSY1 During Fruit Carotenogenesis in Cultivated and Wild Tomato Species (Solanum section Lycopersicon) // Plants. 2020. V. 9: 1169 https://doi.org/10.3390/plants9091169
Slugina M.A., Shchennikova A.V., Kochieva E.Z. TAI vacuolar invertase orthologs: the interspecific variability in tomato plants (Solanum section Lycopersicon) // Mol. Genet. Genomics. 2017. V. 292. P. 1123. https://doi.org/10.1007/s00438-017-1336-y
Slugina M.A., Shchennikova A.V., Kochieva E.Z. LIN7 Cell-Wall Invertase Orthologs in Cultivated and Wild Tomatoes (Solanum Section Lycopersicon) // Plant Mol. Biol. Rep. 2018. V. 36. P. 195. https://doi.org/10.1007/s11105-018-1071-5
Slugina M.A., Shchennikova A.V., Kochieva E.Z. Differences in the sucrose synthase gene SUS1 expression pattern between Solanum lycopersicum and wild tomato species // Theor. Exp. Plant Physiol. 2019. V. 31. P. 455. https://doi.org/10.1007/s40626-019-00158-6
Giovannoni J., Nguyen C., Ampofo B., Zhong S., Fei Z. The epigenome and transcriptional dynamics of fruit ripening // Ann. Rev. Plant Biol. 2017. V. 68. P. 61.
Li Z., Jiang G., Liu X., Ding X., Zhang D., Wang X., Zhou Y., Yan H., Li T., Wu K., Jiang Y., Duan X. Histone demethylase SlJMJ6 promotes fruit ripening by removing H3K27 methylation of ripening-related genes in tomato // New Phytol. 2020. V. 227. P. 1138. https://doi.org/10.1111/nph.16590
Guo J.E., Hu Z., Yu X., Li A., Li F., Wang Y., Tian S., Chen G. A histone deacetylase gene, SlHDA3, acts as a negative regulator of fruit ripening and carotenoid accumulation // Plant Cell Rep. 2018. V. 37. P. 125. https://doi.org/10.1007/s00299-017-2211-3
Liu R., How-Kit A., Stammitti L., Teyssier E., Rolin D., Mortain-Bertrand A., Halle S., Liu M., Kong J., Wu C. A DEMETER-like DNA demethylase governs tomato fruit ripening // PNAS. 2015. V. 112. P. 10804.
Lang Z., Wang Y., Tang K., Tang D., Datsenka T., Cheng J., Zhang Y., Handa A.K., Zhu J.-K. Critical roles of DNA demethylation in the activation of ripening-induced genes and inhibition of ripening-repressed genes in tomato fruit // PNAS. 2017. V. 114. P. 4511.
Cheng J., Niu Q., Zhang B., Chen K., Yang R., Zhu J.K., Zhang Y., Lang Z. Downregulation of RdDM during strawberry fruit ripening // Genome Biol. 2018. V. 19: 212. https://doi.org/10.1186/s13059-018-1587-x
Huang H., Liu R., Niu Q., Tang K., Zhang B., Zhang H., Chen K., Zhu J.K., Lang Z. Global increase in DNA methylation during orange fruit development and ripening // PNAS. 2019 V. 116. P. 1430. https://doi.org/10.1073/pnas.1815441116
Yu W., Peng F., Xiao Y., Wang G., Luo J. Overexpression of PpSnRK1 in Tomato Promotes Fruit Ripening by Enhancing RIPENING INHIBITOR Regulation Pathway // Front. Plant Sci. 2018. V. 9: 1856. https://doi.org/10.3389/fpls.2018.01856
Li G.J., Peng F.T., Zhang L., Shi X.Z., Wang Z.Y. Cloning and characterization of a SnRK1-encoding gene from Malus hupehensis Rehd. and heterologous expression in tomato // Mol. Biol. Rep. 2010. V. 37. P. 947. https://doi.org/10.1007/ s11033-009-9734-9
Wang X.L., Peng F.T., Li M.J., Yang L., Li G.J. Expression of a heterologous SnRK1 in tomato increases carbon assimilation, nitrogen uptake and modifies fruit development // J. Plant Physiol. 2012. V. 169. P. 1173. https://doi.org/10.1016/j.jplph.2012.04.013
Naing A.H., Kyu S.Y., Pe P.P.W., Park K.I., Lee J.M., Lim K.B., Kim C.K. Silencing of the phytoene desaturase (PDS) gene affects the expression of fruit-ripening genes in tomatoes // Plant Methods. 2019. V. 15: 110. https://doi.org/10.1186/s13007-019-0491-z
Sun Q., Zhang N., Wang J., Cao Y., Li X., Zhang H., Zhang L., Tan D.X., Guo Y.D. A label-free differential proteomics analysis reveals the effect of melatonin on promoting fruit ripening and anthocyanin accumulation upon postharvest in tomato // J. Pineal Res. 2016. V. 61. P. 138. https://doi.org/10.1111/jpi.12315
Дополнительные материалы отсутствуют.
Инструменты
Физиология растений


